mRNA 디스플레이
mRNA displaymRNA 디스플레이는 원하는 대상에 결합할 수 있는 분자를 생성하기 위해 시험관내 단백질 및/또는 펩타이드 진화에 사용되는 디스플레이 기법이다. 그 과정은 puromycin 연결을 통해 그들의 mRNA 생성자와 관련된 변환된 펩타이드나 단백질을 만들어낸다. 그런 다음 콤플렉스는 선택 단계(선호도 크로마토그래피)에서 고정된 대상에 결합한다. 잘 결합되는 mRNA-단백질 결합은 역행성 중합효소 연쇄반응을 통해 증폭된 cDNA와 그 염기서열로 역행한다. 결과는 관심 분자에 대한 높은 친화력으로 펩타이드 인코딩하는 뉴클레오티드 시퀀스다.
푸로마이신(Puromycin)은 구조물의 일부가 아데노신 분자를 모방한 티로실-tRNA의 3'끝을 아날로그로, 다른 부분은 티로신 분자를 모방한 것이다. 티로실-tRNA에서 갈라질 수 있는 에스테르 결합에 비해 푸로마이신은 수분이 없는 아미드 결합을 가지고 있다. 그 결과 푸로마이신은 번역을 방해하고, 번역 제품의 조기 출시를 초래한다.

mRNA 디스플레이 기술에 사용되는 모든 mRNA 템플릿은 3'끝에 푸로마이신이 있다. 번역이 진행되면서 리보솜은 mRNA 템플릿을 따라 움직이며, 일단 템플릿의 3의 끝에 도달하면 퓨전 푸로마이신은 리보솜의 A 사이트에 들어가 초기 펩타이드에 통합된다. 그런 다음 mRNA-폴리펩타이드 융합을 리보솜에서 방출한다(그림 1).
mRNA-폴리펩타이드 융합을 합성하기 위해, 퓨전 푸로마이신만이 mRNA 템플릿의 수정이 아니다.[1] 푸로마이신이 A 사이트에 진입할 수 있도록 유연성과 적절한 길이를 제공하기 위해 푸로마이신과 함께 올리고뉴클레오티드 등 스페이서를 모집할 필요가 있다. 이상적으로, mRNA의 3의 끝과 푸로마이신 사이의 링커는 마지막 코돈의 번역에 따라 푸로마이신이 A 사이트에 들어갈 수 있을 정도로 충분히 유연하고 길어야 한다. 이를 통해 고품질의 전신 mRNA 폴리펩타이드 융합을 효율적으로 생산할 수 있다. 리허 류 외 연구진은 3'-푸로마이신 올리고뉴클레오티드 스페이서를 최적화했다. 그들은 dA25가 Spacer 9(Glen Research), 그리고 5' 종단부의 dAdCdCP와 결합한 것이 핵융합 반응에 가장 효과적이라고 보고했다. 그들은 40개 뉴클레오티드가 길고 16개 뉴클레오티드가 짧은 링커는 핵융합 형성의 효율을 크게 떨어뜨린다는 것을 발견했다. 또한 푸로마이신 근처에 제시된 시퀀스 rURUP가 있을 때 융접이 효율적으로 형성되지 않았다.[2]
링커의 폴리 dA 부분은 유연성과 길이뿐만 아니라 dT 셀룰로오스 수지에 대한 친화력이 높아 mRNA-폴리펩타이드 융합을 더욱 정화할 수 있다.[3] mRNA 폴리펩타이드 퓨즈는 끈적임성이 증가하는 여러 라운드에 대해 고정된 선택 대상을 통해 선택할 수 있다. 선정될 때마다 고정된 대상에 묶여 있는 도서관 회원들은 PCR 증폭되고 비빈더들은 씻겨진다.
방법
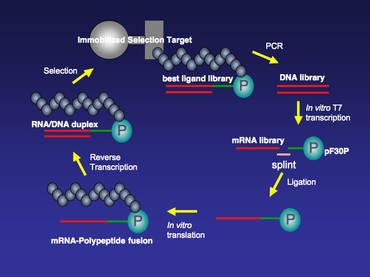
mRNA 디스플레이 라이브러리의 합성은 DNA 라이브러리의 합성으로 시작한다. 관심 단백질이나 작은 펩타이드의 DNA 라이브러리는 고체상 합성에 이어 PCR 증폭에 의해 합성될 수 있다. 보통 이 DNA 라이브러리의 각 구성원은 T7 RNA 중합효소 전사 부지와 5의 끝에 리보솜 결합 부지가 있다. T7 프로모터 영역은 대규모 체외 T7 전사가 DNA 라이브러리를 mRNA 라이브러리로 옮겨서 나중에 체외 번역 반응을 위한 템플릿을 제공한다. 5' 비분산 지역(5' UTR)의 리보솜 결합 부위는 사용할 체외 번역 시스템에 따라 설계된다. 상업적으로 이용 가능한 두 개의 시험관내 번역 시스템이 있다. 하나는 E이다. 리보솜 결합 사이트로 5' UTR의 샤인달가노 시퀀스를 필요로 하는 콜리 S30 추출 시스템(Promega)이며,[4] 다른 하나는 ΔTMV 리보솜 결합 사이트가 필요한 레드 노바 리세이트(Novagen)이다.
mRNA 라이브러리가 생성되면 3의 끝부분에 푸로마이신이 들어 있는 DNA 스페이서 링커에 T4 DNA 리가아제를 사용하여 우레아-PAGE를 정제하고 묶는다. 이 탐색 단계에서, mRNA의 조각은 T4 DNA 리가제의 도움을 받아 하나의 좌초된 DNA로 묶인다. 이것은 두 조각의 이중 좌초된 DNA가 함께 묶여 있는 T4 DNA 리가아제 레깅스 반응의 표준이 아니다. 이 특수 레깅의 수율을 높이기 위해, 단 하나의 좌초된 DNA 부목을 레깅 반응을 돕기 위해 사용할 수 있다. 스플린트의 5' 종단부는 mRNA의 3' 끝단을 보완하도록 설계되었고, 스플린트의 3' 종단부는 DNA 스페이서 링커의 5' 끝단을 보완하도록 설계되었으며, 일반적으로 폴리 dA 뉴클레오티드로 구성된다(그림 2).
묶인 mRNA-DNA-puromycin 라이브러리는 Red Nova Lysate(노바겐) 또는 E로 번역된다. Coly S30 추출 시스템(Promega)으로, cis에서 polyptide가 인코딩 mRNA와 공칭적으로 연결된다. 시험관내 번역은 PURE(재조합 원소를 이용한 단백질 합성) 시스템에서도 할 수 있다. PURE 시스템은 E이다. 필수 번역 성분만 존재하는 대장균 세포 없는 번역 시스템. 아미노산 및 아미노산-tRNA 동기화(AARSs)와 같은 일부 구성요소는 시스템에서 생략할 수 있다. 대신 화학적으로 아틸화 tRNA를 PURE 시스템에 추가할 수 있다. PURE 시스템에서 N-메틸-아미노산 어큐틸화 tRNA와 같은 일부 부자연스러운 아미노산이 펩타이드나 mRNA-폴리펩타이드 퓨전에 통합될 수 있다는 것이 밝혀졌다.[5]
번역 후, 퓨즈의 단일 가닥 mRNA 부분은 원치 않는 RNA 2차 구조를 제거하기 위해 역분해효소에 의해 RNA/DNA의 이질화합물로 변환되며, 핵산 부분을 보다 안정적으로 만들어 줄 것이다. 이 단계는 표준 역전사 반응이다. 예를 들어 제조자의 프로토콜에 따라 Superscript II(GIBCO-BRL)를 사용하면 된다.
mRNA/DNA 폴리펩타이드 퓨즈는 여러 라운드에 대해 고정된 선택 대상을 통해 선택할 수 있다(그림 3). 처음 몇 번의 선택에는 상대적으로 높은 배경이 있을 수 있으며, 이는 대상/융접 결합 기간 동안 염분 농도, 세제 양 및/또는 온도 조절과 같은 선택 끈기를 증가시켜 최소화할 수 있다. 바인딩 선택 후, 고정된 대상에 바인딩된 상태로 있는 라이브러리 구성원은 PCR 증폭된다. PCR 증폭 단계는 고정된 대상에 대한 선호도가 높은 mRNA-디스플레이 라이브러리에서 모집단을 풍부하게 한다. 또한 각 선택 과정 사이에 오류 발생 가능성이 있는 PCR을 수행하여 mRNA-디스플레이 라이브러리의 다양성을 더욱 높이고 선택 배경을 줄일 수 있다.[6]
mRNA 디스플레이를 위한 시간이 덜 걸리는 프로토콜이 최근에 출판되었다.[7]
이점
페이지 디스플레이, 세균 디스플레이, 효모 디스플레이, 리보솜 디스플레이 등 그 외 분자 디스플레이 기술이 많지만 mRNA 디스플레이 기술은 다른 기술보다 장점이 많다.[8] 나열된 처음 3개의 생물학적 표시 라이브러리는 각각의 미생물 표면에 폴리펩타이드나 단백질을 표현하고 있으며, 각 폴리펩타이드나 단백질에 대한 수반되는 코딩 정보는 미생물의 게놈에서 검색할 수 있다. 그러나 이러한 세 가지 체내 디스플레이 시스템의 라이브러리 크기는 각 유기체의 변환 효율성에 의해 제한된다. 예를 들어, 페이지와 박테리아 디스플레이의 라이브러리 크기는 1-10 × 10^9 다른 멤버로 제한된다. 효모 전시용 도서관 크기는 더욱 작다. 더욱이 이러한 세포 기반 디스플레이 시스템은 천연 아미노산을 함유한 펩타이드/단백질만 선별하고 농축할 수 있다. 반대로 mRNA 디스플레이와 리보솜 디스플레이는 체외 선택 방식이다. 그들은 10^15개의 다른 회원들만큼 큰 도서관 크기를 허용한다. 큰 라이브러리 크기는 매우 희귀한 시퀀스를 선택할 확률을 증가시키며, 또한 선택된 시퀀스의 다양성을 향상시킨다. 또한 시험관내 선택방법은 단백질 발현 불량 등 원치 않는 선택압력, 급속한 단백질 분해 등을 제거하여 선택된 시퀀스의 다양성을 떨어뜨릴 수 있다. 마지막으로, 체외 선택 방법은 선택 과정 전반에 걸쳐 체외 돌연변이 유발[9] 및 재결합 기법을 적용할 수 있다.
리보솜 디스플레이와 mRNA 디스플레이 모두 체외 선택 방법이지만, mRNA 디스플레이는 리보솜 디스플레이 기술에 비해 다소 유리하다.[10] mRNA 디스플레이는 푸로마이신을 통해 연결된 공동 mRNA-폴리펩타이드 복합체를 활용하지만, 리보솜 디스플레이는 정지된 비공동 리보솜-mRNA 폴리펩타이드 복합체를 활용한다.[11] 리보솜 디스플레이의 경우, 리보솜-mRNA-폴리펩타이드 콤플렉스가 비코밸런스 리보솜-mRNA-폴리펩타이드 콤플렉스로 인해 리보솜-mRNA-폴리펩타이드 콤플렉스를 단지 내에 보관할 수 있는 선택성이 제한된다. 이는 선택 주기 동안 배경 결합을 줄이는 데 어려움을 초래할 수 있다. 또한 리보솜 디스플레이 시스템에서 선택되고 있는 폴리펩타이드들은 분자량이 200만 Da 이상인 거대한 rRNA-단백질 복합체인 리보솜에 부착되어 있다. 선택 대상과 리보솜 사이에 예측할 수 없는 상호작용이 있을 수 있으며, 이로 인해 선택 주기 동안 잠재적 바인더가 손실될 수 있다. 반면 mRNA 디스플레이 기술에 사용되는 푸로마이신 DNA 스페이서 링커는 리보솜에 비해 훨씬 작다. 이 링커는 고정된 선택 대상과 상호작용할 기회가 적을 수 있다. 따라서 mRNA 디스플레이 기술은 편향된 결과를 덜 줄 가능성이 높다.
적용
1997년, 로버츠와 쇼스탁은 합성 mRNA와 그것의 부호화된 myc 비문 사이의 유전이 면역복제에 의해 mRNA-폴리펩타이드 유착의 풀에서 농축될 수 있다는 것을 보여주었다.[6]
9년 뒤 후쿠다와 동료들은 단일체인 Fv(scFV) 항체 파편의 체외 진화를 위해 mRNA 디스플레이 방식을 선택했다.[12] 그들은 5개의 일치 돌연변이를 가진 6개의 다른 scFv 돌연변이를 선택했다. 그러나 이들 돌연변이에 대한 운동학적 분석을 통해 이들의 항원 특이성이 야생형과 유사한 것으로 나타났다. 그러나 그들은 5개의 합의된 돌연변이 중 2개가 CDR(상보성 결정 지역) 내에 있음을 입증했다. 그리고 그들은 mRNA 디스플레이가 CDR을 최적화함으로써 고선호도 진단 및 치료 항체의 신속한 인공 진화의 잠재력을 가지고 있다고 결론지었다.
로버츠와 동료들은 N-대체 아미노산으로 구성된 부자연스러운 펩타이드 과점체가 mRNA-폴리펩타이드 융합으로 합성될 수 있다는 것을 증명했다.[13] N-대체 아미노산 함유 펩타이드는 양호한 단백질 분해 안정성과 개선된 약동학적 특성과 관련이 있다. 이 연구는 mRNA 디스플레이 기술이 단백질 분해에 내성을 가진 치료적 사용을 위한 약물 유사 펩타이드를 선택할 수 있는 잠재력을 가지고 있음을 보여준다.[14]
참고 항목
참조
- ^ Roberts RW, Szostak JW (1997). "RNA-peptide fusions for the in vitro selection of peptides and proteins". PNAS. 94 (23): 12297–12302. Bibcode:1997PNAS...9412297R. doi:10.1073/pnas.94.23.12297. PMC 24913. PMID 9356443.
- ^ Liu R, Barrick JE, Szostak JW, Roberts RW (2000). "Optimized synthesis of RNA-protein fusions for in vitro protein selection". Methods in Enzymology. 318: 268–93. doi:10.1016/S0076-6879(00)18058-9. ISBN 9780121822194. PMID 10889994.
- ^ Kurz M, Gu K, Lohse PA (2000). "Psoralen photo-crosslinked mRNA–puromycin conjugates: a novel template for the rapid and facile preparation of mRNA–protein fusions". Nucleic Acids Research. 28 (18): 83e–83. doi:10.1093/nar/28.18.e83. PMC 110755. PMID 10982894.
- ^ Mattheakis LC, Bhatt RR, Dower WJ (1994). "An in vitro polysome display system for identifying ligands from very large peptide libraries". Proc Natl Acad Sci USA. 91 (19): 9022–6. Bibcode:1994PNAS...91.9022M. doi:10.1073/pnas.91.19.9022. PMC 44739. PMID 7522328.
- ^ Kawakami T, Murakami H, Suga H (January 2008). "Messenger RNA-programmed incorporation of multiple N-methyl-amino acids into linear and cyclic peptides". Chemistry & Biology. 15 (1): 32–42. doi:10.1016/j.chembiol.2007.12.008. PMID 18215771.
- ^ a b Roberts RW, Szostak JW (1997). "RNA-peptide fusions for the in vitro selection of peptides and proteins". Proc Natl Acad Sci USA. 94 (23): 12297–302. Bibcode:1997PNAS...9412297R. doi:10.1073/pnas.94.23.12297. PMC 24913. PMID 9356443.
- ^ Barendt PA, Ng DT, McQuade CN, Sarkar CA (2013). "Streamlined Protocol for mRNA Display". ACS Combinatorial Science. 15 (2): 77–81. doi:10.1021/co300135r. PMC 3666848. PMID 23305392.
- ^ Roberts RW (June 1999). "Totally in vitro protein selection using mRNA-protein fusions and ribosome display". Current Opinion in Chemical Biology. 3 (3): 268–73. doi:10.1016/S1367-5931(99)80042-8. PMID 10359713.
- ^ Jing D, Li F, Jiang M, Cai J, Wu Y, Xie K, Wu X, Tang C, Liu J, Guo W, Shen G, Luo E (November 2013). "Pulsed Electromagnetic Fields Improve Bone Microstructure And Strength In Ovariectomized Rats". PLoS ONE. 8 (11): e79377. Bibcode:2013PLoSO...879377J. doi:10.1371/journal.pone.0079377. PMC 3828367. PMID 24244491.
- ^ Gold L (April 2001). "mRNA display: diversity matters during in vitro selection". Proc Natl Acad Sci USA. 98 (9): 4825–6. Bibcode:2001PNAS...98.4825G. doi:10.1073/pnas.091101698. PMC 33119. PMID 11320229.
- ^ Andrew Buchanan; Lutz Jermutus. "Ribosome Display or Mrna Display Method With Selection for Increased Stability of the Protein". Google Patents. Retrieved 9 June 2014.
- ^ Fukuda I, Kojoh K, Tabata N, et al. (2006). "In vitro evolution of single-chain antibodies using mRNA display". Nucleic Acids Research. 34 (19): e127. doi:10.1093/nar/gkl618. PMC 1636464. PMID 17012279.
- ^ Frankel A, Millward SW, Roberts RW (November 2003). "Encodamers: unnatural peptide oligomers encoded in RNA" (PDF). Chemistry & Biology. 10 (11): 1043–50. doi:10.1016/j.chembiol.2003.11.004. PMID 14652071.
- ^ White, E. Railey; Sun, Luxin; Ma, Zhong; Beckta, Jason M.; Danzig, Brittany A.; Hacker, David E.; Huie, Melissa; Williams, David C.; Edwards, Ross A. (2015-05-15). "Peptide Library Approach to Uncover Phosphomimetic Inhibitors of the BRCA1 C-Terminal Domain". ACS Chemical Biology. 10 (5): 1198–1208. doi:10.1021/cb500757u. PMC 4433557. PMID 25654734.