중합효소 연쇄반응
Polymerase chain reaction중합효소 연쇄반응(PCR)은 특정 DNA 샘플의 수백만에서 수십억 부(완전 복사 또는 부분 복사)를 빠르게 만드는 데 널리 사용되는 방법으로, 과학자들이 아주 작은 DNA 샘플을 채취해 충분히 상세하게 연구할 수 있을 정도로 크게 증폭시킬 수 있다.PCR은 1983년 미국의 생화학자인 케리 멀리스(Kary Mullis)가 케투스 코퍼레이션에서 발명한 것으로, DNA 조작의 다른 필수적인 방법을 개발한 생화학자 마이클 스미스가 1993년 노벨 화학상을 공동 수상했다.[1]
PCR은 고대의 DNA 샘플 분석과 감염원 식별을 포함한 유전자 검사 및 연구에 사용되는 많은 절차의 기본이다.PCR을 사용하면, 극소량의 DNA 서열의 복사본은 일련의 온도 변화 주기에서 기하급수적으로 증폭된다.PCR은 이제 바이오의학 연구와 범죄 포렌식을 포함한 광범위한 응용을 위해 의료 실험실 연구에서 흔히 사용되는 필수불가결한 기법이 되었다.[2][3]
대부분의 PCR 방법은 열 순환에 의존한다.열 순환은 서로 다른 온도 의존적 반응(특히 DNA 용해 및 효소 기반 DNA 복제)을 허용하기 위해 가열과 냉방의 반복 사이클에 반응제를 노출시킨다.PCR은 두 가지 주요 시약, 즉 프라이머(primer, 표적 DNA 영역에 대한 보완적 시퀀스인 올리고뉴클레오티드라고 알려진 짧은 단일 가닥 DNA 조각)와 DNA 중합효소(dna polyerase)를 사용한다.PCR의 첫 단계에서 DNA 이중나선의 두 가닥은 핵산 변성이라는 과정에서 높은 온도에서 물리적으로 분리된다.두 번째 단계에서는 온도가 낮아지고 프라이머가 DNA의 상호보완적인 배열과 결합한다.그리고 나서 두 개의 DNA 가닥은 DNA의 구성 요소인 자유 뉴클레오티드에서 효소적으로 새로운 DNA 가닥을 조립하는 DNA 중합효소의 템플릿이 된다.PCR이 진행됨에 따라 생성된 DNA는 복제를 위한 템플릿으로 그 자체가 사용되어 원래의 DNA 템플릿이 기하급수적으로 증폭되는 연쇄반응이 일어나게 된다.
거의 모든 PCR 애플리케이션은 열성 박테리아 써머스 아쿠아쿠스로부터 원래 격리된 효소인 Taq 중합효소와 같은 열안정성 DNA 중합효소를 사용한다.사용된 중합효소가 열에 민감하다면 변성 단계의 높은 온도에서 변성될 것이다.Taq 중합효소를 사용하기 전에는 주기마다 DNA 중합효소를 수동으로 첨가해야 했는데, 이는 지루하고 비용이 많이 드는 과정이었다.[4]
기술에 응용 프로그램 서열을 알기 위해 DNA복제, 유전자 복제와 조작, 유전자 돌연변이 유발, 기초로 한 phylogenies 또는 유전자의 기능적인 분석, 진단과 유전적 장애 및 모니터링 대상에 건축이 늦어졌다;고대의 DNA증폭, 유전자 지문의(예를 들, 법 의학적 scienc에 DNA감식에[5]분석을 포함한다.e는nd propage testing); 및 감염성. 진단을 위한 핵산 테스트에서 병원균의 검출.
원칙

PCR은 DNA 가닥의 특정 부위(DNA 표적)를 증폭시킨다.일부 기법은 파편을 최대 40kbp까지 증폭시킬 수 있지만 대부분의 PCR 방법은 0.1~10ko 염기쌍(kbp)의 DNA 파편을 증폭시킨다.[6]증폭된 제품의 양은 반응에서 이용 가능한 기질에 의해 결정되며, 반응이 진행됨에 따라 제한된다.[7]
기본적인 PCR 설정에는 다음과 같은 몇 가지 구성품과 시약이 필요하다.[8]
- 증폭시킬 DNA 표적 부위가 들어 있는 DNA 템플릿
- DNA 중합효소; 새로운 DNA 가닥을 중합하는 효소; 내열성 타크 중합효소는 고온 DNA 변성 과정에서 온전하게 유지될 가능성이 높아 특히 흔하다.[9]
- 는 DNA표적(DNA중합 효소에만 DNA의 이중 나선 지역에서 길게 늘이다;프라이머 없이, 2중 가닥 개시 사이트는은 중합 효소 묶이 묶이)의식과anti-sense 가닥의 3'(3총리)의 끝단에 보완하는 2개 DNA프라이머, 있는 보수[10]특정 프라이머.aryDNA 표적 영역은 사전에 선택되며, 종종 실험실에서 맞춤 제작되거나 상업용 생화학적 공급 업체로부터 구입된다.
- 디옥시뉴클레오시드 3인산염 또는 dNTP(때로는 "데옥시뉴클레오티드 3인산염; 트리인산염을 포함한 뉴클레오티드), DNA 중합효소가 새로운 DNA 가닥을 합성하는 구성 블록
- DNA 중합효소의 최적 활동과 안정성을 위한 적절한 화학적 환경을 제공하는 완충 용액
- Mn 농도가2+ 높을수록 DNA 합성 중 오류율이 증가하며 단발성 양이온(일반적으로 칼륨(K) 이온[better source needed], 마그네슘(Mg) 또는 망간(Mn) 이온, Mg가2+ 가장 일반적이지만 PCR 매개 DNA 돌연변이 유발에 Mn을2+ 사용할 수 있다.[11]
이 반응은 일반적으로 열 사이클러의 작은 반응관(0.2–0.5 mL 부피)에서 10–200 μL의 부피로 수행된다.열 사이클러는 반응의 각 단계에서 요구되는 온도를 달성하기 위해 반응 튜브를 가열하고 냉각시킨다(아래 참조).현대의 많은 열 사이클 선수들은 단순히 전류를 역전시킴으로써 PCR 튜브를 잡고 있는 블록의 가열과 냉각을 모두 허용하는 펠티에어 효과를 이용한다.박벽 반응관은 유리한 열전도도를 허용해 빠른 열평형을 가능하게 한다.대부분의 열 사이클 선수들은 반응관 상단의 응결을 방지하기 위해 열선 뚜껑이 있다.열선내장 뚜껑이 없는 나이든 열성 자전거 사용자들은 반응 혼합물 위에 기름 층을 두거나 튜브 안에 있는 왁스 덩어리들을 필요로 한다.
절차
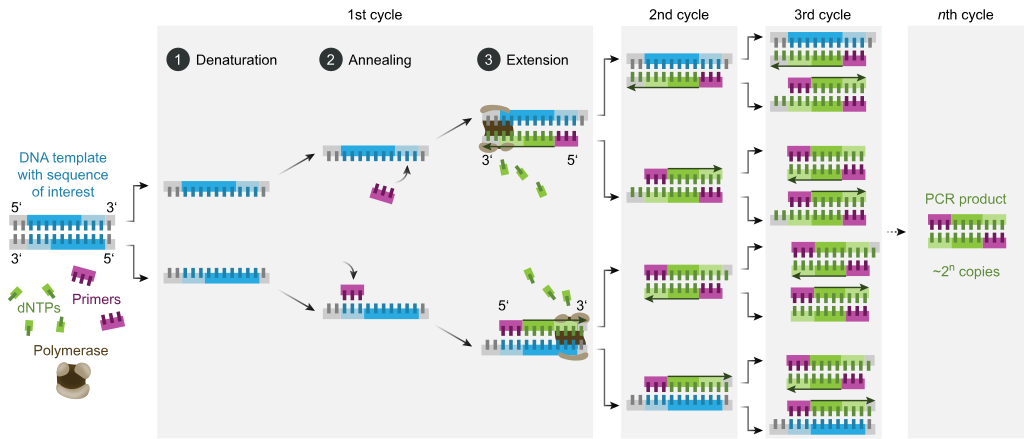
일반적으로 PCR은 열 사이클이라 불리는 일련의 20-40번 반복 온도 변화로 구성되며, 각 사이클은 일반적으로 두 개 또는 세 개의 이산 온도 단계로 구성된다(아래 그림 참조).사이클링은 매우 높은 온도(>90°C(194°F)에서 단일 온도 스텝이 선행되는 경우가 많으며, 최종 제품 확장 또는 간략한 저장을 위해 마지막에 한 번 홀드하는 경우가 많다.각 사이클에서 사용되는 온도와 그것들이 적용되는 시간은 DNA 합성에 사용되는 효소, 반응에서 이온과 dNTP의 농도, 프라이머의 용해 온도(Tm)를 포함한 다양한 파라미터에 따라 달라진다.[12]대부분의 PCR 방법에 공통적인 개별 단계는 다음과 같다.
- 초기화:이 단계는 핫 스타트 PCR에 의한 열 활성화가 필요한 DNA 중합체에만 필요하다.[13]반응 챔버를 94–96°C(201–205°F)의 온도로 가열하거나, 극도로 온도 조절 가능한 중합체를 사용할 경우 98°C(208°F)의 온도로 가열하여 1-10분간 유지한다.
- 변성:이 단계는 첫 번째 정기 사이클링 이벤트로, 반응 챔버를 94–98°C(201–208°F)로 20~30초 동안 가열하는 것으로 구성된다.이것은 보완 염기들 사이의 수소 결합을 깨뜨려 두 개의 단일 가닥 DNA 분자를 생성함으로써 이중 가닥 DNA 템플릿의 DNA 용해 또는 변성을 야기한다.
- Annealing:다음 단계에서는 반응 온도가 20-40초 동안 50–65°C(122–149°F)로 낮아져 프라이머를 각 단일 가닥의 DNA 템플릿에 대해 분해할 수 있다.두 개의 서로 다른 프라이머가 일반적으로 반응 혼합물에 포함된다. 하나는 목표 영역을 포함하는 단일 가닥 보완물이다.프라이머는 단일 가닥 시퀀스 자체지만 대상 영역의 길이보다 훨씬 짧아 각 가닥의 3개 끝에서 매우 짧은 시퀀스만 보완한다.
- 효율성과 특수성은 어닐링 온도의 영향을 강하게 받기 때문에 어닐링 단계에 적절한 온도를 결정하는 것이 중요하다.이 온도는 스트랜드에 프라이머가 혼합될 수 있을 만큼 충분히 낮아야 하지만, 혼합이 구체화될 수 있을 만큼 충분히 높아야 한다. 즉 프라이머는 스트랜드의 완전히 보완되는 부분에만 결합되어야 한다.온도가 너무 낮으면 프라이머가 불완전하게 결합될 수 있다.너무 높으면 프라이머가 전혀 결합되지 않을 수 있다.일반적인 어닐링 온도는 사용되는 프라이머의 T보다m 약 3~5°C 낮다.보완 베이스 간의 안정적인 수소 결합은 프라이머 시퀀스가 템플릿 시퀀스와 매우 밀접하게 일치해야 형성된다.이 단계에서 중합효소는 프라이머-템플릿 하이브리드에 결합되어 DNA 형성을 시작한다.
- 확장/긴축:이 단계의 온도는 사용된 DNA 중합효소에 따라 달라진다. Taq 중합효소의 온도조절 DNA 중합효소에 대한 최적 활성 온도는 약 75-80°C(167–176°F)[14][15]이지만, 이 효소와 함께 일반적으로 72°C(162°F)의 온도가 사용된다.이 단계에서 DNA 중합효소는 5~3' 방향의 템플릿에 보완되는 반응 혼합물로부터 자유 dNTP를 더하고, dNTP의 5'-인산염 그룹을 나스센트(엘론팅) DNA Strand의 끝에 있는 3'-hydroxy 그룹과 응축함으로써 DNA 템플릿 스트랜드에 보완되는 새로운 DNA 가닥을 합성한다.연장에 필요한 정확한 시간은 사용된 DNA 중합효소와 증폭할 DNA 표적 부위의 길이에 따라 달라진다.경험상, 최적의 온도에서 대부분의 DNA 중합체는 분당 천 염기스를 중합한다.최적의 조건(즉, 기판이나 시약 제한으로 인한 제한이 없는 경우), 각 연장/연장 단계에서 DNA 표적 시퀀스의 수는 두 배로 증가한다.각 연속 사이클에 따라 원래 템플릿 가닥과 새로 생성된 모든 가닥이 다음 단계의 연장에 대한 템플릿 가닥이 되어 특정 DNA 표적 부위의 지수(지오메트리) 증폭으로 이어진다.
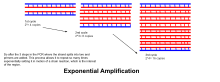
- 변성, 어닐링 및 연장의 과정은 단일 사이클을 구성한다.DNA 표적을 수백만 부까지 증폭시키려면 여러 사이클이 필요하다.주어진 주기 수 후에 형성된 DNA 복사본의 수를 계산하는 데 사용되는 공식은 2이며n, 여기서 n은 주기 수이다.따라서 30 사이클에 대해 설정된 반응은 원래의 이중 가닥 DNA 표적 영역의 사본인30 2, 1,073,741,824의 결과를 낳는다.
- 최종 연장:이 단일 단계는 선택 사항이지만, 마지막 PCR 사이클 이후 5-15분 동안 70–74°C(158–165°F)의 온도(158–165°F)에서 수행되어 나머지 단일 변형 DNA가 완전히 길어지는 것을 보장한다.
- 최종 보류:마지막 단계는 무한정 4–15°C(39–59°F)로 반응 챔버를 냉각시키며, PCR 제품의 단기 보관을 위해 사용할 수 있다.
PCR이 예상 DNA 표적 부위(일명 증폭기 또는 증폭기라고도 함)를 성공적으로 생성했는지 여부를 확인하기 위해 PCR 제품의 크기 분리에 Agarose 젤 전기영향을 사용할 수 있다.PCR 제품의 크기는 알려진 크기의 DNA 파편이 들어 있는 분자 무게 표시기인 DNA 사다리(DNA Ladder)와 비교하여 결정되며, 이는 PCR 제품과 함께 젤 위에서 작동된다.
단계
다른 화학 반응과 마찬가지로 PCR의 반응률과 효율은 제한 인자에 의해 영향을 받는다.따라서 전체 PCR 프로세스는 반응 진도에 따라 세 단계로 더 나눌 수 있다.
- 지수 증폭:주기마다 제품의 양은 두 배로 증가한다(반응 효율 100%로 가정).30주기가 지나면 DNA 한 부씩 최대 100억 부(10억 부)까지 늘릴 수 있다.어떤 의미에서는, DNA의 분리된 가닥의 복제가 통제된 조건에서 튜브 안에서 조작되고 있다.[16]그 반응은 매우 민감하다. DNA의 양은 아주 적어야만 한다.
- 오프 스테이지 레벨링:DNA 중합효소가 활동을 상실하고 dNTP나 프라이머와 같은 시약 섭취가 제한되면서 반응이 느려진다.
- 고원:시약과 효소가 고갈돼 더 이상 제품이 쌓이지 않는다.
최적화
실제로 PCR은 감도나 오염 등 다양한 이유로 실패할 수 있다.[17][18]외부 DNA로 오염되면 가짜 제품을 만들 수 있으며, 잠재적 DNA 오염물질로부터 PCR 혼합물을 분리하는 실험실 프로토콜과 절차로 처리된다.[8]예를 들어 범죄현장의 DNA를 분석하면 실험실 인력의 DNA 분자 1개가 증폭돼 수사를 잘못 이끌 수 있다.따라서 PCR 설정 영역은 다른 PCR 제품의 분석 또는 정화와 분리되어 있으며, 일회용 플라스틱 제품 및 반응 설정 사이의 작업 표면을 철저히 청소할 필요가 있다.
특이성은 가짜 제품이 발생하지 않도록 실험 조건에 의해 조정할 수 있다.프라이머 설계 기법은 PCR 제품 수율을 개선하고 특정되지 않은 제품의 형성을 방지하는 데 중요하다.대체 완충 성분이나 중합효소 효소의 사용은 DNA의 길고 다른 문제 부위의 증폭에 도움이 될 수 있다.예를 들어 Q5 폴리머아제는 Taq 폴리머아제보다 오류 발생률이 약 280배 낮다고 한다.[19][20]실행 매개변수(예: 온도 및 주기 지속시간) 또는 포마미드와 같은 시약 첨가는 PCR의 특수성과 수율을 증가시킬 수 있다.[21]프라이머 설계를 지원하기 위해 이론 PCR 결과(전자 PCR)의 컴퓨터 시뮬레이션을 수행할 수 있다.[22]
적용들
선택적 DNA 격리
PCR은 DNA의 특정 부위의 선택적 증폭에 의해 유전체 DNA로부터 DNA 파편을 분리할 수 있게 한다.PCR의 이러한 사용은 특정 DNA 영역을 대표하는 더 많은 양의 DNA를 필요로 하는 남방 또는 북방 혼성화 및 DNA 복제를 위한 복합화 탐침을 생성하는 것과 같은 여러 가지 방법을 증가시킨다.PCR은 이러한 기법들을 많은 양의 순수한 DNA로 공급하여 아주 적은 양의 시작 물질에서도 DNA 샘플을 분석할 수 있게 한다.
PCR의 다른 응용 프로그램에는 (크기에 따라) 플라스미드, 페이지 또는 코스미드에 DNA 시퀀스를 삽입하는 것과 관련된 재조합 DNA 기술을 촉진하기 위한 DNA 시퀀스(크기에 따라)로 증폭 프라이머 중 하나를 Sanger 시퀀스에 사용할 수 있는 알려지지 않은 PCR 증폭 시퀀스를 결정하기 위한 DNA 시퀀싱이 포함된다.다른 유기체의 종말박테리아 군락(대장균 등)은 정확한 DNA 벡터 구성을 위해 PCR에 의해 신속하게 검사될 수 있다.[23]PCR은 유전자 지문 채취에도 사용될 수 있는데, 이는 다른 PCR 기반 방법을 통해 실험 DNA를 비교함으로써 사람이나 유기체를 식별하는 데 사용되는 법의학 기술이다.
일부 PCR 지문 방식은 차별력이 높아 부모-자녀 또는 형제자매 등 개인 간 유전적 관계를 식별하는 데 사용할 수 있으며, 친자확인 검사(그림 4)에 사용된다.또한 이 기술은 특정 분자시계를 사용할 때 유기체들 사이의 진화적 관계를 결정하는 데 사용될 수 있다(즉, 미생물의 16S rRNA 및 recA 유전자).[24]
DNA 증폭 및 정량화
PCR은 표적이 되는 DNA의 영역을 증폭시키기 때문에 극히 소량의 샘플을 분석하는 데 PCR을 사용할 수 있다.이것은 종종 미량의 DNA만 증거로 사용할 수 있는 법의학 분석에 중요하다.PCR은 수만 년 된 고대 DNA 분석에도 사용될 수 있다.이러한 PCR 기반 기술은 4만 년 된 매머드와 같은 동물과 인간의 DNA에도 성공적으로 사용되었으며, 이집트 미라 분석에서부터 러시아 차르와 영국 왕 리차드 3세의 신체에 대한 확인에 이르는 응용 분야에서도 사용되었다.[25]
정량적 PCR 또는 실시간 PCR(qPCR,[26] RT-PCR과 혼동되지 않음) 방법을 사용하면 샘플에 존재하는 특정 시퀀스의 양을 추정할 수 있는데, 이는 종종 유전자 발현 수준을 정량적으로 결정하는 데 적용되는 기술이다.정량적 PCR은 각 PCR 증폭 후 DNA 제품의 축적을 측정하는 DNA 정량화를 위한 확립된 도구다.
qPCR은 합성 과정이 진행되는 동안 농도를 측정하기 때문에 특정 DNA 시퀀스의 정량화와 검출이 실시간으로 가능하다.동시 검출과 정량화 방법에는 두 가지가 있다.첫 번째 방법은 이중 가닥 사이에 비특이적으로 유지되는 형광 염료를 사용하는 것으로 구성된다.두 번째 방법은 특정 시퀀스에 대해 코드화하고 형광 라벨을 부착하는 프로브를 포함한다.이러한 방법을 이용한 DNA 검출은 보완적인 DNA와 탐침이 혼합된 후에야 알 수 있다.흥미로운 기술 조합은 실시간 PCR과 역전사다.RT-qPCR이라 불리는 이 정교한 기술은 소량의 RNA를 정량화할 수 있게 한다.이 결합 기법을 통해 mRNA를 cDNA로 변환하여, qPCR을 이용하여 추가로 정량화한다.이 기술은 PCR 종점에서의 오류 가능성을 낮춰 암 등 유전질환과 관련된 유전자의 검출 가능성을 높인다.[27][5]실험실은 유전자 조절을 민감하게 측정할 목적으로 RT-qPCR을 사용한다.PCR과[28] RT-qPCR의[29] 신뢰성 있는 정량화를 위한 수학적 기초는 연구, 의료, 진단 및 감염병 애플리케이션에서 실험 데이터의 정확한 적합 절차의 구현을 촉진한다.[30][31][32][33]
의료 및 진단 애플리케이션
예비 부모들은 유전적 보균자라는 이유로 검사를 받을 수도 있고, 아니면 그들의 자녀들이 실제로 병에 걸린 것으로 검사를 받을 수도 있다.[2]산전 검사를 위한 DNA 샘플은 양수, 초리오닉 빌루스 샘플링 또는 심지어 산모의 혈류에서 순환하는 희귀 태아 세포의 분석을 통해 얻을 수 있다.PCR 분석은 또한 발달한 배아의 개별 세포가 돌연변이를 검사하는 이식 전 유전자 진단에도 필수적이다.
- PCR은 또한 장기 이식에 필수적인 조직 입력을 위한 민감한 테스트의 일부로 사용될 수 있다.2008년 현재 혈액형에 대한 기존 항체 기반 검사를 PCR 기반 검사로 대체하자는 제안까지 나오고 있다.[update][34]
- 많은 형태의 암은 종양에 대한 변화를 수반한다.이러한 돌연변이를 연구하기 위해 PCR 기반 테스트를 사용함으로써 치료 요법은 때때로 환자에게 개별적으로 사용자 정의될 수 있다.PCR은 백혈병, 림프종 등 악성 질환에 대한 조기 진단을 허용하는데, 현재 암 연구에서 가장 발달이 높고 이미 일상적으로 사용되고 있다.PCR 검사는 유전체 DNA 검체에 직접 시행해 다른 방법보다 최소 1만배 높은 민감도로 번역특정 악성세포를 검출할 수 있다.[35]PCR은 종양 억제기의 격리와 증폭을 가능하게 하기 때문에 의료 분야에서 매우 유용하다.예를 들어, 정량적 PCR은 DNA, mRNA, 단백질 확인 및 결합을 인식할 뿐만 아니라 단일 세포를 정량화하고 분석하는데 사용될 수 있다.[27]
전염병 응용 프로그램
PCR은 박테리아나 바이러스에 의해 유발되는 감염병을 포함하여 감염병에 대한 신속하고 구체적인 진단을 가능하게 한다.[36]PCR은 또한 조직 배양 검사 및 동물 모델에서 미코박테리아, 혐기성 박테리아 또는 바이러스와 같은 배양 불가능한 또는 느리게 자라는 미생물의 식별을 허용한다.미생물학에서 PCR 진단 어플리케이션의 근거는 감염성 물질을 검출하고 특정 유전자에 의한 병원성 변종과의 비병원성 변종을 구별하는 것이다.[36][37]
감염병 유기체의 특성화와 검출은 다음과 같은 방법으로 PCR에 의해 혁명화되었다.
- 인간면역결핍 바이러스(또는 HIV)는 찾아내고 근절하기 어려운 대상이다.감염에 대한 초기 검사는 혈류에서 순환하는 바이러스에 대한 항체의 존재에 의존했다.그러나 감염 후 수주가 지나야 항체가 나타나고, 산모 항체가 신생아의 감염을 가릴 수 있으며, 감염과 싸우기 위한 치료제는 항체에 영향을 주지 않는다.5만개가 넘는 숙주세포의 DNA 중 1개 정도의 바이러스 게놈을 검출할 수 있는 PCR 테스트가 개발됐다.[38]감염은 조기에 발견될 수 있고 기증된 혈액을 직접 검사해 바이러스를 검사할 수 있으며 신생아는 즉시 감염 여부를 검사할 수 있으며 항바이러스 치료의 효과를 계량화할 수 있다.
- 결핵과 같은 일부 질병 유기체는 환자로부터 샘플을 채취하기 어렵고 실험실에서 재배하는 것이 느리다.PCR 기반 테스트는 편리한 샘플에서 적은 수의 질병 유기체(생존 또는 사망)를 검출할 수 있도록 했다.항생제 내성을 검출하는 데도 세밀한 유전자 분석을 사용할 수 있어 즉각적이고 효과적인 치료가 가능하다.치료의 효과도 즉시 평가할 수 있다.
- 가축이나 야생동물의 개체수를 통한 질병 유기체의 확산은 PCR 테스트로 관찰할 수 있다.많은 경우에, 새로운 치명적인 하위 유형의 출현을 감지하고 감시할 수 있다.초기 전염병을 담당했던 유기체의 하위 유형도 PCR 분석에 의해 결정될 수 있다.
- 바이러스 DNA는 PCR에 의해 검출될 수 있다.사용되는 프라이머는 바이러스의 DNA에서 표적형 배열로 한정되어야 하며 PCR은 바이러스 게놈의 진단 분석이나 DNA 염기서열에 사용될 수 있다.PCR의 높은 민감도는 감염 직후와 심지어 질병이 발생하기 전부터 바이러스 탐지를 허용한다.[36]그러한 조기 발견은 의사에게 치료에 상당한 리드 타임을 줄 수 있다.환자의 바이러스 양("바이러스 부하")도 PCR 기반 DNA 정량화 기법으로 정량화할 수 있다(아래 참조).PCR(RT-PCR)의 변종은 DNA가 아닌 바이러스 RNA를 검출하는 데 사용된다. 이 테스트에서 효소 역분해효소는 바이러스 RNA와 일치하는 DNA 서열을 생성하는 데 사용된다. 그리고 나서 이 DNA는 일반적인 PCR 방법에 따라 증폭된다.RT-PCR은 사스-CoV-2 바이러스 게놈 검출에 널리 사용된다.[39]
- 백일해와 같은 질병은 보르데텔라 백일해균에 의해 발생한다.이 박테리아는 다양한 동물과 인간에게 영향을 미치는 심각한 급성 호흡기 감염으로 특징지어지며 많은 어린 아이들의 죽음으로 이어졌다.백일해독소는 단백질 엑소톡신으로서 두 개의 조광기에 의해 세포 수용체에 결합하고 세포 면역의 역할을 하는 T림프구와 같은 다른 세포 유형과 반응한다.[40]PCR은 백일해독에 대한 유전자 내의 염기서열을 검출할 수 있는 중요한 시험 툴이다.PCR은 독소에 대한 민감도가 높고 전환시간이 빨라 배양에 비해 백일해 진단에 매우 효율적이다.[41]
법의학 응용 프로그램
PCR 기반 유전자(또는 DNA) 지문 채취 프로토콜의 개발은 포렌식 분야에서 광범위하게 적용되고 있다.
- 그것의 가장 차별적인 형태에서, 유전자 지문 채취는 전 세계의 모든 인구로부터 어떤 한 사람이라도 차별할 수 있다.DNA의 미세한 샘플은 범죄현장에서 격리될 수 있으며, 용의자들로부터 또는 이전의 증거나 죄수의 DNA 데이타베이스로부터 비교될 수 있다.이러한 테스트의 간단한 버전은 범죄 수사 중에 용의자를 신속하게 배제하는 데 종종 사용된다.수십 년 된 범죄에서 나온 증거들은 원래 유죄 판결을 받은 사람들을 확인하거나 무죄로 판결할 수 있다.
- 법의학적 DNA 타이핑은 범죄현장에서 발견된 증거의 분석으로 범죄 용의자를 식별하거나 무죄를 선고하는 효과적인 방법이 되어 왔다.인간 게놈은 유전자 배열 내에서나 게놈의 부호화되지 않은 영역에서 발견될 수 있는 반복적인 영역이 많다.구체적으로, 인간 DNA의 최대 40%는 반복적이다.[5]게놈에는 이러한 반복적이고 부호화되지 않는 영역에 대한 두 가지 뚜렷한 범주가 있다.첫 번째 범주는 가변 번호 탠덤 반복(VNTR)으로 불리며, 10–100 기본 쌍 길이, 두 번째 범주는 짧은 탠덤 반복(STR)으로 불리며, 이러한 범주는 반복된 2-10 기본 쌍 섹션으로 구성된다.PCR은 각각의 반복 영역을 측면으로 하는 프라이머를 사용하여 잘 알려진 여러 VNTR과 STR을 증폭하는 데 사용된다.각 STR에 대해 어떤 개인으로부터 얻은 조각의 크기는 어떤 알레르기가 있는지 나타낼 것이다.개인에 대한 여러 STR을 분석함으로써, 각 개인에 대한 일련의 알레르기가 통계적으로 독특할 가능성이 있다는 것을 발견할 것이다.[5]연구원들은 인간 게놈의 전체 염기서열을 밝혀냈다.이 순서는 NCBI 웹사이트를 통해 쉽게 접근할 수 있으며 많은 실제 애플리케이션에서 사용된다.예를 들어, FBI는 신원확인에 사용되는 DNA 표식 사이트 세트를 작성했으며, 이를 연합 DNA 지수 시스템(CODIS) DNA 데이터베이스라고 부른다.[5]이 데이터베이스를 사용하면 DNA 샘플이 일치할 확률을 결정하기 위해 통계 분석을 사용할 수 있다.PCR은 포렌식 DNA 타이핑에 사용할 수 있는 매우 강력하고 중요한 분석 도구로, 연구자들은 분석을 위해 아주 적은 양의 표적 DNA만 필요로 하기 때문이다.예를 들어 모낭이 부착된 사람의 머리카락 한 올은 분석을 실시하기에 충분한 DNA를 가지고 있다.마찬가지로 정자 몇 개, 손톱 밑의 피부 샘플, 또는 소량의 혈액이 결정적인 분석을 위한 충분한 DNA를 제공할 수 있다.[5]
- 덜 차별적인 형태의 DNA 지문 감식은 한 개인이 가까운 친척들과 일치하는 DNA 친자확인 검사에서 도움이 될 수 있다.신원을 알 수 없는 인간 유골의 DNA를 검사할 수 있으며, 가능한 부모, 형제자매 또는 아이들의 DNA와 비교된다.입양된(또는 납치된) 아이의 친부모를 확인하기 위해 유사한 검사를 사용할 수 있다.신생아의 실제 생물학적 아버지도 확인할 수 있다(또는 배제할 수 있다).
- PCR AMGX/AMGY 설계는 극소량의 게놈에서 DNA 서열을 증폭시키는 데만[clarification needed] 도움이 되는 것이 아닌 것으로 밝혀졌다.그러나 법의학 뼈 샘플에서 실시간 성 판별에도 사용할 수 있다.이것은 법의학적 사례와 고대 표본에서 성별을 결정하는 강력하고 효과적인 방법을 제공한다.[42]
연구 응용 프로그램
PCR은 분자 유전학의 많은 연구 분야에 적용되어 왔다.
- PCR은 두 개의 프라이머의 염기서열 이상이 알려져 있지 않더라도 DNA의 짧은 조각들의 빠른 생산을 가능하게 한다.PCR의 이러한 능력은 Southern 또는 Northern Blot Hybridization을 위한 하이브리드화 프로브를 생성하는 것과 같은 많은 방법을 증가시킨다.PCR은 이러한 기법들을 대량의 순수 DNA로 공급하며, 때로는 하나의 가닥으로 제공함으로써 아주 적은 양의 시작 물질에서도 분석이 가능하다.
- DNA 염기서열 분석 작업은 PCR의 도움을 받을 수도 있다.알려진 DNA 조각은 유전병 돌연변이를 가진 환자로부터 쉽게 만들어질 수 있다.증폭 기법을 수정하면 전혀 알려지지 않은 게놈에서 세그먼트를 추출하거나 관심 영역의 한 가닥만 생성할 수 있다.
- PCR은 더 전통적인 DNA 복제 과정에 수많은 응용 프로그램을 가지고 있다.그것은 더 큰 게놈에서 벡터에 삽입하기 위한 세그먼트를 추출할 수 있는데, 이것은 소량으로만 구할 수 있을 것이다.벡터 프라이머 1세트를 이용해 벡터에 이미 삽입된 파편을 분석하거나 추출할 수도 있다.PCR 프로토콜의 일부 변경은 삽입된 파편의 돌연변이(일반 또는 사이트 지향)를 발생시킬 수 있다.
- 시퀀스 태그가 지정된 사이트는 PCR을 특정 클론에서 게놈의 특정 세그먼트가 존재한다는 지표로 사용하는 프로세스다.인간 게놈 프로젝트는 이 응용 프로그램이 그들이 염기서열 분석 중인 코스미드 클론을 매핑하고 다른 실험실의 결과를 조정하는데 필수적이라는 것을 발견했다.
- PCR의 적용은 네안데르탈인의 뼈, 매머드의 냉동 조직 또는 이집트 미라의 뇌에서 발견된 것과 같은 고대 출처에서 나온 DNA의 염기성 분석이다.[16]어떤 경우에는 이러한 출처에서 고도로 분해된 DNA가 증폭 초기 단계에서 재조립될 수 있다.
- PCR의 일반적인 적용은 유전자 발현 패턴에 대한 연구다.조직(또는 개별 세포까지)은 어떤 유전자가 활성화 되었는지, 혹은 어느 유전자가 꺼졌는지 다른 단계에서 분석할 수 있다.또한 이 애플리케이션은 실제 표현 수준을 정량화하기 위해 정량적 PCR을 사용할 수 있다.
- PCR이 개별 정자로부터[43] 여러 로키를 동시에 증폭시키는 능력은 감수분열 후 염색체 교차체를 연구함으로써 유전적 매핑이라는 보다 전통적인 과제를 크게 강화시켰다.수천 개의 개별 정자를 분석하여 매우 가까운 로키 사이의 드문 교차 이벤트를 직접 관찰했다.마찬가지로 수정, 발생 등의 길고 힘든 과정을 기다릴 필요 없이 비정상적인 삭제, 삽입, 변환 또는 뒤집기를 분석할 수 있다.
- 현장주도의 돌연변이 유발: PCR은 과학자들이 임의로 선택한 돌연변이를 가진 돌연변이 유전자를 만드는 데 사용될 수 있다.이러한 돌연변이는 단백질이 어떻게 기능을 수행하는지 이해하고 단백질 기능을 변화시키거나 향상시키기 위해 선택될 수 있다.
이점
PCR은 많은 장점을 가지고 있다.이해와 사용이 상당히 간단하며, 신속하게 결과를 산출한다.이 기술은 시퀀싱, 복제, 분석을 위해 특정 제품의 수백만에서 수십억 개의 복사본을 만들 수 있는 잠재력으로 매우 민감하다. qRT-PCR은 PCR과 동일한 이점을 공유하며, 합성 제품의 정량화라는 추가적인 이점이 있다.따라서, 그것은 종양, 미생물 또는 다른 질병 상태의 유전자 발현 수준의 변화를 분석하기 위해 사용된다.[27]
PCR은 매우 강력하고 실용적인 연구 수단이다.많은 질병의 알려지지 않은 이질들의 염기서열은 PCR에 의해 밝혀지고 있다.이 기술은 이미 알려진 바이러스와 관련된 이전에 알려지지 않은 바이러스의 순서를 식별하는 데 도움을 줄 수 있으며 따라서 질병 자체에 대한 이해를 높일 수 있다.절차를 더욱 간소화하고 민감한 비방사선 검출 시스템을 개발할 수 있다면 PCR은 향후 수년간 임상 실험실에서 중요한 위치를 차지할 것이다.[16]
제한 사항
PCR의 한 가지 주요 제한사항은 선택적 증폭을 허용하는 프라이머를 생성하기 위해 목표 수열에 대한 사전 정보가 필요하다는 것이다.[27]즉, 일반적으로 PCR 사용자는 DNA 중합효소가 프라이머-템플릿 하이브리드에 적절하게 결합되고 이후 DNA 합성 동안 전체 표적 부위를 생성하도록 보장하기 위해 두 개의 단일 가닥 템플릿 각각에서 대상 영역의 업스트림(들)을 정확히 알고 있어야 한다.
모든 효소와 마찬가지로 DNA 중합효소 역시 오류가 발생하기 쉬우며, 이는 결국 생성된 PCR 파편에서 돌연변이를 일으킨다.[44]
PCR의 또 다른 한계는 아주 적은 양의 오염 DNA라도 증폭되어 오도되거나 모호한 결과를 초래할 수 있다는 것이다.오염 가능성을 최소화하기 위해 조사관은 시약 준비, PCR, 제품 분석 등을 위해 별도의 방을 예약해야 한다.시약은 1회용 용액에 넣어야 한다.일회용 플런저와 장시간의 피펫 팁이 있는 피펫터를 정기적으로 사용해야 한다.[16]또한 실험실 설정이 단방향 워크플로우를 따르도록 하는 것이 좋다.PCR 및 분석실에 사용된 재료나 시약은 철저한 제독 없이 PCR 준비실에 반입해서는 안 된다.[45]
험산이 함유된 환경 샘플은 PCR 증폭을 억제하고 부정확한 결과를 초래할 수 있다.
변형
- 알레르 특정 PCR: 단일 뉴클레오티드 변이(SNV를 SNP와 혼동하지 않아야 함)에 기반한 진단 또는 복제 기술(환자의 단일 기저 차이)알레르기의 차이를 포함한 DNA 시퀀스에 대한 사전 지식이 필요하며, 3'의 끝이 SNV(일반적으로 SNV 주위에 기본 쌍 버퍼)를 포함하는 프라이머를 사용한다.엄격한 조건에서의 PCR 증폭은 템플릿과 프라이머 사이의 불일치가 있을 때 훨씬 덜 효율적이므로, SNP별 프라이머 신호로 연속적으로 특정 SNP가 존재할 때 성공적인 증폭을 한다.[46]자세한 내용은 SNP 유전자형을 참조하십시오.
- 조립 PCR 또는 중합효소 사이클 조립체(PCA): 짧은 중첩 세그먼트가 있는 긴 올리고뉴클레오티드의 풀에서 PCR을 수행하여 긴 DNA 시퀀스의 인공 합성.올리고뉴클레오티드는 감각과 반감지 방향을 번갈아 가며, 겹치는 세그먼트가 PCR 파편의 순서를 결정하여 최종 긴 DNA 제품을 선택적으로 생산한다.[47]
- 비대칭 PCR: 이중 가닥 DNA 템플릿에서 한 개의 DNA 가닥을 우선 증폭시킨다.두 개의 보완 가닥 중 하나만 증폭하면 되는 시퀀싱 및 하이브리드화 프로빙에 사용된다.PCR은 평상시처럼 수행되지만 증폭 대상인 스트랜드의 프라이머가 상당히 초과되어 수행된다.제한 프라이머를 다 사용한 후 반응 후 반응이 느리게(산술) 증폭되기 때문에 PCR의 추가 사이클이 필요하다.[48]Linear-After-The-Exponential-PCR(LAT-PCR)로 알려진 이 프로세스에 대한 최근 수정에서는 초과 프라이머보다 용해 온도(Tm)가 높은 제한 프라이머를 사용하여 제한 프라이머 농도가 중간반응을 감소시키므로 반응 효율을 유지한다.[49]
- 대류 PCR: PCR을 수행하는 의사 등온 방식.PCR 혼합물을 반복적으로 가열하고 냉각시키는 대신 용액은 열 경사로를 받는다.그 결과 열 불안정성 구동 대류 흐름은 PCR을 반복적으로 활성화할 수 있는 고온 및 저온 영역에서 PCR 시약을 자동으로 섞는다.[50]열경계 조건과 PCR 인클로저의 기하학적 구조와 같은 매개변수는 혼돈 유장의 출현을 활용함으로써 견고하고 빠른 PCR을 산출하도록 최적화될 수 있다.[51]그러한 대류 PCR 설정은 기기 전력 요구량과 작동 시간을 현저히 감소시킨다.
- 다이얼 아웃 PCR: 유전자 합성을 위한 정확한 DNA 분자를 검색하기 위한 매우 평행한 방법.DNA 분자의 복잡한 라이브러리는 대규모 병렬 배열 전에 고유한 측면 태그로 수정된다.그런 다음 태그 방향 프라이머는 PCR에 의해 원하는 시퀀스를 가진 분자의 검색을 가능하게 한다.[52]
- DPCR(digital PCR): DNA 샘플에서 표적 DNA 시퀀스의 양을 측정하는 데 사용된다.DNA 샘플은 많은 PCR을 병렬로 실행한 후 대상 DNA의 분자를 단 한 개도 받지 못할 정도로 희석성이 높다.목표 DNA 농도는 음성 결과의 비율을 사용하여 계산한다.그래서 '디지털 PCR'이라는 이름이 붙었다.
- 헬리코아제 의존 증폭: 기존 PCR과 유사하지만 변성 및 압연/확장 사이클을 거치지 않고 일정한 온도를 사용한다.DNA를 풀어주는 효소인 DNA 헬리코아제는 열변성 대신 사용된다.[53]
- Hot start PCR: PCR의 초기 설정 단계에서 비특정 증폭을 줄이는 기법이다.중합효소를 첨가하기 전에 반응 성분을 변성 온도(예: 95°C)로 가열하여 수동으로 수행할 수 있다.[54]항체의[13][55] 결합에 의해 또는 고온 활성화 단계 후에만 분리되는 공동 결합 억제제의 존재에 의해 주변 온도에서 중합효소의 활동을 억제하는 전문 효소 시스템이 개발되었다.핫 스타트/콜드 피니시 PCR은 주변 온도에서 비활성화되고 신장 온도에서 즉시 활성화되는 새로운 하이브리드 중합체로 달성된다.
- 실리코 PCR(디지털 PCR, 가상 PCR, 전자 PCR, e-PCR)에서는 서열화된 게놈이나 대본에서 나오는 DNA 서열을 증폭시키기 위해 주어진 프라이머(프로브)를 사용하여 이론적 중합효소 연쇄반응 결과를 계산하는 데 사용되는 계산 도구를 말한다.실리코 PCR은 분자생물학의 교육 도구로 제안되었다.[56]
- ISSR(Intersequence Specific PCR: ISSR): 단순한 시퀀스 사이의 영역을 증폭시키는 DNA 지문 채취용 PCR 방법이 반복되어 증폭된 파편 길이의 고유한 지문을 생성한다.[57]
- Inverse PCR: 게놈 인서트 주위의 플랭킹 시퀀스를 식별하는 데 일반적으로 사용된다.그것은 일련의 DNA 소화와 자기 감시를 포함하며, 그 결과 알 수 없는 염기서열의 양쪽 끝에서 알려진 염기서열을 발생시킨다.[58]
- Large-mediated PCR: 관심의 DNA에 묶인 작은 DNA 링커와 DNA 링커에 연결된 여러 개의 프라이머를 사용한다; 그것은 DNA 염기서열 분석, 게놈 산책, DNA 발자국을 만드는 데 사용되어 왔다.[59]
- 메틸화 특화 PCR(MSP): Stephen Baylin과 James G에 의해 개발되었다. 존스홉킨스 의과대학의 헤르만이며,[60] 게놈 DNA에서 CpG섬의 메틸화를 검출하는 데 사용된다.DNA는 먼저 비메틸화 시토신 염기지를 요라실로 변환하는 비황산나트륨으로 처리되는데, 이는 PCR 프라이머에 의해 티민으로 인식된다.그런 다음 수정된 DNA에 대해 두 개의 PCR이 수행되며, 프라이머 시퀀스 내의 CpG 섬을 제외하고 동일한 프라이머 세트를 사용한다.이러한 시점에서 1개의 프라이머 세트는 시토신(cytosine)으로 DNA를 인식하여 메틸화 DNA를 증폭시키고, 1세트는 우라실이나 티민으로 DNA를 인식하여 미메틸화 DNA를 증폭시킨다.qPCR을 이용한 MSP도 메틸화에 대한 질적 정보가 아닌 정량적 정보를 얻기 위해 수행될 수 있다.
- 미니프리머 PCR: 9~10개의 뉴클레오티드가 짧은 짧은 프라이머("소형 프리머")에서 확장 가능한 자동온도조절 고분자효소(S-Tbr)를 사용한다.이 방법은 더 작은 프라이머 결합 영역을 대상으로 하는 PCR을 허용하며, 16S(또는 진핵종 18S) rRNA 유전자와 같이 보존된 DNA 서열을 증폭시키는 데 사용된다.[61]
- 멀티플렉스 레그 의존형 프로브 증폭(MLPA): 단일 프라이머 쌍으로 다중 대상을 증폭할 수 있으므로 멀티플렉스 PCR의 분해능 한계를 피할 수 있다(아래 참조).
- 멀티플렉스-PCR: 단일 PCR 혼합물 내에 여러 개의 프라이머 세트로 구성되어 서로 다른 DNA 시퀀스에 특정한 다양한 크기의 앰프를 생성한다.한 번에 여러 유전자를 대상으로 지정함으로써, 그렇지 않으면 시약의 몇 배와 더 많은 시간을 필요로 하는 한 번의 테스트 실행으로 추가 정보를 얻을 수 있다.각 프라이머 세트에 대한 어닐링 온도는 단일 반응 및 앰프리콘 크기 내에서 올바르게 작동하도록 최적화되어야 한다.즉, 그들의 기본 쌍 길이는 젤 전기영동으로 시각화할 때 구별되는 띠를 형성할 수 있을 정도로 충분히 달라야 한다.
- 나노입자-지원 PCR(나노PCR): 일부 나노입자(NanoParticle)는 PCR(즉, 나노PCR)의 효율을 높일 수 있고, 심지어 일부 나노입자는 원래의 PCR Enhancer를 능가할 수도 있다.양자점(QD)이 PCR의 특수성과 효율을 향상시킬 수 있다고 보고되었다.단일벽 탄소나노튜브(SWCNT)와 다중벽 탄소나노튜브(MWCNT)는 긴 PCR의 증폭을 강화하는데 효율적이다.탄소나노파우더(CNP)는 반복 PCR과 긴 PCR의 효율을 향상시킬 수 있으며, 산화아연, 이산화티타늄, Ag NP는 PCR 수율을 높이는 것으로 조사되었다.이전 데이터는 비금속 NP가 허용 가능한 증폭 충실도를 유지했음을 나타냈다.많은 NP가 PCR 효율을 높일 수 있다는 점을 감안하면 나노PCR 기술 개선과 제품 개발의 잠재력이 클 것은 분명하다.[62][63]
- 내포 PCR: DNA의 비특정 증폭으로 인한 배경을 줄임으로써 DNA 증폭의 특수성을 높인다.두 세트의 프라이머가 두 개의 연속 PCR에 사용된다.첫 번째 반응에서, DNA 생성에 한 쌍의 프라이머가 사용되는데, 의도된 목표물 외에, 여전히 비특이적으로 증폭된 DNA 조각으로 구성될 수 있다.그런 다음 제품은 첫 번째 반응에 사용된 각 프라이머의 3' 위치에 완전히 또는 부분적으로 결합 부위가 다른 프라이머 세트가 있는 두 번째 PCR에 사용된다.내포된 PCR은 흔히 기존의 PCR보다 긴 DNA 파편을 구체적으로 증폭시키는 데 더 성공하지만, 대상 시퀀스에 대한 보다 상세한 지식이 필요하다.
- 중복 확장 PCR 또는 중복 확장에 의한 스플리싱(SOEing) : 보완적 시퀀스를 포함하는 두 개 이상의 DNA 파편을 결합하는 데 사용되는 유전공학 기법이다.그것은 유전자, 규제 순서 또는 돌연변이를 포함하는 DNA 조각들과 결합하는데 사용된다; 이 기술은 구체적이고 긴 DNA 구조를 만들 수 있다.또한 삭제, 삽입 또는 점 돌연변이를 DNA 시퀀스에 도입할 수 있다.[64][65]
- PAN-AC: 등온 상태를 증폭에 사용하며, 살아있는 세포에 사용할 수 있다.[66][67]
- 정량적 PCR(qPCR): 목표 시퀀스의 수량(일반적으로 실시간) 측정에 사용된다.DNA, cDNA 또는 RNA의 시작 양을 정량적으로 측정한다. 정량적 PCR은 일반적으로 DNA 서열이 샘플에 있는지 여부와 샘플에 있는 복사본의 수를 결정하는 데 사용된다.정량적 PCR은 정밀도가 매우 높다.정량적 PCR 방법은 Sybr Green, EvaGreen 등의 형광 염료나 TaqMan과 같은 불소성분 함유 DNA 탐침을 사용하여 실시간으로 증폭된 제품의 양을 측정한다.또한 RT-PCR(실시간 PCR)로 약칭되기도 하지만 이 약어는 역전사 PCR에만 사용해야 한다. qPCR은 정량적 PCR(실시간 PCR)에 적절한 수축이다.
- Reverse Component PCR(RC-PCR): 하나의 폐쇄 튜브 반응으로 생성된 앰플리콘의 한쪽 끝에 기능 도메인이나 선택 순서를 독립적으로 추가할 수 있다.이 방법은 범용 프라이머(첨부할 대상 시퀀스 또는 도메인을 포함)와 RC 프로브의 상호작용에 의한 반응 내에서 대상 특정 프라이머를 생성한다.
- 역전사 PCR(RT-PCR): RNA로부터 DNA를 증폭시키기 위한 역전사효소 역전사 RNA를 cDNA로 증폭시킨 다음 PCR에 의해 증폭된다.RT-PCR은 표현 프로파일링, 유전자 발현을 결정하거나 전사 시작 및 종료 부지를 포함한 RNA 대본의 순서를 식별하기 위해 널리 사용된다.유전자의 유전체 DNA 서열이 알려지면 RT-PCR을 이용해 유전자 내 엑손과 인트론의 위치를 지도화할 수 있다.유전자의 5' 끝(전사 시작 부지에 대응)은 일반적으로 RACE-PCR(cDNA Ends의 신속한 증폭)로 식별된다.
- RNase H 의존 PCR(rhPCR): 온도 조절식 RNase HII 효소로 제거할 수 있는 3' 확장 블록의 프라이머를 활용하는 PCR의 수정.이 시스템은 프라이머-다이머를 감소시키고 더 많은 프라이머로 멀티플렉스 반응을 수행할 수 있도록 한다.[68]
- 단일 특정 프라이머-PCR(SSP-PCR): 시퀀스 정보가 한쪽 끝에서만 사용 가능한 경우에도 이중 가닥 DNA를 증폭할 수 있다.이 방법은 부분 시퀀스 정보만 가능한 유전자의 증폭을 허용하고, 알려진 염색체 미지의 영역까지 단방향 게놈을 걸어갈 수 있게 한다.[69]
- 솔리드 단계 PCR:, Polony 증폭(어디에 PCR은 식민지가 겔 매트릭스에서는, 예를 들어 도출된다), 브릿지 PCR[70](프라이머 공유는 solid-support 표면에 연관되어 있), 전통적인 솔리드 단계 PCR(어디 비대칭 PCR은 확실한 지지 순서 일치로 입문서 베어링의 존재에 적용된다 등 다양한 의미를 포함한다. 한수용성 프라이머) 및 개선된 솔리드 페이즈 PCR[71](고체 지지 프라이머에 열적 '단계'를 선택적으로 적용하여 기존의 솔리드 페이즈 PCR을 개선할 수 있는 경우).
- 자살 PCR: 일반적으로 잘못된 긍정을 피하고 증폭된 파편의 특수성을 보장하는 고생물학이나 기타 연구에 사용된다.원래는 중세 흑사병 유행 때 페스트에 의해 사망한 것으로 추정되는 사람들의 14세기 무덤에서 얻은 치아 표본에 미생물 예르시니아 페스티스의 존재를 검증하기 위한 연구에서 설명되었다.[72]이 방법은 PCR("자살"이라는 용어를 정의함)에 한 번만 프라이머 조합을 사용하도록 규정하는데, 프라이머는 양성 대조군 PCR 반응에 절대 사용해서는 안 되며 프라이머는 항상 이것 또는 다른 프라이머 세트를 사용하여 연구실에서 이전에 증폭된 적이 없는 게놈 영역을 대상으로 해야 한다.이를 통해 이전의 PCR 반응으로 인한 오염 DNA가 연구소에 존재하지 않도록 보장하고 그렇지 않으면 잘못된 긍정을 발생시킬 수 있다.
- 열 비대칭 인터레이스 PCR(TAIL-PCR): 알려진 시퀀스를 기준으로 알 수 없는 시퀀스의 격리를 위한 것이다.알려진 시퀀스 내에서 TAIL-PCR은 어닐링 온도가 다른 중첩된 쌍의 프라이머를 사용한다. 퇴화 프라이머는 알 수 없는 시퀀스로부터 다른 방향으로 증폭하기 위해 사용된다.[73]
- 터치다운 PCR(Step-down PCR): PCR 사이클이 진행됨에 따라 어닐링 온도를 점진적으로 낮춰 비특정 배경을 줄이는 것을 목표로 하는 PCR의 변종.초기 사이클에서 어닐링 온도는 일반적으로 사용되는 프라이머의 T 위로m 몇 도(3~5 °C) 올라가지만, 이후 사이클에서는 프라이머 T보다m 몇 도(3~5 °C) 낮는다.온도가 높을수록 프라이머 결합에 대한 특수성이 커지며, 온도가 낮을수록 초기 사이클 동안 형성된 특정 제품에서 보다 효율적인 증폭이 가능하다.[74]
- 범용 빠른 걸음걸이: 애벌레 구조 형성과 관련된 메커니즘으로 인해 기존의 '일방' 접근법보다 더 구체적인 '양면' PCR을 사용한 게놈 보행과 유전자 지문 채취(유전자 특정 프라이머와 일반 프라이머 1개만 사용—실제 '소음'[75]으로 이어질 수 있음)UFW의 간소화된 파생상품은 LaNe REGE(유전자 DNA 단부의 신속한 확대를 위한 Lariat-dependent 중첩 PCR),[76] 5'RACE LaNe[77] 및 3'RACE LaNe이다.[78]
역사
중합효소 연쇄반응의 핵심 성분인 내열 효소는 옐로우스톤 버섯샘의 과열수역에 살았던 미생물 형태의 산물로 1960년대에 발견되었다.[79]
Kjell Kleppe와 H. Gobind Khorana의 연구실의 동료들이 1971년 발표한 논문에서는 시험관내 프라이머로 짧은 DNA 템플릿을 복제하기 위해 효소 분석법을 사용하는 방법을 처음으로 설명했다.[80]그러나 이러한 기본적인 PCR 원리의 초기 발현에는 당시에는 큰 주목을 받지 못했고 1983년 중합효소 연쇄반응의 발명은 일반적으로 카리 뮬리스에게 기인한다.[81][page needed]
1983년 뮬리스가 PCR을 개발했을 때, 그는 최초의 생명공학 회사들 중 하나인 Cetus Corporation에서 캘리포니아 에머리빌에서 일하면서 DNA의 짧은 사슬을 합성하는 일을 맡고 있었다.뮬리스는 어느날 밤 자신의 차를 타고 퍼시픽 코스트 하이웨이를 순항하면서 PCR에 대한 아이디어를 생각해냈다고 썼다.[82]그는 DNA의 변화(교정)를 분석하는 새로운 방법으로 마음 속에서 놀고 있었는데, 그 대신 DNA 중합효소에 의해 구동되는 반복적인 중복을 통해 어떤 DNA 영역이라도 증폭시키는 방법을 발명했다는 것을 깨달았다.뮬리스는 사이언티픽 아메리칸에서 "유전자 물질 DNA의 단일 분자로 시작함으로써 PCR은 오후 한 시간 안에 1,000억 개의 유사한 분자를 생성할 수 있다.그 반응은 실행하기 쉽다.검사용 튜브와 간단한 시약 몇 개, 열원만 있으면 된다."[83]DNA 지문 채취는 1988년 처음 친자확인검사에 쓰였다.[84]
Mullis는 LSD를 자신의 PCR 개발에 필수적인 것으로 사용했다고 인정했다: "만약 LSD를 복용하지 않았다면 내가 PCR을 발명했을까?나는 그게 진심으로 의심된다.DNA 분자 위에 앉아서 중합체가 지나가는 걸 지켜볼 수 있어그런 사실은 환각제(sychedelic drugs)에서 어느 정도 배웠다고 말했다.[85]
뮬리스와 생화학자인 마이클 스미스는 1993년 노벨 화학상을 공동 수상했는데, 이는 뮬리스와 그의 동료들이 그의 제안을 처음으로 실행에 옮긴 지 7년 만이다.[1][86]Mullis's 1985 paper with R. K. Saiki and H. A. Erlich, "Enzymatic Amplification of β-globin Genomic Sequences and Restriction Site Analysis for Diagnosis of Sickle Cell Anemia"—the polymerase chain reaction invention (PCR)—was honored by a Citation for Chemical Breakthrough Award from the Division of History of Chemistry of the American Chemical S2017년.[87][2]
PCR 방법의 핵심은 각 복제 주기 후 DNA 이중나선 내 두 DNA 가닥의 분리에 필요한 90°C(194°F) 이상의 고온을 견딜 수 있는 적절한 DNA 중합효소를 사용하는 것이다.PCR을 전제로 한 체외 실험에 처음 사용된 DNA 중합체는 이러한 고온을 견딜 수 없었다.[2]그래서 DNA 복제를 위한 초기 절차는 매우 비효율적이고 시간이 많이 소요되었으며, 그 과정 내내 많은 양의 DNA 중합효소와 지속적인 처리가 필요했다.
1976년 온천 등 고온(50~80°C(122~176°F) 환경에서[14] 자연적으로 서식하는 보온균 테르무스 아쿠아쿠스로부터 정화된 DNA 중합효소인 Taq 중합효소(DNA polyerase)의 발견은 PCR 방식을 획기적으로 개선할 수 있는 길을 열었다.T. 아쿠아틱투스로부터 격리된 DNA 중합효소는 DNA 변성 후에도 활성 상태를 유지하는 고온에서 안정적이기 때문에 [15]각 사이클 후에 새로운 DNA 중합효소를 첨가할 필요가 없다.[3]이것은 DNA 증폭을 위한 자동 열경화기 기반 과정을 가능하게 했다.
특허분쟁
PCR 기법은 캐리 멀리스가 특허를 받아 세투스 코퍼레이션에 배정했는데, 멀리스가 1983년 이 기법을 발명할 때 그곳에서 일했다.Taq 중합효소 효소도 특허에 의해 보호되었다.듀폰이 제기한 소송이 불발되는 등 이 기법과 관련된 몇 차례의 고공 소송이 있었다.스위스의 제약회사 호프만-라 로슈는 1992년에 특허권을 구입했다.상용 PCR 특허 중 마지막은 2017년에 만료되었다.[88]
Taq 중합효소 효소를 둘러싼 관련 특허 싸움은 로슈와 프로메가 사이의 세계 여러 관할 지역에서 여전히 진행 중이다.이 법적 논쟁은 2005년 3월 28일에 만료된 원래의 PCR과 Taq 중합효소 특허의 수명을 넘어 확장되었다.[89]
참고 항목
참조
- ^ a b "The Nobel Prize in Chemistry 1993". NobelPrize.org.
- ^ a b c d Saiki RK, Scharf S, Faloona F, Mullis KB, Horn GT, Erlich HA, Arnheim N (December 1985). "Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia". Science. 230 (4732): 1350–54. Bibcode:1985Sci...230.1350S. doi:10.1126/science.2999980. PMID 2999980.
- ^ a b Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, et al. (January 1988). "Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase". Science. 239 (4839): 487–91. Bibcode:1988Sci...239..487S. doi:10.1126/science.239.4839.487. PMID 2448875.
- ^ Enners, Edward; Porta, Angela R. (2012). "Determining Annealing Temperatures for Polymerase Chain Reaction". The American Biology Teacher. 74 (4): 256–60. doi:10.1525/abt.2012.74.4.9. S2CID 86708426.
- ^ a b c d e f Ninfa, Alexander; Ballou, David; Benore, Marilee (2009). Fundamental Laboratory Approaches for Biochemistry and Biotechnology. United States: Wiley. pp. 408–10. ISBN 978-0-470-08766-4.
- ^ Cheng S, Fockler C, Barnes WM, Higuchi R (June 1994). "Effective amplification of long targets from cloned inserts and human genomic DNA". Proceedings of the National Academy of Sciences of the United States of America. 91 (12): 5695–99. Bibcode:1994PNAS...91.5695C. doi:10.1073/pnas.91.12.5695. PMC 44063. PMID 8202550.
- ^ Carr AC, Moore SD (2012). Lucia A (ed.). "Robust quantification of polymerase chain reactions using global fitting". PLOS ONE. 7 (5): e37640. Bibcode:2012PLoSO...737640C. doi:10.1371/journal.pone.0037640. PMC 3365123. PMID 22701526.
- ^ a b Joseph Sambrook & David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 978-0-879-69576-7. 8장: 중합효소 연쇄반응에 의한 DNA의 체외 증폭
- ^ "Polymerase Chain Reaction (PCR)". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "PCR". Genetic Science Learning Center, University of Utah.
- ^ Pavlov AR, Pavlova NV, Kozyavkin SA, Slesarev AI (May 2004). "Recent developments in the optimization of thermostable DNA polymerases for efficient applications". Trends in Biotechnology. 22 (5): 253–60. doi:10.1016/j.tibtech.2004.02.011. PMID 15109812.
- ^ Rychlik W, Spencer WJ, Rhoads RE (November 1990). "Optimization of the annealing temperature for DNA amplification in vitro". Nucleic Acids Research. 18 (21): 6409–12. doi:10.1093/nar/18.21.6409. PMC 332522. PMID 2243783.
- ^ a b Sharkey DJ, Scalice ER, Christy KG, Atwood SM, Daiss JL (May 1994). "Antibodies as thermolabile switches: high temperature triggering for the polymerase chain reaction". Bio/Technology. 12 (5): 506–09. doi:10.1038/nbt0594-506. PMID 7764710. S2CID 2885453.
- ^ a b Chien A, Edgar DB, Trela JM (September 1976). "Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus". Journal of Bacteriology. 127 (3): 1550–57. doi:10.1128/jb.127.3.1550-1557.1976. PMC 232952. PMID 8432.
- ^ a b Lawyer FC, Stoffel S, Saiki RK, Chang SY, Landre PA, Abramson RD, Gelfand DH (May 1993). "High-level expression, purification, and enzymatic characterization of full-length Thermus aquaticus DNA polymerase and a truncated form deficient in 5' to 3' exonuclease activity". PCR Methods and Applications. 2 (4): 275–87. doi:10.1101/gr.2.4.275. PMID 8324500.
- ^ a b c d Schochetman G, Ou CY, Jones WK (December 1988). "Polymerase chain reaction". The Journal of Infectious Diseases. 158 (6): 1154–57. doi:10.1093/infdis/158.6.1154. JSTOR 30137034. PMID 2461996.
- ^ Borman, Jon; Schuster, David; Li, Wu-bo; Jessee, Joel; Rashtchian, Ayoub (2000). "PCR from problematic templates" (PDF). Focus. 22 (1): 10. Archived from the original (PDF) on 7 March 2017.
- ^ Bogetto, Prachi; Waidne, Lisa; Anderson, Holly (2000). "Helpful tips for PCR" (PDF). Focus. 22 (1): 12. Archived from the original (PDF) on 7 March 2017.
- ^ Biolabs, New England. "Q5® High-Fidelity DNA Polymerase NEB". www.neb.com. Retrieved 4 December 2021.
- ^ Sze, Marc A.; Schloss, Patrick D. (2019). "The Impact of DNA Polymerase and Number of Rounds of Amplification in PCR on 16S rRNA Gene Sequence Data". mSphere. 4 (3): e00163–19. doi:10.1128/mSphere.00163-19. PMC 6531881. PMID 31118299.
- ^ Sarkar G, Kapelner S, Sommer SS (December 1990). "Formamide can dramatically improve the specificity of PCR". Nucleic Acids Research. 18 (24): 7465. doi:10.1093/nar/18.24.7465. PMC 332902. PMID 2259646.
- ^ "Electronic PCR". NCBI – National Center for Biotechnology Information. Retrieved 13 March 2012.
- ^ Pavlov AR, Pavlova NV, Kozyavkin SA, Slesarev AI (2006). "Thermostable DNA Polymerases for a Wide Spectrum of Applications: Comparison of a Robust Hybrid TopoTaq to other enzymes". In Kieleczawa J (ed.). DNA Sequencing II: Optimizing Preparation and Cleanup. Jones & Bartlett. pp. 241–57. ISBN 978-0-7637-3383-4.
- ^ Pombert JF, Sistek V, Boissinot M, Frenette M (October 2009). "Evolutionary relationships among salivarius streptococci as inferred from multilocus phylogenies based on 16S rRNA-encoding, recA, secA, and secY gene sequences". BMC Microbiology. 9: 232. doi:10.1186/1471-2180-9-232. PMC 2777182. PMID 19878555.
- ^ "Chemical Synthesis, Sequencing, and Amplification of DNA (class notes on MBB/BIO 343)". Arizona State University. Archived from the original on 9 October 1997. Retrieved 29 October 2007.
- ^ Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, et al. (April 2009). "The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments" (PDF). Clinical Chemistry. 55 (4): 611–22. doi:10.1373/clinchem.2008.112797. PMID 19246619.
- ^ a b c d Garibyan L, Avashia N (March 2013). "Polymerase chain reaction". The Journal of Investigative Dermatology. 133 (3): 1–4. doi:10.1038/jid.2013.1. PMC 4102308. PMID 23399825.
- ^ Schnell, S.; Mendoza, C. (October 1997). "Theoretical Description of the Polymerase Chain Reaction". Journal of Theoretical Biology. 188 (3): 313–18. Bibcode:1997JThBi.188..313S. doi:10.1006/jtbi.1997.0473. PMID 9344735.
- ^ Schnell, S.; Mendoza, C. (21 February 1997). "Enzymological Considerations for the Theoretical Description of the Quantitative Competitive Polymerase Chain Reaction (QC-PCR)". Journal of Theoretical Biology. 184 (4): 433–40. Bibcode:1997JThBi.184..433S. doi:10.1006/jtbi.1996.0283. ISSN 0022-5193. PMID 9082073.
- ^ Becker, Sven; Böger, Peter; Oehlmann, Ralfh; Ernst, Anneliese (1 November 2000). "PCR Bias in Ecological Analysis: a Case Study for Quantitative Taq Nuclease Assays in Analyses of Microbial Communities". Applied and Environmental Microbiology. 66 (11): 4945–53. Bibcode:2000ApEnM..66.4945B. doi:10.1128/AEM.66.11.4945-4953.2000. ISSN 1098-5336. PMC 92404. PMID 11055948.
- ^ Solomon, Anthony W.; Peeling, Rosanna W.; Foster, Allen; Mabey, David C. W. (1 October 2004). "Diagnosis and Assessment of Trachoma". Clinical Microbiology Reviews. 17 (4): 982–1011. doi:10.1128/CMR.17.4.982-1011.2004. ISSN 0893-8512. PMC 523557. PMID 15489358.
- ^ Ramzy, Reda M.R. (April 2002). "Recent advances in molecular diagnostic techniques for human lymphatic filariasis and their use in epidemiological research". Transactions of the Royal Society of Tropical Medicine and Hygiene. 96: S225–29. doi:10.1016/S0035-9203(02)90080-5. PMID 12055843.
- ^ Sachse, Konrad (2003). "Specificity and performance of diagnostic PCR assays". In Sachse, Konrad; Frey, Joachim (eds.). PCR Detection of Microbial Pathogens. Methods in Molecular Biology. Vol. 216. Totowa, New Jersey: Humana Press. pp. 3–29. doi:10.1385/1-59259-344-5:03. ISBN 978-1-59259-344-6. PMID 12512353.
{{cite book}}:누락 또는 비어 있음title=(도움말) - ^ Quill E (March 2008). "Medicine. Blood-matching goes genetic". Science. 319 (5869): 1478–79. doi:10.1126/science.319.5869.1478. PMID 18339916. S2CID 36945291.
- ^ Tomar, Rukam (2010). Molecular Markers and Plant Biotechnology. Pitman Pura, New Delhi: New India Publishing Agency. p. 188. ISBN 978-93-80235-25-7.
- ^ a b c Cai HY, Caswell JL, Prescott JF (March 2014). "Nonculture molecular techniques for diagnosis of bacterial disease in animals: a diagnostic laboratory perspective". Veterinary Pathology. 51 (2): 341–50. doi:10.1177/0300985813511132. PMID 24569613.
- ^ Salis AD (2009). "Applications in Clinical Microbiology". Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.
- ^ Kwok S, Mack DH, Mullis KB, Poiesz B, Ehrlich G, Blair D, et al. (May 1987). "Identification of human immunodeficiency virus sequences by using in vitro enzymatic amplification and oligomer cleavage detection". Journal of Virology. 61 (5): 1690–94. doi:10.1128/jvi.61.5.1690-1694.1987. PMC 254157. PMID 2437321.
- ^ "Coronavirus: il viaggio dei test". Istituto Superiore di Sanità.
- ^ Finger, Horst; von Koenig, Carl Heinz Wirsing (1996). Baron, Samuel (ed.). Medical Microbiology (4th ed.). Galveston, TX: University of Texas Medical Branch at Galveston. ISBN 978-0-9631172-1-2. PMID 21413270.
- ^ Yeh, Sylvia H.; Mink, ChrisAnna M. (2012). "Bordetella pertussis and Pertussis (Whooping Cough)". Netter's Infectious Diseases. Netter's Infectious Diseases. pp. 11–14. doi:10.1016/B978-1-4377-0126-5.00003-3. ISBN 978-1-4377-0126-5.
- ^ Alonso A, Martín P, Albarrán C, García P, García O, de Simón LF, et al. (January 2004). "Real-Time PCR Designs to Estimate Nuclear and Mitochondrial DNA Copy Number in Forensic and Ancient DNA Studies". Forensic Science International. 139 (2–3): 141–49. doi:10.1016/j.forsciint.2003.10.008. PMID 15040907.
- ^ Boehnke M, Arnheim N, Li H, Collins FS (July 1989). "Fine-structure genetic mapping of human chromosomes using the polymerase chain reaction on single sperm: experimental design considerations". American Journal of Human Genetics. 45 (1): 21–32. PMC 1683385. PMID 2568090.
- ^ Zhou YH, Zhang XP, Ebright RH (November 1991). "Random mutagenesis of gene-sized DNA molecules by use of PCR with Taq DNA polymerase". Nucleic Acids Research. 19 (21): 6052. doi:10.1093/nar/19.21.6052. PMC 329070. PMID 1658751.
- ^ Stursberg, Stephanie (2021). "Setting up a PCR lab from scratch". INTEGRA Biosciences.
{{cite web}}: CS1 maint : url-status (링크) - ^ Newton CR, Graham A, Heptinstall LE, Powell SJ, Summers C, Kalsheker N, et al. (April 1989). "Analysis of any point mutation in DNA. The amplification refractory mutation system (ARMS)". Nucleic Acids Research. 17 (7): 2503–16. doi:10.1093/nar/17.7.2503. PMC 317639. PMID 2785681.
- ^ Stemmer WP, Crameri A, Ha KD, Brennan TM, Heyneker HL (October 1995). "Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides". Gene. 164 (1): 49–53. doi:10.1016/0378-1119(95)00511-4. PMID 7590320.
- ^ Innis MA, Myambo KB, Gelfand DH, Brow MA (December 1988). "DNA sequencing with Thermus aquaticus DNA polymerase and direct sequencing of polymerase chain reaction-amplified DNA". Proceedings of the National Academy of Sciences of the United States of America. 85 (24): 9436–40. Bibcode:1988PNAS...85.9436I. doi:10.1073/pnas.85.24.9436. PMC 282767. PMID 3200828.
- ^ Pierce KE, Wangh LJ (2007). Linear-after-the-exponential polymerase chain reaction and allied technologies Real-time detection strategies for rapid, reliable diagnosis from single cells. Methods in Molecular Medicine. Vol. 132. pp. 65–85. doi:10.1007/978-1-59745-298-4_7. ISBN 978-1-58829-578-1. PMID 17876077.
- ^ Krishnan M, Ugaz VM, Burns MA (October 2002). "PCR in a Rayleigh-Bénard convection cell". Science. 298 (5594): 793. doi:10.1126/science.298.5594.793. PMID 12399582.
- ^ Priye A, Hassan YA, Ugaz VM (November 2013). "Microscale chaotic advection enables robust convective DNA replication". Analytical Chemistry. 85 (21): 10536–41. doi:10.1021/ac402611s. PMID 24083802.
- ^ Schwartz JJ, Lee C, Shendure J (September 2012). "Accurate gene synthesis with tag-directed retrieval of sequence-verified DNA molecules". Nature Methods. 9 (9): 913–15. doi:10.1038/nmeth.2137. PMC 3433648. PMID 22886093.
- ^ Vincent M, Xu Y, Kong H (August 2004). "Helicase-dependent isothermal DNA amplification". EMBO Reports. 5 (8): 795–800. doi:10.1038/sj.embor.7400200. PMC 1249482. PMID 15247927.
- ^ Chou Q, Russell M, Birch DE, Raymond J, Bloch W (April 1992). "Prevention of pre-PCR mis-priming and primer dimerization improves low-copy-number amplifications". Nucleic Acids Research. 20 (7): 1717–23. doi:10.1093/nar/20.7.1717. PMC 312262. PMID 1579465.
- ^ Kellogg DE, Rybalkin I, Chen S, Mukhamedova N, Vlasik T, Siebert PD, Chenchik A (June 1994). "TaqStart Antibody: "hot start" PCR facilitated by a neutralizing monoclonal antibody directed against Taq DNA polymerase". BioTechniques. 16 (6): 1134–37. PMID 8074881.
- ^ San Millán RM, Martínez-Ballesteros I, Rementeria A, Garaizar J, Bikandi J (December 2013). "Online exercise for the design and simulation of PCR and PCR-RFLP experiments". BMC Research Notes. 6: 513. doi:10.1186/1756-0500-6-513. PMC 4029544. PMID 24314313.
- ^ Zietkiewicz E, Rafalski A, Labuda D (March 1994). "Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification". Genomics. 20 (2): 176–83. doi:10.1006/geno.1994.1151. PMID 8020964.
- ^ Ochman H, Gerber AS, Hartl DL (November 1988). "Genetic applications of an inverse polymerase chain reaction". Genetics. 120 (3): 621–23. doi:10.1093/genetics/120.3.621. PMC 1203539. PMID 2852134.
- ^ Mueller PR, Wold B (November 1989). "In vivo footprinting of a muscle specific enhancer by ligation mediated PCR". Science. 246 (4931): 780–86. Bibcode:1989Sci...246..780M. doi:10.1126/science.2814500. PMID 2814500.
- ^ Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB (September 1996). "Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands". Proceedings of the National Academy of Sciences of the United States of America. 93 (18): 9821–26. Bibcode:1996PNAS...93.9821H. doi:10.1073/pnas.93.18.9821. PMC 38513. PMID 8790415.
- ^ Isenbarger TA, Finney M, Ríos-Velázquez C, Handelsman J, Ruvkun G (February 2008). "Miniprimer PCR, a new lens for viewing the microbial world". Applied and Environmental Microbiology. 74 (3): 840–49. Bibcode:2008ApEnM..74..840I. doi:10.1128/AEM.01933-07. PMC 2227730. PMID 18083877.
- ^ Shen C, Yang W, Ji Q, Maki H, Dong A, Zhang Z (November 2009). "NanoPCR observation: different levels of DNA replication fidelity in nanoparticle-enhanced polymerase chain reactions". Nanotechnology. 20 (45): 455103. Bibcode:2009Nanot..20S5103S. doi:10.1088/0957-4484/20/45/455103. PMID 19822925. S2CID 3393115.
- ^ Shen, Cenchao (2013). "An Overview of Nanoparticle-Assisted Polymerase Chain Reaction Technology". An Overview of Nanoparticle‐Assisted Polymerase Chain Reaction Technology. US: Wiley-Blackwell Publishing Ltd. pp. 97–106. doi:10.1002/9781118451915.ch5. ISBN 978-1-118-45191-5.
- ^ Horton RM, Hunt HD, Ho SN, Pullen JK, Pease LR (April 1989). "Engineering hybrid genes without the use of restriction enzymes: gene splicing by overlap extension". Gene. 77 (1): 61–68. doi:10.1016/0378-1119(89)90359-4. PMID 2744488.
- ^ Moller, Simon (2006). PCR: The Basics. US: Taylor & Francis Group. p. 144. ISBN 978-0-415-35547-6.
- ^ David F, Turlotte E (November 1998). "[A method of isothermal gene amplification]" [An Isothermal Amplification Method]. Comptes Rendus de l'Académie des Sciences, Série III. 321 (11): 909–14. Bibcode:1998CRASG.321..909D. doi:10.1016/S0764-4469(99)80005-5. PMID 9879470.
- ^ Fabrice David (September–October 2002). "Utiliser les propriétés topologiques de l'ADN: une nouvelle arme contre les agents pathogènes" (PDF). Fusion. Archived from the original (PDF) on 28 November 2007.(프랑스어로)
- ^ Dobosy JR, Rose SD, Beltz KR, Rupp SM, Powers KM, Behlke MA, Walder JA (August 2011). "RNase H-dependent PCR (rhPCR): improved specificity and single nucleotide polymorphism detection using blocked cleavable primers". BMC Biotechnology. 11: 80. doi:10.1186/1472-6750-11-80. PMC 3224242. PMID 21831278.
- ^ Shyamala, V.; Ferro-Luzzi, Ames G. (1993). Single Specific Primer-Polymerase Chain Reaction (SSP-PCR) and Genome Walking. Methods in Molecular Biology. Vol. 15. pp. 339–48. doi:10.1385/0-89603-244-2:339. ISBN 978-0-89603-244-6. PMID 21400290.
- ^ Bing DH, Boles C, Rehman FN, Audeh M, Belmarsh M, Kelley B, Adams CP (1996). "Bridge amplification: a solid phase PCR system for the amplification and detection of allelic differences in single copy genes". Genetic Identity Conference Proceedings, Seventh International Symposium on Human Identification. Archived from the original on 7 May 2001.
- ^ Khan Z, Poetter K, Park DJ (April 2008). "Enhanced solid phase PCR: mechanisms to increase priming by solid support primers". Analytical Biochemistry. 375 (2): 391–93. doi:10.1016/j.ab.2008.01.021. PMID 18267099.
- ^ Raoult D, Aboudharam G, Crubézy E, Larrouy G, Ludes B, Drancourt M (November 2000). "Molecular identification by "suicide PCR" of Yersinia pestis as the agent of medieval black death". Proceedings of the National Academy of Sciences of the United States of America. 97 (23): 12800–03. Bibcode:2000PNAS...9712800R. doi:10.1073/pnas.220225197. PMC 18844. PMID 11058154.
- ^ Liu YG, Whittier RF (February 1995). "Thermal asymmetric interlaced PCR: automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking". Genomics. 25 (3): 674–81. doi:10.1016/0888-7543(95)80010-J. PMID 7759102.
- ^ Don RH, Cox PT, Wainwright BJ, Baker K, Mattick JS (July 1991). "'Touchdown' PCR to circumvent spurious priming during gene amplification". Nucleic Acids Research. 19 (14): 4008. doi:10.1093/nar/19.14.4008. PMC 328507. PMID 1861999.
- ^ Myrick KV, Gelbart WM (February 2002). "Universal Fast Walking for direct and versatile determination of flanking sequence". Gene. 284 (1–2): 125–31. doi:10.1016/S0378-1119(02)00384-0. PMID 11891053.
- ^ "Full Text – LaNe RAGE: a new tool for genomic DNA flanking sequence determination". www.ejbiotechnology.info.
- ^ Park DJ (January 2005). "A new 5' terminal murine GAPDH exon identified using 5'RACE LaNe". Molecular Biotechnology. 29 (1): 39–46. doi:10.1385/MB:29:1:39. PMID 15668518. S2CID 45702164.
- ^ Park DJ (April 2004). "3' RACE LaNe: a simple and rapid fully nested PCR method to determine 3'-terminal cDNA sequence". BioTechniques. 36 (4): 586–88, 590. doi:10.2144/04364BM04. PMID 15088375.
- ^ "Key ingredient in coronavirus tests comes from Yellowstone's lakes". Science. 31 March 2020. Retrieved 13 May 2020.
- ^ Kleppe K, Ohtsuka E, Kleppe R, Molineux I, Khorana HG (March 1971). "Studies on polynucleotides. XCVI. Repair replications of short synthetic DNA's as catalyzed by DNA polymerases". Journal of Molecular Biology. 56 (2): 341–61. doi:10.1016/0022-2836(71)90469-4. PMID 4927950.
- ^ Rabinow, Paul (1996). Making PCR: A Story of Biotechnology. Chicago: University of Chicago Press. ISBN 978-0-226-70146-2.
- ^ Mullis, Kary (1998). Dancing Naked in the Mind Field. New York: Pantheon Books. ISBN 978-0-679-44255-4.
- ^ Mullis KB (April 1990). "The unusual origin of the polymerase chain reaction". Scientific American. 262 (4): 56–61, 64–65. Bibcode:1990SciAm.262d..56M. doi:10.1038/scientificamerican0490-56. PMID 2315679.
- ^ Patidar M, Agrawal S, Parveen F, Khare P (2015). "Molecular insights of saliva in solving paternity dispute". Journal of Forensic Dental Sciences. 7 (1): 76–79. doi:10.4103/0975-1475.150325. PMC 4330625. PMID 25709326.
- ^ Nichols D, Barker E (2016). "Psychedelics". Pharmacological Reviews. 68 (2): 264–355. doi:10.1124/pr.115.011478. PMC 4813425. PMID 26841800.
- ^ "The Nobel Prize in Chemistry 1993". NobelPrize.org.
- ^ "Citations for Chemical Breakthrough Awards 2017 Awardees". Division of the History of Chemistry. Retrieved 12 March 2018.
- ^ "The effects of business practices, licensing, and intellectual property on development and dissemination of the polymerase chain reaction: case study". 3 July 2006.
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ "Advice on How to Survive the Taq Wars". GEN Genetic Engineering News – Biobusiness Channel. 26 (9). 1 May 2006.
외부 링크
| 라이브러리 리소스 정보 중합효소 연쇄반응 |
| 위키미디어 커먼스는 폴리머아제 연쇄반응과 관련된 미디어를 보유하고 있다. |
- 미국 PCR 특허
- PCR 고원 효과란?유튜브 튜토리얼 비디오
- 스미스소니언 연구소의 중합효소 연쇄반응에 관한 연구