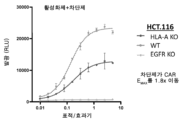
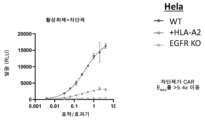
KR20230052289A - Egfr 양성 암을 치료하기 위한 조성물 및 방법 - Google Patents
Egfr 양성 암을 치료하기 위한 조성물 및 방법 Download PDFInfo
- Publication number
- KR20230052289A KR20230052289A KR1020237009181A KR20237009181A KR20230052289A KR 20230052289 A KR20230052289 A KR 20230052289A KR 1020237009181 A KR1020237009181 A KR 1020237009181A KR 20237009181 A KR20237009181 A KR 20237009181A KR 20230052289 A KR20230052289 A KR 20230052289A
- Authority
- KR
- South Korea
- Prior art keywords
- cells
- sequence
- receptor
- hla
- seq
- Prior art date
Links
- 206010028980 Neoplasm Diseases 0.000 title claims abstract description 159
- 201000011510 cancer Diseases 0.000 title claims abstract description 132
- 238000000034 method Methods 0.000 title claims abstract description 105
- 239000000203 mixture Substances 0.000 title description 16
- 102000052116 epidermal growth factor receptor activity proteins Human genes 0.000 title 1
- 108700015053 epidermal growth factor receptor activity proteins Proteins 0.000 title 1
- YOHYSYJDKVYCJI-UHFFFAOYSA-N n-[3-[[6-[3-(trifluoromethyl)anilino]pyrimidin-4-yl]amino]phenyl]cyclopropanecarboxamide Chemical compound FC(F)(F)C1=CC=CC(NC=2N=CN=C(NC=3C=C(NC(=O)C4CC4)C=CC=3)C=2)=C1 YOHYSYJDKVYCJI-UHFFFAOYSA-N 0.000 title 1
- 108020003175 receptors Proteins 0.000 claims abstract description 328
- 210000002865 immune cell Anatomy 0.000 claims abstract description 265
- 108091008042 inhibitory receptors Proteins 0.000 claims abstract description 147
- 239000012190 activator Substances 0.000 claims abstract description 142
- 238000011282 treatment Methods 0.000 claims abstract description 8
- 210000004027 cell Anatomy 0.000 claims description 581
- 102000005962 receptors Human genes 0.000 claims description 327
- 239000000427 antigen Substances 0.000 claims description 254
- 108091007433 antigens Proteins 0.000 claims description 254
- 102000036639 antigens Human genes 0.000 claims description 254
- 102100028972 HLA class I histocompatibility antigen, A alpha chain Human genes 0.000 claims description 236
- 108060006698 EGF receptor Proteins 0.000 claims description 229
- 102000001301 EGF receptor Human genes 0.000 claims description 228
- 108020001756 ligand binding domains Proteins 0.000 claims description 149
- 230000003834 intracellular effect Effects 0.000 claims description 128
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 119
- 108010017736 Leukocyte Immunoglobulin-like Receptor B1 Proteins 0.000 claims description 99
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 claims description 93
- 210000001744 T-lymphocyte Anatomy 0.000 claims description 91
- 230000014509 gene expression Effects 0.000 claims description 88
- 102000040430 polynucleotide Human genes 0.000 claims description 88
- 108091033319 polynucleotide Proteins 0.000 claims description 88
- 239000002157 polynucleotide Substances 0.000 claims description 88
- 108010047041 Complementarity Determining Regions Proteins 0.000 claims description 76
- 108090000623 proteins and genes Proteins 0.000 claims description 66
- 239000013598 vector Substances 0.000 claims description 58
- 108091054437 MHC class I family Proteins 0.000 claims description 55
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 54
- 239000008194 pharmaceutical composition Substances 0.000 claims description 49
- 102000043129 MHC class I family Human genes 0.000 claims description 47
- 229920001184 polypeptide Polymers 0.000 claims description 46
- 102100028971 HLA class I histocompatibility antigen, C alpha chain Human genes 0.000 claims description 32
- 230000002147 killing effect Effects 0.000 claims description 31
- 108010052199 HLA-C Antigens Proteins 0.000 claims description 27
- 108091032973 (ribonucleotides)n+m Proteins 0.000 claims description 23
- 108010081355 beta 2-Microglobulin Proteins 0.000 claims description 23
- 102000015736 beta 2-Microglobulin Human genes 0.000 claims description 22
- 230000002452 interceptive effect Effects 0.000 claims description 21
- 238000006467 substitution reaction Methods 0.000 claims description 21
- 108091027967 Small hairpin RNA Proteins 0.000 claims description 20
- 230000035772 mutation Effects 0.000 claims description 20
- 239000004055 small Interfering RNA Substances 0.000 claims description 20
- 238000012217 deletion Methods 0.000 claims description 19
- 230000037430 deletion Effects 0.000 claims description 19
- 230000000694 effects Effects 0.000 claims description 19
- 108010008553 HLA-B*07 antigen Proteins 0.000 claims description 18
- 108020004999 messenger RNA Proteins 0.000 claims description 17
- 230000004048 modification Effects 0.000 claims description 17
- 238000012986 modification Methods 0.000 claims description 17
- 238000003780 insertion Methods 0.000 claims description 14
- 230000037431 insertion Effects 0.000 claims description 14
- 150000007523 nucleic acids Chemical class 0.000 claims description 14
- 230000002829 reductive effect Effects 0.000 claims description 14
- 230000000295 complement effect Effects 0.000 claims description 13
- 102000039446 nucleic acids Human genes 0.000 claims description 13
- 108020004707 nucleic acids Proteins 0.000 claims description 13
- 230000000415 inactivating effect Effects 0.000 claims description 11
- 208000008839 Kidney Neoplasms Diseases 0.000 claims description 10
- 206010058467 Lung neoplasm malignant Diseases 0.000 claims description 10
- 206010038389 Renal cancer Diseases 0.000 claims description 10
- 201000010982 kidney cancer Diseases 0.000 claims description 10
- 201000005202 lung cancer Diseases 0.000 claims description 10
- 208000020816 lung neoplasm Diseases 0.000 claims description 10
- 230000001939 inductive effect Effects 0.000 claims description 9
- 206010009944 Colon cancer Diseases 0.000 claims description 8
- 206010006187 Breast cancer Diseases 0.000 claims description 7
- 208000026310 Breast neoplasm Diseases 0.000 claims description 7
- 208000001333 Colorectal Neoplasms Diseases 0.000 claims description 7
- 108010058607 HLA-B Antigens Proteins 0.000 claims description 7
- 208000005017 glioblastoma Diseases 0.000 claims description 7
- 201000010536 head and neck cancer Diseases 0.000 claims description 7
- 208000014829 head and neck neoplasm Diseases 0.000 claims description 7
- 230000001404 mediated effect Effects 0.000 claims description 7
- 238000012228 RNA interference-mediated gene silencing Methods 0.000 claims description 6
- 230000000735 allogeneic effect Effects 0.000 claims description 6
- 230000009368 gene silencing by RNA Effects 0.000 claims description 6
- 208000036764 Adenocarcinoma of the esophagus Diseases 0.000 claims description 5
- 206010005003 Bladder cancer Diseases 0.000 claims description 5
- 208000030808 Clear cell renal carcinoma Diseases 0.000 claims description 5
- 201000010915 Glioblastoma multiforme Diseases 0.000 claims description 5
- 206010030137 Oesophageal adenocarcinoma Diseases 0.000 claims description 5
- 206010033128 Ovarian cancer Diseases 0.000 claims description 5
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 5
- 206010060862 Prostate cancer Diseases 0.000 claims description 5
- 208000000236 Prostatic Neoplasms Diseases 0.000 claims description 5
- 206010039491 Sarcoma Diseases 0.000 claims description 5
- 206010041067 Small cell lung cancer Diseases 0.000 claims description 5
- 208000034254 Squamous cell carcinoma of the cervix uteri Diseases 0.000 claims description 5
- 208000005718 Stomach Neoplasms Diseases 0.000 claims description 5
- 208000000728 Thymus Neoplasms Diseases 0.000 claims description 5
- 208000024770 Thyroid neoplasm Diseases 0.000 claims description 5
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 claims description 5
- 201000006612 cervical squamous cell carcinoma Diseases 0.000 claims description 5
- 208000006990 cholangiocarcinoma Diseases 0.000 claims description 5
- 206010073251 clear cell renal cell carcinoma Diseases 0.000 claims description 5
- 208000028653 esophageal adenocarcinoma Diseases 0.000 claims description 5
- 201000006585 gastric adenocarcinoma Diseases 0.000 claims description 5
- 206010017758 gastric cancer Diseases 0.000 claims description 5
- 230000012010 growth Effects 0.000 claims description 5
- 201000007270 liver cancer Diseases 0.000 claims description 5
- 208000014018 liver neoplasm Diseases 0.000 claims description 5
- 230000003211 malignant effect Effects 0.000 claims description 5
- 208000002154 non-small cell lung carcinoma Diseases 0.000 claims description 5
- 208000000587 small cell lung carcinoma Diseases 0.000 claims description 5
- 201000011549 stomach cancer Diseases 0.000 claims description 5
- 201000002510 thyroid cancer Diseases 0.000 claims description 5
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 claims description 5
- 201000005112 urinary bladder cancer Diseases 0.000 claims description 5
- 206010008342 Cervix carcinoma Diseases 0.000 claims description 4
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 claims description 4
- 230000015556 catabolic process Effects 0.000 claims description 4
- 201000010881 cervical cancer Diseases 0.000 claims description 4
- 238000006731 degradation reaction Methods 0.000 claims description 4
- 239000003814 drug Substances 0.000 claims description 4
- 210000002540 macrophage Anatomy 0.000 claims description 4
- 210000000581 natural killer T-cell Anatomy 0.000 claims description 4
- 201000009377 thymus cancer Diseases 0.000 claims description 4
- 108010042407 Endonucleases Proteins 0.000 claims description 3
- 201000010983 breast ductal carcinoma Diseases 0.000 claims description 3
- 231100000221 frame shift mutation induction Toxicity 0.000 claims description 3
- 230000037433 frameshift Effects 0.000 claims description 3
- 210000000277 pancreatic duct Anatomy 0.000 claims description 3
- 230000001131 transforming effect Effects 0.000 claims description 3
- 208000008900 Pancreatic Ductal Carcinoma Diseases 0.000 claims description 2
- 102000009484 Vascular Endothelial Growth Factor Receptors Human genes 0.000 claims description 2
- 108010034265 Vascular Endothelial Growth Factor Receptors Proteins 0.000 claims description 2
- 230000002411 adverse Effects 0.000 claims description 2
- 239000003085 diluting agent Substances 0.000 claims description 2
- 239000003937 drug carrier Substances 0.000 claims description 2
- 210000004475 gamma-delta t lymphocyte Anatomy 0.000 claims description 2
- 230000036210 malignancy Effects 0.000 claims description 2
- 208000037843 metastatic solid tumor Diseases 0.000 claims description 2
- 210000000822 natural killer cell Anatomy 0.000 claims description 2
- 201000008129 pancreatic ductal adenocarcinoma Diseases 0.000 claims description 2
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 2
- 230000000306 recurrent effect Effects 0.000 claims description 2
- 108010075704 HLA-A Antigens Proteins 0.000 claims 35
- 102000004569 Leukocyte Immunoglobulin-like Receptor B1 Human genes 0.000 claims 11
- 102100024952 Protein CBFA2T1 Human genes 0.000 claims 4
- 102100031780 Endonuclease Human genes 0.000 claims 1
- 102100028976 HLA class I histocompatibility antigen, B alpha chain Human genes 0.000 claims 1
- 101710163270 Nuclease Proteins 0.000 claims 1
- 208000002495 Uterine Neoplasms Diseases 0.000 claims 1
- 230000005880 cancer cell killing Effects 0.000 claims 1
- 208000029742 colonic neoplasm Diseases 0.000 claims 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 claims 1
- 206010046766 uterine cancer Diseases 0.000 claims 1
- 101000986086 Homo sapiens HLA class I histocompatibility antigen, A alpha chain Proteins 0.000 description 202
- 239000003446 ligand Substances 0.000 description 118
- 108091008874 T cell receptors Proteins 0.000 description 111
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 111
- 102100025584 Leukocyte immunoglobulin-like receptor subfamily B member 1 Human genes 0.000 description 107
- 230000027455 binding Effects 0.000 description 73
- 102000017420 CD3 protein, epsilon/gamma/delta subunit Human genes 0.000 description 62
- 108050005493 CD3 protein, epsilon/gamma/delta subunit Proteins 0.000 description 61
- 230000002401 inhibitory effect Effects 0.000 description 57
- 230000004913 activation Effects 0.000 description 56
- 235000001014 amino acid Nutrition 0.000 description 53
- 229940024606 amino acid Drugs 0.000 description 50
- 150000001413 amino acids Chemical class 0.000 description 49
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 description 41
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 description 41
- 125000003275 alpha amino acid group Chemical group 0.000 description 39
- 101000889133 Homo sapiens C-X-C motif chemokine 16 Proteins 0.000 description 33
- 102100039396 C-X-C motif chemokine 16 Human genes 0.000 description 32
- 102000004169 proteins and genes Human genes 0.000 description 32
- 235000018102 proteins Nutrition 0.000 description 31
- 101000684673 Homo sapiens Protein APCDD1 Proteins 0.000 description 30
- 230000000139 costimulatory effect Effects 0.000 description 29
- 230000006870 function Effects 0.000 description 29
- 239000011324 bead Substances 0.000 description 28
- 102100023735 Protein APCDD1 Human genes 0.000 description 27
- 230000001086 cytosolic effect Effects 0.000 description 26
- 239000003112 inhibitor Substances 0.000 description 26
- -1 or blockers Substances 0.000 description 23
- 108700028369 Alleles Proteins 0.000 description 22
- 230000004068 intracellular signaling Effects 0.000 description 21
- 101000851370 Homo sapiens Tumor necrosis factor receptor superfamily member 9 Proteins 0.000 description 19
- 102100036856 Tumor necrosis factor receptor superfamily member 9 Human genes 0.000 description 19
- 230000011664 signaling Effects 0.000 description 19
- 101000909528 Homo sapiens Collectin-12 Proteins 0.000 description 18
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 18
- 239000003795 chemical substances by application Substances 0.000 description 18
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 18
- 102100024330 Collectin-12 Human genes 0.000 description 17
- 238000003556 assay Methods 0.000 description 17
- 101000984190 Homo sapiens Leukocyte immunoglobulin-like receptor subfamily B member 1 Proteins 0.000 description 16
- 230000005764 inhibitory process Effects 0.000 description 16
- 241000282414 Homo sapiens Species 0.000 description 15
- 239000012634 fragment Substances 0.000 description 15
- 230000004936 stimulating effect Effects 0.000 description 15
- 210000001519 tissue Anatomy 0.000 description 15
- 108010041397 CD4 Antigens Proteins 0.000 description 13
- 101000946843 Homo sapiens T-cell surface glycoprotein CD8 alpha chain Proteins 0.000 description 13
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 description 13
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 13
- 239000002773 nucleotide Substances 0.000 description 13
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 12
- 125000003729 nucleotide group Chemical group 0.000 description 12
- 239000002245 particle Substances 0.000 description 12
- 102000006390 HLA-B Antigens Human genes 0.000 description 11
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 11
- 108700008625 Reporter Genes Proteins 0.000 description 11
- 238000011467 adoptive cell therapy Methods 0.000 description 11
- 238000003501 co-culture Methods 0.000 description 11
- 230000004044 response Effects 0.000 description 11
- 230000035899 viability Effects 0.000 description 11
- 239000005089 Luciferase Substances 0.000 description 10
- 230000003213 activating effect Effects 0.000 description 10
- 239000012636 effector Substances 0.000 description 10
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 9
- 108060001084 Luciferase Proteins 0.000 description 9
- 108060003951 Immunoglobulin Proteins 0.000 description 8
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 8
- 102100022153 Tumor necrosis factor receptor superfamily member 4 Human genes 0.000 description 8
- 241000700605 Viruses Species 0.000 description 8
- 230000000890 antigenic effect Effects 0.000 description 8
- 201000010099 disease Diseases 0.000 description 8
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 8
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 8
- 102000018358 immunoglobulin Human genes 0.000 description 8
- 210000004881 tumor cell Anatomy 0.000 description 8
- 101710089372 Programmed cell death protein 1 Proteins 0.000 description 7
- 102100040678 Programmed cell death protein 1 Human genes 0.000 description 7
- 238000013459 approach Methods 0.000 description 7
- 239000013604 expression vector Substances 0.000 description 7
- 230000001965 increasing effect Effects 0.000 description 7
- 230000007246 mechanism Effects 0.000 description 7
- 239000002609 medium Substances 0.000 description 7
- 239000012528 membrane Substances 0.000 description 7
- 239000000243 solution Substances 0.000 description 7
- 239000013603 viral vector Substances 0.000 description 7
- 102100038080 B-cell receptor CD22 Human genes 0.000 description 6
- 108020004414 DNA Proteins 0.000 description 6
- 101000884305 Homo sapiens B-cell receptor CD22 Proteins 0.000 description 6
- 101000934341 Homo sapiens T-cell surface glycoprotein CD5 Proteins 0.000 description 6
- 102100025244 T-cell surface glycoprotein CD5 Human genes 0.000 description 6
- 210000003719 b-lymphocyte Anatomy 0.000 description 6
- 238000001727 in vivo Methods 0.000 description 6
- 230000003993 interaction Effects 0.000 description 6
- 210000004698 lymphocyte Anatomy 0.000 description 6
- 238000004519 manufacturing process Methods 0.000 description 6
- 230000000638 stimulation Effects 0.000 description 6
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 6
- 230000008685 targeting Effects 0.000 description 6
- 102100027207 CD27 antigen Human genes 0.000 description 5
- BCCRXDTUTZHDEU-VKHMYHEASA-N Gly-Ser Chemical compound NCC(=O)N[C@@H](CO)C(O)=O BCCRXDTUTZHDEU-VKHMYHEASA-N 0.000 description 5
- 101000914511 Homo sapiens CD27 antigen Proteins 0.000 description 5
- 101000986087 Homo sapiens HLA class I histocompatibility antigen, B alpha chain Proteins 0.000 description 5
- 101000986084 Homo sapiens HLA class I histocompatibility antigen, C alpha chain Proteins 0.000 description 5
- 108010002350 Interleukin-2 Proteins 0.000 description 5
- 102000000588 Interleukin-2 Human genes 0.000 description 5
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 5
- 108010064548 Lymphocyte Function-Associated Antigen-1 Proteins 0.000 description 5
- 108010052285 Membrane Proteins Proteins 0.000 description 5
- 108010038807 Oligopeptides Proteins 0.000 description 5
- 102000015636 Oligopeptides Human genes 0.000 description 5
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 5
- 230000001413 cellular effect Effects 0.000 description 5
- 230000008859 change Effects 0.000 description 5
- 230000003013 cytotoxicity Effects 0.000 description 5
- 231100000135 cytotoxicity Toxicity 0.000 description 5
- 230000007423 decrease Effects 0.000 description 5
- 230000008014 freezing Effects 0.000 description 5
- 238000007710 freezing Methods 0.000 description 5
- 239000000833 heterodimer Substances 0.000 description 5
- 230000005931 immune cell recruitment Effects 0.000 description 5
- 210000000265 leukocyte Anatomy 0.000 description 5
- 210000001616 monocyte Anatomy 0.000 description 5
- 230000002441 reversible effect Effects 0.000 description 5
- 210000002966 serum Anatomy 0.000 description 5
- 208000024891 symptom Diseases 0.000 description 5
- 238000004448 titration Methods 0.000 description 5
- 239000004474 valine Substances 0.000 description 5
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 4
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 4
- 229940045513 CTLA4 antagonist Drugs 0.000 description 4
- 102000004127 Cytokines Human genes 0.000 description 4
- 108090000695 Cytokines Proteins 0.000 description 4
- 102100039498 Cytotoxic T-lymphocyte protein 4 Human genes 0.000 description 4
- 101000934346 Homo sapiens T-cell surface antigen CD2 Proteins 0.000 description 4
- 101000679851 Homo sapiens Tumor necrosis factor receptor superfamily member 4 Proteins 0.000 description 4
- 101000851376 Homo sapiens Tumor necrosis factor receptor superfamily member 8 Proteins 0.000 description 4
- 101000818543 Homo sapiens Tyrosine-protein kinase ZAP-70 Proteins 0.000 description 4
- 102000003839 Human Proteins Human genes 0.000 description 4
- 108090000144 Human Proteins Proteins 0.000 description 4
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 4
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 4
- 241001465754 Metazoa Species 0.000 description 4
- 241000699666 Mus <mouse, genus> Species 0.000 description 4
- 108010029485 Protein Isoforms Proteins 0.000 description 4
- 102000001708 Protein Isoforms Human genes 0.000 description 4
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 4
- 102100025237 T-cell surface antigen CD2 Human genes 0.000 description 4
- 101710165473 Tumor necrosis factor receptor superfamily member 4 Proteins 0.000 description 4
- 102100036857 Tumor necrosis factor receptor superfamily member 8 Human genes 0.000 description 4
- 102100021125 Tyrosine-protein kinase ZAP-70 Human genes 0.000 description 4
- 238000002617 apheresis Methods 0.000 description 4
- 230000000903 blocking effect Effects 0.000 description 4
- 239000000872 buffer Substances 0.000 description 4
- 230000010261 cell growth Effects 0.000 description 4
- 238000002659 cell therapy Methods 0.000 description 4
- 231100000433 cytotoxic Toxicity 0.000 description 4
- 230000001472 cytotoxic effect Effects 0.000 description 4
- 230000001419 dependent effect Effects 0.000 description 4
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 4
- 210000000981 epithelium Anatomy 0.000 description 4
- 230000028993 immune response Effects 0.000 description 4
- 238000000338 in vitro Methods 0.000 description 4
- 208000015181 infectious disease Diseases 0.000 description 4
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 4
- 239000002953 phosphate buffered saline Substances 0.000 description 4
- 210000004986 primary T-cell Anatomy 0.000 description 4
- 230000035755 proliferation Effects 0.000 description 4
- 230000001105 regulatory effect Effects 0.000 description 4
- 230000035945 sensitivity Effects 0.000 description 4
- 102000035160 transmembrane proteins Human genes 0.000 description 4
- 108091005703 transmembrane proteins Proteins 0.000 description 4
- 238000005406 washing Methods 0.000 description 4
- 102000006306 Antigen Receptors Human genes 0.000 description 3
- 108010083359 Antigen Receptors Proteins 0.000 description 3
- 102100027203 B-cell antigen receptor complex-associated protein beta chain Human genes 0.000 description 3
- 102100038078 CD276 antigen Human genes 0.000 description 3
- 102100032937 CD40 ligand Human genes 0.000 description 3
- 102100037904 CD9 antigen Human genes 0.000 description 3
- 108010087819 Fc receptors Proteins 0.000 description 3
- 102000009109 Fc receptors Human genes 0.000 description 3
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 3
- 102100026122 High affinity immunoglobulin gamma Fc receptor I Human genes 0.000 description 3
- 101000914491 Homo sapiens B-cell antigen receptor complex-associated protein beta chain Proteins 0.000 description 3
- 101000738354 Homo sapiens CD9 antigen Proteins 0.000 description 3
- 101000913074 Homo sapiens High affinity immunoglobulin gamma Fc receptor I Proteins 0.000 description 3
- 101000777628 Homo sapiens Leukocyte antigen CD37 Proteins 0.000 description 3
- 101000917858 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 3
- 101000917839 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-B Proteins 0.000 description 3
- 101000934338 Homo sapiens Myeloid cell surface antigen CD33 Proteins 0.000 description 3
- 101001109503 Homo sapiens NKG2-C type II integral membrane protein Proteins 0.000 description 3
- 101000738771 Homo sapiens Receptor-type tyrosine-protein phosphatase C Proteins 0.000 description 3
- 101000914496 Homo sapiens T-cell antigen CD7 Proteins 0.000 description 3
- 101000914484 Homo sapiens T-lymphocyte activation antigen CD80 Proteins 0.000 description 3
- 101000801234 Homo sapiens Tumor necrosis factor receptor superfamily member 18 Proteins 0.000 description 3
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- 241000713666 Lentivirus Species 0.000 description 3
- 102100031586 Leukocyte antigen CD37 Human genes 0.000 description 3
- 102100029185 Low affinity immunoglobulin gamma Fc region receptor III-B Human genes 0.000 description 3
- 102000018697 Membrane Proteins Human genes 0.000 description 3
- 102100025243 Myeloid cell surface antigen CD33 Human genes 0.000 description 3
- 102100022683 NKG2-C type II integral membrane protein Human genes 0.000 description 3
- 108010076504 Protein Sorting Signals Proteins 0.000 description 3
- 102100037422 Receptor-type tyrosine-protein phosphatase C Human genes 0.000 description 3
- 108020004511 Recombinant DNA Proteins 0.000 description 3
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 3
- 230000006044 T cell activation Effects 0.000 description 3
- 102100027208 T-cell antigen CD7 Human genes 0.000 description 3
- 102100027222 T-lymphocyte activation antigen CD80 Human genes 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- 102100033728 Tumor necrosis factor receptor superfamily member 18 Human genes 0.000 description 3
- 102100040245 Tumor necrosis factor receptor superfamily member 5 Human genes 0.000 description 3
- 230000001363 autoimmune Effects 0.000 description 3
- 230000009286 beneficial effect Effects 0.000 description 3
- 238000012258 culturing Methods 0.000 description 3
- 102000003675 cytokine receptors Human genes 0.000 description 3
- 108010057085 cytokine receptors Proteins 0.000 description 3
- 230000002950 deficient Effects 0.000 description 3
- 239000008121 dextrose Substances 0.000 description 3
- 238000010586 diagram Methods 0.000 description 3
- 108010087914 epidermal growth factor receptor VIII Proteins 0.000 description 3
- 102000054430 human APCDD1 Human genes 0.000 description 3
- 210000000987 immune system Anatomy 0.000 description 3
- 238000002955 isolation Methods 0.000 description 3
- FVVLHONNBARESJ-NTOWJWGLSA-H magnesium;potassium;trisodium;(2r,3s,4r,5r)-2,3,4,5,6-pentahydroxyhexanoate;acetate;tetrachloride;nonahydrate Chemical compound O.O.O.O.O.O.O.O.O.[Na+].[Na+].[Na+].[Mg+2].[Cl-].[Cl-].[Cl-].[Cl-].[K+].CC([O-])=O.OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C([O-])=O FVVLHONNBARESJ-NTOWJWGLSA-H 0.000 description 3
- 239000003550 marker Substances 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 239000007758 minimum essential medium Substances 0.000 description 3
- 229910052757 nitrogen Inorganic materials 0.000 description 3
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 3
- 230000006461 physiological response Effects 0.000 description 3
- 230000004043 responsiveness Effects 0.000 description 3
- 230000001177 retroviral effect Effects 0.000 description 3
- 230000019491 signal transduction Effects 0.000 description 3
- 210000001541 thymus gland Anatomy 0.000 description 3
- 230000001988 toxicity Effects 0.000 description 3
- 231100000419 toxicity Toxicity 0.000 description 3
- 238000013518 transcription Methods 0.000 description 3
- 230000035897 transcription Effects 0.000 description 3
- 241001430294 unidentified retrovirus Species 0.000 description 3
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 2
- 102100027205 B-cell antigen receptor complex-associated protein alpha chain Human genes 0.000 description 2
- 101710185679 CD276 antigen Proteins 0.000 description 2
- 210000004366 CD4-positive T-lymphocyte Anatomy 0.000 description 2
- 102100029382 CMRF35-like molecule 6 Human genes 0.000 description 2
- 241000282472 Canis lupus familiaris Species 0.000 description 2
- 241000701022 Cytomegalovirus Species 0.000 description 2
- 102000053602 DNA Human genes 0.000 description 2
- 241000702421 Dependoparvovirus Species 0.000 description 2
- 102000004533 Endonucleases Human genes 0.000 description 2
- 102000004190 Enzymes Human genes 0.000 description 2
- 108090000790 Enzymes Proteins 0.000 description 2
- 108010021472 Fc gamma receptor IIB Proteins 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 2
- 102100028970 HLA class I histocompatibility antigen, alpha chain E Human genes 0.000 description 2
- 108010022070 HLA-A*02 antigen Proteins 0.000 description 2
- 102000006354 HLA-DR Antigens Human genes 0.000 description 2
- 108010058597 HLA-DR Antigens Proteins 0.000 description 2
- 102100029360 Hematopoietic cell signal transducer Human genes 0.000 description 2
- 101000914489 Homo sapiens B-cell antigen receptor complex-associated protein alpha chain Proteins 0.000 description 2
- 101000990034 Homo sapiens CMRF35-like molecule 6 Proteins 0.000 description 2
- 101000986085 Homo sapiens HLA class I histocompatibility antigen, alpha chain E Proteins 0.000 description 2
- 101000990188 Homo sapiens Hematopoietic cell signal transducer Proteins 0.000 description 2
- 101000945490 Homo sapiens Killer cell immunoglobulin-like receptor 3DL2 Proteins 0.000 description 2
- 101000945493 Homo sapiens Killer cell immunoglobulin-like receptor 3DL3 Proteins 0.000 description 2
- 101001109501 Homo sapiens NKG2-D type II integral membrane protein Proteins 0.000 description 2
- 101000946860 Homo sapiens T-cell surface glycoprotein CD3 epsilon chain Proteins 0.000 description 2
- 108091006905 Human Serum Albumin Proteins 0.000 description 2
- 102000008100 Human Serum Albumin Human genes 0.000 description 2
- 241000725303 Human immunodeficiency virus Species 0.000 description 2
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 2
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 2
- 108010002586 Interleukin-7 Proteins 0.000 description 2
- 108010043610 KIR Receptors Proteins 0.000 description 2
- 102000002698 KIR Receptors Human genes 0.000 description 2
- 102100034840 Killer cell immunoglobulin-like receptor 3DL2 Human genes 0.000 description 2
- 102100034834 Killer cell immunoglobulin-like receptor 3DL3 Human genes 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- 102100029205 Low affinity immunoglobulin gamma Fc region receptor II-b Human genes 0.000 description 2
- 108700005089 MHC Class I Genes Proteins 0.000 description 2
- 241000829100 Macaca mulatta polyomavirus 1 Species 0.000 description 2
- 108010024777 Mating Factor Receptors Proteins 0.000 description 2
- 108091092878 Microsatellite Proteins 0.000 description 2
- 102100022680 NKG2-D type II integral membrane protein Human genes 0.000 description 2
- 108091028043 Nucleic acid sequence Proteins 0.000 description 2
- 108091008606 PDGF receptors Proteins 0.000 description 2
- 102000011653 Platelet-Derived Growth Factor Receptors Human genes 0.000 description 2
- RJKFOVLPORLFTN-LEKSSAKUSA-N Progesterone Chemical compound C1CC2=CC(=O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H](C(=O)C)[C@@]1(C)CC2 RJKFOVLPORLFTN-LEKSSAKUSA-N 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 102100035794 T-cell surface glycoprotein CD3 epsilon chain Human genes 0.000 description 2
- 108700019146 Transgenes Proteins 0.000 description 2
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 2
- 238000007792 addition Methods 0.000 description 2
- 239000000654 additive Substances 0.000 description 2
- 210000000612 antigen-presenting cell Anatomy 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 210000004369 blood Anatomy 0.000 description 2
- 239000008280 blood Substances 0.000 description 2
- 210000001772 blood platelet Anatomy 0.000 description 2
- 210000001185 bone marrow Anatomy 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 239000001506 calcium phosphate Substances 0.000 description 2
- 229910000389 calcium phosphate Inorganic materials 0.000 description 2
- 235000011010 calcium phosphates Nutrition 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 230000024245 cell differentiation Effects 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 230000005754 cellular signaling Effects 0.000 description 2
- 238000005119 centrifugation Methods 0.000 description 2
- 230000004186 co-expression Effects 0.000 description 2
- 108091036078 conserved sequence Proteins 0.000 description 2
- 230000001276 controlling effect Effects 0.000 description 2
- 230000016396 cytokine production Effects 0.000 description 2
- 230000001461 cytolytic effect Effects 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 229940119744 dextran 40 Drugs 0.000 description 2
- 231100000371 dose-limiting toxicity Toxicity 0.000 description 2
- 230000003828 downregulation Effects 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 230000008995 epigenetic change Effects 0.000 description 2
- 210000003743 erythrocyte Anatomy 0.000 description 2
- 230000001747 exhibiting effect Effects 0.000 description 2
- 238000000684 flow cytometry Methods 0.000 description 2
- 238000001476 gene delivery Methods 0.000 description 2
- 238000012239 gene modification Methods 0.000 description 2
- 230000030279 gene silencing Effects 0.000 description 2
- 230000005017 genetic modification Effects 0.000 description 2
- 235000013617 genetically modified food Nutrition 0.000 description 2
- 229930004094 glycosylphosphatidylinositol Natural products 0.000 description 2
- 210000003714 granulocyte Anatomy 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 125000001165 hydrophobic group Chemical group 0.000 description 2
- 238000003384 imaging method Methods 0.000 description 2
- 239000012642 immune effector Substances 0.000 description 2
- 229940121354 immunomodulator Drugs 0.000 description 2
- 238000011534 incubation Methods 0.000 description 2
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 description 2
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- FZWBNHMXJMCXLU-BLAUPYHCSA-N isomaltotriose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1OC[C@@H]1[C@@H](O)[C@H](O)[C@@H](O)[C@@H](OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C=O)O1 FZWBNHMXJMCXLU-BLAUPYHCSA-N 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 230000007774 longterm Effects 0.000 description 2
- 238000005259 measurement Methods 0.000 description 2
- 210000003071 memory t lymphocyte Anatomy 0.000 description 2
- 239000000693 micelle Substances 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 230000001537 neural effect Effects 0.000 description 2
- 210000000440 neutrophil Anatomy 0.000 description 2
- 230000005298 paramagnetic effect Effects 0.000 description 2
- 102000054765 polymorphisms of proteins Human genes 0.000 description 2
- 239000002243 precursor Substances 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 238000003757 reverse transcription PCR Methods 0.000 description 2
- 239000000523 sample Substances 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- DAEPDZWVDSPTHF-UHFFFAOYSA-M sodium pyruvate Chemical compound [Na+].CC(=O)C([O-])=O DAEPDZWVDSPTHF-UHFFFAOYSA-M 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 230000001629 suppression Effects 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 230000001225 therapeutic effect Effects 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- 238000010361 transduction Methods 0.000 description 2
- 230000026683 transduction Effects 0.000 description 2
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 2
- 241000701161 unidentified adenovirus Species 0.000 description 2
- 239000003981 vehicle Substances 0.000 description 2
- 230000003612 virological effect Effects 0.000 description 2
- DGVVWUTYPXICAM-UHFFFAOYSA-N β‐Mercaptoethanol Chemical compound OCCS DGVVWUTYPXICAM-UHFFFAOYSA-N 0.000 description 2
- DIGQNXIGRZPYDK-WKSCXVIASA-N (2R)-6-amino-2-[[2-[[(2S)-2-[[2-[[(2R)-2-[[(2S)-2-[[(2R,3S)-2-[[2-[[(2S)-2-[[2-[[(2S)-2-[[(2S)-2-[[(2R)-2-[[(2S,3S)-2-[[(2R)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[2-[[(2S)-2-[[(2R)-2-[[2-[[2-[[2-[(2-amino-1-hydroxyethylidene)amino]-3-carboxy-1-hydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1,5-dihydroxy-5-iminopentylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]hexanoic acid Chemical compound C[C@@H]([C@@H](C(=N[C@@H](CS)C(=N[C@@H](C)C(=N[C@@H](CO)C(=NCC(=N[C@@H](CCC(=N)O)C(=NC(CS)C(=N[C@H]([C@H](C)O)C(=N[C@H](CS)C(=N[C@H](CO)C(=NCC(=N[C@H](CS)C(=NCC(=N[C@H](CCCCN)C(=O)O)O)O)O)O)O)O)O)O)O)O)O)O)O)N=C([C@H](CS)N=C([C@H](CO)N=C([C@H](CO)N=C([C@H](C)N=C(CN=C([C@H](CO)N=C([C@H](CS)N=C(CN=C(C(CS)N=C(C(CC(=O)O)N=C(CN)O)O)O)O)O)O)O)O)O)O)O)O DIGQNXIGRZPYDK-WKSCXVIASA-N 0.000 description 1
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- MZOFCQQQCNRIBI-VMXHOPILSA-N (3s)-4-[[(2s)-1-[[(2s)-1-[[(1s)-1-carboxy-2-hydroxyethyl]amino]-4-methyl-1-oxopentan-2-yl]amino]-5-(diaminomethylideneamino)-1-oxopentan-2-yl]amino]-3-[[2-[[(2s)-2,6-diaminohexanoyl]amino]acetyl]amino]-4-oxobutanoic acid Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCCN MZOFCQQQCNRIBI-VMXHOPILSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 108020005029 5' Flanking Region Proteins 0.000 description 1
- 102000007469 Actins Human genes 0.000 description 1
- 108010085238 Actins Proteins 0.000 description 1
- 229930024421 Adenine Natural products 0.000 description 1
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- 108010031480 Artificial Receptors Proteins 0.000 description 1
- 206010003445 Ascites Diseases 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 241000714230 Avian leukemia virus Species 0.000 description 1
- 102100029822 B- and T-lymphocyte attenuator Human genes 0.000 description 1
- 102100022005 B-lymphocyte antigen CD20 Human genes 0.000 description 1
- 101150076800 B2M gene Proteins 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 102100024263 CD160 antigen Human genes 0.000 description 1
- 210000001266 CD8-positive T-lymphocyte Anatomy 0.000 description 1
- 102100035793 CD83 antigen Human genes 0.000 description 1
- 108010052382 CD83 antigen Proteins 0.000 description 1
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 1
- 108010022366 Carcinoembryonic Antigen Proteins 0.000 description 1
- 102100025475 Carcinoembryonic antigen-related cell adhesion molecule 5 Human genes 0.000 description 1
- 241000700198 Cavia Species 0.000 description 1
- 108010067225 Cell Adhesion Molecules Proteins 0.000 description 1
- 102000016289 Cell Adhesion Molecules Human genes 0.000 description 1
- 102100024046 Cell adhesion molecule 3 Human genes 0.000 description 1
- 101710197433 Cell adhesion molecule 3 Proteins 0.000 description 1
- 102100021396 Cell surface glycoprotein CD200 receptor 1 Human genes 0.000 description 1
- 108010035563 Chloramphenicol O-acetyltransferase Proteins 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 102000004420 Creatine Kinase Human genes 0.000 description 1
- 108010042126 Creatine kinase Proteins 0.000 description 1
- IGXWBGJHJZYPQS-SSDOTTSWSA-N D-Luciferin Chemical compound OC(=O)[C@H]1CSC(C=2SC3=CC=C(O)C=C3N=2)=N1 IGXWBGJHJZYPQS-SSDOTTSWSA-N 0.000 description 1
- CYCGRDQQIOGCKX-UHFFFAOYSA-N Dehydro-luciferin Natural products OC(=O)C1=CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 CYCGRDQQIOGCKX-UHFFFAOYSA-N 0.000 description 1
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 1
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 102100025137 Early activation antigen CD69 Human genes 0.000 description 1
- 101150039808 Egfr gene Proteins 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108050001049 Extracellular proteins Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- BJGNCJDXODQBOB-UHFFFAOYSA-N Fivefly Luciferin Natural products OC(=O)C1CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 BJGNCJDXODQBOB-UHFFFAOYSA-N 0.000 description 1
- 102000003688 G-Protein-Coupled Receptors Human genes 0.000 description 1
- 108090000045 G-Protein-Coupled Receptors Proteins 0.000 description 1
- 206010059024 Gastrointestinal toxicity Diseases 0.000 description 1
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 102000003886 Glycoproteins Human genes 0.000 description 1
- 108090000288 Glycoproteins Proteins 0.000 description 1
- 108010017213 Granulocyte-Macrophage Colony-Stimulating Factor Proteins 0.000 description 1
- 102100039620 Granulocyte-macrophage colony-stimulating factor Human genes 0.000 description 1
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 1
- 102100028966 HLA class I histocompatibility antigen, alpha chain F Human genes 0.000 description 1
- 102100028967 HLA class I histocompatibility antigen, alpha chain G Human genes 0.000 description 1
- 108010074032 HLA-A2 Antigen Proteins 0.000 description 1
- 102000025850 HLA-A2 Antigen Human genes 0.000 description 1
- 102000012153 HLA-B27 Antigen Human genes 0.000 description 1
- 108010061486 HLA-B27 Antigen Proteins 0.000 description 1
- 108010024164 HLA-G Antigens Proteins 0.000 description 1
- 102000001554 Hemoglobins Human genes 0.000 description 1
- 108010054147 Hemoglobins Proteins 0.000 description 1
- 108010034791 Heterochromatin Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 101000777636 Homo sapiens ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase 1 Proteins 0.000 description 1
- 101000864344 Homo sapiens B- and T-lymphocyte attenuator Proteins 0.000 description 1
- 101000897405 Homo sapiens B-lymphocyte antigen CD20 Proteins 0.000 description 1
- 101000761938 Homo sapiens CD160 antigen Proteins 0.000 description 1
- 101000884279 Homo sapiens CD276 antigen Proteins 0.000 description 1
- 101000969553 Homo sapiens Cell surface glycoprotein CD200 receptor 1 Proteins 0.000 description 1
- 101000934374 Homo sapiens Early activation antigen CD69 Proteins 0.000 description 1
- 101000986080 Homo sapiens HLA class I histocompatibility antigen, alpha chain F Proteins 0.000 description 1
- 101000935040 Homo sapiens Integrin beta-2 Proteins 0.000 description 1
- 101001057504 Homo sapiens Interferon-stimulated gene 20 kDa protein Proteins 0.000 description 1
- 101001055144 Homo sapiens Interleukin-2 receptor subunit alpha Proteins 0.000 description 1
- 101000945339 Homo sapiens Killer cell immunoglobulin-like receptor 2DS2 Proteins 0.000 description 1
- 101000971538 Homo sapiens Killer cell lectin-like receptor subfamily F member 1 Proteins 0.000 description 1
- 101100128412 Homo sapiens LILRB1 gene Proteins 0.000 description 1
- 101000984192 Homo sapiens Leukocyte immunoglobulin-like receptor subfamily B member 3 Proteins 0.000 description 1
- 101000633784 Homo sapiens SLAM family member 7 Proteins 0.000 description 1
- 101000738413 Homo sapiens T-cell surface glycoprotein CD3 gamma chain Proteins 0.000 description 1
- 101000738335 Homo sapiens T-cell surface glycoprotein CD3 zeta chain Proteins 0.000 description 1
- 101000835093 Homo sapiens Transferrin receptor protein 1 Proteins 0.000 description 1
- 101000611183 Homo sapiens Tumor necrosis factor Proteins 0.000 description 1
- 101000830565 Homo sapiens Tumor necrosis factor ligand superfamily member 10 Proteins 0.000 description 1
- 101000830594 Homo sapiens Tumor necrosis factor ligand superfamily member 14 Proteins 0.000 description 1
- 101000795169 Homo sapiens Tumor necrosis factor receptor superfamily member 13C Proteins 0.000 description 1
- 101001022129 Homo sapiens Tyrosine-protein kinase Fyn Proteins 0.000 description 1
- 101001047681 Homo sapiens Tyrosine-protein kinase Lck Proteins 0.000 description 1
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 1
- 229920001612 Hydroxyethyl starch Polymers 0.000 description 1
- 229940076838 Immune checkpoint inhibitor Drugs 0.000 description 1
- 108091008026 Inhibitory immune checkpoint proteins Proteins 0.000 description 1
- 102000037984 Inhibitory immune checkpoint proteins Human genes 0.000 description 1
- 108090001061 Insulin Proteins 0.000 description 1
- 102000004877 Insulin Human genes 0.000 description 1
- 102100025390 Integrin beta-2 Human genes 0.000 description 1
- 108010064593 Intercellular Adhesion Molecule-1 Proteins 0.000 description 1
- 102100037877 Intercellular adhesion molecule 1 Human genes 0.000 description 1
- 102100037850 Interferon gamma Human genes 0.000 description 1
- 108010074328 Interferon-gamma Proteins 0.000 description 1
- 102100027268 Interferon-stimulated gene 20 kDa protein Human genes 0.000 description 1
- 102000003814 Interleukin-10 Human genes 0.000 description 1
- 108090000174 Interleukin-10 Proteins 0.000 description 1
- 102000013462 Interleukin-12 Human genes 0.000 description 1
- 108010065805 Interleukin-12 Proteins 0.000 description 1
- 102000003812 Interleukin-15 Human genes 0.000 description 1
- 108090000172 Interleukin-15 Proteins 0.000 description 1
- 108090000978 Interleukin-4 Proteins 0.000 description 1
- 102000004388 Interleukin-4 Human genes 0.000 description 1
- 102000000704 Interleukin-7 Human genes 0.000 description 1
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 1
- 102000004310 Ion Channels Human genes 0.000 description 1
- 102100033630 Killer cell immunoglobulin-like receptor 2DS2 Human genes 0.000 description 1
- 102100021458 Killer cell lectin-like receptor subfamily F member 1 Human genes 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- PWKSKIMOESPYIA-BYPYZUCNSA-N L-N-acetyl-Cysteine Chemical compound CC(=O)N[C@@H](CS)C(O)=O PWKSKIMOESPYIA-BYPYZUCNSA-N 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 1
- 101710145789 Leukocyte immunoglobulin-like receptor subfamily B member 1 Proteins 0.000 description 1
- 101710145805 Leukocyte immunoglobulin-like receptor subfamily B member 3 Proteins 0.000 description 1
- DDWFXDSYGUXRAY-UHFFFAOYSA-N Luciferin Natural products CCc1c(C)c(CC2NC(=O)C(=C2C=C)C)[nH]c1Cc3[nH]c4C(=C5/NC(CC(=O)O)C(C)C5CC(=O)O)CC(=O)c4c3C DDWFXDSYGUXRAY-UHFFFAOYSA-N 0.000 description 1
- 102100034709 Lymphocyte cytosolic protein 2 Human genes 0.000 description 1
- 101710195102 Lymphocyte cytosolic protein 2 Proteins 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- FYYHWMGAXLPEAU-UHFFFAOYSA-N Magnesium Chemical compound [Mg] FYYHWMGAXLPEAU-UHFFFAOYSA-N 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 108010061593 Member 14 Tumor Necrosis Factor Receptors Proteins 0.000 description 1
- 102000003792 Metallothionein Human genes 0.000 description 1
- 108090000157 Metallothionein Proteins 0.000 description 1
- 101150076359 Mhc gene Proteins 0.000 description 1
- 241000713333 Mouse mammary tumor virus Species 0.000 description 1
- 241000714177 Murine leukemia virus Species 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 102000003505 Myosin Human genes 0.000 description 1
- 108060008487 Myosin Proteins 0.000 description 1
- 108091008877 NK cell receptors Proteins 0.000 description 1
- 102000010648 Natural Killer Cell Receptors Human genes 0.000 description 1
- 206010061309 Neoplasm progression Diseases 0.000 description 1
- 108090000590 Neurotransmitter Receptors Proteins 0.000 description 1
- 102000004108 Neurotransmitter Receptors Human genes 0.000 description 1
- 102000007607 Non-Receptor Type 11 Protein Tyrosine Phosphatase Human genes 0.000 description 1
- 108010032107 Non-Receptor Type 11 Protein Tyrosine Phosphatase Proteins 0.000 description 1
- 238000000636 Northern blotting Methods 0.000 description 1
- 108091005461 Nucleic proteins Proteins 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 102000010292 Peptide Elongation Factor 1 Human genes 0.000 description 1
- 108010077524 Peptide Elongation Factor 1 Proteins 0.000 description 1
- 108010089430 Phosphoproteins Proteins 0.000 description 1
- 102000007982 Phosphoproteins Human genes 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 208000002151 Pleural effusion Diseases 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 102000001253 Protein Kinase Human genes 0.000 description 1
- 239000012979 RPMI medium Substances 0.000 description 1
- 239000012980 RPMI-1640 medium Substances 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 241000714474 Rous sarcoma virus Species 0.000 description 1
- 102000014400 SH2 domains Human genes 0.000 description 1
- 108050003452 SH2 domains Proteins 0.000 description 1
- 102100029198 SLAM family member 7 Human genes 0.000 description 1
- XZKQVQKUZMAADP-IMJSIDKUSA-N Ser-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(O)=O XZKQVQKUZMAADP-IMJSIDKUSA-N 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 108010071390 Serum Albumin Proteins 0.000 description 1
- 102000007562 Serum Albumin Human genes 0.000 description 1
- 108010074687 Signaling Lymphocytic Activation Molecule Family Member 1 Proteins 0.000 description 1
- 102000008115 Signaling Lymphocytic Activation Molecule Family Member 1 Human genes 0.000 description 1
- 241000700584 Simplexvirus Species 0.000 description 1
- 206010059516 Skin toxicity Diseases 0.000 description 1
- 238000002105 Southern blotting Methods 0.000 description 1
- 230000005867 T cell response Effects 0.000 description 1
- 102100037911 T-cell surface glycoprotein CD3 gamma chain Human genes 0.000 description 1
- 102100037906 T-cell surface glycoprotein CD3 zeta chain Human genes 0.000 description 1
- 102000002259 TNF-Related Apoptosis-Inducing Ligand Receptors Human genes 0.000 description 1
- 108010000449 TNF-Related Apoptosis-Inducing Ligand Receptors Proteins 0.000 description 1
- 108700012411 TNFSF10 Proteins 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 102000006601 Thymidine Kinase Human genes 0.000 description 1
- 108020004440 Thymidine kinase Proteins 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 102100026144 Transferrin receptor protein 1 Human genes 0.000 description 1
- 108090001012 Transforming Growth Factor beta Proteins 0.000 description 1
- 102000004887 Transforming Growth Factor beta Human genes 0.000 description 1
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 1
- 108060008683 Tumor Necrosis Factor Receptor Proteins 0.000 description 1
- 102100040247 Tumor necrosis factor Human genes 0.000 description 1
- 102100024586 Tumor necrosis factor ligand superfamily member 14 Human genes 0.000 description 1
- 102100029690 Tumor necrosis factor receptor superfamily member 13C Human genes 0.000 description 1
- 102100028785 Tumor necrosis factor receptor superfamily member 14 Human genes 0.000 description 1
- 102100035221 Tyrosine-protein kinase Fyn Human genes 0.000 description 1
- 102100024036 Tyrosine-protein kinase Lck Human genes 0.000 description 1
- 102100021657 Tyrosine-protein phosphatase non-receptor type 6 Human genes 0.000 description 1
- 101710128901 Tyrosine-protein phosphatase non-receptor type 6 Proteins 0.000 description 1
- 229960004308 acetylcysteine Drugs 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 210000005006 adaptive immune system Anatomy 0.000 description 1
- 229960000643 adenine Drugs 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 230000006907 apoptotic process Effects 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 239000000823 artificial membrane Substances 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 239000012298 atmosphere Substances 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 210000003651 basophil Anatomy 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 239000011230 binding agent Substances 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 238000010170 biological method Methods 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 239000002981 blocking agent Substances 0.000 description 1
- 239000011575 calcium Substances 0.000 description 1
- 229910052791 calcium Inorganic materials 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 230000020411 cell activation Effects 0.000 description 1
- 230000030833 cell death Effects 0.000 description 1
- 230000011712 cell development Effects 0.000 description 1
- 230000023549 cell-cell signaling Effects 0.000 description 1
- 230000033077 cellular process Effects 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 239000003638 chemical reducing agent Substances 0.000 description 1
- 230000035605 chemotaxis Effects 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 238000012761 co-transfection Methods 0.000 description 1
- 238000001246 colloidal dispersion Methods 0.000 description 1
- 239000000084 colloidal system Substances 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 230000004940 costimulation Effects 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 210000005220 cytoplasmic tail Anatomy 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 1
- 230000007402 cytotoxic response Effects 0.000 description 1
- 238000002784 cytotoxicity assay Methods 0.000 description 1
- 231100000263 cytotoxicity test Toxicity 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 230000000779 depleting effect Effects 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 230000001627 detrimental effect Effects 0.000 description 1
- 230000018109 developmental process Effects 0.000 description 1
- 238000010494 dissociation reaction Methods 0.000 description 1
- 230000005593 dissociations Effects 0.000 description 1
- 231100000673 dose–response relationship Toxicity 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 230000002708 enhancing effect Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000003979 eosinophil Anatomy 0.000 description 1
- 230000001973 epigenetic effect Effects 0.000 description 1
- 230000004049 epigenetic modification Effects 0.000 description 1
- 230000008029 eradication Effects 0.000 description 1
- 108700021358 erbB-1 Genes Proteins 0.000 description 1
- 238000013401 experimental design Methods 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 210000004700 fetal blood Anatomy 0.000 description 1
- 230000001605 fetal effect Effects 0.000 description 1
- 108091006047 fluorescent proteins Proteins 0.000 description 1
- 102000034287 fluorescent proteins Human genes 0.000 description 1
- 239000012595 freezing medium Substances 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 231100000414 gastrointestinal toxicity Toxicity 0.000 description 1
- 238000003197 gene knockdown Methods 0.000 description 1
- 238000012226 gene silencing method Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 239000003862 glucocorticoid Substances 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 150000002339 glycosphingolipids Chemical class 0.000 description 1
- 239000005090 green fluorescent protein Substances 0.000 description 1
- 239000001963 growth medium Substances 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 210000003494 hepatocyte Anatomy 0.000 description 1
- 229940064366 hespan Drugs 0.000 description 1
- 210000004458 heterochromatin Anatomy 0.000 description 1
- 210000003630 histaminocyte Anatomy 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 102000044811 human COLEC12 Human genes 0.000 description 1
- 102000056727 human CXCL16 Human genes 0.000 description 1
- 102000057041 human TNF Human genes 0.000 description 1
- 102000044949 human TNFSF10 Human genes 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- 230000005965 immune activity Effects 0.000 description 1
- 230000006450 immune cell response Effects 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 239000012274 immune-checkpoint protein inhibitor Substances 0.000 description 1
- 230000036039 immunity Effects 0.000 description 1
- 230000005847 immunogenicity Effects 0.000 description 1
- 229940072221 immunoglobulins Drugs 0.000 description 1
- 230000001506 immunosuppresive effect Effects 0.000 description 1
- 239000007943 implant Substances 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 230000028709 inflammatory response Effects 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 102000006029 inositol monophosphatase Human genes 0.000 description 1
- 229940125396 insulin Drugs 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 108010044426 integrins Proteins 0.000 description 1
- 102000006495 integrins Human genes 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 244000144972 livestock Species 0.000 description 1
- 210000004072 lung Anatomy 0.000 description 1
- 210000001165 lymph node Anatomy 0.000 description 1
- 230000002934 lysing effect Effects 0.000 description 1
- 239000011777 magnesium Substances 0.000 description 1
- 229910052749 magnesium Inorganic materials 0.000 description 1
- 230000005291 magnetic effect Effects 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- MYWUZJCMWCOHBA-VIFPVBQESA-N methamphetamine Chemical compound CN[C@@H](C)CC1=CC=CC=C1 MYWUZJCMWCOHBA-VIFPVBQESA-N 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 239000004005 microsphere Substances 0.000 description 1
- 230000000116 mitigating effect Effects 0.000 description 1
- 230000002438 mitochondrial effect Effects 0.000 description 1
- 230000004898 mitochondrial function Effects 0.000 description 1
- 210000005087 mononuclear cell Anatomy 0.000 description 1
- 108700003805 myo-inositol-1 (or 4)-monophosphatase Proteins 0.000 description 1
- 239000002088 nanocapsule Substances 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 238000012259 partial gene deletion Methods 0.000 description 1
- 230000000149 penetrating effect Effects 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 210000005105 peripheral blood lymphocyte Anatomy 0.000 description 1
- 230000002688 persistence Effects 0.000 description 1
- 210000001539 phagocyte Anatomy 0.000 description 1
- 229910052698 phosphorus Inorganic materials 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 238000000053 physical method Methods 0.000 description 1
- 229940081858 plasmalyte a Drugs 0.000 description 1
- 239000013612 plasmid Substances 0.000 description 1
- 238000010837 poor prognosis Methods 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 210000004990 primary immune cell Anatomy 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 229940002612 prodrug Drugs 0.000 description 1
- 239000000651 prodrug Substances 0.000 description 1
- 239000000186 progesterone Substances 0.000 description 1
- 229960003387 progesterone Drugs 0.000 description 1
- 230000002062 proliferating effect Effects 0.000 description 1
- 230000000069 prophylactic effect Effects 0.000 description 1
- 108060006633 protein kinase Proteins 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 210000003370 receptor cell Anatomy 0.000 description 1
- 108010054624 red fluorescent protein Proteins 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 210000003491 skin Anatomy 0.000 description 1
- 231100000438 skin toxicity Toxicity 0.000 description 1
- 229940054269 sodium pyruvate Drugs 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 210000000952 spleen Anatomy 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 230000008093 supporting effect Effects 0.000 description 1
- 239000004094 surface-active agent Substances 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 238000002626 targeted therapy Methods 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 125000003831 tetrazolyl group Chemical group 0.000 description 1
- 238000010257 thawing Methods 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- 238000002560 therapeutic procedure Methods 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 231100000331 toxic Toxicity 0.000 description 1
- 230000002588 toxic effect Effects 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000001890 transfection Methods 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 238000011269 treatment regimen Methods 0.000 description 1
- 230000005909 tumor killing Effects 0.000 description 1
- 102000003298 tumor necrosis factor receptor Human genes 0.000 description 1
- 230000005751 tumor progression Effects 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- VBEQCZHXXJYVRD-GACYYNSASA-N uroanthelone Chemical compound C([C@@H](C(=O)N[C@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CS)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)C(C)C)[C@@H](C)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)CNC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C1=CC=C(O)C=C1 VBEQCZHXXJYVRD-GACYYNSASA-N 0.000 description 1
- 229940088594 vitamin Drugs 0.000 description 1
- 239000011782 vitamin Substances 0.000 description 1
- 235000013343 vitamin Nutrition 0.000 description 1
- 229930003231 vitamin Natural products 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K35/00—Medicinal preparations containing materials or reaction products thereof with undetermined constitution
- A61K35/12—Materials from mammals; Compositions comprising non-specified tissues or cells; Compositions comprising non-embryonic stem cells; Genetically modified cells
- A61K35/14—Blood; Artificial blood
- A61K35/17—Lymphocytes; B-cells; T-cells; Natural killer cells; Interferon-activated or cytokine-activated lymphocytes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0636—T lymphocytes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K35/00—Medicinal preparations containing materials or reaction products thereof with undetermined constitution
- A61K35/12—Materials from mammals; Compositions comprising non-specified tissues or cells; Compositions comprising non-embryonic stem cells; Genetically modified cells
- A61K35/14—Blood; Artificial blood
- A61K35/15—Cells of the myeloid line, e.g. granulocytes, basophils, eosinophils, neutrophils, leucocytes, monocytes, macrophages or mast cells; Myeloid precursor cells; Antigen-presenting cells, e.g. dendritic cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/461—Cellular immunotherapy characterised by the cell type used
- A61K39/4611—T-cells, e.g. tumor infiltrating lymphocytes [TIL], lymphokine-activated killer cells [LAK] or regulatory T cells [Treg]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/463—Cellular immunotherapy characterised by recombinant expression
- A61K39/4631—Chimeric Antigen Receptors [CAR]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/4643—Vertebrate antigens
- A61K39/4644—Cancer antigens
- A61K39/464402—Receptors, cell surface antigens or cell surface determinants
- A61K39/464403—Receptors for growth factors
- A61K39/464404—Epidermal growth factor receptors [EGFR]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/7051—T-cell receptor (TcR)-CD3 complex
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70539—MHC-molecules, e.g. HLA-molecules
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
- C07K16/2833—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily against MHC-molecules, e.g. HLA-molecules
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2863—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for growth factors, growth regulators
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0693—Tumour cells; Cancer cells
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/60—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments
- C07K2317/62—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments comprising only variable region components
- C07K2317/622—Single chain antibody (scFv)
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/02—Fusion polypeptide containing a localisation/targetting motif containing a signal sequence
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/03—Fusion polypeptide containing a localisation/targetting motif containing a transmembrane segment
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2501/00—Active agents used in cell culture processes, e.g. differentation
- C12N2501/10—Growth factors
- C12N2501/11—Epidermal growth factor [EGF]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2501/00—Active agents used in cell culture processes, e.g. differentation
- C12N2501/50—Cell markers; Cell surface determinants
- C12N2501/515—CD3, T-cell receptor complex
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Immunology (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Medicinal Chemistry (AREA)
- Genetics & Genomics (AREA)
- Cell Biology (AREA)
- Biochemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Biophysics (AREA)
- Molecular Biology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Public Health (AREA)
- Animal Behavior & Ethology (AREA)
- Biomedical Technology (AREA)
- Microbiology (AREA)
- Epidemiology (AREA)
- Biotechnology (AREA)
- Mycology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Toxicology (AREA)
- Gastroenterology & Hepatology (AREA)
- Oncology (AREA)
- Hematology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- General Engineering & Computer Science (AREA)
- Virology (AREA)
- Dermatology (AREA)
- Developmental Biology & Embryology (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
본 개시내용은 제1 활성화제 수용체 및 제2 억제 수용체를 포함하는 면역 세포, 및 암의 치료를 위해 이를 제조하고 사용하는 방법을 제공한다.
Description
관련 출원
본 출원은 2020년 8월 20일에 출원된 미국 가출원 번호 63/068,249; 2020년 10월 26일에 출원된 미국 가출원 번호 63/105,639; 및 2021년 8월 6일에 출원된 미국 가출원 번호 63/230,632에 대한 우선권을 주장하고, 각각의 내용은 전체가 참조로 본원에 포함된다.
기술 분야
본 출원은 일반적으로 세포 요법 적용에 사용하기 위한 조작된 수용체 및 상기 수용체를 포함하는 면역 세포 조성물에 관한 것이다.
서열 목록에 대한 참조
서열 목록 단락 출원서는 EFS-WEB를 통해 ASCII 형식으로 제출된 서열 목록을 포함하며 이로써 전체가 참조로 포함된다. 2021년 8월 17일에 생성된 상기 ASCII 본의 이름은 A2BI_021_02WO_SeqList_ST25.txt이고 675 KB 크기이다.
세포 요법은 다양한 질병, 특히 암 치료를 위한 강력한 도구이다. 기존의 입양 세포 요법에서, 면역 세포는 특정 수용체, 예를 들어 키메라 항원 수용체 (CAR) 또는 T 세포 수용체 (TCR)를 발현하도록 조작되며, 이는 수용체와 표적 세포에 의해 발현되는 리간드의 상호작용을 통해 면역 세포의 활성을 세포 표적으로 유도한다. 많은 표적이 정상 조직에서 발현되기 때문에 적합한 표적 분자를 식별하는 것은 여전히 어려운 일이다. 이 발현은 이식된 세포가 표적 분자를 발현하는 정상 조직을 표적으로 할 때 독성을 유발할 수 있다. 따라서, 입양 세포 요법에 의한 질환, 특히 암의 치료에 유용한 조성물 및 방법이 당업계에 필요하다.
본 개시내용은 입양 세포 요법에 사용되는 면역 세포의 특이성을 증가시키기 위한 조성물 및 방법을 제공한다. 본 개시내용은 표적 항원을 발현하는 표적 세포에 대해 면역 세포의 특이성을 증가시키는 2-수용체 시스템을 포함하는 면역 세포를 제공한다. 면역 세포는 표적 항원에 의한 제1 수용체의 결합에 반응하여 면역 세포를 활성화시키는 제1 활성화제 수용체를 포함한다. 면역 세포는 비-표적 항원에 특이적인 제2 억제 수용체를 추가로 포함한다. 이 제2 수용체는 제1 수용체가 표적 항원과 결합하더라도 제2 수용체가 비-표적 항원과 결합하면 면역세포의 활성화를 억제한다.
한 양태에서, 본 개시내용은 a.) 표피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및 b.) 이형접합성의 소실로 인해 EGFR+ 암에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체를 포함하는 면역 세포를 제공하며, 여기서 제1 수용체는 EGFR에 반응하는 활성화제 수용체이고; 여기서 제2 수용체는 비-표적 항원에 반응하는 억제 수용체이다.
일부 구현예에서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 MHC 단백질의 대립형질 변이체에 특이적으로 결합한다. 일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A, HLA-B, 또는 HLA-C 단백질의 대립형질 변이체에 특이적으로 결합한다. 일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*02에 특이적으로 결합한다. 일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*01, HLA-A*03, HLA-A*11, HLA-C*07, 또는 HLA-B*07에 특이적으로 결합한다.
일부 구현예에서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 표 5에 개시된 바와 같은 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함한다.
일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 서열 번호 101-106 또는 서열 번호 106-112의 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함한다.
일부 구현예에서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 표 4에 개시된 폴리펩티드 서열로부터 선택된 폴리펩티드 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함한다. 일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 서열 번호 89-100 중 하나; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 키메라 항원 수용체 (CAR)이다.
일부 구현예에서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 표 3에 제시된 서열 그룹으로부터 선택된 중쇄 상보성 결정 영역 (HC-CDR) 세트를 포함하는 가변 중쇄 (VH) 부분; 및/또는 표 3에 제시된 서열 그룹의 경쇄 상보성 결정 영역 (LC-CDR) 세트를 포함하는 가변 경쇄 (VL) 부분; 또는 각각의 CDR에서 최대 1, 2, 3, 4개의 치환, 삽입, 또는 결실을 갖는 CDR 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 표 2에 제시된 VH 서열로부터 선택된 서열을 갖는 가변 중쇄 (VH) 부분; 및/또는 표 2에 제시된 서열을 포함하는 가변 경쇄 (VL) 부분; 또는 이에 대해 적어도 70%, 적어도 85%, 적어도 90%, 또는 적어도 95% 동일성을 갖는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 표 1에 제시된 서열 그룹으로부터 선택된 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97%, 또는 적어도 99% 동일성을 갖는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 9-18로 이루어진 그룹으로부터 선택된 scFv 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함한다.
일부 구현예에서, 상기 제2 수용체는 LILRB1 세포 내 도메인 또는 이의 기능적 변이체를 포함한다. 일부 구현예에서, 상기 LILRB1 세포 내 도메인은 서열 번호 129에 대해 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한, 또는 동일한 서열을 포함한다.
일부 구현예에서, 상기 제2 수용체는 LILRB1 막관통 도메인 또는 이의 기능적 변이체를 포함한다. 일부 구현예에서, 상기 LILRB1 막관통 도메인 또는 이의 기능적 변이체는 서열 번호 133에 대해 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함한다.
일부 구현예에서, 상기 제2 수용체는 LILRB1 힌지 도메인 또는 이의 기능적 변이체를 포함한다. 일부 구현예에서, 상기 LILRB1 힌지 도메인은 서열 번호 132, 서열 번호 125, 서열 번호 126에 대해 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함한다.
일부 구현예에서, 상기 제2 수용체는 LILRB1 세포 내 도메인, LILRB1 막관통 도메인, LILRB1 힌지 도메인, 이들 중 임의의 것의 기능적 변이체, 또는 이들의 조합을 포함한다. 일부 구현예에서, 상기 LILRB1 세포 내 도메인 및 LILRB1 막관통 도메인은 서열 번호 128 또는 서열 번호 128에 대해 적어도 95% 동일한 서열을 포함한다.
일부 구현예에서, 상기 EGFR+ 암 세포는 폐암 세포, 소세포 폐암 세포, 비-소세포 폐암 세포, 췌관 암종 세포, 결장직장암 세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포, 또는 자궁암 세포이다.
일부 구현예에서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포이다. 일부 구현예에서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A*02의 소실을 야기하는 HLA-A에서의 이형접합성의 소실에 의해 EGFR+/HLA-A*02+ 세포로부터 유래된다.
일부 구현예에서, 상기 제1 수용체 및 제2 수용체는 함께 이형접합성을 소실한 EGFR/HLA-A*02- 암 세포의 존재 하에 면역 세포를 특이적으로 활성화시킨다.
일부 구현예에서, 상기 제1 수용체 및 제2 수용체는 함께 이형접합성 소실에 의해 HLA-A*02를 소실하지 않은 EGFR+ 세포의 존재 하에 면역 세포를 특이적으로 활성화시키지 않는다.
일부 구현예에서, 상기 면역 세포는 T 세포, 대식세포, NK 세포, iNKT 세포, 또는 감마 델타 T 세포이다. 일부 구현예에서, 상기 T 세포는 CD8+ CD4- T 세포이다.
일부 구현예에서, 상기 면역 세포는 MHC 클래스 I 유전자의 감소된 또는 제거된 발현 및/또는 기능을 추가로 포함한다.
일부 구현예에서, 상기 MHC 클래스 I 유전자는 베타-2-마이크로글로불린 (B2M)이다.
일부 구현예에서, 상기 면역 세포는 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함하고, 상기 간섭 RNA는 B2M mRNA의 서열 (서열 번호 172)에 상보적인 서열을 포함한다. 일부 구현예에서, 상기 간섭 RNA는 B2M mRNA의 RNAi-매개 분해를 유도할 수 있다. 일부 구현예에서, 상기 간섭 RNA는 짧은 헤어핀 RNA (shRNA)이다. 일부 구현예에서, 상기 shRNA는 a.) 5' 말단에서 3' 말단으로 B2M mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및 b.) 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열을 포함하고, 여기서 제1 서열 및 제2 서열은 shRNA를 형성한다.
일부 구현예에서, 상기 면역 세포는 B2M을 암호화하는 서열 (서열 번호 170)에 대한 하나 이상의 변형을 포함하며, 여기서 하나 이상의 변형은 B2M의 발현을 감소시키고/시키거나 기능을 제거한다. 일부 구현예에서, 하나 이상의 변형은 B2M을 암호화하는 내인성 유전자의 하나 이상의 불활성화 돌연변이를 포함한다. 일부 구현예에서, 하나 이상의 불활성화 돌연변이는 결실, 삽입, 치환 또는 프레임이동 돌연변이를 포함한다. 일부 구현예에서, 하나 이상의 불활성화 돌연변이는 B2M을 암호화하는 내인성 유전자의 서열 (서열 번호 170)을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)과의 복합체에서 핵산 유도 엔도뉴클레아제와 함께 도입된다.
일부 구현예에서, 상기 MHC 클래스 I 유전자는 HLA-A*02이다.
일부 구현예에서, 상기 면역 세포는 HLA-A*02 mRNA의 서열 (서열 번호 171)에 상보적인 서열을 포함하는 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함한다. 일부 구현예에서, 상기 간섭 RNA는 HLA-A*02 mRNA의 RNA 간섭 (RNAi)-매개 분해를 유도할 수 있다. 일부 구현예에서, 상기 간섭 RNA는 a.) 5' 말단에서 3' 말단으로 HLA-A*02 mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및 b.) 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열을 포함하는 짧은 헤어핀 RNA (shRNA)이고, 여기서 제1 서열 및 제2 서열은 shRNA를 형성한다.
일부 구현예에서, 상기 면역 세포는 HLA-A*02을 암호화하는 내인성 유전자의 서열 (서열 번호 169)에 대해 하나 이상의 변형을 포함하고, 여기서 하나 이상의 변형은 HLA-A*02의 발현을 감소시키고/시키거나 기능을 제거한다. 일부 구현예에서, 하나 이상의 변형은 HLA-A*02를 암호화하는 유전자의 하나 이상의 불활성화 돌연변이를 포함한다. 일부 구현예에서, 하나 이상의 불활성화 돌연변이는 결실, 삽입, 치환 또는 프레임이동 돌연변이를 포함한다. 일부 구현예에서, 하나 이상의 불활성화 돌연변이는 HLA-A*02를 암호화하는 내인성 유전자의 서열을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)과의 복합체에서 핵산 유도 엔도뉴클레아제와 함께 도입된다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 174 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 174, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 면역 세포는 T2A 자가 절단 펩티드를 추가로 포함하고, 여기서 T2A 자가 절단 펩티드는 서열 번호 178, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 면역 세포는 간섭 RNA를 추가로 포함하고, 여기서 간섭 RNA는 서열 번호 179, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 상기 면역 세포는 자가 세포이다. 일부 구현예에서, 면역 세포는 동종이계이다.
한 양태에서, 본 개시내용은 치료 유효량의 본원에 기재된 면역 세포를 포함하는 약학 조성물을 제공한다. 일부 구현예에서, 상기 면역 세포는 제1 수용체 및 제2 수용체 모두를 발현한다. 일부 구현예에서, 적어도 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 또는 약 95%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현한다. 일부 구현예에서, 적어도 90%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현한다. 일부 구현예에서, 상기 약학 조성물은 약학적으로 허용되는 담체, 희석제 또는 부형제를 추가로 포함한다. 일부 구현예에서, 본원에 기재된 약학 조성물은 EGFR+ 암의 치료에서 약제로 사용하기 위한 것이다.
한 양태에서, 본 개시내용은 다음을 암호화하는 하나 이상의 폴리뉴클레오티드를 포함하는, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템을 제공하며: a.) 내피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및 b.) 이형접합성의 소실로 인해 EGFR+ 암 세포에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체, 여기서 제1 수용체는 EGFR+ 암세포 상의 EGFR에 반응하는 활성화제 수용체이고; 여기서 제2 수용체는 비표적 항원에 반응하는 억제 수용체이다.
한 양태에서, 본 개시내용은 본원에 기재된 면역 세포를 생성하는데 사용하기 위한 제1 수용체 및 제2 수용체를 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하는 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템을 제공한다.
본 개시내용의 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템의 일부 구현예에서, 상기 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 B2M에 특이적인 shRNA를 암호화하는 서열을 포함한다. 일부 구현예에서, 제1 수용체, 제2 수용체, 및 B2M에 특이적인 shRNA를 암호화하는 서열은 동일한 폴리뉴클레오티드에 의해 암호화된다.
한 양태에서, 본 개시내용은 본원에 기재된 하나 이상의 폴리뉴클레오티드를 포함하는 벡터를 제공한다.
한 양태에서, 본 개시내용은 유효량의 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물을 대상체에게 투여하는 것을 포함하는, MHC 클래스 I 유전자좌에서 이형접합성이 소실된 EGFR+ 암 세포를 사멸시키는 방법을 제공한다.
일부 구현예에서, 상기 EGFR+ 암 세포는 폐암 세포, 소세포 폐암 세포, 비-소세포 폐암 세포, 췌관 암종 세포, 결장직장암 세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포, 또는 자궁암 세포이다. 일부 구현예에서, 상기 EGFR+ 암 세포는 폐암 세포이다.
일부 구현예에서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포이다. 일부 구현예에서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A*02의 소실을 야기하는 HLA-A에서 이형접합성의 소실로 인해 EGFR+/HLA-A*02+ 세포로부터 유래된다.
한 양태에서, 본 개시내용은 유효량의 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물을 대상체에게 투여하는 것을 포함하는, 비-표적 항원을 암호화하는 유전자좌에서 이형접합성의 소실을 갖는 EGFR+ 종양을 갖는 대상체에서 EGFR+ 암을 치료하는 방법을 제공한다.
일부 구현예에서, 상기 대상체는 EGFR을 발현하고 HLA-A*02 발현을 상실한 악성종양을 갖는 이형접합 HLA-A*02 환자이다.
일부 구현예에서, 상기 대상체는 EGFR을 발현하고 HLA-A*02 발현을 상실한 재발성 절제 불가능 또는 전이성 고형 종양을 갖는 이형접합 HLA-A*02 환자이다.
한 양태에서, 본 개시내용은 다음 단계를 포함하는 대상체에서 암을 치료하는 방법을 제공한다: a.) 대상체의 비-악성 세포 및 암 세포에서 비-표적 항원의 유전자형 또는 발현 수준을 결정하는 단계; b.) 대상체의 암 세포에서 EGFR의 발현 수준을 결정하는 단계; 및 c.) 비-악성 세포가 비-표적 항원을 발현하고 암 세포가 비-표적 항원을 발현하지 않고, 암세포는 EGFR 양성이면 유효량의 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물을 대상체에게 투여하는 단계.
일부 구현예에서, 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물의 투여는 대상체에서 종양의 크기를 감소시킨다.
일부 구현예에서, 상기 종양은 약 5%, 약 10%, 약 15%, 약 20%, 약 25%, 약 30%, 약 35%, 약 40%, 약 45%, 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 약 95%, 또는 약 100% 만큼 감소된다. 일부 구현예에서, 종양은 제거된다.
일부 구현예에서, 면역 세포 또는 약학 조성물의 투여는 대상체에서 종양의 성장을 정지시킨다. 일부 구현예에서, 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물의 투여는 대상체에서 종양의 수를 감소시킨다.
일부 구현예에서, 면역 세포 또는 약학 조성물의 투여는 암 세포를 선택적으로 사멸시키지만 대상체에서 정상 세포는 사멸시키지 않는다. 일부 구현예에서, 사멸된 세포의 적어도 약 60%는 암세포이고, 사멸된 세포의 적어도 약 65%는 암세포이고, 사멸된 세포의 적어도 약 70%는 암세포이고, 사멸된 세포의 적어도 약 75%는 암세포이고, 사멸된 세포의 적어도 약 80%는 암세포이고, 사멸된 세포의 적어도 약 85%는 암세포이고, 사멸된 세포의 적어도 약 90%는 암세포이고, 사멸된 세포의 적어도 약 95%는 암세포이고, 또는 사멸된 세포의 약 100%가 암세포이다.
일부 구현예에서, 면역 세포 또는 약학 조성물의 투여는 대상체의 암 세포의 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90% 또는 전부를 사멸시킨다. 일부 구현예에서, 상기 암 세포는 폐암 세포, 소세포 폐암 세포, 비-소세포 폐암 세포, 췌관 암종 세포, 결장직장암 세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포, 또는 자궁암 세포를 포함한다. 일부 구현예에서, 면역 세포 또는 약학 조성물의 투여는 제1 활성화제 수용체를 포함하지만 제2 억제 수용체를 포함하지 않는 다른 동등한 면역 세포의 투여보다 대상체에 대해 더 적은 부작용을 초래한다.
한 양태에서, 본 개시내용은 a.) 다수의 면역 세포를 제공하는 단계, 및 b.) 다수의 면역 세포를 본원에 기재된 폴리뉴클레오티드 시스템, 또는 본원에 기재된 벡터로 형질전환시키는 단계를 포함하는, 다수의 면역 세포를 제조하는 방법을 제공한다.
한 양태에서, 본 개시내용은 본원에 기재된 면역 세포 또는 본원에 기재된 약학 조성물을 포함하는 키트를 제공한다. 일부 구현예에서, 상기 키트는 사용 설명서를 추가로 포함한다.
특허 또는 출원 파일에는 컬러로 실행된 도면이 적어도 하나 포함되어 있다. 컬러 드로잉(들)이 포함된 이 특허 또는 특허 출원 공개의 사본은 요청 및 필요한 수수료 지불 시 사무소에서 제공된다.
도 1a는 EGFR CAR 활성화제 및 HLA-A*02 LIR-1 차단제를 발현하는 저캇 세포가 EGFR+/HLA-A*02- HeLa 표적 세포에 의해 활성화되지만 EGFR+/HLA-A*02+ HeLa 표적 세포에 의해 활성화되지 않음을 나타내는 플롯이다.
도 1b는 저캇 세포 상의 활성화제 (CT479) 및 차단제 (C1765) 수용체의 발현을 나타내는 한 쌍의 형광 활성화 세포 분류 (FACS) 플롯이다.
도 1c는 HeLa 세포 상의 활성화제 항원 (EGFR) 및 차단제 항원 (HLA-A*02)의 발현을 나타내는 한 쌍의 플롯이다. HLA-A*02 발현은 BB7.2 항체를 사용하여 검출하였다.
도 2a는 HLA-A*02, 및 HCT116 세포로 형질도입된 HeLa 세포 상의 HLA-A*02의 발현을 나타낸다. HeLa 및 HCT1116 세포는 항-HLA-A2 항체 BB7.2로 표지되고 FAC 분류하였다. 녹색: 표지되지 않은 HeLa; 주황색: 표지되지 않은 HCT116; 파란색: BB7.2로 표지된 야생형 HCT116; 빨간색: HLA-A*02로 형질도입되고 BB7.2로 표지된 HeLa 세포.
도 2b는 HeLa 세포 및 HCT116 세포 상의 EGFR의 발현을 나타낸다. HeLa 및 HCT1116 세포는 항-EGFR 항체로 표지되었다. 녹색: 표지되지 않은 HeLa; 주황색: 표지되지 않은 HCT1116; 파란색: 항-EGFR로 표지된 야생형 HCT116; 빨간색: HLA-A*02로 형질도입되고 항-EGFR로 표지된 HeLa 세포.
도 3a는 HCT116 표적 세포를 공동 배양할 때, EGFR CAR을 발현하는 저캇 세포의 EGFR CAR 활성화를 나타낸다.
도 3b는 저캇 세포의 EGFR CAR 활성화가 HLA-A*02 LIR-1 억제 수용체에 의해 차단될 수 있음을 보여준다. 저캇 세포에 의한 EGFR CAR 및 HLA-A*02 LIR-1 억제 수용체의 공동 발현은 저캇 세포가 EGFR 및 HLA-A*02를 발현하는 HCT116 표적 세포와 함께 제시될 때 대략 1.8x의 CAR EMAX 이동을 유도한다.
도 3c는 HeLa 표적 세포와 함께 공동-배양될 때, EGFR CAR를 발현하는 저캇 세포의 EGFR CAR 활성화를 나타낸다.
도 3d는 저캇 세포의 EGFR CAR 활성화가 HLA-A*02 LIR-1 억제 수용체에 의해 차단될 수 있음을 보여준다. 저캇 세포에 의한 EGFR CAR 및 HLA-A*02 LIR-1 억제 수용체의 공동 발현은 저캇 세포가 EGFR 및 HLA-A*02를 발현하는 HeLa 표적 세포와 함께 제시될 때 대략 5.4x의 CAR EMAX 이동을 유도한다.
도 4a는 활성화제 항원의 최적량을 결정하기 위한 비드 기반 검정에서 활성화제 항원의 적정을 보여준다.
도 4b는 활성화제 대 차단제 항원의 최적 비율을 결정하기 위한 비드 기반 검정에서 차단제 항원의 적정을 보여준다.
도 5는 HLA-A*02 scFv LIR1 억제제가 1차 T 세포에서 상이한 EGFR scFv CAR 활성화제와 함께 사용될 때 상이한 차단 정도가 관찰됨을 보여주는 일련의 플롯 및 표이다.
도 6a는 2개의 상이한 HLA-A*02 scFv LIR1 억제제가 1차 T 세포에서 상이한 EGFR scFv CAR 활성화제와 함께 사용될 때 상이한 차단 정도가 관찰됨을 보여주는 일련의 플롯이다.
도 6b는 상이한 쌍의 활성화 및 억제 수용체가 공동-발현될 때 활성화 및 억제 수용체의 민감도를 나타내는 일련의 플롯이다. 활성화제 또는 비-표적 항원의 양은 mRNA 적정에 의해 표적 세포에서 다양하다.
도 6c는 EGFR 표적화 활성화제 수용체 및 인간화 억제 수용체 또는 마우스 억제 수용체를 발현하는 세포에 의한 표적 세포의 최대 특이적 사멸 (%)을 나타내는 일련의 차트이다. 표적 종양 세포 대 정상 세포의 선택성 비율은 또한 활성화제 및 억제 수용체의 각 쌍에 대해 나타낸다.
도 7은 HeLa 세포와 1:1 비율로 공동 배양된 EGFR scFv CAR, 또는 EGFR scFv Car 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 나타내는 일련의 플롯이다. A 종양 세포: HLA-A*02가 아닌 EGFR를 발현하는 HeLa 세포; AB 정상 세포: HeLa 세포 발현 EGFR 및 HLA-A*02; A-B- 정상 세포: EGFR 또는 HLA-A*02를 발현하지 않는 HeLa 세포; B 정상 세포: EGFR이 아닌 HLA-A*02를 발현하는 HeLa 세포. Y 축은 분석에서 건강한 HeLa 세포에 비례하는 녹색 영역을 나타낸다. UTD: 형질도입되지 않음.
도 8은 차단제 및 활성화제 수용체를 발현하는 1차 T 세포의 농축 (상단 열), EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 이들 T 세포의 세포독성 활성을 나타내는 일련의 플롯이고, EGFR (표적 A), 또는 EGFR 및 HLA-A*02 (표적 AB)를 발현하는 HeLa 세포와 1:1로 혼합했다.
도 9는 EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 보여주는 일련의 플롯이며, 이는 EGFR, HLA-A*02, 또는 둘 다 발현하는 HeLa 세포와 1:1로 혼합되었다. A 종양 세포: HLA-A*02가 아닌 EGFR을 발현하는 HeLa 세포; AB 정상 세포: EGFR 및 HLA-A*02를 발현하는 HeLa 세포; B 정상 세포: EGFR이 아닌 HLA-A*02를 발현하는 HeLa 세포. Y축은 분석에서 건강한 HeLa 세포에 비례하는 녹색 영역을 나타낸다. UTD: 형질도입되지 않음.
도 10은 EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 보여주는 일련의 플롯이며, 이는 EGFR (표적 A) 또는 EGFR 및 HLA-A*02 (표적 AB)를 발현하는 HCT.116 세포와 1:1로 혼합되었다.
도 11a는 상이한 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제제를 발현하는 T 세포와 EGFR 활성화제 단독 (표적 A), 억제제 표적 단독 (표적 B) 또는 활성화제 및 억제제 표적 (표적 AB)을 발현하는 HeLa 세포의 배양 후 T 세포에 의한 EGFR scFv CAR 활성화제 수용체의 발현을 나타내는 일련의 형광 활성화 세포 분류 (FACS) 플롯이다.
도 11b는 표적 세포에 노출되기 전, 및 활성화제 리간드 단독 발현하는 표적 세포 (표적 A), 또는 활성화제 및 차단제 리간드를 모두 발현하는 표적 세포 (표적 AB)와의 공동 배양 120시간 후 정량 활성화제 수용체 발현을 나타내는 플롯이다.
도 12a는 EGFR (표적 A), HLA-A*02 (표적 B)를 발현하는 HeLa 세포의 집단, 동일한 세포 상의 EGFR 및 HLA-A*02의 조합 (표적 AB), 상이한 세포 상에 표적 A 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단, 또는 상이한 세포 상에 표적 B 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단과의 공동 배양 후 EGFR scFv CAR (CT-482) 활성화제 및 HLA-A*02 scFv LIR1 억제제 (C1765)를 발현하는 T 세포 상의 활성화제 수용체의 세포 표면 발현을 나타내는 플롯이다.
도 12b는 EGFR (표적 A), HLA-A*02 (표적 B)를 발현하는 HeLa 세포의 집단, 동일한 세포 상의 EGFR 및 HLA-A*02의 집단 (표적 AB), 상이한 세포 상에 표적 A 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단, 또는 상이한 세포 상에 표적 B 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단과의 공동 배양 후 EGFR scFv CAR (CT-482) 활성화제 및 HLA-A*02 scFv LIR1 억제제 (C1765)를 발현하는 T 세포 상의 억제 수용체의 세포 표면 발현을 나타내는 플롯이다.
도 13은 T 세포에 의한 세포독성 또는 차단 활성이 가역적인지 알아보기 위한 실험도이다.
도 14a는 세포독성 및 차단 활성이 가역적이고 활성화제 표면 발현에 상응함을 보여주는 일련의 플롯이다. T 세포는 1 라운드에서 EGFR+/HLA-A2+ 표적 세포와 공동 배양된 다음, 다음 라운드에서 교대 표적 세포로 전환했다. 상단: T 세포에 의한 표적 HeLa 세포의 사멸 퍼센트가 표시된다. 하단: FACS에 의해 분석된 활성화제 및 억제 수용체 발현.
도 14b는 세포독성 및 차단 활성이 가역적이고 활성화제 표면 발현에 상응함을 보여주는 일련의 플롯이다. T 세포는 1 라운드에서 EGFR+/HLA-A2- 표적 세포와 공동 배양한 다음, 다음 라운드에서 교대 표적 세포로 전환했다. 상단: T 세포에 의한 표적 HeLa 세포의 사멸 퍼센트가 표시된다. 하단: FACS에 의해 분석된 활성화제 및 억제 수용체 발현.
도 15a는 세포 표면 상의 EGFR 활성화제 CAR의 발현이 활성화제 및 억제제 리간드를 모두 발현하는 표적 세포의 존재에 의해 가역적으로 하향 조절됨을 보여주는 플롯이다.
도 15b는 활성화제만 표적 세포가 존재하거나, 활성화제 및 차단제 표적을 발현하는 표적 세포가 존재하지 않을 때 세포 표면 상의 EGFR 활성화제 CAR 발현이 신속하게 회복됨을 보여주는 일련의 플롯이다.
도 16은 비드 기반 검정을 사용하여 EGFR scFv CAR을 발현하는 저캇 세포의 활성화가 활성화제 및 억제제 항원이 시스로 비드 상에 존재하지만, 활성화제 및 억제제 항원이 트랜스로 비드 상에 존재하지 않을 때 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 17a는 EGFR scFv CAR에 의한 저캇 세포의 활성화가 HLA-A*02를 발현하지 않는 SiHa 세포 (SiHa WT)에 의해서가 아니라, HLA-A*02를 발현하는 SiHa 표적 세포 (SiHa A02)를 사용하여 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 17b는 EGFR scFv CAR에 의한 저캇 세포의 활성화가 HLA-A*02를 발현하지 않는 HeLa 세포 (HeLa WT)에 의해서가 아니라, HLA-A*02를 발현하는 HeLa 표적 세포 (HeLa A02)를 사용하여 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 18은 잠재적인 비-표적 항원 후보 유전자를 확인하기 위해 사용되는 생물정보학 검색 과정을 나타낸 도면이다.
도 19는 생체 내 활성화제 수용체 및 억제 수용체를 표적으로 하는 EGFR을 발현하는 면역 세포의 효능 및 선택성을 결정하기 위한 실험 설계를 나타내는 도면이다.
도 20a는 HLA-A*11을 암호화하는 mRNA로 형질감염된 HeLa 세포를 사용하는 mRNA 적정 검정에서 저캇 NFAT 루시퍼라제 (JNL) 세포 억제를 나타내는 플롯이다. HeLa 세포는 연속 희석된 HLA-A*11 mRNA로 형질감염되었고, JNL 세포는 scFv HLA-A*11 #4와 함께 EGFR 활성화 CAR +/- HLA-A*11 억제 수용체로 일시적으로 형질감염되었다. 기능적 반응은 6시간 공동 배양 후 평가되었다.
도 20b는 HLA-A*11 #4를 사용한 억제 수용체의 분자/세포 민감도 (IC50)를 나타낸다.
도 21은 HLA-B*07-scFv-LIR1 억제 수용체가 공동-발현된 EGFR 표적화 활성화제 수용체에 의해 매개되는 면역 세포 활성화를 억제한다는 것을 보여주는 플롯이다.
도 22는 본원에 개시된 벡터를 포함하는 1차 T 세포에서 EGFR 활성화제 수용체 (EGFR-CAR 단독) 및 EGFR 활성화제/HLA-A 억제 수용체 쌍의 발현을 나타내는 일련의 플롯이다. 하나 또는 두 개의 수용체를 발현하는 세포의 백분율은 농축 전 (전 농축)과 농축 후 (후 농축)로 표시되며 농축은 수용체를 발현하는 세포를 선택한다. 수용체 쌍은 두 개의 개별 벡터 (이중 벡터) 또는 하나의 벡터 (단일 벡터)로 표현된다.
도 23a는 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 HeLa 표적 세포 또는 HCT116 표적 세포의 특이적 사멸 백분율을 나타내는 일련의 플롯이다. 표적 세포는 EGFR 항원 (A)을 발현하거나, EGFR 항원 및 HLA-A*02 비-표적 항원 (B) 모두 발현한다. 활성화제/억제제 수용체 쌍을 발현하는 면역 세포를 1:1 비율로 표적 세포와 함께 배양하였다.
도 23b는 암 세포 및 정상 세포의 혼합 배양물에서 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 표적 세포의 특이적 선택적 사멸을 보여주는 플롯 및 일련의 이미지이다. UTD = 활성화제 또는 억제제 수용체가 없는 면역 세포; CAR = 활성화제 수용체만을 발현하는 면역 세포; Tmod = 활성화제 및 억제제 수용체를 모두 발현하는 면역 세포. 오른쪽 이미지에서: 녹색 = 정상 세포; 빨간색 = 종양 세포.
도 24는 EGFR 활성화제 수용체 (CAR) 또는 EGFR 활성화제 수용체 및 HLA-A*02 억제제 수용체 (Tmod)를 발현하는 저캇 세포 (효과기)에서 선택적 세포 활성화를 나타내는 차트이다. 저캇 세포는 EGFR 발현 유무에 관계없이 HLA-A*02를 발현하는 표적 세포주의 패널과 함께 배양되었다. Tmod 저캇 효과기 세포는 HLA-A*02를 발현하는 세포의 EGFR.을 발현하지 않는 표적 세포와 공동 배양했을 때 오프-타겟 활성화를 나타내지 않았다.
도 1a는 EGFR CAR 활성화제 및 HLA-A*02 LIR-1 차단제를 발현하는 저캇 세포가 EGFR+/HLA-A*02- HeLa 표적 세포에 의해 활성화되지만 EGFR+/HLA-A*02+ HeLa 표적 세포에 의해 활성화되지 않음을 나타내는 플롯이다.
도 1b는 저캇 세포 상의 활성화제 (CT479) 및 차단제 (C1765) 수용체의 발현을 나타내는 한 쌍의 형광 활성화 세포 분류 (FACS) 플롯이다.
도 1c는 HeLa 세포 상의 활성화제 항원 (EGFR) 및 차단제 항원 (HLA-A*02)의 발현을 나타내는 한 쌍의 플롯이다. HLA-A*02 발현은 BB7.2 항체를 사용하여 검출하였다.
도 2a는 HLA-A*02, 및 HCT116 세포로 형질도입된 HeLa 세포 상의 HLA-A*02의 발현을 나타낸다. HeLa 및 HCT1116 세포는 항-HLA-A2 항체 BB7.2로 표지되고 FAC 분류하였다. 녹색: 표지되지 않은 HeLa; 주황색: 표지되지 않은 HCT116; 파란색: BB7.2로 표지된 야생형 HCT116; 빨간색: HLA-A*02로 형질도입되고 BB7.2로 표지된 HeLa 세포.
도 2b는 HeLa 세포 및 HCT116 세포 상의 EGFR의 발현을 나타낸다. HeLa 및 HCT1116 세포는 항-EGFR 항체로 표지되었다. 녹색: 표지되지 않은 HeLa; 주황색: 표지되지 않은 HCT1116; 파란색: 항-EGFR로 표지된 야생형 HCT116; 빨간색: HLA-A*02로 형질도입되고 항-EGFR로 표지된 HeLa 세포.
도 3a는 HCT116 표적 세포를 공동 배양할 때, EGFR CAR을 발현하는 저캇 세포의 EGFR CAR 활성화를 나타낸다.
도 3b는 저캇 세포의 EGFR CAR 활성화가 HLA-A*02 LIR-1 억제 수용체에 의해 차단될 수 있음을 보여준다. 저캇 세포에 의한 EGFR CAR 및 HLA-A*02 LIR-1 억제 수용체의 공동 발현은 저캇 세포가 EGFR 및 HLA-A*02를 발현하는 HCT116 표적 세포와 함께 제시될 때 대략 1.8x의 CAR EMAX 이동을 유도한다.
도 3c는 HeLa 표적 세포와 함께 공동-배양될 때, EGFR CAR를 발현하는 저캇 세포의 EGFR CAR 활성화를 나타낸다.
도 3d는 저캇 세포의 EGFR CAR 활성화가 HLA-A*02 LIR-1 억제 수용체에 의해 차단될 수 있음을 보여준다. 저캇 세포에 의한 EGFR CAR 및 HLA-A*02 LIR-1 억제 수용체의 공동 발현은 저캇 세포가 EGFR 및 HLA-A*02를 발현하는 HeLa 표적 세포와 함께 제시될 때 대략 5.4x의 CAR EMAX 이동을 유도한다.
도 4a는 활성화제 항원의 최적량을 결정하기 위한 비드 기반 검정에서 활성화제 항원의 적정을 보여준다.
도 4b는 활성화제 대 차단제 항원의 최적 비율을 결정하기 위한 비드 기반 검정에서 차단제 항원의 적정을 보여준다.
도 5는 HLA-A*02 scFv LIR1 억제제가 1차 T 세포에서 상이한 EGFR scFv CAR 활성화제와 함께 사용될 때 상이한 차단 정도가 관찰됨을 보여주는 일련의 플롯 및 표이다.
도 6a는 2개의 상이한 HLA-A*02 scFv LIR1 억제제가 1차 T 세포에서 상이한 EGFR scFv CAR 활성화제와 함께 사용될 때 상이한 차단 정도가 관찰됨을 보여주는 일련의 플롯이다.
도 6b는 상이한 쌍의 활성화 및 억제 수용체가 공동-발현될 때 활성화 및 억제 수용체의 민감도를 나타내는 일련의 플롯이다. 활성화제 또는 비-표적 항원의 양은 mRNA 적정에 의해 표적 세포에서 다양하다.
도 6c는 EGFR 표적화 활성화제 수용체 및 인간화 억제 수용체 또는 마우스 억제 수용체를 발현하는 세포에 의한 표적 세포의 최대 특이적 사멸 (%)을 나타내는 일련의 차트이다. 표적 종양 세포 대 정상 세포의 선택성 비율은 또한 활성화제 및 억제 수용체의 각 쌍에 대해 나타낸다.
도 7은 HeLa 세포와 1:1 비율로 공동 배양된 EGFR scFv CAR, 또는 EGFR scFv Car 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 나타내는 일련의 플롯이다. A 종양 세포: HLA-A*02가 아닌 EGFR를 발현하는 HeLa 세포; AB 정상 세포: HeLa 세포 발현 EGFR 및 HLA-A*02; A-B- 정상 세포: EGFR 또는 HLA-A*02를 발현하지 않는 HeLa 세포; B 정상 세포: EGFR이 아닌 HLA-A*02를 발현하는 HeLa 세포. Y 축은 분석에서 건강한 HeLa 세포에 비례하는 녹색 영역을 나타낸다. UTD: 형질도입되지 않음.
도 8은 차단제 및 활성화제 수용체를 발현하는 1차 T 세포의 농축 (상단 열), EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 이들 T 세포의 세포독성 활성을 나타내는 일련의 플롯이고, EGFR (표적 A), 또는 EGFR 및 HLA-A*02 (표적 AB)를 발현하는 HeLa 세포와 1:1로 혼합했다.
도 9는 EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 보여주는 일련의 플롯이며, 이는 EGFR, HLA-A*02, 또는 둘 다 발현하는 HeLa 세포와 1:1로 혼합되었다. A 종양 세포: HLA-A*02가 아닌 EGFR을 발현하는 HeLa 세포; AB 정상 세포: EGFR 및 HLA-A*02를 발현하는 HeLa 세포; B 정상 세포: EGFR이 아닌 HLA-A*02를 발현하는 HeLa 세포. Y축은 분석에서 건강한 HeLa 세포에 비례하는 녹색 영역을 나타낸다. UTD: 형질도입되지 않음.
도 10은 EGFR scFv CAR, 또는 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제 수용체를 발현하는 T 세포의 세포독성을 보여주는 일련의 플롯이며, 이는 EGFR (표적 A) 또는 EGFR 및 HLA-A*02 (표적 AB)를 발현하는 HCT.116 세포와 1:1로 혼합되었다.
도 11a는 상이한 EGFR scFv CAR 및 HLA-A*02 scFv LIR1 억제제를 발현하는 T 세포와 EGFR 활성화제 단독 (표적 A), 억제제 표적 단독 (표적 B) 또는 활성화제 및 억제제 표적 (표적 AB)을 발현하는 HeLa 세포의 배양 후 T 세포에 의한 EGFR scFv CAR 활성화제 수용체의 발현을 나타내는 일련의 형광 활성화 세포 분류 (FACS) 플롯이다.
도 11b는 표적 세포에 노출되기 전, 및 활성화제 리간드 단독 발현하는 표적 세포 (표적 A), 또는 활성화제 및 차단제 리간드를 모두 발현하는 표적 세포 (표적 AB)와의 공동 배양 120시간 후 정량 활성화제 수용체 발현을 나타내는 플롯이다.
도 12a는 EGFR (표적 A), HLA-A*02 (표적 B)를 발현하는 HeLa 세포의 집단, 동일한 세포 상의 EGFR 및 HLA-A*02의 조합 (표적 AB), 상이한 세포 상에 표적 A 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단, 또는 상이한 세포 상에 표적 B 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단과의 공동 배양 후 EGFR scFv CAR (CT-482) 활성화제 및 HLA-A*02 scFv LIR1 억제제 (C1765)를 발현하는 T 세포 상의 활성화제 수용체의 세포 표면 발현을 나타내는 플롯이다.
도 12b는 EGFR (표적 A), HLA-A*02 (표적 B)를 발현하는 HeLa 세포의 집단, 동일한 세포 상의 EGFR 및 HLA-A*02의 집단 (표적 AB), 상이한 세포 상에 표적 A 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단, 또는 상이한 세포 상에 표적 B 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단과의 공동 배양 후 EGFR scFv CAR (CT-482) 활성화제 및 HLA-A*02 scFv LIR1 억제제 (C1765)를 발현하는 T 세포 상의 억제 수용체의 세포 표면 발현을 나타내는 플롯이다.
도 13은 T 세포에 의한 세포독성 또는 차단 활성이 가역적인지 알아보기 위한 실험도이다.
도 14a는 세포독성 및 차단 활성이 가역적이고 활성화제 표면 발현에 상응함을 보여주는 일련의 플롯이다. T 세포는 1 라운드에서 EGFR+/HLA-A2+ 표적 세포와 공동 배양된 다음, 다음 라운드에서 교대 표적 세포로 전환했다. 상단: T 세포에 의한 표적 HeLa 세포의 사멸 퍼센트가 표시된다. 하단: FACS에 의해 분석된 활성화제 및 억제 수용체 발현.
도 14b는 세포독성 및 차단 활성이 가역적이고 활성화제 표면 발현에 상응함을 보여주는 일련의 플롯이다. T 세포는 1 라운드에서 EGFR+/HLA-A2- 표적 세포와 공동 배양한 다음, 다음 라운드에서 교대 표적 세포로 전환했다. 상단: T 세포에 의한 표적 HeLa 세포의 사멸 퍼센트가 표시된다. 하단: FACS에 의해 분석된 활성화제 및 억제 수용체 발현.
도 15a는 세포 표면 상의 EGFR 활성화제 CAR의 발현이 활성화제 및 억제제 리간드를 모두 발현하는 표적 세포의 존재에 의해 가역적으로 하향 조절됨을 보여주는 플롯이다.
도 15b는 활성화제만 표적 세포가 존재하거나, 활성화제 및 차단제 표적을 발현하는 표적 세포가 존재하지 않을 때 세포 표면 상의 EGFR 활성화제 CAR 발현이 신속하게 회복됨을 보여주는 일련의 플롯이다.
도 16은 비드 기반 검정을 사용하여 EGFR scFv CAR을 발현하는 저캇 세포의 활성화가 활성화제 및 억제제 항원이 시스로 비드 상에 존재하지만, 활성화제 및 억제제 항원이 트랜스로 비드 상에 존재하지 않을 때 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 17a는 EGFR scFv CAR에 의한 저캇 세포의 활성화가 HLA-A*02를 발현하지 않는 SiHa 세포 (SiHa WT)에 의해서가 아니라, HLA-A*02를 발현하는 SiHa 표적 세포 (SiHa A02)를 사용하여 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 17b는 EGFR scFv CAR에 의한 저캇 세포의 활성화가 HLA-A*02를 발현하지 않는 HeLa 세포 (HeLa WT)에 의해서가 아니라, HLA-A*02를 발현하는 HeLa 표적 세포 (HeLa A02)를 사용하여 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체에 의해 차단될 수 있음을 보여준다.
도 18은 잠재적인 비-표적 항원 후보 유전자를 확인하기 위해 사용되는 생물정보학 검색 과정을 나타낸 도면이다.
도 19는 생체 내 활성화제 수용체 및 억제 수용체를 표적으로 하는 EGFR을 발현하는 면역 세포의 효능 및 선택성을 결정하기 위한 실험 설계를 나타내는 도면이다.
도 20a는 HLA-A*11을 암호화하는 mRNA로 형질감염된 HeLa 세포를 사용하는 mRNA 적정 검정에서 저캇 NFAT 루시퍼라제 (JNL) 세포 억제를 나타내는 플롯이다. HeLa 세포는 연속 희석된 HLA-A*11 mRNA로 형질감염되었고, JNL 세포는 scFv HLA-A*11 #4와 함께 EGFR 활성화 CAR +/- HLA-A*11 억제 수용체로 일시적으로 형질감염되었다. 기능적 반응은 6시간 공동 배양 후 평가되었다.
도 20b는 HLA-A*11 #4를 사용한 억제 수용체의 분자/세포 민감도 (IC50)를 나타낸다.
도 21은 HLA-B*07-scFv-LIR1 억제 수용체가 공동-발현된 EGFR 표적화 활성화제 수용체에 의해 매개되는 면역 세포 활성화를 억제한다는 것을 보여주는 플롯이다.
도 22는 본원에 개시된 벡터를 포함하는 1차 T 세포에서 EGFR 활성화제 수용체 (EGFR-CAR 단독) 및 EGFR 활성화제/HLA-A 억제 수용체 쌍의 발현을 나타내는 일련의 플롯이다. 하나 또는 두 개의 수용체를 발현하는 세포의 백분율은 농축 전 (전 농축)과 농축 후 (후 농축)로 표시되며 농축은 수용체를 발현하는 세포를 선택한다. 수용체 쌍은 두 개의 개별 벡터 (이중 벡터) 또는 하나의 벡터 (단일 벡터)로 표현된다.
도 23a는 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 HeLa 표적 세포 또는 HCT116 표적 세포의 특이적 사멸 백분율을 나타내는 일련의 플롯이다. 표적 세포는 EGFR 항원 (A)을 발현하거나, EGFR 항원 및 HLA-A*02 비-표적 항원 (B) 모두 발현한다. 활성화제/억제제 수용체 쌍을 발현하는 면역 세포를 1:1 비율로 표적 세포와 함께 배양하였다.
도 23b는 암 세포 및 정상 세포의 혼합 배양물에서 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 표적 세포의 특이적 선택적 사멸을 보여주는 플롯 및 일련의 이미지이다. UTD = 활성화제 또는 억제제 수용체가 없는 면역 세포; CAR = 활성화제 수용체만을 발현하는 면역 세포; Tmod = 활성화제 및 억제제 수용체를 모두 발현하는 면역 세포. 오른쪽 이미지에서: 녹색 = 정상 세포; 빨간색 = 종양 세포.
도 24는 EGFR 활성화제 수용체 (CAR) 또는 EGFR 활성화제 수용체 및 HLA-A*02 억제제 수용체 (Tmod)를 발현하는 저캇 세포 (효과기)에서 선택적 세포 활성화를 나타내는 차트이다. 저캇 세포는 EGFR 발현 유무에 관계없이 HLA-A*02를 발현하는 표적 세포주의 패널과 함께 배양되었다. Tmod 저캇 효과기 세포는 HLA-A*02를 발현하는 세포의 EGFR.을 발현하지 않는 표적 세포와 공동 배양했을 때 오프-타겟 활성화를 나타내지 않았다.
암과 정상 세포 사이의 리간드의 유전자 발현 차이에 반응하는 2개의 수용체 시스템을 포함하는 면역 세포를 사용하여 암을 치료하기 위한 조성물 및 방법이 본원에 제공된다. 발현의 이러한 차이는 암 세포의 이형접합성 소실로 인한 것일 수 있다. 대안적으로, 발현의 차이는 유전자 발현이 암세포에서 발현되지 않거나 정상 세포보다 낮은 수준으로 암세포에서 발현되기 때문일 수 있다. 2-수용체 시스템은 면역 세포, 예를 들어 입양 세포 요법에 사용되는 면역 세포에서 발현되며, 이형접합성의 소실 또는 발현 차이를 나타내는 암세포에 대한 이들 면역 세포의 활성을 표적으로 한다. 이 2개의 수용체 시스템에서, 제1 수용체 (활성화제 수용체, 때때로 여기에서 A 모듈로 언급됨)는 면역 세포의 활성화 또는 활성화를 촉진하는 반면, 제2 수용체 (억제 수용체, 때때로 여기에서 차단제, 또는 억제제 수용체, 또는 B 모듈로 언급됨)는 제1 수용체에 의한 면역 세포의 활성화를 억제하는 작용을 한다. 각각의 수용체는 특정 리간드에 결합하는 리간드 결합 도메인 (LBD)을 함유한다. 리간드 결합 시 두 수용체로부터의 신호는 면역 세포에 의해 통합된다. 예를 들어, 암 세포에서 억제 리간드를 암호화하는 유전자좌의 이형접합성 소실 또는 전사 수준의 차이를 통해, 암 및 정상 세포에서 제1 및 제2 수용체에 대한 리간드의 차등적 발현은 제1 활성화제 리간드를 발현하지만 제2 억제 리간드를 발현하지 않는 표적 암 세포에 의한 면역 세포의 활성화를 매개한다.
본원에 제공된 조성물 및 방법의 특정 구현예에서, 본원에 기재된 2개의 수용체 시스템을 포함하는 면역 세포를 사용하여 표피 성장 인자 수용체 (EGFR) 양성 암을 치료한다. 이는 폐암, 교모세포종, 유방암, 두경부암 및 대장암을 포함한다. EGFR-양성 암의 경우, 활성화제 수용체의 표적 항원은 EGFR 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와 복합체에서, 이의 펩티드 항원이다. EGFR은 상피 조직, 간엽 조직, 신경 조직 등 다양한 정상 조직에서 발현되며, 세포 증식 및 분화, 뿐만 아니라 발달과 같은 정상 세포 과정에 중요한 역할을 한다. 특정 종양에서의 발현 때문에, EGFR은 이들 암 세포가 적절한 치료제로 특이적으로 표적화될 수 있다면 EGFR+ 종양의 선택적인 살상을 매개할 수 있는 매력적인 종양 특이적 항원이다. 그러나, 비-암 (비-표적) 세포에서의 정상 EGFR 발현은 입양 세포 요법과 같은 표적 요법을 위한 EGFR의 효과적인 사용을 방해했다. 피부 및 위장 독성은 EGFR 양성 암에서 EGFR의 효과적인 표적화를 방해했다. EGFR 활성화제 수용체를 억제 수용체와 페어링함으로써, 본원에서 제공되는 방법은 EGFR 표적 입양 세포 요법의 특이성을 증가시키고 용량 제한 독성과 같은 이러한 요법과 관련된 유해 효과를 감소시킨다.
일부 구현예에서, 활성화제에 대한 리간드는 MHC 클래스 I과 복합된 EGFR 펩티드이다. 본원에 기술된 방법에서, 이 EGFR 표적 활성화제 수용체는 억제 수용체와 쌍을 이루며, 정상 EGFR-양성 조직에서 세포 용해 효과를 차단함으로써 활성화제의 안전 창을 증가시킨다. 그러나, 활성화제 수용체는 종양 세포가 억제제, 또는 차단제, 수용체에 대한 리간드를 발현하지 않기 때문에, 2-수용체 시스템을 포함하는 면역 세포에 의한 종양 세포의 표적 사멸을 지시한다. 제2, 억제 수용체에 대한 표적은 상피 조직과 같은 EGFR 양성 조직에서 발현되지만, 암 세포에서는 발현되지 않으며, 억제 수용체는 이 "비-표적 항원"을 억제 자극으로 인식한다. 제2 억제 수용체에 대한 예시적인 표적은 폐 상피 조직에 의해 발현되고, 이형접합성 (LOH)으로 인해 EGFR 양성 암 세포에서 소실되어, 억제 수용체 상의 대립유전자 특이적 리간드 결합 도메인을 통해 다른 대립유전자와 구별될 수 있는 암 세포에서 단일 대립유전자 형태를 남긴다. 억제 수용체의 예시적인 표적은 인간 백혈구 항원 A (HLA-A), HLA-B, HLA-C, 및 다른 HLA와 같은 주요 조직적합성 복합체 (MHC) 단백질을 포함하지만, 이에 제한되지 않는다. HLA는 변이 유전자, 예를 들어 HLA-A*01., HLA-A*02, HLA*A03, HLA-C*07, 및 기타에 의해 암호화되고, 이형접합성의 소실을 통해 EGFR 양성 암 세포에서 소실될 수 있다. 대안적으로, 억제 수용체의 추가의 예시적인 표적은 콜렉틴 서브패밀리 구성원 12 COLEC12, APC 하향 조절 1 (APCDD1) 및 C-X-C 모티프 케모카인 리간드 16 (CXCL16)을 포함한다. 이들 각각은 항체에 접근할 수 있는 세포외 도메인의 아미노산 변경이 있는 일반적인 비동의 변이 형태를 가지고 있으며, 이는 EGFR 양성 암 환자를 EGFR 또는 EGFR pMHC 반응 활성화제 수용체와 같은 활성화제 수용체에 의해 활성화되는 조작된 T 세포로 안전하게 치료하도록 설계된 세포 통합체의 B 모듈 표적으로 사용될 수 있다. 본 개시내용의 조성물 및 방법은 정상 조직에 의한 EGFR의 발현에 의해 유발되는 DLT를 감소시키거나 제거할 수 있다. 본 개시내용은 제2 리간드 (EGFR 이외의 리간드, "비-표적 항원"이라고 함)의 존재 하에 입양 면역 세포의 활성화를 차단하는 제2 억제 수용체를 첨가함으로써 입양 세포 요법을 사용하여 EGFR 양성 암을 치료하기 위해 암 세포에서 EGFR을 표적으로 하는 방법을 제공한다. 본원에 기재된 조성물 및 방법을 사용하여, EGFR을 발현하는 종양 세포는 이들 종양 세포가 활성화제 리간드, EGFR만을 발현하기 때문에 2 수용체를 발현하는 입양 면역 세포에 의해 공격받는다. 대조적으로, EGFR 플러스 비-표적 항원 (대안적으로 "차단제 항원"이라고 함)을 발현하는 정상 세포는 입양 면역 세포로부터 보호된다. 정상 세포의 비-표적 항원에 대한 억제 수용체 반응은 그렇게 함으로써 EGFR-표적화 활성화제 수용체에 의한 면역 세포의 활성화를 방지한다. 이 이중 표적 접근법은 EGFR-양성 암 환자에서 EGFR 지시 세포 요법을 안전하고 효과적으로 투약할 수 있는 치료 창을 만든다.
본 개시내용은 온-타겟 독성을 유도하는 강력한 EGFR CAR 및 TCR의 사용을 가능하게 하는 방법 및 조성물을 제공하고, 이들 EGFR 표적 수용체를 이들의 독성을 완화함으로써 치료제로서 유용하게 만든다.
변형으로, 본원에 기재된 조성물 및 방법은 표적 세포를 사멸시키고/거나 비-표적 항원의 발현이 부분 유전자 결실, 후생적 침묵, 점 돌연변이, 절단을 포함하지만, 이에 제한되지 않는 이형접합성의 소실 이외의 원인에 의해 부분적으로 또는 완전히 감소된 대상체를 치료하는 데 사용될 수 있다.
정의
본 개시내용을 보다 상세하게 설명하기 전에, 본원에서 사용되는 특정 용어의 정의를 제공하는 것이 이를 이해하는 데 도움이 될 수 있다.
달리 정의되지 않는 한, 본원에 사용된 모든 기술 및 과학 용어는 본 개시내용이 속하는 기술 분야의 통상의 기술자가 일반적으로 이해하는 것과 동일한 의미를 갖는다. 본원에 기술된 것과 유사하거나 동등한 임의의 방법 및 재료가 특정 구현예의 실시 또는 시험에 사용될 수 있지만, 조성물, 방법 및 재료의 바람직한 구현예가 본원에 기술되어 있다. 본 개시내용의 목적을 위해, 다음 용어는 아래에 정의된다. 추가적인 정의는 본 개시내용 전반에 걸쳐 제시된다.
본원에서 사용되는 바와 같이, 용어 "약" 또는 "대략"은 기준 수량, 수준, 값, 수, 빈도, 백분율, 치수, 크기, 양, 중량 또는 길이에 비해 15%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% 또는 1% 만큼 변하는 수량, 수준, 값, 수, 빈도, 백분율, 치수, 크기, 양, 중량 또는 길이를 지칭한다. 한 구현예에서, 용어 "약" 또는 "대략"은 기준 수량, 수준, 값, 수, 빈도, 백분율, 치수, 크기, 양, 중량 또는 길이에 대해 ± 15%, ± 10%, ± 9%, ± 8%, ± 7%, ± 6%, ± 5%, ± 4%, ± 3%, ± 2%, 또는 ± 1% 수량, 수준, 값, 수, 빈도, 백분율, 치수, 크기, 양, 중량 또는 길이 범위를 지칭한다.
본원에서 사용되는 바와 같이, 용어 "단리된"은 본래의 상태에서 일반적으로 동반하는 성분이 실질적으로 또는 본질적으로 없는 물질을 의미한다. 특정 구현예에서, 용어 "수득된" 또는 "유래된"은 단리된 것과 동의어로 사용된다.
용어 "대상체," "환자" 및 "개체"는 본원에서 척추동물, 바람직하게는 포유동물, 보다 바람직하게는 인간을 지칭하기 위해 상호 교환적으로 사용된다. 조직, 세포, 및 생체 내에서 얻거나 시험관 내에서 배양한 생물학적 독립체의 자손도 포함된다. 본원에서 사용되는 바와 같이, "대상체," "환자" 또는 "개체"는 본원에서 고려되는 벡터, 조성물 및 방법으로 치료될 수 있는 통증을 나타내는 임의의 동물을 포함한다. 적합한 대상체 (예를 들어, 환자)는 실험동물 (예를 들어 마우스, 랫트, 토끼 또는 기니피그), 농장 동물 및 가축 또는 애완동물 (예를 들어 고양이 또는 개)을 포함한다. 비인간 영장류 및 바람직하게는 인간 환자가 포함된다.
본원에서 사용된 "치료" 또는 "치료하는"은 임의의 유익하거나 바람직한 효과를 포함하며, 심지어 증상의 최소한의 개선도 포함할 수 있다. "치료"는 반드시 질환이나 병태 또는 관련 증상의 완전한 박멸이나 치유를 의미하지는 않는다.
본원에서 사용되는 바와 같이, "예방하다," 및 "예방된," "예방하는" 등과 같은 유사한 단어는 질환 증상의 가능성을 예방, 억제 또는 감소시키기 위한 접근법을 나타낸다. 또한, 질환 또는 병태의 발병 또는 재발을 지연시키거나 질환 증상의 발생 또는 재발을 지연시키는 것을 지칭한다. 본원에서 사용되는 바와 같이, "예방" 및 유사한 단어는 또한 개시 또는 재발 전에 질환의 강도, 영향, 증상 및/또는 부담을 감소시키는 것을 포함한다.
본원에서 사용되는 바와 같이, 용어 "양"은 임상 결과를 포함하여 유익하거나 원하는 예방 또는 치료 결과를 달성하기 위한 바이러스의 "유효한 양" 또는 "유효량"을 지칭한다.
바이러스 또는 세포의 "치료적 유효량"은 개체의 질환 상태, 연령, 성별 및 체중, 및 바이러스 또는 세포가 개체에서 원하는 반응을 이끌어내는 능력과 같은 요인에 따라 달라질 수 있다. 치료적 유효량은 또한 바이러스 또는 세포의 임의의 독성 또는 해로운 효과가 치료적으로 유익한 효과보다 더 큰 양이다. 용어 "치료적 유효량"은 대상체 (예를 들어, 환자)를 "치료"하는 데 효과적인 양을 포함한다.
예를 들어, 전기생리학적 활성 또는 세포 활성과 같은 생리학적 반응의 "증가된" 또는 "향상된" 양은 전형적으로 "통계적으로 유의한" 양이며, 처리되지 않은 세포의 1.1, 1.2, 1.5, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 30배 이상 (예를 들어, 500, 1000배) (1 이상의 모든 정수 및 소수점 포함, 예를 들어, 1.5, 1.6, 1.7. 1.8 등) 활성 수준 증가를 포함할 수 있다.
예를 들어, 전기생리학적 활성 또는 세포 활성과 같은 생리학적 반응의 "감소된" 또는 "줄어든" 양은 전형적으로 "통계적으로 유의한" 양이며, 처리되지 않은 세포의 1.1, 1.2, 1.5, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 30배 이상 (예를 들어, 500, 1000배) (1 이상의 모든 정수 및 소수점 포함, 예를 들어, 1.5, 1.6, 1.7. 1.8 등) 활성 수준 감소를 포함할 수 있다.
"유지하다," 또는 "보존하다," 또는 "유지," 또는 "변화없음," 또는 "실질적인 변화없음," 또는 "실질적인 감소 없음"은 일반적으로 두 비히클 또는 대조군 분자/조성물에 의해 유발된 반응에 비교 가능한 생리학적 반응을 지칭한다. 비교 가능한 반응은 기준 반응과 크게 다르지 않거나 측정 가능한 차이가 없는 반응이다.
일반적으로, "서열 동일성" 또는 "서열 상동성"은 각각 2개의 폴리뉴클레오티드 또는 폴리펩티드 서열의 정확한 뉴클레오티드-대-뉴클레오티드 또는 아미노산 대 아미노산 대응을 지칭한다. 전형적으로, 서열 동일성을 결정하기 위한 기술은 폴리뉴클레오티드의 뉴클레오티드 서열 결정 및/또는 그에 의해 암호화된 아미노산 서열 결정, 및 이들 서열을 제2 뉴클레오티드 또는 아미노산 서열과 비교하는 것을 포함한다. 2개 이상의 서열 (폴리뉴클레오티드 또는 아미노산)은 이들의 "퍼센트 동일성"을 결정하여 비교할 수 있다. 핵산 또는 아미노산 서열이든, 두 서열의 퍼센트 동일성은 더 짧은 서열의 길이로 나누고 100을 곱한 2개의 정렬된 서열 간의 정확한 일치 수이다. 퍼센트 동일성은 또한, 예를 들어, 국립보건연구원에서 구할 수 있는 버전 2.2.9를 포함한 고급 BLAST 컴퓨터 프로그램을 사용하는 서열 정보를 비교함으로써 결정할 수 있다. BLAST 프로그램은 Karlin and Altschul, Proc. Natl. Acad. Sci. USA 87:2264-2268 (1990)의 정렬 방법에 기초하고 Altschul, et al., J. Mol. Biol. 215:403-410 (1990); Karlin And Altschul, Proc. Natl. Acad. Sci. USA 90:5873-5877 (1993); 및 Altschul et al., Nucleic Acids Res. 25:3389-3402 (1997)에서 논의된 바와 같다. 간략하게, BLAST 프로그램은 동일하게 정렬된 기호 (일반적으로 뉴클레오티드 또는 아미노산)의 수를 두 서열 중 더 짧은 기호의 총 수로 나눈 것으로 동일성을 정의한다. 이 프로그램은 비교되는 단백질의 전체 길이에 걸쳐 퍼센트 동일성을 결정하는 데 사용될 수 있다. 예를 들어, blastp 프로그램에서 짧은 쿼리 시퀀스로 검색을 최적화하기 위해 기본 매개변수가 제공된다. 이 프로그램은 또한 Wootton and Federhen, Computers and Chemistry 17:149-163 (1993)의 SEG 프로그램에 의해 결정된 바와 같이 쿼리 시퀀스의 세그먼트를 마스크-오프하기 위해 SEG 필터의 사용을 허용한다. 원하는 정도의 서열 동일성의 범위는 약 80% 내지 100%이고 그 사이의 정수 값이다. 전형적으로, 개시된 서열과 청구된 서열 사이의 퍼센트 동일성은 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98%이다.
본원에서 사용되는 바와 같이, "폴리뉴클레오티드 시스템"은 하나 이상의 폴리뉴클레오티드를 지칭한다. 하나 이상의 폴리뉴클레오티드는 특정 적용을 위해 협력하여 작동하도록 또는 원하는 형질전환된 세포를 생성하도록 설계될 수 있다.
용어 "외인성"은 본원에서 유기체 외부로부터 기원하는 핵산, 단백질 또는 펩티드, 소분자 화합물 등을 포함하는 임의의 분자를 지칭하는 데 사용된다. 대조적으로, 용어 "내인성"은 유기체 내부로부터 기원하는 (즉, 유기체에 의해 자연적으로 생성되는) 임의의 분자를 지칭한다.
용어 "MOI"는 감염 다중도를 지칭하는 데 사용되고, 이는 감염 표적 (예를 들어 세포)에 대한 작용제 (예를 들어 바이러스 입자)의 비율이다.
본 명세서에서, 임의의 농도 범위, 백분율 범위, 비율 범위 또는 정수 범위는 달리 명시되지 않는 한, 인용된 범위 내의 임의의 정수 값 및 적절한 경우 그의 분수 (예를 들어 정수의 1/10 및 1/100)를 포함하는 것으로 이해될 것이다.
본원에서 사용되는 바와 같이, "표적 세포"는 입양 세포 요법에 의해 표적화된 세포를 지칭한다. 예를 들어, 표적 세포는 입양 세포 요법의 이식된 T 세포에 의해 사멸될 수 있는 암 세포일 수 있다. 본 개시내용의 표적 세포는 본원에 기재된 바와 같이 표적 항원을 발현하고, 비-표적 항원을 발현하지 않는다.
본원에서 사용되는 바와 같이, "비-표적 세포"는 입양 세포 요법에 의해 표적화되지 않는 세포를 지칭한다. 예를 들어, 암 세포를 표적으로 하는 입양 세포에서, 정상, 건강한 비암성 세포는 비표적 세포이다. 대상체의 일부 또는 모든 비표적 세포는 표적 항원과 비표적 항원을 모두 발현할 수 있다. 대상체의 비-표적 세포는 이러한 세포가 또한 표적 항원을 발현하는지에 관계없이 비표적 항원을 발현할 수 있다.
본원에서 사용되는 바와 같이, "표적 항원"은 항원이라는 용어를 사용하든 특정 항원의 이름을 사용하든 암 세포와 같은 표적 세포에 의해 발현되는 항원을 지칭한다. 표적 항원의 발현은 표적 세포에 국한되지 않는다. 표적 항원은 대상체에서 암 세포 및 정상, 비-암 세포 모두에 의해 발현될 수 있다.
본원에서 사용되는 바와 같이, "비-표적 항원" (또는 "차단제 항원")은 항원이라는 용어를 사용하든 특정 항원의 이름을 사용하든 정상, 비-암 세포에서 발현되고 암 세포에서 발현되지 않는 항원을 지칭한다. 이러한 발현의 차이는 억제 수용체가 비표적 세포의 존재 하에서 면역 세포 활성화를 억제하도록 하지만 표적 세포의 존재 하에서는 그렇지 않다.
다형성은 모집단에서 뉴클레오티드 서열의 두 개 이상의 변이체의 존재를 지칭한다. 다형성은 하나 이상의 염기 변화, 삽입, 반복 또는 결실을 포함할 수 있다. 다형성은 예를 들어 단순 서열 반복 (SSR) 및 단일 뉴클레오티드 다형성 (SNP)을 포함하고, 이는 아데닌 (A), 티민 (T), 시토신 (C) 또는 구아닌 (G)의 단일 뉴클레오티드가 변경될 때 발생하는 변이이다.
본원에서 사용되는 바와 같이, "친화도"는 수용체, 예를 들어 본원에 기재된 임의의 수용체의 항원 결합 도메인에 대한 항원 상의 단일 리간드 결합 부위에 대한 리간드의 결합 강도를 지칭한다. 리간드 결합 도메인은 리간드와 더 약한 상호작용 (낮은 친화도) 또는 더 강한 상호작용 (높은 친화도)을 가질 수 있다.
Kd, 또는 해리 상수는 예를 들어, 수용체와 동족 리간드를 포함하는 거대분자 복합체가 리간드와 수용체로 분리되는 경우와 같이 더 작은 성분으로 가역적으로 분리되는 더 큰 물체의 성향을 측정하는 평형 상수의 한 유형이다. Kd 가 높다는 것은 수용체를 점유하기 위해서는 고농도의 리간드가 필요하고 리간드에 대한 수용체의 친화력이 낮다는 것을 의미한다. 반대로, 낮은 Kd는 리간드가 수용체에 대해 높은 친화도를 갖는다는 것을 의미한다.
본원에서 사용되는 바와 같이, "반응하는" 또는 "에 반응하는" 수용체는 리간드 (즉 항원)에 의해 결합될 때 세포 내 도메인의 알려진 기능에 해당하는 신호를 생성하는 세포 내 도메인을 포함하는 수용체를 지칭한다. 표적 항원에 결합된 활성화제 수용체는 활성화제 수용체를 발현하는 면역 세포의 활성화를 유발하는 신호를 생성한다. 비-표적 항원에 결합된 억제 수용체는 활성화제 수용체를 발현하는 면역 세포의 활성화를 방지하거나 감소시키는 억제 신호를 생성할 수 있다. 수용체의 반응성 및 수용체를 발현하는 면역 세포를 활성화 또는 억제하는 능력은 리포터 분석 및 세포독성 분석을 포함하지만 이에 제한되지 않는 당업계에 공지되고 본원에 기술된 임의의 수단에 의해 분석될 수 있다.
본원에서 사용되는 바와 같이, 면역 세포의 "활성화" 또는 "활성화된" 면역 세포는 면역 반응의 특징적인 하나 이상의 기능을 수행할 수 있는 면역 세포이다. 이러한 기능에는 증식, 사이토카인 방출 및 세포독성, 즉 표적 세포의 사멸이 포함된다. 활성화된 면역 세포는 당업자에게 명백할 마커를 발현한다. 예를 들어, 활성화된 T 세포는 CD69, CD71, CD25 및 HLA-DR 중 하나 이상을 발현할 수 있다. 활성화제 수용체 (예를 들어 EGFR CAR)를 발현하는 면역 세포는 표적 항원 (예를 들어 EGFR)의 결합에 반응하게 될 때 활성화제 수용체에 의해 활성화될 수 있다. "표적 항원"은 또한 "활성화제 항원"으로 지칭될 수 있고 표적 세포에 의해 단리되거나 발현될 수 있다. 억제 수용체를 발현하는 면역 세포의 활성화는 억제 수용체가 비-표적 항원 (예를 들어 HLA-A*02)의 결합에 반응하게 될 때, 활성화제 수용체가 활성화제 리간드에 결합된 경우에도 감소되거나 예방될 수 있다. "비-표적 항원"은 또한 "억제 리간드" 또는 "차단제"로 지칭될 수 있고, 표적 세포에 의해 단리되거나 발현될 수 있다.
면역 세포 상의 수용체 발현은 본원에 기술된 활성화제 수용체 및 억제 수용체의 존재를 보고하는 검정에 의해 확인될 수 있다. 예를 들어, 면역 세포 집단은 표지된 분자 (예를 들어 형광단 표지된 수용체-특이적 항체 또는 형광단 표지된 수용체-특이적 리간드)로 염색될 수 있고, 형광 활성화 세포 분류 (FACS) 유세포 분석을 사용하여 정량화했다. 이 방법은 활성화제 수용체, 억제 수용체, 또는 수용체 모두를 발현하는 것을 특징으로 하는 면역 세포의 집단에서 면역 세포의 백분율을 허용한다. 본원에 기술된 면역 세포에 의해 발현되는 활성화제 수용체 및 억제 수용체의 비율은 예를 들어, 디지털 액적 PCR에 의해 결정될 수 있다. 이들 접근법은 본원에 기술된 면역 세포, 약학 조성물, 및 키트의 생산 및 제조를 위한 세포 집단을 특성화하는 데 사용될 수 있다. 본원에 기술된 면역 세포, 약학 조성물 및 키트의 경우, 활성화제 수용체 및 억제 수용체 모두를 발현하는 면역 세포의 적합한 백분율은 본원에 기술된 방법에 대해 구체적으로 결정되는 것으로 이해된다. 예를 들어, 활성화제 수용체 및 억제 수용체를 모두를 발현하는 면역 세포의 적합한 백분율은 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 또는 적어도 95%일 수 있다. 예를 들어, 활성화제 수용체 및 억제 수용체를 모두를 발현하는 면역 세포의 적합한 백분율은 최대 50%, 최대 55%, 최대 60%, 최대 65%, 최대 70%, 최대 75%, 최대 80%, 최대 85%, 최대 90%, 또는 최대 95%일 수 있다. 예를 들어, 면역 세포에서 활성화제 수용체와 억제 수용체의 적합한 비율은 약 5:1, 약 4:1, 약 3:1, 약 2:1, 약 1:1, 약 1:2, 약 1:3, 약 1:4, 또는 약 1:5일 수 있다. 본원에 기술된 면역 세포, 약학 조성물 및 키트에 대한 적합한 값을 충족시키기 위해 면역 세포 집단에 정제, 농축 및/또는 고갈 단계를 사용할 수 있음을 이해한다.
본원에 기재된 면역 세포에 의해 발현되는 반응성 수용체는 수용체의 세포 내 도메인에 의해 생성될 것으로 예상되는 신호의 생성을 측정하는 검정에 의해 검증될 수 있다. 저캇-루시퍼라제 NFAT 세포 (저캇 세포)와 같은 수용체 세포주는 반응성 수용체를 특성화하는 데 사용할 수 있다. 저캇 세포 T 세포로부터 유래되고 활성화된 T-세포 (NFAT)-유도성 루시퍼라제 리포터 시스템의 안정적으로 통합된 핵 인자를 포함한다. NFAT는 면역 세포 활성화에 필요한 전사 인자 계열이며, 그의 활성화는 T 세포 활성화에 대한 신호 마커로 사용될 수 있다. 저캇 세포는 본원에 기술된 활성화제 수용체 및/또는 억제 수용체로 형질도입되거나 형질감염될 수 있다. 활성화제 수용체는 저캇 세포가 루시퍼라제 리포터 유전자를 발현하는 경우 리간드의 결합에 반응하며, 반응성 수준은 리포터 유전자 발현 수준에 의해 결정될 수 있다. 루시퍼라제의 존재는 루시페린과 같은 임의의 공지된 루시퍼라제 검출 시약을 사용하여 결정할 수 있다. 억제 수용체는 저캇 세포에서 활성화제 수용체와 함께 발현될 때, 정상적으로 반응하는 면역 세포가 활성제 수용체에 대한 반응으로 루시퍼라제를 발현하는 것을 방지하는 경우 리간드의 결합에 반응한다. 예를 들어, 억제 수용체의 반응성은 다음을 관찰함으로써 활성제와 억제제 모두를 발현하는 저캇 세포에서 결정되고 정량화될 수 있다: 1) 저캇 세포는 활성화제 수용체 리간드의 존재 및 억제 수용체 리간드의 부재 하에 루시퍼라제를 발현한다; 및 2) 저캇 세포에서 루시퍼라제 발현은 활성화제 수용체 리간드 및 억제 수용체 리간드 둘 다의 존재 하에 감소되거나 제거된다. 이 접근법은 활성화제 수용체 및 활성화제 수용체와 억제 수용체의 특정 쌍의 민감도, 효능 및 선택성을 결정하는 데 사용할 수 있다. 민감도, 효능 및 선택성은 활성화제 수용체 리간드 및/또는 억제 수용체 리간드가 활성화제 수용체 또는 활성화제와 억제 수용체의 특정 쌍을 발현하는 저캇 세포의 배양물로 적정되는, 용량 반응 실험을 사용하여 EC50 또는 IC50 값으로 정량화될 수 있다. 대안적으로, EC50 및 IC50 값은 활성화제 수용체 또는 활성화제와 억제 수용체의 특정 쌍을 발현하는 면역 세포 (예를 들어 저캇 세포 또는 1차 면역 세포) 및 증가하는 양의 활성화제 리간드 또는 억제제 리간드를 발현하는 표적 세포의 공동 배양물에서 결정될 수 있다. 증가하는 양의 활성화제 리간드 또는 억제제 리간드는 예를 들어, mRNA를 표적 세포로 암호화하는 활성화제 리간드 또는 억제제 리간드의 적정, 또는 상이한 수준의 표적 리간드를 자연적으로 발현하는 표적 세포의 사용에 의해 표적 세포에서 달성될 수 있다. 다양한 양의 표적 및 비-표적 리간드를 발현하는 사용된 표적 세포로 결정된 활성화제 및 억제 수용체에 대한 예시적인 적합한 EC50 및 IC50 값은 활성화제 수용체에 대해 260 백만당 전사체 (TPM) 이하 EC50, 예를 들어 10 내지 260 TPM의 EC50, 및 억제 수용체에 대해 10 TPM 이하의 IC50, 예를 들어 1-5 TPM의 IC50를 포함한다.
활성화제 수용체 또는 활성화제와 억제 수용체의 특정 쌍을 발현하는 본원에 기재된 면역 세포의 활성화는 상기 면역 세포와의 공동 배양 후 표적 세포의 생존력을 측정하는 검정에 의해 추가로 결정될 수 있다. 때때로 효과기 세포로 지칭되는, 면역 세포는 활성화제 수용체 리간드, 억제 수용체 리간드, 또는 활성화제 및 억제 수용체 리간드를 모두 발현하는 표적 세포와 공동 배양된다. 공동 배양 후, 표적 세포의 생존력은 세포 배양물에서 생존력을 측정하기 위한 임의의 방법을 사용하여 측정한다. 예를 들어, 활성 미토콘드리아 효소를 측정하기 위해 테트라졸륨 염 기질을 사용하는 미토콘드리아 기능 분석을 사용하여 생존력을 결정할 수 있다. 생존력은 이미징 기반 방법을 사용하여 결정할 수도 있다. 표적 세포는 녹색 형광 단백질 또는 적색 형광 단백질과 같은 형광 단백질을 발현할 수 있다. 전체 세포 형광의 감소는 표적 세포의 생존력 감소를 나타낸다. 활성화제 수용체 또는 활성화제와 억제 수용체의 특정 쌍을 발현하는 면역 세포와의 배양 후 표적 세포의 생존력 감소는 면역 세포의 표적 세포-매개 활성화로 해석된다. 면역 세포의 선택성의 척도는 또한 이 접근법을 사용하여 결정될 수 있다. 활성화제와 억제 수용체 쌍을 발현하는 면역 세포는 다음이 관찰되는 경우 선택적이다: 1) 억제 수용체 리간드가 아닌 활성화제 수용체 리간드를 발현하는 표적 세포에서 생존력이 감소된다; 2) 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포에서 생존력이 감소하지 않는다. 이들 측정으로부터, 음성 대조군 (활성화제 수용체를 발현하지 않는 면역 세포)의 백분율로서 표적 세포의 생존력 감소에 기초하여 면역 세포 활성화의 백분율을 정량화하는 "특이적 사멸" 값이 도출될 수 있다. 추가로, 이러한 측정으로부터 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포에서 관찰된 특이적 사멸에 대해 억제 수용체 리간드의 부재 하에 활성화제 수용체 리간드를 발현하는 표적 세포에서 관찰된 특이적 사멸의 비율을 나타내는 "선택성 비율" 값이 도출될 수 있다. 이 접근법은 본원에 기술된 면역 세포, 약학 조성물 및 키트의 생산 및 제조를 위한 세포 집단을 특성화하는 데 사용될 수 있다. 면역 세포, 약학 조성물 및 키트에 대해 적합한 특이적 사멸 값은 예를 들어 다음 기준일 수 있다: 1) 면역 세포와 억제 수용체 리간드의 부재 하에 활성화제 수용체 리간드를 발현하는 표적 세포의 48시간 공동-배양 후 적어도 50% 특이적 사멸; 2) 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포의 20% 이하의 특이적 사멸. 추가 예로서, 면역 세포가 6시간, 12시간, 18시간, 24시간, 30시간, 36시간, 42시간, 48시간, 54시간, 또는 60시간의 기간에 걸쳐 억제제 리간드가 아닌 활성화제 리간드를 발현하는 표적 세포의 적어도 30%, 적어도 40%, 적어도 50%, 적어도 60%, 적어도 70%, 적어도 80%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99%를 사멸시킬 수 있는 동안, 동일한 기간 40% 미만, 30% 미만, 20% 미만, 10% 미만, 5% 미만, 3% 미만 또는 1% 미만의 활성화제 및 억제제 리간드를 발현하는 표적 세포를 사멸시킨다.
면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50% 내지 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포에 대해 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50%, 적어도 약 55%, 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 또는 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 최대 약 50%, 최대 약 55%, 최대 약 60%, 최대 약 65%, 최대 약 70%, 최대 약 75%, 최대 약 80%, 최대 약 85%, 최대 약 90%, 또는 최대 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포의 적합한 특이적 사멸 값은 약 50% 미만, 약 45% 미만, 약 40% 미만, 약 35% 미만, 약 30% 미만, 약 25% 미만, 약 20% 미만, 약 15% 미만, 약 10% 미만, 또는 약 5% 미만일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대해 적합한 특이적 사멸 값은 표적 세포와 면역 세포의 약 6시간, 약 12시간, 약 18시간, 약 24, 약 30시간, 약 36시간, 약 42시간, 약 48시간, 약 54시간, 약 60시간, 약 66시간, 또는 약 72시간 공동-배양 후 결정될 수 있다.
면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50% 내지 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50%, 적어도 약 55%, 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 또는 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 최대 약 50%, 최대 약 55%, 최대 약 60%, 최대 약 65%, 최대 약 70%, 최대 약 75%, 최대 약 80%, 최대 약 85%, 최대 약 90%, 또는 최대 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대해 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포의 적합한 특이적 사멸 값은 약 50% 미만, 약 45% 미만, 약 40% 미만, 약 35% 미만, 약 30% 미만, 약 25% 미만, 약 20% 미만, 약 15% 미만, 약 10% 미만, 또는 약 5% 미만일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대해 적합한 특이적 사멸 값은 표적 세포와 면역 세포의 약 6시간, 약 12시간, 약 18시간, 약 24, 약 30시간, 약 36시간, 약 42시간, 약 48시간, 약 54시간, 약 60시간, 약 66시간, 또는 약 72시간의 공동-배양 후 결정될 수 있다.
면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50% 내지 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 적어도 약 50%, 적어도 약 55%, 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 또는 적어도 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대한 억제 리간드 값의 부재 하에 활성화제 리간드를 발현하는 표적 세포의 적합한 특이적 사멸 값은 예를 들어, 최대 약 50%, 최대 약 55%, 최대 약 60%, 최대 약 65%, 최대 약 70%, 최대 약 75%, 최대 약 80%, 최대 약 85%, 최대 약 90%, 또는 최대 약 95%일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대해 활성화제 수용체 리간드 및 억제 수용체 리간드를 모두 발현하는 표적 세포의 적합한 특이적 사멸 값은 약 50% 미만, 약 45% 미만, 약 40% 미만, 약 35% 미만, 약 30% 미만, 약 25% 미만, 약 20% 미만, 약 15% 미만, 약 10% 미만, 또는 약 5% 미만일 수 있다. 면역 세포, 약학 조성물, 및 키트에 대해 적합한 특이적 사멸 값은 표적 세포와 면역 세포의 6시간, 약 12시간, 약 18시간, 약 24, 약 30시간, 약 36시간, 약 42시간, 약 48시간, 약 54시간, 약 60시간, 약 66시간, 또는 약 72시간의 공동-배양 후 결정될 수 있다.
본원에서 사용되는 바와 같이, 용어 "기능적 변이체"는 모 단백질과 비교하여 하나 이상의 아미노산 치환, 삽입 또는 결실을 갖고, 모 단백질의 하나 이상의 원하는 활성을 유지하는 단백질을 지칭한다. 기능적 변이체는 모체 단백질의 하나 이상의 원하는 활성을 유지하는 단백질의 단편 (즉 N- 및/또는 C-말단 결실을 갖는 변이체)일 수 있다.
본원에서 사용되는 바와 같이, "비-표적 대립형질 변이체"는 그 산물이 비-표적 세포에 의해 발현되지만 표적 세포에 의해 발현되지 않는 유전자의 대립유전자를 지칭한다. 예를 들어, 비-표적 대립형질 변이체는 대상체의 정상, 비- 세포에 의해 발현되지만, 대상체의 암 세포에 의해 발현되지 않는 유전자의 대립유전자이다. 비-표적 대립형질 변이체의 발현은 이형접합성의 소실, 돌연변이, 또는 비-표적 대립형질 변이체를 암호화하는 유전자의 후성적 변형을 포함하지만 이에 제한되지 않는 임의의 메커니즘에 의해 암 세포에서 소실될 수 있다.
본원에서 사용되는 바와 같이, 리간드 결합 도메인, 예를 들어 항원 결합 도메인과 관련하여 사용될 때"에 특이적" 또는 "에 특이적으로 결합하는"은 명명된 표적에 대해 높은 특이성을 갖는 리간드 결합 도메인을 지칭한다. 항체 특이성은 리간드 결합 도메인과 상응하는 리간드 사이의 적합도, 또는 리간드 결합 도메인이 유사하거나 유사하지 않은 리간드를 구별하는 능력의 척도로 볼 수 있다. 특이성과 비교하여, 친화도는 리간드 결합 도메인과 리간드 사이의 결합 강도의 척도로서, 저-친화도 리간드 결합 도메인은 약하게 결합하고 고-친화도 리간드 결합 도메인은 견고하게 결합한다. 표적 대립유전자에 특이적인 리간드 결합 도메인은 유전자의 상이한 대립유전자를 구별할 수 있는 것이다. 예를 들어, HLA-A*02에 특이적인 리간드 결합 도메인은 HLA-A*01 또는 HLA-A*03과 같은 다른 HLA-A 대립 유전자에 결합하지 않거나 약하게만 결합한다. 본원에 기술된 활성화제 및 억제 수용체와 관련하여, 리간드 결합 도메인은 면역 세포 반응의 특정 활성화 또는 억제를 매개한다. 당업자는 리간드 결합 도메인이 특정 표적에 특이적이라고 말할 수 있고, 본원에 기술된 수용체 시스템에서 그의 기능에 영향을 미치지 않는 하나 이상의 추가 표적에 여전히 낮은 수준의 결합을 갖는다는 것을 이해할 것이다.
본원에 언급된 모든 간행물 및 특허는 각각의 개별 간행물 또는 특허가 구체적이고 개별적으로 참고로 포함되는 것으로 표시되는 것처럼 전체적으로 참조로 포함된다. 상충하는 경우, 본원의 모든 정의를 포함하여 본 출원이 우선한다. 그러나, 본원에 인용된 모든 참조, 기사, 간행물, 특허, 특허 공개 및 특허 출원에 대한 언급은 그들이 유효한 선행 기술을 구성하거나 세계 어느 국가에서나 일반적인 일반 지식의 일부를 형성한다는 것을 인정하거나 어떤 형태의 제안으로 간주해서는 안 된다.
활성화제 수용체
본 개시내용은 암 세포 특이적 항원, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체에서 이의 펩티드 항원을 포함하는 표적 항원에 특이적인 제1 세포외 리간드 결합 도메인을 포함하는 제1 수용체를 제공한다. 제1 수용체는 활성화제 수용체이고, 제1 수용체의 세포외 리간드 결합 도메인에 의한 표적 항원 결합 시 제1 수용체를 발현하는 면역 세포의 활성화를 매개한다. 제1 수용체는 표적 항원 (즉 활성화제 리간드)에 반응성이다. 예를 들어, 표적 항원이 제1 수용체에 결합하거나 접촉할 때, 제1 반응성이 있고 제1 수용체의 세포외 리간드 결합 도메인에 의한 표적 항원의 결합 시 제1 수용체를 발현하는 면역 세포를 활성화한다. 일부 구현예에서, 제1 수용체는 키메라 항원 수용체 (CAR)이다. 일부 구현예에서, 제1 수용체는 T 세포 수용체 (TCR)이다.
일부 구현예에서, 제1 수용체는 인간화된다. 본원에서 사용되는 바와 같이, "인간화"는 비-인간 종으로부터 단리되거나 유도된 이식유전자 내의 서열 또는 하위서열을 상동적이거나 기능적으로 동등한 인간 서열로 대체하는 것을 지칭한다. 예를 들어, 인간화 항체는 마우스 CDR을 인간 프레임워크 서열에 이식한 다음, 특정 인간 프레임워크 잔기를 공급원 항체의 해당 마우스 잔기로 역치환하여 생성할 수 있다.
활성화제 표적
본 개시내용에 따르면, 제1 수용체에 대한 표적 항원은 표피 성장 인자 수용체 (EGFR), 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체에서 EGFR의 펩티드 항원이다.
주요 조직적합성 복합체 클래스 I (MHC-I)은 면역 체계의 세포에 항원을 표시하여 면역 반응을 유발하는 단백질 복합체이다. MHC-I에 해당하는 인간 백혈구 항원 (HLA)은 HLA-A, HLA-B 및 HLA-C이다.
HLA-A, HLA-B, HLA-C, HLA-E, HLA-F 또는 HLA-G 중 하나를 포함하는 암 세포 특이적 pMHC 항원은 본 개시내용의 범위 내에 있는 것으로 간주된다. 일부 구현예에서, 암 세포 특이적 항원은 HLA-A를 포함한다. HLA-A 수용체는 무거운 α 사슬과 더 작은 β 사슬로 구성된 이종이량체이다. α 사슬은 HLA-A의 변이체에 의해 암호화되는 반면, β 사슬 (β2-마이크로글로불린)은 불변이다. 수천 개의 변이 HLA-A 유전자가 있으며, 이들 모두는 본 개시내용의 범위 내에 속한다. 일부 구현예에서, MHC-I는 인간 백혈구 항원 A*02 대립유전자 (HLA-A*02)를 포함한다.
일부 구현예에서, 암 세포 특이적 항원은 HLA-B를 포함한다. HLA-B 유전자의 수백 가지 버전 (대립유전자)이 알려져 있으며, 각 버전에는 특정 번호가 부여된다 (예를 들어 HLA-B27).
일부 구현예에서, 암 세포 특이적 항원은 HLA-C를 포함한다. HLA-C는 HLA 클래스 I 중쇄 파라로그에 속한다. 이 클래스 I 분자는 중쇄 및 경쇄 (베타-2 마이크로글로불린)로 구성된 이종이량체이다. 100개가 넘는 HLA-C 대립유전자가 당업계에 알려져 있다.
일부 구현예에서, 암 세포 특이적 항원은 폐암 항원, 교모세포종 항원, 유방암 항원, 두경부암 항원 또는 결장직장암 항원이다. 일부 구현예에서, 암 세포 특이적 항원은 결장직장암 항원이다. 일부 구현예에서, 암 세포 특이적 항원은 EGFR 또는 이의 펩티드 항원이다.
일부 구현예에서, 암 세포 특이적 항원은 EGFR, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체에서 이의 펩티드 항원이다. EGFR은 세포외 단백질 리간드의 표피 성장 인자 (EGF) 계열의 수용체 형태 구성원인 막관통 단백질이다. EGFR은 중간엽 조직, 상피 조직 및 신경 조직과 같은 정상 조직에서 광범위하게 발현되며, 세포 증식, 분화 및 발달에 중요한 역할을 한다. EGFR은 또한 다양한 고형 종양에서 높게 발현되며, EGFR 발현은 종양 진행, 화학 요법에 대한 내성 및 불량한 예후와 관련이 있다.
EGFR의 모든 이소형 및 EGFR의 임상적으로 관련된 돌연변이 (예를 들어 EGFRvIII, S468/492R)는 본 개시내용의 암 세포 특이적 항원으로 생각된다. EGFR 이소형 a는 NCBI 기록 번호 NP_005219.2에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR는 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QEILHGAVRF SNNPALCNVE SIQWRDIVSS DFLSNMSMDF
181 QNHLGSCQKC DPSCPNGSCW GAGEENCQKL TKIICAQQCS GRCRGKSPSD CCHNQCAAGC
241 TGPRESDCLV CRKFRDEATC KDTCPPLMLY NPTTYQMDVN PEGKYSFGAT CVKKCPRNYV
301 VTDHGSCVRA CGADSYEMEE DGVRKCKKCE GPCRKVCNGI GIGEFKDSLS INATNIKHFK
361 NCTSISGDLH ILPVAFRGDS FTHTPPLDPQ ELDILKTVKE ITGFLLIQAW PENRTDLHAF
421 ENLEIIRGRT KQHGQFSLAV VSLNITSLGL RSLKEISDGD VIISGNKNLC YANTINWKKL
481 FGTSGQKTKI ISNRGENSCK ATGQVCHALC SPEGCWGPEP RDCVSCRNVS RGRECVDKCN
541 LLEGEPREFV ENSECIQCHP ECLPQAMNIT CTGRGPDNCI QCAHYIDGPH CVKTCPAGVM
601 GENNTLVWKY ADAGHVCHLC HPNCTYGCTG PGLEGCPTNG PKIPSIATGM VGALLLLLVV
661 ALGIGLFMRR RHIVRKRTLR RLLQERELVE PLTPSGEAPN QALLRILKET EFKKIKVLGS
721 GAFGTVYKGL WIPEGEKVKI PVAIKELREA TSPKANKEIL DEAYVMASVD NPHVCRLLGI
781 CLTSTVQLIT QLMPFGCLLD YVREHKDNIG SQYLLNWCVQ IAKGMNYLED RRLVHRDLAA
841 RNVLVKTPQH VKITDFGLAK LLGAEEKEYH AEGGKVPIKW MALESILHRI YTHQSDVWSY
901 GVTVWELMTF GSKPYDGIPA SEISSILEKG ERLPQPPICT IDVYMIMVKC WMIDADSRPK
961 FRELIIEFSK MARDPQRYLV IQGDERMHLP SPTDSNFYRA LMDEEDMDDV VDADEYLIPQ
1021 QGFFSSPSTS RTPLLSSLSA TSNNSTVACI DRNGLQSCPI KEDSFLQRYS SDPTGALTED
1081 SIDDTFLPVP EYINQSVPKR PAGSVQNPVY HNQPLNPAPS RDPHYQDPHS TAVGNPEYLN
1141 TVQPTCVNST FDSPAHWAQK GSHQISLDNP DYQQDFFPKE AKPNGIFKGS TAENAEYLRV
1201 APQSSEFIGA (서열 번호 1).
EGFR 이소형 b는 NCBI 기록 번호 NP_958439.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QEILHGAVRF SNNPALCNVE SIQWRDIVSS DFLSNMSMDF
181 QNHLGSCQKC DPSCPNGSCW GAGEENCQKL TKIICAQQCS GRCRGKSPSD CCHNQCAAGC
241 TGPRESDCLV CRKFRDEATC KDTCPPLMLY NPTTYQMDVN PEGKYSFGAT CVKKCPRNYV
301 VTDHGSCVRA CGADSYEMEE DGVRKCKKCE GPCRKVCNGI GIGEFKDSLS INATNIKHFK
361 NCTSISGDLH ILPVAFRGDS FTHTPPLDPQ ELDILKTVKE ITGFLLIQAW PENRTDLHAF
421 ENLEIIRGRT KQHGQFSLAV VSLNITSLGL RSLKEISDGD VIISGNKNLC YANTINWKKL
481 FGTSGQKTKI ISNRGENSCK ATGQVCHALC SPEGCWGPEP RDCVSCRNVS RGRECVDKCN
541 LLEGEPREFV ENSECIQCHP ECLPQAMNIT CTGRGPDNCI QCAHYIDGPH CVKTCPAGVM
601 GENNTLVWKY ADAGHVCHLC HPNCTYGS (서열 번호 2).
EGFR 이소형 c는 NCBI 기록 번호 NP_958440.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QEILHGAVRF SNNPALCNVE SIQWRDIVSS DFLSNMSMDF
181 QNHLGSCQKC DPSCPNGSCW GAGEENCQKL TKIICAQQCS GRCRGKSPSD CCHNQCAAGC
241 TGPRESDCLV CRKFRDEATC KDTCPPLMLY NPTTYQMDVN PEGKYSFGAT CVKKCPRNYV
301 VTDHGSCVRA CGADSYEMEE DGVRKCKKCE GPCRKVCNGI GIGEFKDSLS INATNIKHFK
361 NCTSISGDLH ILPVAFRGDS FTHTPPLDPQ ELDILKTVKE ITGLS (서열 번호 3).
EGFR 이소형 d는 NCBI 기록 번호 NP_958441.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QEILHGAVRF SNNPALCNVE SIQWRDIVSS DFLSNMSMDF
181 QNHLGSCQKC DPSCPNGSCW GAGEENCQKL TKIICAQQCS GRCRGKSPSD CCHNQCAAGC
241 TGPRESDCLV CRKFRDEATC KDTCPPLMLY NPTTYQMDVN PEGKYSFGAT CVKKCPRNYV
301 VTDHGSCVRA CGADSYEMEE DGVRKCKKCE GPCRKVCNGI GIGEFKDSLS INATNIKHFK
361 NCTSISGDLH ILPVAFRGDS FTHTPPLDPQ ELDILKTVKE ITGFLLIQAW PENRTDLHAF
421 ENLEIIRGRT KQHGQFSLAV VSLNITSLGL RSLKEISDGD VIISGNKNLC YANTINWKKL
481 FGTSGQKTKI ISNRGENSCK ATGQVCHALC SPEGCWGPEP RDCVSCRNVS RGRECVDKCN
541 LLEGEPREFV ENSECIQCHP ECLPQAMNIT CTGRGPDNCI QCAHYIDGPH CVKTCPAGVM
601 GENNTLVWKY ADAGHVCHLC HPNCTYGPGN ESLKAMLFCL FKLSSCNQSN DGSVSHQSGS
661 PAAQESCLGW IPSLLPSEFQ LGWGGCSHLH AWPSASVIIT ASSCH (서열 번호 4).
EGFR 이소형 e는 NCBI 기록 번호 NP_001333826.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QGQKCDPSCP NGSCWGAGEE NCQKLTKIIC AQQCSGRCRG
181 KSPSDCCHNQ CAAGCTGPRE SDCLVCRKFR DEATCKDTCP PLMLYNPTTY QMDVNPEGKY
241 SFGATCVKKC PRNYVVTDHG SCVRACGADS YEMEEDGVRK CKKCEGPCRK VCNGIGIGEF
301 KDSLSINATN IKHFKNCTSI SGDLHILPVA FRGDSFTHTP PLDPQELDIL KTVKEITGFL
361 LIQAWPENRT DLHAFENLEI IRGRTKQHGQ FSLAVVSLNI TSLGLRSLKE ISDGDVIISG
421 NKNLCYANTI NWKKLFGTSG QKTKIISNRG ENSCKATGQV CHALCSPEGC WGPEPRDCVS
481 CRNVSRGREC VDKCNLLEGE PREFVENSEC IQCHPECLPQ AMNITCTGRG PDNCIQCAHY
541 IDGPHCVKTC PAGVMGENNT LVWKYADAGH VCHLCHPNCT YGCTGPGLEG CPTNGPKIPS
601 IATGMVGALL LLLVVALGIG LFMRRRHIVR KRTLRRLLQE RELVEPLTPS GEAPNQALLR
661 ILKETEFKKI KVLGSGAFGT VYKGLWIPEG EKVKIPVAIK ELREATSPKA NKEILDEAYV
721 MASVDNPHVC RLLGICLTST VQLITQLMPF GCLLDYVREH KDNIGSQYLL NWCVQIAKGM
781 NYLEDRRLVH RDLAARNVLV KTPQHVKITD FGLAKLLGAE EKEYHAEGGK VPIKWMALES
841 ILHRIYTHQS DVWSYGVTVW ELMTFGSKPY DGIPASEISS ILEKGERLPQ PPICTIDVYM
901 IMVKCWMIDA DSRPKFRELI IEFSKMARDP QRYLVIQGDE RMHLPSPTDS NFYRALMDEE
961 DMDDVVDADE YLIPQQGFFS SPSTSRTPLL SSLSATSNNS TVACIDRNGL QSCPIKEDSF
1021 LQRYSSDPTG ALTEDSIDDT FLPVPGEWLV WKQSCSSTSS THSAAASLQC PSQVLPPASP
1081 EGETVADLQT Q (서열 번호 5).
EGFR 이소형 f는 NCBI 기록 번호 NP_001333827.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKV CQGTSNKLTQ LGTFEDHFLS LQRMFNNCEV
61 VLGNLEITYV QRNYDLSFLK TIQEVAGYVL IALNTVERIP LENLQIIRGN MYYENSYALA
121 VLSNYDANKT GLKELPMRNL QEILHGAVRF SNNPALCNVE SIQWRDIVSS DFLSNMSMDF
181 QNHLGSCQKC DPSCPNGSCW GAGEENCQKL TKIICAQQCS GRCRGKSPSD CCHNQCAAGC
241 TGPRESDCLV CRKFRDEATC KDTCPPLMLY NPTTYQMDVN PEGKYSFGAT CVKKCPRNYV
301 VTDHGSCVRA CGADSYEMEE DGVRKCKKCE GPCRKVCNGI GIGEFKDSLS INATNIKHFK
361 NCTSISGDLH ILPVAFRGDS FTHTPPLDPQ ELDILKTVKE ITGFLLIQAW PENRTDLHAF
421 ENLEIIRGRT KQHGQFSLAV VSLNITSLGL RSLKEISDGD VIISGNKNLC YANTINWKKL
481 FGTSGQKTKI ISNRGENSCK ATGQVCHALC SPEGCWGPEP RDCVSCRNVS RGRECVDKCN
541 LLEGEPREFV ENSECIQCHP ECLPQAMNIT CTGRGPDNCI QCAHYIDGPH CVKTCPAGVM
601 GENNTLVWKY ADAGHVCHLC HPNCTYGCTG PGLEGCPTNG PKIPSIATGM VGALLLLLVV
661 ALGIGLFMRR RHIVRKRTLR RLLQERELVE PLTPSGEAPN QALLRILKET EFKKIKVLGS
721 GAFGTVYKGL WIPEGEKVKI PVAIKELREA TSPKANKEIL DEAYVMASVD NPHVCRLLGI
781 CLTSTVQLIT QLMPFGCLLD YVREHKDNIG SQYLLNWCVQ IAKGMNYLED RRLVHRDLAA
841 RNVLVKTPQH VKITDFGLAK LLGAEEKEYH AEGGKVPIKW MALESILHRI YTHQSDVWSY
901 GVTVWELMTF GSKPYDGIPA SEISSILEKG ERLPQPPICT IDVYMIMVKC WMIDADSRPK
961 FRELIIEFSK MARDPQRYLV IQGDERMHLP SPTDSNFYRA LMDEEDMDDV VDADEYLIPQ
1021 QGFFSSPSTS RTPLLSSLSA TSNNSTVACI DRNGLQSCPI KEDSFLQRYS SDPTGALTED
1081 SIDDTFLPVP GEWLVWKQSC SSTSSTHSAA ASLQCPSQVL PPASPEGETV ADLQTQ (서열 번호 6).
EGFR 이소형 h는 NCBI 기록 번호 NP_001333829.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MFNNCEVVLG NLEITYVQRN YDLSFLKTIQ EVAGYVLIAL NTVERIPLEN LQIIRGNMYY
61 ENSYALAVLS NYDANKTGLK ELPMRNLQEI LHGAVRFSNN PALCNVESIQ WRDIVSSDFL
121 SNMSMDFQNH LGSCQKCDPS CPNGSCWGAG EENCQKLTKI ICAQQCSGRC RGKSPSDCCH
181 NQCAAGCTGP RESDCLVCRK FRDEATCKDT CPPLMLYNPT TYQMDVNPEG KYSFGATCVK
241 KCPRNYVVTD HGSCVRACGA DSYEMEEDGV RKCKKCEGPC RKVCNGIGIG EFKDSLSINA
301 TNIKHFKNCT SISGDLHILP VAFRGDSFTH TPPLDPQELD ILKTVKEITG FLLIQAWPEN
361 RTDLHAFENL EIIRGRTKQH GQFSLAVVSL NITSLGLRSL KEISDGDVII SGNKNLCYAN
421 TINWKKLFGT SGQKTKIISN RGENSCKATG QVCHALCSPE GCWGPEPRDC VSCRNVSRGR
481 ECVDKCNLLE GEPREFVENS ECIQCHPECL PQAMNITCTG RGPDNCIQCA HYIDGPHCVK
541 TCPAGVMGEN NTLVWKYADA GHVCHLCHPN CTYGCTGPGL EGCPTNGPKI PSIATGMVGA
601 LLLLLVVALG IGLFMRRRHI VRKRTLRRLL QERELVEPLT PSGEAPNQAL LRILKETEFK
661 KIKVLGSGAF GTVYKGLWIP EGEKVKIPVA IKELREATSP KANKEILDEA YVMASVDNPH
721 VCRLLGICLT STVQLITQLM PFGCLLDYVR EHKDNIGSQY LLNWCVQIAK GMNYLEDRRL
781 VHRDLAARNV LVKTPQHVKI TDFGLAKLLG AEEKEYHAEG GKVPIKWMAL ESILHRIYTH
841 QSDVWSYGVT VWELMTFGSK PYDGIPASEI SSILEKGERL PQPPICTIDV YMIMVKCWMI
901 DADSRPKFRE LIIEFSKMAR DPQRYLVIQG DERMHLPSPT DSNFYRALMD EEDMDDVVDA
961 DEYLIPQQGF FSSPSTSRTP LLSSLSATSN NSTVACIDRN GLQSCPIKED SFLQRYSSDP
1021 TGALTEDSID DTFLPVPEYI NQSVPKRPAG SVQNPVYHNQ PLNPAPSRDP HYQDPHSTAV
1081 GNPEYLNTVQ PTCVNSTFDS PAHWAQKGSH QISLDNPDYQ QDFFPKEAKP NGIFKGSTAE
1141 NAEYLRVAPQ SSEFIGA (서열 번호 7).
EGFR 이소형 i는 NCBI 기록 번호 NP_001333870.1에 기재되어 있으며, 그 내용은 본원에 참고로 포함된다. 일부 구현예에서, EGFR은 다음의 아미노산 서열을 포함한다:
1 MRPSGTAGAA LLALLAALCP ASRALEEKKG NYVVTDHGSC VRACGADSYE MEEDGVRKCK
61 KCEGPCRKVC NGIGIGEFKD SLSINATNIK HFKNCTSISG DLHILPVAFR GDSFTHTPPL
121 DPQELDILKT VKEITGFLLI QAWPENRTDL HAFENLEIIR GRTKQHGQFS LAVVSLNITS
181 LGLRSLKEIS DGDVIISGNK NLCYANTINW KKLFGTSGQK TKIISNRGEN SCKATGQVCH
241 ALCSPEGCWG PEPRDCVSCR NVSRGRECVD KCNLLEGEPR EFVENSECIQ CHPECLPQAM
301 NITCTGRGPD NCIQCAHYID GPHCVKTCPA GVMGENNTLV WKYADAGHVC HLCHPNCTYG
361 CTGPGLEGCP TNGPKIPSIA TGMVGALLLL LVVALGIGLF MRRRHIVRKR TLRRLLQERE
421 LVEPLTPSGE APNQALLRIL KETEFKKIKV LGSGAFGTVY KGLWIPEGEK VKIPVAIKEL
481 REATSPKANK EILDEAYVMA SVDNPHVCRL LGICLTSTVQ LITQLMPFGC LLDYVREHKD
541 NIGSQYLLNW CVQIAKGMNY LEDRRLVHRD LAARNVLVKT PQHVKITDFG LAKLLGAEEK
601 EYHAEGGKVP IKWMALESIL HRIYTHQSDV WSYGVTVWEL MTFGSKPYDG IPASEISSIL
661 EKGERLPQPP ICTIDVYMIM VKCWMIDADS RPKFRELIIE FSKMARDPQR YLVIQGDERM
721 HLPSPTDSNF YRALMDEEDM DDVVDADEYL IPQQGFFSSP STSRTPLLSS LSATSNNSTV
781 ACIDRNGLQS CPIKEDSFLQ RYSSDPTGAL TEDSIDDTFL PVPEYINQSV PKRPAGSVQN
841 PVYHNQPLNP APSRDPHYQD PHSTAVGNPE YLNTVQPTCV NSTFDSPAHW AQKGSHQISL
901 DNPDYQQDFF PKEAKPNGIF KGSTAENAEY LRVAPQSSEF IGA (서열 번호 8).
일부 구현예에서, 암 세포 특이적 항원은 EGFR에서 유래된 펩티드 항원이다. 일부 구현예에서, 펩티드 항원은 서열 번호 1-8 중 어느 하나의 서열 또는 하위서열과 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 또는 적어도 99% 동일성을 갖는 서열을 포함한다. 일부 구현예에서, 펩티드 항원은 서열 번호 1-8 또는 서열 번호 918-919 중 어느 하나의 하위서열과 동일한 서열을 포함한다.
세포외 리간드 결합 도메인
본 개시내용은 표적 항원에 특이적인 제1 세포외 리간드 결합 도메인을 포함하는 제1 수용체를 제공한다. 일부 구현예에서, 표적 항원은 암 세포 특이적 항원을 포함한다.
일부 구현예에서, 암 세포 특이적 항원은 EGFR 또는 MHC-I과 복합된 EGFR-유래 펩티드 항원이고, 제1 수용체의 리간드 결합 도메인은 EGFR 항원을 인식하고 결합한다. 일부 구현예에서, 암 세포 특이적 항원은 EGFR의 임상적으로 관련된 돌연변이체이다. 예를 들어, EGFR의 돌연변이 변이체는 치환 돌연변이 (예를 들어 엑손 21의 L858R), 결실 돌연변이 (예를 들어 EGFRvIII 또는 엑손 19 또는 엑손 23의 결실), 또는 EGFR 유전자에 대한 증폭 돌연변이일 수 있다.
리간드 의존적 방식으로 수용체의 활성을 조절할 수 있는 임의의 유형의 리간드 결합 도메인은 본 개시내용의 범위 내에 있는 것으로 생각된다. 일부 구현예에서, 리간드 결합 도메인은 항원 결합 도메인이다. 예시적인 항원 결합 도메인은 특히, scFv, SdAb, Vβ-단독 도메인, 및 TCR α 및 β 사슬 가변 도메인으로부터 유도된 TCR 항원 결합 도메인을 포함한다.
임의의 유형의 항원 결합 도메인은 본 개시내용의 범위 내에 있는 것으로 생각된다.
예를 들어, 제1 세포외 리간드 결합 도메인은 예를 들어, 마우스, 인간화 또는 인간 항체로부터 유래된 Vβ 단독 도메인, 단일 도메인 항체 단편 (sdAb) 또는 중쇄 항체 HCAb, 단쇄 항체 (scFv)를 포함하는 연속 폴리펩티드 사슬의 일부일 수 있다 (Harlow et al., 1999, Using Antibodies: A Laboratory Manual, Cold Spring Harbor Laboratory Press, N.Y.; Harlow et al., 1989, Antibodies: A Laboratory Manual, Cold Spring Harbor, N.Y.; Houston et al., 1988, Proc. Natl. Acad. Sci. USA 85:5879-5883; Bird et al., 1988, Science 242:423-426). 일부 양태에서, 제1 세포외 리간드 결합 도메인은 항체 단편을 포함하는 항원 결합 도메인을 포함한다. 추가 양태에서, 제1 세포외 리간드 결합 도메인은 scFv 또는 sdAb를 포함하는 항체 단편을 포함한다.
본원에서 사용되는 바와 같이 용어 "항체"는 항원에 특이적으로 결합하는 면역글로불린 분자로부터 유래된 단백질 또는 폴리펩티드 서열을 지칭한다. 항체는 폴리클로날 또는 모노클로날 기원의 온전한 면역글로불린, 또는 이의 단편일 수 있고 천연 또는 재조합 공급원으로부터 유래될 수 있다.
용어 "항체 단편" 또는 "항체 결합 도메인"은 항원 결합 도메인, 즉, 항원 및 이의 정의된 에피토프와 같은 표적에 대한 항체 단편의 인식 및 특이적 결합을 부여하기에 충분한 온전한 항체의 항원성 결정 가변 영역을 함유하는 항체, 또는 이의 재조합 변이체의 적어도 하나의 부분을 지칭한다. 항체 단편의 예는 Fab, Fab', F(ab')2, 및 Fv 단편, 단일 사슬 (sc)Fv ("scFv") 항체 단편, 선형 항체, 단일 도메인 항체 (약칭 "sdAb") (VL 또는 VH), 낙타류 VHH 도메인, 및 항체 단편으로부터 형성된 다중특이성 항체를 포함하지만, 이에 제한되지 않는다.
용어 "scFv"는 경쇄의 가변 영역을 포함하는 적어도 하나의 항체 단편 및 중쇄의 가변 영역을 포함하는 적어도 하나의 항체 단편을 포함하는 융합 단백질을 지칭하고, 여기서 경쇄 및 중쇄 가변 영역은 짧은 유연한 폴리펩티드 링커를 통해 연속적으로 연결되고, 단일 폴리펩티드 사슬로 발현될 수 있으며, 여기서 scFv는 그것이 유래된 온전한 항체의 특이성을 유지한다.
항체와 관련하여 "중쇄 가변 영역" 또는 "VH" (또는, 단일 도메인 항체의 경우, 예를 들어, 나노바디, "VHH")는 프레임워크 영역으로 알려진 플랭킹 스트레치 사이에 개재된 개의 CDR을 포함하는 중쇄의 단편을 지칭하고, 이러한 프레임워크 영역은 일반적으로 CDR보다 더 많이 보존되며 CDR을 지지하는 스캐폴드를 형성한다.
명시되지 않는 한, 본원에 사용된 바와 같이 scFv는 예를 들어, 폴리펩티드의 N-말단 및 C-말단과 관련하여, 순서대로 VL 및 VH 가변 영역을 가질 수 있으며, scFv VL-링커-VH를 포함하거나 VH-링커-VL을 포함할 수 있다.
일부 구현예에서, 활성화제 및/또는 억제 수용체의 항원 결합 도메인은 scFv를 포함한다. 일부 구현예에서, scFv는 링커에 의해 연결된 VL 및 VH 영역을 포함한다. 일부 구현예에서, 링커는 글리신 세린 링커, 예를 들어 GGGGSGGGGSGGGGSGG (서열 번호 136)를 포함한다. 일부 구현예에서, scFv는 scFv의 N 말단에서 신호 서열을 추가로 포함한다. 예시적인 신호 서열은 MDMRVPAQLLGLLLLWLRGARC (서열 번호 137)를 포함하고, 이는 ATGGACATGAGGGTCCCCGCTCAGCTCCTGGGGCTCCTGCTACTCTGGCTCCGAGGTGCCAGATGT (서열 번호 138) 또는 ATGGATATGAGAGTGCCTGCCCAGCTGCTCGGACTGCTCCTTCTGTGGTTGAGAGGAGCTCGGTGC (서열 번호 917)에 의해 암호화된다.
용어 "항체 경쇄"는 항체 분자에 자연적으로 발생하는 형태로 존재하는 두 가지 유형의 폴리펩티드 사슬 중 더 작은 것을 지칭한다. 카파 ("κ") 및 람다 ("λ") 경쇄는 2개의 주요 항체 경쇄 아이소타입을 지칭한다.
용어 "재조합 항체"는 예를 들어, 박테리오파지 또는 효모 발현 시스템에 의해 발현된 항체와 같은 재조합 DNA 기술을 사용하여 생성된 항체를 지칭한다. 용어는 또한, 항체를 암호화하는 DNA 분자의 합성에 의해 생성되고 DNA 분자가 항체 단백질을 발현하는 항체 또는 항체를 지정하는 아미노산 서열을 의미하는 것으로 해석되어야 하며, 여기서 DNA 또는 아미노산 서열은 이용 가능하고 당업계에 잘 알려진 재조합 DNA 또는 아미노산 서열 기술을 사용하여 수득된다.
용어 "Vβ 도메인", "Vβ-단독 도메인", "β 사슬 가변 도메인" 또는 "단일 가변 도메인 TCR (svd-TCR)"은 제2 TCR 가변 도메인의 부재 하에 항원에 특이적으로 결합하는 단일 T 세포 수용체 (TCR) 베타 가변 도메인으로 본질적으로 구성된 항원 결합 도메인을 지칭한다. Vβ-단독 도메인은 상보성 결정 영역 (CDR)을 사용하여 항원과 결합한다. 각각의 Vβ-단독 도메인은 3개의 보체 결정 영역 (CDR1, CDR2, 및 CDR3)을 함유한다. 추가 요소는 Vβ 도메인이 제2 TCR 가변 도메인이 없을 때 에피토프에 결합하도록 구성된다면 조합될 수 있다.
일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 항체 단편, 단쇄 Fv 항체 단편 (scFv), 또는 β 사슬 가변 도메인 (Vβ)을 포함한다.
일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 TCR α 사슬 가변 도메인 및 TCR β 사슬 가변 도메인을 포함한다.
일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 scFv 항원 결합 도메인을 포함한다. 예시적인 EGFR scFv는 하기 표 1에 나타낸다.
표 1. EGFR scFv 도메인
일부 구현예에서, 활성화제 리간드는 EGFR 또는 이의 펩티드 항원이고, 활성화제 리간드 결합 도메인은 EGFR 결합 도메인을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 9-18 중 어느 하나의 서열을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 9-18 중 어느 하나와 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, EGFR 리간드 결합 표 1의 서열 그룹으로부터 선택된 서열에 의해 암호화된다. 일부 구현예에서, EGFR 리간드 결합 도메인은 표 1의 서열에 대해 적어도 80% 동일성, 적어도 85% 동일성, 적어도 90% 동일성, 적어도 95% 동일성 또는 적어도 99% 동일성을 갖는 서열에 의해 암호화된다. EGFR에 결합하는 본 개시내용의 scFv 도메인은 EGFR의 하나 이상의 변이체와 교차 반응할 수 있음이 이해된다. 예를 들어, scFv는 적어도 하나의 정상 EGFR, EGFR의 변이체, 또는 돌연변이 EGFR (예를 들어 EGFRvIII)에 결합할 수 있다.
일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 9-18 중 어느 하나에 대해 적어도 80% 동일성, 적어도 85% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 97% 동일성 또는 적어도 99% 동일성을 갖는 scFv 항원 결합 도메인을 포함한다. 일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 9-18 중 어느 하나의 서열을 포함하는 ScFv 항원 결합 도메인을 포함한다. 일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 9-18로 이루어진 그룹으로부터 선택된 서열로 본질적으로 이루어진다.
표 2. EGFR 가변 중쇄 (VH) 및 가변 경쇄 (VL) 도메인
일부 구현예에서, 활성화제 리간드는 EGFR 또는 이의 펩티드 항원이고, 활성화제 리간드 결합 도메인은 EGFR 리간드 결합 도메인을 포함한다. 일부 구현예에서, EGFR 결합 도메인은 표 2에 개시된 그룹으로부터 선택된 VH 및/또는 VL 도메인 또는 이에 대해 적어도 90% 동일성, 적어도 95% 동일성, 적어도 97% 동일성 또는 적어도 99% 동일성을 갖는 서열을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 19-24로 이루어진 그룹으로부터 선택된 VH 도메인을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 19-24로 이루어진 그룹으로부터 선택된 VH 또는 이에 대해 적어도 90%, 적어도 95% 또는 적어도 99% 동일성을 갖는 서열을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 25-30으로 이루어진 그룹으로부터 선택된 VL 도메인을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 25-30으로 이루어진 그룹으로부터 선택된 VL 또는 이에 대해 적어도 90%, 적어도 95% 또는 적어도 99% 동일성을 갖는 서열을 포함한다.
표 3. EGFR 항원 결합 도메인 CDR.
일부 구현예에서, 활성화제 리간드는 EGFR 또는 이의 펩티드 항원이고, 활성화제 리간드 결합 도메인은 EGFR 리간드 결합 도메인이다. 일부 구현예에서, EGFR 결합 도메인은 표 3에 개시된 CDR 그룹에서 선택된 상보성 결정 영역 (CDR)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 표 3에 개시된 CDR에 대해 적어도 95% 서열 동일성을 갖는 CDR을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 31-65로부터 선택된 CDR을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 31-36으로 이루어진 그룹으로부터 선택된 중쇄 CDR 1 (CDR H1)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 37-42로 이루어진 그룹으로부터 선택된 중쇄 CDR 2 (CDR H2)를 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 43-48로 이루어진 그룹으로부터 선택된 중쇄 CDR 3 (CDR H3)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 49-54로 이루어진 그룹으로부터 선택된 경쇄 CDR 1 (CDR L1)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 55-59로 이루어진 그룹으로부터 선택된 경쇄 CDR 2 (CDR L2)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 60-65로 이루어진 그룹으로부터 선택된 경쇄 CDR 3 (CDR L3)을 포함한다. 일부 구현예에서, EGFR 리간드 결합 도메인은 서열 번호 31-36으로부터 선택된 CDR H1, 서열 번호 37-42로부터 선택된 CDR H2, 서열 번호 43-48로부터 선택된 CDR H3, 서열 번호 49-54로부터 선택된 CDR L1, 서열 번호 55-59로부터 선택된 CDR L2, 및 서열 번호 60-65로부터 선택된 CDR L3을 포함한다.
일부 구현예에서, 본원에 제공된 항원 결합 도메인의 CDR에서 하나 이상의 (예를 들어, 1, 2, 3, 4, 5, 또는 6) 아미노산 잔기는 또 다른 아미노산으로 치환된다. 치환은 동일한 패밀리의 아미노산 내의 치환이라는 의미에서 "보존적"일 수 있다. 자연적으로 발생하는 아미노산은 다음 4가지 패밀리로 나눌 수 있으며 이러한 패밀리 내에서 보존적 치환이 일어날 것이다: (1) 염기성 측쇄를 갖는 아미노산: 라이신, 아르기닌, 히스티딘; (2) 산성 측쇄를 갖는 아미노산: 아스파르트산, 글루탐산; (3) 하전되지 않은 극성 측쇄를 갖는 아미노산: 아스파라긴, 글루타민, 세린, 트레오닌, 티로신; 및 (4) 비극성 측쇄를 갖는 아미노산: 글리신, 알라닌, 발린, 류신, 이소류신, 프롤린, 페닐알라닌, 메티오닌, 트립토판, 시스테인. 항체의 CDR의 아미노산 서열을 아미노산의 첨가, 결실 또는 치환에 의해 변화시킴으로써, 표적 항원에 대한 결합 친화력 증가와 같은 다양한 효과를 얻을 수 있다.
키메라 항원 수용체 (CAR)
본 개시내용은 제1, 활성화제 수용체 및 이를 포함하는 면역세포를 제공한다. 일부 구현예에서, 제1 수용체는 키메라 항원 수용체이다.
일부 구현예에서, 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
본원에서 사용되는 바와 같이, 용어 "키메라 항원 수용체 (CAR)"는 T-세포 수용체로부터 유래된 인공 수용체를 지칭할 수 있고 인공 특이성을 특정 면역 효과기 세포에 이식하는 조작된 수용체를 포함한다. CAR은 단일클론 항체의 특이성을 T 세포에 부여하여, 예를 들어, 입양 세포 요법에 사용하기 위해 많은 수의 특정 T 세포가 생성되도록 하는 데 사용될 수 있다. 특정 구현예에서, CAR은 예를 들어 종양 관련 항원에 대한 세포의 특이성을 지시한다. 예시적인 CAR은 세포 내 활성화 도메인, 막관통 도메인, 및 종양 관련 항원 결합 영역을 포함하는 세포외 도메인을 포함한다. 일부 구현예에서, CAR은 힌지 도메인을 추가로 포함한다. 특정 양태에서, CAR은 CD3 막관통 도메인 및 엔도도메인에 융합되는, 단일클론 항체로부터 유도된 단일 사슬 가변 단편 (scFv)을 포함한다. 다른 CAR 디자인의 특이성은 수용체의 리간드 (예를 들어, 펩티드)에서 파생될 수 있다. 특정 경우에, CAR은 추가적인 공동자극 신호전달을 위한 도메인, 예를 들어 CD3, 4-1BB, FcR, CD27, CD28, CD137, DAP10, 및/또는 OX40을 포함한다. 일부 경우에, 공동자극 분자, 영상화를 위한 리포터 유전자, 전구약물 첨가 시 T 세포를 조건부로 제거하는 유전자 산물, 귀소 수용체, 사이토카인 및 사이토카인 수용체를 포함하는 분자가 CAR과 함께 발현될 수 있다.
일부 구현예에서, 제1 수용체의 세포외 리간드 결합 도메인은 CAR의 세포외 도메인에 융합된다.
일부 구현예에서, 본 개시내용의 CAR은 세포외 힌지 영역을 포함한다. 힌지 영역의 통합은 CAR-T 세포의 사이토카인 생산에 영향을 미치고 생체 내 CAR-T 세포의 확장을 향상시킬 수 있다. 예시적인 힌지는 IgD 및 CD8 도메인, 예를 들어 IgG1로부터 단리되거나 유래될 수 있다. 일부 구현예에서, 힌지는 CD8α 또는 CD28로부터 단리되거나 유래된다.
일부 구현예에서, 힌지는 CD8α 또는 CD28로부터 단리되거나 유래된다. 일부 구현예에서, CD8α 힌지는 TTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACD (서열 번호 66)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD8α 힌지는 서열 번호 66을 포함한다. 일부 구현예에서, CD8α 힌지는 서열 번호 66으로 본질적으로 이루어진다. 일부 구현예에서, CD8α 힌지는 ACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGAT (서열 번호 67)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD8α 힌지는 서열 번호 67에 의해 암호화된다.
일부 구현예에서, CD28 힌지는 CTIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKP (서열 번호 68)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD28 힌지는 서열 번호 68을 포함하거나 본질적으로 이루어진다. 일부 구현예에서, CD28 힌지는 TGTACCATTGAAGTTATGTATCCTCCTCCTTACCTAGACAATGAGAAGAGCAATGGAACCATTATCCATGTGAAAGGGAAACACCTTTGTCCAAGTCCCCTATTTCCCGGACCTTCTAAGCCC (서열 번호 69)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD28 힌지는 서열 번호 69에 의해 암호화된다.
본 개시내용의 CAR은 CAR의 세포외 도메인에 융합되는 막관통 도메인을 포함하도록 설계될 수 있다. 일부 구현예에서, CAR의 도메인 중 하나와 자연적으로 연관된 막관통 도메인이 사용된다. 예를 들어, CD28 공동 자극 도메인을 포함하는 CAR은 또한 CD28 막관통 도메인을 사용할 수 있다. 일부 경우에, 막관통 도메인은 수용체 복합체의 다른 구성원과의 상호작용을 최소화하기 위해 동일하거나 상이한 표면 막 단백질의 막관통 도메인에 대한 이러한 도메인의 결합을 피하기 위해 아미노산 치환에 의해 선택되거나 변형될 수 있다.
막관통 도메인은 천연 또는 합성 공급원으로부터 유래될 수 있다. 공급원이 천연인 경우, 도메인은 막 결합 단백질 또는 막관통 단백질에서 유래될 수 있다. 막관통 T-세포 수용체의 알파, 베타 또는 제타 사슬, CD28, CD3 입실론, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154, 또는 IgG4와 같은 면역글로불린의 적어도 막관통 영역(들)로부터 단리되거나 유래될 수 있다 (즉 포함한다. 대안적으로, 막관통 도메인은 합성일 수 있으며, 이 경우 그것은 주로 류신 및 발린과 같은 소수성 잔기를 포함할 것이다. 일부 구현예에서, 페닐알라닌, 트립토판 및 발린의 삼중체는 합성 막관통 도메인의 각 말단에서 발견될 것이다. 임의로, 짧은 올리고- 또는 폴리펩티드 링커, 바람직하게는 2 내지 10개 아미노산 길이가 막관통 도메인과 CAR의 세포질 신호전달 도메인 사이의 연결을 형성할 수 있다. 글리신-세린 이중체는 특히 적합한 링커를 제공한다.
본 개시내용의 CAR의 일부 구현예에서, CAR은 CD28 막관통 도메인을 포함한다. 일부 구현예에서, CD28 막관통 도메인은 FWVLVVVGGVLACYSLLVTVAFIIFWV (서열 번호 70)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD28 막관통 도메인은 서열 번호 70을 포함하거나 본질적으로 이루어진다. 일부 구현예에서, CD28 막관통 도메인은 TTCTGGGTGCTGGTCGTTGTGGGCGGCGTGCTGGCCTGCTACAGCCTGCTGGTGACAGTGGCCTTCATCATCTTTTGGGTG (서열 번호 71)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD28 막관통 도메인은 서열 번호 71에 의해 암호화된다.
본 개시내용의 CAR의 일부 구현예에서, CAR은 IL-2R베타 막관통 도메인을 포함한다. 일부 구현예에서, IL-2R베타 막관통 도메인은 IPWLGHLLVGLSGAFGFIILVYLLI (서열 번호 72)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, IL-2R베타 막관통 도메인은 서열 번호 72을 포함하거나 본질적으로 이루어진다. 일부 구현예에서, IL-2R베타 막관통 도메인은 ATTCCGTGGC TCGGCCACCT CCTCGTGGGC CTCAGCGGGG CTTTTGGCTT CATCATCTTA GTGTACTTGC TGATC (서열 번호 73)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, IL-2R베타 막관통 도메인은 서열 번호 73에 의해 암호화된다.
본 개시내용의 CAR의 세포질 도메인 또는 그렇지 않으면 세포 내 신호전달 도메인은 AR이 배치된 면역 세포의 정상 효과기 기능 중 적어도 하나의 활성화를 담당한다. 용어 "효과기 기능"은 세포의 특화된 기능을 지칭한다. 따라서 용어 "세포 내 신호전달 도메인"은 효과기 기능 신호를 변환하고 세포가 특화된 기능을 수행하도록 지시하는 단백질 부분을 지칭한다. 일반적으로 전체 세포 내 신호전달 도메인이 사용될 수 있지만, 많은 경우에 전체 도메인을 사용할 필요는 없다. 세포 내 신호전달 도메인의 절단된 부분이 사용되는 한, 이러한 절단된 부분은 효과기 기능 신호를 변환하는 한 온전한 사슬 대신에 사용될 수 있다. 일부 경우에, 다수의 세포 내 도메인은 도메인이 조합되어 본 개시내용의 CAR-T 세포의 원하는 기능을 달성할 수 있다. 용어 세포 내 신호전달 도메인은 따라서 전달하기에 충분한 하나 이상의 세포 내 신호전달 도메인의 임의의 절단된 부분을 포함하는 것을 의미한다.
본 개시내용의 CAR에 사용하기 위한 세포 내 신호전달 도메인의 예는 T 세포 수용체 (TCR) 및 항원 수용체 후 신호 전달을 개시하기 위해 함께 작용하는 공동-수용체의 세포질 서열, 뿐만 아니라 이들 서열의 임의의 유도체 또는 변이체 및 동일한 기능적 능력을 갖는 임의의 합성 서열을 포함한다.
따라서, 본 개시내용의 CAR의 세포 내 도메인은 적어도 하나의 세포질 활성화 도메인을 포함한다. 일부 구현예에서, 세포 내 활성화 도메인은 CAR T-세포의 효과기 기능을 활성화하는 데 필요한 T-세포 수용체 (TCR) 신호전달이 있음을 보장한다. 일부 구현예에서, 적어도 하나의 세포질 활성화는 CD247 분자 (CD3ζ) 활성화 도메인, 자극 킬러 면역글로불린 유사 수용체 (KIR) KIR2DS2 활성화 도메인, 또는 12 kDa의 DNAX-활성화 단백질 (DAP12) 활성화 도메인이다.
일부 구현예에서, CD3ζ 활성화 도메인은 RVKFSRSADAPAYKQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (서열 번호 74)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD3ζ 활성화 도메인은 서열 번호 74를 포함하거나 본질적으로 이루어진다. 일부 구현예에서, CD3ζ 활성화 도메인은 AGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACAAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGCGTAGAGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGACTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGC (서열 번호 75)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD3ζ 활성화 도메인은 서열 번호 75에 의해 암호화된다.
TCR 단독으로 생성된 신호는 종종 T 세포의 완전한 활성화에 불충분하며 2차 또는 공동 자극 신호도 필요하다는 것이 알려져 있다. 따라서, T 세포 활성화는 세포질 신호 서열의 2가지 별개 부류에 의해 매개된다고 말할 수 있다: TCR을 통해 항원-의존성 1차 활성화를 개시하는 서열 (1차 세포질 신호전달 서열) 및 항원-비의존적 방식으로 작용하여 2차 또는 공동 자극 신호를 제공하는 서열 (2차 세포질 신호전달 서열).
1차 세포질 신호전달 서열은 자극 방식 또는 억제 방식으로 TCR 복합체의 1차 활성화를 조절한다. 자극 방식으로 작용하는 1차 세포질 신호전달 서열은 면역수용체 티로신-기반 활성화 모티프 또는 ITAM으로 알려진, 신호전달 모티프를 함유할 수 있다. 일부 구현예에서, ITAM은 류신 또는 이소류신으로부터 임의의 2개의 다른 아미노산 (YxxL/I) (서열 번호 157)에 의해 분리된 티로신을 함유한다. 일부 구현예에서, 세포질 도메인은 1, 2, 3, 4 또는 5개의 ITAM을 함유한다. 세포질 도메인을 함유하는 예시적인 ITAM은 CD3ζ 활성화 도메인이다. 본 개시내용의 CAR에 사용될 수 있는 1차 세포질 신호전달 서열을 함유하는 ITAM의 추가 예는 TCRζ, FcRγ, FcRβ, CD3γ, CD3δ, CD3ε, CD3ζ, CD5, CD22, CD79a, CD79b, 및 CD66d로부터 유래된 것들을 포함한다.
일부 구현예에서, 단일 ITAM을 포함하는 CD3ζ 활성화 도메인은 RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLHMQALPPR (서열 번호 76)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD3ζ 활성화 도메인은 서열 번호 76을 포함한다. 일부 구현예에서, 단일 ITAM을 포함하는 CD3ζ 활성화 도메인은 RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLHMQALPPR (서열 번호 76)의 아미노산 서열로 본질적으로 이루어진다. 일부 구현예에서, 단일 ITAM을 포함하는 CD3ζ 활성화 도메인은
AGAGTGAAGT TCAGCAGGAG CGCAGACGCC CCCGCGTACC AGCAGGGCCA GAACCAGCTC
TATAACGAGC TCAATCTAGG ACGAAGAGAG GAGTACGATG TTTTGCACAT GCAGGCCCTG
CCCCCTCGC (서열 번호 77)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD3ζ 활성화 도메인은 서열 번호 77에 의해 암호화된다.
일부 구현예에서, CAR의 세포질 도메인은 그 자체로 CD3ζ 신호전달 도메인을 포함하거나 본 개시내용의 CAR의 맥락에서 유용한 임의의 다른 원하는 세포질 도메인(들)과 조합되도록 설계될 수 있다. 예를 들어, CAR의 세포질 도메인은 CD3ζ 사슬 부분 및 공동 자극 도메인을 포함할 수 있다. 공동 자극 도메인은 공동자극 분자의 세포 내 도메인을 포함하는 CAR의 일부를 지칭한다. 공동자극 분자는 항원에 대한 림프구의 효율적인 반응에 필요한 항원 수용체 또는 그 리간드 이외의 세포 표면 분자이다. 이러한 분자의 예는 IL-2Rβ, Fc 수용체 감마 (FcRγ), Fc 수용체 베타 (FcRβ), CD3g 분자 감마 (CD3γ), CD3δ, CD3ε, CD5 분자 (CD5), CD22 분자 (CD22), CD79a 분자 (CD79a), CD79b 분자 (CD79b), 암 배아 항원 관련 세포 부착 분자 3 (CD66d), CD27 분자 (CD27), CD28 분자 (CD28), TNF 수용체 수퍼패밀리 구성원 9 (4-1BB), TNF 수용체 수퍼패밀리 구성원 4 (OX40), TNF 수용체 수퍼패밀리 구성원 8 (CD30), CD40 분자 (CD40), 프로그램된 세포 사멸 1 (PD-1), 유도성 T 세포 공동자극 (ICOS), 림프구 기능 관련 항원-1 (LFA-1), CD2 분자 (CD2), CD7 분자 (CD7), TNF 수퍼패밀리 구성원 14 (LIGHT), 킬러 세포 렉틴 유사 수용체 C2 (NKG2C) 및 CD276 분자 (B7-H3) c-자극 도메인, 또는 이의 기능적 단편으로 이루어진 그룹으로부터 선택된 공동 자극 도메인을 포함한다. 일부 구현예에서, 본 개시내용의 CAR의 세포 내 도메인은 적어도 하나의 공동 자극 도메인을 포함한다. 일부 구현예에서, 공동 자극 도메인은 CD28로부터 단리되거나 유래된다.
일부 구현예에서, 본 개시내용의 CAR의 세포 내 도메인은 적어도 하나의 공동 자극 도메인을 포함한다. 일부 구현예에서, 공동 자극 도메인은 CD28 로부터 단리되거나 유래된다. 일부 구현예에서, CD28 공동 자극 도메인은 RSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS (서열 번호 78)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, CD28 공동 자극 도메인은 서열 번호 78을 포함하거나 본질적으로 이루어진다. 일부 구현예에서, CD28 공동 자극 도메인은 AGGAGCAAGCGGAGCAGACTGCTGCACAGCGACTACATGAACATGACCCCCCGGAGGCCTGGCCCCACCCGGAAGCACTACCAGCCCTACGCCCCTCCCAGGGATTTCGCCGCCTACCGGAGC (서열 번호 79)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다. 일부 구현예에서, CD28 공동 자극 도메인은 서열 번호 79에 의해 암호화된다. 본 개시내용의 CAR의 세포질 신호전달 부분 내 세포질 도메인은 무작위 또는 지정된 순서로 서로 연결될 수 있다. 임의로, 짧은 올리고- 또는 폴리펩티드 링커, 예를 들어 2 내지 10개 아미노산 길이는 연결을 형성할 수 있다. 글리신-세린 이중체는 적합한 예시를 제공한다.
일부 구현예에서, 공동 자극 도메인은 4-1BB로부터 단리되거나 유래된다. 일부 구현예에서, 4-1BB 공동 자극 도메인은 KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL (서열 번호 159)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, 4-1BB 공동 자극 도메인은 KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL (서열 번호 159)를 포함하거나 본질적으로 이루어진다. 일부 구현예에서, 4-1BB 공동 자극 도메인은 aaacggggcagaaagaaactcctgtatatattcaaacaaccatttatgaggccagtacaaactactcaagaggaagatggctgtagctgccgatttccagaagaagaagaaggaggatgtgaactg (서열 번호 160)의 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖거나 동일한 뉴클레오티드 서열에 의해 암호화된다.
일부 구현예에서, CAR의 세포 내 도메인은 CD28 공동 자극 도메인, 4-1BB 공동자극 도메인, 및 CD3ζ 활성화 도메인을 포함한다. 일부 구현예에서 CAR의 세포 내 도메인은 RSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYKQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (서열 번호 158)의 서열, 또는 이에 대해 적어도 80% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 99% 동일성을 갖는 서열을 포함한다.
본 개시내용의 CAR의 세포질 신호전달 부분 내 세포질 도메인은 무작위 또는 지정된 순서로 서로 연결될 수 있다. 임의로, 짧은 올리고- 또는 폴리펩티드 링커, 예를 들어 2 내지 10개 아미노산 길이는 연결을 형성할 수 있다. 글리신-세린 이중체는 적합한 예시를 제공한다. 예시적인 링커는 GGGGSGGGGSGGGGSGG (서열 번호 136)의 서열을 포함한다.
본 개시내용의 CAR의 세포질 신호전달 부분 내 세포질 도메인은 무작위 또는 지정된 순서로 서로 연결될 수 있다. 임의로, 짧은 올리고- 또는 폴리펩티드 링커, 예를 들어 2 내지 10개 아미노산 길이는 연결을 형성할 수 있다. 글리신-세린 이중체는 적합한 예시를 제공한다.
T 세포 수용체 (TCR)
본 개시내용은 제1, 활성화제 수용체 및 이를 포함하는 면역 세포를 제공한다. 일부 구현예에서, 제1 수용체는 T 세포 수용체 (TCR)이다.
본 개시내용에서 사용하기 위한 세포 내 도메인을 포함하는 예시적인 TCR은 2020년 9월 6일에 출원된 PCT/US2020/045250에 기재되어 있으며, 그 내용은 본원에 참조로 포함된다.
본원에서 사용되는 바와 같이, 때때로 "TCR 복합체" 또는 "TCR/CD3 복합체"라고도 하는 "TCR"은 TCR 알파 사슬, TCR 베타 사슬, 및 때때로 서브유닛으로 지칭되는, 불변 CD3 사슬 (제타, 감마, 델타 및 입실론) 중 하나 이상을 포함하는 단백질 복합체를 지칭한다. TCR 알파 및 베타 사슬은 펩티드-MHC 복합체에 결합하는 이종이량체로서 기능하도록 이황화 연결될 수 있다. TCR 알파/베타 이종이량체가 펩티드-MHC와 결합하면, 관련 불변 CD3 서브유닛에서 TCR 복합체의 구조적 변화가 유도되어, 인산화 및 다운스트림 단백질과의 결합을 유도하여 1차 자극 신호를 전달한다. 예시적인 TCR 복합체에서, TCR 알파 및 TCR 베타 폴리펩티드는 이종이량체를 형성하고, CD3 입실론 및 CD3 델타는 이종이량체를 형성하고, CD3 입실론 및 CD3 감마는 이종이량체를 형성하고, 2개의 CD3 제타는 동종이량체를 형성한다.
임의의 적합한 리간드 결합 도메인은 본원에 기재된 TCR의 세포외 도메인, 힌지 도메인 또는 막관통에 융합될 수 있다. 예를 들어, 리간드 결합 도메인은 항체 또는 TCR의 항원 결합 도메인이거나, 항체 단편, Vβ 단독 도메인, 선형 항체, 단일 사슬 가변 단편 (scFv), 또는 단일 도메인 항체 (sdAb)을 포함할 수 있다.
일부 구현예에서, 리간드 결합 도메인은 하나 이상의 TCR 서브유닛의 하나 이상의 세포외 도메인 또는 막관통 도메인에 융합된다. TCR 서브유닛은 TCR 알파, TCR 베타, CD3 델타, CD3 입실론, CD3 감마 또는 CD3 제타일 수 있다. 예를 들어, 리간드 결합 도메인은 TCR 알파, 또는 TCR 베타에 융합될 수 있거나, 리간드 결합의 일부는 2개의 서브유닛에 융합될 수 있으며, 예를 들어 리간드 결합 도메인의 일부는 TCR 알파 및 TCR 베타 모두에 융합될 수 있다.
TCR 서브유닛은 TCR 알파, TCR 베타, CD3 제타, CD3 델타, CD3 감마 및 CD3 입실론을 포함한다. TCR 알파, TCR 베타 사슬, CD3 감마, CD3 델타, CD3 입실론, 또는 CD3 제타, 또는 이의 단편 또는 유도체 중 임의의 하나 이상은 본 개시내용의 자극 신호를 제공할 수 있는 하나 이상의 도메인에 융합될 수 있으며, 이로써 TCR 기능 및 활동을 향상시킨다.
임의의 공급원으로부터 단리되거나 유래된 TCR 막관통 도메인은 본 개시내용의 범위 내에 있는 것으로 간주된다. 막관통 도메인은 천연 또는 재조합 공급원으로부터 유래될 수 있다. 공급원이 천연인 경우, 도메인은 임의의 막 결합 단백질 또는 막관통 단백질에서 파생될 수 있다.
일부 구현예에서, 막관통 도메인은 TCR 복합체가 표적에 결합할 때마다 세포 내 도메인(들)에 신호 전달할 수 있다. 본 개시내용의 특정 용도의 막관통 도메인은 적어도 예를 들어, TCR의 알파, 베타 또는 제타 사슬, CD3 델타, CD3 입실론 또는 CD3 감마, CD28, CD3 입실론, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154의 막관통 영역(들)을 포함할 수 있다.
일부 구현예에서, 막관통 도메인은 힌지, 예를 들어, 인간 단백질로부터의 힌지를 통해 TCR의 폴리펩티드의 세포외 영역, 예를 들어, TCR 알파 또는 베타 사슬의 항원 결합 도메인에 부착될 수 있다. 예를 들어, 힌지는 인간 면역글로불린 (Ig) 힌지, 예를 들어, IgG4 힌지, 또는 CD8a 힌지일 수 있다. 일부 구현예에서, 힌지는 CD8α 또는 CD28로부터 단리되거나 유래된다.
일부 구현예에서, 세포외 리간드 결합 도메인은 TCR의 하나 이상의 막관통 도메인에 부착된다. 일부 구현예에서, 막관통 도메인은 TCR 알파 막관통 도메인, TCR 베타 막관통 도메인, 또는 둘 모두를 포함한다. 일부 구현예에서, 막관통은 CD3 제타 막관통 도메인을 포함한다.
막관통 도메인 막관통 영역에 인접한 하나 이상의 추가 아미노산, 예를 들어, 막관통이 유래된 단백질의 세포외 영역과 관련된 하나 이상의 아미노산 (예를 들어, 세포외 영역의 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 또는 최대 15개 아미노산) 및/또는 막관통이 유래된 단백질의 세포 내 영역과 관련된 하나 이상의 아미노산 (세포 내 영역의 예를 들어, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 또는 최대 15개 아미노산)을 포함할 수 있다.
일부 구현예에서, 막관통 도메인은 이러한 도메인이 동일하거나 상이한 표면 막 단백질의 막관통 도메인에 결합하는 것을 피하기 위해, 예를 들어, 수용체 복합체의 다른 구성원과의 상호작용을 최소화하기 위해 아미노산 치환에 의해 선택되거나 변형될 수 있다.
존재하는 경우, 막관통 도메인은 천연 TCR 막관통 도메인, 이종 막 단백질로부터의 천연 막관통 도메인, 또는 인공 막관통 도메인일 수 있다. 막관통 도메인은 막 앵커 도메인일 수 있다. 제한 없이, 천연 또는 인공 막관통 도메인은 막관통 도메인은 약 20개 아미노산의 소수성 α-나선을 포함할 수 있으며, 종종 막관통 세그먼트의 측면에 양전하를 가진다. 막관통 도메인은 하나의 막관통 세그먼트 또는 하나 초과의 막관통 세그먼트를 가질 수 있다. 막관통 도메인/세그먼트의 예측은 공개적으로 이용 가능한 예측 도구를 사용하여 이루어질 수 있다 (예를 들어 TMHMM, Krogh et al. Journal of Molecular Biology 2001; 305(3):567-580; 또는 TMpred, Hofmann & Stoffel Biol. Chem. Hoppe-Seyler 1993; 347: 166). 막 앵커 시스템의 비제한적 예는 혈소판 유래 성장 인자 수용체 (PDGFR) 막관통 도메인, 글리코실포스파티딜이노시톨 (GPI) 앵커 (신호 서열에 번역 후 첨가됨) 등을 포함한다.
일부 구현예에서, 막관통 도메인은 TCR 알파 막관통 도메인을 포함한다. 일부 구현예에서, TCR 알파 막관통 도메인은 VIGFRILLLKVAGFNLLMTLRLW (서열 번호 80)의 서열에 대해 적어도 85% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 96% 동일성, 적어도 97% 동일성, 적어도 98% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, TCR 알파 막관통 도메인은 서열 번호 80을 포함하거나, 이로 본질적으로 구성된다. 일부 구현예에서, TCR 알파 막관통 도메인은 GTGATTGGGTTCCGAATCCTCCTCCTGAAAGTGGCCGGGTTTAATCTGCTCATGACGCTGCGGCTGTGG (서열 번호 81)의 서열에 의해 암호화된다.
일부 구현예에서, 막관통 도메인은 TCR 베타 막관통 도메인을 포함한다. 일부 구현예에서, TCR 베타 막관통 도메인은 TILYEILLGKATLYAVLVSALVL (서열 번호 82)의 서열에 대해 적어도 85% 동일성, 적어도 90% 동일성, 적어도 95% 동일성, 적어도 96% 동일성, 적어도 97% 동일성, 적어도 98% 동일성, 적어도 99% 동일성을 갖거나 동일한 아미노산 서열을 포함한다. 일부 구현예에서, TCR 베타 막관통 도메인은 서열 번호 82를 포함하거나, 이로 본질적으로 구성된다. 일부 구현예에서, TCR 베타 막관통 도메인은 ACCATCCTCTATGAGATCTTGCTAGGGAAGGCCACCTTGTATGCCGTGCTGGTCAGTGCCCTCGTGCTG (서열 번호 83)의 서열에 의해 암호화된다.
본 개시내용의 TCR은 하나 이상의 세포 내 도메인을 포함할 수 있다. 일부 구현예에서, 세포 내 도메인은 막관통 도메인에 자극 신호를 제공할 수 있는 하나 이상의 도메인을 포함한다. 일부 구현예에서, 세포 내 도메인은 자극 신호를 제공할 수 있는 제1 세포 내 도메인 및 자극 신호를 제공할 수 있는 제2 세포 내 도메인을 포함한다. 다른 구현예에서, 세포 내 도메인은 자극 신호를 제공할 수 있는 제1, 제2 및 제3 세포 내 도메인을 포함한다. 자극 신호를 제공할 수 있는 세포 내 도메인은 CD28 분자 (CD28) 도메인, LCK 원발암유전자, Src 계열 티로신 키나제 (Lck) 도메인, TNF 수용체 수퍼패밀리 구성원 9 (4-1BB) 도메인, TNF 수용체 수퍼패밀리 구성원 18 (GITR) 도메인, CD4 분자 (CD4) 도메인, CD8a 분자 (CD8a) 도메인, FYN 원발암유전자, Src 계열 티로신 키나제 (Fyn) 도메인, T 세포 수용체 관련 단백질 키나제 70의 제타 사슬 (ZAP70) 도메인, T 세포 (LAT) 도메인의 활성화를 위한 링커, 림프구 세포질 단백질 2 (SLP76) 도메인, (TCR) 알파, TCR 베타, CD3 델타, CD3 감마 및 CD3 입실론 세포 내 도메인으로 이루어진 그룹으로부터 선택된다.
일부 구현예에서, 세포 내 도메인은 적어도 하나의 세포 내 신호전달 도메인을 포함한다. 세포 내 신호전달 도메인은 세포의 기능, 예를 들어 TCR 함유 세포, 예를 들어, TCR- 발현 T-세포의 면역 효과기 기능을 촉진하는 신호를 생성한다. 일부 구현예에서, 본 개시내용의 제1 수용체의 세포 내 도메인은 적어도 하나의 세포 내 신호전달 도메인을 포함한다. 예를 들어, CD3 감마, 델타 또는 입실론의 세포 내 도메인은 신호전달 도메인을 포함한다.
일부 구현예에서, 세포외 도메인, 막관통 도메인 및 세포 내 도메인은 동일한 단백질, 예를 들어 T-세포 수용체 (TCR) 알파, TCR 베타, CD3 델타, CD3 감마, CD3 입실론 또는 CD3 제타로부터 단리되거나 유래된다.
본 개시내용의 활성화제 수용체에서 사용하기 위한 세포 내 도메인의 예는 TCR 알파, TCR 베타, CD3 제타, 및 4-1BB의 세포질 서열, 및 항원 수용체 결합 후 신호 전달을 개시하기 위해 함께 작용하는 세포 내 신호전달 공동-수용체, 뿐만 아니라 이들 서열의 임의의 유도체 또는 변이체 및 동일한 기능적 능력을 갖는 임의의 재조합 서열을 포함한다.
일부 구현예에서, 세포 내 신호전달 도메인은 1차 세포 내 신호전달 도메인을 포함한다. 예시적인 1차 세포 내 신호전달 도메인은 1차 자극, 또는 항원 의존적 자극을 담당하는 단백질로부터 유도된 것들을 포함한다.
일부 구현예에서, 세포 내 도메인은 CD3 델타 세포 내 도메인, CD3 입실론 세포 내 도메인, CD3 감마 세포 내 도메인, CD3 제타 세포 내 도메인, TCR 알파 세포 내 도메인 또는 TCR 베타 세포 내 도메인을 포함한다.
일부 구현예에서, 세포 내 도메인은 TCR 알파 세포 내 도메인을 포함한다. 일부 구현예에서, TCR 알파 세포 내 도메인은 Ser-Ser를 포함한다. 일부 구현예에서, TCR 알파 세포 내 도메인은 TCCAGC의 서열에 의해 암호화된다.
일부 구현예에서, 세포 내 도메인은 TCR 베타 세포 내 도메인을 포함한다. 일부 구현예에서, TCR 베타 세포 내 도메인은 다음 서열에 대해 적어도 80% 동일성, 적어도 90% 동일성을 갖거나 동일한 아미노산 서열을 포함한다: MAMVKRKDSR (서열 번호 84). 일부 구현예에서, TCR 베타 세포 내 도메인은 서열 번호 84를 포함하거나, 이로 본질적으로 구성된다. 일부 구현예에서, TCR 베타 세포 내 도메인은 ATGGCCATGGTCAAGAGAAAGGATTCCAGA (서열 번호 85)의 서열에 의해 암호화된다.
일부 구현예에서, 세포 내 신호전달 도메인은 적어도 하나의 자극 세포 내 도메인을 포함한다. 일부 구현예에서, 세포 내 신호전달 도메인은 1차 세포 내 신호전달 도메인, 예를 들어 CD3 델타, CD3 감마 및 CD3 입실론 세포 내 도메인, 및 하나의 추가 자극 세포 내 도메인, 예를 들어 공동 자극 도메인을 포함한다. 일부 구현예에서, 세포 내 신호전달 도메인은 1차 세포 내 신호전달 도메인, 예를 들어 CD3 델타, CD3 감마 및 CD3 입실론 세포 내 도메인, 및 2개의 추가 자극 세포 내 도메인을 포함한다.
예시적인 공동 자극 세포 내 신호전달 도메인은 공동 자극 신호, 또는 항원 독립적 자극을 담당하는 단백질로부터 유도된 것들을 포함한다. 공동 자극 분자는 MHC 클래스 I 분자, BTLA, Toll 리간드 수용체, 뿐만 아니라 DAP10, DAP12, CD30, LIGHT, OX40, CD2, CD27, CDS, ICAM-1, LFA-1 (CD11a/CD18) 4-1BB (CD137, TNF 수용체 수퍼패밀리 구성원 9), 및 CD28 분자 (CD28)를 포함하지만, 이에 제한되지 않는다. 공동 자극 단백질은 다음 단백질 패밀리로 나타낼 수 있다: TNF 수용체 단백질, 면역글로불린 유사 단백질, 사이토카인 수용체, 인테그린, 신호전달 림프구 c 활성화 분자 (SLAM 단백질), 및 활성화 NK 세포 수용체. 이러한 분자의 예는 CD27, CD28, 4-1BB (CD137), OX40, GITR, CD30, CD40, ICOS, BAFFR, HVEM, 림프구 기능 관련 항원-1 (LFA-1), CD2, CD7, LIGHT, NKG2C, SLAMF7, NKp80, CD160, B7-H3, CD83, CD4와 특이적으로 결합하는 리간드 등을 포함한다. 공동 자극 도메인은 이것이 유래된 분자, 또는 이의 기능적 변이체의 전체 세포 내 부분, 또는 전체 천연 세포 내 신호전달 도메인을 포함할 수 있다.
일부 구현예에서, 자극 도메인은 공동 자극 도메인을 포함한다. 일부 구현예에서, 공동 자극 도메인은 CD28 또는 4-1BB 공동 자극 도메인을 포함한다. CD28 및 4-1BB 완전한 T 세포 활성화에 필요하고 T 세포 효과기 기능을 향상시키는 것으로 알려진 잘 특성화된 공동 자극 분자이다. 예를 들어, CD28 및 4-1BB는 키메라 항원 수용체 (CAR)에 활용되어 CD3 제타 신호전달 도메인만을 함유하는 1세대 CAR에 비해 사이토카인 방출, 세포 용해 기능, 및 지속성을 향상시킨다. 마찬가지로, TCR에 공동 자극 도메인, 예를 들어 CD28 및 4-1BB 도메인을 포함하면 T 세포 효과기 기능을 증가시킬 수 있으며 특히 일반적으로 표면에서 하향 조절되는 공동 자극 리간드가 없는 경우 공동 자극을 허용할 수 있다. 일부 구현예에서, 자극 도메인은 CD28 세포 내 도메인 또는 4-1BB 세포 내 도메인을 포함한다.
억제 수용체
본 개시내용은 암 세포에서 소실된 비-표적 항원, 예를 들어 유전자의 대립형질 변이체에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체를 제공한다. 비-표적 대립형질 변이체는 제한 없이 비-표적 대립형질 변이체 발현에 영향을 미치는 후생유전학적 변화, 비-표적 대립형질 변이체를 암호화하는 유전자에 대한 돌연변이, 비-표적 대립형질 변이체의 발현을 조절하는 세포 신호전달의 파괴, 염색체 소실, 게놈 유전자좌의 부분적 또는 완전한 결실, 핵산 또는 이종염색질의 변형을 통한 유전자 침묵, 또는 다른 메커니즘을 통한 발현 상실과 같은 임의의 메커니즘을 통해 암 세포에서 소실될 수 있다. 본원에 개시된 조성물 및 방법의 변형에서, 치료된 세포 또는 대상체는 비-유전적 변화로 인해 비-표적 대립형질 변이체의 발현 상실을 나타낼 수 있다. 따라서 본 개시내용은 세포를 죽이고/거나 이형접합성의 소실을 포함하지만 이에 제한되지 않는 임의의 원인으로부터 비-표적 항원의 발현이 결여된 개체를 치료하기 위한 조성물 및 방법을 제공한다.
비-표적 항원은 주요 조직적합성 복합체 클래스 I (MHC-I)와 복합체 내의 단백질, 또는 이의 항원 펩티드일 수 있고, 여기서 비-표적 항원은 다형성을 포함한다. 비-표적 항원은 다형성이기 때문에, 암세포에서 이형접합성의 소실을 통해 발생할 수 있는, 비-표적 항원을 암호화하는 유전자의 단일 카피의 소실은 유전자의 다른 다형성 변이체를 유지하는 암 세포를 산출하지만, 비-표적 항원을 소실한다. 예를 들어, HLA 유전자좌에서 HLA-A*02 및 HLA-A*01 대립유전자를 갖는 대상체는 HLA-A*02 대립유전자만 소실되는 암에 걸릴 수 있다. 그러한 대상체에서, 억제제 수용체가 HLA-A*02 (또는 다른 비-표적 항원)에 특이적인 것으로 설계되기 때문에 HLA-A*01 단백질은 존재하지만, 암 세포와 마주치는 면역 세포의 억제 수용체에 의해 인식되지 않는다. 정상 비-악성 세포에서, HLA-A*02 (또는 다른 비-표적 항원)가 존재하고 조작된 면역 세포의 활성화를 억제한다. 이형접합성이 소실된 암세포에서는, HLA-A*02 대립형질 변이체 (또는 다른 비-표적 항원)가 소실된다. 억제 수용체를 발현하도록 조작된 면역 세포는 억제 수용체가 암 세포에 없는 HLA-A*02 (또는 다른 비-표적 항원)에만 반응하기 때문에, 억제 수용체로부터 억제 신호를 받지 않는다. 이 메커니즘에 의해, 면역 세포는 선택적으로 활성화되어, EGFR을 발현하지만 이형 접합성의 소실로 인해 HLA-A*02 (또는 또 다른 비-표적 항원)를 잃은 암세포를 선택적으로 사멸시킨다. HLA-A를 여기서 예로 사용한다. 유사한 다형성 변이가 다른 MHC 유전자 및 다른 비-MHC 유전자 집단에서도 발생한다. 따라서, 일부 구현예에서, 비-표적 항원은 COLEC12, APCDD1 또는 CXCL16, 또는 HLA-A*02의 다형성 변이체를 포함한다. 일부 구현예에서, 비-표적 항원은 HLA 클래스 I 대립유전자 또는 마이너 조직적합성 항원 (MiHA)이다. 일부 구현예에서, HLA 클래스 I 대립유전자는 HLA-A, HLA-B, HLA-C, 또는 HLA-E를 포함한다. 일부 구현예에서, HLA 클래스 I 대립유전자는 HLA-A*02이다. 일부 구현예에서, HLA-A*02 비-표적 항원은 대상체의 건강한 세포에 의해 발현된다. 일부 구현예에서, 비-표적 항원은 비-표적 대립형질 변이체이다. 일부 구현예에서, 비-표적 대상체의 암 세포에서 발현되지 않는다. 일부 구현예에서, 비-표적 항원은 대상체의 종양에서 세포의 일부에서 발현되지 않는다. 일부 구현예에서, 대상체의 암 세포는 비-표적 항원의 발현을 상실했다. 세포에서 비-표적 항원의 발현 상실 또는 발현 부족은 제한 없이, 비-표적 유전자 발현에 영향을 미치는 후생유전학적 변화, 비-표적 항원을 암호화하는 유전자에 대한 돌연변이, 또는 비-표적 유전자의 발현을 조절하는 세포 신호전달의 파괴와 같은 임의의 메커니즘일 수 있다.
일부 구현예에서, 제2 수용체는 억제 키메라 항원 수용체 (즉 억제 수용체)이다. 일부 구현예에서, 제2 수용체는 억제 수용체이다. 일부 구현예에서, 제2 수용체는 인간화된다.
일부 구현예에서, 제2수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 174 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열.
본 개시내용은 비-표적 항원의 단일 아미노산 변이 대립유전자를 구별할 수 있는 세포외 리간드 결합을 포함하는 억제 수용체인, 제2 수용체를 제공한다. 비-표적 항원의 대립형질 변이체를 구별하는 이러한 능력은 제2 수용체가 리간드 결합 도메인에 의해 인식되는 대립유전자를 발현하는 비-표적 세포의 존재 하에 제2 수용체를 포함하는 면역 세포의 활성화를 억제할 수 있게 한다. 그러나, 면역 세포의 활성화는 대립유전자를 소실한 표적 세포, 예를 들어 이형 접합체의 소실을 통해 유전자의 하나의 대립 유전자를 소실한 암 세포의 존재 하에서 억제되지 않는다.
본 개시내용은 비-표적 항원의 상이한 발현 수준을 구별할 수 있는 세포외 리간드 결합을 포함하는 억제 수용체인 제2 수용체를 제공한다. 이는 제2 수용체가 제2 수용체에 대한 리간드를 발현하는 비-표적 세포의 존재 하에 제2 수용체를 포함하는 면역 세포의 활성화를 억제할 수 있게 하지만, 제2 수용체에 대한 리간드를 낮은 수준으로 발현하거나, 발현하지 않는 암 세포의 존재 하에 면역 세포의 활성화를 허용할 수 있다.
억제제 리간드
일부 구현예에서, 비-표적 항원은 표적 세포에 의해 발현되지 않고, 비-표적 세포에 의해 발현된다. 일부 구현예에서, 비-표적 항원은 건강한 세포, 즉 암세포가 아닌 세포에 의해 발현된다. 일부 구현예에서, 표적 세포는 이형접합성 소실 (LOH)을 통해 비-표적 항원의 발현을 상실한 다수의 암 세포이다. 일부 구현예에서, 비-표적 세포는 표적 및 비-표적 항원 모두를 발현하는 복수의 건강한 세포 (즉, 비-암, 정상, 또는 건강한 세포)이다.
표적 세포에 의해 발현되지 않는 비-표적 세포에 의해 발현되는 임의의 세포 표면 분자는 제2 수용체 세포외 리간드 결합 도메인에 대한 적합한 비-표적 항원일 수 있다. 예를 들어, 세포 부착 분자, 세포-세포 신호전달 분자, 세포외 도메인, 화학주성에 관여하는 분자, 당단백질, G 단백질 결합 수용체, 막관통, 신경전달물질 수용체 또는 전압 개폐 이온 채널은 비-표적 항원으로 사용할 수 있다.
일부 구현예에서, 표적 항원은 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내의 암 세포 특이적 항원의 펩티드 항원이다.
일부 구현예에서, 비-표적 항원은 이형접합성의 소실로 인해 암 세포에서 소실된다. 이형접합성의 소실로 인해 암 세포에서 소실된 예시적인 비-표적 항원은 COLEC12, APCDD1, CXCL16 및 HLA-A*02를 포함한다. 일부 구현예에서, 비-표적 항원은 COLEC12, APCDD1 및 CXCL166의 다형성 변이체, 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내의 이의 펩티드 항원, 또는 HLA-A*02로 이루어진 그룹으로부터 선택된다. 일부 구현예에서, 비-표적 항원은 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내의 COLEC12, APCDD1 및 CXCL16의 다형성 잔기를 포함하는 항원 펩티드이다.
HLA-A, HLA-B 또는 HLA-C 임의의 것을 포함하는 비-표적 MHC-1 (MHC) 항원은 본 개시내용의 범위 내에 있는 것으로 간주된다. 일부 구현예에서, 비-표적 항원은 HLA-A를 포함한다. 일부 구현예에서, 비-표적 항원은 인간 백혈구 항원 A*02 대립형질 산물 (HLA-A*02)을 포함한다. 일부 구현예에서, 비-표적 항원은 인간 백혈구 항원 A*69를 포함한다. 일부 구현예에서, 비-표적 항원은 HLA-B를 포함한다. 일부 구현예에서, 비-표적 항원은 HLA-C를 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-A*02를 포함한다.
일부 구현예에서, 비-표적 항원은 C-X-C 모티프 케모카인 리간드 16 (CXCL16) 또는 MHC-I와의 복합체 내 이의 항원 펩티드를 포함한다. 인간 CXCL16 전구체는 NCBI 기록 번호 NP_001094282.1에 기술되어 있으며, 그 내용은 전체적으로 본원에 참조로 포함된다. 일부 구현예에서, CXCL16은 다음의 아미노산 서열을 포함한다:
1 MSGSQSEVAP SPQSPRSPEM GRDLRPGSRV LLLLLLLLLV YLTQPGNGNE GSVTGSCYCG
61 KRISSDSPPS VQFMNRLRKH LRAYHRCLYY TRFQLLSWSV CGGNKDPWVQ ELMSCLDLKE
121 CGHAYSGIVA HQKHLLPTSP P I SQASEGAS SDIHTPAQML LSTLQSTQRP TLPVGSLSSD
181 KELTRPNETT IHTAGHSLA A GPEAGENQKQ PEKNAGPTAR TSATVPVLCL LAIIFILTAA
241 LSYVLCKRRR GQSPQSSPDL PVHYIPVAPD SNT (서열 번호 86).
일부 구현예에서, 비-표적 항원은 CXCL16의 다형성을 포함한다. 예를 들어, 비-표적 항원은 CXCL16의 다형성 잔기를 포함하는 CXCL16으로부터 유래된 펩티드를 포함한다. CXCL16의 다형성 잔기는 서열 번호 86의 위치 142 및 200을 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 142 또는 200을 포함하는 CXCL16의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 200에서 A를 포함하는 CXCL16의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 200에서 V를 포함하는 CXCL16의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 142에서 I를 포함하는 CXCL16의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 142에서 T를 포함하는 CXCL16의 펩티드를 포함한다.
일부 구현예에서, 비-표적 항원은 CXCL16의 다형성을 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 200에서 A를 포함하는 CXCL16의 펩티드를 포함하고, 제2 수용체는 서열 번호 86의 위치 200에서 V를 갖는 CXCL16 리간드보다 서열 번호 86의 위치 200에서 A를 갖는 CXCL16 리간드에 대해 더 높은 친화도를 갖는 리간드 결합 도메인을 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 200에서 V를 포함하는 CXCL16의 펩티드를 포함하고, 제2 수용체는 서열 번호 86의 위치 200에서 A를 갖는 CXCL16 리간드보다 서열 번호 86의 위치 200에서 V를 갖는 CXCL16 리간드에 대해 더 높은 친화도를 갖는 리간드 결합 도메인을 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 142에서 I를 포함하는 CXCL16의 펩티드를 포함하고, 제2 수용체는 서열 번호 86의 위치 142에서 T를 갖는 CXCL16 리간드보다 서열 번호 86의 위치 142에서 I를 갖는 CXCL16 리간드에 대해 더 높은 친화도를 갖는 리간드 결합 도메인을 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 86의 아미노산 142에서 T를 포함하는 CXCL16의 펩티드를 포함하고, 제2 수용체는 서열 번호 86의 위치 142에서 I를 갖는 CXCL16 리간드보다 서열 번호 86의 위치 142에서 T를 갖는 CXCL16 리간드에 대해 더 높은 친화도를 갖는 리간드 결합 도메인을 포함한다.
일부 구현예에서, 비-표적 항원은 콜렉틴 서브패밀리 구성원 12 (COLEC12) 또는 MHC-I와의 복합체 내 이의 항원 펩티드를 포함한다. 인간 COLEC12는 NCBI 기록 번호 NP_569057.2에 기술되어 있고, 그 내용은 전체적으로 참조로 본원에 포함된다. 일부 구현예에서, COLEC12는 다음의 아미노산 서열을 포함한다:
1 MKDDFAEEEE VQSFGYKRFG IQEGTQCTKC KNNWALKFSI ILLYILCALL TITVAILGYK
61 VVEKMDNVTG GMETSRQTYD DKLTAVESDL KKLGDQTGKK AISTNSELST FRSDILDLRQ
121 QLREITEKTS KNKDTLEKLQ ASGDALVDRQ SQLKETLENN SFLITTVNKT LQAYNGYVTN
181 LQQDTSVLQG NLQNQMYSHN VVIMNLNNLN LTQVQQRNLI TNLQRSVDDT SQAIQRIKND
241 FQNLQQVFLQ AKKDTDWLKE KVQSLQTLAA NNSALAKANN DTLEDMNSQL NSFTGQMENI
301 TTISQANEQN LKDLQDLHKD AENRTAIKFN QLEERFQLFE TDIVNIISNI SYTAHHLRTL
361 TSNLNEVRTT CTDTLTKHTD DLTSLNNTLA NIRLDSVSLR MQQDLMRSRL DTEVANLSVI
421 MEEMKLVDSK HGQLIKNFTI LQGPPGPRGP RGDRGSQGPP GPTGNKGQKG EKGEPGPPGP
481 AGERGPIGPA GPPGERGGKG SKGSQGPKGS RGSPGKPGPQ GS S GDPGPPG PPGKEGLPGP
541 QGPPGFQGLQ GTVGEPGVPG PRGLPGLPGV PGMPGPKGPP GPPGPSGAVV PLALQNEPTP
601 APEDNGCPPH WKNFTDKCYY FSVEKEIFED AKLFCEDKSS HLVFINTREE QQWIKKQMVG
661 RESHWIGLTD SERENEWKWL DGTSPDYKNW KAGQPDNWGH GHGPGEDCAG LIYAGQWNDF
721 QCEDVNNFIC EKDRETVLSS AL (서열 번호 87).
일부 구현예에서, 비-표적 항원은 COLEC12의 다형성을 포함한다. 예를 들어, 비-표적 항원은 COLEC12의 다형성 잔기를 포함하는 COLEC12 로부터 유래된 펩티드를 포함한다. COLEC12의 다형성 잔기는 서열 번호 87의 위치 522를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 87의 아미노산 522를 포함하는 COLEC12의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 87의 아미노산 522에서 S를 포함하는 COLEC12의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 87의 아미노산 522에서 P를 포함하는 COLEC12의 펩티드를 포함한다.
일부 구현예에서, 비-표적 항원은 APC 하향 조절 1 (APCDD1) 또는 MHC-I와의 복합체 내 이의 항원 펩티드를 포함한다. 예시적인 인간 APCDD1은 UniProtKB 기록 번호 Q8J025에 기술되어 있으며, 그 내용은 전체적으로 본원에 참조로 포함된다. 일부 구현예에서, APCDD1은 다음의 아미노산 서열을 포함한다:
1 MSWPRRLLLR YLFPALLLHG LGEGSALLHP DSRSHPRSLE KSAWRAFKES QCHHMLKHLH
61 NGARITVQMP PTIEGHWVST GCEVRSGPEF ITRSYRFYHN NTFKAYQFYY GSNRCTNPTY
121 TLIIRGKIRL RQASWIIRGG TEADYQLHN V QVICHTEAVA EKLGQQVNRT CPGFLADGGP
181 WVQDVAYDLW REENGCECTK AVNFAMHELQ LIRVEKQYLH HNLDHLVEEL FLGDIHTDAT
241 QRMFYRPSSY QPPLQNAKNH DHACIACRII YRSDEHHPPI LPPKADLTIG LHGEWVSQRC
301 EVRPEVLFLT RHFIFHDNNN TWEGHYYHYS DPVCKHPTFS IYARGRYSRG VLSSRVMGGT
361 EFVFKVNHMK VTPMDAATAS LLNVFNGNEC GAEGSWQVGI QQDVTHTNGC VALGIKLPHT
421 EYEIFKMEQD ARGRYLLFNG QRPSDGSSPD RPEKRATSYQ MPLVQCASSS PRAEDLAEDS
481 GSSLYGRAPG RHTWSLLLAA LACLVPLLHW NIRR (서열 번호 88).
일부 구현예에서, 비-표적 항원은 APCDD1의 다형성을 포함한다. APCDD1의 예시적인 다형성은 rs3748415를 포함하고, 이는 서열 번호 88의 위치 150에서 V, I 또는 L일 수 있다. 일부 구현예에서, 비-표적 항원은 서열 번호 88의 아미노산 150을 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 88의 아미노산 150에서 V를 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 88의 아미노산 150에서 I를 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 88의 아미노산 150에서 L을 포함하는 APCDD1의 펩티드를 포함한다.
추가의 예시적인 인간 APCDD1은 UniProtKB 기록 번호 V9GY82에 기술되어 있으며, 그 내용은 전체적으로 본원에 참조로 포함된다. 일부 구현예에서, APCDD1은 다음의 아미노산 서열을 포함한다:
1 XDVAYDLWRE ENGCECTKAV NFAMHELQLI RVEKQYLHHN LDHLVEELFL GDIHTDATQR
61 MFYRPSSYQP PLQNAKCAAE SSGSFQILPQ DSSEKEQNGL SHWCLSRPGH QKDWALCAHA
121 GPATAGCPSC LWPPAETGRK AGRTSSKTVH ACPGEAGTSS FELF Y FPNCW SIETKLKISL
181 NAKLSFKPRA SAPLETGHRV KIETLSQLVF LSFIQLCCEV QSPLANK (서열 번호 134).
APCDD1의 예시적인 다형성은 rs1786683을 포함하고, 이는 서열 번호 133의 위치 165에서 Y 또는 S일 수 있다. 일부 구현예에서, 비-표적 항원은 서열 번호 134의 아미노산 165를 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 134의 아미노산 165에서 Y를 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 134의 아미노산 165에서 S를 포함하는 APCDD1의 펩티드를 포함한다.
추가의 예시적인 인간 APCDD1은 UniProt 기록 번호 J3QSE3에 기술되어 있으며, 그 내용은 전체적으로 본원에 참조로 포함된다. 일부 구현예에서, APCDD1은 다음의 아미노산 서열을 포함한다:
1 PEDVLPALQL PAPSAECQVE MGFHHVG Q DG LQLPTSSDPP ALASQSAGIT GVSHRPPGRH
61 LSNDLRTTTM PASPVGSSIG QTSTTLPSCP QRQT (서열 번호 135).
APCDD1의 예시적인 다형성은 rs9952598을 포함하고, 이는 서열 번호 135의 위치 28에서 Q 또는 R일 수 있다. 일부 구현예에서, 비-표적 항원은 서열 번호 135의 아미노산 28을 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 135의 아미노산 28에서 Q를 포함하는 APCDD1의 펩티드를 포함한다. 일부 구현예에서, 비-표적 항원은 서열 번호 135의 아미노산 28에서 R을 포함하는 APCDD1의 펩티드를 포함한다.
일부 구현예에서, APCDD1은 서열 번호 88 또는 134-135 중 어느 하나에 대한 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 적어도 94%, 적어도 97%, 적어도 98%, 또는 적어도 99% 동일성을 공유하는 서열을 포함한다. APCDD1의 다형성 잔기는 서열 번호 88 또는 134-135에서 볼드체로 표시되고 밑줄이 그어져 있다.
일부 구현예에서, 비-표적 항원은 HLA-A*02를 포함한다. HLA-A*02에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
일부 구현예에서, 비-표적 항원은 HLA-B*07을 포함한다. HLA-B*07에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
일부 구현예에서, 비-표적 항원은 HLA-A*11을 포함한다. HLA-A*11에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
일부 구현예에서, 비-표적 항원은 HLA-A*01을 포함한다. HLA-A*01에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
일부 구현예에서, 비-표적 항원은 HLA-A*03을 포함한다. HLA-A*03에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
일부 구현예에서, 비-표적 항원은 HLA-C*07을 포함한다. HLA-C*07에 결합하고 인식하는 당업계에 공지되거나 본원에 개시된 다양한 단일 가변 도메인이 구현예에서 사용하기에 적합하다.
이러한 scFv는 하기 표 4에 제시된 펩티드-독립적 방식으로 예를 들어 제한 없이, 비-표적 항원 (예를 들어 HLA-A*02, HLA-A*01, HLA-A*03, HLA-A*11, HLA-B*07, 또는 HLA-C*07)에 결합하는 다음 마우스 및 인간화 scFv 항체를 포함한다 (표시된 경우, 상보성 결정 영역은 밑줄로 표시됨):
표 4. scFv 결합 도메인
비-표적 리간드 결합 도메인에 대한 예시적인 중쇄 및 경쇄 CDR (각각, CDR-H1, CDR-H2 및 CDR-H3, 또는 CDR-L1, CDR-L2 및 CDR-L3)은 하기 표 5에 나타낸다.
표 5. 항원 결합 도메인에 해당하는 CDR
일부 구현예에서, HLA-A*02를 포함하는 비-표적 항원, 및 제2 수용체의 리간드 결합 도메인은 HLA-A*02 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인은 HLA-A*02를 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA-A*02에 결합한다. 일부 구현예에서, HLA-A*02 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA-A*02 리간드 결합 도메인은 서열 번호 89-100 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA-A*02 리간드 결합 도메인은 서열 번호 89-100 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다.
일부 구현예에서, HLA-A*02 scFv는 서열 번호 101-112 중 어느 하나의 상보성 결정 영역 (CDR)을 포함한다. 일부 구현예에서, scFv는 서열 번호 101-112 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 서열 번호 101-112 중 어느 하나와 동일한 서열을 포함한다. 일부 구현예에서, 항원 결합 도메인의 중쇄는 서열 번호 101-112 중 어느 하나의 중쇄 CDR을 포함하고, 여기서 항원 결합 도메인의 경쇄는 서열 번호 101-112 중 어느 하나의 경쇄 CDR을 포함한다. 일부 구현예에서, HLA-A*02 항원 결합 도메인은 중쇄 및 경쇄를 포함하고, 중쇄는 서열 번호 104-106 및 110-112로부터 선택된 CDR을 포함하고 경쇄는 서열 번호 101-103 및 107-109로부터 선택된 CDR을 포함한다. 임의의 리간드 결합 도메인의 추가 구현예에서, 각각의 CDR 서열은 1, 2, 3개 이상의 치환, 삽입 또는 결실을 가질 수 있다. CDR 서열은 치환, 결실 또는 삽입을 허용할 수 있다. 서열 정렬 도구, 일상적인 실험 및 공지된 검정을 사용하여, 당업자는 과도한 실험 없이 CDR 서열에서 1, 2, 3개 이상의 치환, 삽입 또는 결실을 갖는 변이체 서열을 생성하고 시험할 수 있다.
일부 구현예에서, HLA-A*02 항원 결합 도메인은 중쇄 및 경쇄를 포함하고, 중쇄는 서열 번호 89-100 중 어느 하나의 중쇄 부분과 적어도 95% 동일한 서열을 포함하고, 경쇄는 서열 번호 89-100 중 어느 하나의 경쇄 부분과 적어도 95% 동일한 서열을 포함한다.
일부 구현예에서, 중쇄는 서열 번호 89-100 중 어느 하나의 중쇄 부분과 동일한 서열을 포함하고, 여기서 경쇄는 서열 번호 89-100 중 어느 하나의 경쇄 부분과 동일한 서열을 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-B*07을 포함하고, 제2 수용체의 리간드 결합 도메인은 HLA-B*07 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인은 HLA-B*07을 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA-B*07에 결합한다. 일부 구현예에서, HLA-B*07 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA-B*07 리간드 결합 도메인은 표 4에 표시된 B7 결합 도메인 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA-B*07 리간드 결합 도메인은 표 4에 표시된 HLA-B*07 결합 도메인 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, HLA-B*07 scFv는 표 5에 표시된 HLA-B*07 상보성 결정 영역 (CDR) 중 어느 하나를 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-B*07 scFv 서열 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-B*07 scFv 서열 중 어느 하나와 동일한 서열을 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-A*11을 포함하고, 제2 수용체의 리간드 결합 도메인은 HLA-A*11 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인은 HLA-A*11을 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA-A*11에 결합한다. 일부 구현예에서, HLA-A*11 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA-A*11 리간드 결합 도메인은 표 4에 표시된 HLA-A*11 결합 도메인 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA-A*11 리간드 결합 도메인은 표 4에 표시된 HLA-A*11 결합 도메인 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, HLA-A*11 scFv는 표 5에 표시된 HLA-A*11 상보성 결정 영역 (CDR) 중 어느 하나를 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*11 scFv 서열 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*11 scFv 서열 중 어느 하나와 동일한 서열을 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-A*01을 포함하고, 제2 수용체의 리간드 결합 도메인은 HLA-A*01 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인은 HLA-A*01을 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA-A*01에 결합한다. 일부 구현예에서, HLA-A*01 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA-A*01 리간드 결합 도메인은 표 4에 표시된 HLA-A*01 결합 도메인 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA-A*01 리간드 결합 도메인은 표 4에 표시된 HLA-A*01 결합 도메인 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, HLA-A*01 scFv는 표 5에 표시된 HLA-A*01 상보성 결정 영역 (CDR) 중 어느 하나를 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*01 scFv 서열 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*01 scFv 서열 중 어느 하나와 동일한 서열을 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-A*03을 포함하고, 제2 수용체의 리간드 결합 도메인은 HLA-A*03 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인은 HLA-A*03을 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA-A*03에 결합한다. 일부 구현예에서, HLA-A*03 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA-A*03 리간드 결합 도메인은 표 4에 표시된 HLA-A*03 결합 도메인 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA-A*03 리간드 결합 도메인은 표 4에 표시된 HLA-A*03 결합 도메인 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, HLA-A*03 scFv는 표 5에 표시된 HLA-A*03 상보성 결정 영역 (CDR) 중 어느 하나를 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*03 scFv 서열 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-A*03 scFv 서열 중 어느 하나와 동일한 서열을 포함한다.
일부 구현예에서, 비-표적 항원은 HLA-C*07을 포함하고, 제2 수용체의 리간드 결합 도메인 HLA- C*07 리간드 결합 도메인을 포함한다. 일부 구현예에서, 리간드 결합 도메인 HLA- C*07을 포함하는 pMHC 복합체의 펩티드와 독립적으로 HLA- C*07에 결합한다. 일부 구현예에서, HLA- C*07 리간드 결합 도메인은 scFv 도메인을 포함한다. 일부 구현예에서, HLA- C*07 리간드 결합 도메인은 표 4에 표시된 HLA- C*07 결합 도메인 중 어느 하나의 서열을 포함한다. 일부 구현예에서, HLA- C*07 리간드 결합 도메인은 표 4에 표시된 HLA- C*07 결합 도메인 중 어느 하나의 서열과 적어도 90%, 적어도 95% 또는 적어도 99% 동일한 서열을 포함한다. 일부 구현예에서, HLA- C*07 scFv는 표 5에 표시된 HLA-C*07 상보성 결정 영역 (CDR) 중 어느 하나를 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA- C*07 scFv 서열 중 어느 하나와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, scFv는 표 4에 표시된 HLA-C*07 scFv 서열 중 어느 하나와 동일한 서열을 포함한다.
억제 수용체
본 개시내용은 억제 키메라 항원 수용체 (즉 억제 수용체)인 제2 수용체를 제공한다. 억제 수용체는 MHC-I 복합체에서 비-표적 항원 또는 이의 펩티드 유도체에 결합하고 인식하는 세포외 리간드 결합 도메인을 포함할 수 있다.
예시적인 억제 수용체는 2020년 9월 6일에 출원된 PCT/US2020/045228, 2020년 12월 11일에 출원된 PCT/US2020/064607, 2021년 4월 29일에 출원된 PCT/US2021/029907, 및 2020년 11월 10일에 출원된 PCT/US2020/059856에 기술되어 있고, 각 내용은 본원에 참조로 포함된다.
비-표적 항원은 비-표적 대립형질 변이체일 수 있다. 본원에서 사용된 "비-표적 대립형질 변이체"는 대립유전자 (예를 들어 MHC 클래스 I 유전자의 대립형질 변이체)를 지칭하고, 그의 발현은 이형접합성의 소실, 또는 다른 발현 상실 메커니즘으로 인해 표적 세포 (예를 들어 암 세포)에서 감소되거나 제거되지만, 정상 또는 건강한 세포에서 발현되거나 검출 가능하게 발현된다.
본원에서 사용된 용어 "억제 키메라 항원 수용체" 또는 "억제 수용체"는 면역 세포의 면역 활성을 방해 또는 억제하는 억제 신호를 전달할 수 있는 세포 내 신호전달 도메인에 융합된 항원-결합 도메인을 지칭한다. 억제 수용체는 면역 세포 억제 잠재력을 가지며, 잠재력을 활성화시키는 면역 세포를 갖는 수용체인, CAR과 별개이고 구별된다. 예를 들어, CAR은 세포 내 자극 및/또는 공동 자극 도메인을 포함하기 때문에 활성화 수용체이다. 억제 수용체는 세포 내 억제 도메인을 함유하는 억제 수용체이다.
본원에서 사용된 "억제 신호"는 면역 반응의 억제 (예를 들어, 사이토카인 생성 감소 또는 면역 세포 활성화의 감소)를 유발하는 면역 세포에서의 신호 전달 또는 단백질 발현의 변화를 지칭한다. 면역 세포의 방해 또는 억제는 선택적 및/또는 가역적이거나 비선택적 및/또는 가역적일 수 있다. 억제 수용체는 비-표적 항원 (예를 들어 HLA-A*02)에 반응한다. 예를 들어, 비-표적 항원 (예를 들어 HLA-A*02)이 억제 수용체에 결합하거나 접촉하면, 억제 수용체가 반응하고 억제 수용체의 세포외 리간드 결합 도메인에 의한 비-표적 항원 결합 시 억제 수용체를 발현하는 면역 세포에서 억제 신호를 활성화한다.
본 개시내용의 억제 수용체는 세포외 리간드 결합 도메인을 포함할 수 있다. 리간드 의존적 방식으로 수용체의 활성을 조절할 수 있는 임의의 유형의 리간드 결합 도메인은 본 발명의 범위 내에 있는 것으로 생각된다.
일부 구현예에서, 리간드 결합 도메인은 항원 결합 도메인이다. 예시적인 항원 결합 도메인은 특히, scFv, SdAb, Vβ-단독 도메인, 및 TCR α 및 β 사슬 가변 도메인으로부터 유래된 TCR 항원 결합 도메인을 포함한다.
임의의 유형의 항원 결합 도메인은 본 개시내용의 범위 내에 있는 것으로 생각된다.
일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 주요 조직적합성 복합체 클래스 I (MHC-I), 또는 HLA-A*02와의 복합체 내 COLEC12, APCDD1, CXCL16 또는 이의 항원 펩티드의 다형성 변이체에 결합하고 인식한다. 일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 scFv이다.
일부 구현예에서, 제2 수용체의 세포외 리간드 결합 도메인은 억제 수용체의 세포외 도메인에 융합된다.
일부 구현예에서, 본 개시내용의 억제 수용체는 세포외 힌지 영역을 포함한다. 예시적인 힌지는 IgD 및 CD8 도메인, 예를 들어 IgG1 로부터 단리되거나 유래될 수 있다. 일부 구현예에서, 힌지는 CD8α 또는 CD28로부터 단리되거나 유래된다.
본 개시내용의 억제 수용체는 억제 수용체의 세포외 도메인에 융합된 막관통 도메인을 포함하도록 설계될 수 있다. 일부 경우에, 막관통 도메인은 수용체 복합체의 다른 구성원과의 상호작용을 최소화하기 위해 동일하거나 상이한 표면 막 단백질의 막관통 도메인에 대한 이러한 도메인의 결합을 피하기 위해 아미노산 치환에 의해 선택되거나 변형될 수 있다.
막관통 도메인은 천연 또는 합성 공급원으로부터 유래될 수 있다. 공급원이 천연인 경우, 도메인은 막 결합 단백질 또는 막관통 단백질에서 유래될 수 있다. 막관통 영역은 T-세포 수용체, CD28, CD3 입실론, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154, 또는 IgG4와 같은 면역글로불린의 알파, 베타 또는 제타 사슬로부터 단리되거나 유래 (즉 이의 적어도 막관통 영역(들)을 포함)할 수 있다. 대안적으로, 막관통 도메인은 합성일 수 있으며, 이 경우 그것은 주로 류신 및 발린과 같은 소수성 잔기를 포함할 것이다. 일부 구현예에서, 페닐알라닌, 트립토판 및 발린의 삼중체는 합성 막관통 도메인의 각 말단에서 발견될 것이다. 임의로 짧은 올리고- 또는 폴리펩티드 링커, 바람직하게는 2 내지 10개 아미노산 길이가 막관통 도메인과 억제 수용체의 세포 내 도메인 사이의 연결을 형성할 수 있다. 글리신-세린 이중체는 특히 적합한 링커를 제공한다.
본 개시내용은 세포 내 도메인을 포함하는 억제 수용체를 제공한다. 본 개시내용의 억제 수용체의 세포 내 도메인은 억제 수용체를 포함하는 면역 세포의 활성화 억제를 담당하고, 그렇지 않으면 제1 수용체에 의한 활성화 신호에 대한 반응으로 활성화될 수 있다. 일부 구현예에서, 억제 세포 내 도메인은 면역수용체 티로신계 억제 모티프 (ITIM)를 포함한다. 일부 구현예에서, ITIM을 포함하는 억제 세포 내 도메인은 CTLA-4 및 PD-1과 같은 면역 관문 억제제로부터 단리되거나 유래될 수 있다. CTLA-4 및 PD-1은 T 세포의 표면에 발현된 면역 억제 수용체이고, T 세포 반응을 약화시키거나 종료시키는 데 중추적인 역할을 한다.
일부 구현예에서, 억제 세포 내 도메인은 인간 종양 괴사 인자 관련 세포자멸사 유도 리간드 (TRAIL) 수용체 및 CD200 수용체 1로부터 단리된다. 일부 구현예에서, TRAIL 수용체는 TR10A, TR10B 또는 TR10D를 포함한다.
일부 구현예에서, 억제 세포 내 도메인은 글리코스핑고지질 마이크로도메인 1 (PAG1)을 갖는 인단백질 막 앵커로부터 단리된다. 일부 구현예에서, 억제 세포 내 도메인은 백혈구 면역글로불린 유사 수용체 B1 (LILRB1)로부터 단리된다.
일부 구현예에서, 억제 도메인은 인간 단백질, 예를 들어 인간 TRAIL 수용체, CTLA-4, PD-1, PAG1 또는 LILRB1 단백질로부터 단리되거나 유래된다.
일부 구현예에서, 억제 도메인은 세포 내 도메인, 막관통 또는 이들의 조합을 포함한다. 일부 구현예에서, 억제 도메인은 세포 내 도메인, 막관통 도메인, 힌지 영역 또는 이들의 조합을 포함한다.
일부 구현예에서, 억제 도메인은 킬러 세포 면역글로불린 유사 수용체, 3개의 Ig 도메인 및 긴 세포질 테일 2 (KIR3DL2), 킬러 세포 면역글로불린 유사 수용체, 3개의 Ig 도메인 및 긴 세포질 테일 3 (KIR3DL3), 백혈구 면역글로불린 유사 수용체 B1 (LIR1, LIR-1 및 LILRB1이라고도 함), 프로그램된 세포 사멸 1 (PD-1), Fc 감마 수용체 IIB (FcgRIIB), 킬러 세포 렉틴 유사 수용체 K1 (NKG2D), CTLA-4, 합성 공통 ITIM, ZAP70 SH2 도메인을 함유하는 도메인 (예를 들어, N 및 C 말단 SH2 도메인 중 하나 또는 둘 모두), 또는 ZAP70 KI_K369A (키나제 불활성 ZAP70)로부터 단리되거나 유래된다.
일부 구현예에서, 억제 도메인은 인간 단백질로부터 단리되거나 유래된다.
일부 구현예에서, 제2, 억제 수용체는 억제 도메인을 포함한다. 일부 구현예에서, 제2, 억제 수용체는 억제 세포 내 도메인 및/또는 억제 막관통 도메인을 포함한다. 일부 구현예에서, 억제 세포 내 도메인은 억제 수용체의 세포 내 도메인에 융합된다. 일부 구현예에서, 억제 세포 내 도메인은 억제 수용체의 막관통 도메인에 융합된다.
일부 구현예에서, 제2, 억제 수용체는 동일한 단백질, 예를 들어 ITIM 함유 단백질로부터 단리되거나 유래되거나 단리되거나 유래된 세포질 도메인, 막관통 도메인, 및 세포외 도메인 또는 이의 부분을 포함한다. 일부 구현예에서, 제2, 억제 수용체는 세포 내 도메인 및/또는 막관통 도메인, 예를 들어 ITIM 함유 단백질과 같은 동일한 단백질로부터 단리되거나 유래되거나 단리되거나 유래된 힌지 영역을 포함한다.
일부 구현예에서, 제2 수용체는 억제 도메인을 포함하는 TCR (억제 TCR)이다. 일부 구현예에서, 억제 TCR은 억제 세포 내 도메인 및/또는 억제 막관통 도메인을 포함한다. 일부 구현예에서, 억제 세포 내 도메인은 TCR 알파, TCR 베타, CD3 델타, CD3 감마 또는 CD3 입실론 또는 이의 일부 TCR의 세포 내 도메인에 융합된다. 일부 구현예에서, 억제 세포 내 도메인은 TCR 알파, TCR 베타, CD3 델타, CD3 감마 또는 CD3 입실론의 막관통 도메인에 융합된다.
일부 구현예에서, 제2 수용체는 억제 도메인을 포함하는 TCR (억제 TCR)이다. 일부 구현예에서, 억제 도메인은 LILRB1로부터 단리되거나 유래된다.
LILRB1 억제 수용체
본 개시내용은 LILRB1 억제 도메인, 및 임의로, LILRB1 막관통 및/또는 힌지 도메인, 또는 이의 기능적 변이체를 포함하는 제2, 억제 수용체를 제공한다. 억제 수용체에 LILRB1 막관통 도메인 및/또는 LILRB1 힌지 도메인을 포함하면 다른 막관통 도메인 또는 또 다른 힌지 도메인을 갖는 참조 억제 수용체와 비교하여 억제 수용체에 의해 생성된 억제 신호를 증가시킬 수 있다. LILRB1 억제 도메인을 포함하는 제2, 억제 수용체는 본원에 기재된 바와 같은 CAR 또는 TCR일 수 있다. 본원에 기재된 바와 같은 임의의 적합한 리간드 결합 도메인은 LILRB1-기반 제2, 억제 수용체에 융합될 수 있다.
백혈구 면역글로불린 유사 수용체 B1로도 알려진, 백혈구 면역글로불린 유사 수용체 서브패밀리 B 구성원 1 (LILRB1), 뿐만 아니라 ILT2, LIR1, MIR7, PIRB, CD85J, ILT-2 LIR-1, MIR-7 및 PIR-B는 백혈구 면역글로불린 유사 수용체 (LIR) 계열의 구성원이다. LILRB1 단백질은 LIR 수용체의 서브패밀리 B 클래스에 속한다. 이들 수용체는 2 내지 4개의 세포외 면역글로불린 도메인, 막관통 도메인, 및 2 내지 4개의 세포질 면역수용체 티로신계 억제 모티프 (ITIM)를 함유한다. LILRB1 수용체는 면역 세포에서 발현되며, 항원-제시 세포 상의 MHC 클래스 I 분자에 결합하고 면역 반응의 자극을 억제하는 음성 신호를 전달한다. LILRB1은 세포 독성뿐만 아니라 염증 반응을 조절하고, 자가 반응성을 제한하는 역할을 하는 것으로 생각된다. LILRB1의 상이한 이소형을 암호화하는 다수의 전사 변이체가 존재하며, 이들 모두는 본 발명의 범위 내에 있는 것으로 간주된다.
본원에 기재된 억제 수용체의 일부 구현예에서, 억제 수용체 LILRB1로부터 단리되거나 유래된 하나 이상의 도메인을 포함한다. LILRB1으로부터 단리되거나 유래된 하나 이상의 도메인을 갖는 수용체의 일부 구현예에서, LILRB1의 하나 이상의 도메인은 서열 번호 113의 서열 또는 하위서열과 적어도 80%, 적어도 90%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 적어도 99% 동일한 또는 동일한 아미노산 서열을 포함한다. 일부 구현예에서, LILRB1의 하나 이상의 도메인은 동일한 서열 번호 113의 서열 또는 하위서열과 동일한 아미노산 서열을 포함한다. 일부 구현예에서, LILRB1의 하나 이상의 도메인은 서열 번호 113의 서열 또는 하위서열과 적어도 80%, 적어도 90%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 적어도 99% 동일한 또는 동일한 아미노산 서열로 이루어진다. 일부 구현예에서, LILRB1의 하나 이상의 도메인은 동일한 서열 번호 113의 서열 또는 하위서열과 동일한 아미노산 서열로 이루어진다.
LILRB1으로부터 단리되거나 유래된 하나 이상의 도메인을 갖는 수용체의 일부 구현예에서, LILRB1의 하나 이상의 도메인은 다음 서열 또는 하위서열과 적어도 80%, 적어도 90%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 적어도 99% 동일한 또는 동일한 폴리뉴클레오티드 서열에 의해 암호화된다.
1 aaatgagttt taaaaaggct tgtccaggaa gcacatatgg gagctggtca ctctgcattt
61 tgggccctcc tggaggtgtt tagaccttcc gagagagaaa ctgagacaca tgagagggaa
121 gaaatgactc agtggtgaga ccctgtggag tcccacccac aaccagcaca ctgtgaccca
181 ctgcacaaac ctctagccca cagctcactt cctcctttaa gaagagaaga gaaaagagga
241 gaggagagga ggaacagaaa agaaaagaaa agaaaaagtg ggaaacaaat aatctaagaa
301 tgaggagaaa gcaagaagag tgaccccctt gtgggcactc cattggtttt atggcgcctc
361 tactttctgg agtttgtgta aaacaaaaat attatggtct ttgtgcacat ttacatcaag
421 ctcagcctgg gcggcacagc cagatgcgag atgcgtctct gctgatctga gtctgcctgc
481 agcatggacc tgggtcttcc ctgaagcatc tccagggctg gagggacgac tgccatgcac
541 cgagggctca tccatccaca gagcagggca gtgggaggag acgccatgac ccccatcctc
601 acggtcctga tctgtctcgg gctgagtctg ggcccccgga cccacgtgca ggcagggcac
661 ctccccaagc ccaccctctg ggctgaacca ggctctgtga tcacccaggg gagtcctgtg
721 accctcaggt gtcagggggg ccaggagacc caggagtacc gtctatatag agaaaagaaa
781 acagcaccct ggattacacg gatcccacag gagcttgtga agaagggcca gttccccatc
841 ccatccatca cctgggaaca cacagggcgg tatcgctgtt actatggtag cgacactgca
901 ggccgctcag agagcagtga ccccctggag ctggtggtga caggagccta catcaaaccc
961 accctctcag cccagcccag ccccgtggtg aactcaggag ggaatgtaac cctccagtgt
1021 gactcacagg tggcatttga tggcttcatt ctgtgtaagg aaggagaaga tgaacaccca
1081 caatgcctga actcccagcc ccatgcccgt gggtcgtccc gcgccatctt ctccgtgggc
1141 cccgtgagcc cgagtcgcag gtggtggtac aggtgctatg cttatgactc gaactctccc
1201 tatgagtggt ctctacccag tgatctcctg gagctcctgg tcctaggtgt ttctaagaag
1261 ccatcactct cagtgcagcc aggtcctatc gtggcccctg aggagaccct gactctgcag
1321 tgtggctctg atgctggcta caacagattt gttctgtata aggacgggga acgtgacttc
1381 cttcagctcg ctggcgcaca gccccaggct gggctctccc aggccaactt caccctgggc
1441 cctgtgagcc gctcctacgg gggccagtac agatgctacg gtgcacacaa cctctcctcc
1501 gagtggtcgg cccccagcga ccccctggac atcctgatcg caggacagtt ctatgacaga
1561 gtctccctct cggtgcagcc gggccccacg gtggcctcag gagagaacgt gaccctgctg
1621 tgtcagtcac agggatggat gcaaactttc cttctgacca aggagggggc agctgatgac
1681 ccatggcgtc taagatcaac gtaccaatct caaaaatacc aggctgaatt ccccatgggt
1741 cctgtgacct cagcccatgc ggggacctac aggtgctacg gctcacagag ctccaaaccc
1801 tacctgctga ctcaccccag tgaccccctg gagctcgtgg tctcaggacc gtctgggggc
1861 cccagctccc cgacaacagg ccccacctcc acatctggcc ctgaggacca gcccctcacc
1921 cccaccgggt cggatcccca gagtggtctg ggaaggcacc tgggggttgt gatcggcatc
1981 ttggtggccg tcatcctact gctcctcctc ctcctcctcc tcttcctcat cctccgacat
2041 cgacgtcagg gcaaacactg gacatcgacc cagagaaagg ctgatttcca acatcctgca
2101 ggggctgtgg ggccagagcc cacagacaga ggcctgcagt ggaggtccag cccagctgcc
2161 gatgcccagg aagaaaacct ctatgctgcc gtgaagcaca cacagcctga ggatggggtg
2221 gagatggaca ctcggagccc acacgatgaa gacccccagg cagtgacgta tgccgaggtg
2281 aaacactcca gacctaggag agaaatggcc tctcctcctt ccccactgtc tggggaattc
2341 ctggacacaa aggacagaca ggcggaagag gacaggcaga tggacactga ggctgctgca
2401 tctgaagccc cccaggatgt gacctacgcc cagctgcaca gcttgaccct cagacgggag
2461 gcaactgagc ctcctccatc ccaggaaggg ccctctccag ctgtgcccag catctacgcc
2521 actctggcca tccactagcc caggggggga cgcagacccc acactccatg gagtctggaa
2581 tgcatgggag ctgccccccc agtggacacc attggacccc acccagcctg gatctacccc
2641 aggagactct gggaactttt aggggtcact caattctgca gtataaataa ctaatgtctc
2701 tacaattttg aaataaagca acagacttct caataatcaa tgaagtagct gagaaaacta
2761 agtcagaaag tgcattaaac tgaatcacaa tgtaaatatt acacatcaag cgatgaaact
2821 ggaaaactac aagccacgaa tgaatgaatt aggaaagaaa aaaagtagga aatgaatgat
2881 cttggctttc ctataagaaa tttagggcag ggcacggtgg ctcacgcctg taattccagc
2941 actttgggag gccgaggcgg gcagatcacg agttcaggag atcgagacca tcttggccaa
3001 catggtgaaa ccctgtctct cctaaaaata caaaaattag ctggatgtgg tggcagtgcc
3061 tgtaatccca gctatttggg aggctgaggc aggagaatcg cttgaaccag ggagtcagag
3121 gtttcagtga gccaagatcg caccactgct ctccagcctg gcgacagagg gagactccat
3181 ctcaaattaa aaaaaaaaaa aaaaaagaaa gaaaaaaaaa aaaaaaaaa(서열 번호 114).
LILRB1의 하나 이상의 도메인을 갖는 수용체의 일부 구현예에서, LILRB1의 하나 이상의 도메인은 서열 번호 114의 서열 또는 하위서열과 동일한 폴리뉴클레오티드 서열에 의해 암호화된다.
다양한 구현예에서, 폴리펩티드를 포함하는 억제 수용체가 제공되며, 여기서 폴리펩티드는 다음 중 하나 이상을 포함한다: LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체; LILRB1 막관통 도메인 또는 이의 기능적 변이체; 및 LILRB1 세포 내 도메인 또는 적어도 하나, 또는 적어도 2개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하는 세포 내 도메인, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
본원에서 사용된 "면역수용체 티로신계 억제 모티프" 또는 "ITIM"은 면역계의 많은 억제 수용체의 세포질 꼬리에서 발견되는, S/I/V/LxYxxI/V/L (서열 번호 423) 등의 보존 서열을 갖는 아미노산의 보존된 서열을 지칭한다. ITIM-보유 억제 수용체가 그들의 리간드와 상호작용한 후, ITIM 모티프는 인산화되어, 억제 수용체가 포스포티로신 포스파타제 SHP-1 및 SHP-2, 또는 SHIP로 불리는 이노시톨 포스파타제와 같은 다른 효소를 모집할 수 있다.
일부 구현예에서, 폴리펩티드는 적어도 하나의 면역수용체 티로신계 억제 모티프 (ITIM), 적어도 2개의 ITIM, 적어도 3개의 ITIM, 적어도 4개의 ITIM, 적어도 5개의 ITIM 또는 적어도 6개의 ITIM을 포함하는 세포 내 도메인을 포함한다. 일부 구현예에서, 세포 내 도메인은 1, 2, 3, 4, 5, 또는 6개의 ITIM을 갖는다.
일부 구현예에서, 폴리펩티드는 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로 이루어진 ITIM 그룹으로부터 선택된 적어도 하나의 ITIM을 포함하는 세포 내 도메인을 포함한다.
추가 특정한 구현예에서, 폴리펩티드는 적어도 2개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하는 세포 내 도메인을 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
일부 구현예에서, 세포 내 도메인은 ITIM NLYAAV (서열 번호 115) 및 VTYAEV (서열 번호 116) 모두 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 119와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 119와 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 ITIM VTYAEV (서열 번호 116) 및 VTYAQL (서열 번호 117) 모두 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 120과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 120과 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 ITIM VTYAQL (서열 번호 117) 및 SIYATL (서열 번호 118) 모두 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 121과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 121과 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 ITIM NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), 및 VTYAQL (서열 번호 117)을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 122와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 122와 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 ITIM VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 123과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 123과 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 ITIM NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)을 포함한다. 구현예에서, 세포 내 도메인은 서열 번호 124와 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 서열 번호 124와 동일한 서열을 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 세포 내 도메인은 LILRB1 세포 내 도메인 (서열 번호 129)과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 세포 내 도메인은 LILRB1 세포 내 도메인 (서열 번호 129)과 동일한 서열을 포함하거나 본질적으로 이루어진다.
본 개시내용의 LILRB1 세포 내 도메인 또는 이의 기능적 변이체는 적어도 1, 적어도 2, 적어도 4, 적어도 4, 적어도 5, 적어도 6, 적어도 7, 또는 적어도 8개의 ITIM을 포함할 수 있다. 일부 구현예에서, LILRB1 세포 내 도메인 또는 이의 기능적 변이체는 2, 3, 4, 5, 또는 6개의 ITIM을 갖는다.
특정 구현예에서, 세포 내 도메인은 2개, 3개, 4개, 5개 또는 6개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 적어도 3개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 3개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 4개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 5개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 6개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
특정 구현예에서, 세포 내 도메인은 적어도 7개의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
LILRB1 단백질은 D1, D2, D3 및 D4라고 불리는 4개의 면역글로불린 (Ig) 유사 도메인을 갖는다. 일부 구현예에서, LILRB1 힌지 도메인은 LILRB1 D3D4 도메인 또는 이의 기능적 변이체를 포함한다. 일부 구현예에서, LILRB1 D3D4 도메인은 서열 번호 125와 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 적어도 99% 동일한 또는 동일한 서열을 포함한다. 일부 구현예에서, LILRB1 D3D4 도메인은 서열 번호 125를 포함하거나 본질적으로 이루어진다.
일부 구현예에서, 폴리펩티드는 LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체를 포함한다. 구현예에서, LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체는 서열 번호 132, 서열 번호 125, 또는 서열 번호 126과 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 적어도 99% 동일한 또는 동일한 서열을 포함한다. 구현예에서, LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체는 서열 번호 132, 서열 번호 125, 또는 서열 번호 126과 적어도 95% 동일한 서열을 포함한다.
일부 구현예에서, LILRB1 힌지 도메인은 서열 번호 132, 서열 번호 125, 또는 서열 번호 126과 동일한 서열을 포함한다.
일부 구현예에서, LILRB1 힌지 도메인은 본질적으로 서열 번호 132, 서열 번호 125, 또는 서열 번호 126과 동일한 서열로 이루어진다.
일부 구현예에서, 막관통 도메인은 LILRB1 막관통 도메인 또는 이의 기능적 변이체이다. 일부 구현예에서, LILRB1 막관통 도메인 또는 이의 기능적 변이체는 서열 번호 13에 대해 적어도 95% 동일한, 적어도 96% 동일한, 적어도 97% 동일한, 적어도 98% 동일한 또는 적어도 99%인 서열을 포함한다. 일부 구현예에서, LILRB1 막관통 도메인 또는 이의 기능적 변이체는 서열 번호 133과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, LILRB1 막관통 도메인은 서열 번호 133과 동일한 서열을 포함한다. 구현예에서, LILRB1 막관통 도메인은 본질적으로 서열 번호 133과 동일한 서열로 이루어진다.
일부 구현예에서, 막관통 도메인은 힌지, 예를 들어, 인간 단백질로부터의 힌지를 통해 제2, 억제 수용체, 예를 들어, 항원 결합 도메인 또는 리간드 결합 도메인의 세포외 영역에 부착될 수 있다. 예를 들어, 일부 구현예에서, 힌지는 인간 면역글로불린 (Ig) 힌지, 예를 들어, IgG4 힌지, CD8a 힌지 또는 LILRB1 힌지일 수 있다.
일부 구현예에서, 제2, 억제 수용체는 억제 도메인을 포함한다. 일부 구현예에서, 제2, 억제 수용체는 억제 세포 내 도메인 및/또는 억제 막관통 도메인을 포함한다. 일부 구현예에서, 억제 도메인은 LILR1B로부터 단리되거나 유래된다.
LILRB1 도메인 조합을 포함하는 억제 수용체
일부 구현예에서, 본 개시내용의 LILRB1-기반 억제 수용체는 하나 초과의 LILRB1 도메인 또는 이의 기능적 등가물을 포함한다. 예를 들어, 일부 구현예에서, 억제 수용체는 LILRB1 막관통 도메인 및 세포 내 도메인, 또는 LILRB1 힌지 도메인, 막관통 도메인 및 세포 내 도메인을 포함한다.
특정 구현예에서, 억제 수용체는 LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체, 및 LILRB1 막관통 도메인 또는 이의 기능적 변이체를 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 127과 적어도 95% 동일한, 적어도 96% 동일한, 적어도 97% 동일한, 적어도 98% 동일한, 적어도 99% 동일한 또는 동일한 서열을 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 127과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 127과 동일한 서열을 포함한다.
추가 구현예에서, 억제 수용체는 LILRB1 막관통 도메인 또는 이의 기능적 변이체, 및 LILRB1 세포 내 도메인 및/또는 적어도 하나의 면역수용체 티로신계 억제 모티프 (ITIM)를 포함하는 세포 내 도메인을 포함하고, 여기서 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 선택된다. 일부 구현예에서, 폴리펩티드 LILRB1 막관통 도메인 또는 이의 기능적 변이체, 및 LILRB1 세포 내 도메인 및/또는 적어도 2개의 ITIM을 포함하는 세포 내 도메인을 포함하고, 여기서 각각의 ITIM은 NLYAAV (서열 번호 115), VTYAEV (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
일부 구현예에서, 억제 수용체는 LILRB1 막관통 도메인 및 세포 내 도메인을 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 128과 적어도 95% 동일한, 적어도 96% 동일한, 적어도 97% 동일한, 적어도 98% 동일한, 적어도 99% 동일한 또는 동일한 서열을 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 128과 적어도 95% 동일한 서열을 포함한다. 일부 구현예에서, 폴리펩티드는 서열 번호 128과 동일한 서열을 포함한다.
바람직한 구현예에서, 억제 수용체는 LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체; LILRB1 막관통 도메인 또는 이의 기능적 변이체; 및 LILRB1 세포 내 도메인 및/또는 적어도 2개의 면역수용체 티로신 기반 억제 모티프 (ITIM)를 포함하는 세포 내 도메인을 포함하고, 여기서 각각의 ITIM은 LYAAV (서열 번호 115), VTYAE (서열 번호 116), VTYAQL (서열 번호 117), 및 SIYATL (서열 번호 118)로부터 독립적으로 선택된다.
일부 구현예에서, 억제 수용체는 서열 번호 130 또는 서열 번호 131과 적어도 95% 동일한, 또는 서열 번호 130 또는 서열 번호 131과 적어도 99% 동일한, 또는 서열 번호 130 또는 서열 번호 131과 동일한 서열을 포함한다.
일부 구현예에서, 폴리펩티드는 서열 번호 127과 적어도 99% 동일한, 또는 서열 번호 127과 적어도 99% 동일한, 또는 서열 번호 127과 동일한 서열을 포함한다.
일부 구현예에서, 폴리펩티드는 서열 번호 128과 적어도 99% 동일한, 또는 서열 번호 128과 적어도 99% 동일한, 또는 서열 번호 128과 동일한 서열을 포함한다.
표 6. 예시적인 LILRB1-기반 억제 수용체에 대한 폴리펩티드 서열
폴리뉴클레오티드 및 벡터
본 개시내용은 본 개시내용의 제1 및 제2 수용체의 서열(들)을 암호화하는 서열을 포함하는 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템을 제공한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 shRNA를 추가로 포함한다. 본 개시내용은 본원에 기재된 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템을 포함하는 벡터를 제공한다. 본 개시내용은 본원에 기재된 폴리뉴클레오티드 및 벡터를 포함하는 면역 세포를 제공한다.
일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 180, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 181, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 182, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 183, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 184, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 185, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 187, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 189, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 191 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 서열 번호 193, 또는 그에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 제1 및/또는 제2 수용체의 서열은 프로모터에 작동 가능하게 연결된다. 일부 구현예에서, 제1 수용체를 암호화하는 서열은 제1 프로모터에 작동 가능하게 연결되고, 제2 수용체를 암호화하는 서열은 제2 프로모터에 작동 가능하게 연결된다.
본 개시내용은 본원에 기재된 폴리뉴클레오티드를 포함하는 벡터를 제공한다.
일부 구현예에서, 제1 수용체는 제1 벡터에 의해 암호화되고 제2 수용체는 제2 벡터에 의해 암호화된다. 일부 구현예에서, 두 수용체 모두 단일 벡터에 의해 암호화된다.
일부 구현예에서, 제1 및 제2 수용체는 단일 벡터에 의해 암호화된다. 단일 벡터를 사용하여 다수의 폴리펩티드를 암호화하는 방법은 당업자에게 공지되어 있을 것이며, 특히, 상이한 프로모터의 제어 하에 다수의 폴리펩티드를 암호화하거나, 단일 프로모터를 사용하여 다수의 폴리펩티드의 전사, 내부 리보솜 진입 부위 (IRES) 및/또는 자가 절단 펩티드를 암호화하는 서열의 사용을 제어하는 경우를 포함한다. 예시적인 자가 절단 펩티드는 T2A, P2A, E2A 및 F2A 자가 절단 펩티드를 포함한다. 일부 구현예에서, T2A 자가 절단 펩티드 EGRGSLLTCGDVEENPGP (서열 번호 430)의 서열을 포함한다. 일부 구현예에서, P2A 자가 절단 펩티드는 ATNFSLLKQAGDVEENPGP (서열 번호 431)의 서열을 포함한다. 일부 구현예에서, E2A 자가 절단 펩티드는 QCTNYALLKLAGDVESNPGP (서열 번호 432)의 서열을 포함한다. 일부 구현예에서, F2A 자가 절단 펩티드는 VKQTLNFDLLKLAGDVESNPGP (서열 번호 433)의 서열을 포함한다. 일부 구현예에서, T2A 자가 절단 펩티드는 EGRGSLLTCGDVEENPGP (서열 번호 430)의 서열을 포함한다. 전술한 것 중 임의의 것은 또한 N 말단 GSG 링커를 포함할 수 있다. 예를 들어, T2A 자가 절단 펩티드는 또한 GSGEGRGSLLTCGDVEENPGP (서열 번호 178)의 서열을 포함할 수 있고, 이는 GGATCCGGAGAGGGCAGAGGCAGCCTGCTGACATGTGGCGACGTGGAAGAGAACCCTGGCCCC (서열 번호 434)의 서열에 의해 암호화될 수 있다.
일부 구현예에서, 벡터는 서열 번호 180, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 181, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 182, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 183, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 184, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 벡터는 서열 번호 185, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 187, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 189, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 191, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 벡터는 서열 번호 193, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 벡터는 서열 번호 186, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 폴리펩티드 서열을 암호화한다. 일부 구현예에서, 벡터는 서열 번호 188, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 폴리펩티드 서열을 암호화한다. 일부 구현예에서, 벡터는 서열 번호 190, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 폴리펩티드 서열을 암호화한다. 일부 구현예에서, 벡터는 서열 번호 192, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 폴리펩티드 서열을 암호화한다. 일부 구현예에서, 벡터는 서열 번호 194, 또는 이에 대해 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 폴리펩티드 서열을 암호화한다.
일부 구현예에서, 벡터는 발현 벡터, 즉 적합한 세포에서 제1 및/또는 제2 수용체의 발현을 위한 것이다.
렌티바이러스와 같은 레트로바이러스에서 유래된 벡터는 이식 유전자의 장기적이고 안정적인 통합과 딸 세포에서의 전파를 허용하기 때문에 장기 유전자 전달을 달성하는 데 적합한 도구이다. 렌티바이러스 벡터는 간세포와 같은 비증식 세포를 형질도입할 수 있다는 점에서 쥣과 백혈병 바이러스와 같은 종양-레트로바이러스에서 유래한 벡터에 비해 추가적인 이점이 있다. 그들은 또한 낮은 면역원성이라는 추가적인 이점을 가지고 있다.
수용체를 암호화하는 천연 또는 합성 핵산의 발현은 전형적으로 수용체 또는 그의 일부를 코딩하는 핵산을 프로모터에 작동 가능하게 연결하고 작제물을 발현 벡터에 혼입함으로써 달성된다. 벡터는 복제 및 통합 진핵생물에 적합할 수 있다. 전형적인 클로닝 벡터는 원하는 핵산 서열의 발현 조절에 유용한 전사 및 번역 종결자, 개시 서열 및 프로모터를 함유한다.
수용체를 암호화하는 폴리뉴클레오티드는 여러 유형의 벡터로 클로닝될 수 있다. 예를 들어, 폴리뉴클레오티드는 플라스미드, 파지미드, 파지 유도체, 동물 바이러스 및 코스미드를 포함하지만 이에 제한되지 않는 벡터로 클로닝될 수 있다. 특히 관심있는 벡터에는 발현 벡터, 복제 벡터, 프로브 생성 벡터 및 시퀀싱 벡터가 포함된다.
추가로, 발현 벡터는 바이러스 벡터의 형태로 면역 세포와 같은 세포에 제공될 수 있다. 바이러스 벡터 기술은 당업계에 잘 알려졌으며, 예를 들어, Sambrook et al. (2001, Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory, New York), 및 기타 바이러스학 및 분자생물학 매뉴얼에 기재되어 있다. 벡터로서 유용한 바이러스는 레트로바이러스, 아데노바이러스, 아데노 관련 바이러스, 헤르페스 바이러스 및 렌티바이러스를 포함하지만 이에 제한되지 않는다. 일반적으로, 적합한 벡터는 적어도 하나의 유기체에서 기능적인 복제 기점, 프로모터 서열, 편리한 제한 엔도뉴클레아제 부위 및 하나 이상의 선택가능한 마커를 함유한다 (예를 들어, WO 01/96584; WO 01/29058; 및 미국 특허 번호 6,326,193).
다수의 바이러스 기반 시스템이 포유류 세포로 유전자 전달을 위해 개발되었다. 예를 들어, 레트로바이러스는 유전자 전달 시스템을 위한 편리한 플랫폼을 제공한다. 선택된 유전자는 당업계에 공지된 기술을 사용하여 벡터에 삽입되고 레트로바이러스 입자에 패키징될 수 있다. 이어서 재조합 바이러스는 분리되어 생체 내 또는 생체 외 대상체의 세포로 전달될 수 있다. 다수의 레트로바이러스 시스템이 당업계에 공지되어 있다. 일부 구현예에서, 아데노바이러스 벡터가 사용된다. 다수의 아데노바이러스 벡터가 당업계에 공지되어 있다. 한 구현예에서, 렌티바이러스 벡터가 사용된다.
추가 프로모터 요소, 예를 들어, 인핸서는 전사 개시 빈도를 조절한다. 전형적으로, 이들은 시작 부위의 업스트림 30-110 염기쌍 (bp) 영역에 위치하지만, 최근에는 많은 프로모터가 시작 부위의 다운스트림에도 기능적 요소를 포함하는 것으로 나타났다. 프로모터 요소 사이의 간격은 종종 유연하므로 요소가 반전되거나 서로 상대적으로 이동할 때 프로모터 기능이 보존된다. 티미딘 키나제 (tk) 프로모터에서, 프로모터 요소 사이의 간격은 활성이 감소하기 시작하기 전에 50 bp 간격으로 증가될 수 있다. 프로모터에 따라, 개별 요소가 협력적으로 또는 독립적으로 기능하여 전사를 활성화할 수 있는 것으로 보인다.
적합한 프로모터의 한 예는 즉시 초기 시토메갈로바이러스 (CMV) 프로모터 서열이다. 이 프로모터 서열은 작동 가능하게 연결된 임의의 폴리뉴클레오티드 서열의 높은 수준의 발현을 유도할 수 있는 강력한 구성적 프로모터 서열이다. 적합한 프로모터의 또 다른 예는 신장 성장 인자-1α (EF-1α)이다. 그러나, 유인원 바이러스 40 (SV40) 초기 프로모터, 마우스 유방 종양 바이러스 (MMTV), 인간 면역결핍 바이러스 (HIV) 긴 말단 반복 (LTR) 프로모터, MoMuLV 프로모터, 조류 백혈병 바이러스 프로모터, 엡스테인-바 바이러스 즉시 초기 프로모터, 라우스 육종 바이러스 프로모터, U6 프로모터, 뿐만 아니라 액틴 프로모터, 미오신 프로모터, 헤모글로빈프로모터, 및 크레아틴 키나제 프로모터와 같지만, 이에 제한되지 않는 인간 유전자 프로모터를 포함하지만, 이에 제한되지 않는 다른 구성적 프로모터 서열이 사용될 수 있다. 추가로, 본 개시내용은 구성적 프로모터의 사용에 제한되지 않아야 한다. 유도성 프로모터도 본 발명의 일부로 고려된다. 유도성 프로모터의 사용은 그러한 발현이 요구될 때 작동 가능하게 연결된 폴리뉴클레오티드 서열의 발현을 켜거나 발현이 원하지 않을 때 발현을 끌 수 있는 분자 스위치를 제공한다. 유도성 프로모터의 예는 메탈로티오닌 프로모터, 글루코코르티코이드 프로모터, 프로게스테론 프로모터, 및 테트라사이클린 프로모터를 포함하지만 이에 제한되지 않는다.
수용체의 발현을 평가하기 위해, 세포 내로 도입될 발현 벡터는 또한 바이러스 벡터를 통해 형질감염 또는 감염시키려는 세포 집단으로부터 발현 세포의 식별 및 선택을 용이하게 하기 위해 선별 마커 유전자 또는 리포터 유전자 또는 둘 모두를 함유할 수 있다. 다른 양태에서, 선별 가능한 마커는 별도의 DNA 조각으로 운반될 수 있고 공동-형질감염 절차에 사용될 수 있다. 선별 가능한 마커 및 리포터 유전자 모두는 숙주 세포에서 발현을 가능하게 하는 적절한 조절 서열과 함께 플랭킹될 수 있다. 유용한 선별 마커는 예를 들어, neo 등과 같은 항생제 내성 유전자를 포함한다.
리포터 유전자는 잠재적으로 형질감염되거나 형질도입된 세포를 식별하고 조절 서열의 기능을 평가하는 데 사용된다. 일반적으로, 리포터 유전자는 수용 유기체 또는 조직에 존재하지 않거나 이에 의해 발현되지 않으며 그의 발현이 예를 들어 효소 활성과 같은 쉽게 검출할 수 있는 특성에 의해 나타나는 폴리펩티드를 암호화하는 유전자이다. 리포터 유전자의 발현은 DNA가 수용 세포에 도입된 후 적합한 시간에 분석된다. 적합한 리포터 유전자는 루시퍼라제, 베타-갈락토시다제, 클로람페니콜 아세틸 트랜스퍼라제, 분비된 알칼리 포스파타제 또는 녹색 형광 단백질 유전자를 암호화하는 유전자를 포함할 수 있다 (예를 들어, Ui-Tei et al., 2000 FEBS Letters 479: 79-82). 적합한 발현 시스템은 잘 알려졌고 공지된 기술을 사용하여 제조되거나 상업적으로 입수될 수 있다. 일반적으로, 가장 높은 수준의 리포터 유전자 발현을 나타내는 최소 5' 플랭킹 영역을 갖는 작제물이 프로모터로 식별된다. 이러한 프로모터 영역은 리포터 유전자에 연결될 수 있으며 프로모터-동 전사를 조절하는 능력에 대한 제제를 평가하는 데 사용될 수 있다.
유전자를 세포에 도입하고 발현시키는 방법은 당업계에 공지되어 있다. 발현 벡터와 관련하여, 벡터는 당업계의 임의의 방법에 의해 숙주 세포, 예를 들어 포유동물, 박테리아, 효모 또는 곤충 세포 내로 용이하게 도입될 수 있다. 예를 들어, 발현 벡터는 물리적, 화학적 또는 생물학적 수단에 의해 숙주 세포로 전달될 수 있다.
폴리뉴클레오티드를 숙주 세포에 도입하기 위한 물리적 방법은 인산칼슘 침전법, 리포펙션법, 입자 충격법, 미세주입법, 전기천공법 등을 포함한다 벡터 및/또는 외인성 핵산을 포함하는 세포를 생산하는 방법은 당업계에 잘 알려졌다. 예를 들어, Sambrook et al. (2001, Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory, New York) 참조. 폴리뉴클레오티드를 숙주 세포에 도입하는 한 가지 방법은 인산칼슘 형질감염이다.
관심 폴리뉴클레오티드를 숙주 세포에 도입하기 위한 생물학적 방법은 DNA 및 RNA 벡터의 사용을 포함한다. 바이러스 벡터, 특히 레트로바이러스 벡터는 포유동물, 예를 들어, 인간 세포에 유전자를 삽입하는 데 가장 널리 사용되는 방법이 되었다. 다른 바이러스 벡터는 렌티바이러스, 폭스바이러스, 단순 헤르페스 바이러스 I, 아데노바이러스 및 아데노 관련 바이러스 등으로부터 유래될 수 있다. 예를 들어, 미국 특허 번호 5,350,674 및 5,585,362 참조.
폴리뉴클레오티드를 숙주 세포에 도입하기 위한 화학적 방법은 거대분자 복합체, 나노캡슐, 마이크로스피어, 비드, 및 수중유 에멀젼, 미셀, 혼합 미셀 및 리포솜을 포함하는 지질 기반 시스템과 같은 콜로이드 분산 시스템을 포함한다. 예시적인 시험관 내 및 생체 내 전달 비히클로 사용하기 위한 예시적인 콜로이드 시스템은 리포솜 (예를 들어, 인공 막 소포)이다.
외인성 핵산을 숙주 세포에 도입하는 방법 또는 본 발명의 억제제에 세포를 노출시키는 방법과 상관없이, 숙주 세포 내 재조합 DNA 서열의 존재를 확인하기 위해 다양한 분석을 수행할 수 있다. 이러한 검정은 예를 들어, 서던 및 노던 블로팅, RT-PCR 및 PCR과 같은 당업자에게 잘 알려진 "분자 생물학적" 검정; 예를 들어, 면역학적 수단 (ELISA 및 웨스턴 블롯) 또는 본 개시내용의 범위 내에 속하는 작용제를 확인하기 위한 본원에 기재된 검정에 의해 특정 펩티드의 존재 또는 부재를 검출하는 것과 같은 "생화학적" 검정을 포함한다.
면역 세포
본 개시내용은 본원에 기재된 수용체, 벡터 및 폴리뉴클레오티드를 포함하는 면역 세포를 제공한다.
일부 구현예에서, 면역 세포는 다음을 포함한다: (a) 하기로부터 선택된 표적 항원에 특이적인 제1 세포외 리간드 결합 도메인을 포함하는 제1 수용체: (i) 암 세포 특이적 항원, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 펩티드 항원; 또는 (ii) EGFR, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 펩티드 항원; 및 (b) 그의 발현이 이형접합성의 소실 또는 다른 메커니즘으로 인해 암 세포로부터 소실된 비-표적 항원에 특이적으로 결합하는 제2 세포외 리간드를 포함하는 제2 수용체. 일부 구현예에서, 비-표적 항원은 CXCL16, COLEC12 및 APCDD1, HLA-A*01, HLA-A*02, HLA-A*03, HLA-A*11, HLA-B*07, HLA-C*07, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 항원 펩티드로부터 선택된다. 일부 구현예에서, 비-표적 항원은 HLA-A*02, 또는 이의 대립형질 변이체이다. 일부 구현예에서, 제1 수용체는 CAR 또는 TCR이다. 일부 구현예에서, 제2 수용체는 억제 수용체이다.
일부 구현예에서, 면역 세포는 shRNA를 포함한다. 일부 구현예에서, shRNA는 B2M의 발현을 감소시키거나 제거한다. 일부 구현예에서, shRNA는 HLA-A*02의 발현을 감소시키거나 제거한다.
일부 구현예에서, 면역 세포는 MHC 클래스 I 유전자 또는 B2M 유전자에 대한 변형을 포함한다. 일부 구현예에서, MHC 클래스 I 유전자는 HLA-A*02, 또는 이의 대립형질 변이체이다. 일부 구현예에서, 변형은 MHC 클래스 I 유전자 또는 B2M 유전자의 발현을 감소시키거나 제거한다.
일부 구현예에서, 면역 세포는 본원에 기재된 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 제1 수용체를 암호화하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 제2 수용체를 암호화하는 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 shRNA를 암호화한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 제1 수용체 및 본원에 제2 수용체를 암호화하는 기재된 서열을 포함한다. 일부 구현예에서, 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템은 본원에 기재된 제1 수용체, 본원에 기재된 제2 수용체, 및 본원에 기재된 shRNA를 암호화하는 서열을 포함한다.
본원에서 사용되는 바와 같이, 용어 "면역 세포"는 선천적 또는 후천적 (획득) 면역 시스템에 관여하는 세포를 지칭한다. 예시적인 선천성 면역 세포는 호중구, 단핵구 및 대식세포와 같은 식세포, 자연 살해 (NK) 세포, 호중구 호산구 및 호염기구와 같은 다핵성 백혈구 및 단핵구, 대식세포 및 비만 세포와 같은 단핵 세포를 포함한다. 후천 면역의 역할을 하는 면역세포는 T-세포 및 B-세포와 같은 림프구를 포함한다.
일부 구현예에서, 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 174 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 174, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 제2 수용체는 서열 번호 174, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 면역 세포는 T2A 자가 절단 펩티드를 추가로 포함하고, 여기서 T2A 자가 절단 펩티드는 서열 번호 178, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
일부 구현예에서, 면역 세포는 간섭 RNA를 추가로 포함하고, 여기서 간섭 RNA는 서열 번호 179, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함한다.
본원에서 사용되는 바와 같이, "T-세포"는 흉선에서 발생하는 골수 전구체로부터 유래하는 유형의 림프구를 지칭한다. 흉선으로 이동할 때 발생하는 여러 개별 유형의 T-세포는 헬퍼 CD4+ T-세포, 세포독성 CD8+ T 세포, 기억 T 세포, 조절 CD4+ T-세포, NK T 세포, γδ T 세포, 점막 -관련 불변 T (MAIT) 세포, 및 줄기 기억 T-세포를 포함한다. 상이한 유형의 T-세포는 그들의 마커 발현에 기초하여 통상의 기술자에 의해 구별될 수 있다. T-세포 유형을 구별하는 방법은 통상의 기술자에게 쉽게 자명할 것이다.
일부 구현예에서, 면역 세포는 T 세포, B 세포 및 자연 살해 (NK) 세포로 구성된 그룹으로부터 선택된다. 일부 구현예에서, 면역 세포는 T 세포이다. 일부 구현예에서, 면역 세포는 B 세포이다. 일부 구현예에서, 면역 세포는 자연 살해 (NK) 세포이다. 일부 구현예에서, 면역 세포는 CD8-이다. 일부 구현예에서, 면역 세포는 CD8+이다. 일부 구현예에서, 면역 세포는 CD4+이다. 일부 구현예에서, 면역 세포는 CD4-이다. 일부 구현예에서, 면역 세포는 CD8-/CD4+ 이다. 일부 구현예에서, 면역 세포는 CD8+ CD4- T 세포이다.
일부 구현예에서, 면역 세포는 감마 델타 (γδ) T 세포이다. 일부 구현예에서, 면역 세포는 불변 T 세포이다. 일부 구현예에서, 면역 세포는 불변 자연 살해 T 세포 (iNKT 세포)이다.
일부 구현예에서, 제1 수용체 및 제2 수용체는 함께 표적 세포의 존재 하에 면역 세포를 특이적으로 활성화시킨다.
일부 구현예에서, 면역 세포는 비천연 세포이다. 일부 구현예에서, 면역 세포는 단리된다.
T 세포와 같은, 면역 세포의 집단을 본 개시내용의 벡터로 형질전환시키는 방법은 당업자에게 쉽게 자명할 것이다. 예를 들어, CD3+ T 세포는 제조업체의 지침에 따라 CD3+ T 세포 음성 분리 키트 (Miltenyi)를 사용하여 PBMC로부터 분리할 수 있다. T 세포는 CD3/28 다이나비드 (1:1 세포 대 비드 비율) 및 300 단위/mL의 IL-2 (Miltenyi)의 존재 하에 5% 인간 A/B 혈청 및 1% Pen/strep이 보충된 X-Vivo 15 배지에서 1 x 10^6 세포/mL의 밀도로 배양될 수 있다. 2일 후, 당업계에 공지된 방법을 사용하여 렌티바이러스 벡터와 같은 바이러스 벡터로 T 세포를 형질도입할 수 있다. 일부 구현예에서, 바이러스 벡터는 5의 감염 다중도 (MOI)에서 형질도입된다. 그런 다음 세포는 IL-2 또는 IL-7/15/21의 조합과 같은 다른 사이토카인에서 농축 전 추가 5일 동안 배양될 수 있다. B 세포와 같은 면역 세포의 다른 집단, 또는 다른 T 세포의 다른 집단을 분리하고 배양하는 방법은 당업자에게 쉽게 자명할 것이다. 이 방법은 잠재적인 접근 방식을 설명하지만 이러한 방법론이 빠르게 진화하고 있다는 점에 유의해야 한다. 예를 들어 말초 혈액 단핵 세포의 우수한 바이러스 형질도입은 성장 5일 후에 달성되어 >99% CD3+ 고도로 형질도입된 세포 집단을 생성할 수 있다.
TCR, CAR, 억제 수용체, 수용체 또는 이를 암호화하는 벡터를 포함하는 T 세포 집단을 활성화 및 배양하는 방법은 당업자에게 쉽게 자명할 것이다.
TCR을 발현하기 위한 T 세포의 유전적 변형 전이든 후이든, T 세포는 일반적으로 예를 들어, 미국 특허 번호 6,352,694; 6,534,055; 6,905,680; 6,692,964; 5,858,358; 6,887,466; 6,905,681; 7,144,575; 7,067,318; 7,172,869; 7,232,566; 7,175,843; 5,883,223; 6,905,874; 6,797,514; 6,867,041, 10040846; 및 미국 특허 출원 공개 번호 2006/0121005에 기재된 바와 같은 방법을 사용하여 활성화되고 확장될 수 있다.
일부 구현예에서, 본 개시내용의 T 세포는 시험관 내에서 확장되고 활성화된다. 일반적으로, 본 개시내용의 T 세포는 CD3/TCR 복합체 관련 신호를 자극하는 작용제 및 T 세포의 표면 상의 공동 자극 분자를 자극하는 리간드가 부착된 표면과의 접촉에 의해 시험관 내에서 확장된다. 특히, T 세포 집단은 예를 들어 항-CD3 항체와의 접촉에 의해 본원에 기술된 바와 같이 자극될 수 있다. T 세포의 표면의 보조 분자의 공동 자극을 위해, 보조 분자에 결합하는 리간드가 사용된다. 예를 들어, T 세포의 집단은 T 세포의 증식을 자극하기에 적합한 조건 하에서 항-CD3 항체 및 항-CD28 항체와 접촉할 수 있다. CD4+ T 세포 또는 CD8+ T 세포의 증식을 자극하기 위해, 항-CD3 항체 및 항-CD28 항체를 사용할 수 있다. 항-CD28 항체의 예는 당업계에 일반적으로 알려진 다른 방법을 사용할 수 있는 9.3, B-T3, XR-CD28 (Diaclone, Besancon, France)을 포함한다 (Berg et al., Transplant Proc. 30(8):3975-3977, 1998; Haanen et al., J. Exp. Med. 190(9):13191328, 1999; Garland et al., J. Immunol Meth. 227(1-2):53-63, 1999).
일부 구현예에서, T 세포에 대한 1차 자극 신호 및 공동 자극 신호는 상이한 프로토콜에 의해 제공될 수 있다. 예를 들어, 각 신호를 제공하는 작용제는 용액에 있거나 표면에 결합될 수 있다. 표면에 커플링될 때, 작용제는 동일한 표면 (즉, "시스" 형태) 또는 분리된 표면 (즉, "트랜스" 형태)에 커플링될 수 있다. 대안적으로, 하나의 작용제는 표면에 결합되고 다른 작용제는 용액에 커플링될 수 있다. 일부 구현예에서, 공동 자극 신호를 제공하는 작용제는 세포 표면에 결합되고 1차 활성화 신호를 제공하는 작용제는 용액 중에 있거나 표면에 결합된다. 특정 구현예에서, 두 제제 모두 용액 상태일 수 있다. 또 다른 구현예에서, 작용제는 가용성 형태일 수 있고, 이어서 Fc 수용체를 발현하는 세포 또는 작용제에 결합할 항체 또는 다른 결합제와 같은 표면에 가교 결합될 수 있다. 이와 관련하여, 예를 들어, 본 개시내용에서 T 세포를 활성화 및 확장하는데 사용하기 위해 고려되는 인공 항원 제시 세포 (aAPC)에 대한 미국 특허 출원 공개 번호 20040101519 및 20060034810 참조.
일부 구현예에서, 2개의 작용제는 동일한 비드, 즉, "시스," 또는 별도의 비드, 즉, "트랜스" 상의 비드에 고정화된다. 예로서, 1차 활성화 신호를 제공하는 작용제는 항-CD3 항체 또는 이의 항원-결합 단편이고 공동 자극 신호는 항-CD28 항체 또는 이의 항원-결합 단편이고; 두 작용제는 동일한 분자량으로 동일한 비드에 공동 고정화된다. 한 구현예에서, CD4+ T 세포 확장 및 T 세포 성장을 위해 비드에 결합된 1:1 비율의 각 항체가 사용된다. 일부 구현예에서, 비드에 결합된 CD3:CD28 항체의 비율은 100:1 내지 1:100 범위이고 그 사이의 모든 정수 값이다. 본 개시내용의 한 양태에서, 항-CD3 항체보다 더 많은 항-CD28 항체가 입자에 결합되며, 즉 CD3:CD28의 비율은 1 미만이다. 본 개시내용의 특정 구현예에서, 비드에 결합된 항 CD28 항체 대 항 CD3 항체의 비율은 2:1보다 크다.
1:500 내지 500:1의 입자 대 세포 비율 및 그 사이의 임의의 정수 값을 사용하여 T 세포 또는 다른 표적 세포를 자극할 수 있다. 당업자가 쉽게 알 수 있는 바와 같이, 입자 대 세포의 비율은 표적 세포에 대한 입자 크기에 따라 달라질 수 있다. 예를 들어, 작은 크기의 비드는 몇 개의 세포에만 결합할 수 있는 반면 큰 비드는 많은 세포에 결합할 수 있다. 특정 구현예에서 세포 대 입자의 비율은 1:100 내지 100:1 범위이고 그 사이의 임의의 정수 값이고 추가 구현예에서 비율은 1:9 내지 9:1 및 그 사이의 임의의 정수 값을 포함하고, T 세포를 자극하기 위해 사용될 수 있다. 일부 구현예에서, 1:1 세포 대 비드의 비율이 사용된다. 당업자는 다양한 다른 비율이 본 발명에 사용하기에 적합할 수 있음을 이해할 것이다. 특히, 비율은 입자 크기와 세포 크기 및 유형에 따라 달라질 것이다.
본 개시내용의 추가 구현예에서, 세포, 예를 들어 T 세포는 제제-코팅된 비드와 결합되고, 비드 및 세포는 후속적으로 분리된 다음 세포가 배양된다. 대안적인 구현예에서, 배양 전에 제제-코팅된 비드 및 세포는 분리되지 않고 함께 배양된다. 추가 구현예에서, 비드 및 세포는 자기력과 같은 힘의 적용에 의해 먼저 농축되어 세포 표면 마커의 결찰이 증가되어 세포 자극을 유도한다.
예로서, 세포 표면 단백질은 항-CD3 및 항-CD28가 부착된 상자성 비드가 T 세포와 접촉하도록 함으로써 결찰될 수 있다. 한 구현예에서 세포 (예를 들어, CD4+ T 세포) 및 비드 (예를 들어, 1:1 비율의 DYNABEADS CD3/CD28 T 상자성 비드)는 완충액에서 결합된다. 다시, 당업자는 임의의 세포 농도가 사용될 수 있음을 쉽게 인식할 수 있다. 특정 구현예에서 세포와 입자의 최대 접촉을 보장하기 위해 입자와 세포가 함께 혼합되는 부피를 상당히 감소시키는 것 (즉, 세포의 농도를 증가시키는 것)이 바람직할 수 있다. 예를 들어, 한 구현예에서, 약 20억 세포/ml의 농도가 사용된다. 또 다른 구현예에서, 1억 초과 세포/ml가 사용된다. 추가 구현예에서, 1천, 1천5백, 2천, 2천5백, 3천, 3천5백, 4천, 4천5백, 또는 5천만 세포/ml의 세포 농도가 사용된다. 또 다른 구현예에서, 7천5백, 8천, 8천5백, 9천, 9천5백, 또는 1억 세포/ml의 세포 농도가 사용된다. 추가 구현예에서, 1억 2500만 또는 1억 5000만 세포/ml의 농도가 사용될 수 있다. 일부 구현예에서, 1x106 세포/mL의 밀도로 배양되는 세포가 사용된다.
일부 구현예에서, 혼합물은 수 시간 (약 3시간) 내지 약 14일 또는 그 사이의 임의의 시간당 정수 값 동안 배양될 수 있다. 또 다른 구현예에서, 비드 및 T 세포는 2-3일 동안 함께 배양된다. T 세포 배양에 적합한 조건은 혈청 (예를 들어, 태아 소 또는 인간 혈청), 인터루킨-2 (IL-2), 인슐린, IFN-γ, IL-4, IL-7, GM-CSF, IL-10, IL-12, IL-15, TGFβ, 및 TNF-α 또는 당업자에게 공지된 세포 성장을 위한 임의의 기타 첨가제를 포함하는, 증식 및 생존력을 위해 필수적인 인자를 함유할 수 있는 적절한 배지 (예를 들어, 최소 필수 배지 또는 RPMI 배지 1640 또는, X-vivo 15, (Lonza))를 포함한다. 세포 성장을 위한 기타 첨가제는 계면활성제, 플라스마네이트, 및 N-아세틸-시스테인 및 2-머캅토에탄올과 같은 환원제를 포함되지만 이에 제한되지 않는다. 배지는 RPMI 1640, AIM-V, DMEM, MEM, α-MEM, F-12, X-Vivo 15, 및 X-Vivo 20, 옵티마이저를 포함할 수 있고, 아미노산, 피루브산나트륨 및 비타민이 추가되거나, 무혈청이거나 적절한 양의 혈청(또는 혈장) 또는 정의된 일련의 호르몬, 및/또는 T 세포의 성장 및 확장에 충분한 양의 사이토카인으로 보충된다. 일부 구현예에서, 배지는 5% 인간 A/B 혈청, 1% 페니실린/스트렙토마이신 (pen/strep) 및 300 단위/ml의 IL-2 (Miltenyi)이 보충된 X-VIVO-15 배지를 포함한다.
T 세포는 성장을 지원하는 데 필요한 조건, 예를 들어, 적절한 온도 (예를 들어, 37℃) 및 대기 (예를 들어, 공기 +5% CO2) 하에서 유지된다.
일부 구현예에서, TCR, CAR 및 본 개시내용의 억제 수용체를 포함하는 T 세포는 자가 세포이다. 확장 및 유전적 변형 전에, T 세포의 공급원은 대상체로부터 수득된다. T 세포와 같은 면역 세포는 말초 혈액 단핵 세포, 골수, 림프절 조직, 제대혈, 흉선 조직, 감염 부위의 조직, 복수, 흉수, 비장 조직 및 종양을 포함하는 다수의 공급원으로부터 얻을 수 있다. 본 개시내용의 특정 구현예에서, 당업계에서 이용 가능한 임의의 수의 T 세포주가 사용될 수 있다. 본 개시내용의 특정 구현예에서, T 세포는 Ficoll™ 분리와 같은 당업자에게 공지된 임의의 수의 기술을 사용하여 대상체로부터 수집된 혈액 단위로부터 수득될 수 있다.
일부 구현예에서, 개체의 순환 혈액으로부터의 세포는 성분채집술에 의해 얻어진다. 성분채집 산물은 일반적으로 T 세포, 단핵구, 과립구, B 세포, 기타 유핵 백혈구, 적혈구 및 혈소판을 포함하는 림프구를 포함한다. 일부 구현예에서, 성분채집법에 의해 수집된 세포는 혈장 분획을 제거하고 후속 처리 단계를 위해 적절한 완충액 또는 배지에 세포를 넣기 위해 세척될 수 있다. 일부 구현예에서, 세포는 인산염 완충 식염수 (PBS)로 세척된다. 대안적인 구현예에서, 세척 용액은 칼슘이 부족하고 마그네슘이 부족할 수 있거나 전부는 아니지만 많은 2가 양이온이 부족할 수 있다. 당업자는 세척 단계가 제조업체의 지침에 따라 반자동 "유동식" 원심분리기 (예를 들어, Cobe 2991 cell processor, Baxter CytoMate, 또는 Haemonetics Cell Saver 5)를 사용하여 당업자에게 알려진 방법에 의해 달성될 수 있음을 쉽게 이해할 것이다. 세척 후, 세포는 예를 들어, Ca2+-프리, Mg2+-프리 PBS, PlasmaLyte A, 또는 완충액이 있거나 없는 다른 식염수와 같은 다양한 생체 적합성 완충액에 재현탁될 수 있다. 대안적으로, 성분채집 샘플의 바람직하지 않은 성분을 제거하고 세포를 배양 배지에 직접 재현탁할 수 있다.
일부 구현예에서, T 세포와 같은 면역 세포는 예를 들어, PERCOLL™ 구배를 통한 원심분리 또는 역류 원심 분리에 의해 적혈구를 용해시키고 단핵구를 고갈시킴으로써 말초 혈액 림프구로부터 분리된다. T 세포, B 세포, 또는 CD4+ T 세포와 같은 면역 세포의 특정 하위집단은 양성 또는 음성 선택 기술에 의해 추가로 분리될 수 있다. 예를 들어, 한 구현예에서, T 세포는 원하는 T 세포의 양성 선택에 충분한 시간 동안, 항-CD4 -접합 비드와 함께 배양함으로써 분리된다.
음성 선택에 의한 T 세포 집단과 같은 면역 세포 집단의 농축은 음성 선택 세포에 고유한 표면 마커에 대한 항체의 조합으로 달성될 수 있다. 한 가지 방법은 음성으로 선택된 세포에 존재하는 세포 표면 마커에 대한 단클론 항체 칵테일을 사용하는 음성 자기 면역 부착 또는 유세포 분석을 통한 세포 분류 및/또는 선택이다. 예를 들어, 음성 선택으로 CD4+ 세포를 강화하기 위해, 단일 클론 항체 칵테일은 일반적으로 CD 14, CD20, CD 11b, CD 16, HLA-DR, 및 CD8에 대한 항체를 포함한다.
양성 또는 음성 선택에 의한 원하는 면역 세포 집단의 분리를 위해, 세포 및 표면 (예를 들어, 비드와 같은 입자)의 농도는 다양할 수 있다. 특정 구현예에서, 세포와 비드의 최대 접촉을 보장하기 위해 비드와 세포가 함께 혼합되는 부피를 상당히 감소시키는 것 (즉, 세포의 농도를 증가시키는 것)이 바람직할 수 있다.
일부 구현예에서, 세포는 2-10℃ 또는 실온에서 다양한 속도로 다양한 시간 동안 회전기에서 배양될 수 있다.
자극을 위한 T 세포, 또는 T 세포와 같은 면역 세포가 분리되는 PBMC도 세척 단계 후에 동결될 수 있다. 이론에 얽매이지 않기를 바라면서, 동결 및 후속 해동 단계는 세포 집단에서 과립구 및 어느 정도 단핵구를 제거함으로써 더욱 균일한 산물을 제공한다. 혈장과 혈소판을 제거하는 세척 단계 후 세포를 동결 용액에 현탁할 수 있다. 많은 동결 용액 및 매개변수가 당업계에 알려졌고 이러한 맥락에서 유용할 것이지만, 한 방법은 20% DMSO 및 8% 인간 혈청 알부민을 함유하는 PBS, 또는 10% 덱스트란 40 및 5% 덱스트로스, 20% 인간 혈청 알부민 및 7.5% DMSO, 또는 31.25% Plasmalyte-A, 31.25% 덱스트로스 5%, 0.45% NaCl, 10% 덱스트란 40 및 5% 덱스트로스, 20% 인간 혈청 알부민, 및 7.5% DMSO를 함유하는 배양 배지 또는 예를 들어, Hespan 및 PlasmaLyte A를 함유하는 다른 적합한 세포 동결 배지를 사용하는 것을 포함하고, 이어서 세포는 분당 1°의 속도로 -80℃로 동결되고 액체 질소 저장 탱크의 증기 상태로 저장된다. -20℃ 또는 액체 질소에서 즉시 제어되지 않은 동결뿐만 아니라 제어된 동결의 다른 방법을 사용할 수 있다.
본 개시내용은 본원에 기재된 활성화제 및/또는 억제 수용체를 발현하는 면역 세포를 제공하고, 여기서 면역 세포는 주요 조직적합성 (MHC) 클래스 I 복합체의 감소된 발현 및/또는 기능을 갖는다.
일부 구현예에서, 면역 세포는 자가 세포이다. 예를 들어, 면역 세포는 치료 요법의 일부로서 세포를 수용할 동일한 대상체로부터 분리되거나 유래된다. MHC 클래스 I 항원에 특이적인 억제 수용체로 MHC 클래스 I의 감소된 발현 및/또는 기능을 갖도록 자가 면역 세포를 변형시키는 것이 유리할 수 있다. 이론에 구애됨이 없이, MHC 클래스 I의 감소된 발현 및/또는 기능을 갖도록 하는 자가 면역 세포의 변형은 시스 또는 트랜스로 면역 세포에 의해 발현되는 MHC 클래스 I에 의한 억제 수용체의 결합을 감소시킨다.
일부 구현예에서, 면역 세포는 모두 동종이계이다. 동종이계 면역 세포는 면역 세포가 투여될 대상체 이외의 공여자로부터 유래될 수 있다. 동종이계 면역 세포는 다양한 유전형의 대상체에서 사용하기 위해 동종이계 세포를 준비하고 보관할 가능성 때문에 세포 요법에서 일반적으로 "규격품(off the shelf)" 또는 "보편적"이라고 한다.
MHC 클래스 I 복합체의 발현 및/또는 기능을 감소시키는 임의의 적합한 방법은 본 개시내용의 범위 내에 있는 것으로 간주되며, 특히 MHC 클래스 I 성분을 암호화하는 하나 이상의 RNA를 녹다운시키는 간섭 RNA의 발현, 또는 MHC 클래스 I 성분을 암호화하는 유전자의 변형을 포함한다.
주요 조직적합성 복합체 (MHC)는 적응 면역 체계에 필요한 일련의 폴리펩티드를 암호화하는 척추동물 게놈 상 유전자좌이다. 이들 중에는 HLA-A, HLA-B, 및 HLA-C 및 이의 대립유전자를 포함하는 MHC 클래스 I 폴리펩티드가 있다. MHC 클래스 I 대립유전자는 매우 다형성이 있으며 모든 유핵 세포에서 발현된다. HLA-A, HLA-B, 및 HLA-C 및 이의 대립유전자에 의해 암호화되는 MHC 클래스 I 폴리펩티드는 β마이크로글로불린 (B2M)과 이종이량체를 형성하고 세포 표면에서 항원과 복합체로 존재한다. 본원에서 언급된 바와 같이, MHC 클래스 I 유전자 또는 폴리펩티드는 MHC 또는 상기 폴리펩티드를 암호화하는 상응하는 유전자에서 발견되는 임의의 폴리펩티드를 지칭할 수 있다. 일부 구현예에서, 본 개시내용의 면역 세포는 MHC 클래스 I 폴리펩티드, 예를 들어 HLA-A, HLA-B, 및 HLA-C 및 이의 대립유전자를 포함하는 억제제 리간드에 의해 불활성화된다. HLA-A 대립유전자는 예를 들어 제한 없이, HLA-A*02, HLA-A*02:01, HLA-A*02:01:01, HLA-A*02:01:01:01이고/거나, HLA-A*02 단백질과 동일하거나 유사한 단백질을 암호화하는 임의의 유전자일 수 있다. 따라서, 본원에 기술된 바와 같은 자가분비 신호전달/ 결합을 방지하기 위해, 면역 세포에서 HLA-A, HLA-B, 및 HLA-C 및 이의 대립유전자에 의해 암호화되는 폴리펩티드의 발현을 제거하거나 감소시키는 것이 바람직하다.
MHC 클래스 I 폴리펩티드 발현이 감소된 면역 세포
일부 구현예에서, 본원에 기재된 면역 세포는 내인성 MHC 클래스 I 폴리펩티드의 대립유전자를 암호화하는 내인성 유전자의 발현 또는 기능을 불활성화시키거나 감소 또는 제거하도록 변형된다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A, HLA-B, 및/또는 HLA-C이다. HLA-A, HLA-B 및 HLA-C는 HLA-A, HLA-B 및 HLA-C 유전자좌에 의해 암호화된다. 각각의 HLA-A, HLA-B 및 HLA-C는 많은 변이 대립유전자를 포함하며, 이들 모두는 본 발명의 범위 내에 있는 것으로 간주된다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A이다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A*02이다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A*02:01이다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A*02:01:01이다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자는 HLA-A*02:01:01:01이다.
일부 구현예에서, 본원에 기재된 유전적으로 조작된 면역 세포는 B2M 유전자 산물의 발현을 감소시키거나 제거하도록 변형된다. 베타-2 마이크로글로불린 (B2M) 유전자는 주요 조직적합성 복합체 (MHC) 클래스 I, 즉 MHC-I 복합체와 관련된 단백질을 암호화한다. MHC-I 복합체는 세포 표면에 항원을 제시하는 데 필요하다. MHC -I 복합체는 B2M이 결실될 때 파괴되고 기능하지 않는다 (Wang D et al. Stem Cells Transl Med. 4:1234-1245 (2015)). 또한, B2M 유전자는 당업계에 알려진 유전자 편집 기술을 사용하여 고효율로 파괴될 수 있다 (Ren et al. Clin. Cancer Res. 23:2255-2266 (2017)). B2M을 줄이거나 제거하면 면역 세포의 표면 상의 기능적 MHC I를 줄이거나, 제거할 수 있다.
본 개시내용은 면역 세포에서 내인성 표적 유전자를 편집하기 위한 유전자 편집 시스템을 제공한다. 본 개시내용은 표적 유전자의 서열에 특이적인 간섭 RNA를 제공한다. CRISPR/Cas 시스템, TALEN 및 징크 핑거(zinc finger)와 같은 유전자 편집 시스템을 사용하여 이중 가닥 절단을 생성할 수 있으며, 이는 상동성 지정 복구 또는 비-상동성 말단 접합 (NHEJ)과 같은 유전자 복구 메커니즘을 통해 돌연변이를 도입하는 데 사용할 수 있다. 절단 끝의 절제 후 NHEJ, 또는 부적절한 끝 접합을 사용하여 결실을 도입할 수 있다. 일부 구현예에서, 표적 유전자는 MHC-I 복합체의 서브유닛을 암호화하는 유전자를 포함한다.
표적 유전자 서열은 프로모터, 인핸서, 인트론, 엑손, 인트론/엑손 접합부, 전사 산물 (pre-mRNA, mRNA, 및 스플라이스 변이체), 및/또는 3' 및 5' 비번역 영역 (UTR)을 포함하지만, 이에 제한되지 않는다. 임의의 유전자 요소 또는 유전자 요소의 조합은 본원에 기재된 면역 세포에서 유전자 편집의 목적을 위해 표적화될 수 있다. 표적 유전자에 대한 변형은 표적 유전자 또는 유전자 산물의 발현 또는 기능을 변경하거나 방해하는 표적 유전자를 편집하기 위해 당업계에 공지된 임의의 방법을 사용하여 달성될 수 있다.
일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자를 변형시키는 것은 유전자의 전부 또는 일부를 결실시키는 것을 포함한다. 일부 구현예에서, MHC 클래스 I 폴리펩티드를 암호화하는 유전자를 변형시키는 것은 유전자에 돌연변이를 도입하는 것을 포함한다. 일부 구현예에서, 돌연변이는 결실, 삽입, 치환 또는 프레임이동 돌연변이를 포함한다. 일부 구현예에서, 유전자를 변형시키는 것은 핵산 유도 엔도뉴클레아제를 사용하는 것을 포함한다.
본원에 기재된 표적 유전자에 대한 유전자 서열은 당업계에 공지되어 있다. 서열은 NCBI GenBank 또는 NCBI 뉴클레오티드 데이터베이스와 같은 공용 데이터베이스에서 찾을 수 있다. 서열은 유전자 식별자를 사용하여 찾을 수 있고, 예를 들어, HLA-A 유전자는 NCBI 유전자 ID: 3105를 갖고, HLA-B 유전자는 NCBI 유전자 ID: 3106을 갖고, HLA-C 유전자는 NCBI 유전자 ID: 3107을 갖고, B2M 유전자는 NCBI 유전자 ID: 567 및 NCBI 참조 서열: NC_000015.10을 갖는다. 유전자 서열은 키워드를 사용하여 공개 데이터베이스를 검색하여 찾을 수도 있다. 예를 들어, HLA-A 대립유전자는 키워드, "HLA-A*02", "HLA-A*02:01", "HLA-A*02:01:01" 또는 "HLA-A*02:01:01:01"을 검색하여 NCBI 뉴클레오티드 데이터베이스에서 찾을 수 있다. 이들 서열은 당업계에 공지된 다양한 유전자 편집 기술에서 표적화를 위해 사용될 수 있다. 표 7은 본원에 기재된 변형을 위해 표적화된 HLA-A 대립유전자 및 B2M 유전자 서열의 비제한적 예시적 서열을 제공한다.
표 7. 예시적인 표적 유전자 서열
당업자는 T가 U를 대체하여 서열을 DNA 서열로 또는 그 반대로 전환할 수 있고, 둘 다 본 개시내용의 표적 유전자 서열로 간주됨을 인식할 것이다.
일부 구현예에서, 표적 유전자는 유도된 엔도뉴클레아제를 사용하여 본원에 기술된 면역 세포에서 편집된다. 예시적인 핵산 유도된 엔도뉴클레아제는 CRISPR/Cas9와 같은 클래스 II를 포함한다.
본원에 사용된 "CRISPR" 또는 "CRISPR 유전자 편집"은 군집된 규칙적으로 간격을 둔 짧은 회문 반복 세트 또는 이러한 반복 세트를 포함하는 시스템을 지칭한다. 본원에서 사용되는 바와 같이, "Cas"는 CRISPR-연관 단백질을 지칭한다. "CRISPR/Cas" 시스템은 표적 유전자를 침묵시키거나 녹아웃시키거나 돌연변이시키는 데 사용될 수 있는 CRISPR 및 Cas에서 파생된 시스템을 지칭한다. 이 시스템은 플라스미드 및 파지와 같은 외래 유전 요소에 대한 저항성을 부여하고 일종의 획득 면역을 제공하는 일종의 원핵 면역 시스템이다. CRISPR/Cas 시스템은 유전자 편집에 사용하기 위해 변형되었다. 이는 하나 이상의 특별히 설계된 가이드 핵산 (gNA), 일반적으로 가이드 RNA (gRNA), 및 gNA와 리보핵단백질 복합체를 형성하는 적절한 Cas 엔도뉴클레아제를 진핵 세포에 도입함으로써 달성된다. gNA는 gNA-엔도뉴클레아제 단백질 복합체를 표적 게놈 위치로 유도하고, 엔도뉴클레아제는 표적 게놈 위치에서 가닥 절단을 도입한다. 이러한 이중 가닥 절단은 비-상동 말단 접합 (결실을 야기함) 또는 상동 수리 (삽입을 생성할 수 있음)와 같은 세포 메커니즘에 의해 수리될 수 있으며, 이에 따라 숙주 세포 게놈에 유전적 변형을 도입한다.
CRISPR/Cas 시스템은 클래스 및 유형별로 분류된다. 클래스 2 시스템은 현재 세 가지 유형 (유형 II, V 및 VI)으로 분류되는 단일 간섭 단백질을 나타낸다. 유전자 편집에 적합한 임의의 클래스 2 CRISPR/Cas 시스템, 예를 들어 유형 II, 유형 V 또는 유형 VI 시스템은 본 개시내용의 범위 내에 있는 것으로 생각된다. 예시적인 클래스 2 유형 II CRISPR 시스템은 Cas9, Csn2 및 Cas4를 포함한다. 예시적인 클래스 2, 유형 V CRISPR 시스템은 Cas12, Cas12a (Cpf1), Cas12b (C2c1), Cas12c (C2c3), Cas12d (CasY), Cas12e (CasX), Cas12f, Cas12g, Cas12h, Cas12i 및 Cas12k (C2c5)를 포함한다. 예시적인 클래스 2 유형 VI 시스템은 Cas13, Cas13a (C2c2) Cas13b, Cas13c 및 Cas13d를 포함한다.
CRISPR 유전자좌라고도 하는, CRISPR 서열은 교대 반복과 스페이서로 구성된다. 자연 발생 CRISPR에서, 스페이서는 일반적으로 플라스미드 또는 파지 서열과 같은 박테리아 외래 서열을 포함한다. 본원에 기재된 바와 같이, 스페이서 서열은 또한 "표적화 서열"로 지칭될 수 있다. 유전 공학을 위한 CRISPR/Cas 시스템에서, 스페이서는 표적 유전자 서열 (gNA)에서 파생된다.
예시적인 클래스 2 유형 II CRISPR 시스템은 이중 나선의 각 가닥에 대해 하나씩 2개의 활성 절단 부위를 갖는 뉴클레아제인 단백질 Cas9에 의존한다. Cas9과 변형된 CRISPR 유전자좌 RNA를 조합하면 유전자 편집 시스템에서 사용할 수 있다. Pennisi (2013) Science 341: 833-836. 일부 구현예에서, 면역 세포를 변형시키는 데 사용되는 Cas 단백질은 Cas9이다.
따라서 CRISPR/Cas 시스템은 염기쌍을 추가 또는 삭제하거나 표적의 발현을 감소시키는 조기 중단을 도입함으로써 본원에 기술된 면역 세포에서 편집을 위해 표적화된 유전자와 같은 표적 유전자를 편집하는 데 사용될 수 있다. CRISPR/Cas 시스템은 RNA 간섭처럼 대안적으로 사용되어 가역적인 방식으로 표적 유전자를 차단할 수 있다. 포유류 세포에서, 예를 들어, RNA는 Cas 단백질을 표적 유전자 프로모터로 안내하여 RNA 중합효소를 입체적으로 차단할 수 있다.
Cas 단백질은 임의의 박테리아 또는 고세균 Cas 단백질에서 파생될 수 있다. 임의의 적합한 CRISPR/Cas 시스템은 본 발명의 범위 내에 있는 것으로 생각된다. 다른 양태에서, Cas 단백질은 Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9, Cas10, Cas12a (Cpf1), Cas13, Csy1, Csy2, Csy3, Cse1, Cse2, Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4, CasX, CasY, 이의 상동체, 또는 이의 변형된 버전 중 하나 이상을 포함한다. 일부 구현예에서, Cas 단백질은 Cas9 단백질, Cpf1 단백질, C2c1 단백질, C2c2 단백질, C2c3 단백질, Cas3, Cas3-HD, Cas 5, Cas7, Cas8, Cas10, 또는 이들의 조합 또는 복합체이다. 일부 구현예에서, Cas 단백질은 Cas9 단백질이다.
예를 들어, 미국 공개 번호 20140068797, 및 Cong (2013) Science 339: 819-823에 기술된 당업계에 알려진 기술을 사용하여 표적 유전자를 억제하는 인공 CRISPR/Cas 시스템을 생성할 수 있다. 예를 들어, Tsai (2014) Nature Biotechnol., 32:6 569-576, 미국 특허 번호 8,871,445; 8,865,406; 8,795,965; 8,771,945; 및 8,697,359에 기술된 표적 유전자를 억제하는 당업계에 알려진 다른 인공 CRISPR/Cas 시스템을 또한 생성할 수 있다. 특정 Cas 단백질에 적합한 gNA를 설계하는 방법은 당업자에게 공지될 것이다.
본 개시내용은 표적 핵산 게놈 내의 특정 표적 유전자 서열에 대한 관련 폴리펩티드 (예를 들어, 핵산 유도된 엔도뉴클레아제)의 활성을 지시할 수 있는 유전자-표적화 가이드 핵산 (gNA)을 제공한다. 게놈-표적화 핵산은 RNA일 수 있다. 게놈-표적화 RNA는 본원에서 "가이드 RNA" 또는 "gRNA"로 지칭된다. 가이드 RNA는 적어도 관심 표적 핵산 서열에 혼성화하는 표적화 서열, 및 CRISPR 반복 서열을 포함할 수 있다. 일부 유형 II 시스템에서, gRNA는 또한 본원에서 "스캐폴드" 서열로도 지칭되는, tracrRNA 서열로 불리는 제2 RNA를 포함한다. 유형 II 가이드 RNA (gRNA)에서, CRISPR 반복 서열 및 스캐폴드 서열은 서로 혼성화하여 듀플렉스를 형성한다. 유형 V 가이드 RNA (gRNA)에서, crRNA는 듀플렉스를 형성한다. 두 시스템 모두에서, 듀플렉스는 가이드 RNA와 부위 지정 폴리펩티드가 복합체를 형성하도록 부위 지정 폴리펩티드에 결합할 수 있다. 유전자-표적화 핵산은 부위 지정 폴리펩티드와의 회합에 의해 복합체에 표적 특이성을 제공할 수 있다. 따라서 유전자-표적화 핵산은 부위 지정 폴리펩티드의 활성을 지시할 수 있다.
일부 구현예에서, 본 개시내용은 표적화 서열 및 가이드 RNA 스캐폴드 서열을 포함하는 가이드 RNA를 제공하고, 여기서 표적화 서열은 표적 유전자의 서열에 상보적이다.
예시적인 가이드 RNA는 약 15-20개 염기의 표적화 서열을 포함한다. 당업자에 의해 이해되는 바와 같이, 각각의 gRNA는 그의 게놈 표적 서열에 상보적인 표적 서열을 포함하도록 설계될 수 있다. 예를 들어, 각각의 표적화 서열, 예를 들어, PAM 부위를 나타내는 3개의 3' 뉴클레오티드를 제외한 표 8에 제시된 DNA 서열의 RNA 버전은 단일 RNA 키메라 또는 crRNA에 넣을 수 있다.
유전자 표적화 핵산은 이중-분자 가이드 RNA일 수 있다. 유전자 표적화 핵산은 단일-분자 가이드 RNA일 수 있다. 유전자 표적화 핵산은 예를 들어, 쌍을 이룬 gRNA, 또는 단일 단계에서 사용되는 다중 gRNA를 포함하는 것과 같이 당업계에 알려진 가이드 RNA의 임의의 알려진 구성일 수 있다. 코딩 서열과 스플라이스 접합부가 있는 게놈 서열에서는 분명하지만, 유전자 발현에 필요한 다른 특징은 특이하고 불분명할 수 있다.
이중-분자 가이드 RNA는 RNA의 두 가닥을 포함할 수 있다. 제1 가닥은 5'에서 3' 방향의 서열, 선택적인 스페이서 연장 서열, 표적화 서열 및 최소 CRISPR 반복 서열을 포함한다. 제2 가닥은 최소 tracrRNA 서열 (최소 CRISPR 반복 서열에 상보적), 3' 서열 및 선택적인 tracrRNA 연장 서열을 포함할 수 있다.
유형 II 시스템에서 단일-분자 가이드 RNA (sgRNA)는 5'에서 3' 방향으로, 선택적인 스페이서 연장 서열, 표적화 서열, 최소 CRISPR 반복 서열, 단일-분자 가이드 링커, 최소 tracrRNA 서열, 3' 서열 및 선택적인 tracrRNA 연장 서열을 포함할 수 있다. 선택적 tracrRNA 연장은 가이드 RNA에 대한 추가 기능성 (예를 들어, 안정성)을 제공하는 요소를 포함할 수 있다. 단일-분자 가이드 링커는 최소 CRISPR 반복과 최소 tracrRNA 서열을 연결하여 헤어핀 구조를 형성할 수 있다. 선택적 tracrRNA 연장은 하나 이상의 헤어핀을 포함할 수 있다.
일부 구현예에서, 가이드 RNA 또는 단일-분자 가이드 RNA (sgRNA)는 표적화 서열 및 스캐폴드 서열을 포함할 수 있다. 일부 구현예에서, 스캐폴드 서열은 Cas9 gRNA 서열이다. 일부 구현예에서, 스캐폴드 서열은 GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT (서열 번호 435)과 적어도 90%, 적어도 95%, 적어도 96%, 적어도 97%, 적어도 98%, 또는 적어도 99% 동일성을 공유하는 서열을 포함하는 DNA 서열에 의해 암호화된다. 일부 구현예에서, 스캐폴드 서열은 GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT (서열 번호 435)를 포함하는 DNA 서열에 의해 암호화된다.
일부 구현예에서, 예를 들어 CRISPR/Cas 시스템이 Cas9 시스템인 구현예에서, sgRNA는 sgRNA 서열의 5' 말단에 20개 뉴클레오티드 표적화 서열을 포함할 수 있다. sgRNA 서열의 5' 말단에 20개 미만의 뉴클레오티드 표적화 서열을 포함할 수 있다. sgRNA는 sgRNA 서열의 5' 말단에 20개 초과의 뉴클레오티드 표적화 서열을 포함할 수 있다. sgRNA는 sgRNA 서열의 5' 말단에 17-30개 뉴클레오티드를 갖는 가변 길이 표적화 서열을 포함할 수 있다.
적합한 스캐폴드 서열, 및 스캐폴드 표적화 서열의 배열은 엔도뉴클레아제의 선택에 따라 달라지며 당업자에게 공지될 것이다.
유형 II 시스템, 예를 들어 Cas9의 단일-분자 가이드 RNA (sgRNA)는 5'에서 3' 방향으로 최소 CRISPR 반복 서열 및 표적화 서열을 포함할 수 있다.
예시로서, CRISPR/Cas9 또는 CRISPR/Cpf1 시스템에서 사용되는 가이드 RNA, 또는 다른 더 작은 RNA는 하기 예시되고 당업계에 기재된 바와 같이 화학적 수단에 의해 쉽게 합성될 수 있다. 화학적 합성 절차가 지속적으로 확장되는 동안, 고성능 액체 크로마토그래피 (HPLC, PAGE와 같은 젤의 사용을 피함)와 같은 절차에 의한 그러한 RNA의 정제는 폴리뉴클레오티드 길이가 100개 정도의 뉴클레오티드 이상으로 상당히 증가함에 따라 더욱 어려워지는 경향이 있다. 더 긴 길이의 RNA를 생성하는 데 사용되는 한 가지 접근 방식은 함께 결찰되는 두 개 이상의 분자를 생성하는 것이다. Cas9 또는 Cpf1 엔도뉴클레아제를 암호화하는 것과 같은 훨씬 더 긴 RNA는 효소적으로 더 쉽게 생성된다. 다양한 유형의 RNA 변형은 RNA의 화학적 합성 및/또는 효소적 생성 동안 또는 후에 도입될 수 있으며, 예를 들어, 당업계에 기술된 바와 같이 안정성을 강화하고, 선천적 면역 반응의 가능성 또는 정도를 감소시키고/하거나 다른 속성을 강화하는 변형을 들 수 있다.
gRNA의 표적화 서열은 관심 표적 핵산의 서열에 혼성화한다. 게놈-표적화 핵산의 표적화 서열은 혼성화 (즉, 염기쌍 형성)를 통해 서열-특이적 방식으로 표적 핵산과 상호작용할 수 있다. 표적화 서열의 뉴클레오티드 서열은 관심 표적 핵산의 서열에 따라 다를 수 있다.
본원에 기재된 Cas9 시스템에서, 표적화 서열은 시스템에서 사용되는 Cas9 효소의 PAM의 역 상보체의 5'에 위치하는 표적 핵산에 혼성화하도록 설계될 수 있다. 표적화 서열은 표적 서열과 완벽하게 일치하거나 불일치할 수 있다. 각각의 CRISPR/Cas 시스템 단백질은 표적 DNA에서 인식하는 특정 방향과 위치에 특정 PAM 서열을 가질 수 있다. 예를 들어, S. 피오게네스 Cas9은 표적 핵산에서 서열 5'을 포함하는 PAM을 인식하며, 여기서 R은 A 또는 G를 포함하고, 여기서 N은 임의의 뉴클레오티드이고 N은 표적화 서열에 의해 표적화된 표적 핵산 서열의 바로 3'이다. 적절한 PAM 서열의 선택은 당업자에게 명백할 것이다.
표적 서열은 gRNA의 표적화 서열에 상보적이며, 혼성화한다. 표적 핵산 서열은 20개 뉴클레오티드를 포함할 수 있다. 표적 핵산은 20개 미만의 뉴클레오티드를 포함할 수 있다. 표적 핵산은 20개 초과의 뉴클레오티드를 포함할 수 있다. 표적 핵산은 적어도 5, 10, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30개 이상의 뉴클레오티드를 포함할 수 있다. 일부 구현예에서, 예를 들어 CRISPR/Cas 시스템이 Cas9 시스템인 구현예에서, 표적 핵산 서열은 PAM 서열의 역 상보체의 첫 번째 뉴클레오티드의 5' 바로 옆에 있는 20개의 뉴클레오티드를 포함할 수 있다. 이러한 표적 핵산 서열은 PAM 가닥 또는 표적 가닥으로 종종 지칭되며, 상보적 핵산 서열은 종종 비-PAM 가닥 또는 비-표적 가닥으로 지칭된다. 당업자는 표적화 서열이 표적 핵산의 비-PAM 가닥에 혼성화하는 것을 인식할 것이다, 예를 들어, US20190185849A1 참조.
일부 예에서, 표적화 서열과 표적 핵산 사이의 퍼센트 상보성은 적어도 약 30%, 적어도 약 40%, 적어도 약 50%, 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 97%, 적어도 약 98%, 적어도 약 99%, 또는 100%이다. 일부 예에서, 표적화 서열과 표적 핵산 사이의 퍼센트 상보성은 최대 약 30%, 최대 약 40%, 최대 약 50%, 최대 약 60%, 최대 약 65%, 최대 약 70%, 최대 약 75%, 최대 약 80%, 최대 약 85%, 최대 약 90%, 최대 약 95%, 최대 약 97%, 최대 약 98%, 최대 약 99%, 또는 100%이다. 일부 예에서, 표적화 서열과 표적 핵산 사이의 퍼센트 상보성은 표적 핵산의 상보성 가닥의 표적 서열의 6개의 인접한 5' 대부분 뉴클레오티드에 걸쳐 100%이다. 표적화 서열과 표적 핵산 사이 퍼센트 상보성은 약 20개의 연속 뉴클레오티드에 걸쳐 적어도 60%일 수 있다. 표적화 서열 및 표적 핵산의 길이는 1 내지 6개의 뉴클레오티드만큼 다를 수 있으며, 이는 벌지 또는 벌지들로 생각할 수 있다.
표적화 서열은 당업자에게 공지된 컴퓨터 프로그램을 사용하여 설계되거나 선택될 수 있다. 컴퓨터 프로그램은 예측된 용융 온도, 2차 구조 형성, 예측된 어닐링 온도, 서열 동일성, 게놈 맥락, 염색질 접근성, % GC, 게놈 발생 빈도 (예를 들어, 불일치, 삽입 또는 결실의 결과로 하나 이상의 지점에서 동일하거나 유사하지만 다양한 서열의), 메틸화 상태, SNP의 존재 등과 같은 변수를 사용할 수 있다. 사용 가능한 컴퓨터 프로그램은 NCBI 유전자 ID, 공식 유전자 기호, Ensembl 유전자 ID, 게놈 좌표, 또는 DNA 서열을 입력으로 사용할 수 있으며, 입력으로 지정된 적절한 게놈 영역을 표적으로 하는 sgRNA를 포함하는 출력 파일을 생성할 수 있다. 컴퓨터 프로그램은 또한 표적 유전자에 대한 sgRNA의 온- 및 오프-타겟 결합을 나타내는 통계 및 스코어의 요약을 제공할 수 있다 (Doench et al. Nat Biotechnol. 34:184-191 (2016)). 본 개시내용은 표적화 서열을 포함하는 가이드 RNA를 제공한다. 일부 구현예에서, 가이드 RNA는 가이드 RNA 스캐폴드 서열을 추가로 포함한다. 일부 구현예에서, 표적화 서열은 HLA-A, HLA-B, HLA-C, B2M 또는 이의 대립유전자로 이루어진 그룹으로부터 선택된 표적 유전자의 서열에 상보적이다. 일부 구현예에서, 표적 유전자는 HLA-A 유전자이다. 일부 구현예에서, 표적 유전자는 HLA-B 유전자이다. 일부 구현예에서, 표적 유전자는 HLA-C 유전자이다. 일부 구현예에서 표적 유전자는 HLA-A, HLA-B, HLA-C, 또는 이의 조합이다. 일부 구현예에서, 표적화 서열은 표 7에 개시된 서열과 약 90%, 약 95%, 약 96%, 약 97%, 약 98%, 약 99% 동일성을 공유하거나 동일한 서열을 포함한다.
일부 구현예에서, gNA는 내인성 HLA-A 유전자좌의 서열을 특이적으로 표적화한다. 일부 구현예에서, HLA-A 유전자좌의 서열을 특이적으로 표적화하는 gNA는 표 8에 개시된 서열로부터 선택된 서열과 약 90%, 약 95%, 약 96%, 약 97%, 약 98%, 또는 약 99% 동일성을 공유하는 서열을 포함한다. 일부 구현예에서, HLA-A 유전자좌의 서열을 특이적으로 표적화하는 gNA는 표 8에 개시된 서열로부터 선택된 서열을 포함한다.
일부 구현예에서, gNA는 HLA-A*02 대립유전자의 서열을 특이적으로 표적화한다. 예를 들어, gRNA는 모든 HLA-A*02 대립유전자에 의해 공유되지만, HLA-A*02 및 HLA-A*03 대립유전자에 의해 공유되지 않는 서열을 특이적으로 표적화하고, 혼성화한다. 일부 구현예에서, gNA는 HLA-A*02:01 대립유전자의 서열을 특이적으로 표적화한다. 일부 구현예에서, gNA는 HLA-A*02:01:01 대립유전자의 서열을 특이적으로 표적화한다. 일부 구현예에서, gNA는 HLA-A*02:01:01:01 대립유전자의 서열을 특이적으로 표적화한다. 일부 구현예에서, gNA는 HLA-A*02:01:01:01 대립유전자의 서열을 특이적으로 표적화한다.
일부 구현예에서, gNA는 HLA-A*02의 코딩 DNA 서열을 특이적으로 표적화한다.
일부 구현예에서, gNA는 1000 초과 HLA-A*02 대립유전자에 의해 공유되는 코딩 DNA 서열을 특이적으로 표적화한다. 일부 구현예에서, 1000개 초과 HLA-A*02 대립유전자에서 코딩 DNA 서열을 특이적으로 표적화하는 gNA는 서열 번호 400-465로부터 선택된 서열과 약 90%, 약 95%, 약 96%, 약 97%, 약 98%, 약 99% 동일성을 공유하거나 동일한 서열을 포함한다.
표 8. HLA-A 및 HLA-A 대립유전자를 표적으로 하는 예시적인 서열
표 8에 개시된 서열은 PAM 서열을 포함하는 상응하는 게놈 서열을 포함한다. 당업자는 gRNA의 표적화 서열이 gRNA에 상응하는 PAM 부위를 나타내는, 표 8의 서열의 3개의 3' 말단 뉴클레오티드를 포함하지 않는다는 것을 이해할 것이다.
본 개시내용은 B2M 유전자에 특이적인 표적화 서열을 포함하는 gNA를 제공한다. 일부 구현예에서, gNA는 B2M 유전자의 코딩 서열 (CDS) 서열을 특이적으로 표적화한다. 일부 구현예에서, gNA는 B2M 유전자 프로모터 서열을 표적화하는 서열을 포함한다.
일부 구현예에서 gNA는 표적화 서열 및 gNA 스캐폴드 서열을 포함한다. 일부 구현예에서, 표적화 서열은 표 9에 제시된 서열, 또는 이에 대해 약 90%, 약 95%, 약 96%, 약 97%, 약 98%, 약 99% 동일성을 공유하는 서열을 포함하는 서열을 포함한다.
일부 구현예에서, 표적화 서열은 B2M 유전자의 서열에 대해 상보적이다. 일부 구현예에서, B2M 유전자는 표 7에 제시된 B2M 서열과 약 90%, 약 95%, 약 96%, 약 97%, 약 98%, 약 99% 동일성을 공유하는 서열을 포함한다.
표 9. B2M을 표적화하는 예시적인 서열
일부 구현예에서, 본원에 기재된 면역 세포는 TALEN 유전자 편집을 사용하여 편집된다.
"TALEN" 또는 "TALEN 유전자 편집"은 표적 유전자를 편집하는 데 사용되는 인공 뉴클레아제인, 전사 활성화제-유사 효과기 뉴클레아제를 지칭한다.
TALEN은 TAL 효과기 DNA 결합 도메인을 DNA 절단 도메인에 융합하여 인공적으로 생성된다. 크산토모나스 박테리아에서 파생된 전사 활성화제-유사 효과기 (TALE)는 TCR 서브유닛, MHC 클래스 I 복합체 구성성분, 또는 CD52와 같은 표적 유전자의 일부를 포함하여, 원하는 DNA 서열에 결합하도록 조작될 수 있다. 조작된 TALE을 DNA 절단 도메인과 조합함으로써, 표적 유전자 서열을 포함하는 임의의 원하는 DNA 서열에 특이적인 제한 효소를 생산할 수 있다. 그런 다음 이들은 세포에 도입되어 게놈 편집에 사용될 수 있다.
TALEN을 생산하기 위해, TALE 단백질은 야생형 또는 돌연변이 Fold 엔도뉴클레아제인, 뉴클레아제 (N)에 융합된다. FokI에 대한 여러 돌연변이가 TALEN에서 사용하기 위해 만들어졌고; 예를 들어, 이들은 절단 특이성 또는 활성을 개선한다.
FokI 도메인은 적절한 방향과 간격을 가진 표적 게놈의 부위에 대해 고유한 DNA 결합 도메인이 있는 두 개의 작제물을 필요로 하는 이량체로 기능한다. TALE DNA 결합 도메인과 FokI 절단 도메인 사이의 아미노산 잔기의 수 및 2개의 개별 TALEN 결합 부위 사이의 염기의 수 모두 높은 수준의 활성을 달성하기 위한 중요한 매개변수인 것으로 보인다.
표적 유전자의 서열에 특이적인 TALEN은 모듈 구성 요소를 사용하는 다양한 계획을 포함하여 당업계에 알려진 임의의 방법을 사용하여 구성할 수 있다.
일부 구현예에서, 표적 유전자는 ZFN 유전자 편집을 사용하여 본원에 기재된 면역 세포에서 편집된다.
"ZFN" 또는 "징크 핑거 뉴클레아제" 또는 "ZFN 유전자 편집"은 표적 유전자를 편집하는 데 사용될 수 있는 인공 뉴클레아제인, 징크 핑거 뉴클레아제를 지칭한다.
TALEN과 마찬가지로, ZFN은 DNA-결합 도메인에 융합된 Fold 뉴클레아제 도메인 (또는 이의 유도체)을 포함한다. ZFN의 경우에, DNA-결합 도메인은 하나 이상의 징크 핑거를 포함한다.
징크 핑거는 하나 이상의 아연 이온에 의해 안정화된 작은 단백질 구조 모티프이다. 징크 핑거는 예를 들어, Cys2His2를 포함할 수 있고, 대략 3-bp 서열을 인식할 수 있다. 특이성이 알려진 다양한 징크 핑거를 조합하여 약 6, 9, 12, 15 또는 18-bp 서열을 인식하는 멀티핑거 폴리펩티드를 생산할 수 있다. 파지 디스플레이, 효모 1-하이브리드 시스템, 박테리아 1-하이브리드 및 2-하이브리드 시스템, 포유동물 세포를 포함하는 특정 서열을 인식하는 징크 핑거 (및 이들의 조합)를 생성하기 위해 다양한 선택 및 모듈 조립 기술을 사용할 수 있다.
TALEN과 마찬가지로, ZFN은 DNA를 절단하기 위해 이량체화되어야 한다. 따라서, 비-회문 DNA 부위를 표적으로 삼기 위해서는 한 쌍의 ZFN이 필요하다. 2개의 개별 ZFN은 적절한 간격으로 떨어져 있는 뉴클레아제를 사용하여 DNA의 반대쪽 가닥에 결합해야 한다.
또한 TALEN과 마찬가지로, ZFN은 DNA에 이중-가닥 절단을 생성할 수 있고, 이는 부적절하게 복구될 경우 프레임 이동 돌연변이를 생성하여, 세포의 표적 유전자 또는 유전자 산물의 발현 및 양을 감소시킨다. ZFN은 또한, 상동 재조합과 함께 사용되어 표적 유전자에서 돌연변이를 일으킬 수 있다.
표적 유전자의 서열에 특이적인 ZFN은 당업계에 공지된 임의의 방법을 사용하여 구축할 수 있다.
일부 구현예에서, 하나 이상의 MCH-I 구성성분의 발현 및 기능은 RNA 간섭을 사용하여 감소된다. "RNAi" 또는 "RNA 간섭"은 이중-가닥 RNA (dsRNA)에 의해 매개되는 서열-특이적 전사 후 유전자 침묵 과정을 지칭한다. siRNA (소 간섭 RNA), miRNA (마이크로 RNA), shRNA (짧은 헤어핀 RNA), ddRNA (DNA-지정 RNA), piRNA (Piwi-상호작용 RNA), 또는 rasiRNA (반복 관련 siRNA) 및 이들의 변형된 형태와 같은 듀플렉스 RNA는 모두 RNA 간섭을 매개할 수 있다. 이러한 dsRNA 분자는 상업적으로 이용 가능하거나 알려진 서열 정보를 기반으로 설계 및 제조될 수 있다. 이들 분자의 안티-센스 가닥은 RNA, DNA, PNA, 또는 이의 조합을 포함할 수 있다. DNA/RNA 키메라 폴리뉴클레오티드는 표적 유전자의 발현을 억제하는 DNA 및 RNA로 구성된 이중-가닥 폴리뉴클레오티드를 포함하지만, 이에 제한되지 않는다. dsRNA 분자는 또한 본원에 기재된 바와 같이, 하나 이상의 변형된 뉴클레오티드를 포함할 수 있으며, 이는 가닥 중 하나 또는 둘 다에 통합될 수 있다.
RNAi 유전자 사일런싱 또는 녹다운에서, 표적 유전자의 일부에 상보적인 제1 (안티-센스) 가닥 및 제1 안티-센스 가닥에 완전히 또는 부분적으로 상보적인 제2 (센스) 가닥을 포함하는 dsRNA는 유기체 내로 도입된다. 유기체에 도입된 후, 표적 유전자 특이적 dsRNA는 상대적으로 작은 단편 (siRNA)으로 처리되고 이후 유기체 전체에 분포될 수 있으며, 표적 유전자의 메신저 RNA를 감소시켜, 표적 유전자의 완전한 또는 부분적인 결실로부터 발생하는 표현형과 매우 유사하게 될 수 있는 표현형을 유도한다.
세포의 특정 dsRNA는 Dicer 효소, 리보뉴클레아제 III 효소의 작용을 받을 수 있다. Dicer는 dsRNA를 dsRNA의 더 짧은 조각, 즉 siRNA로 처리할 수 있다. RNAi는 또한 RNA 유도 침묵 복합체 (RISC)로 알려진 엔도뉴클레아제 복합체를 포함한다. Dicer에 의한 절단 후, siRNA는 RISC 복합체에 들어가고 siRNA 듀플렉스의 안티센스 가닥에 상보적인 서열을 갖는 단일 가닥 RNA 표적의 절단을 지시한다. siRNA의 다른 가닥은 패신저 가닥이다. 표적 RNA의 절단은 siRNA 듀플렉스의 안티센스 가닥에 상보적인 영역의 중간에서 일어난다. 따라서 siRNA는 서열-특이적 방식으로 RNA 간섭을 매개함으로써 유전자 발현을 하향 조절하거나 무너뜨릴 수 있다.
RNA 간섭과 관련하여 사용되는 바와 같이, "표적 유전자" 또는 "표적 서열"은 상응하는 RNA가 본원에 기재된 바와 같은 dsRNA 또는 siRNA를 사용하여 RNAi 경로를 통한 분해를 위해 표적화되는 유전자 또는 유전자 서열을 지칭한다. 예시적인 표적 유전자 서열은 표 7에 나타낸다. 예를 들어 siRNA를 사용하여 유전자를 표적화하기 위해, siRNA는 표적 유전자 또는 서열의 적어도 일부에 상보적이거나 실질적으로 상보적인 안티센스 영역, 및 안티-센스 가닥에 상보적인 센스 가닥을 포함한다. 일단 세포에 도입되면, siRNA는 표적 서열을 포함하는 RNA를 절단하도록 RISC 복합체를 지시하여 RNA를 분해한다. 본 개시내용은 간섭 RNA를 제공한다. 본 개시내용의 이중 가닥 RNA 분자는 당업계에 공지된 임의의 유형의 RNA 간섭 분자의 형태일 수 있다. 일부 구현예에서, 이중 가닥 RNA 분자는 소 간섭 RNA (siRNA)이다. 다른 구현예에서, 이중 가닥 RNA 분자는 짧은 헤어핀 RNA (shRNA) 분자이다. 다른 구현예에서, 이중 가닥 RNA 분자는 siRNA를 생성하기 위해 세포에서 처리되는 Dicer 기질이다. 다른 구현예에서 이중 가닥 RNA 분자는 마이크로RNA 전구체 분자의 부분이다.
일부 구현예에서, shRNA는 RISC 활성 siRNA 분자를 생성하기 위해 처리될 수 있는 다이서 기질로서 적합한 길이이다. 예를 들어, Rossi et al., US2005/0244858 참조.
Dicer 기질 이중 가닥 RNA (예를 들어 shRNA)는 활성 siRNA를 생산하기 위해 Dicer에 의해 처리되기에 충분한 길이일 수 있으며 다음 특성 중 하나 이상을 추가로 포함할 수 있다: (i) Dicer 기질 shRNA는 예를 들어, 안티-센스 가닥에 3' 오버행을 갖는 비대칭, (ii) Dicer 기질 shRNA는 센스 가닥에서 변형된 3' 말단을 가질 수 있어 활성 siRNA에 대한 Dicer 결합 및 dsRNA의 방향을 지시할 수 있다, 예를 들어 하나 이상의 DNA 뉴클레오티드의 통합, 및 (iii) Dicer 기질 ds RNA의 제1 및 제2 가닥은 21-30 bp 길이일 수 있다.
일부 구현예에서, 간섭 RNA는 B2M mRNA의 서열에 상보적인 서열을 포함한다. 일부 구현예에서, 간섭 RNA는 B2M mRNA의 RNAi-매개 분해를 유도할 수 있다. 일부 구현예에서, B2M mRNA 서열은 코딩 서열을 포함한다. 일부 구현예에서, B2M mRNA 서열은 비번역 영역을 포함한다.
일부 구현예에서, 간섭 RNA는 HLA-A*02 mRNA의 서열에 상보적인 서열을 포함한다. 일부 구현예에서, 간섭 RNA는 HLA-A*02 mRNA의 RNAi-매개 분해를 유도할 수 있다. 일부 구현예에서, HLA-A*02 mRNA 서열은 코딩 서열을 포함한다. 일부 구현예에서, HLA-A*02 mRNA 서열은 비번역 영역을 포함한다.
일부 구현예에서, 간섭 RNA는 짧은 헤어핀 RNA (shRNA)이다. 일부 구현예에서, shRNA는 5'에서 3' 말단으로 B2M mRNA에 상보적인 서열을 갖는 제1 서열; 및, 5'에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열을 포함하고, 여기서 제1 서열 및 제2 서열은 shRNA를 형성한다.
일부 구현예에서, 제1 서열은 18, 19, 20, 21, 또는 22개 뉴클레오티드이다. 일부 구현예에서, 제1 서열은 표 10 및 11에 제시된 서열로부터 선택된 서열에 상보적이다. 일부 구현예에서, 제1 서열은 25% 이상 60% 미만의 GC 함량을 갖는다. 일부 구현예에서, 제1 서열은 표 10 및 11에 제시된 서열로부터 선택된 서열에 상보적이다. 일부 구현예에서, 제1 서열은 동일한 염기의 4개의 뉴클레오티드 또는 7개의 C 또는 G 뉴클레오티드 염기의 런을 포함하지 않는다. 일부 구현예에서, 제1 서열은 21개 뉴클레오티드이다.
제1 서열에 상보적인 예시적인 표적 B2M 서열은 표 10에 나타낸다.
일부 경우에, 제1 서열은 표적 핵산 서열에 대해 100% 동일성, 즉 완전 동일성, 상동성, 상보성을 가질 수 있다. 다른 경우에, 제1 서열과 표적 핵산 서열 사이에 하나 이상의 불일치가 있을 수 있다. 예를 들어, 센스 영역과 표적 핵산 서열 사이에 1, 2, 3, 4, 5, 6, 또는 7개의 불일치가 있을 수 있다.
표 10에 제시된 서열은 DNA 서열로 제시된다. 표 10에 제시된 모든 서열에서, 티민 (T)은 우라실 (U)로 대체되어 표적 mRNA 서열의 서열에 도달할 수 있다.
표 10. 제1 서열에 상보적인 예시적인 표적 B2M 서열.
일부 구현예에서, 간섭 RNA는 HLA-A*02 mRNA의 서열에 상보적인 서열을 포함한다. 일부 구현예에서, 간섭 RNA는 HLA-A*02 mRNA의 RNAi-매개 분해를 유도할 수 있다. 일부 구현예에서, HLA-A*02 mRNA 서열은 코딩 서열을 포함한다. 일부 구현예에서, HLA-A*02 mRNA 서열은 비번역 영역을 포함한다.
B2M shRNA를 암호화하는 예시적인 서열은 GCACTCAAAGCTTGTTAAGATCGAAATCTTAACAAGCTTTGAGTGC (서열 번호 179)의 서열, 또는 이에 대해 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함한다. B2M shRNA를 암호화하는 추가 예시적인 서열은 GTTAACTTCCAATTTACATACCGAAGTATGTAAATTGGAAGTTAAC (서열 번호 808)의 서열, 또는 이에 대해 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함한다.
일부 구현예에서, 간섭 RNA는 짧은 헤어핀 RNA (shRNA)이다. 일부 구현예에서, shRNA는 5'에서 3' 말단으로 HLA-A*02 mRNA에 상보적인 서열을 갖는 제1 서열; 및 5'에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열, 여기서 제1 서열 및 제2 서열은 shRNA를 형성한다.
제1 서열에 상보적인 예시적인 표적 HLA 서열은 표 11에 나타낸다.
표 11. 제1 서열에 상보적인 예시적인 표적 HLA 서열
일부 구현예에서, 제1 서열 및 제2 서열은 때때로 루프로 지칭되는 링커에 의해 분리된다. 일부 구현예에서, 제1 서열 및 제2 서열 모두 하나의 단일-가닥 RNA 또는 DNA 벡터에 의해 암호화된다. 일부 구현예에서, 루프는 제1 서열과 제2 서열 사이에 있다. 이들 구현예에서, 제1 서열 및 제2 서열은 혼성화하여 듀플렉스 영역을 형성한다. 제1 서열 및 제2 서열은 링커 서열에 의해 결합되어, "헤어핀" 또는 "스템-루프" 구조를 형성한다. shRNA는 단일 가닥 분자의 반대쪽 끝에 상보적인 첫 번째 서열과 두 번째 서열을 가질 수 있으므로, 분자는 상보적인 서열 부분과 듀플렉스 영역을 형성할 수 있고, 가닥은 링커에 의해 듀플렉스 영역의 한쪽 끝에서 연결된다 (즉 루프 서열). 링커, 또는 루프 서열은 뉴클레오티드 또는 비-뉴클레오티드 링커일 수 있다. 링커는 공유 결합 또는 비공유 상호작용을 통해 제1 서열, 선택적으로 제2 서열과 상호작용할 수 있다.
임의의 적합한 뉴클레오티드 루프 서열은 본 개시내용의 범위 내에 있는 것으로 생각된다. 본 개시내용의 shRNA는 shRNA의 제1 서열을 shRNA의 제2 서열에 결합시키는 뉴클레오티드, 비-뉴클레오티드, 또는 혼합된 뉴클레오티드/비-뉴클레오티드 링커를 포함할 수 있다. 뉴클레오티드 루프 서열은 2개 이상의 뉴클레오티드 길이, 예를 들어 약 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 또는 15개 뉴클레오티드 길이일 수 있다. 예시적인 루프 서열은 표 12에 개시되어 있다.
일부 구현예에서, shRNA는 5' 플랭크 서열 및 3' 플랭크 서열을 추가로 포함한다. 일부 구현예에서, 5' 플랭크 서열은 제1 서열의 5' 말단에 연결되고, 3' 플랭크 서열은 제2 서열의 3' 말단에 연결된다.
이론에 얽매이지 않고, 마이크로RNA에서 발견되는 것과 유사한 5' 및 3' 서열을 갖는 플랭킹 shRNA 스템 루프 서열은 shRNA 처리의 효율성을 증가시키면서 내인성 마이크로RNA 처리 기계에 의한 처리를 위해 shRNA를 표적으로 할 수 있다고 생각된다. 대안적으로, 또는 추가로, 플랭킹 서열은 중합효소 II 또는 중합효소 III 프로모터와의 shRNA 호환성을 증가시켜, shRNA 발현의 더욱 효과적인 조절을 유도할 수 있다.
일부 구현예에서, 5' 플랭크 서열은 표 12에 제시된 서열로부터 선택된다. 예시적인 플랭크 서열은 표 12에 나타낸다.
표 12. 예시적인 플랭크 서열
일부 구현예에서, 제1 및 제2 서열은 단일 가닥 폴리뉴클레오티드 상에 존재하고, 여기서 제1 서열 및 제2 서열은 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 또는 15개 뉴클레오티드에 의해 분리되고, 여기서 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 또는 15개 뉴클레오티드는 shRNA에서 루프 영역을 형성한다. 일부 구현예에서, 루프 영역은 표 13에 제시된 서열로부터 선택된 서열을 포함한다.
표 13. 예시적인 루프 영역 서열
본 개시내용의 shRNA는 화학적 합성에 의해, 시험관 내 전사에 의해, 또는 Dicer 또는 유사한 활성을 갖는 또 다른 적절한 뉴클레아제를 사용한 더 긴 이중 가닥 RNA의 절단에 의해 외인성으로 생성될 수 있다. 통상적인 DNA/RNA 합성기를 사용하여 보호된 리보뉴클레오시드 포스포르아미다이트로부터 생성된, 화학적으로 합성된 siRNA는 Millipore Sigma (Houston, Tex.), Ambion Inc. (Austin, Tex.). Invitrogen (Carlsbad, Calif.), 또는 Dharmacon (Lafayette, Colo.)과 같은 상업적 공급자로부터 얻을 수 있다. siRNA는 예를 들어 용매 또는 수지를 사용한 추출, 침전, 전기영동, 크로마토그래피 또는 이들의 조합에 의해 정제될 수 있다. 대안적으로, siRNA는 시료 처리로 인한 소실을 피하기 위해 정제가 거의 없는 경우에 사용할 수 있다.
일부 구현예에서, 본 개시내용의 shRNA는 이중 가닥 RNA를 암호화하는 핵산이 클로닝된 발현 벡터를 사용하여, 예를 들어 적합한 프로모터의 제어 하에 생성될 수 있다.
일부 구현예에서, 면역 세포는 포함한다
약학 조성물
본 개시내용은 본 개시내용의 제1 및 제2 수용체 및 약학적으로 허용 가능한 희석제, 담체 또는 부형제를 포함하는 면역 세포를 포함하는 약학 조성물을 제공한다.
이러한 조성물은 중성 완충 식염수, 인산염 완충 식염수 등과 같은 완충제; 글루코스, 만노스, 수크로즈 또는 덱스트란, 만니톨과 같은 탄수화물; 단백질; 폴리펩티드 또는 글리신과 같은 아미노산; 항산화제; EDTA 또는 글루타티온과 같은 킬레이트제; 및 방부제를 포함할 수 있다.
일부 구현예에서, 면역 세포는 제1 수용체 및 제2 수용체 모두를 발현한다. 일부 구현예에서, 적어도 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 또는 약 95%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현한다. 일부 구현예에서, 적어도 90%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현한다.
암 치료
본 개시내용의 제1 및 제2 수용체를 포함하는 면역 세포를 포함하는 치료적 유효량의 조성물을 대상체에 투여하는 것을 포함하는, 대상체에서 다수의 암 세포를 사멸시키거나 암을 치료하는 방법이 본원에 제공된다. 면역 세포는 동일한 세포에서 두 수용체를 모두 발현한다.
암은 비정상 세포가 통제 없이 분열하여 주변 조직으로 전이되는 질환이다. 일부 구현예에서, 암은 액상 종양 또는 고형 종양을 포함한다. 암은 상피 조직을 포함하여 사실상 신체의 한 기관에서 발생할 수 있다. 다수의 암 세포가 제1 활성제 리간드를 발현하고 제2 억제제 리간드를 발현하지 않는 임의의 암은 본 개시내용의 범위 내에 있는 것으로 간주된다. 예를 들어, 본원에 기재된 방법을 사용하여 치료될 수 있는 EGFR 양성 암은 폐암, 소세포 폐암, 비-소세포 폐암, 췌장관 암종, 결장직장암, 두경부암, 식도 및 위 선암종, 난소암, 다형성 교모세포종, 자궁경부 편평 세포 암종, 신장암, 유두상 신장암, 신장 투명 세포 암종, 방광암, 유방암, 담관암, 간암, 전립선암, 육종, 갑상선암, 흉선암, 위암, 또는 자궁암, 및 기타 모든 EGFR 표적 발현 종양을 포함한다. 본원에 개시된 조성물 및 방법은 재발성, 불응성 및/또는 전이성인 EGFR 양성 암을 치료하는 데 사용될 수 있다.
일부 구현예에서, 다수의 암 세포는 표적 항원을 발현한다. 일부 구현예에서, 대상체의 다수의 암 세포는 EGFR을 발현한다. EGFR 양성 암은 폐암, 소세포 폐암, 비-소세포 폐암, 췌장관 암종, 결장직장암, 두경부암, 식도 및 위 선암종, 난소암, 다형성 교모세포종, 자궁경부 편평 세포 암종, 신장암, 유두상 신장암, 신장 투명 세포 암종, 방광암, 유방암, 담관암, 간암, 전립선암, 육종, 갑상선암, 흉선암, 위암, 또는 자궁암을 포함하지만, 이에 제한되지 않는다.
일부 구현예에서, 다수의 암 세포는 COLEC12, APCDD1 또는 CXCL16의 다형성 대립유전자를 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 비-표적 항원을 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-A*02를 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-A*02의 대립형질 변이체를 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-A*01을 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-A*03을 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-A*11을 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-B*07을 발현하지 않는다. 일부 구현예에서, 다수의 암 세포는 HLA-C*07을 발현하지 않는다. 예를 들어, 암 세포는 해당 유전자좌에서 이형 접합체의 소실을 통해 COLEC12, APCDD1 또는 CXCL16, 또는 HLA-A*02의 대립 유전자를 소실했다.
본원에 기재된 면역 세포 또는 약학 조성물의 투여는 대상체에서 종양의 크기를 감소시킬 수 있다. 일부 구현예에서, 종양의 크기는 면역 세포 또는 약학 조성물의 투여 전 종양의 크기와 비교하여 약 5%, 약 10%, 약 15%, 약 20%, 약 25%, 약 30%, 약 35%, 약 40%, 약 45%, 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 약 95%, 또는 약 100% 감소한다. 일부 구현예에서, 종양은 제거된다.
본원에 기재된 면역 세포 또는 약학 조성물의 투여는 대상체에서 종양의 성장을 정지시킬 수 있다. 예를 들어, 면역 세포 또는 약학 조성물은 종양 세포를 죽일 수 있어 종양이 성장을 멈추거나 크기가 감소한다. 일부 경우에, 면역 세포 또는 약학 조성물은 추가 종양의 형성을 방지하거나 대상체에서 종양의 총 수를 감소시킬 수 있다.
본원에 기재된 면역 세포 또는 약학 조성물의 투여는 대상체에서 정상 세포가 아닌 암 세포의 선택적인 사멸을 초래할 수 있다. 일부 구현예에서, 사멸된 세포의 약 60%는 암세포이고, 사멸된 세포의 약 65%는 암세포이고, 사멸된 세포의 약 70%는 암세포이고, 사멸된 세포의 약 75%는 암세포이고, 사멸된 세포의 약 80%는 암세포이고, 사멸된 세포의 약 85%는 암세포이고, 사멸된 세포의 약 90%는 암세포이고, 사멸된 세포의 약 95%는 암세포이고, 또는 사멸된 세포의 약 100%는 암세포이다.
본원에 기재된 면역 세포 또는 약학 조성물의 투여는 대상체의 암 세포의 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90% 또는 전부의 사멸을 초래할 수 있다.
본원에 기재된 면역 세포 또는 약학 조성물의 투여는 제1 활성화제 수용체를 포함하지만 제2 억제 수용체는 포함하지 않는 등가의 면역 세포를 투여하는 것보다 대상체에 대해 더 적은 부작용을 초래할 수 있다. 예를 들어, 본원에 기재된 면역 세포 또는 약학 조성물의 투여는 제2 억제 수용체 없이 투여된 EGFR CAR, 또는 EGFR TCR에 비해 용량 제한 독성을 감소시킬 수 있다.
본 개시내용은 다음을 포함하는 대상체에서 암을 치료하는 방법을 제공한다: (a) COLEC12의 다형성 유전자좌, APCDD1의 다형성 유전자좌 및 CXCL16의 다형성 유전자좌, 또는 HLA-A*02 유전자좌로 이루어진 그룹으로부터 선택된 다형성 유전자좌에서 대상체의 정상 세포 및 다수의 암 세포의 유전형을 결정하는 단계; (b) 다수의 암 세포에서 EGFR의 발현을 결정하는 단계; 및 (c) 정상 세포가 다형성 유전자좌에 대해 이형접합성이고 다수의 암 세포가 다형성 유전자좌에 대해 반접합성이거나 HLA-A*02를 소실하고, 다수의 암 세포가 EGFR 양성인 경우 다수의 면역 세포를 대상체에 투여하는 단계, 여기서 다수의 면역 세포는 다음을 포함하고: (i) 제1 수용체, 임의로 키메라 항원 수용체 (CAR) 또는 T 세포 수용체 (TCR), EGFR에 특이적인 세포외 리간드 결합 도메인을 포함하는, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 펩티드 항원; 및 (ii) 제2 수용체, 임의로 억제 키메라 항원 수용체 (즉 억제 수용체), COLEC12, APCDD1, 및 CXCL16으로부터 선택된 비-표적 항원에 특이적인 결합을 포함하는 세포외 리간드, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 항원 펩티드, 또는 HLA-A*02, 여기서 비-표적 항원은 다형성을 포함한다.
SNP의 존재 또는 부재에 대해 대상체로부터 암 세포 및 정상 세포를 유전자형 분석하는 방법은 당업자에게 쉽게 자명할 것이다. SNP 유전자형 분석 방법은 특히, 이중 프로브 TaqMan 분석, 어레이 기반 혼성화 방법 및 시퀀싱과 같은 PCR 기반 방법을 포함한다.
암 또는 정상 세포에서 표적 항원의 발현을 측정하는 방법은 당업자에게 쉽게 자명할 것이다. 여기에는 특히 RNA 시퀀싱 및 역전사 중합효소 연쇄 반응 (RT-PCR)과 같은 RNA 발현을 측정하는 방법뿐만 아니라 면역조직화학 기반 방법과 같은 단백질 발현을 측정하는 방법이 포함된다. 다수의 암 세포에서 이형접합성의 소실을 측정하는 방법은 특히 당업계에 공지된 방법을 사용하여 암 세포로부터 추출된 게놈 DNA의 고처리량 시퀀싱을 포함한다.
대상체로부터의 암 또는 정상 세포에서 표적 항원의 발현을 측정하는 방법은 당업자에게 쉽게 자명할 것이다. 여기에는 특히 RNA 시퀀싱 및 역전사 중합효소 연쇄 반응 (RT-PCR)과 같은 RNA 발현을 측정하는 방법뿐만 아니라 면역조직화학 기반 방법과 같은 단백질 발현을 측정하는 방법이 포함된다.
일부 구현예에서, 면역 세포는 T 세포이다.
일부 구현예에서, 면역 세포는 동종 또는 자가이다.
일부 구현예에서, 제2 수용체는 제1 수용체는 발현하지만 제2 수용체는 발현하지 않는 면역 세포와 비교하여 EGFR 양성 암 세포에 대한 면역 세포의 특이성을 증가시킨다. 일부 구현예에서, 면역 세포는 제1 수용체는 발현하지만 제2 수용체는 발현하지 않는 면역 세포와 비교하여 감소된 부작용을 갖는다.
암을 치료하면 종양의 크기가 줄어들 수 있다. 종양 크기의 감소는 "종양 퇴행"이라고도 한다. 바람직하게는, 치료 후, 종양 크기는 치료 전 크기에 비해 5% 이상 감소되고; 보다 바람직하게는, 종양 크기는 10% 이상 감소되고; 보다 바람직하게는, 20% 이상 감소되고; 보다 바람직하게는, 30% 이상 감소되고; 보다 바람직하게는, 40% 이상 감소되고; 더욱 바람직하게는, 50% 이상 감소되고; 가장 바람직하게는, 75% 또는 그 이상 감소된다. 종양의 크기는 재현 가능한 측정 수단으로 측정할 수 있다. 종양의 크기는 종양의 직경으로 측정할 수 있다.
암을 치료하면 종양 부피가 감소할 수 있다. 바람직하게는, 치료 후 종양 부피는 치료 전 크기에 비해 5% 이상 감소되고; 보다 바람직하게는, 종양 부피는 10% 이상 감소되고; 보다 바람직하게는, 20% 이상 감소되고; 보다 바람직하게는, 30% 이상 감소되고; 보다 바람직하게는, 40% 이상 감소되고; 더욱 바람직하게는, 50% 이상 감소되고; 가장 바람직하게는, 75% 또는 그 이상 감소된다. 종양 부피는 재현 가능한 측정 수단으로 측정할 수 있다.
암을 치료하면 종양의 수가 감소한다. 바람직하게는, 치료 후, 종양 수는 치료 전의 수에 비해 5% 이상 감소되고; 보다 바람직하게는, 종양 수는 10% 이상 감소되고; 보다 바람직하게는, 20% 이상 감소되고; 보다 바람직하게는, 30% 이상 감소되고; 보다 바람직하게는, 40% 이상 감소되고; 더욱 바람직하게는, 50% 이상 감소되고; 가장 바람직하게는, 75% 이상 감소된다. 종양의 수는 재현 가능한 측정 수단으로 측정할 수 있다. 종양의 수는 육안으로 또는 특정 배율로 볼 수 있는 종양을 세어 측정할 수 있다. 바람직하게는, 지정된 배율은 2x, 3x, 4x, 5x, 10x, 또는 50x이다.
암을 치료하면 원발성 종양 부위에서 멀리 떨어진 다른 조직이나 기관의 전이성 병변 수가 감소할 수 있다. 바람직하게는, 치료 후, 전이성 병변의 수는 치료 전 수에 비해 5% 이상 감소되고; 보다 바람직하게는, 전이성 병변의 수는 10% 이상 감소되고; 보다 바람직하게는, 20% 이상 감소되고; 보다 바람직하게는, 30% 이상 감소되고; 보다 바람직하게는, 40% 이상 감소되고; 더욱 바람직하게는, 50% 이상 감소되고; 가장 바람직하게는, 75% 이상 감소된다. 전이성 병변의 수는 재현 가능한 측정 수단으로 측정할 수 있다. 전이성 병변의 수는 육안으로 또는 특정 배율로 볼 수 있는 전이성 병변을 세어 측정할 수 있다. 바람직하게는, 지정된 배율은 2x, 3x, 4x, 5x, 10x, 또는 50x이다.
암을 치료하면 보인자만 받는 집단과 비교하여 치료 대상 집단의 평균 생존 시간이 증가할 수 있다. 바람직하게는, 평균 생존 시간은 30일 이상; 보다 바람직하게는, 60일 이상; 보다 바람직하게는, 90일 이상; 가장 바람직하게는, 120일 이상 증가한다. 집단의 평균 생존 시간의 증가는 재현 가능한 모든 수단으로 측정할 수 있다. 집단의 평균 생존 시간의 증가는 예를 들어, 집단에 대해 활성 화합물로 치료를 시작한 후 평균 생존 기간을 계산하여 측정할 수 있다. 집단의 평균 생존 시간의 증가는 또한 예를 들어, 집단에 대해 활성 화합물로 치료의 1차 라운드를 완료한 후 평균 생존 기간을 계산함으로써 측정될 수 있다.
암을 치료하면 치료받지 않은 대상 집단과 비교하여 치료 대상 집단의 평균 생존 시간이 증가할 수 있다. 바람직하게는, 평균 생존 시간은 30일 이상; 보다 바람직하게는, 60일 이상; 보다 바람직하게는, 90일 이상; 가장 바람직하게는, 120일 이상 증가한다. 집단의 평균 생존 시간의 증가는 재현 가능한 모든 수단으로 측정할 수 있다. 집단의 평균 생존 시간의 증가는 예를 들어, 집단에 대해 활성 화합물로 치료를 시작한 후 평균 생존 기간을 계산하여 측정할 수 있다. 집단의 평균 생존 시간의 증가는 또한 예를 들어, 집단에 대해 활성 화합물로 치료의 1차 라운드를 완료한 후 평균 생존 기간을 계산함으로써 측정될 수 있다.
암을 치료하면 본 개시내용의 화합물이 아닌 약물 또는 이의 약학적으로 허용 가능한 염, 전구약물, 대사산물, 유사체 또는 이의 유도체로 단일요법을 받는 집단과 비교하여 치료받는 대상 집단의 평균 생존 시간을 증가시킬 수 있다. 바람직하게는, 평균 생존 시간은 30일 이상; 보다 바람직하게는, 60일 이상; 보다 바람직하게는, 90일 이상; 가장 바람직하게는, 120일 이상 증가한다. 집단의 평균 생존 시간의 증가는 재현 가능한 모든 수단으로 측정할 수 있다. 집단의 평균 생존 시간의 증가는 예를 들어, 집단에 대해 활성 화합물로 치료를 시작한 후 평균 생존 기간을 계산하여 측정할 수 있다. 집단의 평균 생존 시간의 증가는 또한 예를 들어, 집단에 대해 활성 화합물로 치료의 1차 라운드를 완료한 후 평균 생존 기간을 계산함으로써 측정될 수 있다.
암을 치료하면 보인자만을 수용하는 집단과 비교하여 치료 대상 집단의 사망률 감소를 초래할 수 있다. 암을 치료하면 치료되지 않은 집단과 비교하여 치료 대상 집단의 사망률이 감소할 수 있다. 암을 치료하면 본 개시내용의 화합물이 아닌 약물 또는 이의 약학적으로 허용 가능한 염, 전구약물, 대사산물, 유사체 또는 이의 유도체로 단일요법을 받는 집단과 비교하여 치료 대상 집단의 사망률 감소를 초래할 수 있다. 바람직하게는, 사망률이 2% 이상; 보다 바람직하게는, 5% 이상; 보다 바람직하게는, 10% 이상; 가장 바람직하게는, 25% 이상 감소한다. 치료 대상 집단의 사망률 감소는 임의의 재현 가능한 수단으로 측정할 수 있다. 집단의 사망률 감소는 예를 들어, 집단에 대해 활성 화합물로 치료를 시작한 후 단위 시간당 평균 질병 관련 사망 수를 계산하여 측정할 수 있다. 집단의 사망률 감소는 또한 예를 들어, 집단에 대해 활성 화합물로 치료의 1차 라운드를 완료한 후 단위 시간당 평균 질병 관련 사망 수를 계산하여 측정할 수 있다.
암을 치료하면 종양 성장 속도가 감소할 수 있다. 바람직하게는, 치료 후, 종양 성장 속도가 치료 전의 수에 비해 적어도 5% 감소되고; 보다 바람직하게는, 종양 성장 속도가 적어도 10% 감소되고; 보다 바람직하게는, 적어도 20% 감소되고; 보다 바람직하게는, 적어도 30% 감소되고; 보다 바람직하게는, 적어도 40% 감소되고; 보다 바람직하게는, 적어도 50% 감소되고; 더욱 바람직하게는, 적어도 50% 감소되고; 가장 바람직하게는, 적어도 75% 감소된다. 종양 성장 속도는 재현 가능한 측정 수단으로 측정할 수 있다. 단위 시간당 종양 직경의 변화에 따라 종양 성장 속도를 측정할 수 있다.
암을 치료하면 종양 재성장이 감소할 수 있다. 바람직하게는, 치료 후, 종양 재성장은 5% 미만이고; 보다 바람직하게는, 종양 재성장은 10% 미만이고; 보다 바람직하게는, 20% 미만; 보다 바람직하게는, 30% 미만; 보다 바람직하게는, 바람직하게는 40% 미만; 보다 바람직하게는, 50% 미만; 더욱 바람직하게는, 50% 미만; 가장 바람직하게는, 75% 미만이다. 종양 재성장은 임의의 재현 가능한 측정 수단으로 측정할 수 있다. 종양 재성장은 예를 들어, 치료 후 선행 종양 수축 후 종양 직경의 증가를 측정함으로써 측정된다. 종양 재성장의 감소는 치료가 중단된 후 종양이 재발하지 않는 것으로 나타난다.
암을 치료하거나 예방하면 세포 증식 속도가 감소할 수 있다. 바람직하게는, 치료 후, 세포 증식률은 적어도 5%까지; 보다 바람직하게는, 적어도 10%까지; 보다 바람직하게는, 적어도 20%까지; 보다 바람직하게는, 적어도 30%까지; 보다 바람직하게는, 적어도 40%까지; 보다 바람직하게는, 적어도 50%까지; 더욱 바람직하게는, 적어도 50%까지; 가장 바람직하게는, 적어도 75%까지 감소된다. 세포 증식 속도는 재현 가능한 측정 수단으로 측정할 수 있다. 세포 증식 속도는 예를 들어, 단위 시간당 조직 샘플에서 분열하는 세포의 수를 측정함으로써 측정된다.
암을 치료하거나 예방하면 증식하는 세포의 비율이 감소할 수 있다. 바람직하게는, 치료 후, 증식하는 세포의 비율은 적어도 5%까지; 보다 바람직하게는, 적어도 10%까지; 보다 바람직하게는, 적어도 20%까지; 보다 바람직하게는, 적어도 30%까지; 보다 바람직하게는, 적어도 40%까지; 보다 바람직하게는, 적어도 50%까지; 더욱 바람직하게는, 적어도 50%까지; 가장 바람직하게는, 적어도 75%까지 감소된다. 증식 세포의 비율은 재현 가능한 측정 수단으로 측정할 수 있다. 바람직하게는, 증식 세포의 비율은 예를 들어, 조직 샘플에서 분열하지 않는 세포의 수에 대해 분열하는 세포의 수를 정량화함으로써 측정된다. 증식하는 세포의 비율은 유사분열 지수와 동일할 수 있다.
암을 치료하거나 예방하면 세포 증식 영역 또는 영역의 크기가 감소할 수 있다. 바람직하게는, 치료 후, 세포 증식 영역 또는 구역의 크기는 치료 전 크기에 비해 적어도 5% 감소되고; 보다 바람직하게는, 적어도 10% 감소되고; 보다 바람직하게는, 적어도 20% 감소되고; 보다 바람직하게는, 적어도 30% 감소되고; 보다 바람직하게는, 적어도 40% 감소되고; 보다 바람직하게는, 적어도 50% 감소되고; 더욱 바람직하게는, 적어도 50% 감소되고; 가장 바람직하게는, 적어도 75% 감소된다. 세포 증식 영역 또는 영역의 크기는 재현 가능한 측정 수단으로 측정할 수 있다. 세포 증식 영역 또는 영역의 크기는 세포 증식 영역 또는 영역의 직경 또는 폭으로 측정될 수 있다.
암을 치료하거나 예방하면 비정상적인 모양이나 형태를 가진 세포의 수나 비율이 감소할 수 있다. 바람직하게는, 치료 후, 비정상적인 형태를 갖는 세포의 수는 치료 전 크기에 비해 적어도 5% 감소되고; 보다 바람직하게는, 적어도 10% 감소되고; 보다 바람직하게는, 적어도 20% 감소되고; 보다 바람직하게는, 적어도 30% 감소되고; 보다 바람직하게는, 적어도 40% 감소되고; 보다 바람직하게는, 적어도 50% 감소되고; 더욱 바람직하게는, 적어도 50% 감소되고; 가장 바람직하게는, 적어도 75% 감소된다. 비정상적인 세포 외관 또는 형태는 재현 가능한 측정 수단으로 측정할 수 있다. 비정상적인 세포 형태는 현미경, 예를 들어 거꾸로 된 조직 배양 현미경을 사용하여 측정할 수 있다. 비정상적인 세포 형태는 핵 다형성의 형태를 취할 수 있다.
용량 및 투여
본 개시내용의 면역 세포는 국소 또는 전신 치료가 필요한지에 따라 다양한 방식으로 투여될 수 있다.
일반적으로, 투여는 비경구적일 수 있다.
입양 세포 요법을 위한 세포 투여 방법은 공지되어 있으며 제공된 방법 및 조성물과 관련하여 사용될 수 있다. 예를 들어, 입양 T 세포 요법 방법은 예를 들어, Gruenberg et al의 미국 특허 출원 공개 번호 2003/0170238 및 Rosenberg의 미국 특허 번호 4,690,915에 기술되어 있다.
본 개시내용의 조성물은 비경구 투여에 적합하다. 본원에서 사용되는 바와 같이, 약학 조성물의 "비경구 투여"는 대상체의 조직을 물리적으로 파괴하는 것을 특징으로 하는 임의의 투여 경로 및 조직의 파괴를 통한 약학 조성물의 투여를 포함하며, 따라서 일반적으로 혈류, 근육 또는 내부 장기로의 직접 투여를 야기한다. 비경구 투여는 조성물의 주사, 외과적 절개를 통한 조성물의 적용, 조직 관통 비-외과적 상처를 통한 조성물의 적용 등에 의한 약학 조성물의 투여를 포함하지만 이에 제한되지 않는다. 특히, 비경구 투여는 피하, 복강 내, 근육 내, 흉골 내, 정맥 내, 동맥 내, 척수강 내, 심실 내, 요도 내, 두개 내, 종양 내, 활막 내 주사 또는 주입; 및 신장 투석 주입 기술을 포함하지만, 이에 제한되지 않는 것으로 간주된다. 일부 구현예에서, 본 발명의 조성물의 비경구 투여는 정맥 내 또는 동맥 내 투여를 포함한다.
본 개시내용은 본 개시내용의 복수의 면역 세포, 및 약학적으로 허용 가능한 담체, 희석제 또는 부형제를 포함하는 약학 조성물을 제공한다.
비경구 투여에 적합한 약학 조성물의 제형은 전형적으로 일반적으로 멸균수 또는 멸균 등장 식염수와 같은 약학적으로 허용 가능한 담체와 조합된 면역 세포를 포함한다. 이러한 제형은 일시 투여 또는 연속 투여에 적합한 형태로 제조, 포장 또는 판매될 수 있다. 주사용 제제는 방부제를 함유하는 앰플 또는 다중 용량 용기와 같은 단위 용량 형태로 제조, 포장 또는 판매될 수 있다. 비경구 투여용 제제는 현탁액, 용액, 유성 또는 수성 비히클 중의 에멀젼, 페이스트 등을 포함하지만 이에 제한되지 않는다. 이러한 제형은 현탁제, 안정화제 또는 분산제를 포함하지만 이에 제한되지 않는 하나 이상의 추가 성분을 추가로 포함할 수 있다. 비경구 제형은 또한, 염, 탄수화물 및 완충제와 같은 부형제를 함유할 수 있는 수용액을 포함한다. 예시적인 비경구 투여 형태는 멸균 수용액, 예를 들어 수성 프로필렌 글리콜 또는 덱스트로스 용액 중의 용액 또는 현탁액을 포함한다. 이러한 제형은 필요에 따라 적합하게 완충될 수 있다. 비경구 투여용 제제는 즉시 및/또는 변형 방출되도록 제제화될 수 있다. 변형 방출 제형에는 지연, 지속, 펄스, 제어, 표적 및 프로그램 방출이 포함된다.
일부 구현예에서, 면역 세포를 포함하는 제형화된 조성물은 주사를 통한 투여에 적합하다. 일부 구현예에서, 면역 세포를 포함하는 제형화된 조성물은 주입을 통한 투여에 적합하다.
편리하게 단위 투여 형태로 제공될 수 있는 본 개시내용의 약학 조성물은 제약 산업에서 잘 알려진 통상적인 기술에 따라 제조될 수 있다. 이러한 기술은 약학적 담체(들) 또는 액체 담체와 같은 부형제(들)와 면역 세포를 회합시키는 단계를 포함한다.
수성 현탁액은 예를 들어 나트륨 카르복시메틸셀룰로오스, 소르비톨 및/또는 덱스트란을 비롯한 현탁액의 점도를 증가시키는 물질을 추가로 함유할 수 있다. 현탁액에는 안정제가 포함될 수도 있다.
본 개시내용의 조성물은 약학 조성물에서 통상적으로 발견되는 다른 보조 성분을 추가로 함유할 수 있다. 따라서, 예를 들어, 조성물은 예를 들어 항소양제, 수렴제, 국소 마취제 또는 항염증제와 같은 추가의 상용성 약학적 활성 물질을 함유할 수 있거나, 염료, 방부제, 항산화제, 불투명화제, 증점제 및 안정화제와 같은 본 개시내용의 조성물의 다양한 투여 형태를 물리적으로 제형화하는데 유용한 추가 물질을 함유할 수 있다. 그러나 그러한 물질이 첨가될 때 본 개시내용의 조성물의 면역 세포의 생물학적 활성을 부당하게 방해하지 않아야 한다.
제제 또는 조성물은 또한 면역 세포로 치료되는 특정 적응증, 질환 또는 병태에 유용한 하나 이상의 활성 성분을 함유할 수 있으며, 여기서 각각의 활성은 서로 불리하게 영향을 미치지 않는다. 이러한 활성 성분은 의도된 목적에 효과적인 양으로 적합하게 조합되어 존재한다. 따라서, 일부 구현예에서, 약학 조성물은 화학요법제와 같은 다른 약학 활성제 또는 약물을 추가로 포함한다.
일부 양태에서 약학 조성물은 조성물의 전달이 치료될 부위의 민감화를 유발하기에 충분한 시간과 이전에 발생하도록 시간 방출, 지연 방출 및 지속 방출 전달 시스템을 사용할 수 있다. 많은 유형의 방출 전달 시스템이 사용 가능하고 알려졌다. 이러한 시스템은 조성물의 반복 투여를 피할 수 있어 대상체와 의사의 편의성을 높일 수 있다.
투여는 치료 과정 전반에 걸쳐 지속적으로 또는 간헐적으로 1회 용량으로 수행될 수 있다. 단일 또는 다중 투여는 치료 의사에 의해 선택되는 용량 수준 및 패턴으로 수행될 수 있다.
일부 구현예에서 약학 조성물은 치료적 유효량 또는 예방적 유효량과 같이 암을 치료 또는 예방하는데 효과적인 양으로 면역 세포를 함유한다. 일부 구현예에서 치료적 또는 예방적 효능은 치료 대상의 주기적인 평가에 의해 모니터링된다. 수일, 수주 또는 수개월에 걸친 반복 투여의 경우, 상태에 따라 원하는 암 징후 또는 증상 억제가 발생할 때까지 치료를 반복할 수 있다. 그러나 다른 투여 요법이 유용할 수 있고 결정할 수 있다. 원하는 투여량은 조성물의 단일 일시 투여 또는 주입에 의해 또는 조성물의 다중 일시 투여 또는 주입에 의해 전달될 수 있다.
세포 또는 세포 집단은 1회 이상의 투여량으로 투여될 수 있다. 일부 구현예에서, 유효량의 세포가 단일 용량으로 투여될 수 있다. 일부 구현예에서, 유효량의 세포가 일정 기간에 걸쳐 1회 초과 용량으로 투여될 수 있다. 투여 시기는 주치의의 판단 범위 내에 있으며 환자의 임상 상태에 따라 다르다.
세포 또는 세포 집단은 혈액은행, 기증자 또는 환자 자신과 같은 모든 출처에서 얻을 수 있다.
유효량은 치료적 또는 예방적 이점을 제공하는 양을 의미한다. 투여되는 투여량은 수용자의 연령, 건강 및 체중, 동시 치료의 종류 (있는 경우), 치료 빈도 및 원하는 효과의 특성에 따라 달라질 것이다. 일부 구현예에서, 유효량의 세포 또는 이들 세포를 포함하는 조성물은 비경구적으로 투여된다. 일부 구현예에서, 투여는 정맥 내 투여일 수 있다. 일부 구현예에서, 투여는 종양 내 주사에 의해 직접 수행될 수 있다.
본 개시내용의 목적을 위해, 예를 들어 표적 세포가 용해되는 정도 또는 하나 이상의 사이토카인이 수용체를 발현하는 면역 세포에 의해 분비되는 정도를 비교하는 것을 포함하는 검정은 이러한 면역 세포의 주어진 용량을 대상에게 투여시 포유동물 세트 중에서 각각 상이한 용량의 면역 세포가 제공된 포유동물은 포유동물에게 투여될 시작 용량을 결정하는 데 사용될 수 있다.
일부 구현예에서, 세포는 항체 또는 조작된 세포 또는 수용체 또는 제제, 예를 들어 세포독성 제제 또는 치료제와 같은 임의의 순서로 동시에 또는 순차적으로 조합 치료의 일부로서 투여된다. 일부 구현예에서 본 개시내용의 면역 세포는 하나 이상의 추가 치료제와 함께 또는 다른 치료적 개입과 관련하여 임의의 순서로 동시에 또는 순차적으로 공동-투여된다. 일부 상황에서, 면역 세포는 면역 세포 집단이 하나 이상의 추가 치료제의 효과를 향상시키거나 그 반대가 되도록 시간상으로 충분히 가까운 또 다른 치료법과 공동 투여된다. 일부 구현예에서, 면역 세포는 하나 이상의 추가 치료제 전에 투여된다. 일부 구현예에서, 면역 세포는 하나 이상의 추가 치료제 이후에 투여된다.
구현예에서, 림프구 고갈 화학요법은 입양 면역 세포의 투여 (예를 들어, 주입) 전에, 그와 동시에, 또는 그 후에 대상체에게 투여된다. 예에서, 림프구 고갈 화학요법은 면역 세포의 투여 전에 대상체에게 투여된다. 예를 들어, 림프구 고갈 화학요법은 입양 세포 주입 전 1-4일 (예를 들어, 1, 2, 3 또는 4일)에 종료된다. 구현예에서, 예를 들어, 본원에 기재된 바와 같이, 입양 세포의 다중 용량이 투여된다. 구현예에서, 림프구 고갈 화학요법은 본원에 기재된 면역 세포의 투여 (예를 들어, 주입) 전에, 그와 동시에, 또는 그 후에 대상체에게 투여된다. 림프구 고갈의 예는 비골수 림프구 고갈 화학 요법, 골수 파괴 림프구 고갈 화학 요법, 전신 방사선 조사 등을 포함하지만 이에 국한되지 않을 수 있다. 림프구 고갈제의 예는 항흉선 세포 글로불린, 항-CD3 항체, 항-CD4 항체, 항-CD8 항체, 항-CD52 항체, 항-CD2 항체, TCRαβ 차단제, 항-CD20 항체, 항-CD19 항체, 보르테조밉, 리툭시맙, 항-CD 154 항체, 라파마이신, CD3 면역독소, 플루다라빈, 시클로포스파미드, 부설판, 멜팔란, 마브테라, 타크로리무스, 알레파셉트, 알렘투주맙, OKT3, OKT4, OKT8, OKT11, 핑골리모드, 항-CD40 항체, 항-BR3 항체, 캄파트-1H, 항-CD25 항체, 칼시뉴린 억제제, 마이코페놀레이트, 및 스테로이드를 포함하지만, 이에 제한되지 않고, 이는 단독으로 또는 조합하여 사용될 수 있다. 추가의 예로서, 림프구 고갈 요법은 알렘투주맙, 시클로포스파미드, 벤두아무스틴, 리툭시맙, 펜토스타틴 및/또는 플루다라빈의 투여를 포함할 수 있다. 림프구 고갈 요법은 감소된 순환 면역 세포의 원하는 결과가 나올 때까지 하나 이상의 주기로 시행될 수 있다. 일부 구현예에서, 림프구 고갈은 대상체에서 CD52+ 세포를 특이적으로 표적화하고 감소 또는 제거하는 작용제를 투여하는 것을 포함하며, 면역 세포는 CD52 발현을 감소 또는 제거하도록 변형된다.
일부 구현예에서, 면역 자극 요법은 투여 (예를 들어, 주입) 전에, 그와 동시에, 또는 그 후에 대상체에게 투여된다. 일부 구현예에서, 면역 자극 요법은 항상성 사이토카인을 포함한다. 일부 구현예에서, 면역 자극 요법은 면역 자극 분자를 포함한다. 일부 구현예에서, 면역 자극 요법은 IL-2, IL-7, IL-12, IL-15, IL-21, IL-9, 또는 이의 기능적 단편을 포함한다. 일부 구현예에서, 면역 자극 요법은 IL-2, IL-7, IL-12, IL-15, IL-21, IL-9, 또는 이들의 조합을 포함한다. 일부 구현예에서, 면역 자극 요법은 IL-2, 또는 이의 기능적 단편을 포함한다.
자가 세포를 사용하는 양자 세포 치료 방법은 환자 혈액으로부터 면역 세포를 분리하는 것, 본원에 기술된 이중 수용체 시스템을 암호화하는 하나 이상의 벡터로 세포를 형질도입하는 것을 포함하는 분리된 세포에 일련의 변형을 수행하는 것, 및 세포를 환자에게 투여하는 것을 포함한다. 암 또는 혈액학적 악성종양을 앓고 있거나 위험에 처한 대상체로부터 면역 세포를 제공하는 것은 환자의 혈액으로부터 면역 세포를 분리하는 것이 필요하고, 예를 들어, 백혈구 성분채집술에 의해 당업계에 공지된 방법을 통해 달성될 수 있다. 백혈구 성분채집술 동안, 대상체로부터 혈액을 추출하고 말초 혈액 단핵 세포 (PBMC)를 분리하고 나머지 혈액은 대상체의 순환계로 복귀시킨다. PBMC는 면역 세포의 샘플로서 동결 또는 동결보존되어 저장되며 예를 들어 본원에 기재된 변형과 같은 추가 처리 단계를 위해 제공된다.
일부 구현예에서, 본원에 기술된 대상체를 치료하는 방법은 농축 및/또는 고갈, 활성화, 유전적 변형, 확장, 제형화 및 동결보존을 포함하는 일련의 변형을 포함하는 대상체로부터의 면역 세포에 대한 변형을 포함한다.
본 개시내용은 다운스트림 절차, 예를 들어 본원에 기재된 변형을 위한 대상체 PBMC의 제조를 위해 당업계에 공지된 세척 및 분별 방법일 수 있는 강화 및/또는 고갈 단계를 제공한다. 예를 들어, 제한 없이, 방법은 전체 적혈구 및 혈소판 오염물을 제거하는 장치, 단핵구 고갈 및 림프구 분리를 위한 크기 기반 세포 분획 시스템, 및/또는, 예를 들어 CD4+, CD8+, CD25+, 또는 CD62L+ T 세포와 같은 특정 세포의 농축 또는 고갈을 허용하는 시스템을 포함할 수 있다. 농축 단계 후, 면역 세포의 표적 하위 집단이 추가 처리를 위해 대상 PMBC로부터 분리된다. 당업자는 본원에 제공된 농축 단계가 또한 임의의 새롭게 발견된 방법, 장치, 시약 또는 이들의 조합을 포함할 수 있음을 이해할 것이다.
본 개시내용은 생체 외 확장에 필요한 면역 세포, 예를 들어 T 세포의 활성화를 유도하기 위해 당업계에 공지된 임의의 방법일 수 있는 활성화 단계를 제공한다. 예를 들어, 수지상 세포의 존재 하에 대상 면역 세포를 배양하거나, 인공 항원-제시 세포 (AAPC)의 존재 하에 대상 면역 세포를 배양하거나, 조사된 K562-유래 AAPC의 존재 하에 면역 세포를 배양한다. 대상 면역 세포를 활성화하기 위한 다른 방법은 예를 들어, 분리된 활성화 인자 및 조성물, 예를 들어 활성화 인자로 기능화된 비드, 표면, 또는 입자의 존재 하에 면역 세포를 배양하는 것일 수 있다. 활성화 인자는 예를 들어, 항체, 예를 들어 항-CD3 및/또는 항-CD28 항체를 포함할 수 있다. 활성화 인자는 또한 예를 들어, 사이토카인, 예를 들어 인터루킨 (IL)-2 또는 IL-21일 수 있다. 활성화 인자는 예를 들어, CD40, CD40L, CD70, CD80, CD83, CD86, CD137L, ICOSL, GITRL, 및 CD134L과 같은 공동자극 분자일 수 있다. 당업자는 본원에 제공된 활성화 인자가 또한 면역 세포를 활성화할 수 있는 임의의 새롭게 발견된 활성화 인자, 시약, 조성물 또는 이들의 조합을 포함할 수 있음을 이해할 것이다.
본 개시내용은 면역 세포를 변형시키기 위한 유전적 변형 단계를 제공한다. 일부 구현예에서, 유전적 변형은 B2M 또는 HLA-A에 상보적인 본원에 기재된 shRNA를 포함하는 벡터로 면역 세포를 형질도입하는 것을 포함한다. 일부 구현예에서, 유전적 변형은 CRISPR/Cas 매개 게놈 공학을 사용하여 B2M 또는 HLA-A에서 돌연변이를 유도하기 위해 면역 세포의 게놈을 변형하는 것을 포함한다. 일부 구현예에서, 방법은 활성화제 및 억제 수용체를 암호화하는 하나 이상의 벡터로 면역 세포를 형질도입함으로써, 활성화제 및 억제 수용체를 발현하는 면역 세포를 생산하는 것을 포함한다.
본 개시내용은 유전적으로 변형된 대상 면역 세포에 대해 확장 단계를 제공한다. 유전적으로 변형된 대상체 면역 세포는 투여를 위한 치료 용량의 면역 세포를 생성하기 위해 당업계에 공지된 임의의 면역 세포 확장 시스템에서 확장될 수 있다. 예를 들어, 컨트롤러 펌프와 자동 공급 및 폐기물 제거를 허용하는 프로브를 포함하는 시스템에서 사용하기 위한 생물반응기 백을 면역 세포 확장에 사용할 수 있다. 바닥에 기체 투과성 막이 있는 세포 배양 플라스크는 면역 세포 확장에 사용할 수 있다. 있다. 임상적 사용을 위한 면역 세포의 확장을 가능하게 하는 당해 분야에 공지된 임의의 이러한 시스템은 본원에 제공된 확장 단계에 포함된다. 면역 세포는 확장을 위해 특별히 제조된 배지의 배양 시스템에서 확장된다. 확장은 또한 본원에 기술된 바와 같은 활성화 인자의 존재 하에 개시내용의 면역 세포를 배양함으로써 촉진될 수 있다. 당업자는 본원에 제공된 확장 단계가 또한 임의의 새롭게 발견된 배양 시스템, 배지 또는 면역 세포를 확장시키는 데 사용될 수 있는 활성화 인자를 포함할 수 있음을 이해할 것이다.
본 개시내용은 확장된 유전적으로 변형된 대상 면역 세포에 대한 제형 및 동결보존 단계를 제공한다. 제공된 제제화 단계는 예를 들어 본원에 기술된 치료 방법의 면역 세포의 준비 및 확장에 사용된 과잉 성분을 씻어내는 것을 포함한다. 당업계에 공지된 면역 세포와 호환되는 임의의 약학적으로 허용되는 제형 배지 또는 세척 완충액을 사용하여 면역 세포를 세척, 희석/농축하고 투여 용량을 준비할 수 있다. 제형 배지는 예를 들어 정맥 내 주입을 위한 결정질 용액과 같이 면역 세포의 투여에 허용될 수 있다.
동결 보존은 선택적으로 면역 세포를 장기간 보관하는 데 사용할 수 있다. 동결 보존은 예를 들어 동결 보존 성분을 함유하는 동결 보존 배지에 세포를 저장하는 것을 포함하는 당업계에 공지된 방법을 사용하여 달성될 수 있다. 동결 보존 성분은 예를 들어 디메틸 설폭사이드 또는 글리세롤을 포함할 수 있다. 동결보존 배지에 보관된 면역세포는 보관온도를 -80℃ 내지 -196℃로 낮추어 동결보존 가능하다.
일부 구현예에서, 치료 방법은 대상체의 HLA 생식계열 유형을 결정하는 것을 포함한다. 일부 구현예에서, HLA 생식계열 유형은 골수에서 결정된다.
일부 구현예에서, 치료 방법은 EGFR의 발현 수준을 결정하는 것을 포함한다. 일부 구현예에서, EGFR의 발현 수준은 대상체로부터의 종양 조직 샘플에서 결정된다. 일부 구현예에서, EGFR의 발현 수준은 차세대 시퀀싱을 사용하여 결정된다. 일부 구현예에서, EGFR의 발현 수준은 RNA 시퀀싱을 사용하여 결정된다. 일부 구현예에서, EGFR의 수준은 면역조직화학을 사용하여 결정된다.
일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-A*02 억제 수용체를 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-A*02 이형접합성이고 HLA-A*02가 소실된 암 세포를 갖는 것으로 결정된다. 일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-A*01 억제 수용체를 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-A*01 이형접합성이고 HLA-A*01이 소실된 암 세포를 갖는 것으로 결정된다. 일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-A*03을 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-A*03 이형접합성이고 HLA-A*03이 소실된 암 세포를 갖는 것으로 결정된다. 일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-A*07 억제 수용체를 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-A*07 이형접합성이고 HLA-A*07이 소실된 암 세포를 갖는 것으로 결정된다. 일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-C*07 억제 수용체를 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-C*07 이형접합성이고 HLA-C*07이 소실된 암 세포를 갖는 것으로 결정된다. 일부 구현예에서, 치료 방법은 이를 필요로 하는 대상체에 HLA-B*07 억제 수용체를 포함하는 치료 유효량의 면역 세포를 투여하는 것을 포함하며, 여기서 대상체는 HLA 생식계열 HLA-B*07 이형접합성이고 HLA-B*07이 소실된 암 세포를 갖는 것으로 결정된다.
다양한 구현예에서, 본 개시내용은 EGFR을 발현하고 HLA-A*02 발현을 상실한 악성종양을 갖는 이형접합 HLA-A*02 환자의 치료; 및/또는 EGFR을 발현하고 HLA-A*02 발현을 상실한 재발성 절제 불가능 또는 전이성 고형 종양을 갖는 이형접합 HLA-A*02 성인 환자의 치료 방법을 제공한다.
일부 구현예에서, 본원에 기재된 면역 세포의 치료 유효 용량이 투여된다. 일부 구현예에서, 본 개시내용의 면역 세포는 정맥 내 주사에 의해 투여된다. 일부 구현예에서, 본 개시내용의 면역 세포는 복강 내 주사에 의해 투여된다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포, 약 1Х106 세포, 약 2Х106 세포, 약 3Х106 세포, 4Х106 세포, 약 5Х106 세포, 약 6Х106 세포, 약 7Х106 세포, 약 8Х106 세포, 약 9Х106 세포, 약 1Х107, 약 2Х107, 약 3Х107, 약 4Х107, 약 5Х107, 약 6Х107, 약 7Х107, 약 8Х107, 약 9Х107, 약 1Х108 세포, 약 2Х108 세포, 약 3Х108 세포, 약 4Х108 세포, 약 5Х108 세포, 약 6Х108 세포, 약 7Х108 세포, 약 8Х108 세포, 약 9Х108 세포, 약 1Х109 세포, 약 2Х109 세포, 약 3Х109 세포, 약 3Х109 세포, 약 4Х109 세포, 약 5Х109 세포, 약 5Х109 세포, 약 6Х109 세포, 약 7Х109 세포, 약 8Х109 세포, 약 9Х109 세포, 약 1Х1010 세포, 약 2Х1010 세포, 약 3Х1010 세포, 약 4Х1010 세포, 약 5Х1010 세포, 약 6Х1010 세포, 약 7Х1010 세포, 약 8Х1010 세포, 또는 약 9Х1010 세포를 포함한다.
일부 구현예에서 치료 유효 용량은 약 0.5Х106 세포 내지 약 9Х1010 세포, 약 1Х106 세포 내지 약 5Х1010 세포, 약 2Х106 세포 내지 약 5Х109 세포, 약 3Х106 세포 내지 약 5Х109 세포, 약 4Х106 세포 내지 약 3Х109 세포, 약 5Х106 세포 내지 약 2Х109 세포, 약 6Х106 세포 내지 약 1Х109 세포, 0.5Х106 세포 내지 약 6Х109 세포, 약 1Х106 세포 내지 약 5Х109 세포, 약 2Х106 세포 내지 약 5Х109 세포, 약 3Х106 세포 내지 약 4Х109 세포, 약 4Х106 세포 내지 약 3Х109 세포, 약 5Х106 세포 내지 약 2Х109 세포, 약 6Х106 세포 내지 약 1Х109 세포, 0.5Х106 세포 내지 약 6Х108 세포, 약 1Х106 세포 내지 약 5Х108 세포, 약 2Х106 세포 내지 약 5Х108 세포, 약 3Х106 세포 내지 약 4Х108 세포, 약 4Х106 세포 내지 약 3Х108 세포, 약 5Х106 세포 내지 약 2Х108 세포, 약 6Х106 세포 내지 약 1Х108 세포, 약 7Х106 세포 내지 약 9Х108 세포, 약 8Х106 세포 내지 약 8Х108 세포, 약 9Х106 세포 내지 약 7Х108 세포, 약 1Х107 세포 내지 약 6Х108 세포, 약 2Х107 세포 내지 약 5Х108 세포, 약 7Х106 세포 내지 약 9Х107 세포, 약 8Х106 세포 내지 약 8Х107 세포, 약 9Х106 세포 내지 약 7Х107 세포, 약 1Х107 세포 내지 약 6Х107 세포, 또는 약 2Х107 세포 내지 약 5Х107 세포를 포함한다.
일부 구현예에서, 치료 유효 용량은 약 0.5Х105 세포 내지 약 9Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포 내지 약 1Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포 내지 약 5Х109 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포 내지 약 1Х109 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포 내지 약 6Х108 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х106 세포 내지 약 9Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х107 세포 내지 약 1Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х107 세포 내지 약 5Х109 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х107 세포 내지 약 1Х109 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х107 세포 내지 약 6Х108 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х108 세포 내지 약 9Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х108 세포 내지 약 1Х1010 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х108 세포 내지 약 5Х109 세포를 포함한다. 일부 구현예에서, 치료 유효 용량은 약 0.5Х108 세포 내지 약 1Х109 세포를 포함한다. 치료 용량에서 언급되는 용어 "약"은 예를 들어, ± 0.5Х106 세포, ± 0.5Х107 세포, 또는 ± 0.5Х108 세포일 수 있다.
키트 및 제조 물품
본 개시내용은 본원에 기재된 수용체를 암호화하는 폴리뉴클레오티드 및 벡터, 및 본원에 기재된 수용체를 포함하는 면역 세포를 포함하는 키트 및 제조 물품을 제공한다. 일부 구현예에서, 키트는 바이알, 주사기 및 사용 설명서와 같은 물품을 포함한다.
일부 구현예에서, 키트는 본 개시내용의 하나 이상의 수용체를 암호화하는 서열을 포함하는 폴리뉴클레오티드 또는 벡터를 포함한다.
일부 구현예에서, 키트는 본원에 기재된 바와 같은 제1 및 제2 수용체를 포함하는 다수의 면역 세포를 포함한다. 일부 구현예에서, 다수의 면역 세포는 다수의 T 세포를 포함한다.
일부 구현예에서, 키트는 사용 설명서를 추가로 포함한다.
열거된 구현예
구현예 1. 다음을 포함하는 면역 세포:
a.) 표피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및
b.) EGFR+ 암에서 이형접합성의 소실로 인해 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체,
여기서 제1 수용체는 EGFR에 반응하는 활성화제 수용체이고; 여기서 제2 수용체는 비-표적 항원에 반응하는 억제 수용체이다.
구현예 2. 구현예 1에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 MHC 단백질의 대립형질 변이체에 특이적으로 결합하는 면역 세포.
구현예 3. 구현예 1에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A, HLA-B, 또는 HLA-C 단백질의 대립형질 변이체에 특이적으로 결합하는 면역 세포.
구현예 4. 구현예 1에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*02에 특이적으로 결합하는 면역 세포.
구현예 5. 구현예 1에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*01, HLA-A*03, HLA-A*11, HLA-C*07, 또는 HLA-B*07에 특이적으로 결합하는 면역 세포.
구현예 6. 구현예 3 내지 5 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인이 표 5에 개시된 바와 같은 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함하는 면역 세포.
구현예 7. 구현예 3 내지 5 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인이 서열 번호 101-106의 또는 서열 번호 106-112의 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함하는 면역 세포.
구현예 8. 구현예 3 내지 5 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인이 표 4에 개시된 폴리펩티드 서열로부터 선택된 폴리펩티드 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 9. 구현예 4에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인이 서열 번호 89-100 중 어느 하나; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 10. 구현예 1 내지 9 중 어느 하나에 있어서, 상기 제1 수용체가 키메라 항원 수용체 (CAR)인 면역 세포.
구현예 11. 구현예 1 내지 10 중 어느 하나에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인이 표 3에 제시된 서열 그룹으로부터 선택된 가변 중쇄 (VH) 부분 세트를 포함하는 중쇄 상보성 결정 영역 (HC-CDR); 및/또는 표 3에 제시된 서열 그룹의 가변 경쇄 (VL) 부분 세트를 포함하는 경쇄 상보성 결정 영역 (LC-CDR); 또는 각각의 CDR에서 최대 1, 2, 3, 4개의 치환, 삽입, 또는 결실을 갖는 CDR 서열을 포함하는 면역 세포.
구현예 12. 구현예 1 내지 11 중 어느 하나에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인이 표 2에 제시된 VH 서열로부터 선택된 서열을 갖는 가변 중쇄 (VH) 부분; 및/또는 표 2에 제시된 서열을 포함하는 가변 경쇄 (VL) 부분; 또는 이에 대해 적어도 70%, 적어도 85%, 적어도 90%, 또는 적어도 95% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 13. 구현예 1 내지 12 중 어느 하나에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인이 표 1에 제시된 서열의 그룹으로부터 선택된 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97%, 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 14. 구현예 1 내지 13 중 어느 하나에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인이 서열 번호 9-18로 이루어진 그룹으로부터 선택된 scFv 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 15. 구현예 1 내지 14 중 어느 하나에 있어서, 상기 제2 수용체는 LILRB1 세포 내 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 16. 구현예 15에 있어서, 상기 LILRB1 세포 내 도메인이 서열 번호 129에 대해 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한, 또는 동일한 서열을 포함하는 면역 세포.
구현예 17. 구현예 1 내지 16 중 어느 하나에 있어서, 상기 제2 수용체가 LILRB1 막관통 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 18. 구현예 17에 있어서, 상기 LILRB1 막관통 도메인 또는 이의 기능적 변이체가 서열 번호 133과 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함하는 면역 세포.
구현예 19. 구현예 1 내지 18 중 어느 하나에 있어서, 상기 제2 수용체가 LILRB1 힌지 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 20. 구현예 19에 있어서, 상기 LILRB1 힌지 도메인이 서열 번호 132, 서열 번호 125, 서열 번호 126에 대해 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함하는 면역 세포.
구현예 21. 구현예 1 내지 20 중 어느 하나에 있어서, 상기 제2 수용체가 LILRB1 세포 내 도메인, LILRB1 막관통 도메인, LILRB1 힌지 도메인, 이들 중 임의의 것의 기능적 변이체, 또는 이들의 조합을 포함하는 면역 세포.
구현예 22. 구현예 21에 있어서, 상기 LILRB1 세포 내 도메인 및 LILRB1 막관통 도메인이 서열 번호 128 또는 서열 번호 128과 적어도 95% 동일한 서열을 포함하는 면역 세포.
구현예 23. 구현예 1 내지 22 중 어느 하나에 있어서, 상기 EGFR+ 암세포가 폐암 세포, 소세포 폐암 세포, 비-소세포 폐암 세포, 췌관 암종 세포, 대장암 세포, 두경부암 세포, 식도암 및 위선암 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포, 또는 자궁암 세포인 면역 세포.
구현예 24. 구현예 23에 있어서, 상기 EGFR+ 암 세포는 폐암 세포인 면역 세포.
구현예 25. 구현예 1 내지 24 중 어느 하나에 있어서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포인 면역 세포.
구현예 26. 구현예 25에 있어서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A의 이형접합성 소실에 의해 EGFR+/HLA-A*02+ 세포로부터 유도되어 HLA-A*02의 소실을 초래하는 면역 세포.
구현예 27. 구현예 1 내지 26 중 어느 하나에 있어서, 상기 제1 수용체 및 제2 수용체가 함께 이형접합성을 소실한 EGFR/HLA-A*02- 암 세포의 존재 하에 면역 세포를 특이적으로 활성화시키는 면역 세포.
구현예 28. 구현예 1 내지 27 중 어느 하나에 있어서, 상기 제1 수용체 및 제2 수용체가 함께 이형접합성 소실에 의해 HLA-A*02가 소실되지 않은 EGFR+ 세포의 존재 하에 면역 세포를 특이적으로 활성화시키지 않는 면역 세포.
구현예 29. 구현예 1 내지 28 중 어느 하나에 있어서, 상기 면역 세포는 T 세포, 대식세포, NK 세포, iNKT 세포, 또는 감마 델타 T 세포인 면역 세포.
구현예 30. 구현예 29에 있어서, 상기 T 세포는 CD8+ CD4- T 세포인 면역 세포.
구현예 31. 구현예 1 내지 30 중 어느 하나에 있어서, 상기 MHC 클래스 I 유전자의 발현 및/또는 기능이 감소되거나 제거된 면역 세포.
구현예 32. 구현예 30에 있어서, 상기 MHC 클래스 I 유전자는 베타-2-마이크로글로불린 (B2M)인 면역 세포.
구현예 33. 구현예 32에 있어서, 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함하고, 간섭 RNA는 B2M mRNA의 서열 (서열 번호 172)에 상보적인 서열을 포함하는 면역 세포.
구현예 34. 구현예 33에 있어서, 상기 간섭 RNA가 B2M mRNA의 RNAi-매개 분해를 유도할 수 있는 면역 세포.
구현예 35. 구현예 33에 있어서, 상기 간섭 RNA는 짧은 헤어핀 RNA (shRNA)인 면역 세포.
구현예 36. 구현예 35에 있어서, 상기 shRNA는 다음을 포함하고:
a.) 5' 말단에서 3' 말단으로 B2M mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및
b.) 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는, 제2 서열,
여기서 제1 서열 및 제2 서열은 shRNA를 형성하는 면역 세포.
구현예 37. 구현예 32에 있어서, B2M을 암호화하는 서열 (서열 번호 170)에 대한 하나 이상의 변형을 포함하고, 상기 하나 이상의 변형은 B2M의 발현을 감소시키고/시키거나 기능을 제거하는 면역 세포.
구현예 38. 구현예 37에 있어서, 상기 하나 이상의 변형은 B2M을 암호화하는 내인성 유전자의 하나 이상의 불활성화 돌연변이를 포함하는 면역 세포.
구현예 39. 구현예 38에 있어서, 상기 하나 이상의 불활성화 돌연변이는 결실, 삽입, 치환, 또는 프레임이동 돌연변이를 포함하는 면역 세포.
구현예 40. 구현예 38 내지 39 중 어느 하나에 있어서, 상기 하나 이상의 불활성화 돌연변이가 B2M을 암호화하는 내인성 유전자의 서열 (서열 번호 170)을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)과의 복합체에서 핵산 유도 엔도뉴클레아제로 도입되는 면역 세포.
구현예 41. 구현예 31에 있어서, 상기 MHC 클래스 I 유전자는 HLA-A*02인 면역 세포.
구현예 42. 구현예 41에 있어서, 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함하고, HLA-A*02 mRNA의 서열 (서열 번호 171)에 상보적인 서열을 포함하는 면역 세포.
구현예 43. 구현예 42에 있어서, 상기 간섭 RNA는 HLA-A*02 mRNA의 RNA 간섭 (RNAi)-매개 분해를 유도할 수 있는 면역 세포.
구현예 44. 구현예 43에 있어서, 상기 간섭 RNA는 다음을 포함하는 짧은 헤어핀 RNA (shRNA)이고:
a.) 5' 말단에서 3' 말단으로 HLA-A*02 mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및
b.) 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열,
여기서 제1 서열 및 제2 서열은 shRNA를 형성하는 면역 세포.
구현예 45. 구현예 41에 있어서, HLA-A*02를 암호화하는 내인성 유전자의 서열 (서열 번호 169)에 대한 하나 이상의 변형을 포함하고, 여기서 하나 이상의 변형은 HLA-A*02의 발현을 감소시키고/시키거나 기능을 제거하는 면역 세포.
구현예 46. 구현예 45에 있어서, 상기 하나 이상의 변형은 HLA-A*02를 암호화하는 내인성 유전자의 하나 이상의 불활성화 돌연변이를 포함하는 면역 세포.
구현예 47. 구현예 45 또는 구현예 46에 있어서, 상기 하나 이상의 불활성화 돌연변이는 HLA-A*02를 암호화하는 내인성 유전자의 서열을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)을 갖는 복합체에서 핵산 유도 엔도뉴클레아제로 도입되는 면역 세포.
구현예 48. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 174 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 49. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 50. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 174, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 51. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 52. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 53. 구현예 1에 있어서:
a.) 제1 수용체는 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b.) 제2 수용체는 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 54. 구현예 48 내지 53 중 어느 하나에 있어서, T2A 자가 절단 펩티드를 추가로 포함하고, 상기 T2A 자가 절단 펩티드는 서열 번호 178, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 55. 구현예 48 내지 54 중 어느 하나에 있어서, 간섭 RNA를 추가로 포함하고, 상기 간섭 RNA는 서열 번호 179, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 56. 구현예 1 내지 55 중 어느 하나에 있어서, 상기 면역 세포가 자가 세포인 면역 세포.
구현예 57. 구현예 1 내지 55 중 어느 하나에 있어서, 상기 면역 세포가 동종이계인 면역 세포.
구현예 58. 구현예 1 내지 57중 어느 하나의 면역 세포의 치료적 유효량을 포함하는 약학 조성물.
구현예 59. 구현예 58에 있어서, 상기 면역 세포는 제1 수용체 및 제2 수용체를 모두 발현하는 약학 조성물.
구현예 60. 구현예 59에 있어서, 적어도 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 또는 약 95%의 면역 세포는 제1 수용체 및 제2 수용체를 모두 발현하는 약학 조성물.
구현예 61. 구현예 59에 있어서, 적어도 90%의 면역 세포는 제1 수용체 및 제2 수용체를 모두 발현하는 약학 조성물.
구현예 62. 구현예 58 내지 61 중 어느 하나에 있어서, 약학적으로 허용 가능한 담체, 희석제 또는 부형제를 추가로 포함하는 약학 조성물.
구현예 63. 구현예 58 내지 62 중 어느 하나에 있어서, EGFR+ 암 치료에 약제로 사용하기 위한 약학 조성물.
구현예 64. 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템으로서, 다음을 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하고:
a.) 내피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및
b.) 이형접합성의 소실로 인해 EGFR+ 암 세포에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체,
여기서 제1 수용체는 EGFR+ 암 세포 상의 EGFR에 반응하는 활성화제 수용체이고; 여기서 제2 수용체는 비-표적 항원에 반응하는 억제 수용체인 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템.
구현예 65. 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템으로서, 구현예 1 내지 57 중 어느 하나의 면역 세포를 생성하는데 사용하기 위한 제1 수용체 및 제2 수용체를 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하는 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템.
구현예 66. 구현예 64 또는 65의 하나 이상의 폴리뉴클레오티드를 포함하는 벡터.
구현예 67. MHC 클래스 I 유전자좌에서 이형접합성을 소실한 EGFR+ 암 세포를 사멸시키는 방법으로서, 유효량의 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물을 대상체에 투여하는 것을 포함하는 방법.
구현예 68. 구현예 67에 있어서, 상기 EGFR+ 암 세포는 폐암 세포, 소세포 폐암 세포, 비-소세포 폐암 세포, 췌관 암종 세포, 결장직장암 세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포, 또는 자궁암 세포인 방법.
구현예 69. 구현예 67에 있어서, 상기 EGFR+ 암 세포는 폐암 세포인 방법.
구현예 70. 구현예 67에 있어서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포인 방법.
구현예 71. 구현예 70에 있어서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A*02의 소실을 야기하는 HLA-A에서 이형접합성의 소실에 의해 EGFR+/HLA-A*02+ 세포로부터 유래되는 방법.
구현예 72. 비-표적 항원을 암호화하는 유전자좌에서 이형접합성이 소실된 EGFR+ 종양이 있는 대상체에서 EGFR+ 암을 치료하는 방법으로서, 대상체에게 유효량의 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물을 투여하는 것을 포함하는 방법.
구현예 73. 구현예 72에 있어서, 상기 대상체는 EGFR을 발현하고 HLA-A*02 발현을 상실한 악성종양을 갖는 이형접합 HLA-A*02 환자인 방법.
구현예 74. 구현예 72에 있어서, 상기 대상체는 EGFR을 발현하고 HLA-A*02 발현을 상실한 재발성 절제 불가능 또는 전이성 고형 종양을 갖는 이형접합 HLA-A*02 환자인 방법.
구현예 75. 다음 단계를 포함하는 대상체에서 암을 치료하는 방법:
a.) 대상체의 비악성 세포 및 암세포에서 비-표적 항원의 유전자형 또는 발현 수준을 결정하는 단계;
b.) 대상체의 암 세포에서 EGFR의 발현 수준을 결정하는 단계; 및
c.) 비-악성 세포가 비-표적 항원을 발현하고 암 세포는 비-표적 항원을 발현하지 않고, 암 세포가 EGFR 양성인 경우 대상체에게 유효량의 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물을 투여하는 단계.
구현예 76. 구현예 67 내지 75 중 어느 하나에 있어서, 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물의 투여가 대상체에서 종양의 크기를 감소시키는 방법.
구현예 77. 구현예 76에 있어서, 상기 종양이 약 5%, 약 10%, 약 15%, 약 20%, 약 25%, 약 30%, 약 35%, 약 40%, 약 45%, 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 약 95%, 또는 약 100% 감소되는 방법.
구현예 78. 구현예 76에 있어서, 상기 종양이 제거되는 것인 방법.
구현예 79. 구현예 67 내지 75 중 어느 하나에 있어서, 면역 세포 또는 약학 조성물의 투여가 대상체에서 종양의 성장을 정지시키는 방법.
구현예 80. 구현예 67 내지 75 중 어느 하나에 있어서, 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물의 투여가 대상체에서 종양의 수를 감소시키는 방법.
구현예 81. 구현예 67 내지 80 중 어느 하나에 있어서, 상기 면역 세포 또는 약학 조성물의 투여가 대상체에서 정상 세포가 아닌 암 세포의 선택적인 사멸을 초래하는 방법.
구현예 82. 구현예 81에 있어서, 상기 사멸된 세포의 적어도 약 60%가 암세포이고, 사멸된 세포의 적어도 약 65%가 암세포이고, 사멸된 세포의 적어도 약 70%가 암세포이고, 사멸된 세포의 적어도 약 75%가 암세포이고, 사멸된 세포의 적어도 약 80%가 암세포이고, 사멸된 세포의 적어도 약 85%가 암세포이고, 사멸된 세포의 적어도 약 90%가 암세포이고, 사멸된 세포의 적어도 약 95%가 암세포이고, 또는 사멸된 세포의 약 100%가 암세포인 방법.
구현예 83. 구현예 81에 있어서, 상기 면역 세포 또는 약학 조성물의 투여가 대상체의 암세포의 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90% 또는 모두의 사멸을 초래하는 방법.
구현예 84. 구현예 67 내지 83 중 어느 하나에 있어서, 상기 면역 세포 또는 약학 조성물의 투여가 제1 활성화제 수용체를 포함하지만 제2 억제 수용체는 포함하지 않는 달리 동등한 면역 세포의 투여보다 대상체에 대한 부작용이 더 적은 방법.
구현예 85. 다수의 면역 세포를 제조하는 방법으로서, 다음 단계를 포함하는 방법:
a.) 다수의 면역 세포를 제공하는 단계, 및
b.) 다수의 면역 세포를 구현예 64 또는 구현예 65의 폴리뉴클레오티드 시스템, 또는 구현예 66의 벡터로 형질전환하는 단계.
구현예 86. 구현예 1-57 중 어느 하나의 면역 세포 또는 구현예 58-62 중 어느 하나의 약학 조성물을 포함하는 키트.
구현예 87. 구현예 86에 있어서, 사용 설명서를 추가로 포함하는 키트.
구현예 88. 세포에서 이형접합성의 소실에 반응하는 면역 세포로서, 다음을 포함하는 면역 세포:
a. 다음으로부터 선택된 표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체, 임의로 키메라 항원 수용체 (CAR) 또는 T 세포 수용체 (TCR):
i. 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 암 세포 특이적 항원, 또는 이의 펩티드 항원; 또는
ii. 표피 성장 인자 수용체 (EGFR), 또는 주요 조직적합성 복합체 클래스 I (MHC-I)과의 복합체 내 이의 펩티드 항원; 및
b. 이형접합성의 소실로 인해 암 세포에서 소실된 비표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체, 임의로 억제 수용체.
구현예 89. 구현예 88에 있어서, 상기 비-표적 항원은 다형성을 포함하는 방법.
구현예 90. 구현예 89에 있어서, 상기 비-표적 항원은 서열 번호 87과 적어도 95% 동일성을 공유하는 COLEC12 항원을 포함하고 다형성은 서열 번호 87의 위치 522에서 S 또는 P를 포함하는 면역 세포.
구현예 91. 구현예 89에 있어서, 상기 비-표적 항원은 서열 번호 88, 서열 번호 134 또는 서열 번호 135과 적어도 95% 동일성을 공유하는 APCDD1 항원을 포함하고, 다형성은 다음으로 구성된 그룹에서 선택되는 면역 세포:
a. 서열 번호 88의 위치 150에서 A V, I 또는 L;
b. 서열 번호 134의 위치 165에서 Y 또는 S; 및
c. 서열 번호 135의 위치 28에서 A Q 또는 R.
구현예 92. 구현예 89에 있어서, 상기 비-표적 항원은 서열 번호 86과 적어도 95% 동일성을 공유하는 CXCL16 항원을 포함하고 다형성은 다음으로 이루어진 그룹으로부터 선택되는 면역 세포:
a. 서열 번호 86의 위치 142에서 I 또는 T; 및
b. 서열 번호 86의 위치 200에서 A 또는 V.
구현예 93. 구현예 88 또는 89에 있어서, 상기 비-표적 항원은 HLA-A*02를 포함하는 면역 세포.
구현예 94. 구현예 88 내지 93 중 어느 하나에 있어서, 상기 표적 항원이 암 세포에 의해 발현되는 면역 세포.
구현예 95. 구현예 88 내지 94 중 어느 하나에 있어서, 상기 비-표적 항원이 암 세포에 의해 발현되지 않는 면역 세포.
구현예 96. 구현예 88 내지 95 중 어느 하나에 있어서, 상기 비-표적 항원이 비-표적 세포에 의해 발현되는 면역 세포.
구현예 97. 구현예 88 내지 96 중 어느 하나에 있어서, 상기 비-표적 세포가 표적 항원 및 비-표적 항원 모두를 발현하는 면역 세포.
구현예 98. 구현예 88 내지 97 중 어느 하나에 있어서, 상기 표적 항원은 암 세포 특이적 항원인 면역 세포.
구현예 99. 구현예 88 내지 98 중 어느 하나에 있어서, 상기 표적 항원은 주요 조직적합성 복합체 클래스 I (MHC-I)과의 복합체 내의 암 세포 특이적 항원의 펩티드 항원인 면역 세포.
구현예 100. 구현예 88 내지 99 중 어느 하나에 있어서, 상기 암 세포는 폐암 세포, 교모세포종 세포, 유방암 세포, 두경부암 세포 또는 결장직장암 세포인 면역 세포.
구현예 101. 구현예 88 내지 100 중 어느 하나에 있어서, 상기 암 세포가 EGFR을 발현하는 면역 세포.
구현예 102. 구현예 88 내지 101 중 어느 하나에 있어서, 상기 제1 수용체 및 제2 수용체가 함께 암 세포의 존재 하에 면역 세포를 특이적으로 활성화시키는 면역 세포.
구현예 103. 구현예 102에 있어서, 상기 면역 세포는 T 세포, 대식세포, NK 세포, iNKT 세포, 또는 감마 델타 T 세포인 면역 세포.
구현예 104. 구현예 103에 있어서, 상기 T 세포는 CD8+ CD4- T 세포인 면역 세포.
구현예 105. 구현예 88 내지 104 중 어느 하나에 있어서, 상기 EGFR 서열을 서열 번호 1-8 중 어느 하나와 적어도 95% 동일성을 공유하는 서열을 포함하는 면역 세포.
구현예 106. 구현예 88 내지 105 중 어느 하나에 있어서, 상기 제1 수용체는 T 세포 수용체 (TCR)인 면역 세포.
구현예 107. 구현예 88 내지 106 중 어느 하나에 있어서, 상기 제1 수용체는 키메라 항원 수용체 (CAR)인 면역 세포.
구현예 108. 구현예 106 또는 107에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 항체 단편, 단쇄 Fv 항체 단편 (scFv), β 사슬 가변 도메인 (Vβ), 또는 TCR α 사슬 가변 도메인 및 TCR β 사슬 가변 도메인을 포함하는 면역 세포.
구현예 109. 구현예 106 또는 107에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 scFv를 포함하는 면역 세포.
구현예 110. 구현예 109에 있어서, 상기 scFv는 서열 번호 9-18로 이루어진 그룹으로부터 선택된 서열, 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 111. 구현예 109에 있어서, 상기 scFv는 서열 번호 9-18로 이루어진 그룹으로부터 선택된 서열을 포함하거나 본질적으로 이루어지는 면역 세포.
구현예 112. 구현예 106 또는 107에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 VH 및 VL 도메인을 포함하는 면역 세포.
구현예 113. 구현예 112에 있어서, 상기 VH 도메인은 서열 번호 19-24로 이루어진 그룹으로부터 선택된 서열, 또는 표 2에 개시된 VH 서열, 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 114. 구현예 112에 있어서, 상기 VH 도메인은 서열 번호 19-24로 이루어진 그룹으로부터 선택된 서열, 또는 표 2에 개시된 VH 서열을 포함하거나 본질적으로 이루어지는 면역 세포.
구현예 115. 구현예 112 내지 114 중 어느 하나에 있어서, 상기 VL 서열 번호 25-30으로 이루어진 그룹으로부터 선택된 서열, 또는 표 2에 개시된 VH 서열, 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 116. 구현예 112 내지 114 중 어느 하나에 있어서, 상기 VL 도메인은 서열 번호 25-30으로 이루어진 그룹으로부터 선택된 서열, 또는 표 2에 개시된 VL 서열을 포함하거나 본질적으로 이루어지는 세포.
구현예 117. 구현예 105 내지 107 중 어느 하나에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 31-65로 이루어진 그룹으로부터 선택된 상보성 결정 영역 (CDR), 또는 표 3에 개시된 CDR 서열을 포함하는 면역 세포.
구현예 118. 구현예 88 내지 117 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 항체 단편, 단쇄 Fv 항체 단편 (scFv), β 사슬 가변 도메인 (Vβ), 또는 TCR α 사슬 가변 도메인 및 TCR β 사슬 가변 도메인을 포함하는 면역 세포.
구현예 119. 구현예 88 또는 93 내지 118 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 scFv를 포함하는 면역 세포.
구현예 120. 구현예 119에 있어서, 상기 scFv는 서열 번호 89-100 중 어느 하나와 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
구현예 121. 구현예 119에 있어서, 상기 scFv는 서열 번호 89-100 중 어느 하나의 서열을 포함하거나 본질적으로 이루어지는 세포.
구현예 122. 구현예 88 또는 93 내지 118 중 어느 하나에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 서열 번호 101-112 로 이루어진 그룹으로부터 선택된 CDR을 포함하는 면역 세포.
구현예 123. 구현예 88 내지 122 중 어느 하나에 있어서, 상기 제2 수용체는 LILRB1 세포 내 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 124. 구현예 123에 있어서, 상기 LILRB1 세포 내 도메인은 서열 번호 124와 적어도 95% 동일한 서열을 포함하는 면역 세포.
구현예 125. 구현예 88 내지 124 중 어느 하나에 있어서, 상기 제2 수용체는 LILRB1 막관통 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 126. 구현예 125에 있어서, 상기 LILRB1 막관통 도메인 또는 이의 기능적 변이체는 서열 번호 133과 적어도 95% 동일한 서열을 포함하는 면역 세포.
구현예 127. 구현예 88 내지 126 중 어느 하나에 있어서, 상기 제2 수용체는 LILRB1 힌지 도메인 또는 이의 기능적 단편 또는 변이체를 포함하는 면역 세포.
구현예 128. 구현예 127에 있어서, 상기 LILRB1 힌지 도메인은 서열 번호 132, 서열 번호 125 또는 서열 번호 126과 적어도 95% 동일한 서열을 포함하는 면역 세포.
구현예 129. 구현예 88 내지 128 중 어느 하나에 있어서, 상기 제2 수용체는 LILRB1 세포 내 도메인 및 LILRB1 막관통 도메인, 또는 이의 기능적 변이체를 포함하는 면역 세포.
구현예 130. 구현예 129에 있어서, 상기 LILRB1 세포 내 도메인 및 LILRB1 막관통 도메인은 서열 번호 128 또는 서열 번호 128과 적어도 95% 동일한 서열을 포함하는 면역 세포.
구현예 131. 구현예 88 내지 130 중 어느 하나에 있어서, 상기 면역 세포는 T 세포, 대식세포, NK 세포, iNKT 세포, 또는 감마 델타 T 세포인 면역 세포.
구현예 132. 구현예 131에 있어서, 상기 T 세포는 CD8+ CD4- T 세포인 면역 세포.
구현예 133. 치료적 유효량의 구현예 88-132 중 어느 하나의 면역 세포를 포함하는 약학 조성물.
구현예 134. 구현예 133에 있어서, 약학적으로 허용 가능한 담체, 희석제 또는 부형제를 추가로 포함하는 약학 조성물.
구현예 135. 암 치료에 약제로 사용하기 위한 구현예 133 또는 134의 약학 조성물.
구현예 136. 다음을 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하는 폴리뉴클레오티드 시스템:
a. 다음으로부터 선택된 표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체, 임의로 키메라 항원 수용체 (CAR) 또는 T 세포 수용체 (TCR):
i. 암 세포 특이적 항원, 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 펩티드 항원; 또는
ii. 표피 성장 인자 수용체 (EGFR), 또는 주요 조직적합성 복합체 클래스 I (MHC-I)와의 복합체 내 이의 펩티드 항원; 및
b. 이형접합성의 소실로 인해 암 세포에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체, 임의로 억제 수용체.
구현예 137. 구현예 136에 있어서, 비-표적 항원은 다형성을 포함하는 폴리뉴클레오티드 시스템.
구현예 138. 구현예 137에 있어서, 상기 비-표적 항원은 서열 번호 87과 적어도 95% 동일성을 공유하는 COLEC12 항원이고 다형성은 서열 번호 87의 위치 522에서 S 또는 P를 포함하는 폴리뉴클레오티드 시스템.
구현예 139. 구현예 137에 있어서, 상기 비-표적 항원은 서열 번호 88, 서열 번호 134 또는 서열 번호 135와 적어도 95% 동일성을 공유하는 APCDD1 항원이고, 다형성은 다음으로 구성된 그룹에서 선택되는 폴리뉴클레오티드 시스템:
a. 서열 번호 88의 위치 150에서 A V, I 또는 L;
b. 서열 번호 134의 위치 165에서 Y 또는 S; 및
c. 서열 번호 135의 위치 28에서 A Q 또는 R.
구현예 140. 구현예 137에 있어서, 상기 비-표적 항원은 서열 번호 86과 적어도 95% 동일성을 공유하는 CXCL16 항원이고 다형성은 다음으로 구성된 그룹에서 선택되는 폴리뉴클레오티드 시스템:
a. 서열 번호 86의 위치 142에서 I 또는 T; 및
b. 서열 번호 86의 위치 200에서 A 또는 V.
구현예 141. 구현예 136 또는 137에 있어서, 상기 비-표적 항원이 HLA-A*02인 폴리뉴클레오티드 시스템.
구현예 142. 구현예 136-141 중 어느 하나의 하나 이상의 폴리뉴클레오티드를 포함하는 벡터.
구현예 143. 대상체에서 다수의 암세포를 죽이고/거나 암을 치료하는 방법으로서, 대상체에 유효량의 구현예 88-132 중 어느 하나의 면역 세포 또는 구현예 46-48 중 어느 하나의 약학 조성물을 투여하는 것을 포함하는 방법.
구현예 144. 구현예 143에 있어서, 다수의 암 세포가 표적 항원을 발현하는 방법.
구현예 145. 구현예 143 또는 144에 있어서, 다수의 암 세포는 비-표적 항원을 발현하지 않는 방법.
구현예 146. 다수의 면역 세포를 만드는 방법으로서, 다음 단계를 포함하는 방법:
a. 다수의 면역 세포를 생산하는 단계, 및
b. 다수의 면역 세포를 구현예 136-141 중 어느 하나의 폴리뉴클레오티드 시스템, 또는 구현예 142의 벡터로 형질전환하는 단계.
구현예 147. 구현예 88-132 중 어느 하나의 면역 세포 또는 구현예 133-135중 어느 하나의 약학 조성물을 포함하는 키트.
구현예 148. 구현예 147에 있어서, 사용 설명서를 추가로 포함하는 키트.
실시예
다음 실시예는 단지 예시를 위한 것이며 본 개시내용의 범위를 제한하지 않는다. 실시예 전체에서, 용어 "차단제 항원"은 비-표적 항원의 구현예를 기술하기 위해 사용된다.
실시예 1: 이형접합성 소실로 인해 암 세포에서 소실된 차단제 표적의 식별
생물정보학 파이프라인을 사용하여 후보 차단제 표적을 식별했다. 결장직장암에서 높은 이형접합성 소실 (0.5 초과)을 갖는 세포외 도메인에서 공통의 비동의 변이체를 갖는 유전자 세트를 인간 유전자 세트에서 검색하였다. 비동의 변이체를 갖는 유전자는 출판, 인구 빈도, 분자 결과 및 게놈 매핑과 함께 소규모 삽입 및 결실을 포함하는 단일 뉴클레오티드 다형성 데이터베이스인 dbSNP에서 검색되었다. 공통 변이는 적어도 하나의 주요 모집단에서 0.01 이상의 마이너 대립유전자 빈도 (MAF)를 갖고 NCBI에서 마이너 대립유전자를 갖는 적어도 두 명의 무관한 개인으로 정의되었다. 공통 변이에 대한 기준으로 0.1 이상의 MAF. 17번과 18번 염색체는 결장직장암에서 LOH가 높기 때문에 초점이 맞춰졌다. 위에서 기술한 바와 같이, 세포외 도메인에서 막 단백질, 결장 발현 및 일반적인 비동의 변이체에 대해 유전자를 필터링하였다. 검색 프로세스의 요약은 도 18에 나타낸다.
이 분석에 사용된 추가 데이터베이스는 다음과 같다: EMBL-EBI, SIB 및 PIR에서 호스팅하는 단백질 서열 및 주석 데이터에 대한 리소스로 사용되는, Uniprot (The Universal Protein Resource). GTEx (The Genotype-Tissue Expression)는 조직 특이적 유전자 발현 및 조절을 위한 공공 자원으로 사용되었다. 여기에는 거의 1000명의 개체에 걸쳐 54개의 질환이 없는 조직 부위의 샘플이 포함되어 있다. TCGA (The Cancer Genome Atlas)는 20,000개 이상의 원발성 암에 대한 자원으로 사용되었으며 33개 암 유형에 걸친 정상 샘플과 일치했다. TCGA-COADREAD 데이터 세트는 결장 선암종 및 직장 선암종 데이터 세트이다. CCLE (Cancer cell line Encyclopedia)는 57개의 결장직장암 (CRC) 세포주에 대한 정보를 포함한다.
RNASeqDB는 메모리얼 슬론 케터링 암 센터의 비교 연구를 허용하는 동일한 파이프라인을 사용하여 GTEx 및 TCGA에서 처리된 데이터의 데이터베이스이다. GTEx의 372개 TCGA-COADREAD 샘플 및 339개 정상 결장 샘플을 분석했다.
COLEC12, CXCL16 및 APCDD1은 잠재력 차단제 표적으로 식별되었다. 표 14는 결장직장암에서 이들 유전자에 대한 발현 데이터를 요약한 것이다. UCSC Xena 브라우저 (TCGA의 경우) 및 CCLE 샘플의 발현 데이터.
표 14. 발현
표 15는 변이체 및 마이너 대립유전자 빈도를 요약한 것이다.
표 15. 위치, 특성 및 변형
표 16. 다양한 암의 LOH 빈도
실시예 2: 차단제 리간드 결합 도메인의 식별
후보 차단제 항원에 대한 공개적으로 이용 가능한 항체는 CDR 서열이 알려지지 않은 경우 시퀀싱된다. 후보 차단제 표적에 대한 항체가 없는 경우, 정제된 단백질 (예를 들어, COLEC12, CXCL16 및 실시예에 설명된 다른 표적)로 마우스, 랫트 또는 토끼를 면역화하여 이러한 항체를 생성한다. 면역화된 동물의 혈청은 차단제 표적에 결합하기 위해 mAb를 스크리닝하는 데 사용된다. 차단제 표적에 대한 항체도 huTARG 시스템을 사용하여 생성된다. 그런 다음 원하는 특이성을 가진 항체를 분리하고 시퀀싱하여 CDR 서열을 결정한다.
차단제 표적에 대한 항체의 CDR 서열은 표준 분자 생물학 기술을 사용하여 scFv를 생성하는 데 사용된다. 후보 scFv는 억제 수용체 힌지 또는 막관통 도메인에 융합되어 표준 분자 생물학 기술을 사용하여 억제 수용체를 생성한다. 후보 scFv는 또한 활성화제 수용체 힌지 또는 막관통 도메인 (예를 들어, CAR)에 융합되어 표적 항원에 결합하는 scFv에 대한 양성 대조군으로서 사용하기 위한 전장 활성화제 수용체를 생성한다. 억제 수용체와 관련하여 작동하는 후보 scFv의 능력은 NFAT-루시퍼라제 리포터 분석을 사용하여 저캇 세포에서 분석된다.
실시예 3: 저캇 및 1차 T 세포 활성화 실험 방법
세포 배양
NFAT 루시퍼라제 리포터를 암호화하는 저캇 세포는 BPS Bioscience로부터 얻었다. 배양에서, 저캇 세포는 10% FBS, 1% Pen/Strep 및 0.4mg/mL G418/제네티신이 보충된 RPMI 배지에서 유지되었다. 이 연구에 사용된 다른 모든 세포주는 ATCC에서 구입했으며 ATCC에서 제안한 대로 유지했다.
저캇 세포 형질감염
저캇 세포는 다음 설정을 사용하여 제조업체의 프로토콜에 따라 100uL 형식 네온 전기천공 시스템 (Lonza)을 통해 일시적으로 형질감염되었다: 3 펄스, 1500V, 10 msec. 1e6 세포당 1-3ug의 활성화제 CAR 또는 TCR 작제물 및 1-3ug의 차단제 작제물 또는 빈 벡터로 동시형질감염을 수행하고 20% 열 불활성화 FBS 및 0.1% Pen/Strep이 보충된 RPMI 배지에서 회수했다.
저캇-NFAT-루시퍼라제 활성화 연구
저캇 세포를 10% 열 불활성화 FBS 및 0.1% Pen/Strep이 보충된 15uL의 RPMI에서 재현탁하고, 펩티드-로딩된 비드에 첨가하고 6시간 동안 공동 배양하였다. ONE-Step 루시퍼라제 분석 시스템 (BPS Bioscience)을 사용하여 저캇 발광을 평가했다. 검정은 기술적 반복으로 수행되었다.
1차 T 세포 형질도입, 확장 및 농축
동결 PBMC는 37℃ 수조에서 해동하고 1% 인간 혈청을 포함하는 LymphoONE (Takara)에서 1e6 세포/mL로 배양하고 300 IU/ml IL-2가 보충된 1:100의 T 세포 TransAct (Miltenyi)를 사용하여 활성화했다. 24시간 후, 렌티바이러스를 MOI 5에서 PBMC로 첨가하였다. PBMC를 2-3일 더 배양하여 세포가 TransAct 자극 하에 확장되도록 하였다. 확장 후, 활성화제 및 차단제로 형질도입된 1차 T 세포는 제조업체의 지침에 따라 항-PE 마이크로비드 (Miltenyi)를 사용하여 강화되었다. 간략하게, 1차 T 세포를 MACS 완충액 (0.5% BSA + PBS 중 2mM EDTA)에서 실온에서 30분 동안 1:25 희석으로 EGFR-Fc (R&D Systems)와 함께 배양하였다 세포를 MACS 완충액으로 3회 세척하고 MACS 완충액에서 실온에서 30분 동안 2차 항체 (1:1200)에서 배양하였다. 이어서 세포를 항-PE 마이크로비드에서 배양하고 LS 컬럼 (Miltenyi)을 통과시켰다.
1차 T 세포
시험관 내 세포독성 연구
pMHC 표적을 사용한 세포독성 연구를 위해, 농축된 1차 T 세포를 GFP 또는 RFP를 발현하는 HeLa 또는 HCT116 표적 세포와 함께 배양했다. "종양" 세포의 경우, WT HeLa 또는 HLA-A*02 녹아웃 HCT116이 사용되었고 "정상" 세포의 경우, HLA-A*02 형질도입된 HeLa 또는 WT HCT116이 사용되었다. 공동 배양은 IncuCyte 라이브 셀 이미저를 사용하여 이미지화되었고 표적 세포 형광 영역은 살아있는 표적 세포를 정량화하는 데 사용되었다.
실시예 4: HLA-A*02 억제 수용체는 저캇 세포의 EGFR-매개 활성화를 차단한다
EGFR 항원 결합 도메인 (예를 들어 CT479, CT486, 또는 CT489)을 갖는 활성화제 CAR을 발현하는 저캇 세포의 활성화를 차단하는 HLA-A-A*02 항원 결합 도메인 및 LIR-1 ICD (C1765 또는 C2162)를 갖는 억제 수용체의 능력은 이전에 설명한 대로 NFAT-루시퍼라제 리포터 시스템을 사용하여 분석하였다. EGFR+ 및 HLA-A*02-인, 정상 HeLa 종양 세포를 표적 세포로 사용하였다. EGFR+/ HLA-A*02- HeLa 세포는 또한 HLA-A*02+를 암호화하는 폴리뉴클레오티드로 형질도입되고 EGFR+ /HLA-A*02+ HeLa 세포를 생성하여 활성화제 및 차단제 항원을 모두 발현하는 표적 세포로 사용했다.
도 1a에 나타낸 바와 같이, EGFR CAR (CT479)를 발현하는 저캇 세포에서 HLA-A*02 LIR-1 차단제 (C1765)의 발현은 차단제를 발현하지 않는 저캇 세포의 CAR Emax와 비교하여 >5배만큼 CAR EMAX를 이동시킨다.
더욱이, 표적 세포에서 더 낮은 HLA-A2 발현 수준으로 더 낮은 차단이 관찰되었다. 정상 HCT116 세포는 EGFR+ 및 HLA-A*02+이다. EGFR 및 HLA-A*02의 수준은 항-EGFR 항체 및 항-HLA-A*02 항체 (BB7.2)를 사용하여 HLA-A*02 폴리뉴클레오티드를 암호화하는 폴리뉴클레오티드로 형질도입된 HCT116 세포 및 HeLa 세포에서 분석되고 FAC 정렬이 이어진다. 도 2a & 2b에 나타낸 바와 같이, HCT116 세포는 형질도입된 HeLa 세포보다 더 낮은 수준의 차단제 HLA-A*02 항원을 갖는다. EGFR CAR 및 HLA-A*02 LIR-1 차단제를 발현하는 저캇 세포가 EGFR 및 HLA-A*02 항원을 발현하는 HCT116 표적 세포와 함께 제시될 때, HLA-A*02 LIR-1 차단제의 존재는 EGFR CAR의 EMAX를 1.8배 이동시켰다(도 3b). 대조적으로, 더 높은 수준의 HLA-A*02 항원을 발현하는, 형질도입된 HeLa 세포는 >5배의 EGFR CAR EMAX 이동을 매개할 수 있었다 (도 1b, 도 17b, 및 도 3d). 대조군으로서, EGFR 녹아웃 HCT116 세포에 의한 최소 활성화가 있었다 (도 3a).
EGFR CAR 및 HLA-A*02 LIR-1 차단제를 사용하여 50% 차단을 달성하는 데 필요한 차단제 대 활성화제의 비율을 도 4a 및 도 4b에 나타낸, 비드 기반 시스템을 사용하여 검정하였다.
활성화제 항원의 EC50을 결정하기 위해, 활성화제 비드를 상이한 농도의 활성제 항원으로 코팅하여 EC50을 결정하였다. 각 농도별로 무관한 단백질을 첨가하여 총 단백질 농도가 동일하게 되도록 하였으며, EGFR CAR을 발현하는 저캇 효과기 세포에 일정한 양의 비드를 첨가하였다 (도 4a).
차단제 항원 IC50을 결정하기 위해, 비드를 EC50 농도 (도 4a에 결정됨)에서 활성화제 항원으로 코팅하고, 상이한 농도의 차단제 항원으로 코팅하였다. 총 단백질 농도가 동일하게 유지되도록 각 농도에서 무관한 단백질을 첨가하고, EGFR CAR 또는 EGFR CAR 및 HLA-A*02 LIR-1 차단제를 발현하는 저캇 효과기 세포에 일정한 양의 비드를 첨가했다 (도 4b).
실시예 5: 특정 수용체 쌍의 특성화
EGFR scFv CAR 활성화제 (도 5 및 7-9에 나타낸 바와 같은, CT-479, CT-482, CT-486, CT-487, CT-488, 또는 CT-489), 또는 EGFR scFv CAR 활성화제 및 HLA-A*02 PA2.1 scFv LIR1 억제제 (C1765 또는 C2162)로 형질감염된 T 세포를 HeLa 표적 세포와 공동 배양하였다. EGFR을 발현하지만 HLA-A*02를 발현하지 않는 정상 HeLa 세포주는 HLA-A*02 억제 수용체 표적을 발현하도록 형질도입되었다. 세포를 1:1 비율의 효과기 대 표적 (E:T)으로 공동 배양하였다. 도 5의 우측 아래에, 효과기 세포 수용체 발현이 먼저 표시되고, HeLa 세포 발현은 괄호 안에 표시된다. 도 5에 나타낸 바와 같이, 동일한 HLA-A*02 PA2.1 scFv LIR1 억제제를 상이한 EGFR 활성화제 수용체와 함께 사용했을 때 상이한 정도의 차단이 관찰되었다.
EGFR (표적 A) 또는 EGFR 및 HLA-A*02 (표적 AB)를 발현하는 HCT.116 세포와 공동 배양된 T 세포의 EGFR scFv CAR 활성화를 차단하는 HLA-A*02 PA2.1 scFv LIR1 억제제 (C1765)의 능력을 또한 검정하였다 (도 10). HeLa 세포에서와 같이, 동일한 HLA-A*02 PA2.1 scFv LIR1 억제제가 상이한 EGFR 활성화제 수용체와 함께 사용될 때 상이한 차단 정도가 관찰된다.
저캇 세포가 HeLa 표적 세포와 공동 배양되었을 때 저캇 세포의 EGFR scFv CAR 활성화를 차단하는 HLA-A*02 PA2.1 scFv LIR1 억제제 (C1765)의 능력을 또한 검정하였다. 결과를 도 2에 나타낸다. 1차 T 세포의 결과와 유사하게, 동일한 HLA-A*02 PA2.1 scFv LIR1 억제제가 상이한 EGFR 활성화제 수용체와 함께 사용되었을 때 상이한 차단 정도가 관찰되었다.
활성화 리간드만을 발현하는 표적 세포 또는 활성화제 리간드 및 차단 리간드를 모두를 발현하는 표적 세포의 존재 하에 활성화제 또는 특정 활성화제/차단제 쌍을 발현하는 면역 세포에서 면역 세포 활성화를 측정하였다 (도 6a). 선택성 비율 (SR)을 CAR 활성화제 및 차단제의 시험된 쌍에 대해 계산하였다. SR은 활성화 리간드만을 발현하는 표적 세포의 존재 하에 활성화제/차단제 쌍을 발현하는 면역 세포의 활성화 대 활성화제 리간드 및 차단 리간드 모두를 발현하는 표적 세포의 존재 하에 활성화제/차단제 쌍을 발현하는 면역 세포의 활성화의 비율을 계산하여 결정된다. 선택된 EGFR 표적화 CAR 활성화제는 인간화 차단제 (C2162) 또는 마우스 차단제 (C1765)와 한 쌍으로 발현되었다. 인간화 차단제와 페어링된 EGFR 표적화 CAR 활성화제 중에서, CT489에 대한 선택성 비율은 24.2에 도달했고; CT486에 대한 선택성 비율은 17에 도달했고; CT487에 대한 선택성 비율은 5.0에 도달했고; CT487에 대한 선택성 비율은 5.0에 도달했다. 마우스 차단제와 페어링된 EGFR 표적화 CAR 활성화제 중에서, CT489에 대한 선택성 비율은 21.6이었고; CT486에 대한 선택성 비율은 15.7이었고; CT487에 대한 선택성 비율은 18.4이었고; CT487에 대한 선택성 비율은 21.2였다 (도 6a). EGFR 표적화 활성화제 및 억제 수용체 쌍 각각에 대해 최대 특이적 사멸을 또한 결정하였다. 모든 페어링에 대해, 특이적 사멸은 약 60%를 초과했으며, 이는 차단제의 선택이 EGFR 표적화 활성화제 수용체에 의한 T 세포 활성화에 유의한 영향을 미치지 않았음을 나타낸다. 그러나 특정 활성화제 및 특정 차단제 쌍의 페어링은 선택성 비율에 영향을 미쳤으며, 이는 특정 활성화제/억제 수용체 쌍을 촉진하여 오프-타겟 사멸을 예측할 수 없음을 나타낸다.
활성화제 및 차단제 민감도는 CAR 활성화제 단독 또는 CAR 활성화제 및 인간화 차단제 또는 마우스 차단제를 발현하는 면역 세포에서 결정되었다 (도 6b). 활성화제 민감도를 결정하기 위해 면역 세포를 EGFR 음성 표적 세포의 존재 하에 배양하였다. 배양 전, 표적 세포를 EGFR mRNA 로 적정하여, EGFR 발현 수준을 증가시켰다 (도 6b, 왼쪽 패널). 차단제 민감도를 결정하기 위해 면역 세포를 EGFR을 발현하는 HLA-A*02 음성 표적 세포 (세포당 약 53,000개 EGFR 분자)의 존재 하에 배양하였다. 배양 전, 표적 세포를 HLA-A*02 mRNA로 적정하여, HLA-A*02 발현 수준을 증가시켰다 (도 6b, 오른쪽 패널). EC50 값을 상이한 CAR 활성화제와 조합된 각각의 CAR 활성화제 및 차단제에 대해 계산하였다 (표 5.1). 결과는 특정 활성화제 수용체가 억제 수용체 민감도에 영향을 미친다는 것을 추가로 확인한다.
표 5.1
실시예 6: 억제 수용체는 T 세포에서 활성화제 수용체의 표면 수준을 가역적으로 감소시킨다
2명의 HLA-A*02 음성 공여자로부터의 1차 T 세포를 EGFR scFv CAR 활성화제 (CT-479, CT-482, CT-486, CT-487 또는 CT-488) 및 HLA-A*02 PA2.1 scFv LIR1 억제제 (C1765)로 형질도입하였다. 형질도입된 세포는 차단제 및 활성화제 수용체에 대한 FACS 분류, 또는 차단제 및 활성화제 수용체에 대한 이중 컬럼 정제에 의해 강화되었다. 형질도입된 T 세포를 HeLa 표적 세포와 공동 배양하였다. 정상 HeLa 세포주는 EGFR을 발현하지만 HLA-A*02는 발현하지 않지만, HLA-A*02 억제 수용체 표적을 발현하도록 형질도입되었다. 세포는 효과기 대 표적 (E:T)의 1:1 비율로 공동 배양되었다. EGFR CAR 활성화제의 표면 발현을 활성화제 및 억제 수용체에 결합하는 표지된 펩티드, 및 형광 활성화 세포 분류를 사용하여 120시간 후에 검정하였다. 활성화제 및 차단제 리간드 모두를 발현하는 HeLa 세포와의 공동 배양 후 활성화제 표면 수준의 변화는 표적 세포를 사멸시키는 T 세포의 능력에 상응하였다 (도 5 및 도 11a 비교).
CT-482 EGFR scFv CAR 활성화제 및 HLA-A*02 PA2.1 scFv LIR1 억제제 (C1765) 조합을 발현하는 T 세포는 EGFR (표적 A), HLA-A*02 (표적 B), 동일한 세포에서 EGFR 및 HLA-A*02의 조합 (표적 AB), 상이한 세포에서 표적 A 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단, 또는 상이한 세포에서 표적 B 및 표적 AB를 발현하는 HeLa 세포의 혼합 집단을 발현하는 HeLa 세포와 공동 배양되었다 (도 12a-12b). T 세포는 1:1 효과기 세포 대 표적 세포의 비율로 HeLa 표적 세포와 배양되었다. T 세포가 HeLa 세포의 표적 A 플러스 표적 AB 집단과 공동 배양되었을 때, 활성화제의 수준이 감소한 후, 회복되었다 (도 12a). 더욱이, 활성화제 및 차단제 항원은 효과기 T 세포에서 활성화제 표면 발현 상실을 유발하기 위해 동일한 세포에 함께 존재해야 한다. 활성화제와 대조적으로, 차단제 발현은 크게 변하지 않았다 (도 12b).
도 13은 T 세포에 의한 세포독성, 차단 활성, 및 활성화제 수용체의 발현의 상실이 가역적인지 결정하기 위한 실험에 대한 개략도를 보여준다. EGFR scFv CAR 활성화제 수용체 (CT-487) 및 HLA-A*02 PA2.1 scFv LIR1 (C1765) 억제 수용체를 발현하는 T 세포는 활성화제 및 억제 수용체 표적 (AB) 둘 모두를 발현하는 HeLa 표적 세포와 공동 배양하였다. 3일 공동 배양 후, HeLa 세포를 항-EGFR 컬럼을 사용하여 제거하고, T 세포는 활성화제 및 억제 수용체에 대해 염색하거나, EGFR 활성화제 표적만을 발현하는 HeLa 세포와 추가 3일 동안 공동 배양했다. 추가 3일 공동 배양 후, HeLa 세포를 항-EGFR 컬럼을 사용하여 HeLa 세포를 다시 제거하고, T 세포는 활성화제 및 억제 수용체에 대해 염색하거나, 염색 전 EGFR 활성화제 표적만, 또는 활성화제 및 차단제 표적 (AB) 둘 모두 발현하는 HeLa 세포와 추가 3일 동안 공동 배양했다. T 세포를 표지된 EGFR 및 A2 프로브를 사용하여 활성화제 및 억제 수용체 (염색됨)의 존재에 대해 분석하고, 수용체 발현의 형광 활성화 세포 분류를 사용하여 정량화했다. 실험 결과는 도 14a-14b에 나타낸다. 도 14a-14b에 나타낸 바와 같이, T 세포의 활성화제 및 억제제 표적 모두를 발현하는 HeLa 세포와의 공동 배양은 EGFR 활성화제 염색을 감소시킨다 (도 14a-14b, 왼쪽 패널). 2라운드에서 활성화제를 발현하는 HeLa 세포와 공동 배양될 때 (표적 A 단독), EGFR 활성화제의 발현이 증가한다. 따라서, 활성화제 표면 소실은 가역적이며 T 세포 세포독성을 추적한다.
도 15a-15b는 활성화제 수용체 발현의 상실이 빠르게 가역적임을 보여주는 추가 실험의 결과를 보여준다. 활성화제 CAR 세포 표면 발현은 이들 T 세포가 활성화제 및 억제제 리간드 모두를 발현하는 모델 "정상" 세포와 공동 배양될 때 활성화제 및 억제 수용체 모두를 발현하는 T 세포에서 감소된다. 그러나 T 세포가 활성화제 리간드만 발현하는 모델 "암" 세포와의 공동 배양으로 전환되었을 때, T 세포는 활성화제 CAR의 세포 표면 발현을 신속하게 (몇 시간 내에) 복원할 수 있었다. 활성화제 및 억제제 리간드 모두를 발현하는 모델 "정상" 세포의 부재는 또한 종양 세포의 존재에 관계없이 유사한 시간 프레임 내에서, 활성화제 CAR의 세포 표면 발현을 회복시킬 수 있다.
실시예 7: HLA-A*02 LIR-1 기반 억제 수용체는 EGFR CAR 활성화제에 의한 활성화를 차단할 수 있다
EGFR CAR 활성화제 및 pMHC HLA-A*02 scFv LIR-1 기반 억제 수용체 (LIR-1 힌지, 막관통 및 ICD를 포함)를 발현하는 저캇 효과기 세포의 활성화를 실시예 3에 기술된 NFAT 루시퍼라제 검정을 사용하여 검정하였다.
저캇 세포를 활성화제 및 억제 수용체 DNA로 형질감염시키고, 활성화를 무세포 비드 기반 검정에서 검정하였다 (도 16). 비드에는 활성화제 항원, 차단제 항원, 또는 활성화제 및 억제제 항원이 로드되었으며, 비드 내 저캇 세포의 비율은 다양했다. 무세포 비드 기반 검정에서, HLA-A*02 scFv LIR-1 기반 억제 수용체는 세포가 시스로 HLA-A*02 차단제 및 EGFR 활성화제를 운반하는 비드와 접촉했을 때 저캇 세포의 활성화를 차단할 수 있었지만, 트랜스 (상이한 비드 상에서)로 HLA-A*02 차단제 및 EGFR 활성화제를 운반하는 경우에는 그렇지 않는다. 비드 상의 HLA-A*02 차단제의 존재는 EGFR CAR의 EMAX를 9X 이상으로 이동시킬 수 있었다 (도 16).
EGFR CAR 활성화제 및 HLA-A*02 scFv LIR-1 기반 억제 수용체를 발현하는 저캇 세포의 활성화를 또한 표적 세포로서 HeLa 및 SiHa 세포를 사용하여 검정하였다. 정상 HeLa 및 SiHa 세포주는 EGFR을 발현하지만 HLA-A*02 (SiHa WT 및 HeLa WT)는 발현하지 않지만, HLA-A*02 억제 수용체 표적 (SiHa A02 및 HeLa A02)을 발현하도록 형질도입되었다. 도 17a 및 도 17b에서 볼 수 있는 바와 같이, HLA-A*02 scFv LIR-1 기반 억제 수용체는 EGFR EMAX를 SiHa 표적 세포를 사용하여 4X 이상 (도 17a) 및 HeLa 표적 세포를 사용하여 5X 이상 이동시킬 수 있었다 (도 17b).
실시예 8: EGFR CAR 활성화제 및 억제 수용체 발현 T 세포의 생체 내 효능
활성화제 수용체 (EGFR CAR) 및 억제 수용체 (차단제) 쌍을 발현하는 면역 세포에 대한 효능 및 종양 대 정상 세포 선택성은 생체 내 환경에서 확립된다 (도 19). "정상" 세포를 대표하는 인간 EGFR+/HLA-A*02+ HeLa 세포 및 "종양" 세포로서 동질유전자 EGFR+/HLA-A*02(-) 녹아웃 HeLa 세포를 사용하는 NSG (NOD.Cg- PrkdcscidIl2rgtm1Wjl/SzJ)에서 마우스 이종이식 모델. 생체 내 치료 창은 정상 대 종양 세포에 대한 활성화제/억제 수용체 쌍의 효과를 비교함으로써 확립된다. 이러한 데이터는 온-타겟, 오프-종양 치료 창에 대한 생체 내 지원을 제공한다 활성화제/억제 수용체 쌍에 대한 최적 용량이 설정되고 민감도 및 선택성에 대해 최적화된다.
생체 내 모델은 오른쪽 측면 "정상" 세포 및 왼쪽 측면 "종양" 세포의 피하 이종이식을 포함한다. 활성화제 수용체, 활성화제/억제 수용체 쌍을 발현하는 면역 세포, 또는 형질도입되지 않은 대조군 세포를 일정 범위의 시점에서 동물에게 투여한다.
임상 관찰 및 체중에 대한 영향을 통해 동물의 전반적인 건강 상태를 모니터링한다. 종양 이종이식은 캘리퍼 및 이미징에 의해 크기에 대해 평가된다. 각 시점에서, 인간 T 세포는 유동 세포측정법에 의해 열거되고 CD3, CD4, CD8, 활성화제 수용체 발현, 및 억제 수용체 발현을 포함하는 표면 마커에 대해 평가된다. IFNγ 및 인터루킨-2 (IL-2)를 포함하는 혈청 사이토카인 수준이 결정된다. 사후 분석에서, 주사 부위와 종양을 포함한 프로토콜 지정 조직은 ACVP 이사회 인증 병리학자에 의해 종양 침윤 T-세포 및 활성화제 수용체 항원 (예를 들어 EGFR) 및 차단제 항원 발현 (예를 들어 HLA-A*02)에 대한 조직학에 의해 평가된다.
실시예 9: B7 및 HLA-A*11은 억제 수용체 리간드이다
HLA-A*011 억제 수용체는 scFv #4 (표)를 힌지, TM 및 LIR-1로부터 유래된 ICD에 융합하여 만들었다. 유전자 세그먼트는 골든 게이트 클로닝을 사용하여 결합되었고 렌티바이러스 발현 플라스미드에 포함된 인간 EF1알파 프로모터의 다운스트림에 삽입되었다.
저캇 NFAT-루시퍼라제 효과기 세포는 CAR 활성화제, 또는 CAR 활성화제 및 scFv #4 HLA-A*11 억제 (차단제) 수용체로 상기 기술된 바와 같이 형질전환되었다. HLA-A*011 억제 수용체의 서열은 하기 표 9.1에 나타내고, 활성화제 리간드 및 HLA-A*011:01을 발현하는 HeLa 세포와 공동 배양했다. NFAT-루시퍼라제 검정은 T2 표적 세포 대신에 HeLa를 사용하여 수행되었다.
결과를 도 20a 및 도 20b에 나타낸다. 도 20a에 나타낸 바와 같이, LIR-1의 힌지, TM 및 ICD 도메인에 융합될 때, scFv #4는 활성화제 리간드 및 HLA-A*011을 모두 발현하는 표적 세포와 공동 배양된 저캇 효과기 세포의 활성화를 억제할 수 있다.
표 9.1. HLA-A*011 억제 수용체 서열.
저캇 NFAT 루시퍼라제 (JNL) 세포 활성화는 mRNA로 형질감염된 HeLa 세포를 사용하여 mRNA 적정 분석에서 측정되었다. HeLa 세포는 HLA-B*07:02를 암호화하는 연속적으로 희석된 mRNA로 형질감염시켰다. JNL 세포는 EGFR 활성화 CAR 및 HLA-B*07 차단제 (원) 또는 음성 대조군 벡터 (삼각형)로 일시적으로 형질감염시켰다. 기능적 반응은 6시간 공동 배양 후 평가되었다. 도 21에 나타낸 결과는 HLA-B*07 차단제가 적정된 HLA-B*07:02의 존재 하에 EGFR 표적화 CAR의 활성화를 차단하지만 차단제의 부재 하에서는 차단하지 않음을 입증한다.
실시예 10: 1차 T 세포에서 EGFR 활성화제 및 HLA-A*02 억제 수용체 발현의 특성화
EGFR 활성화제를 염색하기 위해, 1차 T 세포를 FACS 완충액 (PBS 중 1% BSA)에서 실온에서 30분 동안 1:25 희석으로 EGFR-Fc (R&D Systems)와 함께 배양했다. 세포를 FACS 완충액으로 2회 세척하고 FACS 완충액에서 실온에서 30분 동안 항-Fc (1:100)에서 배양했다. HLA-A*02 억제 수용체를 염색하기 위해, T 세포를 FACS 완충액에서 실온에서 30분 동안 HLA-A*02 사량체 프로브와 배양했다. 수용체 발현은 염색 후 유동 세포측정법으로 분석하였다.
1차 T 세포는 EGFR 표적화 활성화제 수용체 및 인간 또는 마우스 HLA-A*02 표적화 억제 수용체를 암호화하는 폴리뉴클레오티드로 형질도입되었다. 여러 특정 쌍의 EGFR 표적화 활성화제 및 HLA-A*02 표적화 억제 수용체를 시험했다. 형질도입은 활성화제 수용체 및 억제 수용체를 모두 암호화하는 단일 벡터 또는 2개의 벡터, 활성화제 수용체를 암호화하는 하나 및 억제 수용체를 암호화하는 하나로 수행했다. 활성화제 및 억제제 수용체 모두를 발현하는 세포의 백분율을 결정하기 위해 농축이 있거나 없는 세포를 분석하였다 (도 22).
2개의 벡터, 활성화제 수용체를 암호화하는 하나 및 억제 수용체를 암호화하는 하나로 형질도입된 1차 T 세포에서, 두 수용체를 모두 발현하는 형질도입된 T 세포 집단에서 세포의 백분율은 농축 전에 약 22.5% (도 22a, 왼쪽 패널)였다. 농축 후, 두 수용체를 모두 발현하는 형질도입된 T 세포 집단의 세포 백분율은 약 91.6%로 증가했다.
활성화제 수용체 및 억제 수용체를 모두 암호화하는 단일 벡터로 형질도입된 1차 T 세포에서, 수용체를 모두 발현하는 형질도입된 T 세포 집단에서 세포의 백분율은 농축 전에 약 6.29% 내지 13.2% 범위였다. 농축 후, 두 수용체를 모두 발현하는 세포의 비율은 22.6% 내지 82.8% 범위였다. 두 수용체를 모두 발현하는 세포의 백분율은 특정 활성화제 및 억제 수용체 쌍에 따라 다르다.
실시예 11: 활성화제 및 억제 수용체를 발현하는 1차 T 세포에서 면역 세포 활성화 선택성의 특성화
선택적 세포독성 연구를 위해, 농축된 1차 T 세포를 GFP 또는 RFP를 발현하는 HeLa 또는 HCT116 표적 세포와 함께 배양했다. "종양" 세포의 경우, WT HeLa 또는 HLA-A*02 녹아웃 HCT116이 사용되었고 "정상" 세포의 경우, HLA-A*02 형질도입된 HeLa 또는 WT HCT116이 사용되었다. 공배양물은 IncuCyte 라이브 세포 이미저를 사용하여 이미지화되었고 표적 세포 형광 영역은 살아있는 표적 세포를 정량화하는 데 사용되었다 (도 23a). 혼합 표적 배양에서 선택적 세포독성을 측정하기 위해, "종양" WT HeLa 세포를 발현하는 RFP 및 HLA-A*02로 형질도입된 "정상" HeLa 세포를 발현하는 GFP를 1:1 비율로 혼합했다. 농축된 1차 T 세포를 혼합 표적 배양물에 첨가하고 IncuCyte 라이브 세포 이미저를 사용하여 이미지화하고 표적 세포 영역을 사용하여 살아있는 표적 세포를 정량화하였다 (도 23b).
도 23a는 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 HeLa 표적 세포 또는 HCT116 표적 세포의 특이적 사멸 백분율을 나타내는 일련의 플롯이다. 표적 세포는 EGFR 항원 (A), HLA-A*02 비-표적 항원 (B), 또는 둘 다 (AB)를 발현한다. 활성화제/억제제 수용체 쌍을 발현하는 면역 세포를 1:1 비율로 표적 세포와 함께 배양하였다.
도 23b는 암 세포 및 정상 세포의 혼합 배양에서 EGFR 활성화제 수용체 및 HLA-A*02 scFv LIR1 억제제 수용체를 발현하는 면역 세포에 의한 표적 세포의 특이적이고 선택적인 사멸을 나타내는 플롯 및 일련의 이미지이다. UTD = 활성화제 또는 억제제 수용체가 없는 면역 세포; CAR = 활성화제 수용체만 발현하는 면역 세포; Tmod = 활성화제 및 억제제 수용체 모두를 발현하는 면역 세포.
실시예 12: 활성화제 및 억제 수용체를 발현하는 저캇 세포에서 면역 세포 활성화 선택성의 특성화
도 24는 EGFR 활성화제 수용체 (CAR) 또는 EGFR 활성화제 수용체 및 HLA-A*02 억제제 수용체를 발현하는 저캇 세포 (효과기)에서 면역 세포 활성화를 나타내는 차트이다 (Tmod). 저캇 세포는 EGFR 발현 없이 HLA-A*02를 발현하거나 EGFR 발현과 함께 HLA-A*02를 발현하는 표적 세포주의 패널과 함께 배양되었다. EGFR 음성 세포주는 CRISPR/Cas9를 사용하여 EGFR을 녹아웃하여 생성되었다. Tmod 저캇 효과기 세포는 EGFR 음성 세포와 함께 배양했을 때 오프-타겟 활성화를 나타내지 않았다.
SEQUENCE LISTING
<110> A2 BIOTHERAPEUTICS, INC.
<120> COMPOSITIONS AND METHODS FOR TREATING EGFR POSITIVE CANCERS
<130> A2BI-021/02WO 331656-2067
<150> US 63/230,632
<151> 2021-08-06
<150> US 63/105,639
<151> 2020-10-26
<150> US 63/068,249
<151> 2020-08-20
<160> 919
<170> PatentIn version 3.5
<210> 1
<211> 1210
<212> PRT
<213> Homo sapiens
<400> 1
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
<210> 2
<211> 628
<212> PRT
<213> Homo sapiens
<400> 2
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Ser
625
<210> 3
<211> 405
<212> PRT
<213> Homo sapiens
<400> 3
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Leu Ser
405
<210> 4
<211> 705
<212> PRT
<213> Homo sapiens
<400> 4
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Pro Gly Asn Glu Ser Leu Lys Ala Met Leu Phe Cys Leu
625 630 635 640
Phe Lys Leu Ser Ser Cys Asn Gln Ser Asn Asp Gly Ser Val Ser His
645 650 655
Gln Ser Gly Ser Pro Ala Ala Gln Glu Ser Cys Leu Gly Trp Ile Pro
660 665 670
Ser Leu Leu Pro Ser Glu Phe Gln Leu Gly Trp Gly Gly Cys Ser His
675 680 685
Leu His Ala Trp Pro Ser Ala Ser Val Ile Ile Thr Ala Ser Ser Cys
690 695 700
His
705
<210> 5
<211> 1091
<212> PRT
<213> Homo sapiens
<400> 5
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Gly Gln Lys
130 135 140
Cys Asp Pro Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu
145 150 155 160
Asn Cys Gln Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly
165 170 175
Arg Cys Arg Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala
180 185 190
Ala Gly Cys Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys
195 200 205
Phe Arg Asp Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu
210 215 220
Tyr Asn Pro Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr
225 230 235 240
Ser Phe Gly Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val
245 250 255
Thr Asp His Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu
260 265 270
Met Glu Glu Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys
275 280 285
Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu
290 295 300
Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile
305 310 315 320
Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe
325 330 335
Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr
340 345 350
Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn
355 360 365
Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg
370 375 380
Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile
385 390 395 400
Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val
405 410 415
Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp
420 425 430
Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn
435 440 445
Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu
450 455 460
Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser
465 470 475 480
Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu
485 490 495
Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln
500 505 510
Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly
515 520 525
Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro
530 535 540
His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr
545 550 555 560
Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His
565 570 575
Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro
580 585 590
Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala
595 600 605
Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg
610 615 620
Arg Arg His Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu
625 630 635 640
Arg Glu Leu Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln
645 650 655
Ala Leu Leu Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val
660 665 670
Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro
675 680 685
Glu Gly Glu Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu
690 695 700
Ala Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val
705 710 715 720
Met Ala Ser Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys
725 730 735
Leu Thr Ser Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys
740 745 750
Leu Leu Asp Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr
755 760 765
Leu Leu Asn Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu
770 775 780
Asp Arg Arg Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val
785 790 795 800
Lys Thr Pro Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu
805 810 815
Leu Gly Ala Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro
820 825 830
Ile Lys Trp Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His
835 840 845
Gln Ser Asp Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr
850 855 860
Phe Gly Ser Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser
865 870 875 880
Ile Leu Glu Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile
885 890 895
Asp Val Tyr Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser
900 905 910
Arg Pro Lys Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg
915 920 925
Asp Pro Gln Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu
930 935 940
Pro Ser Pro Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu
945 950 955 960
Asp Met Asp Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln
965 970 975
Gly Phe Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser
980 985 990
Leu Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
995 1000 1005
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1010 1015 1020
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1025 1030 1035
Asp Thr Phe Leu Pro Val Pro Gly Glu Trp Leu Val Trp Lys Gln
1040 1045 1050
Ser Cys Ser Ser Thr Ser Ser Thr His Ser Ala Ala Ala Ser Leu
1055 1060 1065
Gln Cys Pro Ser Gln Val Leu Pro Pro Ala Ser Pro Glu Gly Glu
1070 1075 1080
Thr Val Ala Asp Leu Gln Thr Gln
1085 1090
<210> 6
<211> 1136
<212> PRT
<213> Homo sapiens
<400> 6
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Gly Glu Trp Leu Val Trp Lys Gln
1085 1090 1095
Ser Cys Ser Ser Thr Ser Ser Thr His Ser Ala Ala Ala Ser Leu
1100 1105 1110
Gln Cys Pro Ser Gln Val Leu Pro Pro Ala Ser Pro Glu Gly Glu
1115 1120 1125
Thr Val Ala Asp Leu Gln Thr Gln
1130 1135
<210> 7
<211> 1157
<212> PRT
<213> Homo sapiens
<400> 7
Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn Leu Glu Ile Thr Tyr
1 5 10 15
Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys Thr Ile Gln Glu Val
20 25 30
Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val Glu Arg Ile Pro Leu
35 40 45
Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr Tyr Glu Asn Ser Tyr
50 55 60
Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn Lys Thr Gly Leu Lys
65 70 75 80
Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu His Gly Ala Val Arg
85 90 95
Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu Ser Ile Gln Trp Arg
100 105 110
Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met Ser Met Asp Phe Gln
115 120 125
Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro Ser Cys Pro Asn Gly
130 135 140
Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln Lys Leu Thr Lys Ile
145 150 155 160
Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg Gly Lys Ser Pro Ser
165 170 175
Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys Thr Gly Pro Arg Glu
180 185 190
Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp Glu Ala Thr Cys Lys
195 200 205
Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro Thr Thr Tyr Gln Met
210 215 220
Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly Ala Thr Cys Val Lys
225 230 235 240
Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His Gly Ser Cys Val Arg
245 250 255
Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu Asp Gly Val Arg Lys
260 265 270
Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val Cys Asn Gly Ile Gly
275 280 285
Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys
290 295 300
His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro
305 310 315 320
Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro
325 330 335
Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu
340 345 350
Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu
355 360 365
Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser
370 375 380
Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu
385 390 395 400
Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu
405 410 415
Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly
420 425 430
Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala
435 440 445
Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly
450 455 460
Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg
465 470 475 480
Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe
485 490 495
Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln
500 505 510
Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln
515 520 525
Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala
530 535 540
Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala
545 550 555 560
Gly His Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr
565 570 575
Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser
580 585 590
Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala
595 600 605
Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His Ile Val Arg Lys Arg
610 615 620
Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu Val Glu Pro Leu Thr
625 630 635 640
Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu Arg Ile Leu Lys Glu
645 650 655
Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser Gly Ala Phe Gly Thr
660 665 670
Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu Lys Val Lys Ile Pro
675 680 685
Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser Pro Lys Ala Asn Lys
690 695 700
Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser Val Asp Asn Pro His
705 710 715 720
Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser Thr Val Gln Leu Ile
725 730 735
Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp Tyr Val Arg Glu His
740 745 750
Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn Trp Cys Val Gln Ile
755 760 765
Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg Leu Val His Arg Asp
770 775 780
Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro Gln His Val Lys Ile
785 790 795 800
Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala Glu Glu Lys Glu Tyr
805 810 815
His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp Met Ala Leu Glu Ser
820 825 830
Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp Val Trp Ser Tyr Gly
835 840 845
Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser Lys Pro Tyr Asp Gly
850 855 860
Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu Lys Gly Glu Arg Leu
865 870 875 880
Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr Met Ile Met Val Lys
885 890 895
Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys Phe Arg Glu Leu Ile
900 905 910
Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln Arg Tyr Leu Val Ile
915 920 925
Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro Thr Asp Ser Asn Phe
930 935 940
Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp Asp Val Val Asp Ala
945 950 955 960
Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe Phe Ser Ser Pro Ser Thr
965 970 975
Ser Arg Thr Pro Leu Leu Ser Ser Leu Ser Ala Thr Ser Asn Asn Ser
980 985 990
Thr Val Ala Cys Ile Asp Arg Asn Gly Leu Gln Ser Cys Pro Ile Lys
995 1000 1005
Glu Asp Ser Phe Leu Gln Arg Tyr Ser Ser Asp Pro Thr Gly Ala
1010 1015 1020
Leu Thr Glu Asp Ser Ile Asp Asp Thr Phe Leu Pro Val Pro Glu
1025 1030 1035
Tyr Ile Asn Gln Ser Val Pro Lys Arg Pro Ala Gly Ser Val Gln
1040 1045 1050
Asn Pro Val Tyr His Asn Gln Pro Leu Asn Pro Ala Pro Ser Arg
1055 1060 1065
Asp Pro His Tyr Gln Asp Pro His Ser Thr Ala Val Gly Asn Pro
1070 1075 1080
Glu Tyr Leu Asn Thr Val Gln Pro Thr Cys Val Asn Ser Thr Phe
1085 1090 1095
Asp Ser Pro Ala His Trp Ala Gln Lys Gly Ser His Gln Ile Ser
1100 1105 1110
Leu Asp Asn Pro Asp Tyr Gln Gln Asp Phe Phe Pro Lys Glu Ala
1115 1120 1125
Lys Pro Asn Gly Ile Phe Lys Gly Ser Thr Ala Glu Asn Ala Glu
1130 1135 1140
Tyr Leu Arg Val Ala Pro Gln Ser Ser Glu Phe Ile Gly Ala
1145 1150 1155
<210> 8
<211> 943
<212> PRT
<213> Homo sapiens
<400> 8
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Gly Asn Tyr
20 25 30
Val Val Thr Asp His Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser
35 40 45
Tyr Glu Met Glu Glu Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly
50 55 60
Pro Cys Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp
65 70 75 80
Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr
85 90 95
Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp
100 105 110
Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu
115 120 125
Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro
130 135 140
Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg
145 150 155 160
Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu
165 170 175
Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly
180 185 190
Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile
195 200 205
Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile
210 215 220
Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His
225 230 235 240
Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys
245 250 255
Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys
260 265 270
Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys
275 280 285
Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys
290 295 300
Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp
305 310 315 320
Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn
325 330 335
Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu
340 345 350
Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly
355 360 365
Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val
370 375 380
Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe
385 390 395 400
Met Arg Arg Arg His Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu
405 410 415
Gln Glu Arg Glu Leu Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro
420 425 430
Asn Gln Ala Leu Leu Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile
435 440 445
Lys Val Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp
450 455 460
Ile Pro Glu Gly Glu Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu
465 470 475 480
Arg Glu Ala Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala
485 490 495
Tyr Val Met Ala Ser Val Asp Asn Pro His Val Cys Arg Leu Leu Gly
500 505 510
Ile Cys Leu Thr Ser Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe
515 520 525
Gly Cys Leu Leu Asp Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser
530 535 540
Gln Tyr Leu Leu Asn Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr
545 550 555 560
Leu Glu Asp Arg Arg Leu Val His Arg Asp Leu Ala Ala Arg Asn Val
565 570 575
Leu Val Lys Thr Pro Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala
580 585 590
Lys Leu Leu Gly Ala Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys
595 600 605
Val Pro Ile Lys Trp Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr
610 615 620
Thr His Gln Ser Asp Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu
625 630 635 640
Met Thr Phe Gly Ser Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile
645 650 655
Ser Ser Ile Leu Glu Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys
660 665 670
Thr Ile Asp Val Tyr Met Ile Met Val Lys Cys Trp Met Ile Asp Ala
675 680 685
Asp Ser Arg Pro Lys Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met
690 695 700
Ala Arg Asp Pro Gln Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met
705 710 715 720
His Leu Pro Ser Pro Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp
725 730 735
Glu Glu Asp Met Asp Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro
740 745 750
Gln Gln Gly Phe Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu
755 760 765
Ser Ser Leu Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp
770 775 780
Arg Asn Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln
785 790 795 800
Arg Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
805 810 815
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro Lys
820 825 830
Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln Pro Leu
835 840 845
Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro His Ser Thr
850 855 860
Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln Pro Thr Cys Val
865 870 875 880
Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala Gln Lys Gly Ser His
885 890 895
Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln Gln Asp Phe Phe Pro Lys
900 905 910
Glu Ala Lys Pro Asn Gly Ile Phe Lys Gly Ser Thr Ala Glu Asn Ala
915 920 925
Glu Tyr Leu Arg Val Ala Pro Gln Ser Ser Glu Phe Ile Gly Ala
930 935 940
<210> 9
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-478 EGFR (VH-VL scFv Format)
<400> 9
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Asp Asp Gly Ser Tyr Lys Tyr Tyr Gly Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Ile Thr Met Val Arg Gly Val Met Lys Asp Tyr Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Ala Ile
130 135 140
Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Ser Ala Leu Val
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp
180 185 190
Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Glu
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 10
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-479 EGFR (VL-VH scFv Format)
<400> 10
Ala Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Ser Ala
20 25 30
Leu Val Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Glu Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Val Gln Leu
115 120 125
Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg Ser Leu Arg Leu
130 135 140
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr Gly Met His Trp
145 150 155 160
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Val Ile Trp
165 170 175
Asp Asp Gly Ser Tyr Lys Tyr Tyr Gly Asp Ser Val Lys Gly Arg Phe
180 185 190
Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn
195 200 205
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Gly
210 215 220
Ile Thr Met Val Arg Gly Val Met Lys Asp Tyr Phe Asp Tyr Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 11
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-480 EGFR (VH-VL scFv format)
<400> 11
Gln Ile Gln Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Gly Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Glu Tyr
20 25 30
Pro Ile His Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Trp Met
35 40 45
Gly Met Ile Tyr Thr Asp Ile Gly Lys Pro Thr Tyr Ala Glu Glu Phe
50 55 60
Lys Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Val Arg Asp Arg Tyr Asp Ser Leu Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Val Val Met Thr Gln Thr Pro Leu
130 135 140
Ser Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu His Trp Tyr
165 170 175
Leu Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly
210 215 220
Val Tyr Phe Cys Ser Gln Ser Thr His Val Pro Trp Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Glu Ile Lys
245
<210> 12
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-481 EGFR (VL-VH scFv Format)
<400> 12
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu His Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Thr His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gln Ile Gln Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Gly
130 135 140
Glu Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Glu
145 150 155 160
Tyr Pro Ile His Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Trp
165 170 175
Met Gly Met Ile Tyr Thr Asp Ile Gly Lys Pro Thr Tyr Ala Glu Glu
180 185 190
Phe Lys Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala
195 200 205
Tyr Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Thr Ala Thr Tyr Phe
210 215 220
Cys Val Arg Asp Arg Tyr Asp Ser Leu Phe Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Thr Leu Thr Val Ser Ser
245
<210> 13
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-482 EGFR (VH-VL scFv Format)
<400> 13
Glu Met Gln Leu Val Glu Ser Gly Gly Gly Phe Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Ala Phe Ser His Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Thr Pro Lys Gln Arg Leu Glu Trp Val
35 40 45
Ala Tyr Ile Ala Ser Gly Gly Asp Ile Thr Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Gln Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Ser Ser Leu Lys Ser Glu Asp Thr Ala Met Phe Tyr Cys
85 90 95
Ser Arg Ser Ser Tyr Gly Asn Asn Gly Asp Ala Leu Asp Phe Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Val Val Met Thr Gln
130 135 140
Thr Pro Leu Ser Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser
145 150 155 160
Cys Arg Ser Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu
165 170 175
His Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr
180 185 190
Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu
210 215 220
Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser Thr His Val Leu Thr Phe
225 230 235 240
Gly Ser Gly Thr Lys Leu Glu Ile Lys
245
<210> 14
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-483 EGFR (VL-VH scFv Format)
<400> 14
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu His Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Thr His Val Leu Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Gly
100 105 110
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Glu Met Gln Leu Val Glu Ser Gly Gly Gly Phe Val Lys Pro Gly Gly
130 135 140
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Ala Phe Ser His Tyr
145 150 155 160
Asp Met Ser Trp Val Arg Gln Thr Pro Lys Gln Arg Leu Glu Trp Val
165 170 175
Ala Tyr Ile Ala Ser Gly Gly Asp Ile Thr Tyr Tyr Ala Asp Thr Val
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Gln Asn Thr Leu Tyr
195 200 205
Leu Gln Met Ser Ser Leu Lys Ser Glu Asp Thr Ala Met Phe Tyr Cys
210 215 220
Ser Arg Ser Ser Tyr Gly Asn Asn Gly Asp Ala Leu Asp Phe Trp Gly
225 230 235 240
Gln Gly Thr Ser Val Thr Val Ser Ser
245
<210> 15
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-486 EGFR (VH-VL scFv Format)
<400> 15
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Leu Leu Thr Gln Ser Pro
130 135 140
Val Ile Leu Ser Val Ser Pro Gly Glu Arg Val Ser Phe Ser Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Gly Thr Asn Ile His Trp Tyr Gln Gln Arg Thr
165 170 175
Asn Gly Ser Pro Arg Leu Leu Ile Lys Tyr Ala Ser Glu Ser Ile Ser
180 185 190
Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Ser Ile Asn Ser Val Glu Ser Glu Asp Ile Ala Asp Tyr Tyr Cys
210 215 220
Gln Gln Asn Asn Asn Trp Pro Thr Thr Phe Gly Ala Gly Thr Lys Leu
225 230 235 240
Glu Leu Lys
<210> 16
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-487 EGFR (VH-VL scFv Format)
<400> 16
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser His
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Glu Phe Asn Pro Ser Asn Gly Arg Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Ser Lys Ala Thr Met Thr Val Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Arg Asp Tyr Asp Tyr Asp Gly Arg Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Ser Ala Ser Ser Ser Val Thr Tyr Met Tyr Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp Thr Ser Asn Leu Ala
180 185 190
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr
195 200 205
Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Trp Ser Ser His Ile Phe Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 17
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-488 EGFR (VH-VL scFv Format)
<400> 17
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Thr Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly His Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Ile Asp Thr Ser Lys Thr Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Ile Tyr Tyr
85 90 95
Cys Val Arg Asp Arg Val Thr Gly Ala Phe Asp Ile Trp Gly Gln Gly
100 105 110
Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln
145 150 155 160
Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp Ala Ser Asn Leu Glu Thr
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Phe Cys
210 215 220
Gln His Phe Asp His Leu Pro Leu Ala Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 18
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> CT-489 EGFR scFv
<400> 18
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asp Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Met Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Val Ser Ile Phe Gly Val Gly Thr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Met Thr Gln
130 135 140
Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser
145 150 155 160
Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Asp Ala Ser Asn Arg
180 185 190
Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr
210 215 220
Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Ala Glu Ile Lys
245
<210> 19
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT478, CT479
<400> 19
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Asp Asp Gly Ser Tyr Lys Tyr Tyr Gly Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Ile Thr Met Val Arg Gly Val Met Lys Asp Tyr Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 20
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT480, CT481
<400> 20
Gln Ile Gln Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Gly Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Glu Tyr
20 25 30
Pro Ile His Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Trp Met
35 40 45
Gly Met Ile Tyr Thr Asp Ile Gly Lys Pro Thr Tyr Ala Glu Glu Phe
50 55 60
Lys Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Val Arg Asp Arg Tyr Asp Ser Leu Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser
115
<210> 21
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT482, CT483
<400> 21
Glu Met Gln Leu Val Glu Ser Gly Gly Gly Phe Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Ala Phe Ser His Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Thr Pro Lys Gln Arg Leu Glu Trp Val
35 40 45
Ala Tyr Ile Ala Ser Gly Gly Asp Ile Thr Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Gln Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Ser Ser Leu Lys Ser Glu Asp Thr Ala Met Phe Tyr Cys
85 90 95
Ser Arg Ser Ser Tyr Gly Asn Asn Gly Asp Ala Leu Asp Phe Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 22
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT486
<400> 22
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 23
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT487
<400> 23
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser His
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Glu Phe Asn Pro Ser Asn Gly Arg Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Ser Lys Ala Thr Met Thr Val Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Arg Asp Tyr Asp Tyr Asp Gly Arg Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 24
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT488
<400> 24
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Thr Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly His Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Ile Asp Thr Ser Lys Thr Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Ile Tyr Tyr
85 90 95
Cys Val Arg Asp Arg Val Thr Gly Ala Phe Asp Ile Trp Gly Gln Gly
100 105 110
Thr Met Val Thr Val Ser Ser
115
<210> 25
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT478, CT479
<400> 25
Ala Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Ser Ala
20 25 30
Leu Val Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Glu Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 26
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT480, CT481
<400> 26
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu His Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Thr His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 27
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT482, CT483
<400> 27
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu His Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Thr His Val Leu Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 28
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT486
<400> 28
Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn
20 25 30
Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile
35 40 45
Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser
65 70 75 80
Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 29
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT487
<400> 29
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Val Thr Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser His Ile Phe Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 30
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT488
<400> 30
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln His Phe Asp His Leu Pro Leu
85 90 95
Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 31
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 31
Thr Tyr Gly Met His
1 5
<210> 32
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 32
Glu Tyr Pro Ile His
1 5
<210> 33
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 33
His Tyr Asp Met Ser
1 5
<210> 34
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 34
Asn Tyr Gly Val His
1 5
<210> 35
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 35
Ser His Trp Met His
1 5
<210> 36
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 36
Ser Gly Asp Tyr Tyr Trp Thr
1 5
<210> 37
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 37
Val Ile Trp Asp Asp Gly Ser Tyr Lys Tyr Tyr Gly Asp Ser Val Lys
1 5 10 15
Gly
<210> 38
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 38
Met Ile Tyr Thr Asp Ile Gly Lys Pro Thr Tyr Ala Glu Glu Phe Lys
1 5 10 15
Gly
<210> 39
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 39
Tyr Ile Ala Ser Gly Gly Asp Ile Thr Tyr Tyr Ala Asp Thr Val Lys
1 5 10 15
Gly
<210> 40
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 40
Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr Ser
1 5 10 15
<210> 41
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 41
Glu Phe Asn Pro Ser Asn Gly Arg Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Ser
<210> 42
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 42
His Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 43
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 43
Asp Gly Ile Thr Met Val Arg Gly Val Met Lys Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 44
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 44
Asp Arg Tyr Asp Ser Leu Phe Asp Tyr
1 5
<210> 45
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 45
Ser Ser Tyr Gly Asn Asn Gly Asp Ala Leu Asp Phe
1 5 10
<210> 46
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 46
Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr
1 5 10
<210> 47
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 47
Arg Asp Tyr Asp Tyr Asp Gly Arg Tyr Phe Asp Tyr
1 5 10
<210> 48
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 48
Asp Arg Val Thr Gly Ala Phe Asp Ile
1 5
<210> 49
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 49
Arg Ala Ser Gln Asp Ile Ser Ser Ala Leu Val
1 5 10
<210> 50
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 50
Arg Ser Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu His
1 5 10 15
<210> 51
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 51
Arg Ser Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu His
1 5 10 15
<210> 52
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 52
Arg Ala Ser Gln Ser Ile Gly Thr Asn Ile His
1 5 10
<210> 53
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 53
Ser Ala Ser Ser Ser Val Thr Tyr Met Tyr
1 5 10
<210> 54
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 54
Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 55
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 55
Asp Ala Ser Ser Leu Glu Ser
1 5
<210> 56
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 56
Lys Val Ser Asn Arg Phe Ser
1 5
<210> 57
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 57
Tyr Ala Ser Glu Ser Ile Ser
1 5
<210> 58
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 58
Asp Thr Ser Asn Leu Ala Ser
1 5
<210> 59
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 59
Asp Ala Ser Asn Leu Glu Thr
1 5
<210> 60
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 60
Gln Gln Phe Asn Ser Tyr Pro Leu Thr
1 5
<210> 61
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 61
Ser Gln Ser Thr His Val Pro Trp Thr
1 5
<210> 62
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 62
Ser Gln Ser Thr His Val Leu Thr
1 5
<210> 63
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 63
Gln Gln Asn Asn Asn Trp Pro Thr Thr
1 5
<210> 64
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 64
Gln Gln Trp Ser Ser His Ile Phe Thr
1 5
<210> 65
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 65
Gln His Phe Asp His Leu Pro Leu Ala
1 5
<210> 66
<211> 45
<212> PRT
<213> Unknown
<220>
<223> CD8alpha hinge
<400> 66
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 67
<211> 135
<212> DNA
<213> Unknown
<220>
<223> CD8alpha hinge
<400> 67
accacgacgc cagcgccgcg accaccaaca ccggcgccca ccatcgcgtc gcagcccctg 60
tccctgcgcc cagaggcgtg ccggccagcg gcggggggcg cagtgcacac gagggggctg 120
gacttcgcct gtgat 135
<210> 68
<211> 41
<212> PRT
<213> Unknown
<220>
<223> CD28 hinge
<400> 68
Cys Thr Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys
1 5 10 15
Ser Asn Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser
20 25 30
Pro Leu Phe Pro Gly Pro Ser Lys Pro
35 40
<210> 69
<211> 123
<212> DNA
<213> Unknown
<220>
<223> CD28 hinge
<400> 69
tgtaccattg aagttatgta tcctcctcct tacctagaca atgagaagag caatggaacc 60
attatccatg tgaaagggaa acacctttgt ccaagtcccc tatttcccgg accttctaag 120
ccc 123
<210> 70
<211> 27
<212> PRT
<213> Unknown
<220>
<223> CD28 transmembrane domain
<400> 70
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 71
<211> 81
<212> DNA
<213> Unknown
<220>
<223> CD28 transmembrane domain
<400> 71
ttctgggtgc tggtcgttgt gggcggcgtg ctggcctgct acagcctgct ggtgacagtg 60
gccttcatca tcttttgggt g 81
<210> 72
<211> 25
<212> PRT
<213> Unknown
<220>
<223> IL-2Rbeta transmembrane domain
<400> 72
Ile Pro Trp Leu Gly His Leu Leu Val Gly Leu Ser Gly Ala Phe Gly
1 5 10 15
Phe Ile Ile Leu Val Tyr Leu Leu Ile
20 25
<210> 73
<211> 75
<212> DNA
<213> Unknown
<220>
<223> IL-2Rbeta transmembrane domain
<400> 73
attccgtggc tcggccacct cctcgtgggc ctcagcgggg cttttggctt catcatctta 60
gtgtacttgc tgatc 75
<210> 74
<211> 112
<212> PRT
<213> Unknown
<220>
<223> CD3zeta activation domain
<400> 74
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 75
<211> 336
<212> DNA
<213> Unknown
<220>
<223> CD3zeta activation domain
<400> 75
agagtgaagt tcagcaggag cgcagacgcc cccgcgtaca agcagggcca gaaccagctc 60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gcgtagaggc 120
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 180
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 240
cggaggggca aggggcacga tggcctttac cagggactca gtacagccac caaggacacc 300
tacgacgccc ttcacatgca ggccctgccc cctcgc 336
<210> 76
<211> 43
<212> PRT
<213> Unknown
<220>
<223> CD3zeta activation domain
<400> 76
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu His Met Gln Ala Leu Pro Pro Arg
35 40
<210> 77
<211> 129
<212> DNA
<213> Unknown
<220>
<223> CD3zeta activation domain
<400> 77
agagtgaagt tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc 60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttgcacat gcaggccctg 120
ccccctcgc 129
<210> 78
<211> 41
<212> PRT
<213> Unknown
<220>
<223> CD28 co-stimulatory domain
<400> 78
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 79
<211> 123
<212> DNA
<213> Unknown
<220>
<223> CD28 co-stimulatory domain
<400> 79
aggagcaagc ggagcagact gctgcacagc gactacatga acatgacccc ccggaggcct 60
ggccccaccc ggaagcacta ccagccctac gcccctccca gggatttcgc cgcctaccgg 120
agc 123
<210> 80
<211> 23
<212> PRT
<213> Unknown
<220>
<223> TCR alpha transmembrane domain
<400> 80
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
1 5 10 15
Leu Met Thr Leu Arg Leu Trp
20
<210> 81
<211> 69
<212> DNA
<213> Unknown
<220>
<223> TCR alpha transmembrane domain
<400> 81
gtgattgggt tccgaatcct cctcctgaaa gtggccgggt ttaatctgct catgacgctg 60
cggctgtgg 69
<210> 82
<211> 23
<212> PRT
<213> Unknown
<220>
<223> TCR beta transmembrane domain
<400> 82
Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val
1 5 10 15
Leu Val Ser Ala Leu Val Leu
20
<210> 83
<211> 69
<212> DNA
<213> Unknown
<220>
<223> TCR beta transmembrane domain
<400> 83
accatcctct atgagatctt gctagggaag gccaccttgt atgccgtgct ggtcagtgcc 60
ctcgtgctg 69
<210> 84
<211> 10
<212> PRT
<213> Unknown
<220>
<223> TCR beta intracellular domain
<400> 84
Met Ala Met Val Lys Arg Lys Asp Ser Arg
1 5 10
<210> 85
<211> 30
<212> DNA
<213> Unknown
<220>
<223> TCR beta intracellular domain
<400> 85
atggccatgg tcaagagaaa ggattccaga 30
<210> 86
<211> 273
<212> PRT
<213> Homo sapiens
<400> 86
Met Ser Gly Ser Gln Ser Glu Val Ala Pro Ser Pro Gln Ser Pro Arg
1 5 10 15
Ser Pro Glu Met Gly Arg Asp Leu Arg Pro Gly Ser Arg Val Leu Leu
20 25 30
Leu Leu Leu Leu Leu Leu Leu Val Tyr Leu Thr Gln Pro Gly Asn Gly
35 40 45
Asn Glu Gly Ser Val Thr Gly Ser Cys Tyr Cys Gly Lys Arg Ile Ser
50 55 60
Ser Asp Ser Pro Pro Ser Val Gln Phe Met Asn Arg Leu Arg Lys His
65 70 75 80
Leu Arg Ala Tyr His Arg Cys Leu Tyr Tyr Thr Arg Phe Gln Leu Leu
85 90 95
Ser Trp Ser Val Cys Gly Gly Asn Lys Asp Pro Trp Val Gln Glu Leu
100 105 110
Met Ser Cys Leu Asp Leu Lys Glu Cys Gly His Ala Tyr Ser Gly Ile
115 120 125
Val Ala His Gln Lys His Leu Leu Pro Thr Ser Pro Pro Ile Ser Gln
130 135 140
Ala Ser Glu Gly Ala Ser Ser Asp Ile His Thr Pro Ala Gln Met Leu
145 150 155 160
Leu Ser Thr Leu Gln Ser Thr Gln Arg Pro Thr Leu Pro Val Gly Ser
165 170 175
Leu Ser Ser Asp Lys Glu Leu Thr Arg Pro Asn Glu Thr Thr Ile His
180 185 190
Thr Ala Gly His Ser Leu Ala Ala Gly Pro Glu Ala Gly Glu Asn Gln
195 200 205
Lys Gln Pro Glu Lys Asn Ala Gly Pro Thr Ala Arg Thr Ser Ala Thr
210 215 220
Val Pro Val Leu Cys Leu Leu Ala Ile Ile Phe Ile Leu Thr Ala Ala
225 230 235 240
Leu Ser Tyr Val Leu Cys Lys Arg Arg Arg Gly Gln Ser Pro Gln Ser
245 250 255
Ser Pro Asp Leu Pro Val His Tyr Ile Pro Val Ala Pro Asp Ser Asn
260 265 270
Thr
<210> 87
<211> 742
<212> PRT
<213> Homo sapiens
<400> 87
Met Lys Asp Asp Phe Ala Glu Glu Glu Glu Val Gln Ser Phe Gly Tyr
1 5 10 15
Lys Arg Phe Gly Ile Gln Glu Gly Thr Gln Cys Thr Lys Cys Lys Asn
20 25 30
Asn Trp Ala Leu Lys Phe Ser Ile Ile Leu Leu Tyr Ile Leu Cys Ala
35 40 45
Leu Leu Thr Ile Thr Val Ala Ile Leu Gly Tyr Lys Val Val Glu Lys
50 55 60
Met Asp Asn Val Thr Gly Gly Met Glu Thr Ser Arg Gln Thr Tyr Asp
65 70 75 80
Asp Lys Leu Thr Ala Val Glu Ser Asp Leu Lys Lys Leu Gly Asp Gln
85 90 95
Thr Gly Lys Lys Ala Ile Ser Thr Asn Ser Glu Leu Ser Thr Phe Arg
100 105 110
Ser Asp Ile Leu Asp Leu Arg Gln Gln Leu Arg Glu Ile Thr Glu Lys
115 120 125
Thr Ser Lys Asn Lys Asp Thr Leu Glu Lys Leu Gln Ala Ser Gly Asp
130 135 140
Ala Leu Val Asp Arg Gln Ser Gln Leu Lys Glu Thr Leu Glu Asn Asn
145 150 155 160
Ser Phe Leu Ile Thr Thr Val Asn Lys Thr Leu Gln Ala Tyr Asn Gly
165 170 175
Tyr Val Thr Asn Leu Gln Gln Asp Thr Ser Val Leu Gln Gly Asn Leu
180 185 190
Gln Asn Gln Met Tyr Ser His Asn Val Val Ile Met Asn Leu Asn Asn
195 200 205
Leu Asn Leu Thr Gln Val Gln Gln Arg Asn Leu Ile Thr Asn Leu Gln
210 215 220
Arg Ser Val Asp Asp Thr Ser Gln Ala Ile Gln Arg Ile Lys Asn Asp
225 230 235 240
Phe Gln Asn Leu Gln Gln Val Phe Leu Gln Ala Lys Lys Asp Thr Asp
245 250 255
Trp Leu Lys Glu Lys Val Gln Ser Leu Gln Thr Leu Ala Ala Asn Asn
260 265 270
Ser Ala Leu Ala Lys Ala Asn Asn Asp Thr Leu Glu Asp Met Asn Ser
275 280 285
Gln Leu Asn Ser Phe Thr Gly Gln Met Glu Asn Ile Thr Thr Ile Ser
290 295 300
Gln Ala Asn Glu Gln Asn Leu Lys Asp Leu Gln Asp Leu His Lys Asp
305 310 315 320
Ala Glu Asn Arg Thr Ala Ile Lys Phe Asn Gln Leu Glu Glu Arg Phe
325 330 335
Gln Leu Phe Glu Thr Asp Ile Val Asn Ile Ile Ser Asn Ile Ser Tyr
340 345 350
Thr Ala His His Leu Arg Thr Leu Thr Ser Asn Leu Asn Glu Val Arg
355 360 365
Thr Thr Cys Thr Asp Thr Leu Thr Lys His Thr Asp Asp Leu Thr Ser
370 375 380
Leu Asn Asn Thr Leu Ala Asn Ile Arg Leu Asp Ser Val Ser Leu Arg
385 390 395 400
Met Gln Gln Asp Leu Met Arg Ser Arg Leu Asp Thr Glu Val Ala Asn
405 410 415
Leu Ser Val Ile Met Glu Glu Met Lys Leu Val Asp Ser Lys His Gly
420 425 430
Gln Leu Ile Lys Asn Phe Thr Ile Leu Gln Gly Pro Pro Gly Pro Arg
435 440 445
Gly Pro Arg Gly Asp Arg Gly Ser Gln Gly Pro Pro Gly Pro Thr Gly
450 455 460
Asn Lys Gly Gln Lys Gly Glu Lys Gly Glu Pro Gly Pro Pro Gly Pro
465 470 475 480
Ala Gly Glu Arg Gly Pro Ile Gly Pro Ala Gly Pro Pro Gly Glu Arg
485 490 495
Gly Gly Lys Gly Ser Lys Gly Ser Gln Gly Pro Lys Gly Ser Arg Gly
500 505 510
Ser Pro Gly Lys Pro Gly Pro Gln Gly Ser Ser Gly Asp Pro Gly Pro
515 520 525
Pro Gly Pro Pro Gly Lys Glu Gly Leu Pro Gly Pro Gln Gly Pro Pro
530 535 540
Gly Phe Gln Gly Leu Gln Gly Thr Val Gly Glu Pro Gly Val Pro Gly
545 550 555 560
Pro Arg Gly Leu Pro Gly Leu Pro Gly Val Pro Gly Met Pro Gly Pro
565 570 575
Lys Gly Pro Pro Gly Pro Pro Gly Pro Ser Gly Ala Val Val Pro Leu
580 585 590
Ala Leu Gln Asn Glu Pro Thr Pro Ala Pro Glu Asp Asn Gly Cys Pro
595 600 605
Pro His Trp Lys Asn Phe Thr Asp Lys Cys Tyr Tyr Phe Ser Val Glu
610 615 620
Lys Glu Ile Phe Glu Asp Ala Lys Leu Phe Cys Glu Asp Lys Ser Ser
625 630 635 640
His Leu Val Phe Ile Asn Thr Arg Glu Glu Gln Gln Trp Ile Lys Lys
645 650 655
Gln Met Val Gly Arg Glu Ser His Trp Ile Gly Leu Thr Asp Ser Glu
660 665 670
Arg Glu Asn Glu Trp Lys Trp Leu Asp Gly Thr Ser Pro Asp Tyr Lys
675 680 685
Asn Trp Lys Ala Gly Gln Pro Asp Asn Trp Gly His Gly His Gly Pro
690 695 700
Gly Glu Asp Cys Ala Gly Leu Ile Tyr Ala Gly Gln Trp Asn Asp Phe
705 710 715 720
Gln Cys Glu Asp Val Asn Asn Phe Ile Cys Glu Lys Asp Arg Glu Thr
725 730 735
Val Leu Ser Ser Ala Leu
740
<210> 88
<211> 514
<212> PRT
<213> Homo sapiens
<400> 88
Met Ser Trp Pro Arg Arg Leu Leu Leu Arg Tyr Leu Phe Pro Ala Leu
1 5 10 15
Leu Leu His Gly Leu Gly Glu Gly Ser Ala Leu Leu His Pro Asp Ser
20 25 30
Arg Ser His Pro Arg Ser Leu Glu Lys Ser Ala Trp Arg Ala Phe Lys
35 40 45
Glu Ser Gln Cys His His Met Leu Lys His Leu His Asn Gly Ala Arg
50 55 60
Ile Thr Val Gln Met Pro Pro Thr Ile Glu Gly His Trp Val Ser Thr
65 70 75 80
Gly Cys Glu Val Arg Ser Gly Pro Glu Phe Ile Thr Arg Ser Tyr Arg
85 90 95
Phe Tyr His Asn Asn Thr Phe Lys Ala Tyr Gln Phe Tyr Tyr Gly Ser
100 105 110
Asn Arg Cys Thr Asn Pro Thr Tyr Thr Leu Ile Ile Arg Gly Lys Ile
115 120 125
Arg Leu Arg Gln Ala Ser Trp Ile Ile Arg Gly Gly Thr Glu Ala Asp
130 135 140
Tyr Gln Leu His Asn Val Gln Val Ile Cys His Thr Glu Ala Val Ala
145 150 155 160
Glu Lys Leu Gly Gln Gln Val Asn Arg Thr Cys Pro Gly Phe Leu Ala
165 170 175
Asp Gly Gly Pro Trp Val Gln Asp Val Ala Tyr Asp Leu Trp Arg Glu
180 185 190
Glu Asn Gly Cys Glu Cys Thr Lys Ala Val Asn Phe Ala Met His Glu
195 200 205
Leu Gln Leu Ile Arg Val Glu Lys Gln Tyr Leu His His Asn Leu Asp
210 215 220
His Leu Val Glu Glu Leu Phe Leu Gly Asp Ile His Thr Asp Ala Thr
225 230 235 240
Gln Arg Met Phe Tyr Arg Pro Ser Ser Tyr Gln Pro Pro Leu Gln Asn
245 250 255
Ala Lys Asn His Asp His Ala Cys Ile Ala Cys Arg Ile Ile Tyr Arg
260 265 270
Ser Asp Glu His His Pro Pro Ile Leu Pro Pro Lys Ala Asp Leu Thr
275 280 285
Ile Gly Leu His Gly Glu Trp Val Ser Gln Arg Cys Glu Val Arg Pro
290 295 300
Glu Val Leu Phe Leu Thr Arg His Phe Ile Phe His Asp Asn Asn Asn
305 310 315 320
Thr Trp Glu Gly His Tyr Tyr His Tyr Ser Asp Pro Val Cys Lys His
325 330 335
Pro Thr Phe Ser Ile Tyr Ala Arg Gly Arg Tyr Ser Arg Gly Val Leu
340 345 350
Ser Ser Arg Val Met Gly Gly Thr Glu Phe Val Phe Lys Val Asn His
355 360 365
Met Lys Val Thr Pro Met Asp Ala Ala Thr Ala Ser Leu Leu Asn Val
370 375 380
Phe Asn Gly Asn Glu Cys Gly Ala Glu Gly Ser Trp Gln Val Gly Ile
385 390 395 400
Gln Gln Asp Val Thr His Thr Asn Gly Cys Val Ala Leu Gly Ile Lys
405 410 415
Leu Pro His Thr Glu Tyr Glu Ile Phe Lys Met Glu Gln Asp Ala Arg
420 425 430
Gly Arg Tyr Leu Leu Phe Asn Gly Gln Arg Pro Ser Asp Gly Ser Ser
435 440 445
Pro Asp Arg Pro Glu Lys Arg Ala Thr Ser Tyr Gln Met Pro Leu Val
450 455 460
Gln Cys Ala Ser Ser Ser Pro Arg Ala Glu Asp Leu Ala Glu Asp Ser
465 470 475 480
Gly Ser Ser Leu Tyr Gly Arg Ala Pro Gly Arg His Thr Trp Ser Leu
485 490 495
Leu Leu Ala Ala Leu Ala Cys Leu Val Pro Leu Leu His Trp Asn Ile
500 505 510
Arg Arg
<210> 89
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-001765 PA2.1 scFv (mouse)
<400> 89
Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Ile Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Gly
85 90 95
Ser His Val Pro Arg Thr Ser Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly
130 135 140
Ala Ser Val Arg Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser
145 150 155 160
Tyr His Ile His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp
165 170 175
Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys
180 185 190
Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala
195 200 205
Tyr Met His Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Phe
210 215 220
Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Ser Val Thr Val Ser Ser
245
<210> 90
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002159 PA2.1.8 scFv (humanized)
<400> 90
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Leu Thr Gln Ser Pro Gly
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
210 215 220
Val Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 91
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002160 PA2.1.9 scFv (humanized)
<400> 91
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Val Met Thr Gln Thr Pro Leu
130 135 140
Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly
210 215 220
Val Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 92
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002161 PA2.1.10 scFv (humanized)
<400> 92
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 93
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002162 PA2.1.14 scFv (humanized)
<400> 93
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Ile Thr Ala Asp Glu Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Val Glu Val Lys
245
<210> 94
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002163 PA2.1.18 scFv (humanized)
<400> 94
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Val Gln Met Thr Gln Ser Pro Ser
130 135 140
Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Ser Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Met Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys His Gln Gly Ser His Val Pro Arg Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Val Glu Val Lys
245
<210> 95
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002164 BB7.2 scFv (mouse)
<400> 95
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile Gln Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Thr Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Leu Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Ile Tyr Phe Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Val Leu Met Thr Gln Thr Pro Leu
130 135 140
Ser Leu Pro Val Ser Leu Gly Asp Gln Val Ser Ile Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Leu Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly
210 215 220
Val Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Glu Ile Lys
245
<210> 96
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002165 BB7.2.1 scFv (humanized)
<400> 96
Gln Leu Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile Gln Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Arg Ala Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Asn Leu Asp Ser Val Ser Ala Ala Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Tyr Trp Gly Lys Gly Ser
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala
210 215 220
Thr Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Pro
225 230 235 240
Gly Thr Lys Val Asp Ile Lys
245
<210> 97
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002166 BB7.2.2 scFv (humanized)
<400> 97
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Leu Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile Gln Trp Val Lys Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Val Glu Val Lys
245
<210> 98
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002167 BB7.2.3 scFv (humanized)
<400> 98
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Ile Gln Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Leu Thr Gln Ser Pro Gly
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
210 215 220
Val Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 99
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002168 BB7.2.5 scFv (humanized)
<400> 99
Gln Val Thr Leu Lys Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Val Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Arg Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Val Thr Ile Thr Ala Asp Lys Ser Met Asp Thr Ser Phe
65 70 75 80
Met Glu Leu Thr Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Leu Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Leu Thr Gln Ser Pro Gly
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Ala Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Ser Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
210 215 220
Val Tyr Tyr Cys Gln Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 100
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> C-002169 BB7.2.6 scFv (humanized)
<400> 100
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
His Met His Trp Val Arg Gln Ala Pro Gly Gln Arg Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Val Thr Ile Thr Arg Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Thr Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Val Met Thr Gln Thr Pro Leu
130 135 140
Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser
145 150 155 160
Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Asp Trp Tyr
165 170 175
Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser
180 185 190
Asn Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly
210 215 220
Val Tyr Tyr Cys Met Gln Gly Ser His Val Pro Arg Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 101
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L1
<400> 101
Arg Ser Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu
1 5 10 15
<210> 102
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L2
<400> 102
Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg
1 5 10
<210> 103
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L3
<400> 103
Phe Gln Gly Ser His Val Pro Arg Thr
1 5
<210> 104
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H1
<400> 104
Ala Ser Gly Tyr Thr Phe Thr Ser Tyr His Ile His
1 5 10
<210> 105
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H2
<400> 105
Trp Ile Tyr Pro Gly Asn Val Asn Thr Glu Tyr Asn Glu Lys Phe Lys
1 5 10 15
Gly Lys
<210> 106
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H3
<400> 106
Glu Glu Ile Thr Tyr Ala Met Asp Tyr
1 5
<210> 107
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L1
<400> 107
Arg Ser Ser Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Asp
1 5 10 15
<210> 108
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L2
<400> 108
Lys Val Ser Asn Arg Phe Ser Gly Val Pro Asp Arg
1 5 10
<210> 109
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-L3
<400> 109
Met Gln Gly Ser His Val Pro Arg Thr
1 5
<210> 110
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H1
<400> 110
Ser Gly Tyr Thr Phe Thr Ser Tyr His Met His
1 5 10
<210> 111
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H2
<400> 111
Trp Ile Tyr Pro Gly Asp Gly Ser Thr Gln Tyr Asn Glu Lys Phe Lys
1 5 10 15
Gly
<210> 112
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*02 CDR-H3
<400> 112
Glu Gly Thr Tyr Tyr Ala Met Asp Tyr
1 5
<210> 113
<211> 670
<212> PRT
<213> Unknown
<220>
<223> LILRB1
<400> 113
Met Thr Pro Ile Leu Thr Val Leu Ile Cys Leu Gly Leu Ser Leu Gly
1 5 10 15
Pro Arg Thr His Val Gln Ala Gly His Leu Pro Lys Pro Thr Leu Trp
20 25 30
Ala Glu Pro Gly Ser Val Ile Thr Gln Gly Ser Pro Val Thr Leu Arg
35 40 45
Cys Gln Gly Gly Gln Glu Thr Gln Glu Tyr Arg Leu Tyr Arg Glu Lys
50 55 60
Lys Thr Ala Leu Trp Ile Thr Arg Ile Pro Gln Glu Leu Val Lys Lys
65 70 75 80
Gly Gln Phe Pro Ile Pro Ser Ile Thr Trp Glu His Ala Gly Arg Tyr
85 90 95
Arg Cys Tyr Tyr Gly Ser Asp Thr Ala Gly Arg Ser Glu Ser Ser Asp
100 105 110
Pro Leu Glu Leu Val Val Thr Gly Ala Tyr Ile Lys Pro Thr Leu Ser
115 120 125
Ala Gln Pro Ser Pro Val Val Asn Ser Gly Gly Asn Val Ile Leu Gln
130 135 140
Cys Asp Ser Gln Val Ala Phe Asp Gly Phe Ser Leu Cys Lys Glu Gly
145 150 155 160
Glu Asp Glu His Pro Gln Cys Leu Asn Ser Gln Pro His Ala Arg Gly
165 170 175
Ser Ser Arg Ala Ile Phe Ser Val Gly Pro Val Ser Pro Ser Arg Arg
180 185 190
Trp Trp Tyr Arg Cys Tyr Ala Tyr Asp Ser Asn Ser Pro Tyr Glu Trp
195 200 205
Ser Leu Pro Ser Asp Leu Leu Glu Leu Leu Val Leu Gly Val Ser Lys
210 215 220
Lys Pro Ser Leu Ser Val Gln Pro Gly Pro Ile Val Ala Pro Glu Glu
225 230 235 240
Thr Leu Thr Leu Gln Cys Gly Ser Asp Ala Gly Tyr Asn Arg Phe Val
245 250 255
Leu Tyr Lys Asp Gly Glu Arg Asp Phe Leu Gln Leu Ala Gly Ala Gln
260 265 270
Pro Gln Ala Gly Leu Ser Gln Ala Asn Phe Thr Leu Gly Pro Val Ser
275 280 285
Arg Ser Tyr Gly Gly Gln Tyr Arg Cys Tyr Gly Ala His Asn Leu Ser
290 295 300
Ser Glu Trp Ser Ala Pro Ser Asp Pro Leu Asp Ile Leu Ile Ala Gly
305 310 315 320
Gln Phe Tyr Asp Arg Val Ser Leu Ser Val Gln Pro Gly Pro Thr Val
325 330 335
Ala Ser Gly Glu Asn Val Thr Leu Leu Cys Gln Ser Gln Gly Trp Met
340 345 350
Gln Thr Phe Leu Leu Thr Lys Glu Gly Ala Ala Asp Asp Pro Trp Arg
355 360 365
Leu Arg Ser Thr Tyr Gln Ser Gln Lys Tyr Gln Ala Glu Phe Pro Met
370 375 380
Gly Pro Val Thr Ser Ala His Ala Gly Thr Tyr Arg Cys Tyr Gly Ser
385 390 395 400
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
405 410 415
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
420 425 430
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
435 440 445
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
450 455 460
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
465 470 475 480
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
485 490 495
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
500 505 510
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
515 520 525
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
530 535 540
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
545 550 555 560
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
565 570 575
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
580 585 590
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
595 600 605
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
610 615 620
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
625 630 635 640
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Pro Ser Gln Glu Gly Pro
645 650 655
Ser Pro Ala Val Pro Ser Ile Tyr Ala Thr Leu Ala Ile His
660 665 670
<210> 114
<211> 3229
<212> DNA
<213> Unknown
<220>
<223> LILRB1
<400> 114
aaatgagttt taaaaaggct tgtccaggaa gcacatatgg gagctggtca ctctgcattt 60
tgggccctcc tggaggtgtt tagaccttcc gagagagaaa ctgagacaca tgagagggaa 120
gaaatgactc agtggtgaga ccctgtggag tcccacccac aaccagcaca ctgtgaccca 180
ctgcacaaac ctctagccca cagctcactt cctcctttaa gaagagaaga gaaaagagga 240
gaggagagga ggaacagaaa agaaaagaaa agaaaaagtg ggaaacaaat aatctaagaa 300
tgaggagaaa gcaagaagag tgaccccctt gtgggcactc cattggtttt atggcgcctc 360
tactttctgg agtttgtgta aaacaaaaat attatggtct ttgtgcacat ttacatcaag 420
ctcagcctgg gcggcacagc cagatgcgag atgcgtctct gctgatctga gtctgcctgc 480
agcatggacc tgggtcttcc ctgaagcatc tccagggctg gagggacgac tgccatgcac 540
cgagggctca tccatccaca gagcagggca gtgggaggag acgccatgac ccccatcctc 600
acggtcctga tctgtctcgg gctgagtctg ggcccccgga cccacgtgca ggcagggcac 660
ctccccaagc ccaccctctg ggctgaacca ggctctgtga tcacccaggg gagtcctgtg 720
accctcaggt gtcagggggg ccaggagacc caggagtacc gtctatatag agaaaagaaa 780
acagcaccct ggattacacg gatcccacag gagcttgtga agaagggcca gttccccatc 840
ccatccatca cctgggaaca cacagggcgg tatcgctgtt actatggtag cgacactgca 900
ggccgctcag agagcagtga ccccctggag ctggtggtga caggagccta catcaaaccc 960
accctctcag cccagcccag ccccgtggtg aactcaggag ggaatgtaac cctccagtgt 1020
gactcacagg tggcatttga tggcttcatt ctgtgtaagg aaggagaaga tgaacaccca 1080
caatgcctga actcccagcc ccatgcccgt gggtcgtccc gcgccatctt ctccgtgggc 1140
cccgtgagcc cgagtcgcag gtggtggtac aggtgctatg cttatgactc gaactctccc 1200
tatgagtggt ctctacccag tgatctcctg gagctcctgg tcctaggtgt ttctaagaag 1260
ccatcactct cagtgcagcc aggtcctatc gtggcccctg aggagaccct gactctgcag 1320
tgtggctctg atgctggcta caacagattt gttctgtata aggacgggga acgtgacttc 1380
cttcagctcg ctggcgcaca gccccaggct gggctctccc aggccaactt caccctgggc 1440
cctgtgagcc gctcctacgg gggccagtac agatgctacg gtgcacacaa cctctcctcc 1500
gagtggtcgg cccccagcga ccccctggac atcctgatcg caggacagtt ctatgacaga 1560
gtctccctct cggtgcagcc gggccccacg gtggcctcag gagagaacgt gaccctgctg 1620
tgtcagtcac agggatggat gcaaactttc cttctgacca aggagggggc agctgatgac 1680
ccatggcgtc taagatcaac gtaccaatct caaaaatacc aggctgaatt ccccatgggt 1740
cctgtgacct cagcccatgc ggggacctac aggtgctacg gctcacagag ctccaaaccc 1800
tacctgctga ctcaccccag tgaccccctg gagctcgtgg tctcaggacc gtctgggggc 1860
cccagctccc cgacaacagg ccccacctcc acatctggcc ctgaggacca gcccctcacc 1920
cccaccgggt cggatcccca gagtggtctg ggaaggcacc tgggggttgt gatcggcatc 1980
ttggtggccg tcatcctact gctcctcctc ctcctcctcc tcttcctcat cctccgacat 2040
cgacgtcagg gcaaacactg gacatcgacc cagagaaagg ctgatttcca acatcctgca 2100
ggggctgtgg ggccagagcc cacagacaga ggcctgcagt ggaggtccag cccagctgcc 2160
gatgcccagg aagaaaacct ctatgctgcc gtgaagcaca cacagcctga ggatggggtg 2220
gagatggaca ctcggagccc acacgatgaa gacccccagg cagtgacgta tgccgaggtg 2280
aaacactcca gacctaggag agaaatggcc tctcctcctt ccccactgtc tggggaattc 2340
ctggacacaa aggacagaca ggcggaagag gacaggcaga tggacactga ggctgctgca 2400
tctgaagccc cccaggatgt gacctacgcc cagctgcaca gcttgaccct cagacgggag 2460
gcaactgagc ctcctccatc ccaggaaggg ccctctccag ctgtgcccag catctacgcc 2520
actctggcca tccactagcc caggggggga cgcagacccc acactccatg gagtctggaa 2580
tgcatgggag ctgccccccc agtggacacc attggacccc acccagcctg gatctacccc 2640
aggagactct gggaactttt aggggtcact caattctgca gtataaataa ctaatgtctc 2700
tacaattttg aaataaagca acagacttct caataatcaa tgaagtagct gagaaaacta 2760
agtcagaaag tgcattaaac tgaatcacaa tgtaaatatt acacatcaag cgatgaaact 2820
ggaaaactac aagccacgaa tgaatgaatt aggaaagaaa aaaagtagga aatgaatgat 2880
cttggctttc ctataagaaa tttagggcag ggcacggtgg ctcacgcctg taattccagc 2940
actttgggag gccgaggcgg gcagatcacg agttcaggag atcgagacca tcttggccaa 3000
catggtgaaa ccctgtctct cctaaaaata caaaaattag ctggatgtgg tggcagtgcc 3060
tgtaatccca gctatttggg aggctgaggc aggagaatcg cttgaaccag ggagtcagag 3120
gtttcagtga gccaagatcg caccactgct ctccagcctg gcgacagagg gagactccat 3180
ctcaaattaa aaaaaaaaaa aaaaaagaaa gaaaaaaaaa aaaaaaaaa 3229
<210> 115
<211> 6
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based inhibitory motif
<400> 115
Asn Leu Tyr Ala Ala Val
1 5
<210> 116
<211> 6
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based inhibitory motif
<400> 116
Val Thr Tyr Ala Glu Val
1 5
<210> 117
<211> 6
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based inhibitory motif
<400> 117
Val Thr Tyr Ala Gln Leu
1 5
<210> 118
<211> 6
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based inhibitory motif
<400> 118
Ser Ile Tyr Ala Thr Leu
1 5
<210> 119
<211> 35
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 119
Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val Glu
1 5 10 15
Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr Tyr
20 25 30
Ala Glu Val
35
<210> 120
<211> 58
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 120
Val Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala
1 5 10 15
Ser Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg
20 25 30
Gln Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu
35 40 45
Ala Pro Gln Asp Val Thr Tyr Ala Gln Leu
50 55
<210> 121
<211> 36
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 121
Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg Glu Ala Thr
1 5 10 15
Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val Pro Ser Ile
20 25 30
Tyr Ala Thr Leu
35
<210> 122
<211> 87
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 122
Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val Glu
1 5 10 15
Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr Tyr
20 25 30
Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser Pro Pro
35 40 45
Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln Ala Glu
50 55 60
Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala Pro Gln
65 70 75 80
Asp Val Thr Tyr Ala Gln Leu
85
<210> 123
<211> 88
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 123
Val Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala
1 5 10 15
Ser Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg
20 25 30
Gln Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu
35 40 45
Ala Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg
50 55 60
Arg Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala
65 70 75 80
Val Pro Ser Ile Tyr Ala Thr Leu
85
<210> 124
<211> 117
<212> PRT
<213> Unknown
<220>
<223> intracellular domain
<400> 124
Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val Glu
1 5 10 15
Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr Tyr
20 25 30
Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser Pro Pro
35 40 45
Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln Ala Glu
50 55 60
Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala Pro Gln
65 70 75 80
Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg Glu Ala
85 90 95
Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val Pro Ser
100 105 110
Ile Tyr Ala Thr Leu
115
<210> 125
<211> 20
<212> PRT
<213> Unknown
<220>
<223> LILRB1 D3D4 domain
<400> 125
Tyr Gly Ser Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp
1 5 10 15
Pro Leu Glu Leu
20
<210> 126
<211> 43
<212> PRT
<213> Unknown
<220>
<223> LILRB1 hinge domain
<400> 126
Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly Pro
1 5 10 15
Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly Ser
20 25 30
Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly
35 40
<210> 127
<211> 86
<212> PRT
<213> Unknown
<220>
<223> Hinge-transmembrane
<400> 127
Tyr Gly Ser Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp
1 5 10 15
Pro Leu Glu Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro
20 25 30
Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr
35 40 45
Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val
50 55 60
Val Ile Gly Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu
65 70 75 80
Leu Leu Phe Leu Ile Leu
85
<210> 128
<211> 190
<212> PRT
<213> Unknown
<220>
<223> Transmembrane-intracellular domain
<400> 128
Val Val Ile Gly Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu
1 5 10 15
Leu Leu Leu Phe Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp
20 25 30
Thr Ser Thr Gln Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val
35 40 45
Gly Pro Glu Pro Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala
50 55 60
Ala Asp Ala Gln Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln
65 70 75 80
Pro Glu Asp Gly Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp
85 90 95
Pro Gln Ala Val Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg
100 105 110
Glu Met Ala Ser Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr
115 120 125
Lys Asp Arg Gln Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala
130 135 140
Ala Ser Glu Ala Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu
145 150 155 160
Thr Leu Arg Arg Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro
165 170 175
Ser Pro Ala Val Pro Ser Ile Tyr Ala Thr Leu Ala Ile His
180 185 190
<210> 129
<211> 167
<212> PRT
<213> Unknown
<220>
<223> LILRB1 intracellular domain
<400> 129
Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln Arg Lys Ala
1 5 10 15
Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro Thr Asp Arg
20 25 30
Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln Glu Glu Asn
35 40 45
Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val Glu Met
50 55 60
Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr Tyr Ala
65 70 75 80
Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser Pro Pro Ser
85 90 95
Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln Ala Glu Glu
100 105 110
Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala Pro Gln Asp
115 120 125
Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg Glu Ala Thr
130 135 140
Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val Pro Ser Ile
145 150 155 160
Tyr Ala Thr Leu Ala Ile His
165
<210> 130
<211> 253
<212> PRT
<213> Unknown
<220>
<223> LILRB1 hinge-transmembrane-intracellular domain
<400> 130
Tyr Gly Ser Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp
1 5 10 15
Pro Leu Glu Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro
20 25 30
Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr
35 40 45
Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val
50 55 60
Val Ile Gly Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu
65 70 75 80
Leu Leu Phe Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr
85 90 95
Ser Thr Gln Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly
100 105 110
Pro Glu Pro Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala
115 120 125
Asp Ala Gln Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro
130 135 140
Glu Asp Gly Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro
145 150 155 160
Gln Ala Val Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu
165 170 175
Met Ala Ser Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys
180 185 190
Asp Arg Gln Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala
195 200 205
Ser Glu Ala Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr
210 215 220
Leu Arg Arg Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser
225 230 235 240
Pro Ala Val Pro Ser Ile Tyr Ala Thr Leu Ala Ile His
245 250
<210> 131
<211> 233
<212> PRT
<213> Unknown
<220>
<223> LILRB1 hinge-transmembrane-intracellular domain
<400> 131
Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly Pro
1 5 10 15
Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly Ser
20 25 30
Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly Ile
35 40 45
Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe Leu
50 55 60
Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln Arg
65 70 75 80
Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro Thr
85 90 95
Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln Glu
100 105 110
Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val
115 120 125
Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr
130 135 140
Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser Pro
145 150 155 160
Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln Ala
165 170 175
Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala Pro
180 185 190
Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg Glu
195 200 205
Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val Pro
210 215 220
Ser Ile Tyr Ala Thr Leu Ala Ile His
225 230
<210> 132
<211> 63
<212> PRT
<213> Unknown
<220>
<223> LILRB1 hinge domain
<400> 132
Tyr Gly Ser Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp
1 5 10 15
Pro Leu Glu Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro
20 25 30
Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr
35 40 45
Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly
50 55 60
<210> 133
<211> 23
<212> PRT
<213> Unknown
<220>
<223> LILRB1 transmembrane domain
<400> 133
Val Val Ile Gly Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu
1 5 10 15
Leu Leu Leu Phe Leu Ile Leu
20
<210> 134
<211> 227
<212> PRT
<213> Homo sapiens
<220>
<221> SITE
<222> (1)..(1)
<223> Xaa can be any naturally occurring amino acid
<400> 134
Xaa Asp Val Ala Tyr Asp Leu Trp Arg Glu Glu Asn Gly Cys Glu Cys
1 5 10 15
Thr Lys Ala Val Asn Phe Ala Met His Glu Leu Gln Leu Ile Arg Val
20 25 30
Glu Lys Gln Tyr Leu His His Asn Leu Asp His Leu Val Glu Glu Leu
35 40 45
Phe Leu Gly Asp Ile His Thr Asp Ala Thr Gln Arg Met Phe Tyr Arg
50 55 60
Pro Ser Ser Tyr Gln Pro Pro Leu Gln Asn Ala Lys Cys Ala Ala Glu
65 70 75 80
Ser Ser Gly Ser Phe Gln Ile Leu Pro Gln Asp Ser Ser Glu Lys Glu
85 90 95
Gln Asn Gly Leu Ser His Trp Cys Leu Ser Arg Pro Gly His Gln Lys
100 105 110
Asp Trp Ala Leu Cys Ala His Ala Gly Pro Ala Thr Ala Gly Cys Pro
115 120 125
Ser Cys Leu Trp Pro Pro Ala Glu Thr Gly Arg Lys Ala Gly Arg Thr
130 135 140
Ser Ser Lys Thr Val His Ala Cys Pro Gly Glu Ala Gly Thr Ser Ser
145 150 155 160
Phe Glu Leu Phe Tyr Phe Pro Asn Cys Trp Ser Ile Glu Thr Lys Leu
165 170 175
Lys Ile Ser Leu Asn Ala Lys Leu Ser Phe Lys Pro Arg Ala Ser Ala
180 185 190
Pro Leu Glu Thr Gly His Arg Val Lys Ile Glu Thr Leu Ser Gln Leu
195 200 205
Val Phe Leu Ser Phe Ile Gln Leu Cys Cys Glu Val Gln Ser Pro Leu
210 215 220
Ala Asn Lys
225
<210> 135
<211> 94
<212> PRT
<213> Homo sapiens
<400> 135
Pro Glu Asp Val Leu Pro Ala Leu Gln Leu Pro Ala Pro Ser Ala Glu
1 5 10 15
Cys Gln Val Glu Met Gly Phe His His Val Gly Gln Asp Gly Leu Gln
20 25 30
Leu Pro Thr Ser Ser Asp Pro Pro Ala Leu Ala Ser Gln Ser Ala Gly
35 40 45
Ile Thr Gly Val Ser His Arg Pro Pro Gly Arg His Leu Ser Asn Asp
50 55 60
Leu Arg Thr Thr Thr Met Pro Ala Ser Pro Val Gly Ser Ser Ile Gly
65 70 75 80
Gln Thr Ser Thr Thr Leu Pro Ser Cys Pro Gln Arg Gln Thr
85 90
<210> 136
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> linker
<400> 136
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly
<210> 137
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> signal sequence
<400> 137
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys
20
<210> 138
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<223> signal sequence
<400> 138
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgt 66
<210> 139
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-478 EGFR (VH-VL scFv Format)
<400> 139
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagt acctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatgggatg atggaagtta taaatactat 180
ggagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagatggt 300
attactatgg ttcggggagt tatgaaggac tactttgact actggggcca gggaaccctg 360
gtcaccgtct cctcaggcgg aggtggaagc ggagggggag gatctggcgg cggaggaagc 420
ggaggcgcca tccagttgac ccagtctcca tcctccctgt ctgcatctgt aggagacaga 480
gtcaccatca cttgccgggc aagtcaggac attagcagtg ctttagtctg gtatcagcag 540
aaaccaggga aagctcctaa gctcctgatc tatgatgcct ccagtttgga aagtggggtc 600
ccatcaaggt tcagcggcag tgaatctggg acagatttca ctctcaccat cagcagcctg 660
cagcctgaag attttgcaac ttattactgt caacagttta atagttaccc gctcactttc 720
ggcggaggga ccaaggtgga gatcaaa 747
<210> 140
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-479 EGFR (VL-VH scFv Format)
<400> 140
gccatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca ggacattagc agtgctttag tctggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
aggttcagcg gcagtgaatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag tttaatagtt acccgctcac tttcggcgga 300
gggaccaagg tggagatcaa aggcggaggt ggaagcggag ggggaggatc tggcggcgga 360
ggaagcggag gccaggtgca gctggtggag tctgggggag gcgtggtcca gcctgggagg 420
tccctgagac tctcctgtgc agcgtctgga ttcaccttca gtacctatgg catgcactgg 480
gtccgccagg ctccaggcaa ggggctggag tgggtggcag ttatatggga tgatggaagt 540
tataaatact atggagactc cgtgaagggc cgattcacca tctccagaga caattccaag 600
aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggctgt gtattactgt 660
gcgagagatg gtattactat ggttcgggga gttatgaagg actactttga ctactggggc 720
cagggaaccc tggtcaccgt ctcctca 747
<210> 141
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-480 EGFR (VH-VL scFv format)
<400> 141
cagatccagt tggtgcagtc tggacctgag ctgaagaagc ctggagagac agtcaagatc 60
tcctgcaagg cctctgggta taccttcaca gaatatccaa tacactgggt gaagcaggct 120
ccaggaaagg gtttcaagtg gatgggcatg atatacaccg acattggaaa gccaacatat 180
gctgaagagt tcaagggacg gtttgccttc tctttggaga cctctgccag cactgcctat 240
ttgcagatca acaacctcaa gaatgaggac acggctacat atttctgtgt aagagatcga 300
tatgattccc tctttgacta ctggggccaa ggcaccactc tcacagtctc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgatgt tgtgatgacc 420
caaactccac tctccctgcc tgtcagtctt ggagatcaag cctccatctc ttgcagatct 480
agtcagagcc ttgtacacag taatggaaac acctatttac attggtacct gcagaagcca 540
ggccagtctc caaagctcct gatctacaaa gtttccaacc gattttctgg ggtcccagac 600
aggttcagtg gcagtggatc agggacagat ttcacactca agatcagcag agtggaggct 660
gaggatctgg gagtttattt ctgctctcaa agtacacatg ttccgtggac gttcggtgga 720
ggcaccaagc tggaaatcaa a 741
<210> 142
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-481 EGFR (VL-VH scFv Format)
<400> 142
gatgttgtga tgacccaaac tccactctcc ctgcctgtca gtcttggaga tcaagcctcc 60
atctcttgca gatctagtca gagccttgta cacagtaatg gaaacaccta tttacattgg 120
tacctgcaga agccaggcca gtctccaaag ctcctgatct acaaagtttc caaccgattt 180
tctggggtcc cagacaggtt cagtggcagt ggatcaggga cagatttcac actcaagatc 240
agcagagtgg aggctgagga tctgggagtt tatttctgct ctcaaagtac acatgttccg 300
tggacgttcg gtggaggcac caagctggaa atcaaaggcg gaggtggaag cggaggggga 360
ggatctggcg gcggaggaag cggaggccag atccagttgg tgcagtctgg acctgagctg 420
aagaagcctg gagagacagt caagatctcc tgcaaggcct ctgggtatac cttcacagaa 480
tatccaatac actgggtgaa gcaggctcca ggaaagggtt tcaagtggat gggcatgata 540
tacaccgaca ttggaaagcc aacatatgct gaagagttca agggacggtt tgccttctct 600
ttggagacct ctgccagcac tgcctatttg cagatcaaca acctcaagaa tgaggacacg 660
gctacatatt tctgtgtaag agatcgatat gattccctct ttgactactg gggccaaggc 720
accactctca cagtctcctc a 741
<210> 143
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-482 EGFR (VH-VL scFv Format)
<400> 143
gaaatgcagc tggtggagtc tgggggaggc ttcgtgaagc ctggagggtc cctgaaactc 60
tcatgtgcag cctctggatt cgctttcagt cactatgaca tgtcttgggt tcgccagact 120
ccgaagcaga ggctggagtg ggtcgcatac attgctagtg gtggtgatat cacctactat 180
gcagacactg tgaagggccg attcaccatc tccagagaca atgcccagaa caccctgtac 240
ctgcaaatga gcagtctgaa gtctgaggac acagccatgt tttactgttc acgatcctcc 300
tatggtaaca acggagatgc cctggacttc tggggtcaag gtacctcagt caccgtctcc 360
tcaggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgatgtt 420
gtgatgaccc aaactccact ctccctgcct gtcagtcttg gagatcaagc ctccatctct 480
tgcagatcta gtcagagcct tgttcacagt aatggaaaca cctatttaca ttggtacctg 540
cagaagccag gccagtctcc aaagctcctg atctacaaag tttccaaccg attttctggg 600
gtcccagaca ggttcagtgg cagtggatca gggacagatt tcacactcaa gatcagcaga 660
gtggaggctg aggatctggg agtttatttc tgctctcaaa gtacacatgt tctcacgttc 720
ggctcgggga caaagttgga aataaaa 747
<210> 144
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-483 EGFR (VL-VH scFv Format)
<400> 144
gatgttgtga tgacccaaac tccactctcc ctgcctgtca gtcttggaga tcaagcctcc 60
atctcttgca gatctagtca gagccttgtt cacagtaatg gaaacaccta tttacattgg 120
tacctgcaga agccaggcca gtctccaaag ctcctgatct acaaagtttc caaccgattt 180
tctggggtcc cagacaggtt cagtggcagt ggatcaggga cagatttcac actcaagatc 240
agcagagtgg aggctgagga tctgggagtt tatttctgct ctcaaagtac acatgttctc 300
acgttcggct cggggacaaa gttggaaata aaaggcggag gtggaagcgg agggggagga 360
tctggcggcg gaggaagcgg aggcgaaatg cagctggtgg agtctggggg aggcttcgtg 420
aagcctggag ggtccctgaa actctcatgt gcagcctctg gattcgcttt cagtcactat 480
gacatgtctt gggttcgcca gactccgaag cagaggctgg agtgggtcgc atacattgct 540
agtggtggtg atatcaccta ctatgcagac actgtgaagg gccgattcac catctccaga 600
gacaatgccc agaacaccct gtacctgcaa atgagcagtc tgaagtctga ggacacagcc 660
atgttttact gttcacgatc ctcctatggt aacaacggag atgccctgga cttctggggt 720
caaggtacct cagtcaccgt ctcctca 747
<210> 145
<211> 729
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-486 EGFR (VH-VL scFv Format)
<400> 145
caggtgcagc tgaagcagtc cggccccggc ctggtgcagc cctcccagtc cctgtccatc 60
acctgcaccg tgtccggctt ctccctgacc aactacggcg tgcactgggt gcggcagtcc 120
cccggcaagg gcctggagtg gctgggcgtg atctggtccg gcggcaacac cgactacaac 180
acccccttca cctcccggct gtccatcaac aaggacaact ccaagtccca ggtgttcttc 240
aagatgaact ccctgcagtc caacgacacc gccatctact actgcgcccg ggccctgacc 300
tactacgact acgagttcgc ctactggggc cagggcaccc tggtgaccgt gtccgccggc 360
ggaggtggaa gcggaggggg aggatctggc ggcggaggaa gcggaggcga catcctgctg 420
acccagtccc ccgtgatcct gtccgtgtcc cccggcgagc gggtgtcctt ctcctgccgg 480
gcctcccagt ccatcggcac caacatccac tggtaccagc agcggaccaa cggctccccc 540
cggctgctga tcaagtacgc ctccgagtcc atctccggca tcccctcccg gttctccggc 600
tccggctccg gcaccgactt caccctgtcc atcaactccg tggagtccga ggacatcgcc 660
gactactact gccagcagaa caacaactgg cccaccacct tcggcgccgg caccaagctg 720
gagctgaag 729
<210> 146
<211> 732
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-487 EGFR (VH-VL scFv Format)
<400> 146
caggtgcagc tggtgcagtc cggcgccgag gtgaagaagc ccggcgcctc cgtgaaggtg 60
tcctgcaagg cctccggcta caccttcacc tcccactgga tgcactgggt gcggcaggcc 120
cccggccagg gcctggagtg gatcggcgag ttcaacccct ccaacggccg gaccaactac 180
aacgagaagt tcaagtccaa ggccaccatg accgtggaca cctccaccaa caccgcctac 240
atggagctgt cctccctgcg gtccgaggac accgccgtgt actactgcgc ctcccgggac 300
tacgactacg acggccggta cttcgactac tggggccagg gcaccctggt gaccgtgtcc 360
tccggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgacatc 420
cagatgaccc agtccccctc ctccctgtcc gcctccgtgg gcgaccgggt gaccatcacc 480
tgctccgcct cctcctccgt gacctacatg tactggtacc agcagaagcc cggcaaggcc 540
cccaagctgc tgatctacga cacctccaac ctggcctccg gcgtgccctc ccggttctcc 600
ggctccggct ccggcaccga ctacaccttc accatctcct ccctgcagcc cgaggacatc 660
gccacctact actgccagca gtggtcctcc cacatcttca ccttcggcca gggcaccaag 720
gtggagatca ag 732
<210> 147
<211> 729
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-488 EGFR (VH-VL scFv Format)
<400> 147
caggtgcagc tgcaggagtc cggccccggc ctggtgaagc cctccgagac cctgtccctg 60
acctgcaccg tgtccggcgg ctccgtgtcc tccggcgact actactggac ctggatccgg 120
cagtcccccg gcaagggcct ggagtggatc ggccacatct actactccgg caacaccaac 180
tacaacccct ccctgaagtc ccggctgacc atctccatcg acacctccaa gacccagttc 240
tccctgaagc tgtcctccgt gaccgccgcc gacaccgcca tctactactg cgtgcgggac 300
cgggtgaccg gcgccttcga catctggggc cagggcacca tggtgaccgt gtcctccggc 360
ggaggtggaa gcggaggggg aggatctggc ggcggaggaa gcggaggcga catccagatg 420
acccagtccc cctcctccct gtccgcctcc gtgggcgacc gggtgaccat cacctgccag 480
gcctcccagg acatctccaa ctacctgaac tggtaccagc agaagcccgg caaggccccc 540
aagctgctga tctacgacgc ctccaacctg gagaccggcg tgccctcccg gttctccggc 600
tccggctccg gcaccgactt caccttcacc atctcctccc tgcagcccga ggacatcgcc 660
acctacttct gccagcactt cgaccacctg cccctggcct tcggcggcgg caccaaggtg 720
gagatcaag 729
<210> 148
<211> 735
<212> DNA
<213> Artificial Sequence
<220>
<223> CT-489 EGFR scFv
<400> 148
caggtccagc tccaagagtc aggaccaggt ctggtcaaac cctcacagac gctctctctt 60
acttgcacag tatcaggcgg atccattagt tccggtgatt attactggtc ttggatccgc 120
cagcctcctg gtaaggggct ggagtggatc ggatacatat actacagtgg ttccactgat 180
tataatccgt cactcaagtc ccgagtaact atgtccgtgg atactagcaa aaatcagttc 240
agcctcaaag tcaattccgt aactgccgcc gacaccgcgg tctattattg tgcgcgggtg 300
agcatcttcg gcgtcggtac gttcgattac tggggacaag gaactctcgt aacagtaagc 360
tctggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgagatt 420
gtaatgaccc aaagccctgc cactctttct ctgtctccgg gggaacgagc gactctgtca 480
tgtagggcgt cccagagtgt ctctagctac ctcgcatggt atcagcagaa acccggacaa 540
gcccctcggc tcttgattta cgatgcctca aacagagcaa cggggattcc ggcacggttc 600
agcgggtctg gtagcgggac agacttcaca ctgacgatct cttcactcga accagaagat 660
tttgcagtct actattgcca tcagtatggt tctacgccac ttacatttgg cgggggcacc 720
aaggcggaga taaaa 735
<210> 149
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VH CT489
<400> 149
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asp Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Met Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Val Ser Ile Phe Gly Val Gly Thr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 150
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR VL CT489
<400> 150
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Ala Glu Ile Lys
100 105
<210> 151
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H1
<400> 151
Ser Gly Asp Tyr Tyr Trp Ser
1 5
<210> 152
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H2
<400> 152
Tyr Ile Tyr Tyr Ser Gly Ser Thr Asp Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 153
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR H3
<400> 153
Val Ser Ile Phe Gly Val Gly Thr Phe Asp Tyr
1 5 10
<210> 154
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L1
<400> 154
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 155
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L2
<400> 155
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 156
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> EGFR antigen binding domain CDR L3
<400> 156
His Gln Tyr Gly Ser Thr Pro Leu Thr
1 5
<210> 157
<211> 4
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based activation motif
<220>
<221> SITE
<222> (2)..(3)
<223> Xaa is any amino acid
<220>
<221> SITE
<222> (4)..(4)
<223> Xaa is Leu or Ile
<400> 157
Tyr Xaa Xaa Xaa
1
<210> 158
<211> 195
<212> PRT
<213> Unknown
<220>
<223> CAR intracellular domain
<400> 158
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu
35 40 45
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
50 55 60
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
65 70 75 80
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
85 90 95
Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
100 105 110
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
115 120 125
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
130 135 140
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
145 150 155 160
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
165 170 175
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
180 185 190
Pro Pro Arg
195
<210> 159
<211> 42
<212> PRT
<213> Unknown
<220>
<223> 4-1BB co-stimulatory domain
<400> 159
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 160
<211> 126
<212> DNA
<213> Unknown
<220>
<223> 4-1BB co-stimulatory domain
<400> 160
aaacggggca gaaagaaact cctgtatata ttcaaacaac catttatgag gccagtacaa 60
actactcaag aggaagatgg ctgtagctgc cgatttccag aagaagaaga aggaggatgt 120
gaactg 126
<210> 161
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-001765 PA2.1 scFv (mouse)
<400> 161
gatgttctga tgacccaaac tccactctcc ctgcctgtca gtcttggaga tcaagcctcc 60
atctcttgca gatctagtca gagcattgta catagtaatg gaaacaccta tttagaatgg 120
tacctgcaga agccaggcca gtctccaaag ctgctcatct acaaagtttc caaccgattt 180
tctggggtcc cagacagatt tagcggatct ggctctggga ccgatttcac actcaagatc 240
agtagagtgg aggctgagga tctgggagtt tattactgct ttcaaggttc acatgttcct 300
cggacgtccg gtggaggcac aaagctggaa atcaagggag gtggcggctc tggaggcgga 360
ggtagcggag gtggaggctc tggtggccag gtccagctgc agcagtctgg acctgagctg 420
gtgaagccag gggcttcagt gaggatatcc tgtaaggcct ctggctacac ctttacaagt 480
taccatatac attgggtgaa gcagaggcct ggacagggac tcgaatggat tggatggatt 540
tatcctggaa atgttaatac tgagtacaat gagaagttca agggcaaggc cacactgact 600
gcagacaaat cgtccagcac agcctacatg cacctcagca gcctgacctc tgaggactct 660
gcggtctatt tctgtgccag agaggagatt acctatgcta tggattattg gggtcaagga 720
acctcagtca ccgtgtcctc a 741
<210> 162
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002159 PA2.1.8 scFv (humanized)
<400> 162
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc agtgaaggtt 60
tcctgcaagg cttctggata caccttcact agctatcata tacattgggt gcgccaggcc 120
cccggacaag ggcttgagtg gatgggatgg atctaccctg gcaatgttaa cacagaatat 180
aatgagaagt tcaagggcaa agccaccatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt attactgtgc gagggaggaa 300
attacctacg ctatggacta ctggggccag ggaaccacag tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgagat tgtattgacc 420
cagagcccag gcaccctgag cctctctcca ggagagcggg ccaccctcag ttgtagatcc 480
agtcagagta ttgtacacag taatgggaac acctatttgg aatggtatca gcagaaacca 540
ggtcaagccc caagattgct catctacaaa gtctctaaca gatttagtgg tattccagac 600
aggttcagcg gttccggaag tggtactgat ttcaccctca cgatctccag gctcgagcca 660
gaagatttcg ccgtttatta ctgttttcaa ggttcacatg tgccgcgcac attcggtggg 720
ggtactaaag tagaaatcaa a 741
<210> 163
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002160 PA2.1.9 scFv (humanized)
<400> 163
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc agtgaaggtt 60
tcctgcaagg cttctggata caccttcact agctatcata tacattgggt gcgccaggcc 120
cccggacaag ggcttgagtg gatgggatgg atctaccctg gcaatgttaa cacagaatat 180
aatgagaagt tcaagggcaa agccaccatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt attactgtgc gagggaggaa 300
attacctacg ctatggacta ctggggccag ggaaccacag tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacat tgtaatgacc 420
cagaccccac tcagcctgcc cgtcactcca ggagagccgg ccagcatcag ttgtagatcc 480
agtcagagta ttgtacacag taatgggaac acctatttgg aatggtatct gcagaaacca 540
ggtcaatccc cacaattgct catctacaaa gtctctaaca gatttagtgg tgtaccagac 600
aggttcagcg gttccggaag tggtactgat ttcaccctca agatctccag ggtcgaggca 660
gaagatgtcg gcgtttatta ctgttttcaa ggttcacatg tgccgcgcac attcggtggg 720
ggtactaaag tagaaatcaa a 741
<210> 164
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002161 PA2.1.10 scFv (humanized)
<400> 164
gaggtgcagc tggtggagtc tgggggtggg ctggtgaagc ctgggggctc actgaggctt 60
tcctgcgcgg cttctggata caccttcact agctatcata tacattgggt gcgccaggcc 120
cccggaaaag ggcttgagtg ggtgggatgg atctaccctg gcaatgttaa cacagaatat 180
aatgagaagt tcaagggcag attcaccatt agcagggacg attccaagaa cacactctac 240
ctgcagatga acagcctgaa aactgaagac acggctgtgt attactgtgc gagggaggaa 300
attacctacg ctatggacta ctggggccag ggaaccacag tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacat tcaaatgacc 420
cagagcccat ccagcctgag cgcatctgta ggtgaccggg tcaccatcac ttgtagatcc 480
agtcagagta ttgtacacag taatgggaac acctatttgg aatggtatca gcagaaacca 540
ggtaaagccc caaaattgct catctacaaa gtctctaaca gatttagtgg tgtaccaagc 600
aggttcagcg gttccggaag tggtactgat ttcaccctca cgatctcctc tctccagcca 660
gaagatttcg ccacttatta ctgttttcaa ggttcacatg tgccgcgcac attcggtggg 720
ggtactaaag tagaaatcaa a 741
<210> 165
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002162 PA2.1.14 scFv (humanized)
<400> 165
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc agtgaaggtt 60
tcctgcaagg cttctggata caccttcact agctatcata tacattgggt gcgccaggcc 120
cccggacaag ggcttgagtg gatcggatgg atctaccctg gcaatgttaa cacagaatat 180
aatgagaagt tcaagggcaa agccaccatt accgcggacg aatccacgaa cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt attactgtgc gagggaggaa 300
attacctacg ctatggacta ctggggccag ggaaccctgg tcaccgtgtc ctcaggaggt 360
ggcggctctg gaggcggagg tagcggaggt ggaggctctg gtggcgacat tcaaatgacc 420
cagagcccat ccaccctgag cgcatctgta ggtgaccggg tcaccatcac ttgtagatcc 480
agtcagagta ttgtacacag taatgggaac acctatttgg aatggtatca gcagaaacca 540
ggtaaagccc caaaattgct catctacaaa gtctctaaca gatttagtgg tgtaccagcc 600
aggttcagcg gttccggaag tggtactgaa ttcaccctca cgatctcctc tctccagcca 660
gatgatttcg ccacttatta ctgttttcaa ggttcacatg tgccgcgcac attcggtcag 720
ggtactaaag tagaagtcaa a 741
<210> 166
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002163 PA2.1.18 scFv (humanized)
<400> 166
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc agtgaaggtt 60
tcctgcaagg cttctggata caccttcact agctatcata tgcattgggt gcgccaggcc 120
cccggacaag ggcttgagtg gatcggatac atctaccctg gcaatgttaa cacagaatat 180
aatgagaagt tcaagggcaa agccaccctt accgcggaca aatccacgaa cacagcctac 240
atggagctga gcagcctgag atctgaagac acggctgtgt atttctgtgc gagggaggaa 300
attacctacg ctatggacta ctggggccag ggaaccctgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacgt tcaaatgacc 420
cagagcccat ccaccctgag cgcatctgta ggtgaccggg tcaccatcac ttgtagctcc 480
agtcagagta ttgtacacag taatgggaac acctatatgg aatggtatca gcagaaacca 540
ggtaaagccc caaaattgct catctacaaa gtctctaaca gatttagtgg tgtaccagac 600
aggttcagcg gttccggaag tggtactgaa ttcaccctca cgatctcctc tctccagcca 660
gatgatttcg ccacttatta ctgtcatcaa ggttcacatg tgccgcgcac attcggtcag 720
ggtactaaag tagaagtcaa a 741
<210> 167
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002164 BB7.2 scFv (mouse)
<400> 167
caggtgcagc tgcagcagtc tgggcctgag ctggtgaagc ctggggcctc agtgaagatg 60
tcctgcaagg cttctggata caccttcact agctatcata tccagtgggt gaagcagagg 120
cctggacaag ggcttgagtg gatcggatgg atctaccctg gcgatggtag tacacagtat 180
aatgagaagt tcaagggcaa aaccaccctt accgcggaca aatcctccag cacagcctac 240
atgttgctga gcagcctgac ctctgaagac tctgctatct atttctgtgc gagggagggg 300
acctactacg ctatggacta ctggggccag ggaacctcag tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgatgt tttgatgacc 420
cagactccac tctccctgcc tgtctctctt ggagaccaag tctccatctc ttgtagatcc 480
agtcagagta ttgtacacag taatgggaac acctatttag aatggtatct gcagaaacca 540
ggtcagtctc caaagttgct catctacaaa gtctctaaca gatttagtgg tgtaccagac 600
aggttcagcg gttccggaag tggtactgat ttcaccctca agatctcgag agtggaggct 660
gaggatctgg gagtttatta ctgttttcaa ggttcacatg tgccgcgcac attcggtgga 720
ggtactaaac tggaaatcaa a 741
<210> 168
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002165 BB7.2.1 scFv (humanized)
<400> 168
cagctgcagc tgcaggagtc tgggcccggg ctggtgaagc cttcggaaac gctgagcctc 60
acctgcacgg tttctggata caccttcacc agctatcata tccagtggat ccgacagccc 120
cctggaaaag ggcttgagtg gatcggatgg atctaccctg gcgatggttc aacacagtac 180
aatgagaagt tcaagggcag agccacgatt agcgtggaca catccaagaa ccaattctcc 240
ctgaacctgg acagcgtgag tgctgcggac acggccattt attactgtgc gagagaggga 300
acttactacg ctatggacta ctggggcaaa gggagcacgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacat ccagatgacc 420
cagagcccaa gctccctgag tgcgtccgtg ggcgaccgcg tgaccatcac ttgcagatcc 480
tctcagtcca tcgtgcactc caacggcaac acgtacctcg agtggtacca gcagaagccc 540
gggaaggccc cgaaactgct catctacaag gtgagcaacc ggttctccgg cgtccccagc 600
cgcttctcag ggtccggctc ggggacggat ttcaccttca cgattagcag cttgcagccc 660
gaagacatcg ccacgtacta ctgctttcag ggaagtcacg tgccgcgtac cttcgggccg 720
ggcacgaaag tggatattaa g 741
<210> 169
<211> 3217
<212> DNA
<213> Homo sapiens
<400> 169
atggccgtca tggcgccccg aaccctcgtc ctgctactct cgggggctct ggccctgacc 60
cagacctggg cgggtgagtg cggggtcggg agggaaacgg cctctgtggg gagaagcaac 120
gggcccgcct ggcgggggcg caggacccgg gaagccgcgc cgggaggagg gtcgggcggg 180
tctcagccac tcctcgtccc caggctctca ctccatgagg tatttcttca catccgtgtc 240
ccggcccggc cgcggggagc cccgcttcat cgcagtgggc tacgtggacg acacgcagtt 300
cgtgcggttc gacagcgacg ccgcgagcca gaggatggag ccgcgggcgc cgtggataga 360
gcaggagggt ccggagtatt gggacgggga gacacggaaa gtgaaggccc actcacagac 420
tcaccgagtg gacctgggga ccctgcgcgg ctactacaac cagagcgagg ccggtgagtg 480
accccggccc ggggcgcagg tcacgacctc tcatccccca cggacgggcc aggtcgccca 540
cagtctccgg gtccgagatc cgccccgaag ccgcgggacc ccgagaccct tgccccggga 600
gaggcccagg cgcctttacc cggtttcatt ttcagtttag gccaaaaatc cccccaggtt 660
ggtcggggcg gggcggggct cgggggaccg ggctgaccgc ggggtccggg ccaggttctc 720
acaccgtcca gaggatgtat ggctgcgacg tggggtcgga ctggcgcttc ctccgcgggt 780
accaccagta cgcctacgac ggcaaggatt acatcgccct gaaagaggac ctgcgctctt 840
tgaccgcggc ggacatggca gctcagacca ccaagcacaa gtgggaggcg gcccatgtgg 900
cggagcagtt gagagcctac ctggagggca cgtgcgtgga gtggctccgc agatacctgg 960
agaacgggaa ggagacgctg cagcgcacgg gtaccagggg ccacggggcg cctccctgat 1020
cgcctgtaga tctcccgggc tggcctccca caaggagggg agacaattgg gaccaacact 1080
agaatatcgc cctccctctg gtcctgaggg agaggaatcc tcctgggttt ccagatcctg 1140
taccagagag tgactctgag gttccgccct gctctctgac acaattaagg gataaaatct 1200
ctgaaggaat gacgggaaga cgatccctcg aatactgatg agtggttccc tttgacacac 1260
acaggcagca gccttgggcc cgtgactttt cctctcaggc cttgttctct gcttcacact 1320
caatgtgtgt gggggtctga gtccagcact tctgagtcct tcagcctcca ctcaggtcag 1380
gaccagaagt cgctgttccc tcttcaggga ctagaatttt ccacggaata ggagattatc 1440
ccaggtgcct gtgtccaggc tggtgtctgg gttctgtgct cccttcccca tcccaggtgt 1500
cctgtccatt ctcaagatag ccacatgtgt gctggaggag tgtcccatga cagatgcaaa 1560
atgcctgaat gatctgactc ttcctgacag acgcccccaa aacgcatatg actcaccacg 1620
ctgtctctga ccatgaagcc accctgaggt gctgggccct gagcttctac cctgcggaga 1680
tcacactgac ctggcagcgg gatggggagg accagaccca ggacacggag ctcgtggaga 1740
ccaggcctgc aggggatgga accttccaga agtgggcggc tgtggtggtg ccttctggac 1800
aggagcagag atacacctgc catgtgcagc atgagggttt gcccaagccc ctcaccctga 1860
gatggggtaa ggagggagac gggggtgtca tgtcttttag ggaaagcagg agcctctctg 1920
acctttagca gggtcagggc ccctcacctt cccctctttt cccagagccg tcttcccagc 1980
ccaccatccc catcgtgggc atcattgctg gcctggttct ctttggagct gtgatcactg 2040
gagctgtggt cgctgctgtg atgtggagga ggaagagctc aggtggggaa ggggtgaagg 2100
gtgggtctga gatttcttgt ctcactgagg gttccaagac ccaggtagaa gtgtgccctg 2160
cctcgttact gggaagcacc acccacaatt atgggcctac ccagcctggg ccctgtgtgc 2220
cagcacttac tcttttgtaa agcacctgtt aaaatgaagg acagatttat caccttgatt 2280
acagcggtga tgggacctga tcccagcagt cacaagtcac aggggaaggt ccctgaggac 2340
cttcaggagg gcggttggtc caggacccac acctgctttc ttcatgtttc ctgatcccgc 2400
cctgggtctg cagtcacaca tttctggaaa cttctctgag gtccaagact tggaggttcc 2460
tctaggacct taaggccctg actcctttct ggtatctcac aggacatttt cttcccacag 2520
atagaaaagg agggagctac tctcaggctg caagtaagta tgaaggaggc tgatgcctga 2580
ggtccttggg atattgtgtt tgggagccca tgggggagct cacccacccc acaattcctc 2640
ctctagccac atcttctgtg ggatctgacc aggttctgtt tttgttctac cccaggcagt 2700
gacagtgccc agggctctga tgtgtctctc acagcttgta aaggtgagag cctggagggc 2760
ctgatgtgtg ttgggtgttg ggcggaacag tggacacagc tgtgctatgg ggtttctttc 2820
cattggatgt attgagcatg cgatgggctg tttaaagtgt gacccctcac tgtgacagat 2880
acgaatttgt tcatgaatat ttttttctat agtgtgagac agctgccttg tgtgggactg 2940
agaggcaaga gttgttcctg cccttccctt tgtgacttga agaaccctga ctttgtttct 3000
gcaaaggcac ctgcatgtgt ctgtgttcgt gtaggcataa tgtgaggagg tggggagacc 3060
accccacccc catgtccacc atgaccctct tcccacgctg acctgtgctc cctccccaat 3120
catctttcct gttccagaga ggtggggctg aggtgtctcc atctctgtct caacttcatg 3180
gtgcactgag ctgtaacttc ttccttccct attaaaa 3217
<210> 170
<211> 6654
<212> DNA
<213> Homo sapiens
<400> 170
aagtggaggc gtcgcgctgg cgggcattcc tgaagctgac agcattcggg ccgagatgtc 60
tcgctccgtg gccttagctg tgctcgcgct actctctctt tctggcctgg aggctatcca 120
gcgtgagtct ctcctaccct cccgctctgg tccttcctct cccgctctgc accctctgtg 180
gccctcgctg tgctctctcg ctccgtgact tcccttctcc aagttctcct tggtggcccg 240
ccgtggggct agtccagggc tggatctcgg ggaagcggcg gggtggcctg ggagtgggga 300
agggggtgcg cacccgggac gcgcgctact tgcccctttc ggcggggagc aggggagacc 360
tttggcctac ggcgacggga gggtcgggac aaagtttagg gcgtcgataa gcgtcagagc 420
gccgaggttg ggggagggtt tctcttccgc tctttcgcgg ggcctctggc tcccccagcg 480
cagctggagt gggggacggg taggctcgtc ccaaaggcgc ggcgctgagg tttgtgaacg 540
cgtggagggg cgcttggggt ctgggggagg cgtcgcccgg gtaagcctgt ctgctgcggc 600
tctgcttccc ttagactgga gagctgtgga cttcgtctag gcgcccgcta agttcgcatg 660
tcctagcacc tctgggtcta tgtggggcca caccgtgggg aggaaacagc acgcgacgtt 720
tgtagaatgc ttggctgtga tacaaagcgg tttcgaataa ttaacttatt tgttcccatc 780
acatgtcact tttaaaaaat tataagaact acccgttatt gacatctttc tgtgtgccaa 840
ggactttatg tgctttgcgt catttaattt tgaaaacagt tatcttccgc catagataac 900
tactatggtt atcttctgcc tctcacagat gaagaaacta aggcaccgag attttaagaa 960
acttaattac acaggggata aatggcagca atcgagattg aagtcaagcc taaccagggc 1020
ttttgcggga gcgcatgcct tttggctgta attcgtgcat ttttttttaa gaaaaacgcc 1080
tgccttctgc gtgagattct ccagagcaaa ctgggcggca tgggccctgt ggtcttttcg 1140
tacagagggc ttcctctttg gctctttgcc tggttgtttc caagatgtac tgtgcctctt 1200
actttcggtt ttgaaaacat gagggggttg ggcgtggtag cttacgcctg taatcccagc 1260
acttagggag gccgaggcgg gaggatggct tgaggtccgt agttgagacc agcctggcca 1320
acatggtgaa gcctggtctc tacaaaaaat aataacaaaa attagccggg tgtggtggct 1380
cgtgcctgtg gtcccagctg ctccggtggc tgaggcggga ggatctcttg agcttaggct 1440
tttgagctat catggcgcca gtgcactcca gcgtgggcaa cagagcgaga ccctgtctct 1500
caaaaaagaa aaaaaaaaaa aaagaaagag aaaagaaaag aaagaaagaa gtgaaggttt 1560
gtcagtcagg ggagctgtaa aaccattaat aaagataatc caagatggtt accaagactg 1620
ttgaggacgc cagagatctt gagcactttc taagtacctg gcaatacact aagcgcgctc 1680
accttttcct ctggcaaaac atgatcgaaa gcagaatgtt ttgatcatga gaaaattgca 1740
tttaatttga atacaattta tttacaacat aaaggataat gtatatatca ccaccattac 1800
tggtatttgc tggttatgtt agatgtcatt ttaaaaaata acaatctgat atttaaaaaa 1860
aaatcttatt ttgaaaattt ccaaagtaat acatgccatg catagaccat ttctggaaga 1920
taccacaaga aacatgtaat gatgattgcc tctgaaggtc tattttcctc ctctgacctg 1980
tgtgtgggtt ttgtttttgt tttactgtgg gcataaatta atttttcagt taagttttgg 2040
aagcttaaat aactctccaa aagtcataaa gccagtaact ggttgagccc aaattcaaac 2100
ccagcctgtc tgatacttgt cctcttctta gaaaagatta cagtgatgct ctcacaaaat 2160
cttgccgcct tccctcaaac agagagttcc aggcaggatg aatctgtgct ctgatccctg 2220
aggcatttaa tatgttctta ttattagaag ctcagatgca aagagctctc ttagctttta 2280
atgttatgaa aaaaatcagg tcttcattag attccccaat ccacctcttg atggggctag 2340
tagcctttcc ttaatgatag ggtgtttcta gagagatata tctggtcaag gtggcctggt 2400
actcctcctt ctccccacag cctcccagac aaggaggagt agctgccttt tagtgatcat 2460
gtaccctgaa tataagtgta tttaaaagaa ttttatacac atatatttag tgtcaatctg 2520
tatatttagt agcactaaca cttctcttca ttttcaatga aaaatataga gtttataata 2580
ttttcttccc acttccccat ggatggtcta gtcatgcctc tcattttgga aagtactgtt 2640
tctgaaacat taggcaatat attcccaacc tggctagttt acagcaatca cctgtggatg 2700
ctaattaaaa cgcaaatccc actgtcacat gcattactcc atttgatcat aatggaaagt 2760
atgttctgtc ccatttgcca tagtcctcac ctatccctgt tgtattttat cgggtccaac 2820
tcaaccattt aaggtatttg ccagctcttg tatgcattta ggttttgttt ctttgttttt 2880
tagctcatga aattaggtac aaagtcagag aggggtctgg catataaaac ctcagcagaa 2940
ataaagaggt tttgttgttt ggtaagaaca taccttgggt tggttgggca cggtggctcg 3000
tgcctgtaat cccaacactt tgggaggcca aggcaggctg atcacttgaa gttgggagtt 3060
caagaccagc ctggccaaca tggtgaaatc ccgtctctac tgaaaataca aaaattaacc 3120
aggcatggtg gtgtgtgcct gtagtcccag gaatcacttg aacccaggag gcggaggttg 3180
cagtgagctg agatctcacc actgcacact gcactccagc ctgggcaatg gaatgagatt 3240
ccatcccaaa aaataaaaaa ataaaaaaat aaagaacata ccttgggttg atccacttag 3300
gaacctcaga taataacatc tgccacgtat agagcaattg ctatgtccca ggcactctac 3360
tagacacttc atacagttta gaaaatcaga tgggtgtaga tcaaggcagg agcaggaacc 3420
aaaaagaaag gcataaacat aagaaaaaaa atggaagggg tggaaacaga gtacaataac 3480
atgagtaatt tgatgggggc tattatgaac tgagaaatga actttgaaaa gtatcttggg 3540
gccaaatcat gtagactctt gagtgatgtg ttaaggaatg ctatgagtgc tgagagggca 3600
tcagaagtcc ttgagagcct ccagagaaag gctcttaaaa atgcagcgca atctccagtg 3660
acagaagata ctgctagaaa tctgctagaa aaaaaacaaa aaaggcatgt atagaggaat 3720
tatgagggaa agataccaag tcacggttta ttcttcaaaa tggaggtggc ttgttgggaa 3780
ggtggaagct catttggcca gagtggaaat ggaattggga gaaatcgatg accaaatgta 3840
aacacttggt gcctgatata gcttgacacc aagttagccc caagtgaaat accctggcaa 3900
tattaatgtg tcttttcccg atattcctca ggtactccaa agattcaggt ttactcacgt 3960
catccagcag agaatggaaa gtcaaatttc ctgaattgct atgtgtctgg gtttcatcca 4020
tccgacattg aagttgactt actgaagaat ggagagagaa ttgaaaaagt ggagcattca 4080
gacttgtctt tcagcaagga ctggtctttc tatctcttgt actacactga attcaccccc 4140
actgaaaaag atgagtatgc ctgccgtgtg aaccatgtga ctttgtcaca gcccaagata 4200
gttaagtggg gtaagtctta cattcttttg taagctgctg aaagttgtgt atgagtagtc 4260
atatcataaa gctgctttga tataaaaaag gtctatggcc atactaccct gaatgagtcc 4320
catcccatct gatataaaca atctgcatat tgggattgtc agggaatgtt cttaaagatc 4380
agattagtgg cacctgctga gatactgatg cacagcatgg tttctgaacc agtagtttcc 4440
ctgcagttga gcagggagca gcagcagcac ttgcacaaat acatatacac tcttaacact 4500
tcttacctac tggcttcctc tagcttttgt ggcagcttca ggtatattta gcactgaacg 4560
aacatctcaa gaaggtatag gcctttgttt gtaagtcctg ctgtcctagc atcctataat 4620
cctggacttc tccagtactt tctggctgga ttggtatctg aggctagtag gaagggcttg 4680
ttcctgctgg gtagctctaa acaatgtatt catgggtagg aacagcagcc tattctgcca 4740
gccttatttc taaccatttt agacatttgt tagtacatgg tattttaaaa gtaaaactta 4800
atgtcttcct tttttttctc cactgtcttt ttcatagatc gagacatgta agcagcatca 4860
tggaggtaag tttttgacct tgagaaaatg tttttgtttc actgtcctga ggactattta 4920
tagacagctc taacatgata accctcacta tgtggagaac attgacagag taacatttta 4980
gcagggaaag aagaatccta cagggtcatg ttcccttctc ctgtggagtg gcatgaagaa 5040
ggtgtatggc cccaggtatg gccatattac tgaccctcta cagagagggc aaaggaactg 5100
ccagtatggt attgcaggat aaaggcaggt ggttacccac attacctgca aggctttgat 5160
ctttcttctg ccatttccac attggacatc tctgctgagg agagaaaatg aaccactctt 5220
ttcctttgta taatgttgtt ttattcttca gacagaagag aggagttata cagctctgca 5280
gacatcccat tcctgtatgg ggactgtgtt tgcctcttag aggttcccag gccactagag 5340
gagataaagg gaaacagatt gttataactt gatataatga tactataata gatgtaacta 5400
caaggagctc cagaagcaag agagagggag gaacttggac ttctctgcat ctttagttgg 5460
agtccaaagg cttttcaatg aaattctact gcccagggta cattgatgct gaaaccccat 5520
tcaaatctcc tgttatattc tagaacaggg aattgatttg ggagagcatc aggaaggtgg 5580
atgatctgcc cagtcacact gttagtaaat tgtagagcca ggacctgaac tctaatatag 5640
tcatgtgtta cttaatgacg gggacatgtt ctgagaaatg cttacacaaa cctaggtgtt 5700
gtagcctact acacgcatag gctacatggt atagcctatt gctcctagac tacaaacctg 5760
tacagcctgt tactgtactg aatactgtgg gcagttgtaa cacaatggta agtatttgtg 5820
tatctaaaca tagaagttgc agtaaaaata tgctatttta atcttatgag accactgtca 5880
tatatacagt ccatcattga ccaaaacatc atatcagcat tttttcttct aagattttgg 5940
gagcaccaaa gggatacact aacaggatat actctttata atgggtttgg agaactgtct 6000
gcagctactt cttttaaaaa ggtgatctac acagtagaaa ttagacaagt ttggtaatga 6060
gatctgcaat ccaaataaaa taaattcatt gctaaccttt ttcttttctt ttcaggtttg 6120
aagatgccgc atttggattg gatgaattcc aaattctgct tgcttgcttt ttaatattga 6180
tatgcttata cacttacact ttatgcacaa aatgtagggt tataataatg ttaacatgga 6240
catgatcttc tttataattc tactttgagt gctgtctcca tgtttgatgt atctgagcag 6300
gttgctccac aggtagctct aggagggctg gcaacttaga ggtggggagc agagaattct 6360
cttatccaac atcaacatct tggtcagatt tgaactcttc aatctcttgc actcaaagct 6420
tgttaagata gttaagcgtg cataagttaa cttccaattt acatactctg cttagaattt 6480
gggggaaaat ttagaaatat aattgacagg attattggaa atttgttata atgaatgaaa 6540
cattttgtca tataagattc atatttactt cttatacatt tgataaagta aggcatggtt 6600
gtggttaatc tggtttattt ttgttccaca agttaaataa atcataaaac ttga 6654
<210> 171
<211> 1698
<212> DNA
<213> Homo sapiens
<400> 171
cagaagcaga ggggtcaggg cgaagtccca gggccccagg cgtggctctc agggtctcag 60
gccccgaagg cggtgtatgg attggggagt cccagccttg gggattcccc aactccgcag 120
tttcttttct ccctctccca acctatgtag ggtccttctt cctggatact cacgacgcgg 180
acccagttct cactcccatt gggtgtcggg tttccagaga agccaatcag tgtcgtcgcg 240
gtcgcggttc taaagtccgc acgcacccac cgggactcag attctcccca gacgccgagg 300
atggccgtca tggcgccccg aaccctcgtc ctgctactct cgggggctct ggccctgacc 360
cagacctggg cgggctctca ctccatgagg tatttcttca catccgtgtc ccggcccggc 420
cgcggggagc cccgcttcat cgcagtgggc tacgtggacg acacgcagtt cgtgcggttc 480
gacagcgacg ccgcgagcca gaggatggag ccgcgggcgc cgtggataga gcaggagggt 540
ccggagtatt gggacgggga gacacggaaa gtgaaggccc actcacagac tcaccgagtg 600
gacctgggga ccctgcgcgg ctactacaac cagagcgagg ccggttctca caccgtccag 660
aggatgtatg gctgcgacgt ggggtcggac tggcgcttcc tccgcgggta ccaccagtac 720
gcctacgacg gcaaggatta catcgccctg aaagaggacc tgcgctcttg gaccgcggcg 780
gacatggcag ctcagaccac caagcacaag tgggaggcgg cccatgtggc ggagcagttg 840
agagcctacc tggagggcac gtgcgtggag tggctccgca gatacctgga gaacgggaag 900
gagacgctgc agcgcacgga cgcccccaaa acgcatatga ctcaccacgc tgtctctgac 960
catgaagcca ccctgaggtg ctgggccctg agcttctacc ctgcggagat cacactgacc 1020
tggcagcggg atggggagga ccagacccag gacacggagc tcgtggagac caggcctgca 1080
ggggatggaa ccttccagaa gtgggcggct gtggtggtgc cttctggaca ggagcagaga 1140
tacacctgcc atgtgcagca tgagggtttg cccaagcccc tcaccctgag atgggagccg 1200
tcttcccagc ccaccatccc catcgtgggc atcattgctg gcctggttct ctttggagct 1260
gtgatcactg gagctgtggt cgctgctgtg atgtggagga ggaagagctc agatagaaaa 1320
ggagggagct actctcaggc tgcaagcagt gacagtgccc agggctctga tgtgtctctc 1380
acagcttgta aagtgtgaga cagctgcctt gtgtgggact gagaggcaag agttgttcct 1440
gcccttccct ttgtgacttg aagaaccctg actttgtttc tgcaaaggca cctgcatgtg 1500
tctgtgttcg tgtaggcata atgtgaggag gtggggagac caccccaccc ccatgtccac 1560
catgaccctc ttcccacgct gacctgtgct ccctccccaa tcatctttcc tgttccagag 1620
aggtggggct gaggtgtctc catctctgtc tcaacttcat ggtgcactga gctgtaactt 1680
cttccttccc tattaaaa 1698
<210> 172
<211> 943
<212> RNA
<213> Homo sapiens
<400> 172
auuccugaag cugacagcau ucgggccgag augucucgcu ccguggccuu agcugugcuc 60
gcgcuacucu cucuuucugg ccuggaggcu auccagcgua cuccaaagau ucagguuuac 120
ucacgucauc cagcagagaa uggaaaguca aauuuccuga auugcuaugu gucuggguuu 180
cauccauccg acauugaagu ugacuuacug aagaauggag agagaauuga aaaaguggag 240
cauucagacu ugucuuucag caaggacugg ucuuucuauc ucuuguacua cacugaauuc 300
acccccacug aaaaagauga guaugccugc cgugugaacc augugacuuu gucacagccc 360
aagauaguua agugggaucg agacauguaa gcagcaucau ggagguuuga agaugccgca 420
uuuggauugg augaauucca aauucugcuu gcuugcuuuu uaauauugau augcuuauac 480
acuuacacuu uaugcacaaa auguaggguu auaauaaugu uaacauggac augaucuucu 540
uuauaauucu acuuugagug cugucuccau guuugaugua ucugagcagg uugcuccaca 600
gguagcucua ggagggcugg caacuuagag guggggagca gagaauucuc uuauccaaca 660
ucaacaucuu ggucagauuu gaacucuuca aucucuugca cucaaagcuu guuaagauag 720
uuaagcgugc auaaguuaac uuccaauuua cauacucugc uuagaauuug ggggaaaauu 780
uagaaauaua auugacagga uuauuggaaa uuuguuauaa ugaaugaaac auuuugucau 840
auaagauuca uauuuacuuc uuauacauuu gauaaaguaa ggcaugguug ugguuaaucu 900
gguuuauuuu uguuccacaa guuaaauaaa ucauaaaacu uga 943
<210> 173
<211> 522
<212> PRT
<213> Artificial Sequence
<220>
<223> C1765 (anti-HLA-A*02 chimeric antigen receptor) Prot
<400> 173
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser
20 25 30
Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser
35 40 45
Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu
50 55 60
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn
65 70 75 80
Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
85 90 95
Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val
100 105 110
Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Ser Gly Gly Gly
115 120 125
Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gln Val Gln Leu Gln Gln Ser Gly Pro
145 150 155 160
Glu Leu Val Lys Pro Gly Ala Ser Val Arg Ile Ser Cys Lys Ala Ser
165 170 175
Gly Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Lys Gln Arg Pro
180 185 190
Gly Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn
195 200 205
Thr Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp
210 215 220
Lys Ser Ser Ser Thr Ala Tyr Met His Leu Ser Ser Leu Thr Ser Glu
225 230 235 240
Asp Ser Ala Val Tyr Phe Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met
245 250 255
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His
515 520
<210> 174
<211> 522
<212> PRT
<213> Artificial Sequence
<220>
<223> C2162 (anti-HLA-A*02 chimeric antigen receptor) Protein
<400> 174
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Val Gln Ser Gly Ala Glu
20 25 30
Val Lys Lys Pro Gly Ser Ser Val Lys Val Ser Cys Lys Ala Ser Gly
35 40 45
Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Arg Gln Ala Pro Gly
50 55 60
Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr
65 70 75 80
Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Ile Thr Ala Asp Glu
85 90 95
Ser Thr Asn Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly Asp Arg Val
165 170 175
Thr Ile Thr Cys Arg Ser Ser Gln Ser Ile Val His Ser Asn Gly Asn
180 185 190
Thr Tyr Leu Glu Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Phe Gln Gly Ser His Val
245 250 255
Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Val Lys Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His
515 520
<210> 175
<211> 532
<212> PRT
<213> Artificial Sequence
<220>
<223> CT486 (anti-EGFR chimeric antigen receptor) Protein
<400> 175
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Lys Gln Ser Gly Pro Gly
20 25 30
Leu Val Gln Pro Ser Gln Ser Leu Ser Ile Thr Cys Thr Val Ser Gly
35 40 45
Phe Ser Leu Thr Asn Tyr Gly Val His Trp Val Arg Gln Ser Pro Gly
50 55 60
Lys Gly Leu Glu Trp Leu Gly Val Ile Trp Ser Gly Gly Asn Thr Asp
65 70 75 80
Tyr Asn Thr Pro Phe Thr Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser
85 90 95
Lys Ser Gln Val Phe Phe Lys Met Asn Ser Leu Gln Ser Asn Asp Thr
100 105 110
Ala Ile Tyr Tyr Cys Ala Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe
115 120 125
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
145 150 155 160
Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly Glu Arg
165 170 175
Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn Ile His
180 185 190
Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile Lys Tyr
195 200 205
Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser Glu Asp
225 230 235 240
Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr Thr Phe
245 250 255
Gly Ala Gly Thr Lys Leu Glu Leu Lys Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly Val
305 310 315 320
Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp
325 330 335
Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met
340 345 350
Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala
355 360 365
Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys
370 375 380
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
385 390 395 400
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
405 410 415
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
420 425 430
Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
435 440 445
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
450 455 460
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
465 470 475 480
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
485 490 495
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
500 505 510
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
515 520 525
Leu Pro Pro Arg
530
<210> 176
<211> 532
<212> PRT
<213> Artificial Sequence
<220>
<223> CT488 (anti-EGFR chimeric antigen receptor) Prot
<400> 176
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Gln Glu Ser Gly Pro Gly
20 25 30
Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly
35 40 45
Gly Ser Val Ser Ser Gly Asp Tyr Tyr Trp Thr Trp Ile Arg Gln Ser
50 55 60
Pro Gly Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Tyr Ser Gly Asn
65 70 75 80
Thr Asn Tyr Asn Pro Ser Leu Lys Ser Arg Leu Thr Ile Ser Ile Asp
85 90 95
Thr Ser Lys Thr Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala
100 105 110
Asp Thr Ala Ile Tyr Tyr Cys Val Arg Asp Arg Val Thr Gly Ala Phe
115 120 125
Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
145 150 155 160
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
165 170 175
Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp
195 200 205
Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln His Phe Asp His Leu Pro Leu Ala Phe
245 250 255
Gly Gly Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly Val
305 310 315 320
Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp
325 330 335
Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met
340 345 350
Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala
355 360 365
Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys
370 375 380
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
385 390 395 400
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
405 410 415
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
420 425 430
Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
435 440 445
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
450 455 460
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
465 470 475 480
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
485 490 495
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
500 505 510
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
515 520 525
Leu Pro Pro Arg
530
<210> 177
<211> 534
<212> PRT
<213> Artificial Sequence
<220>
<223> CT489 (anti-EGFR chimeric antigen receptor) Prot
<400> 177
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Gln Glu Ser Gly Pro Gly
20 25 30
Leu Val Lys Pro Ser Gln Thr Leu Ser Leu Thr Cys Thr Val Ser Gly
35 40 45
Gly Ser Ile Ser Ser Gly Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro
50 55 60
Pro Gly Lys Gly Leu Glu Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser
65 70 75 80
Thr Asp Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Met Ser Val Asp
85 90 95
Thr Ser Lys Asn Gln Phe Ser Leu Lys Val Asn Ser Val Thr Ala Ala
100 105 110
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Val Ser Ile Phe Gly Val Gly
115 120 125
Thr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
145 150 155 160
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
165 170 175
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
180 185 190
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
195 200 205
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
225 230 235 240
Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu
245 250 255
Thr Phe Gly Gly Gly Thr Lys Ala Glu Ile Lys Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly
305 310 315 320
Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile
325 330 335
Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met
340 345 350
Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro
355 360 365
Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg
370 375 380
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
385 390 395 400
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
405 410 415
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
420 425 430
Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
435 440 445
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
450 455 460
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
465 470 475 480
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
485 490 495
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
500 505 510
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
515 520 525
Gln Ala Leu Pro Pro Arg
530
<210> 178
<211> 21
<212> PRT
<213> Unknown
<220>
<223> T2A self-cleaving peptide
<400> 178
Gly Ser Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu
1 5 10 15
Glu Asn Pro Gly Pro
20
<210> 179
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> shRNA0089
<400> 179
gcactcaaag cttgttaaga tcgaaatctt aacaagcttt gagtgc 46
<210> 180
<211> 1600
<212> DNA
<213> Artificial Sequence
<220>
<223> CT486 (anti-EGFR chimeric antigen receptor) DNA
<400> 180
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgtcagg tgcagctgaa gcagtccggc cccggcctgg tgcagccctc ccagtccctg 120
tccatcacct gcaccgtgtc cggcttctcc ctgaccaact acggcgtgca ctgggtgcgg 180
cagtcccccg gcaagggcct ggagtggctg ggcgtgatct ggtccggcgg caacaccgac 240
tacaacaccc ccttcacctc ccggctgtcc atcaacaagg acaactccaa gtcccaggtg 300
ttcttcaaga tgaactccct gcagtccaac gacaccgcca tctactactg cgcccgggcc 360
ctgacctact acgactacga gttcgcctac tggggccagg gcaccctggt gaccgtgtcc 420
gccggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgacatc 480
ctgctgaccc agtcccccgt gatcctgtcc gtgtcccccg gcgagcgggt gtccttctcc 540
tgccgggcct cccagtccat cggcaccaac atccactggt accagcagcg gaccaacggc 600
tccccccggc tgctgatcaa gtacgcctcc gagtccatct ccggcatccc ctcccggttc 660
tccggctccg gctccggcac cgacttcacc ctgtccatca actccgtgga gtccgaggac 720
atcgccgact actactgcca gcagaacaac aactggccca ccaccttcgg cgccggcacc 780
aagctggagc tgaagaccac gacgccagcg ccgcgaccac caacaccggc gcccaccatc 840
gcgtcgcagc ccctgtccct gcgcccagag gcgtgccggc cagcggcggg gggcgcagtg 900
cacacgaggg ggctggactt cgcctgtgat ttctgggtgc tggtcgttgt gggcggcgtg 960
ctggcctgct acagcctgct ggtgacagtg gccttcatca tcttttgggt gaggagcaag 1020
cggagcagac tgctgcacag cgactacatg aacatgaccc cccggaggcc tggccccacc 1080
cggaagcact accagcccta cgcccctccc agggatttcg ccgcctaccg gagcaaacgg 1140
ggcagaaaga aactcctgta tatattcaaa caaccattta tgaggccagt acaaactact 1200
caagaggaag atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg 1260
agagtgaagt tcagcaggag cgcagacgcc cccgcgtaca agcagggcca gaaccagctc 1320
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gcgtagaggc 1380
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 1440
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 1500
cggaggggca aggggcacga tggcctttac cagggactca gtacagccac caaggacacc 1560
tacgacgccc ttcacatgca ggccctgccc cctcgctagc 1600
<210> 181
<211> 1566
<212> DNA
<213> Artificial Sequence
<220>
<223> C2162 (anti-HLA-A*02 chimeric antigen receptor) DNA
<400> 181
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgccagg tgcagctggt gcagtctggg gctgaggtga agaagcctgg gtcctcagtg 120
aaggtttcct gcaaggcttc tggatacacc ttcactagct atcatataca ttgggtgcgc 180
caggcccccg gacaagggct tgaatggatt ggctggatct accctggcaa tgttaacaca 240
gaatataatg agaagttcaa gggcaaagcc accattaccg cggacgaatc cacgaacaca 300
gcctacatgg agctgagcag cctgagatct gaagacacgg ctgtgtatta ctgtgcgagg 360
gaggaaatta cctacgctat ggactactgg ggccagggaa ccctggtcac cgtgtcctca 420
ggaggtggcg gctctggagg cggaggtagc ggaggtggag gctctggtgg cgacattcaa 480
atgacccaga gcccatccac cctgagcgca tctgtaggtg accgggtcac catcacttgt 540
agatccagtc agagtattgt acacagtaat gggaacacct atttggaatg gtaccagcag 600
aagccaggta aagccccaaa attgctcatc tacaaagtct ctaacagatt tagtggtgta 660
ccagccaggt tcagcggttc cggaagtggt actgaattca ccctcacgat ctcctctctc 720
cagccagatg atttcgccac ttattactgt tttcaaggtt cacatgtgcc gcgcacattc 780
ggtcagggta ctaaagtaga agtcaaatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccac 1566
<210> 182
<211> 1599
<212> DNA
<213> Artificial Sequence
<220>
<223> CT488 (anti-EGFR chimeric antigen receptor) DNA
<400> 182
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgtcagg tgcagctgca ggagtccggc cccggcctgg tgaagccctc cgagaccctg 120
tccctgacct gcaccgtgtc cggcggctcc gtgtcctccg gcgactacta ctggacctgg 180
atccggcagt cccccggcaa gggcctggag tggatcggcc acatctacta ctccggcaac 240
accaactaca acccctccct gaagtcccgg ctgaccatct ccatcgacac ctccaagacc 300
cagttctccc tgaagctgtc ctccgtgacc gccgccgaca ccgccatcta ctactgcgtg 360
cgggaccggg tgaccggcgc cttcgacatc tggggccagg gcaccatggt gaccgtgtcc 420
tccggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgacatc 480
cagatgaccc agtccccctc ctccctgtcc gcctccgtgg gcgaccgggt gaccatcacc 540
tgccaggcct cccaggacat ctccaactac ctgaactggt accagcagaa gcccggcaag 600
gcccccaagc tgctgatcta cgacgcctcc aacctggaga ccggcgtgcc ctcccggttc 660
tccggctccg gctccggcac cgacttcacc ttcaccatct cctccctgca gcccgaggac 720
atcgccacct acttctgcca gcacttcgac cacctgcccc tggccttcgg cggcggcacc 780
aaggtggaga tcaagaccac gacgccagcg ccgcgaccac caacaccggc gcccaccatc 840
gcgtcgcagc ccctgtccct gcgcccagag gcgtgccggc cagcggcggg gggcgcagtg 900
cacacgaggg ggctggactt cgcctgtgat ttctgggtgc tggtcgttgt gggcggcgtg 960
ctggcctgct acagcctgct ggtgacagtg gccttcatca tcttttgggt gaggagcaag 1020
cggagcagac tgctgcacag cgactacatg aacatgaccc cccggaggcc tggccccacc 1080
cggaagcact accagcccta cgcccctccc agggatttcg ccgcctaccg gagcaaacgg 1140
ggcagaaaga aactcctgta tatattcaaa caaccattta tgaggccagt acaaactact 1200
caagaggaag atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg 1260
agagtgaagt tcagcaggag cgcagacgcc cccgcgtaca agcagggcca gaaccagctc 1320
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gcgtagaggc 1380
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 1440
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 1500
cggaggggca aggggcacga tggcctttac cagggactca gtacagccac caaggacacc 1560
tacgacgccc ttcacatgca ggccctgccc cctcgctag 1599
<210> 183
<211> 1569
<212> DNA
<213> Artificial Sequence
<220>
<223> C1765 (anti-HLA-A*02 chimeric antigen receptor) DNA
<400> 183
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgtgatg ttttgatgac ccaaactcca ctctccctgc ctgtcagtct tggagatcaa 120
gcctccatct cttgcagatc tagtcagagc attgtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaaacc aggccagtct ccaaagctcc tgatctacaa agtttccaac 240
cgattttctg gggtcccaga caggttcagt ggcagtggat cagggacaga tttcacactc 300
aagatcagta gagtggaggc tgaggatctg ggagtttatt actgctttca aggttcacat 360
gttcctcgga cgtccggtgg aggcaccaag ctggaaatca aaggcggagg tggaagcgga 420
gggggaggat ctggcggcgg aggaagcgga ggccaggtcc agctgcagca gtctggacct 480
gagctggtga agcctggggc ttcagtgagg atatcctgca aggcttctgg ctacaccttc 540
acaagttacc atatacattg ggtgaagcag aggcctggac agggacttga gtggattgga 600
tggatttatc ctggaaatgt taatactgag tacaatgaga agttcaaggg caaggccaca 660
ctgactgcag acaaatcgtc cagcacagcc tacatgcacc tcagcagcct gacctctgag 720
gactctgcgg tctatttctg tgccagagag gagattacct atgctatgga ctactggggt 780
caaggaacct cagtcaccgt gtcctcatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccca gtgaccccct ggagctcgtg gtctcaggac cgtctggggg ccccagctcc 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac ccccaccggg 960
tcggatcccc agagtggtct gggaaggcac ctgggggttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctcctcctc ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggac 1260
actcggagcc cacacgatga agacccccag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagga gagaaatggc ctctcctcct tccccactgt ctggggaatt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagcc 1440
ccccaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccactag 1569
<210> 184
<211> 1605
<212> DNA
<213> Artificial Sequence
<220>
<223> CT489 (anti-EGFR chimeric antigen receptor) DNA
<400> 184
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgtcagg tccagctcca agagtcagga ccaggtctgg tcaaaccctc acagacgctc 120
tctcttactt gcacagtatc aggcggatcc attagttccg gtgattatta ctggtcttgg 180
atccgccagc ctcctggtaa ggggctggag tggatcggat acatatacta cagtggttcc 240
actgattata atccgtcact caagtcccga gtaactatgt ccgtggatac tagcaaaaat 300
cagttcagcc tcaaagtcaa ttccgtaact gccgccgaca ccgcggtcta ttattgtgcg 360
cgggtgagca tcttcggcgt cggtacgttc gattactggg gacaaggaac tctcgtaaca 420
gtaagctctg gcggaggtgg aagcggaggg ggaggatctg gcggcggagg aagcggaggc 480
gagattgtaa tgacccaaag ccctgccact ctttctctgt ctccggggga acgagcgact 540
ctgtcatgta gggcgtccca gagtgtctct agctacctcg catggtatca gcagaaaccc 600
ggacaagccc ctcggctctt gatttacgat gcctcaaaca gagcaacggg gattccggca 660
cggttcagcg ggtctggtag cgggacagac ttcacactga cgatctcttc actcgaacca 720
gaagattttg cagtctacta ttgccatcag tatggttcta cgccacttac atttggcggg 780
ggcaccaagg cggagataaa aaccacgacg ccagcgccgc gaccaccaac accggcgccc 840
accatcgcgt cgcagcccct gtccctgcgc ccagaggcgt gccggccagc ggcggggggc 900
gcagtgcaca cgagggggct ggacttcgcc tgtgatttct gggtgctggt cgttgtgggc 960
ggcgtgctgg cctgctacag cctgctggtg acagtggcct tcatcatctt ttgggtgagg 1020
agcaagcgga gcagactgct gcacagcgac tacatgaaca tgaccccccg gaggcctggc 1080
cccacccgga agcactacca gccctacgcc cctcccaggg atttcgccgc ctaccggagc 1140
aaacggggca gaaagaaact cctgtatata ttcaaacaac catttatgag gccagtacaa 1200
actactcaag aggaagatgg ctgtagctgc cgatttccag aagaagaaga aggaggatgt 1260
gaactgagag tgaagttcag caggagcgca gacgcccccg cgtacaagca gggccagaac 1320
cagctctata acgagctcaa tctaggacga agagaggagt acgatgtttt ggacaagcgt 1380
agaggccggg accctgagat ggggggaaag ccgagaagga agaaccctca ggaaggcctg 1440
tacaatgaac tgcagaaaga taagatggcg gaggcctaca gtgagattgg gatgaaaggc 1500
gagcgccgga ggggcaaggg gcacgatggc ctttaccagg gactcagtac agccaccaag 1560
gacacctacg acgcccttca catgcaggcc ctgccccctc gctag 1605
<210> 185
<211> 3234
<212> DNA
<213> Artificial Sequence
<220>
<223> C2162-T2A-CT489 DNA
<400> 185
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgccagg tgcagctggt gcagtctggg gctgaggtga agaagcctgg gtcctcagtg 120
aaggtttcct gcaaggcttc tggatacacc ttcactagct atcatataca ttgggtgcgc 180
caggcccccg gacaagggct tgaatggatt ggctggatct accctggcaa tgttaacaca 240
gaatataatg agaagttcaa gggcaaagcc accattaccg cggacgaatc cacgaacaca 300
gcctacatgg agctgagcag cctgagatct gaagacacgg ctgtgtatta ctgtgcgagg 360
gaggaaatta cctacgctat ggactactgg ggccagggaa ccctggtcac cgtgtcctca 420
ggaggtggcg gctctggagg cggaggtagc ggaggtggag gctctggtgg cgacattcaa 480
atgacccaga gcccatccac cctgagcgca tctgtaggtg accgggtcac catcacttgt 540
agatccagtc agagtattgt acacagtaat gggaacacct atttggaatg gtaccagcag 600
aagccaggta aagccccaaa attgctcatc tacaaagtct ctaacagatt tagtggtgta 660
ccagccaggt tcagcggttc cggaagtggt actgaattca ccctcacgat ctcctctctc 720
cagccagatg atttcgccac ttattactgt tttcaaggtt cacatgtgcc gcgcacattc 780
ggtcagggta ctaaagtaga agtcaaatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccacggat ccggagaggg cagaggcagc ctgctgacat gtggcgacgt ggaagagaac 1620
cctggcccca tggacatgag ggtccccgct cagctcctgg ggctcctgct actctggctc 1680
cgaggtgcca gatgtcaggt ccagctccaa gagtcaggac caggtctggt caaaccctca 1740
cagacgctct ctcttacttg cacagtatca ggcggatcca ttagttccgg tgattattac 1800
tggtcttgga tccgccagcc tcctggtaag gggctggagt ggatcggata catatactac 1860
agtggttcca ctgattataa tccgtcactc aagtcccgag taactatgtc cgtggatact 1920
agcaaaaatc agttcagcct caaagtcaat tccgtaactg ccgccgacac cgcggtctat 1980
tattgtgcgc gggtgagcat cttcggcgtc ggtacgttcg attactgggg acaaggaact 2040
ctcgtaacag taagctctgg cggaggtgga agcggagggg gaggatctgg cggcggagga 2100
agcggaggcg agattgtaat gacccaaagc cctgccactc tttctctgtc tccgggggaa 2160
cgagcgactc tgtcatgtag ggcgtcccag agtgtctcta gctacctcgc atggtatcag 2220
cagaaacccg gacaagcccc tcggctcttg atttacgatg cctcaaacag agcaacgggg 2280
attccggcac ggttcagcgg gtctggtagc gggacagact tcacactgac gatctcttca 2340
ctcgaaccag aagattttgc agtctactat tgccatcagt atggttctac gccacttaca 2400
tttggcgggg gcaccaaggc ggagataaaa accacgacgc cagcgccgcg accaccaaca 2460
ccggcgccca ccatcgcgtc gcagcccctg tccctgcgcc cagaggcgtg ccggccagcg 2520
gcggggggcg cagtgcacac gagggggctg gacttcgcct gtgatttctg ggtgctggtc 2580
gttgtgggcg gcgtgctggc ctgctacagc ctgctggtga cagtggcctt catcatcttt 2640
tgggtgagga gcaagcggag cagactgctg cacagcgact acatgaacat gaccccccgg 2700
aggcctggcc ccacccggaa gcactaccag ccctacgccc ctcccaggga tttcgccgcc 2760
taccggagca aacggggcag aaagaaactc ctgtatatat tcaaacaacc atttatgagg 2820
ccagtacaaa ctactcaaga ggaagatggc tgtagctgcc gatttccaga agaagaagaa 2880
ggaggatgtg aactgagagt gaagttcagc aggagcgcag acgcccccgc gtacaagcag 2940
ggccagaacc agctctataa cgagctcaat ctaggacgaa gagaggagta cgatgttttg 3000
gacaagcgta gaggccggga ccctgagatg gggggaaagc cgagaaggaa gaaccctcag 3060
gaaggcctgt acaatgaact gcagaaagat aagatggcgg aggcctacag tgagattggg 3120
atgaaaggcg agcgccggag gggcaagggg cacgatggcc tttaccaggg actcagtaca 3180
gccaccaagg acacctacga cgcccttcac atgcaggccc tgccccctcg ctag 3234
<210> 186
<211> 1077
<212> PRT
<213> Artificial Sequence
<220>
<223> C2162-T2A-CT489 Prot
<400> 186
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Val Gln Ser Gly Ala Glu
20 25 30
Val Lys Lys Pro Gly Ser Ser Val Lys Val Ser Cys Lys Ala Ser Gly
35 40 45
Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Arg Gln Ala Pro Gly
50 55 60
Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr
65 70 75 80
Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Ile Thr Ala Asp Glu
85 90 95
Ser Thr Asn Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly Asp Arg Val
165 170 175
Thr Ile Thr Cys Arg Ser Ser Gln Ser Ile Val His Ser Asn Gly Asn
180 185 190
Thr Tyr Leu Glu Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Phe Gln Gly Ser His Val
245 250 255
Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Val Lys Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Gly Ser Gly Glu Gly Arg
515 520 525
Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met
530 535 540
Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu
545 550 555 560
Arg Gly Ala Arg Cys Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
565 570 575
Val Lys Pro Ser Gln Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly
580 585 590
Ser Ile Ser Ser Gly Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro
595 600 605
Gly Lys Gly Leu Glu Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr
610 615 620
Asp Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Met Ser Val Asp Thr
625 630 635 640
Ser Lys Asn Gln Phe Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp
645 650 655
Thr Ala Val Tyr Tyr Cys Ala Arg Val Ser Ile Phe Gly Val Gly Thr
660 665 670
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
675 680 685
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Glu
690 695 700
Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu
705 710 715 720
Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu
725 730 735
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
740 745 750
Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly Ser
755 760 765
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
770 775 780
Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu Thr
785 790 795 800
Phe Gly Gly Gly Thr Lys Ala Glu Ile Lys Thr Thr Thr Pro Ala Pro
805 810 815
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
820 825 830
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
835 840 845
Gly Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly
850 855 860
Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe
865 870 875 880
Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn
885 890 895
Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr
900 905 910
Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys
915 920 925
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
930 935 940
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
945 950 955 960
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
965 970 975
Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
980 985 990
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
995 1000 1005
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
1010 1015 1020
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
1025 1030 1035
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
1040 1045 1050
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
1055 1060 1065
Leu His Met Gln Ala Leu Pro Pro Arg
1070 1075
<210> 187
<211> 3234
<212> DNA
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT489 DNA
<400> 187
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgcgatg ttctgatgac ccaaactcca ctctccctgc ctgtcagtct tggagatcaa 120
gcctccatct cttgcagatc tagtcagagc attgtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaagcc aggccagtct ccaaagctgc tcatctacaa agtttccaac 240
cgattttctg gggtcccaga cagatttagc ggatctggct ctgggaccga tttcacactc 300
aagatcagta gagtggaggc tgaggatctg ggagtttatt actgctttca aggttcacat 360
gttcctcgga cgtccggtgg aggcacaaag ctggaaatca agggaggtgg cggctctgga 420
ggcggaggta gcggaggtgg aggctctggt ggccaggtcc agctgcagca gtctggacct 480
gagctggtga agccaggggc ttcagtgagg atatcctgta aggcctctgg ctacaccttt 540
acaagttacc atatacattg ggtgaagcag aggcctggac agggactcga atggattgga 600
tggatttatc ctggaaatgt taatactgag tacaatgaga agttcaaggg caaggccaca 660
ctgactgcag acaaatcgtc cagcacagcc tacatgcacc tcagcagcct gacctctgag 720
gactctgcgg tctatttctg tgccagagag gagattacct atgctatgga ttattggggt 780
caaggaacct cagtcaccgt gtcctcatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccacggat ccggagaggg cagaggcagc ctgctgacat gtggcgacgt ggaagagaac 1620
cctggcccca tggacatgag ggtccccgct cagctcctgg ggctcctgct actctggctc 1680
cgaggtgcca gatgtcaggt ccagctccaa gagtcaggac caggtctggt caaaccctca 1740
cagacgctct ctcttacttg cacagtatca ggcggatcca ttagttccgg tgattattac 1800
tggtcttgga tccgccagcc tcctggtaag gggctggagt ggatcggata catatactac 1860
agtggttcca ctgattataa tccgtcactc aagtcccgag taactatgtc cgtggatact 1920
agcaaaaatc agttcagcct caaagtcaat tccgtaactg ccgccgacac cgcggtctat 1980
tattgtgcgc gggtgagcat cttcggcgtc ggtacgttcg attactgggg acaaggaact 2040
ctcgtaacag taagctctgg cggaggtgga agcggagggg gaggatctgg cggcggagga 2100
agcggaggcg agattgtaat gacccaaagc cctgccactc tttctctgtc tccgggggaa 2160
cgagcgactc tgtcatgtag ggcgtcccag agtgtctcta gctacctcgc atggtatcag 2220
cagaaacccg gacaagcccc tcggctcttg atttacgatg cctcaaacag agcaacgggg 2280
attccggcac ggttcagcgg gtctggtagc gggacagact tcacactgac gatctcttca 2340
ctcgaaccag aagattttgc agtctactat tgccatcagt atggttctac gccacttaca 2400
tttggcgggg gcaccaaggc ggagataaaa accacgacgc cagcgccgcg accaccaaca 2460
ccggcgccca ccatcgcgtc gcagcccctg tccctgcgcc cagaggcgtg ccggccagcg 2520
gcggggggcg cagtgcacac gagggggctg gacttcgcct gtgatttctg ggtgctggtc 2580
gttgtgggcg gcgtgctggc ctgctacagc ctgctggtga cagtggcctt catcatcttt 2640
tgggtgagga gcaagcggag cagactgctg cacagcgact acatgaacat gaccccccgg 2700
aggcctggcc ccacccggaa gcactaccag ccctacgccc ctcccaggga tttcgccgcc 2760
taccggagca aacggggcag aaagaaactc ctgtatatat tcaaacaacc atttatgagg 2820
ccagtacaaa ctactcaaga ggaagatggc tgtagctgcc gatttccaga agaagaagaa 2880
ggaggatgtg aactgagagt gaagttcagc aggagcgcag acgcccccgc gtacaagcag 2940
ggccagaacc agctctataa cgagctcaat ctaggacgaa gagaggagta cgatgttttg 3000
gacaagcgta gaggccggga ccctgagatg gggggaaagc cgagaaggaa gaaccctcag 3060
gaaggcctgt acaatgaact gcagaaagat aagatggcgg aggcctacag tgagattggg 3120
atgaaaggcg agcgccggag gggcaagggg cacgatggcc tttaccaggg actcagtaca 3180
gccaccaagg acacctacga cgcccttcac atgcaggccc tgccccctcg ctaa 3234
<210> 188
<211> 1077
<212> PRT
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT489 Prot
<400> 188
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser
20 25 30
Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser
35 40 45
Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu
50 55 60
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn
65 70 75 80
Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
85 90 95
Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val
100 105 110
Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Ser Gly Gly Gly
115 120 125
Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gln Val Gln Leu Gln Gln Ser Gly Pro
145 150 155 160
Glu Leu Val Lys Pro Gly Ala Ser Val Arg Ile Ser Cys Lys Ala Ser
165 170 175
Gly Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Lys Gln Arg Pro
180 185 190
Gly Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn
195 200 205
Thr Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp
210 215 220
Lys Ser Ser Ser Thr Ala Tyr Met His Leu Ser Ser Leu Thr Ser Glu
225 230 235 240
Asp Ser Ala Val Tyr Phe Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met
245 250 255
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Gly Ser Gly Glu Gly Arg
515 520 525
Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met
530 535 540
Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu
545 550 555 560
Arg Gly Ala Arg Cys Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
565 570 575
Val Lys Pro Ser Gln Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly
580 585 590
Ser Ile Ser Ser Gly Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro
595 600 605
Gly Lys Gly Leu Glu Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr
610 615 620
Asp Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Met Ser Val Asp Thr
625 630 635 640
Ser Lys Asn Gln Phe Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp
645 650 655
Thr Ala Val Tyr Tyr Cys Ala Arg Val Ser Ile Phe Gly Val Gly Thr
660 665 670
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
675 680 685
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Glu
690 695 700
Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu
705 710 715 720
Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu
725 730 735
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
740 745 750
Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly Ser
755 760 765
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
770 775 780
Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu Thr
785 790 795 800
Phe Gly Gly Gly Thr Lys Ala Glu Ile Lys Thr Thr Thr Pro Ala Pro
805 810 815
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
820 825 830
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
835 840 845
Gly Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly
850 855 860
Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe
865 870 875 880
Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn
885 890 895
Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr
900 905 910
Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys
915 920 925
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
930 935 940
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
945 950 955 960
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
965 970 975
Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
980 985 990
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
995 1000 1005
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
1010 1015 1020
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
1025 1030 1035
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
1040 1045 1050
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
1055 1060 1065
Leu His Met Gln Ala Leu Pro Pro Arg
1070 1075
<210> 189
<211> 3228
<212> DNA
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT486 DNA
<400> 189
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgcgatg ttctgatgac ccaaactcca ctctccctgc ctgtcagtct tggagatcaa 120
gcctccatct cttgcagatc tagtcagagc attgtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaagcc aggccagtct ccaaagctgc tcatctacaa agtttccaac 240
cgattttctg gggtcccaga cagatttagc ggatctggct ctgggaccga tttcacactc 300
aagatcagta gagtggaggc tgaggatctg ggagtttatt actgctttca aggttcacat 360
gttcctcgga cgtccggtgg aggcacaaag ctggaaatca agggaggtgg cggctctgga 420
ggcggaggta gcggaggtgg aggctctggt ggccaggtcc agctgcagca gtctggacct 480
gagctggtga agccaggggc ttcagtgagg atatcctgta aggcctctgg ctacaccttt 540
acaagttacc atatacattg ggtgaagcag aggcctggac agggactcga atggattgga 600
tggatttatc ctggaaatgt taatactgag tacaatgaga agttcaaggg caaggccaca 660
ctgactgcag acaaatcgtc cagcacagcc tacatgcacc tcagcagcct gacctctgag 720
gactctgcgg tctatttctg tgccagagag gagattacct atgctatgga ttattggggt 780
caaggaacct cagtcaccgt gtcctcatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccacggat ccggagaggg cagaggcagc ctgctgacat gtggcgacgt ggaagagaac 1620
cctggcccca tggacatgag ggtccccgct cagctcctgg ggctcctgct actctggctc 1680
cgaggtgcca gatgtcaggt gcagctgaag cagtccggcc ccggcctggt gcagccctcc 1740
cagtccctgt ccatcacctg caccgtgtcc ggcttctccc tgaccaacta cggcgtgcac 1800
tgggtgcggc agtcccccgg caagggcctg gagtggctgg gcgtgatctg gtccggcggc 1860
aacaccgact acaacacccc cttcacctcc cggctgtcca tcaacaagga caactccaag 1920
tcccaggtgt tcttcaagat gaactccctg cagtccaacg acaccgccat ctactactgc 1980
gcccgggccc tgacctacta cgactacgag ttcgcctact ggggccaggg caccctggtg 2040
accgtgtccg ccggcggagg tggaagcgga gggggaggat ctggcggcgg aggaagcgga 2100
ggcgacatcc tgctgaccca gtcccccgtg atcctgtccg tgtcccccgg cgagcgggtg 2160
tccttctcct gccgggcctc ccagtccatc ggcaccaaca tccactggta ccagcagcgg 2220
accaacggct ccccccggct gctgatcaag tacgcctccg agtccatctc cggcatcccc 2280
tcccggttct ccggctccgg ctccggcacc gacttcaccc tgtccatcaa ctccgtggag 2340
tccgaggaca tcgccgacta ctactgccag cagaacaaca actggcccac caccttcggc 2400
gccggcacca agctggagct gaagaccacg acgccagcgc cgcgaccacc aacaccggcg 2460
cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg 2520
ggcgcagtgc acacgagggg gctggacttc gcctgtgatt tctgggtgct ggtcgttgtg 2580
ggcggcgtgc tggcctgcta cagcctgctg gtgacagtgg ccttcatcat cttttgggtg 2640
aggagcaagc ggagcagact gctgcacagc gactacatga acatgacccc ccggaggcct 2700
ggccccaccc ggaagcacta ccagccctac gcccctccca gggatttcgc cgcctaccgg 2760
agcaaacggg gcagaaagaa actcctgtat atattcaaac aaccatttat gaggccagta 2820
caaactactc aagaggaaga tggctgtagc tgccgatttc cagaagaaga agaaggagga 2880
tgtgaactga gagtgaagtt cagcaggagc gcagacgccc ccgcgtacaa gcagggccag 2940
aaccagctct ataacgagct caatctagga cgaagagagg agtacgatgt tttggacaag 3000
cgtagaggcc gggaccctga gatgggggga aagccgagaa ggaagaaccc tcaggaaggc 3060
ctgtacaatg aactgcagaa agataagatg gcggaggcct acagtgagat tgggatgaaa 3120
ggcgagcgcc ggaggggcaa ggggcacgat ggcctttacc agggactcag tacagccacc 3180
aaggacacct acgacgccct tcacatgcag gccctgcccc ctcgctaa 3228
<210> 190
<211> 1075
<212> PRT
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT486 Prot
<400> 190
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser
20 25 30
Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser
35 40 45
Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu
50 55 60
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn
65 70 75 80
Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
85 90 95
Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val
100 105 110
Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Ser Gly Gly Gly
115 120 125
Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gln Val Gln Leu Gln Gln Ser Gly Pro
145 150 155 160
Glu Leu Val Lys Pro Gly Ala Ser Val Arg Ile Ser Cys Lys Ala Ser
165 170 175
Gly Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Lys Gln Arg Pro
180 185 190
Gly Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn
195 200 205
Thr Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp
210 215 220
Lys Ser Ser Ser Thr Ala Tyr Met His Leu Ser Ser Leu Thr Ser Glu
225 230 235 240
Asp Ser Ala Val Tyr Phe Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met
245 250 255
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Gly Ser Gly Glu Gly Arg
515 520 525
Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met
530 535 540
Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu
545 550 555 560
Arg Gly Ala Arg Cys Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu
565 570 575
Val Gln Pro Ser Gln Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe
580 585 590
Ser Leu Thr Asn Tyr Gly Val His Trp Val Arg Gln Ser Pro Gly Lys
595 600 605
Gly Leu Glu Trp Leu Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr
610 615 620
Asn Thr Pro Phe Thr Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys
625 630 635 640
Ser Gln Val Phe Phe Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala
645 650 655
Ile Tyr Tyr Cys Ala Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala
660 665 670
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Gly Gly Gly Gly
675 680 685
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Leu
690 695 700
Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly Glu Arg Val
705 710 715 720
Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn Ile His Trp
725 730 735
Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile Lys Tyr Ala
740 745 750
Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
755 760 765
Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser Glu Asp Ile
770 775 780
Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr Thr Phe Gly
785 790 795 800
Ala Gly Thr Lys Leu Glu Leu Lys Thr Thr Thr Pro Ala Pro Arg Pro
805 810 815
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
820 825 830
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
835 840 845
Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly Val Leu
850 855 860
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
865 870 875 880
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
885 890 895
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
900 905 910
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu
915 920 925
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
930 935 940
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
945 950 955 960
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
965 970 975
Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
980 985 990
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
995 1000 1005
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
1010 1015 1020
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
1025 1030 1035
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
1040 1045 1050
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
1055 1060 1065
Met Gln Ala Leu Pro Pro Arg
1070 1075
<210> 191
<211> 3228
<212> DNA
<213> Artificial Sequence
<220>
<223> C2162-T2A-CT486 DNA
<400> 191
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgccagg tgcagctggt gcagtctggg gctgaggtga agaagcctgg gtcctcagtg 120
aaggtttcct gcaaggcttc tggatacacc ttcactagct atcatataca ttgggtgcgc 180
caggcccccg gacaagggct tgagtggatc ggatggatct accctggcaa tgttaacaca 240
gaatataatg agaagttcaa gggcaaagcc accattaccg cggacgaatc cacgaacaca 300
gcctacatgg agctgagcag cctgagatct gaagacacgg ctgtgtatta ctgtgcgagg 360
gaggaaatta cctacgctat ggactactgg ggccagggaa ccctggtcac cgtgtcctca 420
ggaggtggcg gctctggagg cggaggtagc ggaggtggag gctctggtgg cgacattcaa 480
atgacccaga gcccatccac cctgagcgca tctgtaggtg accgggtcac catcacttgt 540
agatccagtc agagtattgt acacagtaat gggaacacct atttggaatg gtatcagcag 600
aaaccaggta aagccccaaa attgctcatc tacaaagtct ctaacagatt tagtggtgta 660
ccagccaggt tcagcggttc cggaagtggt actgaattca ccctcacgat ctcctctctc 720
cagccagatg atttcgccac ttattactgt tttcaaggtt cacatgtgcc gcgcacattc 780
ggtcagggta ctaaagtaga agtcaaatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccacggat ccggagaggg cagaggcagc ctgctgacat gtggcgacgt ggaagagaac 1620
cctggcccca tggacatgag ggtccccgct cagctcctgg ggctcctgct actctggctc 1680
cgaggtgcca gatgtcaggt gcagctgaag cagtccggcc ccggcctggt gcagccctcc 1740
cagtccctgt ccatcacctg caccgtgtcc ggcttctccc tgaccaacta cggcgtgcac 1800
tgggtgcggc agtcccccgg caagggcctg gagtggctgg gcgtgatctg gtccggcggc 1860
aacaccgact acaacacccc cttcacctcc cggctgtcca tcaacaagga caactccaag 1920
tcccaggtgt tcttcaagat gaactccctg cagtccaacg acaccgccat ctactactgc 1980
gcccgggccc tgacctacta cgactacgag ttcgcctact ggggccaggg caccctggtg 2040
accgtgtccg ccggcggagg tggaagcgga gggggaggat ctggcggcgg aggaagcgga 2100
ggcgacatcc tgctgaccca gtcccccgtg atcctgtccg tgtcccccgg cgagcgggtg 2160
tccttctcct gccgggcctc ccagtccatc ggcaccaaca tccactggta ccagcagcgg 2220
accaacggct ccccccggct gctgatcaag tacgcctccg agtccatctc cggcatcccc 2280
tcccggttct ccggctccgg ctccggcacc gacttcaccc tgtccatcaa ctccgtggag 2340
tccgaggaca tcgccgacta ctactgccag cagaacaaca actggcccac caccttcggc 2400
gccggcacca agctggagct gaagaccacg acgccagcgc cgcgaccacc aacaccggcg 2460
cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg 2520
ggcgcagtgc acacgagggg gctggacttc gcctgtgatt tctgggtgct ggtcgttgtg 2580
ggcggcgtgc tggcctgcta cagcctgctg gtgacagtgg ccttcatcat cttttgggtg 2640
aggagcaagc ggagcagact gctgcacagc gactacatga acatgacccc ccggaggcct 2700
ggccccaccc ggaagcacta ccagccctac gcccctccca gggatttcgc cgcctaccgg 2760
agcaaacggg gcagaaagaa actcctgtat atattcaaac aaccatttat gaggccagta 2820
caaactactc aagaggaaga tggctgtagc tgccgatttc cagaagaaga agaaggagga 2880
tgtgaactga gagtgaagtt cagcaggagc gcagacgccc ccgcgtacaa gcagggccag 2940
aaccagctct ataacgagct caatctagga cgaagagagg agtacgatgt tttggacaag 3000
cgtagaggcc gggaccctga gatgggggga aagccgagaa ggaagaaccc tcaggaaggc 3060
ctgtacaatg aactgcagaa agataagatg gcggaggcct acagtgagat tgggatgaaa 3120
ggcgagcgcc ggaggggcaa ggggcacgat ggcctttacc agggactcag tacagccacc 3180
aaggacacct acgacgccct tcacatgcag gccctgcccc ctcgctag 3228
<210> 192
<211> 1075
<212> PRT
<213> Artificial Sequence
<220>
<223> C2162-T2A-CT486 Prot
<400> 192
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Gln Val Gln Leu Val Gln Ser Gly Ala Glu
20 25 30
Val Lys Lys Pro Gly Ser Ser Val Lys Val Ser Cys Lys Ala Ser Gly
35 40 45
Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Arg Gln Ala Pro Gly
50 55 60
Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn Thr
65 70 75 80
Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Ile Thr Ala Asp Glu
85 90 95
Ser Thr Asn Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly Asp Arg Val
165 170 175
Thr Ile Thr Cys Arg Ser Ser Gln Ser Ile Val His Ser Asn Gly Asn
180 185 190
Thr Tyr Leu Glu Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Phe Gln Gly Ser His Val
245 250 255
Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Val Lys Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Gly Ser Gly Glu Gly Arg
515 520 525
Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met
530 535 540
Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu
545 550 555 560
Arg Gly Ala Arg Cys Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu
565 570 575
Val Gln Pro Ser Gln Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe
580 585 590
Ser Leu Thr Asn Tyr Gly Val His Trp Val Arg Gln Ser Pro Gly Lys
595 600 605
Gly Leu Glu Trp Leu Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr
610 615 620
Asn Thr Pro Phe Thr Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys
625 630 635 640
Ser Gln Val Phe Phe Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala
645 650 655
Ile Tyr Tyr Cys Ala Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala
660 665 670
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Gly Gly Gly Gly
675 680 685
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Leu
690 695 700
Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly Glu Arg Val
705 710 715 720
Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn Ile His Trp
725 730 735
Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile Lys Tyr Ala
740 745 750
Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
755 760 765
Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser Glu Asp Ile
770 775 780
Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr Thr Phe Gly
785 790 795 800
Ala Gly Thr Lys Leu Glu Leu Lys Thr Thr Thr Pro Ala Pro Arg Pro
805 810 815
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
820 825 830
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
835 840 845
Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly Val Leu
850 855 860
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
865 870 875 880
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
885 890 895
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
900 905 910
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu
915 920 925
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
930 935 940
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
945 950 955 960
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
965 970 975
Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
980 985 990
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
995 1000 1005
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
1010 1015 1020
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
1025 1030 1035
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
1040 1045 1050
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
1055 1060 1065
Met Gln Ala Leu Pro Pro Arg
1070 1075
<210> 193
<211> 3228
<212> DNA
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT488 DNA
<400> 193
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgcgatg ttctgatgac ccaaactcca ctctccctgc ctgtcagtct tggagatcaa 120
gcctccatct cttgcagatc tagtcagagc attgtacata gtaatggaaa cacctattta 180
gaatggtacc tgcagaagcc aggccagtct ccaaagctgc tcatctacaa agtttccaac 240
cgattttctg gggtcccaga cagatttagc ggatctggct ctgggaccga tttcacactc 300
aagatcagta gagtggaggc tgaggatctg ggagtttatt actgctttca aggttcacat 360
gttcctcgga cgtccggtgg aggcacaaag ctggaaatca agggaggtgg cggctctgga 420
ggcggaggta gcggaggtgg aggctctggt ggccaggtcc agctgcagca gtctggacct 480
gagctggtga agccaggggc ttcagtgagg atatcctgta aggcctctgg ctacaccttt 540
acaagttacc atatacattg ggtgaagcag aggcctggac agggactcga atggattgga 600
tggatttatc ctggaaatgt taatactgag tacaatgaga agttcaaggg caaggccaca 660
ctgactgcag acaaatcgtc cagcacagcc tacatgcacc tcagcagcct gacctctgag 720
gactctgcgg tctatttctg tgccagagag gagattacct atgctatgga ttattggggt 780
caaggaacct cagtcaccgt gtcctcatac ggctcacaga gctccaaacc ctacctgctg 840
actcacccta gtgatcctct ggagctcgtg gtctcaggac cgtctggagg cccaagctct 900
ccgacaacag gccccacctc cacatctggc cctgaggacc agcccctcac acccaccggg 960
tcggatcctc agagtggtct gggaagacac ctgggagttg tgatcggcat cttggtggcc 1020
gtcatcctac tgctcctcct cctgctcctg ctcttcctca tcctccgaca tcgacgtcag 1080
ggcaaacact ggacatcgac ccagagaaag gctgatttcc aacatcctgc aggggctgtg 1140
gggccagagc ccacagacag aggcctgcag tggaggtcca gcccagctgc cgatgcccag 1200
gaagaaaacc tctatgctgc cgtgaagcac acacagcctg aggatggggt ggagatggat 1260
actcggagcc cacacgatga agatccacag gcagtgacgt atgccgaggt gaaacactcc 1320
agacctagaa gggaaatggc ctctcctcct tccccactgt ctggagagtt cctggacaca 1380
aaggacagac aggcggaaga ggacaggcag atggacactg aggctgctgc atctgaagct 1440
cctcaggatg tgacctacgc ccagctgcac agcttgaccc tcagacggga ggcaactgag 1500
cctcctccat cccaggaagg gccctctcca gctgtgccca gcatctacgc cactctggcc 1560
atccacggat ccggagaggg cagaggcagc ctgctgacat gtggcgacgt ggaagagaac 1620
cctggcccca tggacatgag ggtccccgct cagctcctgg ggctcctgct actctggctc 1680
cgaggtgcca gatgtcaggt gcagctgcag gagtccggcc ccggcctggt gaagccctcc 1740
gagaccctgt ccctgacctg caccgtgtcc ggcggctccg tgtcctccgg cgactactac 1800
tggacctgga tccggcagtc ccccggcaag ggcctggagt ggatcggcca catctactac 1860
tccggcaaca ccaactacaa cccctccctg aagtcccggc tgaccatctc catcgacacc 1920
tccaagaccc agttctccct gaagctgtcc tccgtgaccg ccgccgacac cgccatctac 1980
tactgcgtgc gggaccgggt gaccggcgcc ttcgacatct ggggccaggg caccatggtg 2040
accgtgtcct ccggcggagg tggaagcgga gggggaggat ctggcggcgg aggaagcgga 2100
ggcgacatcc agatgaccca gtccccctcc tccctgtccg cctccgtggg cgaccgggtg 2160
accatcacct gccaggcctc ccaggacatc tccaactacc tgaactggta ccagcagaag 2220
cccggcaagg cccccaagct gctgatctac gacgcctcca acctggagac cggcgtgccc 2280
tcccggttct ccggctccgg ctccggcacc gacttcacct tcaccatctc ctccctgcag 2340
cccgaggaca tcgccaccta cttctgccag cacttcgacc acctgcccct ggccttcggc 2400
ggcggcacca aggtggagat caagaccacg acgccagcgc cgcgaccacc aacaccggcg 2460
cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg 2520
ggcgcagtgc acacgagggg gctggacttc gcctgtgatt tctgggtgct ggtcgttgtg 2580
ggcggcgtgc tggcctgcta cagcctgctg gtgacagtgg ccttcatcat cttttgggtg 2640
aggagcaagc ggagcagact gctgcacagc gactacatga acatgacccc ccggaggcct 2700
ggccccaccc ggaagcacta ccagccctac gcccctccca gggatttcgc cgcctaccgg 2760
agcaaacggg gcagaaagaa actcctgtat atattcaaac aaccatttat gaggccagta 2820
caaactactc aagaggaaga tggctgtagc tgccgatttc cagaagaaga agaaggagga 2880
tgtgaactga gagtgaagtt cagcaggagc gcagacgccc ccgcgtacaa gcagggccag 2940
aaccagctct ataacgagct caatctagga cgaagagagg agtacgatgt tttggacaag 3000
cgtagaggcc gggaccctga gatgggggga aagccgagaa ggaagaaccc tcaggaaggc 3060
ctgtacaatg aactgcagaa agataagatg gcggaggcct acagtgagat tgggatgaaa 3120
ggcgagcgcc ggaggggcaa ggggcacgat ggcctttacc agggactcag tacagccacc 3180
aaggacacct acgacgccct tcacatgcag gccctgcccc ctcgctaa 3228
<210> 194
<211> 1075
<212> PRT
<213> Artificial Sequence
<220>
<223> C1765-T2A-CT488 Prot
<400> 194
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser
20 25 30
Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser
35 40 45
Gln Ser Ile Val His Ser Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu
50 55 60
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn
65 70 75 80
Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
85 90 95
Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val
100 105 110
Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Arg Thr Ser Gly Gly Gly
115 120 125
Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gln Val Gln Leu Gln Gln Ser Gly Pro
145 150 155 160
Glu Leu Val Lys Pro Gly Ala Ser Val Arg Ile Ser Cys Lys Ala Ser
165 170 175
Gly Tyr Thr Phe Thr Ser Tyr His Ile His Trp Val Lys Gln Arg Pro
180 185 190
Gly Gln Gly Leu Glu Trp Ile Gly Trp Ile Tyr Pro Gly Asn Val Asn
195 200 205
Thr Glu Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp
210 215 220
Lys Ser Ser Ser Thr Ala Tyr Met His Leu Ser Ser Leu Thr Ser Glu
225 230 235 240
Asp Ser Ala Val Tyr Phe Cys Ala Arg Glu Glu Ile Thr Tyr Ala Met
245 250 255
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Tyr Gly Ser
260 265 270
Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu
275 280 285
Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly
290 295 300
Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly
305 310 315 320
Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly
325 330 335
Ile Leu Val Ala Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe
340 345 350
Leu Ile Leu Arg His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln
355 360 365
Arg Lys Ala Asp Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro
370 375 380
Thr Asp Arg Gly Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln
385 390 395 400
Glu Glu Asn Leu Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly
405 410 415
Val Glu Met Asp Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val
420 425 430
Thr Tyr Ala Glu Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser
435 440 445
Pro Pro Ser Pro Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln
450 455 460
Ala Glu Glu Asp Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala
465 470 475 480
Pro Gln Asp Val Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg
485 490 495
Glu Ala Thr Glu Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val
500 505 510
Pro Ser Ile Tyr Ala Thr Leu Ala Ile His Gly Ser Gly Glu Gly Arg
515 520 525
Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met
530 535 540
Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu
545 550 555 560
Arg Gly Ala Arg Cys Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
565 570 575
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly
580 585 590
Ser Val Ser Ser Gly Asp Tyr Tyr Trp Thr Trp Ile Arg Gln Ser Pro
595 600 605
Gly Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Tyr Ser Gly Asn Thr
610 615 620
Asn Tyr Asn Pro Ser Leu Lys Ser Arg Leu Thr Ile Ser Ile Asp Thr
625 630 635 640
Ser Lys Thr Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp
645 650 655
Thr Ala Ile Tyr Tyr Cys Val Arg Asp Arg Val Thr Gly Ala Phe Asp
660 665 670
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
675 680 685
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
690 695 700
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
705 710 715 720
Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn Trp
725 730 735
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp Ala
740 745 750
Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
755 760 765
Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile
770 775 780
Ala Thr Tyr Phe Cys Gln His Phe Asp His Leu Pro Leu Ala Phe Gly
785 790 795 800
Gly Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro
805 810 815
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
820 825 830
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
835 840 845
Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val Gly Gly Val Leu
850 855 860
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
865 870 875 880
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
885 890 895
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
900 905 910
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu
915 920 925
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
930 935 940
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
945 950 955 960
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
965 970 975
Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
980 985 990
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
995 1000 1005
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
1010 1015 1020
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
1025 1030 1035
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
1040 1045 1050
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
1055 1060 1065
Met Gln Ala Leu Pro Pro Arg
1070 1075
<210> 195
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002166 BB7.2.2 scFv (humanized)
<400> 195
gaggtgcagc tggtgcagtc tggggccgag ctgaagaagc ctgggtcctc ggtgaaggtg 60
tcctgcaagg cttctggata caccttcacc agctatcata tccagtgggt aaaacaggcc 120
cctggacaag ggcttgagtg gatcggatgg atctaccctg gcgatggttc aacacagtac 180
aatgagaagt tcaagggcaa agccacgctt accgtggaca aatccacgaa cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtat attactgtgc gagagaggga 300
acttactacg ctatggacta ctggggccaa gggaccctgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacat ccagatgacc 420
cagagcccat ccaccctgag tgcgtccgtg ggcgaccgcg tgaccatcac ttgcagatcc 480
tctcagtcca tcgtgcactc caacggcaac acgtacctcg agtggtacca gcagaagccc 540
gggaaggccc cgaaactgct catctacaag gtgagcaacc ggttctccgg cgtccccagc 600
cgcttctcag ggtccggctc ggggacggat ttcaccctca cgattagcag cttgcagccc 660
gatgacttcg ccacgtacta ctgctttcag ggaagtcacg tgccgcgtac cttcgggcag 720
ggcacgaaag tggaagttaa g 741
<210> 196
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002167 BB7.2.3 scFv (humanized)
<400> 196
caggtgcagc tggtgcagtc tggggccgag gtgaagaagc ctgggtcctc ggtgaaggtg 60
tcctgcaagg cttctggata caccttcacc agctatcata tccagtgggt acgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atctaccctg gcgatggttc aacacagtac 180
aatgagaagt tcaagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtat attactgtgc gagagaggga 300
acttactacg ctatggacta ctggggccaa gggaccacgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgagat cgtcctgacc 420
cagagcccag ggaccctgag tttgtccccg ggcgagcgcg cgaccctcag ttgcagatcc 480
tctcagtcca tcgtgcactc caacggcaac acgtacctcg agtggtacca gcagaagccc 540
gggcaggccc cgcgactgct catctacaag gtgagcaacc ggttctccgg catccccgac 600
cgcttctcag ggtccggctc ggggacggat ttcaccctca cgattagccg cttggagccc 660
gaagacttcg ccgtgtacta ctgctttcag ggaagtcacg tgccgcgtac cttcgggggg 720
ggcacgaaag tggaaattaa g 741
<210> 197
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002168 BB7.2.5 scFv (humanized)
<400> 197
caggtgaccc tgaagcagtc tggggccgag gtgaagaagc ctgggtcctc ggtgaaggtg 60
tcctgcacgg cttctggata caccttcacc agctatcatg tcagctgggt acgacaggcc 120
cctggacaag ggcttgagtg gttgggaagg atctaccctg gcgatggttc aacacagtac 180
aatgagaagt tcaagggcaa agtcacgatt accgcggaca aatccatgga cacatccttc 240
atggagctga ccagcctgac atctgaggac acggccgtat attactgtgc gagagaggga 300
acttactacg ctatggacct ctggggccaa gggaccctgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgagat cgtcctgacc 420
cagagcccag ggaccctgag tttgtccccg ggcgagcgcg cgaccctcag ttgcagatcc 480
tctcagtcca tcgtgcactc caacggcaac acgtacctcg cgtggtacca gcagaagccc 540
gggcaggccc cgcgactgct catctccaag gtgagcaacc ggttctccgg cgtccccgac 600
cgcttctcag ggtccggctc ggggacggat ttcaccctca cgattagccg cttggagccc 660
gaagacttcg ccgtgtacta ctgccaacag ggaagtcacg tgccgcgtac cttcgggggg 720
ggcacgaaag tggaaattaa g 741
<210> 198
<211> 741
<212> DNA
<213> Artificial Sequence
<220>
<223> C-002169 BB7.2.6 scFv (humanized)
<400> 198
caggtgcagc tggtgcagtc tggggccgag gtgaagaagc ctggggcctc ggtgaaggtg 60
tcctgcaagg cttctggata caccttcacc agctatcata tgcactgggt acgacaggcc 120
cctggacaaa ggcttgagtg gatgggatgg atctaccctg gcgatggttc aacacagtac 180
aatgagaagt tcaagggcaa agtcacgatt acccgggaca catccgcgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtat attactgtgc gagagaggga 300
acttactacg ctatggacta ctggggccaa gggaccctgg tcaccgtgtc ctcaggcgga 360
ggtggaagcg gagggggagg atctggcggc ggaggaagcg gaggcgacat cgtcatgacc 420
cagaccccac tgtccctgcc tgtgaccccg ggcgagcccg cgagcatcag ttgcagatcc 480
tctcagtcca tcgtgcactc caacggcaac acgtacctcg actggtacct gcagaagccc 540
gggcagtccc cgcaactgct catctacaag gtgagcaacc ggttctccgg cgtccccgac 600
cgcttctcag ggtccggctc ggggacggat ttcaccctca agattagccg cgtggaggcc 660
gaagacgtcg gcgtgtacta ctgcatgcag ggaagtcacg tgccgcgtac cttcgggggg 720
ggcacgaaag tggaaattaa g 741
<210> 199
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.10_scFv
<400> 199
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 200
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.9_scFv
<400> 200
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ser Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 201
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.8_scFv
<400> 201
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Leu Thr Gln Ser
130 135 140
Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 202
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.7_scFv
<400> 202
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu Trp
35 40 45
Leu Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Glu Ile Val Leu Thr Gln Ser
130 135 140
Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 203
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.6_scFv
<400> 203
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 204
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.5_scFv
<400> 204
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ser Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 205
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.4_scFv
<400> 205
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 206
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.3_scFv
<400> 206
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Tyr Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 207
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.2_scFv
<400> 207
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Ile Arg Gln His Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 208
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-B*07 antigen binding domain BB7.1.1_scFv
<400> 208
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Tyr Ser Ile Thr Ser Gly
20 25 30
Tyr Ser Trp His Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu Trp
35 40 45
Leu Gly Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu
50 55 60
Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Val Val Ser His Tyr Ala Met Asp Cys Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Thr Tyr Leu Pro
180 185 190
Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln His Phe Trp Val Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 209
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 9
<400> 209
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg His Tyr Tyr Tyr Tyr Ser Met Asp Val Trp Gly Lys Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 210
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 8
<400> 210
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Leu Ile Tyr Trp Asn Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala His Arg His Met Arg Leu Ser Cys Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
180 185 190
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 211
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 7
<400> 211
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Gln Arg Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Ala Gly Asn Gly Asn Thr Lys Tyr Ser Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Arg Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asn Gly Ala Asn Pro Asp Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 212
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 6
<400> 212
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Leu Pro Gly Ser Tyr Trp Tyr Phe Asp Leu Trp Gly Arg Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 213
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 5
<400> 213
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg His Tyr Tyr Tyr Tyr Tyr Leu Asp Val Trp Gly Lys Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 214
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 4
<400> 214
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Val Trp Val
35 40 45
Ser Arg Ile Asn Ser Asp Gly Ser Ser Thr Ser Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Cys Leu Gly Val Leu Leu Tyr Asn Trp Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 215
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 3
<400> 215
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg His Tyr Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
145 150 155 160
Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
180 185 190
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
210 215 220
Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu
225 230 235 240
Ile Lys
<210> 216
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 2
<400> 216
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Leu Ile Tyr Trp Asn Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala His Lys Thr Thr Ser Phe Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 217
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 antigen binding domain 1
<400> 217
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg His Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 218
<211> 240
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-45
<400> 218
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Val Ser Phe Asp Trp Phe Asp Pro Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
130 135 140
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln
145 150 155 160
Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala
165 170 175
Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro
180 185 190
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
195 200 205
Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser
210 215 220
Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
225 230 235 240
<210> 219
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-44
<400> 219
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Arg Ser Ile Ser Pro Tyr Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 220
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-43
<400> 220
Gln Leu Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Ser Tyr Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Ser Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Val Ile Trp Tyr Trp Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Ser Val Leu Thr Gln Pro
130 135 140
Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser
145 150 155 160
Gly Ser Ser Ser Asn Ile Gly Ser Asn Thr Val Asn Trp Tyr Gln Gln
165 170 175
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Ser Asn Asn Gln Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr
210 215 220
Tyr Cys Ala Ala Trp Asp Asp Ser Leu Asn Gly Trp Val Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Thr Val Leu
245
<210> 221
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-42
<400> 221
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Glu Ile Leu Pro Arg Leu Ser Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
180 185 190
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 222
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-41
<400> 222
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Leu Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Thr Asn Thr Gly Asn Pro Thr Tyr Ala Gln Gly Phe
50 55 60
Thr Gly Arg Phe Val Phe Ser Phe Asp Thr Ser Val Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Cys Ser Leu Lys Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Arg Ala His Ser Ser Trp Tyr Phe Asp Leu Trp Gly
100 105 110
Arg Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 223
<211> 251
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-40
<400> 223
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Arg Ile Lys Ile Leu Pro Arg Leu Gly Tyr Tyr Tyr
100 105 110
Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
145 150 155 160
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
165 170 175
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
180 185 190
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
210 215 220
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
225 230 235 240
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 224
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-39
<400> 224
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Thr Val Ile His Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 225
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-38
<400> 225
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Val Ile Val Glu Val Phe Leu Ser Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 226
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-37
<400> 226
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Ile Phe Ile His Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 227
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-36
<400> 227
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Thr Phe Tyr Ser Tyr Ser Pro Tyr Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met
130 135 140
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr
145 150 155 160
Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser
180 185 190
Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 228
<211> 251
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-35
<400> 228
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Trp Ile Lys Ile Leu Pro Arg Leu Gly Tyr Tyr Tyr
100 105 110
Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
145 150 155 160
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
165 170 175
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
180 185 190
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
210 215 220
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
225 230 235 240
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 229
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-34
<400> 229
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Arg Ser Leu Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 230
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-33
<400> 230
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Lys Ile Leu Ala Pro Asn Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 231
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-32
<400> 231
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Lys Ser Trp Lys Tyr Phe Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 232
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-31
<400> 232
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Asn Thr Ser Thr Ile Pro Tyr Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
180 185 190
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 233
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-30
<400> 233
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Asp Val Asp Lys Asn Thr Ser Thr Ile Tyr Tyr Tyr
100 105 110
Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
145 150 155 160
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
165 170 175
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
180 185 190
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
195 200 205
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
210 215 220
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
225 230 235 240
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 234
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-29
<400> 234
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Gly Asp Ile Val Ser Ser Ser Ala Ile Tyr Trp Tyr
100 105 110
Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Ala
130 135 140
Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Ala Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 235
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-28
<400> 235
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Leu Ile Leu Pro Pro Tyr Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 236
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-27
<400> 236
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Thr Trp Ile Lys Ile Leu Pro Arg Tyr Tyr Tyr Tyr
100 105 110
Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
145 150 155 160
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
165 170 175
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
180 185 190
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
195 200 205
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
210 215 220
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
225 230 235 240
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 237
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-26
<400> 237
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Leu Ser Arg Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 238
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-25
<400> 238
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu His Ile Val Leu Cys Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
145 150 155 160
Ser Gln Gly Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro Glu
165 170 175
Lys Ala Pro Lys Ser Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
180 185 190
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
210 215 220
Gln Tyr Asn Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu
225 230 235 240
Ile Lys
<210> 239
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-24
<400> 239
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Lys Ile Leu Pro Arg Pro Tyr Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
180 185 190
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 240
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-23
<400> 240
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Asn Glu Tyr Phe Gln His Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gln Ser Ala Leu Thr Gln Pro Pro Ser Ala
130 135 140
Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly Thr Ser
145 150 155 160
Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln His Pro
165 170 175
Gly Lys Ala Pro Lys Leu Met Ile Tyr Glu Val Ser Lys Arg Pro Ser
180 185 190
Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser
195 200 205
Leu Thr Val Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Ser Ser Tyr Ala Gly Ser Asn Asn Trp Val Phe Gly Gly Gly Thr Lys
225 230 235 240
Leu Thr Val Leu
<210> 241
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-22
<400> 241
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Leu Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Thr Asn Thr Gly Asn Pro Thr Tyr Ala Gln Gly Phe
50 55 60
Thr Gly Arg Phe Val Phe Ser Phe Asp Thr Ser Val Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Cys Ser Leu Lys Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Thr Ser Tyr Trp Tyr Phe Asp Leu Trp Gly Arg Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser
130 135 140
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
145 150 155 160
Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
180 185 190
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
210 215 220
Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu
225 230 235 240
Ile Lys
<210> 242
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-21
<400> 242
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Glu Ile Val Glu Val Phe Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 243
<211> 241
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-20
<400> 243
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Val Asp Asp Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
130 135 140
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
145 150 155 160
Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys
165 170 175
Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val
180 185 190
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
195 200 205
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
210 215 220
Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
225 230 235 240
Lys
<210> 244
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-19
<400> 244
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Val Trp Val
35 40 45
Ser Arg Ile Asn Ser Asp Gly Ser Ser Thr Ser Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Trp Ser Thr Asn Ile Leu Leu Ser Tyr Thr Lys Ala Phe Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met
130 135 140
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr
145 150 155 160
Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser
180 185 190
Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 245
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-18
<400> 245
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Lys Thr Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 246
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-17
<400> 246
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Lys Tyr Phe His Asp Lys Tyr Phe His Asp Tyr Tyr
100 105 110
Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
145 150 155 160
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
165 170 175
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
180 185 190
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
195 200 205
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
210 215 220
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
225 230 235 240
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 247
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-16
<400> 247
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Thr Ser Val Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 248
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-15
<400> 248
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Lys Ile Leu Pro Tyr Tyr Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 249
<211> 257
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-14
<400> 249
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ile Gln Trp Ile Tyr Ile Tyr Ile Asn Pro Arg Gly Phe Ile Phe
100 105 110
Leu His Asp Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val
115 120 125
Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr
145 150 155 160
Pro Gly Gln Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile
165 170 175
Gly Ser Asn Thr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro
180 185 190
Lys Leu Leu Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp
195 200 205
Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser
210 215 220
Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp
225 230 235 240
Asp Ser Leu Asn Gly Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
245 250 255
Leu
<210> 250
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-13
<400> 250
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Thr Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Lys Glu Asp Val Asp Phe His His Asp Ala Phe Asp
100 105 110
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 251
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-12
<400> 251
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Gly Val Asp Lys Asn Thr Ser Thr Ile Tyr Tyr Tyr
100 105 110
Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
145 150 155 160
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
165 170 175
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
180 185 190
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
195 200 205
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
210 215 220
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
225 230 235 240
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 252
<211> 241
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-11
<400> 252
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Arg Gly Tyr Phe Asp Leu Trp Gly Arg Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
130 135 140
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
145 150 155 160
Gln Gly Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro Glu Lys
165 170 175
Ala Pro Lys Ser Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val
180 185 190
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
195 200 205
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
210 215 220
Tyr Asn Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
225 230 235 240
Lys
<210> 253
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-10
<400> 253
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Val Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Met His Trp Val Gln Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Leu Val Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Glu Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Ile His Val Asp Ile Arg Ser Met Glu Asp Trp Phe Asp
100 105 110
Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 254
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-9
<400> 254
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Ile Gly Thr Ser Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 255
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-8
<400> 255
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Val Val Glu Val Phe Leu Tyr Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
180 185 190
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 256
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-7
<400> 256
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Leu Tyr Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 257
<211> 256
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-6
<400> 257
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Ser Trp Lys Tyr Phe Tyr Pro Arg Gly Ser Ile Phe
100 105 110
Ile His Tyr Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
145 150 155 160
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln
165 170 175
Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala
180 185 190
Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro
195 200 205
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
210 215 220
Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser
225 230 235 240
Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250 255
<210> 258
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-5
<400> 258
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Arg Ile Val Glu Val Phe Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 259
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-4
<400> 259
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Lys Tyr Phe His Asp Trp Leu Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 260
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-3
<400> 260
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Leu Val Asp Lys Asn Thr Ser Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 261
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-2
<400> 261
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Gln Asn Glu Tyr Phe Gln His Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gln Ser Ala Leu Thr Gln Pro Pro Ser Ala
130 135 140
Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly Thr Ser
145 150 155 160
Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln His Pro
165 170 175
Gly Lys Ala Pro Lys Leu Met Ile Tyr Glu Val Ser Lys Arg Pro Ser
180 185 190
Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser
195 200 205
Leu Thr Val Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Ser Ser Tyr Ala Gly Ser Asn Asn Trp Val Phe Gly Gly Gly Thr Lys
225 230 235 240
Leu Thr Val Leu
<210> 262
<211> 239
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-C*07 antigen binding domain C7-1
<400> 262
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Asn Trp Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser
130 135 140
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly
145 150 155 160
Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
165 170 175
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
180 185 190
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
195 200 205
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn
210 215 220
Ser Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
225 230 235
<210> 263
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 15
<400> 263
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Arg Val Ser Gln Arg Gly Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
180 185 190
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 264
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 16
<400> 264
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asn Pro Asp Lys Asp Pro Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 265
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 17
<400> 265
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Ser Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Phe Tyr Cys Thr Asn Trp Tyr Phe Asp Leu Trp Gly
100 105 110
Arg Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 266
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 18
<400> 266
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Ser Ser Ser Gly Ser Tyr Trp Tyr Phe Asp Leu Trp Gly Arg
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
180 185 190
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 267
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 19
<400> 267
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Gly Tyr Lys Tyr Asn Leu Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
245 250
<210> 268
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 20
<400> 268
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Asp Leu Ser His Tyr Tyr Tyr Tyr Met Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Thr Val Val Thr
130 135 140
Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr
145 150 155 160
Cys Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Tyr Tyr Pro Asn Trp
165 170 175
Phe Gln Gln Lys Pro Gly Gln Ala Pro Arg Ala Leu Ile Tyr Ser Thr
180 185 190
Ser Asn Lys His Ser Trp Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu
195 200 205
Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu
210 215 220
Ala Glu Tyr Tyr Cys Leu Leu Tyr Tyr Gly Gly Ala Gln Trp Val Phe
225 230 235 240
Gly Gly Gly Thr Lys Leu Thr Val Leu
245
<210> 269
<211> 246
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 21
<400> 269
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asn Arg Arg Tyr Asn Ser Cys Tyr Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr
130 135 140
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
145 150 155 160
Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln
165 170 175
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser
180 185 190
Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
195 200 205
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
210 215 220
Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Glu Ile Lys
245
<210> 270
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 22
<400> 270
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Asp Leu Ser His Tyr Tyr Tyr Tyr Leu Asp Val Trp
100 105 110
Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Thr Val Val Thr
130 135 140
Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr
145 150 155 160
Cys Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Tyr Tyr Pro Asn Trp
165 170 175
Phe Gln Gln Lys Pro Gly Gln Ala Pro Arg Ala Leu Ile Tyr Ser Thr
180 185 190
Ser Asn Lys His Ser Trp Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu
195 200 205
Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu
210 215 220
Ala Glu Tyr Tyr Cys Leu Leu Tyr Tyr Gly Gly Ala Gln Trp Val Phe
225 230 235 240
Gly Gly Gly Thr Lys Leu Thr Val Leu
245
<210> 271
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 23
<400> 271
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Asn
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Ala Thr Leu Leu Ser Leu Ser Tyr Asp Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 272
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 24
<400> 272
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Asp Leu Ser His Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Thr Val Val Thr Gln
130 135 140
Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys
145 150 155 160
Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Tyr Tyr Pro Asn Trp Phe
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Ala Leu Ile Tyr Ser Thr Ser
180 185 190
Asn Lys His Ser Trp Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly
195 200 205
Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala
210 215 220
Glu Tyr Tyr Cys Leu Leu Tyr Tyr Gly Gly Ala Gln Trp Val Phe Gly
225 230 235 240
Gly Gly Thr Lys Leu Thr Val Leu
245
<210> 273
<211> 241
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 25
<400> 273
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Glu Arg Asp Arg Trp Phe Asp Pro Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
130 135 140
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
145 150 155 160
Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys
165 170 175
Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val
180 185 190
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
195 200 205
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
210 215 220
Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
225 230 235 240
Lys
<210> 274
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 26
<400> 274
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Pro Pro Ser Leu Gly Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Ser Ala Leu Thr Gln Pro
130 135 140
Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr
145 150 155 160
Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln
165 170 175
Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Glu Val Ser Lys
180 185 190
Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Asn
195 200 205
Thr Ala Ser Leu Thr Val Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp
210 215 220
Tyr Tyr Cys Ser Ser Tyr Ala Gly Ser Asn Asn Trp Val Phe Gly Gly
225 230 235 240
Gly Thr Lys Leu Thr Val Leu
245
<210> 275
<211> 251
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 27
<400> 275
Gln Leu Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Ser Tyr Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Ser Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Ala Tyr Cys Leu Ser Asp Ser Tyr Trp Tyr Phe Asp
100 105 110
Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Ser Val
130 135 140
Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr
145 150 155 160
Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Thr Val Asn
165 170 175
Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Ser
180 185 190
Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys
195 200 205
Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp
210 215 220
Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp Ser Leu Asn Gly Trp
225 230 235 240
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
245 250
<210> 276
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*03 scFv 28
<400> 276
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Ser Trp Lys Tyr Phe Tyr Pro Arg Gly Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 277
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-9
<400> 277
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Trp Thr Ala Trp Tyr Tyr Tyr Met Asp Val Trp Gly
100 105 110
Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gln Thr Val Val Thr Gln
130 135 140
Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys
145 150 155 160
Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Tyr Tyr Pro Asn Trp Phe
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Ala Leu Ile Tyr Ser Thr Ser
180 185 190
Asn Lys His Ser Trp Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly
195 200 205
Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala
210 215 220
Glu Tyr Tyr Cys Leu Leu Tyr Tyr Gly Gly Ala Gln Trp Val Phe Gly
225 230 235 240
Gly Gly Thr Lys Leu Thr Val Leu
245
<210> 278
<211> 244
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-8
<400> 278
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Lys Tyr Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gln Ser Val Leu Thr Gln Pro Pro Ser Ala
130 135 140
Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser Gly Ser Ser
145 150 155 160
Ser Asn Ile Gly Ser Asn Thr Val Asn Trp Tyr Gln Gln Leu Pro Gly
165 170 175
Thr Ala Pro Lys Leu Leu Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu
195 200 205
Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala
210 215 220
Ala Trp Asp Asp Ser Leu Asn Gly Trp Val Phe Gly Gly Gly Thr Lys
225 230 235 240
Leu Thr Val Leu
<210> 279
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-7
<400> 279
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Gln Val Asp Lys Asn Thr Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 280
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-6
<400> 280
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Cys Gln Leu Ala Glu Tyr Phe Gln His Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln Ser Pro
130 135 140
Ser Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
145 150 155 160
Ala Ser Gln Gly Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro
165 170 175
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
180 185 190
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
195 200 205
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
210 215 220
Gln Gln Ala Asn Ser Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val
225 230 235 240
Glu Ile Lys
<210> 281
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-5
<400> 281
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Arg Val Asp Lys Asn Thr Ser Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 282
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-4
<400> 282
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Asp
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Ser Ser Ser
20 25 30
Asn Trp Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Met Ser Val Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Val Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Val Gln Leu Lys Leu Val His Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 283
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-3
<400> 283
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asp Ile Asn Trp Val Arg Gln Ala Thr Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Met Asn Pro Asn Ser Gly Asn Thr Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asn Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Tyr Tyr Asp Tyr Val Thr Val Phe Tyr Phe Gln His Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 284
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-2
<400> 284
Gln Leu Gln Leu Gln Glu Ser Gly Ser Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Ser Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr His Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Arg Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Glu Ser Tyr Pro Ser Phe Tyr Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile Gln Met Thr Gln
130 135 140
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
145 150 155 160
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
180 185 190
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
195 200 205
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
210 215 220
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Gly Gly Thr
225 230 235 240
Lys Val Glu Ile Lys
245
<210> 285
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*01 scFv A1-1
<400> 285
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Leu Ile Tyr Trp Asn Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala His Ser Asn Met Trp Ser Tyr Ser Leu Asn Asp Tyr Tyr Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
130 135 140
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
145 150 155 160
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
180 185 190
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
195 200 205
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
210 215 220
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 286
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 286
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 287
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 287
Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Tyr Tyr Pro Asn
1 5 10
<210> 288
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 288
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 289
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 289
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Thr Val Asn
1 5 10
<210> 290
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 290
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala
1 5 10
<210> 291
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 291
Arg Ala Ser Gln Gly Ile Ser Ser Ala Leu Ala
1 5 10
<210> 292
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1
<400> 292
Arg Ala Ser Gln Gly Ile Ser Ser Trp Leu Ala
1 5 10
<210> 293
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2
<400> 293
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 294
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2
<400> 294
Ser Thr Ser Asn Lys His Ser
1 5
<210> 295
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2
<400> 295
Glu Val Ser Lys Arg Pro Ser
1 5
<210> 296
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2
<400> 296
Ser Asn Asn Gln Arg Pro Ser
1 5
<210> 297
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2
<400> 297
Ala Ala Thr Tyr Leu Pro Asp
1 5
<210> 298
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 298
Gln Gln Ser Tyr Ser Thr Pro Leu Thr
1 5
<210> 299
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 299
Leu Leu Tyr Tyr Gly Gly Ala Gln Trp Val
1 5 10
<210> 300
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 300
Ser Ser Tyr Ala Gly Ser Asn Asn Trp Val
1 5 10
<210> 301
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 301
Ala Ala Trp Asp Asp Ser Leu Asn Gly Trp Val
1 5 10
<210> 302
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 302
Gln His Phe Trp Val Thr Pro Tyr Thr
1 5
<210> 303
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 303
Gln Gln Tyr Asn Ser Tyr Pro Leu Thr
1 5
<210> 304
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3
<400> 304
Gln Gln Ala Asn Ser Phe Pro Leu Thr
1 5
<210> 305
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 305
Ser Tyr Gly Ile Ser
1 5
<210> 306
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 306
Ser Tyr Ser Met Asn
1 5
<210> 307
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 307
Ser Gly Ser Tyr Tyr Trp Ser
1 5
<210> 308
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 308
Ser Tyr Tyr Trp Ser
1 5
<210> 309
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 309
Ser Tyr Trp Ile Gly
1 5
<210> 310
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 310
Ser Asn Tyr Met Ser
1 5
<210> 311
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 311
Ser Ser Ser Tyr Tyr Trp Gly
1 5
<210> 312
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 312
Ser Gly Gly Tyr Tyr Trp Ser
1 5
<210> 313
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 313
Ser Gly Tyr Ser Trp His
1 5
<210> 314
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 314
Thr Ser Gly Val Gly Val Gly
1 5
<210> 315
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 315
Ser Tyr Trp Met His
1 5
<210> 316
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 316
Ser Tyr Asp Met His
1 5
<210> 317
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 317
Ser Tyr Ala Met His
1 5
<210> 318
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 318
Ser Tyr Ala Met Ser
1 5
<210> 319
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 319
Ser Tyr Ala Met Asn
1 5
<210> 320
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 320
Asp Tyr Tyr Met Ser
1 5
<210> 321
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 321
Ser Asn Ser Ala Ala Trp Asn
1 5
<210> 322
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 322
Asp Tyr Tyr Met His
1 5
<210> 323
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 323
Ser Ser Asn Trp Trp Gly
1 5
<210> 324
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 324
Ser Tyr Asp Ile Asn
1 5
<210> 325
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1
<400> 325
Ser Gly Gly Tyr Ser Trp Ser
1 5
<210> 326
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 326
Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu Gln
1 5 10 15
Gly
<210> 327
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 327
Tyr Ile Ser Ser Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 328
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 328
Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 329
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 329
Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe Gln
1 5 10 15
Gly
<210> 330
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 330
Val Ile Tyr Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 331
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 331
Ser Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 332
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 332
Tyr Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 333
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 333
Tyr Ile His Phe Ser Gly Ser Thr His Tyr His Pro Ser Leu Lys Ser
1 5 10 15
<210> 334
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 334
Leu Ile Tyr Trp Asn Asp Asp Lys Arg Tyr Ser Pro Ser Leu Lys Ser
1 5 10 15
<210> 335
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 335
Arg Ile Asn Ser Asp Gly Ser Ser Thr Ser Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 336
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 336
Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys Gly
1 5 10 15
<210> 337
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 337
Trp Ile Asn Ala Gly Asn Gly Asn Thr Lys Tyr Ser Gln Lys Phe Gln
1 5 10 15
Gly
<210> 338
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 338
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 339
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 339
Trp Ile Asn Thr Asn Thr Gly Asn Pro Thr Tyr Ala Gln Gly Phe Thr
1 5 10 15
Gly
<210> 340
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 340
Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 341
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 341
Arg Thr Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 342
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 342
Leu Val Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Glu Lys Phe Gln
1 5 10 15
Gly
<210> 343
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 343
Trp Met Asn Pro Asn Ser Gly Asn Thr Gly Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 344
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 344
Tyr Ile Tyr His Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 345
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 345
Glu Arg Val Ser Gln Arg Gly Ala Phe Asp Ile
1 5 10
<210> 346
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 346
Gly Asn Pro Asp Lys Asp Pro Phe Asp Tyr
1 5 10
<210> 347
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 347
Asp Phe Tyr Cys Thr Asn Trp Tyr Phe Asp Leu
1 5 10
<210> 348
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 348
Glu Ser Ser Ser Gly Ser Tyr Trp Tyr Phe Asp Leu
1 5 10
<210> 349
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 349
Asp Ser Gly Tyr Lys Tyr Asn Leu Tyr Tyr Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 350
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 350
Gly Gly Asp Leu Ser His Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 351
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 351
Glu Asn Arg Arg Tyr Asn Ser Cys Tyr Tyr Phe Asp Tyr
1 5 10
<210> 352
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 352
Gly Gly Asp Leu Ser His Tyr Tyr Tyr Tyr Leu Asp Val
1 5 10
<210> 353
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 353
Ala Thr Leu Leu Ser Leu Ser Tyr Asp Ala Phe Asp Ile
1 5 10
<210> 354
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 354
Gly Gly Asp Leu Ser His Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 355
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 355
Glu Arg Asp Arg Trp Phe Asp Pro
1 5
<210> 356
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 356
Glu Thr Pro Pro Ser Leu Gly Ala Phe Asp Ile
1 5 10
<210> 357
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 357
Glu Ala Tyr Cys Leu Ser Asp Ser Tyr Trp Tyr Phe Asp Leu
1 5 10
<210> 358
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 358
Glu Ser Trp Lys Tyr Phe Tyr Pro Arg Gly Tyr Met Asp Val
1 5 10
<210> 359
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 359
Gly Gly Val Val Ser His Tyr Ala Met Asp Cys
1 5 10
<210> 360
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 360
His Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5
<210> 361
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 361
Lys Thr Thr Ser Phe Tyr Phe Asp Tyr
1 5
<210> 362
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 362
His Tyr Tyr Tyr Tyr Met Asp Val
1 5
<210> 363
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 363
Gly Val Leu Leu Tyr Asn Trp Phe Asp Pro
1 5 10
<210> 364
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 364
His Tyr Tyr Tyr Tyr Tyr Leu Asp Val
1 5
<210> 365
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 365
Asp Leu Pro Gly Ser Tyr Trp Tyr Phe Asp Leu
1 5 10
<210> 366
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 366
Glu Gly Asn Gly Ala Asn Pro Asp Ala Phe Asp Ile
1 5 10
<210> 367
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 367
Arg His Met Arg Leu Ser Cys Phe Asp Tyr
1 5 10
<210> 368
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 368
His Tyr Tyr Tyr Tyr Ser Met Asp Val
1 5
<210> 369
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 369
Ser Phe Asp Trp Phe Asp Pro
1 5
<210> 370
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 370
Glu Arg Ser Ile Ser Pro Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 371
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 371
Asp Ser Val Ile Trp Tyr Trp Phe Asp Pro
1 5 10
<210> 372
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 372
Glu Glu Ile Leu Pro Arg Leu Ser Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 373
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2
<400> 373
Gly Gly Arg Ala His Ser Ser Trp Tyr Phe Asp Leu
1 5 10
<210> 374
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 374
Asp Arg Ile Lys Ile Leu Pro Arg Leu Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 375
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 375
Asp Thr Val Ile His Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 376
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 376
Asp Val Ile Val Glu Val Phe Leu Ser Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 377
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 377
Asp Ile Phe Ile His Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 378
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 378
Asp Gly Thr Phe Tyr Ser Tyr Ser Pro Tyr Tyr Phe Asp Tyr
1 5 10
<210> 379
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 379
Glu Trp Ile Lys Ile Leu Pro Arg Leu Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 380
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 380
Asp Arg Ser Leu Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 381
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 381
Asp Lys Ile Leu Ala Pro Asn Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 382
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 382
Glu Lys Ser Trp Lys Tyr Phe Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 383
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 383
Glu Asn Thr Ser Thr Ile Pro Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 384
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 384
Glu Asp Val Asp Lys Asn Thr Ser Thr Ile Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 385
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 385
Asp Gly Gly Asp Ile Val Ser Ser Ser Ala Ile Tyr Trp Tyr Phe Asp
1 5 10 15
Leu
<210> 386
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 386
Asp Leu Ile Leu Pro Pro Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 387
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 387
Glu Thr Trp Ile Lys Ile Leu Pro Arg Tyr Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 388
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 388
Asp Leu Ser Arg Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 389
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 389
Glu His Ile Val Leu Cys Phe Asp Tyr
1 5
<210> 390
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 390
Asp Lys Ile Leu Pro Arg Pro Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 391
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 391
Gly Ser Asn Glu Tyr Phe Gln His
1 5
<210> 392
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 392
Gly Thr Ser Tyr Trp Tyr Phe Asp Leu
1 5
<210> 393
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 393
Glu Glu Ile Val Glu Val Phe Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 394
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 394
Val Asp Asp Tyr Tyr Phe Asp Tyr
1 5
<210> 395
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 395
Ser Thr Asn Ile Leu Leu Ser Tyr Thr Lys Ala Phe Asp Ile
1 5 10
<210> 396
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 396
Asp Lys Thr Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 397
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 397
Glu Lys Tyr Phe His Asp Lys Tyr Phe His Asp Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 398
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 398
Asp Thr Ser Val Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 399
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 399
Glu Lys Ile Leu Pro Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 400
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 400
Gln Trp Ile Tyr Ile Tyr Ile Asn Pro Arg Gly Phe Ile Phe Leu His
1 5 10 15
Asp Ala Phe Asp Ile
20
<210> 401
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 401
Glu Asp Val Asp Phe His His Asp Ala Phe Asp Ile
1 5 10
<210> 402
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 402
Glu Gly Val Asp Lys Asn Thr Ser Thr Ile Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 403
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 403
Asp Arg Arg Gly Tyr Phe Asp Leu
1 5
<210> 404
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 404
Gly Ile His Val Asp Ile Arg Ser Met Glu Asp Trp Phe Asp Pro
1 5 10 15
<210> 405
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 405
Asp Ile Gly Thr Ser Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 406
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 406
Glu Val Val Glu Val Phe Leu Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 407
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 407
Asp Leu Tyr Tyr Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 408
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 408
Glu Ser Trp Lys Tyr Phe Tyr Pro Arg Gly Ser Ile Phe Ile His Tyr
1 5 10 15
Tyr Tyr Tyr Met Asp Val
20
<210> 409
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 409
Asp Arg Ile Val Glu Val Phe Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 410
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 410
Glu Lys Tyr Phe His Asp Trp Leu Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 411
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 411
Asp Leu Val Asp Lys Asn Thr Ser Tyr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10 15
<210> 412
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 412
Val Gln Asn Glu Tyr Phe Gln His
1 5
<210> 413
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 413
Ala Asn Trp Phe Asp Pro
1 5
<210> 414
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 414
Gly Gly Trp Thr Ala Trp Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 415
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 415
Ala Lys Tyr Tyr Tyr Met Asp Val
1 5
<210> 416
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 416
Asp Gln Val Asp Lys Asn Thr Tyr Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 417
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 417
Ala Cys Gln Leu Ala Glu Tyr Phe Gln His
1 5 10
<210> 418
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 418
Asp Arg Val Asp Lys Asn Thr Ser Tyr Tyr Tyr Met Asp Val
1 5 10
<210> 419
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 419
Arg Val Gln Leu Lys Leu Val His Trp Phe Asp Pro
1 5 10
<210> 420
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 420
Tyr Tyr Asp Tyr Val Thr Val Phe Tyr Phe Gln His
1 5 10
<210> 421
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 421
Glu Ser Tyr Pro Ser Phe Tyr Ala Phe Asp Ile
1 5 10
<210> 422
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3
<400> 422
Ser Asn Met Trp Ser Tyr Ser Leu Asn Asp Tyr Tyr Phe Asp Tyr
1 5 10 15
<210> 423
<211> 6
<212> PRT
<213> Unknown
<220>
<223> immunoreceptor tyrosine-based inhibitory motif
<220>
<221> SITE
<222> (1)..(1)
<223> Xaa is Ser, Ile, Val or Leu
<220>
<221> SITE
<222> (2)..(2)
<223> Xaa is any amino acid
<220>
<221> SITE
<222> (4)..(5)
<223> Xaa is any amino acid
<220>
<221> SITE
<222> (6)..(6)
<223> Xaa is Ile, Val or Leu
<400> 423
Xaa Xaa Tyr Xaa Xaa Xaa
1 5
<210> 424
<211> 64
<212> PRT
<213> Artificial Sequence
<220>
<223> Hinge (iTIM hinge)
<400> 424
Tyr Gly Ser Gln Ser Ser Lys Pro Tyr Leu Leu Thr His Pro Ser Asp
1 5 10 15
Pro Leu Glu Leu Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro
20 25 30
Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr
35 40 45
Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val
50 55 60
<210> 425
<211> 44
<212> PRT
<213> Artificial Sequence
<220>
<223> Short hinge 2
<400> 425
Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly Pro
1 5 10 15
Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly Ser
20 25 30
Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val
35 40
<210> 426
<211> 67
<212> PRT
<213> Artificial Sequence
<220>
<223> Long hinge 1
<400> 426
Ala Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Pro Val Pro Ser
1 5 10 15
Thr Pro Pro Thr Pro Ser Pro Ser Thr Pro Pro Thr Pro Ser Pro Ser
20 25 30
Gly Gly Ser Gly Asn Ser Ser Gly Ser Gly Gly Ser Pro Val Pro Ser
35 40 45
Thr Pro Pro Thr Pro Ser Pro Ser Thr Pro Pro Thr Pro Ser Pro Ser
50 55 60
Ala Ser Val
65
<210> 427
<211> 75
<212> PRT
<213> Artificial Sequence
<220>
<223> Long hinge 2
<400> 427
Ala Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Pro Val Pro Ser
1 5 10 15
Thr Pro Pro Thr Asn Ser Ser Ser Thr Pro Pro Thr Pro Ser Pro Ser
20 25 30
Pro Val Pro Ser Thr Pro Pro Thr Asn Ser Ser Ser Thr Pro Pro Thr
35 40 45
Pro Ser Pro Ser Pro Val Pro Ser Thr Pro Pro Thr Asn Ser Ser Ser
50 55 60
Thr Pro Pro Thr Pro Ser Pro Ser Ala Ser Val
65 70 75
<210> 428
<211> 85
<212> PRT
<213> Artificial Sequence
<220>
<223> 2x short hinge
<400> 428
Val Val Ser Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly Pro
1 5 10 15
Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly Ser
20 25 30
Asp Pro Gln Ser Gly Leu Gly Arg His Val Val Ser Gly Pro Ser Gly
35 40 45
Gly Pro Ser Ser Pro Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu
50 55 60
Asp Gln Pro Leu Thr Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly
65 70 75 80
Arg His Leu Gly Val
85
<210> 429
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> Hinge (truncated)
<400> 429
Thr Thr Gly Pro Thr Ser Thr Ser Gly Pro Glu Asp Gln Pro Leu Thr
1 5 10 15
Pro Thr Gly Ser Asp Pro Gln Ser Gly Leu Gly Arg His Leu Gly Val
20 25 30
<210> 430
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> T2A self-cleaving peptide
<400> 430
Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro
1 5 10 15
Gly Pro
<210> 431
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> P2A self-cleaving peptide
<400> 431
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1 5 10 15
Pro Gly Pro
<210> 432
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> E2A self-cleaving peptide
<400> 432
Gln Cys Thr Asn Tyr Ala Leu Leu Lys Leu Ala Gly Asp Val Glu Ser
1 5 10 15
Asn Pro Gly Pro
20
<210> 433
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> F2A self-cleaving peptide
<400> 433
Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val
1 5 10 15
Glu Ser Asn Pro Gly Pro
20
<210> 434
<211> 63
<212> DNA
<213> Unknown
<220>
<223> encodes T2A self-cleaving peptide
<400> 434
ggatccggag agggcagagg cagcctgctg acatgtggcg acgtggaaga gaaccctggc 60
ccc 63
<210> 435
<211> 83
<212> DNA
<213> Unknown
<220>
<223> encodes scaffold sequence
<400> 435
gttttagagc tagaaatagc aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt 60
ggcaccgagt cggtgctttt ttt 83
<210> 436
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 436
tggacgacac gcagttcgtg 20
<210> 437
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 437
cagatacctg gagaacggga 20
<210> 438
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 438
tcccgttctc caggtatctg 20
<210> 439
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 439
ccgccgcggt ccaagagcgc 20
<210> 440
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 440
cctgcgctct tggaccgcgg 20
<210> 441
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 441
ggacctgcgc tcttggaccg c 21
<210> 442
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 442
aaggagacgc tgcagcgcac ggg 23
<210> 443
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 443
gaaggagacg ctgcagcgca cgg 23
<210> 444
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 444
gcgggcgccg tggatagagc agg 23
<210> 445
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 445
tgctctatcc acggcgcccg cgg 23
<210> 446
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 446
cgatgaagcg gggctccccg cgg 23
<210> 447
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 447
cgtgtcccgg cccggccgcg ggg 23
<210> 448
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 448
cggctccatc ctctggctcg cgg 23
<210> 449
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 449
gatgtaatcc ttgccgtcgt agg 23
<210> 450
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 450
acagcgacgc cgcgagccag agg 23
<210> 451
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 451
ggatggagcc gcgggcgccg tgg 23
<210> 452
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 452
ggcgccgtgg atagagcagg agg 23
<210> 453
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 453
gcgccgtgga tagagcagga ggg 23
<210> 454
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 454
cggctactac aaccagagcg agg 23
<210> 455
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 455
ctggttgtag tagccgcgca ggg 23
<210> 456
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 456
tactacaacc agagcgaggc cgg 23
<210> 457
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 457
ctacctggag ggcacgtgcg tgg 23
<210> 458
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 458
cacgcacgtg ccctccaggt agg 23
<210> 459
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 459
gcagggtccc caggtccact cgg 23
<210> 460
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 460
gtggacctgg ggaccctgcg cgg 23
<210> 461
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 461
tggagggcac gtgcgtggag tgg 23
<210> 462
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 462
gtatggctgc gacgtggggt cgg 23
<210> 463
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 463
ctgagctgcc atgtccgccg cgg 23
<210> 464
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 464
ggattacatc gccctgaaag agg 23
<210> 465
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 465
caagtgggag gcggcccatg tgg 23
<210> 466
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 466
gtgggaggcg gcccatgtgg cgg 23
<210> 467
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 467
cagttgagag cctacctgga ggg 23
<210> 468
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 468
gcagttgaga gcctacctgg agg 23
<210> 469
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 469
taccaccagt acgcctacga cgg 23
<210> 470
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 470
tgccgtcgta ggcgtactgg tgg 23
<210> 471
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 471
ccagtacgcc tacgacggca agg 23
<210> 472
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 472
ggatgtgaag aaatacctca tgg 23
<210> 473
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 473
atttcttcac atccgtgtcc cgg 23
<210> 474
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 474
aggcgtactg gtggtacccg cgg 23
<210> 475
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 475
cgtactggtg gtacccgcgg agg 23
<210> 476
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 476
gaggatgtat ggctgcgacg tgg 23
<210> 477
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 477
ggatgtatgg ctgcgacgtg ggg 23
<210> 478
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 478
ctcagaccac caagcacaag tgg 23
<210> 479
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 479
tcagaccacc aagcacaagt ggg 23
<210> 480
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 480
caccaagcac aagtgggagg cgg 23
<210> 481
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 481
gaccaccaag cacaagtggg agg 23
<210> 482
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 482
gagccccgct tcatcgcagt ggg 23
<210> 483
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 483
gtagcccact gcgatgaagc ggg 23
<210> 484
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 484
tagcccactg cgatgaagcg ggg 23
<210> 485
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 485
cgtagcccac tgcgatgaag cgg 23
<210> 486
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 486
cttcatcgca gtgggctacg tgg 23
<210> 487
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 487
ggagccccgc ttcatcgcag tgg 23
<210> 488
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 488
cggggagaca cggaaagtga agg 23
<210> 489
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 489
agtattggga cggggagaca cgg 23
<210> 490
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 490
agggtccgga gtattgggac ggg 23
<210> 491
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 491
gagggtccgg agtattggga cgg 23
<210> 492
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 492
ggaccctcct gctctatcca cgg 23
<210> 493
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 493
gtggatagag caggagggtc cgg 23
<210> 494
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 494
agactcaccg agtggacctg ggg 23
<210> 495
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 495
cactcggtga gtctgtgagt ggg 23
<210> 496
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 496
cagactcacc gagtggacct ggg 23
<210> 497
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 497
ccactcacag actcaccgag tgg 23
<210> 498
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 498
ccactcggtg agtctgtgag tgg 23
<210> 499
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 499
tcggactggc gcttcctccg cgg 23
<210> 500
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 500
gcagccatac atcctctgga cgg 23
<210> 501
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 501
tctcaactgc tccgccacat ggg 23
<210> 502
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 502
accctcatgc tgcacatggc agg 23
<210> 503
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 503
acctgccatg tgcagcatga ggg 23
<210> 504
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 504
cacctgccat gtgcagcatg agg 23
<210> 505
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 505
ggaggaccag acccaggaca cgg 23
<210> 506
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 506
ggatggggag gaccagaccc agg 23
<210> 507
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 507
gacctggcag cgggatgggg agg 23
<210> 508
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 508
agatcacact gacctggcag cgg 23
<210> 509
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 509
gatcacactg acctggcagc ggg 23
<210> 510
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 510
aggtcagtgt gatctccgca ggg 23
<210> 511
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 511
aagcccctca ccctgagatg ggg 23
<210> 512
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 512
ctgcggagat cacactgacc tgg 23
<210> 513
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 513
cagcaatgat gcccacgatg ggg 23
<210> 514
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 514
ccagcaatga tgcccacgat ggg 23
<210> 515
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 515
gccagcaatg atgcccacga tgg 23
<210> 516
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 516
ggatggaacc ttccagaagt ggg 23
<210> 517
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 517
gggatggaac cttccagaag tgg 23
<210> 518
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 518
atgcccacga tggggatggt ggg 23
<210> 519
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 519
cagcccacca tccccatcgt ggg 23
<210> 520
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 520
ccagcccacc atccccatcg tgg 23
<210> 521
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 521
gatgcccacg atggggatgg tgg 23
<210> 522
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 522
cagggcccag cacctcaggg tgg 23
<210> 523
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 523
aatgatgccc acgatgggga tgg 23
<210> 524
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 524
ggccctgacc cagacctggg cgg 23
<210> 525
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 525
gacccaggac acggagctcg tgg 23
<210> 526
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 526
acacggagct cgtggagacc agg 23
<210> 527
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 527
cgtggagacc aggcctgcag ggg 23
<210> 528
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 528
tcgtggagac caggcctgca ggg 23
<210> 529
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 529
agctgtgatc actggagctg tgg 23
<210> 530
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 530
aaaaggaggg agctactctc agg 23
<210> 531
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 531
atgtggagga ggaagagctc agg 23
<210> 532
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 532
gtgtctctca cagcttgtaa agg 23
<210> 533
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 533
gagagacaca tcagagccct ggg 23
<210> 534
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 534
ctccgcaggg tagaagctca ggg 23
<210> 535
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 535
ggccctgagc ttctaccctg cgg 23
<210> 536
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 536
gctcagggcc cagcacctca ggg 23
<210> 537
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 537
tatctctgct cctgtccaga agg 23
<210> 538
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 538
agtagcagga cgagggttcg ggg 23
<210> 539
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 539
ccccgagagt agcaggacga ggg 23
<210> 540
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 540
ccctcgtcct gctactctcg ggg 23
<210> 541
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 541
cctcgtcctg ctactctcgg ggg 23
<210> 542
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 542
ctgtggtcgc tgctgtgatg tgg 23
<210> 543
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 543
tcgctgctgt gatgtggagg agg 23
<210> 544
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 544
tggtcgctgc tgtgatgtgg agg 23
<210> 545
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 545
cacagccgcc cacttctgga agg 23
<210> 546
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 546
ccagaagtgg gcggctgtgg tgg 23
<210> 547
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 547
tggaaccttc cagaagtggg cgg 23
<210> 548
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 548
tcacagctcc aaagagaacc agg 23
<210> 549
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 549
ctgaccatga agccaccctg agg 23
<210> 550
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 550
gcaaaccctc atgctgcaca tgg 23
<210> 551
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 551
tgaagccacc ctgaggtgct ggg 23
<210> 552
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 552
ggtgagtcat atgcgttttg ggg 23
<210> 553
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 553
gtgagtcata tgcgttttgg ggg 23
<210> 554
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 554
cttcatggtc agagacagcg tgg 23
<210> 555
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Guide Nucleic Acid Targeting Sequence
<400> 555
tctggccctg acccagacct ggg 23
<210> 556
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 556
cgcgagcaca gctaaggcca 20
<210> 557
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 557
gagtagcgcg agcacagcta 20
<210> 558
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 558
agggtaggag agactcacgc 20
<210> 559
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 559
ctgaatcttt ggagtacctg 20
<210> 560
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 560
tcacgtcatc cagcagagaa 20
<210> 561
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 561
tcctgaattg ctatgtgtct 20
<210> 562
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 562
aagtcaactt caatgtcgga 20
<210> 563
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 563
gtcttttccc gatattcctc 20
<210> 564
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 564
tggagtacct gaggaatatc 20
<210> 565
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 565
cagcccaaga tagttaagtg 20
<210> 566
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 566
acaaagtcac atggttcaca 20
<210> 567
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 567
actctctctt tctggcctgg 20
<210> 568
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 568
tgggctgtga caaagtcaca 20
<210> 569
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 569
ggccgagatg tctcgctccg 20
<210> 570
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 570
cagtaagtca acttcaatgt 20
<210> 571
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 571
actcacgctg gatagcctcc 20
<210> 572
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 572
catactcatc tttttcagtg 20
<210> 573
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 573
cacagcccaa gatagttaag 20
<210> 574
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 574
ttcagacttg tctttcagca 20
<210> 575
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 575
agtcacatgg ttcacacggc 20
<210> 576
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 576
atactcatct ttttcagtgg 20
<210> 577
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 577
ggcatactca tctttttcag 20
<210> 578
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 578
acagcccaag atagttaagt 20
<210> 579
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 579
gctactctct ctttctggcc 20
<210> 580
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 580
tggagagaga attgaaaaag 20
<210> 581
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 581
acttgtcttt cagcaaggac 20
<210> 582
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 582
gaagttgact tactgaagaa 20
<210> 583
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 583
ggccacggag cgagacatct 20
<210> 584
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 584
gcatactcat ctttttcagt 20
<210> 585
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 585
cgtgagtaaa cctgaatctt 20
<210> 586
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 586
ttaccccact taactatctt 20
<210> 587
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 587
ttggagtacc tgaggaatat 20
<210> 588
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 588
acccagacac atagcaattc 20
<210> 589
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 589
tttgactttc cattctctgc 20
<210> 590
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 590
ttcctgaatt gctatgtgtc 20
<210> 591
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 591
ctcaggtact ccaaagattc 20
<210> 592
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 592
cttaccccac ttaactatct 20
<210> 593
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 593
ctcgcgctac tctctctttc 20
<210> 594
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 594
tcgatctatg aaaaagacag 20
<210> 595
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 595
gagacatgta agcagcatca 20
<210> 596
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 596
acatgtaagc agcatcatgg 20
<210> 597
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 597
gaagtcctag aatgagcgcc 20
<210> 598
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 598
gagcgcccgg tgtcccaagc 20
<210> 599
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 599
agcgcccggt gtcccaagct 20
<210> 600
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 600
gcgcccggtg tcccaagctg 20
<210> 601
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 601
ctggggcgcg caccccagat 20
<210> 602
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 602
gggcgcgcac cccagatcgg 20
<210> 603
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 603
ggcgcgcacc ccagatcgga 20
<210> 604
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 604
catcacgaga ctctaagaaa 20
<210> 605
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 605
taagaaaagg aaactgaaaa 20
<210> 606
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 606
aagaaaagga aactgaaaac 20
<210> 607
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 607
gaaagtccct ctctctaacc 20
<210> 608
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 608
ctaacctggc actgcgtcgc 20
<210> 609
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 609
ctggcactgc gtcgctggct 20
<210> 610
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 610
tgcgtcgctg gcttggagac 20
<210> 611
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 611
gctggcttgg agacaggtga 20
<210> 612
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 612
gagacaggtg acggtccctg 20
<210> 613
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 613
agacaggtga cggtccctgc 20
<210> 614
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 614
cctgcgggcc ttgtcctgat 20
<210> 615
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 615
cgggccttgt cctgattggc 20
<210> 616
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 616
gggccttgtc ctgattggct 20
<210> 617
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 617
gggcacgcgt ttaatataag 20
<210> 618
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 618
cacgcgttta atataagtgg 20
<210> 619
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 619
tataagtgga ggcgtcgcgc 20
<210> 620
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 620
aagtggaggc gtcgcgctgg 20
<210> 621
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 621
agtggaggcg tcgcgctggc 20
<210> 622
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 622
ttcctgaagc tgacagcatt 20
<210> 623
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 623
tcctgaagct gacagcattc 20
<210> 624
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 624
gcccgaatgc tgtcagcttc 20
<210> 625
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 625
aaacgcgtgc ccagccaatc 20
<210> 626
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 626
gtgcccagcc aatcaggaca 20
<210> 627
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 627
ccaatcagga caaggcccgc 20
<210> 628
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 628
caatcaggac aaggcccgca 20
<210> 629
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 629
caagccagcg acgcagtgcc 20
<210> 630
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 630
cgcagtgcca ggttagagag 20
<210> 631
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 631
gcagtgccag gttagagaga 20
<210> 632
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 632
gagtctcgtg atgtttaaga 20
<210> 633
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 633
taagaaggca tgcactagac 20
<210> 634
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 634
aagaaggcat gcactagact 20
<210> 635
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 635
tgagtttgct gtctgtacat 20
<210> 636
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 636
tacatcggcg ccctccgatc 20
<210> 637
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 637
acatcggcgc cctccgatct 20
<210> 638
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 638
catcggcgcc ctccgatctg 20
<210> 639
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 639
ctggggtgcg cgccccagct 20
<210> 640
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 640
tggggtgcgc gccccagctt 20
<210> 641
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 641
cgcgccccag cttgggacac 20
<210> 642
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 642
gcgccccagc ttgggacacc 20
<210> 643
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 643
caagtcactt agcatctctg 20
<210> 644
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 644
acagaagttc tccttctgct 20
<210> 645
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 645
attcaaagat cttaatcttc 20
<210> 646
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 646
ttcaaagatc ttaatcttct 20
<210> 647
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 647
ttttctcgaa tgaaaaatgc 20
<210> 648
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 648
tgcaggtccg agcagttaac 20
<210> 649
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 649
ggtccgagca gttaactggc 20
<210> 650
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 650
gtccgagcag ttaactggct 20
<210> 651
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 651
tccgagcagt taactggctg 20
<210> 652
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 652
agcaagtcac ttagcatctc 20
<210> 653
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 653
gcaagtcact tagcatctct 20
<210> 654
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 654
tggggccagt ctgcaaagcg 20
<210> 655
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 655
ggggccagtc tgcaaagcga 20
<210> 656
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 656
gggccagtct gcaaagcgag 20
<210> 657
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 657
ggccagtctg caaagcgagg 20
<210> 658
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 658
ggacaccggg cgctcattct 20
<210> 659
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 659
ggcgctcatt ctaggacttc 20
<210> 660
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 660
ctcattctag gacttcaggc 20
<210> 661
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 661
attctaggac ttcaggctgg 20
<210> 662
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 662
ttcaggctgg aggcacatta 20
<210> 663
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 663
tgccccctcg ctttgcagac 20
<210> 664
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 664
gatgctaagt gacttgctaa 20
<210> 665
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 665
gccccagcca gttaactgct 20
<210> 666
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 666
gcatttttca ttcgagaaaa 20
<210> 667
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 667
tttgaatgct acctagcaga 20
<210> 668
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 668
ttctgtttat aactacagct 20
<210> 669
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M targeting sequence
<400> 669
tctgtttata actacagctt 20
<210> 670
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 670
agagaatgga aagtcaaatt t 21
<210> 671
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 671
atggacatga tcttctttat a 21
<210> 672
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 672
tggacatgat cttctttata a 21
<210> 673
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 673
ggacatgatc ttctttataa t 21
<210> 674
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 674
tgacaggatt attggaaatt t 21
<210> 675
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 675
ttgtggttaa tctggtttat t 21
<210> 676
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 676
tgtggttaat ctggtttatt t 21
<210> 677
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 677
gcagagaatg gaaagtcaaa t 21
<210> 678
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 678
cagagaatgg aaagtcaaat t 21
<210> 679
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 679
gagaatggaa agtcaaattt c 21
<210> 680
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 680
gtcacagccc aagatagtta a 21
<210> 681
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 681
tgcttataca cttacacttt a 21
<210> 682
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 682
gcttatacac ttacacttta t 21
<210> 683
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 683
cttatacact tacactttat g 21
<210> 684
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 684
acatggacat gatcttcttt a 21
<210> 685
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 685
catggacatg atcttcttta t 21
<210> 686
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 686
atcaacatct tggtcagatt t 21
<210> 687
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 687
cttgcactca aagcttgtta a 21
<210> 688
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 688
agttaagcgt gcataagtta a 21
<210> 689
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 689
gcataagtta acttccaatt t 21
<210> 690
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 690
tacatactct gcttagaatt t 21
<210> 691
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 691
acatactctg cttagaattt g 21
<210> 692
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 692
ttgacaggat tattggaaat t 21
<210> 693
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 693
gacaggatta ttggaaattt g 21
<210> 694
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 694
taaggcatgg ttgtggttaa t 21
<210> 695
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 695
gttgtggtta atctggttta t 21
<210> 696
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 696
gttccacaag ttaaataaat c 21
<210> 697
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 697
tccagcgtac tccaaagatt c 21
<210> 698
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 698
tactccaaag attcaggttt a 21
<210> 699
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 699
actccaaaga ttcaggttta c 21
<210> 700
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 700
cacgtcatcc agcagagaat g 21
<210> 701
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 701
ggtttcatcc atccgacatt g 21
<210> 702
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 702
ccgacattga agttgactta c 21
<210> 703
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 703
tgaagaatgg agagagaatt g 21
<210> 704
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 704
gagcattcag acttgtcttt c 21
<210> 705
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 705
ttcagcaagg actggtcttt c 21
<210> 706
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 706
gcaaggactg gtctttctat c 21
<210> 707
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 707
cgtgtgaacc atgtgacttt g 21
<210> 708
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 708
ctttgtcaca gcccaagata g 21
<210> 709
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 709
tcacagccca agatagttaa g 21
<210> 710
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 710
agtgggatcg agacatgtaa g 21
<210> 711
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 711
aggtttgaag atgccgcatt t 21
<210> 712
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 712
ggtttgaaga tgccgcattt g 21
<210> 713
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 713
ttgatatgct tatacactta c 21
<210> 714
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 714
tgagtgctgt ctccatgttt g 21
<210> 715
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 715
tgtctccatg tttgatgtat c 21
<210> 716
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 716
tcaacatctt ggtcagattt g 21
<210> 717
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 717
tcagatttga actcttcaat c 21
<210> 718
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 718
ttcaatctct tgcactcaaa g 21
<210> 719
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 719
ttgcactcaa agcttgttaa g 21
<210> 720
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 720
actcaaagct tgttaagata g 21
<210> 721
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 721
agatagttaa gcgtgcataa g 21
<210> 722
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 722
tgcataagtt aacttccaat t 21
<210> 723
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 723
gttaacttcc aatttacata c 21
<210> 724
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 724
attgacagga ttattggaaa t 21
<210> 725
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 725
gtaaggcatg gttgtggtta a 21
<210> 726
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 726
ggttgtggtt aatctggttt a 21
<210> 727
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 727
ttcctgaagc tgacagcatt c 21
<210> 728
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 728
gctatccagc gtactccaaa g 21
<210> 729
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 729
catccagcag agaatggaaa g 21
<210> 730
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 730
caaatttcct gaattgctat g 21
<210> 731
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 731
attgctatgt gtctgggttt c 21
<210> 732
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 732
gaagatgccg catttggatt g 21
<210> 733
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 733
caatttacat actctgctta g 21
<210> 734
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 734
tatccagcgt actccaaaga t 21
<210> 735
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 735
atccagcgta ctccaaagat t 21
<210> 736
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 736
ctccaaagat tcaggtttac t 21
<210> 737
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 737
tgctatgtgt ctgggtttca t 21
<210> 738
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 738
tttcatccat ccgacattga a 21
<210> 739
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 739
gaagttgact tactgaagaa t 21
<210> 740
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 740
gaagaatgga gagagaattg a 21
<210> 741
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 741
agaatggaga gagaattgaa a 21
<210> 742
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 742
cagcaaggac tggtctttct a 21
<210> 743
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 743
agcaaggact ggtctttcta t 21
<210> 744
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 744
actttgtcac agcccaagat a 21
<210> 745
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 745
ttgtcacagc ccaagatagt t 21
<210> 746
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 746
tgtcacagcc caagatagtt a 21
<210> 747
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 747
cacagcccaa gatagttaag t 21
<210> 748
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 748
gcagcatcat ggaggtttga a 21
<210> 749
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 749
ccgcatttgg attggatgaa t 21
<210> 750
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 750
ttgagtgctg tctccatgtt t 21
<210> 751
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 751
agtgctgtct ccatgtttga t 21
<210> 752
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 752
ctgtctccat gtttgatgta t 21
<210> 753
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 753
tctaggaggg ctggcaactt a 21
<210> 754
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 754
caacatcttg gtcagatttg a 21
<210> 755
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 755
gtcagatttg aactcttcaa t 21
<210> 756
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 756
tcttgcactc aaagcttgtt a 21
<210> 757
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 757
tgcactcaaa gcttgttaag a 21
<210> 758
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 758
gcactcaaag cttgttaaga t 21
<210> 759
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 759
cactcaaagc ttgttaagat a 21
<210> 760
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 760
tcaaagcttg ttaagatagt t 21
<210> 761
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 761
caaagcttgt taagatagtt a 21
<210> 762
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 762
gatagttaag cgtgcataag t 21
<210> 763
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 763
atagttaagc gtgcataagt t 21
<210> 764
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 764
tagttaagcg tgcataagtt a 21
<210> 765
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 765
ttaagcgtgc ataagttaac t 21
<210> 766
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 766
taagcgtgca taagttaact t 21
<210> 767
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 767
atttacatac tctgcttaga a 21
<210> 768
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 768
tttacatact ctgcttagaa t 21
<210> 769
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 769
acaggattat tggaaatttg t 21
<210> 770
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 770
caggattatt ggaaatttgt t 21
<210> 771
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 771
aggcatggtt gtggttaatc t 21
<210> 772
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 772
cagcagagaa tggaaagtca a 21
<210> 773
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 773
tccgacattg aagttgactt a 21
<210> 774
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 774
ctggtctttc tatctcttgt a 21
<210> 775
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 775
ccgtgtgaac catgtgactt t 21
<210> 776
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 776
cccaagatag ttaagtggga t 21
<210> 777
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 777
ggttgctcca caggtagctc t 21
<210> 778
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 778
gctccacagg tagctctagg a 21
<210> 779
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 779
gggagcagag aattctctta t 21
<210> 780
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 780
ggagcagaga attctcttat c 21
<210> 781
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 781
gagcagagaa ttctcttatc c 21
<210> 782
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 782
gagaattctc ttatccaaca t 21
<210> 783
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 783
gaattctctt atccaacatc a 21
<210> 784
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 784
aagtggagca ttcagacttg t 21
<210> 785
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 785
aaggactggt ctttctatct c 21
<210> 786
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 786
aagcttgtta agatagttaa g 21
<210> 787
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 787
aagcgtgcat aagttaactt c 21
<210> 788
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 788
aagatgccgc atttggattg g 21
<210> 789
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 789
aagaatggag agagaattga a 21
<210> 790
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 790
aacatcaaca tcttggtcag a 21
<210> 791
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 791
aaggcatggt tgtggttaat c 21
<210> 792
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 792
aagcagcatc atggaggttt g 21
<210> 793
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 793
aagatgagta tgcctgccgt g 21
<210> 794
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 794
aagttgactt actgaagaat g 21
<210> 795
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 795
aagatagtta agcgtgcata a 21
<210> 796
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 796
aacttccaat ttacatactc t 21
<210> 797
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 797
aacatcttgg tcagatttga a 21
<210> 798
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 798
aactcttcaa tctcttgcac t 21
<210> 799
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 799
aatttcctga attgctatgt g 21
<210> 800
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 800
aatggaaagt caaatttcct g 21
<210> 801
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 801
aaccatgtga ctttgtcaca g 21
<210> 802
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 802
aattgacagg attattggaa a 21
<210> 803
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 803
aattctctta tccaacatca a 21
<210> 804
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 804
aaagtggagc attcagactt g 21
<210> 805
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 805
aaagtcaaat ttcctgaatt g 21
<210> 806
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 806
gttgctccac aggtagctct a 21
<210> 807
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target B2M sequences complement
<400> 807
aatttacata ctctgcttag a 21
<210> 808
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> B2M shRNA
<400> 808
gttaacttcc aatttacata ccgaagtatg taaattggaa gttaac 46
<210> 809
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 809
cttcttcctt ccctattaaa a 21
<210> 810
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 810
tctcactcca tgaggtattt c 21
<210> 811
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 811
ctctcactcc atgaggtatt t 21
<210> 812
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 812
gaggaggaag agctcagata g 21
<210> 813
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 813
gctctcactc catgaggtat t 21
<210> 814
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 814
aggattacat cgccctgaaa g 21
<210> 815
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 815
acaccgtcca gaggatgtat g 21
<210> 816
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 816
agggtccttc ttcctggata c 21
<210> 817
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 817
cctacgacgg caaggattac a 21
<210> 818
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 818
tcactccatg aggtatttct t 21
<210> 819
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 819
ctacgacggc aaggattaca t 21
<210> 820
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 820
ctcactccat gaggtatttc t 21
<210> 821
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 821
ggaggaagag ctcagataga a 21
<210> 822
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 822
cacaccgtcc agaggatgta t 21
<210> 823
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 823
cacgctgtct ctgaccatga a 21
<210> 824
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 824
ctggacagga gcagagatac a 21
<210> 825
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 825
tggaggagga agagctcaga t 21
<210> 826
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 826
ggctctcact ccatgaggta t 21
<210> 827
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 827
catctctgtc tcaacttcat g 21
<210> 828
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 828
tacgacggca aggattacat c 21
<210> 829
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 829
ggattacatc gccctgaaag a 21
<210> 830
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 830
gattacatcg ccctgaaaga g 21
<210> 831
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 831
ctcagaccac caagcacaag t 21
<210> 832
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 832
tcacaccgtc cagaggatgt a 21
<210> 833
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 833
actccatgag gtatttcttc a 21
<210> 834
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 834
cactccatga ggtatttctt c 21
<210> 835
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 835
ccatgaggta tttcttcaca t 21
<210> 836
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 836
acttcttcct tccctattaa a 21
<210> 837
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 837
gtgtctctca cagcttgtaa a 21
<210> 838
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 838
ctgtgttcgt gtaggcataa t 21
<210> 839
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 839
tgtgttcgtg taggcataat g 21
<210> 840
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 840
taacttcttc cttccctatt a 21
<210> 841
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 841
tctggacagg agcagagata c 21
<210> 842
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 842
ttgctggcct ggttctcttt g 21
<210> 843
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 843
tgtctctcac agcttgtaaa g 21
<210> 844
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 844
acttgaagaa ccctgacttt g 21
<210> 845
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 845
gaagaaccct gactttgttt c 21
<210> 846
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 846
tctgtgttcg tgtaggcata a 21
<210> 847
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 847
catggtgcac tgagctgtaa c 21
<210> 848
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 848
gtaacttctt ccttccctat t 21
<210> 849
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 849
catgtgcagc atgagggttt g 21
<210> 850
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 850
ttgttcctgc ccttcccttt g 21
<210> 851
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 851
acccagttct cactcccatt g 21
<210> 852
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 852
gggtttccag agaagccaat c 21
<210> 853
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 853
ttctccctct cccaacctat g 21
<210> 854
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 854
gtctctcaca gcttgtaaag t 21
<210> 855
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 855
tgtgtctctc acagcttgta a 21
<210> 856
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 856
gaggaagagc tcagatagaa a 21
<210> 857
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 857
tgaagaaccc tgactttgtt t 21
<210> 858
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 858
ttgaagaacc ctgactttgt t 21
<210> 859
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 859
gtgttcgtgt aggcataatg t 21
<210> 860
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 860
tggtgcactg agctgtaact t 21
<210> 861
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 861
ctccctctcc caacctatgt a 21
<210> 862
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 862
aggaggaaga gctcagatag a 21
<210> 863
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 863
acctatgtag ggtccttctt c 21
<210> 864
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 864
gggtccttct tcctggatac t 21
<210> 865
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 865
ggtccttctt cctggatact c 21
<210> 866
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 866
gtccttcttc ctggatactc a 21
<210> 867
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 867
aagccaatca gtgtcgtcgc g 21
<210> 868
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 868
aagaggacct gcgctcttgg a 21
<210> 869
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 869
aagtgtgaga cagctgcctt g 21
<210> 870
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 870
aaggcacctg catgtgtctg t 21
<210> 871
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 871
aatcatcttt cctgttccag a 21
<210> 872
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 872
aaaggcacct gcatgtgtct g 21
<210> 873
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 873
aaagaggacc tgcgctcttg g 21
<210> 874
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 874
aaacgcatat gactcaccac g 21
<210> 875
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 875
ggaagagctc agatagaaa 19
<210> 876
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 876
gggagacacg gaaagtgaa 19
<210> 877
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 877
cacctgccat gtgcagcatg a 21
<210> 878
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 878
ggagatcaca ctgacctggc a 21
<210> 879
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 879
ggattacatc gccctgaaag 20
<210> 880
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 880
gcaggagggt ccggagtatt 20
<210> 881
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 881
ggacggggag acacggaaag 20
<210> 882
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 882
gaaagtgaag gcccactca 19
<210> 883
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 883
gatacctgga gaacgggaag 20
<210> 884
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 884
gctgtggtgg tgccttctgg 20
<210> 885
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 885
gctactacaa ccagagcgag 20
<210> 886
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 886
gtggctccgc agatacctg 19
<210> 887
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 887
gccaatcagt gtcgtcgcg 19
<210> 888
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 888
gaggacctgc gctcttgga 19
<210> 889
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 889
gtgtgagaca gctgccttg 19
<210> 890
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 890
ggcacctgca tgtgtctgt 19
<210> 891
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 891
tcatctttcc tgttccaga 19
<210> 892
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 892
aggcacctgc atgtgtctg 19
<210> 893
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 893
agaggacctg cgctcttgg 19
<210> 894
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> target HLA sequences complement
<400> 894
acgcatatga ctcaccacg 19
<210> 895
<211> 2
<212> RNA
<213> Artificial Sequence
<220>
<223> 5' Flank Sequence
<400> 895
gg 2
<210> 896
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> 5' Flank Sequence
<400> 896
acaccauguu gccagucucu agg 23
<210> 897
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> 5' Flank Sequence
<400> 897
ugauagcaau gucagcagug ccu 23
<210> 898
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> 5' Flank Sequence
<400> 898
uauugcuguu gacagugagc gac 23
<210> 899
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> 3' Flank Sequence
<400> 899
uggcgucugg cccaaccaca c 21
<210> 900
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> 3' Flank Sequence
<400> 900
guaagguuga ccauacucua c 21
<210> 901
<211> 4
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 901
cgaa 4
<210> 902
<211> 7
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 902
uucaaga 7
<210> 903
<211> 7
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 903
auauuca 7
<210> 904
<211> 9
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 904
ugugcuguc 9
<210> 905
<211> 6
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 905
cucgag 6
<210> 906
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 906
cuuccuguca ga 12
<210> 907
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 907
cuucccuuug ucaga 15
<210> 908
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 908
guguuauucu ug 12
<210> 909
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 909
gugucuuaau ug 12
<210> 910
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 910
guguuagucu ug 12
<210> 911
<211> 7
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 911
ucaagag 7
<210> 912
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 912
ggacauccag gg 12
<210> 913
<211> 15
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 913
gugaagccac agaug 15
<210> 914
<211> 10
<212> RNA
<213> Artificial Sequence
<220>
<223> Loop Region Sequence
<400> 914
gauucuaaaa 10
<210> 915
<211> 518
<212> PRT
<213> Artificial Sequence
<220>
<223> HLA-A*11 Blocker 4
<400> 915
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Leu Arg Gly Ala Arg Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Thr Phe Ser Ser Tyr Trp Met His Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Val Trp Val Ser Arg Ile Asn Ser Asp Gly Ser Ser Thr
65 70 75 80
Ser Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Cys Leu Gly Val Leu Leu Tyr Asn Trp Phe
115 120 125
Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Ile
145 150 155 160
Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg
165 170 175
Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala
195 200 205
Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
225 230 235 240
Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe
245 250 255
Gly Gly Gly Thr Lys Val Glu Ile Lys Tyr Gly Ser Gln Ser Ser Lys
260 265 270
Pro Tyr Leu Leu Thr His Pro Ser Asp Pro Leu Glu Leu Val Val Ser
275 280 285
Gly Pro Ser Gly Gly Pro Ser Ser Pro Thr Thr Gly Pro Thr Ser Thr
290 295 300
Ser Gly Pro Glu Asp Gln Pro Leu Thr Pro Thr Gly Ser Asp Pro Gln
305 310 315 320
Ser Gly Leu Gly Arg His Leu Gly Val Val Ile Gly Ile Leu Val Ala
325 330 335
Val Ile Leu Leu Leu Leu Leu Leu Leu Leu Leu Phe Leu Ile Leu Arg
340 345 350
His Arg Arg Gln Gly Lys His Trp Thr Ser Thr Gln Arg Lys Ala Asp
355 360 365
Phe Gln His Pro Ala Gly Ala Val Gly Pro Glu Pro Thr Asp Arg Gly
370 375 380
Leu Gln Trp Arg Ser Ser Pro Ala Ala Asp Ala Gln Glu Glu Asn Leu
385 390 395 400
Tyr Ala Ala Val Lys His Thr Gln Pro Glu Asp Gly Val Glu Met Asp
405 410 415
Thr Arg Ser Pro His Asp Glu Asp Pro Gln Ala Val Thr Tyr Ala Glu
420 425 430
Val Lys His Ser Arg Pro Arg Arg Glu Met Ala Ser Pro Pro Ser Pro
435 440 445
Leu Ser Gly Glu Phe Leu Asp Thr Lys Asp Arg Gln Ala Glu Glu Asp
450 455 460
Arg Gln Met Asp Thr Glu Ala Ala Ala Ser Glu Ala Pro Gln Asp Val
465 470 475 480
Thr Tyr Ala Gln Leu His Ser Leu Thr Leu Arg Arg Glu Ala Thr Glu
485 490 495
Pro Pro Pro Ser Gln Glu Gly Pro Ser Pro Ala Val Pro Ser Ile Tyr
500 505 510
Ala Thr Leu Ala Ile His
515
<210> 916
<211> 1554
<212> DNA
<213> Artificial Sequence
<220>
<223> HLA-A*11 Blocker 4
<400> 916
atggacatga gggtccccgc tcagctcctg gggctcctgc tactctggct ccgaggtgcc 60
agatgtgaag tgcagctggt ggaaagcggc ggaggcctgg tgcagcctgg cggcagcctg 120
agactgtctt gcgccgccag cggcttcacc ttcagcagct actggatgca ctgggtccgc 180
caggcccctg gcaagggact ggtctgggtg tctcgaatca acagcgacgg cagcagcacc 240
agctacgccg acagcgtgaa gggccggttc accatcagcc gggacaacgc caagaacacc 300
ctgtacctgc agatgaacag cctgcgggcc gaggacaccg ccgtgtatta ctgttgtttg 360
ggtgttttat tatacaactg gttcgacccc tggggccagg gaaccctggt caccgtgtcc 420
tcaggcggag gtggaagcgg agggggagga tctggcggcg gaggaagcgg aggcgacatc 480
cagatgaccc agtctccatc ctccctgtct gcatctgtag gagacagagt caccatcact 540
tgccgggcaa gtcagagcat tagcagctat ttaaattggt atcagcagaa accagggaaa 600
gcccctaagc tcctgatcta tgctgcatcc agtttgcaaa gtggggtccc atcaaggttc 660
agtggcagtg gatctgggac agatttcact ctcaccatca gcagtctgca acctgaagat 720
tttgcaactt actactgtca acagagttac agtacccctc tcactttcgg cggcggaaca 780
aaggtggaga tcaagtacgg ctcacagagc tccaaaccct acctgctgac tcaccccagt 840
gaccccctgg agctcgtggt ctcaggaccg tctgggggcc ccagctcccc gacaacaggc 900
cccacctcca catctggccc tgaggaccag cccctcaccc ccaccgggtc ggatccccag 960
agtggtctgg gaaggcacct gggggttgtg atcggcatct tggtggccgt catcctactg 1020
ctcctcctcc tcctcctcct cttcctcatc ctccgacatc gacgtcaggg caaacactgg 1080
acatcgaccc agagaaaggc tgatttccaa catcctgcag gggctgtggg gccagagccc 1140
acagacagag gcctgcagtg gaggtccagc ccagctgccg atgcccagga agaaaacctc 1200
tatgctgccg tgaagcacac acagcctgag gatggggtgg agatggacac tcggagccca 1260
cacgatgaag acccccaggc agtgacgtat gccgaggtga aacactccag acctaggaga 1320
gaaatggcct ctcctccttc cccactgtct ggggaattcc tggacacaaa ggacagacag 1380
gcggaagagg acaggcagat ggacactgag gctgctgcat ctgaagcccc ccaggatgtg 1440
acctacgccc agctgcacag cttgaccctc agacgggagg caactgagcc tcctccatcc 1500
caggaagggc cctctccagc tgtgcccagc atctacgcca ctctggccat ccac 1554
<210> 917
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<223> signal sequence
<400> 917
atggatatga gagtgcctgc ccagctgctc ggactgctcc ttctgtggtt gagaggagct 60
cggtgc 66
<210> 918
<211> 1210
<212> PRT
<213> Homo sapiens
<400> 918
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Arg Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
<210> 919
<211> 1210
<212> PRT
<213> Homo sapiens
<400> 919
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Arg Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
Claims (87)
- 하기를 포함하는 면역 세포로서:
a. 표피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및
b. 이형접합성의 소실로 인해 EGFR+ 암에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체,
여기서 제1 수용체는 EGFR에 반응하는 활성화제 수용체이고; 여기서 제2 수용체는 비-표적 항원에 반응하는 억제 수용체인 면역 세포. - 제1항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 MHC 단백질의 대립형질 변이체에 특이적으로 결합하는 면역 세포.
- 제1항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A, HLA-B, 또는 HLA-C 단백질의 대립형질 변이체에 특이적으로 결합하는 면역 세포.
- 제1항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*02에 특이적으로 결합하는 면역 세포.
- 제1항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 HLA-A*01, HLA-A*03, HLA-A*11, HLA-C*07, 또는 HLA-B*07에 특이적으로 결합하는 면역 세포.
- 제3항 내지 제5항 중 어느 한 항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 표 5에 개시된 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함하는 면역 세포.
- 제3항 내지 제5항 중 어느 한 항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 서열 번호 101-106 또는 서열 번호 106-112의 상보성 결정 영역 (CDR) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2, CDR-H3; 또는 표 5의 CDR에 비해 최대 1, 2 또는 3개의 치환, 결실 또는 삽입을 갖는 CDR 서열을 포함하는 면역 세포.
- 제3항 내지 제5항 중 어느 한 항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 표 4에 개시된 폴리펩티드 서열로부터 선택된 폴리펩티드 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
- 제4항에 있어서, 상기 제2 수용체의 세포외 리간드 결합 도메인은 서열 번호 89-100 중 어느 하나; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
- 제1항 내지 제9항 중 어느 한 항에 있어서, 상기 제1 수용체는 키메라 항원 수용체 (CAR)인 면역 세포.
- 제1항 내지 제10항 중 어느 한 항에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인이 표 3에 제시된 서열 그룹으로부터 선택된 중쇄 상보성 결정 영역 (HC-CDR) 세트를 포함하는 가변 중쇄 (VH) 부분; 및/또는 표 3에 제시된 서열 그룹의 경쇄 상보성 결정 영역 (LC-CDR) 세트를 포함하는 가변 경쇄 (VL) 부분; 또는 각각의 CDR에서 최대 1, 2, 3, 4개의 치환, 삽입, 또는 결실을 갖는 CDR 서열을 포함하는 면역 세포.
- 제1항 내지 제11항 중 어느 한 항에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 표 2에 제시된 VH 서열로부터 선택된 서열을 갖는 가변 중쇄 (VH) 부분; 및/또는 표 2에 제시된 서열을 포함하는 가변 경쇄 (VL) 부분; 또는 이에 대해 적어도 70%, 적어도 85%, 적어도 90%, 또는 적어도 95% 동일성을 갖는 서열을 포함하는 면역 세포.
- 제1항 내지 제12항 중 어느 한 항에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 표 1에 제시된 서열 그룹으로부터 선택된 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97%, 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
- 제1항 내지 제13항 중 어느 한 항에 있어서, 상기 제1 수용체의 세포외 리간드 결합 도메인은 서열 번호 9-18로 이루어진 그룹으로부터 선택된 scFv 서열; 또는 이에 대해 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97% 또는 적어도 99% 동일성을 갖는 서열을 포함하는 면역 세포.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 상기 제2 수용체가 LILRB1 세포 내 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
- 제15항에 있어서, 상기 LILRB1 세포 내 도메인은 서열 번호 129와 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한, 또는 동일한 서열을 포함하는 면역 세포.
- 제1항 내지 제16항 중 어느 한 항에 있어서, 상기 제2 수용체가 LILRB1 막관통 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
- 제17항에 있어서, 상기 LILRB1 막관통 도메인 또는 이의 기능적 변이체는 서열 번호 133과 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함하는 면역 세포.
- 제1항 내지 제18항 중 어느 한 항에 있어서, 상기 제2 수용체가 LILRB1 힌지 도메인 또는 이의 기능적 변이체를 포함하는 면역 세포.
- 제19항에 있어서, 상기 LILRB1 힌지 도메인은 서열 번호 132, 서열 번호 125, 서열 번호 126과 적어도 90%, 적어도 95%, 적어도 97%, 적어도 99% 동일한 또는 동일한 서열을 포함하는 면역 세포.
- 제1항 내지 제20항 중 어느 한 항에 있어서, 상기 제2 수용체는 LILRB1 세포 내 도메인, LILRB1 막관통 도메인, LILRB1 힌지 도메인, 이들 중 임의의 것의 기능적 변이체, 또는 이들의 조합을 포함하는 면역 세포.
- 제21항에 있어서, 상기 LILRB1 세포 내 도메인 및 LILRB1 막관통 도메인은 서열 번호 128 또는 서열 번호 128과 적어도 95% 동일한 서열을 포함하는 면역 세포.
- 제1항 내지 제22항 중 어느 한 항에 있어서, 상기 EGFR+ 암 세포는 폐암 세포, 소세포 폐암 세포, 비소세포 폐암 세포, 췌관 암종 세포, 대장암세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포 또는 자궁암 세포인 면역 세포.
- 제23항에 있어서, 상기 EGFR+ 암 세포는 폐암 세포인 면역 세포.
- 제1항 내지 제24항 중 어느 한 항에 있어서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포인 면역 세포.
- 제25항에 있어서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A*02의 소실을 야기하는 HLA-A에서의 이형접합성의 소실로 인해 EGFR+/HLA-A*02+ 세포로부터 유래되는 면역 세포.
- 제1항 내지 제26항 중 어느 한 항에 있어서, 상기 제1 수용체 및 제2 수용체가 함께 이형접합성을 소실한 EGFR/HLA-A*02- 암 세포의 존재 하에서 면역세포를 특이적으로 활성화시키는 것인 면역세포.
- 제1항 내지 제27항 중 어느 한 항에 있어서, 상기 제1 수용체 및 제2 수용체가 함께 이형접합성 소실에 의해 HLA-A*02를 소실하지 않은 EGFR+ 세포의 존재 하에 면역 세포를 특이적으로 활성화시키지 않는 것인 면역 세포.
- 제1항 내지 제28항 중 어느 한 항에 있어서, 상기 면역 세포는 T 세포, 대식세포, NK 세포, iNKT 세포, 또는 감마 델타 T 세포인 면역 세포.
- 제29항에 있어서, 상기 T 세포는 CD8+ CD4- T 세포인 면역 세포.
- 제1항 내지 제30항 중 어느 한 항에 있어서, 상기 MHC 클래스 I 유전자의 발현 및/또는 기능이 감소되거나 제거된 것인 면역 세포.
- 제30항에 있어서, 상기 MHC 클래스 I 유전자가 베타-2-마이크로글로불린 (B2M)인 면역 세포.
- 제32항에 있어서, 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함하고, 간섭 RNA는 B2M mRNA의 서열 (서열 번호 172)에 상보적인 서열을 포함하는 면역 세포.
- 제33항에 있어서, 상기 간섭 RNA는 B2M mRNA의 RNAi-매개 분해를 유도할 수 있는 면역 세포.
- 제33항에 있어서, 상기 간섭 RNA는 짧은 헤어핀 RNA (shRNA)인 면역 세포.
- 제35항에 있어서, 상기 shRNA는 다음을 포함하고:
a. 5' 말단에서 3' 말단으로 B2M mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및
b. 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열,
여기서 제1 서열 및 제2 서열은 shRNA를 형성하는 면역 세포. - 제32항에 있어서, B2M을 암호화하는 서열 (서열 번호 170)에 대해 하나 이상의 변형을 포함하고, 상기 하나 이상의 변형은 B2M의 발현을 감소시키고/시키거나 기능을 제거하는 것인 면역 세포.
- 제37항에 있어서, 상기 하나 이상의 변형이 B2M을 암호화하는 내인성 유전자의 하나 이상의 불활성화 돌연변이를 포함하는 면역 세포.
- 제38항에 있어서, 상기 하나 이상의 불활성화 돌연변이가 결실, 삽입, 치환, 또는 프레임이동 돌연변이를 포함하는 면역 세포.
- 제38항 또는 제39항에 있어서, 상기 하나 이상의 불활성화 돌연변이가 B2M을 암호화하는 내인성 유전자의 서열 (서열 번호 170)을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)과의 복합체 내 핵산 유도 엔도뉴클레아제와 함께 도입되는 것인 면역 세포.
- 제31항에 있어서, 상기 MHC 클래스 I 유전자가 HLA-A*02인 면역 세포.
- 제41항에 있어서, HLA-A*02 mRNA의 서열 (서열 번호 171)에 상보적인 서열을 포함하는 간섭 RNA를 포함하는 폴리뉴클레오티드를 추가로 포함하는 면역 세포.
- 제42항에 있어서, 상기 간섭 RNA가 HLA-A*02 mRNA의 RNA 간섭 (RNAi)-매개 분해를 유도할 수 있는 면역 세포.
- 제43항에 있어서, 상기 간섭 RNA는 다음을 포함하는 짧은 헤어핀 RNA (shRNA)이고:
a. 5' 말단에서 3' 말단으로 HLA-A*02 mRNA의 서열에 상보적인 서열을 갖는 제1 서열; 및
b. 5' 말단에서 3' 말단으로 제1 서열에 상보적인 서열을 갖는 제2 서열,
여기서 제1 서열 및 제2 서열은 shRNA를 형성하는 면역 세포. - 제41항에 있어서, HLA-A*02를 암호화하는 내인성 유전자의 서열 (서열 번호 169)에 대해 하나 이상의 변형을 포함하고, 상기 하나 이상의 변형은 HLA-A*02의 발현을 감소시키고/시키거나 기능을 제거하는 것인 면역 세포.
- 제45항에 있어서, 상기 하나 이상의 변형이 HLA-A*02를 암호화하는 내인성 유전자의 하나 이상의 불활성화 돌연변이를 포함하는 면역 세포.
- 제45항 또는 제46항에 있어서, 상기 하나 이상의 불활성화 돌연변이가 HLA-A*02를 암호화하는 내인성 유전자의 서열을 특이적으로 표적화하는 적어도 하나의 가이드 핵산 (gNA)과의 복합체 내 핵산 유도 엔도뉴클레아제와 함께 도입되는 면역 세포.
- 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 174 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 177, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 174, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 175, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제47항 중 어느 한 항에 있어서:
a. 제1 수용체가 서열 번호 176, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하고; 그리고
b. 제2 수용체가 서열 번호 173, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포. - 제1항 내지 제53항 중 어느 한 항에 있어서, T2A 자가 절단 펩티드를 추가로 포함하고, 상기 T2A 자가 절단 펩티드는 서열 번호 178, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
- 제1항 내지 제54항 중 어느 한 항에 있어서, 간섭 RNA를 추가로 포함하고, 상기 간섭 RNA는 서열 번호 179, 또는 이에 대해 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일성을 공유하는 서열을 포함하는 면역 세포.
- 제1항 내지 제55항 중 어느 한 항에 있어서, 상기 면역 세포가 자가 세포인 면역 세포.
- 제1항 내지 제55항 중 어느 한 항에 있어서, 상기 면역 세포가 동종이계인 면역 세포.
- 치료적 유효량의 제1항 내지 제57항 중 어느 한 항의 면역 세포를 포함하는 약학 조성물.
- 제58항에 있어서, 상기 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현하는 약학 조성물.
- 제59항에 있어서, 적어도 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 또는 약 95%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현하는 약학 조성물.
- 제59항에 있어서, 적어도 90%의 면역 세포가 제1 수용체 및 제2 수용체 모두를 발현하는 약학 조성물.
- 제58항 내지 제61항 중 어느 한 항에 있어서, 약학적으로 허용 가능한 담체, 희석제 또는 부형제를 추가로 포함하는 약학 조성물.
- 제58항 내지 제62항 중 어느 한 항에 있어서, EGFR+ 암 치료에 약제로서 사용하기 위한 약학 조성물.
- 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템으로서, 다음을 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하고:
a. 내피 성장 인자 수용체 (EGFR)에 특이적인 세포외 리간드 결합 도메인을 포함하는 제1 수용체; 및
b. 이형접합성의 소실로 인해 EGFR+ 암 세포에서 소실된 비-표적 항원에 특이적인 세포외 리간드 결합 도메인을 포함하는 제2 수용체,
여기서 제1 수용체는 EGFR+ 암 세포 상의 EGFR에 반응하는 활성화제 수용체이고; 제2 수용체는 비-표적 항원에 반응하는 억제 수용체인 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템. - 제1항 내지 제57항 중 어느 한 항의 면역 세포를 생성하는 데 사용하기 위한 제1 수용체 및 제2 수용체를 암호화하는 폴리뉴클레오티드 서열을 포함하는 하나 이상의 폴리뉴클레오티드를 포함하는 폴리뉴클레오티드 또는 폴리뉴클레오티드 시스템.
- 제64항 또는 제65항의 하나 이상의 폴리뉴클레오티드를 포함하는 벡터.
- MHC 클래스 I 유전자좌에서 이형접합성이 소실된 EGFR+ 암 세포를 사멸시키는 방법으로서, 대상체에게 유효량의 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물을 투여하는 것을 포함하는 방법.
- 제67항에 있어서, 상기 EGFR+ 암 세포는 폐암 세포, 소세포 폐암 세포, 비소세포 폐암 세포, 췌관 암종 세포, 대장암세포, 두경부암 세포, 식도 및 위 선암종 세포, 난소암 세포, 다형성 교모세포종 세포, 자궁경부 편평 세포 암종 세포, 신장암 세포, 유두상 신장암 세포, 신장 투명 세포 암종 세포, 방광암 세포, 유방암 세포, 담관암 세포, 간암 세포, 전립선암 세포, 육종 세포, 갑상선암 세포, 흉선암 세포, 위암 세포 또는 자궁암 세포인 방법.
- 제67항에 있어서, 상기 EGFR+ 암 세포는 폐암 세포인 방법.
- 제67항에 있어서, 상기 EGFR+ 암 세포는 HLA-A*02를 발현하지 않는 EGFR+/HLA-A*02- 암 세포; 또는 HLA-A*02를 발현하지 않는 HLA-A*02+ 개체로부터 유래된 암 세포인 방법.
- 제70항에 있어서, 상기 EGFR+/HLA-A*02- 암 세포는 HLA-A*02의 소실을 야기하는 HLA-A에서의 이형접합성의 소실로 인해 EGFR+/HLA-A*02+ 세포로부터 유래되는 것인 방법.
- 비-표적 항원을 암호화하는 유전자좌에서 이형접합성의 소실을 갖는 EGFR+ 종양을 갖는 대상체에서 EGFR+ 암을 치료하는 방법으로서, 대상체에 유효량의 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물을 투여하는 것을 포함하는 방법.
- 제72항에 있어서, 상기 대상체가 EGFR을 발현하고 HLA-A*02 발현을 상실한 악성종양을 갖는 이형접합 HLA-A*02 환자인 방법.
- 제72항에 있어서, 상기 대상체가 EGFR을 발현하고 HLA-A*02 발현을 상실한 재발성 절제 불가능 또는 전이성 고형 종양을 갖는 이형접합 HLA-A*02 환자인 방법.
- 다음을 포함하는, 대상체에서 암을 치료하는 방법:
a. 대상체의 비-악성 세포 및 암세포에서 비-표적 항원의 유전자형 또는 발현 수준을 결정하는 단계;
b. 대상체의 암 세포에서 EGFR의 발현 수준을 결정하는 단계; 및
c. 비-악성 세포가 비-표적 항원을 발현하고 암세포가 비-표적 항원을 발현하지 않고, 암 세포가 EGFR 양성인 경우 유효량의 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물을 대상체에게 투여하는 단계. - 제67항 내지 제75항 중 어느 한 항에 있어서, 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물의 투여가 대상체에서 종양의 크기를 감소시키는 것인 방법.
- 제76항에 있어서, 상기 종양이 약 5%, 약 10%, 약 15%, 약 20%, 약 25%, 약 30%, 약 35%, 약 40%, 약 45%, 약 50%, 약 55%, 약 60%, 약 65%, 약 70%, 약 75%, 약 80%, 약 85%, 약 90%, 약 95%, 또는 약 100% 감소되는 방법.
- 제76항에 있어서, 상기 종양이 제거되는 방법.
- 제67항 내지 제75항 중 어느 한 항에 있어서, 상기 면역 세포 또는 약학 조성물의 투여가 대상체에서 종양의 성장을 정지시키는 방법.
- 제67항 내지 제75항 중 어느 한 항에 있어서, 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물의 투여가 대상체에서 종양의 수를 감소시키는 것인 방법.
- 제67항 내지 제80항 중 어느 한 항에 있어서, 면역 세포 또는 약학 조성물의 투여가 대상체에서 정상 세포가 아닌 암 세포의 선택적인 사멸을 초래하는 방법.
- 제81항에 있어서, 상기 사멸된 세포의 적어도 약 60%가 암 세포이고, 사멸된 세포의 적어도 약 65%가 암 세포이고, 사멸된 세포의 적어도 약 70%가 암 세포이고, 사멸된 세포의 적어도 약 75%가 암 세포이고, 사멸된 세포의 적어도 약 80%가 암 세포이고, 사멸된 세포의 적어도 약 85%가 암 세포이고, 사멸된 세포의 적어도 약 90%가 암 세포이고, 사멸된 세포의 적어도 약 95%가 암 세포이고, 또는 사멸된 세포의 약 100%가 암 세포인 방법.
- 제81항에 있어서, 면역 세포 또는 약학 조성물의 투여가 대상체의 암 세포의 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90% 또는 전부의 사멸을 초래하는 방법.
- 제67항 내지 제83항 중 어느 한 항에 있어서, 상기 면역 세포 또는 약학 조성물의 투여가 제1 활성화제 수용체를 포함하지만 제2 억제 수용체를 포함하지 않는 다른 동등한 면역 세포의 투여보다 대상체에 대한 부작용이 더 적은 방법.
- 다수의 면역 세포를 제조하는 방법으로서, 다음을 포함하는 방법:
a. 다수의 면역 세포를 제공하는 단계, 및
b. 다수의 면역 세포를 제64항 또는 제65항의 폴리뉴클레오티드 시스템, 또는 제66항의 벡터로 형질전환시키는 단계. - 제1항 내지 제57항 중 어느 한 항의 면역 세포 또는 제58항 내지 제62항 중 어느 한 항의 약학 조성물을 포함하는 키트.
- 제86항에 있어서, 사용 설명서를 추가로 포함하는 키트.
Applications Claiming Priority (7)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US202063068249P | 2020-08-20 | 2020-08-20 | |
| US63/068,249 | 2020-08-20 | ||
| US202063105639P | 2020-10-26 | 2020-10-26 | |
| US63/105,639 | 2020-10-26 | ||
| US202163230632P | 2021-08-06 | 2021-08-06 | |
| US63/230,632 | 2021-08-06 | ||
| PCT/US2021/046733 WO2022040444A1 (en) | 2020-08-20 | 2021-08-19 | Compositions and methods for treating egfr positive cancers |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20230052289A true KR20230052289A (ko) | 2023-04-19 |
Family
ID=77711489
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020237009181A KR20230052289A (ko) | 2020-08-20 | 2021-08-19 | Egfr 양성 암을 치료하기 위한 조성물 및 방법 |
Country Status (19)
| Country | Link |
|---|---|
| US (2) | US11602544B2 (ko) |
| EP (2) | EP4058474B1 (ko) |
| JP (1) | JP2023538116A (ko) |
| KR (1) | KR20230052289A (ko) |
| AU (1) | AU2021328478A1 (ko) |
| BR (1) | BR112023002668A2 (ko) |
| CA (1) | CA3189510A1 (ko) |
| DK (1) | DK4058474T3 (ko) |
| FI (1) | FI4058474T3 (ko) |
| HR (1) | HRP20240903T1 (ko) |
| IL (1) | IL300629A (ko) |
| LT (1) | LT4058474T (ko) |
| MX (1) | MX2023002104A (ko) |
| PL (1) | PL4058474T3 (ko) |
| PT (1) | PT4058474T (ko) |
| RS (1) | RS65802B1 (ko) |
| SI (1) | SI4058474T1 (ko) |
| TW (1) | TW202214846A (ko) |
| WO (1) | WO2022040444A1 (ko) |
Families Citing this family (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20230052289A (ko) | 2020-08-20 | 2023-04-19 | 에이투 바이오쎄라퓨틱스, 인크. | Egfr 양성 암을 치료하기 위한 조성물 및 방법 |
Family Cites Families (68)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4690915A (en) | 1985-08-08 | 1987-09-01 | The United States Of America As Represented By The Department Of Health And Human Services | Adoptive immunotherapy as a treatment modality in humans |
| US6905680B2 (en) | 1988-11-23 | 2005-06-14 | Genetics Institute, Inc. | Methods of treating HIV infected subjects |
| US6534055B1 (en) | 1988-11-23 | 2003-03-18 | Genetics Institute, Inc. | Methods for selectively stimulating proliferation of T cells |
| US5858358A (en) | 1992-04-07 | 1999-01-12 | The United States Of America As Represented By The Secretary Of The Navy | Methods for selectively stimulating proliferation of T cells |
| US6352694B1 (en) | 1994-06-03 | 2002-03-05 | Genetics Institute, Inc. | Methods for inducing a population of T cells to proliferate using agents which recognize TCR/CD3 and ligands which stimulate an accessory molecule on the surface of the T cells |
| US5585362A (en) | 1989-08-22 | 1996-12-17 | The Regents Of The University Of Michigan | Adenovirus vectors for gene therapy |
| US5350674A (en) | 1992-09-04 | 1994-09-27 | Becton, Dickinson And Company | Intrinsic factor - horse peroxidase conjugates and a method for increasing the stability thereof |
| US7175843B2 (en) | 1994-06-03 | 2007-02-13 | Genetics Institute, Llc | Methods for selectively stimulating proliferation of T cells |
| US6692964B1 (en) | 1995-05-04 | 2004-02-17 | The United States Of America As Represented By The Secretary Of The Navy | Methods for transfecting T cells |
| US7067318B2 (en) | 1995-06-07 | 2006-06-27 | The Regents Of The University Of Michigan | Methods for transfecting T cells |
| AU1086501A (en) | 1999-10-15 | 2001-04-30 | Carnegie Institution Of Washington | Rna interference pathway genes as tools for targeted genetic interference |
| US6326193B1 (en) | 1999-11-05 | 2001-12-04 | Cambria Biosciences, Llc | Insect control agent |
| US6867041B2 (en) | 2000-02-24 | 2005-03-15 | Xcyte Therapies, Inc. | Simultaneous stimulation and concentration of cells |
| US6797514B2 (en) | 2000-02-24 | 2004-09-28 | Xcyte Therapies, Inc. | Simultaneous stimulation and concentration of cells |
| IL151287A0 (en) | 2000-02-24 | 2003-04-10 | Xcyte Therapies Inc | A method for stimulation and concentrating cells |
| US7572631B2 (en) | 2000-02-24 | 2009-08-11 | Invitrogen Corporation | Activation and expansion of T cells |
| WO2001096584A2 (en) | 2000-06-12 | 2001-12-20 | Akkadix Corporation | Materials and methods for the control of nematodes |
| US7745140B2 (en) | 2002-01-03 | 2010-06-29 | The Trustees Of The University Of Pennsylvania | Activation and expansion of T-cells using an engineered multivalent signaling platform as a research tool |
| US20030170238A1 (en) | 2002-03-07 | 2003-09-11 | Gruenberg Micheal L. | Re-activated T-cells for adoptive immunotherapy |
| US20050277610A1 (en) | 2004-03-15 | 2005-12-15 | City Of Hope | Methods and compositions for the specific inhibition of gene expression by double-stranded RNA |
| ES2653570T3 (es) | 2004-05-27 | 2018-02-07 | The Trustees Of The University Of Pennsylvania | Células presentadoras de antígeno artificiales novedosas y usos de las mismas |
| US10040846B2 (en) | 2012-02-22 | 2018-08-07 | The Trustees Of The University Of Pennsylvania | Compositions and methods for generating a persisting population of T cells useful for the treatment of cancer |
| ES2636902T3 (es) | 2012-05-25 | 2017-10-10 | The Regents Of The University Of California | Métodos y composiciones para la modificación de ADN objetivo dirigida por ARN y para la modulación de la transcripción dirigida por ARN |
| CN113355357A (zh) | 2012-12-12 | 2021-09-07 | 布罗德研究所有限公司 | 对用于序列操纵的改进的系统、方法和酶组合物进行的工程化和优化 |
| US8697359B1 (en) | 2012-12-12 | 2014-04-15 | The Broad Institute, Inc. | CRISPR-Cas systems and methods for altering expression of gene products |
| WO2014093595A1 (en) | 2012-12-12 | 2014-06-19 | The Broad Institute, Inc. | Crispr-cas component systems, methods and compositions for sequence manipulation |
| SG11201507688VA (en) | 2013-03-15 | 2015-10-29 | Sloan Kettering Inst Cancer | Compositions and methods for immunotherapy |
| WO2014145252A2 (en) | 2013-03-15 | 2014-09-18 | Milone Michael C | Targeting cytotoxic cells with chimeric receptors for adoptive immunotherapy |
| ES2910004T3 (es) | 2013-07-29 | 2022-05-11 | 2Seventy Bio Inc | Proteínas de señalización de múltiples partes y sus usos |
| PL3071223T3 (pl) | 2013-11-21 | 2021-08-16 | Autolus Limited | Komórka |
| EP3102609B1 (en) | 2014-02-04 | 2024-08-28 | The United States of America, as Represented by The Secretary, Department of Health and Human Services | Methods for producing autologous t cells useful to treat b cell malignancies and other cancers and compositions thereof |
| DK3116902T3 (da) * | 2014-03-11 | 2020-04-06 | Cellectis | Fremgangsmåde til generering af T-celler kompatible for allogen transplantation |
| JP7023708B2 (ja) | 2014-11-12 | 2022-03-03 | アロジーン セラピューティクス,インコーポレイテッド | 阻害性キメラ抗原受容体 |
| US20170296623A1 (en) | 2014-12-17 | 2017-10-19 | Cellectis | INHIBITORY CHIMERIC ANTIGEN RECEPTOR (iCAR OR N-CAR) EXPRESSING NON-T CELL TRANSDUCTION DOMAIN |
| WO2016126608A1 (en) | 2015-02-02 | 2016-08-11 | Novartis Ag | Car-expressing cells against multiple tumor antigens and uses thereof |
| EP3262166A4 (en) | 2015-02-24 | 2018-08-15 | The Regents of The University of California | Binding-triggered transcriptional switches and methods of use thereof |
| MX2017011418A (es) | 2015-03-11 | 2017-12-14 | Cellectis | Metodos para manipular geneticamente celula t alogenica para aumentar su persistencia y/o injerto en pacientes. |
| CN107533051B (zh) | 2015-03-27 | 2020-11-13 | 南加利福尼亚大学 | Hla-g作为car t细胞免疫疗法的新靶标 |
| GB201509413D0 (en) | 2015-06-01 | 2015-07-15 | Ucl Business Plc | Fusion protein |
| MA42895A (fr) | 2015-07-15 | 2018-05-23 | Juno Therapeutics Inc | Cellules modifiées pour thérapie cellulaire adoptive |
| MA44314A (fr) | 2015-11-05 | 2018-09-12 | Juno Therapeutics Inc | Récepteurs chimériques contenant des domaines induisant traf, et compositions et méthodes associées |
| WO2017087723A1 (en) | 2015-11-19 | 2017-05-26 | The Regents Of The University Of California | Conditionally repressible immune cell receptors and methods of use thereof |
| JP2018538277A (ja) | 2015-11-23 | 2018-12-27 | トラスティーズ オブ ボストン ユニバーシティ | キメラ抗原受容体に関する方法および組成物 |
| CA3005482A1 (en) | 2015-12-02 | 2017-06-08 | Innovative Targeting Solutions Inc. | Single variable domain t-cell receptors |
| BR112018068354A2 (pt) | 2016-03-11 | 2019-01-15 | Bluebird Bio Inc | células efetoras imunológicas do genoma editado |
| CA3029121A1 (en) | 2016-06-29 | 2018-01-04 | Crispr Therapeutics Ag | Compositions and methods for gene editing |
| EP3263595A1 (en) | 2016-06-30 | 2018-01-03 | Medizinische Hochschule Hannover | Fusion protein for use in the treatment of hvg disease |
| CN109996871A (zh) | 2016-09-28 | 2019-07-09 | 加维什-加利里生物应用有限公司 | 供靶向癌症的新型抗原标志的car疗法用的通用平台 |
| WO2018144535A1 (en) | 2017-01-31 | 2018-08-09 | Novartis Ag | Treatment of cancer using chimeric t cell receptor proteins having multiple specificities |
| EP3579877A4 (en) | 2017-02-09 | 2020-12-09 | The Regents of The University of California | CHEMERIC T-LYMPHOCYTE ANTIGENIC RECEPTORS AND METHODS OF USE |
| US20200188434A1 (en) | 2017-05-15 | 2020-06-18 | Autolus Limited | A cell comprising a chimeric antigen receptor (car) |
| GB201707783D0 (en) | 2017-05-15 | 2017-06-28 | Autolus Ltd | Cell |
| GB201707779D0 (en) | 2017-05-15 | 2017-06-28 | Autolus Ltd | Cell |
| EP3684821A4 (en) | 2017-09-19 | 2021-06-16 | The University Of British Columbia | ANTI-HLA-A2 ANTIBODIES AND METHOD OF USING THEREOF |
| WO2019068007A1 (en) * | 2017-09-28 | 2019-04-04 | Immpact-Bio Ltd. | UNIVERSAL PLATFORM FOR PREPARING AN INHIBITORY CHIMERIC ANTIGEN RECEPTOR (ICAR) |
| WO2019090215A2 (en) | 2017-11-06 | 2019-05-09 | Monticello Daniel J | Dominant negative ligand chimeric antigen receptor systems |
| WO2019241549A1 (en) | 2018-06-15 | 2019-12-19 | A2 Biotherapeutics, Inc. | Foxp3-expressing car-t regulatory cells |
| CA3114736A1 (en) | 2018-09-28 | 2020-04-02 | Immpact-Bio Ltd. | Methods for identifying activating antigen receptor (acar)/inhibitory chimeric antigen receptor (icar) pairs for use in cancer therapies |
| KR20210075117A (ko) | 2018-10-05 | 2021-06-22 | 세인트 안나 킨더크렙스포르슝 | 키메라 항원 수용체(car) 그룹 |
| EP3632461A1 (en) | 2018-10-05 | 2020-04-08 | St. Anna Kinderkrebsforschung | A group of chimeric antigen receptors (cars) |
| WO2021030182A1 (en) | 2019-08-09 | 2021-02-18 | A2 Biotherapeutics, Inc. | Bifunctional single variable domain t cell receptors and uses thereof |
| WO2021030153A2 (en) | 2019-08-09 | 2021-02-18 | A2 Biotherapeutics, Inc. | Engineered t cell receptors and uses thereof |
| EP4010377A4 (en) * | 2019-08-09 | 2023-09-06 | A2 Biotherapeutics, Inc. | CELL SURFACE RECEPTORS THAT RESPOND TO LOSS OF HETEROZYGOSIS |
| CN114585371A (zh) | 2019-08-20 | 2022-06-03 | 森迪生物科学公司 | 嵌合抑制性受体 |
| US20220389075A1 (en) | 2019-11-12 | 2022-12-08 | A2 Biotherapeutics, Inc. | Engineered t cell receptors and uses thereof |
| AU2020401315B2 (en) | 2019-12-11 | 2023-11-09 | A2 Biotherapeutics, Inc. | LILRB1-based chimeric antigen receptor |
| WO2021222576A1 (en) | 2020-05-01 | 2021-11-04 | A2 Biotherapeutics, Inc. | Pag1 fusion proteins and methods of making and using same |
| KR20230052289A (ko) | 2020-08-20 | 2023-04-19 | 에이투 바이오쎄라퓨틱스, 인크. | Egfr 양성 암을 치료하기 위한 조성물 및 방법 |
-
2021
- 2021-08-19 KR KR1020237009181A patent/KR20230052289A/ko active Search and Examination
- 2021-08-19 SI SI202130163T patent/SI4058474T1/sl unknown
- 2021-08-19 PT PT217692912T patent/PT4058474T/pt unknown
- 2021-08-19 HR HRP20240903TT patent/HRP20240903T1/hr unknown
- 2021-08-19 AU AU2021328478A patent/AU2021328478A1/en active Pending
- 2021-08-19 WO PCT/US2021/046733 patent/WO2022040444A1/en active Application Filing
- 2021-08-19 PL PL21769291.2T patent/PL4058474T3/pl unknown
- 2021-08-19 EP EP21769291.2A patent/EP4058474B1/en active Active
- 2021-08-19 CA CA3189510A patent/CA3189510A1/en active Pending
- 2021-08-19 BR BR112023002668A patent/BR112023002668A2/pt unknown
- 2021-08-19 DK DK21769291.2T patent/DK4058474T3/da active
- 2021-08-19 IL IL300629A patent/IL300629A/en unknown
- 2021-08-19 JP JP2023512379A patent/JP2023538116A/ja active Pending
- 2021-08-19 RS RS20240748A patent/RS65802B1/sr unknown
- 2021-08-19 FI FIEP21769291.2T patent/FI4058474T3/fi active
- 2021-08-19 LT LTEPPCT/US2021/046733T patent/LT4058474T/lt unknown
- 2021-08-19 EP EP24168884.5A patent/EP4442815A2/en active Pending
- 2021-08-19 MX MX2023002104A patent/MX2023002104A/es unknown
- 2021-08-19 TW TW110130697A patent/TW202214846A/zh unknown
-
2022
- 2022-05-17 US US17/663,758 patent/US11602544B2/en active Active
-
2023
- 2023-02-07 US US18/165,623 patent/US20230277593A1/en active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| JP2023538116A (ja) | 2023-09-06 |
| MX2023002104A (es) | 2023-07-10 |
| RS65802B1 (sr) | 2024-08-30 |
| EP4058474B1 (en) | 2024-05-08 |
| HRP20240903T1 (hr) | 2024-10-11 |
| EP4442815A2 (en) | 2024-10-09 |
| EP4058474A1 (en) | 2022-09-21 |
| WO2022040444A1 (en) | 2022-02-24 |
| PL4058474T3 (pl) | 2024-09-23 |
| LT4058474T (lt) | 2024-08-26 |
| AU2021328478A1 (en) | 2023-04-20 |
| CA3189510A1 (en) | 2022-02-24 |
| SI4058474T1 (sl) | 2024-09-30 |
| US20230277593A1 (en) | 2023-09-07 |
| BR112023002668A2 (pt) | 2023-05-02 |
| PT4058474T (pt) | 2024-07-09 |
| IL300629A (en) | 2023-04-01 |
| US20220273721A1 (en) | 2022-09-01 |
| US11602544B2 (en) | 2023-03-14 |
| FI4058474T3 (fi) | 2024-07-01 |
| TW202214846A (zh) | 2022-04-16 |
| DK4058474T3 (da) | 2024-07-22 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN110494451B (zh) | 靶向tim-1的嵌合抗原受体 | |
| KR20220053587A (ko) | 이형접합성 소실에 반응성인 세포 표면 수용체 | |
| CN115003802B (zh) | 一种经修饰的免疫效应细胞及其制备方法 | |
| KR20230052291A (ko) | Ceacam 양성 암을 치료하기 위한 조성물 및 방법 | |
| CN114269372A (zh) | 嵌合抗原受体t细胞和nk细胞抑制剂用于治疗癌症的用途 | |
| US20230277590A1 (en) | Compositions and methods for treating mesothelin positive cancers | |
| KR20240027117A (ko) | B7h3을 표적으로 하는 항원 결합 폴리펩티드 및 이의 응용 | |
| KR20230147109A (ko) | Her2 양성 암을 치료하기 위한 조성물 및 방법 | |
| WO2022040470A1 (en) | Compositions and methods for treating ceacam positive cancers | |
| AU2021386366A9 (en) | Adoptive cell therapy for treatment of cancer associated with loss of heterozygosity | |
| US20230277593A1 (en) | Compositions and methods for treating egfr positive cancers | |
| CN116635043A (zh) | 用于治疗egfr阳性癌症的组合物和方法 | |
| JP2024159820A (ja) | Egfr陽性がんを治療するための組成物及び方法 | |
| WO2023172954A2 (en) | Engineered receptors specific to hla-e and methods of use | |
| KR20240155391A (ko) | 메소텔린 양성 암을 치료하기 위한 조성물 및 방법 | |
| WO2023147019A2 (en) | Compositions and methods for treating mesothelin positive cancers | |
| CN116761817A (zh) | 用于治疗间皮素阳性癌症的组合物和方法 | |
| JP2024527537A (ja) | Gd2を標的とするユニバーサルcar-t細胞及びその製造方法並びに使用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| A302 | Request for accelerated examination |