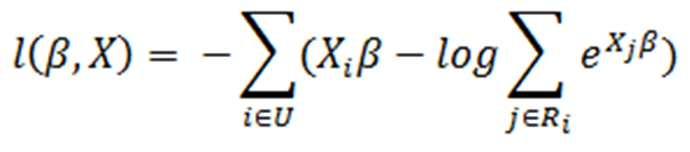KR102336311B1 - Model for Predicting Cancer Prognosis using Deep learning - Google Patents
Model for Predicting Cancer Prognosis using Deep learning Download PDFInfo
- Publication number
- KR102336311B1 KR102336311B1 KR1020190146627A KR20190146627A KR102336311B1 KR 102336311 B1 KR102336311 B1 KR 102336311B1 KR 1020190146627 A KR1020190146627 A KR 1020190146627A KR 20190146627 A KR20190146627 A KR 20190146627A KR 102336311 B1 KR102336311 B1 KR 102336311B1
- Authority
- KR
- South Korea
- Prior art keywords
- prognosis
- cancer
- learning
- predicting
- data
- Prior art date
Links
Images
Classifications
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H50/00—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics
- G16H50/50—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics for simulation or modelling of medical disorders
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H10/00—ICT specially adapted for the handling or processing of patient-related medical or healthcare data
- G16H10/60—ICT specially adapted for the handling or processing of patient-related medical or healthcare data for patient-specific data, e.g. for electronic patient records
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H20/00—ICT specially adapted for therapies or health-improving plans, e.g. for handling prescriptions, for steering therapy or for monitoring patient compliance
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H50/00—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics
- G16H50/30—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics for calculating health indices; for individual health risk assessment
Landscapes
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Medical Informatics (AREA)
- Public Health (AREA)
- Epidemiology (AREA)
- General Health & Medical Sciences (AREA)
- Primary Health Care (AREA)
- Biomedical Technology (AREA)
- Data Mining & Analysis (AREA)
- Databases & Information Systems (AREA)
- Pathology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Abstract
본 발명은 암 환자의 오믹스 데이터로부터 암의 예후를 예측할 수 있는 예후 예측 시스템에 관한 것으로, 상기 시스템에 따르면 복수의 오믹스 데이터를 사용함으로써 분자생물학적 다양성을 통합적으로 고려하여 암 환자의 예후를 예측할 수 있고, 단일 유전자가 암의 예후 예측에 미치는 중요도를 판단할 수 있다.The present invention relates to a prognosis prediction system capable of predicting the prognosis of cancer from omics data of a cancer patient. It is possible to determine the importance of a single gene in predicting the prognosis of cancer.
Description
본 발명은 암 환자의 멀티 오믹스 데이터를 이용하여 딥러닝 기반으로 암의 예후에 영향을 미치는 요인을 파악함으로써 효과적으로 암의 예후를 예측하는 시스템에 관한 것이다.The present invention relates to a system for effectively predicting the prognosis of cancer by identifying factors affecting the prognosis of cancer based on deep learning using multi-omics data of cancer patients.
암의 예후를 예측하는 것, 즉 암 환자의 수술 후 생존기간을 예측하는 것은 암 환자들에게 매우 중요한 요소이다. 과거의 예후 예측 모델은 환자의 나이, 암의 단계(stage), 성별 등 임상정보만을 이용하여 오직 선형적인 관계로 예후를 예측하였다. 그러나 이러한 방법은 같은 암이라도 개별 환자마다 암의 특성이 다르다는 점을 반영하지 못하므로 예측 성능이 떨어지는 한계점이 있었다.Predicting the prognosis of cancer, that is, predicting the survival period of cancer patients after surgery is a very important factor for cancer patients. Past prognosis prediction models used only clinical information such as the patient's age, cancer stage, and sex to predict the prognosis in a linear relationship only. However, this method has a limitation in predicting performance because it does not reflect the fact that the characteristics of each patient are different even for the same cancer.
이러한 문제를 해결하기 위해 최근 암의 다양성에 대한 연구가 진행되고 있다. 2018년 종결된 암 유전체 지도(The Cancer Genome Atlas, TCGA) 프로젝트는 개별 환자의 암 특성을 확인할 수 있는 다양한 오믹스(omics) 데이터를 생산하였다. 최근에는 이런 오믹스 데이터를 암의 예후 예측에 적용함으로써 기존 예후 예측 모델의 한계점을 극복하려는 연구가 진행되고 있다.In order to solve this problem, research on the diversity of cancer is being conducted recently. The Cancer Genome Atlas (TCGA) project, which was completed in 2018, produced various omics data that can confirm the cancer characteristics of individual patients. Recently, studies have been conducted to overcome the limitations of existing prognostic prediction models by applying such omics data to cancer prognosis prediction.
그러나 지금까지 연구된 암의 예후 예측 모델은 일부 한계점이 있다. 첫째는 현재 가장 많이 사용하는 예후 예측 방법은 선형적인 방식이라는 점이다. 이 방법은 직관적으로 예후에 미치는 영향을 확인할 수 있다는 장점이 있지만 생물학적 복잡성을 단순한 선형 모델로 설명하기 어렵다는 한계점이 있고 그로 인해 예후 예측 성능이 조금 떨어진다는 단점이 있다.However, the prognosis prediction model of cancer studied so far has some limitations. First, the most widely used prognostic prediction method is a linear method. Although this method has the advantage of being able to intuitively check the effect on the prognosis, it has a limitation in that it is difficult to explain the biological complexity with a simple linear model, and thus the prognostic performance is slightly lowered.
둘째로 대부분의 암 예후 예측은 단일 오믹스만을 사용한다는 점이다. 최근까지 암의 특징을 확인할 수 있는 다양한 오믹스 방법들이 많이 개발되어 분석에 사용되고 있지만 예후 예측에는 RNA 서열분석과 같이 대부분 유전자의 발현을 확인하는 오믹스 방법만이 사용되고 있다. 하지만 암은 유전자의 발현뿐만 아니라 유전자의 변이 등 복잡한 원인으로 인해 발생하고 예후에 영향을 미친다. 따라서 단일 오믹스만으로는 암의 다양성을 설명하기 힘든 문제가 있다. Second, most cancer prognosis predictions use only a single omics. Until recently, many omics methods for identifying cancer characteristics have been developed and used for analysis, but only the omics method for confirming gene expression, such as RNA sequencing, is mostly used for prognosis prediction. However, cancer is caused by complex causes such as gene mutation as well as gene expression and affects the prognosis. Therefore, there is a problem that it is difficult to explain the diversity of cancer with only a single omics.
셋째는 선행 기술에서는 단일 유전자당 예후에 미치는 중요도 확인을 통해 최종 마커 유전자를 선별하기 어렵다는 것이다. 선행 기술에서는 하나의 유전자당 여러 멀티 오믹스 특징들에 대해 독립적으로 할당한다. 하지만 이 방식으로는 각 오믹스 특징마다 중요한 유전자가 서로 다르기 때문에 최종적으로 어떤 유전자가 예후 예측의 마커로 사용될 수 있을지 선별하기 어렵다는 단점이 존재한다. Third, in the prior art, it is difficult to select a final marker gene by confirming the importance of a single gene on prognosis. In the prior art, multiple multi-omics characteristics per one gene are independently assigned. However, this method has a disadvantage in that it is difficult to select which gene can be used as a marker for prognosis prediction because important genes are different for each omics characteristic.
본 발명자들은 상기 한계점을 해결하기 위해 비선형모델로 생물학적 다양성을 학습하고, 멀티 오믹스를 이용해 암의 분자생물학적 다양성을 통합적으로 고려하며, 단일 유전자당 중요도를 판단할 수 있는 암의 예후 예측용 시스템을 개발하여 본 발명을 완성하였다.In order to solve the above limitations, the present inventors have developed a system for predicting the prognosis of cancer that can learn biological diversity with a non-linear model, comprehensively consider molecular biological diversity of cancer using multi-omics, and determine the importance per single gene. developed and completed the present invention.
본 발명의 목적은 멀티 오믹스 데이터에 기반하여 암의 분자생물학적 다양성을 통합적으로 고려함으로써 암의 예후를 효과적으로 예측할 수 있는 암의 예후 예측용 시스템을 제공하는 것이다.It is an object of the present invention to provide a system for predicting the prognosis of cancer that can effectively predict the prognosis of cancer by integrally considering molecular and biological diversity of cancer based on multi-omics data.
상기 목적을 달성하기 위하여, 본 발명의 일 양상은 하기 단계를 포함하는 딥러닝을 이용한 암의 예후 예측용 시스템을 제공한다:In order to achieve the above object, an aspect of the present invention provides a system for predicting the prognosis of cancer using deep learning comprising the steps of:
(1) 암 환자로부터 수집된 오믹스 데이터와 암의 예후와의 상관관계를 학습하는 학습 모듈;(1) a learning module for learning the correlation between omics data collected from cancer patients and cancer prognosis;
(2) 상기 학습 모듈에서 학습된 암의 예후 예측 알고리즘을 저장하는 저장 모듈; 및(2) a storage module for storing the cancer prognosis prediction algorithm learned in the learning module; and
(3) 분석 정보를 수신하고, 상기 저장 모듈에 저장된 암의 예후 예측 알고리즘을 사용하여 예후를 모르는 암 환자의 예후를 산출하는 예측 모듈.(3) A prediction module for receiving analysis information and calculating a prognosis of a cancer patient whose prognosis is unknown by using the cancer prognosis prediction algorithm stored in the storage module.
본 발명의 일 구체예에 있어서, 상기 암의 예후 예측용 시스템은 암 환자의 치료 후 생존 기간, 또는 외과적 수술 후 생존 기간 예측에 사용될 수 있다.In one embodiment of the present invention, the system for predicting the prognosis of cancer may be used to predict the survival period after treatment or the survival period after surgical operation of a cancer patient.
본 명세서에 사용된 용어, "오믹스(omics)"는 유전체, 전사체, 단백체, 대사체 등 다양한 분자 수준에서 생성된 여러 데이터들을 의미하며, 초고속, 대량분석이 가능한 분자생물학적 기술의 발전, 컴퓨터 산업의 발전에 따른 정보 처리 능력의 비약적 발달에 따라 멀티 오믹스 데이터가 생성되고 있다.As used herein, the term "omics" refers to various data generated at various molecular levels such as genome, transcriptome, proteomic, metabolite, etc. Multi-omics data is being generated according to the rapid development of information processing ability according to the development of industry.
본 발명의 일 구체예에 있어서, 상기 오믹스 데이터는 유전체(genome), 전사체(transcriptome), 단백체(proteome), 대사체(metabolome), 후성유전체 (epigenome), 지질체(lipodome) 및 메틸체(methylome)로 이루어진 군에서 선택될 수 있으나, 이에 제한되지 않는다. 본 발명에서, 상기 오믹스 데이터는 복수개가 사용될 수 있고, 바람직하게는 최소 3개의 오믹스 데이터가 사용될 수 있으며, 예를 들어 유전체, 전사체 및 메틸체 데이터가 사용될 수 있다.In one embodiment of the present invention, the omics data is a genome, a transcriptome, a proteome, a metabolome, an epigenome, a lipodome, and a methyl body. (methylome) may be selected from the group consisting of, but is not limited thereto. In the present invention, a plurality of omics data may be used, and preferably, at least three omics data may be used, for example, genome, transcript, and methyl data may be used.
본 발명의 일 구체예에 있어서, 상기 학습 모듈은 하기 단계를 포함할 수 있다.In one embodiment of the present invention, the learning module may include the following steps.
(a) 예후를 알고 있는 암 환자의 오믹스 데이터로부터 딥러닝용 학습 데이터를 생성하는 학습 데이터 생성부;(a) a learning data generator for generating learning data for deep learning from omics data of cancer patients who know the prognosis;
(b) 생성된 학습 데이터로부터 오믹스 데이터와 암의 예후와의 상관 관계를 학습하는 딥러닝 수행부; 및(b) a deep learning performing unit learning the correlation between the omics data and the cancer prognosis from the generated learning data; and
(c) 상기 딥러닝 수행부의 결과로부터 암의 예후를 예측하는 알고리즘을 생성하는 알고리즘 생성부.(c) an algorithm generating unit for generating an algorithm for predicting the prognosis of cancer from the results of the deep learning performing unit.
본 명세서에 사용된 용어, "딥러닝(deep learning)"은 심층 학습으로도 불리우며, 최근 기계학습 분야에서 대두되고 있는 기술 중 하나로 복수개의 은닉 계층(hidden layer)와 이들에 포함되는 복수 개의 유닛(hidden unit)으로 구성되는 신경망(neural network)이다.As used herein, the term "deep learning" is also called deep learning, and is one of the technologies that are emerging in the field of machine learning recently. A plurality of hidden layers and a plurality of units included in them ( It is a neural network composed of hidden units.
딥러닝 모델에 기본 특성(low level feature)들을 입력하는 경우, 이러한 기본 특성들이 복수 개의 은닉 계층을 통과하면서 예측하고자 하는 문제를 보다 잘 설명할 수 있는 상위 레벨 특성(high level feature)로 변형된다. 이러한 과정에서 전문가의 사전 지식 또는 직관이 요구되지 않기 때문에 특성 추출에서의 주관적 요인을 제거할 수 있으며, 보다 높은 일반화 능력을 갖는 모델을 개발할 수 있게 된다.When low level features are input to the deep learning model, these basic features are transformed into high level features that can better explain the problem to be predicted while passing through a plurality of hidden layers. In this process, since prior knowledge or intuition of an expert is not required, the subjective factor in feature extraction can be removed, and a model with higher generalization ability can be developed.
또한, 딥러닝의 경우 특징 추출과 모델 구축이 하나의 세트로 구성되어 있기 때문에 기존의 기계학습 이론 대비 보다 단순한 과정을 통하여 최종 모델을 형성할 수 있는 장점이 있다.In addition, in the case of deep learning, since feature extraction and model construction are composed of one set, it has the advantage of forming a final model through a simpler process compared to the existing machine learning theory.
본 발명에서, 상기 딥러닝은 심층 신경망(Deep Neural Network, DNN)에 기반하여 수행될 수 있으며, 예를 들어 구글 오픈소스인 텐서플로우(TensorFlow)로 수행될 수 있다.In the present invention, the deep learning may be performed based on a deep neural network (DNN), for example, may be performed with Google open source TensorFlow.
본 발명에서, 상기 학습 데이터 생성부는 예후를 알고 있는 암 환자의 오믹스 데이터를 개별 유전자에 대해 매트릭스 형태로 정렬할 수 있다. 매트릭스 형태로 정렬함으로써 개별 유전자, 단일 오믹스에 대한 정보만으로 암의 예후를 예측하는 것이 아니라, 개별 유전자에 대한 오믹스 데이터들의 공통 특징을 학습하여 비선형적인 방법으로 암의 예후를 예측할 수 있다. 사용되는 유전자는 따로 선별하는 것이 아니라, 오믹스 데이터에 포함된 전체 유전자를 사용할 수 있다.In the present invention, the learning data generating unit may align the omics data of the cancer patient with known prognosis in a matrix form for individual genes. By arranging in a matrix form, the prognosis of cancer is not predicted only with information on individual genes and single omics, but the prognosis of cancer can be predicted in a non-linear way by learning common characteristics of omics data for individual genes. The genes used are not selected separately, but all genes included in the omics data can be used.
본 발명의 일 구체예에 있어서, 상기 딥러닝 수행부는 하기 단계를 포함할 수 있다:In one embodiment of the present invention, the deep learning performing unit may include the following steps:
(b-1) 정렬된 오믹스 데이터로부터 개별 유전자에 대한 오믹스 데이터의 공통 특징을 학습하는 단계;(b-1) learning common characteristics of omics data for individual genes from the aligned omics data;
(b-2) 학습된 개별 유전자에 대한 오믹스 데이터의 공통 특징에서 중요하지 않은 정보를 제거하는 단계;(b-2) removing insignificant information from common features of omics data for individual learned genes;
(b-3) 상기 (b-2)의 결과로부터 개별 유전자당 가장 높은 값을 암의 예후 예측을 위한 대푯값으로 선별하는 단계;(b-3) selecting the highest value per individual gene as a representative value for predicting the prognosis of cancer from the result of (b-2);
(b-4) 상기 (b-3)에서 선별한 개별 유전자당 대푯값을 조합하여 암의 예후 예측의 정확도를 확인하는 단계; 및(b-4) confirming the accuracy of predicting cancer prognosis by combining the representative values for each individual gene selected in (b-3); and
(b-5) 상기 (b-4)의 결과에서 정확도가 가장 높은 대푯값의 조합을 선별하는 단계.(b-5) selecting a representative value combination with the highest accuracy from the result of (b-4).
본 발명의 일 구체예에 있어서, 상기 (b-1) 단계는 정렬된 오믹스 데이터로 컨볼루션(convolution)을 진행하여 수행될 수 있으며, (b-2) 단계는 학습된 개별 유전자에 대한 오믹스 데이터의 공통 특징에 ReLU (rectified linear unit) 함수를 적용하여 수행될 수 있다.In one embodiment of the present invention, step (b-1) may be performed by performing convolution with the aligned omics data, and step (b-2) is an error for each learned individual gene. This may be performed by applying a rectified linear unit (ReLU) function to a common feature of the mix data.
본 발명의 일 구체예에 있어서, 상기 (b-4) 단계는 콕스 비례 위험 모델(Cox proportional hazards model)을 사용하여 개별 유전자당 대푯값과 암 환자의 예후와의 음의 로그 부분 유도(negative log partial likelihood)를 계산하는 것일 수 있다.In one embodiment of the present invention, in the step (b-4), the negative log partial induction of the representative value per individual gene and the prognosis of cancer patients using a Cox proportional hazards model (negative log partial) likelihood) can be calculated.
본 발명의 다른 양상은 상기 암의 예후 예측 시스템을 사용한 암의 예후 예측 방법을 제공한다. 상기 암의 예후 예측 시스템을 사용하면 예후를 모르는 암 환자에서 수집된 멀티 오믹스 데이터로부터 암의 예후에 가장 큰 영향을 미치는 개별 유전자를 선별할 수 있으므로 예후가 불분명한 암 환자의 예후를 효과적으로 예측할 수 있다. 또한, 암의 예후를 예측함으로써 예후가 나쁠 것으로 예상되면 공격적 치료를 수행한다는 등의 방식으로 개인별 맞춤 의료를 제공할 수 있다.Another aspect of the present invention provides a method for predicting the prognosis of cancer using the system for predicting the prognosis of cancer. Using the cancer prognosis prediction system, individual genes that have the greatest impact on cancer prognosis can be selected from multi-omics data collected from cancer patients with unknown prognosis, so the prognosis of cancer patients with unclear prognosis can be effectively predicted. have. In addition, by predicting the prognosis of cancer, if the prognosis is expected to be poor, it is possible to provide personalized medical care in such a way as to perform aggressive treatment.
본 발명의 암의 예후 예측용 시스템에 따르면 복수의 오믹스 데이터를 사용함으로써 암의 분자생물학적 다양성을 통합적으로 고려하여 암 환자의 예후를 예측할 수 있고, 단일 유전자가 암의 예후 예측에 미치는 중요도를 판단할 수 있다.According to the system for predicting cancer prognosis of the present invention, by using a plurality of omics data, the prognosis of a cancer patient can be predicted by considering the molecular and biological diversity of cancer in an integrated manner, and the importance of a single gene in predicting the prognosis of cancer is determined can do.
도 1은 본 발명에 따른 암의 예후 예측 시스템의 구성을 설명하기 위한 블록도이다.
도 2는 본 발명에 따른 암의 예후 예측 시스템에서 학습 모듈의 학습 데이터 생성부의 일 예를 나타낸다.
도 3은 본 발명에 따른 암의 예후 예측 시스템에서 학습 모듈의 딥러닝 수행부의 일 예를 나타낸다.
도 4는 본 발명의 일 실시예에 따른 암의 예후 예측 시스템과 기존의 암 예후 예측 모델의 예후 예측 성능을 비교한 결과를 나타낸다.
도 5는 본 발명의 일 실시예에 따른 암의 예후 예측 시스템과 암의 예후 예측 마커로 알려진 유전자의 예후 예측 성능을 비교한 결과를 나타낸다.1 is a block diagram illustrating the configuration of a cancer prognosis prediction system according to the present invention.
2 shows an example of a learning data generator of a learning module in the cancer prognosis prediction system according to the present invention.
3 shows an example of the deep learning performing unit of the learning module in the cancer prognosis prediction system according to the present invention.
4 shows a result of comparing the prognosis prediction performance of the cancer prognosis prediction system and the existing cancer prognosis prediction model according to an embodiment of the present invention.
5 shows a result of comparing the prognosis prediction system for cancer according to an embodiment of the present invention and the prognosis prediction performance of a gene known as a cancer prognosis predictor marker.
이하에서는 첨부한 도면을 참조하여 본 발명을 설명하기로 한다. 그러나 본 발명은 여러 가지 상이한 형태로 구현될 수 있으며, 따라서 여기에서 설명하는 실시예로 한정되는 것은 아니다. 그리고 도면에서 본 발명을 명확하게 설명하기 위해서 설명과 관계없는 부분은 생략하였으며, 명세서 전체를 통하여 유사한 부분에 대해서는 유사한 도면 부호를 붙였다.Hereinafter, the present invention will be described with reference to the accompanying drawings. However, the present invention may be embodied in several different forms, and thus is not limited to the embodiments described herein. And in order to clearly explain the present invention in the drawings, parts irrelevant to the description are omitted, and similar reference numerals are attached to similar parts throughout the specification.
어떤 부분이 어떤 구성요소를 "포함"한다고 할 때, 이는 특별히 반대되는 기재가 없는 한 다른 구성요소를 제외하는 것이 아니라 다른 구성요소를 추가로 구비할 수 있다는 것을 의미한다.When a part "includes" a certain component, it means that other components may be additionally provided without excluding other components unless otherwise stated.
이하, 첨부된 도면을 참고하여 본 발명을 더욱 상세히 설명한다.Hereinafter, the present invention will be described in more detail with reference to the accompanying drawings.
도 1은 본 발명의 암의 예후 예측 시스템의 구성을 예시적으로 나타낸다.1 exemplarily shows the configuration of the cancer prognosis prediction system of the present invention.
상기 암의 예후 예측 시스템은 The cancer prognosis prediction system
(a) 암 환자로부터 수집된 오믹스 데이터와 암의 예후와의 상관관계를 학습하는 학습 모듈(100);(a) a
(b) 상기 학습 모듈에서 학습된 암의 예후 예측 알고리즘을 저장하는 저장 모듈(200); 및 (b) a
(c) 분석 정보를 수신하고, 상기 저장 모듈에 저장된 암의 예후 예측 알고리즘을 사용하여 예후를 모르는 암 환자의 예후를 산출하는 예측 모듈(300)을 포함한다.and (c) a
상기 (a)의 학습 모듈(100)은 암의 예후, 즉 치료 후 생존 기간을 이미 알고 있는 암 환자의 오믹스 데이터와 암의 예후와의 상관관계를 딥러닝에 의해 학습하는 부분이며, 학습 데이터 생성부(110), 딥러닝 수행부(120) 및 알고리즘 생성부(130)를 포함한다.The
상기 오믹스 데이터는 암 유전체 지도(The Cancer Genome Atlas, TCGA)와 같은 공개 데이터베이스로부터 수집할 수 있으며, 이외에 NCBI (National Center for Biotechnology Information) 등으로부터도 수집할 수 있다. 상기 오믹스 데이터는 암의 분자생물학적 다양성을 통합적으로 위해 복수개가 사용될 수 있으며, 바람직하게는 3개 이상의 오믹스 데이터가 사용될 수 있다.The omics data may be collected from a public database such as the Cancer Genome Atlas (TCGA), and may also be collected from the National Center for Biotechnology Information (NCBI). A plurality of the omics data may be used to integrate the molecular and biological diversity of cancer, and preferably three or more omics data may be used.
상기 학습 데이터 생성부(110)는 암 환자의 오믹스 데이터를 딥러닝용 학습 데이터로 가공하는 부분이며, 도 2에 도시된 바와 같이 각각의 오믹스 데이터를 개별 유전자에 대해 매트릭스 형태로 정렬한다. 매트릭스 형태로 정렬하는 것은 유전자 발현 데이터만을 이용해 암의 예후를 예측했던 기존의 방법과는 달리 유전자의 발현, 유전자 변이 등 암의 다양성을 통합적으로 고려하는 예후 예측 시스템을 구축하기 위함이다.The learning
상기 딥러닝 수행부(120)는 학습 데이터로부터 서로 다른 딥러닝 함수를 사용하여 개별 유전자에 대해 정렬된 오믹스 데이터와 암의 예후와의 상관 관계를 학습하는 부분이며, 도 3에 도시된 바와 같이 하기 단계를 포함할 수 있다:The deep
(b-1) 정렬된 오믹스 데이터로부터 개별 유전자에 대한 오믹스 데이터의 공통 특징을 학습하는 단계;(b-1) learning common characteristics of omics data for individual genes from the aligned omics data;
(b-2) 학습된 개별 유전자에 대한 오믹스 데이터의 공통 특징에서 중요하지 않은 정보를 제거하는 단계;(b-2) removing insignificant information from common features of omics data for individual learned genes;
(b-3) 상기 (b-2)의 결과로부터 개별 유전자당 가장 높은 값을 암의 예후 예측을 위한 대푯값으로 선별하는 단계;(b-3) selecting the highest value per individual gene as a representative value for predicting the prognosis of cancer from the result of (b-2);
(b-4) 상기 (b-3)에서 선별한 개별 유전자당 대푯값을 조합하여 암의 예후 예측의 정확도를 확인하는 단계; 및(b-4) confirming the accuracy of predicting cancer prognosis by combining the representative values for each individual gene selected in (b-3); and
(b-5) 상기 (b-4)의 결과에서 정확도가 가장 높은 대푯값의 조합을 암의 예후 예측용 마커로 선별하는 단계.(b-5) selecting a combination of representative values with the highest accuracy from the results of (b-4) as a marker for predicting cancer prognosis.
상기 (b-1) 단계는 매트릭스 형태로 정렬된 오믹스 데이터로 컨볼루션 (convolution)을 진행하여 수행될 수 있다. 구체적으로 하기 수학식 1에 따라 n개의 유전자에 대해 k개의 커널로 컨볼루션(convolution)을 진행하며, 예를 들어 k=3이라면 k=1, 2, 3 각각에 대해 컨볼루션을 진행하게 된다. 이 과정을 통해 각 커널마다 개별 유전자에 대한 오믹스 데이터들의 공통 특징이 학습된다. 딥러닝이 완료된 후 하기 수학식 1에서 w의 크기는 해당 오믹스 데이터의 예후 예측에 있어서의 중요도를 나타내며, k는 하이퍼파라미터(hyperperparameter) 튜닝(최적화) 과정에서 높은 성능을 나타내는 값을 사용한다. 하이퍼파라미터는 딥러닝을 수행하기 위해 사전에 설정해야 하는 값을 의미한다.Step (b-1) may be performed by performing convolution with the omics data arranged in a matrix form. Specifically, according to Equation 1 below, convolution is performed with k kernels for n genes. For example, if k=3, convolution is performed for each of k=1, 2, and 3. Through this process, common characteristics of omics data for individual genes are learned for each kernel. After the deep learning is completed, in Equation 1 below, the size of w indicates the importance in predicting the prognosis of the omics data, and k is a value indicating high performance in the hyperparameter tuning (optimization) process. Hyperparameter means a value that must be set in advance to perform deep learning.
[수학식 1][Equation 1]
(m=오믹스, n=유전자, Xmn=유전자와 오믹스 특징들로 이루어진 매트릭스, w= 개별 유전자의 멀티 오믹스 데이터와 곱해지는 웨이트(weight), k=컨볼루션을 진행하는 커널(kernel), 및 는 커널 k로 컨볼루션을 진행한 유전자 n에 대한 값을 의미함)(m=omics, n=genes, Xmn =matrix consisting of genes and omics features, w=weight multiplied by multi-omics data of individual genes, k=kernel performing convolution , and denotes the value for gene n convolved with kernel k)
상기 (b-2) 단계는 학습된 개별 유전자에 대한 오믹스 데이터들의 공통 특징에 하기 수학식 2를 적용하여 중요하지 않은 정보를 제거하는 단계이며, 이 과정에서 중요도가 높은 유전자에 대한 결괏값()만 남게 된다.The step (b-2) is a step of removing unimportant information by applying Equation 2 below to the common characteristics of the learned omics data for individual genes, and in this process, the resultant value ( ) will remain.
[수학식 2][Equation 2]
상기 수학식 2에 사용된 ReLU(rectified linear unit) 함수는 딥러닝 과정에서 사용되는 활성화 함수(activation fuction)로 상기 수학식 2에 기재된 바와 같이 x가 0보다 작은 값일 때는 0을 사용하고, 반대로 0보다 큰 값일 때는 해당 값을 그대로 사용하는 함수이다. 또한, 상기 b는 바이어스(bias)를 의미하며, 딥러닝 과정에서 자동적으로 정해진다.The rectified linear unit (ReLU) function used in Equation 2 is an activation function used in a deep learning process. As described in Equation 2, when x is a value less than 0, 0 is used, and vice versa, 0 If it is a larger value, it is a function that uses the value as it is. In addition, b denotes a bias, and is automatically determined in the deep learning process.
상기 (b-3) 단계는 상기 (b-2)에서 나온 중요도가 높은 유전자에 대한 결괏값에 대하여 수학식 3에 따른 맥스 풀링(max pooling)을 진행하여 개별 유전자당 가장 높은 값을 암의 예후 예측을 위한 대푯값()으로 선별하는 단계이다.In the step (b-3), max pooling according to
[수학식 3][Equation 3]
상기 (b-4) 단계는 (b-3)에서 선별한 개별 유전자당 대푯값을 조합하여 암의 예후 예측의 정확도를 확인하는 단계이며, 구체적으로 개별 유전자당 대푯값의 조합으로 예측된 암의 예후와 이미 알고 있는 예후를 비교하여 정확도를 확인한다. 이때 하기 수학식 4에 따른 콕스 비례 위험 모델(Cox proportional hazards model)을 사용하여 개별 유전자당 대푯값과 암 환자의 예후와의 음의 로그 부분 유도(negative log partial likelihood)를 계산하여 학습을 진행하였다.The step (b-4) is a step of confirming the accuracy of predicting the prognosis of cancer by combining the representative values for each individual gene selected in (b-3). Specifically, the prognosis of cancer predicted by the combination of the representative values per individual gene and the Check the accuracy by comparing the known prognosis. At this time, using the Cox proportional hazards model according to Equation 4 below, the negative log partial likelihood between the representative value per individual gene and the prognosis of cancer patients was calculated and learning was carried out.
[수학식 4][Equation 4]
(=매트릭스, =콕스 비례 위험 모델의 파라미터, =검열이 안된 샘플들(uncensored samples) 집단, =검열된 샘플들(at-risk samples) 및 =생존 추적(follow up) 기간으로 보다 더 길 때를 의미함)( = matrix, = parameters of the Cox proportional hazards model, = population of uncensored samples, =at-risk samples and = to follow up period means longer than)
상기 (b-5) 단계는 상기 (b-4)의 결과에서 정확도가 가장 높은 대푯값의 조합을 암의 예후 예측용 마커로 선별하는 단계이며, 이 과정에서 암의 예후 예측에 중요한 영향을 미치는 유전자 또는 유전자들의 조합이 선별된다.Step (b-5) is a step of selecting the combination of representative values with the highest accuracy from the result of (b-4) as a marker for predicting cancer prognosis. or a combination of genes is selected.
상기 저장 모듈(200)은 상기 학습모듈에 의해 학습된 암의 예후 예측 알고리즘을 저장하는 부분으로, 암의 예후 예측 알고리즘 데이터베이스(database, DB)(220)를 포함하여 구성되고, 학습된 개별 유전자당 오믹스 데이터와 암의 예후와의 관련성을 저장하기 위한 오믹스 데이터-예후 관련 DB(210)를 더 포함하여 구성될 수도 있다.The
상기 예측 모듈(300)은 분석할 정보를 수신하는 분석 정보 수신부(310) 및 저장 모듈에 저장된 예후 예측 알고리즘을 사용하여 분석 정보로부터 산출한 예후를 출력하는 예후 결과 출력부(320)를 포함한다.The
상기 분석 정보는 예후가 불확실한 암 환자의 오믹스 데이터일 수 있으며, 상기 예후 결과는 암 치료 후 생존 기간일 수 있다. The analysis information may be omics data of a cancer patient with an uncertain prognosis, and the prognostic result may be a survival period after cancer treatment.
이하, 실시예를 통해 본 발명을 더욱 상세히 기술한다.Hereinafter, the present invention will be described in more detail by way of Examples.
실시예: 암의 예후 예측 시스템 검증Example: Prognosis prediction system validation of cancer
본 발명의 암의 예후 예측 시스템(이하, 예후 예측 시스템으로 기재함)은 TCGA 간암 환자 데이터 중에서 유전제, 전사체 및 메틸체 데이터를 학습에 사용하였다. 구체적으로 TCGA 간암 환자 데이터의 80%는 예후 예측 시스템의 학습, 검증 및 하이퍼파라미터 최적화에 사용하였고, 나머지 20%는 예후 예측 시스템의 테스트에 사용하였다.The cancer prognosis prediction system (hereinafter, referred to as the prognosis prediction system) of the present invention uses genetic agent, transcriptome and methyl data among TCGA liver cancer patient data for learning. Specifically, 80% of the TCGA liver cancer patient data was used for learning, validation, and hyperparameter optimization of the prognostic prediction system, and the remaining 20% was used for testing the prognostic prediction system.
본 발명의 예후 예측 시스템이 학습에 사용된 데이터에 과적합(overfitting)된 것이 아니라 완전히 다른 데이터셋에서도 작동하는 것을 확인하기 위하여 TCGA 데이터뿐만 아니라 완전히 독립적인 데이터셋인 한국인 간암 샘플 데이터에서 성능 확인을 진행했다.In order to confirm that the prognostic prediction system of the present invention does not overfit the data used for learning, but operates on a completely different dataset, the performance was confirmed not only on the TCGA data but also on the Korean liver cancer sample data, which is a completely independent dataset. proceeded
구체적으로 TCGA 간암 환자 211명과 한국인 간암 환자 144명에 대해 유전자 발현, DNA 체세포 변이(somatic mutation), DNA 생식선 변이(germline mutation), 복수체 변이(copy number variation), 발현 조절(DNA methylation)에 해당하는 오믹스 특징들을 사용해 본 발명의 예후 예측 시스템으로 간암 환자의 예후를 예측하고, 선행기술인 Glmnet, RSF(random survival forest) 모델과 성능을 비교하였다.Specifically, gene expression, DNA somatic mutation, DNA germline mutation, copy number variation, and expression regulation (DNA methylation) for 211 TCGA liver cancer patients and 144 Korean liver cancer patients The prognosis of a liver cancer patient was predicted with the prognosis prediction system of the present invention using the omics features of the present invention, and the performance was compared with the prior art Glmnet and RSF (random survival forest) models.
그 결과, 도 4에 나타낸 바와 같이 선형모델을 사용하는 Glmnet 모델은 두 데이터셋 모두에서 성능(concordance index, C-index)이 0.5 근처로 나타나 제일 낮았다. 상기 C-index는 0.0 내지 1.0 사이의 값을 가지며, 0.5에 가까울수록 무작위로 예측하는 것으로 평가하고, 1.0에 가까울수록 정확히 예측한다고 평가한다. 비선형 모델을 사용하는 RSF 모델은 학습에 사용되는 TCGA 간암 샘플에서는 높은 성능을 보였으나, 한국인 간암 샘플에서는 성능이 낮은 것으로 나타나 학습에 사용되는 데이터셋에 과적합되어 있는 현상을 확인할 수 있다. 반면 본 발명의 예후 예측 시스템은 모든 데이터셋에서 다른 모델들에 비해 높은 성능을 나타내었다.As a result, as shown in FIG. 4 , the Glmnet model using a linear model had the lowest performance (concordance index, C-index) near 0.5 in both datasets. The C-index has a value between 0.0 and 1.0, and the closer to 0.5, the more random prediction is evaluated, and the closer to 1.0, the more accurate prediction is evaluated. The RSF model using the nonlinear model showed high performance in the TCGA liver cancer sample used for training, but the performance was low in the Korean liver cancer sample, confirming the phenomenon of overfitting the dataset used for training. On the other hand, the prognostic prediction system of the present invention showed higher performance than other models in all datasets.
또한, 논문에서 간암의 예후를 예측하는 것으로 알려진 3개 유전자 (UPB1, SOCS2 및 RTN3), 11개 유전자 (ACSM3, CXCL14, INTS8, LCAT, MARCO, PAMR1, CRHBP, DNASE1L3, FCN2, MT1X 및 VIPR1) 및 종양 관련 섬유아세포(cancer associated fibroblasts, CAF)와 관련된 12종의 유전자(ACSM3, CXCL14, INTS8, LCAT, MARCO, PAMR1, CRHBP, DNASE1L3, FCN2, MT1X 및 VIPR1)를 확인하고, 상기 유전자들의 간암 예후 예측 성능을 평가하였다. 그 결과, 도 5에 나타낸 바와 같이 논문에서 제시하는 간암의 예후 예측 유전자들은 학습에 사용되는 TCGA 샘플에 과적합되어 있는 현상을 확인할 수 있었다.In addition, 3 genes (UPB1, SOCS2 and RTN3), 11 genes (ACSM3, CXCL14, INTS8, LCAT, MARCO, PAMR1, CRHBP, DNASE1L3, FCN2, MT1X and VIPR1) known to predict the prognosis of liver cancer in the paper and Identification of 12 genes (ACSM3, CXCL14, INTS8, LCAT, MARCO, PAMR1, CRHBP, DNASE1L3, FCN2, MT1X and VIPR1) related to cancer associated fibroblasts (CAF) and prediction of the prognosis of liver cancer by these genes Performance was evaluated. As a result, as shown in FIG. 5 , it was confirmed that the prognosis predicting genes of liver cancer presented in the paper are overfitted to the TCGA sample used for learning.
본 결과를 통해 본 발명의 예후 예측 시스템은 다양한 오믹스 데이터를 활용한 유전자 단위의 통합적인 학습으로 인해 다른 예후 예측 모델과 비교하여 간암의 예후 예측에 현저히 우수한 성능을 보이는 것을 확인할 수 있었다.Through these results, it was confirmed that the prognosis prediction system of the present invention showed significantly superior performance in predicting the prognosis of liver cancer compared to other prognostic prediction models due to the integrated learning of the genetic unit using various omics data.
100: 학습 모듈
110: 학습 데이터 생성부
120: 딥러닝 수행부
130: 알고리즘 생성부
200: 저장 모듈
210: 오믹스 데이터-예후 관련 DB
220: 예후 예측 알고리즘 DB
300: 예측 모듈
310: 분석 정보 수신부
320: 예후 결과 출력부100: learning module
110: training data generation unit
120: deep learning execution unit
130: Algorithm generator
200: storage module
210: omics data-prognosis related DB
220: prognosis prediction algorithm DB
300: prediction module
310: analysis information receiving unit
320: prognostic result output unit
Claims (9)
(2) 상기 학습 모듈에서 생성된 암의 예후 예측 알고리즘을 저장하는 저장 모듈; 및
(3) 분석 정보를 수신하고, 상기 저장 모듈에 저장된 암의 예후 예측 알고리즘을 사용하여 예후를 모르는 암 환자의 예후를 산출하는 예측 모듈;을 포함하고,
상기 학습 모듈은
(a) 예후를 알고 있는 암 환자의 오믹스 데이터를 개별 유전자에 대해 매트릭스 형태로 정렬하여 딥러닝용 학습 데이터를 생성하는 학습 데이터 생성부;
(b) 생성된 학습 데이터로부터 오믹스 데이터와 암의 예후와의 상관 관계를 학습하는 딥러닝 수행부; 및
(c) 상기 딥러닝 수행부의 결과로부터 암의 예후를 예측하는 알고리즘을 생성하는 알고리즘 생성부;를 포함하는 것인, 암의 예후 예측용 시스템.(1) a learning module for learning a correlation between three or more omics data collected from a cancer patient and a cancer prognosis;
(2) a storage module for storing the cancer prognosis prediction algorithm generated by the learning module; and
(3) a prediction module that receives the analysis information and calculates the prognosis of a cancer patient who does not know the prognosis by using the cancer prognosis prediction algorithm stored in the storage module;
The learning module is
(a) a learning data generator for generating learning data for deep learning by aligning omics data of cancer patients with known prognosis in a matrix form for individual genes;
(b) a deep learning performing unit learning the correlation between the omics data and the cancer prognosis from the generated learning data; and
(c) an algorithm generating unit that generates an algorithm for predicting the prognosis of cancer from the results of the deep learning performing unit;
(b-1) 정렬된 오믹스 데이터로부터 개별 유전자에 대한 오믹스 데이터의 공통 특징을 학습하는 단계;
(b-2) 학습된 개별 유전자에 대한 오믹스 데이터의 공통 특징에서 중요하지 않은 정보를 제거하는 단계;
(b-3) 상기 (b-2)의 결과로부터 개별 유전자당 가장 높은 값을 암의 예후 예측을 위한 대푯값으로 선별하는 단계;
(b-4) 상기 (b-3)에서 선별한 개별 유전자당 대푯값을 조합하여 암의 예후 예측의 정확도를 확인하는 단계; 및
(b-5) 상기 (b-4)의 결과에서 정확도가 가장 높은 대푯값의 조합을 암의 예후 예측용 마커로 선별하는 단계;를 포함하는 암의 예후 예측용 시스템.The method of claim 1, wherein the deep learning performing unit
(b-1) learning common characteristics of omics data for individual genes from the aligned omics data;
(b-2) removing insignificant information from common features of omics data for individual learned genes;
(b-3) selecting the highest value per individual gene as a representative value for predicting the prognosis of cancer from the result of (b-2);
(b-4) confirming the accuracy of predicting the prognosis of cancer by combining the representative values for each individual gene selected in (b-3); and
(b-5) selecting a combination of a representative value with the highest accuracy from the result of (b-4) as a cancer prognosis predicting marker; a system for predicting cancer prognosis.
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| KR1020190146627A KR102336311B1 (en) | 2019-11-15 | 2019-11-15 | Model for Predicting Cancer Prognosis using Deep learning |
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| KR1020190146627A KR102336311B1 (en) | 2019-11-15 | 2019-11-15 | Model for Predicting Cancer Prognosis using Deep learning |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| KR20210059325A KR20210059325A (en) | 2021-05-25 |
| KR102336311B1 true KR102336311B1 (en) | 2021-12-08 |
Family
ID=76145766
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020190146627A KR102336311B1 (en) | 2019-11-15 | 2019-11-15 | Model for Predicting Cancer Prognosis using Deep learning |
Country Status (1)
| Country | Link |
|---|---|
| KR (1) | KR102336311B1 (en) |
Families Citing this family (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR102706355B1 (en) * | 2021-12-01 | 2024-09-19 | 부산대학교 산학협력단 | System and Method for classificating Cancer type using deep learning according to the function of gene group |
| CN117334325B (en) * | 2023-09-26 | 2024-04-16 | 中山大学肿瘤防治中心(中山大学附属肿瘤医院、中山大学肿瘤研究所) | Application of LCAT in diagnosis, treatment and recurrence prediction of hepatocellular carcinoma |
Family Cites Families (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA2888125A1 (en) * | 2012-10-09 | 2014-04-17 | Five3 Genomics, Llc | Systems and methods for learning and identification of regulatory interactions in biological pathways |
| WO2018143540A1 (en) * | 2017-02-02 | 2018-08-09 | 사회복지법인 삼성생명공익재단 | Method, device, and program for predicting prognosis of stomach cancer by using artificial neural network |
-
2019
- 2019-11-15 KR KR1020190146627A patent/KR102336311B1/en active IP Right Grant
Also Published As
| Publication number | Publication date |
|---|---|
| KR20210059325A (en) | 2021-05-25 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| Boulesteix et al. | IPF‐LASSO: integrative L1‐penalized regression with penalty factors for prediction based on multi‐omics data | |
| US10339464B2 (en) | Systems and methods for generating biomarker signatures with integrated bias correction and class prediction | |
| KR102190299B1 (en) | Method, device and program for predicting the prognosis of gastric cancer using artificial neural networks | |
| CA2877430C (en) | Systems and methods for generating biomarker signatures with integrated dual ensemble and generalized simulated annealing techniques | |
| Le et al. | Statistical inference relief (STIR) feature selection | |
| Boulesteix et al. | Microarray-based classification and clinical predictors: on combined classifiers and additional predictive value | |
| Meinicke | UProC: tools for ultra-fast protein domain classification | |
| CN111933212B (en) | Clinical histology data processing method and device based on machine learning | |
| US20160364522A1 (en) | Systems and methods for classifying, prioritizing and interpreting genetic variants and therapies using a deep neural network | |
| US20020095260A1 (en) | Methods for efficiently mining broad data sets for biological markers | |
| US20180166170A1 (en) | Generalized computational framework and system for integrative prediction of biomarkers | |
| CN110993113B (en) | LncRNA-disease relation prediction method and system based on MF-SDAE | |
| Zararsiz et al. | voomDDA: discovery of diagnostic biomarkers and classification of RNA-seq data | |
| CN115997255A (en) | Molecular techniques for predicting bacterial phenotypic traits from genome | |
| KR102336311B1 (en) | Model for Predicting Cancer Prognosis using Deep learning | |
| Yu et al. | SANPolyA: a deep learning method for identifying Poly (A) signals | |
| CN115280415A (en) | Application of pathogenicity model and training thereof | |
| CN108427865B (en) | Method for predicting correlation between LncRNA and environmental factors | |
| Shibahara et al. | Deep learning generates custom-made logistic regression models for explaining how breast cancer subtypes are classified | |
| Lagani et al. | Structure-based variable selection for survival data | |
| Gross et al. | A selective approach to internal inference | |
| CN115565610A (en) | Method and system for establishing recurrence transfer analysis model based on multiple sets of mathematical data | |
| US11107555B2 (en) | Methods and systems for identifying a causal link | |
| JP2023547571A (en) | Drug optimization through active learning | |
| Kurz et al. | Isolating cost drivers in interstitial lung disease treatment using nonparametric Bayesian methods |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| E902 | Notification of reason for refusal | ||
| E701 | Decision to grant or registration of patent right |