광분해효소
Photolyase| 크립토크롬/포토화효소, C단자, FAD바인딩 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
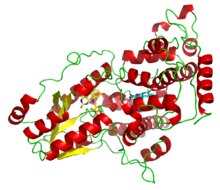 FADH−(노란색)와 8-HDF(시안)의 두 가지 광수확 보조인자를 나타내는 아나시스티스니둘란스산 데자플라빈 광분해효소. | |||||||||||
| 식별자 | |||||||||||
| 기호. | FAD_바인딩_7 | ||||||||||
| 팜 | PF03441 | ||||||||||
| 인터프로 | IPR005101 | ||||||||||
| 프로 사이트 | PDOC00331 | ||||||||||
| SCOP2 | 1qnf / SCOPe / SUPFAM | ||||||||||
| |||||||||||
| 디옥시리보디피리미딘광분해효소(CPD) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
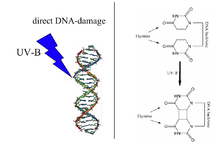 자외선 유도 티민-티민 시클로부탄 이합체(오른쪽)는 DNA 광분해효소에 의해 복구되는 DNA 손상의 일종이다.참고: 구조에는 5-메틸기가 없기 때문에 위의 다이어그램은 티민으로 잘못 표시되어 있습니다. | |||||||||
| 식별자 | |||||||||
| EC 번호 | 4.1.99.3 | ||||||||
| CAS 번호 | 37290-70-3 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
광분해효소(EC 4.1.99.3)는 자외선에 노출되어 발생하는 손상을 복구하는 DNA 복구 효소이다.이러한 효소는 자체 활성화와[1] 실제 DNA [2]복구를 위해 가시광선(스펙트럼의 보라색/파란색 끝)을 필요로 한다.광분해효소를 포함한 DNA 복구 메커니즘은 광재활성화라고 불립니다.주로 피리미딘 이합체를 피리미딘 염기의 정상적인 쌍으로 변환합니다.
기능.
광분해효소는 상보적인 DNA 가닥을 결합하고 동일한 DNA 가닥에 있는 티민 염기 또는 시토신 염기쌍이 공유 결합할 때 발생하는 특정 유형의 피리미딘 이합체를 분해합니다.이합체의 결합 길이는 복제 및 [3]전사를 위한 잘못된 템플릿을 생성하는 일반 B-DNA 구조의 결합 길이보다 짧습니다.보다 일반적인 공유 결합은 사이클로부탄 교량의 형성과 관련이 있다.광분해효소는 이러한 병변에 높은 친화력을 가지며 가역적으로 결합하고 원래 염기로 변환합니다.
진화
포토리아제는 박테리아에서 곰팡이, 식물[4], [5]동물에 이르기까지 많은 종에 존재하고 기능하는 계통학적으로 오래된 효소이다.광분해효소는 식물의 자외선에 의한 손상을 복구하는 데 특히 중요하다.광분해효소 메커니즘은 많은 크립토크롬을 [6]보유하고 있음에도 불구하고 덜 효율적인 뉴클레오티드 제거 복구 메커니즘에 의존하는 인간과 다른 태반 포유류에서는 더 이상 작동하지 않습니다.
광분해효소는 플라보단백질이며 2개의 광수확 보조인자를 포함한다.많은 광분해효소들은 두 번째 보조 인자와 결합하는 N-말단 도메인을 가지고 있다.모든 광분해효소는 2전자 환원− FADH를 포함하고 있으며, 2차 보조인자에 기초하여 2개의 주요 클래스로 분류되며, 이는 엽산 광분해효소의 프테린 메테닐테트라히드로폴레이트(MTHF) 또는 디자플라빈 8-히드록시-7, 8-디데아젤-5-데아자린(lav-lavin-8) 중 하나일 수 있다.촉매 활성에는 FAD만 필요하지만, 두 번째 보조 인자는 저조도 조건에서 반응 속도를 크게 가속화한다.이 효소는 환원된− 플라빈 FADH가 빛 에너지에 의해 활성화되는 전자 전달에 의해 작용하고 [7]피리미딘 이합체를 파괴하는 전자 공여체 역할을 한다.
배열 유사성에 따라 DNA 광분해효소는 몇 가지 [8][9]클래스로 분류할 수 있습니다.
|
- Class 1 CPD 광분해효소는 그램 음성균과 그램 양성균인 할로박테륨 할로박테륨 할로비움으로부터 시클로부탄 피리미딘 이합체(CPD) 병변을 처리하는 효소이다.
- Class 2 CPD 광분해효소는 CPD 병변도 처리한다.그들은 탈레 크레스 아라비도시스 탈리아나와 쌀과 같은 식물에서 발견됩니다.
- 식물과 균류 크립토크롬은 1등급 CPD와 유사하다.청색 빛에 의해 유도되는 유전자 발현과 일주기 리듬의 변조를 매개하는 청색 빛 감광체입니다.
- 클래스 3 CPD 분해효소는 식물 크립토크롬과 자매그룹을 이루며, 크립토크롬은 클래스 1 CPD와 자매그룹이다.
- Cry-DASH 그룹은 단일 가닥 DNA에 매우 특이적인 CPD 분해효소입니다.멤버는 Vibrio cholerae, Xenopus laevis의 X1Cry, Arabidopsis Thaliana의 [5]AtCry3입니다.DASH는 처음에 Drosophila, Arabidopsis, Synechocystis, Human의 이름을 따왔으며, 4개의 분류군은 처음에 이 리아제 과를 옮기는 것으로 생각되었다.그 후 분류가 변경되었습니다.그들의 이름에서 "Cry" 부분은 그들이 크립토크롬이라는 [8]초기 추정 때문이었다.
- 진핵생물(6-4) DNA 광분해효소는 생체 리듬을 조절하는 동물 크립토크롬을 가진 그룹을 형성합니다.그들은 드로소필라와 인간을 포함한 다양한 종에서 발견된다.크립토크롬은 그들만의 세부적인 [9]그룹화를 가지고 있다.
- FeS-BCP 그룹으로 알려진 박테리아 6-4 분해효소(InterPro: IPR007357)는 모든 광분해효소에 대해 자체적인 분해 그룹을 형성합니다.
CPD의 비클래스2 브랜치는 PRINTS(PR00147) 등의 일부 시스템에서는 클래스1로 그룹화되는 경향이 있습니다.소규모 그룹의 구성원들은 동의하지만 방법론의 차이로 인해 계통 발생이 저자마다 크게 다를 수 있으며, 모든 것(예비 FeS-BCP)을 두 가지 등급으로 [9]분류하려고 하는 저자들과 혼동할 수 있다.크립토크롬은 DNA 복구 활성을 잃고 대신 일주기 [8][9]리듬을 조절하는 광분해효소를 포함한 다계통군을 형성합니다.
어플
HeLa 세포에 남조류 Anacystis nidulans의 광분해효소를 첨가함으로써 UVB [10]노출로 인한 DNA 손상을 부분적으로 줄였다.
이 영역을 포함하는 인간 단백질
명명법
이 효소 클래스의 계통명은 디옥시리보시클로부타디피리미딘필리미딘분해효소이다.일반적으로 사용되는 다른 명칭으로는 광활성화효소, DNA 광분해효소, DNA 시클로부탄 디피리미딘 광분해효소, DNA 광분해효소, 디옥시리보핵산 광분해효소, 디옥시리보디피리미딘 광분해효소, 광분해효소, PRE, PrB 광분해효소, 디리보핵산 시클로부티클라아제 등이 있다.올리아제(비감수성), 디옥시리보핵산 피리미딘 이합체(비감수성)이다.이 효소는 리아제 계열, 특히 탄소-탄소 리아제 "포착" 등급에 속합니다.
레퍼런스
- ^ Yamamoto J, Shimizu K, Kanda T, Hosokawa Y, Iwai S, Plaza P, Müller P (October 2017). "Loss of Fourth Electron-Transferring Tryptophan in Animal (6-4) Photolyase Impairs DNA Repair Activity in Bacterial Cells". Biochemistry. 56 (40): 5356–5364. doi:10.1021/acs.biochem.7b00366. PMID 28880077.
- ^ Thiagarajan V, Byrdin M, Eker AP, Müller P, Brettel K (June 2011). "Kinetics of cyclobutane thymine dimer splitting by DNA photolyase directly monitored in the UV". Proceedings of the National Academy of Sciences of the United States of America. 108 (23): 9402–7. Bibcode:2011PNAS..108.9402T. doi:10.1073/pnas.1101026108. PMC 3111307. PMID 21606324.
- ^ Garrett RH, Grisham CM (2010). Biochemistry. Brooks/Cole, Cengage Learning. ISBN 978-0-495-10935-8. OCLC 984382855.
- ^ Teranishi, M., Nakamura, K., Morioka, H.,Yamamoto, K. and Hidema, J. (2008). "The native cyclobutane pyrimidine dimer photolyase of rice is phosphorylated". Plant Physiology. 146 (4): 1941–1951. doi:10.1104/pp.107.110189. PMC 2287361. PMID 18235036.
- ^ a b Selby CP, Sancar A (November 2006). "A cryptochrome/photolyase class of enzymes with single-stranded DNA-specific photolyase activity". Proceedings of the National Academy of Sciences of the United States of America. 103 (47): 17696–700. Bibcode:2006PNAS..10317696S. doi:10.1073/pnas.0607993103. PMC 1621107. PMID 17062752.
- ^ Lucas-Lledó JI, Lynch M (May 2009). "Evolution of mutation rates: phylogenomic analysis of the photolyase/cryptochrome family". Molecular Biology and Evolution. 26 (5): 1143–53. doi:10.1093/molbev/msp029. PMC 2668831. PMID 19228922.
- ^ Sancar A (June 2003). "Structure and function of DNA photolyase and cryptochrome blue-light photoreceptors". Chemical Reviews. 103 (6): 2203–37. doi:10.1021/cr0204348. PMID 12797829.
- ^ a b c d Scheerer P, Zhang F, Kalms J, von Stetten D, Krauß N, Oberpichler I, Lamparter T (May 2015). "The class III cyclobutane pyrimidine dimer photolyase structure reveals a new antenna chromophore binding site and alternative photoreduction pathways". The Journal of Biological Chemistry. 290 (18): 11504–14. doi:10.1074/jbc.M115.637868. PMC 4416854. PMID 25784552.
- ^ a b c d Rivera AS, Ozturk N, Fahey B, Plachetzki DC, Degnan BM, Sancar A, Oakley TH (April 2012). "Blue-light-receptive cryptochrome is expressed in a sponge eye lacking neurons and opsin". The Journal of Experimental Biology. 215 (Pt 8): 1278–86. doi:10.1242/jeb.067140. PMC 3309880. PMID 22442365.
- ^ Kulms D, Pöppelmann B, Yarosh D, Luger TA, Krutmann J, Schwarz T (July 1999). "Nuclear and cell membrane effects contribute independently to the induction of apoptosis in human cells exposed to UVB radiation". Proceedings of the National Academy of Sciences of the United States of America. 96 (14): 7974–9. Bibcode:1999PNAS...96.7974K. doi:10.1073/pnas.96.14.7974. PMC 22172. PMID 10393932.
추가 정보
- Eker AP, Fichtinger-Schepman AM (1975). "Studies on a DNA photoreactivating enzyme from Streptomyces griseus II. Purification of the enzyme". Biochim. Biophys. Acta. 378 (1): 54–63. doi:10.1016/0005-2787(75)90136-7. PMID 804322.
- Sancar GB, Smith FW, Reid R, Payne G, Levy M, Sancar A (1987). "Action mechanism of Escherichia coli DNA photolyase. I. Formation of the enzyme-substrate complex". J. Biol. Chem. 262 (1): 478–85. doi:10.1016/S0021-9258(19)75952-3. PMID 3539939.
- Setlow JK, Bollum FJ (1968). "The minimum size of the substrate for yeast photoreactivating enzyme". Biochim. Biophys. Acta. 157 (2): 233–7. doi:10.1016/0005-2787(68)90077-4. PMID 5649902.
외부 링크
 Wikimedia Commons 포토리아제 관련 미디어
Wikimedia Commons 포토리아제 관련 미디어


