신할라어에 관한 유전학적 연구
Genetic studies on Sinhalese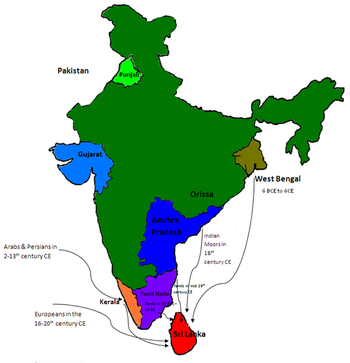
신할라족에 대한 유전 연구는 신할라족의 기원을 조사하는 집단 유전학의 일부입니다.
모든 연구들은 싱할라인과 벵갈인과 남인도 타밀인 사이에 유의한 관계가 있다는 것과 스리랑카 타밀인과 싱할라인 사이에 유의한 유전적 관계가 있다는 것에 동의합니다. 이는 신할리족과 벵골족, 타밀족, 케랄라족 자원봉사자 사이의 유전적 거리 차이가 낮게 나타난 유전적 거리 연구에서도 뒷받침됩니다.[1]
벵갈리스와의 관계

Mastana S(2007)가 싱할라어, 타밀어, 벵골어, 구자라트어(Patel), 펀자브어를 부모 집단으로 사용하여 알루 다형성을 분석한 결과 다음과 같은 유전적 기여 비율을 발견했습니다.[2]
| 통계적 방법 | 벵골어 | 타밀어 | 노스웨스턴 |
|---|---|---|---|
| 점 견적 | 57.49% | 42.5% | - |
| 최대우도법 | 88.07% | - | - |
| 타밀어, 벵골어, 북서부를 부모 인구로 사용 | 50-66% | 11-30% | 20-23% |
| 부모인구 | 벵골어 | 타밀어 | 구자라티 | 펀자브 |
|---|---|---|---|---|
| 타밀어와 벵골어를 부모 인구로 사용하기 | 70.03% | 29.97% | - | |
| 타밀어, 벵골어, 구자라트어를 부모 인구로 사용합니다. | 71.82% | 16.38% | 11.82% | |
| 벵골어, 구자라트어, 펀자브어를 부모 인구로 사용합니다. | 82.09% | - | 15.39% | 2.52% |
Perera et al. (2021)에 의한 X 염색체 STR의 분석 결과, 신할라인(스리랑카 타밀인 및 스리랑카 무슬림뿐만 아니라)은 스리랑카의 인도 타밀인보다 벵골인과 더 밀접한 관련이 있는 것으로 나타났습니다.[3]
커크(Kirk, 1976)의 유전적 거리 분석 결과, 신할라족은 구자라트족이나 판잡족보다 벵골족에 더 가까운 것으로 나타났습니다.[4]
D1S80 대립유전자 빈도(유전자 지문 채취에 인기 있는 대립유전자)도 신할리스와 벵갈리스 사이에 유사하여 두 그룹이 밀접한 관련이 있음을 시사합니다.[5]
신할라인은 또한 대립유전자 MTHFR 677T(13%)의 빈도가 웨스트 벵갈리스(17%)[6][7]와 유사합니다.
Sinhales 사람들 중에서 가장 흔한 y-DNA Haplogroup은 Haplogroup R2 (38.5%)와 Haplogroup R1a (12.8%)를 포함합니다. 하플로그룹 R2는 또한 서벵골주(22.6%)와 텔루구스(20%) 사이에서 중요한 존재이며,[8] 하플로그룹 R1a는 북인도의 더 높은 카스트 집단에서 정점을 찍습니다.[9] Sinhalese 중에서 Haplogroup R2 및 Haplogroup R1a 존재의 총합은 50%[9] 이상입니다.
인도 타밀과의 관계

Kshatriya(1995)의 유전자 혼합 연구에 따르면 싱할라인들은 벵골인들(25.41%+/- 0.51)에 비해 인도 타밀인들(69.86%+/- 0.61)의 기여율이 더 높은 것으로 나타났습니다.[10]
Roychoudhury AK et al. (1985)의 유전적 거리 분석은 싱할라인들이 벵골인들보다 남인도와 서인도의 개체군과 더 밀접한 관련이 있음을 시사했습니다.[11]
커크(Kirk, 1976)의 유전적 거리 분석에 따르면, 신할라인들은 구자라트족이나 판잡족보다 남인도의 타밀족과 케랄라족에 더 가깝다고 합니다.[4]

싱 등이 이전 연구보다 높은 해상도 마커를 사용하여 수행한 2023년 연구에서는 북인도보다 남인도에서 신할라인으로 가는 유전자 흐름이 더 높았으며, 신할라인은 연구된 다른 인도 개체군에 비해 타밀과 혈통별로 가장 높은 동일성을 공유했습니다. 이 연구는 또한 타밀 및 기타 남인도 인구가 부족한 인도 북서부 마라타족과의 공유가 증가했다는 것을 발견했는데, 이는 신할라족 인구에 대한 북인도의 소속 흔적과 일치합니다. 총 Sinhales 표본 크기는 9개체를 사용하였습니다.[12]
북서인도인과의 관계
Mastana S(2007)의 Alu 다형성 분석 결과, 노스웨스트 인디언의 기여가 20-23%[13]로 미미했습니다.
Perera et al., (2011)에 의한 X 염색체 STR의 분석 결과, 스리랑카의 신할라족, 스리랑카 타밀족, 무어족 및 인도 타밀족은 북서 인도의 빌족(Bhil, 원주민 집단)과 친연을 공유하는 것으로 나타났습니다.[14]
스리랑카의 다른 주요 민족과의 관계
싱할라와 스리랑카 타밀 인구에서 FUT2 유전자의 유전적 변이를 조사한 연구는 두 민족 모두에서 유사한 유전적 배경을 발견했으며 다른 이웃 아시아 인구 집단에서는 유전적 흐름이 거의 없었습니다.[15] 연구들은 또한 스리랑카의 신할라족과 다른 민족들 사이의 혈액 그룹, 혈액 유전자 표지자(Saha, 1988) 및 단일 뉴클레오티드 다형성에 있어서 유의미한 차이를 발견하지 못했습니다.[16][17][18] 또 다른 연구에서는 "스리랑카의 주요 민족들 사이에 유의미한 유전적 변이가 없다"는 사실도 발견했습니다.[19] 이것은 인도 타밀, 신할라어, 스리랑카 타밀 및 스리랑카 무어 개체군에서 대립유전자 MTHFR 677T, F220210A & F5 1691A의 매우 유사한 빈도를 발견한 연구에 의해 더욱 뒷받침됩니다.[7]
동아시아와 동남아시아와의 관계
유전학적 연구에 따르면, 신할라인들은 민족적으로 다양하고 이질적인 티베트-부르만 민족과 오스트리아-아시아 민족 등 동아시아와 동남아시아의 이웃 인구로부터 어느 정도 유전적 흐름을 전달받았는데,[20] 이는 그들이 동북 인도와 밀접한 유전적 연관성 때문입니다.[21][22][23] 1985년 Roychoudhury AK와 Nei M이 수행한 연구에 따르면 유전적 거리의 값은 과거에 유전자 교환으로 인해 신할라인들이 몽골로이드 개체군에 약간 더 가깝다는 것을 보여주었습니다.[11][24] 스리랑카 하악 어금니의 근 및 관 형태 비교에 있어서, 몽골로이드 개체군에서 더 멀리 떨어져 있음을 보여주었습니다.[25] 동아시아 인구에서 발견되는 하플로그룹 중 동아시아 mtDNA 하플로그룹의 빈도가 낮은 하플로그룹 중 G는 스리랑카 민족의 주요 mtDNA 하플로그룹인 M 하플로그룹과 함께 하플로그룹 D와 함께 스리랑카 인구에서 발견되었습니다.[26] Y-DNA와 관련하여, 하플로그룹 C-M130은 스리랑카에서 낮은 빈도에서 중간 정도의 빈도로 발견됩니다.[27]
신할라인 중 면역글로불린의 유전자 표지자는 중국 남부 윈난성과 광시성을 기원으로 하는 afb1b3의 높은 빈도를 보입니다.[28] 오디아스, 특정 네팔, 인도 북동부, 한족 남부, 동남아시아, 태평양 제도의 특정 오스트로네시아 개체군 사이에서도 높은 빈도로 발견됩니다.[28] 더 낮은 빈도로, ab3st는 신할라인들 사이에서도 발견되며, 일반적으로 한족 북부, 티베트, 몽골, 한국 및 일본 인구 사이에서 더 높은 빈도로 발견됩니다.[28] 동아시아와 북미 원주민 사이에서 흔한 트랜스페린 TF*Dchi 대립유전자도 신할라인들 사이에서 발견됩니다.[11] HumDN1*4와 HumDN1*5는 신할레족 사이에서 우세한 DNase I 유전자이며 또한 중국 남부 민족과 네팔의 타망족 사이에서 우세한 유전자입니다.[29] N. Saha가 1988년에 수행한 연구에 따르면, 신할라인들 사이의 높은 GC*1F 및 낮은 GC*1S 빈도는 중국인, 일본인, 한국인, 태국인, 말레이시아인, 베트남인, 라오스인 및 티베트인과 유사합니다.[30] 1998년 D.E.가 실시한 연구. 호키는 신할라인들의 치아 형태가 동인도와 북동인도의 오스트리아-아시아 인구의 치아 형태와 밀접한 관련이 있다는 것을 보여주었습니다.[20] 동남아시아에서 시작되어 인구 사이에서 유행하는 정상적인 헤모글로빈의 변종인 헤모글로빈 E도 신할라인들 사이에서 흔하며 스리랑카에서는 최대 40%까지 도달할 수 있습니다.[31]
부계선
Y-DNA of Sinhalese
Sinhalese에서 발견되는 가장 흔한 Y-염색체 DNA 하플로그룹은 Haplogroup R2, Haplogroup R1a 및 Haplogroup L입니다. 하플로그룹 R2는 서벵골주(22.6%)와 텔루구스(20%)[8] 사이에서 상당한 존재감을 보이며, 하플로그룹 R1a는 북인도의 더 높은 카스트 집단에서 정점을 찍습니다.[9] 하플로그룹 L은 카르나타카 해안과 케랄라 북부의 코라가 사람들 사이에서 68%로 최고치를 기록하고 있습니다.[32] 또한 북인도의 자트족 중 가장 큰 단일 남성 계통(36.8%)이며, 구자르족의 잠무족과 카슈미르족 중 16.33%에서 발견됩니다.
| 인구. | n | C | E | F | G | H | I | J | K | L | N | O | P | Q | R | R1 | R1a | R1b | R2 | T | 다른이들 | 언급 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 신할라어 | 39 | 0 | 0 | 10.3% | 0 | 10.3% | 0 | 10.3% | 0 | 18% | 0 | 0 | 0 | 0 | 0 | 0 | 12.8% | 0 | 38.5% | 0 | 키빌트[9] 2003 |
머더 라인
신할라어의 MtDNA
Ranweera et al. (2014)은 싱할라어에서 가장 흔한 mtDNA 하플로그룹은 하플로그룹 M, 하플로그룹 U (U7a) 및 하플로그룹 R (R30b) 및 하플로그룹 G (G3a1'2)인 것을 발견했습니다.[33][34]
하플로그룹 M은 약 60,000년 전 남부 아시아 해안선을 따라 아라비아와 인도를 가로지르는 남부 해안 경로를 따라 호주에 도착한 현대 인류의 분포를 나타냅니다.[35]
하플로그룹 U7은 서유라시아 특유의 mtDNA 하플로그룹으로 여겨지며, 약 30,000년 전 흑해 지역에서 유래된 것으로 추정됩니다. 남아시아에서는 U7이 구자라트에서 약 12% 발생하는 반면, 인도 전체에서는 약 2%, 파키스탄에서는 5%의 빈도를 유지합니다. 스리랑카의 베다족은 13.33%로 가장 높은 빈도를 기록하고 있습니다. 대규모 이민이 이 미토콘드리아 하플로그룹들을 인도로 운반한 것으로 추측됩니다.[36]
Chaubey는 "스리랑카의 상당한 수의 모계 혈통이 인도, 더 정확하게는 인도의 남부 지역과 공유됩니다."라고 말합니다.[37]
참고문헌
- ^ Kirk, R. L. (July 1976). "The legend of Prince Vijaya — a study of Sinhalese origins". American Journal of Physical Anthropology. 45 (1): 91–99. doi:10.1002/ajpa.1330450112.
- ^ Mastana S (2007). "Molecular anthropology: population and forensic genetic applications" (PDF). Anthropologist Special. 3: 373–383.
- ^ 페레라, N., Galhena, G. and Ranawaka, G., 2021. X-염색체 STR 기반 스리랑카 민족의 유전적 다형성 및 인구통계학적 역사 및 세계 인구와의 관계. 과학 보고서, 11(1), 페이지 1-12.
- ^ a b Kirk RL (July 1976). "The legend of Prince Vijaya – a study of Sinhalese origins". American Journal of Physical Anthropology. 45 (1): 91–99. doi:10.1002/ajpa.1330450112.
- ^ Surinder Singh Papiha (1999). 게놈 다양성: 인간 집단 유전학에서의 응용. 런던: 스프링어. 7.
- ^ Mukhopadhyay K, Dutta S, Das Bhomik A (January 2007). "MTHFR gene polymorphisms analyzed in population from Kolkata, West Bengal". Indian Journal of Human Genetics. 13 (1): 38. doi:10.4103/0971-6866.32035. PMC 3168154. PMID 21957342.
- ^ a b Dissanayake VH, Weerasekera LY, Gammulla CG, Jayasekara RW (October 2009). "Prevalence of genetic thrombophilic polymorphisms in the Sri Lankan population--implications for association study design and clinical genetic testing services". Experimental and Molecular Pathology. 87 (2): 159–62. doi:10.1016/j.yexmp.2009.07.002. PMID 19591822.
- ^ a b Poznik GD, Xue Y, Mendez FL, Willems TF, Massaia A, Wilson Sayres MA, et al. (June 2016). "Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences". Nature Genetics. 48 (6): 593–9. doi:10.1038/ng.3559. PMC 4884158. PMID 27111036.
- ^ a b c d Kivisild T, Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (February 2003). "The genetic heritage of the earliest settlers persists both in Indian tribal and caste populations". American Journal of Human Genetics. 72 (2): 313–332. doi:10.1086/346068. PMC 379225. PMID 12536373.
- ^ Kshatriya GK (December 1995). "Genetic affinities of Sri Lankan populations". Human Biology. American Association of Anthropological Genetics. 67 (6): 843–66. PMID 8543296.
- ^ a b c Roychoudhury AK, Nei M (1985). "Genetic relationships between Indians and their neighboring populations". Human Heredity. 35 (4): 201–6. doi:10.1159/000153545. PMID 4029959.
- ^ Prajjval Pratap Singh, Sachin Kumar, Nagarjuna Pasupuletti, Niraj Rai, Gyaneshwer Chaubey, R. Ranasinghe, "ś ī 라 ṅ카의 주요 민족인 싱할라어의 인구 역사 재구성", iScience, 2023년 8월 31일, DOI: https://doi.org/10.1016/j.isci.2023.107797 .
- ^ Mastana S (2007). "Molecular anthropology: population and forensic genetic applications" (PDF). Anthropologist Special. 3: 373–383.
- ^ 페레라, N., Galhena, G. and Ranawaka, G., 2021. X-염색체 STR 기반 스리랑카 민족의 유전적 다형성 및 인구통계학적 역사 및 세계 인구와의 관계. 과학 보고서, 11(1), 페이지 1-12.
- ^ Soejima M, Koda Y (December 2005). "Denaturing high-performance liquid chromatography-based genotyping and genetic variation of FUT2 in Sri Lanka". Transfusion. 45 (12): 1934–9. doi:10.1111/j.1537-2995.2005.00651.x. PMID 16371047. S2CID 10401001.
- ^ Saha N (1988). "Blood genetic markers in Sri Lankan populations--reappraisal of the legend of Prince Vijaya". American Journal of Physical Anthropology. 76 (2): 217–25. doi:10.1002/ajpa.1330760210. PMID 3166342.
- ^ Roberts DF, Creen CK, Abeyaratne KP (1972). "Blood Groups of the Sinhalese". Man. 7 (1): 122–127. doi:10.2307/2799860. JSTOR 2799860.
- ^ Dissanayake VH, Giles V, Jayasekara RW, Seneviratne HR, Kalsheker N, Broughton Pipkin F, Morgan L (April 2009). "A study of three candidate genes for pre-eclampsia in a Sinhalese population from Sri Lanka". The Journal of Obstetrics and Gynaecology Research. 35 (2): 234–42. doi:10.1111/j.1447-0756.2008.00926.x. PMID 19708171. S2CID 24958292.
- ^ Illeperuma RJ, Mohotti SN, De Silva TM, Fernandopulle ND, Ratnasooriya WD (June 2009). "Genetic profile of 11 autosomal STR loci among the four major ethnic groups in Sri Lanka". Forensic Science International. Genetics. 3 (3): e105-6. doi:10.1016/j.fsigen.2008.10.002. PMID 19414153.
- ^ a b Petraglia MD, Allchin B (2007). The Evolution and History of Human Populations in South Asia: Inter-disciplinary Studies in Archaeology, Biological Anthropology, Linguistics and Genetics. Springer Science & Business Media. ISBN 978-1-4020-5562-1.[페이지 필요]
- ^ Soejima M, Koda Y (January 2007). "Population differences of two coding SNPs in pigmentation-related genes SLC24A5 and SLC45A2". International Journal of Legal Medicine. 121 (1): 36–9. doi:10.1007/s00414-006-0112-z. PMID 16847698. S2CID 11192076.
- ^ Kivisild T, Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (February 2003). "The genetic heritage of the earliest settlers persists both in Indian tribal and caste populations". American Journal of Human Genetics. 72 (2): 313–32. doi:10.1086/346068. PMC 379225. PMID 12536373.
- ^ Sengupta S, Zhivotovsky LA, King R, Mehdi SQ, Edmonds CA, Chow CE, et al. (February 2006). "Polarity and temporality of high-resolution y-chromosome distributions in India identify both indigenous and exogenous expansions and reveal minor genetic influence of Central Asian pastoralists". American Journal of Human Genetics. 78 (2): 202–21. doi:10.1086/499411. PMC 1380230. PMID 16400607.
- ^ Bhasin MK (4 September 2017). "Morphology to Molecular Anthropology: Castes and Tribes of India". International Journal of Human Genetics. 9 (3–4): 145–230. doi:10.1080/09723757.2009.11886070. S2CID 53353581.
- ^ Peiris R, Takahashi M, Sasaki K, Kanazawa E (July 2007). "Root and canal morphology of permanent mandibular molars in a Sri Lankan population". Odontology. 95 (1): 16–23. doi:10.1007/s10266-007-0074-8. PMID 17660977. S2CID 8504778.
- ^ Ranaweera L, Kaewsutthi S, Win Tun A, Boonyarit H, Poolsuwan S, Lertrit P (January 2014). "Mitochondrial DNA history of Sri Lankan ethnic people: their relations within the island and with the Indian subcontinental populations". Journal of Human Genetics. 59 (1): 28–36. doi:10.1038/jhg.2013.112. PMID 24196378.
- ^ "Y-DNA Haplogroup C and its Subclades - 2017". International Society of Genetic Genealogy. Retrieved 31 May 2017.
- ^ a b c Matsumoto H (2009). "The origin of the Japanese race based on genetic markers of immunoglobulin G". Proceedings of the Japan Academy. Series B, Physical and Biological Sciences. 85 (2): 69–82. Bibcode:2009PJAB...85...69M. doi:10.2183/pjab.85.69. PMC 3524296. PMID 19212099.
- ^ Fujihara J, Yasuda T, Iida R, Ueki M, Sano R, Kominato Y, et al. (July 2015). "Global analysis of genetic variations in a 56-bp variable number of tandem repeat polymorphisms within the human deoxyribonuclease I gene". Legal Medicine. 17 (4): 283–6. doi:10.1016/j.legalmed.2015.01.005. PMID 25771153.
- ^ Malhotra R (1992). Anthropology of Development: Commemoration Volume in the Honour of Professor I.P. Singh. Mittal Publications. ISBN 978-81-7099-328-5.[페이지 필요]
- ^ Kumar D (2012). Genetic Disorders of the Indian Subcontinent. Springer Science & Business Media. ISBN 978-1-4020-2231-9.[페이지 필요]
- ^ Sengupta, Sanghamitra; Zhivotovsky, Lev A.; King, Roy; Mehdi, S. Q.; Edmonds, Christopher A.; Chow, Cheryl-Emiliane T.; Lin, Alice A.; Mitra, Mitashree; Sil, Samir K.; Ramesh, A.; Usha Rani, M. V.; Thakur, Chitra M.; Cavalli-Sforza, L. Luca; Majumder, Partha P.; Underhill, Peter A. (2006). "Polarity and Temporality of High-Resolution Y-Chromosome Distributions in India Identify Both Indigenous and Exogenous Expansions and Reveal Minor Genetic Influence of Central Asian Pastoralists". American Journal of Human Genetics. 78 (2): 202–221. doi:10.1086/499411. ISSN 0002-9297. PMC 1380230. PMID 16400607.
- ^ Ranaweera, Lanka; Kaewsutthi, Supannee; Win Tun, Aung; Boonyarit, Hathaichanoke; Poolsuwan, Samerchai; Lertrit, Patcharee (January 2014). "Mitochondrial DNA history of Sri Lankan ethnic people: their relations within the island and with the Indian subcontinental populations". Journal of Human Genetics. 59 (1): 28–36. doi:10.1038/jhg.2013.112. PMID 24196378.
- ^ Ranasinghe, Ruwandi; Tennekoon, Kamani H.; Karunanayake, Eric H.; Lembring, Maria; Allen, Marie (November 2015). "A study of genetic polymorphisms in mitochondrial DNA hypervariable regions I and II of the five major ethnic groups and Vedda population in Sri Lanka". Legal Medicine. 17 (6): 539–546. doi:10.1016/j.legalmed.2015.05.007. PMID 26065620.
- ^ Marrero, P.; Abu-Amero, K. K.; Larruga, J. M.; Cabrera, V. M. (2016). "Carriers of human mitochondrial DNA macrohaplogroup M colonized India from southeastern Asia". BMC Evolutionary Biology. 16 (1): 246. doi:10.1186/s12862-016-0816-8. PMC 5105315. PMID 27832758.
- ^ Metspalu M, Kivisild T, Metspalu E, Parik J, Hudjashov G, Kaldma K, et al. (August 2004). "Most of the extant mtDNA boundaries in south and southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans". BMC Genetics. 5: 26. doi:10.1186/1471-2156-5-26. PMC 516768. PMID 15339343.
- ^ 차우베이, G. 언어 분리 및 그들의 유전적 정체성: 스리랑카 민족의 미토콘드리아 DNA 역사에 대한 논평: 섬 내 및 인도 아대륙 인구와의 관계. J Hum Genet 59, 61–63 (2014). https://doi.org/10.1038/jhg.2013.122


