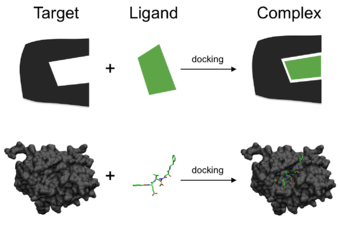
도킹(분자)
Docking (molecular)| 도킹 용어집 |
|---|
|
| 편집하다 |
분자모델링 분야에서 도킹은 배위자와 타깃이 결합해 안정된 [1]착체를 형성할 때 1분자의 바람직한 방향을 1초로 예측하는 방법이다.우선 배향에 대한 지식은 예를 들어 스코어링 함수를 사용하여 두 분자 간의 연관성 또는 결합 친화성의 강도를 예측하기 위해 사용될 수 있다.
단백질, 펩타이드, 핵산, 탄수화물, 지질과 같은 생물학적으로 관련된 분자 사이의 연관성은 신호 전달에서 중심적인 역할을 합니다.또한, 상호작용하는 두 파트너의 상대적 배향은 생성되는 신호 유형에 영향을 미칠 수 있다(예를 들어 작용 대 길항).따라서 도킹은 생성되는 신호의 강도와 유형을 예측하는 데 유용합니다.
분자 도킹은 구조 기반 약물 설계에서 가장 자주 사용되는 방법 중 하나로, 적절한 표적 결합 부위에 대한 작은 분자 리간드의 결합-조합을 예측할 수 있기 때문이다.결합 행동의 특성화는 의약품의 합리적인 설계와 기본적인 생화학적 [2][3]과정을 설명하는 데 중요한 역할을 한다.
문제의 정의
분자 도킹은 "잠금"을 여는 "키"의 올바른 상대 방향을 찾는 "잠금"의 문제라고 생각할 수 있습니다(잠금 표면의 키 구멍은 키를 삽입한 후 돌리는 방향 등).여기서, 단백질은 "잠금"으로 생각될 수 있고, 배위자는 "열쇠"로 생각될 수 있다.분자 도킹은 특정 단백질에 결합하는 배위자의 "최적합" 방향을 설명하는 최적화 문제로 정의될 수 있다.그러나 리간드와 단백질은 모두 유연하기 때문에 "락 앤 키"[4]보다 "핸드 인 글러브" 유추가 더 적절합니다.도킹 프로세스 중에 리간드와 단백질은 전체적인 '최적합'을 달성하기 위해 배위조정을 하며, 이러한 배위조정을 전체적인 결합을 초래하는 것을 '유도적합'[5]이라고 한다.
분자 도킹 연구는 분자 인식 과정을 계산적으로 시뮬레이션하는 데 초점을 맞춥니다.이는 단백질과 배위자 모두에 대한 최적화된 배향과 단백질과 배위자 사이의 상대적 배향을 달성하여 전체 시스템의 자유 에너지가 최소화되도록 하는 것을 목표로 한다.
도킹 어프로치
분자 도킹 커뮤니티에서는 두 가지 접근법이 특히 인기가 있습니다.하나의 접근법은 단백질과 배위자를 상보적인 [6][7][8]표면으로 설명하는 매칭 기술을 사용한다.두 번째 접근법은 배위자-단백질 쌍방향 상호작용 에너지가 [9]계산되는 실제 도킹 과정을 시뮬레이션한다.두 방법 모두 상당한 장점과 몇 가지 제한이 있습니다.이하에 개략을 나타냅니다.
형상보완성
기하학적 매칭/형상보완법은 단백질과 배위자를 도킹할 [10]수 있도록 하는 일련의 특징으로 설명한다.이러한 특징에는 분자 표면/보완 표면 기술자가 포함될 수 있습니다.이 경우 수용체의 분자표면은 용매접근성 표면적, 리간드의 분자표면은 일치하는 표면기술로 기술된다.두 표면 사이의 상호보완성은 표적과 배위자 분자를 도킹하는 상호보완적 자세를 찾는 데 도움이 될 수 있는 설명과 일치하는 형태에 해당합니다.또 다른 접근법은 주쇄 원자의 회전을 이용하여 단백질의 소수성 특징을 설명하는 것이다.또 다른 접근법은 푸리에 형상 기술자 [11][12][13]기술을 사용하는 것이다.형상보완성 기반 접근법은 일반적으로 빠르고 강력하지만, 최근 개발로 인해 리간드 유연성을 조사할 수 있지만 일반적으로 리간드/단백질 구성의 움직임이나 동적 변화를 정확하게 모델링할 수 없다.형상보완성 방법은 수천 개의 리간드를 몇 초 안에 빠르게 스캔할 수 있고 실제로 단백질의 활성 부위에서 결합할 수 있는지 여부를 알아낼 수 있으며, 보통 단백질-단백질 상호작용까지 확장 가능합니다.그들은 또한 최적의 결합을 찾기 위해 배위자의 기하학적 설명을 사용하기 때문에 약초 기반 접근법에 훨씬 더 잘 적응한다.
시뮬레이션
도킹 프로세스의 시뮬레이션은 매우 복잡합니다.이 접근법에서 단백질과 배위자는 물리적인 거리만큼 분리되며, 배위자는 배위 공간에서 일정 수의 "이동" 후 단백질의 활성 부위로 위치를 찾는다.이러한 움직임에는 비틀림 각도 회전을 포함한 리간드 구조의 내부 변경뿐만 아니라 번역 및 회전과 같은 강체 변환이 포함됩니다.이들 각각은 배위자의 배위공간에서 시스템의 총 에너지 비용을 유도한다.따라서 시스템의 총 에너지는 모든 이동 후에 계산됩니다.
도킹 시뮬레이션의 명백한 장점은 리간드의 유연성이 쉽게 통합된다는 것이지만, 형상보완성 기술은 리간드에 유연성을 통합하기 위해 기발한 방법을 사용해야 한다.또한, 그것은 더 정확하게 현실을 모델링하는 반면, 형상 보완 기법은 추상화에 가깝습니다.
시뮬레이션은 계산 비용이 많이 들기 때문에 대규모 에너지 환경을 탐색해야 합니다.그리드 기반 기술, 최적화 방법 및 향상된 컴퓨터 속도를 통해 도킹 시뮬레이션이 더욱 현실적이 되었습니다.
도킹 메커니즘
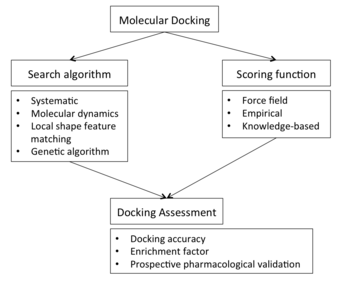
도킹 스크린을 실행하기 위한 첫 번째 요건은 관심 단백질 구조입니다.보통 구조는 X선 결정학, NMR 분광학 또는 크라이오 전자 현미경(cryo-EM)과 같은 생물물리학적 기술을 사용하여 결정되지만, 호몰로지 모델링 구조에서 파생될 수도 있습니다.이 단백질 구조와 잠재적 리간드의 데이터베이스는 도킹 프로그램에 대한 입력으로 기능합니다.도킹 프로그램의 성공은 검색 알고리즘과 스코어링 기능의 두 가지 구성요소에 달려 있습니다.
검색 알고리즘
이론상 탐색 공간은 배위자와 짝을 이룬 단백질의 가능한 모든 방향과 배치로 구성됩니다.그러나, 현재의 계산 자원으로는 탐색 공간을 철저히 탐색하는 것이 불가능하다. 이것은 각 분자의 가능한 모든 왜곡과 (분자는 동적이고 구조 상태의 앙상블에 존재한다) 그리고 상대적인 배위자의 가능한 모든 회전 및 변환 방향을 열거하는 것을 포함할 것이다.특정 입도 수준의 단백질사용 중인 대부분의 도킹 프로그램은 리간드의 전체 배위 공간(플렉시블 리간드)을 설명하고 유연한 단백질 수용체를 모델링하려는 몇 가지 시도를 합니다.쌍의 각 "스냅샷"을 포즈라고 합니다.
배위자 및 수용체에 다양한 배위 탐색 전략이 적용되었다.여기에는 다음이 포함됩니다.
- 회전 가능한 결합에 대한 체계적 또는 확률적 비틀림 탐색
- 분자 역학 시뮬레이션
- 새로운 낮은 에너지 구성을 "고정"하기 위한 유전적 알고리즘과 각 포즈의 점수가 다음 반복을 위해 개인을 선택하는 데 사용되는 피트니스 함수로 작용한다.
리간드 유연성
리셉터가 없는 경우 리간드의 배치가 생성될 수 있으며, 이후[14] 도킹 또는 배치는 리셉터 결합 [15]공동이 존재하는 경우 또는 프래그먼트 기반의 [16]도킹을 사용하여 모든 사면각의 완전한 회전 유연성으로 생성될 수 있다.힘장 에너지 [17]평가는 에너지적으로 합리적인 구성을 선택하기 위해 가장 자주 사용되지만 지식 기반 방법도 [18]사용되어 왔다.
펩타이드는 매우 유연하고 상대적으로 큰 분자이며, 따라서 펩타이드의 유연성을 모델링하는 것이 어려운 작업이다.단백질-펩타이드 [19]도킹 중 펩타이드의 유연성을 효율적으로 모델링할 수 있도록 많은 방법이 개발되었습니다.
수용체 유연성
지난 10년 동안 계산 용량이 급격히 증가하여 컴퓨터 지원 의약품 설계에서 보다 정교하고 계산 집약적인 방법을 사용할 수 있게 되었다.그러나 도킹 방법론에서 리셉터의 유연성을 다루는 것은 여전히 어려운 문제입니다.[20]이러한 어려움의 주된 이유는 이러한 종류의 계산에서 고려해야 할 많은 자유도 때문이다.그러나 경우에 따라서는 이를 무시하면 바인딩 포즈 [21]예측 측면에서 도킹 결과가 불량해질 수 있습니다.
수용체의 유연성을 [22]에뮬레이트하기 위해 다른 구성의 동일한 단백질에 대해 실험적으로 결정된 복수의 정적 구조가 종종 사용된다.대체적이지만 에너지적으로 합리적인 단백질 [23][24]배열을 생성하기 위해 결합강을 둘러싼 아미노산 측쇄의 로타머 라이브러리를 탐색할 수 있다.
스코어링 기능
도킹 프로그램은 단백질과의 충돌로 인해 즉시 거부될 수 있는 많은 잠재적 리간드 포즈를 생성합니다.나머지는 몇 가지 스코어링 함수를 사용하여 평가되며, 이 함수는 포즈가 입력으로 사용되며 포즈가 바람직한 바인딩 상호작용을 나타낼 가능성을 나타내는 숫자를 반환하고 한 배위자를 다른 배위자에 대해 순위를 매긴다.
대부분의 스코어링 함수는 결합 부위 내에서 포즈의 에너지를 추정하는 물리 기반 분자 역학력장이다.결합에 대한 다양한 기여는 가법 방정식으로 나타낼 수 있습니다.
구성 요소는 용매 효과, 단백질 및 배위자의 구조 변화, 단백질-배위자 상호작용에 의한 자유 에너지, 내부 회전, 배위자 및 수용체의 결합 에너지로 구성되어 진동 [25]모드의 변화에 따른 단일 복합 및 자유 에너지를 형성합니다.낮은(음수) 에너지는 안정적인 시스템을 나타내며, 따라서 결합 상호작용이 있을 수 있음을 나타냅니다.
대체 접근법은 알려진 주요 단백질 리간드 [26]상호작용에 기초한 제약조건 또는 단백질 리간드 구조의 대규모 데이터베이스(예: 단백질 데이터 뱅크)[27]에서 관찰된 상호작용에서 파생된 지식 기반 잠재력을 포함하기 위해 수정된 점수 매기기 기능을 사용한다.
단백질과 고친화성 리간드 사이의 복합체에는 X선 결정학의 많은 구조가 있지만, 저친화성 리간드는 후자의 복합체가 덜 안정적이고 따라서 결정화하기가 더 어려운 경향이 있기 때문에 상대적으로 적다.이 데이터로 훈련한 스코어링 함수는 높은 친화력 리간드를 올바르게 도킹할 수 있지만 결합하지 않는 리간드에 대해서도 그럴듯한 도킹 구성을 제공합니다.이것은 많은 양의 잘못된 양성 히트를 줍니다. 즉, 리간드가 시험관에 함께 놓였을 때 실제로 결합하지 않는 단백질과 결합할 것으로 예측됩니다.
잘못된 긍정의 수를 줄이는 한 가지 방법은 Generalized Born 또는 Poisson-Boltzmann [9]방법과 같이 (잠재적으로) 더 정확하지만 계산적으로 더 집약적인 기법을 사용하여 상위 점수 포즈의 에너지를 다시 계산하는 것이다.
도킹 평가
표본 추출과 채점 기능 간의 상호의존성은 새로운 화합물에 대한 그럴듯한 포즈 또는 결합 친화성을 예측하는 도킹 능력에 영향을 미친다.따라서 도킹 프로토콜의 예측 능력을 결정하기 위해서는 일반적으로 (실험 데이터를 이용할 수 있는 경우) 도킹 프로토콜의 평가가 필요하다.도킹 평가는 다음과 같은 다양한 전략을 사용하여 수행할 수 있습니다.
- 도킹 정밀도(DA) 계산
- 도킹 점수와 농축 계수(EF)[28]의 실험 응답 또는 결정 사이의 상관 관계
- 활성 부위의 이온 결합 부분과 이온 사이의 거리
- 유도 적합 모델의 존재
도킹 정밀도
도킹[29][30] 정밀도는 실험적으로 [31]관찰된 리간드의 올바른 자세를 예측하는 능력을 합리화함으로써 도킹 프로그램의 적합성을 정량화하는 하나의 척도를 나타낸다.
농축 계수
도킹 스크린은 또한 추정된 비결합성 "미끼" [28]분자의 대규모 데이터베이스 중에서 알려진 바인더의 주석이 달린 리간드를 농축함으로써 평가할 수 있다.이와 같이 도킹 스크린의 성공은 데이터베이스 내의 훨씬 많은 수의 유인 분자 중에서 화면의 상위 순위에서 소수의 알려진 활성 화합물을 농축하는 능력에 의해 평가된다.Receiver Operation Character(ROC; 수신기 작동 특성) 곡선 아래의 영역은 성능 평가에 널리 사용됩니다.
프로스펙티브
도킹 스크린에서 발생하는 타격은 약리학적 검증을 거친다(예50,: IC 친화력 또는 효력 측정).예비 연구만이 특정 [32]대상에 대한 기법의 적합성에 대한 결정적인 증거를 구성한다.시판 의약품의 30% 이상이 대상인 G단백질결합수용체(GPCR)의 경우 분자 도킹이 500개 이상의 GPCR 리간드를 [33]발견하게 했다.
벤치마킹
X선 결정학에 의해 결정되는 바인딩 모드를 재현하는 도킹 프로그램의 가능성은 다양한 도킹 벤치마크 세트로 평가할 수 있습니다.
소분자의 경우 도킹 및 가상 스크리닝을 위한 여러 벤치마크 데이터 세트가 존재합니다.고품질 단백질 리간드 X선 결정 구조[34] 또는 가상 스크리닝 [28]성능 평가를 위한 DUD(Directory of Use Decoys)로 구성된 Astex Various Set.
도킹 프로그램의 펩타이드 결합 모드 재현 가능성에 대한 평가는 LEADS-PEP([35]도킹 및 스코어링의 효율성 평가 레슨)를 통해 평가할 수 있습니다.
적용들
소분자배위자와 효소단백질 간의 결합상호작용은 효소의 활성화 또는 저해를 초래할 수 있다.단백질이 수용체일 경우 리간드 결합은 작용작용 또는 길항작용을 일으킬 수 있다.도킹은 약물 설계 분야에서 가장 일반적으로 사용됩니다. 대부분의 약은 작은 유기 분자로, 도킹은 다음 분야에 적용될 수 있습니다.
- 히트 식별 – 점수 매기기 기능과 결합된 도킹은 관심 단백질 표적에 결합할 가능성이 있는 분자를 식별하기 위해 실리코에서 잠재적 약물의 대규모 데이터베이스를 신속하게 스크리닝하는 데 사용할 수 있다(가상 스크리닝 참조).역약리학에서는 표적 식별을 위해 도킹을 일상적으로 사용합니다.
- 납 최적화 – 도킹은 배위자가 단백질에 결합하는 위치와 상대적 방향(결합 모드 또는 포즈라고도 함)을 예측하는 데 사용할 수 있습니다.이 정보를 사용하여 보다 강력하고 선택적인 아날로그를 설계할 수 있습니다.
- 생물 조정 – 단백질 배위자 도킹은 [36][37]효소에 의해 분해될 수 있는 오염 물질을 예측하는 데도 사용할 수 있습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Lengauer T, Rarey M (Jun 1996). "Computational methods for biomolecular docking". Current Opinion in Structural Biology. 6 (3): 402–6. doi:10.1016/S0959-440X(96)80061-3. PMID 8804827.
- ^ Kitchen DB, Decornez H, Furr JR, Bajorath J (Nov 2004). "Docking and scoring in virtual screening for drug discovery: methods and applications". Nature Reviews. Drug Discovery. 3 (11): 935–49. doi:10.1038/nrd1549. PMID 15520816. S2CID 1069493.
- ^ Mostashari-Rad, T; Arian, R; Mehridehnavi, A; Fassihi, A; Ghasemi, F (June 13, 2019). "Study of CXCR4 chemokine receptor inhibitors using QSPR andmolecular docking methodologies". Journal of Theoretical and Computational Chemistry. 178 (4). doi:10.1142/S0219633619500184. S2CID 164985789.
- ^ Jorgensen WL (Nov 1991). "Rusting of the lock and key model for protein-ligand binding". Science. 254 (5034): 954–5. Bibcode:1991Sci...254..954J. doi:10.1126/science.1719636. PMID 1719636.
- ^ Wei BQ, Weaver LH, Ferrari AM, Matthews BW, Shoichet BK (Apr 2004). "Testing a flexible-receptor docking algorithm in a model binding site". Journal of Molecular Biology. 337 (5): 1161–82. doi:10.1016/j.jmb.2004.02.015. PMID 15046985.
- ^ Goldman BB, Wipke WT (2000). "QSD quadratic shape descriptors. 2. Molecular docking using quadratic shape descriptors (QSDock)". Proteins. 38 (1): 79–94. doi:10.1002/(SICI)1097-0134(20000101)38:1<79::AID-PROT9>3.0.CO;2-U. PMID 10651041.
- ^ Meng EC, Shoichet BK, Kuntz ID (1992). "Automated docking with grid-based energy evaluation". Journal of Computational Chemistry. 13 (4): 505–524. doi:10.1002/jcc.540130412. S2CID 97778840.
- ^ Morris GM, Goodsell DS, Halliday RS, Huey R, Hart WE, Belew RK, Olson AJ (1998). "Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function". Journal of Computational Chemistry. 19 (14): 1639–1662. CiteSeerX 10.1.1.471.5900. doi:10.1002/(SICI)1096-987X(19981115)19:14<1639::AID-JCC10>3.0.CO;2-B.
- ^ a b Feig M, Onufriev A, Lee MS, Im W, Case DA, Brooks CL (Jan 2004). "Performance comparison of generalized born and Poisson methods in the calculation of electrostatic solvation energies for protein structures". Journal of Computational Chemistry. 25 (2): 265–84. doi:10.1002/jcc.10378. PMID 14648625. S2CID 3191066.
- ^ Shoichet BK, Kuntz ID, Bodian DL (2004). "Molecular docking using shape descriptors". Journal of Computational Chemistry. 13 (3): 380–397. doi:10.1002/jcc.540130311. S2CID 42749294.
- ^ Cai W, Shao X, Maigret B (Jan 2002). "Protein-ligand recognition using spherical harmonic molecular surfaces: towards a fast and efficient filter for large virtual throughput screening". Journal of Molecular Graphics & Modelling. 20 (4): 313–28. doi:10.1016/S1093-3263(01)00134-6. PMID 11858640.
- ^ Morris RJ, Najmanovich RJ, Kahraman A, Thornton JM (May 2005). "Real spherical harmonic expansion coefficients as 3D shape descriptors for protein binding pocket and ligand comparisons". Bioinformatics. 21 (10): 2347–55. doi:10.1093/bioinformatics/bti337. PMID 15728116.
- ^ Kahraman A, Morris RJ, Laskowski RA, Thornton JM (Apr 2007). "Shape variation in protein binding pockets and their ligands". Journal of Molecular Biology. 368 (1): 283–301. doi:10.1016/j.jmb.2007.01.086. PMID 17337005.
- ^ Kearsley SK, Underwood DJ, Sheridan RP, Miller MD (Oct 1994). "Flexibases: a way to enhance the use of molecular docking methods". Journal of Computer-Aided Molecular Design. 8 (5): 565–82. Bibcode:1994JCAMD...8..565K. doi:10.1007/BF00123666. PMID 7876901. S2CID 8834526.
- ^ Friesner RA, Banks JL, Murphy RB, Halgren TA, Klicic JJ, Mainz DT, Repasky MP, Knoll EH, Shelley M, Perry JK, Shaw DE, Francis P, Shenkin PS (Mar 2004). "Glide: a new approach for rapid, accurate docking and scoring. 1. Method and assessment of docking accuracy". Journal of Medicinal Chemistry. 47 (7): 1739–49. doi:10.1021/jm0306430. PMID 15027865.
- ^ Zsoldos Z, Reid D, Simon A, Sadjad SB, Johnson AP (Jul 2007). "eHiTS: a new fast, exhaustive flexible ligand docking system". Journal of Molecular Graphics & Modelling. 26 (1): 198–212. doi:10.1016/j.jmgm.2006.06.002. PMID 16860582.
- ^ Wang Q, Pang YP (September 2007). Romesberg F (ed.). "Preference of small molecules for local minimum conformations when binding to proteins". PLOS ONE. 2 (9): e820. Bibcode:2007PLoSO...2..820W. doi:10.1371/journal.pone.0000820. PMC 1959118. PMID 17786192.
- ^ Klebe G, Mietzner T (October 1994). "A fast and efficient method to generate biologically relevant conformations". Journal of Computer-Aided Molecular Design. 8 (5): 583–606. Bibcode:1994JCAMD...8..583K. doi:10.1007/BF00123667. PMID 7876902. S2CID 206768542.
- ^ Ciemny M, Kurcinski M, Kamel K, Kolinski A, Alam N, Schueler-Furman O, Kmiecik S (May 2018). "Protein-peptide docking: opportunities and challenges". Drug Discovery Today. 23 (8): 1530–1537. doi:10.1016/j.drudis.2018.05.006. PMID 29733895.
- ^ Antunes DA, Devaurs D, Kavraki LE (December 2015). "Understanding the challenges of protein flexibility in drug design" (PDF). Expert Opinion on Drug Discovery. 10 (12): 1301–13. doi:10.1517/17460441.2015.1094458. hdl:1911/88215. PMID 26414598. S2CID 6589810.
- ^ Cerqueira NM, Bras NF, Fernandes PA, Ramos MJ (January 2009). "MADAMM: a multistaged docking with an automated molecular modeling protocol". Proteins. 74 (1): 192–206. doi:10.1002/prot.22146. PMID 18618708. S2CID 36656063.
- ^ Totrov M, Abagyan R (Apr 2008). "Flexible ligand docking to multiple receptor conformations: a practical alternative". Current Opinion in Structural Biology. 18 (2): 178–84. doi:10.1016/j.sbi.2008.01.004. PMC 2396190. PMID 18302984.
- ^ Hartmann C, Antes I, Lengauer T (Feb 2009). "Docking and scoring with alternative side-chain conformations". Proteins. 74 (3): 712–26. doi:10.1002/prot.22189. PMID 18704939. S2CID 36088213.
- ^ Taylor RD, Jewsbury PJ, Essex JW (Oct 2003). "FDS: flexible ligand and receptor docking with a continuum solvent model and soft-core energy function". Journal of Computational Chemistry. 24 (13): 1637–56. CiteSeerX 10.1.1.147.1131. doi:10.1002/jcc.10295. PMID 12926007. S2CID 15814316.
- ^ Murcko MA (Dec 1995). "Computational Methods to Predict Binding Free Energy in Ligand-Receptor Complexes". Journal of Medicinal Chemistry. 38 (26): 4953–67. doi:10.1021/jm00026a001. PMID 8544170.
- ^ Arcon, Juan Pablo; Turjanski, Adrián G.; Martí, Marcelo A.; Forli, Stefano (2021), Ballante, Flavio (ed.), "Biased Docking for Protein–Ligand Pose Prediction", Protein-Ligand Interactions and Drug Design, Methods in Molecular Biology, New York, NY: Springer US, vol. 2266, pp. 39–72, doi:10.1007/978-1-0716-1209-5_3, ISBN 978-1-0716-1209-5, PMID 33759120, S2CID 232340746, retrieved 2021-04-05
- ^ Gohlke, Holger; Hendlich, Manfred; Klebe, Gerhard (2000-01-14). "Knowledge-based scoring function to predict protein-ligand interactions11Edited by R. Huber". Journal of Molecular Biology. 295 (2): 337–356. doi:10.1006/jmbi.1999.3371. ISSN 0022-2836. PMID 10623530.
- ^ a b c Huang N, Shoichet BK, Irwin JJ (Nov 2006). "Benchmarking sets for molecular docking". Journal of Medicinal Chemistry. 49 (23): 6789–801. doi:10.1021/jm0608356. PMC 3383317. PMID 17154509.
- ^ Ballante F, Marshall GR (Jan 2016). "An Automated Strategy for Binding-Pose Selection and Docking Assessment in Structure-Based Drug Design". Journal of Chemical Information and Modeling. 56 (1): 54–72. doi:10.1021/acs.jcim.5b00603. PMID 26682916.
- ^ Bursulaya BD, Totrov M, Abagyan R, Brooks CL (Nov 2003). "Comparative study of several algorithms for flexible ligand docking". Journal of Computer-Aided Molecular Design. 17 (11): 755–63. Bibcode:2003JCAMD..17..755B. doi:10.1023/B:JCAM.0000017496.76572.6f. PMID 15072435. S2CID 12569345.
- ^ Ballante, Flavio (2018). "Protein-Ligand Docking in Drug Design: Performance Assessment and Binding-Pose Selection". Rational Drug Design. Methods in Molecular Biology. Vol. 1824. pp. 67–88. doi:10.1007/978-1-4939-8630-9_5. ISBN 978-1-4939-8629-3. ISSN 1940-6029. PMID 30039402.
- ^ Irwin JJ (2008-02-14). "Community benchmarks for virtual screening". Journal of Computer-Aided Molecular Design. 22 (3–4): 193–9. Bibcode:2008JCAMD..22..193I. doi:10.1007/s10822-008-9189-4. PMID 18273555. S2CID 26260725.
- ^ Ballante, Flavio; Kooistra, Albert J.; Kampen, Stefanie; Graaf, Chris de; Carlsson, Jens (2021-10-01). "Structure-Based Virtual Screening for Ligands of G Protein–Coupled Receptors: What Can Molecular Docking Do for You?". Pharmacological Reviews. 73 (4): 527–565. doi:10.1124/pharmrev.120.000246. ISSN 0031-6997. PMID 34907092.
- ^ Hartshorn MJ, Verdonk ML, Chessari G, Brewerton SC, Mooij WT, Mortenson PN, Murray CW (Feb 2007). "Diverse, high-quality test set for the validation of protein-ligand docking performance". Journal of Medicinal Chemistry. 50 (4): 726–41. doi:10.1021/jm061277y. PMID 17300160.
- ^ Hauser AS, Windshügel B (Dec 2015). "A Benchmark Data Set for Assessment of Peptide Docking Performance". Journal of Chemical Information and Modeling. 56 (1): 188–200. doi:10.1021/acs.jcim.5b00234. PMID 26651532.
- ^ Suresh PS, Kumar A, Kumar R, Singh VP (Jan 2008). "An in silico [correction of insilico] approach to bioremediation: laccase as a case study". Journal of Molecular Graphics & Modelling. 26 (5): 845–9. doi:10.1016/j.jmgm.2007.05.005. PMID 17606396.
- ^ Basharat Z, Yasmin A, Bibi M (2020). "Implications of Molecular Docking Assay for Bioremediation". Data Analytics in Medicine: Concepts, Methodologies, Tools, and Applications. Advances in Environmental Engineering and Green Technologies. IGI Global. pp. 1556–1577. doi:10.4018/978-1-5225-2325-3.ch002. ISBN 978-1799812043.
외부 링크
- Bikadi Z, Kovacs S, Demko L, Hazai E. "Molecular Docking Server - Ligand Protein Docking & Molecular Modeling". Virtua Drug Ltd. Retrieved 2008-07-15.
Internet service that calculates the site, geometry and energy of small molecules interacting with proteins
- Malinauskas T. "Step by step installation of MGLTools 1.5.2 (AutoDockTools, Python Molecular Viewer and Visual Programming Environment) on Ubuntu Linux 8.04". Archived from the original on 2009-02-26. Retrieved 2008-07-15.
- Docking@GRID Conformational Sampling and Docking on Grid 프로젝트: 하나의 목표는 컴퓨터 그리드에 몇 가지 고유한 분산 도킹 알고리즘을 도입하여 Docking@GRID 오픈 소스 Linux 버전을 다운로드하는 것입니다.
- Click2Drug.org - 컴퓨터 의약품 설계 도구 디렉토리.
- 리간드: MOE(분자 동작 환경)를 사용한 리셉터 도킹