KR20240015624A - 게놈 전체 cfdna 단편화 프로파일을 이용한 암을 검출하는 방법 - Google Patents
게놈 전체 cfdna 단편화 프로파일을 이용한 암을 검출하는 방법 Download PDFInfo
- Publication number
- KR20240015624A KR20240015624A KR1020237035747A KR20237035747A KR20240015624A KR 20240015624 A KR20240015624 A KR 20240015624A KR 1020237035747 A KR1020237035747 A KR 1020237035747A KR 20237035747 A KR20237035747 A KR 20237035747A KR 20240015624 A KR20240015624 A KR 20240015624A
- Authority
- KR
- South Korea
- Prior art keywords
- cancer
- cfdna
- subject
- score
- fragments
- Prior art date
Links
- 206010028980 Neoplasm Diseases 0.000 title claims abstract description 201
- 201000011510 cancer Diseases 0.000 title claims abstract description 189
- 238000013467 fragmentation Methods 0.000 title claims description 80
- 238000006062 fragmentation reaction Methods 0.000 title claims description 80
- 238000000034 method Methods 0.000 claims abstract description 153
- 239000012634 fragment Substances 0.000 claims abstract description 145
- 230000004083 survival effect Effects 0.000 claims abstract description 43
- 238000004458 analytical method Methods 0.000 claims abstract description 28
- 238000012544 monitoring process Methods 0.000 claims abstract description 17
- 238000011282 treatment Methods 0.000 claims description 32
- 238000012070 whole genome sequencing analysis Methods 0.000 claims description 12
- 238000012545 processing Methods 0.000 claims description 11
- 238000012163 sequencing technique Methods 0.000 claims description 11
- 206010035226 Plasma cell myeloma Diseases 0.000 claims description 10
- 210000000349 chromosome Anatomy 0.000 claims description 10
- 238000010801 machine learning Methods 0.000 claims description 10
- 206010058467 Lung neoplasm malignant Diseases 0.000 claims description 8
- 238000009169 immunotherapy Methods 0.000 claims description 8
- 201000005202 lung cancer Diseases 0.000 claims description 8
- 208000020816 lung neoplasm Diseases 0.000 claims description 8
- 238000003860 storage Methods 0.000 claims description 8
- 238000002560 therapeutic procedure Methods 0.000 claims description 8
- 206010009944 Colon cancer Diseases 0.000 claims description 6
- 208000029742 colonic neoplasm Diseases 0.000 claims description 6
- 238000004590 computer program Methods 0.000 claims description 6
- 230000008569 process Effects 0.000 claims description 6
- 208000003950 B-cell lymphoma Diseases 0.000 claims description 5
- 206010004593 Bile duct cancer Diseases 0.000 claims description 5
- 206010006187 Breast cancer Diseases 0.000 claims description 5
- 208000026310 Breast neoplasm Diseases 0.000 claims description 5
- 208000000461 Esophageal Neoplasms Diseases 0.000 claims description 5
- 208000028018 Lymphocytic leukaemia Diseases 0.000 claims description 5
- 208000034578 Multiple myelomas Diseases 0.000 claims description 5
- 206010030155 Oesophageal carcinoma Diseases 0.000 claims description 5
- 206010033128 Ovarian cancer Diseases 0.000 claims description 5
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 5
- 206010061902 Pancreatic neoplasm Diseases 0.000 claims description 5
- 206010060862 Prostate cancer Diseases 0.000 claims description 5
- 208000000236 Prostatic Neoplasms Diseases 0.000 claims description 5
- 208000005718 Stomach Neoplasms Diseases 0.000 claims description 5
- 210000001744 T-lymphocyte Anatomy 0.000 claims description 5
- 208000002495 Uterine Neoplasms Diseases 0.000 claims description 5
- 208000026900 bile duct neoplasm Diseases 0.000 claims description 5
- 208000006990 cholangiocarcinoma Diseases 0.000 claims description 5
- 201000004101 esophageal cancer Diseases 0.000 claims description 5
- 201000003444 follicular lymphoma Diseases 0.000 claims description 5
- 206010017758 gastric cancer Diseases 0.000 claims description 5
- 201000011243 gastrointestinal stromal tumor Diseases 0.000 claims description 5
- 208000032839 leukemia Diseases 0.000 claims description 5
- 201000007270 liver cancer Diseases 0.000 claims description 5
- 208000014018 liver neoplasm Diseases 0.000 claims description 5
- 208000003747 lymphoid leukemia Diseases 0.000 claims description 5
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 claims description 5
- 238000013507 mapping Methods 0.000 claims description 5
- 208000025113 myeloid leukemia Diseases 0.000 claims description 5
- 201000000050 myeloid neoplasm Diseases 0.000 claims description 5
- 208000008443 pancreatic carcinoma Diseases 0.000 claims description 5
- 201000011549 stomach cancer Diseases 0.000 claims description 5
- 206010046766 uterine cancer Diseases 0.000 claims description 5
- 201000009030 Carcinoma Diseases 0.000 claims description 4
- 206010025323 Lymphomas Diseases 0.000 claims description 4
- 206010039491 Sarcoma Diseases 0.000 claims description 4
- 238000011226 adjuvant chemotherapy Methods 0.000 claims description 4
- 238000002659 cell therapy Methods 0.000 claims description 4
- 231100000433 cytotoxic Toxicity 0.000 claims description 4
- 230000001472 cytotoxic effect Effects 0.000 claims description 4
- 230000002489 hematologic effect Effects 0.000 claims description 4
- 238000001794 hormone therapy Methods 0.000 claims description 4
- 238000011227 neoadjuvant chemotherapy Methods 0.000 claims description 4
- 238000001959 radiotherapy Methods 0.000 claims description 4
- 238000001356 surgical procedure Methods 0.000 claims description 4
- 238000002626 targeted therapy Methods 0.000 claims description 4
- 201000002528 pancreatic cancer Diseases 0.000 claims description 3
- 230000007423 decrease Effects 0.000 claims 4
- 108091027544 Subgenomic mRNA Proteins 0.000 claims 2
- 238000012252 genetic analysis Methods 0.000 abstract description 3
- 108020004414 DNA Proteins 0.000 description 21
- 239000000523 sample Substances 0.000 description 20
- 238000009826 distribution Methods 0.000 description 19
- 210000004369 blood Anatomy 0.000 description 14
- 239000008280 blood Substances 0.000 description 14
- 230000008685 targeting Effects 0.000 description 14
- 238000001514 detection method Methods 0.000 description 12
- 201000010099 disease Diseases 0.000 description 11
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 11
- 241000124008 Mammalia Species 0.000 description 9
- 238000003556 assay Methods 0.000 description 8
- 238000013459 approach Methods 0.000 description 7
- 239000012472 biological sample Substances 0.000 description 7
- 230000004075 alteration Effects 0.000 description 5
- 238000003745 diagnosis Methods 0.000 description 5
- 210000002381 plasma Anatomy 0.000 description 5
- 238000002512 chemotherapy Methods 0.000 description 4
- 238000002405 diagnostic procedure Methods 0.000 description 4
- 239000012530 fluid Substances 0.000 description 4
- 150000007523 nucleic acids Chemical group 0.000 description 4
- 230000005855 radiation Effects 0.000 description 4
- 230000001225 therapeutic effect Effects 0.000 description 4
- 238000012300 Sequence Analysis Methods 0.000 description 3
- -1 doxyfluridine Chemical compound 0.000 description 3
- 230000006870 function Effects 0.000 description 3
- 229940043355 kinase inhibitor Drugs 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 239000003757 phosphotransferase inhibitor Substances 0.000 description 3
- 238000012360 testing method Methods 0.000 description 3
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 2
- 108091028043 Nucleic acid sequence Proteins 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 239000000090 biomarker Substances 0.000 description 2
- 210000001175 cerebrospinal fluid Anatomy 0.000 description 2
- 239000012829 chemotherapy agent Substances 0.000 description 2
- 230000002759 chromosomal effect Effects 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 238000011156 evaluation Methods 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 150000002500 ions Chemical class 0.000 description 2
- 210000002751 lymph Anatomy 0.000 description 2
- 239000000203 mixture Substances 0.000 description 2
- 210000003097 mucus Anatomy 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 108020004707 nucleic acids Proteins 0.000 description 2
- 102000039446 nucleic acids Human genes 0.000 description 2
- 238000003752 polymerase chain reaction Methods 0.000 description 2
- 238000002271 resection Methods 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 230000035945 sensitivity Effects 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 210000004243 sweat Anatomy 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 210000001138 tear Anatomy 0.000 description 2
- 230000000007 visual effect Effects 0.000 description 2
- FDKXTQMXEQVLRF-ZHACJKMWSA-N (E)-dacarbazine Chemical compound CN(C)\N=N\c1[nH]cnc1C(N)=O FDKXTQMXEQVLRF-ZHACJKMWSA-N 0.000 description 1
- VSNHCAURESNICA-NJFSPNSNSA-N 1-oxidanylurea Chemical compound N[14C](=O)NO VSNHCAURESNICA-NJFSPNSNSA-N 0.000 description 1
- NDMPLJNOPCLANR-UHFFFAOYSA-N 3,4-dihydroxy-15-(4-hydroxy-18-methoxycarbonyl-5,18-seco-ibogamin-18-yl)-16-methoxy-1-methyl-6,7-didehydro-aspidospermidine-3-carboxylic acid methyl ester Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 NDMPLJNOPCLANR-UHFFFAOYSA-N 0.000 description 1
- AOJJSUZBOXZQNB-VTZDEGQISA-N 4'-epidoxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-VTZDEGQISA-N 0.000 description 1
- IDPUKCWIGUEADI-UHFFFAOYSA-N 5-[bis(2-chloroethyl)amino]uracil Chemical compound ClCCN(CCCl)C1=CNC(=O)NC1=O IDPUKCWIGUEADI-UHFFFAOYSA-N 0.000 description 1
- NMUSYJAQQFHJEW-KVTDHHQDSA-N 5-azacytidine Chemical compound O=C1N=C(N)N=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](CO)O1 NMUSYJAQQFHJEW-KVTDHHQDSA-N 0.000 description 1
- WYWHKKSPHMUBEB-UHFFFAOYSA-N 6-Mercaptoguanine Natural products N1C(N)=NC(=S)C2=C1N=CN2 WYWHKKSPHMUBEB-UHFFFAOYSA-N 0.000 description 1
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 1
- 208000023275 Autoimmune disease Diseases 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- COVZYZSDYWQREU-UHFFFAOYSA-N Busulfan Chemical compound CS(=O)(=O)OCCCCOS(C)(=O)=O COVZYZSDYWQREU-UHFFFAOYSA-N 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- GAGWJHPBXLXJQN-UORFTKCHSA-N Capecitabine Chemical compound C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1[C@H]1[C@H](O)[C@H](O)[C@@H](C)O1 GAGWJHPBXLXJQN-UORFTKCHSA-N 0.000 description 1
- GAGWJHPBXLXJQN-UHFFFAOYSA-N Capecitabine Natural products C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1C1C(O)C(O)C(C)O1 GAGWJHPBXLXJQN-UHFFFAOYSA-N 0.000 description 1
- 208000024172 Cardiovascular disease Diseases 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 206010050337 Cerumen impaction Diseases 0.000 description 1
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 description 1
- 208000005443 Circulating Neoplastic Cells Diseases 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-CCXZUQQUSA-N Cytarabine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@@H](O)[C@H](O)[C@@H](CO)O1 UHDGCWIWMRVCDJ-CCXZUQQUSA-N 0.000 description 1
- HTIJFSOGRVMCQR-UHFFFAOYSA-N Epirubicin Natural products COc1cccc2C(=O)c3c(O)c4CC(O)(CC(OC5CC(N)C(=O)C(C)O5)c4c(O)c3C(=O)c12)C(=O)CO HTIJFSOGRVMCQR-UHFFFAOYSA-N 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 108010033040 Histones Proteins 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- XDXDZDZNSLXDNA-TZNDIEGXSA-N Idarubicin Chemical compound C1[C@H](N)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2C[C@@](O)(C(C)=O)C1 XDXDZDZNSLXDNA-TZNDIEGXSA-N 0.000 description 1
- XDXDZDZNSLXDNA-UHFFFAOYSA-N Idarubicin Natural products C1C(N)C(O)C(C)OC1OC1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2CC(O)(C(C)=O)C1 XDXDZDZNSLXDNA-UHFFFAOYSA-N 0.000 description 1
- 229940076838 Immune checkpoint inhibitor Drugs 0.000 description 1
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 1
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 1
- 206010062717 Increased upper airway secretion Diseases 0.000 description 1
- 102000037984 Inhibitory immune checkpoint proteins Human genes 0.000 description 1
- 108091008026 Inhibitory immune checkpoint proteins Proteins 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- GQYIWUVLTXOXAJ-UHFFFAOYSA-N Lomustine Chemical compound ClCCN(N=O)C(=O)NC1CCCCC1 GQYIWUVLTXOXAJ-UHFFFAOYSA-N 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 1
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 1
- 229930012538 Paclitaxel Natural products 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 208000005228 Pericardial Effusion Diseases 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 206010036790 Productive cough Diseases 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 108091008874 T cell receptors Proteins 0.000 description 1
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 1
- BPEGJWRSRHCHSN-UHFFFAOYSA-N Temozolomide Chemical compound O=C1N(C)N=NC2=C(C(N)=O)N=CN21 BPEGJWRSRHCHSN-UHFFFAOYSA-N 0.000 description 1
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 1
- RTJVUHUGTUDWRK-CSLCKUBZSA-N [(2r,4ar,6r,7r,8s,8ar)-6-[[(5s,5ar,8ar,9r)-9-(3,5-dimethoxy-4-phosphonooxyphenyl)-8-oxo-5a,6,8a,9-tetrahydro-5h-[2]benzofuro[6,5-f][1,3]benzodioxol-5-yl]oxy]-2-methyl-7-[2-(2,3,4,5,6-pentafluorophenoxy)acetyl]oxy-4,4a,6,7,8,8a-hexahydropyrano[3,2-d][1,3]d Chemical compound COC1=C(OP(O)(O)=O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](OC(=O)COC=4C(=C(F)C(F)=C(F)C=4F)F)[C@@H]4O[C@H](C)OC[C@H]4O3)OC(=O)COC=3C(=C(F)C(F)=C(F)C=3F)F)[C@@H]3[C@@H]2C(OC3)=O)=C1 RTJVUHUGTUDWRK-CSLCKUBZSA-N 0.000 description 1
- 230000005856 abnormality Effects 0.000 description 1
- SHGAZHPCJJPHSC-YCNIQYBTSA-N all-trans-retinoic acid Chemical compound OC(=O)\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C SHGAZHPCJJPHSC-YCNIQYBTSA-N 0.000 description 1
- 210000004381 amniotic fluid Anatomy 0.000 description 1
- 229960001220 amsacrine Drugs 0.000 description 1
- XCPGHVQEEXUHNC-UHFFFAOYSA-N amsacrine Chemical compound COC1=CC(NS(C)(=O)=O)=CC=C1NC1=C(C=CC=C2)C2=NC2=CC=CC=C12 XCPGHVQEEXUHNC-UHFFFAOYSA-N 0.000 description 1
- 229940124650 anti-cancer therapies Drugs 0.000 description 1
- 238000011319 anticancer therapy Methods 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 210000001742 aqueous humor Anatomy 0.000 description 1
- 210000003567 ascitic fluid Anatomy 0.000 description 1
- 230000001363 autoimmune Effects 0.000 description 1
- 229960002756 azacitidine Drugs 0.000 description 1
- 229960000397 bevacizumab Drugs 0.000 description 1
- 239000013060 biological fluid Substances 0.000 description 1
- 238000001574 biopsy Methods 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 210000001124 body fluid Anatomy 0.000 description 1
- 239000010839 body fluid Substances 0.000 description 1
- 229960002092 busulfan Drugs 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 229960004117 capecitabine Drugs 0.000 description 1
- 229960004562 carboplatin Drugs 0.000 description 1
- 190000008236 carboplatin Chemical compound 0.000 description 1
- 210000004027 cell Anatomy 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 210000002939 cerumen Anatomy 0.000 description 1
- JCKYGMPEJWAADB-UHFFFAOYSA-N chlorambucil Chemical compound OC(=O)CCCC1=CC=C(N(CCCl)CCCl)C=C1 JCKYGMPEJWAADB-UHFFFAOYSA-N 0.000 description 1
- 229960004630 chlorambucil Drugs 0.000 description 1
- 210000004913 chyme Anatomy 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- 238000002790 cross-validation Methods 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 229960000684 cytarabine Drugs 0.000 description 1
- 229960003901 dacarbazine Drugs 0.000 description 1
- 229960000975 daunorubicin Drugs 0.000 description 1
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 229960003668 docetaxel Drugs 0.000 description 1
- 229960004679 doxorubicin Drugs 0.000 description 1
- 238000013399 early diagnosis Methods 0.000 description 1
- 230000002526 effect on cardiovascular system Effects 0.000 description 1
- 230000003511 endothelial effect Effects 0.000 description 1
- 229960001904 epirubicin Drugs 0.000 description 1
- 229960005073 erlotinib hydrochloride Drugs 0.000 description 1
- GTTBEUCJPZQMDZ-UHFFFAOYSA-N erlotinib hydrochloride Chemical compound [H+].[Cl-].C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 GTTBEUCJPZQMDZ-UHFFFAOYSA-N 0.000 description 1
- VJJPUSNTGOMMGY-MRVIYFEKSA-N etoposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@H](C)OC[C@H]4O3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 VJJPUSNTGOMMGY-MRVIYFEKSA-N 0.000 description 1
- 229960005420 etoposide Drugs 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 210000003608 fece Anatomy 0.000 description 1
- 230000012953 feeding on blood of other organism Effects 0.000 description 1
- 229960000961 floxuridine Drugs 0.000 description 1
- ODKNJVUHOIMIIZ-RRKCRQDMSA-N floxuridine Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C(F)=C1 ODKNJVUHOIMIIZ-RRKCRQDMSA-N 0.000 description 1
- 229960000390 fludarabine Drugs 0.000 description 1
- GIUYCYHIANZCFB-FJFJXFQQSA-N fludarabine phosphate Chemical compound C1=NC=2C(N)=NC(F)=NC=2N1[C@@H]1O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O GIUYCYHIANZCFB-FJFJXFQQSA-N 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 238000007672 fourth generation sequencing Methods 0.000 description 1
- 210000004211 gastric acid Anatomy 0.000 description 1
- 230000002496 gastric effect Effects 0.000 description 1
- 210000004051 gastric juice Anatomy 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 230000003862 health status Effects 0.000 description 1
- 210000004251 human milk Anatomy 0.000 description 1
- 235000020256 human milk Nutrition 0.000 description 1
- 229960000908 idarubicin Drugs 0.000 description 1
- HOMGKSMUEGBAAB-UHFFFAOYSA-N ifosfamide Chemical compound ClCCNP1(=O)OCCCN1CCCl HOMGKSMUEGBAAB-UHFFFAOYSA-N 0.000 description 1
- 229960001101 ifosfamide Drugs 0.000 description 1
- 239000012274 immune-checkpoint protein inhibitor Substances 0.000 description 1
- 208000027866 inflammatory disease Diseases 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 229960004768 irinotecan Drugs 0.000 description 1
- UWKQSNNFCGGAFS-XIFFEERXSA-N irinotecan Chemical compound C1=C2C(CC)=C3CN(C(C4=C([C@@](C(=O)OC4)(O)CC)C=4)=O)C=4C3=NC2=CC=C1OC(=O)N(CC1)CCC1N1CCCCC1 UWKQSNNFCGGAFS-XIFFEERXSA-N 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 238000011528 liquid biopsy Methods 0.000 description 1
- 229960002247 lomustine Drugs 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 238000004519 manufacturing process Methods 0.000 description 1
- HAWPXGHAZFHHAD-UHFFFAOYSA-N mechlorethamine Chemical compound ClCCN(C)CCCl HAWPXGHAZFHHAD-UHFFFAOYSA-N 0.000 description 1
- 229960004961 mechlorethamine Drugs 0.000 description 1
- SGDBTWWWUNNDEQ-LBPRGKRZSA-N melphalan Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N(CCCl)CCCl)C=C1 SGDBTWWWUNNDEQ-LBPRGKRZSA-N 0.000 description 1
- 229960001924 melphalan Drugs 0.000 description 1
- GLVAUDGFNGKCSF-UHFFFAOYSA-N mercaptopurine Chemical compound S=C1NC=NC2=C1NC=N2 GLVAUDGFNGKCSF-UHFFFAOYSA-N 0.000 description 1
- 229960001428 mercaptopurine Drugs 0.000 description 1
- 208000037819 metastatic cancer Diseases 0.000 description 1
- 208000011575 metastatic malignant neoplasm Diseases 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 229960001156 mitoxantrone Drugs 0.000 description 1
- KKZJGLLVHKMTCM-UHFFFAOYSA-N mitoxantrone Chemical compound O=C1C2=C(O)C=CC(O)=C2C(=O)C2=C1C(NCCNCCO)=CC=C2NCCNCCO KKZJGLLVHKMTCM-UHFFFAOYSA-N 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 238000013188 needle biopsy Methods 0.000 description 1
- 210000002445 nipple Anatomy 0.000 description 1
- 210000004882 non-tumor cell Anatomy 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- DWAFYCQODLXJNR-BNTLRKBRSA-L oxaliplatin Chemical compound O1C(=O)C(=O)O[Pt]11N[C@@H]2CCCC[C@H]2N1 DWAFYCQODLXJNR-BNTLRKBRSA-L 0.000 description 1
- 229960001756 oxaliplatin Drugs 0.000 description 1
- 229960001592 paclitaxel Drugs 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 229960005079 pemetrexed Drugs 0.000 description 1
- QOFFJEBXNKRSPX-ZDUSSCGKSA-N pemetrexed Chemical compound C1=N[C]2NC(N)=NC(=O)C2=C1CCC1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 QOFFJEBXNKRSPX-ZDUSSCGKSA-N 0.000 description 1
- 210000004912 pericardial fluid Anatomy 0.000 description 1
- 210000004049 perilymph Anatomy 0.000 description 1
- 208000026435 phlegm Diseases 0.000 description 1
- 210000004910 pleural fluid Anatomy 0.000 description 1
- CPTBDICYNRMXFX-UHFFFAOYSA-N procarbazine Chemical compound CNNCC1=CC=C(C(=O)NC(C)C)C=C1 CPTBDICYNRMXFX-UHFFFAOYSA-N 0.000 description 1
- 229960000624 procarbazine Drugs 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 210000004908 prostatic fluid Anatomy 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 108090000623 proteins and genes Proteins 0.000 description 1
- 210000004915 pus Anatomy 0.000 description 1
- 238000000611 regression analysis Methods 0.000 description 1
- 230000029058 respiratory gaseous exchange Effects 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 229930002330 retinoic acid Natural products 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 210000002374 sebum Anatomy 0.000 description 1
- 210000000582 semen Anatomy 0.000 description 1
- 210000002966 serum Anatomy 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 210000003802 sputum Anatomy 0.000 description 1
- 208000024794 sputum Diseases 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 229960001052 streptozocin Drugs 0.000 description 1
- ZSJLQEPLLKMAKR-GKHCUFPYSA-N streptozocin Chemical compound O=NN(C)C(=O)N[C@H]1[C@@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O ZSJLQEPLLKMAKR-GKHCUFPYSA-N 0.000 description 1
- 230000000153 supplemental effect Effects 0.000 description 1
- 238000011477 surgical intervention Methods 0.000 description 1
- 210000001179 synovial fluid Anatomy 0.000 description 1
- 229950003999 tafluposide Drugs 0.000 description 1
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 1
- 229960004964 temozolomide Drugs 0.000 description 1
- NRUKOCRGYNPUPR-QBPJDGROSA-N teniposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@@H](OC[C@H]4O3)C=3SC=CC=3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 NRUKOCRGYNPUPR-QBPJDGROSA-N 0.000 description 1
- 229960001278 teniposide Drugs 0.000 description 1
- 229960003087 tioguanine Drugs 0.000 description 1
- MNRILEROXIRVNJ-UHFFFAOYSA-N tioguanine Chemical compound N1C(N)=NC(=S)C2=NC=N[C]21 MNRILEROXIRVNJ-UHFFFAOYSA-N 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 229960000303 topotecan Drugs 0.000 description 1
- UCFGDBYHRUNTLO-QHCPKHFHSA-N topotecan Chemical compound C1=C(O)C(CN(C)C)=C2C=C(CN3C4=CC5=C(C3=O)COC(=O)[C@]5(O)CC)C4=NC2=C1 UCFGDBYHRUNTLO-QHCPKHFHSA-N 0.000 description 1
- 230000005945 translocation Effects 0.000 description 1
- 238000011269 treatment regimen Methods 0.000 description 1
- 229960001055 uracil mustard Drugs 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 229960000653 valrubicin Drugs 0.000 description 1
- ZOCKGBMQLCSHFP-KQRAQHLDSA-N valrubicin Chemical compound O([C@H]1C[C@](CC2=C(O)C=3C(=O)C4=CC=CC(OC)=C4C(=O)C=3C(O)=C21)(O)C(=O)COC(=O)CCCC)[C@H]1C[C@H](NC(=O)C(F)(F)F)[C@H](O)[C@H](C)O1 ZOCKGBMQLCSHFP-KQRAQHLDSA-N 0.000 description 1
- 229960003048 vinblastine Drugs 0.000 description 1
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 1
- 229960004528 vincristine Drugs 0.000 description 1
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 1
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 1
- 229960004355 vindesine Drugs 0.000 description 1
- UGGWPQSBPIFKDZ-KOTLKJBCSA-N vindesine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(N)=O)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1N=C1[C]2C=CC=C1 UGGWPQSBPIFKDZ-KOTLKJBCSA-N 0.000 description 1
- 229960002066 vinorelbine Drugs 0.000 description 1
- GBABOYUKABKIAF-GHYRFKGUSA-N vinorelbine Chemical compound C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC GBABOYUKABKIAF-GHYRFKGUSA-N 0.000 description 1
- 210000004127 vitreous body Anatomy 0.000 description 1
- 210000004916 vomit Anatomy 0.000 description 1
- 230000008673 vomiting Effects 0.000 description 1
- 230000003442 weekly effect Effects 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
- C12Q1/6886—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material for cancer
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B30/00—ICT specially adapted for sequence analysis involving nucleotides or amino acids
- G16B30/10—Sequence alignment; Homology search
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H50/00—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics
- G16H50/30—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics for calculating health indices; for individual health risk assessment
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2537/00—Reactions characterised by the reaction format or use of a specific feature
- C12Q2537/10—Reactions characterised by the reaction format or use of a specific feature the purpose or use of
- C12Q2537/165—Mathematical modelling, e.g. logarithm, ratio
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/118—Prognosis of disease development
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Physics & Mathematics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Analytical Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Health & Medical Sciences (AREA)
- Medical Informatics (AREA)
- Genetics & Genomics (AREA)
- Biophysics (AREA)
- Biotechnology (AREA)
- Organic Chemistry (AREA)
- Pathology (AREA)
- Bioinformatics & Computational Biology (AREA)
- Theoretical Computer Science (AREA)
- Molecular Biology (AREA)
- Immunology (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Spectroscopy & Molecular Physics (AREA)
- Public Health (AREA)
- Evolutionary Biology (AREA)
- Biomedical Technology (AREA)
- Hospice & Palliative Care (AREA)
- Primary Health Care (AREA)
- Oncology (AREA)
- Microbiology (AREA)
- Epidemiology (AREA)
- Databases & Information Systems (AREA)
- Data Mining & Analysis (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Investigating Or Analysing Biological Materials (AREA)
Abstract
본 개시는 암 상태를 진단하고 예측하기 위해 환자로부터 얻은 샘플에서 무세포 DNA(cfDNA) 단편의 분석을 활용하는 방법 및 시스템을 제공한다. 본 개시는 대상체에서 암을 검출하는 방법을 제공한다. 본 개시는 또한 암이 있는 대상체의 전체 생존을 결정하는 방법을 제공한다. 본 개시는 대상체에서 암을 모니터링하는 방법을 추가로 제공한다. 또한 유전적 분석을 위한 시스템이 제공된다.
Description
관련 출원(들)에 대한 상호 참조
본 출원은 2021년 4월 8일에 출원된 미국 특허 가출원 일련 번호 63/172,493의 35 U.S.C. §119(e)에 따른 우선권의 이익을 주장한다. 선행 출원의 개시는 본 출원의 개시의 일부로 간주되며 참조로 포함된다.
발명의 기술분야
본 발명은 일반적으로 유전적 분석에 관한 것이며, 더욱 구체적으로는 대상체에서 암을 검출하고/검출하거나 대상체의 전체 생존을 평가하기 위한 무세포 DNA(cfDNA) 단편의 분석을 위한 방법 및 시스템에 관한 것이다.
전 세계적으로 인간 암의 이환율과 사망률의 대부분은 치료가 덜 효과적인 이들 질병의 늦은 진단의 결과이다. 불행하게도, 조기 암 환자를 광범위하게 진단하고 치료하는 데 사용될 수 있는 임상적으로 입증된 바이오마커는 널리 이용 가능하지 않다.
무세포 DNA(cfDNA) 분석은 이러한 접근법이 조기 진단 및 치료를 위한 새로운 길을 제공할 수 있음을 시사한다. 순환 종양 DNA(ctDNA) 단편은 비종양 세포로부터의 다른 cfDNA보다 평균적으로 더 짧은 것으로 나타났다. 기존의 작업에서는 단편을 히스톤 코어 또는 링커 단백질(예를 들어, 짧고 긴 또는 상호 배타적인 크기 세트)에 결합하여 발생하는 상이한 크기의 군으로 분리하고 이들 단편의 수를 사용하여 ctDNA를 정량화 하고/하거나 개별 샘플을 종양의 존재/부재로 분류하여 탐구하였다. 그러나 기존의 연구들은 암 진단을 받은 환자의 전체 생존을 결정하는 능력뿐만 아니라 암 검출에 있어 강력한 민감도 및 특이성을 제공하는 능력이 부족하였다.
본 개시는 대상체로부터 얻은 샘플에서 cfDNA 단편의 분석을 통해 얻은 cfDNA 단편화 프로파일을 점수화함으로써 대상체의 전체 생존을 검출하고 예측하기 위한 cfDNA 분석을 활용하는 방법 및 시스템을 제공한다. 점수화 방법론은 대상체의 전체 생존 가능성의 척도를 제공한다.
따라서, 일 구현예에서, 본 발명은 대상체에서 암을 검출하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계로서, cfDNA 단편화 프로파일은 다음에 의해 결정된다:
대상체로부터의 cfDNA 단편을 얻어 분리하는 단계,
cfDNA 단편을 서열 분석하여 서열 분석된 단편을 얻는 단계,
서열 분석된 단편을 게놈에 매핑하여 매핑된 서열의 윈도우를 얻는 단계, 및
매핑된 서열의 윈도우를 분석하여 cfDNA 단편 길이를 결정하고 cfDNA 단편화 프로파일을 생성하는 단계; 및
b) cfDNA 단편화 프로파일을 기반으로 점수를 계산하여 대상체에서 암의 존재 가능성을 나타내는 점수를 계산하여 대상체를 암이 있거나 암이 없는 것으로 분류함으로써 대상체에서 암을 검출하는 단계. 일부 양태에서, 암은 폐암을 제외한다. 일부 양태에서, 화학치료요법제, 방사선, 면역치료요법 또는 기타 치료적 요법이 대상체에게 투여된다.
일부 양태에서, 점수를 계산하는 단계는 다음을 포함한다: i) 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔에 의한 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계.
또 다른 구현예에서, 본 발명은 암이 있는 대상체의 전체 생존을 결정하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계;
b) cfDNA 단편화 프로파일을 기반으로 점수를 계산하는 단계로서, 여기서 점수를 계산하는 단계는 다음을 포함한다: i) 샘플의 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔에 의해 샘플의 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계; 및
c) 점수를 기반으로 대상체의 전체 생존 가능성을 결정함으로써 대상체의 전체 생존을 결정하는 단계.
또 다른 양태에서, 본 발명은 암이 있는 대상체를 치료하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 본 발명의 방법론을 사용하여 대상체에서 암을 검출하거나, 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정하는 단계; 및
b) 대상체에게 암 치료를 투여하여 대상체를 치료하는 단계. 일부 양태에서, 화학치료요법제, 방사선, 면역치료요법 또는 기타 치료적 요법이 대상체에게 투여된다.
또 다른 구현예에서, 본 발명은 대상체에서 암을 모니터링하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 본 발명의 방법론을 사용하여 대상체에서 암을 검출하고/검출하거나 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정하는 단계;
b) 대상체에게 암 치료를 투여하는 단계; 및
c) 암 치료가 투여된 후 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정함으로써 대상체에서 암을 모니터링하는 단계. 일부 양태에서, 화학치료요법제, 방사선, 면역치료요법 또는 기타 치료적 요법이 대상체에게 투여된다.
또 다른 구현예에서, 본 발명은 컴퓨터 프로그램으로 암호화된 비일시적 컴퓨터 판독 가능 저장 매체를 제공한다. 컴퓨터 프로그램은 하나 이상의 프로세서에 의해 실행될 때 하나 이상의 프로세서가 본 발명의 방법을 수행하기 위한 동작을 수행하게 하는 명령을 포함한다.
또 다른 구현예에서, 본 발명은 컴퓨팅 시스템을 제공한다. 시스템은 메모리, 및 메모리에 연결된 하나 이상의 프로세서를 포함하며, 하나 이상의 프로세서는 본 발명의 방법을 구현하는 동작을 수행하도록 구성된다.
또 다른 구현예에서, 본 발명은 유전적 분석 및 암을 평가하기 위한 시스템을 제공하며 이는 다음을 포함한다: (a) 샘플에 대한 전체 게놈 서열 분석(WGS) 데이터 세트를 생성하도록 구성된 서열 분석기; 및 (b) 비일시적 컴퓨터 판독 가능 저장 매체 및/또는 본 발명의 컴퓨터 시스템.
도 1 은 본 발명의 일 구현예에서 본 개시의 방법론을 사용하는 예시적인 DELFI 접근법을 도시하는 개략도이다. 혈액은 건강한 개인과 암이 있는 환자 코호트로부터 수집된다. cfDNA는 혈장 분획에서 추출되어 서열 분석 라이브러리로 처리되고, 전체 게놈 서열 분석을 통해 검사되고, 게놈에 매핑되고, 분석되어 게놈 전체에 걸쳐 cfDNA 단편화 프로필을 결정한다. 기계 학습 접근법을 사용하여 DELFI 점수를 생성하고 개인을 건강한 또는 암이 있는 것으로 분류한다.
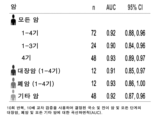
도 2는 암의 비침습적 검출을 위한 cfDNA 단편화 검정의 성과를 나타내는 표이다. 포함 후 3개월 이내에 74명의 환자가 16개의 상이한 고형암 중 1개로 진단받았고, 207명의 환자는 암이 없었다.
도 3은 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 그래프는 암 검출을 위한 cfDNA 단편화 검정의 전체 성과를 나타낸다.
도 4는 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 그래프는 DELFI 점수와 상관관계가 있는 대상체의 생존을 나타낸다. DELFI 점수가 높을수록 암 병기나 기타 임상적 특성과 관계없이 감소된 전체 생존과 연관이 있었다.
도 5는 본 발명의 일 구현예에서 개시된 방법론을 사용하여 생성된 데이터 곡선을 나타내는 일련의 그래픽 플롯이다. 계산된 DELFI 점수는 높은 점수(>0.5) 대 낮은 점수(<0.5)를 정의하는 데 사용된 컷오프 값에 관계없이 암(폐암 제외)이 있는 개인의 묘사된 카플란-마이어 곡선을 분리한다. 각 패널 상단의 숫자는 결정된 컷오프 값을 가리킨다.
도 6은 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 도 6은 두 가지 설정에서 콕스 비례 위험 모델의 결과를 나타낸다. 첫 번째 설정(플롯의 왼쪽 패널)에서 DELFI 점수는 연속적으로 처리된다. 두 번째 설정(플롯의 오른쪽 패널)에서 DELFI 점수는 높음(>0.5) 또는 낮음(<0.5)으로 처리된다. 두 설정 모두에서 DELFI 점수는 채혈 및 병기에서 나이를 조정하더라도 생존에 대한 강력한 예측 변수이다. 병기는 1기에 관한 것임을 유념한다.
도 2는 암의 비침습적 검출을 위한 cfDNA 단편화 검정의 성과를 나타내는 표이다. 포함 후 3개월 이내에 74명의 환자가 16개의 상이한 고형암 중 1개로 진단받았고, 207명의 환자는 암이 없었다.
도 3은 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 그래프는 암 검출을 위한 cfDNA 단편화 검정의 전체 성과를 나타낸다.
도 4는 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 그래프는 DELFI 점수와 상관관계가 있는 대상체의 생존을 나타낸다. DELFI 점수가 높을수록 암 병기나 기타 임상적 특성과 관계없이 감소된 전체 생존과 연관이 있었다.
도 5는 본 발명의 일 구현예에서 개시된 방법론을 사용하여 생성된 데이터 곡선을 나타내는 일련의 그래픽 플롯이다. 계산된 DELFI 점수는 높은 점수(>0.5) 대 낮은 점수(<0.5)를 정의하는 데 사용된 컷오프 값에 관계없이 암(폐암 제외)이 있는 개인의 묘사된 카플란-마이어 곡선을 분리한다. 각 패널 상단의 숫자는 결정된 컷오프 값을 가리킨다.
도 6은 본 발명의 일 구현예에서 본 개시의 방법론을 사용하여 생성된 데이터를 나타내는 그래픽 플롯이다. 도 6은 두 가지 설정에서 콕스 비례 위험 모델의 결과를 나타낸다. 첫 번째 설정(플롯의 왼쪽 패널)에서 DELFI 점수는 연속적으로 처리된다. 두 번째 설정(플롯의 오른쪽 패널)에서 DELFI 점수는 높음(>0.5) 또는 낮음(<0.5)으로 처리된다. 두 설정 모두에서 DELFI 점수는 채혈 및 병기에서 나이를 조정하더라도 생존에 대한 강력한 예측 변수이다. 병기는 1기에 관한 것임을 유념한다.
암의 조기 검출뿐만 아니라 암이 있는 대상체의 전체 생존의 예측을 위한 비침습적 방법이 본원에 설명되어 있다. 혈액 내 cfDNA는 암 환자에게 비침습적 진단 방안을 제공할 수 있다. 본원에서 입증된 바와 같이, 조기 차단을 위한 단편의 DNA 감정(DELFI)을 사용하여 다양한 유형의 암 환자뿐만 아니라 건강한 개인의 cfDNA의 게놈 전체 단편화 패턴을 감정하였다. cfDNA 감정에는 점수화 방법론이 포함되었다. 전체 생존과 상관관계가 있는 주어진 환자 샘플의 cfDNA 단편을 사용하여 얻은 cfDNA 단편화 프로파일에 대해 정의된 점수(본원에서는 'DELFI 점수'라고도 지칭됨)를 결정하였다. 본원에 설명된 방법론을 사용하여 cfDNA를 평가하면 암의 조기 발견 및 평가를 위한 검진 접근법을 제공할 수 있으며, 이는 암이 있는 환자의 성공적인 치료 기회를 증가시킬 수 있다. cfDNA 평가는 또한 암 모니터링을 위한 접근법을 제공할 수 있으며, 이는 성공적인 치료 및 암이 있는 환자의 개선된 결과를 위한 기회를 증가시킬 수 있다.
본 조성물 및 방법을 설명하기 전에, 본 발명은 설명된 특정 방법 및 시스템에 제한되지 않으며, 이러한 방법 및 시스템은 다양할 수 있다는 것이 이해되어야 한다. 본원에서 사용된 전문 용어는 단지 특정 구현예를 설명하기 위한 것이며, 제한하려는 의도가 아닌 것으로 또한 이해되어야 하는데, 이는 본 발명의 범위가 첨부된 청구항에만 제한될 것이기 때문이다.
본 명세서 및 첨부된 청구항에 사용된 바와 같이, 단수형 "a", "an" 및 "the"는 문맥상 명백하게 달리 지시하지 않는 한 복수 지칭을 포함한다. 따라서, 예를 들어, "방법"에 대한 지칭은 본 개시 등을 읽을 때 당업자에게 명백해질 본원에 설명된 유형의 하나 이상의 방법 및/또는 단계를 포함한다.
다르게 정의되지 않는 한, 본원에 사용된 모든 기술 및 과학 용어는 본 발명이 속하는 기술 분야의 숙련자가 일반적으로 이해하는 것과 동일한 의미를 갖는다. 본원에 설명된 것과 유사하거나 등가인 임의의 방법 및 재료가 본 발명의 실시 또는 테스트에 사용될 수 있지만, 이제 바람직한 방법 및 재료가 설명된다.
본 개시는 암을 검출하거나 평가하기 위한 cfDNA의 분석을 위한 혁신적인 방법 및 시스템을 제공한다. 선행 연구에서 가리킨 바와 같이, 평균적으로 암이 없는 개인은 더 긴 cfDNA 단편(평균 크기 167.09bp)을 갖는 반면, 암이 있는 개인은 더 짧은 cfDNA 단편(평균 크기 164.88bp)을 갖는다. 본원에 설명된 방법론은 cfDNA 단편화 패턴의 게놈 전체 분석을 통해 cfDNA의 수많은 이상의 동시 분석을 가능케한다.
따라서, 일 구현예에서, 본 발명은 대상체에서 암을 검출하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계; 및
b) cfDNA 단편화 프로파일을 기반으로 대상체에서 암의 존재 가능성을 나타내는 점수를 계산하여 대상체를 암이 있거나 암이 없는 것으로 분류함으로써, 단 암은 폐암을 포함하지 않는다는 조건으로, 대상체에서 암을 검출하는 단계.
또 다른 구현예에서, 본 발명은 암이 있는 대상체의 전체 생존을 결정하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계;
b) cfDNA 단편화 프로파일을 기반으로 점수를 계산하는 단계로서, 여기서 점수를 계산하는 단계는 다음을 포함한다: i) 샘플의 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔에 의해 샘플의 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계; 및
c) 점수를 기반으로 대상체의 전체 생존 가능성을 결정함으로써 대상체의 전체 생존을 결정하는 단계.
구현예에서, 본 발명은 암이 있는 대상체를 치료하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 본 발명의 방법론을 사용하여 대상체에서 암을 검출하거나, 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정하는 단계; 및
b) 대상체에게 암 치료를 투여하여 대상체를 치료하는 단계. 일부 양태에서, 화학치료요법제, 방사선, 면역치료요법 또는 기타 치료적 요법이 대상체에게 투여된다.
또 다른 구현예에서, 본 발명은 대상체에서 암을 모니터링하는 방법을 제공한다. 방법은 다음을 포함한다:
a) 본 발명의 방법론을 사용하여 대상체에서 암을 검출하거나 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정하는 단계;
b) 대상체에게 암 치료를 투여하는 단계; 및
c) 암 치료가 투여된 후 본 발명의 방법론을 사용하여 대상체의 전체 생존을 결정함으로써 대상체에서 암을 모니터링하는 단계.
본원에 설명된 방법론은 cfDNA 단편화 프로파일을 활용한다. 본원에 사용된 바와 같이, 용어 "단편화 프로파일"은 일부 양태에서, 포유동물에서 cfDNA 단편화 프로파일을 결정하는 것은 포유동물이 암이 있는 것으로 식별하는 데 사용될 수 있다. 예를 들어, 포유동물로부터 얻은 cfDNA 단편(예를 들어, 포유동물로부터 얻은 샘플로부터)은 낮은 적용 범위의 전체 게놈 서열 분석을 적용 받을 수 있으며, 서열 분석된 단편은 게놈에 매핑될 수 있고(예를 들어, 비중첩 윈도우에서) cfDNA 단편화 프로파일을 결정하기 위해 평가될 수 있다. 암이 있는 포유동물의 cfDNA 단편화 프로파일은 건강한 포유동물(예를 들어, 암이 없는 포유동물)의 cfDNA 단편화 프로파일보다 더 이질적이다(예를 들어, 단편 길이).
cfDNA 단편화 프로파일에는 하나 이상의 cfDNA 단편화 패턴이 포함될 수 있다. cfDNA 단편화 패턴은 임의의 적절한 cfDNA 단편화 패턴을 포함할 수 있다. cfDNA 단편화 패턴의 예에는 단편 크기 밀도, 중간 단편 크기, 단편 크기 분포, 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율 및 cfDNA 단편의 적용 범위가 비제한적으로 포함된다. 일부 양태에서, cfDNA 단편화 프로파일은 게놈 전체 cfDNA 프로파일(예를 들어, 게놈 전체 윈도우의 게놈 전체 cfDNA 프로파일)일 수 있다. 일부 양태에서, cfDNA 단편화 프로파일은 표적화 영역 프로파일일 수 있다. 표적화 영역은 게놈의 임의의 적절한 부분(예를 들어 염색체 영역)일 수 있다. cfDNA 단편화 프로파일이 본원에 설명된 바와 같이 결정될 수 있는 염색체 영역의 예에는 염색체의 부분(예를 들어, 2q, 4p, 5p, 6q, 7p, 8q, 9q, 10q, 11q, 12q 및/또는 14q의 부분) 및 염색체 팔(예를 들어, 8q, 13q, 11q 및/또는 3p의 염색체 팔)을 비제한적으로 포함한다. 어떤 경우에는, cfDNA 단편화 프로파일에 두 개 이상의 표적화 영역 프로파일이 포함될 수 있다.
다양한 양태에서, 샘플에서 얻은 cfDNA가 분리되고 특정 크기 범위의 단편이 분석에 활용된다. 일부 양태에서, 분석에서는 약 10, 50, 100 또는 105bp 미만 및 약 220, 250, 300, 350bp 이상의 단편 크기를 제외한다. 일부 양태에서, 분석에서는 105bp 미만 및 170bp 초과의 단편 크기를 제외한다. 일부 양태에서, 분석에서는 약 230, 240, 250, 260bp 미만 및 약 420, 430, 440, 450bp 이상의 단편 크기를 제외한다. 일부 양태에서 분석에서는 260bp 미만 및 440bp 초과의 단편 크기를 제외한다.
일부 양태에서, cfDNA 단편화 프로파일은 다음에 의해 결정될 수 있다: cfDNA 단편을 포함하는 대상체로부터의 샘플을 서열 분석 라이브러리로 처리하는 단계; 서열 분석 라이브러리를 낮은 적용 범위의 전체 게놈 서열 분석에 적용하여 서열 분석된 단편을 얻는 단계; 서열 분석된 단편을 게놈에 매핑하여 매핑된 서열의 윈도우를 얻는 단계; 및 매핑된 서열의 윈도우를 분석하여 cfDNA 단편 길이를 결정하는 단계를 포함한다.
일부 양태에서, cfDNA 단편화 프로파일은 다음에 의해 결정될 수 있다: 대상체로부터 cfDNA 단편을 얻고 분리하는 단계, cfDNA 단편을 서열 분석하여 서열 분석된 단편을 얻는 단계, 서열 분석된 단편을 게놈에 매핑하여 매핑된 서열의 윈도우를 얻는 단계, 매핑된 서열의 윈도우를 분석하여 cfDNA 단편 길이를 결정하고 cfDNA 단편화 프로파일을 생성하는 단계.
본 발명의 방법론은 낮은 적용 범위의 전체 게놈 서열 분석 및 분리된 cfDNA의 분석을 기반으로 한다. 일 양태에서, 본 발명의 방법론을 개발하는 데 사용되는 데이터는 얕은 전체 게놈 서열 데이터(1-2x 적용 범위)를 기반으로 한다.
일부 양태에서, 매핑된 서열은 게놈이 적용되는 비중첩 윈도우에서 분석된다. 개념적으로 윈도우는 크기가 수천에서 수백만 염기 범위에 이를수 있어, 게놈에 수백에서 수천 개의 윈도우를 야기할 수 있다. 5Mb 윈도우는 cfDNA 단편화 패턴을 감정하는 데 사용되고, 이는 제한된 양의 1-2x 게놈 적용 범위에서도 윈도우 당 20,000개 초과의 판독을 제공하기 때문이다. 각 윈도우 내에서, cfDNA 단편의 적용 범위와 크기 분포를 검사하였다. 일부 양태에서, 개인으로부터의 전체 게놈 패턴을 기준 집단과 비교하여 패턴이 건강한 것인지 또는 암에서 유래될 가능성이 있는 것인지를 결정할 수 있다.
특정 양태에서, 매핑된 서열은 수십 내지 수천 개의 게놈 윈도우, 예컨대 10, 50, 100 내지 1,000, 5,000, 10,000개 이상의 윈도우를 포함한다. 이러한 윈도우는 비중첩되거나 중첩될 수 있으며 약 1, 2, 3, 4, 5, 6, 7, 8, 9백만 또는 1천만 개의 염기쌍을 포함할 수 있다.
다양한 양태에서, cfDNA 단편화 프로파일은 각 윈도우 내에서 결정된다. 따라서, 본 발명은 대상체에서(예를 들어, 대상체로부터 얻은 샘플에서) cfDNA 단편화 프로파일을 결정하기 위한 방법을 제공한다.
일부 양태에서, cfDNA 단편화 프로파일은 cfDNA 단편 길이의 변화(예를 들어 변경)를 식별하는 데 사용될 수 있다. 변경은 게놈 전체 변경이거나 하나 이상의 표적화 영역/좌위의 변경일 수 있다. 표적 영역은 하나 이상의 암 특이적 변경을 함유하는 임의의 영역일 수 있다. 일부 양태에서, cfDNA 단편화 프로파일은 약 10개 변경 내지 약 500개 변경(예를 들어, 약 25개 내지 약 500개, 약 50개 내지 약 500개, 약 100개 내지 약 500개, 약 200개 내지 약 500개, 약 300개 내지 약 500개, 약 10개 내지 약 400개, 약 10개 내지 약 300개, 약 10개 내지 약 200개, 약 10개 내지 약 100개, 약 10개 내지 약 50개, 약 20개 내지 약 400개, 약 30개 내지 약 300개, 약 40개 내지 약 200개, 약 50개 내지 약 100개, 약 20개 내지 약 100개, 약 25개 내지 약 75개, 약 50개 내지 약 250개 또는 약 100개 내지 약 200개의 변경)을 식별(예를 들어, 동시에 식별)하는 데 사용될 수 있다.
다양한 양태에서, cfDNA 단편화 프로파일은 cfDNA 단편 크기 패턴을 포함할 수 있다. cfDNA 단편은 임의의 적절한 크기일 수 있다. 예를 들어, 일부 양태에서, cfDNA 단편은 길이로 약 50개 염기쌍(bp) 내지 약 400bp일 수 있다. 본원에 설명된 바와 같이, 암이 있는 대상체는 건강한 대상체의 중앙 cfDNA 단편 크기보다 더 짧은 중앙 cfDNA 단편 크기를 함유하는 cfDNA 단편 크기 패턴을 가질 수 있다. 건강한 대상체(예를 들어, 암이 없는 대상체)는 약 166.6bp 내지 약 167.2bp(예를 들어, 약 166.9bp)의 중앙 cfDNA 단편 크기를 갖는 cfDNA 단편 크기를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 건강한 대상체에서 cfDNA 단편 크기보다 평균적으로 약 1.28bp 내지 약 2.49bp(예를 들어, 약 1.88bp) 더 짧은 cfDNA 단편 크기를 가질 수 있다. 예를 들어, 암이 있는 대상체는 약 164.11bp 내지 약 165.92bp(예를 들어, 약 165.02bp)의 중앙 cfDNA 단편 크기를 갖는 cfDNA 단편 크기를 가질 수 있다.
일부 양태에서, 디뉴클레오솜 cfDNA 단편은 길이로 약 230 염기쌍(bp) 내지 약 450bp일 수 있다. 본원에 설명된 바와 같이, 암이 있는 대상체는 건강한 대상체의 중앙 디뉴클레오솜 cfDNA 단편 크기보다 더 짧은 중앙 디뉴클레오솜 cfDNA 단편 크기를 함유하는 디뉴클레오솜 cfDNA 단편 크기 패턴을 가질 수 있다. 일부 양태에서, 평균적으로, 암이 없는 대상체는 디뉴클레오솜 범위(평균 크기 334.75bp)에서 더 긴 cfDNA 단편을 갖는 반면, 암이 있는 대상체는 더 짧은 디뉴클레오솜 cfDNA 단편(평균 크기 329.6bp)을 갖는다. 따라서, 건강한 대상체(예를 들어, 암이 없는 대상체)는 약 334.75bp의 중앙 cfDNA 단편 크기를 갖는 디뉴클레오솜 cfDNA 단편 크기를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 건강한 대상체의 디뉴클레오솜 cfDNA 단편 크기보다 더 짧은 디뉴클레오솜 cfDNA 단편 크기를 가질 수 있다. 예를 들어, 암이 있는 대상체는 중앙 cfDNA 단편 크기가 약 329.6bp인 디뉴클레오솜 cfDNA 단편 크기를 가질 수 있다.
cfDNA 단편화 프로파일에는 cfDNA 단편 크기 분포가 포함될 수 있다. 본원에 설명된 바와 같이, 암이 있는 대상체는 건강한 대상체의 cfDNA 단편 크기 분포보다 더 가변적인 cfDNA 크기 분포를 가질 수 있다. 일부 양태에서, 크기 분포는 표적화 영역 내에 있을 수 있다. 건강한 대상체(예를 들어, 암이 없는 대상체)는 약 1 또는 약 1 미만의 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 건강한 대상체의 표적화 영역 cfDNA 단편 크기 분포보다 더 긴(예를 들어, 10, 15, 20, 25, 30, 35, 40, 45, 50bp 이상 더 길거나 이들 숫자 사이의 임의의 염기쌍 수) 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 건강한 대상체의 표적화 영역 cfDNA 단편 크기 분포보다 더 짧은(예를 들어, 10, 15, 20, 25, 30, 35, 40, 45, 50bp 이상 더 짧거나 이들 숫자 사이의 임의의 염기쌍 수) 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 건강한 대상체의 표적화 영역 cfDNA 단편 크기 분포보다 약 47bp 더 작거나 약 30bp 더 긴 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 cfDNA 단편의 길이가 평균적으로 10, 11, 12, 13, 14, 15, 15, 17, 18, 19, 20bp 이상 차이인 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 예를 들어, 암이 있는 대상체는 평균적으로 cfDNA 단편의 길이가 약 13bp 차이인 표적화 영역 cfDNA 단편 크기 분포를 가질 수 있다. 일부 양태에서, 크기 분포는 게놈 전체 크기 분포일 수 있다.
cfDNA 단편화 프로파일에는 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율과 단편 비율 대 기준 단편 비율의 상관관계가 포함될 수 있다. 본원에 사용된 바와 같이, 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율과 관련하여, 작은 cfDNA 단편은 길이가 약 100bp 내지 약 150bp일 수 있다. 본원에 사용된 바와 같이, 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율과 관련하여, 큰 cfDNA 단편은 길이가 약 151bp 내지 220bp일 수 있다. 본원에 설명된 바와 같이, 암이 있는 대상체는 건강한 대상체보다 더 낮은(예를 들어, 2배 낮음, 3배 낮음, 4배 낮음, 5배 낮음, 6배 낮음, 7배 낮음, 8배 낮음, 9배 낮음, 10배 낮음 또는 그 이상) 단편 비율의 상관관계(예를 들어, cfDNA 단편 비율 대 기준 DNA 단편 비율, 예컨대 한 명 이상의 건강한 대상체로부터의 DNA 단편 비율의 상관관계)를 가질 수 있다. 건강한 대상체(예를 들어, 암이 없는 대상체)는 약 1(예를 들어, 약 0.96)의 단편 비율의 상관관계(예를 들어, cfDNA 단편 비율 대 기준 DNA 단편 비율, 예컨대 한 명 이상의 건강한 대상체로부터의 DNA 단편 비율의 상관관계)를 가질 수 있다. 일부 양태에서, 암이 있는 대상체는 단편 비율의 상관관계(예를 들어, cfDNA 단편 비율 대 기준 DNA 단편 비율, 예컨대 한 명 이상의 건강한 대상체로부터의 DNA 단편 비율의 상관관계)를 가질 수 있으며, 즉 이는 평균적으로 건강한 대상체의 단편 비율의 상관관계(예를 들어, cfDNA 단편 비율 대 기준 DNA 단편 비율, 예컨대 한 명 이상의 건강한 대상체로부터의 DNA 단편 비율의 상관관계) 보다 약 0.19 내지 약 0.30(예를 들어, 약 0.25) 더 낮다.
본 발명의 방법론은 cfDNA 단편화 프로파일을 기반으로 점수(예를 들어, DELFI 점수)를 계산하는 단계를 추가로 포함한다. 일부 양태에서, 점수를 계산하는 단계는 다음을 포함한다: i) 샘플의 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔에 의해 샘플의 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계. 다양한 양태에서, 점수는 대상체의 전체 생존 가능성을 결정하는 데 활용된다.
하나의 예시적인 예(실시예 1)에서, 다중 암 코호트에서, 발명자들은 각 개인의 5MB 빈에 의한 짧은 단편 대 긴 단편의 비율, 염색체 팔에 의한 Z-점수 및 cfDNA 단편 크기의 혼합 모델을 서열 분석하는 낮은 적용 범위의 전체 게놈으로부터 계산하였다. 이러한 특징을 입력으로 사용하여, 발명자들은 교차 검증된 경사 증폭 기계를 각 사람의 암 상태(암/암 없음)에 맞춘다. 이 모델의 출력은 0에서 1의 범위의 점수이고, 숫자가 높을수록 암의 신호가 강하고 숫자가 낮을수록 비암의 신호와 유사하다는 것을 가리킨다. 완료되면 암으로 진단된 샘플만 유지된다.
일부 양태에서, 출력된 점수를 다음과 같이 분석한다. 추적 시간, 추적 종료 시 환자의 생존 여부, 상기 기계 학습 모델로부터의 점수를 이용하여, cfDNA 단편화와 생존의 관계를 판단하였다. 도 5에서 나타나 있듯이, 카플란-마이어 곡선에서 암이 있는 개인에서 높은 점수 대 낮은 점수로 강력한 분리가 결정되었다. 추가적으로, 이 점수와 기타 임상 특징의 독립성은 콕스 비례 위험 모델을 맞추고 점수, 암 병기 및 환자 나이에 대한 회귀를 통해 평가되었다.
도 5를 참조하면 상기에서 논의된 바와 같이, 계산된 DELFI 점수는 높은 점수(>0.5) 대 낮은 점수(<0.5)를 정의하는 데 사용된 컷오프 값에 관계없이 암(폐암 제외)이 있는 개인의 묘사된 카플란-마이어 곡선을 분리한다. 각 패널 상단의 숫자는 결정된 컷오프 값을 가리킨다.
도 6은 두 가지 설정에서 콕스 비례 위험 모델의 결과를 나타낸다. 첫 번째 설정(플롯의 왼쪽 패널)에서, DELFI 점수는 연속적으로 처리된다. 두 번째 설정(플롯의 오른쪽 패널)에서 DELFI 점수는 높음(>0.5) 또는 낮음(<0.5)으로 처리된다. 두 설정 모두에서, DELFI 점수는 채혈 및 병기에서 나이를 조정하더라도 생존에 대한 강력한 예측 변수이다. 병기는 1기에 관한 것임을 유념한다.
현재 설명된 방법 및 시스템은 대상체의 암 상태를 검출하고, 예측하고, 치료하고/치료하거나 모니터링하는 데 유용하다. 임의의 적절한 대상체, 예컨대 포유동물은 본원에 설명된 바와 같이 평가되고, 모니터링되고/모니터링되거나 치료될 수 있다. 본원에 설명된 바와 같이 평가되고, 모니터링되고/모니터링되거나 치료될 수 있는 일부 포유동물의 예에는 인간, 영장류, 예컨대 원숭이, 개, 고양이, 말, 소, 돼지, 양, 마우스 및 래트가 비제한적으로 포함된다. 예를 들어, 암이 있거나 암이 의심되는 인간은 본원에 설명된 방법을 사용하여 평가될 수 있고, 선택적으로 본원에 설명된 바와 같은 하나 이상의 암 치료로 치료될 수 있다.
임의의 적절한 유형의 암이 있거나 암이 있는 것으로 의심되는 대상체는 본원에 설명된 방법 및 시스템을 사용하여 평가되고/평가되거나 치료될 수 있다(예를 들어, 대상체에게 하나 이상의 암 치료 투여에 의함). 암은 임의의 병기의 암일 수 있다. 일부 양태에서, 암은 조기 암일 수 있다. 일부 양태에서, 암은 무증상 암일 수 있다. 일부 양태에서, 암은 잔여 질병 및/또는 재발(예를 들어, 외과적 절제 후 및/또는 암 치료요법 후)일 수 있다. 암은 임의의 유형의 암일 수 있다. 본원에 설명된 바와 같이 평가되고, 모니터링되고/모니터링되거나 치료될 수 있는 암 유형의 예에는 폐암, 대장암, 전립선암, 유방암, 췌장암, 담관암, 간암, CNS, 위암, 식도암, 위장관 간질 종양(GIST), 자궁암 및 난소암이 비제한적으로 포함된다. 추가적인 유형의 암에는 골수종, 다발성 골수종, B세포 림프종, 여포성 림프종, 림프구성 백혈병, 백혈병 및 골수성 백혈병이 비제한적으로 포함된다. 일부 양태에서, 암은 고형 종양이다. 일부 양태에서, 암은 육종, 암종 또는 림프종이다. 일부 양태에서, 암은 폐암, 대장암, 전립선암, 유방암, 췌장암, 담관암, 간암, CNS, 위암, 식도암, 위장관 간질 종양(GIST), 자궁암 또는 난소암이다. 일부 양태에서, 암은 혈액암이다. 일부 양태에서, 암은 골수종, 다발성 골수종, B 세포 림프종, 여포성 림프종, 림프구성 백혈병, 백혈병 또는 골수성 백혈병이다.
본원에 설명된 바와 같이 암이 있거나 암이 의심되는 대상체를 치료할 때, 대상체에게는 하나 이상의 암 치료가 투여될 수 있다. 암 치료는 임의의 적절한 암 치료일 수 있다. 본원에 설명된 하나 이상의 암 치료는 임의의 적절한 빈도(예를 들어, 며칠 내지 몇 주 범위의 기간에 걸쳐 1회 또는 여러 번)로 대상체에게 투여될 수 있다. 암 치료의 예는 외과적 개입, 보조 화학치료요법, 신보조 화학치료요법, 방사선 치료요법, 호르몬 치료요법, 세포독성 치료요법, 면역치료요법, 입양 T 세포 치료요법(예를 들어, 키메라 항원 수용체 및/또는 야생형 또는 변형된 T 세포 수용체를 갖는 T 세포), 표적화 치료요법, 예컨대 키나제 저해제(예를 들어, 특정 유전적 병변, 예컨대 전좌 또는 돌연변이를 표적으로 하는 키나제 저해제)의 투여(예를 들어, 키나제 저해제, 항체, 이중특이적 항체), 신호 전달 저해제, 이중특이적 항체 또는 항체 단편(예를 들어, BiTE), 단클론 항체, 면역 체크포인트 저해제, 수술(예를 들어, 수술적 절제) 또는 상기의 임의의 조합을 비제한적으로 포함한다. 일부 양태에서, 암 치료는 암의 중증도를 저감시키고, 암의 증상을 저감시키며/저감시키거나 대상체 내에 존재하는 암 세포의 수를 저감시킬 수 있다.
일부 양태에서, 암 치료는 화학치료요법제일 수 있다. 화학치료요법제의 비제한적 예에는 다음이 포함된다: 암사크린, 아자시티딘, 악사티오프린, 베바시주맙(또는 이의 항원 결합 단편), 블레오마이신, 부술판, 카르보플라틴, 카페시타빈, 클로람부실, 시스플라틴, 시클로포스파미드, 시타라빈, 다카르바진, 다우노루비신, 도세탁셀, 독시플루리딘, 독소루비신, 에피루비신, 에를로티닙 하이드로클로라이드, 에토포시드, 피우다라빈, 플록수리딘, 플루다라빈, 플루오로우라실, 젬시타빈, 하이드록시우레아, 이다루비신, 이포스파미드, 이리노테칸, 로무스틴, 메클로레타민, 멜팔란, 메르캅토퓨린, 메토트렉세이트, 미토마이신, 미톡산트론, 옥살리플라틴, 파클리탁셀, 페메트렉스드, 프로카바진, 전 트랜스 레티노산, 스트렙토조신, 타플루포시드, 테모졸로미드, 테니포시드, 티오구아닌, 토포테칸, 우라무스틴, 발루비신, 빈블라스틴, 빈크리스틴, 빈데신, 비노렐빈 및 이들의 조합. 항암 치료요법의 추가적인 예는 당업계에 알려져 있다; 예를 들어 미국임상종양학회(ASCO), 유럽종양학회(ESMO) 또는 국립종합암네트워크(NCCN)의 치료요법 지침을 참조한다.
본원에 설명된 바와 같이 암이 있거나 암이 의심되는 대상체를 모니터링할 때, 모니터링은 암 치료 과정 전, 도중 및/또는 후일 수 있다. 본원에 제공된 모니터링 하는 방법은 하나 이상의 암 치료의 효능을 결정하고/결정하거나 증가된 모니터링을 위한 대상체를 선택하는 데 사용될 수 있다.
일부 양태에서, 모니터링은 하나 이상의 암 치료(예를 들어, 하나 이상의 암 치료의 효능)를 모니터링할 수 있는 기존 기술을 포함할 수 있다. 일부 양태에서, 증가된 모니터링을 위해 선택된 대상체는 증가된 모니터링을 위해 선택되지 않은 대상체에 비해 증가된 빈도로 진단 테스트(예를 들어, 본원에 개시된 진단 테스트 중 임의의 것)를 투여받을 수 있다. 예를 들어, 증가된 모니터링을 위해 선택된 대상체는 매일 2회, 매일, 격주, 매주, 격월, 월간, 분기별, 반년마다, 매년 또는 그 중 임의의 빈도로 진단 테스트가 투여될 수 있다.
다양한 양태에서, DNA는 대상체로부터 채취한 생물학적 샘플에 존재하며 본 발명의 방법론에 사용된다. 생물학적 샘플은 DNA를 포함하는 사실상 임의의 유형의 생물학적 샘플일 수 있다. 생물학적 샘플은 전형적으로 유체, 예컨대 전혈 또는 순환하는 cfDNA가 있는 그의 부분이다. 구현예에서, 샘플에는 종양 또는 액체 생검, 예컨대 비제한적으로 양수, 방수, 유리체액, 혈액, 전혈, 분획 혈액, 혈장, 혈청, 모유, 뇌척수액(CSF), 귀에지(귀지), 유미, 미즙, 내림프, 외림프, 대변, 호흡, 위산, 위액, 림프, 점액(비강 배액 및 가래 포함), 심낭액, 복막액, 흉막액, 고름, 점막 분비물, 타액, 호기 호흡 응축물, 피지, 정액, 가래, 땀, 윤활액, 눈물, 토사물, 전립선액, 유두 흡인액, 눈물액, 땀, 뺨 면봉, 세포 용해물, 위장액, 생검 조직 및 소변 또는 기타 생물학적 체액으로부터의 DNA를 포함한다. 일 양태에서, 샘플은 순환 종양 세포로부터의 DNA를 포함한다.
상기 개시된 바와 같이, 생물학적 샘플은 혈액 샘플일 수 있다. 혈액 샘플은 당업계에 알려진 방법, 예컨대 손가락 찌르기 또는 정맥절개술을 사용하여 얻을 수 있다. 적합하게는, 혈액 샘플은 대략 0.1 내지 20ml, 또는 대안적으로 대략 1 내지 15ml이고 혈액 부피는 대략 10ml이다. 혈액 내 순환하는 유리 DNA뿐만 아니라 소량도 사용될 수 있다. 바늘 생검, 카테터, 배설 또는 DNA를 함유한는 체액 생산에 의한 마이크로샘플링 및 샘플링도 잠재적인 생물학적 샘플원이다.
본 개시의 방법 및 시스템은 핵산 서열 정보를 활용하므로, 핵산 증폭, 중합 효소 연쇄 반응(PCR), 나노포어 서열 분석, 454 서열 분석, 삽입 태그 서열 분석을 포함하는 핵산 서열 분석을 수행하기 위한 임의의 방법 또는 서열 분석 장치를 포함할 수 있다. 일부 양태에서, 본 개시의 방법론 또는 시스템은 시스템, 예컨대 주식회사 일루미나에 의해 제공되는 것들(HiSeqTM X10, HiSeqTM 1000, HiSeqTM 2000, HiSeqTM 2500, Genome AnalyzersTM, MiSeqTM, NextSeq, NovaSeq 6000 시스템을 포함하나 이에 제한되지 않음), 어플라이드 바이오시스템스 라이프 테크놀로지(SOLiDTM System, Ion PGMTM Sequencer, ion ProtonTM Sequencer) 또는 제넵시스 또는 BGI MGI 및 기타 시스템을 활용한다. 핵산 분석은 또한 옥스포드 나노포어 테크놀로지(GridiONTM, MiniONTM) 또는 퍼시픽 바이오사이언스(PacbioTM RS II 또는 후속 I 또는 II)에 의해 제공되는 시스템에 의해 수행될 수 있다.
본 발명은 개시된 방법의 단계를 수행하기 위한 시스템을 포함하며 기능적 구성요소 및 다양한 처리 단계의 관점에서 부분적으로 설명된다. 이러한 기능적 구성요소 및 처리 단계는 특정된 기능을 수행하고 다양한 결과를 달성하도록 구성된 임의의 수의 구성요소, 동작 및 기술에 의해 실현될 수 있다. 예를 들어, 본 발명은 다양한 기능을 수행할 수 있는 다양한 생물학적 샘플, 바이오마커, 요소, 재료, 컴퓨터, 데이터원, 저장 시스템 및 매체, 정보 수집 기술 및 프로세스, 데이터 처리 기준, 통계 분석, 회귀 분석 등을 사용할 수 있다.
따라서, 본 발명은 암을 검출하고, 분석하고/분석하거나 평가하기 위한 시스템을 추가로 제공한다. 다양한 양태에서, 시스템은 다음을 포함한다: (a) 샘플에 대한 낮은 적용 범위의 전체 게놈 서열 분석 데이터 세트를 생성하도록 구성된 서열 분석기; 및 (b) 본 발명의 방법을 수행하는 기능성을 갖춘 컴퓨터 시스템 및/또는 프로세서.
일부 양태에서, 컴퓨터 시스템은 하나 이상의 추가적인 모듈을 추가로 포함한다. 예를 들어, 시스템은 적합한 유전적 구성성분 분석, 예를 들어 특정 크기의 cfDNA 단편을 선택하도록 작동 가능한 하나 이상의 추출 및/또는 분리 유닛을 포함할 수 있다.
일부 양태에서, 컴퓨터 시스템은 시각적 디스플레이 장치를 추가로 포함한다. 시각적 디스플레이 장치는 곡선 맞춤선, 기준 곡선 맞춤선 및/또는 두 가지의 비교를 표시하도록 작동할 수 있다.
본 발명의 다양한 양태에 따른 검출 및 분석을 위한 방법은 예를 들어 컴퓨터 시스템에서 작동하는 컴퓨터 프로그램을 사용하여 임의의 적합한 방식으로 구현될 수 있다. 본원에 논의된 바와 같이, 본 발명의 다양한 양태에 따른 예시적인 시스템은 컴퓨터 시스템, 예를 들어 프로세서 및 랜덤 액세스 메모리, 예컨대 원격 액세스 가능한 애플리케이션 서버, 네트워크 서버, 개인용 컴퓨터 또는 워크스테이션을 포함하는 기존의 컴퓨터 시스템과 함께 구현될 수 있다. 컴퓨터 시스템은 또한 추가적인 메모리 장치 또는 정보 저장 시스템, 예컨대 대용량 저장 시스템 및 사용자 인터페이스, 예를 들어 기존의 모니터, 키보드 및 추적 장치를 적합하게 포함한다. 그러나 컴퓨터 시스템은 임의의 적합한 컴퓨터 시스템 및 연관 장비를 포함할 수 있으며 임의의 적합한 방식으로 구성될 수 있다. 일 구현예에서, 컴퓨터 시스템은 독립형 시스템을 포함한다. 또 다른 구현예에서, 컴퓨터 시스템은 서버와 데이터베이스를 포함하는 컴퓨터의 네트워크의 일부이다.
정보를 수신하고, 처리하고, 분석하는 데 요구되는 소프트웨어는 단일 장치에서 구현될 수도 있고, 복수 개의 장치에서 구현될 수도 있다. 소프트웨어는 정보의 저장 및 처리가 사용자에 대해 원격으로 발생하도록 네트워크를 통해 액세스할 수 있다. 본 발명의 다양한 양태에 따른 시스템과 그 다양한 요소는 검출 및/또는 분석, 예컨대 데이터를 수집하고, 처리하고, 분석하고, 보고하고/보고하거나 진단을 용이하게 하는 기능 및 동작을 제공한다. 예를 들어, 본 양태에서, 컴퓨터 시스템은 인간 게놈 또는 그 영역과 관련된 정보를 수신하고, 저장하고, 검색하고, 분석하고 보고할 수 있는 컴퓨터 프로그램을 실행한다. 컴퓨터 프로그램은 다양한 기능 또는 작업, 예컨대 미가공 데이터를 처리하고 보충 데이터를 생성하기 위한 처리 모듈 및 질병 상태 모델 및/또는 진단 정보의 정량적 평가를 생성하기 위해 미가공 데이터 및 보충 데이터를 분석하기 위한 분석 모듈을 수행하는 다중 모듈을 포함할 수 있다.
시스템에 의해 수행되는 절차는 분석 및/또는 암 진단을 용이하게 하는 임의의 적합한 공정을 포함할 수 있다. 일 구현예에서, 시스템은 질병 상태 모델을 확립하고/확립하거나 환자의 질병 상태를 결정하도록 구성된다. 질병 상태를 결정하거나 식별하는 단계는 질병에 관한 환자의 상태에 관한 임의의 유용한 정보를 생성하는 단계, 예컨대 진단을 수행하고, 진단에 도움이 되는 정보를 제공하고, 질병의 병기 또는 진행을 평가하고, 질병에 대한 취약성을 가리킬 수 있는 상태를 식별하고, 추가 테스트가 권장될 수 있는지 여부를 식별하고, 하나 이상의 치료 프로그램의 효능을 예측하고/예측하거나 평가하고, 질병 상태, 질병 가능성 또는 환자의 기타 건강 양태를 평가하는 것을 포함할 수 있다.
다음의 실시예는 본 발명의 장점과 특징을 더욱 도시하기 위해 제공되는 것이나, 이는 본 발명의 범위를 제한하려는 의도가 아니다. 이 실시예는 사용될 수 있는 것 중 전형적인 것이지만, 당업자에게 알려진 다른 절차, 방법론 또는 기술이 대안적으로 사용될 수 있다.
실시예 1
전향적 진단 코호트에서 게놈 전체 cfDNA 단편화를 사용한 암 검출
게놈 전체 cfDNA 단편화 패턴은 암이 있는 개인과 암이 없는 개인의 혈장 샘플 사이의 높은 민감도와 특이성으로 구분하는 것으로 입증되었다.
본 실시예에서, 본 개시의 방법론은 암을 검출하고 전체 환자 생존을 예측하기 위해 활용되었다.
연구의 목적은 cfDNA 단편화 검정을 혈액 기반 검진 테스트로 감정하여 복수의 상이한 고형 종양을 검출하고 전산 점수 체계를 사용하여 전체 환자 생존을 예측하는 것이었다.
방법
혈장 샘플: 암의 비기관 특이적 징후 및 증상으로 인해 헤르레브 및 겐토프테 병원(덴마크 코펜하겐의 코펜하겐 대학 병원)의 진단 외래 진료소로 의뢰된 281명의 환자로부터 샘플을 수집하였다.
cfDNA 단편화 접근법: cfDNA 단편화 접근법은 도 1에 요약되어 있다. cfDNA는 혈장에서 추출되어 서열 분석 라이브러리로 처리되고 낮은 적용 범위의 전체 게놈 서열 분석(WGS)으로 검사되고 게놈에 매핑되고 분석되어 게놈 전반에 걸쳐 cfDNA 단편화 프로파일을 결정하였다.
기계 학습을 사용하여 DELFI 점수를 생성하고 개인을 건강하거나 암이 있는 것으로 분류하고 전체 환자 생존을 예측하였다.
결과
암의 비침습적 검출을 위한 cfDNA 단편화 검정의 성과: 포함 후 3개월 이내에, 74명의 환자가 16개의 상이한 고형암 중 1개로 진단되었고 207명의 환자는 암이 없었다. 추가적인 결과는 도 2에 나타나 있다. 국소암과 전이암 및 모든 병기의 대장암, 폐암 및 기타 모든 암에 대한 곡선하면적(AUC)은 10회 반복, 10배 교차 검증을 사용하여 결정되었다.
암 검출을 위한 cfDNA 단편화 검정의 전반적인 성과: 결과는 도 3에 요약되어 있다. 1-4기 암이 있는 개인 74명과 비암 대조군 207명의 분석에 대한 수신자 조작 특성(ROC)의 AUC이다.
DELFI 점수에 따른 생존: 도 4에 나타낸 바와 같이 DELFI 점수가 높을수록 암 병기 또는 기타 임상적 특성과 관계없이 감소된 전체 생존과 연관이 있었다. 도 4는 DELFI 점수와 상관관계가 있는 대상체의 생존을 나타낸다. DELFI 점수가 높을수록 암 병기나 기타 임상적 특성과 관계없이 감소된 전체 생존과 연관이 있었다.
결론
전향적으로 등록된 개인에 대한 이 연구는 암이 있는 개인과 암이 없는 개인을 구분하는 cfDNA 단편화 검정의 능력을 입증하였다. 본 발명의 검정은 낮은 적용 범위의 WGS로부터 얻은 단편화 관련 정보만을 사용하여 다중 암 설정에서 높은 성능을 나타냈다.
결과는 기계 학습 모델이 cfDNA 단편화 프로파일을 사용하여 일반적인 비악성 상태(심혈관, 자가면역 또는 염증성 질병 포함)의 존재에도 불구하고 암과 비암 사이를 구분 지을 수 있음을 시사한다. 추가적으로, DELFI 점수가 높은 개인은 다른 특성과 관계없이 예후가 더 나빴다.
이들 데이터는 단일 암과 다중 암 모두의 비침습적 검출을 위한 게놈 전체 cfDNA 단편화 분석의 개발을 지원한다.
본 발명이 상기 실시예를 참조하여 설명되었으나, 수정 및 변형이 본 발명의 사상 및 범위 내에 포괄된다는 것이 이해될 것이다. 따라서, 본 발명은 다음의 청구항에 의해서만 제한된다.
Claims (59)
- 대상체에서 암을 검출하는 방법으로서, 다음을 포함하는, 방법:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계로서, cfDNA 단편화 프로파일은 다음에 의해 결정된다:
대상체로부터의 cfDNA 단편을 얻어 분리하는 단계,
cfDNA 단편을 서열 분석하여 서열 분석된 단편을 얻는 단계,
서열 분석된 단편을 게놈에 매핑하여 매핑된 서열의 윈도우를 얻는 단계, 및
매핑된 서열의 윈도우를 분석하여 cfDNA 단편 길이를 결정하고 cfDNA 단편화 프로파일을 생성하는 단계; 및
b) cfDNA 단편화 프로파일을 기반으로 대상체에서 암의 존재 가능성을 나타내는 점수를 계산하여 대상체를 암이 있거나 암이 없는 것으로 분류함으로써 대상체에서 암을 검출하는 단계. - 제1항에 있어서, 점수를 계산하는 단계는 다음을 포함하는, 방법: i) 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔(arm)에 의한 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계.
- 제2항에 있어서, 점수는 0 내지 1의 범위를 갖는, 방법.
- 제3항에 있어서, 대상체에서 암의 존재 가능성은 점수 값의 증가에 따라 증가하는, 방법.
- 제4항에 있어서, 암이 있는 것으로 분류된 대상체에 대하여, 점수를 기반으로 대상체의 전체 생존 가능성을 결정하는 단계를 추가로 포함하는, 방법.
- 제5항에 있어서, 대상체의 전체 생존 가능성은 점수 값의 증가에 따라 감소하는, 방법.
- 제6항에 있어서, 점수를 높은 점수 또는 낮은 점수로 분류하는 단계를 추가로 포함하고, 여기서 높은 점수는 0.5 초과의 값을 갖고, 낮은 점수는 0.5 미만의 값을 가지며, 높은 점수는 대상체의 감소된 전체 생존을 나타내는, 방법.
- 제1항에 있어서, 서열 분석하는 단계는 cfDNA 단편을 낮은 적용 범위의 전체 게놈 서열분석에 적용하여 서열 분석된 단편을 얻는 단계를 포함하는, 방법.
- 제1항에 있어서, cfDNA 단편을 분리하는 단계는 105bp 미만 및 170bp 초과의 단편 크기를 제외하는 단계를 포함하는, 방법.
- 제1항에 있어서, 매핑된 서열의 윈도우는 수십 내지 수천 개의 윈도우를 포함하는, 방법.
- 제10항에 있어서, 윈도우는 비중첩 윈도우인, 방법.
- 제11항에 있어서, 윈도우 각각은 약 5백만 개의 염기쌍을 포함하는, 방법.
- 제12항에 있어서, cfDNA 단편화 프로파일은 각 윈도우 내에서 결정되는, 방법.
- 제1항에 있어서, cfDNA 단편화 프로파일은 매핑된 서열의 윈도우에서 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율을 포함하는, 방법.
- 제1항에 있어서, cfDNA 단편화 프로파일은 게놈 전체의 윈도우에서 작은 cfDNA 단편 및 큰 cfDNA 단편의 서열 적용 범위를 포함하는, 방법.
- 제1항에 있어서, cfDNA 단편화 프로파일은 전체 게놈에 걸쳐 있는, 방법.
- 제1항에 있어서, cfDNA 단편화 프로파일은 서브게놈 간격에 걸쳐 있는, 방법.
- 제1항에 있어서, 분류하는 단계는 cfDNA 단편화 프로파일을 기준 cfDNA 단편화와 비교하는 단계를 포함하는, 방법.
- 제18항에 있어서, 기준 cfDNA 단편화 프로파일은 건강한 대상체의 cfDNA 단편화 프로파일인, 방법.
- 제1항에 있어서, 암은 고형 종양인, 방법.
- 제20항에 있어서, 암은 육종, 암종 또는 림프종인, 방법.
- 제20항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 대장암, 전립선암, 유방암, 췌장암, 담관암, 간암, CNS, 위암, 식도암, 위장관 간질 종양(GIST), 자궁암 또는 난소암.
- 제1항에 있어서, 암은 혈액암인, 방법.
- 제23항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 골수종, 다발성 골수종, B 세포 림프종, 여포성 림프종, 림프구성 백혈병, 백혈병 또는 골수성 백혈병.
- 제1항에 있어서, 대상체에게 암 치료를 투여하는 단계를 추가로 포함하는, 방법.
- 제25항에 있어서, 암 치료는 수술, 보조 화학치료요법, 신보조 화학치료요법, 방사선 치료요법, 호르몬 치료요법, 세포독성 치료요법, 면역치료요법, 입양 T 세포 치료요법, 표적화 치료요법 또는 이들의 임의의 조합으로 이루어진 군으로부터 선택되는, 방법.
- 암이 있는 대상체의 전체 생존을 결정하는 방법으로서, 다음을 포함하는, 방법:
a) 대상체로부터의 샘플의 무세포 DNA(cfDNA) 단편화 프로파일을 결정하는 단계;
b) cfDNA 단편화 프로파일을 기반으로 점수를 계산하는 단계로서, 여기서 점수를 계산하는 단계는 다음을 포함한다: i) 샘플의 짧은 cfDNA 단편 대 긴 cfDNA 단편의 비율을 결정하는 단계, ii) 염색체 팔에 의해 샘플의 cfDNA 단편에 대한 Z-점수를 결정하는 단계, iii) 전산 혼합 모델 분석을 사용하여 cfDNA 단편 밀도를 정량화하는 단계 및 iv) 기계 학습 모델을 사용하여 i)-iii)의 출력을 처리하여 점수를 정의하는 단계; 및
c) 점수를 기반으로 대상체의 전체 생존 가능성을 결정함으로써 대상체의 전체 생존을 결정하는 단계. - 제27항에 있어서, 점수는 0 내지 1의 범위를 갖는, 방법.
- 제28항에 있어서, 대상체의 전체 생존 가능성은 점수 값의 증가에 따라 감소하는, 방법.
- 제29항에 있어서, 점수를 높은 점수 또는 낮은 점수로 분류하는 단계를 추가로 포함하고, 여기서 높은 점수는 0.5 초과의 값을 갖고, 낮은 점수는 0.5 미만의 값을 가지며, 높은 점수는 대상체의 감소된 전체 생존을 나타내는, 방법.
- 제27항에 있어서, cfDNA 단편화 프로파일은 다음에 의해 결정되는, 방법:
대상체로부터의 cfDNA 단편을 얻어 분리하는 단계,
cfDNA 단편을 서열 분석하여 서열 분석된 단편을 얻는 단계,
서열 분석된 단편을 게놈에 매핑하여 매핑된 서열의 윈도우를 얻는 단계, 및
매핑된 서열의 윈도우를 분석하여 cfDNA 단편 길이를 결정하고 cfDNA 단편화 프로파일을 생성하는 단계. - 제31항에 있어서, 서열 분석하는 단계는 cfDNA 단편을 낮은 적용 범위의 전체 게놈 서열분석에 적용하여 서열 분석된 단편을 얻는 단계를 포함하는, 방법.
- 제31항에 있어서, cfDNA 단편을 분리하는 단계는 105bp 미만 및 170bp 초과의 단편 크기를 제외하는 단계를 포함하는, 방법.
- 제31항에 있어서, 매핑된 서열의 윈도우는 수십 내지 수천 개의 윈도우를 포함하는, 방법.
- 제34항에 있어서, 윈도우는 비중첩 윈도우인, 방법.
- 제35항에 있어서, 윈도우 각각은 약 5백만 개의 염기쌍을 포함하는, 방법.
- 제36항에 있어서, cfDNA 단편화 프로파일은 각 윈도우 내에서 결정되는, 방법.
- 제31항에 있어서, cfDNA 단편화 프로파일은 매핑된 서열의 윈도우에서 작은 cfDNA 단편 대 큰 cfDNA 단편의 비율을 포함하는, 방법.
- 제31항에 있어서, cfDNA 단편화 프로파일은 게놈 전체의 윈도우에서 작은 cfDNA 단편 및 큰 cfDNA 단편의 서열 적용 범위를 포함하는, 방법.
- 제31항에 있어서, cfDNA 단편화 프로파일은 전체 게놈에 걸쳐 있는, 방법.
- 제31항에 있어서, cfDNA 단편화 프로파일은 서브게놈 간격에 걸쳐 있는, 방법.
- 제27항에 있어서, 암은 고형 종양인, 방법.
- 제42항에 있어서, 암은 육종, 암종 또는 림프종인, 방법.
- 제42항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 대장암, 전립선암, 유방암, 췌장암, 담관암, 간암, CNS, 위암, 식도암, 위장관 간질 종양(GIST), 자궁암 또는 난소암.
- 제27항에 있어서, 암은 혈액암인, 방법.
- 제45항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 골수종, 다발성 골수종, B 세포 림프종, 여포성 림프종, 림프구성 백혈병, 백혈병 또는 골수성 백혈병.
- 제27항에 있어서, 대상체에게 암 치료를 투여하는 단계를 추가로 포함하는, 방법.
- 제47항에 있어서, 암 치료는 수술, 보조 화학치료요법, 신보조 화학치료요법, 방사선 치료요법, 호르몬 치료요법, 세포독성 치료요법, 면역치료요법, 입양 T 세포 치료요법, 표적화 치료요법 또는 이들의 임의의 조합으로 이루어진 군으로부터 선택되는, 방법.
- 암이 있는 대상체를 치료하는 방법으로서, 다음을 포함하는, 방법:
a) 제1항 내지 제19항 중 어느 한 항의 방법을 사용하여 대상체에서 암을 검출하거나, 제27항 내지 제41항 중 어느 한 항의 방법을 사용하여 대상체의 전체 생존을 결정하는 단계; 및
b) 대상체에게 암 치료를 투여하여 대상체를 치료하는 단계. - 제49항에 있어서, 암은 고형 종양인, 방법.
- 제50항에 있어서, 암은 육종, 암종 또는 림프종인, 방법.
- 제50항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 폐암, 대장암, 전립선암, 유방암, 췌장암, 담관암, 간암, CNS, 위암, 식도암, 위장관 간질 종양(GIST), 자궁암 또는 난소암.
- 제49항에 있어서, 암은 혈액암인, 방법.
- 제53항에 있어서, 암은 다음으로 구성되는 군으로부터 선택되는 것인, 방법: 골수종, 다발성 골수종, B 세포 림프종, 여포성 림프종, 림프구성 백혈병, 백혈병 또는 골수성 백혈병.
- 제49항에 있어서, 암 치료는 수술, 보조 화학치료요법, 신보조 화학치료요법, 방사선 치료요법, 호르몬 치료요법, 세포독성 치료요법, 면역치료요법, 입양 T 세포 치료요법, 표적화 치료요법 또는 이들의 임의의 조합으로 이루어진 군으로부터 선택되는, 방법.
- 제47항에 있어서, 대상체는 인간인, 방법.
- 대상체에서 암을 모니터링하는 방법으로서, 다음을 포함하는, 방법:
a) 제1항 내지 제19항 중 어느 한 항의 방법을 사용하여 대상체에서 암을 검출하거나 제27항 내지 제41항 중 어느 한 항의 방법을 사용하여 대상체의 전체 생존을 결정하는 단계;
b) 대상체에게 암 치료를 투여하는 단계; 및
c) 암 치료가 투여된 후 제27항 내지 제41항 중 어느 한 항의 방법을 사용하여 대상체의 전체 생존을 결정함으로써 대상체에서 암을 모니터링하는 단계. - 컴퓨터 프로그램으로 암호화된 비일시적 컴퓨터 판독 가능 저장 매체로서, 프로그램은 하나 이상의 프로세서에 의해 실행될 때 하나 이상의 프로세서가 제1항 내지 제24항 또는 제27항 내지 제46항 중 어느 한 항의 방법을 수행하기 위한 동작을 수행하게 하는 명령을 포함하는, 비일시적 컴퓨터 판독 가능 저장 매체.
- 컴퓨팅 시스템으로서, 메모리; 및 메모리에 연결된 하나 이상의 프로세서를 포함하고, 하나 이상의 프로세서는 제1항 내지 제24항 또는 제27항 내지 제46항 중 어느 한 항의 방법을 수행하는 동작을 수행하도록 구성되는, 컴퓨팅 시스템.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US202163172493P | 2021-04-08 | 2021-04-08 | |
| US63/172,493 | 2021-04-08 | ||
| PCT/US2022/023907 WO2022216981A1 (en) | 2021-04-08 | 2022-04-07 | Method of detecting cancer using genome-wide cfdna fragmentation profiles |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20240015624A true KR20240015624A (ko) | 2024-02-05 |
Family
ID=83546571
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020237035747A KR20240015624A (ko) | 2021-04-08 | 2022-04-07 | 게놈 전체 cfdna 단편화 프로파일을 이용한 암을 검출하는 방법 |
Country Status (9)
| Country | Link |
|---|---|
| EP (1) | EP4320277A1 (ko) |
| JP (1) | JP2024515558A (ko) |
| KR (1) | KR20240015624A (ko) |
| CN (1) | CN117561340A (ko) |
| AU (1) | AU2022254718A1 (ko) |
| BR (1) | BR112023020307A2 (ko) |
| CA (1) | CA3214321A1 (ko) |
| IL (1) | IL307524A (ko) |
| WO (1) | WO2022216981A1 (ko) |
Family Cites Families (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20170211143A1 (en) * | 2014-07-25 | 2017-07-27 | University Of Washington | Methods of determining tissues and/or cell types giving rise to cell-free dna, and methods of identifying a disease or disorder using same |
| KR20210045953A (ko) * | 2018-05-18 | 2021-04-27 | 더 존스 홉킨스 유니버시티 | 암의 평가 및/또는 치료를 위한 무 세포 dna |
| GB201818159D0 (en) * | 2018-11-07 | 2018-12-19 | Cancer Research Tech Ltd | Enhanced detection of target dna by fragment size analysis |
| CN113661249A (zh) * | 2019-01-31 | 2021-11-16 | 夸登特健康公司 | 用于分离无细胞dna的组合物和方法 |
| JP2023541368A (ja) * | 2020-08-18 | 2023-10-02 | デルフィ ダイアグノスティックス インコーポレイテッド | がんを評価するための無細胞dna断片サイズ密度のための方法及びシステム |
-
2022
- 2022-04-07 CN CN202280027033.8A patent/CN117561340A/zh active Pending
- 2022-04-07 IL IL307524A patent/IL307524A/en unknown
- 2022-04-07 BR BR112023020307A patent/BR112023020307A2/pt unknown
- 2022-04-07 AU AU2022254718A patent/AU2022254718A1/en active Pending
- 2022-04-07 KR KR1020237035747A patent/KR20240015624A/ko unknown
- 2022-04-07 JP JP2023561634A patent/JP2024515558A/ja active Pending
- 2022-04-07 CA CA3214321A patent/CA3214321A1/en active Pending
- 2022-04-07 WO PCT/US2022/023907 patent/WO2022216981A1/en active Application Filing
- 2022-04-07 EP EP22785477.5A patent/EP4320277A1/en active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| EP4320277A1 (en) | 2024-02-14 |
| AU2022254718A1 (en) | 2023-11-16 |
| CN117561340A (zh) | 2024-02-13 |
| BR112023020307A2 (pt) | 2023-11-21 |
| CA3214321A1 (en) | 2022-10-13 |
| WO2022216981A1 (en) | 2022-10-13 |
| IL307524A (en) | 2023-12-01 |
| JP2024515558A (ja) | 2024-04-10 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7531217B2 (ja) | 癌を査定および/または処置するためのセルフリーdna | |
| JP2023541368A (ja) | がんを評価するための無細胞dna断片サイズ密度のための方法及びシステム | |
| CN105506115B (zh) | 一种检测诊断遗传性心肌病致病基因的dna文库及其应用 | |
| JP2019527544A (ja) | 分子マーカー、参照遺伝子、及びその応用、検出キット、並びに検出モデルの構築方法 | |
| US20210262016A1 (en) | Methods and systems for somatic mutations and uses thereof | |
| CN111833963A (zh) | 一种cfDNA分类方法、装置和用途 | |
| US12000002B2 (en) | Pre-surgical risk stratification based on PDE4D7 expression and pre-surgical clinical variables | |
| CN106399304B (zh) | 一种与乳腺癌相关的snp标记 | |
| CN114807370A (zh) | 一种新型的用于乳腺癌免疫治疗疗效精准预测的模型及其应用 | |
| AU2020364225B2 (en) | Fragment size characterization of cell-free DNA mutations from clonal hematopoiesis | |
| KR20240015624A (ko) | 게놈 전체 cfdna 단편화 프로파일을 이용한 암을 검출하는 방법 | |
| JPWO2021092476A5 (ko) | ||
| AU2023233603A1 (en) | Method of monitoring cancer using fragmentation profiles | |
| WO2021213398A1 (zh) | 评价胶质瘤和/或胃腺癌预后性的试剂盒和系统 | |
| WO2024076769A1 (en) | Incorporating clinical risk into biomarker-based assessment for cancer pre-screening | |
| CN118984879A (zh) | 使用片段化图谱来监测癌症的方法 | |
| WO2024173277A2 (en) | Delfi-derived cell-free dna fragmentation patterns differentiate histologic subtypes of lung cancers in a non-invasive manner | |
| CN106811528B (zh) | 一种乳腺癌治病基因新突变及其应用 | |
| CN118207322A (zh) | 胆汁cfDNA中的HDR信号通路突变在胆管癌预后评估中的应用 | |
| CN117165679A (zh) | 肝癌肝移植术后复发标志物及其应用 |