JP7531217B2 - 癌を査定および/または処置するためのセルフリーdna - Google Patents
癌を査定および/または処置するためのセルフリーdna Download PDFInfo
- Publication number
- JP7531217B2 JP7531217B2 JP2020564491A JP2020564491A JP7531217B2 JP 7531217 B2 JP7531217 B2 JP 7531217B2 JP 2020564491 A JP2020564491 A JP 2020564491A JP 2020564491 A JP2020564491 A JP 2020564491A JP 7531217 B2 JP7531217 B2 JP 7531217B2
- Authority
- JP
- Japan
- Prior art keywords
- cfdna
- cancer
- mammal
- fragmentation profile
- genome
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 206010028980 Neoplasm Diseases 0.000 title claims description 357
- 201000011510 cancer Diseases 0.000 title claims description 311
- 239000012634 fragment Substances 0.000 claims description 228
- 238000013467 fragmentation Methods 0.000 claims description 227
- 238000006062 fragmentation reaction Methods 0.000 claims description 227
- 241000124008 Mammalia Species 0.000 claims description 208
- 238000000034 method Methods 0.000 claims description 77
- 238000012163 sequencing technique Methods 0.000 claims description 21
- 238000012070 whole genome sequencing analysis Methods 0.000 claims description 20
- 238000013507 mapping Methods 0.000 claims description 7
- 108091027544 Subgenomic mRNA Proteins 0.000 claims description 5
- 238000012545 processing Methods 0.000 claims description 4
- 238000011282 treatment Methods 0.000 description 78
- 238000004458 analytical method Methods 0.000 description 55
- 239000000523 sample Substances 0.000 description 53
- 108020004414 DNA Proteins 0.000 description 50
- 238000002405 diagnostic procedure Methods 0.000 description 39
- 238000013459 approach Methods 0.000 description 38
- 238000012544 monitoring process Methods 0.000 description 36
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 32
- 201000005202 lung cancer Diseases 0.000 description 32
- 208000020816 lung neoplasm Diseases 0.000 description 32
- 210000001519 tissue Anatomy 0.000 description 29
- 210000000349 chromosome Anatomy 0.000 description 26
- 239000000463 material Substances 0.000 description 25
- 206010009944 Colon cancer Diseases 0.000 description 23
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 23
- 238000001514 detection method Methods 0.000 description 22
- 230000035772 mutation Effects 0.000 description 20
- 206010033128 Ovarian cancer Diseases 0.000 description 19
- 206010061535 Ovarian neoplasm Diseases 0.000 description 19
- 210000004698 lymphocyte Anatomy 0.000 description 18
- 210000004027 cell Anatomy 0.000 description 17
- 210000004072 lung Anatomy 0.000 description 17
- 206010006187 Breast cancer Diseases 0.000 description 16
- 208000026310 Breast neoplasm Diseases 0.000 description 16
- 108010047956 Nucleosomes Proteins 0.000 description 16
- 210000001623 nucleosome Anatomy 0.000 description 16
- 210000002381 plasma Anatomy 0.000 description 16
- 239000008280 blood Substances 0.000 description 15
- 238000010801 machine learning Methods 0.000 description 15
- 208000005718 Stomach Neoplasms Diseases 0.000 description 14
- 210000004369 blood Anatomy 0.000 description 14
- 208000006990 cholangiocarcinoma Diseases 0.000 description 14
- 206010017758 gastric cancer Diseases 0.000 description 14
- 201000011549 stomach cancer Diseases 0.000 description 14
- 210000000481 breast Anatomy 0.000 description 13
- 230000035945 sensitivity Effects 0.000 description 13
- 108700028369 Alleles Proteins 0.000 description 12
- 230000004075 alteration Effects 0.000 description 11
- 230000002438 mitochondrial effect Effects 0.000 description 11
- 206010004593 Bile duct cancer Diseases 0.000 description 10
- 238000013103 analytical ultracentrifugation Methods 0.000 description 10
- 208000026900 bile duct neoplasm Diseases 0.000 description 10
- 238000002790 cross-validation Methods 0.000 description 10
- 230000001419 dependent effect Effects 0.000 description 10
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 9
- 201000010099 disease Diseases 0.000 description 9
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 9
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 description 9
- 201000002528 pancreatic cancer Diseases 0.000 description 9
- 208000008443 pancreatic carcinoma Diseases 0.000 description 9
- 238000012360 testing method Methods 0.000 description 9
- 210000001744 T-lymphocyte Anatomy 0.000 description 8
- 239000000107 tumor biomarker Substances 0.000 description 8
- 230000002496 gastric effect Effects 0.000 description 7
- 239000000090 biomarker Substances 0.000 description 6
- 238000002659 cell therapy Methods 0.000 description 6
- 230000002759 chromosomal effect Effects 0.000 description 6
- 238000012937 correction Methods 0.000 description 6
- 238000012216 screening Methods 0.000 description 6
- 238000002626 targeted therapy Methods 0.000 description 6
- 238000002560 therapeutic procedure Methods 0.000 description 6
- 101710163270 Nuclease Proteins 0.000 description 5
- 230000005856 abnormality Effects 0.000 description 5
- 238000011226 adjuvant chemotherapy Methods 0.000 description 5
- 230000008901 benefit Effects 0.000 description 5
- 230000002596 correlated effect Effects 0.000 description 5
- 231100000433 cytotoxic Toxicity 0.000 description 5
- 230000001472 cytotoxic effect Effects 0.000 description 5
- 238000001794 hormone therapy Methods 0.000 description 5
- 238000009169 immunotherapy Methods 0.000 description 5
- 238000011227 neoadjuvant chemotherapy Methods 0.000 description 5
- 230000002611 ovarian Effects 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 238000001959 radiotherapy Methods 0.000 description 5
- 238000001356 surgical procedure Methods 0.000 description 5
- 238000012549 training Methods 0.000 description 5
- 238000000342 Monte Carlo simulation Methods 0.000 description 4
- 208000036878 aneuploidy Diseases 0.000 description 4
- 231100001075 aneuploidy Toxicity 0.000 description 4
- 238000013276 bronchoscopy Methods 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 238000002591 computed tomography Methods 0.000 description 4
- 230000009977 dual effect Effects 0.000 description 4
- 230000000694 effects Effects 0.000 description 4
- 210000004602 germ cell Anatomy 0.000 description 4
- 230000003394 haemopoietic effect Effects 0.000 description 4
- 238000007481 next generation sequencing Methods 0.000 description 4
- 238000011160 research Methods 0.000 description 4
- 238000012552 review Methods 0.000 description 4
- 238000004088 simulation Methods 0.000 description 4
- 230000003442 weekly effect Effects 0.000 description 4
- 108010077544 Chromatin Proteins 0.000 description 3
- 229940076838 Immune checkpoint inhibitor Drugs 0.000 description 3
- 102000037984 Inhibitory immune checkpoint proteins Human genes 0.000 description 3
- 108091008026 Inhibitory immune checkpoint proteins Proteins 0.000 description 3
- 108010059724 Micrococcal Nuclease Proteins 0.000 description 3
- 108020005196 Mitochondrial DNA Proteins 0.000 description 3
- 208000008900 Pancreatic Ductal Carcinoma Diseases 0.000 description 3
- 230000002159 abnormal effect Effects 0.000 description 3
- 210000000013 bile duct Anatomy 0.000 description 3
- 210000000601 blood cell Anatomy 0.000 description 3
- 210000003483 chromatin Anatomy 0.000 description 3
- 238000007796 conventional method Methods 0.000 description 3
- 230000001186 cumulative effect Effects 0.000 description 3
- 230000007423 decrease Effects 0.000 description 3
- 239000003814 drug Substances 0.000 description 3
- 238000013399 early diagnosis Methods 0.000 description 3
- 238000005516 engineering process Methods 0.000 description 3
- 230000006870 function Effects 0.000 description 3
- 238000009499 grossing Methods 0.000 description 3
- 230000036541 health Effects 0.000 description 3
- 239000012274 immune-checkpoint protein inhibitor Substances 0.000 description 3
- 229940043355 kinase inhibitor Drugs 0.000 description 3
- 230000000670 limiting effect Effects 0.000 description 3
- 210000004940 nucleus Anatomy 0.000 description 3
- 201000008129 pancreatic ductal adenocarcinoma Diseases 0.000 description 3
- 239000003757 phosphotransferase inhibitor Substances 0.000 description 3
- 229920001184 polypeptide Polymers 0.000 description 3
- 108090000765 processed proteins & peptides Proteins 0.000 description 3
- 102000004196 processed proteins & peptides Human genes 0.000 description 3
- 108090000623 proteins and genes Proteins 0.000 description 3
- 238000002271 resection Methods 0.000 description 3
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 description 2
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 2
- 238000000585 Mann–Whitney U test Methods 0.000 description 2
- 108091093105 Nuclear DNA Proteins 0.000 description 2
- 108091008874 T cell receptors Proteins 0.000 description 2
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 2
- 239000002246 antineoplastic agent Substances 0.000 description 2
- 229950002916 avelumab Drugs 0.000 description 2
- 229910052788 barium Inorganic materials 0.000 description 2
- DSAJWYNOEDNPEQ-UHFFFAOYSA-N barium atom Chemical compound [Ba] DSAJWYNOEDNPEQ-UHFFFAOYSA-N 0.000 description 2
- 239000011324 bead Substances 0.000 description 2
- 238000000876 binomial test Methods 0.000 description 2
- 238000004364 calculation method Methods 0.000 description 2
- 230000006037 cell lysis Effects 0.000 description 2
- 230000001413 cellular effect Effects 0.000 description 2
- 238000005119 centrifugation Methods 0.000 description 2
- 238000006243 chemical reaction Methods 0.000 description 2
- 239000003153 chemical reaction reagent Substances 0.000 description 2
- 238000002052 colonoscopy Methods 0.000 description 2
- 239000003086 colorant Substances 0.000 description 2
- 229940127089 cytotoxic agent Drugs 0.000 description 2
- 239000003599 detergent Substances 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 230000018109 developmental process Effects 0.000 description 2
- 238000003745 diagnosis Methods 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 229950009791 durvalumab Drugs 0.000 description 2
- 239000012530 fluid Substances 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 210000000265 leukocyte Anatomy 0.000 description 2
- 230000000873 masking effect Effects 0.000 description 2
- 230000011987 methylation Effects 0.000 description 2
- 238000007069 methylation reaction Methods 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 230000007935 neutral effect Effects 0.000 description 2
- 229960003301 nivolumab Drugs 0.000 description 2
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 2
- 108020004707 nucleic acids Proteins 0.000 description 2
- 102000039446 nucleic acids Human genes 0.000 description 2
- 150000007523 nucleic acids Chemical class 0.000 description 2
- 239000002773 nucleotide Substances 0.000 description 2
- 125000003729 nucleotide group Chemical group 0.000 description 2
- 230000008520 organization Effects 0.000 description 2
- 230000007170 pathology Effects 0.000 description 2
- 229960002621 pembrolizumab Drugs 0.000 description 2
- 230000003449 preventive effect Effects 0.000 description 2
- 230000008707 rearrangement Effects 0.000 description 2
- 230000004044 response Effects 0.000 description 2
- 230000003068 static effect Effects 0.000 description 2
- 238000007619 statistical method Methods 0.000 description 2
- 239000004094 surface-active agent Substances 0.000 description 2
- 230000009897 systematic effect Effects 0.000 description 2
- 238000002604 ultrasonography Methods 0.000 description 2
- FDKXTQMXEQVLRF-ZHACJKMWSA-N (E)-dacarbazine Chemical compound CN(C)\N=N\c1[nH]cnc1C(N)=O FDKXTQMXEQVLRF-ZHACJKMWSA-N 0.000 description 1
- VSNHCAURESNICA-NJFSPNSNSA-N 1-oxidanylurea Chemical compound N[14C](=O)NO VSNHCAURESNICA-NJFSPNSNSA-N 0.000 description 1
- NDMPLJNOPCLANR-UHFFFAOYSA-N 3,4-dihydroxy-15-(4-hydroxy-18-methoxycarbonyl-5,18-seco-ibogamin-18-yl)-16-methoxy-1-methyl-6,7-didehydro-aspidospermidine-3-carboxylic acid methyl ester Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 NDMPLJNOPCLANR-UHFFFAOYSA-N 0.000 description 1
- AOJJSUZBOXZQNB-VTZDEGQISA-N 4'-epidoxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-VTZDEGQISA-N 0.000 description 1
- IDPUKCWIGUEADI-UHFFFAOYSA-N 5-[bis(2-chloroethyl)amino]uracil Chemical compound ClCCN(CCCl)C1=CNC(=O)NC1=O IDPUKCWIGUEADI-UHFFFAOYSA-N 0.000 description 1
- NMUSYJAQQFHJEW-KVTDHHQDSA-N 5-azacytidine Chemical compound O=C1N=C(N)N=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](CO)O1 NMUSYJAQQFHJEW-KVTDHHQDSA-N 0.000 description 1
- WYWHKKSPHMUBEB-UHFFFAOYSA-N 6-Mercaptoguanine Natural products N1C(N)=NC(=S)C2=C1N=CN2 WYWHKKSPHMUBEB-UHFFFAOYSA-N 0.000 description 1
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 1
- 206010069754 Acquired gene mutation Diseases 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- COVZYZSDYWQREU-UHFFFAOYSA-N Busulfan Chemical compound CS(=O)(=O)OCCCCOS(C)(=O)=O COVZYZSDYWQREU-UHFFFAOYSA-N 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- GAGWJHPBXLXJQN-UORFTKCHSA-N Capecitabine Chemical compound C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1[C@H]1[C@H](O)[C@H](O)[C@@H](C)O1 GAGWJHPBXLXJQN-UORFTKCHSA-N 0.000 description 1
- GAGWJHPBXLXJQN-UHFFFAOYSA-N Capecitabine Natural products C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1C1C(O)C(O)C(C)O1 GAGWJHPBXLXJQN-UHFFFAOYSA-N 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 206010008805 Chromosomal abnormalities Diseases 0.000 description 1
- 208000031404 Chromosome Aberrations Diseases 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-CCXZUQQUSA-N Cytarabine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@@H](O)[C@H](O)[C@@H](CO)O1 UHDGCWIWMRVCDJ-CCXZUQQUSA-N 0.000 description 1
- 238000007400 DNA extraction Methods 0.000 description 1
- 238000007399 DNA isolation Methods 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 241000792859 Enema Species 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- HTIJFSOGRVMCQR-UHFFFAOYSA-N Epirubicin Natural products COc1cccc2C(=O)c3c(O)c4CC(O)(CC(OC5CC(N)C(=O)C(C)O5)c4c(O)c3C(=O)c12)C(=O)CO HTIJFSOGRVMCQR-UHFFFAOYSA-N 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 1
- XDXDZDZNSLXDNA-TZNDIEGXSA-N Idarubicin Chemical compound C1[C@H](N)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2C[C@@](O)(C(C)=O)C1 XDXDZDZNSLXDNA-TZNDIEGXSA-N 0.000 description 1
- XDXDZDZNSLXDNA-UHFFFAOYSA-N Idarubicin Natural products C1C(N)C(O)C(C)OC1OC1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2CC(O)(C(C)=O)C1 XDXDZDZNSLXDNA-UHFFFAOYSA-N 0.000 description 1
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 1
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- GQYIWUVLTXOXAJ-UHFFFAOYSA-N Lomustine Chemical compound ClCCN(N=O)C(=O)NC1CCCCC1 GQYIWUVLTXOXAJ-UHFFFAOYSA-N 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 208000002693 Multiple Abnormalities Diseases 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 1
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 1
- 229930012538 Paclitaxel Natural products 0.000 description 1
- 240000005160 Parkia speciosa Species 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 206010036790 Productive cough Diseases 0.000 description 1
- 239000004365 Protease Substances 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 1
- 208000007660 Residual Neoplasm Diseases 0.000 description 1
- 238000000692 Student's t-test Methods 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- BPEGJWRSRHCHSN-UHFFFAOYSA-N Temozolomide Chemical compound O=C1N(C)N=NC2=C(C(N)=O)N=CN21 BPEGJWRSRHCHSN-UHFFFAOYSA-N 0.000 description 1
- 241000283907 Tragelaphus oryx Species 0.000 description 1
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 1
- 238000001790 Welch's t-test Methods 0.000 description 1
- 210000001766 X chromosome Anatomy 0.000 description 1
- 210000002593 Y chromosome Anatomy 0.000 description 1
- RTJVUHUGTUDWRK-CSLCKUBZSA-N [(2r,4ar,6r,7r,8s,8ar)-6-[[(5s,5ar,8ar,9r)-9-(3,5-dimethoxy-4-phosphonooxyphenyl)-8-oxo-5a,6,8a,9-tetrahydro-5h-[2]benzofuro[6,5-f][1,3]benzodioxol-5-yl]oxy]-2-methyl-7-[2-(2,3,4,5,6-pentafluorophenoxy)acetyl]oxy-4,4a,6,7,8,8a-hexahydropyrano[3,2-d][1,3]d Chemical compound COC1=C(OP(O)(O)=O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](OC(=O)COC=4C(=C(F)C(F)=C(F)C=4F)F)[C@@H]4O[C@H](C)OC[C@H]4O3)OC(=O)COC=3C(=C(F)C(F)=C(F)C=3F)F)[C@@H]3[C@@H]2C(OC3)=O)=C1 RTJVUHUGTUDWRK-CSLCKUBZSA-N 0.000 description 1
- SHGAZHPCJJPHSC-YCNIQYBTSA-N all-trans-retinoic acid Chemical compound OC(=O)\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C SHGAZHPCJJPHSC-YCNIQYBTSA-N 0.000 description 1
- 210000001691 amnion Anatomy 0.000 description 1
- 229940124650 anti-cancer therapies Drugs 0.000 description 1
- 238000011319 anticancer therapy Methods 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 210000003567 ascitic fluid Anatomy 0.000 description 1
- 238000003556 assay Methods 0.000 description 1
- 229960003852 atezolizumab Drugs 0.000 description 1
- 229960002756 azacitidine Drugs 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 229960000397 bevacizumab Drugs 0.000 description 1
- 210000000941 bile Anatomy 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 229960002092 busulfan Drugs 0.000 description 1
- 229960004117 capecitabine Drugs 0.000 description 1
- 229960004562 carboplatin Drugs 0.000 description 1
- 190000008236 carboplatin Chemical compound 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 210000002230 centromere Anatomy 0.000 description 1
- 210000001175 cerebrospinal fluid Anatomy 0.000 description 1
- 239000003795 chemical substances by application Substances 0.000 description 1
- 210000000038 chest Anatomy 0.000 description 1
- JCKYGMPEJWAADB-UHFFFAOYSA-N chlorambucil Chemical compound OC(=O)CCCC1=CC=C(N(CCCl)CCCl)C=C1 JCKYGMPEJWAADB-UHFFFAOYSA-N 0.000 description 1
- 229960004630 chlorambucil Drugs 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- 238000013145 classification model Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 230000008094 contradictory effect Effects 0.000 description 1
- 238000010219 correlation analysis Methods 0.000 description 1
- 230000000875 corresponding effect Effects 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 210000002726 cyst fluid Anatomy 0.000 description 1
- 229960000684 cytarabine Drugs 0.000 description 1
- 229960003901 dacarbazine Drugs 0.000 description 1
- 229960000975 daunorubicin Drugs 0.000 description 1
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 1
- 238000012350 deep sequencing Methods 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 229960003668 docetaxel Drugs 0.000 description 1
- ZWAOHEXOSAUJHY-ZIYNGMLESA-N doxifluridine Chemical compound O[C@@H]1[C@H](O)[C@@H](C)O[C@H]1N1C(=O)NC(=O)C(F)=C1 ZWAOHEXOSAUJHY-ZIYNGMLESA-N 0.000 description 1
- 229950005454 doxifluridine Drugs 0.000 description 1
- 229960004679 doxorubicin Drugs 0.000 description 1
- 238000009547 dual-energy X-ray absorptiometry Methods 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 238000009558 endoscopic ultrasound Methods 0.000 description 1
- 239000007920 enema Substances 0.000 description 1
- 229940095399 enema Drugs 0.000 description 1
- 102000052116 epidermal growth factor receptor activity proteins Human genes 0.000 description 1
- 108700015053 epidermal growth factor receptor activity proteins Proteins 0.000 description 1
- 229960001904 epirubicin Drugs 0.000 description 1
- 229960005073 erlotinib hydrochloride Drugs 0.000 description 1
- GTTBEUCJPZQMDZ-UHFFFAOYSA-N erlotinib hydrochloride Chemical compound [H+].[Cl-].C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 GTTBEUCJPZQMDZ-UHFFFAOYSA-N 0.000 description 1
- 238000002181 esophagogastroduodenoscopy Methods 0.000 description 1
- VJJPUSNTGOMMGY-MRVIYFEKSA-N etoposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@H](C)OC[C@H]4O3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 VJJPUSNTGOMMGY-MRVIYFEKSA-N 0.000 description 1
- 229960005420 etoposide Drugs 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- -1 fiudarabine Chemical compound 0.000 description 1
- 229960000961 floxuridine Drugs 0.000 description 1
- ODKNJVUHOIMIIZ-RRKCRQDMSA-N floxuridine Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C(F)=C1 ODKNJVUHOIMIIZ-RRKCRQDMSA-N 0.000 description 1
- 229960000390 fludarabine Drugs 0.000 description 1
- GIUYCYHIANZCFB-FJFJXFQQSA-N fludarabine phosphate Chemical compound C1=NC=2C(N)=NC(F)=NC=2N1[C@@H]1O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O GIUYCYHIANZCFB-FJFJXFQQSA-N 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 238000011331 genomic analysis Methods 0.000 description 1
- 230000011132 hemopoiesis Effects 0.000 description 1
- 210000004251 human milk Anatomy 0.000 description 1
- 235000020256 human milk Nutrition 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 229960000908 idarubicin Drugs 0.000 description 1
- HOMGKSMUEGBAAB-UHFFFAOYSA-N ifosfamide Chemical compound ClCCNP1(=O)OCCCN1CCCl HOMGKSMUEGBAAB-UHFFFAOYSA-N 0.000 description 1
- 229960001101 ifosfamide Drugs 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 229960005386 ipilimumab Drugs 0.000 description 1
- 229960004768 irinotecan Drugs 0.000 description 1
- UWKQSNNFCGGAFS-XIFFEERXSA-N irinotecan Chemical compound C1=C2C(CC)=C3CN(C(C4=C([C@@](C(=O)OC4)(O)CC)C=4)=O)C=4C3=NC2=CC=C1OC(=O)N(CC1)CCC1N1CCCCC1 UWKQSNNFCGGAFS-XIFFEERXSA-N 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 238000011528 liquid biopsy Methods 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 229960002247 lomustine Drugs 0.000 description 1
- 210000002751 lymph Anatomy 0.000 description 1
- 238000002595 magnetic resonance imaging Methods 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- HAWPXGHAZFHHAD-UHFFFAOYSA-N mechlorethamine Chemical compound ClCCN(C)CCCl HAWPXGHAZFHHAD-UHFFFAOYSA-N 0.000 description 1
- 229960004961 mechlorethamine Drugs 0.000 description 1
- SGDBTWWWUNNDEQ-LBPRGKRZSA-N melphalan Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N(CCCl)CCCl)C=C1 SGDBTWWWUNNDEQ-LBPRGKRZSA-N 0.000 description 1
- 229960001924 melphalan Drugs 0.000 description 1
- GLVAUDGFNGKCSF-UHFFFAOYSA-N mercaptopurine Chemical compound S=C1NC=NC2=C1NC=N2 GLVAUDGFNGKCSF-UHFFFAOYSA-N 0.000 description 1
- 229960001428 mercaptopurine Drugs 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 229960001156 mitoxantrone Drugs 0.000 description 1
- KKZJGLLVHKMTCM-UHFFFAOYSA-N mitoxantrone Chemical compound O=C1C2=C(O)C=CC(O)=C2C(=O)C2=C1C(NCCNCCO)=CC=C2NCCNCCO KKZJGLLVHKMTCM-UHFFFAOYSA-N 0.000 description 1
- 239000000203 mixture Substances 0.000 description 1
- YOHYSYJDKVYCJI-UHFFFAOYSA-N n-[3-[[6-[3-(trifluoromethyl)anilino]pyrimidin-4-yl]amino]phenyl]cyclopropanecarboxamide Chemical compound FC(F)(F)C1=CC=CC(NC=2N=CN=C(NC=3C=C(NC(=O)C4CC4)C=CC=3)C=2)=C1 YOHYSYJDKVYCJI-UHFFFAOYSA-N 0.000 description 1
- 238000009099 neoadjuvant therapy Methods 0.000 description 1
- 210000005170 neoplastic cell Anatomy 0.000 description 1
- DWAFYCQODLXJNR-BNTLRKBRSA-L oxaliplatin Chemical compound O1C(=O)C(=O)O[Pt]11N[C@@H]2CCCC[C@H]2N1 DWAFYCQODLXJNR-BNTLRKBRSA-L 0.000 description 1
- 229960001756 oxaliplatin Drugs 0.000 description 1
- 229960001592 paclitaxel Drugs 0.000 description 1
- 238000009595 pap smear Methods 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 230000001575 pathological effect Effects 0.000 description 1
- 229960005079 pemetrexed Drugs 0.000 description 1
- QOFFJEBXNKRSPX-ZDUSSCGKSA-N pemetrexed Chemical compound C1=N[C]2NC(N)=NC(=O)C2=C1CCC1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 QOFFJEBXNKRSPX-ZDUSSCGKSA-N 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 238000000513 principal component analysis Methods 0.000 description 1
- CPTBDICYNRMXFX-UHFFFAOYSA-N procarbazine Chemical compound CNNCC1=CC=C(C(=O)NC(C)C)C=C1 CPTBDICYNRMXFX-UHFFFAOYSA-N 0.000 description 1
- 229960000624 procarbazine Drugs 0.000 description 1
- 230000002250 progressing effect Effects 0.000 description 1
- 208000037821 progressive disease Diseases 0.000 description 1
- 102000004169 proteins and genes Human genes 0.000 description 1
- 238000011155 quantitative monitoring Methods 0.000 description 1
- 230000002829 reductive effect Effects 0.000 description 1
- 230000008439 repair process Effects 0.000 description 1
- 229930002330 retinoic acid Natural products 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 238000013515 script Methods 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- 210000002966 serum Anatomy 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 230000037439 somatic mutation Effects 0.000 description 1
- 238000010183 spectrum analysis Methods 0.000 description 1
- 210000003802 sputum Anatomy 0.000 description 1
- 208000024794 sputum Diseases 0.000 description 1
- 229960001052 streptozocin Drugs 0.000 description 1
- ZSJLQEPLLKMAKR-GKHCUFPYSA-N streptozocin Chemical compound O=NN(C)C(=O)N[C@H]1[C@@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O ZSJLQEPLLKMAKR-GKHCUFPYSA-N 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 208000024891 symptom Diseases 0.000 description 1
- 238000012353 t test Methods 0.000 description 1
- 229950003999 tafluposide Drugs 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 1
- 229940066453 tecentriq Drugs 0.000 description 1
- 229960004964 temozolomide Drugs 0.000 description 1
- NRUKOCRGYNPUPR-QBPJDGROSA-N teniposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@@H](OC[C@H]4O3)C=3SC=CC=3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 NRUKOCRGYNPUPR-QBPJDGROSA-N 0.000 description 1
- 229960001278 teniposide Drugs 0.000 description 1
- 229960003087 tioguanine Drugs 0.000 description 1
- MNRILEROXIRVNJ-UHFFFAOYSA-N tioguanine Chemical compound N1C(N)=NC(=S)C2=NC=N[C]21 MNRILEROXIRVNJ-UHFFFAOYSA-N 0.000 description 1
- 229960000303 topotecan Drugs 0.000 description 1
- UCFGDBYHRUNTLO-QHCPKHFHSA-N topotecan Chemical compound C1=C(O)C(CN(C)C)=C2C=C(CN3C4=CC5=C(C3=O)COC(=O)[C@]5(O)CC)C4=NC2=C1 UCFGDBYHRUNTLO-QHCPKHFHSA-N 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 230000005945 translocation Effects 0.000 description 1
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 1
- 229940121358 tyrosine kinase inhibitor Drugs 0.000 description 1
- 239000005483 tyrosine kinase inhibitor Substances 0.000 description 1
- 150000004917 tyrosine kinase inhibitor derivatives Chemical class 0.000 description 1
- 229960001055 uracil mustard Drugs 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 229960000653 valrubicin Drugs 0.000 description 1
- ZOCKGBMQLCSHFP-KQRAQHLDSA-N valrubicin Chemical compound O([C@H]1C[C@](CC2=C(O)C=3C(=O)C4=CC=CC(OC)=C4C(=O)C=3C(O)=C21)(O)C(=O)COC(=O)CCCC)[C@H]1C[C@H](NC(=O)C(F)(F)F)[C@H](O)[C@H](C)O1 ZOCKGBMQLCSHFP-KQRAQHLDSA-N 0.000 description 1
- 229960003048 vinblastine Drugs 0.000 description 1
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 1
- 229960004528 vincristine Drugs 0.000 description 1
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 1
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 1
- 229960004355 vindesine Drugs 0.000 description 1
- UGGWPQSBPIFKDZ-KOTLKJBCSA-N vindesine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(N)=O)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1N=C1[C]2C=CC=C1 UGGWPQSBPIFKDZ-KOTLKJBCSA-N 0.000 description 1
- 229960002066 vinorelbine Drugs 0.000 description 1
- GBABOYUKABKIAF-GHYRFKGUSA-N vinorelbine Chemical compound C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC GBABOYUKABKIAF-GHYRFKGUSA-N 0.000 description 1
- 238000012800 visualization Methods 0.000 description 1
- 229940055760 yervoy Drugs 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
- C12Q1/6874—Methods for sequencing involving nucleic acid arrays, e.g. sequencing by hybridisation
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1068—Template (nucleic acid) mediated chemical library synthesis, e.g. chemical and enzymatical DNA-templated organic molecule synthesis, libraries prepared by non ribosomal polypeptide synthesis [NRPS], DNA/RNA-polymerase mediated polypeptide synthesis
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
- C12Q1/6886—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material for cancer
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B30/00—ICT specially adapted for sequence analysis involving nucleotides or amino acids
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B30/00—ICT specially adapted for sequence analysis involving nucleotides or amino acids
- G16B30/10—Sequence alignment; Homology search
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
- G16B40/30—Unsupervised data analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B50/00—ICT programming tools or database systems specially adapted for bioinformatics
- G16B50/20—Heterogeneous data integration
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/118—Prognosis of disease development
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/156—Polymorphic or mutational markers
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F17/00—Digital computing or data processing equipment or methods, specially adapted for specific functions
- G06F17/10—Complex mathematical operations
- G06F17/18—Complex mathematical operations for evaluating statistical data, e.g. average values, frequency distributions, probability functions, regression analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
- G16B40/20—Supervised data analysis
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Organic Chemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Physics & Mathematics (AREA)
- General Health & Medical Sciences (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Genetics & Genomics (AREA)
- Analytical Chemistry (AREA)
- Immunology (AREA)
- Medical Informatics (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Bioinformatics & Computational Biology (AREA)
- Pathology (AREA)
- Theoretical Computer Science (AREA)
- Spectroscopy & Molecular Physics (AREA)
- Evolutionary Biology (AREA)
- Public Health (AREA)
- Oncology (AREA)
- Hospice & Palliative Care (AREA)
- Data Mining & Analysis (AREA)
- Databases & Information Systems (AREA)
- Bioethics (AREA)
- Artificial Intelligence (AREA)
- Evolutionary Computation (AREA)
- Epidemiology (AREA)
- Computer Vision & Pattern Recognition (AREA)
- Software Systems (AREA)
- Biomedical Technology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Medicinal Chemistry (AREA)
Description
本願は、2018年5月18日に出願された米国特許出願第62/673,516号の恩典を主張し、2019年1月23日に出願された米国特許出願第62/795,900号の恩典を主張する。先行出願の開示は、本願の開示の一部と考えられる(および参照により、その中に組み込まれる)。
本発明は、国立衛生研究所からの助成金番号CA121113を受けて、アメリカ合衆国政府の支援を得て為された。アメリカ合衆国政府は、本発明に一定の権利を有する。
本文書は、癌を有する哺乳動物(例えば、ヒト)を査定し、および/または処置するための方法および材料に関する。例えば、本文書は、癌(例えば、限局性癌)を有するとして哺乳動物を特定するための方法および材料を提供する。例えば、本文書は、癌を有する哺乳動物をモニタリングし、および/または処置するための方法および材料を提供する。
全世界におけるヒトの癌の罹患率および死亡率の多くは、処置の有効性がより低いこれらの疾患の診断の遅れの結果である(Torreら、2015 CA Cancer J Clin 65:87;およびWorld Health Organization,2017 Guide to Cancer Early Diagnosis)。残念なことに、患者を広く診断および処置するために使用することができる臨床的に証明された生物マーカーは、広く利用可能でない(Mazzucchelli,2000 Advances in clinical pathology 4:111;Ruibal Morell,1992 The International journal of biological markers 7:160;Galliら、2013 Clinical chemistry and laboratory medicine 51:1369;Sikaris,2011 Heart,lung&circulation 20:634;Linら、2016 in Screening for Colorectal Cancer:A Systematic Review for the U.S.Preventive Services Task Force.(Rockville,MD);Waneboら、1978 N Engl J Med 299:448;およびZauber,2015 Dig Dis Sci 60:681)。
セルフリーDNAの分析は、特定の遺伝子の標的化配列決定に主に焦点を当ててきた。このような研究は、癌を有する患者中の少数の腫瘍特異的変動の検出を可能にするが、全ての患者が、とりわけ、早期ステージの疾患を有する患者が検出可能な変化を有するわけではない。セルフリーDNAの全ゲノム配列決定は、癌患者中の染色体異常および再編成を同定することができるが、このような変動の検出は、1つには、少数の異常な染色体変化を正常な染色体変化と区別することが難しいために困難であった(Learyら、2010 Sci Transl Med 2:20ra14;およびLearyら、2012 Sci Transl Med 4:162ra154)。その他の取り組みは、癌と正常な組織の間で、ヌクレオソームパターンおよびクロマチン構造が異なり得ること、ならびに癌を有する患者中のcfDNAが異常なcfDNA断片サイズおよび位置をもたらし得ることを示唆してきた(Snyderら、2016 Cell 164:57;Jahrら、2001 Cancer Res 61:1659;Ivanovら、2015 BMC Genomics 16(Suppl 13):S1)。しかしながら、cfDNAのヌクレオソームフットプリント分析のために必要とされる配列決定の量は、日常的な分析に対しては実現困難である。
患者および試料の特徴
健康な個体から得た血漿試料ならびに乳癌、肺癌、卵巣癌、結腸直腸癌、胆管癌または胃癌を有する患者から得た血漿および組織試料は、ILSBio/Bioreclamation、オーフス大学、コペンハーゲン大学のヘアレウ病院、ヴィドーヴェ(Hvidovre)病院、ユトレヒト大学の大学附属病院、アムステルダム大学の大学附属病院、オランダ癌研究所および、カリフォルニア大学サンディエゴ校から入手した。全ての試料は、参加機関での研究使用に対するインフォームドコンセントを得て、施設内治験審査委員会によって承認されたプロトコールの下で入手した。健康な個体からの血漿試料は、大腸内視鏡検査またはパパニコロースメアなど日常的なスクリーニングの際に取得した。癌の既往歴がなく、陰性のスクリーニング結果であれば、個体は健康と考えた。
生きて凍結されたリンパ球を、健康な男性(C0618)および女性(D0808-L)から得た白血球から浄化した(Advanced Biotechnologies Inc.,Eldersburg,MD)。EZ Nucleosomal DNA Prep Kit(Zymo Research,Irvine,CA)を用いるヌクレオソームDNA精製のために、1×106細胞の分割試料を使用した。最初に、100μlのNuclei Prep Bufferで細胞を処理し、氷上で5分間インキュベートした。200gで5分間の遠心後、上清を廃棄し、
100μlのAtlantis Digestion Bufferでまたは100μLの小球菌ヌクレアーゼ(MN)Digestion Bufferで、沈降した核を2回処理した。最後に、42℃で20分間、0.5UのAtlantis dsDNアーゼで、または37℃で20分間、1.5UのMNアーゼで、細胞の核DNAを断片化した。5X MN Stop Bufferを用いて反応を停止させ、Zymo-Spin(商標)IIC Columnを用いて、DNAを精製した。Bioanalyzer 2100(Agilent Technologies,Santa Clara,CA)を用いて、溶出された細胞核DNAの濃度および品質を分析した。
全血は、EDTA管中に集めて、直ちにもしくは4℃での保存後1日以内に処理するか、またはモニタリング分析の一員であった3人の癌患者については、Streck管中に集め、収集の2日以内に処理した。800gで、10分間、4℃での遠心によって、血漿および細胞成分を分離した。任意の残存する細胞破片を除去するために、再度、18,000g、室温で、血漿を遠心し、DNA抽出の時点まで-80℃で保存した。Qiagen Circulating Nucleic Acids Kit(Qiagen GmbH)を用いて、血漿からDNAを単離し、LoBind管(Eppendorf AG)中に溶出した。Bioanalyzer 2100(Agilent Technologies)を用いて、cfDNAの濃度および品質を査定した。
cfDNA試料に対する標的化NGSデータの分析は、他の文献に記載されているとおりに実施した(例えば、Phallenら、2017 Sci Transl Med 9:eaan2415参照)。簡潔に述べると、Illumina CASAVA(Consensus Assessment of Sequence and Variation)ソフトウェア(バージョン1.8)を用いて、デュアルインデックスアダプター配列の逆多重化およびマスキングを含む、一次処理を完了した。NovoAlignを用いて、配列読み取りデータをヒト参照ゲノム(バージョンhg18またはhg19)に対して並列し、ニードルマン・ウンシュ法を用いて、選択した領域をさらに再並列した(例えば、Jonesら、2015 Sci Transl Med 7:283ra53参照)。配列変動の位置は、異なるゲノムビルドによって影響を受けなかった。関心対象の標的化領域にわたって、VariantDxを用いて(例えば、Jonesら、2015 Sci Transl Med 7:283ra53参照)(Personal Genome Diagnostics,Baltimore,MD)、点変異、小さな挿入および欠失からなる候補変異を同定した。
Illumina CASAVA(Consensus Assessment of Sequence and Variation)ソフトウェア(バージョン1.8.2)を用いて、デュアルインデックスアダプター配列の逆多重化およびマスキングを含む、cfDNA試料に対する全ゲノムNGSデータの一次処理を行った。配列読み取りデータは、ELANDを用いて、ヒト参照ゲノム(バージョンhg19)に対して並列した。
コピー数変化に対する腕レベルでの統計を開発するために、他の文献に記載されているとおりの血漿中の異数性検出のためのアプローチ(例えば、Learyら、2012 Sci Transl Med 4:162ra154参照)を採用した。このアプローチは、ゲノムを非重複50KBビンに分割し、スパンを3/4とするloessによる補正後に、このビンに対して、GC補正されたlog2リードデプスが得られた。このloessをベースとする補正は、上に概述されているアプローチと同等であったが、より小さなビンにおける外れ値への頑健性を増加させるためにlog2スケールで評価され、断片長によって層別化しない。コピー数変化に対する腕特異的Zスコアを得るために、それぞれ、50の健康な試料の独立した組から得られたGRスコアの平均および標準偏差によって、各腕に対するGC調整された平均リードデプス(GR)を中心配置し、スケール調整した。
ミトコンドリアのゲノムに当初マッピングした全ゲノム配列読み取りデータをbamファイルから抽出し、他の文献に記載されているとおり(例えば、Langmeadら、2012 Nat Methods 9:357-359参照)、Bowtie2を用いて、エンドツーエンドモードで、hg19参照ゲノムに再並列した。両方のメイトが30以上のMAPQでミトコンドリアゲノムに並列するように、得られた並列された読み取りデータにフィルターをかけた。ミトコンドリアゲノムにマッピングする断片の数を計数し、元のbamファイル中の断片の総数の百分率に変換した。
断片化プロファイルを用いて健康者を癌患者と区別するために、確率的勾配ブースティングモデルを使用した(gbm(gradient boosting model);例えば、Friedmanら、2001 Ann Stat 29:1189-1232;およびFriedmanら、2002 Comput Stat Data An 38:367-378参照)。平均0および単位標準偏差を有するように、各試料について、全ての504のビンに対するGC補正された総および短断片カバレッジを中心配置し、スケール調整した。さらなる特徴には、39の常染色体腕およびミトコンドリア表現の各々に対するZスコアが含まれた(log10変換された、ミトコンドリアにマッピングされた読み取りデータの割合)。このアプローチの予測誤差を推定するために、他の文献に記載されているとおりに、10分割交差検証を使用した(例えば、Efronら、1997 J Am Stat Assoc 92,548-560参照)。各交差検証実行において訓練データに対してのみ行われた特徴選択は、高度に相関した(相関>0.9)またはほぼゼロの分散を有するビンを除去した。パラメータをn.trees=150、interaction.depth=3、shrinkage=0.1およびn.minobsinside=10として、Rパッケージ、gbmパッケージを用いて、確率的勾配ブースト機械学習を実行した。分割への患者の無作為化から生じる予測誤差を平均するために、10分割交差検証手順を10回繰り返した。2000ブートストラップの反復から98%に固定された感度および95%特異性に対する信頼区間が得られた。
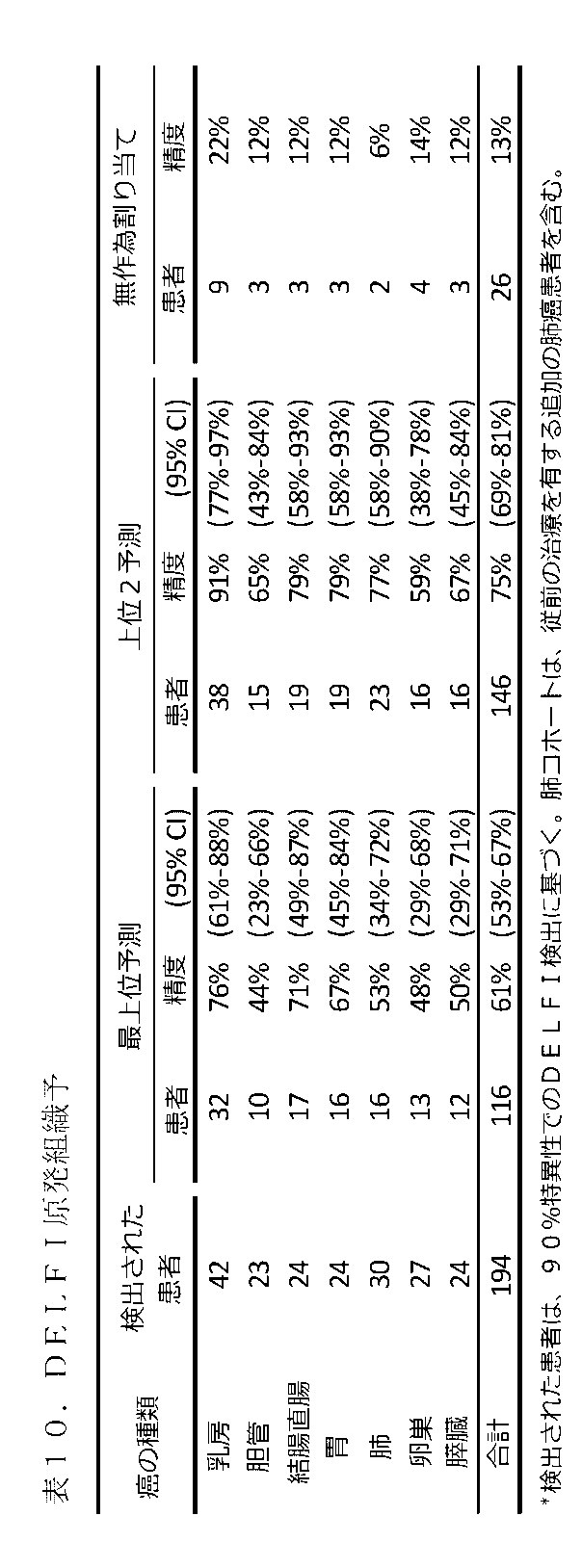
90%の特異性で癌患者として正しく分類される試料に対して(n=174)、原発組織を分類するために、別個の確率的勾配ブースティングモデルを訓練した。予測のために使用した肺試料の数が少ないことを考慮するために、後期ステージ肺癌患者から得た18cfDNAベースライン試料がモニタリング分析から含められた。10分割交差検証を10回反復することによって、モデルの性能特性を評価した。このgbmモデルは、癌分類モデルにおけるのと同じ特徴を用いて訓練された。先述のとおり、交差検証の間に各訓練データセット内で、互いに0.9を上回る相関を示した特徴またはほぼゼロの分散を有した特徴は除去した。組織クラス確率は、各患者につき10回の反復にわたって平均し、最高の確率を有するクラスを予測される組織とみなした。
ヌクレアーゼ処理されたリンパ球から、全ゲノムcfDNA分析に関して記載されているとおりに、断片サイズを5Mbビンで分析した。ヌクレアーゼ処理されたリンパ球細胞株から、ヌクレオソーム位置の全ゲノムマップを構築した。このアプローチによって、循環する断片のカバレッジの局所的なバイアスが特定され、分解から保護された領域を示している。ゲノム中の各塩基対にスコア付けするために、「ウィンドウ・ポジショニング・スコア」(WPS)を使用した(例えば、Snyderら、2016 Cell 164:57参照)。各塩基周囲を中心とした60bpのスライドするウィンドウを用いて、ウィンドウに完全にまたがる断片の数から1つの末端のみがウィンドウ中に存在する断片の数を差し引いたものとして、WPSを計算した。ヌクレオソームから生じる断片は167bpの中央値長を有するので、高いWPSは、ヌクレオソームの位置の可能性があることを示唆した。移動中央値を用いて、WPSスコアをゼロで中央に配置し、コルモゴロフ・ズルベンコフィルターを用いて平滑化した(例えば、Zurbenko,The spectral analysis of time series。North-Holland series in statistics and probability;Elsevier,New York,NY,1986参照)。50~450bpの正のWPSのスパンについては、ヌクレオソームピークは、そのウィンドウにおける中央値を上回るWPSを有する塩基対の組として定義した。9×の配列カバレッジでの、30人の健康な個体から得たcfDNAに対するヌクレオソーム位置の計算を、リンパ球DNAに対するのと同様に決定した。健康なcfDNA中のヌクレオソームが代表的であったことを確保するために、2またはそれを超える個体中に同定されたヌクレオソームのみからなる、ヌクレオソームのコンセンサストラックを定義した。隣接するヌクレオソーム間の距離中央値をコンセンサストラックから計算した。
腫瘍由来の変動を有する分子を検出する確率を推定するために、モンテカルロシミュレーションを使用した。簡潔に述べると、多項分布から100万分子を生成した。m変動でのシミュレーションのために、確率pで野生型分子をシミュレートし、m腫瘍変動の各々を確率(1-p)/mでシミュレートした。次に、g*m分子を復元的に無作為抽出した、ここで、gは、血漿1ml中のゲノム当量の数を表す。腫瘍変動がsまたはそれを超える回数抽出されれば、そのサンプルは癌由来と分類した。シミュレーションを1000回繰り返して、癌指標によって、コンピュータシミュレーションでのサンプルが癌として正しく分類される確率を推定する。g=2000およびs=5に設定して、腫瘍変動の数を2乗ずつ1から256まで、腫瘍由来分子の割合を0.0001%から1%まで変動させた。
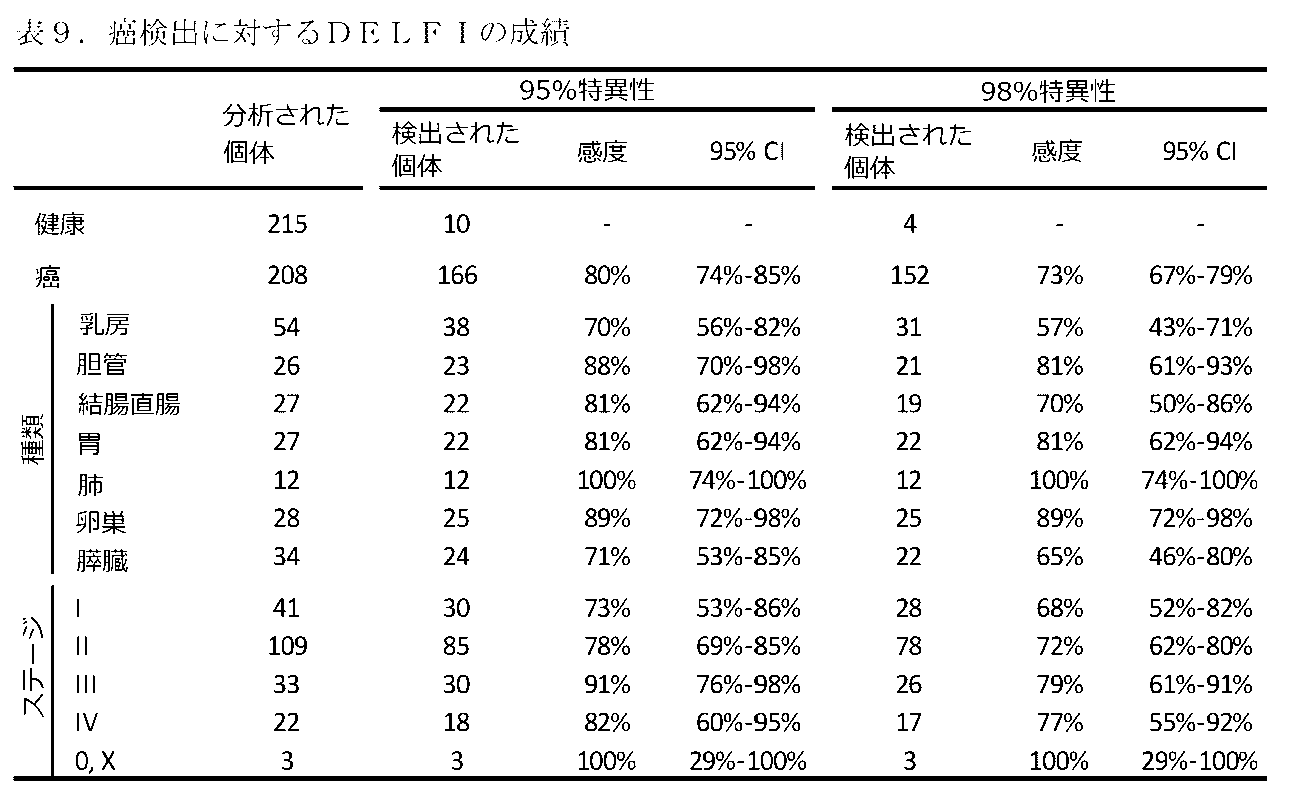
全ての統計解析は、Rバージョン3.4.3を用いて行った。健康対癌および原発組織の分類を実施するために、Rパッケージcaret(バージョン6.0-79)およびgbm(バージョン2.1-4)を使用した。モデル出力からの信頼区間は、pROC(バージョン1.13)Rパッケージを用いて取得した(例えば、Robinら、2011 BMC bioinformatics 12:77参照)。この集団中での診断されていない癌症例の有病率が高い(100人の健康者当たり1または2症例)と仮定すると、0.95の特異性および0.8の感度を有するゲノムアッセイは、有用な操作特性(0.25の陽性的中率およびほぼ1の陰性的中率)を有するであろう。検定力計算は、200人超の癌患者およびほぼ等しい数の健康な対照の分析が、0.95またはそれを超える所望の特異性で、0.06の許容誤差での感度の推定が可能となることを示唆する。
本研究において使用された配列データは、研究アクセッション番号EGAS00001003611およびEGAS00001002577で、European Genome-phenome Archiveに寄託した。分析のためのコードは、github.com/Cancer-Genomics/delfi_scriptsで入手可能である。
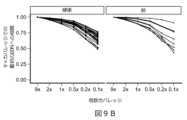
DELFIは、断片化パターンの全ゲノム分析を通じて、cfDNA中の多数の異常の同時分析を可能にする。この方法は、低カバレッジ全ゲノム配列決定および単離されたcfDNAの分析を基礎とする。マッピングされた配列は、ゲノムを覆う非重複ウィンドウ中で分析される。概念的には、ウィンドウのサイズは、数千~数百万塩基の範囲であり得、ゲノム中に、数百~数千のウィンドウがもたらされる。1~2×ゲノムカバレッジという限られた量でさえ、ウィンドウ当たり20,000超の読み取りデータを与えるので、cfDNA断片化パターンを評価するために、5Mbウィンドウを使用した。各ウィンドウ内で、cfDNA断片のカバレッジおよびサイズ分布を調べた。健康な集団および癌集団中での全ゲノム断片化プロファイルの変動を評価するために、このアプローチを使用した(表1;添付書類A)。パターンが健康であるまたは癌に由来する可能性があるかどうかを決定するために、ある個体からの全ゲノムパターンを参照集団と比較することができる。全ゲノムプロファイルは、全般的な断片サイズ分布においては見逃され得る特定組織に付随する位置的差異を明らかにするので、これらのパターンは、cfDNAの組織源も示唆し得る。
野生型配列の当該領域より3番染色体の位置41,266,124で30塩基小さく、11番染色体の位置108,117,753で47塩基大きいという範囲であった(表3;添付書類C)。変異した断片と変異していない断片に対してGC含量は同様であり(図4a)、GC含量と断片長の間に相関は存在しなかった(図4b)。38人の患者から得られた44の生殖系列変動の同様の分析は、異なる対立遺伝子の断片長間に1bp未満の中央値cfDNAサイズ差を特定した(図5、表3(添付書類C))。さらに、同一個体の血漿、バフィーコートおよび腫瘍から得たDNAの以前の配列比較を通じて、クローン性造血に関連する41の変動が特定された。腫瘍由来の断片とは異なり、造血性変動を有する断片と野生型断片との間に有意な差は存在しなかった(図6、表3(添付書類C))。総合すると、癌由来cfDNA断片長は、ある種のゲノム領域において、非癌cfDNA断片と比較して、有意により可変的であった(p<0.001、分散比検定)。これらの差は、高次クロマチン構造の変化の他、癌中でのその他のゲノムおよびエピゲノム異常によるものであり得ること、位置特異的な態様でのcfDNA断片化は、それ故、癌検出のための特有の生物マーカーとしての役割を果たすことが仮定された。
他の実施形態
Claims (13)
- 癌を有するとして哺乳動物を特定する方法であって、
前記哺乳動物から取得された試料から取得されたセルフリーDNA(cfDNA)断片を加工して配列決定ライブラリーにすること;
配列決定された断片を取得するために、前記配列決定ライブラリーを低カバレッジ全ゲノム配列決定に供すること;
マッピングされた配列のウィンドウを取得するために、前記配列決定された断片をゲノムにマッピングすること;および
cfDNA断片長を決定するために、マッピングされた配列の前記ウィンドウを分析し、前記cfDNA断片長を参照cfDNA断片化プロファイルと比較すること、ここで前記参照cfDNA断片化プロファイルは健康な哺乳動物のcfDNA断片化プロファイルである;および
前記哺乳動物から得られた前記cfDNA断片化プロファイルが前記参照cfDNA断片化プロファイルと異なる場合に、癌を有するとして前記哺乳動物を特定すること;
を含む、方法(ただし、ヒトに対する医療行為を除く)。 - 前記マッピングされた配列が数十~数千のウィンドウを含む、請求項1に記載の方法。
- 前記ウィンドウが非重複ウィンドウである、請求項1~2に記載の方法。
- 前記ウィンドウが、それぞれ約500万の塩基対を含む、請求項1~3のいずれか一項に記載の方法。
- cfDNA断片化プロファイルが各ウィンドウ内で決定される、請求項1~4のいずれか一項に記載の方法。
- cfDNA断片化プロファイルが断片サイズ中央値を含む、請求項1~5のいずれか一項に記載の方法。
- cfDNA断片化プロファイルが断片サイズ分布を含む、請求項1~5のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルが、マッピングされた配列の前記ウィンドウ中での、長さ151bp~長さ220bpである大きなcfDNA断片に対する長さ100bp~長さ150bpである小さなcfDNA断片の比率を含む、請求項1~5のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルが、ゲノムにわたるウィンドウ中での小さなcfDNA断片の配列カバレッジを含む、請求項1~5のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルが、ゲノムにわたるウィンドウ中での大きなcfDNA断片の配列カバレッジを含む、請求項1~5のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルが、ゲノムにわたるウィンドウ中での小さなおよび大きなcfDNA断片の配列カバレッジを含む、請求項1~5のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルが全ゲノムにわたる、請求項1~11のいずれか一項に記載の方法。
- 前記cfDNA断片化プロファイルがサブゲノム区間にわたる、請求項1~11のいずれか一項に記載の方法。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2024032720A JP2024069295A (ja) | 2018-05-18 | 2024-03-05 | 癌を査定および/または処置するためのセルフリーdna |
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201862673516P | 2018-05-18 | 2018-05-18 | |
| US62/673,516 | 2018-05-18 | ||
| US201962795900P | 2019-01-23 | 2019-01-23 | |
| US62/795,900 | 2019-01-23 | ||
| PCT/US2019/032914 WO2019222657A1 (en) | 2018-05-18 | 2019-05-17 | Cell-free dna for assessing and/or treating cancer |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2024032720A Division JP2024069295A (ja) | 2018-05-18 | 2024-03-05 | 癌を査定および/または処置するためのセルフリーdna |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2021525069A JP2021525069A (ja) | 2021-09-24 |
| JPWO2019222657A5 JPWO2019222657A5 (ja) | 2022-05-24 |
| JP7531217B2 true JP7531217B2 (ja) | 2024-08-09 |
Family
ID=68541029
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2020564491A Active JP7531217B2 (ja) | 2018-05-18 | 2019-05-17 | 癌を査定および/または処置するためのセルフリーdna |
| JP2024032720A Pending JP2024069295A (ja) | 2018-05-18 | 2024-03-05 | 癌を査定および/または処置するためのセルフリーdna |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2024032720A Pending JP2024069295A (ja) | 2018-05-18 | 2024-03-05 | 癌を査定および/または処置するためのセルフリーdna |
Country Status (9)
| Country | Link |
|---|---|
| US (5) | US20210198747A1 (ja) |
| EP (1) | EP3794348A4 (ja) |
| JP (2) | JP7531217B2 (ja) |
| KR (1) | KR20210045953A (ja) |
| CN (1) | CN112805563A (ja) |
| AU (1) | AU2019269679A1 (ja) |
| BR (1) | BR112020023587A2 (ja) |
| CA (1) | CA3100345A1 (ja) |
| WO (1) | WO2019222657A1 (ja) |
Families Citing this family (20)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20210045953A (ko) | 2018-05-18 | 2021-04-27 | 더 존스 홉킨스 유니버시티 | 암의 평가 및/또는 치료를 위한 무 세포 dna |
| GB201818159D0 (en) * | 2018-11-07 | 2018-12-19 | Cancer Research Tech Ltd | Enhanced detection of target dna by fragment size analysis |
| JP2023507796A (ja) * | 2019-12-19 | 2023-02-27 | カデックス ゲノミクス、コーポレイション | がん特異的無細胞dnaを同定するための異なるフラグメントサイズを使用したマルチプレックスアッセイ |
| CN111088303A (zh) * | 2020-01-08 | 2020-05-01 | 杭州瑞普基因科技有限公司 | 模拟血浆cfDNA及制备方法和测序文库的构建方法 |
| AU2021240092A1 (en) * | 2020-03-20 | 2022-10-27 | Mission Bio, Inc. | Single cell workflow for whole genome amplification |
| CA3173453A1 (en) * | 2020-03-26 | 2021-09-30 | Wai Yi Tsui | Systems and methods for distinguishing pathological mutations from clonal hematopoietic mutations in plasma cell-free dna by fragment size analysis |
| CN115715330A (zh) * | 2020-05-12 | 2023-02-24 | 小利兰·斯坦福大学托管委员会 | 用于从无细胞dna中推断基因表达和起源组织的系统和方法 |
| EP4169025A4 (en) * | 2020-06-22 | 2024-07-10 | Childrens Hospital Med Ct | DE NOVO CHARACTERIZATION OF CELL DNA FRAGMENTATION HOT SPOTS IN HEALTHY AND EARLY STAGE CANCER SUBJECTS |
| WO2022108407A1 (ko) * | 2020-11-23 | 2022-05-27 | 주식회사 녹십자지놈 | 핵산 길이 비를 이용한 암 진단 및 예후예측 방법 |
| CN117561340A (zh) * | 2021-04-08 | 2024-02-13 | 德尔菲诊断公司 | 使用全基因组cfDNA片段化图谱检测癌症的方法 |
| CA3214391A1 (en) * | 2021-04-08 | 2022-10-13 | Gavin HA | Cell-free dna sequence data analysis method to examine nucleosome protection and chromatin accessibility |
| KR20220160806A (ko) | 2021-05-28 | 2022-12-06 | 주식회사 지씨지놈 | 세포유리 핵산단편 말단 서열 모티프 빈도 및 크기를 이용한 암 진단 및 암 종 예측방법 |
| KR20230059423A (ko) | 2021-10-26 | 2023-05-03 | 주식회사 지씨지놈 | 메틸화된 무세포 핵산을 이용한 암 진단 및 암 종 예측방법 |
| KR20230064172A (ko) | 2021-11-03 | 2023-05-10 | 주식회사 지씨지놈 | 세포유리 핵산단편 위치별 서열 빈도 및 크기를 이용한 암 진단 방법 |
| WO2023200404A2 (en) * | 2022-04-13 | 2023-10-19 | Lucence Life Sciences Pte. Ltd. | Method for determining cfdna fragment size ratio and fragment size distribution |
| WO2023220414A1 (en) * | 2022-05-12 | 2023-11-16 | Delfi Diagnostics, Inc. | Use of cell-free dna fragmentomes in the diagnostic evaluation of patients with signs and symptoms suggestive of cancer |
| CN115558716B (zh) * | 2022-09-29 | 2023-11-03 | 南京医科大学 | 一种用于预测癌症的cfDNA片段特征组合、系统及应用 |
| KR20240051739A (ko) * | 2022-10-13 | 2024-04-22 | 인하대학교 산학협력단 | cfDNA의 구조 변이 서열 검출을 통한 미세잔존질환 진단 방법 |
| CN118028462A (zh) * | 2022-11-02 | 2024-05-14 | 深圳湾实验室 | 一种泛癌种血液诊断标志物及其应用 |
| WO2024098073A1 (en) * | 2022-11-06 | 2024-05-10 | The Johns Hopkins University | Detecting liver cancer using cell-free dna fragmentation |
Family Cites Families (19)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20090280479A1 (en) | 2005-05-27 | 2009-11-12 | John Wayne Cancer Institute | Use of free circulating dna for diagnosis, prognosis, and treatment of cancer funding |
| BR122021026173B1 (pt) | 2010-12-09 | 2023-12-05 | The Trustees Of The University Of Pennsylvania | Composição farmacêutica |
| WO2012082841A2 (en) | 2010-12-14 | 2012-06-21 | University Of Maryland, Baltimore | Universal anti-tag chimeric antigen receptor-expressing t cells and methods of treating cancer |
| CN103748236B (zh) | 2011-04-15 | 2018-12-25 | 约翰·霍普金斯大学 | 安全测序系统 |
| WO2013096843A1 (en) * | 2011-12-21 | 2013-06-27 | Myriad Genetics, Inc. | Methods and materials for assessing loss of heterozygosity |
| US8904664B2 (en) | 2012-08-15 | 2014-12-09 | Mimedx Group, Inc. | Dehydration device and methods for drying biological materials |
| TWI654206B (zh) | 2013-03-16 | 2019-03-21 | 諾華公司 | 使用人類化抗-cd19嵌合抗原受體治療癌症 |
| WO2014201092A1 (en) | 2013-06-11 | 2014-12-18 | Dana-Farber Cancer Institute, Inc. | Non-invasive blood based monitoring of genomic alterations in cancer |
| US9499870B2 (en) | 2013-09-27 | 2016-11-22 | Natera, Inc. | Cell free DNA diagnostic testing standards |
| US20170211143A1 (en) * | 2014-07-25 | 2017-07-27 | University Of Washington | Methods of determining tissues and/or cell types giving rise to cell-free dna, and methods of identifying a disease or disorder using same |
| CN107209190B (zh) * | 2014-10-29 | 2021-07-16 | 比利时意志有限责任公司 | 用于富集循环肿瘤dna的方法 |
| EP3502273B1 (en) * | 2014-12-12 | 2020-07-08 | Verinata Health, Inc. | Cell-free dna fragment |
| US10364467B2 (en) * | 2015-01-13 | 2019-07-30 | The Chinese University Of Hong Kong | Using size and number aberrations in plasma DNA for detecting cancer |
| PT3967775T (pt) * | 2015-07-23 | 2023-10-10 | Univ Hong Kong Chinese | Análise de padrões de fragmentação de adn sem células |
| WO2017190067A1 (en) * | 2016-04-28 | 2017-11-02 | Impact Genomics, Inc. | Methods of assessing and monitoring tumor load |
| WO2018027176A1 (en) * | 2016-08-05 | 2018-02-08 | The Broad Institute, Inc. | Methods for genome characterization |
| US11168356B2 (en) * | 2017-11-02 | 2021-11-09 | The Chinese University Of Hong Kong | Using nucleic acid size range for noninvasive cancer detection |
| US20190352695A1 (en) * | 2018-01-10 | 2019-11-21 | Guardant Health, Inc. | Methods for fragmentome profiling of cell-free nucleic acids |
| KR20210045953A (ko) | 2018-05-18 | 2021-04-27 | 더 존스 홉킨스 유니버시티 | 암의 평가 및/또는 치료를 위한 무 세포 dna |
-
2019
- 2019-05-17 KR KR1020207036527A patent/KR20210045953A/ko unknown
- 2019-05-17 US US17/056,726 patent/US20210198747A1/en active Pending
- 2019-05-17 BR BR112020023587-3A patent/BR112020023587A2/pt unknown
- 2019-05-17 CA CA3100345A patent/CA3100345A1/en active Pending
- 2019-05-17 JP JP2020564491A patent/JP7531217B2/ja active Active
- 2019-05-17 EP EP19803533.9A patent/EP3794348A4/en active Pending
- 2019-05-17 WO PCT/US2019/032914 patent/WO2019222657A1/en active Application Filing
- 2019-05-17 CN CN201980047828.3A patent/CN112805563A/zh active Pending
- 2019-05-17 AU AU2019269679A patent/AU2019269679A1/en active Pending
- 2019-12-30 US US16/730,949 patent/US10975431B2/en active Active
- 2019-12-30 US US16/730,938 patent/US10982279B2/en active Active
-
2021
- 2021-03-17 US US17/204,892 patent/US20210254152A1/en active Pending
-
2022
- 2022-06-17 US US17/842,893 patent/US20220325343A1/en active Pending
-
2024
- 2024-03-05 JP JP2024032720A patent/JP2024069295A/ja active Pending
Non-Patent Citations (2)
| Title |
|---|
| CHANDRANANDA D., et al.,Investigating and Correcting Plasma DNA Sequencing Coverage Bias to Enhance Aneuploidy Discovery,PLOS ONE, 2014, vol. 9, no. 1, p. e86993 |
| MA X. et al.,Cell-Free DNA Provides a Good Representation of the Tumor Genome Despite Its Biased Fragmentation Patterns,PLOS ONE, 2017, vol. 12, no. 1, p. e0169231 |
Also Published As
| Publication number | Publication date |
|---|---|
| US20200149118A1 (en) | 2020-05-14 |
| WO2019222657A1 (en) | 2019-11-21 |
| KR20210045953A (ko) | 2021-04-27 |
| US20220325343A1 (en) | 2022-10-13 |
| US10982279B2 (en) | 2021-04-20 |
| JP2024069295A (ja) | 2024-05-21 |
| CA3100345A1 (en) | 2019-11-21 |
| US10975431B2 (en) | 2021-04-13 |
| EP3794348A4 (en) | 2022-03-09 |
| BR112020023587A2 (pt) | 2021-02-09 |
| JP2021525069A (ja) | 2021-09-24 |
| CN112805563A (zh) | 2021-05-14 |
| US20210198747A1 (en) | 2021-07-01 |
| AU2019269679A1 (en) | 2020-12-17 |
| US20200131571A1 (en) | 2020-04-30 |
| EP3794348A1 (en) | 2021-03-24 |
| US20210254152A1 (en) | 2021-08-19 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7531217B2 (ja) | 癌を査定および/または処置するためのセルフリーdna | |
| CA2784613C (en) | Diagnostic methods based on somatically acquired rearrangement | |
| JP2024105502A (ja) | がんの非侵襲的検出のためのdnaメチル化マーカーとその使用 | |
| CN111876478A (zh) | 肺结节诊断标志物及应用 | |
| WO2021173722A2 (en) | Methods of analyzing cell free nucleic acids and applications thereof | |
| CN116631508B (zh) | 肿瘤特异性突变状态的检测方法及其应用 | |
| US20210087638A1 (en) | Next-generation sequencing assay for genomic characterization and minimal residual disease detection in the bone marrow, peripheral blood, and urine of multiple myeloma and smoldering myeloma patients | |
| WO2012125712A2 (en) | Lung tumor classifier for current and former smokers | |
| EP3095056B1 (en) | Combined cytology and molecular testing for early detection of esophageal adenocarcinoma | |
| Liu et al. | A noninvasive multianalytical approach for lung cancer diagnosis of patients with pulmonary nodules | |
| Chen et al. | Cell-free DNA detection of tumor mutations in heterogeneous, localized prostate cancer via targeted, multiregion sequencing | |
| US20220098677A1 (en) | Method for determining rcc subtypes | |
| CN111968702B (zh) | 一种基于循环肿瘤dna的恶性肿瘤早期筛查系统 | |
| BR112020012280A2 (pt) | composições e métodos para diagnosticar cânceres de pulmão usando perfis de expressão de gene | |
| Oben et al. | Whole genome sequencing provides evidence of two biologically and clinically distinct entities of asymptomatic monoclonal gammopathies: progressive versus stable myeloma precursor condition | |
| WO2021041968A1 (en) | Systems and methods for predicting and monitoring treatment response from cell-free nucleic acids | |
| Pei et al. | Classification of multiple primary lung cancer in patients with multifocal lung cancer: assessment of a machine learning approach using multidimensional genomic data | |
| EP2959298B1 (en) | Methods | |
| CN118406766B (zh) | 预测ii期和iii期结直肠癌预后临床结果的生物标志物、引物组及检测试剂盒 | |
| WO2024098073A1 (en) | Detecting liver cancer using cell-free dna fragmentation | |
| WO2024118500A2 (en) | Methods for detecting and treating ovarian cancer | |
| Gregson | Characterisation of Copy Number Changes in the Progression of Barrett’s Oesophagus | |
| CN118186078A (zh) | 一种新型多靶点肺癌辅助诊断的标志物组合及其应用 | |
| WO2019215223A1 (en) | Method of cancer prognosis by assessing tumor variant diversity by means of establishing diversity indices |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220516 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20220516 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20230616 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20230919 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20231106 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20240305 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20240423 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20240628 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20240723 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7531217 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |