CN108026556B - 在具有经改造的输入/输出的微生物宿主中人乳寡糖的产生 - Google Patents
在具有经改造的输入/输出的微生物宿主中人乳寡糖的产生 Download PDFInfo
- Publication number
- CN108026556B CN108026556B CN201680052611.8A CN201680052611A CN108026556B CN 108026556 B CN108026556 B CN 108026556B CN 201680052611 A CN201680052611 A CN 201680052611A CN 108026556 B CN108026556 B CN 108026556B
- Authority
- CN
- China
- Prior art keywords
- host cell
- export
- protein
- lacto
- nucleic acid
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 229920001542 oligosaccharide Polymers 0.000 title claims abstract description 119
- 150000002482 oligosaccharides Chemical class 0.000 title claims abstract description 119
- 238000004519 manufacturing process Methods 0.000 title claims abstract description 28
- 230000000813 microbial effect Effects 0.000 title claims description 31
- 235000020256 human milk Nutrition 0.000 title claims description 10
- 210000004251 human milk Anatomy 0.000 title claims description 10
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 158
- 102000004169 proteins and genes Human genes 0.000 claims abstract description 91
- 150000007523 nucleic acids Chemical group 0.000 claims abstract description 71
- 108091028043 Nucleic acid sequence Proteins 0.000 claims abstract description 58
- 238000000034 method Methods 0.000 claims abstract description 53
- 108700023372 Glycosyltransferases Proteins 0.000 claims abstract description 18
- 230000001580 bacterial effect Effects 0.000 claims abstract description 16
- 102000051366 Glycosyltransferases Human genes 0.000 claims abstract description 15
- 241000588724 Escherichia coli Species 0.000 claims description 65
- AXQLFFDZXPOFPO-UNTPKZLMSA-N beta-D-Galp-(1->3)-beta-D-GlcpNAc-(1->3)-beta-D-Galp-(1->4)-beta-D-Glcp Chemical compound O([C@@H]1O[C@H](CO)[C@H](O)[C@@H]([C@H]1O)O[C@H]1[C@@H]([C@H]([C@H](O)[C@@H](CO)O1)O[C@H]1[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O1)O)NC(=O)C)[C@H]1[C@H](O)[C@@H](O)[C@H](O)O[C@@H]1CO AXQLFFDZXPOFPO-UNTPKZLMSA-N 0.000 claims description 61
- AXQLFFDZXPOFPO-UHFFFAOYSA-N UNPD216 Natural products O1C(CO)C(O)C(OC2C(C(O)C(O)C(CO)O2)O)C(NC(=O)C)C1OC(C1O)C(O)C(CO)OC1OC1C(O)C(O)C(O)OC1CO AXQLFFDZXPOFPO-UHFFFAOYSA-N 0.000 claims description 60
- USIPEGYTBGEPJN-UHFFFAOYSA-N lacto-N-tetraose Natural products O1C(CO)C(O)C(OC2C(C(O)C(O)C(CO)O2)O)C(NC(=O)C)C1OC1C(O)C(CO)OC(OC(C(O)CO)C(O)C(O)C=O)C1O USIPEGYTBGEPJN-UHFFFAOYSA-N 0.000 claims description 60
- 230000014509 gene expression Effects 0.000 claims description 42
- 239000002243 precursor Substances 0.000 claims description 30
- 108010078791 Carrier Proteins Proteins 0.000 claims description 27
- 230000000694 effects Effects 0.000 claims description 24
- 230000001965 increasing effect Effects 0.000 claims description 20
- 239000001963 growth medium Substances 0.000 claims description 19
- 235000000346 sugar Nutrition 0.000 claims description 19
- IEQCXFNWPAHHQR-UHFFFAOYSA-N lacto-N-neotetraose Natural products OCC1OC(OC2C(C(OC3C(OC(O)C(O)C3O)CO)OC(CO)C2O)O)C(NC(=O)C)C(O)C1OC1OC(CO)C(O)C(O)C1O IEQCXFNWPAHHQR-UHFFFAOYSA-N 0.000 claims description 18
- 102000039446 nucleic acids Human genes 0.000 claims description 18
- 108020004707 nucleic acids Proteins 0.000 claims description 18
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 claims description 17
- 230000002018 overexpression Effects 0.000 claims description 17
- 102100021700 Glycoprotein-N-acetylgalactosamine 3-beta-galactosyltransferase 1 Human genes 0.000 claims description 16
- 101000896564 Homo sapiens Glycoprotein-N-acetylgalactosamine 3-beta-galactosyltransferase 1 Proteins 0.000 claims description 16
- 108010046068 N-Acetyllactosamine Synthase Proteins 0.000 claims description 16
- 238000000855 fermentation Methods 0.000 claims description 16
- 230000004151 fermentation Effects 0.000 claims description 16
- 239000008103 glucose Substances 0.000 claims description 14
- 101150049349 setA gene Proteins 0.000 claims description 13
- 150000004043 trisaccharides Chemical class 0.000 claims description 13
- 101150020448 yjhB gene Proteins 0.000 claims description 13
- OVRNDRQMDRJTHS-UHFFFAOYSA-N N-acelyl-D-glucosamine Natural products CC(=O)NC1C(O)OC(CO)C(O)C1O OVRNDRQMDRJTHS-UHFFFAOYSA-N 0.000 claims description 12
- MBLBDJOUHNCFQT-LXGUWJNJSA-N N-acetylglucosamine Natural products CC(=O)N[C@@H](C=O)[C@@H](O)[C@H](O)[C@H](O)CO MBLBDJOUHNCFQT-LXGUWJNJSA-N 0.000 claims description 12
- HSCJRCZFDFQWRP-UHFFFAOYSA-N Uridindiphosphoglukose Natural products OC1C(O)C(O)C(CO)OC1OP(O)(=O)OP(O)(=O)OCC1C(O)C(O)C(N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-UHFFFAOYSA-N 0.000 claims description 10
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 claims description 9
- LFTYTUAZOPRMMI-CFRASDGPSA-N UDP-N-acetyl-alpha-D-glucosamine Chemical compound O1[C@H](CO)[C@@H](O)[C@H](O)[C@@H](NC(=O)C)[C@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(NC(=O)C=C2)=O)O1 LFTYTUAZOPRMMI-CFRASDGPSA-N 0.000 claims description 9
- LFTYTUAZOPRMMI-UHFFFAOYSA-N UNPD164450 Natural products O1C(CO)C(O)C(O)C(NC(=O)C)C1OP(O)(=O)OP(O)(=O)OCC1C(O)C(O)C(N2C(NC(=O)C=C2)=O)O1 LFTYTUAZOPRMMI-UHFFFAOYSA-N 0.000 claims description 9
- 238000012217 deletion Methods 0.000 claims description 9
- 102000004190 Enzymes Human genes 0.000 claims description 8
- 108090000790 Enzymes Proteins 0.000 claims description 8
- 241000660147 Escherichia coli str. K-12 substr. MG1655 Species 0.000 claims description 8
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 claims description 8
- 150000002016 disaccharides Chemical class 0.000 claims description 8
- 108010060845 lactose permease Proteins 0.000 claims description 8
- 101100159740 Escherichia coli (strain K12) yebQ gene Proteins 0.000 claims description 7
- OVRNDRQMDRJTHS-FMDGEEDCSA-N N-acetyl-beta-D-glucosamine Chemical compound CC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-FMDGEEDCSA-N 0.000 claims description 7
- 241000606856 Pasteurella multocida Species 0.000 claims description 7
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims description 7
- 230000008676 import Effects 0.000 claims description 7
- 229950006780 n-acetylglucosamine Drugs 0.000 claims description 7
- 229940051027 pasteurella multocida Drugs 0.000 claims description 7
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 claims description 6
- 241001293415 Mannheimia Species 0.000 claims description 6
- 229910052799 carbon Inorganic materials 0.000 claims description 6
- 229940062780 lacto-n-neotetraose Drugs 0.000 claims description 6
- RBMYDHMFFAVMMM-PLQWBNBWSA-N neolactotetraose Chemical compound O([C@H]1[C@H](O)[C@H]([C@@H](O[C@@H]1CO)O[C@@H]1[C@H]([C@H](O[C@H]([C@H](O)CO)[C@H](O)[C@@H](O)C=O)O[C@H](CO)[C@@H]1O)O)NC(=O)C)[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O RBMYDHMFFAVMMM-PLQWBNBWSA-N 0.000 claims description 6
- 241000193744 Bacillus amyloliquefaciens Species 0.000 claims description 5
- 101100052570 Bacillus subtilis (strain 168) ydeA gene Proteins 0.000 claims description 5
- 101100043029 Escherichia coli (strain K12) sotB gene Proteins 0.000 claims description 5
- 101100106940 Escherichia coli (strain UTI89 / UPEC) yubL gene Proteins 0.000 claims description 5
- 108060003306 Galactosyltransferase Proteins 0.000 claims description 5
- 102000030902 Galactosyltransferase Human genes 0.000 claims description 5
- 101100512780 Pseudomonas putida mdeA gene Proteins 0.000 claims description 5
- 108090000141 Sialyltransferases Proteins 0.000 claims description 5
- 102000003838 Sialyltransferases Human genes 0.000 claims description 5
- 230000009467 reduction Effects 0.000 claims description 5
- 241000588698 Erwinia Species 0.000 claims description 4
- 241000606766 Haemophilus parainfluenzae Species 0.000 claims description 4
- 241001465754 Metazoa Species 0.000 claims description 4
- 108010093077 N-Acetylglucosaminyltransferases Proteins 0.000 claims description 4
- 102000002493 N-Acetylglucosaminyltransferases Human genes 0.000 claims description 4
- 241000588650 Neisseria meningitidis Species 0.000 claims description 4
- 102000009569 Phosphoglucomutase Human genes 0.000 claims description 4
- 235000014897 Streptococcus lactis Nutrition 0.000 claims description 4
- 229930182830 galactose Natural products 0.000 claims description 4
- 230000002779 inactivation Effects 0.000 claims description 4
- 108091000115 phosphomannomutase Proteins 0.000 claims description 4
- 230000002829 reductive effect Effects 0.000 claims description 4
- 101100533919 Bacillus subtilis (strain 168) spoVB gene Proteins 0.000 claims description 3
- 101100212709 Bacillus subtilis (strain 168) yabM gene Proteins 0.000 claims description 3
- 241000196324 Embryophyta Species 0.000 claims description 3
- 101100256741 Escherichia coli (strain K12) setA gene Proteins 0.000 claims description 3
- 108010019236 Fucosyltransferases Proteins 0.000 claims description 3
- 102000006471 Fucosyltransferases Human genes 0.000 claims description 3
- 108010056664 N-acetyllactosaminide beta-1,6-N-acetylglucosaminyltransferase Proteins 0.000 claims description 3
- 241000589516 Pseudomonas Species 0.000 claims description 3
- 241000589774 Pseudomonas sp. Species 0.000 claims description 3
- QUOQJNYANJQSDA-MHQSSNGYSA-N Sialyllacto-N-tetraose a Chemical compound O1C([C@H](O)[C@H](O)CO)[C@H](NC(=O)C)[C@@H](O)C[C@@]1(C(O)=O)O[C@@H]1[C@@H](O)[C@H](OC2[C@H]([C@H](OC3[C@H]([C@H](O[C@H]([C@H](O)CO)[C@H](O)[C@@H](O)C=O)O[C@H](CO)[C@@H]3O)O)O[C@H](CO)[C@H]2O)NC(C)=O)O[C@H](CO)[C@@H]1O QUOQJNYANJQSDA-MHQSSNGYSA-N 0.000 claims description 3
- SFMRPVLZMVJKGZ-JRZQLMJNSA-N Sialyllacto-N-tetraose b Chemical compound O1[C@@H]([C@H](O)[C@H](O)CO)[C@H](NC(=O)C)[C@@H](O)C[C@@]1(C(O)=O)OC[C@@H]1[C@@H](O)[C@H](O[C@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O2)O)[C@@H](NC(C)=O)[C@H](O[C@@H]2[C@H]([C@H](O[C@H]([C@H](O)CO)[C@H](O)[C@@H](O)C=O)O[C@H](CO)[C@@H]2O)O)O1 SFMRPVLZMVJKGZ-JRZQLMJNSA-N 0.000 claims description 3
- 241000191967 Staphylococcus aureus Species 0.000 claims description 3
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 claims description 3
- 229930006000 Sucrose Natural products 0.000 claims description 3
- 102000003929 Transaminases Human genes 0.000 claims description 3
- 108090000340 Transaminases Proteins 0.000 claims description 3
- 108010061048 UDPacetylglucosamine pyrophosphorylase Proteins 0.000 claims description 3
- 101100315377 Yersinia pestis tsgA gene Proteins 0.000 claims description 3
- 241000029538 [Mannheimia] succiniciproducens Species 0.000 claims description 3
- FZIVHOUANIQOMU-YIHIYSSUSA-N alpha-L-Fucp-(1->2)-beta-D-Galp-(1->3)-beta-D-GlcpNAc-(1->3)-beta-D-Galp-(1->4)-D-Glcp Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@H]1O[C@H]1[C@H](O[C@@H]2[C@H]([C@H](O[C@@H]3[C@H]([C@H](O[C@@H]4[C@H](OC(O)[C@H](O)[C@H]4O)CO)O[C@H](CO)[C@@H]3O)O)O[C@H](CO)[C@H]2O)NC(C)=O)O[C@H](CO)[C@H](O)[C@@H]1O FZIVHOUANIQOMU-YIHIYSSUSA-N 0.000 claims description 3
- 108010005774 beta-Galactosidase Proteins 0.000 claims description 3
- 238000012258 culturing Methods 0.000 claims description 3
- 101150092956 fucP gene Proteins 0.000 claims description 3
- 108010084034 glucosamine-1-phosphate acetyltransferase Proteins 0.000 claims description 3
- 108010022717 glucosamine-6-phosphate isomerase Proteins 0.000 claims description 3
- 102000045442 glycosyltransferase activity proteins Human genes 0.000 claims description 3
- 108700014210 glycosyltransferase activity proteins Proteins 0.000 claims description 3
- FZIVHOUANIQOMU-UHFFFAOYSA-N lacto-N-fucopentaose I Natural products OC1C(O)C(O)C(C)OC1OC1C(OC2C(C(OC3C(C(OC4C(OC(O)C(O)C4O)CO)OC(CO)C3O)O)OC(CO)C2O)NC(C)=O)OC(CO)C(O)C1O FZIVHOUANIQOMU-UHFFFAOYSA-N 0.000 claims description 3
- 230000008569 process Effects 0.000 claims description 3
- 239000005720 sucrose Substances 0.000 claims description 3
- 101710098620 Alpha-1,2-fucosyltransferase Proteins 0.000 claims description 2
- 241000751139 Beauveria bassiana Species 0.000 claims description 2
- TXCIAUNLDRJGJZ-UHFFFAOYSA-N CMP-N-acetyl neuraminic acid Natural products O1C(C(O)C(O)CO)C(NC(=O)C)C(O)CC1(C(O)=O)OP(O)(=O)OCC1C(O)C(O)C(N2C(N=C(N)C=C2)=O)O1 TXCIAUNLDRJGJZ-UHFFFAOYSA-N 0.000 claims description 2
- TXCIAUNLDRJGJZ-BILDWYJOSA-N CMP-N-acetyl-beta-neuraminic acid Chemical compound O1[C@@H]([C@H](O)[C@H](O)CO)[C@H](NC(=O)C)[C@@H](O)C[C@]1(C(O)=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(N=C(N)C=C2)=O)O1 TXCIAUNLDRJGJZ-BILDWYJOSA-N 0.000 claims description 2
- GSXOAOHZAIYLCY-UHFFFAOYSA-N D-F6P Natural products OCC(=O)C(O)C(O)C(O)COP(O)(O)=O GSXOAOHZAIYLCY-UHFFFAOYSA-N 0.000 claims description 2
- 241001198387 Escherichia coli BL21(DE3) Species 0.000 claims description 2
- LQEBEXMHBLQMDB-UHFFFAOYSA-N GDP-L-fucose Natural products OC1C(O)C(O)C(C)OC1OP(O)(=O)OP(O)(=O)OCC1C(O)C(O)C(N2C3=C(C(N=C(N)N3)=O)N=C2)O1 LQEBEXMHBLQMDB-UHFFFAOYSA-N 0.000 claims description 2
- LQEBEXMHBLQMDB-JGQUBWHWSA-N GDP-beta-L-fucose Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C3=C(C(NC(N)=N3)=O)N=C2)O1 LQEBEXMHBLQMDB-JGQUBWHWSA-N 0.000 claims description 2
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 claims description 2
- TVVLIFCVJJSLBL-SEHWTJTBSA-N Lacto-N-fucopentaose V Chemical compound O[C@H]1C(O)C(O)[C@H](C)O[C@H]1OC([C@@H](O)C=O)[C@@H](C(O)CO)O[C@H]1[C@H](O)[C@@H](OC2[C@@H](C(OC3[C@@H](C(O)C(O)[C@@H](CO)O3)O)[C@H](O)[C@@H](CO)O2)NC(C)=O)[C@@H](O)[C@@H](CO)O1 TVVLIFCVJJSLBL-SEHWTJTBSA-N 0.000 claims description 2
- 102100021436 UDP-glucose 4-epimerase Human genes 0.000 claims description 2
- 108010075202 UDP-glucose 4-epimerase Proteins 0.000 claims description 2
- 108010082433 UDP-glucose-hexose-1-phosphate uridylyltransferase Proteins 0.000 claims description 2
- 230000009471 action Effects 0.000 claims description 2
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 claims description 2
- HXXFSFRBOHSIMQ-VFUOTHLCSA-N alpha-D-glucose 1-phosphate Chemical compound OC[C@H]1O[C@H](OP(O)(O)=O)[C@H](O)[C@@H](O)[C@@H]1O HXXFSFRBOHSIMQ-VFUOTHLCSA-N 0.000 claims description 2
- CMQZRJBJDCVIEY-JEOLMMCMSA-N alpha-L-Fucp-(1->3)-[beta-D-Galp-(1->4)]-beta-D-GlcpNAc-(1->3)-beta-D-Galp-(1->4)-D-Glcp Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@H]1O[C@H]1[C@H](O[C@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O2)O)[C@@H](CO)O[C@@H](O[C@@H]2[C@H]([C@H](O[C@@H]3[C@H](OC(O)[C@H](O)[C@H]3O)CO)O[C@H](CO)[C@@H]2O)O)[C@@H]1NC(C)=O CMQZRJBJDCVIEY-JEOLMMCMSA-N 0.000 claims description 2
- DUKURNFHYQXCJG-JEOLMMCMSA-N alpha-L-Fucp-(1->4)-[beta-D-Galp-(1->3)]-beta-D-GlcpNAc-(1->3)-beta-D-Galp-(1->4)-D-Glcp Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@H]1O[C@H]1[C@H](O[C@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O2)O)[C@@H](NC(C)=O)[C@H](O[C@@H]2[C@H]([C@H](O[C@@H]3[C@H](OC(O)[C@H](O)[C@H]3O)CO)O[C@H](CO)[C@@H]2O)O)O[C@@H]1CO DUKURNFHYQXCJG-JEOLMMCMSA-N 0.000 claims description 2
- 108010001671 galactoside 3-fucosyltransferase Proteins 0.000 claims description 2
- 229950010772 glucose-1-phosphate Drugs 0.000 claims description 2
- GSXOAOHZAIYLCY-HSUXUTPPSA-N keto-D-fructose 6-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)[C@H](O)COP(O)(O)=O GSXOAOHZAIYLCY-HSUXUTPPSA-N 0.000 claims description 2
- FKADDOYBRRMBPP-UHFFFAOYSA-N lacto-N-fucopentaose II Natural products OC1C(O)C(O)C(C)OC1OC1C(OC2C(C(O)C(O)C(CO)O2)O)C(NC(C)=O)C(OC2C(C(OC(C(O)CO)C(O)C(O)C=O)OC(CO)C2O)O)OC1CO FKADDOYBRRMBPP-UHFFFAOYSA-N 0.000 claims description 2
- CMQZRJBJDCVIEY-UHFFFAOYSA-N lacto-N-fucopentaose III Natural products OC1C(O)C(O)C(C)OC1OC1C(OC2C(C(O)C(O)C(CO)O2)O)C(CO)OC(OC2C(C(OC3C(OC(O)C(O)C3O)CO)OC(CO)C2O)O)C1NC(C)=O CMQZRJBJDCVIEY-UHFFFAOYSA-N 0.000 claims description 2
- 241000194035 Lactococcus lactis Species 0.000 claims 2
- HSCJRCZFDFQWRP-ABVWGUQPSA-N UDP-alpha-D-galactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-ABVWGUQPSA-N 0.000 claims 2
- 241000223679 Beauveria Species 0.000 claims 1
- 101100004297 Caenorhabditis elegans bet-1 gene Proteins 0.000 claims 1
- 102000014914 Carrier Proteins Human genes 0.000 claims 1
- 241000043250 Cedecea neteri Species 0.000 claims 1
- 241000186660 Lactobacillus Species 0.000 claims 1
- 102100023315 N-acetyllactosaminide beta-1,6-N-acetylglucosaminyl-transferase Human genes 0.000 claims 1
- 230000009056 active transport Effects 0.000 claims 1
- 229940039696 lactobacillus Drugs 0.000 claims 1
- 210000004027 cell Anatomy 0.000 description 110
- 235000018102 proteins Nutrition 0.000 description 61
- 108090000765 processed proteins & peptides Proteins 0.000 description 31
- 239000013612 plasmid Substances 0.000 description 29
- 102000004196 processed proteins & peptides Human genes 0.000 description 28
- 229920001184 polypeptide Polymers 0.000 description 27
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 25
- 239000008101 lactose Substances 0.000 description 25
- 230000015572 biosynthetic process Effects 0.000 description 21
- 238000004809 thin layer chromatography Methods 0.000 description 18
- 238000010367 cloning Methods 0.000 description 17
- 239000013598 vector Substances 0.000 description 17
- 108020004414 DNA Proteins 0.000 description 16
- IEQCXFNWPAHHQR-YKLSGRGUSA-N beta-D-Gal-(1->4)-beta-D-GlcNAc-(1->3)-beta-D-Gal-(1->4)-D-Glc Chemical compound O([C@H]1[C@H](O)[C@H]([C@@H](O[C@@H]1CO)O[C@@H]1[C@H]([C@H](O[C@@H]2[C@H](OC(O)[C@H](O)[C@H]2O)CO)O[C@H](CO)[C@@H]1O)O)NC(=O)C)[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O IEQCXFNWPAHHQR-YKLSGRGUSA-N 0.000 description 15
- 238000011534 incubation Methods 0.000 description 15
- 238000004458 analytical method Methods 0.000 description 14
- 101710161145 Sugar efflux transporter Proteins 0.000 description 13
- 239000012634 fragment Substances 0.000 description 13
- 239000002609 medium Substances 0.000 description 13
- 239000000047 product Substances 0.000 description 13
- 108091033319 polynucleotide Proteins 0.000 description 11
- 102000040430 polynucleotide Human genes 0.000 description 11
- 239000002157 polynucleotide Substances 0.000 description 10
- 239000006228 supernatant Substances 0.000 description 10
- 238000003786 synthesis reaction Methods 0.000 description 10
- 108010020764 Transposases Proteins 0.000 description 9
- 102000008579 Transposases Human genes 0.000 description 9
- 125000003275 alpha amino acid group Chemical group 0.000 description 9
- 239000000284 extract Substances 0.000 description 9
- 229910052500 inorganic mineral Inorganic materials 0.000 description 9
- 239000011707 mineral Substances 0.000 description 9
- 235000010755 mineral Nutrition 0.000 description 9
- 239000000758 substrate Substances 0.000 description 9
- 108020004705 Codon Proteins 0.000 description 8
- 238000007792 addition Methods 0.000 description 8
- 150000001875 compounds Chemical class 0.000 description 8
- 238000004128 high performance liquid chromatography Methods 0.000 description 8
- 150000002500 ions Chemical class 0.000 description 8
- 150000003839 salts Chemical class 0.000 description 8
- USAZACJQJDHAJH-KDEXOMDGSA-N [[(2r,3s,4r,5s)-5-(2,4-dioxo-1h-pyrimidin-6-yl)-3,4-dihydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2r,3r,4s,5r,6r)-3,4,5-trihydroxy-6-(hydroxymethyl)oxan-2-yl] hydrogen phosphate Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](C=2NC(=O)NC(=O)C=2)O1 USAZACJQJDHAJH-KDEXOMDGSA-N 0.000 description 7
- 239000012228 culture supernatant Substances 0.000 description 7
- 230000037430 deletion Effects 0.000 description 7
- 230000010354 integration Effects 0.000 description 7
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 6
- 102000053602 DNA Human genes 0.000 description 6
- 150000001720 carbohydrates Chemical class 0.000 description 6
- 235000014633 carbohydrates Nutrition 0.000 description 6
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 6
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 6
- 241000894006 Bacteria Species 0.000 description 5
- 238000004977 Hueckel calculation Methods 0.000 description 5
- OVRNDRQMDRJTHS-RTRLPJTCSA-N N-acetyl-D-glucosamine Chemical compound CC(=O)N[C@H]1C(O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-RTRLPJTCSA-N 0.000 description 5
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 5
- 229960000723 ampicillin Drugs 0.000 description 5
- 239000002773 nucleotide Substances 0.000 description 5
- 125000003729 nucleotide group Chemical group 0.000 description 5
- 238000013518 transcription Methods 0.000 description 5
- 230000035897 transcription Effects 0.000 description 5
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 5
- ZKHQWZAMYRWXGA-KQYNXXCUSA-J ATP(4-) Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)[C@H]1O ZKHQWZAMYRWXGA-KQYNXXCUSA-J 0.000 description 4
- ZKHQWZAMYRWXGA-UHFFFAOYSA-N Adenosine triphosphate Natural products C1=NC=2C(N)=NC=NC=2N1C1OC(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)C(O)C1O ZKHQWZAMYRWXGA-UHFFFAOYSA-N 0.000 description 4
- 241001646716 Escherichia coli K-12 Species 0.000 description 4
- 101100511168 Haemophilus influenzae (strain ATCC 51907 / DSM 11121 / KW20 / Rd) lex1 gene Proteins 0.000 description 4
- 150000001413 amino acids Chemical class 0.000 description 4
- 101150046612 gatD gene Proteins 0.000 description 4
- 230000001976 improved effect Effects 0.000 description 4
- 230000001939 inductive effect Effects 0.000 description 4
- 230000004060 metabolic process Effects 0.000 description 4
- 244000005700 microbiome Species 0.000 description 4
- 230000004048 modification Effects 0.000 description 4
- 238000012986 modification Methods 0.000 description 4
- 238000002703 mutagenesis Methods 0.000 description 4
- 231100000350 mutagenesis Toxicity 0.000 description 4
- 229930101283 tetracycline Natural products 0.000 description 4
- 238000012546 transfer Methods 0.000 description 4
- 230000003612 virological effect Effects 0.000 description 4
- 229940062827 2'-fucosyllactose Drugs 0.000 description 3
- HWHQUWQCBPAQQH-UHFFFAOYSA-N 2-O-alpha-L-Fucosyl-lactose Natural products OC1C(O)C(O)C(C)OC1OC1C(O)C(O)C(CO)OC1OC(C(O)CO)C(O)C(O)C=O HWHQUWQCBPAQQH-UHFFFAOYSA-N 0.000 description 3
- HWHQUWQCBPAQQH-BWRPKUOHSA-N 2-fucosyllactose Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@H]([C@H](O)CO)[C@H](O)[C@@H](O)C=O HWHQUWQCBPAQQH-BWRPKUOHSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 108010052285 Membrane Proteins Proteins 0.000 description 3
- 108091034117 Oligonucleotide Proteins 0.000 description 3
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 3
- SNFSYLYCDAVZGP-UHFFFAOYSA-N UNPD26986 Natural products OC1C(O)C(O)C(C)OC1OC1C(OC2C(OC(O)C(O)C2O)CO)OC(CO)C(O)C1O SNFSYLYCDAVZGP-UHFFFAOYSA-N 0.000 description 3
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 3
- KJCBCYNGQDISNK-UHFFFAOYSA-N acetic acid;butan-1-ol;propan-2-one Chemical compound CC(C)=O.CC(O)=O.CCCCO KJCBCYNGQDISNK-UHFFFAOYSA-N 0.000 description 3
- 230000003321 amplification Effects 0.000 description 3
- 239000011324 bead Substances 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 230000018109 developmental process Effects 0.000 description 3
- -1 emrO Proteins 0.000 description 3
- 230000004927 fusion Effects 0.000 description 3
- 101150002054 galE gene Proteins 0.000 description 3
- 238000012239 gene modification Methods 0.000 description 3
- 230000005017 genetic modification Effects 0.000 description 3
- 235000013617 genetically modified food Nutrition 0.000 description 3
- 239000011521 glass Substances 0.000 description 3
- 101150117187 glmS gene Proteins 0.000 description 3
- 230000003834 intracellular effect Effects 0.000 description 3
- 229930027917 kanamycin Natural products 0.000 description 3
- 229960000318 kanamycin Drugs 0.000 description 3
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 3
- 229930182823 kanamycin A Natural products 0.000 description 3
- 230000007246 mechanism Effects 0.000 description 3
- 239000012528 membrane Substances 0.000 description 3
- 150000002772 monosaccharides Chemical class 0.000 description 3
- 238000003199 nucleic acid amplification method Methods 0.000 description 3
- 239000012071 phase Substances 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 239000000741 silica gel Substances 0.000 description 3
- 229910002027 silica gel Inorganic materials 0.000 description 3
- 241000894007 species Species 0.000 description 3
- 229960005322 streptomycin Drugs 0.000 description 3
- 150000008163 sugars Chemical class 0.000 description 3
- 150000004044 tetrasaccharides Chemical class 0.000 description 3
- 230000009466 transformation Effects 0.000 description 3
- 230000005945 translocation Effects 0.000 description 3
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- AUNPEJDACLEKSC-ZAYDSPBTSA-N 3-fucosyllactose Chemical compound O[C@H]1[C@H](O)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](O)[C@H](O[C@@H]2[C@H](OC(O)[C@H](O)[C@H]2O)CO)O[C@H](CO)[C@@H]1O AUNPEJDACLEKSC-ZAYDSPBTSA-N 0.000 description 2
- DBTMGCOVALSLOR-UHFFFAOYSA-N 32-alpha-galactosyl-3-alpha-galactosyl-galactose Natural products OC1C(O)C(O)C(CO)OC1OC1C(O)C(OC2C(C(CO)OC(O)C2O)O)OC(CO)C1O DBTMGCOVALSLOR-UHFFFAOYSA-N 0.000 description 2
- WJPIUUDKRHCAEL-UHFFFAOYSA-N 3FL Natural products OC1C(O)C(O)C(C)OC1OC1C(OC2C(C(O)C(O)C(CO)O2)O)C(CO)OC(O)C1O WJPIUUDKRHCAEL-UHFFFAOYSA-N 0.000 description 2
- 101100061328 Acinetobacter baumannii (strain ATCC 19606 / DSM 30007 / JCM 6841 / CCUG 19606 / CIP 70.34 / NBRC 109757 / NCIMB 12457 / NCTC 12156 / 81) craA gene Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 101100310361 Bacillus subtilis (strain 168) skfE gene Proteins 0.000 description 2
- 101150049556 Bcr gene Proteins 0.000 description 2
- 108010006654 Bleomycin Proteins 0.000 description 2
- 241000186226 Corynebacterium glutamicum Species 0.000 description 2
- RXVWSYJTUUKTEA-UHFFFAOYSA-N D-maltotriose Natural products OC1C(O)C(OC(C(O)CO)C(O)C(O)C=O)OC(CO)C1OC1C(O)C(O)C(O)C(CO)O1 RXVWSYJTUUKTEA-UHFFFAOYSA-N 0.000 description 2
- SHZGCJCMOBCMKK-UHFFFAOYSA-N D-mannomethylose Natural products CC1OC(O)C(O)C(O)C1O SHZGCJCMOBCMKK-UHFFFAOYSA-N 0.000 description 2
- 101100279821 Escherichia coli (strain K12) entS gene Proteins 0.000 description 2
- 101100401434 Escherichia coli (strain K12) mhpT gene Proteins 0.000 description 2
- 101100149325 Escherichia coli (strain K12) setC gene Proteins 0.000 description 2
- 101100544652 Escherichia coli (strain K12) ynfM gene Proteins 0.000 description 2
- 101100545036 Escherichia coli (strain K12) yuaE gene Proteins 0.000 description 2
- 241000606790 Haemophilus Species 0.000 description 2
- 241000590002 Helicobacter pylori Species 0.000 description 2
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical compound C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 2
- PNNNRSAQSRJVSB-UHFFFAOYSA-N L-rhamnose Natural products CC(O)C(O)C(O)C(O)C=O PNNNRSAQSRJVSB-UHFFFAOYSA-N 0.000 description 2
- 102000003939 Membrane transport proteins Human genes 0.000 description 2
- 108090000301 Membrane transport proteins Proteins 0.000 description 2
- 229910019142 PO4 Inorganic materials 0.000 description 2
- 241000235070 Saccharomyces Species 0.000 description 2
- 241001138501 Salmonella enterica Species 0.000 description 2
- 244000057717 Streptococcus lactis Species 0.000 description 2
- 102000003673 Symporters Human genes 0.000 description 2
- 108090000088 Symporters Proteins 0.000 description 2
- 239000004098 Tetracycline Substances 0.000 description 2
- 108091023040 Transcription factor Proteins 0.000 description 2
- 102000040945 Transcription factor Human genes 0.000 description 2
- DRTQHJPVMGBUCF-XVFCMESISA-N Uridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-XVFCMESISA-N 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- 241000607734 Yersinia <bacteria> Species 0.000 description 2
- HXXFSFRBOHSIMQ-FPRJBGLDSA-N alpha-D-galactose 1-phosphate Chemical compound OC[C@H]1O[C@H](OP(O)(O)=O)[C@H](O)[C@@H](O)[C@H]1O HXXFSFRBOHSIMQ-FPRJBGLDSA-N 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 230000003115 biocidal effect Effects 0.000 description 2
- 229960001561 bleomycin Drugs 0.000 description 2
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 229960005091 chloramphenicol Drugs 0.000 description 2
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 2
- 230000002759 chromosomal effect Effects 0.000 description 2
- 101150113191 cmr gene Proteins 0.000 description 2
- 230000002860 competitive effect Effects 0.000 description 2
- 238000001514 detection method Methods 0.000 description 2
- 238000010586 diagram Methods 0.000 description 2
- 238000013467 fragmentation Methods 0.000 description 2
- 238000006062 fragmentation reaction Methods 0.000 description 2
- 150000008195 galaktosides Chemical class 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 229940037467 helicobacter pylori Drugs 0.000 description 2
- 101150018810 lgtB gene Proteins 0.000 description 2
- 238000004895 liquid chromatography mass spectrometry Methods 0.000 description 2
- FYGDTMLNYKFZSV-UHFFFAOYSA-N mannotriose Natural products OC1C(O)C(O)C(CO)OC1OC1C(CO)OC(OC2C(OC(O)C(O)C2O)CO)C(O)C1O FYGDTMLNYKFZSV-UHFFFAOYSA-N 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 101150008808 mdfA gene Proteins 0.000 description 2
- 101150067217 mdtD gene Proteins 0.000 description 2
- 108020004999 messenger RNA Proteins 0.000 description 2
- 238000012269 metabolic engineering Methods 0.000 description 2
- 150000002972 pentoses Chemical class 0.000 description 2
- 239000010452 phosphate Substances 0.000 description 2
- 229920001282 polysaccharide Polymers 0.000 description 2
- 239000013641 positive control Substances 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 101150011264 setB gene Proteins 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 238000006467 substitution reaction Methods 0.000 description 2
- 238000010189 synthetic method Methods 0.000 description 2
- 229960002180 tetracycline Drugs 0.000 description 2
- OFVLGDICTFRJMM-WESIUVDSSA-N tetracycline Chemical compound C1=CC=C2[C@](O)(C)[C@H]3C[C@H]4[C@H](N(C)C)C(O)=C(C(N)=O)C(=O)[C@@]4(O)C(O)=C3C(=O)C2=C1O OFVLGDICTFRJMM-WESIUVDSSA-N 0.000 description 2
- 235000019364 tetracycline Nutrition 0.000 description 2
- 150000003522 tetracyclines Chemical class 0.000 description 2
- 230000017105 transposition Effects 0.000 description 2
- 241001515965 unidentified phage Species 0.000 description 2
- 101150060554 ydeE gene Proteins 0.000 description 2
- 101150021406 ydhC gene Proteins 0.000 description 2
- 101150102516 yfcJ gene Proteins 0.000 description 2
- 101150103854 yhhS gene Proteins 0.000 description 2
- FYGDTMLNYKFZSV-BYLHFPJWSA-N β-1,4-galactotrioside Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@H](CO)O[C@@H](O[C@@H]2[C@@H](O[C@@H](O)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O FYGDTMLNYKFZSV-BYLHFPJWSA-N 0.000 description 2
- CILYIEBUXJIHCO-UHFFFAOYSA-N 102778-91-6 Natural products O1C(C(O)C(O)CO)C(NC(=O)C)C(O)CC1(C(O)=O)OC1C(O)C(OC2C(C(O)C(O)OC2CO)O)OC(CO)C1O CILYIEBUXJIHCO-UHFFFAOYSA-N 0.000 description 1
- KZDCMKVLEYCGQX-UDPGNSCCSA-N 2-(diethylamino)ethyl 4-aminobenzoate;(2s,5r,6r)-3,3-dimethyl-7-oxo-6-[(2-phenylacetyl)amino]-4-thia-1-azabicyclo[3.2.0]heptane-2-carboxylic acid;hydrate Chemical compound O.CCN(CC)CCOC(=O)C1=CC=C(N)C=C1.N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 KZDCMKVLEYCGQX-UDPGNSCCSA-N 0.000 description 1
- DVGKRPYUFRZAQW-UHFFFAOYSA-N 3 prime Natural products CC(=O)NC1OC(CC(O)C1C(O)C(O)CO)(OC2C(O)C(CO)OC(OC3C(O)C(O)C(O)OC3CO)C2O)C(=O)O DVGKRPYUFRZAQW-UHFFFAOYSA-N 0.000 description 1
- 108700016155 Acyl transferases Proteins 0.000 description 1
- 102000057234 Acyl transferases Human genes 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 102100031317 Alpha-N-acetylgalactosaminidase Human genes 0.000 description 1
- KLSJWNVTNUYHDU-UHFFFAOYSA-N Amitrole Chemical compound NC1=NC=NN1 KLSJWNVTNUYHDU-UHFFFAOYSA-N 0.000 description 1
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 1
- 241000203069 Archaea Species 0.000 description 1
- 241000423333 Bacteroides fragilis NCTC 9343 Species 0.000 description 1
- 241000772835 Beauveria bassiana D1-5 Species 0.000 description 1
- 235000002566 Capsicum Nutrition 0.000 description 1
- BVKZGUZCCUSVTD-UHFFFAOYSA-L Carbonate Chemical compound [O-]C([O-])=O BVKZGUZCCUSVTD-UHFFFAOYSA-L 0.000 description 1
- 108020004638 Circular DNA Proteins 0.000 description 1
- 241000182144 Coptotermes niger Species 0.000 description 1
- 108010084372 D-arabinose isomerase Proteins 0.000 description 1
- 241000588694 Erwinia amylovora Species 0.000 description 1
- 101100011534 Escherichia coli (strain K12) emrD gene Proteins 0.000 description 1
- 101100375798 Escherichia coli (strain K12) ybhJ gene Proteins 0.000 description 1
- 108091029865 Exogenous DNA Proteins 0.000 description 1
- 108010046276 FLP recombinase Proteins 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 102100041034 Glucosamine-6-phosphate isomerase 1 Human genes 0.000 description 1
- 241000606768 Haemophilus influenzae Species 0.000 description 1
- 101150040736 K12 gene Proteins 0.000 description 1
- 241001454354 Kingella Species 0.000 description 1
- 241001147746 Lactobacillus delbrueckii subsp. lactis Species 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 108050004064 Major facilitator superfamily Proteins 0.000 description 1
- 102000015841 Major facilitator superfamily Human genes 0.000 description 1
- 241000863420 Myxococcus Species 0.000 description 1
- CILYIEBUXJIHCO-UITFWXMXSA-N N-acetyl-alpha-neuraminyl-(2->3)-beta-D-galactosyl-(1->4)-beta-D-glucose Chemical compound O1[C@@H]([C@H](O)[C@H](O)CO)[C@H](NC(=O)C)[C@@H](O)C[C@@]1(C(O)=O)O[C@@H]1[C@@H](O)[C@H](O[C@H]2[C@@H]([C@@H](O)[C@H](O)O[C@@H]2CO)O)O[C@H](CO)[C@@H]1O CILYIEBUXJIHCO-UITFWXMXSA-N 0.000 description 1
- 108010069483 N-acetylglucosamine-6-phosphate deacetylase Proteins 0.000 description 1
- OIZGSVFYNBZVIK-UHFFFAOYSA-N N-acetylneuraminosyl-D-lactose Natural products O1C(C(O)C(O)CO)C(NC(=O)C)C(O)CC1(C(O)=O)OC1C(O)C(OC(C(O)CO)C(O)C(O)C=O)OC(CO)C1O OIZGSVFYNBZVIK-UHFFFAOYSA-N 0.000 description 1
- 241000588653 Neisseria Species 0.000 description 1
- 241000529648 Neisseria meningitidis MC58 Species 0.000 description 1
- HDCBDWMGGHHXEI-GGCCYKIWSA-N O=C[C@@H](O)[C@H](O)[C@H](O)[C@@H](O)C.OCC(=O)[C@H](O)[C@H](O)[C@@H](O)C Chemical compound O=C[C@@H](O)[C@H](O)[C@H](O)[C@@H](O)C.OCC(=O)[C@H](O)[C@H](O)[C@@H](O)C HDCBDWMGGHHXEI-GGCCYKIWSA-N 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 239000006002 Pepper Substances 0.000 description 1
- 235000016761 Piper aduncum Nutrition 0.000 description 1
- 235000017804 Piper guineense Nutrition 0.000 description 1
- 244000203593 Piper nigrum Species 0.000 description 1
- 235000008184 Piper nigrum Nutrition 0.000 description 1
- 102000001253 Protein Kinase Human genes 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 102000018120 Recombinases Human genes 0.000 description 1
- 108010091086 Recombinases Proteins 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 241001374020 Staphylococcus aureus Bmb9393 Species 0.000 description 1
- 108050005268 Sugar efflux Proteins 0.000 description 1
- 108700005078 Synthetic Genes Proteins 0.000 description 1
- HSCJRCZFDFQWRP-JZMIEXBBSA-N UDP-alpha-D-glucose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-JZMIEXBBSA-N 0.000 description 1
- OIRDTQYFTABQOQ-UHTZMRCNSA-N Vidarabine Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@@H]1O OIRDTQYFTABQOQ-UHTZMRCNSA-N 0.000 description 1
- 108020005202 Viral DNA Proteins 0.000 description 1
- 108010084455 Zeocin Proteins 0.000 description 1
- 239000000370 acceptor Substances 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 239000012491 analyte Substances 0.000 description 1
- 230000000844 anti-bacterial effect Effects 0.000 description 1
- OIRDTQYFTABQOQ-UHFFFAOYSA-N ara-adenosine Natural products Nc1ncnc2n(cnc12)C1OC(CO)C(O)C1O OIRDTQYFTABQOQ-UHFFFAOYSA-N 0.000 description 1
- 101150035354 araA gene Proteins 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- DRTQHJPVMGBUCF-PSQAKQOGSA-N beta-L-uridine Natural products O[C@H]1[C@@H](O)[C@H](CO)O[C@@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-PSQAKQOGSA-N 0.000 description 1
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 210000002421 cell wall Anatomy 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 150000001793 charged compounds Chemical class 0.000 description 1
- 238000001311 chemical methods and process Methods 0.000 description 1
- 238000007385 chemical modification Methods 0.000 description 1
- 230000008131 children development Effects 0.000 description 1
- RPKLZQLYODPWTM-KBMWBBLPSA-N cholanoic acid Chemical compound C1CC2CCCC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@@H](CCC(O)=O)C)[C@@]1(C)CC2 RPKLZQLYODPWTM-KBMWBBLPSA-N 0.000 description 1
- 238000013375 chromatographic separation Methods 0.000 description 1
- 239000013611 chromosomal DNA Substances 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 238000007796 conventional method Methods 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000037213 diet Effects 0.000 description 1
- 239000003480 eluent Substances 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 235000019197 fats Nutrition 0.000 description 1
- 101150054547 galM gene Proteins 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 230000002496 gastric effect Effects 0.000 description 1
- 238000012215 gene cloning Methods 0.000 description 1
- 108091008053 gene clusters Proteins 0.000 description 1
- 101150073660 glmM gene Proteins 0.000 description 1
- 101150111330 glmU gene Proteins 0.000 description 1
- 150000004676 glycans Chemical class 0.000 description 1
- 239000000937 glycosyl acceptor Substances 0.000 description 1
- 125000003147 glycosyl group Chemical group 0.000 description 1
- 229940047650 haemophilus influenzae Drugs 0.000 description 1
- 238000003306 harvesting Methods 0.000 description 1
- 150000002402 hexoses Chemical class 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 230000000415 inactivating effect Effects 0.000 description 1
- 230000006698 induction Effects 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000010039 intracellular degradation Effects 0.000 description 1
- 238000005342 ion exchange Methods 0.000 description 1
- 238000010829 isocratic elution Methods 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 101150066555 lacZ gene Proteins 0.000 description 1
- 238000011031 large-scale manufacturing process Methods 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 230000033001 locomotion Effects 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 101150106875 malE gene Proteins 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 239000000203 mixture Substances 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 230000036457 multidrug resistance Effects 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 101150070589 nagB gene Proteins 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 238000002414 normal-phase solid-phase extraction Methods 0.000 description 1
- 235000020939 nutritional additive Nutrition 0.000 description 1
- 230000003204 osmotic effect Effects 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 210000001322 periplasm Anatomy 0.000 description 1
- CWCMIVBLVUHDHK-ZSNHEYEWSA-N phleomycin D1 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC[C@@H](N=1)C=1SC=C(N=1)C(=O)NCCCCNC(N)=N)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C CWCMIVBLVUHDHK-ZSNHEYEWSA-N 0.000 description 1
- 229920001339 phlorotannin Polymers 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 108010032867 phosphoglucosamine mutase Proteins 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 230000008092 positive effect Effects 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 235000013406 prebiotics Nutrition 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 230000001902 propagating effect Effects 0.000 description 1
- 108060006633 protein kinase Proteins 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 230000008439 repair process Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 230000009469 supplementation Effects 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 231100000167 toxic agent Toxicity 0.000 description 1
- 239000003440 toxic substance Substances 0.000 description 1
- 239000011573 trace mineral Substances 0.000 description 1
- 235000013619 trace mineral Nutrition 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 238000001890 transfection Methods 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- DRTQHJPVMGBUCF-UHFFFAOYSA-N uracil arabinoside Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-UHFFFAOYSA-N 0.000 description 1
- 229940045145 uridine Drugs 0.000 description 1
- 239000011782 vitamin Substances 0.000 description 1
- 235000013343 vitamin Nutrition 0.000 description 1
- 229940088594 vitamin Drugs 0.000 description 1
- 229930003231 vitamin Natural products 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/18—Preparation of compounds containing saccharide radicals produced by the action of a glycosyl transferase, e.g. alpha-, beta- or gamma-cyclodextrins
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
- C07K14/24—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria from Enterobacteriaceae (F), e.g. Citrobacter, Serratia, Proteus, Providencia, Morganella, Yersinia
- C07K14/245—Escherichia (G)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/67—General methods for enhancing the expression
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1048—Glycosyltransferases (2.4)
- C12N9/1051—Hexosyltransferases (2.4.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/04—Polysaccharides, i.e. compounds containing more than five saccharide radicals attached to each other by glycosidic bonds
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y204/00—Glycosyltransferases (2.4)
- C12Y204/01—Hexosyltransferases (2.4.1)
- C12Y204/01146—Beta-1,3-galactosyl-O-glycosyl-glycoprotein beta-1,3-N-acetylglucosaminyltransferase (2.4.1.146)
Landscapes
- Chemical & Material Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- Molecular Biology (AREA)
- Biophysics (AREA)
- Medicinal Chemistry (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Gastroenterology & Hepatology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Communicable Diseases (AREA)
Abstract
本发明涉及用于在经遗传修饰的细菌宿主细胞中产生寡糖的方法,以及涉及所述方法中使用的经遗传修饰的宿主细胞。所述经遗传修饰的宿主细胞包含至少一种重组的糖基转移酶和至少一种编码蛋白的核酸序列,所述蛋白使得能够输出寡糖。
Description
背景技术
人乳被认为是婴儿发育的最佳饮食。它由脂肪、蛋白质、维生素、矿物质、微量元素和复合碳水化合物混合物组成,所述复合碳水化合物混合物包括乳糖和约150种结构不同的寡糖(人乳寡糖,HMO)。
通过化学方法或通过生物技术方法产生HMO的尝试主要引起了普遍的关注,这是由于它们对婴儿的胃肠道菌群发育的有益影响,因此提倡将它们用作营养添加剂。除了这些益生元特性,迄今为止可观察到HMO的许多其他积极的作用,这扩展了它们的应用领域。
然而,大量的科学研究需要纯的单一化合物,这几乎不可实现。对于复合的游离中性和酸性寡糖而言尤其如此,对于所述寡糖而言仍缺少有竞争力的大规模生产方法(例如,乳-N-四糖(Gal(β1-3)GlcNAc(β1-3)Gal(β1-4)Gluc)、乳-N-新四糖(Gal(β1-4)GlcNAc(β1-3)Gal(β1-4)Gluc)、乳-N-岩藻戊糖I(Fuc(α1-2)Gal(β1-3)GlcNAc(β1-3)Gal(β1-4)Gluc)、乳-N-新岩藻戊糖I(Fuc(α1-2)Gal(β1-4)GlcNAc(β1-3)Gal(β1-4)Gluc)(乳-N-唾液酰戊糖a(LST-a;Neu5Ac(α2-3)Gal(β1-3)GlcNAc(β1-3)Gal(β1-4)Gluc))。用于产生这些化合物的微生物的代谢改造代表了最有希望的方法,因为化学方法不能产生数吨规模的这些分子。
对于结构上更简单的HMO(例如2’-岩藻基乳糖、3-岩藻基乳糖或3’-唾液酰乳糖),已经开发了数种发酵方法,这些方法主要使用经代谢改造的大肠杆菌(Escherichia coli)菌株。
然而,大规模的量仅可通过以下方式来实现:增加寡糖向细菌细胞外的输出,因此(i)增加产率和(ii)允许从培养液中回收所需寡糖。解决输出问题的需求似乎随着所产生的糖的大小而增大。此外,使用目前可获得的发酵方法,在产生更复杂的寡糖时,发生不想要的寡糖前体从生产细胞的输出的问题,导致在发酵培养基中产物和前体寡糖的不想要的混合。尽管已知多种转运蛋白跨膜转移单糖或二糖,但是几乎不存在任何关于更大的寡糖(例如三糖和更大的寡糖)的转运的知识。
例如,通常使用的发酵模式生物大肠杆菌的基因组编码多于500种不同的转运蛋白(Busch and Saier,Crit Rev Biochem Mol Biol.2002;37(5):287-337)。这些膜转运蛋白的分类是非常不同的,并且亚组在易位(translocation)机制、蛋白结构或进化起源方面可不同。
典型的能量驱动的主动转运蛋白逆着底物浓度或电化学梯度进行底物移动,而底物流经通道的动力学和方向主要遵循这样的梯度。根据用于易位的能量来源,泵主要可分为初级主动转运蛋白和次级主动转运蛋白,其分别利用代谢能量如ATP或电化学电势(Davidson and Maloney,Trends Microbiol.2007Oct;15(10):448-55;Forrest et al,Biochim Biophys Acta.201 1Feb;1807(2):167-88)。尽管对于数种允许能量产生、碳水化合物输入以及蛋白质和抗菌物质流出的膜蛋白获得了深入的知识,然而迄今为止仅获得了对少数所注释的细菌转运蛋白的机理过程或天然或可能底物的信息的敏锐的深刻了解。
大肠杆菌乳糖通透酶LacY可能代表最充分表征的溶质转运蛋白(Guan andKaback,Annu Rev Biophys Biomol Struct.2006;35:67-91),并且是大的且特别多样的主要易化子超家族(major facilitator superfamily,MFS)的成员,所述超家族属于次级主动转运蛋白类别,其通过单向转运、同向转运或反向转运来转运糖类、药物、疏水分子、肽、有机离子等(Saier et al.,J Mol Microbiol Biotechnol.1999Nov;1(2):257-79)。除少数例外,MFS转运蛋白的共有结构特征是两个横跨胞质膜的六螺旋亚结构域。在相关H+-偶联的MFS同向转运蛋白之间功能上同源的氨基酸位置的存在进一步表明类似的动力学机制,如针对乳糖通透酶所测定的(Madej and Kaback,Proc Natl Acad Sci U S A.2013Dec10;1 10(50):E4831-8)。
几十年来,可获得大量关于将碳水化合物输入到细菌中的知识。但是关于碳水化合物的输出,特别是关于非表面相关的分子,仅可获得很少的信息。这不是出乎预料的,因为糖类实际上是有利的碳源和能源,因此一旦在细胞中,它们不应当被释放到竞争性环境中。
然而,糖转运蛋白的天然功能可能涉及渗透压或糖磷酸盐胁迫的降低,这可能意味着灵活的底物范围。有趣的是,对于所谓的糖流出转运蛋白家族(SET)(属于MFS转运蛋白组)的成员,示出了多种半乳糖苷类如IPTG、TMG和乳糖的输出(Liu et al.,J BiolChem.1999Aug 13;274(33):22977-84;Liu et al.,Mol Microbiol.1999Mar;31(6):1845-51)。
大肠杆菌转运蛋白SetA甚至被描述为转运人乳寡糖3-岩藻基乳糖,引起在过表达setA的重组大肠杆菌菌株的发酵过程中所述化合物的产生增加(参见申请人的国际专利申请WO 2010/142305)。类似地,耶尔森氏菌属(Yersinia)的糖流出转运蛋白的表达证明能够将人乳寡糖2’-岩藻基乳糖输出至经改造的大肠杆菌生产菌株之外。
除此之外,从机理和能量角度来看,只有离子梯度驱动的转运系统具有同时在两个方向上跨膜转移溶质的潜力。这对于上述LacY(一种半乳糖苷/H+同向转运蛋白)而言是尤其(exemplarily)正确的,所述转运蛋白是允许在大肠杆菌中进行乳糖代谢的细菌乳糖操纵子的一部分。该通透酶主要将乳糖输送到细胞中,但是其也能够以相反方向转移其底物。
除了代表最大的转运蛋白组的主要易化子超家族,细菌还具有其他排出溶质的机制——通常总结于多药流出泵类别中。类似于MFS,小的多药抗性超家族(SMR)、多药和毒性化合物排出超家族(MATE)和抗性-结瘤-细胞分裂超家族(RND)的活性依赖于电化学梯度。第五类是腺苷三磷酸(ATP)-结合盒超家族(ABC),其使用ATP作为能源以从细胞驱动分子。如同MFS,SMR、MATE、RND和ABC的成员转运在结构上不同的分子。另外,迄今为止所鉴定的它们的底物中的大多数不是天然存在的,因此它们的偏好难以预测。
尽管人乳寡糖的化学合成方法是已知的,这些方法的成本非常高并且不会产生令人满意的量。另一方面,使用经遗传修饰的微生物的发酵方法仍然具有以下缺陷:更大寡糖(四糖、戊糖、己糖)的输出代表了建立成本有效的生产方法的主要限制。因此,仍需要改进的用于产生大规模的人寡糖的方法。
发明内容
根据本发明,该目的和其他目的是通过所附权利要求中要求保护的方法和微生物宿主细胞来解决的。
使用本发明的方法和宿主细胞,可产生所需寡糖,优选在未经修饰的宿主细胞中不产生的寡糖,还优选属于人乳寡糖的寡糖,所述寡糖可大量从培养基中获得。因此,可以说所述寡糖可以游离形式从培养基中获得;它不结合于表面蛋白或膜蛋白或宿主细胞表面的其他蛋白。
根据本发明,提供了通过经遗传修饰的微生物宿主细胞产生所需寡糖的方法,所述方法包括以下步骤:a)提供经遗传修饰的微生物宿主细胞,其包含至少一种重组糖基转移酶,并且具有至少一种内源性糖输出蛋白的表达或活性,所述蛋白被修饰,从而使得与未经遗传修饰的宿主细胞相比,所述糖输出蛋白的表达或活性(i)增加或(ii)减少或失活,从而与未经遗传修饰的宿主细胞相比,(i)寡糖至培养基的输出减少或消除,或(ii)所需寡糖的转运增加;b)在允许产生所需寡糖的条件下在培养基中培养宿主细胞,由此将所需寡糖转运到培养基中。所述方法可还包括步骤c)从培养基中获得所需寡糖。
在本发明的方法中,优选地,所需寡糖是人乳寡糖,其包括乳-N-三糖II(LNT-II;GlcNAc(β-3)Gal(β-4)Gluc)作为核心三糖。在这方面,具有“核心三糖”的寡糖意指包含代表所需寡糖的还原端的特定的三糖,以及包含(视情况而定)额外的糖部分,所述特定的三糖代表主要部分。
因此,在本发明的方法和宿主细胞的一个实施方案中,所需寡糖选自乳-N-三糖II、乳-N-四糖、乳-N-新四糖、乳-N-岩藻戊糖I、乳-N-岩藻戊糖II、乳-N-岩藻戊糖III、乳-N-岩藻戊糖V、乳-N-二岩藻基己糖I、乳-N-二岩藻基己糖II、乳-N-唾液酰戊糖LSTa、LSTb、LSTc、二唾液酰乳-N-四糖、二唾液酰乳-N-新四糖。
为了克服上述的寡糖输出受限的缺陷,进一步通过本发明的方法来解决所述目标,其中所述宿主细胞包括:至少一种同源或异源核酸序列,所述序列编码使得能够将所需寡糖输出到培养基中的蛋白,其中所述宿主细胞已经修饰,从而使得所述同源或异源核酸序列的表达为过表达的或在使得能够过表达所述核酸序列的启动子的控制下;和/或至少一种编码输出蛋白的内源性核酸序列的缺失、破坏、减少或失活,所述输出蛋白将所需寡糖的前体输出到所述宿主细胞外;和/或至少一种编码蛋白的同源或异源核酸序列,所述蛋白介导所需寡糖的前体输入到所述宿主细胞中,其中优选地所述核酸序列为过表达的,并且其中优选地所述前体大于二糖。
将包含本文所述特征的经遗传修饰的微生物宿主细胞在存在葡萄糖、蔗糖、甘油或其组合的情况下(使用这些底物作为碳源和能源)以及在存在乳糖或大于二糖的寡糖(例如LNT-II)的情况下培养。
在该方法和宿主细胞的一个优选实施方案中,所述使得能够输出所需寡糖的蛋白属于次级主动转运蛋白类别,并且更优选地进行包含至少三个部分的寡糖的输出。
根据优选的实施方案,为了输出所需寡糖,除负责胞内寡糖生物合成的基因以外,还表达适合的输出蛋白。
根据本发明的方法和宿主细胞的一方面,编码使得能够输出所需寡糖的蛋白的至少一种核酸序列是内源性核酸或重组核酸。
在本发明的方法和宿主细胞的优选实施方案中,编码使得能够输出所需寡糖的蛋白的核酸序列是细菌、古细菌、酵母或动物来源;优选地,至少一种编码使得能够输出所需寡糖的蛋白的核酸序列是选自以下的基因:来自大肠杆菌的yebQ和yjhB,来自产琥珀酸曼氏杆菌(Mannheimia succiniciproducens)的proP,以及来自奈氏西地西菌(Cedeceaneteri)的setA,或它们的功能性片段。
优选地,所述寡糖输出蛋白是选自以下中的至少一种的蛋白:大肠杆菌的SetA、SetB、SetC、YdeA、Cmr、YnfM、MdtD、YfcJ、YhhS、EmrO、YdhC、YbdA、YdeE、MhpT、YebQ、YjhB、Bcr和YdeA,来自产琥珀酸曼氏杆菌的proP以及来自奈氏西地西菌的setA,或它们的变体或同源物。
在另一个优选的实施方案中,所述重组的糖基转移酶选自以下中的至少一种:半乳糖基转移酶、唾液酰转移酶、N-乙酰葡糖胺转移酶和岩藻糖基转移酶,并优选地选自以下中的至少一种:β-1,3-N-乙酰葡糖胺转移酶、β-1,3-半乳糖基转移酶、β-1,4-半乳糖基转移酶、β-1,6-半乳糖基转移酶、α-2,3-唾液酰转移酶、α-2,6-唾液酰转移酶、α-1,2-岩藻糖基转移酶或α-1,3-岩藻糖基转移酶。
本发明的方法和宿主细胞的优选实施方案涉及宿主细胞或其供应,其中所述宿主细胞包含:(i)β-1,3-N-乙酰葡糖胺转移酶和(ii)β-1,3-半乳糖基转移酶或β-1,4-半乳糖基转移酶来作为糖基转移酶。在这方面,优选地,所述β-1,3-N-乙酰葡糖胺转移酶具有将N-乙酰葡糖胺连接至乳糖产生乳-N-三糖II的活性,以及所述β-1,3-半乳糖基转移酶或所述β-1,4-半乳糖基转移酶分别具有半乳糖基化乳-N-三糖II由此分别产生乳-N-四糖或乳-N-新四糖的活性。通过表达其他糖基转移酶,此处开发的系统易于被改造为适应于甚至更复杂的寡糖。
使用本发明的微生物细胞和方法,可大量发酵所需寡糖(特别是包含LNT-II作为核心结构的寡糖),以及从培养液中回收寡糖。
在一个优选的实施方案中,所述β-1,3-N-乙酰葡糖胺转移酶属于脑膜炎奈瑟氏菌(Neisseria meningitides)的IgtA或多杀性巴氏杆菌(Pasteurella multocida)的PmnagT或它们的变体的类别。
优选地,糖基转移酶选自半乳糖基转移酶、唾液酰转移酶、N-乙酰葡糖胺转移酶和岩藻糖基转移酶。
在另一个优选的实施方案中,产生乳-N-四糖的β-1,3-半乳糖基转移酶是WbdO或其功能变体。根据本发明的一方面,所述β-1,3-半乳糖基转移酶是源自肠道沙门氏菌(Salmonella enterica)的β-1,3-半乳糖基转移酶(wbdO,登录号AY730594),并且优选地由选自以下的基因编码:来自大肠杆菌055:H7的wbgO或来自Lutiella nitroferrum的furA或它们的功能性片段。
本发明还涉及经遗传修饰的微生物宿主细胞,优选如上文所述的微生物宿主细胞,其中内源性β-半乳糖苷酶基因失活或缺失并且其中存在功能性乳糖通透酶基因。
因此,在本发明的方法和宿主细胞的优选实施方案中,提供了经遗传修饰的宿主细胞,其中在适用的情况下,内源性β-半乳糖苷酶基因和葡糖胺-6-磷酸脱氨酶基因失活或缺失,并且其中所述经遗传修饰的宿主细胞包含编码功能性乳糖通透酶蛋白(优选LacY)的核酸序列。
在一个优选的实施方案中,与未经遗传修饰的宿主细胞相比,经遗传修饰的宿主细胞包含提高的UDP-N-乙酰葡糖胺和UDP-半乳糖、GDP-岩藻糖或CMP-N-乙酰神经氨酸的产生能力。
在本发明的方法和宿主细胞的该实施方案的改进方案中,所述提高的UDP-N-乙酰葡糖胺和UDP-半乳糖产生能力包括编码蛋白的一种或多种基因的过表达,所述蛋白包含以下酶的活性:L-谷氨酰胺:D-果糖-6-磷酸氨基转移酶、N-乙酰葡糖胺-1-磷酸尿苷酰转移酶/葡糖胺-1-磷酸乙酰转移酶、磷酸葡糖胺变位酶、UDP-半乳糖-4-差向异构酶、葡糖磷酸变位酶、葡萄糖-1-磷酸尿苷酰基转移酶。
对于例如LNT的合成而言,需要UDP-半乳糖和UDP-N-乙酰葡糖胺。UDP半乳糖可通过经由发酵培养基将半乳糖加料到产HMO的细菌宿主细胞而获得。半乳糖随后被细胞吸收,磷酸化为半乳糖-1-磷酸,之后被转化为UDP-半乳糖。编码这些酶活性的基因是文献中公知的(Grossiord et al.,J.Bacteriol 2003 185(3)870-878)。UDP-半乳糖的供应还可从细胞自身代谢中获得,并且可通过进一步的遗传修饰来改进代谢,例如UDP-半乳糖-4’-差向异构酶的过表达或UDP-半乳糖-4’-差向异构酶与葡萄糖-1-磷酸-1-尿苷酰转移酶的过表达。UDP-N-乙酰葡糖胺还可从细菌宿主细胞自身的UDP-N-乙酰葡糖胺代谢中获得。用于合成包含N-乙酰葡糖胺的寡糖的UDP-N-乙酰葡糖胺的供应可通过失活生产细胞内的N-乙酰葡糖胺的分解代谢来改进。
根据本发明的一方面,在存在葡萄糖、蔗糖、甘油或其组合的情况下培养经遗传修饰的宿主细胞,但是不添加或不存在N-乙酰葡糖胺或半乳糖,也不存在其组合。
在本发明的方法和宿主细胞的优选实施方案中,所需寡糖为乳-N-三糖II,其通过在宿主细胞中通过异源表达的糖基转移酶β-1,4-半乳糖基转移酶和β-1,3-N-乙酰葡糖氨基转移酶的作用,从单一碳源(simple carbon source)的完全发酵(total fermentation)而产生。
如上文已经提及的,本发明还涉及用于产生所需寡糖的经遗传修饰的宿主细胞,所述寡糖包括乳-N-三糖II(LNT-II;GlcNAc(β1-3)Gal(β1-4)Gluc)作为核心三糖,其中所述宿主细胞包含至少一种重组的糖基转移酶(所述糖基转移酶优选地选自半乳糖基转移酶、唾液酰转移酶和N-乙酰葡糖氨基转移酶),并且具有至少内源性糖转运蛋白的表达或活性,所述内源性糖转运蛋白被修饰,从而使得所述内源性糖转运蛋白的表达或活性对于所需寡糖的前体的输出而言是功能性失活的。
一个优选实施方案涉及如上文所述的宿主细胞,其包括(i)异源表达的β-1,3-N-乙酰葡糖氨基转移酶和(ii)异源表达的β-1,3-半乳糖基转移酶或异源表达的β-1,4-半乳糖基转移酶来作为糖基转移酶,其中所述宿主细胞还优选地包含至少一种编码蛋白的同源或异源核酸序列,所述蛋白使得能够将寡糖输出到培养宿主细胞的培养基中,其中所述使得能够输出所需寡糖的蛋白属于次级主动转运蛋白类别,其中所述宿主细胞已经被修饰,从而使得所述同源或异源核酸序列的表达为过表达的或在使得能够过表达所述核酸序列的启动子的控制下。在宿主细胞的优选实施方案中,所述至少一种编码使得能够输出所需寡糖的+蛋白的核酸序列为内源性核酸序列或重组核酸序列。
如本发明的方法已经概述的,还优选地在本发明的宿主细胞中,所述编码使得能够输出所需寡糖的蛋白的核酸序列为细菌、古细菌、植物、酵母或动物来源。
根据本发明的另一方面,如上文所述的宿主细胞还包含至少一种内源性核酸序列的缺失、破坏、减少或失活,所述核酸序列编码将所需寡糖的前体输出到宿主细胞外的输出蛋白;和/或至少一种同源或异源核酸序列,其编码使得能够将所需寡糖的前体输入到所述宿主细胞中的蛋白,其中优选地所述核酸序列为过表达的,并且优选地所述前体大于二糖。
通过编码使得能够将所需寡糖的前体输入到所述宿主细胞中的蛋白的至少一种同源或异源核酸序列的过表达,可将所需寡糖的前体加料到培养基中,所述前体被输入到宿主细胞中,例如LNT-II。
根据本发明的一方面,在宿主细胞中,所述至少一种编码使得能够输出所需寡糖的蛋白的核酸序列为选自以下的基因:来自大肠杆菌的yebQ和yjhB,来自产琥珀酸曼氏杆菌的proP,以及来自奈氏西地西菌的setA。或它们的功能性片段。
根据另一个优选实施方案,所需寡糖为乳-N-三糖II,所述使得能够将所述寡糖输出到培养宿主细胞的培养基的蛋白是来自大肠杆菌的YjhB,来自产琥珀酸曼氏杆菌的ProP,以及来自奈氏西地西菌的SetA,或它们的功能性片段。
根据一个优选的实施方案,本发明的微生物宿主被进一步修饰以不表达输出所需寡糖的前体的蛋白。
在宿主细胞的一个优选实施方案中,所需寡糖为乳-N-四糖,所述前体为乳-N-三糖II,并且所述宿主细胞具有缺失、破坏或失活的至少一种核酸序列,所述核酸序列编码使得能够将乳-N-三糖II输出到宿主细胞外的输出蛋白。
在这方面,优选地,使得能够输出乳-N-四糖的蛋白选自来自大肠杆菌BL21(DE3)的YebQ、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)的SpoVB、沙梨欧文氏菌(Erwiniapyrilfolia)的YabM、大肠杆菌MG1655的Bcr、大肠杆菌MG1655的YdeA、副流感嗜血杆菌(Haemophilus parainfluenzae)的ProP2、胡萝卜软腐果胶杆菌(Pectobacteriumcarotovorum)的SetA、大肠杆菌MG1655的FucP、金黄色葡萄球菌(Staphylococcus aureus)Bmb9393的MdeA、乳酸乳球菌(Lactococcus lactis)的ImrA、假单胞菌属种(Pseudomonassp.)MT-1的SetA和巴西安白僵菌(Beauveria bassiana)D1-5的SetA。
优选地,所述寡糖输出蛋白为选自以下中的至少一种的蛋白:大肠杆菌的SetA、SetB、SetC、YdeA、Cmr、YnfM、MdtD、YfcJ、YhhS、EmrD、YdhC、YbdA、YdeE、MhpT、YebQ、YjhB、Bcr和YdeA,或来自产琥珀酸曼氏杆菌的ProP,以及来自奈氏西地西菌的SetA,或她们的变体或同源物。
目前,术语“核酸”是指单链或双链脱氧核糖核苷酸或核糖核苷酸大分子,并且包括与所需核酸杂交且编码某一多肽的已知类似物或天然或合成产生的核苷酸。
本文使用的关于微生物宿主细胞的术语“重组的”或“遗传修饰的”表明微生物宿主细胞复制异源核酸或表达由异源核酸(即“对所述细胞而言是外来的”序列)编码的肽或蛋白。重组细胞可包含在细胞的天然(非重组)形式中不存在的基因。重组细胞还可包含在细胞的天然形式中存在的基因,其中所述基因通过人工方法被修饰并重新引入到细胞中。该术语还包括这样的细胞,其包含已经修饰的对细胞而言是内源性核酸,而不从细胞中除去所述核酸;这样的修饰包括通过基因置换、位点特异性突变和相关技术获得的那些。因此,“重组多肽”是通过重组细胞产生的多肽。本文使用的“异源序列”或“异源核酸”是源自对特定宿主细胞而言是外来的来源(例如源自不同的物种)的序列或核酸,或者如果来自相同来源,是从其原始形式修饰而来的。因此,与启动子可操作地连接的异源核酸的来源不同于启动子的来源,或者如果来自相同来源,则是从其原始形式修饰而来的。可以(例如通过转染、转化、接合或转导)将异源序列稳定引入到宿主微生物宿主细胞,代表经遗传修饰的宿主细胞。可根据待引入所述序列的宿主细胞来应用技术。各种技术是本领域技术人员已知的,并且例如在Sambrook et al.,Molecular Cloning:A Laboratory Manual,第2版,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.(1989)中公开。
因此,“微生物宿主细胞”目前被理解为已被转化或转染或者能够被外源多核苷酸序列转化或转染的微生物(优选细菌)细胞。
因此,本发明中使用的核酸序列可例如被包含在载体中,所述载体待被稳定转化/转染或者以其他方式被引入到宿主微生物细胞中。
目前,本文使用的术语“可操作地连接的”应意指核酸表达控制序列(例如启动子、信号序列或转录因子结合位点阵列)和第二核酸序列之间的功能性连接,其中所述表达控制序列影响对应于第二序列的核酸的转录和/或翻译。因此,术语“启动子”指通常在DNA聚合物中的基因“之前”并提供起始向mRNA的转录的位点的DNA序列。“调控子”指通常也位于给定DNA聚合物中的基因的“上游”(即之前)的结合决定转录起始频率(或速率)的蛋白的DNA序列。这些序列统称为“启动子/调控子”或“控制”DNA序列,其位于功能性DNA聚合物中的所选基因(或基因系列)之前,协作以确定基因的转录(和最终表达)是否发生。在DNA聚合物中位于基因“之后”并提供终止向mRNA的转录的信号的DNA序列被称为转录“终止子”序列。
非常多的表达系统均可用于产生本发明的多肽。这样的载体尤其包括染色体载体、游离型载体和病毒衍生载体,例如源自细菌质粒、源自噬菌体、源自转座子、源自酵母游离体、源自插入元件、源自酵母染色体元件、源自病毒的载体,以及源自其组合的载体,例如源自质粒和噬菌体遗传元件(例如粘粒和噬菌粒)的那些。表达系统构建体可包含调节以及产生表达的控制区。通常,在这方面,任何适于在宿主中维持、增殖或表达多核苷酸以及合成多肽的系统或载体都可用于表达。通过多种公知和常规技术中的任一种(例如Sambrooket al.(见上文)中所述的那些)可将适合的DNA序列插入到表达系统中。
现有技术中有许多涉及“重组DNA”方法学的专利和文献出版物,所述方法学用于分离、合成、纯化和扩增用于所选宿主生物体的转化的遗传材料。因此,使用包含所选外源(即外来的或“异源”)DNA序列的“杂合”病毒或环状质粒DNA来转化宿主生物体是公知常识。本领域已知的方法首先包括通过酶促切割环状病毒或质粒DNA以形成线性DNA链而产生转化载体。通过使用相同/相似的酶,以制备线性形式的所选择的外源DNA链,其通常包含编码所需蛋白产物的序列。在存在能够进行恢复过程的连接酶的情况下,孵育线性病毒或质粒DNA与外源DNA,并且形成“杂合”载体,其包含“被剪接”到病毒或环状DNA质粒中的所选择的外源DNA片段。
本文使用的术语“培养”意指在培养基中并且在允许和适于产生所需寡糖的条件下使细菌细胞生长。在阅读本发明的公开内容之后,结合技术人员的技术和专业背景,本领域技术人员易于获得几种适合的细菌宿主细胞以及培养基和它们的培养条件。
本文使用的术语“回收”或“获得”意指从宿主细胞培养物中分离(isolate)、收获、纯化、收集或分开(separate)由本发明的宿主细胞产生的寡糖。
本发明的“微生物”宿主细胞按照通常的理解,意指任何微生物,包括细菌、真菌和古细菌,其通常适于大量培养并且可根据本发明进行遗传修饰以产生所需寡糖。优选的微生物为细菌,例如大肠杆菌、谷氨酸棒状杆菌(Corynebacterium glutamicum)和酵母酵母属种(Saccharomyces sp.),其优势在于这些微生物可在实验室环境中容易且廉价地生长,并且细菌和酵母已经被深入研究许多年。
通常以及在本发明通篇中,术语“糖基转移酶活性”或“糖基转移酶”表示且包括负责二糖、寡糖和多糖的生物合成的酶,它们催化单糖部分从活化的核苷酸单糖/糖(“糖基供体”)转移到糖基受体分子。
通常以及在本发明通篇中,术语“输出蛋白(exporter或exporter protein)”或“使得能够输出所需寡糖的蛋白”在本文中作为同义词使用,指这样的一种或多种多肽,其单独地或作为多蛋白复合物的一部分,将寡糖从细菌细胞的胞内环境转移到所述细胞的周质或培养物上清液中,因此使得所述寡糖能够穿过所述细胞的细胞膜和/或细胞壁。
在本发明的范围内,那些术语还包括核酸/多核苷酸和多肽多态变体、等位基因、突变体和种间同源物,其具有这样的氨基酸序列,即所述氨基酸序列相对于野生型糖基转移酶活性或寡糖输出显示蛋白编码的多肽,优选在至少约25、50、100、200、500、1000或更多个氨基酸的区域上,具有大于约60%的氨基酸序列同一性,65%、70%、75%、80%、85%、90%、优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更高的氨基酸序列同一性。
本文使用的术语“变体”是分别不同于参照多核苷酸或多肽,但是保留了必需特性的多核苷酸或多肽。多核苷酸的典型变体在核苷酸序列上不同于另一种参照多核苷酸。变体核苷酸序列的变化可改变或不改变由参照多核苷酸编码的多肽的氨基酸序列。核苷酸变化可在由参照序列编码的多肽中导致氨基酸取代、添加、缺失、融合和截短,如下文所讨论的。多肽的典型变体在氨基酸序列上不同于另一种参照多肽。通常,差异是有限的,从而使得参照多肽和变体的序列在整体上非常类似并且在许多区域中是相同的。变体和参照多肽在氨基酸序列上的区别在于任意组合的一种或多种取代、添加、缺失。取代的或插入的氨基酸残基可以是或不是遗传密码编码的残基。多核苷酸或多肽的变体可以是天然存在的,例如等位基因变体,或者其可以是已知不是天然存在的变体。多核苷酸和多肽的非天然变体可通过诱变技术、通过直接合成,以及通过本领域技术人员已知的其他重组方法来制备。
因此,本文公开的任何基因/蛋白的“功能性片段”意指仍然保留了与衍生出各个片段的基因或蛋白的活性相同或稍微更小的活性的基因/蛋白的序列变体。
在这方面,术语“编码……的核酸序列”通常是指任何多核糖核苷酸或多脱氧核糖核苷酸,其可以是未经修饰的RNA或DNA或经修饰的RNA或DNA,并且通常代表编码某种多肽或蛋白的基因。
在本文的上下文中,术语“多肽”是指包含两个或多个氨基酸的任何肽或蛋白,所述氨基酸通过肽键或经修饰的肽键彼此连接。“多肽”既指短链(通常指肽、寡肽和低聚物),也指长链(通常指“蛋白”)。多肽可包含20种基因编码的氨基酸以外的氨基酸。“多肽”包括通过天然过程(例如加工和其他翻译后修饰),以及通过化学修饰技术修饰的那些。应理解,相同类型的修饰可以以相同或不同程度存在于给定多肽的若干位点,而基本上不改变多肽的活性。同样,给定的多肽可包含许多类型的修饰。修饰可发生在多肽的任何位置,包括肽主链、氨基酸侧链,以及氨基末端或羧基末端。
另外,表述“前体”包括在本发明的寡糖的生物合成途径中涉及的化合物或在宿主细胞中产生和天然存在的化合物。
“大于二糖的前体”在本文中被理解为包含多于两个单糖残基的糖部分。
术语“所需寡糖”是指由至少三个部分组成的糖聚合物,因此包括三糖、四糖、戊糖等,优选选自以下的至少一种的寡糖:乳-N-三糖II、乳-N-四糖、乳-N-新四糖、乳-N-岩藻戊糖I、乳-N-岩藻戊糖II、乳-N-岩藻戊糖III、乳-N-岩藻戊糖V、乳-N-二岩藻基己糖I、乳-N-二岩藻基己糖II、乳-N-唾液酰戊糖LSTa、LSTb、LSTc、二唾液酰乳-N-四糖、二唾液酰乳-N-新四糖。
在本文中以及如相关领域中通常理解的,表述“同源的”是指编码特定的产物并且来自相同物种(其中插入所述核酸序列)的核酸序列。因此,术语“异源的”是指编码特定的产物并且来自其中插入所述核酸序列的那些物种以外的物种的核酸序列。
本文和通常本领域内的术语“内源性”意指编码目的酶的核酸源自细菌宿主细胞并且尚未被引入到所述宿主细胞中,而“重组的”核酸已被引入到所述宿主细胞中并且不源自所述宿主细胞。
表述“过表达的”或“过表达”或“在使得能够过表达所述核酸序列的启动子序列的控制下”目前以及通常在本领域中意指基因表达量大于正常量,即表达量增加,因此导致所述核酸序列编码的蛋白的量增加。
在一些实施方案中,所述核酸序列被置于诱导型启动子的控制下,所述启动子是指导基因表达的启动子,其中表达水平可被环境或发育因素(例如温度、pH、厌氧或有氧条件、光、转录因子和化学品)改变的。这样的启动子在本文中被称为“诱导型”启动子,其允许控制用于本发明的蛋白的表达时机。对于大肠杆菌和其他微生物宿主细胞,诱导型启动子是本领域技术人员已知的。
根据说明书和附图,其他优势是显而易见的。
应理解,上文提及的特征和将在下文中解释的那些特征不仅可以用于所示的各个组合中,还可以用于其他组合或单独使用,而不偏离本发明的上下文。
将在实施例和附图中详细描述本发明,其中:
图1示出了在培养基中培养的宿主细胞中产生乳-N-三糖II或乳-N-四糖的示意图。
图2示出了产乳-N-三糖II(LNT II)的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析结果,所述菌株过表达β-1,3-N-乙酰葡糖氨基转移酶基因PmnagT(13,14);
图3示出了产乳-N-四糖(LNT)的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析结果,所述菌株过表达β-1,4-半乳糖基转移酶编码基因BfgalT2(1)、PmgalT7(3)、MsgalT8(6)、gatD(7)、lex1(9)、lgtB(11)或lsgD(13);
图4示出了产乳-N-四糖(LNT)的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析结果,所述菌株过表达β-1,4-半乳糖基转移酶编码基因KdgalT10(1)、cpsl14J(7)、cpslaJ(8,9)、HpgalT(12);
图5示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析结果,所述菌株过表达β-1,4-半乳糖基转移酶编码基因waaX(5);
图6示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析结果,所述菌株过表达β-1,3-半乳糖基转移酶编码基因wbdO或furA;
图7示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的HPLC分析结果。(A)孵育24h之后,在存在葡萄糖和乳糖的情况下生长的大肠杆菌BL21(DE3)1353和1431的上清液。(B)孵育48h之后,在存在葡萄糖和乳糖的情况下生长的大肠杆菌BL21(DE3)1353和1431的上清液。
图8示出了描绘了与对照菌株1353相比,过表达糖流出转运蛋白的大肠杆菌BL21(DE3)菌株的上清液中乳-N-四糖的相对浓度的图;以及
图9示出了描绘了过表达糖流出转运蛋白TP11(2)、YjhB(3)或TP70(4)的大肠杆菌BL21(DE3)菌株的上清液中乳-N-三糖II的浓度的图。
实施例
图1示出了本发明的示例性宿主细胞10的示意图,输入乳糖并合成乳-N-三糖II(LNT II)和乳-N-四糖(LNT)。乳糖经由转运蛋白1从培养宿主细胞的培养基输入到细胞中。所述酶N-乙酰葡糖氨基转移酶NacGlcT将N-乙酰葡萄糖胺连接至受体底物乳糖,从而产生LNT-II。LNT-II经由输出蛋白20从细胞输中出。由于LNT-II是LNT或LNnT的前体,输出LNT-II的输出蛋白代表输出后者寡糖的前体的输出蛋白。如从图1中可进一步看出的,细胞包含具有β-1,3-半乳糖基转移酶活性的蛋白,使得能够进行LNT-II的半乳糖基化以在胞内产生LNT;细胞还可以和/或替代地包含β-1,4-半乳糖基转移酶活性,使得能够进行LNT-II的半乳糖基化以在胞内产生乳-N-新四糖LNnt。之后LNT(或者可以是LNnt)经由寡糖输出蛋白从细胞中被输出到培养细胞的培养基中。
输出蛋白是结合于膜的,并且它们的表达可为过表达的,在LNT-II输出蛋白过表达的情况下,这会导致LNT-II输出增加且LNT输出减少,而当输出LNT-II的输出蛋白缺失或失活时,这导致LNT输出增加。LNT-II输出蛋白优选地为内源性输出蛋白,而LNT输出蛋白优选地为异源的输出蛋白。
实施例1
大肠杆菌乳-N-三糖II生产菌株的开发
大肠杆菌BL21(DE3)用于构建乳-N-三糖II(LNT-2)生产菌株。代谢改造分别包括特定基因的诱变和缺失,以及异源基因的基因组整合。使用错配寡核苷酸,通过诱变使基因lacZ和araA失活,如Ellis et al.,"High efficiency mutagenesis,repair,andengineering of chromosomal DNA using single-stranded oligonucleotides",Proc.Natl.Acad.Sci.USA 98:6742-6746(2001)中所记载的。
根据Datsenko和Warner(Proc.Natl.Acad.Sci.USA 97:6640-6645(2000))的方法进行基因组缺失。为了防止N-乙酰葡萄糖胺的胞内降解,使大肠杆菌菌株BL21(DE3)的基因组缺失编码N-乙酰葡萄糖胺-6-磷酸脱乙酰酶的基因(nagA)和葡萄糖胺-6-磷酸脱氨酶的基因(nagB)。还缺失了wzxC-wcaJ基因。WcaJ编码UDP-葡萄糖:十一异戊二烯基磷酸葡萄糖-1-磷酸转移酶,其催化荚膜异多糖酸(colanic acid)合成中的第一步(Stevenson et al.,J.Bacteriol.1996,178:4885-4893)。此外,去除了分别编码L-岩藻糖异构酶和L-墨角藻糖(fuculose)激酶的基因fucl和fucK。
通过转座进行异源基因的基因组整合。将EZ-Tn5TM转座酶(Epicentre,USA)用于整合线性DNA片段,或将水手转座酶Himar1的活性过强的C9突变体(Lampe et al.,Proc.Natl.Acad.Sci.1999,USA 96:11428-11433)用于转座。为产生EZ-Tn5转座体,使用引物1119和1120(所有使用的引物列于以下表3中)来扩增目的基因以及侧翼有FRT位点的抗生素抗性标记物;所得PCR产物在两个位置上携带EZ-Tn5转座酶的19-bp嵌合末端(mosaicend)识别位点。对于使用Himar1转座酶进行整合,将目的表达构建体(操纵子)以及侧翼有FRT位点的抗生素抗性标记物类似地克隆到pEcomar载体中。pEcomar载体编码在阿拉伯糖诱导型启动子ParaB控制下的水手转座酶Himar1的活性过强的C9突变体。通过使用EZ-Tn5转座酶来整合表达片段<Ptet-lacY-FRT-aadA-FRT>(SeqID1)。在成功整合来自大肠杆菌K12TG1的乳糖输入蛋白LacY(登录号ABN72583)的基因之后,通过在质粒pCP20上编码的FLP重组酶从链霉素抗性克隆中除去抗性基因(Datsenko and Warner,Proc.Natl.Acad.Sci.2000,USA 97:6640-6645)。对来自脑膜炎奈瑟氏菌MC58的N-乙酰葡糖氨基转移酶基因lgtA(登录号NP_274923)进行密码子优化以在大肠杆菌中表达并通过基因合成来以合成方法制备。使用质粒pEcomar-lgtA-galT,通过转座插入lgtA以及编码来自大肠杆菌K-12亚菌株MG1655的半乳糖-1-磷酸尿苷酰基转移酶的基因galT(登录号NP_415279)(其类似地通过基因合成获得)(SeqID2)。为了增加UDP-N-乙酰葡萄糖胺的从头合成,对编码来自大肠杆菌K-12亚菌株MG1655的L-谷氨酰胺:D-岩藻糖-6-磷酸氨基转移酶的基因(glmS)、编码来自大肠杆菌K-12亚菌株MG1655的磷酸葡糖胺变位酶的基因(glmM)和编码来自大肠杆菌K-12亚菌株MG1655的N-乙酰葡糖胺-1-磷酸尿苷酰转移酶/葡糖胺-1-磷酸乙酰转移酶的基因(glmU)(登录号分别为NP_418185、NP_417643、NP_418186)进行密码子优化并通过基因合成获得。将操纵子glmUM克隆在组成型四环素启动子Ptet的控制下,同时将glmS克隆在组成型PT5启动子的控制下。将转座盒<Ptet-glmUM-PT5-glmS-FRT-dhfr-FRT>(SeqID3)(其侧翼为被水手样元件Himar1转座酶特异性识别的反向末端重复)从pEcomar-glmUM-glmS(其显示为乳-N-三糖II生产菌株)插入。另外,通过使用EZ-Tn5转座酶整合表达片段<Ptet-lacY(6HIS)-FRT-aadA-FRT>(SeqID4)。
使用引物605和606,从大肠杆菌K12TG1(SeqID6)扩增gal-操纵子(galETKM),并通过同源重组将其插入到大肠杆菌BL21(DE3)菌株的galM ybhJ基因座中,所述同源重组通过使用红色重组酶辅助质粒pKD46来辅助进行(Datsenko aand Warner,Proc.Natl.Acad.Sci.2000,USA 97:6640-6645)。异源基因和基因簇的序列记录在附录1中。
实施例2
筛选多种β-1,3-N-乙酰-糖氨基转移酶的大肠杆菌BL21(DE3)707的分批发酵
对来自多杀性巴氏杆菌多杀亚种(Pasteurella multocida subsp.multocida)菌株HN06的β-1,3-N-乙酰-葡糖氨基转移酶的基因PmnagT(登录号为PMCN06_0022)进行密码子优化并通过GenScript Cooperation(Piscataway,USA)以合成方法合成。通过不依赖序列和连接的克隆将基因克隆到质粒pET-DUET(Merck KGaA,Darmstadt,德国)中。用于克隆的全部引物列于以下表3中。
含有编码β-1,3-N-乙酰葡糖氨基转移酶的质粒pET-PmnagT的大肠杆菌BL21(DE3)707(下表2)在30℃下生长在补充有2%(重量/体积)葡萄糖和氨苄青霉素100μg ml-1的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中。当培养物达到0.1的OD660nm时,通过添加0.3mM IPTG来诱导基因表达。孵育4小时之后添加1.5mM乳糖。30℃下在摇瓶中另外孵育24小时之后收集细胞。通过薄层色谱法检测LNT-2。因此,使用玻璃小珠在确定的体积下机械破碎细胞。随后,将样品施加到TLC硅胶60F254(Merck KGaA,Darmstadt,德国)上。流动相由丙酮:丁醇:乙酸:水(35:35:7:23)组成。
TLC分析结果示于图2。当基因PmnagT过表达时,可观察到显示出与三糖标准品LNT-II相同的迁移率的化合物的形成。LNT-II生产菌株724充当对照(19)。描绘了乳糖(1)和LNT-II(2)的标准品。通过星号标记出样品中的LNT-II产物形成。
实施例3
过表达同源糖流出转运蛋白的大肠杆菌乳-N-三糖II生产菌株的产生
大肠杆菌中产生的寡糖的输出被证明是发酵过程中的限制因素。然而,三糖如2’-岩藻基乳糖和LNT-2在一定程度上被易位到培养上清液中,因此可能编码发挥作用的糖流出转运蛋白。为了增加乳-N-三糖II(LNT-II;GluNAc(β1-3)Gal(β1-4)Glc)的流出,将大肠杆菌BL21(DE3)菌株1326(下表2)用于筛选糖流出转运蛋白(SET)的文库。从大肠杆菌BL21(DE3)的基因组DNA扩增推定的大肠杆菌SET蛋白,并通过不依赖序列和连接的克隆将其整合到载体pINT中。以基因yjhB为例,使用引物2567、2568、2526和2443,产生质粒pINT-yjhB。用于克隆的引物序列列于下表3中。
含有编码20种不同的大肠杆菌转运蛋白的质粒的大肠杆菌BL21(DE3)1326在30℃下生长在补充有2%(重量/体积)葡萄糖、氨苄青霉素100μg ml-1和博莱霉素(zeocin)40μgml-1的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中。当培养物达到0.1的OD660nm时,通过添加200ng/ml无水四环素来诱导基因的基因表达。孵育4小时之后添加2.5mM乳糖。30℃下在摇瓶中另外孵育24和48小时之后,通过LC-MS测定上清液中LNT-II浓度。
使用LC三重四极杆MS检测系统,通过特征碎片离子检测来进行质量分析。选择前体离子并在四极杆1中进行分析,碎片化发生在碰撞室,使用氮气作为CID气体,碎片离子的选择是在四极杆3中进行。
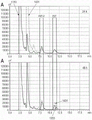
以ESI正离子化模式分析乳-N-四糖(LNT(Gal(β1-3)GlcNAc(β1-3)Gal(β1-4)Glc))、LNT-II和麦芽三糖(用于定量的内标)。LNT形成m/z 708.3[M+H+]的离子,LNT-II形成m/z 546.1[M+H+]的离子,麦芽三糖形成m/z 522.0[M+NH4+]的离子。该碳水化合物与铵离子(NH4+)形成加合物[m/z 504.0],导致质量偏移+18。因此,对于麦芽三糖而言,选择m/z522.0的前体离子。前体离子在碰撞室中进一步破碎成特征碎片离子m/z 487.1、m/z 325.0和m/z 163.2。LNT的分子离子(m/z 708.3)破碎成m/z 546.3、m/z 528.3、m/z 366.2和m/z204.0。LNT-II(m/z 546.1)破碎成m/z 204.2、186.0、138.0和126.0(参见方法说明)。
LNT和LNT-II的色谱分离是在Luna NH2HPLC柱(Phenomenex,Aschaffenburg,德国)上进行。这是必需的,因为在离子化过程中LNT的部分碎片化产生LNT-II信号,影响单独的碳水化合物的定量结果。
仅对于表达基因yjhB的菌株,观察到培养物上清液中LNT-2的量增加(参见下表1)。
表1:在过表达yjhB的大肠杆菌BL21(DE3)菌株和参照菌株的培养物上清液中LNT-II的计算浓度。
| 样品 | 孵育24h之后的计算浓度[μM] | 孵育48h之后的计算浓度[μM] | 分析物RT |
| 1326 | 751 | 1265 | 0.616 |
| 1326pINT-yjhB | 413 | 1975 | 0.609 |
实施例4
筛选多种β-1,4-半乳糖基转移酶的大肠杆菌BL21(DE3)724的分批发酵
对来自嗜沫凝聚杆菌(Aggregatibacter aphrophilus)的β-1,4-半乳糖基转移酶lex1的基因(登录号YP_003008647)、来自多杀性巴氏杆菌多杀亚种菌株HN06的PmgalT7(登录号PMCN06_0021)、来自叶柄粘球菌(Myxococcus stipitatu)DSM14675的MsgalT8(登录号MYSTI_04346)、来自脱氮金氏菌(Kingella denitrificans)ATCC 33394的KdgalT10(登录号HMPREF9098_2407)、来自多杀性巴氏杆菌M1404的gatD(登录号GQ444331)、来自脆弱拟杆菌(Bacterioidis fragilis)NCTC9343的BfgalT2(登录号BF9343_0585)、来自流感嗜血菌(Haemophilus influenza)的IsgD(登录号AAA24981)和来自幽门螺杆菌(Helicobacterpylori)的HpgalT(登录号AB035971)进行密码子优化并通过GenScript Cooperation(Piscataway,USA)以合成方法合成。通过不依赖序列和连接的克隆来进行基因克隆(Liand Elledge,Nat Methods.2007Mar;4(3):251-6)。因此,使用包含在无水四环素诱导型启动子控制下的malE基因的质粒pINT,使得使得能够产生β-1,4-半乳糖基转移酶基因与malE的N末端融合。单独地,将来自黑腐果胶杆菌(Pectobacterium atrosepticum)JG10-08的β-1,4-半乳糖基转移酶编码基因waaX(登录号ECA0154)克隆到质粒pACYC-Duet(Merck KGaA,Darmstadt,德国)中。用于克隆的所有引物均列于下表3中。
含有质粒pCDF-galE和编码malE与β-1,4-半乳糖基转移酶的基因融合的质粒的大肠杆菌BL21(DE3)724在30℃下生长在补充有2%(重量/体积)葡萄糖、氨苄青霉素100μgml-1和博莱霉素40μg ml-1的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中。当培养物达到0.1的OD660nm时,通过添加0.3mM IPTG和200ng/ml无水四环素来诱导galE基因和β-1,4-半乳糖基转移酶的基因表达。含有质粒pET-lgtA、pCOLA-glmUM-glmS、pCDF-galT-galE和pACYC-waaX的大肠杆菌BL21(DE3)534(下表2)在30℃下生长在补充有2%(重量/体积)葡萄糖、氨苄青霉素100μg ml-1、氯霉素34μg ml-1、链霉素50μg ml-1和卡那霉素30μg ml-1的矿物盐培养基中。当培养物达到0.1的OD660nm时,通过添加0.3mM IPTG来诱导基因表达。诱导基因表达之后4小时添加2mM乳糖。30℃下在摇瓶中另外孵育48小时之后收集细胞并进行机械破碎。通过薄层色谱法检测乳-N-新四糖(LNnT(Gal(β1-4)GlcNAc(β1-3)Gal(β1-4)Glc))。因此,使用玻璃小珠在确定的体积下机械破碎细胞。随后,将样品施加在TLC硅胶60F254(Merck KGaA,Darmstadt,德国)上。流动相由丙酮:丁醇:乙酸:水(35:35:7:23)组成。
TLC分析结果示于图3-5。图3示出了产乳-N-四糖(LNT)的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析,所述菌株过表达β-1,4-半乳糖基转移酶编码基因BfgalT2(1)、PmgalT7(3)、MsgalT8(6)、gatD(7)、lex1(9)、lgtB(11)或lsgD(13)。描绘了乳糖(15)、LNT-II(16)和LNnT(17)的标准品。通过星号标记出样品中LNnT产物形成。
图4示出了产乳-N-四糖(LNT)的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析,所述菌株过表达β-1,4-半乳糖基转移酶编码基因KdgalTIO(1)、cpsl14J(7)、cpslaJ(8,9)、HpgalT(12)。描绘了乳糖(3,15)、LNT-II(4,16)和LNnT(5,17)的标准品。通过星号标记出样品中LNnT产物形成。
图5示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析,所述菌株过表达β-1,4-半乳糖基转移酶编码基因waaX(5)。描绘了乳糖(1)、LNT-II和LNnT(2)的标准品。通过星号标记出样品中LNnT产物形成。
当以下基因过表达时,可观察到显示出与三糖标准品LNnT相同的迁移率的化合物形成:lex1、PmgalT7、MsgalT8、BfgalT2、gatD、IsgD、KdgalT10、HpgalT、wax。
从文献中已知产生LNnT的β-1,4-半乳糖基转移酶cpslaJ和cpsl14J(Watanabe etal.,J Biochem.2002Feb;131(2):183-91;Kolkman et al,J Bacteriol.1996Jul;178(13):3736-41)也包含在活性筛选中并且作为阳性对照。使用所述表达系统,通过CpslaJ和Cpsl14J可观察到LNnT形成(图3)。总共观察到30种测试基因中的11种从LNT-II和UDP-半乳糖产生LNnT。
实施例5
筛选不同的β-1,3-半乳糖基转移酶的大肠杆菌BL21(DE3)534的分批发酵
使用大肠杆菌K12DH5α的基因组作为模板,使用引物1163和1162来扩增galE。纯化PCR产物,用限制性核酸内切酶NdeI和XhoI进行限制性切割,并连接到用相同的酶切割的载体pCDFDuet(Merck KGaA,Darmstadt,德国)的第二多克隆位点中。从IPTG诱导型T7启动子表达GalE。使用引物991-994,从基因组DNA扩增大肠杆菌K12基因galT,并通过不依赖序列和连接的克隆将其整合到质粒pCDF-galE中,产生质粒pCDF-galT-galE。
使用密码子优化的IgtA的基因作为模板,使用引物688和689进行扩增。纯化PCR产物,用限制性核酸内切酶NdeI和AatII进行限制性切割,并连接到用相同的酶切割的载体pCDFDuet(Merck KGaA,Darmstadt,德国)的多克隆位点中,产生质粒pET-lgtA。
使用引物848-851,通过不依赖序列和连接的克隆,将密码子优化的glmUM基因构建体克隆到质粒pCOLA-Duet(Merck KGaA,Darmstadt,德国)中。使用引物852和853来扩增glmS的密码子优化形式。纯化PCR产物,用限制性核酸内切酶NdeI和AatII进行限制性切割,并连接到用相同的酶切割的载体pCOLA-glmUM的第二多克隆位点中,产生质粒pCOLA-glmUM-glmS。
还对来自肠道沙门氏菌萨拉姆亚种(Salmonella enterica subsp.salamae)血清型Greenside的β-1,3-半乳糖基转移酶的基因wbdO(登录号AY730594)和来自Lutiellanitroferrum 2002的furA(FuraDRAFT_0419)进行密码子优化,并通过GenScriptCooperation(Piscataway,USA)以合成方法合成。通过不依赖序列和连接的克隆,将所述基因克隆到质粒pACYC-Duet(Merck KGaA,Darmstadt,德国)。所有用于克隆的引物均列于下表3中。
含有质粒pET-lgtA、pCOLA-glmUM-glmS、pCDF-galT-galE和编码β-1,3-半乳糖基转移酶的质粒pACYC-furA或pACYC-wbdO的大肠杆菌BL21(DE3)534在30℃下生长在补充有2%(w/v)葡萄糖、氨苄青霉素100μg ml-1、氯霉素34μg ml-1、链霉素50μg ml-1和卡那霉素30μg ml-1的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中。当培养物达到0.1的OD660nm时,通过添加0.3mM IPTG来诱导基因表达。孵育4小时之后添加2mM乳糖。30℃下在摇瓶中另外孵育48小时之后收集细胞。通过薄层色谱法检测LNT。因此,使用玻璃小珠机械破碎细胞。随后,将样品施加到TLC硅胶60F254(Merck KGaA,Darmstadt,德国)上。流动相由丙酮:丁醇:乙酸:水(35:35:7:23)组成。
TLC分析的结果示于图6,其示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的培养提取物的TLC分析,所述菌株过表达β-1,3-半乳糖基转移酶编码基因wbdO或furA。标记出了样品中LNT产物形成。在12种测试的推定β-1,3-半乳糖基转移酶中,仅当基因wbdO或furA过表达时可观察到显示出与四糖标准品LNT相同迁移率的化合物的形成。
实施例6
改进的无质粒的大肠杆菌乳-N-四糖生产菌株的开发
将大肠杆菌BL21(DE3)菌株724用于构建乳-N-四糖(LNT)生产菌株。代谢改造包括转座子盒
<Ptet-wbdO-PT5-galE-FRT-cat-FRT>(SeqlD5)的基因组整合,所述转座子盒的侧翼为被水手样元件Himar1转座酶特异性识别的反向末端重复,其从pEcomar-wbdO-galE插入。对所得菌株1353进行进一步代谢改造以显示出增加的胞内LNT-II池,使LNT产量增加。因此,从大肠杆菌菌株的基因组中缺失主要易化子超家族转运蛋白yjhB(登录号YP_003001824),产生菌株1431(以下表2)。
30℃下在包含2%(重量/体积)葡萄糖(作为唯一碳源和能源)的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中进行大肠杆菌BL21(DE3)菌株1353和1431的分批发酵48小时。当培养物达到0.5的OD660nm时,添加2.5mM乳糖。通过高效液相色谱法(HPLC)检测培养上清液中LNT-II和LNT的存在。
使用连接至HPLC系统(Shimadzu,Duisburg,德国)的折射率检测仪(RID-10A)(Shimadzu,Duisburg,德国)和ReproSil Carbohydrate,5μm(250mm×4.6mm)(Dr.MaischGmbH,德国)进行HPLC分析。在35℃下,以1.4ml/min的流速,使用乙腈:H2O(68/32(v/v))作为洗脱剂,进行等强度洗脱。将40μl样品施加于柱上。通过在离子交换基质上的固相萃取(Strata ABW,Phenomenex,Aschaffenburg,德国)过滤样品(0.22μm孔径)并使之澄清。
HPLC分析的结果示于图7,示出了产乳-N-四糖的大肠杆菌BL21(DE3)菌株的培养物上清液的HPLC分析。(A)孵育24h之后,在存在葡萄糖和乳糖的情况下生长的大肠杆菌BL21(DE3)1353(黑色图)和1431(粉色图)的上清液。(B)孵育48h之后,在存在葡萄糖和乳糖的情况下生长的大肠杆菌BL21(DE3)1353(蓝色图)和1431(褐色图)的上清液。从HPLC分析中可以看出,产LNT菌株中yjhB的缺失导致培养物上清液中LNT的积累增加。
实施例7
过表达糖流出转运蛋白的大肠杆菌乳-N-四糖生产菌株的产生
由于乳-N-四糖向培养基中的输出对于生产菌株而言仅为中等程度,因此进行糖流出转运蛋白文库的筛选。根据实施例3,从大肠杆菌基因组DNA扩增推定的SET蛋白,或对其进行密码子优化并通过GenScript Cooperation(Piscataway,USA)以合成方法合成。扩增之后,通过不依赖序列和连接的克隆将基因整合到载体pINT中。根据实施例3设计用于克隆大肠杆菌基因的引物。使用引物2527、2444、2526和2443,使用标准化核苷酸突出物(overhang)合成合成基因,同样将其整合到表达载体中。用于克隆的引物序列列于下表3中。
含有编码66种不同的转运蛋白的质粒的大肠杆菌BL21(DE3)1353(下表2)在30℃下生长在补充有3%(w/v)葡萄糖、5g l-1NH4Cl2、氨苄青霉素100μg ml-1和卡那霉素15μgml-1的矿物盐培养基(Samain et al.,J.Biotech.1999,72:33-47)中。在含有200μl总体积的96孔板中进行预培养。30℃下通过连续振荡孵育24h之后,将50μl/孔转移到96孔深孔板中,所述板包含总体积为400μl的矿物盐培养基,所述培养基额外补充有200ng ml-1无水四环素和10mM乳糖。在持续孵育24至48小时之后,通过LC-MS测定上清液中的LNT浓度。如实施例3中所述进行质量分析。
图8示出了与对照菌株1353相比,过表达糖流出转运蛋白的大肠杆菌BL21(DE3)菌株的上清液中乳-N-四糖的相对浓度。将菌株1353的LNT效价设定为100%。如图8所示,66种基因中的11种的过表达导致LNT产量翻倍。在这些中,还证明大肠杆菌BL21(DE3)的基因组中编码的蛋白增加LNT输出(TP37,yebQ,登录号NC_012971)。YebQ是预测的MFS转运蛋白,推定其参与多药物流出,其可代表在菌株1353发酵过程中实现所观察到的LNT基础流出的负责的转运蛋白。
此外,由以下基因编码的转运蛋白在大肠杆菌生产菌株1353中过表达时导致LNT产生增加:解淀粉芽孢杆菌的spoVB(TP1,登录号AFJ60154)、沙梨欧文氏菌的yabM(TP2,登录号CAY73138)、大肠杆菌MG1655的bcr(TP18,登录号AAC75243)、大肠杆菌MG1655的ydeA(TP20,登录号AAC74601)、副流感嗜血杆菌的proP2(TP54,登录号EGC72107)、胡萝卜软腐果胶杆菌的setA(TP55,登录号ZP_03829909)、大肠杆菌MG1655的fucP(TP59,登录号AIZ90162)、金黄色葡萄球菌Bmb9393的mdeA(TP61,登录号SABB_01261)、乳酸乳球菌的ImrA(TP62,登录号L116532)、假单胞菌属种MT-1的setA(TP72,登录号BAP78849)和巴西安白僵菌D1-5的setA(TP73,登录号KGQ13398)。
实施例8
通过过表达异源糖流出转运蛋白产生大肠杆菌乳-N-三糖II生产菌株
值得注意的是,实施例6中所述的LNT输出蛋白筛选公开了两种蛋白:来自产琥珀酸曼氏杆菌MBEL55E的TP11(proP,登录号AAU37785)和来自奈氏西地西菌M006的TP70(setA,登录号WP_039290253),所述蛋白的过表达导致LNT-II的产生显著增加以及因此LNT产生减少(数据未示出)。这种观察结果在实施例3中所述的实验方案中得到确认。糖流出转运蛋白YjhB的过表达作为阳性对照。TP11和TP70的过表达导致LNT-II产量约增加4倍,该产量甚至略高于YjhB:图9示出的图标显示了过表达糖流出转运蛋白TP11(2)、YjhB(3)或TP70(4)的大肠杆菌BL21(DE3)菌株的上清液中乳-N-三糖II的浓度。含有空对照质粒的菌株1326作为对照(1)。因此,鉴定了三种糖流出转运蛋白,其靶向LNT-II以进行输出并且其过表达可有用于改造LNT-II生产菌株。
表2:菌株和质粒
表3:用于PCR的寡核苷酸
Claims (30)
1.用于通过经遗传修饰的微生物宿主细胞产生包括作为核心三糖的乳-N-三糖II的所需寡糖的方法,包括以下步骤:
-提供经遗传修饰的微生物宿主细胞,所述宿主细胞
-包含至少一种重组的糖基转移酶,
-具有至少一种内源性糖输出蛋白的表达或活性,所述蛋白被修饰从而使得与未经遗传修饰的宿主细胞相比,所述糖输出蛋白的表达或活性减少或失活,从而使得与未经遗传修饰的宿主细胞相比,所需寡糖的前体向培养基中的输出减少或消除,
-具有至少一种内源性糖输出蛋白的表达或活性,所述蛋白被修饰从而使得与未经遗传修饰的宿主细胞相比,所述糖输出蛋白的表达或活性增加,从而使得与未经遗传修饰的宿主细胞相比,所需寡糖的输出增加,
-在允许产生所需寡糖的条件下在培养基中培养所述宿主细胞,由此将所需寡糖转运到培养基中,并且
-从所述培养基中获得所需寡糖。
2.权利要求1的方法,其中所述寡糖为人乳寡糖。
3.权利要求1或2的方法,其中所述寡糖选自乳-N-三糖II、乳-N-四糖、乳-N-新四糖、乳-N-岩藻戊糖I、乳-N-岩藻戊糖II、乳-N-岩藻戊糖III、乳-N-岩藻戊糖V、乳-N-二岩藻基己糖I、乳-N-二岩藻基己糖II、乳-N-唾液酰戊糖LSTa、LSTb、LSTc、二唾液酰乳-N-四糖、二唾液酰乳-N-新四糖。
4.权利要求1或2的方法,其中所述宿主细胞包含:
-至少一种同源或异源核酸序列,所述序列编码使得能够将所需寡糖输出到培养基中的蛋白,其中所述宿主细胞已经修饰,从而使得所述同源或异源核酸序列的表达为过表达的;和
-至少一种编码输出蛋白的内源性核酸序列的缺失、破坏、减少或失活,所述输出蛋白将所需寡糖的前体输出到宿主细胞外;和/或
-至少一种编码蛋白的同源或异源核酸序列的过表达,所述蛋白介导所需寡糖的前体输入到所述宿主细胞中,其中所述前体大于二糖。
5.权利要求4的方法,其中所述使得能够输出所需寡糖的蛋白属于次级主动转运蛋白的类别。
6.权利要求4的方法,其中所述使得能够输出所需寡糖的蛋白进行包含至少三个部分的寡糖的输出。
7.权利要求6的方法,其中所述使得能够输出所需寡糖的蛋白进行包含作为核心三糖的乳-N-三糖II的寡糖的输出。
8.权利要求4的方法,其中所述至少一种编码使得能够输出所需寡糖的蛋白的核酸序列是内源性核酸或重组核酸。
9.权利要求4的方法,其中所述编码使得能够输出所需寡糖的蛋白的核酸序列为细菌、古细菌、植物、酵母或动物来源。
10.权利要求4的方法,其中所述至少一种编码使得能够输出所需寡糖的蛋白的核酸序列是选自以下的基因:来自大肠杆菌(Escherichia coli)的yebQ和yjhB,来自产琥珀酸曼氏杆菌(Mannheimia succiniciproducens)的proP,以及来自奈氏西地西菌(Cedeceaneteri)的setA。
11.权利要求1的方法,其中所述重组的糖基转移酶选自以下中的至少一种:半乳糖基转移酶、唾液酰转移酶、N-乙酰葡糖胺转移酶和岩藻糖基转移酶。
12.权利要求11的方法,其中所述重组的糖基转移酶选自以下中的至少一种:β-1,3-N-乙酰葡糖胺转移酶、β-1,3-半乳糖基转移酶、β-1,4-半乳糖基转移酶、β-1,6-半乳糖基转移酶、α-2,3-唾液酰转移酶、α-2,6-唾液酰转移酶、α-1,2-岩藻糖基转移酶或α-1,3-岩藻糖基转移酶。
13.权利要求1的方法,其中所述宿主细胞包含(i)β-1,3-N-乙酰葡糖胺转移酶和(ii)β-1,3-半乳糖基转移酶或β-1,4-半乳糖基转移酶来作为糖基转移酶。
14.权利要求12或13中任一项的方法,其中所述β-1,3-N-乙酰葡糖氨基转移酶属于脑膜炎奈瑟氏菌(Neisseria meningitides)的lgtA或多杀性巴氏杆菌(Pasteurellamultocida)的PmnagT的类别。
15.权利要求13的方法,其中产生乳-N-四糖的β-1,3-半乳糖基转移酶是WbdO。
16.权利要求1的方法,其中在适用的情况下,在所述经遗传修饰的宿主细胞中内源性β-半乳糖苷酶基因和葡糖胺-6-磷酸脱氨酶基因失活或缺失,并且其中所述经遗传修饰的宿主细胞包含编码功能性乳糖通透酶蛋白的核酸序列。
17.权利要求1的方法,其中与未经遗传修饰的宿主细胞相比,所述经遗传修饰的宿主细胞包括提高的UDP-N-乙酰葡糖胺和UDP-半乳糖或GDP-岩藻糖或CMP-N-乙酰神经氨酸的产生能力。
18.权利要求17的方法,其中所述提高的UDP-N-乙酰葡糖胺和UDP-半乳糖产生能力包括编码蛋白的一种或多种基因的过表达,所述蛋白包含以下酶的活性:L-谷氨酰胺:D-果糖-6-磷酸氨基转移酶、N-乙酰葡糖胺-1-磷酸尿苷酰转移酶/葡糖胺-1-磷酸乙酰转移酶、磷酸葡糖胺变位酶、UDP-半乳糖-4-差向异构酶、葡糖磷酸变位酶、葡萄糖-1-磷酸尿苷酰基转移酶。
19.权利要求16或17的方法,其中所述经遗传修饰的微生物宿主细胞是在存在葡萄糖、蔗糖、甘油或其组合的情况下培养,但是不添加或不存在N-乙酰葡萄糖胺或半乳糖,也不存在其组合。
20.权利要求1的方法,其中所需寡糖为乳-N-三糖II,其是通过在宿主细胞中通过异源表达的糖基转移酶β-1,4-半乳糖基转移酶和β-1,3-N-乙酰葡糖氨基转移酶的作用,从单一碳源的完全发酵而产生。
21.用于产生所需寡糖的经遗传修饰的微生物宿主细胞,所述寡糖包括作为核心三糖的乳-N-三糖II,其中所述宿主细胞
-包含至少一种重组的糖基转移酶,
-具有至少内源性糖转运蛋白的表达或活性,所述内源性糖转运蛋白被修饰,从而使得所述内源性糖转运蛋白的表达或活性对于所需寡糖的前体的输出而言是功能性失活的,并且
-具有至少一种内源性糖输出蛋白的表达或活性,所述蛋白被修饰从而使得与未经遗传修饰的宿主细胞相比,所述糖输出蛋白的表达或活性增加,从而使得与未经遗传修饰的宿主细胞相比,所需寡糖的输出增加。
22.权利要求21的微生物宿主细胞,其中所述糖基转移酶选自半乳糖基转移酶、唾液酰转移酶和N-乙酰葡糖氨基转移酶。
23.权利要求21的微生物宿主细胞,其包含(i)异源表达的β-1,3-N-乙酰葡糖氨基转移酶和(ii)异源表达的β-1,3-半乳糖基转移酶或异源表达的β-1,4-半乳糖基转移酶来作为糖基转移酶。
24.权利要求21或23的微生物宿主细胞,其还包含
-至少一种同源或异源核酸序列,所述序列编码使得能够将寡糖输出到培养宿主细胞的培养基中的蛋白,其中所述使得能够输出所需寡糖的蛋白属于次级主动转运蛋白的类别,并且其中所述编码使得能够输出所需寡糖的蛋白的同源或异源核酸序列为过表达的或在使得能够过表达所述核酸序列的启动子序列的控制下;和
-至少一种编码输出蛋白的内源性核酸序列的缺失、破坏、减少或失活,所述输出蛋白将所需寡糖的前体输出到宿主细胞外;和/或
-至少一种编码蛋白的同源或异源核酸序列的过表达,所述蛋白介导所需寡糖的前体输入到所述宿主细胞中,其中所述前体大于二糖。
25.权利要求24的微生物宿主细胞,其中所述编码使得能够输出所需寡糖的蛋白的核酸序列为细菌、古细菌、植物、酵母或动物来源。
26.权利要求24的微生物宿主细胞,其中所述至少一种编码使得能够输出所需寡糖的蛋白的核酸序列为内源性核酸序列或重组的核酸序列。
27.权利要求24的微生物宿主细胞,其中所述至少一种编码使得能够输出所需寡糖的蛋白的核酸序列是选自以下的基因:来自大肠杆菌的yebQ和yjhB,来自产琥珀酸曼氏杆菌的proP以及来自奈氏西地西菌的setA。
28.权利要求27的微生物宿主细胞,其中所需寡糖为乳-N-三糖II。
29.权利要求25或27中任一项的微生物宿主细胞,其中所需寡糖为乳-N-四糖,所述前体为乳-N-三糖II,其中所述宿主细胞具有缺失、破坏或失活的至少一种编码输出蛋白的核酸序列,所述输出蛋白能够将乳-N-三糖II输出到宿主细胞外。
30.权利要求29的微生物宿主细胞,其中所述使得能够输出乳-N-四糖的蛋白选自来自大肠杆菌BL21(DE3)的YebQ、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)的SpoVB、沙梨欧文氏菌(Erwinia pyrilfolia)的YabM、大肠杆菌MG1655的Bcr、大肠杆菌MG1655的YdeA、副流感嗜血杆菌(Haemophilus parainfluenzae)的ProP2、胡萝卜软腐果胶杆菌(Pectobacterium carotovorum)的SetA、大肠杆菌MG1655的FucP、金黄色葡萄球菌(Staphylococcus aureus)Bmb9393的MdeA、乳酸乳球菌(Lactoccoccus lactis)的ImrA、假单胞菌属种(Pseudomonas sp.)MT-1的SetA和巴西安白僵菌(Beauveria bassiana)D1-5的SetA。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| EP15184968.4A EP3141610A1 (en) | 2015-09-12 | 2015-09-12 | Production of human milk oligosaccharides in microbial hosts with engineered import / export |
| EP15184968.4 | 2015-09-12 | ||
| PCT/EP2016/071420 WO2017042382A1 (en) | 2015-09-12 | 2016-09-12 | Production of human milk oligosaccharides in microbial hosts with engineered import / export |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| CN108026556A CN108026556A (zh) | 2018-05-11 |
| CN108026556B true CN108026556B (zh) | 2023-01-20 |
Family
ID=54148344
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201680052611.8A Active CN108026556B (zh) | 2015-09-12 | 2016-09-12 | 在具有经改造的输入/输出的微生物宿主中人乳寡糖的产生 |
Country Status (11)
| Country | Link |
|---|---|
| US (4) | US11046985B2 (zh) |
| EP (2) | EP3141610A1 (zh) |
| JP (4) | JP7069007B2 (zh) |
| KR (2) | KR102700050B1 (zh) |
| CN (1) | CN108026556B (zh) |
| AU (1) | AU2016319304B2 (zh) |
| BR (1) | BR112018004725A2 (zh) |
| MX (2) | MX2018003037A (zh) |
| PH (1) | PH12018550020A1 (zh) |
| RU (1) | RU2763170C2 (zh) |
| WO (1) | WO2017042382A1 (zh) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US11875220B2 (en) * | 2018-09-04 | 2024-01-16 | Tencent Technology (Shenzhen) Company Limited | Method, apparatus, and storage medium for generating network representation for neural network |
Families Citing this family (55)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| PL2896628T3 (pl) | 2014-01-20 | 2019-03-29 | Jennewein Biotechnologie Gmbh | Sposób wydajnego oczyszczania obojętnych oligosacharydów ludzkiego mleka (HMO) z fermentacji mikrobiologicznej |
| EP3141610A1 (en) | 2015-09-12 | 2017-03-15 | Jennewein Biotechnologie GmbH | Production of human milk oligosaccharides in microbial hosts with engineered import / export |
| AU2018305183A1 (en) * | 2017-07-26 | 2020-02-13 | Chr. Hansen HMO GmbH | Sialyltransferases and their use in producing sialylated oligosaccharides |
| EP3784772A1 (en) | 2018-04-23 | 2021-03-03 | DuPont Nutrition Biosciences ApS | Increasing activity of 2' fucosyllactose transporters endogenous to microbial cells |
| EP3790413A1 (en) | 2018-05-07 | 2021-03-17 | Jennewein Biotechnologie GmbH | A SIMPLE METHOD FOR THE PURIFICATION OF LACTO-N-NEOTETRAOSE (LNnT) FROM CARBOHYDRATES OBTAINED BY MICROBIAL FERMENTATION |
| CN109554385B (zh) * | 2018-07-24 | 2022-04-12 | 石家庄葛兰德生物科技有限公司 | 一种基因工程菌制备2-岩藻糖基乳糖的方法 |
| EP3861123A4 (en) * | 2018-10-02 | 2022-08-10 | Zimitech, Inc. | USE OF SUBSTRATE IMPORTERS FOR EXPORT OF OLIGOSACCHARIDS |
| CN109370948B (zh) * | 2018-11-26 | 2020-08-04 | 江南大学 | 一株高产6-磷酸-β-半乳糖苷酶的乳酸乳球菌及其应用 |
| CN109735480B (zh) * | 2019-02-27 | 2021-11-12 | 光明乳业股份有限公司 | 一种合成乳酰-n-新四糖的重组枯草芽孢杆菌及其构建方法与应用 |
| CN109825465B (zh) * | 2019-02-27 | 2021-11-12 | 光明乳业股份有限公司 | 基于平衡udp-糖供给合成乳酰-n-新四糖的重组枯草芽孢杆菌及其构建方法与应用 |
| CA3135164A1 (en) * | 2019-04-02 | 2020-10-08 | Spiber Inc. | Method for producing recombinant protein |
| EP3751003A1 (en) | 2019-06-12 | 2020-12-16 | Jennewein Biotechnologie GmbH | Production of fucosylated oligosaccharides in bacillus |
| EP3999635A1 (en) * | 2019-07-19 | 2022-05-25 | Inbiose N.V. | Production of fucosyllactose in host cells |
| MX2022001814A (es) * | 2019-08-13 | 2022-06-27 | Amyris Inc | Produccion mejorada de oligosacaridos en levadura. |
| EP3789495A1 (en) | 2019-09-03 | 2021-03-10 | Jennewein Biotechnologie GmbH | Production of sialylated oligosaccharides in bacillus cells |
| WO2021019104A2 (en) | 2019-09-18 | 2021-02-04 | Basf Se | A production host for producing human milk oligosaccharides |
| JP2022548785A (ja) | 2019-09-24 | 2022-11-21 | プロラクタ バイオサイエンス,インコーポレイテッド | 炎症性疾患及び免疫疾患の治療のための組成物及び方法 |
| BR112022011904A2 (pt) * | 2019-12-18 | 2022-09-06 | Inbiose Nv | Produção de oligossacarídeo sialilado em células hospedeiras |
| EP3848471A1 (en) * | 2020-01-10 | 2021-07-14 | Chr. Hansen HMO GmbH | Sequential fermentative production of oligosaccharides |
| AU2021209395A1 (en) | 2020-01-23 | 2022-07-14 | Glycom A/S | New major facilitator superfamily (MFS) protein (Fred) in HMO production |
| CN114981291A (zh) * | 2020-01-23 | 2022-08-30 | 格礼卡姆股份公司 | Hmo产生 |
| CN115003813A (zh) * | 2020-01-23 | 2022-09-02 | 格礼卡姆股份公司 | Hmo生产中的新的主要易化因子超家族(mfs)蛋白质(fred) |
| BR112022014414A2 (pt) * | 2020-01-23 | 2022-09-13 | Glycom As | Produção de hmo |
| WO2021148614A1 (en) * | 2020-01-23 | 2021-07-29 | Glycom A/S | Hmo production |
| WO2021202883A1 (en) * | 2020-04-01 | 2021-10-07 | Zimitech, Inc. | Methods for production of oligosaccharides |
| EP3929300A1 (en) | 2020-06-26 | 2021-12-29 | Chr. Hansen HMO GmbH | Improved export of oligosaccharides from bacterial cells |
| BR112023002758A2 (pt) | 2020-08-14 | 2023-05-02 | Prolacta Bioscience Inc | Composições de oligossacarídeos de leite humano para uso com bacterioterapias |
| CN111979168B (zh) * | 2020-08-17 | 2022-07-22 | 江南大学 | 一种提高乳酰-n-三糖ii产量的基因工程菌及生产方法 |
| EP4263851A1 (en) * | 2020-12-17 | 2023-10-25 | Amyris, Inc. | MODIFIED ß-1, 3-N-ACETYLGLUCOSAMINYLTRANSFERASE POLYPEPTIDES |
| DK180952B1 (en) | 2020-12-22 | 2022-08-10 | Glycom As | A dfl-producing strain |
| KR20230131228A (ko) | 2021-01-12 | 2023-09-12 | 프롤랙타 바이오사이언스, 인코포레이티드 | 신바이오틱 치료 요법 |
| JP2024503868A (ja) * | 2021-01-17 | 2024-01-29 | ジェノマティカ, インコーポレイテッド | アミド化合物を生成する方法および組成物 |
| EP4281577A1 (en) * | 2021-01-20 | 2023-11-29 | Inbiose N.V. | Production of oligosaccharides comprising ln3 as core structure in host cells |
| WO2022157280A1 (en) | 2021-01-22 | 2022-07-28 | Glycom A/S | New major facilitator superfamily (mfs) protein (fred) in production of sialylated hmos |
| CN116848248A (zh) * | 2021-02-08 | 2023-10-03 | 协和发酵生化株式会社 | 具有含有岩藻糖的糖质的转运活性的蛋白和含有岩藻糖的糖质的制造方法 |
| CN115197954B (zh) * | 2021-04-14 | 2024-09-17 | 上海凯赛生物技术股份有限公司 | 用于发酵生产1,5-戊二胺的重组dna、菌株及其用途 |
| WO2022219188A1 (en) * | 2021-04-16 | 2022-10-20 | Inbiose N.V. | Cellular production of sialylated di- and/or oligosaccharides |
| DK181497B1 (en) | 2021-05-17 | 2024-03-12 | Dsm Ip Assets Bv | ENHANCING FORMATION OF THE HMOS LNT AND/OR LNnT BY MODIFYING LACTOSE IMPORT IN THE CELL |
| DK181242B1 (en) | 2021-05-17 | 2023-05-30 | Dsm Ip Assets Bv | GENETICALLY ENGINEERED CELLS COMPRISING A RECOMBINANT NUCLEIC ACID SEQUNCE ENCODING AN α-1,2-FUCOSYLTRANSFERASE CAPABLE OF PRODUCING LNFP-I, NUCLEIC ACID SEQUENCES ENCODING SAME AND METHODS FOR USE OF SAME |
| DK181310B1 (en) * | 2021-05-17 | 2023-08-08 | Dsm Ip Assets Bv | Cell factories for lnt-ii production |
| EP4341277A1 (en) | 2021-05-17 | 2024-03-27 | DSM IP Assets B.V. | Microbial strain expressing an invertase/sucrose hydrolase |
| BR112023024033A2 (pt) | 2021-05-20 | 2024-02-06 | Chr Hansen As | Processo para a produção de um oligossacarídeo desejado e processo para a produção de lacto-n-neotetraose |
| CN113684163B (zh) * | 2021-08-04 | 2023-07-25 | 江南大学 | 一种提高乳酰-n-四糖产量的基因工程菌及其生产方法 |
| CN113684164B (zh) * | 2021-08-06 | 2023-08-25 | 江南大学 | 一种高产乳酰-n-新四糖的微生物的构建方法及应用 |
| CN118541471A (zh) | 2021-12-21 | 2024-08-23 | 协和发酵生化株式会社 | 含有乳糖-n-三糖ii作为核心三糖的糖质的制造方法和该糖质的晶体的制造方法 |
| WO2023168441A1 (en) * | 2022-03-04 | 2023-09-07 | The Regents Of The University Of California | Production of milk oligosaccharides in plants |
| CN116042562B (zh) * | 2022-03-11 | 2023-10-20 | 山东恒鲁生物科技有限公司 | 重组酵母菌及其应用 |
| CN117003803A (zh) | 2022-05-07 | 2023-11-07 | 山东恒鲁生物科技有限公司 | 一种三糖的新晶型 |
| WO2023219437A1 (ko) * | 2022-05-11 | 2023-11-16 | (주)에이피테크놀로지 | 코리네박테리움 글루타미쿰을 이용한 락토-n-테트라오스 및 락토-n-네오테트라오스의 생산 방법 |
| CN115873051B (zh) | 2022-05-17 | 2024-06-25 | 山东恒鲁生物科技有限公司 | 三糖的新晶型 |
| DK202200689A1 (en) | 2022-07-15 | 2024-02-27 | Dsm Ip Assets Bv | New fucosyltransferases for in vivo synthesis of lnfp-iii |
| WO2024089131A1 (en) * | 2022-10-25 | 2024-05-02 | Inbiose N.V. | Saccharide importers for lacto-n-triose |
| CN118056902A (zh) * | 2022-11-21 | 2024-05-21 | 山东恒鲁生物科技有限公司 | 合成HMOs的成熟多肽序列及应用 |
| WO2024128827A1 (ko) * | 2022-12-14 | 2024-06-20 | (주)에이피테크놀로지 | 코리네박테리움 글루타미쿰을 이용한 락토-n-푸코펜타오스의 생산 방법 |
| WO2024130119A2 (en) | 2022-12-16 | 2024-06-20 | Prolacta Bioscience, Inc. | Synbiotic compositions for short chain fatty acid production |
Family Cites Families (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| FR2796082B1 (fr) * | 1999-07-07 | 2003-06-27 | Centre Nat Rech Scient | Procede de production d'oligosaccharides |
| MXPA05012413A (es) | 2003-05-22 | 2006-02-13 | Genencor Int | Cepas bacterianas disenadas metabolicamente que tienen transportadores de gluconato endogenos no funcionales. |
| US20070202578A1 (en) * | 2005-08-26 | 2007-08-30 | Centre National De La Recherche Scientifique (Cnrs) | Production of globosides oligosaccharides using metabolically engineered microorganisms |
| DK1991690T3 (da) * | 2006-03-09 | 2014-04-14 | Centre Nat Rech Scient | Fremgangsmåde til fremstilling af sialylerede oligosaccharider |
| GB0909284D0 (en) * | 2009-05-29 | 2009-07-15 | Micromass Ltd | Acquisition system and method for mass spectrometer data |
| AU2009347610B2 (en) | 2009-06-08 | 2013-02-21 | Chr. Hansen HMO GmbH | HMO synthesis |
| WO2012168495A1 (es) * | 2011-06-07 | 2012-12-13 | Hero Ag | Obtención de oligosacaridos mediante un procedimiento biotecnológico |
| CN103060252B (zh) | 2012-12-25 | 2015-04-15 | 江南大学 | 一种高产乙酰氨基葡萄糖的枯草芽孢杆菌工程菌及其应用 |
| US10017804B2 (en) | 2013-02-11 | 2018-07-10 | Evolva Sa | Efficient production of steviol glycosides in recombinant hosts |
| WO2015032413A1 (en) * | 2013-09-06 | 2015-03-12 | Glycom A/S | Fermentative production of oligosaccharides |
| PL2896628T3 (pl) | 2014-01-20 | 2019-03-29 | Jennewein Biotechnologie Gmbh | Sposób wydajnego oczyszczania obojętnych oligosacharydów ludzkiego mleka (HMO) z fermentacji mikrobiologicznej |
| EP2905341B1 (en) * | 2014-02-07 | 2020-04-29 | Jennewein Biotechnologie GmbH | Methods for producing GDP-fucose using nucleotide sugar transporters and recombinant microorganism host cells used therefor |
| EP3461890A1 (en) | 2014-03-31 | 2019-04-03 | Jennewein Biotechnologie GmbH | Total fermentation of oligosaccharides |
| EP3141610A1 (en) | 2015-09-12 | 2017-03-15 | Jennewein Biotechnologie GmbH | Production of human milk oligosaccharides in microbial hosts with engineered import / export |
-
2015
- 2015-09-12 EP EP15184968.4A patent/EP3141610A1/en active Pending
-
2016
- 2016-09-12 CN CN201680052611.8A patent/CN108026556B/zh active Active
- 2016-09-12 MX MX2018003037A patent/MX2018003037A/es unknown
- 2016-09-12 KR KR1020187007254A patent/KR102700050B1/ko active IP Right Grant
- 2016-09-12 WO PCT/EP2016/071420 patent/WO2017042382A1/en active Application Filing
- 2016-09-12 EP EP16763821.2A patent/EP3347483A1/en active Pending
- 2016-09-12 BR BR112018004725A patent/BR112018004725A2/pt active Search and Examination
- 2016-09-12 RU RU2018109987A patent/RU2763170C2/ru active
- 2016-09-12 US US15/758,653 patent/US11046985B2/en active Active
- 2016-09-12 JP JP2018512972A patent/JP7069007B2/ja active Active
- 2016-09-12 AU AU2016319304A patent/AU2016319304B2/en active Active
- 2016-09-12 KR KR1020247028456A patent/KR20240134049A/ko active Search and Examination
-
2018
- 2018-02-22 PH PH12018550020A patent/PH12018550020A1/en unknown
- 2018-03-09 MX MX2022003994A patent/MX2022003994A/es unknown
-
2021
- 2021-05-18 US US17/323,737 patent/US12037622B2/en active Active
- 2021-05-18 US US17/323,769 patent/US20210277436A1/en active Pending
-
2022
- 2022-04-28 JP JP2022074228A patent/JP7362831B2/ja active Active
- 2022-04-28 JP JP2022074229A patent/JP2022105119A/ja active Pending
-
2024
- 2024-05-27 JP JP2024085361A patent/JP2024105715A/ja active Pending
- 2024-06-04 US US18/733,787 patent/US20240318216A1/en active Pending
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US11875220B2 (en) * | 2018-09-04 | 2024-01-16 | Tencent Technology (Shenzhen) Company Limited | Method, apparatus, and storage medium for generating network representation for neural network |
Also Published As
| Publication number | Publication date |
|---|---|
| PH12018550020A1 (en) | 2018-08-13 |
| KR20180043297A (ko) | 2018-04-27 |
| JP2018526019A (ja) | 2018-09-13 |
| US20180305724A1 (en) | 2018-10-25 |
| US20240318216A1 (en) | 2024-09-26 |
| RU2018109987A (ru) | 2019-10-16 |
| EP3347483A1 (en) | 2018-07-18 |
| US11046985B2 (en) | 2021-06-29 |
| RU2021135111A (ru) | 2021-12-30 |
| MX2018003037A (es) | 2019-01-10 |
| US20210277436A1 (en) | 2021-09-09 |
| JP7362831B2 (ja) | 2023-10-17 |
| BR112018004725A2 (pt) | 2018-09-25 |
| KR20240134049A (ko) | 2024-09-05 |
| JP2024105715A (ja) | 2024-08-06 |
| RU2018109987A3 (zh) | 2020-01-27 |
| CN108026556A (zh) | 2018-05-11 |
| AU2016319304A1 (en) | 2018-03-08 |
| RU2763170C2 (ru) | 2021-12-28 |
| JP7069007B2 (ja) | 2022-05-17 |
| US12037622B2 (en) | 2024-07-16 |
| JP2022105119A (ja) | 2022-07-12 |
| AU2016319304B2 (en) | 2021-04-29 |
| KR102700050B1 (ko) | 2024-08-29 |
| MX2022003994A (es) | 2022-04-26 |
| WO2017042382A1 (en) | 2017-03-16 |
| US20210277435A1 (en) | 2021-09-09 |
| EP3141610A1 (en) | 2017-03-15 |
| JP2022105118A (ja) | 2022-07-12 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN108026556B (zh) | 在具有经改造的输入/输出的微生物宿主中人乳寡糖的产生 | |
| US12098403B2 (en) | Metabolically engineered organisms for the production of added value bio-products | |
| AU2018296557B2 (en) | Fucosyltransferases and their use in producing fucosylated oligosaccharides | |
| JP2024010049A (ja) | シアリルトランスフェラーゼ及びシアリル化オリゴ糖の生産におけるその使用 | |
| JP7565801B2 (ja) | シアリル化糖の発酵産生 | |
| EP2927316A1 (en) | Total fermentation of oligosaccharides | |
| RU2818835C2 (ru) | Фукозилтрансферазы и их применение для получения фукозилированных олигосахаридов |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PB01 | Publication | ||
| PB01 | Publication | ||
| SE01 | Entry into force of request for substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| CB02 | Change of applicant information |
Address after: Bright Bach, Rhine, Germany Applicant after: Kohansen breast milk oligosaccharides Co.,Ltd. Address before: Bright Bach, Rhine, Germany Applicant before: JENNEWEIN BIOTECHNOLOGIE GmbH |
|
| CB02 | Change of applicant information | ||
| GR01 | Patent grant | ||
| GR01 | Patent grant |