PAS 도메인
PAS domain| PAS 폴드 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
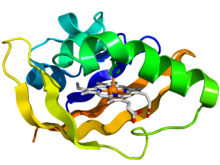 세균산소센서 단백질 fixL의 [1]PAS 도메인의 결정구조.단백질은 무지개색 만화(N-terminus = blue, C-terminus = red)로 표시되고, 헴 배위자는 막대기(탄소 = 흰색, 질소 = blue, 산소 = 적색, 철분 = 오렌지)로 나타난다. | |||||||||||
| 식별자 | |||||||||||
| 기호. | PAS | ||||||||||
| 팜 | PF00989 | ||||||||||
| 인터프로 | IPR013767 | ||||||||||
| 스마트 | PAS | ||||||||||
| 프로 사이트 | PDOC50112 | ||||||||||
| SCOP2 | 2phy/SCOPe/SUPFAM | ||||||||||
| CDD | cd00130 | ||||||||||
| |||||||||||
Per-Arnt-Sim(PAS) 도메인은 모든 생명의 [2]왕국에서 발견되는 단백질 도메인입니다.일반적으로 PAS 도메인은 분자 센서로 작용하며, PAS [3][4][5]도메인의 결합을 통해 작은 분자와 다른 단백질이 결합한다.이러한 감지 능력으로 인해, PAS 도메인은 일주기 클럭의 단백질-단백질 상호작용에 관여하는 주요 구조적 모티프로서 보여졌으며, 또한 시그널링 [6][7]센서로서 기능하는 시그널링 단백질에서 발견되는 일반적인 모티프이기도 하다.
검출
PAS 도메인은 박테리아에서 포유류에 이르는 많은 유기체에서 발견된다.PAS 도메인은 처음 [8]발견된 세 가지 단백질에서 이름을 따왔다.
- Per – 기간 전반 단백질
- Arnt – 아릴 탄화수소 수용체 핵전달체 단백질
- Sim – 일편단심 단백질
PAS 도메인의 최초 발견 이후, 다량의 PAS 도메인 결합 부위가 박테리아와 진핵 생물에서 발견되었다.PAS LOV 단백질이라고 불리는 서브셋은 산소, 빛, [9]전압에 반응합니다.
구조.
PAS 도메인은 어느 정도의 시퀀스 변동성을 나타내지만, PAS 도메인 코어의 3차원 구조는 대체로 [10]보존되어 있다.이 코어는 5가닥 반평행 β-시트와 여러 개의 α-헬리로 구성된다.신호 전달의 결과로서 구조 변화는 주로 β 시트 내에서 발생한다.이러한 신호는 코어의 α-헬리스를 통해 공유 결합 이펙터 [11]도메인으로 전파된다.1998년, PAS 도메인 코어 아키텍처는 할로호도스피라 [10]할로필라의 광활성 황단백질(PYP) 구조에서 처음 특징지어졌습니다.많은 단백질에서, PAS 도메인의 이합체가 필요하며, 한 개는 배위자를 결합하고 다른 하나는 다른 [5]단백질과의 상호작용을 매개한다.
유기체에서의 PAS 예시
기존의 PAS 도메인은 20% 미만의 평균 쌍방향 시퀀스 ID를 공유합니다.즉,[10] 이들 도메인은 놀라울 정도로 다릅니다.PAS 도메인은 다른 환경 감지 메커니즘을 가진 단백질에서 자주 발견된다.또한 많은 PAS 도메인이 광수용 [12]세포에 부착되어 있다.
박테리아
종종 박테리아의 왕국에서 PAS 도메인은 센서 히스티딘 키나아제, 고리형-di-GMP 신타아제 및 가수분해효소, 메틸수용 화학축성 [10]단백질과 같은 시그널링 단백질의 아미노 말단에 위치한다.
뉴로스포라
빛이 비치는 경우 WC-1(White Collar-1) 및 White Collar-2(WC-2)는 PAS 도메인에 의한 조정에 의해 감광되어 [13]FRQ 변환이 활성화됩니다.
드로소필라속
빛이 비추면 CLK와 CYC가 PAS 도메인을 통해 접속되어 PER의 변환이 활성화되고 PER PAS 도메인을 통해 Tim과 관련지어집니다.PER, Tim, CLK, CYC 유전자는 PAS 결합 도메인을 포함합니다.
아라비도시스속
PAS 도메인은 ZTL 및 NPH1 유전자에서 발견된다.이러한 도메인은 Neurospora 일주기 관련 단백질 WC-1에서 [14]발견되는 PAS 도메인과 매우 유사하다.
포유동물
현재 포유류가 이해할 수 있는 일주기 시계는 빛이 BMAL1과 CLK를 활성화하여 PAS 도메인을 통해 결합할 때 시작됩니다.이 액티베이터 콤플렉스는 모두 크립토크롬1 및 2(CRI 1.2 패밀리)에 바인드하기 위해 사용되는PAS 도메인을 가지는 Per1, Per2, 및 Per3 를 규제합니다.다음 포유류의 유전자는 PAS 결합 도메인을 포함합니다: Per1, Per2, Per3, Cry1, Cry2, Bmal, Clk, Pasd1.
기타 포유동물 PAS 역할
포유동물에서는 두 PAS 도메인이 모두 중요한 역할을 합니다.PAS A는 다른 PAS 도메인 단백질과의 단백질-단백질 상호작용을 담당하며, PAS B는 더 다양한 역할을 한다.그것은 샤페로닌과 다이옥신과 같은 다른 작은 분자들과의 상호작용을 매개하지만, 드로소필라 clk 유전자의 상동체인 NPAS2의 PAS B 도메인과 저산소 유도인자(HIF) 또한 리간드 [12]결합을 매개하는 데 도움을 준다.또한 NPAS2 단백질을 포함한 PAS 도메인은 Clock 유전자가 [15]완전히 결여된 돌연변이 생쥐에서 Clock 유전자의 대체물로 나타났다.
PAS 도메인은 BHLH와도 직접 대화합니다.이것은 일반적으로 BHLH 단백질의 C-Terminus에 위치합니다.BHLH 단백질을 포함한 PAS 도메인은 일반적으로 HIF에서 발견되고 인코딩되는 BHLH-Pas 단백질을 형성하며, PAS 도메인과 BHLH 도메인과 Clock [16][17][18]유전자를 모두 필요로 한다.
레퍼런스
- ^ PDB: 1y28;Dunham CM, Dioum EM, Tuckerman JR, Gonzalez G, Scott WG, Gilles-Gonzalez MA (July 2003). "A distal arginine in oxygen-sensing heme-PAS domains is essential to ligand binding, signal transduction, and structure". Biochemistry. 42 (25): 7701–8. doi:10.1021/bi0343370. PMID 12820879.
- ^ Henry, Jonathan T.; Crosson, Sean (1 January 2011). "Ligand-binding PAS domains in a genomic, cellular, and structural context". Annual Review of Microbiology. 65: 261–286. doi:10.1146/annurev-micro-121809-151631. PMC 3298442. PMID 21663441.
- ^ Liu, Yu C.; Machuca, Mayra A.; Beckham, Simone A.; Gunzburg, Menachem J.; Roujeinikova, Anna (1 October 2015). "Structural basis for amino-acid recognition and transmembrane signalling by tandem Per-Arnt-Sim (tandem PAS) chemoreceptor sensory domains". Acta Crystallographica Section D. 71 (10): 2127–2136. doi:10.1107/S139900471501384X. PMID 26457436.
- ^ Möglich, Andreas; Ayers, Rebecca A.; Moffat, Keith (14 October 2009). "Structure and signaling mechanism of Per-ARNT-Sim domains". Structure. 17 (10): 1282–1294. doi:10.1016/j.str.2009.08.011. PMC 3092527. PMID 19836329.
- ^ a b Hennig, Sven; Strauss, Holger M.; Vanselow, Katja; Yildiz, Özkan; Schulze, Sabrina; Arens, Julia; Kramer, Achim; Wolf, Eva (28 April 2009). "Structural and Functional Analyses of PAS Domain Interactions of the Clock Proteins Drosophila PERIOD and Mouse PERIOD2". PLOS Biology. 7 (4): e1000094. doi:10.1371/journal.pbio.1000094. PMC 2671562. PMID 19402751.
- ^ Ponting CP, Aravind L (November 1997). "PAS: a multi-functional domain family comes to light". Curr. Biol. 7 (11): R674–7. doi:10.1016/S0960-9822(06)00352-6. PMID 9382818. S2CID 14105830.
- ^ Hefti MH, Françoijs KJ, de Vries SC, Dixon R, Vervoort J (March 2004). "The PAS fold. A redefinition of the PAS domain based upon structural prediction". Eur. J. Biochem. 271 (6): 1198–208. doi:10.1111/j.1432-1033.2004.04023.x. PMID 15009198.
- ^ Möglich, Andreas; Ayers, Rebecca A.; Moffat, Keith (14 October 2009). "Structure and Signaling Mechanism of Per-ARNT-Sim Domains". Structure. 17 (10): 1282–1294. doi:10.1016/j.str.2009.08.011. PMC 3092527. PMID 19836329.
- ^ Rosato, Ezio; Tauber, Eran; Kyriacou, Charalambos P. (1 January 2006). "Molecular genetics of the fruit-fly circadian clock". European Journal of Human Genetics. 14 (6): 729–738. doi:10.1038/sj.ejhg.5201547. PMID 16721409.
- ^ a b c d Henry, Jonathan T.; Crosson, Sean (1 January 2011). "Ligand-Binding PAS Domains in a Genomic, Cellular, and Structural Context". Annual Review of Microbiology. 65: 261–286. doi:10.1146/annurev-micro-121809-151631. PMC 3298442. PMID 21663441.
- ^ Möglich, A; Ayers, RA; Moffat, K (2009). "Structure and Signaling Mechanism of Per-ARNT-Sim Domains". Structure. 17 (10): 1282–94. doi:10.1016/j.str.2009.08.011. PMC 3092527. PMID 19836329.
- ^ a b McIntosh, Brian; Hogenesch, John; Bradfield, Christopher (2010). "Mammalian Per-Arnt-Sim Proteins in Environmental Adaptation". Annual Review of Physiology. 72: 625–645. doi:10.1146/annurev-physiol-021909-135922. PMID 20148691.
- ^ Harmer, Stacey L.; Panda, Satchidananda; Kay, Steve A. (28 November 2003). "Molecular Bases of Circadian Rhythms". Annual Review of Cell and Developmental Biology. 17: 215–253. doi:10.1146/annurev.cellbio.17.1.215. PMID 11687489.
- ^ Somers, David; Schultz, Thomas; Kay, Steve; Milnamow, Maureen (2000). "ZEITLUPE Encodes a Novel Clock-Associated PAS Protein from Arabidopsis". Cell. 101 (3): 319–329. doi:10.1016/S0092-8674(00)80841-7. PMID 10847686. S2CID 3013788.
- ^ Debruyne JP, Noton E, Lambert CM, Maywood ES, Weaver DR, Reppert SM (May 2006). "A clock shock: mouse CLOCK is not required for circadian oscillator function". Neuron. 50 (3): 465–77. doi:10.1016/j.neuron.2006.03.041. PMID 16675400. S2CID 19028601.
- ^ Jones, Susan (1 January 2004). "An overview of the basic helix-loop-helix proteins". Genome Biology. 5 (6): 226. doi:10.1186/gb-2004-5-6-226. PMC 463060. PMID 15186484.
- ^ Ke, Qingdong; Costa, Max (1 November 2006). "Hypoxia-Inducible Factor-1 (HIF-1)". Molecular Pharmacology. 70 (5): 1469–1480. doi:10.1124/mol.106.027029. PMID 16887934. S2CID 2522614.
- ^ Wang, G. L.; Jiang, B. H.; Rue, E. A.; Semenza, G. L. (6 June 1995). "Hypoxia-inducible factor 1 is a basic-helix-loop-helix-PAS heterodimer regulated by cellular O2 tension". Proceedings of the National Academy of Sciences of the United States of America. 92 (12): 5510–5514. Bibcode:1995PNAS...92.5510W. doi:10.1073/pnas.92.12.5510. PMC 41725. PMID 7539918.


