KR20240007760A - 항-icos 항체의 용도 - Google Patents
항-icos 항체의 용도 Download PDFInfo
- Publication number
- KR20240007760A KR20240007760A KR1020237043292A KR20237043292A KR20240007760A KR 20240007760 A KR20240007760 A KR 20240007760A KR 1020237043292 A KR1020237043292 A KR 1020237043292A KR 20237043292 A KR20237043292 A KR 20237043292A KR 20240007760 A KR20240007760 A KR 20240007760A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- amino acid
- acid sequence
- antibody
- sequence seq
- Prior art date
Links
- 206010028980 Neoplasm Diseases 0.000 claims abstract description 227
- 230000027455 binding Effects 0.000 claims abstract description 201
- 239000000427 antigen Substances 0.000 claims abstract description 182
- 108091007433 antigens Proteins 0.000 claims abstract description 180
- 102000036639 antigens Human genes 0.000 claims abstract description 180
- 239000012634 fragment Substances 0.000 claims abstract description 123
- 210000003289 regulatory T cell Anatomy 0.000 claims abstract description 109
- 201000011510 cancer Diseases 0.000 claims abstract description 87
- 210000003162 effector t lymphocyte Anatomy 0.000 claims abstract description 49
- 125000003275 alpha amino acid group Chemical group 0.000 claims description 753
- 238000011282 treatment Methods 0.000 claims description 157
- 241000282414 Homo sapiens Species 0.000 claims description 148
- 238000000034 method Methods 0.000 claims description 146
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 57
- 101001042104 Homo sapiens Inducible T-cell costimulator Proteins 0.000 claims description 55
- 201000010099 disease Diseases 0.000 claims description 54
- 229960003852 atezolizumab Drugs 0.000 claims description 45
- 239000003814 drug Substances 0.000 claims description 45
- 230000004044 response Effects 0.000 claims description 44
- 230000001965 increasing effect Effects 0.000 claims description 43
- 101100179075 Mus musculus Icos gene Proteins 0.000 claims description 30
- 229940124597 therapeutic agent Drugs 0.000 claims description 28
- 108010047041 Complementarity Determining Regions Proteins 0.000 claims description 26
- 208000000102 Squamous Cell Carcinoma of Head and Neck Diseases 0.000 claims description 17
- 201000000459 head and neck squamous cell carcinoma Diseases 0.000 claims description 17
- 201000001441 melanoma Diseases 0.000 claims description 16
- 230000000779 depleting effect Effects 0.000 claims description 15
- 208000006265 Renal cell carcinoma Diseases 0.000 claims description 8
- 208000002154 non-small cell lung carcinoma Diseases 0.000 claims description 8
- 206010008342 Cervix carcinoma Diseases 0.000 claims description 6
- 206010061902 Pancreatic neoplasm Diseases 0.000 claims description 6
- 208000005718 Stomach Neoplasms Diseases 0.000 claims description 6
- 208000003721 Triple Negative Breast Neoplasms Diseases 0.000 claims description 6
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 claims description 6
- 201000010881 cervical cancer Diseases 0.000 claims description 6
- 206010017758 gastric cancer Diseases 0.000 claims description 6
- 206010073071 hepatocellular carcinoma Diseases 0.000 claims description 6
- 231100000844 hepatocellular carcinoma Toxicity 0.000 claims description 6
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 claims description 6
- 201000002528 pancreatic cancer Diseases 0.000 claims description 6
- 208000008443 pancreatic carcinoma Diseases 0.000 claims description 6
- 201000011549 stomach cancer Diseases 0.000 claims description 6
- 208000022679 triple-negative breast carcinoma Diseases 0.000 claims description 6
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 claims description 6
- 206010030155 Oesophageal carcinoma Diseases 0.000 claims description 5
- 230000003442 weekly effect Effects 0.000 claims description 5
- 208000000461 Esophageal Neoplasms Diseases 0.000 claims description 4
- 208000032271 Malignant tumor of penis Diseases 0.000 claims description 4
- 208000002471 Penile Neoplasms Diseases 0.000 claims description 4
- 206010034299 Penile cancer Diseases 0.000 claims description 4
- 201000004101 esophageal cancer Diseases 0.000 claims description 4
- 208000037819 metastatic cancer Diseases 0.000 claims description 4
- 208000011575 metastatic malignant neoplasm Diseases 0.000 claims description 4
- 230000001225 therapeutic effect Effects 0.000 abstract description 46
- 238000009097 single-agent therapy Methods 0.000 abstract description 26
- 238000002648 combination therapy Methods 0.000 abstract description 20
- 210000000987 immune system Anatomy 0.000 abstract description 6
- 230000001105 regulatory effect Effects 0.000 abstract description 6
- 102000053646 Inducible T-Cell Co-Stimulator Human genes 0.000 description 246
- 108700013161 Inducible T-Cell Co-Stimulator Proteins 0.000 description 246
- 210000004027 cell Anatomy 0.000 description 177
- 150000007523 nucleic acids Chemical group 0.000 description 164
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 121
- 108091028043 Nucleic acid sequence Proteins 0.000 description 114
- 230000014509 gene expression Effects 0.000 description 85
- 235000001014 amino acid Nutrition 0.000 description 69
- 241001465754 Metazoa Species 0.000 description 68
- 239000012636 effector Substances 0.000 description 63
- 230000000694 effects Effects 0.000 description 63
- 229940024606 amino acid Drugs 0.000 description 58
- 102000005962 receptors Human genes 0.000 description 58
- 108020003175 receptors Proteins 0.000 description 58
- 150000001413 amino acids Chemical class 0.000 description 57
- 210000001744 T-lymphocyte Anatomy 0.000 description 56
- 102000039446 nucleic acids Human genes 0.000 description 54
- 108020004707 nucleic acids Proteins 0.000 description 54
- 238000003556 assay Methods 0.000 description 50
- 238000002560 therapeutic procedure Methods 0.000 description 49
- 235000014966 Eragrostis abyssinica Nutrition 0.000 description 47
- 239000000203 mixture Substances 0.000 description 47
- 102000043396 human ICOS Human genes 0.000 description 42
- 230000006870 function Effects 0.000 description 41
- 108090000623 proteins and genes Proteins 0.000 description 41
- 101000716102 Homo sapiens T-cell surface glycoprotein CD4 Proteins 0.000 description 38
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 description 38
- 239000003795 chemical substances by application Substances 0.000 description 37
- 230000035772 mutation Effects 0.000 description 36
- 108090000765 processed proteins & peptides Proteins 0.000 description 36
- 238000006467 substitution reaction Methods 0.000 description 35
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 31
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 description 31
- 238000012360 testing method Methods 0.000 description 30
- 239000002773 nucleotide Substances 0.000 description 29
- 125000003729 nucleotide group Chemical group 0.000 description 29
- 210000004881 tumor cell Anatomy 0.000 description 29
- 241000699670 Mus sp. Species 0.000 description 28
- 230000008901 benefit Effects 0.000 description 27
- 241000699666 Mus <mouse, genus> Species 0.000 description 26
- 229920001184 polypeptide Polymers 0.000 description 25
- 102000004196 processed proteins & peptides Human genes 0.000 description 25
- 102100034980 ICOS ligand Human genes 0.000 description 24
- 238000004519 manufacturing process Methods 0.000 description 24
- 101710093458 ICOS ligand Proteins 0.000 description 23
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 23
- 230000000259 anti-tumor effect Effects 0.000 description 22
- 230000005888 antibody-dependent cellular phagocytosis Effects 0.000 description 22
- 230000036961 partial effect Effects 0.000 description 22
- 210000001519 tissue Anatomy 0.000 description 22
- 230000005867 T cell response Effects 0.000 description 20
- 102000004169 proteins and genes Human genes 0.000 description 20
- 239000003446 ligand Substances 0.000 description 18
- 235000018102 proteins Nutrition 0.000 description 18
- 102000004127 Cytokines Human genes 0.000 description 17
- 108090000695 Cytokines Proteins 0.000 description 17
- 102100027581 Forkhead box protein P3 Human genes 0.000 description 17
- 101000861452 Homo sapiens Forkhead box protein P3 Proteins 0.000 description 17
- 239000000556 agonist Substances 0.000 description 17
- 230000000875 corresponding effect Effects 0.000 description 17
- 238000001727 in vivo Methods 0.000 description 17
- 241000894007 species Species 0.000 description 17
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 16
- 238000000338 in vitro Methods 0.000 description 16
- 239000008194 pharmaceutical composition Substances 0.000 description 16
- 108010074708 B7-H1 Antigen Proteins 0.000 description 15
- 102000008096 B7-H1 Antigen Human genes 0.000 description 15
- 230000006044 T cell activation Effects 0.000 description 15
- 210000003719 b-lymphocyte Anatomy 0.000 description 15
- 230000001419 dependent effect Effects 0.000 description 15
- 238000002347 injection Methods 0.000 description 15
- 239000007924 injection Substances 0.000 description 15
- 241000124008 Mammalia Species 0.000 description 14
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 14
- 102000000852 Tumor Necrosis Factor-alpha Human genes 0.000 description 14
- 230000008484 agonism Effects 0.000 description 14
- 238000001574 biopsy Methods 0.000 description 14
- 210000004369 blood Anatomy 0.000 description 14
- 239000008280 blood Substances 0.000 description 14
- 230000037396 body weight Effects 0.000 description 14
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 14
- 230000002601 intratumoral effect Effects 0.000 description 14
- 108010087819 Fc receptors Proteins 0.000 description 13
- 102000009109 Fc receptors Human genes 0.000 description 13
- 108060003951 Immunoglobulin Proteins 0.000 description 13
- 229940079593 drug Drugs 0.000 description 13
- 238000009472 formulation Methods 0.000 description 13
- 230000028993 immune response Effects 0.000 description 13
- 102000018358 immunoglobulin Human genes 0.000 description 13
- 230000001976 improved effect Effects 0.000 description 13
- 230000006698 induction Effects 0.000 description 13
- 239000000523 sample Substances 0.000 description 13
- 241000700159 Rattus Species 0.000 description 12
- 230000004913 activation Effects 0.000 description 12
- 238000012217 deletion Methods 0.000 description 12
- 230000037430 deletion Effects 0.000 description 12
- 210000004602 germ cell Anatomy 0.000 description 12
- 239000011780 sodium chloride Substances 0.000 description 12
- 230000009261 transgenic effect Effects 0.000 description 12
- 241000282412 Homo Species 0.000 description 11
- 230000001900 immune effect Effects 0.000 description 11
- 208000015181 infectious disease Diseases 0.000 description 11
- 230000003472 neutralizing effect Effects 0.000 description 11
- 230000004614 tumor growth Effects 0.000 description 11
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 11
- 125000000539 amino acid group Chemical group 0.000 description 10
- 230000030833 cell death Effects 0.000 description 10
- 210000002865 immune cell Anatomy 0.000 description 10
- 230000001506 immunosuppresive effect Effects 0.000 description 10
- 238000007920 subcutaneous administration Methods 0.000 description 10
- 230000004083 survival effect Effects 0.000 description 10
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 description 9
- -1 IFNγ Proteins 0.000 description 9
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 9
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 9
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 description 9
- 230000002411 adverse Effects 0.000 description 9
- 239000011324 bead Substances 0.000 description 9
- 239000000872 buffer Substances 0.000 description 9
- 230000008859 change Effects 0.000 description 9
- 238000003780 insertion Methods 0.000 description 9
- 230000037431 insertion Effects 0.000 description 9
- 230000001404 mediated effect Effects 0.000 description 9
- 230000004048 modification Effects 0.000 description 9
- 238000012986 modification Methods 0.000 description 9
- 230000003285 pharmacodynamic effect Effects 0.000 description 9
- 229940002612 prodrug Drugs 0.000 description 9
- 239000000651 prodrug Substances 0.000 description 9
- 230000002829 reductive effect Effects 0.000 description 9
- 239000002904 solvent Substances 0.000 description 9
- 230000000638 stimulation Effects 0.000 description 9
- 230000001052 transient effect Effects 0.000 description 9
- 238000002255 vaccination Methods 0.000 description 9
- 241000725303 Human immunodeficiency virus Species 0.000 description 8
- 238000007792 addition Methods 0.000 description 8
- 230000001270 agonistic effect Effects 0.000 description 8
- 238000013459 approach Methods 0.000 description 8
- 230000009286 beneficial effect Effects 0.000 description 8
- 239000006285 cell suspension Substances 0.000 description 8
- 238000002868 homogeneous time resolved fluorescence Methods 0.000 description 8
- 230000036210 malignancy Effects 0.000 description 8
- 102000040430 polynucleotide Human genes 0.000 description 8
- 108091033319 polynucleotide Proteins 0.000 description 8
- 239000002157 polynucleotide Substances 0.000 description 8
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 8
- 208000035473 Communicable disease Diseases 0.000 description 7
- 108020004414 DNA Proteins 0.000 description 7
- 102100040678 Programmed cell death protein 1 Human genes 0.000 description 7
- 230000006023 anti-tumor response Effects 0.000 description 7
- 230000022534 cell killing Effects 0.000 description 7
- 230000001413 cellular effect Effects 0.000 description 7
- 230000009260 cross reactivity Effects 0.000 description 7
- 210000004408 hybridoma Anatomy 0.000 description 7
- 229940127130 immunocytokine Drugs 0.000 description 7
- 229960005386 ipilimumab Drugs 0.000 description 7
- 230000001394 metastastic effect Effects 0.000 description 7
- 206010061289 metastatic neoplasm Diseases 0.000 description 7
- 244000052769 pathogen Species 0.000 description 7
- 239000000546 pharmaceutical excipient Substances 0.000 description 7
- 238000002360 preparation method Methods 0.000 description 7
- 230000009467 reduction Effects 0.000 description 7
- 208000024891 symptom Diseases 0.000 description 7
- 229960005486 vaccine Drugs 0.000 description 7
- 102100022005 B-lymphocyte antigen CD20 Human genes 0.000 description 6
- 238000012413 Fluorescence activated cell sorting analysis Methods 0.000 description 6
- 108010017213 Granulocyte-Macrophage Colony-Stimulating Factor Proteins 0.000 description 6
- 241000711549 Hepacivirus C Species 0.000 description 6
- 101000897405 Homo sapiens B-lymphocyte antigen CD20 Proteins 0.000 description 6
- 101001023379 Homo sapiens Lysosome-associated membrane glycoprotein 1 Proteins 0.000 description 6
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 6
- 241000701806 Human papillomavirus Species 0.000 description 6
- 102100035133 Lysosome-associated membrane glycoprotein 1 Human genes 0.000 description 6
- 206010027476 Metastases Diseases 0.000 description 6
- 101710089372 Programmed cell death protein 1 Proteins 0.000 description 6
- 230000009471 action Effects 0.000 description 6
- 210000000612 antigen-presenting cell Anatomy 0.000 description 6
- 230000001472 cytotoxic effect Effects 0.000 description 6
- 206010012818 diffuse large B-cell lymphoma Diseases 0.000 description 6
- 238000010494 dissociation reaction Methods 0.000 description 6
- 230000005593 dissociations Effects 0.000 description 6
- 238000005516 engineering process Methods 0.000 description 6
- 238000002474 experimental method Methods 0.000 description 6
- 230000002519 immonomodulatory effect Effects 0.000 description 6
- 230000005764 inhibitory process Effects 0.000 description 6
- 239000000463 material Substances 0.000 description 6
- 230000007246 mechanism Effects 0.000 description 6
- 230000004660 morphological change Effects 0.000 description 6
- 239000013642 negative control Substances 0.000 description 6
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 6
- 230000003389 potentiating effect Effects 0.000 description 6
- 230000000770 proinflammatory effect Effects 0.000 description 6
- 238000001959 radiotherapy Methods 0.000 description 6
- 230000004936 stimulating effect Effects 0.000 description 6
- 229910052717 sulfur Inorganic materials 0.000 description 6
- 238000011269 treatment regimen Methods 0.000 description 6
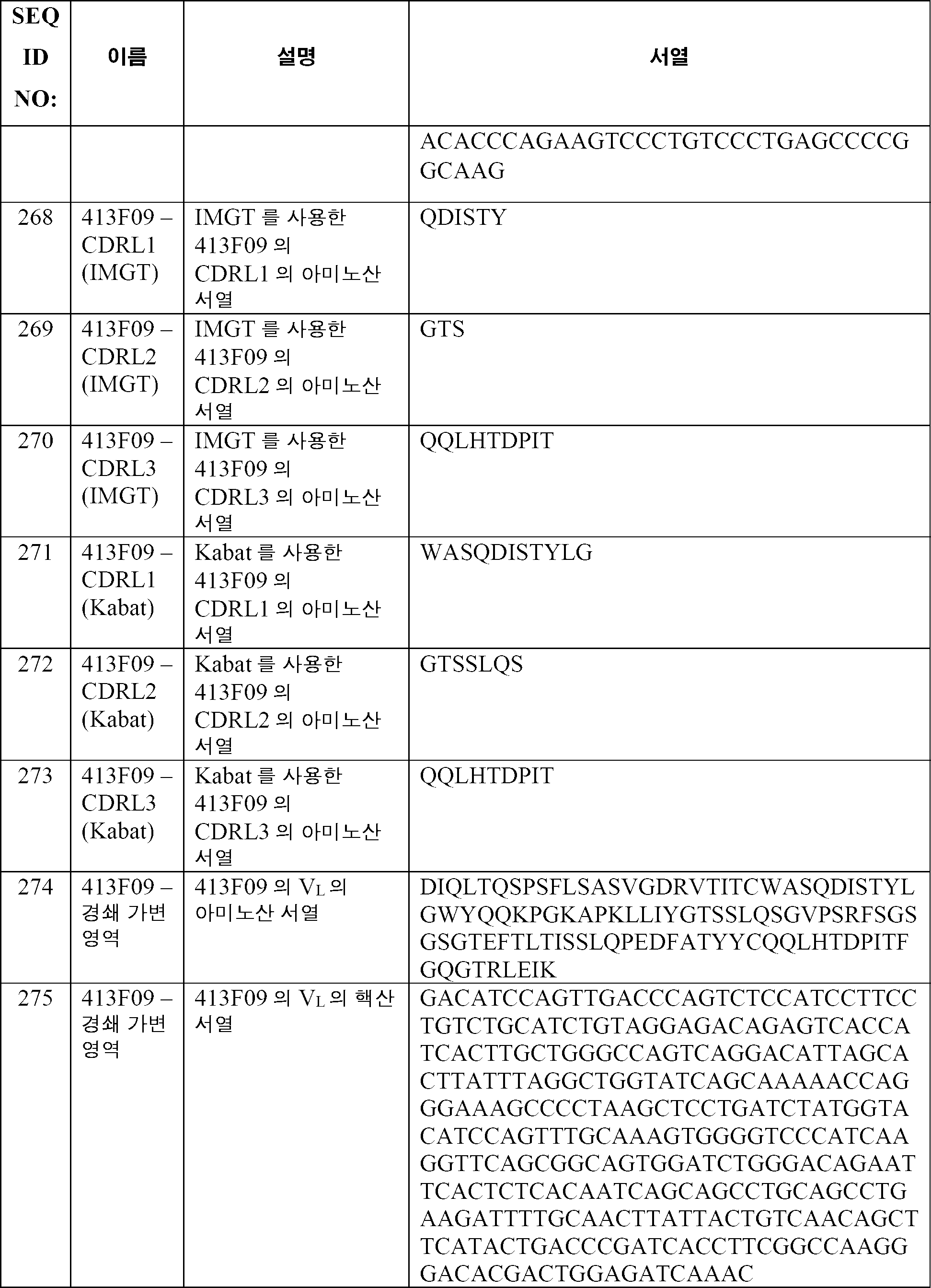
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 5
- 208000003950 B-cell lymphoma Diseases 0.000 description 5
- 102100039620 Granulocyte-macrophage colony-stimulating factor Human genes 0.000 description 5
- 101001117317 Homo sapiens Programmed cell death 1 ligand 1 Proteins 0.000 description 5
- 208000031671 Large B-Cell Diffuse Lymphoma Diseases 0.000 description 5
- 206010035226 Plasma cell myeloma Diseases 0.000 description 5
- 239000002671 adjuvant Substances 0.000 description 5
- 239000000611 antibody drug conjugate Substances 0.000 description 5
- 238000009175 antibody therapy Methods 0.000 description 5
- 229940049595 antibody-drug conjugate Drugs 0.000 description 5
- 230000015572 biosynthetic process Effects 0.000 description 5
- 210000003169 central nervous system Anatomy 0.000 description 5
- 238000012512 characterization method Methods 0.000 description 5
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 5
- 230000004940 costimulation Effects 0.000 description 5
- 229940127089 cytotoxic agent Drugs 0.000 description 5
- 239000002254 cytotoxic agent Substances 0.000 description 5
- 230000007423 decrease Effects 0.000 description 5
- 239000002552 dosage form Substances 0.000 description 5
- 230000009977 dual effect Effects 0.000 description 5
- 230000004927 fusion Effects 0.000 description 5
- 238000003364 immunohistochemistry Methods 0.000 description 5
- 239000002955 immunomodulating agent Substances 0.000 description 5
- 229940121354 immunomodulator Drugs 0.000 description 5
- 238000009169 immunotherapy Methods 0.000 description 5
- 230000006872 improvement Effects 0.000 description 5
- 230000003993 interaction Effects 0.000 description 5
- 238000011813 knockout mouse model Methods 0.000 description 5
- 230000003902 lesion Effects 0.000 description 5
- 230000007774 longterm Effects 0.000 description 5
- 239000003550 marker Substances 0.000 description 5
- 238000005259 measurement Methods 0.000 description 5
- 201000000050 myeloid neoplasm Diseases 0.000 description 5
- 230000001737 promoting effect Effects 0.000 description 5
- 239000007787 solid Substances 0.000 description 5
- 210000000952 spleen Anatomy 0.000 description 5
- 239000006228 supernatant Substances 0.000 description 5
- 230000002459 sustained effect Effects 0.000 description 5
- 230000009885 systemic effect Effects 0.000 description 5
- MZOFCQQQCNRIBI-VMXHOPILSA-N (3s)-4-[[(2s)-1-[[(2s)-1-[[(1s)-1-carboxy-2-hydroxyethyl]amino]-4-methyl-1-oxopentan-2-yl]amino]-5-(diaminomethylideneamino)-1-oxopentan-2-yl]amino]-3-[[2-[[(2s)-2,6-diaminohexanoyl]amino]acetyl]amino]-4-oxobutanoic acid Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCCN MZOFCQQQCNRIBI-VMXHOPILSA-N 0.000 description 4
- 238000011725 BALB/c mouse Methods 0.000 description 4
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 4
- 102000008203 CTLA-4 Antigen Human genes 0.000 description 4
- 229940045513 CTLA4 antagonist Drugs 0.000 description 4
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 4
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 4
- 101001019455 Homo sapiens ICOS ligand Proteins 0.000 description 4
- 108010074328 Interferon-gamma Proteins 0.000 description 4
- 206010027480 Metastatic malignant melanoma Diseases 0.000 description 4
- 102100024216 Programmed cell death 1 ligand 1 Human genes 0.000 description 4
- 239000004480 active ingredient Substances 0.000 description 4
- 230000004075 alteration Effects 0.000 description 4
- 239000000562 conjugate Substances 0.000 description 4
- 238000013270 controlled release Methods 0.000 description 4
- 230000016396 cytokine production Effects 0.000 description 4
- 231100000433 cytotoxic Toxicity 0.000 description 4
- 210000003743 erythrocyte Anatomy 0.000 description 4
- 210000003527 eukaryotic cell Anatomy 0.000 description 4
- 238000011156 evaluation Methods 0.000 description 4
- 230000007717 exclusion Effects 0.000 description 4
- 230000013595 glycosylation Effects 0.000 description 4
- 238000006206 glycosylation reaction Methods 0.000 description 4
- 238000002513 implantation Methods 0.000 description 4
- 238000001802 infusion Methods 0.000 description 4
- 239000003112 inhibitor Substances 0.000 description 4
- 238000007912 intraperitoneal administration Methods 0.000 description 4
- 238000001990 intravenous administration Methods 0.000 description 4
- 230000002147 killing effect Effects 0.000 description 4
- 210000001165 lymph node Anatomy 0.000 description 4
- 239000002609 medium Substances 0.000 description 4
- 108020004999 messenger RNA Proteins 0.000 description 4
- 208000021039 metastatic melanoma Diseases 0.000 description 4
- 229960002621 pembrolizumab Drugs 0.000 description 4
- 229920001223 polyethylene glycol Polymers 0.000 description 4
- 229910052700 potassium Inorganic materials 0.000 description 4
- 229960004641 rituximab Drugs 0.000 description 4
- 102200072304 rs1057519530 Human genes 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- 239000000758 substrate Substances 0.000 description 4
- 208000002918 testicular germ cell tumor Diseases 0.000 description 4
- 238000002054 transplantation Methods 0.000 description 4
- 208000024827 Alzheimer disease Diseases 0.000 description 3
- 241000239290 Araneae Species 0.000 description 3
- 208000023275 Autoimmune disease Diseases 0.000 description 3
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 3
- 206010006187 Breast cancer Diseases 0.000 description 3
- 208000026310 Breast neoplasm Diseases 0.000 description 3
- 206010061818 Disease progression Diseases 0.000 description 3
- 101100043696 Drosophila melanogaster Stim gene Proteins 0.000 description 3
- 102000004190 Enzymes Human genes 0.000 description 3
- 108090000790 Enzymes Proteins 0.000 description 3
- 108010021472 Fc gamma receptor IIB Proteins 0.000 description 3
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 3
- 241000700721 Hepatitis B virus Species 0.000 description 3
- 208000017604 Hodgkin disease Diseases 0.000 description 3
- 208000021519 Hodgkin lymphoma Diseases 0.000 description 3
- 208000010747 Hodgkins lymphoma Diseases 0.000 description 3
- 101001057504 Homo sapiens Interferon-stimulated gene 20 kDa protein Proteins 0.000 description 3
- 101001055144 Homo sapiens Interleukin-2 receptor subunit alpha Proteins 0.000 description 3
- 101000738771 Homo sapiens Receptor-type tyrosine-protein phosphatase C Proteins 0.000 description 3
- 102000037982 Immune checkpoint proteins Human genes 0.000 description 3
- 108091008036 Immune checkpoint proteins Proteins 0.000 description 3
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 3
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 3
- 206010062016 Immunosuppression Diseases 0.000 description 3
- 102100026878 Interleukin-2 receptor subunit alpha Human genes 0.000 description 3
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical group C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 3
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 3
- 102100029205 Low affinity immunoglobulin gamma Fc region receptor II-b Human genes 0.000 description 3
- 241000282567 Macaca fascicularis Species 0.000 description 3
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 3
- 206010033128 Ovarian cancer Diseases 0.000 description 3
- 206010061535 Ovarian neoplasm Diseases 0.000 description 3
- 208000037581 Persistent Infection Diseases 0.000 description 3
- DNIAPMSPPWPWGF-UHFFFAOYSA-N Propylene glycol Chemical compound CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 3
- 102100037422 Receptor-type tyrosine-protein phosphatase C Human genes 0.000 description 3
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 3
- 108010088160 Staphylococcal Protein A Proteins 0.000 description 3
- 210000000662 T-lymphocyte subset Anatomy 0.000 description 3
- 241000700605 Viruses Species 0.000 description 3
- 235000004279 alanine Nutrition 0.000 description 3
- 230000001580 bacterial effect Effects 0.000 description 3
- BVKZGUZCCUSVTD-UHFFFAOYSA-N carbonic acid Chemical compound OC(O)=O BVKZGUZCCUSVTD-UHFFFAOYSA-N 0.000 description 3
- 230000003915 cell function Effects 0.000 description 3
- 230000003833 cell viability Effects 0.000 description 3
- 201000006612 cervical squamous cell carcinoma Diseases 0.000 description 3
- 238000006243 chemical reaction Methods 0.000 description 3
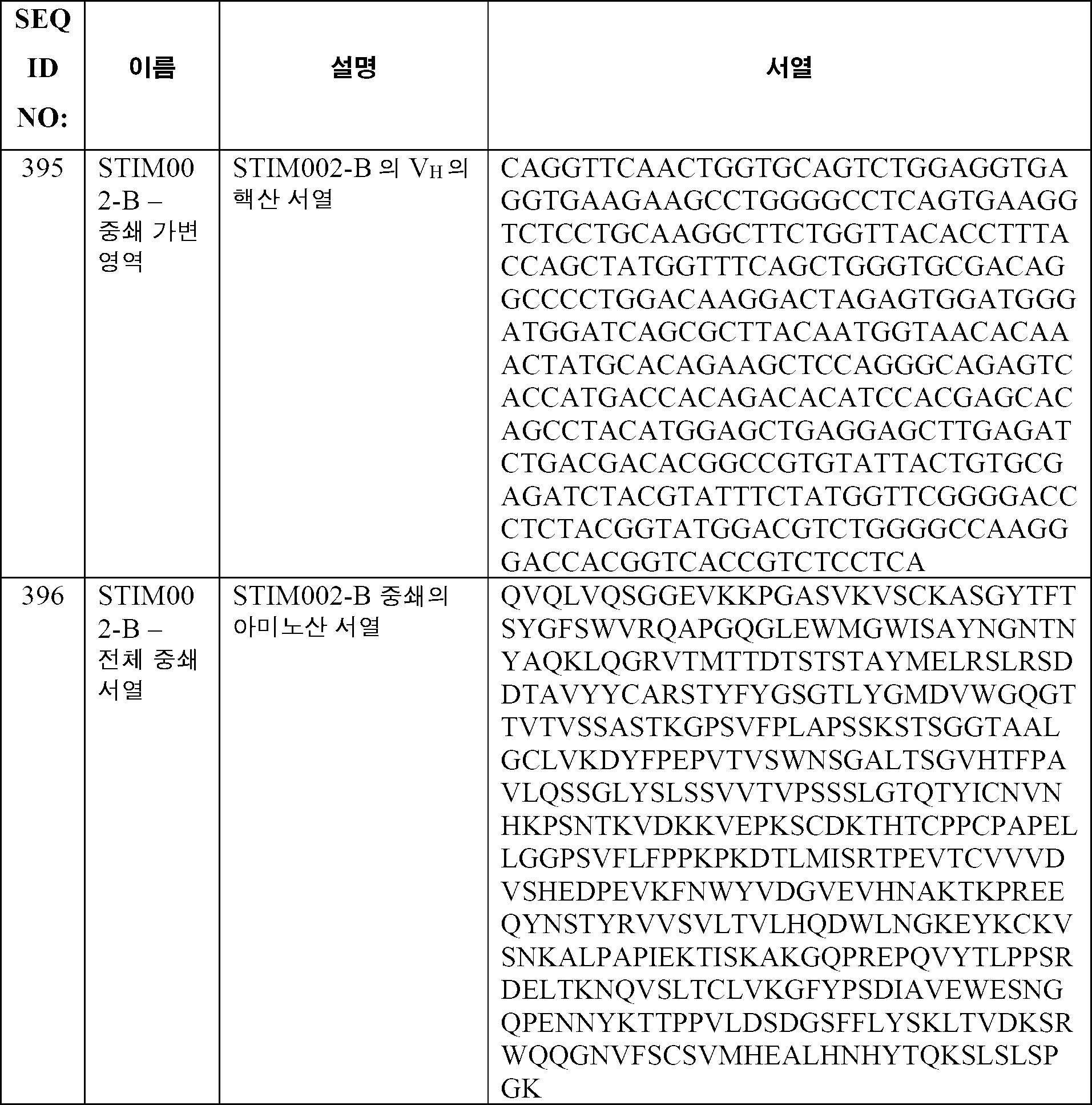
- 208000029742 colonic neoplasm Diseases 0.000 description 3
- 230000000295 complement effect Effects 0.000 description 3
- 231100000599 cytotoxic agent Toxicity 0.000 description 3
- 210000004443 dendritic cell Anatomy 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 230000018109 developmental process Effects 0.000 description 3
- 239000000539 dimer Substances 0.000 description 3
- 230000005750 disease progression Effects 0.000 description 3
- 208000035475 disorder Diseases 0.000 description 3
- 231100000673 dose–response relationship Toxicity 0.000 description 3
- 229940088598 enzyme Drugs 0.000 description 3
- 230000002349 favourable effect Effects 0.000 description 3
- 239000012894 fetal calf serum Substances 0.000 description 3
- 229910052731 fluorine Inorganic materials 0.000 description 3
- 108010037389 glutamyl-cysteinyl-lysine Proteins 0.000 description 3
- 201000010536 head and neck cancer Diseases 0.000 description 3
- 208000014829 head and neck neoplasm Diseases 0.000 description 3
- 108010045383 histidyl-glycyl-glutamic acid Proteins 0.000 description 3
- 230000003053 immunization Effects 0.000 description 3
- 229940072221 immunoglobulins Drugs 0.000 description 3
- 239000012678 infectious agent Substances 0.000 description 3
- 230000010354 integration Effects 0.000 description 3
- 238000010212 intracellular staining Methods 0.000 description 3
- 108010060857 isoleucyl-valyl-tyrosine Proteins 0.000 description 3
- 150000002632 lipids Chemical class 0.000 description 3
- 239000002502 liposome Substances 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 210000004698 lymphocyte Anatomy 0.000 description 3
- 229960005558 mertansine Drugs 0.000 description 3
- 230000001717 pathogenic effect Effects 0.000 description 3
- 229910052698 phosphorus Inorganic materials 0.000 description 3
- 239000013612 plasmid Substances 0.000 description 3
- 208000037920 primary disease Diseases 0.000 description 3
- 230000002035 prolonged effect Effects 0.000 description 3
- 238000000159 protein binding assay Methods 0.000 description 3
- 238000011002 quantification Methods 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 210000002966 serum Anatomy 0.000 description 3
- 150000003431 steroids Chemical class 0.000 description 3
- 230000008685 targeting Effects 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- 238000005303 weighing Methods 0.000 description 3
- NHBKXEKEPDILRR-UHFFFAOYSA-N 2,3-bis(butanoylsulfanyl)propyl butanoate Chemical compound CCCC(=O)OCC(SC(=O)CCC)CSC(=O)CCC NHBKXEKEPDILRR-UHFFFAOYSA-N 0.000 description 2
- 208000031261 Acute myeloid leukaemia Diseases 0.000 description 2
- 208000010507 Adenocarcinoma of Lung Diseases 0.000 description 2
- WUHJHHGYVVJMQE-BJDJZHNGSA-N Ala-Leu-Ile Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WUHJHHGYVVJMQE-BJDJZHNGSA-N 0.000 description 2
- DHONNEYAZPNGSG-UBHSHLNASA-N Ala-Val-Phe Chemical compound C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 DHONNEYAZPNGSG-UBHSHLNASA-N 0.000 description 2
- 102100036475 Alanine aminotransferase 1 Human genes 0.000 description 2
- 108010082126 Alanine transaminase Proteins 0.000 description 2
- 108010088751 Albumins Proteins 0.000 description 2
- 102000009027 Albumins Human genes 0.000 description 2
- 108010032595 Antibody Binding Sites Proteins 0.000 description 2
- QQEWINYJRFBLNN-DLOVCJGASA-N Asn-Ala-Phe Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 QQEWINYJRFBLNN-DLOVCJGASA-N 0.000 description 2
- MYOHQBFRJQFIDZ-KKUMJFAQSA-N Asp-Leu-Tyr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O MYOHQBFRJQFIDZ-KKUMJFAQSA-N 0.000 description 2
- XWSIYTYNLKCLJB-CIUDSAMLSA-N Asp-Lys-Asn Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O XWSIYTYNLKCLJB-CIUDSAMLSA-N 0.000 description 2
- 108010003415 Aspartate Aminotransferases Proteins 0.000 description 2
- 102000004625 Aspartate Aminotransferases Human genes 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- BPYKTIZUTYGOLE-IFADSCNNSA-N Bilirubin Chemical compound N1C(=O)C(C)=C(C=C)\C1=C\C1=C(C)C(CCC(O)=O)=C(CC2=C(C(C)=C(\C=C/3C(=C(C=C)C(=O)N\3)C)N2)CCC(O)=O)N1 BPYKTIZUTYGOLE-IFADSCNNSA-N 0.000 description 2
- 206010005003 Bladder cancer Diseases 0.000 description 2
- 101150100936 CD28 gene Proteins 0.000 description 2
- 210000001266 CD8-positive T-lymphocyte Anatomy 0.000 description 2
- 241000283707 Capra Species 0.000 description 2
- 208000020446 Cardiac disease Diseases 0.000 description 2
- 102000014914 Carrier Proteins Human genes 0.000 description 2
- 102000005600 Cathepsins Human genes 0.000 description 2
- 108010084457 Cathepsins Proteins 0.000 description 2
- 208000006545 Chronic Obstructive Pulmonary Disease Diseases 0.000 description 2
- 206010009944 Colon cancer Diseases 0.000 description 2
- 101150097493 D gene Proteins 0.000 description 2
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 2
- 241000196324 Embryophyta Species 0.000 description 2
- 208000032320 Germ cell tumor of testis Diseases 0.000 description 2
- CYTSBCIIEHUPDU-ACZMJKKPSA-N Gln-Asp-Ala Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(O)=O CYTSBCIIEHUPDU-ACZMJKKPSA-N 0.000 description 2
- ISXJHXGYMJKXOI-GUBZILKMSA-N Glu-Cys-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CS)NC(=O)[C@@H](N)CCC(O)=O ISXJHXGYMJKXOI-GUBZILKMSA-N 0.000 description 2
- QQLBPVKLJBAXBS-FXQIFTODSA-N Glu-Glu-Asn Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O QQLBPVKLJBAXBS-FXQIFTODSA-N 0.000 description 2
- UJMNFCAHLYKWOZ-DCAQKATOSA-N Glu-Lys-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O UJMNFCAHLYKWOZ-DCAQKATOSA-N 0.000 description 2
- JHSRJMUJOGLIHK-GUBZILKMSA-N Glu-Met-Glu Chemical compound CSCC[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@H](CCC(=O)O)N JHSRJMUJOGLIHK-GUBZILKMSA-N 0.000 description 2
- IALQAMYQJBZNSK-WHFBIAKZSA-N Gly-Ser-Asn Chemical compound [H]NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O IALQAMYQJBZNSK-WHFBIAKZSA-N 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- YAALVYQFVJNXIV-KKUMJFAQSA-N His-Leu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC1=CN=CN1 YAALVYQFVJNXIV-KKUMJFAQSA-N 0.000 description 2
- BRQKGRLDDDQWQJ-MBLNEYKQSA-N His-Thr-Ala Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O BRQKGRLDDDQWQJ-MBLNEYKQSA-N 0.000 description 2
- 101000917826 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-a Proteins 0.000 description 2
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 2
- 101000946843 Homo sapiens T-cell surface glycoprotein CD8 alpha chain Proteins 0.000 description 2
- 108010073807 IgG Receptors Proteins 0.000 description 2
- 102000009490 IgG Receptors Human genes 0.000 description 2
- 102100026120 IgG receptor FcRn large subunit p51 Human genes 0.000 description 2
- 101710177940 IgG receptor FcRn large subunit p51 Proteins 0.000 description 2
- BALLIXFZYSECCF-QEWYBTABSA-N Ile-Gln-Phe Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N BALLIXFZYSECCF-QEWYBTABSA-N 0.000 description 2
- RENBRDSDKPSRIH-HJWJTTGWSA-N Ile-Phe-Met Chemical compound N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCSC)C(=O)O RENBRDSDKPSRIH-HJWJTTGWSA-N 0.000 description 2
- YCKPUHHMCFSUMD-IUKAMOBKSA-N Ile-Thr-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N YCKPUHHMCFSUMD-IUKAMOBKSA-N 0.000 description 2
- 229940076838 Immune checkpoint inhibitor Drugs 0.000 description 2
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 2
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 2
- 102100040061 Indoleamine 2,3-dioxygenase 1 Human genes 0.000 description 2
- 102000037984 Inhibitory immune checkpoint proteins Human genes 0.000 description 2
- 108091008026 Inhibitory immune checkpoint proteins Proteins 0.000 description 2
- 102100037850 Interferon gamma Human genes 0.000 description 2
- 108010038453 Interleukin-2 Receptors Proteins 0.000 description 2
- 102000010789 Interleukin-2 Receptors Human genes 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- MMEDVBWCMGRKKC-GARJFASQSA-N Leu-Asp-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N1CCC[C@@H]1C(=O)O)N MMEDVBWCMGRKKC-GARJFASQSA-N 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
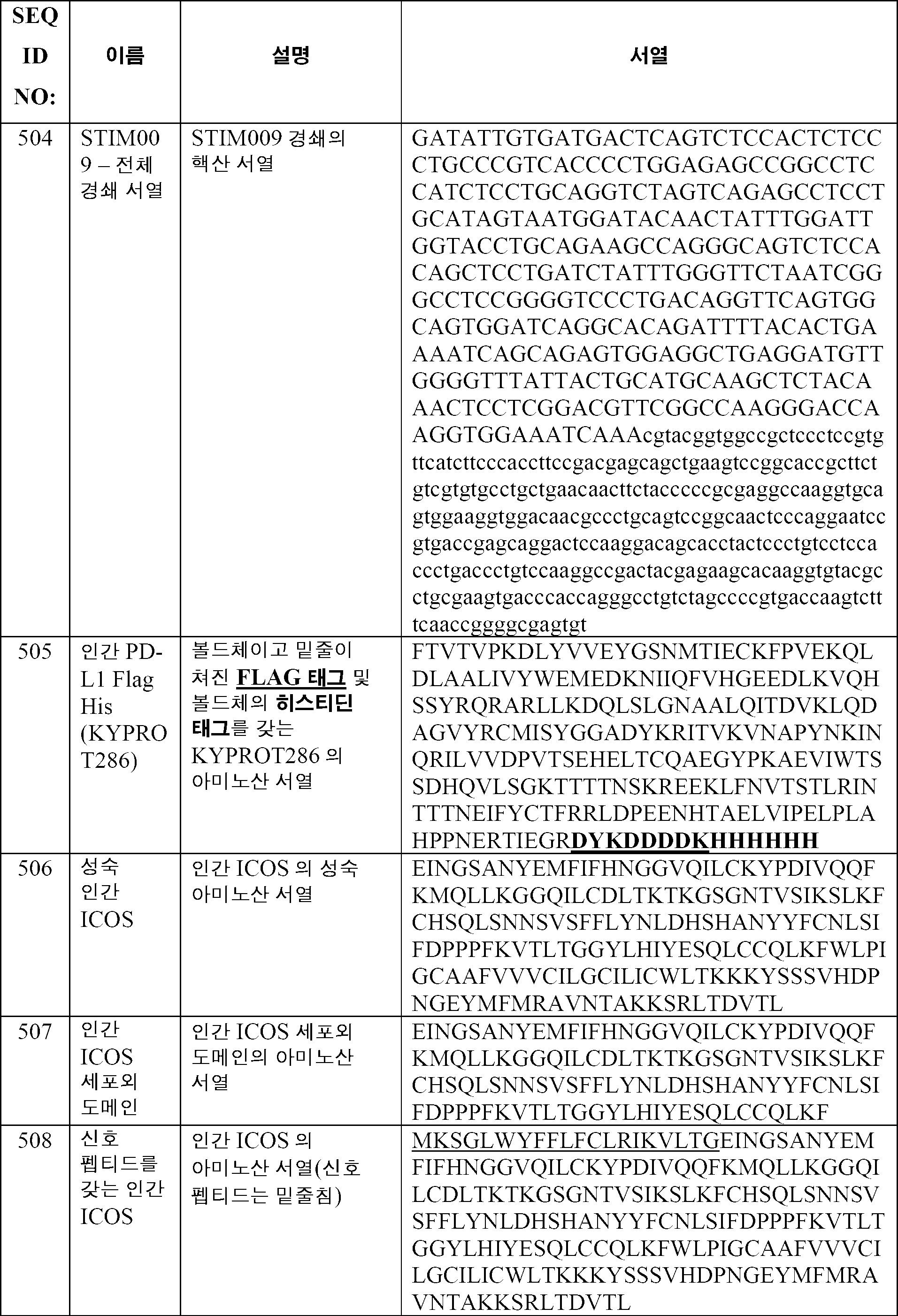
- 102100029193 Low affinity immunoglobulin gamma Fc region receptor III-A Human genes 0.000 description 2
- 101710099301 Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 2
- 206010025323 Lymphomas Diseases 0.000 description 2
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 2
- ZEDVFJPQNNBMST-CYDGBPFRSA-N Met-Arg-Ile Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O ZEDVFJPQNNBMST-CYDGBPFRSA-N 0.000 description 2
- WXJLBSXNUHIGSS-OSUNSFLBSA-N Met-Thr-Ile Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WXJLBSXNUHIGSS-OSUNSFLBSA-N 0.000 description 2
- 241000699660 Mus musculus Species 0.000 description 2
- 101001042093 Mus musculus ICOS ligand Proteins 0.000 description 2
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 description 2
- 101100342977 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) leu-1 gene Proteins 0.000 description 2
- 208000015914 Non-Hodgkin lymphomas Diseases 0.000 description 2
- 108091034117 Oligonucleotide Proteins 0.000 description 2
- ZLAKUZDMKVKFAI-JYJNAYRXSA-N Phe-Pro-Val Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O ZLAKUZDMKVKFAI-JYJNAYRXSA-N 0.000 description 2
- 208000007452 Plasmacytoma Diseases 0.000 description 2
- 206010035664 Pneumonia Diseases 0.000 description 2
- 239000004698 Polyethylene Substances 0.000 description 2
- 239000002202 Polyethylene glycol Substances 0.000 description 2
- 108091008611 Protein Kinase B Proteins 0.000 description 2
- 102000005765 Proto-Oncogene Proteins c-akt Human genes 0.000 description 2
- 101100179076 Rattus norvegicus Icos gene Proteins 0.000 description 2
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 2
- 108020004511 Recombinant DNA Proteins 0.000 description 2
- 206010038019 Rectal adenocarcinoma Diseases 0.000 description 2
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- BQCADISMDOOEFD-UHFFFAOYSA-N Silver Chemical compound [Ag] BQCADISMDOOEFD-UHFFFAOYSA-N 0.000 description 2
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 2
- PXIPVTKHYLBLMZ-UHFFFAOYSA-N Sodium azide Chemical compound [Na+].[N-]=[N+]=[N-] PXIPVTKHYLBLMZ-UHFFFAOYSA-N 0.000 description 2
- 208000034254 Squamous cell carcinoma of the cervix uteri Diseases 0.000 description 2
- 230000020385 T cell costimulation Effects 0.000 description 2
- 201000008754 Tenosynovial giant cell tumor Diseases 0.000 description 2
- AAZOYLQUEQRUMZ-GSSVUCPTSA-N Thr-Thr-Asn Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CC(N)=O AAZOYLQUEQRUMZ-GSSVUCPTSA-N 0.000 description 2
- SJPDTIQHLBQPFO-VLCNGCBASA-N Thr-Tyr-Trp Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O SJPDTIQHLBQPFO-VLCNGCBASA-N 0.000 description 2
- KZTLZZQTJMCGIP-ZJDVBMNYSA-N Thr-Val-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KZTLZZQTJMCGIP-ZJDVBMNYSA-N 0.000 description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 2
- 239000004473 Threonine Substances 0.000 description 2
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 2
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 description 2
- PTFPUAXGIKTVNN-ONGXEEELSA-N Val-His-Gly Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)NCC(=O)O)N PTFPUAXGIKTVNN-ONGXEEELSA-N 0.000 description 2
- ZRSZTKTVPNSUNA-IHRRRGAJSA-N Val-Lys-Leu Chemical compound CC(C)C[C@H](NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)C(C)C)C(O)=O ZRSZTKTVPNSUNA-IHRRRGAJSA-N 0.000 description 2
- BGXVHVMJZCSOCA-AVGNSLFASA-N Val-Pro-Lys Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)O)N BGXVHVMJZCSOCA-AVGNSLFASA-N 0.000 description 2
- OFTXTCGQJXTNQS-XGEHTFHBSA-N Val-Thr-Ser Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](C(C)C)N)O OFTXTCGQJXTNQS-XGEHTFHBSA-N 0.000 description 2
- LMVWCLDJNSBOEA-FKBYEOEOSA-N Val-Tyr-Trp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CNC3=CC=CC=C32)C(=O)O)N LMVWCLDJNSBOEA-FKBYEOEOSA-N 0.000 description 2
- NLNCNKIVJPEFBC-DLOVCJGASA-N Val-Val-Glu Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CCC(O)=O NLNCNKIVJPEFBC-DLOVCJGASA-N 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 description 2
- 108010045023 alanyl-prolyl-tyrosine Proteins 0.000 description 2
- 108010005233 alanylglutamic acid Proteins 0.000 description 2
- 108010047495 alanylglycine Proteins 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 238000010171 animal model Methods 0.000 description 2
- 239000005557 antagonist Substances 0.000 description 2
- 230000009830 antibody antigen interaction Effects 0.000 description 2
- 238000011394 anticancer treatment Methods 0.000 description 2
- 230000030741 antigen processing and presentation Effects 0.000 description 2
- 239000012911 assay medium Substances 0.000 description 2
- 229950002916 avelumab Drugs 0.000 description 2
- 244000052616 bacterial pathogen Species 0.000 description 2
- SESFRYSPDFLNCH-UHFFFAOYSA-N benzyl benzoate Chemical compound C=1C=CC=CC=1C(=O)OCC1=CC=CC=C1 SESFRYSPDFLNCH-UHFFFAOYSA-N 0.000 description 2
- 108091008324 binding proteins Proteins 0.000 description 2
- 230000000975 bioactive effect Effects 0.000 description 2
- 210000000481 breast Anatomy 0.000 description 2
- KQNZDYYTLMIZCT-KQPMLPITSA-N brefeldin A Chemical compound O[C@@H]1\C=C\C(=O)O[C@@H](C)CCC\C=C\[C@@H]2C[C@H](O)C[C@H]21 KQNZDYYTLMIZCT-KQPMLPITSA-N 0.000 description 2
- JUMGSHROWPPKFX-UHFFFAOYSA-N brefeldin-A Natural products CC1CCCC=CC2(C)CC(O)CC2(C)C(O)C=CC(=O)O1 JUMGSHROWPPKFX-UHFFFAOYSA-N 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 230000005880 cancer cell killing Effects 0.000 description 2
- 238000002619 cancer immunotherapy Methods 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 210000004970 cd4 cell Anatomy 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 210000003679 cervix uteri Anatomy 0.000 description 2
- 239000003153 chemical reaction reagent Substances 0.000 description 2
- 210000000349 chromosome Anatomy 0.000 description 2
- 230000004087 circulation Effects 0.000 description 2
- 201000010897 colon adenocarcinoma Diseases 0.000 description 2
- 238000011284 combination treatment Methods 0.000 description 2
- 238000007596 consolidation process Methods 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 230000000139 costimulatory effect Effects 0.000 description 2
- DDRJAANPRJIHGJ-UHFFFAOYSA-N creatinine Chemical compound CN1CC(=O)NC1=N DDRJAANPRJIHGJ-UHFFFAOYSA-N 0.000 description 2
- 238000004132 cross linking Methods 0.000 description 2
- 238000012258 culturing Methods 0.000 description 2
- 208000030381 cutaneous melanoma Diseases 0.000 description 2
- 238000004163 cytometry Methods 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 238000001514 detection method Methods 0.000 description 2
- 208000035647 diffuse type tenosynovial giant cell tumor Diseases 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 239000003085 diluting agent Substances 0.000 description 2
- 230000008034 disappearance Effects 0.000 description 2
- 208000037765 diseases and disorders Diseases 0.000 description 2
- 231100000371 dose-limiting toxicity Toxicity 0.000 description 2
- 230000002222 downregulating effect Effects 0.000 description 2
- 239000003937 drug carrier Substances 0.000 description 2
- 229950009791 durvalumab Drugs 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 230000008030 elimination Effects 0.000 description 2
- 238000003379 elimination reaction Methods 0.000 description 2
- 201000003683 endocervical adenocarcinoma Diseases 0.000 description 2
- 230000002708 enhancing effect Effects 0.000 description 2
- 102000052116 epidermal growth factor receptor activity proteins Human genes 0.000 description 2
- 108700015053 epidermal growth factor receptor activity proteins Proteins 0.000 description 2
- 201000006585 gastric adenocarcinoma Diseases 0.000 description 2
- 238000010199 gene set enrichment analysis Methods 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- 125000000291 glutamic acid group Chemical group N[C@@H](CCC(O)=O)C(=O)* 0.000 description 2
- 108010057083 glutamyl-aspartyl-leucine Proteins 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- 230000009036 growth inhibition Effects 0.000 description 2
- 208000019622 heart disease Diseases 0.000 description 2
- 201000005787 hematologic cancer Diseases 0.000 description 2
- 208000002672 hepatitis B Diseases 0.000 description 2
- 102000046492 human ICOSLG Human genes 0.000 description 2
- 229910052739 hydrogen Inorganic materials 0.000 description 2
- 229940126546 immune checkpoint molecule Drugs 0.000 description 2
- 239000012274 immune-checkpoint protein inhibitor Substances 0.000 description 2
- 238000002649 immunization Methods 0.000 description 2
- 230000002163 immunogen Effects 0.000 description 2
- 230000002584 immunomodulator Effects 0.000 description 2
- 238000002650 immunosuppressive therapy Methods 0.000 description 2
- 230000001771 impaired effect Effects 0.000 description 2
- 230000001939 inductive effect Effects 0.000 description 2
- 230000002401 inhibitory effect Effects 0.000 description 2
- 208000024312 invasive carcinoma Diseases 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 2
- 210000003292 kidney cell Anatomy 0.000 description 2
- 210000004185 liver Anatomy 0.000 description 2
- 201000005249 lung adenocarcinoma Diseases 0.000 description 2
- 208000019420 lymphoid neoplasm Diseases 0.000 description 2
- 239000012139 lysis buffer Substances 0.000 description 2
- 108010003700 lysyl aspartic acid Proteins 0.000 description 2
- 108010056787 lysyl-arginyl-glutamyl-glutamic acid Proteins 0.000 description 2
- 210000002540 macrophage Anatomy 0.000 description 2
- 238000012423 maintenance Methods 0.000 description 2
- 201000004792 malaria Diseases 0.000 description 2
- 210000004962 mammalian cell Anatomy 0.000 description 2
- 230000010534 mechanism of action Effects 0.000 description 2
- ANZJBCHSOXCCRQ-FKUXLPTCSA-N mertansine Chemical compound CO[C@@H]([C@@]1(O)C[C@H](OC(=O)N1)[C@@H](C)[C@@H]1O[C@@]1(C)[C@@H](OC(=O)[C@H](C)N(C)C(=O)CCS)CC(=O)N1C)\C=C\C=C(C)\CC2=CC(OC)=C(Cl)C1=C2 ANZJBCHSOXCCRQ-FKUXLPTCSA-N 0.000 description 2
- 210000001616 monocyte Anatomy 0.000 description 2
- 238000010172 mouse model Methods 0.000 description 2
- 210000004877 mucosa Anatomy 0.000 description 2
- LWGJTAZLEJHCPA-UHFFFAOYSA-N n-(2-chloroethyl)-n-nitrosomorpholine-4-carboxamide Chemical compound ClCCN(N=O)C(=O)N1CCOCC1 LWGJTAZLEJHCPA-UHFFFAOYSA-N 0.000 description 2
- YOHYSYJDKVYCJI-UHFFFAOYSA-N n-[3-[[6-[3-(trifluoromethyl)anilino]pyrimidin-4-yl]amino]phenyl]cyclopropanecarboxamide Chemical compound FC(F)(F)C1=CC=CC(NC=2N=CN=C(NC=3C=C(NC(=O)C4CC4)C=CC=3)C=2)=C1 YOHYSYJDKVYCJI-UHFFFAOYSA-N 0.000 description 2
- 238000006386 neutralization reaction Methods 0.000 description 2
- 229960003301 nivolumab Drugs 0.000 description 2
- 231100000252 nontoxic Toxicity 0.000 description 2
- 230000003000 nontoxic effect Effects 0.000 description 2
- 239000003921 oil Substances 0.000 description 2
- 230000014207 opsonization Effects 0.000 description 2
- 210000000056 organ Anatomy 0.000 description 2
- 230000001575 pathological effect Effects 0.000 description 2
- 230000007170 pathology Effects 0.000 description 2
- 230000002093 peripheral effect Effects 0.000 description 2
- 230000002085 persistent effect Effects 0.000 description 2
- 238000002823 phage display Methods 0.000 description 2
- ORMNNUPLFAPCFD-DVLYDCSHSA-M phenethicillin potassium Chemical compound [K+].N([C@@H]1C(N2[C@H](C(C)(C)S[C@@H]21)C([O-])=O)=O)C(=O)C(C)OC1=CC=CC=C1 ORMNNUPLFAPCFD-DVLYDCSHSA-M 0.000 description 2
- 108010082795 phenylalanyl-arginyl-arginine Proteins 0.000 description 2
- 210000004986 primary T-cell Anatomy 0.000 description 2
- 239000000047 product Substances 0.000 description 2
- 238000004393 prognosis Methods 0.000 description 2
- 210000001236 prokaryotic cell Anatomy 0.000 description 2
- 230000002062 proliferating effect Effects 0.000 description 2
- 230000035755 proliferation Effects 0.000 description 2
- 230000000069 prophylactic effect Effects 0.000 description 2
- 244000079416 protozoan pathogen Species 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 201000001281 rectum adenocarcinoma Diseases 0.000 description 2
- 208000029922 reticulum cell sarcoma Diseases 0.000 description 2
- 230000035945 sensitivity Effects 0.000 description 2
- 238000012163 sequencing technique Methods 0.000 description 2
- 229910052709 silver Inorganic materials 0.000 description 2
- 239000004332 silver Substances 0.000 description 2
- 201000003708 skin melanoma Diseases 0.000 description 2
- 230000009870 specific binding Effects 0.000 description 2
- 230000002269 spontaneous effect Effects 0.000 description 2
- 238000010561 standard procedure Methods 0.000 description 2
- 210000002784 stomach Anatomy 0.000 description 2
- 238000001356 surgical procedure Methods 0.000 description 2
- 229940124598 therapeutic candidate Drugs 0.000 description 2
- 208000008732 thymoma Diseases 0.000 description 2
- 231100000331 toxic Toxicity 0.000 description 2
- 230000002588 toxic effect Effects 0.000 description 2
- 230000001988 toxicity Effects 0.000 description 2
- 231100000419 toxicity Toxicity 0.000 description 2
- 238000011830 transgenic mouse model Methods 0.000 description 2
- 108010029384 tryptophyl-histidine Proteins 0.000 description 2
- 239000000439 tumor marker Substances 0.000 description 2
- 108010017949 tyrosyl-glycyl-glycine Proteins 0.000 description 2
- 231100000402 unacceptable toxicity Toxicity 0.000 description 2
- 241000701447 unidentified baculovirus Species 0.000 description 2
- 201000005112 urinary bladder cancer Diseases 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- 229960004295 valine Drugs 0.000 description 2
- 239000013598 vector Substances 0.000 description 2
- 244000052613 viral pathogen Species 0.000 description 2
- 230000003612 virological effect Effects 0.000 description 2
- 229940121351 vopratelimab Drugs 0.000 description 2
- 230000036642 wellbeing Effects 0.000 description 2
- XOOUIPVCVHRTMJ-UHFFFAOYSA-L zinc stearate Chemical compound [Zn+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O XOOUIPVCVHRTMJ-UHFFFAOYSA-L 0.000 description 2
- MHVJRKBZMUDEEV-APQLOABGSA-N (+)-Pimaric acid Chemical compound [C@H]1([C@](CCC2)(C)C(O)=O)[C@@]2(C)[C@H]2CC[C@](C=C)(C)C=C2CC1 MHVJRKBZMUDEEV-APQLOABGSA-N 0.000 description 1
- MHVJRKBZMUDEEV-UHFFFAOYSA-N (-)-ent-pimara-8(14),15-dien-19-oic acid Natural products C1CCC(C(O)=O)(C)C2C1(C)C1CCC(C=C)(C)C=C1CC2 MHVJRKBZMUDEEV-UHFFFAOYSA-N 0.000 description 1
- AXFMEGAFCUULFV-BLFANLJRSA-N (2s)-2-[[(2s)-1-[(2s,3r)-2-amino-3-methylpentanoyl]pyrrolidine-2-carbonyl]amino]pentanedioic acid Chemical compound CC[C@@H](C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O AXFMEGAFCUULFV-BLFANLJRSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- QFVHZQCOUORWEI-UHFFFAOYSA-N 4-[(4-anilino-5-sulfonaphthalen-1-yl)diazenyl]-5-hydroxynaphthalene-2,7-disulfonic acid Chemical compound C=12C(O)=CC(S(O)(=O)=O)=CC2=CC(S(O)(=O)=O)=CC=1N=NC(C1=CC=CC(=C11)S(O)(=O)=O)=CC=C1NC1=CC=CC=C1 QFVHZQCOUORWEI-UHFFFAOYSA-N 0.000 description 1
- 102100022464 5'-nucleotidase Human genes 0.000 description 1
- 102000007471 Adenosine A2A receptor Human genes 0.000 description 1
- 108010085277 Adenosine A2A receptor Proteins 0.000 description 1
- BLGHHPHXVJWCNK-GUBZILKMSA-N Ala-Gln-Leu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O BLGHHPHXVJWCNK-GUBZILKMSA-N 0.000 description 1
- PAIHPOGPJVUFJY-WDSKDSINSA-N Ala-Glu-Gly Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(O)=O PAIHPOGPJVUFJY-WDSKDSINSA-N 0.000 description 1
- YHKANGMVQWRMAP-DCAQKATOSA-N Ala-Leu-Arg Chemical compound C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N YHKANGMVQWRMAP-DCAQKATOSA-N 0.000 description 1
- SUMYEVXWCAYLLJ-GUBZILKMSA-N Ala-Leu-Gln Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O SUMYEVXWCAYLLJ-GUBZILKMSA-N 0.000 description 1
- OYJCVIGKMXUVKB-GARJFASQSA-N Ala-Leu-Pro Chemical compound C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N OYJCVIGKMXUVKB-GARJFASQSA-N 0.000 description 1
- MEFILNJXAVSUTO-JXUBOQSCSA-N Ala-Leu-Thr Chemical compound C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MEFILNJXAVSUTO-JXUBOQSCSA-N 0.000 description 1
- CQJHFKKGZXKZBC-BPNCWPANSA-N Ala-Pro-Tyr Chemical compound C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 CQJHFKKGZXKZBC-BPNCWPANSA-N 0.000 description 1
- WNHNMKOFKCHKKD-BFHQHQDPSA-N Ala-Thr-Gly Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O WNHNMKOFKCHKKD-BFHQHQDPSA-N 0.000 description 1
- 201000004384 Alopecia Diseases 0.000 description 1
- 101100339431 Arabidopsis thaliana HMGB2 gene Proteins 0.000 description 1
- SGYSTDWPNPKJPP-GUBZILKMSA-N Arg-Ala-Arg Chemical compound NC(=N)NCCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O SGYSTDWPNPKJPP-GUBZILKMSA-N 0.000 description 1
- VSPLYCLMFAUZRF-GUBZILKMSA-N Arg-Cys-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CCCN=C(N)N)N VSPLYCLMFAUZRF-GUBZILKMSA-N 0.000 description 1
- GIVWETPOBCRTND-DCAQKATOSA-N Arg-Gln-Arg Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O GIVWETPOBCRTND-DCAQKATOSA-N 0.000 description 1
- FRMQITGHXMUNDF-GMOBBJLQSA-N Arg-Ile-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N FRMQITGHXMUNDF-GMOBBJLQSA-N 0.000 description 1
- HJDNZFIYILEIKR-OSUNSFLBSA-N Arg-Ile-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O HJDNZFIYILEIKR-OSUNSFLBSA-N 0.000 description 1
- LVMUGODRNHFGRA-AVGNSLFASA-N Arg-Leu-Arg Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O LVMUGODRNHFGRA-AVGNSLFASA-N 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- 208000002109 Argyria Diseases 0.000 description 1
- XWFPGQVLOVGSLU-CIUDSAMLSA-N Asn-Gln-Arg Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N XWFPGQVLOVGSLU-CIUDSAMLSA-N 0.000 description 1
- MSBDSTRUMZFSEU-PEFMBERDSA-N Asn-Glu-Ile Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O MSBDSTRUMZFSEU-PEFMBERDSA-N 0.000 description 1
- YYSYDIYQTUPNQQ-SXTJYALSSA-N Asn-Ile-Ile Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O YYSYDIYQTUPNQQ-SXTJYALSSA-N 0.000 description 1
- JWKDQOORUCYUIW-ZPFDUUQYSA-N Asn-Lys-Ile Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JWKDQOORUCYUIW-ZPFDUUQYSA-N 0.000 description 1
- UGXYFDQFLVCDFC-CIUDSAMLSA-N Asn-Ser-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O UGXYFDQFLVCDFC-CIUDSAMLSA-N 0.000 description 1
- HRGGPWBIMIQANI-GUBZILKMSA-N Asp-Gln-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O HRGGPWBIMIQANI-GUBZILKMSA-N 0.000 description 1
- QHHVSXGWLYEAGX-GUBZILKMSA-N Asp-His-Gln Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)O)N QHHVSXGWLYEAGX-GUBZILKMSA-N 0.000 description 1
- DINOVZWPTMGSRF-QXEWZRGKSA-N Asp-Pro-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O DINOVZWPTMGSRF-QXEWZRGKSA-N 0.000 description 1
- UTLCRGFJFSZWAW-OLHMAJIHSA-N Asp-Thr-Asn Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)O)N)O UTLCRGFJFSZWAW-OLHMAJIHSA-N 0.000 description 1
- BJDHEININLSZOT-KKUMJFAQSA-N Asp-Tyr-Lys Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCCN)C(O)=O BJDHEININLSZOT-KKUMJFAQSA-N 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 206010006002 Bone pain Diseases 0.000 description 1
- 208000011691 Burkitt lymphomas Diseases 0.000 description 1
- 229940116741 CD137 agonist Drugs 0.000 description 1
- 102000017420 CD3 protein, epsilon/gamma/delta subunit Human genes 0.000 description 1
- 108050005493 CD3 protein, epsilon/gamma/delta subunit Proteins 0.000 description 1
- HFOBENSCBRZVSP-LKXGYXEUSA-N C[C@@H](O)[C@H](NC(=O)N[C@@H](CC(N)=O)c1nc(no1)[C@@H](N)CO)C(O)=O Chemical compound C[C@@H](O)[C@H](NC(=O)N[C@@H](CC(N)=O)c1nc(no1)[C@@H](N)CO)C(O)=O HFOBENSCBRZVSP-LKXGYXEUSA-N 0.000 description 1
- 101100463133 Caenorhabditis elegans pdl-1 gene Proteins 0.000 description 1
- UXVMQQNJUSDDNG-UHFFFAOYSA-L Calcium chloride Chemical compound [Cl-].[Cl-].[Ca+2] UXVMQQNJUSDDNG-UHFFFAOYSA-L 0.000 description 1
- 102100023073 Calcium-activated potassium channel subunit alpha-1 Human genes 0.000 description 1
- 102100029968 Calreticulin Human genes 0.000 description 1
- 108090000549 Calreticulin Proteins 0.000 description 1
- BVKZGUZCCUSVTD-UHFFFAOYSA-L Carbonate Chemical compound [O-]C([O-])=O BVKZGUZCCUSVTD-UHFFFAOYSA-L 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 208000017897 Carcinoma of esophagus Diseases 0.000 description 1
- ZAMOUSCENKQFHK-UHFFFAOYSA-N Chlorine atom Chemical compound [Cl] ZAMOUSCENKQFHK-UHFFFAOYSA-N 0.000 description 1
- 241000251730 Chondrichthyes Species 0.000 description 1
- 208000030808 Clear cell renal carcinoma Diseases 0.000 description 1
- 241001327965 Clonorchis sinensis Species 0.000 description 1
- 241000699800 Cricetinae Species 0.000 description 1
- 241000699802 Cricetulus griseus Species 0.000 description 1
- 239000004971 Cross linker Substances 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- IZUNQDRIAOLWCN-YUMQZZPRSA-N Cys-Leu-Gly Chemical compound CC(C)C[C@@H](C(=O)NCC(=O)O)NC(=O)[C@H](CS)N IZUNQDRIAOLWCN-YUMQZZPRSA-N 0.000 description 1
- NLDWTJBJFVWBDQ-KKUMJFAQSA-N Cys-Lys-Phe Chemical compound NCCCC[C@H](NC(=O)[C@@H](N)CS)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 NLDWTJBJFVWBDQ-KKUMJFAQSA-N 0.000 description 1
- PEZINYWZBQNTIX-NAKRPEOUSA-N Cys-Met-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)N PEZINYWZBQNTIX-NAKRPEOUSA-N 0.000 description 1
- JTEGHEWKBCTIAL-IXOXFDKPSA-N Cys-Thr-Phe Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CS)N)O JTEGHEWKBCTIAL-IXOXFDKPSA-N 0.000 description 1
- 230000007067 DNA methylation Effects 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 238000002965 ELISA Methods 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 208000010201 Exanthema Diseases 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 1
- 108700007698 Genetic Terminator Regions Proteins 0.000 description 1
- RZSLYUUFFVHFRQ-FXQIFTODSA-N Gln-Ala-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O RZSLYUUFFVHFRQ-FXQIFTODSA-N 0.000 description 1
- MWLYSLMKFXWZPW-ZPFDUUQYSA-N Gln-Arg-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@@H](N)CCC(N)=O MWLYSLMKFXWZPW-ZPFDUUQYSA-N 0.000 description 1
- ICDIMQAMJGDHSE-GUBZILKMSA-N Gln-His-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CO)C(O)=O ICDIMQAMJGDHSE-GUBZILKMSA-N 0.000 description 1
- QBLMTCRYYTVUQY-GUBZILKMSA-N Gln-Leu-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O QBLMTCRYYTVUQY-GUBZILKMSA-N 0.000 description 1
- YPMDZWPZFOZYFG-GUBZILKMSA-N Gln-Leu-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YPMDZWPZFOZYFG-GUBZILKMSA-N 0.000 description 1
- QFXNFFZTMFHPST-DZKIICNBSA-N Gln-Phe-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)[C@H](CCC(=O)N)N QFXNFFZTMFHPST-DZKIICNBSA-N 0.000 description 1
- UTOQQOMEJDPDMX-ACZMJKKPSA-N Gln-Ser-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(O)=O UTOQQOMEJDPDMX-ACZMJKKPSA-N 0.000 description 1
- VEYGCDYMOXHJLS-GVXVVHGQSA-N Gln-Val-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O VEYGCDYMOXHJLS-GVXVVHGQSA-N 0.000 description 1
- SRZLHYPAOXBBSB-HJGDQZAQSA-N Glu-Arg-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SRZLHYPAOXBBSB-HJGDQZAQSA-N 0.000 description 1
- JVSBYEDSSRZQGV-GUBZILKMSA-N Glu-Asp-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CCC(O)=O JVSBYEDSSRZQGV-GUBZILKMSA-N 0.000 description 1
- HJIFPJUEOGZWRI-GUBZILKMSA-N Glu-Asp-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCC(=O)O)N HJIFPJUEOGZWRI-GUBZILKMSA-N 0.000 description 1
- NKLRYVLERDYDBI-FXQIFTODSA-N Glu-Glu-Asp Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O NKLRYVLERDYDBI-FXQIFTODSA-N 0.000 description 1
- LGYZYFFDELZWRS-DCAQKATOSA-N Glu-Glu-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CCC(O)=O LGYZYFFDELZWRS-DCAQKATOSA-N 0.000 description 1
- JGHNIWVNCAOVRO-DCAQKATOSA-N Glu-His-Glu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(O)=O JGHNIWVNCAOVRO-DCAQKATOSA-N 0.000 description 1
- WTMZXOPHTIVFCP-QEWYBTABSA-N Glu-Ile-Phe Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 WTMZXOPHTIVFCP-QEWYBTABSA-N 0.000 description 1
- UGSVSNXPJJDJKL-SDDRHHMPSA-N Glu-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCC(=O)O)N UGSVSNXPJJDJKL-SDDRHHMPSA-N 0.000 description 1
- GJBUAAAIZSRCDC-GVXVVHGQSA-N Glu-Leu-Val Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O GJBUAAAIZSRCDC-GVXVVHGQSA-N 0.000 description 1
- JSIQVRIXMINMTA-ZDLURKLDSA-N Glu-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H](N)CCC(O)=O JSIQVRIXMINMTA-ZDLURKLDSA-N 0.000 description 1
- HAGKYCXGTRUUFI-RYUDHWBXSA-N Glu-Tyr-Gly Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)NCC(=O)O)NC(=O)[C@H](CCC(=O)O)N)O HAGKYCXGTRUUFI-RYUDHWBXSA-N 0.000 description 1
- FGGKGJHCVMYGCD-UKJIMTQDSA-N Glu-Val-Ile Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FGGKGJHCVMYGCD-UKJIMTQDSA-N 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- RLFSBAPJTYKSLG-WHFBIAKZSA-N Gly-Ala-Asp Chemical compound NCC(=O)N[C@@H](C)C(=O)N[C@@H](CC(O)=O)C(O)=O RLFSBAPJTYKSLG-WHFBIAKZSA-N 0.000 description 1
- UXJHNZODTMHWRD-WHFBIAKZSA-N Gly-Asn-Ala Chemical compound [H]NCC(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(O)=O UXJHNZODTMHWRD-WHFBIAKZSA-N 0.000 description 1
- CCQOOWAONKGYKQ-BYPYZUCNSA-N Gly-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)CN CCQOOWAONKGYKQ-BYPYZUCNSA-N 0.000 description 1
- VIIBEIQMLJEUJG-LAEOZQHASA-N Gly-Ile-Gln Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCC(N)=O)C(O)=O VIIBEIQMLJEUJG-LAEOZQHASA-N 0.000 description 1
- FXGRXIATVXUAHO-WEDXCCLWSA-N Gly-Lys-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CCCCN FXGRXIATVXUAHO-WEDXCCLWSA-N 0.000 description 1
- LYZYGGWCBLBDMC-QWHCGFSZSA-N Gly-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)CN)C(=O)O LYZYGGWCBLBDMC-QWHCGFSZSA-N 0.000 description 1
- IZVICCORZOSGPT-JSGCOSHPSA-N Gly-Val-Tyr Chemical compound [H]NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O IZVICCORZOSGPT-JSGCOSHPSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 208000012766 Growth delay Diseases 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 108700010013 HMGB1 Proteins 0.000 description 1
- 101150021904 HMGB1 gene Proteins 0.000 description 1
- 102000001554 Hemoglobins Human genes 0.000 description 1
- 108010054147 Hemoglobins Proteins 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 102100037907 High mobility group protein B1 Human genes 0.000 description 1
- FIMNVXRZGUAGBI-AVGNSLFASA-N His-Glu-Leu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O FIMNVXRZGUAGBI-AVGNSLFASA-N 0.000 description 1
- HAPWZEVRQYGLSG-IUCAKERBSA-N His-Gly-Glu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(O)=O HAPWZEVRQYGLSG-IUCAKERBSA-N 0.000 description 1
- 101000678236 Homo sapiens 5'-nucleotidase Proteins 0.000 description 1
- 101000834898 Homo sapiens Alpha-synuclein Proteins 0.000 description 1
- 101100166600 Homo sapiens CD28 gene Proteins 0.000 description 1
- 101000913074 Homo sapiens High affinity immunoglobulin gamma Fc receptor I Proteins 0.000 description 1
- 101000961156 Homo sapiens Immunoglobulin heavy constant gamma 1 Proteins 0.000 description 1
- 101000917824 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor II-b Proteins 0.000 description 1
- 101000917858 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 1
- 101001117312 Homo sapiens Programmed cell death 1 ligand 2 Proteins 0.000 description 1
- 101000611936 Homo sapiens Programmed cell death protein 1 Proteins 0.000 description 1
- 101000611643 Homo sapiens Protein phosphatase 1 regulatory subunit 15A Proteins 0.000 description 1
- 101000652359 Homo sapiens Spermatogenesis-associated protein 2 Proteins 0.000 description 1
- 101000914484 Homo sapiens T-lymphocyte activation antigen CD80 Proteins 0.000 description 1
- 101000801234 Homo sapiens Tumor necrosis factor receptor superfamily member 18 Proteins 0.000 description 1
- 101000851370 Homo sapiens Tumor necrosis factor receptor superfamily member 9 Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 241000714260 Human T-lymphotropic virus 1 Species 0.000 description 1
- 206010020751 Hypersensitivity Diseases 0.000 description 1
- XDXDZDZNSLXDNA-TZNDIEGXSA-N Idarubicin Chemical compound C1[C@H](N)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2C[C@@](O)(C(C)=O)C1 XDXDZDZNSLXDNA-TZNDIEGXSA-N 0.000 description 1
- XDXDZDZNSLXDNA-UHFFFAOYSA-N Idarubicin Natural products C1C(N)C(O)C(C)OC1OC1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2CC(O)(C(C)=O)C1 XDXDZDZNSLXDNA-UHFFFAOYSA-N 0.000 description 1
- NCSIQAFSIPHVAN-IUKAMOBKSA-N Ile-Asn-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N NCSIQAFSIPHVAN-IUKAMOBKSA-N 0.000 description 1
- HPCFRQWLTRDGHT-AJNGGQMLSA-N Ile-Leu-Leu Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O HPCFRQWLTRDGHT-AJNGGQMLSA-N 0.000 description 1
- IOVUXUSIGXCREV-DKIMLUQUSA-N Ile-Leu-Phe Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 IOVUXUSIGXCREV-DKIMLUQUSA-N 0.000 description 1
- DSDPLOODKXISDT-XUXIUFHCSA-N Ile-Leu-Val Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O DSDPLOODKXISDT-XUXIUFHCSA-N 0.000 description 1
- UYNXBNHVWFNVIN-HJWJTTGWSA-N Ile-Phe-Arg Chemical compound NC(N)=NCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)[C@@H](C)CC)CC1=CC=CC=C1 UYNXBNHVWFNVIN-HJWJTTGWSA-N 0.000 description 1
- WLRJHVNFGAOYPS-HJPIBITLSA-N Ile-Ser-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N WLRJHVNFGAOYPS-HJPIBITLSA-N 0.000 description 1
- QSXSHZIRKTUXNG-STECZYCISA-N Ile-Val-Tyr Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 QSXSHZIRKTUXNG-STECZYCISA-N 0.000 description 1
- 102000018071 Immunoglobulin Fc Fragments Human genes 0.000 description 1
- 108010091135 Immunoglobulin Fc Fragments Proteins 0.000 description 1
- 108700005091 Immunoglobulin Genes Proteins 0.000 description 1
- 102000013463 Immunoglobulin Light Chains Human genes 0.000 description 1
- 108010065825 Immunoglobulin Light Chains Proteins 0.000 description 1
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 1
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 1
- 102100039345 Immunoglobulin heavy constant gamma 1 Human genes 0.000 description 1
- 206010051792 Infusion related reaction Diseases 0.000 description 1
- 108090001005 Interleukin-6 Proteins 0.000 description 1
- 101150008942 J gene Proteins 0.000 description 1
- 208000007766 Kaposi sarcoma Diseases 0.000 description 1
- AHLPHDHHMVZTML-BYPYZUCNSA-N L-Ornithine Chemical compound NCCC[C@H](N)C(O)=O AHLPHDHHMVZTML-BYPYZUCNSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- 150000008575 L-amino acids Chemical class 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- 239000005411 L01XE02 - Gefitinib Substances 0.000 description 1
- 239000005551 L01XE03 - Erlotinib Substances 0.000 description 1
- 108010017739 LAGA Proteins 0.000 description 1
- 108010092555 Large-Conductance Calcium-Activated Potassium Channels Proteins 0.000 description 1
- PBCHMHROGNUXMK-DLOVCJGASA-N Leu-Ala-His Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 PBCHMHROGNUXMK-DLOVCJGASA-N 0.000 description 1
- TWQIYNGNYNJUFM-NHCYSSNCSA-N Leu-Asn-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O TWQIYNGNYNJUFM-NHCYSSNCSA-N 0.000 description 1
- OXRLYTYUXAQTHP-YUMQZZPRSA-N Leu-Gly-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)NCC(=O)N[C@@H](C)C(O)=O OXRLYTYUXAQTHP-YUMQZZPRSA-N 0.000 description 1
- LXKNSJLSGPNHSK-KKUMJFAQSA-N Leu-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N LXKNSJLSGPNHSK-KKUMJFAQSA-N 0.000 description 1
- RZXLZBIUTDQHJQ-SRVKXCTJSA-N Leu-Lys-Asp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O RZXLZBIUTDQHJQ-SRVKXCTJSA-N 0.000 description 1
- LZHJZLHSRGWBBE-IHRRRGAJSA-N Leu-Lys-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O LZHJZLHSRGWBBE-IHRRRGAJSA-N 0.000 description 1
- KWLWZYMNUZJKMZ-IHRRRGAJSA-N Leu-Pro-Leu Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(O)=O KWLWZYMNUZJKMZ-IHRRRGAJSA-N 0.000 description 1
- RGUXWMDNCPMQFB-YUMQZZPRSA-N Leu-Ser-Gly Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O RGUXWMDNCPMQFB-YUMQZZPRSA-N 0.000 description 1
- LINKCQUOMUDLKN-KATARQTJSA-N Leu-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC(C)C)N)O LINKCQUOMUDLKN-KATARQTJSA-N 0.000 description 1
- VQHUBNVKFFLWRP-ULQDDVLXSA-N Leu-Tyr-Val Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](C(C)C)C(O)=O)CC1=CC=C(O)C=C1 VQHUBNVKFFLWRP-ULQDDVLXSA-N 0.000 description 1
- FMFNIDICDKEMOE-XUXIUFHCSA-N Leu-Val-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FMFNIDICDKEMOE-XUXIUFHCSA-N 0.000 description 1
- 102100029204 Low affinity immunoglobulin gamma Fc region receptor II-a Human genes 0.000 description 1
- 101710177649 Low affinity immunoglobulin gamma Fc region receptor III Proteins 0.000 description 1
- 206010025327 Lymphopenia Diseases 0.000 description 1
- PNPYKQFJGRFYJE-GUBZILKMSA-N Lys-Ala-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O PNPYKQFJGRFYJE-GUBZILKMSA-N 0.000 description 1
- GQZMPWBZQALKJO-UWVGGRQHSA-N Lys-Gly-Arg Chemical compound [H]N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O GQZMPWBZQALKJO-UWVGGRQHSA-N 0.000 description 1
- KYNNSEJZFVCDIV-ZPFDUUQYSA-N Lys-Ile-Asn Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(O)=O KYNNSEJZFVCDIV-ZPFDUUQYSA-N 0.000 description 1
- XIZQPFCRXLUNMK-BZSNNMDCSA-N Lys-Leu-Phe Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CCCCN)N XIZQPFCRXLUNMK-BZSNNMDCSA-N 0.000 description 1
- CAVRAQIDHUPECU-UVOCVTCTSA-N Lys-Thr-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O CAVRAQIDHUPECU-UVOCVTCTSA-N 0.000 description 1
- MDDUIRLQCYVRDO-NHCYSSNCSA-N Lys-Val-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN MDDUIRLQCYVRDO-NHCYSSNCSA-N 0.000 description 1
- XABXVVSWUVCZST-GVXVVHGQSA-N Lys-Val-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN XABXVVSWUVCZST-GVXVVHGQSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 208000002030 Merkel cell carcinoma Diseases 0.000 description 1
- MHQXIBRPDKXDGZ-ZFWWWQNUSA-N Met-Gly-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)CNC(=O)[C@@H](N)CCSC)C(O)=O)=CNC2=C1 MHQXIBRPDKXDGZ-ZFWWWQNUSA-N 0.000 description 1
- JKXVPNCSAMWUEJ-GUBZILKMSA-N Met-Met-Asp Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC(O)=O)C(O)=O JKXVPNCSAMWUEJ-GUBZILKMSA-N 0.000 description 1
- LQTGGXSOMDSWTQ-UNQGMJICSA-N Met-Phe-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)[C@H](CCSC)N)O LQTGGXSOMDSWTQ-UNQGMJICSA-N 0.000 description 1
- 206010050513 Metastatic renal cell carcinoma Diseases 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 241001529936 Murinae Species 0.000 description 1
- 101100369989 Mus musculus Tnfaip3 gene Proteins 0.000 description 1
- 201000002481 Myositis Diseases 0.000 description 1
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 1
- SITLTJHOQZFJGG-UHFFFAOYSA-N N-L-alpha-glutamyl-L-valine Natural products CC(C)C(C(O)=O)NC(=O)C(N)CCC(O)=O SITLTJHOQZFJGG-UHFFFAOYSA-N 0.000 description 1
- RRMJMHOQSALEJJ-UHFFFAOYSA-N N-[5-[[4-[4-[(dimethylamino)methyl]-3-phenylpyrazol-1-yl]pyrimidin-2-yl]amino]-4-methoxy-2-morpholin-4-ylphenyl]prop-2-enamide Chemical compound CN(C)CC=1C(=NN(C=1)C1=NC(=NC=C1)NC=1C(=CC(=C(C=1)NC(C=C)=O)N1CCOCC1)OC)C1=CC=CC=C1 RRMJMHOQSALEJJ-UHFFFAOYSA-N 0.000 description 1
- 125000000729 N-terminal amino-acid group Chemical group 0.000 description 1
- 208000002454 Nasopharyngeal Carcinoma Diseases 0.000 description 1
- 206010061306 Nasopharyngeal cancer Diseases 0.000 description 1
- 206010061309 Neoplasm progression Diseases 0.000 description 1
- 206010029266 Neuroendocrine carcinoma of the skin Diseases 0.000 description 1
- 244000061176 Nicotiana tabacum Species 0.000 description 1
- 235000002637 Nicotiana tabacum Nutrition 0.000 description 1
- 102220596838 Non-structural maintenance of chromosomes element 1 homolog_R38A_mutation Human genes 0.000 description 1
- 241000242726 Opisthorchis viverrini Species 0.000 description 1
- AHLPHDHHMVZTML-UHFFFAOYSA-N Orn-delta-NH2 Natural products NCCCC(N)C(O)=O AHLPHDHHMVZTML-UHFFFAOYSA-N 0.000 description 1
- UTJLXEIPEHZYQJ-UHFFFAOYSA-N Ornithine Natural products OC(=O)C(C)CCCN UTJLXEIPEHZYQJ-UHFFFAOYSA-N 0.000 description 1
- 206010031096 Oropharyngeal cancer Diseases 0.000 description 1
- 206010057444 Oropharyngeal neoplasm Diseases 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 206010033109 Ototoxicity Diseases 0.000 description 1
- 102000038030 PI3Ks Human genes 0.000 description 1
- 108091007960 PI3Ks Proteins 0.000 description 1
- 108090000526 Papain Proteins 0.000 description 1
- 102000057297 Pepsin A Human genes 0.000 description 1
- 108090000284 Pepsin A Proteins 0.000 description 1
- BWTKUQPNOMMKMA-FIRPJDEBSA-N Phe-Ile-Phe Chemical compound C([C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=CC=C1 BWTKUQPNOMMKMA-FIRPJDEBSA-N 0.000 description 1
- CVAUVSOFHJKCHN-BZSNNMDCSA-N Phe-Tyr-Cys Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CS)C(O)=O)C1=CC=CC=C1 CVAUVSOFHJKCHN-BZSNNMDCSA-N 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000224016 Plasmodium Species 0.000 description 1
- 241000223960 Plasmodium falciparum Species 0.000 description 1
- 229920003171 Poly (ethylene oxide) Polymers 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- OBVCYFIHIIYIQF-CIUDSAMLSA-N Pro-Asn-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O OBVCYFIHIIYIQF-CIUDSAMLSA-N 0.000 description 1
- OFGUOWQVEGTVNU-DCAQKATOSA-N Pro-Lys-Ala Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O OFGUOWQVEGTVNU-DCAQKATOSA-N 0.000 description 1
- ZLXKLMHAMDENIO-DCAQKATOSA-N Pro-Lys-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O ZLXKLMHAMDENIO-DCAQKATOSA-N 0.000 description 1
- GFHOSBYCLACKEK-GUBZILKMSA-N Pro-Pro-Asn Chemical compound [H]N1CCC[C@H]1C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(N)=O)C(O)=O GFHOSBYCLACKEK-GUBZILKMSA-N 0.000 description 1
- QHSSUIHLAIWXEE-IHRRRGAJSA-N Pro-Tyr-Asn Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O QHSSUIHLAIWXEE-IHRRRGAJSA-N 0.000 description 1
- IMNVAOPEMFDAQD-NHCYSSNCSA-N Pro-Val-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O IMNVAOPEMFDAQD-NHCYSSNCSA-N 0.000 description 1
- 102100024213 Programmed cell death 1 ligand 2 Human genes 0.000 description 1
- 206010060862 Prostate cancer Diseases 0.000 description 1
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 1
- 102000001253 Protein Kinase Human genes 0.000 description 1
- 102100040714 Protein phosphatase 1 regulatory subunit 15A Human genes 0.000 description 1
- 206010037660 Pyrexia Diseases 0.000 description 1
- 208000037323 Rare tumor Diseases 0.000 description 1
- 102100029986 Receptor tyrosine-protein kinase erbB-3 Human genes 0.000 description 1
- 101710100969 Receptor tyrosine-protein kinase erbB-3 Proteins 0.000 description 1
- 241000242683 Schistosoma haematobium Species 0.000 description 1
- 206010040047 Sepsis Diseases 0.000 description 1
- COAHUSQNSVFYBW-FXQIFTODSA-N Ser-Asn-Met Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCSC)C(O)=O COAHUSQNSVFYBW-FXQIFTODSA-N 0.000 description 1
- ICHZYBVODUVUKN-SRVKXCTJSA-N Ser-Asn-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O ICHZYBVODUVUKN-SRVKXCTJSA-N 0.000 description 1
- SFZKGGOGCNQPJY-CIUDSAMLSA-N Ser-Asp-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CO)N SFZKGGOGCNQPJY-CIUDSAMLSA-N 0.000 description 1
- INCNPLPRPOYTJI-JBDRJPRFSA-N Ser-Cys-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CO)N INCNPLPRPOYTJI-JBDRJPRFSA-N 0.000 description 1
- XNCUYZKGQOCOQH-YUMQZZPRSA-N Ser-Leu-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)NCC(O)=O XNCUYZKGQOCOQH-YUMQZZPRSA-N 0.000 description 1
- NNFMANHDYSVNIO-DCAQKATOSA-N Ser-Lys-Arg Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O NNFMANHDYSVNIO-DCAQKATOSA-N 0.000 description 1
- CRJZZXMAADSBBQ-SRVKXCTJSA-N Ser-Lys-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CO CRJZZXMAADSBBQ-SRVKXCTJSA-N 0.000 description 1
- FHXGMDRKJHKLKW-QWRGUYRKSA-N Ser-Tyr-Gly Chemical compound OC[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CC1=CC=C(O)C=C1 FHXGMDRKJHKLKW-QWRGUYRKSA-N 0.000 description 1
- 108091008874 T cell receptors Proteins 0.000 description 1
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 1
- 102100027222 T-lymphocyte activation antigen CD80 Human genes 0.000 description 1
- HATRDXDCPOXQJX-UHFFFAOYSA-N Thapsigargin Natural products CCCCCCCC(=O)OC1C(OC(O)C(=C/C)C)C(=C2C3OC(=O)C(C)(O)C3(O)C(CC(C)(OC(=O)C)C12)OC(=O)CCC)C HATRDXDCPOXQJX-UHFFFAOYSA-N 0.000 description 1
- UTCFSBBXPWKLTG-XKBZYTNZSA-N Thr-Cys-Gln Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N)O UTCFSBBXPWKLTG-XKBZYTNZSA-N 0.000 description 1
- AYCQVUUPIJHJTA-IXOXFDKPSA-N Thr-His-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC(C)C)C(O)=O AYCQVUUPIJHJTA-IXOXFDKPSA-N 0.000 description 1
- XTCNBOBTROGWMW-RWRJDSDZSA-N Thr-Ile-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@H]([C@@H](C)O)N XTCNBOBTROGWMW-RWRJDSDZSA-N 0.000 description 1
- AMXMBCAXAZUCFA-RHYQMDGZSA-N Thr-Leu-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O AMXMBCAXAZUCFA-RHYQMDGZSA-N 0.000 description 1
- XHWCDRUPDNSDAZ-XKBZYTNZSA-N Thr-Ser-Glu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N)O XHWCDRUPDNSDAZ-XKBZYTNZSA-N 0.000 description 1
- AHERARIZBPOMNU-KATARQTJSA-N Thr-Ser-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O AHERARIZBPOMNU-KATARQTJSA-N 0.000 description 1
- WPSKTVVMQCXPRO-BWBBJGPYSA-N Thr-Ser-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WPSKTVVMQCXPRO-BWBBJGPYSA-N 0.000 description 1
- RVMNUBQWPVOUKH-HEIBUPTGSA-N Thr-Ser-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O RVMNUBQWPVOUKH-HEIBUPTGSA-N 0.000 description 1
- NDZYTIMDOZMECO-SHGPDSBTSA-N Thr-Thr-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O NDZYTIMDOZMECO-SHGPDSBTSA-N 0.000 description 1
- COYHRQWNJDJCNA-NUJDXYNKSA-N Thr-Thr-Thr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O COYHRQWNJDJCNA-NUJDXYNKSA-N 0.000 description 1
- PWONLXBUSVIZPH-RHYQMDGZSA-N Thr-Val-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N)O PWONLXBUSVIZPH-RHYQMDGZSA-N 0.000 description 1
- HRKOLWXWQSDMSK-XIRDDKMYSA-N Trp-Glu-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)N HRKOLWXWQSDMSK-XIRDDKMYSA-N 0.000 description 1
- VMXLNDRJXVAJFT-JYBASQMISA-N Trp-Thr-Ser Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)N)O VMXLNDRJXVAJFT-JYBASQMISA-N 0.000 description 1
- 102100033728 Tumor necrosis factor receptor superfamily member 18 Human genes 0.000 description 1
- 102100036856 Tumor necrosis factor receptor superfamily member 9 Human genes 0.000 description 1
- 206010050283 Tumour ulceration Diseases 0.000 description 1
- HKIUVWMZYFBIHG-KKUMJFAQSA-N Tyr-Arg-Gln Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N)O HKIUVWMZYFBIHG-KKUMJFAQSA-N 0.000 description 1
- WOAQYWUEUYMVGK-ULQDDVLXSA-N Tyr-Lys-Arg Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O WOAQYWUEUYMVGK-ULQDDVLXSA-N 0.000 description 1
- 208000025865 Ulcer Diseases 0.000 description 1
- 206010046865 Vaccinia virus infection Diseases 0.000 description 1
- SLLKXDSRVAOREO-KZVJFYERSA-N Val-Ala-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C)NC(=O)[C@H](C(C)C)N)O SLLKXDSRVAOREO-KZVJFYERSA-N 0.000 description 1
- QPZMOUMNTGTEFR-ZKWXMUAHSA-N Val-Asn-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](C(C)C)N QPZMOUMNTGTEFR-ZKWXMUAHSA-N 0.000 description 1
- DDNIHOWRDOXXPF-NGZCFLSTSA-N Val-Asp-Pro Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N1CCC[C@@H]1C(=O)O)N DDNIHOWRDOXXPF-NGZCFLSTSA-N 0.000 description 1
- HQYVQDRYODWONX-DCAQKATOSA-N Val-His-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CO)C(=O)O)N HQYVQDRYODWONX-DCAQKATOSA-N 0.000 description 1
- JZWZACGUZVCQPS-RNJOBUHISA-N Val-Ile-Pro Chemical compound CC[C@H](C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](C(C)C)N JZWZACGUZVCQPS-RNJOBUHISA-N 0.000 description 1
- MYLNLEIZWHVENT-VKOGCVSHSA-N Val-Ile-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](C(C)C)N MYLNLEIZWHVENT-VKOGCVSHSA-N 0.000 description 1
- YMTOEGGOCHVGEH-IHRRRGAJSA-N Val-Lys-Lys Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(O)=O YMTOEGGOCHVGEH-IHRRRGAJSA-N 0.000 description 1
- HTONZBWRYUKUKC-RCWTZXSCSA-N Val-Thr-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O HTONZBWRYUKUKC-RCWTZXSCSA-N 0.000 description 1
- JXCOEPXCBVCTRD-JYJNAYRXSA-N Val-Tyr-Arg Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N JXCOEPXCBVCTRD-JYJNAYRXSA-N 0.000 description 1
- 238000004833 X-ray photoelectron spectroscopy Methods 0.000 description 1
- 238000012452 Xenomouse strains Methods 0.000 description 1
- IEDXPSOJFSVCKU-HOKPPMCLSA-N [4-[[(2S)-5-(carbamoylamino)-2-[[(2S)-2-[6-(2,5-dioxopyrrolidin-1-yl)hexanoylamino]-3-methylbutanoyl]amino]pentanoyl]amino]phenyl]methyl N-[(2S)-1-[[(2S)-1-[[(3R,4S,5S)-1-[(2S)-2-[(1R,2R)-3-[[(1S,2R)-1-hydroxy-1-phenylpropan-2-yl]amino]-1-methoxy-2-methyl-3-oxopropyl]pyrrolidin-1-yl]-3-methoxy-5-methyl-1-oxoheptan-4-yl]-methylamino]-3-methyl-1-oxobutan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]-N-methylcarbamate Chemical compound CC[C@H](C)[C@@H]([C@@H](CC(=O)N1CCC[C@H]1[C@H](OC)[C@@H](C)C(=O)N[C@H](C)[C@@H](O)c1ccccc1)OC)N(C)C(=O)[C@@H](NC(=O)[C@H](C(C)C)N(C)C(=O)OCc1ccc(NC(=O)[C@H](CCCNC(N)=O)NC(=O)[C@@H](NC(=O)CCCCCN2C(=O)CCC2=O)C(C)C)cc1)C(C)C IEDXPSOJFSVCKU-HOKPPMCLSA-N 0.000 description 1
- 238000002679 ablation Methods 0.000 description 1
- 239000002250 absorbent Substances 0.000 description 1
- 230000002745 absorbent Effects 0.000 description 1
- 238000010521 absorption reaction Methods 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 230000003213 activating effect Effects 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 208000009956 adenocarcinoma Diseases 0.000 description 1
- 230000009824 affinity maturation Effects 0.000 description 1
- 108010041407 alanylaspartic acid Proteins 0.000 description 1
- 150000001298 alcohols Chemical class 0.000 description 1
- 108700025316 aldesleukin Proteins 0.000 description 1
- 229960005310 aldesleukin Drugs 0.000 description 1
- 208000026935 allergic disease Diseases 0.000 description 1
- 231100000360 alopecia Toxicity 0.000 description 1
- KOSRFJWDECSPRO-UHFFFAOYSA-N alpha-L-glutamyl-L-glutamic acid Natural products OC(=O)CCC(N)C(=O)NC(CCC(O)=O)C(O)=O KOSRFJWDECSPRO-UHFFFAOYSA-N 0.000 description 1
- 229940035676 analgesics Drugs 0.000 description 1
- 125000000129 anionic group Chemical group 0.000 description 1
- 239000000730 antalgic agent Substances 0.000 description 1
- 229940045799 anthracyclines and related substance Drugs 0.000 description 1
- 230000003466 anti-cipated effect Effects 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 210000000628 antibody-producing cell Anatomy 0.000 description 1
- 239000003080 antimitotic agent Substances 0.000 description 1
- 230000005975 antitumor immune response Effects 0.000 description 1
- 210000000436 anus Anatomy 0.000 description 1
- 230000006907 apoptotic process Effects 0.000 description 1
- 239000012736 aqueous medium Substances 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 210000004507 artificial chromosome Anatomy 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 229940009098 aspartate Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 239000012131 assay buffer Substances 0.000 description 1
- 229940090047 auto-injector Drugs 0.000 description 1
- 229960002903 benzyl benzoate Drugs 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 238000007622 bioinformatic analysis Methods 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 206010005084 bladder transitional cell carcinoma Diseases 0.000 description 1
- 201000001528 bladder urothelial carcinoma Diseases 0.000 description 1
- 230000017531 blood circulation Effects 0.000 description 1
- GXJABQQUPOEUTA-RDJZCZTQSA-N bortezomib Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)B(O)O)NC(=O)C=1N=CC=NC=1)C1=CC=CC=C1 GXJABQQUPOEUTA-RDJZCZTQSA-N 0.000 description 1
- 229960001467 bortezomib Drugs 0.000 description 1
- 239000001110 calcium chloride Substances 0.000 description 1
- 229910001628 calcium chloride Inorganic materials 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 230000004611 cancer cell death Effects 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 235000014633 carbohydrates Nutrition 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 229940097217 cardiac glycoside Drugs 0.000 description 1
- 239000002368 cardiac glycoside Substances 0.000 description 1
- 239000004359 castor oil Substances 0.000 description 1
- 235000019438 castor oil Nutrition 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 125000002091 cationic group Chemical group 0.000 description 1
- 230000020411 cell activation Effects 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 230000006041 cell recruitment Effects 0.000 description 1
- 238000002659 cell therapy Methods 0.000 description 1
- 230000019522 cellular metabolic process Effects 0.000 description 1
- 230000036755 cellular response Effects 0.000 description 1
- 229960005395 cetuximab Drugs 0.000 description 1
- 238000010516 chain-walking reaction Methods 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- 229910052801 chlorine Inorganic materials 0.000 description 1
- 239000000460 chlorine Substances 0.000 description 1
- 230000002759 chromosomal effect Effects 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 206010073251 clear cell renal cell carcinoma Diseases 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 238000011198 co-culture assay Methods 0.000 description 1
- 230000004186 co-expression Effects 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 230000002301 combined effect Effects 0.000 description 1
- 230000024203 complement activation Effects 0.000 description 1
- 239000002299 complementary DNA Substances 0.000 description 1
- 238000013329 compounding Methods 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 238000012937 correction Methods 0.000 description 1
- 239000003246 corticosteroid Substances 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 229940109239 creatinine Drugs 0.000 description 1
- 238000002425 crystallisation Methods 0.000 description 1
- 230000008025 crystallization Effects 0.000 description 1
- 208000035250 cutaneous malignant susceptibility to 1 melanoma Diseases 0.000 description 1
- 208000017763 cutaneous neuroendocrine carcinoma Diseases 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 230000002939 deleterious effect Effects 0.000 description 1
- 239000003599 detergent Substances 0.000 description 1
- FSXRLASFHBWESK-UHFFFAOYSA-N dipeptide phenylalanyl-tyrosine Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FSXRLASFHBWESK-UHFFFAOYSA-N 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 150000002019 disulfides Chemical class 0.000 description 1
- 230000003828 downregulation Effects 0.000 description 1
- 230000007783 downstream signaling Effects 0.000 description 1
- 229960004679 doxorubicin Drugs 0.000 description 1
- 230000001804 emulsifying effect Effects 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 208000030172 endocrine system disease Diseases 0.000 description 1
- 239000012645 endogenous antigen Substances 0.000 description 1
- 210000002889 endothelial cell Anatomy 0.000 description 1
- 229960001433 erlotinib Drugs 0.000 description 1
- AAKJLRGGTJKAMG-UHFFFAOYSA-N erlotinib Chemical compound C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 AAKJLRGGTJKAMG-UHFFFAOYSA-N 0.000 description 1
- 201000005619 esophageal carcinoma Diseases 0.000 description 1
- 210000003238 esophagus Anatomy 0.000 description 1
- 230000017188 evasion or tolerance of host immune response Effects 0.000 description 1
- 230000003203 everyday effect Effects 0.000 description 1
- 201000005884 exanthem Diseases 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 238000000684 flow cytometry Methods 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 230000003325 follicular Effects 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 244000053095 fungal pathogen Species 0.000 description 1
- 108010063718 gamma-glutamylaspartic acid Proteins 0.000 description 1
- 229960002584 gefitinib Drugs 0.000 description 1
- XGALLCVXEZPNRQ-UHFFFAOYSA-N gefitinib Chemical compound C=12C=C(OCCCN3CCOCC3)C(OC)=CC2=NC=NC=1NC1=CC=C(F)C(Cl)=C1 XGALLCVXEZPNRQ-UHFFFAOYSA-N 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- 210000001280 germinal center Anatomy 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 229930195712 glutamate Natural products 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- ZEMPKEQAKRGZGQ-XOQCFJPHSA-N glycerol triricinoleate Natural products CCCCCC[C@@H](O)CC=CCCCCCCCC(=O)OC[C@@H](COC(=O)CCCCCCCC=CC[C@@H](O)CCCCCC)OC(=O)CCCCCCCC=CC[C@H](O)CCCCCC ZEMPKEQAKRGZGQ-XOQCFJPHSA-N 0.000 description 1
- XBGGUPMXALFZOT-UHFFFAOYSA-N glycyl-L-tyrosine hemihydrate Natural products NCC(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 XBGGUPMXALFZOT-UHFFFAOYSA-N 0.000 description 1
- 108010015792 glycyllysine Proteins 0.000 description 1
- 108010087823 glycyltyrosine Proteins 0.000 description 1
- 208000035474 group of disease Diseases 0.000 description 1
- 230000035876 healing Effects 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 230000004217 heart function Effects 0.000 description 1
- 210000002443 helper t lymphocyte Anatomy 0.000 description 1
- 208000024200 hematopoietic and lymphoid system neoplasm Diseases 0.000 description 1
- 229940022353 herceptin Drugs 0.000 description 1
- 231100000086 high toxicity Toxicity 0.000 description 1
- 231100000171 higher toxicity Toxicity 0.000 description 1
- 239000000710 homodimer Substances 0.000 description 1
- 239000005556 hormone Substances 0.000 description 1
- 229940088597 hormone Drugs 0.000 description 1
- 229940062714 humalog mix Drugs 0.000 description 1
- 102000048776 human CD274 Human genes 0.000 description 1
- 230000028996 humoral immune response Effects 0.000 description 1
- 230000008348 humoral response Effects 0.000 description 1
- 150000007857 hydrazones Chemical group 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- MPGWGYQTRSNGDD-UHFFFAOYSA-N hypericin Chemical compound OC1=CC(O)=C(C2=O)C3=C1C1C(O)=CC(=O)C(C4=O)=C1C1=C3C3=C2C(O)=CC(C)=C3C2=C1C4=C(O)C=C2C MPGWGYQTRSNGDD-UHFFFAOYSA-N 0.000 description 1
- 229940005608 hypericin Drugs 0.000 description 1
- PHOKTTKFQUYZPI-UHFFFAOYSA-N hypericin Natural products Cc1cc(O)c2c3C(=O)C(=Cc4c(O)c5c(O)cc(O)c6c7C(=O)C(=Cc8c(C)c1c2c(c78)c(c34)c56)O)O PHOKTTKFQUYZPI-UHFFFAOYSA-N 0.000 description 1
- 230000009610 hypersensitivity Effects 0.000 description 1
- 229960000908 idarubicin Drugs 0.000 description 1
- 238000003384 imaging method Methods 0.000 description 1
- 230000005934 immune activation Effects 0.000 description 1
- 230000008004 immune attack Effects 0.000 description 1
- 230000008073 immune recognition Effects 0.000 description 1
- 230000036039 immunity Effects 0.000 description 1
- 238000003018 immunoassay Methods 0.000 description 1
- 230000005847 immunogenicity Effects 0.000 description 1
- 230000016784 immunoglobulin production Effects 0.000 description 1
- 230000002055 immunohistochemical effect Effects 0.000 description 1
- 238000013388 immunohistochemistry analysis Methods 0.000 description 1
- 210000000428 immunological synapse Anatomy 0.000 description 1
- 210000005008 immunosuppressive cell Anatomy 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 108020004201 indoleamine 2,3-dioxygenase Proteins 0.000 description 1
- 102000006639 indoleamine 2,3-dioxygenase Human genes 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 238000011081 inoculation Methods 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000010255 intramuscular injection Methods 0.000 description 1
- 239000007927 intramuscular injection Substances 0.000 description 1
- 239000007928 intraperitoneal injection Substances 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 239000000644 isotonic solution Substances 0.000 description 1
- 235000015110 jellies Nutrition 0.000 description 1
- 238000012004 kinetic exclusion assay Methods 0.000 description 1
- 238000002372 labelling Methods 0.000 description 1
- YJAGIIHSFUDVBG-OOEBKATBSA-N laga peptide Chemical compound C([C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](NC(=O)[C@H](C)N)CC(C)C)C(=O)N[C@@H](CCC(=O)OC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CCC(=O)OC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)N)C(=O)OC(=O)CC[C@H](NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)N)C(=O)N[C@@H](C)C(=O)OC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CCC(O)=O)C1C=NC=N1 YJAGIIHSFUDVBG-OOEBKATBSA-N 0.000 description 1
- 231100000518 lethal Toxicity 0.000 description 1
- 230000001665 lethal effect Effects 0.000 description 1
- 125000001909 leucine group Chemical group [H]N(*)C(C(*)=O)C([H])([H])C(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- 108010073472 leucyl-prolyl-proline Proteins 0.000 description 1
- 208000032839 leukemia Diseases 0.000 description 1
- 229940124590 live attenuated vaccine Drugs 0.000 description 1
- 229940023012 live-attenuated vaccine Drugs 0.000 description 1
- 201000007270 liver cancer Diseases 0.000 description 1
- 208000014018 liver neoplasm Diseases 0.000 description 1
- 201000005243 lung squamous cell carcinoma Diseases 0.000 description 1
- 231100001023 lymphopenia Toxicity 0.000 description 1
- 238000002826 magnetic-activated cell sorting Methods 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 231100000682 maximum tolerated dose Toxicity 0.000 description 1
- WKPWGQKGSOKKOO-RSFHAFMBSA-N maytansine Chemical class CO[C@@H]([C@@]1(O)C[C@](OC(=O)N1)([C@H]([C@@H]1O[C@@]1(C)[C@@H](OC(=O)[C@H](C)N(C)C(C)=O)CC(=O)N1C)C)[H])\C=C\C=C(C)\CC2=CC(OC)=C(Cl)C1=C2 WKPWGQKGSOKKOO-RSFHAFMBSA-N 0.000 description 1
- 238000010999 medical injection Methods 0.000 description 1
- 210000001806 memory b lymphocyte Anatomy 0.000 description 1
- 210000003071 memory t lymphocyte Anatomy 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 108091070501 miRNA Proteins 0.000 description 1
- 239000002679 microRNA Substances 0.000 description 1
- 231100000782 microtubule inhibitor Toxicity 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- KKZJGLLVHKMTCM-UHFFFAOYSA-N mitoxantrone Chemical compound O=C1C2=C(O)C=CC(O)=C2C(=O)C2=C1C(NCCNCCO)=CC=C2NCCNCCO KKZJGLLVHKMTCM-UHFFFAOYSA-N 0.000 description 1
- 229960001156 mitoxantrone Drugs 0.000 description 1
- 108010093470 monomethyl auristatin E Proteins 0.000 description 1
- 230000000877 morphologic effect Effects 0.000 description 1
- 210000002200 mouth mucosa Anatomy 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 230000000869 mutational effect Effects 0.000 description 1
- 210000001989 nasopharynx Anatomy 0.000 description 1
- 201000011216 nasopharynx carcinoma Diseases 0.000 description 1
- 230000010807 negative regulation of binding Effects 0.000 description 1
- 230000001613 neoplastic effect Effects 0.000 description 1
- 230000004770 neurodegeneration Effects 0.000 description 1
- 208000015122 neurodegenerative disease Diseases 0.000 description 1
- 230000002314 neuroinflammatory effect Effects 0.000 description 1
- 210000000440 neutrophil Anatomy 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 210000004882 non-tumor cell Anatomy 0.000 description 1
- 239000002736 nonionic surfactant Substances 0.000 description 1
- 201000011330 nonpapillary renal cell carcinoma Diseases 0.000 description 1
- 230000009871 nonspecific binding Effects 0.000 description 1
- 238000010606 normalization Methods 0.000 description 1
- 230000005257 nucleotidylation Effects 0.000 description 1
- 235000019198 oils Nutrition 0.000 description 1
- 239000002674 ointment Substances 0.000 description 1
- 229960003104 ornithine Drugs 0.000 description 1
- 210000003300 oropharynx Anatomy 0.000 description 1
- 201000006958 oropharynx cancer Diseases 0.000 description 1
- 230000001151 other effect Effects 0.000 description 1
- 231100000262 ototoxicity Toxicity 0.000 description 1
- 201000010302 ovarian serous cystadenocarcinoma Diseases 0.000 description 1
- 210000001672 ovary Anatomy 0.000 description 1
- DWAFYCQODLXJNR-BNTLRKBRSA-L oxaliplatin Chemical compound O1C(=O)C(=O)O[Pt]11N[C@@H]2CCCC[C@H]2N1 DWAFYCQODLXJNR-BNTLRKBRSA-L 0.000 description 1
- 229960001756 oxaliplatin Drugs 0.000 description 1
- NMHMNPHRMNGLLB-UHFFFAOYSA-N p-hydroxybenzene propanoic acid Natural products OC(=O)CCC1=CC=C(O)C=C1 NMHMNPHRMNGLLB-UHFFFAOYSA-N 0.000 description 1
- 238000004806 packaging method and process Methods 0.000 description 1
- 238000002638 palliative care Methods 0.000 description 1
- 239000006072 paste Substances 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 229940090048 pen injector Drugs 0.000 description 1
- 230000035515 penetration Effects 0.000 description 1
- 210000003899 penis Anatomy 0.000 description 1
- 210000005259 peripheral blood Anatomy 0.000 description 1
- 239000011886 peripheral blood Substances 0.000 description 1
- 208000033808 peripheral neuropathy Diseases 0.000 description 1
- 229940021222 peritoneal dialysis isotonic solution Drugs 0.000 description 1
- 230000002688 persistence Effects 0.000 description 1
- 230000000144 pharmacologic effect Effects 0.000 description 1
- WVDDGKGOMKODPV-ZQBYOMGUSA-N phenyl(114C)methanol Chemical compound O[14CH2]C1=CC=CC=C1 WVDDGKGOMKODPV-ZQBYOMGUSA-N 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- 108010018625 phenylalanylarginine Proteins 0.000 description 1
- VGEREEWJJVICBM-UHFFFAOYSA-N phloretin Chemical compound C1=CC(O)=CC=C1CCC(=O)C1=C(O)C=C(O)C=C1O VGEREEWJJVICBM-UHFFFAOYSA-N 0.000 description 1
- 150000003906 phosphoinositides Chemical class 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000002186 photoactivation Effects 0.000 description 1
- 239000002504 physiological saline solution Substances 0.000 description 1
- 229950010773 pidilizumab Drugs 0.000 description 1
- 239000006187 pill Substances 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 239000000244 polyoxyethylene sorbitan monooleate Substances 0.000 description 1
- 235000010482 polyoxyethylene sorbitan monooleate Nutrition 0.000 description 1
- 229920000053 polysorbate 80 Polymers 0.000 description 1
- 229940068968 polysorbate 80 Drugs 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 238000012805 post-processing Methods 0.000 description 1
- 208000017805 post-transplant lymphoproliferative disease Diseases 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 238000007781 pre-processing Methods 0.000 description 1
- 150000003141 primary amines Chemical class 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 230000007112 pro inflammatory response Effects 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 108010070643 prolylglutamic acid Proteins 0.000 description 1
- 238000011321 prophylaxis Methods 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 238000002818 protein evolution Methods 0.000 description 1
- 108060006633 protein kinase Proteins 0.000 description 1
- SSKVDVBQSWQEGJ-UHFFFAOYSA-N pseudohypericin Natural products C12=C(O)C=C(O)C(C(C=3C(O)=CC(O)=C4C=33)=O)=C2C3=C2C3=C4C(C)=CC(O)=C3C(=O)C3=C(O)C=C(O)C1=C32 SSKVDVBQSWQEGJ-UHFFFAOYSA-N 0.000 description 1
- 230000005855 radiation Effects 0.000 description 1
- 206010037844 rash Diseases 0.000 description 1
- 238000003259 recombinant expression Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 208000016691 refractory malignant neoplasm Diseases 0.000 description 1
- 230000008929 regeneration Effects 0.000 description 1
- 238000011069 regeneration method Methods 0.000 description 1
- 230000000284 resting effect Effects 0.000 description 1
- 210000001525 retina Anatomy 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000002702 ribosome display Methods 0.000 description 1
- 102200124087 rs76057237 Human genes 0.000 description 1
- 239000012146 running buffer Substances 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 238000009094 second-line therapy Methods 0.000 description 1
- 238000011519 second-line treatment Methods 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 239000008159 sesame oil Substances 0.000 description 1
- 235000011803 sesame oil Nutrition 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- 231100000046 skin rash Toxicity 0.000 description 1
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 1
- 239000008247 solid mixture Substances 0.000 description 1
- 239000000243 solution Substances 0.000 description 1
- 230000000392 somatic effect Effects 0.000 description 1
- 239000003549 soybean oil Substances 0.000 description 1
- 235000012424 soybean oil Nutrition 0.000 description 1
- 125000006850 spacer group Chemical group 0.000 description 1
- 238000009987 spinning Methods 0.000 description 1
- 230000006641 stabilisation Effects 0.000 description 1
- 238000011105 stabilization Methods 0.000 description 1
- 238000010186 staining Methods 0.000 description 1
- 238000011272 standard treatment Methods 0.000 description 1
- 238000001370 static light scattering Methods 0.000 description 1
- 229930002534 steroid glycoside Natural products 0.000 description 1
- 150000008143 steroidal glycosides Chemical class 0.000 description 1
- 238000010254 subcutaneous injection Methods 0.000 description 1
- 239000007929 subcutaneous injection Substances 0.000 description 1
- 150000005846 sugar alcohols Polymers 0.000 description 1
- 125000000472 sulfonyl group Chemical group *S(*)(=O)=O 0.000 description 1
- 239000000829 suppository Substances 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 208000011580 syndromic disease Diseases 0.000 description 1
- 238000009121 systemic therapy Methods 0.000 description 1
- 239000003826 tablet Substances 0.000 description 1
- 230000002123 temporal effect Effects 0.000 description 1
- 230000002381 testicular Effects 0.000 description 1
- 150000003568 thioethers Chemical class 0.000 description 1
- 230000000699 topical effect Effects 0.000 description 1
- 125000002088 tosyl group Chemical group [H]C1=C([H])C(=C([H])C([H])=C1C([H])([H])[H])S(*)(=O)=O 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 108700012359 toxins Proteins 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 238000011222 transcriptome analysis Methods 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 102000035160 transmembrane proteins Human genes 0.000 description 1
- 108091005703 transmembrane proteins Proteins 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 229950007217 tremelimumab Drugs 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- 230000001960 triggered effect Effects 0.000 description 1
- 201000008827 tuberculosis Diseases 0.000 description 1
- 230000005751 tumor progression Effects 0.000 description 1
- 229910052721 tungsten Inorganic materials 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 231100000397 ulcer Toxicity 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 241001430294 unidentified retrovirus Species 0.000 description 1
- 230000003827 upregulation Effects 0.000 description 1
- 208000007089 vaccinia Diseases 0.000 description 1
- 210000001215 vagina Anatomy 0.000 description 1
- 238000010200 validation analysis Methods 0.000 description 1
- OPUPHQHVRPYOTC-UHFFFAOYSA-N vgf3hm1rrf Chemical compound C1=NC(C(=O)C=2C3=CC=CN=2)=C2C3=NC=CC2=C1 OPUPHQHVRPYOTC-UHFFFAOYSA-N 0.000 description 1
- 230000009278 visceral effect Effects 0.000 description 1
- 230000000007 visual effect Effects 0.000 description 1
- 210000003905 vulva Anatomy 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
- 239000001993 wax Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
- C07K16/2818—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily against CD28 or CD152
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
- C07K16/2827—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily against B7 molecules, e.g. CD80, CD86
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
- A61K2039/507—Comprising a combination of two or more separate antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/545—Medicinal preparations containing antigens or antibodies characterised by the dose, timing or administration schedule
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/57—Medicinal preparations containing antigens or antibodies characterised by the type of response, e.g. Th1, Th2
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/74—Inducing cell proliferation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/75—Agonist effect on antigen
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
Landscapes
- Health & Medical Sciences (AREA)
- Immunology (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Medicinal Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- General Health & Medical Sciences (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Biophysics (AREA)
- Biochemistry (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Peptides Or Proteins (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
Abstract
예를 들어, 항-PD-L1 항체 또는 이의 항원 결합 단편을 사용하여, 단독치료법 또는 조합 치료법으로서, 조절 T 세포와 효과기 T 세포 사이의 비를 조절하고/하거나, 환자의 면역계를 자극하고/하거나 종양 또는 암을 치료하기 위한 항-ICOS 항체 또는 이의 항원 결합 단편의 치료 용도 및 투약 요법.
Description
본 출원은 2021년 5월 18일에 출원된 미국 특허 가출원 일련 번호 제63/190,016호의 이익을 주장하며, 전체 개시내용이 본원에 참조로 포함된다.
ASCII 텍스트 파일(이름: 728466-SA9-642PC_SL_ST25.txt; 크기: 290.6 KB; 생성일: 2022년 5월 17일)로 전자적으로 제출된 서열 목록의 내용은 그 전체가 본원에 참조로 포함된다.
1.2. 발명의 분야
본 발명은 포유동물 면역 반응, 특히 T 세포 반응을 자극하기 위한 항-ICOS 항체(전장 항체 또는 이의 항원-결합 단편을 포함할 수 있음)를 포함하는 조성물에 관한 것이다. 본 발명은 또한 환자의 항-종양 T 세포 반응의 촉진에 의한 항-종양 치료법을 포함하는 면역종양학에서의 이러한 조성물의 의학적 용도뿐만 아니라, 예를 들어 효과기 T 세포의 자극 및/또는 조절 T 세포의 고갈을 통해, 효과기 T 세포 활성에 유리하게 효과기 T 세포와 조절 T 세포 사이의 균형을 조절하여 치료적 이점을 갖는 다른 질환 및 질병에서의 조성물의 용도에 관한 것이다. 일부 구현예에서, 본 발명은 단독치료법으로서의 항-ICOS 항체에 관한 것이다. 다른 구현예에서, 본 발명은, 예를 들어 항-PD -L1 항체(전장 항체 또는 이의 항원-결합 단편을 포함할 수 있음)를 추가로 포함하는, 조합 치료법의 일부로서의 항-ICOS 항체에 관한 것이다. 본 발명은 또한 대상체에서 포유동물 면역 반응, 예를 들어 항-종양 T 세포 반응을 자극하는 데 놀랍도록 효과적인 항-ICOS 항체(단독치료법으로서 또는 조합 치료법의 일환으로서)의 투약량 및/또는 빈도에 관한 것이다.
1.3. 배경
ICOS(유도성 T 세포 공동-자극인자)는 면역 반응, 특히 체액성 면역 반응 조절에 관여하는 CD28 유전자 패밀리의 구성원으로, 1999년에 처음 확인되었다[1]. 이는 55 kDa의 막관통 단백질로, 두 개의 차별적으로 글리코실화된 서브유닛을 갖는 이황화 연결 동종이량체로 존재한다. ICOS는 T 림프구 상에서만 독점적으로 발현되며 다양한 T 세포 하위세트 상에서 발견된다. 이는 나이브 T 림프구에 낮은 수준으로 존재하지만 면역 활성화 시 그 발현이 빠르게 유도되며, TCR의 참여 및 CD28를 사용하는 공동 자극과 같은 친염증성 자극에 반응하여 상향조절된다[2, 3]. ICOS는 T 세포 활성화의 후기 단계, 기억 T 세포 형성에서 역할을 하며, 중요하게는 T 세포 의존적 B 세포 반응을 통한 체액성 반응의 조절에서 역할을 한다[4, 5]. 세포내에서 ICOS는 PI3K에 결합하고 포스포이노시티드 의존적 키나제 1(PDK1) 및 단백질 키나제 B(PKB)인, 키나제를 활성화한다. ICOS의 활성화는 세포 사멸을 예방하고 세포 대사를 상향조절한다. ICOS가 부재하거나(ICOS 녹아웃) 항-ICOS 중화 항체가 존재하는 경우 친염증성 반응이 억제될 수 있다.
ICOS는 B 세포 및 항원 제시 세포(APC) 상에 발현된 ICOS 리간드(ICOSL)에 결합한다[6, 7]. 공동 자극 분자로서 이는 TCR 매개 면역 반응 및 항원에 대한 항체 반응을 조절하는 역할을 한다. 이 세포 유형이 암 세포의 면역감시에서 부정적인 역할을 한다는 것이 제안되었으므로 - 난소암에서 이에 대한 새로운 증거가 있어서 - T 조절 세포 상에서 ICOS의 발현은 중요할 수 있다[8]. 중요하게는 ICOS 발현은 종양 미세환경에 존재하는 CD4+ 및 CD8+ 효과기 세포와 비교하여 종양내 조절 T 세포(TReg) 상에서 더 높은 것으로 보고되었다. Fc 매개 세포 효과기 기능을 갖는 항체를 사용한 TReg의 고갈은 전임상 모델에서 강력한 항-종양 유효성을 실증하였다[9]. ICOS가 동물 모델뿐만 아니라 면역 체크포인트 억제제로 치료된 환자 둘 모두에서의 항-종양 효과에 연루된다는 증거가 늘어나고 있다. ICOS 또는 ICOSL이 결핍된 마우스에서는 항-CTLA4 치료법의 항-종양 효과가 감소되는 반면[10] 정상 마우스에서는 ICOS 리간드가 흑색종 및 전립샘암에서 항-CTLA4 치료의 효과를 증가시킨다[11]. 더욱이, 인간에서의 진행성 흑색종 환자의 후향적 연구는 이필리무맙(항-CTLA4) 치료 후 증가된 ICOS 수준을 나타내었다[12]. 또한, ICOS 발현은 항-CTLA4로 치료된 방광암 환자에서 상향조절된다[13]. 또한 항-CTLA4 치료법으로 치료된 암 환자에서 종양 특이적 IFN□ 생성 CD4 T 세포의 대부분은 ICOS 양성인 반면 ICOS 양성 CD4 T 세포의 지속적인 증가는 생존과 상관관계가 있는 것으로 관찰되었다[12, 13, 14].
WO2016/120789는 항-ICOS 항체를 기재하고 T 세포 활성화 및 암, 감염성 질환 및/또는 패혈증 치료를 위한 이의 용도를 제안했다. 다수의 쥣과 항-ICOS 항체가 생성되었으며, 그 중 하위세트가 인간 ICOS 수용체의 효능제인 것으로 보고되었다. 항체 "422.2"가 선도 항-ICOS 항체로 선택되었고 인간화되어 "H2L5"로 지정된 인간 "IgG4PE" 항체를 생성했다. H2L5는 인간 ICOS에 대해 1.34 nM 및 게잡이원숭이 ICOS에 대해 0.95 nM의 친화도를 갖고, T 세포에서 사이토카인 생산을 유도하고, CD3 자극과 함께 T 세포 활성화 마커를 상향조절하는 것으로 보고되었다. 그러나 이식된 인간 흑색종 세포를 보유하는 마우스는 대조 처리군과 비교하여 H2L5 hIgG4PE로 치료될 때 최소 종양 성장 지연 또는 생존 증가만을 나타내는 것으로 보고되었다. 또한 이 항체는 이필리무맙 또는 펨브롤리주맙 단독치료법과 비교하여 이필리무맙(항-CTLA-4) 또는 펨브롤리주맙(항-PD-1)과의 조합 실험에서 종양 성장의 유의한 추가 억제를 일으키지 못했다. 마지막으로, 이식된 결장암 세포(CT26)를 보유하는 마우스에서 이필리무맙 또는 펨브롤리주맙의 마우스 대리물과 조합된 낮은 용량의 마우스 교차반응성 대리물 H2L5는 항-CTLA4 및 항-PD1 단독 치료법과 비교하여 전체 생존을 약간만 개선하였다. 이식된 EMT6 세포를 보유하는 마우스에서 강력한 치료적 이점의 유사한 결여가 나타났다.
WO2016/154177은 항-ICOS 항체의 추가 예를 기재하였다. 이들 항체는 효과기 CD8+ T 세포(TEff)를 포함한 CD4+ T 세포의 효능제이고 T 조절 세포(TReg)를 고갈시키는 것으로 보고되었다. TEff 대 TReg 세포에 대한 항체의 선택적 효과가 기재되었으며, 이에 따라 항체는 더 낮은 수준의 ICOS를 발현하는 TEff에 최소 영향을 미치면서 TReg를 우선적으로 고갈시킬 수 있었다. 항-ICOS 항체는 암 치료에서 사용하기 위해 제안되었으며, 항-PD-1 또는 항-PD-L1 항체와의 조합 치료법이 기재되었다.
1.4. 발명의 요약
효과기 T 세포 활성을 증가시키는 작용을 하는 ICOS에 대한 항체는 면역종양학 및 다양한 질환 및 질병 그리고 백신접종 요법을 포함하는 CD8+ T 세포 반응이 유익한 다른 의학적 상황에서의 치료적 접근을 나타낸다. 면역 성분이 관여되는 많은 질환 및 질병에서는 CD8+ T 세포 면역 반응을 발휘하는 효과기 T 세포(TEff)와 TEff를 하향조절하여 면역 반응을 억제하는 조절 T 세포(TReg) 사이에 균형이 존재한다. 본 발명은 효과기 T 세포 활성에 유리하게 이러한 TEff/TReg 균형을 조절하는 항체에 관한 것이다. ICOS 고도 양성 조절 T 세포의 고갈을 유발하는 항체는 TEff의 억제를 완화(relieve)시키고, 따라서 효과기 T 세포 반응을 촉진하는 순 효과를 가질 것이다. 항-ICOS 항체에 대한 추가적 또는 보완적 메커니즘은 ICOS 수용체 수준에서의 효능작용 활성을 통해 효과기 T 세포 반응을 자극하는 것이다.
조절 T 세포(TReg)와 비교하여 효과기 T 세포(TEff) 상에서의 ICOS의 상대적 발현, 및 이들 세포 집단의 상대적 활성은 생체내 항-ICOS 항체의 전체 효과에 영향을 미칠 것이다. 예상되는 작용 방식은 효과기 T 세포의 효능작용과 ICOS 양성 조절 T 세포의 고갈을 조합한다. 이들 두 개의 상이한 T 세포 집단에 대한 차별적이고 심지어 반대되는 효과는 이의 상이한 수준의 ICOS 발현으로 인해 달성 가능할 수 있다. 항-ICOS 항체의 가변 영역 및 불변 영역 각각의 이중 조작은 CD8/TReg 비에 영향을 줌으로써 효과기 T 세포 반응에 긍정적 순 효과를 발휘하는 분자를 제공할 수 있다. ICOS 수용체를 활성화하는 효능제 항체의 항원-결합 도메인은 항체가 결합된 고도 발현 세포의 하향조절 및/또는 제거를 촉진하는 항체 불변(Fc) 영역과 조합될 수 있다. 효과기 양성 불변 영역은 예를 들어 항체 의존적 세포 매개 세포독성(ADCC) 또는 항체 의존적 세포 식세포작용(ADCP)을 촉진하기 위해 표적 세포(TReg)에 대해 세포 효과기 기능을 동원하기 위해 사용될 수 있다. 따라서 항체는 효과기 T 세포 활성화를 촉진하고 면역억제 T 조절 세포를 하향조절하는 역할 둘 모두를 할 수 있다. ICOS는 TEff보다 TReg 상에서 더 고도 발현되므로 TReg가 고갈되는 동안 Teff 기능이 촉진되어 T 세포 면역 반응(예를 들어, 항-종양 반응 또는 다른 치료적으로 유익한 T 세포 반응)의 순 증가를 초래하는 치료 균형이 달성될 수 있다.
몇몇 전임상 및 임상 연구는 종양 미세환경(TME)에서 높은 효과기 T-세포 대 T-reg 세포 비와 전체 생존 사이에 강한 양의 상관관계를 나타내었다. 난소암 환자에서 CD8:T-reg 세포의 비는 우수한 임상 결과의 지표인 것으로 보고되었다[15]. 이필루무맙(ipilumumab)을 제공받은 후 전이성 흑색종 환자에서 유사한 관찰이 이루어졌다[16]. 또한 전임상 연구에서 TME의 높은 효과기 세포:T-reg 비가 항-종양 반응과 관련된 것으로 나타났다[43].
본 발명은 놀랍게도 낮은 용량에서 유효성을 갖는 것들을 포함하여 인간 ICOS에 결합하는 항체를 제공한다. 항체는 ICOS 세포외 도메인을 표적화하고, 이에 따라 ICOS를 발현하는 T 세포에 결합한다. IFNγ 발현 및 분비를 증가시키는 능력에 의해 표시되는 바와 같이, ICOS에 대한 효능작용 효과를 갖도록 설계되고 이에 따라 효과기 T 세포의 기능을 향상시키는 항체의 예가 제공된다. 주지된 바와 같이, 항-ICOS 항체는 또한 이들이 결합하는 세포를 고갈시키도록 조작될 수 있으며, 이는 조절 T 세포를 우선적으로 하향조절하는 효과를 가질 것이며, 효과기 T 세포 반응에 대한 이들 세포의 억제 효과를 높이고 이에 따라 전체 효과기 T 세포 반응을 촉진한다. 이의 작용 메커니즘과 관계없이, 실시예에 나타낸 바와 같이, 본 발명에 따른 항-ICOS 항체가 T 세포 반응을 자극하고 생체내에서 항-종양 효과를 갖는다는 것이 경험적으로 실증되었다. 원하는 수준의 Fc 효과기 기능을 갖는 불변 영역을 포함하거나 적절한 경우 이러한 효과기 기능이 없는 것들과 같은 적절한 항체 형식의 선택을 통해 항-ICOS 항체는 효과기 T 세포 반응이 유익한 및/또는 조절 T 세포의 억제가 바람직한 질환 및 질병의 치료를 포함하는 다양한 의학적 상황에서의 사용을 위해 맞춤화될 수 있다.
예시적인 항체에는 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009가 포함되며, 그 서열은 본원에 제시되어 있다.
일부 구현예에서, 본 발명은 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시키는 것이 필요한 대상체에서 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법을 제공하며, 방법은 인간 및/또는 마우스 ICOS의 세포외 도메인에 결합하는 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함하고, 항-ICOS 항체 또는 이의 항원-결합 단편은 약 0.8 mg 내지 240 mg의 용량으로 대상체에 투여된다.
일부 양태에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법에서 사용되는 항-ICOS 항체 또는 이의 항원-결합 단편이 중쇄 상보성 결정 영역(HCDR) HCDR1, HCDR2 및 HCDR3, 그리고 경쇄 상보성 결정 영역(LCDR) LCDR1, LCDR2 및 LCDR3을 포함하고, (a) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 363, SEQ ID NO: 364, 및 SEQ ID NO: 365와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 370, SEQ ID NO: 371, SEQ ID NO: 372와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (b) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 377, SEQ ID NO: 378, 및 SEQ ID NO: 379와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 384, SEQ ID NO: 385, SEQ ID NO: 386과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (c) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 391, SEQ ID NO: 392, 및 SEQ ID NO: 393과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 398, SEQ ID NO: 399, SEQ ID NO: 400과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (d) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 405, SEQ ID NO: 406, 및 SEQ ID NO: 407과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 412, SEQ ID NO: 413, SEQ ID NO: 414와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (e) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 419, SEQ ID NO: 420, 및 SEQ ID NO: 421과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 426, SEQ ID NO: 427, SEQ ID NO: 428과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (f) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 435, SEQ ID NO: 436, 및 SEQ ID NO: 437과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 442, SEQ ID NO: 443, SEQ ID NO: 444와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (g) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 449, SEQ ID NO: 450, 및 SEQ ID NO: 451과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 456, SEQ ID NO: 457, SEQ ID NO: 458과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (h) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 463, SEQ ID NO: 464, 및 SEQ ID NO: 465와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 470, SEQ ID NO: 471, SEQ ID NO: 472와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (i) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 477, SEQ ID NO: 478, 및 SEQ ID NO: 479와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 484, SEQ ID NO: 485, SEQ ID NO: 486과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나, (j) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 491, SEQ ID NO: 492, 및 SEQ ID NO: 493과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 498, SEQ ID NO: 499, SEQ ID NO: 500과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함한다. 일부 구현예에서, (a) HCDR1은 아미노산 서열 SEQ ID NO: 363을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 364를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 365를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 370을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 371을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 372를 포함하거나; (b) HCDR1은 아미노산 서열 SEQ ID NO: 377을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 378을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 379를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 384를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 385를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 386을 포함하거나; (c) HCDR1은 아미노산 서열 SEQ ID NO: 391을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 392를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 393을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 398을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 399를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 400을 포함하거나; (d) HCDR1은 아미노산 서열 SEQ ID NO: 405를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 406을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 407을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 412를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 413을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 414를 포함하거나; (e) HCDR1은 아미노산 서열 SEQ ID NO: 419를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 420을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 421을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 426을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 427을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 428을 포함하거나; (f) HCDR1은 아미노산 서열 SEQ ID NO: 435를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 436을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 437을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 442를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 443을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 444를 포함하거나; (g) HCDR1은 아미노산 서열 SEQ ID NO: 449를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 450을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 451을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 456을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 457을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 458을 포함하거나; (h) HCDR1은 아미노산 서열 SEQ ID NO: 463을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 464를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 465를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 470을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 471을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 472를 포함하거나; (i) HCDR1은 아미노산 서열 SEQ ID NO: 477을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 478을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 479를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 484를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 485를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 486을 포함하거나; (j) HCDR1은 아미노산 서열 SEQ ID NO: 491을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 492를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 493을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 498을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 499를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 500을 포함한다.
또 다른 구현예에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 아미노산 서열 SEQ ID NO: 405를 포함하는 HCDR1, 아미노산 서열 SEQ ID NO: 406을 포함하는 HCDR2, 아미노산 서열 SEQ ID NO: 407을 포함하는 HCDR3, 아미노산 서열 SEQ ID NO: 412를 포함하는 LCDR1, 아미노산 서열 SEQ ID NO: 413을 포함하는 LCDR2, 및 아미노산 서열 SEQ ID NO: 414를 포함하는 LCDR3을 포함하는, 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함한다.
또 다른 구현예에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하고, (a) VH 도메인은 아미노산 서열 SEQ ID NO: 366과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 373과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (b) VH 도메인은 아미노산 서열 SEQ ID NO: 380과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 387과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (c) VH 도메인은 아미노산 서열 SEQ ID NO: 394와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 401과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (d) VH 도메인은 아미노산 서열 SEQ ID NO: 408과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 415와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (e) VH 도메인은 아미노산 서열 SEQ ID NO: 422와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 429와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (f) VH 도메인은 아미노산 서열 SEQ ID NO: 438과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 445와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (g) VH 도메인은 아미노산 서열 SEQ ID NO: 452와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 459와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (h) VH 도메인은 아미노산 서열 SEQ ID NO: 467과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 473과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (i) VH 도메인은 아미노산 서열 SEQ ID NO 481:과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 488과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (j) VH 도메인은 아미노산 서열 SEQ ID NO: 494와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 501과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하는, 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, (a) VH 도메인은 아미노산 서열 SEQ ID NO: 366을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 373을 포함하거나; (b) VH 도메인은 아미노산 서열 SEQ ID NO: 380을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 387을 포함하거나; (c) VH 도메인은 아미노산 서열 SEQ ID NO: 394를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 401을 포함하거나; (d) VH 도메인은 아미노산 서열 SEQ ID NO: 408을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 415를 포함하거나; (e) VH 도메인은 아미노산 서열 SEQ ID NO: 422를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 429를 포함하거나; (f) VH 도메인은 아미노산 서열 SEQ ID NO: 438을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 445를 포함하거나; (g) VH 도메인은 아미노산 서열 SEQ ID NO: 452를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 459를 포함하거나; (h) VH 도메인은 아미노산 서열 SEQ ID NO: 467을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 473을 포함하거나; (i) VH 도메인은 아미노산 서열 SEQ ID NO: 480을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 487을 포함하거나; (j) VH 도메인은 아미노산 서열 SEQ ID NO: 494를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 501을 포함한다.
또 다른 구현예에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 SEQ ID NO: 408과 적어도 95% 서열 동일성을 갖는 서열을 포함하는 VH 도메인 및 SEQ ID NO: 415와 적어도 95% 서열 동일성을 갖는 서열을 포함하는 VL 도메인을 포함하는, 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, VH 도메인은 아미노산 서열 SEQ ID NO: 408을 포함하고, VL 도메인은 아미노산 서열 SEQ ID NO: 415를 포함한다.
또 다른 구현예에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 중쇄 및 경쇄를 포함하고, (a) 중쇄는 아미노산 서열 SEQ ID NO: 368과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 375와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (b) 중쇄는 아미노산 서열 SEQ ID NO: 385와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 389와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (c) 중쇄는 아미노산 서열 SEQ ID NO: 396과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 403과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (d) 중쇄는 아미노산 서열 SEQ ID NO: 410과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 417과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (e) 중쇄는 아미노산 서열 SEQ ID NO: 424와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 432와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (f) 중쇄는 아미노산 서열 SEQ ID NO: 440과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 447과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (g) 중쇄는 아미노산 서열 SEQ ID NO: 454와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 461과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (h) 중쇄는 아미노산 서열 SEQ ID NO: 468과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 475와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (i) 중쇄는 아미노산 서열 SEQ ID NO: 482와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 489와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나; (j) 중쇄는 아미노산 서열 SEQ ID NO: 496과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 503과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하는, 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, (a) 중쇄는 아미노산 서열 SEQ ID NO: 368을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 375를 포함하거나; (b) 중쇄는 아미노산 서열 SEQ ID NO: 382를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 389를 포함하거나; (c) 중쇄는 아미노산 서열 SEQ ID NO: 396을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 403을 포함하거나; (d) 중쇄는 아미노산 서열 SEQ ID NO: 410을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 417을 포함하거나; (e) 중쇄는 아미노산 서열 SEQ ID NO: 424를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 432를 포함하거나; (f) 중쇄는 아미노산 서열 SEQ ID NO: 440을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 447을 포함하거나; (g) 중쇄는 아미노산 서열 SEQ ID NO: 454를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 461을 포함하거나; (h) 중쇄는 아미노산 서열 SEQ ID NO: 468을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 475를 포함하거나; (i) 중쇄는 아미노산 서열 SEQ ID NO: 482를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 489를 포함하거나; (j) 중쇄는 아미노산 서열 SEQ ID NO: 496을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 503을 포함한다.
또 다른 구현예에서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 SEQ ID NO: 410과 적어도 95% 서열 동일성을 갖는 서열을 포함하는 중쇄 및 SEQ ID NO: 417과 적어도 95% 서열 동일성을 갖는 서열을 포함하는 경쇄를 포함하는, 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 중쇄는 아미노산 서열 SEQ ID NO: 410을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 417을 포함한다.
또 다른 구현예에서, 방법은 인간 IgG1 항체인 항-ICOS 항체를 투여하는 단계를 포함한다.
또 다른 구현예에서, 방법은 KY1044를 투여하는 단계를 포함한다.
또 다른 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 0.5 mg 내지 약 10 mg의 용량으로 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 0.8 mg 내지 약 8 mg의 용량으로 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 8 mg 미만의 용량으로(예를 들어, 7.5 mg 이하의 용량, 7 mg 이하의 용량으로) 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 0.8 mg 내지 약 2.4 mg의 용량으로 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 2.4 mg 내지 약 8 mg의 용량으로 대상체에 투여하는 단계를 포함한다.
또 다른 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 0.8 mg의 용량으로 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 2.4 mg의 용량으로 대상체에 투여하는 단계를 포함한다. 일부 구현예에서, 방법은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)을 약 8 mg의 용량으로 대상체에 투여하는 단계를 포함한다.
또 다른 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 2 내지 6주마다, 예를 들어 2주마다, 3주마다, 4주마다, 5주마다, 또는 6주마다 대상체에 투여된다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 3주마다 투여된다. 일부 구현예에서 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 6주마다 투여된다. 일부 구현예에서 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 매월 투여된다.
또 다른 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 1회 투여된다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 1회 초과로 투여된다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 적어도 6개월 동안, 예를 들어 6개월, 12개월, 또는 12개월 초과 동안 투여된다.
또 다른 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 단독치료법으로 투여된다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)은 조합 치료법으로 투여된다. 예를 들어, 일부 구현예에서 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법은 제2 치료제를 대상체에 투여하는 단계를 추가로 포함한다.
또 다른 구현예에서, 제2 치료제는 항-PD-L1 항체 또는 이의 항원 결합 단편을 포함한다. 일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙이다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 약 1200 mg의 용량으로 대상체에 투여된다.
또 다른 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 2 내지 6주마다, 예를 들어 2주마다, 3주마다, 4주마다, 5주마다 또는 6주마다 대상체에 투여된다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 3주마다 투여된다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 6주마다 투여된다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 매월 투여된다.
또 다른 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 1회 투여된다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 1회 초과로 투여된다. 일부 구현예에서, 항-PD-L1 항체 또는 이의 항원 결합 단편(예를 들어, 아테졸리주맙)은 적어도 6개월 동안, 예를 들어 6개월, 12개월, 또는 12개월 초과 동안 투여된다. 또 다른 구현예에서, 항-PD-L1 항체 또는 이의 항원-결합 단편은 항-ICOS 항체 또는 이의 항원-결합 단편과 함께 3주마다 대상체에 공동-투여된다.
또 다른 구현예에서, 항-PD-L1 항체 또는 이의 항원-결합 단편(예를 들어, 아테졸리주맙)은 항-ICOS 항체 또는 이의 항원-결합 단편(예를 들어, KY1044)과 교대 용량으로 대상체에 투여되며, 예를 들어 항-PD-L1 항체 또는 이의 항원-결합 단편은 3주마다 투여되고, 항-ICOS 항체 또는 이의 항원-결합 단편은 6주마다 투여된다.
또 다른 구현예에서, 방법은 종양을 치료하는 단계를 포함한다. 일부 구현예에서, 방법은 암을 치료하는 단계를 포함한다. 일부 구현예에서, 암은 진행성 및/또는 전이성 암을 포함한다. 일부 구현예에서, 암은 삼중 음성 유방암, 두경부 편평 세포 암종, 음경암, 췌장암, 비소세포 폐암, 간세포 암종, 식도암, 위암, 흑색종, 신장 세포 암종 및/또는 자궁경부암을 포함한다.
항체를 포함하는 약학적 조성물도 제공된다.
교차반응성 항체를 생성하기 위해 ICOS 녹아웃 동물이 사용되었다. 특히, ICOS 녹아웃 마우스에서 강한 역가가 얻어졌으며, 바람직한 교차반응성 항체를 포함하여, 항체 레퍼토리 중에서 고기능성 항체가 단리되었다. WO 2018/029474 A2를 참고한다(본원에 그 전체가 참조로 포함됨).
본 발명의 예시적인 구현예는 도면, 아래의 설명 및 첨부된 청구범위에 제시되어 있다.
1.5. 도면의 간략한 설명
이제 본 발명의 특정 양태 및 구현예가 첨부 도면을 참조하여 더 상세히 기재될 것이다.
도 1, 도 2, 도 3, 도 4: 실시예 1에 기재된 연구에 대해 마우스에서의 시간 경과에 따른 A20 종양의 부피를 나타내는 그래프. 각 처리군은 개별 동물의 종양 크기를 나타내는 스파이더 플롯으로 표시되며, 그룹당 n = 8이다. 각 그룹에 대해 종양의 징후가 없는(질환이 치유되었음을 도시함) 동물의 수는 그래프의 왼쪽 하단에 표시된다. 투약은 종양 세포 이식 후 제8일, 제11일, 제15일, 제18일, 제22일, 제25일 및 제29일에 수행되었으며 투약 시간은 회색 음영 영역으로 표시된다. 대조군(도 1) 및 항-PD-L1 처리군(도 2)과 비교하여, STIM001 mIgG2a(도 3) 및 STIM003 mIgG2a(도 4) 처리군은 A20 종양 성장의 유의한 억제를 나타내었다.
도 5: STIM001, STIM002B 및 관련 항체 CL-61091, CL-64536, CL-64837, CL-64841 및 CL-64912 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM002 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다.
도 6: 관련 항체 CL-71642 및 CL-74570 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM003 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다. 시퀀싱으로부터 얻은 항체 CL-71642의 VL 도메인은 N 말단 잔기 없이 본원에 나타낸다. 정렬로부터 전체 VH 도메인 서열이 N 말단 글루탐산을 포함할 것임을 알 수 있다.
도 7: STIM008 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM007 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다.
도 8: J558 동계 모델에서의 STIM003(항-ICOS) 및 AbW(항-PD-L1) mIgG2a 항체의 효과. 각 처리군은 개별 동물의 종양 크기를 나타내는 "스파이더 플롯"으로 표시된다(그룹당 n=10 또는 n=8). STIM003 단독치료법은 일부 유효성을 실증했고 8마리 중 3마리의 동물은 질환이 치유되었다. 유사하게 항-PDL1은 이 모델에서 효과적이었으며 8마리 중 6마리의 동물은 제37일까지 질환이 치유되었다. 항-PDL1 항체와 조합될 때 STIM003 mIgG2는 종양 성장을 완전히 억제하고 치유받은 동물의 생존을 개선하였다. 각 그룹에 대해 이의 질환이 치유된 동물의 수는 각각의 그래프의 오른쪽 하단에 표시된다. 투약일은 점선으로 표시된다(제11일, 제15일, 제18일, 제22일, 제25일 및 제29일).
도 9: 종양 조직 내의 상이한 TILS 세포 하위유형에 대한 ICOS 발현(양성 세포의 백분율 및 상대적 발현/dMFI)의 정량. (a) ICOS 발현에 대해 양성인 면역 세포 하위유형% 및 (b) 식염수 또는 항-PD-L1 또는 항-PD-1 대리물 항체로 처리된 동물의 면역 세포 하위유형의 ICOS dMFI(ICOS 양성 세포 상의 상대적 ICOS 발현). 제0일에 마우스에 1 x 106개의 생활성 세포/ml를 이식했다(n=7 또는 n=8). 제13일 및 제15일에 동물에 130 ug의 항체를 i.p. 투약했다. 조직 샘플을 제16일에 단리하고 분석했다. CD4+/FOXP3+ 세포는 TReg 집단(오른쪽 끝 그래프)에만 포함되었으며 모두 Foxp3 음성인 "효과기" CD4 세포(왼쪽 끝 그래프)로부터 제외되었다. 실시예 3을 참고한다.
도 10: A20 생체내 유효성 연구로부터의 데이터. 각 처리군은 개별 동물의 종양 크기를 나타내는 "스파이더 플롯"으로 표시된다(그룹당 n=10). 각 그룹에 대해 이의 질환이 치유된 동물의 수는 각각의 그래프에 표시된다. 다중 용량의 경우, 점선으로 표시된 제8일, 제11일, 제15일, 제18일, 제22일 및 제25일에 투약했다. 단일 용량의 경우, 동물은 제8일에만 IP 주사를 제공받았다. (도 10a) 식염수; (도 10b) STIM003 mIgG2a 다중 용량; (도 10c) STIM003 mIgG2a 단일 용량. 실시예 4를 참고한다.
도 11: STIM003 mIgG2a 60 μg 고정 용량을 사용하는 실시예 4에서 보고된 연구에 대한 카플란-마이어(Kaplan-Meier) 곡선. SD = 단일 용량, 제8일. MD = 제8일로부터의 다중 용량 BIW.
도 12: 식염수를 투약한 CT26 종양 보유 동물(시점당 n=4)로부터의 주요 T 세포 하위세트(T-reg[CD4+/FoxP3+], CD4 Eff[CD4+/FoxP3-] 세포 및 CD8+) 상의 ICOS 발현. 면역 세포 표현형 분석은 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 수행되었으며 모든 시점에 모든 조직에서 ICOS 발현에 대해 염색되었다. 도 12a 내지 도 12d는 4개의 상이한 조직에서 모든 시점에서의 ICOS 양성 세포의 백분율을 나타낸다. 도 12e 내지 도 12h는 4개의 상이한 조직 모두에서 모든 시점의 ICOS dMFI(상대적 발현)를 나타낸다. 실시예 5를 참고한다.
도 13: STIM003 mIgG2a 항체에 반응한 TME에서 T-reg 고갈을 실증하는 FACS 분석. CT-26 종양 보유 동물은 종양 세포 이식 후 제12일에 STIM003의 단일 용량(6, 60 또는 200 μg)으로 처리되었다. 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 FACS 분석을 위해 수확된 조직(시점당 n=4). 전체 종양내 T-reg 세포(CD4+CD25+Foxp3+)의 백분율(도 13a) 및 혈액 내 T-reg 세포의 백분율(도 13b)이 상이한 시점에 나타난다. 실시예 5를 참고한다.
도 14: STIM003 mIgG2a에 반응한 CD8:T-reg 및 CD4 eff:T-reg 비의 증가. CT-26 종양 보유 동물은 종양 세포 이식 후 제12일에 STIM003 mIgG2a의 단일 용량(6, 60 또는 200 μg)을 제공받았다. 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 FACS 분석을 위해 조직(시점당 n=4)이 수확되었고 T eff 대 T-reg 비가 계산되었다. (도 14a) 및 (도 14b), 종양 및 혈액내 CD8:T-reg 비, (도 14c) 및 (도 14d) 종양 및 혈액내 CD4-eff:T-reg 비. 실시예 5를 참고한다.
도 15: STIM003 처리는 TIL에 의한 증가된 탈과립화 및 Th1 사이토카인 생산과 상관관계가 있다. 처리 후 제8일에 TIL이 단리되고 FACS 분석이 수행되어 CD4 및 CD8 T 세포(도 15a 및 도 15b) 상의 CD107a 발현을 검출했다. 동시에, 해리된 종양의 세포가 브레펠딘-A의 존재 하에 4시간 동안 휴지되고, 세포가 T 세포 마커에 대해 염색되고 세포내 염색을 위해 투과화되어 IFN-γ 및 TNF-α(도 15c 내지 도 15h)를 검출했다. 실시예 5를 참고한다.
도 16a: ICOS 양성 CD4 기억 세포(ICOS+CD3+CD4+FoxP3-CD45RA-로 정의됨)에 대한 KY1044 표적 참여의 증거. Y축은 샘플 수집 날짜의 함수로 CD4 기억 세포의 점유 백분율을 측정한다. 혈액 샘플은 주기 1 제1일(C1D1), 주기 1 제8일(C1D8), 주기 2 제1일(C2D1) 및 주기 2 제8일(C2D8)에 수집되었다. 용량 수준 1 = 0.8 mg. 용량 수준 2 = 2.4 mg. 선은 동일한 환자의 데이터 포인트를 연결한다.
도 16b: ICOS 양성 CD4 기억 세포(ICOS+CD3+CD4+FoxP3-CD45RA-로 정의됨)에 대한 KY1044 표적 참여의 증거. Y축은 샘플 수집 날짜의 함수로 CD4 기억 세포의 점유 백분율을 측정한다. 혈액 샘플은 주기 1 제1일(C1D1), 주기 1 제8일(C1D8), 주기 2 제1일(C2D1) 및 주기 2 제8일(C2D8)에 수집되었다. 용량 수준 3 = 8 mg. 용량 수준 4 = 24 mg. 용량 수준 5 = 80 mg. 용량 수준 6 = 240 mg. 선은 동일한 환자의 데이터 포인트를 연결한다.
도 17a: 순환 사이토카인 수준을 측정함으로써 평가된 KY1044 의존적 효능작용. 실선 플롯은 평균을 표시하고 음영 영역은 KY1044로 처리된 환자에 대한 GM-CSF의 방문과 기준선 측정값 간 비의 95% 신뢰도 구간을 표시한다. 밝은 회색 데이터 포인트는 더 낮은 KY1044 용량 수준(0.8 mg 및 2.4 mg)을 받은 환자(n = 27)로부터 얻은 것이며, 불완전한 수용체 점유를 초래하였다. 진한 회색 데이터 포인트는 더 높은 KY1044 용량 수준(8 mg 이상)을 받은 환자(n = 14)로부터 얻은 것이며, 완전한 수용체 점유를 초래했다.
도 17b: 순환 사이토카인 수준을 측정함으로써 평가된 KY1044 의존적 효능작용. 실선 플롯은 평균을 표시하고 음영 영역은 KY1044로 처리된 환자에 대한 TNFα의 방문과 기준선 측정값 간 비의 95% 신뢰도 구간을 표시한다. 밝은 회색 데이터 포인트는 더 낮은 KY1044 용량 수준(0.8 mg 및 2.4 mg)을 받은 환자(n = 27)로부터 얻은 것이며, 불완전한 수용체 점유를 초래하였다. 진한 회색 데이터 포인트는 더 높은 KY1044 용량 수준(8 mg 이상)을 받은 환자(n = 14)로부터 얻은 것이며, 완전한 수용체 점유를 초래했다.
도 18a: 치료 지속기간에 관한 I/II상 임상 시험의 중간 결과. 등록된 모든 환자에 대한 치료 지속기간 중앙값은 9주였다.
도 18b: 치료법 요법 및 부분적 또는 완전 수용체 점유와 관련된 치료 지속기간을 나타내는, I/II상 임상 시험의 중간 결과.
도 18c: ICOS 수용체 점유와 관련된 치료 지속기간을 나타내는 I/II상 임상 시험의 중간 결과.
이제 본 발명의 특정 양태 및 구현예가 첨부 도면을 참조하여 더 상세히 기재될 것이다.
도 1, 도 2, 도 3, 도 4: 실시예 1에 기재된 연구에 대해 마우스에서의 시간 경과에 따른 A20 종양의 부피를 나타내는 그래프. 각 처리군은 개별 동물의 종양 크기를 나타내는 스파이더 플롯으로 표시되며, 그룹당 n = 8이다. 각 그룹에 대해 종양의 징후가 없는(질환이 치유되었음을 도시함) 동물의 수는 그래프의 왼쪽 하단에 표시된다. 투약은 종양 세포 이식 후 제8일, 제11일, 제15일, 제18일, 제22일, 제25일 및 제29일에 수행되었으며 투약 시간은 회색 음영 영역으로 표시된다. 대조군(도 1) 및 항-PD-L1 처리군(도 2)과 비교하여, STIM001 mIgG2a(도 3) 및 STIM003 mIgG2a(도 4) 처리군은 A20 종양 성장의 유의한 억제를 나타내었다.
도 5: STIM001, STIM002B 및 관련 항체 CL-61091, CL-64536, CL-64837, CL-64841 및 CL-64912 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM002 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다.
도 6: 관련 항체 CL-71642 및 CL-74570 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM003 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다. 시퀀싱으로부터 얻은 항체 CL-71642의 VL 도메인은 N 말단 잔기 없이 본원에 나타낸다. 정렬로부터 전체 VH 도메인 서열이 N 말단 글루탐산을 포함할 것임을 알 수 있다.
도 7: STIM008 및/또는 인간 생식계열의 상응하는 서열이 상이한 잔기를 나타내는, STIM007 VH(상단) 및 VL(하단) 도메인 아미노산 서열. 서열 넘버링은 IMGT에 따른다.
도 8: J558 동계 모델에서의 STIM003(항-ICOS) 및 AbW(항-PD-L1) mIgG2a 항체의 효과. 각 처리군은 개별 동물의 종양 크기를 나타내는 "스파이더 플롯"으로 표시된다(그룹당 n=10 또는 n=8). STIM003 단독치료법은 일부 유효성을 실증했고 8마리 중 3마리의 동물은 질환이 치유되었다. 유사하게 항-PDL1은 이 모델에서 효과적이었으며 8마리 중 6마리의 동물은 제37일까지 질환이 치유되었다. 항-PDL1 항체와 조합될 때 STIM003 mIgG2는 종양 성장을 완전히 억제하고 치유받은 동물의 생존을 개선하였다. 각 그룹에 대해 이의 질환이 치유된 동물의 수는 각각의 그래프의 오른쪽 하단에 표시된다. 투약일은 점선으로 표시된다(제11일, 제15일, 제18일, 제22일, 제25일 및 제29일).
도 9: 종양 조직 내의 상이한 TILS 세포 하위유형에 대한 ICOS 발현(양성 세포의 백분율 및 상대적 발현/dMFI)의 정량. (a) ICOS 발현에 대해 양성인 면역 세포 하위유형% 및 (b) 식염수 또는 항-PD-L1 또는 항-PD-1 대리물 항체로 처리된 동물의 면역 세포 하위유형의 ICOS dMFI(ICOS 양성 세포 상의 상대적 ICOS 발현). 제0일에 마우스에 1 x 106개의 생활성 세포/ml를 이식했다(n=7 또는 n=8). 제13일 및 제15일에 동물에 130 ug의 항체를 i.p. 투약했다. 조직 샘플을 제16일에 단리하고 분석했다. CD4+/FOXP3+ 세포는 TReg 집단(오른쪽 끝 그래프)에만 포함되었으며 모두 Foxp3 음성인 "효과기" CD4 세포(왼쪽 끝 그래프)로부터 제외되었다. 실시예 3을 참고한다.
도 10: A20 생체내 유효성 연구로부터의 데이터. 각 처리군은 개별 동물의 종양 크기를 나타내는 "스파이더 플롯"으로 표시된다(그룹당 n=10). 각 그룹에 대해 이의 질환이 치유된 동물의 수는 각각의 그래프에 표시된다. 다중 용량의 경우, 점선으로 표시된 제8일, 제11일, 제15일, 제18일, 제22일 및 제25일에 투약했다. 단일 용량의 경우, 동물은 제8일에만 IP 주사를 제공받았다. (도 10a) 식염수; (도 10b) STIM003 mIgG2a 다중 용량; (도 10c) STIM003 mIgG2a 단일 용량. 실시예 4를 참고한다.
도 11: STIM003 mIgG2a 60 μg 고정 용량을 사용하는 실시예 4에서 보고된 연구에 대한 카플란-마이어(Kaplan-Meier) 곡선. SD = 단일 용량, 제8일. MD = 제8일로부터의 다중 용량 BIW.
도 12: 식염수를 투약한 CT26 종양 보유 동물(시점당 n=4)로부터의 주요 T 세포 하위세트(T-reg[CD4+/FoxP3+], CD4 Eff[CD4+/FoxP3-] 세포 및 CD8+) 상의 ICOS 발현. 면역 세포 표현형 분석은 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 수행되었으며 모든 시점에 모든 조직에서 ICOS 발현에 대해 염색되었다. 도 12a 내지 도 12d는 4개의 상이한 조직에서 모든 시점에서의 ICOS 양성 세포의 백분율을 나타낸다. 도 12e 내지 도 12h는 4개의 상이한 조직 모두에서 모든 시점의 ICOS dMFI(상대적 발현)를 나타낸다. 실시예 5를 참고한다.
도 13: STIM003 mIgG2a 항체에 반응한 TME에서 T-reg 고갈을 실증하는 FACS 분석. CT-26 종양 보유 동물은 종양 세포 이식 후 제12일에 STIM003의 단일 용량(6, 60 또는 200 μg)으로 처리되었다. 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 FACS 분석을 위해 수확된 조직(시점당 n=4). 전체 종양내 T-reg 세포(CD4+CD25+Foxp3+)의 백분율(도 13a) 및 혈액 내 T-reg 세포의 백분율(도 13b)이 상이한 시점에 나타난다. 실시예 5를 참고한다.
도 14: STIM003 mIgG2a에 반응한 CD8:T-reg 및 CD4 eff:T-reg 비의 증가. CT-26 종양 보유 동물은 종양 세포 이식 후 제12일에 STIM003 mIgG2a의 단일 용량(6, 60 또는 200 μg)을 제공받았다. 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 FACS 분석을 위해 조직(시점당 n=4)이 수확되었고 T eff 대 T-reg 비가 계산되었다. (도 14a) 및 (도 14b), 종양 및 혈액내 CD8:T-reg 비, (도 14c) 및 (도 14d) 종양 및 혈액내 CD4-eff:T-reg 비. 실시예 5를 참고한다.
도 15: STIM003 처리는 TIL에 의한 증가된 탈과립화 및 Th1 사이토카인 생산과 상관관계가 있다. 처리 후 제8일에 TIL이 단리되고 FACS 분석이 수행되어 CD4 및 CD8 T 세포(도 15a 및 도 15b) 상의 CD107a 발현을 검출했다. 동시에, 해리된 종양의 세포가 브레펠딘-A의 존재 하에 4시간 동안 휴지되고, 세포가 T 세포 마커에 대해 염색되고 세포내 염색을 위해 투과화되어 IFN-γ 및 TNF-α(도 15c 내지 도 15h)를 검출했다. 실시예 5를 참고한다.
도 16a: ICOS 양성 CD4 기억 세포(ICOS+CD3+CD4+FoxP3-CD45RA-로 정의됨)에 대한 KY1044 표적 참여의 증거. Y축은 샘플 수집 날짜의 함수로 CD4 기억 세포의 점유 백분율을 측정한다. 혈액 샘플은 주기 1 제1일(C1D1), 주기 1 제8일(C1D8), 주기 2 제1일(C2D1) 및 주기 2 제8일(C2D8)에 수집되었다. 용량 수준 1 = 0.8 mg. 용량 수준 2 = 2.4 mg. 선은 동일한 환자의 데이터 포인트를 연결한다.
도 16b: ICOS 양성 CD4 기억 세포(ICOS+CD3+CD4+FoxP3-CD45RA-로 정의됨)에 대한 KY1044 표적 참여의 증거. Y축은 샘플 수집 날짜의 함수로 CD4 기억 세포의 점유 백분율을 측정한다. 혈액 샘플은 주기 1 제1일(C1D1), 주기 1 제8일(C1D8), 주기 2 제1일(C2D1) 및 주기 2 제8일(C2D8)에 수집되었다. 용량 수준 3 = 8 mg. 용량 수준 4 = 24 mg. 용량 수준 5 = 80 mg. 용량 수준 6 = 240 mg. 선은 동일한 환자의 데이터 포인트를 연결한다.
도 17a: 순환 사이토카인 수준을 측정함으로써 평가된 KY1044 의존적 효능작용. 실선 플롯은 평균을 표시하고 음영 영역은 KY1044로 처리된 환자에 대한 GM-CSF의 방문과 기준선 측정값 간 비의 95% 신뢰도 구간을 표시한다. 밝은 회색 데이터 포인트는 더 낮은 KY1044 용량 수준(0.8 mg 및 2.4 mg)을 받은 환자(n = 27)로부터 얻은 것이며, 불완전한 수용체 점유를 초래하였다. 진한 회색 데이터 포인트는 더 높은 KY1044 용량 수준(8 mg 이상)을 받은 환자(n = 14)로부터 얻은 것이며, 완전한 수용체 점유를 초래했다.
도 17b: 순환 사이토카인 수준을 측정함으로써 평가된 KY1044 의존적 효능작용. 실선 플롯은 평균을 표시하고 음영 영역은 KY1044로 처리된 환자에 대한 TNFα의 방문과 기준선 측정값 간 비의 95% 신뢰도 구간을 표시한다. 밝은 회색 데이터 포인트는 더 낮은 KY1044 용량 수준(0.8 mg 및 2.4 mg)을 받은 환자(n = 27)로부터 얻은 것이며, 불완전한 수용체 점유를 초래하였다. 진한 회색 데이터 포인트는 더 높은 KY1044 용량 수준(8 mg 이상)을 받은 환자(n = 14)로부터 얻은 것이며, 완전한 수용체 점유를 초래했다.
도 18a: 치료 지속기간에 관한 I/II상 임상 시험의 중간 결과. 등록된 모든 환자에 대한 치료 지속기간 중앙값은 9주였다.
도 18b: 치료법 요법 및 부분적 또는 완전 수용체 점유와 관련된 치료 지속기간을 나타내는, I/II상 임상 시험의 중간 결과.
도 18c: ICOS 수용체 점유와 관련된 치료 지속기간을 나타내는 I/II상 임상 시험의 중간 결과.
1.6. 상세한 설명
1.6.1. ICOS
본 발명에 따른 항체는 인간 ICOS의 세포외 도메인에 결합한다. 따라서 항체는 ICOS 발현 T 림프구에 결합한다. 본원에 언급된 "ICOS" 또는 "ICOS 수용체"는 문맥상 달리 지시하지 않는 한, 인간 ICOS일 수 있다. 인간, 게잡이원숭이 및 마우스 ICOS의 서열은 첨부된 서열 목록에 나타나며 NCBI로부터 인간 NCBI ID: NP_036224.1, 마우스 NCBI ID: NP_059508.2 및 게잡이원숭이 GenBank ID: EHH55098.1로 이용 가능하다.
1.6.2. 교차반응성
본 발명에 따른 항체는 바람직하게는 교차반응성이고, 예를 들어 마우스 ICOS의 세포외 도메인뿐만 아니라 인간 ICOS에도 결합할 수 있다. 항체는 게잡이원숭이와 같은 영장류의 ICOS를 포함하여 다른 비인간 ICOS에 결합할 수 있다. 인간에서 치료 용도로 의도된 항-ICOS 항체는 인간 ICOS에 결합해야 하는 반면, 다른 종의 ICOS에 대한 결합은 인간 임상 상황에서 직접적인 치료 관련성을 갖지 않을 것이다. 그럼에도 불구하고, 본원의 데이터는 인간 및 마우스 ICOS 둘 모두에 결합하는 항체가 이를 효능제 및 고갈 분자로서 특히 적합하게 만드는 특성을 가짐을 표시한다. 이는 교차반응성 항체에 의해 표적화되는 하나 이상의 특정 에피토프로부터 발생할 수 있다. 그러나 기저 이론에 관계없이 교차반응성 항체는 가치가 높으며 전임상 및 임상 연구를 위한 치료 분자로서 탁월한 후보이다.
실험예에 기재된 바와 같이, 본원에 기재된 STIM 항체는 마우스 ICOS의 발현이 결여되도록(ICOS 녹아웃) 마우스를 조작한 Kymouse™ 기술을 사용하여 생성되었다. ICOS 녹아웃 트랜스제닉 동물 및 교차반응성 항체를 생성하기 위한 이의 용도는 본 발명의 추가 양태이다.
항체의 종 교차반응성 정도를 정량하는 한 가지 방식은 또 다른 종의 항원과 비교하여 항원 또는 한 종에 대한 그 친화도의 배율-차이, 예를 들어 인간 ICOS 대 마우스 ICOS에 대한 친화도의 배율 차이이다. 친화도는 본원의 다른 곳에서 기재된 바와 같이 Fab 형식의 항체를 사용한 SPR에 의해 결정된 항체-항원 반응의 평형 해리 상수를 참조하여, KD로 정량될 수 있다. 종 교차반응성 항-ICOS 항체는 인간 및 마우스 ICOS 결합에 대한 친화도에서 30배 이하, 25배 이하, 20배 이하, 15배 이하, 10배 이하 또는 5배 이하의 배율-차이를 가질 수 있다. 다르게 말하면, 인간 ICOS의 세포외 도메인 결합 KD는 마우스 ICOS의 세포외 도메인의 결합 KD의 30배, 25배, 20배, 15배, 10배 또는 5배 이내일 수 있다. 두 종의 항원에 대한 결합 KD가 역치 값을 충족하는 경우, 예를 들어 인간 ICOS의 결합 KD 및 마우스 ICOS의 결합 KD 둘 모두는 10 mM 이하, 바람직하게는 5 mM 이하, 보다 바람직하게는 1 mM 이하인 경우 항체는 또한 교차반응성인 것으로 간주될 수 있다. KD는 10 nM 이하, 5 nM 이하, 2 nM 이하, 또는 1 nM 이하일 수 있다. KD는 0.9 nM 이하, 0.8 nM 이하, 0.7 nM 이하, 0.6 nM 이하, 0.5 nM 이하, 0.4 nM 이하, 0.3 nM 이하, 0.2 nM 이하, 또는 0.1 nM 이하일 수 있다.
인간 ICOS 및 마우스 ICOS 결합에 대한 교차반응성의 대안적인 측정은 HTRF 검정에서와 같이 ICOS 수용체에 결합하는 ICOS 리간드를 중화시키는 항체의 능력이다(미국 특허 제9,957,323호의 실시예 8 참고). STIM001, STIM002, STIM002-B, STIM003, STIM005 및 STIM006을 포함하는 종 교차반응성 항체의 예가 본원에 제공되며, 이들 각각은 HTRF 검정에서 인간 ICOS에 대한 인간 B7-H2(ICOS 리간드)의 결합 중화 및 마우스 ICOS에 대한 마우스 B7-H2의 결합 중화로 확인되었다. 인간 및 마우스 ICOS에 대해 교차반응하는 항체가 요망되는 경우 임의의 이들 항체 또는 이의 변이체가 선택될 수 있다. 종 교차반응성 항-ICOS 항체는 인간 ICOS 수용체에 대한 인간 ICOS의 결합을 억제하는 IC50이 HTRF 검정에서 결정된 바와 같이 마우스 ICOS 수용체로의 마우스 ICOS 수용체의 억제에 대한 IC50의 25배, 20배, 15배, 10배 또는 5배 이내일 수 있다. 인간 ICOS 수용체에 대한 인간 ICOS의 결합을 억제하는 IC50 및 마우스 ICOS 수용체에 대한 마우스 ICOS의 결합을 억제하는 IC50 둘 모두가 1 mM 이하, 바람직하게는 0.5 mM 이하, 예를 들어 30 nM 이하, 20 nM 이하, 10 nM 이하인 경우 항체가 또한 교차반응성인 것으로 간주될 수 있다. IC50은 5 nM 이하, 4 nM 이하, 3 nM 이하 또는 2 nM 이하일 수 있다. 일부 경우에는 IC50이 적어도 0.1 nM, 적어도 0.5 nM 또는 적어도 1 nM일 것이다.
1.6.3. 특이성
본 발명에 따른 항체는 바람직하게는 ICOS에 특이적이다. 즉, 항체는 표적 단백질 ICOS(인간 ICOS, 바람직하게는 위에서 주지된 바와 같이 마우스 및/또는 게잡이원숭이 ICOS) 상의 그의 에피토프에 결합하지만, CD28 유전자 패밀리 내의 다른 분자를 포함하여 해당 에피토프를 제시하지 않는 분자에 대해서는 유의한 결합을 나타내지 않는다. 본 발명에 따른 항체는 바람직하게는 인간 CD28에 결합하지 않는다. 항체는 바람직하게는 또한 마우스 또는 게잡이원숭이 CD28에 결합하지 않는다.
CD28은 TCR을 통한 항원 인식과 관련하여 전문 항원 제시 세포 상의 그의 리간드 CD80 및 CD86에 의해 참여될 때 T 세포 반응을 공동 자극한다. 본원에 기재된 항체의 다양한 생체내 용도에 있어서, CD28에 대한 결합의 회피가 유리한 것으로 간주된다. CD28에 대한 항-ICOS 항체의 비결합은 CD28이 그 천연 리간드와 상호작용하고 T 세포 활성화를 위해 적절한 공동자극 신호를 생성하도록 허용해야 한다. 추가로, CD28에 대한 항-ICOS 항체의 비결합은 과효능작용의 위험을 회피한다. CD28의 과자극은 TCR을 통한 동족 항원의 인식을 위한 일반적인 요구사항 없이 휴지 T 세포에서 증식을 유도할 수 있고, 잠재적으로, 특히 인간 대상체에서, T 세포의 폭주(runaway) 활성화 및 결과적인 사이토카인 방출 증후군을 야기할 수 있다. 따라서 본 발명에 따른 항체에 의한 CD28의 비인식은 인간에서 이의 안전한 임상적 사용 측면에서의 이점을 표시한다.
본원의 다른 곳에서 논의된 바와 같이, 본 발명은 다중특이적 항체(예를 들어, 이중특이적 항체)까지 확장된다. 다중특이적(예를 들어, 이중특이적) 항체는 (i) ICOS에 대한 항체 항원 결합 부위 및 (ii) 다른 항원(예를 들어 PD-L1)을 인식하는 추가 항원 결합 부위(선택적으로 본원에 기재된 바와 같은 항체 항원 결합 부위)를 포함할 수 있다. 개별 항원 결합 부위의 특이적 결합이 결정될 수 있다. 따라서, ICOS에 특이적으로 결합하는 항체에는 ICOS에 특이적으로 결합하는 항원 결합 부위를 포함하는 항체가 포함되며, 선택적으로 ICOS에 대한 항원 결합 부위는 하나 이상의 다른 항원에 대한 하나 이상의 추가 결합 부위를 추가로 포함하는 항원-결합 분자, 예를 들어, ICOS 및 PD-L1에 결합하는 이중특이적 항체 내에 포함된다.
1.6.4. 친화도
ICOS에 대한 항체의 결합 친화도가 결정될 수 있다. 항원에 대한 항체의 친화도는 항체-항원 상호작용의 평형 해리 상수 KD, 결합(association) 또는 온 속도(Ka)와 해리 또는 오프 속도(kd)의 비 Ka/Kd의 측면에서 정량될 수 있다. 항체-항원 결합에 대한 Kd, Ka 및 Kd는 표면 플라즈몬 공명(SPR)을 사용하여 측정될 수 있다.
본 발명에 따른 항체는 10 mM 이하, 바람직하게는 5 mM 이하, 더욱 바람직하게는 1 mM 이하의 KD로 인간 ICOS의 EC 도메인에 결합할 수 있다. KD는 50 nM 이하, 10 nM 이하, 5 nM 이하, 2 nM 이하, 또는 1 nM 이하일 수 있다. KD는 0.9 nM 이하, 0.8 nM 이하, 0.7 nM 이하, 0.6 nM 이하, 0.5 nM 이하, 0.4 nM 이하, 0.3 nM 이하, 0.2 nM 이하, 또는 0.1 nM 이하일 수 있다. KD는 적어도 0.001 nM, 예를 들어 적어도 0.01 nM 또는 적어도 0.1 nM일 수 있다.
친화도 정량은 Fab 형식의 항체를 사용하는, SPR을 사용하여 수행될 수 있다. 적합한 프로토콜은 하기와 같다:
1. 예컨대 1차 아민 커플링에 의해 항-인간(또는 종 일치된 다른 항체 불변 영역) IgG를 바이오센서 칩(예를 들어, GLM 칩)에 커플링하고;
2. 항-인간 IgG(또는 다른 일치된 종 항체)를 예를 들어, Fab 형식의 시험 항체에 노출시켜 시험 항체를 칩 상에 포획하고;
3. 시험 항원을 다양한 농도, 예를 들어 5000 nM, 1000 nM, 200 nM, 40 nM, 8 nM 및 2 nM, 그리고 0 nM(즉, 완충액 단독)로 칩의 포획 표면 위로 통과시키고;
4. 25℃에서 SPR을 사용하여 시험 항원에 대한 시험 항체의 결합 친화도를 결정한다. 완충액은 pH 7.6, 150 mM NaCl, 0.05% 세제(예를 들어, P20) 및 3 mM EDTA일 수 있다. 완충액은 선택적으로 10 mM HEPES를 함유할 수 있다. HBS-EP는 수행 완충액으로 사용될 수 있다. HBS-EP는 Teknova Inc(California, 카탈로그 번호 H8022)로부터 이용 가능하다.
포획 표면의 재생은 pH 1.7에서 10 mM 글리신을 사용하여 수행될 수 있다. 이는 포획된 항체를 제거하고 표면이 또 다른 상호작용에 사용될 수 있게 된다. 결합 데이터는 표준 기술을 사용하여, 예를 들어 ProteOn XPR36TM 분석 소프트웨어에 고유한 모델을 사용하여 고유한 1:1 모델에 피팅될 수 있다.
다양한 SPR 장비, 예컨대 Biacore™, ProteOn XPR36™(Bio-Rad®), 및 KinExA®(Sapidyne Instruments, Inc)가 알려져 있다.
기재된 바와 같이, 친화도는 Fab 형식의 항체를 사용하며, 항원이 칩 표면에 커플링되고 시험 항체가 용액 중 Fab 형식의 칩 위로 통과되는 SPR에 의해 결정되어, 단량체 항체-항원 상호작용의 친화도를 결정할 수 있다. 친화도는 임의의 원하는 pH(예를 들어, pH 5.5 또는 pH 7.6) 및 임의의 원하는 온도(예를 들어, 25℃ 또는 37℃)에서 결정될 수 있다.
ICOS에 대한 항체의 결합을 측정하는 다른 방식은 예를 들어 ICOS의 외인성 표면 발현을 갖는 세포(예를 들어, CHO 세포) 또는 내인성 수준의 ICOS를 발현하는 활성화 1차 T 세포를 사용하는 형광 활성화 세포 분류(FACS)를 포함한다. FACS에 의해 측정된 ICOS-발현 세포에 대한 항체 결합은 항체가 ICOS의 세포외(EC) 도메인에 결합할 수 있음을 표시한다.
1.6.5. ICOS 수용체 효능작용
ICOS 리간드(ICOSL, B7-H2로도 알려짐)는 ICOS 수용체에 결합하는 세포 표면 발현 분자이다[17]. 이러한 세포간 리간드-수용체 상호작용은 T 세포 표면 상에서 ICOS의 다량체화를 촉진하여 수용체를 활성화하고 T 세포의 하류 신호전달을 자극한다. 효과기 T 세포에서 이 수용체 활성화는 효과기 T 세포 반응을 자극한다.
항-ICOS 항체는 ICOS의 효능제로서 작용하여 수용체 상의 천연 ICOS 리간드의 이러한 자극 효과를 모방하거나 심지어 능가할 수 있다. 이러한 효능작용은 T 세포 상에서 ICOS의 다량체화를 촉진하는 항체의 능력으로부터 발생할 수 있다. 이에 대한 한 가지 메커니즘은 항체가 T 세포 표면 상의 ICOS와 Fc 수용체와 같은 인접 세포(예를 들어, B 세포, 항원 제시 세포 또는 다른 면역 세포) 상의 수용체 사이에 세포간 가교를 형성하는 것이다. 또 다른 메커니즘은 다중(예를 들어, 2개의) 항원-결합 부위(예를 들어, 2개의 VH-VL 도메인 쌍)를 갖는 항체가 다중 ICOS 수용체 분자를 가교하여 다량체화를 촉진하는 것이다. 이러한 메커니즘의 조합이 발생할 수 있다.
효능작용은 가교제를 포함하거나 제외하는 가용성 형태(예를 들어, 면역글로불린 형식 또는 공간적으로 분리된 2개의 항원 결합 부위, 예를 들어 2개의 VH-VL 쌍을 포함하는 다른 항체 형식)의 항체를 사용하거나 항원 결합 부위의 테더링된 배열을 제공하기 위해 고체 표면에 결합된 항체를 사용하여 시험관내 T 세포 활성화 검정에 대해 시험할 수 있다. 효능작용 검정은 이러한 검정에서의 활성화를 위한 표적 T 세포로서 MJ 세포(ATCC CRL-8294)와 같은 인간 ICOS 양성 T 림프구 세포주를 사용할 수 있다. T 세포 활성화의 하나 이상의 측정은 시험 항체에 대해 결정되고 참조 분자 또는 음성 대조군과 비교되어 참조 분자 또는 대조군에 비해 시험 항체에 의해 영향을 받는 T 세포 활성화에 통계적으로 유의한(p<0.05) 차이가 존재하는지 여부를 결정할 수 있다. T 세포 활성화의 한 가지 적합한 측정은 사이토카인, 예를 들어 IFNγ, TNFα 또는 IL-2의 생산이다. 당업자는 시험 항체와 대조군 사이의 검정 조건을 표준화하는 적절한 대조를 적절하게 포함할 것이다. 적합한 음성 대조군은 ICOS에 결합하지 않는 동일한 형식(예를 들어, 이소형 대조군)의 항체, 예를 들어 검정 시스템에 존재하지 않는 항원에 특이적인 항체이다. 검정의 동적 범위 내에서 동족 이소형 대조군에 비해 시험 항체에 대해 유의한 차이가 관찰된다는 것은 항체가 해당 검정에서 ICOS 수용체의 효능제로서 작용함을 나타낸다.
효능제 항체는 T 세포 활성화 검정에서 시험될 때 다음과 같은 것으로 정의될 수 있다:
대조군 항체와 비교하여 IFNγ 생산 유도에 대한 EC50이 유의하게 더 낮고/낮거나;
대조군 항체와 비교하여 유의하게 더 높은 최대 IFNγ 생산을 유도하고/하거나;
ICOSL-Fc와 비교하여 IFNγ 생산 유도에 대한 EC50이 유의하게 더 낮고/낮거나;
ICOSL-Fc와 비교하여 유의하게 더 높은 최대 IFNγ 생산을 유도하고/하거나;
참조 항체 C398.4A와 비교하여 IFNγ 생산 유도에 대한 EC50이 유의하게 더 낮고/낮거나;
참조 항체 C398.4A와 비교하여 유의하게 높은 최대 IFNγ 생산을 유도한다.
예시적인 시험관내 T 세포 검정에는 미국 특허 제9,957,323호의 실시예 13 내지 15에 개시된 바와 같은 비드 결합 검정, 플레이트 결합 검정 및 가용성 형태 검정이 포함된다.
유의하게 더 낮거나 유의하게 더 높은 값은 참조 또는 대조군 값과 비교하여, 예를 들어 최대 0.5배 상이할 수 있거나, 최대 0.75배 상이할 수 있거나, 최대 2배 상이할 수 있거나, 최대 3배 상이할 수 있거나, 최대 4배 상이할 수 있거나 최대 5배 상이할 수 있다.
따라서, 한 예에서, 본 발명에 따른 항체는 대조군과 비교하여 비드 결합 형식의 항체를 사용한 MJ 세포 활성화 검정에서 IFNγ 유도에 대한 EC50이 유의하게 더 낮다, 예를 들어 적어도 2배 더 낮다.
비드 결합 검정은 비드 표면에 결합된 항체(및 대조군 또는 참조 실험의 경우 대조군 항체, 참조 항체 또는 ICOSL-Fc)를 사용한다. 자성 비드가 사용될 수 있으며, 다양한 종류가 시판되고 있고, 예를 들어 토실-활성화 DYNABEADS M-450(DYNAL Inc, 5 Delaware Drive, Lake Success, N.Y. 11042 Prod No. 140.03, 140.04)이 있다. 비드는 코팅될 수 있거나, 일반적으로 코팅 물질을 카보네이트 완충액(pH 9.6, 0.2 M)에 용해하거나 당업계에 알려진 다른 방법으로 처리될 수 있다. 비드의 사용은 비드 표면에 결합된 단백질의 양을 매우 정확하게 측정할 수 있게 하여 편리하다. 표준 Fc-단백질 정량 방법은 비드 상에 커플링된 단백질 정량을 위해 사용될 수 있다. DELFIA, ELISA 또는 다른 방법을 포함하는 임의의 적합한 방법이 검정의 동적 범위 내에서 관련 표준을 참조하여 사용될 수 있다.
항체의 효능작용 활성은 또한 생체외 1차 인간 T 림프구에서 측정될 수 있다. 이러한 T 세포에서 IFNγ의 발현을 유도하는 항체의 능력은 ICOS 효능작용을 나타낸다. 바람직하게는, 항체는 T 세포 활성화 검정 1 및/또는 T 세포 활성화 검정 2에서 대조군 항체와 비교하여 5 μg/ml에서 IFNγ의 유의한(p<0.05) 유도를 나타낼 것이다. 위에서 주지된 바와 같이, 항-ICOS 항체는 이러한 검정에서 ICOS-L 또는 C398.4보다 더 높은 정도로 T 세포 활성화를 자극할 수 있다. 따라서, 항체는 T 세포 활성화 검정 1 또는 2에서 대조군 또는 참조 항체와 비교하여 5 μg/ml에서 IFNγ의 유의하게(p<0.05) 더 큰 유도를 나타낼 수 있다. TNFα 또는 IL-2 유도는 대안적 검정 판독으로 측정될 수 있다.
항-ICOS 항체의 효능작용은 생체내, 예를 들어 종양 미세환경과 같은 병리 부위에서 TEff 세포에 유리하게 TReg와 TEff 세포 집단 사이의 균형을 변화시키는 능력에 기여할 수 있다. 활성화 ICOS-양성 효과기 T 세포에 의한 종양 세포 사멸을 향상시키는 항체의 능력은 본원의 다른 곳에서 논의된 바와 같이 결정될 수 있다.
더 낮은 용량에서의 ICOS 수용체 효능작용 및 치료 유효성
본 발명은 부분적으로 대상체로의 더 낮은 용량의 투여로부터 발생하는 더 낮은 항-ICOS 항체 농도가 더 높은 용량으로부터 발생하는 더 높은 항-ICOS 농도와 비교하여 임상 유효성을 개선할 수 있다는 발견에 기반한다. 놀랍게도, 본원에 제시된 데이터에 나타난 바와 같이, 단지 부분적 수용체/일시적 점유를 초래하는 항-ICOS 항체 용량이 완전 수용체 점유를 초래하는 항-ICOS 항체 용량과 비교하여 치료 후 더 강력한 GM-CSF 및 TNFα 신호를 유도할 수 있다.
이론에 의해 제한되지 않고, KY1044와 같은 항-ICOS 항체는 T 세포 상에서 ICOS의 다량체화를 촉진함으로써 ICOS의 효능제로서 작용할 수 있다. ICOS 수용체는 동종이량체로 구성되는 성향을 갖는다. 따라서, ICOS에 대한 다중 항원 결합 부위를 갖는 항체는 다중 ICOS 수용체 분자를 가교할 수 있고 리간드-유도 클러스터링 또는 다량체화를 초래할 수 있다. 이러한 리간드 가교는 리간드-수용체 상호작용의 증가된 안정성을 통해 결합력 효과를 매개하는 것으로 제안되었다.
ICOS 수용체의 다량체화는 부분적으로 항체 농도와 수용체의 화학양론적 비에 좌우될 수 있다. 예를 들어, 이론에 의해 제한되지 않고, 항체의 농도가 이용 가능한 수용체의 수보다 유의하게 더 큰 경우, 이는 2개의 상이한 항체에 결합된 단리된 수용체의 형성을 선호하고 FcγR 의존적 자극을 감소시킬 것이지만, 수용체의 수가 존재하는 항체의 수를 크게 초과하면 리간드를 가교가 발생할 가능성이 낮고 또한 결과적으로 FcγR 의존적 자극의 감소를 야기할 것이다. 일부 구현예에서, 동일한 농도의 항체 및 수용체가 존재하고 다량체성 복합체의 형성을 촉진하고 FcγR 의존적 자극을 최대로 유도하여 더 큰 친염증성 사이토카인의 방출을 초래한다. 또한 이론에 의해 제한되지 않고, 높은 항-ICOS 항체 옵소닌화는 클러스터링이 없고/없거나 면역학적 시냅스가 불량하고 공동 자극이 없을 수 있는 반면, 낮은 항-ICOS 항체 옵소닌화는 클러스터링을 개선하여 FcγR 의존적 공동 자극을 초래할 수 있다.
일부 구현예에서, 부분적 ICOS 수용체/일시적 점유를 생성하고/하거나, 클러스터링을 개선하고/하거나 공동자극을 개선하는 데 효과적인 용량으로 투여되는 항-ICOS 항체는 KY1044의 CDR을 포함한다. 또 다른 구현예에서, 항-ICOS 항체는 KY1044의 중쇄 및 경쇄 가변 도메인과 적어도 85%, 90% 또는 95% 서열 동일성을 갖는 중쇄 및 경쇄 가변 도메인을 포함한다. 일부 이러한 구현예에서, KY1044의 중쇄 및 경쇄 가변 도메인과 적어도 85%, 90% 또는 95% 서열 동일성을 갖는 중쇄 및 경쇄 가변 도메인은 KY1044의 CDR을 포함한다. 또 다른 구현예에서, 항-ICOS 항체는 KY1044의 중쇄 및 경쇄 가변 도메인을 포함한다. 일부 구현예에서, 약 8 mg의 항-ICOS 항체 용량은 완전한 ICOS 수용체 점유를 생성한다. 따라서, 일부 구현예에서, 부분적 ICOS 수용체/일시적 점유를 생성하고/하거나, 클러스터링을 개선하고/하거나 공동자극을 개선하는 데 효과적인 항-ICOS 항체 용량은 약 8 mg 미만, 예를 들어 약 7 mg, 약 6 mg, 약 5 mg, 약 4 mg, 약 3 mg, 약 2 mg, 약 1 mg, 또는 약 1 mg 미만이다. 한 구현예에서, 항-ICOS 항체의 용량은 약 2.4 mg이다. 또 다른 구현예에서, 항-ICOS 항체의 용량은 약 0.8 mg이다.
또 다른 구현예에서, 부분적인 ICOS 수용체/일시적 점유를 생성하고/하거나, 클러스터링을 개선하고/하거나 공동자극을 개선하는 데 효과적인 용량으로 투여되는 항-ICOS 항체는 KY1044의 중쇄 및 경쇄와 적어도 85%, 90% 또는 95% 서열 동일성을 갖는 중쇄 및 경쇄를 포함한다. 일부 이러한 구현예에서, KY1044의 중쇄 및 경쇄와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 중쇄 및 경쇄는 KY1044의 CDR을 포함한다. 또 다른 구현예에서, 항-ICOS 항체는 KY1044의 중쇄 및 경쇄를 포함한다. 또 다른 구현예에서, 항-ICOS 항체는 KY1044이다. 일부 구현예에서, 약 8 mg의 항-ICOS 항체 용량은 완전 ICOS 수용체 점유를 생성한다. 따라서, 일부 구현예에서, 부분적 ICOS 수용체/일시적 점유를 생성하고/하거나, 클러스터링을 개선하고/하거나 공동자극을 개선하는 데 효과적인 항-ICOS 항체 용량은 약 8 mg 미만, 예를 들어 약 7 mg, 약 6 mg, 약 5 mg, 약 4 mg, 약 3 mg, 약 2 mg, 약 1 mg, 또는 약 1 mg 미만이다. 한 구현예에서, 항-ICOS 항체의 용량은 약 2.4 mg이다. 또 다른 구현예에서, 항-ICOS 항체의 용량은 약 0.8 mg이다.
일부 구현예에서, 종양내 ICOS+ Treg 고갈(ICOS+FOXP3+ 세포의 감소)은 항-ICOS 항체(예를 들어, KY1044) 약 8 mg에서 가장 높다. 일부 구현예에서, 항-ICOS 항체에 의해 생성된 종양 미세환경에서의 CD8/ICOS+FOXP3+ Treg 비의 개선은 항-ICOS 항체(예를 들어, KY1044)의 약 8 mg 이상의 용량에서 정체(plateau)된다.
일부 구현예에서, ICOS 효능작용은 약 8 mg 미만의 항-ICOS 항체(예를 들어, KY1044)의 용량에서 가장 명백하다. 일부 구현예에서, 항-ICOS 항체(예를 들어, KY1044)의 효능작용 활성은 약 2.4 내지 8 mg에서 효과적이다. 일부 구현예에서, 항-ICOS 항체(예를 들어, KY1044)의 효능작용 활성은 약 0.8 내지 2.4 mg에서 효과적이다. 일부 구현예에서, 항-ICOS 항체(예를 들어, KY1044)의 효능작용 활성은 약 2.4 mg에서 효과적이다. 또 다른 구현예에서, 항-ICOS 항체(예를 들어, KY1044)의 효능작용 활성은 약 0.8 mg에서 효과적이다.
1.6.6. T 세포 의존적 사멸
종양 세포가 관련 면역 세포와 함께 인큐베이션되어 면역 세포 의존적 살해를 유발하며, TEff에 의한 종양 세포 사멸에 대한 항-ICOS 항체의 효과가 관찰되는, 효과기 T 세포 기능은 시험관내 공동 배양 검정을 사용하여 생물학적으로 관련된 맥락에서 결정될 수 있다.
활성화 ICOS-양성 효과기 T 세포에 의한 종양 세포 사멸을 향상시키는 항체의 능력이 결정될 수 있다. 항-ICOS 항체는 대조군 항체와 비교하여 유의하게 더 큰(p<0.05) 종양 세포 사멸을 자극할 수 있다. 항-ICOS 항체는 ICOS 리간드 또는 C398.4 항체와 같은 참조 분자와 비교하여 이러한 검정에서 유사하거나 더 큰 종양 세포 사멸을 자극할 수 있다. 유사한 정도의 종양 세포 사멸은 시험 항체에 대한 검정 판독이 참조 분자에 대한 판독과 2배 미만으로 상이한 것으로 표시될 수 있다.
1.6.7. ICOS 리간드-수용체 중화 효능
본 발명에 따른 항체는 ICOS가 그의 리간드 ICOSL에 결합하는 것을 억제하는 것일 수 있다.
항체가 ICOS 수용체의 그의 리간드로의 결합을 억제하는 정도가 그의 리간드-수용체 중화 효능으로 지칭된다. 효능은 달리 명시되지 않는 한 일반적으로 IC50 값(pM)으로 표현된다. 리간드 결합 연구에서 IC50은 수용체 결합을 최대 특이적 결합 수준의 50%까지 감소시키는 농도이다. IC50은 항체 농도 로그의 함수로서 특이적 수용체 결합%를 플롯팅하고, 프리즘(GraphPad)과 같은 소프트웨어 프로그램을 사용하여 S자형 함수를 데이터에 피팅해서 IC50 값을 생성함으로써 계산될 수 있다. 중화 효능은 미국 특허 제9,957,323호의 실시예 8에 개시된 바와 같이 HTRF 검정에서 결정될 수 있다.
IC50 값은 복수의 측정값의 평균을 나타낼 수 있다. 따라서, 예를 들어, 3회 반복 실험 결과로부터 IC50 값을 얻을 수 있으며, 이어서 평균 IC50 값을 계산할 수 있다.
항체는 리간드-수용체 중화 검정에서 1 mM 이하, 예를 들어 0.5 mM 이하의 IC50을 가질 수 있다. IC50은 30 nM 이하, 20 nM 이하, 10 nM 이하, 5 nM 이하, 4 nM 이하, 3 nM 이하 또는 2 nM 이하일 수 있다. IC50은 적어도 0.1 nM, 적어도 0.5 nM 또는 적어도 1 nM일 수 있다.
1.6.8. 항체
미국 특허 제9,957,323호의 실시예에 기재된 바와 같이, 본 발명자들은 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009로 지정된 특정 관심 항체를 단리하고 특성화하였다. 본 발명의 다양한 양태에서, 문맥상 달리 지시하지 않는 한, 항체는 임의의 이들 항체로부터 또는 STIM001, STIM002, STIM003, STIM004 및 STIM005의 하위세트로부터 선택될 수 있다. 이들 항체 각각의 서열은 첨부된 서열 목록에 제공되며, 각 항체에 대해 다음 서열이 각각 표시된다: VH 도메인을 암호화하는 뉴클레오티드 서열; VH 도메인의 아미노산 서열; VH CDR1 아미노산 서열, VH CDR2 아미노산 서열; VH CDR3 아미노산 서열; VL 도메인을 암호화하는 뉴클레오티드 서열; VL 도메인의 아미노산 서열; VL CDR1 아미노산 서열; VL CDR2 아미노산 서열; 및 VL CDR3 아미노산 서열. 본 발명은 첨부된 서열 목록 및/또는 도면에 표시된 모든 항체의 VH 및/또는 VL 도메인 서열을 갖는 항-ICOS 항체뿐만 아니라 그러한 항체의 HCDR 및/또는 LCDR을 포함하고, 선택적으로 전체 중쇄 및/또는 전체 경쇄 아미노산 서열을 갖는 항체를 포괄한다.
STIM001은 Seq ID No: 363의 CDRH1 아미노산 서열, Seq ID No: 364의 CDRH2 아미노산 서열, 및 Seq ID No: 365의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 366의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 367이다. STIM001은 Seq ID No: 370의 CDRL1 아미노산 서열, Seq ID No: 371의 CDRL2 아미노산 서열, 및 Seq ID No: 372의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 373의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 374이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 368(중쇄 핵산 서열 Seq ID No: 369)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 375(경쇄 핵산 서열 Seq ID No: 376)이다.
STIM002는 Seq ID No: 377의 CDRH1 아미노산 서열, Seq ID No: 378의 CDRH2 아미노산 서열, 및 Seq ID No: 379의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 380의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 381이다. STIM002는 Seq ID No: 384의 CDRL1 아미노산 서열, Seq ID No: 385의 CDRL2 아미노산 서열, 및 Seq ID No: 386의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 387의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 388 또는 Seq ID No: 519이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 382(중쇄 핵산 서열 Seq ID No: 383)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 389(경쇄 핵산 서열 Seq ID No: 390 또는 Seq ID No: 520)이다.
STIM002-B는 Seq ID No: 391의 CDRH1 아미노산 서열, Seq ID No: 392의 CDRH2 아미노산 서열, 및 Seq ID No: 393의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 394의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 395이다. STIM002-B는 Seq ID No: 398의 CDRL1 아미노산 서열, Seq ID No: 399의 CDRL2 아미노산 서열, 및 Seq ID No: 400의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 401의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 402이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 396(중쇄 핵산 서열 Seq ID No: 397)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 403(경쇄 핵산 서열 Seq ID No: 404)이다.
KY1044로 본원에서 상호교환적으로 지칭되는 STIM003은 Seq ID No: 405의 CDRH1 아미노산 서열, Seq ID No: 406의 CDRH2 아미노산 서열, 및 Seq ID No: 407의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 408의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 409 또는 Seq ID No: 521이다. STIM003은 Seq ID No: 412의 CDRL1 아미노산 서열, Seq ID No: 413의 CDRL2 아미노산 서열, 및 Seq ID No: 414의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 415의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 4416이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 410(중쇄 핵산 서열 Seq ID No: 411 또는 Seq ID No: 522)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 417(경쇄 핵산 서열 Seq ID No: 418)이다.
STIM004는 Seq ID No: 419의 CDRH1 아미노산 서열, Seq ID No: 420의 CDRH2 아미노산 서열, 및 Seq ID No: 421의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 422의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 423이다. STIM004는 Seq ID No: 426의 CDRL1 아미노산 서열, Seq ID No: 427의 CDRL2 아미노산 서열, 및 Seq ID No: 428의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 429의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 430 또는 Seq ID No: 431이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 424(중쇄 핵산 서열 Seq ID No: 425)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 432(경쇄 핵산 서열 Seq ID No: 433 또는 Seq ID no: 434)이다.
STIM005는 Seq ID No: 435의 CDRH1 아미노산 서열, Seq ID No: 436의 CDRH2 아미노산 서열, 및 Seq ID No: 437의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 438의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 439이다. STIM005는 Seq ID No: 442의 CDRL1 아미노산 서열, Seq ID No: 443의 CDRL2 아미노산 서열, 및 Seq ID No: 444의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 445의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 446이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 440(중쇄 핵산 서열 Seq ID No: 441)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 447(경쇄 핵산 서열 Seq ID No: 448)이다.
STIM006은 Seq ID No: 449의 CDRH1 아미노산 서열, Seq ID No: 450의 CDRH2 아미노산 서열, 및 Seq ID No: 451의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 452의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 453이다. STIM006은 Seq ID No: 456의 CDRL1 아미노산 서열, Seq ID No: 457의 CDRL2 아미노산 서열, 및 Seq ID No: 458의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 459의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 460이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 454(중쇄 핵산 서열 Seq ID No: 455)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 461(경쇄 핵산 서열 Seq ID No: 462)이다.
STIM007은 Seq ID No: 463의 CDRH1 아미노산 서열, Seq ID No: 464의 CDRH2 아미노산 서열, 및 Seq ID No: 465의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 466의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 467이다. STIM007은 Seq ID No: 470의 CDRL1 아미노산 서열, Seq ID No: 471의 CDRL2 아미노산 서열, 및 Seq ID No: 472의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 473의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 474이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 468(중쇄 핵산 서열 Seq ID No: 469)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 475(경쇄 핵산 서열 Seq ID No: 476)이다.
STIM008은 Seq ID No: 477의 CDRH1 아미노산 서열, Seq ID No: 478의 CDRH2 아미노산 서열, 및 Seq ID No: 479의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 480의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 481이다. STIM008은 Seq ID No: 484의 CDRL1 아미노산 서열, Seq ID No: 485의 CDRL2 아미노산 서열, 및 Seq ID No: 486의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 487의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 488이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 482(중쇄 핵산 서열 Seq ID No: 483)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 489(경쇄 핵산 서열 Seq ID No: 490)이다.
STIM009는 Seq ID No: 491의 CDRH1 아미노산 서열, Seq ID No: 492의 CDRH2 아미노산 서열, 및 Seq ID No: 493의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 494의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 495이다. STIM009는 Seq ID No: 498의 CDRL1 아미노산 서열, Seq ID No: 499의 CDRL2 아미노산 서열, 및 Seq ID No: 500의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 501의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 502이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 496(중쇄 핵산 서열 Seq ID No: 497)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 503(경쇄 핵산 서열 Seq ID No: 504)이다.
추가의 예시적인 항-ICOS 항체에는 37A10S713(보프라텔리맙 또는 JTX-2011로도 지칭됨)(예를 들어, 미국 특허 제10023635호 및 제11,292,840호; WO2017070423; WO 2016154177 참고); XMAb23104(XmAb104로도 지칭됨)(미국 특허 제10981992호 참고); 314.8 mAb(Icos 314-8로도 지칭됨)(WO2014033327A1, WO2012131004A2; 미국 특허 제11180556호), JMab-136(IC009로도 지칭됨)(예를 들어, WO2008137915; 미국 특허 제9193789호, 제US20110243929A1호 참고) 및 ICOS.33 IgG1f S267E(미국 특허 제10898556호)가 포함되지만 이에 제한되지 않는다. ICOS에 대한 항체 및 질환 치료에서의 사용 방법은 WO2019222188A1 및 미국 특허 제11292840호에 기재되어 있다. ICOS에 대한 항체는 또한 EP1374902, EP1374901 및 EP1125585에 개시된다. ICOS에 대한 효능제 항체는 또한 US20210340250A1; WO2018222711A2; WO2021209356A1; WO2016120789; US20160215059A1; 및 WO2012131004A2에 개시된다.
37A10S713의 중쇄 및 경쇄의 서열은 SEQ ID NO: 611 및 612로 개시된다.
37A10S713 중쇄: (SEQ ID NO: 611)
37A10S713 경쇄: (SEQ ID NO: 612)
한 구현예에서, ICOS 결합 단백질은 보프라텔리맙이다. 한 구현예에서, ICOS 결합 단백질은 JTX-2011이다.
XMAb23104의 중쇄 및 경쇄의 서열은 SEQ ID NO: 613 및 614로 개시된다.
XmAb23104 중쇄: (SEQ ID NO: 613)
XmAb23104 경쇄: (SEQ ID NO: 614)
314.8 mAb의 중쇄 및 경쇄 가변 영역의 서열은 SEQ ID NO: 615 및 616으로 개시된다.
314.8 mAb 중쇄 가변 영역: (SEQ ID NO: 615)
314.8 mAb 경쇄 가변 영역: (SEQ ID NO: 616)
JMab-136의 중쇄 및 경쇄 가변 영역의 서열은 SEQ ID NO: 617 및 618로 개시된다.
JMab-136 중쇄 가변 영역: (SEQ ID NO: 617)
JMab-136 경쇄 가변 영역: (SEQ ID NO: 618)
ICOS.33 IgG1f S267E의 중쇄 및 경쇄의 서열은 SEQ ID NO: 619 및 620으로 개시된다.
ICOS.33 IgG1f S267E 중쇄: SEQ ID NO: 619.
ICOS.33 IgG1f S267E 경쇄: SEQ ID NO: 620.
용어 "항체"는 (전장) 항체뿐만 아니라 이의 항원 결합 단편도 지칭한다. 본 발명에 따른 항체는 자연 또는 부분적으로 또는 전체적으로 합성에 의해 생성된 면역글로불린 또는 면역글로불린 도메인을 포함하는 분자이다. 항체는 항체를 자연적으로 생성하는 임의의 종으로부터 유래되거나 재조합 DNA 기술에 의해 생성되거나, 혈청, B-세포, 하이브리도마, 트랜스펙토마, 효모, 또는 박테리아로부터 단리되는 IgG, IgM, IgA, IgD, 또는 IgE 분자 또는 이의 항원 특이적(항원-결합) 항체 단편(예컨대 비제한적으로, Fab, F(ab')2, Fv, 이황화 연결된 Fv, scFv, 단일 도메인 항체, 폐쇄 형태의 다중특이적 항체, 이황화 연결된 scfv, 디아바디)일 수 있다. 항체는 일상적인 기술을 사용하여 인간화될 수 있다. 용어 항체는 항체 항원 결합 부위를 포함하는 모든 폴리펩티드 또는 단백질을 포괄한다. 항원 결합 부위(파라토프)는 그의 표적 항원(예를 들어, ICOS)의 에피토프에 결합하고 이에 대해 상보적인 항체의 일부이다.
용어 "에피토프"는 항체에 의해 결합된 항원의 영역을 지칭한다. 에피토프는 구조적 또는 기능적으로 정의될 수 있다. 기능적 에피토프는 일반적으로 구조적 에피토프의 하위세트이고 상호작용의 친화도에 직접적으로 기여하는 잔기를 갖는다. 에피토프는 또한 입체형태적일 수 있으며, 즉 비선형 아미노산으로 이루어질 수 있다. 특정 구현예에서, 에피토프는 아미노산, 당 측쇄, 포스포릴기, 또는 설포닐기와 같은 분자의 화학적 활성 표면 그룹인 결정인자를 포함할 수 있고, 특정 구현예에서, 특이적 삼차원 구조 특징 및/또는 특이적 전하 특징을 가질 수 있다.
항원 결합 부위는 항체의 하나 이상의 CDR을 포함하고 항원에 결합할 수 있는 폴리펩티드 또는 도메인이다. 예를 들어, 폴리펩티드는 CDR3(예를 들어, HCDR3)을 포함한다. 예를 들어, 폴리펩티드는 항체의 가변 도메인의 CDR 1 및 2(예를 들어, HCDR 1 및 2) 또는 CDR 1 내지 3(예를 들어, HCDR 1 내지 3)을 포함한다.
항체 항원 결합 부위는 하나 이상의 항체 가변 도메인에 의해 제공될 수 있다. 한 예에서, 항체 결합 부위는 단일 가변 도메인, 예를 들어 중쇄 가변 도메인(VH 도메인) 또는 경쇄 가변 도메인(VL 도메인)에 의해 제공된다. 다른 예에서, 결합 부위는 VH/VL 쌍 또는 2개 이상의 이러한 쌍을 포함한다. 따라서, 항체 항원 결합 부위는 VH 및 VL을 포함할 수 있다.
항체는 불변 영역을 포함하는 전체 면역글로불린일 수 있거나, 항체 단편, 예를 들어 항체의 항원 결합 단편일 수 있다. 항체 단편은 예를 들어 온전한 항체의 항원 결합 및/또는 가변 영역을 포함하는 온전한 항체의 일부이다. 항체 단편의 예에는 다음이 포함된다:
(i) VL, VH, CL 및 CH1 도메인으로 구성된 1가 단편인 Fab 단편;
(ii) 힌지 영역에서 이황화 가교에 의해 연결된 2개의 Fab 단편을 포함하는 2가 단편인 F(ab')2 단편;
(iii) VH 및 CH1 도메인으로 구성된 Fd 단편;
(iv) 항체의 단일 아암의 VL 및 VH 도메인으로 구성된 Fv 단편,
(v) VH 또는 VL 도메인으로 구성된 dAb 단편(문헌[Ward et al., (1989) Nature 341:544-546]; 이는 그 전체가 본원에 참조로 포함됨); 및
(vi) 특이적 항원 결합 기능을 유지하는 단리된 상보성 결정 영역(CDR).
항체의 추가 예는 중쇄(5'-VH-(임의 힌지)-CH2-CH3-3')의 이량체를 포함하고 경쇄가 결여된 H2 항체이다.
단일쇄 항체(예를 들어, scFv)는 일반적으로 사용되는 단편이다. 다중특이적 항체는 항체 단편으로부터 형성될 수 있다. 본 발명의 항체는 적절한 경우 임의의 이러한 형식을 사용할 수 있다.
선택적으로, 항체 면역글로불린 도메인은 추가적인 폴리펩티드 서열 및/또는 표지, 태그, 독소, 또는 다른 분자에 융합되거나 접합될 수 있다. 항체 면역글로불린 도메인은 하나 이상의 상이한 항원 결합 영역에 융합되거나 접합되어 ICOS 외에 제2 항원에 결합할 수 있는 분자를 제공할 수 있다. 본 발명의 항체는 (i) ICOS에 대한 항체 항원 결합 부위 및 (ii) 다른 항원(예를 들어, PD-L1)을 인식하는 추가 항원 결합 부위(선택적으로 본원에 기재된 바와 같은 항체 항원 결합 부위)를 포함하는 다중특이적 항체, 예를 들어 이중특이적 항체일 수 있다.
항체는 일반적으로 항체 VH 및/또는 VL 도메인을 포함한다. 항체의 단리된 VH 및 VL 도메인 또한 본 발명의 일부이다. 항체 가변 도메인은 상보성 결정 영역(CDR, 즉 CDR1, CDR2, 및 CDR3) 및 프레임워크 영역(FR)의 아미노산 서열을 포함하는 항체의 경쇄 및 중쇄 부분이다. 따라서, 각각의 VH 및 VL 도메인 내에 CDR 및 FR이 있다. VH 도메인은 HCDR의 세트를 포함하고, VL 도메인은 LCDR의 세트를 포함한다. VH는 중쇄의 가변 도메인을 지칭한다. VL은 경쇄의 가변 도메인을 지칭한다. 각각의 VH 및 VL은 전형적으로 아미노 말단에서 카복시 말단으로 하기 순서로 배열된 3개의 CDR 및 4개의 FR로 이루어진다: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4. 본 발명에서 사용된 방법에 따르면, CDR 및 FR에 할당된 아미노산 위치는 Kabat(Sequences of Proteins of Immunological Interest (National Institutes of Health, Bethesda, Md., 1987 and 1991)) 또는 IMGT 명명법에 따라 정의될 수 있다. 항체는 VH CDR1, CDR2 및 CDR3 그리고 프레임워크를 포함하는 항체 VH 도메인을 포함할 수 있다. 이는 대안적으로, 또는 또한 VL CDR1, CDR2 및 CDR3 그리고 프레임워크를 포함하는 항체 VL 도메인을 포함할 수 있다. 본 발명에 따른 항체 VH 및 VL 도메인과 CDR의 예는 본 개시내용의 일부를 형성하는 첨부된 서열 목록에 나열되어 있다. 서열 목록에 나타낸 CDR은 IMGT 시스템에 따라 정의된다[18]. 본원에 개시된 모든 VH 및 VL 서열, CDR 서열, CDR 세트 및 HCDR 세트 및 LCDR 세트는 본 발명의 양태 및 구현예를 나타낸다. 본원에 기재된 바와 같이, "CDR 세트"는 CDR1, CDR2, 및 CDR3을 포함한다. 따라서, HCDR 세트는 HCDR1, HCDR2, 및 HCDR3을 지칭하고, LCDR 세트는 LCDR1, LCDR2, 및 LCDR3을 지칭한다. 달리 명시되지 않는 한, "CDR 세트"는 HCDR 및 LCDR을 포함한다.
본 발명의 항체는 본원에 기재된 하나 이상의 CDR, 예를 들어 CDR3, 및 선택적으로 CDR1 및 CDR2를 또한 포함하여 CDR 세트를 형성할 수 있다. CDR 또는 CDR 세트는 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009 중 임의의 것의 CDR 또는 CDR 세트일 수 있거나, 본원에 기재된 바와 같은 이의 변이체일 수 있다.
본 발명은 항체 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009 중 임의의 것의 HCDR1, HCDR2 및/또는 HCDR3 및/또는 이러한 항체 중 하나의 LCDR1, LCDR2 및/또는 LCDR3(예를 들어, CDR 세트)을 포함하는 항체를 제공한다. 항체는 이들 항체 중 하나의 VH CDR 세트를 포함할 수 있다. 선택적으로 이는 또한 이들 항체 중 하나의 VL CDR 세트를 포함할 수 있으며, VL CDR은 VH CDR과 동일하거나 상이한 항체로부터 유래될 수 있다.
개시된 HCDR 세트를 포함하는 VH 도메인 및/또는 개시된 LCDR 세트를 포함하는 VL 도메인이 또한 본 발명에 의해 제공된다.
전형적으로, VH 도메인은 VL 도메인과 쌍을 이루어 항체 항원-결합 부위를 제공하지만, 아래에서 추가로 논의되는 바와 같이 VH 또는 VL 도메인 단독이 항원에 결합하는 데 사용될 수 있다. STIM003 VH 도메인은 STIM003 VL 도메인과 쌍을 이루어 STIM003 VH 및 VL 도메인 둘 모두를 포함하는 항체 항원 결합 부위가 형성될 수 있다. 유사한 구현예가 본원에 개시된 다른 VH 및 VL 도메인에 대해 제공된다. 또 다른 구현예에서, STIM003 VH는 STIM003 VL 이외의 VL 도메인과 쌍을 이룬다. 경쇄 난잡함은 상응하는 분야에서 잘 확립되어 있다. 다시 말하면, 본 발명에 개시된 다른 VH 및 VL 도메인에 대한 유사한 구현예가 본 발명에 의해 제공된다.
따라서, 임의의 항체 STIM001, STIM002, STIM003, STIM004 및 STIM005의 VH는 임의의 항체 STIM001, STIM002, STIM003, STIM004 및 STIM005의 VL과 쌍을 이룰 수 있다. 또한, 임의의 항체 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009의 VH는 임의의 항체 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 또는 STIM009의 VL과 쌍을 이룰 수 있다.
항체는 항체 프레임워크 내에 하나 이상의 CDR, 예를 들어 CDR의 세트를 포함할 수 있다. 프레임워크 영역은 인간 생식계열 유전자 절편 서열일 수 있다. 따라서, 항체는 인간 생식계열 프레임워크에 HCDR 세트를 포함하는 VH 도메인을 갖는 인간 항체일 수 있다. 일반적으로, 항체는 또한 예를 들어 인간 생식계열 프레임워크에 LCDR의 세트를 포함하는 VL 도메인을 갖는다. 항체 "유전자 절편", 예를 들어 VH 유전자 절편, D 유전자 절편, 또는 JH 유전자 절편은 항체의 상응하는 부분이 유래된 핵산 서열을 갖는 올리고뉴클레오티드를 지칭하며, 예를 들어 VH 유전자 절편은 FR1에서부터 CDR3의 일부까지의 폴리펩티드 VH 도메인에 해당하는 핵산 서열을 포함하는 올리고뉴클레오티드이다. 인간 V, D, 및 J 유전자 절편은 재조합되어 VH 도메인을 생성하고, 인간 V 및 J 절편은 재조합되어 VL 도메인을 생성한다. D 도메인 또는 영역은 항체 쇄의 다양성 도메인 또는 영역을 의미한다. J 도메인 또는 영역은 항체 쇄의 연결 도메인 또는 영역을 의미한다. 체세포성 과돌연변이는 상응하는 유전자 절편과 정확하게 일치하지 않거나 정렬되지 않는 프레임워크 영역을 갖는 항체 VH 또는 VL 도메인을 생성할 수 있지만, 서열 정렬을 사용하여 가장 가까운 유전자 절편을 식별하여 특정 VH 또는 VL 도메인이 유래되는 유전자 절편의 특정 조합을 확인할 수 있다. 항체 서열을 유전자 절편과 정렬하는 경우, 항체 아미노산 서열은 유전자 절편에 의해 암호화된 아미노산 서열과 정렬될 수 있거나, 항체 뉴클레오티드 서열은 유전자 절편의 뉴클레오티드 서열과 직접 정렬될 수 있다.
관련 항체 및 인간 생식계열 서열에 대한 STIM 항체 VH 및 VL 도메인 서열의 정렬을 도 5, 도 6 및 도 7에 나타낸다.
본 발명의 항체는 인간 항체일 수 있거나, 인간 가변 영역 및 비인간(예를 들어, 마우스) 불변 영역을 포함하는 키메라 항체일 수 있다. 예를 들어, 본 발명의 항체는 인간 가변 영역을 갖고, 선택적으로 인간 불변 영역을 또한 갖는다.
따라서, 항체는 선택적으로 불변 영역 또는 이의 일부, 예를 들어 인간 항체 불변 영역 또는 이의 일부를 포함한다. 예를 들어, VL 도메인은 이의 C-말단에서 항체 경쇄 카파 또는 람다 불변 도메인에 부착될 수 있다. 마찬가지로, 항체 VH 도메인은 이의 C-말단에서 임의의 항체 이소형, 예를 들어 IgG, IgA, IgE, 및 IgM 및 임의의 이소형 하위클래스, 예컨대 IgG1 또는 IgG4로부터 유래된 면역글로불린 중쇄 불변 영역의 전체 또는 일부(예를 들어, CH1 도메인 또는 Fc 영역)에 부착될 수 있다.
인간 중쇄 불변 영역의 예를 표 S1에 나타낸다.
본 발명의 항체의 불변 영역은 대안적으로 비인간 불변 영역일 수 있다. 예를 들어, 항체가 트랜스제닉 동물에서 생성되는 경우(예는 본원의 다른 곳에서 기재됨), 인간 가변 영역 및 비인간(숙주 동물) 불변 영역을 포함하는 키메라 항체가 생성될 수 있다. 일부 트랜스제닉 동물은 완전 인간 항체를 생성한다. 다른 것들은 키메라 중쇄 및 완전 인간 경쇄를 포함하는 항체를 생성하도록 조작되었다. 항체가 하나 이상의 비인간 불변 영역을 포함하는 경우, 이는 인간 불변 영역으로 대체되어, 면역원성이 감소됨으로써 치료 조성물로서 인간에 투여하기 보다 적합한 항체를 제공할 수 있다.
파파인 효소로 항체를 소화하면 "Fab" 단편으로도 알려진 두 개의 동일한 항원 결합 단편 및 항원 결합 활성은 없지만 결정화 능력이 있는 "Fc" 단편이 생성된다. 본원에 사용된 "Fab"은 각각의 중쇄 및 경쇄의 하나의 불변 도메인 및 하나의 가변 도메인을 포함하는 항체의 단편을 지칭한다. 본원에서 용어 "Fc 영역"은 자연 서열 Fc 영역 및 변이체 Fc 영역을 포함하는 면역글로불린 중쇄의 C-말단 영역을 정의하기 위해 사용된다. "Fc 단편"은 이황화물에 의해 함께 고정된 두 H 쇄의 카복시 말단 부분을 지칭한다. 항체의 효과기 기능은 특정 유형의 세포에서 발견되는 Fc 수용체(FcR)에 의해 인식되는 Fc 영역의 서열에 의해 결정된다. 효소 펩신으로 항체를 소화하면 항체 분자의 두 아암이 연결된 상태로 유지되고 두 개의 항원 결합 부위를 포함하는 F(ab')2 단편이 생성된다. F(ab')2 단편은 항원을 가교시키는 능력을 갖는다.
본원에 사용된 "Fv"는 항원 인식 부위 및 항원 결합 부위를 모두 보유하는 항체의 적어도 단편을 지칭한다. 이 영역은 단단하고, 비공유적인 또는 공유적인 회합으로 1개의 중쇄 및 1개의 경쇄 가변 도메인의 이량체로 구성된다. 이러한 구성에서, 각각의 가변 도메인의 3개의 CDR이 상호작용하여 VH-VL 이량체의 표면 상의 항원 결합 부위를 정의한다. 전체적으로, 6개의 CDR이 항체에 항원 결합 특이성을 부여한다. 그러나 단일 가변 도메인(또는 항원에 특이적인 3개의 CDR만을 포함하는 Fv의 절반)도 항원을 인식하고 결합하는 능력을 갖지만, 전체 결합 부위에 비해 친화도가 낮다.
본원에 개시된 항체는 혈청 반감기를 증가시키거나 감소시키도록 변형될 수 있다. 한 구현예에서, 항체의 생물학적 반감기를 증가시키기 위해 하기 돌연변이: T252L, T254S, 또는 T256F 중 하나 이상이 도입된다. 미국 특허 제5,869,046호 및 제6,121,022호(문헌에 기재된 변형은 본원에 참조로 포함됨)에 기재된 바와 같이, 생물학적 반감기는 또한 IgG의 Fc 영역의 CH2 도메인의 2개 루프로부터 취한 회수(salvage) 수용체 결합 에피토프를 함유하도록 중쇄 불변 영역 CH1 도메인 또는 CL 영역을 변경함으로써 증가될 수 있다. 다른 구현예에서, 본 발명의 항체 또는 항원 결합 단편의 Fc 힌지 영역은 항체 또는 단편의 생물학적 반감기를 감소시키도록 돌연변이된다. 항체 또는 단편이 자연 Fc-힌지 도메인 스타필로코커스 단백질 A(SpA) 결합에 비해 손상된 SpA 결합을 갖도록 하나 이상의 아미노산 돌연변이가 Fc-힌지 단편의 CH2-CH3 도메인 인터페이스 영역 내로 도입된다. 혈청 반감기를 증가시키는 다른 방법은 당업자에게 알려져 있다. 따라서, 한 구현예에서, 항체 또는 단편은 PEG화된다. 다른 구현예에서, 항체 또는 단편은 알부민 결합 도메인, 예를 들어 알부민 결합 단일 도메인 항체(dAb)에 융합된다. 다른 구현예에서, 항체 또는 단편은 PAS화된다(즉, 큰 유체역학적 부피를 갖는 비전하 무작위 코일 구조를 형성하는 PAS로 구성된 폴리펩티드 서열의 유전적 융합(XL-Protein GmbH)). 다른 구현예에서, 항체 또는 단편은 XTEN화(XTENylated)®/rPEG화된다(즉, 치료 펩티드에 대한 정확하지 않은 반복 펩티드 서열의 유전적 융합(Amunix, Versartis)). 다른 구현예에서, 항체 또는 단편은 ELP화된다(즉, ELP 반복 서열에 대한 유전적 융합(PhaseBio)). 이러한 다양한 반감기 연장 융합은 문헌[Strohl, BioDrugs (2015) 29:215-239]에 보다 상세히 기재되어 있으며, 예를 들어 표 2 및 6에 기재된 융합은 본원에 참조로 포함된다.
항체는 안정성을 증가시키는 변형된 불변 영역을 가질 수 있다. 따라서, 한 구현예에서, 중쇄 불변 영역은 Ser228Pro 돌연변이를 포함한다. 다른 구현예에서, 본원에 개시된 항체 및 단편은 시스테인 잔기의 수를 변경하도록 변형된 중쇄 힌지 영역을 포함한다. 이러한 변형은 경쇄 및 중쇄의 조립을 촉진하거나 항체의 안정성을 증가 또는 감소시키기 위해 사용될 수 있다.
1.6.9. Fc 효과기 기능, ADCC, ADCP 및 CDC
위에서 논의된 바와 같이, 항-ICOS 항체는 다양한 이소형 및 상이한 불변 영역으로 제공될 수 있다. 인간 IgG 항체 중쇄 불변 영역 서열의 예를 표 S1에 나타낸다. 항체의 Fc 영역은 주로 Fc 결합, 항체 의존적 세포 매개 세포독성(ADCC) 활성, 보체 의존적 세포독성(CDC) 활성 및 항체 의존적 세포 식세포작용(ADCP) 활성과 관련하여 그의 효과기 기능을 결정한다. 효과기 T 세포 기능과 구별되는 이러한 "세포 효과기 기능"은 Fc 수용체를 보유하는 세포의 표적 세포 부위로의 모집에 관여하여 항체-결합 세포의 사멸을 초래한다. ADCC 및 CDC 외에도 ADCP 메커니즘은 항체-결합 T 세포를 고갈시키고 이에 따라 결실을 위해 높은 ICOS 발현 TReg를 표적화하는 수단을 표시한다[19].
세포 효과기 기능 ADCC, ADCP 및/또는 CDC는 또한 Fc 영역이 결여된 항체에 의해 나타날 수 있다. 항체는 다수의 상이한 항원-결합 부위를 포함할 수 있으며, 하나는 ICOS에 대한 것이고 또 다른 하나는 표적 분자의 참여가 ADCC, ADCP 및/또는 CDC를 유도하는 표적 분자에 대한 것, 예를 들어 링커에 의해 연결된 2개의 scFv 영역을 포함하는 항체를 포함할 수 있으며, 하나의 scFv는 효과기 세포와 결합할 수 있다.
본 발명에 따른 항체는 ADCC, ADCP 및/또는 CDC를 나타내는 것일 수 있다. 대안적으로, 본 발명에 따른 항체는 ADCC, ADCP 및/또는 CDC 활성이 결여될 수 있다. 두 경우 모두, 본 발명에 따른 항체는 하나 이상의 유형의 Fc 수용체에 결합하는 Fc 영역을 포함할 수 있거나 선택적으로 이것이 결여될 수 있다. 상이한 항체 형식의 사용, 그리고 FcR 결합 및 세포 효과기 기능의 존재 또는 부재는 항체가 본원의 다른 곳에서 논의된 바와 같이 특정 치료 목적에서 사용하기 위해 맞춤화될 수 있게 한다.
일부 치료 적용에 적합한 항체 형식은 야생형 인간 IgG1 불변 영역을 사용한다. 불변 영역은 선택적으로 ADCC 및/또는 CDC 및/또는 ADCP 활성을 갖는, 효과기-활성화 IgG1 불변 영역일 수 있다. 적합한 야생형 인간 IgG1 불변 영역 서열은 SEQ ID NO: 340(IGHG1*01)이다. 인간 IgG1 불변 영역의 추가 예를 표 S1에 나타낸다.
인간 질환의 마우스 모델에서 후보 치료 항체를 시험하기 위해, 효과기 양성 인간 불변 영역 대신 마우스 IgG2a(mIgG2a)와 같은 효과기 양성 마우스 불변 영역이 포함될 수 있다.
불변 영역은 향상된 ADCC 및/또는 CDC 및/또는 ADCP에 대해 조작될 수 있다.
Fc 매개 효과의 효능은 다양한 확립 기술에 의해 Fc 도메인을 조작함으로써 향상될 수 있다. 이러한 방법은 특정 Fc-수용체에 대한 친화도를 증가시키고, 이에 따라 잠재적으로 다양한 활성화 향상 프로파일을 생성한다. 이는 하나의 또는 몇몇 아미노산 잔기 변형에 의해 달성될 수 있다[20]. 잔기 Asn297 상에 특정 돌연변이 또는 변경된 글리코실화를 포함하는(예를 들어, N297Q, EU 인덱스 넘버링) 인간 IgG1 불변 영역은 Fc 수용체에 대한 결합을 향상시키는 것으로 나타났다. 예시적 돌연변이는 인간 IgG1 불변 영역에 대해 239, 332 및 330(또는 다른 IgG 이소형의 동등한 위치)으로부터 선택되는 잔기 중 하나 이상이다. 따라서 항체는 N297Q, S239D, I332E 및 A330L(EU 인덱스 넘버링)로부터 독립적으로 선택된 하나 이상의 돌연변이를 갖는 인간 IgG1 불변 영역을 포함할 수 있다. FcRn에 대한 결합을 향상시키기 위해 삼중 돌연변이(M252Y/S254T/T256E)가 사용될 수 있으며, FcRn 결합에 영향을 미치는 다른 돌연변이는 [21]의 표 2에 논의되어 있고, 이들 중 임의의 것이 본 발명에서 사용될 수 있다.
Fc 수용체에 대한 증가된 친화도는 또한 예를 들어 저푸코실화된 또는 탈푸코실화된 변이체 하에서 생성되어 Fc 도메인의 천연 글리코실화 프로파일을 변경함으로써 달성될 수 있다[22]. 푸코실화되지 않은 항체는 푸코스 잔기가 없는 Fc의 복합체 유형 N-글리칸의 트리-만노실 코어 구조를 보유한다. Fc N-글리칸의 코어 푸코스 잔기가 결여된 이러한 당조작된 항체는 FcγRIIIa 결합 능력의 향상으로 인해 푸코실화된 등가물보다 강력한 ADCC를 나타낼 수 있다. 예를 들어, ADCC를 증가시키기 위해 힌지 영역의 잔기가 변경되어 Fc-감마 RIII에 대한 결합을 증가시킬 수 있다[23]. 따라서, 항체는 야생형 인간 IgG 중쇄 불변 영역의 변이체인 인간 IgG 중쇄 불변 영역을 포함할 수 있으며, 변이체 인간 IgG 중쇄 불변 영역은 야생형 인간 IgG 중쇄 불변 영역이 인간 Fcγ 수용체에 결합하는 것보다 높은 친화도로 FcyRIIB 및 FcyRIIA로 구성된 군으로부터 선택된 인간 Fcγ 수용체에 결합한다. 항체는 야생형 인간 IgG 중쇄 불변 영역의 변이체인 인간 IgG 중쇄 불변 영역을 포함할 수 있으며, 변이체 인간 IgG 중쇄 불변 영역은 야생형 인간 IgG 중쇄 불변 영역이 인간 FcγRIIB에 결합하는 것보다 높은 친화도로 인간 FcγRIIB에 결합한다. 변이체 인간 IgG 중쇄 불변 영역은 변이체 인간 IgG1, 변이체 인간 IgG2, 또는 변이체 인간 IgG4 중쇄 불변 영역일 수 있다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 G236D, P238D, S239D, S267E, L328F 및 L328E(EU 인덱스 넘버링 시스템)로부터 선택된 하나 이상의 아미노산 돌연변이를 포함한다. 또 다른 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 S267E 및 L328F; P238D 및 L328E; P238D 및 E233D, G237D, H268D, P271G 및 A330R로 구성된 군으로부터 선택된 하나 이상의 치환; P238D, E233D, G237D, H268D, P271G 및 A330R; G236D 및 S267E; S239D 및 S267E; V262E, S267E 및 L328F; 및 V264E, S267E 및 L328F(EU 인덱스 넘버링 시스템)로 구성된 군으로부터 선택된 아미노산 돌연변이의 세트를 포함한다. CDC의 향상은 전통적인 보체 활성화 캐스케이드의 첫 번째 구성요소인 C1q에 대한 친화도를 증가시키는 아미노산 변화에 의해 달성될 수 있다[24]. 또 다른 접근은 C1q에 대한 IgG3의 더 높은 친화도를 이용하는 인간 IgG1 및 인간 IgG3 절편으로부터 생성된 키메라 Fc 도메인을 생성하는 것이다[25]. 본 발명의 항체는 C1q 결합 및/또는 감소 또는 폐지된 CDC 활성을 변경하기 위해 잔기 329, 331 및/또는 322에 돌연변이된 아미노산을 포함할 수 있다. 또 다른 구현예에서, 본원에 개시된 항체 또는 항체 단편은 잔기 231 및 239에 변형을 갖는 Fc 영역을 함유할 수 있으며, 이에 따라 아미노산이 대체되어 보체를 고정하는 항체의 능력을 변경한다. 한 구현예에서, 항체 또는 단편은 E345K, E430G, R344D 및 D356R로부터 선택된 하나 이상의 돌연변이, 특히 R344D 및 D356R(EU 인덱스 넘버링 시스템)을 포함하는 이중 돌연변이를 포함하는 불변 영역을 갖는다.
WO2008/137915는 향상된 효과기 기능을 갖는 변형된 Fc 영역을 갖는 항-ICOS 항체를 기재한다. 항체는 VH 및 VK 도메인 그리고 야생형 Fc 영역을 포함하는 모 항체에 의해 매개되는 ADCC 활성 수준과 비교하여 향상된 ADCC 활성을 매개하는 것으로 보고되었다. 본 발명에 따른 항체는 본원에 기재된 바와 같은 효과기 기능을 갖는 이러한 변이체 Fc 영역을 사용할 수 있다.
항체의 ADCC 활성은 WO2008/137915에 개시된 검정과 같은 검정에서 결정될 수 있다. 항-ICOS 항체의 ADCC 활성은 미국 특허 제9,957,323호의 실시예 10에 기재된 바와 같이 ICOS 양성 T 세포주를 사용하여 시험관내에서 결정될 수 있다. 항-PD-L1 항체의 ADCC 활성은 PD-L1 발현 세포를 사용한 ADCC 검정에서 시험관내에서 결정될 수 있다.
특정 적용의 경우(예를 들어, 예방접종의 측면에서) Fc 효과기 기능이 없는 항체를 사용하는 것이 바람직할 수 있다. 항체는 불변 영역 없이 또는 Fc 영역 없이 제공될 수 있다 - 이러한 항체 형식의 예는 본원의 다른 곳에서 기재된다. 대안적으로, 항체는 효과기 비포함(null)인 불변 영역을 가질 수 있다. 항체는 Fcγ 수용체에 결합하지 않는 중쇄 불변 영역을 가질 수 있으며, 예를 들어 불변 영역은 Leu235Glu 돌연변이를 포함할 수 있다(즉, 야생형 류신 잔기가 글루탐산 잔기로 돌연변이되는 경우). 중쇄 불변 영역에 대한 또 다른 선택적 돌연변이는 Ser228Pro이며, 이는 안정성을 증가시킨다. 중쇄 불변 영역은 Leu235Glu 돌연변이 및 Ser228Pro 돌연변이 둘 모두를 포함하는 IgG4일 수 있다. 이 "IgG4-PE" 중쇄 불변 영역은 효과기 비포함이다.
대안적인 효과기 비포함 인간 불변 영역은 비활성화 IgG1이다. 비활성화 IgG1 중쇄 불변 영역은 위치 235 및/또는 237(EU 인덱스 넘버링)에 알라닌을 함유할 수 있으며, 예를 들어 이는 L235A 및/또는 G237A 돌연변이("LAGA")를 포함하는 IgG1*01 서열일 수 있다.
변이체 인간 IgG 중쇄 불변 영역은 인간 FcγRIIIA, 인간 FcγRIIA 또는 인간 FcγRI에 대한 IgG의 친화도를 감소시키는 하나 이상의 아미노산 돌연변이를 포함할 수 있다. 한 구현예에서, FcγRIIB는 대식구, 단핵구, B 세포, 수지상 세포, 내피 세포 및 활성화 T 세포로 구성된 군으로부터 선택된 세포 상에서 발현된다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 하기 아미노산 돌연변이 G236A, S239D, F243L, T256A, K290A, R292P, S298A, Y300L, V305I, A330L, I332E, E333A, K334A, A339T 및 P396L(EU 인덱스 넘버링 시스템) 중 하나 이상을 포함한다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 S239D; T256A; K290A; S298A; I332E; E333A; K334A; A339T; S239D 및 I332E; S239D, A330L 및 I332E; S298A, E333A 및 K334A; G236A, S239D 및 I332E; 그리고 F243L, R292P, Y300L, V305I 및 P396L(EU 인덱스 넘버링 시스템)로 구성된 군으로부터 선택된 아미노산 돌연변이의 세트를 포함한다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 S239D, A330L 또는 I332E 아미노산 돌연변이(EU 인덱스 넘버링 시스템)를 포함한다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 S239D 및 I332E 아미노산 돌연변이(EU 인덱스 넘버링 시스템)를 포함한다. 한 구현예에서, 변이체 인간 IgG 중쇄 불변 영역은 S239D 및 I332E 아미노산 돌연변이(EU 인덱스 넘버링 시스템)를 포함하는 변이체 인간 IgG1 중쇄 불변 영역이다. 한 구현예에서, 항체 또는 단편은 비푸코실화된 Fc 영역을 포함한다. 다른 구현예에서, 항체 또는 이의 단편은 탈푸코실화된다. 다른 구현예에서, 항체 또는 단편은 저푸코실화된다.
항체는 Fc 수용체의 하나 이상의 유형에 결합하지만 세포 효과기 기능을 유도하지 않는, 즉 ADCC, CDC, 또는 ADCP 활성을 매개하지 않는 중쇄 불변 영역을 가질 수 있다. 이러한 불변 영역은 ADCC, CDC, 또는 ADCP 활성을 유발하는 특정 Fc 수용체(들)에 결합하지 못할 수 있다.
1.6.10. 항체 생성 및 변형
항체를 식별하고 제조하는 방법은 잘 알려져 있다. 항체는 ICOS 또는 이의 단편 또는 관심 ICOS 서열 모티프를 포함하는 합성 펩티드로 면역화된 트랜스제닉 마우스(예를 들어, Kymouse™, Velocimouse®, Omnimouse®, Xenomouse®, HuMab Mouse® 또는 MeMo Mouse®), 래트(예를 들어, Omnirat®), 낙타류, 상어, 토끼, 닭 또는 다른 비인간 동물을 사용하고, 이어서 선택적으로 불변 영역 및/또는 가변 영역을 인간화하여 인간 또는 인간화 항체를 생성함으로써 생성될 수 있다. 한 예로, 당업자에게 자명할 디스플레이 기술, 예컨대 효모, 파지, 또는 리보솜 디스플레이를 사용할 수 있다. 예를 들어 디스플레이 기술을 사용하는 표준 친화도 성숙은 트렌스제닉 동물, 파지 디스플레이 라이브러리, 또는 다른 라이브러리로부터 항체 선도를 단리한 후 추가 단계에서 수행될 수 있다. 적합한 기술의 대표적인 예는 US20120093818(Amgen, Inc), 예를 들어 문단 [0309] 내지 [0346]에 제시된 방법에 기재되어 있으며, 이의 전체가 본원에 참조로 포함된다.
인간 ICOS 항원을 사용하는 ICOS 녹아웃 비인간 동물의 면역화는 인간 및 비인간 ICOS 둘 모두를 인식하는 항체의 생성을 촉진한다. 본원에 기재되고 실시예에 예시된 바와 같이, ICOS 녹아웃 마우스는 인간 ICOS를 발현하는 세포로 면역화되어 마우스에서 인간 및 마우스 ICOS에 대한 항체의 생산을 자극할 수 있으며, 이는 회수되고 인간 ICOS 및 마우스 ICOS에 대한 결합에 대해 시험될 수 있다. 따라서 교차반응성 항체가 선택될 수 있으며, 이는 본원에 기재된 바와 같이 다른 바람직한 특성에 대해 스크리닝될 수 있다. 동물에서 내인성 항원(예를 들어, 내인성 마우스 항원)의 발현이 녹아웃된 항원을 사용하는 동물 면역화를 통해 항원(예를 들어, 인간 항원)에 대한 항체를 생성하는 방법은 인간 가변 도메인을 포함하는 항체를 생성할 수 있는 동물에서 수행될 수 있다. 이러한 동물의 게놈은 인간 가변 영역 유전자 절편, 및 선택적으로 내인성 불변 영역 또는 인간 불변 영역을 암호화하는 인간 또는 인간화 면역글로불린 유전자좌를 포함하도록 조작될 수 있다. 인간 가변 영역 유전자 절편의 재조합은 인간 항체를 생성하며, 이는 비인간 또는 인간 불변 영역을 가질 수 있다. 비인간 불변 영역은 이어서 항체가 인간의 생체내 사용을 위해 의도되는 경우 인간 불변 영역으로 대체될 수 있다. 이러한 방법 및 녹아웃 트랜스제닉 동물은 WO2013/061078에 기재되어 있다.
일반적으로 Kymouse™, VELOCIMMUNE® 또는 다른 마우스 또는 래트(주지된 바와 같이 선택적으로 ICOS 녹아웃 마우스 또는 래트)는 관심 항원으로 유발접종될 수 있으며 림프 세포(예컨대, B 세포)가 항체를 발현하는 마우스로부터 회수된다. 림프 세포는 골수종 세포주와 융합되어 불멸 하이브리도마 세포주를 제조할 수 있으며, 이러한 하이브리도마 세포주가 스크리닝되고 선택되어 관심 항원에 특이적인 항체를 생산하는 하이브리도마 세포주를 식별한다. 중쇄 및 경쇄의 가변 영역을 암호화하는 DNA가 단리되고 중쇄 및 경쇄의 원하는 이소형 불변 영역에 연결될 수 있다. 이러한 항체 단백질은 CHO 세포와 같은 세포에서 생산될 수 있다. 대안적으로, 항원 특이적 키메라 항체 또는 경쇄 및 중쇄의 가변 도메인을 암호화하는 DNA가 항원 특이적 림프구로부터 직접적으로 단리될 수 있다.
처음에, 인간 가변 영역 및 마우스 불변 영역을 갖는 고친화도 키메라 항체가 단리된다. 항체는 친화도, 선택성, 효능작용, T 세포 의존적 사멸, 중화 효능, 에피토프 등을 포함하는 원하는 특성에 대해 특성규명되고 선택된다. 마우스 불변 영역은 선택적으로 원하는 인간 불변 영역으로 대체되어 본 발명의 완전 인간 항체, 예를 들어 야생형 또는 변형된 IgG1 또는 IgG4(예를 들어 US2011/0065902(그 전체가 본원에 참조로 포함됨)의 SEQ ID NO: 751, 752, 753)를 생성한다. 선택된 불변 영역은 특정 용도, 고친화도 항원 결합 및 가변 영역에 존재하는 표적 특이성 특성에 따라 달라질 수 있다.
따라서, 추가 양태에서, 본 발명은 인간 또는 인간화 면역글로불린 유전자좌를 포함하는 게놈을 갖는 트랜스제닉 비인간 포유류를 제공하며, 포유류는 ICOS를 발현하지 않는다. 포유류는 예를 들어 녹아웃 마우스 또는 래트, 또는 다른 실험 동물 종일 수 있다. Kymouse™와 같은 트랜스제닉 마우스는 상응하는 내인성 마우스 면역글로불린 유전자좌에 삽입된 인간 중쇄 및 경쇄 면역글로불린 유전자좌를 함유한다. 본 발명에 따른 트랜스제닉 포유류는 이러한 표적화된 삽입을 포함하는 것일 수도 있고, 인간 중쇄 및 경쇄 면역글로불린 유전자좌 또는 그의 게놈에 무작위로 삽입되거나, 내인성 Ig 유전자좌 이외의 유전자좌에 삽입되거나 추가 염색체 또는 염색체 단편에 제공되는 면역글로불린 유전자를 함유할 수도 있다.
본 발명의 추가 양태는 ICOS에 대한 항체를 생산하기 위한 이러한 비-인간 포유류의 용도, 및 이러한 포유류에서 항체 또는 항체 중쇄 및/또는 경쇄 가변 도메인을 생산하는 방법이다.
인간 및 비-인간 ICOS의 세포외 도메인에 결합하는 항체를 생산하는 방법은 인간 또는 인간화 면역글로불린 유전자좌를 포함하는 게놈을 갖는 트랜스제닉 비-인간 포유류를 제공하는 단계로서, 포유류는 ICOS를 발현하지 않는, 단계, 및
(a) 포유류를 인간 ICOS 항원(예를 들어, 인간 ICOS를 발현하는 세포 또는 정제된 재조합 ICOS 단백질)으로 면역화하는 단계;
(b) 포유류에 의해 생성된 항체를 단리하는 단계;
(c) 인간 ICOS 및 비-인간 ICOS에 결합하는 능력에 대해 항체를 시험하는 단계; 및
(d) 인간 및 비-인간 ICOS 둘 모두에 결합하는 하나 이상의 항체를 선택하는 단계를 포함할 수 있다.
인간 ICOS 및 비-인간 ICOS에 결합하는 능력에 대한 시험은 표면 플라즈몬 공명, HTRF, FACS 또는 본원에 기재된 임의의 다른 방법을 사용하여 수행될 수 있다. 선택적으로, 인간 및 마우스 ICOS에 대한 결합 친화도가 결정된다. 인간 ICOS 및 마우스 ICOS에 대한 결합의 친화도 또는 친화도 배율-차이가 결정될 수 있고, 따라서 종 교차반응성을 표시하는 항체가 선택될 수 있다(선택 기준으로 사용될 수 있는 친화도 역치 및 배율 차이는 본원의 다른 곳에서 예시됨). 각각 인간 및 마우스 ICOS 수용체에 대한 인간 및 마우스 ICOS 리간드 결합을 억제하는 항체의 중화 효능, 또는 중화 효능의 배율-차이가 또한 또는 대안적으로 교차반응성 항체를 스크리닝하기 위한 방식으로서, 예를 들어 HTRF 검정에서 결정될 수 있다. 다시, 선택 기준으로 사용될 수 있는 가능한 역치 및 배율-차이가 본원의 다른 곳에서 예시된다.
방법은 면역화된 포유류와 동일한 종 또는 상이한 종으로부터의 비-인간 ICOS에 결합하는 능력에 대해 항체를 시험하는 단계를 포함할 수 있다. 따라서, 트랜스제닉 포유류가 마우스(예를 들어, Kymouse™)인 경우, 항체는 마우스 ICOS에 결합하는 능력에 대해 시험될 수 있다. 트랜스제닉 포유류가 래트인 경우, 항체는 래트 ICOS에 결합하는 능력에 대해 시험될 수 있다. 그러나 또 다른 종의 비인간 ICOS에 대한 단리된 항체의 교차반응성을 결정하는 것이 동일하게 유용할 수 있다. 따라서, 염소에서 생성된 항체가 래트 또는 마우스 ICOS에 대한 결합에 대해 시험될 수 있다. 선택적으로 염소 ICOS에 대한 결합이 대신 또는 추가적으로 결정될 수 있다.
다른 구현예에서, 트랜스제닉 비인간 포유류는 인간 ICOS 대신에 비-인간 ICOS, 선택적으로 동일한 포유동물 종의 ICOS로 면역화될 수 있다(예를 들어, ICOS 녹아웃 마우스가 마우스 ICOS로 면역화될 수 있음). 그런 다음 인간 ICOS 및 비인간 ICOS로의 결합에 대한 단리된 항체의 친화도가 동일한 방식으로 결정되고, 인간 및 비인간 ICOS 둘 모두에 결합하는 항체가 선택된다.
선택된 항체의 항체 중쇄 가변 도메인 및/또는 항체 경쇄 가변 도메인을 암호화하는 핵산이 단리될 수 있다. 이러한 핵산은 전장 항체 중쇄 및/또는 경쇄, 또는 관련된 불변 영역(들)이 없는 가변 도메인(들)을 암호화할 수 있다. 주지된 바와 같이, 암호화 뉴클레오티드 서열은 마우스의 항체 생산 세포로부터 직접 얻을 수 있거나, B 세포가 불멸화되거나 융합되어 항체를 발현하고 이러한 세포로부터 얻은 핵산을 암호화하는 하이브리도마를 생성할 수 있다. 이어서 선택적으로, 가변 도메인(들)을 암호화하는 핵산은 인간 중쇄 불변 영역 및/또는 인간 경쇄 불변 영역을 암호화하는 뉴클레오티드 서열에 접합되어 인간 항체 중쇄 및/또는 인간 항체 경쇄를 암호화하는, 예를 들어 중쇄 및 경쇄 둘 모두를 포함하는 항체를 암호화하는 핵산을 제공한다. 본원의 다른 곳에서 기재된 바와 같이, 이 단계는 면역화된 포유류가 비-인간 불변 영역을 갖는 키메라 항체를 생산하는 경우 특히 유용하며, 이는 바람직하게는 인간 불변 영역으로 대체되어 인간에게 약제로서 투여될 때 면역원성이 더 적을 항체를 생성한다. 특정 인간 이소형 불변 영역의 제공이 또한 항체의 효과기 기능을 결정하는 데 유의하며, 여러 적합한 중쇄 불변 영역이 본원에서 논의된다.
항체 중쇄 및/또는 경쇄 가변 도메인을 암호화하는 핵산에 대한 다른 변경, 예컨대 본원에 기재된 바와 같은 잔기의 돌연변이 및 변이체의 생성이 수행될 수 있다.
단리된(선택적으로 돌연변이된) 핵산은 논의된 바와 같이 숙주 세포, 예를 들어 CHO 세포 내로 도입될 수 있다. 그런 다음 숙주 세포는 임의의 원하는 항체 형식으로 항체 또는 항체 중쇄 및/또는 경쇄 가변 도메인의 발현을 위한 조건 하에 배양된다. 일부 가능한 항체 형식, 예를 들어 전체 면역글로불린, 항원 결합 단편 및 다른 설계가 본원에 기재되어 있다.
서열이 본원에 구체적으로 개시된 임의의 VH 및 VL 도메인 또는 CDR의 가변 도메인 아미노산 서열 변이체가 논의된 바와 같이 본 발명에 따라 사용될 수 있다.
대규모 제조를 위해 항체 서열을 개선하거나, 정제를 용이하게 하거나, 안정성을 향상시키거나, 원하는 약학적 제형에 포함되도록 적합성을 개선하는 것을 포함하여, 변이체를 생성하는 것이 바람직할 수 있는 여러 이유가 있다. 예를 들어 1개의 아미노산을 대안적인 아미노산으로 치환시키고 (선택적으로, 이 위치에서 가능하게는 Cys 및 Met를 제외한 모든 자연 발생 아미노산을 함유하는 변이체를 생성함), 최상의 치환을 결정하도록 기능 및 발현에 대한 영향을 모니터링하기 위해, 항체 서열의 하나 이상의 표적 잔기에서 단백질 조작 작업을 수행할 수 있다. 일부 경우에는, 잔기를 Cys 또는 Met로 치환시키거나, 또는 이들 잔기를 서열에 도입하는 것이 바람직하지 않을 수 있으며, 그렇게 하는 경우 예를 들어 새로운 분자내 또는 분자간 시스테인-시스테인 결합의 형성을 통해 제조에 어려움이 생길 수 있기 때문이다. 선도 후보가 선택되고, 제조 및 임상 개발을 위해 변경되는 경우, 일반적으로 이의 항원 결합 성질을 가능한 한 적게 변화시키거나, 적어도 모 분자의 친화도 및 효능을 유지하는 것이 바람직할 것이다. 그러나 친화도, 교차반응성, 또는 중화 효능과 같은 주요 항체 특성을 조절하기 위해 변이체를 또한 생성할 수 있다.
항체는 개시된 H 및/또는 L CDR 세트 내에 하나 이상의 아미노산 돌연변이를 갖는 개시된 임의의 항체의 H 및/또는 L CDR 세트를 포함할 수 있다. 돌연변이는 아미노산 치환, 결실, 또는 삽입일 수 있다. 따라서, 예를 들어 개시된 H 및/또는 L CDR 세트 내에 하나 이상의 아미노산 치환이 있을 수 있다. 예를 들어, H 및/또는 L CDR의 세트 내에 최대 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 또는 2개의 돌연변이, 예를 들어 치환이 있을 수 있다. 예를 들어, HCDR3에서 최대 6, 5, 4, 3, 또는 2개의 돌연변이, 예를 들어 치환이 있을 수 있고/있거나, LCDR3에서 최대 6, 5, 4, 3, 또는 2개의 돌연변이, 예를 들어 치환이 있을 수 있다. 항체는 본원의 임의의 STIM 항체에 대해 제시된 HCDR, LCDR 세트 또는 6개의(H 및 L) CDR 세트를 포함할 수 있거나 1개 또는 2개의 보존적 치환을 갖는 CDR 세트를 포함할 수 있다.
하나 이상의 아미노산 돌연변이는 선택적으로 본원에 개시된 항체 VH 또는 VL 도메인의 프레임워크 영역에서 만들어질 수 있다. 예를 들어, 상응하는 인간 생식계열 절편 서열과 상이한 하나 이상의 잔기는 생식계열로 복귀될 수 있다. 예시적인 항-ICOS 항체의 VH 및 VL 도메인에 상응하는 인간 생식계열 유전자 절편 서열이 표 E12-1, 표 E12-2 및 표 E12-3에 나타내며, 상응하는 생식계열 서열에 대한 항체 VH 및 VL 도메인의 정렬을 도면에 나타낸다.
항체는 첨부된 서열 목록에 제시된 임의의 항체의 VH 도메인과 적어도 60, 70, 80, 85, 90, 95, 98, 또는 99%의 아미노산 서열 동일성을 갖는 VH 도메인을 포함할 수 있고/있거나 임의의 항체의 VL 도메인과 적어도 60, 70, 80, 85, 90, 95, 98, 또는 99%의 아미노산 서열 동일성을 갖는 VL 도메인을 포함할 수 있다. 두 아미노산 서열의 동일성(%)을 계산하는 데 사용할 수 있는 알고리즘에는 예를 들어 BLAST, FASTA, 또는 Smith-Waterman 알고리즘(예를 들어, 디폴트 파라미터를 사용)이 포함된다. 특정 변이체에는 하나 이상의 아미노산 서열 변경(아미노산 잔기의 부가, 결실, 치환, 및/또는 삽입)이 포함될 수 있다.
하나 이상의 프레임워크 영역 및/또는 하나 이상의 CDR에서 변경이 이루어질 수 있다. 변이체는 CDR 돌연변이 유발에 의해 선택적으로 제공된다. 변경은 일반적으로 기능 손실을 초래하지 않으므로, 변경된 아미노산 서열을 포함하는 항체는 ICOS에 결합하는 능력을 유지할 수 있다. 이는 예를 들어 본원에 기재된 검정에서 측정되는 바와 같이, 변경이 이루어지지 않은 항체와 동일한 정량적 결합 능력을 유지할 수 있다. 이렇게 변형된 아미노산 서열을 포함하는 항체는 ICOS에 결합하고/하거나 이를 억제하는 개선된 능력을 가질 수 있다.
변경은 하나 이상의 아미노산 잔기를 비자연 발생 또는 비표준 아미노산으로 대체하거나, 하나 이상의 아미노산 잔기를 비자연 발생 또는 비표준 형태로 변경시키거나, 하나 이상의 비자연 발생 또는 비표준 아미노산을 서열에 삽입하는 것을 포함할 수 있다. 본 발명의 서열에서 변경의 개수 및 위치의 예는 본원의 다른 곳에 기재되어 있다. 자연 발생 아미노산에는 표준 단일-문자 코드에 의해 G, A, V, L, I, M, P, F, W, S, T, N, Q, Y, C, K, R, H, D, E로 식별되는 20개의 "표준" L-아미노산이 포함된다. 비표준 아미노산에는 폴리펩티드 골격에 통합될 수 있거나 기존 아미노산 잔기의 변형에 의해 생성될 수 있는 다른 잔기가 포함된다. 비표준 아미노산은 자연 발생 또는 비자연 발생일 수 있다.
본원에서 사용된 바와 같이, 용어 "변이체"는 하나 이상의 아미노산 또는 핵산 결실, 치환, 또는 부가에 의해, 모 폴리펩티드 또는 핵산과 상이하지만 모 분자의 하나 이상의 특이적 기능 또는 생물학적 활성을 유지하는 펩티드 또는 핵산을 지칭한다. 아미노산 치환에는 아미노산이 상이한 자연 발생 아미노산 잔기로 대체되는 변형이 포함된다. 이러한 치환은 "보존적인" 것으로 분류될 수 있고, 이 경우에 폴리펩티드에 함유된 아미노산 잔기는 극성, 측쇄 관능성 또는 크기와 관련하여 유사한 특징을 갖는 또 다른 자연 발생 아미노산으로 대체된다. 이러한 보존적 치환은 당업계에 잘 알려져 있다. 본 발명에 포함되는 치환은 또한 "비보존적"일 수 있고, 펩티드에 존재하는 아미노산 잔기가 상이한 성질을 갖는 아미노산, 예컨대 상이한 그룹에서 유래된 자연 발생 아미노산으로 치환되거나(예를 들어, 하전된 또는 소수성 아미노산이 알라닌으로 치환됨), 또는 대안적으로, 자연 발생 아미노산이 비-통상적인 아미노산으로 치환된다. 일부 구현예에서, 아미노산 치환은 보존적이다. 폴리뉴클레오티드 또는 폴리펩티드와 관련하여 사용될 때 변이체라는 용어에, 각각 기준 폴리뉴클레오티드 또는 폴리펩티드와 비교하여(예를 들어, 야생형 폴리뉴클레오티드 또는 폴리펩티드와 비교하여) 일차, 이차, 또는 삼차 구조가 다를 수 있는 폴리뉴클레오티드 또는 폴리펩티드가 포함된다.
일부 양태에서, 당업계에 널리 알려진 방법을 사용하여 단리되거나 생성되는 "합성 변이체", "재조합 변이체", 또는 "화학적으로 변형된" 폴리뉴클레오티드 변이체 또는 폴리펩티드 변이체를 사용할 수 있다. "변형된 변이체"는 하기 기재된 바와 같이 보존적 또는 비보존적 아미노산 변화를 포함할 수 있다. 폴리뉴클레오티드 변화는 기준 서열에 의해 암호화된 폴리펩티드에서 아미노산 치환, 부가, 결실, 융합, 및 절단을 초래할 수 있다. 일부 양태 용도는 변이체의 기반인 펩티드 서열에서 일반적으로 발생하지 않는 아미노산 및 다른 분자의 삽입 및 치환을 비롯하여, 예를 들어 인간 단백질에서 일반적으로 발생하지 않는 오르니틴의 삽입으로 제한되지 않는 아미노산의 치환을 갖는 삽입 변이체, 결실 변이체, 또는 치환된 변이체를 포함한다. 폴리펩티드를 설명할 때 용어 "보존적 치환"은 폴리펩티드의 활성을 실질적으로 변경시키지 않는 폴리펩티드의 아미노산 조성에서의 변화를 지칭한다. 예를 들어, 보존적 치환은 아미노산 잔기를 유사한 화학적 성질(예를 들어, 산성, 염기성, 양으로 또는 음으로 하전된, 극성 또는 비극성 등)을 갖는 상이한 아미노산 잔기로 치환하는 것을 지칭한다. 보존적 아미노산 치환에는 류신에서 이소류신 또는 발린으로, 아스파르테이트에서 글루타메이트로, 또는 트레오닌에서 세린으로의 대체가 포함된다. 기능적으로 유사한 아미노산을 제공하는 보존적 치환 표는 당업계에 잘 알려져 있다. 예를 들어, 각각의 하기 6가지 그룹은 서로 보존적 치환인 아미노산을 함유한다: 1) 알라닌(A), 세린(S), 트레오닌(T); 2) 아스파르트산(D), 글루탐산(E); 3) 아스파라긴(N), 글루타민(Q); 4) 아르기닌(R), 리신(K); 5) 이소류신(I), 류신(L), 메티오닌(M), 발린(V); 및 6) 페닐알라닌(F), 티로신(Y), 트립토판(W)(또한, 문헌[Creighton, Proteins, W. H. Freeman and Company (1984)] 참고, 이 문헌은 전체가 본원에 참조로 포함됨). 일부 구현예에서, 단일 아미노산 또는 적은 백분율의 아미노산을 변경시키거나, 부가하거나 또는 결실시키는 개별 치환, 결실 또는 부가가 펩티드의 활성을 감소시키지 않는 경우 "보존적 치환"으로 간주될 수 있다. 삽입 또는 결실은 일반적으로 약 1 내지 5개 범위의 아미노산이다. 보존적 아미노산의 선택은 펩티드에서 치환되는 아미노산의 위치, 예를 들어 아미노산이 펩티드의 외부에 있어서 용매에 노출되는지 또는 내부에 있어서 용매에 노출되지 않는지를 기반으로 하여 선택될 수 있다.
용매에 대한 노출을 비롯하여 기존 아미노산의 위치를 기반으로 하여(즉, 용매에 노출되지 않은 내부에 국소화된 아미노산과 비교하여 아미노산이 용매에 노출되거나 또는 펩티드 또는 폴리펩티드의 외부 표면 상에 존재하는 경우) 기존 아미노산을 치환시키는 아미노산을 선택할 수 있다. 이러한 보존적 아미노산 치환의 선택은 예를 들어 문헌[Dordo et al, J. MoI Biol, 1999, 217, 721-739 및 Taylor et al, J. Theor. Biol. 119(1986);205-218 및 S. French and B. Robson, J. Mol. Evol. 19(1983)171]에 개시된 바와 같이 당업계에 잘 알려져 있다. 따라서, 단백질 또는 펩티드의 외부에 있는 아미노산(즉, 용매에 노출된 아미노산)에 적합한 보존적 아미노산 치환을 선택할 수 있으며, 예를 들어, 비제한적으로 하기 치환이 사용될 수 있다: Y가 F로, T가 S 또는 K로, P가 A로, E가 D 또는 Q로, N이 D 또는 G로, R이 K로, G가 N 또는 A로, T가 S 또는 K로, D가 N 또는 E로, I가 L 또는 V로, F가 Y로, S가 T 또는 A로, R이 K로, G가 N 또는 A로, K가 R로, A가 S, K 또는 P로 치환됨.
대안적 구현예에서, 단백질 또는 펩티드의 내부에 있는 아미노산에 적합한 보존적 아미노산 치환을 선택할 수 있으며, 예를 들어 단백질 또는 펩티드의 내부에 있는 아미노산(즉, 아미노산이 용매에 노출됨)에 적합한 보존적 치환을 사용할 수 있으며, 예를 들어 비제한적으로, 하기의 보존적 치환을 사용할 수 있다: Y가 F로, T가 A 또는 S로, I가 L 또는 V로, W가 Y로, M이 L로, N이 D로, G가 A로, T가 A 또는 S로, D가 N으로, I가 L 또는 V로, F가 Y 또는 L로, S가 A 또는 T로 및 A가 S, G, T 또는 V로 치환됨. 일부 구현예에서, 비보존적 아미노산 치환은 또한 변이체라는 용어 내에 포괄된다.
본 발명에는 첨부된 서열 목록에 나타낸 항체 VH 및/또는 VL 도메인의 VH 및/또는 VL 도메인 변이체를 함유하는 항체를 생산하는 방법이 포함된다. 이러한 항체는 다음 단계를 포함하는 방법에 의해 생산될 수 있다:
(i) 모 항체 VH 도메인의 아미노산 서열에 하나 이상의 아미노산의 부가, 결실, 치환 또는 삽입에 의해, 모 항체 VH 도메인의 아미노산 서열 변이체인 항체 VH 도메인을 제공하는 단계로서,
모 항체 VH 도메인은 임의의 항체 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009의 VH 도메인 또는 임의의 이들 항체의 중쇄 상보성 결정 영역을 포함하는 VH 도메인인, 단계,
(ii) 선택적으로 이렇게 제공된 VH 도메인을 VL 도메인과 조합하여 VH/VL 조합을 제공하는 단계, 및
(iii) 이렇게 제공된 VH 도메인 또는 VH/VL 도메인 조합을 시험하여 하나 이상의 원하는 특성을 갖는 항체를 식별하는 단계.
바람직한 특성에는 인간 ICOS에 대한 결합, 마우스 ICOS에 대한 결합, 및 게잡이원숭이 ICOS와 같은 다른 비인간 ICOS에 대한 결합이 포함된다. 인간 및/또는 마우스 ICOS에 대해 필적하거나 더 높은 친화도를 갖는 항체가 식별될 수 있다. 다른 원하는 특성에는 면역억제성 TReg의 고갈을 통해 간접적으로 또는 T 효과기 세포에 대한 ICOS 신호전달 활성화를 통해 직접적으로 효과기 T 세포 기능을 증가시키는 것이 포함된다. 원하는 특성을 갖는 항체를 식별하는 단계는 그 친화도, 교차반응성, 특이성, ICOS 수용체 효능작용, 중화 효능 및/또는 T 세포 의존적 사멸의 촉진과 같은 본원에 기재된 기능적 속성을 갖는 항체를 식별하는 것을 포함할 수 있으며, 이들 중 임의의 것이 본원에 기재된 바와 같은 검정에서 결정될 수 있다.
VL 도메인이 방법에 포함되는 경우, VL 도메인은 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 또는 STIM009 중 임의의 것의 VL 도메인일 수 있거나, 모 VL 도메인의 아미노산 서열에 하나 이상의 아미노산의 부가, 결실, 치환 또는 삽입에 의해 제공되는 변이체일 수 있고, 모 VL 도메인은 STIM001, STIM002, STIM002-B, STIM003, STIM004, STIM005, STIM006, STIM007, STIM008 및 STIM009 중 임의의 것의 VL 도메인 또는 이들 항체 중 임의의 것의 경쇄 상보성 결정 영역을 포함하는 VL 도메인이다.
변이체 항체를 생성하는 방법은 선택적으로 항체 또는 VH/VL 도메인 조합의 카피를 생성하는 단계를 포함할 수 있다. 방법은 생성된 항체를 발현시키는 단계를 추가로 포함할 수 있다. 선택적으로 하나 이상의 발현 벡터에서 원하는 항체 VH 및/또는 VL 도메인에 상응하는 뉴클레오티드 서열을 생성하는 것이 가능하다. 숙주 세포에서의 재조합 발현을 포함하는 적합한 발현 방법이 본원에 상세히 제시되어 있다.
1.6.11. 암호화 핵산 및 발현 방법
본 발명에 따른 항체를 암호화하는 단리된 핵산이 제공될 수 있다. 핵산은 DNA 및/또는 RNA일 수 있다. 합성 기원의 게놈 DNA, cDNA, mRNA, 또는 다른 RNA, 또는 이들의 임의의 조합이 항체를 암호화할 수 있다.
본 발명은 상기와 같은 하나 이상의 폴리뉴클레오티드를 포함하는 플라스미드, 벡터, 전사, 또는 발현 카세트 형태의 작제물을 제공한다. 예시적인 뉴클레오티드 서열은 서열 목록에 포함되어 있다. 본원에 제시된 뉴클레오티드 서열에 대한 언급은 문맥상 달리 요구되지 않는 한 특정 서열을 갖는 DNA 분자를 포함하며, U가 T로 치환된 특정 서열을 갖는 RNA 분자를 포함한다.
본 발명은 또한 항체를 암호화하는 하나 이상의 핵산을 포함하는 재조합 숙주 세포를 제공한다. 암호화된 항체를 생산하는 방법은 예를 들어, 핵산을 함유하는 재조합 숙주 세포를 배양함으로써 핵산으로부터 발현시키는 단계를 포함할 수 있다. 따라서 항체가 얻어질 수 있고, 임의의 적합한 기술을 사용하여 단리 및/또는 정제된 후 적절하게 사용될 수 있다. 생성 방법은 적어도 하나의 추가의 구성요소, 예를 들어, 약학적으로 허용가능한 부형제를 포함하는 조성물로 생성물을 제형화하는 단계를 포함할 수 있다.
다양한 상이한 숙주 세포에서 폴리펩티드의 클로닝 및 발현을 위한 시스템이 널리 알려져 있다. 적합한 숙주 세포에는 박테리아, 포유동물 세포, 식물 세포, 사상성 진균, 효모 및 바쿨로바이러스 시스템, 및 트랜스제닉 식물 및 동물이 포함된다.
원핵생물 세포에서 항체 및 항체 단편의 발현은 당업계에 널리 확립되어 있다. 일반적인 박테리아 숙주는 대장균(E. coli)이다. 배양물에서 진핵생물 세포의 발현은 또한 생성을 위한 옵션으로서 당업자에게 이용 가능하다. 이종성 폴리펩티드의 발현을 위해 당업계에서 이용 가능한 포유동물 세포주에는 중국 햄스터 난소(CHO) 세포, HeLa 세포, 새끼 햄스터 신장 세포, NSO 마우스 흑색종 세포, YB2/0 래트 골수종 세포, 인간 배아 신장 세포, 인간 배아 망막 세포, 및 다른 다수가 포함된다.
벡터는 적절한 경우 프로모터 서열, 종결자 서열, 폴리아데닐화 서열, 인핸서 서열, 마커 유전자 및 다른 서열을 포함하는 적절한 조절 서열을 함유할 수 있다. 항체를 암호화하는 핵산은 숙주 세포에 도입될 수 있다. 핵산은 인산칼슘 형질감염, DEAE-덱스트란, 전기천공, 리포솜 매개 형질감염, 및 레트로바이러스 또는 다른 바이러스, 예를 들어 백시니아, 또는 곤충 세포의 경우, 바쿨로바이러스를 사용하는 형질도입을 포함하는 다양한 방법에 의해 진핵생물 세포에 도입될 수 있다. 숙주 세포, 특히 진핵생물 세포에서 핵산의 도입은 바이러스 또는 플라스미드 기반 시스템을 사용할 수 있다. 플라스미드 시스템은 에피솜으로 유지될 수 있거나 숙주 세포 또는 인공 염색체에 통합될 수 있다. 통합은 단일 또는 다중 유전자좌에서 하나 이상의 카피를 무작위로 통합하거나 표적화하여 통합할 수 있다. 박테리아 세포의 경우, 적합한 기술에는 염화칼슘 형질전환, 전기천공, 및 박테리오파지를 사용한 형질감염이 포함된다. 도입 후에, 예를 들어 유전자 발현을 위한 조건 하에 숙주 세포를 배양함으로써 핵산을 발현시킨 후, 선택적으로 항체를 단리하거나 정제할 수 있다.
본 발명의 핵산은 숙주 세포의 게놈(예를 들어 염색체) 내로 통합될 수 있다. 통합은 표준 기술에 따라 게놈과의 재조합을 촉진하는 서열을 포함함으로써 촉진될 수 있다.
본 발명은 또한 항체를 발현하기 위해 발현 시스템에서 본원에 기재된 핵산을 사용하는 단계를 포함하는 방법을 제공한다.
1.6.12. 치료 용도
본원에 기재된 항체(예를 들어, 전장 항체 또는 이의 항원 결합 단편)는 치료법에 의한 인간 또는 동물 신체의 치료 방법에 사용될 수 있다. 항체는 효과기 T 세포 반응을 증가시키는 데 사용되며, 이는 암 또는 고형 종양 치료를 포함하며 백신접종 상황에서의 다양한 질환 또는 질병에 대하여 이점을 갖는다. 증가된 Teff 반응은 Teff 활성에 유리하게 Teff와 Treg 사이의 균형 또는 비를 조절하는 항체를 사용하여 달성될 수 있다.
항-ICOS 항체는 환자의 조절 T 세포를 고갈시키고/시키거나 효과기 T 세포 반응을 증가시키는 데 사용될 수 있으며, 조절 T 세포를 고갈시키고/시키거나 효과기 T 세포 반응을 증가시켜 치료법에 적합한 질환 또는 질병을 치료하기 위해 환자에 투여될 수 있다.
본 발명의 항체, 또는 이러한 항체 분자 또는 이를 암호화하는 핵산을 포함하는 조성물은 임의의 이러한 방법에서 사용되거나 사용을 위해 제공될 수 있다. 임의의 이러한 방법에서 사용하기 위한 약제의 제조를 위한 항체, 또는 항체 또는 항체를 암호화하는 핵산을 포함하는 조성물의 용도도 예상된다. 방법은 전형적으로 항체 또는 조성물을 포유류에 투여하는 단계를 포함한다. 적합한 제형물 및 투여 방법은 본원의 다른 곳에 기재되어 있다.
항체의 예상되는 치료 용도 중 하나는 암의 치료이다. 암은 고형 종양, 예를 들어 신장 세포암(선택적으로 신장 세포 암종, 예를 들어 투명 세포 신장 세포 암종), 두경부암, 흑색종(선택적으로 악성 흑색종), 비소세포 폐암(예를 들어 샘암종), 방광암, 난소암, 자궁경부암, 위암, 간암, 췌장암, 유방암, 고환 생식 세포 암종, 또는 나열된 것과 같은 고형 종양의 전이일 수 있거나 액체 혈액학적 종양, 예를 들어 림프종(예컨대 호지킨 림프종 또는 비호지킨 림프종, 예를 들어 미만성 거대 B세포 림프종, DLBCL) 또는 백혈병(예를 들어 급성 골수성 백혈병)일 수 있다. 항-ICOS 항체는 흑색종, 두경부암, 비소세포 폐암 그리고 중등도 내지 높은 돌연변이 부하를 갖는 다른 암에서 종양 제거를 향상시킬 수 있다[26]. 일부 구현예에서, 암은 유방암이다. 일부 구현예에서, 암은 삼중 음성 유방암이다. 일부 구현예에서, 암은 두경부 편평 세포 암종이다. 일부 구현예에서, 암은 음경암이다. 일부 구현예에서, 암은 췌장암이다. 일부 구현예에서, 암은 비소세포 폐암이다. 일부 구현예에서, 암은 간세포 암종이다. 일부 구현예에서, 암은 식도암이다. 일부 구현예에서, 암은 위암이다. 일부 구현예에서, 암은 흑색종이다. 일부 구현예에서, 암은 신장 세포 암종이다. 일부 구현예에서, 암은 자궁경부암이다. 일부 구현예에서, 암은 진행성 암이다. 일부 구현예에서, 암은 전이성 암이다.
일부 구현예에서, 하나 이상의 용량의 항-ICOS 항체 또는 이의 항원-결합 단편을 사용한 치료는 부분적인 항-종양 반응을 초래한다. 일부 구현예에서, 하나 이상의 용량의 항-ICOS 항체 또는 이의 항원-결합 단편을 사용한 치료는 완전한 항-종양 반응을 초래한다. 일부 구현예에서, 하나 이상의 용량의 항-ICOS 항체 또는 이의 항원 결합 단편 및 하나 이상의 용량의 항-PD-L1 항체 또는 이의 항원-결합 단편을 사용한 치료는 부분적인 항-종양 반응을 초래한다. 일부 구현예에서, 하나 이상의 용량의 항-ICOS 항체 또는 이의 항원 결합 단편 및 하나 이상의 용량의 항-PD-L1 항체 또는 이의 항원-결합 단편을 사용한 치료는 완전한 항-종양 반응을 초래한다.
일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편은 KY1044의 CDR 서열을 포함한다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편은 KY1044의 CDR 서열을 포함한다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편은 KY1044의 중쇄 및 경쇄 가변 도메인 서열을 포함한다. 일부 구현예에서, 항-ICOS 항체 또는 이의 항원-결합 단편은 KY1044의 중쇄 및 경쇄 서열을 포함한다. 일부 구현예에서, 항-ICOS 항체는 KY1044이다. 일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙이다. 일부 구현예에서, KY1044를 사용한 치료는 항-PD-L1 항체(예를 들어, 아테졸리주맙)의 유효성을 촉진한다.
환자의 종양성 병변에 대한 환자의 면역 반응을 향상시킴으로써 항-ICOS 항체를 사용한 면역치료법은 잠재적으로 말기 질환의 상황에서도 지속적인 치유 또는 장기 관해 가능성을 제공한다.
암은 다양한 질환 그룹이지만, 항-ICOS 항체는 암세포를 정상 조직과 구별하는 돌연변이체 또는 과발현된 에피토프의 인식을 통하여 임의의 암 세포를 죽일 수 있는 잠재력을 가진, 환자 자신의 면역계를 활용하여 다양한 상이한 암을 치료하는 가능성을 제공한다. Teff/Treg 균형을 조절함으로써 항-ICOS 항체는 면역 인식 및 암 세포의 사멸을 가능하게 하고/하거나 촉진할 수 있다. 따라서 항-ICOS 항체는 매우 다양한 암에 유용한 치료제이지만, 항-ICOS 치료법이 특히 적합하고/하거나 다른 치료제가 효과적이지 않을 때 항-ICOS 치료법이 효과적일 수 있는 특정 범주의 암이 존재한다.
이러한 그룹 중 하나는 ICOS 리간드 발현에 양성인 암이다. 흑색종에 대해 기재된 바와 같이[27], 암 세포는 ICOS 리간드의 발현을 획득할 수 있다. ICOS 리간드의 발현은 표면 발현된 리간드가 Treg 상의 ICOS에 결합하여 Treg의 증식 및 활성화를 촉진하고 이에 따라 암에 대한 면역 반응을 억제하므로 세포에 선택적인 장점을 제공할 수 있다. ICOS 리간드를 발현하는 암 세포는 이의 생존을 위해 Treg에 의한 면역계의 이러한 억제에 의존할 수 있고, 이에 따라 Treg를 표적화하는 항-ICOS 항체를 사용한 치료에 취약할 수 있을 것이다. 이는 ICOS 리간드를 자연적으로 발현하는 세포로부터 유래된 암에도 적용된다. 이들 세포에 의한 ICOS 리간드의 지속적인 발현은 면역 억제를 통해 생존 장점을 다시 제공한다. ICOS 리간드를 발현하는 암은 B 세포, 수지상 세포 및 단핵구와 같은 항원 제시 세포로부터 유래될 수 있고, 본원에 언급된 것들과 같은 액체 혈액학적 종양일 수 있다. 흥미롭게도 이러한 유형의 암은 ICOS 및 FOXP3 발현도 높은 것으로 나타났다(TCGA 데이터) - 실시예 6을 참고한다. 본원의 실시예 1은 ICOS 리간드를 발현하는 암성 B 세포(A20 동계 세포)로부터 유래된 종양을 치료하는 데 있어서 예시적인 항-ICOS 항체의 유효성을 실증한다.
따라서, 항-ICOS 항체는 ICOS 리간드의 발현에 대해 양성인 암을 치료하는 방법에서 사용될 수 있다. 추가로, 본 발명에 따른 항-ICOS 항체로 치료될 암은 ICOS 및/또는 FOXP3의 발현에 대해 양성이고 선택적으로 또한 ICOS 리간드를 발현하는 암일 수 있다.
환자는 예를 들어 환자로부터 시험 샘플(예를 들어, 종양 생검)을 채취하고 관심 단백질의 발현을 결정함으로써 암이 관심 단백질(예를 들어, ICOS 리간드, ICOS 및/또는 FOXP3)의 발현에 대해 양성인지 여부를 결정하기 위한 시험을 거칠 수 있다. 암이 하나, 둘 또는 모든 이러한 관심 단백질의 발현에 대해 양성인 것으로 특성규명된 환자가 항-ICOS 항체를 사용한 치료를 위해 선택된다. 본원의 다른 곳에서 논의된 바와 같이, 항-ICOS 항체는 단독치료법으로 또는 하나 이상의 다른 치료제와의 조합으로 사용될 수 있다.
항-ICOS 항체는 또한 이들의 암이 CTLA-4, PD-1, PD-L1, CD137, GITR 또는 CD73과 같은 면역 체크포인트 분자에 대한 항체 또는 다른 약물을 사용한 치료에 불응성인 환자에게 희망을 제공한다. 이러한 면역치료법은 일부 암에 대해 효과적이지만 일부 경우에는 암이 반응하지 않을 수 있거나 항체를 사용한 지속적인 치료에 반응하지 않게 될 수 있다. 면역 체크포인트 억제제에 대한 항체와 마찬가지로 항-ICOS 항체는 환자의 면역계를 조절한다 - 그럼에도 불구하고 항-ICOS 항체는 이러한 다른 항체가 실패하는 경우 성공할 수 있다. A20 B 세포 림프종을 보유하는 동물은 항-ICOS 항체로 치료되어 종양의 성장을 감소시키고, 종양을 축소시키며 실제로 신체에서 종양을 제거할 수 있는 반면, 항-PD-L1 항체를 사용한 치료는 대조군보다 낫지 않음이 본원에 나타난다. A20 세포주도 항-CTLA-4에 내성이 있는 것으로 보고되었다[28].
따라서, 항-ICOS 항체는 하나 이상의 면역치료법, 예컨대 항-CTLA-4 항체, 항-PD1 항체, 항-PD1 L1 항체, 항-CD137 항체, 항-GITR 항체 또는 항-CD73 항체(중 임의의 것 또는 전부)를 사용한 치료에 불응성인 암을 치료하는 방법에서 사용될 수 있다. 항체나 약물을 사용한 치료가 암의 성장을 유의하게 감소시키지 않는 경우, 예를 들어, 종양이 계속 성장하거나 크기가 감소하지 않는 경우 또는 반응 기간 후 종양이 그 성장을 다시 시작하는 경우 암은 항체 또는 다른 약물을 사용한 치료에 불응성인 것으로 특성규명될 수 있다. 치료제에 대한 무반응은 암 세포 사멸 또는 성장 억제에 대해 샘플(예를 들어, 종양 생검 샘플)을 시험함으로써 생체외에서, 및/또는 임상 환경에서 치료법으로 치료된 환자가 치료에 반응하지 않음을 관찰함으로써(예를 들어, MRI를 포함한 영상화 기술을 사용하여) 결정될 수 있다. 환자의 암이 이러한 면역치료법을 사용한 치료에 불응성인 것으로 특성규명된 환자가 항-ICOS 항체를 사용한 치료를 위해 선택된다.
또한, 항-ICOS 항체는 항-CD20 항체를 사용한 치료에 내성이 있는 B 세포 유래 암을 치료하기 위해 사용될 수 있다. 항-ICOS 항체는 리툭시맙과 같은 항-CD20 항체를 사용한 치료법에 반응하지 않거나 내성이 된 암에 대한 치료법을 표시한다. 항-ICOS 항체는 이러한 암에 대한 2선(또는 추가 또는 추가적인) 치료로 사용될 수 있다. 항-CD20 항체 내성 암은 B 세포 암, 예를 들어 B 세포 림프종, 예컨대 미만성 거대 B 세포 림프종일 수 있다. 항-CD20에 대한 암의 내성은 항-CD20 항체에 의한 암 세포 사멸 또는 성장 억제에 대해 샘플(예를 들어, 종양 생검 샘플)을 시험함으로써 생체외에서, 및/또는 임상 환경에서 항-CD20 항체로 치료된 환자가 치료에 반응하지 않음을 관찰함으로써 결정될 수 있다. 대안적으로 또는 추가적으로, 암(예를 들어, 종양 생검 샘플)은 CD20의 발현을 평가하기 위해 시험될 수 있으며, CD20 발현의 부재 또는 낮은 수준은 항-CD20 항체에 대한 민감성 상실을 표시한다.
따라서 환자로부터 얻은 샘플이 시험되어 관심 단백질, 예를 들어 ICOS 리간드, ICOS, FOXP3 및/또는 또 다른 치료제(예를 들어, 항수용체 항체)가 유도되는 표적 수용체의 표면 발현을 결정할 수 있다. 표적 수용체는 CD20(리툭시맙과 같은 항-CD20 항체 치료법이 유도됨) 또는 PD1, EGFR, HER2 또는 HER3과 같은 또 다른 수용체일 수 있다. ICOS 리간드, ICOS, FOXP3의 표면 발현 및/또는 표적 수용체의 표면 발현의 결여 또는 상실은 암이 항-ICOS 항체 치료법에 민감하다는 표시이다. 항-ICOS 항체는 환자의 암이 ICOS 리간드, ICOS, FOXP3의 표면 발현 및/또는 표적 수용체의 표면 발현의 결여 또는 상실을 특징으로 하는 환자에 대한 투여를 위해 제공될 수 있으며, 선택적으로 환자는 이전에 항-CTLA4, 항-PD1, 항-PD-L1 또는 표적 수용체에 대한 항체로 치료되었고 예를 들어, 지속적인 또는 갱신된 암 세포 성장, 예를 들어 종양 크기의 증가에 의해 측정되는, 해당 항체를 사용한 치료에 반응하지 않았거나 반응을 중단하였다.
암 세포가 ICOS 리간드, CD20 또는 본원에 언급된 다른 표적 수용체와 같은 단백질의 표면 발현에 대해 양성 반응을 보이는지 여부를 결정하기 위해 임의의 적합한 방법이 사용될 수 있다. 전형적인 방법은 면역조직화학이며, 세포 샘플(예를 들어, 종양 생검 샘플)이 관심 단백질에 대한 항체와 접촉되고 항체의 결합이 표지된 시약 - 전형적으로 1차 항체의 Fc 영역을 인식하며 형광 마커와 같은 검출 가능한 표지를 운반하는 2차 항체를 사용하여 검출된다. 세포 염색 또는 다른 표지 검출에 의해 시각화된 바와 같이 적어도 5%의 세포가 표지된 경우 샘플은 양성 반응을 보인 것으로 선언될 수 있다. 선택적으로 10% 또는 25%와 같은 더 높은 컷오프가 사용될 수 있다. 항체는 일반적으로 과량으로 사용될 것이다. 관심 분자에 대한 시약 항체가 이용 가능하거나 간단한 방법으로 생성될 수 있다. ICOS 리간드를 시험하기 위해 항체 MAB1651은 인간 ICOS 리간드를 인식하는 마우스 IgG로 현재 R&D systems에서 이용 가능하다. CD20 발현을 시험하기 위해 리툭시맙이 사용될 수 있다. ICOS 리간드 또는 관심 표적 수용체의 mRNA 수준 검출은 대안적 기술이다[27].
종양이 항-ICOS 항체를 사용한 치료에 반응할 것이라는 추가 표시는 종양 미세환경에서의 Treg 존재이다. 활성화 Treg는 ICOS-고 및 Foxp3-고 표면 발현을 특징으로 한다. 종양에서 특히 상승된 수의, Treg의 존재는 환자가 항-ICOS 항체를 사용한 치료를 위해 선택될 수 있는 추가 기반을 제공한다. Treg는 예를 들어 면역조직화학(상술한 바와 같이 표적 단백질에 대한 항체를 사용한 후 표지를 검출하는, Foxp3 및 ICOS 둘 모두의 공동 발현에 대한 검정)에 의해 또는 ICOS 및 Foxp3에 대한 표지 항체를 사용하는 FACS에서 사용하기 위한 샘플의 단일 세포 분산에 의해, 종양 생검 샘플에서 생체외 검출될 수 있다.
항-ICOS 항체는 바이러스-유도 암과 같은 감염원과 관련된 암을 치료하는 데 사용될 수 있다. 이 범주에는 두경부 편평 세포 암종, 자궁경부암, 메르켈 세포 암종 등이 있다. 암과 관련된 바이러스에는 HBV, HCV, HPV(자궁경부암, 구인두암) 및 EBV(버키트 림프종, 위암, 호지킨 림프종, 다른 EBV 양성 B 세포 림프종, 비인두 암종 및 이식 후 림프증식성 질환)가 포함된다. 국제 암 연구 기관(Monograph 100B)은 감염원과 관련된 하기 주요 암 부위를 확인했다:
· 위부(stomach)/위: 헬리오박터 파일로리(Heliobacter pylori)
· 간: B형 간염 바이러스, C형 간염 바이러스(HCV), 오피스토르키스 비베리니(Opisthorchis viverrini), 클로노르키스 시넨시스(Clonorchis sinensis)
· 자궁경: HIV를 갖거나 갖지 않는 인유두종 바이러스(HPV)
· 항문생식기(음경, 외음부, 질, 항문): HIV를 갖거나 갖지 않는 HPV
· 비인두: 엡스타인-바 바이러스(EBV)
· 구인두: 담배 또는 알코올 소비를 갖거나 갖지 않는 HPV
· 카포시 육종: HIV를 갖거나 갖지 않는 인간 헤르페스 바이러스 8형
· 비호지킨 림프종: H. 파일로리, HIV를 갖거나 갖지 않는 EBV, HCV, 인간 T 세포 림프친화 바이러스 1형
· 호지킨 림프종: HIV를 갖거나 갖지 않는 EBV
· 방광: 스키스토소마 해마토비움(Schistosoma haematobium).
본 발명에 따른 항체는 상기 특정된 암과 같은 임의의 이들 감염원과 관련되거나 이에 의해 유도된 암을 치료하는 데 사용될 수 있다.
효과기 T 세포 반응의 자극은 또한 감염성 질환에 대한 면역성 및/또는 환자의 감염성 질환으로부터의 회복에 기여할 수 있다. 따라서, 항-ICOS 항체는 환자에게 항체를 투여함으로써 감염성 질환을 치료하는 데 사용될 수 있다.
감염성 질환에는 병원체, 예를 들어 박테리아, 진균, 바이러스 또는 원생동물 병원체에 의해 유발되는 질환이 포함되며, 치료는 병원체 감염에 대해 환자의 면역 반응을 촉진하는 것일 수 있다. 세균성 병원체의 예는 결핵이다. 바이러스성 병원체의 예는 B형 간염 및 HIVdl다. 원생동물 병원체의 예로는 P. 팔시파럼(P. falciparum)과 같이 말라리아를 유발하는 플라스모디움 종이 있다.
항체는 감염, 예를 들어 본원에 언급된 임의의 병원체에 의한 감염을 치료하는 데 사용될 수 있다. 감염은 지속적인 또는 만성 감염일 수 있다. 감염은 국소적 또는 전신적일 수 있다. 병원체와 면역계 사이의 장기간 접촉은 표시된 병원체 항원의 진화 및 변형을 통해 병원체에 의한 면역계의 소진 또는 내성 발달(예를 들어 Treg 수준의 증가 및 Treg에 유리하게 기울어진 Treg:Teff 균형을 통해 나타남) 및/또는 면역 회피를 야기할 수 있다. 이러한 특징은 암에서 발생하는 것으로 여겨지는 유사한 프로세스를 반영한다. 항-ICOS 항체는 Teff 및/또는 본원에 기재된 다른 효과에 유리한 Treg:Teff 비의 조절을 통해 병원체에 의한 감염, 예를 들어 만성 감염을 치료하기 위한 치료적 접근을 제시한다.
치료는 감염성 질환 또는 감염을 갖는 것으로 진단된 환자에 대한 것일 수 있다. 대안적으로, 치료는 예방적일 수 있고, 예를 들어 본원의 다른 곳에서 기재된 바와 같이 백신으로서, 질환에 걸리는 것을 방지하기 위해 환자에 투여될 수 있다.
또한 면역 반응, 특히 IFNγ 의존적 전신 면역 반응이 알츠하이머병 및 신경염증 구성요소를 일부로서 공유하는 다른 CNS 병리의 치료에 유익할 수 있다고 제안되었다[29]. WO2015/136541에서는 항-PD-1 항체를 사용한 알츠하이머병의 치료를 제안했다. 항-ICOS 항체는 선택적으로 하나 이상의 다른 면역조절제(예를 들어, PD-1에 대한 항체)와의 조합으로, 알츠하이머병 또는 다른 신경변성 질환의 치료에 사용될 수 있다.
1.6.13. 조합 치료법
항-CTLA4, 항-PD1 또는 항-PDL1과 같은 면역조절 항체, 특히 Fc 효과기 기능이 있는 항체를 사용한 치료는 면역억제 세포를 고도로 발현하는 ICOS의 추가 고갈이 유익한 환경을 생성할 수 있다. 그 치료 효과를 향상시키기 위해 항-ICOS 항체를 이러한 면역조절제와 조합하는 것이 유리할 수 있다.
면역조절 항체(예를 들어, 항-PDL-1, 항-PD-1, 항-CTLA-4)로 치료된 환자는 항-ICOS 항체로의 치료로부터 특히 이점을 얻을 수 있다. 이에 대한 한 가지 이유는 면역조절 항체가 환자에서 ICOS 양성 Treg(예를 들어, 종양내 Treg)의 수를 증가시킬 수 있다는 것이다. 이 효과는 재조합 IL-2와 같은 다른 특정 치료제로도 관찰된다. 항-ICOS 항체는 환자를 또 다른 치료제로 치료함으로써 초래되는 ICOS+ Treg(예를 들어, 종양내 Treg)의 급증 또는 상승을 감소 및/또는 역전시킬 수 있다. 따라서 항-ICOS 항체를 사용한 치료를 위해 선택된 환자는 제1 치료제로 이미 치료를 받은 환자일 수 있으며, 제1 치료제는 항체(예를 들어, 면역조절제 항체) 또는 환자의 ICOS+ Treg 수를 증가시키는 다른 제제(예를 들어, IL-2)이다.
항-ICOS 항체가 조합될 수 있는 면역조절제에는 PDL1(예를 들어, 아벨루맙), PD-1(예를 들어, 펨브롤리주맙 또는 니볼루맙) 또는 CTLA-4(예를 들어, 이필리무맙 또는 트레멜리무맙) 중 임의의 것에 대한 항체가 포함된다. 항-ICOS 항체는 피딜리주맙과 조합될 수 있다. 다른 구현예에서, 항-ICOS 항체는 항-CTLA-4 항체와 조합으로 투여되지 않고/않거나 선택적으로 항-CTLA-4 항체가 아닌 치료 항체와의 조합으로 투여된다.
예를 들어, 항-ICOS 항체는 항-PDL1 항체와 조합 치료법에서 사용될 수 있다. 바람직하게는, 항-ICOS 항체는 ADCC, ADCP 및/또는 CDC를 매개하는 항체이다. 바람직하게는, 항-PDL1 항체는 ADCC, ADCP 및/또는 CDC를 매개하는 항체이다. 이러한 조합 치료법의 한 예는 항-ICOS 항체를 항-PDL1 항체와의 투여이며, 두 항체 모두 효과기 양성 불변 영역을 갖는다. 따라서, 항-ICOS 항체 및 항-PDL1 항체 둘 모두는 ADCC, CDC 및/또는 ADCP를 매개할 수 있다. Fc 효과기 기능 및 불변 영역의 선택은 본원의 다른 곳에 상세히 기재되어 있지만, 한 예로서 항-ICOS 인간 IgG1이 항-PD-L1 인간 IgG1과 조합될 수 있다. 항-ICOS 항체 및/또는 항-PD-L1 항체는 야생형 인간 IgG1 불변 영역을 포함할 수 있다. 대안적으로, 항체의 효과기 양성 불변 영역은 향상된 효과기 기능, 예를 들어 향상된 CDC, ADCC 및/또는 ADCP를 위해 조작된 것일 수 있다. 야생형 인간 IgG1 서열 및 효과기 기능을 변경하는 돌연변이를 포함하는 예시적인 항체 불변 영역은 본원의 다른 곳에서 상세히 논의된다.
항-ICOS 항체와 조합될 수 있는 항-PDL1 항체에는 다음이 포함된다:
· PD-1의 PDL1에 대한 결합을 억제하고/하거나 선택적으로 효과기 양성 인간 IgG1로서 PDL1을 억제하는 항-PDL1 항체;
· PD-1의 PDL1 및/또는 PDL2에 대한 결합을 억제하는 항-PD-1 항체;
· PD-1이 PDL-1에 결합하는 것을 억제하는 인간 IgG1 항체인 아벨루맙. WO2013/079174를 참고한다;
· 돌연변이 L234A, L235A 및 331을 갖는 변이체 인간 IgG1 항체인 두르발루맙(또는 "MEDI4736"). WO2011/066389를 참고한다;
· 돌연변이 N297A, D356E 및 L358M을 갖는 변이체 인간 IgG1 항체인 아테졸리주맙. US2010/0203056을 참고한다;
· 돌연변이 S228P를 포함하는 인간 IgG4 항체인 BMS-936559. WO2007/005874를 참고한다.
일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙을 포함한다. 일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙이다.
항-PD-L1 항체의 여러 추가 예가 본원에 개시되어 있으며 다른 것들은 당업계에 알려져 있다. 본원에 언급된 많은 항-PD-L1 항체에 대한 특성규명 데이터는 US9,567,399 및 US9,617,338에 공개되었으며, 둘 모두는 본원에 참조로 포함된다. 예시적인 항-PD-L1 항체는 US9,567,399 또는 US9,617,338에 제시된 바와 같은 1D05, 84G09, 1D05 HC 돌연변이체 1, 1D05 HC 돌연변이체 2, 1D05 HC 돌연변이체 3, 1D05 HC 돌연변이체 4, 1D05 LC 돌연변이체 1, 1D05 LC 돌연변이체 2, 1D05 LC 돌연변이체 3, 411B08, 411C04, 411D07, 385F01, 386H03, 389A03, 413D08, 413G05, 413F09, 414B06 또는 416E01 중 임의의 것의 HCDR 및/또는 LCDR을 포함하는 VH 및/또는 VL 도메인을 갖는다. 항체는 임의의 이들 항체의 VH 및 VL 도메인을 포함할 수 있고, 선택적으로 임의의 이들 항체의 중쇄 및/또는 경쇄 아미노산 서열을 갖는 중쇄 및/또는 경쇄를 포함할 수 있다. 이러한 항-PD-L1 항체의 VH 및 VL 도메인은 본원의 다른 곳에서 추가로 기재된다.
추가의 예시적인 항-PD-L1 항체는 WO2017/034916, WO2017/020291, WO2017/020858, WO2017/020801, WO2016/111645, WO2016/197367, WO2016/061142, WO2016/149201, WO2016/000619, WO2016/160792, WO2016/022630, WO2016/007235, WO2015/179654, WO2015/173267, WO2015/181342, WO2015/109124, WO2015/112805, WO2015/061668, WO2014/159562, WO2014/165082, WO2014/100079, WO2014/055897, WO2013/181634, WO2013/173223, WO2013/079174, WO2012/145493, WO2011/066389, WO2010/077634, WO2010/036959, WO2010/089411 및 WO2007/005874 중 임의의 것에 개시된 KN-035, CA-170, FAZ-053, M7824, ABBV-368, LY-3300054, GNS-1480, YW243.55.S70, REGN3504, 또는 항-PD-L1 항체의 HCDR 및/또는 LCDR을 포함하는 VH 및/또는 VL 도메인을 갖는다. 항체는 이들 항체 중 임의의 것의 VH 및 VL 도메인을 포함할 수 있고, 선택적으로 이들 항체 중 임의의 것의 중쇄 및/또는 경쇄 아미노산 서열을 갖는 중쇄 및/또는 경쇄를 포함할 수 있다. 항-PD-L1과의 조합 치료법에서 사용되는 항-ICOS 항체는 본원에 개시된 바와 같은 본 발명의 항체일 수 있다. 대안적으로, 항-ICOS 항체는 임의의 하기 공보에 개시된 항-ICOS 항체의 CDR, 또는 VH 및/또는 VL 도메인을 포함할 수 있다:
WO2016154177, US2016304610 - 예를 들어, 항체 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710, 또는 35A9S89 중 임의의 것;
WO16120789, US2016215059 - 예를 들어 422.2 및/또는 H2L5로 알려진 항체;
WO14033327, EP2892928, US2015239978 - 예를 들어 314-8로 알려진 및/또는 하이브리도마 CNCM I-4180으로부터 생산된 항체;
WO12131004, EP2691419, US9376493, US20160264666 - 예를 들어 항체 Icos145-1 및/또는 하이브리도마 CNCM I-4179에 의해 생산된 항체;
WO10056804 - 예를 들어 항체 JMAb 136 또는 "136";
WO9915553, EP1017723B1, US7259247, US7132099, US7125551, US7306800, US7722872, WO05103086, EP1740617, US8318905, US8916155 - 예를 들어 항체 MIC-944 또는 9F3;
WO983821, US7932358B2, US2002156242, EP0984023, EP1502920, US7030225, US7045615, US7279560, US7226909, US7196175, US7932358, US8389690, WO02070010, EP1286668, EP1374901, US7438905, US7438905, WO0187981, EP1158004, US6803039, US7166283, US7988965, WO0115732, EP1125585, US7465445, US7998478 - 예를 들어 임의의 JMAb 항체, 예를 들어 JMAb-124, JMAb-126, JMAb-127, JMAb-128, JMAb-135, JMAb-136, JMAb-137, JMAb-138, JMAb-139, JMAb-140, JMAb-141 중 임의의 것, 예를 들어, JMAb136;
WO2014/089113 - 예를 들어 항체 17G9;
WO12174338;
US2016145344;
WO11020024, EP2464661, US2016002336, US2016024211, US8840889;
US8497244.
항-ICOS 항체는 선택적으로 WO2016154177에 개시된 바와 같이 37A10S713의 CDR을 포함한다. 이는 37A10S713의 VH 및 VL 도메인을 포함할 수 있고, 선택적으로 37A10S713의 항체 중쇄 및 경쇄를 가질 수 있다.
항-ICOS 항체와 면역조절제의 조합은 단독치료법과 비교하여 증가된 치료 효과를 제공할 수 있고, 더 낮은 용량의 면역조절제(들)로 치료 이익을 달성할 수 있도록 할 수 있다. 따라서, 예를 들어 항-ICOS 항체와 조합으로 사용되는 항체(예를 들어 항-PD-L1 항체, 선택적으로 이필리무맙 또는 아테졸리주맙)는 보다 일반적인 10 mg/kg의 용량보다는 3 mg/kg으로 투약될 수 있다. 항-PD-L1 항체 또는 다른 항체의 투여 요법에는 총 4회 용량에 대해 3주마다 90분 기간에 걸친 정맥내 투여가 관여할 수 있다.
항-ICOS 항체는 항-PD-L1 항체를 사용한 치료에 대한 종양의 민감도를 증가시키기 위해 사용될 수 있으며, 이는 항-PD-L1 항체가 치료적 이점을 발휘하는 용량의 감소로 인식될 수 있다. 따라서, 항-ICOS 항체는 환자의 암 또는 종양을 치료하는 데 효과적인 항-PD-L1 항체의 용량을 감소시키기 위해 환자에 투여될 수 있다. 항-ICOS 항체의 투여는 항-PD-L1 항체가 항-ICOS 없이 투여될 때의 투여량과 비교하여 해당 환자에 대한 항-PD-L1 항체 투여의 권장되거나 요구되는 투여량을 예를 들어 75%, 50%, 25%, 20%, 10% 이하로 감소시킬 수 있다. 환자는 본원에 기재된 바와 같은 조합 치료법에서 항-ICOS 항체 및 항-PD-L1 항체의 투여에 의해 치료될 수 있다.
항-PD-L1과 항-ICOS를 조합하는 것의 이점은 단독치료법으로 사용하는 경우와 비교될 때 각 제제의 투여량을 줄이는 데까지 확장될 수 있다. 항-PD-L1 항체는 항-ICOS 항체가 치료적 이점을 발휘하는 용량을 감소시키기 위해 사용될 수 있으며, 이에 따라 환자에 투여되어 환자의 암 또는 종양을 치료하는 데 효과적인 항-ICOS 항체의 용량을 감소시킬 수 있다. 따라서, 항-PD-L1 항체는 항-ICOS 항체가 항-PD-L1 없이 투여될 때의 투여량과 비교하여 해당 환자에 대한 항-ICOS 항체 투여의 권장되거나 요구되는 투여량을, 예를 들어, 75%, 50%, 25%, 20%, 10% 이하로 감소시킬 수 있다. 환자는 본원에 기재된 바와 같은 조합 치료법에서 항-ICOS 항체 및 항-PD-L1 항체의 투여에 의해 치료될 수 있다.
본원의 다른 곳에서 논의된 바와 같이, 항-PD-L1 항체, 특히 효과기 양성 Fc를 갖는 항체를 사용한 치료는 Teff 세포 상에서 ICOS의 발현을 증가시키지 않는 것으로 보인다. 이는 이러한 항체를 효과기 양성 항-ICOS 항체와 조합으로 투여할 때 유리하며, Teff 상의 ICOS 발현의 증가는 바람직하지 않게 이들 세포를 항-ICOS 항체에 의한 고갈에 더 민감하게 만들 것이다. 따라서 항-PD-L1과의 조합으로. 항-ICOS 치료법은 Treg와 비교하여 Teff 상에서 ICOS의 차별적 발현을 활용하여 고갈에 대해 ICOS-고 Treg를 우선적으로 표적화할 수 있다. 이는 결과적으로 TEff의 억제를 완화하고 환자의 효과기 T 세포 반응을 촉진하는 순 효과를 갖는다. T 세포 상의 ICOS의 발현에 대한 면역 체크포인트 분자를 표적화하는 효과도 이전에 연구되었다 - 참고문헌 [30]의 도 S6C를 참고하며(보충 자료), CTLA-4 항체 및/또는 항-PD-1 항체를 사용한 치료는 ICOS를 발현하는 CD4+ Treg의 백분율을 증가시키는 것으로 보고되었다. Treg 및 Teff에서 ICOS 발현에 대한 치료제의 효과는 항-ICOS 항체와 조합으로 사용하기 위한 적절한 제제를 선택하는 데 있어서 요인이 될 수 있으며, 항-ICOS 항체의 효과는 Treg 대 Teff 상의 ICOS의 큰 차별적 발현이 존재하는 조건 하에 향상될 수 있음을 주지한다.
본원에 기재된 바와 같이, 항-ICOS 항체의 단일 용량은 특히 항-PD-L1 항체와 같은 다른 치료제와의 조합으로, 치료 효과를 제공하기에 충분할 수 있다. 종양 치료법에서, 이러한 단일 용량 이점에 대한 기저 근거는 항-ICOS 항체가 종양을 면역 공격 및/또는 언급된 것들과 같은 다른 면역조절제의 효과에 더 민감하게 만들기에 충분하도록 종양의 미세환경을 재설정하거나 변경함으로써 적어도 부분적으로 그 효과를 매개한다는 것일 수 있다. 종양 미세환경 재설정은 예를 들어 ICOS 양성 종양 침윤 T-reg의 고갈을 통해 유발된다. 따라서, 예를 들어, 환자는 단일 용량의 항-ICOS 항체에 이어 하나의 또는 다중 용량의 항-PD-L1 항체로 치료될 수 있다. 치료 지속기간, 예를 들어 6개월 또는 1년에 걸쳐, 항-ICOS 항체는 단일 용량으로 투여될 수 있는 반면, 다른 효능제, 예를 들어 항-PD-L1 항체는 선택적으로 해당 치료 지속기간에 걸쳐 여러 번 투여되며, 바람직하게는 항-ICOS 항체를 사용한 치료 후에 적어도 하나의 이러한 용량이 투여된다.
조합 치료법의 추가 예에는 항-ICOS 항체와 다음의 조합이 포함된다:
- 아데노신 A2A 수용체의 길항제("A2AR 억제제");
- CD137 효능제(예를 들어, 효능제 항체);
- 트립토판의 분해를 촉매하는 효소 인돌아민-2,3 디옥시게나제의 길항제("IDO 억제제"). IDO는 수지상 세포 및 대식구에서 활성화되는 면역 체크포인트로, 면역 억제/관용성에 기여한다.
항-ICOS 항체는 IL-2(예를 들어, 알데스류킨과 같은 재조합 IL-2)와의 조합 치료법에서 사용될 수 있다. IL-2는 높은 용량(HD)으로 투여될 수 있다. 전형적인 HD IL-2 치료법은 치료법 주기당 500,000 IU/kg 초과의 볼루스 주입, 예를 들어 600,000 또는 720,000 IU/kg의 볼루스 주입이 관여되며, 10 내지 15회의 이러한 볼루스 주입이 5 내지 10시간의 간격으로, 예를 들어 8시간마다 최대 15회 볼루스 주입이 주어지고, 최대 6 내지 8주기 동안 대략 14 내지 21일마다 치료법 주기를 반복한다. HD IL-2 치료법은 종양, 특히 흑색종(예를 들어, 전이성 흑색종) 및 신장 세포 암종을 치료하는 데 성공적이었지만, 그 사용은 중증 유해 효과를 유발할 수 있는 IL-2의 높은 독성으로 제한된다.
높은 용량의 IL-2를 사용한 치료는 암 환자에서 ICOS 양성 Treg의 집단을 증가시키는 것으로 나타났다[31]. HD IL-2 치료법의 첫 번째 주기 후 ICOS+ TReg의 이러한 증가는 더 나쁜 임상 결과와 상관관계가 있는 것으로 보고되었다 - ICOS+ Treg의 수가 높을수록 예후가 더 나쁘다. IL-2 변이체 F42K는 ICOS+ Treg 세포의 이러한 바람직하지 않은 증가를 회피하기 위한 대안적 치료법으로 제안되었다[32]. 그러나 또 다른 접근은 본 발명에 따른 항체를 2선 치료제로 사용하여 ICOS+ T reg의 증가를 활용할 것이다.
IL-2 치료법을 항-ICOS 항체와 조합하여 ICOS를 고도로 발현하는 TReg를 표적화하는 항-ICOS 항체의 능력을 활용하고 이들 세포를 억제하고 IL-2 치료법을 받는 환자의 예후를 개선하는 것이 유익할 수 있다. IL-2와 항-ICOS 항체의 병용 투여는 치료되는 환자 집단에서 유해 사례를 회피하거나 감소시키면서 반응률을 증가시킬 수 있다. 이 조합은 IL-2 단독치료법과 비교하여 IL-2를 더 낮은 용량으로 사용할 수 있도록 허용하여 IL-2 치료법으로부터 발생하는 유해 사례의 위험 또는 수준을 감소시키면서 임상적 이점(예를 들어, 종양 성장 감소, 고형 종양의 제거 및/또는 전이의 감소)을 유지하거나 향상시킬 수 있다. 이러한 방식으로 항-ICOS의 추가는 높은 용량(HD) IL-2이든 낮은 용량(LD) IL-2이든 관계없이 IL-2를 제공받는 환자의 치료를 개선할 수 있다.
따라서, 본 발명의 한 양태는 환자에게 항-ICOS 항체를 투여함으로써 환자를 치료하는 방법을 제공하며, 환자는 또한 IL-2, 예를 들어 HD IL-2로 치료된다. 본 발명의 또 다른 양태는 환자를 치료하는 데 사용하기 위한 항-ICOS 항체이며, 환자는 또한 IL-2, 예를 들어 HD IL-2로 치료된다. 항-ICOS 항체는 2선 치료법으로 사용될 수 있다. 따라서, 환자는 IL-2로 치료되었고, 예를 들어 적어도 1주기의 HD IL-2 치료법을 제공받았고 ICOS+ Treg의 수준이 증가된 환자일 수 있다. ICOS, Foxp3, ICOSL 및 선택적으로 하나 이상의 추가 관심 마커에 대해 양성인 세포를 검출하기 위해 본원의 다른 곳에서 기재된 바와 같이 면역조직화학 또는 FACS를 사용하여 암 세포 샘플, 예를 들어 종양 생검 샘플에 대해 검정이 수행될 수 있다. 방법은 IL-2 치료 후 환자가 증가된 수준의 ICOS+ Treg을 갖는지(예를 들어, 말초혈 또는 종양 생검에서) 결정하는 단계를 포함할 수 있으며, 증가된 수준은 환자가 항-ICOS 항체를 사용한 치료로부터 이익을 얻을 것임을 나타낸다. Treg의 증가는 대조군(치료되지 않은) 개체 또는 IL-2 치료법 전 환자와 대비될 수 있다. 상승된 Treg를 갖는 이러한 환자는 지속적인 IL-2 치료 단독으로는 이익을 얻지 못할 수 있지만 항-ICOS 항체와 IL-2 치료법의 조합 또는 항-ICOS 항체만으로의 치료가 치료 이익을 제공하는 그룹을 나타낸다. 따라서, 환자가 증가된 수준의 ICOS+ Treg을 갖는 긍정적인 결정 이후, 항-ICOS 항체 및/또는 추가 IL-2 치료법이 투여될 수 있다. 항-ICOS 항체를 사용한 치료는 이러한 환자의 다른 T 세포 집단에 비해 ICOS+ Treg를 선택적으로 표적화하고 고갈시킬 수 있다. 이는 이들 세포에 의해 매개되는 면역억제를 완화하고 이에 따라 표적 세포, 예를 들어 종양 세포 또는 감염된 세포에 대한 Teff의 활성을 향상시킴으로써 치료 효과를 제공한다.
항-ICOS 항체 및 IL-2와의 조합 치료법은 본원에 기재된 임의의 치료 적응증에 대해, 특히 종양, 예를 들어 전이성 흑색종과 같은 흑색종 또는 신장 세포 암종을 치료하기 위해 사용될 수 있다. 따라서, 한 예에서, 항-ICOS 항체로 치료된 환자는 전이성 흑색종을 제시하고 IL-2, 예를 들어 HD IL-2 치료법 또는 LD IL-2 치료법으로 치료된 환자이다.
일반적으로, 항-ICOS 항체가 제1 치료제(예를 들어, 면역조절제 항체) 또는 다른 제제(예를 들어, IL-2)로 치료를 받은 환자에 투여되는 경우, 항-ICOS 항체는 예를 들어 제1 치료제 투여 후 24시간, 48시간, 72시간, 1주 또는 2주의 최소 기간 후에 투여될 수 있다. 항-ICOS 항체는 제1 치료제의 투여 후 2, 3, 4 또는 5주 이내에 투여될 수 있다. 환자의 순응도를 높이고 비용을 절감하기 위해, 투여되는 치료 횟수를 최소화하는 것이 바람직할 수 있지만 이는 언제든지 어느 한 제제의 추가 투여를 배제하지 않는다. 오히려, 투여의 상대적 시기는 이의 조합 효과를 향상시키기 위해 선택될 것이며, 제1 치료제는 항-ICOS 항체의 효과가 특히 유리한 면역학적 환경(예를 들어, 상승된 ICOS+ Treg 또는 아래 논의된 항원 방출)을 생성한다. 따라서, 제1 치료제 및 이어서 항-ICOS 항체의 순차적 투여는 제1 제제가 작용할 시간을 허용하여 항-ICOS 항체가 그 향상된 효과를 나타낼 수 있는 생체내 조건을 생성할 수 있다. 동시 또는 순차적 조합 치료를 포함한 다양한 투여 요법이 본원에 기재되어 있으며 적절하게 활용될 수 있다. 제1 치료제가 환자에서 ICOS+ Treg의 수를 증가시키는 경우, 환자에 대한 치료 요법은 환자가 증가된 수의 ICOS+ Treg를 가짐을 결정하고 그 다음 항-ICOS 항체를 투여하는 것을 포함할 수 있다.
주지된 바와 같이, 조합 치료법에서 항-ICOS 항체의 사용은 치료제의 유효 용량을 감소시키고/시키거나 환자에서 ICOS+ Treg을 증가시키는 치료제의 유해 효과에 대응하는 장점을 제공할 수 있다. "면역학적 세포 사멸"을 통해 표적 세포로부터 항원의 방출을 유발하는 제1 치료제를 선택하고 제1 치료제를 항-ICOS 항체와 조합으로 투여하는 것을 통해 추가 치료 이점이 달성될 수 있다. 주지된 바와 같이, 항-ICOS 항체의 투여는 제1 치료제의 투여를 순차적으로 따를 수 있으며, 2개의 제제의 투여는 상기 논의된 바와 같이 특정 시간 윈도우에 의해 분리된다.
면역학적 세포 사멸은 아폽토시스와 대조되는, 인식되는 세포 사멸 방식이다. 이는 세포로부터 ATP 및 HMGB1의 방출 및 원형질막 상의 칼레티쿨린의 노출을 특징으로 한다[33, 34].
표적 조직 또는 표적 세포에서의 면역학적 세포 사멸은 항원 제시 세포에 의한 세포의 포식작용(engulfment)을 촉진하여 표적 세포로부터의 항원 표시를 초래하고, 이는 결과적으로 항원 특이적 Teff 세포를 유도한다. 항-ICOS 항체는 Teff 세포 상에서 ICOS의 효능제로 작용하여 Teff 반응의 규모 및/또는 지속기간을 증가시킬 수 있다. 또한, 항-ICOS 항체에서 Fc 효과기 기능이 활성화된 경우(예를 들어, 인간 IgG1 항체), 항-ICOS 항체는 항원-특이적 Treg의 고갈을 유발할 수 있다. 따라서 이러한 효과 중 하나 또는 둘 모두의 조합을 통해 Teff와 Treg 세포 사이의 균형이 Teff 활성 향상에 유리하게 조절된다. 항-ICOS 항체를 표적 조직 또는 세포 유형, 예컨대 종양 또는 암 세포에서 면역학적 세포 사멸을 유도하여 표적 조직 또는 세포에 대한 환자의 면역 반응을 촉진하는 치료와 조합하여 백신 항원이 생체내에서 생성되는 백신접종의 한 형태를 표시한다.
따라서, 본 발명의 한 양태는 이의 암 세포에 대한 환자의 생체내 백신접종에 의해 환자의 암을 치료하는 방법이다. 본 발명의 또 다른 양태는 이러한 방법에서 사용하기 위한 항-ICOS 항체이다. 항-ICOS 항체는 다음 단계를 포함하는 방법에서 사용될 수 있다:
암 세포의 면역학적 세포 사멸을 유발하는 치료법으로 환자를 치료하여 항원 특이적 효과기 T 세포에 대한 항원 제시를 초래하는 단계, 및
항-ICOS 항체를 환자에 투여하는 단계로서, 항-ICOS 항체는 암 세포에 대한 항원-특이적 효과기 T 세포 반응을 향상시키는, 단계.
면역학적 세포 사멸을 유도하는 치료법에는 방사선(예를 들어, UVC 광선 또는 γ선을 사용한 세포의 이온화 조사), 화학치료제(예를 들어, 옥살리플라틴, 독소루비신, 이다루비신 또는 미톡산트론과 같은 안트라사이클린, 플로레틴 또는 피마르산과 같은 BK 채널 효능제, 보르테조밉, 강심 배당체, 사이클로포스파미드, 미토마이신을 포함하는 GADD34/PP1 억제제, 히페리신을 포함하는 PDT, 폴리이노신-폴리시티딜산, 5-플루오로우라실, 젬시타빈, 게피티닙, 에를로티닙 또는 시스플라틴을 포함하는 탑시가르긴) 및 종양 관련 항원에 대한 항체가 포함된다. 종양 관련 항원은 동일한 조직의 비종양 세포에 비해 종양 세포에 의해 과발현되는 임의의 항원, 예를 들어 HER2, CD20, EGFR일 수 있다. 적합한 항체에는 허셉틴(항-HER2), 리툭시맙(항-CD20) 또는 세툭시맙(항-EGFR)이 포함된다.
따라서, 일부 구현예에서, 항-ICOS 항체를 하나 이상의 이러한 치료와 조합하는 것이 유리하다. 선택적으로, 항-ICOS 항체는 이미 이러한 치료를 받은 환자에 투여된다. 항-ICOS 항체는 일정 기간 후, 예를 들어 면역학적 세포 사멸을 유도하는 치료 24시간, 48시간, 72시간, 1주 또는 2주 후, 예를 들어 치료 24 내지 72시간 후에 투여될 수 있다. 항-ICOS 항체는 치료 후 2, 3, 4 또는 5주 이내에 투여될 수 있다. 조합 치료법을 위한 다른 요법은 본원의 다른 곳에서 논의된다.
"생체내 백신접종"이 상술되었지만, 종양 세포를 치료하여 생체외에서 면역학적 세포 사멸을 유도하는 것도 가능하며, 그 후 세포가 환자에 재도입될 수 있다. 면역학적 세포 사멸을 유도하는 제제 또는 치료를 환자에 직접 투여하는 것보다는, 치료된 종양 세포가 환자에 투여된다. 환자의 치료는 상술된 투여 요법에 따를 수 있다.
이미 주지된 바와 같이, 항-ICOS 항체의 단일 용량은 치료 이점을 제공하기에 충분할 수 있다. 따라서, 본원에 기재된 치료 방법에서, 항-ICOS 항체는 선택적으로 단일 용량으로 투여된다. 항-ICOS 항체의 단일 용량은 환자의 Treg를 고갈시킬 수 있으며, 그 결과 암과 같은 질환에서 유익한 효과를 갖는다. Treg의 일시적인 절제는 종양 진행 감소, 확립된 종양 및 전이 치료, 그리고 생존 연장을 포함하는 항-종양 효과를 가지며 종양 조사의 치료 효과를 향상시킬 수 있음이 이전에 보고되었다[35]. 단일 용량의 항-ICOS 투여는 이러한 Treg 고갈을 제공할 수 있으며, 방사선 치료법과 같이 조합으로 사용되는 다른 치료 접근의 효과를 향상시키기 위해 사용될 수 있다.
1.6.14. PD-L1에 대한 항체
별도 치료제로서 또는 본원에 기재된 바와 같은 다중특이적 항체에서 항-ICOS 항체와 조합으로 사용하기 위한 PD-L1에 대한 항체는 임의의 항-PD-L1 항체의 항원-결합 부위를 포함할 수 있다. 항-PD-L1 항체의 여러 예가 본원에 개시되어 있고 다른 것들은 당업계에 알려져 있다. 본원에 언급된 여러 항-PD-L1 항체에 대한 특성규명 데이터는 US9,567,399 및 US9,617,338에 공개되었으며, 둘 모두 본원에 참조로 포함된다.
1D05는 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 33의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 34이다. 1D05는 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 43의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 35(중쇄 핵산 서열 Seq ID No: 36)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
84G09는 Seq ID No: 7(IMGT) 또는 Seq ID No: 10(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 8(IMGT) 또는 Seq ID No: 11(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 9(IMGT) 또는 Seq ID No: 12(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 13의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 14이다. 84G09는 Seq ID No: 17(IMGT) 또는 Seq ID No: 20(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 18(IMGT) 또는 Seq ID No: 21(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 19(IMGT) 또는 Seq ID No: 22(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 23의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 24이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 15(중쇄 핵산 서열 Seq ID No: 16)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 25(경쇄 핵산 서열 Seq ID No: 26)이다.
1D05 HC 돌연변이체 1은 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 47의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. 1D05 HC 돌연변이체 1은 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 43의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
1D05 HC 돌연변이체 2는 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 48의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. 1D05 HC 돌연변이체 2는 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 43의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
1D05 HC 돌연변이체 3은 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 49의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. 1D05 HC 돌연변이체 3은 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 43의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
1D05 HC 돌연변이체 4는 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 342의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. 1D05 HC 돌연변이체 4는 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 43의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
1D05 LC 돌연변이체 1은 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 33의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 34이다. 1D05 LC 돌연변이체 1은 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 50의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. 1D05 LC 돌연변이체 1의 CDRL2 서열은 Seq ID No: 50의 VL 서열로부터 Kabat 또는 IMGT 시스템에 의해 정의된 바와 같다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205 또는 Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 35(중쇄 핵산 서열 Seq ID No: 36)이다.
1D05 LC 돌연변이체 2는 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 33의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 34이다. 1D05 LC 돌연변이체 2는 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 38(IMGT) 또는 Seq ID No: 41(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 51의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 35(중쇄 핵산 서열 Seq ID No: 36)이다.
1D05 LC 돌연변이체 3은 Seq ID No: 27(IMGT) 또는 Seq ID No: 30(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 28(IMGT) 또는 Seq ID No: 31(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 29(IMGT) 또는 Seq ID No: 32(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 33의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 34이다. 1D05 LC 돌연변이체 3은 Seq ID No: 37(IMGT) 또는 Seq ID No: 40(Kabat)의 CDRL1 아미노산 서열, 및 Seq ID No: 39(IMGT) 또는 Seq ID No: 42(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 298의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. 1D05 LC 돌연변이체 3의 CDRL2 서열은 Seq ID No: 298의 VL 서열로부터 Kabat 또는 IMGT 시스템에 의해 정의된 바와 같다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 44이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205 또는 Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 35(중쇄 핵산 서열 Seq ID No: 36)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 45(경쇄 핵산 서열 Seq ID No: 46)이다.
411B08은 Seq ID No: 52(IMGT) 또는 Seq ID No: 55(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 53(IMGT) 또는 Seq ID No: 56(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 54(IMGT) 또는 Seq ID No: 57(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 58의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 59이다. 411B08은 Seq ID No: 62(IMGT) 또는 Seq ID No: 65(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 63(IMGT) 또는 Seq ID No: 66(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 64(IMGT) 또는 Seq ID No: 67(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 68의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 69이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 60(중쇄 핵산 서열 Seq ID No: 61)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 70(경쇄 핵산 서열 Seq ID No: 71)이다.
411C04는 Seq ID No: 72(IMGT) 또는 Seq ID No: 75(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 73(IMGT) 또는 Seq ID No: 76(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 74(IMGT) 또는 Seq ID No: 77(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 78의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 79이다. 411C04는 Seq ID No: 82(IMGT) 또는 Seq ID No: 85(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 83(IMGT) 또는 Seq ID No: 86(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 84(IMGT) 또는 Seq ID No: 87(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 88의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 89이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 80(중쇄 핵산 서열 Seq ID No: 81)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 90(경쇄 핵산 서열 Seq ID No: 91)이다.
411D07은 Seq ID No: 92(IMGT) 또는 Seq ID No: 95(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 93(IMGT) 또는 Seq ID No: 96(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 94(IMGT) 또는 Seq ID No: 97(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 98의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 99이다. 411D07은 Seq ID No: 102(IMGT) 또는 Seq ID No: 105(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 103(IMGT) 또는 Seq ID No: 106(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 104(IMGT) 또는 Seq ID No: 107(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 108의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 109이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 100(중쇄 핵산 서열 Seq ID No: 101)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 110(경쇄 핵산 서열 Seq ID No: 111)이다.
385F01은 Seq ID No: 112(IMGT) 또는 Seq ID No: 115(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 113(IMGT) 또는 Seq ID No: 116(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 114(IMGT) 또는 Seq ID No: 117(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 118의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 119이다. 385F01은 Seq ID No: 122(IMGT) 또는 Seq ID No: 125(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 123(IMGT) 또는 Seq ID No: 126(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 124(IMGT) 또는 Seq ID No: 127(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 128의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 129이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 120(중쇄 핵산 서열 Seq ID No: 121)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 130(경쇄 핵산 서열 Seq ID No: 131)이다.
386H03은 Seq ID No: 152(IMGT) 또는 Seq ID No: 155(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 153(IMGT) 또는 Seq ID No: 156(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 154(IMGT) 또는 Seq ID No: 157(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 158의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 159이다. 386H03은 Seq ID No: 162(IMGT) 또는 Seq ID No: 165(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 163(IMGT) 또는 Seq ID No: 166(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 164(IMGT) 또는 Seq ID No: 167(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 168의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 169이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 160(중쇄 핵산 서열 Seq ID No: 161)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 170(경쇄 핵산 서열 Seq ID No: 171)이다.
389A03은 Seq ID No: 172(IMGT) 또는 Seq ID No: 175(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 173(IMGT) 또는 Seq ID No: 176(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 174(IMGT) 또는 Seq ID No: 177(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 178의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 179이다. 389A03은 Seq ID No: 182(IMGT) 또는 Seq ID No: 185(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 183(IMGT) 또는 Seq ID No: 186(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 184(IMGT) 또는 Seq ID No: 187(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 188의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 189이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 180(중쇄 핵산 서열 Seq ID No: 181)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 190(경쇄 핵산 서열 Seq ID No: 191)이다.
413D08은 Seq ID No: 132(IMGT) 또는 Seq ID No: 135(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 133(IMGT) 또는 Seq ID No: 136(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 134(IMGT) 또는 Seq ID No: 137(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 138의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 139이다. 413D08은 Seq ID No: 142(IMGT) 또는 Seq ID No: 145(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 143(IMGT) 또는 Seq ID No: 146(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 144(IMGT) 또는 Seq ID No: 147(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 148의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 149이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 140(중쇄 핵산 서열 Seq ID No: 141)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 150(경쇄 핵산 서열 Seq ID No: 151)이다.
413G05는 Seq ID No: 238(IMGT) 또는 Seq ID No: 241(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 239(IMGT) 또는 Seq ID No: 242(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 240(IMGT) 또는 Seq ID No: 243(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 244의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 245이다. 413G05는 Seq ID No: 248(IMGT) 또는 Seq ID No: 251(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 249(IMGT) 또는 Seq ID No: 252(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 250(IMGT) 또는 Seq ID No: 253(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 254의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 255이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 246(중쇄 핵산 서열 Seq ID No: 247)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 256(경쇄 핵산 서열 Seq ID No: 257)이다.
413F09는 Seq ID No: 258(IMGT) 또는 Seq ID No: 261(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 259(IMGT) 또는 Seq ID No: 262(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 260(IMGT) 또는 Seq ID No: 263(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 264의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 265이다. 413F09는 Seq ID No: 268(IMGT) 또는 Seq ID No: 271(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 269(IMGT) 또는 Seq ID No: 272(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 270(IMGT) 또는 Seq ID No: 273(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 274의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 275이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 266(중쇄 핵산 서열 Seq ID No: 267)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 276(경쇄 핵산 서열 Seq ID No: 277)이다.
414B06은 Seq ID No: 278(IMGT) 또는 Seq ID No: 281(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 279(IMGT) 또는 Seq ID No: 282(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 280(IMGT) 또는 Seq ID No: 283(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 284의 중쇄 가변(VH) 영역 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 285이다. 414B06은 Seq ID No: 288(IMGT) 또는 Seq ID No: 291(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 289(IMGT) 또는 Seq ID No: 292(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 290(IMGT) 또는 Seq ID No: 293(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 294의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 295이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 286(중쇄 핵산 서열 Seq ID No: 287)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 296(경쇄 핵산 서열 Seq ID No: 297)이다.
416E01은 Seq ID No: 343(IMGT) 또는 Seq ID No: 346(Kabat)의 CDRH1 아미노산 서열, Seq ID No: 344(IMGT) 또는 Seq ID No: 347(Kabat)의 CDRH2 아미노산 서열, 및 Seq ID No: 345(IMGT) 또는 Seq ID No: 348(Kabat)의 CDRH3 아미노산 서열을 포함하는 Seq ID No: 349의 중쇄 가변 영역(VH) 아미노산 서열을 갖는다. VH 도메인의 중쇄 핵산 서열은 Seq ID No: 350이다. 416E01은 Seq ID No: 353(IMGT) 또는 Seq ID No: 356(Kabat)의 CDRL1 아미노산 서열, Seq ID No: 354(IMGT) 또는 Seq ID No: 357(Kabat)의 CDRL2 아미노산 서열, 및 Seq ID No: 355(IMGT) 또는 Seq ID No: 358(Kabat)의 CDRL3 아미노산 서열을 포함하는 Seq ID No: 359의 경쇄 가변 영역(VL) 아미노산 서열을 갖는다. VL 도메인의 경쇄 핵산 서열은 Seq ID No: 360이다. VH 도메인은 본원에 기재된 임의의 중쇄 불변 영역 서열, 예를 들어 Seq ID No: 193, Seq ID No: 195, Seq ID No: 197, Seq ID No: 199, Seq ID No: 201, Seq ID No: 203, Seq ID No: 205, Seq ID No: 340, Seq ID No: 524, Seq ID No: 526, Seq ID No: 528, Seq ID No: 530, Seq ID No: 532 또는 Seq ID No: 534와 조합될 수 있다. VL 도메인은 본원에 기재된 임의의 경쇄 불변 영역 서열, 예를 들어 Seq ID No: 207, 209, 211, 213, 215, 217, 219, 221, 223, 225, 227, 229, 231, 233, 235, 237, 536 및 538과 조합될 수 있다. 전장 중쇄 아미노산 서열은 Seq ID No: 351(중쇄 핵산 서열 Seq ID No: 352)이다. 전장 경쇄 아미노산 서열은 Seq ID No: 361(경쇄 핵산 서열 Seq ID No: 362)이다.
일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙을 포함한다. 일부 구현예에서, 항-PD-L1 항체는 아테졸리주맙이다.
1.6.15. 항체-약물 접합체
항-ICOS 항체는 Treg를 표적화하기 위해 세포독성제의 담체로 사용될 수 있다. 종양 미세환경(TME)에 위치한 Treg는 ICOS를 강력하게 발현한다(미국 특허 제9,957,323호 참고). ICOS는 종양내 Teff 또는 말초 Treg 상에서보다 종양내 Treg 상에서 더 강력하게 발현된다. 따라서 독성 약물 또는 전구약물로 표지된 항-ICOS 항체는 독성 페이로드를 전달하기 위해 TME의 Treg를 우선적으로 표적화하여 이들 세포를 선택적으로 억제할 수 있다. 세포독성제의 이러한 표적화는 Treg의 면역 억제 효과를 제거하는 추가 경로를 제공하여 Treg:Teff 균형을 Teff 활성에 유리하게 변경하며 본원에 논의된 다른 치료 접근(예를 들어, Treg의 Fc 효과기-매개 억제, 효과기 T 세포의 효능작용) 중 임의의 하나 이상에 대한 대안으로 또는 이와 조합으로 사용될 수 있다.
따라서, 본 발명은 세포독성 약물 또는 전구약물에 접합된 항-ICOS 항체를 제공한다. 전구약물의 경우, 전구약물은 TME 또는 다른 치료 활성의 표적 부위에서 활성화 가능하여 세포독성제를 생성한다. 활성화는 예를 들어 광흡수 접합체를 활성화하기 위해 근적외선을 사용하는, 광활성화와 같은 유발제에 반응하여 이루어질 수 있다[36]. 전구약물의 공간 선택적 활성화는 항체-약물 접합체의 세포독성 효과를 추가로 향상시켜, 종양내 Treg 상의 높은 ICOS 발현과 조합하여 이들 세포에 대해 고도로 선택적인 세포독성 효과를 제공한다.
항체-약물 접합체에서 사용하기 위해, 세포독성 약물 또는 전구약물은 혈액 내 항체-약물 접합체의 순환 동안 바람직하게는 비면역원성 및 무독성(휴면 또는 불활성)이다. 바람직하게는 세포독성 약물(또는 활성화될 때, 전구약물)은 강력하다 - 예를 들어 약물의 2 내지 4개 분자가 표적 세포를 사멸시키기에 충분할 수 있다. 광활성화 가능 전구약물은 실리카프탈로시아닌 염료(IRDye 700 DX)로, 이는 근적외선 노출 후 세포막에 치명적인 손상을 유도한다. 세포독성 약물에는 모노메틸 아우리스타틴 E와 같은 항유사분열제 및 메이탄신 유도체, 예를 들어, 메르탄신, DM1, 엠탄신과 같은 미세소관 억제제가 포함된다.
항체에 대한 약물(또는 전구약물)의 접합은 일반적으로 링커를 통해 이루어질 것이다. 링커는 절단 가능 링커, 예를 들어 이황화물, 하이드라존 또는 펩티드 링크일 수 있다. 카텝신 절단 가능 링커가 사용되어 약물이 종양 세포에서 카텝신에 의해 방출될 수 있다. 대안적으로, 절단 불가능 링커, 예를 들어 티오에테르 연결이 사용될 수 있다. 추가 부착기 및/또는 스페이서도 포함될 수 있다.
항체-약물 접합체의 항체는 Fab'2 또는 본원에 기재된 다른 항원-결합 단편과 같은 항체 단편일 수 있는데, 이는 이러한 단편의 작은 크기가 조직 부위(예를 들어, 고형 종양)로의 침투를 보조할 수 있기 때문이다.
본 발명에 따른 항-ICOS 항체는 면역사이토카인으로 제공될 수 있다. 항-ICOS 항체는 또한 조합 치료법에서 면역사이토카인과 함께 투여될 수 있다. 항-ICOS와의 조합 치료법에서 사용하기 위한 항체의 다수의 예가 본원에 기재되며, 이들 중 임의의 것(예를 들어, 항-PD-L1 항체)이 본 발명에서 사용하기 위한 면역사이토카인으로 제공될 수 있다. 면역사이토카인은 IL-2와 같은 사이토카인에 접합된 항체 분자를 포함한다. 따라서 항-ICOS:IL-2 접합체 및 항-PD-L1:IL-2 접합체는 본 발명의 추가 양태이다.
IL-2 사이토카인은 고(aβγ)친화도 IL-2 수용체 및/또는 중간 친화도(aβ) IL-2 수용체에서 활성을 가질 수 있다. 면역사이토카인에 사용되는 IL-2는 인간 야생형 IL-2 또는 하나 이상의 아미노산 결실, 치환 또는 부가를 갖는 변이체 IL-2 사이토카인, 예를 들어 N-말단에 1 내지 10개의 아미노산 결실을 갖는 IL-2일 수 있다. 다른 IL-2 변이체에는 돌연변이 R38A 또는 R38Q가 포함된다.
예시적인 항-PD-L1 면역사이토카인은 면역글로불린 중쇄 및 면역글로불린 경쇄를 포함하며, 중쇄는 N-말단에서 C-말단 방향으로:
a) CDRH1, CDRH2 및 CDRH3을 포함하는 VH 도메인; 및
b) 중쇄 불변 영역;
을 포함하고, 경쇄는 N-에서 C-말단 방향으로:
c) CDRL1, CDRL2 및 CDRL3을 포함하는 VL 도메인;
d) 경쇄 불변 영역(CL) ;
e) 선택적으로, 링커(L); 및
f) IL-2 사이토카인;
을 포함하고, VH 도메인 및 VL 도메인은 인간 PD-L1에 특이적으로 결합하는 항원-결합 부위에 포함되고;
면역사이토카인은 모티프 X1GSGX2YGX3X4FD(SEQ ID NO: 609)를 포함하는 CDRH3을 포함하는 VH 도메인을 포함하며, X1, X2 및 X3은 독립적으로 임의의 아미노산이고, X4는 존재하거나 부재하며, 존재하는 경우 임의의 아미노산일 수 있다.
VH 및 VL 도메인은 본원에 언급된 임의의 항-PD-L1 항체의 VH 및 VL 도메인, 예를 들어 1D05 VH 및 VL 도메인일 수 있다.
IL-2는 인간 야생형 또는 변이체 IL-2일 수 있다.
1.6.16. 백신접종
항-ICOS 항체는 백신 조성물에서 제공되거나 백신 조제물과 공동-투여될 수 있다. ICOS는 T 여포 헬퍼 세포 형성 및 배중심 반응에 관여한다[37]. 따라서 효능제 ICOS 항체는 백신 유효성을 향상시키는 분자 보조제로서 잠재적인 임상적 유용성을 갖는다. 항체는 B형 간염, 말라리아, HIV에 대한 백신과 같은 다양한 백신의 보호 유효성을 증가시키기 위해 사용될 수 있다.
백신접종 상황에서, 항-ICOS 항체는 일반적으로 Fc 효과기 기능이 결여되고, 따라서 ADCC, CDC 또는 ADCP를 매개하지 않는 항체일 것이다. 항체는 Fc 영역이 결여되거나 효과기 비포함 불변 영역을 갖는 형식으로 제공될 수 있다. 선택적으로, 항-ICOS 항체는 하나 이상의 유형의 Fc 수용체에 결합하지만 ADCC, CDC 또는 ADCP 활성을 유도하지 않거나 야생형 인간 IgG1과 비교하여 더 낮은 ADCC, CDC 및 ADCP 활성을 나타내는 중쇄 불변 영역을 가질 수 있다. 이러한 불변 영역은 ADCC, CDC 또는 ADCP 활성을 유발하는 역할을 하는 특정 Fc 수용체(들)에 결합할 수 없거나 더 낮은 친화도로 결합할 수 있다. 대안적으로, 세포 효과기 기능이 백신접종 상황에서 허용가능하거나 바람직한 경우, 항-ICOS 항체는 Fc 효과기 기능이 양성인 중쇄 불변 영역을 포함할 수 있다. 임의의 IgG1, IgG4 및 IgG4.PE 형식이 예를 들어 백신접종 요법에서 항-ICOS 항체를 위해 사용될 수 있으며, 적합한 이소형 및 항체 불변 영역의 다른 예는 본원의 다른 곳에서 더 상세히 제시되어 있다.
1.6.17. 제형 및 투여
항체는 모노클로날 또는 폴리클로날일 수 있지만, 바람직하게는 치료 용도를 위해 모노클로날 항체로 제공된다. 이는 선택적으로 상이한 결합 특이성의 항체를 포함하는 다른 항체 혼합물의 일부로 제공될 수 있다.
본 발명에 따르며 핵산을 암호화하는 항체는 일반적으로 단리된 형태로 제공될 것이다. 따라서, 항체, VH 및/또는 VL 도메인, 및 핵산은 이의 자연 환경 또는 이의 생산 환경으로부터 정제되어 제공될 수 있다. 단리된 항체 및 단리된 핵산에는 이들과 자연적으로 결합된 물질, 예컨대 생체내에서 이들과 함께 발견되거나, 제조가 시험관내 재조합 DNA 기술에 의해 이루어지는 경우 이들이 제조되는 환경(예를 들어, 세포 배양물)에서 발견되는 다른 폴리펩티드 또는 핵산이 없거나 또는 실질적으로 없을 것이다. 선택적으로, 단리된 항체 또는 핵산은 (1) 일반적으로 함께 발견되는 적어도 일부 다른 단백질이 없는 것, (2) 동일한 공급원(예를 들어, 동일한 종)에서 유래된 다른 단백질이 본질적으로 없는 것, (3) 상이한 종의 세포에 의해 발현되는 것, (4) 자연에서 이와 결합되는 폴리뉴클레오티드, 지질, 탄수화물, 또는 다른 물질의 적어도 약 50%로부터 분리된 것, (5) 자연에서 이와 결합되지 않는 폴리펩티드와 (공유 또는 비공유 상호작용에 의해) 작동 가능하게 결합된 것, 또는 (6) 자연에서 존재하지 않는 것이다.
항체 또는 핵산은 희석제 또는 보조제와 함께 제형화될 수 있고, 여전히 실용적인 목적을 위해 단리될 수 있으며, 예를 들어 이들은 면역검정에서 사용하기 위해 미세역가 플레이트를 코팅하는 데 사용될 때 담체와 혼합될 수 있고, 치료법에서 사용되는 경우 약학적으로 허용가능한 담체 또는 희석제와 혼합될 수 있다. 본원의 다른 곳에서 기재된 바와 같이, 다른 활성 성분이 또한 치료 조제물에 포함될 수 있다. 항체는 생체내에서 자연적으로 또는 이종성 진핵생물 세포, 예컨대 CHO 세포의 시스템에 의해 글리코실화될 수 있거나, 이들은(예를 들어 원핵생물 세포에서 발현에 의해 생성되는 경우) 글리코실화되지 않을 수 있다. 본 발명은 변형된 글리코실화 패턴을 갖는 항체를 포괄한다. 일부 적용에서는 바람직하지 않은 글리코실화 부위를 제거하기 위한 변형, 또는 예를 들어 ADCC 기능을 증가시키기 위한 푸코스 모이어티의 제거가 유용할 수 있다[38]. 다른 적용에서는 CDC를 변형하기 위해 갈락토실화의 변형이 이루어질 수 있다.
전형적으로, 단리된 생성물은 주어진 샘플의 적어도 약 5%, 적어도 약 10%, 적어도 약 25%, 또는 적어도 약 50%를 구성한다. 항체는 치료, 진단, 예방(prophylactic), 연구, 다른 용도에 방해될 수 있는, 자연 또는 생산 환경에서 발견되는 단백질 또는 폴리펩티드 또는 다른 오염물질이 실질적으로 없을 수 있다.
항체는 그 생산 환경의 구성요소로부터(예를 들어, 자연적으로 또는 재조합적으로) 식별, 분리 및/또는 회수되었을 수 있다. 단리된 항체는 예를 들어 항체가 FDA 승인 가능하거나 승인된 표준에 따라 단리되었도록, 그 생산 환경의 모든 다른 구성요소와 결합되지 않을 수 있다. 재조합 형질감염 세포에서 발생하는 것과 같은 그 생산 환경의 오염 구성요소는 전형적으로 항체에 대한 연구, 진단 또는 치료 용도를 방해할 물질이며, 효소, 호르몬 및 다른 단백질성 또는 비단백질성 용질이 포함될 수 있다. 일부 구현예에서, 항체는: (1) 예를 들어 로우리(Lowry) 방법에 의해 결정된 95중량% 초과, 일부 구현예에서는 99중량% 초과의 항체; (2) 회전 컵 서열분석기를 사용하여 N-말단 또는 내부 아미노산 서열의 적어도 15개의 잔기를 얻기에 충분한 정도로, 또는 (3) 쿠마시 블루 또는 은 염색을 사용하여 비환원 또는 환원 조건 하에서 SDS-PAGE에 의한 균질성까지 정제될 것이다. 단리된 항체에는 항체의 자연 환경 중 적어도 하나의 구성요소가 존재하지 않을 것이므로 재조합 세포 내의 제자리 항체가 포함된다. 그러나 일반적으로 단리된 항체 또는 그의 암호화 핵산은 적어도 하나의 정제 단계에 의해 제조될 것이다.
본 발명은 본원에 기재된 항체를 포함하는 치료 조성물을 제공한다. 이러한 항체를 암호화하는 핵산을 포함하는 치료 조성물도 제공된다. 암호화 핵산은 본원의 다른 곳에서 더 상세히 기재되어 있으며 DNA 및 RNA, 예를 들어, mRNA가 포함된다. 본원에 기재된 치료 방법에서, 항체를 암호화하는 핵산 및/또는 이러한 핵산을 함유하는 세포의 사용이 항체 자체를 포함하는 조성물에 대한 대안으로(또는 이에 추가적으로) 사용될 수 있다. 항체를 암호화하는 핵산을 함유하는 세포는, 선택적으로 핵산이 게놈 내로 안정하게 통합되고, 따라서 환자의 치료 용도를 위한 약제를 표시한다. 항-ICOS 항체를 암호화하는 핵산은 인간 B 림프구, 선택적으로 의도된 환자로부터 유래되고 생체외에서 변형된 B 림프구 내로 도입될 수 있다. 선택적으로 기억 B 세포가 사용된다. 환자에 대한 암호화 핵산을 함유하는 세포 투여는 항-ICOS 항체를 발현할 수 있는 세포 저장소를 제공하며, 이는 단리된 핵산 또는 단리된 항체의 투여와 비교하여 더 긴 기간에 걸쳐 치료 이점을 제공할 수 있다.
조성물은 적절한 담체, 부형제, 및 개선된 수송, 전달, 관용성 등을 제공하기 위하여 제형 내에 포함되는 다른 제제를 함유할 수 있다. 모든 약제사에게 알려진 처방집[Remington's Pharmaceutical Sciences, Mack Publishing Company, Easton, Pa]에서 다수의 적절한 제형이 확인될 수 있다. 이러한 제형에는, 예를 들어 산제, 페이스트, 연고, 젤리, 왁스, 오일, 지질, 지질(양이온성 또는 음이온성) 함유 소포체(예컨대, LIPOFECTINT™), DNA 접합체, 무수 흡수 페이스트, 수중유 및 유중수 에멀젼, 에멀젼형 카보왁스(다양한 분자량의 폴리에틸렌 글리콜), 반고체 겔, 및 카보왁스를 함유한 반고체 혼합물이 포함된다. 또한, 문헌[Powell et al. "Compendium of excipients for parenteral formulations" PDA (1998) J Pharm Sci Technol 52:238-311]을 참고한다. 조성물은 의료용 주사 완충액 및/또는 보조제와 조합된 항체 또는 핵산을 포함할 수 있다.
항체 또는 이를 암호화하는 핵산은 환자에 대한 원하는 투여 경로를 위해, 예를 들어 주사용 액체(선택적으로 수용액)에 제형화될 수 있다. 다양한 전달 시스템이 알려져 있으며 본 발명의 약학적 조성물을 투여하기 위해 사용될 수 있다. 투여 방법에는 피내, 근육내, 복강내, 정맥내, 피하, 비강내, 경막외, 및 경구 경로가 포함되지만 이에 제한되지 않는다. 피하 투여용 항체를 제형화하는 것은 전형적으로 정맥내 조제물과 비교하여 항체를 더 작은 부피로의 농축을 필요로 한다. 본 발명에 따른 항체의 높은 효능은 피하 제형을 실용화하기 위해 충분히 낮은 용량(sufficiently low dose)으로 사용할 수 있도록 하여, 덜 강력한 항-ICOS 항체와 비교하여 장점을 나타낼 수 있다.
조성물은 임의의 편리한 경로에 의해, 예를 들어 주입 또는 볼루스 주사에 의해, 상피 또는 점막피부의 내벽(예컨대, 구강 점막, 직장 점막, 및 내장 점막 등)을 통한 흡수에 의해 투여될 수 있으며, 다른 생물학적 활성제와 함께 투여될 수 있다. 투여는 전신적 또는 국소적일 수 있다.
또한, 약학적 조성물은 소포, 특히 리포솜으로 전달될 수 있다(문헌[Langer (1990) Science 249:1527-1533; Treat et al. (1989) in Liposomes in the Therapy of Infectious Disease and Cancer, Lopez Berestein and Fidler (eds.), Liss, New York, pp. 353-365; Lopez-Berestein, ibid., pp. 317-327; 일반적으로 동일 문헌 참고).
특정 상황에서, 약학적 조성물은 제어 방출 시스템으로 전달될 수 있다. 하나의 구현예에서, 펌프가 사용될 수 있다(문헌[Langer, 상기 문헌; Sefton (1987) CRC Crit. Ref. Biomed. Eng. 14:201] 참고). 다른 구현예에서, 중합체성 물질이 사용될 수 있다; 문헌[Medical Applications of Controlled Release, Langer and Wise (eds.), CRC Pres., Boca Raton, Fla. (1974)]을 참고한다. 다른 구현예에서, 제어 방출 시스템이 조성물 표적의 인근에 배치될 수 있고, 따라서 전신 용량의 일부만을 필요로 한다(예를 들어, 문헌[Goodson, in Medical Applications of Controlled Release, supra, vol. 2, pp. 115-138, 1984] 참고).
주사용 조제물(injectable preparation)은 정맥내, 피하, 피내, 및 근육내 주사, 점적 주입 등을 위한 투여형을 포함할 수 있다. 이러한 주사용 조제물은 알려진 방법에 의해 제조될 수 있다. 예를 들어, 주사용 조제물은, 예를 들어 주사용으로 통상적으로 사용되는 멸균 수성 매질 또는 유성 매질에 상기 항체 또는 이의 염을 용해, 현탁, 또는 유화시킴으로써 제조될 수 있다. 주사용 수성 매질로서, 예를 들어 생리 식염수, 글루코스 및 다른 보조제를 함유하는 등장성 용액 등이 있으며, 이들은 알코올(예를 들어, 에탄올), 폴리알코올(예를 들어, 프로필렌 글리콜, 폴리에틸렌 글리콜), 비이온성 계면활성제(예를 들어, 폴리소르베이트 80, HCO-50(수소화된 피마자유의 폴리옥시에틸렌(50 mol) 부가물)) 등과 같은 적절한 가용화제와 조합하여 사용될 수 있다. 유성 매질로서, 예를 들어 참깨유, 대두유 등이 사용될 수 있으며, 이들은 벤질 벤조에이트, 벤질 알코올 등과 같은 가용화제와 조합하여 사용될 수 있다. 이렇게 제조된 주사제는 일반적으로 적절한 앰플에 채워질 수 있다. 본 발명의 약학적 조성물은 표준 주사바늘 및 시린지를 사용하여 피하 또는 정맥내 전달될 수 있다. 치료는 클리닉 내 사용으로만 제한되지 않을 것으로 고려된다. 따라서 주사바늘 없는 디바이스를 이용한 피하주사에도 유리하다. 또한, 피하 전달과 관련하여, 펜 전달 디바이스는 본 발명의 약학적 조성물을 전달하는 데 용이하게 활용된다. 이러한 펜 전달 디바이스는 재사용이 가능하거나 일회용일 수 있다. 재사용이 가능한 펜 전달 디바이스는 일반적으로 약학적 조성물을 함유하는 교체 가능한 카트리지를 이용한다. 카트리지 내의 약학적 조성물이 전부 투여되어 카트리지가 비게 되면, 빈 카트리지는 용이하게 폐기되고, 약학적 조성물을 함유하는 새로운 카트리지로 교체될 수 있다. 이후, 펜 전달 디바이스는 재사용될 수 있다. 일회용 펜 전달 디바이스에는, 교체 가능한 카트리지가 없다. 대신, 일회용 펜 전달 디바이스는 디바이스 내의 저장소에 보유된 약학적 조성물로 미리 채워진다. 약학적 조성물의 저장소가 비게 되면, 전체 디바이스가 폐기된다. 다수의 재사용 가능 펜 및 자동주사기 전달 디바이스가 본 발명의 약학적 조성물의 피하 전달에 활용된다. 몇 가지만 예를 들면, AUTOPEN™(Owen Mumford, Inc., Woodstock, UK), DISETRONIC™ 펜(Disetronic Medical Systems, Burghdorf, Switzerland), HUMALOG MIX 75/25™ 펜, HUMALOG™ 펜, HUMALIN 70/30™ 펜(Eli Lilly and Co., Indianapolis, Ind.), NOVOPEN™I, II 및 III(Novo Nordisk, Copenhagen, Denmark), NOVOPEN JUNIOR™(Novo Nordisk, Copenhagen, Denmark), BD™ 펜(Becton Dickinson, Franklin Lakes, N.J.), OPTIPENT™, OPTIPEN PRO™, OPTIPEN STARLET™, 및 OPTICLIKT™(Sanofi-Aventis, Frankfurt, Germany)이 있으나, 이에 제한되지 않음이 명확하다. 본 발명의 약학적 조성물의 피하 전달에 적용되는 일회용 펜 전달 디바이스의 예로는 SOLOSTAR™ 펜(Sanofi-Aventis), FLEXPEN™(Novo Nordisk), 및 KWIKPEN™(Eli Lilly)이 있으나, 이에 제한되지 않음이 명확하다.
유리하게는, 상기 기재된 경구 또는 비경구용 약학적 조성물은 활성 성분의 용량을 맞추기에 적합한 단위 용량의 투여형으로 제조된다. 단위 용량의 이러한 투여형은, 예를 들어 정제, 환제, 캡슐제, 주사제(앰플), 좌제 등을 포함한다. 포함된 상기 항체의 양은 일반적으로 단위 용량; 특히 주사 형태의 투여형 당 약 5 내지 약 500 mg이고, 상기 항체는 다른 투여형의 경우 약 5 내지 약 100 mg 및 약 10 내지 약 250 mg으로 포함될 수 있다.
항체, 핵산, 또는 이를 포함하는 조성물은 약병, 시린지, IV 용기, 또는 주사 디바이스와 같은 의료 용기에 함유될 수 있다. 한 예에서, 항체, 핵산, 또는 조성물은 시험관내에 있고, 멸균성 용기에 담길 수 있다. 한 예에서, 항체, 패키징, 및 본원에 기재된 치료 방법에서 사용하기 위한 지침을 포함하는 키트가 제공된다.
본 발명의 한 양태는 본 발명의 항체 또는 핵산 및 하나 이상의 약학적으로 허용가능한 부형제를 포함하는 조성물이며, 그 예는 상기 나열되어 있다. "약학적으로 허용가능한"은 인간을 포함하여 동물에서 사용하기 위해 미국 연방 정부 또는 주 정부의 관리 기관에 의해 승인되거나 승인 가능하거나 미국 약전, 또는 다른 일반적으로 인정되는 약전에 열거된 것을 의미한다. 약학적으로 허용가능한 담체, 부형제, 또는 보조제는 제제, 예를 들어 본원에 기재된 임의의 항체 또는 항체 사슬과 함께 환자에 투여될 수 있고, 이의 약리 활성을 파괴하지 않으며, 치료량의 제제가 전달하기에 충분한 용량으로 투여될 때 무독성이다.
일부 구현예에서, 항체는 본 발명에 따른 조성물에서 단독 활성 성분일 것이다. 따라서, 조성물은 항체로 구성되거나 항체와 하나 이상의 약학적으로 허용가능한 부형제로 구성될 수 있다. 그러나, 본 발명에 따른 조성물에는 선택적으로 하나 이상의 추가의 활성 성분이 포함된다. 항-ICOS 항체와 조합될 수 있는 제제에 대한 상세한 설명은 본원의 다른 곳에서 제공된다. 선택적으로, 조성물은 조합 조제물, 예를 들어 항-ICOS 항체 및 하나 이상의 다른 항체를 포함하는 단일 제형물에 다중 항체(또는 암호화 핵산)를 함유한다. 본 발명에 따른 항체 또는 핵산과 함께 투여하는 것이 바람직할 수 있는 다른 치료제에는 진통제가 포함된다. 임의의 이러한 제제 또는 제제의 조합이 조합된 또는 별도 조제물로서 본 발명에 따른 항체 또는 핵산과 조합되어 투여되거나 이와의 조성물로 제공될 수 있다. 본 발명에 따른 항체 또는 핵산은 별도로 및 순차적으로, 또는 동시에 및 선택적으로 조합된 조제물로서, 언급된 것들과 같은 다른 치료제 또는 제제와 함께 투여될 수 있다.
특정 치료 적응증에 사용하기 위한 항-ICOS 항체는 허용된 표준 치료와 조합될 수 있다. 따라서, 항암 치료를 위해, 항체 치료법은 예를 들어 화학치료법, 수술 및/또는 방사선 치료법이 또한 포함되는 치료 요법에서 사용될 수 있다. 방사선치료법은 단일 용량 또는 분할 용량으로 영향을 받은 조직에 직접 또는 전신으로 전달될 수 있다.
다수의 조성물이 별도로 또는 동시에 투여될 수 있다. 별도 투여는 두 조성물을 상이한 시간에, 예를 들어 적어도 10, 20, 30, 또는 10 내지 60분의 간격으로, 또는 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12시간의 간격으로 투여하는 것을 지칭한다. 또한 조성물은 24시간의 간격으로 또는 심지어 더 긴 간격으로 투여할 수 있다. 대안적으로, 2개 이상의 조성물은 동시에, 예를 들어 10분 미만 또는 5분 미만의 간격으로 투여될 수 있다. 동시에 투여되는 조성물은 일부 양태에서 각각의 구성요소에 대한 유사한 또는 상이한 시간 방출 메커니즘을 갖거나 갖지 않고, 혼합물로서 투여될 수 있다.
항체, 및 이의 암호화 핵산은 치료제로서 투여될 수 있다. 본원에서 환자는 일반적으로 포유류, 전형적으로 인간이다. 항체 또는 핵산은 포유류에게 예를 들어 본원에서 언급된 임의의 투여 경로에 의해 투여될 수 있다.
투여는 일반적으로 "치료 유효량"이며, 이는 투여시 목적하는 효과를 생성하고 환자에게 유익을 나타내는 데 충분한 양이다. 정확한 양은 치료 목적에 따라 달라질 것이며, 알려진 기술을 사용하여 당업자에 의해 확인될 수 있을 것이다(예를 들어, 문헌[Lloyd (1999) The Art, Science and Technology of Pharmaceutical Compounding] 참고). 치료의 처방, 예를 들어 용량 등의 결정은 일반 개업의 및 그밖의 의사의 책임 내에 있으며, 치료할 질환의 증상 및/또는 진행의 중증도에 따라 달라질 수 있다. 항체 또는 핵산의 치료 유효량 또는 적합한 용량은 동물 모델에서 이의 시험관내 활성 및 생체내 활성을 비교함으로써 측정될 수 있다. 마우스 및 다른 시험 동물에서 효과적인 투여량을 인간에게 외삽하는 방법은 알려져 있다.
본원의 실시예에 기재된 생체내 연구에 의해 나타난 바와 같이, 항-ICOS 항체는 놀랍도록 낮은 용량을 포함하는 다양한 용량에서 효과적일 수 있다. 놀랍게도, 이러한 전임상 연구를 고려할 때, 체중당 낮은 용량의 항-ICOS 항체(예를 들어, KY1044) 또는 낮은 고정 용량의 항-ICOS 항체(예를 들어, KY1044)가 다양한 암에 걸쳐 인간 환자에서 부분적 또는 완전 항-종양 활성을 생성하는 데 효과적이었다.
항-ICOS 항체(예를 들어, 전장 항체 또는 이의 항원 결합 단편)는 용량당 하기 값 또는 범위 중 하나의 양으로 대상체에 투여될 수 있다:
약 10 μg/kg 체중 내지 약 3 mg/kg 체중,
약 10 μg/kg 체중 내지 약 1 mg/kg 체중,
약 10 μg/kg 체중 내지 약 0.3 mg/kg 체중,
약 10 μg/kg 체중 내지 약 0.1 mg/kg 체중, 또는
약 10 μg/kg 체중 내지 약 30 μg/kg 체중.
성인 인간에서의 고정 투약의 경우, 적합한 용량은 약 10 mg 이하, 9 mg 이하, 또는 약 8 mg 이하, 예를 들어 약 8 mg, 약 7 mg, 약 6 mg, 약 5 mg, 약 4 mg, 약 3 mg, 약 2.4 mg, 약 2 mg, 약 1 mg, 약 0.8 mg, 또는 약 0.5 mg, 또는 그 사이의 임의의 값일 수 있다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 0.5 내지 10 mg으로 투여된다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 0.5 내지 8 mg으로 투여된다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 0.8 내지 8 mg으로 투여된다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 0.8 내지 2.4 mg으로 투여된다.
일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 8 mg으로 투여된다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 2.4 mg으로 투여된다. 일부 구현예에서, 대상체에는 항-ICOS 항체(예를 들어, KY1044)가 용량당 약 0.8 mg으로 투여된다.
본원에 기재된 치료 방법에서, 하나 이상의 용량이 투여될 수 있다. 일부 경우에, 단일 용량은 장기 이익을 달성하는 데 효과적일 수 있다. 따라서, 방법은 항체, 그 암호화 핵산, 또는 조성물의 단일 용량을 투여하는 단계를 포함할 수 있다. 대안적으로, 다중 용량은 일반적으로 순차적으로 및 수일, 수주, 또는 수개월 기간으로 분리되어 투여될 수 있다. 항-ICOS 항체는 2 내지 6주 간격으로, 예를 들어 2주마다, 3주마다, 4주마다, 5주마다 또는 6주마다 대상체에 반복적으로 투여될 수 있다. 일부 구현예에서, 항-ICOS 항체는 3주마다 대상체에 투여된다. 일부 구현예에서, 항-ICOS 항체는 6주마다 대상체에 투여된다. 일부 구현예에서, KY1044는 3주마다 대상체에 투여된다. 일부 구현예에서, KY1044는 6주마다 대상체에 투여된다. 선택적으로, 항-ICOS 항체는 대상체에 한 달에 한 번, 또는 덜 빈번하게, 예를 들어 2개월마다 또는 3개월마다 투여될 수 있다. 따라서, 환자를 치료하는 방법은 대상체에 항-ICOS 항체의 단일 용량을 투여하고, 적어도 1개월, 적어도 2개월, 적어도 3개월 동안 투여를 반복하지 않는, 선택적으로 적어도 12개월 동안 투여를 반복하지 않는 단계를 포함할 수 있다.
일부 구현예에서 항-ICOS 항체, 예를 들어 KY1044는 대상체에 적어도 6개월 동안 투여된다. 일부 구현예에서 항-ICOS 항체, 예를 들어 KY1044는 대상체에 6개월 동안 투여된다. 일부 구현예에서 항-ICOS 항체, 예를 들어 KY1044는 대상체에 적어도 12개월 동안 투여된다. 일부 구현예에서 항-ICOS 항체, 예를 들어 KY1044는 대상체에 12개월 동안 투여된다. 일부 구현예에서 항-ICOS 항체, 예를 들어 KY1044는 12개월 초과 동안 대상체에 투여된다.
일부 구현예에서, KY1044는 적어도 6개월 동안, 예를 들어 6개월 동안, 12개월 동안, 또는 12개월 초과 동안 3주마다 대상체에 투여된다.
항-ICOS 항체의 하나의 또는 다중 용량을 사용하여 필적하는 치료 효과를 얻을 수 있으며, 이는 단일 용량의 항체가 종양 미세환경을 재설정하는 데 효과적인 결과일 수 있다. 의사는 항-ICOS 항체가 조합되는 질환 상태 및 임의의 다른 치료제 또는 치료 조치(예를 들어, 수술, 방사선치료법 등)를 고려하여 질환 및 치료법을 거치는 환자에 대한 항-ICOS 항체의 투여 요법을 맞춤화할 수 있다. 일부 구현예에서, 항-ICOS 항체의 유효 용량은 한 달에 1회보다 더 자주, 예를 들어 3주 1회, 2주 1회, 또는 매주 1회 투여된다. 항-ICOS 항체를 사용한 치료에는 적어도 1개월, 적어도 6개월 또는 적어도 1년의 기간에 걸쳐 투여되는 다중 용량이 포함될 수 있다. 다중 용량은 동일할 수도 있고 상이할 수도 있다.
본원에서 사용된 바와 같이, 용어 "치료하다", "치료", "치료하는", 또는 "완화"는 치료적 처치를 지칭하며, 질환 또는 장애와 관련된 질병의 진행 또는 중증도를 역전시키거나, 경감시키거나, 완화하거나, 억제하거나, 늦추거나, 중단시키는 것을 목적으로 한다. 용어 "치료하는"에는 질병, 질환, 또는 장애의 적어도 하나의 유해 효과 또는 증상을 감소시키거나 경감시키는 것이 포함된다. 하나 이상의 증상 또는 임상적 마커가 감소되는 경우, 치료는 일반적으로 "효과적"이다. 대안적으로, 질환의 진행이 감소되거나 멈추는 경우, 치료는 "효과적"이다. 즉, "치료"에는 증상 또는 마커의 개선뿐만 아니라, 치료의 부재시 예상될 것과 비교하여 증상의 진행 또는 악화의 중단 또는 적어도 늦춤이 포함된다. 유익한 또는 목적하는 임상 결과에는 하나 이상의 증상(들)의 경감, 질환 정도의 감소, 질환의 안정화된(즉, 악화되지 않는) 상태, 질환 진행의 지연 또는 늦춤, 질환 상태의 완화 또는 경감, 관해(부분적 또는 전체적), 및/또는 감소된 사망률(검출 가능하거나 검출 불가능함)이 포함되지만 이에 제한되지 않는다. 용어 질환의 "치료"에는 또한 질환의 증상 또는 부작용의 경감(완화 치료 포함)을 제공하는 것이 포함된다. 효과적인 치료를 위해, 완전 치유는 고려되지 않는다. 특정 양태에서, 방법에는 치유가 또한 포함된다. 본 발명의 맥락에서, 치료는 예방적 치료일 수 있다.
일부 구현예에서, "치료하는"은 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 것을 포함한다. 본원에서 사용된 바와 같이, 이러한 질환 또는 질병에는 종양 및/또는 암이 포함되지만 이에 제한되지 않는다. 일부 구현예에서, 암은 진행성 및/또는 전이성 암이다.
본원에서 사용된, mg 단위의 투약량의 언급에서 "약"은 명시된 값이 1.5 mg 미만인 경우 명시된 값의 ±0.1 mg을 지칭하고, 명시된 값이 적어도 1.5 mg인 경우 명시된 값의 ±0.5 mg을 지칭한다.
1.6.18. T 세포 치료법
WO2011/097477은 T 세포 집단을 1차 활성화 신호를 제공하는 제1 제제(예를 들어, 항-CD3 항체) 및 ICOS를 활성화하는 제2 제제(예를 들어, 항-ICOS 항체)와, 선택적으로 IL-1β, IL-6과 같은 Th17 극성화제, 중화 항-IFNγ 및/또는 항-IL-4의 존재 하에 접촉시킴으로써 T 세포를 생성하고 증식시키기 위한 항-ICOS 항체의 용도를 기재했다. 본원에 기재된 항-ICOS 항체는 T 세포 집단을 제공하기 위해 이러한 방법에서 사용될 수 있다. 치료 활성(예를 들어, 항-종양 활성)을 갖는 배양된 증식 T 세포 집단이 생성될 수 있다. WO2011/097477에 기재된 바와 같이, 이러한 T 세포는 면역치료법에 의해 환자를 치료하는 방법에서 치료적으로 사용될 수 있다.
1.6.19. 치료 후보로서의 항-ICOS 항체에 대한 형태학적 검정
후보 치료 항-ICOS 항체가 고체 표면에 커플링되어 ICOS-발현 T 세포와 접촉될 때 세포에서 형태학적 변화를 유도할 수 있음이 관찰되었다. 항-ICOS 항체로 내부 코팅된 웰에 ICOS+ T 세포를 첨가할 때, 세포는 이의 초기의 둥근 모양으로부터 변화하여 스핀들 모양을 채택하고 확산되어 항체 코팅 표면에 부착하는 것으로 나타났다. 이러한 형태학적 변화는 대조군 항체로는 관찰되지 않았다. 더욱이, 효과는 용량 의존적인 것으로 확인되었으며, 표면의 항체 농도가 증가함에 따라 더 빠르고/빠르거나 더 뚜렷한 형태 변화가 발생했다. 형태 변화는 T 세포가 ICOS에 결합하는 것 및/또는 항-ICOS 항체에 의한 효능작용의 대리 지표를 제공한다. 검정은 T 세포 표면 상에서 ICOS의 다량체화를 촉진하는 항체를 식별하기 위해 사용될 수 있다. 이러한 항체는 치료 후보 효능제 항체를 나타낸다. 편리하게도 이 검정에 의해 제공되는 시각적 지표는 특히 많은 수의, 항체 또는 세포를 스크리닝하는 간단한 방법이다. 검정은 자동화되어 고처리량 시스템에서 수행될 수 있다.
따라서, 본 발명의 한 양태는 ICOS에 결합하는 항체를 선택하기 위한, 선택적으로 ICOS 효능제 항체를 선택하기 위한 검정이고, 검정은 다음을 포함한다:
시험 웰의 기판에 고정화된(부착 또는 접착된) 항체 어레이를 제공하고;
ICOS 발현 세포(예를 들어 활성화 1차 T 세포 또는 MJ 세포)를 시험 웰에 첨가하고;
세포의 형태를 관찰하고;
웰 내의 기판에 대해 둥근 모양으로부터 편평한 모양으로 세포의 모양 변화를 검출하고; 형태 변화는 항체가 ICOS에 결합하는 항체, 선택적으로 ICOS 효능제 항체임을 나타내고,
시험 웰로부터 항체를 선택함.
검정은 각각 시험을 위해 상이한 항체를 함유하는 여러 시험 웰을 사용하여, 선택적으로 병렬로, 예를 들어 96웰 플레이트 형식으로 수행될 수 있다. 기판은 바람직하게는 웰의 내부 표면이다. 따라서, 세포의 편평화가 관찰될 수 있는 2차원 표면이 제공된다. 예를 들어, 웰의 바닥 및/또는 벽이 항체로 코팅될 수 있다. 기판에 대한 항체의 테더링은 항체의 불변 영역을 통해 이루어질 수 있다.
ICOS에 결합하지 않는 것으로 알려진 항체, 바람직하게는 사용될 ICOS-발현 세포의 표면 상 항원에 결합하지 않는 항체와 같은 음성 대조군이 포함될 수 있다. 검정은 형태학적 변화의 정도를 정량하고, 다수의 항체가 시험되는 경우 하나 이상의 다른 시험 항체보다 더 큰 형태학적 변화를 유도하는 항체를 선택하는 것을 포함할 수 있다.
항체의 선택은 관심 시험 웰에 존재하는 항체를 암호화하는 핵산을 발현하거나 해당 항체의 CDR 또는 항원 결합 도메인을 포함하는 항체를 발현하는 것을 포함할 수 있다. 항체는 예를 들어 선택된 항체의 항원 결합 도메인을 포함하는 항체, 예를 들어 항체 단편, 또는 상이한 불변 영역을 포함하는 항체를 제공하기 위해 선택적으로 재형식화될 수 있다. 선택된 항체에는 바람직하게는 인간 IgG1 불변 영역 또는 본원에 기재된 바와 같은 다른 불변 영역이 제공된다. 선택된 항체는 하나 이상의 추가 성분을 포함하는 조성물로 추가 제형화될 수 있으며 - 적합한 약학적 제형은 본원의 다른 곳에서 논의된다.
본 발명의 다양한 추가 양태 및 실시예는 본 개시내용을 고려하여 당업자에게 자명할 것이다. 지칭된 임의의 특허 또는 특허 출원의 공개된 미국 대응물을 포함하여 본 명세서에서 언급된 모든 문서는 그 전체가 본원에 참조로 포함된다.
1.7. 실험예
항-ICOS 항체의 생성, 특성규명 및 성능은 이전에 미국 특허 제9,957,323호에 개시되었다.
1.8. 실시예 1: 마우스에서 A20 종양 성장에 대한 항-ICOS Ab의 단독치료 유효성
항-ICOS 항체 STIM001 mIgG2a 및 STIM003 mIgG2a는 각각 마우스 A20 동계 모델에서 생체내 단독치료법으로 사용되었을 때 강력한 항-종양 유효성을 나타냈다.
1.8.1. 재료 및 방법
유효성 연구를 피하 A20 망상 세포 육종 모델(ATCC, TIB-208)을 사용하여 BALB/c 마우스에서 수행하였다. A20 세포주는 늙은 BALB/cAnN 마우스에서 발견된 자발적 망상 세포 신생물에서 유래된 BALB/c B 세포 림프종 세포주이다. 이 세포주는 ICOSL에 대해 양성인 것으로 보고되었다.
18 그램 초과의 BALB/c 마우스를 Charles River UK에의해 공급받았으며 특정 병원체가 없는 조건 하에 수용하였다. 총 5x10e5개 A20 세포(P20 미만의 계대 수)를 마우스의 오른쪽 옆구리에 피하 주사했다. A20 세포를 시험관내에서 계대배양하고 PBS로 2회 세척하고 10% 송아지 태아 혈청이 보충된 RPMI에 재현탁하였다. 종양세포 주사시 세포 생활성은 85% 초과로 확인되었다. 달리 명시되지 않는 한, 항체 또는 이소형 투여는 종양 세포 주사 제8일 후부터 개시하였다.
STIM001 및 STIM003 항-ICOS 항체를 마우스 IgG2a 이소형 형식으로 생성하였다. 마우스 교차반응성 항-PD-L1 항체(AbW)도 동일한 이소형 형식(마우스 IgG2a)으로 생성하였다. STIM001, STIM003 및 항-PD-L1 항체를 종양 세포 이식 후 제8일로부터 시작하여 주 2회 각 항체 200 μg을 복강내(IP) 투약했다(제8일 내지 제29일에 3주 동안 투약). 동물 체중 및 종양 부피를 종양 세포 주사일로부터 주 3회 측정하였다. 종양 부피를 변형된 타원체 공식 1/2(길이 × 너비2)을 사용하여 계산하였다. 마우스의 종양이 평균 직경 12 mm에 도달할 때까지 마우스를 계속 연구했다. 실험은 종양 세포 이식 후 제43일에 중단하였다. 종양 성장을 모니터링하고 이소형 대조(mIgG2a) 항체로 처리된 동물의 종양과 비교했다. 처리군을 아래 표 E20에 나타낸다.
[표 E20]
A20 연구를 위한 처리군.
1.8.2. 결과
A20 종양 모델에서 STIM001 또는 STIM003(mIgG2a)의 단독치료법 투여는 완전 항-종양 반응을 일으켰다(도 3, 도 4). STIM001 또는 STIM003이 투여된 모든 동물은 질환이 치유되었다. 이는 이소형 대조군 및 PD-L1 mIgG2a 그룹의 결과와 대조된다(도 1, 도 2). 드물게 이소형 대조군(자발적 퇴행) 및 항-PDL-1 그룹의 일부 동물에 대하여 종양의 퇴행이 관찰되었지만 항-ICOS 항체를 사용한 처리는 유의하게 더 큰 유효성을 일으켰다. 연구 종료 시, 대조군 동물 8마리 중 3마리 및 항-PDL-1 처리 동물 8마리 중 2마리는 종양을 갖지 않았다. 그러나 STIM001 또는 STIM003으로 처리된 모든 동물은 연구 종료 시 종양이 없어서(두 그룹 모두에서 마우스 8마리 중 8마리) 항-ICOS 항체를 사용한 100% 치유를 나타냈다.
1.9. 실시예 2: 항-ICOS 항체와 항-PD-L1 항체의 조합에 대한 J558 골수종 동계 모델에서의 생체내 강력한 항-종양 유효성
항-ICOS 항체 STIM003 mIgG2a 및 항-PD-L1 항체 AbW mIgG2a를 J558 종양 모델에 개별적으로 및 조합으로 투여하였다. 이는 골수종의 동계 마우스 모델이다. 항-ICOS 항체는 단독치료법으로 또는 항-PD-L1과의 조합으로 투약될 때 종양 성장을 억제하는 것으로 밝혀졌다.
1.9.1. 재료 및 방법
항-종양 유효성 연구를 피하 J558 형질세포종:골수종 세포주(ATCC, TIB-6)를 사용하여 Balb/c 마우스에서 수행하였다. Balb/c 마우스는 6 내지 8주령 및 18 g 초과로 Charles River UK로부터 공급받았으며, 이를 특정 병원체가 없는 조건 하에 수용하였다. 총 5 x 106개의 세포(P15 미만 계대 수)를 마우스의 오른쪽 옆구리에 피하 주사했다(100 μl). 달리 명시되지 않는 한, 종양 세포 주사 후 제11일에, 동물을 종양 크기에 기반하여 무작위화하고 처리를 개시했다. J558 세포를 TrypLE™ 익스프레스 효소(Thermofisher)를 사용하여 시험관내에 계대배양하고, PBS로 2회 세척하여 10% 송아지 태아 혈청이 보충된 DMEM에 재현탁하였다. 종양 세포 주사시 세포 생활성은 90% 초과로 확인되었다.
종양이 약 140 mm^3의 평균 부피에 도달했을 때 처리를 개시하였다. 그런 다음 동물을 유사한 평균 종양 크기를 갖는 4개 그룹에 할당했다(투약군에 대해서는 표 E-21 참고). 동물이 복지(희귀) 또는 종양 크기로 인해 연구에서 제외되어야 하지 않는 한, 마우스 교차반응성인 두 항체 모두를 제11일(종양 세포 이식 후)부터 3주 동안 주 2회 IP 투약하였다(도 8). 대조군으로서 하나의 동물 그룹(n=10)에 식염수 용액을 사용하여 동시에 투약했다. 조합 그룹의 경우, STIM003 및 항-PDL1 항체를 둘 모두를 각각 60 μg 및 200 μg(0.9% 식염수 중)으로 동시에 IP 투약했다. 종양 성장을 37일에 걸쳐 모니터링하고 식염수로 처리된 동물의 종양과 비교했다. 동물 체중 및 종양 부피를 종양 세포 주사일로부터 주 3회 측정하였다. 종양 부피는 변형된 타원체 공식 1/2(길이 x 너비2)을 사용하여 계산하였다. 마우스의 종양이 평균 직경 12 mm3에 도달할 때까지 또는 드물게 종양 궤양 발생이 관찰될 때까지(복지) 마우스를 계속 연구했다.
[표 E21]
J558 유효성 연구를 위한 처리군.
1.9.2. 결과
J558 동계 종양은 매우 공격적이었고 식염수 대조군(n=10)의 모든 동물을 종양 크기로 인해 제21일까지 연구에서 제외해야 했다. 항-STIM003 mIgG2a 및 항-PDL1 mIgG2a 둘 모두는 이 모델에서 단독치료법으로서 우수한 유효성을 실증했으며 각각 37.5% 및 75%의 동물이 질환을 치유했다. 중요하게는, 두 항체의 조합으로 제37일까지 동물의 100%에서 형질세포종 종양의 거부가 초래되었다. 데이터를 도 8에 나타낸다.
1.10. 실시예 3: 항-PD1의 투여가 항-PD-L1 항체보다 TIL 상의 ICOS 발현을 유의하게 더 증가시킴
종양 침윤 림프구(TIL) 하위세트 상의 ICOS 발현에 대한 항-PD-L1 또는 항-PD-1 항체를 사용하는 처리의 효과를 평가하기 위해 확립된 CT26 종양을 보유하는 동물에서 약력학 연구를 수행했다. 하기 항체를 비교했다:
· 항-PD-L1 AbW mIgG1[제한된 효과기 기능]
· 항-PD-L1 AbW mIgG2a[효과기 기능 있음]
· 항-PD-L1 10F9.G2 래트 IgG2b[효과기 기능 있음]
· 항-PD1 항체 RMT1-14 래트 IgG2a[효과기 비포함].
처리된 마우스의 종양을 단리하고, 단일 세포로 해리하고, CD45, CD3, CD4, CD8, FOXP3 및 ICOS에 대해 염색했다.
1.10.1. 재료 및 방법
래트 항-PD-1 RMP1-14 IgG2a(BioXCell; 카탈로그 번호: BE0146), 래트 항-PD-L1 10F9.G2 IgG2b(Bio-Legend; 카탈로그 번호: 124325) 그리고 항-PD-L1 AbW mIgG1 및 mIgG2a를 종양 세포 이식 후 제13일 및 제15일에 130 μg으로 i.p. 투약하여 CT26 종양 모델에서 시험했다. 제16일에 동물을 도태시키고 FACS 분석을 위해 마우스 종양을 수확했다. 마우스 종양 해리 키트(Miltenyi Biotec)를 사용하여 종양을 해리하고 균질화했다. 생성된 세포 현탁액을 70 μM 필터를 통해 정화하고, 펠릿화하고 96웰 플레이트에서 200만 개 세포/웰로 FACS 완충액에 재현탁했다. 세포 현탁액을 항-16/32 mAb(eBioscience)와 함께 인큐베이션하고 모두 eBioscience Ltd.에서 얻은 CD3(17A2), CD45(30-F11), CD4(RM4-5), CD8(53-6.7) 및 ICOS(7E.17G9)에 특이적인 FACS 항체로 염색했다. 세포를 또한 라이브데드(LiveDead) 황색 고정 가능한 생활성 염료(Life Technologies)로 염색하였다. Foxp3 세포내 염색을 위해 샘플을 고정하고 투과화하여 Foxp3에 특이적인 항체(eBioscience, FJK-16s)로 염색했다. 샘플을 PBS에 재현탁하고 Attune 유세포 분석기(Invitrogen)에서 데이터를 획득하고 FlowJo V10 소프트웨어(Treestar)를 사용하여 분석했다.
1.10.2. 결과
항-PD1 및 항-PD-L1 항체를 사용한 처리는 측정된 시점에 ICOS를 발현하는 CD8 세포 및 T Reg 백분율의 약간의 증가만 초래했다. 그러나, 항-PD1 래트 IgG2a에 반응한, ICOS+ve CD8 세포의 표면 상에서 ICOS 발현(증가된 dMFI)의 명확하고 유의한 증가(식염수 처리군 대비)가 관찰되었다. ICOS 발현은 또한 CD4 효과기 및 CD4 T Reg 세포 상에서 상향조절됨이 주지되었으나 이는 통계적 유의성에는 도달하지 못했다. 이 항-PD1 항체는 항-PD-L1 mIgG2a로는 거의 나타나지 않던 CD8 효과기 세포 상의 ICOS 발현의 현저한 증가를 유도했다. 유사하게, 항-PD-L1 항체의 상이한 형식을 비교할 때, 처리된 동물 중 일부에서 가장 낮은 효과기 기능을 갖는 항체(mIgG1)는 이를 거의 나타내지 않고 효과기 기능을 갖는 항체(mIgG2a 및 래트IgG2b)와 비교될 때 효과기 CD8 및 CD4 세포 상에서 더 높은 ICOS 발현과 관련됨이 관찰되었다. 도 9를 참고한다.
효과기 CD8/CD4 T 세포 상의 ICOS 발현의 증가는 이들 세포를 항-ICOS 항체에 의한 고갈에 더 민감하게 만드는 효과를 가질 수 있다(예를 들어, STIM003 mIgG2a로의 마우스 처리 시). 효과기 CD8 및 CD4 T 세포에서 더 낮은 ICOS 유도를 나타내는 항체가 항-ICOS 항체와 조합으로 사용하기 위해 바람직할 수 있다. 본 연구로부터의 데이터는 항-PD-L1 효과기 양성 항체가 항-ICOS 효과기 양성 항체와의 조합에 특히 적합할 수 있음을 표시하여, 본원의 다른 실시예에 보고된 항-PDL1 mIgG2a와 STIM003 mIgG2a를 조합할 때 관찰된 항-종양 유효성을 반영한다.
1.11. 실시예 4: B 세포 림프종 동계 모델에서 생체내 단일 용량 항-ICOS 항체 단독치료법의 강력한 항-종양 유효성
이 실험은 단독치료법으로서 STIM003 mIgG2a의 항-종양 유효성을 확인시켜 준다. STIM003 mIgG2a의 짧은 노출 후 강력한 항-종양 유효성이 실증되었다.
1.11.1. 재료 및 방법
피하 A20 망상 세포 육종 모델(ATCC 번호 CRL-TIB-208)을 사용하여 BALB/c 마우스에서 유효성 연구를 수행했다. BALB/c 마우스는 6 내지 8주령 및 18 g 초과로 Charles River UK로부터 공급되었으며 특정 병원체가 없는 조건 하에 수용하였다. 총 5x10E5개 A20 세포(P20 미만 계대 수)를 마우스의 오른쪽 옆구리에 피하 주사했다. 아래 표에 나타낸 바와 같이 종양 세포 주사 후 제8일에 처리를 개시하였다. A20 세포를 TrypLE™ 익스프레스 효소(Thermofisher)를 사용하여 시험관내 계대배양하고, PBS로 2회 세척하여 10% 송아지 태아 혈청이 보충된 RPMI에 재현탁하였다. 종양 세포 주사시 세포 생활성은 85% 초과로 확인되었다. STIM003 mIgG2a는 60 μg(20 g 동물의 경우 3 mg/kg에 해당)의 단일 용량(SD) 또는 60 μg의 다중 용량(MD, 3주 동안 주 2회)으로 사용하였다. 두 가지 일정에 대하여 관찰된 항-종양 유효성을 식염수(MD, 3주 동안 주 2회)로 "처리된" 동물의 유효성과 비교했다. 항체를 0.9% 식염수 중 1 mg/ml로 복강내(IP) 투약했다. 동물 체중 및 종양 부피를 종양 세포 주사일로부터 주 3회 측정하였다. 종양 부피를 변형된 타원체 공식 1/2(길이 x 너비2)을 사용하여 계산하였다. 마우스의 종양이 평균 직경 12 mm에 도달할 때까지 또는 드물게 종양 궤양의 발생이 관찰될 때까지(복지) 마우스를 계속 연구했다.
[표 E23-1]
처리군.
1.11.2. 결과
STIM003 mIgG2a의 다중 및 단일 용량 둘 모두는 종점(제41일)에 종양 성장의 징후가 없는 동물 수에 의해 나타나듯이 강력하고 유의한 단독치료법 항-종양 유효성을 초래했다. SD는 동물 10마리 중 7마리에서 질환 치유를 초래한 반면, 다중 용량은 A20 B 세포 림프모세포가 주사된 동물 10마리 중 9마리를 치유하였다. 식염수 처리군의 모든 동물은 종양 크기로 인해 제40일까지 연구에서 제외해야 했다. 도 10을 참고한다.
인간적 종점 생존 통계를 GraphPad Prism V7.0을 사용하여 카플란-마이어 곡선(도 11)으로부터 계산하였다. 이 접근은 처리가 개선된 생존과 관련되는지를 결정하기 위해 사용하였다. 위험 비(만텔-핸젤(Mantel-Haenszel)) 값 및 이의 관련 P 값(로그 순위 만텔-콕스(Log-Rank Mantel-Cox))을 아래 표에 나타낸다.
[표 E23-1]
도 11 카플란-마이어 곡선에 상응하는 위험 비(만텔-핸젤) 값 및 이의 관련 P 값(로그 순위 만텔 콕스).
1.12. 실시예 5: CT-26 종양 보유 동물에서 항-ICOS 항체의 시간 및 용량 의존적 효과
이 실시예는 CT-26 종양을 보유하는 마우스의 면역 세포에 대한 항-ICOS 항체의 효과를 평가하는 약력학 연구 결과를 제시한다. 상이한 조직으로부터의 T 및 B 세포 하위유형을 STIM003 mIgG2a의 단일 용량 후 FACS로 분석했다.
1.12.1. 방법
CT-26 종양 보유 동물에 종양 세포 이식 후 제12일에 식염수 또는 STIM003 200 μg, 60 μg 또는 6 μg을 i.p. 투약했다. 종양 조직, 혈액, 종양 배액 림프절(TDLN) 및 비장을 처리 후 제1일, 제2일, 제3일, 제4일 및 제8일에 수확했다. 마우스 종양 해리 키트(Miltenyi Biotec)를 사용해서 종양을 해리하여 단일 세포 현탁액을 만들었다. 온건한 MACS 해리를 사용하여 비장 조직을 해리하고, RBC 용해 완충액을 사용하여 적혈구를 용해했다. 종양 배액 림프절을 기계적으로 분해하여 단일 세포 현탁액을 만들었다. 생성된 세포 현탁액을 조직에 따라 70 μM 또는 40 μM 필터를 통해 정화한 후, 세포를 RMPI 완전 배지에서 2회 세척하고 마지막으로 빙냉 FACS 완충액에 재현탁하였다. 전체 혈액을 혈장 튜브 내에 수집하고 적혈구를 RBC 용해 완충액을 사용하여 용해했으며, 세포를 RMPI 완전 배지에서 2회 세척하고 마지막으로 빙냉 FACS 완충액에 재현탁하였다. FACS 분석을 위해 모든 조직으로부터의 단일 세포 현탁액을 96 깊은 웰 플레이트에 분배했다. 세포를 라이브 데드 고정 가능한 황색 생활성 염료(Life Technologies)로 염색했다. 세포 현탁액을 항-CD16/CD32 mAb(eBioscience)와 함께 인큐베이션하고 모두 eBioscience Ltd.로부터 얻은 CD3(17A2), CD45(30-F11), CD4(RM4-5), CD8(53-6.7), CD25(PC61.5), ICOSL(HK5.3), B220(RA3-6B2), Ki-67(SolA15), CD107a(eBio1D4B), IFN-γ(XMG1.2), TNF-α(MP6-XT22), Foxp3(FJK-16s) 및 ICOS(7E.17G9)에 특이적인 FACS 항체로 염색하였다. FACS에 의한 사이토카인 판독을 위해 종양으로부터의 단일 세포 현탁액을 브레펠딘-A의 존재 하에 4시간 동안 24웰 플레이트에 플레이팅했다. 세포내 염색을 위해 샘플을 고정하고 투과화하고 특이적 항체로 염색했다. 샘플을 최종적으로 PBS에 재현탁하고 Attune 유세포 분석기(Invitrogen)에서 데이터를 획득하고 FlowJo V10 소프트웨어(Treestar)를 사용하여 분석했다.
결과가 아래에 제시되고 논의된다.
1.12.2. ICOS 발현이 CT26 모델의 종양내 T-reg 상에서 높음.
ICOS를 발현하는 종양 침윤 림프구(TIL)의 백분율을 비장, 혈액 및 TDLN의 면역 세포의 백분율과 비교했을 때, 본 발명자들은 CT-26 종양의 미세 환경에서 더 많은 면역 세포가 다른 조직에 비해 ICOS를 발현함을 실증했다. 더 중요하게는, 모든 조직 및 모든 시점에서 ICOS 양성 T-reg 세포의 백분율이 ICOS에 양성인 CD4 또는 CD8 효과기 T 세포의 백분율보다 높았다. 중요하게는, ICOS에 대한 dMFI(상대적 발현)도 유사한 발현 순위를 따랐으며 다른 TIL 하위유형에 비해 ICOS 발현에 대해 종양내 T-reg가 고도 양성이었다. 흥미롭게도 이 실험 시기 내에 ICOS+ TIL의 백분율에는 눈에 띄는 변화가 없었다. 비장 및 TDLN에서도 유사한 결과가 나타났다. 반면, 혈액에서는 ICOS 발현이 T 효과기 세포 상에서 상대적으로 안정적이지만 실험 과정 동안 T-reg 상에서는 증가했다. 전체적으로 데이터는 종양 미세환경에서 더 많은 세포가 ICOS를 발현하며 이들 양성 세포가 또한 이의 표면 상에 더 많은 ICOS 분자를 발현함을 실증했다. 더 중요하게는 TIL의 T reg는 ICOS에 대해 고도 양성이다. 도 12를 참고한다.
1.12.3. STIM003 투여에 반응한 종양내 T-reg 세포의 강력한 고갈
STIM003 mIgG2a 항체에 반응하여 TME에서 T-reg 세포(CD4+CD25+Foxp3)가 강력하고 빠르게 고갈되었다. T-reg는 다른 T 세포 하위세트와 비교하여 높은 ICOS 발현을 가지므로 효과기 기능을 갖는 항-ICOS 항체가 이들 세포를 우선적으로 고갈시킬 것으로 예상된다. 더 낮은 용량의 STIM003(20 g 동물의 경우 0.3 mg/kg에 상응하는 6 μg)으로는 T-reg가 지속적으로 고갈되었으며 제3일까지 대부분의 T-reg가 TME로부터 고갈되었다. 흥미롭게도, 제8일까지 T-reg 세포가 TME를 재증식한 후 식염수 처리 동물에서 관찰된 것보다 약간 높은 수준에 도달한다. 더 낮은 용량에서 T-reg 세포의 재증식은 Ki-67+ CD4 T 세포의 관찰된 증가에 의해 입증된 바와 같이 TME에서 증식하는 CD4 T 세포의 증가에 기인할 수 있다. 6 μg보다 높은 용량에서는 이 연구에서 분석된 마지막 시점(제8일)까지 전체 T Reg 고갈에 의해 나타난 바와 같이 TME에서 T-reg 세포가 장기간 고갈되었다. 반면 혈액에서는 모든 용량에서, T-reg 세포가 일시적으로 고갈되었다. 중요하게는 제8일까지 모든 처리 동물이 식염수 처리 동물과 비교하는 경우 유사한(또는 6 μg 용량의 경우 더 높은) 혈액 내 T-reg 세포 수준을 가졌다. 데이터를 도 13에 나타낸다. 특히 CTLA-4 항체 고갈에 대해 이전에 공개된 데이터와 유사하게 비장 또는 TDLN 조직의 T-reg 세포의 백분율에는 큰 변화가 없고, T-reg 세포가 이 기관에서 고갈로부터 보호될 수 있음을 제시하였다.
요약하면, TME에서 T-reg 세포의 강력한 고갈은 동물당 6 μg만큼 낮은 용량으로 CT-26 모델에서 달성되었다. 그러나 60 μg 용량은 STIM003 mIgG2a 주사 후 최대 8일까지 장기 고갈을 초래했다. 이는 더 높은 용량(200 μg)을 사용해도 개선되지 않았다.
1.12.4. STIM003 mIgG2a가 CD8:T Reg 및 CD4:T Reg 비를 증가시킴
T-eff:T-reg 비에 대한 STIM003의 효과를 도 14에 나타낸다.
STIM003 mIgG2a는 CD8:T-reg 비뿐만 아니라 CD4 eff:T-reg 비도 증가시켰다. 모든 치료 용량이 T-eff 대 T-reg 비의 증가와 관련되지만 중간 용량인 60 μg(20 g 동물의 경우 3 mg/kg에 해당)이 처리 후 제8일까지 가장 높은 비와 관련되었다.
흥미롭게도 6 μg 용량에서는 비가 제4일까지 높았지만 처리 후 제8일까지는 식염수 처리 동물의 비와 일치했다. 이는 치료 후 제8일까지 이 용량에 대해 관찰된 TReg의 재증식에 의해 설명될 수 있다. 반면, 60 또는 200 μg의 용량에서는 Teff 대 T-reg 비가 모든 시점에서 높게 유지되었다. 이는 이러한 용량에서의 Treg의 장기 고갈에 의해 설명된다. 특히, 더 높은 용량(200 μg)에서는 장기 Treg 고갈에도 불구하고 제8일까지 비가 중등도로만 개선되었다. 이는 고농도의 STIM003에서의 ICOSINT 효과기 세포의 일부 고갈에 의해 설명될 수 있다.
전체적으로, 데이터는 시험된 모든 용량에서 TReg 고갈 및 증가된 효과기:T reg 비를 실증하였다. 그러나 60 μg(약 3 mg/kg)의 용량은 T-reg의 장기 고갈뿐만 아니라 항-종양 면역 반응을 개시하기 위한 가장 유리한 면역 상황과 관련될 가장 높은 T-eff 대 T-reg 비 둘 모두를 달성했다. 흥미롭게도 유사한 패턴이 혈액에서 관찰되었으며, 60 μg의 중간 용량이 가장 높은 T-eff 대 T-reg 비와 관련되었다. 중요하게는, 혈액에서 비의 개선은 더 이른 시점(제3일 내지 제4일)에 관찰되었다.
1.12.5. STIM003에 반응한 효과기 세포의 활성화
종양 침윤 T 효과기 세포 상에서 CD107a의 표면 발현은 활성화되어 세포독성 활성을 발휘하는 세포에 대한 신뢰할 수 있는 마커로 이전에 확인되었다[39]. 본 연구에서는 STIM003이 T-reg를 고갈시키는 것 외에도 TME에서 효과기 T 세포의 세포독성 활성을 자극할 수 있음을 확인하기 위해 이 마커를 사용했다. 흥미롭게도 처리 후 제8일에, 모든 용량의 STIM003에서 CD4 및 CD8 효과기 T 세포 구획 둘 모두에서 CD107a의 표면 발현이 증가했다. 더욱이, CD4 및 CD8 T 세포 둘 모두의 표면 상에서 CD107a 발현의 이러한 상향조절은 200 μg 투약에서는 개선이 나타나지 않았으므로 동물에 60 μg이 투약될 때 정체되는 것으로 드러났다.
TME에서 효과기 세포의 활성화를 추가 실증하기 위해 CD4 및 CD8 TIL에 의한 사이토카인 방출을 FACS에 의해 분석했다. 예상대로, 그리고 본원의 이전 실시예에 제시된 시험관내 효능작용 데이터와 일치하게, 모든 용량에서 STIM003 mIgG2a는 효과기 CD4 및 CD8 T 세포에 의한 친염증성 사이토카인 IFN-γ 및 TNF-α 생산을 촉진했다. 친염증성 사이토카인 생산의 유도는 60 μg의 용량에서 높게 나타났다. 실제로, 60 μg의 STIM003은 CD4 T 세포에 의한 사이토카인 생산을 유의하게 증가시켰다. TME의 효과기 CD8 T 세포에 의한 친염증성 사이토카인 IFN-γ 및 TNF-α 생산에 대해서도 유사한 경향이 나타났다. 데이터를 도 15에 나타낸다.
요약하면, 모든 용량의 STIM003은 (1) 이의 표면 상에 탈과립 마커 CD107a의 존재 및 (2) T 세포에 의한 Th1 사이토카인(IFNγ 및 TNFα)의 생산에 의해 나타난 바와 같이 TME에서 T 세포 활성화를 초래했다. 이는 STIM003이 TME의 면역 환경에 강력하게 영향을 미치고 Treg 세포를 고갈시키고 T 효과기 세포의 사멸 활성을 자극하는 이중 역할을 한다는 것을 나타낸다.
1.12.6. 인간 용량 추정
마우스에서 나타난 전임상 유효성 데이터에 기반하여, 상응하는 생물학적 표면적(BSA)에 기반하여 인간 환자에게 적절한 임상 용량에 대한 초기 예측을 할 수 있다[40].
예를 들어, 마우스의 항-ICOS IgG 용량을 3 mg/kg(60 μg)으로 취하고, 참고문헌 [40]의 방법을 따르면, 인간에 대해 상응하는 용량은 0.25 mg/kg이다.
모스텔러(Mosteller) 공식을 사용하면 60 kg 및 1.70 m의 개체에 있어서 BSA는 1.68 m2이다. mg/kg 단위의 용량에 35.7(60/1.68)을 곱하면 고정 용량 15 mg이 된다. 80 kg 개인의 경우 상응하는 고정 용량은 20 mg일 것이다.
안전하고 효과적인 치료 요법을 결정하기 위해 임상 시험에서 인간 치료법에 맞게 용량을 조정할 수 있다.
1.13. 실시예 6: 종양 샘플로부터의 데이터의 생물정보학적 분석
본 발명에 따른 암의 한 표적 그룹은 비교적 높은 수준의 ICOS+ 면역억제 Treg와 관련된 암이다.
높은 함량의 Treg와 관련된 암 유형을 식별하기 위해 암 게놈 아틀라스(TCGA) 공개 데이터세트로부터 전사체 데이터를 얻고 ICOS 및 FOXP3 발현 수준에 대해 분석했다. TCGA는 여러 상이한 유형의 암에 대해 축적된 카탈로그화 유전체 게놈 및 전사체 데이터를 갖고 돌연변이, 카피 수 변이, mRNA 및 miRNA 유전자 발현, 그리고 DNA 메틸화와 함께 상당한 샘플 메타데이터룰 포함하는 대규모 연구이다.
유전자 세트 농축 분석(GSEA)을 하기와 같이 수행하였다. TCGA 컨소시엄의 일환으로 수집된 유전자 발현 RNA seq 데이터를 UCSC Xena 기능적 게노믹스 브라우저에서 log2(정규화_카운트+1)로 다운로드하였다. 비종양 조직 샘플을 데이터세트에서 제거하여 9732개 샘플로부터 20530개 유전자에 대한 데이터가 남았다. 특정된 유전자 세트 내의 유전자에 대한 농축 점수를 계산하는 [41]으로부터의 알고리즘 및 [42]의 이의 구현을 사용하여 유전자 수준 카운트를 각 샘플에 대한 유전자 세트 점수로 전치했다. 관심 유전자 세트를 ICOS 및 FOXP3 둘 모두를 함유하는 것으로 정의하였다. 샘플을 1차 질환별로 그룹화하고 각 그룹에 대한 ssGSEA 점수를 33개 1차 질환 그룹에 걸쳐 비교했다. 가장 높은 점수 중앙값을 나타낸 질환 그룹은 림프성 신생물 미만성 거대 b-세포 림프종, 가슴샘종, 두경부 편평 세포 암종인 것으로 나타났으나 미만성 거대 b-세포 림프종은 하위세트를 갖는 점수는 높고 나머지 점수는 그룹 중앙값보다 낮은 다중모드 분포를 나타내었다.
ICOS 및 FOXP3 발현에 대해 ssGSEA 점수가 가장 높은 것부터 가장 낮은 것까지 순위를 매기면 상위 15개 암 유형은 다음과 같았다:
DLBC(n=48)
림프성 신생물 미만성 거대 b-세포 림프종
THYM (n=120)
가슴샘종
HNSC(n = 522)
두경부 편평 세포 암종
TGCT(n = 156)
고환 생식 세포 종양
STAD(n = 415)
위부 샘암종
SKCM(n = 473)
표피 피부 흑색종
CESC(n = 305)
자궁경부 편평 세포 암종 및 자궁경내 샘암종
LUAD(n = 517)
폐 샘암종
LAML(n = 173)
급성 골수성 백혈병
ESCA(n = 185)
식도 암종
LUSC(n = 502)
폐 편평 세포 암종
READ(n = 95)
직장 샘암종
COAD(n = 288)
결장 샘암종
BRCA(n = 1104)
유방 침습성 암종
LIHC(n = 373)
간 간세포 암종
n은 TCGA 데이터세트에서 해당 암 유형에 대한 환자 샘플 수이다. 본원에 기재된 항-ICOS 항체를 이들 암 및 다른 암의 치료를 위해 사용할 수 있다.
상대적으로 높은 수준의 ICOS+ 면역억제 Treg와 관련되고 PD-L1을 추가 발현하는 암은 항-ICOS 항체 및 항-PD-L1 항체의 조합을 사용한 치료에 특히 잘 반응할 수 있다. 이러한 목적을 위해 적절한 치료 요법 및 항체는 상기 설명에서 이미 상세하게 기재되었다.
이전과 같이 TCGA 데이터세트를 사용하여 ICOS 및 FOXP3에 대한 농축 점수를 스피어만(Spearman) 순위 상관관계를 사용하여 PD-L1의 발현 수준과 상관시키고 1차 질환 적응증별로 그룹화했다. P-값을 각 그룹에 대해 계산하였으며 0.05의 p-값 (본페로니(Bonferroni) 다중 비교 보정 사용)이 통계적으로 유의한 것으로 간주되었다. ICOS/FOXP3와 PD-L1 발현 사이에서 가장 높은 상관관계를 갖는 질환 그룹은 다음과 같았다:
TGCT(n = 156)
고환 생식 세포 종양
COAD(n = 288)
결장 샘암종
READ(n = 95)
직장 샘암종
BLCA(n = 407)
방광 요로상피 암종
OV(n = 308)
난소 장액성 낭샘암종
BRCA(n = 1104)
유방 침습성 암종
SKCM(n = 473)
표피 피부 흑색종
CESC(n = 305)
자궁경부 편평 세포 암종 및 자궁경내 샘암종
STAD(n = 415)
위부 샘암종
LUAD(n = 517)
폐 샘암종
환자의 암이 ICOS+ 면역억제 Treg 및 PD-L1 발현과 관련됨을 결정하는 검정 후 환자를 치료를 위해 선택할 수 있다. 위와 같이 상관관계 점수가 높은 암 유형의 경우, ICOS+ 면역억제 Treg 및 PD-L1 발현 중 하나가 존재하는지(예를 들어, 역치 값 초과)를 결정하는 것으로 충분할 수 있다. PD-L1 면역조직화학 검정을 이러한 맥락에서 사용할 수 있다.
1.14. 실시예 7: 추가 항-ICOS 항체의 평가
CL-74570 및 CL-61091 항체 서열을 합성하고 HEK 세포에서 IgG1 형식으로 발현하였다.
BCT 상청액보다는 정제된 IgG1을 사용하기 위해 검정을 조정하도록 변형된, 이전에 기재된 바와 같은 HTRF 검정(예를 들어, 미국 특허 제9,957,323호의 실시예 6 참고)을 사용하여 이들 항체의 기능적 특성규명을 수행하였다. HEK 세포로부터 발현된 인간 IgG1 항체를 함유하는 상청액 5 μL를 BCT 상청액 대신 사용했으며, 이전과 같이 HTRF 완충액을 사용하여 총 부피를 웰당 최대 20 μl로 만들었다. 인간 IgG1 항체를 음성 대조군으로 사용했다. 두 항체 모두 공식 1을 사용하여 계산된 바와 같이 인간 및 마우스 ICOS로의 결합에 대해 5% 초과의 효과를 나타냈고, 따라서 이 검정에서 양성 반응을 보인 것을 확인하였다.
CHO-S 세포의 표면 상에 발현된 인간 및 마우스 ICOS에 결합하는 이들 항체의 능력을 미러볼(Mirrorball) 검정을 사용하여 추가 확인하였다. 이 검정에서, 항-ICOS IgG1을 함유하는 5 μl 상청액을 384개의 미러볼 블랙 플레이트(Corning)의 각 웰로 옮겼다. 검정 완충액(PBS + 1% BSA + 0.1% 아지드화나트륨)에 희석된 10 μl의 염소 항-인간 488(Jackson Immunoresearch)을 0.8 mg/ml의 농도로 모든 웰에 첨가하여 항-ICOS 항체의 결합을 검출했다.
양성 대조군 웰의 경우 검정 배지에서 2.2 μg/mL로 희석된 5 μL 참조 항체를 플레이트에 첨가했다. 음성 대조군 웰의 경우 검정 배지에서 2.2 μg/mL로 희석된 5 μl의 하이브리드 대조군 IgG1을 플레이트에 첨가했다. 10 μM의 DRAQ5(Thermoscientific)를 검정 완충액에 재현탁된 0.4 X 106개/ml 세포에 첨가하고 5 μl를 모든 웰에 첨가했다. 플레이트를 4℃에서 2시간 동안 인큐베이션하였다.
형광 강도를 미러볼 플레이트 판독기(TTP Labtech)를 사용하여 500 내지 700개의 단일 세포 집단으로부터의 Alexaflur 488(여기 493 nm, 방출 519 nm)을 측정하여 측정했다. 검정 신호를 평균 강도 중앙값(FL2) 으로 측정하였다.
총 결합을 2.2 μg/mL의 검정 농도에서 참조 항체를 사용하여 정의하였다. 비특이적 결합은 2.22 μg/mL의 검정 농도에서 하이브리드 대조군 hIgG1을 사용하여 정의하였다. 두 항체 모두 1% 초과 효과를 나타냈고, 이에 따라 이 검정에서 양성 반응을 보인 것을 확인하였다.
CL-74570 및 CL-61091 각각은 또한 유세포 분석에 의해 결정된 바와 같이 CHO-S 세포 상에서 발현된 인간 및 마우스 ICOS에 대한 결합을 실증했다. BCT 상청액보다는 정제된 IgG1을 사용하여 FACS 스크리닝을 수행하였다. 두 항체 모두 hICOS, mICOS 및 WT CHO 세포에 결합하는 음성 대조군의 기하 평균(geomean)의 평균보다 10배를 초과하는 높은 결합을 나타냈다.
[표 E26-1]
CL-74570 및 CL-61091의 기능적 특성규명.
1.15. 실시예 8: KY1044의 임상 시험 I/II상 공개 표지 연구.
단일 제제로 및 아테졸리주맙과의 조합으로, 이중 작용 메커니즘을 갖는 항-ICOS 항체인 KY1044의 I/II상 공개 표지 연구를 진행성 악성종양을 갖는 성인 환자에서 수행하였다. 참가자에는 고형 종양의 반응 평가 기준 버전 1.1(RECIST 1.1)에 의해 결정된 측정 가능 질환(측정 불가능 질환은 I상에서만 허용되었음)을 가졌던 진행성/전이성 악성종양을 갖는 환자가 포함되었고 국가 종합 암 네트워크(NCCN) 지침에 따라 이의 질환에 대해 임상적 이점을 부여하는 것으로 알려진 이용 가능한 치료법이 없거나 이들이 이러한 이용 가능한 옵션을 모두 소진한 경우, 적격이었다.
1.15.1. 방법
연구 아암:
KY1044 단독치료법 I상: KY1044 단독치료법 용량 증량
KY1044 및 아테졸리주맙 I상: KY1044 및 아테졸리주맙 조합 용량 증량
KY1044 단독치료법 II상: KY1044 단독치료법
KY1044 및 아테졸리주맙 II상: KY1044 및 아테졸리주맙 조합
I상: 진행성/전이성 악성종양 및 선호 적응증(비소세포 폐암(NSCLC), 두경부 편평 세포 암종(HNSCC), 간세포 암종(HCC), 흑색종, 자궁경부암, 식도암, 위암, 신장 세포 암종, 췌장암 및 삼중 음성 유방암)을 갖는 참가자.
II상 KY1044 단일 제제: 단일 제제로서 KY1044의 용량 증량 동안 항-종양 활성(완전 반응(CR), 부분적 반응(PR) 또는 PR에 적합하지 않은 종양 수축을 갖는 지속성 안정 질환(SD))의 징후가 나타난 적응증에서 진행성/전이성 악성종양을 갖는 참가자.
아테졸리주맙과 조합된 II상 KY1044: 아래 선택된 적응증 및/또는 I상에서 유망한 활성을 나타낸 적응증에서 진행성/전이성 악성종양을 갖는 참가자:
NSCLC(항-PD-(L)1 치료법 경험이 없고 사전 치료됨)
위(항-PD-(L)1 치료법 경험이 없고 사전 치료됨)
HNSCC(항-PD-(L)1 치료법 경험이 없고 사전 치료됨)
식도(항-PD-(L)1 치료법 경험이 없고 사전 치료됨)
자궁경부(항-PD-(L)1 치료법 경험이 없고 사전 치료됨)
아테졸리주맙과 조합된 KY1044를 사용하여 I상에서 항-종양 활성의 징후가 관찰된 적응증.
진행성/전이성 악성종양을 갖는 환자는 질환이 진행되거나 허용 불가능한 독성이 나타날 때까지 KY1044의 증량 용량을 단일 제제로 또는 항-PD-L1 항체인 아테졸리주맙 1200 mg과의 조합으로 3주마다 IV 주입에 의해 제공받았다. 용량 증량은 변형된 독성 확률 간격 설계에 의해 안내받았다. 1차 목적은 안전성, 내약성, 최대 내약 용량을 결정하는 것이었다. 내약된 코호트는 나중에 더 많은 대상체로 강화하였다. 유해 사례(AE)를 유해 사례에 대한 공통 용어 기준 버전 5(CTCAE v5)에 따라 분류하였으며, 유효성 측정을 고형 종양의 반응 평가 기준 버전 1.1(RECIST v1.1)에 따라 처음 16주 동안은 8주마다, 그 다음에는 12주마다 수행하였다.
1.15.2. 환자 포함 기준:
참가자는 하기 추가적 포함 기준을 모두 충족했어야 한다:
1. 사전 항-PD-(L)1 및/또는 항-PD-L1 지향 치료법에 기인한 임의의 독성이 치료법의 중단을 야기하지 않은 한, 항-PD-(L)1 및/또는 항-PD-L1 억제제를 사용한 시전 치료법은 허용되었다.
2. 동부 협력 종양 그룹(ECOG) 수행 상태 0 내지 1;
3. 기대 수명 12주 초과; 및
4. 치료 기관의 지침에 따라 생검에 적합하고 종양 생검의 후보가 되는 질환 부위.
1.15.3. 환자 제외 기준:
환자는 하기 제외 기준 중 하나라도 가져서는 안 된다:
1. 증상이 있는 중추 신경계(CNS) 전이 또는 국소 CNS 지향 치료법이 필요한 CNS 전이의 존재, 또는 연구 치료의 첫 번째 투약 전 2주 이내에 코르티코스테로이드 용량의 증가,
2. 다른 모노클로날 항체 및/또는 이의 부형제에 대한 중증 과민반응 이력;
3. 중화 항-아테졸리주맙 항체의 알려진 존재(이전에 아테졸리주맙으로 치료된 환자의 경우);
4. 범위를 벗어난 실험실 수치를 가짐: 크레아티닌, 빌리루빈, 알라닌 아미노트랜스퍼라제(ALT), 아스파테이트 아미노트랜스퍼라제(AST), 절대 호중구 카운트(ANC), 혈소판 카운트, 헤모글로빈;
5. 손상된 심장 기능 또는 임상적으로 유의한 심장 질환;
6. 알려진 인간 면역결핍 바이러스(HIV), 활성 B형 간염 바이러스(HBV) 또는 활성 C형 간염 바이러스(HCV) 감염;
7. 이 연구에서 치료되는 질환 이외의 악성 질환;
8. 연구자의 판단에 따라 안전성 문제, 임상 연구 절차의 준수 또는 연구 결과의 해석으로 인해 임상 연구 참가를 방해할 임의의 의학적 질병;
9. 활성 자가면역 질환 또는 기록된 자가면역 질환 이력;
10. 이전에 항-PD-(L)1 치료에 노출되었으나 피부 발진에 대해 적절한 치료를 받지 않았거나 내분비병증에 대한 대체 치료법을 받은 적이 없는 참가자는 제외해야 한다;
11. 약물 유도 폐렴의 이력 또는 현재 폐렴을 앓고 있는 참가자;
12. 전신 스테로이드 치료법 또는 임의의 면역억제 치료법. 국소, 흡입, 비강 및 안과용 스테로이드는 금지되지 않는다;
13. 연구 치료의 첫 번째 투약 4주 이내에 감염성 질환에 대한 약독화 생백신의 사용;
14. 연구 치료의 첫 번째 투약 4주 이내에 항-CTLA4, 항-PD-(L)1 치료;
15. T 세포 공동자극 또는 체크포인트 경로를 특이적으로 표적화하는 임의의 다른 항체 또는 약물과 조합된 항-CTLA4 항체를 사용한 이전 치료;
16. 사전 암 치료로 인한, 유해 사례에 대한 공통 용어 기준 버전 5(CTCAE v5)의 2등급 이상의 독성의 존재(CTCAE v5 3등급 이상인 경우 제외되는 탈모증, 말초 신경병증 및 내이독성 제외);
17. 뼈 통증 또는 중심적으로 통증이 있는 종양 덩어리의 치료와 같은 제한된 분야에 대한 완화적 방사선치료법을 제외하고, 연구 치료의 첫 번째 투약 2주 이내에 방사선치료법. 치료에 대한 반응 평가를 허용하기 위해, II상 부분에 등록된 참가자는 방사선 조사를 받지 않은 측정 가능 질환이 남아 있어야 한다; 및
18. 임산부 또는 수유부.
1.15.4. 투약
KY1044를 3주마다 0.8 mg, 2.4 mg, 8 mg, 24 mg, 80 mg, 또는 240 mg의 용량으로 단일 제제로 또는 아테졸리주맙 1200 mg과의 조합으로 투여하였다.
1.15.5. 중간 결과
103명의 환자가 연구에 등록하였다(단독치료법으로 6개 코호트에서 0.8 내지 240 mg 범위의 용량으로 38명의 환자, 및 아테졸리주맙과의 조합으로 5개 코호트에서 0.8 내지 80 mg 용량으로 65명). 환자의 63% 및 55%가 단일 제제 및 조합 코호트에서 각각 4회 이상의 사전 항암 치료법을 제공받았다.
모든 코호트는 치료 첫 21일 동안 용량 제한 독성(DLT) 없이 완료되었다. KY1044 단일 제제 코호트에서는 환자의 47.4%가 치료 관련 AE(TRAE)를 경험했으며 모두 1등급 또는 2등급이었다. 조합 코호트에서는 환자의 58%에서 TRAE가 관찰되었다. 대부분의 TRAE는 1등급 또는 2등급이었으며, 8% 미만의 환자에서 3등급 이상의 TRAE가 8회 발생했다. 주입 관련 반응, 발열 및 림프구감소증이 환자의 10% 이상에서 가장 흔히 발생하는 TRAE였다. 용량 중단을 야기하는 TRAE는 단일 제제 코호트에서는 1명의 환자 그리고 조합 코호트에서는 4명의 환자에서 발생했다. 단 1명의 환자만 조합과 관련된 것으로 간주되는 근염으로 인해 치료를 중단하였다.
69명의 환자로부터의 예비 KY1044 데이터는 약동학(PK) 모델 예측과 일치했다.
1.15.6. 결론:
이러한 결과는 KY1044가 단일 제제로서 그리고 아테졸리주맙과의 조합에서 내약성이 우수하였음을 나타낸다.
1.16. 실시예 9: I/II상 다기관 시험으로부터의 임상 시험 예비 약력학 마커.
종방향 혈액 샘플을 사용하여 KY1044 표적 참여 수준을 순환에서의 약력학적(PD) 특성과 상관시켰다.
1.16.1. 방법:
실시예 8에 기재된 I/II상 대상체가 KY1044의 효과를 3주마다 단독치료법으로(80 내지 240 mg), 및 3주마다 아테졸리주맙(1200 mg)과의 조합으로(0.8 내지 80 mg) 평가하기 위해 용량 증량 및 강화 코호트에 등록하였다. 말초 혈액 단핵 세포(PBMC), 혈장 및 종양 생검을 처음 3주기에 걸쳐 수집하여 표적 참여 및 KY1044 작용 방법(MoA)을 확인했다. 샘플 분석에는 다음이 포함되었다: 칩 세포계측에 의한 순환 T 세포 수용체 점유; PBMC 및 종양 샘플 처리-전 및 -후 전사체 분석; 및 순환 사이토카인(예를 들어, GM-CSF 및 TNFα)의 평가.
1.16.2. 중간 결과:
PBMC에서 평가된 바와 같이, T 세포에 대한 전체/연장된 ICOS 표적 참여가 8 내지 240 mg의 KY1044라는 더 높은 균일 용량을 제공받은 대상체에서 확인되었으며, 부분/일시적 포화가 더 낮은 균일 용량(0.8 내지 2.4 mg)을 제공받은 대상체에서 관찰되었다. ICOS 표적 참여는 환자 혈장 샘플로부터 칩 세포계측에 의해 측정된 바와 같이 CD4 기억 세포의 점유 백분율로 정량하였다. 표적 참여는 아테졸리주맙에 의해 영향받지 않았다. 도 16a 및 도 16b는 상이한 용량 수준을 제공받은 환자의 CD4 기억의 점유 백분율을 나타낸다.
KY1044 의존적 효능작용을 순환 사이토카인 수준을 측정하여 간접적으로 평가하였다. GM-CSF 및 TNFα 수준을 처음 3주기에 걸쳐 평가하였으며 기준선에서의 값과 비교하였다. 투약 후 GM-CSF의 일시적 유도는 0.8 mg 및 2.4 mg KY1044가 투약된 대상체에서 명백했지만, 8 mg 이상의 용량에서는 최소 유도가 관찰되었다. 도 17a를 참고한다. 투약 후 TNFα의 일시적 유도가 또한 0.8 및 2.4 mg 용량으로 KY1044가 투약된 대상체에서 명백했지만, 8 mg 이상의 용량에서는 최소 유도가 관찰되었다. 도 17b를 참고한다. 치료와 IFNγ 수준 간 관련은 관찰되지 않았다.
1.16.3. 결론:
부분적 수용체 점유를 초래하는 더 낮은 용량 KY1044(0.8 mg 및 2.4 mg)는 처리 후 더 강력한 GM-SCF 및 TNFα 신호를 유도했다. 따라서, 완전 미만의 수용체 점유를 달성하는 양으로 KY1044를 투약하는 것은 펄스형 사이토카인 반응을 생성하는 한 유리할 수 있으며, 더 높은 용량 수준과 비교하여 더 낮은 용량 수준의 반복 투여 시 사이토카인 수준의 투약 후 피크가 더 높다.
1.17. 실시예 10: 종방향 약력학 데이터가 예상되는 KY1044 작용 방법을 확인시켜줌.
종방향 샘플을 사용하여 종양 미세환경(TME)에서의 KY1044 표적 참여 수준을 약력학적(PD) 특성(예를 들어, 이중 작용 방법)과 상관시켰다.
1.17.1. 방법
실시예 8에 기재된 I/II상 대상체가 KY1044의 효과를 3주마다 단독치료법으로(0.8 내지 240 mg), 및 3주마다 아테졸리주맙(1200 mg)과의 조합으로(0.8 내지 80 mg) 평가하기 위해 용량 증량 및 강화 코호트에 등록하였다. 말초 혈액 단핵 세포(PBMC), 혈장 및 종양 생검을 처음 3주기에 걸쳐 수집하여 표적 참여 및 KY1044 작용 방법(MoA)을 확인했다. 샘플 분석에는 다음이 포함되었다: 종양 샘플의 면역조직화학(IHC)(ICOS, FOXP3 및 CD8) 및 순환 T 세포 면역프로파일링.
1.17.2. 중간 결과:
면역 세포 프로파일링은 일부 집단에서 변화를 나타냈으나 말초 ICOS+ 세포의 상당한 고갈은 존재하지 않았다. 대조적으로, 종양 생검에서 ICOS+/FOXP3+ 세포의 처리-전 및 -후 IHC 분석으로 TME에서 ICOS+ Treg의 KY1044 용량 의존적 감소 및 CD8+ T 세포의 유지를 확인하였으며, 가장 높은 종양내 ICOS+ Treg 고갈은 8 mg 이상 용량으로 관찰되었다. KY1044는 TME에서 모든 시험된 용량으로 ICOS+ Treg를 감소시키고 CD8 대 ICOS+ Treg의 비를 개선했으며, 8 mg 이상의 KY1044 용량을 제공받은 대상체에서 정체되었다. 이러한 결과는 KY1044 지향 효능작용이 부분적인 ICOS 수용체 점유를 달성한 용량인 더 낮은 용량(0.8 mg 및 2.4 mg)에서 가장 명백함을 나타낸다.
1.17.3. 결론:
종방향 PD 데이터는 KY1044 작용 방법, 즉 ICOS+ Treg 고갈 및 TME에서의 CD8+/ICOS+ Treg 비 증가뿐만 아니라 T 세포 공동 자극을 확인시켜 주었다. 실시예 9에 보고된 결과와 함께 이러한 결과는 KY1044의 이중 작용 방법을 뒷받침한다. 이론에 얽매이지 않으면서, KY1044의 더 낮은 용량(예를 들어, 8 mg 미만, 예를 들어, 2.4 mg 또는 0.8 mg)은 사이토카인 반응의 증가(친염증성 사이토카인 GM-CSF 및 TNFα의 증가)를 자극하고 동시에 ICOS+ Treg에서 종양내 감소를 매개하고 CD8 대 ICOS+ Treg의 비를 개선할 수 있다.
1.18. 실시예 11: I/II상 시험으로부터의 중간 결과: 조합 치료법에 대한 부분적 및 완전 반응.
1.18.1. 방법
사용된 방법을 실시예 8에서 인용한다.
1.18.2. 환자 포함 기준:
환자는 실시예 8에 기재된 바와 같이 등록하였다.
1.18.3. 중간 결과:
I/II상으로부터의 중간 결과는 항-종양 활성의 징후를 나타낸다. 부분적 반응(PR) 또는 완전 반응(CR)이 시험에서 관찰되었다. 문서화된 객관적 반응에는 다음이 포함된다:
삼중 음성 유방암(TNBC)의 CR
2.4 mg KY1044 + 1200 mg 아테졸리주맙
TNBC의 PR
2.4 mg KY1044 + 1200 mg 아테졸리주맙
두경부 편평 세포 암종의 PR
8 mg KY1044 + 1200 mg 아테졸리주맙
음경암의 PR
24 mg KY1044 + 1200 mg 아테졸리주맙
췌장암의 PR
0.8 mg KY1044 + 1200 mg 아테졸리주맙
이들 환자에 투여된 KY1044 용량(아테졸리주맙 1200 mg 포함)이 표시된다.
완전 반응을 RECIST 1.1 및 irRESIST에 따라 하기와 같이 정의하였다: 완전 반응(CR): 모든 표적 병변의 소실. 모든 병리학적 림프절(표적이건 비표적이건)이 10 mm 미만의 단축으로 감소해야 한다. CR: 모든 비표적 병변의 소실 및 (해당되는 경우) 종양 표지 수준의 정상화). 모든 림프절은 크기가 비병리적이어야 한다(10 mm 미만의 단축).
부분적 반응을 RECIST 1.1 및 iRESIST에 따라 하기와 같이 정의하였다: 부분적 반응(PR): 기준선 직경의 합을 참조하여 표적 병변 직경의 합의 적어도 30% 감소. 비-CR/비-PD: 하나 이상의 비표적 병변(들)의 지속 및/또는 (해당되는 경우) 정상 한계를 초과하는 종양 표지 수준의 유지.
1.18.4. 결론:
항-ICOS 항체 KY1044는 항-PD-L1 항체 치료법의 유효성을 촉진한다.
1.19. 실시예 12: I/II상 시험으로부터의 중간 결과: 치료 지속기간.
1.19.1. 방법
사용된 방법은 실시예 8에서 인용한다. 간략하게, 진행성/전이성 악성종양을 갖는 환자는 질환 진행 또는 허용 불가능한 독성까지 3주마다 IV 주사에 의해 단일 제제로서 또는 항-PD-L1 항체 아테졸리주맙 1200 mg과의 조합으로 KY1044의 용량을 받았다.
1.19.2. 환자 포함 기준:
환자를 실시예 8에 기재된 바와 같이 등록하였다.
1.19.3. 중간 결과:
모든 등록된 환자에 있어서 치료 지속기간 중앙값은 9주였다. 16주 이상 치료 지속기간이 각각 단일 제제 및 조합 코호트에서 24%(9/38) 및 27%(17/64) 환자에서 관찰되었다. 단독치료법 및 조합 치료법의 치료 지속기간에 대한 추가 데이터를 도 18a에 제공한다. 예를 들어, 도 18a는 단일 제제로서 KY1044를 사용하여 치료된 환자의 18%(7/38) 및 조합 치료법으로 치료된 환자의 10%(11/110)에서 20주 이상의 치료 지속기간이 관찰되었음을 나타낸다.
도 18b에서, 도 18a의 데이터를 각각 더 낮은(0.8 또는 2.4 mg) 또는 더 높은(8 mg 이상) 용량의 KY1044에 의해 얻은 부분적 또는 완전 포화(수용체 점유)에 따라 추가로 계층화한다. 단일 제제로서 더 낮은 용량(0.8 mg 또는 2.4 mg)의 KY1044를 제공받은 환자의 22%(2/9)에서 20주 이상의 치료 지속기간이 관찰되었으며, 이는 부분적인 수용체 점유를 초래했다. 단일 제제로서 더 높은 용량(8 mg 이상)의 KY1044를 제공받은 환자의 17%(5/29)에서 20주 이상의 치료 지속기간이 관찰되었다. 아테졸리주맙(1200 mg)과 조합된 더 낮은 용량(0.8 mg 또는 2.4 mg)의 KY1044를 제공받은 환자의 8%(4/49)에서 20주 이상의 치료 지속기간이 관찰되었다. 아테졸리주맙(1200 mg)과 조합된 더 높은 용량(8 mg 이상)의 KY1044를 제공받은 환자의 11%(7/61)에서 20주 이상의 치료 지속기간이 관찰되었다. 도 18b를 참고한다. 또한, 부분적 수용체 점유를 초래하는 용량(0.8 mg 또는 2.4 mg KY1044)으로 KY1044를 제공받은 환자의 10%(6/58)에서 20주 이상의 치료 지속기간이 관찰되었다. 완전 수용체 점유를 초래하는 용량(8 mg 이상)으로 KY1044를 제공받은 환자의 13%(12/90)에서 20주 이상의 치료 지속기간이 관찰되었다. 도 18c를 참고한다.
1.19.4. 결론:
이들 데이터는 특히 항-PD-L1 항체 치료법과의 조합으로, 항-ICOS 항체 KY1044의 더 낮은 용량(예를 들어, 0.8 mg, 2.4 mg)의 놀라운 유효성을 뒷받침한다.
1.20. 실시예 13: HNSCC 환자에서 아테졸리주맙과 조합된 KY1044
1.20.1. 방법
연구의 이 단계에서는 두경부 편평 세포 암종(HNSCC)을 갖는 환자에서 2상 코호트(2개 코호트, PD-L1 경험이 없고 사전 치료됨)를 개시한다. 각 코호트에 대략 40명의 환자를 등록할 것이다. 사용된 방법은 실시예 8에서 인용한다. 유효성 측정은 고형 종양의 반응 평가 기준 버전 1.1(RECIST 1.1)에 따라 처음 16주 동안은 8주마다, 그 다음에는 12주마다 수행할 것인 한편, 유해 사례(AE)를 유해 사례에 대한 공통 용어 기준 버전 5(CTCAE v5)에 따라 분류할 것이다.
1.20.2. 포함/제외 기준
주요 포함 기준: 항-PD-L1 치료법 경험이 없고 사전 치료된, 진행성 질환에 대한 1 내지 2선 사전 전신 치료법, 조직학적으로 문서화된 진행성/전이성 악성종양, RECIST 1.1에 의해 측정 가능한 질환, 생검이 적합한 질환 부위.
주요 제외 기준: CNS 전이, 활성 자가면역 질환, 유의한 심장 질환 및/또는 QT 연장, 스테로이드 치료법 또는 임의의 면역억제 치료법.
1.20.3. 중간 결과:
KY1044는 내약성이 우수했으며 HNSCC 치료에 대한 초기 활성 징후를 나타내었다. 연구의 1상 단계에서, 사전 5선 치료법(니볼루맙 포함)에서 진행된 HPV+ HNSCC를 갖는 59세 남성 환자는 부분적 반응(42% 종양 수축)을 경험했으며, 이는 이후에도 주기 26 제1일(C26D1) 상태를 유지하고 있고, 20개월 초과 치료 중이다(2022년 2월 10일 이후).
1.20.4. 결론:
두경부암의 종양내 Treg 상의 ICOS의 강력한 발현 문헌[(Sainson R et al. Cancer Immunol Res. 8, 2020:1568-82)]뿐만 아니라 치료 지속기간이 20개월을 초과하는 1명의 HNSCC 환자에서 유망한 임상 활성 문헌[(Patel MR et al. J Clinical Oncology 39, 2021(suppl 15; abstract 2624))은 HNSCC가 KY1044(SAR445256)에 대해 유리한 적응증임을 제시한다.
참고문헌
1.21. 서열
1.21.1.1. 항체 STIM001
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 367
VH 도메인 아미노산 서열: SEQ ID NO: 366
VH CDR1 아미노산 서열: SEQ ID NO: 363
VH CDR2 아미노산 서열: SEQ ID NO: 364
VH CDR3 아미노산 서열: SEQ ID NO: 365
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 374
VL 도메인 아미노산 서열: SEQ ID NO: 373
VL CDR1 아미노산 서열: SEQ ID NO: 370
VL CDR2 아미노산 서열: SEQ ID NO: 371
VL CDR3 아미노산 서열: SEQ ID NO: 372
1.21.1.2. 항체 STIM002
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 381
VH 도메인 아미노산 서열: SEQ ID NO: 380
VH CDR1 아미노산 서열: SEQ ID NO: 377
VH CDR2 아미노산 서열: SEQ ID NO: 378
VH CDR3 아미노산 서열: SEQ ID NO: 379
VL 도메인 뉴클레오티드 서열: 388
수정된 STIM002 VL 도메인 뉴클레오티드 서열: SEQ ID NO: 519
VL 도메인 아미노산 서열: SEQ ID NO: 387
VL CDR1 아미노산 서열: SEQ ID NO: 384
VL CDR2 아미노산 서열: SEQ ID NO: 385
VL CDR3 아미노산 서열: SEQ ID NO: 386
1.21.1.3. 항체 STIM002-B
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 395
VH 도메인 아미노산 서열: SEQ ID NO: 394
VH CDR1 아미노산 서열: SEQ ID NO: 391
VH CDR2 아미노산 서열: SEQ ID NO: 392
VH CDR3 아미노산 서열: SEQ ID NO: 393
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 402
VL 도메인 아미노산 서열: SEQ ID NO: 401
VL CDR1 아미노산 서열: SEQ ID NO: 398
VL CDR2 아미노산 서열: SEQ ID NO: 399
VL CDR3 아미노산 서열: SEQ ID NO: 400
1.21.1.4. 항체 STIM003
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 409
수정된 STIM003 VH 도메인 뉴클레오티드 서열: SEQ ID NO: 521
VH 도메인 아미노산 서열: SEQ ID NO: 408
VH CDR1 아미노산 서열: SEQ ID NO: 405
VH CDR2 아미노산 서열: SEQ ID NO: 406
VH CDR3 아미노산 서열: SEQ ID NO: 407
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 416
VL 도메인 아미노산 서열: SEQ ID NO: 415
VL CDR1 아미노산 서열: SEQ ID NO: 412
VL CDR2 아미노산 서열: SEQ ID NO: 413
VL CDR3 아미노산 서열: SEQ ID NO: 414
1.21.1.5. 항체 STIM004
VH 도메인 뉴클레오티드 서열:
VH 도메인 아미노산 서열:
VH CDR1 아미노산 서열: SEQ ID NO: 419
VH CDR2 아미노산 서열: SEQ ID NO: 420
VH CDR3 아미노산 서열: SEQ ID NO: 421
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 431
상기 VL 도메인 뉴클레오티드 서열에 의해 암호화되는 VL 도메인 아미노산 서열.
수정된 VL 도메인 뉴클레오티드 서열: SEQ ID NO: 430
수정된 VL 도메인 아미노산 서열: SEQ ID NO: 432
VL CDR1 아미노산 서열: SEQ ID NO: 426
VL CDR2 아미노산 서열: SEQ ID NO: 427
VL CDR3 아미노산 서열: SEQ ID NO: 428
1.21.1.6. 항체 STIM005
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 439
VH 도메인 아미노산 서열: SEQ ID NO: 438
VH CDR1 아미노산 서열: SEQ ID NO: 435
VH CDR2 아미노산 서열: SEQ ID NO: 436
VH CDR3 아미노산 서열: SEQ ID NO: 437
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 446
VL 도메인 아미노산 서열: SEQ ID NO: 445
VL CDR1 아미노산 서열: SEQ ID NO: 442
VL CDR2 아미노산 서열: SEQ ID NO: 443
VL CDR3 아미노산 서열: SEQ ID NO: 444
1.21.1.7. 항체 STIM006
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 453
VH 도메인 아미노산 서열: SEQ ID NO: 454
VH CDR1 아미노산 서열: SEQ ID NO: 449
VH CDR2 아미노산 서열: SEQ ID NO: 450
VH CDR3 아미노산 서열: SEQ ID NO: 451
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 460
VL 도메인 아미노산 서열: SEQ ID NO: 459
VL CDR1 아미노산 서열: SEQ ID NO: 456
VL CDR2 아미노산 서열: SEQ ID NO: 457
VL CDR3 아미노산 서열: SEQ ID NO: 458
1.21.1.8. 항체 STIM007
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 467
VH 도메인 아미노산 서열: SEQ ID NO: 466
VH CDR1 아미노산 서열: SEQ ID NO: 463
VH CDR2 아미노산 서열: SEQ ID NO: 464
VH CDR3 아미노산 서열: SEQ ID NO: 465
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 474
VL 도메인 아미노산 서열: SEQ ID NO: 473
VL CDR1 아미노산 서열: SEQ ID NO: 470
VL CDR2 아미노산 서열: SEQ ID NO: 471
VL CDR3 아미노산 서열: SEQ ID NO: 472
1.21.1.9. 항체 STIM008
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 481
VH 도메인 아미노산 서열: SEQ ID NO: 480
VH CDR1 아미노산 서열: SEQ ID NO: 477
VH CDR2 아미노산 서열: SEQ ID NO: 478
VH CDR3 아미노산 서열: SEQ ID NO: 479
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 488
VL 도메인 아미노산 서열: SEQ ID NO: 489
VL CDR1 아미노산 서열: SEQ ID NO: 484
VL CDR2 아미노산 서열: SEQ ID NO: 485
VL CDR3 아미노산 서열: SEQ ID NO: 486
1.21.1.10. 항체 STIM009
VH 도메인 뉴클레오티드 서열: SEQ ID NO: 495
VH 도메인 아미노산 서열: SEQ ID NO: 494
VH CDR1 아미노산 서열: SEQ ID NO: 491
VH CDR2 아미노산 서열: SEQ ID NO: 492
VH CDR3 아미노산 서열: SEQ ID NO: 493
VL 도메인 뉴클레오티드 서열: SEQ ID NO: 502
VL 도메인 아미노산 서열: SEQ ID NO: 501
VL CDR1 아미노산 서열: SEQ ID NO: 498
VL CDR2 아미노산 서열: SEQ ID NO: 499
VL CDR3 아미노산 서열: SEQ ID NO: 500
1.21.1.11. 표 S1. SEQ ID NO: 1 내지 342
1.21.1.12. 표 S2. SEQ ID NO: 343 내지 538
1.21.1.13. 표 S3. SEQ ID NO: 539 내지 562
1.21.1.14. 표 S4: 추가 클론으로부터 얻은 항체 중쇄 가변 영역의 서열
CDR은 IMGT에 따라 정의된다.
1.21.1.15. 표 S5: 추가 클론으로부터 얻은 항체 경쇄 가변 영역의 서열
CL-71642에서의 N 말단 E 및 5' 뉴클레오티드 부가를 볼드체로 나타낸다. 이들은 시퀀싱에서 회수되지 않았지만 도 6에 나타낸 바와 같이 관련 클론과의 비교에 의해 서열에 존재하는 것으로 결정되었다. CDR은 IMGT에 따라 정의된다.
SEQUENCE LISTING
<110> Kymab Limited
<120> Uses of Anti-ICOS Antibodies
<130> P211136WO/AH
<150> US 63/190,016
<151> 2021-05-18
<160> 620
<170> PatentIn version 3.5
<210> 1
<211> 290
<212> PRT
<213> Homo Sapiens
<400> 1
Met Arg Ile Phe Ala Val Phe Ile Phe Met Thr Tyr Trp His Leu Leu
1 5 10 15
Asn Ala Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val Glu Tyr
20 25 30
Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys Gln Leu
35 40 45
Asp Leu Ala Ala Leu Ile Val Tyr Trp Glu Met Glu Asp Lys Asn Ile
50 55 60
Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His Ser Ser
65 70 75 80
Tyr Arg Gln Arg Ala Arg Leu Leu Lys Asp Gln Leu Ser Leu Gly Asn
85 90 95
Ala Ala Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr
100 105 110
Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Val
115 120 125
Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu Val Val
130 135 140
Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu Gly Tyr
145 150 155 160
Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val Leu Ser
165 170 175
Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu Phe Asn
180 185 190
Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Thr Asn Glu Ile Phe Tyr
195 200 205
Cys Thr Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala Glu Leu
210 215 220
Val Ile Pro Glu Leu Pro Leu Ala His Pro Pro Asn Glu Arg Thr His
225 230 235 240
Leu Val Ile Leu Gly Ala Ile Leu Leu Cys Leu Gly Val Ala Leu Thr
245 250 255
Phe Ile Phe Arg Leu Arg Lys Gly Arg Met Met Asp Val Lys Lys Cys
260 265 270
Gly Ile Gln Asp Thr Asn Ser Lys Lys Gln Ser Asp Thr His Leu Glu
275 280 285
Glu Thr
290
<210> 2
<211> 241
<212> PRT
<213> Cynomologus
<400> 2
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Met Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val
20 25 30
Glu Tyr Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys
35 40 45
Gln Leu Asp Leu Thr Ser Leu Ile Val Tyr Trp Glu Met Glu Asp Lys
50 55 60
Asn Ile Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His
65 70 75 80
Ser Asn Tyr Arg Gln Arg Ala Gln Leu Leu Lys Asp Gln Leu Ser Leu
85 90 95
Gly Asn Ala Ala Leu Arg Ile Thr Asp Val Lys Leu Gln Asp Ala Gly
100 105 110
Val Tyr Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile
115 120 125
Thr Val Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu
130 135 140
Val Val Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu
145 150 155 160
Gly Tyr Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val
165 170 175
Leu Ser Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu
180 185 190
Leu Asn Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Ala Asn Glu Ile
195 200 205
Phe Tyr Cys Ile Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala
210 215 220
Glu Leu Val Ile Pro Glu Leu Pro Leu Ala Leu Pro Pro Asn Glu Arg
225 230 235 240
Thr
<210> 3
<211> 245
<212> PRT
<213> Homo Sapiens
<400> 3
Met Arg Ile Phe Ala Val Phe Ile Phe Met Thr Tyr Trp His Leu Leu
1 5 10 15
Asn Ala Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val Glu Tyr
20 25 30
Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys Gln Leu
35 40 45
Asp Leu Ala Ala Leu Ile Val Tyr Trp Glu Met Glu Asp Lys Asn Ile
50 55 60
Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His Ser Ser
65 70 75 80
Tyr Arg Gln Arg Ala Arg Leu Leu Lys Asp Gln Leu Ser Leu Gly Asn
85 90 95
Ala Ala Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr
100 105 110
Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Val
115 120 125
Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu Val Val
130 135 140
Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu Gly Tyr
145 150 155 160
Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val Leu Ser
165 170 175
Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu Phe Asn
180 185 190
Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Thr Asn Glu Ile Phe Tyr
195 200 205
Cys Thr Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala Glu Leu
210 215 220
Val Ile Pro Glu Leu Pro Leu Ala His Pro Pro Asn Glu Arg Thr His
225 230 235 240
His His His His His
245
<210> 4
<211> 475
<212> PRT
<213> Homo Sapiens
<400> 4
Met Arg Ile Phe Ala Val Phe Ile Phe Met Thr Tyr Trp His Leu Leu
1 5 10 15
Asn Ala Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val Glu Tyr
20 25 30
Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys Gln Leu
35 40 45
Asp Leu Ala Ala Leu Ile Val Tyr Trp Glu Met Glu Asp Lys Asn Ile
50 55 60
Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His Ser Ser
65 70 75 80
Tyr Arg Gln Arg Ala Arg Leu Leu Lys Asp Gln Leu Ser Leu Gly Asn
85 90 95
Ala Ala Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr
100 105 110
Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Val
115 120 125
Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu Val Val
130 135 140
Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu Gly Tyr
145 150 155 160
Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val Leu Ser
165 170 175
Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu Phe Asn
180 185 190
Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Thr Asn Glu Ile Phe Tyr
195 200 205
Cys Thr Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala Glu Leu
210 215 220
Val Ile Pro Glu Leu Pro Leu Ala His Pro Pro Asn Glu Arg Thr Ile
225 230 235 240
Glu Gly Arg Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
245 250 255
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
260 265 270
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
275 280 285
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
290 295 300
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
305 310 315 320
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
325 330 335
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
340 345 350
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
355 360 365
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
370 375 380
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
385 390 395 400
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
405 410 415
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
420 425 430
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
435 440 445
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
450 455 460
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 5
<211> 249
<212> PRT
<213> Cynomologus
<400> 5
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Met Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val
20 25 30
Glu Tyr Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys
35 40 45
Gln Leu Asp Leu Thr Ser Leu Ile Val Tyr Trp Glu Met Glu Asp Lys
50 55 60
Asn Ile Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His
65 70 75 80
Ser Asn Tyr Arg Gln Arg Ala Gln Leu Leu Lys Asp Gln Leu Ser Leu
85 90 95
Gly Asn Ala Ala Leu Arg Ile Thr Asp Val Lys Leu Gln Asp Ala Gly
100 105 110
Val Tyr Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile
115 120 125
Thr Val Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu
130 135 140
Val Val Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu
145 150 155 160
Gly Tyr Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val
165 170 175
Leu Ser Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu
180 185 190
Leu Asn Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Ala Asn Glu Ile
195 200 205
Phe Tyr Cys Ile Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala
210 215 220
Glu Leu Val Ile Pro Glu Leu Pro Leu Ala Leu Pro Pro Asn Glu Arg
225 230 235 240
Thr Asp Tyr Lys Asp Asp Asp Asp Lys
245
<210> 6
<211> 405
<212> PRT
<213> Homo Sapiens
<400> 6
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Leu Asp Ser Pro Asp Arg Pro Trp Asn Pro Pro Thr Phe
20 25 30
Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp Asn Ala Thr Phe Thr
35 40 45
Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val Leu Asn Trp Tyr Arg
50 55 60
Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala Ala Phe Pro Glu Asp
65 70 75 80
Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg Val Thr Gln Leu Pro
85 90 95
Asn Gly Arg Asp Phe His Met Ser Val Val Arg Ala Arg Arg Asn Asp
100 105 110
Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala Gln
115 120 125
Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val Thr Glu Arg Arg Ala
130 135 140
Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro Arg Pro Ala Gly Gln
145 150 155 160
Lys Leu Glu Asn Leu Tyr Phe Gln Gly Ile Glu Gly Arg Met Asp Glu
165 170 175
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
180 185 190
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
195 200 205
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
210 215 220
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
225 230 235 240
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
245 250 255
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
260 265 270
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
275 280 285
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
290 295 300
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
305 310 315 320
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
325 330 335
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
340 345 350
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
355 360 365
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
370 375 380
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
385 390 395 400
Leu Ser Leu Ser Pro
405
<210> 7
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 7
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 8
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 8
Ile Ser Trp Lys Ser Asn Ile Ile
1 5
<210> 9
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 9
Ala Arg Asp Ile Thr Gly Ser Gly Ser Tyr Gly Trp Phe Asp Pro
1 5 10 15
<210> 10
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 10
Asp Tyr Ala Met His
1 5
<210> 11
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 11
Gly Ile Ser Trp Lys Ser Asn Ile Ile Gly Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 12
<211> 13
<212> PRT
<213> Homo Sapiens
<400> 12
Asp Ile Thr Gly Ser Gly Ser Tyr Gly Trp Phe Asp Pro
1 5 10
<210> 13
<211> 122
<212> PRT
<213> Homo Sapiens
<400> 13
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Thr Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Lys Ser Asn Ile Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Ile Thr Gly Ser Gly Ser Tyr Gly Trp Phe Asp Pro Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 14
<211> 508
<212> DNA
<213> Homo Sapiens
<400> 14
caagaaaaag cttgccgcca ccatggagtt tgggctgagc tggattttcc ttttggctat 60
tttaaaaggt gtccagtgtg aagtacaatt ggtggagtcc gggggaggct tggtacagcc 120
tggcaggtcc ctgagactct cctgtgcagc ctctggattc acctttgatg attatgccat 180
gcactgggtc cgacaaactc cagggaaggg cctggagtgg gtctcaggta taagttggaa 240
gagtaatatc ataggctatg cggactctgt gaagggccga ttcaccatct ccagagacaa 300
cgccaagaac tccctgtatc tgcaaatgaa cagtctgaga gctgaggaca cggccttgta 360
ttattgtgca agagatataa cgggttcggg gagttatggc tggttcgacc cctggggcca 420
gggaaccctg gtcaccgtct cctcagccaa aacgacaccc ccatctgtct atccactggc 480
ccctgaatct gctaaaactc agcctccg 508
<210> 15
<211> 449
<212> PRT
<213> Homo Sapiens
<400> 15
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Thr Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Lys Ser Asn Ile Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Ile Thr Gly Ser Gly Ser Tyr Gly Trp Phe Asp Pro Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr
210 215 220
Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp
260 265 270
Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
435 440 445
Lys
<210> 16
<211> 1356
<212> DNA
<213> Homo Sapiens
<400> 16
gaagtgcagc tggtggaatc tggcggcgga ctggtgcagc ctggcagatc cctgagactg 60
tcttgtgccg cctccggctt caccttcgac gactacgcta tgcactgggt gcgacagacc 120
cctggcaagg gcctggaatg ggtgtccggc atctcctgga agtccaacat catcggctac 180
gccgactccg tgaagggccg gttcaccatc tcccgggaca acgccaagaa ctccctgtac 240
ctgcagatga acagcctgcg ggccgaggac accgccctgt actactgcgc cagagacatc 300
accggctccg gctcctacgg atggttcgat ccttggggcc agggcaccct cgtgaccgtg 360
tcctctgcca gcaccaaggg cccctctgtg ttccctctgg ccccttccag caagtccacc 420
tctggcggaa cagccgctct gggctgcctc gtgaaggact acttccccga gcctgtgacc 480
gtgtcctgga actctggcgc tctgaccagc ggagtgcaca ccttccctgc tgtgctgcag 540
tcctccggcc tgtactccct gtcctccgtc gtgaccgtgc cttccagctc tctgggcacc 600
cagacctaca tctgcaacgt gaaccacaag ccctccaaca ccaaggtgga caagaaggtg 660
gaacccaagt cctgcgacaa gacccacacc tgtccccctt gtcctgcccc tgaactgctg 720
ggcggacctt ccgtgttcct gttcccccca aagcccaagg acaccctgat gatctcccgg 780
acccccgaag tgacctgcgt ggtggtggat gtgtcccacg aggaccctga agtgaagttc 840
aattggtacg tggacggcgt ggaagtgcac aacgccaaga ccaagcctag agaggaacag 900
tacaactcca cctaccgggt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 960
ggcaaagagt acaagtgcaa ggtgtccaac aaggccctgc ctgcccccat cgaaaagacc 1020
atctccaagg ccaagggcca gccccgggaa ccccaggtgt acacactgcc ccctagcagg 1080
gacgagctga ccaagaacca ggtgtccctg acctgtctcg tgaaaggctt ctacccctcc 1140
gatatcgccg tggaatggga gtccaacggc cagcctgaga acaactacaa gaccaccccc 1200
cctgtgctgg actccgacgg ctcattcttc ctgtacagca agctgacagt ggacaagtcc 1260
cggtggcagc agggcaacgt gttctcctgc tccgtgatgc acgaggccct gcacaaccac 1320
tacacccaga agtccctgtc cctgagcccc ggcaag 1356
<210> 17
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 17
Gln Ser Ile Ser Ser Tyr
1 5
<210> 18
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 18
Val Ala Ser
1
<210> 19
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 19
Gln Gln Ser Tyr Ser Asn Pro Ile Thr
1 5
<210> 20
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 20
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 21
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 21
Val Ala Ser Ser Leu Gln Ser
1 5
<210> 22
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 22
Gln Gln Ser Tyr Ser Asn Pro Ile Thr
1 5
<210> 23
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 23
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Pro Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Ser Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Asn Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 24
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 24
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagcccct gatctatgtt gcatccagtt tgcaaagtgg ggtcccatca 180
agtttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta atccgatcac cttcggccaa 300
gggacacgac tggagatcaa a 321
<210> 25
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 25
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Pro Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Ser Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Asn Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 26
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 26
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagcccct gatctatgtt gcatccagtt tgcaaagtgg ggtcccatca 180
agtttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta atccgatcac cttcggccaa 300
gggacacgac tggagatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 27
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 27
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 28
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 28
Ile Ser Trp Ile Arg Thr Gly Ile
1 5
<210> 29
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 29
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
1 5 10 15
<210> 30
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 30
Asp Tyr Ala Met His
1 5
<210> 31
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 31
Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 32
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 32
Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
1 5 10
<210> 33
<211> 123
<212> PRT
<213> Homo Sapiens
<400> 33
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 34
<211> 482
<212> DNA
<213> Homo Sapiens
<400> 34
aagcttgccg ccaccatgga gtttgggctg agctggattt tccttttggc tattttaaaa 60
ggtgtccagt gtgaagtgca gctggtggag tctgggggag gcttggtgca gcctggcagg 120
tccctgagac tctcctgtgc agcctctgga ttcacctttg atgattatgc catgcactgg 180
gtccggcaag ttccagggaa gggcctggaa tgggtctcag gcattagttg gattcgtact 240
ggcataggct atgcggactc tgtgaagggc cgattcacca ttttcagaga caacgccaag 300
aattccctgt atctgcaaat gaacagtctg agagctgagg acacggcctt gtattactgt 360
gcaaaagata tgaagggttc ggggacttat ggggggtggt tcgacacctg gggccaggga 420
accctggtca ccgtctcctc agccaaaaca acagccccat cggtctatcc actggcccct 480
gc 482
<210> 35
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 35
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
195 200 205
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
210 215 220
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
260 265 270
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
435 440 445
Gly Lys
450
<210> 36
<211> 1359
<212> DNA
<213> Homo Sapiens
<400> 36
gaagtgcagc tggtggaatc tggcggcgga ctggtgcagc ctggcagatc cctgagactg 60
tcttgtgccg cctccggctt caccttcgac gactacgcta tgcactgggt gcgacaggtg 120
ccaggcaagg gcctggaatg ggtgtccggc atctcttgga tccggaccgg catcggctac 180
gccgactctg tgaagggccg gttcaccatc ttccgggaca acgccaagaa ctccctgtac 240
ctgcagatga acagcctgcg ggccgaggac accgccctgt actactgcgc caaggacatg 300
aagggctccg gcacctacgg cggatggttc gatacttggg gccagggcac cctcgtgacc 360
gtgtcctctg ccagcaccaa gggcccctct gtgttccctc tggccccttc cagcaagtcc 420
acctctggcg gaacagccgc tctgggctgc ctcgtgaagg actacttccc cgagcctgtg 480
accgtgtcct ggaactctgg cgctctgacc agcggagtgc acaccttccc tgctgtgctg 540
cagtcctccg gcctgtactc cctgtcctcc gtcgtgaccg tgccttccag ctctctgggc 600
acccagacct acatctgcaa cgtgaaccac aagccctcca acaccaaggt ggacaagaag 660
gtggaaccca agtcctgcga caagacccac acctgtcccc cttgtcctgc ccctgaactg 720
ctgggcggac cttccgtgtt cctgttcccc ccaaagccca aggacaccct gatgatctcc 780
cggacccccg aagtgacctg cgtggtggtg gatgtgtccc acgaggaccc tgaagtgaag 840
ttcaattggt acgtggacgg cgtggaagtg cacaacgcca agaccaagcc tagagaggaa 900
cagtacaact ccacctaccg ggtggtgtcc gtgctgaccg tgctgcacca ggattggctg 960
aacggcaaag agtacaagtg caaggtgtcc aacaaggccc tgcctgcccc catcgaaaag 1020
accatctcca aggccaaggg ccagccccgg gaaccccagg tgtacacact gccccctagc 1080
agggacgagc tgaccaagaa ccaggtgtcc ctgacctgtc tcgtgaaagg cttctacccc 1140
tccgatatcg ccgtggaatg ggagtccaac ggccagcctg agaacaacta caagaccacc 1200
ccccctgtgc tggactccga cggctcattc ttcctgtaca gcaagctgac agtggacaag 1260
tcccggtggc agcagggcaa cgtgttctcc tgctccgtga tgcacgaggc cctgcacaac 1320
cactacaccc agaagtccct gtccctgagc cccggcaag 1359
<210> 37
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 37
Gln Ser Ile Ser Ser Tyr
1 5
<210> 38
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 38
Val Ala Ser
1
<210> 39
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 39
Gln Gln Ser Tyr Ser Thr Pro Ile Thr
1 5
<210> 40
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 40
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 41
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 41
Val Ala Ser Ser Leu Gln Ser
1 5
<210> 42
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 42
Gln Gln Ser Tyr Ser Thr Pro Ile Thr
1 5
<210> 43
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 43
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 44
<211> 418
<212> DNA
<213> Homo Sapiens
<400> 44
aaagcttgcc gccaccatga ggctccctgc tcagcttctg gggctcctgc tactctggct 60
ccgaggtgcc agatgtgaca tccagatgac ccagtctcca tcctccctgt ctgcatctgt 120
aggagacaga gtcaccatca cttgccgggc aagtcagagc attagcagct atttaaattg 180
gtatcagcag aaaccaggga aagcccctaa actcctgatc tatgttgcat ccagtttgca 240
aagtggggtc ccatcaaggt tcagtggcag tggatctggg acagatttca ctctcactat 300
cagcagtctg caacctgaag attttgcaac ttactactgt caacagagtt acagtacccc 360
gatcaccttc ggccaaggga cacgtctgga gatcaaacgt acggatgctg caccaact 418
<210> 45
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 45
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 46
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 46
gacatccaga tgacccagtc cccctccagc ctgtctgctt ccgtgggcga cagagtgacc 60
atcacctgtc gggcctccca gtccatctcc tcctacctga actggtatca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgtg gccagctctc tgcagtccgg cgtgccctct 180
agattctccg gctctggctc tggcaccgac tttaccctga ccatcagctc cctgcagccc 240
gaggacttcg ccacctacta ctgccagcag tcctactcca cccctatcac cttcggccag 300
ggcacccggc tggaaatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 47
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 47
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
195 200 205
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
210 215 220
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Leu Ala Gly Ala
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
260 265 270
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
435 440 445
Gly Lys
450
<210> 48
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 48
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
195 200 205
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
210 215 220
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Leu Ala Gly Ala
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
260 265 270
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
435 440 445
Gly Lys
450
<210> 49
<211> 448
<212> PRT
<213> Homo Sapiens
<400> 49
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
195 200 205
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
210 215 220
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Val Ala Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
260 265 270
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 50
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 50
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 51
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 51
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Phe Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 52
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 52
Gly Phe Thr Phe Ser Ser Tyr Trp
1 5
<210> 53
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 53
Ile Lys Glu Asp Gly Ser Glu Lys
1 5
<210> 54
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 54
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn
1 5 10
<210> 55
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 55
Ser Tyr Trp Met Ser
1 5
<210> 56
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 56
Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val Lys
1 5 10 15
Gly
<210> 57
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 57
Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn
1 5 10
<210> 58
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 58
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 59
<211> 358
<212> DNA
<213> Homo Sapiens
<400> 59
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac atcaaagaag atggaagtga gaaatactat 180
gtcgactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagaaatcga 300
ctctacagtg acttccttga caactggggc cagggaaccc tggtcaccgt ctcctcag 358
<210> 60
<211> 449
<212> PRT
<213> Homo Sapiens
<400> 60
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 61
<211> 1347
<212> DNA
<213> Homo Sapiens
<400> 61
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac atcaaagaag atggaagtga gaaatactat 180
gtcgactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagaaatcga 300
ctctacagtg acttccttga caactggggc cagggaaccc tggtcaccgt ctcctcagcc 360
agcaccaagg gcccctctgt gttccctctg gccccttcca gcaagtccac ctctggcgga 420
acagccgctc tgggctgcct cgtgaaggac tacttccccg agcctgtgac cgtgtcctgg 480
aactctggcg ctctgaccag cggagtgcac accttccctg ctgtgctgca gtcctccggc 540
ctgtactccc tgtcctccgt cgtgaccgtg ccttccagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gccctccaac accaaggtgg acaagaaggt ggaacccaag 660
tcctgcgaca agacccacac ctgtccccct tgtcctgccc ctgaactgct gggcggacct 720
tccgtgttcc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 780
gtgacctgcg tggtggtgga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaactcc 900
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 960
tacaagtgca aggtgtccaa caaggccctg cctgccccca tcgaaaagac catctccaag 1020
gccaagggcc agccccggga accccaggtg tacacactgc cccctagcag ggacgagctg 1080
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1140
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gactccgacg gctcattctt cctgtacagc aagctgacag tggacaagtc ccggtggcag 1260
cagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctgt ccctgagccc cggcaag 1347
<210> 62
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 62
Gln Gly Val Ser Ser Trp
1 5
<210> 63
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 63
Gly Ala Ser
1
<210> 64
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 64
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 65
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 65
Arg Ala Ser Gln Gly Val Ser Ser Trp Leu Ala
1 5 10
<210> 66
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 66
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 67
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 67
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 68
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 68
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 69
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 69
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agctggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 70
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 70
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 71
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 71
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agctggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 72
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 72
Gly Phe Thr Phe Ser Ser Tyr Trp
1 5
<210> 73
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 73
Ile Lys Glu Asp Gly Ser Glu Lys
1 5
<210> 74
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 74
Ala Arg Val Arg Leu Tyr Ser Asp Phe Leu Asp Tyr
1 5 10
<210> 75
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 75
Ser Tyr Trp Met Ser
1 5
<210> 76
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 76
Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Leu Lys
1 5 10 15
Gly
<210> 77
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 77
Val Arg Leu Tyr Ser Asp Phe Leu Asp Tyr
1 5 10
<210> 78
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 78
Glu Val Gln Leu Val Asp Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Leu
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Val Arg Leu Tyr Ser Asp Phe Leu Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 79
<211> 358
<212> DNA
<213> Homo Sapiens
<400> 79
gaggtgcagc tggtggactc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccaggaaagg ggctggagtg ggtggccaac ataaaagaag atggaagtga gaaatactat 180
gtagactctt tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagagttcga 300
ctctacagtg acttccttga ctactggggc cagggaaccc tggtcaccgt ctcctcag 358
<210> 80
<211> 449
<212> PRT
<213> Homo Sapiens
<400> 80
Glu Val Gln Leu Val Asp Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Leu
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Val Arg Leu Tyr Ser Asp Phe Leu Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 81
<211> 1347
<212> DNA
<213> Homo Sapiens
<400> 81
gaggtgcagc tggtggactc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccaggaaagg ggctggagtg ggtggccaac ataaaagaag atggaagtga gaaatactat 180
gtagactctt tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagagttcga 300
ctctacagtg acttccttga ctactggggc cagggaaccc tggtcaccgt ctcctcagcc 360
agcaccaagg gcccctctgt gttccctctg gccccttcca gcaagtccac ctctggcgga 420
acagccgctc tgggctgcct cgtgaaggac tacttccccg agcctgtgac cgtgtcctgg 480
aactctggcg ctctgaccag cggagtgcac accttccctg ctgtgctgca gtcctccggc 540
ctgtactccc tgtcctccgt cgtgaccgtg ccttccagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gccctccaac accaaggtgg acaagaaggt ggaacccaag 660
tcctgcgaca agacccacac ctgtccccct tgtcctgccc ctgaactgct gggcggacct 720
tccgtgttcc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 780
gtgacctgcg tggtggtgga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaactcc 900
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 960
tacaagtgca aggtgtccaa caaggccctg cctgccccca tcgaaaagac catctccaag 1020
gccaagggcc agccccggga accccaggtg tacacactgc cccctagcag ggacgagctg 1080
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1140
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gactccgacg gctcattctt cctgtacagc aagctgacag tggacaagtc ccggtggcag 1260
cagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctgt ccctgagccc cggcaag 1347
<210> 82
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 82
Gln Gly Val Ser Ser Trp
1 5
<210> 83
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 83
Gly Ala Ser
1
<210> 84
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 84
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 85
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 85
Arg Ala Ser Gln Gly Val Ser Ser Trp Leu Ala
1 5 10
<210> 86
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 86
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 87
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 87
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 88
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 88
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Ser Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 89
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 89
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agttggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcctccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca gcatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 90
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 90
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Ser Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 91
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 91
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agttggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcctccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca gcatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 92
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 92
Gly Gly Ser Ile Ile Ser Ser Asp Trp
1 5
<210> 93
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 93
Ile Phe His Ser Gly Arg Thr
1 5
<210> 94
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 94
Ala Arg Asp Gly Ser Gly Ser Tyr
1 5
<210> 95
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 95
Ser Ser Asp Trp Trp Asn
1 5
<210> 96
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 96
Glu Ile Phe His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 97
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 97
Asp Gly Ser Gly Ser Tyr
1 5
<210> 98
<211> 115
<212> PRT
<213> Homo Sapiens
<400> 98
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Ile Val Ser Gly Gly Ser Ile Ile Ser Ser
20 25 30
Asp Trp Trp Asn Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Phe His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Ile Asp Lys Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Ser Gly Ser Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 99
<211> 346
<212> DNA
<213> Homo Sapiens
<400> 99
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggggac cctgtccctc 60
acctgcattg tctctggtgg ctccatcatc agtagtgact ggtggaattg ggtccgccag 120
cccccaggga aggggctgga gtggattgga gaaatctttc atagtgggag gaccaactac 180
aacccgtccc tcaagagtcg agtcaccata tcaatagaca agtccaagaa tcagttctcc 240
ctgaggctga gctctgtgac cgccgcggac acggccgtgt attactgtgc gagagatggt 300
tcggggagtt actggggcca gggaaccctg gtcaccgtct cctcag 346
<210> 100
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 100
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Ile Val Ser Gly Gly Ser Ile Ile Ser Ser
20 25 30
Asp Trp Trp Asn Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Phe His Ser Gly Arg Thr Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Ile Asp Lys Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Ser Gly Ser Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 101
<211> 1335
<212> DNA
<213> Homo Sapiens
<400> 101
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggggac cctgtccctc 60
acctgcattg tctctggtgg ctccatcatc agtagtgact ggtggaattg ggtccgccag 120
cccccaggga aggggctgga gtggattgga gaaatctttc atagtgggag gaccaactac 180
aacccgtccc tcaagagtcg agtcaccata tcaatagaca agtccaagaa tcagttctcc 240
ctgaggctga gctctgtgac cgccgcggac acggccgtgt attactgtgc gagagatggt 300
tcggggagtt actggggcca gggaaccctg gtcaccgtct cctcagccag caccaagggc 360
ccctctgtgt tccctctggc cccttccagc aagtccacct ctggcggaac agccgctctg 420
ggctgcctcg tgaaggacta cttccccgag cctgtgaccg tgtcctggaa ctctggcgct 480
ctgaccagcg gagtgcacac cttccctgct gtgctgcagt cctccggcct gtactccctg 540
tcctccgtcg tgaccgtgcc ttccagctct ctgggcaccc agacctacat ctgcaacgtg 600
aaccacaagc cctccaacac caaggtggac aagaaggtgg aacccaagtc ctgcgacaag 660
acccacacct gtcccccttg tcctgcccct gaactgctgg gcggaccttc cgtgttcctg 720
ttccccccaa agcccaagga caccctgatg atctcccgga cccccgaagt gacctgcgtg 780
gtggtggatg tgtcccacga ggaccctgaa gtgaagttca attggtacgt ggacggcgtg 840
gaagtgcaca acgccaagac caagcctaga gaggaacagt acaactccac ctaccgggtg 900
gtgtccgtgc tgaccgtgct gcaccaggat tggctgaacg gcaaagagta caagtgcaag 960
gtgtccaaca aggccctgcc tgcccccatc gaaaagacca tctccaaggc caagggccag 1020
ccccgggaac cccaggtgta cacactgccc cctagcaggg acgagctgac caagaaccag 1080
gtgtccctga cctgtctcgt gaaaggcttc tacccctccg atatcgccgt ggaatgggag 1140
tccaacggcc agcctgagaa caactacaag accacccccc ctgtgctgga ctccgacggc 1200
tcattcttcc tgtacagcaa gctgacagtg gacaagtccc ggtggcagca gggcaacgtg 1260
ttctcctgct ccgtgatgca cgaggccctg cacaaccact acacccagaa gtccctgtcc 1320
ctgagccccg gcaag 1335
<210> 102
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 102
Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr
1 5 10
<210> 103
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 103
Trp Ala Ser
1
<210> 104
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 104
Gln Gln Tyr Tyr Ser Asn Arg Ser
1 5
<210> 105
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 105
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 106
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 106
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 107
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 107
Gln Gln Tyr Tyr Ser Asn Arg Ser
1 5
<210> 108
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 108
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Ser Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Thr Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asn Arg Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 109
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 109
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ttacttagct 120
tggtaccagc agaaatcagg acagcctcct aagttgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcagactga agatgtggca gtttattact gtcagcaata ttatagtaat 300
cgcagttttg gccaggggac caagctggag atcaaac 337
<210> 110
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 110
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Ser Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Thr Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asn Arg Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 111
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 111
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ttacttagct 120
tggtaccagc agaaatcagg acagcctcct aagttgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcagactga agatgtggca gtttattact gtcagcaata ttatagtaat 300
cgcagttttg gccaggggac caagctggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 112
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 112
Gly Phe Thr Phe Ser Ser Tyr Trp
1 5
<210> 113
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 113
Ile Lys Glu Asp Gly Ser Glu Lys
1 5
<210> 114
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 114
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn
1 5 10
<210> 115
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 115
Ser Tyr Trp Met Ser
1 5
<210> 116
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 116
Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val Lys
1 5 10 15
Gly
<210> 117
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 117
Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn
1 5 10
<210> 118
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 118
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 119
<211> 358
<212> DNA
<213> Homo Sapiens
<400> 119
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac atcaaagaag atggaagtga gaaatactat 180
gtcgactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagaaatcga 300
ctctacagtg acttccttga caactggggc cagggaaccc tggtcaccgt ctcctcag 358
<210> 120
<211> 449
<212> PRT
<213> Homo Sapiens
<400> 120
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Glu Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ser Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Arg Leu Tyr Ser Asp Phe Leu Asp Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 121
<211> 1347
<212> DNA
<213> Homo Sapiens
<400> 121
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagt agctattgga tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac atcaaagaag atggaagtga gaaatactat 180
gtcgactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acgtctgtgt attactgtgc gagaaatcga 300
ctctacagtg acttccttga caactggggc cagggaaccc tggtcaccgt ctcctcagcc 360
agcaccaagg gcccctctgt gttccctctg gccccttcca gcaagtccac ctctggcgga 420
acagccgctc tgggctgcct cgtgaaggac tacttccccg agcctgtgac cgtgtcctgg 480
aactctggcg ctctgaccag cggagtgcac accttccctg ctgtgctgca gtcctccggc 540
ctgtactccc tgtcctccgt cgtgaccgtg ccttccagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gccctccaac accaaggtgg acaagaaggt ggaacccaag 660
tcctgcgaca agacccacac ctgtccccct tgtcctgccc ctgaactgct gggcggacct 720
tccgtgttcc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 780
gtgacctgcg tggtggtgga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaactcc 900
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 960
tacaagtgca aggtgtccaa caaggccctg cctgccccca tcgaaaagac catctccaag 1020
gccaagggcc agccccggga accccaggtg tacacactgc cccctagcag ggacgagctg 1080
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1140
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gactccgacg gctcattctt cctgtacagc aagctgacag tggacaagtc ccggtggcag 1260
cagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctgt ccctgagccc cggcaag 1347
<210> 122
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 122
Gln Gly Val Ser Ser Trp
1 5
<210> 123
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 123
Gly Ala Ser
1
<210> 124
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 124
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 125
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 125
Arg Ala Ser Gln Gly Val Ser Ser Trp Leu Ala
1 5 10
<210> 126
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 126
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 127
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 127
Gln Gln Ala Asn Ser Ile Pro Phe Thr
1 5
<210> 128
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 128
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 129
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 129
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agctggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 130
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 130
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Val Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Ile Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Ile Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 131
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 131
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtgttagc agctggttag cctggtatca gcagaaatca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tgcaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagag ttcattctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagta tcccattcac tttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 132
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 132
Gly Phe Thr Phe Arg Ile Tyr Gly
1 5
<210> 133
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 133
Ile Trp Tyr Asp Gly Ser Asn Lys
1 5
<210> 134
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 134
Ala Arg Asp Met Asp Tyr Phe Gly Met Asp Val
1 5 10
<210> 135
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 135
Ile Tyr Gly Met His
1 5
<210> 136
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 136
Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 137
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 137
Asp Met Asp Tyr Phe Gly Met Asp Val
1 5
<210> 138
<211> 118
<212> PRT
<213> Homo Sapiens
<400> 138
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ile Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Asp Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Met Asp Tyr Phe Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 139
<211> 355
<212> DNA
<213> Homo Sapiens
<400> 139
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttccgt atttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa taaatactat 180
gctgactccg tgaagggccg attcaccatc tccagagaca attccgacaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagatatg 300
gactacttcg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc ctcag 355
<210> 140
<211> 448
<212> PRT
<213> Homo Sapiens
<400> 140
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ile Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Asp Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Met Asp Tyr Phe Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 141
<211> 1344
<212> DNA
<213> Homo Sapiens
<400> 141
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttccgt atttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa taaatactat 180
gctgactccg tgaagggccg attcaccatc tccagagaca attccgacaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagatatg 300
gactacttcg gtatggacgt ctggggccaa gggaccacgg tcaccgtctc ctcagccagc 360
accaagggcc cctctgtgtt ccctctggcc ccttccagca agtccacctc tggcggaaca 420
gccgctctgg gctgcctcgt gaaggactac ttccccgagc ctgtgaccgt gtcctggaac 480
tctggcgctc tgaccagcgg agtgcacacc ttccctgctg tgctgcagtc ctccggcctg 540
tactccctgt cctccgtcgt gaccgtgcct tccagctctc tgggcaccca gacctacatc 600
tgcaacgtga accacaagcc ctccaacacc aaggtggaca agaaggtgga acccaagtcc 660
tgcgacaaga cccacacctg tcccccttgt cctgcccctg aactgctggg cggaccttcc 720
gtgttcctgt tccccccaaa gcccaaggac accctgatga tctcccggac ccccgaagtg 780
acctgcgtgg tggtggatgt gtcccacgag gaccctgaag tgaagttcaa ttggtacgtg 840
gacggcgtgg aagtgcacaa cgccaagacc aagcctagag aggaacagta caactccacc 900
taccgggtgg tgtccgtgct gaccgtgctg caccaggatt ggctgaacgg caaagagtac 960
aagtgcaagg tgtccaacaa ggccctgcct gcccccatcg aaaagaccat ctccaaggcc 1020
aagggccagc cccgggaacc ccaggtgtac acactgcccc ctagcaggga cgagctgacc 1080
aagaaccagg tgtccctgac ctgtctcgtg aaaggcttct acccctccga tatcgccgtg 1140
gaatgggagt ccaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
tccgacggct cattcttcct gtacagcaag ctgacagtgg acaagtcccg gtggcagcag 1260
ggcaacgtgt tctcctgctc cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgtccc tgagccccgg caag 1344
<210> 142
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 142
Gln Gly Ile Arg Asn Asp
1 5
<210> 143
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 143
Ala Ala Ser
1
<210> 144
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 144
Leu Gln His Asn Ser Tyr Pro Arg Thr
1 5
<210> 145
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 145
Arg Ala Ser Gln Gly Ile Arg Asn Asp Leu Gly
1 5 10
<210> 146
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 146
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 147
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 147
Leu Gln His Asn Ser Tyr Pro Arg Thr
1 5
<210> 148
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 148
Asp Leu Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 149
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 149
gacctccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacag cataatagtt accctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 150
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 150
Asp Leu Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 151
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 151
gacctccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacag cataatagtt accctcggac gttcggccaa 300
gggaccaagg tggaaatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 152
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 152
Gly Gly Ser Ile Ser Ser Ser Asp Trp
1 5
<210> 153
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 153
Ile Phe His Ser Gly Asn Thr
1 5
<210> 154
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 154
Val Arg Asp Gly Ser Gly Ser Tyr
1 5
<210> 155
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 155
Ser Ser Asp Trp Trp Ser
1 5
<210> 156
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 156
Glu Ile Phe His Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 157
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 157
Asp Gly Ser Gly Ser Tyr
1 5
<210> 158
<211> 115
<212> PRT
<213> Homo Sapiens
<400> 158
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Asp Trp Trp Ser Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Phe His Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser Lys Asn Gln Ile Ser
65 70 75 80
Leu Arg Leu Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Arg Asp Gly Ser Gly Ser Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 159
<211> 346
<212> DNA
<213> Homo Sapiens
<400> 159
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggggac cctgtccctc 60
acctgcgctg tctctggtgg ctccatcagc agtagtgact ggtggagttg ggtccgccag 120
cccccaggga aggggctgga gtggattggg gaaatctttc atagtgggaa caccaactac 180
aacccgtccc tcaagagtcg agtcaccata tcagtagaca agtccaagaa ccagatctcc 240
ctgaggctga actctgtgac cgccgcggac acggccgtgt attactgtgt gagagatggt 300
tcggggagtt actggggcca gggaaccctg gtcaccgtct cctcag 346
<210> 160
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 160
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Asp Trp Trp Ser Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Phe His Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser Lys Asn Gln Ile Ser
65 70 75 80
Leu Arg Leu Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Arg Asp Gly Ser Gly Ser Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 161
<211> 1335
<212> DNA
<213> Homo Sapiens
<400> 161
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggggac cctgtccctc 60
acctgcgctg tctctggtgg ctccatcagc agtagtgact ggtggagttg ggtccgccag 120
cccccaggga aggggctgga gtggattggg gaaatctttc atagtgggaa caccaactac 180
aacccgtccc tcaagagtcg agtcaccata tcagtagaca agtccaagaa ccagatctcc 240
ctgaggctga actctgtgac cgccgcggac acggccgtgt attactgtgt gagagatggt 300
tcggggagtt actggggcca gggaaccctg gtcaccgtct cctcagccag caccaagggc 360
ccctctgtgt tccctctggc cccttccagc aagtccacct ctggcggaac agccgctctg 420
ggctgcctcg tgaaggacta cttccccgag cctgtgaccg tgtcctggaa ctctggcgct 480
ctgaccagcg gagtgcacac cttccctgct gtgctgcagt cctccggcct gtactccctg 540
tcctccgtcg tgaccgtgcc ttccagctct ctgggcaccc agacctacat ctgcaacgtg 600
aaccacaagc cctccaacac caaggtggac aagaaggtgg aacccaagtc ctgcgacaag 660
acccacacct gtcccccttg tcctgcccct gaactgctgg gcggaccttc cgtgttcctg 720
ttccccccaa agcccaagga caccctgatg atctcccgga cccccgaagt gacctgcgtg 780
gtggtggatg tgtcccacga ggaccctgaa gtgaagttca attggtacgt ggacggcgtg 840
gaagtgcaca acgccaagac caagcctaga gaggaacagt acaactccac ctaccgggtg 900
gtgtccgtgc tgaccgtgct gcaccaggat tggctgaacg gcaaagagta caagtgcaag 960
gtgtccaaca aggccctgcc tgcccccatc gaaaagacca tctccaaggc caagggccag 1020
ccccgggaac cccaggtgta cacactgccc cctagcaggg acgagctgac caagaaccag 1080
gtgtccctga cctgtctcgt gaaaggcttc tacccctccg atatcgccgt ggaatgggag 1140
tccaacggcc agcctgagaa caactacaag accacccccc ctgtgctgga ctccgacggc 1200
tcattcttcc tgtacagcaa gctgacagtg gacaagtccc ggtggcagca gggcaacgtg 1260
ttctcctgct ccgtgatgca cgaggccctg cacaaccact acacccagaa gtccctgtcc 1320
ctgagccccg gcaag 1335
<210> 162
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 162
Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr
1 5 10
<210> 163
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 163
Trp Ala Ser
1
<210> 164
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 164
Gln Gln Tyr Tyr Ser Thr Arg Ser
1 5
<210> 165
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 165
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 166
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 166
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 167
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 167
Gln Gln Tyr Tyr Ser Thr Arg Ser
1 5
<210> 168
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 168
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Arg Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 169
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 169
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aaactgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
cgcagttttg gccaggggac caagctggag atcaaac 337
<210> 170
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 170
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Arg Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 171
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 171
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aaactgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
cgcagttttg gccaggggac caagctggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 172
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 172
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 173
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 173
Ile Tyr Ser Thr Gly Tyr Thr
1 5
<210> 174
<211> 13
<212> PRT
<213> Homo Sapiens
<400> 174
Ala Ile Ser Thr Ala Ala Gly Pro Glu Tyr Phe His Arg
1 5 10
<210> 175
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 175
Ser Ser Ser Tyr Tyr Cys Gly
1 5
<210> 176
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 176
Ser Ile Tyr Ser Thr Gly Tyr Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 177
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 177
Ser Thr Ala Ala Gly Pro Glu Tyr Phe His Arg
1 5 10
<210> 178
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 178
Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu
1 5 10 15
Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Ser Ser Tyr
20 25 30
Tyr Cys Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Asp Trp Ile
35 40 45
Gly Ser Ile Tyr Ser Thr Gly Tyr Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Cys
65 70 75 80
Leu Ile Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ile Ser Thr Ala Ala Gly Pro Glu Tyr Phe His Arg Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 179
<211> 361
<212> DNA
<213> Homo Sapiens
<400> 179
cagctgcagg agtcgggccc aggcctggtg aagccttcgg agaccctgtc cctcacctgc 60
actgtctctg gtggctccat cagcagtagt agttattact gcggctggat ccgccagccc 120
cctgggaagg ggctggactg gattgggagt atctattcta ctgggtacac ctactacaac 180
ccgtccctca agagtcgagt caccatttcc atagacacgt ccaagaacca gttctcatgc 240
ctgatactga cctctgtgac cgccgcagac acggctgtgt attactgtgc gataagtaca 300
gcagctggcc ctgaatactt ccatcgctgg ggccagggca ccctggtcac cgtctcctca 360
g 361
<210> 180
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 180
Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu
1 5 10 15
Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Ser Ser Tyr
20 25 30
Tyr Cys Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Asp Trp Ile
35 40 45
Gly Ser Ile Tyr Ser Thr Gly Tyr Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Cys
65 70 75 80
Leu Ile Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ile Ser Thr Ala Ala Gly Pro Glu Tyr Phe His Arg Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 181
<211> 1350
<212> DNA
<213> Homo Sapiens
<400> 181
cagctgcagg agtcgggccc aggcctggtg aagccttcgg agaccctgtc cctcacctgc 60
actgtctctg gtggctccat cagcagtagt agttattact gcggctggat ccgccagccc 120
cctgggaagg ggctggactg gattgggagt atctattcta ctgggtacac ctactacaac 180
ccgtccctca agagtcgagt caccatttcc atagacacgt ccaagaacca gttctcatgc 240
ctgatactga cctctgtgac cgccgcagac acggctgtgt attactgtgc gataagtaca 300
gcagctggcc ctgaatactt ccatcgctgg ggccagggca ccctggtcac cgtctcctca 360
gccagcacca agggcccctc tgtgttccct ctggcccctt ccagcaagtc cacctctggc 420
ggaacagccg ctctgggctg cctcgtgaag gactacttcc ccgagcctgt gaccgtgtcc 480
tggaactctg gcgctctgac cagcggagtg cacaccttcc ctgctgtgct gcagtcctcc 540
ggcctgtact ccctgtcctc cgtcgtgacc gtgccttcca gctctctggg cacccagacc 600
tacatctgca acgtgaacca caagccctcc aacaccaagg tggacaagaa ggtggaaccc 660
aagtcctgcg acaagaccca cacctgtccc ccttgtcctg cccctgaact gctgggcgga 720
ccttccgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatctc ccggaccccc 780
gaagtgacct gcgtggtggt ggatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaacgcc aagaccaagc ctagagagga acagtacaac 900
tccacctacc gggtggtgtc cgtgctgacc gtgctgcacc aggattggct gaacggcaaa 960
gagtacaagt gcaaggtgtc caacaaggcc ctgcctgccc ccatcgaaaa gaccatctcc 1020
aaggccaagg gccagccccg ggaaccccag gtgtacacac tgccccctag cagggacgag 1080
ctgaccaaga accaggtgtc cctgacctgt ctcgtgaaag gcttctaccc ctccgatatc 1140
gccgtggaat gggagtccaa cggccagcct gagaacaact acaagaccac cccccctgtg 1200
ctggactccg acggctcatt cttcctgtac agcaagctga cagtggacaa gtcccggtgg 1260
cagcagggca acgtgttctc ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgtccctgag ccccggcaag 1350
<210> 182
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 182
Gln Ser Val Leu Tyr Ser Ser Asn Ser Lys Asn Phe
1 5 10
<210> 183
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 183
Trp Ala Ser
1
<210> 184
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 184
Gln Gln Tyr Tyr Ser Thr Pro Arg Thr
1 5
<210> 185
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 185
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Ser Lys Asn Phe Leu
1 5 10 15
Ala
<210> 186
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 186
Trp Ala Ser Thr Arg Gly Ser
1 5
<210> 187
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 187
Gln Gln Tyr Tyr Ser Thr Pro Arg Thr
1 5
<210> 188
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 188
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Ser Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Phe Ile Tyr Trp Ala Ser Thr Arg Gly Ser Gly Val
50 55 60
Pro Asp Arg Ile Ser Gly Ser Gly Ser Gly Thr Asp Phe Asn Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 189
<211> 340
<212> DNA
<213> Homo Sapiens
<400> 189
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acagtaagaa cttcttagct 120
tggtaccagc agaaaccggg acagcctcct aagctgttca tttactgggc atctacccgg 180
ggatccgggg tccctgaccg aatcagtggc agcgggtctg ggacagattt caatctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcaacaata ttatagtact 300
cctcggacgt tcggccaagg gaccaaggtg gagatcaaac 340
<210> 190
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 190
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Ser Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Phe Ile Tyr Trp Ala Ser Thr Arg Gly Ser Gly Val
50 55 60
Pro Asp Arg Ile Ser Gly Ser Gly Ser Gly Thr Asp Phe Asn Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 191
<211> 660
<212> DNA
<213> Homo Sapiens
<400> 191
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acagtaagaa cttcttagct 120
tggtaccagc agaaaccggg acagcctcct aagctgttca tttactgggc atctacccgg 180
ggatccgggg tccctgaccg aatcagtggc agcgggtctg ggacagattt caatctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcaacaata ttatagtact 300
cctcggacgt tcggccaagg gaccaaggtg gagatcaaac gtacggtggc cgctccctcc 360
gtgttcatct tcccaccttc cgacgagcag ctgaagtccg gcaccgcttc tgtcgtgtgc 420
ctgctgaaca acttctaccc ccgcgaggcc aaggtgcagt ggaaggtgga caacgccctg 480
cagtccggca actcccagga atccgtgacc gagcaggact ccaaggacag cacctactcc 540
ctgtcctcca ccctgaccct gtccaaggcc gactacgaga agcacaaggt gtacgcctgc 600
gaagtgaccc accagggcct gtctagcccc gtgaccaagt ctttcaaccg gggcgagtgt 660
<210> 192
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 192
gcttccacca agggcccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagccg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc 300
aaatatggtc ccccatgccc atcatgccca gcacctgagt tcctgggggg accatcagtc 360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat 480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac 540
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccgtcc tccatcgaga aaaccatctc caaagccaaa 660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 840
gacggctcct tcttcctcta cagcaggcta accgtggaca agagcaggtg gcaggagggg 900
aatgtcttct catgctccgt gatgcatgag gctctgcaca accactacac acagaagagc 960
ctctccctgt ctctgggtaa a 981
<210> 193
<211> 327
<212> PRT
<213> Homo Sapiens
<400> 193
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 194
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 194
gcttccacca agggcccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagccg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc 300
aaatatggtc ccccgtgccc atcatgccca gcacctgagt tcctgggggg accatcagtc 360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat 480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac 540
cgtgtggtca gcgtcctcac cgtcgtgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccgtcc tccatcgaga aaaccatctc caaagccaaa 660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 840
gacggctcct tcttcctcta cagcaggcta accgtggaca agagcaggtg gcaggagggg 900
aatgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 960
ctctccctgt ctctgggtaa a 981
<210> 195
<211> 327
<212> PRT
<213> Homo Sapiens
<400> 195
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 196
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 196
gcttccacca agggcccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagccg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc 300
aaatatggtc ccccatgccc atcatgccca gcacctgagt tcctgggggg accatcagtc 360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat 480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac 540
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccgtcc tccatcgaga aaaccatctc caaagccaaa 660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 840
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcaggagggg 900
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 960
ctctccctgt ctctgggtaa a 981
<210> 197
<211> 327
<212> PRT
<213> Homo Sapiens
<400> 197
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 198
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 198
gcctccacca agggcccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacggccg ccctgggctg cctggtcaag gactacttcc ccgaaccagt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc 300
aaatatggtc ccccatgccc accatgccca gcgcctgaat ttgagggggg accatcagtc 360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat 480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac 540
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccgtca tcgatcgaga aaaccatctc caaagccaaa 660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 840
gacggatcct tcttcctcta cagcaggcta accgtggaca agagcaggtg gcaggagggg 900
aatgtcttct catgctccgt gatgcatgag gctctgcaca accactacac acagaagagc 960
ctctccctgt ctctgggtaa a 981
<210> 199
<211> 327
<212> PRT
<213> Homo Sapiens
<400> 199
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
100 105 110
Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 200
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 200
gcctccacca agggacctag cgtgttccct ctcgccccct gttccaggtc cacaagcgag 60
tccaccgctg ccctcggctg tctggtgaaa gactactttc ccgagcccgt gaccgtctcc 120
tggaatagcg gagccctgac ctccggcgtg cacacatttc ccgccgtgct gcagagcagc 180
ggactgtata gcctgagcag cgtggtgacc gtgcccagct ccagcctcgg caccaaaacc 240
tacacctgca acgtggacca caagccctcc aacaccaagg tggacaagcg ggtggagagc 300
aagtacggcc ccccttgccc tccttgtcct gcccctgagt tcgagggagg accctccgtg 360
ttcctgtttc cccccaaacc caaggacacc ctgatgatct cccggacacc cgaggtgacc 420
tgtgtggtcg tggacgtcag ccaggaggac cccgaggtgc agttcaactg gtatgtggac 480
ggcgtggagg tgcacaatgc caaaaccaag cccagggagg agcagttcaa ttccacctac 540
agggtggtga gcgtgctgac cgtcctgcat caggattggc tgaacggcaa ggagtacaag 600
tgcaaggtgt ccaacaaggg actgcccagc tccatcgaga agaccatcag caaggctaag 660
ggccagccga gggagcccca ggtgtatacc ctgcctccta gccaggaaga gatgaccaag 720
aaccaagtgt ccctgacctg cctggtgaag ggattctacc cctccgacat cgccgtggag 780
tgggagagca atggccagcc cgagaacaac tacaaaacaa cccctcccgt gctcgatagc 840
gacggcagct tctttctcta cagccggctg acagtggaca agagcaggtg gcaggagggc 900
aacgtgttct cctgttccgt gatgcacgag gccctgcaca atcactacac ccagaagagc 960
ctctccctgt ccctgggcaa g 981
<210> 201
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 201
gccagcacca agggcccttc cgtgttcccc ctggcccctt gcagcaggag cacctccgaa 60
tccacagctg ccctgggctg tctggtgaag gactactttc ccgagcccgt gaccgtgagc 120
tggaacagcg gcgctctgac atccggcgtc cacacctttc ctgccgtcct gcagtcctcc 180
ggcctctact ccctgtcctc cgtggtgacc gtgcctagct cctccctcgg caccaagacc 240
tacacctgta acgtggacca caaaccctcc aacaccaagg tggacaaacg ggtcgagagc 300
aagtacggcc ctccctgccc tccttgtcct gcccccgagt tcgaaggcgg acccagcgtg 360
ttcctgttcc ctcctaagcc caaggacacc ctcatgatca gccggacacc cgaggtgacc 420
tgcgtggtgg tggatgtgag ccaggaggac cctgaggtcc agttcaactg gtatgtggat 480
ggcgtggagg tgcacaacgc caagacaaag ccccgggaag agcagttcaa ctccacctac 540
agggtggtca gcgtgctgac cgtgctgcat caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtca gcaataaggg actgcccagc agcatcgaga agaccatctc caaggctaaa 660
ggccagcccc gggaacctca ggtgtacacc ctgcctccca gccaggagga gatgaccaag 720
aaccaggtga gcctgacctg cctggtgaag ggattctacc cttccgacat cgccgtggag 780
tgggagtcca acggccagcc cgagaacaat tataagacca cccctcccgt cctcgacagc 840
gacggatcct tctttctgta ctccaggctg accgtggata agtccaggtg gcaggaaggc 900
aacgtgttca gctgctccgt gatgcacgag gccctgcaca atcactacac ccagaagtcc 960
ctgagcctgt ccctgggaaa g 981
<210> 202
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 202
gcctccacca agggcccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacggccg ccctgggctg cctggtcaag gactacttcc ccgaaccagt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc 300
aaatatggtc ccccatgccc accatgccca gcgcctccag ttgcgggggg accatcagtc 360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg 420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat 480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac 540
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaacggcaa ggagtacaag 600
tgcaaggtct ccaacaaagg cctcccgtca tcgatcgaga aaaccatctc caaagccaaa 660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag 720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag 780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 840
gacggatcct tcttcctcta cagcaggcta accgtggaca agagcaggtg gcaggagggg 900
aatgtcttct catgctccgt gatgcatgag gctctgcaca accactacac acagaagagc 960
ctctccctgt ctctgggtaa a 981
<210> 203
<211> 327
<212> PRT
<213> Homo Sapiens
<400> 203
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 204
<211> 990
<212> DNA
<213> Homo Sapiens
<400> 204
gcctccacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agtggagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cgcgggggca 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 720
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc tccgggtaaa 990
<210> 205
<211> 330
<212> PRT
<213> Homo Sapiens
<400> 205
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 206
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 206
cgtacggtgg ccgctccctc cgtgttcatc ttcccacctt ccgacgagca gctgaagtcc 60
ggcaccgctt ctgtcgtgtg cctgctgaac aacttctacc cccgcgaggc caaggtgcag 120
tggaaggtgg acaacgccct gcagtccggc aactcccagg aatccgtgac cgagcaggac 180
tccaaggaca gcacctactc cctgtcctcc accctgaccc tgtccaaggc cgactacgag 240
aagcacaagg tgtacgcctg cgaagtgacc caccagggcc tgtctagccc cgtgaccaag 300
tctttcaacc ggggcgagtg t 321
<210> 207
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 207
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 208
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 208
cgaactgtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
tggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggag 180
agcaaggaca gcacctacag cctcagcagc accctgacgc tgagcaaagc agactacgag 240
aaacacaaag tctacgccgg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg t 321
<210> 209
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 209
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Glu Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Gly Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 210
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 210
cgaactgtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
cggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggag 180
agcaaggaca gcacctacag cctcagcagc accctgacgc tgagcaaagc agactacgag 240
aaacacaaag tctacgcctg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg t 321
<210> 211
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 211
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Arg Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Glu Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 212
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 212
cgaactgtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
tggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggac 180
agcaaggaca gcacctacag cctcagcagc accctgacgc tgagcaaagc agactacgag 240
aaacacaaac tctacgcctg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg t 321
<210> 213
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 213
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Leu Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 214
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 214
cgaactgtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
tggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggac 180
agcaaggaca gcacctacag cctcagcaac accctgacgc tgagcaaagc agactacgag 240
aaacacaaag tctacgcctg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg c 321
<210> 215
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 215
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Asn Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 216
<211> 312
<212> DNA
<213> Homo Sapiens
<400> 216
cccaaggcca accccacggt cactctgttc ccgccctcct ctgaggagct ccaagccaac 60
aaggccacac tagtgtgtct gatcagtgac ttctacccgg gagctgtgac agtggcttgg 120
aaggcagatg gcagccccgt caaggcggga gtggagacga ccaaaccctc caaacagagc 180
aacaacaagt acgcggccag cagctacctg agcctgacgc ccgagcagtg gaagtcccac 240
agaagctaca gctgccaggt cacgcatgaa gggagcaccg tggagaagac agtggcccct 300
acagaatgtt ca 312
<210> 217
<211> 104
<212> PRT
<213> Homo Sapiens
<400> 217
Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
1 5 10 15
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
20 25 30
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro Val Lys
35 40 45
Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn Lys Tyr
50 55 60
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
65 70 75 80
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
85 90 95
Thr Val Ala Pro Thr Glu Cys Ser
100
<210> 218
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 218
ggtcagccca aggccaaccc cactgtcact ctgttcccgc cctcctctga ggagctccaa 60
gccaacaagg ccacactagt gtgtctgatc agtgacttct acccgggagc tgtgacagtg 120
gcctggaagg cagatggcag ccccgtcaag gcgggagtgg agaccaccaa accctccaaa 180
cagagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcccga gcagtggaag 240
tcccacagaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacag aatgttca 318
<210> 219
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 219
Gly Gln Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 220
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 220
ggtcagccca aggccaaccc cactgtcact ctgttcccgc cctcctctga ggagctccaa 60
gccaacaagg ccacactagt gtgtctgatc agtgacttct acccgggagc tgtgacagtg 120
gcctggaagg cagatggcag ccccgtcaag gcgggagtgg agaccaccaa accctccaaa 180
cagagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcccga gcagtggaag 240
tcccacagaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacag aatgttca 318
<210> 221
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 221
ggccagccta aggccgctcc ttctgtgacc ctgttccccc catcctccga ggaactgcag 60
gctaacaagg ccaccctcgt gtgcctgatc agcgacttct accctggcgc cgtgaccgtg 120
gcctggaagg ctgatagctc tcctgtgaag gccggcgtgg aaaccaccac cccttccaag 180
cagtccaaca acaaatacgc cgcctcctcc tacctgtccc tgacccctga gcagtggaag 240
tcccaccggt cctacagctg ccaagtgacc cacgagggct ccaccgtgga aaagaccgtg 300
gctcctaccg agtgctcc 318
<210> 222
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 222
ggccagccta aagctgcccc cagcgtcacc ctgtttcctc cctccagcga ggagctccag 60
gccaacaagg ccaccctcgt gtgcctgatc tccgacttct atcccggcgc tgtgaccgtg 120
gcttggaaag ccgactccag ccctgtcaaa gccggcgtgg agaccaccac accctccaag 180
cagtccaaca acaagtacgc cgcctccagc tatctctccc tgacccctga gcagtggaag 240
tcccaccggt cctactcctg tcaggtgacc cacgagggct ccaccgtgga aaagaccgtc 300
gcccccaccg agtgctcc 318
<210> 223
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 223
Gly Gln Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 224
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 224
ggtcagccca aggctgcccc ctcggtcact ctgttcccgc cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcata agtgacttct acccgggagc cgtgacagtg 120
gcctggaagg cagatagcag ccccgtcaag gcgggagtgg agaccaccac accctccaaa 180
caaagcaaca acaagtacgc ggccagcagc tatctgagcc tgacgcctga gcagtggaag 240
tcccacagaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacag aatgttca 318
<210> 225
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 225
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 226
<211> 312
<212> DNA
<213> Homo Sapiens
<400> 226
cccaaggctg ccccctcggt cactctgttc ccaccctcct ctgaggagct tcaagccaac 60
aaggccacac tggtgtgtct cataagtgac ttctacccgg gagccgtgac agttgcctgg 120
aaggcagata gcagccccgt caaggcgggg gtggagacca ccacaccctc caaacaaagc 180
aacaacaagt acgcggccag cagctacctg agcctgacgc ctgagcagtg gaagtcccac 240
aaaagctaca gctgccaggt cacgcatgaa gggagcaccg tggagaagac agttgcccct 300
acggaatgtt ca 312
<210> 227
<211> 104
<212> PRT
<213> Homo Sapiens
<400> 227
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
1 5 10 15
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
20 25 30
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
35 40 45
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
50 55 60
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
65 70 75 80
Lys Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
85 90 95
Thr Val Ala Pro Thr Glu Cys Ser
100
<210> 228
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 228
ggtcagccca aggctgcccc ctcggtcact ctgttcccac cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcata agtgacttct acccggggcc agtgacagtt 120
gcctggaagg cagatagcag ccccgtcaag gcgggggtgg agaccaccac accctccaaa 180
caaagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcctga gcagtggaag 240
tcccacaaaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacgg aatgttca 318
<210> 229
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 229
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Pro Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Lys Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 230
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 230
ggtcagccca aggctgcccc ctcggtcact ctgttcccac cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcata agtgacttct acccgggagc cgtgacagtg 120
gcctggaagg cagatagcag ccccgtcaag gcgggagtgg agaccaccac accctccaaa 180
caaagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcctga gcagtggaag 240
tcccacaaaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacag aatgttca 318
<210> 231
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 231
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Lys Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 232
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 232
ggtcagccca aggctgcccc ctcggtcact ctgttcccgc cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcata agtgacttct acccgggagc cgtgacagtg 120
gcctggaagg cagatagcag ccccgtcaag gcgggagtgg agaccaccac accctccaaa 180
caaagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcctga gcagtggaag 240
tcccacagaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctacag aatgttca 318
<210> 233
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 233
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 234
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 234
ggtcagccca aggctgcccc atcggtcact ctgttcccgc cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgcctgatc agtgacttct acccgggagc tgtgaaagtg 120
gcctggaagg cagatggcag ccccgtcaac acgggagtgg agaccaccac accctccaaa 180
cagagcaaca acaagtacgc ggccagcagc tacctgagcc tgacgcctga gcagtggaag 240
tcccacagaa gctacagctg ccaggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctgcag aatgttca 318
<210> 235
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 235
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Lys Val Ala Trp Lys Ala Asp Gly Ser Pro
35 40 45
Val Asn Thr Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Ala Glu Cys Ser
100 105
<210> 236
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 236
ggtcagccca aggctgcccc atcggtcact ctgttcccac cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcgta agtgacttct acccgggagc cgtgacagtg 120
gcctggaagg cagatggcag ccccgtcaag gtgggagtgg agaccaccaa accctccaaa 180
caaagcaaca acaagtatgc ggccagcagc tacctgagcc tgacgcccga gcagtggaag 240
tcccacagaa gctacagctg ccgggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctgcag aatgctct 318
<210> 237
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 237
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Val Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro
35 40 45
Val Lys Val Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Arg Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Ala Glu Cys Ser
100 105
<210> 238
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 238
Gly Phe Thr Phe Ser Asp Tyr Tyr
1 5
<210> 239
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 239
Ile Ser Thr Ser Gly Ser Thr Ile
1 5
<210> 240
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 240
Ala Arg Gly Ile Thr Gly Thr Asn Phe Tyr His Tyr Gly Leu Gly Val
1 5 10 15
<210> 241
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 241
Asp Tyr Tyr Met Ser
1 5
<210> 242
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 242
Tyr Ile Ser Thr Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 243
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 243
Gly Ile Thr Gly Thr Asn Phe Tyr His Tyr Gly Leu Gly Val
1 5 10
<210> 244
<211> 123
<212> PRT
<213> Homo Sapiens
<400> 244
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Thr Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Ala Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ile Thr Gly Thr Asn Phe Tyr His Tyr Gly Leu Gly Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 245
<211> 370
<212> DNA
<213> Homo Sapiens
<400> 245
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactactaca tgagctggat ccgccaggtt 120
ccagggaagg ggctggagtg ggtttcatac attagtacta gtggtagtac catatactac 180
gcagactctg tgaagggccg attcaccatc tccagggaca acgccaagaa ctcactgtat 240
ctacaaatga acagcctgag agccgaggac gcggccgtgt atcactgtgc gagaggtata 300
actggaacta acttctacca ctacggtttg ggcgtctggg gccaagggac cacggtcacc 360
gtctcctcag 370
<210> 246
<211> 453
<212> PRT
<213> Homo Sapiens
<400> 246
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Thr Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Ala Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ile Thr Gly Thr Asn Phe Tyr His Tyr Gly Leu Gly Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 247
<211> 1359
<212> DNA
<213> Homo Sapiens
<400> 247
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactactaca tgagctggat ccgccaggtt 120
ccagggaagg ggctggagtg ggtttcatac attagtacta gtggtagtac catatactac 180
gcagactctg tgaagggccg attcaccatc tccagggaca acgccaagaa ctcactgtat 240
ctacaaatga acagcctgag agccgaggac gcggccgtgt atcactgtgc gagaggtata 300
actggaacta acttctacca ctacggtttg ggcgtctggg gccaagggac cacggtcacc 360
gtctcctcag ccagcaccaa gggcccctct gtgttccctc tggccccttc cagcaagtcc 420
acctctggcg gaacagccgc tctgggctgc ctcgtgaagg actacttccc cgagcctgtg 480
accgtgtcct ggaactctgg cgctctgacc agcggagtgc acaccttccc tgctgtgctg 540
cagtcctccg gcctgtactc cctgtcctcc gtcgtgaccg tgccttccag ctctctgggc 600
acccagacct acatctgcaa cgtgaaccac aagccctcca acaccaaggt ggacaagaag 660
gtggaaccca agtcctgcga caagacccac acctgtcccc cttgtcctgc ccctgaactg 720
ctgggcggac cttccgtgtt cctgttcccc ccaaagccca aggacaccct gatgatctcc 780
cggacccccg aagtgacctg cgtggtggtg gatgtgtccc acgaggaccc tgaagtgaag 840
ttcaattggt acgtggacgg cgtggaagtg cacaacgcca agaccaagcc tagagaggaa 900
cagtacaact ccacctaccg ggtggtgtcc gtgctgaccg tgctgcacca ggattggctg 960
aacggcaaag agtacaagtg caaggtgtcc aacaaggccc tgcctgcccc catcgaaaag 1020
accatctcca aggccaaggg ccagccccgg gaaccccagg tgtacacact gccccctagc 1080
agggacgagc tgaccaagaa ccaggtgtcc ctgacctgtc tcgtgaaagg cttctacccc 1140
tccgatatcg ccgtggaatg ggagtccaac ggccagcctg agaacaacta caagaccacc 1200
ccccctgtgc tggactccga cggctcattc ttcctgtaca gcaagctgac agtggacaag 1260
tcccggtggc agcagggcaa cgtgttctcc tgctccgtga tgcacgaggc cctgcacaac 1320
cactacaccc agaagtccct gtccctgagc cccggcaag 1359
<210> 248
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 248
Gln Gly Ile Asn Ser Trp
1 5
<210> 249
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 249
Ala Ala Ser
1
<210> 250
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 250
Gln Gln Val Asn Ser Phe Pro Leu Thr
1 5
<210> 251
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 251
Arg Ala Ser Gln Gly Ile Asn Ser Trp Leu Ala
1 5 10
<210> 252
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 252
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 253
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 253
Gln Gln Val Asn Ser Phe Pro Leu Thr
1 5
<210> 254
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 254
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Asn Ser Phe Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 255
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 255
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattaac agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtgggtc tggggcagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gttaacagtt tcccgctcac tttcggcgga 300
gggaccaagg tggagatcaa ac 322
<210> 256
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 256
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Asn Ser Phe Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 257
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 257
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattaac agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtgggtc tggggcagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gttaacagtt tcccgctcac tttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 258
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 258
Gly Phe Thr Phe Ser Tyr Tyr Ala
1 5
<210> 259
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 259
Ile Ser Gly Gly Gly Gly Asn Thr
1 5
<210> 260
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 260
Ala Lys Asp Arg Met Lys Gln Leu Val Arg Ala Tyr Tyr Phe Asp Tyr
1 5 10 15
<210> 261
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 261
Tyr Tyr Ala Met Ser
1 5
<210> 262
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 262
Thr Ile Ser Gly Gly Gly Gly Asn Thr His Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 263
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 263
Asp Arg Met Lys Gln Leu Val Arg Ala Tyr Tyr Phe Asp Tyr
1 5 10
<210> 264
<211> 123
<212> PRT
<213> Homo Sapiens
<400> 264
Glu Val Pro Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Asp Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Gly Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Met Lys Gln Leu Val Arg Ala Tyr Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 265
<211> 370
<212> DNA
<213> Homo Sapiens
<400> 265
gaggtgccgc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagc tactatgcca tgagctgggt ccgtcaggct 120
ccagggaagg ggctggactg ggtctcaact attagtggtg gtggtggtaa cacacactac 180
gcagactccg tgaagggccg attcactata tccagagaca attccaagaa cacgctgtat 240
ctgcacatga acagcctgag agccgaagac acggccgtct attactgtgc gaaggatcgg 300
atgaaacagc tcgtccgggc ctactacttt gactactggg gccagggaac cctggtcacc 360
gtctcctcag 370
<210> 266
<211> 453
<212> PRT
<213> Homo Sapiens
<400> 266
Glu Val Pro Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Asp Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Gly Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Met Lys Gln Leu Val Arg Ala Tyr Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 267
<211> 1359
<212> DNA
<213> Homo Sapiens
<400> 267
gaggtgccgc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacgtttagc tactatgcca tgagctgggt ccgtcaggct 120
ccagggaagg ggctggactg ggtctcaact attagtggtg gtggtggtaa cacacactac 180
gcagactccg tgaagggccg attcactata tccagagaca attccaagaa cacgctgtat 240
ctgcacatga acagcctgag agccgaagac acggccgtct attactgtgc gaaggatcgg 300
atgaaacagc tcgtccgggc ctactacttt gactactggg gccagggaac cctggtcacc 360
gtctcctcag ccagcaccaa gggcccctct gtgttccctc tggccccttc cagcaagtcc 420
acctctggcg gaacagccgc tctgggctgc ctcgtgaagg actacttccc cgagcctgtg 480
accgtgtcct ggaactctgg cgctctgacc agcggagtgc acaccttccc tgctgtgctg 540
cagtcctccg gcctgtactc cctgtcctcc gtcgtgaccg tgccttccag ctctctgggc 600
acccagacct acatctgcaa cgtgaaccac aagccctcca acaccaaggt ggacaagaag 660
gtggaaccca agtcctgcga caagacccac acctgtcccc cttgtcctgc ccctgaactg 720
ctgggcggac cttccgtgtt cctgttcccc ccaaagccca aggacaccct gatgatctcc 780
cggacccccg aagtgacctg cgtggtggtg gatgtgtccc acgaggaccc tgaagtgaag 840
ttcaattggt acgtggacgg cgtggaagtg cacaacgcca agaccaagcc tagagaggaa 900
cagtacaact ccacctaccg ggtggtgtcc gtgctgaccg tgctgcacca ggattggctg 960
aacggcaaag agtacaagtg caaggtgtcc aacaaggccc tgcctgcccc catcgaaaag 1020
accatctcca aggccaaggg ccagccccgg gaaccccagg tgtacacact gccccctagc 1080
agggacgagc tgaccaagaa ccaggtgtcc ctgacctgtc tcgtgaaagg cttctacccc 1140
tccgatatcg ccgtggaatg ggagtccaac ggccagcctg agaacaacta caagaccacc 1200
ccccctgtgc tggactccga cggctcattc ttcctgtaca gcaagctgac agtggacaag 1260
tcccggtggc agcagggcaa cgtgttctcc tgctccgtga tgcacgaggc cctgcacaac 1320
cactacaccc agaagtccct gtccctgagc cccggcaag 1359
<210> 268
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 268
Gln Asp Ile Ser Thr Tyr
1 5
<210> 269
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 269
Gly Thr Ser
1
<210> 270
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 270
Gln Gln Leu His Thr Asp Pro Ile Thr
1 5
<210> 271
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 271
Trp Ala Ser Gln Asp Ile Ser Thr Tyr Leu Gly
1 5 10
<210> 272
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 272
Gly Thr Ser Ser Leu Gln Ser
1 5
<210> 273
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 273
Gln Gln Leu His Thr Asp Pro Ile Thr
1 5
<210> 274
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 274
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Asp Ile Ser Thr Tyr
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Thr Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu His Thr Asp Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 275
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 275
gacatccagt tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca ggacattagc acttatttag gctggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gatctatggt acatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttcatactg acccgatcac cttcggccaa 300
gggacacgac tggagatcaa ac 322
<210> 276
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 276
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Asp Ile Ser Thr Tyr
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Thr Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu His Thr Asp Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 277
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 277
gacatccagt tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca ggacattagc acttatttag gctggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gatctatggt acatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttcatactg acccgatcac cttcggccaa 300
gggacacgac tggagatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 278
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 278
Gly Phe Thr Phe Ser Ser Tyr Trp
1 5
<210> 279
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 279
Ile Lys Gln Asp Gly Ser Glu Lys
1 5
<210> 280
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 280
Ala Arg Val Arg Gln Trp Ser Asp Tyr Ser Asp Tyr
1 5 10
<210> 281
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 281
Ser Tyr Trp Met Asn
1 5
<210> 282
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 282
Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val Lys
1 5 10 15
Gly
<210> 283
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 283
Val Arg Gln Trp Ser Asp Tyr Ser Asp Tyr
1 5 10
<210> 284
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 284
Glu Val His Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Arg Gln Trp Ser Asp Tyr Ser Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Pro Val Thr Val Ser Ser
115
<210> 285
<211> 358
<212> DNA
<213> Homo Sapiens
<400> 285
gaggtgcacc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagt agctattgga tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac ataaagcaag atggaagtga gaaatactat 180
gtggactctg tgaagggccg cttcaccgtc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagttcga 300
caatggtccg actactctga ctactggggc cagggaaccc cggtcaccgt ctcctcag 358
<210> 286
<211> 449
<212> PRT
<213> Homo Sapiens
<400> 286
Glu Val His Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Trp Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Arg Gln Trp Ser Asp Tyr Ser Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Pro Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 287
<211> 1347
<212> DNA
<213> Homo Sapiens
<400> 287
gaggtgcacc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagt agctattgga tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac ataaagcaag atggaagtga gaaatactat 180
gtggactctg tgaagggccg cttcaccgtc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagttcga 300
caatggtccg actactctga ctactggggc cagggaaccc cggtcaccgt ctcctcagcc 360
agcaccaagg gcccctctgt gttccctctg gccccttcca gcaagtccac ctctggcgga 420
acagccgctc tgggctgcct cgtgaaggac tacttccccg agcctgtgac cgtgtcctgg 480
aactctggcg ctctgaccag cggagtgcac accttccctg ctgtgctgca gtcctccggc 540
ctgtactccc tgtcctccgt cgtgaccgtg ccttccagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gccctccaac accaaggtgg acaagaaggt ggaacccaag 660
tcctgcgaca agacccacac ctgtccccct tgtcctgccc ctgaactgct gggcggacct 720
tccgtgttcc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 780
gtgacctgcg tggtggtgga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaactcc 900
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 960
tacaagtgca aggtgtccaa caaggccctg cctgccccca tcgaaaagac catctccaag 1020
gccaagggcc agccccggga accccaggtg tacacactgc cccctagcag ggacgagctg 1080
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1140
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gactccgacg gctcattctt cctgtacagc aagctgacag tggacaagtc ccggtggcag 1260
cagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctgt ccctgagccc cggcaag 1347
<210> 288
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 288
Gln Gly Ile Ser Ser Trp
1 5
<210> 289
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 289
Ala Ala Ser
1
<210> 290
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 290
Gln Gln Ala Asn Ser Phe Pro Phe Thr
1 5
<210> 291
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 291
Arg Ala Ser Gln Gly Ile Ser Ser Trp Leu Ala
1 5 10
<210> 292
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 292
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 293
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 293
Gln Gln Ala Asn Ser Phe Pro Phe Thr
1 5
<210> 294
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 294
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 295
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 295
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 296
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 296
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 297
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 297
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 298
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 298
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 299
<211> 453
<212> PRT
<213> Homo Sapiens
<400> 299
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 300
<211> 347
<212> PRT
<213> Homo Sapiens
<400> 300
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr
210 215 220
Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn
225 230 235 240
Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Arg Met Leu Thr Phe
245 250 255
Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys
260 265 270
Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln
275 280 285
Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn
290 295 300
Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu
305 310 315 320
Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile
325 330 335
Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
340 345
<210> 301
<211> 133
<212> PRT
<213> Homo Sapiens
<400> 301
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 302
<211> 586
<212> PRT
<213> Homo Sapiens
<400> 302
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln
450 455 460
Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly
465 470 475 480
Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys
485 490 495
Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu
500 505 510
Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser
515 520 525
Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val
530 535 540
Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr
545 550 555 560
Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr
565 570 575
Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585
<210> 303
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 303
Ala Pro Thr Ser Thr Gln Leu Gln Leu Glu Leu Leu Leu Asp
1 5 10
<210> 304
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 304
Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 305
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 305
Ala Pro Thr Ser Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu
1 5 10 15
Asp
<210> 306
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 306
Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu
1 5 10 15
Leu Leu Asp
<210> 307
<211> 18
<212> PRT
<213> Homo Sapiens
<400> 307
Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu
1 5 10 15
Leu Asp
<210> 308
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 308
Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu
1 5 10 15
Asp
<210> 309
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 309
Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 310
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 310
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 311
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 311
Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 312
<211> 13
<212> PRT
<213> Homo Sapiens
<400> 312
Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 313
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 313
Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 314
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 314
Ala Pro Thr Ser Ser Ser Thr Lys Thr Gln Leu Gln Leu Glu His Leu
1 5 10 15
Leu Leu Asp
<210> 315
<211> 18
<212> PRT
<213> Homo Sapiens
<400> 315
Ala Pro Thr Ser Ser Ser Thr Thr Gln Leu Gln Leu Glu His Leu Leu
1 5 10 15
Leu Asp
<210> 316
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 316
Ala Pro Thr Ser Ser Ser Thr Gln Leu Gln Leu Glu His Leu Leu Leu
1 5 10 15
Asp
<210> 317
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 317
Ala Pro Thr Ser Ser Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 318
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 318
Ala Pro Thr Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 319
<211> 13
<212> PRT
<213> Homo Sapiens
<400> 319
Ala Pro Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 320
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 320
Ala Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10
<210> 321
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 321
Ala Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 322
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 322
Ala Pro Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 323
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 323
Ala Pro Thr Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp
1 5 10 15
<210> 324
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 324
Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu
1 5 10 15
Thr Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu
20 25 30
Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu
35 40 45
Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp
50 55 60
Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu
65 70 75 80
Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu
85 90 95
Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu
100 105 110
Thr
<210> 325
<211> 290
<212> PRT
<213> Mus Musculus
<400> 325
Met Arg Ile Phe Ala Gly Ile Ile Phe Thr Ala Cys Cys His Leu Leu
1 5 10 15
Arg Ala Phe Thr Ile Thr Ala Pro Lys Asp Leu Tyr Val Val Glu Tyr
20 25 30
Gly Ser Asn Val Thr Met Glu Cys Arg Phe Pro Val Glu Arg Glu Leu
35 40 45
Asp Leu Leu Ala Leu Val Val Tyr Trp Glu Lys Glu Asp Glu Gln Val
50 55 60
Ile Gln Phe Val Ala Gly Glu Glu Asp Leu Lys Pro Gln His Ser Asn
65 70 75 80
Phe Arg Gly Arg Ala Ser Leu Pro Lys Asp Gln Leu Leu Lys Gly Asn
85 90 95
Ala Ala Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr
100 105 110
Cys Cys Ile Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Leu
115 120 125
Lys Val Asn Ala Pro Tyr Arg Lys Ile Asn Gln Arg Ile Ser Val Asp
130 135 140
Pro Ala Thr Ser Glu His Glu Leu Ile Cys Gln Ala Glu Gly Tyr Pro
145 150 155 160
Glu Ala Glu Val Ile Trp Thr Asn Ser Asp His Gln Pro Val Ser Gly
165 170 175
Lys Arg Ser Val Thr Thr Ser Arg Thr Glu Gly Met Leu Leu Asn Val
180 185 190
Thr Ser Ser Leu Arg Val Asn Ala Thr Ala Asn Asp Val Phe Tyr Cys
195 200 205
Thr Phe Trp Arg Ser Gln Pro Gly Gln Asn His Thr Ala Glu Leu Ile
210 215 220
Ile Pro Glu Leu Pro Ala Thr His Pro Pro Gln Asn Arg Thr His Trp
225 230 235 240
Val Leu Leu Gly Ser Ile Leu Leu Phe Leu Ile Val Val Ser Thr Val
245 250 255
Leu Leu Phe Leu Arg Lys Gln Val Arg Met Leu Asp Val Glu Lys Cys
260 265 270
Gly Val Glu Asp Thr Ser Ser Lys Asn Arg Asn Asp Thr Gln Phe Glu
275 280 285
Glu Thr
290
<210> 326
<211> 226
<212> PRT
<213> Mus Musculus
<400> 326
Phe Thr Ile Thr Ala Pro Lys Asp Leu Tyr Val Val Glu Tyr Gly Ser
1 5 10 15
Asn Val Thr Met Glu Cys Arg Phe Pro Val Glu Arg Glu Leu Asp Leu
20 25 30
Leu Ala Leu Val Val Tyr Trp Glu Lys Glu Asp Glu Gln Val Ile Gln
35 40 45
Phe Val Ala Gly Glu Glu Asp Leu Lys Pro Gln His Ser Asn Phe Arg
50 55 60
Gly Arg Ala Ser Leu Pro Lys Asp Gln Leu Leu Lys Gly Asn Ala Ala
65 70 75 80
Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr Cys Cys
85 90 95
Ile Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Leu Lys Val
100 105 110
Asn Ala Pro Tyr Arg Lys Ile Asn Gln Arg Ile Ser Val Asp Pro Ala
115 120 125
Thr Ser Glu His Glu Leu Ile Cys Gln Ala Glu Gly Tyr Pro Glu Ala
130 135 140
Glu Val Ile Trp Thr Asn Ser Asp His Gln Pro Val Ser Gly Lys Arg
145 150 155 160
Ser Val Thr Thr Ser Arg Thr Glu Gly Met Leu Leu Asn Val Thr Ser
165 170 175
Ser Leu Arg Val Asn Ala Thr Ala Asn Asp Val Phe Tyr Cys Thr Phe
180 185 190
Trp Arg Ser Gln Pro Gly Gln Asn His Thr Ala Glu Leu Ile Ile Pro
195 200 205
Glu Leu Pro Ala Thr His Pro Pro Gln Asn Arg Thr His His His His
210 215 220
His His
225
<210> 327
<211> 251
<212> PRT
<213> Homo Sapiens
<400> 327
Glu Leu Cys Asp Asp Asp Pro Pro Glu Ile Pro His Ala Thr Phe Lys
1 5 10 15
Ala Met Ala Tyr Lys Glu Gly Thr Met Leu Asn Cys Glu Cys Lys Arg
20 25 30
Gly Phe Arg Arg Ile Lys Ser Gly Ser Leu Tyr Met Leu Cys Thr Gly
35 40 45
Asn Ser Ser His Ser Ser Trp Asp Asn Gln Cys Gln Cys Thr Ser Ser
50 55 60
Ala Thr Arg Asn Thr Thr Lys Gln Val Thr Pro Gln Pro Glu Glu Gln
65 70 75 80
Lys Glu Arg Lys Thr Thr Glu Met Gln Ser Pro Met Gln Pro Val Asp
85 90 95
Gln Ala Ser Leu Pro Gly His Cys Arg Glu Pro Pro Pro Trp Glu Asn
100 105 110
Glu Ala Thr Glu Arg Ile Tyr His Phe Val Val Gly Gln Met Val Tyr
115 120 125
Tyr Gln Cys Val Gln Gly Tyr Arg Ala Leu His Arg Gly Pro Ala Glu
130 135 140
Ser Val Cys Lys Met Thr His Gly Lys Thr Arg Trp Thr Gln Pro Gln
145 150 155 160
Leu Ile Cys Thr Gly Glu Met Glu Thr Ser Gln Phe Pro Gly Glu Glu
165 170 175
Lys Pro Gln Ala Ser Pro Glu Gly Arg Pro Glu Ser Glu Thr Ser Cys
180 185 190
Leu Val Thr Thr Thr Asp Phe Gln Ile Gln Thr Glu Met Ala Ala Thr
195 200 205
Met Glu Thr Ser Ile Phe Thr Thr Glu Tyr Gln Val Ala Val Ala Gly
210 215 220
Cys Val Phe Leu Leu Ile Ser Val Leu Leu Leu Ser Gly Leu Thr Trp
225 230 235 240
Gln Arg Arg Gln Arg Lys Ser Arg Arg Thr Ile
245 250
<210> 328
<211> 525
<212> PRT
<213> Homo Sapiens
<400> 328
Ala Val Asn Gly Thr Ser Gln Phe Thr Cys Phe Tyr Asn Ser Arg Ala
1 5 10 15
Asn Ile Ser Cys Val Trp Ser Gln Asp Gly Ala Leu Gln Asp Thr Ser
20 25 30
Cys Gln Val His Ala Trp Pro Asp Arg Arg Arg Trp Asn Gln Thr Cys
35 40 45
Glu Leu Leu Pro Val Ser Gln Ala Ser Trp Ala Cys Asn Leu Ile Leu
50 55 60
Gly Ala Pro Asp Ser Gln Lys Leu Thr Thr Val Asp Ile Val Thr Leu
65 70 75 80
Arg Val Leu Cys Arg Glu Gly Val Arg Trp Arg Val Met Ala Ile Gln
85 90 95
Asp Phe Lys Pro Phe Glu Asn Leu Arg Leu Met Ala Pro Ile Ser Leu
100 105 110
Gln Val Val His Val Glu Thr His Arg Cys Asn Ile Ser Trp Glu Ile
115 120 125
Ser Gln Ala Ser His Tyr Phe Glu Arg His Leu Glu Phe Glu Ala Arg
130 135 140
Thr Leu Ser Pro Gly His Thr Trp Glu Glu Ala Pro Leu Leu Thr Leu
145 150 155 160
Lys Gln Lys Gln Glu Trp Ile Cys Leu Glu Thr Leu Thr Pro Asp Thr
165 170 175
Gln Tyr Glu Phe Gln Val Arg Val Lys Pro Leu Gln Gly Glu Phe Thr
180 185 190
Thr Trp Ser Pro Trp Ser Gln Pro Leu Ala Phe Arg Thr Lys Pro Ala
195 200 205
Ala Leu Gly Lys Asp Thr Ile Pro Trp Leu Gly His Leu Leu Val Gly
210 215 220
Leu Ser Gly Ala Phe Gly Phe Ile Ile Leu Val Tyr Leu Leu Ile Asn
225 230 235 240
Cys Arg Asn Thr Gly Pro Trp Leu Lys Lys Val Leu Lys Cys Asn Thr
245 250 255
Pro Asp Pro Ser Lys Phe Phe Ser Gln Leu Ser Ser Glu His Gly Gly
260 265 270
Asp Val Gln Lys Trp Leu Ser Ser Pro Phe Pro Ser Ser Ser Phe Ser
275 280 285
Pro Gly Gly Leu Ala Pro Glu Ile Ser Pro Leu Glu Val Leu Glu Arg
290 295 300
Asp Lys Val Thr Gln Leu Leu Leu Gln Gln Asp Lys Val Pro Glu Pro
305 310 315 320
Ala Ser Leu Ser Ser Asn His Ser Leu Thr Ser Cys Phe Thr Asn Gln
325 330 335
Gly Tyr Phe Phe Phe His Leu Pro Asp Ala Leu Glu Ile Glu Ala Cys
340 345 350
Gln Val Tyr Phe Thr Tyr Asp Pro Tyr Ser Glu Glu Asp Pro Asp Glu
355 360 365
Gly Val Ala Gly Ala Pro Thr Gly Ser Ser Pro Gln Pro Leu Gln Pro
370 375 380
Leu Ser Gly Glu Asp Asp Ala Tyr Cys Thr Phe Pro Ser Arg Asp Asp
385 390 395 400
Leu Leu Leu Phe Ser Pro Ser Leu Leu Gly Gly Pro Ser Pro Pro Ser
405 410 415
Thr Ala Pro Gly Gly Ser Gly Ala Gly Glu Glu Arg Met Pro Pro Ser
420 425 430
Leu Gln Glu Arg Val Pro Arg Asp Trp Asp Pro Gln Pro Leu Gly Pro
435 440 445
Pro Thr Pro Gly Val Pro Asp Leu Val Asp Phe Gln Pro Pro Pro Glu
450 455 460
Leu Val Leu Arg Glu Ala Gly Glu Glu Val Pro Asp Ala Gly Pro Arg
465 470 475 480
Glu Gly Val Ser Phe Pro Trp Ser Arg Pro Pro Gly Gln Gly Glu Phe
485 490 495
Arg Ala Leu Asn Ala Arg Leu Pro Leu Asn Thr Asp Ala Tyr Leu Ser
500 505 510
Leu Gln Glu Leu Gln Gly Gln Asp Pro Thr His Leu Val
515 520 525
<210> 329
<211> 347
<212> PRT
<213> Homo Sapiens
<400> 329
Leu Asn Thr Thr Ile Leu Thr Pro Asn Gly Asn Glu Asp Thr Thr Ala
1 5 10 15
Asp Phe Phe Leu Thr Thr Met Pro Thr Asp Ser Leu Ser Val Ser Thr
20 25 30
Leu Pro Leu Pro Glu Val Gln Cys Phe Val Phe Asn Val Glu Tyr Met
35 40 45
Asn Cys Thr Trp Asn Ser Ser Ser Glu Pro Gln Pro Thr Asn Leu Thr
50 55 60
Leu His Tyr Trp Tyr Lys Asn Ser Asp Asn Asp Lys Val Gln Lys Cys
65 70 75 80
Ser His Tyr Leu Phe Ser Glu Glu Ile Thr Ser Gly Cys Gln Leu Gln
85 90 95
Lys Lys Glu Ile His Leu Tyr Gln Thr Phe Val Val Gln Leu Gln Asp
100 105 110
Pro Arg Glu Pro Arg Arg Gln Ala Thr Gln Met Leu Lys Leu Gln Asn
115 120 125
Leu Val Ile Pro Trp Ala Pro Glu Asn Leu Thr Leu His Lys Leu Ser
130 135 140
Glu Ser Gln Leu Glu Leu Asn Trp Asn Asn Arg Phe Leu Asn His Cys
145 150 155 160
Leu Glu His Leu Val Gln Tyr Arg Thr Asp Trp Asp His Ser Trp Thr
165 170 175
Glu Gln Ser Val Asp Tyr Arg His Lys Phe Ser Leu Pro Ser Val Asp
180 185 190
Gly Gln Lys Arg Tyr Thr Phe Arg Val Arg Ser Arg Phe Asn Pro Leu
195 200 205
Cys Gly Ser Ala Gln His Trp Ser Glu Trp Ser His Pro Ile His Trp
210 215 220
Gly Ser Asn Thr Ser Lys Glu Asn Pro Phe Leu Phe Ala Leu Glu Ala
225 230 235 240
Val Val Ile Ser Val Gly Ser Met Gly Leu Ile Ile Ser Leu Leu Cys
245 250 255
Val Tyr Phe Trp Leu Glu Arg Thr Met Pro Arg Ile Pro Thr Leu Lys
260 265 270
Asn Leu Glu Asp Leu Val Thr Glu Tyr His Gly Asn Phe Ser Ala Trp
275 280 285
Ser Gly Val Ser Lys Gly Leu Ala Glu Ser Leu Gln Pro Asp Tyr Ser
290 295 300
Glu Arg Leu Cys Leu Val Ser Glu Ile Pro Pro Lys Gly Gly Ala Leu
305 310 315 320
Gly Glu Gly Pro Gly Ala Ser Pro Cys Asn Gln His Ser Pro Tyr Trp
325 330 335
Ala Pro Pro Cys Tyr Thr Leu Lys Pro Glu Thr
340 345
<210> 330
<211> 152
<212> PRT
<213> Homo Sapiens
<400> 330
Asp Cys Asp Ile Glu Gly Lys Asp Gly Lys Gln Tyr Glu Ser Val Leu
1 5 10 15
Met Val Ser Ile Asp Gln Leu Leu Asp Ser Met Lys Glu Ile Gly Ser
20 25 30
Asn Cys Leu Asn Asn Glu Phe Asn Phe Phe Lys Arg His Ile Cys Asp
35 40 45
Ala Asn Lys Glu Gly Met Phe Leu Phe Arg Ala Ala Arg Lys Leu Arg
50 55 60
Gln Phe Leu Lys Met Asn Ser Thr Gly Asp Phe Asp Leu His Leu Leu
65 70 75 80
Lys Val Ser Glu Gly Thr Thr Ile Leu Leu Asn Cys Thr Gly Gln Val
85 90 95
Lys Gly Arg Lys Pro Ala Ala Leu Gly Glu Ala Gln Pro Thr Lys Ser
100 105 110
Leu Glu Glu Asn Lys Ser Leu Lys Glu Gln Lys Lys Leu Asn Asp Leu
115 120 125
Cys Phe Leu Lys Arg Leu Leu Gln Glu Ile Lys Thr Cys Trp Asn Lys
130 135 140
Ile Leu Met Gly Thr Lys Glu His
145 150
<210> 331
<211> 133
<212> PRT
<213> Homo Sapiens
<400> 331
Gly Ile His Val Phe Ile Leu Gly Cys Phe Ser Ala Gly Leu Pro Lys
1 5 10 15
Thr Glu Ala Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu
20 25 30
Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser
35 40 45
Asp Val His Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu
50 55 60
Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp
65 70 75 80
Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn
85 90 95
Gly Asn Val Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu
100 105 110
Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met
115 120 125
Phe Ile Asn Thr Ser
130
<210> 332
<211> 133
<212> PRT
<213> Homo Sapiens
<400> 332
Gln Gly Gln Asp Arg His Met Ile Arg Met Arg Gln Leu Ile Asp Ile
1 5 10 15
Val Asp Gln Leu Lys Asn Tyr Val Asn Asp Leu Val Pro Glu Phe Leu
20 25 30
Pro Ala Pro Glu Asp Val Glu Thr Asn Cys Glu Trp Ser Ala Phe Ser
35 40 45
Cys Phe Gln Lys Ala Gln Leu Lys Ser Ala Asn Thr Gly Asn Asn Glu
50 55 60
Arg Ile Ile Asn Val Ser Ile Lys Lys Leu Lys Arg Lys Pro Pro Ser
65 70 75 80
Thr Asn Ala Gly Arg Arg Gln Lys His Arg Leu Thr Cys Pro Ser Cys
85 90 95
Asp Ser Tyr Glu Lys Lys Pro Pro Lys Glu Phe Leu Glu Arg Phe Lys
100 105 110
Ser Leu Leu Gln Lys Met Ile His Gln His Leu Ser Ser Arg Thr His
115 120 125
Gly Ser Glu Asp Ser
130
<210> 333
<211> 127
<212> PRT
<213> Homo Sapiens
<400> 333
Ala Pro Ala Arg Ser Pro Ser Pro Ser Thr Gln Pro Trp Glu His Val
1 5 10 15
Asn Ala Ile Gln Glu Ala Arg Arg Leu Leu Asn Leu Ser Arg Asp Thr
20 25 30
Ala Ala Glu Met Asn Glu Thr Val Glu Val Ile Ser Glu Met Phe Asp
35 40 45
Leu Gln Glu Pro Thr Cys Leu Gln Thr Arg Leu Glu Leu Tyr Lys Gln
50 55 60
Gly Leu Arg Gly Ser Leu Thr Lys Leu Lys Gly Pro Leu Thr Met Met
65 70 75 80
Ala Ser His Tyr Lys Gln His Cys Pro Pro Thr Pro Glu Thr Ser Cys
85 90 95
Ala Thr Gln Ile Ile Thr Phe Glu Ser Phe Lys Glu Asn Leu Lys Asp
100 105 110
Phe Leu Leu Val Ile Pro Phe Asp Cys Trp Glu Pro Val Gln Glu
115 120 125
<210> 334
<211> 171
<212> PRT
<213> Homo Sapiens
<400> 334
Cys Asp Leu Pro Gln Asn His Gly Leu Leu Ser Arg Asn Thr Leu Val
1 5 10 15
Leu Leu His Gln Met Arg Arg Ile Ser Pro Phe Leu Cys Leu Lys Asp
20 25 30
Arg Arg Asp Phe Arg Phe Pro Gln Glu Met Val Lys Gly Ser Gln Leu
35 40 45
Gln Lys Ala His Val Met Ser Val Leu His Glu Met Leu Gln Gln Ile
50 55 60
Phe Ser Leu Phe His Thr Glu Arg Ser Ser Ala Ala Trp Asn Met Thr
65 70 75 80
Leu Leu Asp Gln Leu His Thr Glu Leu His Gln Gln Leu Gln His Leu
85 90 95
Glu Thr Cys Leu Leu Gln Val Val Gly Glu Gly Glu Ser Ala Gly Ala
100 105 110
Ile Ser Ser Pro Ala Leu Thr Leu Arg Arg Tyr Phe Gln Gly Ile Arg
115 120 125
Val Tyr Leu Lys Glu Lys Lys Tyr Ser Asp Cys Ala Trp Glu Val Val
130 135 140
Arg Met Glu Ile Met Lys Ser Leu Phe Leu Ser Thr Asn Met Gln Glu
145 150 155 160
Arg Leu Arg Ser Lys Asp Arg Asp Leu Gly Ser
165 170
<210> 335
<211> 177
<212> PRT
<213> Homo Sapiens
<400> 335
Gly Pro Gln Arg Glu Glu Phe Pro Arg Asp Leu Ser Leu Ile Ser Pro
1 5 10 15
Leu Ala Gln Ala Val Arg Ser Ser Ser Arg Thr Pro Ser Asp Lys Pro
20 25 30
Val Ala His Val Val Ala Asn Pro Gln Ala Glu Gly Gln Leu Gln Trp
35 40 45
Leu Asn Arg Arg Ala Asn Ala Leu Leu Ala Asn Gly Val Glu Leu Arg
50 55 60
Asp Asn Gln Leu Val Val Pro Ser Glu Gly Leu Tyr Leu Ile Tyr Ser
65 70 75 80
Gln Val Leu Phe Lys Gly Gln Gly Cys Pro Ser Thr His Val Leu Leu
85 90 95
Thr His Thr Ile Ser Arg Ile Ala Val Ser Tyr Gln Thr Lys Val Asn
100 105 110
Leu Leu Ser Ala Ile Lys Ser Pro Cys Gln Arg Glu Thr Pro Glu Gly
115 120 125
Ala Glu Ala Lys Pro Trp Tyr Glu Pro Ile Tyr Leu Gly Gly Val Phe
130 135 140
Gln Leu Glu Lys Gly Asp Arg Leu Ser Ala Glu Ile Asn Arg Pro Asp
145 150 155 160
Tyr Leu Asp Phe Ala Glu Ser Gly Gln Val Tyr Phe Gly Ile Ile Ala
165 170 175
Leu
<210> 336
<211> 197
<212> PRT
<213> Homo Sapiens
<400> 336
Arg Asn Leu Pro Val Ala Thr Pro Asp Pro Gly Met Phe Pro Cys Leu
1 5 10 15
His His Ser Gln Asn Leu Leu Arg Ala Val Ser Asn Met Leu Gln Lys
20 25 30
Ala Arg Gln Thr Leu Glu Phe Tyr Pro Cys Thr Ser Glu Glu Ile Asp
35 40 45
His Glu Asp Ile Thr Lys Asp Lys Thr Ser Thr Val Glu Ala Cys Leu
50 55 60
Pro Leu Glu Leu Thr Lys Asn Glu Ser Cys Leu Asn Ser Arg Glu Thr
65 70 75 80
Ser Phe Ile Thr Asn Gly Ser Cys Leu Ala Ser Arg Lys Thr Ser Phe
85 90 95
Met Met Ala Leu Cys Leu Ser Ser Ile Tyr Glu Asp Leu Lys Met Tyr
100 105 110
Gln Val Glu Phe Lys Thr Met Asn Ala Lys Leu Leu Met Asp Pro Lys
115 120 125
Arg Gln Ile Phe Leu Asp Gln Asn Met Leu Ala Val Ile Asp Glu Leu
130 135 140
Met Gln Ala Leu Asn Phe Asn Ser Glu Thr Val Pro Gln Lys Ser Ser
145 150 155 160
Leu Glu Glu Pro Asp Phe Tyr Lys Thr Lys Ile Lys Leu Cys Ile Leu
165 170 175
Leu His Ala Phe Arg Ile Arg Ala Val Thr Ile Asp Arg Val Met Ser
180 185 190
Tyr Leu Asn Ala Ser
195
<210> 337
<211> 306
<212> PRT
<213> Homo Sapiens
<400> 337
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser
305
<210> 338
<211> 103
<212> PRT
<213> Homo Sapiens
<400> 338
Thr Pro Val Val Arg Lys Gly Arg Cys Ser Cys Ile Ser Thr Asn Gln
1 5 10 15
Gly Thr Ile His Leu Gln Ser Leu Lys Asp Leu Lys Gln Phe Ala Pro
20 25 30
Ser Pro Ser Cys Glu Lys Ile Glu Ile Ile Ala Thr Leu Lys Asn Gly
35 40 45
Val Gln Thr Cys Leu Asn Pro Asp Ser Ala Asp Val Lys Glu Leu Ile
50 55 60
Lys Lys Trp Glu Lys Gln Val Ser Gln Lys Lys Lys Gln Lys Asn Gly
65 70 75 80
Lys Lys His Gln Lys Lys Lys Val Leu Lys Val Arg Lys Ser Gln Arg
85 90 95
Ser Arg Gln Lys Lys Thr Thr
100
<210> 339
<211> 77
<212> PRT
<213> Homo Sapiens
<400> 339
Val Pro Leu Ser Arg Thr Val Arg Cys Thr Cys Ile Ser Ile Ser Asn
1 5 10 15
Gln Pro Val Asn Pro Arg Ser Leu Glu Lys Leu Glu Ile Ile Pro Ala
20 25 30
Ser Gln Phe Cys Pro Arg Val Glu Ile Ile Ala Thr Met Lys Lys Lys
35 40 45
Gly Glu Lys Arg Cys Leu Asn Pro Glu Ser Lys Ala Ile Lys Asn Leu
50 55 60
Leu Lys Ala Val Ser Lys Glu Arg Ser Lys Arg Ser Pro
65 70 75
<210> 340
<211> 330
<212> PRT
<213> Homo Sapiens
<400> 340
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 341
<211> 996
<212> DNA
<213> Homo Sapiens
<400> 341
gccagcacca agggcccctc tgtgttccct ctggcccctt ccagcaagtc cacctctggc 60
ggaacagccg ctctgggctg cctcgtgaag gactacttcc ccgagcctgt gaccgtgtcc 120
tggaactctg gcgctctgac cagcggagtg cacaccttcc ctgctgtgct gcagtcctcc 180
ggcctgtact ccctgtcctc cgtcgtgacc gtgccttcca gctctctggg cacccagacc 240
tacatctgca acgtgaacca caagccctcc aacaccaagg tggacaagaa ggtggaaccc 300
aagtcctgcg acaagaccca cacctgtccc ccttgtcctg cccctgaact gctgggcgga 360
ccttccgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatctc ccggaccccc 420
gaagtgacct gcgtggtggt ggatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 480
tacgtggacg gcgtggaagt gcacaacgcc aagaccaagc ctagagagga acagtacaac 540
tccacctacc gggtggtgtc cgtgctgacc gtgctgcacc aggattggct gaacggcaaa 600
gagtacaagt gcaaggtgtc caacaaggcc ctgcctgccc ccatcgaaaa gaccatctcc 660
aaggccaagg gccagccccg ggaaccccag gtgtacacac tgccccctag cagggacgag 720
ctgaccaaga accaggtgtc cctgacctgt ctcgtgaaag gcttctaccc ctccgatatc 780
gccgtggaat gggagtccaa cggccagcct gagaacaact acaagaccac cccccctgtg 840
ctggactccg acggctcatt cttcctgtac agcaagctga cagtggacaa gtcccggtgg 900
cagcagggca acgtgttctc ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 960
cagaagtccc tgtccctgag ccccggcaag tgatga 996
<210> 342
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 342
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ile Arg Thr Gly Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Lys Gly Ser Gly Thr Tyr Gly Gly Trp Phe Asp Thr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
195 200 205
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
210 215 220
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Leu Ala Gly Ala
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
260 265 270
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
435 440 445
Gly Lys
450
<210> 343
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 343
Gly Phe Thr Phe Ser Asn Tyr Ala
1 5
<210> 344
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 344
Ile Ser Phe Ser Gly Gly Thr Thr
1 5
<210> 345
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 345
Ala Lys Asp Glu Ala Pro Ala Gly Ala Thr Phe Phe Asp Ser
1 5 10
<210> 346
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 346
Asn Tyr Ala Met Ser
1 5
<210> 347
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 347
Ala Ile Ser Phe Ser Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 348
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 348
Asp Glu Ala Pro Ala Gly Ala Thr Phe Phe Asp Ser
1 5 10
<210> 349
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 349
Glu Val Gln Leu Ala Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Thr Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Phe Ser Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Asn Ser Leu Arg Ala Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Ala Pro Ala Gly Ala Thr Phe Phe Asp Ser Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 350
<211> 364
<212> DNA
<213> Homo Sapiens
<400> 350
gaagtgcaac tggcggagtc tgggggaggc ttggtacagc cgggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc aactatgcca tgagttgggt ccgccagact 120
ccaggaaagg ggctggagtg ggtctcagct attagtttta gtggtggtac tacatactac 180
gctgactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ttgcacatga acagcctgag agccgatgac acggccgtat attactgtgc gaaagatgag 300
gcaccagctg gcgcaacctt ctttgactcc tggggccagg gaacgctggt caccgtctcc 360
tcag 364
<210> 351
<211> 448
<212> PRT
<213> Homo Sapiens
<400> 351
Glu Val Gln Leu Ala Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Thr Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Phe Ser Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Asn Ser Leu Arg Ala Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Ala Pro Ala Gly Ala Thr Phe Phe Asp Ser Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly
210 215 220
Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
260 265 270
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 352
<211> 1344
<212> DNA
<213> Homo Sapiens
<400> 352
gaagtgcaac tggcggagtc tgggggaggc ttggtacagc cgggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc aactatgcca tgagttgggt ccgccagact 120
ccaggaaagg ggctggagtg ggtctcagct attagtttta gtggtggtac tacatactac 180
gctgactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ttgcacatga acagcctgag agccgatgac acggccgtat attactgtgc gaaagatgag 300
gcaccagctg gcgcaacctt ctttgactcc tggggccagg gaacgctggt caccgtctcc 360
tcagccagca ccaagggccc ttccgtgttc cccctggccc cttgcagcag gagcacctcc 420
gaatccacag ctgccctggg ctgtctggtg aaggactact ttcccgagcc cgtgaccgtg 480
agctggaaca gcggcgctct gacatccggc gtccacacct ttcctgccgt cctgcagtcc 540
tccggcctct actccctgtc ctccgtggtg accgtgccta gctcctccct cggcaccaag 600
acctacacct gtaacgtgga ccacaaaccc tccaacacca aggtggacaa acgggtcgag 660
agcaagtacg gccctccctg ccctccttgt cctgcccccg agttcgaagg cggacccagc 720
gtgttcctgt tccctcctaa gcccaaggac accctcatga tcagccggac acccgaggtg 780
acctgcgtgg tggtggatgt gagccaggag gaccctgagg tccagttcaa ctggtatgtg 840
gatggcgtgg aggtgcacaa cgccaagaca aagccccggg aagagcagtt caactccacc 900
tacagggtgg tcagcgtgct gaccgtgctg catcaggact ggctgaacgg caaggagtac 960
aagtgcaagg tcagcaataa gggactgccc agcagcatcg agaagaccat ctccaaggct 1020
aaaggccagc cccgggaacc tcaggtgtac accctgcctc ccagccagga ggagatgacc 1080
aagaaccagg tgagcctgac ctgcctggtg aagggattct acccttccga catcgccgtg 1140
gagtgggagt ccaacggcca gcccgagaac aattataaga ccacccctcc cgtcctcgac 1200
agcgacggat ccttctttct gtactccagg ctgaccgtgg ataagtccag gtggcaggaa 1260
ggcaacgtgt tcagctgctc cgtgatgcac gaggccctgc acaatcacta cacccagaag 1320
tccctgagcc tgtccctggg aaag 1344
<210> 353
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 353
Gln Gly Ile Arg Arg Trp
1 5
<210> 354
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 354
Gly Ala Ser
1
<210> 355
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 355
Gln Gln Ala Asn Ser Phe Pro Ile Thr
1 5
<210> 356
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 356
Arg Ala Ser Gln Gly Ile Arg Arg Trp Leu Ala
1 5 10
<210> 357
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 357
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 358
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 358
Gln Gln Ala Asn Ser Phe Pro Ile Thr
1 5
<210> 359
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 359
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ile Ile Thr Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 360
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 360
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagg aggtggttag cctggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctctggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca tcattaccag tctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccgatcac cttcggccaa 300
gggacacgac tggagatcaa ac 322
<210> 361
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 361
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ile Ile Thr Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 362
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 362
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagg aggtggttag cctggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctctggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca tcattaccag tctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccgatcac cttcggccaa 300
gggacacgac tggagatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 363
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 363
Gly Tyr Thr Phe Ser Thr Phe Gly
1 5
<210> 364
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 364
Ile Ser Ala Tyr Asn Gly Asp Thr
1 5
<210> 365
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 365
Ala Arg Ser Ser Gly His Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 366
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 366
Gln Val Gln Val Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Thr Phe
20 25 30
Gly Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asp Thr Asn Tyr Ala Gln Asn Leu
50 55 60
Gln Gly Arg Val Ile Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly His Tyr Tyr Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 367
<211> 363
<212> DNA
<213> Homo Sapiens
<400> 367
caggttcagg tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta caccttttcc acctttggta tcacctgggt gcgacaggcc 120
cctggacaag ggcttgaatg gatgggatgg atcagcgctt acaatggtga cacaaactat 180
gcacagaatc tccagggcag agtcatcatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgttt attactgtgc gaggagcagt 300
ggccactact actactacgg tatggacgtc tggggccaag ggaccacggt caccgtctcc 360
tca 363
<210> 368
<211> 451
<212> PRT
<213> Homo Sapiens
<400> 368
Gln Val Gln Val Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Thr Phe
20 25 30
Gly Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asp Thr Asn Tyr Ala Gln Asn Leu
50 55 60
Gln Gly Arg Val Ile Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly His Tyr Tyr Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 369
<211> 1359
<212> DNA
<213> Homo Sapiens
<400> 369
caggttcagg tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta caccttttcc acctttggta tcacctgggt gcgacaggcc 120
cctggacaag ggcttgaatg gatgggatgg atcagcgctt acaatggtga cacaaactat 180
gcacagaatc tccagggcag agtcatcatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgttt attactgtgc gaggagcagt 300
ggccactact actactacgg tatggacgtc tggggccaag ggaccacggt caccgtctcc 360
tcagccagca ccaagggccc ctctgtgttc cctctggccc cttccagcaa gtccacctct 420
ggcggaacag ccgctctggg ctgcctcgtg aaggactact tccccgagcc tgtgaccgtg 480
tcctggaact ctggcgctct gaccagcgga gtgcacacct tccctgctgt gctgcagtcc 540
tccggcctgt actccctgtc ctccgtcgtg accgtgcctt ccagctctct gggcacccag 600
acctacatct gcaacgtgaa ccacaagccc tccaacacca aggtggacaa gaaggtggaa 660
cccaagtcct gcgacaagac ccacacctgt cccccttgtc ctgcccctga actgctgggc 720
ggaccttccg tgttcctgtt ccccccaaag cccaaggaca ccctgatgat ctcccggacc 780
cccgaagtga cctgcgtggt ggtggatgtg tcccacgagg accctgaagt gaagttcaat 840
tggtacgtgg acggcgtgga agtgcacaac gccaagacca agcctagaga ggaacagtac 900
aactccacct accgggtggt gtccgtgctg accgtgctgc accaggattg gctgaacggc 960
aaagagtaca agtgcaaggt gtccaacaag gccctgcctg cccccatcga aaagaccatc 1020
tccaaggcca agggccagcc ccgggaaccc caggtgtaca cactgccccc tagcagggac 1080
gagctgacca agaaccaggt gtccctgacc tgtctcgtga aaggcttcta cccctccgat 1140
atcgccgtgg aatgggagtc caacggccag cctgagaaca actacaagac caccccccct 1200
gtgctggact ccgacggctc attcttcctg tacagcaagc tgacagtgga caagtcccgg 1260
tggcagcagg gcaacgtgtt ctcctgctcc gtgatgcacg aggccctgca caaccactac 1320
acccagaagt ccctgtccct gagccccggc aagtgatga 1359
<210> 370
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 370
Gln Ser Leu Leu His Ser Asn Glu Tyr Asn Tyr
1 5 10
<210> 371
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 371
Leu Gly Ser
1
<210> 372
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 372
Met Gln Ser Leu Gln Thr Pro Leu Thr
1 5
<210> 373
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 373
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Glu Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Thr Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ser
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 374
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 374
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg aatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct ttttgggttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
accagagtgg aggctgagga tgttggaatt tattactgca tgcaatctct acaaactccg 300
ctcactttcg gcggagggac caaggtggag atcaaa 336
<210> 375
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 375
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Glu Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Thr Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ser
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 376
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 376
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg aatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct ttttgggttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
accagagtgg aggctgagga tgttggaatt tattactgca tgcaatctct acaaactccg 300
ctcactttcg gcggagggac caaggtggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 377
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 377
Gly Tyr Thr Phe Thr Ser Tyr Gly
1 5
<210> 378
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 378
Ile Ser Ala Tyr Asn Gly Asn Thr
1 5
<210> 379
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 379
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
1 5 10 15
Val
<210> 380
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 380
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 381
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 381
caggttcaac tggtgcagtc tggaggtgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt attactgtgc gagatctacg 300
tatttctatg gttcggggac cctctacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct ca 372
<210> 382
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 382
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 383
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 383
caggttcaac tggtgcagtc tggaggtgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt attactgtgc gagatctacg 300
tatttctatg gttcggggac cctctacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 384
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 384
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Tyr
1 5 10
<210> 385
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 385
Leu Gly Ser
1
<210> 386
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 386
Met Gln Ala Leu Gln Thr Pro Leu Ser
1 5
<210> 387
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 387
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 388
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 388
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaa 336
<210> 389
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 389
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 390
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 390
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 391
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 391
Gly Tyr Thr Phe Thr Ser Tyr Gly
1 5
<210> 392
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 392
Ile Ser Ala Tyr Asn Gly Asn Thr
1 5
<210> 393
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 393
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
1 5 10 15
Val
<210> 394
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 394
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 395
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 395
caggttcaac tggtgcagtc tggaggtgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt attactgtgc gagatctacg 300
tatttctatg gttcggggac cctctacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct ca 372
<210> 396
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 396
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Phe Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 397
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 397
caggttcaac tggtgcagtc tggaggtgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt attactgtgc gagatctacg 300
tatttctatg gttcggggac cctctacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 398
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 398
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Cys
1 5 10
<210> 399
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 399
Leu Gly Ser
1
<210> 400
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 400
Met Gln Ala Leu Gln Thr Pro Cys Ser
1 5
<210> 401
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 401
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 402
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 402
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaa 336
<210> 403
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 403
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 404
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 404
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 405
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 405
Gly Val Thr Phe Asp Asp Tyr Gly
1 5
<210> 406
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 406
Ile Asn Trp Asn Gly Gly Asp Thr
1 5
<210> 407
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 407
Ala Arg Asp Phe Tyr Gly Ser Gly Ser Tyr Tyr His Val Pro Phe Asp
1 5 10 15
Tyr
<210> 408
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 408
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Val Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Asn Gly Gly Asp Thr Asp Tyr Ser Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Phe Tyr Gly Ser Gly Ser Tyr Tyr His Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Ile Leu Val Thr Val Ser Ser
115 120
<210> 409
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 409
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgtag cctctggagt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggartg ggtctctggt attaattgga atggtggcga cacagattat 180
tcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctacaaatga atagtctgag agccgaggac acggccttgt attactgtgc gagggatttc 300
tatggttcgg ggagttatta tcacgttcct tttgactact ggggccaggg aatcctggtc 360
accgtctcct ca 372
<210> 410
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 410
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Val Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Asn Gly Gly Asp Thr Asp Tyr Ser Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Phe Tyr Gly Ser Gly Ser Tyr Tyr His Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Ile Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 411
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 411
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgtag cctctggagt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggartg ggtctctggt attaattgga atggtggcga cacagattat 180
tcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctacaaatga atagtctgag agccgaggac acggccttgt attactgtgc gagggatttc 300
tatggttcgg ggagttatta tcacgttcct tttgactact ggggccaggg aatcctggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 412
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 412
Gln Ser Val Ser Arg Ser Tyr
1 5
<210> 413
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 413
Gly Ala Ser
1
<210> 414
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 414
His Gln Tyr Asp Met Ser Pro Phe Thr
1 5
<210> 415
<211> 108
<212> PRT
<213> Homo Sapiens
<400> 415
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Arg Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Asp Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Asp Met Ser Pro
85 90 95
Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 416
<211> 324
<212> DNA
<213> Homo Sapiens
<400> 416
gaaattgtgt tgacgcagtc tccagggacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agaagctact tagcctggta ccagcagaaa 120
cgtggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcgatgg gtctgggaca gacttcactc tctccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcac cagtatgata tgtcaccatt cactttcggc 300
cctgggacca aagtggatat caaa 324
<210> 417
<211> 215
<212> PRT
<213> Homo Sapiens
<400> 417
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Arg Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Asp Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Asp Met Ser Pro
85 90 95
Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 418
<211> 645
<212> DNA
<213> Homo Sapiens
<400> 418
gaaattgtgt tgacgcagtc tccagggacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agaagctact tagcctggta ccagcagaaa 120
cgtggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcgatgg gtctgggaca gacttcactc tctccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcac cagtatgata tgtcaccatt cactttcggc 300
cctgggacca aagtggatat caaacgtacg gtggccgctc cctccgtgtt catcttccca 360
ccttccgacg agcagctgaa gtccggcacc gcttctgtcg tgtgcctgct gaacaacttc 420
tacccccgcg aggccaaggt gcagtggaag gtggacaacg ccctgcagtc cggcaactcc 480
caggaatccg tgaccgagca ggactccaag gacagcacct actccctgtc ctccaccctg 540
accctgtcca aggccgacta cgagaagcac aaggtgtacg cctgcgaagt gacccaccag 600
ggcctgtcta gccccgtgac caagtctttc aaccggggcg agtgt 645
<210> 419
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 419
Gly Leu Thr Phe Asp Asp Tyr Gly
1 5
<210> 420
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 420
Ile Asn Trp Asn Gly Asp Asn Thr
1 5
<210> 421
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 421
Ala Arg Asp Tyr Tyr Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
1 5 10 15
Tyr
<210> 422
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 422
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Asn Gly Asp Asn Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 423
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 423
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgcag cctctggact cacctttgat gattatggca tgagctgggt ccgccaagtt 120
ccagggaagg ggctggagtg ggtctctggt attaattgga atggtgataa cacagattat 180
gcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agccgaggac acggccttgt attactgtgc gagggattac 300
tatggttcgg ggagttatta taacgttcct tttgactact ggggccaggg aaccctggtc 360
accgtctcct ca 372
<210> 424
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 424
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Asn Gly Asp Asn Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 425
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 425
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgcag cctctggact cacctttgat gattatggca tgagctgggt ccgccaagtt 120
ccagggaagg ggctggagtg ggtctctggt attaattgga atggtgataa cacagattat 180
gcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agccgaggac acggccttgt attactgtgc gagggattac 300
tatggttcgg ggagttatta taacgttcct tttgactact ggggccaggg aaccctggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 426
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 426
Gln Ser Val Ser Ser Ser Tyr
1 5
<210> 427
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 427
Gly Ala Ser
1
<210> 428
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 428
Gln Gln Tyr Gly Ser Ser Pro Phe
1 5
<210> 429
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 429
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Phe Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 430
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 430
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatatat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag aagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gttcaccatt cttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 431
<211> 323
<212> DNA
<213> Homo Sapiens
<400> 431
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatatat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag aagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gttcaccatt cacttcggcc 300
ctgggaccaa agtggatatc aaa 323
<210> 432
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 432
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Phe Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 433
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 433
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatatat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag aagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gttcaccatt cttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 434
<211> 644
<212> DNA
<213> Homo Sapiens
<400> 434
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatatat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag aagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gttcaccatt cacttcggcc 300
ctgggaccaa agtggatatc aaacgtacgg tggccgctcc ctccgtgttc atcttcccac 360
cttccgacga gcagctgaag tccggcaccg cttctgtcgt gtgcctgctg aacaacttct 420
acccccgcga ggccaaggtg cagtggaagg tggacaacgc cctgcagtcc ggcaactccc 480
aggaatccgt gaccgagcag gactccaagg acagcaccta ctccctgtcc tccaccctga 540
ccctgtccaa ggccgactac gagaagcaca aggtgtacgc ctgcgaagtg acccaccagg 600
gcctgtctag ccccgtgacc aagtctttca accggggcga gtgt 644
<210> 435
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 435
Gly Tyr Thr Phe Asn Ser Tyr Gly
1 5
<210> 436
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 436
Ile Ser Val His Asn Gly Asn Thr
1 5
<210> 437
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 437
Ala Arg Ala Gly Tyr Asp Ile Leu Thr Asp Phe Ser Asp Ala Phe Asp
1 5 10 15
Ile
<210> 438
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 438
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Asn Ser Tyr
20 25 30
Gly Ile Ile Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Val His Asn Gly Asn Thr Asn Cys Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Thr Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Gly Tyr Asp Ile Leu Thr Asp Phe Ser Asp Ala Phe Asp
100 105 110
Ile Trp Gly His Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 439
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 439
caggttcagt tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttaat agttatggta tcatctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgttc acaatggtaa cacaaactgt 180
gcacagaagc tccagggtag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag aactgacgac acggccgtgt attactgtgc gagagcgggt 300
tacgatattt tgactgattt ttccgatgct tttgatatct ggggccacgg gacaatggtc 360
accgtctctt ca 372
<210> 440
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 440
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Asn Ser Tyr
20 25 30
Gly Ile Ile Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Val His Asn Gly Asn Thr Asn Cys Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Thr Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Gly Tyr Asp Ile Leu Thr Asp Phe Ser Asp Ala Phe Asp
100 105 110
Ile Trp Gly His Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 441
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 441
caggttcagt tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttaat agttatggta tcatctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgttc acaatggtaa cacaaactgt 180
gcacagaagc tccagggtag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag aactgacgac acggccgtgt attactgtgc gagagcgggt 300
tacgatattt tgactgattt ttccgatgct tttgatatct ggggccacgg gacaatggtc 360
accgtctctt cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 442
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 442
Gln Asn Ile Asn Asn Phe
1 5
<210> 443
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 443
Ala Ala Ser
1
<210> 444
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 444
Gln Gln Ser Tyr Gly Ile Pro Trp
1 5
<210> 445
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 445
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Asn Asn Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Glu Gly Lys Gly Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Arg Gly Ile Pro Ser Thr Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Ile Cys Gln Gln Ser Tyr Gly Ile Pro Trp
85 90 95
Val Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 446
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 446
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gaacattaat aactttttaa attggtatca gcagaaagaa 120
gggaaaggcc ctaagctcct gatctatgca gcatccagtt tgcaaagagg gataccatca 180
acgttcagtg gcagtggatc tgggacagac ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacat ctgtcaacag agctacggta tcccgtgggt cggccaaggg 300
accaaggtgg aaatcaaa 318
<210> 447
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 447
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Asn Asn Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Glu Gly Lys Gly Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Arg Gly Ile Pro Ser Thr Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Ile Cys Gln Gln Ser Tyr Gly Ile Pro Trp
85 90 95
Val Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 448
<211> 639
<212> DNA
<213> Homo Sapiens
<400> 448
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gaacattaat aactttttaa attggtatca gcagaaagaa 120
gggaaaggcc ctaagctcct gatctatgca gcatccagtt tgcaaagagg gataccatca 180
acgttcagtg gcagtggatc tgggacagac ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacat ctgtcaacag agctacggta tcccgtgggt cggccaaggg 300
accaaggtgg aaatcaaacg tacggtggcc gctccctccg tgttcatctt cccaccttcc 360
gacgagcagc tgaagtccgg caccgcttct gtcgtgtgcc tgctgaacaa cttctacccc 420
cgcgaggcca aggtgcagtg gaaggtggac aacgccctgc agtccggcaa ctcccaggaa 480
tccgtgaccg agcaggactc caaggacagc acctactccc tgtcctccac cctgaccctg 540
tccaaggccg actacgagaa gcacaaggtg tacgcctgcg aagtgaccca ccagggcctg 600
tctagccccg tgaccaagtc tttcaaccgg ggcgagtgt 639
<210> 449
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 449
Gly Phe Thr Phe Ser Asp Tyr Phe
1 5
<210> 450
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 450
Ile Ser Ser Ser Gly Ser Thr Ile
1 5
<210> 451
<211> 21
<212> PRT
<213> Homo Sapiens
<400> 451
Ala Arg Asp His Tyr Asp Gly Ser Gly Ile Tyr Pro Leu Tyr Tyr Tyr
1 5 10 15
Tyr Gly Leu Asp Val
20
<210> 452
<211> 128
<212> PRT
<213> Homo Sapiens
<400> 452
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Phe Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Tyr Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp His Tyr Asp Gly Ser Gly Ile Tyr Pro Leu Tyr Tyr Tyr
100 105 110
Tyr Gly Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 453
<211> 384
<212> DNA
<213> Homo Sapiens
<400> 453
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactacttca tgagctggat ccgccaggcg 120
ccagggaagg ggctggagtg gatttcatac attagttcta gtggtagtac catatactac 180
gcagactctg tgaggggccg attcaccatc tccagggaca acgccaagta ctcactgtat 240
ctgcaaatga acagcctgag atccgaggac acggccgtgt attactgtgc gagagatcac 300
tacgatggtt cggggattta tcccctctac tactattacg gtttggacgt ctggggccag 360
gggaccacgg tcaccgtctc ctca 384
<210> 454
<211> 458
<212> PRT
<213> Homo Sapiens
<400> 454
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Phe Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Tyr Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp His Tyr Asp Gly Ser Gly Ile Tyr Pro Leu Tyr Tyr Tyr
100 105 110
Tyr Gly Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
130 135 140
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
195 200 205
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
225 230 235 240
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
245 250 255
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
260 265 270
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
275 280 285
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
290 295 300
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
305 310 315 320
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
325 330 335
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
340 345 350
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
355 360 365
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
370 375 380
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
385 390 395 400
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
405 410 415
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
420 425 430
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
435 440 445
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 455
<211> 1380
<212> DNA
<213> Homo Sapiens
<400> 455
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactacttca tgagctggat ccgccaggcg 120
ccagggaagg ggctggagtg gatttcatac attagttcta gtggtagtac catatactac 180
gcagactctg tgaggggccg attcaccatc tccagggaca acgccaagta ctcactgtat 240
ctgcaaatga acagcctgag atccgaggac acggccgtgt attactgtgc gagagatcac 300
tacgatggtt cggggattta tcccctctac tactattacg gtttggacgt ctggggccag 360
gggaccacgg tcaccgtctc ctcagccagc accaagggcc cctctgtgtt ccctctggcc 420
ccttccagca agtccacctc tggcggaaca gccgctctgg gctgcctcgt gaaggactac 480
ttccccgagc ctgtgaccgt gtcctggaac tctggcgctc tgaccagcgg agtgcacacc 540
ttccctgctg tgctgcagtc ctccggcctg tactccctgt cctccgtcgt gaccgtgcct 600
tccagctctc tgggcaccca gacctacatc tgcaacgtga accacaagcc ctccaacacc 660
aaggtggaca agaaggtgga acccaagtcc tgcgacaaga cccacacctg tcccccttgt 720
cctgcccctg aactgctggg cggaccttcc gtgttcctgt tccccccaaa gcccaaggac 780
accctgatga tctcccggac ccccgaagtg acctgcgtgg tggtggatgt gtcccacgag 840
gaccctgaag tgaagttcaa ttggtacgtg gacggcgtgg aagtgcacaa cgccaagacc 900
aagcctagag aggaacagta caactccacc taccgggtgg tgtccgtgct gaccgtgctg 960
caccaggatt ggctgaacgg caaagagtac aagtgcaagg tgtccaacaa ggccctgcct 1020
gcccccatcg aaaagaccat ctccaaggcc aagggccagc cccgggaacc ccaggtgtac 1080
acactgcccc ctagcaggga cgagctgacc aagaaccagg tgtccctgac ctgtctcgtg 1140
aaaggcttct acccctccga tatcgccgtg gaatgggagt ccaacggcca gcctgagaac 1200
aactacaaga ccaccccccc tgtgctggac tccgacggct cattcttcct gtacagcaag 1260
ctgacagtgg acaagtcccg gtggcagcag ggcaacgtgt tctcctgctc cgtgatgcac 1320
gaggccctgc acaaccacta cacccagaag tccctgtccc tgagccccgg caagtgatga 1380
<210> 456
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 456
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 457
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 457
Leu Gly Ser
1
<210> 458
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 458
Met Gln Ala Leu Gln Thr Pro Arg Ser
1 5
<210> 459
<211> 111
<212> PRT
<213> Homo Sapiens
<400> 459
Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly Glu
1 5 10 15
Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser Asn
20 25 30
Gly Tyr Asn Tyr Leu Asp Tyr Tyr Leu Gln Lys Pro Gly Gln Ser Pro
35 40 45
Gln Leu Leu Ile Tyr Leu Gly Ser Tyr Arg Ala Ser Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
65 70 75 80
Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala Leu
85 90 95
Gln Thr Pro Arg Ser Phe Gly Gln Gly Thr Thr Leu Glu Ile Lys
100 105 110
<210> 460
<211> 333
<212> DNA
<213> Homo Sapiens
<400> 460
attgtgatga ctcagtctcc actctcccta cccgtcaccc ctggagagcc ggcctccatc 60
tcctgcaggt ctagtcagag cctcctgcat agtaatggat acaactattt ggattattac 120
ctgcagaagc cagggcagtc tccacagctc ctgatctatt tgggttctta tcgggcctcc 180
ggggtccctg acaggttcag tggcagtgga tcaggcacag attttacact gaaaatcagc 240
agagtggagg ctgaggatgt tggggtttat tactgcatgc aagctctaca aactcctcgc 300
agttttggcc aggggaccac gctggagatc aaa 333
<210> 461
<211> 218
<212> PRT
<213> Homo Sapiens
<400> 461
Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly Glu
1 5 10 15
Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser Asn
20 25 30
Gly Tyr Asn Tyr Leu Asp Tyr Tyr Leu Gln Lys Pro Gly Gln Ser Pro
35 40 45
Gln Leu Leu Ile Tyr Leu Gly Ser Tyr Arg Ala Ser Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
65 70 75 80
Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala Leu
85 90 95
Gln Thr Pro Arg Ser Phe Gly Gln Gly Thr Thr Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 462
<211> 654
<212> DNA
<213> Homo Sapiens
<400> 462
attgtgatga ctcagtctcc actctcccta cccgtcaccc ctggagagcc ggcctccatc 60
tcctgcaggt ctagtcagag cctcctgcat agtaatggat acaactattt ggattattac 120
ctgcagaagc cagggcagtc tccacagctc ctgatctatt tgggttctta tcgggcctcc 180
ggggtccctg acaggttcag tggcagtgga tcaggcacag attttacact gaaaatcagc 240
agagtggagg ctgaggatgt tggggtttat tactgcatgc aagctctaca aactcctcgc 300
agttttggcc aggggaccac gctggagatc aaacgtacgg tggccgctcc ctccgtgttc 360
atcttcccac cttccgacga gcagctgaag tccggcaccg cttctgtcgt gtgcctgctg 420
aacaacttct acccccgcga ggccaaggtg cagtggaagg tggacaacgc cctgcagtcc 480
ggcaactccc aggaatccgt gaccgagcag gactccaagg acagcaccta ctccctgtcc 540
tccaccctga ccctgtccaa ggccgactac gagaagcaca aggtgtacgc ctgcgaagtg 600
acccaccagg gcctgtctag ccccgtgacc aagtctttca accggggcga gtgt 654
<210> 463
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 463
Gly Phe Ser Leu Ser Thr Thr Gly Val Gly
1 5 10
<210> 464
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 464
Ile Tyr Trp Asp Asp Asp Lys
1 5
<210> 465
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 465
Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp Val
1 5 10 15
<210> 466
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 466
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Thr
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Val Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Phe
85 90 95
Cys Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 467
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 467
cagatcacct tgaaggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ctcactcagc actactggag tgggtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcagtcattt attgggatga tgataagcgc 180
tacagcccat ctctgaagag cagactcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacat ggaccctgtg gacacagcca catatttctg tacacacgga 300
tatggttcgg cgagttatta ccactacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct ca 372
<210> 468
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 468
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Thr
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Val Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Phe
85 90 95
Cys Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 469
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 469
cagatcacct tgaaggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ctcactcagc actactggag tgggtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcagtcattt attgggatga tgataagcgc 180
tacagcccat ctctgaagag cagactcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacat ggaccctgtg gacacagcca catatttctg tacacacgga 300
tatggttcgg cgagttatta ccactacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 470
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 470
Gln Ser Val Thr Asn Tyr
1 5
<210> 471
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 471
Asp Ala Ser
1
<210> 472
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 472
Gln His Arg Ser Asn Trp Pro Leu Thr
1 5
<210> 473
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 473
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Asn Tyr
20 25 30
Leu Ala Trp His Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 474
<211> 322
<212> DNA
<213> Homo Sapiens
<400> 474
gaaattgtat tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttacc aactacttag cctggcacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcac cgtagcaact ggcctctcac tttcggcgga 300
gggaccaagg tggagatcaa ac 322
<210> 475
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 475
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Asn Tyr
20 25 30
Leu Ala Trp His Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 476
<211> 643
<212> DNA
<213> Homo Sapiens
<400> 476
gaaattgtat tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttacc aactacttag cctggcacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcac cgtagcaact ggcctctcac tttcggcgga 300
gggaccaagg tggagatcaa accgtacggt ggccgctccc tccgtgttca tcttcccacc 360
ttccgacgag cagctgaagt ccggcaccgc ttctgtcgtg tgcctgctga acaacttcta 420
cccccgcgag gccaaggtgc agtggaaggt ggacaacgcc ctgcagtccg gcaactccca 480
ggaatccgtg accgagcagg actccaagga cagcacctac tccctgtcct ccaccctgac 540
cctgtccaag gccgactacg agaagcacaa ggtgtacgcc tgcgaagtga cccaccaggg 600
cctgtctagc cccgtgacca agtctttcaa ccggggcgag tgt 643
<210> 477
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 477
Gly Phe Ser Leu Ser Thr Ser Gly Val Gly
1 5 10
<210> 478
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 478
Ile Tyr Trp Asp Asp Asp Lys
1 5
<210> 479
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 479
Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp Val
1 5 10 15
<210> 480
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 480
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Val Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Phe
85 90 95
Cys Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 481
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 481
cagatcacct tgaaggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ctcactcagc actagtggag tgggtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcagtcattt attgggatga tgataagcgc 180
tacagcccat ctctgaagag caggctcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacat ggaccctgtg gacacagcca catatttctg tacacacgga 300
tatggttcgg cgagttatta ccactacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct ca 372
<210> 482
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 482
Gln Ile Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Val Ile Tyr Trp Asp Asp Asp Lys Arg Tyr Ser Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Thr Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Phe
85 90 95
Cys Thr His Gly Tyr Gly Ser Ala Ser Tyr Tyr His Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 483
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 483
cagatcacct tgaaggagtc tggtcctacg ctggtgaaac ccacacagac cctcacgctg 60
acctgcacct tctctgggtt ctcactcagc actagtggag tgggtgtggg ctggatccgt 120
cagcccccag gaaaggccct ggagtggctt gcagtcattt attgggatga tgataagcgc 180
tacagcccat ctctgaagag caggctcacc atcaccaagg acacctccaa aaaccaggtg 240
gtccttacaa tgaccaacat ggaccctgtg gacacagcca catatttctg tacacacgga 300
tatggttcgg cgagttatta ccactacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 484
<211> 6
<212> PRT
<213> Homo Sapiens
<400> 484
Gln Ser Val Thr Asn Tyr
1 5
<210> 485
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 485
Asp Ala Ser
1
<210> 486
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 486
Gln Gln Arg Ser Asn Trp Pro Leu Thr
1 5
<210> 487
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 487
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Asn Tyr
20 25 30
Leu Ala Trp His Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 488
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 488
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttacc aactacttag cctggcacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag cgtagcaact ggcctctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 489
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 489
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Asn Tyr
20 25 30
Leu Ala Trp His Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 490
<211> 642
<212> DNA
<213> Homo Sapiens
<400> 490
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttacc aactacttag cctggcacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag cgtagcaact ggcctctcac tttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gccgctccct ccgtgttcat cttcccacct 360
tccgacgagc agctgaagtc cggcaccgct tctgtcgtgt gcctgctgaa caacttctac 420
ccccgcgagg ccaaggtgca gtggaaggtg gacaacgccc tgcagtccgg caactcccag 480
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgtcctc caccctgacc 540
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gcgaagtgac ccaccagggc 600
ctgtctagcc ccgtgaccaa gtctttcaac cggggcgagt gt 642
<210> 491
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 491
Gly Phe Thr Phe Ser Asp Tyr Tyr
1 5
<210> 492
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 492
Ile Ser Ser Ser Gly Ser Thr Ile
1 5
<210> 493
<211> 20
<212> PRT
<213> Homo Sapiens
<400> 493
Ala Arg Asp Phe Tyr Asp Ile Leu Thr Asp Ser Pro Tyr Phe Tyr Tyr
1 5 10 15
Gly Val Asp Val
20
<210> 494
<211> 127
<212> PRT
<213> Homo Sapiens
<400> 494
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Ile Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Phe Tyr Asp Ile Leu Thr Asp Ser Pro Tyr Phe Tyr Tyr
100 105 110
Gly Val Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 495
<211> 381
<212> DNA
<213> Homo Sapiens
<400> 495
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactactaca tgagctggat ccgccaggct 120
ccagggaagg ggctggagtg ggtttcatac attagtagta gtggtagtac catatactac 180
gcagactctg tgaagggccg attcaccatc tccagggaca acgccaagaa ctcactgtat 240
ctgcaaatta acagcctgag agccgaggac acggccgtgt attactgtgc gagagatttt 300
tacgatattt tgactgatag tccgtacttc tactacggtg tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc a 381
<210> 496
<211> 457
<212> PRT
<213> Homo Sapiens
<400> 496
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Ile Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Phe Tyr Asp Ile Leu Thr Asp Ser Pro Tyr Phe Tyr Tyr
100 105 110
Gly Val Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 497
<211> 1377
<212> DNA
<213> Homo Sapiens
<400> 497
caggtgcagc tggtggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactactaca tgagctggat ccgccaggct 120
ccagggaagg ggctggagtg ggtttcatac attagtagta gtggtagtac catatactac 180
gcagactctg tgaagggccg attcaccatc tccagggaca acgccaagaa ctcactgtat 240
ctgcaaatta acagcctgag agccgaggac acggccgtgt attactgtgc gagagatttt 300
tacgatattt tgactgatag tccgtacttc tactacggtg tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc agccagcacc aagggcccct ctgtgttccc tctggcccct 420
tccagcaagt ccacctctgg cggaacagcc gctctgggct gcctcgtgaa ggactacttc 480
cccgagcctg tgaccgtgtc ctggaactct ggcgctctga ccagcggagt gcacaccttc 540
cctgctgtgc tgcagtcctc cggcctgtac tccctgtcct ccgtcgtgac cgtgccttcc 600
agctctctgg gcacccagac ctacatctgc aacgtgaacc acaagccctc caacaccaag 660
gtggacaaga aggtggaacc caagtcctgc gacaagaccc acacctgtcc cccttgtcct 720
gcccctgaac tgctgggcgg accttccgtg ttcctgttcc ccccaaagcc caaggacacc 780
ctgatgatct cccggacccc cgaagtgacc tgcgtggtgg tggatgtgtc ccacgaggac 840
cctgaagtga agttcaattg gtacgtggac ggcgtggaag tgcacaacgc caagaccaag 900
cctagagagg aacagtacaa ctccacctac cgggtggtgt ccgtgctgac cgtgctgcac 960
caggattggc tgaacggcaa agagtacaag tgcaaggtgt ccaacaaggc cctgcctgcc 1020
cccatcgaaa agaccatctc caaggccaag ggccagcccc gggaacccca ggtgtacaca 1080
ctgcccccta gcagggacga gctgaccaag aaccaggtgt ccctgacctg tctcgtgaaa 1140
ggcttctacc cctccgatat cgccgtggaa tgggagtcca acggccagcc tgagaacaac 1200
tacaagacca ccccccctgt gctggactcc gacggctcat tcttcctgta cagcaagctg 1260
acagtggaca agtcccggtg gcagcagggc aacgtgttct cctgctccgt gatgcacgag 1320
gccctgcaca accactacac ccagaagtcc ctgtccctga gccccggcaa gtgatga 1377
<210> 498
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 498
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 499
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 499
Leu Gly Ser
1
<210> 500
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 500
Met Gln Ala Leu Gln Thr Pro Arg Thr
1 5
<210> 501
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 501
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 502
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 502
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactcct 300
cggacgttcg gccaagggac caaggtggaa atcaaa 336
<210> 503
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 503
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 504
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 504
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactcct 300
cggacgttcg gccaagggac caaggtggaa atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 505
<211> 239
<212> PRT
<213> Homo Sapiens
<400> 505
Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val Glu Tyr Gly Ser
1 5 10 15
Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys Gln Leu Asp Leu
20 25 30
Ala Ala Leu Ile Val Tyr Trp Glu Met Glu Asp Lys Asn Ile Ile Gln
35 40 45
Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His Ser Ser Tyr Arg
50 55 60
Gln Arg Ala Arg Leu Leu Lys Asp Gln Leu Ser Leu Gly Asn Ala Ala
65 70 75 80
Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr Arg Cys
85 90 95
Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Val Lys Val
100 105 110
Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu Val Val Asp Pro
115 120 125
Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu Gly Tyr Pro Lys
130 135 140
Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val Leu Ser Gly Lys
145 150 155 160
Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu Phe Asn Val Thr
165 170 175
Ser Thr Leu Arg Ile Asn Thr Thr Thr Asn Glu Ile Phe Tyr Cys Thr
180 185 190
Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala Glu Leu Val Ile
195 200 205
Pro Glu Leu Pro Leu Ala His Pro Pro Asn Glu Arg Thr Ile Glu Gly
210 215 220
Arg Asp Tyr Lys Asp Asp Asp Asp Lys His His His His His His
225 230 235
<210> 506
<211> 179
<212> PRT
<213> Homo Sapiens
<400> 506
Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val Gln Gln Phe Lys
20 25 30
Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Thr Val Ser Ile Lys Ser Leu Lys Phe Cys His
50 55 60
Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Tyr Asn Leu Asp
65 70 75 80
His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu His Ile Tyr Glu
100 105 110
Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro Ile Gly Cys Ala
115 120 125
Ala Phe Val Val Val Cys Ile Leu Gly Cys Ile Leu Ile Cys Trp Leu
130 135 140
Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro Asn Gly Glu Tyr
145 150 155 160
Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser Arg Leu Thr Asp
165 170 175
Val Thr Leu
<210> 507
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 507
Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val Gln Gln Phe Lys
20 25 30
Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Thr Val Ser Ile Lys Ser Leu Lys Phe Cys His
50 55 60
Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Tyr Asn Leu Asp
65 70 75 80
His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu His Ile Tyr Glu
100 105 110
Ser Gln Leu Cys Cys Gln Leu Lys Phe
115 120
<210> 508
<211> 199
<212> PRT
<213> Homo Sapiens
<400> 508
Met Lys Ser Gly Leu Trp Tyr Phe Phe Leu Phe Cys Leu Arg Ile Lys
1 5 10 15
Val Leu Thr Gly Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile
20 25 30
Phe His Asn Gly Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val
35 40 45
Gln Gln Phe Lys Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp
50 55 60
Leu Thr Lys Thr Lys Gly Ser Gly Asn Thr Val Ser Ile Lys Ser Leu
65 70 75 80
Lys Phe Cys His Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu
85 90 95
Tyr Asn Leu Asp His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser
100 105 110
Ile Phe Asp Pro Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu
115 120 125
His Ile Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro
130 135 140
Ile Gly Cys Ala Ala Phe Val Val Val Cys Ile Leu Gly Cys Ile Leu
145 150 155 160
Ile Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro
165 170 175
Asn Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser
180 185 190
Arg Leu Thr Asp Val Thr Leu
195
<210> 509
<211> 33
<212> PRT
<213> Homo Sapiens
<400> 509
Lys Tyr Ser Ser Ser Val His Asp Pro Asn Gly Glu Tyr Met Phe Met
1 5 10 15
Arg Ala Val Asn Thr Ala Lys Lys Ser Arg Leu Thr Asp Val Thr Leu
20 25 30
Met
<210> 510
<211> 128
<212> PRT
<213> Mus Musculus
<400> 510
Glu Ile Asn Gly Ser Ala Asp His Arg Met Phe Ser Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Ser Cys Lys Tyr Pro Glu Thr Val Gln Gln Leu Lys
20 25 30
Met Arg Leu Phe Arg Glu Arg Glu Val Leu Cys Glu Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Ala Val Ser Ile Lys Asn Pro Met Leu Cys Leu
50 55 60
Tyr His Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Asn Asn Pro Asp
65 70 75 80
Ser Ser Gln Gly Ser Tyr Tyr Phe Cys Ser Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Gln Glu Arg Asn Leu Ser Gly Gly Tyr Leu His Ile Tyr
100 105 110
Glu Ser Gln Leu Cys Cys Gln Leu Lys Ile Val Val Gln Val Thr Glu
115 120 125
<210> 511
<211> 121
<212> PRT
<213> Mus Musculus
<400> 511
Glu Ile Asn Gly Ser Ala Asp His Arg Met Phe Ser Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Ser Cys Lys Tyr Pro Glu Thr Val Gln Gln Leu Lys
20 25 30
Met Arg Leu Phe Arg Glu Arg Glu Val Leu Cys Glu Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Ala Val Ser Ile Lys Asn Pro Met Leu Cys Leu
50 55 60
Tyr His Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Asn Asn Pro Asp
65 70 75 80
Ser Ser Gln Gly Ser Tyr Tyr Phe Cys Ser Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Gln Glu Arg Asn Leu Ser Gly Gly Tyr Leu His Ile Tyr
100 105 110
Glu Ser Gln Leu Cys Cys Gln Leu Lys
115 120
<210> 512
<211> 147
<212> PRT
<213> Mus Musculus
<400> 512
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Ile Asn Gly Ser Ala Asp His Arg Met Phe Ser Phe
20 25 30
His Asn Gly Gly Val Gln Ile Ser Cys Lys Tyr Pro Glu Thr Val Gln
35 40 45
Gln Leu Lys Met Arg Leu Phe Arg Glu Arg Glu Val Leu Cys Glu Leu
50 55 60
Thr Lys Thr Lys Gly Ser Gly Asn Ala Val Ser Ile Lys Asn Pro Met
65 70 75 80
Leu Cys Leu Tyr His Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Asn
85 90 95
Asn Pro Asp Ser Ser Gln Gly Ser Tyr Tyr Phe Cys Ser Leu Ser Ile
100 105 110
Phe Asp Pro Pro Pro Phe Gln Glu Arg Asn Leu Ser Gly Gly Tyr Leu
115 120 125
His Ile Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Ile Val Val Gln
130 135 140
Val Thr Glu
145
<210> 513
<211> 199
<212> PRT
<213> Cynomologus
<400> 513
Met Lys Ser Gly Leu Trp Tyr Phe Phe Leu Phe Cys Leu His Met Lys
1 5 10 15
Val Leu Thr Gly Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile
20 25 30
Phe His Asn Gly Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val
35 40 45
Gln Gln Phe Lys Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp
50 55 60
Leu Thr Lys Thr Lys Gly Ser Gly Asn Lys Val Ser Ile Lys Ser Leu
65 70 75 80
Lys Phe Cys His Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu
85 90 95
Tyr Asn Leu Asp Arg Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser
100 105 110
Ile Phe Asp Pro Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu
115 120 125
His Ile Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro
130 135 140
Ile Gly Cys Ala Thr Phe Val Val Val Cys Ile Phe Gly Cys Ile Leu
145 150 155 160
Ile Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Thr Val His Asp Pro
165 170 175
Asn Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser
180 185 190
Arg Leu Thr Gly Thr Thr Pro
195
<210> 514
<211> 120
<212> PRT
<213> Cynomologus
<400> 514
Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val Gln Gln Phe Lys
20 25 30
Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Lys Val Ser Ile Lys Ser Leu Lys Phe Cys His
50 55 60
Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Tyr Asn Leu Asp
65 70 75 80
Arg Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu His Ile Tyr Glu
100 105 110
Ser Gln Leu Cys Cys Gln Leu Lys
115 120
<210> 515
<211> 240
<212> PRT
<213> Homo Sapiens
<400> 515
Asp Thr Gln Glu Lys Glu Val Arg Ala Met Val Gly Ser Asp Val Glu
1 5 10 15
Leu Ser Cys Ala Cys Pro Glu Gly Ser Arg Phe Asp Leu Asn Asp Val
20 25 30
Tyr Val Tyr Trp Gln Thr Ser Glu Ser Lys Thr Val Val Thr Tyr His
35 40 45
Ile Pro Gln Asn Ser Ser Leu Glu Asn Val Asp Ser Arg Tyr Arg Asn
50 55 60
Arg Ala Leu Met Ser Pro Ala Gly Met Leu Arg Gly Asp Phe Ser Leu
65 70 75 80
Arg Leu Phe Asn Val Thr Pro Gln Asp Glu Gln Lys Phe His Cys Leu
85 90 95
Val Leu Ser Gln Ser Leu Gly Phe Gln Glu Val Leu Ser Val Glu Val
100 105 110
Thr Leu His Val Ala Ala Asn Phe Ser Val Pro Val Val Ser Ala Pro
115 120 125
His Ser Pro Ser Gln Asp Glu Leu Thr Phe Thr Cys Thr Ser Ile Asn
130 135 140
Gly Tyr Pro Arg Pro Asn Val Tyr Trp Ile Asn Lys Thr Asp Asn Ser
145 150 155 160
Leu Leu Asp Gln Ala Leu Gln Asn Asp Thr Val Phe Leu Asn Met Arg
165 170 175
Gly Leu Tyr Asp Val Val Ser Val Leu Arg Ile Ala Arg Thr Pro Ser
180 185 190
Val Asn Ile Gly Cys Cys Ile Glu Asn Val Leu Leu Gln Gln Asn Leu
195 200 205
Thr Val Gly Ser Gln Thr Gly Asn Asp Ile Gly Glu Arg Asp Lys Ile
210 215 220
Thr Glu Asn Pro Val Ser Thr Gly Glu Lys Asn Ala Ala Thr Trp Ser
225 230 235 240
<210> 516
<211> 302
<212> PRT
<213> Homo Sapiens
<400> 516
Met Arg Leu Gly Ser Pro Gly Leu Leu Phe Leu Leu Phe Ser Ser Leu
1 5 10 15
Arg Ala Asp Thr Gln Glu Lys Glu Val Arg Ala Met Val Gly Ser Asp
20 25 30
Val Glu Leu Ser Cys Ala Cys Pro Glu Gly Ser Arg Phe Asp Leu Asn
35 40 45
Asp Val Tyr Val Tyr Trp Gln Thr Ser Glu Ser Lys Thr Val Val Thr
50 55 60
Tyr His Ile Pro Gln Asn Ser Ser Leu Glu Asn Val Asp Ser Arg Tyr
65 70 75 80
Arg Asn Arg Ala Leu Met Ser Pro Ala Gly Met Leu Arg Gly Asp Phe
85 90 95
Ser Leu Arg Leu Phe Asn Val Thr Pro Gln Asp Glu Gln Lys Phe His
100 105 110
Cys Leu Val Leu Ser Gln Ser Leu Gly Phe Gln Glu Val Leu Ser Val
115 120 125
Glu Val Thr Leu His Val Ala Ala Asn Phe Ser Val Pro Val Val Ser
130 135 140
Ala Pro His Ser Pro Ser Gln Asp Glu Leu Thr Phe Thr Cys Thr Ser
145 150 155 160
Ile Asn Gly Tyr Pro Arg Pro Asn Val Tyr Trp Ile Asn Lys Thr Asp
165 170 175
Asn Ser Leu Leu Asp Gln Ala Leu Gln Asn Asp Thr Val Phe Leu Asn
180 185 190
Met Arg Gly Leu Tyr Asp Val Val Ser Val Leu Arg Ile Ala Arg Thr
195 200 205
Pro Ser Val Asn Ile Gly Cys Cys Ile Glu Asn Val Leu Leu Gln Gln
210 215 220
Asn Leu Thr Val Gly Ser Gln Thr Gly Asn Asp Ile Gly Glu Arg Asp
225 230 235 240
Lys Ile Thr Glu Asn Pro Val Ser Thr Gly Glu Lys Asn Ala Ala Thr
245 250 255
Trp Ser Ile Leu Ala Val Leu Cys Leu Leu Val Val Val Ala Val Ala
260 265 270
Ile Gly Trp Val Cys Arg Asp Arg Cys Leu Gln His Ser Tyr Ala Gly
275 280 285
Ala Trp Ala Val Ser Pro Glu Thr Glu Leu Thr Gly His Val
290 295 300
<210> 517
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 517
Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu
1 5 10 15
Thr Ala Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu
20 25 30
Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu
35 40 45
Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp
50 55 60
Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu
65 70 75 80
Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu
85 90 95
Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu
100 105 110
Thr
<210> 518
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 518
Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu
1 5 10 15
Thr Gln Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu
20 25 30
Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu
35 40 45
Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp
50 55 60
Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu
65 70 75 80
Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu
85 90 95
Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu
100 105 110
Thr
<210> 519
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 519
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
ctcagttttg gccaggggac caagctggag atcaaa 336
<210> 520
<211> 657
<212> DNA
<213> Homo Sapiens
<400> 520
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtgatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
ctcagttttg gccaggggac caagctggag atcaaacgta cggtggccgc tccctccgtg 360
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 420
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 480
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 540
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 600
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgt 657
<210> 521
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 521
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgtag cctctggagt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggagtg ggtctctggt attaattgga atggtggcga cacagattat 180
tcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctacaaatga atagtctgag agccgaggac acggccttgt attactgtgc gagggatttc 300
tatggttcgg ggagttatta tcacgttcct tttgactact ggggccaggg aatcctggtc 360
accgtctcct ca 372
<210> 522
<211> 1368
<212> DNA
<213> Homo Sapiens
<400> 522
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgtag cctctggagt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggagtg ggtctctggt attaattgga atggtggcga cacagattat 180
tcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctacaaatga atagtctgag agccgaggac acggccttgt attactgtgc gagggatttc 300
tatggttcgg ggagttatta tcacgttcct tttgactact ggggccaggg aatcctggtc 360
accgtctcct cagccagcac caagggcccc tctgtgttcc ctctggcccc ttccagcaag 420
tccacctctg gcggaacagc cgctctgggc tgcctcgtga aggactactt ccccgagcct 480
gtgaccgtgt cctggaactc tggcgctctg accagcggag tgcacacctt ccctgctgtg 540
ctgcagtcct ccggcctgta ctccctgtcc tccgtcgtga ccgtgccttc cagctctctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccct ccaacaccaa ggtggacaag 660
aaggtggaac ccaagtcctg cgacaagacc cacacctgtc ccccttgtcc tgcccctgaa 720
ctgctgggcg gaccttccgt gttcctgttc cccccaaagc ccaaggacac cctgatgatc 780
tcccggaccc ccgaagtgac ctgcgtggtg gtggatgtgt cccacgagga ccctgaagtg 840
aagttcaatt ggtacgtgga cggcgtggaa gtgcacaacg ccaagaccaa gcctagagag 900
gaacagtaca actccaccta ccgggtggtg tccgtgctga ccgtgctgca ccaggattgg 960
ctgaacggca aagagtacaa gtgcaaggtg tccaacaagg ccctgcctgc ccccatcgaa 1020
aagaccatct ccaaggccaa gggccagccc cgggaacccc aggtgtacac actgccccct 1080
agcagggacg agctgaccaa gaaccaggtg tccctgacct gtctcgtgaa aggcttctac 1140
ccctccgata tcgccgtgga atgggagtcc aacggccagc ctgagaacaa ctacaagacc 1200
accccccctg tgctggactc cgacggctca ttcttcctgt acagcaagct gacagtggac 1260
aagtcccggt ggcagcaggg caacgtgttc tcctgctccg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagtc cctgtccctg agccccggca agtgatga 1368
<210> 523
<211> 990
<212> DNA
<213> Homo Sapiens
<400> 523
gcctccacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 720
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc cccgggtaaa 990
<210> 524
<211> 330
<212> PRT
<213> Homo Sapiens
<400> 524
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
225 230 235 240
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 525
<211> 990
<212> DNA
<213> Homo Sapiens
<400> 525
gcctccacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 720
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acatcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc tccgggtaaa 990
<210> 526
<211> 330
<212> PRT
<213> Homo Sapiens
<400> 526
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Ile Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 527
<211> 978
<212> DNA
<213> Homo Sapiens
<400> 527
gcctccacca agggcccatc ggtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagccg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgctctgac cagcggcgtg cacaccttcc cagctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcaacttcgg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
gtggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt tgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 960
tccctgtctc cgggtaaa 978
<210> 528
<211> 326
<212> PRT
<213> Homo Sapiens
<400> 528
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 529
<211> 978
<212> DNA
<213> Homo Sapiens
<400> 529
gcctccacca agggcccatc ggtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgctctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgacctcca gcaacttcgg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
atggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt cgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 960
tccctgtctc cgggtaaa 978
<210> 530
<211> 326
<212> PRT
<213> Homo Sapiens
<400> 530
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Thr Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Met Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 531
<211> 978
<212> DNA
<213> Homo Sapiens
<400> 531
gcctccacca agggcccatc ggtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgctctgac cagcggcgtg cacaccttcc cagctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
gtggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt tgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 960
tccctgtctc cgggtaaa 978
<210> 532
<211> 326
<212> PRT
<213> Homo Sapiens
<400> 532
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 533
<211> 978
<212> DNA
<213> Homo Sapiens
<400> 533
gcctccacca agggcccatc ggtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgctctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcaacttcgg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
gtggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt cgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatctc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 960
tccctgtctc cgggtaaa 978
<210> 534
<211> 326
<212> PRT
<213> Homo Sapiens
<400> 534
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ser Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 535
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 535
ggtcagccca aggctgcccc ctcggtcact ctgttcccac cctcctctga ggagcttcaa 60
gccaacaagg ccacactggt gtgtctcgta agtgacttca acccgggagc cgtgacagtg 120
gcctggaagg cagatggcag ccccgtcaag gtgggagtgg agaccaccaa accctccaaa 180
caaagcaaca acaagtatgc ggccagcagc tacctgagcc tgacgcccga gcagtggaag 240
tcccacagaa gctacagctg ccgggtcacg catgaaggga gcaccgtgga gaagacagtg 300
gcccctgcag aatgctct 318
<210> 536
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 536
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Val Ser Asp
20 25 30
Phe Asn Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro
35 40 45
Val Lys Val Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Arg Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Ala Glu Cys Ser
100 105
<210> 537
<211> 990
<212> DNA
<213> Homo Sapiens
<400> 537
gcctccacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 720
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc tccgggtaaa 990
<210> 538
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 538
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 539
<211> 663
<212> PRT
<213> Homo Sapiens
<400> 539
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
210 215 220
Leu Thr Gln Pro Gly Lys Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly
225 230 235 240
Phe Thr Phe Ser Ser Phe Thr Met His Trp Val Arg Gln Ser Pro Gly
245 250 255
Lys Gly Leu Glu Trp Val Ala Phe Ile Arg Ser Gly Ser Gly Ile Val
260 265 270
Phe Tyr Ala Asp Ala Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn
275 280 285
Ala Lys Asn Leu Leu Phe Leu Gln Met Asn Asp Leu Lys Ser Glu Asp
290 295 300
Thr Ala Met Tyr Tyr Cys Ala Arg Arg Pro Leu Gly His Asn Thr Phe
305 310 315 320
Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
325 330 335
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
340 345 350
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
355 360 365
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
370 375 380
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
385 390 395 400
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
405 410 415
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
420 425 430
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
435 440 445
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
450 455 460
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
465 470 475 480
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
485 490 495
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
500 505 510
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
515 520 525
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
530 535 540
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
545 550 555 560
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
565 570 575
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
580 585 590
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
595 600 605
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
610 615 620
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
625 630 635 640
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
645 650 655
Leu Ser Leu Ser Pro Gly Lys
660
<210> 540
<211> 215
<212> PRT
<213> Homo Sapiens
<400> 540
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe
20 25 30
Tyr Met Ala Trp Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val
50 55 60
Met Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Lys Val
210 215
<210> 541
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 541
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 542
<211> 667
<212> PRT
<213> Homo Sapiens
<400> 542
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
210 215 220
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu Lys Leu
225 230 235 240
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe Tyr Met Ala Trp
245 250 255
Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val Ala Ser Ile Ser
260 265 270
Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val Met Gly Arg Phe
275 280 285
Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr Leu Gln Met Asn
290 295 300
Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Gln Arg
305 310 315 320
Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met Val Thr Val Ser
325 330 335
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
340 345 350
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
355 360 365
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
370 375 380
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
385 390 395 400
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
405 410 415
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
420 425 430
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
435 440 445
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
450 455 460
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
465 470 475 480
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
485 490 495
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
500 505 510
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
515 520 525
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
530 535 540
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
545 550 555 560
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
565 570 575
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
580 585 590
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
595 600 605
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
610 615 620
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
625 630 635 640
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
645 650 655
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
660 665
<210> 543
<211> 217
<212> PRT
<213> Homo Sapiens
<400> 543
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Thr Gln Pro Gly Lys
1 5 10 15
Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Thr Met His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Ser Gly Ser Gly Ile Val Phe Tyr Ala Asp Ala Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Leu Leu Phe
65 70 75 80
Leu Gln Met Asn Asp Leu Lys Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Arg Pro Leu Gly His Asn Thr Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val
210 215
<210> 544
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 544
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 545
<211> 663
<212> PRT
<213> Homo Sapiens
<400> 545
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Asp Ala Ala
100 105 110
Pro Thr Val Ser Ile Phe Pro Pro Ser Ser Glu Gln Leu Thr Ser Gly
115 120 125
Gly Ala Ser Val Val Cys Phe Leu Asn Asn Phe Tyr Pro Lys Asp Ile
130 135 140
Asn Val Lys Trp Lys Ile Asp Gly Ser Glu Arg Gln Asn Gly Val Leu
145 150 155 160
Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp Ser Thr Tyr Ser Met Ser
165 170 175
Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr Glu Arg His Asn Ser Tyr
180 185 190
Thr Cys Glu Ala Thr His Lys Thr Ser Thr Ser Pro Ile Val Lys Ser
195 200 205
Phe Asn Arg Asn Glu Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
210 215 220
Leu Thr Gln Pro Gly Lys Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly
225 230 235 240
Phe Thr Phe Ser Ser Phe Thr Met His Trp Val Arg Gln Ser Pro Gly
245 250 255
Lys Gly Leu Glu Trp Val Ala Phe Ile Arg Ser Gly Ser Gly Ile Val
260 265 270
Phe Tyr Ala Asp Ala Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn
275 280 285
Ala Lys Asn Leu Leu Phe Leu Gln Met Asn Asp Leu Lys Ser Glu Asp
290 295 300
Thr Ala Met Tyr Tyr Cys Ala Arg Arg Pro Leu Gly His Asn Thr Phe
305 310 315 320
Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Lys Thr
325 330 335
Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys Gly Asp Thr Thr
340 345 350
Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly Tyr Phe Pro Glu
355 360 365
Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser Ser Gly Val His
370 375 380
Thr Phe Pro Ala Val Leu Gln Ser Asp Leu Tyr Thr Leu Ser Ser Ser
385 390 395 400
Val Thr Val Thr Ser Ser Thr Trp Pro Ser Gln Ser Ile Thr Cys Asn
405 410 415
Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys Lys Ile Glu Pro
420 425 430
Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro Ala Pro
435 440 445
Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Ile Lys
450 455 460
Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val Val Val
465 470 475 480
Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe Val Asn
485 490 495
Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu Asp Tyr
500 505 510
Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp
515 520 525
Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu
530 535 540
Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser Val Arg
545 550 555 560
Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met Thr Lys
565 570 575
Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro Glu Asp
580 585 590
Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys
595 600 605
Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser
610 615 620
Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser
625 630 635 640
Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr Lys Ser
645 650 655
Phe Ser Arg Thr Pro Gly Lys
660
<210> 546
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 546
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe
20 25 30
Tyr Met Ala Trp Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val
50 55 60
Met Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met
100 105 110
Val Thr Val Ser Ser Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu
115 120 125
Ala Pro Val Cys Gly Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys
130 135 140
Leu Val Lys Gly Tyr Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser
145 150 155 160
Gly Ser Leu Ser Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Asp Leu Tyr Thr Leu Ser Ser Ser Val Thr Val Thr Ser Ser Thr Trp
180 185 190
Pro Ser Gln Ser Ile Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr
195 200 205
Lys Val Asp Lys Lys Ile
210
<210> 547
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 547
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Ser
115 120 125
Glu Gln Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu Asn Asn
130 135 140
Phe Tyr Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly Ser Glu
145 150 155 160
Arg Gln Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr
180 185 190
Glu Arg His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr Ser Thr
195 200 205
Ser Pro Ile Val Lys Ser Phe Asn Arg Asn Glu Cys
210 215 220
<210> 548
<211> 667
<212> PRT
<213> Homo Sapiens
<400> 548
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro Ser Ser
115 120 125
Glu Gln Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu Asn Asn
130 135 140
Phe Tyr Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly Ser Glu
145 150 155 160
Arg Gln Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr
180 185 190
Glu Arg His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr Ser Thr
195 200 205
Ser Pro Ile Val Lys Ser Phe Asn Arg Asn Glu Cys Glu Val Gln Leu
210 215 220
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu Lys Leu
225 230 235 240
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe Tyr Met Ala Trp
245 250 255
Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val Ala Ser Ile Ser
260 265 270
Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val Met Gly Arg Phe
275 280 285
Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr Leu Gln Met Asn
290 295 300
Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Gln Arg
305 310 315 320
Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met Val Thr Val Ser
325 330 335
Ser Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro Val Cys
340 345 350
Gly Asp Thr Thr Gly Ser Ser Val Thr Leu Gly Cys Leu Val Lys Gly
355 360 365
Tyr Phe Pro Glu Pro Val Thr Leu Thr Trp Asn Ser Gly Ser Leu Ser
370 375 380
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Asp Leu Tyr Thr
385 390 395 400
Leu Ser Ser Ser Val Thr Val Thr Ser Ser Thr Trp Pro Ser Gln Ser
405 410 415
Ile Thr Cys Asn Val Ala His Pro Ala Ser Ser Thr Lys Val Asp Lys
420 425 430
Lys Ile Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys
435 440 445
Cys Pro Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro
450 455 460
Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr
465 470 475 480
Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser
485 490 495
Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His
500 505 510
Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile
515 520 525
Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn
530 535 540
Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys
545 550 555 560
Gly Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu
565 570 575
Glu Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe
580 585 590
Met Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu
595 600 605
Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr
610 615 620
Phe Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg
625 630 635 640
Asn Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His
645 650 655
Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
660 665
<210> 549
<211> 216
<212> PRT
<213> Homo Sapiens
<400> 549
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Thr Gln Pro Gly Lys
1 5 10 15
Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Thr Met His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Ser Gly Ser Gly Ile Val Phe Tyr Ala Asp Ala Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Leu Leu Phe
65 70 75 80
Leu Gln Met Asn Asp Leu Lys Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Arg Pro Leu Gly His Asn Thr Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Lys Thr Thr Ala Pro Ser Val Tyr
115 120 125
Pro Leu Ala Pro Val Cys Gly Asp Thr Thr Gly Ser Ser Val Thr Leu
130 135 140
Gly Cys Leu Val Lys Gly Tyr Phe Pro Glu Pro Val Thr Leu Thr Trp
145 150 155 160
Asn Ser Gly Ser Leu Ser Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Asp Leu Tyr Thr Leu Ser Ser Ser Val Thr Val Thr Ser Ser
180 185 190
Thr Trp Pro Ser Gln Ser Ile Thr Cys Asn Val Ala His Pro Ala Ser
195 200 205
Ser Thr Lys Val Asp Lys Lys Ile
210 215
<210> 550
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 550
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Asp Ala Ala
100 105 110
Pro Thr Val Ser Ile Phe Pro Pro Ser Ser Glu Gln Leu Thr Ser Gly
115 120 125
Gly Ala Ser Val Val Cys Phe Leu Asn Asn Phe Tyr Pro Lys Asp Ile
130 135 140
Asn Val Lys Trp Lys Ile Asp Gly Ser Glu Arg Gln Asn Gly Val Leu
145 150 155 160
Asn Ser Trp Thr Asp Gln Asp Ser Lys Asp Ser Thr Tyr Ser Met Ser
165 170 175
Ser Thr Leu Thr Leu Thr Lys Asp Glu Tyr Glu Arg His Asn Ser Tyr
180 185 190
Thr Cys Glu Ala Thr His Lys Thr Ser Thr Ser Pro Ile Val Lys Ser
195 200 205
Phe Asn Arg Asn Glu Cys
210
<210> 551
<211> 659
<212> PRT
<213> Homo Sapiens
<400> 551
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
210 215 220
Leu Thr Gln Pro Gly Lys Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly
225 230 235 240
Phe Thr Phe Ser Ser Phe Thr Met His Trp Val Arg Gln Ser Pro Gly
245 250 255
Lys Gly Leu Glu Trp Val Ala Phe Ile Arg Ser Gly Ser Gly Ile Val
260 265 270
Phe Tyr Ala Asp Ala Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn
275 280 285
Ala Lys Asn Leu Leu Phe Leu Gln Met Asn Asp Leu Lys Ser Glu Asp
290 295 300
Thr Ala Met Tyr Tyr Cys Ala Arg Arg Pro Leu Gly His Asn Thr Phe
305 310 315 320
Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
325 330 335
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
340 345 350
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
355 360 365
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
370 375 380
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
385 390 395 400
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
405 410 415
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
420 425 430
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Asn Leu Leu Gly
435 440 445
Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met
450 455 460
Ile Ser Leu Ser Pro Ile Val Thr Cys Val Val Val Asp Val Ser Glu
465 470 475 480
Asp Asp Pro Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val
485 490 495
His Thr Ala Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu
500 505 510
Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly
515 520 525
Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile
530 535 540
Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser Val Arg Ala Pro Gln Val
545 550 555 560
Tyr Val Leu Pro Pro Pro Glu Glu Glu Met Thr Lys Lys Gln Val Thr
565 570 575
Leu Thr Cys Met Val Thr Asp Phe Met Pro Glu Asp Ile Tyr Val Glu
580 585 590
Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys Asn Thr Glu Pro
595 600 605
Val Leu Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val
610 615 620
Glu Lys Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser Cys Ser Val Val
625 630 635 640
His Glu Gly Leu His Asn His His Thr Thr Lys Ser Phe Ser Arg Thr
645 650 655
Pro Gly Lys
<210> 552
<211> 215
<212> PRT
<213> Homo Sapiens
<400> 552
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe
20 25 30
Tyr Met Ala Trp Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val
50 55 60
Met Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Lys Val
210 215
<210> 553
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 553
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 554
<211> 663
<212> PRT
<213> Homo Sapiens
<400> 554
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
210 215 220
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu Lys Leu
225 230 235 240
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe Tyr Met Ala Trp
245 250 255
Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val Ala Ser Ile Ser
260 265 270
Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val Met Gly Arg Phe
275 280 285
Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr Leu Gln Met Asn
290 295 300
Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Gln Arg
305 310 315 320
Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met Val Thr Val Ser
325 330 335
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
340 345 350
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
355 360 365
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
370 375 380
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
385 390 395 400
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
405 410 415
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
420 425 430
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
435 440 445
Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Ile Lys
450 455 460
Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val Val Val
465 470 475 480
Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe Val Asn
485 490 495
Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu Asp Tyr
500 505 510
Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp
515 520 525
Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu
530 535 540
Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser Val Arg
545 550 555 560
Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met Thr Lys
565 570 575
Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro Glu Asp
580 585 590
Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys
595 600 605
Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser
610 615 620
Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser
625 630 635 640
Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr Lys Ser
645 650 655
Phe Ser Arg Thr Pro Gly Lys
660
<210> 555
<211> 217
<212> PRT
<213> Homo Sapiens
<400> 555
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Thr Gln Pro Gly Lys
1 5 10 15
Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Thr Met His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Ser Gly Ser Gly Ile Val Phe Tyr Ala Asp Ala Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Leu Leu Phe
65 70 75 80
Leu Gln Met Asn Asp Leu Lys Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Arg Pro Leu Gly His Asn Thr Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val
210 215
<210> 556
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 556
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 557
<211> 659
<212> PRT
<213> Homo Sapiens
<400> 557
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly
210 215 220
Leu Thr Gln Pro Gly Lys Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly
225 230 235 240
Phe Thr Phe Ser Ser Phe Thr Met His Trp Val Arg Gln Ser Pro Gly
245 250 255
Lys Gly Leu Glu Trp Val Ala Phe Ile Arg Ser Gly Ser Gly Ile Val
260 265 270
Phe Tyr Ala Asp Ala Val Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn
275 280 285
Ala Lys Asn Leu Leu Phe Leu Gln Met Asn Asp Leu Lys Ser Glu Asp
290 295 300
Thr Ala Met Tyr Tyr Cys Ala Arg Arg Pro Leu Gly His Asn Thr Phe
305 310 315 320
Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
325 330 335
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
340 345 350
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
355 360 365
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
370 375 380
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
385 390 395 400
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
405 410 415
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
420 425 430
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Asn Leu Leu Gly
435 440 445
Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met
450 455 460
Ile Ser Leu Ser Pro Ile Val Thr Cys Val Val Val Asp Val Ser Glu
465 470 475 480
Asp Asp Pro Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val
485 490 495
His Thr Ala Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu
500 505 510
Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly
515 520 525
Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile
530 535 540
Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser Val Arg Ala Pro Gln Val
545 550 555 560
Tyr Val Leu Pro Pro Pro Glu Glu Glu Met Thr Lys Lys Gln Val Thr
565 570 575
Leu Thr Cys Met Val Thr Asp Phe Met Pro Glu Asp Ile Tyr Val Glu
580 585 590
Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys Asn Thr Glu Pro
595 600 605
Val Leu Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val
610 615 620
Glu Lys Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser Cys Ser Val Val
625 630 635 640
His Glu Gly Leu His Asn His His Thr Thr Lys Ser Phe Ser Arg Thr
645 650 655
Pro Gly Lys
<210> 558
<211> 215
<212> PRT
<213> Homo Sapiens
<400> 558
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe
20 25 30
Tyr Met Ala Trp Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val
50 55 60
Met Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Lys Val
210 215
<210> 559
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 559
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 560
<211> 663
<212> PRT
<213> Homo Sapiens
<400> 560
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Lys Ser Ser Gln Ser Leu Tyr Tyr Ser
20 25 30
Gly Val Lys Glu Asn Leu Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Ile Arg Phe Thr Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Met Gly Gln Tyr Phe Cys Gln Gln
85 90 95
Gly Ile Asn Asn Pro Leu Thr Phe Gly Asp Gly Thr Lys Leu Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
210 215 220
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu Lys Leu
225 230 235 240
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Phe Tyr Met Ala Trp
245 250 255
Val Arg Gln Ala Pro Lys Lys Gly Leu Glu Trp Val Ala Ser Ile Ser
260 265 270
Tyr Glu Gly Ser Ser Thr Tyr Tyr Gly Asp Ser Val Met Gly Arg Phe
275 280 285
Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr Leu Gln Met Asn
290 295 300
Ser Leu Arg Ser Glu Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Gln Arg
305 310 315 320
Glu Ala Asn Trp Glu Asp Trp Gly Gln Gly Val Met Val Thr Val Ser
325 330 335
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
340 345 350
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
355 360 365
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
370 375 380
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
385 390 395 400
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
405 410 415
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
420 425 430
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
435 440 445
Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys Ile Lys
450 455 460
Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val Val Val
465 470 475 480
Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe Val Asn
485 490 495
Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu Asp Tyr
500 505 510
Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His Gln Asp
515 520 525
Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys Asp Leu
530 535 540
Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser Val Arg
545 550 555 560
Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met Thr Lys
565 570 575
Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro Glu Asp
580 585 590
Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys
595 600 605
Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser
610 615 620
Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser
625 630 635 640
Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr Lys Ser
645 650 655
Phe Ser Arg Thr Pro Gly Lys
660
<210> 561
<211> 217
<212> PRT
<213> Homo Sapiens
<400> 561
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Thr Gln Pro Gly Lys
1 5 10 15
Ser Leu Lys Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Thr Met His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Ser Gly Ser Gly Ile Val Phe Tyr Ala Asp Ala Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Leu Leu Phe
65 70 75 80
Leu Gln Met Asn Asp Leu Lys Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Arg Pro Leu Gly His Asn Thr Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val
210 215
<210> 562
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 562
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Thr Val Thr Ile Gln Cys Arg Ala Ser Glu Asp Ile Tyr Ser Gly
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Ser Ser Met Gln Thr
65 70 75 80
Glu Asp Glu Gly Val Tyr Phe Cys Gln Gln Gly Leu Lys Tyr Pro Pro
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 563
<211> 364
<212> DNA
<213> Homo Sapiens
<400> 563
caggttcaac tgatgcagtc tggaactgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaaga cttctggtta cacctttacc acctatggta tcacttgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acagtggtga cacagactat 180
gcacagaagt tccagggcag agtcaccgtg acaacagaca catccacgaa cacagcctac 240
atggagttga ggagcctgaa atctgacgac acggccgtgt attattgtgc gagaagtagt 300
ggctggcccc accactacgg tatggacgtc tggggccaag ggaccacggt caccgtctcc 360
tcag 364
<210> 564
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 564
Gln Val Gln Leu Met Gln Ser Gly Thr Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Thr Phe Thr Thr Tyr
20 25 30
Gly Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Ser Gly Asp Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Val Thr Thr Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Trp Pro His His Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 565
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 565
Gly Tyr Thr Phe Thr Thr Tyr Gly
1 5
<210> 566
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 566
Ile Ser Ala Tyr Ser Gly Asp Thr
1 5
<210> 567
<211> 14
<212> PRT
<213> Homo Sapiens
<400> 567
Ala Arg Ser Ser Gly Trp Pro His His Tyr Gly Met Asp Val
1 5 10
<210> 568
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 568
caggttcaac tggtgcagtc tggaggtgag gtgaaaaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtctccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt atttctgtgc gcgatctacg 300
tcttactatg gttcggggac cctatacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cag 373
<210> 569
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 569
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Ser Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Thr Ser Tyr Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 570
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 570
Ala Arg Ser Thr Ser Tyr Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
1 5 10 15
Val
<210> 571
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 571
caggttcaac tggtgcagtc tggaggtgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtctccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt attactgtgc gcgatctacg 300
tcttactatg gttcggggac cctctacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cag 373
<210> 572
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 572
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Ser Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Ser Tyr Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 573
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 573
caggttcaac tggtgcagtc tggaggtgag gtgaaaaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggtt tcagctgggt gcgacaggcc 120
cctggacaag gactagagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtctccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt atttctgtgc gcgatctacg 300
tcttactatg gttcggggac cctatacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cag 373
<210> 574
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 574
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Phe Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Ser Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Thr Ser Tyr Tyr Gly Ser Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 575
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 575
caggttcaac tggtgcagtc tggaggtgag gtgaaaaagc ctcgggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatgtgt tcagctgggt gcgacatgcc 120
gctggacaag gactagagtg gatgggatgg atcagcggtt acaatggtaa cacaaactat 180
gcacagaagc tccagtgcgg agtctcgatg accgcagaca catccacgag cacagcctac 240
atggagctga ggagcttgag atctgacgac acggccgtgt atttctgtgc gcgatctacg 300
tcttactatg gtgcggggac cctatacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cag 373
<210> 576
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 576
Gln Val Gln Leu Val Gln Ser Gly Gly Glu Val Lys Lys Pro Arg Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Val Phe Ser Trp Val Arg His Ala Ala Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Gly Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Cys Gly Val Ser Met Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Thr Ser Tyr Tyr Gly Ala Gly Thr Leu Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 577
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 577
Gly Tyr Thr Phe Thr Ser Tyr Val
1 5
<210> 578
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 578
Ile Ser Gly Tyr Asn Gly Asn Thr
1 5
<210> 579
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 579
Ala Arg Ser Thr Ser Tyr Tyr Gly Ala Gly Thr Leu Tyr Gly Met Asp
1 5 10 15
Val
<210> 580
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 580
gaggtgcagc tggtggagtc tgggggaggt gtggtacggc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggagtg ggtctctggt attaattgga atggtggtag cacaggttat 180
gcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agccgaggac acggccttgt attactgtgc ggccgattac 300
tatggttcgg ggagttatta taacgtcccc tttgactact ggggccaggg aaccctggtc 360
accgtctcct cag 373
<210> 581
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 581
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Asn Gly Gly Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Ala Asp Tyr Tyr Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 582
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 582
Gly Phe Thr Phe Asp Asp Tyr Gly
1 5
<210> 583
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 583
Ile Asn Trp Asn Gly Gly Ser Thr
1 5
<210> 584
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 584
Ala Ala Asp Tyr Tyr Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
1 5 10 15
Tyr
<210> 585
<211> 373
<212> DNA
<213> Homo Sapiens
<400> 585
gaggtgcagc tggtggagtc tgggggaggt gtgatacggc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatggca tgagctgggt ccgccaagct 120
ccagggaagg ggctggagtg ggtctctggt attaattgga ttggtgataa cacagattat 180
gcagactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctatat 240
ctgcaaatga acagtctgag agccgaggac acggccttgt attactgtgc gagagattac 300
tttggttcgg ggagttatta taacgttccc tttgactact ggggccaggg aaccctggtc 360
accgtctcct cag 373
<210> 586
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 586
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Ile Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asn Trp Ile Gly Asp Asn Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Phe Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 587
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 587
Ile Asn Trp Ile Gly Asp Asn Thr
1 5
<210> 588
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 588
Ala Arg Asp Tyr Phe Gly Ser Gly Ser Tyr Tyr Asn Val Pro Phe Asp
1 5 10 15
Tyr
<210> 589
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 589
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gattcaacta tttcgattgg 120
tacctgcaga agccaggaca gtctccacag ctcctgatct ttttggtttc taatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttgggatt tattactgca tgcaagctct acaaactccg 300
ctcactttcg gcggagggac caaggtggag atcaaac 337
<210> 590
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 590
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Phe Asn Tyr Phe Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Val Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 591
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 591
Gln Ser Leu Leu His Ser Asn Gly Phe Asn Tyr
1 5 10
<210> 592
<211> 3
<212> PRT
<213> Homo Sapiens
<400> 592
Leu Val Ser
1
<210> 593
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 593
Met Gln Ala Leu Gln Thr Pro Leu Thr
1 5
<210> 594
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 594
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaac 337
<210> 595
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 595
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 596
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 596
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Cys
1 5 10
<210> 597
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 597
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaac 337
<210> 598
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 598
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 599
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 599
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattctac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaac 337
<210> 600
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 600
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Ser Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 601
<211> 337
<212> DNA
<213> Homo Sapiens
<400> 601
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaactg tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tactcgggcc 180
tccgggttcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactccg 300
tgcagttttg gccaggggac caagctggag atcaaac 337
<210> 602
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 602
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Cys Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Phe Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Cys Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 603
<211> 325
<212> DNA
<213> Homo Sapiens
<400> 603
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcaccttt cactttcggc 300
cctgggacca aagtggatat caaac 325
<210> 604
<211> 108
<212> PRT
<213> Homo Sapiens
<400> 604
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 605
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 605
Gln Gln Tyr Gly Ser Ser Pro Phe Thr
1 5
<210> 606
<211> 325
<212> DNA
<213> Homo Sapiens
<400> 606
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggaa 240
cctgaagatt ttgcagtata ttactgtcac cagtatggta attcaccatt cactttcggc 300
cctgggacca aagtggatat caaac 325
<210> 607
<211> 108
<212> PRT
<213> Homo Sapiens
<400> 607
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Asn Ser Pro
85 90 95
Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 608
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 608
His Gln Tyr Gly Asn Ser Pro Phe Thr
1 5
<210> 609
<211> 11
<212> PRT
<213> Homo Sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> X = any amino acid.
<220>
<221> MISC_FEATURE
<222> (5)..(5)
<223> X = any amino acid.
<220>
<221> MISC_FEATURE
<222> (8)..(8)
<223> X = any amino acid.
<220>
<221> MISC_FEATURE
<222> (9)..(9)
<223> X = is either present or absent, and if present, may be any amino
acid.
<400> 609
Xaa Gly Ser Gly Xaa Tyr Gly Xaa Xaa Phe Asp
1 5 10
<210> 610
<211> 478
<212> PRT
<213> Artificial Sequence
<220>
<223> ICOSL-Fc fusion protein
<400> 610
Asp Thr Gln Glu Lys Glu Val Arg Ala Met Val Gly Ser Asp Val Glu
1 5 10 15
Leu Ser Cys Ala Cys Pro Glu Gly Ser Arg Phe Asp Leu Asn Asp Val
20 25 30
Tyr Val Tyr Trp Gln Thr Ser Glu Ser Lys Thr Val Val Thr Tyr His
35 40 45
Ile Pro Gln Asn Ser Ser Leu Glu Asn Val Asp Ser Arg Tyr Arg Asn
50 55 60
Arg Ala Leu Met Ser Pro Ala Gly Met Leu Arg Gly Asp Phe Ser Leu
65 70 75 80
Arg Leu Phe Asn Val Thr Pro Gln Asp Glu Gln Lys Phe His Cys Leu
85 90 95
Val Leu Ser Gln Ser Leu Gly Phe Gln Glu Val Leu Ser Val Glu Val
100 105 110
Thr Leu His Val Ala Ala Asn Phe Ser Val Pro Val Val Ser Ala Pro
115 120 125
His Ser Pro Ser Gln Asp Glu Leu Thr Phe Thr Cys Thr Ser Ile Asn
130 135 140
Gly Tyr Pro Arg Pro Asn Val Tyr Trp Ile Asn Lys Thr Asp Asn Ser
145 150 155 160
Leu Leu Asp Gln Ala Leu Gln Asn Asp Thr Val Phe Leu Asn Met Arg
165 170 175
Gly Leu Tyr Asp Val Val Ser Val Leu Arg Ile Ala Arg Thr Pro Ser
180 185 190
Val Asn Ile Gly Cys Cys Ile Glu Asn Val Leu Leu Gln Gln Asn Leu
195 200 205
Thr Val Gly Ser Gln Thr Gly Asn Asp Ile Gly Glu Arg Asp Lys Ile
210 215 220
Thr Glu Asn Pro Val Ser Thr Gly Glu Lys Asn Ala Ala Thr Trp Ser
225 230 235 240
Asp Ile Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 611
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> 37A10S713 Heavy Chain
<400> 611
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Trp Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Val Trp Val
35 40 45
Ser Asn Ile Asp Glu Asp Gly Ser Ile Thr Glu Tyr Ser Pro Phe Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Trp Gly Arg Phe Gly Phe Asp Ser Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
<210> 612
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> 37A10S713 Light Chain
<400> 612
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Leu Leu Ser Gly
20 25 30
Ser Phe Asn Tyr Leu Thr Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Tyr Ala Ser Thr Arg His Thr Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys His His His Tyr
85 90 95
Asn Ala Pro Pro Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 613
<211> 453
<212> PRT
<213> Artificial Sequence
<220>
<223> XMAb23104 Heavy Chain
<400> 613
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro His Ser Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Tyr Tyr Tyr Asp Thr Ser Gly Tyr Tyr His Asp Ala Phe
100 105 110
Asp Val Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Ile Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asp Thr Lys Val Asp Lys Lys Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Lys His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Glu Tyr Asn
290 295 300
Ser Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Asp Val Ser Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asp Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Glu Gln Gly Asp Val Phe Ser Cys Ser
420 425 430
Val Leu His Glu Ala Leu His Ser His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 614
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> XmAb23104 Light Chain
<400> 614
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Arg Leu
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 615
<211> 142
<212> PRT
<213> Artificial Sequence
<220>
<223> 314.8 mAb Heavy Chain Variable Region
<400> 615
Met Gly Trp Arg Cys Ile Ile Leu Phe Leu Val Ser Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Gln Gln Pro Gly Thr Glu Leu Met Lys
20 25 30
Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Thr Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Glu Ile Asp Pro Ser Asp Ser Tyr Val Asn Tyr Asn
65 70 75 80
Gln Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser
85 90 95
Thr Ala Tyr Ile Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val
100 105 110
Tyr Phe Cys Ala Arg Ser Pro Asp Tyr Tyr Gly Thr Ser Leu Ala Trp
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Thr
130 135 140
<210> 616
<211> 132
<212> PRT
<213> Artificial Sequence
<220>
<223> 314.8 mAb Light Chain Variable Region
<400> 616
Met Arg Cys Leu Ala Glu Phe Leu Gly Leu Leu Val Leu Trp Ile Pro
1 5 10 15
Gly Val Ile Gly Asp Ile Val Met Thr Gln Ala Ala Pro Ser Val Pro
20 25 30
Val Thr Pro Gly Glu Ser Val Ser Ile Ser Cys Arg Ser Ser Lys Ser
35 40 45
Pro Leu His Ser Asn Gly Asn Ile Tyr Leu Tyr Trp Phe Leu Gln Arg
50 55 60
Pro Gly Gln Ser Pro Gln Leu Leu Ile Tyr Arg Met Ser Asn Leu Ala
65 70 75 80
Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Thr Phe
85 90 95
Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr
100 105 110
Cys Met Gln His Leu Glu Tyr Pro Tyr Thr Phe Gly Gly Gly Thr Lys
115 120 125
Leu Glu Ile Lys
130
<210> 617
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> JMab-136 Heavy Chain Variable Region
<400> 617
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro His Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr His Asp Ala Phe
100 105 110
Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120 125
<210> 618
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> JMab-136 Light Chain Variable Region
<400> 618
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Arg Leu
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 619
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> ICOS.33 IgGlf S267E heavy chain
<400> 619
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Phe Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Val Ile Asp Thr Lys Ser Phe Asn Tyr Ala Thr Tyr Tyr Ser Asp
50 55 60
Leu Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Ala Thr Ile Ala Val Pro Tyr Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Glu His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly
450
<210> 620
<211> 213
<212> PRT
<213> Artificial Sequence
<220>
<223> ICOS.33 IgGlf S267E light chain
<400> 620
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Asn Leu Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asn Tyr Arg Thr
85 90 95
Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
Claims (42)
- 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시키는 것이 필요한 대상체에서 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병을 치료하는 방법으로서, 인간 및/또는 마우스 ICOS의 세포외 도메인에 결합하는 항-ICOS 항체 또는 이의 항원-결합 단편을 대상체에 투여하는 단계를 포함하고, 여기서 항-ICOS 항체 또는 이의 항원-결합 단편은 약 0.8 mg 내지 240 mg의 용량으로 대상체에 투여되는, 방법.
- 제1항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 중쇄 상보성 결정 영역(HCDR) HCDR1, HCDR2 및 HCDR3, 그리고 경쇄 상보성 결정 영역(LCDR) LCDR1, LCDR2, 및 LCDR3을 포함하고,
(a) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 363, SEQ ID NO: 364, 및 SEQ ID NO: 365와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 370, SEQ ID NO: 371, SEQ ID NO: 372와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(b) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 377, SEQ ID NO: 378, 및 SEQ ID NO: 379와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 384, SEQ ID NO: 385, SEQ ID NO: 386과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(c) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 391, SEQ ID NO: 392, 및 SEQ ID NO: 393과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 398, SEQ ID NO: 399, SEQ ID NO: 400과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(d) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 405, SEQ ID NO: 406, 및 SEQ ID NO: 407과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 412, SEQ ID NO: 413, SEQ ID NO: 414와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(e) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 419, SEQ ID NO: 420, 및 SEQ ID NO: 421과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 426, SEQ ID NO: 427, SEQ ID NO: 428과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(f) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 435, SEQ ID NO: 436, 및 SEQ ID NO: 437과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 442, SEQ ID NO: 443, SEQ ID NO: 444와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(g) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 449, SEQ ID NO: 450, 및 SEQ ID NO: 451과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 456, SEQ ID NO: 457, SEQ ID NO: 458과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(h) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 463, SEQ ID NO: 464, 및 SEQ ID NO: 465와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 470, SEQ ID NO: 471, SEQ ID NO: 472와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(i) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 477, SEQ ID NO: 478, 및 SEQ ID NO: 479와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 484, SEQ ID NO: 485, SEQ ID NO: 486과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나,
(j) HCDR1, HCDR2, 및 HCDR3은 아미노산 서열 SEQ ID NO: 491, SEQ ID NO: 492, 및 SEQ ID NO: 493과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고, LCDR1, LCDR2, 및 LCDR3은 아미노산 서열 SEQ ID NO: 498, SEQ ID NO: 499, SEQ ID NO: 500과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하는, 방법. - 제1항 또는 제2항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 중쇄 상보성 결정 영역(HCDR) HCDR1, HCDR2, 및 HCDR3, 그리고 경쇄 상보성 결정 영역(LCDR) LCDR1, LCDR2 및 LCDR3을 포함하고,
(a) HCDR1은 아미노산 서열 SEQ ID NO: 363을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 364를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 365를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 370을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 371을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 372를 포함하거나;
(b) HCDR1은 아미노산 서열 SEQ ID NO: 377을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 378을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 379를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 384를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 385를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 386을 포함하거나;
(c) HCDR1은 아미노산 서열 SEQ ID NO: 391을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 392를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 393을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 398을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 399를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 400을 포함하거나;
(d) HCDR1은 아미노산 서열 SEQ ID NO: 405를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 406을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 407을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 412를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 413을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 414를 포함하거나;
(e) HCDR1은 아미노산 서열 SEQ ID NO: 419를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 420을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 421을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 426을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 427을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 428을 포함하거나;
(f) HCDR1은 아미노산 서열 SEQ ID NO: 435를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 436을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 437을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 442를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 443을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 444를 포함하거나;
(g) HCDR1은 아미노산 서열 SEQ ID NO: 449를 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 450을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 451을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 456을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 457을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 458을 포함하거나;
(h) HCDR1은 아미노산 서열 SEQ ID NO: 463을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 464를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 465를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 470을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 471을 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 472를 포함하거나;
(i) HCDR1은 아미노산 서열 SEQ ID NO: 477을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 478을 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 479를 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 484를 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 485를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 486을 포함하거나;
(j) HCDR1은 아미노산 서열 SEQ ID NO: 491을 포함하고, HCDR2는 아미노산 서열 SEQ ID NO: 492를 포함하고, HCDR3은 아미노산 서열 SEQ ID NO: 493을 포함하고, LCDR1은 아미노산 서열 SEQ ID NO: 498을 포함하고, LCDR2는 아미노산 서열 SEQ ID NO: 499를 포함하고, LCDR3은 아미노산 서열 SEQ ID NO: 500을 포함하는, 방법. - 제3항에 있어서,
HCDR1은 아미노산 서열 SEQ ID NO: 405를 포함하고,
HCDR2는 아미노산 서열 SEQ ID NO: 406을 포함하고,
HCDR3은 아미노산 서열 SEQ ID NO: 407을 포함하고,
LCDR1은 아미노산 서열 SEQ ID NO: 412를 포함하고,
LCDR2는 아미노산 서열 SEQ ID NO: 413을 포함하고,
LCDR3은 아미노산 서열 SEQ ID NO: 414를 포함하는, 방법. - 제1항 내지 제4항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 중쇄 가변(VH) 도메인 및 경쇄 가변(VL) 도메인을 포함하고,
(a) VH 도메인은 아미노산 서열 SEQ ID NO: 366과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 373과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(b) VH 도메인은 아미노산 서열 SEQ ID NO: 380과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 387과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(c) VH 도메인은 아미노산 서열 SEQ ID NO: 394와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 401과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(d) VH 도메인은 아미노산 서열 SEQ ID NO: 408과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 415와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(e) VH 도메인은 아미노산 서열 SEQ ID NO: 422와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 429와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(f) VH 도메인은 아미노산 서열 SEQ ID NO: 438과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 445와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(g) VH 도메인은 아미노산 서열 SEQ ID NO: 452와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 459와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(h) VH 도메인은 아미노산 서열 SEQ ID NO: 467과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 473과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(i) VH 도메인은 아미노산 서열 SEQ ID NO 481:과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 488과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(j) VH 도메인은 아미노산 서열 SEQ ID NO: 494와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 501과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하는, 방법. - 제5항에 있어서, VH 도메인이 SEQ ID NO: 408과 적어도 95% 서열 동일성을 갖는 서열을 포함하고 VL 도메인이 SEQ ID NO: 415와 적어도 95% 서열 동일성을 갖는 서열을 포함하는, 방법.
- 제5항에 있어서,
(a) VH 도메인은 아미노산 서열 SEQ ID NO: 366을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 373을 포함하거나;
(b) VH 도메인은 아미노산 서열 SEQ ID NO: 380을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 387을 포함하거나;
(c) VH 도메인은 아미노산 서열 SEQ ID NO: 394를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 401을 포함하거나;
(d) VH 도메인은 아미노산 서열 SEQ ID NO: 408을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 415를 포함하거나;
(e) VH 도메인은 아미노산 서열 SEQ ID NO: 422를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 429를 포함하거나;
(f) VH 도메인은 아미노산 서열 SEQ ID NO: 438을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 445를 포함하거나;
(g) VH 도메인은 아미노산 서열 SEQ ID NO: 452를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 459를 포함하거나;
(h) VH 도메인은 아미노산 서열 SEQ ID NO: 467을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 473을 포함하거나;
(i) VH 도메인은 아미노산 서열 SEQ ID NO: 480을 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 487을 포함하거나;
(j) VH 도메인은 아미노산 서열 SEQ ID NO: 494를 포함하고 VL 도메인은 아미노산 서열 SEQ ID NO: 501을 포함하는, 방법. - 제5항 내지 제7항 중 어느 한 항에 있어서, VH 도메인이 아미노산 서열 SEQ ID NO: 408을 포함하고 VL 도메인이 아미노산 서열 SEQ ID NO: 415를 포함하는, 방법.
- 제1항 내지 제8항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원 결합 단편이 중쇄 및 경쇄를 포함하고,
(a) 중쇄는 아미노산 서열 SEQ ID NO: 368과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 375와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(b) 중쇄는 아미노산 서열 SEQ ID NO: 385와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 389와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(c) 중쇄는 아미노산 서열 SEQ ID NO: 396과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 403과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(d) 중쇄는 아미노산 서열 SEQ ID NO: 410과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 417과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(e) 중쇄는 아미노산 서열 SEQ ID NO: 424와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 432와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(f) 중쇄는 아미노산 서열 SEQ ID NO: 440과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 447과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(g) 중쇄는 아미노산 서열 SEQ ID NO: 454와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 461과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(h) 중쇄는 아미노산 서열 SEQ ID NO: 468과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 475와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(i) 중쇄는 아미노산 서열 SEQ ID NO: 482와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 489와 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하거나;
(j) 중쇄는 아미노산 서열 SEQ ID NO: 496과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 503과 적어도 85%, 90%, 또는 95% 서열 동일성을 갖는 서열을 포함하는, 방법. - 제9항에 있어서, 중쇄가 SEQ ID NO: 410과 적어도 95% 서열 동일성을 갖는 서열을 포함하고 경쇄가 SEQ ID NO: 417과 적어도 95% 서열 동일성을 갖는 서열을 포함하는, 방법.
- 제9항에 있어서,
(a) 중쇄는 아미노산 서열 SEQ ID NO: 368을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 375를 포함하거나;
(b) 중쇄는 아미노산 서열 SEQ ID NO: 382를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 389를 포함하거나;
(c) 중쇄는 아미노산 서열 SEQ ID NO: 396을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 403을 포함하거나;
(d) 중쇄는 아미노산 서열 SEQ ID NO: 410을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 417을 포함하거나;
(e) 중쇄는 아미노산 서열 SEQ ID NO: 424를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 432를 포함하거나;
(f) 중쇄는 아미노산 서열 SEQ ID NO: 440을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 447을 포함하거나;
(g) 중쇄는 아미노산 서열 SEQ ID NO: 454를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 461을 포함하거나;
(h) 중쇄는 아미노산 서열 SEQ ID NO: 468을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 475를 포함하거나;
(i) 중쇄는 아미노산 서열 SEQ ID NO: 482를 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 489를 포함하거나;
(j) 중쇄는 아미노산 서열 SEQ ID NO: 496을 포함하고 경쇄는 아미노산 서열 SEQ ID NO: 503을 포함하는, 방법. - 제9항 내지 제11항 중 어느 한 항에 있어서, 중쇄가 아미노산 서열 SEQ ID NO: 410을 포함하고 경쇄가 아미노산 서열 SEQ ID NO: 417을 포함하는, 방법.
- 제1항 내지 제12항 중 어느 한 항에 있어서, 항-ICOS 항체가 인간 IgG1 항체인, 방법.
- 제1항 내지 제13항 중 어느 한 항에 있어서, 항-ICOS 항체가 KY1044인, 방법.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 0.5 mg 내지 약 10 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 0.8 mg 내지 약 8 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 대상체에 약 8 mg 미만의 용량으로(예를 들어, 7.5 mg 이하, 7 mg 이하의 용량으로) 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 0.8 mg 내지 약 2.4 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 2.4 mg 내지 약 8 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 0.8 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 2.4 mg의 용량으로 대상체에 투여되는, 방법.
- 제15항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 약 8 mg의 용량으로 대상체에 투여되는, 방법.
- 제1항 내지 제22항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 2 내지 6주마다, 예를 들어 2주마다, 3주마다, 4주마다, 5주마다 또는 6주마다 대상체에 투여되는, 방법.
- 제1항 내지 제23항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 3주마다 대상체에 투여되는, 방법.
- 제1항 내지 제23항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 6주마다 대상체에 투여되는, 방법.
- 제1항 내지 제22항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 매월 대상체에 투여되는, 방법.
- 제1항 내지 제26항 중 어느 한 항에 있어서, 항-ICOS 항체 또는 이의 항원-결합 단편이 대상체에 적어도 6개월 동안, 예를 들어 6개월, 12개월, 또는 12개월 초과 동안 투여되는, 방법.
- 제1항 내지 제27항 중 어느 한 항에 있어서, 대상체에 제2 치료제를 투여하는 단계를 추가로 포함하는, 방법.
- 제28항에 있어서, 제2 치료제가 항-PD-L1 항체 또는 이의 항원 결합 단편을 포함하는, 방법.
- 제29항에 있어서, 항-PD-L1 항체가 아테졸리주맙인, 방법.
- 제29항 또는 제30항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 약 1200 mg의 용량으로 대상체에 투여되는, 방법.
- 제29항 내지 제31항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 대상체에 2 내지 6주마다, 예를 들어 2주마다, 3주마다, 4주마다, 5주마다 또는 6주마다 투여되는, 방법.
- 제29항 내지 제32항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 3주마다 대상체에 투여되는, 방법.
- 제29항 내지 32항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 6주마다 대상체에 투여되는, 방법.
- 제29항 내지 제31항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 매월 대상체에 투여되는, 방법.
- 제29항 내지 제35항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원 결합 단편이 적어도 6개월 동안, 예를 들어 6개월, 12개월, 또는 12개월 초과 동안 대상체에 투여되는, 방법.
- 제29항 내지 제31항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원-결합 단편이 3주마다 항-ICOS 항체 또는 이의 항원-결합 단편과 함께 대상체에 공동-투여되는, 방법.
- 제29항 내지 제31항 중 어느 한 항에 있어서, 항-PD-L1 항체 또는 이의 항원-결합 단편이 항-ICOS 항체 또는 이의 항원-결합 단편과 교대 용량으로 대상체에 투여되고, 예를 들어 항-PD-L1 항체 또는 이의 항원-결합 단편은 3주마다 투여되고, 항-ICOS 항체 또는 이의 항원-결합 단편은 6주마다 투여되는, 방법.
- 제1항 내지 제38항 중 어느 한 항에 있어서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병이 종양을 포함하는, 방법.
- 제1항 내지 제39항 중 어느 한 항에 있어서, 조절 T 세포(Treg)를 고갈시키고/시키거나 효과기 T 세포(Teff) 반응을 증가시킴으로써 치료법에 적합한 질환 또는 질병이 암을 포함하는, 방법.
- 제40항에 있어서, 암이 진행성 및/또는 전이성 암을 포함하는, 방법.
- 제40항 또는 제41항에 있어서, 암이 삼중 음성 유방암, 두경부 편평 세포 암종, 음경암, 췌장암, 비소세포 폐암, 간세포 암종, 식도암, 위암, 흑색종, 신장 세포 암종, 및/또는 자궁경부암을 포함하는, 방법.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US202163190016P | 2021-05-18 | 2021-05-18 | |
| US63/190,016 | 2021-05-18 | ||
| PCT/EP2022/063450 WO2022243378A1 (en) | 2021-05-18 | 2022-05-18 | Uses of anti-icos antibodies |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20240007760A true KR20240007760A (ko) | 2024-01-16 |
Family
ID=89324631
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020237043292A KR20240007760A (ko) | 2021-05-18 | 2022-05-18 | 항-icos 항체의 용도 |
Country Status (6)
| Country | Link |
|---|---|
| EP (1) | EP4340943A1 (ko) |
| JP (1) | JP2024518843A (ko) |
| KR (1) | KR20240007760A (ko) |
| CN (1) | CN117460529A (ko) |
| AU (1) | AU2022276349A1 (ko) |
| IL (1) | IL308603A (ko) |
-
2022
- 2022-05-18 AU AU2022276349A patent/AU2022276349A1/en active Pending
- 2022-05-18 IL IL308603A patent/IL308603A/en unknown
- 2022-05-18 KR KR1020237043292A patent/KR20240007760A/ko unknown
- 2022-05-18 JP JP2023571502A patent/JP2024518843A/ja active Pending
- 2022-05-18 CN CN202280036196.2A patent/CN117460529A/zh active Pending
- 2022-05-18 EP EP22731078.6A patent/EP4340943A1/en active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| IL308603A (en) | 2024-01-01 |
| EP4340943A1 (en) | 2024-03-27 |
| AU2022276349A1 (en) | 2024-01-04 |
| CN117460529A (zh) | 2024-01-26 |
| JP2024518843A (ja) | 2024-05-07 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR102473028B1 (ko) | 항-tim-3 항체 및 조성물 | |
| RU2769282C2 (ru) | Анти-PD-L1 и IL-2 цитокины | |
| KR102493282B1 (ko) | Tim3에 대한 항체 및 그의 용도 | |
| TWI822101B (zh) | 靶向pd-1、tim-3及lag-3的組合療法 | |
| CN108137687B (zh) | 抗ox40抗体及其用途 | |
| KR102569813B1 (ko) | Cd73에 대항한 항체 및 그의 용도 | |
| CN109689688B (zh) | 抗icos抗体 | |
| KR20230038311A (ko) | 항-cd73 항체와의 조합 요법 | |
| KR20200135830A (ko) | Mica 및/또는 micb에 대한 항체 및 그의 용도 | |
| KR20200044899A (ko) | 암의 치료 및 진단을 위한 tim-3 길항제 | |
| JP2023055904A (ja) | 癌免疫療法のための併用療法での多重特異性抗体 | |
| KR20200108870A (ko) | Tim3에 대한 항체 및 그의 용도 | |
| CN115380049A (zh) | 对生理性铁过载的治疗 | |
| KR20230165285A (ko) | Gdf15 신호전달의 치료적 억제제 | |
| KR20240007760A (ko) | 항-icos 항체의 용도 | |
| KR20240017054A (ko) | 항-icos 항체를 사용한 pd-l1 음성 또는 저발현 암의 치료 | |
| RU2795232C2 (ru) | Комбинированные лекарственные средства, нацеленные на pd-1, tim-3 и lag-3 | |
| NZ789986A (en) | Antibodies against tim3 and uses thereof |