KR101016198B1 - 종자 수확량 증가 방법 - Google Patents
종자 수확량 증가 방법 Download PDFInfo
- Publication number
- KR101016198B1 KR101016198B1 KR1020077011516A KR20077011516A KR101016198B1 KR 101016198 B1 KR101016198 B1 KR 101016198B1 KR 1020077011516 A KR1020077011516 A KR 1020077011516A KR 20077011516 A KR20077011516 A KR 20077011516A KR 101016198 B1 KR101016198 B1 KR 101016198B1
- Authority
- KR
- South Korea
- Prior art keywords
- eif
- plant
- plants
- dhs
- seq
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8261—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/415—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from plants
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A40/00—Adaptation technologies in agriculture, forestry, livestock or agroalimentary production
- Y02A40/10—Adaptation technologies in agriculture, forestry, livestock or agroalimentary production in agriculture
- Y02A40/146—Genetically Modified [GMO] plants, e.g. transgenic plants
Landscapes
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Organic Chemistry (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- Zoology (AREA)
- Molecular Biology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Biophysics (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Physics & Mathematics (AREA)
- Microbiology (AREA)
- Plant Pathology (AREA)
- Cell Biology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Botany (AREA)
- Medicinal Chemistry (AREA)
- Gastroenterology & Hepatology (AREA)
- Breeding Of Plants And Reproduction By Means Of Culturing (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Agricultural Chemicals And Associated Chemicals (AREA)
- Pretreatment Of Seeds And Plants (AREA)
- Peptides Or Proteins (AREA)
- Enzymes And Modification Thereof (AREA)
Abstract
Description
본 출원은 2004년 12월 3일 출원된 미국 잠정출원 제60/632,567호, 2005년 6월 14일 출원된 미국 잠정출원 제60/690,160호, 2005년 8월 25일 출원된 미국 잠정출원 제60/710,999호, 2005년 10월 21일 출원된 미국 잠정출원 제60/728,737호의 우선권을 주장하고 그 참고사항을 포함한다. 본 출원은 그 전체가 참고사항으로 포함된 1999년 7월 6일 출원되고 현재 포기된 미국 특허출원 제09/348,675호의 부분연속출원인 2000년 6월 19일 출원된 미국 특허출원 제09/597,771호(현재 미국특허 제6,538,182호)의 부분연속출원인 2000년 11월 29일 출원된 미국 특허출원 제09/725,019호의 부분연속출원인 2004년 6월 8일 출원된 미국 특허출원 제10/862,440호의 부분연속출원이다.
본 발명은 진핵성 개시 인자 5A("eIF-5A")의 특정 이소폼 및 eIF-5A를 인코드하는 폴리뉴클레오타이드 및 디옥시하이푸신 신타제("DHS") 및 DHS를 인코드하는 폴리뉴클레오타이드 및 이소폼 eIF-5A 및 DHS의 발현 조절 관련 방법에 관한 것이 다.
노화는 식물의 수명에 있어서 생물적 발달의 말기 현상이다. 이는 죽음의 조짐이고, 전체 식물, 기관, 꽃과 열매, 조직 및 개체 세포를 포함한 생물적 조직화의 다양한 레벨에서 발생한다.
노화의 착수는 내부적 및 외부적 모두의 다른 인자에 의해 유도될 수 있다. 노화는 식물의 수명에 있어서 열매, 꽃 및 잎과 같이 복잡하고, 매우 조절된 발달 단계이다. 노화는 세포 막과 거대 분자의 통합된 파괴 및 식물의 다른 부분으로 대사물질의 수반된 이동을 초래한다.
정상적인 식물 발달 동안 발생하는 프로그램화된 노화 이외에도 세포와 조직의 사멸 및 뒤이은 대사물질의 재이동이 외부, 환경적 인자에 대한 통합된 반응으로서 발생한다. 괴사(necrosis) 또는 아폽토시스(apoptosis)로도 표현되는 노화의 조기 개시를 유도하는 외부 인자는 병원균 공격뿐만 아니라 온도, 가뭄, 부족한 빛 또는 영양 공급과 같은 환경적 스트레스를 포함한다. 식물 조직은 환경적 스트레스에 노출되고, 스트레스 에틸렌이라고 일반적으로 알려진 에틸렌을 생성한다(Buchana-Wollaston, V., 1997, J. Exp. Botany, 48:181-199; Wright, M., 1974, Plant, 120:63-69). 에틸렌은 일부 식물에서 노화를 유발하는 것으로 알려져 있다.
노화는 수동성 과정이 아니라 오히려 특별한 유전자의 통합된 발현을 포함한 능동적으로 조절된 과정이다. 노화동안 총 RNA 수주의 감소 및 많은 유전자의 발현이 스위치 오프된다(Bate et al., 1991, J. Exper. Botany, 42, 801-11; Hensel et al., 1993, The Plant Cell, 5, 553-64). 그러나 노화 과정이 핵 유전자의 디 노보(de novo) 전사에 따라 달라진다는 증가하는 증거가 있다. 예를 들어 노화는 mRNA의 개시자 및 단백질 합성 및 핵의 제거(enucleation)에 의해 봉쇄된다. 노화 잎과 푸른 입을 이용한 시험관내에서의 번역 실험의 분자적 연구는 노화 잎에서의 잎 단백질 생성물의 변화된 패턴을 나타낸다(Thomas et al., 1992, J. Plant Physiol., 139, 403-12). 차별적 스크리닝 및 마이너스의(subtractive) 하이브리디제이션 기술을 이용하여 노화-유도된 유전자를 나타내는 많은 cDNA 클론이 아라비돕시스, 옥수수, 오이, 아스파라거스, 토마토, 쌀 및 감자와 같은 단자엽식물 및 쌍자엽식물 모두를 포함한 다른 식물 범위로부터 확인되었다. 노화 동안 특이적으로 발현되는 유전자의 확인은 진행되는 노화에 대한 디 노보 전사에 대한 필요성의 어려운 증거이다.
노화 동안 발생하는 이벤트는 매우 통합되어 괴사 및 사멸이 발생하기 전에 세포성 구성성분의 최대 이용을 가능하게 하는 것으로 보인다. 특이적인 시그널 의 인식과 유전자 발현의 케스케이드(cascade)의 유도를 포함한 복잡한 상호관계가 이러한 과정을 조절하기 위해 발생한다. 노화 관련 단백질을 인코딩하는 유전자의 발현은 일반적인 활성체 단백질을 통해 조절되고 이어서 호르몬 시그널에 의해 직접적으로 또는 간접적으로 활성된다. 초기 시그널링 또는 과정의 수반된 공동-작용에 포함된 메카니즘에 대해 알려진 것이 거의 없다.
통합된 유전자 발현은 개시 인자를 포함하여 전사 및 번역에 포함된 인자를 필요로 한다. 번역 개시 인자 유전자는 식물을 포함한 다양한 생물체에서 분리되고 특성화되었다. 번역 개시 인자는 mRNA 집단이 세포핵으로부터 이동되는 속도, 리보솜과 관련된 속도를 조절할 수 있고, 어느 정도까지 특정 mRNA의 안정성에 영향을 미칠 수 있다(Zuk et al., EMBO J. 17:2914-2925(1998). 실제로 전체적인 벙녁 활성에 필요 없는 이러한 번역 개시 인자 중의 하나는 번역을 위해 세포핵으로부터 세포질로 mRNA 특정 서브세트를 왕복하는 것으로 여겨진다(Jao et al., Cell Biochem. 86:590-600(2002); Wang et al., J. Biol. Chem. 276:17541017549(2001); Rosorius et al., J. Cell Sci., 112:2369-2380(1999)). 이러한 번역 인자는 진핵성 개시 인자 5A(eIF-5A)로 알려져 있고 아미노산 하이푸신을 포함한 것으로 알려진 유일한 단백질이다(Park et al., J. Biol. Chem. 263:15264-15269(1988)).
진핵성 번역 개시 인자 5A(elF-5A)는 진핵 세포 단백질 합성의 개시에 포함 된 약 17KDa 크기의 필수 단백질 인자이다. 이는 elF-5A에만 존재하는 것으로 알려진 독특한 변형된 아미노산인 하이푸신(hypusine)[N-(4-아미노-2-하이드록시부틸)리신]의 존재에 의해 특성화된다. 하이푸신은 폴리아민(polyamine), 스퍼미딘(spermidine)으로부터의 부틸아미노 그룹의 elF-5A의 특이적 리신 잔기의 아미노 그룹의 사이드 체인으로의 전달 및 하이드록실레이션(hydroxylation)을 통해 후-번역적으로 형성된다. elF-5A의 활성화는 스퍼미딘의 부틸아민 잔기의 elF-5A의 리신으로의 전달을 포함하고, 하이푸신 및 활성화 elF-5A를 형성한다. 진핵세포에서 디옥시하이푸신 신타제(DHS)는 elF-5A 내의 하이푸신의 후-번역적 합성을 매개한다. 상응하는 DHS 유전자는 식물에서 확인되지 않았으나 식물 elF-5A은 하이푸신을 포함하는 것으로 알려져 있다. 하이푸신 매개는 메티오닐-프로마이신(methionyl-puromycin) 에세이를 이용한 시험관 내에서의 elF-5A 활성에 필수적인 것으로 나타났다.
하이푸신은 디옥시하이푸신 신타제(DHS; EC 1.1.1.249) 및 디옥시하이푸신 수산화효소(DHH; EC 1.14.99.29)의 작용에 의해 보존된 리신 잔기의 전환을 통해 후-번역적으로 eIF-5A 상에 형성된다. DHS는 토마토(유전자은행 접근번호 AF296077), 아라비돕시스 탈리아나(Arabidopsis thaliana)(AT-DHS; 유전자은행 접근번호 AF296078), 담배(Ober and Hartmann, 1999), 카네이션(유전자은행 접근번호 AF296079) 및 바나나(유전자접근번호 AF296080)을 포함한 여러 식물종으로부터 분리된 반면 DHH에 대한 유전자는 인식되지 않았다.
DHS는 스퍼미딘으로부터 유래된 부틸아민기의 첨가를 통해 eIF-5A의 보존된 리신 잔기를 디옥시하이푸신으로 전환시킨다. 이후 이러한 eIF-5A의 중간물 형태는 DHH에 의해 수산화되어 하이푸신이 된다(Park et al., Biol. Signals 6:115-123(1997). eIF-5A의 디옥시하이푸신 및 하이푸신 형태 모두는 생체 내에서 mRNA에 결합할 수 있다(Liu et al., Biol. Signals 6:166-174(1997)). eIF-5A의 기능이 충분히 이해되지 않았더라도 이들이 세포 분열(Park et al., J. Biol. Chem. 263:15264-15269(1998); Tome et al., Biol. Signals 6:150-156(1997)) 및 노화(Wang et al., J. Biol. Chem. 276(20):17541-17549(2001))를 조절한다는 일부 증거가 있다. 참고문헌에 포함된 미국특허 제6,538,182호 및 미국출원 제09/725,019도 참조. 일부 생물체는 하나 이상의 eIF-5A 이소폼을 지닌 것으로 알려져 있고, 이는 각 이소폼이 세포 분열 및 노화와 같은 과정에 관련된 특정한 한벌의 mRNA에 대한 특정 셔틀이라는 전제에 적합하다.
Wang et al.은 증가된 수준의 DHS mRNA는 토마토의 열매 연화 및 자연적 및 스트레스-유도된 잎 노화와 상호관련됨을 증명하였다(Wang et al., J. Biol. Chem. 276(20):17541-17549(2001)). 더욱이 DHS 발현이 구성적 프로모터의 조절하에 DHS 안티센스 cDNA 단편을 도입함으로서 형질전환 토마토 식물 내에서 억제된 경우 이들 형질전환 식물로부터의 토마토 열매는 지연된 열매 연화 및 부패에 의해 증거가 된 극적으로 지연된 노화를 나타내었다. 참고문헌에 포함된 미국특허 제 6,538,182호 및 2003년 11월 29일에 출원된 미국출원 제09/725,019도 참조. 또한 이는 구성적 프로모터의 조절 하에 전체 길이 또는 3'UTR cDNA의 안티센스에 의해 아라비돕시스 탈리아나("AT") 내의 DHS의 하향-조절을 통해 증명되었다. 아라비돕시스 탈리아나 DHS("AT-DHS") 발현을 하향 조절하고 eIF-5A 활성화에 덜 유용하게 함으로서 노화는 약 2주까지 지연되었다(미국특허 제6,538,182호 참조). 노화가 지연되었을 뿐만 아니라 종자 수확량이 증가되었고, 스트레스 내성의 증가 및 생물량의 증가 형질전환 식물 내에서 관찰되었고, 각 표현형의 정도는 DHS의 하향-조절 정도에 의해 측정되었다. 서던 블럿에 의해 나타난 바와 같이(Wang et al., 2001)) 세포핵 밖으로의 노화 전사체의 왕복에 중요한 특정 eIF-5A 이소폼을 타겟하기 위해 토마토 및 아라비돕시스 탈리아나가 그들의 게놈 내에 단지 하나의 복제를 지니기 때문에 eIF-5A의 노화 특이적 이소폼은 노화-유도된 eIF-5A의 안티센스 컨스트럭트(3'UTR의)를 통해 또는 과발현된 유전자(즉, 센스 폴리뉴클레오타이드의 이용을 통한 과발현의 생성)의 하향-조절에 대한 식물의 자연적 능력을 이용함으로서 확인되고 특이적으로 하향-조절된다.
식물은 면역 체계가 없고 따라서 동시-억제라고 불리는 바이러스를 처리하는 유일한 방법을 지니고, 이는 바이러스성 RNA의 서열-특이적 분해를 유발한다. 트랜스유전자가 콜리플라워 모자이크 바이러스 이중 35S 프로모터와 같은 강한 구성적 프로모터의 조절 하에 있는 경우 이는 식물에 대한 바이러스 전사체로 여겨지고 노화 특이적 분해가 발생하나 트랜스유전자뿐만이 아니라 내생적 유전자도 마찬가 지이다(Fagard and Vaucheret, Annual Review. Plant Physiol. Plant Mol. Biol., June;51:167-194(2000)에서 조사됨). 내생적 유전자의 하향-조절에 대한 발현의 안티센스 억제보다 더욱 효과적이지 않은 경우 동시-억제가 효과적이라는 일부 증거가 있다.
발명의 요약
본 발명은 진핵성 개시 인자 5A("eIF-5A")의 이소폼: 노화-유도된 eIF-5A; 손상/병원체-유도된 eIF-5A; 생장 eIF-5A; 및 스트레스 eIF-5A 뿐만 아니라 이들 인자를 인코드하는 폴리뉴클레오타이드를 제공한다. 본 발명은 eIF-5A 이소폼의 안티센스 및 센스 폴리뉴클레오타이드를 제공한다. 또한 본 발명은 eIF-5A 이소폼의 센스 및 안티센스 폴리뉴클레오타이드를 포함한 발현 벡터를 제공한다. 또한 본 발명은 eIF-5A의 변형된 발현을 지닌 식물 및 그의 식물 부분 및 자손 이외에 이들 인자 발현의 조절(증가/상향-조절 또는 억제/하향-조절) 관련 방법에 관한 것이다. 또한 본 발명은 이들 eIF-5A 이소폼의 넉다운(knock down) 돌연변이도 고려한다.
또한 본 발명은 디옥시하이푸신 신타제("DHS") 및 DHS를 인코드하는 폴리뉴클레오타이드에 관한 것이다. 또한 본 발명은 DHS의 안티센스 및 센스 폴리뉴클레오타이드를 제공한다. 또한 본 발명은 DHS의 센스 및 안티센스 폴리뉴클레오타 이드를 포함한 발현 벡터를 제공한다. 또한 본 발명은 DHS의 변형된 발현을 지닌 식물 및 그의 식물 부분 및 자손 이외에 DHS 발현의 조절(증가/상향-조절 또는 억제/하향-조절) 관련 방법에 관한 것이다. 또한 본 발명은 이들 DHS 이소폼의 넉다운 돌연변이도 고려한다.
여기에 사용된 바와 같이 "식물"이라는 용어는 전체 식물, 식물 부분, 식물 세포 또는 식물 세포군을 나타낸다. 본 발명의 방법에 사용될 수 있는 식물의 타입은 예를 들어 에틸렌-민감성 및 에틸렌-불감성 식물을 포함하나 이에 한정적인 것은 아니다 : 살구, 사과, 오렌지, 바나나, 자몽, 배, 토마토, 딸기, 아보카도 등과 같은 열매 맺는 식물 ; 당근, 완두콩, 상추, 양배추, 순무, 감자, 브로콜리, 아스파라거스 등과 같은 야채 ; 카네이션, 장미, 국화 등과 같은 꽃 ; 옥수수, 쌀, 대두, 자주개자리 등과 같은 농작식물 ; 및 일반적으로 본 발명의 DNA 분자를 흡수하고 발현할 수 있는 어떤 식물. 이는 반수체, 이배체, 사배체 및 다배체를 포함한 다양한 배수성 레벨의 식물을 포함한다. 식물은 단자엽식물 또는 쌍자엽식물이다.
형질전환 식물은 여기서 게놈 내로 이형성 또는 상동성 노화-유도된 elF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS를 도입시킨 식물을 포함하나 이에 한정적인 것은 아닌 방법으로 유전적으로 변형된 식물이다. 변화된 유전적 물질은 단백질을 인코드하고, 조절적 또는 제어 서열로 구성되거나 안티센스 서열 또는 센스 서열이거나 이를 포함하거나 식물의 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS DNA 또는 mRNA 서열 또는 그의 일부에 대해 안티센스 또는 센스인 안티센스 RNA 또는 센스 RNA를 인코드한다.
여기에 사용된 "하이브리디제이션"이라는 용어는 일반적으로 프로브 서열과 타겟 서열의 성질에 따라 당업자에게 용이하게 명백한 적당한 스트린젠시 조건에서의 핵산의 하이브리디제이션을 의미하는데 사용된다. 하이브리디제이션 및 세척의 조건은 당 분야에 잘 알려져 있고, 인큐베이션 시간, 온도 및/또는 용액의 이온 강도를 변화시킴으로서 원하는 스트린젠시(stringency)에 따라 다른 조건의 조정이 용이하게 성취된다. 예를 들어 Sambrook, J. et al., Molecular Cloning : A Laboratory Manual, 2nd ed., Cold Spring Harbor Press, Cold Spring Harbor, NY, 1989 참조. 조건의 선택은 하이브리다이즈되는 서열의 길이, 특히 프로브 서열의 길이, 핵산의 상대 G-C 함유량 및 허용되는 미스매치(mismatch)의 양에 의해 규정된다. 더 낮은 정도의 상보성을 지닌 스트랜드 사이의 부분적인 하이브리디제이션이 요구될 때는 낮은 스트린젠시 조건이 바람직하다. 완벽한 또는 거의 완벽한 상보성이 요구될 때는 높은 스트린젠시 조건이 바람직하다. 일반적인 높은 스트린젠시 조건의 경우 하이브리디제이션 용액은 6X S.S.C, 0.01M EDTA, 1X Denhardt's 용액 및 0.5% SDS를 포함한다. 하이브리디제이션은 클론된 DNA의 단편의 경우 약 3∼4시간 동안, 진핵세포성 DNA의 경우 12∼16시간 동안 68℃에서 수행된다. 더 낮은 스트린젠시의 경우 하이브리디제이션의 온도는 이중나선의 녹는점(TM)인 42℃ 이하로 감소된다. TM은 용액의 이온 강도뿐만 아니라 G-C 함량 및 이중나선의 길이의 기능인 것으로 알려져 있다.
여기에 사용된 바와 같이 "실질적인 서열 동일성" 또는 "실질적인 상동성"이라는 용어는 뉴클레오타이드 서열 또는 아미노산 서열이 다른 뉴클레오타이드 또는 아미노산 서열과 실질적인 구조적 또는 기능적 동일성을 나타냄을 의미하는데 사용된다. 실질적인 서열 동일성 또는 실질적인 상동성을 지닌 서열 사이의 구조적 또는 기능적 차이점은 최소수준(de minimis)이 될 것이다 ; 즉 서열의 능력이 원하는 적용에 나타난 바와 같은 기능을 하게 할 수 없을 것이다. 차이점은 예를 들어 다른 종 사이의 코돈 용법(usage)의 고유한 변이에 기인한다. 2 이상의 다른 서열 사이에 서열 중복 또는 유사성의 유의적인 양이 있거나 또는 다른 서열이 길이나 구조에 있어 다르더라도 유사한 물리적 특성을 나타내는 경우 구조적 차이점은 최소수준으로 여겨진다. 이러한 특성은 예를 들어 한정된 조건하에서 하이브리다이즈하는 능력 또는 단백질의 경우에 있어서 면역적 교차반응성, 유사한 효소 활성 등을 포함한다. 이들 특성의 각각은 당 분야 알려진 방법에 의해 당업자에 의해 용이하게 측정될 수 있다.
더욱이 2개의 뉴클레오타이드 서열이 그들 사이에 적어도 약 70%, 더욱 바람직하게는 80%, 가장 바람직하게는 90% 이상의 서열 유사성을 지니는 경우 "실질적으로 상보적"이다. 2개의 아미노산 서열은 폴리펩타이드의 활성 부분 사이에 적어도 약 50%, 바람직하게는 70% 이상의 유사성을 지니는 경우 실질적으로 상동성이다.
여기에 사용된 바와 같이 DNA 또는 RNA 분자의 "상응하는 부분에 하이브리다이즈하는"이라는 구문은 올리고뉴클레오타이드, 폴리뉴클레오타이드 도는 어떤 뉴클레오타이드 서열을 하이브리다이즈하고(센스 또는 안티센스 방향으로), 거의 동일한 크기의 또다른 핵산 분자의 서열을 인식하고 하이브리다이즈하고, 적당한 조건하에서 하이브리디제이션에 효과를 나타내는 것에 충분한 서열 유사성을 지닌 분자를 의미한다. 예를 들어 토마토 손상/병원체 유도된 eIF-5A의 3' 코딩 또는 비-코딩 구역으로부터의 100 뉴클레오타이드 길이 안티센스 분자는 2개의 서열 사이에 약 70% 이상의 서열 유사성이 있는 한 각각 AT 손상/병원체 유도된 eIF-5A 유전자 또는 어떠한 다른 식물 손상/병원체 유도된 eIF-5A 유전자의 3' 코딩 또는 비-코딩 구역 내의 뉴클레오타이드 서열의 거의 100 뉴클레오타이드 부분을 인식하고 하이브리다이즈할 것이다. "상응하는 부분"의 크기는 하이브리디제이션에서의 일부 미스매치를 허용하여 "상응하는 부분"이 하이브리다이즈 하는 분자보다 더 크거나 더 작은, 예를 들어 20∼30% 더 크거나 또는 더 작은, 바람직하게는 약 12∼15% 더 크거나 더 작게 될 것이다.
여기서 사용된 핵산(또는 폴리뉴클레오타이드)의 "기능적 유도체"라는 용어는 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A를 인코드하는 유전자 또는 뉴클레오타이드 서열 또는 DHS를 인코드하는 DNA의 단편, 변이체, 상동체 또는 아날로그를 의미한다. 기능적 유도체는 본 발명에 따른 사용을 가능하게 하는 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A를 인코드하는 유전자 또는 뉴클레오타이드 서열 또는 DHS를 인코드하는 DNA의 기능의 적어도 일부를 보유한다. 이러한 기능은 고유의 분리된 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS 또는 또다른 식물 유래의 실질적인 상동성 DNA 또는 그의 mRNA 전사체와 높은 스트린전시 조건 하에서 하이브리다이즈할 수 있는 능력을 포함하고, 안티센스 방향의 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS는 노화-유도된 eIF-5A, 손상/병원체 유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS의 발현을 저해한다.
유전자 또는 DNA 서열의 "단편"은 분자의 서브세트 즉, 더 짧은 폴리뉴클레오타이드 또는 올리고뉴클레오타이드를 나타낸다. "변이체"는 하나 이상의 치환된 뉴클레오타이드를 지닌 뉴클레오타이드 치환 변이체와 같은 전체 유전자 또는 그의 단편을 나타내나 이는 특정한 유전자와 하이브리다이즈할 수 있고, 고유의 DNA와 하이브리다이즈하는 mRNA 전사체를 인코드할 수 있는 능력을 유지한다. "상동체"는 다른 식물속 또는 종으로부터의 단편 또는 변이체를 나타낸다. "아날로그"는 실질적으로 유사하거나 또는 전체 분자, 그의 변이체 또는 단편에 대해 기능하는 비-자연적 분자를 나타낸다.
"발현 조절"에 의해 발현이 억제되거나 상향-조절됨이 의미된다. "발현의 억제"는 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS와 같은 타겟 유전자로부터의 단백질 및/또는 mRNA 생성물의 부재 또는 검출 가능한 수준 감소를 나타낸다. "상향-조절" 또는 "과발현"은 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS와 같은 타겟 유전자로부터의 단백질 및/또는 mRNA 생성물 수준의 검출 가능한 증가를 나타낸다. DHS 또는 eIF-5A의 어떠한 이소폼 활성의 감소도 발현 조절 의미에 포함된다. 예를 들어 식물 내 DHS의 넉다운 돌연변이체를 생성하고 수득된 식물은 야생형 DHS보다 더 낮은 수치 또는 효율로 eIF-5A를 활성화시키는 DHS를 지닐 것이다. 또한 발현 조절은 DHS 또는 eIF-5A 활성을 저해하거나 감소시키는 화학물질 또는 약물의 이용도 포함한다.
본 발명의 분리된 폴리뉴클레오타이드 및 펩타이드는 자연적 원료로부터 분리되거나 재조합적으로 생성되거나 합성된 것을 포함한다. 본 발명의 분리된 단백질은 바람직하게는 말토즈 결합 단백질과 융합된 eIF-5A 또는 DHS를 포함하는 융합 단백질로서 발현되는 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A 또는 DHS를 포함한다.
본 발명의 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A, 스트레스 eIF-5A 또는 DHS 펩타이드의 "기능적 유도체"는 eIF-5A 이소폼 또는 DHS에 대해 특이적인 항체와의 적어도 일부분 이상의 활성 또는 면역 교차 반응성을 보유한 단편, 변이체, 아날로그 또는 화학적 유도체를 포함한다. eIF-5A 또는 DHS 펩타이드의 단편은 어떠한 분자 서브세트도 나타낸다. 변이체 펩타이드는 예를 들어 당분야에 잘 알려진 방법을 이용하여 직접적인 화학 합성에 의해 제조된다. eIF-5A 또는 DHS 펩타이드의 아날로그는 전체 단백질 또는 그의 단편과 실질적으로 유사한 비-천연 단백질을 나타낸다. eIF-5A 또는 DHS의 화학적 유도체는 일반적으로 펩타이드 또는 펩타이드 단편의 일부가 아닌 추가적인 화학적 모이어티를 포함한다. 선택된 측쇄 또는 말단 잔기와 반응할 수 있는 유기 유도제와 펩타이드의 타겟된 아미노산 잔기를 반응시킴으로서 변형이 펩타이드 또는 그의 단편 내로 도입된다.
본 발명에 따른 eIF-5A 또는 DHS는 본 발명의 뉴클레오타이드 서열로 형질전환된(센스 방향으로) 세포를 배양하고, 세포가 단백질을 합성하게 한 후 사용된 클로닝 프로토콜에 따라 배양액 배지로부터 또는 세포 추출물로부터 본 단백질을 자유 단백질 또는 융합 단백질로서 분리함으로서 생성된다. 대안으로, 본 단백질은 무-세포 시스템 내에서 생성될 수 있다(Ranu et al., Meth. Eznymol., 60:459-484(1979)).
플라스미드 DNA의 제조, 제한 효소 분해, DNA의 아가로스 겔 전기영동, 단백질의 폴리아크릴아미드 겔 전기영동, PCR, RT-PCR, 서던 블럿, 노던 블럿, DNA 라이게이션 및 박테리아 형질전환이 당분야에 잘 알려진 통상의 방법을 이용하여 수행되었다. 예를 들어 Sambrook, J. et al., Molecular Cloning : A Laboratory Manual, 2nd ed., Cold Spring Harbor Press, Cold Spring Harbor, NY, 1989 참조. 핵산 하이브리디제이션 기술은 Sambrook에 의해 개시되었다.
본 발명에 따른 재조합 뉴클레오타이드 분자를 구축하기 위한 절차는 Sambrook et al.,에서 개시되었다: 참고문헌에 포함된 Molecular Cloning : A Laboratory Manual, 2nd ed., Cold Spring Harbor Press, Cold Spring Harbor, NY, (1989) 및 Maniatis, T. et al, Molecular mechanisms in the Control of Gene expression, eds., Nierlich et al., eds., Acad. Press, N.Y.(1976).
본 발명에 따라 제조된 형질전환 식물은 당분야에 알려진 어떠한 식물 형질전환 방법도 이용하여 DNA 형질전환에 의해 제조된다. 식물 형질전환 방법은 아그로박테리움 튜메파시엔스(Agrobacterium tumefaciens)와 식물, 조직 또는 세포의 직접적인 동시-배양 또는 직접적인 감염(Miki et al., Meth. in Plant Mol. Biol. and Biotechnology, (1993), p.67-88); 원형질체 내로의 직접적인 유전자 전이 또는 원형질체 흡수(Paszkowski et al., 유도된 J., 12:2717 (1984)); 일렉트로포레이션(electroporation)(Fromn et al., Mature, 319:710 (1986)); 입자 포격(particle bombardment)(Klein et al., BioTechnology, 6:559-563 (1988)); 묘목 또는 식물의 분열조직 내로의 주입(De LePena et al., Nature, 325:274-276 (1987)); 배양된 세포 및 조직의 프로토플라스트 내로의 주입(Reich et al., BioTechnology, 4:1001-1004 (1986))을 포함한다.
일반적으로 완전한 식물은 형질전환 과정으로부터 수득된다. 식물은 프로토플라스트, 캘러스, 조직 부분 또는 외식편 등으로부터 재생된다. 잎, 꽃, 열매, 종자 등과 같이 노화-유도된 DHS의 발현이 변화된 재생된 식물로부터 수득된 식물부가 여기에 사용된 바와 같이 "식물"의 정의에 포함된다. 또한 재생된 식물의 자손, 변이체 및 돌연변이도 "식물"의 정의에 포함된다.
일반적 eIF-5A
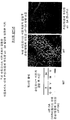
본 발명은 eIF-5A(진핵성 번역 개시 인자 5A)에 관한 것이다. eIF-5A는 잎, 꽃 및 열매의 자연적 노화시 프로그램화된 세포 사멸; 스트레스-유도된 조숙한 노화, 물관생성 관련 세포 사멸 및 병원체 침입 관련 세포 사멸을 조절한다. eIF-5A는 2개의 효소 디옥시하이푸신 신타제(DHS) 및 디옥시하이푸신 가수분해효소(DHH)에 의해 후번역적으로 활성화된다. 도 111 참조. eIF-5A는 통상적인 번역 개시 인자가 아니라 오히려 셔틀(shuttle) 단백질로 기능한다. 3 또는 4개의 별개의 eIF-5A 이소폼이 확인되었다. 3개의 다른 이소폼은 번역을 위해 세포핵에서 리보솜으로 다른 하위 집단의 mRNA를 선택적으로 이동시킨다. 하나의 이소폼은 세포 분열 및 생장을 조절하고 2 또는 3개의 이소폼은 세포 사멸을 조절한다. 예를 들어 도 1은 아라비돕시스에서 분리된 3개의 다른 eIF-5A 이소폼을 나타낸다. 도 8∼10은 eIF-5A에 대한 이소폼 특이적 항체로 프로브된 웨스턴 블럿을 나타낸다("At"는 아라비돕시스를 의미함). 이들 도면은 아라비돕시스 내 3개의 다른 이소폼의 발현 수치 및 다른 생명주기 및 이벤트를 나타낸다. 더 큰 게놈을 지닌 식물 즉 농작 식물 내에는 4개의 eIF-5A 이소폼이 존재한다. 하나의 이소폼은 세포 분열 및 세포 생장에 관련되고 3개의 이소폼은 세포 사멸에 관련된다.
본 발명은 다른 eIF-5A 이소폼에 관한 것이다: 노화-유도된 eIF-5A; 손상 유도된 eIF-5A; 스트레스 eIF-5A 및 생장 eIF-5A. 노화-유도된 eIF-5A는 식물 또는 식물 조직 수명 종말시 노화를 유도한다. 또한 스트레스 eIF-5A는 환경 스트레스 즉 가뭄 스트레스에 반응하여 노화를 조숙하게 유도하는 사멸 이소폼이다. 질병/손상 eIF-5A는 병원체 침입 또는 손상 이벤트에 반응하여 세포 사멸을 유도하는 사멸 이소폼이다. 생장 eIF-5A는 식물 생장을 증진시킨다. eIF-5A는 하나의 지위에서 생장을 조절하고 다른 하나의 지위에서 사멸을 조절하는 생물학적 스위치로서 기능하는 것으로 판단된다. 사멸 지위에서 하나 이상의 사멸 이소폼이 강하게 발현되고 생장 이소폼은 약하게만 발현되거나 전혀 발현되지 않는다. 생장 지위에서 생장 이소폼이 강하게 발현되고 사멸 이소폼은 약하게만 발현되거나 전혀 발현되지 않는다.
3∼4개 eIF-5A 이소폼이 존재하나 단지 하나의 DHS 이소폼이 존재하는 것으로 판단되고 이러한 단일 이소폼은 모든 eIF-5A 이소폼을 활성화시킨다(하이푸신화).
본 발명은 다양한 식물종으로부터 분리된 다양한 eIF-5A 이소폼 및 다양한 eIF-5A 이소폼을 분리하는 방법을 제공한다. 또한 본 발명은 이들 본 발명의 다양한 eIF-5A 이소폼을 인코드하는 폴리뉴클레오타이드를 제공한다. 또한 본 발명은 eIF-5A 이소폼의 안티센스 폴리뉴클레오타이드 및 이러한 폴리뉴클레오타이드 또는 안티센스 폴리뉴클레오타이드를 포함한 발현 벡터를 제공한다. 일부의 실시태양에서 식물을 형질전환하기 위해 eIF-5A 이소폼의 안티센스 폴리뉴클레오타이드를 포함한 발현 벡터의 이용을 통해 내생적 eIF-5A의 발현을 억제하는 방법이 제공된다. 일부 실시태양에서 센스 방향으로 eIF-5A 이소폼의 폴리뉴클레오타이드를 포함한 발현 벡터를 제공함으로서 내생적 eIF-5A 이소폼을 상향-조절하는 방법이 제공된다.
다른 이소폼은 식물의 수명 단계 또는 식물의 조건에 따라 다르게 자연적으로 상향 또는 하향-조절된다. 예를 들어 노화 조직에서 노화-유도된 eIF-5A 이소폼은 상향-조절된다. 노화-유도된 eIF-5A는 번역을 위해 세포핵으로부터 세포질로 mRNA의 특정 서브세트를(노화 경로에 관련된) 왕복시킴으로서 식물 또는 식물 조직의 추가적인 노화에 관여되는 것으로 여겨진다. 노화-유도된 eIF-5A의 발현을 하향 조절하거나 억제함으로서 노화가 식물 및/또는 식물 조직 내에서 지연될 수 있다. 지연된 노화는 비-형질전환된 또는 야생형 식물과 비교시 더 큰 생물량, 증가된 열매 저장 수명, 증가된 꽃 저장 수명, 증가된 종자 크기 및 증가된 종자 수확량을 지님으로서 형질전환 식물 내에서 명백해진다.
식물 및/또는 식물 조직이 냉기, 탈수 또는 기계적 힘과 같은 손상 이벤트에 노출되거나 병원체에 노출되는 경우 손상/병원체-유도된 eIF-5A 이소폼이 상향-조절된다. 손상/병원체-유도된 eIF-5A의 발현을 하향 조절함으로서 비-형질전환된 또는 야생형 식물의 독성 손상에 대한 저항성과 비교시 병원균 이입으로 인한 독성 손상에 대한 증가된 저항성이 식물에 부여된다.
식물이 생장 단계에 있는 경우 생장 eIF-5A 이소폼이 상향-조절된다. 생장 eIF-5A를 상향-조절함으로서 수득된 형질전환 식물은 증가된 종자 크기, 증가된 생물량 및 증가된 종자 수확량을 지닌다.
도 1은 아라비돕시스 탈리아나("At")로부터 분리된 3가지 eIF-5A 이소폼의 정렬을 나타낸다. 도 2는 이들 3가지 이소폼의 코딩 구역의 정렬을 나타낸다. 도 305는 3가지 이소폼의 게놈 서열을 제공한다.
웨스턴 블럿(도 8 참조)은 다른 식물 수명 단계에서의 이들 3가지 이소폼 내의 발현을 나타낸다. 도 8은 노화-유도된 인자 eIF-5A 이소폼의 양이 잎 연령의 증가로서 증가됨을 나타낸다. 이는 개화되지 않은 꽃봉오리, 장각과 또는 줄기에서 나타나지 않고 흡수된 종자 내에 나타난다. 흡수된 종자내에 생장하는 배아뿐만 아니라 자엽 조직도 있다. 따라서 자엽 조직은 배아가 생장함에 따라 노화되기 때문에 노화-유도된 eIF-5A는 흡수된 종자 내에 존재한다. 손상/병원체-유도된 eIF-5A는 이들 조직의 수확이 일부 손상을 유도할 때 장각과, 종자 및 줄기에 나타난다.
다른 이소폼 사이 또는 다른 식물종 내의 다른 이소폼 사이의 높은 정도의 상동성(약 85%)이 존재하나 다른 이소폼은 3'UTR 내에서 서로 다르다. 종간뿐만아니라 이소폼 사이에서 높게 보존된 구역은 하이푸신 사이트로 여겨지는 지역이다. 하이푸신 사이트는 하기 아미노산으로 여겨진다: 5'-CKVVEVSTSKTGKHGHAKCHFV-3'(서열번호: 32). 다양한 eIF-5A 이소폼 및 여러 식물종의 정렬에 대해 도 85 참조.
노화-유도된 eIF-5A
노화-유도된 eIF-5A는 노화 조직 내에서 발현된다. 도 116 참조. 본 발명은 아라비돕시스 탈리아나, 토마토, 카네이션, 상추 및 자주개자리 식물을 포함한 다양한 식물 내 노화-유도된 eIF-5A의 발견에 관한 것이다. 노화-유도된 eIF-5A는 노화 조직에서 상향-조절되고 식물 및 식물 조직 내 형태 변화에 관련된 노화의 유도에 관련된다. 노화 이소폼이 하향 조절시 자연적 노화가 지연된다. 노화-유도된 eIF-5A의 하향 조절(동시-억제를 달성하기 위한 노화-유도된 eIF-5A의 안티센스 컨스트럭트의 이용, 센스 컨스트럭트의 이용, 노화-유도된 eIF-5A의 넉다운 돌연변이체, 감소된 DHS 발현(eIF-5A를 활성화시키는데 필수적), DHS의 넉다운 돌연변이체 또는 DHS 저해제의 이용을 통한)은 증가된 식물 생물량, 지연된 열매 연화 또는 부패, 상추 잎과 같은 절단 꽃 또는 식물 조직의 지연된 갈변, 증가된 종자 수확량 및 증가된 종자 크기를 포함한 형질전환 식물 내 다양한 형태적 변화를 유발한다. 도 35, 36 및 117(아라비돕시스 내 지연된 잎 노화), 도 112 및 113(절단 카네이션 꽃의 더 긴 저장 수명), 도 115(토마토의 지연된 부패) 및 도 119(지연된 물관부 형성) 참조.
노화-유도된 eIF-5A가 노화-유도된 eIF-5A의 센스 컨스트럭트로 상향-조절되는 경우 기능-획득 표현형이 유발된다. 예를 들어 물관부 형성의 증가가 주목된다. 도 120 참조.
DHS 및 노화-유도된 eIF-5A의 역할을 더욱 해명하기 위해 DHS 및 노화-유도된 eIF-5A 모두의 프로모터 활성이 리포터 유전자로서 GUS를 사용하여 형질전환 아라비돕시스 식물의 생장 및 발달 동안 특성화되었다. 도 118은 노화-유도된 eIF-5A이 도관 조직에서 발현됨을 나타낸다. DHS::GUS 및 노화-유도된 eIF-5A::GUS 발현 수치는 잎 노화 착수시 강하게 상향 조절되었고, 이러한 높은 발현은 잎 노화가 종결될 때까지 유지되었다. 더욱이 DHS 및 노화-유도된 eIF-5A는 발달하는 자엽, 로제트 잎 및 줄기의 도관 조직에서 일시적이고 동시-좌표적으로 발현되었다. GUS 발현의 시간조절 및 위치는 물관생성시 노화-유도된 eIF-5A의 역할을 의미한다. 더욱이 DHS 및 노화-유도된 eIF-5A는 화분 생성의 퇴화 단계 동안 꽃밥 조직에서 동시-좌표적으로 발현되었다. 따라서 이들 결과는 DHS 및 노화-유도된 eIF-5A가 아라비돕시스 내의 물관생성에 관련된 프로그램화된 세포 사멸, 잎 노화 및 꽃 노화를 조절함을 나타낸다.
또한 노화-유도된 eIF-5A는 발달하는 물관부 내에서 발현된다. 노화-유도된 eIF-5A가 물관부 생성에 중요한 역할을 하는지 여부를 측정하기 위해 전체-길이 cDNA가 형질전환 아라비돕시스 식물 내에서 지속적으로 과발현되었다. 이들 식물은 대조군 식물과 비교시 더 큰 로제트 잎뿐만 아니라 더 크고 두꺼운 화서 줄기를 지녔고, 이는 더 빠른 생장을 반영한다. 더욱이 그의 주요 화서 줄기는 물관부 발달시 유의적인 증가를 나타내었다. 도관 및 총생내 영역 모두에서 세포층 및 조직 두께는 라인에 따라 다르게 각각 80% 또는 60%까지 증가되었다. 증가된 물관부 생장은 1차 물관부보다 2차 물관부에서 더 많이 표명되었다. 더욱이 노화-유도된 eIF-5A의 구성적 안티센스 억제는 대조군 식물과 비교시 발육 저지된 신장 및 지연된 잎 노화뿐만 아니라 물관부 조직 내 ∼30% 감소를 유발하였다.
eIF-5A는 보존된 리신의 하이푸신으로의 전환에 의해 후-번역적으로 변형된다. 이들이 물관생성을 조절하는 노화-유도된 eIF-5A의 하이푸신화 형태임을 확증하기 위해 하이푸신화될 수 없는 노화-유도된 eIF-5A의 돌연변이 형태의 구성적 과발현을 지닌 형질전환 아라비돕시스를 생성하였다. 돌연변이는 보존된 리신에 대한 AAG를 GCG(알라닌용)로 코돈을 변화시킴으로서 이루어졌고, 따라서 노화-유도된 eIF-5A의 하이푸신화가 불가능하게 되었다. 돌연변이 집단에서 대조군 식물 대비 물관부 발달시 어떠한 변화도 없었다. 따라서 노화-유도된 eIF-5A는 변절 요소의 프로그램화된 사멸을 촉진시킴으로서 물관생성을 양성적으로 조절하는 것으로 판단되고, 이러한 기능은 하이푸신화를 통한 단백질의 활성화를 필요로 한다.
따라서 본 발명의 하나의 실시태양은 아라비돕시스 탈리아나로부터 분리된 노화-유도된 eIF-5A이다. 아미노산 서열은 도 59에 제공되고 서열번호: 16이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 59에 제공되고 서열번호: 15이다.
본 발명의 또다른 실시태양은 토마토로부터 분리된 노화-유도된 eIF-5A이다. 아미노산 서열(서열번호: 12, 127) 및 이를 인코드하는 폴리뉴클레오타이드(각각 서열번호: 11, 126)은 도 57(서열번호: 11∼12) 및 86(서열번호: 126∼127)에 나타나있다.
본 발명의 또다른 실시태양은 카네이션으로부터 분리된 노화-유도된 eIF-5A이다. 아미노산 서열은 도 58에 제공되고 서열번호: 14이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 58 및 86에 제공되고 서열번호: 13이다.
본 발명의 또다른 실시태양은 자주개자리로부터 분리된 노화-유도된 eIF-5A이다. 아미노산 서열은 도 138에 제공되고 서열번호: 175이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 138에 제공되고 서열번호: 174이다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 서열 상동성을 지니고 높은 스트린젠시 조건 하에서 열거된 서열번호의 상보체에 하이브리다이즈하고 노화-유도된 eIF-5A를 인코드하는 노화-유도된 eIF-5A의 분리된 폴리뉴클레오타이드 제공한다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 아미노산 서열 상동성을 지니는 노화-유도된 eIF-5A의 노화-유도된 eIF-5A 분리된 폴리펩타이드를 제공한다.
또한 본 발명은 노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드를 제공한다. 안티센스 폴리뉴클레오타이드는 발현을 억제할 수 있는 길이이면 된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하고 다른 특정하게 바람직한 실시태양에서 eIF-5A의 다른 이소폼이 3'UTR에서 이소폼 내의 다양한 정도의 변이를 지니기 때문에 안티센스 폴리뉴클레오타이드는 3'UTR로 지시된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 5'-비-코딩 서열로 지시된다. 5'-비-코딩 서열에 아마도 상보적인 안티센스 폴리뉴클레오타이드는 전사 인자를 인코드하는 유전자 발현의 효과적인 억제제로 알려져 있다(Branch, M.A., Molec. Cell Biol., 13:4282-4290(1993)).
여기서 사용된 "노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드"라는 용어는 열거된 서열번호의 상보체의 100% 상보성을 공유하는 안티센스 폴리뉴클레오타이드를 포함할 뿐만 아니라 기능적 변이체인 안티센스 폴리뉴클레오타이드도 포함한다. 기능적 변이체는 노화-유도된 eIF-5A의 상응하는 부분에 적어도 80% 이상의 서열 상동성을 지니고 높은 스트린젠시 조건 하에서 이에 하이브리다이즈하는 자연적 또는 인공적 변이체이다. 또한 변이체는 발현 벡터 내로 도입되고 이러한 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합되는 경우 내생적 노화-유도된 eIF-5A의 발현을 조절할 수 있는 본 발명에 의해 의도된 기능을 지녀야 한다. 당업자는 전사체에 결합하고 유전자 발현을 감소시키거나 억제하기 위해 안티센스 폴리뉴클레오타이드의 능력에 극적으로 영향을 미치지 않는 서열 내에 실질적이지 않는 변화가 생성될 수 있음을 인식할 수 있다. 따라서 "안티센스 폴리뉴클레오타이드"라는 용어는 전사체에 실질적으로 상보적이고 전사체에 특이적으로 결합하고 유전자 발현을 억제하거나 감소시키는 능력을 유지하는 폴리뉴클레오타이드를 포함한다. 안티센스의 일반적인 논의를 위해 Albert et al., Molecular Biology of the Cell, 2nd ed., Garland Publishing, Inc. New York, New York, 1989(특히 페이지 195-196, 참고사항으로 여기에 포함됨) 참조.
본 발명의 하나의 실시태양은 노화-유도된 eIF-5A 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드(상기 기술된 본 발명의)를 제공한다. 벡터는 플라스미드, 바람직하게는 바이러스성 또는 식물 세포 또는 박테리아 세포 내에서 인크드되는 유전자를 복제하고 발현하는 당분야에 알려진 다른 벡터이다. 벡터는 염색체적으로 통합되어 노화-유도된 eIF-5A RNA의 원하는 안티센스 폴리뉴클레오타이드를 생성하도록 전사될 수 있다. 이러한 플라스미드 또는 바이러스성 벡터는 당분야에서 일반적인 재조합 DNA 기술에 의해 구축될 수 있다. 예를 들어 벡터는 원핵성 숙주 및 본 발명에 따른 안티센스 폴리뉴클레오타이드 내에서 기능적인 복제 시스템을 포함한 플라스미드 벡터이다. 대안으로 벡터는 아그로벡테리움 및 본 발명에 따른 안티센스 폴리뉴클레오타이드 내에서 기능적인 복제 시스템을 포함한 플라스미드이다. 아그로박테리움 내에서 복제할 수 플라스미드는 당분야에 잘 알려져 있다. Miki et al., Procedures for Introducing Foreign DNA Into Plants, Methods in Plant Molecular Biology and Biotechnology, Eds. B.R. Glick and J.E. Thompson. CRC Press (1993) pp.67-83 참조.
또한 벡터는 폴리뉴클레오타이드을 발현하게 하도록 폴리뉴클레오타이드에 실시가능하게 결합된 조절 서열을 포함한다. 조절 서열은 형질전환된 식물 세포 내에서 기능적인 프로모터를 포함한다. 프로모터는 유도성, 구성적 또는 조직 특이적이다. 이러한 프로모터는 당업자에 의해 잘 알려져 있다.
유전자의 센스 또는 안티센스 전사체를 생성하는 노화-유도된 DHS 유전자와의 결합에 유용한 프로모터 조절 요소는 일반적으로 어떤 식물 프로모터 및 더욱 특별하게는 피그 와트 모자이크 바이러스 35S 프로모터, 콜리플라워 모자이크 바이러스 프로모터, CaMV35S 프로모터 또는 MAS 프로모터와 같은 구성적 프로모터 또는 카네이션 화판 GST1 프로모터 또는 아라비돕시스 SAG12 프로모터와 같은 조직-특이적 또는 노화-유도된 프로모터를 포함한다(예를 들어 J.C. Palaqui et al., Plant Physiol., 112:1447-1456 (1996); Morton et al., Molecular Breeding, 1:123-132 (1995); Forbert et al., Plant Journal, 6:567-577 (1994); Gan et al., Plant Physiol., 113:313 (1997) 참조). 바람직하게는 본 발명에 사용된 프로모터는 구성적 프로모터이다. 노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드를 사용하는 경우 SAG12 프로모터가 바람직하다. 실시예 23 참조.
본 발명에 유용한 프로모터로부터의 발현 수준은 예를 들어 리포터 유전자 생성물 즉, 프로모터/리포터 유전자가 도입된 형질전환 식물의 잎, 꽃, 열매 또는 다른 조직의 추출물 내의 단백질 또는 mRNA의 수준을 측정함으로서 통상의 발현 시스템을 이용하여 시험될 수 있다. 예시적인 리포터 유전자는 GUS이다.
선택적으로는, 조절 서열은 5'-비-번역 리더 서열 또는 폴리아데닐레이션 신호 또는 인핸서를 포함한다. 또한 본 발명은 당업자에 알려진 다른 조절 서열을 고려한다.
또한 본 발명은 센스 또는 안티센스 방향의 노화-유도된 eIF-5A의 폴리뉴클레오타이드를 포함한 본 발명의 벡터 또는 벡터의 결합으로 형질전환된 형질전환 식물 세포, 형질전환 묘목 또는 이러한 세포에서 생성된 성숙 형질전환 식물 또는 꽃, 열매, 잎, 종자 등과 같은 식물 부분을 제공한다.
또한 본 발명은 내생적 노화-유도된 eIF-5A의 발현을 억제하는 방법을 제공한다. 이들 방법은 적어도 하나 이상의 식물 세포의 게놈 내로 노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드를 포함한 본 발명의 발현 벡터를 통합시키는 것을 포함한다. 노화-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드는 전사되고 내생적 노화-유도된 eIF-5A의 발현을 억제한다.
내생적 노화-유도된 eIF-5A의 발현을 억제하는 또다른 방법에서 센스 방향으로 본 발명의 노화-유도된 eIF-5A 폴리뉴클레오타이드를 포함한 발현 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합된다. 노화-유도된 eIF-5A의 폴리뉴클레오타이드는 전사되고 수득된 외생적 노화-유도된 eIF-5A의 동시-발현은 내생적 노화-유도된 eIF-5A의 하향-조절 또는 억제를 유발한다.
본 발명은 자주개자리, 토마토 및 상추를 포함한 다양한 식물 내 스트레스-유도된 eIF-5A의 발견에 관한 것이다. eIF-5A 스트레스 이소폼을 하향 조절하는 것은 스트레스-유도된 노화를 개선한다. eIF-5A 스트레스 이소폼의 하향 조절은 스트레스에 대한 증가된 내성을 제공하고 증가된 생장을 유발한다. 도 121∼123은 형질전환 식물(스트레스-유도된 eIF-5A의 감소된 발현을 지닌)이 증가된 가뭄 저항성을 나타냄을 나타낸다. 도 124는 형질전환 식물(스트레스-유도된 eIF-5A의 감소된 발현을 지닌)이 대조군(야생형) 식물 대비 증가된 종자 수확량가뭄 저항성을 나타냄을 나타낸다.
따라서 본 발명의 하나의 실시태양은 자주개자리에서 분리된 스트레스 eIF-5A이다. 아미노산 서열은 도 139에 제공되고 서열번호: 177이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 139에 제공되고 서열번호: 176이다.
따라서 본 발명의 하나의 실시태양은 토마토에서 분리된 스트레스 eIF-5A이다. 아미노산 서열은 도 146에 제공되고 서열번호: 191이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 146에 제공되고 서열번호: 190이다.
또한 본 발명은 상기 열거된 서열번호에 65∼100% 서열 상동성을 지니고, 높은 스트린전시 조건 하에서 열거된 서열번호의 상보체에 하이브리다이즈하고 스트레스 eIF-5A를 인코드하는 스트레스 eIF-5A의 분리된 폴리뉴클레오타이드를 제공한다.
또한 본 발명은 상기 열거된 서열번호에 65∼100% 아미노산 서열 상동성을 지니는 스트레스 eIF-5A의 스트레스 eIF-5A 분리된 폴리펩타이드를 제공한다.
또한 본 발명은 스트레스 eIF-5A의 안티센스 폴리뉴클레오타이드를 제공한다. 안티센스 폴리뉴클레오타이드는 발현을 저해할 수 있는 길이이다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하고 또다른 특히 바람직한 실시태양에서 다른 eIF-5A 이소폼이 3'UTR에서 더 높은 정도의 변이를 지니기 때문에 안티센스 폴리뉴클레오타이드는 3'UTR에 지시된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 5'-비-코딩 서열에 지시된다. 5'-비-코딩 서열에 주로 상보적인 안티센스 폴리뉴클레오타이드는 전사 인자를 인코드하는 유전자 발현의 효과적인 저해제로 알려져 있다. Branch, M.A., Molec. Cell Biol., 13:4284-4290 (1993).
여기서 사용되고 청구된 "스트레스 eIF-5A의 안티센스 폴리뉴클레오타이드"라는 용어는 열거된 서열번호의 상보체와 100% 상동성을 공유한 안티센스 폴리뉴클레오타이드를 포함할 뿐만 아니라 기능적 변이체인 안티센스 폴리뉴클레오타이드를 포함한다. 기능적 변이체는 스트레스 eIF-5A의 상응하는 부분에 적어도 80% 이상의 서열 상동성을 지니고 높은 스트린전시 조건 하에서 이에 하이브리다이즈하는 천연 또는 인공적 변이체이다. 또한 변이체는 본 발명에 의해 의도된 기능을 지녀야 하고 즉, 발현 벡터 내로 도입되고 이러한 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합되는 경우 내생적 스트레스 eIF-5A의 발현을 조절할 수 있는 것이다. 당업자는 비실질적 변화가 전사체에 결합하고 유전자 발현을 감소시키거나 저해할 수 있는 안티센스 폴리뉴클레오타이드의 능력에 해롭게 영향을 미치지 않는 서열 내에서 이루어질 수 있음을 인식할 수 있다. 따라서 "안티센스 폴리뉴클레오타이드"라는 용어는 전사체에 실질적으로 상보적이고 전사체에 특이적으로 결합하고 유전자 발현을 저해하거나 감소시킬 수 있는 능력을 여전히 유지하는 폴리뉴클레오타이드를 포함한다. 안티센스의 일반적인 논의를 위해 Alberts, et al., Molecular Biology of the Cell, 2nd ed., Garland Publishing, Inc. New York, New York ,1989(특히 페이지 195∼196, 참고사항으로 여기에 포함됨) 참조.
본 발명의 하나의 실시태양은 스트레스 eIF-5A 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 스트레스 eIF-5A의 안티센스 폴리뉴클레오타이드(상기 기술된 본 발명의)를 제공한다. 벡터는 플라스미드가 될 수 있고, 바람직하게는 바이러스성 또는 식물 세포 또는 박테리아 세포 내에서 인코드된 유전자를 복제하고 발현하는 것으로 당분야에 알려진 또다른 벡터도 된다. 벡터는 원하는 스트레스 eIF-5A RNA의 안티센스 폴리뉴클레오타이드를 생성하도록 전사될 수 있도록 염색체로 도입된다. 이러한 플라스미드 또는 바이러스 벡터는 당분야에서 표준인 재조합 DAN 기술 방법에 의해 구축될 수 있다. 예를 들어 벡터는 원핵성 숙주에서 기능적인 복제 시스템 및 본 발명에 따른 안티센스 폴리뉴클레오타이드를 포함한 플라스미드 벡터이다. 대안으로 벡터는 아그로벡테리움에서 기능적인 복제 시스템 및 본 발명의 따른 안티센스 폴리뉴클레오타이드를 포함한 플라스미드이다. 아그로박테리움 내에서 복제하는 것이 가능한 플라스미드는 당분야에 잘 알려져 있다. Miki, et al., Procedures for Introducing Foreign DNA Into Plants, Methods in Plant Molecular Biology and Biotechnology,, Eds. B.R. Glick and J.E. Thompson. CRC Press (1993), PP. 67-83 참조.
벡터는 상기 논의된 조절 서열 및 프로모터 조절 요소를 더욱 포함한다.
또한 본 발명은 센스 또는 안티센스 방향으로 스트레스 eIF-5A의 폴리뉴클레오타이드를 포함한 본 발명의 벡터 또는 벡터의 결합으로 형질전환된 형질전환 식물 세포, 형질전환 묘목 또는 이러한 세포에서 생성된 형질전환 식물, 또는 형질전환 식물의 꽃, 열매, 잎, 종자 등과 같은 식물 부분을 제공한다.
또한 본 발명은 내생적 스트레스 eIF-5A의 발현을 저해하는 방법을 제공한다. 이들 방법은 적어도 하나 이상의 식물 세포의 게놈 내로 스트레스 eIF-5A의 안티센스 폴리뉴클레오타이드를 포함한 본 발명의 발현 벡터를 도입시키는 것을 포함한다. 스트레스 eIF-5A의 안티센스 폴리뉴클레오타이드가 전사되고 내생적 스트레스 eIF-5A의 발현을 저해한다.
내생적 스트레스 eIF-5A의 발현을 저해하는 또다른 방법에서 센스 방향으로 본 발명의 스트레스 eIF-5A 폴리뉴클레오타이드를 포함한 발현 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 도입된다. 스트레스 eIF-5A의 폴리뉴클레오타이드가 전사되고 수득된 내생적 스트레스 eIF-5A의 동시-발현은 내생적 스트레스 eIF-5A 발현의 하향-조절 또는 저해를 유발한다.
손상/병원체-유도된 eIF-5A
손상/병원체-유도된 eIF-5A(eIF-5A3으로 표기됨)는 손상된 조직 및 감염된 조직에서 발현된다. 본 발명은 아라비돕시스 탈리아나, 토마토, 자주개자리 및 상추를 포함한 다양한 식물 내의 손상/병원체-유도된 eIF-5A의 발견에 관한 것이다. 본 발명자는 이러한 이소폼이 식물에 대한 손상 이벤트 또는 병원체 이입 동안 상향 조절됨을 발견하였다. 도 125는 손상/병원체 유도된 eIF-5A는 슈도모나스 시린게(Pseudomonas syringae)로의 감염 후 상향-조절됨을 나타낸다. 상향-조절은 전사 수준에서 발생한다. 또한 이는 독성 감염 후 오직 단백질 수준에서 상향-조절된 후 세포 사멸을 유발하고 손상/병원체 유도된 eIF-5A가 병원체 이입 이벤트시 세포 사멸을 추진한다는 추론을 유도한다.
손상/병원체-유도된 eIF-5A 이소폼의 하향 조절은 병원체-유도된 세포 사멸을 저해한다. 도 126은 손상/병원체-유도된 eIF-5A 이소폼의 안티센스 억제가 독성 병원체에 의한 감염을 저해함을 나타낸다. 형질전환 식물은 대조군 식물 대비 감염 저해의 99% 이상의 증가를 나타내었다. 도 127∼130은 형질전환 카놀라(안티센스/병원체-유도된 eIF-5A 이소폼)가 스클레로티니아 스클레로티오룸(Sclerotinia sclerotiorum)에 의한 감염 저항성을 나타냄을 나타낸다.
손상/병원체 유도된 eIF-5A는 살생생물 박테리아성 및 진균성 병원체에 의한 감염을 수반한 프로그램화된 세포 사멸을 조절한다. 독성 슈도모나스 시린게 pv 토마토 DC 3000으로의 감염 72시간 이내에 손상/병원체 유도된 eIF-5A 단백질은 아라비돕시스 야생형 식물 잎에서 강하게 상향-조절된다. 상향-조절은 질병 진행의 가시적 증후와 일치한다. 더욱이 손상/병원체 유도된 eIF-5A는 감염 후 증가된 발현을 나타내는 단지 eIF-5A 이소폼이다. 더욱이 상향-조절은 후-전사적으로 조절되는 것으로 나타난다. 질병 발달시 손상/병원체 유도된 eIF-5A의 역할을 시험하기 위해 지속적으로 억제된 손상/병원체 유도된 eIF-5A 발현을 지닌 형질전환 아라비돕시스 식물이 안티센스 T-DNA 삽입을 이용하여 개발되었다. 억제된 식물은 일부 라인이 야생형 식물 대비 박테리아 적재의 99% 감소를 나타냄으로서 독성 슈도모나스 시린게 pv 토마토 DC 3000에 대한 현저한 저항성을 나타내었다. 이러한 병원체 생장의 억제는 질병 증후의 부재와 상호관련된다. 또한 동일한 라인은 살생생물 진균성 병원체인 스클레로티니아 스클레로티오룸에 의한 감염에 대해 강한 저항성도 나타내었다. 이러한 결과는 손상/병원체 유도된 eIF-5A가 살생생물 병원체에 의해 유도되는 프로그램화된 세포 사멸을 조절하고 손상/병원체 유도된 eIF-5A의 억제가 감염시 숙주 세포 사멸을 저해하고 병원체의 생장을 정상적으로 지지하는 영양분의 방출이 발생됨을 나타내는 것으로 해석되었다.
도 9는 노화-유도된 eIF-5A가 대조군 식물, 모의 처리된 식물, Avr 처리된 식물 및 Vir 처리된 식물 내에서 일정하게 유지됨을 나타낸다(식물이 4주령인 것으로 검출됨). 손상/병원체-유도된 eIF-5A는 Vir 처리된 식물에서 상향-조절된다.
도 10은 식물의 수준이 지혈기로 손상된 실험 결과를 나타낸다. 아라비돕시스 탈리아나("At") 내의 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A 및 생장 eIF-5A의 수준은 손상 직후, 1시간 후 및 9시간 후 측정되었다. 노던 블럿은 노화-유도된 eIF-5A가 일정하게 유지되나 손상/병원체-유도된 eIF-5A의 발현 수준의 현저한 증가가 있음을 나타낸다. 생장 eIF-5A의 발현 수준은 손상 이벤트시 감소하기 시작하였다.
본 발명자는 손상/병원체-유도된 eIF-5A가 상향-조절되고 손상 이벤트가 식물에 부과될 때(묘목이 이식될 때 발생하는 것과 같은) 이러한 손상은 생장 eIF-5A의 매우 강한 억제를 유발함을 증명하였다. 도 14-17 참조. 수득된 식물은 매우 왜소한 생장을 지닌다. 그러나 종자가 카나마이신 내에 침지되고 토양 내로 직접 심어지는 경우(이식이 필요없고 따라서 이식 손상이 없음) 종자는 정상 크기의 식물로 발달한다.
모두 통합된 센스 손상/병원체-유도된 eIF-5A 컨스트럭트를 지닌 다양한 시험 식물 사이에서 나타나는 차이(도 15)는 손상/병원체-유도된 eIF-5A 발현의 다양한 정도에 기인한다. 당업자는 유전자가 도입될 때(센스 또는 안티센스) 유전자 다양한 정도의 상향-조절 또는 하향-조절을 지님을 인식할 것이다. 정도의 차이는 유전자가 어디에 통합되는지 및 얼마나 많은 복제가 통합되는지에 따라 달라진다. 발현 정도를 변화시킴으로서 다양한 표현형을 유전자 발현에 상호관련시킬 수 있다. 원하는 표현형이 생성되면 그 식물이 채취되어 원하는 자손을 생성하는데 이용될 수 있다. 따라서 도 15에서 손상/병원체-유도된 eIF-5A에 대해 강하게 상향-조절된 식물이 손상 이벤트 후 거의 생장하지 않았으나(식물 태그 10), 다소 잘 생장한 식물(그러나 야생형만틈 양질은 아닌)은 강하게 상향-조절되지 않았다.
본 발명의 하나의 실시태양은 아라비돕시스 탈리아나로부터의 분리된 손상/병원체-유도된 eIF-5A이다. 아미노산 서열은 도 41에 제공되어 있고 서열번호: 55이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 41에 제공되어 있고 서열번호: 54이다.
본 발명의 또다른 실시태양은 토마토로부터의 분리된 손상/병원체-유도된 eIF-5A이다. 아미노산 서열은 도 103에 제공되어 있고 서열번호: 57이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 103에 제공되어 있고 서열번호: 56이다.
본 발명의 또다른 실시태양은 자주개자리로부터의 분리된 손상/병원체-유도된 eIF-5A이다. 아미노산 서열은 도 137에 제공되어 있고 서열번호: 173이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 137에 제공되어 있고 서열번호: 172이다.
본 발명의 또다른 실시태양은 자주개자리로부터의 분리된 손상/병원체-유도된 eIF-5A이다. 아미노산 서열은 도 137에 제공되어 있고 서열번호: 173이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 137에 제공되어 있고 서열번호: 172이다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 서열 상동성을 지니고, 높은 스트린젠시 조건 하에서 열거된 서열번호의 상보체에 하이브리다이즈하고 손상/병원체-유도된 eIF-5A를 인코드하는 손상/병원체-유도된 eIF-5A의 분리된 폴리뉴클레오타이드를 제공한다.
또한 본 발명은 상기 열거된 서열번호에 65∼100% 아미노산 서열 상동성을 지닌 손상/병원체-유도된 eIF-5A의 손상/병원체 유도된 eIF-5A 분리된 폴리펩타이드를 제공한다.
또한 본 발명은 손상/병원체-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드를 제공한다. 안티센스 폴리뉴클레오타이드는 발현을 억제할 수 있는 길이면 된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하고 다른 특정하게 바람직한 실시태양에서 eIF-5A의 다른 이소폼이 3'UTR에서 더 큰 정도의 이소폼 변이를 지니기 때문에 안티센스 폴리뉴클레오타이드는 3'UTR로 지시된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 5'-비-코딩 서열로 지시된다. 5'-비-코딩 서열에 상보적인 안티센스 폴리뉴클레오타이드는 전사 인자를 인코드하는 유전자 발현의 효과적인 억제제로 알려져 있다(Branch, M.A., Molec. Cell Biol., 13:4282-4290(1993)).
여기서 사용된 "손상/병원체 유도된 eIF-5A의 안티센스 폴리뉴클레오타이드"라는 용어는 열거된 서열번호의 상보체의 100% 상보성을 공유하는 안티센스 폴리뉴클레오타이드를 포함할 뿐만 아니라 기능적 변이체인 안티센스 폴리뉴클레오타이드도 포함한다. 기능적 변이체는 높은 스트린전시 조건 하에서 손상/병원체 유도된 eIF-5A의 상응하는 부분과 적어도 80% 이상의 서열 상동성을 지니고 이에 하이브리다이즈하는 천연 또는 인공 변이체이다. 또한 변이체는 발현 벡터 내로 도입되고 이러한 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합되는 경우 내생적 손상/병원체-유도된 eIF-5A의 발현을 조절할 수 있는 본 발명에 의해 의도된 바와 같은 기능을 지녀야 한다. 당업자는 전사체에 결합하고 유전자 발현을 감소시키거나 저해할 수 있는 안티센스 폴리뉴클레오타이드의 능력에 악영향을 미치지 않는 서열 내에 비실질적 변화가 생성될 수 있음을 인식할 수 있다. 따라서 "안티센스 폴리뉴클레오타이드"라는 용어는 전사체에 실질적으로 상보적이고 전사체에 특이적으로 결합하고 유전자 발현을 저해하거나 감소시킬 수 있는 능력을 여전히 유지하는 폴리뉴클레오타이드를 포함한다.
본 발명의 하나의 실시태양은 손상/병원체-유도된 eIF-5A 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 손상/병원체-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드(상기 기술된 본 발명의)를 포함한 발현 벡터를 제공한다. 벡터, 조절 서열 및 프로모터 조절 요소는 상기 기술된 바와 같다.
또한 본 발명은 센스 또는 안티센스 방향으로 손상/병원체-유도된 eIF-5A의 폴리펩타이드를 포함한 본 발명의 벡터 또는 벡터 결합으로 형질전환된 형질전환 식물 세포, 형질전환 묘목 또는 이러한 세포로부터 생성된 숙성 형질전환 식물 또는 꽃, 열매, 잎, 종자와 같은 형질전환 식물의 식물 일부를 제공한다.
또한 본 발명은 내생적 손상/병원체-유도 eIF-5A의 발현을 억제하는 방법을 제공한다. 이들 방법은 식물의 적어도 하나 이상의 세포의 게놈 내로 손상/병원체-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드를 포함한 본 발명의 발현 벡터를 통합시키는 것을 포함한다. 손상/병원체-유도된 eIF-5A의 안티센스 폴리뉴클레오타이드는 전사되고 내생적 손상/병원체-유도된 eIF-5A의 발현을 억제한다.
내생적 손상/병원체-유도된 eIF-5A의 발현을 억제하는 또다른 방법에 있어서, 센스 방향의 본 발명의 손상/병원체-유도된 eIF-5A 폴리뉴클레오타이드를 포함한 발현 벡터는 식물의 적어도 하나 이상의 세포 게놈 내로 통합된다. 손상/병원체-유도된 eIF-5A의 폴리뉴클레오타이드는 전사되고 수득된 내생적 손상/병원체-유도된 eIF-5A의 동시 발현은 내생적 손상/병원체-유도된 eIF-5A의 발현의 하향-조절 또는 억제를 유발한다.
내생적 손상/병원체 유도된 eIF-5A의 발현을 억제함으로서 수득된 형질전환 식물은 병원균 이입에 의해 기인한 독성적 손상에 대한 증가된 저항성을 지닌다. 병원체는 바나나 내 블랙 스가토카(Black Sigatoka), 스클레로티니아 스클레로티오룸(상추 및 잔디의 주요 진균성 질병) 및 슈도모나스 시린게를 포함하나 이에 한정적인 것은 아니다. 실시예 16 및 도 43 및 44 참조.
생장 eIF-5A
또한 본 발명은 생장 eIF-5A에 관한 것이다. 생장 eIF-5A는 생장 조직 내에서 발현된다. eIF-5A이 센스 방향으로 생장 eIF-5A의 폴리뉴클레오타이드로 상향-조절되면 3가지 표현형적 변화가 나타난다: 증가된 종자 크기, 증가된 생물량 및 증가된 종자 수확량. 도 132-134 참조.
본 발명의 하나의 실시태양은 아라비돕시스 탈리아나로부터의 분리된 생장 eIF-5A이다. 아미노산 서열은 도 1에 제공되어 있고 서열번호: 60이다. 아미노산의 폴리뉴클레오타이드는 도 2에 제공되어 있고 서열번호: 63이다.
본 발명의 또다른 실시태양은 토마토로부터의 분리된 생장 eIF-5A이다. 아미노산 서열은 도 101에 제공되어 있고 서열번호: 65이다. 아미노산의 폴리뉴클레오타이드는 도 101에 제공되어 있고 서열번호: 64이다.
본 발명의 또다른 실시태양은 카놀라로부터의 분리된 생장 eIF-5A이다. 아미노산 서열은 도 95에 제공되어 있고 서열번호: 67이다. 아미노산의 폴리뉴클레오타이드는 도 95에 제공되어 있고 서열번호: 66이다.
본 발명의 또다른 실시태양은 자주개자리로부터의 분리된 생장 eIF-5A이다. 아미노산 서열은 도 140에 제공되어 있고 서열번호: 179이다. 아미노산의 폴리뉴클레오타이드는 도 140에 제공되어 있고 서열번호: 178이다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 서열 상동성을 지니고 높은 스트린젠시 조건 하에서 열거된 서열번호의 상보체에 하이브리다이즈하고 생장 eIF-5A를 인코드하는 생장 eIF-5A의 폴리뉴클레오타이드를 제공한다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 아미노산 서열 상동성을 지니는 생장 eIF-5A의 분리된 폴리펩타이드를 제공한다.
또한 본 발명은 생장 eIF-5A의 안티센스 폴리뉴클레오타이드를 제공한다. 안티센스 폴리뉴클레오타이드는 발현을 억제할 수 있는 길이면 된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하고 다른 특정하게 바람직한 실시태양에서 eIF-5A의 다른 이소폼이 3'UTR에서 더 큰 정도의 이소폼 변이를 지니기 때문에 안티센스 폴리뉴클레오타이드는 3'UTR로 지시된다. 일부 실시태양에서 안티센스 폴리뉴클레오타이드는 5'-비-코딩 서열로 지시된다. 5'-비-코딩 서열에 상보적인 안티센스 폴리뉴클레오타이드는 전사 인자를 인코드하는 유전자 발현의 효과적인 억제제로 알려져 있다(Branch, M.A., Molec. Cell Biol., 13:4282-4290(1993)).
여기서 사용된 "생장 eIF-5A의 안티센스 폴리뉴클레오타이드"라는 용어는 열거된 서열번호의 상보체의 100% 상보성을 공유하는 안티센스 폴리뉴클레오타이드를 포함할 뿐만 아니라 기능적 변이체인 안티센스 폴리뉴클레오타이드도 포함한다. 기능적 변이체는 상기 기술된 것이다. 기능적 변이체는 높은 스트린전시 조건 하에서 생장 eIF-5A의 상응하는 부분과 적어도 80% 이상의 상동성을 지니고 이에 하이브리다이즈하는 천연 또는 인공 변이체이다. 또한 변이체는 발현 벡터 내로 도입되고 이러한 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합되는 경우 내생적 생장 eIF-5A의 발현을 조절할 수 있는 본 발명에 의해 의도된 바와 같은 기능을 지녀야 한다. 당업자는 전사체에 결합하고 유전자 발현을 감소시키거나 저해할 수 있는 안티센스 폴리뉴클레오타이드의 능력에 악영향을 미지지 않는 서열 내에서 비실질적 변화가 생성될 수 있음을 이식할 수 있다. 따라서 "안티센스 폴리뉴클레오타이드"라는 용어는 전사체에 실질적으로 상보적이고 전사체에 특이적으로 결합하고 유전자 발현을 저해하거나 감소시킬 수 있는 능력을 여전히 유지하는 폴리뉴클레오타이드를 포함한다.
본 발명의 하나의 실시태양은 생장 eIF-5A 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 생장 eIF-5A의 안티센스 폴리뉴클레오타이드(상기 기술된 본 발명의)를 포함한 발현 벡터를 제공한다. 벡터, 조절 서열 및 프로모터 조절 요소는 상기 기술된 바와 같다.
또한 본 발명은 센스 또는 안티센스 방향으로 생장 eIF-5A의 폴리펩타이드를 포함한 벡터 또는 벡터 결합으로 형질전환된 형질전환 식물 세포, 형질전환 묘목 또는 이러한 세포로부터 생성된 숙성 형질전환 식물 또는 꽃, 열매, 잎, 종자와 같은 형질전환 식물의 식물 일부를 제공한다.
또한 본 발명은 내생적 생장 eIF-5A의 발현을 억제하는 방법을 제공한다. 이들 방법은 식물의 적어도 하나 이상의 세포의 게놈 내로 생장 eIF-5A의 안티센스 폴리뉴클레오타이드를 포함한 본 발명의 발현 벡터를 통합시키는 것을 포함한다. 생장 eIF-5A의 안티센스 폴리뉴클레오타이드는 전사되고 내생적 생장 eIF-5A의 발현을 억제한다.
내생적 생장 eIF-5A의 발현을 억제하는 또다른 방법에 있어서, 센스 방향의 본 발명의 생장 eIF-5A 폴리뉴클레오타이드를 포함한 발현 벡터는 식물의 적어도 하나 이상의 세포 게놈 내로 통합된다. 생장 eIF-5A의 폴리뉴클레오타이드는 전사되고 수득된 내생적 생장 eIF-5A의 동시 발현은 내생적 생장 eIF-5A의 발현의 하향-조절 또는 억제를 유발한다.
본 발명의 또다른 실시태양에서 생장 eIF-5A의 발현을 상향-조절하는 방법이 제공된다. 센스 방향으로 본 발명의 생장 eIF-5A 폴리뉴클레오타이드를 포함한 발현 벡터는 식물의 적어도 하나 이상의 세포 게놈 내로 통합된다. 생장 eIF-5A의 폴리뉴클레오타이드는 전사되고 수득된 외생적 생장 eIF-5A의 동시-발현은 세포가 비-형질전환 세포보다 더 많은 생장 eIF-5A를 발현하도록 한다.
도 19는 생장 eIF-5A에 대해 상향-조절된 식물이 대조군 식물보다 증가된 생물량을 지녔음을 나타낸다. 생장 eIF-5A의 발현을 상향-조절하기 위해 생장 eIF-5A이 센스 방향으로 아라비돕시스 탈리아나 내로 삽입되었다. 16개 모계(1-16)는 일반 수준의 생장 eIF-5A 발현을 측정하는데 분석되었다. 각각의 모계 8로부터 자매 라인이 생성되었다(A-H). 각 모계 내의 생장 eIF-5A의 발현 수준이 시험되었고 결과는 도 20에 나타나 있다. 다양한 정도의 발현이 전체 모계에 나타나 있다. 예를 들어 라인 2 및 10은 높은 수준의 발현을 지니는 반면 라인 11 및 16은 매우 낮은 발현을 지니거나 전혀 없다.
도 21 및 22는 라인 1 및 2로부터의 식물을 나타낸다. 이들 식물은 대조군 식물보다 더 크다. 생장 eIF-5A이 세포-분열 이소폼이고 구성적으로 발현되기 때문에 세포 분열이 증가된다. 식물이 생장 모드로 잠기고 노화 경로로 스위치될 수 없기 때문에 노화의 감소가 발생한다.
도 23 및 24는 중간 수준의 생장 eIF-5A 발현을 라인으로부터의 것이다. 이들은 더 큰 잎 및 지연된 노화를 나타낸다.
도 25 및 26은 낮은 수준의 상향-조절을 지닌 라인으로부터의 것이다. 이들은 큰 잎 및 큰 로제트를 지닌다.
도 27 및 28은 상향-조절이 없는 라인으로부터의 것이다(유전자의 동시-억제에 기인함). 식물이 카나마이신 저항적이기 때문에 식물이 배지 상에서 생장하도록 하기 위해 본 유전자가 존재해야 한다. 또한 노화-유도된 eIF-5A가 동시-억제되고 따라서 크기의 증가를 유발함이 나타난다.
증가된 생물량에 더하여 생장 eIF-5A 상향-조절된 식물 내에서 증가된 종자 크기를 보인다. 모든 라인의 종자 크기가 측정되었다. 가장 높은 수준의 생장 eIF-5A 발현을 지닌 라인에서 종자 크기 내의 3x 이상의 증가가 나타났다. 이는 생장 eIF-5A의 상향-조절이 세포 분열을 증가시켜 종자 크기를 증가시켰기 때문이다.
상기 예에서의 생장 eIF-5A(아라비돕시스 탈리아나 유래)는 구성적으로 발현된 것으로 즉, 보편적인 프로모터의 이용을 통해 식물 내 어디에서나 발현된다. 반대로 조직 특이적 프로모터를 이용함으로서 특정 조직 내로 상향-조절이 지시되기도 한다. 예를 들어 종자 특이적 프로모터를 이용함으로서 생장 eIF-5A는 종자 내에서만 상향-조절되어 잎이 정상적으로 생장하도록 하나 종자의 양의 증가를 생성한다. 따라서 특정 프로모터를 이용하여 생장 eIF-5A가 원하는 식물 부분에서 상향-조절되어 원하는 표현형을 수득할 수 있다.
생장 eIF-5A를 상향-조절함으로서 3가지 표현형을 유발하나 - 증가된 생물량, 증가된 종자 수확량 또는 증가된 종자 크기- 3가지 표현형 모구가 동시에(또는 동일 식물 내에) 존재하지는 않는다. 예를 들어 식물이 종자 크기의 증가를 나타내는 경우 더 작은 식물이 존재할 것이다. 생장 eIF-5A의 가장 높은 상향-조절을 지닌 식물 라인에서 가장 큰 종자가 생성되었으나 세포 확대(더 큰 잎에 필요한)의 비용인 전체 식물에 걸쳐 진행된 대규모 세포 분열이 존재하기 때문에 식물은 더 작았다. 생장 eIF-5A 발현의 낮은 수준의 상향-조절에서 종자를 충격을 주지 않고 잎(더 큰) 상의 충격을 보인다. 따라서 조직 특이적 발현을 이용하고 원하는 표현형을 취한다. 예를 들어 생성되는 물관부 양의 증가를 달성하기 위해 생장 eIF-5A를 물관부 특이적 프로모터 아래에 생장 eIF-5A를 위치시킨다. 따라서 어떠한 원하는 프로모터도 원하는 조직-특이적 상향-조절을 달성하는데 이용된다.
다른 eIF-5A 이소폼뿐만 아니라 생장 eIF-5A는 식물종간에 높게 보존된다. 도 85는 하이푸신 영역이 매우 높게 보존됨을 여러 식물종 유래의 다양한 eIF-5A 이소폼의 아미노산 정렬을 나타낸다. 실제로 eIF-5A는 매우 높게 보존되어 하나의 식물 생장 eIF-5A를 인코드하는 분리된 폴리뉴클레오타이드는 센스 방향으로 또다른 식물종 내에 삽입되어 생장 eIF-5A의 상향-조절을 달성할 수 있다. 도 162는 아라비돕시스 내로의 센스 방향의 미루나무 생장 eIF-5A의 도입 효과를 나타낸다. 도 162는 7주령 T1 식물을 나타낸다. 더 큰 식물은 2개의 다른 형질전환 라인에서 유래된다. 더 작은 식물은 대조군이다. 도 163은 3개의 개별적 라인(표지된 라인 2, 3 및 4) 유래의 T2 식물을 나타내고 "BIN"으로 표지된 식물은 빈 벡터의 대조군을이다. 본 식물은 28일령이다. 형질전환 아라비돕시스는 대조군 식물과 비교시 증가된 생장률을 지닌다. 도 164는 대조군 식물 대비 32일령의 T2 식물(라인 3)을 나타낸다.
생장 eIF-5A의 상호교환가능성의 또다른 증거는 도 165에 나타나 있다. 생장 인자 eIF-5A는 35S 프로모터의 조절 하에서 센스 방향의 카놀라 생장 인자 eIF-5A 또는 아라비돕시스 생장 eIF-5A(전체 길이)에 의해 카놀라 내에서 상향-조절되었다. 수득된 형질전환 식물은 야생형 대조군 식물 대비 종자 수확량 증가를 지녔다. 센스 방향으로 아라비돕시스 생장 eIF-5A를 지닌 형질전환 카놀라 식물 및 센스 방향으로 외인성 카놀라 생장 eIF-5A을 지닌 형질전환 카놀라 식물 내 종자 수확량의 증가는 매우 유사하다.
DHS
DHS는 eIF-5A의 활성화에 필수적이고 노화 조직에서 발현된다. 따라서 본 발명은 아라비돕시스 탈리아나, 토마토, 카네이션, 카놀라, 상추, 자주개자리, 바나나, 미루나무 및 미코스파에렐라(mycosphaerella)로부터의 분리된 DHS를 제공한다.
따라서 본 발명의 하나의 실시태양은 아라비돕시스 탈리아나로부터 분리된 DHS이다. 아미노산 서열은 도 46에 제공되어 있고 서열번호: 6이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 46에 제공되어 있고 서열번호: 5이다.
본 발명의 또다른 실시태양은 토마토로부터 분리된 DHS이다. 아미노산 서열은 도 45A 및 B에 제공되어 있고 서열번호: 2이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 45A 및 B에 제공되어 있고 서열번호: 1이다.
본 발명의 또다른 실시태양은 카네이션으로부터 분리된 DHS이다. 아미노산 서열은 도 54에 제공되어 있고 서열번호: 10이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 54에 제공되어 있고 서열번호: 9이다.
본 발명의 또다른 실시태양은 카놀라로부터 분리된 DHS이다. 아미노산 서열은 도 97에 제공되어 있고 서열번호: 71이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 97에 제공되어 있고 서열번호: 70이다.
본 발명의 또다른 실시태양은 상추로부터 분리된 DHS이다. 도 105는 상추 DHS 폴리뉴클레오타이드 서열의 일부를 제공한다.
본 발명의 또다른 실시태양은 자주개자리로부터 분리된 DHS이다. 아미노산 서열은 도 107A 및 B에 제공되어 있고 서열번호: 73이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 107A 및 B에 제공되어 있고 서열번호: 72이다.
본 발명의 또다른 실시태양은 바나나로부터 분리된 DHS이다. 아미노산 서열은 도 108A 및 B에 제공되어 있고 서열번호: 75이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 108A 및 B에 제공되어 있고 서열번호: 74이다.
본 발명의 또다른 실시태양은 미루나무로부터의 분리된 DHS이다. 아미노산 서열은 도 109A 및 B에 제공되어 있고 서열번호: 76이다. 아미노산을 인코드하는 폴리뉴클레오타이드는 도 109A 및 B에 제공되어 있고 서열번호: 77이다.
본 발명의 또다른 실시태양은 미코스파에렐라로부터 분리된 DHS이다. 서열은 도 110에 제공된다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 서열 상동성을 지니고 높은 스트린전시 조건 하에서 열거된 서열번호의 상보체에 하이브리다이즈하고 DHS를 인코드하는 DHS의 분리된 폴리뉴클레오타이드을 제공한다.
또한 본 발명은 상기 열거된 서열번호와 65∼100% 아미노산 서열 상동성을 지니는 DHS의 분리된 폴리펩타이드를 제공한다. 도 47 및 도 148(a) 및 (b)는 종간 DHS의 높게 보존된 특성을 나타내는 다양한 DHS의 아미노산 정렬을 제공한다.
또한 본 발명은 DHS의 안티센스 폴리뉴클레오타이드를 제공한다. 안티센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하거나 3'UTR로 지시되거나 5'-비-코딩 서열로 지시된다. 5'-비-코딩 서열에 상보적인 안티센스 폴리뉴클레오타이드는 전사 인자를 인코드하는 유전자 발현의 효과적인 억제제로 알려져 있다(Branch, M.A., Molec. Cell Biol., 13:4282-4290(1993)). 안티센스 폴리뉴클레오타이드는 발현을 억제할 수 있는 어떠한 길이도 된다.
여기서 사용된 "DHS의 안티센스 폴리뉴클레오타이드"라는 용어는 열거된 서열번호의 상보체의 100% 상보성을 공유하는 안티센스 폴리뉴클레오타이드를 포함할 뿐만 아니라 기능적 변이체인 안티센스 폴리뉴클레오타이드도 포함한다. 기능적 변이체는 상기 기술된 것이다. 변이체는 발현 벡터 내로 도입되고 이러한 벡터가 적어도 하나 이상의 식물 세포의 게놈 내로 통합되는 경우 내생적 DHS의 발현을 조절할 수 있는 본 발명에 의해 의도된 바와 같이 기능한다.
또한 본 발명은 DHS의 센스 폴리뉴클레오타이드를 제공한다. 센스 폴리뉴클레오타이드는 전체 길이 코딩 서열을 포함하거나 3'UTR로 지시되거나 5'-비-코딩 서열로 지시된다. 센스 폴리뉴클레오타이드는 동시-억제를 통해 발현을 하향-조절하거나 또는 대안으로 발현을 상향-조절할 수 있는 어떠한 길이도 된다.
본 발명의 하나의 실시태양은 DHS 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 DHS의 안티센스 폴리뉴클레오타이드(상기 기술된 본 발명의) 또는 DHS의 센스 폴리뉴클레오타이드(상기 기술된 본 발명의)를 포함한 발현 벡터를 제공한다. 벡터는 상기 기술된 바와 같다.
또한 본 발명은 센스 또는 안티센스 방향으로 DHS의 폴리펩타이드를 포함한 벡터 또는 벡터 결합으로 형질전환된 형질전환 식물 세포, 형질전환 묘목 또는 이러한 세포로부터 생성된 숙성 형질전환 식물 또는 꽃, 열매, 잎, 종자와 같은 형질전환 식물의 식물 일부를 제공한다.
또한 본 발명은 내생적 DHS의 발현을 억제/감소하는 방법을 제공한다. 이들 방법은 식물의 적어도 하나 이상의 세포의 게놈 내로 DHS의 안티센스 폴리뉴클레오타이드를 포함한 본 발명의 발현 벡터를 통합시키는 것을 포함한다. DHS의 안티센스 폴리뉴클레오타이드는 전사되고 내생적 DHS의 발현을 억제/감소한다. 수득된 식물은 증가된 생물량, 증가된 종자 수확량 및 더 긴 저장수명을 지닌다.
예를 들어 도 65∼68은 안티센스 컨스트럭트의 사용에 의해 야기된 DHS의 하향-조절을 지닌 아라비돕시스 식물과 비교된 야생형 아라비돕시스 식물의 사진을 나타낸다. 안티센스 아라비돕시스 DHS 컨스트럭트로 하향-조절된 DHS를 지닌 아라비돕시스 식물은 야생형 아라비돕시스(또는 DHS의 하향-조절을 지니지 않는)와 비교시 증가된 생물량을 지닌다. 도 153 및 154 참조. 또한 도 69 및 155는 하향-조절된 DHS 아라비돕시스가 야생형 식물 대비 증가된 종자 수확량을 지님을 나타낸다. 도 156은 DHS의 하향 조절이 뿌리 발달 증가를 유발함을 나타낸다. 센스 방향의 아라비돕시스 DHS(서열번호: 30)의 전체 길이 또는 3'영역으로 아라비도비스를 형질전환시키는데 사용된 다양한 컨스트럭트 설명의 실시예 13 참조.
실시예 14 및 15는 안티센스 방향의 토마토 DHS의 전체-길이 또는 3' 영역으로의 토마토 식물의 형질전환을 설명한다. 도 70∼79는 수득된 토마토 식물이 야생형 토마토 식물과 비교시 토마토의 지연된 연화 및 부패를 지님을 나타낸다. 도 84(a) 및 84(b)는 안티센스 방향으로 토마토 DHS를 형질전환시킨 토마토가 감소된 꽃 말단 부패 발생률을 지님을 나타내고, 이는 DHS의 발현 감소가 생리적 질병에서 기인한 조직 및 세포 사멸의 착수를 방지함을 나타내는 토마토 열매의 사진이다.
카놀라 내 DHS 발현 억제는 식물 생물량을 증가시키고 종자 수확량을 증가시킨다. 실시예 28 및 도 157∼161 참조. 도 157은 형질전환 카놀라가 증가된 잎 중량에 이해 입증되는 바와 같이 증가된 생물량을 지님을 나타낸다. 도 158은 카놀라의 형질전환 라인 중 하나에서 DHS의 부분적 하향-조절을 나타내는 웨스턴 블럿의 결과를 나타낸다. 도 159는 카놀라 내 DHS의 하향-조절이 더 큰 장각과를 유발함을 나타낸다. 도 160은 카놀라 내 DHS의 하향-조절이 증가된 종자 수확량을 유발함을 나타낸다. 도 161(a)는 트리아실글리세롤의 총 산출량이 "종자 중량 당"을 기초로 표현되었음을 나타낸다. 이는 종자 당 오일 함량의 변화가 존재하지 않음을 입증한다. 따라서 종자 수확량의 ∼65% 증가는 종자 오일의 ∼65 증가로 해석된다. 도 161(b)는 DHS 하향 조절된 카놀라 식물 내 오일의 지방산 비교시 변화가 존재하지 않음을 나타낸다.
도 99는 작은 화분에서 생장되고 식물에 적은 양의(치사-이하) 스트레스가 가해진 카놀라 식물의 실험 결과를 나타낸다. DHS가 하향-조절되는 경우 종자 수확량의 증가가 관찰될 수 있다. 카놀라 DHS의 3'UTR이 안티센스 뉴클레오타이드로 사용되었다. 안티센스 올리고뉴클레오타이드는 35S 프로모터를 사용하여 지속적으로 발현되었다. 구성적 프로모터를 사용하여 DHS 안티센스 폴리뉴클레오타이드는 식물 전체 생장 및 발달 동안 발현되었다. 식물이 치사-이하 스트레스 에피소드(너무 작은 화분에서 생장되거나 최적이하의 생장 조건 기간 등)에 노출시 식물은 노화 경로에 진입한다. 그러나 식물이 감소된 DHS 발현을 지닌 형질전환 식물인 경우 식물은 치사-이하 스트레스 조건시 노화 모드에 진입하지 않는다. 도 99는 하향-조절된 DHS를 지닌 식물 내 카놀라 수확량의 증가를 나타낸다. 도 99는 T1∼T4 세대 및 특정 식물 라인의 2개의 특정 T4 세대("P" 및 "T" 라인으로 명명) 나타낸다.
카네이션 내 DHS 하향 조절은 꽃이 연장된 화병 수명을 지니게 한다. 실시예 29는 안티센스 컨스트럭트의 사용뿐만 아니라 DHS 반응의 저해제의 사용에 의해 야기된 하향 조절이 83%까지 꽃의 저장 수명을 연장시킴을 나타낸다.
안티센스 DHS를 지닌 형질전환 바나나는 야생형과 비교시 더 큰 식물체로 입증된 바와 같이 증가된 생물량을 나타낸다. 또한 형질전환 식물 유래의 열매는 야생형과 비교시 지연된 부패를 나타낸다. 도 114 참조. 바나나 DHS를 인코하는 뉴클레오타이드는 35S 프로모터 또는 열매 특이적 프로모터(pPDK)의 조절 하에 안티센스 방향으로 벡터에 삽입되었다. 전체 길이 DHS 또는 3'UTR이 사용되었다.
통상적으로-이용 가능한 미리-포장된 샐러드는 제품의 저장 수명을 연장시키기 위해 산소 수치가 그의 대기 농도 이하로 크게 감소된 조절된 대기 조건 하에서 일반적으로 저장된다. 미리-포장된 샐러드의 대부분 일반적 부패 징후는 상추의 절단 표면 상의 갈변이다. 조절된 대기 포장이 갈변 지연을 달성하더라도 이는 악취 및 불쾌취를 유발할 수 있다. DHS의 하향-조절은 상추 절단 표면 상의 갈변을 지연시키기 위한 대안적 전략으로서의 잠재력을 지니는 것으로 나타났다. DHS는 mRNA 집단을 선택하기 위해 핵세포질성 셔틀 단백질로 작용하는 진핵성 번역 개시 인자 5A(eIF-5A)의 활성화를 촉매한다. DHS 발현의 억제(안티센스 기술에 의한)가 대기 조건 하에서의 갈변 착수의 유의적인 지연을 유발하는 만큼 DHS는 절단 상추의 갈변에 중요한 역할을 하는 것으로 판단된다. 특히 야생형 상추 식물의 절단 분절의 80%는 절단 6일 후 갈변을 나타낸 반면 5개의 분리 라인 유래의 형질전환 식물의 절단 분절의 평균 27%가 동일한 기간에 갈변되었고 일부 개체 식물은 0% 갈변을 나타내었다. 안티센스 컨스트럭트의 사용을 통한 상추 내 DHS 발현의 억제는 야생형 상추와 비교시 절단 상추의 갈변 착수 지연을 유발한다. 실시예 27 및 도 149∼151 참조.
자트로파 쿠르카스(Jatropha curcas L.)는 높은 오일 함량으로 잘 알려진 식물이고 바이오-연료의 원료로 가능하다. 종자로부터 압착되는 투명한 오일은 조명 및 광택에 사용되었고 더욱 최근에는 에너지원 목적으로 제안되었고, 1톤의 견과는 70 kg의 정제 석유, 40 kg "가스오일 레거(gasoil leger)"(광 연료 오일), 40 kg 일반 연료 오일, 34 kg 건조 타르/피치/로진, 270 kg 코크-유사 목탄 및 200 kg 암모니아처리수, 천연 가스, 크레오소트 등을 산출할 수 있다. 따라서 종자 수확량(따라서 오일 함량)을 증가시킬 수 있는 능력은 유용한 것으로 증명된다. 종자 수확량 증가는 3가지 전략을 통해 달성될 수 있다. 첫 번째 전략은 생장 eIF-5A를 지속적으로 상향-조절하는 것을 포함한다. 바람직하게는 이는 동시-억제의 가망을 최소화하기 위해 생장 eIF-5A의 이형적 이소폼으로 달성되고(즉, 카놀라 또는 아라비돕시스 생장 eIF-5A를 이용), 이를 활성화(하이푸신화)시키는데 유용한 DHS보다 더 많은 eIF-5A가 생성되기 때문에 비하이푸신화된 노화 eIF-5A의 상향-조절이 존재한다. 어떠한 식물의 eIF-5A도 사용되고, 예를 들어 토마토 또는 미루나무 생장 eIF-5A가 자트로파에서 사용될 수 있다. 두 번째 전략은 일반적으로 하이푸신화되는 보존된 리신이 알라닌으로 전환된 돌연변이화 노화 eIF-5A를 지속적으로 상향-조절하는 것이다. 이러한 돌연변이화 eIF-5A는 하이푸신화(활성화)될 수 없고, 이러한 비하이푸신화 형티는 생장을 증진시킨다. 돌연변이화 노화 eIF-5A를 지닌 형질전환 아라비돕시스 식물이 야생형 아라비돕시스 식물보다 더 큼을 나타내는 도 166 및 167 참조. 더 큰 식물은 더 많은 볼트 및 더 많은 종자를 지닌다. 또한 비하이푸신화 노화 eIF-5A는 식물에 증가된 질소 이용 효율을 부여하고, 따라서 토양 내 낮은 질소 조건 하에서 식물 조절을 수행할 수 있게 한다. 세 번째 전략은 DHS를 지속적으로 하향-조절하는 것을 포함한다. 이는 식물이 적당하게 기능하기 위해 일부 DHS를 필요로 하기 때문에 DHS의 부분적 하향-조절이 되어야 한다. 그러나 상기 나타난 바와 같이 DHS의 부분적 하향-조절은 아마도 비하이푸신화 노화 eIF-5A의 축적으로 인해 생장을 실질적으로 증진시키고 종자 수확량을 증가시킨다.
도 47 및 148(a) 및 (b)는 다양한 DHS의 아미노산 정렬을 나타내고, 이는 DHS가 높게 보존됨을 나타낸다. 도 148(a) 및 (b)는 DHS의 매우 높게 보존된 리신뿐만 아니라 다른 영역도 나타낸다. 실제로 DHS는 매우 높게 보존되어 하나의 식물 DHS를 인코드하는 분리된 폴리뉴클레오타이드가 안티센스 방향으로 또다른 식물종 내로 삽입되어 DHS의 하향-조절을 달성할 수 있게 된다.
도 100은 카놀라 수확량(식물 생장 능력을 증가시킴으로서 이루어진)을 측정한 막대 그래프를 나타낸다. 이러한 도면은 생장 eIF-5A 발현을 증가시키거나(센스 컨스트럭트의 이용) 또는 DHS의 발현을 증가시킴으로써(센스 컨스트럭트의 이용) 수확량을 증가시킬 수 있음을 나타낸다. "카놀라"로 표시된 두 번째 막대는 센스 방향으로 카놀라 DHS를 사용함으로써 카놀라 DHS를 상향조절시킬 때 증가가 달성되었음을 나타낸다. 네 번째 막대는 카놀라 식물 내에 센스 방향의 토마토 DHS의 사용에 의한 카놀라 수확량의 증가 나타낸다.
내생적 DHS의 발현을 억제하는 또다른 방법에 있어서, 센스 방향의 본 발명의 DHS 폴리뉴클레오타이드를 포함한 발현 벡터는 식물의 적어도 하나 이상의 세포 게놈 내로 통합된다. DHS의 폴리뉴클레오타이드는 전사되고 수득된 내생적 DHS의 동시-발현은 내생적 DHS의 발현의 하향-조절 또는 억제를 유발한다.
내생적 DHS의 발현을 억제함으로서 수득된 형질전환 식물은 eIF-5A를 활성화시키는 DHS를 전혀 지니지 않거나 실질적으로 거의 지니지 않는다. 앞서 논의된 바와 같이 eIF-5A는 활성화되어 생물학적으로 유용하게 되어야 한다. 따라서 안티센스 폴리뉴클레오타이드에 의해 또는 센스 폴리펩타이드로의 동시-억제에 의해 DHS 발현을 억제하거나 감소시킴으로서 수득된 형질전환 식물은 활성 eIF-5A를 지니지 않거나 활성 eIF-5A를 감소시킬 것이다. 이들 형질전환 식물은 식물의 생물량의 증가, 증가된 종자 수확량 및/또는 증가된 종자 크기를 나타낼 것이다. DHS의 안티센스 폴리뉴클레오타이드를 지닌 형질전환 식물은 광합성의 증가를 나타내고 또한 증가된 전분 함량을 지닌다. 실시예 24 및 25 참조.
또한 본 발명은 DHS의 상향-조절을 제공한다. 상향-조절을 유발하기 위해 센스 DHS 뉴클레오타이드를 식물에 제공함으로서 수득된 식물은 증가된 함량의 DHS를 지닌다. 수득된 형질전환 식물은 증가된 생물량 및 종자 수확량을 지닌다.
DHS 및 eIF-5A가 노화에 조절적인 역할을 한다는 논쟁을 지지하는 또다른 증거는 DHS에 특이적인 억제제로 카네이션 꽃을 처리함으로서 제공되었다. 스퍼미딘 및 eIF-5A는 DHS 반응의 기질이다(Park et al., 1993; Park et al., 1997). 스퍼미딘과 유사한 구조적 특징을 지닌 일부 모노, 디- 및 폴리아민이 시험관 내에서 DHS 활성을 억제한다(Jakus et al., 1993). 스퍼미딘, 푸트레신(putrescine) 및 스퍼민과 같은 일부 폴리아민이 카네이션 꽃병 수명을 연장시키는데 일반적으로 사용되었다(Wang and Baker, 1980). 다른 농도의 다른 폴리아민으로의 처리를 통해 Wang et al(발표되지 않음)은 2배까지 카네이션 꽃의 꽃병 수명을 연장시킬 수 있었다. DHS를 하향-조절하기 위해 일시적인 감염 시스템을 이용한 연구는 진행중이다. 초기 데이터는 생존율 백분율이 비처리된 꽃보다 안티센스 DHS를 발현하는 일시적 감염 시스템으로 진공 침윤된 카네이션 절단 내에서 8일에 거의 4배 이상 높음을 나타낸다(Wang et al, 발표되지 않음).
스트레스에 의한 생장 손실 이외의 농업 내의 또다른 주요 손실은 수확-후 스트레스-유도된 노화이다(McCabe et al., 2001). 이는 절단된 상추와 같이 부분적으로 처리된 식물에 특히 맞다. 절단 상추의 증후는 페놀 생성의 결과인 갈변이다(Matile et al, 1999). 상추 eIF-5A의 안티센스 폴리뉴클레오타이드 또는 안티센스 전체 길이 DHS로의 상추의 시도는 형질전환 상추가 대조군 상추보다 절단 후 갈변에 유의적으로 더 저항적임을 증명하였다. 이는 수확에 의한 스트레스 유도된 노화가 별도의 회로이나(Page et al., 2001) 갈변 및 다른 노화의 번역적 조절 업스트림은 DHS 및 eIF-5A에 의해 적어도 부분적으로 조절됨을 나타낸다. 노화 조절의 다운스트림은 실행 유전자이다. 이들은 노화의 이펙터이고 노화 증후군을 야기하는 대사적 변화를 유발한다. 이는 eIF-5A 및 DHS가 하향-조절될 때 노화의 의해 유발된 광범위한 징후를 감소시킬 수 있음을 나타낸다.
실시예 1은 다양한 식물 내의 DHS 분리 및 이를 인코드하는 mRNA를 기술한다. 실시예 2는 토마토 잎을 솔비톨로 처리함으로서 토마토 내 노화-유도된 DHS의 유도를 기술한다. 도 52는 및 62는 잎이 솔비톨로 처리될 때 DHS 발현이 증가됨을 나타낸다. 실시예 3 및 도 50 및 63은 DHS가 노화 꽃에서 상향-조절됨을 나타낸다. 실시예 4 및 도 51 및 61은 DHS가 숙성 열매에서 상향-조절됨을 나타낸다.
또한 본 발명은 eIF-5A의 이소폼(노화-유도된 eIF-5A);(손상-유도된 eIF-5A) 및 (생장 eIF-5A)을 인식하는 항체에 관한 것이다.
또한 본 발명은 다른 식물 및 진균 내에서 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A 또는 DHS를 확인하는 방법을 제공한다. 여기에 기술된 방법 및 제공된 서열을 이용함으로서 프로브는 원하는 이소폼 또는 DHS를 분리/확인하도록 고안된다. eIF-5A의 이소폼(노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A, 생장 eIF-5A)이 코딩 구역 내에서 종종 매우 상동적이기 때문에(도 2 참조) 원하는 이소폼의 확인을 확신하고 증폭을 변화시키기 위해 바람직하게는 프로브 또는 프라이머는 5'UTR의 시작점 및 3'UTR의 말단으로부터 고안된다(도 3, 4 및 5 참조). 손상/병원체-유도된 eIF-5A의 증폭을 위한 바람직한 프라이머 또는 손상/병원체-유도된 eIF-5A의 확인을 위한 바람직한 프라이머의 세트는 하기와 같다. 다운스트림 프라이머는 5' GAG CTC AAG AAT AAC ATC TCA TAA GAAAC 3'(서열번호: 33)이다. 프라이머의 업스트림은 5' CTC GAG TGC TCA CTT CTC TCT CTT AGG 3'(서열번호: 34)이다.
식물 또는 식물 부분으로부터 손상/병원체-유도된 eIF-5A를 분리하기 전에 식물이 손상/병원체-유도된 eIF-5A를 발현하기 시작하도록 손상 이벤트를 도입하는 것이 최선이다. 어떠한 손상 이벤트도 수용 가능하고 예시적인 손상 이벤트는 중앙 엽맥에서 잎을 압착시키는 것을 포함한다. 유사하게는 노화-유도된 eIF-5A를 분리하기 전에 식물 조직이 노화를 유도하도록 하는 것이 최선이다.
일반적으로 여기에 기술된 바와 같이 동일한 것이 실례에 의해 제공된 하기 실시예에 대한 참조를 통해 쉽게 이해될 것이고, 이는 본 발명에 한정적이지 않다.
도 1은 eIF-5A 분리된 아라비돕시스 탈리아나(Arabidopsis thaliana) 노화-유도된 eIF-5A(서열번호: 58; 라인 1)(미국특허 제6,538,182호 및 미국출원 제09/725,019호에서 앞서 기술됨); 손상/병원체-유도된 eIF-5A(서열번호: 59; 라인 2); 및 생장 eIF-5A(서열번호: 60; 라인 3)의 3가지 이소폼의 정렬을 나타낸 것이다. 동일한 아미노산은 점선으로 강조되고 펩타이드 고안에 사용된 영역은 직선으로 나타내었다. 각각의 펩타이드는 컨쥬게이션이 KLH와 함께 발생하도록 하기 위해 N-말단의 추가적인 시스테인 잔기뿐만 아니라 eIF-5A 서열 유래 11개 아미노산을 포함한다.
도 2는 3가지 아라비돕시스 탈리아나 이소폼의 코딩 구역의 정렬을 나타낸 것이다. 라인 1은 노화-유도된 eIF-5A(서열번호: 61)이다. 라인 2는 손상/병원체-유도된 eIF-5A(서열번호: 62)이다. 라인 3은 생장 eIF-5A(서열번호: 63)이다. 3개 이소폼 모두에서 동일한 염기쌍은 사각형으로 나타내었다. 서열은 메티오닌(ATG)에서 종결 코돈까지의 코딩 영역만을 포함한다.
도 3은 아라비돕시스 탈리아나의 노화-유도된 eIF-5A의 게놈 서열을 제공한다(서열번호: 78). 점선 밑줄은 프라이머가 고안된 영역을 나타낸다. 또한 이원 벡터 내로 라이게이트시 적당한 방향을 보증하기 위해 5' 말단 프라이머는 HindⅢ 제한 사이트를 포함하였고 3' 말단 프라이머는 SacⅠ 제한 사이트를 포함하였다. 사각형 영역은 노던 블럿용 프로브로 사용된 3' 말단을 나타낸다.
도 4는 아라비돕시스 탈리아나의 손상/병원체-유도된 eIF-5A의 게놈 서열을 제공한다(서열번호: 79). 점선 밑줄은 프라이머가 고안된 영역을 나타낸다. 또한 이원 벡터 내로 라이게이트시 적당한 방향을 보증하기 위해 5' 말단 프라이머는 XhoⅠ 제한 사이트를 포함하였고 3' 말단 프라이머는 SacⅠ 제한 사이트를 포함하였다. 사각형 영역은 노던 블럿용 프로브로 사용된 3' 말단을 나타낸다.
도 5는 아라비돕시스 탈리아나의 생장 eIF-5A의 게놈 서열을 제공한다(서열번호: 52). 점선 밑줄은 프라이머가 고안된 영역을 나타낸다. 또한 이원 벡터 내로 라이게이트시 적당한 방향을 보증하기 위해 5' 말단 프라이머는 XhoⅠ 제한 사이트를 포함하였고 3' 말단 프라이머는 SacⅠ 제한 사이트를 포함하였다. 사각형 영역은 노던 블럿용 프로브로 사용된 3' 말단을 나타낸다.
도 6은 이원 벡터 pKYLX71-35S2의 지도이다(서열번호: 80). 이원 벡터 pKYLX71-35S2는 E. coli 내 형질전환체 선택을 위한 테트라사이클린 저항성 및 카나마이신을 함유한 MS 플레이트 상에서의 종자 형질전환체 선택을 위한 카나마이신 저항성을 포함한다. 프로모터는 단일 35S 보다 더 높은 수치의 발현을 제공하도록 작용하는 이중 35S 프로모터이다. RbcS 3'은 리불로스-1,5-비스포스페이트 카르복실라제의 UTR이다.
도 7은 이원 벡터 pGEM-T EASY Vector의 지도이다. 다수의 클로닝 사이트 중간의 T 돌출부는 PCR 생성물의 삽입 사이트를 제공한다. Ampr 유전자는 암피실린 존재시 생장에 기반한 형질전환체 선별에 유용하다.
도 8은 콜롬비아 생태형의 아라비돕시스 탈리아나 야생형의 다른 조직 내의 eIF-5A의 3가지 이소폼의 웨스턴 블럿을 나타낸 것이다. 레인 설명은 하기와 같다: 2, 3, 4, 5, 6, 7로 표지된 레인은 2, 3, 4, 5, 6, 7주령에서 채취된 총 로제트 잎이고; Tr은 5% PEG로 처리된 식물 유래 잎이고; U는 물로 급수된 PEG 대조군 식물 유래 잎이고; B는 폐쇄된 비개화 꽃봉오리이고; F1은 폐쇄 봉오리부터 노화 꽃까지에 걸친 모든 주령의 꽃이고; Si는 6주에 채취된 장각과이고; Se는 1일간 섭취된 종자이고; St는 6주에 채취된 줄기이다.
도 9는 콜롬비아 생태형의 아라비돕시스 탈리아나 야생형의 감염된 잎의 72시간 후 노화-유도된 eIF-5A 및 손상/병원체-유도된 eIF-5A에 대한 웨스턴 블럿이다. 노화-유도된 AteIF-5A의 발현 수치는 식물이 4주령일때 일정하게 유지된다. 손상-유도된 AteIF-5A의 발현은 독성 처리된 식물에서 증가된다. 생장 AteIF-5A의 발현은 검출 가능하지 않았고 따라서 도면에 포함되지 않았다.
도 10은 콜롬비아 생태형의 아라비돕시스 탈리아나 야생형의 손상된 잎의 72시간 후 eIF-5A의 3가지 이소폼의 노던 블럿이다. 잎은 지혈기로 손상되었고 0시간, 처리 직후, 손상 1시간 후 및 손상 9시간 후에 채취되었다. 손상-유도된 AteIF-5A 전사체의 발현은 손상-9시간 후에 증가된 것 하나뿐이다. 생장 AteIF-5A의 발현은 낮게 시작되었으나 손상 이벤트시 감소된다.
도 11은 각각 라인 1, 2 및 3 내의 노화-유도된 eIF-5A(AteIF-5A), 손상/병원체-유도된 AteIF-5A 및 생장 AteIF-5A의 게놈 DNA로부터의 PCR 생성물을 나타낸 것이다. 단일 상단 밴드가 절단되고 pGEM 내로의 라이게이션을 위해 정제되었다.
도 12는 pGEM 내의 노화-유도된 AteIF-5A, 손상/병원체-유도된 AteIF-5A 및 생장 AteIF-5A로의 아가로스 겔을 나타낸 것이다. 적당한 크기의 삽입물을 포함한 양성 형질전환체 콜로니를 확인하기 위해 pGEM: 노화-유도된 AteIF-5A, pGEM: 순상-유도된 AteIF-5A 및 pGEM: 생장 AteIF-5A가 EcoRI로 분해되었다. 이후 이들 콜로니는 식물 내 과발현에 대한 적합성을 확인하기 위해 서열 분석되었다.
도 13은 pKYLX71 내의 손상/병원체-유도된 AteIF-5A, 생장 AteIF-5A, 게놈 서열로의 아가로스 겔을 나타낸 것이다. 테트라사이클린 함유 플레이트 상에서 생장할 수 있는 콜로니는 적당한 효소 및 대응 프라이머를 지닌 PCR(P)로의 이중 분해(D)를 통해 손상-유도된 AteIF-5A 삽입물 또는 생장 AteIF-5A 삽입물로 선별되었다.
도 14는 센스 손상/병원체-유도된 AteIF-5A를 지닌 컨스트럭트로 형질전환된 식물에 대한 T1 식물의 사진이다. 이러한 플레이트 상의 2개의 형질전환체는 흑색 원이고 레인 13 및 14에 해당한다. 야생형 대조군은 백색 원이다.
도 15는 4주령의 센스 손상/병원체-유도된 AteIF-5A로 형질전환된 T1 식물의 사진이다. 형질전환 라인은 P 태그로 나타내고 야생형 식물은 W 태그로 나타내고 이원 벡터 대조군 식물은 Y 태그로 나타내었다. 레인 6, 8, 10, 13 및 14는 종자를 생성하지 않았다.
도 16은 5.5주령의 센스 손상/병원체-유도된 AteIF-5A로 형질전환된 T1 식물의 사진이다. 매우 작은 라인만이 도면에 포함된다. 라인 1, 4 및 12는 모두 종자를 생성하였고 나머지는 실질적으로 종자 생성 없이 사멸되었다.
도 17은 씨뿌리기 10일 후의 센스 손상/병원체-유도된 AteIF-5A로 형질전환된 T2 식물의 사진이다. 모든 T2 라인은 카나마이신 저항성(어두운 식물)과 카나마이신 저항성이 부재한 비-형질전환체(밝은 식물)의 혼합으로 나타난 바와 같이 이형접합성이다. 야생형 대조군 식물은 백색 원으로 나타내었다. 라인 12는 하나의 형질전환체 생장만을 포함하고 이식되어야 하기 때문에 도면에 포함되지 않았다.
도 18은 씨뿌리기 10일 후의 센스 생장 AteIF-5A로 형질전환된 T1 식물의 사진이다. 3개 형질전환체는 이러한 플레이트 대해 흑색 원으로 나타내었고 라인 6, 7 및 8에 해당된다. 야생형 대조군 식물은 백색 원으로 나타내었다.
도 19는 4주령의 센스 생장 AteIF-5A로 형질전환된 T1 식물의 사진이다. 형질전환 라인은 B 태그로, 야생형 대조군은 W 태그 또는 태그 없이 나타내었다. 빈 이원 대조군(Y 태그)는 야생형과 어떠한 차이도 나타내지 않음을 나타내는 도면의 하단에 포함되어 있다.
도 20은 센스 생장 AteIF-5A 라인으로 형질전환된 T2 식물의 웨스턴 블럿이다. 각 모계의 대표는 각 라인 발현의 일반적 수치를 측정하는데 사용되었다.
도 21은 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 1A-1D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그로(회색 원), 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 1A(흑색 사각형으로 나타냄)은 T3을 통해 운반될 것이다.
도 22는 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 2A-1D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 2D(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 23은 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 4A-D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 4D(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 24는 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 15A-D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 15A(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 25는 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 8A-D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 8D(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 26은 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 9E-H)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 9H(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 27은 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 11A-D)로 형질전환된 T2 식물이다. 형질전환 라인은 청색 태그로, 야생형 대조군은 백색 태그로 나타내었다. 빈 이원 벡터 대조군은 황색 태그로 나타내었다. 라인 11C(청색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 28은 4주령(상단), 5주령(좌측 하단) 및 6주령(우측 하단)의 센스 생장 AteIF-5A(라인 16A-D)로 형질전환된 T2 식물이다. 형질전환 라인은 B 태그(회색 원)로, 야생형 대조군은 W 태그(백색 타원)로 나타내었다. 빈 이원 벡터 대조군은 Y 태그(흑색 원)로 나타내었다. 라인 16C(흑색 사각형으로 나타냄)는 T3을 통해 운반될 것이다.
도 29는 다양한 식물 라인(야생형 대조군 및 센스 생장 AteIF-5A로 형질전환된 식물 라인을 포함한)으로부터의 아라비돕시스 탈리아나 종자의 사진이다. 라인 11C 및 16C는 평균 야생형 종자 크기의 88 및 87%인 반면 라인 2D 및 2H는 각각 야생형보다 273 및 299% 더 크다.
도 30은 센스 생장 AteIF-5A로 형질전환된 각 식물 서브라인의 평균 종자 크기의 막대 그래프이다. 각 라인은 도면에서 개별적으로 표지되지 않은 A-H 서브라인을 지닌다. 이원 대조군 및 야생형 대조군은 마지막 2개 막대에 해당된다. 오차 막대로 표시된 표준 오차는 n=10으로 계산되었다.
도 31은 센스 생장 AteIF-5A로 형질전환된 각 식물 서브라인에 대한 개체 종자 중량의 막대 그래프이다. 각 라인은 서브라인 A-H를 지닌다. 이원 대조군 및 야생형 대조군은 마지막 2개 막대에 해당된다.
도 32는 개체 종자 중량과 개체 종자 부피 사이의 비례적 관계를 나타낸 그래프이다.
도 33은 센스 생장 AteIF-5A로 형질전환된 각 식물 서브라인에 대한 식물 당 종자 수확량을 나타낸 막대 그래프이다. 각 라인은 서브라인 A-H를 지닌다. 이원 대조군 및 야생형 대조군은 마지막 2개 막대에 해당된다.
도 34는 센스 생장 AteIF-5A 식물에 나타난 표현형의 요약이다. 표현형은 웨스턴 블럿팅에 의해 측정된 발현 수치에 기반하여 분류된다. 높은 수치의 발현을 입증한 라인은 직교(crosshatching)로 표시되고 중간 수치의 발현을 입증한 라인은 빗금쳐 있고 낮거가 발현을 나타내지 않음을 입증한 라인은 백색으로 나타내었다. 발현을 나타내지 않은 라인은 동시억제에 의한 것이다.
도 35는 형질전환 아라비돕시스 식물(안티센스 전체 길이 노화-유도된 eIF-5A로 형질전환된)과 야생형 식물의 비교를 나타낸 것이다. 형질전환 식물은 지연된 노화를 나타낸다. 형질전환 라인은 왜소하고 증가된 수의 작은 로제트 잎을 지니고 지연된 노화를 나타낸다.
도 36∼38은 식물(안티센스 생장 eIF-5A로 형질전환된)의 사진을 나타낸다.
도 39는 안티센스 아라비돕시스 탈리아나 3'DHS를 생성하기 위한 벡터를 구축하는데 사용된 프라이머(서열번호: 81∼82)를 나타낸 것이다. 증폭된 아라비돕시스 서열은 각각 나타난 순서대로 서열번호: 83∼84에 개시되어 있다.
도 40은 벡터 컨스트럭트를 나타낸 것이다.
도 41은 아라비돕시스로부터 분리된 손상/병원체 인자 eIF-5A에 대한 서열(서열번호: 54에 개시된 DNA 서열; 서열번호: 55에 개시된 단백질) 및 안티센스 컨스트럭트의 위치를 나타낸 것이다. 서열번호: 85∼86에 개시된 프라이머.
도 42는 벡터 컨스트럭트(각각 나타난 순서대로 서열번호: 87∼89)를 나타낸 것이다.
도 43은 슈도모나스(pseudomanas)로 접종된 잎 디스크의 식물 카운트를 나타낸 것이다.
도 44는 야생형에 대한 안티센스 형질전환 식물 내 CFU의 그래프를 나타낸 것이다.
도 45는 토마토 잎 DHS cDNA 서열의 뉴클레오타이드 서열(서열번호: 1) 및 토마토 잎 cDNA 라이브러리로부터 수득된 유도된 아미노산 서열(서열번호: 2)을 나타낸 것이다.
도 46은 밑줄친 게놈 서열을 지닌 토마토 DHS 서열을 아라비돕시스 유전자 은행(서열번호: 5; 서열번호: 6에 개시된 단백질) 내의 확인되지 않은 게놈 서열에 정렬시킴으로서 수득된 아라비돕시스 DHS 유전자의 뉴클레오타이드 서열을 나타낸 것이다. 아미노산 서열 사이의 갭은 추정되는 인트론이다. 도 46B는 유도된 아라비돕시스 DHS 아미노산 서열(서열번호: 6)을 나타낸 것이다. 도 46C는 PCR에 의해 수득된 600 염기쌍의 아라비돕시스 DHS cDNA의 뉴클레오타이드 서열을 나타낸 것이다(서열번호: 26). 도 46D는 아라비돕시스 DHS cDNA 단편의 아미노산 서열을 나타낸 것이다(서열번호: 92).
도 47은 유도된 전체 길이 토마토 잎 DHS 아미노산 서열(서열번호: 2의 잔기 1∼369) 및 유도된 전체 길이 아라미돕시스 노화-유도된 DHS 아미노산 서열(서열번호: 6)과 인간(서열번호: 3), 효모(서열번호: 45), 진균(서열번호: 44) 및 알케오박테리아(Archaeobacteria)(서열번호: 46)의 DHS 단백질의 서열과의 정렬이다. 3 또는 4의 서열 사이의 동일한 아미노산이 박스쳐 있다.
도 48은 토마토 DHS cDNA의 제한 지도이다.
도 49는 토마토 잎으로부터 분리되고, 32P-dCTP-표지된 전체 길이 토마토 DHS cDNA로 프로브된 게놈 DNA의 서던 블럿이다.
도 50은 발달의 다른 단계에서의 토마토 꽃으로부터 분리된 RNA의 노던 블럿이다. 상부 패널은 총 RNA의 에티듐 브로마이드 염색된 겔이다. 각각의 레인은 10㎍의 RNA를 함유한다. 하부 패널은 32P-dCTP-표지된 전체 길이 토마토 DHS cDNA로 프로브된 노던 블럿의 방사능 사진이다.
도 51은 32P-dCTP-표지된 전체 길이 토마토 DHS cDNA로 프로브된 다양한 숙성 단계에서의 토마토 열매로부터 분리된 RNA의 노던 블럿이다. 각각의 레인은 10㎍의 RNA를 함유하였다.
도 52는 6시간 동안 2M 솔비톨로 처리함으로서 가뭄-스트레스 받은 토마토 잎으로부터 분리된 RNA의 노던 블럿이다. 각각의 레인은 10㎍의 RNA를 함유한다. 블럿은 32P-dCTP-표지된 전체 길이 토마토 DHS cDNA로 프로브되었다.
도 53은 냉기 온도에 노출된 토마토 잎으로부터 분리된 RNA의 노던 블럿이다. 도 53A는 총 RNA의 에티듐 브로마이드 염색된 겔이다. 각각의 레인은 10㎍의 RNA를 함유한다. 도 53B는 32P-dCTP-표지된 전체 길이 토마토 DHS cDNA로 프로브된 노던 블럿의 방사능 사진이다. 도 53C는 잎 확산체의 전도율로서 측정된 상응하는 누출 데이터를 나타낸다.
도 54는 폴리A 꼬리 및 5' 말단 비-코딩 구역을 포함하지 않은 카네이션 DHS 전체길이(1384 염기쌍) cDNA 클론 뉴클레오타이드 서열(서열번호: 9)이다. 유도된 아미노산 서열은 뉴클레오타이드 서열 아래에 나타나 있다(373 아미노산)(서열번호: 10).
도 55는 32P-dCTP-표지된 전체 길이 아라비돕시스 DHS cDNA로 프로브된 노화 아라비돕시스 잎으로부터의 총 RNA의 노던 블럿이다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 56은 다양한 단계의 카네이션 꽃의 화판으로부터 분리된 총 RNA의 노던 블럿이다. 블럿은 32P-dCTP-표지된 전체-길이 카네이션 DHS cDNA로 프로브되었다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 57은 토마토 열매 노화-유도된 elF-5A 유전자의 뉴클레오타이드(상)(서열번호: 11) 및 유도된 아미노산 서열(하)(서열번호: 12)이다.
도 58은 카네이션 노화-유도된 elF-5A 유전자의 뉴클레오타이드(상)(서열번호: 13) 및 유도된 아미노산 서열(하)(서열번호: 14)이다.
도 59는 아라비돕시스 노화-유도된 elF-5A 유전자의 뉴클레오타이드(상)(서열번호: 15) 및 유도된 아미노산 서열(하)(서열번호: 16)이다.
도 60은 다양한 발단 단계에서의 아라비돕시스 식물의 잎으로부터 분리된 총 RNA의 노던 블럿이다. 블럿은 32P-dCTP-표지된 전체-길이 아라비돕시스 DHS cDNA 및 전체-길이 노화-유도된 elF-5A로 프로브되었다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 61은 브레이커(breaker)(BK), 레드-펌(red-firm, RF) 및 레드-소프트(RS) 발달 단계에서의 토마토 열매로부터 분리된 총 RNA의 노던 블럿이다. 블럿은 32P-dCTP-표지된 전체-길이 DHS cDNA 및 전체-길이 노화-유도된 elF-5A로 프로브되었다. DHS 및 elF-5A는 열매 숙성과 일치하게 레드-소프트 열매에 병행하여 상향-조절된다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 62는 가뭄 스트레스를 유도하기 위해 솔비톨로 처리된 토마토 잎으로부터 분리된 총 RNA의 노던 블럿이다. C는 대조군이고; S는 솔비톨 처리된 것이다. 블럿은 32P-dCTP-표지된 전체-길이 DHS cDNA 및 전체-길이 노화-유도된 elF-5A로 프로브되었다. elF-5A와 DHS 모두 가뭄 스트레스에 대해 상향-조절된다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 63은 토마토 식물의 꽃봉오리 및 개화된 노화 꽃으로부터 분리된 총 RNA의 노던 블럿이다. 블럿은 32P-dCTP-표지된 전체-길이 DHS cDNA 및 전체-길이 노 화-유도된 elF-5A로 프로브되었다. elF-5A와 DHS 모두 개화/노화 꽃에서 상향-조절되었다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 64는 냉기(chill)-손상된 토마토 잎으로부터 분리된 총 RNA의 노던 블럿이다. 블럿은 32P-dCTP-표지된 전체-길이 DHS cDNA 및 전체-길이 노화-유도된 elF-5A로 프로브되었다. elF-5A와 DHS 모두 다시 따뜻해지는 동안 냉기 손상의 발달로 상향-조절되었다. 방사능 사진이 상위이고, 에티듐 염색된 겔이 하위이다.
도 65는 3.1주령 아라비돕시스 야생형(좌측) 및 형질전환 식물에서 증가된 잎 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타남)을 발현하는 형질전환 식물의 사진이다.
도 66은 4.6주령 아라비돕시스 야생형(좌측) 및 형질전환 식물에서 증가된 잎 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타남)을 발현하는 형질전환 식물의 사진이다.
도 67은 5.6주령 아라비돕시스 야생형(좌측) 및 형질전환 식물에서 증가된 잎 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타 남)을 발현하는 형질전환 식물의 사진이다.
도 68은 6.1주령 아라비돕시스 야생형(좌측) 및 형질전환 식물에서 증가된 잎 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타남)을 발현하는 형질전환 식물의 사진이다.
도 69는 안티센스 방향으로 노화-유도된 DHS 유전자를 발현하는 3개의 T1 형질전환 아라비돕시스 식물 라인으로부터 수확된 종자의 증가를 나타내는 그래프이다. 종자 수확량은 종자의 부피로 표현된다. n = 30에 대한 SE는 야생형 식물에 대해 나타난다.
도 70은 형질전환 식물에서 증가된 잎 크기와 증가된 식물체 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타남)을 발현하는 형질전환 토마토 식물(좌측) 및 야생형 식물(우측)의 사진이다. 사진은 묘목을 토양으로 옮긴지 18일 후에 찍혔다.
도 71은 형질전환 식물에서 증가된 잎 크기와 증가된 식물체 크기를 나타내는 안티센스 방향으로 DHS 유전자의 3'-말단(서열은 도 80에 나타남)을 발현하는 형질전환 토마토 식물(좌측) 및 야생형 식물(우측)의 사진이다. 사진은 묘목을 토양으로 옮긴지 32일 후에 찍혔다.
도 72∼79는 야생형(상위 패널) 및 안티센스 방향으로 전체-길이 DHS 유전자를 발현하는 형질전환 식물(하위 패널)로부터의 토마토 열매의 사진이다. 열매는 발달의 브레이커 단계에서 수확되어 그로쓰 챔버(growth chamber)에서 숙성되었다. 수확후 날짜가 각 패널의 상부 좌측 모서리에 나타나 있다.
도 80은 식물을 형질전환시키기 위해 안티센스 방향으로 사용된 아라비돕시스 노화-유도된 DHS 유전자의 3'-말단의 뉴클레오타이드(상)(서열번호: 30) 및 유도된 아미노산(하)(서열번호: 90) 서열이다.
도 81은 식물을 형질전환시키기 위해 안티센스 방향으로 사용된 토마토 DHS 유전자의 3'-말단의 뉴클레오타이드(상)(서열번호: 31) 및 유도된 아미노산(하)(서열번호: 91) 서열이다.
도 82는 전체-길이 아라비돕시스 유전자를 분리하기 위해 사용된 600 염기쌍의 아라비돕시스 DHS 프로브의 뉴클레오타이드(상)(서열번호: 26) 및 유도된 아미노산(하)(서열번호: 92) 서열이다.
도 83은 전체-길이 카네이션 유전자를 분리하기 위해 사용된 483 염기쌍의 카네이션 DHS 프로브의 뉴클레오타이드(상)(서열번호: 27) 및 유도된 아미노산(하) (서열번호: 93)서열이다.
도 84(a) 및 (b)는 안티센스 방향으로 DHS 유전자의 3'-말단을 발현하는 형질전환 토마토 식물로부터의 토마토 열매(좌측) 및 야생형 식물로부터의 토마토 열매(우측)의 사진이다. 야생형 열매는 꽃 말단 부패를 나타내는 반면 형질전환 열매는 그렇지 않다.
도 85는 일부 식물종 유래의 다양한 eIF-5A 이소폼의 정렬을 나타낸 것이다. 이는 또한 하이푸신 보존 구역의 정렬을 제공한다. 각각 나타난 순서대로 서열번호: 4, 94∼106 및 108∼125가 나타나 있다.
도 86은 토마토 노화-유도된 eIF-5A 폴리뉴클레오타이드(서열번호: 126) 및 아미노산(서열번호: 127) 서열을 제공한다.
도 87은 아라비돕시스 노화-유도된 eIF-5A 및 pKYLX71-센스 노화-유도된 eIF-5A의 구축을 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 128∼129로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 130∼132로 개시되어 있다.
도 88은 토마토 노화-유도된 eIF-5A 및 pKYLX71-센스 노화-유도된 eIF-5A의 구축을 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 133∼134로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 135∼137로 개시되어 있다.
도 89는 아라비돕시스 탈리아나 대조군과 센스 폴리뉴클레오타이드 노화-유도 eIF-5A를 포함한 형질전환 식물의 비교 사진을 제공한다. 형질전환 식물은 대조군 식물보다 두꺼운 화서 줄기를 지닌다.
도 90 및 91은 센스 폴리뉴클레오타이드 노화-유도 eIF-5A(도 90 - 아라비돕시스 및 도 91 - 토마토)를 포함한 형질전환 식물이 형질전환 식물 내 증가된 물관부에 의해 나타난 증가된 물관생성을 지님을 나타낸 것이다. 물관부 구역은 플로로구시놀-HCl로 회색으로 염색되었다. 막대 = 100 ㎛.
도 92는 아라비돕시스 탈리아나 대조군과 센스 폴리뉴클레오타이드 노화-유도 eIF-5A를 포함한 아라비돕시스 탈리아나 형질전환 식물의 비교 사진을 제공한다. 토마토 센스 폴리뉴클레오타이드 노화-유도 eIF-5A가 아라비돕시스 탈리아나에 사용되었다. 형질전환 식물은 대조군 식물보다 두꺼운 화서 줄기를 지닌다.
도 93 및 94는 센스 폴리뉴클레오타이드 노화-유도 eIF-5A를 포함한 형질전환 식물 내 증가된 물관생성을 나타내는 막대 그래프이다. 도 94는 토마토 센스 폴리뉴클레오타이드 노화-유도된 eIF-5A에 관한 것이다.
도 95는 카놀라 생장 eIF-5A 아미노산(서열번호: 67) 및 폴리뉴클레오타이드(서열번호: 66) 서열을 제공한다.
도 96은 카놀라 생장 eIF-5A 및 pKYLX71-센스 생장 eIF-5A의 구축을 제공한다. 프라이머는 서열번호: 138로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 139∼141로 개시되어 있다.
도 97은 카놀라 DHS 아미노산(서열번호: 71) 및 폴리뉴클레오타이드(서열번호: 70) 서열을 제공한다.
도 98은 카놀라 DHS 및 pKYLX71-센스 DHS의 구축을 제공한다. 3'-UTR 서열은 서열번호: 142로 개시되어 있다. 벡터 서열은 각각 서열번호: 143∼145로 개시되어 있다.
도 99는 막대 그래프 형태 내에서 DHS 발현의 억제가 카놀라 내 종자 수확량을 증가시킴을 나타낸다.
도 100은 막대 그래프 형태 내에서 좌측으로부터 아라비돕시스, 카놀라, 토 마토의 생장 eIF-5A 이소폼의 상향 조절 및 토마토 DHS의 상향 조절을 나타낸다.
도 101은 토마토 생장 eIF-5A 아미노산(서열번호: 65) 및 폴리뉴클레오타이드(서열번호: 64) 서열을 제공한다.
도 102는 생장 eIF-5A 및 pKYLX71-센스 토마토 생장 eIF-5A의 구축을 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 146∼147로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 148∼150에 나타나 있다.
도 103은 손상/병원체-유도된 eIF-5A 아미노산(서열번호: 57) 및 폴리뉴클레오타이드(서열번호: 56) 서열을 제공한다.
도 104a 및 b는 토마토 손상/병원체-유도된 eIF-5A 및 pKYLX71-센스 토마토 손상/병원체-유도된 eIF-5A의 구축을 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 151∼152로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 153∼155에 나타나 있다.
도 105는 상추 DHS 폴리뉴클레오타이드 서열의 일부를 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 156∼157로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 158∼159에 나타나 있다.
도 106은 pTA7001-3'UTR 안티센스 상추 DHS의 컨스트럭트를 제공한다.
도 107A 및 B는 자주개자리 DHS 아미노산 및 폴리뉴클레오타이드 서열을 제공한다. 도 107A는 서열번호: 73을 코드하는 서열번호: 72의 뉴클레오타이드 1∼1861을 개시한다. 도 107B는 서열번호: 72(뉴클레오타이드) 및 73(단백질)을 개시한다.
도 108A 및 B는 바나나 DHS 아미노산(서열번호: 75) 및 폴리뉴클레오타이드(서열번호: 74) 서열을 제공한다.
도 109A 및 B는 미루나무 DHS 아미노산(서열번호: 77) 및 폴리뉴클레오타이드(서열번호: 76) 서열을 제공한다.
도 110은 미코스파에렐라 피지엔시스(Mycosphaerella fijiensis) DHS 아미노산 및 폴리뉴클레오타이드 서열을 제공한다. 도면은 각각 나타난 순서대로 서열번호: 164/53, 47(프라이머), 107 및 53을 코드하는 서열번호: 68, 160, 69, 161-163, 165, 163을 개시한다.
도 111은 eIF-5A 활성화의 개략도를 나타낸다.
도 112는 카네이션 노화 및 eIF-5A 발현의 노던 분석이다.
도 113은 형질전환 카네이션(안티센스 노화-유도된 eIF-5A)가 절단 꼿의 더 긴 저장 수명을 나타냄을 나타낸다.
도 114는 바나나의 지연된 부패를 나타낸다(안티센스 DHS를 지닌 형질전환 식물).
도 115는 토마토의 지연된 부패를 나타낸다(안티센스 DHS를 지닌 형질전환 식물).
도 116은 자엽, 잎 및 꽃 내 아라비돕시스 노화-유도된 eIF-5A(AteIF-5A1)의 발현을 나타낸다.
도 117은 형질전환 아라비돕시스(아라비돕시스 안티센스 노화-유도된 eIF-5A)(AteIF-5A1) 내 아라비돕시스 잎의 지연된 노화를 나타낸다.
도 118은 첫 번째 진성 잎쌍 내 아라비돕시스 노화-유도된 eIF-5A::GUS의 발현을 나타낸다.
도 119는 형질전환 식물(안티센스 노화-유도된 eIF-5A)이 야생형 식물보다 더 적은 물관부를 지님을 나타낸다.
도 120은 형질전환 식물(노화-유도된 eIF-5A의 센스 컨스트럭트) 내 증가된 물관부 형성을 나타낸다.
도 121은 형질전환 식물(안티센스 컨스트럭트를 통한 스트레스-유도된 eIF-5A의 감소된 발현) 내 가뭄 저항성을 나타낸다.
도 122는 야생형 및 형질전환 아라비돕시스의 가뭄 스트레스 하에서의 생존율을 나타내는 막대 그래프이다. 형질전환 식물(안티센스 컨스트럭트를 통한 스트레스-유도된 eIF-5A의 감소된 발현)은 형질전환 카놀라가 증가된 생물량을 지님을 지닌다.
도 123은 야생형 카놀라와 형질전환 카놀라(안티센스 컨스트럭트를 통한 스트레스-유도된 eIF-5A의 감소된 발현)의 비교를 나타내고 형질전환 카놀라가 증가된 생물량을 지님을 나타낸다.
도 124는 형질전환 카놀라 라인(안티센스 컨스트럭트를 통한 스트레스-유도 된 eIF-5A의 감소된 발현)이 야생형 식물보다 증가된 종자 수확량을 지님을 나타낸다.
도 125는 손상/병원체 유도된 eIF-5A가 슈도모나스 시린게(Pseudomanas syringae)로의 감염 후 상향-조절됨을 나타낸다.
도 126은 eIF-5A의 손상/병원체-유도된 이소폼의 안티센스 억제가 독성 병원체에 의한 감염을 저해함을 나타낸다. 형질전환 식물은 대조군 식물보다 99% 이상의 감염 저해 증가를 나타내었다.
도 127∼130은 형질전환 카놀라(eIF-5A의 안티센스 손상/병원체-유도된 이소폼)가 스클레로티니아 스클레로티오룸(Sclerotinia sclerotiorum)에 의한 감염 저항성을 나타낸다. "WT"는 야생형 식물 유래 잎이다. "TG-1" 및 "TG-5"는 형질전환 식물 유래 잎이다.
도 131은 형질전환 바나나(안티센스 DHS)(좌측) 및 야생형 바나나(우측)의 사진을 나타내고 형질전환 바나나 식물이 야생형 대조군보다 더 큰 생물량을 지님을 나타낸다.
도 132는 형질전환 토마토(센스 방향의 생장 eIF-5A)(좌측)가 야생형 대조군 식물(우측)보다 증가된 생물량을 지님을 나타낸다.
도 133은 형질전환 카놀라(센스 방향의 생장 eIF-5A)가 야생형 대조군 식물보다 증가된 생물량을 지님을 나타낸다.
도 134는 형질전환 카놀라(센스 방향의 생장 eIF-5A)가 야생형 대조군 식물보다 증가된 종자 수확량을 지님을 나타낸다.
도 135는 상추 DHS 서열을 제공한다. 핵산은 서열번호: 166이고 아미노산 서열은 서열번호: 167이다.
도 136은 페튜니아 DHS 뉴클레오타이드(서열번호: 170) 및 아미노산(서열번호: 171) 일부 서열뿐만 아니라 DHS 서열을 분리하는데 사용된 2개의 프라이머(서열번호: 168∼169)를 제공한다.
도 137은 손상/병원체 eIF-5A에 대한 자주개자리 핵산 서열(서열번호: 172) 및 아미노산 서열(서열번호: 173)을 제공한다.
도 138은 노화 eIF-5A에 대한 자주개자리 핵산 서열(서열번호: 174) 및 아미노산 서열(서열번호: 175)을 제공한다.
도 139는 스트레스 eIF-5A에 대한 자주개자리 핵산 서열(서열번호: 176) 및 아미노산 서열(서열번호: 177)을 제공한다.
도 140은 생장 eIF-5A에 대한 자주개자리 핵산 서열(서열번호: 178) 및 아미노산 서열(서열번호: 179)을 제공한다.
도 141∼144는 상추 내 eIF-5A의 다른 이소폼에 대한 부분적 서열을 제공한다(LF5A-1, LF5A-3, LFA-2 및 LFA-1). 도 141은 서열번호: 180(뉴클레오타이드) 및 181(단백질)을 개시한다. 도 142는 서열번호: 182(뉴클레오타이드) 및 183(단백질)을 개시한다. 도 143은 서열번호: 184(뉴클레오타이드) 및 185(단백질)을 개시한다. 도 144는 각각 나타난 순서대로 서열번호: 186∼187(뉴클레오타이드) 및 210∼211(단백질)을 개시한다.
도 145는 토마토에 대한 생장 eIF-5A 서열(핵산 - 서열번호: 188) 및 아미노산(서열번호: 189)에 대한 서열을 제공한다.
도 146은 토마토 내 스트레스 eIF-5A 서열(핵산 - 서열번호: 190) 및 아미노산(서열번호: 191)에 대한 서열을 제공한다.
도 147A-C는 미루나무 유래 4개 eIF-5A에서 분리된 핵산 및 아미노산 eIF-5A 서열을 제공한다("F1"(서열번호: 193을 코드하는 서열번호: 192), "F3"(서열번호: 195를 코드하는 서열번호: 194), "F3"(서열번호: 196을 코드하는 서열번호: 197) 및 "F4"(서열번호: 199를 코드하는 서열번호: 198)).
도 148(a) 및 (b)는 종간 DHS의 높게 보존되는 특성을 나타내는 다양한 DHS의 아미노산 정렬을 제공한다. 본 도면은 각각 나타난 순서대로 서열번호: 45, 2, 200, 73, 77, 201, 10, 167, 75, 202 및 3을 개시한다.
도 149는 DHS의 하향-조절을 지닌 상추 생장에 사용된 물재배 조건을 나타낸다.
도 150은 하향-조절된 상추 DHS 발현을 지닌 절단 상추 내 지연된 갈변을 나타낸다.
도 151은 상추 DHS의 안티센스 3'말단을 지닌 형질전환 상추가 감소된 DHS 발현을 나타냄을 나타내는 웨스턴 블럿을 나타낸다.
도 152는 DHS의 안티센스 3'말단으로 아라비돕시스를 형질전환시키는데 사용하고자 구축된 벡터를 나타낸다.
도 153은 하향 조절된 DHS를 지닌 아라비돕시스 식물이 야생형 식물보다 증가된 생장률/생물량을 지님을 나타낸다(도 152에 나타난 벡터로 생성된 형질전환 아라비돕시스).
도 154는 하향 조절된 DHS를 지닌 아라비돕시스 식물이 야생형 식물보다 증가된 생장률/생물량을 지님을 나타낸다(도 152에 나타난 벡터로 생성된 형질전환 아라비돕시스).
도 155는 하향 조절된 DHS를 지닌 아라비돕시스 식물이 야생형 식물보다 증가된 종자 수확량을 지님을 나타낸다(도 152에 나타난 벡터로 생성된 형질전환 아라비돕시스).
도 156은 하향 조절된 DHS를 지닌 아라비돕시스 식물이 야생형 식물보다 증가된 뿌리 발달을 지님을 나타낸다(도 152에 나타난 벡터로 생성된 형질전환 아라비돕시스).
도 157은 하향 조절된 DHS를 지닌 카놀라 식물이 야생형 식물보다 증가된 생장률/생물량을 지님을 나타낸다.
도 158은 안티센스 DHS가 감소된 DHS 발현을 유발함을 나타내는 웨스턴 블럿을 나타낸다.
도 159는 하향 조절된 DHS를 지닌 카놀라 식물이 야생형 식물보다 증가된 생장각 크기를 지님을 나타낸다.
도 160은 하향 조절된 DHS를 지닌 카놀라 식물이 야생형 식물보다 증가된 종자 생물량을 지님을 나타낸다.
도 161(a) 및 (b)는 하향 조절된 DHS를 지닌 카놀라 식물에서 종자 당 오일 양의 변화가 존재하지 않고 오일 내 지방산 조성의 변화가 존재하지 않음을 나타낸다.
도 162는 생장 eIF-5A의 상향-조절을 달성하기 위해 센스 방향으로 아라비돕시스 내에 미루나무 생장 eIF-5A를 도입시키는 효과를 나타낸다. 수득된 식물은 증가된 생장률/생물량을 지닌다.
도 163은 생장 eIF-5A의 상향-조절을 달성하기 위해 센스 방향으로 아라비돕시스 내에 미루나무 생장 eIF-5A를 도입시키는 효과를 나타낸다. 수득된 식물은 증가된 생장률/생물량을 지닌다.
도 164는 생장 eIF-5A의 상향-조절을 달성하기 위해 센스 방향으로 아라비돕시스 내에 미루나무 생장 eIF-5A를 도입시키는 효과를 나타낸다. 수득된 식물은 증가된 생장률/생물량을 지닌다.
도 165는 카놀라 또는 아라비도빗스 생장 eIF-5A가 카놀라 식물에서 센스 방향으로 사용될 때 카놀라 식물 내 종자 수확량이 증가됨을 나타낸다.
도 166은 돌연변이 노화 eIF-5A(하이푸신화될 수 없음)로 형질전환된 형질전환 아라비돕시스가 야생형 아라비돕시스보다 증가된 생물량을 지님을 나타낸다.
도 167은 돌연변이 노화 eIF-5A(하이푸신화될 수 없음)로 형질전환된 형질전환 아라비돕시스가 야생형 아라비돕시스보다 증가된 생물량을 지님을 나타낸다.
도 168은 아라비돕시스에서 센스 방향으로 발현된 미루나무(Populas deltoides)에서 분리된 생장 eIF-5A가 대조군 색물보다 증가된 종자 수확량을 유발함을 나타낸다.
도 169는 센스 방향으로 발현된 미루나무 생장인자 eIF-5A를 지닌 다른 3개의 형질전환 아라비돕시스 라인 유래의 종자 수확량의 비교를 나타내고, 3개 라인 모두는 증가된 종자 수확량을 지녔다.
도 170은 포풀루스 델토이데스(Populas deltoides)(미루나무) 생장 eIF-5A의 전체 길이 cDNA(서열번호: 203) 및 단백질의 전체 길이 아미노산 서열(서열번호: 204)을 제공한다. 도면 부호는 서열번호: 203의 뉴클레오타이드 40∼57 및 530∼550을 개시한다.
도 171은 센스 방향으로 미루나무 생장 eIF-5A로 아라비돕시스를 형질전환시키는데 사용되는 벡터 컨스트럭트를 제공한다. 프라이머 서열은 각각 나타난 순서대로 서열번호: 205∼206로 개시되어 있다. 벡터 서열은 각각 나타난 순서대로 서열번호: 207∼209로 개시되어 있다.
도 172 및 173은 형질전환 아라비돕시스 식물(도 171에 나타난 벡터로 형질전환된)이 최적, 최적이하 및 최적이상 농도를 포함한 다른 수치의 화학비료로 처리시 대조군 식물보다 증가된 생물량을 지님을 나타낸다.
도 174 및 175는 형질전환 아라비돕시스 식물(돌연변이 노화-유도된 eIF-5A로 형질전환된)이 최적, 최적이하 및 최적이상 농도를 포함한 다른 수치의 화학비료로 처리시 대조군 식물보다 증가된 생물량을 지님을 나타낸다.
(실시예 1)
메신저 RNA(mRNA) 분리
총 RNA는 다양한 발단 단계의 토마토 꽃 및 토마토 열매 및 잎으로부터 분리되었다(비처리, 냉기화후 또는 솔비톨 처리). 간단히 조직(5 g)이 액체 질소 내에서 갈아졌다. 갈린 분말은 30 ml의 구아니디늄 완충액(4 M 구아니디늄 이소티오시아테이트(guanidinium isothiocyanate), 2.5 mM NaOAc pH 8.5, 8.0%의 β-머캅토에탄올(mercaptoethanol)로 혼합되었다. 혼합물은 4겹의 치즈천으로 여과되었고 4℃에서 30분간 10,000×g로 원심분리되었다. 이후 상청액은 20시간 동안 26,000×g에서 염화세슘 밀도 기울기 원심분리되었다. 펠렛화된 RNA는 75% 에탄올로 세척되었고, 600 ㎕의 DEPC-처리된 물에 재현탁되었고, RNA는 -70℃에서 0.75 ml의 95% 에탄올과 30 ㎕의 3 M NaOAc로 침전되었다. 10 ㎍의 RNA는 1.2%의 변성 포름알데하이드 아가로스 겔 상에서 분획화되었고, 나일론 멤브레인으로 옮겨졌다. 무작위로 프라임된 32P-dCTP-표지된 전체 길이 DHS cDNA(서열번호: 1)가 42℃에서 밤새 멤브레인을 프로브하는데 사용되었다. 멤브레인은 상온에서 15분간 0.1%의 SDS를 함유한 1X SSC 내에서 1회 세척되었고, 65℃에서 15분간 0.1%의 SDS를 함유한 0.2X SSC 내에서 3회 세척되었다. 멤브레인은 밤새 -70℃에서 X-선 필름에 노 출되었다.
폴리A+ mRNA는 Promega사의 폴리A+ 트랙(tract) mRNA 분리 시스템을 이용하여 총 RNA로부터 분리되었다. 폴리A+ mRN는 Stratagene(La Jolla, Calif)사의 ZAP Express cDNA 합성 시스템을 이용하여 cDNA 합성을 위한 주형으로서 이용되었다.
토마토 잎 cDNA 라이브러리 스크리닝
2 M의 솔비톨에 6시간 동안 노출된 Match F1 하이브리드 토마토 잎으로부터 분리된 mRNA를 이용하여 제조된 cDNA 라이브러리가 약 5 ×106 PFU/ml로 희석되었다. cDNA 라이브러리는 32P-표지된 600 bp RT-PCR 단편을 이용하여 선별되었다. 3개의 양성 cDNA 클론이 절제되었고, 제조사의 지침 내에 있는 방법을 이용하여 pBK-CMV(Stratagene) 파지미드(phagemid) 내로 재순환되었다. 전체 길이 cDNA가 pBK-CMV 벡터 내로 삽입되었다.
플라스미드 DNA 분리, DNA 서열분석
Sambrook et al.,(상동)에 의해 기술된 알칼리성 분해 방법이 플라스미드 DNA를 분리하는데 이용되었다. 전체 길이 양성 cDNA 클론은 디디옥시(dideoxy) 서열분석 방법을 이용하여 서열분석되었다(Sanger et al., Proc. Natl. Acad. Sci. USA, 74:5463-5467). 오픈 리딩 프레임이 편집되었고, BLAST 검색(GenBank, Bethesda, MD)을 이용하여 분석되었고, 인코드된 유전자의 유도된 아미노산 서열과의 5개의 가장 상동적인 단백질의 정렬은 BCM Search Launcher를 이용하여 이루어졌다: Multiple Sequence Alignments Pattern-Induced Multiple Alignment Method(F. Corpet, Nuc. Acids Res., 16:10881-10890 (1987) 참조). 유도된 아미노산 서열 내에 존재하는 기능적인 모티프는 MultiFinder에 의해 확인되었다
토마토 RNA의 노던 블럿 하이브리디제이션
다양한 단계의 토마토 꽃(봉오리 및 꽃 및 넓게 개화된 노화 화판 또는 건조), 토마토 잎 및 다양한 숙성 단계의 토마토 열매(브레이커 즉, 10% 이하의 붉은 색을 지닌 푸른색 열매, 핑크색 즉, 전체 열매가 오렌지색이거나 핑크색인 및 부드럽거나 단단한 붉은색)로부터 분리된 총 RNA의 10 ㎍이 1%의 변성된 포름알데히드 아가로스 겔 상에서 분리되었고, 나일론 멤브레인 상에 고정되었다. 무작위 프라이머 키트(Boehringer Mannuheim)를 이용하여 32P-dCTP로 표지된 전체 길이 토마토 cDNA가 필터를 프로브하는데 이용되었다(7 ×107 cpm). 필터는 상온에서 1x SSC, 0.1% SDS로 1회 세척되었고, 65℃에서 0.2x SSC, 0.1% SDS로 3회 세척되었다. 필 터는 건조되었고, -70℃에서 밤새 X-선 필름에 노출되었다. 결과는 도 50∼52에 나타나 있다.
아라비돕시스 RNA의 노던 블럿 하이브리디제이션
5주령(레인 1), 6주령(레인 2) 및 7주령(레인 3)의 아라비돕시스 식물의 잎으로부터 분리된 총 RNA가 상기와 같이 분리되었고, 1%의 변성된 포름알데히드 아가로스 겔 상에서 분리되었고, 나일론 멤브레인 상에 고정되었다. 무작위 프라이머 키트(Boehringer Mannuheim)를 이용하여 32P-dCTP로 표지된 전체-길이 아라비돕시스 노화-유도된 DHS cDNA가 필터를 프로브하는데 이용되었다(7 ×107 cpm). 필터는 상온에서 1x SSC, 0.1% SDS로 1회 세척되었고, 65℃에서 0.2x SSC, 0.1% SDS로 3회 세척되었다. 필터는 건조되었고, -70℃에서 밤새 X-선 필름에 노출되었다. 결과는 도 55에 나타나 있다.
카네이션 RNA의 노던 블럿 하이브리디제이션
다양한 발달 단계 즉, 단단한-봉오리 꽃(레인 1), 개화를 시작한 꽃(레인 2), 충분히 개화한 꽃(레인 3), 인롤링 화판을 지닌 꽃(레인 4)의 카네이션 식물의 화판으로부터 분리된 총 RNA가 상기와 같이 분리되었고, 1%의 변성된 포름알데히드 아가로스 겔 상에서 분리되었고, 나일론 멤브레인 상에 고정되었다. 무작위 프라이머 키트(Boehringer Mannuheim)를 이용하여 32P-dCTP로 표지된 전체 길이 카네이션 노화-유도된 DHS cDNA가 필터를 프로브하는데 이용되었다(7 ×107 cpm). 필터는 상온에서 1x SSC, 0.1% SDS로 1회 세척되었고, 65℃에서 0.2x SSC, 0.1% SDS로 3회 세척되었다. 필터는 건조되었고, -70℃에서 밤새 X-선 필름에 노출되었다. 결과는 도 56에 나타나 있다.
(실시예 2)
토마토 노화-유도된 DHS 유전자의 솔비톨 유도
토마토 잎은 6시간 동안 밀폐된 챔버 안에서 2 M 솔비톨로 처리되었다. RNA는 하기와 같이 솔비톨 처리된 잎으로부터 추출되었다.
잎(5 g)은 액체 질소 내에서 갈아졌다. 갈린 분말은 30 ml의 구아니디늄 완충액(4 M 구아니디늄 이소티오시아테이트, 2.5 mM NaOAc pH 8.5, 8.0%의 β-머캅토에탄올로 혼합되었다. 혼합물은 4겹의 치즈천으로 여과되었고 4℃에서 30분간 10,000 ×g로 원심분리되었다. 이후 상청액은 20시간 동안 26,000 ×g에서 염화세슘 밀도 기울기 원심분리되었다. 펠렛화된 RNA는 75% 에탄올로 세척되었고, 600 ㎕의 DEPC-처리된 물에 재현탁되었고, RNA는 -70℃에서 0.75 ml의 95% 에탄올 과 30 ㎕의 3 M NaOAc로 침전되었다. 10 ㎍의 RNA는 1.2%의 변성 포름알데하이드 아가로스 겔 상에서 분획화되었고, 나일로 멤브레인으로 옮겨졌다. 무작위로 프라임된 32P-dCTP-표지된 전체 길이 DHS cDNA(서열번호: 1)가 42℃에서 밤새 멤브레인을 프로브하는데 사용되었다. 멤브레인은 상온에서 15분간 0.1%의 SDS를 함유한 1x SSC 내에서 1회 세척되었고, 65℃에서 15분간 0.1%의 SDS를 함유한 0.2x SSC 내에서 3회 세척되었다. 멤브레인은 밤새 -70℃에서 X-선 필름에 노출되었다.
결과는 도 52에 나타나 있다. 나타난 바와 같이 DHS의 전사는 솔비톨에 의해 잎에서 유도된다.
(실시예 3)
노화 꽃에서의 토마토 DHS 유전자의 유도
토마토 식물의 단단한 꽃봉오리 및 개화, 노화 꽃이 수확되었고, RNA가 실시예 2에서와 같이 분리되었다. 10 ㎍의 RNA가 1.2%의 변성 포름알데하이드 아가로스 겔 상에서 분획화되었고, 나일로 멤브레인으로 옮겨졌다. 무작위로 프라임된 32P-dCTP-표지된 전체 길이 DHS cDNA(서열번호: 1)가 42℃에서 밤새 멤브레인을 프로브하는데 사용되었다. 멤브레인은 상온에서 15분간 0.1%의 SDS를 함유한 1x SSC 내에서 1회 세척되었고, 65℃에서 15분간 0.1%의 SDS를 함유한 0.2x SSC 내에 서 3회 세척되었다. 멤브레인은 밤새 -70℃에서 X-선 필름에 노출되었다.
결과는 도 50에 나타나 있다. 나타난 바와 같이 DHS의 전사는 노화 꽃에서 유도된다.
(실시예 4)
숙성 열매에서의 토마토 DHS 유전자의 유도
브레이커, 핑크 및 숙성 여래로부터의 RNA가 실시예 2에서와 같이 분리되었다. 10 ㎍의 RNA가 1.2%의 변성 포름알데하이드 아가로스 겔 상에서 분획화되었고, 나일로 멤브레인으로 옮겨졌다. 무작위로 프라임된 32P-dCTP-표지된 전체 길이 DHS cDNA(서열번호: 1)(도 45)가 42℃에서 밤새 멤브레인을 프로브하는데 사용되었다. 멤브레인은 상온에서 15분간 0.1%의 SDS를 함유한 1x SSC 내에서 1회 세척되었고, 65℃에서 15분간 0.1%의 SDS를 함유한 0.2x SSC 내에서 3회 세척되었다. 멤브레인은 밤새 -70℃에서 X-선 필름에 노출되었다.
결과는 도 51에 나타나 있다. 나타난 바와 같이 DHS의 전사는 부패를 유도하는 노화의 착수 바로 전 숙성, 붉은 열매에서 가장 강하다.
(실시예 5)
냉기에 의한 토마토 노화-유도된 DHS 유전자의 유도
화분 내의 토마토 식물(7∼8주령)이 6℃, 그로쓰 챔버에서 2일, 3일 또는 6일간 노출되었다. 광 사이클은 8시간 동안 암조건 16시간 동안 광조건으로 맞춰졌다. 식물은 비닐하우스로 다시 옮김으로서 다시 따뜻해졌다. 다시 따뜻해지지 않은 식물은 그로쓰 챔버로부터 제거된 후 바로 수확되었다. RNA는 하기와 같이 잎으로부터 추출되었다.
잎(5 g)은 액체 질소 내에서 갈아졌다. 갈린 분말은 30 ml의 구아니디늄 완충액(4 M 구아니디늄 이소티오시아테이트, 2.5 mM NaOAc pH 8.5, 8.0%의 β-멀캅토에탄올로 혼합되었다. 혼합물은 4겹의 치즈천으로 여과되었고 4℃에서 30분간 10,000 ×g로 원심분리되었다. 이후 상청액은 20시간 동안 26,000 ×g에서 염화세슘 밀도 기울기 원심분리되었다. 펠렛화된 RNA는 75% 에탄올로 세척되었고, 600 ㎕의 DEPC-처리된 물에 재현탁되었고, RNA는 -70℃에서 0.75 ml의 95% 에탄올과 30 ㎕의 3 M NaOAc로 침전되었다. 10 ㎍의 RNA는 1.2%의 변성 포름알데하이드 아가로스 겔 상에서 분획화되었고, 나일론 멤브레인으로 옮겨졌다. 무작위로 프라임된 32P-dCTP-표지된 전체 길이 DHS cDNA(서열번호: 1)가 42℃에서 밤새 멤브레인을 프로브하는데 사용되었다. 멤브레인은 상온에서 15분간 0.1%의 SDS를 함유 한 1x SSC 내에서 1회 세척되었고, 65℃에서 15분간 0.1%의 SDS를 함유한 0.2x SSC 내에서 3회 세척되었다. 멤브레인은 밤새 -70℃에서 X-선 필름에 노출되었다.
결과는 도 53에 나타나 있다. 나타난 바와 같이 DHS의 전사는 냉기 온도에노출하고 뒤이어 다시 따뜻하게 함으로서 잎에서 유도되고, 증가된 전사는 멤브레인의 누출로서 측정된 냉기 손상과 관련된다.
(실시예 6)
확인되지 않은 아라비돕시스 게놈 서열에 기반한 프라이머를 이용한 아라비돕시스 PCR 생성물의 생성
아라비돕시스 cDNA 주형으로부터의 일부 길이의 노화-유도된 DHS 서열이 아라비돕시스 게놈 서열로부터 고안된 올리고뉴클레오타이드 프라이머 쌍을 이용한 PCR에 의해 생성되었다. 5' 프라이머는 서열 5'-GGTGGTGTTGAGGAAGATC(서열번호: 7)를 지닌 19-머(mer)이고 ; 3' 프라이머는 서열 3'-GGTGCACGCCCTGATGAAGC(서열번호: 8)를 지닌 20-머이다. Expand High Fidelity PCR System(Boehringer Mannheim)과 주형으로서 아라비돕시스 노화 잎 cDNA 라이브러리를 이용한 중합효소 연쇄 반응이 하기와 같이 수행되었다.
반응 구성성분 :
cDNA 1 ㎕(5 ×107 pfu)
dNTP (각각 10 mM) 1 ㎕
MgCl2 (5 mM) + 10x 완충액 5 ㎕
프라이머 1 및 2 (각각 100 ㎕) 2 ㎕
Expand High Fidelity DNA 중합효소 1.75 ㎕
반응 부피 50 ㎕
반응 파라미터 :
94℃ 3분간
94℃/1분, 58℃/1분, 72℃/2분, 45 사이클
72℃ 15분간
(실시예 7)
게놈 DNA의 분리 및 서던 분석
게놈 DNA는 액제 질소 내에서 토마토 잎 조직을 미세한 분말로 갊으로서 토마토 잎으로부터 추출되었다. 60℃에서 예열된 25 ml의 균질화 완충액[100mM 트리스-HCl, pH 8.0, 100 mM EDTA, 250 mM NaCl, 1% 살코실(sarkosyl), 1% 2-머캅토에탄올, 10 ㎍/ml RNase 및 12.5 ml 페놀]을 함유한 37.5ml의 혼합물이 분쇄 조직 내로 첨가되었다. 혼합물은 15분간 진탕되었다. 추가 12.5 ml의 클로로포름/이소아밀알코올(24:1)이 혼합물에 첨가되어 다시 15분간 진탕되었다. 혼합물은 원심분리되고, 수성 상(phase)은 25ml의 페놀/클로로포름/이소아밀알코올(25:24:1)과 클로로포름/이소아밀알코올(24:1)로 재추출되었다. 핵산은 상온에서 15 ml의 이소프로판올 침전시킴으로서 회수되었다. 침전물은 1 ml의 물에 재현탁되었다.
게놈 DNA는 하기와 같이 제한효소 분해되었다. 10 ㎍의 게놈 DNA, 40 ㎕의 10X 반응 완충액 및 100 U 제한효소(Xbal, EcoRI, EcoRV 또는 HinDⅢ)가 400 ㎕의 총 부피로 5∼6시간 동안 반응되었다. 이후 혼합물은 페놀-추출되고, 에탄올-추출되었다. 분해된 DNA는 약 15시간 동안 15 추대로 0.8%의 아가로스 겔 상에서 아가로스 겔 전기영동되었다. 겔은 천천히 교반하면서 30분간 변성 완충액[87.66 g NaCl 및 20 g NaOH/리터] 내에 침지되었고, 증류수로 세척되었고, 천천히 교반하면서 30분간 중화 완충액[87.66 g NaCl 및 60.55 g 트리스-HCl, pH 7.5/리터]내에 침지되었다. DNA는 모세관 블럿팅에 의해 Hybond-N+ 나일론 멤브렌인으로 옮겨졌다.
하이브리디제이션은 1 ×106 cpm/ml의 32P-dCTP-표지된 전체 길이 DHS cDNA 또는 DHS cDNA의 3'-비-코딩 구역을 이용하여 42℃에서 밤새 수행되었다. 프리하이브리디제이션(prehybridization)과 하이브리디제이션은 50%의 포름아마이드, 6X SSC, 5X Denhardt's 용액, 0.1%의 SDS 및 100 mg/ml의 변성된 연어 정자 DNA를 함유한 완충액 내에서 수행되었다. 멤브레인은 2∼4시간 동안 프리하이브리다이즈되었고, 하이브리디제이션은 밤새 수행되었다.
하이브리디제이션이 완성된 후 멤브레인은 상온에서 2X SSC 및 0.1% SDS로 세척된 후 15분간 2X SSC 및 0.1% SDS로, 15분간 0.2X SSC 및 0.1% SDS로 세척되었다. 멤브레인은 -80℃에서 밤새 X-선 필름에 노출되었다. 결과는 도 49에 나타나 있다.
(실시예 8)
아라비돕시스로부터의 노화-유도된 elF-5A 유전자의 분리
아라비돕시스 잎에서 발현된 노화-유도된 elF-5A 유전자의 전체-길이 cDNA 클론은 주형으로서 아라비돕시스 노화 잎 cDNA 라이브러리를 이용한 PCR에 의해 수득되었다. 먼저 유전자의 5'- 및 3'- 말단에 상응하는 PCR 생성물은 벡터 T7 프라이머 <AATACGACTCACTATAG>(서열번호: 18)와 쌍을 이룬 퇴화한 업스트림 프라이머 <AAARRYCGMCCYTGCAAGGT>(서열번호: 17) 및 벡터 T3 프라이머 <ATTAACCCTCACTAAAG>(서열번호: 20)와 쌍을 이룬 퇴화한 다운스트림 프라이머 <TCYTTNCCYTCMKCTAAHCC>(서열번호: 19)를 이용하여 제조되었다. PCR 생성물은 서열분석을 위해 pBluescript 내로 서브클론되었다. 이후 전체-길이 cDNA는 3'-특 이적 프라이머 <AGAAGAAGTATAAAAACCATC>(서열번호: 22)와 쌍을 이룬 5'-특이적 프라이머 <CTGTTACCAAAAAATCTGTACC>(서열번호: 21)를 이용하여 수득되었고, 서열분석을 위해 pBluescript 내로 서브클론되었다.
(실시예 9)
토마토 열매로부터의 노화-유도된 elF-5A 유전자의 분리
토마토 열매에서 발현된 노화-유도된 elF-5A 유전자의 전체-길이 cDNA 클론이 주형으로서 토마토 열매 cDNA 라이브러리르 이용한 PCR에 의해 수득되었다. 먼저 유전자의 5'- 및 3'- 말단에 상응하는 PCR 생성물은 벡터 T7 프라이머 <AATACGACTCACTATAG>(서열번호: 18)와 쌍을 이룬 퇴화한 업스트림 프라이머 <AAARRYCGMCCYTGCAAGGT>(서열번호: 17) 및 벡터 T3 프라이머 <ATTAACCCTCACTAAAG>(서열번호: 20)와 쌍을 이룬 퇴화한 다운스트림 프라이머 <TCYTTNCCYTCMKCTAAHCC>(서열번호: 19)를 이용하여 제조되었다. PCR 생성물은 서열분석을 위해 pBluescript 내로 서브클론되었다. 이후 전체-길이 cDNA는 벡터 T7 프라이머 <AATACGACTCACTATAG>(서열번호: 18)와 쌍을 이룬 5'-특이적 프라이머 <AAAGAATCCTAGAGAGAGAAAGG>(서열번호: 23)를 이용하여 수득되었고, 서열분석을 위해 pBluescript 내로 서브클론되었다.
(실시예 10)
카네이션으로부터의 노화-유도된 elF-5A 유전자의 분리
카네이션 꽃에서 발현된 노화-유도된 elF-5A 유전자의 전체-길이 cDNA 클론은 주형으로서 카네이션 노화 꽃 cDNA 라이브러리를 이용한 PCR에 의해 수득되었다. 먼저 유전자의 5'- 및 3'- 말단에 상응하는 PCR 생성물은 벡터 T7 프라이머(서열번호: 18)와 쌍을 이룬 퇴화한 업스트림 프라이머(서열번호: 17) 및 벡터 T3 프라이머(서열번호: 20)와 쌍을 이룬 퇴화한 다운스트림 프라이머(서열번호: 19)를 이용하여 제조되었다. PCR 생성물은 서열분석을 위해 pBluescript 내로 서브클론되었다. 이후 전체-길이 cDNA는 3'-특이적 프라이머 <ACCAAAACCTGTGTTATAACTCC>(서열번호: 25)와 쌍을 이룬 5'-특이적 프라이머 <TTTTACATCAATCGAAAA>(서열번호: 24)를 이용하여 수득되었고, 서열분석을 위해 pBluescript 내로 서브클론되었다.
(실시예 11)
아라비돕시스로부터의 노화-유도된 DHS 유전자의 분리
아라비돕시스 잎에서 발현된 노화-유도된 DHS 유전자의 전체-길이 cDNA 클론은 아라비돕시스 노화 잎 cDNA 라이브러리를 선별함으로서 수득되었다. 선별을 위해 사용된 프로브의 서열(서열번호: 26)은 도 82에 나타나 있다. 프로브는 주형으로서 노화 잎 cDNA 라이브러리 및 유전자은행의 확인되지 않은 게놈 서 열(AB017060)로부터 고안된 프라이머를 이용한 PCR에 의해 수득되었다. PCR 생성물은 서열분석을 위해 pBluescript 내로 서브클론되었다.
(실시예 12)
카네이션으로부터의 노화-유도된 DHS 유전자의 분리
카네이션 화판에서 발현된 노화-유도된 DHS 유전자의 전체-길이 cDNA 클론은 카네이션 노화 화판 cDNA 라이브러리를 선별함으로서 수득되었다. 선별을 위해 사용된 프로브의 서열(서열번호: 27)은 도 83에 나타나 있다. 프로브는 주형으로서 노화 화판 cDNA 라이브러리 및 퇴화한 프라이머(업스트림: 5' TTG ARG AAG ATY CAT MAA RTG CCT 3'(서열번호: 28); 다운스트림: 5' CCA TCA AAY T CY TGK GCR GTG TT 3'(서열번호: 29))를 이용한 PCR에 의해 수득되었다. PCR 생성물은 서열분석을 위해 pBluescript 내로 서브클론되었다.
(실시예 13)
안티센스 방향의 아라비돕시스 DHS의 전체-길이 또는 3' 구역으로의 아라비돕시스의 형질전환
아그로박테라아가 이중 35S 프로모터의 조절하에서 안티센스 형상으로 발현되는 전체-길이 노화-유도된 아라비돕시스 DHS cDNA 서열 또는 DHS 유전자의 3' 말 단(서열번호: 30)(도 80)을 포함한 이원(binary) 벡터, pKYLX71로 형질전환되었다. 아라비돕시스 식물은 진공 침윤에 의해 형질전환된 아그로박테리아로 형질전환되었고 결과적인 T0 식물로부터의 형질전환 종자가 암피실린에 대해 선택되었다.
도 65∼68은 형질전환된 식물 내에서 안티센스 방향의 그의 DHS 유전자 또는 3' 말단의 발현이 증가된 생물량 즉, 더 큰 잎과 증가된 식물 크기를 초래함을 나타내는 형질전환된 아라비돕시스 식물의 사진이다. 도 69는 형질전환 아라비돕시스 식물이 종자 수확량을 증가시켰음을 나타낸다.
또다른 실험에서 벡터는 아라비돕시스 루비스코(Rubisco) 프로모터(At-rbcS 프로모터)의 조절 하에 안티센스 형태로 아라비돕시스 DHS 유전자(서열번호: 30)의 3'말단을 이용하여 구축되었다. 벡터 컨스트럭트는 플라스미드 pBI101을 사용하였다. GUS 유전자는 제거되었으나 NOS-ter는 잔존하였다. 루스비소(Rusbiso) 수탁반허는 X14564이다. 사용된 프로모터 서열은 뉴클레오타이드 2821∼4780이다. 컨스트럭트를 위해 도 152 참조. 이러한 벡터는 아라비돕시스를 형질전환시키는데 사용되었다. 도 153 및 154는 수득된 형질전환 식물이 야생형 대조군 및 빈 이원 벡터 대조군과 비교시 증가된 생물량을 지님을 나타낸다. 도 155는 형질전환 라인이 종자 수확량의 약 ∼100% 증가를 지님을 나타낸다. 도 156은 형질전환 라인이 증가된 뿌리 발달을 지님을 나타낸다.
(실시예 14)
안티센스 방향의 토마토 DHS의 전체-길이 또는 3' 구역으로의 토마토 식물의 형질전환
아그로박테라아가 이중 35S 프로모터의 조절하에서 안티센스 형상으로 발현되는 전체-길이 노화-유도된 토마토 DHS cDNA 서열 또는 DHS 유전자의 3' 말단(서열번호: 31)(도 81)을 포함한 이원 벡터, pKYLX71로 형질전환되었다. 토마토 잎 외식편은 이들 아그로박테리아로 형질전환되었고, 형질전환된 캘러스 및 묘목이 생성되었고, 표준 조직 배양 방법에 의해 선택되었다. 형질전환된 묘목은 온실 조건하에서 열매-생산 T1 식물을 숙성시키도록 생장되었다.
도 70∼79는 형질전환된 아라비돕시스 식물에서 나타난 바와 같이 형질전환된 식물 내에서 노화-유도된 토마토 DHS 유전자의 감소된 발현이 토마토 열매의 지연된 연화 및 부패뿐만 아니라 증가된 생물량 즉, 더 큰 잎과 증가된 식물 크기를 초래함을 나타내는 사진이다.
(실시예 15)
안티센스 방향의 토마토 DHS의 3' 구역으로의 토마토 식물의 형질전환
아그로박테라아가 이중 35S 프로모터의 조절하에서 안티센스 형상으로 발현되는 DHS 유전자의 3' 말단(도 81)을 포함한 이원 벡터, pKYLX71로 형질전환되었다. 토마토 잎 외식편은 이들 아그로박테리아로 형성되었고, 형질환된 캘러스 및 묘목이 생성되었고, 표준 조직 배양 방법에 의해 선택되었다. 형질전환된 묘목은 온실 조건하에서 열매 생산 T1 식물을 숙성시키도록 생장되었다.
감소된 DHS 발현을 지닌 이들 형질전환 식물로부터의 열매는 대조군 식물의 약 33%의 열매가 발명되는 조건하에서 꽃 말단 부패를 전혀 나타내지 않았다. 꽃 말단 부패는 열매의 기부(꽃) 말단이 노화하거나 부패하는 것을 유발하는 영양분 스트레스에 의한 생리적 질병이다. 도 84(A) 및 84(B)는 꽃 말단 부패를 나타내는 대조군 열매 및 꽃 말단 부패를 나타내지 않는 형질전환 열매를 나타내는 사진이다.
결과는 DHS 발현의 감소가 생리적 질병에 기인한 조직 및 세포의 사멸 착수를 방지함을 나타낸다.
(실시예 16)
야생형 콜롬비아 - 식물 재료 - 내의 아라비돕시스 탈리아나 번역 개시 인자 5A(AteIF-5A) 이소폼의 발현
생태형 콜롬비아(Columbia)인 아라비돕시스 탈리아나의 종자가 6-인치 화분내의 Promix BX 토양(Premier Brands, Brampton, ON, Canada)에서 생장되었다. 신선하기 씨뿌려진 화분은 4℃에서 2일간 유지된 후 16-h 광/8-h 암 사이클로 22℃에서 작동하는 그로쓰 챔버로 옮겨졌다. 150 μmol radiation m-2.s-2에서의 조명이 냉-백 형광 전구에 의해 제공되었다. 전체 로제트가 2주령에서 7주령 사이에 1주일 간격으로 수집되었고 줄기에서 나는 잎은 5주에 수집되었고, 줄기, 장각과, 싹 및 꽃은 6주에 수집되었고 흡수된 종자(물에 24시간)도 수집되어 액체 질소로 순간 동결되어 -80℃에 보관되었다.
슈도모나스 시린게(
Pseudomanas syringae
)로의 아라비돕시스 탈리아나 식물의 감염
아라비돕시스 탈리아나 생태형 콜롬비아 종자가 64 생장 세포를 포함한 평면에서 Promix BX 토양에 뿌려졌다. 씨뿌려진 평면은 4℃에서 2일간 유지되었고 9-h 광/15-h 암의 광주기를 지닌 그로쓰 챔버로 옮겨졌다. 감소된 광주기에 생리적으로 기인하여 발달이 더 느려진 것으로 보이나 모든 식물은 4주령에 처리되었다.
4-주령 식물의 로제트 잎은 Dr. Robin Cameron(토론토대학, 토론토, 캐나다)로부터 획득된 무독성(avr) 및 독성(vir) 균주 슈도모나스 시린게 pv. 토마토 DC 3000으로 감염되었다. 각 식물의 로제트 잎의 배축 표면은 바늘이 없는 1 ml 주사기를 이용하여 접종되었다. 식물은 4가지 처리 중의 하나를 이용하여 처리되었다: 무접종, 10 mM MgCl2로의 모의-접종, avr P. syringae 균주로의 접종(106 cfu/ml 10 mM MgCl2) 또는 vir P. syringae 균주로의 접종(106 cfu/ml 10 mM MgCl2). 충분한 양의 박테리아가 침윤되어 avr 처리에서 전신으로 획득된 저항성을 유도하도록 침투되었는지 확인하기 위해 2개의 박테리아 카운트가 이루어졌고 하나는 접종 직후이고 두 번째는 3일 후이다. 접종된 잎은 이후의 분석을 위해 예비측정된 시점에 채취되었다.
감소된 DHS 또는 손상/병원체-유도된 eIF-5A 발현을 지닌 식물은 유전자에 대한 안티센스 T-DNA 삽입을 이용하여 발달되었다. 이들 식물 라인은 야생형 식물에 비해 박테리아 부하의 99%까지의 감소를 나타내는 형질전환 라인을 지닌 슈도모나스 시린게 pv. 토마토 DC 3000에 대한 두드러진 저항성을 나타내었다. 도 43 및 44 참조. 농작물 식물을 이용한 데이터도 증가된 병원균 저항성을 나타내었다.
지혈기로의 아라비돕시스 탈리아나 식물의 손상
일반적인 조명 조건 하에서 생장된 4-주령 식물이 Stotz et al(2000)에 따라 중앙 엽맥을 따라(잎 표면의 약 10%) 지혈기로 압축시킴으로서 손상되었다. 조직은 0분, 1시간 및 9시간에 채취되었고 액체 질소에서 즉시 동결되었고 추가 분석을 위해 -80℃에서 보관되었다.
RNA 분리 및 노던 불럿팅
노던 블럿 분석을 위한 총 RNA가 Davis et al.(1986)에 따라 아라비돕시스 탈리아나 로제트로부터 분리되었다. RNA는 1% 아가로스 겔 상에 분획되었고 나일론 멤브레인으로 옮겨졌다(Davis et al.,1986). 고정된 RNA는 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A 또는 생장 eIF-5A의 방사성동위원소표지된 3'UTR 부분으로 42℃에서 밤새 하이브리다이즈되었다. 3'UTR은 무작위 프라이머 세트(Boehringer Manheim)를 사용하여 [α-32P]-dCTP로 표지되었다. 하이브리다이즈된 멤브레인은 42℃에서 15분간 0.1% SDS를 함유한 2X SSC로 2회 세척되었고 42℃에서 30분간 0.1% SDS를 함유한 1X SSC로 2회 세척되었다. 하이브리디제이션은 -80℃에서 밤새 노출 후 방사능 사진에 의해 시각화되었다.
항체 생성 및 정제
아라비돕시스 탈리아나(At)의 진핵성 번역 개시 인자 5A(eIF-5A)는 아미노산 수준 특히 단백질의 N-말단 구역 및 중심 구역에서 매우 상동적이다(도 1). 특정 이소폼이 될 항체를 수득하기 위해 펩타이드는 서로 특정한 것으로 나타난 AteIF-5A의 이소폼 내의 구역에 대해 고안된다. 추가적인 시스테인 잔기가 KLH와의 컨쥬게이션을 위해 N-말단에서 각 펩타이드에 첨가된다. 사용된 서열은: 노화-유도된 AteIF-5A의 경우 CNDDTLLQQIKS (서열번호: 35)이고, 손상/병원체-유도된 AteIF-5A의 경우 CTDDGLTAQMRL (서열번호: 36)이고, 생장 AteIF-5A의 경우 CTDEALLTQLKN (서열번호: 37)이다. 이들 서열이 단백질 BLAST(짧은 거의 정확한 서열; 아라비돕시스 탈리아나에 국한; 예측 개수 20000; 문자 크기 2; Matrix PAM90; Gap cost 91)에 제출시 데이터베이스 내에서 발견된 유의적인 서열은 다른 것이 아닌 AteIF-5A에만 일치되었다. 펩타이드는 University of Western Ontario Peptide Synthesis 시설에서 합성되었다. 캐리어 단백질, Keyhole Limpet Hemocyanin(Sigma)이 Drenckhaln et al.(1993) 및 Collawn 및 Patterson(1999)에 따라 m-말레이미도벤조일-N-하이드록시숙신이미드 에스테르를 이용하여 펩타이드의 N-말단 시스테인에 컨쥬게이트되었다. 토끼는 결합된 펩타이드로 2-주 간격으로 4회 주사되었다. 최종 주사 2주 후 혈액이 토끼의 방혈에 의해 수집되었고 항혈청을 모으기 위해 수집된 혈액을 응혈시켰다.
단백질 분획화 및 웨스턴 블럿팅
상기 조직 리스트는 작은 막자를 지닌 에펜도르프 튜브 또는 큰 모르타르 및 막자 내의 완충액(50 mM EPPS, pH 7.4, 0.25 M 솔비톨, 10 mM EDTA, 2 mM EGTA, 1 mM DTT, 10 mM 아미노-n-카프론산, Protease Inhibitor Cocktail for Plant tissues(Sigma)) 내에서 균질화되었다(∼0.5 g/ml). 균질체는 최대 속도로 마이크로원심분리기에서 간단히 원심분리되었고 펠렛이 제거되었다. 총 단백질은 Ghosh et al.(1988)에 따라 정량화되었다. SDS-PAGE가 Mini protein Dual Slab cells(BioRad, Mississauga, Ontario) 상에서 수행되었고 겔(12% 폴리아크릴아미드)은 쿠마시 브릴리언트 블루 R250으로 염색되거나(Fairbanks et al., 1971) 반-건조 이동 방법(semi-dry transfer cell, Bio-Rad, Hercules, CA)을 이용하여 멤브레인으로 옮겨졌다. 블럿은 1 mg/ml 폴리비닐알코올 내에서 30초간(Miranda et al., 1993) 및 0.1%(v/v) Tween 20 및 5%(w/v) 분말화된 우유를 함유한 인산-완충 식염수 내에서 1시간 동안 블록되었다. 1차 항체(두 번째 주사후 방혈로부터의)는 0.1%(v/v) Tween 20 및 1%(w/v) 분말화된 우유를 함유한 PBS 내에서 1:50으로 희석되었다. 항원은 알칼리성 포스파타제(Bioshop, Burlington, Ontario) 및 포스파타제 기질, NBT 및 BCIP(BioRad, Mississauga, ON)에 결합된 토끼 항체에 대한 염소 내에서 제조된 2차 항체를 이용하여 시각화되었다.
(실시예 17)
3가지 eIF-5A 이소폼을 과발현하는 형질전환된 아라비돕시스 탈리아나의 생성
프라이머 디자인
아라비돕시스 탈리아나(At)의 진핵성 번역 개시 인지 5A(eIF-5A) 이소폼은 코딩 구역에서 크게 상동적이다(도 2). 정확한 유전자의 증폭 문제를 방지하기 위해 노화-유도된 eIF-5A, 손상/병원체-유도된 eIF-5A 또는 생장 eIF-5A에 대한 프라이머는 각각 도 3, 4 및 5에 나타난 바와 같이 5'UTR의 시작점 및 3'UTR 말단으로부터 고안되었다. 5'UTR 및 3'UTR은 EST 정보 및 유전자은행 데이터베이스 내의 다른 서열 정보에 기반하여 추정되었다. 적당한 제한 사이트가 pKYLX71 이원 벡터 내에서 센스 방향으로 라이게이션을 위한 프라이머의 말단에 첨가되었다(도 1). 노화-유도된 AteIF-5A의 경우 업스트림 프라이머는 5' AAGCTT GATCGTGGTCAACTTCCTCTGTTACC 3'(서열번호: 38)이고, 다운스트림 프라이머는 5' GAGCT CAGAAGAAGTATAAAAACCATC 3'(서열번호: 39)이다. 손상/병원체-유도된 AteIF-5A의 업스트림 프라이머는 5' CTC GAGTGCTCACTTCTCTCTCTTAGG 3'(서열번호: 40)이고, 다운스트림 프라이머는 5' GAGCTCA AGAATAACATCTCATAAGAAAC 3'(서열번호: 41)이다. 생장 AteIF-5A에 대한 업스트림 프라이머는 5' CTC GAGCTAAACTCCATTCGCTGACTTCGC 3'(서열번호: 42)이고, 다운스트림 프라이머는 5' GAGC TCTAGTAAATATAAGAGTGTCTTGC 3'(서열번호: 43)이다. 프라이머에 첨가된 제한 사이트는 상기 목록된 프라이머 내에 밑줄침으로서 나타난 바와 가팅 노화-손상된 AteIF-5A의 경우 HindⅢ 및 SacI이고, 손상/병원체-유도된 AteIF-5의 경우 XhoI 및 SacI이고, 생장 AteIF-5A의 경우 XhoI 및 SacI이다.
아라비돕시스 탈리아나로부터의 게놈 DNA의 분리
게놈 DNA는 3-주령 로제트 잎으로부터 분리되었다. 조직은 추출 완충액(200 mM Tris pH 7.5, 250 mM NaCl, 25 mM EDTA, 0.5% SDS) 내에서 균질화되었고 수득된 균질체는 15초간 볼텍스되었다. 잔여 파편은 1분간 최대 속도로 마이크로원심분리기로의 원심분리에 의해 제거되었다. 상청액이 수집되었고 이소프로판올과 1:1 비율로 혼합되었고, 볼텍스되었고 2분간 상온에 정치되었다. 펠렛은 5분간 최대 속도로 마이크로원심분리기로의 원심분리에 의해 수집되었고, 70% 에탄올로 세척되었고 2분간 진공 건조되었다. 건조된 펠렛은 물에 재현탁되었고 클로로포름의 1:1 부피로 처리되었고 볼텍스되었다. 2분간 마이크로원심분리기로의 원심분리 후 상층이 수집되었고 침전을 위해 -20℃에서 30분간 20 ㎕(3 M 아세트산나트륨) 및 2 부피의 에탄올로 처리되었다. 이후 정제된 게놈 DNA는 마이크로원심분리기 내에서 30분간 최대 속도로 원심분리되었고, 건조되었고 PCR을 위해 물에 재현탁되었다.
게놈 DNA로부터의 PCR
PCR은 상기 기술된 프라이머로 수행되었다. PCR 반응 혼합물은 1x Tsg 또는 Taq 중합효소 반응 완충액, 1U의 Tsg 또는 Taq 중합효소, 0.2 mM dNTP, 2 mM MgCl2 및 15 pmol의 각 특정 프라이머를 포함하였다. 반응은 95℃에서 10분간 hot start로 시작되었고 첫 번째 사이클은 95℃의 변성 온도 1분, 55℃의 어닐링 온도 2분 및 72℃의 확장 온도 2분으로 구성되었다. 이후 다음의 29 사이클이 어닐링 온도가 사이클 당 0.5℃까지 감소되는 터치다운 프로그램을 진행시켰고, 최종 사이클은 40℃의 어닐링 온도를 지녔다. 72℃의 최종 확장은 10분간 유지되었다. PCR 생성물은 지침에 따라 1% 아가로스 겔 전기영동에 의해 분리되었고 Millipore Ultrafree-DA for DNA Extraction from Agarose spin columns(Millipore Corporation, Bedford, MA)에 의해 수행되고 회수되었다.
pGEM-T Easy 내로의 라이게이션
정제된 PCR 생성물은 Promega에 의해 제공된 지침에 따라 pGEM-T Easy Vector 내로 라이게이트되었다(도 7). 간단히, PCR 생성물은 Promega pGEM-T Easy Vector System(Promega Corporation, Madison WI)에서 제공된 Rapid Ligation Buffer(30 mM Tris-HCl, 10 mM MgCl2, 10 mM DTT, 1 mM ATP 및 5% 폴리에틸렌글리콜(MW8000, ACS Grade) pH 7.8) 내의 pGEM-T Easy Vector, 3 Weiss Units T4 DNA 리가제와 3:1의 비율로 혼합되었다. 라이게이션 반응은 15℃에서 밤새 인큐베이 트되었고 적합한 E. coli DH5-α 세포 현탁액(RbCl/CaCl을 이용하여 적합하게 제조; Kushner, 1978) 내로 옮겨졌다. 형질전환 혼합물은 먼저 30분간 얼음 위에서 인큐베이트되었고, 42℃에서 90초간 열충격이 가해졌고, 1 ml 2xYT 브로쓰의 첨가 후 1시간 동안 37℃에서 회복되었다. 형질전환된 세포는 펠렛되었고, 적은 부피의 2xYT 브로쓰 내에서 재현탁되었고 선택을 위해 50 ㎍/ml 암피실린을 함유한 한천 플레이트 상에 놓였다. pGEM-T Easy Vector가 세포에 암피실린 저항성을 제공하기 때문에 형질전환체만 암피실린 함유 플레이트 상에서 생장할 수 있다. 형질전환체는 pGEM-T Easy Vector 내로 라이게이트된 PCR 생성물 삽입에 대해 선택되고 선별되었다.
제한효소 분해를 통한 pGEM-T Easy Vector 내의 PCR 생성물 삽입의 선별
선택 배지 상에서 생장된 콜로니는 37℃에서 밤새 50 ㎍/ml 암피실린을 함유한 5 ml 2xYT 브로쓰 내에서 생장되었다. 선택된 콜로니로부터의 재조합 플라스미드는 Wizard Prep DNA Purification Kit(Promega)를 이용하여 정제되었다. 플라스미드 DNA는 37℃에서 1시간 동안 EcoRI로 분해되었고 AteIF-5A 삽입 크기가 존재한다는 증명을 위해 1% 아가로스 겔 상에서 시각화되었다. 이후 양성 플라스미드는 본 서열이 식물체 직접(in planta) 과발현에 적당하다는 확인을 위해 Core Molecular Biology Facility(University of Waterloo, Waterloo, ON)에 의해 서열분석되었다.
pKYLX71 내로의 라이게이션
pGEM:손상/병원체-유도된 AteIF-5A 및 pGEM:생장 AteIF-5A의 컨스트럭트가 XhoI 및 SacI로 이중 분해되었고 XhoI 및 SacI로 역시 분해된 이원 벡터 pKYLX71 내로 서브-클론되었다. 이들 효소 분해는 손상/병원체-유도된 AteIF-5A 및 생장 AteIF-5A이 콜리플라워 모자이크 바이러스 이중 35S 프로모터의 조절 하에서 이원 벡터 pKYLX71 내로 센스 방향으로 삽입됨을 확인시켰다. 라이게이션 반응은 1 ㎍의 이원 벡터 및 3 ㎍의 손상/병원체-유도된 AteIF-5A 또는 생장 AteIF-5A를 이용하였다. 라이게이션은 3 Weiss units of T4 DNA Ligase(Fermentas)로 라이게이션 완충액(30 mM Tris-HCl, 10 mM MgCl2, 10 mM DTT, 1 mM ATP 및 5% 폴리에틸렌글리콜(MW8000, ACS Grade) pH 7.8) 내에서 발생되었다. 라이게이션 반응은 15℃에서 밤새 인큐베이트되었고 적합한 E. coli DH5-α 세포 현탁액(RbCl/CaCl을 이용하여 적합하게 제조; Kushner, 1978) 내로 옮겨졌다. 형질전환 혼합물은 먼저 30분간 얼음 위에서 인큐베이트되었고, 42℃에서 90초간 열충격이 가해졌고, 1 ml 2xYT 브로쓰의 첨가 후 1시간 동안 37℃에서 회복되었다. 형질전환된 세포는 펠렛되었고, 적은 부피의 2xYT 브로쓰 내에서 재현탁되었고 선택을 위해 50 ㎍/ml 테트라사이클린을 함유한 한천 플레이트 상에 놓였다. pGEM-T Easy Vector가 세포에 테트라사이클린 저항성을 제공하기 때문에 형질전환체만 테트라사이클린 함유 플레이트 상에서 생장할 수 있다. 형질전환체는 PCR 및 XhoI 및 SacI로의 이중 분해에 의해 손상/병원체-유도된 AteIF-5A 또는 생장 AteIF-5A에 대해 선택되고 선별되었다. PCR 증폭(상기 설명된 게놈 DNA에서 수행된 것과 동일) 및 분해 후 생성물은 정확한 크기의 삽입의 확인을 위해 1% 아가로스 전기영동을 이용하여 분리되었다.
아그로박테리움 일렉트로포레이션(electroporation) 및 선택
pKYLX71:손상/병원체-유도된 AteIF-5A 및 pKYLX71:생장 AteIF-5A 컨스트럭트가 적합한 아그로박테리움 투메파시엔스(Agrobacterium tumefaciens) GV3010 내로 일렉트로포레이트되었다. 적합한 아그로박테리움 세포 단일 콜로니의 제조는 50 ㎍/ml의 리팜피신(rifampicin) 및 50 ㎍/ml 겐타마이신(gentamycin)을 함유한 2xYT 브로쓰 5ml 내에 접종되었다. 이는 280 rpm으로 Forma Scientific Orbital Shaker(Fisher Scientific) 내에서 28℃에서 밤새 생장되었고 다양한 희석(1:500, 1:1000, 1:2000)으로 50 ㎍/ml의 리팜피신 및 50 ㎍/ml의 겐타마이신을 지닌 30 ml의 2xYT 배양액을 접종시키는데 사용되었다. 새롭게 접종된 배양액은 냉각 전까지 OD600이 0.5와 0.8 사이에 있을 때까지 생장되었고 15분간 2000 g에서 SS-34 rotor(Sorvall) 내에서 원심분리되었다. 펠렛은 50 ml의 얼음-냉각수에 재현탁되었고 15분간 2000 g에서 원심분리되었다. 이러한 세척 절차는 배양액으로부터 염 및 사멸된 세포를 제거하기 위해 총 4회 반복되었다. 최종 펠렛은 40 ml 얼음 냉각된 10%(v/v) 글리세롤 내에 재현탁되었고 15분간 2000 g로 원심분리되었고 1회 반복되었다. 이후 펠렛은 100 ㎕ 얼음-냉각된 10% 글리세롤 내에 재현탁되었고 잘 혼합되었다. 세포는 100 ㎕의 일정량으로 분할되었고 얼음 위에 보관되었다.
적합한 아그로박테리움 세포 내로의 DNA 컨스트럭트의 일렉트로포레이션을 위해 100 ㎕ 알리쿼트가 500 ng의 DNA 컨스트럭트와 잘 혼합되었다. 이후 박테리아:벡터 혼합물은 미리-냉각된 일렉트로포레이션 큐벳으로 옮겨졌고 하기 셋팅에 조정된 Gene Pulser(Biorad)에 위치되었다: 2.5 kV, 25 μF 및 200Ω. 일렉트로포레이션 후 1 ml의 2xYT 브로쓰가 첨가되었고 전체 현탁액은 배양액 튜브로 옮겨졌다. 일렉트로포레이트된 배양액은 회복되게 하기 위해 3시간 동안 28℃에서 280 rpm으로 인큐베이트된 후 50 ㎍/ml의 리팜피신 및 50 ㎍/ml 겐타마이신뿐만 아니라 2ml 2xYT 브로쓰가 첨가되었다. 배양액 내에서의 생장 2일 후 일렉트로포레이트된 세포는 테트라사이클린, 겐타마이신 및 리팜피신(모두 50 ㎍/ml) 상에서 플레이트되었고 첨가 2일 후 콜로니가 생장되었다. 수득된 콜로니는 PCR 및 SacI 및 XhoI로의 이중 분해에 의해 pKYLX71:손상/병원체-유도된 AteIF-5A 또는 pKYLX71:생장 AteIF-5A에 대해 선별되었고, 1% 아가로스 겔 상에서의 분리에 의해 시각화되었다.
식물 형질전환
pKYLX71:손상/병원체-유도된 AteIF-5A 또는 pKYLX71:생장 AteIF-5A을 포함한 아그로박테리움 투메파시엔스 GV3010의 양성 콜로니가 야생형 아라비돕시스 탈리아나 생태형 콜롬비아의 형질전환을 위해 사용되었다. 식물 형질전환을 위해 사용된 박테리아 슬러리의 제조시 pKYLX71:손상/병원체-유도된 AteIF-5A 또는 pKYLX71:생장 AteIF-5A 컨스트럭트에 대해 양성인 단일 콜로니가 50 ㎍/ml의 테트라사이클린, 50 ㎍/ml의 리팜피신 및 50 ㎍/ml의 겐타마이신을 함유한 5 ml의 2xYT 브로쓰 내에서 접종되었다. 이는 280 rpm으로 Forma Scientific Orbital Shaker(Fisher Scientific) 내에서 28℃에서 2일간 생장되었고 50 ㎍/ml의 리팜피신 및 50 ㎍/ml의 겐타마이신을 지닌 35 ml의 2xYT를 접종시키는데 사용되었다. 35 ml 배양액은 28℃에서 280 rpm으로 밤새 생장되었고 50 ㎍/ml의 리팜피신 및 50 ㎍/ml의 겐타마이신을 지닌 535 ml(총)의 2xYT를 접종시키는데 사용되었다. 다시 배양액은 약 2의 OD600가 되도록 28℃에서 280 rpm으로 밤새 생장되었다.
배양액은 GSA rotor(Sorvall) 내의 4℃에서 1945 g으로 15분간 원심분리 전에 2개의 250 ml의 튜브로 옮겨졌다. 펠렛은 500 ml의 침투 배지(1.1 g MS 염, 25 g 슈크로즈, 0.25 g MES, pH 5.7 with KOH, 100 ng/ml 벤질아미노퓨린 및 50 ㎕ Vac-In-Stuff(Silwet L-77; Lehle Seeds)) 내에 재현탁되었고 4개의 큰 고무 마개를 지닌 진공 건조기 내의 큰 플라스틱 접시에 놓였다. 각각의 해당 발단 단계의 8개 식물체를 포함한 5개 화분이 침투를 위해 연속적으로 사용되었다. 각각의 화분은 느슨한 토양을 제거하기 위해 먼저 쓰레기통 위에서 전도된 후 유리 건조기 내의 플라스틱 컨테이너 내로 옮겨져서(여전히 전도됨) 4개의 큰 고무 마개가 전도된 화분에 대한 스탠드로서 작용하게 하여 추대가 로제트가 아닌 아그로박테리움 슬러리 내로 떨어지도록 하였다. 이후 식물은 10분간 전도된 상태로 진공처리되었다(400 mmHg). 이후 진공 침투된 식물은 회수되었고 식물 재료 섹션 내에서 설명된 그로쓰 챔버 조건에서와 같이 통상적으로 생장되었다. 몇주 후 장각과가 건조되고 종자가 숙성되면 각 화분이 함께 풀되어 종자가 수집되었다.
식물 형질전환체의 선택 및 분리(segregation) 분석
초기 형질전환체를 확인하기 위해 진공-침투된 식물로부터의 종자가 로테이터(Barnstead/Thermolyne) 상에서 20분간 1%(v/v) 차아염소산나트륨 및 0.1%(v/v) Tween 80 수용액 내에서 표면 멸균되었고 멸균수로 4회 세척되었고 멸균된 0.8% 한천 내에 재현탁되었다. 이후 재현탁된 종자는 1%(w/v) 슈크로즈, 0.5 g/L 2-[N-몰폴리노]에탄술폰산(MES), 0.7%(w/v) 박테리아성 한천 및 40∼50 ㎍/ml 카나마이신로 보충된 멸균된 반-강도 Murashige and Skoog(MS) 배지(2.2 g/L) 상에 이식되었다(Murashige and Skoog, 1962). 이원 벡터가 형질전환체 묘목에 카나마이신 저항성 유전자를 제공하기 때문에 형질전환체만이 카나마이신-함유 플레이트 상에서 생장할 수 있다(도 6). 이원 벡터를 품지 않는 묘목은 카나마이신 저항성 유전자가 없기 때문에 황화되고 사멸하게 된다. 야생형 묘목은 대조군으로 사용되었고 배지에 첨가되는 카나마이신이 없는 MS 배지 상에 이식되었고, 또한 빈 pKYLX71 벡터를 포함한 상동접합성 라인으로부터의 종자가 카나마이신 함유 플레이트 상에서 대조군으로서 씨뿌려졌다. 빈 벡터 대조군은 아라비돕시스 탈리아나의 게놈 내로의 이원 벡터의 무작위 통합의 효과뿐만 아니라 카나마이신 효과가 묘목 생장에 영햐을 미침을 증명하는데 유용하다. 적은 양의 야생형 종자는 MS 배지 및 40∼50 ㎍/ml 카나마이신을 포함한 각 플레이트의 작은 지역 상에 플레이트되었다. 이는 배지가 형질전환체에 충분히 선택적인지를 확인하고 카나마이신의 강도를 시험하기 위해 수행되었다.
씨뿌려진 플레이트는 발아를 동시 발생시키기 위해 3일 간 4℃에서 유지되었다. 3일 후 플레이트는 20±2℃에서 16-h 광/8-h 암 사이클 조건 하에서 추가 7일간 생장되는 그로쓰 챔버로 옮겨졌다. 조명은 150 μmol radiation m-2.s-1에서 유지되었고 냉-백 형광 전구에 의해 제공되었다. pKYLX71:손상/병원체-유도된 AteIF-5A 및 pKYLX71:생장 AteIF-5A 벡터로의 아라비돕시스 탈리아나 식물의 형질전환 효율이 측정되었다.
씨뿌리기 총 10일 후 센스 손상/병원체-유도된 AteIF-5A 및 센스 생장 AteIF-5A 각각에 대한 14개 형질전환체 또는 16개 형질전환체가 32 세포를 포함한 플랫 내에서 Promix BX 토양(Premier Brands, Brampton, ON, Canada)로 이식되었다. 이후 이들 이식된 T1 세대 식물은 16-h 광/8-암 사이클로 22℃에서 작동하는 또다른 그로쓰 챔버 내로 옮겨졌다. 150 μmol radiation m-2.s-1에서의 조명이 냉-백 형광 전구에 의해 제공되었다. T1 세대 식물은 생장하여 성숙되었고 T2 세대 종자를 생성하였다. 이들은 수확되었고 추가 선별이 수행될 때까지 -20℃에서 보관되었다. T1 세대는 1, 2, 3으로 명명되었다. 센스 생장 AteIF-5A 식물의 모든 16개 라인은 생존하였고 종자를 생성하였으나 센스 손상/병원체-유도된 AteIF-5A 식물의 14개 형질전환체 중 단지 9개만이 생존하였고 종자를 생성하였다.
T2 세대 형질전환체의 선택은 T1 세대 형질전환체와 동일한 방법으로 수행되었다. 센스 생장 AteIF-5A 식물의 12개 라인은 선택 가능한 배지 상에서 어떠한 형질전환체도 생성하지 않았고 어떠한 추가 작업에 포함되지 않았다. 센스 생장 AteIF-5A 식물의 1 내지 16개 라인(마이너스 라인 12) 각각은 운반되는 8개 서브라인을 지녔다. 이들은 A 내지 H로 명명되어 예를 들어 T1 라인 1 내에서 T2 세대 식물은 1A, 1B, 1C 등으로 명명되었다. 센스 손상/병원체-유도된 AteIF-5A 식물 라인 1, 2, 3, 4, 5, 7, 9 및 11 각각은 운반되는 8개 서브라인(A-H)를 지녔다. 라인 12 T1 식물은 약 30개 T2 종자만을 생성하였고 T2 세대 내의 단지 1개 서브라인만이 운반될 것이다. 센스 손상/병원체-유도된 AteIF-5A 식물의 T2 식물은 여전히 생장하고 있고 특성화되고 있다. 센스 생장 AteIF-5A 식물에 대한 T2 식물은 숙성하였고 종자를 생성하여 수확되었고 추가 분석까지 -20℃에서 보관되었다.
센스 생장 AteIF-5A의 T3 세대 형질전환체의 선택은 T2와 동일한 방식으로 수행되었다. 8개 라인이 센스 생장 AteIF-5A의 과발현 정도뿐만 아니라 표현형 분석에 기반하여 선택되었다. 발현 수준은 4개 카테고리로 분류되었다: 높은-수준 발현, 중간-수준 발현, 낮은-수준 발현 및 무발현(동시-억제에 기인함). 2개 라인이 각각의 발현 수준에 대해 선택되었고 각 라인으로부터의 12개 식물이 이식되었다. 이들 4개 발현 수준에 대해 상응하는 라인은: 1A, 2D, 4D, 15A, 8D, 9H, 11C 및 16C이다. 센스 생장 AteIF-5A 식물에 대한 T3 세대는 여전히 생장하고 있고 특성화되고 있다.
(실시예 18)
센스 손상/병원체-유도된 AteIF-5A 및 센스 생장 AteIF-5A의 표현형 분석:
사진 기록
센스 손상/병원체-유도된 AteIF-5A 및 센스 생장 AteIF-5A 라인의 형태적 표현형은 상응하는 대조군 야생형 식물(아라비돕시스 탈리아나 생태형 콜롬비아) 및 빈 이원 벡터 pKYLX71로 형질전환 식물의 표현형과 같이 분리동안 사진으로 기록되었다.
종자 측정
센스 생장 AteIF-5A의 T2 식물로부터 수집된 T3 종자는 총 종자 수확량(중량 및 부피), 종자 크기(길이 및 폭) 대해 측정되었고, 생성된 종자의 개체 중량 및 부피를 계산하였다. 중량에 의한 총 종자 수확량은 봉공근 분석 디지털화 저울 상에서 측정되었고 부피는 각 식물에 의해 산출된 총 종자를 100 ㎕ 마다 누진된 유리 1 ml 주사기 내로 쏟아 붓고 채워 넣음으로서 측정되었다. 길이, 폭에 의해 종자 크기를 측정하고 부피를 계산하기 위해 종자는 마이크로미터를 포함한 슬라이드 상에 놓였고 Olympus BX51 현미경 상에서 관찰되었다. 마이크로미터 상의 종자 사진이 Compaq Evo D500(Compaq Company Corporation; Intel Pentium 4 CPU 1.7GHz, 262 MG RAM, running Windows 2000)에 부착된 Spot Insight Color Camera(Diagnostic Instruments Inc.)로 촬영되었다. Image-Pro Express Version 4.0 for Windows를 이용하여 크기 교정을 위한 이미지 내 마이크로미터를 이용하여 각 서브라인 내의 10개 종자의 측정이 이루어졌다. 측정은 Microsoft Excel로 입수되어 표준 오차 및 부피와 같은 계산이 수행되었다.
(실시예 19)
센스 손상/병원체-유도된 AteIF-5A 및 센스 생장 AteIF-5A의 생화학적 분석 - 단백질 분획화 및 웨슨턱 블럿팅
센스 생장 AteIF-5A T2 식물의 각 서브라인으로부터의 첫 번째 줄기에 나는 잎이 수집되었고 단백질이 상기 기술된 바와 같이 추출되었다. 라인 1A, 2A로부 터 16A까지의 총 단백질이 12% SDS-PAGE에 의해 분획화되었고 PVDF 멤브레인으로 옮겨졌다. 블럿은 1:50 희석으로 생장 αAteIF-5A로 프로브되었다. 대조군 총 단백질은 야생형으로부터의 첫 번째 줄기에 난 잎 및 빈 이원 벡터 대조군 식물로부터 추출되었다.
(실시예 20)
야생형 콜롬비아 내의 아라비돕시스 탈리아나 번역 개시 인자 5A(AteIF-5A) 이소폼의 발현
일부 조직이 다른 발달 단계에서 수집되었고 이들 조직으로부터 추출된 단백질은 웨스턴 블럿팅에 사용되었다. 도 8 내의 웨스턴 블럿은 노화-유도된 eIF-5A이 2 주령 로제트 잎 내에 존재하지 않으나 3 주령 로제트 잎 내에서 상향조절되고 5주까지 풍부하게 증가되고 감소되나 7주에도 여전히 존재함을 증명한다. 노화 AteIF-5A는 PEG 처리된 식물 또는 대조군에서 검출되지 않았으나 꽃 레인(노화 꽃 포함) 및 자엽 조직의 노화를 반영하는 흡수된 종자 레인 내에 존재하였다. 블럿이 손상/병원체-유도된 αAteIF-5A 항체로 프로브되면 희미한 밴드가 장각과, 흡수된 종자 및 줄기 레인에서 나타났다. 장각과 및 줄기 레인 내에서 나타난 밴드는 조직의 수집으로 발생된 손상에 기인한다. 장각과 및 줄기를 수집하기 어렵기 때문에 AteIF-5A의 손상/병원체-유도된 이소폼의 일부 상향-조절을 가능하게 하는 직시 순간 동결되지 않았다. 블럿이 생장 αAteIF-5A로 프로브될 때 나타난 밴드만 이 흡수된 종자이고 이것이 세포 분열에 관련된 이소폼이라는 견해를 유지시킨다.
비처리되거나 MgCl2로의 avr P. syringae 또는 vir P. syringae로의 모의 접종으로 처리된 식물은 병원균 침입 동안 AteIF-5A의 발현을 분석하기 위해 여러 시점에서 수집되었다. avr 균주는 식물에 의해 인식 가능하고 감염 구역 내의 세포사멸 및 괴사를 유도하는 고감도 반응을 유도하고, 따라서 질병을 유발하는 병원균을 불허한다. 더욱이 위치화된 반응은 추가 침입으로부터 식물을 보호하기 위해 전신성 반응이 된다. 이는 발병 반응(PR) 유전자로 알려진 한 벌의 유전자 발현을 포함한 전신성 획득 저항성(SAR)로 알려져 있다. 반대로 vir 균주는 식물에 의해 인식되지 않을 것이고 고감도 반응을 유도하지 않을 것이고 질병을 유발하지 않을 것이다. 아라비돕시스 탈리아나의 병에 걸린 상태는 감염 며칠 후 황화 잎 및 세포 사멸을 포함한다. 처리 72시간 후 대조군 식물, 모의 처리된 식물, avr 처리된 식물 및 vir 처리된 식물이 3개의 αAteIF-5A 항체로의 웨스턴 블럿팅을 위해 수집되었다(도 9). 이 시점에서 SAR 및 질병 모두는 avr 처리된 및 vir 처리된 식물 각각에서 관찰할 수 있었다. 노화-유도된 αAteIF-5A 항체로 프로브된 경우 모든 표본에서 매우 동일한 밴드가 관찰되었다. 모든 식물이 4주령이고 때문에 이는 놀라운 것도 아니고, 도 8에서 노화 이소폼은 3주에 나타나기 시작하였다. 블럿이 손상/병원체-유도된 αAteIF-5A 항체로 프로브된 경우 희미한 밴드가 비처리된, 모의 처리된 및 avr 처리된 식물에서 검출 가능하였고 vir 처리된 식물 내에서 검출되는 강한 밴드가 존재하였다. 이러한 손상 이소폼의 상향 조절은 질병에 의해 유발된 세포 사멸에 기인한다(또한 세포적 손상의 형태). 생장 αAteIF-5A로 프로브된 블럿은 어떠한 밴드도 나타내지 않았고 따라서 도면에 포함되지 않았다. 노화-유도된 AteIF-5A가 이들 처리 동안 발현 내 변화를 나타내지 않았기 때문에 자연적 노화에 대한 특이성을 증명한다. 손상/병원체-유도된 AteIF-5A 발현 내의 증가는 손상에 기인한 사멸에 대한 그의 특이성을 증명한다. 이러한 가능성을 더욱 조사하기 위해 아라비돕시스 탈리아나의 손상 잎으로 실험이 수행되었다.
손상 실험은 발명 실험과 동일한 결과를 나타내었다(도 10). 노던 블럿이 노화-유도된 AteIF-5A, 손상/병원체-유도된 AteIF-5A 및 생장 AteIF-5A의 전사적 변화를 나타내는데 사용되었다. 프로브는 각각의 AteIF-5A에 특이적이고 각각 3'UTR로 구성되었다. 이들은 4-주령 식물이고 표본은 9시간 간격으로만 채취되었기 때문에 발병 실험과 같이 노화-유도 AteIF-5A 발현은 어떠한 변화도 나타내지 않음이 관찰되었다. 이는 다시 노화-유도된 AteIF-5A이 자연적 노화 특이적 이소폼이라는 사실과 일치한다. 그러나 손상/병원체-유도된 AteIF-5A의 발현은 9시간 후 증가되지 않았다. 전사체가 완전히 구성적인 것으로 나타날 때(도 10) 발생되는 번역 조절이 있으나 유도되지 않은 경우(도 9) 단백질은 크게 발현되는 것으로 나타나지 않았다. 생장 AteIF-5A의 전사체는 모든 표본에서 거의 검출되지 않았고 손상 후 발현 내 감소를 나타낸다.
(실시예 21)
3가지 eIF-5A 이소폼을 과발현하는 형질전환된 아라비돕시스 탈리아나 식물의 생성
AteIF-5A는 PCR에 의해 게놈 DNA로부터 분리되었다(도 11). 생성물은 pGEM 내에 라이게이트되었고 서열은 인 플란타에서 과-발현에 대한 적합성에 대해 검증되었다. 손상/병원체-유도된 AteIF-5A 및 생장 AteIF-5A는 XhoI 및 SacI로 pGEM으로부터 이중 분해되었고 pKYLX71 내에서 콜리플라워 모자이크 바이러스 35S2 프로모터 뒤에 센스 방향으로 라이게이트되었다. 양성 라이게이션은 분해 및 PCR에 의해 확인되었다(도 13). 이후 pKYLX71:노화-유도된 AteIF-5A 및 pKYLX71:생장 AteIF-5A은 형질전환을 위해 생태형 콜롬비아의 아라비돕시스 탈리아나 야생형의 진공 침투를 통해 아그로박테리움 투메파시엔스 GV3010 내로 일렉트로포레이트되었다. 식물 형질전환후 종자가 수집되었고 카나마이신 함유 MS 플레이트 사에서 형질전환가 선택되었다.
손상/병원체-유도된 AteIF-5A(센스 손상/병원체-유도된 AteIF-5A)를 과발현하는 아라리돕시스 탈리아나 식물
T1 세대 식물은 50 ㎍/ml 카나마이신을 함유한 MS 플레이트 상에서 씨뿌려졌고 4℃에서 3일간 보관되었고 7일간 그로쓰 챔버에서 보관되었다(도 14). 토양으로 이식된 14개 형질전환가 있었다. 이들 14개 T1 세대 식물의 일반적 표현형은 왜소한 생장이었다. 라인 1, 4, 6, 8, 10, 11, 12, 13 및 14는 생장면에서 매우 왜소하였고 6, 8, 10, 13 및 14는 종자를 생성하지 않았다. 라인 2 및 3은 다소 왜소한 반면 라인 5, 7, 9는 야생형 식물과 유사하게 생장하였다(도 15 및 16). 센스 손상/병원체-유도된 AteIF-5A 식물의 T1 세대 내에서 관찰된 일부 또다른 표현형은 황화 잎, 보라색 자엽, 말아 올려진 잎 및 꽃 형태의 차이를 포함하였다. 왜소한 생장의 양상은 식물이 토양으로 이식되기 전까지는 관찰되지 않았다는 점을 주지하는 것이 흥미롭다. 이의 가능한 설명은 이식 동안 뿌리가 다소 손상되고(피할 수 없이 이식의 결과로) 회복될 수 없었다는 것이다. 사실상 이식이 없다면 뿌리 손상이 야기되지 않기 때문에 카나마이신 용액 내에 침지되고 토양에 직접 씨뿌려진 예비 실험에서 왜소한 식물은 발견되지 않았다(반면 식물의 약 70%가 어느 정도 왜소증을 나타냄).
라인 1, 2, 3, 4, 5, 7, 8, 11 및 12는 T2 종자를 생성하였고 견뎌내었다(도 17). 각 T2 라인은 하나의 형질전환체만을 생장시키고 현재 분석 중인 12를 제외하고는 서브라인 A-H를 지닌다.
생장 AteIF-5A(센스 생장 AteIF-5A)을 과발현하는 아라비돕시스 탈리아나 식 물
센스 생장 AteIF-5A의 T1 세대 종자가 선택 배지 상에서 생장되었고 16개 형질전환체가 생장되었다(도 18). 형질전환체는 그의 수명에 대해 사진촬영되었다. 표현형은 야생형과 동일한 것부터(라인 1, 2, 5, 6, 7, 8, 10, 11, 12, 13, 14, 15 및 16) 다소 왜소하고 황화된 것(라인 2, 4 및 9; 도 19)까지 다양하였다. 모든 라인은 T2로 되었고 각 라인은 A-H로 표지된 8개의 서브라인을 지녔다. 라인 12는 T2의 형질전환체를 생성하지 않았고 야생형으로 간주되었다. T2 세대 식물은 T1 세대 식물보다 더욱 더 과장된 표현형을 지녔다. T3까지 진행된 라인은 더욱 상세히 논의될 것이다.
센스 생장 AteIF-5A T2 세대 라인은 생장 AteIF-5A 트랜스유전자의 발현 수준에 따른 그룹으로 특성화되었다. 웨스턴 블럿이 각 라인으로부터의 줄기에서 난 잎으로부터 추출된 단백질에 대해 수행되었다(도 20). 대부분의 서브라인 A-H가 라인 내의 유사한 표현형을 증명하였기 때문에 웨스턴 블럿은 생장 AteIF-5A의 발현 수준의 일반적은 개요를 획득하기 위해 각 라인의 서브라인 A에 대해서만 수행되었다. 야생형 식물 및 빈 이원 벡터를 포함한 식물의 줄기에서 난 잎으로부터의 단백질이 겔 상에서 대조군으로 사용되었다. 이들 서브라인에서 관찰된 발현 수준은 고(라인 1, 2, 3, 10, 13), 중(라인 4, 5, 6, 15) 저(라인 7, 8, 9, 14) 또는 무(라인 11, 16, 야생형 및 이원 대조군)로 분류될 수 있다. 또한 블럿은 노화-유도된 AteIF-5A 및 손상/병원체-유도된 AteIF-5A에 대한 항체로 프로브되었다. 이들 웨스턴은 다른 이소폼 양의 유의적인 양이 검출되지 않았기 때문에 센스 생장 AteIF-5A 라인 내에서의 발현 증가가 다른 AteIF-5A 이소폼의 일반적인 상향 조절이 아닌 생장 AteIF-5A에 기인함을 나타내었다. 또한 이는 이소폼 특이적 항체의 특이성이 수용 가능함을 증명하였다.
T3까지 관철된 센스 생장 AteIF-5A 라인은 생장 AteIF-5A의 발현 수준뿐만 아니라 표현형에 기반하여 선택되었다(각 라인의 표현형 요약에 대한 표 1 참조). 발현 수준의 각 카테고리로부터의 2개 라인이 선택되었다. 관철될 라인은 1A, 2D, 4D, 15A, 8D, 9H, 11C 및 16C이다.
도 20에서 웨스턴 블럿에 따른 라인 1은 생장 AteIF-5A 발현의 높은 수준을 지닌다. 이들 식물은 야생형 식물과 비교시 완전히 원형인 잎을 지닌 크고 암록색의 로제트를 지녔다(도 21). 또한 라인 1으로부터의 로제트는 잎이 모두 동일한 방향으로 감긴 윤생 표현형을 지녔다. 이들 센스 생장 AteIF-5A 식물은 야생형보다 다소 후에 추대되었다. 또한 라인 2는 높은 수준의 생장 AteIF-5A 발현을 증명하였으나 이들 식물이 작고 황화되었다는 점에서 라인 1과 달랐다(도 22). 또한 라인 2 식물은 야생형 및 이원 대조군 식물보다 더 늦게 추대되었고 또한 더 작은 추대(약 반 크기) 및 더 적은 장각과를 생성하였다.
중간 수준의 발현 라인 중에서 라인 4는 야생형 및 이원 대조군 식물보다 바로 며칠 전에 추대됨을 나타내었으나 잎/로제트 크리 및 추대 크기면에서 야생형과 동일함을 나타내었다. 생장 AteIF-5A의 중간 수준 발현을 지닌 두 번째 라인은 라인 15이다. 라인 4와 같이 이들 식물은 야생형과 매우 유사하나 로제트가 확보한 지역이 대조군보다 더 컸다(도 23 및 24). 또한 로제트의 잎은 대조군보다 말단에서 더 둥글게 나타났다. 그러나 추대는 뚜렷한 표현형을 지닌 것으로 나타내지 않았다.
T3까지 진행될 낮은 발현 센스 생장 AteIF-5A 라인은 라인 8 및 9까지이다. 라인 8은 대조군 식물과 비교시 매우 큰 잎 및 큰 로제트를 지녔다(도 25). 또한 잎은 대조군 식물보다 더 넓고 둥글게 보였다. 추대 시간, 추대 크기 및 수는 대조군과 일치하는 것으로 보였다. 센스 생장 AteIF-5A 라인 9는 라인 8와 유사한 잎 형태를 지녔으나 더욱 더 노란색이고 작았다(도 26). 라인 2에서와 같이(높은 발현 라인 중의 하나) 이들 식물은 왜소한 생장, 짧은 추대를 나타내었으나 라인 2와 달리 대조군 식물과 동일하게 추대되었다.
웨스턴 블럿에 따른 센스 생장 AteIF-5A 식물의 2개 라인 11 및 16(도 20)은 생장 AteIF-5A의 상향 조절된 발현을 지니지 않는다. 이는 내생적 유전자뿐만 아니라 트랜스유전자의 동시 억제에 기인한다. 이들 식물이 대조군과 유사하게 보이지 않으나(도 27 및 도 28) 트랜스유전자가 여러 이유로 라인 11 및 16의 게놈 내로 통합된 것으로 여겨진다. 첫 번째로 이들은 카나마이신 함유 MS 플레이트 상에서의 선택성에 의해 증명된 카나마이신 저항성을 지닌다. 두 번째로 라인 16의 로제트 크기, 잎 크기 및 추대 크기(도 28)는 대조군보다 적어도 50% 이상 크다. 그러나 가장 강력한 증거는 그들이 생성한 T 3 종자의 크기 및 조성이다.
T3 종자는 센스 생장 AteIF-5A 식물의 모든 라인으로부터 측정되었다. 사진이 각 라인에서 촬영되었고(도 29에서 가장 큰 것과 가장 작은 것이 하이라이트되어 있음), 측정은 교정을 위해 사용된 사진에서 마이크로미터로 인 실리코(in silico)로 이루어졌다. 각 라인 및 대조군에 있어서 가장 큰 종자의 10개가 측정되었고 교정에 사용되었다. 높은 발현 라인 2는 야생형 및 이원 대조군보다 3배 큰 종자를 지님이 발견되었다. 반면에 가장 낮은 발현을 증명한 라인(라인 11 및 16)은 야생형 및 이원 대조군 종자 크기의 약 88% 정도로 가장 작은 종자를 지녔다. 각 라인의 평균 종자 크기는 nm3으로 표현되었고 거의 타원체인 아라비돕시스 탈리아나로부터의 종자로서 타원체 부피에 대한 방정식을 이용하여 계산되었다. 대조군 종자의 측정 크기는 Boyes et al(2001)에 의해 측정된 발표된 가이드라인과 맞았다. 개체 종자의 측정 크기 및 총 종자 수확량(중량 및 부피 모두)로부터 평균 개체 종자 중량이 계산되었고 플롯되었다(도 31). 대조군 종자와 다른 크기를 증명한 대부분의 라인이 개제 종자 중량에서도 동일한 경향을 지님이 나타났다. 실제로 종자 중량이 종자 크기(부피)에 대해 플롯될 때 상관관계는 R2=0.7412로 거 의 직선이었다. 증가된 밀도(이들 중 3개) 또는 감소된 밀도(이들 중 2개)을 지닌 이상값인 5개 라인이 존재하였다. 증가된 밀도를 지닌 이들 라인의 하나는 8D이고 T3 세대까지 진행될 것이다. 모든 T2 세대 식물로부터의 총 종자 수확량은 몇가지 경향을 지닌 매우 가변적이다. 그러나 하나의 주목할 만한 라인은 중간 발현 센스 생장 AteIF-5A 라인 4D이고, 이는 가장 많은 종자(중량 및 부피 모두)를 생성하였다. 실제로 4D는 대조군 식물보다 2.5배 이상을 생성하였고 T3으로 진행될 것이다.
T3 종자는 앞서 기술된 바와 같이 선택 배지 상에 플레이트되었다. 라인 1A, 2D, 4D, 15A, 8D, 8H, 11C 및 16C가 토양으로 이식되었다. 센스 생장 AteIF-5A의 일부 다른 서브라인은 발아되지 않았을 뿐만 아니라 모든 서브라인 중에서 가장 큰 종자를 지닌 라인 2H도 발아되지 않았다. 라인 11로부터의 식물(동시 억제 라인 중의 하나)은 이렇나 시기에 일반적으로 발견되는 바와 같이 건강하지 않았다. 또한 이들 종자는 가장 작게 측정된 것 중의 하나였다. 이들 라인은 여전히 비-카나마이신 저항성 식물일 뿐만 아니라 종자가 모든 라인으로부터 발아하지 않기 때문에 여전히 분리하고 있는 것으로 보인다. 동일한 방식으로 처리된 대조군 종자 모두 발아되었기 때문에 이는 기술이 아닌 트랜스유전자의 부작용이다.
(실시예 22)
아라비돕시스 노화-유도된 eIF-5A의 특성화
전체 길이 아라비돕시스 노화-유도된 eIF-5A를 수득하기 위한 방법론
아라비돕시스 cDNA 라이브러리로부터의 eIF-5A 유전자를 PCR하기 위해 벡터 프라이머 T3 & T7와 결합하는, 일부 식물 eIF-5A 유전자에 기반한 퇴화 프라이머가 사용되었다. 특히 eIF-5A 유전자의 5' 구역이 T3 프라이머(라이브러리 벡터 내 F5A 유전자의 업스트림에 위치)와 다운스트림(역-방향) 퇴화 프라이머의 중의 하나를 이용한 PCR 반응으로부터 수득되었다. 이와 유사하게 유전자의 3' 구역은 T7 프라이머(라이브러리 벡터 내 eIF-5A 유전자의 다운스트림에 위치)와 업스트림(전방-방향) 퇴화 프라이머 중의 하나를 이용한 PCR 반응으로부터 수득되었다. 전체-길이 eIF-5A 유전자는 유전자의 5' 구역 및 3' 구역의 정렬 분석으로부터 유래되었다.
각 PCR 반응에 대한 2∼3개 주요 생성물이 존재한다. 이들 단편은 pBluescript 플라스미드 내로 클론되었고 시퀀스되었다. 유전자 맵핑 분석 eIF-5A 양성 PCR 단편이 확인되었다. 아라비돕시스에 대해 단 하나의 업스트림 및 다운스트림 양성 eIF-5A PCR 단편이 존재한다.
아라비돕시스 eIF-5A에 대한 특정 5'- 및 3'-말단 프라이머가 5' 및 3' PCR 단편 시퀀싱 결과에 따라 고안되었다. 전체-길이 아라비돕시스 eIF-5A 유전자는 그들의 특정 5'- 및 3'-말단 프라이머 및 주형으로서 상응하는 cDNA 라이브러리를 이용한 PCR 반응으로부터 수득되었다. 전체-길이 유전자는 시퀀싱에 의해 추가 확인되었다. 결국 노화-유도된 eIF-5A로 명명된 하나의 아라비돕시스 eIF-5A 이소폼 유전자를 클론하였다.
T3 및 T7 프라이머:
T3: 5' - ATT AAC CCT CAC TAA AG - 3'(서열번호: 20)
T7: 5' - AAT ACG ACT CAC TAT AG - 3'(서열번호: 18)
아라비돕시스 eIF-5A에 대한 퇴화 프라이머:
전방(업스트림) 프라이머: 5' - AAA RRY CGM CCY TGC AAG GT - 3'(서열번호: 17)
역방(다운스트림) 프라이머: 5' - TCY TTN CCY TCM KCT AAH CC - 3'(서열번호: 19)
pKYLX71 벡터(SAG12 프로모터를 포함한) 내로의 아라비돕시스 안티센스 전체-길이 노화-유도된 eIF-5A의 서브클로닝
아라비돕시스 노화-유도된 eIF-5A에 특이적인(상동적) 프라이머, 안티센스 전체-길이 컨스트럭트: 전방 전체-길이 노화-유도된 eIF-5A 프라이머(30-머): 5'-CCGAGCTCCTGTTACCAAAAAATCTGTACC-3'(서열번호: 48)(주지: 밑줄친 부분은 pBluescript의 멀티플 클로닝 사이트(MCS) 내의 SacI 사이트 내로 PCR 단편의 5'-말단을 라이게이트하는데 사용된 SacI 인식 서열). 역방 전체-길이 노화-유도된 eIF-5A 프라이머(36-머): 5'-ACCTCGAGCGGCCGCAGAAGAAGTATAAAAACCATC-3'(서열번호: 49)(주지: 밑줄친 부분은 pBluescript의 MCS 내로의 라이게이션에 사용된 NotI 인식 서열).
pBluescript 벡터의 MCS 내 SacI및 NotI의 방향은 유전자가 이의 안티센스 방향으로 서브클론되게 하는 방향이다(즉, NotI 사이트는 SacI 사이트의 업스트림임).
(실시예 23)
SAG 12 프로모터가 안티센스 노화-유도된 아라비돕시스 전체-길이 eIF-5A를 발현하는데 사용됨
실험 증거는 "노화-관련 유전자" 또는 SAG 세트의 전사가 노화의 착수 동안 증가됨을 나타낸다(Lohman et al., 1994; Weaver et al., 1998). 실제로, 노화는 새로운 mRNA의 합성 및 아마도 다른 mRNA의 하향-조절과 함께 시작하는 것으로 보이고, 이는 단백질의 선택적 합성은 노화에 필수적임을 나타낸다(Nooden, 1988). 잎 노화 문제가 유전자 발현 내의 변화에 의해 달성된다는 것은 번역 가능한 mRNA 집단 내에서 발생하는 변화를 검출하기 위해 시험관 내 번역 후 겔 전기영동을 이 용한 Watanabe and Imaseki(1982)에 의해 먼저 증명되었다. 이러한 초기 연구 및 시험관 내에서 번역된 단백질의 이후의 분석은 대부분의 많은 mRNA가 노화의 진행 동안 유의적으로 감소하고 다른 번역 가능한 mRNA는 증가함을 나타내었다(Watanabe and Imaseki, 1982; Davies and Grierson, 1989; Becker and Apel, 1993; Buchanan-Wollaston, 1994; Smart et al., 1995). 또한 노화 잎 조직의 mRNA로부터 제조된 cDNA 라이브러리의 차별적 선별은 노화 동안 많은 유전자 발현이 하향-조절된 반면 다른 유전자의 발현이 상향-조절됨이 증명하였다. SAG는 아라비돕시스(Hensel et al., 1993; Taylor et al., 1993; Lohman et al., 1994; Oh et al, 1996), 아스파라거스(King et al., 1995), 보리(Becker and Apel, 1993), 브라시카 나푸스(Brassica napus)(Buchanana-Wollaston, 1994), 옥수수(Smart et al., 1995), 무(Azumi and Watanabe, 1991) 및 토마토(Davies and Grierson, 1989; Drake et al., 1996)을 포함한 다양한 식물종으로부터 확인되었다.
노화는 잎의 말단에서 시작하여 마지막으로 엽맥까지 도달하는 특성적으로 패턴화된 잎 황화로서 형태학적으로 확인될 수 있다(Weaver et al., 1998). 아라비돕시스 탈리아나 로제트 잎에서 관찰 가능한 노화는 발아 약 21일 후 그때에 SAG 12의 극적인 상향 조절로 나타난다. 어린 잎에서 검출 가능한 발현이 없는 경우 SAG 12는 ∼20% 황화된 후 노후한 잎에서 유도되나 황화 잎을 유도하지 않는 처리에 의해 유도될 수 없다(Weaver et al., 1998). 자연적 노화에 대한 높은 정도의 특이성은 SAG 12의 유전자 생성물이 시스테인 프로테아제와의 유사성을 나타낸다는 사실에 의해 설명될 수 있고 이는 노화 동안 단백질 전도에 관련된다(Lohman et al., 1994; Weaver et al., 1998).
형질전환 식물의 설명
형질전환 아라비돕시스 식물은 발아 약 21일 후 스트레스-유도된 노화의 이벤트가 아닌 자연적 잎 노화의 착수시 활성화되는(Noh and Amasino, 1994) SAG 12(잎 노화-특이적) 프로모터의 조절 하에서 전체-길이 안티센스 노화-유도된 eIF-5A 트랜스유전자를 발현하여 생성되었다. 이 시점에서 형질전환 식물은 억제된 전체-길이 노화-유도된 eIF-5A 발현의 표현형 특성을 발현한다. 로제트 잎은 3∼8주령 형질전환 아라비돕시스 안티센스 전체-길이 노화-유도된 eIF-5A 식물로부터 수확되었다.
SAG 12 프로모터의 조절 하의 상동적 형질전환 안티센스 노화-유도된 eIF-5A 아라비돕시스 탈리아나 식물의 생성을 위한 방법론
pKYLX71 내로 SAG 12-안티센스-전체-길이 노화-유도된 eIF-5A 컨스트럭트의 삽입
먼저, 이중 35S 프로모터를 제거하기 위해 플라스미드 pKYLX71이 EcoRI 및 HindⅢ로 절단되었고, 수득된 점착성 말단은 Klenow 효소로 채워져서 무딘(blunt) 말단을 생성하였다. 이후 프로모터가 없는 pKYLX71는 라이게이트되어 플라스미드를 재순환시켰다.
두 번째로 아라비돕시스 SAG 12 프로모터는 하기 기술된 바와 같이 SalI 및 XbaI를 포함한 프라이머를 이용하여 PCR에 의해 게놈 DNA로부터 증폭되었다. 이후 이러한 프로모터 서열은 제한 효소 SalI 및 XbaI를 이용하여 pBluescript의 멀티플 클로닝 사이트(MCS) 내로 삽입된 후 T4 DNA 리가제로 라이게이션되었다.
전방 SAG 12 프라이머는 5'-GGC CGTCGACGATATCTCTTTTTATATTCAAAC-3'(서열번호: 50)(밑줄친 부분은 pBluescript의 멀티플 클로닝 사이트(MCS) 내의 SalI 내로 PCR 단편의 5'-말단을 라이게이트하는데 사용된 SalI 인식 사이트). 역방 SAG 12 프랄이머는 5'-CGTCTAGACATTGTTTTAGGAAAGTTAAATGA-3'(서열번호: 51)(밑줄친 부분은 pBluescript의 멀티플 클로닝 사이트(MCS) 내의 SacI 내로 PCR 단편의 5'-말단을 라이게이트하는데 사용된 XbaI 인식 사이트).
세 번째로 pBlueScript-SAG 12:안티센스-전체 길이-노화-유도된 eIF-5A 컨스트럭트를 생성하기 위해 전체 길이 노화-유도된 eIF-5A가 하기에 개요된 바와 같이 SacI 및 NotI 제한 사이트를 지닌 프라이머를 이용하여 아라비돕시스 cDNA 라이브러리로부터 PCR에 의해 증폭되었고 앞선 단락에 기술된 pBluscript-SAG 12 내로 서브클론되었다. MCS 내의 SacI 및 NotI의 방향은 유전자가 그의 안티센스 방향으 로 서브클론되는 방향임(즉, NotI 사이트가 SacI 사이트의 업스트림)이 주지되어야 한다.
전방 전체-길이 노화-유도된 eIF-5A 프라이머는 5'-CCGAGCTCCTGTTACCAAAAAATCTGTACC-3'(서열번호: 48)(주지: 밑줄친 부분은 pBluescript-SAG 12 벡터의 멀티플 클로닝 사이트(MCS) 내의 SacI 내로 PCR 단편의 5'-말단을 라이게이트하는데 사용된 SacI 인식 사이트). 역방 전체-길이 노화-유도된 eIF-5A 프라이머는 5'-ACCTCGAGCGGCCGCAGAAGAAGTATAAAAACCATC-3'(서열번호: 49)(주지: 밑줄친 부분은 pBluescript-SAG 12 벡터의 멀티플 클로닝 사이트(MCS) 내로의 라이게이션에 사용된 NotI 인식 사이트).
마지막으로 원하는 컨스트럭트가 SacI 및 XhoI로 pKYLX71을 분해하고 SalI 및 SacI를 지닌 pBluescript로부터 SAG 12: 전체-길이 노화-유도된 eIF-5A 카세트를 절단함으로서 이원 벡터 pKYLX71을 생성하였다.
XhoI 및 SalI 점착성 말단은 부분적으로 상보적이다. 따라서 분해된 돌출의 이들 2 세트(특히, SacI를 지닌 SacI 및 SalI를 지닌 XhoI)는 T4 DNA 리가제와 함께 라이게이트되어 최종 컨스트럭트를 생성할 수 있었다(pKYLX71 내의 SAG 12:안티센스 노화-유도된 eIF-5A).
형질전환 및 T1 종자 수확
pKYLX71-SAG 12:안티센스-eIF-5A 컨스트럭트는 E. coli DHα 세포 내에서 정제되었고, 분리되었고 적합한 아그로박테리움 균주 내로 일렉트로포레이트되었다. 이후 박테리아는 4.5 주령 야생형 아라비돕시스 식물을 침투하였고 수득된 침투된 식물은 "T0" 식물로 명명되었으며, 이후 이는 그이 수명까지 생장되었다. 종자가 수확되었고 수집되었고 T1 종자로 명명되었다. T1 종자의 10개 플레이트가 대조군으로서 야생형과 함께 카나마이신 저항성에 대해 선별되었고(1/2 MS 염 및 50 ㎍ 카나마이신/ml); pKYLX71-SAG 12-안티센스-eIF-5A 컨스트럭트를 포함한 종자만이 카나마이신(K50) 배지 상에서 생존하고 생장하였다. 24개 T1 묘목이 이들 플레이트로부터 선택되어 토양으로 옮겨졌다. T1 형질전환 식물로부터 수확된 종자는 T2 종자로 표지되었다. 각 묘목은 하나의 식물 라인을 산출하였다(#1= 1 식물을 포함한 1 라인, #2= 1 식물을 포함한 1 라인 등).
표현형의 선별 및 확인
카나마이신 저항성 T1 종자가 확인되면 T2, T3 및 T4 식물의 성공적인 세대가 생장되었다. K50 배지 상에서 종자를 선별함으로서 유전적 컨스트럭트를 유전받 고 컨스트럭트에 상동접합성인 식물을 구별하는 것이 가능하였다. 왜소한 생장의 표현형적 발현은 토양에서 생장시 하나의 T3 식물 라인에서 관찰되었다. 그러나 동일한 세트의 종자가 동일한 조건에서 재-생장되었고 표현형은 관찰되지 않았다.
24개 T1 식물로부터 4개 라인이 높은 종자 수확량에 기초하여 선택되었고(라인 T2.14, T2.18, T2.19 및 T2.23) 대조군으로서 야생형 종자와 함께 K50 배지 상에 플레이트되었다. 각 라인으로부터의 약 75% 종자가 K50 배지 상에서 생존하였고 소, 중, 대의 크기 카테고리에 포함되었다. 각 라인으로부터 소, 중, 대 묘목이 플레이트로부터 제거되었고 토양에 심어졌다. 온실 조건 하에서 소 묘목은 중 및 대 대응물만큼 빨리 회복되지 않았다. 6주에 소 식물은 추대의 사인을 나타내지 시작한 반면 다른 식물은 추대되고 개화되었다. 전체적으로 6개 형질전환 T2 식물(총 3 라인 X 8 식물 = 96 형질전환 식물)이 추대 내에서의 극적인 지연을 증명하였고 "늦은 추대" 식물로 간주되었다. 이들 식물의 종자 수확량은 다른 형질전환체보다 극적으로 낮았다.
96개 T2 식물로부터 3개 라인이 T3 식물을 생성하는데 선택되었다(늦은 추대인 T3.19.S8 및 T3.14.L7; 및 늦은 추대가 아닌 T3.23.S3). K50 배지 상에 놓였을 때 이들 라인은 상동접합성 생존을 나타내었다. 13개 묘목이 토양으로 이식되었다(화분 당 10개 묘목). 이들 식물 세트로부터 극적인 왜소증 표현형이 T3.14.L7 식물 라인에서 관찰되었다. T4 종자가 수집되었고 낮은 종자 수확량이 본 라인엔서 관찰되었다. 조밀한 생장(조밀한 장각과 생장, 많은 가지) 표현형이 T3.19.S8에서 관찰된 반면 야생형과 유사한 표현형은 T3.23.S3에서 관찰되었다. 3개 형질전환 라인으로부터의 종자 크기가 비교되었으나 통계적으로 유의적인 차이는 측정되지 않았다. 또한 엽록소 수준이 분석되었으나 야생형 대조군과 통계적으로 유의적인 차이가 측정되지 않았다.
다음 세대 식물을 수득하기 위해 라인 T3.19.S8, T3.14.L7 및 T3.23.S3의 T4 종자가 선별되었고 사멸된 야생형-종자와 비교시 유전된 유전자 컨스트럭트의 증거를 나타내었다. 그러나 개별적인 평면 내에 심어졌을 때 왜소증 표현형은 표현되지 않았고 이는 eIF-5A 안티센스 트랜스유전자가 유실되었음을 나타내었다. 마지막으로 모든 T5 식물로부터 수집된 종자는 K50 플레이트 상에서 선별되었고 카나마이신 저항성의 증거를 나타내었다. 안티센스 트랜스유전자가 유실되었는지를 확인하기 위해 연구가 진행중이고, 이들 T4 식물은 쌍을 이루지 않는다.
8개 딸 라인이 모계 T2.14, T2.19 및 T2.23으로부터 선택되었고 대조군으로서 야생형 종자와 함께 K50 배지 상에서 선별되었다. 낮은 종자 수확량에 기반하여 3개 라인이 선택되었다: T3.14.L8, T3.14.S8 및 T3.23.S1. 선택된 다른 5개 라인은: T3.18.S7, T3.18.S2, T3.19.S1, T3.19.S5 및 T3.23.S6이다. K50 배지 상 에서 선별된 모든 라인은 상동접합성 생존을 나타낸 반면 T3.14.L8, T3.14.S8 및 T3.23.S6은 이형접합성 생존을 나타내었다. 생존한 라인 T3.14.L8 및 T3.14.S8로부터의 묘목은 녹색 도관 조직을 지닌 백색인 반면 생존한 T3.23.S6로부터의 묘목은 전체적으로 암록색이었다. 이들 묘목은 이식을 위해 선택되었다. 전체적으로 각 라인으로부터의 28개 묘목이 세포 내로 이식되었고 온실 조건으로 생장되었다.
T3.14.L8 및 T3.23.S1 및 T3.18.S7, T3.18.S2, T3.19.S1, T3.19.S5, T3.23.S1 및 T3.23.S6 내의 일부 식물을 제외하고 3주에 모든 라인이 추대하기 시작하였다. 불규칙적인 로제트 잎 형태(두 번째 쌍 잎 표현형의 신장)가 T3.14.L8 및 T3.14.S8 라인에서 관찰되었다. 5주에 증가된 수의 로제트 잎의 추가적인 불규칙적 잎 형태 및 오그라든-말단 로제트 잎 표현형도 T3.18.S7 및 T3.23.S6에서 관찰되었다. 야생형보다 작은 로제트가 라인 T3.23.S1, T3.19.S1 및 T3.19.S5에서 관찰되었다. 7주에 가늘고 긴 줄기 및 줄기 신장이 없는 표현형이 라인 T3.18.S7, T3.18.S2, T3.19.S1, T3.119.S5, T3.23.S1 및 T3.23.S6에서 관찰되었다. 각 식물의 첫 번째 및 두 번째 줄기에서 난 잎이 노화 eIF-5A 단백질 발현의 조사를 위해 각각 5주 및 6주에 수집되었다.
(실시예 24)
산소 방출의 측정
잎이 수확되었고 무게 측정 전에 면적이 측정되었다. 잎은 막자사발과 막자로 1 ml의 차가운 가스 제거된 분쇄 완충액을 이용하여 미세 분말로 분쇄되었다. 이후 균질체는 에펜도르프 튜브로 옮겨졌고 얼음에 즉시 놓였다. 토마토 잎의 경우 분리된 균질체는 Miracloth 조각을 통해 여과될 필요가 있었다.
모든 표본으로부터의 50 ㎕의 균질체가 5 ml의 분쇄 완충액 및 25 ㎕ DCPIP(2,6-디클로로페놀 인도페놀)을 포함한 10 ml 시험관에 첨가되었다. 표본은 잘 진탕되었고 이후 표본 세트 하나는 램프 쌍에 의한 조명 하에 15분간 놓였고 두 번째 표본 세트는 15분간 암조건에 놓였다. 반응을 정지시키기 위해 15분 인큐베이션 후 50 ㎕의 DCMU(3-(3,4-디클로로페닐)-1,1 디메틸우레아)가 두 표본 세트에 첨가된 후 14,000 g로 2분간 마이크로원심분리기에 의해 원심분리되었다. 수집된 상청액의 흡광도는 블랭크로서 분쇄 완충액을 이용하여 590 nm에서 판독되었다.
이러한 분석의 몰 흡광 계수는 16 x 103이고, 즉, 리터 당 1 몰 농도의 변화가 DCPIP 감소된/h/ml의 16 x 103 μmol = (흡광도 차이) x [1/16 x 103(mole/l)] x [반응 부피(ml)/ 103(ml/l)] x [106(μmole/mole)] x [60(분/시)/반응시간(분)] x [1/표본 부피(ml)]에 의해 용액의 흡광도를 변화시킨다.
감소된 DCPIP의 모든 2 몰에 대해 1 몰의 O2가 생성된다. 참조: Allen J.F. and Holmes N.G., 1986 Electron Transport and Redox Titrations in Photosynthesis: Energy Transduction. Edited by M.F. Hipkins & N.R. Baker., IRL Press, Oxford pp 107-108.
(실시예 25)
전분의 정량적 측정
토마토 줄기 내의 전분 함량이 Lustinec et al. Quantitative determination of starch, amylose, and amylopectin in plant tissues using glass fiber pepper. Anal. Biochem. 132:265-271(1983).로부터 채택된 방법을 이용하여 측정되었다. 토마토 줄기 조직은 Omnimixer(각각 5초의 12 반복)를 이용 하여 3 부피의 물에 균질화된 후 Polystron 균질기(30초)로 균질화되었다. 균질체는 분석전에 -20℃에서 10 ml 알리쿼트로 보관되었다. 분석을 위해 10 ml 균질체가 해동되었고 동일 부피의 농축된 과염소산(HClO4, 70% w/w)으로 혼합되었고 20분간 상온에서 인큐베이트되어 전분을 용해하였다. 동시에 감자 전분의 일부 용액(0.1∼1.0 mg/ml 범위)은 표준 곡선을 생성하기 위해 토마토 줄기 표본과 나란히 처리되었다. 균질체(또는 감자 전분 표분 용액)가 교반되었고 흡입기에 부착된 진공 플라스크를 이용하여 Whatman GF/A 유리 마이크로필터 종이(9.0 cm 지름)를 통해 여과되었다. 여과물 1 ml가 3 ml의 요오드 용액 A(8 mM I2, 17 mM KI, 514 mM NaCl)과 혼합되었고 4℃에서 30분간 인큐베이트되어 전분-요오드 침전물을 형성하였다. 침전물은 흡입기에 부착된 진공 플라스크를 이용하여 Whatman GF/A 유리 마이크로필터 종이(9.0 cm 지름) 상에 수집된 후 하기 용액으로 여과물을 세척하였다: 10 ml 요오드 용액 B(83 mM I2, 180 mM KI, 8% 과염소산[HClO4])로 1회; 5 ml 에탄올-NaCl 용액(67% 에탄올, 342 mM NaCl)로 1회; 3 ml 에탄올-NaOH 용액(67% 에탄올, 250 mM NaOH)으로 2회. 에탄올이 증발되면 마이크로필터 종이가 흡입기로부터 제거되었고 나사-마개 유리 튜브로 삽입되었다. 황산[H2SO4](9 ml의 0.75 M 용액)이 튜브에 첨가되었고 튜브는 30분간 끓는 물에서 인큐베이트되었다. 용출액 1 ml-알리쿼트 3개가 유리 시험관 내로 피펫팅되었고 1 ml의 5% 페놀과 혼합되었고 5 ml의 농축 H2SO4과 빨리 혼합되었다. 튜브는 볼텍스되었고 30분간 상온에서 인큐베이트되어 발색되었다. 동시에 분광광도계 측정을 위한 블랭크가 1 m의 0.75 M H2SO4를 1 ml의 5% 페놀과 혼합시키고 5 ml의 농축 H2SO4을 빨리 첨가시킴으로서 제조되었고; 블랭크도 상온에서 30분간 인큐베이트되었다. 분광광도계는 블랭크를 이용하여 480 nm에서 보정되었고, 모든 표본 및 감자 전분 표준의 O.D가 측정되고 기록되었다. 표준 곡선은 감자 전분 용액을 이용하여 준비되었고 각 표본 내 전분 양을 삽입(interpolate)시키는데 이용되었다.
(실시예 26)
아라비돕시스 탈리아나(콜롬비아 생태형)는 아라비돕시스 탈리아나 센스 노화-유도된 eIF-5A(At-eIF-5A) 및 토마토 센스 노화-유도된 eIF-5A 유전자에 의해 독립적으로 형질전환되었다. 이들 유전자는 형질전환 식물의 생활환에서 구성적으로 발현되었다. 이들 식물의 화서 줄기는 물관부 발달의 유의적인 증가를 나타내었다. 도 89∼94 참조.
형질전환 및 대조군 식물의 종자는 1/2 MS 배지 한천 플레이트 상에 씨뿌려졌고 9일간 22℃, 80% rh 및 16 h 광/일로 그로쓰 챔버에서 유지되었다. 이후 묘목이 통상의 토양을 지닌 32-웰 플랫으로 옮겨졌고 48일간 상기와 동일한 조건으로 유지되었다. 주요 화서 줄기가 현미경 관찰을 위해 선택되었다. 횡단면은 로제트로부터 2 mm 위의 줄기 기부로부터 손으로 절단되었다. 섹션은 플로로글루시놀(phloroglucinol)-HCl 방법으로 염색되었다. 이러한 연령에서 줄기 물관부가 최대 발달을 달성하였음이 발견하였다. 물관부 크기면에서의 형질전환과 대조군 식물 사이의 비교가 이루어졌다. 더욱이 형질전환 및 대조군 식물 모두에서 체관부 및 속에 대한 측정이 수행되었다.
조직 면적에 대한 측정은 하기와 같았다. 횡단면은 Zeiss 현미경으로 사진촬영되었고 현미경 사진은 Photoshop을 이용하여 디지털화되었다. 이들 이미지는 종이로 인쇄되었고 다른 조직이 절단되었고 그의 면적이 면적-측정기에 의해 측정되었다. 각 조직의 실제 면적을 계산하기 위해 하기 식이 이용되었다: 실제 면적 = (종이 상의 개체 조직의 면적)/(배율)2
따라서 노화-유도된 eIF-5A도 물관생성과 관련된 프로그램된 세포 사멸에 관련됨을 나타낸다. 아라비돕시스 내의 노화-유도된 AteIF-5A 구성적 안티센스 억제는 물관부 세포층의 수뿐만 아니라 화서 줄기의 두께를 감소시켰다. 반대로 아라비돕시스 또는 토마토 노화-유도된 eIF-5A가 구성적으로 과발현된 식물의 화서 줄기는 상응하는 야생형 식물보다 평균 1.7-배 더 두꺼웠고 화서 줄기 횡단면 당 총 물관부 면적은 2배 이상이었다. 과발현 형질전환 식물은 매우 증가된 로제트 잎 생물량을 지녔고 야생형 식물보다 더 빨리 생장되었고, 이는 증가된 영양분 흡수를 반영한다. 동일한 표현형은 토마토로부터의 노화-유도된 eIF-5A 이소폼이 아라비돕시스 식물 내에서 과발현될 때 관찰되었다. 이들 결과는 노화-유도된 eIF-5A 이소폼이 잎 및 꽃 노화를 조절할 뿐만 아니라 물관생성에도 관련됨을 종합적으로 나타낸다.
(실시예 27)
디옥시하이푸신 신타제의 억제는 대기 내에서 미리-포장된 절단 상추의 갈변을 지연시킴
상추는 글루코코르티코이드-유도성 프로모터에 의한 트랜스유전자(안티센스 DHS)의 제어되고 지속적인 유도를 획득하는데 이상적인 수경재배로 생장되었다. 유도제인 덱사메타손이 수경재배 용액에 직접 적용되었다.
도 149(a)는 채취 직전 6주령 식물을 나타낸다. 도 149(b)는 화분 내 에어 스톤(air stone)을 나타낸다. 도 149(c)는 식물을 지지하는 3개의 오프닝을 지닌 덮개 및 센터 내 에어라인을 나타낸다. 도 150은 대기 내 절단 상추의 갈변을 나타낸다.
도 150(a)는 3개의 형질전환 라인의 시간 인자에 따라 구분된 갈변 착수를 나타낸다: 라인 13(n=4), 라인 21 (n=5) 및 라인 34 (n=7), 안티센스 방향의 상추 DHS의 3'UTR뿐만 아니라 야생형 로메인 상추(Green Towers 품종; n=12)를 발현. 트랜스유전자는 글루코코르티코이드-유도성 프로모터의 조절 하에서 발현되었다. 상추 두부는 수경재배로 생장되고 채취되고 작은 분절로 절단되었다. 절단된 상추는 구멍이 난 지퍼백 내에 6일간 7.5℃로 유지되었다. 절단 상추의 갈변은 평가 1이 갈변의 착수로 정의된 0∼4의 측정척도로 평가되었다. 표준 오차 막대가 나타나 있다. 도 150(b)는 6일간 연구 동안 0% 갈변을 나타낸 라인 21의 2개의 식물 개체의 결과를 나타낸다. 도 150(c)는 야생형 상추가 갈변된 동일한 기간 동안 갈변되지 않은 라인 34의 식물 개체 중 하나의 결과를 나타낸다.
도 151은 DHS 단백질의 감소된 발현을 나타내는 형질전환 상추의 웨스턴 블럿을 나타낸다. 상추 DHS의 안티센스 3'UTR을 발현하는 4개의 상추 식물 개체뿐반 아니라 유전자가 T2 세대로 분리시 트랜스유전자를 소실한 식물("무효") 및 야생형 식물 유래의 총 단백질이 분리되었고 각 표본의 20 ㎕가 쿠마시 블루로 염색된 14% 아크릴아미드 겔 상에 로드되었다. 겔은 PVDF 멤브레인 상에서 블럿되었고 토마토 DHS에 대해 유발된 폴리클론 항체로 프로브되었다. 블럿은 색채적으로 발달되었고 형질전환 식물 내 DHS의 발현은 무효 및 야생형 식물 대비 감소되었다.
(실시예 28)
카놀라 내의 디옥시하이푸신 신타제 발현의 억제는 종자 수확량을 증가시킴
디옥시하이푸신 신타제(DHS)는 불활성 진핵성 번역 개시 인자-5A(eIF-5A)를 번역을 촉진시킬 수 있는 활성 형태로 전환시키는 2개 효소 반응의 첫 번째를 매개한다. 카놀라(Brassica napus cv Westar) DHS를 인코드하는 DHS 전체-길이 cDNA 클론이 노화 잎으로부터 제조된 cDNA 발현 라이브러리로부터 분리되었다. DHS는 구성적 콜리플라워 모자이크 바이러스(CaMV-35S) 프로모터의 조절 하에서 카놀라 DHS cDNA의 안티센스 3'-UTR를 발현함으로서 형질전환 카놀라 식물 내에서 억제되었다. DHS 단백질의 감소된 발현을 나타내는 도 158 참조. 형질전환 카놀라 식물은 아라비돕시스에 대해 개발된 프로토콜을 변형함으로서 카놀라 화서의 진공 침 윤에 의해 수득되었다. 형질전환 효율은 식물이 진공-침윤된 후 형성된 새로운 꽃을 제거함으로서 증진되었고, 50∼60%의 형질전환률이 일반적으로 수득되었다. 형질전환 식물은 잎 DHS 단백질의 수치를 감소시켰고 지연된 천연 잎 노화를 나타내었다. 또한 DHS의 억제는 1.5∼2배까지 로제트 잎 크기를 증가시켰고(도 157 참조), 65%까지 종자 수확량을 증가시켰다(도 160 참조). 이는 라인에 따라 다르게 야생형 장각과보다 평균 18∼26% 더 긴 장각과 크기의 증가에 부분적으로 기여한다(도 159 참조). 야생형 및 형질전환 식물이 6-인치 화분에서 생장시 DHS 억제에서 야기된 종자 수확량의 증가는 식물이 12 인치 화분에서 생장되는 경우보다 ∼4.5-배 더 컸다. 따라서 DHS의 억제는 작은 컨테이너 내에서의 생장에 의해 유발된 치사-이하 스트레스 효과를 개선시키는 것으로 판단된다. 형질전환 식물 내 종자 수확량의 증가는 트리아실글리세롤 측정에 기반한 종자 오일 함량의 상응하는 증가로 해석되고, 형질전환 종자 내 오일의 지방산 조성의 변화가 존재하지 않았다. 도 161(a) 및 (b) 참조.
(실시예 29)
디옥시하이푸신 신타제 억제제의 투여 및 디옥시하이푸신 신타제의 안티센스에 의한 카네이션 꽃의 화병 수명의 연장
디옥시하이푸신 신타제(DHS)를 인코드하는 전체-길이 cDNA 클론(AF296079)은 카네이션 화판으로부터 분리되었다. DHS는 불활성 진핵성 번역 개시 인자- 5A(eIF-5A)를 번역을 촉진시킬 수 있는 활성 형태로 전환시키는 2개 효소 반응의 첫 번째를 매개한다. 노던 분석은 DHS 발현은 카네이션 꽃 화판의 노화와 상호관련됨을 나타내었다. 절단 카네이션 꽃의 디아미노푸탄(푸트레신), 디아미노프로판, 디아미노헥산, 디아미노옥탄 및 스퍼미딘을 포함한 DHS 반응의 억제제로의 처리는 83%까지 꽃의 화병 수명을 연장시켰다. 카네이션 꽃 노화에서 DHS의 역할을 더욱 한정적으로 평가하기 위해 단백질 발현은 아그로박테리움 형질전환을 통해 구성적 콜리플라워 모자이크 바이러스 프로모터의 조절 하에서 카네이션 DHS cDNA의 안티센스 3'-UTR을 도입시킴으로서 형질전환 식물 내에서 억제되었다. 감소된 DHS 발현을 지닌 형질전환 꽃의 3개 라인이 분석되었고 야생형-꽃에 비해 더 긴 화병-수명을 지니는 것으로 나타났다. 실제로 라인 중의 하나는 >100%의 화병 수명의 증가를 나타내었다. 이들 발견은 DHS가 꽃 노화에 중심 역할을 함을 나타낸다.
(실시예 30)
아라비돕시스 내 디옥시하이푸신 신타제의 억제에 의해 유도된 지연된 추대 표현형은 GA3의 처리에 의해 감소될 수 있음
디옥시하이푸신 신타제(DHS)는 진핵성 번역 개시 인자 5A(eIF-5A)의 후-번역 활성화에 필수적인 편재하는 효소이고 정상 식물 생장 및 발달에 필수적인 것으로 나타난다. DHS는 노화-유도된 SAG 12 프로모터의 조절 하에서 형질전환 식물 내 에서 전체-길이 안티센스 아라비돕시스 DHS cDNA를 발현함으로서 아라비돕시스 내에서 억제되었다. 트랜스유전자를 발현하는 식물은 잎 DHS 단백질 수준을 감소시켰고, 지연된 추대 및 잎 노화 착수의 선고된 지연(2∼5주)을 나타내었다. 또한 생물량 또는 종자 수확량 내의 감소를 유발하지 않더라도 추대는 더욱 짧았다. GA3로의 형질전환 식물의 처리는 지연된 추대 표현형을 나타내었다. 유사한 표현형은 덱사메타손(dexamethasone, DEX)을 투여함으로서 달성될 수 있는 글루카코티코이드(glucacorticoid)-유도성 프로모터(GCI)의 조절 하에서 DHS의 안티센스 억제에 의해 수득되었다. 다시, GA3의 투여는 이러한 표현형을 감소시켰다; 즉, GA3-처리된 형질전환 식물은 정상적으로 추대되었고, 추대는 정상 크기의 것이었고 잎 노화의 착수의 지연이 없었다. 이들 결과는 아라비돕시스 내 3가지 eIF-5A 이소폼의 하나 이상의 활성화를 통해 DHS가 GA 대사에 영향을 미침을 종합적으로 나타낸다.
(실시예 31)
생물량을 증가시키기 위한 미루나무 유래 센스 생장 eIF-5A로의 아라비돕시스 식물의 형질전환
4주령 아라비돕시스 식물이 pKYLX71-센스-포플라 생장-eIF-5A3을 포함한 아그로박테리움 투메파시엔스 GV3101로 침투되었다(도 171 참조).
형질전환뿐만 아니라 야생형(카나마이신 저항성만을 부여한 빈 pKYLX71 벡터로 형질전환된) 식물의 첫 번째 세대(T1)의 종자가 카나마이신이 보충된 1/2 MS 배지 한천 플레이트 상에 파종되었다. 플레이트는 9일간 22℃, 80% rh 및 16 시간 광/일로 그로쓰 챔버 내에 유지되었다. 이후 모종은 통상적인 토양 혼합물을 지닌 32-평평-웰에 옮겨졌고, T2 종자가 수확될 때까지 상기와 동일한 조건 하에 유지되었다. 다른 라인의 T2 종자뿐만 아니라 대조군 식물이 다시 1/2 MS 배지 한천 플레이트 상에서 선별되었다. 모종은 토양으로 옮겨졌고 T1 식물과 동일한 조건 하에서 유지되었다.
최상의 표현형을 지닌 형질전환 라인 3이 본 실험에 선택되었다. eIF-5A 단백질은 웨스턴 분석에 의해 과-발현되는 것으로 확인되었다. 형질전환 라인 3 식물은 6주째에 벡터-유일 대조군 식물 대비 화서 줄기 신장의 1.5-배 증가를 나타내었고 대조군 식물보다 4-배 더 많은 종자 수확량을 생성하였다.
형질전환 라인 3 유래의 종자는 과발현 포플라 eIF5A3을 지닌 형질전환 모종을 선별하기 위해 1/2 MS 배지(카나마이신으로 보충된)를 지닌 플레이트 상에 파종되었다.
1-주령 모종은 토양인 선샤인 LC1/LG3, 1:1(기본 토양, 비료 비첨가)로 옮겨졌다. 일반적으로 사용되는 더 풍부한 토양 상표(Promix BS)와 비교된 본 토양 상표에 대한 토양 분석에 대한 논의를 위해 Wang et al. (2003) Plant Mol Biol 53: 1223-1235 참조.
급수가 요구되는 경우(약 3일 후) 0 g/L, 0.5 g/L, 1 g/L, 3 g/L 및 5 g/L의 일련의 비료(20:20:20)가 토양이 적셔질 때까지 적용되었다(식물의 개별 플랫으로). 최적 생장을 위한 추천되는 비료 함량은 1 g/L이다. 비료는 주 당 1회로 재적용되었다.
형태 및 표현형은 잎 크기 및 색채, 추대 시기 및 화서 줄기 신장, 장각과 크기 및 수 등에 대해 관찰되었다. 수득된 형질전환 식물은 최적, 최적이하 및 최적이상 농도의 비료에 대해 증가된 생물량을 지녔다. 도 172 및 173 참조.
(실시예 32)
생물량을 증가시키기 위한 센스 방향의 돌연변이 노화 eIF-5A로의 아라비돕시스 형질전환
아라비돕시스 식물은 상기 기술된 바와 같이(하이푸신화될 수 없도록) 돌연변이 노화 eIF-5A로 형질전환되었다. 수득된 형질전환 식물은 최적, 최적이하 및 최적이상 농도의 비료에 대해 증가된 생물량을 지녔다. 도 174 및 175 참조.
참고문헌
Azumi, Y and Watanabe, A (1991) Evidence for a senescence-associated gene induced by darkness. Plant Physiol 95: 577-583.
Becker W, Apel K (1993) Differences in gene expression between natural and artificially induced leaf senescence. Planta 189: 74-79
Bevec , D., Jaksche, H., Oft, M., Wohl, T., Himmelspach, M., Pacher, A., Schebesta, M., Koettnitz, K., Dobrovnik, M., Csonga, R., Lottspeich, F., and Hauber, J (1996) Inhibition of HIV-1 replication in lymphocytes by mutants of the Rev cofactor of eIF-5A. Science 271:1858-1860.
Bevec, D, and Hauber, J (1997) Eukaryotic initiation factor 5A activity and HIV-1 Rev function. Biol Signals 6:124-133.
Bleecker, A.B., and Patterson, SE (1997) Last exit: Senescence, abscission, and meristem arrest in Arabidopsis. Plant Cell. 9:1169-1179.
Buchanan-Wollaston, V (1997) The molecular biology of leaf senescence. J Exp Bot 48:181-191.
Buchanan-Wollaston V (1994) Isolation of cDNA clones for genes that are expressed during leaf senescence in Brassica napus. Plant Physiol 105: 839-846
Chen, KY, and Liu, AYC. (1997) Biochemistry and function of hypusine formation on eukaryotic initiation factor 5A. Biol Signals 6:105-109.
Davies KM, and Grierson D (1989) Identification of cDNA clones for tomato (Lycopersicon esculentum Mill.) mRNAs that accumulate during fruit ripening and leaf senescence in response to ethylene. Plant Cell 179: 73-80.
Drake R, John I, Farrell A, Cooper W, Schuch W, and Grierson D (1996) Isolation and analysis of cDNAs encoding tomato cysteine proteases expressed during leaf senescence. Plant Mol Biol 30: 755-767
Gan, S and Amasino, RM (1997). Molecular genetic regulation and manipulation of leaf senescence. Plant Physiol 113: 313-319.
Gan, S and Amasino, RM (1995) Inhibition of leaf senescence by autoregulated production of cytokinin. Science 270: 1986-1988.
Hanauske-Abel, H.M., Park, M.H., Hanauske, A.-R., Popowicz, A.M., Lalande, M., and Folk, J.E. Inhibition of G1-S transition by inhibitors of deoxyhypusine hydroxylation. Biochem Biophys Acta 1221: 115-124, 1994.
Henderson, BR, and Percipalle, P. (1997) Interactions between HIV Rev and nuclear import and export factors: the Rev nuclear localization signal mediate specific binding to human importin-beta. J Mol Biol 274: 693-707.
Hensel, L.L., V. Grbic, D.B. Baumgarten, and A.B. Bleecker. (1993). Developmental and age-related processes that influence the longevity and senescence of photosynthetic tissues in Arabidopsis. Plant Cell 5:553-564.
Jakus, J., Wolff, E.C., Park, M.H., and Folk, J.E. Features of the spermidine-binding site of deoxyhypusine synthase as derived from inhibition studies: effective inhibition by bis-and mono-guanylated diamines and polyamines. J Biol Chem 268:13151-13159, 1993.
Kang, HA, and Hershey, JWB (1994) Effect of initiation factor eIF-5A depletion on protein synthesis and proliferation of Saccharomyces cerevisiae. J Biol Chem 269: 3934-3940.
Katahira, J, Ishizaki, T, Sakai, H, Adachi, A, Yamamoto, K, and Shida, H. (1995) Effects of translation initiation factor eIF-5A on the functioning of human T-cell leukemia virus type I Rex and human immunodeficiency virus Rev inhibited trans dominantly by a Rex mutant deficient in RNA binding. J Virol 69: 3125-3133
Kemper, WM, Berry, KW, and Merrick, WC (1976) Purification and properties of rabbit reticulocyte protein synthesis initiation factors M2Bα and M2Bβ. J Biol Chem 251: 5551-5557.
Lohman, KN, Gan, S, John, MC and Amasino, R (1994) Molecular analysis of natural leaf senescence in Arabidopsis thaliana. Phys Plant 92: 322:328.
Lipowsky, G., Bischoff, F.R., Schwarzmaier, P, Kraft, R., Kostka, S., Hartmann, E., Kutay, U., and Gorlich, D. (2000) Exportin 4: a mediator of a novel nuclear export pathway in higher eukaryotes. EMBO J. 19:4362-4371.
Liu, YP, Nemeroff, M, Yan, YP, and Chen, KY. (1997) Interaction of eukaryotic initiation facto 5A with the human immunodeficiency virus type 1 Rev response element RNA and U6 snRNA requires deoxyhypusine or hypusine modification. Biol Signals 6:166-174.
Martinez-Zapater, JM and Salinas, J (1994) Arabidopsis Protocols. Humana Press: p. 197.
Mattaj, I.W., and Englmeier, L. (1998) Nucleocytoplasmic transport: The soluble phase. Annu Rev Biochem 67: 265-306
Mehta, AM, Saftner, RA, Mehta, RA, and Davies, PJ (1994) Identification of posttranslationally modified 18-kilodalton protein from rice as eukaryotic translation initiation factor 5A. Plant Physiol 106:1413-1419.
Noh, Y-S and Amasino, R (1999) Identification of a promoter region responsible for the senescence-specific expression of SAG12. Plant Mol Biol 41:181-194.
Nooden, LD, Guaimet, JJ and John, I (1997) Senescence mechanisms. Physiol Plant 101: 746-753.
Nooden, LD and Leopold, AC (eds) (1988) The phenomena of senescence and aging. Senescence and Aging in Plants. Academic Press: pp.1-50.
Ober, D and Hartmann, T (1999) Deoxyhypusine synthase from tobacco. J Biol Chem 274: 32040-32047.
Oh SA, Lee SY, Chung IK, Lee CH, and Nam HG (1996) A senescence-associated gene of Arabidopsis thaliana is distinctively regulated during natural and artificially induced leaf senescence. Plant Mol Biol 30: 739-754
Page, DR and Grossniklaus, U (2002) The art and design of genetic screens: Arabidopsis Thaliana. Nature Reviews Genetics 3:124-136
Park, MH, Joe, YA, and Kang KR (1998) Deoxyhypusine synthase activity is essential for cell viability in the yeast Saccharomyces cerevisiae. J Biol Chem 16:1677-1683.
Park, MH, Wolff, EC, Lee, YB and Folk, JE (1994) Antiproliferative effects of inhibitors of deoxyhypusine synthase: inhibition of growth of Chinese hamster ovary cells by guanyl diamines. J Biol Chem 269:27827-27832.
Park, M.H., Wolff E.C., and Folk J.E. (1993) Hypusine: its post-translational formation in eukaryotic initiation factor 5A and its potential role in cellular regulation. BioFactors 4:95-104.
Park, MH, Wolff, EC and Folk, JE (1993) Is hypusine essential for eukaryotic cell proliferation? Trends Biochem Sci 18:475-479.
Park, MH, Wolff, EC, Smit-McBride, Z, Hershey, JWB and Folk, JE (1991) Comparison of the activities of variant forms of eIF-4D: the requirement for hypusine or deoxyhypusine. J Biol Chem 266:7988-7994, 1991.
Park, MH and Wolff, EC (1988) Cell-free synthesis of deoxyhypusine. J Biol Chem 263:15264-15269.
Quirino, BF, Noh, Y-S, Himelblau, E and Amasino, R (2000) Molecular aspects of leaf senescence. Trends Plant Sci 5:278-282.
Rosorius, O., Reichart, B., Kratzer, F., Heger, P., Dabauvalle, M.C. & Hauber, J. (1999) Nuclear pore localization & nucleocytoplasmic transport of eIF-5A: evidence for direct interaction with the export receptor CRM1. J.Cell Sci. 112, 2369-2380.
Ruhl, M., Himmelspach, M., Bahr, G.M., Hammerschmid, F., Jaschke, H., Wolff, B., Aschauer, H., Farrington, G.K., Probst, H., Bevec, D., and Hauber, J. (1993) Eukaryotic initiation factor 5A is a cellular target of the human immunodeficiency virus type 1 Rev activation domain mediating transactivation. J Cell Biol 123:1309-1320.
Schardl, CL, Byrd, AD, Bension, G, Altschuler, MS, Hildebrand, DF and Hunt, AG (1987) Design and construction of a versatile system for the expression of foreign genes in plants. Genes 61: 1-11.
Schnier J, Schwelberger HG, Smit-McBride Z, Kang HA and Hershey JWB (1991) Translation initiation factor 5A and its hypusine modification are essential for cell viability in the yeast Saccharomyces cerevisiae. Mol Cell Biol11:3105-3114
Smart CM, Hosken SE, Thomas H, Greaves JA, Blair BG, Schuch W (1995) The timing of maize leaf senescence and characterization of senescence-related cDNAs. Physiol Plant 93: 673-682
Smart , CM (1994) Gene expression during leaf senescence. New Phytologist 126:419-448.
Taylor CB, Bariola PA, Delcardayre SB, Raines RT, Green RT (1993) RNS2: A senescence-associated RNase of Arabidopsis that diverged from the S-RNases before speciation. Proc Natl Acad Sci USA 90: 5118-5122
Tome, M., Fiser, S.M., Payne, C.M., and Gerner, EW. (1997) Excess putrescine accumulation inhibits the formation of modified eukaryotic initiation factor 5A (eIF-5A) and induces apoptosis. Biochem. J. 328: 847-854.
Tome, M., and Gerner, EW. (1997) Cellular eukaryotic initation factor 5A content as a mediator of polyamine effects on growth and apoptosis. Biol Signals 6:150- 156.
Walbot V (2000) A green chapter in the book of life. Nature 408: 794-795.
Wang, T-W, Lu, L, Wang, D, and Thompson, JE (2001) Isolation and characterization of senescence-induced cDNAs encoding deoxyhypusine synthase and eucaryotic translation initiation factor 5A from tomato. J Biol Chem 276:17541-17549.
Watanabe, A and Imaseki, H (1982) Changes in translatable mRNA in senescing wheat leaves. Plant Cell Physiol 23:489-497.
Weaver, L.M., Gan, S, Quirino, B and Amasino, R (1998) A comparison of the expression patterns of several senescence-associated genes in response to stress and hormone treatment. Plant Mol Biol 37:455-469.
Xu, A. and Chen, K.Y. (2001) Hypusine is required for a sequence-specific interaction of eukaryotic initiation factor 5A with post-SELEX RNA. J. Biol Chem. 276:2555-2561.
Zuk, D., and Jacobson, A. (1998) A single amino acid substitution in yeast eIF-5A results in mRNA stabilization. EMBO J. 17:2914 -2925.
Boyes, D.C., A.M. Zayed, R. Ascenzi, A.J. McCaskill, N.E. Hoffman, K.R. Davis, and J. Goerlach. 2001. Growth stage-based phenotypic analysis of Arabidopsis: A model for high throughput functional genomics in plants. Plant Cell, 13: 1499-1510.
Collawn, J.F., and Y. Patterson. 1999. Production of antipeptide antibodies. In FM Ausubel, R Brent, RE Kingston, DD Moore, JA Smith, JG Seidman, K Struhl, eds, Current Protocols in Molecular Biology on CD. John Wiley & Sons, New York.
Chamot, D. and C. Kuhlemeier. 1992. Differential expression of genes encoding the hypusine-containing translation initiation factor, eIF-5A, in tobacco. Nucleic Acids Res 20: 665-669.
Clemens, M. J., and U.-A. Bommer. 1999. Translational control: The cancer connection. Int. J. of Biochem. Cell Biol. 31: 1-23.
Davis, L.G., M.D. Dibner, and J. B. Battey. 1986. Basic methods in molecular biology. Elsevier Science Publishing Co., Inc, New York, pp. 130-5.
Drenckhahn, D., T. Jons, and F. Schmitz. 1993. Production of polyclonal antibodies against proteins and peptides. In DJ Asai, ed, Methods in Cell Biology, Vol 37. Academic Press, New York, pp 7-56.
Dresselhaus, T., C. Simone, and H. Lorz. 1999. A transcript encoding translation factor eIF-5A is stored in unfertilized egg cells of maize. Plant Mol. Biol. 39: 1063-1071.
Fagard, M. and H. Vaucheret. 2000. (Trans)Gene silencing in plants: How many mechanisms? Annu. Rev. Plant Physiol. Mol. Biol. 51: 167-94.
Fairbanks, G., T.L. Steck, and D.F.H. Wallach. 1971. Coomassie blue R250 used in isopropanol-acetic acid. Biochem 10: 2606-2618.
Ghosh, S., S. Gepstein, J.J. Heikkila, and E.B. Dumbroff. 1988. Use of a scanning densitometer or an ELISA plate reader for measurement of nanogram amounts of protein in crude extracts from biological tissues. Anal. Biochem. 169: 227-233.
Jao, D.L.-E., and K.Y. Chen. 2002. Subcellular localization of the hypusin-containing eukaryotic initiation factor 5A by immunofluorescent staining and green fluorescent protein tagging. J. Cell. Biochem. 86: 590-600.
Jakus, J., E.C. Wolff, M.H. Park, and J.E. Folk. 1993. Features of the spermidine-binding site of deoxyhypusine synthase as derived from inhibition studies: effective inhibition by bis- and mono-guanylated diamines and polyamines. J. Biol. Chem. 268, 13151-13159.
Kushner, S. R. 1978. An improved method for transformation of Escherichia coli with ColE1 derived plasmids. In: Genetic Engineering: Proceedings of the International Symposium on Genetic Engineering (H.W. Boyer and S. Nicosia, eds.), Elsevier/North-Holland Press, New York. Pp. 17-23.
Liu, Y.P., M. Nemeroff, Y.P. Yan, and K.Y. Chen. 1997. Interaction of eukaryotic initiation factor 5A with the human immunodeficiency virus type 1 Rev response element RNA and U6 snRNA requires deoxyhypusine or hypusine modification. Biol. Signals 6(3), 166-74.
Matile, P., S. Hoertensteiner, and H. Thomas. 1999. Chlorophyll degradation. Annu. Rev. Plant Physiol. Plant Mol. Biol. 50: 67-95.
McCabe, M.S, L.C. Garratt, F. Schepers, W.J.R.M. Jordi, G.M. Stoopen, E. Davelaar, J.H.A. van Rhijn, J.B. Power, and M.R. Davey. 2001. Effects of PSAG12-IPT gene expression on development and senescence in transgenic lettuce. Plant Physiol. 127: 505-516.
Miranda, P.V., A. Brandelli, and J. G. Tezon. 1993. Instantaneous blocking for immunoblots. Anal. Biochem. 209: 376-377.
Murashige, T. and F. Skoog. 1962. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol. Plant. 15:473-497.
Nowack, L.N. 2002 personal communications.
Ober, D., and T. Hartmann. 1999. Deoxyhypusine synthase from tobacco. J. Biol. Chem. 274(45): 32040-32047.
Page, T., G. Griffiths, and V. Buchanan-Wollaston. 2001. Molecular and biochemical characterization of postharvest senescence in broccoli. Plant Physiol. 125: 718-727.
Park, J.-H., S.A. Oh, Y.H. Kim, H.R. Woo, and H.G. Nam. 1998. Differential expression of senescence-associated mRNAs during leaf senescence induced by different senescence-inducing factors in Arabidopsis. Plant Mol. Biol. 37: 445-454.
Park, M.H., Y.B. Lee, and Y.A . Joe. 1997. Hypusine is essential for eukaryotic cell proliferation. Biol. Signals 6, 115-123.
Park, M.H., E.C. Wolff, and J.E. Folk. 1993. Hypusine: its post-translational formation in eukaryotic initiation factor 5A and its potential role in cellular regulation. BioFactors 4(2), 95-104.
Park, M.H., and E.C. Wolff. 1988. Cell-free synthesis of deoxyhypusine. J. Biol. Chem. 263(30): 15264-15269.
Rosorius, O., B. Reicher, F. Kraetzer, P. Heger, M.-C. Dabauvalle, and J. Hauber. 1999. Nuclear pore localization and nucleocytoplasmic transport of eIF-5A: evidence for direct interaction with the export receptor CRM1. J. Cell Sci. 112: 2369-2380.
Stotz 2000.
Tome, M.E., and E.W. Gerner. 1997. Cellular eukaryotic initiation factor 5A content as a mediator of polyamine effects of growth and apoptosis. Biol. Signals 6, 150-156.
Wang, C.Y. and J.E. Baker. 1980. Extending vase life of carnations with minooxyacetic acid, polyamines, EDU, and CCCP. HortSci. 15, 805-806.
Wang, T.-W., L. Lu, C.-G. Zhang, C.A. Taylor, and J.E. Thompson. Unpublished a. Suppression of deoxyhypusine synthase expression in Arabidopsis thaliana delays leaf senescence.
Wang, T.-W., W. Wu, C. Taylor, and J.E. Thompson. Unpublished b. Characterization of eukaryotic translation initiation factor-5A cDNA isoforms from tomato.
Wang, T.-W., C.-G. Zhang, L. Lu, L.N. Nowack, and J. E. Thompson. Unpublished c. Extending vase life of carnation by deoxyhypusine synthase inhibitors and transient transfection with antisense deoxyhypusine synthase.
Wang, T.-W., L. Lu, D. Wang, and J.E. Thompson. 2001. Isolation and characterization of senescence-induced cDNAs ecoding deoxyhypusine synthase and eucaryotic translation initiation factor 5A from tomato. J. Biol. Chem. 276(20): 17541-17549.
Zuk, D., and A. Jacobson. 1998. A single amino acid substitution in yeast eIF-5A results in mRNA stabilization. EMBO J. 17(10): 2914-2925.
SEQUENCE LISTING
<110> THOMPSON, JOHN E.
WANG, TZANN-WEI
NOWACK, LINDA
WU, WENDY
<120> POLYNUCLEOTIDES OF DHA AND ISOFORMS OF EIF-5A AND
METHODS OF USING SAME
<130> 10799/206
<140> 11/293,392
<141> 2005-12-05
<150> 60/632,567
<151> 2004-12-03
<150> 60/690,160
<151> 2005-06-14
<150> 60/710,999
<151> 2005-08-25
<150> 60/728,737
<151> 2005-10-21
<150> 10/862,440
<151> 2004-06-08
<150> 09/725,019
<151> 2000-11-29
<150> 09/597,771
<151> 2000-06-19
<150> 09/348,675
<151> 1999-07-06
<160> 211
<170> PatentIn Ver. 3.3
<210> 1
<211> 1584
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (54)..(1196)
<400> 1
cgcagaaact cgcggcggca gtcttgttcc gtacataatc ttggtctgca ata atg 56
Met
1
gga gaa gct ctg aag tac agt atc atg gac tca gta aga tcg gta gtt 104
Gly Glu Ala Leu Lys Tyr Ser Ile Met Asp Ser Val Arg Ser Val Val
5 10 15
ttc aaa gaa tcc gaa aat cta gaa ggt tct tgc act aaa atc gag ggc 152
Phe Lys Glu Ser Glu Asn Leu Glu Gly Ser Cys Thr Lys Ile Glu Gly
20 25 30
tac gac ttc aat aaa ggc gtt aac tat gct gag ctg atc aag tcc atg 200
Tyr Asp Phe Asn Lys Gly Val Asn Tyr Ala Glu Leu Ile Lys Ser Met
35 40 45
gtt tcc act ggt ttc caa gca tct aat ctt ggt gac gcc att gca att 248
Val Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile Ala Ile
50 55 60 65
gtt aat caa atg cta gat tgg agg ctt tca cat gag ctg ccc acg gag 296
Val Asn Gln Met Leu Asp Trp Arg Leu Ser His Glu Leu Pro Thr Glu
70 75 80
gat tgc agt gaa gaa gaa aga gat gtt gca tac aga gag tcg gta acc 344
Asp Cys Ser Glu Glu Glu Arg Asp Val Ala Tyr Arg Glu Ser Val Thr
85 90 95
tgc aaa atc ttc ttg ggg ttc act tca aac ctt gtt tct tct ggt gtt 392
Cys Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val
100 105 110
aga gac act gtc cgc tac ctt gtt cag cac cgg atg gtt gat gtt gtg 440
Arg Asp Thr Val Arg Tyr Leu Val Gln His Arg Met Val Asp Val Val
115 120 125
gtt act aca gct ggt ggt att gaa gag gat ctc ata aag tgc ctc gca 488
Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu Ala
130 135 140 145
cca acc tac aag ggg gac ttc tct tta cct gga gct tct cta cga tcg 536
Pro Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly Ala Ser Leu Arg Ser
150 155 160
aaa gga ttg aac cgt att ggt aac tta ttg gtt cct aat gac aac tac 584
Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr
165 170 175
tgc aaa ttt gag aat tgg atc atc cca gtt ttt gac caa atg tat gag 632
Cys Lys Phe Glu Asn Trp Ile Ile Pro Val Phe Asp Gln Met Tyr Glu
180 185 190
gag cag att aat gag aag gtt cta tgg aca cca tct aaa gtc att gct 680
Glu Gln Ile Asn Glu Lys Val Leu Trp Thr Pro Ser Lys Val Ile Ala
195 200 205
cgt ctg ggt aaa gaa att aat gat gaa acc tca tac ttg tat tgg gct 728
Arg Leu Gly Lys Glu Ile Asn Asp Glu Thr Ser Tyr Leu Tyr Trp Ala
210 215 220 225
tac aag aac cgg att cct gtc ttc tgt cct ggc ttg acg gat gga tca 776
Tyr Lys Asn Arg Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly Ser
230 235 240
ctt ggt gac atg cta tac ttc cat tct ttc aaa aag ggt gat cca gat 824
Leu Gly Asp Met Leu Tyr Phe His Ser Phe Lys Lys Gly Asp Pro Asp
245 250 255
aat cca gat ctt aat cct ggt cta gtc ata gac att gta gga gat att 872
Asn Pro Asp Leu Asn Pro Gly Leu Val Ile Asp Ile Val Gly Asp Ile
260 265 270
agg gcc atg aat ggt gaa gct gtc cat gct ggt ttg agg aag aca gga 920
Arg Ala Met Asn Gly Glu Ala Val His Ala Gly Leu Arg Lys Thr Gly
275 280 285
atg att ata ctg ggt gga ggg ctg cct aag cac cat gtt tgc aat gcc 968
Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Val Cys Asn Ala
290 295 300 305
aat atg atg cgc aat ggt gca gat ttt gcc gtc ttc att aac acc gca 1016
Asn Met Met Arg Asn Gly Ala Asp Phe Ala Val Phe Ile Asn Thr Ala
310 315 320
caa gag ttt gat ggt agt gac tct ggt gcc cgt cct gat gaa gct gta 1064
Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Val
325 330 335
tca tgg gga aag ata cgt ggt ggt gcc aag act gtg aag gtg cat tgt 1112
Ser Trp Gly Lys Ile Arg Gly Gly Ala Lys Thr Val Lys Val His Cys
340 345 350
gat gca acc att gca ttt ccc ata tta gta gct gag aca ttt gca gct 1160
Asp Ala Thr Ile Ala Phe Pro Ile Leu Val Ala Glu Thr Phe Ala Ala
355 360 365
aag agt aag gaa ttc tcc cag ata agg tgc caa gtt tgaacattga 1206
Lys Ser Lys Glu Phe Ser Gln Ile Arg Cys Gln Val
370 375 380
ggaagctgtc cttccgacca cacatatgaa ttgctagctt ttgaagccaa cttgctagtg 1266
tgcagcacca tttattctgc aaaactgact agagagcagg gtatattcct ctaccccgag 1326
ttagacgaca tcctgtatgg ttcaaattaa ttatttttct ccccttcaca ccatgttatt 1386
tagttctctt cctcttcgaa agtgaagagc ttagatgttc ataggttttg aattatgttg 1446
gaggttggtg ataactgact agtcctctta ccatatagat aatgtatcct tgtactatga 1506
gattttgggt gtgtttgata ccaaggaaaa atgtttattt ggaaaacaat tggattttta 1566
atttattttt tcttgttt 1584
<210> 2
<211> 381
<212> PRT
<213> Lycopersicon esculentum
<400> 2
Met Gly Glu Ala Leu Lys Tyr Ser Ile Met Asp Ser Val Arg Ser Val
1 5 10 15
Val Phe Lys Glu Ser Glu Asn Leu Glu Gly Ser Cys Thr Lys Ile Glu
20 25 30
Gly Tyr Asp Phe Asn Lys Gly Val Asn Tyr Ala Glu Leu Ile Lys Ser
35 40 45
Met Val Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile Ala
50 55 60
Ile Val Asn Gln Met Leu Asp Trp Arg Leu Ser His Glu Leu Pro Thr
65 70 75 80
Glu Asp Cys Ser Glu Glu Glu Arg Asp Val Ala Tyr Arg Glu Ser Val
85 90 95
Thr Cys Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly
100 105 110
Val Arg Asp Thr Val Arg Tyr Leu Val Gln His Arg Met Val Asp Val
115 120 125
Val Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu
130 135 140
Ala Pro Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly Ala Ser Leu Arg
145 150 155 160
Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn
165 170 175
Tyr Cys Lys Phe Glu Asn Trp Ile Ile Pro Val Phe Asp Gln Met Tyr
180 185 190
Glu Glu Gln Ile Asn Glu Lys Val Leu Trp Thr Pro Ser Lys Val Ile
195 200 205
Ala Arg Leu Gly Lys Glu Ile Asn Asp Glu Thr Ser Tyr Leu Tyr Trp
210 215 220
Ala Tyr Lys Asn Arg Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly
225 230 235 240
Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe Lys Lys Gly Asp Pro
245 250 255
Asp Asn Pro Asp Leu Asn Pro Gly Leu Val Ile Asp Ile Val Gly Asp
260 265 270
Ile Arg Ala Met Asn Gly Glu Ala Val His Ala Gly Leu Arg Lys Thr
275 280 285
Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Val Cys Asn
290 295 300
Ala Asn Met Met Arg Asn Gly Ala Asp Phe Ala Val Phe Ile Asn Thr
305 310 315 320
Ala Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala
325 330 335
Val Ser Trp Gly Lys Ile Arg Gly Gly Ala Lys Thr Val Lys Val His
340 345 350
Cys Asp Ala Thr Ile Ala Phe Pro Ile Leu Val Ala Glu Thr Phe Ala
355 360 365
Ala Lys Ser Lys Glu Phe Ser Gln Ile Arg Cys Gln Val
370 375 380
<210> 3
<211> 369
<212> PRT
<213> Homo sapiens
<400> 3
Met Glu Gly Ser Leu Glu Arg Glu Ala Pro Ala Gly Ala Leu Ala Ala
1 5 10 15
Val Leu Lys His Ser Ser Thr Leu Pro Pro Glu Ser Thr Gln Val Arg
20 25 30
Gly Tyr Asp Phe Asn Arg Gly Val Asn Tyr Arg Ala Leu Leu Glu Ala
35 40 45
Phe Gly Thr Thr Gly Phe Gln Ala Thr Asn Phe Gly Arg Ala Val Gln
50 55 60
Gln Val Asn Ala Met Ile Glu Lys Lys Leu Glu Pro Leu Ser Gln Asp
65 70 75 80
Glu Asp Gln His Ala Asp Leu Thr Gln Ser Arg Arg Pro Leu Thr Ser
85 90 95
Cys Thr Ile Phe Leu Gly Tyr Thr Ser Asn Leu Ile Ser Ser Gly Ile
100 105 110
Arg Glu Thr Ile Arg Tyr Leu Val Gln His Asn Met Val Asp Val Leu
115 120 125
Val Thr Thr Ala Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala
130 135 140
Pro Thr Tyr Leu Gly Glu Phe Ser Leu Arg Gly Lys Glu Leu Arg Glu
145 150 155 160
Asn Gly Ile Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Glu Asn Tyr
165 170 175
Cys Lys Phe Glu Asp Trp Leu Met Pro Ile Leu Asp Gln Met Val Met
180 185 190
Glu Gln Asn Thr Glu Gly Val Lys Trp Thr Pro Ser Lys Met Ile Ala
195 200 205
Arg Leu Gly Lys Glu Ile Asn Asn Pro Glu Ser Val Tyr Tyr Trp Ala
210 215 220
Gln Lys Asn His Ile Pro Val Phe Ser Pro Ala Leu Thr Asp Gly Ser
225 230 235 240
Leu Gly Asp Met Ile Phe Phe His Ser Tyr Lys Asn Pro Gly Leu Val
245 250 255
Leu Asp Ile Val Glu Asp Leu Arg Leu Ile Asn Thr Gln Ala Ile Phe
260 265 270
Ala Lys Cys Thr Gly Met Ile Ile Leu Gly Gly Gly Val Val Lys His
275 280 285
His Ile Ala Asn Ala Asn Leu Met Arg Asn Gly Ala Asp Tyr Ala Val
290 295 300
Tyr Ile Asn Thr Ala Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg
305 310 315 320
Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Val Asp Ala Gln Pro
325 330 335
Val Lys Val Tyr Ala Asp Ala Ser Leu Val Phe Pro Leu Leu Val Ala
340 345 350
Glu Thr Phe Ala Gln Lys Met Asp Ala Phe Met His Glu Lys Asn Glu
355 360 365
Asp
<210> 4
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide fragment
<400> 4
Thr Gly Lys His Gly His
1 5
<210> 5
<211> 2272
<212> DNA
<213> Arabidopsis thaliana
<220>
<221> CDS
<222> (68)..(265)
<220>
<221> CDS
<222> (348)..(536)
<220>
<221> CDS
<222> (624)..(842)
<220>
<221> CDS
<222> (979)..(1065)
<220>
<221> CDS
<222> (1154)..(1258)
<220>
<221> CDS
<222> (1575)..(1862)
<400> 5
gaactcccaa aaccctctac tactacactt tcagatccaa ggaaatcaat tttgtcattc 60
gagcaac atg gag gat gat cgt gtt ttc tct tcg gtt cac tca aca gtt 109
Met Glu Asp Asp Arg Val Phe Ser Ser Val His Ser Thr Val
1 5 10
ttc aaa gaa tcc gaa tca ttg gaa gga aag tgt gat aaa atc gaa gga 157
Phe Lys Glu Ser Glu Ser Leu Glu Gly Lys Cys Asp Lys Ile Glu Gly
15 20 25 30
tac gat ttc aat caa gga gta gat tac cca aag ctt atg cga tcc atg 205
Tyr Asp Phe Asn Gln Gly Val Asp Tyr Pro Lys Leu Met Arg Ser Met
35 40 45
ctc acc acc gga ttt caa gcc tcg aat ctc ggc gaa gct att gat gtc 253
Leu Thr Thr Gly Phe Gln Ala Ser Asn Leu Gly Glu Ala Ile Asp Val
50 55 60
gtc aat caa atg gttcgtttct cgaattcatc aaaaataaaa attccttctt 305
Val Asn Gln Met
65
tttgttttcc tttgttttgg gtgaattagt aatgacaaag ag ttt gaa ttt gta 359
Phe Glu Phe Val
70
ttg aag cta gat tgg aga ctg gct gat gaa act aca gta gct gaa gac 407
Leu Lys Leu Asp Trp Arg Leu Ala Asp Glu Thr Thr Val Ala Glu Asp
75 80 85
tgt agt gaa gag gag aag aat cca tcg ttt aga gag tct gtc aag tgt 455
Cys Ser Glu Glu Glu Lys Asn Pro Ser Phe Arg Glu Ser Val Lys Cys
90 95 100
aaa atc ttt cta ggt ttc act tca aat ctt gtt tca tct ggt gtt aga 503
Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg
105 110 115
gat act att cgt tat ctt gtt cag cat cat atg gtttgtgatt tttgctttat 556
Asp Thr Ile Arg Tyr Leu Val Gln His His Met
120 125
caccctgctt ttttatagat gttaaaattt tcgagcttta gttttgattt caatggtttt 616
tctgcag gtt gat gtt ata gtc acg aca act ggt ggt gtt gag gaa gat 665
Val Asp Val Ile Val Thr Thr Thr Gly Gly Val Glu Glu Asp
130 135 140
ctc ata aaa tgc ctt gca cct aca ttt aaa ggt gat ttc tct cta cct 713
Leu Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe Ser Leu Pro
145 150 155
gga gct tat tta agg tca aag gga ttg aac cga att ggg aat ttg ctg 761
Gly Ala Tyr Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu
160 165 170 175
gtt cct aat gat aac tac tgc aag ttt gag gat tgg atc att ccc atc 809
Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile
180 185 190
ttt gac gag atg ttg aag gaa cag aaa gaa gag gtattgcttt atctttcctt 862
Phe Asp Glu Met Leu Lys Glu Gln Lys Glu Glu
195 200
tttatatgat ttgagatgat tctgtttgtg cgtcactagt ggagatagat tttgattcct 922
ctcttgcatc attgacttcg ttggtgaatc cttctttctc tggtttttcc ttgtag aat 981
Asn
gtg ttg tgg act cct tct aaa ctg tta gca cgg ctg gga aaa gaa atc 1029
Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg Leu Gly Lys Glu Ile
205 210 215
aac aat gag agt tca tac ctt tat tgg gca tac aag gtatccaaaa 1075
Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys
220 225 230
ttttaacctt tttagttttt taatcatcct gtgaggaact cggggattta aattttccgc 1135
ttcttgtggt gtttgtag atg aat att cca gta ttc tgc cca ggg tta aca 1186
Met Asn Ile Pro Val Phe Cys Pro Gly Leu Thr
235 240
gat ggc tct ctt ggg gat atg ctg tat ttt cac tct ttt cgt acc tct 1234
Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe Arg Thr Ser
245 250 255
ggc ctc atc atc gat gta gta caa ggtacttctt ttactcaata agtcagtgtg 1288
Gly Leu Ile Ile Asp Val Val Gln
260 265
ataaatattc ctgctacatc tagtgcagga atattgtaac tagtagtgca ttgtagcttt 1348
tccaattcag caacggactt tactgtaagt tgatatctaa aggttcaaac gggagctagg 1408
agaatagcat aggggcattc tgatttaggt ttggggcact gggttaagag ttagagaata 1468
ataatcttgt tagttgttta tcaaactctt tgatggttag tctcttggta atttgaattt 1528
tatcacagtg tttatggtct ttgaaccagt taatgtttta tgaaca gat atc aga 1583
Asp Ile Arg
gct atg aac ggc gaa gct gtc cat gca aat cct aaa aag aca ggg atg 1631
Ala Met Asn Gly Glu Ala Val His Ala Asn Pro Lys Lys Thr Gly Met
270 275 280 285
ata atc ctt gga ggg ggc ttg cca aag cac cac ata tgt aat gcc aat 1679
Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn
290 295 300
atg atg cgc aat ggt gca gat tac gct gta ttt ata aac acc ggg caa 1727
Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr Gly Gln
305 310 315
gaa ttt gat ggg agc gac tcg ggt gca cgc cct gat gaa gcc gtg tct 1775
Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Val Ser
320 325 330
tgg ggt aaa att agg ggt tct gct aaa acc gtt aag gtc tgc ttt tta 1823
Trp Gly Lys Ile Arg Gly Ser Ala Lys Thr Val Lys Val Cys Phe Leu
335 340 345
att tct tca cat cct aat tta tat ctc act cag tgg ttt tgagtacata 1872
Ile Ser Ser His Pro Asn Leu Tyr Leu Thr Gln Trp Phe
350 355 360
tttaatattg gatcattctt gcaggtatac tgtgatgcta ccatagcctt cccattgttg 1932
gttgcagaaa catttgccac aaagagagac caaacctgtg agtctaagac ttaagaactg 1992
actggtcgtt ttggccatgg attcttaaag atcgttgctt tttgatttta cactggagtg 2052
accatataac actccacatt gatgtggctg tgacgcgaat tgtcttcttg cgaattgtac 2112
tttagtttct ctcaacctaa aatgatttgc agattgtgtt ttcgtttaaa acacaagagt 2172
cttgtagtca ataatccttt gccttataaa attattcagt tccaacaaca cattgtgatt 2232
ctgtgacaag tctcccgttg cctatgttca cttctctgcg 2272
<210> 6
<211> 362
<212> PRT
<213> Arabidopsis thaliana
<400> 6
Met Glu Asp Asp Arg Val Phe Ser Ser Val His Ser Thr Val Phe Lys
1 5 10 15
Glu Ser Glu Ser Leu Glu Gly Lys Cys Asp Lys Ile Glu Gly Tyr Asp
20 25 30
Phe Asn Gln Gly Val Asp Tyr Pro Lys Leu Met Arg Ser Met Leu Thr
35 40 45
Thr Gly Phe Gln Ala Ser Asn Leu Gly Glu Ala Ile Asp Val Val Asn
50 55 60
Gln Met Phe Glu Phe Val Leu Lys Leu Asp Trp Arg Leu Ala Asp Glu
65 70 75 80
Thr Thr Val Ala Glu Asp Cys Ser Glu Glu Glu Lys Asn Pro Ser Phe
85 90 95
Arg Glu Ser Val Lys Cys Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu
100 105 110
Val Ser Ser Gly Val Arg Asp Thr Ile Arg Tyr Leu Val Gln His His
115 120 125
Met Val Asp Val Ile Val Thr Thr Thr Gly Gly Val Glu Glu Asp Leu
130 135 140
Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe Ser Leu Pro Gly
145 150 155 160
Ala Tyr Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val
165 170 175
Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe
180 185 190
Asp Glu Met Leu Lys Glu Gln Lys Glu Glu Asn Val Leu Trp Thr Pro
195 200 205
Ser Lys Leu Leu Ala Arg Leu Gly Lys Glu Ile Asn Asn Glu Ser Ser
210 215 220
Tyr Leu Tyr Trp Ala Tyr Lys Met Asn Ile Pro Val Phe Cys Pro Gly
225 230 235 240
Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe Arg
245 250 255
Thr Ser Gly Leu Ile Ile Asp Val Val Gln Asp Ile Arg Ala Met Asn
260 265 270
Gly Glu Ala Val His Ala Asn Pro Lys Lys Thr Gly Met Ile Ile Leu
275 280 285
Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg
290 295 300
Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr Gly Gln Glu Phe Asp
305 310 315 320
Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys
325 330 335
Ile Arg Gly Ser Ala Lys Thr Val Lys Val Cys Phe Leu Ile Ser Ser
340 345 350
His Pro Asn Leu Tyr Leu Thr Gln Trp Phe
355 360
<210> 7
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 7
ggtggtgttg aggaagatc 19
<210> 8
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 8
ggtgcacgcc ctgatgaagc 20
<210> 9
<211> 1660
<212> DNA
<213> Dianthus caryophyllus
<220>
<221> CDS
<222> (256)..(1374)
<400> 9
gtcattacaa tgcataggat cattgcacat gctaccttcc tcattgcact tgagcttgcc 60
atacttttgt ttttgacgtt tgataataat actatgaaaa tattatgttt tttcttttgt 120
gtgttggtgt ttttgaagtt gtttttgata agcagaaccc agttgtttta cacttttacc 180
attgaactac tgcaattcta aaactttgtt tacattttaa ttccatcaaa gattgagttc 240
agcataggaa aaagg atg gag gat gct aat cat gat agt gtg gca tct gcg 291
Met Glu Asp Ala Asn His Asp Ser Val Ala Ser Ala
1 5 10
cac tct gca gca ttc aaa aag tcg gag aat tta gag ggg aaa agc gtt 339
His Ser Ala Ala Phe Lys Lys Ser Glu Asn Leu Glu Gly Lys Ser Val
15 20 25
aag att gag ggt tat gat ttt aat caa ggt gta aac tat tcc aaa ctc 387
Lys Ile Glu Gly Tyr Asp Phe Asn Gln Gly Val Asn Tyr Ser Lys Leu
30 35 40
ttg caa tct ttc gct tct aat ggg ttt caa gcc tcg aat ctt gga gat 435
Leu Gln Ser Phe Ala Ser Asn Gly Phe Gln Ala Ser Asn Leu Gly Asp
45 50 55 60
gcc att gaa gta gtt aat cat atg cta gat tgg agt ctg gca gat gag 483
Ala Ile Glu Val Val Asn His Met Leu Asp Trp Ser Leu Ala Asp Glu
65 70 75
gca cct gtg gac gat tgt agc gag gaa gag agg gat cct aaa ttc aga 531
Ala Pro Val Asp Asp Cys Ser Glu Glu Glu Arg Asp Pro Lys Phe Arg
80 85 90
gaa tct gtg aag tgc aaa gtg ttc ttg ggc ttt act tca aat ctt att 579
Glu Ser Val Lys Cys Lys Val Phe Leu Gly Phe Thr Ser Asn Leu Ile
95 100 105
tcc tct ggt gtt cgt gac aca att cgg tat ctc gtg caa cat cat atg 627
Ser Ser Gly Val Arg Asp Thr Ile Arg Tyr Leu Val Gln His His Met
110 115 120
gtt gac gtg ata gta acg aca acc gga ggt ata gaa gaa gat cta ata 675
Val Asp Val Ile Val Thr Thr Thr Gly Gly Ile Glu Glu Asp Leu Ile
125 130 135 140
aaa gga aga tcc atc aag tgc ctt gca ccc act ttc aaa ggc gat ttt 723
Lys Gly Arg Ser Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe
145 150 155
gcc tta cca gga gct caa tta cgc tcc aaa ggg ttg aat cga att ggt 771
Ala Leu Pro Gly Ala Gln Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly
160 165 170
aat ctg ttg gtt ccg aat gat aac tac tgt aaa ttt gag gat tgg atc 819
Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile
175 180 185
att cca att tta gat aag atg ttg gaa gag caa att tca gag aaa atc 867
Ile Pro Ile Leu Asp Lys Met Leu Glu Glu Gln Ile Ser Glu Lys Ile
190 195 200
tta tgg aca cca tcg aag ttg att ggt cga tta gga aga gaa ata aac 915
Leu Trp Thr Pro Ser Lys Leu Ile Gly Arg Leu Gly Arg Glu Ile Asn
205 210 215 220
gat gag agt tca tac ctt tac tgg gcc ttc aag aac aat att cca gta 963
Asp Glu Ser Ser Tyr Leu Tyr Trp Ala Phe Lys Asn Asn Ile Pro Val
225 230 235
ttt tgc cca ggt tta aca gac ggc tca ctc gga gac atg cta tat ttt 1011
Phe Cys Pro Gly Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe
240 245 250
cat tct ttt cgc aat ccg ggt tta atc gtc gat gtt gtg caa gat ata 1059
His Ser Phe Arg Asn Pro Gly Leu Ile Val Asp Val Val Gln Asp Ile
255 260 265
aga gca gta aat ggc gag gct gtg cac gca gcg cct agg aaa aca ggc 1107
Arg Ala Val Asn Gly Glu Ala Val His Ala Ala Pro Arg Lys Thr Gly
270 275 280
atg att ata ctc ggt gga ggg ttg cct aag cac cac atc tgc aac gca 1155
Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala
285 290 295 300
aac atg atg aga aat ggc gcc gat tat gct gtt ttc atc aac act gcc 1203
Asn Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr Ala
305 310 315
gaa gag ttt gac ggc agt gat tct ggt gct cgc ccc gat gag gct att 1251
Glu Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Ile
320 325 330
tca tgg ggc aaa att agc gga tct gct aag act gtg aag gtg cat tgt 1299
Ser Trp Gly Lys Ile Ser Gly Ser Ala Lys Thr Val Lys Val His Cys
335 340 345
gat gcc acg ata gct ttc cct cta cta gtc gct gag aca ttt gca gca 1347
Asp Ala Thr Ile Ala Phe Pro Leu Leu Val Ala Glu Thr Phe Ala Ala
350 355 360
aaa aga gaa aaa gag agg aag agc tgt taaaactttt ttgattgttg 1394
Lys Arg Glu Lys Glu Arg Lys Ser Cys
365 370
aaaaatctgt gttatacaag tctcgaaatg cattttagta attgacttga tcttatcatt 1454
tcaatgtgtt atctttgaaa atgttggtaa tgaaacatct cacctcttct atacaacatt 1514
gttgatccat tgtactccgt atcttgtaat tttggaaaaa aaaaaccgtc tattgttacg 1574
agagagtaca tttttgaggt aaaaatatag gatttttgtg cgatgcaaat gctggttatt 1634
cccttgaaaa aaaaaaaaaa aaaaaa 1660
<210> 10
<211> 373
<212> PRT
<213> Dianthus caryophyllus
<400> 10
Met Glu Asp Ala Asn His Asp Ser Val Ala Ser Ala His Ser Ala Ala
1 5 10 15
Phe Lys Lys Ser Glu Asn Leu Glu Gly Lys Ser Val Lys Ile Glu Gly
20 25 30
Tyr Asp Phe Asn Gln Gly Val Asn Tyr Ser Lys Leu Leu Gln Ser Phe
35 40 45
Ala Ser Asn Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile Glu Val
50 55 60
Val Asn His Met Leu Asp Trp Ser Leu Ala Asp Glu Ala Pro Val Asp
65 70 75 80
Asp Cys Ser Glu Glu Glu Arg Asp Pro Lys Phe Arg Glu Ser Val Lys
85 90 95
Cys Lys Val Phe Leu Gly Phe Thr Ser Asn Leu Ile Ser Ser Gly Val
100 105 110
Arg Asp Thr Ile Arg Tyr Leu Val Gln His His Met Val Asp Val Ile
115 120 125
Val Thr Thr Thr Gly Gly Ile Glu Glu Asp Leu Ile Lys Gly Arg Ser
130 135 140
Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe Ala Leu Pro Gly
145 150 155 160
Ala Gln Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val
165 170 175
Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Leu
180 185 190
Asp Lys Met Leu Glu Glu Gln Ile Ser Glu Lys Ile Leu Trp Thr Pro
195 200 205
Ser Lys Leu Ile Gly Arg Leu Gly Arg Glu Ile Asn Asp Glu Ser Ser
210 215 220
Tyr Leu Tyr Trp Ala Phe Lys Asn Asn Ile Pro Val Phe Cys Pro Gly
225 230 235 240
Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe Arg
245 250 255
Asn Pro Gly Leu Ile Val Asp Val Val Gln Asp Ile Arg Ala Val Asn
260 265 270
Gly Glu Ala Val His Ala Ala Pro Arg Lys Thr Gly Met Ile Ile Leu
275 280 285
Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg
290 295 300
Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr Ala Glu Glu Phe Asp
305 310 315 320
Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Ile Ser Trp Gly Lys
325 330 335
Ile Ser Gly Ser Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile
340 345 350
Ala Phe Pro Leu Leu Val Ala Glu Thr Phe Ala Ala Lys Arg Glu Lys
355 360 365
Glu Arg Lys Ser Cys
370
<210> 11
<211> 780
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (43)..(522)
<400> 11
aaagaatcct agagagagaa agggaatcct agagagagaa gc atg tcg gac gaa 54
Met Ser Asp Glu
1
gaa cac cat ttt gag tca aag gca gat gct ggt gcc tca aaa act ttc 102
Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser Lys Thr Phe
5 10 15 20
cca cag caa gct gga acc atc cgt aag aat ggt tac atc gtt atc aaa 150
Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val Ile Lys
25 30 35
ggc cgt ccc tgc aag gtt gtt gag gtc tcc act tca aaa act gga aaa 198
Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys
40 45 50
cac gga cat gct aaa tgt cac ttt gtg gca att gac att ttc aat gga 246
His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe Asn Gly
55 60 65
aag aaa ctg gaa gat atc gtt ccg tcc tcc cac aat tgt gat gtg cca 294
Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp Val Pro
70 75 80
cat gtt aac cgt acc gac tat cag ctg att gat atc tct gaa gat ggt 342
His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly
85 90 95 100
ttt gtc tca ctt ctt act gaa agt gga aac acc aag gat gac ctc agg 390
Phe Val Ser Leu Leu Thr Glu Ser Gly Asn Thr Lys Asp Asp Leu Arg
105 110 115
ctt ccc acc gat gaa aat ctg ctg aag cag gtt aaa gat ggg ttc cag 438
Leu Pro Thr Asp Glu Asn Leu Leu Lys Gln Val Lys Asp Gly Phe Gln
120 125 130
gaa gga aag gat ctt gtg gtg tct gtt atg tct gcg atg ggc gaa gag 486
Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly Glu Glu
135 140 145
cag att aac gcc gtt aag gat gtt ggt acc aag aat tagttatgtc 532
Gln Ile Asn Ala Val Lys Asp Val Gly Thr Lys Asn
150 155 160
atggcagcat aatcactgcc aaagctttaa gacattatca tatcctaatg tggtactttg 592
atatcactag attataaact gtgttatttg cactgttcaa aacaaaagaa agaaaactgc 652
tgttatggct agagaaagta ttggctttga gcttttgaca gcacagttga actatgtgaa 712
aattctactt tttttttttt gggtaaaata ctgctcgttt aatgttttgc aaaaaaaaaa 772
aaaaaaaa 780
<210> 12
<211> 160
<212> PRT
<213> Lycopersicon esculentum
<400> 12
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Phe Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Ser Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Glu Asn Leu Leu Lys Gln Val Lys
115 120 125
Asp Gly Phe Gln Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Asn Ala Val Lys Asp Val Gly Thr Lys Asn
145 150 155 160
<210> 13
<211> 812
<212> DNA
<213> Dianthus caryophyllus
<220>
<221> CDS
<222> (67)..(546)
<400> 13
ctcttttaca tcaatcgaaa aaaaattagg gttcttattt tagagtgaga ggcgaaaaat 60
cgaacg atg tcg gac gac gat cac cat ttc gag tca tcg gcc gac gcc 108
Met Ser Asp Asp Asp His His Phe Glu Ser Ser Ala Asp Ala
1 5 10
gga gca tcc aag act tac cct caa caa gct ggt aca atc cgc aag agc 156
Gly Ala Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Ser
15 20 25 30
ggt cac atc gtc atc aaa aat cgc cct tgc aag gtg gtt gag gtt tct 204
Gly His Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser
35 40 45
acc tcc aag act ggc aag cac ggt cat gcc aaa tgt cac ttt gtt gcc 252
Thr Ser Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala
50 55 60
att gac att ttc aac ggc aag aag ctg gaa gat att gtc ccc tca tcc 300
Ile Asp Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser
65 70 75
cac aat tgt gat gtt cca cat gtc aac cgt gtc gac tac cag ctg ctt 348
His Asn Cys Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Leu
80 85 90
gat atc act gaa gat ggc ttt gtt agt ctg ctg act gac agt ggt gac 396
Asp Ile Thr Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Asp
95 100 105 110
acc aag gat gat ctg aag ctt cct gct gat gag gcc ctt gtg aag cag 444
Thr Lys Asp Asp Leu Lys Leu Pro Ala Asp Glu Ala Leu Val Lys Gln
115 120 125
atg aag gag gga ttt gag gcg ggg aaa gac ttg att ctg tca gtc atg 492
Met Lys Glu Gly Phe Glu Ala Gly Lys Asp Leu Ile Leu Ser Val Met
130 135 140
tgt gca atg gga gaa gag cag atc tgc gcc gtc aag gac gtt agt ggt 540
Cys Ala Met Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Val Ser Gly
145 150 155
ggc aag tagaagcttt tgatgaatcc aatactacgc ggtgcagttg aagcaatagt 596
Gly Lys
160
aatctcgaga acattctgaa ccttatatgt tgaattgatg gtgcttagtt tgttttggaa 656
atctctttgc aattaagttg taccaaatca atggatgtaa tgtcttgaat ttgttttatt 716
tttgttttga tgtttgctgt gattgcatta tgcattgtta tgagttatga cctgttataa 776
cacaaggttt tggtaaaaaa aaaaaaaaaa aaaaaa 812
<210> 14
<211> 160
<212> PRT
<213> Dianthus caryophyllus
<400> 14
Met Ser Asp Asp Asp His His Phe Glu Ser Ser Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Ser Gly His
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Leu Asp Ile
85 90 95
Thr Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Asp Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Ala Asp Glu Ala Leu Val Lys Gln Met Lys
115 120 125
Glu Gly Phe Glu Ala Gly Lys Asp Leu Ile Leu Ser Val Met Cys Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Val Ser Gly Gly Lys
145 150 155 160
<210> 15
<211> 702
<212> DNA
<213> Arabidopsis thaliana
<220>
<221> CDS
<222> (56)..(529)
<400> 15
ctgttaccaa aaaatctgta ccgcaaaatc ctcgtcgaag ctcgctgctg caacc atg 58
Met
1
tcc gac gag gag cat cac ttt gag tcc agt gac gcc gga gcg tcc aaa 106
Ser Asp Glu Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser Lys
5 10 15
acc tac cct caa caa gct gga acc atc cgt aag aat ggt tac atc gtc 154
Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val
20 25 30
atc aaa aat cgt ccc tgc aag gtt gtt gag gtt tca acc tcg aag act 202
Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr
35 40 45
ggc aag cat ggt cat gct aaa tgt cat ttt gta gct att gat atc ttc 250
Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe
50 55 60 65
acc agc aag aaa ctc gaa gat att gtt cct tct tcc cac aat tgt gat 298
Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
70 75 80
gtt cct cat gtc aac cgt act gat tat cag ctg att gac att tct gaa 346
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
85 90 95
gat gga tat gtc agt ttg ttg act gat aac ggt agt acc aag gat gac 394
Asp Gly Tyr Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys Asp Asp
100 105 110
ctt aag ctc cct aat gat gac act ctg ctc caa cag atc aag agt ggg 442
Leu Lys Leu Pro Asn Asp Asp Thr Leu Leu Gln Gln Ile Lys Ser Gly
115 120 125
ttt gat gat gga aaa gat cta gtg gtg agt gta atg tca gct atg gga 490
Phe Asp Asp Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly
130 135 140 145
gag gaa cag atc aat gct ctt aag gac atc ggt ccc aag tgagactaac 539
Glu Glu Gln Ile Asn Ala Leu Lys Asp Ile Gly Pro Lys
150 155
aaagcctccc ctttgttatg agattcttct tcttctgtag gcttccatta ctcgtcggag 599
attatcttgt ttttgggtta ctcctatttt ggatatttaa acttttgtta ataatgccat 659
cttcttcaac cttttccttc tagatggttt ttatacttct tct 702
<210> 16
<211> 158
<212> PRT
<213> Arabidopsis thaliana
<400> 16
Met Ser Asp Glu Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile
20 25 30
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
Glu Asp Gly Tyr Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Asn Asp Asp Thr Leu Leu Gln Gln Ile Lys Ser
115 120 125
Gly Phe Asp Asp Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met
130 135 140
Gly Glu Glu Gln Ile Asn Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 17
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 17
aaarrycgmc cytgcaaggt 20
<210> 18
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 18
aatacgactc actatag 17
<210> 19
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<220>
<221> modified_base
<222> (6)
<223> a, t, c, g, unknown or other
<400> 19
tcyttnccyt cmkctaahcc 20
<210> 20
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 20
attaaccctc actaaag 17
<210> 21
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 21
ctgttaccaa aaaatctgta cc 22
<210> 22
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 22
agaagaagta taaaaaccat c 21
<210> 23
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 23
aaagaatcct agagagagaa agg 23
<210> 24
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 24
ttttacatca atcgaaaa 18
<210> 25
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 25
accaaaacct gtgttataac tcc 23
<210> 26
<211> 581
<212> DNA
<213> Arabidopsis thaliana
<220>
<221> CDS
<222> (1)..(579)
<400> 26
ggt ggt gtt gag gaa gat ctc ata aaa tgc ctt gca cct aca ttt aaa 48
Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro Thr Phe Lys
1 5 10 15
ggt gat ttc tct cta cct gga gct tat tta agg tca aag gga ttg aac 96
Gly Asp Phe Ser Leu Pro Gly Ala Tyr Leu Arg Ser Lys Gly Leu Asn
20 25 30
cga att ggg aat ttg ctg gtt cct aat gat aac tac tgc aag ttt gag 144
Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu
35 40 45
gat tgg atc att ccc atc ttt gac gag atg ttg aag gaa cag aaa gaa 192
Asp Trp Ile Ile Pro Ile Phe Asp Glu Met Leu Lys Glu Gln Lys Glu
50 55 60
gag aat gtg ttg tgg act cct tct aaa ctg tta gca cgg ctg gga aaa 240
Glu Asn Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg Leu Gly Lys
65 70 75 80
gaa atc aac aat gag agt tca tac ctt tat tgg gca tac aag atg aat 288
Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys Met Asn
85 90 95
att cca gta ttc tgc cca ggg tta aca gat ggc tct ctt agg gat atg 336
Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly Ser Leu Arg Asp Met
100 105 110
ctg tat ttt cac tct ttt cgt acc tct ggc ctc atc atc gat gta gta 384
Leu Tyr Phe His Ser Phe Arg Thr Ser Gly Leu Ile Ile Asp Val Val
115 120 125
caa gat atc aga gct atg aac ggc gaa gct gtc cat gca aat cct aaa 432
Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His Ala Asn Pro Lys
130 135 140
aag aca ggg atg ata atc ctt gga ggg ggc ttg cca aag cac cac ata 480
Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile
145 150 155 160
tgt aat gcc aat atg atg cgc aat ggt gca gat tac gct gta ttt ata 528
Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile
165 170 175
aac acc ggg caa gaa ttt gat ggg agc gac tcg ggt gca cgc cct gat 576
Asn Thr Gly Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp
180 185 190
gaa gc 581
Glu
<210> 27
<211> 523
<212> DNA
<213> Dianthus caryophyllus
<220>
<221> CDS
<222> (1)..(522)
<400> 27
mga aga tcc atc aag tgc ctt gca ccc act ttc aaa ggc gat ttt gcc 48
Arg Arg Ser Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe Ala
1 5 10 15
tta cca gga gct caa tta cgc tcc aaa ggg ttg aat cga att ggt aat 96
Leu Pro Gly Ala Gln Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn
20 25 30
ctg ttg gtt ccg aat gat aac tac tgt aaa ttt gag gat tgg atc att 144
Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile
35 40 45
cca att tta gat aag atg ttg gaa gag caa att tca gag aaa atc tta 192
Pro Ile Leu Asp Lys Met Leu Glu Glu Gln Ile Ser Glu Lys Ile Leu
50 55 60
tgg aca cca tcg aag ttg att ggt cga tta gga aga gaa ata aac gat 240
Trp Thr Pro Ser Lys Leu Ile Gly Arg Leu Gly Arg Glu Ile Asn Asp
65 70 75 80
gag agt tca tac ctt tac tgg gcc ttc aag aac aat att cca gta ttt 288
Glu Ser Ser Tyr Leu Tyr Trp Ala Phe Lys Asn Asn Ile Pro Val Phe
85 90 95
tgc cca ggt tta aca gac ggc tca ctc gga gac atg cta tat ttt cat 336
Cys Pro Gly Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His
100 105 110
tct ttt cgc aat ccg ggt tta atc atc gat gtt gtg caa gat ata aga 384
Ser Phe Arg Asn Pro Gly Leu Ile Ile Asp Val Val Gln Asp Ile Arg
115 120 125
gca gta aat ggc gag gct gtg cac gca gcg cct agg aaa aca ggc atg 432
Ala Val Asn Gly Glu Ala Val His Ala Ala Pro Arg Lys Thr Gly Met
130 135 140
att ata ctc ggt gga ggg ttg cct aag cac cac atc tgc aac gca aac 480
Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn
145 150 155 160
atg atg aga aat ggc gcc gat tat gct gtt ttc atc aac acc g 523
Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr
165 170
<210> 28
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 28
ttgargaaga tycatmaart gcct 24
<210> 29
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 29
ccatcaaayt cytgkgcrgt gtt 23
<210> 30
<211> 484
<212> DNA
<213> Arabidopsis thaliana
<220>
<221> CDS
<222> (2)..(112)
<400> 30
t gca cgc cct gat gaa gct gtg tct tgg ggt aaa att agg ggt tct gct 49
Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser Ala
1 5 10 15
aaa acc gtt aag gtc tgc ttt tta att tct tca cat cct aat tta tat 97
Lys Thr Val Lys Val Cys Phe Leu Ile Ser Ser His Pro Asn Leu Tyr
20 25 30
ctc act cag tgg ttt tgagtacata tttaatattg gatcattctt gcaggtatac 152
Leu Thr Gln Trp Phe
35
tgtgatgcta ccatagcctt cccattgttg gttgcagaaa catttgccac aaagagagac 212
caaacctgtg agtctaagac ttaagaactg actggtcgtt ttggccatgg attcttaaag 272
atcgttgctt tttgatttta cactggagtg accatataac actccacatt gatgtggctg 332
tgacgcgaat tgtcttcttg cgaattgtac tttagtttct ctcaacctaa aatgatttgc 392
agattgtgtt ttcgtttaaa acacaagagt cttgtagtca ataatccttt gccttataaa 452
attattcagt tccaacaaaa aaaaaaaaaa aa 484
<210> 31
<211> 559
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (1)..(156)
<220>
<221> modified_base
<222> (542)
<223> a, t, c, g, unknown or other
<400> 31
ggt gct cgt cct gat gaa gct gta tca tgg gga aag ata cgt ggt ggt 48
Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Gly
1 5 10 15
gcc aag act gtg aag gtg cat tgt gat gca acc att gca ttt ccc ata 96
Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile Ala Phe Pro Ile
20 25 30
tta gta gct gag aca ttt gca gct aag agt aag gaa ttc tcc cag ata 144
Leu Val Ala Glu Thr Phe Ala Ala Lys Ser Lys Glu Phe Ser Gln Ile
35 40 45
agg tgc caa gtt tgaacattga ggaagctgtc cttccgacca cacatatgaa 196
Arg Cys Gln Val
50
ttgctagctt ttgaagccaa cttgctagtg tgcagcacca tttattctgc aaaactgact 256
agagagcagg gtatattcct ctaccccgag ttagacgaca tcctgtatgg ttcaaattaa 316
ttatttttct ccccttcaca ccatgttatt tagttctctt cctcttcgaa agtgaagagc 376
ttagatgttc ataggttttg aattatgttg gaggttggtg ataactgact agtcctctta 436
ccatatagat aatgtatcct tgtactatga gattttgggt gtgtttgata ccaaggaaaa 496
atgtttattt ggaaaacaat tggattttta atttaaaaaa aattgnttaa aaaaaaaaaa 556
aaa 559
<210> 32
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide fragment
<400> 32
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 33
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 33
gagctcaaga ataacatctc ataagaaac 29
<210> 34
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 34
ctcgagtgct cacttctctc tcttagg 27
<210> 35
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 35
Cys Asn Asp Asp Thr Leu Leu Gln Gln Ile Lys Ser
1 5 10
<210> 36
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 36
Cys Thr Asp Asp Gly Leu Thr Ala Gln Met Arg Leu
1 5 10
<210> 37
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 37
Cys Thr Asp Glu Ala Leu Leu Thr Gln Leu Lys Asn
1 5 10
<210> 38
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 38
aagcttgatc gtggtcaact tcctctgtta cc 32
<210> 39
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 39
gagctcagaa gaagtataaa aaccatc 27
<210> 40
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 40
ctcgagtgct cacttctctc tcttagg 27
<210> 41
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 41
gagctcaaga ataacatctc ataagaaac 29
<210> 42
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 42
ctcgagctaa actccattcg ctgacttcgc 30
<210> 43
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 43
gagctctagt aaatataaga gtgtcttgc 29
<210> 44
<211> 353
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Fungi sequence
<400> 44
Met Ala Asp Asn Gln Ile Pro Ser Ser Val Ala Asp Ala Val Leu Val
1 5 10 15
Lys Ser Ile Glu Met Pro Glu Gly Ser Gln Lys Val Glu Glu Leu Asp
20 25 30
Phe Asn Lys Phe Lys Gly Arg Pro Ile Thr Val Asp Asp Leu Leu Gln
35 40 45
Gly Met Lys His Met Gly Phe Gln Ala Ser Ser Met Cys Glu Ala Val
50 55 60
Arg Ile Ile Asn Glu Met Arg Ala Tyr Arg Asp Pro Thr Thr Ser Glu
65 70 75 80
Lys Thr Thr Ile Phe Leu Gly Tyr Thr Ser Asn Leu Ile Ser Ser Gly
85 90 95
Leu Arg Gly Thr Leu Arg Tyr Leu Val Gln His Lys His Val Ser Ala
100 105 110
Ile Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Phe Ile Lys Cys Leu
115 120 125
Gly Asp Thr Tyr Met Ser Ser Phe Ser Ala Val Gly Ala Asp Leu Arg
130 135 140
Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Val Val Pro Asn Ser Asn
145 150 155 160
Tyr Cys Ala Phe Glu Asp Trp Val Val Pro Ile Leu Asp Lys Met Leu
165 170 175
Glu Glu Gln Glu Ala Ser Arg Gly Thr Glu Asn Glu Ile Asn Trp Thr
180 185 190
Pro Ser Lys Val Ile His Arg Leu Gly Lys Glu Ile Asn Asp Glu Arg
195 200 205
Ser Val Tyr Tyr Trp Ala Trp Lys Asn Asp Ile Pro Val Phe Cys Pro
210 215 220
Ala Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Thr Phe
225 230 235 240
Lys Ala Ser Pro Glu Gln Leu Arg Ile Asp Ile Val Glu Asp Ile Arg
245 250 255
Lys Ile Asn Thr Ile Ala Val Arg Ala Lys Arg Ala Gly Met Ile Ile
260 265 270
Leu Gly Gly Gly Ile Val Lys His His Ile Ala Asn Ala Cys Leu Met
275 280 285
Arg Asn Gly Ala Glu Ser Ala Val Tyr Ile Asn Thr Ala Gln Glu Phe
290 295 300
Asp Gly Ser Asp Ala Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly
305 310 315 320
Lys Ile Lys Val Gly Ala Asp Ala Val Lys Val Tyr Met Glu Ala Thr
325 330 335
Ala Ala Phe Pro Phe Ile Val Ala Asn Thr Phe Ala Lys Glu Asp Gly
340 345 350
Leu
<210> 45
<211> 387
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Yeast sequence
<400> 45
Met Ser Asp Ile Asn Glu Lys Leu Pro Glu Leu Leu Gln Asp Ala Val
1 5 10 15
Leu Lys Ala Ser Val Pro Ile Pro Asp Asp Phe Val Lys Val Gln Gly
20 25 30
Ile Asp Tyr Ser Lys Pro Glu Ala Thr Asn Met Arg Ala Thr Asp Leu
35 40 45
Ile Glu Ala Met Lys Thr Met Gly Phe Gln Ala Ser Ser Val Gly Thr
50 55 60
Ala Cys Glu Ile Ile Asp Ser Met Arg Ser Trp Arg Gly Lys His Ile
65 70 75 80
Asp Glu Leu Asp Asp His Glu Lys Lys Gly Cys Phe Asp Glu Glu Gly
85 90 95
Tyr Gln Lys Thr Thr Ile Phe Met Gly Tyr Thr Ser Asn Leu Ile Ser
100 105 110
Ser Gly Val Arg Glu Thr Leu Arg Tyr Leu Val Gln His Lys Met Val
115 120 125
Asp Ala Val Val Thr Ser Ala Gly Gly Val Glu Glu Asp Leu Ile Lys
130 135 140
Cys Leu Ala Pro Thr Tyr Leu Gly Glu Phe Ala Leu Lys Gly Lys Ser
145 150 155 160
Leu Arg Asp Gln Gly Met Asn Arg Ile Gly Asn Leu Leu Val Pro Asn
165 170 175
Asp Asn Tyr Cys Lys Phe Glu Glu Trp Ile Val Pro Ile Leu Asp Lys
180 185 190
Met Leu Glu Glu Gln Asp Glu Tyr Val Lys Lys His Gly Ala Asp Cys
195 200 205
Leu Glu Ala Asn Gln Asp Val Asp Ser Pro Ile Trp Thr Pro Ser Lys
210 215 220
Met Ile Asp Arg Phe Gly Lys Glu Ile Asn Asp Glu Ser Ser Val Leu
225 230 235 240
Tyr Trp Ala His Lys Asn Lys Ile Pro Ile Phe Cys Pro Ser Leu Thr
245 250 255
Asp Gly Ser Ile Gly Asp Met Leu Phe Phe His Thr Phe Lys Ala Ser
260 265 270
Pro Lys Gln Leu Arg Val Asp Ile Val Gly Asp Ile Arg Lys Ile Asn
275 280 285
Ser Met Ser Met Ala Ala Tyr Arg Ala Gly Met Ile Ile Leu Gly Gly
290 295 300
Gly Leu Ile Lys His His Ile Ala Asn Ala Cys Leu Met Arg Asn Gly
305 310 315 320
Ala Asp Tyr Ala Val Tyr Ile Asn Thr Gly Gln Glu Tyr Asp Gly Ser
325 330 335
Asp Ala Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Lys
340 345 350
Ala Glu Ala Lys Ser Val Lys Leu Phe Ala Asp Val Thr Thr Val Leu
355 360 365
Pro Leu Ile Val Ala Ala Thr Phe Ala Ser Gly Lys Pro Ile Lys Lys
370 375 380
Val Lys Asn
385
<210> 46
<211> 370
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Archaebacterial
sequence
<400> 46
Met Arg Asp Ile Lys Asp Asn Pro Ile Arg Arg Gly Ile Ala Glu Gln
1 5 10 15
Ser Glu Ala Met His Pro Gly Tyr Thr Asn Arg Ala Lys Pro Tyr Gly
20 25 30
Cys Lys Arg Asp Pro Lys Asp Ile Val Leu Lys Glu Ser Glu Asp Ile
35 40 45
Glu Gly Ile Ala Ile Glu Gly Pro Trp Leu Glu Asp Asp Ile Ser Leu
50 55 60
Glu Glu Ile Ile Lys Lys Tyr Tyr Leu Lys Ile Gly Phe Gln Ala Ser
65 70 75 80
His Ile Gly Lys Ala Ile Lys Ile Trp Lys His Ile Glu Glu Lys Arg
85 90 95
Lys Lys Gly Asp Glu Ile Thr Val Phe Phe Gly Tyr Thr Ser Asn Ile
100 105 110
Val Ser Ser Gly Leu Arg Glu Ile Ile Ala Tyr Leu Val Lys His Lys
115 120 125
Lys Ile Asp Ile Ile Val Thr Thr Ala Gly Gly Val Glu Glu Asp Phe
130 135 140
Ile Lys Cys Leu Lys Pro Phe Ile Leu Gly Asp Trp Glu Val Asp Gly
145 150 155 160
Lys Met Leu Arg Glu Lys Gly Ile Asn Arg Ile Gly Asn Ile Phe Val
165 170 175
Pro Asn Asp Arg Tyr Ile Ala Phe Glu Glu Tyr Met Met Glu Phe Phe
180 185 190
Glu Glu Ile Leu Asn Leu Gln Arg Glu Thr Gly Lys Ile Ile Thr Ala
195 200 205
Ser Glu Phe Cys Tyr Lys Leu Gly Glu Phe Met Asp Lys Lys Leu Lys
210 215 220
Ser Lys Glu Lys Glu Lys Ser Ile Leu Tyr Trp Ala Tyr Lys Asn Asn
225 230 235 240
Ile Pro Ile Phe Cys Pro Ala Ile Thr Asp Gly Ser Ile Gly Asp Met
245 250 255
Leu Tyr Phe Phe Lys Lys Tyr Asn Lys Asp Glu Glu Leu Lys Ile Asp
260 265 270
Val Ala Asn Asp Ile Val Lys Leu Asn Asp Ile Ala Ile Asn Ser Lys
275 280 285
Glu Thr Ala Cys Ile Val Leu Gly Gly Ser Leu Pro Lys His Ser Ile
290 295 300
Ile Asn Ala Asn Leu Phe Arg Glu Gly Thr Asp Tyr Ala Ile Tyr Val
305 310 315 320
Thr Thr Ala Leu Pro Trp Asp Gly Ser Leu Ser Gly Ala Pro Pro Glu
325 330 335
Glu Gly Val Ser Trp Gly Lys Ile Gly Ala Lys Ala Asp Tyr Val Glu
340 345 350
Ile Trp Gly Asp Ala Thr Ile Ile Phe Pro Leu Leu Val Tyr Cys Val
355 360 365
Met Lys
370
<210> 47
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<220>
<221> modified_base
<222> (3)
<223> a, t, c, g, unknown or other
<220>
<221> modified_base
<222> (15)
<223> a, t, c, g, unknown or other
<400> 47
gcngarttyg ayggntccga yca 23
<210> 48
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 48
ccgagctcct gttaccaaaa aatctgtacc 30
<210> 49
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 49
acctcgagcg gccgcagaag aagtataaaa accatc 36
<210> 50
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 50
cgtcgacgat atctcttttt atattcaaac 30
<210> 51
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 51
cgtctagaca ttgttttagg aaagttaaat ga 32
<210> 52
<211> 1235
<212> DNA
<213> Arabidopsis thaliana
<400> 52
accctagatc gctttcttca gtgttctata aaaactaaac tccattcgct gacttcgcaa 60
agaagaacac tttctctctg aaatctcaaa ttcatctctt ctcttccgat ttcgctgaat 120
catgtcagac gacgagcatc acttcgaatc cagcgacgcc ggagcttcta agacttatcc 180
tcaacaagcc ggtaacattc gtaaaggtgg tcacatcgtc atcaagggac gtccctgcaa 240
ggttttgtct ctgatttgat tattattgat tttagaggaa tcatcttcat ggattgtatt 300
aaagcagtgt tccgttacct gatcgttgtg aatttttgag gtttagtgat tctggattgt 360
gatctggtgt ttagtgttga gaaaaacctc tgtttttgaa gtttatggat ttatagggtt 420
tttaaatcta taatagggtt taattcaatt ggtgatatgt ggggtttatg atataggtgg 480
ttgaggtatc gacttcgaag actgggaagc atggtcacgc caagtgtcac tttgttgcca 540
ttgatatctt tacttctaag aagcttgaag atatcgttcc ttcttcccac aattgtgatg 600
tgagtcttgt gaatggatta gaaacgttat acaaagtcta taatttttga ctcacaacac 660
aaaactgttt cctttttatt ggcacaggtt ccacatgtga atcgtgttga ttatcagttg 720
attgatatct ctgaagatgg ctttgtatgt catcttcttt ttcactagtt cagctttgtg 780
ttttgtcttt gcccatatgg ttgaattaga gggttttgtt ctttgattac atttacaggt 840
tagtcttctt actgataatg gtagcactaa ggatgatctg aagctgccaa cagatgaagc 900
tttactcaca cagctcaaga atggatttga ggagggtaag gatattgttg tgtctgtcat 960
gtctgcaatg ggagaggagc agatgtgtgc tctcaaggaa gttggtccca agtaataata 1020
ataagtaagc attctctctt ttacagaggc tatgtattat caagtttgac agagtcaaat 1080
gttataagaa caaagtttgg tccttttttt tggtcttctt agtataattt aagcccacat 1140
gtgtttccca tgcaagacac tcttatattt actagtatat cttactattg gttttggttg 1200
tggagaagtt actgttgaca gttccaaacc tctac 1235
<210> 53
<211> 161
<212> PRT
<213> Mycosphaerella fijiensis
<400> 53
Gly Leu Asn Arg Ile Gly Asn Phe Leu Val Pro Asn Asp Asn Tyr Cys
1 5 10 15
Arg Phe Glu Asp Trp Val Met Pro Ile Leu Asp Thr Met Leu Glu Glu
20 25 30
Gln Glu Ala Cys Lys Gly Ser Gly Glu Ala Ile His Trp Thr Pro Ser
35 40 45
Lys Ile Ile Asn Arg Leu Gly Lys Glu Val Asn Asp Glu Ser Ser Val
50 55 60
Tyr Tyr Trp Ala Trp Lys Asn Asp Ile Pro Val Phe Cys Pro Ala Leu
65 70 75 80
Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Thr Phe Lys Ser
85 90 95
Ser Pro Gln Gln Leu Arg Val Asp Ile Val Glu Asp Ile Arg Lys Ile
100 105 110
Asn Thr Leu Ala Val Arg Ala Lys Arg Thr Gly Met Ile Ile Leu Gly
115 120 125
Gly Gly Ile Val Lys His His Ile Ala Asn Ala Asn Leu Met Arg Asn
130 135 140
Gly Ala Glu Ser Ala Val Tyr Ile Asn Thr Ala Glu Phe Asp Gly Ser
145 150 155 160
Asp
<210> 54
<211> 861
<212> DNA
<213> Arabidopsis sp.
<400> 54
tttttccctt ctcccaatct catcttctcc gaaaaccttt cttctctcaa atttctgtga 60
aaacatgtct gacgacgagc accactttga ggccagcgaa tccggagctt ccaagaccta 120
tcctcaatca gccggtaaca tccgtaaagg tggtcacatc gtcatcaaaa accgtccctg 180
caaggttgtt gaggtttcga cttccaaaac tggcaagcac ggtcacgcca aatgtcactt 240
tgttgctatt gatatcttca ctgctaagaa gcttgaagat attgttccat cttcccacaa 300
ttgtgatgtt ccacatgtga accgtgttga ttaccagttg attgatatca ctgaggatgg 360
cttcgtgagc cttctcactg acagtggtgg caccaaggat gatctcaagc ttcccaccga 420
tgatggtctc accgcccaga tgaggcttgg attcgatgag ggaaaggata ttgtggtgtc 480
tgtcatgtct tccatgggag aggagcagat ctgtgccgtc aaggaagttg gtggtggcaa 540
gtaaacaagt atcattcgat atattattac cagtttgaca acggacgtca atgttataag 600
aaccaaaaga tgtttttctt tttcctaatt tagacccttt gtgtgtgttt cttgttgcaa 660
gacaaccata tctattggtt ttggattgtt ggaaaagttt gtgttgaaac attcaaagtt 720
tcttatgaga tgttattctt aaaaccactt tttgtttgtt cactggatat gtttgttcat 780
gaagcttgtt ttaagcaact ctttacatga tattcattgc tatttgcacg attcaagagt 840
gaaatataca ttttatttaa c 861
<210> 55
<211> 159
<212> PRT
<213> Arabidopsis thaliana
<400> 55
Met Ser Asp Asp Glu His His Phe Glu Ala Ser Glu Ser Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Ser Ala Gly Asn Ile Arg Lys Gly Gly His Ile
20 25 30
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Ile Asp Ile Thr
85 90 95
Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Gly Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Thr Asp Asp Gly Leu Thr Ala Gln Met Arg Leu
115 120 125
Gly Phe Asp Glu Gly Lys Asp Ile Val Val Ser Val Met Ser Ser Met
130 135 140
Gly Glu Glu Gln Ile Cys Ala Val Lys Glu Val Gly Gly Gly Lys
145 150 155
<210> 56
<211> 698
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (52)..(528)
<400> 56
cttcctgaat ttttctcctt ctccttctcc gttcaatcga atttttcagc c atg tct 57
Met Ser
1
gac gag gag cat caa ttt gag tct aag gct gat gcc gga gca tca aaa 105
Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser Lys
5 10 15
act tac cct caa caa gct ggt act att cgt aag aac ggt tat atc gtc 153
Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val
20 25 30
atc aaa ggc cgt cca tgc aag gtt gtg gaa gtc tct aca tcc aaa act 201
Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr
35 40 45 50
ggc aag cac ggt cac gcc aaa tgt cat ttc gtt gct att gac atc ttc 249
Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe
55 60 65
act ggg aag aag ctt gag gat att gtc ccc tct tca cac aat tgt gat 297
Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
70 75 80
gtg ccc cat gtt aat cgt aca gat tat cag ctt att gac atc tct gaa 345
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
85 90 95
gat gga ttt gtg agt ctg ctt act gac aat ggt aac acc aag gat gac 393
Asp Gly Phe Val Ser Leu Leu Thr Asp Asn Gly Asn Thr Lys Asp Asp
100 105 110
ctc agg ctt cct act gat gaa aat ctg ctt tca ctg atc aag gac ggg 441
Leu Arg Leu Pro Thr Asp Glu Asn Leu Leu Ser Leu Ile Lys Asp Gly
115 120 125 130
ttt gcc gag ggt aag gac ctc gtt gtg tct gtt atg tca gct atg ggt 489
Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly
135 140 145
gag gaa cag att aat gct ttg aag gat att ggc ccc aag tgatctcttg 538
Glu Glu Gln Ile Asn Ala Leu Lys Asp Ile Gly Pro Lys
150 155
attggatgga ttgcttgacg cgatggttct ttacgacctt gagtgagata gatatttata 598
gtcatggaaa aaaattgtga tcttatggaa tattcgtatc atgatttatg gaccattgtg 658
agttagattt ttatttatgt tgttttaaat tgtggtattc 698
<210> 57
<211> 159
<212> PRT
<213> Lycopersicon esculentum
<400> 57
Met Ser Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Glu Asn Leu Leu Ser Leu Ile Lys
115 120 125
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Asn Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 58
<211> 158
<212> PRT
<213> Arabidopsis thaliana
<400> 58
Met Ser Asp Glu Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile
20 25 30
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
Glu Asp Gly Tyr Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Asn Asp Asp Thr Leu Leu Gln Gln Ile Lys Ser
115 120 125
Gly Phe Asp Asp Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met
130 135 140
Gly Glu Glu Gln Ile Asn Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 59
<211> 159
<212> PRT
<213> Arabidopsis thaliana
<400> 59
Met Ser Asp Asp Glu His His Phe Glu Ala Ser Glu Ser Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Ser Ala Gly Asn Ile Arg Lys Gly Gly His Ile
20 25 30
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Ile Asp Ile Thr
85 90 95
Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Gly Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Thr Asp Asp Gly Leu Thr Ala Gln Met Arg Leu
115 120 125
Gly Phe Asp Glu Gly Lys Asp Ile Val Val Ser Val Met Ser Ser Met
130 135 140
Gly Glu Glu Gln Ile Cys Ala Val Lys Glu Val Gly Gly Gly Lys
145 150 155
<210> 60
<211> 158
<212> PRT
<213> Arabidopsis thaliana
<400> 60
Met Ser Asp Asp Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Gln Ala Gly Asn Ile Arg Lys Gly Gly His Ile
20 25 30
Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Thr Asp Glu Ala Leu Leu Thr Gln Leu Lys Asn
115 120 125
Gly Phe Glu Glu Gly Lys Asp Ile Val Val Ser Val Met Ser Ala Met
130 135 140
Gly Glu Glu Gln Met Cys Ala Leu Lys Glu Val Gly Pro Lys
145 150 155
<210> 61
<211> 477
<212> DNA
<213> Arabidopsis thaliana
<400> 61
atgtccgacg aggagcatca ctttgagtcc agtgacgccg gagcgtccaa aacctaccct 60
caacaagctg gaaccatccg taagaatggt tacatcgtca tcaaaaatcg tccctgcaag 120
gttgttgagg tttcaacctc gaagactggc aagcatggtc atgctaaatg tcattttgta 180
gctattgata tcttcaccag caagaaactc gaagatattg ttccttcttc ccacaattgt 240
gatgttcctc atgtcaaccg tactgattat cagctgattg acatttctga agatggatat 300
gtcagtttgt tgactgataa cggtagtacc aaggatgacc ttaagctccc taatgatgac 360
actctgctcc aacagatcaa gagtgggttt gatgatggaa aagatctagt ggtgagtgtg 420
atgtcagcta tgggagagga acagatcaat gctcttaagg acatcggtcc caagtga 477
<210> 62
<211> 480
<212> DNA
<213> Arabidopsis thaliana
<400> 62
atgtctgacg acgagcacca ctttgaggcc agcgaatccg gagcttccaa gacctatcct 60
caatcagccg gtaacatccg taaaggtggt cacatcgtca tcaaaaaccg tccctgcaag 120
gttgttgagg tttcgacttc caaaactggc aagcacggtc acgccaaatg tcactttgtt 180
gctattgata tcttcactgc taagaagctt gaagatattg ttccatcttc ccacaattgt 240
gatgttccac atgtgaaccg tgttgattac cagttgattg atatcactga ggatggcttc 300
gtgagccttc tcactgacag tggtggcacc aaggatgatc tcaagcttcc caccgatgat 360
ggtctcaccg cccagatgag gcttggattc gatgagggaa aggatattgt ggtgtctgtc 420
atgtcttcca tgggagagga gcagatctgt gccgtcaagg aagttggtgg tggcaagtaa 480
<210> 63
<211> 477
<212> DNA
<213> Arabidopsis thaliana
<400> 63
atgtcagacg acgagcatca cttcgaatcc agcgacgccg gagcttctaa gacttatcct 60
caacaagccg gtaacattcg taaaggtggt cacatcgtca tcaagggacg tccctgcaag 120
gtggttgagg tatcgacttc gaagactggg aagcatggtc acgccaagtg tcactttgtt 180
gccattgata tctttacttc taagaagctt gaagatatcg ttccttcttc ccacaattgt 240
gatgttccac atgtgaatcg tgttgattat cagttgattg atatctctga agatggcttt 300
gttagtcttc ttactgataa tggtagcact aaggatgatc tgaagctgcc aacagatgaa 360
gctttactca cacagctcaa gaatggattt gaggagggta aggatattgt tgtgtctgtc 420
atgtctgcaa tgggagagga gcagatgtgt gctctcaagg aagttggtcc caagtaa 477
<210> 64
<211> 700
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (56)..(532)
<400> 64
aaatttctcc ttctccttaa tcctctccac cggcgaaccg gcgaagatca aaacg atg 58
Met
1
tcg gac gaa gag cac cac ttc gaa tcc aag gcc gat gcc gga gct tca 106
Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser
5 10 15
aag acg tat cct caa caa gct ggt act att cgt aaa ggt ggt cac atc 154
Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His Ile
20 25 30
gtc ata aaa aat cgt cct tgc aag gtg gtt gaa gtt tca act tcc aag 202
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
aca ggc aag cac ggt cat gct aaa tgt cac ttc gtg gca att gac att 250
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60 65
ttc act gga aag aaa ctt gag gat att gtt ccc tct tct cac aat tgt 298
Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
70 75 80
gat gtt cct cat gtg aat agg act gat tat caa ctt att gat atc tct 346
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
gag gat ggc ttt gtg agt ctg ttg act gaa aat ggt aac acc aag gat 394
Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp
100 105 110
gac ttg aga ctc cca act gat gat act ctt ctg gct cag gtc aaa gat 442
Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Ala Gln Val Lys Asp
115 120 125
ggt ttt gct gag ggg aaa gac ctg gtt cta tca gtg atg tct gcc atg 490
Gly Phe Ala Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala Met
130 135 140 145
gga gag gag cag att tgt ggt atc aag gac att ggc cct aag 532
Gly Glu Glu Gln Ile Cys Gly Ile Lys Asp Ile Gly Pro Lys
150 155
tagctgcaga tggtattggt gtatgtttac agagtttcta taaaagatgt attaagaacc 592
aaaacttctt tactttctct tgcagttgct ctatataact gccatttaac tattattata 652
tgtgttgtga ttagattctt gtctcactac agtatttcct ttactctg 700
<210> 65
<211> 159
<212> PRT
<213> Lycopersicon esculentum
<400> 65
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Ala Gln Val Lys
115 120 125
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Gly Ile Lys Asp Ile Gly Pro Lys
145 150 155
<210> 66
<211> 658
<212> DNA
<213> Brassica napus
<220>
<221> CDS
<222> (1)..(477)
<400> 66
atg tct gac gag gag cac cac ttc gag tcc agc gac gcc gga gct tcc 48
Met Ser Asp Glu Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser
1 5 10 15
aaa acc tac cct cag cag gct ggt aac atc cgc aag ggt ggt cac atc 96
Lys Thr Tyr Pro Gln Gln Ala Gly Asn Ile Arg Lys Gly Gly His Ile
20 25 30
gtc atc aag ggc cgt ccc tgc aag gtt gtt gag gtt tcg act tcg aag 144
Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
act ggg aag cac ggt cac gca aag tgt cac ttt gtt gct atc gac atc 192
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
ttc act gct aag aag ctc gag gat att gtt ccc tct tcc cac aat tgt 240
Phe Thr Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
gat gtt ccc cat gtg aac cgt att gac tac cag ttg att gat atc tct 288
Asp Val Pro His Val Asn Arg Ile Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
gag aat ggc ttt gtt agc ctt ttg acc gac agt ggt ggc acc aag gac 336
Glu Asn Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Gly Thr Lys Asp
100 105 110
gac ctc aag ctt ccc acc gat gat aat ctc agc gct ctg atg aag agt 384
Asp Leu Lys Leu Pro Thr Asp Asp Asn Leu Ser Ala Leu Met Lys Ser
115 120 125
gga ttc gag gag gga aag gat gtg gtg gtg tct gtc atg tct tcc atg 432
Gly Phe Glu Glu Gly Lys Asp Val Val Val Ser Val Met Ser Ser Met
130 135 140
gga gag gag cag atc tgt gcc gtc aag gaa gtt ggt ggt ggc aag 477
Gly Glu Glu Gln Ile Cys Ala Val Lys Glu Val Gly Gly Gly Lys
145 150 155
taaaacccat tctctgagag aggataatct tattaccagt ggtcaatgtt ataagaacaa 537
gaacttgttt tttttccttt ttctaattta gatcatttgt gttgtgtttc tttgttgcaa 597
gacaaccatt atctattatt ggttttggat tgtttaaaaa aaaaaaaaaa aaaaaaaaaa 657
a 658
<210> 67
<211> 159
<212> PRT
<213> Brassica napus
<400> 67
Met Ser Asp Glu Glu His His Phe Glu Ser Ser Asp Ala Gly Ala Ser
1 5 10 15
Lys Thr Tyr Pro Gln Gln Ala Gly Asn Ile Arg Lys Gly Gly His Ile
20 25 30
Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60
Phe Thr Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
65 70 75 80
Asp Val Pro His Val Asn Arg Ile Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
Glu Asn Gly Phe Val Ser Leu Leu Thr Asp Ser Gly Gly Thr Lys Asp
100 105 110
Asp Leu Lys Leu Pro Thr Asp Asp Asn Leu Ser Ala Leu Met Lys Ser
115 120 125
Gly Phe Glu Glu Gly Lys Asp Val Val Val Ser Val Met Ser Ser Met
130 135 140
Gly Glu Glu Gln Ile Cys Ala Val Lys Glu Val Gly Gly Gly Lys
145 150 155
<210> 68
<211> 8
<212> PRT
<213> Mycosphaerella fijiensis
<400> 68
Gly Leu Asn Arg Ile Gly Asn Leu
1 5
<210> 69
<211> 8
<212> PRT
<213> Mycosphaerella fijiensis
<400> 69
Ala Glu Phe Asp Gly Ser Asp Gln
1 5
<210> 70
<211> 1335
<212> DNA
<213> Brassica napus
<220>
<221> CDS
<222> (57)..(1160)
<400> 70
cttgctagaa ccctaaaact ccctcccaaa actctccaca tcttccgaga aagaag atg 59
Met
1
gag gag gat cgt gtt ctc tcg tct gtc cac tca acg gtc ttc aag gaa 107
Glu Glu Asp Arg Val Leu Ser Ser Val His Ser Thr Val Phe Lys Glu
5 10 15
tcc gaa tcg ttg gaa gga aag tgc gac aaa atc gaa gga tac gat ttc 155
Ser Glu Ser Leu Glu Gly Lys Cys Asp Lys Ile Glu Gly Tyr Asp Phe
20 25 30
aac caa gga gta aac tac ccg aag ctc ctc cga tcc atg ctc aca acc 203
Asn Gln Gly Val Asn Tyr Pro Lys Leu Leu Arg Ser Met Leu Thr Thr
35 40 45
ggc ttc caa gcc tca aac ctc ggc gac gta att gat gtc gtt aat caa 251
Gly Phe Gln Ala Ser Asn Leu Gly Asp Val Ile Asp Val Val Asn Gln
50 55 60 65
atg cta gag tgg aga ctc tct gat gaa act ata gca cct gaa gac tgt 299
Met Leu Glu Trp Arg Leu Ser Asp Glu Thr Ile Ala Pro Glu Asp Cys
70 75 80
agt gaa gag gag aag gat cca gcg tat aga gag tcc gtg aag tgt aaa 347
Ser Glu Glu Glu Lys Asp Pro Ala Tyr Arg Glu Ser Val Lys Cys Lys
85 90 95
atc ttt cta ggc ttc act tcg aat ctt gtt tcc tct ggt gtt aga gag 395
Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg Glu
100 105 110
act att cga tac ctt gtt cag cat cat atg gtt gat gtt ata gtt act 443
Thr Ile Arg Tyr Leu Val Gln His His Met Val Asp Val Ile Val Thr
115 120 125
aca act ggt ggc gta gag gaa gat ctc atc aaa tgc ctt gct cct act 491
Thr Thr Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro Thr
130 135 140 145
ttc aaa ggt gat ttc tct cta ccg ggt gcg tat ctt cgg tca aag gga 539
Phe Lys Gly Asp Phe Ser Leu Pro Gly Ala Tyr Leu Arg Ser Lys Gly
150 155 160
ttg aac cgg atc ggg aac ttg ctt gtt cct aat gat aac tac tgc aag 587
Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys
165 170 175
ttt gag gat tgg atc att ccc atc ttt gac cag atg ttg aag gaa cag 635
Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln Met Leu Lys Glu Gln
180 185 190
aaa gaa gag agt gtg ttg tgg acg cct tct aaa ttg tta gcg cgg ctg 683
Lys Glu Glu Ser Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg Leu
195 200 205
ggg aaa gaa ata aat aat gag agt tca tat ctt tat tgg gca tac aag 731
Gly Lys Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys
210 215 220 225
atg aat att cca gtg ttc tgc cgg ggg tta acc gat ggc tct ctc ggt 779
Met Asn Ile Pro Val Phe Cys Arg Gly Leu Thr Asp Gly Ser Leu Gly
230 235 240
gat atg ttg tat ttt cac tca ttt cgt acc tct ggc ctt gtc atc gat 827
Asp Met Leu Tyr Phe His Ser Phe Arg Thr Ser Gly Leu Val Ile Asp
245 250 255
gtt gtg caa gat att aga gct atg aac ggt gaa gca gtc cat gcg act 875
Val Val Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His Ala Thr
260 265 270
cca aga aag aca ggg atg ata atc ctt gga ggg ggc ttg ccg aag cac 923
Pro Arg Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His
275 280 285
cac ata tgt aat gcc aac atg atg cgt aac ggt gcg gat tac gct gtg 971
His Ile Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp Tyr Ala Val
290 295 300 305
ttt atc aac acc ggg caa gag ttt gat gga agt gac tcg ggt gca cgc 1019
Phe Ile Asn Thr Gly Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg
310 315 320
cct gat gaa gca gtg tct tgg ggt aaa ata agg gga tct gct aaa act 1067
Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser Ala Lys Thr
325 330 335
gtc aag gtg tac tgt gat gct acc ata gcc ttc cct ttg ttg gtt gct 1115
Val Lys Val Tyr Cys Asp Ala Thr Ile Ala Phe Pro Leu Leu Val Ala
340 345 350
gaa aca ttt gcc tcc aag aga gaa caa agc tgt gag cac aag acc 1160
Glu Thr Phe Ala Ser Lys Arg Glu Gln Ser Cys Glu His Lys Thr
355 360 365
taagcccaag aaagcttacg tctcttttat cggtttgttc ttccatcttg ttgttgtacc 1220
ctttgtcctg ctttacataa cattcatctc taaaacaata ctacctcctt ttgacaaaaa 1280
ataaaaaaaa ttggaaaaat ggtttcacaa gaataaaaaa aaaaaaaaaa aaaaa 1335
<210> 71
<211> 368
<212> PRT
<213> Brassica napus
<400> 71
Met Glu Glu Asp Arg Val Leu Ser Ser Val His Ser Thr Val Phe Lys
1 5 10 15
Glu Ser Glu Ser Leu Glu Gly Lys Cys Asp Lys Ile Glu Gly Tyr Asp
20 25 30
Phe Asn Gln Gly Val Asn Tyr Pro Lys Leu Leu Arg Ser Met Leu Thr
35 40 45
Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Val Ile Asp Val Val Asn
50 55 60
Gln Met Leu Glu Trp Arg Leu Ser Asp Glu Thr Ile Ala Pro Glu Asp
65 70 75 80
Cys Ser Glu Glu Glu Lys Asp Pro Ala Tyr Arg Glu Ser Val Lys Cys
85 90 95
Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg
100 105 110
Glu Thr Ile Arg Tyr Leu Val Gln His His Met Val Asp Val Ile Val
115 120 125
Thr Thr Thr Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro
130 135 140
Thr Phe Lys Gly Asp Phe Ser Leu Pro Gly Ala Tyr Leu Arg Ser Lys
145 150 155 160
Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys
165 170 175
Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln Met Leu Lys Glu
180 185 190
Gln Lys Glu Glu Ser Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg
195 200 205
Leu Gly Lys Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr
210 215 220
Lys Met Asn Ile Pro Val Phe Cys Arg Gly Leu Thr Asp Gly Ser Leu
225 230 235 240
Gly Asp Met Leu Tyr Phe His Ser Phe Arg Thr Ser Gly Leu Val Ile
245 250 255
Asp Val Val Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His Ala
260 265 270
Thr Pro Arg Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys
275 280 285
His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp Tyr Ala
290 295 300
Val Phe Ile Asn Thr Gly Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala
305 310 315 320
Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser Ala Lys
325 330 335
Thr Val Lys Val Tyr Cys Asp Ala Thr Ile Ala Phe Pro Leu Leu Val
340 345 350
Ala Glu Thr Phe Ala Ser Lys Arg Glu Gln Ser Cys Glu His Lys Thr
355 360 365
<210> 72
<211> 1862
<212> DNA
<213> Medicago sativa
<220>
<221> CDS
<222> (293)..(1396)
<400> 72
gaaaccttct tcttctggag caaagtcgcc attccctacc tccttcttca ttcttattct 60
ctataacaaa cggtccgacc ggatccaagt tgcaccggtt cgaaccgctt tagttactac 120
taacggttcg aaccgttatt tttcaacccg tgacaaacgt ggaaggcttc gttgtttctt 180
cttcttcttc ttaattacca tgcgtttttg tttttctttt gagtcattga agtcttgttt 240
tttgtcgtgt ttctgtcttg agaccgtgaa agagaaaaca aagagtacga ga atg agt 298
Met Ser
1
gaa aca aag caa gaa gat gat aca att atg tcc tca gtt cac tcc act 346
Glu Thr Lys Gln Glu Asp Asp Thr Ile Met Ser Ser Val His Ser Thr
5 10 15
gtc ttc aaa gaa tcc gaa aat ctc gca gga aag tgt gtc caa atc gag 394
Val Phe Lys Glu Ser Glu Asn Leu Ala Gly Lys Cys Val Gln Ile Glu
20 25 30
ggt tat gat ttc aac cgc ggc gtc gat tat caa cag ctt ctc aaa tca 442
Gly Tyr Asp Phe Asn Arg Gly Val Asp Tyr Gln Gln Leu Leu Lys Ser
35 40 45 50
atg ctc aca act ggt ttt caa gct tcc aac ttt ggt gat gcc gtt aaa 490
Met Leu Thr Thr Gly Phe Gln Ala Ser Asn Phe Gly Asp Ala Val Lys
55 60 65
gtc gtt aat caa atg cta gat tgg agg ttg gtt gat gaa cca att gat 538
Val Val Asn Gln Met Leu Asp Trp Arg Leu Val Asp Glu Pro Ile Asp
70 75 80
gag gat tgt gat gaa gat aag aag gat ttg gag tat agg aaa tct gtt 586
Glu Asp Cys Asp Glu Asp Lys Lys Asp Leu Glu Tyr Arg Lys Ser Val
85 90 95
aca tgc aaa gtg ttt ttg ggt ttc act tct aat ctt atc tct tct ggt 634
Thr Cys Lys Val Phe Leu Gly Phe Thr Ser Asn Leu Ile Ser Ser Gly
100 105 110
gtt aga gat gtt gtt cgt tac ctt tgt cag cat cac atg gtt cat gta 682
Val Arg Asp Val Val Arg Tyr Leu Cys Gln His His Met Val His Val
115 120 125 130
gtt gtt aca act aca ggt ggt att gaa gaa gat ctt ata aag tgc ctt 730
Val Val Thr Thr Thr Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu
135 140 145
gca cca aca tat aaa gga gag ttc tct ttg ccc gga gct tat ctt cgc 778
Ala Pro Thr Tyr Lys Gly Glu Phe Ser Leu Pro Gly Ala Tyr Leu Arg
150 155 160
tca aaa gga ttg aat cga atc ggt aat tta ttg gtc cct aat gaa aat 826
Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Glu Asn
165 170 175
tat tgc aaa ttt gag gac tgg att att cct att ttt gat caa atg ttg 874
Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln Met Leu
180 185 190
aag gag caa aag gaa gag aaa gtg ctg tgg aca ccg tct aag tta ata 922
Lys Glu Gln Lys Glu Glu Lys Val Leu Trp Thr Pro Ser Lys Leu Ile
195 200 205 210
gct cga ttg gga aaa gag atc aac aat gaa aac tcc tac ctt tac tgg 970
Ala Arg Leu Gly Lys Glu Ile Asn Asn Glu Asn Ser Tyr Leu Tyr Trp
215 220 225
gca tat aag aac aat att cca gtt tat tgt cca gga tta acc gat ggc 1018
Ala Tyr Lys Asn Asn Ile Pro Val Tyr Cys Pro Gly Leu Thr Asp Gly
230 235 240
tca ctg ggt gac atg ctg tac ttc cat tcc ttc cac aac cct ggt ctg 1066
Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe His Asn Pro Gly Leu
245 250 255
att gtg gac ata gtg caa gat ata agg gcc atg aat ggt gaa gct gta 1114
Ile Val Asp Ile Val Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val
260 265 270
cat gca aat cct agc aag acg ggc atg att att tta gga ggc ggc ctt 1162
His Ala Asn Pro Ser Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu
275 280 285 290
cca aaa cat cac att tgc aat gcc aat atg atg cgc aat ggt gca gac 1210
Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp
295 300 305
tat gcg gtt ttt att aat act gca caa gaa ttt gat gga agt gat tct 1258
Tyr Ala Val Phe Ile Asn Thr Ala Gln Glu Phe Asp Gly Ser Asp Ser
310 315 320
gga gct cgt cca gat gag gct gtt tca tgg ggg aaa ata cga gga tct 1306
Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser
325 330 335
gct aaa act gtt aag gta cat tgt gat gca acg ata gca ttc cct ctg 1354
Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile Ala Phe Pro Leu
340 345 350
ctg gtt gct gaa aca ttt gcc tca aga acg tca ccc ctt aat 1396
Leu Val Ala Glu Thr Phe Ala Ser Arg Thr Ser Pro Leu Asn
355 360 365
tgataaaggt ccaccgtcaa aagtaaaagg tgtggctggg aagtgtttta ccgcagctcc 1456
acttgtgagt gccaaatgtt ttggtatgta acttataaga ccaaggtcgg ctgtatgtca 1516
tacttgagtt gaggtcaaag ttcatttgca atgcagtgtg tttgaggatc ttgatggacc 1576
agtttgccat tgacttttaa tttgactgtc ttgttattcg caaggtccac ataacaagca 1636
tttttaccat ttagaaacaa tttattagtc ctgaaggaat tgagagtcat gaattcagat 1696
gtaaattatg caatgctaac tatatttttt tggaactgtg gtttctctta gatttgaggt 1756
gttgaaaact gtaatatcta gagcaaataa gactagaaaa gtttatcaac tattactgat 1816
cagttatagt atcttcaata ttttccagaa aaaaaaaaaa aaaaaa 1862
<210> 73
<211> 368
<212> PRT
<213> Medicago sativa
<400> 73
Met Ser Glu Thr Lys Gln Glu Asp Asp Thr Ile Met Ser Ser Val His
1 5 10 15
Ser Thr Val Phe Lys Glu Ser Glu Asn Leu Ala Gly Lys Cys Val Gln
20 25 30
Ile Glu Gly Tyr Asp Phe Asn Arg Gly Val Asp Tyr Gln Gln Leu Leu
35 40 45
Lys Ser Met Leu Thr Thr Gly Phe Gln Ala Ser Asn Phe Gly Asp Ala
50 55 60
Val Lys Val Val Asn Gln Met Leu Asp Trp Arg Leu Val Asp Glu Pro
65 70 75 80
Ile Asp Glu Asp Cys Asp Glu Asp Lys Lys Asp Leu Glu Tyr Arg Lys
85 90 95
Ser Val Thr Cys Lys Val Phe Leu Gly Phe Thr Ser Asn Leu Ile Ser
100 105 110
Ser Gly Val Arg Asp Val Val Arg Tyr Leu Cys Gln His His Met Val
115 120 125
His Val Val Val Thr Thr Thr Gly Gly Ile Glu Glu Asp Leu Ile Lys
130 135 140
Cys Leu Ala Pro Thr Tyr Lys Gly Glu Phe Ser Leu Pro Gly Ala Tyr
145 150 155 160
Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn
165 170 175
Glu Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln
180 185 190
Met Leu Lys Glu Gln Lys Glu Glu Lys Val Leu Trp Thr Pro Ser Lys
195 200 205
Leu Ile Ala Arg Leu Gly Lys Glu Ile Asn Asn Glu Asn Ser Tyr Leu
210 215 220
Tyr Trp Ala Tyr Lys Asn Asn Ile Pro Val Tyr Cys Pro Gly Leu Thr
225 230 235 240
Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe His Asn Pro
245 250 255
Gly Leu Ile Val Asp Ile Val Gln Asp Ile Arg Ala Met Asn Gly Glu
260 265 270
Ala Val His Ala Asn Pro Ser Lys Thr Gly Met Ile Ile Leu Gly Gly
275 280 285
Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly
290 295 300
Ala Asp Tyr Ala Val Phe Ile Asn Thr Ala Gln Glu Phe Asp Gly Ser
305 310 315 320
Asp Ser Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg
325 330 335
Gly Ser Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile Ala Phe
340 345 350
Pro Leu Leu Val Ala Glu Thr Phe Ala Ser Arg Thr Ser Pro Leu Asn
355 360 365
<210> 74
<211> 1363
<212> DNA
<213> Musa acuminata
<220>
<221> CDS
<222> (93)..(1220)
<400> 74
ggcacgagcg cgcggcgccc gcaacgaata ttgcagagag taagaaggat cctcgccttt 60
gtcaccaaac ccttggtttc cagcgaggcg ac atg gaa ggc ggc gcc gcg gga 113
Met Glu Gly Gly Ala Ala Gly
1 5
ggg cag cga gac cgg gaa acc ctg gac gcg gtg cgg tcg gtg gtg ttt 161
Gly Gln Arg Asp Arg Glu Thr Leu Asp Ala Val Arg Ser Val Val Phe
10 15 20
aag cct tcc gta tcc ttg gag gag aag cgg ttc ccg agg gtc cag ggg 209
Lys Pro Ser Val Ser Leu Glu Glu Lys Arg Phe Pro Arg Val Gln Gly
25 30 35
tac gac ttc aac cgg ggt tgt gac ctc atc ggc ctc ctc gat tcc atc 257
Tyr Asp Phe Asn Arg Gly Cys Asp Leu Ile Gly Leu Leu Asp Ser Ile
40 45 50 55
tcc tct acc ggg ttc caa gct tcc aac ctc ggc gac gcc atc gat gtc 305
Ser Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile Asp Val
60 65 70
atc aat cag atg att gac tgg agg ctc tcc cat gat gcg ccc acg gaa 353
Ile Asn Gln Met Ile Asp Trp Arg Leu Ser His Asp Ala Pro Thr Glu
75 80 85
gat tgc agc gag gaa gag cgc aat ctg gct tac agg caa tcg gtc acg 401
Asp Cys Ser Glu Glu Glu Arg Asn Leu Ala Tyr Arg Gln Ser Val Thr
90 95 100
tgc aag atc ttt ctg ggc ttc act tcg aac ctt gta tct tct ggc atc 449
Cys Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Ile
105 110 115
agg gag ata att cgg ttt ctt gtg cag cac cga atg gtt gaa gtt tta 497
Arg Glu Ile Ile Arg Phe Leu Val Gln His Arg Met Val Glu Val Leu
120 125 130 135
gtc aca act gct ggc ggc att gaa gaa gat tta atc aaa tgc ctt gct 545
Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu Ala
140 145 150
cca aca tat aag ggt gac ttt tct ttg cct gga tcg tat ctg cgt tca 593
Pro Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly Ser Tyr Leu Arg Ser
155 160 165
aaa gga ttg aat cgg ata gga aac ctt ctt gtc cct aat gac aat tac 641
Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr
170 175 180
tgc aaa ttc gag gac tgg atc atg cca att ctg gac cag atg tta ctt 689
Cys Lys Phe Glu Asp Trp Ile Met Pro Ile Leu Asp Gln Met Leu Leu
185 190 195
gaa cag act aca gag aat gta gtt tgg aca cca tct aaa gtg att gcg 737
Glu Gln Thr Thr Glu Asn Val Val Trp Thr Pro Ser Lys Val Ile Ala
200 205 210 215
cgc ctt gga aaa gaa ata aat gat gaa agt tca tac ctg tac tgg gca 785
Arg Leu Gly Lys Glu Ile Asn Asp Glu Ser Ser Tyr Leu Tyr Trp Ala
220 225 230
tac aag aac aat gtt tct gtc tat tgc ccg gca ttg act gat gga tca 833
Tyr Lys Asn Asn Val Ser Val Tyr Cys Pro Ala Leu Thr Asp Gly Ser
235 240 245
ttg ggg gat atg ttg tac tgc cat tca gtg cgg aat cct ggt tta ctt 881
Leu Gly Asp Met Leu Tyr Cys His Ser Val Arg Asn Pro Gly Leu Leu
250 255 260
att gat att gtg caa gac ata cga gca atg aat gga gaa gct gta cat 929
Ile Asp Ile Val Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His
265 270 275
gtg ggt ctg aga aag act ggg gtc ata att ctt ggt ggg ggc ctc cca 977
Val Gly Leu Arg Lys Thr Gly Val Ile Ile Leu Gly Gly Gly Leu Pro
280 285 290 295
aag cac cat ata tgt aat gcc aac atg ttt cgg aat ggt gca gat tat 1025
Lys His His Ile Cys Asn Ala Asn Met Phe Arg Asn Gly Ala Asp Tyr
300 305 310
gct gtt tat gtc aac act gca cag gaa ttt gat gga agt gat tct gga 1073
Ala Val Tyr Val Asn Thr Ala Gln Glu Phe Asp Gly Ser Asp Ser Gly
315 320 325
gca gag cct gat gag gcg att tca tgg gga aag ata aaa ggt tct gcg 1121
Ala Glu Pro Asp Glu Ala Ile Ser Trp Gly Lys Ile Lys Gly Ser Ala
330 335 340
aag act att aaa gtt cat tgt gat gca act att gct ttt cct cta ttg 1169
Lys Thr Ile Lys Val His Cys Asp Ala Thr Ile Ala Phe Pro Leu Leu
345 350 355
gta gct gca aca ttt gca aga aag ttt cag gaa aga aac aac aaa tta 1217
Val Ala Ala Thr Phe Ala Arg Lys Phe Gln Glu Arg Asn Asn Lys Leu
360 365 370 375
gcc tgatgggggt gcaaaaggtg atcatcttat ttggattcaa ataccttaat 1270
Ala
gtaatctgct aacatctgca gatgctgtat tcttgcaaac caaaaattta atattagata 1330
accgagagcc tacagagggt cctttcaaaa aaa 1363
<210> 75
<211> 376
<212> PRT
<213> Musa acuminata
<400> 75
Met Glu Gly Gly Ala Ala Gly Gly Gln Arg Asp Arg Glu Thr Leu Asp
1 5 10 15
Ala Val Arg Ser Val Val Phe Lys Pro Ser Val Ser Leu Glu Glu Lys
20 25 30
Arg Phe Pro Arg Val Gln Gly Tyr Asp Phe Asn Arg Gly Cys Asp Leu
35 40 45
Ile Gly Leu Leu Asp Ser Ile Ser Ser Thr Gly Phe Gln Ala Ser Asn
50 55 60
Leu Gly Asp Ala Ile Asp Val Ile Asn Gln Met Ile Asp Trp Arg Leu
65 70 75 80
Ser His Asp Ala Pro Thr Glu Asp Cys Ser Glu Glu Glu Arg Asn Leu
85 90 95
Ala Tyr Arg Gln Ser Val Thr Cys Lys Ile Phe Leu Gly Phe Thr Ser
100 105 110
Asn Leu Val Ser Ser Gly Ile Arg Glu Ile Ile Arg Phe Leu Val Gln
115 120 125
His Arg Met Val Glu Val Leu Val Thr Thr Ala Gly Gly Ile Glu Glu
130 135 140
Asp Leu Ile Lys Cys Leu Ala Pro Thr Tyr Lys Gly Asp Phe Ser Leu
145 150 155 160
Pro Gly Ser Tyr Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu
165 170 175
Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Met Pro
180 185 190
Ile Leu Asp Gln Met Leu Leu Glu Gln Thr Thr Glu Asn Val Val Trp
195 200 205
Thr Pro Ser Lys Val Ile Ala Arg Leu Gly Lys Glu Ile Asn Asp Glu
210 215 220
Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys Asn Asn Val Ser Val Tyr Cys
225 230 235 240
Pro Ala Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Cys His Ser
245 250 255
Val Arg Asn Pro Gly Leu Leu Ile Asp Ile Val Gln Asp Ile Arg Ala
260 265 270
Met Asn Gly Glu Ala Val His Val Gly Leu Arg Lys Thr Gly Val Ile
275 280 285
Ile Leu Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met
290 295 300
Phe Arg Asn Gly Ala Asp Tyr Ala Val Tyr Val Asn Thr Ala Gln Glu
305 310 315 320
Phe Asp Gly Ser Asp Ser Gly Ala Glu Pro Asp Glu Ala Ile Ser Trp
325 330 335
Gly Lys Ile Lys Gly Ser Ala Lys Thr Ile Lys Val His Cys Asp Ala
340 345 350
Thr Ile Ala Phe Pro Leu Leu Val Ala Ala Thr Phe Ala Arg Lys Phe
355 360 365
Gln Glu Arg Asn Asn Lys Leu Ala
370 375
<210> 76
<211> 1451
<212> DNA
<213> Populus deltoides
<220>
<221> CDS
<222> (8)..(1126)
<400> 76
gggattt atg aca ggc aaa aaa caa tgg gag gaa gat ttg cta tca tca 49
Met Thr Gly Lys Lys Gln Trp Glu Glu Asp Leu Leu Ser Ser
1 5 10
gta cgg acc aca gtg ttt aaa gaa tca gaa gct ctt gat ggg aaa tgc 97
Val Arg Thr Thr Val Phe Lys Glu Ser Glu Ala Leu Asp Gly Lys Cys
15 20 25 30
att aaa att gaa ggt tat gat ttt aat caa gga gtg aac tac tct aag 145
Ile Lys Ile Glu Gly Tyr Asp Phe Asn Gln Gly Val Asn Tyr Ser Lys
35 40 45
ctt ctc aaa tcc atg gtc tct acc ggg ttt caa gct tcc aac ctt gga 193
Leu Leu Lys Ser Met Val Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly
50 55 60
gat gcc att caa gtt gtt aat aac atg cta gac tgg agg ctt gct gat 241
Asp Ala Ile Gln Val Val Asn Asn Met Leu Asp Trp Arg Leu Ala Asp
65 70 75
gaa gag ata aca gaa gat tgt agt gat gag gag agg gag ttg gcc tat 289
Glu Glu Ile Thr Glu Asp Cys Ser Asp Glu Glu Arg Glu Leu Ala Tyr
80 85 90
aga gag tct gtg aga tgc aaa ctg ttc ttg ggt ttt aca tca aat ctt 337
Arg Glu Ser Val Arg Cys Lys Leu Phe Leu Gly Phe Thr Ser Asn Leu
95 100 105 110
gtt tct tca ggt gtc aga gat aca att cga tat ctt gtt cag cat cat 385
Val Ser Ser Gly Val Arg Asp Thr Ile Arg Tyr Leu Val Gln His His
115 120 125
atg gtt gat gta gtg gtt aca acg gca ggt ggc ata gaa gaa gat ctt 433
Met Val Asp Val Val Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu
130 135 140
ata aaa tgc ctg gca cca aca tac aaa ggt gac ttt tct cta ccc ggg 481
Ile Lys Cys Leu Ala Pro Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly
145 150 155
gct caa tta cga tca aaa ggg ttg aat cga att ggt aac ttg ttg gta 529
Ala Gln Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val
160 165 170
cct aat gac aac tac tgc aaa ttt gag gat tgg atc att cca atc ttt 577
Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe
175 180 185 190
gac caa atg ttg aag gaa caa att gaa gag aat atc acc tgg aca cct 625
Asp Gln Met Leu Lys Glu Gln Ile Glu Glu Asn Ile Thr Trp Thr Pro
195 200 205
tct aaa tta ata gct cgc atg ggg aaa gaa ata aat aat gag agt tca 673
Ser Lys Leu Ile Ala Arg Met Gly Lys Glu Ile Asn Asn Glu Ser Ser
210 215 220
tac ctt tat tgg gca tat aag aac gac att cca gta ttc tgt cca ggc 721
Tyr Leu Tyr Trp Ala Tyr Lys Asn Asp Ile Pro Val Phe Cys Pro Gly
225 230 235
tta aca gat ggt tct cta ggg gac atg cta tac ttt cat tcc ttc cac 769
Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe His
240 245 250
aac cct ggt cta att gtt gcc ata gtc caa gat att aga gcc atg aat 817
Asn Pro Gly Leu Ile Val Ala Ile Val Gln Asp Ile Arg Ala Met Asn
255 260 265 270
ggt gaa gct gtc cac gca agt cct aga aaa act ggt atc atc att ctt 865
Gly Glu Ala Val His Ala Ser Pro Arg Lys Thr Gly Ile Ile Ile Leu
275 280 285
gga ggt ggg ctt cct aag cat cat ata tgc aat gcc aat atg atg cgt 913
Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg
290 295 300
aac ggt gca gat tat gct gta ttc atc aat aca gca caa gaa ttt gat 961
Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr Ala Gln Glu Phe Asp
305 310 315
ggg agt gat tct gga gct cat cct gat gag gct gta tcg tgg ggg aaa 1009
Gly Ser Asp Ser Gly Ala His Pro Asp Glu Ala Val Ser Trp Gly Lys
320 325 330
ata cga ggt tct gct aaa act gtt aag gtc cac tgt gat gca act att 1057
Ile Arg Gly Ser Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile
335 340 345 350
gct ttt cct ctc cta gtt gct gaa aca ttt gcc cct agg agg aac aga 1105
Ala Phe Pro Leu Leu Val Ala Glu Thr Phe Ala Pro Arg Arg Asn Arg
355 360 365
ttc tgc agc agt act caa agc tagggctgtg tgcagttctt ggccagaaaa 1156
Phe Cys Ser Ser Thr Gln Ser
370
ttgattcatt tttatttgta ttatgactga acgatccgca ggatgggtag tgggctccat 1216
tgatgccata aacttctttt tttttcccct cagaattaag ggatccgcca gaacacactg 1276
ctctcagccc caaaccattg ttgcctctac tgggagtagc ataaccaatt gaattgcgct 1336
cctccaagca gcgcctctta gttgcgttat ttattgtaag tagcgcaacc aactaaatta 1396
tgctagttcc cacatttatt gactgctatt ttcaaaagaa aaaaaaaaaa aaaaa 1451
<210> 77
<211> 373
<212> PRT
<213> Populus deltoides
<400> 77
Met Thr Gly Lys Lys Gln Trp Glu Glu Asp Leu Leu Ser Ser Val Arg
1 5 10 15
Thr Thr Val Phe Lys Glu Ser Glu Ala Leu Asp Gly Lys Cys Ile Lys
20 25 30
Ile Glu Gly Tyr Asp Phe Asn Gln Gly Val Asn Tyr Ser Lys Leu Leu
35 40 45
Lys Ser Met Val Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala
50 55 60
Ile Gln Val Val Asn Asn Met Leu Asp Trp Arg Leu Ala Asp Glu Glu
65 70 75 80
Ile Thr Glu Asp Cys Ser Asp Glu Glu Arg Glu Leu Ala Tyr Arg Glu
85 90 95
Ser Val Arg Cys Lys Leu Phe Leu Gly Phe Thr Ser Asn Leu Val Ser
100 105 110
Ser Gly Val Arg Asp Thr Ile Arg Tyr Leu Val Gln His His Met Val
115 120 125
Asp Val Val Val Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu Ile Lys
130 135 140
Cys Leu Ala Pro Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly Ala Gln
145 150 155 160
Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn
165 170 175
Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln
180 185 190
Met Leu Lys Glu Gln Ile Glu Glu Asn Ile Thr Trp Thr Pro Ser Lys
195 200 205
Leu Ile Ala Arg Met Gly Lys Glu Ile Asn Asn Glu Ser Ser Tyr Leu
210 215 220
Tyr Trp Ala Tyr Lys Asn Asp Ile Pro Val Phe Cys Pro Gly Leu Thr
225 230 235 240
Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe His Asn Pro
245 250 255
Gly Leu Ile Val Ala Ile Val Gln Asp Ile Arg Ala Met Asn Gly Glu
260 265 270
Ala Val His Ala Ser Pro Arg Lys Thr Gly Ile Ile Ile Leu Gly Gly
275 280 285
Gly Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly
290 295 300
Ala Asp Tyr Ala Val Phe Ile Asn Thr Ala Gln Glu Phe Asp Gly Ser
305 310 315 320
Asp Ser Gly Ala His Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg
325 330 335
Gly Ser Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile Ala Phe
340 345 350
Pro Leu Leu Val Ala Glu Thr Phe Ala Pro Arg Arg Asn Arg Phe Cys
355 360 365
Ser Ser Thr Gln Ser
370
<210> 78
<211> 1488
<212> DNA
<213> Arabidopsis thaliana
<400> 78
acaataaggc tttaaagccc ataaaaccct taaatatatc aaagcccaaa agaaacgcct 60
tttgcgcttt cccgatcgtg gtcaacttcc tctgttacca aaaaatctgt accgcaaaat 120
cctcgtcgaa gctcgctgct gcaaccatgt ccgacgagga gcatcacttt gagtccagtg 180
acgccggagc gtccaaaacc taccctcaac aagctggaac catccgtaag aatggttaca 240
tcgtcatcaa aaatcgtccc tgcaaggttt cgttctcaaa catttctcca ctctcttcct 300
ctgatcttat tagatctgtt cattacttag attcctcaga ttcttttttt tgtcacctcc 360
acgatgttcg actgatattt gttcttgtca tcattgttaa attcacattt tattgcactt 420
ttgttttagc gaaattatta aattggtcat cttcagtttt gttcgattag ataagtttta 480
ggattttttc ttacacaagt tactggatca gctgctaaat gtcattttgt gtcgcaggtt 540
gttgaggttt caacctcgaa gactggcaag catggtcatg ctaaatgtca ttttgtagct 600
attgatatct tcaccagcaa gaaactcgaa gatattgttc cttcttccca caattgtgat 660
gtatgtgaaa aaagctcctt tgatcacttt catttcttgt ttgtttcttt caagtcccat 720
ttgagatttt gtttttgttg aattgggttt caggttcctc atgtcaaccg tactgattat 780
cagctgattg acatttctga agatggatat gtatgtgttc ttaaatagca cttgttcctt 840
tatatggttt agttacttgt tctgttttgt aatcattttg caggtcagtt tgttgactga 900
taacggtagt accaaggatg accttaagct ccctaatgat gacactctgc tccaacaggt 960
taagttttgc atgttcatca cattaaatgt tgctagttaa ttaaaatcaa ctctatgtcg 1020
atttctgaaa atggaagaaa aagtgcagag taatgagtga cctgattgtg ttaatgaaac 1080
agatcaagag tgggtttgat gatggaaaag atctagtggt gagtgtgatg tcagctatgg 1140
gagaggaaca gatcaatgct cttaaggaca tcggtcccaa gtgagactaa caaagcctcc 1200
cctttgttat gagattcttc ttcttcttct gtaggcttcc attactcatc ggagattatc 1260
ttgtttttgg gtgactccta ttttggatat ttaaactttt gttaataatg ccatcttctt 1320
caaccttttc cttctagatg gtttttatac ttcttctaat tgattgattc tttatggttg 1380
tccaagtgtc aaagtgttcc acccatatga ttctaacctt ttgatgagcg aagtctttac 1440
tcgtgcgtta tgtagagacg tagaagcaat accacaaaag agtataat 1488
<210> 79
<211> 1566
<212> DNA
<213> Arabidopsis thaliana
<400> 79
aggataataa tacagtaacc ctagaaaggt ttcctccacc ttcctcttcc cctcctatat 60
aaaaaaaatc gacatcgctt ttgctcactt ctctctctta ggtttttttt cccttctccc 120
aatctcatct tctccgaaaa cctttcttct ctcaaatttc tgtgaaaaca tgtctgacga 180
cgagcaccac tttgaggcca gcgaatccgg agcttccaag acctatcctc aatcagccgg 240
taacatccgt aaaggtggtc acatcgtcat caaaaaccgt ccctgcaagg tctgatttct 300
atttcatcat caaacatcgt tctcgatctc tttttcctga ttctagatct cgtctctgta 360
tagtagctcc ttgattttgt ttttatcctc ggatttgacc tggttctgtt tagtttgaat 420
ttttcttata gatcgctact tagatgaata tgatgaatct tatcctgtta ttttgatggt 480
ggtacctctc tagattcgtg gaattttggg aaatgaaaat gaaaaatgga tagaaatcaa 540
gcaatatcag acgacgcctt ttgtgatttt gaatctaagt agtctattga ttgatttgat 600
ttaaacgttt atggagaaca tagatttgat tttgatattt tggttttgat taggttgttg 660
aggtttcgac ttccaaaact ggcaagcacg gtcacgccaa atgtcacttt gttgctattg 720
atatcttcac tgctaagaag cttgaagata ttgttccatc ttcccacaat tgtgatgtaa 780
gttactacac aaactatgta gattcatttt cacagtattt gatatgattg tgtgatctga 840
ctcaaatatt gttcctttct ctttttttct caggttccac atgtgaaccg tgttgattac 900
cagttgattg atatcactga ggatggcttc gtatgttttt ctttatactc actttcctca 960
tcactccagc tttatttatc tattcttgcc ataacttttg tacttgttta cattataggt 1020
gagccttctc actgacagtg gtggcaccaa ggatgatctc aagcttccca ccgatgatgg 1080
tctcaccgcc caggttattt tcttgtcttt tcatactcgc acacaaatga cttgactttg 1140
tattcatctc tcgaattgtg atattgaaaa cagttgttgt gttttgttaa tgcagatgag 1200
gcttggattc gatgagggaa aggatattgt ggtgtctgtc atgtcttcca tgggagagga 1260
gcagatctgt gccgtcaagg aagttggtgg tggcaagtaa acaagtatca ttcgatatat 1320
tattaccagt ttgacaacgg acgtcaatgt tataagaacc aaaagatgtt tttctttttc 1380
ctaatttaga ccctttgtgt gtgtttcttg ttgcaagaca accatatcta ttggttttgg 1440
attgttggaa aagtttgtgt tgaaacattc aaagtttctt atgagatgtt attcttaaaa 1500
ccactttttg tttgttcact ggatatgttt gttcatgaag cttgttttaa gcaactcttt 1560
acatga 1566
<210> 80
<211> 319
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
promoter sequence
<400> 80
aagacgttcc aaccacgtct tcaaagcaag tggattgatg tgatatctcc actgacgtaa 60
gggatgacgc acaatcccac tatccttcgc aagacccttc ctctatataa ggaagttcat 120
ttcatttgga gaggacacgc tgaaatcacc agtctctctc tcaagcttgg atcctcgagt 180
actagttcag ggagctcgaa ttgatcctct agagctttcg ttcgtatcat cggtttcgac 240
aacgttcgtc aagttcaatg catcagtttc attgcgcaca caccagaatc ctactgagtt 300
tgagtattat ggcattggg 319
<210> 81
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 81
gggagggact agtgtgcacg cc 22
<210> 82
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 82
gcgaagcggc catggctcga gttttttttt tttttttt 38
<210> 83
<211> 521
<212> DNA
<213> Arabidopsis thaliana
<400> 83
gggagggact agtgtgcacg ccctgatgaa gctgtgtctt ggggtaaaat taggggttct 60
gctaaaaccg ttaaggtata ctgtgatgct accatagcct tcccattgtt ggttgcagaa 120
acatttgcca caaagagaga ccaaacctgt gagtctaaga cttaagaact gactggttcg 180
tacctctggc ctcatcatcg atgtagtaca agatatcaga gctatgaacg gcgaagctgt 240
ccatgcaaat cctaaaaaga caggcgtttt ggccatggat tcttaaagat cgttgctttt 300
tgattttaca ctggagtgac catataacac tccacattga tgtggctgtg acgcgaattg 360
tcttcttgcg aattgtactt tagtttctct caacctaaaa tgatttgcag attgtgtttt 420
cgtttaaaac acaagagtct tgtagtcaat aatcctttgc cttataaaat tattcagttc 480
caacaaaaaa aaaaaaaaaa ctcgagccat ggccgcttcg c 521
<210> 84
<211> 497
<212> DNA
<213> Arabidopsis thaliana
<400> 84
ctagtgtgca cgccctgatg aagctgtgtc ttggggtaaa attaggggtt ctgctaaaac 60
cgttaaggta tactgtgatg ctaccatagc cttcccattg ttggttgcag aaacatttgc 120
cacaaagaga gaccaaacct gtgagtctaa gacttaagaa ctgactggtt cgtacctctg 180
gcctcatcat cgatgtagta caagatatca gagctatgaa cggcgaagct gtccatgcaa 240
atcctaaaaa gacaggcgtt ttggccatgg attcttaaag atcgttgctt tttgatttta 300
cactggagtg accatataac actccacatt gatgtggctg tgacgcgaat tgtcttcttg 360
cgaattgtac tttagtttct ctcaacctaa aatgatttgc agattgtgtt ttcgtttaaa 420
acacaagagt cttgtagtca ataatccttt gccttataaa attattcagt tccaacaaaa 480
aaaaaaaaaa aactcga 497
<210> 85
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 85
ctcgagaaga ataacatctc ataagaaac 29
<210> 86
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 86
gagctcggca agtaaacaag tatcattcg 29
<210> 87
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 87
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 88
<211> 205
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 88
aagaataaca tctcataaga aactttgaat gtttcaacac aaacttttcc aacaatccaa 60
aaccaataga tatggttgtc ttgcaacaag aaacacacac aaagggtcta aattaggaaa 120
aagaaaaaca tcttttggtt cttataacat tgacgtccgt tgtcaaactg gtaataatat 180
atcgaatgat acttgtttac ttgcc 205
<210> 89
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 89
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 90
<211> 37
<212> PRT
<213> Arabidopsis thaliana
<400> 90
Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser Ala
1 5 10 15
Lys Thr Val Lys Val Cys Phe Leu Ile Ser Ser His Pro Asn Leu Tyr
20 25 30
Leu Thr Gln Trp Phe
35
<210> 91
<211> 52
<212> PRT
<213> Lycopersicon esculentum
<400> 91
Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Gly
1 5 10 15
Ala Lys Thr Val Lys Val His Cys Asp Ala Thr Ile Ala Phe Pro Ile
20 25 30
Leu Val Ala Glu Thr Phe Ala Ala Lys Ser Lys Glu Phe Ser Gln Ile
35 40 45
Arg Cys Gln Val
50
<210> 92
<211> 193
<212> PRT
<213> Arabidopsis thaliana
<400> 92
Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro Thr Phe Lys
1 5 10 15
Gly Asp Phe Ser Leu Pro Gly Ala Tyr Leu Arg Ser Lys Gly Leu Asn
20 25 30
Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu
35 40 45
Asp Trp Ile Ile Pro Ile Phe Asp Glu Met Leu Lys Glu Gln Lys Glu
50 55 60
Glu Asn Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg Leu Gly Lys
65 70 75 80
Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys Met Asn
85 90 95
Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly Ser Leu Arg Asp Met
100 105 110
Leu Tyr Phe His Ser Phe Arg Thr Ser Gly Leu Ile Ile Asp Val Val
115 120 125
Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His Ala Asn Pro Lys
130 135 140
Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile
145 150 155 160
Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile
165 170 175
Asn Thr Gly Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp
180 185 190
Glu
<210> 93
<211> 174
<212> PRT
<213> Dianthus caryophyllus
<400> 93
Arg Arg Ser Ile Lys Cys Leu Ala Pro Thr Phe Lys Gly Asp Phe Ala
1 5 10 15
Leu Pro Gly Ala Gln Leu Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn
20 25 30
Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile
35 40 45
Pro Ile Leu Asp Lys Met Leu Glu Glu Gln Ile Ser Glu Lys Ile Leu
50 55 60
Trp Thr Pro Ser Lys Leu Ile Gly Arg Leu Gly Arg Glu Ile Asn Asp
65 70 75 80
Glu Ser Ser Tyr Leu Tyr Trp Ala Phe Lys Asn Asn Ile Pro Val Phe
85 90 95
Cys Pro Gly Leu Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His
100 105 110
Ser Phe Arg Asn Pro Gly Leu Ile Ile Asp Val Val Gln Asp Ile Arg
115 120 125
Ala Val Asn Gly Glu Ala Val His Ala Ala Pro Arg Lys Thr Gly Met
130 135 140
Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Ile Cys Asn Ala Asn
145 150 155 160
Met Met Arg Asn Gly Ala Asp Tyr Ala Val Phe Ile Asn Thr
165 170
<210> 94
<211> 22
<212> PRT
<213> Arabidopsis thaliana
<400> 94
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 95
<211> 22
<212> PRT
<213> Arabidopsis thaliana
<400> 95
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 96
<211> 22
<212> PRT
<213> Arabidopsis thaliana
<400> 96
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 97
<211> 22
<212> PRT
<213> Brassica napus
<400> 97
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 98
<211> 22
<212> PRT
<213> Dianthus caryophyllus
<400> 98
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 99
<211> 22
<212> PRT
<213> Lycopersicon esculentum
<400> 99
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 100
<211> 22
<212> PRT
<213> Lycopersicon esculentum
<400> 100
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 101
<211> 22
<212> PRT
<213> Lycopersicon esculentum
<400> 101
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 102
<211> 22
<212> PRT
<213> Medicago sativa
<400> 102
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 103
<211> 22
<212> PRT
<213> Medicago sativa
<400> 103
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 104
<211> 22
<212> PRT
<213> Medicago sativa
<400> 104
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 105
<211> 22
<212> PRT
<213> Lactuca sativa
<400> 105
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 106
<211> 22
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree sequence
peptide
<400> 106
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 107
<211> 488
<212> DNA
<213> Mycosphaerella fijiensis
<400> 107
gggttaaatc gtattggaaa cttcttagtg ccaaacgaca attactgccg ctttgaagac 60
tgggtgatgc caatcctcga cacaatgctc gaagaacagg aagcatgcaa gggttcgggc 120
gaagcaatcc actggactcc cagcaaaatc atcaaccggc ttggcaagga ggtcaacgac 180
gaatcgtccg tgtactactg ggcatggaag aacgacattc cagtgttctg tccggcgctt 240
actgatggca gtctcggaga catgctgtac ttccacacgt tcaaatcctc accgcagcag 300
cttcgagtcg acattgtgga agacatccga aagatcaaca ccctcgccgt ccgagccaag 360
cgcactggca tgatcattct cggaggcggc attgtcaagc accacatcgc aaatgccaac 420
ctgatgcgca atggcgcgga aagcgcagtg tacatcaata ccgcgccgga attcgacgga 480
tccgacca 488
<210> 108
<211> 22
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree sequence
peptide
<400> 108
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 109
<211> 22
<212> PRT
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree sequence
peptide
<400> 109
Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His
1 5 10 15
Ala Lys Cys His Phe Val
20
<210> 110
<211> 66
<212> DNA
<213> Arabidopsis thaliana
<400> 110
tgcaaggttg ttgaggtttc aacctcgaag actggcaagc atggtcatgc taaatgtcat 60
tttgta 66
<210> 111
<211> 66
<212> DNA
<213> Arabidopsis thaliana
<400> 111
tgcaaggttg ttgaggtttc gacttccaaa actggcaagc acggtcacgc caaatgtcat 60
tttgtt 66
<210> 112
<211> 66
<212> DNA
<213> Arabidopsis thaliana
<400> 112
tgcaaggtgg ttgaggtatc gacttcgaag actgggaagc atggtcacgc caagtgtcac 60
tttgtt 66
<210> 113
<211> 66
<212> DNA
<213> Brassica napus
<400> 113
tgcaaggttg ttgaggtttc gacttcgaag actgggaagc acggtcacgc aaagtgtcac 60
tttgtt 66
<210> 114
<211> 66
<212> DNA
<213> Dianthus caryophyllus
<400> 114
tgcaaggtgg ttgaggtttc tacctccaag actggcaagc acggtcatgc caaatgtcac 60
tttgtt 66
<210> 115
<211> 66
<212> DNA
<213> Lycopersicon esculentum
<400> 115
tgcaaggtgg ttgaggtttc aacttccaag acaggcaagc acggtcatgc taaatgtcac 60
tttgtt 66
<210> 116
<211> 66
<212> DNA
<213> Lycopersicon esculentum
<400> 116
tgcaaggttg tggaagtctc tacatccaaa actggcaagc acggtcacgc caaatgtcat 60
ttcgtt 66
<210> 117
<211> 66
<212> DNA
<213> Lycopersicon esculentum
<400> 117
tgcaaggttg ttgaggtctc cacttccaaa actggcaagc atggacatgc aaaatgtcac 60
tttgtg 66
<210> 118
<211> 66
<212> DNA
<213> Medicago sativa
<400> 118
tgcaaggtgg ttgaagtttc gacttcgaag accgggaagc atggacatgc caagtgtcat 60
tttgtt 66
<210> 119
<211> 66
<212> DNA
<213> Medicago sativa
<400> 119
tgcaaggttg ttgaggtttc tacttcaaaa acaggaaaac atggacatgc aaagtgtcac 60
tttgtt 66
<210> 120
<211> 66
<212> DNA
<213> Medicago sativa
<400> 120
tgcaaggtag ttgaagtttc aacttctaaa actggaaagc atggacatgc aaagtgtcac 60
tttgtt 66
<210> 121
<211> 66
<212> DNA
<213> Lactuca sativa
<400> 121
tgcaaggttg ttgaagtttc tacctccaag actgggaagc atgggcatgc taagtgtcac 60
tttgtc 66
<210> 122
<211> 66
<212> DNA
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree
oligonucleotide sequence
<400> 122
tgcaaggtcg tggaggtttc aacctctaaa actggcaagc atggccatgc taaatgtcac 60
tttgtt 66
<210> 123
<211> 66
<212> DNA
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree
oligonucleotide sequence
<400> 123
tgcaaggttg ttgaggtttc cacctcaaag acaggcaagc acggacatgc taagtgccac 60
tttgtg 66
<210> 124
<211> 66
<212> DNA
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree
oligonucleotide sequence
<400> 124
tgcaaggttg tggaggtttc tacctctaaa actggcaagc acggccatgc caaatgtcac 60
tttgtt 66
<210> 125
<211> 66
<212> DNA
<213> Unknown Organism
<220>
<223> Description of Unknown Organism: Tree
oligonucleotide sequence
<400> 125
tgcaaggttg ttgaggtttc cacctcaaag acaggcaagc atggacatgc taagtgccac 60
tttgtg 66
<210> 126
<211> 745
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (55)..(534)
<400> 126
ttctccacag caaacacaga gaagttcata gcagaagaag agagagattt agct atg 57
Met
1
tct gat gaa gaa cac cat ttt gag tcc aaa gct gat gct ggt gcc tca 105
Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser
5 10 15
aaa act tac cct caa caa gct ggt act att cgc aag aat ggt tat ata 153
Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile
20 25 30
gtt atc aaa ggc aga cct tgc aag gtt gtt gag gtc tcc act tcc aaa 201
Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
act ggc aag cat gga cat gca aaa tgt cac ttt gtg gca atc gac att 249
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60 65
ttc aat gca aaa aag ctt gaa gat att gtt cct tca tcc cac aat tgt 297
Phe Asn Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
70 75 80
gat gtg cca cat gtc aat cgt act gac tat cag ctg att gac ata tct 345
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
gaa gat ggt ttt gtg tct ctt ctt act gaa aat gga aac acc aaa gac 393
Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp
100 105 110
gac ctc aga ctt ccc acc gat gac acc ctg ttg aac cag gtt aaa ggt 441
Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Asn Gln Val Lys Gly
115 120 125
gga ttt gag gaa gga aag gat ctc gtt ctg tct gtg atg tct gca atg 489
Gly Phe Glu Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala Met
130 135 140 145
ggt gaa gag cag atc tgt gct gtg aag gac att ggt acc aag acc 534
Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Ile Gly Thr Lys Thr
150 155 160
tagttgtgtg cattctgcag cataaataat tgctttttag cgaagacgtt ttatatcttg 594
ttatcgtggt acctttgcaa tctgttttat cgtgaaaaca ccttatatct attggcatgg 654
cttgaatagt tgaaactcta atagtttctg tttggcataa ggcaatgaac tggatttgat 714
agcagaagta atctacatgt cacttttttt t 745
<210> 127
<211> 160
<212> PRT
<213> Lycopersicon esculentum
<400> 127
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Ala Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Asn Gln Val Lys
115 120 125
Gly Gly Phe Glu Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Ile Gly Thr Lys Thr
145 150 155 160
<210> 128
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 128
gaagctcgag gctgcaacca tgtcc 25
<210> 129
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 129
ggggagctct tgttagtctc acttgg 26
<210> 130
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 130
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcaccaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 131
<211> 495
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 131
gctgcaacca tgtccgacga ggagcatcac tttgagtcca gtgacgccgg agcgtccaaa 60
acctaccctc aacaagctgg aaccatccgt aagaatggtt acatcgtcat caaaaatcgt 120
ccctgcaagg ttgttgaggt ttcaacctcg aagactggca agcatggtca tgctaaatgt 180
cattttgtag ctattgatat cttcaccagc aagaaactcg aagatattgt tccttcttcc 240
cacaattgtg atgttcctca tgtcaaccgt actgattatc agctgattga catttctgaa 300
gatggatatg tcagtttgtt gactgataac ggtagtacca aggatgacct taagctccct 360
aatgatgaca ctctgctcca acagatcaag agtgggtttg atgatggaaa agatctagtg 420
gtgagtgtaa tgtcagctat gggagaggaa cagatcaatg ctcttaagga catcggtccc 480
aagtgagact aacaa 495
<210> 132
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 132
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 133
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 133
gcgctcgagc tatgtctgat gaagaacacc 30
<210> 134
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 134
tttgagctcc agaatgcaca caactagg 28
<210> 135
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 135
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 136
<211> 499
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 136
ctatgtctga tgaagaacac cattttgagt ccaaagctga tgctggtgcc tcaaaaactt 60
accctcaaca agctggtact attcgcaaga atggttatat agttatcaaa ggcagacctt 120
gcaaggttgt tgaggtctcc acttccaaaa ctggcaagca tggacatgca aaatgtcact 180
ttgtggcaat cgacattttc aatgcaaaaa agcttgaaga tattgttcct tcatcccaca 240
attgtgatgt gccacatgtc aatcgtactg actatcagct gattgacata tctgaagatg 300
gttttgtgtc tcttcttact gaaaatggaa acaccaaaga cgacctcaga cttcccaccg 360
atgacaccct gttgaaccag gttaaaggtg gatttgagga aggaaaggat ctcgttctgt 420
ctgtgatgtc tgcaatgggt gaagagcaga tctgtgctgt gaaggacatt ggtaccaaga 480
cctagttgtg tgcattctg 499
<210> 137
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 137
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 138
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 138
gcatgtcgac atgtctgacg aggagcacc 29
<210> 139
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 139
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 140
<211> 661
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 140
tcgacatgtc tgacgaggag caccacttcg agtccagcga cgccggagct tccaaaacct 60
accctcagca ggctggtaac atccgcaagg gtggtcacat cgtcatcaag ggccgtccct 120
gcaaggttgt tgaggtttcg acttcgaaga ctgggaagca cggtcacgca aagtgtcact 180
ttgttgctat tgacatcttc actgctaaga agctcgagga tattgttccc tcttcccaca 240
attgtgatgt tccccatgtg aaccgtattg actaccagtt gattgatatc tctgagaatg 300
gctttgttag ccttttgacc gacagtggtg gcaccaagga cgacctcaag cttcccaccg 360
atgataatct cagcgctctg atgaagagtg gattcgagga gggaaaggat gtggtggtgt 420
ctgtcatgtc ttccatggga gaggagcaga tctgtgccgt caaggaagtt ggtggtggca 480
agtaaaaccc attctctgag agaggataat cttattacca gtggtcaatg ttataagaac 540
aagaacttgt tttttttcct ttttctaatt tagatcattt gtgttgtgtt tctttgttgc 600
aagacaacca ttatctatta ttggttttgg attgtttaaa aaaaaaaaaa aaaaaaaaaa 660
a 661
<210> 141
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 141
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 142
<211> 325
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 142
ggtgcacgcc ctgatgaagc agtgtcttgg ggtaaaataa ggggatctgc taaaactgtc 60
aaggtgtact gtgatgctac catagccttc cctttgttgg ttgctgaaac atttgcctcc 120
aagagagaac aaagctgtga gcacaagacc taagcccaag aaagcttacg tctcttttat 180
cggtttgttc ttccatcttg ttgttgtacc ctttgtcctg ctttacataa cattcatctc 240
taaaacaata ctacctcctt ttgacaaaaa ataaaaaaaa ttggaaaaat ggtttcacaa 300
gaataaaaaa aaaaaaaaaa aaaaa 325
<210> 143
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 143
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 144
<211> 325
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 144
tttttttttt tttttttttt tattcttgtg aaaccatttt tccaattttt tttatttttt 60
gtcaaaagga ggtagtattg ttttagagat gaatgttatg taaagcagga caaagggtac 120
aacaacaaga tggaagaaca aaccgataaa agagacgtaa gctttcttgg gcttaggtct 180
tgtgctcaca gctttgttct ctcttggagg caaatgtttc agcaaccaac aaagggaagg 240
ctatggtagc atcacagtac accttgacag ttttagcaga tccccttatt ttaccccaag 300
acactgcttc atcagggcgt gcacc 325
<210> 145
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 145
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtgc aaatggaaat ggatggagaa gagttaatga atgatatggt 240
ccttttgttc attctcaaat taatattatt tgttttttct cttatttgtt gtgtgttgaa 300
tttgaaatta taagagatat gcaaacattt tgttttgagt aaaaatgtgt caaatcgtgg 360
cctctaatga ccgaagttaa tatgaggagt aaaacacttg tagttgtacc attatgctta 420
ttcactaggc aacaaatata ttttcagacc tagaaaagct gaaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 146
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 146
aagctcgaga tgtcggacga agagcacc 28
<210> 147
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 147
gtagagctcc accaatacca tctgcagc 28
<210> 148
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 148
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 149
<211> 497
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 149
atgtcggacg aagagcacca cttcgaatcc aaggccgatg ccggagcttc aaagacgtat 60
cctcaacaag ctggtactat tcgtaaaggt ggtcacatcg tcataaaaaa tcgtccttgc 120
aaggtggttg aagtttcaac ttccaagaca ggcaagcacg gtcatgctaa atgtcactcg 180
tggcaattga cattttcact ggaaagaaac ttgaggatat tgttccctct tctcacaatt 240
gtgatgttcc tcatgtgaat aggactgatt atcaacttat tgatatctct gaggatggct 300
ttgtgagtct gttgactgaa aatggtaaca ccaaggatga cttgagactc ccaactgatg 360
atactcttct ggctcaggtc aaagatggtt ttgctgaggg gaaagacctg gttctatcag 420
tgatgtctgc catgggagag gagcagattt gtggtatcaa ggacattggc cctaagtagc 480
tgcagatggt attggtg 497
<210> 150
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 150
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 151
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 151
cgactcgagc agccatgtct gacgagg 27
<210> 152
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 152
atcgagctca tcacttgggg ccaatatcc 29
<210> 153
<211> 906
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 153
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 154
<211> 486
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 154
cagccatgtc tgacgaggag catcaatttg agtctaaggc tgatgccgga gcatcaaaaa 60
cttaccctca acaagctggt actattcgta agaacggtta tatcgtcatc aaaggccgtc 120
catgcaaggt tgtggaagtc tctacatcca aaactggcaa gcacggtcac gccaaatgtc 180
atttcgttgc tattgacatc ttcactggga agaagcttga ggatattgtc ccctcttcac 240
acaattgtga tgtgccccat gttaatcgta cagattatca gcttattgac atctctgaag 300
atggatttgt gagtctgctt actgacaatg gtaacaccaa ggatgacctc aggcttccta 360
ctgatgaaaa tctgctttca ctgatcaagg acgggtttgc cgagggtaag gacctcgttg 420
tgtctgttat gtcagctatg ggtgaggaac agattaatgc tttgaaggat attggcccca 480
agtgat 486
<210> 155
<211> 662
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
construct
<400> 155
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 156
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 156
cactgctcac tagtttgatg gc 22
<210> 157
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<400> 157
gcgaagcggc catggctcga gttttttttt tttttttt 38
<210> 158
<211> 413
<212> DNA
<213> Lactuca sativa
<400> 158
cactgctcac tagtttgatg gcagtgattc tggtgctcga cctgatgaag ctgtctcctg 60
ggggaaaata cgtggttctg ctaaatctgt caaggtgcac tgtgatgcaa ctatcgcgtt 120
ccctttactt gttgcagaaa catttgctgc aaagagagag ggggagatga aaaatgttga 180
gtcaaccaaa gctttggttt aaaaaggtgg aacagtgtag gacagggact catttttgat 240
attttgtttg ctaaaaaatg gtctttggaa gaatattgat gcacacaaac aaggagacaa 300
tgttactgat cttggagagt gtaatgtaaa atgtctaaat aatttcaaag cttctcacaa 360
caaatcaaac tttaaaaaaa aaaaaaaaaa aactcgagcc atggccgctt cgc 413
<210> 159
<211> 388
<212> DNA
<213> Lactuca sativa
<400> 159
ctagtttgat ggcagtgatt ctggtgctcg acctgatgaa gctgtctcct gggggaaaat 60
acgtggttct gctaaatctg tcaaggtgca ctgtgatgca actatcgcgt tccctttact 120
tgttgcagaa acatttgctg caaagagaga gggggagatg aaaaatgttg agtcaaccaa 180
agctttggtt taaaaaggtg gaacagtgta ggacagggac tcatttttga tattttgttt 240
gctaaaaaat ggtctttgga agaatattga tgcacacaaa caaggagaca atgttactga 300
tcttggagag tgtacatgta aaatgtctaa ataatttcaa agcttctcac aacaaatcaa 360
acttaaaaaa aaaaaaaaaa aaactcga 388
<210> 160
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<220>
<221> modified_base
<222> (3)
<223> a, t, c, g, unknown or other
<220>
<221> modified_base
<222> (12)
<223> a, t, c, g, unknown or other
<220>
<221> modified_base
<222> (18)
<223> a, t, c, g, unknown or other
<400> 160
ggnttraayc gnathggnaa ytt 23
<210> 161
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
Primer
<220>
<221> modified_base
<222> (9)
<223> a, t, c, g, unknown or other
<220>
<221> modified_base
<222> (21)
<223> a, t, c, g, unknown or other
<400> 161
tgrtcgganc crtcraaytc ngc 23
<210> 162
<211> 108
<212> DNA
<213> Mycosphaerella fijiensis
<400> 162
cgccaagcta tttaggtgac actatagaat actcaagcta tgcatccaac gcgttgggag 60
ctctcccata tggtcgacct gcaggcggcc gcgaattcac tagtgatt 108
<210> 163
<211> 487
<212> DNA
<213> Mycosphaerella fijiensis
<220>
<221> CDS
<222> (1)..(486)
<400> 163
ggg tta aat cgt att gga aac ttc tta gtg cca aac gac aat tac tgc 48
Gly Leu Asn Arg Ile Gly Asn Phe Leu Val Pro Asn Asp Asn Tyr Cys
1 5 10 15
cgc ttt gaa gac tgg gtg atg cca atc ctc gac aca atg ctc gaa gaa 96
Arg Phe Glu Asp Trp Val Met Pro Ile Leu Asp Thr Met Leu Glu Glu
20 25 30
cag gaa gca tgc aag ggt tcg ggc gaa gca atc cac tgg act ccc agc 144
Gln Glu Ala Cys Lys Gly Ser Gly Glu Ala Ile His Trp Thr Pro Ser
35 40 45
aaa atc atc aac cgg ctt ggc aag gag gtc aac gac gaa tcg tcc gtg 192
Lys Ile Ile Asn Arg Leu Gly Lys Glu Val Asn Asp Glu Ser Ser Val
50 55 60
tac tac tgg gca tgg aag aac gac att cca gtg ttc tgt ccg gcg ctt 240
Tyr Tyr Trp Ala Trp Lys Asn Asp Ile Pro Val Phe Cys Pro Ala Leu
65 70 75 80
act gat ggc agt ctc gga gac atg ctg tac ttc cac acg ttc aaa tcc 288
Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Thr Phe Lys Ser
85 90 95
tca ccg cag cag ctt cga gtc gac att gtg gaa gac atc cga aag atc 336
Ser Pro Gln Gln Leu Arg Val Asp Ile Val Glu Asp Ile Arg Lys Ile
100 105 110
aac acc ctc gcc gtc cga gcc aag cgc act ggc atg atc att ctc gga 384
Asn Thr Leu Ala Val Arg Ala Lys Arg Thr Gly Met Ile Ile Leu Gly
115 120 125
ggc ggc att gtc aag cac cac atc gca aat gcc aac ctg atg cgc aat 432
Gly Gly Ile Val Lys His His Ile Ala Asn Ala Asn Leu Met Arg Asn
130 135 140
ggc gcg gaa agc gca gtg tac atc aat acc gcg ccg aat tcg acg gat 480
Gly Ala Glu Ser Ala Val Tyr Ile Asn Thr Ala Pro Asn Ser Thr Asp
145 150 155 160
ccg acc a 487
Pro Thr
<210> 164
<211> 162
<212> PRT
<213> Mycosphaerella fijiensis
<400> 164
Gly Leu Asn Arg Ile Gly Asn Phe Leu Val Pro Asn Asp Asn Tyr Cys
1 5 10 15
Arg Phe Glu Asp Trp Val Met Pro Ile Leu Asp Thr Met Leu Glu Glu
20 25 30
Gln Glu Ala Cys Lys Gly Ser Gly Glu Ala Ile His Trp Thr Pro Ser
35 40 45
Lys Ile Ile Asn Arg Leu Gly Lys Glu Val Asn Asp Glu Ser Ser Val
50 55 60
Tyr Tyr Trp Ala Trp Lys Asn Asp Ile Pro Val Phe Cys Pro Ala Leu
65 70 75 80
Thr Asp Gly Ser Leu Gly Asp Met Leu Tyr Phe His Thr Phe Lys Ser
85 90 95
Ser Pro Gln Gln Leu Arg Val Asp Ile Val Glu Asp Ile Arg Lys Ile
100 105 110
Asn Thr Leu Ala Val Arg Ala Lys Arg Thr Gly Met Ile Ile Leu Gly
115 120 125
Gly Gly Ile Val Lys His His Ile Ala Asn Ala Asn Leu Met Arg Asn
130 135 140
Gly Ala Glu Ser Ala Val Tyr Ile Asn Thr Ala Pro Asn Ser Thr Asp
145 150 155 160
Pro Thr
<210> 165
<211> 214
<212> DNA
<213> Mycosphaerella fijiensis
<400> 165
aatcgaattc ccgcggccgc catggcggcc gggagcatgc gacgtcgggc ccaattcgcc 60
ctatagtgag tcgtattaca attcactggc cgtcgtttta caacgtcgtg actgggaaaa 120
cctggcggta cccaacttaa tcgccttgca gcacatcccc ctttcgccag ctggcgtaat 180
agcgaagagg cccgcaccga tcgcccttcc aaca 214
<210> 166
<211> 1490
<212> DNA
<213> Lactuca sativa
<400> 166
gctcagcatt tcgtcgccgg tacctccccg ccgcctctcc ctaccggttc atcttcccca 60
ccggcgctcc ctctccttca ccggctcgac aatatctttt tcgccctagt gttctacaga 120
aacagcccct tagtagcgtg agagtgcagc agcccaagtt gcagatatgg gggaagtcgt 180
taaagaggca ggggtgattg agaacttaag gtcggtggtg ttcaaggaat cagagagttt 240
agaaggcagc tgtgctaaaa tccagggcta cgatttcaac actgggatta attattcgca 300
gattctcaaa tccctaattt ccactggatt ccaagcttct aacctcggtg atgccattga 360
aaccgttaat caaatgctag actggaggct ttctcatgag caagtaaccg aagattgcag 420
tgaggaagag agtaatccag cttacagaga gtctgtcaag tgcaaaatct ttctagggtt 480
cacttcaaac ctcatttctt ctggtgttcg agacattatt cgctatctaa ctcaacatca 540
tatggttgat gtgattgtga cgacaaccgg tggcattgag gaagatctga taaaatgtct 600
tgcagacaca tacagaggtg aattttctct acctggtgct gccttaaggt caaaaggtct 660
taatcgtatt ggtaacttat tggtgccaaa tgataactac tgtaaatttg aggattggat 720
tatcccaata ttcgaccaaa tgcttcaaga acaaaataca cagcatgtgt tatggactcc 780
atcaaaagta atatcacgtt tggggaagga gattaataat gagagttctt atttatattg 840
ggcatataag aacgacattc ctgtgttttg cccaggctta acagacgggt ctctagggga 900
catgttatat ttccattcat ttcgcaatcc gggtcttgtt attgatgtag tacaagatat 960
acgcgcgatt aacagcgagg ctgtgcatgc aaaccctaga aagacaggga tgataattct 1020
aggaggaggg ttaccaaaac atcacatttg taatgcgaat atgatgagaa atggtgcgga 1080
ttatgctgtt ttcatcaaca ctgctcagga gtttgatggc agtgattctg gtgctcgacc 1140
tgatgaagct gtctcctggg ggaaaatacg tggttctgct aaatctgtca aggtgcactg 1200
tgatgcaact atcgcgttcc ctttacttgt tgcagaaaca tttgctgcaa agagagaggg 1260
ggagatgaaa aatgttgagt caaccaaagc tttggtttaa aaaggtggaa cagtgtagga 1320
cagggactca tttttgatat tttgtttgct aaaaaatggt ctttggaaga atattgatgc 1380
acacaaacaa ggagacaatg ttactgatct tggagagtgt acatgtaaaa tgtctaaata 1440
atttcaaagc ttctcacaac aaatcaaact taaaaaaaaa aaaaaaaaaa 1490
<210> 167
<211> 377
<212> PRT
<213> Lactuca sativa
<400> 167
Met Gly Glu Val Val Lys Glu Ala Gly Val Ile Glu Asn Leu Arg Ser
1 5 10 15
Val Val Phe Lys Glu Ser Glu Ser Leu Glu Gly Ser Cys Ala Lys Ile
20 25 30
Gln Gly Tyr Asp Phe Asn Thr Gly Ile Asn Tyr Ser Gln Ile Leu Lys
35 40 45
Ser Leu Ile Ser Thr Gly Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile
50 55 60
Glu Thr Val Asn Gln Met Leu Asp Trp Arg Leu Ser His Glu Gln Val
65 70 75 80
Thr Glu Asp Cys Ser Glu Glu Glu Ser Asn Pro Ala Tyr Arg Glu Ser
85 90 95
Val Lys Cys Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Ile Ser Ser
100 105 110
Gly Val Arg Asp Ile Ile Arg Tyr Leu Thr Gln His His Met Val Asp
115 120 125
Val Ile Val Thr Thr Thr Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys
130 135 140
Leu Ala Asp Thr Tyr Arg Gly Glu Phe Ser Leu Pro Gly Ala Ala Leu
145 150 155 160
Arg Ser Lys Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp
165 170 175
Asn Tyr Cys Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Gln Met
180 185 190
Leu Gln Glu Gln Asn Thr Gln His Val Leu Trp Thr Pro Ser Lys Val
195 200 205
Ile Ser Arg Leu Gly Lys Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr
210 215 220
Trp Ala Tyr Lys Asn Asp Ile Pro Val Phe Cys Pro Gly Leu Thr Asp
225 230 235 240
Gly Ser Leu Gly Asp Met Leu Tyr Phe His Ser Phe Arg Asn Pro Gly
245 250 255
Leu Val Ile Asp Val Val Gln Asp Ile Arg Ala Ile Asn Ser Glu Ala
260 265 270
Val His Ala Asn Pro Arg Lys Thr Gly Met Ile Ile Leu Gly Gly Gly
275 280 285
Leu Pro Lys His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly Ala
290 295 300
Asp Tyr Ala Val Phe Ile Asn Thr Ala Gln Glu Phe Asp Gly Ser Asp
305 310 315 320
Ser Gly Ala Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly
325 330 335
Ser Ala Lys Ser Val Lys Val His Cys Asp Ala Thr Ile Ala Phe Pro
340 345 350
Leu Leu Val Ala Glu Thr Phe Ala Ala Lys Arg Glu Gly Glu Met Lys
355 360 365
Asn Val Glu Ser Thr Lys Ala Leu Val
370 375
<210> 168
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<220>
<221> modified_base
<222> (9)
<223> a, c, g, t, unknown or other
<220>
<221> modified_base
<222> (12)
<223> a, c, g, t, unknown or other
<220>
<221> modified_base
<222> (18)
<223> a, c, g, t, unknown or other
<400> 168
tgtgatgcna cnathgcntt ycc 23
<210> 169
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 169
gactgcagtc gacatcgatt tttttttttt ttttt 35
<210> 170
<211> 431
<212> DNA
<213> Petunia sp.
<220>
<221> CDS
<222> (1)..(87)
<400> 170
tgt gat gca acg atc gcg ttc ccc ata tta gta gct gag aca ttt gca 48
Cys Asp Ala Thr Ile Ala Phe Pro Ile Leu Val Ala Glu Thr Phe Ala
1 5 10 15
gct aag aaa aac gaa ttc tca cac ata agg tgc caa gtt tgacaattga 97
Ala Lys Lys Asn Glu Phe Ser His Ile Arg Cys Gln Val
20 25
gaagctgcgt ccaatcacat atctgaatgg tctattcttg ttagttgcaa catcaattat 157
tctgcgcaaa ctgactagaa agggtacatt caagtgccct gagtttagag aacatttatg 217
gttctaataa ttatttttcc cttcccatca tgttatttgt gggaggagcc atatttagtt 277
ctctccctgt gctcgagtgg agagtctctg atgttgaatt tgagaataga gtagctggtc 337
tggaggtgtg gccatagttc ttagtgtgca taggttttga accttactgt attaatggct 397
ctgggttttg gctcaaaaaa aaaaaaaaaa aaaa 431
<210> 171
<211> 29
<212> PRT
<213> Petunia sp.
<400> 171
Cys Asp Ala Thr Ile Ala Phe Pro Ile Leu Val Ala Glu Thr Phe Ala
1 5 10 15
Ala Lys Lys Asn Glu Phe Ser His Ile Arg Cys Gln Val
20 25
<210> 172
<211> 753
<212> DNA
<213> Medicago sativa
<220>
<221> CDS
<222> (65)..(544)
<400> 172
gccgaaccaa atctctgttc tcattatcgc tcttcggcac cgccaccgcc actcctgaat 60
caat atg tct gac gaa gag cac caa ttt gaa tct aag gcc gat gcc ggt 109
Met Ser Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly
1 5 10 15
gct tcc aaa act tat cct caa caa gct ggt acc atc cgt aag aat gga 157
Ala Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly
20 25 30
tac ata gtt atc aaa aac cgt cca tgc aag gtg gtt gaa gtt tcg act 205
Tyr Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr
35 40 45
tcg aag acc ggg aag cat gga cat gcc aag tgt cat ttt gtt gct att 253
Ser Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile
50 55 60
gat ata ttc act tca aag aag ctt gag gat atc gtg ccg tct tca cat 301
Asp Ile Phe Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His
65 70 75
aat tgt gat gtt cct cac gtg aat cgt acc gat tat cag ctt att gac 349
Asn Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp
80 85 90 95
att tct gaa gat gga ttt gtg agc ttg ctc act gaa aat gga aac act 397
Ile Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr
100 105 110
aag gat gac ctc aaa ctt ccc acc gat gac agt ctg ctc act cag atc 445
Lys Asp Asp Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile
115 120 125
aag gat gga ttt gct gag gga aag gac ctt gtt gta tcc gtg atg tct 493
Lys Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser
130 135 140
gct atg ggt gag gag cag atc tgt gct ttg aag gat att ggt gga aag 541
Ala Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Gly Lys
145 150 155
aac taacttattt ctagctattt gttttgtttg ctatgatcat ttctctaagc 594
Asn
160
tttcacattg ttttgtttaa gagatctgtc ttatagattc ttgtgagact agtggtgttg 654
gtggtgttta tggatttttg ttggtttatc tgttactgtt tttctgttgt cgtgtgactt 714
cttcaaacga gtatttcctt aaaaaaaaaa aaaaaaaaa 753
<210> 173
<211> 160
<212> PRT
<213> Medicago sativa
<400> 173
Met Ser Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Thr Ser Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys
115 120 125
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Gly Lys Asn
145 150 155 160
<210> 174
<211> 743
<212> DNA
<213> Medicago sativa
<220>
<221> CDS
<222> (51)..(527)
<400> 174
gctcacacat aacacattac ccacgcgcgc agagaaagca tcaatcaatc atg tct 56
Met Ser
1
gac gaa gaa cat cac ttt gaa ccc gct gcc gat gcc gga gca tcc aaa 104
Asp Glu Glu His His Phe Glu Pro Ala Ala Asp Ala Gly Ala Ser Lys
5 10 15
acc tac cct caa cag gct ggt act att cgc aag aat ggt tac ata gtc 152
Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val
20 25 30
atc aag tcc agg cct tgc aag gtt gtt gag gtt tct act tca aaa aca 200
Ile Lys Ser Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr
35 40 45 50
gga aaa cat gga cat gca aag tgt cac ttt gtt gct att gat att ttc 248
Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe
55 60 65
aat ggg aaa aaa ctt gaa gat att gtt cct tca tcc cac aat tgt gat 296
Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
70 75 80
gtt cct cat gtg aat cgt act gat tat cag ttg att gat att tct gaa 344
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
85 90 95
gat gga ttt gtg agt ttg ttg act gac aat gga agt acc aaa gat gat 392
Asp Gly Phe Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys Asp Asp
100 105 110
ctg aag ctt cct act gat gat tca cta ctt act cag att aaa gat gga 440
Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys Asp Gly
115 120 125 130
ttt gct gat gga aaa gat ctc gtg gtt tct gtt atg tct gcg atg ggt 488
Phe Ala Asp Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly
135 140 145
gaa gag cag ata tgt gcc ctg aag gac att ggt cct aag tagtaatttt 537
Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
150 155
tcgtatgatt gtgtgctcaa taaacttgtc aatgtggaaa agagcttgaa actcataaaa 597
catggtgtgg agaaaaggac catatttatc ttgtctcttt aattattgaa ctctctggtt 657
ttggccagat gaattcttta agtgtccctt gtaactttat atttttgttg agtttaatat 717
tgaaccctaa aaaaaaaaaa aaaaaa 743
<210> 175
<211> 159
<212> PRT
<213> Medicago sativa
<400> 175
Met Ser Asp Glu Glu His His Phe Glu Pro Ala Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Ser Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Asp Asn Gly Ser Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys
115 120 125
Asp Gly Phe Ala Asp Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 176
<211> 535
<212> DNA
<213> Medicago sativa
<220>
<221> CDS
<222> (2)..(313)
<400> 176
c aag tgt cac ttt gtt gga atc gat att ttc act tcc aaa aag ctc gaa 49
Lys Cys His Phe Val Gly Ile Asp Ile Phe Thr Ser Lys Lys Leu Glu
1 5 10 15
gat att gtg ccc tct tcc cac aat tgt gat gtt ccc cat gtg aat cgt 97
Asp Ile Val Pro Ser Ser His Asn Cys Asp Val Pro His Val Asn Arg
20 25 30
acc gac tat caa ttg atc gat att tct gaa gat gga ttt gtg agt ctg 145
Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly Phe Val Ser Leu
35 40 45
ctt act gaa aat ggt aac acc aag gat gac ttg aag ctt cct acc gat 193
Leu Thr Glu Asn Gly Asn Thr Lys Asp Asp Leu Lys Leu Pro Thr Asp
50 55 60
gac aat ctg ctt act cag atc aag gaa ggt ttt gct gag ggt aag gac 241
Asp Asn Leu Leu Thr Gln Ile Lys Glu Gly Phe Ala Glu Gly Lys Asp
65 70 75 80
ctt gtt gtg tcc gtg atg tct gct atg ggt gag gag cag att aat gcc 289
Leu Val Val Ser Val Met Ser Ala Met Gly Glu Glu Gln Ile Asn Ala
85 90 95
ctg aaa gat att ggt cca aag aac tagcttcaac tattactgcc tatttgttcc 343
Leu Lys Asp Ile Gly Pro Lys Asn
100
ttctatttaa gcaaaagata tttttgtatt gtcttattgt tcaacccttt ggttatggac 403
actggtcaga ctagtcttac caactgtgtt tatggatgtg acacaaacta tttagcattt 463
ttctgtttcc atgctgtccc gagttttttt tataaaaaat acaagtttgt taaaaaaaaa 523
aaaaaaaaaa aa 535
<210> 177
<211> 104
<212> PRT
<213> Medicago sativa
<400> 177
Lys Cys His Phe Val Gly Ile Asp Ile Phe Thr Ser Lys Lys Leu Glu
1 5 10 15
Asp Ile Val Pro Ser Ser His Asn Cys Asp Val Pro His Val Asn Arg
20 25 30
Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly Phe Val Ser Leu
35 40 45
Leu Thr Glu Asn Gly Asn Thr Lys Asp Asp Leu Lys Leu Pro Thr Asp
50 55 60
Asp Asn Leu Leu Thr Gln Ile Lys Glu Gly Phe Ala Glu Gly Lys Asp
65 70 75 80
Leu Val Val Ser Val Met Ser Ala Met Gly Glu Glu Gln Ile Asn Ala
85 90 95
Leu Lys Asp Ile Gly Pro Lys Asn
100
<210> 178
<211> 774
<212> DNA
<213> Medicago sativa
<220>
<221> CDS
<222> (1)..(477)
<400> 178
atg tcg gat gac gag cac cat ttt gag tcc aaa gca gat gct gga gca 48
Met Ser Asp Asp Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
tca aag aca tat cca caa caa gct ggt act atc cgc aaa aat ggt cac 96
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly His
20 25 30
att gtt atc aaa aac aga ccc tgc aag gta gtt gaa gtt tca act tct 144
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
aaa act gga aag cat gga cat gca aag tgt cac ttt gtt ggt att gat 192
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Gly Ile Asp
50 55 60
atc ttc act ggc aaa aaa ctt gaa gat att gtt cct tct tcc cat aac 240
Ile Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
tgt gat gtt cct cat gtt aat cgc act gat tat cag ctc att gat ata 288
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
tct gag gat gga ttt gtg agt ttg ctg gct gag agt ggt ggt act aag 336
Ser Glu Asp Gly Phe Val Ser Leu Leu Ala Glu Ser Gly Gly Thr Lys
100 105 110
gat gac ctt aag ctt cca acc gat gag agt ctg ctt tcg cag atc aag 384
Asp Asp Leu Lys Leu Pro Thr Asp Glu Ser Leu Leu Ser Gln Ile Lys
115 120 125
gat gga ttt ggt gag ggt aaa gat ctg att gtg tct gtc atg tct gct 432
Asp Gly Phe Gly Glu Gly Lys Asp Leu Ile Val Ser Val Met Ser Ala
130 135 140
atg ggt gag gag cag att tgt ggc ctc aag gac att ggc ccc aag 477
Met Gly Glu Glu Gln Ile Cys Gly Leu Lys Asp Ile Gly Pro Lys
145 150 155
taataaatcc atgaatgctt cttgaaaagg agatgatata tttaatattt gtgtgtgatt 537
gttgtgtaaa aggaccaatc taagtttgtg caaattctta caagatttgg taatggttaa 597
ctttattgag tctttgaccc cttatatttt tgtggtctaa gatcaatatt ggaagcatta 657
agactgttat atgttgtgaa gaagttacat tggctactgg ccagtaacat ttaatcatgt 717
gctaatataa gaccctctaa catttctact ataaaaaaaa aaaaaaaaaa aaaaaaa 774
<210> 179
<211> 159
<212> PRT
<213> Medicago sativa
<400> 179
Met Ser Asp Asp Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly His
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Gly Ile Asp
50 55 60
Ile Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Ala Glu Ser Gly Gly Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Thr Asp Glu Ser Leu Leu Ser Gln Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Ile Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Gly Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 180
<211> 598
<212> DNA
<213> Lactuca sativa
<220>
<221> CDS
<222> (1)..(477)
<400> 180
atg tcg gac gag gaa cac cag ttt gaa tca aag gcc gac gcc ggt gct 48
Met Ser Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
tcc aag acc tac cct cag caa gcc ggt act atc cgc aag ggc ggc cac 96
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His
20 25 30
atc gtc atc aaa aat cga gct tgc aag gtt gtt gaa gtt tct acc tcc 144
Ile Val Ile Lys Asn Arg Ala Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
aag act ggg aag cat ggg cat gct aag tgt cac ttt gtc gcc att gat 192
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
atc ttc aat ggt aaa aag ctt gaa gat att gtt ccc tca tcc cat aac 240
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
tgt gat gtt cct cat gtc aac cgc act gac tac cag cta att gat att 288
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
tct gag gat gga ttt gtg agt ttg ctg act gaa aat gga aac act aaa 336
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
gat gac ctc aag ctc cca act gat gat gct ctc ctg act cag atc aag 384
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile Lys
115 120 125
gat gga ttt gca gag gga aag gac ttg gtg gtg agt gtg atg tca gca 432
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
atg gga gag gag cag atc tgt gct ctt aag gat atc ggc cca aag 477
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
tagtaatcta cttctactaa gtctataaat gaatccataa acacttgttg cttgcattat 537
cagcagaatg ttttctgtta taatagtagc aatctactct accaaaaaaa aaaaaaaaaa 597
a 598
<210> 181
<211> 159
<212> PRT
<213> Lactuca sativa
<400> 181
Met Ser Asp Glu Glu His Gln Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His
20 25 30
Ile Val Ile Lys Asn Arg Ala Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile Lys
115 120 125
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 182
<211> 614
<212> DNA
<213> Lactuca sativa
<220>
<221> CDS
<222> (1)..(327)
<400> 182
ggg aag cac gga cac gcc aag tgt cat ttc gtt gcc att gat atc ttc 48
Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe
1 5 10 15
aat gga aaa aag ctt gaa gat atc gtt ccc tct tcc cac aat tgt gat 96
Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
20 25 30
gtt cca cat gtc aac cga act gac tac cag ctc att gac att tct gaa 144
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
35 40 45
gat gga ttt gtg agt ctg ttg act gaa aat ggt ggc act aag gat gat 192
Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Gly Thr Lys Asp Asp
50 55 60
ctc aag ctt cca acc gat gat agt ctg ctt aca cag att aag gat gga 240
Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys Asp Gly
65 70 75 80
ttt gct gaa gga aag gat ctt gtg gtg aca gtg atg tct gcg atg gga 288
Phe Ala Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ala Met Gly
85 90 95
gag gag cag atc tgt gct ctt aag gat att ggt ccc aag taactttacc 337
Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
100 105
ttgtgtggat tgagagagat tttatatgaa gaactgcgtt tgctagattt gtggtgatgg 397
ttatttaaga aaaaaaaact ttattaggtt tcttttttct attttacaaa tttttgtttt 457
gtttttggtt attgttgatg tgctaaaaaa gtcttgaaat ttgggcttgg ggagaggatg 517
ggcaatatgt acgtacactt tgtatctcga tatgtttggg atgcgacttg tgaacttatg 577
gagtggttat tctgttaaaa aaaaaaaaaa aaaaaaa 614
<210> 183
<211> 109
<212> PRT
<213> Lactuca sativa
<400> 183
Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe
1 5 10 15
Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
20 25 30
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
35 40 45
Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Gly Thr Lys Asp Asp
50 55 60
Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys Asp Gly
65 70 75 80
Phe Ala Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ala Met Gly
85 90 95
Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
100 105
<210> 184
<211> 536
<212> DNA
<213> Lactuca sativa
<220>
<221> CDS
<222> (1)..(339)
<400> 184
tcg aag act ggt aag cac ggt cat gcg aag tgt cac ttt gtt gca atc 48
Ser Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile
1 5 10 15
gac atc ttc aat gga aaa aag ctt gaa gat atc gtc cca tct tcc cat 96
Asp Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His
20 25 30
aac tgt gat gtc cct cat gtc aac cgc act gac tac cag cta att gac 144
Asn Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp
35 40 45
atc tct gaa gat gga ttt gtg agt ttg ttg act gaa aat ggc aac acc 192
Ile Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr
50 55 60
aag gat gac ctg aaa ctg cca acc gat gat gct ctt ttg act cag atc 240
Lys Asp Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile
65 70 75 80
aag gag gga ttt gga gag ggg aag gat ttg gtt gtg agt gtg atg tct 288
Lys Glu Gly Phe Gly Glu Gly Lys Asp Leu Val Val Ser Val Met Ser
85 90 95
gcc atg gga gag gag cag ata tgt gct gtt aag gac att tct ggt ccg 336
Ala Met Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Ile Ser Gly Pro
100 105 110
aag tagactgatc tttttatatc aacagaaagg tatgttcttg ttcttgttgt 389
Lys
tgttgttgtt gtaatagtag cagcatttta ctttgtgcac tactatgact agctcatttg 449
ggctacgaac agtgtatttt attggtttgt gtactttgaa atttgaattc ctccatgttg 509
tcaagtgtgc aaaaaaaaaa aaaaaaa 536
<210> 185
<211> 113
<212> PRT
<213> Lactuca sativa
<400> 185
Ser Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile
1 5 10 15
Asp Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His
20 25 30
Asn Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp
35 40 45
Ile Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr
50 55 60
Lys Asp Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile
65 70 75 80
Lys Glu Gly Phe Gly Glu Gly Lys Asp Leu Val Val Ser Val Met Ser
85 90 95
Ala Met Gly Glu Glu Gln Ile Cys Ala Val Lys Asp Ile Ser Gly Pro
100 105 110
Lys
<210> 186
<211> 471
<212> DNA
<213> Lactuca sativa
<400> 186
tcgaagacag ggaagcacgg tcatgccaag tgtcactttg ttgcaatcga catttttaac 60
gggaaaaagc ttgaagatat tgttccatca tcccataatt gtgatgtccc ccatgtcaat 120
cgcaccgatt accagctaat tgatatttct gaagatggat ttgtgagttt gctgactgaa 180
aatggtaaca ccaaagatga cctcaaactc cccaccgatg atgctctcct cactcagata 240
aaggatggat ttggagaggg gaagggcctg gttgtgactg ttatgtcagc aatgggagag 300
gaacagatat gtgctctcaa ggacattggc cccaaataac ttttcaaaaa tccacaaaaa 360
cacacactat aatattatta taacatttgg attaacagaa aacaagttgt gtttttattt 420
gttttatcat gtacagatgt tatggattaa aaaaaaaaaa aaaaaaaaaa a 471
<210> 187
<211> 112
<212> PRT
<213> Lactuca sativa
<400> 187
Ser Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile
1 5 10 15
Asp Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His
20 25 30
Asn Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp
35 40 45
Ile Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr
50 55 60
Lys Asp Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile
65 70 75 80
Lys Asp Gly Phe Gly Glu Gly Lys Gly Leu Val Val Thr Val Met Ser
85 90 95
Ala Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
100 105 110
<210> 188
<211> 700
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (56)..(532)
<400> 188
aaatttctcc ttctccttaa tcctctccac cggcgaaccg gcgaagatca aaacg atg 58
Met
1
tcg gac gaa gag cac cac ttc gaa tcc aag gcc gat gcc gga gct tca 106
Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser
5 10 15
aag acg tat cct caa caa gct ggt act att cgt aaa ggt ggt cac atc 154
Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His Ile
20 25 30
gtc ata aaa aat cgt cct tgc aag gtg gtt gaa gtt tca act tcc aag 202
Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys
35 40 45
aca ggc aag cac ggt cat gct aaa tgt cac ttc gtg gca att gac att 250
Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile
50 55 60 65
ttc act gga aag aaa ctt gag gat att gtt ccc tct tct cac aat tgt 298
Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
70 75 80
gat gtt cct cat gtg aat agg act gat tat caa ctt att gat atc tct 346
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
85 90 95
gag gat ggc ttt gtg agt ctg ttg act gaa aat ggt aac acc aag gat 394
Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp
100 105 110
gac ttg aga ctc cca act gat gat act ctt ctg gct cag gtc aaa gat 442
Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Ala Gln Val Lys Asp
115 120 125
ggt ttt gct gag ggg aaa gac ctg gtt cta tca gtg atg tct gcc atg 490
Gly Phe Ala Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala Met
130 135 140 145
gga gag gag cag att tgt ggt atc aag gac att ggc cct aag 532
Gly Glu Glu Gln Ile Cys Gly Ile Lys Asp Ile Gly Pro Lys
150 155
tagctgcaga tggtattggt gtatgtttac agagtttcta taaaagatgt attaagaacc 592
aaaacttctt tactttctct tgcagttgct ctatataact gccatttaac tattattata 652
tgtgttgtga ttagattctt gtctcactac agtatttcct ttactctg 700
<210> 189
<211> 159
<212> PRT
<213> Lycopersicon esculentum
<400> 189
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Gly Gly His
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Thr Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Asp Thr Leu Leu Ala Gln Val Lys
115 120 125
Asp Gly Phe Ala Glu Gly Lys Asp Leu Val Leu Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Gly Ile Lys Asp Ile Gly Pro Lys
145 150 155
<210> 190
<211> 764
<212> DNA
<213> Lycopersicon esculentum
<220>
<221> CDS
<222> (43)..(522)
<400> 190
aaagaatcct agagagagaa agggaatcct agagagagaa gc atg tcg gac gaa 54
Met Ser Asp Glu
1
gaa cac cat ttt gag tca aag gca gat gct ggt gcc tca aaa act ttc 102
Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser Lys Thr Phe
5 10 15 20
cca cag caa gct gga acc atc cgt aag aat ggt tac atc gtt atc aaa 150
Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val Ile Lys
25 30 35
ggc cgt ccc tgc aag gtt gtt gag gtc tcc act tca aaa act gga aaa 198
Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys
40 45 50
cac gga cat gct aaa tgt cac ttt gtg gca att gac att ttc aat gga 246
His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe Asn Gly
55 60 65
aag aaa ctg gaa gat atc gtt ccg tcc tcc cac aat tgt gat gtg cca 294
Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp Val Pro
70 75 80
cat gtt aac cgt acc gac tat cag ctg att gat atc tct gaa gat ggt 342
His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly
85 90 95 100
ttt gtc tca ctt ctt act gaa agt gga aac acc aag gat gac ctc agg 390
Phe Val Ser Leu Leu Thr Glu Ser Gly Asn Thr Lys Asp Asp Leu Arg
105 110 115
ctt ccc acc gat gaa aat ctg ctg aag cag gtt aaa gat ggg ttc cag 438
Leu Pro Thr Asp Glu Asn Leu Leu Lys Gln Val Lys Asp Gly Phe Gln
120 125 130
gaa gga aag gat ctt gtg gtg tct gtt atg tct gcg atg ggc gaa gag 486
Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly Glu Glu
135 140 145
cag att aac gcc gtt aag gat gtt ggt acc aag aat tagttatgtc 532
Gln Ile Asn Ala Val Lys Asp Val Gly Thr Lys Asn
150 155 160
atggcagcat ataatcactg ccaaagcttt aagacattat catatcctaa tgtggtactt 592
tgatatcact agattataaa ctgtgttatt tgcactgttc aaaacaaaag aaagaaaact 652
gctgttatgg ctagagaaag tattggcttt gagcttttga cagcacagtt gaactatgtg 712
aaaattctac tttttttttt ttgggtaaaa tactgctcgt ttaatgtttt gc 764
<210> 191
<211> 160
<212> PRT
<213> Lycopersicon esculentum
<400> 191
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Phe Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Gly Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Ser Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Glu Asn Leu Leu Lys Gln Val Lys
115 120 125
Asp Gly Phe Gln Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Asn Ala Val Lys Asp Val Gly Thr Lys Asn
145 150 155 160
<210> 192
<211> 825
<212> DNA
<213> Populus deltoides
<220>
<221> CDS
<222> (60)..(536)
<400> 192
ggtactctct gtgttttttt attgttagtg ttttctagtt ctcttgatca atcgccgcc 59
atg tct gac gag gag cat cac ttc gag tca aag gcc gat gct gga gct 107
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
tca aaa act ttt cct cag caa gct ggt acc att cgc aag aat ggt tac 155
Ser Lys Thr Phe Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
ata gtc atc aag aat cgc cct tgc aag gtc gtg gag gtt tca acc tct 203
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
aaa act ggc aag cat ggc cat gct aaa tgt cac ttt gtt gcg att gat 251
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
att ttc aat gga aaa aag ctt gaa gat att gtt cct tct tcc cac aac 299
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
tgt gat gtt ccc cac gtc acc cgt act gac tac cag ctg att gat atc 347
Cys Asp Val Pro His Val Thr Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
tct gag gat gga ttt gtg agc tta ctg act gag aat ggc aat act aag 395
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
gat gac ctg aaa ctc cca act gat gac agt ctc ctc act cag att aag 443
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys
115 120 125
gat gga ttt ggt gag ggg aaa gat ctt gtt gtg acg gtg atg tct tcc 491
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ser
130 135 140
atg gga gag gag cag atc tgc gcc ctc aag gac att ggt ccc aag 536
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
taatctgcgc ctggctgcaa gcagcgcatg cccgaggttg actgctgata gttgtagttt 596
tagcatgggg ttttatgtgg gtttgagaac tgggcccatt taagtaacgt tggcggcctt 656
atatgaagac ttgccattat ttaagtggtt ttttatttgt aatgaaggat attcttgtgg 716
ttcttgttgt ggagaaagtt tgttctgcca ttaagactgt atccaccttc ttgccccctt 776
gggtggtttc aaccaatttc cagtttttat taaaaaaaaa aaaaaaaaa 825
<210> 193
<211> 159
<212> PRT
<213> Populus deltoides
<400> 193
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Phe Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Thr Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Lys Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ser
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
145 150 155
<210> 194
<211> 846
<212> DNA
<213> Populus deltoides
<220>
<221> CDS
<222> (101)..(580)
<220>
<221> modified_base
<222> (729)
<223> a, c, g, t, unknown or other
<400> 194
gtgttggtgt cctcttgtgt cttcagagtc atcgtcctct ttcacttgtt cttctttttt 60
ctgcttgaga gagaatcgat ccaagcctaa ctaaaccatc atg tcg gac gag gag 115
Met Ser Asp Glu Glu
1 5
cac cac ttt gaa tcc aag gct gat gca ggc gcc tcc aag act tac cct 163
His His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser Lys Thr Tyr Pro
10 15 20
cag caa gct ggt acc atc cgc aag aat ggt tac ata gtc atc aag aac 211
Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr Ile Val Ile Lys Asn
25 30 35
cgc cct tgc aag gtt gtt gag gtt tcc acc tca aag aca ggc aag cac 259
Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His
40 45 50
gga cat gct aag tgc cac ttt gtg gga att gat ata ttt aat ggg aaa 307
Gly His Ala Lys Cys His Phe Val Gly Ile Asp Ile Phe Asn Gly Lys
55 60 65
aag ctt gaa gat att gtt ccc tca tct cac aac tgt gat gtt ccc cat 355
Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp Val Pro His
70 75 80 85
gtt aat cgt gtt gat tat caa ttg att gat atc tct gaa gat ggt ttt 403
Val Asn Arg Val Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly Phe
90 95 100
gtg agt ctt ttg act gaa aat gga aat acc aag gat gat ctg agg ctt 451
Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp Asp Leu Arg Leu
105 110 115
ccc act gat gac agt cta ctt acc cag att aaa gat gga ttt ggc gaa 499
Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys Asp Gly Phe Gly Glu
120 125 130
ggg aag gac ctt gtg gtg tct gtc atg tct gcc atg gga gag gag cag 547
Gly Lys Asp Leu Val Val Ser Val Met Ser Ala Met Gly Glu Glu Gln
135 140 145
atc tgt gcc ctt aag gac att ggc cca aaa aat tgaatatgat gctatagctg 600
Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys Asn
150 155 160
tagctgtagc tgatggcaaa acgatttctt ttatgttgct aacaaggatt tttatccact 660
atgcatgtgt acaacaaact tataagacgg tattttagct atggaattga aactcgcaag 720
gcatgtaang gaggaggtgg gaaggacccc ggaaatgctg attccctgat gaagggaact 780
tgtgttatca gcaggtggga atttatatat atttctattt cgctaaaaaa aaaaaaaaaa 840
aaaaaa 846
<210> 195
<211> 160
<212> PRT
<213> Populus deltoides
<400> 195
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Gly Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Val Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Asp Ser Leu Leu Thr Gln Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Ser Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys Asn
145 150 155 160
<210> 196
<211> 824
<212> DNA
<213> Populus deltoides
<220>
<221> CDS
<222> (48)..(524)
<400> 196
ggtattttgt ttgttttgtg attgcgactc tcttatcaat cgcggcc atg tct gac 56
Met Ser Asp
1
gag gag cag cac ttc gag tca aaa gct gat gcg gga gct tcg aaa act 104
Glu Glu Gln His Phe Glu Ser Lys Ala Asp Ala Gly Ala Ser Lys Thr
5 10 15
tac cct caa caa gct ggt acc att cgc aag agc ggt tac att gtc atc 152
Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Ser Gly Tyr Ile Val Ile
20 25 30 35
aag aat cgc cct tgc aag gtt gtg gag gtt tct acc tct aaa act ggc 200
Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser Lys Thr Gly
40 45 50
aag cac ggc cat gcc aaa tgt cac ttt gtt gca att gat atc ttc aat 248
Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp Ile Phe Asn
55 60 65
gga aaa aag ctt gaa gat att gtt cct tct tcc cac aac tgt gat gtt 296
Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp Val
70 75 80
ccc cat gtc acc cgt act gac tat cag ctg att gat atc tca gag gat 344
Pro His Val Thr Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp
85 90 95
gga ttt gtg agc ttg ctg act gag aat ggc aat acc aag gat gac ctg 392
Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp Asp Leu
100 105 110 115
agg ctc cca act gat gag agt ctc ctc tct cag atc aag gat gga ttt 440
Arg Leu Pro Thr Asp Glu Ser Leu Leu Ser Gln Ile Lys Asp Gly Phe
120 125 130
ggc gag ggt aaa gat ctt gtt gtg act gtg atg tcc tcc atg gga gag 488
Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ser Met Gly Glu
135 140 145
gag cag atc tgc gcc ctc aag gac gtt ggc ccg aag taaattgtac 534
Glu Gln Ile Cys Ala Leu Lys Asp Val Gly Pro Lys
150 155
cctgcttcat gcaggcatgc ccaaggttga cggctgatag ttgtagctta gcaagtggtt 594
ctatgtgggt ttgagaattg gtcccgtttc aggaacattc cgtactagat gcggccttcc 654
cttatatgaa gacttgccat tatttgagtg gcgttttatc cgtgatgata tttttgtggt 714
tcttggtgtg gggaagttta ttctgccagt aagactgcat acacttcttg gcacctgtgg 774
tggtttcaaa caattacagt ttttatttcc ttaaaaaaaa aaaaaaaaaa 824
<210> 197
<211> 159
<212> PRT
<213> Populus deltoides
<400> 197
Met Ser Asp Glu Glu Gln His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Ser Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Thr Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Glu Ser Leu Leu Ser Gln Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ser
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Val Gly Pro Lys
145 150 155
<210> 198
<211> 898
<212> DNA
<213> Populus deltoides
<220>
<221> CDS
<222> (85)..(564)
<400> 198
gctgtcctct tctgtctcga gaagtcatca tcctcttcca ttattttttc cttttctgct 60
cgagagagaa ccgaaccaac caac atg tcg gac gag gag cac cac ttt gaa 111
Met Ser Asp Glu Glu His His Phe Glu
1 5
tcg aag gct gat gca gga gcc tcc aag act tac cct cag caa gct ggt 159
Ser Lys Ala Asp Ala Gly Ala Ser Lys Thr Tyr Pro Gln Gln Ala Gly
10 15 20 25
act atc cgc aag aat ggc tat ata gtc atc aag aac cgt cct tgc aag 207
Thr Ile Arg Lys Asn Gly Tyr Ile Val Ile Lys Asn Arg Pro Cys Lys
30 35 40
gtt gtt gag gtt tcc acc tca aag aca ggc aag cat gga cat gct aag 255
Val Val Glu Val Ser Thr Ser Lys Thr Gly Lys His Gly His Ala Lys
45 50 55
tgc cac ttt gtg ggg att gat ata ttt aat ggg aaa aag ctt gaa gat 303
Cys His Phe Val Gly Ile Asp Ile Phe Asn Gly Lys Lys Leu Glu Asp
60 65 70
att gtt cct tca tct cac aac tgt gat gtt ccc cat gtt aat cgt act 351
Ile Val Pro Ser Ser His Asn Cys Asp Val Pro His Val Asn Arg Thr
75 80 85
gat tat cag ctg att gat atc tct gaa gat ggc ttt gtg agt ctt ttg 399
Asp Tyr Gln Leu Ile Asp Ile Ser Glu Asp Gly Phe Val Ser Leu Leu
90 95 100 105
act gaa aat gga aat acc aag gat gat ctg agg ctt ccc act gat gat 447
Thr Glu Asn Gly Asn Thr Lys Asp Asp Leu Arg Leu Pro Thr Asp Asp
110 115 120
agt ctg ctt act ccg att aaa gat ggg ttt ggt gaa gga aag gac ctt 495
Ser Leu Leu Thr Pro Ile Lys Asp Gly Phe Gly Glu Gly Lys Asp Leu
125 130 135
gtg gtg act gtc atg tct gcc atg gga gag gag cag atc tgt gcc ctt 543
Val Val Thr Val Met Ser Ala Met Gly Glu Glu Gln Ile Cys Ala Leu
140 145 150
aag gac att ggt cca aaa aat tgaatatgat gctgtagcag cagctatagc 594
Lys Asp Ile Gly Pro Lys Asn
155 160
tgatggcgaa gcgagtttta tgttgctaac aaggattttt atgcactatg cgttgtgtac 654
aacaaactta taagacagta ttttagatat gtcattaaaa ctcgcaaggc atgtcatgga 714
ggtggaagag gacacaggag atgcttattc cctgatgaag ggaactaggt tttcagcagg 774
atggaattta tatatatata tctacttcac atttgtctgt gctacttaaa gataaataaa 834
ggtttgtgct caaccctttt cctttcaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 894
aaaa 898
<210> 199
<211> 160
<212> PRT
<213> Populus deltoides
<400> 199
Met Ser Asp Glu Glu His His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Asn Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Gly Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Asp Ser Leu Leu Thr Pro Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ala
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys Asn
145 150 155 160
<210> 200
<211> 379
<212> PRT
<213> Nicotiana tabacum
<400> 200
Met Gly Glu Ala Leu Asn Val Met Glu Ser Val Arg Ser Ile Val Phe
1 5 10 15
Lys Glu Ser Glu Asn Leu Glu Gly Ser Ala Thr Lys Ile Glu Gly Tyr
20 25 30
Asp Phe Asn Lys Gly Val Asn Tyr Ala Glu Leu Phe Lys Ser Met Ala
35 40 45
Ser Thr Gly Phe Gln Ala Ala Asn Leu Gly Asp Ala Ile Gln Ile Val
50 55 60
Asn Gln Met Leu Asp Trp Arg Leu Ser His Glu Gln Pro Met Glu Asp
65 70 75 80
Cys Ser Glu Glu Glu Arg Asp Val Ala Tyr Arg Glu Ser Val Thr Cys
85 90 95
Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg
100 105 110
Asp Thr Ile Arg Tyr Leu Val Gln His Arg Met Val Asp Val Val Val
115 120 125
Thr Thr Ala Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro
130 135 140
Thr Tyr Lys Gly Asp Phe Ser Leu Pro Gly Ala Val Leu Arg Ser Lys
145 150 155 160
Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys
165 170 175
Lys Phe Glu Asn Trp Ile Ile Pro Ile Phe Asp Gln Met Tyr Glu Glu
180 185 190
Gln Ile Lys Glu Lys Val Leu Trp Thr Pro Ser Lys Val Ile Ala Arg
195 200 205
Leu Ala Lys Glu Ile Asn Asp Glu Thr Ser Tyr Leu Tyr Trp Ala Tyr
210 215 220
Lys Asn Arg Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly Ser Leu
225 230 235 240
Gly Asp Met Leu Tyr Phe His Ser Phe Lys Lys Gly Asp Pro Asp Asn
245 250 255
Pro Asp Leu Asn Pro Gly Leu Ile Ile Asp Ile Val Gly Asp Ile Arg
260 265 270
Ala Met Asn Ser Glu Ala Val His Ala Gly Ser Arg Lys Thr Gly Met
275 280 285
Ile Ile Leu Gly Gly Gly Leu Pro Lys His His Val Cys Asn Ala Asn
290 295 300
Met Met Arg Asn Gly Ala Asp Phe Ala Val Tyr Ile Asn Thr Ala Gln
305 310 315 320
Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu Ala Val Ser
325 330 335
Trp Gly Lys Ile Arg Gly Gly Ala Lys Thr Val Lys Val His Cys Asp
340 345 350
Ala Thr Ile Ala Phe Pro Ile Leu Val Ala Glu Thr Phe Ala Ala Lys
355 360 365
Arg Lys Glu Leu Ser His Ile Arg Cys Gln Val
370 375
<210> 201
<211> 368
<212> PRT
<213> Arabidopsis sp.
<400> 201
Met Glu Asp Asp Arg Val Phe Ser Ser Val His Ser Thr Val Phe Lys
1 5 10 15
Glu Ser Glu Ser Leu Glu Gly Lys Cys Asp Lys Ile Gln Gly Tyr Asp
20 25 30
Phe Asn Gln Gly Val Asp Tyr Pro Lys Leu Met Arg Ser Met Leu Thr
35 40 45
Thr Gly Phe Gln Ala Ser Asn Leu Gly Glu Ala Ile Asp Val Val Asn
50 55 60
Gln Met Leu Asp Trp Arg Leu Ala Asp Glu Thr Thr Val Ala Glu Asp
65 70 75 80
Cys Ser Glu Glu Glu Lys Asn Pro Ser Phe Arg Glu Ser Val Lys Cys
85 90 95
Lys Ile Phe Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg
100 105 110
Asp Thr Ile Arg Tyr Leu Val Gln His His Met Val Asp Val Ile Val
115 120 125
Thr Thr Thr Gly Gly Val Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro
130 135 140
Thr Phe Lys Gly Asp Phe Ser Leu Pro Gly Ala Tyr Leu Arg Ser Lys
145 150 155 160
Gly Leu Asn Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys
165 170 175
Lys Phe Glu Asp Trp Ile Ile Pro Ile Phe Asp Glu Met Leu Lys Glu
180 185 190
Gln Lys Glu Glu Asn Val Leu Trp Thr Pro Ser Lys Leu Leu Ala Arg
195 200 205
Leu Gly Lys Glu Ile Asn Asn Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr
210 215 220
Lys Met Asn Ile Pro Val Phe Cys Pro Gly Leu Thr Asp Gly Ser Leu
225 230 235 240
Gly Asp Met Leu Tyr Phe His Ser Phe Arg Thr Ser Gly Leu Ile Ile
245 250 255
Asp Val Val Gln Asp Ile Arg Ala Met Asn Gly Glu Ala Val His Ala
260 265 270
Asn Pro Lys Lys Thr Gly Met Ile Ile Leu Gly Gly Gly Leu Pro Lys
275 280 285
His His Ile Cys Asn Ala Asn Met Met Arg Asn Gly Ala Asp Tyr Ala
290 295 300
Val Phe Ile Asn Pro Gly Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala
305 310 315 320
Arg Pro Asp Glu Ala Val Ser Trp Gly Lys Ile Arg Gly Ser Ala Lys
325 330 335
Thr Val Lys Val Tyr Cys Asp Ala Thr Ile Ala Phe Pro Leu Leu Val
340 345 350
Ala Glu Thr Phe Ala Thr Lys Arg Asp Gln Thr Cys Glu Ser Lys Thr
355 360 365
<210> 202
<211> 379
<212> PRT
<213> Zea mays
<400> 202
Met Gly Ala Glu Leu Gly Glu Ala Gly Val Pro Ala Arg Asp Met Val
1 5 10 15
Asp Ala Leu Glu Gly Val Arg Ser Ile Leu Leu Lys Pro Ser Glu Arg
20 25 30
Leu Asp Asp Lys Arg Phe Thr Arg Ile Ala Gly Ala Asp Phe Asp Asp
35 40 45
Ala Gly Leu Gly Leu Ala Gly Leu Leu Gly Ser Leu Ala Ser Thr Gly
50 55 60
Phe Gln Ala Ser Asn Leu Gly Asp Ala Ile Asp Val Val Asn Gln Met
65 70 75 80
Leu Asp Trp Arg Leu Ser His Glu Lys Pro Ser Asp Asp Cys Ala Glu
85 90 95
Ala Glu Leu Asp Pro Ala Tyr Arg Glu Ser Val Lys Cys Lys Ile Phe
100 105 110
Leu Gly Phe Thr Ser Asn Leu Val Ser Ser Gly Val Arg Glu Ile Ile
115 120 125
Arg Phe Leu Val Gln His Arg Met Val Asp Val Ile Val Thr Thr Ala
130 135 140
Gly Gly Ile Glu Glu Asp Leu Ile Lys Cys Leu Ala Pro Thr Tyr Arg
145 150 155 160
Gly Asp Phe Thr Leu Pro Gly Ala Leu Leu Arg Ser Arg Gly Leu Asn
165 170 175
Arg Ile Gly Asn Leu Leu Val Pro Asn Asp Asn Tyr Cys Lys Phe Glu
180 185 190
His Trp Ile Met Pro Ile Leu Asp Lys Met Leu Leu Glu Gln Ser Thr
195 200 205
Gln Asn Val Trp Thr Pro Ser Lys Val Ile Ala Arg Leu Gly Lys Glu
210 215 220
Ile Asn Asp Glu Ser Ser Tyr Leu Tyr Trp Ala Tyr Lys Asn Asn Ile
225 230 235 240
Pro Val Phe Cys Pro Ala Leu Thr Asp Gly Ser Ile Gly Asp Met Leu
245 250 255
Phe Cys His Ser Val His Ser Pro Gly Leu Val Val Asp Ile Val Gln
260 265 270
Asp Val Arg Leu Met Asn Ala Glu Thr Ile His Ala Ser Pro Arg Lys
275 280 285
Thr Gly Ile Ile Leu Leu Gly Gly Gly Leu Pro Lys His His Ile Cys
290 295 300
Asn Ala Asn Met Leu Arg Asp Gly Ala Asp Tyr Ala Val Tyr Val Asn
305 310 315 320
Thr Ala Gln Glu Phe Asp Gly Ser Asp Ser Gly Ala Arg Pro Asp Glu
325 330 335
Ala Val Ser Trp Gly Lys Ile Lys Ser Ser Ala Arg Thr Val Lys Val
340 345 350
His Cys Asp Ala Thr Ile Ala Phe Pro Leu Leu Val Ala Ala Thr Phe
355 360 365
Ala Arg Lys Val His Gly Thr Lys Ser Thr Asn
370 375
<210> 203
<211> 820
<212> DNA
<213> Populus deltoides
<400> 203
gaattcggca cgagggtatt ttgtttgttt tgtgattgcg actctcttat caatcgcggc 60
catgtctgac gaggagcagc acttcgagtc aaaagctgat gcgggagctt cgaaaactta 120
ccctcaacaa gctggtacca ttcgcaagag cggttacatt gtcatcaaga atcgcccttg 180
caaggttgtg gaggtttcta cctctaaaac tggcaagcac ggccatgcca aatgtcactt 240
tgttgcaatt gatatcttca atggaaaaaa gcttgaagat attgttcctt cttcccacaa 300
ctgtgatgtt ccccatgtca cccgtactga ctatcagctg attgatatct cagaggatgg 360
atttgtgagc ttgctgactg agaatggcaa taccaaggat gacctgaggc tcccaactga 420
tgagagtctc ctctctcaga tcaaggatgg atttggcgag ggtaaagatc ttgttgtgac 480
tgtgatgtcc tccatgggag aggagcagat ctgcgccctc aaggacgttg gcccgaagta 540
aattgtaccc tgcttcatgc aggcatgccc aaggttgacg gctgatagtt gtagcttagc 600
aagtggttct atgtgggttt gagaattggt cccgtttcag gaacattccg tactagatgc 660
ggccttccct tatatgaaga cttgccatta tttgagtggc gttttatccg tgatgatatt 720
tttgtggttc ttggtgtggg gaagtttatt ctgccagtaa gactgcatac acttcttggc 780
acctgtggtg gtttcaaaca attacagttt ttatttcctt 820
<210> 204
<211> 159
<212> PRT
<213> Populus deltoides
<400> 204
Met Ser Asp Glu Glu Gln His Phe Glu Ser Lys Ala Asp Ala Gly Ala
1 5 10 15
Ser Lys Thr Tyr Pro Gln Gln Ala Gly Thr Ile Arg Lys Ser Gly Tyr
20 25 30
Ile Val Ile Lys Asn Arg Pro Cys Lys Val Val Glu Val Ser Thr Ser
35 40 45
Lys Thr Gly Lys His Gly His Ala Lys Cys His Phe Val Ala Ile Asp
50 55 60
Ile Phe Asn Gly Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn
65 70 75 80
Cys Asp Val Pro His Val Thr Arg Thr Asp Tyr Gln Leu Ile Asp Ile
85 90 95
Ser Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys
100 105 110
Asp Asp Leu Arg Leu Pro Thr Asp Glu Ser Leu Leu Ser Gln Ile Lys
115 120 125
Asp Gly Phe Gly Glu Gly Lys Asp Leu Val Val Thr Val Met Ser Ser
130 135 140
Met Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Val Gly Pro Lys
145 150 155
<210> 205
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 205
gaagctcgag gactctctta tcaatcgc 28
<210> 206
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 206
ggggagctca gggtacaatt tacttcgggc c 31
<210> 207
<211> 906
<212> DNA
<213> Populus deltoides
<400> 207
cactgaatca aaggccatgg agtcaaagat tcaaatagag gacctaacag aactcgccgt 60
aaagactggc gaacagttca tacagagtct cttacgactc aatgacaaga agaaaatctt 120
cgtcaacatg gtggagcacg acacgcttgt ctacctccaa aaatatcaaa gatacagtct 180
cagaagacca aagggaattg agacttttca acaaagggta atatccggaa acctcctcgg 240
attccattgc ccagctatct gtcactttat tgtgaagata gtggaaaagg aaggtggctc 300
ctacaaatgc catcattgcg ataaaggaaa ggccatcgtt gaagatgcct ctgccgacag 360
tggtcccaaa gatggacccc cacccacgag gagcatcgtg gaaaaagaag acgttccaac 420
cacgtcttca aagcaagtgg attgatgtga taacatggtg gagcacgaca cgcttgtcta 480
cctccaaaaa tatcaaagat acagtctcag aagaccaaag ggaattgaga cttttcaaca 540
aagggtaata tccggaaacc tcctcggatt ccattgccca gctatctgtc actttattgt 600
gaagatagtg gaaaaggaag gtggctccta caaatgccat cattgcgata aaggaaaggc 660
catcgttgaa gatgcctctg ccgacagtgg tcccaaagat ggacccccac ccacgaggag 720
catcgtggaa aaagaagacg ttccaaccac gtcttcaaag caagtggatt gatgtgatat 780
ctccactgac gtaagggatg acgcacaatc ccactatcct tcgcaagacc cttcctctat 840
ataaggaagt tcatttcatt tggagaggac acgctgaaat caccagtctc tctctaagct 900
tggatc 906
<210> 208
<211> 512
<212> DNA
<213> Populus deltoides
<400> 208
gactctctta tcaatcgcgg ccatgtctga cgaggagcag cacttcgagt caaaagctga 60
tgcgggagct tcgaaaactt accctcaaca agctggtacc attcgcaaga gcggttacat 120
tgtcatcaag aatcgccctt gcaaggttgt ggaggtttct acctctaaaa ctggcaagca 180
cggccatgcc aaatgtcact ttgttgcaat tgatatcttc aatggaaaaa agcttgaaga 240
tattgttcct tcttcccaca actgtgatgt tccccatgtc acccgtactg actatcagct 300
gattgatatc tcagaggatg gatttgtgag cttgctgact gagaatggca ataccaagga 360
tgacctgagg ctcccaactg atgagagtct cctctctcag atcaaggatg gatttggcga 420
gggtaaagat cttgttgtga ctgtgatgtc ctccatggga gaggagcaga tctgcgccct 480
caaggacgtt ggcccgaagt aaattgtacc ct 512
<210> 209
<211> 662
<212> DNA
<213> Populus deltoides
<400> 209
gaattgatcc tctagagctt tcgttcgtat catcggtttc gacaacgttc gtcaagttca 60
atgcatcagt ttcattgcgc acacaccaga atcctactga gttcgagtat tatggcattg 120
ggaaaactgt ttttcttgta ccatttgttg tgcttgtaat ttactgtgtt ttttattcgg 180
ttttcgctat cgaactgtga aatggaaatg gatggagaag agttaatgaa tgatatggtc 240
cttttgttca ttctcaaatt aatattattt gttttttctc ttatttgttg tgtgttgaat 300
ttgaaattat aagagatatg caaacatttt gttttgagta aaaatgtgtc aaatcgtggc 360
ctctaatgac cgaagttaat atgaggagta aaacacttgt agttgtacca ttatgcttat 420
tcactaggca acaaatatat tttcagacct agaaaagctg caaatgttac tgaatacaag 480
tatgtcctct tgtgttttag acatttatga actttccttt atgtaatttt ccagaatcct 540
tgtcagattc taatcattgc tttataatta tagttatact catggatttg tagttgagta 600
tgaaaatatt ttttaatgca ttttatgact tgccaattga ttgacaacat gcatcaatcg 660
at 662
<210> 210
<211> 466
<212> DNA
<213> Lactuca sativa
<220>
<221> CDS
<222> (3)..(329)
<400> 210
cg cat ttg cgt cgt cgt ggg ata ttt cga ttt ggg gaa tcg aca ttt 47
His Leu Arg Arg Arg Gly Ile Phe Arg Phe Gly Glu Ser Thr Phe
1 5 10 15
tta atc tgg aaa aag ctt gaa gat att gtt cca tca tcc cat aat tgt 95
Leu Ile Trp Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys
20 25 30
gat gtc ccc cat gtc aat cgc acc gat tac cag cta att gat att tct 143
Asp Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser
35 40 45
gaa gat gga ttt gtg agt ttg ctg act gaa aat ggt aac acc aaa gat 191
Glu Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp
50 55 60
gac ctc aaa ctc ccc acc gat gat gct ctc ctc act cag ata aag gat 239
Asp Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile Lys Asp
65 70 75
gga ttt gga gag ggg aag ggc ctg gtt gtg act gtt atg tca gca atg 287
Gly Phe Gly Glu Gly Lys Gly Leu Val Val Thr Val Met Ser Ala Met
80 85 90 95
gga gag gaa cag ata tgt gct ctc aag gac att ggc ccc aaa 329
Gly Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
100 105
taacttttca aaaatccaca aaaacacaca ctataatatt attataacat ttggattaac 389
agaaaacaag ttgtgttttt atttgtttta tcatgtacag atgttatgga ttaaaaaaaa 449
aaaaaaaaaa aaaaaaa 466
<210> 211
<211> 109
<212> PRT
<213> Lactuca sativa
<400> 211
His Leu Arg Arg Arg Gly Ile Phe Arg Phe Gly Glu Ser Thr Phe Leu
1 5 10 15
Ile Trp Lys Lys Leu Glu Asp Ile Val Pro Ser Ser His Asn Cys Asp
20 25 30
Val Pro His Val Asn Arg Thr Asp Tyr Gln Leu Ile Asp Ile Ser Glu
35 40 45
Asp Gly Phe Val Ser Leu Leu Thr Glu Asn Gly Asn Thr Lys Asp Asp
50 55 60
Leu Lys Leu Pro Thr Asp Asp Ala Leu Leu Thr Gln Ile Lys Asp Gly
65 70 75 80
Phe Gly Glu Gly Lys Gly Leu Val Val Thr Val Met Ser Ala Met Gly
85 90 95
Glu Glu Gln Ile Cys Ala Leu Lys Asp Ile Gly Pro Lys
100 105
Claims (22)
- 삭제
- 생장 eIF-5A의 센스 폴리뉴클레오타이드 및 상기 센스 폴리뉴클레오타이드의 전사를 제공하기 위해 센스 폴리뉴클레오타이드에 실시가능하게 연결된 조절 서열을 포함한 벡터를 식물의 적어도 하나 이상의 세포의 게놈 내로 도입시켜 상기 센스 폴리뉴클레오타이드의 전사가 식물 내 생장 eIF-5A의 발현을 증가시키는 단계를 포함하고, 상기 증가된 생장 eIF-5A의 발현은 야생형 식물과 비교시 식물 내 종자 수확량을 증가시키거나 식물의 생물량을 증가시킴을 특징으로 하는 식물 내 종자 수확량 증가 방법
- 제 2항에 있어서, 상기 식물은 카놀라임을 특징으로 하는 방법
- 제 2항에 있어서, 상기 생장 eIF-5A 폴리뉴클레오타이드는 미루나무 생장 eIF-5A를 암호화하는 폴리뉴클레오타이드, 카놀라 생장 eIF-5A를 암호화하는 폴리뉴클레오타이드, 자주개자리 생장 eIF-5A를 암호화하는 폴리뉴클레오타이드, 토마토 생장 eIF-5A를 암호화하는 폴리뉴클레오타이드 및 아라비돕시스 생장 eIF-5A를 암호화하는 폴리뉴클레오타이드로 구성된 군에서 선택됨을 특징으로 하는 방법
- 제 2항에 있어서, 상기 식물은 카놀라이고 상기 생장 eIF-5A는 카놀라 생장 eIF-5A임을 특징으로 하는 방법
- 제 5항에 있어서, 상기 카놀라 생장 eIF-5A 폴리펩타이드는 서열번호: 66을 지님을 특징으로 하는 방법
- 삭제
- 제 4항에 있어서, 상기 미루나무 생장 eIF-5A는 서열번호: 203을 지님을 특징으로 하는 방법
- 제 4항에 있어서, 상기 자주개자리 생장 eIF-5A는 서열번호: 178을 지님을 특징으로 하는 방법
- 제 4항에 있어서, 상기 토마토 생장 eIF-5A는 서열번호: 188을 지님을 특징으로 하는 방법
- 제 4항에 있어서, 상기 아라비돕시스 생장 eIF-5A는 서열번호: 63 또는 서열번호: 52를 지님을 특징으로 하는 방법
- 서열번호: 179를 지닌 분리된 자주개자리 생장 이소폼 eIF-5A 단백질
- 제 12항의 자주개자리 생장 이소폼 eIF-5A 단백질을 암호화하는 분리된 폴리뉴클레오타이드
- 서열번호: 178을 지닌 자주개자리 생장 이소폼 eIF-5A를 암호화하는 분리된 폴리뉴클레오타이드
- 서열번호: 204를 지닌 분리된 미루나무 생장 이소폼 eIF-5A 단백질
- 제 15항의 미루나무 생장 이소폼 eIF-5A 단백질을 암호화하는 분리된 폴리뉴클레오타이드
- 서열번호: 203을 지닌 미루나무 생장 이소폼 eIF-5A를 암호화하는 분리된 폴리뉴클레오타이드
- 삭제
- 생장 eIF-5A의 센스 폴리뉴클레오타이드 및 상기 센스 폴리뉴클레오타이드의 전사를 제공하기 위해 센스 폴리뉴클레오타이드에 실시가능하게 연결된 조절 서열을 포함한 벡터를 식물의 적어도 하나 이상의 세포의 게놈 내로 도입시켜 상기 센스 폴리뉴클레오타이드의 전사가 식물 내 생장 eIF-5A의 발현을 증가시키는 단계를 포함하고, 상기 증가된 생장 eIF-5A의 발현은 야생형 식물과 비교시 형질전환 식물 내 종자 수확량을 증가시킴을 특징으로 하는 증가된 종자 수확량을 나타내는 형질전환 식물의 생성 방법
- 제 19항에 따라 생성된 식물
- 제 20항에 있어서, 상기 식물은 카놀라임을 특징으로 하는 식물
- 제 20항 또는 제 21항의 식물의 자손, 식물 일부 또는 종자
Applications Claiming Priority (8)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US63256704P | 2004-12-03 | 2004-12-03 | |
| US60/632,567 | 2004-12-03 | ||
| US69016005P | 2005-06-14 | 2005-06-14 | |
| US60/690,160 | 2005-06-14 | ||
| US71099905P | 2005-08-25 | 2005-08-25 | |
| US60/710,999 | 2005-08-25 | ||
| US72873705P | 2005-10-21 | 2005-10-21 | |
| US60/728,737 | 2005-10-21 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| KR20070079602A KR20070079602A (ko) | 2007-08-07 |
| KR101016198B1 true KR101016198B1 (ko) | 2011-02-24 |
Family
ID=36466270
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020077011516A KR101016198B1 (ko) | 2004-12-03 | 2005-12-05 | 종자 수확량 증가 방법 |
Country Status (11)
| Country | Link |
|---|---|
| EP (2) | EP1824982A2 (ko) |
| JP (2) | JP5205058B2 (ko) |
| KR (1) | KR101016198B1 (ko) |
| CN (2) | CN101166826B (ko) |
| AU (1) | AU2005311640B2 (ko) |
| CA (1) | CA2588330A1 (ko) |
| HK (2) | HK1119200A1 (ko) |
| IL (2) | IL183586A0 (ko) |
| NZ (2) | NZ556235A (ko) |
| TW (1) | TWI452137B (ko) |
| WO (1) | WO2006060699A2 (ko) |
Families Citing this family (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2011242553A1 (en) * | 2010-04-22 | 2012-11-15 | Senesco Technologies, Inc. | Transgenic algae with enhanced oil expression |
| CN103060373B (zh) * | 2013-01-25 | 2014-10-15 | 北京市农林科学院 | 杨树蛋白PtoeIF5A1及其编码基因在调控植物生长中的应用 |
| US10973187B2 (en) | 2017-12-28 | 2021-04-13 | Agribody Technologies, Inc. | Plants with modified deoxyhypusine synthase genes |
Family Cites Families (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6878860B1 (en) | 1999-07-06 | 2005-04-12 | Senesco, Inc. | DNA encoding a plant deoxyhypusine synthase, a plant eukaryotic initiation factor 5A, transgenic plants and a method for controlling senescence programmed and cell death in plants |
| US6538182B1 (en) | 1999-07-06 | 2003-03-25 | Senesco, Inc. | DNA encoding a plant deoxyhypusine synthase, a plant eukaryotic initiation factor 5A, transgenic plants and a method for controlling senescence programmed and cell death in plants |
| US7358418B2 (en) * | 1999-07-06 | 2008-04-15 | Senesco Technologies, Inc. | Isoforms of eIF-5A: senescence-induced eLF5A; wounding-induced eIF-4A; growth eIF-5A; and DHS |
| DE60144056D1 (de) * | 2000-11-29 | 2011-03-31 | Senesco Technologies Inc | Dna, welche für eine pflanzliche deoxyhypusine synthase und einen pflanzlichen eukaryotischen initiationsfaktor 5a kodiert; transgene pflanzen und verfahren zum kontrollieren der seneszenz und des programmierten zelltods in pflanzen |
| US7968523B2 (en) * | 2001-07-23 | 2011-06-28 | Senesco Technologies, Inc. | Method for inducing apoptosis using apoptosis-specific EIF5-A |
| US7217517B2 (en) * | 2001-07-23 | 2007-05-15 | Senesco Technologies, Inc. | Nucleic acids, polypeptides, and methods for modulating apoptosis |
| NZ542665A (en) * | 2003-03-05 | 2008-05-30 | Senesco Technologies Inc | Use of antisense oligonucleotides or siRNA to suppress expression of eIF-5A1 |
-
2005
- 2005-12-05 KR KR1020077011516A patent/KR101016198B1/ko not_active IP Right Cessation
- 2005-12-05 TW TW094142842A patent/TWI452137B/zh not_active IP Right Cessation
- 2005-12-05 NZ NZ556235A patent/NZ556235A/en not_active IP Right Cessation
- 2005-12-05 CN CN2005800416482A patent/CN101166826B/zh not_active Expired - Fee Related
- 2005-12-05 EP EP05852821A patent/EP1824982A2/en not_active Withdrawn
- 2005-12-05 AU AU2005311640A patent/AU2005311640B2/en not_active Ceased
- 2005-12-05 CN CN201310573295.7A patent/CN103911391A/zh active Pending
- 2005-12-05 EP EP08013335A patent/EP2019142A3/en not_active Withdrawn
- 2005-12-05 CA CA002588330A patent/CA2588330A1/en not_active Abandoned
- 2005-12-05 NZ NZ587138A patent/NZ587138A/en not_active IP Right Cessation
- 2005-12-05 JP JP2007544560A patent/JP5205058B2/ja not_active Expired - Fee Related
- 2005-12-05 WO PCT/US2005/043708 patent/WO2006060699A2/en active Application Filing
-
2007
- 2007-05-31 IL IL183586A patent/IL183586A0/en not_active IP Right Cessation
-
2008
- 2008-08-21 HK HK08109377.6A patent/HK1119200A1/xx not_active IP Right Cessation
-
2012
- 2012-08-30 IL IL221703A patent/IL221703A0/en unknown
- 2012-12-20 JP JP2012278669A patent/JP2013135669A/ja active Pending
-
2015
- 2015-01-07 HK HK15100120.6A patent/HK1199742A1/xx unknown
Non-Patent Citations (1)
| Title |
|---|
| GenBank accession number AAG53647.1 |
Also Published As
| Publication number | Publication date |
|---|---|
| KR20070079602A (ko) | 2007-08-07 |
| TW200634155A (en) | 2006-10-01 |
| CN103911391A (zh) | 2014-07-09 |
| EP2019142A3 (en) | 2009-04-01 |
| IL221703A0 (en) | 2012-10-31 |
| CN101166826B (zh) | 2013-12-18 |
| EP2019142A2 (en) | 2009-01-28 |
| CN101166826A (zh) | 2008-04-23 |
| CA2588330A1 (en) | 2006-06-08 |
| WO2006060699A9 (en) | 2006-08-24 |
| NZ587138A (en) | 2012-06-29 |
| EP1824982A2 (en) | 2007-08-29 |
| EP2019142A9 (en) | 2009-05-13 |
| WO2006060699A3 (en) | 2007-01-18 |
| WO2006060699A2 (en) | 2006-06-08 |
| JP2008521445A (ja) | 2008-06-26 |
| IL183586A0 (en) | 2007-09-20 |
| HK1119200A1 (en) | 2009-02-27 |
| JP2013135669A (ja) | 2013-07-11 |
| HK1199742A1 (en) | 2015-07-17 |
| AU2005311640B2 (en) | 2011-08-18 |
| JP5205058B2 (ja) | 2013-06-05 |
| AU2005311640A1 (en) | 2006-06-08 |
| NZ556235A (en) | 2010-08-27 |
| TWI452137B (zh) | 2014-09-11 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US8563285B2 (en) | Isoforms of eIF-5A: senescence-induced eIF5A; wounding-induced eIF-5A; growth eIF-5A; and DHS | |
| US20090235390A1 (en) | Isoforms of eIF-5A: senescence-induced eIF-5A; wounding-induced eIF-5A; growth eIF-5A: and DHS | |
| KR101016198B1 (ko) | 종자 수확량 증가 방법 | |
| US20100333233A1 (en) | Cottonwood Growth eIF-5A | |
| US8232455B2 (en) | Polynucleotides encoding canola DHS and antisense polynucleotides thereof | |
| AU2011250877B2 (en) | Methods of Increasing Seed Yield | |
| AU2011265387B2 (en) | Isoforms of eIF-5A: Senescence-induced eIF-5A; Wounding-induced eIF-5A; Growth eIF-5A; and DHS | |
| AU2014202022A1 (en) | Isoforms of elF-5A: Senescence-induced elF-5A; Wounding-induced elF-5A; Growth elF-5A; and DHS | |
| MX2007006597A (en) | Polynucleotides of dha and isoforms of eif-5a and methods of using same |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E902 | Notification of reason for refusal | ||
| E701 | Decision to grant or registration of patent right | ||
| GRNT | Written decision to grant | ||
| FPAY | Annual fee payment |
Payment date: 20140212 Year of fee payment: 4 |
|
| FPAY | Annual fee payment |
Payment date: 20150209 Year of fee payment: 4 |
|
| LAPS | Lapse due to unpaid annual fee |