CN112662606A - 用于治疗苯丙酮尿症的工程益生菌 - Google Patents
用于治疗苯丙酮尿症的工程益生菌 Download PDFInfo
- Publication number
- CN112662606A CN112662606A CN202110013298.XA CN202110013298A CN112662606A CN 112662606 A CN112662606 A CN 112662606A CN 202110013298 A CN202110013298 A CN 202110013298A CN 112662606 A CN112662606 A CN 112662606A
- Authority
- CN
- China
- Prior art keywords
- stla
- pfnrs
- phep
- gene
- phenylalanine
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 201000011252 Phenylketonuria Diseases 0.000 title claims abstract description 41
- 239000006041 probiotic Substances 0.000 title claims abstract description 41
- 235000018291 probiotics Nutrition 0.000 title claims abstract description 41
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 claims abstract description 59
- 229960005190 phenylalanine Drugs 0.000 claims abstract description 48
- 230000000529 probiotic effect Effects 0.000 claims abstract description 27
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 18
- 241000894006 Bacteria Species 0.000 claims abstract description 17
- 108700023158 Phenylalanine ammonia-lyases Proteins 0.000 claims abstract description 14
- 241001302654 Escherichia coli Nissle 1917 Species 0.000 claims abstract description 11
- 239000003814 drug Substances 0.000 claims abstract description 10
- 150000008575 L-amino acids Chemical class 0.000 claims abstract description 9
- 238000000034 method Methods 0.000 claims description 43
- 101150066555 lacZ gene Proteins 0.000 claims description 41
- 101150038370 yicS gene Proteins 0.000 claims description 41
- 101100378572 Escherichia coli (strain K12) agaI gene Proteins 0.000 claims description 38
- 101100244111 Dictyostelium discoideum stlA gene Proteins 0.000 claims description 25
- 101100352026 Escherichia coli (strain K12) pheP gene Proteins 0.000 claims description 21
- 101100465553 Dictyostelium discoideum psmB6 gene Proteins 0.000 claims description 14
- 101100169519 Pyrococcus abyssi (strain GE5 / Orsay) dapAL gene Proteins 0.000 claims description 14
- 101150011371 dapA gene Proteins 0.000 claims description 14
- RZVAJINKPMORJF-UHFFFAOYSA-N Acetaminophen Chemical compound CC(=O)NC1=CC=C(O)C=C1 RZVAJINKPMORJF-UHFFFAOYSA-N 0.000 claims description 12
- 108010069013 Phenylalanine Hydroxylase Proteins 0.000 claims description 10
- 102100038223 Phenylalanine-4-hydroxylase Human genes 0.000 claims description 8
- 108091000044 4-hydroxy-tetrahydrodipicolinate synthase Proteins 0.000 claims description 5
- 108010078791 Carrier Proteins Proteins 0.000 claims description 4
- 101150023358 stlA gene Proteins 0.000 claims description 4
- 238000010356 CRISPR-Cas9 genome editing Methods 0.000 claims description 3
- 238000010362 genome editing Methods 0.000 claims description 3
- 230000010354 integration Effects 0.000 claims description 3
- 238000009472 formulation Methods 0.000 claims description 2
- 239000000203 mixture Substances 0.000 claims description 2
- 230000017105 transposition Effects 0.000 claims description 2
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 claims 1
- 206010064571 Gene mutation Diseases 0.000 claims 1
- 101710118538 Protease Proteins 0.000 claims 1
- 238000004519 manufacturing process Methods 0.000 claims 1
- 238000002360 preparation method Methods 0.000 abstract description 10
- 229940079593 drug Drugs 0.000 abstract description 3
- 239000013612 plasmid Substances 0.000 description 86
- 239000012634 fragment Substances 0.000 description 69
- 210000004027 cell Anatomy 0.000 description 42
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 38
- 238000012408 PCR amplification Methods 0.000 description 24
- 238000007702 DNA assembly Methods 0.000 description 18
- 238000011144 upstream manufacturing Methods 0.000 description 15
- 239000007787 solid Substances 0.000 description 14
- 241000699670 Mus sp. Species 0.000 description 12
- 238000010276 construction Methods 0.000 description 11
- 239000000243 solution Substances 0.000 description 11
- UNFWWIHTNXNPBV-WXKVUWSESA-N spectinomycin Chemical compound O([C@@H]1[C@@H](NC)[C@@H](O)[C@H]([C@@H]([C@H]1O1)O)NC)[C@]2(O)[C@H]1O[C@H](C)CC2=O UNFWWIHTNXNPBV-WXKVUWSESA-N 0.000 description 11
- 229960000268 spectinomycin Drugs 0.000 description 11
- 239000008280 blood Substances 0.000 description 9
- 210000004369 blood Anatomy 0.000 description 9
- 239000010931 gold Substances 0.000 description 9
- 229910052737 gold Inorganic materials 0.000 description 9
- 239000000047 product Substances 0.000 description 9
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 8
- 230000000694 effects Effects 0.000 description 7
- 238000000338 in vitro Methods 0.000 description 7
- WBYWAXJHAXSJNI-VOTSOKGWSA-M .beta-Phenylacrylic acid Natural products [O-]C(=O)\C=C\C1=CC=CC=C1 WBYWAXJHAXSJNI-VOTSOKGWSA-M 0.000 description 6
- 241000588724 Escherichia coli Species 0.000 description 6
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 6
- 238000002474 experimental method Methods 0.000 description 6
- 101150094644 rhtC gene Proteins 0.000 description 6
- WBYWAXJHAXSJNI-VOTSOKGWSA-N trans-cinnamic acid Chemical compound OC(=O)\C=C\C1=CC=CC=C1 WBYWAXJHAXSJNI-VOTSOKGWSA-N 0.000 description 6
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 5
- 201000010099 disease Diseases 0.000 description 5
- KPGXRSRHYNQIFN-UHFFFAOYSA-N 2-oxoglutaric acid Chemical compound OC(=O)CCC(=O)C(O)=O KPGXRSRHYNQIFN-UHFFFAOYSA-N 0.000 description 4
- 241000660147 Escherichia coli str. K-12 substr. MG1655 Species 0.000 description 4
- 101150063416 add gene Proteins 0.000 description 4
- 230000015556 catabolic process Effects 0.000 description 4
- 238000006731 degradation reaction Methods 0.000 description 4
- 238000001514 detection method Methods 0.000 description 4
- 238000011161 development Methods 0.000 description 4
- 239000003651 drinking water Substances 0.000 description 4
- 235000020188 drinking water Nutrition 0.000 description 4
- 230000008030 elimination Effects 0.000 description 4
- 238000003379 elimination reaction Methods 0.000 description 4
- 210000001035 gastrointestinal tract Anatomy 0.000 description 4
- 238000010353 genetic engineering Methods 0.000 description 4
- 229930027917 kanamycin Natural products 0.000 description 4
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 4
- 229960000318 kanamycin Drugs 0.000 description 4
- 229930182823 kanamycin A Natural products 0.000 description 4
- BTNMPGBKDVTSJY-UHFFFAOYSA-N keto-phenylpyruvic acid Chemical compound OC(=O)C(=O)CC1=CC=CC=C1 BTNMPGBKDVTSJY-UHFFFAOYSA-N 0.000 description 4
- 101150076563 malP gene Proteins 0.000 description 4
- 239000002609 medium Substances 0.000 description 4
- 238000010369 molecular cloning Methods 0.000 description 4
- 230000035772 mutation Effects 0.000 description 4
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 241000196324 Embryophyta Species 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 230000000593 degrading effect Effects 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 238000001727 in vivo Methods 0.000 description 3
- 239000007788 liquid Substances 0.000 description 3
- 238000010172 mouse model Methods 0.000 description 3
- 239000002773 nucleotide Substances 0.000 description 3
- 125000003729 nucleotide group Chemical group 0.000 description 3
- 102000004169 proteins and genes Human genes 0.000 description 3
- 239000000126 substance Substances 0.000 description 3
- 208000024891 symptom Diseases 0.000 description 3
- 230000001225 therapeutic effect Effects 0.000 description 3
- 239000001903 2-oxo-3-phenylpropanoic acid Substances 0.000 description 2
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 2
- XEEYBQQBJWHFJM-UHFFFAOYSA-N Iron Chemical compound [Fe] XEEYBQQBJWHFJM-UHFFFAOYSA-N 0.000 description 2
- XUMBMVFBXHLACL-UHFFFAOYSA-N Melanin Chemical compound O=C1C(=O)C(C2=CNC3=C(C(C(=O)C4=C32)=O)C)=C2C4=CNC2=C1C XUMBMVFBXHLACL-UHFFFAOYSA-N 0.000 description 2
- 241000699666 Mus <mouse, genus> Species 0.000 description 2
- 241001148064 Photorhabdus luminescens Species 0.000 description 2
- 241001110331 Proteus mirabilis HI4320 Species 0.000 description 2
- DEDGUGJNLNLJSR-UHFFFAOYSA-N alpha-hydroxycinnamic acid Natural products OC(=O)C(O)=CC1=CC=CC=C1 DEDGUGJNLNLJSR-UHFFFAOYSA-N 0.000 description 2
- HWXBTNAVRSUOJR-UHFFFAOYSA-N alpha-hydroxyglutaric acid Natural products OC(=O)C(O)CCC(O)=O HWXBTNAVRSUOJR-UHFFFAOYSA-N 0.000 description 2
- 229940009533 alpha-ketoglutaric acid Drugs 0.000 description 2
- 238000010171 animal model Methods 0.000 description 2
- XZNUGFQTQHRASN-XQENGBIVSA-N apramycin Chemical compound O([C@H]1O[C@@H]2[C@H](O)[C@@H]([C@H](O[C@H]2C[C@H]1N)O[C@@H]1[C@@H]([C@@H](O)[C@H](N)[C@@H](CO)O1)O)NC)[C@@H]1[C@@H](N)C[C@@H](N)[C@H](O)[C@H]1O XZNUGFQTQHRASN-XQENGBIVSA-N 0.000 description 2
- 229950006334 apramycin Drugs 0.000 description 2
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 description 2
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 2
- 230000015572 biosynthetic process Effects 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 239000002158 endotoxin Substances 0.000 description 2
- 235000013305 food Nutrition 0.000 description 2
- 239000008103 glucose Substances 0.000 description 2
- 230000002440 hepatic effect Effects 0.000 description 2
- 238000004128 high performance liquid chromatography Methods 0.000 description 2
- 238000002347 injection Methods 0.000 description 2
- 239000007924 injection Substances 0.000 description 2
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 2
- 229920006008 lipopolysaccharide Polymers 0.000 description 2
- 210000004185 liver Anatomy 0.000 description 2
- 208000030159 metabolic disease Diseases 0.000 description 2
- 244000005700 microbiome Species 0.000 description 2
- 230000003278 mimic effect Effects 0.000 description 2
- 238000011160 research Methods 0.000 description 2
- 239000000523 sample Substances 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 235000019640 taste Nutrition 0.000 description 2
- GMKMEZVLHJARHF-UHFFFAOYSA-N (2R,6R)-form-2.6-Diaminoheptanedioic acid Natural products OC(=O)C(N)CCCC(N)C(O)=O GMKMEZVLHJARHF-UHFFFAOYSA-N 0.000 description 1
- 229920001817 Agar Polymers 0.000 description 1
- 208000004429 Bacillary Dysentery Diseases 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- 206010009900 Colitis ulcerative Diseases 0.000 description 1
- 208000029767 Congenital, Hereditary, and Neonatal Diseases and Abnormalities Diseases 0.000 description 1
- 208000011231 Crohn disease Diseases 0.000 description 1
- 206010012735 Diarrhoea Diseases 0.000 description 1
- 206010012742 Diarrhoea infectious Diseases 0.000 description 1
- 208000012661 Dyskinesia Diseases 0.000 description 1
- 208000022559 Inflammatory bowel disease Diseases 0.000 description 1
- FEMXZDUTFRTWPE-DZSWIPIPSA-N L-erythro-7,8-dihydrobiopterin Chemical compound N1C(N)=NC(=O)C2=C1NCC([C@@H](O)[C@@H](O)C)=N2 FEMXZDUTFRTWPE-DZSWIPIPSA-N 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- 239000012880 LB liquid culture medium Substances 0.000 description 1
- 239000007993 MOPS buffer Substances 0.000 description 1
- 241000186359 Mycobacterium Species 0.000 description 1
- 108090000854 Oxidoreductases Proteins 0.000 description 1
- 102000004316 Oxidoreductases Human genes 0.000 description 1
- 208000001431 Psychomotor Agitation Diseases 0.000 description 1
- 102000018120 Recombinases Human genes 0.000 description 1
- 108010091086 Recombinases Proteins 0.000 description 1
- 206010038743 Restlessness Diseases 0.000 description 1
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 1
- 201000006704 Ulcerative Colitis Diseases 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 230000003044 adaptive effect Effects 0.000 description 1
- 210000004100 adrenal gland Anatomy 0.000 description 1
- 239000008272 agar Substances 0.000 description 1
- 239000012131 assay buffer Substances 0.000 description 1
- 244000052616 bacterial pathogen Species 0.000 description 1
- 230000008827 biological function Effects 0.000 description 1
- 238000010241 blood sampling Methods 0.000 description 1
- 230000037396 body weight Effects 0.000 description 1
- 229940041514 candida albicans extract Drugs 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 230000010307 cell transformation Effects 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 238000006243 chemical reaction Methods 0.000 description 1
- 239000002299 complementary DNA Substances 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000037213 diet Effects 0.000 description 1
- 102000038379 digestive enzymes Human genes 0.000 description 1
- 108091007734 digestive enzymes Proteins 0.000 description 1
- 208000016097 disease of metabolism Diseases 0.000 description 1
- 208000035475 disorder Diseases 0.000 description 1
- 208000001848 dysentery Diseases 0.000 description 1
- 238000013399 early diagnosis Methods 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 238000001976 enzyme digestion Methods 0.000 description 1
- 206010015037 epilepsy Diseases 0.000 description 1
- 239000003797 essential amino acid Substances 0.000 description 1
- 235000020776 essential amino acid Nutrition 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 210000003608 fece Anatomy 0.000 description 1
- 235000019138 food restriction Nutrition 0.000 description 1
- 230000006870 function Effects 0.000 description 1
- 238000001415 gene therapy Methods 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 230000037308 hair color Effects 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 208000013403 hyperactivity Diseases 0.000 description 1
- 230000004957 immunoregulator effect Effects 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 210000002490 intestinal epithelial cell Anatomy 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 229910052742 iron Inorganic materials 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000008099 melanin synthesis Effects 0.000 description 1
- GMKMEZVLHJARHF-SYDPRGILSA-N meso-2,6-diaminopimelic acid Chemical compound [O-]C(=O)[C@@H]([NH3+])CCC[C@@H]([NH3+])C([O-])=O GMKMEZVLHJARHF-SYDPRGILSA-N 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- 239000002207 metabolite Substances 0.000 description 1
- 108010079904 microcin Proteins 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 210000005036 nerve Anatomy 0.000 description 1
- 239000002547 new drug Substances 0.000 description 1
- 208000024335 physical disease Diseases 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 208000020016 psychiatric disease Diseases 0.000 description 1
- FNKQXYHWGSIFBK-RPDRRWSUSA-N sapropterin Chemical compound N1=C(N)NC(=O)C2=C1NC[C@H]([C@@H](O)[C@@H](O)C)N2 FNKQXYHWGSIFBK-RPDRRWSUSA-N 0.000 description 1
- 229960004617 sapropterin Drugs 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 238000010254 subcutaneous injection Methods 0.000 description 1
- 239000007929 subcutaneous injection Substances 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 239000013595 supernatant sample Substances 0.000 description 1
- 208000011580 syndromic disease Diseases 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 210000001685 thyroid gland Anatomy 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 239000012137 tryptone Substances 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 241000701161 unidentified adenovirus Species 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 238000012795 verification Methods 0.000 description 1
- 239000012138 yeast extract Substances 0.000 description 1
Images
Landscapes
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
本发明公开了一种用于治疗苯丙酮尿症的工程益生菌及其制备方法,其为大肠杆菌Nissle 1917衍生菌,在基因组上整合了外源L‑苯丙氨酸解氨酶基因、L‑苯丙氨酸内运蛋白基因和L‑氨基酸脱氨酶基因。本发明的工程益生菌能够用于制备苯丙酮尿症治疗药物。
Description
技术领域
本发明属于基因工程领域和医药领域,具体地说,涉及一种用于治疗苯丙酮尿症的工程益生菌、其制备方法及其在制备苯丙酮尿症治疗药物中的应用。
背景技术
苯丙酮尿症(phenylketonuria,PKU)是一种先天性的苯丙氨酸代谢障碍疾病。在中国,PKU在新生儿中的发病率大概在1/11000,PKU患儿出生若不及时诊治,将会出现高苯丙酸血症,严重影响患儿的智力发育,引起癫痫和运动障碍等症状,并伴随黑色素合成障碍。
苯丙酮尿症是因肝脏苯丙氨酸羟化酶(phenylalanine hydroxylase,PAH)缺乏或四氢生物喋呤合成酶、二氢生物喋呤还原酶突变导致。PAH主要是在肝脏中表达,可将苯丙氨酸转化为酪氨酸,从而合成甲状腺、肾上腺和黑色素等。PAH突变,导致苯丙氨酸在肝脏中出现代谢紊乱,无法转化为酪氨酸,而是苯丙氨酸与α-酮戊二酸在血液与组织中堆积并被排泄到尿液中,另外,其代谢产物在中枢神经中蓄积会产生毒性,从而诱发患儿出现兴奋不安、多动和精神异常等。
苯丙氨酸作为人体必需氨基酸之一,主要从食物中获取,对于PKU患儿不能完全不摄入苯丙氨酸,所以目前对PKU患儿的唯一治疗方法就是采取低苯丙氨酸饮食的食物限制策略,但是这种策略对于青少年的发育和孕妇具有极大的副作用,而且由于该类食品口感差,病人依从性差。另外,目前也有一些针对PKU治疗的策略尚在实验阶段,例如,通过体外重组表达具有苯丙氨酸降解能力的苯丙氨酸解氨酶(phenylalanine ammonia-lase,stlA),通过口服蛋白降低血液中苯丙氨酸,但缺点是直接口服蛋白易被肠道内的消化酶分解。科学家也在尝试基因疗法,将携带表达PAH基因的cDNA重组腺病毒置于小鼠体内,以恢复肝脏PAH活性,但目前主要存在转运效率低和疗效不长久的问题。
目前,国际上使用较多的苯丙酮尿症动物模型主要是上世纪90年代初由国外实验室通过化学诱变得到的Pahenu1和Pahenu2两种疾病模型,这两种疾病模型虽然能够模拟苯丙酮尿症的疾病表型,但是这两种动物模型的突变型并不能代表绝大多数的病人,因此并不具有临床代表性。从苯丙酮尿症突变型数据库中,可以发现目前临床上最具代表性的突变型为PAHR408W,该突变型患病人数占21.4%,是目前患者携带人数最多的突变型。因此,使用PahR408W突变的PKU小鼠模型能够更好地模拟临床病人的疾病表征,并且该PahR408W的小鼠模型在PKU治疗的研究领域中也有相关报道。
发明内容
益生菌是一大类药物,主要通过口服活菌制剂达到治疗疾病和康复保健效果。益生菌药物制剂的优点在于给药方便,口感一般较好,病人乐于接受,顺从性高,更重要的是能够持续地在肠道内增殖从而稳定地发挥治疗作用。
大肠杆菌属Nissle 1917(简称EcN)是一种非致病性大肠杆菌,是1917年从一次志贺菌痢大爆发时未出现腹泻的士兵的粪便中分离得到的。EcN是非乳酸益生菌中研究最多的益生菌,血清型为O6:K5:H1,具有独特的基因组、半粗糙O6-脂多糖(LPS)表型、K5型荚膜、3种不同的菌毛(F1A、F1C和卷曲菌毛)。EcN具有小菌素和铁摄取系统等特殊适应性因子,在与其他微生物竞争中起关键作用。EcN能长期稳定定植于肠道,与肠上皮细胞相互作用,被广泛用于预防传染性腹泻、炎性肠疾病如溃疡性结肠炎和克罗恩病,防止新生儿消化道内病原菌定殖等,和发挥免疫调节生物学功能。
为了寻求新的苯丙酮尿症治疗药物,发明人利用基因工程技术来改造大肠杆菌Nissle1917(EcN),使其能够降解苯丙氨酸,构建出了一株经动物实验证明具有降解苯丙氨酸效应、因此有潜力用于治疗苯丙酮尿症的工程益生菌。具体而言,本发明包括如下技术方案:
一种用于治疗苯丙酮尿症的工程益生菌,其为大肠杆菌Nissle 1917衍生菌,在基因组上整合了外源L-苯丙氨酸解氨酶基因比如stlA、L-苯丙氨酸内运蛋白基因pheP和L-氨基酸脱氨酶基因比如pma。
优选地,所述L-苯丙氨酸解氨酶(Genbank号KGM29850.1)基因是stlA,核苷酸序列为SEQ ID NO:1;
所述L-苯丙氨酸内运蛋白(Genbank号QPA14453.1)基因是pheP,核苷酸序列为SEQID NO:2;
所述L-氨基酸脱氨酶(Genbank号AAA86752.1)基因是pma,核苷酸序列为SEQ IDNO:3。
进一步地,上述大肠杆菌Nissle 1917衍生菌的基因组中还可以敲除二氢吡啶二羧酸合酶基因dapA。
在一种实施方式中,上述工程益生菌的基因型为Nissle 1917(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma,△dapA),或者表示为EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma,△dapA),本文中命名为CIBT4564。
本发明的第二个方面提供了一种构建上述工程益生菌的方法,包括以下步骤:
A.以大肠杆菌Nissle 1917为底盘菌,分别在基因组的yicS位点、malPT位点、malE位点、exo位点和rhtCB位点中的一个以上位点、优选两个以上位点、优选三个以上位点、优选四个以上位点、优选五个位点敲入L-苯丙氨酸解氨酶基因比如stlA优选SEQ ID NO:1,获得stlA因整合菌株;
B.对于步骤A中获得的stlA因整合菌株,在其基因组的lacZ位点和agaI位点中的一个以上位点、优选两个位点敲入L-苯丙氨酸内运蛋白基因pheP比如SEQ ID NO:2,得到stlA+pheP整合菌株;
C.对于步骤B中获得的stlA+pheP整合菌株,在其基因组的araBD位点敲入L-氨基酸脱氨酶基因比如pma优选SEQ ID NO:3,得到stlA+pheP+pma整合菌株。
优选地,上述方法还可以包括以下步骤:
对于步骤C中获得的stlA+pheP+pma整合菌株,敲除其基因组中的二氢吡啶二羧酸合酶基因dapA,得到stlA+pheP+pma△dapA菌株或称stlA+pheP+pma整合△dapA菌株。
在一种优选的实施方式中,上述基因stlA、pheP、pma的敲入和基因dapA的敲除通过基因编辑技术实施,所述基因编辑采用CRISPR-Cas9系统、CRISPR-Cpf1系统、CRISPR-Cas相关的转座系统INTEGRATE系统或者CAST系统。
本发明的第三个方面提供了上述工程益生菌在制备苯丙酮尿症治疗药物中的应用。
上述苯丙酮尿症可以是苯丙氨酸羟化酶基因(Pah)突变比如PahR408W突变引起的。
该药物中,所述工程益生菌可降解苯丙氨酸。
在一种实施方式中,上述药物是口服制剂,通过口服给药。
本发明构建的工程益生菌能够有效地降解苯丙氨酸,动物实验证明,该工程菌可有效降解苯丙酮尿症小鼠(PahR408W)的体内苯丙氨酸,显示出良好的应用前景。
附图说明
图1是原始菌株EcN和工程益生菌CIBT4564体外降解苯丙氨酸生成反式肉桂酸的实验结果柱形图。
图2苯丙酮尿症小鼠口服生理盐水(saline)、原始菌株EcN和工程益生菌CIBT4564后血液中苯丙氨酸浓度的检测结果柱形图。
具体实施方式
本发明的工程益生菌是通过对大肠杆菌Nissle 1917进行基因工程改造而产生的基因工程衍生菌,通过对原始大肠杆菌Nissle 1917的基因组进行改造,使得衍生菌株能够有效降解苯丙氨酸,从而达到治疗苯丙酮尿症的效果。
为简要起见,本文中有时将“用于治疗苯丙酮尿症的工程益生菌”简称为“工程益生菌”、“重组益生菌”、“(基因)工程菌”、“益生菌”或者CIBT4564,它们表示相同的意义,可以互换使用。
本文中,基因的“敲入”与“插入”表示同一概念,这是本领域技术人员容易理解的。
在本文中,为了描述简便,有时会将某种蛋白比如苯丙氨酸羟化酶(PAH)与其编码基因Pah名称混用,本领域技术人员应能理解它们在不同描述场合表示不同的物质。本领域技术人员根据语境和上下文容易理解它们的含义。
在一种具体实施方式中,本发明是在益生菌E.coli Nissle 1917基因组上引入了利用组成型启动子Pj23119调控来源于发光分枝杆菌(Photorhabdus luminescens)的苯丙氨酸解氨酶(stlA)和增强了大肠杆菌MG1655来源苯丙氨酸特异转运体(pheP),可有效提高胞内苯丙氨酸的运输,并将转化为反式肉桂酸;同时引入了来源于奇异变形杆菌(Proteusmirabilis HI4320)的L-氨基酸脱氨酶(pma),可将苯丙氨酸降解为苯丙酮酸。
应理解,在构建本发明的基因工程菌的具体操作中,步骤A、步骤B、步骤C和步骤D的排序并非完全根据英文字母顺序由前到后地固定不变,它们可以交叉、颠倒地操作,只要每个步骤能实现各自的功能、完成宿主细胞基因型的定向改变即可。同样地,同一个基因比如stlA的多次敲入的不同位点的排序可以交叉、颠倒地操作。
为了考察工程菌降解苯丙氨酸的效果,通过微生物体外催化化学反应、并且在PahR408W突变的PKU小鼠模型中口服工程菌,验证了本发明所构建的工程益生菌CIBT4564能够在体外、体内均能有效降解苯丙氨酸,有望应用于治疗苯丙酮尿症。
以下结合具体实施例对本发明做进一步详细说明。应理解,以下实施例仅用于说明本发明而非用于限定本发明的范围。
本文中涉及到多种物质的添加量、含量及浓度,其中所述的百分含量,除特别说明外,皆指质量百分含量。
实施例
材料和方法
本文中的全基因合成、引物合成及测序皆由南京金瑞斯生物科技有限公司完成。
实施例中的分子生物学实验包括质粒构建、酶切、感受态细胞制备、转化等主要参照《分子克隆实验指南》(第三版),J.萨姆布鲁克,D.W.拉塞尔(美)编著,黄培堂等译,科学出版社,北京,2002)进行。比如感受态细胞转化方法及感受态制备方法均参照《分子克隆实验指南》(第三版)第1章96页进行。必要时可以通过简单试验确定具体实验条件。
主要培养基:
LB培养基:10g/L胰蛋白胨,5g/L酵母提取物,10g/L氯化钠。(固体培养基另加20g/L琼脂粉。)
原始大肠杆菌Nissle 1917、pTargetF质粒(Addgene:62226)、pCas质粒(Addgene:62225)和pMDIAI质粒(addgene:51655)由中国科学院分子植物科学卓越创新中心杨晟课题组保存,任何单位和个人都可以获得该质粒及相关质粒和细菌用于验证本发明,但未经中国科学院分子植物科学卓越创新中心允许不得用作其他用途,包括开发利用、科学研究和教学。
工程益生菌的构建参考Isabella,V.等的文献(Development of a syntheticlive bacterial therapeutic for the human metabolic diseasephenylketonuria.Nat Biotechnol 36,857–864,2018.)。利用Jiang Y等的文献的Crispr-Cas9方法(Multigene Editing in the Escherichia coli Genome via the CRISPR-Cas9System,Appl Environ Microbiol,2015)进行益生菌基因组改造。
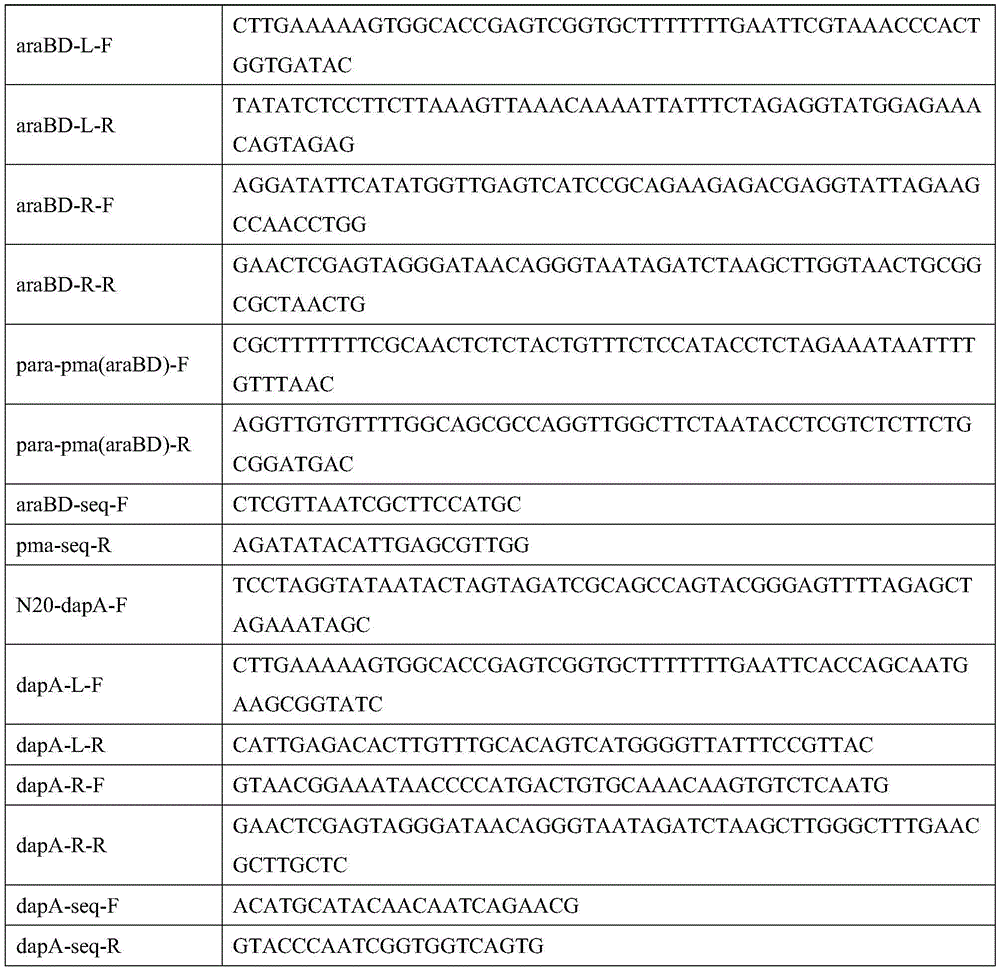
以下实施例中使用的引物序列信息如表1所示。
表1、实施例中使用的引物列表
实施例1工程益生菌CIBT4564构建
对菌株E.coli Nissle 1917基因组进行改造,包括如下步骤:
1.1在菌株E.coli Nissle 1917基因组上yicS位点敲入苯丙氨酸解氨酶stlA
(1)pTargetN20-pfnrs-stlA(yicS)质粒构建
以pTargetF(Addgene:62226)质粒为模板,N20-yicS-F/pTargetF-R为引物,PCR扩增获得N20-yicS片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-yicS质粒。
以大肠杆菌Nissle 1917(EcN)基因组为模板,分别以yicS-L-F/yicS-L-R和yicS-R-F/yicS-R-R为引物,PCR扩增分别获得上游同源臂yicS-L和下游同源臂yicS-R,分别约610bp。
以大肠杆菌MG1655基因组为模板,yicS-MG1655-F1/yicS-MG1655-R为引物,PCR扩增yicS-MG1655-1片段,约260bp;以yicS-MG1655-1片段为模板,yicS-MG1655-F2/yicS-MG1655-R为引物,PCR扩增yicS-MG1655片段,约300bp。
以pUC-pfnrs-stlA质粒为模板(由金斯瑞合成),pfnrs-stlA(yicS)-F/pfnrs-stlA(yicS)-R为引物,PCR扩增pfnrs-sltA(yicS)片段,约2.2kb。
yicS-L、yicS-MG1655、pfnrs-stlA(yicS)和yicS-R片段利用DNA assembly方法(试剂盒DNA assembly Kit购自全式金)克隆入pTargetN20-yicS质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-pfnrs-stlA(yicS)质粒转化子。
(2)将pCas(addgene:62225)质粒转化至EcN化学感受态细胞中,30℃条件下在含卡那霉素(终浓度50μg/mL)的LB固体平板上进行筛选(化学转化感受态细胞制备方法参考《分子克隆实验指南》(第三版))。
(3)挑取EcN/pCas阳性转化子,制备电转感受态细胞,在离心收集菌体前1小时加入终浓度为10mM的阿拉伯糖诱导RED重组酶表达。
(4)电转化转入pTargetN20-pfnrs-stlA(yicS)辅助质粒(电转电压为2.5kV),30℃、220rpm条件下复苏1小时,菌液涂布于含卡那霉素(50μg/mL)和壮观霉素(50μg/mL)双抗LB固体平板,30℃过夜培养后对克隆子利用yicS-seq-F/stlA-seq-R进行菌落PCR验证(电转化感受态细胞制备方法参考《分子克隆实验指南》(第三版))。
(5)辅助质粒消除:挑取阳性克隆子于LB液体(含卡那霉素50μg/mL),加入1mMIPTG,培养过夜,划线分单菌,并在含壮观霉素(50μg/mL)LB固体平板上验证辅助质粒的消除,获得EcN(yicS::pfnrs-stlA)/pCas。
1.2malPT位点敲入苯丙氨酸解氨酶stlA
(1)pTargetN20-pfnrs-stlA(malPT)质粒构建
以pTargetF质粒为模板,N20-malP-F/pTagetF-R为引物,PCR扩增获得N20-malPT片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-malPT质粒。
以EcN基因组为模板,分别以malP-L-F/malP-L-R和malP-R-F/malP-R-R为引物,PCR扩增分别获得上游同源臂malPT-L和下游同源臂malPT-R,分别约610bp。
以pTargetN20-pfnrs-stlA(yicS)质粒为模板,pfnrs-stlA(malP)-F/pfnrs-stlA(malP)-R为引物,PCR扩增pfnrs-stlA(malP)片段,约2.3kb。
malPT-L、pfnrs-stlA(malP)和malPT-R片段利用DNA assembly方法(试剂盒DNAassembly Kit购自全式金)克隆入pTargetN20-malPT质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-pfnrs-stlA(malPT)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-pfnrs-stlA(malPT)质粒电转入EcN(yicS::pfnrs-stlA)/pCas感受态细胞,转化子利用malP-seq-F/stlA-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA)/pCas。
1.3malE位点敲入苯丙氨酸解氨酶stlA
(1)malE位点敲入安普霉素抗性盒
以pMDIAI质粒(addgene:51655)为模板,malE-Apr-F/malE-Apr-R为引物,PCR扩增Apr(malE)片段,约1.4kb。以EcN基因组为模板,分别以malE-L-F1/malE-L-R1和malE-R-F1/malE-R-R1为引物,分别PCR扩增malE-L1和malE-R1片段,分别约600bp。以malE-L1/Apr(malE)/malE-R1片段为模板,malE-L1/malE-R1为引物,overlap PCR扩增malE-Apr片段,约2.7kb。
参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA)/pCas电转感受态细胞,并参考1.1(4)方法将malE-Apr片段电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA)/pCas感受态细胞,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::Apr)/pCas。
(2)pTargetN20-pfnrs-stlA(malE)质粒构建
以pTargetF质粒为模板,N20-Apr-F/pTargetF--R为引物,PCR扩增获得N20-Apr片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-Apr质粒。
以EcN基因组为模板,分别以malE-L-F/malE-L-R和malE-R-F/malE-R-R为引物,PCR扩增分别获得上游同源臂malE-L和下游同源臂malE-R,分别约600bp和630bp。
以pTargetN20-pfnrs-stlA(malPT)质粒为模板,pfnrs-stlA(malE)-F/pfnrs-stlA(malE)-R为引物,PCR扩增pfnrs-stlA(malE)片段,约2.2kb。
malE-L、pfnrs-stlA(malE)和malE-R片段利用DNA assembly方法(试剂盒DNAassembly Kit购自全式金)克隆入pTargetN20-Apr质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-pfnrs-stlA(malE)质粒转化子。
(3)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::Apr)/pCas电转感受态细胞。
(4)参考1.1(4)方法将pTargetN20-pfnrs-stlA(malE)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::Apr)/pCas感受态细胞,转化子利用malE-seq-F/stlA-seq-R进行菌落PCR验证。
(5)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA)/pCas。
1.4lacZ位点敲入L-苯丙氨酸内运蛋白pheP
(1)pTargetN20-pfnrs-pheP(lacZ)质粒构建
以pTargetF质粒为模板,N20-lacZ-F/pTargetF-R为引物,PCR扩增获得N20-lacZ片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-lacZ质粒。
以EcN基因组为模板,lacZ-L-F(phep)/lacZ-L-R(phep)和lacZ-R-F(phep)/lacZ-R-R(phep)分别为引物,PCR扩增分别获得上游同源臂lacZ-L和下游同源臂lacZ-R,分别约710bp和730bp。
以pTargetN20-pfnrs-stlA(yicS)质粒为模板,分别以pfnrs-phep(lacZ)-F/pfnrs-phep(lacZ)-R、pfnrs-phep(lacZ)-F2/pfnrs-phep(lacZ)-R2为引物,PCR扩增pfnrs-phep(lacZ)-1和pfnrs-phep(lacZ)-2片段,分别约300bp和470bp。以大肠杆菌MG1655基因为模板,pfnrs-phep(lacZ)-F1/pfnrs-phep(lacZ)-R1为引物,PCR扩增pfnrs-phep片段,约1.4kb。
lacZ-L、pfnrs-phep(lacZ)-1、pfnrs-phep、pfnrs-phep(lacZ)-2和lacZ-R片段利用DNA assembly方法(试剂盒DNA assembly Kit购自全式金)克隆入pTargetN20-lacZ质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-pfnrs-pheP(lacZ)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-pfnrs-pheP(lacZ)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA)/pCas感受态细胞,转化子利用lacZ-seq-F/phep-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP)/pCas。
1.5agaI位点敲入L-苯丙氨酸内运蛋白pheP
(1)agaI位点敲入安普霉素抗性盒
以pMDIAI质粒(addgene:51655)为模板,Apr-F/Apr-R为引物,PCR扩增Apr片段,约1.4kb。以EcN基因组为模板,分别以agaI-L-F/Apr-agaI-L-R和Apr-agaI-R-F/agaI-R-R为引物,分别PCR扩增Apr-agaI-L和Apr-agaI-R片段,分别约600bp。以Apr-agaI-L/Apr/Apr-agaI-R片段为模板,agaI-L-F/agaI-R-R为引物,overlap PCR扩增获得agaI-Apr片段,约2.7kb。
参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP)/pCas转感受态细胞,并参考1.1(4)方法将agaI-Apr片段电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP)/pCas感受态细胞,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::Apr)/pCas。
(2)pTargetN20-pfnrs-phep(agaI)质粒构建
以EcN基因组为模板,agaI-L-F/agaI-L-R和agaI-R-F/agaI-R-R分别为引物,PCR扩增分别获得上游同源臂agaI-L和下游同源臂agaI-R,分别约620bp。
以pTargetN20-pfnrs-stlA(malE)质粒为模板,pfnrs-phep(agaI)-F/pfnrs-phep(lacZ)-R为引物,PCR扩增pfnrs(agaI)片段,约600bp。
以pTargetN20-pfnrs-pheP(lacZ)质粒为模板,pfnrs-phep(lacZ)-F1/pfnrs-phep(agaI)-R为引物,PCR扩增phep(agaI)-1片段,约1.4kb;以phep(agaI)-1片段为模板,pfnrs-phep(lacZ)-F1/pfnrs-phep(agaI)-R1为引物,PCR扩增phep(agaI)-2片段,约1.4kb;以phep(agaI)-2片段为模板,pfnrs-phep(lacZ)-F1/pfnrs-phep(agaI)-R2为引物,PCR扩增phep(agaI)片段,约1.5kb。
agaI-L、pfnrs(agaI)、phep(agaI)和agaI-R片段利用DNA assembly方法(试剂盒DNA assembly Kit购自全式金)克隆入pTargetN20-Apr质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-pfnrs-phep(agaI)质粒转化子。
(3)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::Apr)/pCas电转感受态细胞。
(4)参考1.1(4)方法将pTargetN20-pfnrs-phep(agaI)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::Apr)/pCas感受态细胞,转化子利用agaI-seq-F/phep-seq-R进行菌落PCR验证。
(5)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP)/pCas。
1.6exo位点敲入苯丙氨酸解氨酶stlA
(1)pTargetN20-ptac-stlA(exo)构建
以pTargetF质粒为模板,N20-exo-F/pTargetF-R为引物,PCR扩增获得N20-exo片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-exo质粒。
以EcN基因组为模板,exo-L-F/exo-L-R和exo-R-F/exo-R-R分别为引物,PCR扩增分别获得上游同源臂exo-L和下游同源臂exo-R,分别约620bp和630bp。
以pTargetN20-pfnrs-phep(agaI)质粒为模板,ptac-stlA(exo)-F/ptac-stlA(exo)-R为引物,PCR扩增ptac(exo)-1片段,约500bp;依次以ptac-stlA(exo)-F/ptac-stlA(exo)-R6、ptac-stlA(exo)-F/ptac-stlA(exo)-R7为引物,前一PCR片段为模板,依次PCR扩增获得ptac(exo)-2、ptac(exo)片段,分别约520bp、550bp。以pTrc99a质粒为模板,分别以ptac-stlA(exo)-F1/ptac-stlA(exo)-R1、ptac-stlA(exo)-F2/ptac-stlA(exo)-R2、ptac-stlA(exo)-F3/ptac-stlA(exo)-R3、ptac-stlA(exo)-F4/ptac-stlA(exo)-R4为引物,分别PCR扩增lac-1、lac-2、lac-3、lac-4片段,分别约190bp、170bp、400bp、550bp;以lac-1/lac-2/lac-3/lac-4片段为模板,ptac-stlA(exo)-F1/ptac-stlA(exo)-R4为引物,PCR扩增lac-14片段,约1.2kb;依次以ptac-stlA(exo)-F5/ptac-stlA(exo)-R5、ptac-stlA(exo)-F6/ptac-stlA(exo)-R5、ptac-stlA(exo)-F7/ptac-stlA(exo)-R5、ptac-stlA(exo)-F8/ptac-stlA(exo)-R8为引物,前一PCR片段为模板,依次PCR扩增lac-5、lac-56、lac-57和lac片段,分别约1.2kb、1.2kb、1.3kb和1.4kb。以pTargetN20-pfnrs-stlA(malE)质粒为模板,ptac-stlA(exo)-F9/ptac-stlA(exo)-R9为引物,PCR扩增ptac-stlA-1片段,约1.8kb;依次以ptac-stlA(exo)-F10/ptac-stlA(exo)-R10、ptac-stlA(exo)-F11/ptac-stlA(exo)-R11、ptac-stlA(exo)-F12/ptac-stlA(exo)-R12为引物,前一PCR片段为模板,依次PCR扩增ptac-stlA-2、ptac-stlA-3、ptac-stlA-4片段,分别约1.8kb、1.9kb、2.0kb;以ptac-stlA-4片段为模板,ptac-stlA(exo)-F13/ptac-stlA(exo)-R12为引物,PCR扩增ptac-stlA(exo)片段,约2kb。
exo-L、ptac(exo)、lac、ptac-stlA(exo)和exo-R片段利用DNA assembly方法(试剂盒DNA assembly Kit购自全式金)克隆入pTargetN20-exo质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-ptac-stlA(exo)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-ptac-stlA(exo)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP)/pCas感受态细胞,转化子利用exo-seq-F/stlA-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA)/pCas。
1.7rhtCB位点敲入苯丙氨酸解氨酶stlA
(1)pTargetN20-ptac-stlA(rhtCB)构建
以pTargetF质粒为模板,N20-rhtC-F/pTargetF-R为引物,PCR扩增获得N20-rhtC片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-rhtC质粒。
以EcN基因组为模板,rhtC-L-F/rhtC-L-R和rhtC-R-F/rhtC-R-R分别为引物,PCR扩增分别获得上游同源臂rhtC-L和下游同源臂rhtC-R,分别约620bp。
以pTargetN20-ptac-stlA(exo)质粒为模板,ptac-stlA(rhtC)-F/ptac-stlA(rhtC)-R为引物,PCR扩增ptac-stlA(rhtC)片段,约3.2kb。
rhtC-L、ptac-stlA(rhtC)和rhtC-R片段利用DNA assembly方法(试剂盒DNAassembly Kit购自全式金)克隆入pTargetN20-rhtC质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-ptac-stlA(rhtC)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-ptac-stlA(rhtC)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA)/pCas感受态细胞,转化子利用rhtC-seq-F/stlA-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA)/pCas。
1.8araBD位点敲入L-氨基酸脱氨酶pma
(1)pTargetN20-para-pma(araBD)质粒构建
以pTargetF质粒为模板,N20-araBD-F/pTargetF-R为引物,PCR扩增获得N20-araBD片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-araBD质粒。
以EcN基因组为模板,分别以araBD-L-F/araBD-L-R和araBD-R-F/araBD-R-R为引物,PCR扩增分别获得上游同源臂araBD-L和下游同源臂araBD-R,分别约600bp。
以pUC-Para-pma质粒为模板,para-pma(araBD)-F/para-pma(araBD)-R为引物,PCR扩增para-pma(araBD)片段,约1.6kb。
araBD-L、para-pma(araBD)和araBD-R片段利用DNA assembly方法(试剂盒DNAassembly Kit购自全式金)克隆入pTargetN20-araBD质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-para-pma(araBD)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-para-pma(araBD)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA)/pCas感受态细胞,转化子利用araBD-seq-F/pma-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma)/pCas。
1.9敲除二氢吡啶二羧酸合酶dapA
(1)pTargetN20-dapA质粒构建
以pTargetF质粒为模板,N20-dapA-F/pTargetF-R为引物,PCR扩增获得N20-dapA片段约2.2kb,DpnI消化PCR片段后自连,获得pTargetN20-dapA质粒。
以EcN基因组为模板,分别以dapA-L-F/dapA-L-R和dapA-R-F/dapA-R-R为引物,PCR扩增分别获得上游同源臂dapA-L和下游同源臂dapA-R,分别约620bp。
dapA-L和dapA-R片段利用DNA assembly方法(试剂盒DNA assembly Kit购自全式金)克隆入pTargetN20-dapA质粒的EcoRI/HindIII位点,连接产物转化入DH5α化学感受态细胞,复苏液涂布于含壮观霉素(终浓度50μg/mL)LB固体平板,获得含pTargetN20-dapA(Ho)质粒转化子。
(2)参考1.1(3)方法制备EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma)/pCas电转感受态细胞。
(3)参考1.1(4)方法将pTargetN20-dapA(Ho)质粒电转入EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma)/pCas感受态细胞,转化子利用dapA-seq-F/dapA-seq-R进行菌落PCR验证。
(4)参考1.1(5)方法将辅助质粒消除,获得EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma,△dapA)/pCas。
(5)pCas质粒消除:挑取辅助质粒消除阳性克隆子,直接在LB液体培养基中37℃过夜摇床培养,吸取少量菌液于LB固体平板上划线分单菌。单菌落在含卡那霉素(50μg/mL)抗性LB平板上进行鉴定,验证pCas质粒消除,获得基因工程菌EcN(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma,△dapA),命名为CIBT4564。
实施例2工程益生菌CIBT4564对苯丙氨酸体外降解考察
2.1体外测活
(1)将EcN和CIBT4564菌株分别接种于LB培养基中(CIBT4564培养时需添加终浓度为100μg/mL二氨基庚二酸),37℃、250rpm过夜培养。以1%v/v接种量转接于30mL LB培养基中,37℃、250rpm条件下培养1.5小时,1.5小时后时添加10mM阿拉伯糖和1mM IPTG继续培养3小时。
(2)4000rpm条件下离心收集菌体,M9培养基(含0.5%葡萄糖)重悬菌体,将菌浓OD600调至1.0。分别取0.4mL菌液于2mL离心管中,4500rpm离心10min,去上清,用5mL分析缓冲液(M9培养基、5g/L葡萄糖,50mM MOPS,4mM L-苯丙氨酸)悬浮菌体于普通试管中,定位T=0h,放于37℃,250rpm培养3小时取样1mL。13000rpm,离心10min,收集上清样品进行HPLC检测。
3.1苯丙氨酸、苯丙酮酸及反式肉桂酸HPLC检测方法
色谱柱ZORBAX SB-C18色谱柱(150mm×4.6mm,5μm);柱温40℃;以1.5%乙酸和乙腈为流动相,进行梯度洗脱;采用紫外检测器,检测波长260nm,进样量5μL。检测结果见图1。
从图1可知,添加原始菌株EcN,无反式肉桂酸生成;而添加工程益生菌CIBT4564则有反式肉桂酸生成,表明CIBT4564降解苯丙氨酸生成了反式肉桂酸。
实施例3工程益生菌CIBT4564在苯丙酮尿症小鼠体内对苯丙氨酸的降解考察
苯丙酮尿症小鼠PahR408W的制备方法参照文献Yin,S.,et al.(2020).Enhancedgenome editing to ameliorate a genetic metabolic liver disease through co-delivery of adeno-associated virus receptor.Sci China Life Sci 63。该模型8-12周龄,由华东师范大学构建,具备苯丙酮尿症的典型症状,在正常饲料喂养下,PahR408W苯丙酮尿症小鼠在出生6周后血液中的苯丙氨酸含量都在1200μmol/L以上,并且随着饲养时间延长,血液中的苯丙氨酸含量持续增加,8周龄小鼠的苯丙氨酸含量都将超过1500μmol/L。此外,PahR408W苯丙酮尿症小鼠的体重和体型都明显偏小,随着时间推移,小鼠的毛色也逐渐表现出棕黄色。
8-12周龄的小鼠利用无苯丙氨酸(Phe-deficient)饲料喂养(购自Deyts),含苯丙氨酸(0.5g/L)的饮用水作为苯丙氨酸补给。动物实验开始时(T=0h),停止含苯丙氨酸饮用水补给,更换为普通饮用水,采血检测T=0h血液中苯丙氨酸浓度。接着,小鼠皮下注射0.1mg/g(BW)苯丙氨酸溶液,在皮下注射后第1、2、3小时分别灌胃300μL(即5×1010cfu/只小鼠)益生菌EcN或CIBT4564或生理盐水,在注射后第4小时采血检测血液中苯丙氨酸的浓度。结果如图2所示。
从图2可知,口服工程益生菌CIBT4564后,苯丙酮尿症小鼠血液中苯丙氨酸浓度与口服生理盐水和EcN相比有明显下降。
综上所述,本发明所构建的工程益生菌CIBT4564能够在体外、体内对苯丙氨酸均能有效降解,有望应用于治疗苯丙酮尿症。
序列表
<110> 中国科学院分子植物科学卓越创新中心
华东师范大学
<120> 用于治疗苯丙酮尿症的工程益生菌
<130> SHPI2010645
<160> 3
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1599
<212> DNA
<213> Photorhabdus luminescens
<400> 1
atgaaagcta aagatgttca gccaaccatt attattaata aaaatggcct tatctctttg 60
gaagatatct atgacattgc gataaaacaa aaaaaagtag aaatatcaac ggagatcact 120
gaacttttga cgcatggtcg tgaaaaatta gaggaaaaat taaattcagg agaggttata 180
tatggaatca atacaggatt tggagggaat gccaatttag ttgtgccatt tgagaaaatc 240
gcagagcatc agcaaaatct gttaactttt ctttctgctg gtactgggga ctatatgtcc 300
aaaccttgta ttaaagcgtc acaatttact atgttacttt ctgtttgcaa aggttggtct 360
gcaaccagac caattgtcgc tcaagcaatt gttgatcata ttaatcatga cattgttcct 420
ctggttcctc gctatggctc agtgggtgca agcggtgatt taattccttt atcttatatt 480
gcacgagcat tatgtggtat cggcaaagtt tattatatgg gcgcagaaat tgacgctgct 540
gaagcaatta aacgtgcagg gttgacacca ttatcgttaa aagccaaaga aggtcttgct 600
ctgattaacg gcacccgggt aatgtcagga atcagtgcaa tcaccgtcat taaactggaa 660
aaactattta aagcctcaat ttctgcgatt gcccttgctg ttgaagcatt acttgcatct 720
catgaacatt atgatgcccg gattcaacaa gtaaaaaatc atcctggtca aaacgcggtg 780
gcaagtgcat tgcgtaattt attggcaggt tcaacgcagg ttaatctatt atctggggtt 840
aaagaacaag ccaataaagc ttgtcgtcat caagaaatta cccaactaaa tgatacctta 900
caggaagttt attcaattcg ctgtgcacca caagtattag gtatagtgcc agaatcttta 960
gctaccgctc ggaaaatatt ggaacgggaa gttatctcag ctaatgataa tccattgata 1020
gatccagaaa atggcgatgt tctacacggt ggaaatttta tggggcaata tgtcgcccga 1080
acaatggatg cattaaaact ggatattgct ttaattgcca atcatcttca cgccattgtg 1140
gctcttatga tggataaccg tttctctcgt ggattaccta attcactgag tccgacaccc 1200
ggcatgtatc aaggttttaa aggcgtccaa ctttctcaaa ccgctttagt tgctgcaatt 1260
cgccatgatt gtgctgcatc aggtattcat accctcgcca cagaacaata caatcaagat 1320
attgtcagtt taggtctgca tgccgctcaa gatgttttag agatggagca gaaattacgc 1380
aatattgttt caatgacaat tctggtagtt tgtcaggcca ttcatcttcg cggcaatatt 1440
agtgaaattg cgcctgaaac tgctaaattt taccatgcag tacgcgaaat cagttctcct 1500
ttgatcactg atcgtgcgtt ggatgaagat ataatccgca ttgcggatgc aattattaat 1560
gatcaacttc ctctgccaga aatcatgctg gaagaataa 1599
<210> 2
<211> 1377
<212> DNA
<213> Escherichia coli MG1655
<400> 2
atgaaaaacg cgtcaaccgt atcggaagat actgcgtcga atcaagagcc gacgcttcat 60
cgcggattac ataaccgtca tattcaactg attgcgttgg gtggcgcaat tggtactggt 120
ctgtttcttg gcattggccc ggcgattcag atggcgggtc cggctgtatt gctgggctac 180
ggcgtcgccg ggatcatcgc tttcctgatt atgcgccagc ttggcgaaat ggtggttgag 240
gagccggtat ccggttcatt tgcccacttt gcctataaat actggggacc gtttgcgggc 300
ttcctctctg gctggaacta ctgggtaatg ttcgtgctgg tgggaatggc agagctgacc 360
gctgcgggca tctatatgca gtactggttc ccggatgttc caacgtggat ttgggctgcc 420
gccttcttta ttatcatcaa cgccgttaac ctggtgaacg tgcgcttata tggcgaaacc 480
gagttctggt ttgcgttgat taaagtgctg gcaatcatcg gtatgatcgg ctttggcctg 540
tggctgctgt tttctggtca cggcggcgag aaagccagta tcgacaacct ctggcgctac 600
ggtggtttct tcgccaccgg ctggaatggg ctgattttgt cgctggcggt aattatgttc 660
tccttcggcg gtctggagct gattgggatt actgccgctg aagcgcgcga tccggaaaaa 720
agcattccaa aagcggtaaa tcaggtggtg tatcgcatcc tgctgtttta catcggttca 780
ctggtggttt tactggcgct ctatccgtgg gtggaagtga aatccaacag tagcccgttt 840
gtgatgattt tccataatct cgacagcaac gtggtagctt ctgcgctgaa cttcgtcatt 900
ctggtagcat cgctgtcagt gtataacagc ggggtttact ctaacagccg catgctgttt 960
ggcctttctg tgcagggtaa tgcgccgaag tttttgactc gcgtcagccg tcgcggtgtg 1020
ccgattaact cgctgatgct ttccggagcg atcacttcgc tggtggtgtt aatcaactat 1080
ctgctgccgc aaaaagcgtt tggtctgctg atggcgctgg tggtagcaac gctgctgttg 1140
aactggatta tgatctgtct ggcgcatctg cgttttcgtg cagcgatgcg acgtcagggg 1200
cgtgaaacac agtttaaggc gctgctctat ccgttcggca actatctctg cattgccttc 1260
ctcggcatga ttttgctgct gatgtgcacg atggatgata tgcgcttgtc agcgatcctg 1320
ctgccggtgt ggattgtatt cctgtttatg gcatttaaaa cgctgcgtcg gaaataa 1377
<210> 3
<211> 1422
<212> DNA
<213> Proteus mirabilis HI4320
<400> 3
atgaacattt caaggagaaa gctactttta ggtgttggtg ctgcgggcgt tttagcaggt 60
ggtgcggctt tagttccaat ggttcgccgt gacggcaaat ttgtggaagc taaatcaaga 120
gcatcatttg ttgaaggtac gcaaggggct cttcctaaag aagcagatgt agtgattatt 180
ggtgccggta ttcaagggat catgaccgct attaaccttg ctgaacgtgg tatgagtgtc 240
actatcttag aaaagggtca gattgccggt gagcaatcag gccgtgcata cagccaaatt 300
attagttacc aaacatcgcc agaaatcttc ccattacacc attatgggaa aatattatgg 360
cgtggcatga atgagaaaat tggtgcggat accagttatc gtactcaagg tcgtgtagaa 420
gcgctggcag atgaaaaagc attagataaa gctcaagcgt ggatcaaaac agctaaagaa 480
gcggcaggtt ttgatacacc attaaatact cgcatcatta aaggtgaaga gctatcaaat 540
cgcttagtcg gtgctcaaac gccatggact gttgctgcat ttgaagaaga ttcaggctct 600
gttgatcctg aaacaggcac acctgcactc gctcgttatg ccaaacaaat cggtgtgaaa 660
atttatacca actgtgcagt aagaggtatt gaaactgcgg gtggtaaaat ctctgatgtg 720
gtgagtgaga aaggggcgat taaaacgtct caagttgtac tcgctggggg tatctggtcg 780
cgtttattta tgggcaatat gggtattgat atcccaacgc tcaatgtata tctatcacaa 840
caacgtgtct caggggttcc tggtgcacca cgtggtaatg tgcatttacc taatggtatt 900
catttccgcg aacaagcgga tggtacttat gccgttgcac cacgtatctt tacgagttca 960
atagtcaaag atagcttcct gctagggcct aaatttatgc acttattagg tggcggagag 1020
ttaccgttgg aattctctat tggtgaagat ctatttaatt catttaaaat gccgacctct 1080
tggaatttag atgaaaaaac accattcgaa caattccgag ttgccacggc aacacaaaat 1140
acgcaacact tagatgctgt tttccaaaga atgaaaacag aattcccagt atttgaaaaa 1200
tcagaagttg ttgaacgttg gggtgccgtt gtgagtccaa catttgatga attacctatc 1260
atttctgagg tcaaagaata cccaggctta gtgattaaca cggcaacagt gtggggtatg 1320
acagaaggcc cggcagcggg tgaagtgacc gctgatattg tcatgggcaa gaaacctgtt 1380
attgatccaa cgccgtttag tttggatcgt tttaagaagt aa 1422
Claims (10)
1.一种用于治疗苯丙酮尿症的工程益生菌,其为大肠杆菌Nissle 1917衍生菌,在基因组上整合了外源L-苯丙氨酸解氨酶基因、L-苯丙氨酸内运蛋白基因和L-氨基酸脱氨酶基因。
2.如权利要求1所述的工程益生菌,其特征在于,所述L-苯丙氨酸解氨酶基因是stlA(SEQ ID NO:1);所述L-苯丙氨酸内运蛋白基因是pheP(SEQ ID NO:2);所述L-氨基酸脱氨酶基因是pma(SEQ ID NO:3)。
3.如权利要求1所述的工程益生菌,其特征在于,所述大肠杆菌Nissle 1917衍生菌的基因组中敲除二氢吡啶二羧酸合酶基因dapA。
4.如权利要求3所述的工程益生菌,其特征在于,其基因型为Nissle 1917(yicS::pfnrs-stlA,malPT::pfnrs-stlA,malE::pfnrs-stlA,lacZ::pfnrs-pheP,agaI::pfnrs-pheP,exo::ptac-stlA,rhtCB::ptac-stlA,araBD::para-pma,△dapA)。
5.一种构建如权利要求1-4中任一项所述工程益生菌的方法,其特征在于,包括以下步骤:
A.以大肠杆菌Nissle 1917为底盘菌,分别在基因组的yicS位点、malPT位点、malE位点、exo位点和rhtCB位点中的一个以上位点敲入L-苯丙氨酸解氨酶基因,获得stlA因整合菌株;
B.对于步骤A中获得的stlA因整合菌株,在其基因组的lacZ位点和agaI位点中的一个以上位点、优选两个位点敲入L-苯丙氨酸内运蛋白基因pheP,得到stlA+pheP整合菌株;
C.对于步骤B中获得的stlA+pheP整合菌株,在其基因组的araBD位点敲入L-氨基酸脱氨酶基因,得到stlA+pheP+pma整合菌株。
6.如权利要求5所述的方法,其特征在于,还包括以下步骤:
对于步骤C中获得的stlA+pheP+pma整合菌株,敲除其基因组中的二氢吡啶二羧酸合酶基因dapA,得到stlA+pheP+pma△dapA菌株。
7.如权利要求4或5所述的方法,其特征在于,基因stlA、pheP、pma的敲入和基因dapA的敲除通过基因编辑技术实施,所述基因编辑采用CRISPR-Cas9系统、CRISPR-Cpf1系统、CRISPR-Cas相关的转座系统INTEGRATE系统或者CAST系统。
8.如权利要求1-3中任一项所述的工程益生菌在制备苯丙酮尿症治疗药物中的应用。
9.如权利要求8所述的应用,其特征在于,所述苯丙酮尿症是苯丙氨酸羟化酶基因突变PahR408W引起的。
10.如权利要求8所述的应用,其特征在于,所述药物是口服制剂。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN202110013298.XA CN112662606A (zh) | 2021-01-06 | 2021-01-06 | 用于治疗苯丙酮尿症的工程益生菌 |
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN202110013298.XA CN112662606A (zh) | 2021-01-06 | 2021-01-06 | 用于治疗苯丙酮尿症的工程益生菌 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN112662606A true CN112662606A (zh) | 2021-04-16 |
Family
ID=75413296
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN202110013298.XA Pending CN112662606A (zh) | 2021-01-06 | 2021-01-06 | 用于治疗苯丙酮尿症的工程益生菌 |
Country Status (1)
| Country | Link |
|---|---|
| CN (1) | CN112662606A (zh) |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN113969292A (zh) * | 2021-11-15 | 2022-01-25 | 山东第一医科大学(山东省医学科学院) | 治疗苯丙酮尿症的工程益生菌及其构建方法与应用 |
| WO2022148362A1 (zh) * | 2021-01-07 | 2022-07-14 | 上海陶宇晟生物技术有限责任公司 | 具备表面展示苯丙氨酸解氨酶的工程益生菌 |
| WO2023165521A1 (zh) * | 2022-03-02 | 2023-09-07 | 和度生物技术(上海)有限公司 | 一种用于治疗高苯丙氨酸血症的工程微生物及其用途 |
Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN102753566A (zh) * | 2010-02-04 | 2012-10-24 | 生物马林药物股份有限公司 | 原核苯丙氨酸解氨酶变异体的组合物以及利用其组合物的方法 |
| WO2014066945A1 (en) * | 2012-11-01 | 2014-05-08 | The Sydney Children's Hospital Network (Randwick & Westmead) | Genetically-modified probiotic for treatment of phenylketonuria |
| WO2016183532A1 (en) * | 2015-05-13 | 2016-11-17 | Synlogic, Inc. | Bacteria engineered to treat a disease or disorder |
| CA2985819A1 (en) * | 2015-05-13 | 2016-11-17 | Synlogic, Inc. | Bacteria engineered to reduce hyperphenylalaninemia |
| US20190282628A1 (en) * | 2016-01-11 | 2019-09-19 | Synlogic Inc. | Recombinant bacteria engineered to treat diseases and disorders associated with amino acid metabolism and methods of use thereof |
-
2021
- 2021-01-06 CN CN202110013298.XA patent/CN112662606A/zh active Pending
Patent Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN102753566A (zh) * | 2010-02-04 | 2012-10-24 | 生物马林药物股份有限公司 | 原核苯丙氨酸解氨酶变异体的组合物以及利用其组合物的方法 |
| WO2014066945A1 (en) * | 2012-11-01 | 2014-05-08 | The Sydney Children's Hospital Network (Randwick & Westmead) | Genetically-modified probiotic for treatment of phenylketonuria |
| WO2016183532A1 (en) * | 2015-05-13 | 2016-11-17 | Synlogic, Inc. | Bacteria engineered to treat a disease or disorder |
| CA2985819A1 (en) * | 2015-05-13 | 2016-11-17 | Synlogic, Inc. | Bacteria engineered to reduce hyperphenylalaninemia |
| US20190282628A1 (en) * | 2016-01-11 | 2019-09-19 | Synlogic Inc. | Recombinant bacteria engineered to treat diseases and disorders associated with amino acid metabolism and methods of use thereof |
Non-Patent Citations (2)
| Title |
|---|
| VINCENT M ISABELLA ET AL.: ""Development of a synthetic live bacterial therapeutic for the human metabolic disease phenylketonuria"", 《NATURE BIOTECHNOLOGY》 * |
| 蔡朱男等: ""一种可用于治疗苯丙酮尿症的工程菌株的建立"", 《中国病理生理杂志》 * |
Cited By (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022148362A1 (zh) * | 2021-01-07 | 2022-07-14 | 上海陶宇晟生物技术有限责任公司 | 具备表面展示苯丙氨酸解氨酶的工程益生菌 |
| CN113969292A (zh) * | 2021-11-15 | 2022-01-25 | 山东第一医科大学(山东省医学科学院) | 治疗苯丙酮尿症的工程益生菌及其构建方法与应用 |
| CN113969292B (zh) * | 2021-11-15 | 2023-07-07 | 山东第一医科大学(山东省医学科学院) | 治疗苯丙酮尿症的工程益生菌及其构建方法与应用 |
| WO2023165521A1 (zh) * | 2022-03-02 | 2023-09-07 | 和度生物技术(上海)有限公司 | 一种用于治疗高苯丙氨酸血症的工程微生物及其用途 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN112501098B (zh) | 具有降解苯丙氨酸能力的工程益生菌 | |
| Bron et al. | Genetic characterization of the bile salt response in Lactobacillus plantarum and analysis of responsive promoters in vitro and in situ in the gastrointestinal tract | |
| Denou et al. | Identification of genes associated with the long-gut-persistence phenotype of the probiotic Lactobacillus johnsonii strain NCC533 using a combination of genomics and transcriptome analysis | |
| JP7494345B2 (ja) | 高フェニルアラニン血症を低減させるように操作された細菌 | |
| CN112662606A (zh) | 用于治疗苯丙酮尿症的工程益生菌 | |
| KR20170121291A (ko) | 감소된 창자 염증 및/또는 강화된 창자 점막 장벽으로부터 이익을 얻는 질병을 치료하기 위해 공학처리된 박테리아 | |
| EP2274422B1 (en) | Expression vector | |
| CN110747157B (zh) | 一种能在肠道中降解尿酸的工程益生菌及其制备方法与应用 | |
| Saraoui et al. | A unique in vivo experimental approach reveals metabolic adaptation of the probiotic Propionibacterium freudenreichii to the colon environment | |
| WO2022148362A1 (zh) | 具备表面展示苯丙氨酸解氨酶的工程益生菌 | |
| US20230105474A1 (en) | Recombinant bacteria engineered to treat diseases associated with uric acid and methods of use thereof | |
| US8741622B2 (en) | Stress tolerant Bifidobacteria | |
| US20230090705A1 (en) | Recombinant bacteria engineered to treat diseases associated with methionine metabolism and methods of use thereof | |
| US20240122993A1 (en) | Genetically gengineered bacterium for hangover and liver disease prevention and/or treatment | |
| CN114774338A (zh) | 一种产丁酸的益生菌及其构建方法和应用 | |
| CN113969292B (zh) | 治疗苯丙酮尿症的工程益生菌及其构建方法与应用 | |
| US20240175039A1 (en) | Engineering probiotic for degrading uric acid, and construction method therefor and use thereof | |
| CN114561333B (zh) | 一种转化支链氨基酸的工程菌及在制备治疗枫糖尿症产品中的应用 | |
| CN111518801A (zh) | 一种组成型乳酸菌启动子、重组载体及其重组菌和应用 | |
| WO2023165521A1 (zh) | 一种用于治疗高苯丙氨酸血症的工程微生物及其用途 | |
| CN107513516B (zh) | 不溶血无乳链球菌WC1535△cyl及其构建和应用 | |
| US20220047654A1 (en) | Pharmabiotic treatments for metabolic disorders | |
| CN107603933B (zh) | 一种无乳链球菌WC1535△cps及其构建和应用 | |
| CN117417872A (zh) | 一种用于治疗苯丙酮尿症的细胞或菌株、药物及应用 | |
| Ghatani et al. | Bifidobacterial Genome Editing for Potential Probiotic Development |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PB01 | Publication | ||
| PB01 | Publication | ||
| SE01 | Entry into force of request for substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| RJ01 | Rejection of invention patent application after publication |
Application publication date: 20210416 |
|
| RJ01 | Rejection of invention patent application after publication |