CN102787105A - 棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 - Google Patents
棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 Download PDFInfo
- Publication number
- CN102787105A CN102787105A CN2012102457652A CN201210245765A CN102787105A CN 102787105 A CN102787105 A CN 102787105A CN 2012102457652 A CN2012102457652 A CN 2012102457652A CN 201210245765 A CN201210245765 A CN 201210245765A CN 102787105 A CN102787105 A CN 102787105A
- Authority
- CN
- China
- Prior art keywords
- gene
- cotton
- ugd6
- arabidopis thaliana
- glucose dehydrogenase
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Granted
Links
- 108090000623 proteins and genes Proteins 0.000 title claims abstract description 100
- 229920000742 Cotton Polymers 0.000 title claims abstract description 83
- 102000004169 proteins and genes Human genes 0.000 title abstract description 17
- 108010054269 Uridine Diphosphate Glucose Dehydrogenase Proteins 0.000 title abstract description 9
- 239000000835 fiber Substances 0.000 claims abstract description 29
- 125000003275 alpha amino acid group Chemical group 0.000 claims abstract description 15
- 150000001413 amino acids Chemical class 0.000 claims abstract description 7
- 239000002773 nucleotide Substances 0.000 claims abstract description 7
- 125000003729 nucleotide group Chemical group 0.000 claims abstract description 7
- HSCJRCZFDFQWRP-RDKQLNKOSA-N UDP-D-glucose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)OC1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-RDKQLNKOSA-N 0.000 claims description 20
- 235000001014 amino acid Nutrition 0.000 claims description 6
- 230000014509 gene expression Effects 0.000 abstract description 28
- 229920002678 cellulose Polymers 0.000 abstract description 26
- 239000001913 cellulose Substances 0.000 abstract description 25
- 210000002421 cell wall Anatomy 0.000 abstract description 21
- 241000219194 Arabidopsis Species 0.000 abstract description 6
- 230000015572 biosynthetic process Effects 0.000 abstract description 6
- 238000011161 development Methods 0.000 abstract description 6
- 102000057144 Uridine Diphosphate Glucose Dehydrogenase Human genes 0.000 abstract description 5
- 239000002243 precursor Substances 0.000 abstract description 5
- 239000000126 substance Substances 0.000 abstract description 2
- 229920002488 Hemicellulose Polymers 0.000 abstract 1
- 230000001131 transforming effect Effects 0.000 abstract 1
- 244000299507 Gossypium hirsutum Species 0.000 description 63
- 210000004027 cell Anatomy 0.000 description 22
- 230000008859 change Effects 0.000 description 21
- 239000013612 plasmid Substances 0.000 description 19
- 108090000790 Enzymes Proteins 0.000 description 17
- 102000004190 Enzymes Human genes 0.000 description 17
- 241000196324 Embryophyta Species 0.000 description 16
- 239000013604 expression vector Substances 0.000 description 14
- 235000018102 proteins Nutrition 0.000 description 14
- 230000000694 effects Effects 0.000 description 12
- 239000000284 extract Substances 0.000 description 11
- 238000000034 method Methods 0.000 description 10
- 229920001282 polysaccharide Polymers 0.000 description 10
- 239000005017 polysaccharide Substances 0.000 description 10
- 150000004804 polysaccharides Chemical class 0.000 description 10
- 238000004458 analytical method Methods 0.000 description 9
- 230000004927 fusion Effects 0.000 description 9
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 9
- 230000009261 transgenic effect Effects 0.000 description 9
- 241000588724 Escherichia coli Species 0.000 description 8
- 240000000047 Gossypium barbadense Species 0.000 description 8
- 235000009429 Gossypium barbadense Nutrition 0.000 description 8
- 239000012634 fragment Substances 0.000 description 8
- HDYANYHVCAPMJV-LXQIFKJMSA-K UDP-alpha-D-glucuronate(3-) Chemical compound C([C@@H]1[C@H]([C@H]([C@@H](O1)N1C(NC(=O)C=C1)=O)O)O)OP([O-])(=O)OP([O-])(=O)O[C@H]1O[C@H](C([O-])=O)[C@@H](O)[C@H](O)[C@H]1O HDYANYHVCAPMJV-LXQIFKJMSA-K 0.000 description 7
- 239000000047 product Substances 0.000 description 7
- 101710088194 Dehydrogenase Proteins 0.000 description 6
- 238000006243 chemical reaction Methods 0.000 description 6
- 238000001514 detection method Methods 0.000 description 6
- 238000005516 engineering process Methods 0.000 description 6
- 230000001939 inductive effect Effects 0.000 description 6
- 239000002777 nucleoside Substances 0.000 description 6
- 125000003835 nucleoside group Chemical group 0.000 description 6
- 230000035939 shock Effects 0.000 description 6
- 230000000692 anti-sense effect Effects 0.000 description 5
- 230000001580 bacterial effect Effects 0.000 description 5
- 230000018109 developmental process Effects 0.000 description 5
- 230000012010 growth Effects 0.000 description 5
- 230000006872 improvement Effects 0.000 description 5
- 229920001277 pectin Polymers 0.000 description 5
- 239000001814 pectin Substances 0.000 description 5
- 235000010987 pectin Nutrition 0.000 description 5
- 238000003753 real-time PCR Methods 0.000 description 5
- 238000012216 screening Methods 0.000 description 5
- 238000012360 testing method Methods 0.000 description 5
- 230000009466 transformation Effects 0.000 description 5
- 101150034129 ugd gene Proteins 0.000 description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 4
- 108700026244 Open Reading Frames Proteins 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 238000010276 construction Methods 0.000 description 4
- 238000010586 diagram Methods 0.000 description 4
- 230000002068 genetic effect Effects 0.000 description 4
- 238000004519 manufacturing process Methods 0.000 description 4
- 239000000463 material Substances 0.000 description 4
- 239000002245 particle Substances 0.000 description 4
- 230000008719 thickening Effects 0.000 description 4
- 210000001519 tissue Anatomy 0.000 description 4
- 241000589158 Agrobacterium Species 0.000 description 3
- 244000291564 Allium cepa Species 0.000 description 3
- 235000002732 Allium cepa var. cepa Nutrition 0.000 description 3
- 238000006555 catalytic reaction Methods 0.000 description 3
- 239000003153 chemical reaction reagent Substances 0.000 description 3
- 238000013461 design Methods 0.000 description 3
- 230000029087 digestion Effects 0.000 description 3
- 238000001962 electrophoresis Methods 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 230000001965 increasing effect Effects 0.000 description 3
- 230000002018 overexpression Effects 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 230000009465 prokaryotic expression Effects 0.000 description 3
- 238000011084 recovery Methods 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 239000010902 straw Substances 0.000 description 3
- 230000004960 subcellular localization Effects 0.000 description 3
- 235000018322 upland cotton Nutrition 0.000 description 3
- 238000011144 upstream manufacturing Methods 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- CSCPPACGZOOCGX-UHFFFAOYSA-N Acetone Chemical compound CC(C)=O CSCPPACGZOOCGX-UHFFFAOYSA-N 0.000 description 2
- 101100101638 Arabidopsis thaliana UGD1 gene Proteins 0.000 description 2
- 101100539395 Arabidopsis thaliana UGD3 gene Proteins 0.000 description 2
- 101100539397 Arabidopsis thaliana UGD4 gene Proteins 0.000 description 2
- 241000620209 Escherichia coli DH5[alpha] Species 0.000 description 2
- 244000068988 Glycine max Species 0.000 description 2
- 235000010469 Glycine max Nutrition 0.000 description 2
- NYHBQMYGNKIUIF-UUOKFMHZSA-N Guanosine Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O NYHBQMYGNKIUIF-UUOKFMHZSA-N 0.000 description 2
- 206010021703 Indifference Diseases 0.000 description 2
- 108091026898 Leader sequence (mRNA) Proteins 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 108091036066 Three prime untranslated region Proteins 0.000 description 2
- HSCJRCZFDFQWRP-JZMIEXBBSA-N UDP-alpha-D-glucose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1OP(O)(=O)OP(O)(=O)OC[C@@H]1[C@@H](O)[C@@H](O)[C@H](N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-JZMIEXBBSA-N 0.000 description 2
- HSCJRCZFDFQWRP-UHFFFAOYSA-N Uridindiphosphoglukose Natural products OC1C(O)C(O)C(CO)OC1OP(O)(=O)OP(O)(=O)OCC1C(O)C(O)C(N2C(NC(=O)C=C2)=O)O1 HSCJRCZFDFQWRP-UHFFFAOYSA-N 0.000 description 2
- 229920002000 Xyloglucan Polymers 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- RJGDLRCDCYRQOQ-UHFFFAOYSA-N anthrone Chemical compound C1=CC=C2C(=O)C3=CC=CC=C3CC2=C1 RJGDLRCDCYRQOQ-UHFFFAOYSA-N 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 239000000470 constituent Substances 0.000 description 2
- 235000012343 cottonseed oil Nutrition 0.000 description 2
- 238000011033 desalting Methods 0.000 description 2
- 238000007598 dipping method Methods 0.000 description 2
- 210000002615 epidermis Anatomy 0.000 description 2
- 238000010195 expression analysis Methods 0.000 description 2
- 210000000630 fibrocyte Anatomy 0.000 description 2
- 238000010230 functional analysis Methods 0.000 description 2
- 102000037865 fusion proteins Human genes 0.000 description 2
- 108020001507 fusion proteins Proteins 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 230000005764 inhibitory process Effects 0.000 description 2
- 239000002609 medium Substances 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 238000003757 reverse transcription PCR Methods 0.000 description 2
- 239000000758 substrate Substances 0.000 description 2
- 229920001221 xylan Polymers 0.000 description 2
- 150000004823 xylans Chemical class 0.000 description 2
- MVMSCBBUIHUTGJ-UHFFFAOYSA-N 10108-97-1 Natural products C1=2NC(N)=NC(=O)C=2N=CN1C(C(C1O)O)OC1COP(O)(=O)OP(O)(=O)OC1OC(CO)C(O)C(O)C1O MVMSCBBUIHUTGJ-UHFFFAOYSA-N 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 241000219195 Arabidopsis thaliana Species 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 240000008564 Boehmeria nivea Species 0.000 description 1
- 241001504766 Bovichtus Species 0.000 description 1
- 102000004031 Carboxy-Lyases Human genes 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- MIKUYHXYGGJMLM-GIMIYPNGSA-N Crotonoside Natural products C1=NC2=C(N)NC(=O)N=C2N1[C@H]1O[C@@H](CO)[C@H](O)[C@@H]1O MIKUYHXYGGJMLM-GIMIYPNGSA-N 0.000 description 1
- AVGPOAXYRRIZMM-UHFFFAOYSA-N D-Apiose Natural products OCC(O)(CO)C(O)C=O AVGPOAXYRRIZMM-UHFFFAOYSA-N 0.000 description 1
- ASNHGEVAWNWCRQ-LJJLCWGRSA-N D-apiofuranose Chemical compound OC[C@@]1(O)COC(O)[C@@H]1O ASNHGEVAWNWCRQ-LJJLCWGRSA-N 0.000 description 1
- ASNHGEVAWNWCRQ-UHFFFAOYSA-N D-apiofuranose Natural products OCC1(O)COC(O)C1O ASNHGEVAWNWCRQ-UHFFFAOYSA-N 0.000 description 1
- AEMOLEFTQBMNLQ-YMDCURPLSA-N D-galactopyranuronic acid Chemical compound OC1O[C@H](C(O)=O)[C@H](O)[C@H](O)[C@H]1O AEMOLEFTQBMNLQ-YMDCURPLSA-N 0.000 description 1
- NYHBQMYGNKIUIF-UHFFFAOYSA-N D-guanosine Natural products C1=2NC(N)=NC(=O)C=2N=CN1C1OC(CO)C(O)C1O NYHBQMYGNKIUIF-UHFFFAOYSA-N 0.000 description 1
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- IAJILQKETJEXLJ-UHFFFAOYSA-N Galacturonsaeure Natural products O=CC(O)C(O)C(O)C(O)C(O)=O IAJILQKETJEXLJ-UHFFFAOYSA-N 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- 240000002024 Gossypium herbaceum Species 0.000 description 1
- 102000005455 Monosaccharide Transport Proteins Human genes 0.000 description 1
- 108010006769 Monosaccharide Transport Proteins Proteins 0.000 description 1
- GRYLNZFGIOXLOG-UHFFFAOYSA-N Nitric acid Chemical compound O[N+]([O-])=O GRYLNZFGIOXLOG-UHFFFAOYSA-N 0.000 description 1
- 108091092724 Noncoding DNA Proteins 0.000 description 1
- 101100539399 Oryza sativa subsp. japonica UGD5 gene Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 238000002123 RNA extraction Methods 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 240000000111 Saccharum officinarum Species 0.000 description 1
- 235000007201 Saccharum officinarum Nutrition 0.000 description 1
- 241000124033 Salix Species 0.000 description 1
- 229920005654 Sephadex Polymers 0.000 description 1
- 239000012507 Sephadex™ Substances 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- LPQOADBMXVRBNX-UHFFFAOYSA-N ac1ldcw0 Chemical compound Cl.C1CN(C)CCN1C1=C(F)C=C2C(=O)C(C(O)=O)=CN3CCSC1=C32 LPQOADBMXVRBNX-UHFFFAOYSA-N 0.000 description 1
- 230000035508 accumulation Effects 0.000 description 1
- 238000009825 accumulation Methods 0.000 description 1
- SRBFZHDQGSBBOR-LECHCGJUSA-N alpha-D-xylose Chemical compound O[C@@H]1CO[C@H](O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-LECHCGJUSA-N 0.000 description 1
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 description 1
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 1
- 238000009835 boiling Methods 0.000 description 1
- 239000006227 byproduct Substances 0.000 description 1
- 230000024245 cell differentiation Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 108010040093 cellulose synthase Proteins 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 238000004737 colorimetric analysis Methods 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- 230000018044 dehydration Effects 0.000 description 1
- 238000006297 dehydration reaction Methods 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- XOAGKSFNHBWACO-RAUZPKMFSA-L disodium;[[(2r,3s,4r,5r)-5-(2-amino-6-oxo-3h-purin-9-yl)-3,4-dihydroxyoxolan-2-yl]methoxy-oxidophosphoryl] [(2r,3s,4s,5s,6r)-3,4,5-trihydroxy-6-(hydroxymethyl)oxan-2-yl] phosphate Chemical compound [Na+].[Na+].C([C@H]1O[C@H]([C@@H]([C@@H]1O)O)N1C=NC=2C(=O)N=C(NC=21)N)OP([O-])(=O)OP([O-])(=O)O[C@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@@H]1O XOAGKSFNHBWACO-RAUZPKMFSA-L 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 1
- 238000001952 enzyme assay Methods 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 229940050410 gluconate Drugs 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 239000003292 glue Substances 0.000 description 1
- 229940029575 guanosine Drugs 0.000 description 1
- 238000005286 illumination Methods 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 230000000968 intestinal effect Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 230000033001 locomotion Effects 0.000 description 1
- 239000002075 main ingredient Substances 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000002503 metabolic effect Effects 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- 210000001724 microfibril Anatomy 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 239000000203 mixture Substances 0.000 description 1
- 229910017604 nitric acid Inorganic materials 0.000 description 1
- 210000000633 nuclear envelope Anatomy 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 230000000050 nutritive effect Effects 0.000 description 1
- 210000001672 ovary Anatomy 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 230000000452 restraining effect Effects 0.000 description 1
- 230000005070 ripening Effects 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 210000003491 skin Anatomy 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 238000002798 spectrophotometry method Methods 0.000 description 1
- 230000001954 sterilising effect Effects 0.000 description 1
- 238000004659 sterilization and disinfection Methods 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 230000002123 temporal effect Effects 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 210000003934 vacuole Anatomy 0.000 description 1
- 230000002792 vascular Effects 0.000 description 1
- 239000013598 vector Substances 0.000 description 1
- 229910052902 vermiculite Inorganic materials 0.000 description 1
- 235000019354 vermiculite Nutrition 0.000 description 1
- 239000010455 vermiculite Substances 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 229960003487 xylose Drugs 0.000 description 1
Images
Landscapes
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
本发明涉及棉花尿苷二磷酸葡萄糖脱氢酶基因UGD6、其编码蛋白及应用。棉花尿苷二磷酸葡萄糖脱氢酶具有如SEQ ID No.2所示的氨基酸序列或该序列经替换、缺失或添加一个或几个氨基酸且具有同等功能的由SEQ ID NO.2衍生的氨基酸序列。棉花尿苷二磷酸葡萄糖脱氢酶基因具有如SEQ ID No.1所示的核苷酸序列。通过研究该基因在纤维发育不同时期的表达模式及转化拟南芥,表明该基因在形成细胞壁纤维素和半纤维素前体物中起着重要作用,而这些物质又直接参与棉纤维的形成,因此该基因为棉纤维品质改良提供了新的候选基因,具有重大的应用价值。
Description
技术领域
本发明涉及基因工程领域,特别是涉及棉花尿苷二磷酸葡萄糖脱氢酶基因、其编码蛋白及应用。
背景技术
棉纤维是已知纤维素纯度最高的天然资源之一。棉纤维品质主要包括纤维长度、强度、马克隆值等性状,这些性状主要受本身基因型控制。我国棉纤维存在内在品质相对较差,纤维强度比美棉低等问题。因此,开展棉纤维发育的分子生物学及相关基因表达调控的研究,对于利用基因工程改良棉花纤维品质具有重要意义。
棉花纤维实质上是种皮毛,由位于子房内的胚珠外被上的表皮细胞发育分化而成。从形态上讲,棉花纤维细胞的发育过程是细胞超常伸长和细胞壁超常加厚的过程。棉花纤维细胞的形成一般可以分为四个相互有重叠的时期:纤维原始细胞分化突起、伸长期、次生壁加厚期、脱水成熟期。伸长期和次生壁加厚期是棉纤维品质形成的关键时期。初生细胞壁的合成是细胞伸长期的重要活动之一,与纤维长度直接相关。在伸长期结束之前,纤维细胞开始次生细胞壁的合成,主要是纤维素的淀积,纤维细胞次生壁的加厚决定纤维强力。
棉纤维细胞壁主要包括纤维素和木葡聚糖、木聚糖、果胶多糖等非纤维素成分。纤维素含量约占次生壁干重的95%;非纤维素物质则构成初生壁的主要成分。尿苷二磷酸葡萄糖(UDP-Glc)是纤维素合成的底物,UDP-Glc在纤维素合酶作用下合成纤维素。非纤维素多糖由不同的单糖聚合而成,只有当它们被激活形成核苷糖后,才能作为非纤维素多糖合成的底物。核苷糖的合成由一整套酶来完成,研究发现几乎所有的核苷糖都是以UDP-Glc和GDP-Man(鸟苷二磷酸甘露糖)为前体经过相关代谢酶催化合成的,因此,核苷糖转换相关的酶对棉纤维细胞壁纤维素和非纤维素的合成发挥着重要作用。
尿苷二磷酸葡萄糖醛酸(UDP-GlcA)是许多核苷糖合成中的关键前体物,据推测,大约50%的细胞壁物质来源于前体UDP-GlcA,它是尿苷二磷酸葡萄糖脱氢酶(UDP-glucose dehydrogenase,UGD)催化UDP-Glc形成的产物。因此,尿苷二磷酸葡萄糖脱氢酶是形成非纤维素多糖和果胶的关键酶。在植物上已有多篇有关UGD基因的克隆报道。UGD基因首先在大豆中克隆出来,随后,在拟南芥、甘蔗、玉米中分别克隆出此基因。研究表明,UGD在不同的物种和组织中,其表达水平有较大差异。在杨树中UGD主要在幼叶和未成熟的木质部中表达,在成熟韧皮部中几乎无表达。然而在拟南芥幼苗中主要在根部表达,而成熟的植株中主要在维管系统中表达。苎麻UGD主要在茎中表达,其次在韧皮部和叶中,根中表达最少。大豆UGD在主根尖和侧根中有高量表达,在主根、成熟叶中则只有微量表达。说明UGD在幼嫩的组织有非常高的表达,而在植物成熟组织中表达量很少,这说明此酶在形成细胞壁纤维素和半纤维素前体物的过程中起着非常重要的作用,UGD的表达将对细胞壁纤维素和非纤维素多糖含量产生影响,并进而影响到纤维素微纤丝和非纤维素交联。综上所述,UGD基因对于揭示棉纤维品质形成的内在机理有重要意义,在纤维品质遗传改良中具有良好的应用前景。UGD在棉纤维合成过程中起着重要作用,但至今在棉花中仍无深入研究。
发明内容
本发明目的是提供一种棉花尿苷二磷酸葡萄糖脱氢酶基因(UGD6)及其编码蛋白,以用于提高棉纤维品质。
本发明另一目的是提供含有上述基因的载体。
本发明又一目的是提供含有上述基因或载体的宿主细胞。
本发明再一目的是提供棉花尿苷二磷酸葡萄糖脱氢酶或棉花UGD6基因在提高棉纤维品质中的应用。
本发明所述棉花UGD6基因编码的蛋白,具有SEQ ID NO.2所示的氨基酸序列或该序列经替换一个或几个氨基酸形成的具有同等功能的氨基酸序列。
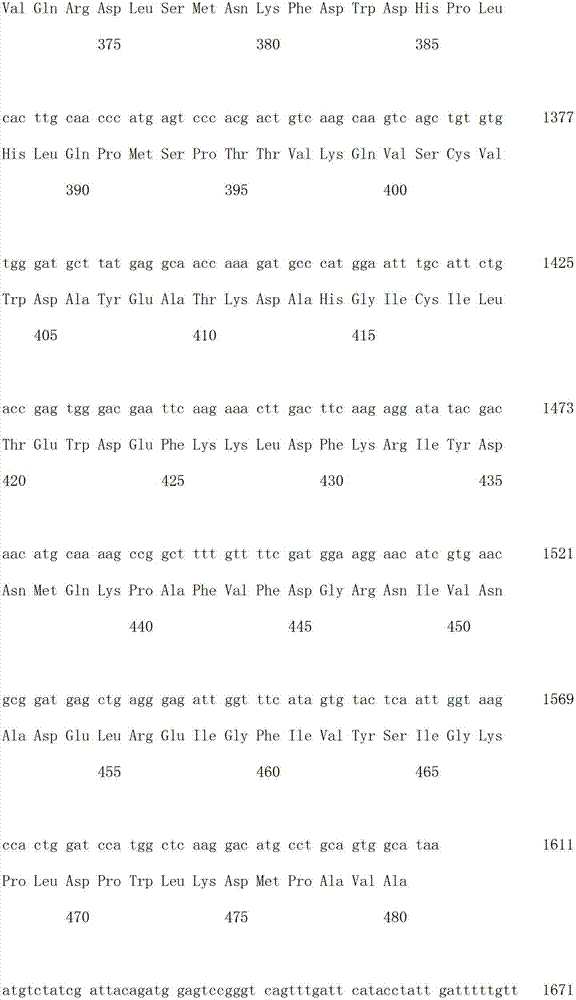
本发明所述棉花UGD6基因的核苷酸序列如SEQ ID No.1所示,全长1912bp,ORF长1443bp,5’UTR长168bp,3’UTR长301bp。
具体地,本发明基于拟南芥四条尿苷二磷酸葡萄糖脱氢酶(At1g26570,AT3g29360,At5g15490和At5g39320)的编码序列,利用BLASTN在美国国立生物技术信息中心网站(https://www.ncbi.nlm.nih.gov/)棉花的表达序列标签数据库中搜索相似性较高的EST序列,结合电子拼接、RT-PCR以及RACE技术克隆得到棉花尿苷二磷酸葡萄糖脱氢酶基因,命名为UGD6。
应当理解,考虑到密码子的简并性,例如可在其编码区,在不改变氨基酸序列的条件下,或在其非编码区在不影响蛋白表达的条件下,对编码上述蛋白的基因序列进行修改。因此,本发明还包含对编码上述蛋白的基因序列进行的替换、添加和/或缺失一个或多个核苷酸,具有与上述编码基因具有相同功能的核苷酸序列。本发明还包括基于所述基因的正义序列或反义序列,包括含有所述核苷酸序列或其片段的克隆载体或表达载体、含有所述载体的宿主细胞,利用所述宿主细胞制备棉花尿苷二磷酸葡萄糖脱氢酶的方法等。
所述该序列经替换一个或几个氨基酸形成的具有同等功能的氨基酸序列,是指该序列的活性功能域之外的位点进行有限氨基酸的保护替换所得的序列仍能保持原来的活性。本发明氨基酸序列的活性功能域为209~306位,在此位点外可替换一个或几个氨基酸得到具有同等功能的氨基酸序列。比如,SEQ ID NO.3所示为海岛棉UGD7基因的核苷酸序列,SEQ ID NO.4所示为UGD7基因编码的氨基酸序列,与UGD6氨基酸序列相比,第26、40、56、60、64、149、193、202、368位发生了9个氨基酸的替换,且具有同样的功能效果。
利用Real-time PCR检测UGD6基因在陆地棉“中棉所8号”和海岛棉“Pima90-53”棉纤维发育不同时期及幼苗根茎叶中的表达量,发现此基因在两个品种中表达量的变化趋势相似,在开花后20~30d的纤维中持续高效表达,30d之后呈下降趋势,说明它对棉纤维次生壁的起始和合成具有一定作用。15d幼苗中UGD6基因在“中棉所8号”和“Pima90-53”的幼苗根、下胚轴和叶的组织中,均有类似的表达量,表达量差异为:茎>根>叶。
将UGD6基因与原核表达载体pET-32a连接,转化大肠杆菌DH5α感受态细胞,获得含有重组质粒的大肠杆菌工程菌株。在IPTG诱导条件下,表达融合蛋白,并进行SDS-PAGE电泳分析。pET32a-UGD6重组质粒在大约68.4kDa的位置上出现特异条带,说明此基因可成功在原核细胞中表达蛋白。
将UGD6基因与融合表达载体pCamE∷GFP连接,转入大肠杆菌DH5α感受态细胞,获得含有重组质粒的大肠杆菌工程菌株。通过基因枪轰击洋葱表皮细胞。暗培养后在显微镜(蓝光激发)下观察,确定该基因编码的蛋白定位于细胞质膜系统。
采用农杆菌浸花法将S-UGD6(正义)及A-UGD6(反义)基因表达载体转入Columbia型拟南芥中,转S-UGD6和A-UGD6拟南芥中的UGD酶活性相应增高和降低。转A-UGD6拟南芥植株纤维素含量与野生型相比显著增高,而转S-UGD6拟南芥则显著下降。转A-UGD6基因拟南芥茎长和茎粗均显著小于野生型,茎秆强度显著变小,而转S-UGD拟南芥表型无明显变化。
提取野生型和转基因拟南芥RNA,进行Real-time PCR分析,发现UGD基因的导入和敲除,显著影响了拟南芥内源UGD基因的表达水平,而UGD作为UDP-葡萄糖代谢的关键酶,会对核苷糖合成乃至纤维素和果胶的合成产生影响。
本发明的有益效果:
(1)本发明提供的棉花UGD6基因为棉纤维品质改良基因工程提供了新的候选基因。
(2)本发明提供的棉花UGD6基因在纤维强力不同的棉种中,表达量变化趋势有差异,UGD6基因在棉纤维发育伸长期上调表达,其表达高峰在开花后20~30天,可用于初生壁的非纤维素多糖及次生壁纤维素合成研究。
(3)本发明提供的棉花UGD6基因与棉花纤维发育有重要关系,可以利用基因工程手段进一步转化棉花从而提高棉纤维品质。
附图说明
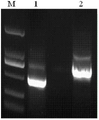
图1为本发明棉花UGD6基因的开放读码框(open reading frame,ORF)RT-PCR扩增结果,其中,M为DL5000Marker,1为UGD6 ORF扩增产物。
图2为本发明在NCBI上寻找棉花UGD6基因保守结构域的结果。
图3为本发明棉花UGD6基因5’RACE及3’RACE扩增产物,其中,M为DL2000Marker,1为5’RACE扩增产物,2为3’RACE扩增产物。
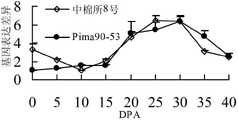
图4为本发明棉花UGD6基因在纤维发育不同时期的表达分析结果;横坐标DPA为开花后的不同天数,纵坐标为UGD6基因的表达量。
图5为本发明棉花UGD6基因在根茎叶中的表达分析结果。
图6为转棉花UGD6基因的E.coli BL21(DE3)诱导前和诱导后目的融合蛋白表达的SDS-PAGE检测结果,箭头所示为目的融合蛋白,其中,M:蛋白标准分子量,1:BL21(DE3)plys空菌株未经IPTG诱导的总蛋白,2:pET-32a(+)空载体未经IPTG诱导的总蛋白;3:UGD6-pET-32a(+)未经IPTG诱导的总蛋白;4:BL21(DE3)plys空菌株经IPTG诱导6小时后的总蛋白;5:pET-32a(+)空载体经IPTG诱导6小时后的总蛋白;6:UGD6-pET-32a(+)经IPTG诱导6小时后的总蛋白。
图7为本发明UGD6基因亚细胞定位的结果,其中,A:pCamE-GFP蓝光激发下的细胞;B:A的可见光下的细胞;C、D、F、G:pCamEUGD6∷GFP蓝光激发下的细胞;E:C和D可见光下的细胞。H:F和G的可见光下的细胞。
图8为本发明棉花UGD6基因真核表达载体构建示意图,其中,A:质粒pBI121结构;B:pBI121-SUGD6结构;C:pBI121-AUGD6结构。
图9为本发明棉花UGD6基因转化拟南芥的幼苗,其中,A:转S-UGD6拟南芥;B:转A-UGD6拟南芥。
图10为生长10天的转A-UGD6 T3代与野生型拟南芥植株个体比较,其中,WT:野生型拟南芥对照,AU:转A-UGD6拟南芥植株。
图11为生长10天的转A-UGD6基因T3代与野生型拟南芥的根长比较,其中,WT:野生型拟南芥对照,AU:转A-UGD6拟南芥植株。
图12为生长35天的转A-UGD6基因T3代与野生型拟南芥的表型比较,其中,WT:野生型拟南芥对照,AU:转A-UGD6拟南芥植株。
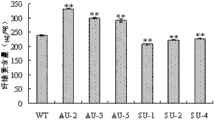
图13为转S-UGD6和S-UGD6拟南芥株系与野生型纤维素含量的比较图,其中,WT:野生型拟南芥对照,SU-:转S-UGD6拟南芥T3代不同株系,AU-:转A-UGD6拟南芥T3代不同株系。
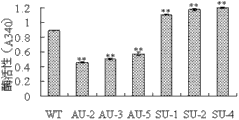
图14为转S-UGD6和A-UGD6拟南芥株系与野生型UGD酶活性的比较图,其中,WT:野生型拟南芥对照,SU-:转S-UGD6拟南芥T3代不同株系,AU-:转A-UGD6拟南芥T3代不同株系。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。
本发明的载体,菌株,质粒均可市售获得。
实施例1棉花UGD6基因的克隆
(1)首先利用拟南芥四条尿苷二磷酸葡萄糖醛酸脱羧酶(At1g26570,AT3g29360,At5g15490和At5g39320)的编码序列,利用tBLASTN在NCBI棉花表达序列标签数据库中搜索相似性较高的EST序列,利用DNASTAR软件对检索获得的EST序列进行电子拼接,获得具有完整开放阅读框的拼接序列UGD6。
(2)依据拼接序列设计特异性引物,同时在上、下游引物分别引入KpnI和SalI酶切位点(下划线部分)。UGD6基因的上、下游引物分别为:
上游引物:5’-GGTACCATGGTGAAGATCTGTTGC-3’
下游引物:5’-GTCGACTTATGCCACTGCAGGC-3’
(3)利用设计的特异性引物分别以陆地棉“中棉所8号”和海岛棉“Pima90-53”(购自中国农业科学院棉花研究所国家棉花种质资源中期库)开花后20天的棉纤维cDNA为模板进行PCR扩增,获得UGD6基因目的条带(图1);
(4)将回收得到的UGD6基因的PCR产物与pGM-T连接,采用天根生化科技(北京)有限公司的pGM-T克隆试剂盒克隆目的片段,连接产物采用热激转化法转入大肠杆菌TOP10感受态细胞(购自天根生化科技有限公司),并筛选阳性克隆,经上海生物工程有限公司测序,所测序列与预期结果一致,且两棉种间基因序列无差异。UGD6基因ORF序列见SEQ ID NO.1(长1443bp),对应氨基酸序列为SEQ IDNO.2。
实施例2棉花UGD6基因编码蛋白的相似性分析和保守结构域分析
利用DNAMAN对棉花UGD6基因、棉花GhUGD1~UGD5以及4个拟南芥UDP-葡萄糖脱氢酶基因编码蛋白的氨基酸序列进行相似性分析,结果显示棉花UGD6基因与棉花UDP-葡萄糖脱氢酶基因编码蛋白的氨基酸序列相似性分布在89.0%~99.6%之间,与拟南芥UDP-葡萄糖脱氢酶基因编码蛋白的氨基酸序列相似性分布在84.4%~92.7%之间,说明棉花UGD6基因属于UDP-葡萄糖酸脱氢酶基因家族。
利用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)CDD数据库进行在线保守域BLAST分析,发现了UDP-葡萄糖/GDP-甘露糖脱氢酶基因家族保守区(图2),说明本发明的UGD6基因属于UDP-葡萄糖脱氢酶基因家族。
实施例3棉花UGD6基因5’和3’非翻译区(UTR)序列的克隆
以棉花UGD6基因ORF序列为基础,按Clontech RACE试剂盒要求,分别设计5’RACE及3’RACE引物,具体引物序列如下:
5’RACE引物:
GSP:5'-CAGCTCCGAGCCCCCGAGTCTTGGT-3'
NGSP:5'-TCACTGTTCCAGGCAGCGATCCTAG-3'
3’RACE引物:
GSP:5'-ATATATGACCCGCAGGTGACCGAAG-3'
NGSP:5'-GCAACCCATGAGTCCCACGACTGTC-3'
以陆地棉“中棉所8号”和海岛棉“Pima90-53”开花后20天的棉纤维cDNA为材料,按Clontech RACE试剂盒操作步骤完成5’端及3’端非翻译区序列扩增,获得目的条带,UGD6 5’RACE长420bp,3’RACE长530bp(图3)。对PCR产物进行测序,最后分析得UGD6 5’UTR长168bp,3’UTR长301bp。将测序结果与UGD6基因开放阅读框拼接,获得全长cDNA序列,且两棉种间基因序列无差异(SEQ ID NO.1)。
实施例4棉花UGD6基因的时空表达模式分析
使用天根生化科技(北京)有限公司的RNAplant提取试剂盒提取“中棉所8号”和海岛棉“Pima90-53”开花当天胚珠以及开花后5、10、15、20、25、30、35、40天(days post anthesis,DPA)的纤维RNA。棉花根、茎和叶RNA的提取使用北京奥莱博生物技术公司的植物总RNA提取试剂盒。
采用大连宝生物公司的RT reagent Kit With gDNAEraser(Perfect Real Time)试剂盒合成以上9个时期的单链cDNA,浓度用Beckman DU800分光光度计测定。对棉花UGD6基因在棉纤维发育不同时期的表达进行Real-time PCR分析。内参选用EF1α,其引物序列如下:
EF1αF:5′-GCTGAGATGAACAAGAGGTCATTC-3′
EF1αR:5′-GGAATCAATAATCAAAACAGCACAG-3′
Real-timePCR结果(图4)显示,棉花UGD6基因在20~30DPA的纤维中持续高效表达,30d之后呈下降趋势,说明UGD6对棉纤维次生壁的起始和合成具有一定作用;15d幼苗中UGD6基因在棉花根茎叶中均有表达,在茎中表达量最高,根中次之,叶中最低(图5)。
实施例5UGD6基因原核表达载体的构建及诱导表达
(1)原核表达载体的构建和转化子的获得
利用KpnI和SalI分别对实施例1中克隆得到的UGD6基因的ORF和原核表达载体pET-32a(+)质粒(购自Novagen公司)进行双酶切并回收目的片段。
将表达载体与目的基因片段进行连接,转化大肠杆菌DH5α,经过PCR和酶切检测,筛选含重组质粒的阳性克隆,利用热激法将含有阳性克隆的质粒转入BL21(DE3)菌株(购自天根生化科技有限公司)中,并进一步挑取阳性克隆,经质粒提取、酶切检测获得含有重组质粒pET-32a(+)-UGD6的大肠杆菌工程菌株。
(2)重组蛋白的诱导表达与鉴定
将含有以上各类型的重组质粒和含有空质粒(对照)的BL21(DE3)菌株在28℃,IPTG终浓度为1.0mmol/L的条件下诱导6h后,进行SDS-PAGE垂直式凝胶电泳,其中分离胶为12%,浓缩胶为5%。
结果如图6所示,经IPTG诱导的含有去掉跨膜区重组质粒pET-32a(+)-UGD6的大肠杆菌BL21(DE3)与含空质粒对照相比,在68.4KDa出现一条表达量明显增高的差异带。生物信息学预测可表达的UGD6目的融合蛋白包括UGD6蛋白(约48KDa)和pET-32a标签蛋白(约20.4KDa),分子量大约为68.4KDa。因此,该差异带是pET-32a(+)-UGD6基因在大肠杆菌BL21(DE3)中表达的目的融合蛋白。
实施例6UGD6融合表达载体构建及融合蛋白的亚细胞定位
(1)融合表达载体的构建
设计用于构建融合表达载体的引物。在UGD6基因的ORF两端添加酶切位点SalI和KpnⅠ(下划线部分),引物序列如下:
PUF:5’-GTCGACATGGTGAAGATCTGTTGC-3’
PUR:5’-GGTACCTGCCACTGCAGGCAT-3’
以该引物进行PCR反应,并回收目的片段,与pGM-T载体连接,热击法转化大肠杆菌感受态细胞DH5α,挑取阳性克隆,提取质粒进行酶切检测、测序,得到中间载体pGM-UGD6。
将中间载体pGM-UGD6和表达载体pCamE∷GFP分别用SalI和KpnⅠ双酶切,电泳检测酶切产物,回收目的片段进行连接,热击法转化大肠杆菌感受态细胞DH5α,挑取阳性克隆,进行质粒提取,酶切检测和测序,获得融合表达载体pCamUGD6∷GFP。
(2)基因枪介导的亚细胞定位
在倒好的MS培养基平板上铺一层灭菌滤纸,将洋葱内表皮平铺在平板的中央,通过基因枪轰击转化融合表达载体。培养室暗培养1-3d后在显微镜(蓝光激发)下观察并照相。
显微镜下观察基因枪轰击后的洋葱表皮细胞,发现不含外源基因的载体pCamE-GFP定位在细胞核和细胞膜上(图7A-7B),与前人定位结果一致。PCamE-UGD6∷GFP绿色荧光出现在细胞膜、核膜、液泡膜以及内质网等结构上,另外观察到了明显的膜泡运动,说明UGD6蛋白定位在细胞膜系统上(图7C-H)。
实施例7棉花UGD6基因真核表达载体构建和拟南芥遗传转化
(1)真核表达载体的构建
真核表达载体构建示意图见图8。设计用于构建植物表达载体的引物,在UGD6基因的ORF两端添加酶切位点SacI和XbaⅠ(下划线部分),序列如下:
SUF:5’-TCTAGAATGGTGAAGATCTGTTGC-3’
SUR:5’-GAGCTCTTATGCCACTGCAGG-3’
AUF:5’-GAGCTCATGGTGAAGATCTGTTGC-3’
AUR:5’-TCTAGATTATGCCACTGCAGGCAT-3’
以该引物进行PCR反应,并回收目的片段,与pGM-T载体连接,热击法转化大肠杆菌感受态细胞DH5α(天根生化科技有限公司产品),挑取阳性克隆提取质粒,经酶切检测并测序,得到中间载体pGM-SUGD6和pGM-AUGD6。
将中间载体和真核表达载体pBI121p35S分别用SacI和XbaⅠ双酶切,电泳酶切产物,回收目的片段,然后进行连接,热击法转化大肠杆菌感受态细胞DH5α,挑取阳性克隆,进行质粒提取,经酶切检测和测序,获得真核表达载体pBI121p35S::SUGD6和pBI121p35S::AUGD6。
(2)拟南芥遗传转化及UGD6的功能分析
1)拟南芥遗传转化:将质粒pBI121p35S::SUGD6和pBI121p35S::AUGD6转化农杆菌菌株GV3101感受态细胞,采用农杆菌浸花法将S-UGD6和A-UGD6载体转化Columbia型拟南芥。将转化拟南芥植株花序浸泡于含有目的基因的转化介质,几分钟后取出,保湿暗培养24h后浇足营养液,恢复正常光照培养,待拟南芥种子成熟,收取种子进行干燥并春化后,种植于MS筛选培养基上(卡那霉素浓度为100mg/L),筛选得到转基因阳性苗(图9),移栽于蛭石中培养,并进行PCR检测进一步确定转基因阳性植株。之后,再经2轮的种植、筛选直至获得T3代阳性拟南芥转基因株系,进行表型和细胞壁成分分析。
2)表型分析:转基因拟南芥与野生型拟南芥相比,表型发生显著变化,转反义UGD6拟南芥发育迟缓,根长显著变短(图10,图11),抽薹时间较野生型晚(图12),茎长和茎粗均显著短于野生型,茎秆强度亦显著变小(表1)。WT为野生型拟南芥对照,AU-为转A-UGD6拟南芥T3代不同株系。而转正义UGD6拟南芥表型无明显变化。
表1转A-UGD6拟南芥株系与野生型表型指标的比较
| 株系 | WT | AU-2 | AU-3 | AU-5 |
| 茎长/cm | 26.78±1.07 | 14.98±0.80** | 17.43±0.67** | 18.25±1.29** |
| 茎粗/mm | 1.05±0.05 | 0.73±0.08** | 0.77±0.07** | 0.77±0.08** |
| 茎秆强度/N | 12.1±0.92 | 11.3±0.81** | 9.9±0.68** | 9.5±0.63** |
3)细胞壁成分分析:为进一步确定UGD6基因在细胞壁多糖转化中的功能,提取转基因和野生型拟南芥的细胞壁,采用蒽酮比色法测定拟南芥细胞壁中纤维素的含量。
具体步骤为:采用乙酸、硝酸和水(8∶1∶2)的混合溶液沸水浴提取细胞壁中纤维素;ddH2O和丙酮洗涤后真空抽干;用72%的硫酸重新溶解纤维素;加入蒽酮试剂并充分反应后测定A620,对比标准曲线既得细胞壁纤维素含量。
结果显示转反义UGD6拟南芥株系(AU)纤维素含量与野生型相比显著增高,而转正义UGD6拟南芥(SU)则显著下降(图13)。
4)UGD酶活性分析:提取转基因及野生型拟南芥的UGD酶,按株系取生长六周的拟南芥,提取蛋白酶;材料称重,加入两倍体积的蛋白提取液(50mM Tris-HCl(pH7.5);2mM EDTA,用前加5mMDTT),充分研磨;4℃,11,200g离心2min,弃掉细胞壁碎片,上清再次离心2min,即为粗酶液。UGD酶的纯化使用上海生物工程有限公司的重力型Sephadex脱盐柱(BSP090)进行蛋白的脱盐纯化。纯化步骤严格按照说明书进行。
转反义UGD6拟南芥株系酶活性与野生型相比显著降低,而转正义UGD6拟南芥植株则显著增高(图14)。
UGD6的功能分析结果表明:UGD6基因的过量表达显著提高了拟南芥UGD酶活性,导致大量的尿苷二磷酸葡萄糖(UDP-Glc)被消耗,而细胞壁主要组分的纤维素是由葡聚糖结合所形成,UGD的过量表达造成了纤维素含量的降低。
UGD基因的抑制表达,一方面造成UDP-Glc的大量累积,引起纤维素含量的增高,另一方面也直接导致了由UGD催化葡萄糖的产物尿苷二磷酸葡萄糖醛酸(UDP-GlcA)含量的减少,在半纤维素和果胶中大部分糖(树胶醛醣、芹菜糖、半乳糖醛酸和木糖等)直接或间接来源于UDP-GlcA,打破了原来植物细胞壁纤维素和非纤维素多糖含量的平衡,影响了细胞壁结构,从而导致一系列的表型变化。
5)转基因拟南芥内源UGD基因的表达分析:
提取野生型和转基因拟南芥的RNA,进行Real-time PCR反应,结果发现外源基因S-UGD6和A-UGD6均在拟南芥中高效转录,无论是转S-UGD6或A-UGD6基因,拟南芥内源UGD基因的转录水平都大幅降低。
结果表明UGD6基因的过量表达对拟南芥内源UGD基因的表达产生了共抑制作用,但是由于35S启动子的强启动能力,外源UGD6基因得到了很好的表达,内源和外源基因的共同作用,并未引起植株表型的明显变化。UGD6基因的抑制表达,则打破了拟南芥中UGD表达的平衡,造成一系列的表型差异。
因此,UGD6基因的过量和低量表达,导致棉纤维细胞壁中纤维素含量大幅变动,同时可能引起非纤维素多糖物质即木葡聚糖、木聚糖或果胶多糖含量的变化,形成类似于拟南芥的表型特征变化,进而影响到棉纤维长度和强度,影响棉纤维品质。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
Claims (6)
1.一种棉花尿苷二磷酸葡萄糖脱氢酶,其特征在于,具有如SEQID NO.2所示的氨基酸序列或该序列经替换、缺失或添加一个或几个氨基酸且具有同等功能的由SEQ ID NO.2衍生的氨基酸序列。
2.编码权利要求1所述棉花尿苷二磷酸葡萄糖脱氢酶的基因。
3.根据权利要求2所述基因,其特征在于,具有如SEQ ID No.1所示的核苷酸序列。
4.含有权利要求2所述基因的载体。
5.含有权利要求2或3所述基因或权利要求4所述载体的宿主细胞。
6.权利要求1所述棉花尿苷二磷酸葡萄糖脱氢酶或权利要求2或3所述基因在提高棉纤维品质中的应用。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN2012102457652A CN102787105B (zh) | 2012-07-16 | 2012-07-16 | 棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 |
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN2012102457652A CN102787105B (zh) | 2012-07-16 | 2012-07-16 | 棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| CN102787105A true CN102787105A (zh) | 2012-11-21 |
| CN102787105B CN102787105B (zh) | 2013-11-27 |
Family
ID=47152696
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN2012102457652A Expired - Fee Related CN102787105B (zh) | 2012-07-16 | 2012-07-16 | 棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 |
Country Status (1)
| Country | Link |
|---|---|
| CN (1) | CN102787105B (zh) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN103352044A (zh) * | 2013-07-05 | 2013-10-16 | 安徽农业大学 | 一种Spot-9-UDP-葡萄糖脱氢酶基因片段的构建获得方法 |
| CN111518825A (zh) * | 2020-04-30 | 2020-08-11 | 浙江工业大学 | 一种多基因组合表达制备蛹虫草多糖的方法 |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2004065571A2 (en) * | 2003-01-23 | 2004-08-05 | Texas Tech University | Dna molecules encoding a cotton chitinase and promoter |
| CN102154258A (zh) * | 2010-12-17 | 2011-08-17 | 河南省农业科学院 | 加速植物生长及增加纤维素量和/或产量的方法 |
-
2012
- 2012-07-16 CN CN2012102457652A patent/CN102787105B/zh not_active Expired - Fee Related
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2004065571A2 (en) * | 2003-01-23 | 2004-08-05 | Texas Tech University | Dna molecules encoding a cotton chitinase and promoter |
| CN102154258A (zh) * | 2010-12-17 | 2011-08-17 | 河南省农业科学院 | 加速植物生长及增加纤维素量和/或产量的方法 |
Non-Patent Citations (1)
| Title |
|---|
| PANG,C.等: "ACJ11712.1 UDP-D-glucose dehydrogenase [Gossypium hirsutum]", 《GENBANK》, 1 December 2009 (2009-12-01) * |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN103352044A (zh) * | 2013-07-05 | 2013-10-16 | 安徽农业大学 | 一种Spot-9-UDP-葡萄糖脱氢酶基因片段的构建获得方法 |
| CN111518825A (zh) * | 2020-04-30 | 2020-08-11 | 浙江工业大学 | 一种多基因组合表达制备蛹虫草多糖的方法 |
Also Published As
| Publication number | Publication date |
|---|---|
| CN102787105B (zh) | 2013-11-27 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US10597669B2 (en) | Method of reducing acetylation in plants to improve biofuel production | |
| AU2007335706A2 (en) | Polynucleotides and polypeptides involved in plant fiber development and methods of using same | |
| CN104480118A (zh) | 花生lrr-rlk基因及在烟草抗青枯病中的应用 | |
| Day et al. | ESTs from the fibre-bearing stem tissues of flax (Linum usitatissimum L.): expression analyses of sequences related to cell wall development | |
| CN102766610B (zh) | 一种植物抗旱相关蛋白PvSnRK2.3及其编码基因和应用 | |
| CN101280007A (zh) | 一种与植物耐冷性相关的蛋白及其编码基因与应用 | |
| CN103103169B (zh) | 一种植物抗旱相关蛋白EeSnRK2.4及其编码基因和应用 | |
| CN103497940B (zh) | 一种植物抗旱相关蛋白TaSnRK2.6及其编码基因和应用 | |
| CN102787105B (zh) | 棉花尿苷二磷酸葡萄糖脱氢酶基因ugd6、其编码蛋白及应用 | |
| CN101712718B (zh) | 一种与植物抗旱相关的蛋白及其编码基因与应用 | |
| CN103172716A (zh) | 植物抗热基因及其应用 | |
| CN101704882B (zh) | 一种植物黄矮病抗性相关蛋白及其编码基因和应用 | |
| AU2011239486B2 (en) | Plants with altered cell wall biosynthesis and methods of use | |
| CN102851300B (zh) | 棉花黄萎病抗病相关基因GbVdr3及其应用 | |
| CN102344927B (zh) | 水稻茎秆机械强度和粒重控制基因bc14及其应用 | |
| CN105296443A (zh) | 一种植物抗旱、耐盐相关蛋白EeSAPK7及其编码基因和应用 | |
| CN104975028A (zh) | 一种芒草wrky转录因子在提高植物纤维生物量中的应用 | |
| CN101578370A (zh) | 编码c3hc4家族植物蛋白质的核酸分子和改变植物纤维素和木素含量的方法 | |
| CN102351950A (zh) | 水稻抗旱相关转录因子基因OsWTF1及其编码蛋白与应用 | |
| US20170107542A1 (en) | Transgenic plants having altered expression of a xylan xylosyltransferase and methods of using same | |
| CN102260661B (zh) | 棉花GhUXS3基因、其编码蛋白及应用 | |
| Chen et al. | Identification of the CAD gene from Eucalyptus urophylla GLU4 and its functional analysis in transgenic tobacco | |
| CN101280008B (zh) | 一种与植物耐冷性相关的蛋白及其编码基因与应用 | |
| CN114480479B (zh) | 一种与植物抗病性相关的基因OsERF65的应用 | |
| US20140331363A1 (en) | Plants with altered glucuronoxylan methyl transferase activity and methods of use |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| C14 | Grant of patent or utility model | ||
| GR01 | Patent grant | ||
| CF01 | Termination of patent right due to non-payment of annual fee | ||
| CF01 | Termination of patent right due to non-payment of annual fee |
Granted publication date: 20131127 Termination date: 20160716 |