Listagem de Sequência
[001] O presente pedido está sendo depositado juntamente comuma Listagem de Sequências em formato eletrônico. A listagem de sequência é fornecida como um arquivo intitulado BIOL025- 4WOSEQ_ST25.txt criado 28 de abril de 2015, que tem um tamanho de 0,98 MB. As informações no formato eletrônico da listagem de sequência são incorporadas a este documento por referência em sua totalidade.
CAMPO DA INVENÇÃO
[002] São fornecidos neste documento métodos, compostos ecomposições para reduzir a expressão do mRNA de angiopoietina do tipo 3 (ANGPTL3) e da proteína em um animal. Além disso, são previstos aqui métodos, compostos e composições com um inibidor de ANGPTL3 para redução de doenças ou condições médicas relacionadas a ANGPTL3 em um animal. Tais métodos, compostos e composições são úteis, por exemplo, para tratar, prevenir, retardar ou aliviar uma ou mais dentre: doenças cardiovasculares ou síndromes metabólicas, ou sintomas destas, em um animal.
FUNDAMENTOS DA INVENÇÃO
[003] O diabetes e a obesidade (algumas vezes coletivamentereferidas como "diabesidade") estão inter-relacionados no que a obesidade é conhecida por exacerbar a patologia do diabetes e mais de 60% dos diabéticos são obesos. A maior parte da obesidade humana está associada à resistência à insulina e à resistência à leptina. De fato, foi sugerido que a obesidade pode ter um maior impacto sobre a ação da insulina do que o diabetes em si (Sindelka et al., Physiol Res., 2002, 51, 85-91). Adicionalmente, vários compostos no mercado para o tratamento do diabetes são conhecidos por induzir o ganho de peso, um efeito colateral bem indesejável para o tratamento desta doença.
[004] A doença cardiovascular também está inter-relacionada àobesidade e diabetes. A doença cardiovascular inclui uma ampla variedade de etiologias e tem uma variedade igualmente ampla de agentes causadores e protagonistas inter-relacionados. Muitos agentes causadores contribuem para os sintomas, como níveis elevados de plasma de colesterol, incluindo colesterol de lipoproteína de baixa densidade (não-HDL-C), assim como outros distúrbios relacionados a lipídios. Esses distúrbios relacionados a lipídios, geralmente denominados dislipidemia, incluem hiperlipidemia, hipercolesterolemia e hipertrigliceridemia, entre outras indicações. O colesterol não-HDL elevado está associado a aterogênese e suas sequelas, incluindo doenças cardiovasculares, tais como aterosclerose, doença arterial coronariana, infarto do miocárdio, acidente vascular cerebral isquêmico e outras formas de doença cardíaca. Estas estão entre os tipos mais prevalentes de doença nos países industrializados. Com efeito, estima- se que 12 milhões de pessoas nos Estados Unidos sofram de doença arterial coronariana e cerca de 36 milhões necessitem de tratamento para elevados níveis de colesterol.
[005] Evidências epidemiológicas e experimentais demonstraramque níveis elevados de triglicerídeos em circulação (TG) podem contribuir para a doença cardiovascular e uma miríade de distúrbios metabólicos (Valdivielso et al., 2009, Atherosclerosis Zhang et al., 2008, Circ Res. 1;102(2):250-6). Os TG derivados de fontes exógenas ou endógenas são incorporados e secretados em quilomicrons a partir do intestino ou em lipoproteínas de densidade muito baixa (VLDL) a partir do fígado. Uma vez em circulação, os TG são hidrolisados pela lipase de lipoproteína (LPL) e os ácidos graxos livres resultantes podem então ser absorvidos pelos tecidos locais e usados como fonte de energia. Devido ao efeito profundo que a LpL tem sobre os TG do plasma e o metabolismo em geral, a descoberta e o desenvolvimento de compostos que afetam a atividade da LpL são de grande interesse.
[006] A síndrome metabólica é uma combinação de distúrbiosmédicos que aumentam o risco de se desenvolver doença cardiovascular e diabetes. Os sintomas, incluindo alta pressão sanguínea, triglicerídeos altos, HDL diminuído e obesidade, tendem a aparecer juntos em alguns indivíduos. Eles afetam um amplo número de pessoas de maneira agregada. Em alguns estudos, a prevalência nos EUA é calculada como sendo de até 25% da população. A síndrome metabólica é conhecida sob vários outros nomes, tais como síndrome X (metabólica), síndrome de resistência à insulina, síndrome de Reaven ou CHAOS. Com a alta prevalência de distúrbios cardiovasculares e distúrbios metabólicas continua havendo uma necessidade de melhores abordagens para tratar essas condições médicas.
[007] As angiopoietinas são uma família de fatores de crescimentosegregados. Juntamente com os seus respectivos receptores específicos de endotélio, as angiopoietinas desempenham papéis importantes na angiogênese. Um membro da família, angiopoietina do tipo 3 (também conhecida como a proteína do tipo angiopoietina 3, ANGPT5, ANGPTL3 ou angiopoietina 5), é predominantemente expresso no fígado e, acredita-se, desempenha um papel na regulação do metabolismo lipídico (Kaplan et al., J. Lipid Res., 2003, 44, 136-143). As varreduras de associação de genoma amplo (GWAS) que analisam o genoma quanto às variantes comuns associadas com as concentrações de plasma de HDL, LDL e triglicerídeos encontraram uma associação entre os triglicerídeos e os polimorfismos de nucleotídeo simples (SNPs) próxima à ANGPTL3 (Willer et al., Nature Genetics, 2008, 40(2):161-169). Os indivíduos com mutações de perda de função de ANGPTL3 homozigótica presentes com baixos níveis de lipídios de plasma ateorogênico e lipoproteínas, como colesterol total (TC) e TG), colesterol de lipoproteína de baixa densidade (LDL-C), apoliproteína B (apoB), não-HDL-C, bem como HDL-C (Romeo et al. 2009, J Clin Invest, 119(1):70-79; Musunuru et al. 2010 N Engl J Med, 363:2220-2227; Martin-Campos et al. 2012, Clin Chim Acta, 413:552555; Minicocci et al. 2012, J Clin Endocrinol Metab, 97:e1266-1275; Noto et al. 2012, Arterioscler Thromb Vasc Biol, 32:805-809; Pisciotta et al. 2012, Circulation Cardiovasc Genet, 5:42-50). Esse fenótipo clínico foi denominado hipolipidemia combinada familiar (FHBL2). Apesar redução da secreção de VLDL, os indivíduos com FHBL2 não apresentam um conteúdo de gordura hepática aumentado. Eles também parecem ter níveis de insulina e glicose de plasma mais baixos, tanto o diabetes quanto a doença cardiovascular parecem estar ausentes desses indivíduos. Nenhum fenótipo clínico adverso foi relatado até o momento (Minicocci et al. 2013, J of Lipid Research, 54:3481-3490). Mostrou-se que a redução de ANGPTL3 leva a uma redução nos níveis de TG, colesterol e LDL em modelos animais (US número de série 13 / 520,997; Publicação de PCT WO 2011/085271). Os camundongos deficientes na ANGPTL3 têm níveis de colesterol e triglicerídeos (TG) de plasma muito baixos, enquanto que a superexpressão produz efeitos contrários (Koishi et al. 2002; Koster 2005; Fujimoto 2006). Por conseguinte, a função potencial da ANGPTL3 no metabolismo lipídico faz dela um alvo atraente para interveção terapêutica.
[008] Até à data, as estratégias terapêuticas para tratar a doençacardiometabólica direcionando diretamente níveis de ANGPTL3 foram limitadas. Os fragmentos polipeptídicos de ANGPTL3 (U.S. número de série 12 / 128,545), os anticorpos anti-ANGPTL3 (U.S. número de série 12/001012) e os inibidores de ácido nucleico da ANGPTL3, incluindo oligonucleotídeos antisense (U.S. número de série 13 / 520,997; Publicação de PCT WO 2011/085271; incorporados aqui por referência em sua totalidade) foram sugeridos ou desenvolvidos anteriormente, mas nenhum dos compostos diretamente direcionado à ANGPTL3 foi aprovado para tratar doenças cardiometabólicas. Assim, há uma necessidade não correspondida de compostos toleráveis e com maior potência para inibir ANGPTL3. A invenção aqui descrita relaciona-se com a descoberta de novos inibidores, altamente potentes de expressão de ANGPTL3 e sua utilização em tratamentos.
Sumário da invenção
[009] São proporcionados aqui composições e métodos para amodulação da expressão de mRNA de ANGPTL3 e proteína. Em certas modalidades, a composição é um inibidor específico de ANGPTL3. Em certas modalidades, o inibidor específico de ANGPTL3 diminui a expressão da proteína e do mRNA de ANGPTL3.
[0010] Em certas modalidades, a composição é um inibidorespecífico de ANGPTL3. Em certas modalidades, o inibidor específico de ANGPTL3 é um ácido nucleico. Em certas modalidades, o ácido nucleico é um composto antisense. Em certas modalidades, o composto antisense é um oligonucleotídeo modificado. Em certas modalidades, o composto antisense é um oligonucleotídeo modificado com um grupo de conjugados acoplado.
[0011] Em certas modalidades, o inibidor específico de ANGPTL3 éum oligonucleotídeo modificado com um grupo de conjugados, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e tem uma sequência de nucleobase compreendendo pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19, ou 20 nucleobases contíguas da sequência de nucleobase da SEQ ID NO: 77.
[0012] Em certas modalidades, o inibidor específico de ANGPTL3 é um oligonucleotídeo modificado com um grupo de conjugado, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases compreendendo uma porção de pelo menos 8 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases 1140-1159 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1.
[0013] Em certas modalidades, o inibidor específico de ANGPTL3 éum oligonucleotídeo modificado com um grupo de conjugado, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases compreendendo uma porção de pelo menos 8 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases 9715-9734 of SEQ ID NO: 2, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 2
[0014] Em certas modalidades, o inibidor específico de ANGPTL3 éum oligonucleotídeo modificado por um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas da SEQ ID NO:77, onde o oligonucleotídeo modificado compreende: (a) um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; (b) um segmento de asa 5' consistindo em cinco nucleosídeos ligados; (c) um segmento de asa 3' consistindo em cinco nucleosídeos ligados; e em que o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3', em que cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila, em que cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5- metilcitosina.
[0015] Em certas modalidades, o inibidor específico de ANGPTL3 é um oligonucleotídeo modificado por um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas da SEQ ID NO:77, onde o oligonucleotídeo modificado consiste em: (a) um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; (b) um segmento de asa 5' consistindo em cinco nucleosídeos ligados; (c) um segmento de asa 3' consistindo em cinco nucleosídeos ligados; e em que o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3', em que cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila, em que cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5- metilcitosina.
[0016] Em determinadas modalidades, a presente divulgaçãofornece compostos antisense conjugados. Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados, compreendendo o oligonucleotídeo antisense complementar a um transcrito de ácido nucleico. Em determinadas modalidades, a presente divulgação fornece métodos que compreendem o contato de uma célula com um composto antisense conjugado compreendendo um oligonu- cleotídeo antisense complementar a um transcrito de ácido nucleico. Em determinadas modalidades, a presente divulgação fornece métodos que compreendem o contato de uma célula com um composto antisense conjugado compreendendo um oligonucleotídeo antisense e reduzindo a quantidade ou a atividade de um transcrito de ácido nucleico em uma célula.
[0017] O receptor da asialoglicoproteína (ASGP-R) foi descritoanteriormente. Veja por exemplo,Park et al., PNAS vol. 102, No. 47, pp 17125-17129 (2005). Esses receptores são expressos em células do fígado, particularmente hepatócitos. Além disso, verificou-se que compostos compreendendo aglomerados de três ligantes de N- acetilgalactosamina (GalNAc) são capazes de ligação ao ASGP-R, resultando em absorção do composto para a célula. Ver, por exemplo, Khorev et al., Bioorganic and Medicinal Chemistry, 16, 9, pp 5216-5231 (maio de 2008). Nesse sentido, os conjugados compreendendo esses agrupamentos de GalNAc têm sido utilizado para facilitar a absorção de certos compostos nas células do fígado, especificamente nos hepatócitos. Por exemplo, foi mostrado que certos conjugados contendo GalNAc aumentam a atividade de compostos de siRNA duplex nas células do fígado in vivo. Em tais casos, o conjugado contendo GalNAc está normalmente ligado à fita sense do siRNA duplex. Uma vez que a fita sense é descartada antes que a fita antisense se hibridize, enfim, no ácido nucleico alvo, há pouca preocupação de que o conjugado interfira na atividade. Normalmente, o conjugado é ligado à extremidade 3' da fita sense do siRNA. Ver, por exemplo, a Patente U.S. 8.106.022. Certos grupos de conjugados descritos neste documento são mais ativos e/ou mais fáceis de sintetizar do que os grupos de conjugados descritos anteriormente.
[0018] Em determinadas modalidades da presente invenção, osconjugados são ligados aos compostos antisense de fita única, incluindo, mas não se limitando a compostos antisense baseados em RNase H e compostos antisense que alteram o splicing de um ácido nucleico alvo de pré-mRNA. Em tais modalidades, o conjugado deve permanecer ligado ao composto antisense tempo suficiente para fornecer o benefício (absorção melhorada nas células) mas em seguida deve ser clivado, ou de outra forma para não interferir nas etapas subsequentes necessárias para a atividade, tais como a hibridização de um ácido nucleico alvo e a interação com a RNase H ou com enzimas associadas ao splicing ou à modulação por splice. Esse equilíbrio de propriedades é mais importante na configuração dos compostos antisense de fita única do que nos compostos de siRNA, onde o conjugado pode simplesmente estar ligado à fita sense. Divulgados neste documento estão os compostos antisense de fita única conjugados tendo potência melhorada nas células do fígado in vivo em comparação com o mesmo composto antisense em que falta o conjugado. Dado o equilíbrio necessário das propriedades para esses compostos, tal potência melhorada é surpreendente.
[0019] Em determinadas modalidades, os grupos de conjugadoscompreendem, neste documento, uma fração clivável. Como observado, sem querer estar vinculado pelo mecanismo, é lógico que o conjugado deve permanecer no composto tempo suficiente para proporcionar melhoria na absorção, mas após isso, é desejável que uma alguma parte ou, idealmente, a totalidade do conjugado seja clivada, liberando o composto de origem (por exemplo, composto antisense) em sua forma mais ativa. Em determinadas modalidades, a fração clivável é um nucleosídeo clivável. Tais modalidades tiram proveito de nucleases endógenas na célula, ligando o resto do conjugado (o agrupamento) ao oligonucleotídeo antisense através de um nucleosídeo através de uma ou mais ligações cliváveis, tais como aquelas de uma ligação de fosfodiéster. Em determinadas modalidades, o agrupamento está ligado ao nucleosídeo clivável através de uma ligação fosfodiéster. Em determinadas modalidades, o nucleosídeo clivável está ligado ao oligonucleotídeo antisense (composto antisense) por uma ligação de fosfodiéster. Em determinadas modalidades, o grupo conjugado pode compreender dois ou três nucleosídeos cliváveis. Em tais modalidades, tais nucleosídeos cliváveis estão ligados um ao outro, ao composto antisense e/ou ao agrupamento por meio de ligações cliváveis (tais como aquelas de uma ligação fosfodiéster). Certos conjugados neste documento não compreendem um nucleosídeo clivável e, em vez disso, compreendem uma ligação clivável. É mostrado que a clivagem suficiente do conjugado a partir do oligonucleotídeo é proporcionada por pelo menos uma ligação que é vulnerável à clivagem na célula (uma ligação clivável).
[0020] Em determinadas modalidades, os compostos antisenseconjugados são pró-drogas. Essas pró-drogas são administradas a um animal e são, em última análise, metabolizadas em uma forma mais ativa. Por exemplo, os compostos antisense conjugados são clivados para remover a totalidade ou parte do conjugado resultando na forma ativa (ou mais ativa) do composto antisense em que falta a totalidade ou uma parte do conjugado.
[0021] Em determinadas modalidades, os conjugados são ligados naextremidade 5' de um oligonucleotídeo. Alguns desses conjugados 5' são clivados mais eficientemente do que as contrapartes tendo um grupo de conjugado semelhante ligado à extremidade 3'. Em determinadas modalidades, a atividade melhorada pode se correlacionar com a clivagem melhorada. Em determinadas modalidades, os oligonucleotídeos compreendendo um conjugado na extremidade 5' têm maior eficácia do que os oligonucleotídeos compreendendo um conjugado na extremidade 3' (ver, por exemplo, Exemplos, 56, 81, 83 e 84). Além disso, a ligação em 5' permite uma síntese mais simples do oligonucleotídeo. Normalmente, os oligonucleotídeos são sintetizados em um suporte sólido na direção 3' para 5'. Para fazer o oligonucleotídeo conjugado em 3', normalmente, ligase um nucleosídeo pré-conjugado 3' ao suporte sólido e então monta-se o oligonucleotídeo como de costume. Todavia, a ligação desse nucleosídeo conjugado ao suporte sólido adiciona uma complicação à síntese. Além disso, usando essa abordagem, o conjugado está então presente em toda a síntese do oligonucleotídeo e pode tornar-se degradado durante as etapas subsequentes ou pode limitar os tipos de reações e reagentes que podem ser usados. Usando as estruturas e as técnicas descritas neste documento para oligonucleotídeos conjugados em 5', pode-se sintetizar o oligonucleotídeo pelo uso de técnicas convencionais automatizadas e introduzir o conjugado com o nucleosídeo (mais extremo em 5') final ou depois do oligonucleotídeo ter sido clivado do suporte sólido.
[0022] Tendo em vista a técnica e a presente divulgação, umversado na técnica pode facilmente fabricar qualquer um dos conjugados e oligonucleotídeos conjugados neste documento. Além disso, a síntese de tais conjugados e oligonucleotídeos conjugados divulgados neste documento é mais fácil e/ou requer algumas etapas, e é, portanto, menos caro do que a dos conjugados anteriormente divulgados, oferecendo vantagens na fabricação. Por exemplo, a síntese de certos grupos de conjugado consiste em menos etapas sintéticas, resultando em aumento de rendimento, em relação aos grupos de conjugados descritos anteriormente. Os grupos de conjugado, tais como GalNAc3-10 no Exemplo 46 e GalNAc3-7 no Exemplo 48 são muito mais simples do os conjugados descritos anteriormente, tais como os descritos em U.S. 8.106.022 ou U.S. 7.262.177 que exigem o conjunto de mais intermediários químicos. Por conseguinte, esses e outros conjugados descritos neste documento têm vantagens sobre os compostos descritos anteriormente para uso com qualquer oligonucleotídeo, incluindo oligonucleotídeos de fita única e qualquer fita de oligonucleotídeos de fita dupla (por exemplo, siRNA).
[0023] De forma semelhante, são divulgados neste documentogrupos de conjugado tendo apenas um ou dois ligantes de GalNAc. Como mostrado, tais grupos de conjugados melhoram a atividade dos compostos antisense. Tais compostos são mais fáceis de preparar do que os conjugados que compreendem três ligantes de GalNAc. Os grupos de conjugado que compreendem um ou dois ligantes de GalNAc podem estar ligados a quaisquer compostos antisense, incluindo oligonucleotídeos de fita única e qualquer fita de oligonucleotídeos de fita dupla (por exemplo, siRNA).
[0024] Em determinadas modalidades, os conjugados destedocumento não alteram substancialmente certas medidas de tolerabilidade. Por exemplo, é mostrado neste documento que os compostos antisense conjugados não são mais imunogênicos do que os compostos de origem não conjugados. Uma vez que a potência é melhorada, as modalidades, em que a tolerabilidade permanece a mesma (ou de fato, mesmo se a tolerabilidade piorar apenas ligeiramente em comparação com os ganhos de potência), têm propriedades melhoradas para a terapia.
[0025] Em determinadas modalidades, a conjugação permite alteraros compostos antisense de formas a ter consequências menos atraentes na ausência de conjugação. Por exemplo, em determinadas modalidades, a substituição de uma ou mais ligações de fosforotioato de um composto antisense totalmente de fosforotioato com ligações de fosfodiéster resulta na melhoria de algumas medidas de tolerabilidade. Por exemplo, em certos casos, tais compostos antisense, que têm um ou mais fosfodiéster, são menos imunogênicos do que o mesmo composto, em que cada ligação é um fosforotioato. No entanto, em certos casos, conforme mostrado no Exemplo 26, essa mesma substituição de uma ou mais ligações de fosforotioato com ligações de fosfodiéster também resulta numa absorção celular reduzida e/ou perda de potência. Em determinadas modalidades, os compostos antisense conjugados descritos neste documento toleram tal mudança em ligações com pouca ou nenhuma perda na absorção e potência quando comparados com a contraparte de fosforotioato inteira conjugada. Na verdade, em determinadas modalidades, por exemplo, nos Exemplos 44, 57, 59 e 86, os oligonucleotídeos compreendendo um conjugado e, pelo menos, uma ligação internucleosídica de fosfodiéster, na verdade, exibem um aumento de potência in vivo, até mesmo em relação a uma contraparte de fosforotioato completo compreendendo também o mesmo conjugado. Além disso, uma vez que conjugação resulta em aumentos substanciais na absorção/potência uma pequena perda em que o ganho substancial pode ser aceitável para alcançar tolerância melhorada. Por conseguinte, em determinadas modalidades, compostos antisense conjugados compreendem pelo menos uma ligação de fosfodiéster.
[0026] Em determinadas modalidades, a conjugação de compostosantisense resulta, neste documento, em maior distribuição, absorção e atividade nos hepatócitos. Assim, mais composto é distribuído para o tecido hepático. No entanto, em determinadas modalidades, a distribuição aumentada sozinha não explica o aumento todo na atividade. Em algumas dessas modalidades, mais compostos entram nos hepatócitos. Em determinadas modalidades, mesmo que o aumento da absorção de hepatócitos não explique o aumento total na atividade. Em tais modalidades, a absorção produtiva do composto conjugado é aumentada. Por exemplo, como mostrado no Exemplo 102, determinadas modalidades dos conjugados contendo GalNAc aumentam o enriquecimento dos oligonucleotídeos antisense em hepatócitos versus células não parenqui- matosas. Este enriquecimento é benéfico para os oligonucleotídeos que têm como alvo genes que são expressos em hepatócitos.
[0027] Em determinadas modalidades, os compostos antisenseconjugados mencionados neste documento resultam na exposição renal reduzida. Por exemplo, como mostrado no Exemplo 20, as concentrações dos oligonucleotídeos antisense compreendendo determinadas modalidades de conjugados contendo GalNAc são inferiores no rim do que a dos oligonucleotídeos antisense sem um conjugado contendo GalNAc. Há várias implicações terapêuticas benéficas. Para indicações terapêuticas em que não se busca a atividade no rim, a exposição do rim arrisca a toxicidade renal, sem benefício correspondente. Além disso, a alta concentração no rim geralmente resulta na perda do composto na urina resultando em uma depuração mais rápida Nesse sentido, para alvos não renais, o acúmulo renal é indesejado.
[0028] Em determinadas modalidades, a presente divulgaçãofornece compostos antisense conjugados representados pela fórmula:
[0029] em que
[0030] A é o oligonucleotídeo antisense;
[0031] B é a porção clivável
[0032] C é o ligante conjugado
[0033] D é o grupo de ramificação
[0034] cada E é uma corrente;
[0035] cada F é um ligante; e
[0036] q é um número inteiro entre 1 e 5.
[0037] No diagrama acima e nos diagramas similares contidosneste documento, o grupo de ramificação "D" ramifica-se tantas vezes quanto for necessário para acomodar o número dos grupos (E-F), conforme indicado por "q". Assim, onde q = 1, a fórmula é:
[0038] onde q = 2, a fórmula é:
[0039] em que q = 3, a fórmula é:
[0040] onde q = 4, a fórmula é:
[0041] onde q = 5, a fórmula é:
[0042] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[0043] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[0044] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[0045] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[0046] Em modalidades que têm mais de uma variável específica(por exemplo, mais de um "m" ou "n"), salvo indicação em contrário, cada uma dessas variáveis específicas é selecionada independentemente. Assim, para uma estrutura que tem mais de um n, cada n é selecionado de forma independente assim, eles podem ser iguais ou diferentes uns dos outros.
[0047] Em determinadas modalidades, a presente divulgaçãofornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado ISIS 563580 com 5'-X, onde X é um grupo conjugado que compreende GalNAc. Em determinadas modalidades, o composto antisense consiste o oligonucleotídeo modificado ISIS 563580 com 5'-X, onde X é um grupo conjugado que compreende GalNAc.
[0048] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado conjugado ISIS 703801. Em determinadas modalidades, o composto antisense consiste no oligonucleotídeo modificado conjugado ISIS 703801.
[0049] Em determinadas modalidades, a presente divulgaçãofornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado conjugado ISIS 703802. Em determinadas modalidades, o composto antisense consiste no oligonucleotídeo modificado conjugado ISIS 703802.
[0050] Em determinadas modalidades, a presente divulgaçãofornece compostos antisense conjugados representados pela estrutura a seguir. Em certas modalidades, o composto antisense compreende um oligonucleotídeo modificado com a sequência de nucleobases da SEQ ID NO: 77 com uma 5'-GalNAc com variabilidade nas modificações de açúcar das asas. Em certas modalidades, o composto antisense consiste em um oligonucleotídeo modificado com a sequência de nucleobases da SEQ ID NO: 77 com uma 5'-GalNAc com variabilidade nas modificações de açúcar das asas.
[0051] em que ou R1 é -OCH2CH2OCH3 (MOE) e R2 é H; ou R1 e R2em conjunto, formam uma ponte, em que R1 é -O- e R2 é -CH2-, -CH (CH3) -, ou -CH2CH2-, e R1 e R2 estão diretamente ligados, de tal modo que a ponte resultante é selecionada a partir de: -O-CH2-, -O-CH(CH3)- , and -O-CH2CH2-;
[0052] e para cada par de R3 e R4 no mesmo anel,independentemente para cada anel: R3 é selecionado dentre H e - OCH2CH2OCH3 e R4 é H; ou R3 e R4 formam, em conjunto, uma ponte, onde R3 is -O- e R4 é -CH2-, -CH(CH3)- ou -CH2CH2- e R3 e R4 estão diretamente conectados, de maneira que a ponte resultante é selecionada dentre: -O-CH2-, -O-CH(CH3)- e -O-CH2CH2-;
[0053] e R5 é selecionado a partir de H e -CH3;
[0054] e Z é selecionado dentre S- e O-.
[0055] Em certas modalidades, é fornecida uma composiçãocompreendendo um composto antisense conjugado descrito neste documento, ou um sal da mesma, e um carregador ou diluente farmaceuticamente aceitável.
[0056] Em certas modalidades, a modulação da expressão deANGPTL3 ocorre em uma célula ou um tecido. Em certas modalidades, as modificações ocorrem em uma célula ou um tecido em um animal. Em determinadas modalidades, o animal é um ser humano. Em certas modalidades, a modulação é uma redução no nível de mRNA de ANGPTL3. Em certas modalidades, a modulação é uma redução no nível de proteína de ANGPTL3. Em certas modalidades, os níveis do mRNA e da proteína de ANGPTL3 são reduzidos. Essa redução pode ocorrer em uma forma dependente do tempo e em uma forma dependente da dose.
[0057] Certas modalidades fornecem composições e métodos parauso na terapia. Certas modalidades fornecem composições e métodos para impedir, tratar, retardar, reduzir a progressão e/ou melhorar doenças, distúrbios e condições relacionadas à ANGPTL3. Em certas modalidades, tais doenças, distúrbios e condições médias são doenças, distúrbios e condições cardiovasculares e/ou metabólicos. Em certas modalidades, as composições e os métodos para a terapia incluem a administração de um inibidor específico de ANGPTL3 a um indivíduo em necessidade do mesmo. Em certas modalidades, o inibidor específico de ANGPTL3 é um ácido nucleico. Em certas modalidades, o ácido nucleico é um composto antisense. Em certas modalidades, o composto antisense é um oligonucleotídeo modificado. Em certas modalidades, o composto antisense é um oligonucleotídeo modificado com um grupo de conjugados acoplado.
DESCRIÇÃO DETALHADA DA INVENÇÃO
[0058] Deve ser compreendido que tanto a descrição geral anteriorquanto a descrição detalhada seguinte são exemplares e explicativas apenas e não são restritivas da invenção, conforme reivindicado. Aqui, o uso do singular inclui o plural, salvo quando houver outra especificação. Conforme usado aqui, o uso de "ou" significa "e/ou", salvo quando houver outra especificação. Ademais, o uso do termo "incluindo", bem como outras formas, como "que inclui" ou "incluído", não é limitativo. Além disso, termos como "elemento" ou "componente" abrangem elementos e componentes que compreendem uma unidade e elementos e componentes que compreendem mais de uma subunidade, salvo quando se estabelecer de outra forma.
[0059] Os títulos das seções usados aqui são para finsorganizacionais apenas e não devem ser interpretados como limitadores do objeto descrito. Todos os documentos, ou partes de documentos, citados neste pedido, incluindo, mas não se limitando a, patentes, pedidos de patente, artigos, livros, e tratados, são expressamente incorporados por meio deste para as partes do documento discutidas neste documento, bem como em sua totalidade. Definições
[0060] A menos que as definições específicas sejam fornecidas, anomenclatura utilizada em conexão com, e os procedimentos e técnicas de, química analítica, química orgânica sintética, e químicas farmacêutica e medicinal descritos neste documento são aqueles bem- conhecidos e comumente usados na técnica. Técnicas convencionais podem ser usadas para a síntese química e análise química. Determinadas técnicas e procedimentos podem ser encontrados, por exemplo, em "Carbohydrate Modifications in Antisense Research", editado por Sangvi e Cook, American Chemical Society, Washington D.C., 1994; "Remington's Pharmaceutical Sciences," Mack Publishing Co., Easton, Pa., 21a edição, 2005; e "Antisense Drug Technology, Principles, Strategies, and Applications", editado por Stanley T. Crooke, CRC Press, Boca Raton, Flórida; e Sambrook et al.,"Molecular Cloning, A laboratory Manual," 2a Edição, Cold Spring Harbor Laboratory Press, 1989, que são incorporados por referência por meio deste para qualquer finalidade. Onde permitido, todas as patentes, pedidos, pedidos publicados e outras publicações de jornal, Números de acessoGENBANK e informação de sequência associada podem ser obtidos através de bancos de dados tais como National Center forBiotechnology Information (NCBI) e outros dados referidos ao longo da divulgação neste documento são incorporados por referência nas partes do documento discutido aqui, bem como em sua totalidade.
[0061] Salvo indicado de outra forma, os termos a seguir têm osseguintes significados:
[0062] Como usado neste documento, "nucleosídeo" se refere a umcomposto que compreende uma fração de nucleobase e uma fração de açúcar. Os nucleosídeos incluem, mas não estão limitados a, nucleosídeos de ocorrência natural (como encontrado no DNA e RNA) e nucleosídeos modificados. Os nucleosídeos podem estar ligados a uma fração de fosfato.
[0063] Como usado neste documento, o termo "modificaçãoquímica" significa uma diferença química em um composto quando comparado a uma contraparte de ocorrência natural. As modificações químicas dos oligonucleotídeos incluem modificações de nucleosídeos (incluindo modificações da fração de açúcar e modificações da nucleobase) e modificações da ligação internucleosídica. Em referência a um oligonucleotídeo, a modificação química não inclui diferenças apenas na sequência de nucleobase.
[0064] Como usado neste documento, o termo "furanosil" refere-sea uma estrutura que compreende um anel de 5 membros compreendendo quatro átomos de carbono e um átomo de oxigênio.
[0065] Como usado neste documento, "fração de açúcar deocorrência natural" se refere a um ribofuranosil como é encontrado no RNA de ocorrência natural ou a um desoxirribofuranosil como é encontrado no DNA de ocorrência natural.
[0066] Como usado neste documento, "fração de açúcar" significa uma fração de açúcar de ocorrência natural ou a uma fração de açúcar modificada de um nucleosídeo.
[0067] Como usado neste instrumento, a expressão "fração deaçúcar modificado" se refere a uma fração de açúcar substituída ou a um substituto do açúcar.
[0068] Como usado neste documento, "fração de açúcarsubstituída" significa um furanosil que não é uma fração de açúcar de ocorrência natural. As frações de açúcar substituídas incluem, mas não estão limitadas a furanosis que compreendem substituintes na posição 2', posição 3', posição 5' e/ou posição 4'. Determinadas frações de açúcar substituídas são frações de açúcar bicíclico.
[0069] Como usado neste documento, a expressão "fração deaçúcar substituída em 2'" se refere a um furanosil que compreende um substituinte na posição 2' diferente de H ou OH. Salvo indicação em contrário, uma fração de açúcar substituída em 2' não é uma fração de açúcar bicíclica (isto é, substituinte em 2' de uma fração de açúcar substituída em 2' não forma uma ponte para outro átomo do anel de furanosil).
[0070] Como usado neste documento, "MOE" se refere a -OCH2CH2OCH3.
[0071] Conforme usado neste documento, "nucleosídeo 2'-F" serefere a um nucleosídeo que compreende um açúcar compreendendo um flúor na posição 2'. Salvo indicação em contrário, o flúor em um nucleosídeo 2'-F está na posição ribo (substituindo o OH de uma ribose natural).
[0072] Como usado neste documento, o termo "substituto deaçúcar" significa uma estrutura que não compreende um furanosil e que é capaz de substituir a fração de açúcar de ocorrência natural de um nucleosídeo, tal que as subunidades do nucleosídeo resultantes sejam capazes de se ligar e/ou de se ligar a outros nucleosídeos para formar um composto oligomérico que seja capaz de se hibridizar em um composto oligomérico complementar. Essas estruturas incluem anéis que compreendem um número diferente de átomos do que o furanosil (por exemplo, anéis com 4, 6 ou 7 membros); substituição do oxigênio de um furanosil por um átomo de não-oxigênio (por exemplo, carbono, enxofre ou nitrogênio); ou uma mudança no número de átomos e uma substituição do oxigênio. Essas estruturas também podem compreender substituições correspondentes àquelas descritas para frações de açúcar substituídas (por exemplo, substitutos de açúcar bicíclico carbocíclico de 6 membros compreendendo opcionalmente substituintes adicionais). Substitutos do açúcar também incluem substituições de açúcar mais complexo (por exemplo, sistemas de não-anel do ácido nucleico do peptídeo). Os substitutos do açúcar incluem, sem limitação, morfolino, ciclohexenil e ciclohexitol.
[0073] Como usado neste documento, a expressão "fração deaçúcar bicíclico" significa uma fração de açúcar modificada compreendendo um anel de 4 a 7 membros (incluindo, mas não se limitando a um furanosil) compreendendo uma ponte que conecta dois átomos do anel de 4 a 7 membros para formar um segundo anel, resultando numa estrutura bicíclica. Em determinadas modalidades, o anel de 4 a 7 membros é um anel de açúcar. Em determinadas modalidades, o anel de 4 a 7 membros é um furanosil. Em certas modalidades, a ponte conecta o carbono 2' e o carbono 4' do furanosil.
[0074] Como usado neste documento, "nucleotídeo" significa umnucleosídeo que compreende ainda um grupo de ligação fosfato. Como usado neste documento, "nucleosídeos ligados" podem ou não estar ligados por ligações fosfato e, assim, incluem, mas não estão limitados a, "nucleotídeos ligados". Como usado neste documento, "nucleosídeos ligados" são nucleosídeos que estão conectados a uma sequência contínua (isto é, nenhum nucleosídeo adicional está presente entre aqueles que estão ligados).
[0075] Como usado neste documento, o termo "nucleobase" serefere a um grupo de átomos que pode estar ligado a uma fração de açúcar para criar um nucleosídeo que seja capaz de se incorporar em um oligonucleotídeo, e em que o grupo de átomos seja capaz de se ligar a uma nucleobase de ocorrência natural complementar de outro oligonucleotídeo ou ácido nucleico. As nucleobases podem ocorrer naturalmente ou podem ser modificadas. "Sequência de nucleobases" significa a ordem de nucleobases contíguas independentes de qualquer açúcar, ligação ou modificação de nucleobase.
[0076] Como usados neste documento, "nucleobase nãomodificada" ou "nucleobase de ocorrência natural" significam nucleobases heterocíclicas de ocorrência natural do RNA ou DNA: as bases purínicas adenina (A) e guanina (G) e as bases pirimidínicas timina (T), citosina (C) (incluindo 5-metil C) e uracila (U).
[0077] Como usado neste instrumento, o termo "nucleobasemodificada" se refere a qualquer nucleobase que não tenha ocorrência natural.
[0078] Como usado neste instrumento, o termo "nucleosídeomodificado" se refere a um nucleosídeo que consiste em pelo menos uma modificação química em comparação com os nucleosídeos do RNA ou DNA de ocorrência natural. Os nucleosídeos modificados compõem uma fração de açúcar modificada e/ou uma nucleobase modificada.
[0079] Como usado neste instrumento, "nucleosídeo bicíclico" ou"BNA" se refere a um nucleosídeo que consiste em uma fração de açúcar bicíclica.
[0080] Como usado neste instrumento, "nucleosídeo de etil restrito"ou "cEt" refere-se a um nucleosídeo que compreende uma fração de açúcar bicíclico compreendendo uma ponte 4'-CH(CH3)-O-2'.
[0081] Como usado neste instrumento, o termo "nucleosídeo de ácido nucleico bloqueado" ou "LNA" se refere a um nucleosídeo que compreende uma fração de açúcar bicíclico compreendendo uma ponte de 4'-CH2-O-2'.
[0082] Como usado neste documento, "nucleosídeo substituído em2'" significa a um nucleosídeo que compreende um substituinte na posição 2' diferente de H ou OH. Salvo indicação em contrário, um nucleosídeo substituído em 2' não é um nucleosídeo bicíclico.
[0083] Como usado neste documento, "desoxirribonucleosídeo"significa um nucleosídeo que compreende uma fração de açúcar de furanosil 2'-H, como encontrado nos desoxirribonucleosídeos de ocorrência natural (DNA). Em determinadas modalidades, um 2- desoxinucleosídeo pode compreender uma nucleobase modificada ou pode compreender uma nucleobase de RNA (por exemplo, uracil).
[0084] Conforme usado neste documento, "oligonucleotídeo"significa um composto compreendendo uma pluralidade de nucleosídeos ligados. Em certas modalidades, um oligonucleotídeo compreende um ou mais ribonucleosídeos (RNA) não modificados e/ou desoxirribonucleosídeos (DNA) não modificados e/ou um ou mais nucleosídeos modificados.
[0085] Como usado neste documento, "oligonucleosídeo" se referea um oligonucleotídeo em que nenhuma das ligações de internucleosídeo contém um átomo de fósforo. Como usado neste documento, oligonucleotídeos incluem oligonucleosídeos.
[0086] Como usado neste documento, "oligonucleotídeomodificado" significa um oligonucleotídeo que compreende pelo menos um nucleosídeo modificado e/ou pelo menos uma ligação internucleosídica modificada.
[0087] Como usado neste documento, "ligação" ou "grupo deligação" significa um grupo de átomos que se ligam a dois ou mais grupos de átomos.
[0088] Como usado neste documento, "ligação internucleosídica"significa uma ligação covalente entre nucleosídeos adjacentes em um oligonucleotídeo.
[0089] Como usado neste documento, "ligação internucleosídica deocorrência natural" significa uma ligação de fosfodiéster 3' a 5'.
[0090] Como usado neste documento, "ligação internucleosídicamodificada" significa qualquer ligação internucleosídica diferente da ligação internucleosídica de ocorrência natural.
[0091] Como usado neste documento, "ligação internucleosídicaterminal" significa a ligação entre os últimos dois nucleosídeos de um oligonucleotídeo ou da respectiva região definida.
[0092] Como usado neste documento, o "grupo de ligação defósforo" significa um grupo de ligação que consiste em um átomo de fósforo. Grupos de ligação de fósforo incluem, sem limitação, grupos que têm a fórmula:
[0093] em que:
[0094] Ra e Rd são cada um, independentemente, O, S, CH2, NH,ou NJ1 em que J1 é alquil substituído C1-C6 ou alquil C1-C6 ;
[0095] Rb é O ou S;
[0096] Rc é OH, SH, alquil C1-C6 , alquil substituído C 1-C6 , alcoxiC1-C6, alcoxi substituído, amino ou amino substituído C 1-C6e
[0097] J1 is Rb é O ou S.
[0098] Grupos de ligação de fósforo incluem, sem limitação,fosfodiéster, fosforotioato, fosforoditioato, fosfonato, fosforamidato, fosforotioamidato, tionoalquilfosfonato, fosfotriésteres,tionoalquilfosfotriéster e boranofosfato.
[0099] Como usado neste documento, "grupo de ligação de fósforo" diz respeito a um grupo de ligação de fósforo que liga diretamente dois nucleosídeos.
[00100] Como usado neste instrumento, "grupo de ligação fósforonão-internucleosídeo" se refere a um grupo de ligação fósforo que não se liga diretamente a dois nucleosídeos. Em determinadas modalidades, um grupo de ligação fósforo não-internucleosídeo liga um nucleosídeo a um grupo diferente de um nucleosídeo. Em determinadas modalidades, um grupo de ligação fósforo não-internucleosídeo liga dois grupos, sendo que nenhum deles é um nucleosídeo.
[00101] Como usado neste instrumento, "grupo de ligação neutro" se refere a um grupo de ligação não é carregado. Os grupos de ligações neutras incluem, sem limitação, fosfotriésteres, metilfosfonatos, MMI (CH2-N (CH3) -O-), Amida-3 (-CH2-C (= O) -N (H) -), amida-4 (-CH2-N (H) -C (= O) -), formacetal (-O-CH2-O-) e tioformacetal (-S-CH2-O-). Além disso, os grupos de ligação incluem ligações não iônicas que compreendem siloxano (dialquilsiloxano), éster carboxilato, carboxamida, sulfeto, éster sulfonato e amidas (ver, por exemplo: Carbohydrate Modifications in Antisense Research; Y.S. Sanghvi e P.D. Cook Eds. ACS Symposium Series 580; Capítulos 3 e 4, (pp. 40-65)). Outros grupos de ligação neutros incluem ligações não-iônicas, que compreendem partes do componente misturadas N, O, S e CH2.
[00102] Como usado neste documento, "grupo de ligação neutro de internucleosídeo" se refere a um grupo de ligação neutro que liga diretamente dois nucleosídeos.
[00103] Como usado neste instrumento, "grupo de ligação neutro de não-internucleosídeo" se refere a um grupo de ligação neutro que não se liga diretamente a dois nucleosídeos. Em determinadas modalidades, um grupo de ligação neutro de não-internucleosídeo liga um nucleosídeo a um grupo diferente de um nucleosídeo. Em determinadas modalidades, um grupo de ligação neutro de não- internucleosídeo liga dois grupos, sendo que nenhum deles é um nucleosídeo.
[00104] Como usado neste instrumento, "composto oligoméricos" se refere a uma estrutura polimérica que compreende duas ou mais subestruturas. Em determinadas modalidades, um composto oligomérico compreende um oligonucleotídeo. Em determinadas modalidades, um composto oligomérico compreende um ou mais grupos conjugados e/ou grupos terminais. Em determinadas modalidades, um composto oligomérico consiste em um oligonucleotídeo. Compostos oligoméricos incluem também ácidos nucleicos de ocorrência natural. Em determinadas modalidades, um composto oligomérico compreende uma estrutura principal de uma ou mais subunidades monoméricas ligadas onde cada subunidade monomérica está direta ou indiretamente afixada a uma fração de base heterocíclica. Em determinadas modalidades, os compostos oligoméricos também podem incluir subunidades monoméricas que não estão ligadas a uma fração de base heterocíclica fornecendo, assim, sítios abásicos. Em determinadas modalidades, as ligações juntam subunidades monoméricas, frações ou substitutos de açúcar e as frações de base heterocíclica podem ser independentemente modificadas. Em determinadas modalidades, a unidade de açúcar de ligação, que pode ou não incluir uma base heterocíclica, pode ser substituída por um mimético como monômeros em ácidos nucleicos de peptídeo.
[00105] Como usado neste documento, "grupo terminal" se refere a um ou mais átomo incorporado à extremidade 3' ou à extremidade 5' ou a ambas extremidades de um oligonucleotídeo. Em determinadas modalidades, um grupo terminal é um grupo conjugado. Em determinadas modalidades, um grupo terminal compreende um ou mais nucleosídeos do grupo terminal.
[00106] Como usado neste instrumento, "conjugado" ou "grupo conjugado" se refere a um átomo ou grupo de átomos ligados a um oligonucleotídeo ou composto oligomérico. Em geral, grupos conjugados modificam uma ou mais propriedades do composto ao qual estão afixados, incluindo, entre outros, propriedades de farmacodinâmica, farmacocinética, absorção, distribuição celular, absorção celular, carga e/ou depuração.
[00107] Como usado neste documento, "ligador conjugado" ou "ligador" no contexto de um grupo conjugado representa uma parte de um grupo conjugado que consiste em qualquer átomo ou grupo de átomos e que liga covalentemente (1) um oligonucleotídeo a outra parte do grupo conjugado ou (2) duas ou mais partes do grupo conjugado.
[00108] Os grupos conjugados são mostrados neste instrumento como radicais, fornecendo uma ligação para a formação de ligação covalente com um composto oligomérico como um oligonucleotídeo antisense. Em determinadas modalidades, o ponto de ligação em um composto oligomérico é o átomo de oxigênio 3' do grupo 3'-hidroxil do nucleosídeo terminal 3' do composto oligomérico. Em determinadas modalidades, o ponto de ligação ao composto oligomérico é o átomo de oxigênio 5' do grupo 5'-hidroxil do nucleosídeo terminal 5' do composto oligomérico. Em determinadas modalidades, a ligação para a formação da ligação com um composto oligomérico é uma ligação clivável. Em certas modalidades, tal ligação clivável constitui a totalidade ou parte de uma porção clivável.
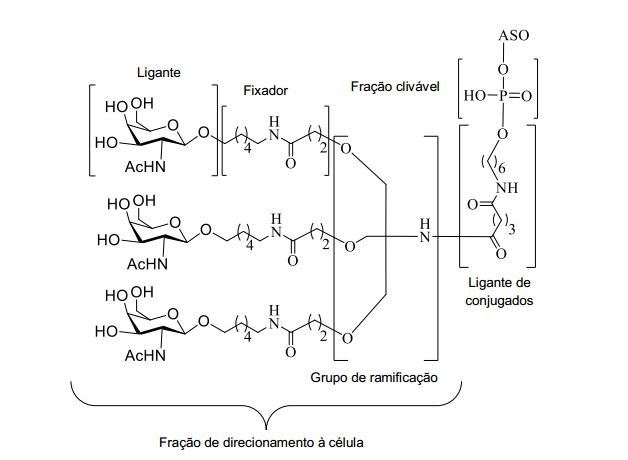
[00109] Em determinadas modalidades, os grupos conjugados compreendem uma fração clivável (por exemplo, uma ligação clivável ou um nucleosídeo clivável) e uma parte do agrupamento de carboidrato, como uma parte do agrupamento GalNAc. Essa parte do agrupamento do carboidrato compreende: uma fração dedirecionamento e, opcionalmente, um ligador de conjugado. Em determinadas modalidades, a parte do agrupamento de carboidrato é identificada pelo número e identidade do ligante. Por exemplo, em determinadas modalidades, a parte do agrupamento do carboidratocompreende 3 grupos GalNAc e é designada "GalNAc3". Em determinadas modalidades, a parte do agrupamento de carboidratocompreende 4 grupos GalNAc e é designada "GalNAc4". Partes do agrupamento de carboidrato específicas (tendo ligante específico, grupos de ramificação e conjugados) são descritas neste documento e designadas pelo numeral romano seguido por subscrito "a". Portanto, "GalNac3-1a" se refere a uma parte do agrupamento de carboidrato específico de um grupo conjugado tendo 3 grupos GalNac e ligante identificado especificamente grupos de ramificação e grupos de ligação. Esse fragmento de agrupamento do carboidrato está afixado a um composto oligomérico via fração clivável, como uma ligação clivável ou nucleosídeo clivável.
[00110] Como usado neste documento, "fração clivável" se refere a uma ligação ou grupo que é capaz de ser dividido em condições fisiológicas. Em determinadas modalidades, uma fração clivável está no interior de compartimentos celulares ou subcelulares, como um lisossomo. Em determinadas modalidades, uma fração clicável é clivada por enzimas endógenas, como as nucleases. Em determinadas modalidades, uma fração clivável compreende um grupo de átomos com uma, duas, três, quatro ou mais de quatro ligações cliváveis.
[00111] Como usado neste instrumento, "ligação clivável" se refere a qualquer ligação química capaz de ser dividida. Em determinadas modalidades, a ligação clivável é selecionada dentre: uma amida, uma poliamida, um éster, um éter, um ou ambos ésteres de fosfodiéster, um éster de fosfato, um carbamato, um dissulfeto ou um peptídeo.
[00112] Tal como aqui utilizado, "agrupamento de carboidratos" denota um composto com um ou mais resíduos de carboidtrados unidos a um grupo ligante ou de suporte (ver, por exemplo, Maier et al., "Synthesis of Antisense Oligonucleotides Conjugated to a Multivalent Carbohydrate Cluster for Cellular Targeting," Bioconjugate Chemistry, 2003, (14): 18-29, que é incorporado aqui por referência em suatotalidade, ou Rensen et al., "Design and Synthesis of Novel N- Acetylgalactosamine-Terminated Glycolipids for Targeting of Lipoproteins to the Hepatic Asiaglycoprotein Receptor," J. Med. Chem. 2004, (47): 5798-5808, para exemplos de agrupamentos de conjugados de carboidratos).
[00113] Como usado neste documento, "carboidrato modificado" se refere a qualquer carboidrato que tem uma ou mais modificações químicas relativas a carboidratos de ocorrência natural.
[00114] Como usado neste instrumento, "derivado de carboidrato" se refere a qualquer composto que pode ser sintetizado usando um carboidrato como matéria-prima ou intermediário.
[00115] Como usado neste instrumento, "carboidrato" se refere a um carboidrato de ocorrência natural, um carboidrato modificado ou um derivado do carboidrato.
[00116] Como usado neste instrumento, "grupo de proteção" se refere a qualquer composto ou grupo de proteção conhecido pelos versados na técnica. Exemplos não limitantes de grupos de proteção podem ser encontrados em "Protective Groups in Organic Chemistry", T. W. Greene, P. G. M. Wuts, ISBN 0-471-62301-6, John Wiley & Sons, Inc, Nova York, o qual é incorporado a este documento por referência na sua totalidade.
[00117] Como usado neste documento, "de fita única" se refere a um composto oligomérico que não é hibridizado ao seu complemento e que não possui auto-complementaridade suficiente para formar um duplex auto-estável.
[00118] Como usado neste documento, "de fita dupla" se refere a um par de compostos oligoméricos que são hibridizados a um outro ou um composto oligomérico auto-complementar único que forma uma estrutura hairpin. Em determinadas modalidades, um composto oligomérico de fita dupla compreende um primeiro e um segundo composto oligomérico.
[00119] Como usado neste instrumento, "composto antisense" se refere a um composto que compreende ou que consiste em um oligonucleotídeo, sendo que, pelo menos uma parte dele é complementar ao ácido nucleico alvo que é capaz de hibridizar, resultando em pelo menos uma atividade antisense.
[00120] Como usado neste instrumento, "atividade antisense" se refere a qualquer atividade detectável e/ou mudança mensurável atribuível à hibridização de um composto antisense para seu ácido nucleico alvo. Em determinadas modalidades, a atividade antisense inclui modulação da quantidade ou atividade de uma transcrição do ácido nucleico alvo (por exemplo, mRNA). Em determinadas modalidades, a atividade antisense inclui modulação do splicing do pré- mRNA.
[00121] Como usado neste instrumento, "composto antisense à base de RNase H" se refere a um composto antisense em que pelo menos alguma atividade antisense do composto antisense é atribuível à hibridação do composto antisense para um ácido nucleico alvo e clivagem subsequente do ácido nucleico alvo pela RNase H.
[00122] Como usado neste instrumento, "composto antisense à base de RISC" se refere a um composto antisense em que pelo menos alguma atividade antisense do composto antisense é atribuída ao Complexo de Silenciamento Induzido do RNA (RISC).
[00123] Como usado neste instrumento, "detectar" ou "medir" significa que um teste ou ensaio para detecção ou medição é realizado. Essa detecção e/ou medição pode resulta em um valor zero. Assim, se um teste para detecção ou medição resultar na constatação de nenhuma atividade (atividade zero), a etapa de detecção ou medição da atividade, no entanto, foi realizada.
[00124] Como usado neste instrumento, "atividade detectável e/ou mensurável" se refere a uma atividade estatisticamente significativa que não seja equivalente a zero.
[00125] Como usado neste instrumento, "essencialmente inalterado" se refere a pouca ou nenhuma alteração em um determinado parâmetro, especificamente em relação a outro parâmetro que muda muito mais. Em determinadas modalidades, um parâmetro é essencialmente inalterado quando muda em menos de 5%. Em determinadas modalidades, um parâmetro é essencialmente inalterado se ele muda menos de duas vezes enquanto outro parâmetro muda pelo menos dez vezes. Por exemplo, em determinadas modalidades, uma atividade antisense é uma mudança na quantidade de um ácido nucleico alvo. Em determinadas modalidades, a quantidade de ácido nucleico não-alvo é essencialmente inalterada se ela muda muito menos do que o ácido nucleico alvo, mas a mudança não precisa ser zero.
[00126] Como usado neste documento, "expressão" se refere ao processo pelo qual um gene resulta, em última análise, em uma proteína. Expressão inclui, entre outros, transcrição, modificação pós- transcricional (por exemplo, splicing, poliadenilação, adição de 5'-cap) e tradução.
[00127] Como usado neste instrumento, "ácido nucleico alvo" se refere a uma molécula de ácido nucleico à qual um composto antisense destina-se a hibridizar para resultar em uma atividade antisense desejada. Oligonucleotídeos antisense têm complementaridade suficiente para seus ácidos nucleicos alvo para permitir a hibridação em condições fisiológicas.
[00128] Como usado neste documento, "complementaridade da nucleobase" ou "complementaridade" quando em referência às nucleobases se refere a uma nucleobase capaz de se emparelhar com outra nucleobase. Por exemplo, no DNA, a adenina (A) é complementar à timina (T). Por exemplo, no RNA, a adenina (A) é complementar a uracil (U). Em certas modalidades, nucleobase complementar se refere a uma nucleobase de um composto antisense que é capaz de se emparelhar com uma nucleobase do seu ácido nucleico alvo. Por exemplo, se uma nucleobase em uma determinada posição de um composto antisense for capaz de fazer ligação de hidrogênio com uma nucleobase em uma determinada posição de um ácido nucleico alvo, então a posição da ligação de hidrogênio entre o oligonucleotídeo e o ácido nucleico alvo são considerados complementares nesse par de nucleobase. As nucleobases que compreendem determinadas modificações podem manter a capacidade de se emparelhar com uma nucleobase equivalente e, assim, são ainda aptos à complementaridade de base.
[00129] Como usado neste instrumento, "não complementar", em relação às nucleobases, se refere a um par de nucleobases que não forma ligações de hidrogênio com outro.
[00130] Como usado neste documento, "complementar", em relação aos compostos oligoméricos (por exemplo, nucleosídeos ligados, oligonucleotídeos ou ácidos nucleicos) se refere à capacidade desses compostos oligoméricos ou suas regiões para hibridização para um outro composto oligomérico ou sua região através da complementaridade da nucleobase. Os compostos oligoméricos complementares não precisam apresentar complementaridade de nucleobase em cada nucleosídeo. Pelo contrário, algumas incompatibilidades são toleradas. Em determinadas modalidades, compostos oligoméricos complementares ou regiões são complementares em 70% das nucleobases (70% complementares). Em determinadas modalidades, os compostos oligoméricos complementares ou regiões são 80% complementares. Em determinadas modalidades, os compostos oligoméricos complementares ou regiões são 90% complementares. Em determinadas modalidades, os compostos oligoméricos complementares ou regiões são 95% complementares. Em determinadas modalidades, os compostos oligoméricos complementares ou regiões são 100% complementares.
[00131] Como usado neste documento, "incompatibilidade" se refere a uma nucleobase de um primeiro composto oligoméricos que não é capaz de se emparelhar com uma nucleobase em uma posição correspondente de um segundo composto oligomérico, quando o primeiro e o segundo composto oligomérico estão alinhados. Um ou ambos do primeiro e segundo compostos oligoméricos podem ser oligonucleotídeos.
[00132] Como usado neste documento, "hibridização" se refere ao emparelhamento de compostos oligoméricos complementares (por exemplo, um composto antisense e seu ácido nucleico alvo). Enquanto não houver limitação a um mecanismo específico, o mecanismo mais comum de emparelhamento envolve a ligação de hidrogênio, que pode ser ligação de hidrogênio Watson-Crick, Hoogsteen ou Hoogsteen reversa entre nucleobases complementares.
[00133] Como usado neste documento, "hibridiza especificamente" se refere à capacidade de um composto oligomérico de hibridizar em um sítio de ácido nucleico com maior afinidade do que hibridizar em outro sítio de ácido nucleico.
[00134] Como usado neste documento, "totalmente complementar", em relação a um oligonucleotídeo ou à fração do mesmo, significa que cada base do oligonucleotídeo ou da fração do mesmo é capaz de se emparelhar com uma nucleobase de um ácido nucleico complementar ou fração contígua do mesmo. Assim, uma região totalmente complementar não compreende incompatibilidades nem nucleobases não hibridizadas em qualquer fita.
[00135] Como usado neste documento, "complementaridade percentual" se refere à porcentagem de nucleobases de um composto oligomérico que seja complementar a uma parte de comprimento equivalente de ácido nucleico alvo. a complementaridade percentual é calculada pela divisão do número de nucleobases do composto oligomérico que seja complementar às nucleobases nas posições correspondentes no ácido nucleico alvo pelo comprimento total do composto oligomérico.
[00136] Como usado neste documento, "identidade percentual" se refere ao número de nucleobases em um primeiro ácido nucleico que é do mesmo tipo (independente de modificação química) que as nucleobases nas posições correspondentes em um segundo ácido nucleico, dividido pelo número total de nucleobases no primeiro ácido nucleico.
[00137] Como usado neste documento, "modulação" se refere a uma mudança da quantidade ou qualidade de uma molécula, função ou atividade, em comparação com a quantidade ou qualidade de uma molécula, função ou atividade antes da modulação. Por exemplo, a modulação inclui a mudança, ou o aumento (estímulo ou indução) ou a diminuição (inibição ou redução) da expressão gênica. Como outro exemplo, a modulação da expressão pode incluir uma alteração na seleção do sítio de splice do processamento de pré-mRNA, resultando em uma alteração na quantidade absoluta ou relativa de um determinado variante de splice em comparação com a quantidade na ausência de modulação.
[00138] Como usado neste documento, "motivo químico" se refere a um padrão de modificações químicas em um oligonucleotídeo ou em uma região do mesmo. Os motivos podem ser definidos pelas modificações em determinados nucleosídeos e/ou certos grupos de ligação de um oligonucleotídeo.
[00139] Como usado neste documento, "motivo do nucleosídeo" serefere a um padrão de modificações do nucleosídeo em um oligonucleotídeo ou em uma região do mesmo. As ligações desse oligonucleotídeo podem ser modificadas ou não modificadas. Salvo indicação em contrário, os motivos descritos neste documento apenas nucleosídeos destinados a serem motivos de nucleosídeos. Assim, nesses casos, as ligações não são limitadas.
[00140] Como usado neste documento, "motivo do açúcar" se refere a um padrão de modificações do açúcar em um oligonucleotídeo ou em uma região do mesmo.
[00141] Como usado neste documento, "motivo da ligação" se refere a um padrão de modificações da ligação em um oligonucleotídeo ou em uma região do mesmo. Os nucleosídeos desse oligonucleotídeo podem ser modificados ou não modificados. Salvo indicação em contrário, os motivos descritos neste documento são apenas ligações destinadas a serem motivos de ligação. Assim, nesses casos, os nucleosídeos não são limitados.
[00142] Como usado neste documento, "motivo de modificação da nucleobase" se refere a um padrão de modificações de nucleobases ao longo de um oligonucleotídeo. Salvo indicação em contrário, um motivo de modificação da nucleobase é independente da sequência de nucleobase.
[00143] Como usado neste documento, "motivo de sequência" se refere a um padrão de nucleobases dispostos ao longo de um oligonucleotídeo ou parte do mesmo. Salvo indicação em contrário, um motivo de sequência é independente de modificações químicas e, portanto, pode ter qualquer combinação de modificações químicas, inclusive não ter modificações químicas.
[00144] Como usado neste documento, "tipo de modificação", em relação a um nucleosídeo ou um nucleosídeo de um "tipo" se refere à modificação química de um nucleosídeo e inclui nucleosídeos modificados e não modificados. Portanto, salvo indicação em contrário, um "nucleosídeo com uma modificação do primeiro tipo" pode ser um nucleosídeo não modificado.
[00145] Como usado neste documento, "diferentemente modificado" se refere às modificações químicas ou substituintes químicos que são diferentes uns dos outros, incluindo ausência de modificações. Assim, por exemplo, um nucleosídeo MOE e um nucleosídeo de DNA não modificado são "diferentemente modificados", embora o nucleosídeo do DNA não seja modificado. Da mesma forma, DNA e RNA são "diferentemente modificados", embora ambos sejam nucleosídeos não modificados de ocorrência natural. Os nucleosídeos que são idênticos, mas por compreenderem nucleobases diferentes, não são diferentemente modificados. Por exemplo, um nucleosídeo que compreende um açúcar modificado 2'-OMe e uma nucleobase adenina não modificada e um nucleosídeo compreendendo um açúcar modificado 2'-OMe e uma nucleobase timina não modificada não são diferentemente modificados.
[00146] Como usado neste documento, "o mesmo tipo de modificações" se refere às modificações que são iguais entre si, incluindo a ausência de modificações. Assim, por exemplo, dois nucleosídeos de DNA não modificados têm "o mesmo tipo de modificação", embora o nucleosídeo de DNA não seja modificado. Esses nucleosídeos que têm o mesmo tipo de modificação podem incluir nucleobases diferentes.
[00147] Como usado neste instrumento, "regiões separadas" se refere às partes de um oligonucleotídeo em que as modificações químicas ou o motivo das modificações químicas de quaisquer partes vizinhas incluem pelo menos uma diferença para permitir que regiões separadas se distinguam uma da outra.
[00148] Como usado neste documento, "transportadora ou diluente farmaceuticamente aceitável" se refere a qualquer substância apropriada para uso na administração de um animal. Em determinadas modalidades, um transportador ou diluente farmaceuticamente aceitável é uma solução salina estéril. Em determinadas modalidades, essa solução salina é uma solução salina de qualidade farmacêutica.
[00149] Como usado neste documento, o termo "distúrbio metabólico" se refere a uma doença ou condição caracterizada principalmente pela desregulação do metabolismo - o conjunto complexo de reações químicas associadas à degradação de alimentos para produzir energia.
[00150] Como usado neste instrumento, os termos "distúrbio cardiovascular" ou "doença cardiovascular" se referem a uma doença ou condição caracterizada principalmente pela função prejudicada do coração ou dos vasos sanguíneos. Exemplos de doenças ou distúrbios cardiovasculares incluem, mas não são limitados a, aneurisma, angina, arritmia, aterosclerose, doença cerebrovascular (derrame), doença cardíaca coronariana, hipertensão, dislipidemia, hiperlipidemia, hipertrigliceridemia e hipercolesterolemia.
[00151] Conforme usado neste documento, o termo "sistema de anel mono ou policíclico" deve incluir todos os sistemas de anel selecionados a partir de sistemas de anel de radical simples ou policíclico em que os anéis são fundidos ou ligados e deve incluir também sistemas de anel simples e mistos individualmente selecionados a partir de aril alifático, alicíclico, heteroaril, aralquil, arilalquil, heterocíclico, heteroaril, heteroaromático e heteroarilalquil. Essas estruturas mono e policíclicas podem conter anéis que tem o mesmo nível de saturação ou tendo cada um, de maneira independente, diferentes graus de saturação incluindo totalmente saturados, parcialmente saturados ou totalmente insaturados. Cada anel pode compreender átomos do anel selecionados a partir de C, N, O e S para dar origem a anéis heterocíclicos, bem como anéis que compreendem apenas anel apenas com átomos de C que podem estar presentes em um motivo misto, como por exemplo, benzimidazol, em que um anel tem apenas átomos de carbono anel e o anel fundido tem dois átomos de nitrogênio. O sistema de anel mono ou policíclico pode ser substituído também por grupos substituintes, como por exemplo, ftalimida que tem dois grupos =O afixados a um dos anéis. Os sistemas de anel mono ou policíclico podem ser afixados a moléculas de origem usando várias estratégias como diretamente através de um átomo do anel, fundido através de vários átomos do anel, através de um grupo substituinte ou através de uma fração de ligação bifuncional.
[00152] Como usado neste documento, "pró-droga" se refere a uma forma menos ativa ou inativa de um composto que, quando administrado a um assunto, é metabolizada para formar o composto ativo ou mais ativo (por exemplo, drogas).
[00153] Como usado neste documento, "substituinte" e "grupo substituinte" se refere a um átomo ou grupo que substitui o átomo ou grupo de um composto nomeado de origem. Por exemplo, um substituinte de um nucleosídeo modificado é qualquer átomo ou grupo que difere do átomo ou do grupo encontrado em um nucleosídeo de ocorrência natural (por exemplo, um substituinte 2' modificado é qualquer átomo ou grupo na posição 2' de um nucleosídeo diferente de H ou OH). Os grupos substituintes podem ser protegidos ou desprotegidos. Em determinadas modalidades, os compostos de presente divulgação têm substituintes em uma ou em mais de uma posição do composto de origem. Os substituintes também podem ser substituídos ainda por outros grupos substituintes e podem ser afixados diretamente ou através de um grupo de ligação como um grupo alquil ou hidrocarbil a um composto de origem.
[00154] Da mesma forma, como usado neste documento, "substituinte", em relação a um grupo funcional químico, se refere a um átomo ou grupo de átomos que difere do átomo ou do grupo de átomos normalmente presente no grupo funcional mencionado. Em determinadas modalidades, um substituinte substitui um átomo de hidrogênio do grupo funcional (por exemplo, em determinadas modalidades, o substituinte de um grupo metil substituído é um átomo ou grupo diferente do hidrogênio que substitui um dos átomos de hidrogênio de um grupo metil não substituído). A menos que indicado de outra forma, os grupos susceptíveis a serem utilizados como substituintes incluem, sem limitação, halogênio, hidroxil, alquil, alcenil, alcinil, acil(-C(O)Raa), carboxil(-C(O)ORaa), grupos alifáticos, grupos alicíclicos, alcoxi, oxi substituído(-ORaa), aril, aralquil, heterociclo, heteroaril, heteroarilalquil, amino(-N (Rbb)(Rcc)), imino(= NRbb), amido(- C(O)N (Rbb)(Rcc) ou -N(Rbb)C(O)Raa), azido (-N3), nitro (-NO2), ciano(- CN), carbamido(-OC (O) N (Rbb) (Rcc) ou -N(Rbb)C(O)ORaa), ureído (- N(Rbb)C(O)N(Rbb) (Rcc)), tioureído(-N (Rbb)C(S)N(Rbb) (Rcc)), guanidinil(- N (Rbb)C(= NRbb)N(Rbb) (Rcc)), amidinil(-C (= NRbb)N(Rbb)(Rcc) ou -N(Rbb) C(=NRbb)(Raa)), tiol (-SRbb), sulfinil (-S(O)Rbb), sulfonil (-S(O)2Rbb) e sulfonamidil (-S(O)2N(Rbb)(Rcc) ou -N(Rbb)S(O)2Rbb). Onde Raa, Rbb e Rcc é, independentemente, H, um grupo funcional químico opcionalmente ligado ou um outro grupo substituinte com uma lista preferencial incluindo, entre outros, alquil, alquenil, alquinil, alifático, alcóxi, acil, aril, aralquil, heteroaril, alicíclico, heterocíclico e heteroarilalquil. Os substituintes selecionados nos compostos descritos neste documento estão presentes em grau recursivo.
[00155] Tal como aqui utilizado, "alquil", como usado neste documento, "alquil" se refere um radical hidrocarboneto saturado linear ou ramificado que contém até vinte e quatro átomos de carbono. Exemplos de grupos alquil incluem, entre outros, metil, etil, propil, butil, isopropil, n-hexil, octil, decil, dodecil e similares. Os grupos alquil incluem tipicamente de 1 a cerca de 24 átomos de carbono, mais tipicamente de 1 a cerca de 12 átomos de carbono (alquil C1-C12), sendo que de 1 a cerca de 6 átomos de carbono é mais preferencial. Como usado neste documento, "alquenil" se refere a um radical de uma cadeia de hidrocarboneto linear ou ramificada que contém até vinte e quatro átomos de carbono e que tem pelo menos uma ligação dupla de carbono-carbono. Exemplos de grupos alquenil incluem, entre outros, etenil, propenil, butenil, 1-metil-2-buten-1-il, dienos como 1,3-butadieno e afins. Os grupos alquenil incluem tipicamente de 2 a cerca de 24 átomos de carbono, mais tipicamente de 2 a cerca de 12 átomos de carbono, sendo que de 2 a cerca de 6 átomos de carbono é mais preferencial. Grupos alquenil usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00156] Como usado neste documento, "alquinil" se refere a um radical de uma cadeia de hidrocarboneto linear ou ramificada que contém até vinte e quatro átomos de carbono e que tem pelo menos uma ligação tripla de carbono-carbono. Exemplos de grupos alquinil incluem, entre outros, etinil, 1-propinil, 1-butinil e similares. Os grupos alquinil incluem tipicamente de 2 a cerca de 24 átomos de carbono, mais tipicamente de 2 a cerca de 12 átomos de carbono, sendo que de 2 a cerca de 6 átomos de carbono é mais preferencial. Grupos alquinil usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00157] Como usado neste documento, "acil" se refere a um radical formado pela remoção de um grupo hidroxil de um ácido orgânico e que tem fórmula geral -C(O)-X onde X é normalmente alifático, alicíclico ou aromático. Os exemplos incluem carbonilas alifáticas, carbonilas aromáticas, sulfonilas alifáticas, sulfinilas aromáticas, sulfinilas alifáticas, fosfatos aromáticos, fosfatos alifáticos e similares. Grupos acil usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00158] Como usado neste documento, "alicíclico" se refere a um sistema de anel cíclico, em que o anel é alifático. O sistema de anel pode compreender um ou mais anéis, em que pelo menos um anel é alifático. Alicíclicos preferenciais incluem anéis com cerca de 5 a cerca de 9 átomos de carbono no anel. Alicíclicos usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00159] Como usado neste documento, "alifático" se refere a um radical de hidrocarboneto linear ou ramificado contendo até vinte e quatro átomos de carbono, em que a saturação entre quaisquer dois átomos de carbono é uma ligação simples, dupla ou tripla. O grupo alifático contém, preferencialmente, de 1 a cerca de 24 átomos de carbono, mais tipicamente de 1 a cerca de 12 átomos de carbono, sendo que de 1 a cerca de 6 átomos de carbono é mais preferencial. A cadeia linear ou ramificada de um grupo alifático pode ser interrompida por um ou mais heteroátomos que incluem nitrogênio, oxigênio, enxofre e fósforo. Esses grupos alifáticos interrompidos por heteroátomos incluem, entre outros, polialcóxis, como polialcaleno glicóis, poliaminas e poliminas. Grupos alifáticos usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00160] Como usado neste documento, "alcóxi" se refere a um radical formado entre um grupo alquil e um átomo de oxigênio, em que o átomo de oxigênio é usado para afixar o grupo alcóxi a uma molécula de origem. Exemplos de grupos alcóxi incluem, sem limitação, metóxi, etóxi, n-propóxi, isopropóxi, n-butóxi, sec-butóxi, terc-butóxi, n-pentóxi, neopentóxi, n-hexóxi e similares. Grupos alcóxi usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00161] Como usado neste documento, "aminoalquil" se refere um radical alquil C1-C12 substituído por amina. A parte da alquil do radical forma uma ligação covalente com uma molécula de origem. O grupo amina pode estar localizado em qualquer posição e o grupo de aminoalquil pode ser substituído por um grupo substituinte nas partes alquil e/ou amina.
[00162] Como usado neste instrumento, "aralquil" e "arilalquil" se refere a um grupo aromático que é ligado covalentemente a um radical alquil C1-C12. A parte do radical alquil do grupo aralquil (ou arilalquil) resultante forma uma ligação covalente com uma molécula de origem. Os exemplos incluem, entre outros, benzil, fenetil e similares. Os grupos aralquil usados neste documento podem incluir ainda, opcionalmente, grupos substituintes afixados a alquil, aril ou a ambos os grupos que formam o grupo radical.
[00163] Como usados neste documento, "aril" e "aromático" se referem a radicais de um sistema de anel carboxílico mono ou policíclico com um ou mais anéis aromáticos. Os exemplos de grupos aril incluem, entre outros, fenil, naftil, tetrahidronaftil, indanil, idenil e similares. Os sistemas de anel aril preferenciais têm cerca de 5 a cerca de 20 átomos de carbono em um ou mais anéis. Grupos aril usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00164] Como usados neste documento, os termos "halo" e "halogênio" se referem a um átomo selecionado do flúor, cloro, bromo e iodo.
[00165] Como usado neste documento, "heteroaril" e "hetero- aromático" se referem a um radical que compreende um sistema de anel aromático mono ou policíclico, um sistema de anel ou um sistema de anel fundido em que pelo menos um dos anéis seja aromático e inclua um ou mais heteroátomos. Heteroaril também deve incluir sistemas de anel fundidos incluindo sistemas em que um ou mais anéis fundidos não contêm heteroátomos. Os grupos heteroaril incluem tipicamente um átomo do anel selecionado do enxofre, nitrogênio ou oxigênio. Exemplos de grupos de heteroaril incluem, entre outros, piridinil, pirazinil, pirimidinil, pirrolil, pirazolil, imidazolil, tiazolil, oxazolil, isooxazolil, tiadiazolil, oxadiazolil, tiofenil, furanil, quinolinil, isoquinolinil, benzimi- dazolil, benzooxazolil, quinoxalinil e similares. Os radicais heteroaril podem ser afixados a uma molécula de origem diretamente ou através de uma fração de ligação como um grupo alifático ou heteroátomo. Grupos heteroaril usados neste instrumento podem incluir ainda, opcionalmente, um ou mais grupos substituintes.
[00166] Como usado neste documento, "composto conjugado" se refere a quaisquer átomos, grupo de átomos ou grupo de átomos ligados adequados para serem usados como um grupo conjugado. Em determinadas modalidades, os compostos conjugados podem possuir ou conferir uma ou mais propriedades do composto ao qual estão afixados, incluindo, entre outros, propriedades de farmacodinâmica, farmacocinética, absorção, distribuição celular, absorção celular, carga e/ou depuração.
[00167] Como usado neste documento, salvo se indicado ou modificado de outro modo, o termo "de fita dupla" se refere a dois compostos oligoméricos separados que são hibridizados a outro. Esses compostos de fita dupla podem ter um ou mais nucleosídeos não hibridizantes em uma ou ambas as extremidades de uma ou ambas as fitas (overhangs) e/ou um ou mais nucleosídeos não-hibridizantes internos (incompatibilidades) desde que exista complementaridade suficiente para manter a hibridização em condições fisiologicamente relevantes.
[00168] Tal como aqui utilizado, "2'-O-metoxietil" (também 2'-MOE e 2'-O-(CH2)2-OCH3) refere-se a uma modificação de O-metoxi-etil de posição 2' de um anel de furosil. Um açúcar modificado em 2'-O- metoxietil é um açúcar modificado.
[00169] Conforme usado neste documento, "nucleotídeo de 2'-O- metoxietil" denota um nucleotídeo que compreende uma fração de açúcar modificada em 2'-O-metoxietil.
[00170] "Local alvo 3'" ou "local de interrupção 3'" referem-se ao nucleotídeo de um ácido nucleico alvo que é complementar ao nucleotídeo mais próximo de 3' de um composto anti-sense particular.
[00171] Como usados neste documento, o termo "local alvo 5'" ou "local de início 5" referem-se ao nucleotídeo de um ácido nucleico alvo que é complementar ao nucleotídeo mais extremo em 5' de um composto antisense específico.
[00172] Conforme usado neste documento, "5-metilcitosina" significa uma citosina modificada por um grupo metila ligado à posição 5'. Uma 5-metilcitosina é uma nucleobase modificada.
[00173] Como usado neste documento, o termo "cerca de" se refere a ± 10% de um valor. Por exemplo, se se constatar, "um marcador pode ser aumentado em cerca de 50%", estará implícito que o marcador pode ser aumentado entre 45% a 55%.
[00174] Conforme usado aqui, "agente farmacêutico ativo" significa a substância ou substâncias em uma composição farmacêutica que fornecem um benefício terapêutico quando administradas a um indivíduo. Por exemplo, em determinadas modalidades um oligonu- cleotídeo antisense direcionado para ANGPTL3 é um agente farmacêutico ativo.
[00175] Conforme usados aqui, "região alvo ativa" ou "região alvo" significam uma região à qual um ou mais compostos antisense ativos estão ligados.
[00176] Conforme usado aqui, "compostos antisense ativos" denota compostos antisense que reduzem os níveis de ácido nucleico alvo ou os níveis de proteína.
[00177] Conforme usado aqui, "adipogênese" significa que o desenvolvimento de células de gordura a partir de pré-adipócitos. "Lipogênese" significa a produção ou a formação de gordura, tanto degeneração gordurosa quanto infiltração gordurosa.
[00178] Como usados aqui, "adiposidade" ou "obesidade" se referem ao estado de ser obeso ou uma quantidade excessivamente alta de gordura corporal ou tecido adiposo em relação à massa magra corporal. A quantidade de gordura corporal inclui preocupação com ambos a distribuição de gordura em todo o corpo e o tamanho e massa dos depósitos de tecido adiposo. A distribuição de gordura corporal pode ser estimada por medidas de pregas cutâneas, razões da circunferência da cintura-quadril, ou técnicas tal como ultrassom, tomografia computa-dorizada, ou imageamento por ressonância magnética. De acordo com o Centro de Controle e Prevenção de Doenças, os indivíduos com um índice de massa corporal (IMC) igual ou superior a 30 são considerados obesos. O termo "Obesidade" como usado neste documento inclui condições onde há um aumento da gordura corporal, além da exigência física como resultado do acúmulo em excesso de tecido adiposo no corpo. O termo "obesidade" inclui, mas não está limitado a, as seguintes condições: obesidade do adulto; obesidade alimentar; obesidade endógena ou metabólica; obesidade endócrina; obesidade familiar; obesidade hiperinsulinar; obesidade hiperplásica-hipertrófica; hipogo- nadismo; hipotireoidismo; obesidade ao longo da vida; obesidade mórbida e obesidade exógena.
[00179] Conforme usado aqui, "administrado concomitantemente" refere-se à coadministração de dois agentes de qualquer forma na qual os efeitos farmacológicos de ambos são manifestados no paciente ao mesmo tempo. A administração concomitante não exige que os ambos os agentes sejam administrados numa única composição farmacêutica, na mesma forma de dosagem ou pela mesma via de administração. Os efeitos de ambos os agentes não precisam se manisfestar ao mesmo tempo. Os efeitos precisam apenas ser sobrepostos por um período de tempo e não precisam ser co-extensivos.
[00180] Conforme usado neste documento, "administrar" significa fornecer um agente a um animal e inclui, mas não está limitado a, administração por um profissional médico e autoadministração.
[00181] Como usado neste instrumento, "agente" significa uma substância ativa que pode fornecer um benefício terapêutico quando administrado a um animal. "Primeiro agente" significa um composto terapêutico da invenção. Por exemplo, um primeiro agente pode ser um oligonucleotídeo antisense que direciona ANGPTL3. "Segundo agente" significa um segundo composto terapêutico da invenção (por exemplo, um segundo oligonucleotídeo antisense que direciona ANGPTL3) e/ou um composto terapêutico de não ANGPTL3.
[00182] Como usado aqui, "melhora" refere-se a uma diminuição de pelo menos um indicador, sinal ou sintoma de uma doença, distúrbio ou condição associada. A gravidade dos indicadores pode ser determinada por medidas subjetivas ou objetivas, que são conhecidas pelos versados na técnica.
[00183] Tal como aqui utilizado, "ANGPTL3" significa qualquer ácido nucleico ou proteína de ANGPTL3.
[00184] Tal como aqui utilizado, "expressão de ANGPTL3" denota o nível de mRNA transcrito a partir do gene que codifica ANGPTL3 ou o nível de proteína traduzida a partir do mRNA. A expressão de ANGPTL3 pode ser determinada pelos métodos conhecidos na técnica tais como Northern ou Western blot.
[00185] Tal como aqui utilizado, "ácido nucleico de ANGPTL3" significa qualquer ácido nucleico que codifica ANGPTL3. Por exemplo, em determinadas modalidades, um ácido nucleico de ANGPTL3 inclui uma sequência de DNA que codifica ANGPTL3, uma sequência de RNA transcrita do DNA que codifica ANGPTL3 (incluindo o DNA genômico, compreendendo os íntrons e éxons), e uma sequência de mRNA que codifica ANGPTL3. "mRNA de ANGPTL3" significa um mRNA que codifica uma proteína de ANGPTL3.
[00186] Conforme utilizado aqui, "animal" refere-se a um humano ou animal não-humano, incluindo, mas não limitado a, camundongos, ratos, coelhos, cães, gatos, porcos e primatas não-humanos, incluindo, mas não se limitando a, macacos e chimpanzés.
[00187] Tal como aqui utilizado, "lipoproteína contendo apoB" significa qualquer lipoproteína que tenha apolipoproteína B como seu componente proteico e entende-se que inclua LDL, VLDL, IDL e a lipoproteína(a) e pode ser geralmente alvo do agente de redução de lípidos e terapias. "LDL contendo apoB-100" significa a LDL que contém a isoforma apoB-100.
[00188] Como usado neste documento, "aterosclerose" significa um endurecimento das artérias que afeta artérias de tamanho médio e grande e é caracterizado pela presença de depósitos graxos. Os depósitos graxos são denominados "ateromas" ou "placas", que consistem principalmente em colesterol e outras gorduras, cálcio e tecido da cicatriz, e danificam o forro das artérias.
[00189] Tal como aqui utilizado, "doença cardiometabólica" ou "distúrbio cardiometabólico" são as doenças ou distúrbios relativos tanto ao sistema cardiovascular quanto ao sistema metabólico. Exemplos de doenças ou distúrbios cardiometabólicos incluem, mas não se limitam a, diabetes e dislipidemia.
[00190] Tal como aqui utilizado, "co-administração" significa a administração de dois ou mais agentes a um indivíduo. Os dois ou mais agentes podem estar numa única composição farmacêutica, ou podem estar em composições farmacêuticas separadas. Cada um dos dois ou mais agentes pode ser administrado através de vias de administração iguais ou diferentes. A co-administração engloba a administração sequencial ou paralela.
[00191] Conforme usado aqui, "colesterol" é uma molécula de esterol encontrada nas membranas celulares de todos os tecidos animais. O colesterol deve ser transportado no plasma do sangue de um animal por lipoproteínas incluindo lipoproteína de densidade muito baixa (VLDL), lipoproteína de densidade intermediária (IDL), lipoproteína de baixa densidade (LDL) e lipoproteína de alta densidade (HDL). "Colesterol do plasma" se refere à soma de todo colesterol esterificado e/ou não- esterificado das lipoproteínas (VDL, IDL, LDL e HDL) presentes no plasma ou no soro.
[00192] Conforme usado aqui, "inibição de absorção de colesterol" significa um agente que inibe a absorção de colesterol exógeno obtido a partir de dieta.
[00193] Como usado neste instrumento, "doença cardíaca coronariana (CHD)" significa estreitar os vasos sanguíneos pequenos que fornecem sangue e oxigênio ao coração, que é frequentemente um resultado da aterosclerose.
[00194] Conforme usado aqui, "diabetes mellitus" ou "diabetes" é uma síndrome caracterizada por metabolismo desordenado e teor de açúcar no sangue altamente elevado (hiperglicemia) resultantes de níveis insuficientes de insulina ou sensibilidade à insulina reduzida. Os sintomas característicos são a produção de urina excessiva (poliúria) devido aos altos níveis de glicose no sangue, sede excessiva e ingestão de líquidos aumentada (polidipsia) tentativa de compensar o aumento da frequência urinária, visão turva devido a efeitos do alto nível de glicose no sangue elevada na óptica do olho, perda de peso inexplicada e letargia.
[00195] Conforme usado aqui, "dislipidemia diabética" ou "diabetes tipo 2 com dislipidemia" significa uma condição caracterizada pelo diabetes tipo 2, HDL-C reduzido, triglicerídeos elevados, e pequenas partículas de LDL densas elevadas.
[00196] Conforme usado aqui, "diluente" significa um ingrediente em uma composição que não tem atividade farmacológica, mas é farmaceuticamente necessário ou desejável. Por exemplo, o diluente em uma composição injetada pode ser um líquido, por exemplo, solução salina.
[00197] Como usado neste instrumento, "dislipidemia" refere-se a um distúrbio do metabolismo lipídico e/ou de lipoproteína, incluindo a superprodução ou deficiência de lipídios e/ou de lipoproteína. As dislipidemias podem se manifestar pela elevação de lipídios tais como colesterol e triglicerídeos, bem como lipoproteínas, tal como colesterol da lipoproteína de baixa densidade (LDL).
[00198] Como usado neste documento, "unidade de dosagem" significa uma forma na qual um agente farmacêutico é fornecido, por exemplo, pílula, comprimido, ou outra unidade de dosagem conhecida na técnica. Em determinadas modalidades, uma unidade de dosagem é um frasco contendo oligonucleotídeo antisense liofilizado. Em determinadas modalidades, uma unidade de dosagem é um frasco contendo oligonucleotídeo antisense reconstituído.
[00199] Conforme usado aqui, "dose" significa uma quantidade especificada de um agente farmacêutico fornecido numa administração única ou em um período de tempo especificado. Em certas modalidades, uma dose pode ser administrada em um, dois, ou mais bolos, comprimidos ou injeções. Por exemplo, em certas modalidades onde a administração subcutânea é desejada, a dose desejada requer um volume não facilmente acomodado por uma única injeção, portanto, duas ou mais injeções podem ser usadas para atingir a dose desejada. Em determinadas modalidades, o agente farmacêutico é administrado por infusão durante um período de tempo estendido ou continuamente. As doses podem ser indicadas como a quantidade de agente farmacêutico por hora, dia, semana ou mês. As doses podem ser expressas em mg/kg ou g/kg.
[00200] Conforme usado aqui, "quantidade eficaz" ou "quantidade terapeuticamente eficaz" significa a quantidade de agente farmacêutico ativo suficiente para efetuar um resultado fisiológico desejado em um indivíduo em necessidade do agente. A quantidade eficaz pode variar entre os indivíduos dependendo da saúde e condição física do indivíduo a ser tratado, o grupo taxonômico dos indivíduos a serem tratados, a formulação da composição, a avaliação da condição médica do indivíduo, e outros fatores relevantes.
[00201] Como usado neste instrumento, "glicose" é um monossacarídeo usado pelas células como uma fonte de energia e intermediário metabólico. "Glicose do plasma" se refere à glicose presente no plasma.
[00202] Como usado neste instrumento, "lipoproteína-C de alta densidade" ou "HDL-C" significa colesterol associado a partículas de lipoproteína de alta densidade. A concentração de HDL-C no soro (ou plasma) é normalmente quantificada em mg/dL ou nmol/L. "HDL-C do soro" e "HDL-C do plasma" significam HDL-C no soro e no plasma, respectivamente.
[00203] Como usado neste instrumento, "inibidor da HMG-CoA redutase" significa um agente que age através da inibição da enzima HMG-CoA redutase, tal como atorvastatina, rosuvastatina, fluvastatina, lovastatina, pravastatina e sinvastatina.
[00204] Como usado neste documento, "hipercolesterolemia" significa uma condição caracterizada pelo colesterol elevado ou colesterol circulante (do plasma), LDL-colesterol e VLDL-colesterol, como por diretrizes do Relatório de Painel Perito do Programa Educacional de Colesterol Nacional (NCEP) de Detecção, Avaliação do Tratamento de colesterol alto em adultos (vide, Arch. Int. Med. (1988) 148, 36-39).
[00205] Como usado neste instrumento, "hiperlipidemia" ou "hiperlipidemia" é uma condição caracterizada por lipídios séricos elevados ou lipídios circulantes (plasma). Esta condição manifesta uma concentração anormalmente alta de gorduras. As frações de lipídio no sangue circulante são colesterol, lipoproteínas de baixa densidade, lipoproteínas de densidade muito baixa e triglicerídeos.
[00206] Como usado neste documento, "hipertrigliceridemia" significa uma condição caracterizada por níveis elevados de triglicerídeos.
[00207] Conforme utilziado neste documento, "identificar" ou "selecionar um indivíduo com doença metabólica ou cardiovascular" significa identificar ou selecionar um sujeito propenso a ou que foi diagnosticado com uma doença metabólica, uma doença cardiovascular, ou uma síndrome metabólica; ou identificar ou selecionar um sujeito que tem qualquer sintoma de uma doença metabólica, doença cardiovascular, ou síndrome metabólica incluindo, mas não limitado a, hipercolesterolemia, hiperglicemia, hiperlipidemia, hipertrigliceridemia, resistência à insulina aumentada pela hipertensão, sensibilidade à insulina reduzida, peso corporal acima do normal e/ou teor de gordura corporal acima do normal ou qualquer combinação dos mesmos. Essa identificação pode ser realizada por qualquer método, incluindo, mas não limitado a, testes ou avaliações clínicas padrão, tais como medição do soro ou colesterol circulante (plasma), medição do soro ou glicose no sangue circulante (plasma), medição do soro ou triglicerídeos circulantes (plasma), medição da pressão arterial, medição do teor de gordura corporal, medição do peso corporal e similares.
[00208] Tal como aqui utilizado, "identificar" ou "seleção de um indivíduo diabético" significa identificar ou selecionar um indivíduo que foi identificado como diabético ou identificar ou selecionar um indivíduo com algum sintoma de diabetes (tipo 1 ou 2), como, mas sem se limitar a, glicemia em jejum de pelo menos 110 mg/Dl, glicosúria, poliúria, polidipsia, resistência a insulina aumentada e/ou sensibilidade a insulina diminuída.
[00209] Como usado aqui, "identificar" ou "selecionar um indivíduo obeso" significa identificar ou selecionar um indivíduo diagnosticado como obeso ou identificar ou selecionar um indivíduo com um IMC acima de 30 e/ou com uma circunferência de cintura superior a 102 cm em homens e superior a 88 cm em mulheres.
[00210] Tal como aqui utilizado, "identificar" ou "selecionar um indivíduo com dislipidemia" significa identificar ou selecionar um indivíduo diagnosticado com um distúrbio de metabolismo de lipídios e/ou liproproteínas, incluindo superprodução ou deficiência de lipídios e/ou lipoproteínas. As dislipidemias podem se manifestar pela elevação de lipídios tais como colesterol e triglicerídeos, bem como lipoproteínas, tal como colesterol da lipoproteína de baixa densidade (LDL).
[00211] Tal como aqui utilizado, "identificar" ou "selecionar um indivíduo com adiposidade aumentada" significa identificar ou selecionar um indivíduo com uma quantidade aumentada de gordura corporal (ou adiposidade) que inclui o cuidado com um ou ambos dentre a distribuição de gordura pelo corpo e o tamanho e a massa dos depósitos no tecido adiposo. A distribuição de gordura corporal pode ser estimada por medidas de pregas cutâneas, razões da circunferência da cintura-quadril, ou técnicas tal como ultrassom, tomografia computadorizada ou imageamento por ressonância magnética. De acordo com o Centro de Controle e Prevenção de Doenças, os indivíduos com um índice de massa corporal (IMC) igual ou superior a 30 são considerados obesos.
[00212] Como usado neste documento, "resultado cardiovascular melhorado" significa uma redução na ocorrência de eventos cardiovasculares adversos, ou o risco da mesma. Exemplos de eventos cardiovasculares adversos incluem, sem limitação, morte, reinfarto, derrame, choque cardiogênico, edema pulmonar, parada cardíaca e disritmia atrial.
[00213] Conforme usado aqui, "imediatamente adjacente" significa que não há nenhum elemento interveniente entre os elementos imediatamente adjacentes.
[00214] Conforme usados aqui, "indivíduo", "paciente" ou "animal" denota um animal humano ou não-humano selecionado para tratamento ou terapia.
[00215] Como usado neste documento, "resistência à insulina" é definida como a condição na qual quantidades normais de insulina são inadequadas para produzir uma resposta normal à insulina a partir das células, por exemplo: de gordura, dos músculos e/ou do fígado. Resistência à insulina nas células de gordura resulta na hidrólise de triglicerídeos armazenados, que eleva os ácidos graxos livres no plasma sanguíneo. Resistência à insulina no músculo reduz a absorção de glicose, considerando que a resistência à insulina no fígado reduz o armazenamento de glicose, com ambos os efeitos, servindo para elevar a glicose no sangue. Altos níveis plasmáticos de insulina e glicose devido à resistência à insulina, muitas vezes, leva à síndrome metabólica e diabetes tipo 2.
[00216] Como usado neste documento, "sensibilidade à insulina" é uma medida do quão eficazmente um indivíduo processa a glicose. Um indivíduo que tem sensibilidade à insulina alta efetivamente processa a glicose, considerando que um indivíduo com sensibilidade à insulina baixa não processa efetivamente a glicose.
[00217] Tal como aqui utilizado, "administração intravenosa" significa a administração em uma veia.
[00218] Como usado neste documento, "redução de lipídios" significa uma redução em um ou mais lipídios em um indivíduo. A redução de lipídios pode ocorrer com uma ou mais doses ao longo do tempo.
[00219] Tal como aqui utilizado, "agente de redução de lípidos" significa um agente, por exemplo, um modulador específico de ANGPTL3, fornecido a um indivíduo para atingir uma redução de lípidos no paciente. Em determinadas modalidades, um agente de redução de lipídios é fornecido para reduzir um ou mais dentre apoB, apoC-III, colesterol total, LDL-C, VLDL-C, IDL-C, não-HDL-C, triglicerídeos, pequenas partículas densas de LDL e LP (a) em um indivíduo.
[00220] Como usado neste documento, "terapia de redução de lipídios" significa um regime terapêutico fornecido a um sujeito para reduzir um ou mais lipídios em um sujeito. Em determinadas modalidades, uma terapia de redução de lipídios é fornecida para reduzir uma ou mais das apoB, apoC-III, colesterol total, LDL-C, VLDL-C, IDL-C, não-HDL-C, triglicerídeos, pequenas partículas densas de LDL e LP (a) em um indivíduo.
[00221] Como usado neste documento, "lipoproteína", tais comoVLDL, LDL e HDL, se refere a um grupo de proteínas encontradas no soro, no plasma e no linfonodo e é importante para o transporte de lipídios. A composição química de cada lipoproteína difere pelo fato de que a HDL tem uma proporção mais alta de proteína versus lipídio, considerando que a VLDL tem uma proporção mais baixa de proteína versus lipídio.
[00222] Como usado neste documento, "lipoproteína-colesterol de baixa densidade (LDL-C)" significa colesterol transportado em partículas de lipoproteína de baixa densidade. A concentração de LDL-C no soro (ou plasma) é normalmente quantificada em mg/dL ou nmol/L. "LDL-C do soro" e "LDL-C do plasma" significam LDL-C no soro e no plasma, respectivamente.
[00223] Como usado neste documento, "principais fatores de risco" referem-se aos fatores que contribuem para um alto risco de uma doença ou condição específica. Em determinadas modalidades, os principais fatores de risco para doença cardíaca coronariana incluem, sem limitação, tabagismo, hipertensão, HDL-C baixo, histórico familiar de doença cardíaca coronariana, idade, e outros fatores divulgados neste documento.
[00224] Como usado neste documento, "distúrbio metabólico" ou "doença metabólica" se refere a uma condição caracterizada por uma alteração ou distúrbio na função metabólica. "Doença metabólica" e "metabolismo" são termos conhecidos na técnica e geralmente incluem toda a gama de processos bioquímicos que ocorrem dentro de um organismo vivo. Distúrbios metabólicos incluem, mas não estão limitados a, hiperglicemia, pré-diabetes, diabetes (tipo 1 e tipo 2), obesidade, resistência à insulina, síndrome metabólica e dislipidemia devido ao diabetes tipo 2.
[00225] Como usado neste documento, "síndrome metabólica" significa uma condição caracterizada por um agrupamento de lipídios e fatores de risco cardiovasculares não-lipídicos de origem metabólica. Em certas modalidades, a síndrome metabólica é identificada pela presença de 3 dos seguintes fatores: circunferência da cintura maior do que 102 cm em homens ou maior do que 88 cm em mulheres; triglicerídeo do soro de pelo menos 150 mg/dL; HDL-C inferior a 40 mg/dL em homens ou inferior a 50 mg/dL em mulheres; pressão arterial de pelo menos 130/85 mmHg; e glicemia de jejum de pelo menos 110 mg/dL. Estes determinantes podem ser facilmente medidos na prática clínica (JAMA, 2001, 285: 2486-2497).
[00226] Conforme usado aqui, "dislipidemia mista" significa umacondição caracterizada por colesterol elevado e triglicerídeos elevados.
[00227] Conforme usado aqui, "inibidor de MTP" significa um agente que inibe a enzima, proteína de transferência de triglicerídeos microssômicos.
[00228] Conforme usados aqui, "doença hepática gordurosa não alcoólica" ou "NAFLD" significam uma condição caracterizada pela inflamação gordurosa do fígado que não é devido ao uso excessivo de álcool (por exemplo, consumo de álcool, de mais de 20 g/dia). Em determinadas modalidades, NAFLD está relacionada à resistência à insulina e síndrome metabólica. NAFLD engloba um espectro de doença que vai desde o acúmulo de triglicerídeo simples em hepatócitos (esteatose hepática) à esteatose hepática com inflamação (esteato-hepatite), fibrose e cirrose.
[00229] Conforme usado aqui, "esteato-hepatite não alcoólica" (NASH) ocorre a partir da progressão de NAFLD além da deposição de triglicerídeos. Um "segundo hit" capaz de induzir necrose, inflamação, e fibrose é necessário para o desenvolvimento de NASH. Candidatos para o segundo hit podem ser agrupados em categorias amplas: fatores que causam um aumento no estresse oxidativo e fatores que promovem a expressão de citocinas pró-inflamatórias. Sugeriu-se que o aumento de triglicerídeos hepáticos leva ao aumento do estresse oxidativo em hepatócitos de animais e humanos, indicando uma potencial relação de causa e efeito entre a acumulação hepática de triglicerídeos, estresse oxidativo e a progressão da esteatose hepática para NASH (Browning e Horton, J Clin Invest, 2004, 114, 147-152). A hipertrigliceridemia e a hipergraxoacidemia podem levar à acumulação de triglicerídeos nos tecidos periféricos (Shimamura et al., Biochem Biophys Res Commun, 2004, 322, 10801085).
[00230] Conforme utilizado aqui, o termo "ácido nucleico" refere-se a moléculas compostas por nucleotídeos monoméricos. Um ácido nucleico inclui ácidos ribonucleicos (RNA), ácidos desoxirribonucleicos (DNA), ácidos nucleicos de fita única, ácidos nucleicos de fita dupla, pequenos ácidos ribonucleicos de interferência (siRNA) e microRNAs (miRNA). Um ácido nucleico também pode compreender uma combinação destes elementos em uma única molécula.
[00231] Tal como aqui utilizado, "administração parentérica" significa administração por outra forma que não através do trato digestivo. A administração parentérica inclui administração tópica, administração subcutânea, administração intravenosa, administração intramuscular, administração intra-arterial, administração intraperitoneal, ou administração intracraniana, por exemplo, administração intratecal ou intracerebroventricular. A administração pode ser contínua ou crônica ou curta ou intermitente.
[00232] Como usado neste documento, "agente farmacêutico" significa uma substância que proporciona um benefício terapêutico quando administrada a um indivíduo. Por exemplo, em determinadas modalidades um oligonucleotídeo antisense direcionado para ANGPTL3 é um agente farmacêutico.
[00233] Conforme usado aqui, "composição farmacêutica" ou "composição" significa uma mistura de substâncias adequadas para a administração a um indivíduo. Por exemplo, uma composição farmacêutica pode compreender um ou mais agentes ativos e uma solução aquosa estéril.
[00234] Conforme usado aqui, "transportador farmaceuticamente aceitável" significa um meio ou diluente que não interfere na estrutura ou na função do oligonucleotídeo. Alguns desses carreadores permitem que as composições farmacêuticas sejam formuladas como, por exemplo, comprimidos, pílulas, drágeas, cápsulas, líquidos, géis, xaropes, pastas fluidas, suspensão e pastilhas para a ingestão oral por parte de um indivíduo. Alguns desses carreadores permitem que composições farmacêuticas sejam formuladas para injeção ou infusão. Por exemplo, um transportador farmacêuticamente aceitável pode ser uma solução aquosa estéril.
[00235] Conforme usado aqui, "sais farmaceuticamente aceitáveis" significa sais fisiologicamente e farmaceuticamente aceitáveis de compostos antisense, ou seja, sais que mantêm a atividade biológica desejada do composto pai e não transmitem efeitos toxicológicos indesejáveis aos mesmos.
[00236] Conforme usado aqui, "porção" significa um número definido de nucleobases contíguas (isto é, ligadas) de um ácido nucleico. Em determinadas modalidades, uma porção é um número definido de nucleobases contíguas de um ácido nucleico alvo. Em determinadas modalidades, uma porção é um número definido de nucleobases contíguas de um composto antisense.
[00237] Conforme usado aqui, "prevenir" ou "prevenção" se refere ao retardamento ou prevenção do aparecimento ou do desenvolvimento de uma doença, distúrbio, ou condição por um período de tempo de minutos a indefinidamente. Prevenir também significa reduzir o risco de desenvolvimento de uma doença, distúrbio, ou condição.
[00238] Conforme usado aqui, "efeitos colaterais" significa respostas fisiológicas atribuíveis a um tratamento diferente dos efeitos desejados. Em determinadas modalidades, os efeitos colaterais incluem reações no local de injeção, anormalidades do teste de função hepática, anormalidades da função renal, toxicidade hepática, toxicidade renal, anormalidades do sistema nervoso central, miopatias e mal-estar. Por exemplo, níveis de aminotransferase aumentados no soro podem indicar toxicidade hepática ou anormalidade da função hepática. Por exemplo, a bilirrubina aumentada pode indicar toxicidade hepática ou anormalidade da função hepática.
[00239] Como usado neste documento, "estatina" significa um agente que inibe a atividade da redutase HMG-CoA.
[00240] Como usado neste documento, "administração subcutânea" significa a administração logo abaixo da pele.
[00241] Como usado neste documento, "direcionamento" ou "direcionado" significa o processo de projeto e seleção de um composto antisense que se hibridizará em um ácido nucleico alvo e induzir um efeito desejado.
[00242] Conforme usados aqui, "ácido nucleico alvo", "RNA alvo" e "transcrição de RNA alvo", todos se referem a um ácido nucleico capaz de ser direcionado por compostos antisense.
[00243] Como usado neste documento, "região alvo" é definida como uma porção do ácido nucleico alvo que tem pelo menos uma estrutura, função ou característica identificável.
[00244] Tal como aqui utilizado, "segmento alvo" significa a sequência de nucleotídeos de um ácido nucleico alvo à qual um ou mais compostos anti-sense tem como alvo. "Local alvo 5'" ou "local de início 5'" referem-se ao nucleotídeo mais próximo de 5' de um segmento alvo. "Local alvo 3'" ou "local de interrupção 3'" referem-se ao nucleotídeo mais próximo de 3' de um segmento alvo.
[00245] Conforme usado neste documento, "quantidade terapeuticamente eficaz" significa uma quantidade de um agente farmacêutico que fornece um benefício terapêutico a um indivíduo.
[00246] Como usado neste documento, "mudança terapêutica do estilo de vida" significa mudanças na dieta e no estilo de vida pretendidas para diminuir a massa de gordura/tecido adiposo e/ou o colesterol. Essa mudança pode reduzir o risco de desenvolvimento de doença cardíaca e pode incluir recomendações para ingestão de calorias diárias totais, gordura total, gordura saturada, gordura poli- insaturada, gordura monoinsaturada, carboidrato, proteína, colesterol, fibra insolúvel, bem como as recomendações para atividade física.
[00247] Como usado neste documento, "triglicerídeo" significa um lipídio ou gordura neutra consistindo em glicerol combinado com três moléculas de ácido graxo.
[00248] Conforme usados aqui, "diabetes tipo 2," (também conhecido como "diabetes mellitus tipo 2" ou "diabetes mellitus, tipo 2", e antigamente denominado "diabetes mellitus tipo 2", "diabetes não dependente de insulina (NIDDM)", "diabetes", ou "diabetes do adulto") é um distúrbio metabólico que é principalmente caracterizado por resistência à insulina, deficiência de insulina relativa e hiperglicemia.
[00249] Conforme usado aqui, "tratar" se refere à administração de uma composição farmacêutica para efetuar uma alteração ou melhora de uma doença, distúrbio ou condição.
Determinadas Modalidades
[00250] Em certas modalidades aqui divulgadas, ANGPTL3 tem a sequência conforme estabelecido no GenBank com n° de acesso NM_014495.2 (incorporado aqui como SEQ ID NO: 1). Em certas modalidades, ANGPTL3 possui a sequência estebelecida no GenBank com n° de acesso NT_032977.9 nucleotídeos 33032001 a 33046000 (aqui incorporados como SEQ ID NO: 2).
[00251] Certas modalidades divulgadas aqui preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos com uma sequência de nucleobase compreendendo ao menos 8 nucleobases contínuas complementares a uma porção de comprimento igual das SEQ ID NOs: 1- 2.
[00252] Em determinadas modalidades, um composto compreende um siRNA ou um oligonucleotídeo antisense direcionado à ANGPTL3 conhecidA na técnica e a um grupo de conjugados aqui descrito. Exemplos de oligonucleotídeos antisense direcionados à ANGPTL3 adequados para conjugação incluem, entre outros, aqueles divulgados em US 8,653,047 (WO 2011/085271), que é incorporado por referência na sua totalidade. Em determinadas modalidades, um composto compreende um oligonucleotídeo antisense que possui uma sequência de quaisquer SEQ ID NOs 34-111 divulgadas em US 8.653.047 e um grupo conjugado aqui descrito. Em determinadas modalidades, um composto compreende uma fita antisense ou sense de siRNA que possui uma sequência de quaisquer SEQ ID NOs 34-111 divulgadas em US 8.653.047 e um grupo conjugado aqui descrito. As sequências de nucleobases de todas as SEQ ID NOs referenciadas anteriormente estão incorporadas por referência neste documento.
[00253] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados em um comprimento direcionado à ANGPTL3. A ANGPTL3 alvo pode ter uma sequência selecionada a partir de qualquer uma das SEQ ID NOs: 1 -2.
[00254] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases compreendendo uma porção de pelo menos 8 nucleobases contíguas complementares a uma porção de comprimento igual das nucleobases de 1140-1159 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1. Em certas modalidades, o oligonucleotídeo modificado é de pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases de 1140 a 1159 da SEQ ID NO: 1.
[00255] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases complementares às nucleobases de 1140 a 1159 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1.
[00256] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases compreendendo uma porção de pelo menos 8 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases de 1907-1926 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1. Em certas modalidades, o oligonucleotídeo modificado é de pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases de 1907 a 1926 da SEQ ID NO: 1.
[00257] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases complementares às nucleobases de 1907 a 1926 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1.
[00258] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases compreendendo uma porção de pelo menos 8 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases de 147-162 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1. Em certas modalidades, o oligonucleotídeo modificado é de pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15 ou 16 nucleobases contíguas complementares a uma porção de comprimento igual de nucleobases de 147 a 162 da SEQ ID NO: 1.
[00259] Certas modalidades divulgadas aqui preveem compostos ou composições compreendendo um oligonucleotídeo modificado e um grupo de conjugados, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreendendo uma sequência de nucleobases complementares às nucleobases de 147 a 162 da SEQ ID NO: 1, em que a sequência de nucleobases do oligonucleotídeo modificado é pelo menos 80% complementar à SEQ ID NO: 1.
[00260] Em certas modalidades, o oligonucleotídeo modificado consiste em 12 a 30, 15 a 30, 18 a 24, 19 a 22, 13 a 25, 14 a 25, 15 a 25 ou 16 a 24 nucleosídeos ligados. Em certas modalidades, o oligonucleotídeo modificado é constituído por 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 ou 30 nucleosídeos ligados ou a um intervalo definido por algum desses dois valores. Em certas modalidades, o oligonucleotídeo modificado tem comprimento de 16 nucleosídeos ligados. Em certas modalidades, o oligonucleotídeo modificado tem comprimento de 20 nucleosídeos ligados.
[00261] Em certas modalidades, o oligonucleotídeo modificado compreende uma sequência de nucleobases que compreende uma porção de pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19, ou 20 nucleobases contíguas complementares a uma porção de comprimento igual da SEQ ID NO: 1 ou 2.
[00262] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, em que o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e tendo uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, menos 19 ou 20 nucleobases contíguas de uma sequência de nucleobases selecionada a partir de qualquer uma das ID SEQ NOs: 15-27, 30-73, 75-85, 87-232, 238, 240-243, 245-247, 249-262, 264397, 399-469, 471-541, 543-600, 604-760, 762-819, 821-966, 968971, 973-975, 977-990, 992-1110, 1112-1186, 1188- 1216, 12181226, 1228-1279, 1281-1293, 1295-1304, 1306-1943, 1945-1951,1953-1977, 1979-1981, 1983-2044, 2046-2097, 2099-2181, 21832232, 2234-2238, 2240-2258, 2260-2265, 2267-2971, 2973-2976,2978-4162, 4164-4329, 4331-4389, 4391-4394, 4396-4877.
[00263] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas das sequências de nucleobases da SEQ ID NO: 77. Em certas modalidades, o composto compreende ISIS 563580 e um grupo de conjugados. Em certas modalidades, o composto consiste em ISIS 563580 e um grupo conjugado.
[00264] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado ISIS 563580 com 5'-X, onde X é um grupo conjugado que compreende GalNAc. Em determinadas modalidades, o composto antisense consiste no oligonucleotídeo modificado ISIS 563580 com 5'-X, onde X é um grupo conjugado que compreende GalNAc.
[00265] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado conjugado ISIS 703801. Em determinadas modalidades, o composto antisense consiste no oligonucleotídeo modificado conjugado ISIS 703801.
[00266] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela estrutura a seguir. Em determinadas modalidades, o composto antisense compreende o oligonucleotídeo modificado conjugado ISIS 703802. Em determinadas modalidades, o composto antisense consiste no oligonucleotídeo modificado conjugado ISIS 703802.
[00267] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela estrutura a seguir. Em certas modalidades, o composto antisense compreende um oligonucleotídeo modificado com a sequência de nucleobases da SEQ ID NO: 77 com uma 5'-GalNAc com variabilidade nas modificações de açúcar das asas. Em certas modalidades, o composto antisense consiste em um oligonucleotídeo modificado com a sequência de nucleobases da SEQ ID NO: 77 com uma 5'-GalNAc com variabilidade nas modificações de açúcar das asas.
[00268] em que ou R1 é -OCH2CH2OCH3 (MOE) e R2 é H; ou R1 e R2 em conjunto, formam uma ponte, em que R1 é -O- e R2 é -CH2-, -CH (CH3) -, ou -CH2CH2-, e R1 e R2 estão diretamente ligados, de tal modo que a ponte resultante é selecionada a partir de: -O-CH2-, -O-CH(CH3)- , e -O-CH2CH2-;
[00269] e para cada par de R3 e R4 no mesmo anel, independentemente para cada anel: R3 é selecionado dentre H e - OCH2CH2OCH3 e R4 é H; ou R3 e R4 formam, em conjunto, uma ponte, onde R3 is -O- e R4 é -CH2-, -CH(CH3)- ou -CH2CH2- e R3 e R4 estão diretamente conectados, de maneira que a ponte resultante é selecionada dentre: -O-CH2-, -O-CH(CH3)- e -O-CH2CH2-;
[00270] e R5 é selecionado a partir de H e -CH3;
[00271] e Z é selecionado dentre S- e O-.
[00272] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 20. Em certas modalidades, o composto compreende ISIS 544199 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 544199 e um grupo conjugado.
[00273] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 35. Em certas modalidades, o composto compreende ISIS 560400 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 560400 e um grupo conjugado.
[00274] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 90. Em certas modalidades, o composto compreende ISIS 567233 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 567233 e um grupo conjugado.
[00275] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 93. Em certas modalidades, o composto compreende ISIS 567320 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 567320 e um grupo conjugado.
[00276] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15, pelo menos 16, pelo menos 17, pelo menos 18, pelo menos 19 ou 20 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 94. Em certas modalidades, o composto compreende ISIS 567321 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 567321 e um grupo conjugado.
[00277] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15 ou pelo menos 16 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 110. Em certas modalidades, o composto compreende ISIS 559277 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 559277 e um grupo conjugado.
[00278] Certas modalidades aqui divulgadas preveem compostos ou composições que compreendem um oligonucleotídeo modificado e um grupo conjugado, onde o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e com uma sequência de nucleobases que compreende pelo menos 8, pelo menos 9, pelo menos 10, pelo menos 11, pelo menos 12, pelo menos 13, pelo menos 14, pelo menos 15 ou pelo menos 16 nucleobases contíguas da sequência de nucleobases da SEQ ID NO: 114. Em certas modalidades, o composto compreende ISIS 561011 e um grupo conjugado. Em certas modalidades, o composto consiste em ISIS 561011 e um grupo conjugado.
[00279] Em certas modalidades, a sequência de nucleobases do oligonucleótido modificado é pelo menos 70%, 75%, 80%, 85%, 90%, 95% ou 100% complementar a qualquer uma dentre as SEQ ID NO: 12, conforme medido na totalidade do oligonucleotídeo modificado.
[00280] Em certas modalidades, o composto aqui descrito é um oligonucleotídeo de cadeia simples. Em certas modalidades, o composto aqui descrito é um oligonucleotídeo de cadeia simples modificado.
[00281] Em certas modalidades, pelo menos uma ligação internucleosídica do referido oligonucleotídeo modificado é uma ligação de internucleosídeos modificada. Em certas modalidades, a ligação internucleosídica modificada é uma ligação internucleosídica de fosforotioato. Em determinadas modalidades, pelo menos 1, pelo menos 2, pelo menos 3, pelo menos 4, pelo menos 5, 6, pelo menos, pelo menos 7, 8, pelo menos, pelo menos 9 ou pelo menos 10 ligações internucleosídicas do referido oligonucleotídeo modificado são ligações internucleosídicas de fosforotioato. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato. Em determinadas modalidades, o oligonucleotídeo modificado compreende pelo menos 1, pelo menos 2, pelo menos 3, pelo menos 4, pelo menos 5, 6, pelo menos, pelo menos 7, 8, pelo menos, pelo menos 9 ou pelo menos 10 ligações internucleosídicas do referido oligonucleotídeo modificado são ligações internucleosídicas de fosforo fosforotioato. Em determinadas modalidades, cada ligação internucleosídica do oligonucleotídeo modificado é selecionada de uma ligação internucleosídica de fosfodiéster e uma ligação internucleosídica de fosforotioato.
[00282] Em determinadas modalidades, pelo menos um nucleosídeo o oligonucleotídeo modificado compreende um açúcar modificado. Em certas modalidades, pelo menos o açúcar modificado é um açúcar bicíclico. Em certas modalidades, pelo menos um açúcar modificado compreende uma 2'-O-metoxietila, uma etila restrita, um 3'-flúor-HNA ou uma ponte de 4'-(CH ) -O-2', em que n é 1 ou 2.
[00283] Em determinadas modalidades, pelo menos um nucleosídeo do referido oligonucleotídeo modificado compreende uma nucleobase modificada. Em certas modalidades, a nucleobase modificada é uma 5- metilcitosina.
[00284] Determinadas modalidades aqui divulgadas proporcionam compostos ou composições que compreendem um oligonucleotídeo e um grupo de conjugados, onde o oligonucleotídeo politicado possui: a) um segmento de lacuna consistindo em desoxinucleosídeos ligados; b) segmento de asa 5' que consiste em nucleosídeos ligados; e c) um segmento 3' de asa que consiste em nucleosídeos ligados. O segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3' e cada nucleosídeo de cada segmento de asa compreende um açúcar modificado.
[00285] Em certas modalidades, o oligonucleotídeo modificado consiste em 12 a 30 nucleosídeos ligados e compreende: um segmento de lacuna consistindo em desoxinucleosídeos ligados; um segmento 5' de asa que consiste de nucleosídeos ligados; um 3' segmento de asa que consiste de nucleosídeos ligados, em que o segmento gap está posicionado imediatamente entre as extremidades 5' e o segmento de asa 3' e em que cada nucleosídeo de cada segmento de asa compreende um açúcar modificado.
[00286] Em certas modalidades, os compostos ou composições aqui divulgados compreendem um oligonucleotídeo modificado por um grupo conjugado, onde o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas da SEQ ID NO:1-2, onde o oligonucleotídeo modificado compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00287] Em certas modalidades, o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5- metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00288] Em certas modalidades, os compostos ou composições aqui divulgados compreendem um oligonucleotídeo modificado por um grupo conjugado, onde o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas de uma sequência de nucleobases selecionadas da SEQ ID NO:77, onde o oligonucleotídeo modificado compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O- metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00289] Em certas modalidades, o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados com a sequência de nucleobases da SEQ ID NO: 77 e compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00290] Em certas modalidades, os compostos ou composições aqui divulgados compreendem é um oligonucleotídeo modificado por um grupo conjugado, onde o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas de uma sequência de nucleobases selecionadas da SEQ ID NO:20, onde o oligonucleotídeo modificado compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O- metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00291] Em certas modalidades, o oligonucleotídeo modificado consiste em 20 nucleosídeos ligados com a sequência de nucleobases da SEQ ID NO: 20 e compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em cinco nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada nucleosídeo de cada segmento de asa compreende um açúcar de 2'-O-metoxietila; onde cada ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00292] Em certas modalidades, os compostos ou composições aqui divulgados compreendem um oligonucleotídeo modificado por um grupo conjugado, onde o oligonucleotídeo modificado consiste em 16 nucleosídeos ligados e com uma sequência de nucleobases consistindo em pelo menos 8 nucleobases contíguas de uma sequência de nucleobases da SEQ ID NO:110, onde o oligonucleotídeo modificado compreende: um segmento de lacuna consistindo em dezdesoxinucleosídeos ligados; um segmento de asa 5' consistindo em cinco nucleosídeos ligados; e um segmento de asa 3' consistindo em três nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada segmento de asa compreende pelo menos um açúcar de 2'-O- metoxietila e pelo menos um açucar de cEt; onde pelo menos uma ligação internucleosídica é uma ligação de fosforotioato e em que cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00293] Em certas modalidades, o oligonucleotídeo modificado consiste em 16 nucleosídeos ligados com a sequência de nucleobases da SEQ ID NO: 110 e compreende: um segmento de lacuna consistindo em dez desoxinucleosídeos ligados; um segmento de asa 5' consistindo em três nucleosídeos ligados; e um segmento de asa 3' consistindo em três nucleosídeos ligados; onde o segmento de lacuna é posicionado entre o segmento de asa 5' e o segmento de asa 3'; onde cada segmento de asa compreende pelo menos um açúcar de 2'-O- metoxietila e pelo menos um açúcar de cEt; onde pelo menos uma ligação internucleosídica é uma ligação de fosforotioato e onde cada resíduo de citosina é uma 5-metilcitosina. Em certas modalidades, cada ligação internucleosídica é uma ligação internucleosídica de fosforotioato.
[00294] Em certas modalidades, o grupo conjugado está ligado ao oligonucleotídeo modificado na extremidade 5' do oligonucleotídeo modificado. Em certas modalidades, o grupo de conjugado está ligado ao oligonucleotídeo modificado na extremidade 3' do oligonucleotídeo modificado.
[00295] Em certas modalidades, o grupo conjugado compreende exatamente um ligante. Em determinadas modalidades, um grupo conjugado compreende um ou mais ligantes. Em certas modalidades, o grupo conjugado compreende exatamente dois ligantes. Em determinadas modalidades, um grupo conjugado compreende dois ou mais ligantes. Em determinadas modalidades, um grupo conjugado compreende três ou mais ligantes. Em certas modalidades, o grupo conjugado compreende exatamente três ligantes. Em determinadas modalidades, cada ligante é selecionado dentre: um polissacarídeo, um polissacarídeo modificado, manose, galactose, um derivado de manose, um derivado de galactose, D-manopiranose, L-Manopiranose, D-Arabinose, L-Galactose, D-xilofuranose, L-xilofuranose, D-glicose, L-glicose, D-Galactose, L-Galactose a-D-Manofuranose, β-D-Manofuranose, a-D-Manopyranose, β-D-Manopyranose, a-D-Glicopiranose, β-D-Glicopiranose, a-D-Glicofuranose, β-D-Glicofuranose, a-D-fructofuranose, a-D-fructopiranose, a-D-Galactopiranose, β -D-Galactopiranose, a-D-Galactofuranose, β -D-Galactofuranose, glucosamina, ácido siálico, α-D-galactosamina, N-acetilgalactosamina, 2-Amino - 3- O -[(R )-1-carboxietil]-2-s-β-D-glicopiranose, 2-Desoxi-2-metilamino-L-glicopiranose , 4,6-Didesoxi-4- formamido-2,3-di-O-metil-D-manopiranose, 2-Desoxi-2-sulfoamino-D- glicopiranose, ácido N- Glicoloil-a-neuramínico, 5-tio-β-D-glicopiranose, metil 2, 3,4-tri-O-acetil-1-tio-6- O etil - tritil-a-D-glicopiranósido, 4-Tio-β-D-galactopiranose, 3,4,6,7-tetra-O-acetil-2-desoxi-1,5-ditio-a-D-glico-heptopiranósido, 2,5-anidro-D-alononitrilo, ribose, D-ribose, D-4- tiorribose, L-ribose, L-4-tiorribose. Em determinadas modalidades, cada ligante é N-acetil galactosamina.
[00296] Em determinadas modalidades, o grupo de conjugadoscompreende:
[00297] Em determinadas modalidades, compreende:
[00298] Em determinadas modalidades, o grupo compreende:
[00299] Em determinadas modalidades, o grupo compreende:
[00300] Em determinadas modalidades, o grupo de conjugados compreende:
[00301] Em determinadas modalidades, o grupo de conjugados compreende pelo menos um grupo de ligação de fósforo ou grupo de ligação neutro.
[00302] Em determinadas modalidades, o grupo de conjugados compreende uma estrutura selecionada dentre:
[00303] em que n é de 1 a 12; e
[00304] em que m é de 1 a 12.
[00305] Em determinadas modalidades, o grupo de conjugados tem um fixador com uma estrutura selecionada entre:
[00306] em que L é um grupo de ligação de fósforo ou um grupo de ligação neutro;
[00307] Z1 é C(=O)O-R2;
[00308] Z2 é H, alquil C1-C6 ou alquil C1-C6 substituído;
[00309] R2 é H, alquil C1-C6 ou alquil C1-C6 substituído; e
[00310] cada m1 é, independentemente, de 0 a 20 em que pelo menos um m1 é maior que 0 para cada corrente.
[00311] Em determinadas modalidades, o grupo de conjugados tem um fixador com uma estrutura selecionada entre:
[00312] em que Z2 é H ou CH3; e
[00313] cada m1 é, independentemente, de 0 a 20 em que pelo menos um m1 é maior que 0 para cada corrente.
[00314] Em determinadas modalidades, o grupo conjugado tem um fixador com uma estrutura selecionada entre:
[00315] em que n é de 1 a 12; e
[00316] em que m é de 1 a 12.
[00317] Em determinadas modalidades, o grupo conjugado está covalentemente anexado ao oligonucleotídeo modificado.
[00318] Em determinadas modalidades, o composto tem estrutura representada pela fórmula:
[00319] em que
[00320] A é o oligonucleotídeo modificado;
[00321] B é a porção clivável
[00322] C é o ligante conjugado
[00323] D é o grupo de ramificação
[00324] cada E é uma corrente;
[00325] cada M é um ligante; e
[00326] q é um número inteiro entre 1 e 5.
[00327] Em determinadas modalidades, o composto tem estrutura representada pela fórmula:
[00328] em que:
[00329] A é o oligonucleotídeo modificado;
[00330] B é a porção clivável
[00331] C é o ligante conjugado
[00332] D é o grupo de ramificação
[00333] cada E é uma corrente;
[00334] cada F é um ligante;
[00335] cada n é, independentemente, 0 ou 1; e
[00336] q é um número inteiro entre 1 e 5.
[00337] Em determinadas modalidades, o composto tem estrutura representada pela fórmula:
[00338] em que
[00339] A é o oligonucleotídeo modificado;
[00340] B é a fração clivável;
[00341] C é o ligante conjugado;
[00342] cada E é uma corrente;
[00343] cada F é um ligante; e
[00344] q é um número inteiro entre 1 e 5.
[00345] Em determinadas modalidades, o composto tem uma estrutura representada pela fórmula:
[00346] em que
[00347] A é o oligonucleotídeo modificado;
[00348] C é o ligante conjugado;
[00349] D é o grupo de ramificação;
[00350] cada E é uma corrente;
[00351] cada F é um ligante; e
[00352] q é um número inteiro entre 1 e 5.
[00353] Em determinadas modalidades, o composto tem uma estrutura representada pela fórmula:
[00354] em que
[00355] A é o oligonucleotídeo modificado;
[00356] C é o ligante conjugado;
[00357] cada E é uma corrente;
[00358] cada F é um ligante; e
[00359] q é um número inteiro entre 1 e 5.
[00360] Em determinadas modalidades, o composto tem uma estrutura representada pela fórmula:
[00361] em que
[00362] A é o oligonucleotídeo modificado;
[00363] B é a fração clivável;
[00364] D é o grupo de ramificação;
[00365] cada E é uma corrente;
[00366] cada F é um ligante; e
[00367] q é um número inteiro entre 1 e 5.
[00368] Em determinadas modalidades, o composto estrutura representada pela fórmula:
[00369] em que
[00370] A é o oligonucleotídeo modificado;
[00371] B é a fração clivável;
[00372] cada E é uma corrente;
[00373] cada F é um ligante; e
[00374] q é um número inteiro entre 1 e 5.
[00375] Em determinadas modalidades, o composto estrutura representada pela fórmula:
[00376] em que
[00377] A é o oligonucleotídeo modificado;
[00378] D é o grupo de ramificação;
[00379] cada E é uma corrente;
[00380] cada F é um ligante; e
[00381] q é um número inteiro entre 1 e 5.
[00382] Em determinadas modalidades, um ligador do tem uma estrutura selecionada entre:
[00383] em que cada L é, independentemente, um grupo de ligação de fósforo ou um grupo de ligação neutro; e
[00384] cada n é, independentemente, de 1 a 20.
[00385] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00386] Em determinadas modalidades, o ligante conjugado tem aseguinte estrutura:
[00387] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00388] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00389] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00390] Em determinadas modalidades, ligador de conjugado compreende uma pirrolidina. Em determinadas modalidades, o ligador do conjugado não compreende uma pirrolidina.
[00391] Em determinadas modalidades, o ligador de conjugadocompreende um PEG.
[00392] Em determinadas modalidades, o ligador conjugadocompreende uma amida. Em determinadas modalidades, o ligador de conjugado compreende pelo menos duas amidas. Em determinadas modalidades, o ligador de conjugado não compreende uma amida. Em determinadas modalidades, o ligador de conjugado compreende uma poliamida.
[00393] Em determinadas modalidades, o ligador de conjugadocompreende uma amina.
[00394] Em determinadas modalidades, o ligador de conjugadocompreende uma ou mais ligações de bissulfeto.
[00395] Em determinadas modalidades, o ligador conjugado compreende uma porção ligante de proteínas. Em determinadas modalidades, uma fração ligante de proteína compreende um lipídio. Em determinadas modalidades, a fração ligante de proteínas é selecionada dentre: colesterol, ácido cólico, ácido acético de adamantano, ácido 1-pireno butírico, dihidrotestosterona, 1,3-Bis- O(hexadecil)glicerol, grupo geraniloxihexil, hexadecilglicerol, borneol, mentol, 1,3-propanodiol, grupo heptadecil, ácido palmítico, ácido mirístico, ácido O3-(oleoil)litocólico , ácido O3-(oleoil) colênico, dimetoxitritil ou fenoxazina), uma vitamina (por exemplo, folato, vitamina A, vitamina E, biotina, piridoxal), um peptídeo, um carboidrato (por exemplo, monossacarídeo, dissacarídeo, trissacarídeo, tetrassacarídeo, oligossacarídeo, polissacarídeo), um componente endossomolítico, um esteroide (por exemplo, uvaol, hecigenina, diosgenina), um terpeno (por exemplo, triterpeno, por exemplo, sarsasapogenina, friedelina, ácido litocólico derivatizado de epifriedelanol) ou um lipídio catiônico. Em determinadas modalidades, a fração ligante de proteínas é selecionada entre: um ácido graxo saturado ou insaturado de cadeia longa de C16 a C22, colesterol, ácido cólico, vitamina E, adamantano ou 1- pentafluoropropil.
[00396] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00397] em que cada n é, i pendentemente, de 1 a 20; e p é de 1a 6.
[00398] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00399] em que n é, independentemente, de 1 a 20.
[00400] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00401] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00402] em que n é de 1 a 20.
[00403] Em determinadas modalidades, um ligador do conjugado tem uma estrutura selecionada entre:
[00404] Em determinadas modalidades, um ligador do conjugadotem uma estrutura selecionada entre:
[00405] onde cada n é, independentemente, 0, 1, 2, 3, 4, 5, 6 ou 7.
[00406] Em determinadas modalidades, o ligador de conjugado tema seguinte estrutura:
[00407] Em determinadas modalidades, o grupo de ramificação temuma das seguintes estruturas:
[00408] em que cada A1 é, independentemente, O, S, C=O ou NH; e
[00409] cada n é, independentemente, de 1 a 20.
[00410] Em determinadas modalidades, o grupo de ramificação tem uma das seguintes estruturas:
[00411] em que cada A1 é, independentemente, O, S, C=O ou NH; e
[00412] cada n é, independentemente, de 1 a 20.
[00413] Em determinadas modalidades, o grupo de ramificação tem a seguinte estrutura:
[00414] Em determinadas modalidades, o grupo de ramificação tem a seguinte estrutura:
[00415] Em determinadas modalidades, o grupo de ramificação tem a seguinte estrutura:
[00416] Em determinadas modalidades, o grupo de ramificação tem a seguinte estrutura:
[00417] Em determinadas modalidades, o grupo de ramificação compreende um éter.
[00418] Em determinadas modalidades, o grupo de ramificação tem
[00419] cada n é, independentemente, de 1 a 20; e
[00420] m é de 2 a 6.
[00421] Em determinadas modalidades, o grupo de ramificação tema seguinte estrutura:
[00422] Em determinadas modalidades, o grupo de ramificação tem a seguinte estrutura:
[00423] Em determinadas modalidades, o grupo de ramificaçãocompreende:
[00424] em que cada j é um número inteiro de 1 a 3; e
[00425] em que cada n é um número inteiro de 1 a 20.
[00426] Em determinadas modalidades, o grupo de ramificaçãocompreende:
[00427] Em determinadas modalidades, cada fixador é selecionado entre:
[00428] em que L é selecionado a partir de um grupo de ligação de fósforo e um grupo de ligação neutro;
[00429] Z1 é C(=O)O-R2;
[00430] Z2 é H, alquil C1-C6 ou alquil substituído C1-C6;
[00431] R2 é alquil H, C1-C6 ou alquil substituído C1-C6 ; e
[00432] cada m1 é, independentemente, de 0 a 20 em que pelo menos um m1 é maior que 0 para cada corrente.
[00433] Em determinadas modalidades, cada fixador é selecionado entre:
[00434] em que Z2 é H ou CH3; e
[00435] cada m2 é, independentemente, de 0 a 20 em que pelo menos um m2 é maior que 0 para cada corrente.
[00436] Em determinadas modalidades, cada fixador é selecionado entre:
[00437] em que n é de 1 a 12; e
[00438] em que m é de 1 a 12.
[00439] Em determinadas modalidades, pelo menos uma corrente compreende etilenoglicol.
[00440] Em determinadas modalidades, cada corrente compreende uma amida. Em determinadas modalidades, pelo menos uma corrente compreende uma poliamida.
[00441] Em determinadas modalidades, cada corrente compreende uma amina.
[00442] Em determinadas modalidades, pelo menos duas correntes são diferentes uma da outra. Em determinadas modalidades, as correntes são as mesmas.
[00443] Em determinadas modalidades, cada fixador é selecionado entre:
[00444] em que n é, independentemente, de 1 a 20; e
[00445] cada p é de 1 a cerca de 6.
[00446] Em determinadas modalidades, cada fixador é selecionado entre:
[00447] Em determinadas modalidades, cada corrente tem a seguinte estrutura:
[00448] em que n é, independentemente, de 1 a 20.
[00449] Em determinadas modalidades, cada corrente tem a seguinte estrutura:
[00450] Em determinadas modalidades, a corrente tem uma estrutura selecionada entre:
[00451] em que cada n é, independentemente, 0, 1, 2, 3, 4, 5, 6 ou 7.
[00452] Em determinadas modalidades, a corrente tem umaestrutura selecionada entre:
[00453] Em determinadas modalidades, o ligante é a galactose.
[00454] Em determinadas modalidades, o ligante é manose-6- fosfato.
[00455] Em determinadas modalidades, cada ligante é selecionado entre:
[00456] em que R1 é selecionado de OH e NHCOOH.
[00457] Em determinadas modalidades, cada ligante é selecionadoentre:
[00458] Em determinadas modalidades, cada ligante tem a seguinte estrutura:
[00459] Em determinadas modalidades, cada ligante tem a seguinte estrutura:
[00460] Em determinadas modalidades, o grupo de conjugado compreende uma fração de direcionamento de célula.
[00461] Em determinadas modalidades, o grupo de conjugado com-preende uma fração de direcionado de célula com a seguinte estrutura:
[00462] em que n é, independentemente, de 1 a 20.
[00463] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00464] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00465] em que n é, independentemente, de 1 a 20.
[00466] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00467] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00468] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00469] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00470] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00471] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00472] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00473] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00474] Em determinadas modalidades, a porção de direcionamento de célula compreende:
[00475] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00476] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00477] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00478] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00479] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00480] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00481] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00482] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00483] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00484] Em determinadas modalidades, a fração de direcionamentode célula compreende:
[00485] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00486] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00487] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00488] Em determinadas modalidades, a fração de direcionamento de célula compreende:
[00489] em que cada Y é selecionado de O, S, um alquil C1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil.
[00490] Em determinadas modalidades, o grupo de conjugados compreende:
[00491] em que cada Y é selecionado de O, S, um alquil C1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil.
[00492] Em determinadas modalidades, a fração de direcionamento de célula tem a seguinte estrutura:
[00493] em que cada Y é selecionado de O, S, um alquil C1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil.
[00494] Em determinadascompreende:
[00495] Em determinadas modalidades, o grupo de conjugados compreende:
[00496] Em determinadas modalidades, o grupo de conjugado compreende:
[00497] Em determinadas modalidades, o grupo de conjugados compreende:
[00498] Em determinadas modalidades, o grupo de conjugado compreende uma fração clivável selecionada entre: um fosfodiéster, uma amida ou um éster.
[00499] Em determinadas modalidades, o grupo conjugado compreende uma fração clivável de fosfodiéster.
[00500] Em determinadas modalidades, o grupo do conjugado não compreende uma fração clivável, e em que o grupo do conjugado compreende uma ligação de fosforotioato entre o grupo do conjugado e o oligonucleotídeo.
[00501] Em determinadas modalidades, o grupo conjugadocompreende uma fração clivável de amida.
[00502] Em determinadas modalidades, o grupo conjugadocompreende uma fração clivável de éster.
[00503] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00504] em que n é, independentemente, de 1 a 20;
[00505] Q13 é H ou O(CH2)2-OCH3;
[00506] A é o oligonucleotide modificado; e
[00507] Bx é uma porção de base heterocíclica.
[00508] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00509] em que n é, independentemente, de 1 a 20;
[00510] Q13 é H ou O(CH2)2-OCH3;
[00511] A é o oligonucleotide modificado; e
[00512] Bx é uma porção de base heterocíclica.
[00513] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00514] em que n é, independentemente, de 1 a 20;
[00515] Q13 é H ou O(CH2)2-OCH3;
[00516] Uma é o oligonucleotídeo modificado;
[00517] Z é H ou um suporte sólido ligado; e
[00518] Bx é uma porção de base heterocíclica.
[00519] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00520] em que n é, independentemente, de 1 a 20;
[00521] Q13 é H ou O(CH2)2-OCH3;
[00522] Uma é o oligonucleotídeo modificado;
[00523] Z é H ou um suporte sólido ligado; e
[00524] Bx é uma porção de base heterocíclica.
[00525] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00526] em que Q13 é H ou O(CH2)2-OCH3;
[00527] A é o oligonucleotídeo modificado; e
[00528] Bx é uma porção de base heterocíclica.
[00529] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00530] em que Q13 é H ou O(CH2)2-OCH3;
[00531] A é o oligonucleotídeo modificado; e
[00532] Bx é uma porção de base heterocíclica.
[00533] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00534] em que Q13 é H ou O(CH2)2-OCH3;
[00535] A é o oligonucleotide modificado; e
[00536] Bx é uma porção de base heterocíclica.
[00537] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00538] em que Q13 é H ou O(CH2)2-OCH3;
[00539] A é o oligonucleotide modificado; e
[00540] Bx é uma porção de base heterocíclica.
[00541] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00542] em que Q13 é H ou O(CH2)2-OCH3;
[00543] A é o oligonucleotide modificado; e
[00544] Bx é uma porção de base heterocíclica.
[00545] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00546] em que Q13 é H ou O(CH2)2-OCH3;
[00547] A é o oligonucleotide modificado; e
[00548] Bx é uma porção de base heterocíclica.
[00549] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00550] em que Q13 é H ou O(CH2)2-OCH3;
[00551] A é o oligonucleotide modificado; e
[00552] Bx é uma porção de base heterocíclica.
[00553] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00554] em que Q13 é H ou O(CH2)2-OCH3;
[00555] A é o oligonucleotide modificado; e
[00556] Bx é uma porção de base heterocíclica.
[00557] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00558] em que Q13 é H ou O(CH2)2-OCH3;
[00559] A é o oligonucleotide modificado; e
[00560] Bx é uma porção de base heterocíclica.
[00561] Em determinadas modalidades, o composto tem a seguinte estrutura:
[00562] em que Q13 é H ou O(CH2)2-OCH3;
[00563] A é o oligonucleotide modificado; e
[00564] Bx é uma porção de base heterocíclica.
[00565] Em determinadas modalidades, o composto tem a seguinteestrutura:
[00566] em que Q13 é H ou O(CH2)2-OCH3;
[00567] A é o oligonucleotide modificado; e
[00568] Bx é uma porção de base heterocíclica.
[00569] Em determinadas modalidades, o grupo de conjugadoscompreende:
[00570] em que Q13 é H ou O(CH2)2-OCH3;
[00571] A é o oligonucleotide modificado; e
[00572] Bx é uma porção de base heterocíclica.
[00573] Em determinadas modalidades, o grupo de conjugados compreende:
[00574] em que Q13 é H ou O(CH2)2-OCH3;
[00575] A é o oligonucleotide modificado; e
[00576] Bx é uma porção de base heterocíclica.
[00577] Em determinadas modalidades, o grupo de conjugados compreende:
[00578] em que Q13 é H ou O(CH2)2-OCH3;
[00579] A é o oligonucleotide modificado; e
[00580] Bx é uma porção de base heterocíclica.
[00581] Em determinadas modalidades, Bx é selecionado entre adenina, guanina, timina, uracila ou citosina, ou 5-metil-citosina. Em certas modalidades, BX é adenina. Em certas modalidades, Bx é timina. Em certas modalidades, Q13 é O (CH2)2-OCH3. Em certas modalidades, Q13 é H.
[00582] Certas modalidades da invenção fornecem uma pró-droga que compreende composições ou compostos divulgados neste documento. Certas modalidades proporcionam métodos de utilização dos compostos antisense conjugados e composições aqui descritos para inibir a expressão de ANGPTL3. Em certas modalidades, os compostos antisense conjugados ou composições inibem ANGPTL3 em pelo menos 5%, pelo menos 10%, pelo menos 20%, pelo menos 30%,pelo menos 35%, pelo menos 40%, pelo menos 45%, pelo menos 50%,pelo menos 55%, pelo menos 60%, pelo menos 65%, pelo menos 70%,pelo menos 75%, pelo menos 80%, pelo menos 85%, pelo menos 90%ou pelo menos 95%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 50%. Numa modalidade preferida, o composto anti-sense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 55%. Numa modalidade preferida, o composto anti-sense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 60%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 65%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 70%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 75%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 80%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 85%. Numa modalidade preferida, o composto anti-sense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 90%. Numa modalidade preferida, o composto antisense que compreende um oligonucleotídeo modificado e um grupo conjugado diminui ANGPTL3 por, pelo menos, 95%.
[00583] Em certas modalidades, os compostos conjugados antisense ou composições aqui divulgados têm um IC50 inferior a 20 μM, inferiores a 10 μM, menos de 8 μM, menos do que 5 μM, menos de 2 μM, menos de 1 μM, ou inferior a 0,8 μM, quando testadas células humanas, por exemplo, na linhagem celular de Hep3B como descrita nos Exemplos 2-3 e 7-10.
[00584] Em certas modalidades, os compostos antisense conjugados como descritos neste documento são eficazes em virtude de terem uma viscosidade de menos do que 40 cP, menos do que 35 cP, menos do que 30 cP, menos do que 25 cP, menos do que 20 cP ou menos do que 15 cP quando medidos pelos parâmetros, como descrito no Exemplo 13.
[00585] Em certas modalidades, os compostos anti-sense conjugados ou composições aqui divulgados são altamente toleráveis, tal como demonstrado pelas medições de tolerabilidadein vivo descritas nos Exemplos. Em certas modalidades, os compostos anti-sense conjugados como aqui descritos são altamente toleráveis, tal como demonstrado por ter um aumento de valor ALT e/ou AST de não mais do que 4 vezes, 3 vezes, 2 vezes ou 1,5 vezes mais em animais tratados com solução salina.
[00586] Certas modalidades aqui descritas proporcionam um sal dos compostos anti-sense conjugados aqui revelados. Em certas modalidades, os compostos ou composições aqui descritos compreendem um sal do oligonucleotídeo modificado com o grupo conjugado.
[00587] Em determinadas modalidades, os compostos conjugados antisense ou composições divulgados neste documento compreendem um carreador ou diluente farmaceuticamente aceitável.
[00588] Em determinadas modalidades, o animal é um ser humano.
[00589] Certas modalidades descritas aqui proporcionam métodos compreendendo a administração a um animal dos compostos antisense conjugados ou composições aqui divulgados. Em certas modalidades, a administração do composto anti-sense conjugado ou composição é terapêutica. Em certas modalidades, a administração do composto antisense conjugado ou composição trata, previne ou retarda a progressão de uma doença relacionada com ANGPTL3. Em certas modalidades, a doença está relacionada com ANGPTL3 elevada. Em certas modalidades, a administração do composto antisense conjugado ou composição previne, trata, melhora ou retarda a progressão de um distúrbio cardiovascular e/ou uma doença metabólica.
[00590] Certas modalidades aqui descritoas proporcionam métodos para o tratamento de um ser humano com uma doença cardiovascular e/ou metabólica, compreendendo a identificação de um ser humano com doença cardiovascular e/ou metabólica e a administração a um humano de uma quantidade terapeuticamente eficaz de qualquer um dos compostos antisense conjugados ou composições aqui divulgados, para assim tratar o ser humano e sua doença cardiovascular e/ou metabólica.
[00591] Certas modalidades proporcionam compostos antisense conjugados e composições aqui descritos para utilização em terapia. Em certas modalidades, a terapia é utilizada no tratamento, prevenção, ou retardo da progressão de uma doença relacionada com ANGPTL3. Em certas modalidades, a terapia é utilizada no tratamento, prevenção, ou retardo da progressão de uma doença relacionada com ANGPTL3 elevada.
[00592] Em determinadas modalidades, a doença é doença, distúrbio ou condição cardiovascular e/ou metabólica. Em certas modalidades, o metabolismo e/ou doença cardiovascular inclui, mas não está limitado a obesidade, diabetes, resistência a insulina, aterosclerose, dislipidemia, lipodistrofia, doença cardíaca coronária, a doença não alcoólica do fígado graxo (esteatose hepática), hiperacidemia de excesso de ácido graxo, esteato-hepatite não alcoólica (NASH ) ou síndrome metabólica, ou uma combinação dos mesmos. A dislipidemia pode ser hiperlipidemia. A hiperlipidemia pode ser hiperlipidemia combinada (CHL), hipercolesterolemia, hipertrigliceridemia, ou ambos hipercolesterolemia e hipertrigliceridemia. A hiperlipidemia combinada familiar pode ser ou não familiar. A hipercolesterolemia pode ser hipercolesterolemia familiar homozigótica (HFHo), hipercolesterolemia heterozigótica familiar (HeFH). A hipertrigliceridemia pode ser síndrome de quilomicronemia familiar (FCS) ou hiperlipoproteinemia tipo IV. A DHGNA pode ser esteatose hepática ou esteato-hepatite. A diabetes pode ser diabetes tipo 2 ou diabetes tipo 2 com dislipidemia. A resistência à insulina pode ser a resistência à insulina com dislipidemia.
[00593] Em certas modalidades, os compostos conjugados antisense ou composições aqui descritos são designados como um primeiro agente e os métodos ou utilizações aqui descritos compreendem ainda a administração de um segundo agente. Em certas modalidades, o primeiro agente e o segundo agente são co-administrados. Em certas modalidades, o primeiro agente e o segundo agente são co- administrados sequencialmente ou concomitantemente.
[00594] Em certas modalidades, o segundo agente é um agente redutor de glicose. O agente redutor de glicose pode incluir, mas não está limitado a, uma mudança terapêutica do estilo de vida, agonista PPAR, um inibidor da dipeptidil peptidase (IV), um análogo de GLP-1, insulina ou um análogo de insulina, uma secretagogo de insulina, um inibidor de SGLT2, um análogo da amilina humana, uma biguanida, um inibidor da alfa-glicosidase ou uma combinação destes. O agente redutor de glicose pode incluir, mas não está limitado a metformina, sulfonilureia, rosiglitazona, meglitinida, tiazolidinediona, inibidor da alfa- glicosidase ou uma combinação destes. A sulfonilureia pode ser acetohexamida, clorpropamida, tolbutamida, tolazamida, glimepirida, uma glipizida, uma gliburida, ou uma gliclazida. A meglitinida pode ser nateglinida ou repaglinida. A tiazolidinediona pode ser pioglitazona ou rosiglitazona. A alfa-glicosidase pode ser acarbose ou miglitol.
[00595] Em certas modalidades, o segundo agente é uma terapia de redução de lípidos. Em certas modalidades, a terapia de redução de lípidos pode incluir, mas não está limitado a uma alteração terapêutica de estilo de vida, inibidor de HMG-CoA-redutase, inibidor da absorção de colesterol, um inibidor de MTP (por exemplo, uma pequena molécula, polipéptido, anticorpo ou um composto antisense dirigido para MTP) , inibidor de ApoB (por exemplo, uma pequena molécula, polipéptido, anticorpo ou composto antisense dirigido a ApoB), inibidor de APOC3 (por exemplo, uma pequena molécula, polipéptido, anticorpo ou composto antisense dirigido para APOC3), inibidor de PCSK9 (por exemplo, uma molécula pequena, polipéptido, anticorpo ou um composto antisense dirigido para PCSK9), inibidor de CETP (por exemplo, uma pequena molécula, polipéptido, anticorpo ou composto antisense dirigido para CETP), fibrato, óleo benéfico (por exemplo, krill ou óleos de peixe (por exemplo, VascepaR), óleo de linhaça, ou outros óleos ricos em ácidos graxos de omega-3 tais como o ácido a-linolênico (ALA), ácido docosahexaenóico (DHA) ou ácido eicosapentaenóico (EPA)), ou qualquer combinação dos mesmos. O inibidor da HMG-CoA redutase pode ser atorvastatina, rosuvastatina, fluvastatina, lovastatina, pravastatina ou sinvastatina. O inibidor de absorção de colesterol pode ser ezetimiba. O fibrato pode ser fenofibrato, bezafibrato, ciprofibrato, clofibrato, gemfibrozil e outros semelhantes.
[00596] Em certas modalidades, a administração compreende a administração parentérica. Em certas modalidades, a administração compreende a administração subcutânea.
[00597] Em certas modalidades, a administração de um composto antisense conjugado aqui divulgado resulta numa diminuição dos níveis de lípidos, incluindo os níveis de triglicéridos, os níveis de colesterol, resistência à insulina, os níveis de glucose ou uma combinação dos mesmos. Um ou mais dos níveis pode ser independentemente reduzida em pelo menos 5%, pelo menos 10%, pelo menos 20%, pelo menos 30%, pelo menos 35%, pelo menos 40%, pelo menos 45%, pelo menos50% , pelo menos 55%, pelo menos 60%, pelo menos 65%, pelo menos70%, pelo menos 75%, pelo menos 80%, pelo menos 85%, pelo menos90% ou pelo menos 95%. A administração do composto antisenseconjugado pode resultar no aumento da sensibilidade à insulina ou a sensibilidade à insulina hepática. Administração do composto antisense conjugado aqui divulgado pode resultar numa redução em placas ateroscleróticas, obesidade, glicose, lípidos, resistência à glucose, colesterol, ou uma melhoria na sensibilidade à insulina ou qualquer combinação dos mesmos.
[00598] Certas modalidades proporcionam o uso de um composto conjugado antisense como aqui descrito, na fabricação de um medicamento para tratar, melhorar, retardar ou prevenir uma ou mais de uma doença relacionada com ANGPTL3. Certas modalidades proporcionam o uso de um composto conjugado antisense como aqui descrito, na fabricação de um medicamento para tratar, melhorar, retardar ou prevenir um ou mais de uma doença metabólica ou uma doença cardiovascular.
[00599] Certas modalidades proporcionam um kit para o tratamento, prevenção, ou melhoramento de um ou mais de uma doença metabólica ou uma doença cardiovascular, como aqui descrito, em que o kit compreende: a) um composto conjugado antisense tal como aqui descrito; e opcionalmente b) um agente ou terapia adicional, tal como aqui descrito. O kit pode ainda incluir instruções ou uma etiqueta para a utilização do kit para tratar, prevenir, ou melhorar um ou mais de uma doença metabólica ou uma doença cardiovascular.
Compostos Antisense
[00600] Compostos oligoméricos incluem, mas não estão limitados a, oligonucleotídeos, oligonucleosídeos, análogos de oligonucleotídeo, miméticos de oligonucleotídeo, compostos antisense, oligonucleotídeos antisense e siRNAs. Um composto oligomérico pode ser "antisense" para um ácido nucleico alvo, significando que um composto oligomérico é capaz de sofrer hibridização de um ácido nucleico alvo através da ligação de hidrogênio.
[00601] Em certas modalidades, um composto antisense tem uma sequência de nucleobases que, quando escrita na direção 5' para 3', compreende o complemento inverso do segmento alvo de um ácido nucleico alvo ao qual este é direcionado. Em algumas dessas modalidades, um oligonucleotídeo antisense tem uma sequência de nucleobases que, quando escrita na direção 5' para 3', compreende o complemento inverso do segmento alvo de um ácido nucleico alvo ao qual este é direcionado.
[00602] Em determinadas modalidades, um composto antisense direcionado para um ácido nucleico de ANGPTL3 é de 10 a 30 nucleotídeos de comprimento. Em outras palavras, os compostos antisense são de 10 a 30 nucleobases ligadas. Em outras modalidades, o composto antisense compreende um oligonucleotídeo modificado que consiste em 8 a 80, 10 a 80, 12 a 30, 12 a 50, 15 a 30, 18 a 24, 19 a 22, ou 20 nucleobases ligadas. Em algumas dessas modalidades, o composto antisense compreende um oligonucleotídeo modificado consistindo em 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22,23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41,42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79 ou 80 nucleobases ligadas de comprimento, ou uma faixa definida por qualquer um dos dois valores acima.
[00603] Em certas modalidades, o composto antisense compreende um oligonucleotídeo modificado encurtado ou truncado. O oligonucleotídeo modificado encurtado ou truncado pode ter um único nucleosídeo excluído da extremidade 5' (truncamento em 5'), ou alternativamente da extremidade 3' (truncamento em 3'). Um oligonucleotídeo encurtado ou truncado pode ter dois ou mais nucleosídeos excluídos da extremidade 5', ou alternativamente pode ter dois ou mais nucleosídeos deletados da extremidade 3'. Alternativamente, os nucleosídeos deletados podem ser dispersos ao longo do oligonucleotídeo modificado, por exemplo, em um composto antisense tendo um ou mais nucleosídeos deletado da extremidade 5' e um ou mais nucleosídeos deletados da extremidade 3'.
[00604] Quando um único nucleosídeo adicional estiver presente em um oligonucleotídeo alongado, o nucleosídeo adicional poderá ser localizado na extremidade 5' ou 3' ou porção central do oligonucleotídeo. Quando dois ou mais nucleosídeos adicionais estiverem presentes, os nucleosídeos adicionados poderão estar adjacentes uns aos outros, por exemplo, em um oligonucleotídeo tendo dois nucleosídeos adicionados à extremidade 5' (adição em 5'), ou alternativamente à extremidade 3' (adição em 3'), ou porção central, do oligonucleotídeo. Em alternativa, o nucleosído adicionado pode ser disperso por todo o composto antisense, por exemplo, em um oligonucleotídeo possuindo um ou mais nucleosído adicionado à extremidade 5', um ou mais nucleosído adicionado à extremidade 3', e/ou um ou mais nucleosído adicionado para a porção central.
[00605] É possível aumentar ou diminuir o comprimento de um composto antisense, tal como um oligonucleotídeo antisense, e/ou apresentar bases incompatíveis sem eliminar a atividade. Por exemplo, em Woolf et al. (Proc. Natl. Acad. Sci. USA 89:7305-7309, 1992), uma série de oligonucleotídeos antisense de 13 a 25 nucleobases de comprimento foi testada quanto à sua capacidade de induzir a clivagem de um RNA alvo em um modelo de injeção de oócito. Os oligonucleotídeos antisense de 25 nucleobases de comprimento com 8 ou 11 bases de incompatibilidade perto das extremidades dos oligonucleotídeos antisense foram capazes de direcionar a clivagem específica do mRNA alvo, ainda que em uma menor extensão do que os oligonucleotídeos antisense que não continham nenhuma incompatibilidade. De forma similar, a clivagem específica alvo foi obtida usando 13 oligonucleotídeos antisense de nucleobase, incluindo aqueles com 1 ou 3 incompatibilidades.
[00606] Gautschi et al. (J. Natl. Cancer Inst. 93:463-471, March 2001) demonstrou a capacidade do oligonucleotídeo ter 100% de complementaridade com o mRNA de bcl-2 e ter 3 incompatibilidades com o mRNA de bcl-xL para reduzir a expressão de ambos bcl-2 e bcl- xLin vitro e in vivo. Além disso, este oligonucleotídeo demonstrou atividade antitumor potente in vivo.
[00607] Maher and Dolnick (Nuc. Acid. Res. 16:3341-3358.1988) testou uma série de oligonucleotídeos antisense de 14 nucleobases em tandem e oligonucleotídeos antisense de 28 e 42 nucleobases compostos da sequência de dois ou três dos oligonucleotídeos antisense em tandem, respectivamente, quanto à sua capacidade de suspender a tradução de DHFR humana em um ensaio com reticulócitos de coelho. Cada um dos três oligonucleotídeos antisense de 14 nucleobases sozinho foi capaz de inibir a tradução, embora a um nível mais baixo do que os oligonucleotídeos antisense de 28 ou 42 nucleobases.
Determinados motivos e mecanismos do composto antisense
[00608] Em determinadas modalidades, os compostos antisense têm subunidades quimicamente modificadas dispostas em padrões, ou motivos, para conferir às propriedades dos compostos antisense, como atividade inibitória intensificada, afinidade de ligação aumentada para um ácido nucleico alvo, ou resistência à degradação por nucleases in vivo.
[00609] Compostos antisense quiméricos normalmente contêm pelo menos uma região modificada com a finalidade de conferir resistência aumentada à degradação da nuclease, absorção celular aumentada, afinidade de ligação aumentada para o ácido nucleico alvo, e/ou atividade inibitória aumentada. Uma segunda região de um composto antisense quimérico pode conferir outra propriedade desejada, por exemplo, servir como um substrato para a RNase H da endonuclease celular, que cliva a fita do RNA de um RNA:DNA duplex.
[00610] A atividade antisense pode resultar de qualquer mecanismo envolvendo a hibridização do composto antisense (por exemplo, oligonucleotídeos) com um ácido nucleico alvo, em que a hibridização resulta, em última análise, em um efeito biológico. Em certas modalidades, a quantidade e/ou a atividade do ácido nucleico alvo é modulada. Em certas modalidades, a quantidade e/ou a atividade do ácido nucleico alvo é reduzida. Em certas modalidades, a hibridização do composto antisense para o ácido nucleico alvo resulta, em última análise, em uma degradação do ácido nucleico alvo. Em certas modalidades, a hibridização do composto antisense para o ácido nucleico não resulta na degradação do ácido nucleico alvo. Em algumas dessas modalidades, a presença do composto antisense hibridizado com o ácido nucleico alvo (ocupação) resulta em uma modulação da atividade antisense. Em certas modalidades, os compostos antisense com um motivo ou padrão químico particular de modificações químicas são particularmente adequados para explorar um ou mais mecanismos. Em determinadas modalidades, os compostos antisense funcionam através de mais de um mecanismo e/ou através de mecanismos que não foram elucidados. Por conseguinte, os compostos antisense descritos aqui não são limitados por um mecanismo particular.
[00611] Os mecanismos antisense incluem, sem limitação, antisense mediado por RNase H; mecanismos de RNAi, que utilizam vias de RISC e incluem, sem limitação, mecanismos de siRNA, ssRNA e microRNA; e mecanismos baseados em ocupação. Certos compostos antisense podem atuar através de mais de um mecanismo desses e/ou através de mecanismos adicionais.
Antisense mediado por RNase H
[00612] Em certas modalidades, os resultados da atividade antisense, pelo menos em parte, a partir da degradação do RNA alvo por RNase H. RNase H é uma endonuclease celular que cliva a fita de RNA de um duplex de RNA :DNA. É conhecido na técnica que os compostos antisense de fita única que são "similares ao DNA" eliciam atividade de RNase H nas células de mamíferos. Por conseguinte, os compostos antisense compreendendo pelo menos uma porção dos nucleosídeos de DNA ou semelhantes a DNA podem ativar a RNAse H, resultando na clivagem do ácido nucleico alvo. Em determinadas modalidades, os compostos antisense que utilizam RNAse H compreendem um ou mais nucleosídeos modificados. Em certas modalidades, esses compostos antisense compreendem pelo menos um bloco de 1-8 nucleosídeos modificados. Em certas modalidades, os nucleosídeos modificados não sustentam a atividade da RNAse H. Em certas modalidades, tais compostos antisense são gapmers, conforme descrito aqui. Em certas modalidades, o intervalo do gapmer compreende nucleosídeos de DNA. Em certas modalidades, o intervalo do gapmer compreende nucleosídeos semelhantes a DNA. Em certas modalidades, o intervalo do gapmer compreende nucleosídeos de DNA e nucleosídeos semelhantes a DNA.
[00613] Determinados compostos antisense com um motivo de gapmer são considerados compostos antisense quiméricos. Em um gapmer uma região interna que tem uma pluralidade de nucleotídeos que suporta a clivagem de RNaseH está posicionada entre as regiões externas que têm uma pluralidade de nucleotídeos que são quimicamente diferentes dos nucleosídeos da região interna. No caso de um oligonucleotídeo antisense que tem um motivo de gapmer, o segmento de lacuna geralmente serve como o substrato para a clivagem da endonuclease, enquanto os segmentos de asa compreendem nucleosídeos modificados. Em certas modalidades, as regiões de um gapmer são diferenciadas pelos tipos de frações de açúcar compreendendo cada região distinta. Os tipos de frações de açúcar que são usados para diferenciar as regiões de um gapmer podem em algumas modalidades incluir β-D-ribonucleosídeos, β-D-desoxirribonucleosídeos, nucleosídeos modificados em 2' (tais nucleosídeos modificados em 2' podem incluir 2'-MOE e 2'-O-CH3, entre outros) e nucleosídeos modificados pelo açúcar bicíclico (tais nucleosídeos modificados pelo açúcar bicíclico podem incluir aqueles que têm uma etila restrita). Em certas modalidades, os nucleosídeos nas asas podem incluir diversas porções de açúcar modificado, incluindo, por exemplo, porções de açúcar bicíclico e 2'-MOE, como etil limitado ou LNA. Em determinadas modalidades, as asas podem incluir várias porções de açúcar modificado e não-modificado. Em certas modalidades, as asas podem incluir várias combinações de nucleosídeos de 2'-MOE, porções de açúcar bicíclico, como nucleosídeos de etil limitado ou nucleosídeos de LNA, e 2'- deoxinucleosídeos.
[00614] Cada região distinta pode compreender frações de açúcar uniformes, variantes, ou frações de açúcar alternadas. O motivo de asa- lacuna-asa é frequentemente descrito como "X-Y-Z", onde "X" representa o comprimento de 5'-asa, "Y" representa o comprimento da lacuna, e "Z" representa o comprimento de 3'-asa. "X" e "Z" podem incluir frações de açúcar uniformes, variantes, ou alternadas. Em determinadas modalidades, "X" e "Y" podem incluir um ou mais 2'- desoxinucleosídeos. "Y" pode compreender 2'-desoxinucleosídeos. Como usado neste documento, um gapmer descrito como "X-Y-Z" tem uma configuração de modo que a lacuna esteja posicionada imediatamente adjacente a cada 5'-asa e 3'-asa. Desse modo, não há nenhuma intervenção de nucleotídeos entre 5'-asa e a lacuna, ou a lacuna e 3'-asa. Qualquer um dos compostos antisense descritos neste documento pode ter um motivo de gapmer. Em determinadas modalidades, "X" e "Z" são iguais; em outras modalidades, eles são diferentes. Em determinadas modalidades, "Y" tem entre 8 e 15 nucleosídeos. X, Y ou Z podem ter 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30 ou mais nucleosídeos.
[00615] Em certas modalidades, o composto antisense direcionado a um ácido nucleico de ANGPTL3 tem um motivo de gapmer em que o intervalo consiste em 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, ou 16nucleosídeos ligados.
[00616] Em certas modalidades, o oligonucleotídeo antisense tem um motivo de açúcar descrito pela Fórmula A, conforme segue:(J)m-(B)n-(J)p-(B)r-(A)t-(D)g-(A)v-(B)w-(J)x-(B)y-(J)z
[00617] em que:
[00618] cada A é independentemente um nucleosídeo substituído em 2';
[00619] cada B é independentemente um nucleosídeo bicíclico;
[00620] cada J representa, independentemente, um nucleosídeo substituído em 2' ou um 2'-desoxinucleosídeo;
[00621] cada D é um 2'-desoxinucleosídeo;
[00622] m é 0-4; n é 0-2; p é 0-2; r é 0-2; t é 0-2; v é 0-2; w é 0-4; x é 0-2; y é 0-2; Z é 0-4; g é 6-14;
[00623] contanto que:
[00624] pelo menos um de m, n, e r é outro que não 0;
[00625] pelo menos um de W e Y é diferente de 0;
[00626] a soma de m, n, p, r, e t é 2 a 5; e
[00627] a soma de v, w, x, y, e z é de 2 a 5.Compostos de RNAi
[00628] Em certas modalidades, os compostos antisense são compostos de RNA de intervenção (RNAi), que incluem compostos de RNA de fita supla (também referidos como RNA de interferência breve ou siRNA) e compostos de RNAi de fira simples (ou ssRNA). Esses compostos funcionam, pelo menos em parte, através da via de RISC para degradar e/ou sequestrar um ácido nucleico alvo (e, assim, incluir compostos de microRNA/mímicos de microRNA). Em certas modalidades, os compostos antisense compreendem modificações que os tornam particularmente apropriados para tais mecanismos.1. Compostos de ssRNA
[00629] Em certas modalidades, os compostos antisense, incluindo aqueles particularmente apropriados para uso como compostos de RNAi de fita simples (ssRNA), compreendem uma terminação modificada 5'-terminal. Em algumas dessas modalidades, a exremidade do terminal 5' compreende uma porção de fosfato modificado. Em certas modalidades, tal fosfato modificado é estabilizado (por exemplo, resistente à degradação/clivagem em comparação ao 5'-fosfato não- modificado). Em certas modalidades, esses nucleosídeos 5'-terminais estabilizam a porção de 5'-fosforosa. Certos nucleosídeos 5'-terminais podem ser encontrados na técnica, por exemplo, no documento WO/2011/139702.
[00630] Em certas modalidades, o 5'-nucleosídeo de um composto de ssRNA tem a fórmula IIc:
[00631] em que:
[00632] Ti é uma porção de fósforo opcionalmente protegido;
[00633] T2 é um grupo de ligação de internucleosídeos que liga ocomposto de Fórmula IIc ao composto oligomérico;
[00634] A tem uma das fórmulas:
[00635] Q1 e Q2 são cada um, independentemente, H, halogênio, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6, alcoxi substituído C1C6, alquenil C2-C6, alquenil substituído C2-C6, alquinil C2-C6, alquinil substituído C2-C6 ou N (R3) (R4);
[00636] Q3 é O, S, N(R5) ou C(R6)(R7);
[00637] cada R3, R4 R5, R6 e R7 é, independentemente, H, alquil C1C6 , alquil substituído C1-C6 ou alcoxi C1-C6;
[00638] M3 é O, S, NR14, C(R15)(R16), C(R15)(R16)C(R17)(R18),C(R15)=C(R17), OC(R15)(R16) ou OC(R15)(Bx2);
[00639] R14 é H, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6,alcoxi substituído C1-C6, alquenil C2-C6, alquenil substituído C2-C6, alquinil substituído C2-C6 ou alquinil C2-C6;
[00640] R15, R16, R17 e R18 são cada um, independentemente, H,halogênio, alquil C1-C6 , alquil substituído C1-C6 , alcoxi C1-C6 , alcoxi substituído C1-C6 , alquenil C2-C6 , C alquenil substituído C2-C6 , alquinil C2-C6 ou alquinil substituído C2-C6 ;
[00641] Bx1 é uma porção de base heterocíclica;
[00642] ou se Bx2 está presente, em seguida, Bx2 é um radical de base heterocíclica e Bx1 é H, halogênio, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6, alcoxi substituído C1-C6, alquenil C2-C6, alquenil substituído C2-C6, alquinil C2-C6, ou alquinil substituído C2-C6;
[00643] J4, J5, J6 e J7 são cada um, independentemente, H,halogênio, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6, alcoxi substituído C1-C6, alquenil C2-C6, C alquenil substituído C2-C6, alquinil C2-C6 ou alquinil substituído C2-C6;
[00644] ou J4 forma uma ponte com um dos J5 ou J7 em que a dita ponte compreende desde 1 a 3 grupos selecionados ligados birradical a partir de O, S, NR19, C (R20) (R21), C (R20) = C (R21), C [= C (R20) (R21)] e C (= O) e os outros dois de J5, J6 e J7 são cada um, independentemente, H, halogênio, alquil C1-C6, alquil substituído C1-C6 , alcoxi C1-C6, alcoxi substituído C1-C6, alquenil C2-C6, alquenil substituído C2-C6 , alquinil C2-C6, ou alquinil substituído C2-C6;
[00645] cada R19, R20 e R21 é, independentemente, H, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6, alcoxi substituído C1-C6, alquenil C2-C6 , C alquenil substituído C2-C6, alquinil C2-C6 ou alquinil substituído C2-C6;
[00646] G é H, OH, halogênio ou O-[C(R8)(R9)]n-[(C=O)m-X1]j-Z;
[00647] cada R8 e R9 é, independentemente, H, halogênio, alquil C1C6 ou alquil substituído C1-C6;
[00648] X1 é O, S ou N (E1);
[00649] Z é H, halogênio, alquil C1-C6, alquil substituído C1-C6, alquenil C2-C6, alquenil substituído C2-C6, alquinil C2-C6, alquinil substituído C2-C6 ou N (E2) (E3);
[00650] E1, E2 and E3 são, cada um, independentemente, H, alquil C1-C6 ou alquil substituído C1-C6;
[00651] n é de 1 a cerca de 6;
[00652] m é 0 ou 1;
[00653] j é 0 ou 1;
[00654] cada grupo substituído compreende um ou mais grupos substituintes opcionalmente protegidos independentemente selecionados a partir de halogênio, JO1, N (J1) (J2), = NJ1, SJ1, N3, CN, OC (= X2) J1, OC (= X2) N (J1) (J2) e C (= X2) N (J1) (J2);
[00655] x2 é O, S ou NJ3;
[00656] cada J1, J2 e J3 is, independentemente, H ou alquil C1-C6 ;
[00657] quando j é 1, então Z é diferente de halogênio ou N (E2) (E3);e
[00658] onde o referido composto oligomérico compreende de 8 a 40 subunidades monoméricas e for hibridizável em pelo menos uma porção de um ácido nucleico alvo.
[00659] Em certas modalidades, M3 é O, CH = CH, OCH2 ou OC (H) (Bx2). Em certas modalidades, M3 é O.
[00660] Em certas modalidades, J4, J5, J6 e J7 são cada um H. Em certas modalidades, J4 forma uma ponte com um dos J5 ou J7.
[00661] Em certas modalidades, A tem uma das fórmulas:
[00662] em que:
[00663] Q1 e Q2 são cada um, independentemente, H, halogênio, alquil C1-C6 , alquil substituído C1-C6 , alcoxil C1-C6 ou alcoxi C1-C6 . Em certas modalidades, Q1 e Q2 são cada um H. Em certas modalidades, Q1 e Q2 são cada um, independentemente, H ou halogênio. Em certas modalidades, Q1 e Q2 é H e o outro de Q1 e Q2 é F, CH3 ou OCH3.
[00664] Em certas modalidades, T1 tem a fórmula:
[00665] em que:
[00666] Ra e Rc são cada um, independentemente, hidroxil protegido, tiol protegido, alquil C1-C6, alquil substituído C1-C6, alcoxi C1-C6, alcoxi substituído1-C6, amino protegido ou amino substituído; e
[00667] Rb é O ou S. Em certas modalidades, Rb é O e Ra e Rc são cada um, independentemente, OCH3, OCH2CH3 ou CH (CH3)2.
[00668] Em determinadas incorporações, G é halogênio , OCH3, OCH2F, OCHF2, OCF3, OCH2CH3, O(CH2)2F, OCH2CHF2, OCH2CF3, OCH2-CH=CH2, O(CH2)2-OCH3, O(CH2)2-SCH3, O(CH2)2-OCF3,O(CH2)3-N(R10)(R11), O(CH2)2-ON(R10)(R11), O(CH2)2-O(CH2)2-N(R10)(R11), OCH2C(=O)-N(R10)(R11), OCH2C(=O)-N(R12)-(CH2)2-N(R10)(R11) or O(CH2)2-N(R12)-C(=NR13)[N(R10)(R11)] em que R10, R-11, R12 e R13 cada um, são independentes, H ou alquil C1- C6. Em certas modalidades, G é halogênio, OCH3, OCF3, OCH2CH3, OCH2CF3, OCH2- CH = CH2, O (CH2)2-OCH3, O (CH2)2-O (CH2)2-N (CH3)2, OCH2C (= O) - N (H) CH3, OCH2C (= O) -N (H) - (CH2)2-N (CH3)2 ou OCH2-N (H) -C (= NH) NH2. Em certas modalidades, G é F, OCH3 ou O (CH2)2-OCH3. Em certas modalidades, G é O(CH2)2-OCH3.
[00669] Em certas modalidades, o nucleosídeo 5'-terminal tem a Fórmula IIe:
[00670] Em determinadas modalidades, os compostos antisense, incluindo aqueles particularmente apropriados para o ssRNA, compreendem um ou mais tipos de porções de açúcar modificadas e/ou frações de açúcar de ocorrência natural dispostas ao longo do oligonucleotídeo ou da sua região em um padrão definido ou motivo de modificação do açúcar. Esses motivos podem incluir qualquer uma das modificações de açúcar discutidas neste documento e/ou outras modificações de açúcar conhecidas.
[00671] Em certas modalidades, os oligonucleotídeos compreendem ou consistem em uma região com modificações de açúcar uniforme. Em certas modalidades, cada nucleosídeo da região compreende a mesma modificação de açúcar semelhante a RNA. Em certas modalidades, cada nucleosídeo da região é um nucleosídeo 2'-F. Em certas modalidades, cada nucleosídeo da região é um nucleosídeo 2'-OMe. Em certas modalidades, cada nucleosídeo da região é um nucleosídeo 2'-MOE. Em certas modalidades, cada nucleosídeo da região é um nucleosídeo cEt. Em certas modalidades, cada nucleosídeo da região é um nucleosídeo de LNA. Em certas modalidades, a região uniforme constitui todos ou essencialmente todos os oligonucleotídeos. Em certas modalidades, a região constitui todos os oligonucleotídeos, exceto os nucleosídeos terminais 1-4.
[00672] Em certas modalidades, os oligonucleotídeos compreendem uma ou mais regiões de modificações de açúcar alternadas, em que os nucleotídeos alternam entre os nucleotídeos com uma modificação de açúcar de um primeiro tipo e nucleotídeos com uma modificação de açúcar de um segundo tipo. Em certas modalidades, os nucleotídeos de ambos os tipos são nucleosídeos semelhantes a RNA. Em certas modalidades, os nucleosídeos alternados são selecionados a partir de: 2'-OMe, 2'-F, 2'-MOE, LNA e cEt. Em certas modalidades, asmodificações alternadas são 2'-F e 2'-OMe. Essas regiões podem ser contíguas ou podem ser interrompidas por nucleosídeos modificados de maneira diferente ou por nucleosídeos conjugados.
[00673] Em certas modalidades, a região alternada das modificações alternadas consistem em um único nucleosídeo (isto é, o padrão é (AB)xAy onde A é um nucleosídeo com uma modificação de açúcar de um primeiro tipo e B é um nucleosídeo com uma modificação de açúcar de um segundo tipo; x é 1-20 e y é 0 ou 1). Em certas modalidades, uma ou mais regiões alternadas em um motivo alternado incluem mais de um único nucleosídeo de um tipo. Por exemplo, os oligonucleotídeos podem incluir uma ou mais regiões de qualquer um dos seguintes motivos de nucleosídeos:
[00674] AABBAA;
[00675] ABBABB;
[00676] AABAAB;
[00677] ABBABAABB;
[00678] ABABAA;
[00679] AABABAB;
[00680] ABABAA;
[00681] ABBAABBABABAA;
[00682] BABBAABBABABAA; ou
[00683] ABABBAABBABABAA;
[00684] em que A é um nucleosídeo de um primeiro tipo e B é umnucleosídeo de um segundo tipo. Em certas modalidades, A e B são, cada um, selecionados a partir de 2'-F, 2'-OMe, BNA e MOE.
[00685] Em certas modalidades, os oligonucleotídeos com esse motivo alternado compreendem ainda um nucleosídeo terminal 5' modificado, como aqueles da fórmula IIc ou IIe.
[00686] Em certas modalidades, os oligonucleotídeos compreendem uma região com um motivo 2-2-3. Essas regiões compreendem o seguinte motivo:-(A)2- (B)x-(A)2- (C)y-(A)3-
[00687] em que: A é um primeiro tipo de nucleosídeo modificado;
[00688] B e C, são nucleosídeos que são modificados de maneira diferente do que A, no entanto, B e C podem ter a mesma ou diferentes modificações;
[00689] x e y são de 1 a 15.
[00690] Em certas modalidades, A é um nucleotídeo modificado em 2'-OMe. Em certas modalidades, B e C são, ambos, nucleosídeos modificados em 2'-F. Em certas modalidades, A é um nucleosídeo modificado em 2'OMe e B e C são, ambos, nucleosídeos modificados em 2'-F.
[00691] Em certas modalidades, os oligonucleosídeos têm o seguinte motivo de açúcar:5'-(Q)-(AB)xAy-(D)z
[00692] em que:
[00693] Q é um nucleosídeo que compreende uma porção de fosfato estabilizada. Em certas modalidade, Q é um nucleosídeo tendo uma Fórmula IIc ou IIe;
[00694] A é um primeiro tipo de nucleosídeo modificado;
[00695] B é um segundo tipo de nucleosídeo modificado;
[00696] D é um nucleosídeo modificado compreendendo uma modificação diferente do nucleosídeo adjacente a ela. Assim, se y for 0, então D deve ser modificado de forma diferente de B e, se y for 1, então D deve ser modificado de forma diferente de A. Em certas modalidades, D difere de ambos A e B.
[00697] X é 5-15;
[00698] y é 0 ou 1;
[00699] Z é 0-4.
[00700] Em certas modalidades, os oligonucleosídeos têm o seguintemotivo de açúcar:5'- (Q)- (A)x-(D)z
[00701] em que:
[00702] Q é um nucleosídeo que compreende uma porção de fosfato estabilizada. Em certas modalidade, Q é um nucleosídeo tendo uma Fórmula IIc ou IIe;
[00703] A é um primeiro tipo de nucleosídeo modificado;
[00704] D é um nucleosídeo modificado que compreende uma modificação diferente de A.
[00705] X é 11-30;
[00706] Z é 0-4.
[00707] Em certas modalidades, A, B, C e D nos motivos acima são selecionados a partir de: 2'-OMe, 2'-F, 2'-MOE, LNA e cEt. Em certas modalidades, D representa nucleosídeos terminais. Em certas modalidades, tais nucleosídeos terminais não são concebidos para hibridizar para o ácido nucleico alvo (embora um ou mais possam hibridizar por acaso). Em certas modalidades, a nucleobase de cada nucleosídeo D é adenina, independentemente da identidade da nucleobase na posição correspondente do ácido nucleico alvo. Em certas modalidades, a nucleobase de cada nucleosídeo D é timina.
[00708] Em certas modalidades, os compostos antisense, incluindo aqueles particularmente apropriados para uso como ssRNA, compreendem ligações de internucleosídeos modificadas juntamente com oligonucleotídeos ou uma região sua em um padrão definido ou um motivo de ligação de internucleosídeos modificados. Em determinadas modalidades, os oligonucleotídeos compreendem uma região com um motivo de ligação de internucleosídeo alternado. Em determinadas modalidades, os oligonucleotídeos compreendem uma região de ligações de internucleosídeo uniformemente modificadas. Em algumas dessas modalidades, o oligonucleotídeo compreende uma região que é uniformemente ligada por ligações de internucleosídeo de fosforotioato. Em determinadas modalidades, o oligonucleotídeo é uniformemente ligado por ligações de internucleosídeo de fosforotioato. Em determinadas modalidades, cada ligação de internucleosídeo dooligonucleotídeo é selecionada de fosfodiéster e fosforotioato. Em determinadas modalidades, cada liagação de internucleosídeo dooligonucleotídeo é selecionado de fosfodiéster e fosforotioato e pelo menos uma ligação de internucleosídeo é fosforotioato.
[00709] Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 6 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 8 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 10 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 6 ligações consecutivas de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 8 ligações consecutivas de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 10 ligações consecutivas de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 12 ligações consecutivas de internucleosídeo fosforotioato. Nestas determinadas modalidades, pelo menos um destes blocos está localizado na extremidade 3' do oligonucleotídeo. Em determinadas modalidades, pelo menos um destes blocos está localizado em 3 nucleosídeos da extremidade 3' do oligonucleotídeo.
[00710] Os oligonucleotídeos com um de vários motivos de açúcar descritos aqui podem ter qualquer motivo de ligação. Por exemplo, os oligonucleotídeos, incluindo mas não se limitando aos descritos acima, podem ter um motivo de ligação selecionado a partir da tabela não- limitativa abaixo:
2. Compostos de siRNA
[00711] Em certas modalidades, os compostos antisense são compostos de RNAi de fita dupla (siRNA). Nessas modalidades, uma ou ambas as fitas podem compreender qualquer motivo de modificação descrito acima para o ssRNA. Em certas modalidades, os compostos de ssRNA podem ser RNA não-modificado. Em certas modalidades, os compostos de siRNA podem compreender nucleosídeos de RNA não- modificado, mas ligações de internucleosídeos modificados.
[00712] Várias modalidades se relacionam a composições de fita dupla, onde cada fita compreende um motivo definido pelo local de um ou mais nucleosídeos modificados ou não-modificados. Em certas modalidades, composições são fornecida compreendendo um primeiro e um segundo composto oligomérico que são completamente ou pelo menos parcialmente hibridizados de modo a formar uma região duplex e compreendendo ainda uma região que é complementar ao e hibridiza um alvo de ácido nucleico. É apropriado que essa composição compreende um primeiro composto oligomérico que seja uma fita antisense com complementaridade total ou parcial a um alvo de ácido nucleico e um segundo composto oligomérico que seja uma fita sense com uma ou mais regiões de complementaridade a e que forme pelo menos uma região duplex com o primeiro composto oligomérico.
[00713] As composições de várias modalidades modulam a expressão do gene hibridizando a um alvo de ácido nucleico que resulta na perda de sua função normal. Em algumas modalidades, o alvo de ácido nucleico é ANGPTL3. Em certas modalidades, a degradação do ANGPTL3 alvo é facilitada por um complexo de RISC ativado que é formado com as composições divulgadas neste documento.
[00714] Diversas modalidades são direcionadas a composições de fita dupla, onde uma das fitas é útil, por exemplo, influenciando o carregamento preferencial da fita oposta no complexo de RISC (ou clivagem). As composições são úteis para direcionar a moléculas de ácido nucleico selecionadas e para modular a expressão de um ou mais genes. Em algumas modalidades, as composições da presente invenção hibridizam para uma porção de um RNA alvo, resultando na perda da função normal do RNA alvo.
[00715] Certas modalidades são atraídas a composições de fita dupla, onde ambas as fitas compreendem um motivo de hemímero, um motivo completamente modificado, um motivo modificado posicional- mente ou um motivo alternado. Cada fita das composições da presente invenção pode ser modificada para preencher uma função particular, por exemplo, na via de siRNA. O uso de um motivo diferente em cada fita ou o mesmo motivo com diferentes modificações químicas em cada fita permite direcionar a fita antisense para o complexo de RISC, ao mesmo tempo em que inibe a incorporação da fita sense. Neste modelo, cada fita pode ser modificada de maneira independente, de modo que seja potencializada para essa função particular. A fita antisense pode ser modificada na terminação 5' para potencializar sua função em uma região do RISC, enquanto 3' pode ser modificado de maneira diferente, de modo a potencializar sua função em uma região diferente do RISC.
[00716] As moléculas de fita dupla de oligonucleotídeos podem ser uma molécula de polinucleotídeo de fita dupla que compreende regiões antisense e sense auto-complementar, onde a região antisense compreende a sequência de nucleotídeos que é complementar à sequência de nucleotídeos em uma molécula de ácido nucleico alvo ou uma porção sua e a região sense com uma sequência de nucleotídeos correspondente à sequência de ácido nucleico alvo ou uma porção sua. As moléculas de fita dupla do oligonucleotídeo podem ser montadas de dois oligonucleotídeos separados, onde uma fita é a fita de sense e o outro é a fita antisense, onde as fitas antisenses e sense são auto- complementares (ou seja, cada fita é composta pela sequência de nucleotídeos que é complementar à sequência de nucleotídeos em outra fita; tais como onde a fita antisense e fita sense formam uma estrutura duplex ou fita dupla, por exemplo no qual a região de fita dupla é cerca de 15 a 30, por exemplo, cerca de 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 ou 30 pares de base; a fita antisense compreende a sequência de nucleotídeos que é complementar à seqüência de nucleotídeos em uma molécula de ácido nucleico alvo ou uma fração e a fita sense compreende sequência nucleotídica correspondente para a sequência do ácido nucleico alvo ou uma fração (por exemplo, cerca de 15 a com 25 ou mais nucleotídeos da molécula de fita dupla do oligonucleotídeo são complementares para o ácido nucleico alvo ou uma parte dele). Alternativamente, o oligonucleotídeo de fita dupla é montado a partir de um único oligonucleotídeo, em que as regiões antsense e auto-complementar do siRNA estão ligados por meio de um ácido nucleico com base ou ligante à base de ácido não nucleico (s).
[00717] O oligonucleotídeo de fita dupla pode ser um polinucleotídeo com um duplex, duplex assimétrica,hairpin ou uma estrutura secundária em hairpin assimétrica, tendo regiões de sense e antisense de auto- complementar, em que a região antisense compreende a sequência de nucleotídeos que é complementar a sequência de nucleotídeos na molécula do ácido nucleico alvo separado ou uma porção da mesma e a sequência de nucleotídeos possuindo região sense corresponde à sequência de ácido nucleico alvo ou uma sua porção. O oligonucle- otídeo de fita dupla pode ser um polinucletídeo de fita única circular tendo duas ou mais estrutura em alça e uma haste compreendendo as regiões autocomplementares sense e antisense, em que a região antisense compreende uma sequência nucleotídica que é complementar à sequência nucleotídica numa molécula de ácido nucleico alvo ou uma porção desta e a região sense tendo uma sequência nucleotídica correspondente à sequência do ácido nucleico alvo ou uma porção desta, e em que o polinucleotídeo circular pode ser processado in vivo ou in vitro para gerar uma molécula de siRNA ativa capaz de mediar o RNAi.
[00718] Em certas modalidades, o oligonucleotídeo de fita dupla compreende sequências ou regiões sense e antisense separadas, onde as regiões sense e antisense são covalentemente ligadas por moléculas ligantes nucleotídicas ou não nucleotídicas conforme conhecido na técnica, ou são, alternativamente, ligadas não covalentemente por interações iônicas, ligação de hidrogênio, interações de van der Waals, interações hidrofóbicas e/ou interações por empilhamento. Em certas modalidades, o oligonucleotídeo de fita dupla compreende a sequência de nucleotídeos que é complementar à sequência de nucleotídeos de um gene alvo. Numa outra modalidade, o oligonucleotídeo de fita dupla interage com a sequência de um gene alvo de um modo que faz com que a inibição da expressão do gene alvo.
[00719] Tal como aqui utilizado, os oligonucleotíiedos de fita dupla não necessita de ser limitada a essas moléculas contendo apenas RNA, mas engloba ainda nucleotídeos quimicamente modificados e não- nucleotídeos. Em certas modalidades, a curta interferência das moléculas de ácido nucleico sem 2'-hidroxi (2'-OH) contendo nucleotídeos. Em certas modalidades de curta interferência dos ácidos nucleicos opcionalmente não incluem quaisquer ribonucleotídeos (por exemplo, tendo um grupo 2'-OH). Tais oligonucleotídeos de fita dupla que não requerem a presença de ribonucleotídeos no interior da molécula para suportar RNAi pode, contudo, ter um ligante ou ligantes anexado ou outros grupos ligados ou associados, unidades, cadeias ou contendo um ou mais nucleotídeos com grupos 2'-OH. Opcionalmente, os oligonucleotídeos de fita dupla pode compreender ribonucleotídeos a cerca de 5, 10, 20, 30, 40, ou 50% das posições de nucleotídeos. Tal como aqui utilizado, o termo de siRNA pretende ser equivalente a outros termos utilizados para descrever moléculas de ácido nucleico que são capazes de mediar a sequência de RNAi específico, por exemplo interferência curta de RNA (siRNA), RNA de fita dupla (dsRNA), micro- RNA (miRNA), RNA de hairpin curto (shRNA), oligonucleotídeo de interferência curta, ácido nucleico de interferência curta, oligonucleotídeo modificado de interferência curta, quimicamente modificados siRNA, gene silenciamento pós-transcricional RNA (ptgsRNA), e outros. Além disso, tal como aqui utilizado, o termo RNAi pretende ser equivalente a outros termos utilizados para descrever a sequência de RNA de interferência específica, tal como o silenciamento pós-transcricional de genes, a inibição de translação, ou epigenética. Por exemplo, os oligonucleotídeos de fita dupla pode ser utilizado para silenciar genes epigeneticamente tanto ao nível pós-transcricional e o nível pré-transcricional. Num exemplo não limitativo, a regulação da expressão do gene epigenética por moléculas de siRNA da invenção pode resultar a partir de modificação siRNA mediada da estrutura da cromatina ou padrão de metilação para alterar a expressão de genes (ver, por exemplo, Verdel et al., 2004, Science, 303, 672-676; Pal- Bhadra et al., 2004, Science, 303, 669-672; Allshire, 2002, Science, 297, 1818-1819; Volpe et al., 2002, Science, 297, 1833-1837; Jenuwein, 2002, Science, 297, 2215-2218; and Hall et al., 2002, Science, 297, 2232-2237).
[00720] Está contemplado que os compostos e as composições de diversas modalidades aqui proporcionadas podem alvejar ANGPTL3 por um silenciamento de genes mediado por dsRNA ou mecanismo de RNAi, incluindo, por exemplo, "hairpin" ou ansa pedunculada moléculas de cadeia dupla de RNA efetoras em que uma única cadeia de RNA com sequências de auto-complementar é capaz de assumir uma conformação de cadeia dupla, ou moléculas efetoras dsRNA duplex compreendendo duas cadeias separadas de RNA. Em várias modalidades, o dsRNA consiste inteiramente de ribonucleotídeos ou é constituído por uma mistura de ribonucleotídeos e desoxirribonucl- eotídeos, tais como as de RNA/DNA híbridos descritos, por exemplo, pelo documento WO 00/63364, depositado em abril 19, 2000, ou US Ser. No. 60/130.377, depositado em Abril 21, 1999. A molécula efetora de dsRNA ou dsRNA pode ser uma única molécula com uma região de auto-complementaridade tal que nucleotídeos de um segmento da molécula de pares de bases com nucleotídeos num outro segmento da molécula. Em várias modalidades, um dsRNA que consiste de uma única molécula consiste inteiramente de ribonucleotídeos ou inclui uma região de ribonucleotídeos que é complementar a uma região de desoxirribonucleotídeos. Alternativamente, o dsRNA pode incluir dois fios diferentes que têm uma região de complementaridade para o outro.
[00721] Em várias modalidades, ambas as fitas são inteiramente constituídas por ribonucleotídeos, uma cadeia consiste inteiramente de ribonucleotídeos e uma fita de desoxirribonucleotídeos consiste inteiramente, ou uma ou ambas as fitas contém uma mistura de ribonucleotídeos e desoxirribonucleotídeos. Em certas modalidades, as regiões de complementaridade são, pelo menos, 70, 80, 90, 95, 98, ou 100% complementares um ao outro e a uma sequência de ácido nucleico alvo. Em certas modalidades, a região do dsRNA que está presente numa conformação de fita dupla inclui, pelo menos, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 50, 75,100, 200 , 500, 1000, 2000 ou 5000 nucleotídeos ou inclui todos os nucleotídeos de um cDNA ou sequência de ácido nucleico alvo a ser representada outra no dsRNA. Em algumas modalidades, o dsRNA não contém quaisquer regiões de fita simples, tais como as extremidades de fita simples, ou o dsRNA é um hairpin. Em outras modalidades, o dsRNA tem uma ou mais regiões de fita simples ou saliências. Em certas modalidades, o RNA/DNA híbridos incluem uma fita ou região de DNA que é uma fita ou da região antisense (por exemplo, tem, pelo menos, 70, 80, 90, 95, 98, ou 100% de complementaridade de um ácido nucleico alvo) e uma fita de RNA ou região que é uma fita ou região sense (por exemplo, tem, pelo menos, 70, 80, 90, 95, 98, ou 100% de identidade com um ácido nucleico alvo), e vice-versa.
[00722] Em várias modalidades, o RNA/DNA híbrido é feito in vitro utilizando métodos de síntese químicas ou enzimáticas, tais como os aqui descritos ou os descritos no documento WO 00/63364, depositado em abril 19, 2000, ou US Ser. No. 60/130.377, depositado em Abril 21, 1999. Em outras modalidades, uma cadeia de DNA sintetizada in vitro é complexado com uma fita de RNA feito in vivo ou in vitro antes, depois, ou em simultâneo com a transformação da fita de DNA na célula. Em ainda outras modalidades, o dsRNA é um único ácido nucleico circular contendo um sense e uma região antisense, ou o dsRNA inclui um ácido nucleico circular e quer um segundo ácido nucleico circular ou um ácido nucleico linear (ver, por exemplo, o documento WO 00/63364, apresentado abril 19, 2000, ou US n° de série 60/130.377, depositado em 21de Abril de 1999.) Ácidos nucleicos circulares exemplares incluem estruturas lariat em que o grupo fosforil 5' livre de um nucleotídeo torna- se ligados ao 2' grupo hidroxil de outro nucleotídeo em um loop de volta fashion.
[00723] Em outras modalidades, o dsRNA inclui um ou mais nucleotídeos modificados no qual a posição 2' no açúcar contém um átomo de halogênio (tal como um grupo flúor) ou contém um grupo alcoxi (tal como um grupo metoxi), que aumenta a semi-vida do dsRNA in vivo ou in vitro em comparação com o dsRNA correspondente no qual a posição correspondente 2' contém um átomo de hidrogênio ou um grupo hidroxil. Em ainda outras modalidades, o dsRNA inclui uma ou mais ligações entre outros do que uma ligação fosfodiéster de ocorrência natural de nucleotídeos adjacentes. Exemplos de tais ligações incluem ligações fosforamida, fosforotioato e fosforoditioato. O dsRNA pode também ser moléculas do ácido nucleico modificadas quimicamente, como ensinado em US Pat. No. 6.673.661. Em outras modalidades, o dsRNA contém uma ou duas fitas tampadas, como divulgado, por exemplo, pelo documento WO 00/63364, depositado em abril 19, 2000, ou US Ser. No. 60/130.377, depositado em 21 de Abril de 1999.
[00724] Em outras modalidades, o dsRNA pode ser qualquer uma das moléculas de dsRNA, pelo menos, parcialmente divulgados no documento WO 00/63364, bem como qualquer uma das moléculas de dsRNA descritos em US Aplicação Provisória 60/399.998; e US Pedido Provisório 60/419.532, e PCT/US2003/033466, cujo ensinamento é aqui incorporado por referência. Qualquer um dos dsRNA podem ser expressos in vitro ou in vivo utilizando os métodos aqui descritos ou métodos padrão, tais como os descritos no documento WO 00/63364. Ocupantes
[00725] Em certas modalidades, os compostos antisense não são esperados para resultar na clivagem ou o ácido nucleico alvo através de RNase H ou para resultar na clivagem ou sequestro através da via RISC. Em algumas destas modalidades, a atividade antisense pode resultar de ocupação, em que a presença do composto antisense hibridizado interrompe a atividade do ácido nucleico alvo. Em algumas destas modalidades, o composto antisense pode ser modificado de modo uniforme ou podem compreender uma mistura de modificações e/ou nucleosídeos modificados e não modificados.Ácidos Nucleicos Alvo, Regiões Alvo e Sequências Nucleotídicas
[00726] As sequências de nucleotídeos que codificam ANGPTL3 incluem, sem limitação, as seguintes: a sequência humana como descrita no GenBank N° de Acesso NM_014495.2 (aqui incorporado como SEQ ID NO: 1) ou GenBank N° de Acesso NT_032977.9 nucleotídeos 33032001 a 33046000 (aqui incorporados como SEQ ID NO: 2). Entende-se que a sequência estabelecida em cada SEQ ID NO nos exemplos contidos neste documento é independente de qualquer modificação de uma fração de açúcar, uma ligação internucleosídeos ou uma nucleobase. Como tais, os compostos antisense definidos por uma SEQ ID NO podem compreender, independentemente, uma ou mais modificações para uma fração de açúcar, uma ligação internucleosídeos ou uma nucleobase. Os compostos antisense descritos pelo Número de Isis (N° de Isis) indicam uma combinação da sequência de nucleobase e motivo.
[00727] Em determinadas modalidades, uma região alvo é uma região estruturalmente definida do ácido nucleico alvo. Por exemplo, uma região alvo pode abranger uma 3' UTR, uma 5' UTR, um éxon, um íntron, uma junção éxon/íntron, uma região de codificação, uma região de iniciação da tradução, uma região de terminação da tradução, ou outra região de ácido nucleico definida. As regiões estruturalmente definidas para ANGPTL3 podem ser obtidas pelo número de acesso de bancos de dados de sequência tal como NCBI e tais informações são incorporadas neste documento por referência. Em cercas modalidades, uma região alvo pode abranger a sequência de um sítio alvo 5' de um segmento alvo dentro da região alvo para um sítio alvo 3' de outro segmento alvo dentro da região alvo.
[00728] Em certas modalidades, um "um segmento alvo" é uma subporção menor de uma região alvo dentro de um ácido nucleico. Por exemplo, um segmento alvo pode ser a sequência de nucleotídeos de um ácido nucleico alvo à qual uma ou mais composto antisense se destina. "Sítio alvo 5'" ou "sítio de início 5'" refere-se ao nucleotídeo mais a 5' de um segmento alvo. "Sítio alvo 3'" ou "sítio de terminação 3'" refere-se ao nucleotídeo mais a 3' de um segmento alvo.
[00729] O direcionamento inclui a determinação de, pelo menos, um segmento alvo ao qual um composto antisense hibridiza, de modo que ocorra um efeito desejado. Em certas modalidades, o efeito desejado é uma redução nos níveis de ácido nucleico alvo do mRNA. Em certas modalidades, o efeito desejado é a redução dos níveis de proteína codificada pelo ácido nucleico alvo ou uma alteração fenotípica associada ao ácido nucleico alvo.
[00730] Uma região alvo pode conter um ou mais segmentos alvo. Múltiplos segmentos alvo dentro de uma região alvo podem estar sobrepostos. Alternativamente, eles podem estar não sobrepostos. Em certas modalidades, os segmentos alvo dentro de uma região alvo são separados por não mais do que cerca de 300 nucleotídeos. Em certas modalidades, os segmentos alvo dentro de uma região alvo são separados por um número de nucleotídeos que é, é cerca de, se não mais do que, se não mais do que cerca de, 250, 200, 150, 100, 90, 80, 70, 60, 50, 40, 30, 20, ou 10 nucleotídeos no ácido nucleico alvo, ou é uma faixa definida por quaisquer dois dos valores precedentes. Em certas modalidades, os segmentos alvo dentro de uma região alvo são separados por não mais do que, ou não mais do que cerca de, 5 nucleotídeos no ácido nucleico alvo. Em certas modalidades, os segmentos alvo são contíguos. Contempla-se regiões alvo definidas por uma faixa tendo um ácido nucleico de iniciação que é qualquer um dos sítios alvo 5' e sítios alvo 3' listados neste documento.
[00731] Os segmentos alvo adequados podem ser encontrados dentro de uma 5' UTR, uma região de codificação, uma 3' UTR, um íntron, um éxon, ou uma junção de éxon/íntron. Os segmentos alvo contendo um códon de iniciação ou um códon de parada também são segmentos alvo adequados. Um segmento alvo adequado pode especificamente excluir uma determinada região estruturalmente definida, tal como o códon de iniciação ou o códon de parada.
[00732] A determinação dos segmentos alvo adequados pode incluir uma comparação da sequência de um ácido nucleico alvo com outras sequências ao longo do genoma. Por exemplo, o algoritmo BLAST pode ser usado para identificar regiões de similaridade entre ácidos nucleicos diferentes. Esta comparação pode impedir a seleção de sequências do composto antisense que podem hibridizar de forma não específica sequências diferentes de um ácido nucleico alvo selecionado (isto é, sequências não alvo ou fora do alvo).
[00733] Pode haver variação na atividade (por exemplo, conforme definido pela redução percentual dos níveis de ácido nucleico alvo) dos compostos antisense dentro de uma região alvo ativa. Em determinadas modalidades, reduções nos níveis de mRNA de ANGPTL3 são indicativas de inibição da expressão de ANGPTL3. As reduções nos níveis de uma proteína de ANGPTL3 também são indicativas de inibição da expressão de mRNA alvo. Além disso, as alterações fenotípicas, tais como a redução do nível de colesterol, colesterol LDL, triglicérido, ou glicose, podem ser indicativas de inibição de mRNA e/ou expressão da proteína de ANGPTL3 .
Hibridização
[00734] Em algumas modalidades, a hibridização ocorre entre um composto antisense divulgado neste documento e um ácido nucleico de ANGPTL3. O mecanismo mais comum de hibridização envolve a ligação de hidrogênio (por exemplo, ligação de hidrogênio de Watson-Crick, Hoogsteen ou Hoogsteen invertida) entre as nucleobases complementares das moléculas de ácido nucleico.
[00735] A hibridização pode ocorrer sob condições variáveis. Condições rigorosas são dependentes da sequência e são determinadas pela natureza e composição das moléculas de ácido nucleico a serem hibridizadas.
[00736] Os métodos para determinar se uma sequência é especificamente hibridizável com um ácido nucleico alvo são bem conhecidos na técnica (Sambrook e Russell, Molecular Cloning: A Laboratory Manual, 3a Ed., 2001). Em determinadas modalidades, os compostos antisense fornecidos neste documento são especificamente hibridizáveis com um ácido nucleico de ANGPTL3.
Complementaridade
[00737] Um composto antisense e um ácido nucleico alvo são complementares uns aos outros quando um número suficiente de nucleobases do composto antisense pode fazer uma ligação de hidrogênio com as nucleobases correspondentes do ácido nucleico alvo, de modo que ocorra um efeito desejado (por exemplo, inibição antisense de um ácido nucleico alvo, tal como um ácido nucleico de ANGPTL3).
[00738] Um composto antisense pode hibridizar em um ou mais segmentos de ácido nucleico de ANGPTL3 de modo que os segmentos adjacentes ou intermediários não estejam envolvidos no evento de hibridização (por exemplo, uma estrutura de loop, estrutura de incompatibilidade ou hairpin).
[00739] Em determinadas modalidades, os compostos antisense fornecidos neste documento, ou uma porção especificada dos mesmos, são, são pelo menos, 70%, 75%, 80%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, ou 100% complementares a um ácido nucleico de ANGPTL3, uma região alvo, segmento alvo, ou porção especificada dos mesmos. Em certas modalidades, os compostos antisense aqui fornecidos, ou uma porção específica dos mesmos, são, ou são pelo menos, 70%, 75%, 80%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, ou 100% complementares à sequência de uma ou mais das SEQ ID NOs: 1-2. A complementaridade percentual de um composto antisense com um ácido nucleico alvo pode ser determinada usando os métodos de rotina.
[00740] Por exemplo, um composto antisense, no qual 18 de 20 nucleobases do composto antisense são complementares para uma região alvo e, portanto, se hibridizaria especificamente, representaria 90 por cento de complementaridade. Neste exemplo, as nucleobases não complementares restantes podem ser agrupadas ou intercaladas com nucleobases complementares e não precisam ser contíguas umas às outras ou às nucleobases complementares. Como tal, um composto antisense o qual tem 18 nucleobases de comprimento, com 4 (quatro) nucleobases não complementares que são flanqueadas por duas regiões de complementaridade completa com o ácido nucleico alvo teria 77,8% de complementaridade geral com o ácido nucleico alvo e, assim, estaria dentro do escopo da presente invenção. A complementaridade percentual de um composto antisense com uma região de um ácido nucleico alvo pode ser determinada rotineiramente, usando os programas BLAST (ferramentas de busca de alinhamento local básico) e os programas PowerBLAST conhecidos na técnica (Altschul et al., J. Mol. Biol., 1990, 215, 403 410; Zhang e Madden, Genome Res., 1997, 7, 649 656). A homologia percentual, a identidade de sequência ou a complementaridade podem ser determinadas, por exemplo, pelo programa Gap (Wisconsin Sequence Analysis Package, Versão 8 para Unix, Genetics Computer Group, University Research Park, Madison Wis.), usando as configurações padrão, que usa o algoritmo de Smith e Waterman (Adv. Appl. Math., 1981, 2, 482 489).
[00741] Em certas modalidades, os compostos antisense aqui proporcionados, ou porções dos mesmos especificado, são completa-mente complementares (isto é. 100% complementares) a um ácido nucleico alvo, ou porção especificada do mesmo. Por exemplo, um composto antisense pode ser totalmente complementar a um ácido nucleico de ANGPTL3, ou a uma região alvo, ou a um segmento alvo ou a uma sequência alvo deste. Conforme usado neste documento, "totalmente complementar" significa que cada nucleobase de um composto antisense é capaz de parear a base precisamente com as nucleobases correspondentes de um ácido nucleico alvo. Por exemplo, um composto antisense 20 de nucleobases é totalmente complementar para uma sequência alvo que é de 400 nucleobases de comprimento, desde que haja uma parte 20 de nucleobases correspondente do ácido nucleico alvo que é totalmente complementar ao composto antisense. Totalmente complementar também pode ser usado em referência a uma porção especificada do primeiro e/ou do segundo ácido nucleico. Por exemplo, uma porção de 20 nucleobases de um composto antisense de 30 nucleobases pode ser "totalmente complementar" a uma sequência alvo que tem 400 nucleobases de comprimento. A porção de 20 nucleobases do oligonucleotídeo de 30 nucleobases é totalmente complementar à sequência alvo, se a sequência alvo tiver uma porção correspondente de 20 nucleobases, em que cada nucleobase é complementar à porção de 20 nucleobases do composto antisense. Ao mesmo tempo, o composto antisense de 30 nucleobases inteiro pode ou não ser totalmente complementar à sequência alvo, dependendo de se as 10 nucleobases restantes do composto antisense também forem complementares à sequência alvo.
[00742] A localização de uma nucleobase não complementar pode estar na extremidade 5' ou na extremidade 3' do composto antisense. Alternativamente, a nucleobase ou nucleobases não complementar pode estar numa posição interna do composto antisenso. Quando duas ou mais nucleobases não complementares estão presentes, elas podem ser contíguas (isto é. ligada) ou não contíguas. Em uma modalidade, uma nucleobase não complementar está localizada no segmento de asa de um oligonucleotídeo antisense de gapmer.
[00743] Em certas modalidades, os compostos antisense que têm, ou têm até 10, 12, 13, 14, 15, 16, 17, 18, 19 ou 20 nucleobases de comprimento compreendem não mais que 4, não mais que 3, não mais que 2 ou não mais que 1 nucleobase(s) não complementar(es) em relação a um ácido nucleico alvo, como um ácido nucleico de ANGPTL3 ou sua porção especificada.
[00744] Em certas modalidades, os compostos antisense que têm, ou têm até 10, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, ou 30 nucleobases de comprimento compreendem não mais do que 6, não mais do que 5, não mais do que 4, não mais do que 3, não mais do que 2, ou não mais do que 1 nucleobase(s) não complementar(es) em relação a um ácido nucleico alvo, tal como um ácido nucleico de ANGPTL3, ou porção especificada do mesmo.
[00745] Os compostos antisense fornecidos neste documento também incluem aqueles que são complementares a uma porção de um ácido nucleico alvo. Conforme usado neste documento, "porção" se refere a um número definido de nucleobases contíguas (ou seja, ligadas) dentro de uma região ou segmento de um ácido nucleico alvo. Uma "porção" também pode se referir a um número definido de nucleobases contíguas de um composto antisense. Em certas modalidades, os compostos antisense são complementares a pelo menos uma porção de 8 nucleobases de um segmento alvo. Em certas modalidades, os compostos antisense são complementares a pelomenos uma porção de 10 nucleobases de um segmento alvo. Em certas modalidades, os compostos antisense são complementares a pelomenos uma porção de 15 nucleobases de um segmento alvo. Também são contemplados os compostos antisense que são complementares a pelo menos uma porção de 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20,ou mais nucleobases de um segmento alvo, ou um intervalo definido por quaisquer dois desses valores.
Identidade
[00746] Os compostos antisense fornecidos neste documento também podem ter uma identidade percentual definida para uma sequência de nucleotídeo específica, SEQ ID NO, ou sequência de um composto representado por um número de Isis específico, ou porção do mesmo. Conforme usado neste documento, um composto antisense é idêntico à sequência divulgada neste documento se tiver a mesma capacidade de pareamento de nucleobase. Por exemplo, um RNA que contém uracil no lugar de timidina em uma sequência de DNA divulgada seria considerado idêntico à sequência de DNA, uma vez que ambos, uracil e timidina, pareiam com a adenina. Versões encurtadas e alongadas dos compostos antisense descritos neste documento, bem como compostos tendo bases não idênticas em relação aos compostos antisense fornecidos neste documento, também são contempladas. As bases não idênticas podem ser adjacentes umas às outras ou dispersas por todo o composto antisense. A identidade percentual de um composto antisense é calculada de acordo com o número de bases que têm pareamento de bases idêntico em relação à sequência a qual está sendo comparado.
[00747] Em certas modalidades, os compostos antisense, ou porções dos mesmos, são pelo menos 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% ou 100% idênticos a um ou mais dos compostos antisense ou SEQ ID NOs, ou uma porção dos mesmos, divulgados neste documento.
Modificações
[00748] Um nucleosídeo é uma combinação de base-açúcar. A porção de nucleobase (também conhecida como base) do nucleosídeo é normalmente uma fração de base heterocíclica. Os nucleotídeos são nucleosídeos que incluem adicionalmente um grupo fosfato covalentemente ligado à porção de açúcar do nucleosídeo. Para os nucleosídeos que incluem um açúcar de pentofuranosil, o grupo fosfato pode ser ligado à fração de hidroxil em 2', 3' ou 5' do açúcar. Os oligonucleotídeos são formados através da ligação covalente dos nucleosídeos adjacentes um ao outro, para formar um oligonucleotídeo polimérico linear. Dentro da estrutura do oligonucleotídeo, os grupos fosfato são comumente referidos como formadores das ligações internucleosídeos do oligonucleotídeo.
[00749] As modificações dos compostos antisense englobam substituições ou mudanças das ligações internucleosídeos, frações de açúcar, ou nucleobases. Os compostos antisense modificados são frequentemente preferidos sobre as formas nativas devido a propriedades desejáveis, tais como, por exemplo, maior absorção celular, maior afinidade para o ácido nucleico alvo, maior estabilidade na presença de nucleases, ou maior atividade inibitória.
[00750] Os nucleosídeos quimicamente modificados também podem ser empregados para aumentar a afinidade de ligação de um oligonucleotídeo anisense encurtado ou truncado a seu ácido nucleico alvo. Consequentemente, resultados comparáveis podem ser frequentemente obtidos com compostos antisense menores que possuem esses nucleosídeos quimicamente modificados.Ligações Internucleosídeos Modificadas
[00751] A ligação internucleosídeos de ocorrência natural de RNA e DNA é uma ligação de fosfodiéster 3' para 5'. Compostos antisense possuindo um ou mais modificado, isto é. de ocorrência não natural, são frequentemente selecionados sobre os compostos antisense que têm ligações internucleosídeos de ocorrência natural, devido às propriedades desejáveis, tais como, por exemplo, maior absorção celular, maior afinidade para os ácidos nucleicos alvos, e maior estabilidade na presença de nucleases.
[00752] Os oligonucleotídeos tendo ligações internucleosídeos modificadas incluem ligações internucleosídeos que mantêm um átomo de fósforo, bem como as ligações internucleosídeos que não têm um átomo de fósforo. As ligações internucleosídeos representativas contendo fósforo incluem, mas não estão limitadas a, fosfodiésteres, fosfotriésteres, metilfosfonatos, fosforamidato, e fosforotioatos. Os métodos de preparação das ligações contendo fósforo e não contendo fósforo são bem conhecidos.
[00753] Em certas modalidades, os compostos antisense direcionados para um ácido nucleico de ANGPTL3 compreendem uma ou mais ligações internucleosídeos modificadas. Em certas modalidades, as ligações internucleosídeos modificadas são ligações de fosforotioato. Em certas modalidades, cada ligação internucleosídeos de um composto antisense é uma ligação internucleosídeos de fosforotioato.
[00754] Em determinadas modalidades, oligonucleotídeos compõem ligações de internucleosídeo modificado dispostas ao longo dooligonucleotídeo, ou região do mesmo, em um padrão definido ou motivo de ligação de internucleosídeo modificado. Em certasmodalidades, as ligações internucleosídicas são dispostas em um motivo incompleto. Nessas modalidades, as ligações de internucleo- sídeo em cada uma das duas regiões de asa são diferentes das ligações de internucleosídeo na região da lacuna. Em certas modalidades, as ligações de internucleosídeo nas asas são fosfodiéster e as ligações de internucleosídeo na lacuna são fosforotioato. O motivo nucleosídeo é selecionado, independentemente, de modo que esses oligonucleotí- deos que têm uma ligação de internucleosídeo incompleta possam ou não possam ter um motivo de modificação nucleosídeo incompleto e, se ele tiver um motivo nucleosídeo incompleto, os comprimentos da asa e da lacuna podem ou não ser os mesmos.
[00755] Em determinadas modalidades, os oligonucleotídeos compreendem uma região com um motivo de ligação de internucleo- sídeo alternado. Em determinadas modalidades, os oligonucleotídeos da presente invenção compreendem uma região de ligações de internucleosídeo uniformemente modificadas. Em algumas dessas modalidades, o oligonucleotídeo compreende uma região que é uniformemente ligada por ligações de internucleosídeo de fosforotioato. Em determinadas modalidades, o oligonucleotídeo é uniformemente ligado por ligações de internucleosídeo de fosforotioato. Em determinadas modalidades, cada ligação de internucleosídeo dooligonucleotídeo é selecionada de fosfodiéster e fosforotioato. Em determinadas modalidades, cada liagação de internucleosídeo dooligonucleotídeo é selecionado de fosfodiéster e fosforotioato e pelo menos uma ligação de internucleosídeo é fosforotioato.
[00756] Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 6 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 8 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos 10 ligações de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 6 ligações consecutivas de internucleosídeo fosforotioato. Em determi-nadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 8 ligações consecutivas de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 10 ligações consecutivas de internucleosídeo fosforotioato. Em determinadas modalidades, o oligonucleotídeo compreende pelo menos um bloco de, pelo menos, 12 ligações consecutivas de internucleosídeo fosforotioato. Nestas determinadas modalidades, pelo menos um destes blocos está localizado na extremidade 3' do oligonucleotídeo. Em determinadas modalidades, pelo menos um destes blocos está localizado em 3 nucleosídeos da extremidade 3' do oligonucleotídeo.
[00757] Em certas modalidades, os oligonucleotídeos compreendem uma ou mais ligações de metilfosfonato. Em certas modalidades, os oligonucleotídeos tendo um gapmer motivonucleosídeo compreende um motivo de ligação compreendendo todas as ligações fosforotioato, exceto por uma ou duas ligações metilfosfonato. Em certas modalidades, uma ligação metilfosfonato está na abertura central de um oligonucleotídeo possuindo um gapmer motivo nucleosídeo.
[00758] Em certas modalidades, é desejável providenciar o número de ligações internucleosídicas de fosforotioato e ligações internucleosídeos fosfodiéster para manter a resistência à nuclease. Em certas modalidades, é desejável providenciar o número e posição de ligações internucleosídicas de fosforotioato e número e posição das ligações de internucleosídeos fosfodiéster para manter a resistência à nuclease. Em certas modalidades, o número de ligações de internucleosídeos de fosforotioato pode ser diminuído e o número de ligações de internucleosídeos de fosfodiéster pode ser aumentado. Em certas modalidades, o número de ligações de internucleosídeos de fosforotioato pode ser diminuído e o número de ligações de internucleosídeos de fosfodiéster pode ser aumentado enquanto ainda mantém a resistência nuclease. Em certas modalidades, é desejável diminuir o número de ligações de internucleosídeos fosforotioato, mantendo a resistência a nucleases. Em certas modalidades, é desejável aumentar o número de ligações de internucleosídeos fosfodiéster, mantendo a resistência a nucleases.Frações de Açúcar Modificadas
[00759] Os compostos antisense da invenção podem opcionalmente conter um ou mais nucleosídeos, em que o grupo de açúcar foi modificado. Esses nucleosídeos de açúcar modificado podem conferir maior estabilidade à nuclease, maior afinidade de ligação, ou alguma outra propriedade biológica benéfica para os compostos antisense. Em certas modalidades, os nucleosídeos compreendem frações de anel de ribofuranose quimicamente modificado. Exemplos de anéis de ribofuranose quimicamente modificados incluem, sem limitação, a adição de grupos substituintes (incluindo grupos substituintes em 5' e 2', ligações em ponte de átomos de aneis não geminais para formar ácidos nucleicos bicíclicos (BNA), a substituição do átomo de oxigênio do anel de ribosil por S, N(R), ou C(R1)(R2) (R, R1 e R2 são cada umindependentemente H, alquil C1-C12 ou um grupo protetor) e suas combinações. Exemplos de açúcares modificados incluem 2'-F 5'-metil- nucleosídeo substituído (ver Pedido PCT Internacional WO 2008/101157 publicado em 8/21/08 para outros descritos nucleosídicos 5 ', 2'-bis substituído) ou a substituição do ribosilo átomo de oxigénio do anel com S com substituição adicional na posição 2' (ver publicação US Pedido de Patente US2005-0130923, publicado em 16 de Junho, 2005) ou, alternativamente, 5'-substituição de um BNA (ver Pedido PCT Internacional WO 2007/134181 publicado em 11/22/07, em que o LNA é substituído com, por exemplo, um 5'-metil ou um grupo 5'-vinil).
[00760] Os exemplos de nucleosídeos que têm porções de açúcar modificadas incluem, sem limitação de nucleosídeos compreendendo grupos substituintes 5'-vinil, 5'-metil (R ou S), 4'-S, 2'-F, 2'-OCH3, 2'- OCH2CH3, 2'-OCH2CH2F e 2'-O (CH2)2OCH3 . O substituinte na posição 2' pode também ser selecionado a partir de alilo, amino, azido, tio, O- alil, alquil O-C1-C10 , OCF3, OCH2F, O (CH2)2SCH3, O (CH2)2-ON (Rm) (Rn), O-CH2-C (= O) -N (Rm) (Rn), e O-CH2-C (= O) -N (Rl)-(CH2)2-N (Rm) (Rn), em que cada Rl, Rm e Rn é, independentemente, H ou alquil substituído ou não substituído C1-C10.
[00761] Conforme usado neste documento, os "nucleosídeos bicíclicos" se referem aos nucleosídeos modificados que compreendem uma fração de açúcar bicíclico. Exemplos de ácidos nucleicos bicíclicos (BNAs) incluem, sem limitação, os nucleosídeos que compreendem uma ponte entre os átomos 4' e 2' do anel de ribosila. Em determinadas modalidades, compostos antisense aqui fornecidos incluem um ou mais nucleosídeos BNA onde a ponte é composta por uma das fórmulas: 4'- (CH2)-O-2' (LNA); 4'-(CH2)-S-2'; 4'-(CH2)2-O-2' (ENA); 4'-CH(CH3)-O-2' and 4'-CH(CH2OCH3)-O-2' (e respectivos análogos respectivos, ver patente U.S. 7.399.845, publicada em 15 de julho de 2008); 4'-C(CH3)(CH3)-O-2' (e respectivos análogos respectivos, ver PCT/US2008/068922 publicado como WO 2009/006478, publicado em 8 de janeiro de 2009); 4'-CH2- N(OCH3)-2' (e respectivos análogos respectivos, ver PCT/US2008/064591 publicado como WO/2008/150729, publicado em 11 de dezembro de 2008); 4'-CH2-O-N(CH3)-2' (ver pedido de patente U.S. US2004-0171570, publicado em 2 de setembro de 2004); 4'-CH2-N(R)-O-2', onde R é H, alquil C1- C12 ou um grupo protetor (ver patente U.S. 7.427.672, publicada em 23 de setembro de 2008); 4'-CH2-C(H)(CH3)-2' (ver Zhou et al, J. Org. Chem., 2009, 74, 118-134); e 4'-CH2-C(=CH2)-2' (e seus análogos, ver PCT/US2008/066154 publicada como WO 2008/154401, publicada em 8 de dezembro de 2008).
[00762] Mais nucleosídos bicíclicos têm sido relatados na literatura (ver, por exemplo: Srivastava et al., J. Am. Chem. Soc., 2007, 129(26) 8362-8379; Frieden et al., Nucleic Acids Research, 2003, 21, 63656372; Elayadi et al., Curr. Opinion Invens. Drugs, 2001, 2, 558-561; Braasch et al., Chem. Biol., 2001, 8, 1-7; Orum et al., Curr. Opinion Mol. Ther., 2001, 3, 239-243; Wahlestedt et al., Proc. Natl. Acad. Sci. U. S. A., 2000, 97, 5633-5638; Singh et al., Chem. Commun., 1998, 4, 455456; Koshkin et al., Tetrahedron, 1998, 54, 3607-3630; Kumar et al., Bioorg. Med. Chem. Lett., 1998, 8, 2219-2222; Singh et al., J. Org. Chem., 1998, 63, 10.035-10.039; Patentes US N°s .: 7.741.457;7.399.845; 7.053.207; 7.034.133; 6.794.499; 6.770.748; 6.670.461;6.525.191; 6.268.490; Publicação de Patente US N°s US2008-0039618; US2007-0287831; US2004-0171570; Pedidos de Patentes U.S., números de série .: 61/097.787; 61/026,995; e pedidos internacionais: WO 2009/006478; WO 2008/154401; WO 2008/150729; WO 2009/100320; WO 2011/017521; WO 2009/067647; WO 2010/036698; WO 2007/134181; WO 2005/021570; WO 2004/106356; WO 99/14226. Cada um dos nucleosídeos bicíclicos anteriores pode ser preparado tendo uma ou mais configurações de açúcar estereoquímico, incluindo, por exemplo, α-L-ribofuranose e β-D-ribofuranose (vide pedido internacional PCT PCT/DK98/00393, publicado em 25 de Março de 1999 como WO 99/14226).
[00763] Conforme usado neste documento, "nucleosídeos monocí- clicos" se refere aos nucleosídeos que compreendem frações de açúcar modificado que não sejam frações de açúcar bicíclico. Em certas modalidades, a fração de açúcar, ou análogo da fração de açúcar, de um nucleosídeo pode ser modificado ou substituído em qualquer posição.
[00764] Conforme usado neste documento, "nucleosídeo bicíclico 4'2'" ou "nucleosídeo bicíclico 4' para 2'" se refere a um nucleosídeo bicíclico compreendendo um anel de furanose compreendendo uma ponte que conecta dois átomos de carbono do anel de furanose conecta o átomo de carbono 2' e o átomo de carbono 4' do anel de açúcar.
[00765] Em certas modalidades, porções de açúcar bicíclicos de nucleosídeos BNA incluem, mas não estão limitados aos compostos possuindo pelo menos uma ponte entre o 4' e da posição 2' da porção de açúcar pentofuranosil em que essas pontes compreendem, independentemente, 1 ou de 1 a 4 grupos ligados selecionados independentemente a partir de -[C(Ra)(Rb)]n-, -C(Ra)=C(Rb)-, -C(Ra)=N- , -C(=NRa)-, -C(=O)-, -C(=S)-, -O-, -Si(Ra)2-, -S(=O)x-, e -N(Ra)-; em que: x é 0, 1, ou 2; n é 1, 2, 3, ou 4; cada Ra e Rb é, independentemente, H, um grupo protetor, hidroxil, alquil C1-C12, alquil C1-C12 substituído, alquenil C2-C12, alquenil C2-C12 substituído, alquinil C2-C12, alquinil C2C12 substituído, aril C5-C20, aril C5-C20, radical heterocíclico, radical heterocíclico substituído, heteroaril, heteroaril substituído, radical alicíclico C5-C7, radical alicíclico C5-C7 substituído, halogênio, OJ1, NJ1J2, SJ1, N3, COOJ1, acill (C(=O)-H), acil substituído, CN, sulfonil (S(=O)2-J1), ou sulfoxil (S(=O) -J1); e
[00766] cada J1 e J2 é, independentemente, H, alquil C1-C12 , alquil substituído C1-C12 , alquenil C2-C12 , alquenil substituído C2-C12 , alquinil C2-C12 , alquinil substituído C2-C12 , aril C5-C20 , aril substituído C5-C20 , acil (C (= O) -H), acil substituído, um radical heterocíclico, um radical heterociclo substituído, aminoalquil C1-C12 , aminoalquil substituído C1-C12 ou um grupo protetor.
[00767] Em certas modalidades, a ponte de uma porção de açúcar bicíclico é -[C(Ra)(Rb)]n-, -[C(Ra)(Rb)]n-O-, -C(RaRb)-N(R)-O- ou -C(RaRb)-O-N(R)-. Em certas modalidades, a ponte é 4'-CH2-2', 4'- (CH2)2-2', 4'-(CH2)3-2', 4'-CH2-O-2', 4'-(CH2)2-O-2', 4'-CH2-O-N(R)-2' e 4'-CH2-N(R)-O-2'- em que cada R é, independentemente, H, um grupo protetor ou alquil C1-C12.
[00768] Em certas modalidades, os nucleosídeos bicíclicos sãodefinidos ainda pela configuração isomérica. Por exemplo, um nucleosído que compreende um agente de formação de ponte de 4'- (CH2)-O-2‘, pode estar na configuração a-L ou na configuração β-D. Anteriormente, o BNA de a-L-metileno-óxi (4'-CH2-O-2‘) foram incorporados aos oligonucleotídeos antisense que mostraram atividade antisense (Frieden et al., Nucleic Acids Research,2003, 21, 6365-6372).
[00769] Em certas modalidades, nucleosídos bicíclicos incluem os que têm uma ponte de 4 'a 2', em que essas pontes incluem, sem limitação,a-L-4'-(CH2)-O-2', β-D-4'-CH2-O-2', 4'-(CH2)2-O-2', 4'-CH2- O-N(R)-2', 4'-CH2-N(R)-O-2', 4'-CH(CH3)-O-2', 4'-CH2-S-2', 4'-CH2- N(R)-2', 4'-CH2-CH(CH3)-2', and 4'-(CH2)3-2', em que R é H, um grupo protetor ou alquil C1-C12.
[00770] Em certos modo de realização, nucleosídos bicíclicos têm a fórmula:
[00771] em que:
[00772] Bx é uma fração de base heterocíclica;
[00773] -Qa-Qb-Qc- é -CH2-N(Rc)-CH2-, -C(=O)-N(Rc)-CH2-, -CH2-O-N(Rc)-, -CH2-N(Rc)-O- ou -N(Rc)-O-CH2;
[00774] Rc é alquil C1-C12 ou um grupo protetor de amino; e
[00775] Ta e Tb são, cada um, independentemente, H, um grupo de proteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte.
[00776] Em certas modalidades, nucleosídos bicíclicos têm a fórmula:
[00777] em que:
[00778] Bx é uma fração de base heterocíclica;
[00779] Ta e Tb são, cada um, independentemente, H, um grupo deproteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte;
[00780] Za é alquil C1-C6 , alquenil C2-C6 , alquinil C2-C6 , alquilsubstituído C1-C6 , alquenil substituído C2-C6 , alquinil substituído C2-C6 , acil, acil substituído, amida substituída, tiol ou tio substituído.
[00781] Numa modalidade, cada um dos grupos substituídos é, independentemente, mono ou poli substituído com grupos substituintes independentemente selecionados a partir de halogênio, oxo, hidroxil, JOc, NJcJd, SJc, N3, OC (= X) Jc, e NJeC (= X) NJcJd, em que cada símbolo Jc, Jd e Je é, independentemente, H, alquil C1-C6 , ou alquil substituído C1-C6 e X é O ou NJc.
[00782] Em certas modalidades, nucleosídos bicíclicos têm a fórmula:
[00783] em que:
[00784] Bx é uma fração de base heterocíclica;
[00785] Ta e Tb são, cada um, independentemente, H, um grupo de proteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte;
[00786] Zb é alquil C1-C6 , alquenil C2-C6 , alquinil C2-C6 , alquil substituído C1-C6 , alquenil substituído C2-C6 , alquinil substituído C2-C6 ou acil substituído (C (= O) -).
[00787] Em certas modalidades, nucleosídos bicíclicos têm a fórmula:
[00788] em que:
[00789] Bx é uma fração de base heterocíclica;
[00790] Ta e Tb são, cada um, independentemente, H, um grupo deproteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte;
[00791] Rd é alquil C1-C6 , alquil substituído C1-C6 ,alquenil C2-C6 , alquenil substituído C2-C6 , alquinil C2-C6 ou alquinil substituído C2-C6 ;
[00792] cada qa, qb, qc e qd é, independentemente, H, halogênio, alquil C1-C6 , alquil substituído C1-C6 , alquenil C2-C6 , alquenilsubstituído C2-C6 , alquinil C2-C6 ou alquinil substituído C2-C6 , alcoxil C1-C6 , alcoxil substituído C1-C6 , acil, acil substituído C1-C6 aminoalquil substituídos ou aminoalquil C1-C6 ;
[00793] Em certas modalidades, nucleosídos bicíclicos têm a fórmula:
[00794] em que:
[00795] Bx é uma fração de base heterocíclica;
[00796] Ta e Tb são, cada um, independentemente, H, um grupo deproteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte;
[00797] qa, qb, qe e Qf são cada um, independentemente, hidrogênio, halogêneo, alquil C1-C12 , alquil substituído C1-C12 , alquenil C2-C12, alquenil substituído C2-C12 , alquinil C2-C12 , alquinil substituído C2-C12, alcoxi C1-C12 , alcoxi substituído C1-C12 , OJj, SJj, SOJj, SO2Jj, NJjJk, N3, CN, C(=O)OJj, C(=O)NJjJk, C(=O)Jj, O-C(=O)NJjJk, N(H)C(=NH)NJjJk, N(H)C(=O)NJjJkor N(H)C(=S)NJjJk;
[00798] ou qe e qf em conjunto são = C(Qg)(qh);
[00799] qg e qh são cada um, independentemente, alquil de H,halogênio, C1-C12 ou alquil substituído de C1-C12 .
[00800] A síntese e a preparação de nucleosídos bicíclicos deadenina, citosina, guanina, 5-metil-citosina, timina e uracil tendo uma ponte 4'-CH2-O-2', juntamente com a sua oligomerização, e propriedades de reconhecimento de ácido nucleico foram descritas (Koshkin et al., Tetraedro, 1998, 54, 3607-3630). A síntese denucleosídos bicíclicos também foi descrita em WO 98/39352 e WO 99/14226.
[00801] Os análogos de nucleosídos diferentes bicíclicos que têm grupos de ponte 4 'para 2', tais como 4'-CH2-O-2 'e 4'-CH2-S-2 ', foram também preparados (Kumar et al., Bioorg. Med. Chem. Lett., 1998, 8, 2219-2222). Preparação de duplexes de oligodesoxirribonucleotídeo compreendendo nucleosídos bicíclicos para utilização como substratos para as polimerases de ácido nucleico também foi descrita (Wengel et al., WO 99/14226). Além disso, a síntese de 2'-amino-BNA, um novo análogo de oligonucleotídeo de alta afinidade conformacionalmente restrito, foi descrita na técnica (Singh et al.,J. Org. Chem., 1998, 63, 10035-10039). Além disso, 2'-amino - e 2'-metilamino-BNA's foram preparados e a estabilidade térmica dos seus duplexes com fitas de RNA e DNA complementares foi relatada anteriormente.
[00802] Em certas modalidades, nucleosídos bicíclicos têm a fórmula:
[00803] em que:
[00804] Bx é uma fração de base heterocíclica;
[00805] Ta e Tb são, cada um, independentemente, H, um grupo deproteção de hidroxila, um grupo conjugado, um grupo de fósforo reativo, uma fração de fósforo ou uma ligação covalente a um meio de suporte;
[00806] cada qi, qj, qk e ql é, independentemente, Hidrogênio, halogêneo, alquil C1-C12, alquil substituído C1-C12, alquenil C2-C12, alquenil substituído C2-C12, alquinil C2-C12, alquinil substituído C2-C12, alcoxi C1-C12, alcoxi substituído C1-C12, OJj, SJj, SOJj, SO2Jj, NJjJk, N3, CN, C(=O)OJj, C(=O)NJjJk, C(=O)Jj, O-C(=O)NJjJk, N(H)C(=NH)NJjJk, N(H)C(=O)NJjJkou N(H)C(=S)NJjJk; e
[00807] qi e qj ou ql e qk em conjunto são = C(qg)(qh), em que qg e qh são cada um, independentemente, H, halogêneo, alquil C1-C12 ou alquil substituído C1-C12 .
[00808] Um nucleosídeo carbocíclico bicíclico tendo uma ponte 4'- (CH2)3-2' e o alquenil analógico da ponte 4'-CH = CH-CH2-2' foram descritos (Frier et al., Nucleic Acids Research, 1997, 25(22), 4429-4443 and Albaek et al., J. Org. Chem., 2006, 71, 7731-7740). A síntese e a preparação dos nucleosídeos bicíclicos carbocíclicos juntamente com seus estudos bioquímicos e de oligomerização também foram descritos (Srivastava et al., J. Am. Chem. Soc., 2007, 129(26), 8362-8379).
[00809] Em certas modalidades, nucleosídeos bicíclicos incluem, mas não estão limitados a, (a) a-L-metileno-oxi (4'-CH2-O-2‘) BNA, (B)D-β-metileno-oxi (4'-CH2-O-2') BNA, (C) etileno-oxi (4' - (CH)2-O-2') BNA, (D) amino-oxi (4'-CH2-ON (R) -2') BNA, (E) oxiamino (4'-CH2-N (R) -O-2 ') BNA, (F) metil (metileno-oxi) (4'-CH (CH3) -O-2') BNA (também referido como etil restrito, ou cEt), (G) metileno-tio (4'-CH2-S-2') BNA, (H) metileno-amino (4'-CH2-N (R) -2 ') BNA, (I) carbocíclico metil (4'-CH2- CH (CH3) -2') BNA, (J) carbocíclico propileno (4' - (CH2)3-2') BNA e (K) vinil BNA como representado abaixo.
[00810] em que Bx é a porção base e R é, independentemente, H, um grupo protetor, alquil C1-C6 ou alcoxi C1-C6.
[00811] Conforme usado neste documento, o termo "nucleosídeo de tetraidropirano modificado" ou "nucleosídeo de THP modificado" significa um nucleosídeo tendo um "açúcar" de tetraidropirano de seis membros substituído pelo resíduo de pentofuranosil nos nucleosídeos normais e pode ser referido como um substituto do açúcar. Os nucleosídeos de THP modificados incluem, mas não estão limitados a, o que é referido na técnica como ácido nucleico de hexitol (HNA), ácido nucleico de anitol (ANA), ácido nucleico de manitol (MNA) (vide Leumann, Bioorg. Med. Chem., 2002, 10, 841-854) ou HNA flúor (F- HNA), que tem um sistema em anel tetra-hidropiranil, tal como ilustrado abaixo.
[00812] Em certos modo de realização, substitutos de açúcar são seleccionados possuindo a fórmula:
[00813] em que:
[00814] Bx é uma fração de base heterocíclica;
[00815] T3 e T4são cada um, independentemente, um grupo de ligação de internucleosídeo ligando o análogo de nucleosídeo de tetrahidropiano a um composto oligomérico ou um dentre T3 e T4 é um grupo de ligação de internucleosídeo ligando o análogo de nucleosídeo de tetrahidropirano a um composto oligomérico e o outro dentre T3 e T4 é H, um grupo protetor de hidroxil, um grupo conjugado ligado, ou um grupo 5' ou 3'-terminal;
[00816] q1, q2, q3, q4, q5, q6 e q7 são cada um, independentemente, H, alquil C1-C6, alquil C1-C6 substituído, alquenil C2-C6, alquenil C2-C6 substituído, aquinil C2-C6substituído ou alquinil C2-C6; e
[00817] um de R1 e R2 é hidrogênio e o outro é seleccionado a partir de halogênio, alcoxi substituído ou não substituído, NJ1J2, SJ1, N3, OC(=X)J1, OC(=X)NJ1J2, NJ3C(=X)NJ1J2 e CN, em que X é O, S ou NJ1 e cada símbolo J1, J2 e J3 é, independentemente, H ou alquil C1-C6.
[00818] Em certas modalidades, q1, q2, q3, q4, q5, q6e q7 são cada um H. Em certas modalidades, pelo menos um de q1, q2, q3, q4, q5, q6e q7 é outro que não H. Em certas modalidades, pelo menos um de q1, q2, q3, q4, q5, q6e q7 é metil. Em certas modalidades, nucleosídos de THP são proporcionados em que um de R1 e R2 é F. Em certas modalidades, R1 é flúor e R2 é H; R1 representa um grupo metoxi e R2 é H, e R1 é metoxietoxi e R2 é H.
[00819] Em determinadas modalidades, os substitutos de açúcar compreendem anéis com mais de 5 átomos e mais de um heteroátomo. Por exemplo, os nucleosídos que compreendem porções de açúcar morfolino e a sua utilização em compostos oligoméricos foram relatados (ver por exemplo: Braasch et al., Biochemistry, 2002, 41, 4503-4510; e as Patentes US 5,698,685; 5.166.315; 5.185.444; e 5.034.506). Como usado neste documento, o termo "morfolino" se refere a um substituto de açúcar com a seguinte fórmula:
[00820] Em determinadas modalidades, os morfolinos podem ser modificados, por exemplo, pelo acréscimo ou alteração de vários grupos substituintes da estrutura do morfolino acima. Esses substitutos do açúcar são mencionados neste documento como "morfolinos modificados"
[00821] Combinações de modificações também são fornecidas sem limitação, tais como nucleosídeos de metil substituído 2'-F-5' (ver Pedido PCT Internacional WO 2008/101157 publicado em 8/21/08 para outros descritos nucleosídeos 5', 2'-bis substituído) e substituição do anel de ribosil átomo de oxigênio com S e outra substituição na posição 2' (ver Pedido de Patente Publicado US US2005-0130923, publicado em 16 de Junho, 2005) ou, alternativamente, 5'-substituição de um ácido nucleico bicíclico (ver Pedido PCT Internacional WO 2007/134181, publicado em 11/22/07, em que uma 4'-CH2-O-2 'nucleosídeo bicíclico é ainda substituído na posição 5' com um 5'-metil ou um grupo 5'-vinil). A síntese e preparação de nucleosídeos bicíclicos carbocíclicos juntamente com seus estudos bioquímicos e de oligomerização também foram descritos (ver, por exemplo, Srivastava et al., J. Am. Chem. Soc. 2007, 129(26), 8362-8379).
[00822] Em certas modalidades, os compostos antisenses compreendem um ou mais nucleosídeos ciclohexenil modificados, que é um nucleosídeo possuindo um ciclohexenil seis membros no lugar do resíduo pentofuranosil em nucleosídeos que ocorrem naturalmente. Nucleosídeos ciclohexenil modificados incluem, mas não estão limitados aos descritos na técnica (ver, por exemplo, de propriedade comum, pedido PCT publicado WO 2010/036696, publicado em de Abril, 10, 2010, Robeyns et al., J. Am. Chem. Soc., 2008, 130(6), 1979-1984; Horváth et al., Tetrahedron Letters, 2007, 48, 3621-3623; Nauwelaerts et al., J. Am. Chem. Soc., 2007, 129(30), 9340-9348; Gu et al.,,Nucleosides, Nucleotides & Nucleic Acids, 2005, 24(5-7), 993-998;Nauwelaerts et al., Nucleic Acids Research, 2005, 33 (8), 2452-2463; Robeyns et al., Acta Crystallographica, Section F: Structural Biology and Crystallization Communications, 2005, F61(6), 585-586; Gu et al.,Tetrahedron, 2004, 60(9), 2111-2123; Gu et al., Oligonucleotides, 2003, 13(6), 479-489; Wang et al., J. Org. Chem., 2003, 68, 4499-4505; Verbeure et al., Nucleic Acids Research, 2001, 29(24), 4941-4947; Wang et al., J. Org. Chem., 2001, 66, 8478-82; Wang et al., Nucleosides, Nucleotides & Nucleic Acids, 2001, 20(4-7), 785-788; Wang et al., J. Am. Chem., 2000, 122, 8595-8602; Pedido PCT publicado, documento WO 06/047842; e Pedido PCT publicado WO 01/049687; o texto de cada um é aqui incorporado por referência, na sua totalidade). Nucleosídeos de ciclohexenil modificados têm a Fórmula X.

[00823] em que, independentemente, para cada um dentre dito pelo menos um análogo de nucleosídeo de ciclohexenil da Fórmula X:
[00824] Bx é uma fração de base heterocíclica;
[00825] T3 e T4 são cada um, independentemente, um grupo de ligação de internucleosídeo ligando o análogo de nucleosídeo de ciclohexenil a um composto antisense ou um dentre T3 e T4 é um grupo de ligação de internucleosídeo ligando o análogo de nucleosídeo de tetrahidropirano a um composto antisense e o outro deentre T3 e T4 é H, um grupo protector de hidroxil, um grupo conjugado ligado, ou um grupo 5'- ou 3'-terminal; e
[00826] q1, q2, q3, q4, q5, q6, q7, q8 e q9 são cada um,independentemente, H, alquil C1-C6, alquil substituído C1-C6, alquenil C2-C6, alquenil substituído C2-C6, alquinil C2-C6, alquinil substituído C2C6 ou outro grupo substituinte de açúcar.
[00827] Muitos outros sistemas monocíclicos, bicíclicos e tricíclicos de anel são conhecidos na técnica e são adequados como substitutos de açúcar que podem ser utilizados para modificar nucleosídos para incorporação em compostos oligoméricos como aqui proporcionados (ver por exemplo o artigo de revisão: Leumann, Christian J. Bioorg. & Med. Chem., 2002, 10, 841-854). Tais sistemas de anel podem sesubmeter a várias substituições adicionais para intensificar ainda mais a sua atividade.
[00828] Conforme usado neste documento, "açúcar modificado 2'" significa um açúcar de furanosila modificado na posição 2'. Em certas modalidades, essas modificações incluem substituintes selecionados a partir de: um haleto, incluindo, mas não se limitando a alcóxi substituído e e não substituído, tioalquila substituída e não substituída, amino alquila substituída e não substituída, alquila substituída e não substituída, alila substituída e não substituída, e alquinila substituída e não substituída. Em certas modalidades, modificações 2' são seleccionadas a partir de substituintes incluindo, mas não se limitando a: O[(CH2)nO]mCH3, O(CH2)nNH2, O(CH2)nCH3, O(CH2)nF,O(CH2)nONH2, OCH2C(=O)N(H)CH3, e O(CH2)nON[(CH2)nCH3]2, em que n e m estão entre 1 e cerca de 10. Outros 2' grupos substituintes podem ser selecionados de: alquil C1-C12, alquil substituído, alquenil, alquinil, alcaril, aralquil, O-alcaril ou O-aralquil, SH, SCH3, OCN, Cl, Br, CN, F, CF3, OCF3, SOCH3, SO2CH3, ONO2, NO2, N3, NH2, heterocicloalquilo, heterocicloalcaril, aminoalquilamino, polialquilamino, sililo substituído, um grupo de clivagem do RNA, um grupo repórter, um intercalador, um grupo para melhorar as propriedades farmacocinéticas, ou um grupo para melhorar as propriedades farmacocinéticas de um composto antisense, e outros substituintes tendo propriedades semelhantes. Em certas modalidades, os nucleosídeos modificados compreendem uma cadeia lateral 2'-MOE (Baker et al., J. Biol. Chem., 1997, 272, 1194412000). Essa substituição de 2'-MOE foi descrita como tendo melhor afinidade de ligação em comparação com os nucleosídeos não modificados e com outros nucleosídeos modificados, tais como 2'-O- metil, O-propil, eO-aminopropil. Os oligonucleotídeos tendo o substituinte 2'-MOE também foram mostrados como sendo inibidores antisense da expressão do gene com características promissoras para uso in vivo use (Martin, Helv. Chim. Acta, 1995, 78, 486-504; Altmann et al., Chimia, 1996, 50, 168-176; Altmann et al., Biochem. Soc. Trans., 1996, 24, 630-637; e Altmann et al., Nucleosides Nucleotides, 1997, 16, 917-926).
[00829] Conforme usado neste documento, "modificado em 2'" ou "substituído em 2'" se refere a um nucleosídeo compreendendo um açúcar compreendendo um substituinte na posição 2' diferente de H ou OH. nucleosídeos 2'-modificados, incluem, mas não estão limitados aos nucleosídeos bicíclicos em que a ponte de ligação entre dois átomos de carbono do anel de açúcar conecta o carbono 2' e do outro carbono do anel de açúcar; e nucleosídeos com 2' substituíntes sem ponte, como aliI, amino, azido, tio, O-alil, alquil O-C1-C10 , -OCF3, O- (CH2)2-O-CH3, 2'-O (CH2)2SCH3, O- (CH2)2-ON (Rm) (Rn), Ou O-CH2-C (= O) -N (Rm) (Rn), em que cada Rm e Rn é, independentemente, H ou alquil substituído ou não substituído C1-C10 . Os nucleosídeos modificados em 2' podem compreender ainda outras modificações, por exemplo, em outras posições do açúcar e/ou na nucleobase.
[00830] Conforme usado neste documento, "2'-F" se refere a um nucleosídeo compreendendo um açúcar compreendendo um grupo flúor na posição 2' do anel de açúcar.
[00831] Conforme usado neste documento, "2'-OMe" ou "2'-OCH3, "2'-O-metil" ou "2'-O-metoxi" cada um se refere a um nucleosídeo compreendendo um açúcar compreendendo um grupo -OCH3 na posição 2' do anel de açúcar.
[00832] Como usado neste documento, "MOE" ou "2'- OCH2CH2OCH3" ou " 2'-O-metoxietil", cada um, refere-se a um nucleosídeo compreendendo um açúcar compreendendo um grupo - OCH2CH2OCH3 na posição 2' do anel de açúcar.
[00833] Os métodos para as preparações de açúcares modificados são bem conhecidos por aqueles versados na técnica. Algumas patentes representativas dos Estados Unidos que ensinam a preparação de tais açúcares modificados incluem, sem limitação, U.S.: 4.981.957; 5.118.800; 5.319.080; 5.359.044; 5.393.878; 5.446.137;5.466.786; 5.514.785; 5.519.134; 5.567.811; 5.576.427; 5.591.722;5.597.909; 5.610.300; 5.627.053; 5.639.873; 5.646.265; 5.670.633;5.700.920; 5.792.847 e 6.600.032 e Pedido InternacionalPCT/US2005/019219, depositado em 2 de junho de 2005 e publicado como WO 2005/121371 em 22 de dezembro de 2005, e cada uma das quais é neste documento incorporada por referência na sua totalidade.
[00834] Como usado neste documento, "oligonucleotídeo" se refere a um composto compreendendo uma pluralidade de nucleosídeos ligados. Em certas modalidades, um ou mais da pluralidade de nucleosídeos são modificados. Em certas modalidades, um oligonucleotídeo compreende um ou mais ribonucleosídeos (RNA) e/ou desoxirribonucleosídeos (DNA).
[00835] Nos nucleotídeos tendo frações de açúcar modificado, as frações de nucleobase (natural, modificada ou uma combinação destas) são mantidas para a hibridização com um alvo de ácido nucleico apropriado.
[00836] Em certas modalidades, os compostos antisense compreendem um ou mais nucleosídeos tendo frações de açúcar modificado. Em certas modalidades, a fração de açúcar modificado é 2'- MOE. Em certas modalidades, os nucleosídeos modificados em 2'-MOE são dispostos em um motivo de gapmer. Em certas modalidades, a fração de açúcar modificado é um nucleosídeo bicíclico tendo um grupo de ponte (4'-CH(CH3)-O-2'). Em certas modalidades, os nucleosídeos modificados de (4'-CH(CH3)-O-2') são dispostos ao longo das asas de um motivo de gapmer.
Nucleobases Modificadas
[00837] As modificações ou substituições de nucleobase (ou base) são estruturalmente distinguíveis, e funcionalmente permutáveis com, nucleobases não modificadas sintéticas ou de ocorrência natural. As nucleobases naturais e modificadas são capazes de participar da ligação de hidrogênio. Essas modificações de nucleobase podem conferir estabilidade à nuclease, afinidade de ligação ou alguma outra propriedade biológica benéfica para os compostos antisense. As nucleobases modificadas incluem nucleobases sintéticas e naturais, tal como, por exemplo, 5-metilcitosina (5-me-C). Determinadas substituições de nucleobase, incluindo substituições de 5-metilcitosina, são particularmente úteis para aumentar a afinidade de ligação de um composto antisense por um ácido nucleico alvo. Por exemplo, as substituições de 5-metilcitosina demonstraram aumentar a estabilidade do duplex de ácido nucleico por 0,6-1,2°C (Sanghvi, YS, Crooke, S.T. and Lebleu, B., eds., Antisense Research and Applications, CRC Press, Boca Raton, 1993, pp. 276-278).
[00838] As nucleobases modificadas adicionais incluem 5- hidroximetil citosina, xantina, hipoxantina, 2-aminoadenina, 6-metila e outros derivados de alquila de adenina e guanina, 2-propila e outros derivados de alquila de adenina e guanina, 2-tiouracila, 2-tiotimina e 2- tiocitosina, 5-halouracila e citosina, 5-propinil (-C=C-CH3) uracila e citosina e outros derivados de alquinila de bases de pirimidina, 6-azo uracila, citosina e timina, 5-uracil(pseudouracila), 4-tiouracila, 8-halo, 8- amino, 8-tiol, 8-tioalquila, 8-hidroxila e outras adeninas e guaninas 8- substituídas, 5-halo, particularmente, 5-bromo, 5-trifluorometila e outras uracilas e citosinas 5-substituídas, 7-metilguanina e 7-metiladenina, 2- F-adenina, 2-amino-adenina, 8-azaguanina e 8-aza-adenina, 7- deazaguanina e 7-deaza-adenina e 3-deazaguanina e 3-deaza- adenina.
[00839] As frações de base heterocíclicas também podem incluir aquelas em que a base purina ou pirimidina é substituída por outros heterocíclicos, por exemplo, 7-deaza-adenina, 7-deazaguanosina, 2- aminopiridina e 2-piridona. As nucleobases que são particularmente úteis para aumentar a afinidade de ligação dos compostos antisense incluem pirimidinas 5-substituídas, 6-azapirimidinas e purinas substituídas N-2, N-6 e 6-O, incluindo 2 aminopropiladenina, 5-propiniluracila e 5-propinilcitosina.
[00840] Em certas modalidades, os compostos antisense direcionados para um ácido nucleico de ANGPTL3 compreendem uma ou mais nucleobases modificadas. Em certas modalidades, os oligonucleotídeos antisense de lacuna ampliada ou encurtada ou direcionados para um ácido nucleico de ANGPTL3 compreendem uma ou mais nucleobases modificadas. Em certas modalidades, a nucleobase modificada é uma 5-metilcitosina. Em certas modalidades, cada citosina é uma 5-metilcitosina.Composições e Métodos para a Formulação de Composições Farmacêuticas
[00841] Os oligonucleotídeos antisense podem ser misturados com uma substância farmaceuticamente aceitável ativa ou inerte para a preparação de composições ou formulações farmacêuticas. As composições e métodos para a formulação de composições farmacêuticas são dependentes de uma série de critérios, incluindo, mas não limitado a, via de administração, extensão da doença, ou dose a ser administrada.
[00842] O composto antisense direcionado para um ácido nucleico de ANGPTL3 pode ser utilizado em composições farmacêuticas, combinando o composto antisense com um diluente ou carreador farmaceuticamente aceitável adequado. Um diluentefarmaceuticamente aceitável inclui solução salina tamponada com fosfato (PBS). O PBS é um diluente adequado para uso em composições a serem distribuídas por via parenteral. Nesse sentido, em uma modalidade, empregada nos métodos descritos neste documento, está uma composição farmacêutica que compreende um composto antisense direcionado para um ácido nucleico de DANGPTL3 e um diluente farmaceuticamente aceitável. Em certas modalidades, o diluente farmaceuticamente aceitável é o PBS. Em certas modalidades, o composto antisense é um oligonucleotídeo antisense.
[00843] As composições farmacêuticas que compreendem compostos antisense englobam quaisquer sais farmaceuticamente aceitáveis, ésteres e sais desses ésteres, ou qualquer outro oligonucleotídeo que, após a administração a um animal, incluindo um humano, seja capaz de fornecer (direta ou indiretamente) o metabólito biologicamente ativo ou resíduo deste. Assim, por exemplo, a divulgação é concebida também para sais farmaceuticamente aceitáveis dos compostos antisense, pró-drogas, sais farmaceuticamente aceitáveis dessas pró-drogas e outros bioequivalentes. Os sais farmaceuticamente aceitáveis adequados incluem, entre outros, sais de sódio e de potássio.
[00844] Uma pró-droga pode incluir a incorporação de nucleosídeos adicionais em uma ou em ambas as extremidades de um composto antisense que são clivados por endonucleases dentro do corpo, para formar o composto antisense ativo.
Compostos Antisense Conjugados
[00845] Os compostos antisense podem ser covalentemente ligados a uma ou mais frações ou conjugados que intensificam a atividade, a distribuição celular ou a absorção celular dos oligonucleotídeos antisense resultantes. Os grupos conjugados típicos incluem frações de colesterol e frações de lipídio. Os grupos conjugados adicionais incluem carboidratos, fosfolipídios, biotina, fenazina, folato, fenantridina, antraquinona, acridina, fluoresceínas, rodaminas, cumarinas e corantes.
[00846] Os compostos antisense também podem ser modificados para ter um ou mais grupos de estabilização que estão geralmente ligados a um ou a ambos os terminais dos compostos antisense para intensificar as propriedades, tal como, por exemplo, a estabilidade à nuclease. Incluídas nos grupos de estabilização estão as estruturas de revestimento. Estas modificações terminais protegem o composto antisense tendo um ácido nucleico terminal a partir da degradação pela exonuclease, e podem ajudar na distribuição e/ou localização dentro de uma célula. O revestimento pode estar presente no 5'- terminal (5'-revestimento), ou no 3'-terminal (3'-revestimento), ou pode estar presente em ambos os terminais. As estruturas de revestimento são bem conhecidas na técnica e incluem, por exemplo, revestimentos abásicos de desoxi invertido. Além disso, os grupos de estabilização 3' e 5' que podem ser usados para revestir uma ou ambas as extremidades de um composto antisense para transmitir estabilidade de nuclease, incluem aqueles divulgados em WO 03/004602 publicado em 16 de janeiro de 2003.
[00847] Em determinadas modalidades, a presente divulgação fornece compostos antisense conjugados representados pela fórmula:
[00848] em que
[00849] A é o oligonucleotídeo antisense;
[00850] B é a porção clivável
[00851] C é o ligante conjugado
[00852] D é o grupo de ramificação
[00853] cada E é uma corrente;
[00854] cada M é um ligante; e
[00855] q é um número inteiro entre 1 e 5.
[00856] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[00857] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[00858] Em determinadas modalidades, compostos antisenseconjugados são fornecidos com a estrutura:
[00859] A presente divulgação apresenta as seguintes modalidadesnão-limitantes numeradas:
[00860] em que:
[00861] T2 é um nucleosídeo, um nucleotídeo, uma subunidademonomérica, ou um composto oligomérico.
[00862] Em modalidades que têm mais de uma variável específica (por exemplo, mais de um "m" ou "n"), salvo indicação em contrário, cada uma dessa variável específica é selecionada independentemente. Assim, para uma estrutura que tem mais de um n, cada n é selecionado de forma independente assim, eles podem ser iguais ou diferentes uns dos outros.i. Determinadas Frações Cliváveis
[00863] Em determinadas modalidades, uma fração clivável é uma ligação clivável. Em determinadas modalidades, uma fração clivável compreende uma ligação clivável. Em determinadas modalidades, o grupo conjugado compreende uma fração clivável. Nestas determinadas modalidades, a fração clivável é afixada ao oligonucleotídeo antisense. Nestas determinadas modalidades, a fração clivável é afixada diretamente a fração de direcionamento de célula. Nestas determinadas modalidades, a fração clivável é afixada ao ligante conjugado. Em determinadas modalidades, a fração compreende um fosfato ou fosfodiéster. Em determinadas modalidades, a fração clivável é um nucleosídeo clivável ou nucleosídeo análogo. Em determinadas modalidades, o nucleosídeo ou nucleosídeo análogo compreende uma base heterocíclica opcionalmente protegida, selecionada a partir de uma purina, purina substituída, pirimidina ou pirimidina substituída. Em determinadas modalidades, a fração clivável é um nucleosídeo que compreende uma base heterocíclica opcionalmente protegida selecionada de uracil, timina, citosina, 4-N- benzoilcitosina, 5-metilcitosina, 4-N-benzoil-5-metilcitosina, adenina, 6-N-benzoiladenina, guanina e 2-N-isobutirilguanina. Em determinadas modalidades, a fração clivável é 2'-deoxinucleosídeo que é afixado à posição 3' do oligonucleotídeo antisense por uma ligação fosfodiéster e é afixado ao ligante por uma ligação de fosfodiéster ou fosforotioato. Em determinadas modalidades, a fração clivável é 2'-deoxi adenosina que é afixada à posição 3' do oligonucleotídeo antisense por uma ligação de fosfodiéster e é afixada ao ligante por uma ligação fosfodiéster ou fosforotioato. Em determinadas modalidades, a fração clivável é 2'-deoxi adenosina que é afixado à posição 3' do oligonucleotídeo antisense por uma ligação de fosfodiéster e é afixado ao ligante por uma ligação de fosfodiéster.
[00864] Em determinadas modalidades, a fração clivável é afixada à posição 3' do oligonucleotídeo antisense. Nestas determinadas modalidades, a fração clivável é afixada à posição 5' do oligonucleotídeo antisense. Em determinadas modalidades, a fração clivável é afixada à posição 2' do oligonucleotídeo antisense. Em determinadas modalidades, a fração clivável é anexado ao oligonucleotídeo antisense por uma ligação fosfodiéster. Em determinadas modalidades, a fração clivável é afixada ao ligante por um ligação fosforotioato ou um fosfodiéster. Em determinadas modalidades, a fração clivável é afixado ao ligante por uma ligação de fosfodiéster. Em determinadas modalidades, o grupo conjugado não inclui uma fração clivável.
[00865] Em determinadas modalidades, a fração clivável é clivada depois que o complexo foi administrado a um animal somente após ser interiorizado por uma célula alvo. Dentro da célula a fração clivável é clivada liberando, desta forma, o oligonucleotídeo antisense ativo. Embora não seja destinado a ser limitado pela teoria, acredita- se que a fração clivável é clivada por uma ou mais nucleases dentro da célula. Em determinadas modalidades, a uma ou mais nucleases clivam a ligação de fosfodiéster entre a fração clivável e o ligante. Em determinadas modalidades, a fração clivável tem uma estrutura selecionada dentre os seguintes:
[00866] em que cada um dos Bx, Bx1, Bx2, e Bx3 é independentemente uma fração de heterocíclica. Em determinadas modalidades, a fração clivável tem uma estrutura selecionada dentre os seguintes:
[00867] Em determinadas modalidades, os grupos de conjugado compreendem um ligante. Em algumas dessas modalidades, o ligante está covalentemente ligado à fração clivável. Em algumas dessas modalidades, o ligante está covalentemente ligado ao oligonucleotídeo antisense. Em determinadas modalidades, o ligante está covalente- mente ligado a uma fração da célula-alvo. Em determinadas modalidades, o ligante compreende ainda uma ligação covalente com um suporte sólido. Em determinadas modalidades, o ligante compreende ainda uma ligação covalente com uma fração de ligação de proteínas. Em determinadas modalidades, o ligante compreende ainda uma ligação covalente com um suporte sólido e compreende ainda uma fração de ligação de proteínas. Em determinadas modalidades, o ligante inclui várias posições para anexação dos ligantes fixados. Em determinadas modalidades, o ligante inclui várias posições para anexação dos ligantes fixados e não está anexado a um grupo de ramificação. Em determinadas modalidades, o ligante compreende ainda uma ou mais ligação clivável. Em determinadas modalidades, o grupo conjugado não inclui um ligante.
[00868] Em determinadas modalidades, o ligante inclui pelo menos um grupo linear compreendendo grupos selecionados de alquil, amida, bissulfeto, polietilenoglicol, éter, tioéter (-S-) e hidroxilamina (grupos-O- N(H)-). Em determinadas modalidades, o grupo linear compreende grupos selecionados a partir de grupos alquil, amida e éter. Em determinadas modalidades, o grupo linear compreende grupos selecionados a partir de grupos alquil e éter. Em determinadas modalidades, o grupo linear compreende pelo menos um grupo de ligação fósforo. Em determinadas modalidades, o grupo linear compreende pelo menos um grupo fosfodiéster. Em determinadas modalidades, o grupo linear inclui pelo menos um grupo ligante neutro. Em determinadas modalidades, o grupo linear está covalentemente anexado à fração de célula-alvo e à fração clivável. Em determinadas modalidades, o grupo linear está covalentemente anexado à fração de célula-alvo e ao oligonucleotídeo antisense. Em determinadas modalidades, o grupo linear está covalentemente anexado à fração de célula-alvo, à fração clivável e a um suporte sólido. Em determinadas modalidades, o grupo linear está covalentemente anexado à fração de célula-alvo, à fração clivável, a um suporte sólido a uma fração ligante de proteína. Em determinadas modalidades, o grupo linear inclui uma ou mais ligações cliváveis.
[00869] Em determinadas modalidades, o ligante inclui o grupo linear covalentemente anexado a um grupo andaime (scaffold). Em determina-das modalidades, o andaime inclui um grupo alifático ramificado compre-endendo grupos selecionados de alquil amida, bissulfeto, polietilenoglicol, éter, grupos tioéter e hidroxilamina. Em determinadas modalidades, o andaime inclui um grupo alifático ramificado compreendendo grupos selecionados a partir de grupos alquil, amida e éter. Em determinadas modalidades, o andaime inclui pelo menos um sistema de anéis mono ou policíclico. Em determinadas modalidades, o andaime inclui pelo menos dois sistemas de anel mono ou policíclico. Em determinadas modalidades, o grupo linear é covalentemente anexado ao grupo andaime e o grupo andaime é covalentemente anexado à fração clivável e ao ligante. Em determinadas modalidades, o grupo linear está covalentemente anexado ao grupo andaime e o grupo andaime está covalentemente anexado à fração clivável, ao ligante e ao suporte sólido. Em determinadas modalidades, o grupo linear está covalentemente anexado ao grupo andaime e o grupo andaime está covalentemente anexado à fração e a um grupo ligante de proteína. Em determinadas modalidades, o grupo linear está covalentemente ligado ao grupo andaime e o grupo andaime está covalentemente anexado à fração clivável, ao ligante, à fração ligante de proteína e a um suporte sólido. Em determinadas modalidades, o grupo linear inclui uma ou mais ligações cliváveis.
[00870] Em determinadas modalidades, o ligante inclui uma fração ligante de proteínas. Em determinadas modalidades, a fração ligante de proteínas é um lipídio como, por exemplo, incluindo, entre outros, colesterol, ácido cólico, ácido acético de adamantano, ácido 1-pireno butírico, dihidrotestosterona, 1,3-Bis-O(hexadecil)glicerol, grupo geranilo- xihexil, hexadecilglicerol, borneol, mentol, 1,3-propanodiol, grupo heptadecil, ácido palmítico, ácido mirístico, ácido O3-(oleoil)litocólico , ácido O3- (oleoil) colênico, dimetoxitritil ou fenoxazina), uma vitamina (por exemplo, folato, vitamina A, vitamina E, biotina, piridoxal), um peptídeo, um carboidrato (por exemplo, monossacarídeo, dissacarídeo, trissacarídeo, tetrassacarídeo, oligossacarídeo, polissacarídeo), um componente endos- somolítico, um esteroide (por exemplo, uvaol, hecigenina, diosgenina), um terpeno (por exemplo, triterpeno, por exemplo, sarsasapogenina, friedelina, ácido litocólico derivatizado de epifriedelanol) ou um lipídio catiônico. Em determinadas modalidades, a fração ligante de proteínas é um ácido graxo saturado ou insaturado de cadeia longa de C16 a C22, colesterol, ácido cólico, vitamina E, adamantano ou 1-pentafluoropropil.
[00871] Em determinadas modalidades, um ligante tem uma conjugados são fornecidos com a estrutura:
[00872] onde cada n é, independentemente, de 1 a 20; e p é de 1a 6.
[00873] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00874] onde n é, independentemente, de 1 a 20.
[00875] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00876] onde n é de 1 a 20.
[00877] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00878] onde cada L é, de forma independente, um grupo ligante fósforo ou um grupo ligante neutro; e
[00879] onde n é, independentemente, de 1 a 20.
[00880] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00881] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00882] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00883] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00884] onde n é de 1 a 20.
[00885] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00886] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00887] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00888] Em determinadas modalidades, o ligante conjugado tem a estrutura:
[00889] Em determinadas modalidades, o ligante conjugado tem a estrutura:
[00890] Em determinadas modalidades, um ligante tem umaestrutura selecionada entre:
[00891] Em determinadas modalidades, um ligante tem uma estrutura selecionada entre:
[00892] onde cada n é independente, 0, 1, 2, 3, 4, 5, 6 ou 7.ii. Determinadas Frações de Célula-Alvo
[00893] Em determinadas modalidades, os grupos conjugados compreendem frações de célula-alvo. Determinadas frações de célula- alvo aumentam a absorção celular de compostos antisense. Em determinadas modalidades, as frações de célula-alvo compreendem um grupo de ramificação, um ou mais fixador e um ou mais ligante. Em determinadas modalidades, frações de célula-alvo compreendem um grupo de ramificação, um ou mais ligantes e uma ou mais ligação clivável. 1. Determinados Grupos de Ramificação
[00894] Em determinadas modalidades, os grupos conjugados compreendem uma fração-alvo compreendendo um grupo de ramificação e pelo menos dois ligantes fixados. Em determinadas modalidades, o grupo de ramificação anexa o ligante conjugado. Em determinadas modalidades, o grupo de ramificação anexa a fração clivável. Em determinadas modalidades, o grupo de ramificação anexa o oligonucleotídeo antisense. Em determinadas modalidades, o grupo de ramificação está covalentemente anexado ao vinculador e aos ligantes fixados. Em determinadas modalidades, o grupo de ramificação compreende um grupo alifático ramificado compreendendo grupos selecionados de alquil amida, bissulfeto, polietilenoglicol, éter, grupos tioéter e hidroxilamina. Em determinadas modalidades, o grupo de ramificação compreende grupos selecionados a partir de grupos alquil, amida e éter. Em determinadas modalidades, o grupo de ramificação compreende grupos selecionados a partir de grupos alquil e éter. Em determinadas modalidades, o grupo de ramificação compreende um sistema de anéis mono ou policíclicos. Em determinadas modalidades, o grupo de ramificação compreende uma ou mais ligação clivável. Em determinadas modalidades, o grupo conjugado não inclui um grupo de ramificação.
[00895] Em determinadas modalidades, um grupo de ramificação tem uma estrutura selecionada entre:
[00896] onde n é, independentemente, de 1 a 20.
[00897] j é de 1 a 3; e
[00898] m é de 2 a 6.
[00899] Em determinadas modalidades, um grupo de ramificação tem uma estrutura selecionada entre:
[00900] onde n é, independentemente, de 1 a 20; e
[00901] m é de 2 a 6.
[00902] Em determinadas modalidades, um grupo de ramificação tem uma estrutura selecionada entre:
[00903] Em determinadas modalidades, um grupo de ramificaçãotem uma estrutura selecionada entre:
[00904] onde A1 é independente, O, S, C=O ou NH; e
[00905] onde n é, independentemente, de 1 a 20.
[00906] Em determinadas modalidades, um grupo de ramificaçãotem uma estrutura selecionada entre:
[00907] onde A1 é, independentemente, O, S, C=O ou NH; e
[00908] onde n é, independentemente, de 1 a 20.
[00909] Em determinadas modalidades, um grupo tem uma estrutura selecionada entre:
[00910] onde A1 é O, S, C=O ou NH; e
[00911] onde n é, independentemente, de 1 a 20.
[00912] Em determinadas modalidades, um grupo de ramificaçãotem uma estrutura selecionada entre:
[00913] Em determinadas modalidades, um grupo de ramificaçãotem uma estrutura selecionada entre:
[00914] Em determinadas modalidades, um grupo de ramificaçãotem uma estrutura selecionada entre:
[00915] Em determinadas modalidades, os grupos conjugados compreendem um ou mais fixadores covalentemente anexados ao grupo de ramificação. Em determinadas modalidades, os grupos conjugados compreendem um ou mais fixadores covalentemente anexados ao grupo de ligação. Em determinadas modalidades, cada fixador é um grupo alifático linear, compreendendo um ou mais grupos selecionados de alquil, éter, tioéter, bissulfeto, amida e grupos de polietilenoglicol em qualquer combinação. Em determinadas modalidades, cada fixador é um grupo linear, compreendendo um ou mais grupos selecionados de alquil, éter, tioéter, bissulfeto, amida, fosfodiéster e grupos de polietilenoglicol em qualquer combinação. Em determinadas modalidades, cada fixador é um grupo linear, compreendendo um ou mais grupos selecionados de alquil, éter e amida em qualquer combinação. Em determinadas modalidades, cada fixador é um grupo alifático linear compreendendo um ou mais grupos selecionados de alquil, fosfodiéster, éter e grupos amida em qualquer combinação. Em determinadas modalidades, cada fixador é um grupo alifático linear compreendendo um ou mais grupos selecionados de alquil e fosfodiéster em qualquer combinação. Em determinadas modalidades, cada fixador compreende pelo menos um grupo de ligação fósforo ou grupo de ligação neutro.
[00916] Em determinadas modalidades, o fixador inclui uma ou mais ligações cliváveis. Em determinadas modalidades, a corrente é anexada ao grupo de ramificação através de ou uma Amida ou um grupo éter. Em determinadas modalidades, o fixador é anexado ao grupo ramificação por meio de um grupo fosfodiéster. Em determinadas modalidades, o fixador é anexado ao grupo de ramificação por meio de um grupo de ligação fósforo ou grupo de ligação neutro. Em determinadas modalidades, o fixador é anexado ao grupo ramificação por meio de um um grupo éter. Em determinadas modalidades, a corrente é anexada ao ligante por meio de um um grupo amida ou éter. Em determinadas modalidades, a corrente é anexada ao ligante por meio de um grupo éter. Em determinadas modalidades, a corrente é anexada ao ligante por meio de um um grupo amida ou éter. Em determinadas modalidades, a corrente é anexada ao ligante por meio de um grupo éter.
[00917] Em determinadas modalidades, cada fixador compreende de cerca de 8 a cerca de 20 átomos no comprimento da cadeia entre o ligante e o grupo de ramificação. Em determinadas modalidades, cada grupo fixador compreende de cerca de 10 a cerca de 18 átomos no comprimento da cadeia entre o ligante e o grupo de ramificação. Em determinadas modalidades, cada grupo fixador compreende de cerca de 13 átomos no comprimento da cadeia.
[00918] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00919] onde n é, independentemente, de 1 a 20; e
[00920] e p é de 1 a 6.
[00921] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00922] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00923] onde n é, independentemente, de 1 a 20.
[00924] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00925] onde L é um grupo de ligação fósforo ou um grupo de ligação neutro;
[00926] Z1 é C(=O)O-R2;
[00927] Z2 é H, C1-C6 alquil ou C substituído1-C6 alqui;
[00928] R2 é H, alquil C1-C6 or alquil C1-C6 substituído; e
[00929] m1 é, independentemente, de 0 a 20 onde pelo menos um m1 é maior que 0 para cada fixador.
[00930] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00931] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00932] onde Z2 é H ou CH3; e
[00933] m1 é, independentemente, de 0 a 20 onde pelo menos um m1 é maior que 0 para cada fixador.
[00934] Em determinadas modalidades, uma corrente tem uma estrutura selecionada entre:
[00935] onde n é, independentemente, 0, 1,2, 3, 4, 5, 6, ou 7.
[00936] Em determinadas modalidades, uma corrente compreende um grupo de ligação de fósforo. Em determinadas modalidades, uma corrente não compreende nenhuma ligação amida. Em determinadas modalidades, uma corrente compreende um grupo de ligação de fósforo e não compreende nenhuma ligação amida.3. Certos Ligantes
[00937] Em determinadas modalidades, a presente divulgação provê ligantes em que cada ligante é anexado covalentemente a uma corrente. Em determinadas modalidades, cada ligante é selecionado para ter uma afinidade para pelo menos um tipo de receptor em uma célula-alvo. Em determinadas modalidades, os ligantes são selecionados para que tenham afinidade para pelo menos um tipo de receptor na superfície de uma célula hepática de mamífero. Em determinadas modalidades, os ligantes são selecionados para que tenham afinidade para pelo menos um receptor hepático de asialoglicoproteína. Em determinadas modalidades, cada ligante é um carboidrato. Em determinadas modalidades, cada ligante é, selecionados independentemente da galactose, N-acetil galactosamina, manose, glicose, glicosamina e fucose. Em determinadas modalidades, cada ligante é N-acetil galactosamina (GalNAc). Em determinadas modalidades, a fração-alvo compreende 2 a 6 ligantes. Em determinadas modalidades, a fração- alvo compreende 3 ligantes. Em determinadas modalidades, a fração- alvo compreende 3 ligantes N-acetil galactosamina.
[00938] Em determinadas modalidades, o ligante é um carboidrato, derivado de carboidrato, carboidrato modificado, aglomerado de carboidrato multivalente, polissacarídeo, polissacarídeo modificado ou derivado de polissacarídeo. Em determinadas modalidades, o ligante é um açúcar aminado ou açúcar tio. Por exemplo, açúcares aminados podem ser selecionados de qualquer número de compostos conhecidos na técnica, por exemplo glicosamina, ácido siálico, a-D-galactosamina, N-acetilgalactosamina, 2-acetamido-2-deóxi-D-galactopiranose(GalNAc), 2-Amina-3- O -[(R )-1-carboxietil]-2-de0xi-β-D-glicopiranose (β- ácido murâmico), 2-Deóxi-2-metilamina-L-glicopiranose, 4,6-Dideóxi-4- formamida-2,3-di-O-metil-D-manopiranose, 2-Deóxi-2-sulfoamina-D-glicopiranose e N- sulfo-D-glicosamina e N- Glicoloil-a-ácido neuramínico. Por exemplo, açúcares tiolados podem ser selecionados do grupo que consiste em 5-Tio-β-D-glicopiranose, Metil 2, 3,4-tri-O- acetil-1-tio-6-O-tritil-a-D-glicopiranosida, 4-Tio-β-D-galactopiranose e etil 3,4,6,7-tetra-O-acetil-2-deóxi-1,5-ditio-a-D-glico- heptopiranosida.
[00939] Em determinadas modalidades, "GalNac" ou "Gal-NAc" se refere a 2-(Acetilamina)-2-deóxi-D-galactopiranose, comumente referido na literatura como N-acetil galactosamina . Em determinadas modalidades, "N-acetil galactosamina" se refere a 2-(Acetylamina)-2- deóxi-D-galactopiranose. Em determinadas modalidades, "GalNac" ou "Gal-NAc" se refere a 2-(Acetilamina)-2-deóxi-D-galactopiranose. Em determinadas modalidades, "GalNac" ou "Gal-NAc" se refere a 2- (Acetilamina)-2-deóxi-D-galactopiranose, que inclui a forma β : 2-(Acetilamina)-2-Deóxi^-D-galactopiranose e forma a: 2-(Acetilamina)- 2-Deóxi-D-galactopiranose. Em determinadas modalidades, a forma β : 2-(Acetilamina)-2-Deóxi^-D-galactopiranose e forma a: 2-(Acetilamina)-2-deóxi-D-galactopiranose pode ser usado de forma intercambiável. Portanto, nas estruturas em que uma forma é retratada, essas estruturas destinam-se a incluir a outra forma também. Por exemplo, se a estrutura para a forma a: 2-(Acetilamina)-2-Deóxi-D- galactopiranose é mostrado, esta estrutura destina-se a incluir a outra forma também. Em determinadas modalidades, em determinadas modalidades preferenciais, a forma β 2-(Acetylamina)-2-deóxi-D- galactopiranose é a modalidade preferencial.

[00940] 2-(Acetilamina)-2-deóxi-D-galactopiranose.
[00941] 2-(Acetilamina)-2-de0xi-β-D-galactopiranose.
[00942] 2-(Acetilamina)-2-deóxi-a-D-galactopiranose.
[00943] Em determinadas modalidades, um ou mais ligantes têm uma estrutura selecionada entre:
[00944] onde R1 é selecionado de OH e NHCOOH.
[00945] Em determinadas modalidades, um ou mais ligantes têm uma estrutura selecionada entre:
[00946] Em determinadas modalidades, um ou mais ligantes têm uma estrutura selecionada entre:
[00947] Em determinadas modalidades, um ou mais ligantes têm uma estrutura selecionada entre:
[00948] Em determinadas modalidades, os grupos conjugados compreendem as características estruturais acima. Em determinadas modalidades, os grupos conjugados compreendem a seguinte estrutura:
[00949] onde n é, independentemente, de 1 a 20.
[00950] Em determinadas modalidades, os grupos conjugados compreendem a seguinte estrutura:
[00951] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00952] onde n é, independentemente, de 1 a 20.
[00953] Z é H ou um suporte sólido ligado;
[00954] Q é um composto antisense;
[00955] X é O ou S; e
[00956] BX é uma fração de base heterocíclica.
[00957] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00958] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00959] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00960] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00961] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00962] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00963] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00964] Em determinadas modalidades, os grupos conjugados têm a seguinte estrutura:
[00965] Em determinadas modalidades, os conjugados não compreendem uma pirrolidina.b. Certos compostos antisense conjugados
[00966] Em determinadas modalidades, os conjugados estão ligados a um nucleosídeo do oligonucleotídeo antisense nas posições 2', 3', 5' do nucleosídeo. Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[00967] onde
[00968] A é o oligonucleotídeo antisense;
[00969] B é a fração clivável
[00970] C é o ligador conjugado
[00971] D é o grupo de ramificação
[00972] E é uma corrente;
[00973] F é um ligante; e
[00974] q é um número inteiro entre 1 e 5.
[00975] Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[00976] onde
[00977] A é o oligonucleotídeo antisense;
[00978] C é o ligador conjugado
[00979] D é o grupo de ramificação
[00980] E é uma corrente;
[00981] F é um ligante; e
[00982] q é um número inteiro entre 1 e 5.
[00983] Em determinadas modalidades, o ligador conjugadocompreende pelo menos uma ligação clivável.
[00984] Em determinadas modalidades, o grupo de ramificaçãocompreende pelo menos uma ligação clivável.
[00985] Em determinadas modalidades, cada corrente compreende pelo menos uma ligação clivável.
[00986] Em determinadas modalidades, os conjugados estão ligados a um nucleosídeo do oligonucleotídeo antisense nas posições 2', 3', 5' do nucleosídeo.
[00987] Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[00988] onde
[00989] A é o oligonucleotídeo antisense;
[00990] B é a fração clivável
[00991] C é o ligador conjugado
[00992] E é uma corrente;
[00993] F é um ligante; e
[00994] q é um número inteiro entre 1 e 5.
[00995] Em determinadas modalidades, os conjugados estão ligados a um nucleosídeo do oligonucleotídeo antisense nas posições 2', 3', 5' do nucleosídeo. Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[00996] onde
[00997] A é o oligonucleotídeo antisense;
[00998] C é o ligador conjugado
[00999] E é uma corrente;
[001000] F é um ligante; e
[001001] q é um número inteiro entre 1 e 5.
[001002] Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[001003] onde
[001004] A é o oligonucleotídeo antisense;
[001005] B é a fração clivável
[001006] D é o grupo de ramificação
[001007] E é uma corrente;
[001008] F é um ligante; e
[001009] q é um número inteiro entre 1 e 5.
[001010] Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[001011] onde
[001012] A é o oligonucleotídeo antisense;
[001013] D é o grupo de ramificação
[001014] E é uma corrente;
[001015] F é um ligante; e
[001016] q é um número inteiro entre 1 e 5.
[001017] Em determinadas modalidades, o ligador conjugado compreende pelo menos uma ligação clivável.
[001018] Em determinadas modalidades, cada corrente compreende pelo menos uma ligação clivável.
[001019] Em determinadas modalidades, um composto antisenseconjugado tem uma estrutura selecionada entre os itens a seguir:
[001020] Em determinadas modalidades, um composto antisense conjugado tem uma estrutura selecionada entre os itens a seguir:
[001021] Em determinadas modalidades, um composto antisense conjugado tem uma estrutura selecionada entre os itens a seguir:
[001022] Em determinadas modalidades, um composto antisense conjugado tem a seguinte estrutura:
[001023] As patentes dos Estados Unidos representativas, as publicações do pedido de patente dos Estados Unidos e publicações do pedido de patente internacional que ensinam a preparação de certos conjugados observados acima, compostos antisense conjugados, fixadores, grupos de ramificação, ligantes, frações cliváveis, bem como outras modificações incluem, sem limitação, U.S. N° 5.994.517, U.S. N° 6.300.319, U.S. N° 6.660.720, U.S. N° 6.906.182, U.S. N° 7.262.177, U.S. N° 7.491.805, U.S. N° 8.106.022, U.S. N° 7.723.509, U.S. N° 2006/0148740, U.S. N° 2011/0123520, WO 2013/033230 e WO 2012/037254, sendo que cada um é incorporado por referência a este documento na íntegra.
[001024] Publicações representativas que ensinam a preparação de certos conjugados observados acima, compostos antisense conjugados, correntes, ligadores, grupos de ramificação, ligantes, frações cliváveis, bem como outras modificações incluem, sem limitação, BIESSEN et al, "The Cholesterol Derivative of a Triantennary Galactoside with High Affinity for the Hepatic Asialoglycoprotein Receptor: a Potent Cholesterol Lowering Agent" J. Med. Chem. (1995) 38:1846-1852, BIESSEN et al., "Synthesis of Cluster Galactosides with High Affinity for the Hepatic Asialoglycoprotein Receptor" J. Med. Chem. (1995) 38:1538-1546, LEE et al., "New and more efficient multivalent glyco-ligands for asialoglycoprotein receptor of mammalian hepatocytes" Bioorganic & Medicinal Chemistry (2011) 19:2494-2500, RENSEN et al., "Determination of the Upper Size Limit for Uptake and Processing of Ligands by the Asialoglycoprotein Receptor on Hepatocytes in Vitro and in Vivo" J. Biol. Chem. (2001) 276(40):37577- 37584, RENSEN et al., "Design and Synthesis of Novel N- Acetylgalactosamine-Terminated Glycolipids for Targeting of Lipoproteins to the Hepatic Asialoglycoprotein Receptor" J. Med. Chem. (2004) 47:5798-5808, SLIEDREGT et al., "Design and Synthesis of Novel Amphiphilic Dendritic Galactosides for Selective Targeting of Liposomes to the Hepatic Asialoglycoprotein Receptor" J. Med. Chem. (1999) 42:609-618, and Valentijn et al., "Solid-phase synthesis of lysine- based cluster galactosides with high affinity for the Asialoglycoprotein Receptor" Tetrahedron, 1997, 53(2), 759-770, sendo que cada um desses itens é incorporado por referência a este documento em sua integralidade.
[001025] Em determinadas modalidades, os compostos antisense conjugados compreendem um oligonucleotídeo baseado em RNase H (como gapmer) ou um oligonucleotídeo modulador de união (como oligonucleotídeo totalmente modificado) e qualquer grupo conjugado compreendendo pelo menos um, dois ou três grupos GalNAc. Em determinadas modalidades, um composto antisense conjugado compreende qualquer grupo conjugado encontrado em qualquer uma das seguintes referências: Lee, Carbohydr Res, 1978, 67, 509-514; Connolly et al., J Biol Chem, 1982, 257, 939-945; Pavia et al., Int J Pep Protein Res, 1983, 22, 539-548; Lee et al., Biochem, 1984, 23, 42554261; Lee et al., Glycoconjugate J, 1987, 4, 317-328; Toyokuni et al., Tetrahedron Lett, 1990, 31, 2673-2676; Biessen et al., J Med Chem, 1995, 38, 1538-1546; Valentijn et al., Tetrahedron, 1997, 53, 759-770; Kim et al., Tetrahedron Lett, 1997, 38, 3487-3490; Lee et al., Bioconjug Chem, 1997, 8, 762-765; Kato et al., Glycobiol, 2001, 11, 821-829; Rensen et al., J Biol Chem, 2001, 276, 37577-37584; Lee et al., Methods Enzymol, 2003, 362, 38-43; Westerlind et al., Glycoconj J, 2004, 21, 227-241; Lee et al., Bioorg Med Chem Lett, 2006, 16(19), 5132-5135; Maierhofer et al., Bioorg Med Chem, 2007, 15, 7661-7676; Khorev et al., Bioorg Med Chem, 2008, 16, 5216-5231; Lee et al., Bioorg Med Chem, 2011, 19, 2494-2500; Kornilova et al., Analyt Biochem, 2012, 425, 4346; Pujol et al., Angew Chemie Int Ed Engl, 2012, 51, 7445-7448; Biessen et al., J Med Chem, 1995, 38, 1846-1852; Sliedregt et al., J Med Chem, 1999, 42, 609-618; Rensen et al., J Med Chem, 2004, 47, 57985808; Rensen et al., Arterioscler Thromb Vasc Biol, 2006, 26, 169-175; van Rossenberg et al., Gene Ther, 2004, 11, 457-464; Sato et al., J Am Chem Soc, 2004, 126, 14013-14022; Lee et al., J Org Chem, 2012, 77, 7564-7571; Biessen et al., FASEB J, 2000, 14, 1784-1792; Rajur et al., Bioconjug Chem, 1997, 8, 935-940; Duff et al., Methods Enzymol, 2000, 313, 297-321; Maier et al., Bioconjug Chem, 2003, 14, 18-29; Jayaprakash et al., Org Lett, 2010, 12, 5410-5413; Manoharan,Antisense Nucleic Acid Drug Dev, 2002, 12, 103-128; Merwin et al., Bioconjug Chem, 1994, 5, 612-620; Tomiya et al., Bioorg Med Chem, 2013, 21, 5275-5281;Pedidos internacionais WO1998/013381;WO2011/038356; WO1997/046098; WO2008/098788;WO2004/101619; WO2012/037254; WO2011/120053; WO2013/166121; WO2013/165816; Patentes U.S. 4,751,219;8.552.163; 6.908.903; 7.262.177; 5,994,517; 6,300,319; 8,106,022;7,491,805; 7,491,805; 7,582,744; 8,137,695; 6,383,812; 6,525,031;6,660,720; 7,723,509; 8,541,548; 8,344,125; 8,313,772; 8,349,308;8,450,467; 8,501,930; 8,158,601; 7,262,177; 6,906,182; 6,620,916;8,435,491; 8,404,862; 7,851,615; Publicações de Pedido de PatenteU.S Publicados US2011/0097264; US2011/0097265; US2013/0004427;US2005/0164235; US2006/0148740; US2008/0281044;US2010/0240730; US2003/0119724; US2006/0183886;US2008/0206869; US2011/0269814; US2009/0286973;US2011/0207799; US2012/0136042; US2012/0165393;US2008/0281041; US2009/0203135; US2012/0035115;US2012/0095075; US2012/0101148; US2012/0128760;US2012/0157509; US2012/0230938; US2013/0109817;US2013/0121954; US2013/0178512; US2013/0236968;US2011/0123520; US2003/0077829; US2008/0108801; eUS2009/0203132; cada uma das quais é aqui incorporada porreferência na sua totalidade.Cultura Celular e Tratamento de Compostos Antisense
[001026] Os efeitos de compostos antisense no nível, atividade ou expressão de ácidos nucleicos ANGPTL3 podem ser testados in vitro em uma variedade de tipos de células. Os tipos de células usados para essas análises estão disponíveis a partir de fornecedores comerciais (por exemplo, American Type Culture Collection, Manassus, VA; Zen- Bio, Inc., Research Triangle Park, NC; Clonetics Corporation, Walkersville, MD) e as células são cultivadas de acordo com as instruções do fornecedor, usando os reagentes comercialmente disponíveis (por exemplo, Invitrogen Life Technologies, Carlsbad, CA). Tipos de células ilustrativos incluem, mas não estão limitados a, células HepG2, células Hep3B, células Huh7 (carcinoma hepatocelular), hepatócitos primários, células A549, fibroblastos GM04281 e células LLC-MK2.Testes in vitro de oligonucleotídeos antisense
[001027] São descritos neste documento os métodos para o tratamento de células com oligonucleotídeos antisense que podem ser modificados apropriadamente para o tratamento com outros compostos antisense.
[001028] Em geral, as células são tratadas com oligonucleotídeos antisense quando as células atingem aproximadamente 60 a 80% de confluência na cultura.
[001029] Um reagente comumente usado para introduzir os oligonucleotídeos antisense nas células cultivadas inclui o reagente de transfecção lipídica catiônica LIPOFECTIN® (Invitrogen, Carlsbad, CA). Os oligonucleotídeos antisense são misturados com LIPOFECTIN® em OPTI-MEM® 1 (Invitrogen, Carlsbad, CA) para atingir a concentração final desejada do oligonucleotídeo antisense e uma concentração de LIPOFECTIN® que normalmente varia de 2 a 12 ug/mL por 100 nM de oligonucleotídeo antisense.
[001030] Outro reagente usado para introduzir os oligonucleotídeos antisense nas células cultivadas inclui LIPOFECTAMINE 2000® (Invitrogen, Carlsbad, CA). O oligonucleotídeo antisense é misturado com LIPOFECTAMINE 2000® no meio de soro reduzido de OPTI- MEM® 1 (Invitrogen, Carlsbad, CA) para atingir a concentração desejada do oligonucleotídeo antisense e uma concentração de LIPOFECTAMINE® que normalmente varia de 2 a 12 ug/mL por 100 nM de oligonucleotídeo antisense.
[001031] Outro reagente usado para introduzir os oligonucleotídeos antisense nas células cultivadas inclui Cytofectin® (Invitrogen, Carlsbad, CA). O oligonucleotídeo antisense é misturado com Cytofectin® no meio de soro reduzido de OPTI-MEM® 1 (Invitrogen, Carlsbad, CA) para atingir a concentração desejada do oligonucleotídeo antisense e uma concentração de Cytofectin® que normalmente varia de 2 a 12 ug/mL por 100 nM de oligonucleotídeo antisense.
[001032] Outro reagente usado para introduzir os oligonucleotídeos antisense nas células cultivadas inclui Oligofectamine™ (Invitrogen Life Technologies, Carlsbad, CA). O oligonucleotídeo antisense é misturado com Oligofectamine™ no meio de soro reduzido de Opti-MEM™-1 (Invitrogen Life Technologies, Carlsbad, CA) para obter a concentração desejada de oligonucleotídeo com uma razão de Oligofectamine™ para oligonucleotídeo de cerca de 0,2 a 0,8 μL por 100 nM.
[001033] Outro reagente usado para introduzir oligonucleotídeos antisense em células cultivadas inclui FuGENE 6 (Roche Diagnostics Corp., Indianapolis, IN). O composto oligomérico antisense foi misturado com FuGENE 6 em 1 mL de RPMI livre de soro, para obter a concentração desejada do oligonucleotídeo com uma razão de FuGENE 6 para composto oligomérico de 1 a 4 μL de FuGENE 6 por 100 nM.
[001034] Outra técnica utilizada para introduzir oligonucleotídeos antisense em células cultivadas inclui eletroporação (Sambrook e Russell, Molecular Cloning: A Laboratory Manual, 3a Ed., 2001).
[001035] As células são tratadas com os oligonucleotídeos antisense por métodos de rotina. As células são normalmente colhidas 16-24 horas após o tratamento com o oligonucleotídeo antisense, tempo no qual o RNA ou os níveis de proteína dos ácidos nucleicos alvos são medidos por métodos conhecidos na técnica e descritos neste documento. Em geral, quando os tratamentos são executados em múltiplas réplicas, os dados são apresentados como a média dos tratamentos replicados.
[001036] A concentração do oligonucleotídeo antisense usada varia de linhagem celular para linhagem celular. Os métodos para determinar a concentração de oligonucleotídeo antisense ideal para uma linhagem celular particular são bem conhecidos na técnica. Os oligonucleotídeos antisense são normalmente usados em concentrações variando de 1 nM a 300 nM quando transfectados com LIPOFECTAMINE2000®, Lipofectin ou Cytofectin. Os oligonucleotídeos antisense são usados em concentrações superiores variando de 625 a 20.000 nM, quando transfectados usando eletroporação.
Isolamento de RNA
[001037] A análise de RNA pode ser executada no RNA celular total ou poli(A) + mRNA. Os métodos de isolamento de RNA são bem conhecidos na técnica (Sambrook e Russell, Molecular Cloning: A Laboratory Manual, 3a Ed., 2001). O RNA é preparado usando os métodos conhecidos na técnica, por exemplo, usando o Reagente TRIZOL® (Invitrogen, Carlsbad, CA) de acordo com os protocolos recomendados pelo fabricante.
Análise da inibição de níveis ou expressão alvo
[001038] A inibição dos níveis de expressão ou de um ácido nucleico ANGPTL3 pode ser ensaiada em uma variedade de formas conhecidas na técnica (Sambrook e Russell, Molecular Cloning: A Laboratory Manual, 3a Ed., 2001). Por exemplo, os níveis de ácido nucleico alvo podem ser quantificados, por exemplo, pela análise de Northern blot, reação em cadeia de polimerase competitiva (PCR), ou PCR em tempo real quantitativa. A análise de RNA pode ser executada no RNA celular total ou poli(A) + mRNA. Os métodos de isolamento de RNA são bem conhecidos na técnica. A análise de Northern blot também é rotina na técnica. O PCR em tempo real quantitativo pode ser convenientemente realizado, usando o comercialmente disponível Sequence Detection System 7600, 7700, ou 7900 da ABI PRISM® disponível pela PE- Applied Biosystems, Foster City, CA e usado de acordo com as instruções do fabricante.Análise de PCR Quantitativa em Tempo Real de Níveis de RNA Alvo
[001039] A quantificação dos níveis de RNA alvo pode ser realizada pela PCR em tempo real quantitativa, usando o Sequence Detection System 7600, 7700, ou 7900 de ABI PRISM® (PE-Applied Biosystems, Foster City, CA) de acordo com as instruções do fabricante. Métodos de PCR quantitativa em tempo real são bem conhecidos na técnica.
[001040] Antes da PCR em tempo real, o RNA isolado é submetido a uma reação de transcriptase reversa (RT), que produz o DNA complementar (cDNA) que é então usado como o substrato para a amplificação de PCR em tempo real. A RT e as reações de PCR em tempo real são executadas sequencialmente no mesmo poço da amostra. A RT e os reagentes de PCR em tempo real são obtidos a partir de Invitrogen (Carlsbad, CA). A RT e as reações de PCR em tempo real são realizadas por métodos bem conhecidos por aqueles versados na técnica.
[001041] As quantidades alvo de gene (ou RNA) obtidas por PCR em tempo real poem ser normalizadas usando o nível de expressão de um gene cuja expressão é constante, tal como ciclofilina A ou GADPH por quantificação de RNA total usando RIBOGREEN® (Life TechnologiesTM, Inc. Carlsbad, CA). A expressão da ciclofilina A ou GADPH pode ser quantificada por PCR em tempo real, sendo executada simultaneamente com o alvo, por multiplexação, ou separadamente. O RNA total pode ser quantificado utilizando o reagente de quantificação de RNA RIBOGREEN®. Os métodos de quantificação de RNA por RIBOGREEN® são ensinados em Jones, L.J., et al., (Analytical Biochemistry, 1998, 265, 368-374). um instrumento CYTOFLUOR® 4000 (PE Applied Biosystems) pode ser usado para medir a fluorescência por RIBOGREEN®.
[001042] Os métodos para projetar as sondas de PCR em tempo real e os iniciadores são bem conhecidos na técnica, e podem incluir o uso de software, tal como o Software PRIMER EXPRESS® (Applied Biosystems, Foster City, CA). As sondas e os iniciadores utilizados em PCR em tempo real foram concebidos para hibridar com sequências específicas de ANGPTL3 e são descritos nos Exemplos abaixo. As sondas de PCR específicas de alvo podem ter FAM covalentemente ligado à extremidade 5' e TAMRA ou MGB ligado covalentemente à extremidade 3', onde FAM é o corante fluorescente e TAMRA ou MGB é o corante extintor.
Análise de Níveis de Proteína
[001043] A inibição antisense de ácidos nucleicos ANGPTL3 pode ser avaliada ao medir níveis de proteína ANGPTL3. Os níveis de proteína de ANGPTL3 podem ser avaliados ou quantificados em uma variedade de formas bem conhecidas na técnica, tais como imunoprecipitação, análise por Western blot (immunoblotting), ensaio imunoabsorvente ligado à enzima (ELISA), ensaios de proteína quantitativos, ensaios de atividade de proteína (por exemplo, ensaios de atividade de caspase), imunohistoquímica, imunocitoquímica ou separação de células ativada por fluorescência (FACS) (Sambrooke and Russell in Molecular Cloning: A Laboratory Manual, 3a Ed., 2001). Os anticorpos direcionados a um alvo podem ser identificados e obtidos a partir de uma variedade de fontes, tais como o catálogo MSRS de anticorpos (Aerie Corporation, Birmingham, MI), ou podem ser preparados por meio de métodos de geração de anticorpos monoclonais ou policlonais convencionais bem conhecidos na técnica. Testes in vivo de compostos antisense
[001044] Os compostos antisense, por exemplo, oligonucleotídeos antisense, são testados em animais para avaliar sua capacidade para inibir a expressão de ANGPTL3 e produzir alterações fenotípicas. Os testes podem ser realizados em animais normais, ou em modelos de doença experimentais. Para a administração em animais, os oligonucleotídeos antisense são formulados em um diluente farmaceuticamente aceitável, tal como salina tamponada com fosfato. A administração inclui as vias parenterais de administração. Após um período de tratamento com os oligonucleotídeos antisense, o RNA é isolado do tecido e alterações na expressão do ácido nucleico de ANGPTL3 são medidas. Alterações nos níveis de proteína de ANGPTL3 também são medidas.
Determinadas Indicações
[001045] Em certas modalidades, são fornecidos neste documento métodos de tratamento de um indivíduo compreendendo a administração de uma ou mais composições farmacêuticas, conforme descrito neste documento. Em certas modalidades, o indivíduo tem uma doença metabólica e/ou doença cardiovascular. Em determinadas incorporações, o indivíduo tem hiperlipidemia combinada (por exemplo, familiar ou não-familiar), hipercolesterolemia (por exemplo, hipercolesterolemia homozigótica familiar (HoFH), hipercolesterolemia familiar de heterozigoto (HeFH)), dislipidemia, lipodistrofia, hipertrigliceridemia (por exemplo, deficiência de LPL de heterozigoto, deficiência LPL de homozigoto), doença arterial coronariana (DAC), síndrome de quilomicronemia familiar (FCS), hiperlipoproteinemia tipo IV), síndrome metabólica, doença hepática gordurosa não alcoólica (NAFLD), esteatohepatite não-alcoólica (NASH), diabetes (por exemplo, diabetes tipo 2, diabetes de tipo 2 com dislipidemia) , resistência à insulina (por exemplo, a resistência de insulina com dislipidemia), espessamento da parede vascular, hipertensão arterial (por exemplo, hipertensão arterial pulmonar), esclerose (por exemplo, aterosclerose, esclerose sistêmica, esclerose progressiva da pele e vasculopatia obliterativa proliferativa como úlceras digitais e envolvimento vascular pulmonar) ou uma combinação destes.
[001046] Em certas modalidades, os compostos direcionados a ANGPTL3 aqui descritos modulam lípidos e/ou metabolismo de energia num animal. Em certas modalidades, os compostos direccionados para ANGPTL3 aqui descritos modulam marcadores fisiológicos ou fenótipos de hipercolesterolemia, dislipidemia, lipodistrofia, hipertrigliceridemia, síndrome metabólica, DHGNA, Nash e/ou diabetes. Por exemplo, a administração dos compostos a animais pode modular um ou mais de VLDL, ácidos graxos não esterificados (NEFA), LDL, colesterol, triglicerídeos, glicose, insulina ou níveis de ANGPTL3. Em certas modalidades, a modulação dos marcadores fisiológicos ou fenótipos pode estar associada à inibição de ANGPTL3 pelos compostos.
[001047] Em certas modalidades, os compostos direcionados a ANGPTL3 aqui descritos reduzem e/ou evitam que um ou mais de acumulação de TG hepática (isto é, esteatose hepática), aterosclerose, espessamento da parede vascular (por exemplo, o espessamento íntima-média arterial), hiperlipidemia combinada (por exemplo, familiar ou não familiar), hipercolesterolemia (por exemplo, hipercolesterolemia familiar homozigótica (HFHo), hipercolesterolemia familiar heterozigótica (HeFH)), dislipidemia, lipodistrofia, hipertrigliceridemia (por exemplo, deficiência de LPL heterozigótica, a deficiência de LPL homozigótica, síndrome de quilomicronemia familiar (FCS), hiperlipoproteinemia tipo IV), síndrome metabólica, doença hepática gordurosa não alcoólica (DHGNA), esteato-hepatite não alcoólica (NASH), diabetes (por exemplo, diabetes tipo 2, diabetes tipo 2 com dislipidemia), resistência à insulina (por exemplo, resistência à insulina com dislipidemia), hipertensão arterial e a esclerose múltipla, ou qualquer combinação dos mesmos. Em certas modalidades, os compostos direcionados a ANGPTL3 aqui descritos melhoram a sensibilidade à insulina.
[001048] Em certas modalidades, a administração de um composto antisense dirigido a um ácido nucleico de ANGPTL3 resulta na redução da expressão de ANGPTL3 em cerca de pelo menos 15%, pelo menos20%, pelo menos 25%, pelo menos 30%, pelo menos 35%, pelo menos40%, pelo menos 45%, pelo menos 50%, pelo menos 55%, pelo menos60%, pelo menos 65%, pelo menos 70%, pelo menos 75%, pelo menos80%, pelo menos 85%, pelo menos 90%, pelo menos 95% ou pelo menos 99%, ou uma gama definida por dois quaisquer destes valores.
[001049] Em determinadas modalidades, as composições farmacêuticas que compreendem um composto antisense direcionado ao ANGPTL3 são usadas para a preparação de um medicamento para o tratamento de um paciente sofrendo ou suscetível à doença metabólica ou cardiovascular relacionada. Em certas modalidades, as composições farmacêuticas que compreendem um composto antisense direccionados para ANGPTL3 são utilizadas na preparação de um medicamento para o tratamento de um doente que sofre de, ou é susceptível a um ou mais de hiperlipidemia combinada (por exemplo, familiar ou não familiar), hipercolesterolemia (por exemplo, hipercolesterolemia familiar homozigótica (HFHo), hipercolesterolemia familiar heterozigótica (HeFH)), dislipidemia, lipodistrofia, hipertrigliceridemia (por exemplo, síndrome de quilomicronemia familiar (FCS), hiperlipoproteinemia tipo IV), síndrome metabólica, doença hepática gordurosa não alcoólica (DHGNA), esteato-hepatite não alcoólica (NASH), diabetes (por exemplo, diabetes tipo 2, diabetes tipo 2 com dislipidemia), resistência à insulina (por exemplo, resistência à insulina com dislipidemia), hipertensão arterial e a esclerose múltipla, ou qualquer combinação dos mesmos.
Administração
[001050] Em certas modalidades, os compostos e as composições tal como aqui descritos são administrados por via parentérica.
[001051] Em certas modalidades, a administração parentérica é por infusão. A infusão pode ser crônica ou contínua ou intermitente ou curta. Em certas modalidades, os agentes farmacêuticos infundidos são entregues com uma bomba.
[001052] Em certas modalidades, a administração parentérica é por injecção. A injeção pode ser fornecida com uma seringa ou uma bomba. Em certas modalidades, a injecção é uma injecção em bolus. Em certas modalidades, a injecção é administrada directamente a um tecido ou órgão. Em certas modalidades, a injecção é subcutânea.
Determinadas Terapias de Combinação
[001053] Em certas modalidades, um primeiro agente que compreende o oligonucleotídeo modificado divulgado aqui é co- administrado com um ou mais agentes secundários. Em certas modalidades, tais agentes secundários são destinados a tratar a mesma doença, distúrbio, ou condição como o agente primário descrito neste documento. Em certas modalidades, tais agentes secundários são projetados para tratar uma doença, distúrbio, ou condição diferente como o agente primário descrito neste documento. Em certas modalidades, esses agentes secundários são projetados para tratar um efeito colateral indesejado de uma ou mais composições farmacêuticas, conforme descrito neste documento. Em certas modalidades, os agentes secundários são coadministrados com o agente primário para tratar um efeito indesejado do agente primário. Em certas modalidades, os agentes secundários são coadministrados com o agente primário para produzir um efeito combinacional. Em certas modalidades, os agentes secundários são coadministrados com o agente primário para produzir um efeito sinérgico.
[001054] Em determinadas modalidades, um primeiro agente e um ou mais segundos agentes são administrados ao mesmo tempo. Em determinadas modalidades, o primeiro agente e um ou mais segundos agentes são administrados em horários diferentes. Em determinadas modalidades, o primeiro agente e um ou mais segundos agentes são preparados juntos em uma única formulação farmacêutica. Em determinadas modalidades, o primeiro agente e um ou mais segundos agentes são preparados separadamente.
[001055] Em certas modalidades, segundo agentes incluem, mas não estão limitados a um agente de redução de glicose ou de um agente de redução de lípidos. O agente redutor de glicose pode incluir, mas não está limitado a, uma mudança terapêutica do estilo de vida, agonista PPAR, um inibidor da dipeptidil peptidase (IV), um análogo de GLP-1, insulina ou um análogo de insulina, uma secretagogo de insulina, um inibidor de SGLT2, um análogo da amilina humana, uma biguanida, um inibidor da alfa-glicosidase ou uma combinação destes. O agente redutor de glicose pode incluir, mas não está limitado a metformina, sulfonilureia, rosiglitazona, meglitinida, tiazolidinediona, inibidor da alfa- glicosidase ou uma combinação destes. A sulfonilureia pode ser acetohexamida, clorpropamida, tolbutamida, tolazamida, glimepirida, uma glipizida, uma gliburida, ou uma gliclazida. A meglitinida pode ser nateglinida ou repaglinida. A tiazolidinediona pode ser pioglitazona ou rosiglitazona. A alfa-glicosidase pode ser acarbose ou miglitol. Em certos enquadramentos, a terapia de redução de lípidos pode incluir, mas não está limitado a uma alteração terapêutica de estilo de vida, niacina, inibidor de HMG-CoA-redutase, inibidor da absorção de colesterol, um inibidor de MTP (por exemplo, uma pequena molécula, polipéptido, anticorpo ou um composto antisense dirigido a MTP), fibratos, inibidores de PCSK9 (por exemplo, anticorpos de PCSK9, polipéptidos, compostos de ácido nucleico de molécula pequena direcionados a PCSK9), inibidor de CETP (por exemplo, moléculas pequenas tais como torcetrapibe e anacetrapibe, polipéptidos, anticorpos ou compostos de ácido nucleico direcionados para CETP), inibidor de apoC-III (por exemplo, uma pequena molécula, polipéptido, anticorpo ou compostos de ácido nucleico direcionados para apoC-III), inibidor de apoB (por exemplo, uma molécula pequena, um polipéptido, anticorpo ou compostos de ácidos nucleicos direcionados para a apoB), ácidos graxos de óleos ricos em ômega-3 benéficos, ácidos graxos de ômega-3, ou qualquer combinação dos mesmos. O inibidor da HMG- CoA redutase pode ser atorvastatina, rosuvastatina, fluvastatina, lovastatina, pravastatina, sinvastatina e similares. O inibidor de absorção de colesterol pode ser ezetimiba. O fibrato pode ser fenofibrato, bezafibrato, ciprofibrato, clofibrato, gemfibrozil e outros semelhantes. Os ácidos graxos de óleo benéfico ricos em ômega-3 pode ser de krill, peixes (por exemplo, VascepaR), óleo de linhaça e semelhantes. O ácido graxo de ômega-3 pode ser ALA, DHA, EPA e outros semelhantes.
Determinados Compostos
[001056] Oligonucleotídeos antisense direcionados a ANGPTL3 humana foram descritos numa publicação anterior (ver Publicação de Patente PCT No. O documento WO 2011/085271 publicada em 14 de julho de 2011, aqui incorporada por referência, na sua totalidade). Vários oligonucleotídeos (233676, 233690, 233710, 233717, 233721, 233722, 337459, 337460, 337474, 337477, 337478, 337479, 337481,337484, 337487, 337488, 337490, 337491, 337492, 337497, 337498,337503, 337505, 337506 , 337508, 337513, 337514, 337516, 337520,337521, 337525, 337526 e 337528) aqui descritos, incluindo os dezcompostos antisense mais potentes in vitro foram utilizados como referência ao longos de triagens seletas in vitro para os compostos antisense descritos aqui a seguir e em Número de Série U.S. 61/920.652. Dentre os compostos mais potentes descritos no documento WO 2011/085271, o ISIS 233722 verificou-se ser altamente variável na sua capacidade de inibir ANGPTL3. Segundo, embora inicialmente incluído em alguns estudos in vitro, 233722 não foiselecionado como um ponto de referência para estudos posteriores. Dos potentes e previamente identificado compostos de referência in vitro, cinco (233710, 233717, 337477, 337478, 337479 e 337487) foram selecionados para testes in vivo, Tal como descrito aqui a seguir, em camundongos transgénicos huANGPTL3 para avaliar o mais potente na redução da transcrição do mRNA humano e a expressão da proteína (Exemplo 126). O oligonucleotídeo antisense com a mais alta potência inicial in vivo na redução dos níveis de ANGPTL3 (233710) foi utilizado como referência para avaliação in vivo dos novos compostos antisense descritos aqui a seguir.
[001057] Em certas modalidades, os compostos antisense aqui descritos se beneficiam de uma ou mais propriedades melhoradas em relação aos compostos antisense descritos em WO 2011/085271 e em Número de Série US 61/920.652. Estas propriedades melhoradas são demonstradas nos exemplos aqui, e um resumo não exaustivo dos exemplos é fornecido abaixo para facilitar a referência.
[001058] Numa primeira triagem aqui descrita, cerca de 3000 recém concebidos compostos antisense de espaçâmero de 5-10-5 MOE direcionados a ANGPTL3 humana foram testados em células Hep3B para o seu efeito no mRNA humano de ANGPTL3 in vitro (Exemplo 116). Os níveis de inibição de mRNA dos novos compostos antisense foram avaliados com alguns compostos antisense previamente concebidos (233717, 337484, 337487, 337492 e 337516) utilizados como referência em estudos seletos. Dos cerca de 3000 compostos anti-sense recém-concebidos desta primeira triagem, foram selecionados cerca de 85 compostos anti-sense para estudos de inibição in vitro dependentes da dose para determinar a sua concentração inibitória meia-máxima (IC50) (Exemplos 117-118). Dos cerca de 85 novos compostos antisense testados para a sua concentração inibitória meio-máxima (IC50), cerca de 38 compostos antisense que demonstraram redução dependente da dose potente de ANGPTL3 foram seleccionados para teste in vivo de potência e tolerabilidade (ALT e AST) em camundongos (Exemplos 126-127) com composto anti-sense 233710 usado como um ponto de referência.
[001059] Em uma segunda triagem aqui descrita, cerca de 2.000 compostos antisense concebidos recentemente direcionados a ANGPTL3 humana com um motivo espaçâmero MOE ou um motivo misto (desoxi, 5-10-5 MOE e espaçâmetos cET ) foram também testados em células Hep3B para o seu efeito sobre mRNA de ANGPTL3 humano in vitro (Exemplos 119-121). Os níveis de inibição dos novos compostos antisense foram avaliados com alguns compostos antisense previamente concebidos (233717, 337484, 337487, 337492 e 337516) utilizados como referência em estudos seletos. Dos cerca de 2000 compostos anti-sense recém-concebidos desta segunda triagem, foram selecionados cerca de 147 compostos anti-sense para estudos de inibição in vitro dependentes da dose para determinar a sua concentração inibitória meia-máxima (IC50) (Exemplos 122-125). Dos cerca de 147cnovos compostos antisense testados para a sua concentração inibitória meio-máxima (IC50), cerca de 73 compostos antisense que demonstraram redução dependente da dose potente de ANGPTL3 foram seleccionados para teste in vivo de potência e tolerabilidade (por exemplo, ALT e AST) em camundongos (Exemplos 126-127) com composto anti-sense 233710 usado como um ponto de referência.
[001060] Das cerca de 111 compostos antisense a partir de triagens um e dois que foram testadas para a potência e a tolerância em camundongos, 24 foram seleccionados para testes mais extensivos tolerabilidade em camundongos por meio da avaliação de marcadores metabólicos hepáticos, como a alanina-transaminase (ALT), aspartato transaminase (AST) , albumina e bilirrubina, bem como marcadores metabólicos de rim de BUN e de creatinina e de órgãos peso (Exemplo 127).
[001061] Em paralelo com os estudos murinos in vivo dezessete compostos antisense foram seleccionadas para o ensaio de viscosidade (Exemplo 128). Geralmente, os compostos antisense que não foram ideais para a viscosidade não foram levados para a frente em estudos posteriores.
[001062] Com base nos resultados do estudo de tolerabilidade de camundongos, foram seleccionados vinte compostos antisense para testes de tolerabilidade em camundongos in vivo (Exemplo 129). Nos ratos, os marcadores do metabolismo do fígado, tais como ALT, AST, albumina e bilirrubina, do corpo e dos órgãos pesos, bem como marcadores metabólicos nos rins, tais como ureia, creatinina e razão total de proteína/creatinina, foram medidos para determinar a tolerabilidade de uma composto in vivo.
[001063] Os vinte compostos antisense testados nos ratos foram também avaliados quanto a reactividade cruzada com uma sequência de gene de macaco rhesus ANGPTL3 (Exemplo 130). Embora os compostos antisense neste estudo foram testados em macacos Cynomolgus, a sequência ANGPTL3 de macaco cynomolgus não estava disponível para comparação das sequências dos compostos de comprimento total, por conseguinte, as sequências de compostos antisense foram comparadas com a do macaco rhesus intimamente relacionado . As sequências de oito compostos antisense foram encontradas com 0-2 erros de emparelhamento com a sequência do gene ANGPTL3 de rhesus e foram ainda estudadas em macacos cynomolgus (Exemplo 130). Os oito compostos anti-sense (ISIS 563580, ISIS 560400, ISIS 567320, ISIS 567321, ISIS 544199, ISIS 567233, ISIS 561011 e ISIS 559277) foram testados para a inibição do mRNA de ANGPTL3 e expressão da proteína, bem como a tolerabilidade nos macacos. Nos estudos de tolerabilidade, pesos corporais, marcadores metabólicos do fígado (ALT, AST e bilirrubina), marcadores metabólicos renais (uréia e creatinina), parâmetros hematológicos (contagem de células sanguíneas, hemoglobina e hematócrito), e marcadores pró-inflamatórios (CRP e C3) foram medidos. Além disso, a concentração de oligonucleotídeo de comprimento completo presente no fígado e no rim foi medida e foi calculada a razão de oligonucleotídeo de comprimento completo no rim/fígado.
[001064] A sequência antisense de um composto potente e tolerável, ISIS 563580, avaliado em macacos Cynomolgus foi ainda modificada com um conjugado de GalNAc e/ou alterações na química espinha dorsal, como mostrado nos Exemplos 113-115 e 131 e avaliados para aumentar a potência.
[001065] Consequentemente, são aqui proporcionados os compostos antisense com qualquer uma ou mais características melhoradas, por exemplo, melhoradas em relação aos compostos antisense descritos em WO 2011/085271 e em US Número de Série 61/920.652. Em certas modalidades, são aqui proporcionados os compostos antisense que compreendem um oligonucleotídeo modificado como aqui descrito orientada para, ou especificamente hibridar com, uma região de nucleotídeos de qualquer uma de SEQ ID NOs: 1-2.
[001066] Em certas modalidades, certos compostos antisense, como aqui descritos, são eficazes devido à sua potência para inibirem a expressão de ANGPTL3. Em certas modalidades, os compostos ou composições inibem ANGPTL3 em pelo menos 40%, pelo menos 45%, pelo menos 50%, pelo menos 55%, pelo menos 60%, pelo menos 65%, pelo menos 70%, pelo menos 75% , pelo menos 80%, pelo menos 85%, pelo menos 90% ou pelo menos 95%.
[001067] Em certas modalidades, certos compostos antisense, como aqui descritos são eficazes em virtude de umin vitro IC50 de menos de 20 μM, menos de 10 μM, menos de 8 μM, menos do que 5 μM, menos de 2 μM, menos de 1 μM, menos de 0,9 μM, menos de 0,8 μM, menos de 0,7 μM, menos do que 0,6 μM, ou inferior a 0,5 μM, quando testada em células humanas, por exemplo, na linhagem celular de Hep3B (tal como descrito nos Exemplos 117-118 e 122-125). Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <1,0 μM incluem SEQ ID NOs: 15, 20, 24, 34, 35, 36, 37, 42, 43, 44, 47, 50, 51, 57, 58, 60, 77, 79, 82, 87, 88, 90, 91, 93, 94, 100, 101, 104, 110, 111, 112, 113, 114, 115, 116, 117, 118, 119, 120, 122,123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136,137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 147, 148, 149, 150,151, 152, 153, 154, 155, 156, 157, 158, 169, 170, 177, 188, 209, 210,211, 212, 213, 214, 215, 216, 217, 218, 219, 220, 221, 222, 223, 224,225, 226, 227, 228, 229, 230, 231 e 232. Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <0,9 μM incluem SEQ ID NOs: 15, 20, 35, 36, 42, 43, 44, 50, 57, 60, 77, 79, 87, 88, 90, 91, 93, 94, 101, 104, 110, 111, 112, 113, 114, 115, 116, 117, 118, 119, 122, 123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133,134, 135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 147,148, 149, 150, 151, 152, 153, 154, 155, 156, 157, 158, 177, 209, 210,211, 212, 213, 214, 215, 216, 217, 218, 219, 220, 221, 222, 223, 224,225, 226, 227, 228, 229, 230, 231, e 232. Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <0,8 μM incluem SEQ ID NOs: 15, 20, 35, 36, 42, 43, 44, 50, 57, 60, 77, 79, 87, 88, 90, 91, 93, 94, 101, 104, 110, 111, 112, 113, 114, 115, 116, 117, 118, 122, 123, 124, 125, 126, 127, 128, 129, 130, 131, 132, 133, 134,135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 147, 148,149, 150, 151, 152, 153, 154, 155, 156, 157, 158, 177, 209, 210, 211,212, 213, 214, 215, 217, 218, 219, 220, 221, 222, 223, 224, 225, 228,229, 230, 231 e 232. Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <0,7 μM incluem SEQ ID NOs: 15, 20, 36, 42, 43, 57, 60, 114, 117, 127, 131, 177, 209, 210, 211, 212, 213, 214, 215, 217, 218, 219, 220, 221, 222, 223, 224, 225, 228, 229, 230, 231 e 232. Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <0,6 μM incluem SEQ ID NOs: 15, 20, 36, 42, 43, 57, 60, 114, 117, 127, 131, 177, 209, 210, 211, 212, 213, 215, 217, 218, 219, 220, 221, 222, 224, 225, 228, 229, 230, 231 e 232. Em certas modalidades, os compostos antisense preferidos, que possuem um valor de IC50 <0,5 μM incluem SEQ ID NOS: 43, 114, 117, 127, 131, 177, 209, 210, 211, 212, 215, 217, 218, 219, 220, 221, 222, 229, 230, e 232.
[001068] Em certas modalidades, certos compostos antisense, como aqui descritos, são eficazes em virtude de ter uma viscosidade inferior a 40 cP, inferior a 35 cP, inferior a 30 cP, inferior a 25 cP, inferior a 20 cP, inferior a 15 cP, ou inferior a 10 cP quando medida por um ensaio (como descrito no Exemplo 128). Os oligonucleotídeos com uma viscosidade maior do que 40 cP teria menos do que a viscosidade ótima. Em certas modalidades, os compostos antisense preferidos possuindo uma viscosidade <20 cP incluem SEQ ID NOs: 16, 18, 20, 34, 35, 36, 38, 49, 77, 90, 93, e 94. Em certas modalidades, os compostos antisense preferidos possuindo uma viscosidade <15 cP incluem SEQ ID NOs: 16, 18, 20, 34, 35, 38, 49, 90, 93, e 94. Em certas modalidades, os compostos antisense preferidos possuindo uma viscosidade <10 cP incluem SEQ ID NOs: 18, 34, 35, 49, 90, 93, e 94.
[001069] Em certas modalidades, certos compostos antisense, como aqui descritos são altamente tolerável, tal como demonstrado pelas medições de tolerabilidade in vivo descritas nos Exemplos. Em certas modalidades, os compostos anti-sense como aqui descritos são altamente toleráveis, tal como demonstrado por ter um aumento de valor ALT e/ou AST de não mais do que 3 vezes, 2 vezes ou 1,5 vezes mais em animais tratados com solução salina.
[001070] Em certas modalidades, certos compostos antisense, como aqui descritos são eficazes em virtude de ter um ou mais de uma potência de inibição superior a 50%, um in vitro IC50 de menos do que 1 μM, uma viscosidade inferior a 20 cP, e não mais do que um aumento de 3 vezes na ALT e/ou AST.
[001071] Em certas modalidades, ISIS 563580 (SEQ ID NO: 77) é preferido. Este composto verificou-se ser um inibidor potente em camundongos transgênicos ANGPTL3 e o composto antisense mais tolerável. Ele tinha uma viscosidade aceitável de cerca de 16,83 cP e um IC50 valor de <0,8 mM in vitro. Nos camundongos que não tinham mais do que um aumento de 3 vezes na ALT e/ou níveis de AST sobre os animais tratados com solução salina. Além disso, em macacos, foi entre os compostos mais toleráveis e potentes em inibir ANGPTL3 e tinha a melhor relação de concentração de oligonucleotídeo de comprimento completo.
[001072] Em certas modalidades, ISIS 544199 (SEQ ID NO: 20) é preferido. Este composto verificou-se ser um composto antisense potente e tolerável. Ele tinha uma viscosidade aceitável de 1,7 cP e um valor IC50 de <0,5 μM in vitro. Nos camundongos que não tinham mais do que um aumento de 3 vezes na ALT e/ou níveis de AST sobre os animais tratados com solução salina. Além disso, em macacos, foi entre os compostos mais potentes em inibir ANGPTL3 e tinha a melhor relação de concentração de oligonucleotídeo de comprimento completo.
[001073] Em certas modalidades, ISIS 559277 (SEQ ID NO: 110) é preferido. Este composto verificou-se ser um composto antisense potente e tolerável. Ele tinha um IC50 valor de <0,8 μM in vitro. Nos camundongos que não tinham mais do que um aumento de 3 vezes na ALT e/ou níveis de AST sobre os animais tratados com solução salina. Além disso, em macacos, foi entre os compostos mais potentes em inibir ANGPTL3 e tinha a melhor relação de concentração de oligonucleotídeo de comprimento completo.
[001074] Em certas modalidades, um composto antisense conjugado GalNAc, ISIS 658501 (SEQ ID NO: 4912), é o preferido. Este composto antisense foi descoberto mais potente do que o seu composto parental ISIS 563580 (SEQ ID NO: 77) como mostrado pelos níveis de inibição.
[001075] Em certas modalidades, um composto antisense conjugado GalNAc, ISIS 703801 (SEQ ID NO: 77), é o preferido. Este composto antisense foi descoberto sendo várias vezes mais potente do que o seu composto parental ISIS 563580 (SEQ ID NO: 77). ISIS 703801 tinha um valor de ID50 de 1 enquanto ISIS 563580 teve valor ID50 de 6.
[001076] Em certas modalidades, um composto antisense conjugado GalNAc, ISIS 703802 (SEQ ID NO: 77), é o preferido. Este composto antisense foi descoberto sendo várias vezes mais potente do que o seu composto parental ISIS 563580 (SEQ ID NO: 77). ISIS 703802 tinha um valor de ID50 de 0,3, enquanto ISIS 563580 tinha um valor de ID50 de 6.
EXEMPLOS
[001077] Os exemplos a seguir ilustram determinadas modalidades da presente divulgação e não são limitantes. Além disso, se modalidades específicas forem providas, os inventores contemplaram um pedido genérico dessas modalidades específicas. Por exemplo, a divulgação de um oligonucleotídeo que tem um motivo específico provê suporte razoável para oligonucleotídeos adicionais que têm o mesmo motivo ou motivo semelhante. E, por exemplo, se uma modificação específica de alta afinidade surgir em uma posição específica, outras modificações de alta afinidade na mesma posição são consideradas adequadas, salvo indicação contrária.Exemplo 1: Método Geral para a Preparação de Fosforamiditos, Compostos 1, 1a e 2
[001078] Os compostos 1, 1a e 2 foram preparados de acordo com procedimentos bem conhecidos na técnica, descritos na especificação aqui contida (ver Seth et al., Bioorg. Med. Chem., 2011, 21(4), 11221125, J. Org. Chem., 2010, 75(5), 1569-1581, Nucleic Acids Symposium Series, 2008, 52(1), 553-554).; e também publicado nos PedidosInternacionais PCT (WO 2011/115818, WO 2010/077578, WO2010/036698, WO2009/143369, WO 2009/006478 e WO2007/090071) e patente US N° 7569686).Exemplo 2: Preparação do Composto 7
[001079] Os compostos 3 (2-acetamida-1,3,4,6-tetraO-acetil-2- desóxi-p-Dgalactopiranose ou galactosamina pentaacetato) estão comercialmente disponíveis. O composto 5 foi preparado de acordo com procedimentos publicados (Weber et al., J. Med. Chem., 1991, 34, 2692).Exemplo 3: Preparação do Composto 11
[001080] Os compostos 8 e 9 estão comercialmente disponíveis.Exemplo 4: Preparação do Composto 18
[001081] O composto 11 foi preparado de acordo com os procedimentos ilustrados no Exemplo 3. O composto 14 está comercialmente disponível. O composto 17 foi preparado usando procedimentos similares relatados por Rensen et al., J. Med. Chem., 2004, 47, 5798-5808.Exemplo 5: Preparação do Composto 23
[001082] Os compostos 19 e 21 estão comercialmente disponíveis.Exemplo 6: Preparação do Composto 24
[001083] Os compostos 18 e 23 foram preparados de acordo com os procedimentos ilustrados nos Exemplos 4 e 5. Exemplo 7: Preparação do Composto 25
[001084] O composto 11 foi preparado de acordo com os procedimentos ilustrados no Exemplo 6.Exemplo 8: Preparação do Composto 26
[001085] O composto 24 é preparado de acordo com osprocedimentos ilustrados no Exemplo 6.Exemplo 9: Preparação geral de ASOs conjugadoscompreendendo GalNAc3-1 no terminal 3', Composto 29
[001086] Onde GalNAc3-1 protegido tem a estrutura:
[001087] A parte do agrupamento de GalNAc3 do grupo conjugado GalNAc3-1 (GalNAc3-1a) pode ser combinada com qualquer fração clivável para prover uma variedade de grupos conjugados. Onde GalNAc3-1a tem a fórmula:
[001088] O suporte sólido ligou GalNAc3-1 protegido, Composto 25, foi preparado de acordo com os procedimentos ilustrados no Exemplo 7. Composto 29 oligomérico compreendendo GalNAc3-1 no terminal 3' foi preparado utilizando procedimentos padrão em síntese de DNA/RNA automatizado (ver Dupouy et al., Angew. Chem. Int. Ed., 2006, 45, 3623-3627). Blocos de construção de fosforamidita, Compostos 1 e 1a foram preparados de acordo com os procedimentos ilustrados no Exemplo 1. As fosforamiditas ilustradas pretendem ser representativas e não pretendem ser limitantes como outros blocos de construção de fosforamidita podem ser utilizados para preparar compostos oligoméricos com uma sequência e composição predeterminada. A ordem e quantidade de fosforamiditas adicionadas ao suporte sólido podem ser ajustadas para preparar compostos oligoméricos com lacunas como descrito neste instrumento. Esses compostos oligoméricos lacunados podem ter uma composição predeterminada e uma sequência de base mencionada por um determinado alvo.Exemplo 10: ASOs conjugados de preparação geral compreendendo GalNAc3-1 no terminal 5', Composto 34

[001089] O UnylinkerTM 30 está comercialmente disponível. Composto 34 oligomérico compreendendo um aglomerado deGalNAc3-1 no terminal 5' é preparado utilizando procedimentos padrão em síntese automatizada de DNA/RNA (ver Dupouy et al., Angew. Chem. Int. Ed., 2006, 45, 3623-3627). Blocos de construção de fosforamidita, Compostos 1 e 1a foram preparados de acordo com os procedimentos ilustrados no Exemplo 1. As fosforamiditas ilustradas pretendem ser representativas e não pretendem ser limitantes como outros blocos de construção de fosforamidita podem ser utilizados para preparar compostos oligoméricos com uma sequência e composição predeterminada. A ordem e quantidade de fosforamiditas adicionadas ao suporte sólido podem ser ajustadas para preparar compostos oligoméricos com lacunas como descrito neste instrumento. Esses compostos oligoméricos lacunados podem ter uma composição predeterminada e uma sequência de base mencionada por um determinado alvo.Exemplo 11: Preparação do Composto 39


[001090] Os compostos 4, 13 e 23 foram preparados de acordo com os procedimentos ilustrados nos Exemplos 2, 4 e 5. Composto 35 é preparado utilizando processos semelhantes publicados em Rouchaud et al., Eur. J. Org. Chem., 2011, 12, 2346-2353.Exemplo 12: Preparação do Composto 40
[001091] O composto 38 é preparado de acordo com os procedimentos ilustrados no Exemplo 11.Exemplo 13: Preparação do Composto 44
[001092] Os compostos 23 e 36 são preparados de acordo com os procedimentos ilustrados nos exemplos 5 e 11. Composto 41 é preparado utilizando processos semelhantes publicados em WO 2009082607.Exemplo 14: Preparação do Composto 45
[001093] O composto 43 é preparado de acordo com os procedimentos ilustrados no Exemplo 13.Exemplo 15: Preparação do Composto 47
[001094] O composto 14 está comercialmente disponível. Exemplo 16: Preparação do Composto 53
[001095] Compostos 48 e 49 estão comercialmente disponíveis. Os compostos 17 e 47 são preparados de acordo com os procedimentos ilustrados nos Exemplos 4 e 15. Exemplo 17: Preparação do Composto 54
[001096] O composto 53 é preparado de acordo com os procedimentos ilustrados no Exemplo 16.Exemplo 18: Preparação do Composto 55
[001097] O composto 53 é preparado de acordo com osprocedimentos ilustrados no Exemplo 16.Exemplo 19: Método geral para a preparação de ASOs conjugados compreendendo GalNAc3-1 na posição 3' por meio de técnicas em fase sólida (preparação de ISIS 647535, 647536 e 651900)
[001098] Salvo indicação em contrário, todos os reagentes e soluções utilizadas para a síntese dos compostos oligoméricos são adquiridas de fontes comerciais. Blocos de construção de fosforamidita padrão e suporte sólido são usados para a incorporação de resíduos de nucleosídeos que incluem, por exemplo, T, A, G, e mResíduos C. Uma solução de fosforamidita a 0,1 M em anidro acetonitrila foi utilizada para β-D-2'-desoxinbonucleosideo e 2'-MOE.
[001099] As sínteses ASO foram realizadas em sintetizador ABI 394 (escala de 1-2 nmol) ou em sintetizador oligopiloto GE Healthcare Bioscience ÃKTA (escala de 40-200 mmol) pelo método de acoplamento de fosforamidita em GalNAc3-1 suporte sólido VIMAD carregado (110 mmol/g, Guzaev et al., 2003) empacotada na coluna. Para a etapa de acoplamento, as fosforamiditas foram entregues 4 vezes em excesso sobre a carga sobre o suporte sólido e a condensação de fosforamidita foi realizada por 10 min. Todas as outras etapas seguiram os protocolos padrão fornecidos pelo fabricante. Uma solução de ácido dicloroacético a 6% em tolueno foi utilizada para remover o grupo de dimetoxitritilo (DMT) do grupo 5'- hidroxila do nucleotídeo. 4,5-Dicianoimidazol (0,7 M) em anidro CH3CN foi usado como ativador passo durante a etapa de acoplamento. As ligações de fosforotioato foram introduzidas por sulfurização com solução a 0,1 M de hidreto de xantano em piridina/CH3CN 1:1 para um tempo de contato de 3 minutos. Uma solução de 20% terc-butilhidroperóxido em CH3CN contendo 6% de água foi utilizada como um agente oxidante para prover ligações internucleosídeas de fosfodiéster com um tempo de contato de 12 minutos.
[001100] Depois da sequência desejada foi montada, os grupos protetores de cianoetil fosfato foram desprotegidos utilizando uma mistura 1: 1 (v/v) de uma mistura de trietilamina e acetonitrila, com um tempo de contato de 45 minutos. ASOs de ligação de suporte sólido foram suspensos em amônia aquosa (28-30% em peso) e aquecidos a a 55°C por 6 h.
[001101] Os ASO não ligados foram então filtrada e a amônia foi fervida. O resíduo foi purificado por cromatografia líquida de alta pressão numa coluna de troca aniônica forte (GE Healthcare Bioscience, Fonte 30Q, 30 μm, com 2,54 x 8 cm, A = 100 mM acetato de amônia em 30% de CH 3CN aquoso, B = 1,5 M de NaBr, em A, 040% de B em 60 min, fluxo 14 mL min-1, À = 260 nm). O resíduo foi dessalinizado por HPLC numa coluna de fase inversa para produzir ASOs desejados com um rendimento isolado de 15-30% com base na carga inicial sobre o suporte sólido. ASOs foram caracterizados pela análise MS acoplado íon-par-HPLC com sistema Agilent 1100 MSD.
[001102] Os oligonucleotídeos antisense que não compreendem um conjugado foram sintetizados utilizando procedimentos de síntese de oligonucleotídeos convencionais bem conhecidos na técnica.
[001103] Usando esses métodos, três compostos antisense distintos direcionados à ApoC III estavam preparados. Como resumido na Tabela 17, abaixo, três compostos antisense que direcionam a ApoC III tinham a mesma sequência de nucleobases; ISIS 304801 é um gamper 5-10-5 MOE com todas as ligações de fósforotioato; ISIS 647535 é o mesmo que ISIS 304.801, salvo se tinha GalNAc3-1 conjugado na extremidade 3'; e ISIS 647536 é o mesmo que ISIS 647535 salvo se determinadas ligações internucleosídeas desse composto forem ligações fosfodiéster. Como resumido ainda na Tabela 17, dois compostos antisense separados que direcionam SRB-1 foram sintetizados. ISIS 440762 foi um gapmer 2-10-2 CET com todas as ligações internucleosídeas fósforotioato; ISIS 651900 é o mesmo que ISIS 440762, exceto se incluiu um GalNAc3-1 na sua extremidade 3'.Tabela 17ASO modificado direcionando ApoC III e SRB-1
[001104] Subscritos: "e" indicam nucleosídeo 2'-MOE modificado; "d" indica β-D-2'-desoximbonucleosideo; "k" indica 6' (S) CH3 nucleosídeo bicíclico (por exemplo, cEt); "s" indica ligaçõesinternucleosídeas de fosforotioato (PS); "o" indica ligaçõesinternucleosídeas de fosfodiéster (PO); e "o" indica -O-P (=O)(OH)-. O subscrito "m" indica 5-metilcitosinas. "GalNAc3-1" indica um grupo conjugado com a estrutura mostrada anteriormente no Exemplo 9. Observe que GalNAc3-1 compreende uma adenosina clivável que liga o ASO ao restante do conjugado, que é designado "GalNAc3-1a. " Esta nomenclatura é usada na tabela acima, para mostrar a sequência de nucleobases completo, incluindo a adenosina, que faz parte do conjugado. Assim, na tabela acima, as sequências também poderiam ser listadas com uma terminação "GalNAc3-1 " com "Ado" omitido. Esta convenção de uso do subscrito "a" para indicar a parte de um grupo conjugado desprovido de nucleosídeo clivável ou fração clivável é usada nesses Exemplos. Esta parte de um grupo conjugado desprovido de fração clivável é denominada "agrupamento" ou "agrupamento conjugado" ou agrupamento "GalNAc3". Emdeterminados casos, é conveniente descrever um grupo conjugado apresentando, separadamente, seu agrupamento e sua fração clivável.Exemplo 20: Inibição antisense dependente de dose de ApoC III humana em camundongos transgênicos huApoC III
[001105] ISIS 304801 e ISIS 647535, cada um direcionando ApoC III humana e descrita acima, foram testados separadamente e avaliados em um estudo dependente da dose para sua capacidade de inibir ApoC III humana em camundongos transgênicos ApoC III humana.
Tratamento
[001106] Camundongos transgênicos APOCIII humana foram mantidos num ciclo de luz/escuro de 12 horas e foram alimentados ad libitum Teklad lab Chow. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. ASOs foram preparados em PBS e esterilizados pela filtração através de um filtro de 0,2 mícron. ASOs foram dissolvidos em PBS 0,9% para injeção.
[001107] Camundongos transgênicos ApoC III humana foi injetada por via intraperitoneal uma vez por semana por duas semanas com ISIS 304801 ou 647535 em 0,08, 0,25. 0,75, 2,25 ou 6,75 μmol/kg ou com PBS como controle. Cada grupo de tratamento consistiu em 4 animais. Quarenta e oito horas após a administração da última dose, foi retirado sangue de cada camundongo e os camundongos foram sacrificados e os tecidos foram coletados.
Análise de ApoC III mRNA
[001108] Os níveis de mRNA de ApoC III nos fígados dos camundongos foram determinados usando PCR em tempo real e reagente de quantificação RIBOGREEN® RNA (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis de mRNA de ApoC III foram determinados em relação ao RNA total (usando Ribogreen), antes da normalização para controle tratado com PBS. Os resultados abaixo são apresentados como a média percentual dos níveis de mRNA de ApoC III para cada grupo de tratamento, normalizado para o controle tratado com PBS e é indicado como "% PBS". A metade da dosagem máxima eficaz (ED50) de cada ASO é também apresentada na Tabela 18, abaixo.
[001109] Como ilustrado, ambos os compostos antisense reduziram o RNA da ApoC III em relação ao controle com PBS. Além disso, o composto antisense conjugado a GalNAc3-1 (ISIS 647535) era substancialmente mais potentes do que o composto antisense desprovido do conjugado GalNAc3-1 (ISIS 304.801).Tabela 18Efeito do tratamento de ASO nos níveis de mRNA de ApoC III em camundongos transgênicos com ApoC III humana
[001110] Análise da proteína ApoC III do plasma foi determinada usando procedimentos relatados por Graham et al, Circulation Research, publicado online antes da impressão em 29 de março de 2013.
[001111] Cerca de 100 μl de plasma isolado de camundongos foram analisados sem diluição, usando um Analisador Clínico Olympus e um ensaio de ApoC III turbidométrico comercialmente disponível (Kamiya, Cat N° KAI-006, Kamiya biomédica, Seattle, WA). O protocolo do ensaio foi realizado como descrito pelo fornecedor.
[001112] Como mostrado na Tabela 19 abaixo, ambos os compostos antisense reduziram a proteína ApoC III em relação ao controle com PBS. Além disso, o composto antisense conjugado a GalNAc3-1 (ISIS 647535) era substancialmente mais potentes do que o composto antisense desprovido do conjugado GalNAc3-1 (ISIS 304801).Tabela 19Efeito do tratamento de ASO nos níveis da proteína do plasma ApoC III em camundongos transgênicos ApoC III humana
[001113] Triglicerídeos e colesterol do plasma foram extraídos pelo método de Bligh e Dyer (Bligh, E.G. e Dyer, W.J. Can. J. Biochem. Physiol. 37: 911-917, 1959)(Bligh, E and Dyer, W, Can J BiochemPhysiol, 37, 911-917, 1959)(Bligh, E and Dyer, W, Can J BiochemPhysiol, 37, 911-917, 1959) e medidos pelo uso do analisador clínico Beckmann Coulter e reagentes comercialmente disponíveis.
[001114] Os níveis de triglicerídeo foram medidos em relação aos camundongos injetados com PBS e são indicadas como "% PBS". Os resultados são apresentados na Tabela 20. Como ilustrado, ambos os compostos antisense reduziram os níveis de triglicerídeo. Além disso, o composto antisense conjugado a GalNAc3-1 (ISIS 647535) era substancialmente mais potente do que o composto antisense desprovido do conjugado GalNAc3-1 (ISIS 304801).Tabela 20Efeito do tratamento com ASO nos níveis de triglicerídeo em camundongos transgênicos
[001115] As amostras de plasma foram analisadas por HPLC para determinar a quantidade de colesterol total e as diferentes frações de colesterol (HDL e LDL). Os resultados são apresentados nas Tabelas 21 e 22. Como ilustrado, ambos os compostos antisense reduziram os níveis de colesterol total; ambos reduziram o LDL; e ambos aumentaram o HDL. Além disso, o composto antisense conjugado a GalNAc3-1 (ISIS 647535) era substancialmente mais potente do que o composto antisense desprovido do conjugado the GalNAc3-1 (ISIS 304801). Um aumento nos níveis de HDL e uma diminuição nos níveis de LDL é um efeito benéfico cardiovascular de inibição antisense de ApoC IIITabela 21Efeito do tratamento com ASO nos níveis de colesterol total em camundongos transgênicos
Tabela 22Efeito do tratamento de ASO em níveis de colesterol HDL e LDL em camundongos transgênicos
[001116] A PK do ASO também foi avaliada. As amostras de fígado e de rim foram picadas e extraídas utilizando protocolos convencionais. As amostras foram analisadas em MSD1 utilizando IP-HPLC-MS. O nível do tecido (μg/g) de comprimento total e ISIS 304801 e 647535 foi medido e os resultados estão apresentados na Tabela 23. Como ilustrado, as concentrações do fígado dos compostos antisense totais de comprimento completo eram semelhantes para os dois compostos antisense. Assim, mesmo que o composto antisense conjugado de GalNAc3-1 seja mais ativo no fígado (como demonstrado pelos dados do RNA e da proteína acima), ele não está presente em concentrações substancialmente elevadas no fígado. De fato, o EC50 calculado (apresentado na Tabela 23) confirma que o aumento observado na potência do composto conjugado não pode ser inteiramente atribuído ao aumento do acúmulo. Este resultado sugere que o conjugado melhorou a potência por um mecanismo diferente da acumulação no fígado sozinho, possivelmente pela melhoria da absorção produtiva do composto antisense nas células.
[001117] Os resultados também mostram que a concentração do composto antisense conjugado GalNAc3-1 no rim é menor do que o do composto antisense desprovido do conjugado de GalNAc. Há várias implicações terapêuticas benéficas. Para indicações terapêuticas em que a atividade no rim não é pedida, a exposição aos riscos de toxicidade renal, sem benefício correspondente. Além disso, a alta concentração no rim geralmente resulta na perda do compostos na urina resultando em uma depuração mais rápida Portanto, para alvos não renais, o acúmulo no rim não é desejado. Estes dados sugerem que a conjugação de GalNAc3-1 reduz o acúmulo no rim. Tabela 23Análise PK do tratamento com ASO em camundongos transgênicos
[001118] Os metabólitos de ISIS 647535 também foram identificados e suas massas foram confirmadas pela análise de espectrometria de massa de alta resolução. Os locais de clivagem e as estruturas dos metabólitos observados são mostrados abaixo. % relativa de ASO de comprimento completo foi calculado utilizando procedimentos convencionais e os resultados são apresentados na Tabela 23a. O metabólito principal de ISIS 647535 foi ASO de comprimento completo desprovido do conjugado inteiro (ou seja, ISIS 304.801), que resulta da clivagem no sítio de clivagem A, mostrado abaixo. Além disso, metabólitos adicionais resultantes de outros sítios de clivagem também foram observados. Estes resultados sugerem que a introdução de outras ligações cliváveis como ésteres, peptídeos, dissulfuretos, fosforamidatos ou acil-hidrazonas entre o açúcar GalNAc3-1 e ASO, que podem ser clivados pelas enzimas dentro da célula, ou que podem clivar no ambiente redutivo do citosol, ou que são lábeis ao pH ácido no interior dos endossomos e lisossomos também pode ser úteis.Tabela 23aMetabólitos de comprimento completo de ISIS 647535


Exemplo 21: Inibição antisense da ApoC III humana em camundongos transgênicos ApoC III humana em estudo de administração única
[001119] ISIS 304801, 647535 e 647536 cada direcionando ApoC III humana e descrito na Tabela 17, foram ainda avaliados num estudo de administração única pela sua capacidade de inibir a ApoC III humana em camundongos transgênicos ApoC III humana.
Tratamento
[001120] Camundongos transgênicos APOCIII humana foram mantidos num ciclo de luz/escuro de 12 horas e foram alimentados ad libitum Teklad lab Chow. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. ASOs foram preparados em PBS e esterilizados pela filtração através de um filtro de 0,2 mícron. ASOs foram dissolvidos em PBS 0,9% para injeção.
[001121] Camundongos transgênicos ApoC III humana foram injetados por via intraperitoneal em uma dosagem mostrada abaixo com ISIS 304801, 647535 ou 647536 ou com controle tratado com PBS. O grupo de tratamento consistiu em 3 animais e o grupo controle consistiu em 4 animais. Antes do tratamento, bem como após a última dose, o sangue foi retirado de cada camundongo e as amostras de plasma foram analisadas. Os camundongos foram sacrificados 72 horas após a última administração.
[001122] As amostras foram coletadas e analisadas para determinar o mRNA da ApoC III e os níveis proteicos no fígado; triglicerídeos plasmáticos; e colesterol, incluindo as frações HDL e LDL foram avaliadas como descrito acima (Exemplo 20). Os dados dessas análises são apresentados nas Tabelas 24-28 abaixo. Os níveis de transaminase hepática, alanina aminotransferase (ALT) e aspartato aminotransferase (AST), no soro, foram medidos em relação aos camundongos injetados com solução salina usando protocolos padrão. A níveis de ALT e AST mostrou que os compostos antisense foram bem tolerados em todas as doses administradas.
[001123] Estes resultados mostram a melhoria da potência para os compostos antisense compreendendo um conjugadoGalNAc3-1no terminal 3' (ISIS 647535 e 647536) em comparação com o composto antisense desprovido do conjugadoGalNAc3-1 (ISIS 304.801). Além disso, o ISIS 647536, que compreende um conjugado GalNAc3-1 e algumas ligações fosfodiéster eram tão potentes quanto ISIS 647535, as quais compreendem o mesmo conjugado e todas as ligações internucleosídeas no ASO são fósforotioatoTabela 24
[001124] Efeito do tratamento de ASO nos níveis de mRNA de ApoCIII em camundongos transgênicos com ApoC III humana
Tabela 25Efeito do tratamento de ASO nos níveis da proteína do plasma ApoC III em camundongos transgênicos ApoC III humana
Tabela 26Efeito do tratamento com ASO nos níveis de triglicerídeo em camundongos transgênicos
Tabela 27Efeito do tratamento com ASO nos níveis de colesterol total em camundongos transgênicos
Tabela 28Efeito do tratamento de ASO em níveis de colesterol HDL e LDL em camundongos transgênicos
[001125] Estes resultados coni firmam que o conjugadoGalNAcβ-1aumenta a potência de um composto antisense. Os resultados também mostram uma potência igual de compostos antisense conjuga- dosGalNAc3-1onde os oligonucleotídeos antisense misturaram ligações (ISIS 647536 que tem seis ligações fosfodiéster) e uma versão completa de fósforotioato versão do mesmo composto antisense (ISIS 647535).
[001126] As ligações fosforotioato proveem várias propriedades para compostos antisense. Por exemplo, elas resistem à digestão da nuclease e ligam proteínas resultando no acúmulo do composto no fígado, em vez de no rim/urina. Estas são as propriedades desejáveis, especialmente quando tratam de uma indicação no fígado. No entanto, as ligações de fósforotioato também foram associadas a uma resposta inflamatória. Por conseguinte, a redução do número de ligações fósforotioato em um composto é esperada para reduzir o risco de inflamação, mas também para diminuir a concentração do composto no fígado, aumentar a concentração na urina e rim, diminuir a estabilidade na presença de nucleases, e reduzir a potência global. Os presentes resultados mostram que um composto antisense conjugadoGalNAc3-1 onde determinadas ligações de fósforotioato foram substituídas por ligações fosfodiéster são tão potentes contra um alvo no fígado como um homólogo tendo ligações completas de fósforotioato. Espera-se que esses sejam menos pró-inflamatórios (ver Exemplo 24 que descreve uma experiência que mostra a redução de resultados PS no efeito inflamatório reduzido). Exemplo 22: Efeito do ASO modificado do conjugado GalNAc3-1 direcionando SRB-1 in vivo
[001127] ISIS 440762 e 651900, cada um direcionando SRB-1 e descritos na Tabela 17, foram avaliados num estudo dependente da dose quanto à sua capacidade de inibir a SRB-1 em camundongos Balb/c.
Tratamento
[001128] Camundongos Balb/c machos de seis semanas de idade (Laboratório Jackson, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 440762, 651900 ou com controle tratado com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 48 horas após a administração final para determinar os níveis de mRNA de SRB- 1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis de mRNA de SRB-1 foram determinados em relação ao RNA total (usando Ribogreen), antes da normalização para controle tratado com PBS. Os resultados abaixo são apresentados como a média percentual dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizado para o controle tratado com PBS e é indicado como "% PBS".
[001129] Como ilustrado na Tabela 29, ambos os compostos antisense reduziram os níveis de mRNA do SRB-1. Além disso, o composto antisense compreendendo conjugado GalNAc3-1 (ISIS 651900) era substancialmente mais potente do que o composto antisense desprovido do conjugado GalNAc3-1 (ISIS 440762). Estes resultados demonstram que o benefício da potência dos conjugdos de GalNAc3-1 é observado utilizando oligonucleotídeos antisense complementares a um alvo diferente e com nucleosídeos quimicamente diferentes, neste caso nucleosídeos modificados compreendem frações de açúcar etílico (uma fração de açúcar bicíclico).Tabela 29Efeito do tratamento com ASO sobre os níveis de mRNA de SRB-1 em camundongos Balb/c
Exemplo 23: Protocolo do Ensaio de Células Mononucleares do Sangue Periférico Humano (hPBMC)
[001130] O ensaio de hPBMC foi realizado utilizando o método do BD Vautainer CPT. Uma amostra de todo o sangue de doadores voluntários com consentimento informado na clínica US HealthWorks (Faraday & El Camino Real, Carlsbad) foi obtida e recolhidos em tubos 4-15 BD Vacutainer CPT 8 ml (VWR Cat. N° BD362753). O volume de sangue total inicial aproximado de sangue total nos tubos CPT para cada doador foi registrado utilizando a ficha de dados de ensaio PBMC.
[001131] A amostra de sangue foi misturada novamente imediatamente antes da centrifugação, invertendo suavemente os tubos de 8-10 vezes. Tubos CPT foram centrifugados à temperatura ambiente (18-25 oC) em um rotor horizontal (swing-out) por 30 min. em 1500-1800 RCF com pausa (2700 RPM Beckman Allegra 6R). As células foram recuperadas a partir da interface buffy coat (entre Ficoll e camadas de gel de polímero); transferidas para um tubo cônico estéril de 50 ml e reunidos em tubos 5 CPT/50 ml tubo/doador cônico. As células foram então lavadas duas vezes com PBS (Ca++, Mg++ livre; GIBCO). Os tubos foram enchidos até 50 ml e misturados pela inversão diversas vezes. A amostra foi então centrifugada em 330 x g durante 15 minutos à temperatura ambiente (1215 rpm, em Beckman Allegra 6R) e foram aspirados sobrenadantes tanto quanto possíveis, sem perturbar o sedimento. O sedimento celular foi desalojado por agitação suave do tubo e as células foram ressuspensas em RPMI+10% FBS+pen/strep (~1 ml/10 ml volume de sangue total inicial). Uma amostra de 60 μl foi pipetada em um frasco de amostras (Beckman Coulter) com 600 μl de reagente VersaLyse (Beckman Coulter N° A09777) e foi suavemente agitada por 10-15 seg. Foi permitido que a amostra fosse incubada por 10 min. à temperatura ambiente, sendo novamente misturada antes da contagem. A suspensão de células foi contada no analisador de viabilidade da célula Vicell XR (Beckman Coulter), utilizando o tipo de célula PBMC (fator de diluição de 1:11 foi armazenado com outros parâmetros). Foi registrada a célula viva/ml e a viabilidade. A suspensão de células foi diluída para 1 x 107 PBMC vivo/ml em RPMI+10% FBS+pen/strep.
[001132] As células foram plaqueadas a 5 x 105 em 50 uL / poço placa de cultura do tecido de 96 poços (Falcon Microtest). 50 μl/poço de 2x concentração de oligos/controles diluídos em RPMI+10% FBS+pen/strep foram adicionados conforme modelo experimental (100 μl/poço total). As placas foram colocadas no agitador e foram deixadas para misturar por aprox. 1 min. Após serem incubadas por 24 horas a 37 oC; 5% de CO2, as placas foram centrifugadas a 400 x g por 10 minutos antes da remoção do sobrenadante para o ensaio de citocina MSD (ou seja, IL-6, IL-10, IL-8 e MCP-1 humana).Exemplo 24: Avaliação dos Efeitos Pró-inflamatórios no Ensaio de hPBMC para ASOs conjugados GalNAc3-1
[001133] Os oligonucleotídeos antisense (ASOs) listados na Tabela 30 foram avaliados quanto ao efeito pró-inflamatório no ensaio de hPBMC utilizando o protocolo descrito no Exemplo 23. ISIS 353512 é um padrão interno conhecido por ser um respondedor elevado para liberação de IL-6 no ensaio. Os hPBMCs foram isolados de doadores novos e voluntários e foram tratados com ASO em concentrações de 0, 0,0128, 0,064, 0,32, 1,6, 8, 40 e 200 μM. Após um tratamento de 24 horas, os níveis de citocinas foram medidos.
[001134] Os níveis de IL-6 foram utilizados como leitura primária. A EC50 e Emáx foram calculados utilizando procedimentos padrão. Os resultados são expressos como a razão média de Emáx/EC50 de dois doadores e são denotados como "Emáx/EC50." A razão mais baixa indica uma diminuição relativa na resposta pró-inflamatória e a razão mais elevada indica um aumento relativo na resposta pró-inflamatória.
[001135] No que diz respeito aos compostos de teste, o composto menos pró-inflamatório foi o ASO ligado ao PS/PO (ISIS 616468). O ASO conjugado GalNAc3-1 ISIS 647535 era ligeiramente menos pró- inflamatório do que sua contraparte não-conjugada ISIS 304801. Estes resultados indicam que a incorporação de algumas ligações PO reduz a reação pró-inflamatória e adição de um conjugadoGalNAc3-1 não torna um composto mais pró-inflamatório e pode reduzir a resposta pró- inflamatória. Por conseguinte, seria de esperar que um composto antisense compreendendo ligações PS/PO mistas e um conjugadoGalNAc3-1 produziria respostas pró-inflamatórias mais baixas em relação ao composto antisense ligado ao PS completo, com ou sem um conjugadoGalNAc3-1. Estes resultados demonstram que compotos antisense conjugadosGalNAc3-1 , particularmente aqueles conjugados com teor reduzido de PS são menos pró-inflamatórios.
[001136] Juntos, estes resultados sugerem que um composto conjugadoGalNAc3-1, particularmente um com teor reduzido de PS, pode ser administrado a uma dose mais elevada do que um composto antisense do PS completo da contraparte desprovido do conjugado GalNAc3-1. Uma vez que não se espera que a meia-vida seja substancialmente diferente para estes compostos, essa administração resultaria em uma dosagem menos frequente. Na verdade essa administração poderia ser ainda menos frequente, porque os compostos conjgados GalNAc3-1 são mais potentes (Ver Exemplos 20-22) e a nova dosagem é necessária uma vez que a concentração de um composto ficou abaixo de um nível desejado, onde esse nível desejado se baseia na potência.Tabela 30ASOs Modificados
[001137] Subscritos: "e" indicam nucleosídeo 2'-M OE modif ficado; "d"indica β-D-2'-desoximbonucleosideo; "k" indica 6' (S) CH3 nucleosídeo bicíclico (por exemplo, cEt); "s" indica ligações internucleosídeas de fosforotioato (PS); "o" indica ligações internucleosídeas de fosfodiéster (PO); e "o" indica -O-P (=O)(OH)-. O subscrito m" indica 5-metilcitosinas. "Ado'-GalNAc3-1a" indica um conjugado com a estrutura GalNAc3-1 mostrada no Exemplo 9 anexado à extremidade 3'do oligonucleotídoe antisense, como indicado.Tabela 31Efeito de Pró-inflamatório dos ASOs direcionando ApoC III no ensaio de hPBMC
Exemplo 25: Efeito do ASO modil Ficado do conjugado GalNAc3-1direcionando ApoC III humana in vitro
[001138] ISIS 304801 e 647535 descritos acima foram testados in vitro. As células de hepatócitos primários de camundongos transgênicos em uma densidade de 25.000 células por poço, foram tratados com concentrações de 0,03,0,08, 0,24, 0,74, 2,22, 6,67 e 20 μM deoligonucleotídeos modificados. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis de mRNA foram medidos por PCR em tempo real e os níveis de mRNA de hApoC III foram ajustados de acordo com o conteúdo total de RNA total, conforme medido por RIBOGREEN®.
[001139] A IC50 foi calculada usando métodos padrão e os resultados são apresentados na Tabela 32. Como ilustrado, uma potência comparável foi observada em células tratadas com ISIS 647535 em comparação com o controle, ISIS 304801.Tabela 32ASO Modificado que possui como alvo ApoC III humano em hepatócitos primários
[001140] Neste experimento, os grandes benefícios de potência da conjugação GalNAc3-1 que são observados in vivo não foram observados in vitro. Experimentos de absorção livres subsequentes em hepatócitos primários in vitro mostraram aumento da potência de oligonucletoídeos compreendendo vários conjugados GalNAc relativos aos oligonucleotídeos desprovidos do conjugado GalNAc. (ver Exemplos 60, 82 e 92)Exemplo 26: Efeito das ligações PO/PS na Atividade do ASO da ApoC III
[001141] Camundongos transgênicos ApoC III humana foram injetados por via intraperitoneal uma vez em 25 mg/kg de ISIS 304801 ou 616468 (descritos acima) ou com controle tratado com PBS uma vez por semana por duas semanas. O grupo de tratamento consistiu em 3 animais e o grupo controle consistiu em 4 animais. Antes do tratamento, bem como após a última dose, o sangue foi retirado de cada camundongo e as amostras de plasma foram analisadas. Os camundongos foram sacrificados 72 horas após a última administração.
[001142] As amostras foram coletadas e analisadas para determinar os níveis da proteína ApoC III no fígado descritos acima (Exemplo 20). Os dados dessas análises são apresentados na Tabela 33 abaixo.
[001143] Estes resultados mostram redução na potência de compostos antisense com PO/PS (ISIS 616468) nas asas relativas ao PS completo (ISIS 304801).Tabela 33Efeito do tratamento com ASO nos níveis da proteína ApoC III em camundongos transgênicos com ApoC III humana
[001144] Composto 56 está comercialmente disponível de GlenResearch, ou pode ser preparados de acordo com procedimentos publicados relatados por Shchepinov et al., Nucleic Acids Research, 1997, 25(22), 4447-4454.Exemplo 28: Preparação do Composto 60
[001145] O composto 4 foi preparado de acordo com os procedimentos ilustrados no Exemplo 2. O composto 57 está comercialmente disponível. Composto 60 foi confirmado por análise estrutural.
[001146] Composto 57 destina-se a ser representativo e não destina- se a ser limitante como outro monoalquil diol protegido substituído ou não substituído, incluindo, entre outros, aqueles apresentados na especificação aqui contida podem ser utilizados para preparar fosforamiditas com uma composição predeterminada. Exemplo 29: Preparação do Composto 63
[001147] Os compostos 61 e 62 são preparados utilizandoprocedimentos semelhantes aos relatados por Tober et al., Eur. J. Org.Chem., 2013, 3, 566-577; e Jiang et al., Tetrahedron, 2007, 63(19),3982-3988.
[001148] Alternativamente, o Composto 63 é preparado utilizando procedimentos semelhantes aos relatados na literatura científica e de patentes por Kim et al., Synlett, 2003, 12, 1838-1840; e Kim et al.,Pedido Internacional PCT publicado, WO 2004063208.Exemplo 30: Preparação do Composto 63b
[001149] Composto 63a é preparado utilizando procedimentos semelhantes aos relatados por Hanessian et al., Canadian Journal ofChemistry, 1996, 74 (9), 1731-1737.Exemplo 31: Preparação de Composto 63d
[001150] Composto 63c é preparado utilizando procedimentos semelhantes aos relatados por Chen et al., Chinese Chemical Letters, 1998, 9 (5), 451-453. Exemplo 32: Preparação do Composto 67
[001151] O composto 64 foi preparado de acordo com os procedimentos ilustrados no Exemplo 2. Composto 65 é preparado utilizando procedimentos semelhantes aos relatados por Or et al., Pedido Internacional PCT publicado, WO 2009/003009. Os grupos protetores utilizados para o Composto 65 são destinados a serem representativos e não pretendem ser limitantes como outros grupos protetores, incluindo, entre outros, aqueles apresentados na especificação aqui contida podem ser utilizados.Exemplo 33: Preparação do Composto 70
[001152] O composto 64 foi preparado de acordo com os procedimentos ilustrados no Exemplo 2. O composto 68 está comercialmente disponível. Os grupos protetores utilizados para o Composto 68 são destinados a serem representativos e não pretendem ser limitantes como outros grupos protetores, incluindo, entre outros, aqueles apresentados na especificação aqui contida podem ser utilizados.Exemplo 34: Preparação do Composto 75a
[001153] Composto 75 é preparado de acordo com os procedimentos publicados relatados por Shchepinov et al., Nucleic Acids Research, 1997, 25 (22), 4447-4454.Exemplo 35: Preparação do Composto 79
[001154] Composto 76 foi preparado de acordo com os procedimentos publicados relatados por Shchepinov et al., Nucleic Acids Research, 1997, 25(22), 4447-4454. Exemplo 36: Preparação do Composto 79a
[001155] O composto 77 é preparado de acordo com os procedimentos ilustrados no Exemplo 35.Exemplo 37: Método geral para a preparação do composto oligomérico conjugado 82 compreendendo um conjugado GalNAc3-2 ligado ao fosfodiéster no terminal 5 ' via suporte sólido (Método I)
[001156] onde GalNAc3-2 tem a estrutura:
[001157] A parte do agrupamento de GalNAc3 do grupo conjugado GalNAc3-2 (GalNAc3-2a) pode ser combinada com qualquer fração clivável para prover uma variedade de grupos conjugados. Onde GalNAc3-2a tem a fórmula:
[001158] Composto oligomérico ligado ao VIMAD 79B foi preparado utilizando procedimentos padrão para síntese de DNA/RNA automatizado (ver Dupouy et al., Angew. Chem. Int. Ed., 2006, 45, 36233627). Os compostos de fosforamidita 56 e 60 foram preparados de acordo com os procedimentos ilustrados nos Exemplos 27 e 28, respectivamente. As fosforamiditas ilustradas pretendem ser representativas e não pretendem ser limitantes como outros blocos de construção de fosforamiditas, incluindo, entre outros, aquelas apresentadas na especificação aqui contida podem ser usadas para preparar compostos oligoméricos com um grupo conjugado ligado do fosfodiéster no terminal 5'. A ordem e quantidade de fosforamiditas adicionadas ao suporte sólido podem ser ajustadas para preparar compostos oligoméricos descritos neste instrumento com qualquer sequência ou composição predeterminada.Exemplo 38: Método alternativo para a preparação do composto oligomérico conjugado 82 compreendendo um conjugado GalNAc3-2 ligado ao fosfodiéster no terminal 5' (Método II)

[001159] Composto oligomérico ligado ao VIMAD 79B foi preparado utilizando procedimentos padrão para síntese de DNA/RNA automatizado (ver Dupouy et al., Angew. Chem. Int. Ed., 2006, 45, 3623-3627). Fosforamidita do agrupamento GalNAc3-2 , Composto 79 foi preparado de acordo com os procedimentos ilustrados no Exemplo 35. Este método alternativo permite uma instalação de uma etapa do do conjugado GalNAc3-2 ligado ao fosfodiéster para o composto oligomérico na etapa final da síntese. As fosforamiditas ilustradas pretendem ser representativas e não pretendem ser limitantes como outros blocos de construção de fosforamiditas, incluindo, entre outros, aquelas apresentadas na especificação aqui contida podem ser usadas para preparar compostos oligoméricos com um grupo conjugado ligado do fosfodiéster no terminal 5'. A ordem e quantidade de fosforamiditas adicionadas ao suporte sólido podem ser ajustadas para preparar compostos oligoméricos descritos neste instrumento com qualquer sequência ou composição predeterminada.Exemplo 39: Método geral para a preparação de composto oligomérico de 83h compreendendo Conjugado GalNAc3-3 no terminal 5' (GalNAc3-1 modificado para anexação da extremidade 5') via Suporte Sólido


[001160] O composto 18 foi preparado de acordo com os procedimentos ilustrados no Exemplo 4. Os compostos 83a e 83b estão disponíveis comercialmente. O composto oligomérico 83e, compreendendo uma hexilamina ligada ao fosfodiéster foi preparada usando procedimentos de síntese de oligonucleotídeo padrão. O tratamento do composto oligomérico protegido com amônia aquosa forneceu composto oligomérico conjugado 5'-GalNAc3-3 (83h).
[001161] Onde GalNAc3-3 tem a estrutura:
[001162] A parte do agrupamento de GalNAc3 do grupo conjugado GalNAc3-3 (GalNAc3-3a) pode ser combinada com qualquer fração clivável para prover uma variedade de grupos conjugados. Onde GalNAc3-3a tem a fórmula:
Exemplo 40: Método alternativo para a preparação do composto oligomérico conjugado 89 compreendendo um conjugado GalNAc3-4 ligado ao fosfodiéster no terminal 3' via suporte sólido
[001163] Onde GalNAc3-4 tem a estrutura:
[001164] Onde CM é uma fração clivável. Em determinadas modalidades, a fração clivável é:
[001165] A parte do agrupamento de GalNAc3 do grupo conjugado GalNAc3-4 (GalNAc3-4a) pode ser combinada com qualquer fração clivável para prover uma variedade de grupos conjugados. OndeGalNAc3-4a tem a fórmula:
[001166] O Composto 30 do suporte sólido funcionalizado do Unylinker 30 protegido está comercialmente disponível. Composto 84 é preparado utilizando procedimentos semelhantes aos relatados na literatura (ver Shchepinov et al., Nucleic Acids Research, 1997, 25 (22), 4447-4454; Shchepinov et al., Nucleic Acids Research, 1999, 27, 30353041; e Hornet et al., Nucleic Acids Research, 1997, 25, 4842-4849).
[001167] Os blocos de construção de fosforamidita, Compostos 60 e 79a são preparados de acordo com os procedimentos ilustrados nos Exemplos 28 e 36. As fosforamiditas ilustradas pretendem ser representativas e não pretendem ser limitantes como outros blocos de construção de fosforamiditas podem ser utilizados para preparar compostos oligoméricos com um conjugado ligado ao fosfodiéster no terminal 3' com uma sequência e composição predeterminadas. A ordem e quantidade de fosforamiditas adicionadas ao suporte sólido podem ser ajustadas para preparar compostos oligoméricos descritos neste instrumento com qualquer sequência ou composição predeterminada. Exemplo 41: Método geral para a preparação de ASOs compreendendo um conjugado GalNAc3-2 ligado ao fosfodiéster (Ver Exemplo 37, Bx é adenina) na posição 5' via técnicas de fase sólida (preparação do ISIS 661134)
[001168] Salvo indicação em contrário, todos os reagentes e soluções utilizadas para a síntese dos compostos oligoméricos são adquiridas de fontes comerciais. Blocos de construção de fosforamidita padrão e suporte sólido são usados para a incorporação de resíduos de nucleosídeos que incluem, por exemplo, T, A, G, e mResíduos C. Compostos de fosforamidita 56 e 60 foram utilizados para sintetizar o conjugado GalNAc3-2 ligado ao fosfodiéster no terminal 5'. Uma solução de fosforamidita a 0,1 M em anidro acetonitrila foi utilizada para β -D-2'- desoxinbonucleosídeo e 2'-MOE.
[001169] As sínteses ASO foram realizadas em sintetizador ABI 394 (escala de 1-2 μmol) ou em sintetizador oligopiloto GE Healthcare Bioscience ÃKTA (escala de 40-200 μmol) pelo método de acoplamento de fosforamidita em suporte sólido VIMAD (110 μmol/g, Guzaev et al., 2003) empacotada na coluna. Para a etapa de acoplamento, as fosforamiditas foram entregues 4 vezes em excesso sobre a carga sobre a carga inicial do suporte sólido e acoplamento da fosforamidita foi realizada por 10 min. Todas as outras etapas seguiram os protocolos padrão fornecidos pelo fabricante. Uma solução de ácido dicloroacético a 6% em tolueno foi utilizada para remover o grupo de dimetoxitritila (DMT) dos grupos 5'-hidroxila do nucleotídeo. 4,5-Dicianoimidazol (0,7 M) em anidro CH3CN foi usado como ativador durante a etapa de acoplamento. As ligações de fosforotioato foram introduzidas por sulfurização com solução a 0,1 M de hidreto de xantano em piridina/CH3CN 1:1 para um tempo de contato de 3 minutos. Uma solução de 20% terc-butilhidroperóxido em CH3CN contendo 6% de água foi utilizada como um agente oxidante para prover ligações internucleosídeas de fosfodiéster com um tempo de contato de 12 minutos.
[001170] Depois da sequência desejada ter sido montada, os grupos protetores de cianoetil fosfato foram desprotegidos usando uma dietilamina a 20% em tolueno (v/v) com um tempo de contato de 45 minutos. ASOs de ligação de suporte sólido foram suspensos em amônia aquosa (28-30% em peso) e aquecidos a a 55 oC por 6 h.
[001171] Os ASO não ligados foram então filtrada e a amônia foi fervida. O resíduo foi purificado por cromatografia líquida de alta pressão numa coluna de troca aniônica forte (GE Healthcare Bioscience, Fonte 30Q, 30 μm, com 2,54 x 8 cm, A = 100 mM acetatode amônia em 30% de CH 3CN aquoso, B = 1,5 M de NaBr, em A, 040% de B em 60 min, fluxo 14 mL min-1, À = 260 nm). O resíduo foidessalinizado por HPLC numa coluna de fase inversa para produzirASOs desejados com um rendimento isolado de 15-30% com base na carga inicial sobre o suporte sólido. ASOs foram caracterizados pela análise MS acoplado íon-par-HPLC com sistema Agilent 1100 MSD.Tabela 34ASO compreendendo um conjugado GalNAc3-2 ligado ao fosfodiéster na posição 5' direcionando SRB-1
[001172] Subscritos: "e" indicam nucleosídeo 2'-MOE modificado; "d" indica β-D-2'-desoximbonucleosideo; "k" indica 6' (S) CH3 nucleosídeo bicíclico (por exemplo, cEt); "s" indica ligações internucleosídeas de fosforotioato (PS); "o" indica ligações internucleosídeas de fosfodiéster (PO); e "o" indica -O-P (=O)(OH)-. O subscrito "m" indica 5- metilcitosinas. A estrutura de GalNAc3-2a é apresentada no Exemplo 37. Exemplo 42: Método geral para a preparação de ASOcompreendendo conjugado GalNAc3-3 na posição 5' via técnicas em fase sólida (preparação do ISIS 661166)
[001173] A síntese para ISIS 661.166 foi realizada usando procedimentos semelhantes ilustrados nos Exemplos 39 e 41.
[001174] ISIS 661166 é um gapmer 5-10-5 MOE, em que a posição 5'compreende um conjugado GalNAc3-3. ASO foi caracterizado pela análise MS acoplado íon-par-HPLC com sistema Agilent 1100 MSD.Tabela 34aASO compreendendo um conjugado GalNAc3-3 na posição 5' via ligação de fosfodiéster hexilamina direcionando Malat-1
[001175] Subscritos: "e" indica o nucleosídeo 2'-MOE modificado; "d" indica o β-D-2'-desoximbonucleosideo; "s" indica as ligações internucleosídeos de fosforotioato (PS); "o" indica as ligaçõesinternucleosídeos de fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. O subscrito "m" indica 5-metilcitosinas. A estrutura de "5'-GalNAc3-3a " é mostrada no Exemplo 39.Exemplo 43: Estudo dependente de dose de GalNAc3-2 ligado a fosfodiéster (ver os exemplos 37 e 41, Bx é adenina) no terminal 5' direcionado ao SRB-1 in vivo
[001176] ISIS 661134 (ver o Exemplo 41) que compreende um conjugado GalNAc3-2 ligado a fosfodiéster no terminal 5' fio testado em um estudo dependente de dose para a inbição antisense de SRB-1 em camundongos. O ISIS 440762 e 651900 não conjugados (GalNAc3-1 conjugado no terminal 3', ver o Exemplo 9) foram incluídos no estudo para comparação e são descritos anteriormente na Tabela 17.
Tratamento
[001177] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 440762, 651900, 661134 ou com controle tratado com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis de mRNA de SRB-1 foram determinados em relação ao RNA total (usando Ribogreen), antes da normalização para controle tratado com PBS. Os resultados abaixo são apresentados como a média percentual dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizado para o controle tratado com PBS e é indicado como "% PBS". Os ED50s foram medidos usando métodos semelhantes, conforme descrito anteriormente e são apresentados a seguir.
[001178] Conforme ilustrado na Tabela 35, o tratamento com oligonucleotídeos antisense reduziu os níveis de mRNA de SRB-1 de uma forma dependente da dose. De fato, os oligonucleotídeos antisense que compreendem o conjugado de GalNAc3-2 ligado ao fosfodiéster no terminal 5' (ISIS 661134) ou o conjugado de GalNAc3-1 ligado no teminal 3' (ISIS 651900) mostraram melhora substancial de potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 440762). Além disso, ISIS 661134, que compreende o conjugado GalNAc3-2 ligado ao fosfodiéster no terminal 5' foi equipotente em comparação ao ISIS 651900, que compreende o conjugado de GalNAc3-1 no terminal 3'. Tabela 35ASOs contendo GalNAc3-1 ou GalNAc3-2 direcionado ao SRB-1
[001179] As estruturas para o 3' GalNAc3-1 e 5' GalNAc3-2 foram descritas anteriormente nos Exemplos 9 e 37.Análise farmacocinética (PK)
[001180] A PK dos ASOs do grupo de dose alta (7 mg/kg) foi examinada e avaliada da mesma forma como ilustrado no Exemplo 20. A amostra de fígado foi cortada e extraída usando protocolos padrão. Os metabólitos de comprimento total de 661134 (5' GalNAc3-2) e ISIS 651900 (3' GalNAc3-1) foram identificados e suas massas foramconfirmadas por análise de espectrometria de massa de alta resolução. Os resultados mostraram que o maior metabólito detectado para o ASO compreendendo um conjugado de GalNAc3-2 ligado ao fosfodiéster no terminal 5' (ISIS 661134) foi ISIS 440762 (dados não mostrados). Nenhum metabólito adicional, num nível detectável, foi observado. Ao contrário de sua contraparte, os metabólitos adicionais semelhantes àqueles relatados anteriormente na Tabela 23a foram observados para o ASO tendo o conjugado de GalNAc3-1 no terminal 3' (ISIS 651900). Esses resultados sugerem que ter o conjugado de GalNAc3-1 ou GalNAc3-2 pode melhorar o perfil da PK dos ASOs sem comprometer sua potência.Exemplo 44: Efeito de ligações de PO/PS na inibição antisense de ASOs compreendendo o conjugado de GalNAc3-1 (ver o Exemplo 9) no terminal 3' direcionado ao SRB-1
[001181] ISIS 655861 e 655862 compreendendo um conjugado de GalNAc3-1 no terminal 3', cada um se direcionando ao SRB-1, foram testados num estudo de administração única para sua capacidade em inibir o SRB-1 em camundongos. O composto não conjugado de origem, ISIS 353382 foi incluído no estudo para comparação.
[001182] Os ASOs são gapmeros 5-10-5 MOE, em que a região de lacuna compreende dez 2'-desoxirribonucleosídeos e cada região de asa compreende cinco nucleosídeos 2'-MOE modificados. Os ASOs foram preparados usando métodos semelhantes conforme ilustrado anteriormente no Exemplo 19 e são descritos na Tabela 36 abaixo.Tabela 36Os ASOs modificados compreendendo o conjugado de GalNAc3-1 no terminal 3' se direcionando ao SRB-1
[001183] Subscritos: "e" indica o nucleosídeo 2'-MOE modificado; "d" indica o β-D-2'-desoximbonucleosideo; "s" indica as ligações internucleosídeos de fosforotioato (PS); "o" indica as ligaçõesinternucleosídeos de fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. O sobrescrito "m" indica 5-metilcitosinas. A estrutura de "GalNAc3-1" é mostrada no Exemplo 9.
Tratamento
[001184] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 353382, 655861, 655862 ou com controle tratado com PBS. Cada grupo de tratamento consistiu em 4 animais. Antes do tratamento, bem como após a última dose, o sangue foi retirado de cada camundongo e as amostras de plasma foram analisadas. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis de mRNA de SRB-1 foram determinados em relação ao RNA total (usando Ribogreen), antes da normalização para controle tratado com PBS. Os resultados abaixo são apresentados como a média percentual dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizado para o controle tratado com PBS e é indicado como "% PBS". Os ED50s foram medidos usando métodos semelhantes, conforme descrito anteriormente e são apresentados a seguir.
[001185] Conforme ilustrado na Tabela 37, o tratamento com oligonucleotídeos antisense reduziu os níveis de mRNA de SRB-1 de uma forma dependente de dose em comparação ao controle tratado com PBS. De fato, os oligonucleotídeos antisense que compreendem o conjugado de GalNAc3-1 no terminal 3' (ISIS 655861 e 655862) mostraram melhora substancial de potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 353382). Além disso, o ISIS 655862 com ligações de PS/PO mistas mostrou uma melhora na potência em relação ao PS completo (ISIS 655861).Tabela 37Efeito de ligações de PO/PS na inibição antisense de ASOs compreendendo o conjugado de GalNAc3-1 no terminal 3' se direcionando ao SRB-1
[001186] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Os pesos dos órgãos também foram avaliados. Os resultados demonstraram que não foi observada nenhuma elevação nos níveis de transaminase (Tabela 38) ou pesos dos órgãos (dados não mostrados) em camundongos tratados com ASOs em comparação ao controle com PBS. Além disso, o ASO com ligações de PS/PO mistas (ISIS 655862) mostraram níveis de transaminase semelhantes em comparação ao P completo (ISIS 655861).Tabela 38Efeito de ligações de PO/PS sobre os níveis de transaminase de ASOs compreendendo o conjugado de GalNAc3-1 no terminal 3' se direcionando ao SRB-1
[001187] O composto 4 (9,5 g, 28,8 mmoles) foi tratado com o composto 103a ou 103b (38 mmoles), individualmente, e TMSOTf (0,5 eq.) e peneiras moleculares em diclorometano (200 mL), e agitado por 16 horas à temperatura ambiente. Nesse tempo, a camada orgânicas foi filtrada através de celite, em seguida lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida sob pressão reduzida. O óleo resultando foi purificado por cromatografia em gel de sílica (2%-->10% metanol/diclorometano) para gerar os compostos 104a e 104b em >80% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001188] Os compostos 104a e 104b foram tratados nas mesmas condições como para os compostos 100a-d (Exemplo 47), para gerar os compostos 105a e 105b em >90% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001189] Os compostos 105a e 105b foram tratados, individualmente, com o composto 90 sob as mesmas condições que os compostos 901a- d, para gerar os compostos 106a (80%) e 106b (20%). LCMS e NMR de prótons foram consistentes com a estrutura.
[001190] Os compostos 106a e 106b foram tratados nas mesmas condições que os compostos 96a-d (Exemplo 47), para gerar o 107a (60%) e 107b (20%). LCMS e NMR de prótons foram consistentes com a estrutura.
[001191] Os compostos 107a e 107b foram tratados nas mesmas condições que os compostos 97a-d (Exemplo 47), para gerar os compostos 108a e 108b em 40-60% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001192] Os compostos 108a (60%) e 108b (40%) foram tratados nas mesmas condições para os compostos 100a-d (Exemplo 47), para gerar os compostos 109a e 109b em >80% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001193] O composto 109a foi tratado nas mesmas condições que os compostos 101a-d (Exemplo 47), para gerar o Composto 110a em 3060% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura. Alternativamente, o Composto 110b pode ser preparado de forma semelhante, começando com o Composto 109b.Exemplo 46: Procedimento Geral para Conjugação com Ésteres de PFP (Oligonucleotídeo 111); Preparação de ISIS 666881 (GalNAc3- 10)
[001194] Um oligonucleotídeo modificado 5'-hexilamino foi sintetizado e purificado usando procedimentos de oligonucleotídeo de fase sólida padrão. O oligonucleotídeo modificado 5'-hexilamino foi dissolvido em tetraborato de sódio 0,1 M, pH 8,5 (200 μL) e 3 equivalentes de um cluster de GalNAc3 esterificado com PFP selecionado dissolvido em DMSO (50 μL) foi adicionado. Se o éster de PFP precipitou após a adição na solução de ASO, DMSO foi adicionado até que todo o éster de PFP estivesse na solução. A reação foi concluída após cerca de 16 h de mistura à temperatura ambiente. A solução resultante foi diluída com água a 12 mL e, em seguida, rotacionada em 3000 rpm em um filtro de rotação com um limite de massa de 3000 Da. Este processo foi repetido duas vezes para remover as impurezas de pequenas moléculas. A solução foi então liofilizada à secura e redissolvida em amônia aquosa concentrada e misturada à temperatura ambiente por 2,5 h seguido por concentração in vacuo para remover a maior parte da amônia. O oligonucleotídeo conjugado foi purificado e dessalinizado por RP-HPLC e liofilizado para fornecer o oligonucleotídeo conjugado a GalNAc3.

[001195] O oligonucleotídeo 111 é conjugado com o GalNAc3-10. A parte de cluster de GalNAc3 do GalNAc3-10 de grupo do conjugado (GalNAc3-10a) pode ser combinado com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)- como mostrado no oligonucleotídeo (ISIS 666881) sintetizado com o GalNAc3- 10 abaixo. A estrutura de GalNAc3-10 (GalNAc3-10a-CM-) é mostrada abaixo:
[001196] Após este procedimento geral, o ISIS 666881 foi preparado. O oligonucleotídeo modificado 5'-hexilamino, ISIS 660254, foi sintetizado e purificado usando procedimentos de oligonucleotídeo de fase sólida padrão. O ISIS 660254 (40 mg, 5,2 μmol) foi dissolvido em tetraborato de sódio 0,1 M, pH 8,5 (200 μL) e 3 equivalentes, foi adicionado éster de PFP (Composto 110a) dissolvido em DMSO (50 μL). O éster de PFP precipitou após a adição na solução de ASO, exigindo DMSO adicional (600 μL) para dissolver completamente o éster de PFP. A reação foi concluída após cerca de 16 h de mistura à temperatura ambiente. A solução resultante foi diluída com água a 12 mL de volume total e rotacionada em 3000 rpm em um filtro de rotação com um limite de massa de 3000 Da. Este processo foi repetido duas vezes para remover as impurezas de pequenas moléculas. A solução foi liofilizada à secura e redissolvido em amônia aquosa concentrada com mistura à temperatura ambiente por 2,5 h seguido por concentração in vacuo para remover a maior parte da amônia. O oligonucleotídeo conjugado foi purificado e dessalinizado por RP-HPLC e liofilizado para gerar o ISIS 666881 em 90% de rendimento em peso (42 mg, 4,7 μmol).oligonucleotídeo conjugado a GalNAc3-10

[001197] Letras maiúsculas indicam a nucleo base para cadanucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2-desoximbo- nucleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O- P(=O)(OH)-. Os grupos de conjugado estão em negrito. Exemplo 47: Preparação do Oligonucleotídeo 102 CompreendendoGalNAc3-8
[001198] O triácido 90 (4 g, 14,43 mmol) foi dissolvido em DMF (120 mL) e N,N-Di-isopropiletilamina (12,35 mL, 72 mmoles).Trifluoroacetato de pentafluorofenil (8,9 mL, 52 mmoles) foi adicionado em gotas, sob argônio, e a reação foi deixada agitar à temperatura ambiente por 30 minutos. Boc-diamina 91a ou 91b (68,87 mmol) foi adicionada, juntamente com N,N-Di-isopropiletilamina (12,35 mL, 72 mmoles), e a reação foi deixada agitar à temperatura ambiente por 16 horas. Nesse tempo, o DMF foi reduzido em >75% sob pressão reduzida e, em seguida, a mistura foi dissolvida em diclorometano. A camada orgânica foi lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida a um óleo sob pressão reduzida. O óleo resultante foi purificado por cromatografia em gel de sílica (2%-->10% metanol/diclorometano) para gerar os compostos 92a e 92b em um rendimento de aproximadamente 80%. LCMS e NMR de prótons foram consistentes com a estrutura.
[001199] O composto 92a ou 92b (6,7 mmoles) foi tratado com 20 mL de diclorometano e 20 mL de ácido trifluoroacético à temperatura ambiente por 16 horas. A solução resultante foi evaporada e, em seguida, dissolvida em metanol e tratada com resina DOWEX-OH por 30 minutos. A solução resultante foi filtrada e reduzida a um óleo sob pressão reduzida para gerar 85-90% de rendimento dos compostos 93a e 93b.
[001200] O composto 7 ou 64 (9,6 mmoles) foram tratados com HBTU (3,7 g, 9,6 mmoles) e N,N-Di-isopropiletilamina (5 mL) em DMF (20 mL) por 15 minutos. A isto foi adicionado qualquer um do composto 93a ou 93b (3 mmoles), e deixado agitar à temperatura ambiente por 16 horas. Nesse tempo, o DMF foi reduzido em >75% sob pressão reduzida e, em seguida, a mistura foi dissolvida em diclorometano. A camada orgânica foi lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida a um óleo sob pressão reduzida. O óleo resultante foi purificado por cromatografia em gel de sílica (5%-->20% metanol/diclorometano) para gerar os compostos 96a-d em 20-40% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001201] Os compostos 96a-d (0,75 mmoles), individualmente,foram hidrogenados com Níquel de Raney por 3 horam em Etanol (75 mL). Nesse tempo, o catalisador foi removido por filtração através de celite, e o etanol removido sob pressão reduzida para gerar os compostos 97a-d em 80-90% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001202] O composto 23 (0,32 g, 0,53 mmoles) foi tratado com HBTU (0,2 g, 0,53 mmoles) e N,N-Di-isopropiletilamina (0,19 mL, 1,14 mmoles) em DMF (30mL) por 15 minutos. A isto foram adicionados os compostos 97a-d (0,38 mmoles), individualmente, e deixadosagitar à temperatura ambiente por 16 horas. Nesse tempo, o DMF foi reduzido em >75% sob pressão reduzida e, em seguida, a mistura foi dissolvida em diclorometano. A camada orgânica foi lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida a um óleo sob pressão reduzida. O óleo resultante foi purificado por cromatografia em gel de sílica (2%-->20% metanol/diclorometano) para gerar os compostos 98a-d em 30-40% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001203] O composto 99 (0,17 g, 0,76 mmoles) foi tratado com HBTU (0,29 g, 0,76 mmoles) e N,N-Di-isopropiletilamina (0,35 mL, 2,0 mmoles) em DMF (50mL) por 15 minutos. A isto foram adicionados os compostos 97a-d (0,51 mmoles), individualmente, e deixadosagitar à temperatura ambiente por 16 horas. Nesse tempo, o DMF foi reduzido em >75% sob pressão reduzida e, em seguida, a mistura foi dissolvida em diclorometano. A camada orgânica foi lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida a um óleo sob pressão reduzida. O óleo resultante foi purificado por cromatografia em gel de sílica (5%-->20% metanol/diclorometano) para gerar os compostos 100a-d em 40-60% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001204] Os compostos 100a-d (0,16 mmoles), individualmente,foram hidrogenados com 10% Pd(OH)2/C por 3 horas em metanol/acetato de etil (1:1, 50 mL). Nesse tempo, o catalisador foi removido por filtração através de celite, e os compostos orgânicos removidos sob pressão reduzida para gerar os compostos 101a-d em 80-90% de rendimento. LCMS e NMR de prótons foram consistentes com a estrutura.
[001205] Os compostos 101a-d (0,15 mmoles), individualmente,foram dissolvidos em DMF (15 mL) e piridina (0,016 mL, 0,2 mmoles). Trifluoroacetato de pentafluorofenil (0,034 mL, 0,2 mmoles) foi adicionado em gotas, sob argônio, e a reação foi deixada agitar à temperatura ambiente por 30 minutos. Nesse tempo, o DMF foi reduzido em >75% sob pressão reduzida e, em seguida, a mistura foi dissolvida em diclorometano. A camada orgânica foi lavada com bicarbonato de sódio, água e salmoura. A camada orgânica foi então separada e seca com sulfato de sódio, filtrada e reduzida a um óleo sob pressão reduzida. O óleo resultante foi purificado por cromatografia em gel de sílica (2%-->5% metanol/diclorometano) para gerar os compostos 102a-d num rendimento aproximado de 80%. LCMS e NMR de prótons foram consistentes com a estrutura.
[001206] O composto oligomérico 102, compreendendo um grupo de conjugado de GalNAc3-8, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo do conjugado de GalNAc3-8 (GalNAc3-8a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Numa modalidade preferencial, a fração clivável é - P(=O)(OH)-Ad-P(=O)(OH)-.
[001207] A estrutura de GalNAc3-8 (GalNAc3-8a-CM-) é mostrada abaixo:
Exemplo 48: Preparação do Oligonucleotídeo 119 Compreendendo
[001208] O composto 112 foi sintetizado após o procedimento descrito na literatura (J. Med. Chem. 2004, 47, 5798-5808).
[001209] O composto 112 (5 g, 8,6 mmol) foi dissolvido em 1:1 de metanol/acetato de etil (22 mL/22 mL). Foi adicionado hidróxido de paládio em carbono (0,5 g). A mistura de reação foi agitada àtemperatura ambiente sob hidrogênio por 12 h. A mistura de reação foi filtrada através de uma camada de celite e lavada a camada com 1:1 de metanol/acetato de etil. O filtrado e as lavagens foram combinadas e concentradas à secura para produzir o Compostos 105a (quantitativo). A estrutura foi confirmada por LCMS.
[001210] O composto 113 (1,25 g, 2,7 mmol), HBTU (3,2 g, 8,4 mmol) e DIEA (2,8 mL, 16,2 mmol) foram dissolvidos em DMF (17 mL) e a mistura de reação foi agitada à temperatura ambiente por 5 min. A isto, uma solução do Composto 105a (3,77 g, 8,4 mmol) em DMF anidro (20 mL) foi adicionada. A reação foi agitada à temperatura ambiente por 6 h. O solvente foi removido sob pressão reduzida para obter um óleo. O resíduo foi dissolvido em CH2Cl2 (100 mL) e lavado com solução de NaHCO aquoso saturada3 (100 mL) e salmoura (100 mL). A fase orgânica foi separada, seca (Na2SO4), filtrada e evaporada. O resíduo foi purificado por cromatografia de coluna em gel de sílica e eluído com MeOH 10 a 20 % em diclorometano para produzir o Composto 114 (1,45 g, 30%). A estrutura foi confirmada por LCMS e análise 1H NMR.
[001211] O composto 114 (1,43 g, 0,8 mmol) foi dissolvido em 1:1 de metanol/acetato de etil (4 mL/4 mL). Foi adicionado paládio em carbono (úmido, 0,14 g). A mistura de reação foi carregada com hidrogênio e agitada à temperatura ambiente sob hidrogênio por 12 h. A mistura de reação foi filtrada através de uma camada de celite. A camada de celite foi lavada com metanol/acetato de etil (1:1). O filtrado e as lavagens foram combinadas e evaporadas sob pressão reduzida para produzir o Composto 115 (quantitativo). A estrutura foi confirmada por LCMS e análise 1H NMR.
[001212] O composto 83a (0,17 g, 0,75 mmol), HBTU (0,31 g, 0,83 mmol) e DIEA (0,26 mL, 1,5 mmol) foram dissolvidos em DMF anidro (5 mL) e a mistura de reação foi agitada à temperatura ambiente por 5 min. A isto, uma solução do Composto 115 (1,22 g, 0,75 mmol) em DMF anidro foi adicionada e a reação foi agitada à temperatura ambiente por 6 h. O solvente foi removido sob pressão reduzida e o resíduo foi dissolvido em CH2Cl2. A camada orgânica foi lavada com solução de NaHCO3 aquosa saturada e salmoura e seca com Na2SO4 anidro e filtrada. A camada orgânica foi concentrada à secura e o resíduo obtido foi purificado por cromatografia de coluna em gel de sílica e eluído com MeOH 3 a 15% em diclorometano para produzir o Composto 116 (0,84 g, 61%). A estrutura foi confirmada por LCMS e análise 1H NMR.
[001213] O composto 116 (0,74 g, 0,4 mmol) foi dissolvido em 1:1 de metanol/acetato de etil (5 mL/5 mL). Foi adicionado paládio em carbono (úmido, 0,074 g). A mistura de reação foi carregada com hidrogênio e agitada à temperatura ambiente sob hidrogênio por 12 h. A mistura de reação foi filtrada através de uma camada de celite. A camada de celite foi lavada com metanol/acetato de etil (1:1). O filtrado e as lavagens foram combinadas e evaporadas sob pressão reduzida para produzir o composto 117 (0,73 g, 98%). A estrutura foi confirmada por LCMS e análise 1H NMR.
[001214] O composto 117 (0,63 g, 0,36 mmol) foi dissolvido em DMF anidro (3 mL). A esta solução, N, N -Di-isopropiletilamina (70 μL, 0,4 mmol) e trifluoroacetato de pentafluorofenil (72 μL, 0,42 mmol) foram adicionados. A mistura de reação foi agitada à temperatura ambiente por 12 h e vertida numa solução de NaHCO3aquosa saturada. A mistura foi extraída com diclorometano, lavada com salmoura e seca com Na2SO4anidro. A solução de diclorometano foi concentrada àsecura e purificada com cromatografia de coluna em gel de sílica e eluída com MeOH 5 a 10% em diclorometano para produzir ocomposto 118 (0,51 g, 79%). A estrutura foi confirmada por LCMS e1H e 1H e 19F NMR.
[001215] O Composto Oligomérico 119, compreendendo um grupo de conjugado de GalNAc3-7, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado de GalNAc3-7 (GalNAc3-7a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é - P(=O)(OH)-Ad-P(=O)(OH)-.
[001216] A estrutura de GalNAc3-7 (GalNAc3-7a-CM-) é mostrada abaixo:
Exemplo 49: Preparação do Oligonucleotídeo 132Compreendendo GalNAc3-5
[001217] O composto 120 (14,01 g, 40 mmol) e HBTU (14,06 g, 37 mmol) foram dissolvidos em DMF anidro (80 mL). Trietilamina (11,2 mL, 80,35 mmol) foi adicionada e agitada por 5 min. A mistura de reação foi resfriada em banho gelado e uma solução do composto 121 (10 g, mmol) em DMF anidro (20 mL) foi adicionada. Trietilamina adicional (4,5 mL, 32,28 mmol) foi adicionada e a mistura de reação foi agitada por 18 h sob atmosfera de argônio. A reação foi monitorada por TLC (acetato de etil:hexano; 1:1; Rf = 0,47). O solvente foi removido sob pressão reduzida. O resíduo foi absorvido em EtOAc (300 mL) e lavado com 1M NaHSO4 (3 x 150 mL), solução de NaHCO3 aquosa saturada (3 x 150 mL) e salmoura (2 x 100 mL). Camada orgânica foi seca com Na2SO4. O agente de secagem foi removido por filtração e a camada orgânica foi concentrada por evaporação giratória. A mistura crua foi purificada por cromatografia de coluna em gel de sílica e eluída, usando-se EtOAc 35 - 50% em hexano para produzir um composto 122 (15,50 g, 78,13%). A estrutura foiconfirmada por LCMS e análise 1H NMR. Massa m/z 589,3 [M + H]+.
[001218] Uma solução de LiOH (92,15 mmol) em água (20 mL) e THF (10 mL) foi adicionada a uma solução resfriada do Composto 122 (7,75 g, 13,16 mmol) dissolvido em metanol (15 mL). A mistura de reação foi agitada à temperatura ambiente por 45 min e monitorada por TLC (EtOAc:hexano; 1:1). A mistura de reação foi concentrada à metade do volume sob pressão reduzida. A solução restante foi resfriada em banho gelado e neutralizado, adicionando-se HCl concentrado. A mistura de reação foi diluída, extraída com EtOAc (120 mL) e lavada com salmoura (100 mL). Uma emulsão foi formada e limpa durante a noite. A camada orgânica foi separada, seca (Na2SO4), filtrada e evaporada para produzir o Composto 123 (8,42 g). O sal residual é a causa provável de excesso de massa. LCMS é consistente com a estrutura. O produto foi usado sem nenhuma purificação adicional. M.W.cal:574,36; M.W.fd:575,3 [M + H]+.
[001219] O composto 126 foi sintetizado após o procedimento descrito a literatura (J. Am. Chem. Soc. 2011, 133, 958-963).
[001220] O composto 123 (7,419 g, 12,91 mmol), HOBt (3,49 g, 25,82 mmol) e o composto 126 (6,33 g, 16,14 mmol) foram dissolvidos em DMF (40 mL) e a mistura de reação resultante foi resfriada em banho gelado. A isto, N,N-Di-isopropiletilamina (4,42 mL, 25,82 mmol), PyBop (8,7 g, 16,7 mmol) seguido por agente de acoplamento Bop (1,17 g, 2,66 mmol) foram adicionados sob uma atmosfera de argônio. O banho gelado foi removido e a solução foi deixada aquecer à temperatura ambiente. A reação foi concluída 1 h como determinado por TLC (DCM:MeOH:AA; 89:10:1). A mistura de reação foi concentrada sob pressão reduzida. O resíduo foi dissolvido em EtOAc (200 mL) e lavado com 1 M NaHSO4 (3x100 mL), NaHCO3aquoso saturado (3x100 mL) e salmoura (2x100 mL). A fase orgânica foi separada, seca (Na2SO4), filtrada e concentrada. O resíduo foi purificado por cromatografia de coluna em gel de sílica com um gradiente de 50% hexanos/EtOAC a 100% EtOAc para produzir o Composto 127 (9,4 g) como uma espuma branca. LCMS e 1H NMR foram consistentes com a estrutura. Massa m/z 778,4 [M + H] +.
[001221] Ácido trifluoroacético (12 mL) foi adicionado a uma solução do composto 127 (1,57 g, 2,02 mmol) em diclorometano (12 mL) e agitado à temperatura ambiente por 1 h. A mistura de reação foi coevaporada com tolueno (30 mL) sob pressão reduzida à secura. O resíduo obtido foi coevaporado duas vezes com acetonitrila (30 mL) e tolueno (40 mL) para produzir o Composto 128 (1,67 g) como sal de trifluoro acetato para a etapa seguinte sem purificação adicional. LCMS e 1H NMR foram consistentes com a estrutura. Massa m/z 478,2 [M + H] +.
[001222] O composto 7 (0,43 g, 0,963 mmol), HATU (0,35 g, 0,91 mmol), e HOAt (0,035 g, 0,26 mmol) foram combinados e secos por 4 h com P2O5 sob pressão reduzida em um frasco de fundo redondo e, em seguida, dissolvidos em DMF anidro (1 mL) e agitados por 5 min. A isto, uma solução do composto 128 (0,20 g, 0,26 mmol) em DMF anidro (0,2 mL) e N,N-Di-isopropiletilamina (0,2 mL) foi adicionada. A mistura de reação foi agitada à temperatura ambiente sob uma atmosfera de argônio. A reação foi concluída após 30 min como determinado por LCMS e TLC (7% MeOH/DCM). A mistura de reação foi concentrada sob pressão reduzida. O resíduo foi dissolvido em DCM (30 mL) e lavado com 1 M NaHSO4 (3x20 mL), NaHCO3 aquoso saturado (3 x 20 mL) e salmoura (3x20 mL). A fase orgânica foi separada, seca com Na2SO4, filtrada e concentrada. O resíduo foi purificado por cromatografia de coluna em gel de sílica usando 5-15% de MeOH em diclorometano para produzir o Composto 129 (96,6 mg). LCMS e1H NMR são consistentes com a estrutura. Massa m/z 883,4 [M + 2H]+.
[001223] O composto 129 (0,09 g, 0,051 mmol) foi dissolvido em metanol (5 mL) em 20 mL de frasco de cintilação. A isto, foi adicionada uma pequena quantidade de 10% Pd/C (0,015 mg) e o recipiente de reação foi carregado com gás H2. A mistura de reação foi agitada à temperatura ambiente sob atmosfera de H2 por 18 h. A mistura de reação foi filtrada através de uma camada de Celite e a camada de Celite foi lavada com metanol. As lavagens do filtrado foram agrupadas e concentradas sob pressão reduzida para produzir o Composto 130 (0,08 g). LCMS e 1H NMR foram consistentes com a estrutura. O produto foi usado sem nenhuma purificação adicional. Massa m/z 838,3 [M + 2H]+.
[001224] A um frasco de fundo redondo pontudo de 10 mL foram adicionados o composto 130 (75,8 mg, 0,046 mmol), 0,37 M depiridina/DMF (200 μL) e uma barra de agitação. A esta solução, foi adicionada 0,7 M de trifluoroacetato de pentafluorofenil/DMF (100 μL) em gotas com agitação. A reação foi concluída após 1 hora, como determinado por LCMS. O solvente foi removido sob pressão reduzida e o resíduo foi dissolvido em CHCl3 (~10 mL). A camada orgânica foi particionada contra NaHSO4 (1 M, 10 mL), NaHCO3 aquosa saturada (10 mL) e salmoura (10 mL) três vezes cada. A fase orgânica foi separada e seca com Na2SO4, filtrada e concentrada para produzir o Composto 131 (77,7 mg). LCMS é consistente com a estrutura. Usado sem purificação adicional. Massa m/z 921,3 [M + 2H]+.
[001225] O composto oligomérico 132, compreendendo um grupo de conjugado de GalNAc3-5, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado de GalNAc3-5 (GalNAc3-5a) pode ser combinada com qualquer fração clivável para fornecer uma variedade dos grupos de conjugado. Em determinadas modalidades, a fração clivável é - P(=O)(OH)-Ad-P(=O)(OH)-.
[001226] A estrutura de GalNAc3-5 (GalNAc3-5a-CM-) é mostradaabaixo:
Exemplo 50: Preparação do Oligonucleotídeo 144 CompreendendoGalNAc4-11
[001227] Síntese do Composto 134. A um frasco de Merrifield foi adicionada resina de aminometil VIMAD (2,5 g, 450 μmol/g) que foi lavada com acetonitrila, dimetilformamida, diclorometano e acetonitrila.A resina foi inchada em acetonitrila (4 mL). O composto 133 foi pré- ativado em um frasco de fundo redondo de 100 mL, adicionando-se 20 (1,0 mmol, 0,747 g), TBTU (1,0 mmol, 0,321 g), acetonitrila (5 mL) e DIEA (3,0 mmol, 0,5 mL). Esta solução foi deixada agitar por 5 min e foi então adicionada ao frasco de Merrifield com agitação. A suspensão foi deixada agitar por 3 h. A mistura de reação foi drenada e a resina foi lavada com acetonitrila, DMF e DCM. Um novo carregamento de resina foi quantificado, medindo-se a absorbância do cátion de DMT em 500 nm (coeficiente de extinção = 76000) em DCM e determinado para ser 238 μmol/g. A resina foi revestida, suspendendo-se em uma solução de anidrido acético por dez minutos três vezes.
[001228] O composto ligado ao suporte sólido 141 foi sintetizado usando métodos iterativos de síntese de peptídeo de fase sólida à base de Fmoc. Uma pequena quantidade de suporte sólido foi retirada e suspensa em amônia aquosa (28-30% em peso) por 6 h. O composto clivado foi analisado por LC-MS e a massa observada foi consistente com a estrutura. Massa m/z 1063,8 [M + 2H]+.
[001229] O composto ligado ao suporte sólido 142 foi sintetizado usando os métodos de síntese de peptídeo de fase sólida.
[001230] O composto ligado ao suporte sólido 143 foi sintetizado usando síntese de fase sólida padrão em um sintetizador de DNA.
[001231] O composto ligado ao suporte sólido 143 foi suspenso em amônia aquosa (28-30% em peso) e aquecido a 55 oC por 16 h. A solução foi resfriada e o suporte sólido foi filtrado. O filtrado foi concentrado e o resíduo dissolvido em água e purificado por HPLC numa coluna de troca de ânion forte. As frações contendo o composto de comprimento total 144 foram agrupadas e dessalinizadas. O composto oligomérico conjugado a GalNAc4-11 foi analisado por LC-MS e a massa observada foi consistente com a estrutura.
[001232] A parte de cluster de GalNAc4 do grupo do conjugado de GalNAc4-11 (GalNAc4-11a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad- P(=O)(OH)-.
[001233] A estrutura de GalNAc4-11 (GalNAc4-11a-CM) é mostrada abaixo:
Exemplo 51: Preparação do Oligonucleotídeo 155 CompreendendoGalNAc3-6
[001234] O composto 146 foi sintetizado como descrito na literatura (Analytical Biochemistry 1995, 229, 54-60).
[001235] O composto 4 (15 g, 45,55 mmol) e o composto 35b (14,3 gramas, 57 mmol) foram dissolvidos em CH2Cl2 (200 ml). Peneiras moleculares ativadas (4 Â. 2 g, em pó) foram adicionadas, e a reação foi deixada agitar por 30 minutos sob atmosfera de nitrogênio. TMS- OTf foi adicionado (4,1 ml, 22,77 mmol) e a reação foi deixada agitar à temperatura ambiente durante a noite. Após conclusão, a reação foi extinta vertendo-se numa solução de NaHCO 3aquoso saturado (500 ml) e gelo esmagado (~150 g). A camada orgânica foi separada, lavada com salmoura, seca com MgSO4, filtrada, e foi concentrada em um óleo laranja sob pressão reduzida. O material bruto foi purificado por cromatografia de coluna em gel de sílica e eluído com 2-10 % MeOH em CH2Cl2 para produzir o Composto 112 (16,53 g, 63%). LCMS e 1H NMR foram consistentes com o composto esperado.
[001236] O composto 112 (4,27 g, 7,35 mmol) foi dissolvido em 1:1 MeOH/EtOAc (40 ml). A mistura de reação foi purgada, ebulindo-se um fluxo de argônio através da solução por 15 minutos. Um catalisador de Pearlman (hidróxido de paládio em carbono, 400 mg) foi adicionado, e o gás hidrogênio foi ebulido através da solução por 30 minutos. Após a conclusão (TLC 10% de MeOH em CH2Cl2, e LCMS), o catalisador foi removido por filtração através de uma camada de celite. O filtrado foi concentrado por evaporação giratória, e foi seco brevemente sob alto vácuo para produzir o Composto 105a (3,28 g). LCMS e 1H NMR foram consistentes com o produtodesejado.
[001237] O composto 147 (2,31 g, 11 mmol) foi dissolvido em DMF anidro (100 mL). N,N-Di-isopropiletilamina (DIEA, 3,9 mL, 22 mmol) foi adicionada, seguido por HBTU (4 g, 10,5 mmol). A mistura de reação foi deixada agitar por ~15 minutos sob nitrogênio. A isto, uma solução do composto 105a (3,3 g, 7,4 mmol) em DMF seco foi adicionada e agitada por 2 h sob atmosfera de nitrogênio. A ração foi diluída com EtOAc e lavada com NaHCO aquoso saturado3 e salmoura. A fase orgânica foi separada, seca (MgSO4), filtrada e concentrada em um xarope laranja. O material bruto foi purificado por cromatografia de coluna 2-5 % MeOH em CH2Cl2 para produzir o Composto 148 (3,44 g, 73 %). LCMS e1H NMR foram consistentes com o produto esperado.
[001238] O composto 148 (3,3 g, 5,2 mmol) foi dissolvido em 1:1 MeOH/EtOAc (75 ml). A mistura de reação foi purgada, ebulindo-se um fluxo de argônio através da solução por 15 minutos. Um catalisador de Pearlman (hidróxido de paládio em carbono) foi adicionado (350 mg). O gás hidrogênio foi ebulido através da solução por 30 minutos. Após a conclusão (TLC 10% de MeOH em DCM, e LCMS), o catalisador foi removido por filtração através de uma camada de celite. O filtrado foi concentrado por evaporação giratória, e foi seco brevemente sob alto vácuo para produzir o Composto 149 (2,6 g). A LCMS foi consistente com o produto desejado. O resíduo foi dissolvido em DMF seco (10 ml) e foi usado imediatamente na etapa seguinte.
[001239] O composto 146 (0,68 g, 1,73 mmol) foi dissolvido em DMF seco (20 ml). A isto, DIEA (450 μL, 2,6 mmol, 1,5 eq.) e HBTU (1,96 g, 0.5.2 mmol) foram adicionados. A mistura de reação foi deixada agitar por 15 minutos à temperatura ambiente sob nitrogênio. Uma solução do composto 149 (2,6 g) em DMF anidro (10 mL) foi adicionada. O pH da reação foi ajustado para pH = 9-10 pela adição de DIEA (se necessário). A reação foi deixada agitar à temperatura ambiente sob nitrogênio por 2 h. Após a conclusão, a reação foi diluída com EtOAc (100 mL), e lavada com NaHCO3aquoso saturado, seguido por salmoura. A fase orgânica foi separada, seca com MgSO4, filtrada e concentrada. O resíduo foi purificado por cromatografia de coluna em gel de sílica e eluída com 210 % de MeOH em CH2Cl2 para produzir o Composto 150 (0,62 g, 20%). LCMS e1H NMR foram consistentes com o produto desejado.
[001240] O composto 150 (0,62 g) foi dissolvido em 1:1 MeOH/EtOAc (5 L). A mistura de reação foi purgada, ebulindo-se um fluxo de argônio através da solução por 15 minutos. Um catalisador de Pearlman (hidróxido de paládio em carbono) foi adicionado (60 mg). O gás hidrogênio foi ebulido através da solução por 30 minutos. Após a conclusão (TLC 10% de MeOH em DCM, e LCMS), o catalisador foi removido por filtração (filtro de Teflon na ponta da seringa, 0,45 μm). O filtrado foi concentrado por evaporação giratória, e foi seco brevemente sob alto vácuo para produzir o Composto 151 (0,57 g). A LCMS foi consistente com o produto desejado. O produto foi dissolvido em 4 mL de DMF seco e foi usado imediatamente na etapa seguinte.
[001241] O composto 83a (0,11 g, 0,33 mmol) foi dissolvido em DMF anidro (5 mL) e N,N-Di-isopropiletilamina (75 μL, 1 mmol) e PFP-TFA (90 μL, 0,76 mmol) foram adicionados. A mistura de reação se tornou magenta após o contato, e se torou gradualmente laranja durante os próximos 30 minutos. O progresso da reação foi monitorado por TLC e LCMS. Após a conclusão (formação de éster de PFP), uma solução do composto 151 (0,57 g, 0,33 mmol) em DMF foi adicionada. O pH da reação foi ajustado para pH = 9-10 pela adição de N,N-Di-isopropiletilamina (se necessário). A mistura de reação foi agitada sob nitrogênio por ~30 min. Após a conclusão, a maior parte do solvente foi removida sob pressão reduzida. O resíduo foi diluído com CH2Cl2 e lavado com NaHCO3aquoso saturado, seguido por salmoura. A fase orgânica foi separada, seca com MgSO4, filtrada e concentrada num xarope laranja. O resíduo foi purificado por cromatografia de coluna em gel de sílica (2-10 % de MeOH em CH2Cl2) para produzir o Composto 152 (0,35 g, 55%). LCMS e1H NMR foram consistentes com o produto desejado.
[001242] O composto 152 (0,35 g, 0,182 mmol) foi dissolvido em 1:1 MeOH/EtOAc (10 mL). A mistura de reação foi purgada, ebulindo-se um fluxo de argônio através da solução por 15 minutos. Um catalisador de Pearlman (hidróxido de paládio em carbono) foi adicionado (35 mg). O gás hidrogênio foi ebulido através da solução por 30 minutos. Após a conclusão (TLC 10% de MeOH em DCM, e LCMS), o catalisador foi removido por filtração (filtro de Teflon na ponta da seringa, 0,45 μm). O filtrado foi concentrado por evaporação giratória, e foi seco brevemente sob alto vácuo para produzir o Composto 153 (0,33 g, quantitativo). A LCMS foi consistente com o produto desejado.
[001243] O composto 153 (0,33 g, 0,18 mmol) foi dissolvido em DMF anidro (5 mL) com agitação sob nitrogênio. A isto, N,N-Di- isopropiletilamina (65 μL, 0,37 mmol) e PFP-TFA (35 μL, 0,28 mmol) foram adicionados. A mistura de reação foi agitada sob nitrogênio por ~30 min. A mistura de reação se tornou magenta após o contato, e gradualmente se tornou laranja. O pH da mistura de reação foi mantida em pH = 9-10, adicionando-se mais N,-Di-isopropiletilamina. Oprogresso da reação foi monitorado por TLC e LCMS. Após a conclusão, a maior parte do solvente foi removido sob pressão reduzida. O resíduo foi diluído com CH2Cl2 (50 mL), e lavado com NaHCO3aquoso saturado, seguido por salmoura. A camada orgânica foi seca com MgSO4, filtrada e concentrada num xarope laranja. O resíduo foi purificado por cromatografia de coluna e eluído com 2-10% MeOH em CH2Cl2 para produzir o Composto 154 (0,29 g, 79%). LCMS e1H NMR foramconsistentes com o produto desejado.
[001244] O composto oligomérico 155, compreendendo um grupo do conjugado de GalNAc3-6, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo do conjugado de GalNAc3-6 (GalNAc3-6a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é- P(=O)(OH)-Ad-P(=O)(OH)-.
[001245] A estrutura de GalNAc3-6 (GalNAc3-6a-CM-) é mostrada abaixo:
Exemplo 52: Preparação do Oligonucleotídeo 160 CompreendendoGalNAc3-9
[001246] O composto 156 foi sintetizado após o procedimento descrito na literatura (J. Med. Chem. 2004, 47, 5798-5808).
[001247] O composto 156, (18,60 g, 29,28 mmol) foi dissolvido em metanol (200 mL). Paládio em carbono (6,15 g, 10% em peso, carregamento (base seca), pó de carbono de matriz, úmido) foi adicionado. A mistura de reação foi agitada à temperatura ambiente sob hidrogênio por 18 h. A mistura de reação foi enchida através de uma camada de celite e a camada de celite foi lavada completamente com metanol. O filtrado combinado foi lavado e concentrado à secura.O resíduo foi purificado por cromatografia de coluna em gel de sílica e eluído com 5-10% de metanol em diclorometano para produzir o Composto 157 (14,26 g, 89%). Massa m/z 544,1 [M-H]-.
[001248] O composto 157 (5 g, 9,17 mmol) foi dissolvido em DMF anidro (30 mL). HBTU (3,65 g, 9,61 mmol) e N,N-Di-isopropiletilamina (13,73 mL, 78,81 mmol) foram adicionados e a mistura de reação foi agitada à temperatura ambiente por 5 minutos. A isto, foi adicionada uma solução do composto 47 (2,96 g, 7,04 mmol). A reação foi agitada à temperatura ambiente por 8 h. A mistura de reação foi vertida numa solução aquosa de NaHCO3 saturado. A mistura foi extraída com acetato de etil e a camada orgânica foi lavada com salmoura e seca (Na2SO4), filtrada e evaporada. O resíduo foi purificado por cromatografia de coluna em gel de sílica e eluído com 50% de acetato de etil em hexano para produzir o Composto 158 (8,25 g, 73,3%). A estrutura foi confirmada por MS e análise 1H NMR.
[001249] O composto 158 (7,2 g, 7,61 mmol) foi seco com P2O5 sob pressão reduzida. O composto seco foi dissolvido em DMF anidro (50 mL). A isto, 1H-tetrazol (0,43 g, 6,09 mmol) e N-metilimidazol (0,3 mL, 3,81 mmol) e fosforodiamidita de 2-cianoetil-N,N,N',N'-tetraisopropil (3,65 mL, 11,50 mmol) foram adicionados. A mistura de reação foi agitada sob atmosfera de argônio por 4 h. A mistura de reação foi diluída com acetato de etil (200 mL). A mistura de reação foi lavada com NaHCO3saturado e salmoura. A fase orgânica foi separada, seca (Na2SO4), filtrada e evaporada. O resíduo foi purificado por cromatografia de coluna em gel de sílica e eluído com 50-90% e acetato de etil em hexano para produzir o Composto 159 (7,82 g, 80,5%). A estrutura foi confirmada por LCMS e análise31P NMR.
[001250] O composto oligomérico 160, compreendendo um grupo do conjugado de GalNAc3-9, foi preparado usando procedimentos de síntese de oligonucleotídeo padrão. Três unidades do composto 159 foram acopladas ao suporte sólido, seguido por fosforamiditas nucleotídicas. O tratamento do composto oligomérico protegido com amônia aquosa produziu o composto 160. A parte de cluster de GalNAc3 do grupo do conjugado de GalNAc3-9 (GalNAc3-9a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é- P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-9 (GalNAc3-9a-CM) é mostrada abaixo:
Exemplo 53: Procedimento alternativo para preparação do Composto 18 (GalNAc3-1a e GalNAc3-3a)
[001251] A lactona 161 foi reagida com diamino propano (3-5 eq) ou diamino propano protegido por Mono-Boc (1 eq) para fornecer o álcool 162a ou 162b. Quando a propanodiamina não protegida foi usada para a reação acima, o excesso de diamina foi removido por evaporação sob alto vácuo e o grupo amino livre em 162a foi protegido usando CbzCl para fornecer o 162b como um sólido branco após a purificação por cromatografia de coluna. O álcool 162b foi ainda reagido com o composto 4 na presença de TMSOTf para fornecer o 163a que foi convertido em 163b pela remoção do grupo Cbz usando hidrogenação catalítica. O éster de pentafluorofenil (PFP) 164 foi preparado, reagindo- se o triácido 113 (ver o Exemplo 48) com PFPTFA (3,5 eq) e piridina (3,5 eq) em DMF (0,1 a 0,5 M). O triéster 164 foi diretamente reagido com a amina 163b (3-4 eq) e DIPEA (3-4 eq) para fornecer o Composto 18. O método acima facilita grandemente a purificação dos intermediários e minimiza a formação de subprodutos que são formados usando o procedimento descrito no Exemplo 4.Exemplo 54: Procedimento alternativo para preparação do Composto 18 (GalNAc3-1a e GalNAc3-3a)

[001252] O éster triPFP 164 foi preparado a partir do ácido 113 usando o procedimento delineado no exemplo 53 acima e reagido com diamina protegida por mono-Boc para fornecer o 165 num rendimento essencialmente quantitativo. Os grupos Boc foram removidos com ácido clorídrico ou ácido trifluoroacético para fornecer a triamina que foi reagida com o ácido ativado por PFP 166 na presença de uma base adequada, tal como DIPEA para fornecer o Composto 18.
[001253] O ácido de Gal-NAc protegido por PFP 166 foi preparado a partir do ácido correspondente pelo tratamento com PFPTFA (1-1,2 eq) e piridina (1-1,2 eq) em DMF. O ácido precursor, por sua vez, foi preparado a partir do álcool correspondente por oxidação usando TEMPO (0,2 eq) e BAIB em acetonitrila e água. O álcool precursor foi preparado a partir do intermediário de açúcar 4 pela reação com 1,6- hexanodiol (ou 1,5-pentanodiol ou outro diol para outros n valores) (2-4 eq) e TMSOTf usando as condições descritas anteriormente no exemplo 47.Exemplo 55: Estudo dependente de dose de oligonucleotídeos compreendendo tanto um grupo de conjugado em 3' quanto em 5' (comparação de GalNAc3-1, 3, 8 e 9) direcionados ao SRB-1 in vivo
[001254] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. ISIS 353382 não conjugado foi incluído como um padrão. Cada um dos diversos grupos de conjugado de GalNAc3 foi ligado tanto ao terminal 3' quanto ao 5' do oligonucleotídeos respectivo por um nucleosídeo de 2'-desoxiadenosina ligado a fosfodiéster (fração clivável).Tabela 39ASO modificado que se direciona ao SRB-1
[001255] Le tras maiúsculas indicam a nucleo base para cadanucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica umnucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonucleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001256] A estrutura de GalNAc3-1a foi mostrada anteriormente noExemplo 9. A estrutura de GalNAc3-9 foi mostrada anteriormente noExemplo 52. A estrutura de GalNAc3-3 foi mostrada anteriormente noExemplo 39. A estrutura de GalNAc3-8 foi mostrada anteriormente noExemplo 47.
Tratamento
[001257] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 353382, 655861, 664078, 661161, 665001 ou com salina. Cada grupo detratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB- 1 para cada grupo de tratamento, normalizados para o controle com salina.
[001258] Conforme ilustrado na Tabela 40, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. De fato, os oligonucleotídeos que compreendem os conjugados de GalNAc3-1 e GalNAc3-9 ligados a fosfodiéster no terminal 3' (ISIS 655861 e ISIS 664078) e os conjugados de GalNAc3-3 e GalNAc3-8 ligados no terminal 5' (ISIS 661161 e ISIS 665001) mostraram melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 353382). Além disso, o ISIS 664078, que compreende um conjugado de GalNAc3-9 no terminal 3' foi essencialmente equipotente comparado ao ISIS 655861, que compreende um conjugado de GalNAc3-1 no terminal 3'. Os oligonucleotídeos antisense conjugados em 5', ISIS 661161 e ISIS 665001, que compreendem um GalNAc3-3 ou GalNAc3-9,respectivamente, tiveram maior potência em comparação aos oligonucleotídeos antisense conjugados em 3' (ISIS 655861 e ISIS 664078).Tabela 40ASOs contendo GalNAc3-1, 3, 8 ou 9 direcionado ao SRB-1


[001259] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. A alteração nos pesos corporais foi avaliada sem nenhuma alteração significativa do grupo com salina. Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na tabela abaixo.
Exemplo 56: Estudo dependente de dose de oligonucleotídeos compreendendo tanto um grupo de conjugado em 3' quanto em 5' (comparação de GalNAc3-1,2, 3, 5, 6, 7 e 10) direcionados ao SRB- 1 in vivo
[001260] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. ISIS 353382 não conjugado foi incluído como um padrão. Cada um dos diversos grupos de conjugado de GalNAc3 foi ligado ao terminal 5' do respectivo oligonucleotídeo por um nucleosídeo de 2'- desoxiadenosina ligado a fosfodiéster (fração clivável), exceto pelo ISIS 655861 que teve o grupo de conjugado de GalNAc3 ligado no terminal 3'.Tabela 42ASO modificado direcionado ao SRB-1
[001261] Le tras maiúsculas indicam a nucleo base para cadanucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoximbonu- cleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O- P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001262] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9. A estrutura de GalNAc3-2a foi mostrado anteriormente no Exemplo 37. A estrutura de GalNAc3-3a foi mostrada anteriormente noExemplo 39. A estrutura de GalNAc3-5a foi mostrada anteriormente noExemplo 49. A estrutura de GalNAc3-6a foi mostrada anteriormente noExemplo 51. A estrutura de GalNAc3-7afoi mostrada anteriormente noExemplo 48. A estrutura de GalNAc3-10a foi mostrada anteriormente no Exemplo 46. Tratamento
[001263] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados uma vez na dosagem mostrada abaixo com ISIS 353382, 655861, 664507, 661161, 666224, 666961, 666981, 666881 ou com salina. Cada grupo detratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB- 1 para cada grupo de tratamento, normalizados para o controle com salina.
[001264] Conforme ilustrado na Tabela 43, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. De fato, os oligonucleotídeos antisense mostraram melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 353382). Os oligonucleotídeos antisense conjugados em 5' mostraram um ligeiro aumento na potência em comparação ao oligonucleotídeo antisense conjugado em 3'.
[001265] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. A alteração nos pesos corporais foi avaliada sem nenhuma alteração significativa do grupo com salina. Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 44 abaixo.
Exemplo 57: Duração do estudo de ação de oligonucleotídeos compreendendo um grupo de conjugado em 3' direcionado à ApoC III in vivo
[001266] Camundongos foram injetados uma vez que as doses indicadas abaixo e monitorados durante o curso de 42 dias para os níveis de ApoC-III e de triglicerídeos plasmáticos (TG plasmático). O estudo foi realizado usando 3 camundongos transgênicos que expressam a APOC-III humana em cada grupo.Tabela 45ASO modificado direcionado à ApoC III
[001267] Letras maiúsculas indicam a nucleobase para cada nucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonucleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001268] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9.Tabela 46Níveis de mRNA de ApoC III (% de Salina no Dia 1) e TG Plasmático (% de Salina no Dia 1)
[001269] Como pode ser visto na tabela acima, a duração da ação aumentou com a adição do grupo de conjugado em 3' em comparação ao oligonucleotídeo não conjugado. Houver um aumento adicional na duração da ação para o oligonucleotídeo de PO/PS misto conjugado 647536 em comparação ao oligonucleotídeo PS completo conjugado 647535.Exemplo 58: Estudo dependente de dose de oligonucleotídeos compreendendo um grupo de conjugado em 3' (comparação de GalNAc3-1 e GalNAc4-11) direcionados ao SRB-1 in vivo
[001270] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. ISIS 440762 não conjugado foi incluído como um padrão não conjugado. Cada um dos grupos de conjugado foi ligado no terminal 3' do respectivo oligonucleotídeo por uma fração clivável do nucleosídeo de 2'-desoxiadenosina ligado a fosfodiéster.
[001271] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9. A estrutura de GalNAc3-11a foi mostrada anteriormente no Exemplo 50.Tratamento
[001272] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 440762, 651900, 663748 ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizados para o controle com salina.
[001273] Conforme ilustrado na Tabela 47, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. Os oligonucleotídeos antisense que compreendem os conjugados de GalNAc3-1 ligado a fosfodiéster e GalNAc4-11 no terminal 3' (ISIS 651900 e ISIS 663748) mostraram melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 440762). Os dois oligonucleotídeos conjugados, GalNAc3-1 e GalNAc4-11, foram equipotentes.Tabela 47ASO modificado direcionado ao SRB-1
[001274] Letras maiúsculas indicam a nucleobase para cada nucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "k" indica o nucleosídeo bicíclico 6'-(S)- CH3; "d" indica um β-D-2'-desoximbonucleosideo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucle- osídica fosfodiéster(PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001275] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. A alteração nos pesos corporais foi avaliada sem nenhuma alteração significativa do grupo com salina. Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 48 abaixo.
Exemplo 59: Efeitos de ASOs conjugados com GalNAc3-1direcionados ao FXI in vivo
[001276] Os oligonucleotídeos listados abaixo foram testados em um estudo de doses múltiplas para a inibição antisense de FXI em camundongos. ISIS 404071 foi incluído como um padrão não conjugado. Cada um dos grupos de conjugado foi ligado no terminal 3' do respectivo oligonucleotídeo por uma fração clivável de nucleosídeo de 2'-desoxiadenosina ligado a fosfodiéster.Tabela 49ASOs modificados direcionados ao FXI
[001277] Letras maiúsculas indicam a nucleobase para cada nucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonu- cleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica - O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001278] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9.
Tratamento
[001279] Camundongos Balb/c machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea duas vezes por semana por 3 semanas na dosagem mostrada abaixo com ISIS 404071, 656172, 656173 ou controle tratado com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de FXI no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis da proteína FXI plasmáticos também foram medidos usando ELISA. Os níveis de mRNA de FXI foram determinados em relação ao RNA total (usando RIBOGREEN®), antes da normalização para controles tratados com PBS. Os resultados são apresentados a seguir como a porcentagem média dos níveis de mRNA de FXI para cada grupo de tratamento. Os dados foram normalizados para os controles tratados com PBS e é denotado como "% de PBS". As ED50s foram medidas usando métodos semelhantes conforme descrito anteriormente e são apresentadas abaixo.Tabela 50 mRNA do Fator XI (% de Salina)

[001280] Conforme ilustrado na Tabe a 50, o tra tamento comoligonucleotídeos antisense diminuiu os níveis de mRNA de FXI de uma forma dependente da dose. Os oligonucleotídeos que compreendem um grupo de conjugado de 3'-GalNAc3-1 mostrou melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 404071). Entre os dois oligonucleotídeos conjugados, uma melhora na potência foi ainda fornecida, substituindo-se algumas das ligações PS por PO (ISIS 656173).
[001281] Conforme ilustrado na Tabela 50a, o tratamento com oligonucleotídeos antisense diminuiu os níveis de da proteína FXI de uma forma dependente da dose. Os oligonucleotídeos que compreendem um grupo de conjugado de 3'-GalNAc3-1 mostraram melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 404071). Entre os dois oligonucleotídeos conjugados, uma melhora na potência foi ainda fornecida, substituindo- se algumas das ligações PS por PO (ISIS 656173).Tabela 50aProteína Fator XI (% de Salina)
[001282] Os níveis de transaminase, alanina aminotransferase (ALT)e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Bilirrubina total, albumina total, CRE e BUN também foram avaliados. A alteração nos pesos corporais foi avaliada sem nenhuma alteração significativa do grupo com salina. Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na tabela abaixo.
Exemp o 60: El Feitos de ASO is conjugados direcionados ao SRB-1in vitro
[001283] Os oligonucleotídeos listados abaixo foram testados em um estudo de doses múltiplas para inibição antisense de SRB-1 em hepatócitos de camundongo primários. ISIS 353382 foi incluído como um padrão não conjugado. Cada um dos grupos de conjugado foi ligado no terminal 3' ou 5' do respectivo oligonucleotídeo por uma fração clivável de nucleosídeo de 2'-desoxiadenosina ligado a fosfodiéster.Tabela 52ASO modificado direcionado ao SRB-1
[001284] Letras maiúsculas indicam a nucleobase para cada nucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonu- cleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica - O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001285] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9. A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39. A estrutura de GalNAc3-8a foi mostrada anteriormente noExemplo 47. A estrutura de GalNAc3-9a foi mostrada anteriormente noExemplo 52. A estrutura de GalNAc3-6a foi mostrada anteriormente noExemplo 51. A estrutura de GalNAc3-2a foi mostrada anteriormente noExemplo 37. A estrutura de GalNAc3-10a foi mostrada anteriormente no Exemplo 46. A estrutura de GalNAc3-5a foi mostrada anteriormente noExemplo 49. A estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48.
Tratamento
[001286] Os oligonucleotídeos listados acima foram testados in vitro em células hepatócitos primários de camundongo colocadas numa densidade de 25.000 células por poço e tratadas com 0,03, 0,08, 0,24, 0,74, 2,22, 6,67 ou 20 nM de oligonucleotídeo modificado. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis de mRNA foram medidos por PCR em tempo real e os níveis de mRNA de SRB-1 foram ajustados de acordo com o conteúdo total de RNA, conforme medido por RIBOGREEN®.
[001287] A IC50 foi calculada usando métodos padrão e os resultados são apresentados na Tabela 53. Os resultados mostram que, sob condições de absorção livre nas quais nenhum reagente ou nenhuma técnica de eletroporação são usados para promover artificialmente a entrada dos oligonucleotídeos nas células, os oligonucleotídeos que compreendem um conjugado de GalNAc foram significativamente mais potentes nos hepatócitos do que o oligonucleotídeo de origem (ISIS 353382) que não compreende um conjugado de GalNAc.
aMédia de várias execuções.Exemplo 61: Preparação do composto oligomérico 175compreendendo GalNAc3-12
[001288] O composto 169 está disponível comercialmente. Ocomposto 172 foi preparado pela adição de benzil (perfluorofenil) glutarato ao composto 171. O benzil (perfluorofenil) glutarato foipreparado, adicionando-se PFP-TFA e DIEA ao ácido 5-(benziloxi)-5- oxopentanoico em DMF. O composto oligomérico 175, compreendendo um grupo de conjugado de GalNAc3-12, foi preparado a partir do composto 174 usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado de GalNAc3- 12 (GalNAc3-12a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-12 (GalNAc3-12a-CM-) é mostrada abaixo:
Exemplo 62: Preparação do composto oligomérico 180compreendendo GalNAc3-13
[001289] O composto 176 foi preparado usando o procedimento geral mostrado no Exemplo 2. O composto oligomérico 180, que compreende um grupo de conjugado de GalNAc3-13, foi preparado a partir do composto 177 usando os procedimentos gerais ilustrados no Exemplo 49. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-13 (GalNAc3-13a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-13 (GalNAc3-13a-CM-) é mostrada abaixo:
Exemplo 63: Preparação do composto oligomérico 188 compreendendo GalNAc3-14
[001290] Os compostos 181 e 185 estão disponíveis comercialmente. O composto oligomérico 188, que compreende um grupo de conjugado de GalNAc3-14, foi preparado a partir do composto 187 usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-14 (GalNAc3-14a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-14 (GalNAc3-14a-CM-) é mostrada abaixo:
Exemplo 64: Preparação do composto oligomérico 197compreendendo GalNAc3-15
[001291] O composto 189 está disponível comercialmente. O composto 195 foi preparado usando o procedimento geral mostrado no Exemplo 31. O composto oligomérico 197, que compreende um grupo de conjugado de GalNAc3-15, foi preparado a partir dos compostos 194 e 195 usando procedimentos de síntese de oligonucleotídeo padrão. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-15 (GalNAc3-15a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-15 (GalNAc3-15a-CM-) é mostrada abaixo:
Exemplo 65: Estudo dependente de dose de oligonucleotídeos compreendendo um grupo de conjugado em 5' (comparação de GalNAc3-3, 12, 13, 14 e 15) direcionados ao SRB-1 in vivo
[001292] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. ISIS 353382 não conjugado foi incluído como um padrão. Cada um dos grupos de conjugado de GalNAc3 foi ligado no terminal 5' do respectivo oligonucleotídeo por um nucleosídeo de 2'- desoxiadenosina ligado a fosfodiéster (fração clivável).Tabela 54ASOs modificados direcionados a SRB-1
[001293] Letras maiúsculas indicam a nucleobase para cadanucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica umnucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonucleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001294] A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39. A estrutura de GalNAc3-12a foi mostrada anteriormente noExemplo 61. A estrutura de GalNAc3-13a foi mostrada anteriormente noExemplo 62. A estrutura de GalNAc3-14a foi mostrada anteriormente noExemplo 63. A estrutura de GalNAc3-15a foi mostrada anteriormente noExemplo 64.Tratamento
[001295] Camundongos C57bl6 de seis a oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma ou duas vezes na dosagem mostrada abaixo com ISIS 353382, 661161, 671144, 670061, 671261, 671262, ou com salina. Os camundongos que foram dosados duas vezes receberam a segunda dose três dias após a primeira dose. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizados para o controle com salina.
[001296] Conforme ilustrado na Tabela 55, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. Nenhuma diferença significativa no knockdown alvo foi observada entre os animais que receberam uma dose única e os animais que receberam duas doses (ver ISIS 353382, dosagens 30 e 2 x 15 mg/kg; e ISIS 661161 dosagens 5 e 2 x 2,5 mg/kg). Os oligonucleotídeos que compreendem os conjugados de GalNAc3-3, 12, 13, 14 e 15 ligados a fosfodiéster mostraram melhora substancial na potência em comparação ao oligonucleotídeo antisense não conjugado (ISIS 335382).Tabela 55mRNA de SRB-1 (% de Salina)
[001297] Os níveis de transaminase, alanina aminotransferase (ALT)e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. As alterações dos pesos corporais foram avaliadas sem nenhuma diferença significativa do grupo com salina (dados não mostrados). Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 56 abaixo.
Exemplo 66: Efeito de diversas frações cliváveis sobre a inibição antisense in vivo pelos oligonucleotídeos direcionados ao SRB-1 compreendendo um cluster de 5'-GalNAc3
[001298] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. Cada um dos grupos de conjugado de GalNAc3 foi ligado no terminal 5' do respectivo oligonucleotídeo por um nucleosídeo ligado a fosfodiéster (fração clivável (CM)).Tabela 57ASOs modificados direcionados a SRB-1
[001299] Letras maiúsculas indicam a nucleobase para cada nucleosídeo emC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'-desoxirribonu- cleosídeo; "s" indica uma ligação internucleosídica fosforotioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001300] A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39. A estrutura de GalNAc3-13a foi mostrada anteriormente no Exemplo 62.Tratamento
[001301] Camundongos C57bl6 de seis a oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com ISIS 661161, 670699, 670700, 670701, 671165, ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 no fígado usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizados para o controle com salina.
[001302] Conforme ilustrado na Tabela 58, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. Os oligonucleotídeos antisense compreendendo diversas frações cliváveis todos mostraram potências semelhantes.Tabela 58mRNA de SRB-1 (% de Salina)
[001303] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. As alterações dos pesos corporais foram avaliadas sem nenhuma diferença significativa do grupo com salina (dados não mostrados). Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 56 abaixo.
[001304] O composto oligomérico 199, compreendendo um grupo de conjugado de GalNAc3-16, é preparado usando os procedimentos gerais ilustrados nos Exemplos 7 e 9. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-16 (GalNAc3-16a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-16 (GalNAc3-16a-CM-) é mostrada abaixo:
Exemplo 68: Preparação do composto oligomérico 200 compreendendo GalNAc3-17
[001305] O composto oligomérico 200, compreendendo um grupo do conjugado de GalNAc3-17, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupos de conjugado GalNAc3-17 (GalNAc3-17a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é- P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-17 (GalNAc3-17a- CM-) é mostrada abaixo:
Exemplo 69: Preparação do composto oligomérico 201compreendendo GalNAc3-18
[001306] O composto oligomérico 201, compreendendo um grupo do conjugado de GalNAc3-18, foi preparado usando os procedimentos gerais ilustrados no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-18 (GalNAc3-18a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é - P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-18 (GalNAc3-18a- CM-) é mostrada abaixo:
Exemplo 70: Preparação do composto oligomérico 204compreendendo GalNAc3-19
[001307] O composto oligomérico 204, compreendendo um grupo de conjugado de GalNAc3-19, foi preparado a partir do composto 64 usando os procedimentos gerais ilustrados no Exemplo 52. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-19 (GalNAc3-19a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é-P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-19 (GalNAc3-19a-CM-) é mostrada abaixo:
Exemplo 71: Preparação do composto oligomérico 210 compreendendo GalNAc3-20
[001308] O composto 205 foi preparado, adicionando-se PFP-TFA e DIEA ao ácido 6-(2,2,2-trifluoroacetamido)hexanoico em acetonitrila, que foi preparada pela adição de anidrido tríflico ao ácido 6- aminohexanoico. A mistura de ração foi aquecida a 80 oC, em seguida abaixada para rt (temperatura ambiente). O composto oligomérico 210, compreendendo um grupo de conjugado de GalNAc3-20, foi preparado a partir do composto 208 usando os procedimentos gerais ilustrados no Exemplo 52. A parte de cluster GalNAc3 do grupo de conjugado GalNAc3-20 (GalNAc3-20a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é - P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-20 (GalNAc3-20a- CM-) é mostrada abaixo:
[001309] O composto 211 está disponível comercialmente. O composto oligomérico 215, compreendendo um grupo de conjugado de GalNAc3-21, foi preparado a partir do composto 213 usando os procedimentos gerais ilustrados no Exemplo 52. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-21 (GalNAc3-21a) pode ser combinada com qualquer fração clivável para fornecer uma variedades de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad-P(=O)(OH)-. A estrutura de GalNAc3-21 (GalNAc3-21a-CM-) é mostrada abaixo:
Exemplo 73: Preparação do composto oligomérico 221compreendendo GalNAc3-22
[001310] O composto 220 foi preparado a partir do composto 219 usando tetrazolida de di-isopropilamônio. O composto oligomérico 221, compreendendo um grupo de conjugado de GalNAc3-21, é preparado a partir do composto 220 usando o procedimento geral ilustrado no Exemplo 52. A parte de cluster de GalNAc3 do grupo de conjugado GalNAc3-22 (GalNAc3-22a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, a fração clivável é -P(=O)(OH)-Ad- P(=O)(OH)-. A estrutura de GalNAc3-22 (GalNAc3-22a-CM-) é mostrada abaixo:
Exemplo 74: Efeito de diversas frações cliváveis sobre a inibição antisense in vivo pelos oligonucleotídeos direcionados ao SRB-1 compreendendo um conjugado de 5'-GalNAc3
[001311] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. cada um dos grupos de conjugado de GalNAc3 foi ligado no terminal 5' do respectivo oligonucleotídeo.Tabela 60ASOs modificados direcionados a SRB-1
[001312 Em todas as tabelas, as letras maiúsculas indicam a nucle-obase para cada nucleosídeo e mC indica uma 5-metil citosina. Subscritos: "e" indica um nucleosídeo 2'-MOE modificado; "d" indica um β-D-2'- desoxirribonucleosídeo; "s" indica uma ligação internucleosídica fosforo- tioato (PS); "o" indica uma ligação internucleosídica fosfodiéster (PO); e "o'" indica -O-P(=O)(OH)-. Os grupos de conjugado estão em negrito.
[001313] A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39. A estrutura de GalNAc3-17a foi mostrada anteriormente no Exemplo 68, e a estrutura de GalNAc3-18a foi mostrada no Exemplo 69. Tratamento
[001314] Camundongos C57BL/6 de seis a oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por vis subcutânea uma vez na dosagem mostrada abaixo com um oligonucleotídeo listado na Tabela 60 ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizados para o controle com salina.
[001315] Conforme ilustrado na Tabela 61, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose. Os oligonucleotídeos antisense que compreendem um conjugado de GalNAc mostraram potências semelhantes e foram significativamente mais potentes do que o oligonucleotídeo de origem em que faltava um conjugado de GalNAc.Tabela 61mRNA de SRB-1 (% de Salina)
[001316] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro foram medidos em relação aos camundongos injetados com salina usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. A alteração nos pesos corporais foi avaliada sem nenhuma alteração significativa do grupo com salina (dados não mostrados). Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 62 abaixo.
Exemplo 75: Análise farmacocinética de oligonucleotídeos compreendendo um grupo de conjugado em 5'
[001317] A PK dos ASOs nas Tabelas 54, 57 e 60 acima foi avaliada usando amostras de fígado que foram obtidas após os procedimentos de tratamento descritos nos Exemplos 65, 66 e 74. As amostras de fígado foram cortadas e extraídas usando protocolos padrão e analisadas por IP-HPLC-MS juntamente com um padrão interno. O nível de tecido combinado (μg/g) de todos os metabólitos foi medido pela integração dos picos de UV apropriados, e o nível de tecido do ASO de comprimento total em que faltava o conjugado ("de origem", que tem N° Isis 353382 neste caso) foi medido usando os cromatogramas de íon extraído apropriados (EIC).Tabela 63Análise PK no Fígado
[001318] Os resultados na Tabela 63 acima mostram que houve maiores níveis de tecido hepático dos oligonucleotídeos compreendendo um grupo de conjugado de GalNAc3 do que do oligonucleotídeo de origem que não compreende um grupo de conjugado de GalNAc3 (ISIS 353382) 72 horas após a administração do oligonucleotídeo, particularmente ao levar em consideração as diferenças na dosagem entre os oligonucleotídeos com e sem um grupo de conjugado de GalNAc3. Além disso, em 72 horas, 40-98% de cada oligonucleotídeo compreendendo um grupo de conjugado de GalNAc3 foram metabolizados no composto de origem, indicando que os grupos de conjugado de GalNAc3 foram clivados a partir dos oligonucleotídeos. Exemplo 76: Preparação do composto oligomérico 230 compreendendo GalNAc3-23
[001319] O composto 222 está disponível comercialmente. 44,48 ml (0,33 mol) do composto 222 foram tratados com cloreto de tosil (25,39 g, 0,13 mol) em piridina (500mL) por 16 horas. A reação foi então evaporada para um óleo, dissolvida em EtOAc e lavada com água, NaHCO3saturado, salmoura e seca com Na2SO4. O acetato de etil foi concentrado à secura e purificado por cromatografia de coluna, eluído com EtOAc/hexanos (1:1) seguido por metanol 10% em CH2Cl2 para gerar o composto 223 como um óleo incolor. LCMS e NMR foram consistentes com a estrutura. 10 g (32,86 mmol) de 1-Tosiltrietileno glicol (composto 223) foi tratado com azida sódica (10,68 g, 164,28 mmol) em DMSO (100 mL) à temperatura ambiente por 17 horas. A mistura de reação foi então vertida em água, e extraída com EtOAc. A camada orgânica foi lavada com água três vezes e seca com Na2SO4. A camada orgânica foi concentrada à secura para gerar 5,3 g do composto 224 (92%). LCMS e NMR foram consistentes com a estrutura. 1-Azidotrietileno glicol (composto 224, 5,53 g, 23,69 mmol) e ocomposto 4 (6 g, 18,22 mmol) foram tratados com peneiras moleculares 4A (5 g), e TMSOTf (1,65 ml, 9,11 mmol) em diclorometano (100 mL) sob uma atmosfera inerte. Após 14 horas, a reação foi filtrada para remover as peneiras, e a camada orgânica foi lavada com NaHCO3saturado, água, salmoura e seca com Na2SO4. A camada orgânica foi concentrada à secura e purificada por cromatografia de coluna, eluída com um gradiente de metanol 2 a 4% em diclorometano para gerar o composto 225. LCMS e NMR foram consistentes com a estrutura. O composto 225 (11,9 g, 23,59 mmol) foi hidrogenado em EtOAc/Metanol (4:1, 250 mL) com catalisador de Pearlman. Após 8 horas, o catalisador foi removido por filtração e os solventes removidos à secura para gerar o composto 226. LCMS e NMR foram consistentes com a estrutura.
[001320] A fim de gerar o composto 227, uma solução de ácido nitrometanotrispropiônico (4,17 g, 15,04 mmol) e base de Hunig (10,3 ml, 60,17 mmol) em DMF (100 mL) foram tratados em gotas com acetato de pentaflourotrifluoro (9,05 ml, 52,65 mmol). Após 30 minutos, a reação foi vertida em água gelada e extraída com EtOAc. A camada orgânica foi lavada com água, salmoura e seca com Na2SO4. A camada orgânica foi concentrada à secura e então recristalizada a partir de heptano para gerar o composto 227 como um sólido branco. LCMS e NMR foram consistentes com a estrutura. O composto 227 (1,5 g, 1,93 mmol) e o composto 226 (3,7 g, 7,74 mmol) foram agitados à temperaturaambiente em acetonitrila (15 mL) por 2 horas. A reação foi então evaporada à secura e purificada por cromatografia de coluna, eluindo- se com um gradiente de metanol 2 a 10% em diclorometano para gerar o composto 228. LCMS e NMR foram consistentes com a estrutura. O composto 228 (1,7 g, 1,02 mmol) foi tratado com níquel de Raney (cerca de 2 g úmido) em etanol (100mL) numa atmosfera de hidrogênio. Após 12 horas, o catalisador foi removido por filtração e a camada orgânica foi evaporada a um sólido que foi usado diretamente na etapa seguinte. LCMS e NMR foram consistentes com a estrutura. Este sólido (0,87 g, 0,53 mmol) foi tratado com ácido benzilglutárico (0,18 g, 0,8 mmol), HBTU (0,3 g, 0,8 mmol) e DIEA (273,7 μl, 1,6 mmol) em DMF (5 mL). Após 16 horas, o DMF foi removido sob pressão reduzida a 65°C a um óleo, e o óleo foi dissolvido em diclorometano. A camada orgânica foi lavada com solução de NaHCO3saturado, salmoura, e seca com Na2SO4. Após a evaporação da camada orgânica, o composto foi purificado por cromatografia de coluna e eluído com um gradiente de metanol 2 a 20% em diclorometano para gerar o produto acoplado. LCMS e NMR foram consistentes com a estrutura. O éster benzílico foi desprotegido com catalisador de Pearlman sob uma atmosfera de hidrogênio por 1 hora. O catalisador foi então removido por filtração e os solventes removidos à secura para gerar o ácido. LCMS e NMR foram consistentes com a estrutura. O ácido (486 mg, 0,27 mmol) foi dissolvido em DMF seco (3 mL). Piridina (53,61 μl, 0,66 mmol) foi adicionada e a reação foi purgada com argônio. Acetato de pentaflourotriflouro (46,39 μl, 0,4 mmol) foi lentamente adicionado à mistura de reação. A cor da reação mudou de amarelo pálido para vinho, e gerou uma fumaça clara que foi dissipada com um fluxo de argônio. A reação foi deixada agitar à temperatura ambiente por uma hora (a conclusão da reação foi confirmada por LCMS). O solvente foi removido sob pressão reduzida (rotovap (evaporador rotativo)) a 70°C. O resíduo foi diluído com DCM e lavado com 1N NaHSO4, salmoura, bicarbonato de sódio saturado e salmoura novamente. Os produtos orgânicos foram secos com Na2SO4, filtrados, e foram concentrados à secura para gerar 225 mg do composto 229 como uma espuma amarela frágil. LCMS e NMR foram consistentes com a estrutura.
[001321] O composto oligomérico 230, compreendendo um grupo de conjugado de GalNAc3-23, foi preparado a partir do composto 229 usando o procedimento geral ilustrado no Exemplo 46. A parte de cluster de GalNAc3 do grupo de conjugado de GalNAc3-23 (GalNAc3-23a) pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAc3-23 (GalNAc3-23a-CM) é mostrada abaixo:
Exemplo 77: Inibição antisense in vivo por oligonucleotídeos direcionados ao SRB-1 compreendendo um conjugado de GalNAc3
[001322] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos.Tabela 64ASOs modificados direcionados a SRB-1
[001323] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-9a foi mostrado no Exemplo 52, GalNAc3-10a foi mostrado no Exemplo 46, GalNAc3-19a foi mostrado no Exemplo 70, GalNAc3-20a foi mostrado no Exemplo 71, e GalNAc3-23a foi mostrado no Exemplo 76.Tratamento
[001324] Camundongos C57BL/6 de seis a oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram, cada um, injetados por via subcutânea uma vez numa dosagem mostrada abaixo com um oligonucleotídeo listado na Tabela 64 ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final para determinar os níveis de mRNA de SRB-1 usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento, normalizados para o controle com salina.
[001325] Conforme ilustrado na Tabela 65, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose.Tabela 65mRNA de SRB-1 (% de Salina)
[001326] Os níveis de transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas no soro também foram medidos usando protocolos padrão. Também foram avaliadas a bilirrubina total e BUN. As alterações dos pesos corporais foram avaliadas sem nenhuma diferença significativa do grupo com salina (dados não mostrados). Os valores de ALTs, ASTs, bilirrubina total e BUN são mostrados na Tabela 66 abaixo.
Exemplo 78: Inibição antisense in vivo por oligonucleotídeos direcionados ao angiotensinogênio compreendendo um conjugado de GalNAc3
[001327] Os oligonucleotídeos listados abaixo foram testados em um estudo dependente de dose para inibição antisense do Angioten- sinogênio (AGT) em ratos Sprague Dawley normotensos.Tabela 67ASOs modificados direcionados a AGT
[001328] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9.Tratamento
[001329] Ratos Sprague Dawley machos de seis semanas de idade foram, cada um, injetados por via subcutânea uma vez por semana numa dosagem mostrada abaixo, para um total de três doses, com um oligonucleotídeo listado na Tabela 67 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Os ratos foram sacrificados 72 horas após a dose final. Os níveis de mRNA hepáticos de AGT foram medidos usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis plasmáticos da proteína AGT foram medidos usando o ELISA de Angiotensinogênio Total (Catálogo # JP27412, IBL International, Toronto, ON) com plasma diluído 1:20.000. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de AGT no fígado ou níveis da proteína AGT no plasma para cada grupo de tratamento, normalizados para o controle com PBS.
[001330] Conforme ilustrado na Tabela 68, o tratamento com oligonucleotídeos antisense diminuiu os níveis da proteína plasmática e do mRNA hepático de AGT de uma forma dependente da dose, e o oligonucleotídeo compreendendo um conjugado de GalNAc foi significativamente mais potente do que o oligonucleotídeo de origem em que faltava um conjugado de GalNAc.Tabela 68Níveis da proteína plasmáticos e do mRNA hepático de AGT
[001331] Os níveis da transaminase, alanina aminotransferase (ALT) e aspartato aminotransferase (AST) hepáticas, no plasma e os pesos corporais também foram medidos no momento do sacrifício usando protocolos padrão. Os resultados são mostrados na Tabela 69 abaixo.Tabela 69Níveis de transaminase hepática e pesos corporais dos ratos
Exemplo 79: Duração da ação in vivo de oligonucleotídeos direcionados à APOC-III compreendendo um conjugado de GalNAc3
[001332] Os oligonucleotídeos listados na Tabela 70 abaixo foram testados em um estudo de dose única para a duração da ação em camundongos.Tabela 70ASOs modificados direcionados a APOC-III
[001333] A estrutura de GalNAc3-1a foi mostrada anl eriormente noExemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-7a foi mostrado no Exemplo 48, GalNAc3-10a foi mostrado no Exemplo 46, e GalNAc3-13a foi mostrado no Exemplo 62.Tratamento
[001334] Camundongos transgênicos de seis a oito semanas de idade que expressam APOC-III humana foram, cada um, injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 70 ou com PBS. Cada grupo de tratamento consistiu em 3 animais. O sangue foi retirado antes da dosagem para determinar a base de referência e em 72 horas, 1 semana, 2 semanas, 3 semanas, 4 semanas, 5 semanas e 6 semanas após a dose. Os níveis de triglicerídeos plasmáticos e da proteína APOC-III foram medidos conforme descrito no Exemplo 20. Os resultados abaixo são apresentados como a porcentagem média dos níveis de triglicerídeos plasmáticos e de APOC-III para cada grupo de tratamento, normalizados para os níveis de base de referência, mostrando que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc exibiam uma duração mais longa de ação do que o oligonucle- otídeo de origem sem um grupo de conjugado (ISIS 304801), mesmo com a dosagem do original sendo três vezes a dosagem dos oligonucleotídeos que compreendem um grupo de conjugado de GalNAc.Tabela 71Níveis de triglicerídeos plasmáticos e da proteína APOC-III em camundongos transgênicos


Exemplo 80: Inibição antisense in vivo por oligonucleotídeos direcionados à Alfa-1 Antitripsina (A1AT) compreendendo um conjugado de GalNAc3
[001335] Os oligonucleotídeos listados na Tabela 72 abaixo foram testados em um estudo para a inibição dependente de dose de A1AT em camundongos.Tabela 72ASOs modificados direcionados à A1AT
[001336] A estrutura de GalNAc3-1a foi mostrada anl eriormente noExemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-7a foi mostrado no Exemplo 48, GalNAc3-10a foi mostrado no Exemplo 46, e GalNAc3-13a foi mostrado no Exemplo 62.Tratamento
[001337] Camundongos C57BL/6 machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram, cada um, injetados por vis subcutânea uma vez por semana numa dosagem mostrada abaixo, para um total de três doses, com um oligonucleotídeo listado na Tabela 72 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final. Os níveis de mRNA hepáticos de A1AT foram medidos usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis da proteína plasmática A1AT foram determinados usando o ELISA de Alfa-1 Antitripsina de Camundongo (catálogo # 41-A1AMS- E01, Alpco, Salem, NH). Os resultados abaixo são apresentados como a porcentagem média dos níveis da proteína plasmática e do mRNA hepático de A1AT para cada grupo de tratamento, normalizados para o controle com PBS.
[001338] Conforme ilustrado na Tabela 73, o tratamento com oligonucleotídeos antisense diminuiu os níveis da proteína plasmática A1AT e do mRNA hepático de A1AT de uma forma dependente da dose. Os oligonucleotídeos compreendendo um conjugado de GalNAc foram significativamente mais potentes do que o original (ISIS 476366).Tabela 73Níveis da proteína plasmática e do mRNA hepático de A1AT
[001339] Os níveis de BUN e transaminase hepática no plasma foram medidos no momento de sacrifício usando protocolos padrão. Os pesos corporais e os pesos dos órgãos também foram medidos. Os resultados são mostrados na Tabela 74 abaixo. O peso corporal é mostrado como % em relação à base de referência. Os pesos dos órgãos são mostrados como % de peso corporal em relação ao grupo controle com PBS.
Exemplo 81: Duração da ação in vivo de oligonucleotídeos direcionados à A1AT compreendendo um cluster de GalNAc3
[001340] Os oligonucleotídeos listados na Tabela 72 foram testados em um estudo de dose única para duração da ação em camundongos. Tratamento
[001341] Os camundongos C57BL/6 machos de seis semanas de idade foram, cada um, injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 72 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. O sangue foi retirado o dia antes da dosagem para determinar a base de referência e em 5, 12, 19 e 25 dias após a dose. Os níveis da proteína A1AT plasmática foram medidos através de ELISA (ver o Exemplo 80). Os resultados abaixo são apresentados como a porcentagem média dos níveis da proteína A1AT plasmática para cada grupo de tratamento, normalizados para os níveis de base de referência. Os resultados mostram que os oligonucleotídeos que compreendem um conjugado de GalNAc eram mais potentes e tinham maior duração de ação do que o original em que faltava um conjugado de GalNAc (ISIS 476366). Além disso, os oligonucleotídeos que compreendem um conjugado de 5'-GalNAc (ISIS 678381, 678382, 678383 e 678384) eram geralmente ainda mais potentes com maior duração de ação do que o oligonucleotídeo que compreende um conjugado de 3'-GalNAc (ISIS 656326).Tabela 75Níveis da proteína A1AT plasmática em camundongos


Exemplo 82: Inibição antisense in vitro por oligonucleotídeos direcionados ao SRB-1 compreendendo um conjugado de GalNAc3
[001343] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-5a foi mostrado no Exemplo 49, GalNAc3-6a foi mostrado no Exemplo 51, GalNAc3-7a foi mostrado no Exemplo 48, GalNAc3-8a foi mostrado no Exemplo 47, GalNAc3-9a foi mostrado no Exemplo 52, GalNAc3-10a foi mostrado no Exemplo 46, GalNAc3-12a foi mostrado no Exemplo 61, GalNAc3-13a foi mostrado no Exemplo 62, GalNAc3-14a foi mostrado no Exemplo 63, GalNAc3-15a foi mostrado no Exemplo 64, GalNAc3-17a foi mostrado no Exemplo 68, GalNAc3-18a foi mostrado no Exemplo 69, GalNAc3-19a foi mostrado no Exemplo 70, GalNAc3-20a foi mostrado no Exemplo 71, e GalNAc3-23a foi mostrado no Exemplo 76.Exemplo 83: Inibição antisense in vivo por oligonucleotídeos direcionados ao Fator XI compreendendo um cluster de GalNAc3
[001344] Os oligonucleotídeos listados na Tabela 77 abaixo foram testados em um estudo para inibição dependente de dose do Fator XI em camundongos.Tabela 77Oligonucleotídeos modificados direcionados ao Fator XI
[001345] A estrutura de GalNAc3-1a foi mostrada anteriormente noExemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-7a foi mostrado no Exemplo 48, GalNAc3-10a foi mostrado no Exemplo 46, e GalNAc3-13a foi mostrado no Exemplo 62.Tratamento
[001346] Camundongos de seis a oito semanas de idade foram, cada um, injetados por via subcutânea uma vez por semana numa dosagem mostrada abaixo, para um total de três doses, com um oligonucleotídeo listado abaixo ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a dose final. Os níveis de mRNA hepático do Fator XI foram medidos usando PCR em tempo real e normalizados para ciclofilina de acordo com protocolos padrão. Transaminases hepáticas, BUN e bilirrubina também foram medidos. Os resultados abaixo são apresentados como a porcentagem média para cada grupo de tratamento, normalizados para o controle com PBS.
[001347] Conforme ilustrado na Tabela 78, o tratamento com oligonucleotídeos antisense diminuiu o mRNA hepático do Fator XI de uma forma dependente de dose. Os resultados mostram que os oligonucleotídeos compreendendo um conjugado de GalNAc foi mais potente do que o original em que falta um conjugado de GalNAc (ISIS 404071). Além disso, os oligonucleotídeos compreendendo um conjugado de 5'-GalNAc (ISIS 663086, 678347, 678348 e 678349) foram ainda mais potentes do que o oligonucleotídeo que compreende um conjugado de 3'-GalNAc (ISIS 656173).Tabela 78Níveis de mRNA hepático do Fator XI, transaminase hepática, BUN e bilirrubina
Exemplo 84: Duração da ação in vivo de oligonucleotídeos direcionados ao Fator XI compreendendo um conjugado de GalNAc3
[001348] Os oligonucleotídeos listados na Tabela 77 foram testados em um estudo de dose única para duração da ação em camundongos. Tratamento
[001349] Camundongos de seis a oito semanas foram, cada um, injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 77 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. O sangue foi retirado por sangramentos da cauda no dia antes da dosagem para determinar a base de referência e em 3, 10 e 17 dias após a dose. Os níveis da proteína Fator XI plasmático foram medidos por ELISA usando anticorpos de captura do Fator XI e de detecção biotinilados da R & D Systems, Minneapolis, MN (catálogo # AF2460 e # BAF2460, respectivamente) e o Conjunto B do Reagente OptEIA (Catálogo # 550534, BD Biosciences, San Jose, CA). Os resultados abaixo são apresentados como a porcentagem média dos níveis da proteína Fator XI plasmático para cada grupo de tratamento, normalizados para os níveis de base de referência. Os resultados mostram que os oligonucleotídeos que compreendem um conjugado de GalNAc eram mais potentes com maior duração de ação do que o original em que faltava um conjugado de GalNAc (ISIS 404071). Além disso, os oligonucleotídeos que compreendem um conjugado de 5'- GalNAc (ISIS 663086, 678347, 678348 e 678349) foram ainda mais potentes com uma maior duração de ação do que o oligonucleotídeo que compreende um conjugado de 3'-GalNAc (ISIS 656173).Tabela 79Níveis da proteína Fator XI plasmático em camundongos

Exemplo 85: Inibição antisense in vivo por oligonucleotídeos direcionados ao SRB-1 compreendendo um conjugado de GalNAc3
[001350] Os oligonucleotídeos listados na Tabela 76 foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos.Tratamento
[001351] Camundongos de seis a oito semanas de idade C57BL/6 foram, cada um, injetados por via subcutânea uma vez por semana numa dosagem mostrada abaixo, para um total de três doses, com um oligonucleotídeo listado na Tabela 76 ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 48 horas após a administração final para determinar os níveis de mRNA de SRB-1 usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 hepáticos para cada grupo de tratamento, normalizados para o controle com salina.
[001352] Conforme ilustrado nas Tabelas 80 e 81, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente da dose.Tabela 80mRNA de SRB-1 no fígado
[001353] Os níveis de transaminases hepá ticas, bilirrubina total, BUN,e pesos corporais também foram medidos usando protocolos padrão. Os valores médios para cada grupo de tratamento são mostrados na Tabela 82 abaixo.
Exemplo 86: Inibição antisense in vivo por oligonucleotídeos direcionados a TTR compreendendo um cluster de GalNAc3
[001354] Os oligonucleotídeos listados na Tabela 83 abaixo foram testados em um estudo dependente de dose para a inibição antisense da transtirretina humana (TTR) em camundongos transgênicos que expressam o gene da TTR humano.Tratamento
[001355] Camundongos transgênicos para TTR de oito semanas de idade foram, cada um, injetados por via subcutânea uma vez por semana por três semanas, para um total de três doses, com um oligonucleotídeo e dosagem listados nas tabelas abaixo ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final. Os sangramentos da cauda foram realizados em vários momentos por todo o experimento, e os níveis plasmáticos de proteína TTR, ALT e AST foram medidos e relatados nas Tabelas 85-87. Após os animais serem sacrificados, os níveis plasmáticos de ALT, AST e TTR humana foram medidos, assim como os pesos corporais, pesos dos órgãos, e níveis hepáticos de mRNA de TTR humana. Os níveis da proteína TTR foram medidos usando um analisador clínico (AU480, Beckman Coulter, CA). PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) Foram usados de acordo com protocolos padrão para determinar os níveis hepáticos de mRNA de TTR humana. Os resultados apresentados nas Tabelas 84-87 são os valores médios para cada grupo de tratamento. Os níveis de mRNA são os valores médios relativos à média para o grupo com PBS. Os níveis plasmáticos da proteína são os valores médios relativos ao valor médio para o grupo com PBS na base de referência. Os pesos corporais são a mudança de peso percentual média da base de referência até o sacrifício para cada grupo de tratamento individual. Os pesos dos órgãos mostrados são normalizados para o peso do corpo do animal, e o peso do órgão normalizado médio para cada grupo de tratamento é então apresentado em relação ao peso do órgão normalizado médio para o grupo com PBS.
[001356] Nas Tabelas 84-87, "BL" indica base de referência, as medições que foram tomadas imediatamente antes da primeira dose. Conforme ilustrado nas Tabelas 84 e 85, o tratamento com oligonucleotídeos antisense diminuiu o nível de expressão de TTR de uma forma dependente de dose. Os oligonucleotídeos compreendendo um conjugado de GalNAc foram mais potentes que o de origem em que faltava o conjugado de GalNAc (ISIS 420915). Além disso, os oligonucleotídeos que compreendem um conjugado de GalNAc e ligações internucleosídicas PS/PO mistas foram ainda mais potentes que o oligonucleotídeo que compreende um conjugado de GalNAc e ligações PS totais.Tabela 83Oligonucleotídeos direcionados à TTR humana
[001357] A legenda para a Tabela 85 pode ser encontrada no Exemplo 74. A estrutura de GalNAc3-1 foi mostrada no Exemplo 9. A estrutura de GalNAc3-3a foi mostrada no Exemplo 39. A estrutura de GalNAc3-7a foi mostrada no Exemplo 48. A estrutura de GalNAc3-10a foi mostrada no Exemplo 46. A estrutura de GalNAc3-13a foi mostrada no Exemplo 62. A estrutura de GalNAc3-19a foi mostrada no Exemplo 70.Tabela 84Inibição antisense de TTR humana in vivo
Tabela 85Inibição antisense de TTR humana in vivo
Tabela 87Níveis de transaminases, alterações do peso corporal e peso dos
Exemplo 87: Duração da ação in vivo por doses únicas de oligonucleotídeos direcionados à TTR compreendendo um cluster de GalNAc3
[001358] Os números ISIS 420915 e 660261 (ver a Tabela 83) foram testados em um estudo de dose única para a duração da ação em camundongos. Os números ISIS 420915, 682883 e 682885 (ver a Tabela 83) também foram testados em um estudo de dose única para duração da ação em camundongos.Tratamento
[001359] Camundongos transgênicos machos de oito semanas de idade que expressam TTR humana foram, cada um, injetados por via subcutânea uma vez com 100 mg/kg de N°. ISIS 420915 ou 13,5 mg/kg de N°. ISIS 660261. Cada grupo de tratamento consistiu em 4 animais. Os sangramentos da cauda foram realizados antes da dosagem para determinar a base de referência e nos dias 3, 7, 10, 17, 24 e 39 após a dose. Os níveis plasmáticos da proteína TTR foram medidos conforme descrito o Exemplo 86. Os resultados abaixo são apresentados como a porcentagem média dos níveis plasmáticos da proteína TTR para cada grupo de tratamento, normalizados para os níveis de base de referência.Tabela 88Níveis plasmáticos da proteína TTR
[001360] Camundongos transgênicos fêmeas que expressam a TTR humana foram, cada um, injetados por via subcutânea uma vez com 100 mg/kg de N°. ISIS 420915, 10,0 mg/kg de N°. ISIS 682883, ou 10,0 mg/kg de 682885. Cada grupo de tratamento consistiu em 4 animais. Os sangramentos da cauda foram realizados antes da dosagem para determinar a base de referência e nos dias 3, 7, 10, 17, 24 e 39 após a dose. Os níveis plasmáticos da proteína TTR foram medidos conforme descrito o Exemplo 86. Os resultados abaixo são apresentados como a porcentagem média dos níveis plasmáticos da proteína TTR para cada grupo de tratamento, normalizados para os níveis de base de referência.Tabela 89Níveis plasmáticos da proteína TTR
[001361] Os resultados na T Fabela 88 e 89 mostram que osoligonucleotídeos que compreendem um conjugado de GalNAc são mais potentes com uma duração de ação maior que o oligonucleotídeo de origem e que falta um conjugado (ISIS 420915).Exemplo 88: Modulação por splicing in vivo por oligonucleotídeos direcionados a SMN compreendendo um conjugado de GalNAc3
[001362] Os oligonucleotídeos listados na Tabela 90 foram testados para a modulação por splicing da Sobrevivência do Neurônio Motor humana (SMN) em camundongos.Tabela 90ASOs modificados direcionados a SMN
[001363] A estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48. "X" indica uma amina primária em 5' gerada por Gene Tools (Philomath, OR), e GalNAc3-7b indica a estrutura de GalNAc3-7a em que falta a porção -NH-C6-O do ligador como mostrado abaixo:
[001364] Os números ISIS 703421 e 703422 são oligonucleotídeos de morfolino, em que cada nucleotídeo dos dois oligonucleotídeos é um nucleotídeo de morfolino.Tratamento
[001365] Camundongos transgênicos de seis semanas de idade que expressam SMN humana foram injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 91 ou com salina. Cada grupo de tratamento consistiu em 2 machos e 2 fêmeas. Os camundongos foram sacrificados 3 dias após a dose para determinar os níveis hepáticos de mRNA de SMN humana com e sem o éxon 7 usando PCR em tempo real, de acordo com protocolos padrão. O RNA total foi medido usando o reagente Ribogreen. Os níveis de mRNA da SMN foram normalizados para mRNA total, e normalizados ainda para as médias para o grupo tratado com salina. As razões médias resultantes entre mRNA de SMN incluindo o éxon 7 e mRNA de SMN faltando o éxon 7 são mostradas na Tabela 91. Os resultados mostram que os oligonucleotídeos totalmente modificados que modulam o splicing e compreendem um conjugado de GalNAc são significativamente mais potentes em alterar o splicing no fígado do que os oligonucleotídeos de origem em que falta o conjugado de GalNAc. Além disso, esta tendência é mantida para várias químicas de modificação, incluindo 2'-MOE e oligonucleotídeos modificados por morfolino.Tabela 91Efeito de oligonucleotídeos direcionados à SMN humana in vivo


Exemplo 89: Inibição antisense in vivo por oligonucleotídeos direcionados à Apolipoproteína A (Apo(A)) compreendendo um conjugado de GalNAc3
[001366] Os oligonucleotídeos listados na Tabela 92 abaixo foram testados em um estudo para a inibição dependente de dose de Apo(a) em camundongos transgênicos.Tabela 92ASOs modificados direcionados à Apo(a)
[001367] A estrutura de GalNAc3-7a foi mostrada no Exemplo 48.Tratamento
[001368] Camundongos C57BL/6 fêmeas de oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram, cada um, injetados por via subcutânea uma vez por semana numa dosagem mostrada abaixo, para um total de seis doses, com um oligonucleotídeo listado na Tabela 92 ou com PBS. Cada grupo de tratamento consistiu em 3-4 animais. Os sangramentos da cauda foram realizados no dia anterior à primeira dose e semanalmente após cada dose para determinar os níveis plasmáticos da proteína Apo(a). Os camundongos foram sacrificados dois dias após a administração final. Os níveis de mRNA hepáticos de Apo(a) foram determinados usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os níveis plasmáticos da proteína Apo(a) foram determinados usando ELISA, e os níveis hepáticos da transaminase foram determinados. Os resultados da proteína plasmática e do mRNA na Tabela 93 são apresentados como a porcentagem média do grupo de tratamento em relação ao grupo tratado com PBS. Os níveis da proteína plasmática foram ainda normalizados para o valor de base de referência (BL) para o grupo com PBS. Os níveis de transaminase absolutos médios e os pesos corporais (% em relação às médias de base de referência) são relatados na Tabela 94.
[001369] Como ilustrado na Tabela 93, o tratamento com os oligonucle- otídeos diminuiu os níveis da proteína plasmática e do mRNA hepático da Apo(a) de uma forma dependente de dose. Além disso, o oligonucleotídeo que compreende o conjugado de GalNAc foi significativamente mais po-tente com uma duração maior de ação do que o oligonucleotídeo de origem em que falta o conjugado de GalNAc. Como ilustrado na Tabela 94, os níveis de transaminase e os pesos corporais não foram afetados pelo oligo- nucleotídeos, indicando que os oligonucleotídeos foram bem tolerados.Tabela 93Níveis de proteína plasmática e de mRNA hepático de Apo(a)
Exemplo 90: Inibição antisense in vivo por oligonucleotídeos direcionados a TTR compreendendo um cluster de GalNAc3
[001370] Os oligonucleotídeos listados na Tabela 95 abaixo foram testados em um estudo dependente de dose para a inibição antisense da transtirretina humana (TTR) em camundongos transgênicos que expressam o gene da TTR humano.Tratamento
[001371] Camundongos transgênicos para TTR foram, cada um, injetados por via subcutânea uma vez por semana por três semanas, para um total de três doses, com um oligonucleotídeo e dosagem listados na Tabela 96 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Antes da primeira dose, um sangramento da cauda foi realizado para determinar os níveis plasmáticos da proteína TTR na base de referência (BL). Os camundongos foram sacrificados 72 horas após a administração final. Os níveis da proteína TTR foram medidos usando um analisador clínico (AU480, Beckman Coulter, CA). PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) Foram usados de acordo com protocolos padrão para determinar os níveis hepáticos de mRNA de TTR humana. Os resultados apresentados na Tabela 96 são os valores médios para cada grupo de tratamento. Os níveis de mRNA são os valores médios relativos à média para o grupo com PBS. Os níveis plasmáticos da proteína são os valores médios relativos ao valor médio para o grupo com PBS na base de referência. "BL" indica base de referência, as medições que foram tomadas imediatamente antes da primeira dose. Conforme ilustrado na Tabela 96, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de FXI de uma forma dependente da dose.de expressão de TTR de uma forma dependente da dose. Os oligonucleotídeos que compreendem um conjugado de GalNAc foram mais potentes do que o de origem em que falta um conjugado de GalNAc (ISIS 420915), e os oligonucleotídeos que compreendem uma fração clivável de fosfodiéster ou desoxiadenosina mostraram melhoras significativas na potência em comparação ao de origem em que falta um conjugado (ver os números ISIS 682883 e 666943 vs 420915 e ver os Exemplo 86 e 87). Tabela 95Oligonucleotídeos direcionados à TTR humana


[001372] A legenda para a Tabela 95 pode ser encontrada no Exemplo 74. A estrutura de GalNAc3-3a foi mostrada no Exemplo 39. A estrutura de GalNAc3-7a foi mostrada no Exemplo 48. A estrutura de GalNAc3-10a foi mostrada no Exemplo 46. A estrutura de GalNAc3-13a foi mostrada no Exemplo 62.Tabela 96Inibição antisense de TTR humana in vivo
Exemplo 91: Inibição antisense in vivo por oligonucleotídeos direcionados ao Fator VII compreendendo um conjugado de GalNAc3 em primatas não humanos
[001373] Os oligonucleotídeos listados na Tabela 97 abaixo foram testados em um estudo de aumento de dose não terminal para a inibição antisense do Fator VII em macacos.Tratamento
[001374] Macacos expostos (não virgens) foram, cada um, injetados por via subcutânea nos dias 0, 15 e 29 com doses crescentes de um oligonucleotídeo listado na Tabela 97 ou com PBS. Cada grupo de tratamento consistiu em 4 machos e 1 fêmea. Antes da primeira dose e em vários momentos após isso, as retiradas de sangue foram realizadas para determinar os níveis plasmáticos da proteína Fator VII. Os níveis da proteína Fator VII foram medidos por ELISA. Os resultados apresentados na Tabela 98 são os valores médios para cada grupo de tratamento em relação ao valor médio para o grupo com PBS na base de referência (BL), as medidas tomadas imediatamente antes da primeira dose. Como ilustrado na Tabela 98, o tratamento com oligonucleotídeos antisense diminuiu os níveis de expressão do Fator VII de uma forma dependente de dose, e o oligonucleotídeo compreendendo o conjugado de GalNAc foi significativamente mais potente em macacos em comparação ao oligonucleotídeo em que falta um conjugado de GalNAcTabela 97Oligonucleotídeos direcionados ao Fator VII

[001375] A legenda para a Tabela 97 pode ser encontrada no Exemplo 74. A estrutura de GalNAc3-10a foi mostrada no Exemplo 46.Tabela 98Níveis plasmáticos da proteína Fator VII
Exemplo 92: Inibição antisense em hepatócitos primários por oligonucleotídeos antisense direcionados à Apo-CIIIcompreendendo um conjugado de GalNAc3
[001376] Os hepatócitos primários de camundongo foram semeados em placas de 96 poços em 15.000 células por poço, e os oligonucleotídeos listados na Tabela 99, direcionados à ApoC-III, foram adicionados em 0,46, 1,37, 4,12, ou 12,35, 37,04, 111,11, ou 333,33 nM ou 1,00 μM. Após a incubação com os oligonucleotídeos por 24 horas, as células foram lisadas e o RNA total foi purificado usando RNeasy (Qiagen). Os níveis de mRNA da ApoC-III foram determinados usando PCR em tempo real e reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR) de acordo com protocolos padrão. Os valores de IC50 foram determinados usando o software Prism 4 (GraphPad). Os resultados mostram que independentemente de se a fração clivável era um fosfodiéster ou uma adenosina ligada a fosfodiéster, os oligonucleotídeos que compreendem um conjugado de GalNAc foram significativamente mais potentes do que o oligonucleotídeo de origem em que falta um conjugado.Tabela 99Inibição da expressão de APOC-III de camundongo em hepatócitos primários de camundongo

[001377] A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9, GalNAc3-3a foi mostrado no Exemplo 39, GalNAc3-7a foi mostrado no Exemplo 48, GalNAc3-10a foi mostrado no Exemplo 46, GalNAc3-13a foi mostrado no Exemplo 62, e GalNAc3-19a foi mostrado no Exemplo 70.Exemplo 93: Inibição antisense in vivo por oligonucleotídeos direcionados a SRB-1 compreendendo asas mistas e um conjugado de 5'-GalNAc3
[001378] Os oligonucleotídeos listados na Tabela 100 foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos.Tabela 100ASOs modificados direcionados a SRB-1
[001379] A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39, e a estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48. Subscritos: "e" indica o nucleosídeo modificado por 2'- MOE; "d" indica β-D-2'-desoximbonucleosideo; "k" indica nucleosídeo bicíclico 6'-(S)-CH3 (cEt); "s" indica ligações internucleosídicas de fosforotioato (PS); "o" indica ligações internucleosídicas de fosfodiéster (PO). O sobrescrito "m" indica 5-metilcitosinas.Tratamento
[001380] Camundongos C57BL/6 de seis a oito semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez na dosagem mostrada abaixo com um oligonucleotídeo listado na Tabela 100 ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final. Os níveis hepáticos de mRNA de SRB-1 foram medidos usando PCR em tempo real. Os níveis de mRNA de SRB-1 foram normalizados para os níveis de mRNA de ciclofilina, de acordo com protocolos padrão. Os resultados são apresentados como a porcentagem média dos níveis de mRNA de SRB-1 para cada grupo de tratamento em relação ao grupo controle com salina. Como ilustrado na Tabela 101, o tratamento com oligonucleotídeos antisense diminuiu os níveis de mRNA de SRB-1 de uma forma dependente de dose, e os oligonucleotídeos do gapmer que compreendem um conjugado de GalNAc e têm asas que eram modificações de cEt total ou de açúcar misto foram significativamente mais potentes do que o oligonucleotídeo de origem em que falta um conjugado e que compreende asas modificadas por cEt total.
[001381] Os pesos corporais, transaminases hepáticas, bilirrubina total, e BUN também foram medidos, e os valores médios para cada grupo de tratamento são mostrados na Tabela 101. O peso corporal é mostrado como o peso corporal percentual médio relativo ao peso do corpo de base de referência (% BL) medido imediatamente antes da dose de oligonucleotídeo. Tabela 101Níveis de mRNA de SRB-1, ALT, AST, BUN e bilirrubina total e pesos corporais
Exemplo 94: inibição antisense in vivo por oligonucleotídeos direcionados a SRB-1 compreendendo modificações por 2'- açúcar e um conjugado de 5'-GalNAc3
[001382] Os oligonucleotídeos listados na Tabela 102 foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos. Tabela 102ASOs modificados direcionados a SRB-1
[001383] O subscrito "m" indica um nucleosídeo modificado por 2'-O-metil. Ver o Exemplo 74 para a legenda completa da tabela. A estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39, e a estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48.Tratamento
[001384] O estudo foi completado usando o protocolo descrito no Exemplo 93. Os resultados são mostrados na Tabela 103 abaixo e mostram que os oligonucleotídeos modificados por 2'-MOE e 2'-OMe compreendendo um conjugado de GalNAc foram significativamente mais potentes do que os respectivos oligonucleotídeos de origem em que faltam um conjugado. Os resultados das medições de pesos corporais, transaminases hepáticas, bilirrubina total, e BUN indicam que os compostos foram todos bem tolerados. Tabela 103mRNA de SRB-1
Exemplo 95: Inibição antisense in vivo por oligonucleotídeo direcionados a SRB-1 compreendendo nucleosídeos bicíclicos e um conjugado de 5'-GalNAc3
[001385] Os oligonucleotídeos listados na Tabela 104 foram testados em um estudo dependente de dose para a inibição antisense de SRB-1 em camundongos.Tabela 104ASOs modificados direcionados a SRB-1
[001386] O subscrito "g" indica um nucleosídeo de fluoro-HNA, o subscrito "l" indica um nucleosídeo bloqueado compreendendo uma ponte de 2'-O-CH2-4'. Veja a legenda da tabela do Exemplo 74 para outras abreviações. A estrutura de GalNAc3-1a foi mostrada anteriormente no Exemplo 9, a estrutura de GalNAc3-3a foi mostrada anteriormente no Exemplo 39, e a estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48.Tratamento
[001387] O estudo foi completado usando o protocolo descrito no Exemplo 93. Os resultados são mostrados na Tabela 105 abaixo e mostram que os oligonucleotídeos que compreendem um conjugado de GalNAc e várias modificações de nucleosídeo bicíclico foram significativamente mais potentes do que o oligonucleotídeo de origem em que falta um conjugado e que compreende modificações de nucleosídeo bicíclico. Além disso, o oligonucleotídeo que compreende um conjugado de GalNAc e modificações de fluoro-HNA foi significativamente mais potente do que o de origem em que falta um conjugado e que compreende modificações de fluoro-HNA. Os resultados das medições de pesos corporais, transaminases hepáticas, bilirrubina total, e BUN indicam que os compostos foram todos bem tolerados.Tabela 105Níveis de mRNA de SRB-1, ALT, AST, BUN e bilirrubina total e pesos corporais
Exemplo 96: Ligação de proteína plasmática de oligonucleotídeos antisense que compreendem um grupo de conjugado de GalNAc3
[001388] Os oligonucleotídeos listados na Tabela 70 direcionados àApoC-III e os oligonucleotídeos na Tabela 106 direcionados à Apo(a) foram testados em um ensaio de ultra-filtração a fim de avaliar a ligação da proteína plasmática.Tabela 106Oligonucleotídeos modificados direcionados à Apo(a)
[001389] Veja o Exemplo 74 para a legenda da tabela. A estrutura de GalNAc3-7a foi mostrada anteriormente no Exemplo 48.
[001390] Unidades de ultrafiltração Ultrafree-MC (30.000 NMWL, membrana de celulose regenerada de baixa ligação, Millipore, Bedford, MA) foram pré-condicionadas com 300 μL de 0,5% de Tween 80 e centrifugadas a 2000 g por 10 minutos, em seguida com 300μL de uma solução de 300 μg/mL de um oligonucleotídeo controle em H2O e centrifugadas em 2000 g por 16 minutos. A fim de avaliar a ligação não específica aos filtros de cada oligonucleotídeo de teste das Tabelas 70 e 106 a serem usados nos estudos, 300 μL de uma solução de 250 ng/mL de oligonucleotídeo em H2O em pH 7,4 foram colocados nos filtros pré-condicionados e centrifugados a 2000 g por 16 minutos. As amostras não filtradas e filtradas foram analisadas por um ensaio ELISA para determinar as concentrações do oligonucleotídeo. Três réplicas foram usadas para se obter uma concentração média para cada amostra. A concentração da amostra filtrada em relação à amostra não filtrada é usada para determinar a porcentagem de oligonucleotídeo que é recuperada através do filtro na ausência de plasma (% de recuperação).
[001391] Amostras de plasma inteiras congeladas coletadas em K3- EDTA de voluntários humanos sem drogas normais, macacos cynomolgus e camundongos CD-1 foram compradas da Bioreclamation LLC (Westbury, NY). Os oligonucleotídeos de teste foram adicionados a alíquotas de 1,2 mL de plasma em duas concentrações (5 e 150 μg/mL). Uma alíquota (300 μL) de cada amostra de plasma enriquecida foi colocada em uma unidade de filtro pré-condicionada e incubada a 37°C por 30 minutos, imediatamente após por centrifugação a 2000 g por 16 minutos. As alíquotas das amostras de plasma enriquecidas filtrada e não filtrada foram analisadas por ELISA para determinar a concentração de oligonucleotídeo em cada amostra. Três replicatas por concentração foram usadas para determinar a porcentagem média de oligonucleotídeo ligado e não ligado em cada amostra. A concentração média da amostra filtrada em relação à concentração da amostra não filtrada é usada é usada para determinar a porcentagem de oligonucleotídeo no plasma que não tá ligada às proteínas plasmáticas (% não ligado). Os valores de oligonucleotídeo não ligado final são corrigidos para a ligação não específica, dividindo-se o % não ligado pelo % de recuperação para cada oligonucleotídeo. Os valores de % de oligonucleotídeo ligado final são determinados, subtraindo-se os valores de % não ligado de 100. Os resultados são mostrados na Tabela 107 para as duas concentrações de oligonucleotídeo testadas (5 e 150 μg/mL) em cada espécie de plasma. Os resultados mostram que os grupos de conjugados de GalNAc não têm um impacto significativo sobre a ligação às proteínas plasmáticas. Além disso, os oligonucleotídeos com ligações internucleosídicas PS totais e ligações PO/PS mistas ligam proteínas plasmáticas, e aqueles com ligações PS totais ligam proteínas plasmáticas em um grau um pouco maior do que aqueles com ligações PO/PS mistas.Tabela 107Porcentagem de oligonucleotídeo modificado ligado às proteínas plasmáticas

Exemplo 97: Oligonucleotíd eos modificados d irecionados à TTRcompreendendo um grupo de conjugado de GalNAc3
[001392] Os oligonucleotídeos mostrados na Tabela 108 que compreendem um conjugado de GalNAc foram projetados para se direcionar à TTR. Tabela 108Oligonucleotídeos modificados direcionados à TTR
[001393] A legenda para a Tabela 108 pode ser encontrada no Exemplo 74. A estrutura de GalNAc3-1 foi mostrada no Exemplo 9. A estrutura de GalNAc3-3a foi mostrada no Exemplo 39. A estrutura de GalNAc3-7a foi mostrada no Exemplo 48. A estrutura de GalNAc3-10a foi mostrada no Exemplo 46. A estrutura de GalNAc3-13a foi mostrada no Exemplo 62. A estrutura de GalNAc3-19a foi mostrada no Exemplo 70.Exemplo 98: Avaliação dos efeitos pro-inflamatórios deoligonucleotídeos compreendendo um conjugado de GalNAc no ensaio hPMBC
[001394] Os oligonucleotídeos são listados na Tabela 109 e foram testados para os efeitos pró-inflamatórios em um ensaio hPMBC conforme descrito nos Exemplos 23 e 24. (Veja as Tabelas 30, 83, 95 e 108 para descrições dos oligonucleotídeos.) ISIS 353512 é um alto respondedor usado como um controle positivo, e os outros oligonucleotídeos são descritos nas Tabelas 83, 95 e 108. Os resultados mostrados na Tabela 109 foram obtidos usando sangue de um doador voluntário. Os resultados mostram que os oligonucleotídeos que compreendem ligações internucleosídicas PO/PS mistas produziram respostas pró-inflamatórias significativamente menores em comparação aos mesmos oligonucleotídeos com ligações PS totais. Além disso, o grupo de conjugado de GalNAc não teve um efeito significativo neste ensaio.
Exemplo 99: Afinidades de ligação dos oligonucleotídeos que compreendem um conjugado de GalNAc para o receptor de asialoglicoproteína
[001395] As afinidades de ligação dos oligonucleotídeos listados na Tabela 110 (ver a Tabela 76 para as descrições dos oligonucleotídeos) para o receptor de asialoglicoproteína foram testadas em um ensaio de ligação de receptor competitivo. O ligante competidor, al-glicoproteína ácida (AGP), foi incubada em 50 mM de tampão de acetato de sódio (pH 5) com 1 U de neuraminidase-agarose por 16 horas em 37°C, e > 90% de desialilação foram confirmados pelo ensaio de ácido siálico ou cromatografia de exclusão por tamanho (SEC). Monocloreto de iodo foi usado para iodar o AGP de acordo com o mesmo procedimento por Atsma et al. (ver J Lipid Res. 1991 Jan; 32(1):173-81.) Neste método, a a1- glicoproteína ácida desialilatada (de-AGP) foi adicionada a 10 mM de cloreto de iodo, Na125I, e 1 M de glicina em NaOH 0,25 M. Após a incubação por 10 minutos à temperatura ambiente, de-AGP marcado com125I foi separado de 125I livre, concentrando-se a mistura duas vezes utilizando uma coluna de giro 3 KDMWCO. A proteína foi testada para eficiência de marcação e pureza em um sistema de HPLC equipado com uma coluna Agilent SEC-3 (7,8x300mm) e um contador β-RAM. Experimentos de competição utilizando de-AGP marcado com 125I e vários clusters de GalNAc contendo ASOs foram realizados conforme se segue. Células HepG2 humanas (106 células/ml) foram colocadas em placas de 6 poços em 2 ml de meio de crescimento apropriado. Foram usados o meio MEM complementado com 10% de soro bovino fetal (FBS), 2 mM de L- Glutamina e 10mM de HEPES. As células foram incubadas 16-20 horas em 37°C com 5% e 10% de CO2 respectivamente. As células foram lavadas com meio sem FBS antes do experimento. As células foram incubadas por 30 min em 37°C com 1 ml de mix de competição contendo meio de crescimento apropriado com 2% de FBS, 10-8 M de de-AGP marcado com125I e cluster de GalNAc contendo ASOs em concentrações variando de 10-11 a 10-5 M. Ligações não específicas foram determinadas na presença de 10-2 M de açúcar de GalNAc. As células foram lavadas duas vezes com meio sem FBS para remover o de-AGP marcado com 125I não ligado e o competidor ASO de GalNAc. As células foram lisadas usando um tampão RLT da Qiagen contendo 1% de β-mercaptoetanol. Os lisados foram transferidos para tubos de ensaio de fundo redondo após um breve ciclo de congelamento/descongelamento de 10 min e analisados em um contador y. As ligações não específicas foram subtraídas antes de dividir as contagens de proteína com 125I pelo valor das contagens de concentração mais baixas de GalNAc-ASO. As curvas de inibição foram ajustadas para uma única equação de ligação de competição de sítio usando um algoritmo de regressão não linear para calcular as afinidades de ligação (KD's).
[001396] Os resultados na Tabela 110 foram obtidos a partir de experimentos realizados em cinco dias diferentes. Os resultados para os oligonucleotídeos marcados com sobrescrito "a" são a média dos experimentos realizados em dois dias diferentes. Os resultados mostram que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc na extremidade 5' ligavam-se ao receptor de asialoglicoproteína nas células HepG2 humanas com afinidade 1,5 a 16 vezes maior do que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc na extremidade 3'. Tabela 110Resultados do ensaio de ligação ao receptor de asialoglicoproteína
Exemplo 100: Inibição antisense in vivo por oligonucleotídeos compreendendo um grupo de conjugado de GalNAc direcionados à Apo(a) in vivo
[001397] Os oligonucleotídeos listados na Tabela 111a abaixo foram testados em um estudo de dose única para a duração da ação em camundongos.Tabela 111aASOs modificados direcionados à APO(a)
[001398] A estrutura de GalNAc3-7a foi mostrada no Exemplo 48. Tratamento
[001399] Camundongos transgênicos fêmeas que expressam a Apo(a) humana foram, cada um, injetados por via subcutânea uma vez por semana, por um total de 6 doses, com um oligonucleotídeo e a dosagem listada na Tabela 111b ou com PBS. Cada grupo de tratamento consistiu em 3 animais. O sangue foi retirado no dia antes da dosagem para determinar os níveis de base de referência da proteína Apo(a) no plasma e em 72 horas, 1 semana e 2 semanas após a primeira dose. Retiradas adicionais de sangue ocorrerão em 3 semanas, 4 semanas, 5 semanas e 6 semanas após a primeira dose. Os níveis da proteína Apo(a) foram medidos usando um ELISA. Os resultados na Tabela 111b são apresentados como a porcentagem média dos níveis da proteína Apo(a) no plasma para cada grupo de tratamento, normalizados para os níveis de base de referência (% BL). Os resultados mostram que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc exibiam potente redução na expressão de Apo(a). Este potente efeito foi observado para o oligonucleotídeo que compreende ligações internucleosídicas PS totais e para o oligonucleotídeo que compreende ligações PO e PS mistas.Tabela 111bNíveis plasmáticos da proteína Apo(a)


Exemplo 101: Inibição antisense por oligonucleotídeoscompreendendo um cluster de GalNAc ligado através de uma fração estável
[001400] Os oligonucleotídeos listados na Tabela 112 foram testados para a inibição da expressão de APOC-III de camundongos in vivo. Camundongos C57Bl/6 foram, cada um, injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 112 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Cada camundongo tratado com ISIS 440670 recebeu uma dose de 2, 6, 20, ou 60 mg/kg. Cada camundongo tratado com ISIS 680772 ou 696847 recebeu 0,6, 2, 6, ou 20 mg/kg. O grupo de conjugado de GalNAc de ISIS 696847 é ligado através de uma fração estável, uma ligação fosforotioato em vez de uma ligação contendo fosfodiéster facilmente clivável. Os animais foram sacrificados 72 horas após a dose. Os níveis hepáticos de mRNA de APOC-III foram medidos usando PCR em tempo real. Os níveis de mRNA de APOC-III foram normalizados para os níveis de mRNA de ciclofilina, de acordo com protocolos padrão. Os resultados são apresentados na Tabela 112 como a porcentagem média dos níveis de mRNA de APOC-III para cada grupo de tratamento em relação ao grupo controle com salina. Os resultados mostram que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAC foram significativamente mais potentes do que o oligonucleotídeo em que falta um grupo de conjugado. Além disso, o oligonucleotídeo que compreende um grupo de conjugado de GalNAc ligado ao oligonucleotídeo através de uma fração clivável(ISIS 680772) foi até mais potente do que o oligonucleotídeo que compreende um grupo de conjugado de GalNAc ligado ao oligonucleotídeo através de uma fração estável (ISIS 696847).Tabela 112Oligonucleotídeos modificados direcionados à APOC-III de camundongo

[001401] A estrutura de GalNAc3-7a foi mostrada no Exemplo 48.Exemplo 102: Distribuição no fígado de oligonucleotídeos antisense compreendendo um conjugado de GalNAc
[001402] A distribuição no fígado de ISIS 353382 (ver a Tabela 36) que não compreende um conjugado de GalNAc e ISIS 655861 (ver a Tabela 36) que compreende um conjugado de GalNAc foi avaliada. Camundongos balb/c machos foram injetados por via subcutânea uma vez com ISIS 353382 ou 655861 numa dosagem listada na Tabela 113. Cada grupo de tratamento consistiu em 3 animais, exceto para o grupo de 18 mg/kg para ISIS 655861, que consistiu em 2 animais. Os animais foram sacrificados 48 horas após a dose para determinar a distribuição no fígado dos oligonucleotídeos. A fim de medir o número de moléculas de oligonucleotídeo antisense por célula, um marcador de tris-bipiridina de rutênio(II) (MSD TAG, Meso Scale Discovery) foi conjugado a uma sonda de oligonucleotídeo usada para detectar os oligonucleotídeos antisense. Os resultados apresentados na Tabela 113 são as concentrações médias de oligonucleotídeo para cada grupo de tratamento em unidades de milhões de moléculas de oligonucleotídeo por célula. Os resultados mostram que, em doses equivalentes, o oligonucleotídeo que compreende um conjugado de GalNAc esteve presente em concentrações maiores o fígado total e em hepatócitos do que o oligonucleotídeo que não compreende um conjugado de GalNAc. Além disso, o oligonucleotídeo que compreende um conjugado de GalNAc esteve presente em concentrações mais baixas em células hepáticas não-parenquimatosas do que o oligonucleotídeo que não compreende um conjugado de GalNAc. E, embora as concentrações de ISIS 655861 em hepatócitos e em células hepáticas não- parenquimatosas sejam semelhantes por célula, o fígado é aproximadamente 80% de hepatócitos em volume. Assim, a maioria do oligonucleotídeo ISIS 655861 que esteve presente no fígado foi encontrada nos hepatócitos, considerando que a maioria do oligonucleotídeo ISIS 353382 que esteve presente no fígado foi encontrada em células hepáticas não-parenquimatosas.

Exemplo 103: Duração da ação in vivo de o igonucleotídeosdirecionados à APOC-III compreendendo um conjugado de GalNAc3
[001403] Os oligonucleotídeos listados na Tabela 114 abaixo foram testados em um estudo de dose única para a duração da ação em camundongos.Tabela 114ASOs modificados direcionados a APOC-III
[001404] A estrutura de GalNAc3-3a foi mostrada no Exemplo 39, e GalNAc3-19a foi mostrado no Exemplo 70.Tratamento
[001405] Camundongos transgênicos fêmeas que expressam a APOC-III humana foram, cada um, injetados por via subcutânea uma vez com um oligonucleotídeo listado na Tabela 114 ou com PBS. Cada grupo de tratamento consistiu em 3 animais. O sangue foi retirado antes da dosagem para determinar a base de referência e em 3, 7, 14, 21, 28, 35 e 42 dias após a dose. Os níveis de triglicerídeos plasmáticos e da proteína APOC-III foram medidos conforme descrito no Exemplo 20. Os resultados na Tabela 115 são apresentados como a percentagem média dos níveis de triglicerídeos plasmáticos e APOC-III para cada grupo de tratamento, normalizados para os níveis de base de referência. Uma comparação dos resultados na Tabela 71 do Exemplo 79 com os resultados na Tabela 115 abaixo mostra que os oligonucleotídeos que compreendem uma mistura de ligações internucleosídicas de fosfodiéster e fosforotioato exibiram maior duração de ação do que os oligonucleotídeos equivalentes que compreendem apenas ligações internucleosídicas de fosforotioato. Tabela 115Níveis de triglicerídeos plasmáticos e da proteína APOC-III em camundongos transgênicos

Exemplo 104: Síntese de oligonucleotídeos compreendend o umconjugado de 5'-GalNAC2
[001406] O composto 120 está disponível comercialmente, e a síntese do composto 126 é descrita no Exemplo 49. O composto 120 (1 g, 2,89 mmol), HBTU (0,39 g, 2,89 mmol), e HOBt (1,64 g, 4,33 mmol) foram dissolvidos em DMF (10 mL) e N,N-di-isopropiletilamina (1,75 mL, 10,1 mmol) foi adicionada. Após cerca de 5 min, éster benzílico de ácido aminohexanoico (1,36 g, 3,46 mmol) foi adicionado à reação. Após 3 h, a mistura de reação foi vertida em 100 mL de 1 M de NaHSO4 e extraída com 2 x 50 mL de acetato de etil. As camadas orgânicas foram combinadas e lavadas com 3 x 40 mL de NaHCO3 saturado e 2 x de salmoura, secas com Na2SO4, filtradas e concentradas. O produto foi purificado por cromatografia de coluna em gel de sílica (DCM:EA:Hex, 1:1:1) para produzir o composto 231. LCMS e NMR foram consistentes com a estrutura. O composto 231 (1,34 g, 2,438 mmol) foi dissolvido em diclorometano (10 mL) e ácido trifluoracético (10 mL) foi adicionado. Após agitação em temperatura ambiente por 2 h, a mistura de reação foi concentrada sob pressão reduzida e co-evaporada com tolueno (3 x 10 mL). O resíduo foi seco sob pressão reduzida para produzir o composto 232 como sal de trifluoroacetato. A síntese do composto 166 é descrita no Exemplo 54. O composto 166 (3,39 g, 5,40 mmol) foi dissolvido em DMF (3 mL). Uma solução do composto 232 (1,3 g, 2,25 mmol) foi dissolvida em DMF (3 mL) e N,N-di-isopropiletilamina (1,55 mL) foi adicionada. A reação foi agitada à temperatura ambiente por 30 minutos, em seguida, vertida em água (80 mL) e a camada aquosa foi extraída com EtOAc (2x100 mL). A fase orgânica foi separada e lavada com NaHCO3 aquoso saturado (3 x 80 mL), 1 M de NaHSO4 (3 x 80 mL) e salmoura (2 x 80 mL), em seguida, seca (Na2SO4), filtrada e concentrada. O resíduo foi purificado por cromatografia de coluna em gel de sílica para produzir o composto 233. LCMS e NMR foram consistentes com a estrutura. O composto 233 (0,59 g, 0,48 mmol) foi dissolvido em metanol (2,2 mL) e acetato de etil (2,2 mL). Paládio em carbono (10% em peso de Pd/C, úmido, 0,07 g) foi adicionado, e a mistura de reação foi agitada sob atmosfera de hidrogênio por 3 h. A mistura de reação foi filtrada através de uma camada de Celite e concentrada para produzir o ácido carboxílico. O ácido carboxílico (1,32 g, 1,15 mmol, ácido sem cluster) foi dissolvido em DMF (3,2 mL). A isto, N,N-di- isopropiletilamina (0,3 mL, 1,73 mmol) e PFPTFA (0,30 mL, 1,73 mmol) foram adicionados. Após 30 min de agitação à temperatura ambiente, a mistura de reação foi vertida em água (40 mL) e extraída com EtOAc (2 x 50 mL). Um acompanhamento padrão foi concluído conforme descrito acima para produzir o composto 234. LCMS e NMR foram consistentes com a estrutura. O oligonucleotídeo 235 foi preparado usando o procedimento geral descrito no Exemplo 46. A parte de cluster de GalNAC2 (GalNAC2-24a) do grupo de conjugado GalNAC2- 24 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC2-24 (GalNAC2-24a-CM) é mostrada abaixo:

Exemplo 105: Síntese de oligonucleotídeos compreendendo um conjugado de GalNACi-25
[001407] A síntese do composto 166 é descrita no Exemplo 54. O oligonucleotídeo 236 foi preparado usando o procedimento geral descrito no Exemplo 46.
[001408] Alternativamente, o oligonucleotídeo 236 foi sintetizado usando o esquema abaixo, e o composto 238 foi usado para formar o oligonucleotídeo 236 usando os procedimentos descritos no Exemplo 10.
[001409] A parte de cluster de GalNAC1 (GalNAC1-25a) do grupo de conjugado GalNAC1-25 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC1-25 (GalNAC1-25a-CM) é mostrada abaixo:
Exemplo 106: inibição antisense in vivo por oligonucleotídeos direcionados a SRB-1 compreendendo um conjugado de 5'- GalNAC2 ou de 5'-GalNAc3
[001410] Os oligonucleotídeos listados nas Tabelas 116 e 117 foram testados em estudos dependentes de dose para uma inibição antisense de SRB-1 em camundongos.Tratamento
[001411] Camundongos C57BL/6 machos de seis semanas de idade (Jackson Laboratory, Bar Harbor, ME) foram injetados por via subcutânea uma vez com 2, 7, ou 20 mg/kg de N°. ISIS 440762; ou com 0,2, 0,6, 2, 6, ou 20 mg/kg de N°. ISIS 686221, 686222, ou 708561; ou com salina. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados 72 horas após a administração final. Os níveis hepáticos de mRNA de SRB-1 foram medidos usando PCR em tempo real. Os níveis de mRNA de SRB-1 foram normalizados para os níveis de mRNA de ciclofilina, de acordo com protocolos padrão. Os oligonucleotídeos antisense diminuíram os níveis de mRNA de SRB-1 de uma forma dependente de dose, e os resultados de ED50 são apresentados nas Tabelas 116 e 117. Embora estudos anteriores mostrem que os oligonucleotídeos conjugados a GalNAc trivalentes foram significativamente mais potentes do que os oligonucleotídeos conjugados a GalNAc, que foram, por sua vez, significativamente mais potentes do que os oligonucleotídeos conjugados a GalNAc monovalentes (ver, por exemplo, Khorev et al., Bioorg. & Med. Chem., Vol. 16, 5216-5231 (2008)), o tratamento com oligonucleotídeosantisense compreendendo clusters de GalNAc monovalentes, divalentes e trivalentes diminuiu os níveis de mRNA de SRB-1 com potências semelhantes, como mostrado nas Tabelas 116 e 117.Tabela 116Oligonucleotídeos modificados direcionados a SRB-1

[001412] Veja o Exemplo 93 para a legenda da tabe a. A estrutura deGalNAc3-13a foi mostrada no Exemplo 62, e a estrutura de GalNAC2- 24a foi mostrada no Exemplo 104.Tabela 117Oligonucleotídeos modificados direcionados a SRB-1
[001413] Veja o Exemplo 93 para a legenda da tabe a. A estrutura deGalNAC1-25a foi mostrada no Exemplo 105.
[001414] As concentrações dos oligonucleotídeos nas Tabelas 116 e 117 no fígado também foram avaliadas, usando os procedimentos descritos no Exemplo 75. Os resultados mostrados nas Tabelas 117a e 117b abaixo são os níveis médios totais teciduais do oligonucleotídeo antisense para cada grupo de tratamento, conforme medido por UV em unidades de μg de oligonucleotídeo por grama de tecido hepático. Os resultados mostram que os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc acumulado no fígado em níveis significativamente mais altos do que a mesma dose do oligonucleotídeo em que falta um grupo de conjugado de GalNAc. Além disso, os oligonucleotídeos antisense que compreendem um, dois ou três ligantes de GalNAc em seus respectivos grupos de conjugado todos se acumularam o fígado em níveis semelhantes. Este resultado é surpreendente em vista da referência da literatura de Khorev et al. citada acima e é consistente com os dados de atividade mostrados nas Tabelas 116 e 117 acima.Tabela 117aConcentrações hepáticas de oligonucleotídeos compreendendo um grupo de conjugado de GalNAC2 ou de GalNAc3

Tabela 117bConcentrações hepáticas de oligonucleotídeos compreendendo um grupo de conjugado de GalNAC1
Exemplo 107: síntese de oligonucleotídeos compreendendo um conjugado de GalNAci-26 ou de GalNAci-27
[001415] O oligonucleotídeo 239 é sintetizado através do acoplamento do composto 47 (ver o Exemplo 15) ao ácido 64 (ver o Exemplo 32) usando HBTU e DIEA em DMF. O composto contendo amida resultante é fosfitilado, em seguida, adicionado à extremidade 5' de um oligonucleotídeo usando os procedimentos descritos no Exemplo 10. A parte de cluster de GalNAC1 (GalNAC1-26a) do grupo de conjugado GalNAC1-26 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC1-26 (GalNAC1-26a-CM) é mostrada abaixo:
[001416] A fim de adicionar o grupo de conjugado de GalNAC1 à extremidade 3' de um oligonucleotídeo, a amida formada a partir da reação dos compostos 47 e 64 é adicionada a um suporte sólido usando os procedimentos descritos no Exemplo 7. A síntese do oligonucleotídeo é então concluída usando os procedimentos descritos no Exemplo 9 a fim de formar o oligonucleotídeo 240.
[001417] A parte de cluster de GalNAC1 (GalNAC1-27a) do grupo de conjugado GalNAC1-27 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC1-27 (GalNAC1-27a-CM) é mostrada abaixo:
Exemplo 108: Inibição antisense in vivo por oligonucleotídeos compreendendo um grupo de conjugado de GalNAc direcionados à Apo(a) in vivo
[001418] Os oligonucleotídeos listados na Tabela 118 abaixo foram testados em um estudo de dose única em camundongos. Tabela 118ASOs modificados direcionados à APO(a)
[001419] A estrutura de GalNAc3-7a foi mostrada no Exemplo 48. Tratamento
[001420] Camundongos transgênicos machos que expressam a Apo(a) humana foram, cada um injetados por via subcutânea uma vez com um oligonucleotídeo e dosagem listados na Tabela 119 ou com PBS. Cada grupo de tratamento consistiu em 4 animais. O sangue foi retirado no dia antes da dosagem para determinar os níveis de base de referência da proteína Apo(a) no plasma e em 1 semana após a primeira dose. Retiradas adicionais de sangue ocorrerão semanalmente por aproximadamente 8 semanas. Os níveis da proteína Apo(a) foram medidos usando um ELISA. Os resultados na Tabela 119 são apresentados como a porcentagem média dos níveis da proteína Apo(a) plasmática para cada grupo de tratamento, normalizados para os níveis de base de referência (% BL). Os resultados mostram que os oligonucleotídeos antisense reduziram a expressão da proteína Apo(a). Além disso, os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc exibiram uma redução até mais potente da expressão de Apo(a) do que os oligonucleotídeos que não compreendem um grupo de conjugado.Tabela 119Níveis plasmáticos da proteína Apo(a)

Exemplo 109 1: Síntese de oligonucleotídeos compreendendo umconjugado de GalNAci-28 ou de GalNAci-29
[001421] O oligonucleotídeo 241 é sintetizado usando procedimentos semelhantes àqueles descritos no Exemplo 71 para formar o intermediário fosforamidita, seguido pelos procedimentos descritos no Exemplo 10 para sintetizar o oligonucleotídeo. A parte de cluster de GalNAC1 (GalNAC1-28a) do grupo de conjugado GalNAC1-28 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC1-28 (GalNAC1-28a-CM) é mostrada abaixo:
[001422] A fim de adicionar o grupo de conjugado de GalNAC1 à extremidade 3' de um oligonucleotídeo, procedimentos semelhantes àqueles descritos no Exemplo 71 são usados para formar o intermediário hidroxil, que é então adicionado ao suporte sólido usando os procedimentos descritos no Exemplo 7. A síntese do oligonucleotídeo é então concluída usando os procedimentos descritos no Exemplo 9 a fim de formar o oligonucleotídeo 242.
[001423] A parte de cluster de GalNAC1 (GalNAC1-29a) do grupo de conjugado GalNAC1-29 pode ser combinada com qualquer fração clivável presente no oligonucleotídeo para fornecer uma variedade de grupos de conjugado. A estrutura de GalNAC1-29 (GalNAC1-29a-CM) é mostrada abaixo:
Exemplo 110: Síntese de oligonucleotídeos compreendendo umconjugado de GalNAci-30
[001424] O oligonucleotídeo 246 compreendendo um grupo de conjugado de GalNAC1-30, em que Y é selecionado de O, S, um alquil C1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil, é sintetizado como mostrado acima. A parte de cluster de GalNAC1 (GalNAC1-30a) do grupo de conjugado de GalNAC1- 30 pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, o símbolo Y é parte da fração clivável. Em determinadas modalidades, Y é parte de uma fração estável, e a fração clivável está presente no oligonucleotídeo. A estrutura de GalNAC1-30a é mostrada abaixo:
Exemplo 111: A síntese de oligonucleotídeos compreendendo um conjugado de GalNAc2-31 ou de GalNAc2-32
[001425] O oligonucleotídeo 250 compreendendo um grupo de conjugado de GalNAC2-31, em que Y é selecionado de O, S, um alquil C1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil, é sintetizado como mostrado acima. A parte de cluster de GalNAC2 (GalNAC2-31a) do grupo de conjugado GalNAC2-31 pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, o grupo contendo Y diretamente adjacente à extremidade 5' do oligonucleotídeo é parte da fração clivável. Em determinadas modalidades, o grupo contendo Y diretamente adjacente à extremidade 5' do oligonucleotídeo é parte de uma fração estável, e a fração clivável está presente no oligonucleotídeo. A estrutura de GalNAC2-31a é mostrada abaixo:
[001426] A síntese de um oligonucleotídeo compreendendo um conjugado de GalNAC2-32 é mostrada abaixo.
[001427] O oligonucleotídeo 252 compreendendo um grupo de conjugado de GalNAC2-32, em que Y é selecionado de O, S, um alquilC1-C10 substituído ou não substituído, amino, amino substituído, azido, alquenil ou alquinil, é sintetizado como mostrado acima. A parte de cluster de GalNAC2 (GalNAC2-32a) do grupo de conjugado GalNAC2-32 pode ser combinada com qualquer fração clivável para fornecer uma variedade de grupos de conjugado. Em determinadas modalidades, o grupo contendo Y diretamente adjacente à extremidade 5' do oligonucleotídeo é parte da fração clivável. Em determinadas modalidades, o grupo contendo Y diretamente adjacente à extremidade 5' do oligonucleotídeo é parte de uma fração estável, e a fração clivável está presente no oligonucleotídeo. A estrutura de GalNAC2-32a é mostrada abaixo:
Exemplo 112: Oligonucleotídeo modificados compreendendo um conjugado de GalNAci
[001428] Os oligonucleotídeos na Tabela 120 direcionados a SRB-1 foram sintetizados com um grupo de conjugado de GalNAC1 a fim de testar ainda mais a potência dos oligonucleotídeos que compreendem grupos de conjugado que contêm um ligante de GalNAc.
Exemplo 113: Oligonucleotídeos antisense que direcionam semelhante à angiopoietina 3 e compreendem um grupo conjugado GalNac
[001429] Os oligonucleotídeos listados na Tabela 121 foram projetados para direcionar semelhante à angiopoietina 3 (ANGPTL3).
Exemplo 114: Inibição anti-sense in vivo por oligonucleotídeoscompreendendo um grupo GalNAc conjugado de segmentação humana ANGPTL3
[001430] Camundongos transgênicos C57BL/6 do sexo masculino de seis semanas de idade, que expressam ANGPTL3 humano foram cada um injetados intraperitonealmente uma vez por semana a uma dosagem mostrada abaixo, para um total de duas doses, com um oligonucleotídeo listados na Tabela 122 (e descrito na Tabela 121) ou com PBS. Cada grupo de tratamento consistiu em 4 animais. Os camundongos foram sacrificados em dois dias após a última dose. Os níveis de mRNA de fígado ANGPTL3 foram medidos utilizando PCR em tempo real e a reagente de quantificação de RNA RIBOGREEN® (Molecular Probes, Inc. Eugene, OR), de acordo com protocolos padrão. Os resultados abaixo são apresentados como a porcentagem média de níveis mRNA ANGPTL3 no fígado para cada grupo de tratamento, normalizados para o controle com PBS.Tal como ilustrado na Tabela 122, tratamentocom oligonucleotídeos anti-sense com os reduzido os níveis de mRNA de fígado ANGPTL3 de uma forma dependente da dose, e o oligonucleotídeo que compreende um conjugado de GalNAc foi significativamente mais potente do que o oligonucleotídeo de origem sem um conjugado GalNAc. Tabela 122Os níveis de mRNA do fígado ANGPTL3

[001431] Níveis de alanina aminotransferase do fígado (ALT) também foram medidos na altura do sacrifício utilizando protocolos padrão. Os resultados têm mostrado que nenhum dos grupos de tratamento tinham níveis elevados de ALT, indicando que os oligonucleotídeos foram bem tolerados.Exemplo 115: Inibição anti-sense in vivo por oligonucleotídeos compreendendo um grupo GalNAc conjugado de segmentação do camundongo ANGPTL3
[001432] Os oligonucleotídeos listados na Tabela 123 abaixo foram testados em um estudo dependente da dose em camundongos.
[001433] A estrutura de GalNAc3-7a foi apresentada no Exemplo 48.
[001434] Receptor de lipoproteína de baixa densidade dos camundongos de knockout (LDLR-/-) foram alimentados com uma dieta ocidental durante 1 semana antes de ser injetado por via intraperitoneal uma vez por semana a uma dosagem mostrada abaixo com um oligonucleotídeo listado na Tabela 123 ou com PBS. Cada grupo de tratamento consistiu em 5 animais. O sangue foi colhido antes da primeira dose ser administrada, a fim de determinar os níveis de linha de base de triglicerídeos no plasma e às 2 semanas após a primeira dose. Os resultados apresentados na Tabela 124 são apresentados como a porcentagem média dos níveis de triglicerídeos no plasma para cada grupo de tratamento, normalizados para níveis de linha de base (BL%), os resultados mostram que os oligonucleotídeos anti-sense reduziu os triglicerídeos de uma maneira dependente da dose. Além disso, os oligonucleotídeos que compreendem um grupo de conjugado de GalNAc exibiram uma redução até mais potente nos triglicerídeos do que os oligonucleotídeos que não compreendem um grupo de conjugado. Tabela 124Níveis de triglicerídeos plasmáticos (TG)

Exemplo 116: Inibição anti-sense de Angiopoietina-do tipo 3 humana como em células Hep3B por gapmeros MOE
[001435] Oligonucleotídeos anti-sense foram concebidos segmentando um ácido nucleico (ANGPTL3) de angiopoietina-do tipo 3 e foram testados quanto aos seus efeitos sobre o mRNA ANGPTL3 in vitro. Os oligonucleotídeos antisense foram testados em uma série de experimentos que tinham condições de cultura semelhantes. Os resultados para cada experimento são apresentados em tabelas separadas mostradas abaixo. As células Hep3B cultivadas em uma densidade de 20.000 células por poço foram transfectadas usando eletroporação com 4.500 nM do oligonucleotídeo antisense. Após um período de tratamento de aproximadamente 24 horas, o RNA foi isolado das células e os níveis de mRNA de ANGPTL3 foram medidos por PCR quantitativa em tempo real. Conjunto de sonda humana de iniciadores RTS3492_MGB (sequência inicial CCGTGGAAGACCAATATAAACAATT, aqui designada como SEQ ID NO:4; AGTCCTTCTGAGCTGATTT- TCTATTTCT; sequência inversa, aqui designada como SEQ ID NO: 5; sequência da sonda AACCAACAGCATAGTCAAATA, aqui designada como SEQ ID NO: 6) foi utilizada para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.Os oligonucleotídeos antisense quiméricos recém-concebidos nas Tabelas abaixo foram concebidos como 5-10-5 gapmers MOE. Os gapmers 5-10-5 MOE têm 20 nucleosídeos de comprimento, nos quais o segmento de lacuna central compreende dez 2'-desoxinucleosídeos e é flanqueado por segmentos de asa na direção 5' e direção 3' que compreende cinco nucleosídeos cada. Cada nucleosídeo no segmento 5' wing e cada nucleosídeo no segmento 3' wing tem uma modificação em 2'-MOE. As ligações internucleosídicas ao longo de cada gapmer são ligações fosforotioato (P=S). Todos os resíduos de citosina ao longo de cada gapmer são 5-metilcitosinas. "Sítio de iniciação" indica o nucleosídeo mais extremo em 5' para o qual o gapmer é direcionado na sequência do gene humano. "Sítio de parada" indica o 3'- nucleosídeos a mais ao qual o gapmer é direcionado na sequência do gene humano. Cada gapmer listado nas tabelas abaixo é voltado para o mRNA, quer ANGPTL3 humana, aqui designado como SEQ ID NO: 1 (número de acesso GenBank NM_014495.2) ou a sequência genômica ANGPTL3 humana, aqui designada como SEQ ID NO: 2 (No. de Acesso GenBank NT_032977.9 truncado de nucleotídeos 33032001-33.046.000). 'N/a' indica que o oligonucleotídeo anti-sense não segmentado da sequência de gene particular com 100% de complementaridade. Tabela 125Inibição de mRNA ANGPTL3 pela segmentação de gapmers 5-10-5MOE SEQ ID NO: 1 e 2










































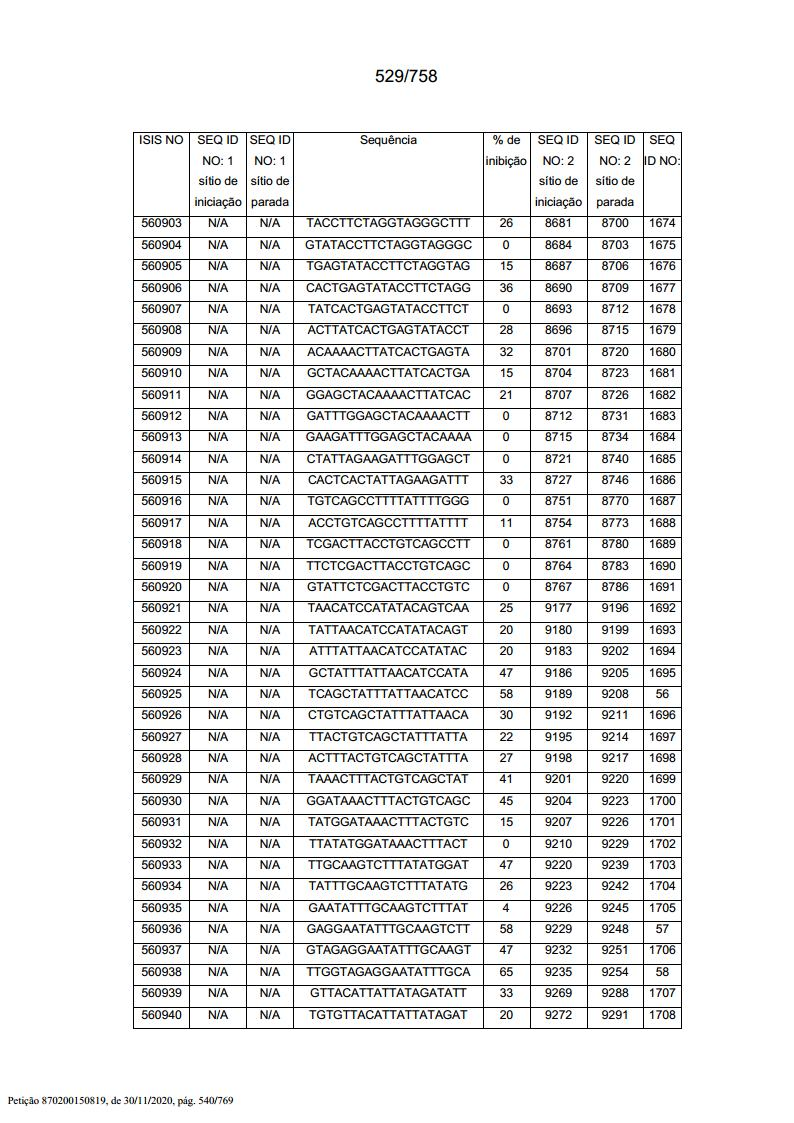






































Exemplo 117: Inibição anti-sense de dose-dependente deANGPTL3 humano em células Hep3B por gapmers MOE
[001436] Gapmers 5-10-5 MOE a partir dos estudos descritos acima apresentam significativa inibiçãoin vitro de ANGPTL3 de mRNA foram selecionados e testados em diversas doses em células Hep3B. As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de0.75 μM, 1.50 μM, 3.00 μM, 6.00 μM e 12.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001437] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucleotídeo anti-sense.
Exemplo 118: Inibição anti-sense de dose-dependente deANGPTL3 humano em células Hep3B por gapmers MOE
[001438] Gapmers 5-10-5 MOE a partir dos estudos descritos acima apresentam significativa inibiçãoin vitro de ANGPTL3 de mRNA foram selecionados e testados em diversas doses em células Hep3B. As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de 0.813 μM, 1.625 μM, 3.25 μM, 6.50 μM e 13.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001439] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucleotídeo anti-sense.
Exemplo 119: Inibição anti-sense de ANGPTL3 humano em células Hep3B por desoxi, MOE e (S)-cEt gapmers
[001440] Os oligonucleotídeos antisense adicionais foram projetados para se direcionar para um ácido nucleico de ANGPTL3 e foram testados para seus efeitos no mRNA do ANGPTL3 in vitro. As células Hep3B cultivadas em uma densidade de 20.000 células por poço foram transfectadas usando eletroporação com 4.500 nM do oligonucleotídeo antisense. Após um período de tratamento de aproximadamente 24 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001441] Os recém-concebidos oligonucleotídeos antisenses quiméricos nas Tabelas abaixo foram concebidos como oligonucleotídeos desoxi, MOE e (S)-cEt. Os oligonucleotídeos (S)-cEt, MOE e deoxi são 16 nucleosídeos de comprimento, nos quais o nucleosídeo tem uma modificação de açúcar MOE, uma modificação de açúcar (S)-cEt, ou um resíduo desoxi de açúcar. As modificações de açúcar de cada oligonucleotídeo antisense é descrita como 'eek-d10-KKE', onde 'k' indica uma (S)-cEt modificação de açúcar; 'd' indica desoxirribose; o número indica o número de resíduos de açúcares desoxirribose; e 'e' indica uma modificação de açúcar MOE. As ligações internucleosídicas ao longo de cada oligonucleotídeo são ligações fosforotioato (P=S). Todos os resíduos de citosina ao longo de cada oligonucletídeo são 5- metilcitosinas. "Sítio de partida" indica o nucleosídeo mais extremo em 5' para o qual o oligonucleotídeo é direcionado na sequência do gene humano. "Sítio de parada" indica o nucleosídeo mais extremo em 3' para o qual o oligonucleotídeo é sequência do gene humano segmentada. Cada oligonucleotídeo listado nas tabelas abaixo é voltado para o mRNA, quer ANGPTL3 humana, aqui designado como SEQ ID NO: 1 (número de acesso GenBank NM_014495.2) ou a sequência genômica ANGPTL3 humana, aqui designada como SEQ ID NO: 2 (No. de Acesso GenBank NT_032977.9 truncado de nucleotídeos 3303200133.046.000). 'N/a' indica que o oligonucleotídeo anti-sense não segmentado da sequência de gene particular com 100% de complementaridade.Tabela 148Inibição de mRNA ANGPTL3 por desoxi, MOE e cET oligonucleotídeos alvo SEQ ID NO: 1 e 2











Exemplo 120: Inibição anti-sense de ANGPTL3 humano em cé ulasHep3B por desoxi, MOE e (S)-cEt gapmers
[001442] Os oligonucleotídeos antisense adicionais foram projetados para se direcionar para um ácido nucleico de ANGPTL3 e foram testados para seus efeitos no mRNA do ANGPTL3 in vitro. ISIS 337487 e ISIS 233717, que são gapmers MOE 5-10-5, foram também incluídos no ensaio como referência de oligonucleotídeos. As células Hep3B cultivadas em uma densidade de 20.000 células por poço foram transfectadas usando eletroporação com 4.500 nM do oligonucleotídeo antisense. Após um período de tratamento de aproximadamente 24 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001443] Os recém-concebidos oligonucleotídeos antisenses quiméricos nas Tabelas abaixo foram concebidos como oligonucleotídeos desoxi, MOE e (S)-cEt ou gapmers MOE 5-10-5. Os oligonucleotídeos (S)-cEt, MOE e deoxi são 16 nucleosídeos de comprimento, nos quais o nucleosídeo tem uma modificação de açúcar MOE, uma modificação de açúcar (S)-cEt, ou um resíduo desoxi de açúcar. As modificações de açúcar de cada oligonucleotídeo antisense é descrita como 'eek-d10- KKE', onde 'k' indica uma (S)-cEt modificação de açúcar; 'd' indica desoxirribose; o número indica o número de resíduos de açúcares desoxirribose; e 'e' indica uma modificação MOE. Os gapmers 5-10-5 MOE têm 20 nucleosídeos de comprimento, nos quais o segmento de lacuna central compreende dez 2'-desoxinucleosídeos e é flanqueado por segmentos de asa na direção 5' e direção 3' que compreende cinco nucleosídeos cada. As ligações internucleosídicas ao longo de cada oligonucleotídeo são ligações fosforotioato (P=S). Todos os resíduos de citosina ao longo de cada oligonucletídeo são 5-metilcitosinas. "Sítio de partida" indica o nucleosídeo mais extremo em 5' para o qual ooligonucleotídeo é direcionado na sequência do gene humano. "Sítio de parada" indica o nucleosídeo mais extremo em 3' para o qual ooligonucleotídeo é sequência do gene humano segmentada. Cada oligonucleotídeo listado nas tabelas abaixo é voltado para o mRNA, quer ANGPTL3 humana, aqui designado como SEQ ID NO: 1 (número de acesso GenBank NM_014495.2) ou a sequência genômica ANGPTL3 humana, aqui designada como SEQ ID NO: 2 (No. de Acesso GenBank NT_032977.9 truncado de nucleotídeos 33032001-33.046.000). 'N/a' indica que o oligonucleotídeo anti-sense não segmentado da sequência de gene particular com 100% de complementaridade.Tabela 150Inibição de mRNA ANGPTL3 por oligonucleotídeos alvo SEQ ID NO: 1 e 2




















































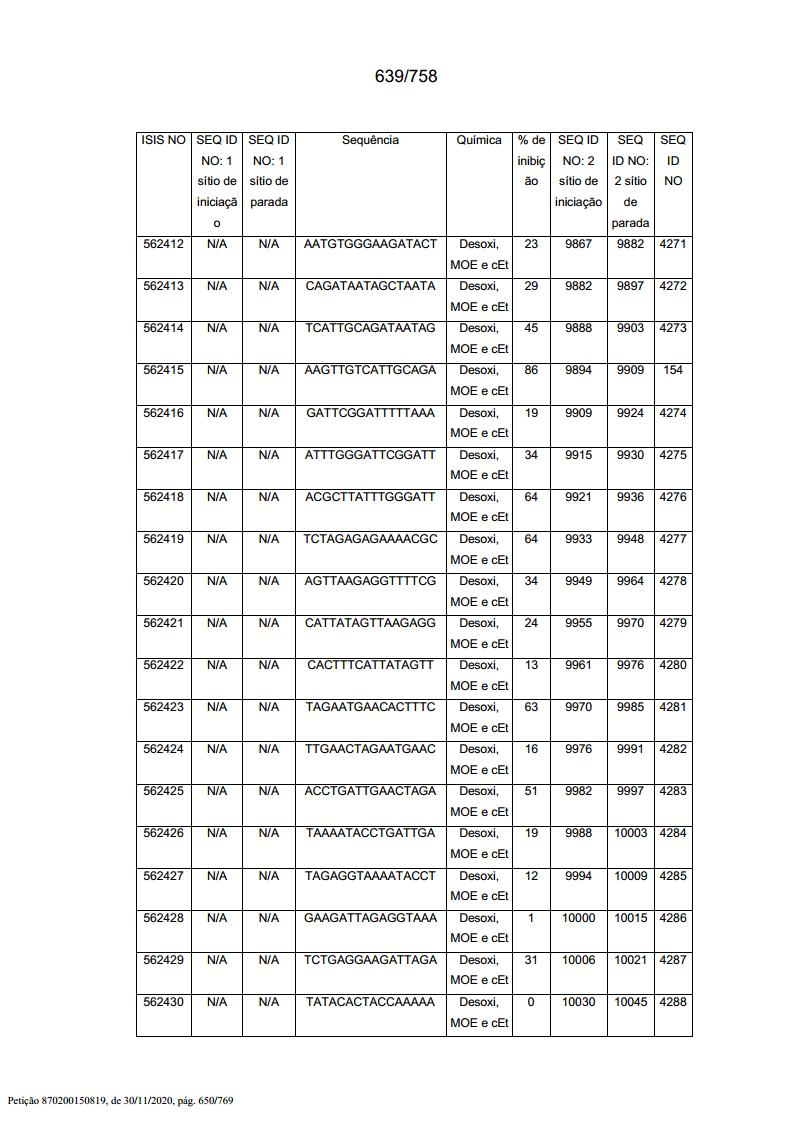






























[001444] Os oligonucleotídeos antisense adicionais foram projetados para se direcionar para um ácido nucleico de ANGPTL3 e foram testados para seus efeitos no mRNA do ANGPTL3 in vitro. As células Hep3B cultivadas em uma densidade de 20.000 células por poço foram transfectadas usando eletroporação com 4.500 nM do oligonucleotídeo antisense. Após um período de tratamento de aproximadamente 24 horas, o RNA foi isolado das células e os níveis de mRNA de ANGPTL3 foram medidos por PCR quantitativa em tempo real. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001445] Os oligonucleotídeos antisense quiméricos designados recentemente nas Tabelas abaixo foram concebidos como 5-10-5 MOE ou 3-10-4 MOE gapmers. Os gapmers 5-10-5 MOE têm 20 nucleosídeos de comprimento, nos quais o segmento de lacuna central compreende dez 2'- desoxinucleosídeos e é flanqueado por segmentos de asa na direção 5' e direção 3' que compreende cinco nucleosídeos cada. Os gapmers 3-10-4 MOE têm 17 nucleosídeos de comprimento, nos quais o segmento de lacuna central compreende dez 2'-desoxinucleosídeos e é flanqueado por segmentos de asa na direção 5' e na direção 3' que compreendem três nucleosídeos e quatro nucleosídeos, respectivamente. Cada nucleosídeo no segmento 5' wing e cada nucleosídeo no segmento 3' wing tem uma modificação em 2'-MOE. Cada nucleosídeo no segmento 5' wing e cada nucleosídeo no segmento 3' wing tem uma modificação em 2'-MOE. As ligações internucleosídicas ao longo de cada gapmer são ligações fosforotioato (P=S). Todos os resíduos de citosina ao longo de cada gapmer são 5-metilcitosinas. "Sítio de iniciação" indica o nucleosídeo mais extremo em 5' para o qual o gapmer é direcionado na sequência do gene humano. "Sítio de parada" indica o 3'- nucleosídeos a mais ao qual o gapmer é direcionado na sequência do gene humano. Cada gapmer listado nas tabelas abaixo é voltado para o mRNA, quer ANGPTL3 humana, aqui designado como SEQ ID NO: 1 (número de acesso GenBank NM_014495.2) ou a sequência genômica ANGPTL3 humana, aqui designada como SEQ ID NO: 2 (No. de Acesso GenBank NT_032977.9 truncado de nucleotídeos 33032001-33.046.000). 'N/a'indica que o oligonucleotídeo anti-sense não segmentado da sequência de gene particular com 100% de complementaridade.Tabela 155Inibição de mRNA ANGPTL3 pela segmentação de gapmers MOE SEQ ID NO: 1 e 2



Exemplo 122: Inibição anti-sense de dose-dependente deANGPTL3 humano em células Hep3B
[001446] Oligonucleotídeos desoxi, MOE e cET dos estudos descritos acima exibindo significativa inibição in vitro de mRNA ANGPTL3 foram selecionados e testados em várias doses em células Hep3B. ISIS 233717 e ISIS 337847, ambos gapmers MOE 5-10-5, também foram incluídos nos estudos. Os oligonucleotídeos antisense foram testados em uma série de experimentos que tinham condições de cultura semelhantes. Os resultados para cada experimento são apresentados em tabelas separadas mostradas abaixo.
[001447] As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de 0.813 μM, 1.625 μM, 3.25 μM, 6.500 μM e 13.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001448] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucle- otídeo anti-sense.Tabela 156
Exemplo 123: Inibição anti-sense de dose-dependente deANGPTL3 humano em células Hep3B
[001449] Oligonucleotídeos desoxi, MOE e cET dos estudos descritos acima exibindo significativa inibição in vitro de mRNA ANGPTL3 foram selecionados e testados em várias doses em células Hep3B. ISIS 337847, um gapmer MOE 5-10-5, também foi incluído nos estudos. Os oligonucleotídeos antisense foram testados em uma série de experimentos que tinham condições de cultura semelhantes. Os resultados para cada experimento são apresentados em tabelas separadas mostradas abaixo.
[001450] As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de0.160 μM, 0.481 μM, 1.444 μM, 4.333 μM e 13.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001451] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucle- otídeo anti-sense.
Exemplo 124: Inibição anti-sense de dose-dependente deANGPTL3 humano em células Hep3B por gapmers MOE
[001452] Gapmers MOE a partir dos Exemplos acima apresentam significativa inibiçãoin vitro de mRNA de ANGPTL3 foram selecionados e testados em diversas doses em células Hep3B. As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de0.160 μM, 0.481 μM, 1.444 μM, 4.333 μM e 13.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001453] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucleo- tídeo anti-sense.
Exemplo 125: Inibição anti-sense da dose-dependente do ser humano ANGPTL3 nas células Hep3B por oligonucleotídeos desoxi, MOE e cET
[001454] Oligonucleotídeos desoxi, MOE e cET dos estudos descritos acima exibindo significativa inibição in vitro de mRNA ANGPTL3 foram selecionados e testados em várias doses em células Hep3B. As células foram colocadas em placas em uma densidade de 20.000 por poço e transfectadas com eletroporação com concentrações de0.111 μM, 0.333 μM, 1.00 μM, 3.00 μM e 9.00 μM do oligonucleotídeo antisense, conforme especificado nas Tabelas abaixo. Após um período de tratamento de aproximadamente 16 horas, o RNA foi isolado das células e os níveis do mRNA de ANGPTL3 foram medidos por PCR em tempo real quantitativa. O conjunto de sonda de primer humano RTS3492_MGB foi usado para medir os níveis de mRNA. Os níveis de mRNA de ANGPTL3 foram ajustados de acordo com o conteúdo de RNA total, como medido pelo RIBOGREEN®. Os resultados são apresentados como inibição percentual de ANGPTL3, em relação às células de controle não tratadas.
[001455] A concentração do fármaco que induz metade do efeito máximo (IC50) de cada oligonucleotídeo também é apresentada. Os níveis de mRNA de ANGPTL3 foram significativamente reduzidos de maneira dependente de dose em células tratadas com o oligonucleotídeo anti-sense.
Exemplo 126: Inibição anti-sense de ANGPTL3 humano em camundongos transgênicos huANGPTL3
[001456] Oligonucleotídeos anti-sense descritos nos estudos acima foram ainda avaliados quanto à sua capacidade de reduzir ANGPTL3 transcrito de mRNA humano em camundongos C57BL/6 com o transgene humano ANGPTL3 (camundongos Tg).Estudo 1
[001457] Camundongos Tg fêmeas foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001458] Grupos de camundongos receberam injeções intraperito- neais de gapmers MOE 5-10-5 a uma dose de 50 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001459] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com RTS3492_MGB. Os níveis de mRNA foram também medidos com um conjunto de sonda humana de iniciadores RTS1984 (sequência inicial TCTTCAATGAAACGTGGGAGAACT, aqui designada como SEQ ID NO: 7; sequência reversaTCTCTAGGCCCAACCAAAATTC, aqui designada como SEQ ID NO: 8; sequência da sonda AAATATGGTTTTGGGAGGCTTGAT, aqui designada como SEQ ID NO: 9 ). Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.
[001460] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com oligonucleotídeo ISIS resultou na diminuição dos níveis de proteína ANGPTL3.Tabela 169A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001461] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 10, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 170Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 10
[001462] Camundongos Tg machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001463] Grupos de camundongos receberam injeções intraperiton- eais de gapmers MOE 5-10-5 a uma dose de 50 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. Os grupos PBS injetados serviram como grupos de controle aos quais foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001464] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com RTS1984. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 171A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001465] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com oligonucleotídeo ISIS resultou na diminuição dos níveis de proteína ANGPTL3.Tabela 172A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001466] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 8, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 173Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 8
[001467] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001468] Grupos de camundongos receberam injeções intraperito- neais de gapmers MOE 5-10-5 a uma dose de 2.5 mg/kg, 12.5 mg/kg, ou 25 mg/kg uma vez por semana durante 3 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. Os grupos PBS injetados serviram como grupos de controle aos quais foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001469] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022 (sequência inicial AAATTTTAGCCAATGGCCTCC, aqui designada como SEQ ID NO: 10; sequência reversa TGTCATTAATTTGGCCCTTCG, aqui designada como SEQ ID NO: 11; sequência da sondaTCAGTTGGGACATGGTCTTAAAGACTTTGTCC , aqui designada como SEQ ID NO: 12). Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS. O ED50 de cada gapmer também é apresentado na Tabela abaixo. 'N.d' indica que o ED50 não foi possível determinar.Tabela 174A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001470] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com oligonucleotídeos ISIS resultou em níveis de proteína ANGPTL3 reduzidas. 'N.d' indica que o ED50 não foi possível determinar.Tabela 175A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001471] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 17, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 176Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 17
[001472] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001473] Grupos de camundongos receberam injeções intraperitoneais de gapmers MOE 5-10-5 a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001474] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 177A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001475] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 10, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 178Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 10
[001476] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001477] Grupos de camundongos receberam injeções intraperit- oneais de gapmers MOE 5-10-5, ou desoxi e gapmers cET a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001478] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com RTS1984. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 179A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001479] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 9, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 180Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 9
[001480] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001481] Grupos de camundongos receberam injeções intraperi- toneais de oligonucleotídeos desoxi, MOE, e cET a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. ISIS 233710, um gapmer MOE 5-10-5, também foi incluído como referência. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001482] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com vários oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 181
[001483] A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001484] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando oprotocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com vários oligonucleotídeo ISIS resultou na diminuição dos níveis de proteína ANGPTL3.Tabela 182A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001485] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 10, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 183Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 10
[001486] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001487] Grupos de camundongos receberam injeções intraperito- neais de oligonucleotídeos desoxi, MOE, e cET a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. ISIS 233710, um gapmer MOE 5-10-5, também foi incluído como referência. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001488] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS. Tabela 184A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001489] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com alguns oligonucleotídeo ISIS resultou na diminuição dos níveis de ANGPTL3. Neste caso, '0' valor implica que o tratamento com o oligonucleotídeo ISIS não inibiram a expressão; em alguns casos, níveis aumentados de expressão podem ter sido gravados.Tabela 185A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001490] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 9, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 186Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 9
[001491] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001492] Grupos de camundongos receberam injeções intraperito- neais de oligonucleotídeos desoxi, MOE, e cET a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. ISIS 233710, um gapmer MOE 5-10-5, também foi incluído como referência. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001493] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 187A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001494] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando oprotocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com oligonucleotídeo ISIS resultou na diminuição dos níveis de ANGPTL3.Tabela 188A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001495] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 8, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 189Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 8
[001496] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001497] Grupos de camundongos receberam injeções intraperito- neais de oligonucleotídeos desoxi, MOE, e cET a uma dose de 25 mg/kg uma vez por semana durante 2 semanas. ISIS 233710, um gapmer MOE 5-10-5, também foi incluído como referência. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001498] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com alguns oligonucleotídeos antisense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS. Neste caso, '0' valor implica que o tratamento com o oligonucleotídeo ISIS não inibiram a expressão; em alguns casos, níveis aumentados de expressão podem ter sido gravados.Tabela 190A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001499] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Os resultados indicam que o tratamento com alguns oligonucleotídeo ISIS resultou na diminuição dos níveis de ANGPTL3. Neste caso, '0' valor implica que o tratamento com o oligonucleotídeo ISIS não inibiram a expressão; em alguns casos, níveis aumentados de expressão podem ter sido gravados.Tabela 191A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
[001500] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 9, os níveis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 192Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 9
[001501] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001502] Grupos de camundongos receberam injeções intraperito- neais de gapmers MOE 5-10-5 ou oligonucleotídeos desoxi, MOE e cET a uma dose de 5 mg/kg, 12.5 mg/kg, ou 25 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001503] No final do período de tratamento, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com hANGPTL3_LTS01022, e também com RTS3492_MGB. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com alguns oligonucleotídeos anti-sense ISIS resultou na redução do mRNA de ANGPTL3, em comparação com o controle de PBS.Tabela 193A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001504] Para avaliar o efeito dos oligonucleotídeos ISIS no dia 8, os ní-veis plasmáticos das transaminases (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligo- nucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 194Os níveis de transaminases no plasma (IU/L) em camundongos transgênicos no dia 8
Exemplo 127: A tolerabilidade dos oligonucleotídeos anti-sense segmentando ANGPTL3 humano em camundongos CD1
[001505] Os camundongos CD1® (Charles River, MA) são um modelo de camundongos para múltiplas finalidades, frequentemente utilizado para testes de segurança e eficácia. Os camundongos foram tratados com oligonucleotídeos antisense ISIS, selecionados a partir dos estudos descritos acima e avaliados para alterações nos níveis de diversos marcadores químicos do plasma.
Estudo 1
[001506] Camundongos CD1 machos (um animal por grupo de tratamento) foram injetados intraperitonealmente com uma dose única de 200 mg/kg de oligonucleotídeo desoxi, MOE, e cET. Um camundongo CD1 macho foi injetado por via subcutânea com uma dose única de PBS. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.Marcadores plasmáticos químicos
[001507] Para avaliar o efeito dos oligonucleotídeos ISIS nos níveis plasmáticos das transaminases no dia 4 (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 195Níveis de transaminases no plasma em camundongos plasmáticos CD1 no dia 4
[001508] Os pesos corporais foram medidos um dia após a dose única de oligonucleotídeo ISIS, e são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 196Os pesos corporais (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
[001509] Camundongos CD1 machos (um animal por grupo de tratamento) foram injetados intraperitonealmente com uma dose única de 200 mg/kg de oligonucleotídeos desoxi, MOE e cET. Um camundongo CD1 macho foi injetado por via subcutânea com uma dose única de PBS. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.
Marcadores plasmáticos químicos
[001510] Para avaliar o efeito dos oligonucleotídeos ISIS nos níveis plasmáticos das transaminases no dia 5 (ALT e AST) foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 197Níveis de transaminases no plasma em camundongos plasmáticos CD1 no dia 5
[001511] Os pesos corporais foram medidos um dia 5 após a dose única de oligonucleotídeo ISIS, e são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 198Os pesos corporais (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
[001512] Camundongos CD1 machos (quatro animais por grupo de tratamento) foram injetados intraperitonealmente com 100 mg/kg de gapmers MOE 5-10-5 administrado uma vez por semana durante 6 semanas. Um grupo de quatro camundongos machos CD1 foi injetado intraperitonealmente com PBS administrado uma vez por semana durante 6 semanas. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.
Marcadores plasmáticos químicos
[001513] Para avaliar o efeito dos oligonucleotídeos ISIS, os níveis plasmáticos de vários marcadores da função hepática e renal foram medidos no dia 45 utilizando um analisador de química clínica automático (Hitachi AU400e Olympus, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 199Níveis de marcadores plasmáticos químicos no plasma de camundongos CD1 no dia 45
[001514] Os pesos corporais foram medidos no dia 43 e são apresentados na Tabela abaixo. Pesos de rim, fígado e baço foram medidos no fim do estudo no dia 45. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 200Os pesos (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
[001515] Camundongos CD1 machos (quatro animais por grupo de tratamento) foram injetados intraperitonealmente com 50 mg/kg ou 100 mg/kg de gapmers MOE 5-10-5 ou desoxi, oligonucleotídeos MOE e cET dadas uma vez por semana durante 6 semanas. Um grupo de quatro camundongos machos CD1 foi injetado intraperitonealmente com PBS administrado uma vez por semana durante 6 semanas. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.
Marcadores plasmáticos químicos
[001516] Para avaliar o efeito dos oligonucleotídeos ISIS, os níveis plasmáticos de vários marcadores da função hepática e renal foram medidos no dia 46 utilizando um analisador de química clínica automático (Hitachi AU400e Olympus, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 201Níveis de marcadores plasmáticos químicos no plasma de camundongos CD1 no dia 45
Pesos corporais
[001517] Os pesos corporais foram medidos no dia 44 e são apresentados na Tabela abaixo. Pesos de rim, fígado e baço foram medidos no fim do estudo no dia 46. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 202Os pesos (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
[001518] Camundongos CD1 machos (quatro animais por grupo de tratamento) foram injetados intraperitonealmente com 50 mg/kg de oligonucleotídeos desoxi, MOE e cET dadas uma vez por semana durante 6 semanas. Um grupo de quatro camundongos machos CD1 foi injetado intraperitonealmente com PBS administrado uma vez por semana durante 6 semanas. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.
Marcadores plasmáticos químicos
[001519] Para avaliar o efeito dos oligonucleotídeos ISIS, os níveis plasmáticos de vários marcadores da função hepática e renal foram medidos no dia 43 utilizando um analisador de química clínica automático (Hitachi AU400e Olympus, Melville, NY). Os resultados são apresentados na Tabela abaixo. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um destes marcadores fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 203Níveis de marcadores plasmáticos químicos no plasma de camundongos CD1 no dia 43
[001520] Os pesos corporais foram medidos no dia 41 e são apresentados na Tabela abaixo. Pesos de rim, fígado e baço foram medidos no fim do estudo no dia 43. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 204Os pesos (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
Exemplo 128: Medição da viscosidade dos oligonucleotídeos antisense ISIS que se direcionam para o ANGPTL3 humano
[001521] A viscosidade dos oligonucleotídeos antisense selecionados a partir dos estudos descritos acima foi medida com o objetivo de filtrar os oligonucleotídeos antisense que têm uma viscosidade maior do que 40 centipoise (cP). Os oligonucleotídeos com uma viscosidade maior do que 40 cP teria menos do que a viscosidade ótima.
[001522] Oligonucleotídeos ISIS (32-35 mg) foram pesados para um frasco de vidro, 120 μL de água foi adicionado e o oligonucleotídeo antisense foi dissolvido em solução aquecendo o frasco a 50oC. Parte (75 μL) da amostra pré-aquecida foi pipetada para um micro- viscosímetro (Cambridge). A temperatura do micro-viscosímetro foi definida como 250C e a viscosidade da amostra foi medida. Outra parte (20 μL) da amostra pré-aquecida foi pipetada em 10 mL de água para leitura em UV em 260 nM a 85oC (instrumento Cary UV). Os resultados são apresentados na Tabela seguinte, onde a concentração de cada um dos oligonucleotídeos anti-sense foi de 350 mg/ml, e indicam que a maioria das soluções de oligonucleotídeos anti-sense são ótimas na sua viscosidade sob o critério indicado anteriormente. Tabela 205Viscosidade dos oligonucleotídeos antisense ISIS que se direcionam para o ANGPTL3 humano

Exemplo 129: Tolerabilidade dos oligonucleotídeos antisense que se direcionam para o ANGPTL3 humano em ratos Sprague-Dawley
[001523] Os camundongos Sprague-Dawley são um modelo para múltiplas finalidades usado para avaliações de segurança e eficácia. Os camundongos foram tratados com oligonucleotídeos antisense ISIS a partir dos estudos descritos nos Exemplos acima e avaliados para alterações nos níveis de diversos marcadores plasmáticos químicos. Estudo 1
[001524] Os camundongos Sprague-Dawley machos foram mantidos em um ciclo de claro/escuro de 12 horas e alimentados ad libitum com Purina chow de camundongo normal, dieta 5001. Injetou-se, por via subcutânea uma vez por semana durante 6 semanas, em grupos de 4 camundongos Sprague-Dawley, com PBS ou com 100 mg/kg de gapmers 5-10-5 MOE. Quarenta e oito horas após a última dose, realizou-se a eutanásia dos camundongos e os órgãos e o plasma foram colhidos para análise posterior.
Função hepática
[001525] Para avaliar o efeito dos oligonucleotídeos ISIS na função hepática, os níveis plasmáticos das transaminases foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os níveis plasmáticos da ALT (alanina transaminase) e AST (aspartato transaminase) foram medidos no dia 45 e os resultados são apresentados na Tabela abaixo, expressos em IU/L. Os níveis plasmáticos de bilirrubina também foram medidos, usando o mesmo analisador de química clínica e os resultados também são apresentados na Tabela abaixo expressa em mg/dL. Os oligonucleo- tídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 206Marcadores de função hepática em camundongos Sprague-Dawley
[001526] Para avaliar o efeito dos oligonucleotídeos ISIS na função renal, os níveis plasmáticos do nitrogênio da ureia sanguínea (BUN) e creatinina foram medidos, usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo, expressos em mg/dL. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função renal fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores. Os níveis de proteína total de urina e creatinina na urina foram medidos, e a proporção de proteína na urina total a creatinina foi avaliada. Os resultados são apresentados na Tabela abaixo.Tabela 207Marcadores plasmáticos da função renal (mg/dL) em camundongosSprague Dawley
Tabela 208Marcadores de urina da função renal em camundongos SpragueDawley
[001527] Os pesos corporais foram medidos no dia 42 e apresentados na Tabela abaixo. Os pesos do fígado, baço e rins foram medidos no final do estudo no dia 45, e são apresentados na tabela abaixo. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 209Os pesos corporais e órgãos (g) de camundongos Sprague Dawley
[001528] Os camundongos Sprague-Dawley machos foram mantidos em um ciclo de claro/escuro de 12 horas e alimentados ad libitum com Purina chow de camundongo normal, dieta 5001. Grupos de 4 camundongos Sprague-Dawley foram injetados subcutaneamente cada uma vez por semana durante 6 semanas, com PBS ou com 50 mg/kg ou 100 mg/kg de 5-10-5 MOE gapmers ou oligonucleotídeos desoxi, MOE e cET. Quarenta e oito horas após a última dose, realizou-se a eutanásia dos camundongos e os órgãos e o plasma foram colhidos para análise posterior.
Função hepática
[001529] Para avaliar o efeito dos oligonucleotídeos ISIS na função hepática, os níveis plasmáticos das transaminases foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os níveis plasmáticos da ALT (alanina transaminase) e AST (aspartato transaminase) foram medidos no dia 44 e os resultados são apresentados na Tabela abaixo, expressos em IU/L. Os níveis plasmáticos de bilirrubina também foram medidos, usando o mesmo analisador de química clínica e os resultados também são apresentados na Tabela abaixo expressa em mg/dL. Os oligonucle- otídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 210Marcadores de função hepática em camundongos Sprague-Dawley
[001530] Para avaliar o efeito dos oligonucleotídeos ISIS na função renal, os níveis plasmáticos do nitrogênio da ureia sanguínea (BUN) e creatinina foram medidos no dia 44, usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo, expressos em mg/dL. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função renal fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores. Os níveis de proteína total de urina e creatinina na urina foram medidos, e a proporção de proteína na urina total a creatinina foi avaliada. Os resultados são apresentados na Tabela abaixo.Tabela 211Marcadores plasmáticos da função renal (mg/dL) em camundongosSprague Dawley
Tabela 212Marcadores de urina da função renal em camundongos SpragueDawley
[001531] Os pesos corporais foram medidos no dia 42 e apresentados na Tabela abaixo. Os pesos do fígado, baço e rins foram medidos no final do estudo no dia 44, e são apresentados na tabela abaixo. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 213Os pesos corporais e órgãos (g) de camundongos Sprague Dawley
[001532] Os camundongos Sprague-Dawley machos foram mantidos em um ciclo de claro/escuro de 12 horas e alimentados ad libitum com Purina chow de camundongo normal, dieta 5001. Injetou-se, por via subcutânea uma vez por semana durante 6 semanas, em grupos de 4 camundongos Sprague-Dawley, com PBS ou 50 mg/kg de oligonucleotídeos desoxi, MOE e cEt. Quarenta e oito horas após a última dose, realizou-se a eutanásia dos camundongos e os órgãos e o plasma foram colhidos para análise posterior.
Função hepática
[001533] Para avaliar o efeito dos oligonucleotídeos ISIS na função hepática, os níveis plasmáticos das transaminases foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os níveis plasmáticos da ALT (alanina transaminase) e AST (aspartato transaminase) foram medidos no dia 44 e os resultados são apresentados na Tabela abaixo, expressos em IU/L. Os níveis plasmáticos de bilirrubina também foram medidos, usando o mesmo analisador de química clínica e os resultados também são apresentados na Tabela abaixo expressa em mg/dL. Os oligonu- cleotídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função hepática fora do intervalo esperado para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 214Marcadores de função hepática em camundongos Sprague-Dawley
[001534] Para avaliar o efeito dos oligonucleotídeos ISIS na função renal, os níveis plasmáticos do nitrogênio da ureia sanguínea (BUN) e creatinina foram medidos no dia 44, usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os resultados são apresentados na Tabela abaixo, expressos em mg/dL. Os oligonucleotídeos ISIS que provocaram as alterações nos níveis de qualquer um dos marcadores de função renal fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores. Os níveis de proteína total de urina e creatinina na urina foram medidos, e a proporção de proteína na urina total a creatinina foi avaliada. Os resultados são apresentados na Tabela abaixo.Tabela 215Marcadores plasmáticos da função renal (mg/dL) em camundongosSprague Dawley
Tabela 216Marcadores de urina da função renal em camundongos SpragueDawley
[001535] Os pesos corporais foram medidos no dia 42 e apresentados na Tabela abaixo. Os pesos do fígado, baço e rins foram medidos no final do estudo no dia 44, e são apresentados na tabela abaixo. Os oligonucleotídeos ISIS que provocaram quaisquer alterações nos pesos dos órgãos fora da faixa esperada para os oligonucleotídeos antisense foram excluídos em estudos posteriores.Tabela 217Os pesos corporais e órgãos (g) de camundongos Sprague Dawley
Exemplo 130: Efeito dos oligonucleotídeos anti-sense ISIS que têm como alvo ANGPTL3 humana em macacos-cinomolgos
[001536] Os macacos cynomolgus foram tratados com os oligonucleo- tídeos antisense ISIS selecionados dos estudos descritos nos Exemplos acima. A eficácia e a tolerabilidade do oligonucleotídeo antisense, bem como seu perfil farmacocinético no fígado e rins, foram avaliados.
[001537] Este estudo foi realizado no momento que, a sequência genômica de macaco cynomolgus não estava disponível no banco de dados do National Center for Biotechnology Information (NCBI); portanto, a reatividade cruzada com a sequência de gene de macaco cynomolgus não poderia ser confirmada. Em vez disso, as sequências dos oligonucleotídeos antisense ISIS usadas em macacos cynomolgus foram comparadas com uma sequência de macaco rhesus para a homologia. Espera-se que os oligonucleotídeos ISIS com homologia para a sequência de macaco rhesus sejam totalmente reativos de forma cruzada com a sequência de macaco cynomolgus também. Os oligonucleotídeos antisense humanos testados são reativos cruzadamente com a sequência genômica de rhesus (No. de acesso GENBANK NW_001108682.1 truncado de nucleotídeos 3.049.001 a 3.062.000, aqui designados como SEQ ID NO: 3). Quanto maior for a complementaridade entre o oligonucleotídeo humano e a sequência do macaco Rhesus, mais provável que o oligonucleotídeo humano possa reagir de forma cruzada com a sequência do macaco Rhesus. Os sítios de arranque e paragem de cada oligonucleotídeo da SEQ ID NO: 3 está apresentado na Tabela abaixo. "Sítio de iniciação" indica o nucleosídeo mais extremo em 5' para o qual o gapmer é direcionado na sequência gênica de macaco rhesus. 'Incompatibilidades' indicam o número de nucleobases no oligonucleotídeo humano que é incompatível com a sequência genômica de rhesus.Tabela 218Oligonucleótidos antisense complementares à sequência genômica deANGPTL3 de Rhesus (SEQ ID NO: 3)

[001538] Antes do estudo, os macacos foram mantidos em quarentena por pelo menos um período de 30 dias, durante o qual os animais foram observados diariamente para a saúde geral. Os macacos tinham 2-4 anos de idade e pesavam entre 2 e 4 kg. Nove grupos de 5 macacos cynomolgus machos distribuídos aleatoriamente cada foram injetados, por via subcutânea, com o oligonucleotídeos ISIS ou PBS em quatro locais nas costas em uma rotação horária (isto é, esquerda, parte superior, direita e parte inferior), um local por dose. Aos macacos foram dadas doses de carga de PBS ou 40 mg/kg de oligonucleotídeo ISIS de dois em dois dias durante a primeira semana (dias 1, 3, 5, e 7) e foram posteriormente doseados uma vez por semana durante 12 semanas (dias 14, 21, 28, 35, 42, 49, 56, 63, 70, 77 e 84) com PBS ou 40 mg/kg de oligonucleotídeo ISIS.
[001539] Durante o período do estudo, os macacos foram observados duas vezes ao dia para sinais de doença ou sofrimento. Qualquer animal experimentando mais que momentâneo ou ligeira dor ou sofrimento devido ao tratamento, à lesão ou à doença foi tratado pela equipe de veterinários com analgésicos ou agentes aprovados para aliviar a dor, após consulta com o Diretor do estudo. Qualquer animal com saúde frágil ou em uma possível condição moribunda era identificado para monitoramento posterior e possível eutanásia. Por exemplo, um animal no grupo de tratamento ISIS 567321 foi encontrado moribundo no dia 45 e foi terminado. Programada a eutanásia dos animais foi realizada no dia 86 (aproximadamente 48 horas após a dose final) por exsanguinação após anestesia de cetamina/xilazina e induzida por administração de pentobarbital de sódio. Os protocolos descritos no Exemplo foram aprovados pelo Institutional Animal Care and Use Committee (IACUC - Comitê Institucional de Uso e Cuidados Animais). Redução no Alvo Hepático Análise de RNA
[001540] No dia 86, o RNA foi extraído a partir do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Como mostrado na Tabela abaixo, o tratamento com oligonucleotídeos anti-sense ISIS resultou na redução significativa do mRNA de ANGPTL3, em comparação com o controle de PBS. A análise dos níveis de mRNA ANGPTL3 revelou que o ISIS 544199 e ISIS 559277, que são ambos totalmente reação cruzada com a sequência de rhesus, os níveis de expressão significativamente reduzidos. Outros oligonucleotídeos ISIS, que direcionados a sequência de macaco com desfasamentos, também foram capazes de reduzir os níveis de mRNA ANGPTL3.Tabela 219Inibição percentual do mRNA de ANGPTL3 no fígado de macacos cinomolgos em relação ao controle de PBS
[001541] Aproximadamente 1 mL de sangue foi coletado de todos os animais disponíveis no dia 85 e colocado em tubos contendo o sal de potássio de EDTA. As amostras de sangue foram colocadas em gelo e centrifugadas (3000 rpm durante 10 min a 4oC) para obter plasma.
[001542] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Análise de ANGPTL3 Plasma revelou que ISIS 563580, 544199 e ISIS 559277 reduziu os níveis de proteína de uma forma sustentada. Outros oligonucleotídeos ISIS também foram capazes de reduzir os níveis de ANGPTL3.Tabela 220Níveis de proteína no plasma (ng/mL) nos macacos cynomolgus
[001543] Para avaliar o efeito dos oligonucleotídeos ISIS sobre a saúde geral dos animais, corpo e pesos foram medidos foram medidos e são apresentados na Tabela abaixo. Os resultados indicam que o efeito do tratamento com os oligonucleotídeos antisense nos pesos corporais estava dentro do intervalo esperado para os oligonucleotídeos antisense. Especificamente, o tratamento com ISIS 563580 foi bem tolerado em termos dos pesos corporais dos macacos. Tabela 221Pesos corporais finais (g) em macacos cynomolgus
[001544] Para avaliar o efeito dos oligonucleotídeos ISIS na função hepática, amostras de sangue foram coletadas de todos os grupos de estudo. As amostras de sangue foram coletadas das veias cefálica, safena ou femoral 48 horas após a dosagem. Os macacos foram deixados em jejum durante a noite antes da coleta de sangue. O sangue foi coletado em tubos sem anticoagulante para a separação do soro. Os tubos foram mantidos em temperatura ambiente por um período mínimo de 90 min e então centrifugados (aproximadamente 3.000 rpm por 10 minutos) para obter o soro. Os níveis de diversos marcadores de função hepática foram medidos, usando um analisador de química Toshiba 200FR NEO (Toshiba Co., Japão). Os níveis plasmáticos da ALT e da AST foram medidos e os resultados são apresentados na Tabela abaixo, expressos em IU/L. A bilirrubina, um marcador da função hepática, foi medido de forma semelhante e é apresentada na Tabela abaixo expressa em mg/dL. Os resultados indicam que a maioria dos oligonucleotídeos antisense não tiveram efeito na função hepática fora do intervalo esperado para os oligonucleotídeos antisense. Especifica-mente, o tratamento com ISIS 563580 foi bem tolerado em termos da função hepática nos macacos.Tabela 222Os níveis de ALT (UI/L) no plasma dos macacos cynomolgus

Tabela 223Os níveis de AST (UI/L) no plasma dos macacos cynomolgus
Tabela 224Os níveis de bilirrubina (mg/dL) no plasma do macaco cynomolgus
[001545] Para avaliar o efeito dos oligonucleotídeos ISIS na função renal, amostras de sangue foram coletadas de todos os grupos de estudo. As amostras de sangue foram coletadas das veias cefálica, safena ou femoral 48 horas após a dosagem. Os macacos foram deixados em jejum durante a noite antes da coleta de sangue. O sangue foi coletado em tubos sem anticoagulante para a separação do soro. Os tubos foram mantidos em temperatura ambiente por um período mínimo de 90 min e então centrifugados (aproximadamente 3.000 rpm por 10 minutos) para obter o soro. Os níveis de BUN e creatinina foram medidos, usando um analisador de química Toshiba 200FR NEO (Toshiba Co., Japão). Os resultados são apresentados na Tabela abaixo, expressos em mg/dL.
[001546] Os dados químicos do plasma indicam que a maioria dos oligonucleotíde ISIS não tem qualquer efeito sobre a função renal fora do intervalo esperado para oligonucleotídeos antisense. Especificamente, o tratamento com ISIS 563580 foi bem tolerado em termos da função renal dos macacos.
Tabela 226Níveis de creatinina do plasma (mg/dL) em macacos cynomolgus
[001547] Para avaliar qualquer efeito dos ologonucleotídeos ISIS nos macacos cynomolgus sobre os parâmetros hematológicos, amostras de sangue de aproximadamente 0,5 mL de sangue foram coletadas de cada um dos animais do estudo disponíveis em tubos contendo K2- EDTA. As amostras foram analisadas para a contagem de células vermelhas sanguíneas (RBC), contagem de células brancas sangue- neas (WBC), contagens de células brancas sanguíneas individuais, tais como de monócitos, neutrófilos, linfócitos, bem como a contagem de plaquetas, conteúdo de hemoglobina e hematócrito, usando um analisador hematológico ADVIA120 (Bayer, EUA). Os dados são apresentados nas Tabelas abaixo.
[001548] Os dados indicam que os oligonucleotídeos não provocaram quaisquer alterações nos parâmetros hematológicos fora do intervalo esperado para os oligonucleotídeos antisense nesta dose. Especifica-mente, o tratamento com ISIS 563580 foi bem tolerado em termos dos parâmetros hematológicos dos macacos.Tabela 227Contagens de células sanguíneas em macacos cynomolgus
[001549] Para avaliar qualquer efeito inflamatório de oligonucle- otídeos ISIS em macacos cynomolgus, amostras de sangue foram colhidas para análise de proteína C reativa e os níveis de C3 no dia 84 de pré-dose. Cerca de 1,5 mL de sangue foi coletado de cada animal e colocado nos tubos sem anticoagulante para separação do soro. Os tubos foram mantidos à temperatura ambiente por um período mínimo de 90 min e então centrifugados a 3.000 rpm durante 10 minutos à temperatura ambiente para obter o soro. A proteína C reativa (CRP) e complemento C3, que servem como marcadores de inflamação, foram medidos usando um Toshiba 200FR NEO Chemistry Analyzer (Toshiba Co., Japão). Os resultados indicam que o tratamento com ISIS 563580 foi tolerável em macacos.Tabela 229Níveis de proteína C-reativa (mg/L) no plasma de macaco cynomolgus
Tabela 230Níveis C3 (mg/dL) no plasma de macaco cynomolgus
[001550] A concentração do oligonucleotídeo do comprimento completo foi medido. O método usado é uma modificação dos métodos publicados anteriormente (Leeds et al., 1996; Geary et al., 1999) que consiste em uma extração de fenol-clorofórmio (líquido-líquido) seguida por uma extração em fase sólida. Um padrão interno (ISIS 355868, um oligonucleotídeo de fosforotioato modificado por 2'-O-metoxietila 27- mer, GCGTTTGCTCTTCTTCTTGCGTTTTTT, designado neste documento como SEQ ID NO: 13) foi adicionado antes da extração. Concentrações de amostra de tecido foram calculadas utilizando as curvas de calibração, com um limite inferior de quantificação (LLOQ) de cerca de 1,14 μg/g. Os resultados são apresentados na Tabela abaixo, expressos como μg/g de fígado ou tecido renal. Calculou-se a proporção das concentrações de oligonucleotídeos de comprimento total no rim em relação ao fígado. A proporção de concentrações de oligonucleotídeos de comprimento total no rim contra o fígado após o tratamento com ISIS 563580 foi encontrada para ser mais otimizada em comparação com outros compostos avaliados.Tabela 231Concentração de comprimento total de oligonucleootídeo

Exemplo 131: Comparação da inibição anti-sense de ANGPTL3 humano em camundongos transgênicos huANGPTL3 por oligonucleotídeos ISIS compreendendo um grupo GalNAc conjugado e oligonucleotídeos ISIS que não compreendem um grupo conjugado GalNAc
[001551] Oligonucleotídeos anti-sense compreendendo GalNAc3-7a foram avaliados em comparação com os seus homólogos não conjugados para a sua capacidade de reduzir um transcrito mRNA ANGPTL3 humano nos camundongos Tg. Os gapmers, que têm como alvo SEQ ID NO: 1, estão descritos na Tabela a seguir e na Tabela 121. Os símbolos da coluna Química Principal são as seguintes: 's' denota éster tioato e 'o' denota éster de fosfato. Tabela 232oligonucleotídeos ISIS
[001552] Camundongos Tg fêmeas e machos foram mantidos com um ciclo de 12 horas claro/escuro. Os animais foram aclimatados por pelo menos 7 dias no centro de investigação antes do início do experimento. Os oligonucleotídeos anti-sense (ASOs) foram preparados em solução salina tamponada (PBS) e esterilizado por filtração através de um filtro de 0,2 mícron. Oligonucleotídeos foram dissolvidos em PBS 0,9% para injeção.
[001553] Um grupo de 4 camundongos receberam injeções subcutâneas de ISIS 563580 em doses de 5 mg/kg, 10 mg/kg, 15 mg/kg, ou 30 mg/kg uma vez por semana durante 2 semanas. Grupos de quatro camundongos cada receberam injeções intraperitoneais de ISIS 703801 ou ISIS 703802 em doses de 0,3 mg/kg, 1 mg/kg, 3 mg/kg, ou 10 mg/kg uma vez por semana durante 2 semanas. Um grupo de camundongos receberam injeções subcutâneas de PBS uma vez por semana durante 2 semanas. O grupo PBS injetado serviu como grupo de controle ao qual foram comparados os grupos tratados com oligonucleotídeos correspondentes.
Análise de RNA
[001554] No final do período de tratamento, o RNA foi extraído a partir do tecido do fígado para análise em tempo real de PCR de medição da expressão de mRNA de ANGPTL3 com conjunto da sonda de primer hANGPTL3_LTS01022. Os resultados são apresentados como alteração da porcentagem de mRNA, em relação ao controle de PBS, normalizado com RIBOGREEN®. Um valor de zero indica que apenas o oligonucleotídeo anti-sense não inibiu a expressão num nível mensurável.
[001555] Os resultados demonstram que os compostos conjugados são muito mais potente na redução da expressão ANGPTL3 do que a sua contraparte não conjugada, como é evidente a partir da inibição percentual e valores ID50. O oligonucleotídeo conjugado com a química principal mista (703802) foi mais potente na inibição da expressão do que o oligonucleotídeo conjugado com a química principal de fosforotioato completo (703801).Tabela 233A porcentagem de inibição de mRNA ANGPTL3 no fígado de camundongo transgênico relativo para o controle PBS
[001556] Os níveis de proteína ANGPTL3 humanos foram quantificados utilizando um kit ELISA disponível comercialmente (Catálogo#DANL30 por R&D Systems, Minneapolis, MN) com amostras de plasma transgênica diluída 1: 20000 utilizando o protocolo descrito do fabricante. Os resultados são apresentados na Tabela abaixo. Um valor de zero indica que apenas o oligonucleotídeo anti-sense não inibiu a expressão num nível mensurável.
[001557] Os resultados demonstram que os compostos conjugados são mais potente na redução da expressão ANGPTL3 do que a sua contraparte não conjugada, como é evidente a partir da inibição percentual e valores. O oligonucleotídeo conjugado com a química principal mista (703802) foi mais potente na inibição da expressão do que o oligonucleotídeo conjugado com a química principal de fosforotioato completo (703801).Tabela 234A porcentagem de inibição dos níveis de proteína de plasma no camundongo transgênico
Exemplo 132: A tolerabilidade de um oligonucleotídeo anti-sense conjugado GalNAc segmentando ANGPTL3 humano em camundongos CD1
[001558] Camundongos CD1 machos (quatro animais por grupo de tratamento) foram injetados por via subcutânea com várias doses de ISIS 703802, conforme descrito na tabela abaixo para 6 semanas (nos dias 1, 3, 5, 8, 14, 21, 28, 35 e 42). Um grupo de quatro camundongos CD1 macho foi injetado subcutaneamente com PBS durante 6 semanas. Os camundongos sofreram eutanásia 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior.
Marcadores plasmáticos químicos
[001559] Para avaliar o efeito dos ISIS 703802, os níveis plasmáticos de vários marcadores da função hepática e renal foram medidos no dia 44 utilizando um analisador de química clínica automático (Hitachi AU400e Olympus, Melville, NY). Os resultados são apresentados na Tabela abaixo. ISIS 703802 foi mostrado como sendo um composto tolerável, mesmo em doses elevadas.Tabela 235Níveis de marcadores plasmáticos químicos no plasma de camundongos CD1 no dia 44
[001560] Pesos corporais, de rim, fígado e baço foram medidos no fim do estudo no dia 44. ISIS 703802 não se alterou significativamente de órgãos do corpo e pesos, mesmo quando administrado em doses elevadas.Tabela 236Os pesos (g) de camundongos CD1 após o tratamento de oligonucleotídeo anti-sense
Exemplo 133: A tolerabilidade de um oligonucleotídeo anti-sense conjugado GalNAc segmentando ANGPTL3 humano em camundongos Sprague-Dawley
[001561] Os camundongos Sprague-Dawley são um modelo para múltiplas finalidades usado para avaliações de segurança e eficácia. Os camundongos Sprague-Dawley machos foram mantidos em um ciclo de claro/escuro de 12 horas e alimentados ad libitum com Purina chow de camundongo normal, dieta 5001. Grupos de 4 camundongos Sprague- Dawley cada foram injetados por via subcutânea com várias doses de ISIS 703802, conforme descrito na Tabela abaixo para 6 semanas (nos dias 1, 3, 5, 8, 14, 21, 28, 35 e 42). Um grupo de 4 camundongos foi injetado subcutaneamente com PBS durante 6 semanas. Realizou-se a eutanásia dos ratos 48 horas após a última dose, e os órgãos e o plasma foram colhidos para análise posterior. Função hepática e renal
[001562] Para avaliar o efeito dos ISIS 703802 na função hepática, os níveis plasmáticos das transaminases foram medidos usando um analisador de química clínica automatizado (Hitachi Olympus AU400e, Melville, NY). Os níveis plasmáticos de ALT (alanina transaminase) e AST (aspartato transaminase) foram medidos no dia 44 e os resultados são apresentados na Tabela abaixo expressos em IU/L.
[001563] Para avaliar o efeito de ISIS 703802 sobre a função renal, os níveis plasmáticos de albumina, ureia no sangue de nitrogênio (BUN), creatina e bilirrubina foram medidas utilizando o mesmo analisador de química clínica e os resultados são apresentados na Tabela abaixo, expresso em g/dL ou mg/dL.
[001564] Para avaliar ainda o efeito dos ISIS 703802 na função renal, os níveis de proteína na urina e de creatinina na urina foram medidos, e a razão entre proteína total na urina e creatinina foi avaliada. Os resultados são apresentados na Tabela abaixo.
[001565] ISIS 703802 foi mostrado como sendo um composto tolerável, mesmo em doses elevadas.Tabela 237Marcadores de função hepática e renal em plasma de camundongoSprague-Dawley no dia 44
Tabela 238Marcadores de urina da função renal (mg/dL) no camundongo Sprague-Dawley no dia 44
[001566] Os pesos corporais, do fígado, baço e rins foram medidos no final do estudo no dia 44 e são apresentados na tabela abaixo. ISIS 703802 não alterou significativamente os pesos corporais, de rins e de fígado mesmo quando administrado em doses elevadas.Tabela 239Os pesos corporais e órgãos (g) de camundongos Sprague Dawley