WO2011007876A1 - Hmgb1結合核酸分子およびその用途 - Google Patents
Hmgb1結合核酸分子およびその用途 Download PDFInfo
- Publication number
- WO2011007876A1 WO2011007876A1 PCT/JP2010/062104 JP2010062104W WO2011007876A1 WO 2011007876 A1 WO2011007876 A1 WO 2011007876A1 JP 2010062104 W JP2010062104 W JP 2010062104W WO 2011007876 A1 WO2011007876 A1 WO 2011007876A1
- Authority
- WO
- WIPO (PCT)
- Prior art keywords
- hmgb1
- nucleic acid
- acid molecule
- binding
- seq
- Prior art date
Links
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
- A61K31/7105—Natural ribonucleic acids, i.e. containing only riboses attached to adenine, guanine, cytosine or uracil and having 3'-5' phosphodiester links
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P29/00—Non-central analgesic, antipyretic or antiinflammatory agents, e.g. antirheumatic agents; Non-steroidal antiinflammatory drugs [NSAID]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/04—Antineoplastic agents specific for metastasis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/10—Drugs for disorders of the cardiovascular system for treating ischaemic or atherosclerotic diseases, e.g. antianginal drugs, coronary vasodilators, drugs for myocardial infarction, retinopathy, cerebrovascula insufficiency, renal arteriosclerosis
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/46—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates
- C07K14/47—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals
- C07K14/4701—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans from vertebrates from mammals not used
- C07K14/4702—Regulators; Modulating activity
- C07K14/4703—Inhibitors; Suppressors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/115—Aptamers, i.e. nucleic acids binding a target molecule specifically and with high affinity without hybridising therewith ; Nucleic acids binding to non-nucleic acids, e.g. aptamers
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/68—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing involving proteins, peptides or amino acids
- G01N33/6863—Cytokines, i.e. immune system proteins modifying a biological response such as cell growth proliferation or differentiation, e.g. TNF, CNF, GM-CSF, lymphotoxin, MIF or their receptors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/16—Aptamers
Definitions
- the present invention relates to a nucleic acid molecule that binds to HMGB1 protein and its use.
- HMGB1 High-mobility group box 1 protein
- Non-patent Document 1 HMGB1 protein
- Non-patent Document 2 HMGB1 protein essential for survival
- Non-patent Document 3 HMGB1 protein essential for survival
- Non-patent Document 4 HMGB1 becomes a target substance and a diagnostic marker for the treatment of these diseases.
- HMGB1 expression level is increased as compared with normal tissues because it is involved in proliferation and metastasis invasion of breast cancer, colon cancer, melanoma, prostate cancer, pancreatic cancer, lung cancer and the like.
- An object of the present invention is to provide, for example, a nucleic acid molecule capable of binding to HMGB1 as a substance that can be used for elucidating the onset mechanism of a disease caused by HMGB1 and for diagnosing and treating the disease, and uses thereof. It is in.
- the HMGB1-binding nucleic acid molecule of the present invention is a binding nucleic acid molecule capable of binding to HMGB1, characterized by having a dissociation constant of HMGB1 protein (hereinafter referred to as HMGB1) of 5 ⁇ 10 ⁇ 7 or less.
- composition of the present invention is a composition comprising the HMGB1-binding nucleic acid molecule of the present invention.
- the detection reagent of the present invention is an HMGB1 detection reagent for detecting HMGB1, characterized in that it comprises the HMGB1-binding nucleic acid molecule of the present invention.
- the HMGB1-binding nucleic acid molecule of the present invention can bind to HMGB1. For this reason, according to the HMGB1-binding nucleic acid molecule of the present invention, for example, by binding to HMGB1 and inhibiting its function, the above-mentioned diseases caused by HMGB1 can be prevented and treated. In addition, according to the HMGB1-binding nucleic acid molecule of the present invention, for example, HMGB1 can be detected by confirming the presence or absence of binding to HMGB1, thereby enabling early diagnosis of the disease.
- HMGB1-binding nucleic acid molecule of the present invention by expressing the HMGB1-binding nucleic acid molecule of the present invention in cultured cells, gene transcription inhibition experiments can be performed, and the HMGB1 binding nucleic acid molecule of the present invention and its receptor are used.
- the HMGB1-binding nucleic acid molecule of the present invention can also be used for elucidating the function of HMGB1, such as allowing binding inhibition experiments with the body. Therefore, the HMGB1-binding nucleic acid molecule of the present invention is also useful as a new research tool.
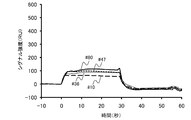
- FIG. 1 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 1 of the present invention.
- FIG. 2 is a graph showing the binding ability of each RNA aptamer to HMGB1 in Example 1 of the present invention.
- FIG. 3 is a graph showing the binding ability of each RNA aptamer to the His-tag-added MIF protein in Example 1 of the present invention.
- FIG. 4 is a graph showing the binding ability of each RNA aptamer to HMGB1 under physiological conditions in Example 1 of the present invention.
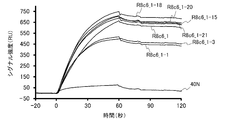
- FIG. 5 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 2 of the present invention.
- FIG. 1 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 1 of the present invention.
- FIG. 2 is a graph showing the binding ability of each RNA aptamer to HMGB
- FIG. 6 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 3 of the present invention.
- FIG. 7 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 4 of the present invention.
- FIG. 8 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 5 of the present invention.
- FIG. 9 is a photograph of a pull-down assay showing binding between HMGB1 and TLR-2 in Example 6 of the present invention.
- FIG. 10 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 7 of the present invention.
- FIG. 11 is a diagram showing the predicted secondary structures of RNA aptamers R8c6_1 and R8c6_18 in the present invention.
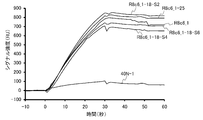
- FIG. 12 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 9 of the present invention.
- FIG. 13 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 9 of the present invention.
- FIG. 14 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 10 of the present invention.
- FIG. 15 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 11 of the present invention.
- FIG. 12 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 9 of the present invention.
- FIG. 13 is a graph showing the binding ability of each RNA
- FIG. 16 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 12 of the present invention.
- FIG. 17 is a graph showing the binding ability of each RNA aptamer to His-tag-added HMGB1 in Example 12 of the present invention.
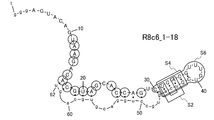
- FIG. 18 is a diagram showing a predicted secondary structure of RNA aptamer R8c6_1 in the present invention.
- FIG. 19 is a diagram showing a predicted secondary structure of RNA aptamer R8c6_1-18 in the present invention.
- FIG. 20 is a diagram showing a predicted secondary structure of RNA aptamer R8c6_1-25-S6 in the present invention.
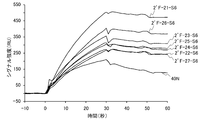
- FIG. 21 is a graph showing the interaction between HMGB1 and RNA aptamer in Example 13 of the present invention.
- the HMGB1-binding nucleic acid molecule of the present invention is a nucleic acid molecule capable of binding to HMGB1, which has a dissociation constant of 5 ⁇ 10 ⁇ 7 or less with HMGB1.
- the HMGB1-binding nucleic acid molecule of the present invention is also referred to as, for example, an HMGB1 aptamer.
- “possible to bind to HMGB1” means, for example, having a binding ability to HMGB1 or having a binding activity to HMGB1 (HMGB1 binding activity).
- the HMGB1-binding nucleic acid molecule of the present invention specifically binds to, for example, HMGB1.
- the binding between the HMGB1-binding nucleic acid molecule and HMGB1 can be determined by, for example, surface plasmon resonance molecular interaction analysis. For the analysis, for example, Biacore X (trade name, GE Healthcare UK Ltd.) can be used.
- the HMGB1-binding nucleic acid molecule of the present invention may have a dissociation constant of 5 ⁇ 10 ⁇ 7 or less with HMGB1, and other configurations are not limited.
- the dissociation constant for HMGB1 is not particularly limited, but the upper limit thereof is, for example, 10 ⁇ 8 order, more preferably 10 ⁇ 10 order, and further preferably 10 ⁇ 12 order.
- the range of the dissociation constant is, for example, on the order of 10 ⁇ 14 to 10 ⁇ 8 , preferably on the order of 10 ⁇ 14 to 10 ⁇ 10 , and more preferably on the order of 10 ⁇ 14 to 10 ⁇ 12 .
- the structural unit of the HMGB1-binding nucleic acid molecule of the present invention is not particularly limited.
- the structural unit is, for example, a nucleotide residue, and examples of the nucleotide residue include a ribonucleotide residue and a deoxyribonucleotide residue.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, RNA composed of ribonucleotide residues or DNA composed of deoxyribonucleotide residues, preferably RNA.
- the HMGB1-binding nucleic acid molecule of the present invention may contain, for example, both deoxyribonucleotides, which are constituent units of DNA, and ribonucleotides, which are constituent units of RNA.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, RNA containing deoxyribonucleotide residues or RNA containing ribonucleotide residues.
- the nucleotide residue may be, for example, a modified nucleotide residue.
- the modified nucleotide residue include those in which a sugar residue in the nucleotide residue is modified.
- examples of the sugar residue include a ribose residue and a deoxyribose residue.
- the modification site in the nucleotide residue is not particularly limited, and examples thereof include the 2 'position and / or the 4' position of the sugar residue. Examples of the modification include methylation, fluorination, amination, and thiolation.
- the modified nucleotide residue is, for example, a modified nucleotide residue having a pyrimidine base (pyrimidine nucleus) as a base, or a modified nucleotide residue having a purine base (purine nucleus) as a base.
- the former is preferred.
- a nucleotide residue having a pyrimidine base is referred to as a pyrimidine nucleotide residue
- a modified pyrimidine nucleotide residue is referred to as a modified pyrimidine nucleotide residue
- a nucleotide residue having a purine base is referred to as a purine nucleotide residue.
- the purified purine nucleotide residue is referred to as a modified purine nucleotide residue.
- the pyrimidine nucleotide residues include uracil nucleotide residues having uracil, cytosine nucleotide residues having cytosine, thymine nucleotide residues having thymine, and the like.
- the modified nucleotide residue when the base is a pyrimidine base, for example, the 2'-position and / or the 4'-position of the sugar residue is preferably modified.
- modified nucleotide residue examples include, for example, 2′-methyluracil (2′-methylated-uracil nucleotide residue), 2′-methylcytosine (2′-modified ribose residue) 2'-methylated-cytosine nucleotide residues), 2'-fluorouracil (2'-fluorinated-uracil nucleotide residues), 2'-fluorocytosine (2'-fluorinated-cytosine nucleotide residues), 2'- Aminouracil (2′-aminated uracil-nucleotide residue), 2′-aminocytosine (2′-aminated-cytosine nucleotide residue), 2′-thiouracil (2′-thiolated-uracil nucleotide residue), 2'-thiocytosine (2'-thiolated-cytosine nucleotide residue) and the like.
- the HMGB1-binding nucleic acid molecule of the present invention includes monomer residues such as PNA (peptide nucleic acid), LNA (Locked Nucleic Acid), ENA (2′-O, 4′-C-Ethylenebridged Nucleic Acids) and the like as the structural unit. Can be given.
- Examples of the HMGB1-binding nucleic acid molecule of the present invention include RNA or DNA containing at least one monomer residue of PNA, LNA and ENA.
- the HMGB1-binding nucleic acid molecule of the present invention contains the monomer residue, the number is not particularly limited.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, a single-stranded nucleic acid or a double-stranded nucleic acid.
- the single-stranded nucleic acid include single-stranded RNA and single-stranded DNA.
- the double-stranded nucleic acid include double-stranded RNA, double-stranded DNA, and double-stranded nucleic acid of RNA and DNA.
- the HMGB1-binding nucleic acid molecule of the present invention is a double-stranded nucleic acid, it may be made into a single strand by denaturation or the like prior to use, for example.
- each base may be a natural base (non-artificial nucleic acid) of, for example, adenine (a), cytosine (c), guanine (g), thymine (t), and uracil (u).
- An artificial base (unnatural base) may be used.
- the artificial base include a modified base and a modified base, and preferably have the same function as the natural base (a, c, g, t, or u).
- the artificial base having the same function is, for example, an artificial base capable of binding to cytosine (c) instead of guanine (g), an artificial base capable of binding to guanine (g) instead of cytosine (c), Instead of adenine (a), an artificial base capable of binding to thymine (t) or uracil (u), instead of thymine (t), an artificial base capable of binding to adenine (a), instead of uracil (u) And an artificial base capable of binding to adenine (a).
- the modified base include a methylated base, a fluorinated base, an aminated base, and a thiolated base.
- the modified base include, for example, 2′-methyluracil, 2′-methylcytosine, 2′-fluorouracil, 2′-fluorocytosine, 2′-aminouracil, 2′-aminocytosine, 2-thiouracil, 2-thiocytosine and the like.
- the bases represented by a, g, c, t and u include the meaning of the artificial base having the same function as each of the natural bases in addition to the natural base.
- the HMGB1-binding nucleic acid molecule of the present invention is preferably resistant to nucleases, for example.
- the nuclease is not particularly limited, and examples thereof include exonuclease and endonuclease. Specific examples include, for example, ribonuclease (RNase) that is an RNA-degrading enzyme, deoxyribonuclease (DNase) that is a DNA-degrading enzyme, RNA and Examples include nucleases that act on both DNA.
- RNase ribonuclease
- DNase deoxyribonuclease
- RNA examples include nucleases that act on both DNA.
- the HMGB1-binding nucleic acid molecule of the present invention is preferably RNA.
- the HMGB1-binding nucleic acid molecule of the present invention is RNA, it is preferably resistant to, for example, RNase, that is, RNase.
- the method for making the nuclease resistant is not particularly limited, and examples thereof include a method of modifying nucleotide residues constituting the nucleic acid molecule.
- the nucleotide residue or a part of the nucleotide residues constituting the nucleic acid molecule is preferably a modified nucleotide residue.
- the modified nucleotide residue include the aforementioned modified nucleotide residues.
- modified nucleotide residue examples include the methylated nucleotide residue, the fluorinated nucleotide residue, the aminated nucleotide residue, the thiolated nucleotide residue, and the like. preferable.
- the modified nucleotide residue is, for example, the pyrimidine nucleotide residue having a pyrimidine base as a base, and the sugar residue (ribose residue or deoxyribose residue) is preferably modified.
- the method for making the nuclease resistant includes, for example, a method of converting a nucleotide residue constituting the nucleic acid molecule into LNA.
- the method of making the nuclease resistant includes, for example, a method of converting nucleotide residues constituting RNA, which is the nucleic acid molecule, into DNA.
- HMGB1-binding nucleic acid molecule of the present invention is RNA
- all or part of nucleotide residues having uracil are converted to nucleotide residues having thymine. Specifically, it may be substituted with a deoxyribonucleotide residue having the thymine.
- the nucleic acid molecule is RNA
- all or some of the nucleotide residues constituting the RNA may be deoxyribonucleotide residues and the LNA residues.
- the method for making the nuclease resistant includes, for example, a method in which polyethylene glycol (PEG) or deoxythymidine is bonded to the 5 'end and / or the 3' end.
- PEG polyethylene glycol
- the PEG is preferably tens of kDa, for example.
- the length of the HMGB1-binding nucleic acid molecule of the present invention is not particularly limited, but the total length is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100 bases. is there.
- HMGB1-binding nucleic acid molecule of the present invention examples include any one of the following (A1), (A2), (B1) and (B2) nucleic acid molecules.
- A1 Nucleic acid molecule comprising the base sequence represented by any of SEQ ID NOs: 1 to 42 (A2) In the base sequence represented by any of SEQ ID NOs: 1-42, one or more bases are substituted, deleted, Any of nucleic acid molecules (B2) comprising a base sequence represented by any one of nucleic acid molecules (B1) SEQ ID NOs: 45 to 81, which contain an added or inserted base sequence and can bind to HMGB1
- the nucleic acid molecule (A1) will be described.
- the nucleic acid molecule (A1) is referred to as HMGB1-binding nucleic acid molecule (A1).
- the base sequences represented by SEQ ID NOs: 1-42 are also referred to as base sequences (A1).
- the nucleotide sequence (A1) represented by SEQ ID NOs: 1-42 and the HMGB1-binding nucleic acid molecule (A1) containing the nucleotide sequence (A1) are represented by the names shown before the sequence numbers as shown below. There is also.
- (A1) a nucleic acid molecule comprising the base sequence represented by any of SEQ ID NOs: 1-42
- SEQ ID NO: 1 “m” is adenine or cytosine, “h” is adenine, cytosine, thymine, or uracil, and “b” is guanine, cytosine, thymine, or uracil.
- Examples of the base sequence represented by SEQ ID NO: 1 include the base sequences represented by SEQ ID NOs: 2 to 5.
- uracil (u) may be thymine (t).
- one or more uracils may be thymine and all uracils may be thymine.
- the nucleic acid molecule which consists of a base sequence containing thymine or the nucleic acid molecule which contains the said base sequence can be illustrated as a nucleic acid molecule of (A2) mentioned later, for example.
- the HMGB1-binding nucleic acid molecule (A1) may be a nucleic acid molecule consisting of the base sequence (A1) represented by any one of the sequence numbers 1 to 42, or a nucleic acid molecule containing the base sequence (A1). .
- the base sequence (A1) may be an X region, and may further have a Y region and / or a Y ′ region.
- the X region, the Y region, and the Y ′ region are as described later, for example.
- the Y region is not particularly limited, and examples thereof include a sequence containing the base sequence represented by SEQ ID NO: 43 or 115 and a sequence consisting of the base sequence.
- the Y ′ region is not particularly limited, and examples thereof include a sequence containing the base sequence represented by SEQ ID NO: 44 and a sequence consisting of the base sequence. These sequences are merely examples, and do not limit the present invention.
- the Y region is preferably bound to the 5 ′ side of the base sequence (A1), for example.
- the HMGB1-binding nucleic acid molecule (A1) includes the Y ′ region, the Y ′ region is preferably bound to the 3 ′ side of the base sequence (A1), for example.
- the base sequence (A1), the Y region, and the Y ′ region may be directly bonded or may be bonded via an intervening sequence, for example.
- the HMGB1-binding nucleic acid molecule (A1) includes any one of the base sequences (A1) of SEQ ID NOs: 1-42, for example, the nucleic acid molecule comprising the base sequence represented by any of SEQ ID NOs: 45-81 or the above Examples include nucleic acid molecules containing a base sequence.
- the base sequences of SEQ ID NOs: 45 to 81 shown below each include the base sequence (A1) of SEQ ID NOs: 6 to 42, and the regions represented by the underlined portions are the base sequences of SEQ ID NOs: 6 to 42, respectively. Equivalent to.
- the base sequence of SEQ ID NOs: 45 to 81 and the HMGB1-binding nucleic acid molecule (A1) containing the base sequence may be represented by names shown before the sequence numbers as shown below.
- # 04 (SEQ ID NO: 45) gggacgcucacguacgcuca ucccaugauuguucaggcacggccuuucgguucccucaau ucagugccuggacgugcagu # 08 (SEQ ID NO: 46) gggacgcucacguacgcuca agucccuugacacguccguuuucuaacuggaauagaggcc ucagugccuggacgugcagu # 12 (SEQ ID NO: 47) gggacgcucacguacgcuca gggcugcaccucuccgcuacguugucguuggaggcaccau ucagugccuggacgugcagu # 43 (SEQ ID NO: 48) ggga
- the HMGB1-binding nucleic acid molecule (A1) is, for example, a nucleic acid consisting of a base sequence from the fourth base on the 5 ′ side to the terminal base on the 3 ′ side in any one of the base sequences (A1) of SEQ ID NOs: 45 to 81 It may be a molecule or a nucleic acid molecule containing the base sequence. That is, the HMGB1-binding nucleic acid molecule (A1) may have, for example, the 5 ′ terminal ggg deleted in any one of the nucleotide sequences (A1) of SEQ ID NOs: 45 to 81.
- the base sequence (A1) may include a motif sequence represented by SEQ ID NO: 114, for example.
- n is adenine (a), cytosine (c), guanine (g), uracil (u), thymine (t), and the fifth base n is adenine (a), the 13th base n is preferably cytosine (c), and n at the 17th base is preferably adenine (a).
- Examples of the base sequence (A1) containing the motif sequence include # 47 (SEQ ID NO: 65), # 80 (SEQ ID NO: 67), R8c6_1 (SEQ ID NO: 74), and R8c6_14 (SEQ ID NO: 75).
- Motif sequence (SEQ ID NO: 114) uaagncacguagnaccng
- each base is the same as described above, for example. That is, the base may be, for example, the natural base (non-artificial base) of adenine (a), cytosine (c), guanine (g), thymine (t) and uracil (u), or the artificial base (non-artificial base) Natural base).
- the artificial base is, for example, as described above.
- the bases represented by a, g, c, t and u mean the artificial base having the same function as each of the natural bases in addition to the natural bases. Including.
- the structural unit of the HMGB1-binding nucleic acid molecule (A1) is not particularly limited, and is the same as described above. That is, the structural unit is, for example, a nucleotide residue, and examples of the nucleotide residue include a ribonucleotide residue and a deoxyribonucleotide residue.
- the HMGB1-binding nucleic acid molecule (A1) may be, for example, RNA composed of ribonucleotide residues or DNA composed of deoxyribonucleotide residues, and is preferably RNA.
- the HMGB1-binding nucleic acid molecule (A1) may include both deoxyribonucleotides, which are DNA constituent units, and ribonucleotides, which are RNA constituent units.
- the base sequences of SEQ ID NO: 1-42 and SEQ ID NO: 45-81 may be any sequence as long as the bases are continuous as described above, for example, may be RNA composed of ribonucleotide residues, or deoxyribonucleotides It may be DNA composed of residues, RNA containing deoxyribonucleotide residues, or RNA containing ribonucleotide residues.
- examples of the structural unit include monomer residues such as PNA, LNA, and ENA.
- examples of the HMGB1-binding nucleic acid molecule (A1) include RNA or DNA containing at least one monomer residue of PNA, LNA and ENA.
- the number is not particularly limited.
- the HMGB1-binding nucleic acid molecule (A1) is preferably resistant to nucleases as described above, for example.
- the method for making the nuclease resistant is not particularly limited, and is the same as described above.
- the HMGB1-binding nucleic acid molecule (A1) is preferably RNA.
- the HMGB1-binding nucleic acid molecule (A1) is RNA, it is preferably resistant to RNase, for example.
- the technique for making the RNase resistant is not particularly limited and is the same as described above.
- HMGB1-binding nucleic acid molecule (A1) is RNA
- the modified nucleotide residue include the aforementioned modified nucleotide residues.
- all or some of the nucleotide residues constituting the RNA may be the deoxyribonucleotide residue and / or the LNA residue. It is preferably a group.
- the HMGB1-binding nucleic acid molecule (A1) is RNA, for example, among the nucleotide residues constituting the RNA, all or part of the nucleotide residues having uracil are left as nucleotide residues having thymine. It may be substituted with a group, specifically, it may be substituted with a deoxyribonucleotide residue having the thymine.
- the HMGB1-binding nucleic acid molecule (A1) is RNA, for example, it is preferable that the PEG or the deoxythymidine is present at the 5 'end and / or the 3' end as described above.
- the nucleic acid molecule which consists of a base sequence substituted by thymine or the nucleic acid molecule containing the said base sequence can be illustrated as a nucleic acid molecule of (A2) mentioned later, for example.
- the length of the HMGB1-binding nucleic acid molecule (A1) is not particularly limited, but the total length thereof is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100 bases. It is.
- HMGB1-binding nucleic acid molecule A2
- A2 a nucleic acid molecule comprising one or a plurality of bases substituted, deleted, added or inserted in the base sequence represented by any of SEQ ID NOs: 1-42 and capable of binding to HMGB1
- the HMGB1-binding nucleic acid molecule (A2) may be a nucleic acid molecule containing the substituted base sequence or a nucleic acid molecule composed of the substituted base sequence.
- the substituted base sequence is also referred to as a base sequence (A2).
- “One or more” is not particularly limited. “One or more” is, for example, 1 to 5, preferably 1 to 4, more preferably 1 to 3, in any one of the nucleotide sequences of SEQ ID NOs: 1 to 42, One or two is preferable, and one is particularly preferable. Further, “one or more” is, for example, 1 to 5, preferably 1 to 4, more preferably 1 to 3, in the full-length sequence of the HMGB1-binding nucleic acid molecule (A1). More preferably, it is one or two, and particularly preferably one.
- the base used for the substitution, addition or insertion is not particularly limited, and may be, for example, the natural base or the artificial base. For the substitution, addition or insertion of the base, for example, the nucleotide residue may be used, or the monomer residue may be used.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, the following nucleic acid molecule (A3).
- the nucleic acid molecule (A3) is referred to as HMGB1-binding nucleic acid molecule (A3).
- A3) a nucleic acid molecule comprising a base sequence having 60% or more homology in the base sequence represented by any of SEQ ID NOs: 1-42 and capable of binding to HMGB1
- the HMGB1-binding nucleic acid molecule (A3) may be a nucleic acid molecule containing the homologous base sequence or a nucleic acid molecule consisting of the homologous base sequence.
- the base sequence having the homology is also referred to as a base sequence (A3).
- the homology is, for example, 70% or more, preferably 80% or more, more preferably 90% or more, still more preferably 95% or more, and particularly preferably 99% or more.
- the HMGB1-binding nucleic acid molecule (A3) may be, for example, a nucleic acid molecule that includes a base sequence having 60% or more homology in the full-length sequence of the HMGB1-binding nucleic acid molecule (A1) and can bind to HMGB1.
- the homology is, for example, 70% or more, preferably 80% or more, more preferably 90% or more, still more preferably 95% or more, and particularly preferably 99% or more.
- the homology can be calculated, for example, by calculating under default conditions using BLAST or the like.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, the following (A4) nucleic acid molecule.
- the nucleic acid molecule (A4) is referred to as HMGB1-binding nucleic acid molecule (A4).
- Nucleic acid comprising a base sequence that hybridizes with the base sequence represented by any of SEQ ID NOs: 1-42 under stringent conditions or a base sequence complementary to the base sequence and capable of binding to HMGB1 molecule
- the HMGB1-binding nucleic acid molecule (A4) may be a nucleic acid molecule consisting of the hybridizing base sequence or a nucleic acid molecule containing the base sequence.
- the HMGB1-binding nucleic acid molecule (A4) may be a nucleic acid molecule comprising the complementary base sequence or a nucleic acid molecule comprising the complementary base sequence.
- the hybridizing base sequence and the complementary base sequence are also referred to as a base sequence (A4).
- HMGB1-binding nucleic acid molecule “hybridizes under stringent conditions” is, for example, well-known experimental conditions for hybridization by those skilled in the art.
- stringent conditions refers to, for example, hybridization at 60 to 68 ° C. in the presence of 0.7 to 1 mol / L NaCl, and then 0.1 to 2 times the SSC solution. Used refers to conditions that can be identified by washing at 65-68 ° C. 1 ⁇ SSC consists of 150 mmol / L NaCl and 15 mmol / L sodium citrate.
- the HMGB1-binding nucleic acid molecule (A4) includes, for example, a nucleic acid molecule that includes a base sequence that hybridizes under stringent conditions with the full-length base of the HMGB1-binding nucleic acid molecule (A1) and is capable of binding to HMGB1. But you can.
- HMGB1-binding nucleic acid molecules (A2) to (A4) are the same as the HMGB1-binding nucleic acid molecule (A1) unless otherwise indicated, for example, base, constitutional unit, length, nuclease resistance and the like.
- the nucleic acid molecule (B1) is referred to as HMGB1-binding nucleic acid molecule (B1).
- the base sequence represented by SEQ ID NOs: 45 to 81 is also referred to as a base sequence (B1).
- B1 a nucleic acid molecule comprising the base sequence represented by any of SEQ ID NOs: 45 to 81
- the base sequences represented by SEQ ID NOs: 45 to 81 are as described above.
- the HMGB1-binding nucleic acid molecule (B1) containing the base sequence represented by SEQ ID NOs: 45 to 81 may be represented by the names shown before the aforementioned SEQ ID NOs.
- the HMGB1-binding nucleic acid molecule (B1) may be, for example, a nucleic acid molecule consisting of the base sequence (B1) represented by any one of SEQ ID NOs: 45 to 81, or a nucleic acid molecule containing the base sequence (B1). Good.
- the HMGB1-binding nucleic acid molecule (B1) is, for example, a nucleic acid comprising a base sequence from the 4th base on the 5 ′ side to the terminal base on the 3 ′ side in any one of the base sequences (B1) of SEQ ID NOs: 45 to 81 It may be a molecule or a nucleic acid molecule containing the base sequence. That is, the HMGB1-binding nucleic acid molecule (B1) may lack, for example, the 5 ′ terminal ggg in any one of the base sequences (A1) of SEQ ID NOs: 45 to 81.
- the base sequence (B1) may include a motif sequence represented by SEQ ID NO: 114, for example.
- n is adenine (a), cytosine (c), guanine (g), uracil (u), thymine (t), and the fifth base n is adenine (a), the 13th base n is preferably cytosine (c), and n at the 17th base is preferably adenine (a).
- Examples of the base sequence (B1) including the motif sequence include # 47 (SEQ ID NO: 65), # 80 (SEQ ID NO: 67), R8c6_1 (SEQ ID NO: 74), and R8c6_14 (SEQ ID NO: 75).
- Motif sequence (SEQ ID NO: 114) uaagncacguagnaccng
- the predicted secondary structure of R8c6_1 (SEQ ID NO: 74) is shown in FIG.
- the circled base corresponds to the motif sequence.
- the base corresponding to “n” in the motif sequence is omitted from the circle.
- each base is the same as described above, for example. That is, the base may be, for example, the natural base (non-artificial base) of adenine (a), cytosine (c), guanine (g), thymine (t) and uracil (u), or the artificial base (non-artificial base) Natural base).
- the artificial base is, for example, as described above.
- the bases represented by a, g, c, t and u mean the artificial base having the same function as each of the natural bases in addition to the natural bases. Including.
- the structural unit of the HMGB1-binding nucleic acid molecule (B1) is not particularly limited, and is the same as described above. That is, the structural unit is, for example, a nucleotide residue, and examples of the nucleotide residue include a ribonucleotide residue and a deoxyribonucleotide residue.
- the HMGB1-binding nucleic acid molecule (A1) may be, for example, RNA composed of ribonucleotide residues or DNA composed of deoxyribonucleotide residues, and is preferably RNA.
- the HMGB1-binding nucleic acid molecule (A1) may include both deoxyribonucleotides, which are DNA constituent units, and ribonucleotides, which are RNA constituent units.
- the base sequences of SEQ ID NOs: 45 to 81 may be any sequence as long as the bases are continuous as described above, for example, may be RNA composed of ribonucleotide residues, or composed of deoxyribonucleotide residues. It may be DNA, RNA containing deoxyribonucleotide residues, or RNA containing ribonucleotide residues.
- examples of the structural unit include monomer residues such as PNA, LNA, and ENA.
- examples of the HMGB1-binding nucleic acid molecule (B1) include RNA or DNA containing at least one monomer residue of PNA, LNA and ENA.
- the number is not particularly limited.
- the HMGB1-binding nucleic acid molecule (B1) is preferably resistant to nucleases as described above, for example.
- the method for making the nuclease resistant is not particularly limited, and is the same as described above.
- the HMGB1-binding nucleic acid molecule (B1) is preferably RNA.
- the HMGB1-binding nucleic acid molecule (B1) is RNA, it is preferably resistant to RNase, for example.
- the technique for making the RNase resistant is not particularly limited and is the same as described above.
- HMGB1-binding nucleic acid molecule (B1) is RNA
- the modified nucleotide residue include the aforementioned modified nucleotide residues.
- the HMGB1-binding nucleic acid molecule (B1) is RNA, for example, as described above, all or some of the nucleotide residues constituting the RNA are the deoxyribonucleotide residues and / or the LNA residues. It is preferably a group.
- the HMGB1-binding nucleic acid molecule (B1) is RNA, as described above, for example, among nucleotide residues constituting RNA, all or part of nucleotide residues having uracil have nucleotide residues having thymine. It may be substituted with a group, specifically, it may be substituted with a deoxyribonucleotide residue having the thymine.
- the HMGB1-binding nucleic acid molecule (B1) is RNA, for example, it is preferable that the PEG or the deoxythymidine is present at the 5 'end and / or 3' end, as described above.
- the nucleic acid molecule which consists of a base sequence substituted by thymine or the nucleic acid molecule containing the said base sequence can be illustrated as a nucleic acid molecule of (B2) mentioned later, for example.
- the length of the HMGB1-binding nucleic acid molecule (B1) is not particularly limited, but the total length is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100 bases. It is.
- the HMGB1-binding nucleic acid molecule (B1) is preferably the nucleic acid molecule (b1) below.
- the nucleic acid molecule (b1) is also referred to as HMGB1-binding nucleic acid molecule (b1).
- the HMGB1 nucleic acid molecule (b1) may be, for example, a nucleic acid molecule having the base sequence of SEQ ID NO: 74 or a nucleic acid molecule having the base sequence.
- (B1) a nucleic acid molecule comprising the base sequence represented by SEQ ID NO: 74
- HMGB1-binding nucleic acid molecule B2
- HMGB1-binding nucleic acid molecule B2
- a nucleic acid molecule comprising a base sequence in which one or more bases are substituted, deleted, added or inserted in the base sequence represented by any of SEQ ID NOs: 45 to 81 and capable of binding to HMGB1
- the HMGB1-binding nucleic acid molecule (B2) may be a nucleic acid molecule containing the substituted base sequence or a nucleic acid molecule composed of the substituted base sequence.
- the substituted base sequence is also referred to as a base sequence (B2).
- “One or more” is not particularly limited as long as the HMGB1-binding nucleic acid molecule (B2) can bind to HMGB1.
- the number of the substituted bases is, for example, 1 to 5, preferably 1 to 4, more preferably 1 to 3, in any one of the nucleotide sequences of SEQ ID NOS: 45 to 81, More preferably, it is one or two, and particularly preferably one.
- the number of added or inserted bases is, for example, 1 to 5, preferably 1 to 4, more preferably 1 to 3, in any of the base sequences of SEQ ID NOs: 45 to 81. Yes, more preferably one or two, and particularly preferably one.
- the number of deleted bases is not particularly limited. For example, in the base sequence of any of SEQ ID NOs: 45 to 81, 1 to 46, 1 to 43, 1 to 21 and ⁇ 1 to 18 1 to 4, 1 to 3, 2 or 1.
- the length of the HMGB1-binding nucleic acid molecule (B2) is not particularly limited, but the total length thereof is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100 bases. It is.
- the nucleic acid molecule is a nucleic acid molecule obtained by downsizing the HMGB1-binding nucleic acid molecule (B1).
- the miniaturized nucleic acid molecule is also referred to as a miniaturized HMGB1-binding nucleic acid molecule (B2).
- the miniaturized HMGB1-binding nucleic acid molecule has not only one or more bases deleted in the base sequence represented by any of SEQ ID NOs: 45 to 81, but also, for example, 1 Alternatively, it may be a nucleic acid molecule containing a base sequence in which a plurality of bases are substituted, added or inserted, and capable of binding to HMGB1.
- the miniaturized HMGB1 binding nucleic acid molecule (B2) may be a nucleic acid molecule containing the deleted base sequence, or a nucleic acid molecule consisting of the deleted base sequence.
- the deleted base sequence is also referred to as a miniaturized base sequence.
- the number of bases deleted is not particularly limited, and is as described above, for example.
- the miniaturized HMGB1-binding nucleic acid molecule (B2) is, for example, a nucleic acid molecule consisting of a base sequence from the 4 ′ base on the 5 ′ side to the terminal base on the 3 ′ side in the base sequence of SEQ ID NOs: 45 to 81 or the base sequence And nucleic acid molecules containing. That is, the miniaturized HMGB1-binding nucleic acid molecule (B2) is, for example, a nucleic acid molecule comprising a base sequence in which the 5 ′ terminal ggg is deleted in the base sequence (B1) of any of SEQ ID NOs: 45 to 81 Examples include nucleic acid molecules that contain sequences.
- the length of the miniaturized HMGB1 binding nucleic acid molecule (B2) is not particularly limited, but the total length is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100. Base length.
- the miniaturized HMGB1-binding nucleic acid molecule (B2) for example, in the base sequence represented by SEQ ID NO: 74, one or a plurality of bases contains a deleted base sequence and can be bound to HMGB1 A molecule is preferable, and specifically, a nucleic acid molecule of the following (b2) is preferable.
- the nucleic acid molecule (b2) is also referred to as a miniaturized HMGB1-binding nucleic acid molecule (b2).
- (B2) a nucleic acid molecule comprising a partial sequence of 11 or more consecutive bases in the base sequence represented by SEQ ID NO: 74 and capable of binding to HMGB1
- the continuous partial sequence of 11 bases or more is hereinafter also referred to as a continuous partial sequence.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) may be a nucleic acid molecule composed of the continuous partial sequence or a nucleic acid molecule containing the continuous partial sequence.
- HMGB1-binding nucleic acid molecule for example, as described above, in the base sequence of SEQ ID NO: 74, not only one or more bases are deleted, but also one or more bases are It may be a nucleic acid molecule comprising a substituted, added or inserted nucleotide sequence and capable of binding to HMGB1.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) may include, for example, one sequence of the continuous partial sequence of 11 or more bases in the base sequence of SEQ ID NO: 74, or two or more sequences.
- the length of the continuous partial sequence is 11 bases or more as described above.
- the length of the continuous partial sequence is not particularly limited, and may be, for example, 12 bases or more, 14 bases or more, or 16 bases or more.
- the upper limit of the length of the continuous partial sequence is not particularly limited, and is 80 bases or less, preferably 79 bases or less.
- the continuous partial sequence is not particularly limited, and examples thereof include the following y sequence, x sequence, and y ′ sequence.
- Examples of the y sequence include the first to twentieth and fourth to twentieth regions in the base sequence of SEQ ID NO: 74.
- the x sequence is, for example, 21st to 60th, 22nd to 60th, 24th to 60th, 25th to 60th, 29th to 60th, 34th to 60th in the base sequence of SEQ ID NO: 74.
- the y ′ sequence is, for example, the 61st to 80th, the 61st to 79th, the 61st to 77th, the 61st to 76th, the 61st to 78th, the 64th to the 64th sequence in the base sequence of SEQ ID NO: 74.
- Examples include the 80th, 65th to 80th, 70th to 80th, and 64th to 77th areas.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) may be, for example, a nucleic acid molecule consisting of the base sequence (b2) or a nucleic acid molecule containing the base sequence (b2).
- Examples of the base sequence (b2) include a base sequence including the x sequence and the y ′ sequence, and a base sequence including the y sequence, the x sequence, and the y ′ sequence.
- the base sequence (b2) preferably includes, for example, the y sequence on the 5 'side of the x sequence and the y' sequence on the 3 'side of the x sequence.
- the base sequence (b2) may have, for example, the 5 'terminal ggg in the y sequence deleted.
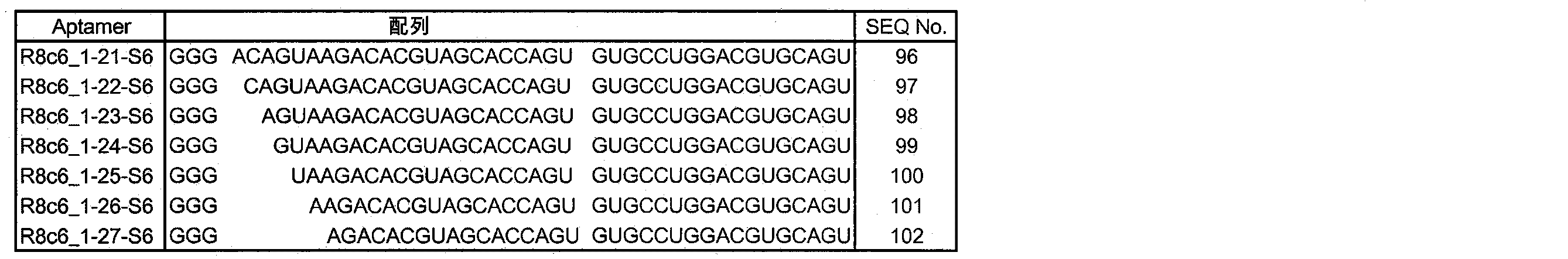
- Examples of the base sequence (b2) include base sequences represented by SEQ ID NOs: 83 to 113. These base sequences are shown in Table 1 below. In Table 1 below, each base sequence is shown to correspond to the base sequence of SEQ ID NO: 74. For each base sequence, as compared with the base sequence of SEQ ID NO: 74, the deleted portion is indicated by a blank, and different bases are underlined.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) containing the nucleotide sequences of SEQ ID NOs: 83 to 113 may be represented by names shown in Table 1 below, respectively.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) may be, for example, a nucleic acid molecule having a base sequence represented by any of SEQ ID NOs: 83 to 113, or any one of SEQ ID NOs: 83 to 113. It may be a nucleic acid molecule containing the represented base sequence.
- the miniaturized HMGB1-binding nucleic acid molecule (b2) is, for example, from the fourth base on the 5 ′ side to the terminal base on the 3 ′ side in any one of the base sequences (b2) of SEQ ID NO: 74 and SEQ ID NOS: 83 to 113 Or a nucleic acid molecule comprising the base sequence. That is, in the miniaturized HMGB1-binding nucleic acid molecule (b2), for example, the 5 ′ terminal ggg may be deleted in any one of the nucleotide sequences of SEQ ID NO: 74 and SEQ ID NOs: 83 to 113.
- the base sequence (b2) may include a motif sequence represented by SEQ ID NO: 114, for example.
- n is adenine (a), cytosine (c), guanine (g), uracil (u), thymine (t), and the fifth base n is adenine (a), the 13th base n is preferably cytosine (c), and n at the 17th base is preferably adenine (a).
- the base sequence (b2) containing the motif sequence is, for example, R8c6_1-1 (SEQ ID NO: 83), R8c6_1-3 (SEQ ID NO: 84), R8c6_1-4 (SEQ ID NO: 85), R8c6_1-15 (SEQ ID NO: 86), R8c6_1-18 (SEQ ID NO: 87), R8c6_1-20 (SEQ ID NO: 88), R8c6_1-21 (SEQ ID NO: 89), R8c6_1-25 (SEQ ID NO: 90), R8c6_1-18-S2 (SEQ ID NO: 93), R8c6_1-18 -S4 (SEQ ID NO: 94), R8c6_1-18-S6 (SEQ ID NO: 95), R8c6_1-21-S6 (SEQ ID NO: 96), R8c6_1-22-S6 (SEQ ID NO: 97), R8c6_1-23-S6 (SEQ ID NO: 98)
- the base sequence (B1) including a part of the motif sequence is, for example, R8c6_1-30 (SEQ ID NO: 91), R8c6_1-34CC (SEQ ID NO: 92), R8c6_1-26-S6 (SEQ ID NO: 101), R8c6_1- 25-S6A2 (SEQ ID NO: 104), R8c6_1-25-S6C (SEQ ID NO: 106) and the like.
- Motif sequence (SEQ ID NO: 114) uaagncacguagnaccng
- the predicted secondary structure of R8c6_1-18 (SEQ ID NO: 74) is shown in FIG.
- the circled base corresponds to the motif sequence.
- the base corresponding to “n” in the motif sequence is omitted from the circle.
- the predicted secondary structure of R8c6_1-25-S6 (SEQ ID NO: 100) is shown in FIG.
- the circled base corresponds to the motif sequence.
- the base corresponding to “n” in the motif sequence is omitted from the circle.
- the length of the miniaturized HMGB1-binding nucleic acid molecule (b2) is not particularly limited, but the total length is, for example, 20 to 160 bases, preferably 30 to 120 bases, more preferably 40 to 100. Base length.
- the base sequence (b2) is not particularly limited in the total length, but the lower limit thereof is, for example, 20 base length or more, 30 base length or more, 34 base length As mentioned above, it is 37 base length or more and 40 base length or more, The upper limit is 160 bases or less, 120 bases or less, 100 bases or less, 80 bases or less, for example, Preferably it is 79 bases or less.
- the miniaturized HMGB1-binding nucleic acid molecule (B2) is, for example, a base sequence in which one or more bases are substituted, deleted, added or inserted in the continuous partial sequence of the miniaturized HMGB1-binding nucleic acid molecule (b2). And a nucleic acid molecule capable of binding to HMGB1.
- the base sequence represented by any one of SEQ ID NOs: 83 to 113 one or a plurality of bases includes a base sequence in which substitution, deletion, addition or insertion is included, and binds to HMGB1 Possible nucleic acid molecules are mentioned.
- the number of bases for the substitution and the like is not particularly limited, and in the base sequence of any of SEQ ID NOs: 83 to 113, for example, 1 to 5, preferably 1 to 4, more preferably 1 to The number is 3, more preferably 1 or 2, and particularly preferably 1.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, the following (B3) nucleic acid molecule.
- the nucleic acid molecule (B3) is referred to as HMGB1-binding nucleic acid molecule (B3).
- (B3) a nucleic acid molecule comprising a base sequence having 60% or more homology in the base sequence represented by any of SEQ ID NOs: 45 to 81 and capable of binding to HMGB1
- the HMGB1-binding nucleic acid molecule (B3) may be a nucleic acid molecule containing the homologous base sequence or a nucleic acid molecule consisting of the homologous base sequence.
- the homology is, for example, 70% or more, preferably 80% or more, more preferably 90% or more, still more preferably 95% or more, and particularly preferably 99% or more.
- the HMGB1-binding nucleic acid molecule (B3) may be, for example, a nucleic acid molecule that includes a base sequence having 60% or more homology in the full-length sequence of the HMGB1-binding nucleic acid molecule (B1) and can bind to HMGB1.
- the homology is, for example, 70% or more, preferably 80% or more, more preferably 90% or more, still more preferably 95% or more, and particularly preferably 99% or more.
- the homology can be calculated, for example, by calculating under default conditions using BLAST or the like.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, the following nucleic acid molecule (B4).
- the nucleic acid molecule (B4) is referred to as HMGB1-binding nucleic acid molecule (B4).
- (B4) a nucleic acid comprising a base sequence that hybridizes with the base sequence represented by any of SEQ ID NOs: 45 to 81 under stringent conditions or a base sequence complementary to the base sequence and capable of binding to HMGB1 molecule
- the HMGB1-binding nucleic acid molecule (B4) may be a nucleic acid molecule consisting of the hybridizing base sequence or a nucleic acid molecule containing the base sequence. Further, the HMGB1-binding nucleic acid molecule (B4) may be a nucleic acid molecule composed of the complementary base sequence or a nucleic acid molecule containing the complementary base sequence.
- HMGB1-binding nucleic acid molecule B4
- “hybridizes under stringent conditions” is, for example, well-known experimental conditions for hybridization by those skilled in the art.
- stringent conditions refers to, for example, hybridization at 60 to 68 ° C. in the presence of 0.7 to 1 mol / L NaCl, and then 0.1 to 2 times the SSC solution. Used refers to conditions that can be identified by washing at 65-68 ° C. 1 ⁇ SSC consists of 150 mmol / L NaCl and 15 mmol / L sodium citrate.
- the HMGB1-binding nucleic acid molecule (B4) includes, for example, a nucleic acid molecule that includes a base sequence that hybridizes under stringent conditions with the full-length base of the HMGB1-binding nucleic acid molecule (B1) and is capable of binding to HMGB1. But you can.
- HMGB1-binding nucleic acid molecules (B2) to (B4) are the same as the HMGB1-binding nucleic acid molecule (B1) unless otherwise indicated, for example, base, constitutional unit, length, nuclease resistance and the like.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, a single-stranded nucleic acid or a double-stranded nucleic acid.
- the HMGB1-binding nucleic acid molecule of the present invention is the double-stranded nucleic acid, for example, one single strand is any one of the nucleic acid molecules (A1) to (A4) and (B1) to (B4).
- the MGB1-binding nucleic acid molecule of the present invention may be further combined with a linker sequence such as polyadenine within the range that does not affect the binding property to HMGB1.
- a linker sequence such as polyadenine within the range that does not affect the binding property to HMGB1.
- the production method of the HMGB1-binding nucleic acid molecule of the present invention is not limited at all, and can be synthesized by a known method such as a nucleic acid synthesis method using chemical synthesis.
- the HMGB1-binding nucleic acid molecule of the present invention may be, for example, a nucleic acid molecule consisting of the base sequence represented by any one of SEQ ID NOs: 1 to 42 as described above, or a nucleic acid molecule containing the base sequence. . In the latter case, the HMGB1-binding nucleic acid molecule of the present invention may contain, for example, a base sequence other than the base sequence represented by any one of SEQ ID NOs: 1-42.
- HMGB1-binding nucleic acid molecule of the present invention includes, for example, a Y region, an X region and a Y ′ region, and the Y region, the X region and the Y ′ region are linked from the 5 ′ end.
- the X region includes a base sequence represented by any one of SEQ ID NOs: 1 to 42, and the Y region and the Y ′ region each have an arbitrary base sequence. Preferably it consists of.
- the number of bases in the X region is not particularly limited, but is, for example, 10 to 60 bases, preferably 15 to 50 bases, and more preferably 20 to 40 bases.
- the number of bases in the Y region and Y ′ region is not particularly limited, but is, for example, 10 to 50 bases, preferably 15 to 40 bases, and more preferably 20 to 30 bases.
- the total number of bases of the HMGB1-binding nucleic acid molecule of the present invention is not particularly limited, but is, for example, 20 to 160 bases, preferably 30 to 120 bases, and more preferably 40 to 100 bases.
- the base sequence of the Y region and the base sequence of the Y ′ region are not particularly limited, but preferably include, for example, a primer binding sequence that can be annealed by a primer, a polymerase recognition sequence that can be recognized by a polymerase, and the like.
- a primer binding sequence that can be annealed by a primer
- a polymerase recognition sequence that can be recognized by a polymerase, and the like.
- the HMGB1-binding nucleic acid molecule of the present invention when amplified by a nucleic acid amplification method, includes, for example, a primer binding sequence that can hybridize with a primer and a polymerase recognition sequence that can be recognized by a polymerase. Is preferred.
- the HMGB1-binding nucleic acid molecule of the present invention has, for example, the primer binding to at least one of 5 ′ upstream of the X region, that is, the Y region, and 3 ′ downstream of the X region, that is, the Y ′ region. It preferably includes a sequence and a polymerase recognition sequence.
- the polymerase recognition region can be appropriately determined according to, for example, the type of polymerase used in nucleic acid amplification.
- the polymerase recognition sequence is preferably, for example, a DNA-dependent RNA polymerase recognition sequence (hereinafter also referred to as “RNA polymerase recognition sequence”), and specific examples include T7 RNA. Examples include the T7 promoter which is a recognition sequence for polymerase.
- the 5′-side Y region comprises the RNA polymerase recognition sequence and the primer-binding sequence (hereinafter also referred to as “5′-side primer region”).
- the X region is preferably connected to the 3 'side of the Y region. Furthermore, it is preferable that the Y ′ region is linked to the 3 ′ side of the X region, and the Y ′ region includes a primer binding sequence (hereinafter also referred to as “3 ′ side primer region”).
- the 5 ′ primer region in the RNA is capable of binding to a sequence complementary to the 3 ′ side of a DNA antisense strand synthesized using, for example, the RNA, that is, the 3 ′ side of the antisense strand.
- the sequence is preferably the same as that of the primer.
- the HMGB1-binding nucleic acid molecule of the present invention may further have a region that assists binding to HMGB1, for example.
- the Y region and the X region, and the X region and the Y ′ region may be directly adjacent to each other, or indirectly via an intervening sequence. May be adjacent to
- the method for preparing the HMGB1-binding nucleic acid molecule of the present invention by nucleic acid amplification is not particularly limited.
- the HMGB1-binding nucleic acid molecule of the present invention is RNA, for example, it can be prepared using DNA as a template.
- a DNA strand that is a template for RNA is also referred to as an antisense strand
- a DNA strand that includes a sequence in which uracil (u) of RNA is replaced with thymine (t) is also referred to as a sense strand.
- the template DNA includes, for example, DNA (antisense strand) in which uracil (u) of the complementary region of the X region in the RNA is replaced with thymine (t), and uracil (u) of the X region is converted to thymine (t). It is preferable to include any one of DNAs (sense strands) containing the sequence substituted in (). Using these DNAs as templates and performing nucleic acid amplification using DNA-dependent DNA polymerase, using the obtained DNA amplification products as templates, and further using the DNA-dependent RNA polymerase to transcribe RNA, RNA can be amplified.
- RNA is prepared by reverse transcription using an RNA-dependent DNA polymerase, DNA is amplified by PCR using the cDNA as a template, and the resulting DNA amplification product is used as a template.
- the RNA may be amplified by transcribing the RNA using a DNA-dependent RNA polymerase.
- the HMGB1-binding nucleic acid molecule of the present invention is DNA
- the DNA can be amplified by a polymerase chain reaction (PCR) method or the like.
- the X region can be exemplified by the x sequence described above, for example.
- the base sequences of the Y region and Y ′ region are not particularly limited and can be arbitrarily determined.
- the sequence of the Y region include, for example, a sequence including the base sequence represented by any of SEQ ID NO: 43 and SEQ ID NO: 115 and a sequence consisting of the base sequence.
- the Y region can be exemplified by the above-described y arrangement, for example. These sequences are merely examples, and do not limit the present invention.
- sequence of the Y ′ region examples include, for example, a sequence containing the base sequence represented by SEQ ID NO: 44 and a sequence consisting of the base sequence.
- the Y ′ region can be exemplified by the y ′ sequence described above, for example. These sequences are merely examples, and do not limit the present invention. ucagugccuggacgugcagu (SEQ ID NO: 44)
- HMGB1-binding nucleic acid molecule of the present invention may have a secondary structure by self-annealing, for example.
- the secondary structure include a stem loop structure.
- the stem loop structure may be formed, for example, by forming any of the Y region, the X region, and the Y ′ region into a double strand.
- a part of the Y region may form a stem-loop structure by forming a double strand with a part of the X region, or a part of the Y ′ region may A stem loop structure may be formed by forming a double strand with a part of the X region.
- a part of the Y region and a part of the Y ′ region may form a stem-loop structure by forming a double strand with a part of the X region, respectively.
- a stem loop structure may be formed by forming a double strand inside the Y region, or a stem loop structure may be formed by forming a double strand inside the Y ′ region.
- a stem loop structure may be formed inside the Y region and the Y ′ region.
- HMGB1-binding nucleic acid molecule of the present invention can bind to HMGB1, it can be used, for example, as a neutralizing agent that neutralizes the function of HMGB1 by binding to HMGB1.
- HMGB1-binding nucleic acid molecule of the present invention can bind to HMGB1 as described above, it can be used, for example, as an inhibitor that inhibits the function of HMGB1 by binding to HMGB1.
- HMGB1-binding nucleic acid molecule of the present invention can bind to HMGB1 as described above, it can be used as a pharmaceutical for preventing or treating a disease caused by the expression of HMGB1.
- the medicament of the present invention can be used as, for example, an anticancer agent, an anti-inflammatory agent, an anti-stroke agent and the like.
- the neutralizing agent of the present invention, the inhibitor of the present invention, and the pharmaceutical agent of the present invention only need to contain the HMGB1-binding nucleic acid molecule of the present invention, and other configurations are not limited at all.
- the neutralizing agent of the present invention, the inhibitor of the present invention, and the pharmaceutical agent of the present invention may each contain, for example, a carrier.
- a carrier for example, the same as the composition shown below Can be used in the same manner.
- composition of the present invention comprises the HMGB1-binding nucleic acid molecule of the present invention.
- the composition of this invention should just contain the HMGB1 binding nucleic acid molecule of the said this invention, and another structure is not restrict
- composition of the present invention can bind to HMGB1 as described above, it can be used, for example, as a neutralizing agent that neutralizes the function of HMGB1 by binding to HMGB1.
- composition of the present invention can bind to HMGB1 as described above, it can be used, for example, as an inhibitor that inhibits the function of HMGB1 by binding to HMGB1.
- composition of the present invention can bind to HMGB1 as described above, it can be used as a pharmaceutical for preventing or treating a disease caused by the expression of HMGB1.
- the medicament of the present invention can be used as, for example, an anticancer agent, an anti-inflammatory agent, an anti-stroke agent and the like.
- the application target of the composition of the present invention is not particularly limited, and can be appropriately determined according to the use.
- Examples of the application target include cells, tissues, and living bodies.
- the origin of the cells and tissues and the type of living body are not particularly limited.
- Examples of the living body include organisms having the HMGB1 gene and / or the HMGB1 ortholog gene, and specific examples include animals such as humans, non-human mammals other than humans, birds, and fish.
- the administration method is not particularly limited, and examples thereof include oral administration and parenteral administration.
- the parenteral administration include intravenous administration, arterial administration, administration to lymphatic vessels, intramuscular administration, subcutaneous administration, rectal administration, transdermal administration, intraperitoneal administration, and local administration.
- composition of the present invention may contain, for example, various additives in addition to the HMGB1-binding nucleic acid molecule of the present invention.
- the additive is not particularly limited, and can be appropriately determined depending on the use of the composition of the present invention, for example.
- the composition of the present invention preferably further contains a carrier as the additive.
- the carrier is not particularly limited, and examples thereof include nanoparticles, liposomes, micelles, reverse micelles, polycations, cell membrane permeable peptides, magnetic particles, and calcium phosphate.
- the nanoparticles are not particularly limited, and examples thereof include nanocarbons such as carbon nanohorns and carbon nanotubes. Any one kind of these carriers may be used, or two or more kinds may be used in combination.
- the additive include a buffer, a metal salt, and a surfactant.
- the detection reagent of the present invention is the HMGB1 detection reagent for detecting HMGB1, comprising the HMGB1-binding nucleic acid molecule of the present invention.
- the present invention is not limited as long as it contains the HMGB1-binding nucleic acid molecule of the present invention.
- the HMGB1 binding nucleic acid molecule of the present invention can bind to HMGB1. Therefore, for example, HMGB1 in a sample can be qualitatively or quantified by confirming the presence or absence of binding between the HMGB1 binding nucleic acid molecule of the present invention and HMGB1 using the detection reagent of the present invention.
- the method for confirming the presence or absence of binding between the HMGB1-binding nucleic acid molecule and HMGB1 is not particularly limited, and a known method for detecting binding between a nucleic acid and a protein can be used. Thus, if the detection reagent of this invention is used, since HMGB1 can be detected easily, it is useful, for example in the field of biochemistry or clinical.
- the therapeutic method of the present invention comprises a step of administering the HMGB1-binding nucleic acid molecule of the present invention to a subject having a disease associated with the HMGB1.
- the disease involving HMGB1 is not particularly limited, and examples thereof include at least one disease selected from the group consisting of cancer, inflammation, and stroke. Examples of the cancer include breast cancer, colon cancer, melanoma, prostate cancer, pancreatic cancer and lung cancer.
- the treatment method of the present invention for example, prevention of the disease, suppression of progression of the disease, treatment of the disease, and the like are possible.
- the treatment method of the present invention includes the meaning of a prevention method, and may include a step of administering the HMGB1-binding nucleic acid molecule of the present invention to a subject at risk of the disease.
- the administration method, administration conditions, etc. of the HMGB1-binding nucleic acid molecule of the present invention are not particularly limited and are as described above.
- the administration subject for example, patient
- the living body include organisms having the HMGB1 gene and / or the HMGB1 ortholog gene, and specific examples include animals such as humans, non-human mammals other than humans, birds, and fish.
- the composition of the present invention may be administered.
- the present invention is characterized in that it is a nucleic acid molecule for use in the treatment of a disease involving HMGB1.
- the nucleic acid molecule is the HMGB1-binding nucleic acid molecule of the present invention.
- the HMGB1-binding nucleic acid molecule of the present invention is as described above.
- the present invention is also characterized in that it is a composition for use in the treatment of a disease involving HMGB1.
- the composition is the composition of the present invention comprising the HMGB1-binding nucleic acid molecule of the present invention.
- the composition of the present invention is as described above.
- RNA aptamers capable of binding to HMGB1 were prepared, and the binding ability to HMGB1 was confirmed for each RNA aptamer.
- RNA aptamer # 47 (SEQ ID NO: 65), # 80 (SEQ ID NO: 67), # 06 (SEQ ID NO: 56), # 36 (SEQ ID NO: 60), # 34 (SEQ ID NO: 63), # 08 (SEQ ID NO: 46), each RNA aptamer of # 10 (SEQ ID NO: 58) was prepared by a known nucleic acid synthesis method and used as an RNA aptamer in the examples.
- An RNA library (40N) containing a plurality of RNAs consisting of the oligonucleotide represented by SEQ ID NO: 82 including a 40-base long random sequence was used as a comparative RNA (hereinafter the same).
- n is adenine, guanine, cytosine, thymine or uracil.
- 40N SEQ ID NO: 82
- a BIACORE dedicated sensor chip (trade name Sensor Chip SA, manufactured by GE Healthcare) was set on the BIACORE (registered trademark) X. 5 ⁇ mol / L of biotinylated deoxythymidine was injected into the flow cell 2 of the sensor chip using a running buffer and allowed to bind until the signal intensity (RU: Resonance Unit) was about 1000 RU.
- RU Resonance Unit
- the biotinylated deoxythymidine a 20-base deoxythymidine having a biotinylated 5 ′ end was used.
- RNA aptamer was injected into the flow cells 1 and 2 of the chip for 1 minute at a flow rate of 20 ⁇ L / min using a running buffer, and was bound until the signal intensity reached about 1000 RU.
- 675 nmol / L of the His-tag-added HMGB1 was injected with the running buffer at a flow rate of 20 ⁇ L / min for 30 seconds, followed by washing with the running buffer flowing under the same conditions.
- the signal intensity was measured.
- the composition of the running buffer was 20 mmol / L HEPES, 500 mmol / L NaCl, 0.1 mmol / L MgCl 2 , 0.1% Triton X-100 (registered trademark), and its pH was 7.2. Further, tRNA was used at a concentration of 1 mg / mL as a blocking agent for suppressing nonspecific binding.
- the signal intensity was measured in the same manner except that the RNA of the comparative example was used instead of the RNA aptamer of the above example.
- FIG. 1 is a graph showing the binding ability of each RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (second).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 30 seconds is the His- The tag addition HMGB1 injection time
- after 30 seconds is the washing time with the running buffer.
- the dissociation constant between each RNA aptamer and the His-tag added HMGB1 was determined from the obtained signal intensity.
- Table 2 the RNA of the comparative example has a dissociation constant of 1.04 ⁇ 10 ⁇ 6
- each RNA aptamer of the above example has a dissociation constant of 10 ⁇ 13 to 10 ⁇ .
- An excellent value of 7 orders was shown, and it was found that the binding ability to the His-tag added HMGB1 was excellent.
- the # 06 and # 08 RNA aptamers have dissociation constants on the order of 10 ⁇ 10 and 10 ⁇ 11 , respectively, which are well below the general order of antibodies (10 ⁇ 9 order). High binding ability was shown.
- the # 47 and # 80 RNA aptamers show 10 ⁇ 12 and 10 ⁇ 13 orders, respectively, which are orders not reported at the time of filing.
- FIG. 2 is a graph showing the binding ability of each RNA aptamer to HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the HMGB1 injection start time
- 0 to 60 seconds is the HMGB1 injection time.
- the time after 60 seconds is the time for washing with the running buffer.
- each RNA aptamer of the Example also showed high binding ability for HMGB1 to which no His-tag was added. From this result, it was found that the RNA aptamer of Example was not an His-tag but an RNA aptamer that binds to HMGB1.
- FIG. 3 is a graph showing the binding ability of each RNA aptamer to the His-tag added MIF protein.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added MIF protein
- 0 seconds to 30 seconds is the His.
- -Injection time of tag-added MIF protein, and after 30 seconds is the time for washing with the running buffer.
- each RNA aptamer of the example showed a slight increase in signal intensity during the injection of His-tag-added MIF protein (0 to 30 seconds), but it increases with time. However, after 30 seconds of washing, a rapid decrease in signal intensity was observed, and the signal intensity eventually became zero. From this, it was confirmed that each RNA aptamer of Example does not bind to His-tag.
- FIG. 4 is a graph showing the binding ability of each RNA aptamer to HMGB1 under physiological conditions.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -20 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the HMGB1 injection start time
- 0 seconds to 45 seconds is the HMGB1 injection time.
- the time after 45 seconds is the time for washing with the running buffer.
- each RNA aptamer of the Example showed high binding ability to the HMGB1 even under physiological conditions. From this result, it was found that the RNA aptamer of Example was an aptamer that binds to HMGB1 even under physiological conditions. Therefore, it can be said that the RNA aptamer of the present invention can bind to HMGB1 in the living body with excellent binding ability even when administered to the living body, for example.
- RNA aptamers capable of binding to HMGB1 were prepared, and the binding ability to HMGB1 was confirmed for each RNA aptamer.
- RNA aptamer of R8c6_1 SEQ ID NO: 74
- R8c6_14 SEQ ID NO: 75
- FIG. 5 is a graph showing the binding ability of each RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -20 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 60 seconds is the His- The tag addition HMGB1 injection time
- 60 seconds is the washing time with the running buffer.
- RNA aptamer As shown in FIG. 5, according to each RNA aptamer of the Example, since the increase in signal intensity was observed, it was found that the RNA aptamer was bound to the HMGB1. In addition, the dissociation constants of the RNA aptamers R8c6_1 and R8c6_14 were extremely low, 7.52 ⁇ 10 ⁇ 14 and 1.06 ⁇ 10 ⁇ 13 , respectively, indicating very excellent binding ability.
- RNA aptamer of R8c6_1 (SEQ ID NO: 74) was further miniaturized and the binding ability to HMGB1 was confirmed.
- RNA aptamer Each RNA aptamer shown in the following Table 3 was prepared by a known nucleic acid synthesis method, and used as an RNA aptamer in Examples.
- the RNA aptamer had a nucleotide sequence in which the 5 ′ region or the 3 ′ region of R8c6_1 was deleted.
- FIG. 6 is a graph showing the binding ability of each RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -20 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 60 seconds is the His- The tag addition HMGB1 injection time
- 60 seconds is the washing time with the running buffer.
- each RNA aptamer in which R8c6_1 was miniaturized was bound to the HMGB1 because an increase in signal intensity was observed.
- R8c6_1-15, R8c6_1-18, R8c6_1-20, and R8c6_1-21, in which the 5 ′ side of R8c6_1 was deleted, showed binding ability superior to R8c6_1.
- RNA aptamer of R8c6_1 (SEQ ID NO: 74) was fluorinated to confirm the binding ability to HMGB1.
- RNA aptamer Using 2′-fluoro-CTP and 2′-fluoro-UTP (hereinafter the same) in which the 2 ′ position of the ribose residue is fluorinated, each of those shown in Table 3 of Example 3 is used.
- An RNA aptamer was prepared by a known nucleic acid synthesis method and used as a fluorinated RNA aptamer in the examples.
- fluorinated RNA aptamer cytosine nucleotide residues and uracil nucleotide residues are fluorinated in the base sequences shown in Table 3 above.
- Each fluorinated RNA aptamer is represented by 2'F-R8c6_1, 2'F-R8c6_1-1, 2'F-R8c6_1-4, 2'F-R8c6_1-15, 2'F-R8c6_1-18, 2'F, respectively. This is represented as -R8c6_1-21.
- N40 the cytosine nucleotide residue and uracil nucleotide residue were fluorinated to produce fluorinated N40 (2′F-N40), which was used as a comparative example (the same applies hereinafter).
- FIG. 7 is a graph showing the binding ability of each fluorinated RNA aptamer to HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -20 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the HMGB1 injection start time
- 0 to 60 seconds is the HMGB1 injection time.
- the time after 60 seconds is the time for washing with the running buffer.
- each of the fluorinated RNA aptamers in which R8c6_1 was miniaturized was found to be bound to the HMGB1 because an increase in signal intensity was observed.
- 2'F-R8c6_1-18 exhibited excellent binding ability equivalent to unmodified R8c6_1.
- Fluorinated RNA aptamers are generally known to exhibit RNase resistance. For this reason, since the fluorinated RNA aptamer having the binding ability to HMGB1 is difficult to be decomposed even when administered in vivo, for example, it can be said that it is useful for pharmaceuticals and the like.
- R8c6_1 (SEQ ID NO: 74) and its miniaturized RNA aptamer R8c6_1-18 (SEQ ID NO: 87) were each fluorinated to confirm the binding ability to HMGB1.
- RNA aptamer Using the 2′-fluoro-CTP and the 2′-fluoro-UTP, the RNA aptamers of R8c6_1 and R8c6_1-18 were prepared by a known nucleic acid synthesis method, and It was used as a modified RNA aptamer.
- fluorinated RNA aptamer cytosine nucleotide residues and uracil nucleotide residues are fluorinated in the base sequences of R8c6_1 (SEQ ID NO: 74) and R8c6_1-18 (SEQ ID NO: 87).
- Each fluorinated RNA aptamer is represented as 2'F-R8c6_1, 2'F-R8c6_1-18, respectively.
- FIG. 8 is a graph showing the binding ability of each fluorinated RNA aptamer to the His-tag added HMGB1.
- the vertical axis indicates the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis indicates the analysis time (seconds).
- -20 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 60 seconds is the His- The tag addition HMGB1 injection time
- 60 seconds is the washing time with the running buffer.
- each of the fluorinated RNA aptamers was found to be bound to the HMGB1 since an increase in signal intensity was observed.
- 2'F-R8c6_1-18 exhibited excellent binding ability equivalent to unmodified R8c6_1.
- FIG. 9 is a photograph of a pull-down assay showing the binding between HMGB1 and TLR-2.
- tRNA “ ⁇ ” indicates the use of the binding buffer without tRNA added
- tRNA “+” indicates the use of the binding buffer with tRNA added.
- lane “M” is a molecular weight marker
- lane “ ⁇ ” is a result of tRNA not added and RNA aptamer not added
- lane “+” is a result of tRNA added and RNA aptamer not added
- lane “40N” indicates the results of addition amounts of 4 ⁇ g, 2 ⁇ g, and 1 ⁇ g, respectively
- lane “R8c6_1-18” indicates the results of addition amounts of 4 ⁇ g, 2 ⁇ g, and 1 ⁇ g, respectively.
- the relative value with the detected HMGB1 amount corrected by the TLR-2 amount and the correction value of lane “+” as 100 is shown below each lane.
- HMGB1-binding nucleic acid molecule of the present invention is useful for the treatment of diseases involving HMGB1, for example.
- RNA aptamer was fluorinated to confirm the binding ability to HMGB1.
- RNA aptamer Using the 2′-fluoro-CTP and the 2′-fluoro-UTP, each RNA aptamer shown in the following Table 4 was prepared by a known nucleic acid synthesis method, and the fluorinated RNA aptamer of Example Used as. In the fluorinated RNA aptamer, cytosine nucleotide residues and / or uracil nucleotide residues are fluorinated in the base sequences shown in Table 4 below.
- RNA aptamers in which uracil nucleotide residues and cytosine nucleotide residues are fluorinated are converted into 2′F-CU-R8c6_1, 2′F-CU- # 06, 2′F-CU-CU # 80, 2′F, respectively. This is represented as -CU-R4_9068.
- RNA aptamers in which only cytosine nucleotide residues are fluorinated are designated as 2'FC-R8c6_1, 2'FC- # 06, 2'FC- # 80, and 2'FC-R4_9068, respectively. Represent.
- RNA aptamers in which only uracil nucleotide residues are fluorinated are designated as 2′F-U-R8c6_1, 2′F-U- # 06, 2′F-U- # 80, and 2′F-U-R4 — 9068, respectively. Represent.
- FIG. 10 is a graph showing the binding ability of each fluorinated RNA aptamer to the His-tag added HMGB1.
- the vertical axis indicates the signal intensity (RU) measured with the BIACORE (registered trademark) X at the end of the injection of the His-tag added HMGB1.
- the graph shows the results of 40N, # 06, # 80, R8c6_1, and HMGB1R4_9068 from the left.
- “2′F-CU-modified” is the result of RNA aptamer in which both cytosine nucleotide residues and uracil nucleotide residues are fluorinated
- “2′F-C-modified” is cytosine.
- RNA aptamer in which only uracil nucleotide residues are fluorinated
- non-modified is the result of RNA aptamer in which only nucleotide residues are fluorinated.
- each fluorinated RNA aptamer showed an increase in signal intensity higher than that of the unmodified 40N of the comparative example, indicating that it was bound to the HMGB1.
- 2′F-CU-R8c6_1, 2′F-C-R8c6_1 and 2′F-U-R8c6_1 show excellent binding ability, and in particular, 2′F-CU-R8c6_1 is an unmodified R8c6_1 and The binding ability was equivalent.
- Example 8 The secondary structures of R8c6_1 (SEQ ID NO: 74) and R8c6_1-18 (SEQ ID NO: 87) were estimated. These secondary structures are shown in FIG.
- RNA aptamer of R8c6_1 (SEQ ID NO: 74) was further miniaturized and the binding ability to HMGB1 was confirmed.
- RNA aptamer Each RNA aptamer shown in Table 5 and Table 6 below was prepared by a known nucleic acid synthesis method, and used as an RNA aptamer in Examples.
- the RNA aptamer in Table 5 is an aptamer obtained by miniaturizing R8c6_1.
- FIG. 18 shows a schematic diagram of a presumed secondary structure of R8c6_1
- FIG. 19 shows a predicted secondary structure of R8c6_1-18.
- R8c6_1-25 and R8c6_1-18 have base sequences in which the 5 'region is deleted from R8c6_1.
- R8c6_1-18-S2, R8c6_1-18-S4 and R8c6_1-18-S6 have their respective 5′-region deleted from R8c6_1 as in R8c6_1-18, and S2 shown in FIG.
- the nucleotide sequence was deleted from the S4 or S6 region.
- S2 and S4 are each a base pair region forming a stem structure
- S6 is a region of a stem loop structure.
- RNA aptamer in Table 6 is an aptamer obtained by further reducing the size of R8c6_1-25-S6 in Table 5.
- FIG. 20 shows a schematic diagram of the predicted secondary structure of R8c6_1-25-S6.
- R8c6_1-25-S6A2, R8c6_1-25-S6C, and R8c6_1-25-S8 each had a nucleotide sequence lacking the regions A2, C, and S8 shown in FIG.
- R8c6_1-26-S6 was a base sequence in which one base on the 5 'side of R8c6_1-25-S6 was deleted.
- FIGS. 12 and 13 are graphs showing the binding ability of each RNA aptamer to the His-tag added HMGB1, respectively.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 30 seconds is the His- The tag addition HMGB1 injection time
- after 30 seconds is the washing time with the running buffer.
- each RNA aptamer having a reduced R8c6_1 size was found to be bound to the HMGB1 because an increase in signal intensity was observed.
- R8c6_1-18-S2 and R8c6_1-25 showed binding ability superior to R8c6_1.
- RNA aptamer was fluorinated to confirm the binding ability to HMGB1.
- RNA aptamer Using the 2′-fluoro-CTP and the 2′-fluoro-UTP, each RNA aptamer shown in the following Table 7 was prepared by a known nucleic acid synthesis method, and the fluorinated RNA aptamer of Example Used as. In the fluorinated RNA aptamer, cytosine nucleotide residues and uracil nucleotide residues are fluorinated in the base sequences shown in Table 7.
- RNA aptamers in which uracil nucleotide residues and cytosine nucleotide residues are fluorinated are converted into 2'F-R8c6_1-18-S6, 2'F-R8c6_1-25, 2'F-R8c6_1-25-S6, 2 ', respectively.
- the N40 was also fluorinated using the 2′-fluoro-CTP and the 2′-fluoro-UTP.
- FIG. 14 is a graph showing the binding ability of each fluorinated RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 30 seconds is the His- The tag addition HMGB1 injection time
- after 30 seconds is the washing time with the running buffer.
- each of the miniaturized fluorinated RNA aptamers was found to be bound to the HMGB1 because an increase in signal intensity was observed.
- 2'F-R8c6_1-18-S6 and 2'-R8c6_1-25 showed excellent binding ability.
- RNA aptamer of R8c6_1 (SEQ ID NO: 74) was further miniaturized and the binding ability to HMGB1 was confirmed.
- RNA aptamer Each RNA aptamer shown in the following Table 8 was prepared by a known nucleic acid synthesis method, and used as an RNA aptamer in Examples.
- RNA aptamer in Table 8 is an aptamer obtained by miniaturizing R8c6_1.
- R8c6_1-21-S6, R8c6_1-22-S6, R8c6_1-23-S6, and R8c6_1-24-S6 each had a nucleotide sequence in which the 5 'region was deleted from R8c6_1-18-S6.
- R8c6_1-21-S8, R8c6_1-22-S8, R8c6_1-23-S8, R8c6_1-24-S8 are arranged on the 5 ′ side as R8c6_1-21-S6, R8c6_1-22-S6, R8c6_1-, respectively.
- 23-S6 and R8c6_1-24-S6, and the nucleotide sequence lacking the same region as S8 shown in FIG. 20 was used.
- FIG. 15 is a graph showing the binding ability of each RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (second).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 30 seconds is the His- The tag addition HMGB1 injection time
- after 30 seconds is the washing time with the running buffer.
- each of the RNA aptamers in which R8c6_1 was miniaturized was found to be bound to the HMGB1 because an increase in signal intensity was observed.
- RNA aptamer was fluorinated to confirm the binding ability to HMGB1.
- RNA aptamer Using the 2′-fluoro-CTP and the 2′-fluoro-UTP, each RNA aptamer shown in Table 9 and Table 10 below was prepared by a known nucleic acid synthesis method. Used as fluorinated RNA aptamer. In the fluorinated RNA aptamer, cytosine nucleotide residues and uracil nucleotide residues are fluorinated in the base sequences shown in Table 9 and Table 10.
- RNA aptamers in which uracil nucleotide residues and cytosine nucleotide residues are fluorinated are converted to 2′F-R8c6_1-21-S6, 2′F-R8c6_1-12-S6, 2′F-R8c6_1-23-S6, respectively.
- FIGS. 16 and 17 are graphs showing the binding ability of each fluorinated RNA aptamer to the His-tag added HMGB1.
- the vertical axis represents the signal intensity (RU) measured by the BIACORE (registered trademark) X
- the horizontal axis represents the analysis time (seconds).
- -10 seconds to 0 seconds is the pre-washing time with the running buffer
- 0 seconds is the injection start time of the His-tag added HMGB1
- 0 seconds to 30 seconds is the His- The tag addition HMGB1 injection time
- after 30 seconds is the washing time with the running buffer.
- each of the miniaturized fluorinated RNA aptamers was found to be bound to the HMGB1 because an increase in signal intensity was observed.
- 2'F-R8c6_1-21-S6 and 2'F-R8c6_1-21-S8 showed excellent binding ability.
- RNA aptamer R8c6_1-18-S6 SEQ ID NO: 95
- HMGB1 HMGB1
- Anti-his antibody (product name: Penta / His Antibody: QIAGEN) was diluted to 4 ug / mL using a carbonate buffer (pH 9.0). 50 ⁇ L of this diluted antibody solution was dispensed into each well of a 96-well plate (product name: 96-well Elisa plate: Asahi Techno Glass) and allowed to stand at room temperature for 2 hours. Furthermore, 50 ⁇ L of 1% BSA / TBS was dispensed into each well of the plate and allowed to stand overnight. The composition of TBS was 20 mmol / L Tris-HCl (pH 7.6) and 0.9% NaCl. In this way, the plate was blocked.
- the well of the plate subjected to the blocking treatment was washed with 200 ⁇ L of buffer three times.
- the composition of the buffer was 10 mmol / L HEPES, 500 mmol / L NaCl, 0.1 mmol / L MgCl 2 , 0.1% Triton X-100 (registered trademark), and the pH was 7.2.
- HMGB1 (product name: HMG-1: Sigma) was diluted with the buffer containing tRNA to a predetermined concentration (1 ⁇ g / mL, 2 ⁇ g / mL) to prepare the HMGB1 diluted solution.
- the RNA aptamer was diluted with the buffer, and the RNA aptamer dilution was heated at 95 ° C. for 5 minutes, and then returned to room temperature.
- the RNA aptamer dilution was mixed with an RNase inhibitor (product name: RNase Inhibitor: TOYOBO) and biotinylated deoxythymidine to prepare an RNA aptamer mixture.
- the biotinylated deoxythymidine used was a 20-base deoxythymidine with a biotinylated 5 'end.
- the concentration of the RNA aptamer was 10 ⁇ g / mL and 20 ⁇ g / mL.
- the RNA aptamer mixed solution was set so that the RNase inhibitor was 0.2 ⁇ L and the biotinylated deoxythymidine was 0.2 ⁇ L per 50 ⁇ L (that is, per well).
- HMGB1 diluted solution 50 ⁇ L was dispensed into each well of the washed plate, allowed to stand at 4 ° C. for 2 hours, and then washed 3 times with 200 ⁇ L of the buffer.
- 50 ⁇ L of the RNA aptamer mixture was further dispensed into each well and reacted at 4 ° C. for 2 hours.
- the ratio between the concentration of the HMGB1 diluted solution (50 ⁇ L) added to each well and the concentration of the RNA aptamer mixed solution (50 ⁇ L) was 1:20, 2:20, 1:10, 2:10.
- FIG. 21 is a graph showing the interaction between HMGB1 and the RNA aptamer, and the vertical axis shows the absorbance.
- the absorbance indicating the binding between HMGB1 and the RNA aptamer increased, it was confirmed that the RNA aptamer bound to HMGB1.
- the absorbance increased by increasing the HMGB1 concentration relative to the RNA aptamer concentration. From this, it can be said that HMGB1 can be quantified according to the HMGB1-binding nucleic acid molecule of the present invention.
- HMGB1 can be detected by confirming the presence or absence of binding to HMGB1, and it can also be used for elucidating the function of HMGB1, so that it is also useful as a new research tool. is there.
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- General Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Medicinal Chemistry (AREA)
- Biomedical Technology (AREA)
- Genetics & Genomics (AREA)
- Veterinary Medicine (AREA)
- Public Health (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Biochemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Immunology (AREA)
- Biotechnology (AREA)
- Zoology (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Urology & Nephrology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- General Engineering & Computer Science (AREA)
- Hematology (AREA)
- Microbiology (AREA)
- Wood Science & Technology (AREA)
- Epidemiology (AREA)
- Cell Biology (AREA)
- Food Science & Technology (AREA)
- Analytical Chemistry (AREA)
- Toxicology (AREA)
- Pathology (AREA)
- Plant Pathology (AREA)
- General Physics & Mathematics (AREA)
- Gastroenterology & Hepatology (AREA)
- Heart & Thoracic Surgery (AREA)
Abstract
Description
本発明のHMGB1結合核酸分子は、前述のように、HMGB1との解離定数が5×10-7以下であることを特徴とする、HMGB1に結合可能な核酸分子である。本発明のHMGB1結合核酸分子は、例えば、HMGB1アプタマーともいう。
(A1)配列番号1~42のいずれかで表わされる塩基配列を含む核酸分子
(A2)配列番号1~42のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(B1)配列番号45~81のいずれかで表わされる塩基配列を含む核酸分子
(B2)配列番号45~81のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(A1)配列番号1~42のいずれかで表わされる塩基配列を含む核酸分子
mmhbuaagmcacguagmaccag
C_1(配列番号2)
accguaagacacguagaaccag
C_2(配列番号3)
aauuuaagccacguagaaccag
C_3(配列番号4)
acaguaagacacguagcaccag
C_4(配列番号5)
cauguaagccacguagaaccag
#04(配列番号6)
ucccaugauuguucaggcacggccuuucgguucccucaau
#08(配列番号7)
agucccuugacacguccguuuucuaacuggaauagaggcc
#12(配列番号8)
gggcugcaccucuccgcuacguugucguuggaggcaccau
#43(配列番号9)
gguauuaaaacucccucguaggucauccgcccggccuagc
#49(配列番号10)
cauccuuaucacauggucauccgcccggccaugcaauguu
#32(配列番号11)
cauucuaaauucuaucaagggucauccgcccggcccgcau
#58(配列番号12)
cauucuaaauucuaucaagggucauccgcccggccgcgcucgccaguca
#01(配列番号13)
uggcauccuugcucacuccaggcuaaaccucucgguuccc
#26(配列番号14)
ccaagcacuucaucgucuaggcaauugccucucgguaccc
#73(配列番号15)
ccacaagcucgcacuaguuccaggcuuccucucgguaccc
#77(配列番号16)
cauguauuucugcacguuccagagaauccucucgguaccc
#06(配列番号17)
uacacugcacgcuccgcuuugaacaucaauggaggcccug
#22(配列番号18)
gcgcucgcucauagucaaggugaaaacccccauagagacu
#10(配列番号19)
uagucaaggugaaaacccccauagagacu
#21(配列番号20)
ggccugugcuaacaugagucauccguccggcucgcaacuc
#36(配列番号21)
ccuagcacguccguuucuggaucugucaguuagaggccua
#15(配列番号22)
gcaucaaccucuguaagagcgcgcuuugcuucaccaaaaa
#23(配列番号23)
acgguccuuaaaaucuuccuuaaccacgcccaggaucuua
#34(配列番号24)
auucaccucagcauguccgcuugugacgauggaggcaccu
#40(配列番号25)
gguccuuaaaaucuuccaaucuaaacgauccagacacggc
#47(配列番号26)
aaaaacuacugccgaaccguaagacacguagaaccaggca
#79(配列番号27)
gaccagguuccugacaucucugaacuauaccuccaaaacg
#80(配列番号28)
caucugaauuuaagccacguagaaccaggcccuccacgcg
#82(配列番号29)
uaauacgacucacuauagggacgcucacguacgcucagug
R4_1(配列番号30)
aaugagggcccacuuccggaucuuugguuugcuuccuugc
R4_4(配列番号31)
ucgcuuauggaugcccacuuccacucacuguccugcgcaa
R4_10(配列番号32)
uauuaauaccucagcccucuucucuuagucuggugccgau
R4_11(配列番号33)
ucucuuuucgaauuccguucuggcucacuccuuggguauu
R4_12(配列番号34)
cugacaucuuuuacacugauuucuguuggcccacuucugu
R8c6_1(配列番号35)
gaguacaguaagacacguagcaccagucugacguuugucg
R8c6_14(配列番号36)
ugccaucaccauguaagccacguagaaccagcacuacuag
R8c9_1(配列番号37)
ugagucuuauagccguccguuuacguuugucuagaggcca
R8c9_6(配列番号38)
gcuucuugcauuguccgcuuaguuucuauggaggcauagu
R8c9_10(配列番号39)
ccgaauauuuuugcaccguccgauugccaugcauugaggc
HMGB1R4_9068(配列番号40)
ugauauuuaaauuuggccgcguuuaaaacauccccuacga
HMGB1R4_2478(配列番号41)
gauuccguugcccuuccguugaacugugccaggcuuuuug
HMGB1R4_5108(配列番号42)
accuuugccgcaucucacccacgucuugucaggccguuuc
gggacgcucacguacgcuca(配列番号43)
acgcucacguacgcuca(配列番号115)
ucagugccuggacgugcagu(配列番号44)
#04(配列番号45)
gggacgcucacguacgcucaucccaugauuguucaggcacggccuuucgguucccucaauucagugccuggacgugcagu
#08(配列番号46)
gggacgcucacguacgcucaagucccuugacacguccguuuucuaacuggaauagaggccucagugccuggacgugcagu
#12(配列番号47)
gggacgcucacguacgcucagggcugcaccucuccgcuacguugucguuggaggcaccauucagugccuggacgugcagu
#43(配列番号48)
gggacgcucacguacgcucagguauuaaaacucccucguaggucauccgcccggccuagcucagugccuggacgugcagu
#49(配列番号49)
gggacgcucacguacgcucacauccuuaucacauggucauccgcccggccaugcaauguuucagugccuggacgugcagu
#32(配列番号50)
gggacgcucacguacgcucacauucuaaauucuaucaagggucauccgcccggcccgcauucagugccuggacgugcagu
#58(配列番号51)
gggacgcucacguacgcucacauucuaaauucuaucaagggucauccgcccggccgcgcucgccagucaucagugccuggacgugcagu
#01(配列番号52)
gggacgcucacguacgcucauggcauccuugcucacuccaggcuaaaccucucgguucccucagugccuggacgugcagu
#26(配列番号53)
gggacgcucacguacgcucaccaagcacuucaucgucuaggcaauugccucucgguacccucagugccuggacgugcagu
#73(配列番号54)
gggacgcucacguacgcucaccacaagcucgcacuaguuccaggcuuccucucgguacccucagugccuggacgugcagu
#77(配列番号55)
gggacgcucacguacgcucacauguauuucugcacguuccagagaauccucucgguacccucagugccuggacgugcagu
#06(配列番号56)
gggacgcucacguacgcucauacacugcacgcuccgcuuugaacaucaauggaggcccugucagugccuggacgugcagu
#22(配列番号57)
gggacgcucacguacgcucagcgcucgcucauagucaaggugaaaacccccauagagacuucagugccuggacgugcagu
#10(配列番号58)
gggacgcucacguacgcucauagucaaggugaaaacccccauagagacuucagugccuggacgugcagu
#21(配列番号59)
gggacgcucacguacgcucaggccugugcuaacaugagucauccguccggcucgcaacucucagugccuggacgugcagu
#36(配列番号60)
gggacgcucacguacgcucaccuagcacguccguuucuggaucugucaguuagaggccuaucagugccuggacgugcagu
#15(配列番号61)
gggacgcucacguacgcucagcaucaaccucuguaagagcgcgcuuugcuucaccaaaaaucagugccuggacgugcagu
#23(配列番号62)
gggacgcucacguacgcucaacgguccuuaaaaucuuccuuaaccacgcccaggaucuuaucagugccuggacgugcagu
#34(配列番号63)
gggacgcucacguacgcucaauucaccucagcauguccgcuugugacgauggaggcaccuucagugccuggacgugcagu
#40(配列番号64)
gggacgcucacguacgcucagguccuuaaaaucuuccaaucuaaacgauccagacacggcucagugccuggacgugcagu
#47(配列番号65)
gggacgcucacguacgcucaaaaaacuacugccgaaccguaagacacguagaaccaggcaucagugccuggacgugcagu
#79(配列番号66)
gggacgcucacguacgcucagaccagguuccugacaucucugaacuauaccuccaaaacgucagugccuggacgugcagu
#80(配列番号67)
gggacgcucacguacgcucacaucugaauuuaagccacguagaaccaggcccuccacgcgucagugccuggacgugcagu
#82(配列番号68)
gggacgcucacguacgcucauaauacgacucacuauagggacgcucacguacgcucagugccuggacgugcagu
R4_1(配列番号69)
gggacgcucacguacgcucaaaugagggcccacuuccggaucuuugguuugcuuccuugcucagugccuggacgugcagu
R4_4(配列番号70)
gggacgcucacguacgcucaucgcuuauggaugcccacuuccacucacuguccugcgcaaucagugccuggacgugcagu
R4_10(配列番号71)
gggacgcucacguacgcucauauuaauaccucagcccucuucucuuagucuggugccgauucagugccuggacgugcagu
R4_11(配列番号72)
gggacgcucacguacgcucaucucuuuucgaauuccguucuggcucacuccuuggguauuucagugccuggacgugcagu
R4_12(配列番号73)
gggacgcucacguacgcucacugacaucuuuuacacugauuucuguuggcccacuucuguucagugccuggacgugcagu
R8c6_1(配列番号74)
gggacgcucacguacgcucagaguacaguaagacacguagcaccagucugacguuugucgucagugccuggacgugcagu
R8c6_14(配列番号75)
gggacgcucacguacgcucaugccaucaccauguaagccacguagaaccagcacuacuagucagugccuggacgugcagu
R8c9_1(配列番号76)
gggacgcucacguacgcucaugagucuuauagccguccguuuacguuugucuagaggccaucagugccuggacgugcagu
R8c9_6(配列番号77)
gggacgcucacguacgcucagcuucuugcauuguccgcuuaguuucuauggaggcauaguucagugccuggacgugcagu
R8c9_10(配列番号78)
gggacgcucacguacgcucaccgaauauuuuugcaccguccgauugccaugcauugaggcucagugccuggacgugcagu
HMGB1R4_9068(配列番号79)
gggacgcucacguacgcucaugauauuuaaauuuggccgcguuuaaaacauccccuacgaucagugccuggacgugcagu
HMGB1R4_2478(配列番号80)
gggacgcucacguacgcucagauuccguugcccuuccguugaacugugccaggcuuuuugucagugccuggacgugcagu
HMGB1R4_5108(配列番号81)
gggacgcucacguacgcucaaccuuugccgcaucucacccacgucuugucaggccguuucucagugccuggacgugcagu
モチーフ配列(配列番号114)
uaagncacguagnaccng
(A2)配列番号1~42のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(A3)配列番号1~42のいずれかで表わされる塩基配列において、60%以上の相同性を有する塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(A4)配列番号1~42のいずれかで表わされる塩基配列とストリンジェントな条件下でハイブリダイズする塩基配列または前記塩基配列に相補的な塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(B1)配列番号45~81のいずれかで表わされる塩基配列を含む核酸分子
モチーフ配列(配列番号114)
uaagncacguagnaccng
(b1)配列番号74で表わされる塩基配列を含む核酸分子
(B2)配列番号45~81のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(b2)配列番号74で表わされる塩基配列における連続する11塩基以上の部分配列を含み、且つ、HMGB1に結合可能である核酸分子
モチーフ配列(配列番号114)
uaagncacguagnaccng
(B3)配列番号45~81のいずれかで表わされる塩基配列において、60%以上の相同性を有する塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
(B4)配列番号45~81のいずれかで表わされる塩基配列とストリンジェントな条件下でハイブリダイズする塩基配列または前記塩基配列に相補的な塩基配列を含み、且つ、HMGB1に結合可能である核酸分子
gggacgcucacguacgcuca(配列番号43)
acgcucacguacgcuca(配列番号115)
ucagugccuggacgugcagu(配列番号44)
本発明の組成物は、前述のように、前記本発明のHMGB1結合核酸分子を含むことを特徴とする。本発明の組成物は、前記本発明のHMGB1結合核酸分子を含んでいればよく、その他の構成は、何ら制限されない。
本発明の検出試薬は、前記本発明のHMGB1結合核酸分子を含むことを特徴とする、前記HMGB1を検出するためのHMGB1検出試薬である。本発明は、前記本発明のHMGB1結合核酸分子を含んでいればよく、その他の構成は何ら制限されない。
本発明の治療方法は、前記HMGB1が関与する疾患の対象に、本発明のHMGB1結合核酸分子を投与する工程を含むことを特徴とする。前記HMGB1が関与する疾患は、特に制限されないが、例えば、癌、炎症および脳卒中からなる群から選択された少なくとも一つの疾患があげられる。前記癌は、例えば、乳癌、大腸癌、メラノーマ、前立腺癌、膵臓癌および肺癌等があげられる。本発明の治療方法によれば、例えば、前記疾患の予防、前記疾患の進行の抑制または前記疾患の治療等が可能である。本発明の治療方法は、予防方法の意味も含み、前記疾患の危険性がある対象に、本発明のHMGB1結合核酸分子を投与する工程を含んでもよい。本発明のHMGB1結合核酸分子の投与方法、投与条件等は、特に制限されず、前述の通りである。また、前記投与対象(例えば、患者)も、特に制限されない。前記生体は、例えば、HMGB1遺伝子および/またはHMGB1オーソログ遺伝子を有する生物があげられ、具体例としては、例えば、ヒト、ヒトを除く非ヒト哺乳類、鳥類、魚類等の動物があげられる。前記投与工程において、例えば、本発明の組成物を投与してもよい。
HMGB1に結合可能なRNAアプタマーを作製し、各RNAアプタマーについて、HMGB1に対する結合能を確認した。
#47(配列番号65)、#80(配列番号67)、#06(配列番号56)、#36(配列番号60)、#34(配列番号63)、#08(配列番号46)、#10(配列番号58)の各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のRNAアプタマーとして使用した。40塩基長のランダム配列を含む下記配列番号82で表わされるオリゴヌクレオチドからなるRNAを複数含むRNAライブラリー(40N)を、比較例のRNAとした(以下、同様)。配列番号82において、「n」は、アデニン、グアニン、シトシン、チミンまたはウラシルである。
40N(配列番号82)
gggacgcucacguacgcucannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnucagugccuggacgugcagu
前記各RNAアプタマーについて、N末端にHis-tagが付加されたHMGB1(His-tag付加HMGB1)に対する結合能を確認した。前記結合能の解析には、BIACORE(登録商標)X(GEヘルスケア社製)を、その使用説明書にしたがって使用した。
前記各RNAアプタマーについて、N末端にHis-tagが付加されていないHMGB1に対する結合能を確認した。前記結合能の解析は、前記His-tag付加HMGB1に代えて、1μmol/Lの前記HMGB1を使用し、前記HMGB1のインジェクション時間を60秒とした以外は、前記(2)と同様にして、結合能の解析を行った。これらの結果を、図2に示す。
前記各RNAアプタマーについて、His-tagに対する結合能を確認した。前記結合能の解析は、前記His-tag付加HMGB1に代えて、300nmol/Lの前記His-tag付加MIFタンパク質を使用した以外は、前記(2)と同様にして、結合能の解析を行った。これらの結果を、図3に示す。
生理条件下でのHMGB1に対する前記各RNAアプタマーの結合能を確認した。前記ランニングバッファーにおけるNaCl濃度500mmol/Lを、生体の生理条件である150mmol/Lに設定し、前記His-tag付加HMGB1のインジェクション時間を45秒とした以外は、前記(2)と同様にして、結合能の解析を行った。これらの結果を、図4に示す。
HMGB1に結合可能なRNAアプタマーを作製し、各RNAアプタマーについて、HMGB1に対する結合能を確認した。
R8c6_1(配列番号74)およびR8c6_14(配列番号75)の各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のRNAアプタマーとして使用した。
前記(1)で調製したRNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を1μmol/L、インジェクション時間を60秒とした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。これらの結果を、図5に示す。
R8c6_1(配列番号74)のRNAアプタマーを、さらに小型化し、HMGB1に対する結合能を確認した。
下記表3に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のRNAアプタマーとして使用した。前記RNAアプタマーは、それぞれ、R8c6_1の5’側の領域または3’側の領域を欠失した塩基配列とした。
前記RNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を635nmol/Lとし、前記His-tag付加HMGB1のインジェクション時間を60秒とした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。また、比較例として、前記40Nについても、同様に解析を行った。これらの結果を、図6に示す。
R8c6_1(配列番号74)の小型化RNAアプタマーをフルオロ化して、HMGB1に対する結合能を確認した。
リボース残基の2’位がフルオロ化された2’-フルオロ-CTPおよび2’-フルオロ-UTP(以下、同様)を用いて、前記実施例3の前記表3に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のフルオロ化RNAアプタマーとして使用した。前記フルオロ化RNAアプタマーは、前記表3に示す塩基配列において、シトシンヌクレオチド残基およびウラシルヌクレオチド残基がフルオロ化されている。各フルオロ化RNAアプタマーは、それぞれ、2’F-R8c6_1、2’F-R8c6_1-1、2’F-R8c6_1-4、2’F-R8c6_1-15、2’F-R8c6_1-18、2’F-R8c6_1-21と表わす。また、前記N40について、シトシンヌクレオチド残基およびウラシルヌクレオチド残基がフルオロ化し、フルオロ化N40(2’F-N40)を作製し、比較例として使用した(以下、同様)。
前記フルオロ化RNAアプタマーを使用し、His-tag付加HMGB1の濃度を200nmol/Lとし、前記His-tag付加HMGB1のインジェクション時間を60秒とした以外は、前記実施例1の前記(2)と同様にして、HMGB1に対する結合能の解析を行った。また、前記2’F-N40、フルオロ化していない未修飾のR8c6_1についても、同様に解析を行った。これらの結果を、図7に示す。
R8c6_1(配列番号74)およびその小型化RNAアプタマーであるR8c6_1-18(配列番号87)を、それぞれフルオロ化して、HMGB1に対する結合能を確認した。
前記2’-フルオロ-CTPおよび前記2’-フルオロ-UTPを用いて、前記R8c6_1および前記R8c6_1-18の各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のフルオロ化RNAアプタマーとして使用した。前記フルオロ化RNAアプタマーは、R8c6_1(配列番号74)およびR8c6_1-18(配列番号87)の塩基配列において、シトシンヌクレオチド残基およびウラシルヌクレオチド残基がフルオロ化されている。各フルオロ化RNAアプタマーは、それぞれ、2’F-R8c6_1、2’F-R8c6_1-18と表わす。
前記フルオロ化RNAアプタマーを使用し、His-tag付加HMGB1の濃度を200nmol/Lとし、前記ランニングバッファーにおけるNaClの濃度を150mmol/Lとし、前記His-tag付加HMGB1のインジェクション時間を60秒とした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。また、前記2’F-N40、フルオロ化していない未修飾のR8c6_1についても、同様に解析を行った。これらの結果を、図8に示す。
R8c6_1-18(配列番号87)による、HMGB1とレセプターTLR-2との結合阻害を確認した。
N末端にHis-tagが付加されたTLR-2(His-tag付加TLR-2)を使用した。2μgのHis-tag付加TLR-2、2μgのHMGB1および所定量のR8c6_1-18を、50μLのバインディングバッファーに添加し、この混合液を、4℃で20分間インキュベートした。R8c6_1-18の添加量は、1μg、2μgおよび4μgとした。前記バインディングバッファーの組成は、前記ランニングバッファーと同じであり、1mg/mL tRNA添加またはtRNA未添加とした。前記混合液に、Co2+が結合したアガロースビーズ1μL(製品名BD TALON(登録商標)Metal Affinity Resin:BD バイオサイエンス社製)を加えて、4℃で20分間インキュベートした。インキュベート後、前記アガロースビーズを洗浄し、抗HMGB1抗体(製品名Rabbit polyclonal to HMGB1:abcam社製)を用いて、ウェスタンブロッティングを行った。また、比較例として前記40Nについても、同様に解析を行った。これらの結果を、図9に示す。
RNAアプタマーをフルオロ化して、HMGB1に対する結合能を確認した。
前記2’-フルオロ-CTPおよび前記2’-フルオロ-UTPを用いて、下記表4に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のフルオロ化RNAアプタマーとして使用した。前記フルオロ化RNAアプタマーは、下記表4に示す塩基配列において、シトシンヌクレオチド残基および/またはウラシルヌクレオチド残基がフルオロ化されている。ウラシルヌクレオチド残基およびシトシンヌクレオチド残基がフルオロ化されたRNAアプタマーを、それぞれ、2’F-CU-R8c6_1、2’F-CU-#06、2’F-CU-CU#80、2’F-CU-R4_9068と表わす。シトシンヌクレオチド残基のみがフルオロ化されたRNAアプタマーを、それぞれ、2’F-C-R8c6_1、2’F-C-#06、2’F-C-#80、2’F-C-R4_9068と表わす。ウラシルヌクレオチド残基のみがフルオロ化されたRNAアプタマーを、それぞれ、2’F-U-R8c6_1、2’F-U-#06、2’F-U-#80、2’F-U-R4_9068と表わす。
前記フルオロ化RNAアプタマーを使用し、前記His-tag付加HMGB1(Sigma社製)の濃度を200nmol/Lとし、前記ランニングバッファーにおけるNaClの濃度を150mmol/Lとし、前記His-tag付加HMGB1のインジェクション時間を60秒とした以外は、前記実施例1の前記(2)と同様にして、前記His-tag付加HMGB1に対する結合能の解析を行った。また、フルオロ化していない未修飾のR8c6_1についても、同様に解析を行った。また、比較例として、フルオロ化していない前記40Nについても、同様に解析を行った。これらの結果を、図10に示す。
R8c6_1(配列番号74)およびR8c6_1-18(配列番号87)の二次構造を推定した。これらの二次構造を、図11に示す。
R8c6_1(配列番号74)のRNAアプタマーを、さらに小型化し、HMGB1に対する結合能を確認した。
下記表5および下記表6に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のRNAアプタマーとして使用した。
前記RNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を160nmol/Lとした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。また、比較例として、前記40Nについても、同様に解析を行った。これらの結果を、図12および図13に示す。
RNAアプタマーをフルオロ化して、HMGB1に対する結合能を確認した。
前記2’-フルオロ-CTPおよび前記2’-フルオロ-UTPを用いて、下記表7に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のフルオロ化RNAアプタマーとして使用した。前記フルオロ化RNAアプタマーは、前記表7に示す塩基配列において、シトシンヌクレオチド残基およびウラシルヌクレオチド残基がフルオロ化されている。ウラシルヌクレオチド残基およびシトシンヌクレオチド残基がフルオロ化されたRNAアプタマーを、それぞれ、2’F-R8c6_1-18-S6、2’F-R8c6_1-25、2’F-R8c6_1-25-S6、2’F-R8c6_1-25-S6A、2’F-R8c6_1-25-S6A2、2’F-R8c6_1-25-S6A3、2’F-R8c6_1-25-S6C、2’F-R8c6_1-25-S6C2、2’F-R8c6_1-25-S8と表わす。また、前記N40についても、同様に、前記2’-フルオロ-CTPおよび前記2’-フルオロ-UTPを用いてフルオロ化を行った。
前記RNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を150500nmol/Lとした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。これらの結果を、図14に示す。
R8c6_1(配列番号74)のRNAアプタマーを、さらに小型化し、HMGB1に対する結合能を確認した。
下記表8に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のRNAアプタマーとして使用した。
前記RNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を150nmol/Lとした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。また、比較例として、前記40Nについても、同様に解析を行った。これらの結果を、図15に示す。
RNAアプタマーをフルオロ化して、HMGB1に対する結合能を確認した。
前記2’-フルオロ-CTPおよび前記2’-フルオロ-UTPを用いて、下記表9および下記表10に示す各RNAアプタマーを、公知の核酸合成方法により作製し、実施例のフルオロ化RNAアプタマーとして使用した。前記フルオロ化RNAアプタマーは、前記表9および前記表10に示す塩基配列において、シトシンヌクレオチド残基およびウラシルヌクレオチド残基がフルオロ化されている。ウラシルヌクレオチド残基およびシトシンヌクレオチド残基がフルオロ化されたRNAアプタマーを、それぞれ、2’F-R8c6_1-21-S6、2’F-R8c6_1-22-S6、2’F-R8c6_1-23-S6、2’F-R8c6_1-24-S6、2’F-R8c6_1-25-S6、2’F-R8c6_1-26-S6、2’F-R8c6_1-27-S6、2’F-R8c6_1-21-S8、2’F-R8c6_1-22-S8、2’F-R8c6_1-23-S8、2’F-R8c6_1-24-S8、2’F-R8c6_1-25-S8、2’F-R8c6_1-25-S8CAと表わす。
前記RNAアプタマーを使用し、前記His-tag付加HMGB1の濃度を150nmol/Lとした以外は、前記実施例1の前記(2)と同様にして、His-tag付加HMGB1に対する結合能の解析を行った。また、比較例として、未修飾の前記40Nについても、同様に解析を行った。これらの結果を、図16および図17に示す。図16および図17において、各RNAアプタマーの名称は、R8c6_1を省略して表わした。
ELISA法により、RNAアプタマーR8c6_1-18-S6(配列番号95)とHMGB1との相互作用を確認した。
Claims (26)
- HMGB1タンパク質との解離定数が5×10-7以下の核酸分子である、HMGB1タンパク質に結合可能なHMGB1結合核酸分子。
- 下記(A1)、(A2)、(B1)および(B2)のいずれかの核酸分子である、請求項1記載のHMGB1結合核酸分子。
(A1)配列番号1~42のいずれかで表わされる塩基配列を含む核酸分子
(A2)配列番号1~42のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1タンパク質に結合可能である核酸分子
(B1)配列番号45~81のいずれかで表わされる塩基配列を含む核酸分子
(B2)配列番号45~81のいずれかで表わされる塩基配列において、1または複数の塩基が、置換、欠失、付加または挿入された塩基配列を含み、且つ、HMGB1タンパク質に結合可能である核酸分子 - 前記(B2)の核酸分子が、配列番号45~81のいずれかで表わされる塩基配列において、5’側の4塩基目から3’側の末端塩基までの塩基配列を含む核酸分子である、請求項2記載のHMGB1結合核酸分子。
- 前記(B1)の核酸分子が、下記(b1)の核酸分子であり、前記(B2)の核酸分子が、下記(b2)の核酸分子である、請求項2または3記載のHMGB1結合核酸分子。
(b1)配列番号74で表わされる塩基配列を含む核酸分子
(b2)配列番号74で表わされる塩基配列における連続する11塩基以上の部分配列を含み、且つ、HMGB1タンパク質に結合可能である核酸分子 - 前記(b2)の核酸分子が、配列番号83~113のいずれかで表わされる塩基配列を含む核酸分子である、請求項4記載のHMGB1結合核酸分子。
- 前記(b2)の核酸分子が、配列番号83~113のいずれかで表わされる塩基配列において、5’側の4塩基目から3’側の末端塩基までの塩基配列を含む核酸分子である、請求項4記載のHMGB1結合核酸分子。
- 前記核酸分子が、修飾化ヌクレオチド残基を含む、請求項1から6のいずれか一項に記載のHMGB1結合核酸分子。
- 前記修飾化ヌクレオチド残基が、メチル化ヌクレオチド残基、フルオロ化ヌクレオチド残基、アミノ化ヌクレオチド残基およびチオ化ヌクレオチド残基からなる群から選択された少なくとも一つである、請求項7記載のHMGB1結合核酸分子。
- 前記修飾化ヌクレオチド残基が、シトシンを有するヌクレオチド残基およびウラシルを有するヌクレオチド残基の少なくとも一方である、請求項7または8記載のHMGB1結合核酸分子。
- 前記修飾化ヌクレオチド残基が、リボース残基が修飾されたヌクレオチド残基である、請求項7から9のいずれか一項に記載のHMGB1結合核酸分子。
- Y領域、X領域およびY’領域を含み、
5’末端から前記Y領域、前記X領域および前記Y’領域が連結し、
前記X領域が、前記配列番号1~42のいずれかの配列番号で表わされる塩基配列を含み、
前記Y領域および前記Y’領域が、それぞれ任意の塩基配列からなる、請求項1~10のいずれか一項に記載のHMGB1結合核酸分子。 - 前記X領域の塩基数が、20~40塩基である、請求項11記載のHMGB1結合核酸分子。
- 前記Y領域の塩基数および前記Y’領域の塩基数が、それぞれ、20~30塩基である、請求項11または12記載のHMGB1結合核酸分子。
- 前記Y領域の配列が、配列番号43および配列番号115のいずれかで表わされる塩基配列を含む、請求項11から13のいずれか一項に記載のHMGB1結合核酸分子。
gggacgcucacguacgcuca(配列番号43)
acgcucacguacgcuca(配列番号115) - 前記Y’の配列が、配列番号44で表わされる塩基配列を含む、請求項11から14のいずれか一項に記載のHMGB1結合核酸分子。
ucagugccuggacgugcagu(配列番号44) - 前記核酸分子の全塩基数が、40~100塩基である、請求項1から15のいずれか一項に記載のHMGB1結合核酸分子。
- 前記核酸分子が、ステムループ構造を有する、請求項1から16のいずれか一項に記載のHMGB1結合核酸分子。
- 前記Y領域および前記Y’領域の少なくとも一方において、一部が、前記X領域の一部と二本鎖を形成することにより、ステムループ構造を形成している、請求項10から17のいずれか一項に記載のHMGB1結合核酸分子。
- 前記Y’領域および前記Y’領域の少なくとも一方において、内部で二本鎖を形成することにより、ステムループ構造を形成している、請求項10から18のいずれか一項に記載のHMGB1結合核酸分子。
- 請求項1から19のいずれか一項に記載のHMGB1結合核酸分子を含み、HMGB1タンパク質と前記HMGB1結合核酸分子との結合により、HMGB1タンパク質の機能を中和することを特徴とする中和剤。
- 請求項1から19のいずれか一項に記載のHMGB1結合核酸分子を含み、HMGB1タンパク質と前記HMGB1結合核酸分子との結合により、HMGB1タンパク質の機能を阻害することを特徴とする阻害剤。
- 請求項1から19のいずれか一項に記載のHMGB1結合核酸分子を含むことを特徴とする医薬品。
- 前記医薬品が、抗癌剤、抗炎症剤、抗脳卒中剤からなる群から選択された少なくとも一つである、請求項22記載の医薬品。
- 請求項1から19のいずれか一項に記載のHMGB1結合核酸分子を含む、組成物。
- さらに、キャリアーを含む、請求項24記載の組成物。
- 請求項1から19のいずれか一項に記載のHMGB1結合核酸分子を含む、HMGB1タンパク質を検出するためのHMGB1検出試薬。
Priority Applications (4)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2011522874A JP5582580B2 (ja) | 2009-07-16 | 2010-07-16 | Hmgb1結合核酸分子およびその用途 |
| CN201080032095.5A CN102625836B (zh) | 2009-07-16 | 2010-07-16 | Hmgb1结合核酸分子及其用途 |
| EP10799934A EP2455469A4 (en) | 2009-07-16 | 2010-07-16 | NUCLEIC ACID MOLECULE CAPABLE OF BINDING TO HMGB1 AND USE THEREOF |
| US13/383,826 US9278108B2 (en) | 2009-07-16 | 2010-07-16 | HMGB1 binding nucleic acid molecule and applications thereof |
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2009167622 | 2009-07-16 | ||
| JP2009-167622 | 2009-07-16 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| WO2011007876A1 true WO2011007876A1 (ja) | 2011-01-20 |
Family
ID=43449486
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| PCT/JP2010/062104 WO2011007876A1 (ja) | 2009-07-16 | 2010-07-16 | Hmgb1結合核酸分子およびその用途 |
Country Status (5)
| Country | Link |
|---|---|
| US (1) | US9278108B2 (ja) |
| EP (1) | EP2455469A4 (ja) |
| JP (1) | JP5582580B2 (ja) |
| CN (1) | CN102625836B (ja) |
| WO (1) | WO2011007876A1 (ja) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20130123350A1 (en) * | 2010-07-26 | 2013-05-16 | Kanagawa Prefectural Hospital Organization | NUCLEIC ACID MOLECULE CAPABLE OF BINDING TO c-MET AND USE THEREOF |
| JP2022517742A (ja) * | 2018-12-28 | 2022-03-10 | ディセルナ ファーマシューティカルズ インコーポレイテッド | Hmgb1発現を阻害するための組成物及び方法 |
Families Citing this family (6)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP6338191B2 (ja) * | 2013-07-01 | 2018-06-06 | Necソリューションイノベータ株式会社 | 属性推定システム |
| EP3405577B8 (en) | 2016-01-20 | 2023-02-15 | 396419 B.C. Ltd. | Compositions and methods for inhibiting factor d |
| WO2018136827A1 (en) | 2017-01-20 | 2018-07-26 | Vitrisa Therapeutics, Inc. | Stem-loop compositions and methods for inhibiting factor d |
| KR20200023427A (ko) | 2017-06-29 | 2020-03-04 | 다이서나 파마수이티컬, 인크. | Hmgb1 발현을 억제하기 위한 조성물 및 방법 |
| CN111304202B (zh) * | 2020-02-26 | 2023-03-28 | 西安交通大学 | 一种用于识别且增强hmgb1生物学活性的核酸适配子及其应用 |
| CN114807150B (zh) * | 2022-06-28 | 2022-10-14 | 中国科学院基础医学与肿瘤研究所(筹) | 靶向和拮抗hmgb1分子的核酸适体 |
Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP3876325B1 (ja) | 2005-10-24 | 2007-01-31 | 国立大学法人 岡山大学 | 脳梗塞抑制剤 |
| WO2007076200A2 (en) * | 2005-11-28 | 2007-07-05 | Medimmune, Inc. | Antagonists of hmgb1 and/or rage and methods of use thereof |
| JP2008520552A (ja) | 2004-10-22 | 2008-06-19 | メディミューン,インコーポレーテッド | Hmgb1に対する高親和性の抗体およびその使用法 |
Family Cites Families (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| ITMI20010562A1 (it) | 2001-03-16 | 2002-09-16 | Marco E Bianchi | Inibitori o antagonisti della proteina hmg1 per il trattamento di disordini vascolari |
| JP4792392B2 (ja) | 2003-09-11 | 2011-10-12 | コーナーストーン セラピューティクス インコーポレイテッド | Hmgb1に対するモノクローナル抗体 |
| US8129130B2 (en) | 2004-10-22 | 2012-03-06 | The Feinstein Institute For Medical Research | High affinity antibodies against HMGB1 and methods of use thereof |
| US20100172909A1 (en) | 2005-10-24 | 2010-07-08 | Masahiro Nishibori | Cerebral edema suppressant |
-
2010
- 2010-07-16 CN CN201080032095.5A patent/CN102625836B/zh not_active Expired - Fee Related
- 2010-07-16 JP JP2011522874A patent/JP5582580B2/ja active Active
- 2010-07-16 EP EP10799934A patent/EP2455469A4/en not_active Withdrawn
- 2010-07-16 WO PCT/JP2010/062104 patent/WO2011007876A1/ja active Application Filing
- 2010-07-16 US US13/383,826 patent/US9278108B2/en not_active Expired - Fee Related
Patent Citations (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2008520552A (ja) | 2004-10-22 | 2008-06-19 | メディミューン,インコーポレーテッド | Hmgb1に対する高親和性の抗体およびその使用法 |
| JP3876325B1 (ja) | 2005-10-24 | 2007-01-31 | 国立大学法人 岡山大学 | 脳梗塞抑制剤 |
| WO2007076200A2 (en) * | 2005-11-28 | 2007-07-05 | Medimmune, Inc. | Antagonists of hmgb1 and/or rage and methods of use thereof |
Non-Patent Citations (12)
| Title |
|---|
| ANDERSSON, U. ET AL., J. LEUKOC. BIOL., vol. 72, 2002, pages 1084 - 1091 |
| BUSTIN, M., MOL. CELL BIOL., vol. 19, 1999, pages 523 - 5246 |
| CALOGERO, S. ET AL., NAT. GENET., vol. 22, 1999, pages 276 - 280 |
| ELLERMAN, J. E. ET AL., CLIN. CANCER RES., vol. 13, 2007, pages 2836 - 2848 |
| JAOUEN S. ET AL.: "Determinants of Specific Binding of HMGB1 Protein to Hemicatenated DNA Loops.", J. MOL. BIOL., vol. 353, no. 4, 4 November 2005 (2005-11-04), pages 822 - 837, XP005118793 * |
| JAYASENA, S.: "Aptamers: an emerging class of molecules that rival antibodies in diagnostics.", CLIN. CHEM., vol. 45, no. 9, July 1999 (1999-07-01), pages 1628 - 1650, XP008067440 * |
| KEEFE, A.D. ET AL.: "SELEX with modified nucleotides.", CURR. OPIN. CHEM. BIOL., vol. 12, no. 4, August 2008 (2008-08-01), pages 448 - 456, XP024528065 * |
| KLUNE, J. R. ET AL., MOL. MED., vol. 14, 2008, pages 476 - 484 |
| LIU, K. ET AL., FASEB J., vol. 21, 2007, pages 3904 - 16 |
| OHTSU, T. ET AL.: "RNA aptamers against HMGB1 - the possibility to inhibit the cancer cell proliferation.", PROCEEDINGS OF THE JAPANESE CANCER ASSOCIATION, vol. 68TH, 31 August 2009 (2009-08-31), pages 494 * |
| See also references of EP2455469A4 * |
| WEBB M. ET AL.: "Structural requirements for cooperative binding of HMG1 to DNA minicircles.", J. MOL. BIOL., vol. 309, no. 1, 25 May 2001 (2001-05-25), pages 79 - 88, XP004464218 * |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20130123350A1 (en) * | 2010-07-26 | 2013-05-16 | Kanagawa Prefectural Hospital Organization | NUCLEIC ACID MOLECULE CAPABLE OF BINDING TO c-MET AND USE THEREOF |
| US8822667B2 (en) * | 2010-07-26 | 2014-09-02 | Nec Solution Innovators, Ltd. | Nucleic acid molecule capable of binding to c-Met and use thereof |
| JP2022517742A (ja) * | 2018-12-28 | 2022-03-10 | ディセルナ ファーマシューティカルズ インコーポレイテッド | Hmgb1発現を阻害するための組成物及び方法 |
Also Published As
| Publication number | Publication date |
|---|---|
| JPWO2011007876A1 (ja) | 2012-12-27 |
| JP5582580B2 (ja) | 2014-09-03 |
| EP2455469A1 (en) | 2012-05-23 |
| CN102625836B (zh) | 2015-12-16 |
| EP2455469A4 (en) | 2013-03-13 |
| US20120208867A1 (en) | 2012-08-16 |
| US9278108B2 (en) | 2016-03-08 |
| CN102625836A (zh) | 2012-08-01 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5582580B2 (ja) | Hmgb1結合核酸分子およびその用途 | |
| AU2014284836B2 (en) | Respiratory disease-related gene specific siRNA, double-helical oligo RNA structure containing siRNA, compositon containing same for preventing or treating respiratory disease | |
| JP5291129B2 (ja) | 細胞および/または組織および/または疾患フェーズ特異的な薬剤の製造方法 | |
| TW202136508A (zh) | 用於編輯目標rna之附加有功能性領域之反義型嚮導rna | |
| JP5537812B2 (ja) | Mcp−1に結合する核酸 | |
| JP2006508688A (ja) | 2’−置換核酸のインビトロ選択のための方法 | |
| JP6591392B2 (ja) | Il−6に結合するアプタマー及びil−6介在性状態の治療または診断におけるそれらの使用 | |
| WO2019196887A1 (zh) | 一种新型小激活rna | |
| Wang et al. | In vitro selection of DNA aptamers against renal cell carcinoma using living cell-SELEX | |
| US7960102B2 (en) | Regulated aptamer therapeutics | |
| KR101900878B1 (ko) | TNF-α 결합 압타머 및 그것의 치료적 용도 | |
| WO2012014890A1 (ja) | c-Met結合核酸分子およびその用途 | |
| JP5249998B2 (ja) | オステオポンチンsiRNA | |
| JP4589794B2 (ja) | オステオポンチンsiRNA | |
| JP5777240B2 (ja) | 新規オリゴヌクレオチド誘導体及びそれから成るNF−κBデコイ | |
| US20240336922A1 (en) | Inhibitors of expression and/or function | |
| WO2023224102A1 (ja) | 新規Staple核酸 | |
| JP6934235B2 (ja) | 遺伝子発現制御のための発現制御核酸分子およびその用途 | |
| WO2022266042A1 (en) | Treatment of mst1r related diseases and disorders | |
| Liu | Selecting and improving the functionality of DNAzymes | |
| WO2010004816A1 (ja) | 肥満細胞の脱顆粒抑制剤 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WWE | Wipo information: entry into national phase |
Ref document number: 201080032095.5 Country of ref document: CN |
|
| 121 | Ep: the epo has been informed by wipo that ep was designated in this application |
Ref document number: 10799934 Country of ref document: EP Kind code of ref document: A1 |
|
| NENP | Non-entry into the national phase |
Ref country code: DE |
|
| WWE | Wipo information: entry into national phase |
Ref document number: 2011522874 Country of ref document: JP |
|
| REEP | Request for entry into the european phase |
Ref document number: 2010799934 Country of ref document: EP |
|
| WWE | Wipo information: entry into national phase |
Ref document number: 2010799934 Country of ref document: EP |
|
| WWE | Wipo information: entry into national phase |
Ref document number: 13383826 Country of ref document: US |