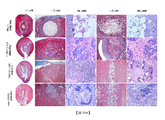
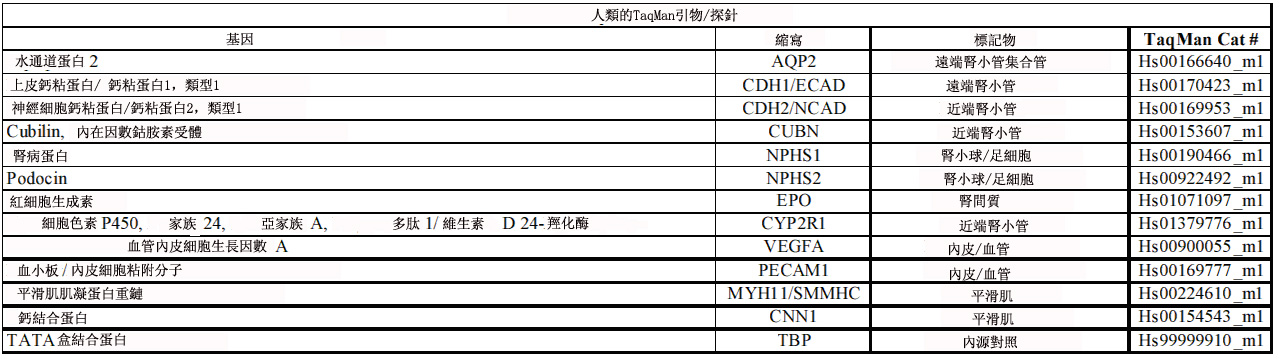
TWI690320B - 生物活性腎細胞 - Google Patents
生物活性腎細胞 Download PDFInfo
- Publication number
- TWI690320B TWI690320B TW107113584A TW107113584A TWI690320B TW I690320 B TWI690320 B TW I690320B TW 107113584 A TW107113584 A TW 107113584A TW 107113584 A TW107113584 A TW 107113584A TW I690320 B TWI690320 B TW I690320B
- Authority
- TW
- Taiwan
- Prior art keywords
- mir
- cells
- cell
- kidney
- cell population
- Prior art date
Links
- 230000000975 bioactive effect Effects 0.000 title abstract description 19
- 210000004027 cell Anatomy 0.000 claims description 884
- 102000003951 Erythropoietin Human genes 0.000 claims description 156
- 108090000394 Erythropoietin Proteins 0.000 claims description 156
- 229940105423 erythropoietin Drugs 0.000 claims description 150
- OXCMYAYHXIHQOA-UHFFFAOYSA-N potassium;[2-butyl-5-chloro-3-[[4-[2-(1,2,4-triaza-3-azanidacyclopenta-1,4-dien-5-yl)phenyl]phenyl]methyl]imidazol-4-yl]methanol Chemical compound [K+].CCCCC1=NC(Cl)=C(CO)N1CC1=CC=C(C=2C(=CC=CC=2)C2=N[N-]N=N2)C=C1 OXCMYAYHXIHQOA-UHFFFAOYSA-N 0.000 claims description 149
- 239000000203 mixture Substances 0.000 claims description 138
- 210000003734 kidney Anatomy 0.000 claims description 88
- 208000017169 kidney disease Diseases 0.000 claims description 68
- 241000282414 Homo sapiens Species 0.000 claims description 60
- 230000008929 regeneration Effects 0.000 claims description 56
- 238000011069 regeneration method Methods 0.000 claims description 56
- 239000002679 microRNA Substances 0.000 claims description 51
- 210000003904 glomerular cell Anatomy 0.000 claims description 41
- 238000001727 in vivo Methods 0.000 claims description 37
- 108700011259 MicroRNAs Proteins 0.000 claims description 32
- 230000000694 effects Effects 0.000 claims description 27
- -1 miR-130a Proteins 0.000 claims description 27
- 210000005167 vascular cell Anatomy 0.000 claims description 27
- 239000003814 drug Substances 0.000 claims description 26
- 230000002829 reductive effect Effects 0.000 claims description 25
- 210000001808 exosome Anatomy 0.000 claims description 23
- 238000002360 preparation method Methods 0.000 claims description 21
- 108091070501 miRNA Proteins 0.000 claims description 20
- 230000001172 regenerating effect Effects 0.000 claims description 14
- 101000979342 Homo sapiens Nuclear factor NF-kappa-B p105 subunit Proteins 0.000 claims description 13
- 102100023050 Nuclear factor NF-kappa-B p105 subunit Human genes 0.000 claims description 13
- 108060008682 Tumor Necrosis Factor Proteins 0.000 claims description 13
- 230000001404 mediated effect Effects 0.000 claims description 13
- 108091063344 miR-30b stem-loop Proteins 0.000 claims description 13
- 102000000852 Tumor Necrosis Factor-alpha Human genes 0.000 claims description 12
- 230000007423 decrease Effects 0.000 claims description 11
- 108091037327 miR-449 stem-loop Proteins 0.000 claims description 11
- 108091040525 miR-449a stem-loop Proteins 0.000 claims description 11
- 230000009467 reduction Effects 0.000 claims description 11
- 108091062762 miR-21 stem-loop Proteins 0.000 claims description 9
- 108091041631 miR-21-1 stem-loop Proteins 0.000 claims description 9
- 108091044442 miR-21-2 stem-loop Proteins 0.000 claims description 9
- 108091072763 miR-151 stem-loop Proteins 0.000 claims description 8
- 229940079593 drug Drugs 0.000 claims description 7
- 230000004054 inflammatory process Effects 0.000 claims description 7
- 108091032320 miR-146 stem-loop Proteins 0.000 claims description 7
- 108091024530 miR-146a stem-loop Proteins 0.000 claims description 7
- 108091092722 miR-23b stem-loop Proteins 0.000 claims description 7
- 108091031298 miR-23b-1 stem-loop Proteins 0.000 claims description 7
- 108091082339 miR-23b-2 stem-loop Proteins 0.000 claims description 7
- 206010061218 Inflammation Diseases 0.000 claims description 6
- 108091056924 miR-124 stem-loop Proteins 0.000 claims description 6
- 108091035155 miR-10a stem-loop Proteins 0.000 claims description 5
- 108091084882 miR-10a-1 stem-loop Proteins 0.000 claims description 5
- 108091062170 Mir-22 Proteins 0.000 claims description 4
- 108091088205 miR-10a-2 stem-loop Proteins 0.000 claims description 4
- 108091027766 Mir-143 Proteins 0.000 claims description 3
- 108091028684 Mir-145 Proteins 0.000 claims description 3
- 108091059916 miR-7a stem-loop Proteins 0.000 claims description 3
- 108091060270 miR-7a-3 stem-loop Proteins 0.000 claims description 3
- 108091080310 miR-7a-4 stem-loop Proteins 0.000 claims description 3
- 108091049334 miR-7a-5 stem-loop Proteins 0.000 claims description 3
- 108010057466 NF-kappa B Proteins 0.000 claims description 2
- 102000003945 NF-kappa B Human genes 0.000 claims description 2
- 102100040247 Tumor necrosis factor Human genes 0.000 claims description 2
- 108091061920 miR-7b stem-loop Proteins 0.000 claims description 2
- 108091050724 let-7b stem-loop Proteins 0.000 claims 1
- 108091030917 let-7b-1 stem-loop Proteins 0.000 claims 1
- 108091082924 let-7b-2 stem-loop Proteins 0.000 claims 1
- 238000000034 method Methods 0.000 abstract description 150
- 230000014509 gene expression Effects 0.000 description 150
- 210000003292 kidney cell Anatomy 0.000 description 126
- 108090000623 proteins and genes Proteins 0.000 description 98
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 87
- 239000001301 oxygen Substances 0.000 description 87
- 229910052760 oxygen Inorganic materials 0.000 description 87
- 102000004169 proteins and genes Human genes 0.000 description 76
- 235000018102 proteins Nutrition 0.000 description 65
- 239000000523 sample Substances 0.000 description 62
- 210000001519 tissue Anatomy 0.000 description 62
- 239000003636 conditioned culture medium Substances 0.000 description 58
- 239000012620 biological material Substances 0.000 description 54
- 239000000047 product Substances 0.000 description 45
- 208000020832 chronic kidney disease Diseases 0.000 description 44
- 230000003907 kidney function Effects 0.000 description 43
- 208000007502 anemia Diseases 0.000 description 41
- 239000003550 marker Substances 0.000 description 41
- 238000012360 testing method Methods 0.000 description 41
- 239000002245 particle Substances 0.000 description 40
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 38
- 210000005084 renal tissue Anatomy 0.000 description 37
- 210000000130 stem cell Anatomy 0.000 description 37
- 210000003743 erythrocyte Anatomy 0.000 description 36
- KIUKXJAPPMFGSW-DNGZLQJQSA-N (2S,3S,4S,5R,6R)-6-[(2S,3R,4R,5S,6R)-3-Acetamido-2-[(2S,3S,4R,5R,6R)-6-[(2R,3R,4R,5S,6R)-3-acetamido-2,5-dihydroxy-6-(hydroxymethyl)oxan-4-yl]oxy-2-carboxy-4,5-dihydroxyoxan-3-yl]oxy-5-hydroxy-6-(hydroxymethyl)oxan-4-yl]oxy-3,4,5-trihydroxyoxane-2-carboxylic acid Chemical compound CC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O[C@H]1[C@H](O)[C@@H](O)[C@H](O[C@H]2[C@@H]([C@@H](O[C@H]3[C@@H]([C@@H](O)[C@H](O)[C@H](O3)C(O)=O)O)[C@H](O)[C@@H](CO)O2)NC(C)=O)[C@@H](C(O)=O)O1 KIUKXJAPPMFGSW-DNGZLQJQSA-N 0.000 description 35
- 206010021143 Hypoxia Diseases 0.000 description 35
- 230000007812 deficiency Effects 0.000 description 35
- 229920002674 hyaluronan Polymers 0.000 description 35
- 229960003160 hyaluronic acid Drugs 0.000 description 35
- 238000013059 nephrectomy Methods 0.000 description 34
- 241000700159 Rattus Species 0.000 description 31
- 239000002609 medium Substances 0.000 description 29
- 238000000926 separation method Methods 0.000 description 29
- 239000002953 phosphate buffered saline Substances 0.000 description 28
- 230000004083 survival effect Effects 0.000 description 26
- 241001465754 Metazoa Species 0.000 description 24
- 238000004458 analytical method Methods 0.000 description 24
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 24
- 238000000338 in vitro Methods 0.000 description 24
- 230000001146 hypoxic effect Effects 0.000 description 22
- 238000002513 implantation Methods 0.000 description 21
- 238000002955 isolation Methods 0.000 description 21
- 210000002966 serum Anatomy 0.000 description 21
- 238000004393 prognosis Methods 0.000 description 20
- 238000004113 cell culture Methods 0.000 description 19
- 238000002474 experimental method Methods 0.000 description 19
- 230000001434 glomerular Effects 0.000 description 19
- 239000000463 material Substances 0.000 description 19
- 108010088751 Albumins Proteins 0.000 description 18
- 102000009027 Albumins Human genes 0.000 description 18
- 239000013068 control sample Substances 0.000 description 18
- 239000000017 hydrogel Substances 0.000 description 18
- 241000283984 Rodentia Species 0.000 description 17
- 239000000872 buffer Substances 0.000 description 17
- 239000002771 cell marker Substances 0.000 description 17
- 238000005119 centrifugation Methods 0.000 description 17
- 230000032258 transport Effects 0.000 description 17
- 102100024616 Platelet endothelial cell adhesion molecule Human genes 0.000 description 16
- 230000006872 improvement Effects 0.000 description 16
- 230000001965 increasing effect Effects 0.000 description 16
- 239000000243 solution Substances 0.000 description 16
- 241000282465 Canis Species 0.000 description 15
- 210000004204 blood vessel Anatomy 0.000 description 15
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 15
- 238000002347 injection Methods 0.000 description 15
- 239000007924 injection Substances 0.000 description 15
- 238000004519 manufacturing process Methods 0.000 description 15
- 229920000642 polymer Polymers 0.000 description 15
- 230000004044 response Effects 0.000 description 15
- 210000004739 secretory vesicle Anatomy 0.000 description 15
- 230000002792 vascular Effects 0.000 description 15
- 102000000905 Cadherin Human genes 0.000 description 14
- 108050007957 Cadherin Proteins 0.000 description 14
- 238000006243 chemical reaction Methods 0.000 description 14
- DDRJAANPRJIHGJ-UHFFFAOYSA-N creatinine Chemical compound CN1CC(=O)NC1=N DDRJAANPRJIHGJ-UHFFFAOYSA-N 0.000 description 14
- 238000001514 detection method Methods 0.000 description 14
- 210000003890 endocrine cell Anatomy 0.000 description 14
- 238000005534 hematocrit Methods 0.000 description 14
- 108090000765 processed proteins & peptides Proteins 0.000 description 14
- 206010016654 Fibrosis Diseases 0.000 description 13
- 238000011529 RT qPCR Methods 0.000 description 13
- 210000003850 cellular structure Anatomy 0.000 description 13
- 201000010099 disease Diseases 0.000 description 13
- 230000002124 endocrine Effects 0.000 description 13
- 230000004761 fibrosis Effects 0.000 description 13
- NBQNWMBBSKPBAY-UHFFFAOYSA-N iodixanol Chemical compound IC=1C(C(=O)NCC(O)CO)=C(I)C(C(=O)NCC(O)CO)=C(I)C=1N(C(=O)C)CC(O)CN(C(C)=O)C1=C(I)C(C(=O)NCC(O)CO)=C(I)C(C(=O)NCC(O)CO)=C1I NBQNWMBBSKPBAY-UHFFFAOYSA-N 0.000 description 13
- 229960004359 iodixanol Drugs 0.000 description 13
- 108020004999 messenger RNA Proteins 0.000 description 13
- 230000003076 paracrine Effects 0.000 description 13
- 102000004196 processed proteins & peptides Human genes 0.000 description 13
- 229940124597 therapeutic agent Drugs 0.000 description 13
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 12
- 239000006143 cell culture medium Substances 0.000 description 12
- 230000001413 cellular effect Effects 0.000 description 12
- 230000025215 erythrocyte homeostasis Effects 0.000 description 12
- 230000007954 hypoxia Effects 0.000 description 12
- 239000012528 membrane Substances 0.000 description 12
- 230000030648 nucleus localization Effects 0.000 description 12
- 230000037361 pathway Effects 0.000 description 12
- 229920001184 polypeptide Polymers 0.000 description 12
- 210000002700 urine Anatomy 0.000 description 12
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 12
- 238000001262 western blot Methods 0.000 description 12
- 101710204736 Platelet endothelial cell adhesion molecule Proteins 0.000 description 11
- 210000004369 blood Anatomy 0.000 description 11
- 239000008280 blood Substances 0.000 description 11
- 239000006285 cell suspension Substances 0.000 description 11
- 230000006870 function Effects 0.000 description 11
- 230000024924 glomerular filtration Effects 0.000 description 11
- 239000011159 matrix material Substances 0.000 description 11
- 210000004379 membrane Anatomy 0.000 description 11
- 230000006641 stabilisation Effects 0.000 description 11
- 238000011105 stabilization Methods 0.000 description 11
- 238000010186 staining Methods 0.000 description 11
- 101000610551 Homo sapiens Prominin-1 Proteins 0.000 description 10
- 101710162479 Podocin Proteins 0.000 description 10
- 102100036037 Podocin Human genes 0.000 description 10
- 102100040120 Prominin-1 Human genes 0.000 description 10
- 239000012472 biological sample Substances 0.000 description 10
- 239000000090 biomarker Substances 0.000 description 10
- 210000004748 cultured cell Anatomy 0.000 description 10
- 238000011161 development Methods 0.000 description 10
- 230000018109 developmental process Effects 0.000 description 10
- 230000029087 digestion Effects 0.000 description 10
- 230000001976 improved effect Effects 0.000 description 10
- 238000011694 lewis rat Methods 0.000 description 10
- 230000001105 regulatory effect Effects 0.000 description 10
- 230000001225 therapeutic effect Effects 0.000 description 10
- 238000012546 transfer Methods 0.000 description 10
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 9
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 9
- 102000001554 Hemoglobins Human genes 0.000 description 9
- 108010054147 Hemoglobins Proteins 0.000 description 9
- 239000000560 biocompatible material Substances 0.000 description 9
- 239000003795 chemical substances by application Substances 0.000 description 9
- 238000009826 distribution Methods 0.000 description 9
- 238000000684 flow cytometry Methods 0.000 description 9
- 239000012530 fluid Substances 0.000 description 9
- 239000007788 liquid Substances 0.000 description 9
- 210000003584 mesangial cell Anatomy 0.000 description 9
- 210000005239 tubule Anatomy 0.000 description 9
- 108010035532 Collagen Proteins 0.000 description 8
- 102000008186 Collagen Human genes 0.000 description 8
- 101000777245 Homo sapiens Undifferentiated embryonic cell transcription factor 1 Proteins 0.000 description 8
- 102100040206 Hyaluronan synthase 2 Human genes 0.000 description 8
- 101710197056 Hyaluronan synthase 2 Proteins 0.000 description 8
- 102100039285 Hyaluronidase-2 Human genes 0.000 description 8
- 101710199674 Hyaluronidase-2 Proteins 0.000 description 8
- 108091030146 MiRBase Proteins 0.000 description 8
- 238000001574 biopsy Methods 0.000 description 8
- 230000008859 change Effects 0.000 description 8
- 229920001436 collagen Polymers 0.000 description 8
- 238000004891 communication Methods 0.000 description 8
- 230000010437 erythropoiesis Effects 0.000 description 8
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 8
- 239000000499 gel Substances 0.000 description 8
- 230000000977 initiatory effect Effects 0.000 description 8
- 238000011862 kidney biopsy Methods 0.000 description 8
- 230000010412 perfusion Effects 0.000 description 8
- 102000005962 receptors Human genes 0.000 description 8
- 108020003175 receptors Proteins 0.000 description 8
- 108091006146 Channels Proteins 0.000 description 7
- 101000884271 Homo sapiens Signal transducer CD24 Proteins 0.000 description 7
- 239000004698 Polyethylene Substances 0.000 description 7
- 102100038081 Signal transducer CD24 Human genes 0.000 description 7
- GLNADSQYFUSGOU-GPTZEZBUSA-J Trypan blue Chemical compound [Na+].[Na+].[Na+].[Na+].C1=C(S([O-])(=O)=O)C=C2C=C(S([O-])(=O)=O)C(/N=N/C3=CC=C(C=C3C)C=3C=C(C(=CC=3)\N=N\C=3C(=CC4=CC(=CC(N)=C4C=3O)S([O-])(=O)=O)S([O-])(=O)=O)C)=C(O)C2=C1N GLNADSQYFUSGOU-GPTZEZBUSA-J 0.000 description 7
- 102100031278 Undifferentiated embryonic cell transcription factor 1 Human genes 0.000 description 7
- 230000009102 absorption Effects 0.000 description 7
- 238000010521 absorption reaction Methods 0.000 description 7
- 239000011324 bead Substances 0.000 description 7
- 239000003153 chemical reaction reagent Substances 0.000 description 7
- 229940109239 creatinine Drugs 0.000 description 7
- 238000012258 culturing Methods 0.000 description 7
- 201000005206 focal segmental glomerulosclerosis Diseases 0.000 description 7
- 231100000854 focal segmental glomerulosclerosis Toxicity 0.000 description 7
- 238000011534 incubation Methods 0.000 description 7
- 239000004615 ingredient Substances 0.000 description 7
- 210000004940 nucleus Anatomy 0.000 description 7
- 230000028327 secretion Effects 0.000 description 7
- 230000003827 upregulation Effects 0.000 description 7
- 239000003981 vehicle Substances 0.000 description 7
- 102000004190 Enzymes Human genes 0.000 description 6
- 108090000790 Enzymes Proteins 0.000 description 6
- 108010067306 Fibronectins Proteins 0.000 description 6
- 102000016359 Fibronectins Human genes 0.000 description 6
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 6
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 6
- 102000004887 Transforming Growth Factor beta Human genes 0.000 description 6
- 108090001012 Transforming Growth Factor beta Proteins 0.000 description 6
- 230000002238 attenuated effect Effects 0.000 description 6
- 210000001185 bone marrow Anatomy 0.000 description 6
- 239000000284 extract Substances 0.000 description 6
- 239000006260 foam Substances 0.000 description 6
- 239000008103 glucose Substances 0.000 description 6
- 239000001963 growth medium Substances 0.000 description 6
- 238000003306 harvesting Methods 0.000 description 6
- 238000003384 imaging method Methods 0.000 description 6
- 230000006698 induction Effects 0.000 description 6
- 108010003082 intrinsic factor-cobalamin receptor Proteins 0.000 description 6
- 230000007246 mechanism Effects 0.000 description 6
- 229910052757 nitrogen Inorganic materials 0.000 description 6
- 230000003248 secreting effect Effects 0.000 description 6
- ZRKFYGHZFMAOKI-QMGMOQQFSA-N tgfbeta Chemical group C([C@H](NC(=O)[C@H](C(C)C)NC(=O)CNC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](CC(C)C)NC(=O)CNC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](NC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CCSC)C(C)C)[C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](C)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N1[C@@H](CCC1)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O)C1=CC=C(O)C=C1 ZRKFYGHZFMAOKI-QMGMOQQFSA-N 0.000 description 6
- 238000002560 therapeutic procedure Methods 0.000 description 6
- 108010036221 Aquaporin 2 Proteins 0.000 description 5
- 102100034414 Aquaporin-2 Human genes 0.000 description 5
- 102100031096 Cubilin Human genes 0.000 description 5
- 101150002621 EPO gene Proteins 0.000 description 5
- 101150043640 FXYD4 gene Proteins 0.000 description 5
- 108010010803 Gelatin Proteins 0.000 description 5
- 101500027610 Homo sapiens Neurokinin A Proteins 0.000 description 5
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 5
- 108050000637 N-cadherin Proteins 0.000 description 5
- 230000004913 activation Effects 0.000 description 5
- 125000000129 anionic group Chemical group 0.000 description 5
- 230000015572 biosynthetic process Effects 0.000 description 5
- 208000022831 chronic renal failure syndrome Diseases 0.000 description 5
- 239000003937 drug carrier Substances 0.000 description 5
- 210000002889 endothelial cell Anatomy 0.000 description 5
- 229940088598 enzyme Drugs 0.000 description 5
- 238000011156 evaluation Methods 0.000 description 5
- 230000007717 exclusion Effects 0.000 description 5
- 210000002950 fibroblast Anatomy 0.000 description 5
- 229920000159 gelatin Polymers 0.000 description 5
- 239000008273 gelatin Substances 0.000 description 5
- 235000019322 gelatine Nutrition 0.000 description 5
- 235000011852 gelatine desserts Nutrition 0.000 description 5
- 230000028993 immune response Effects 0.000 description 5
- 150000002500 ions Chemical class 0.000 description 5
- 238000005259 measurement Methods 0.000 description 5
- 210000000056 organ Anatomy 0.000 description 5
- 239000008188 pellet Substances 0.000 description 5
- 239000008194 pharmaceutical composition Substances 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 230000000750 progressive effect Effects 0.000 description 5
- 230000035755 proliferation Effects 0.000 description 5
- 238000011160 research Methods 0.000 description 5
- 210000001995 reticulocyte Anatomy 0.000 description 5
- 238000003757 reverse transcription PCR Methods 0.000 description 5
- 239000011734 sodium Substances 0.000 description 5
- 229910052708 sodium Inorganic materials 0.000 description 5
- 239000011780 sodium chloride Substances 0.000 description 5
- 238000010561 standard procedure Methods 0.000 description 5
- 239000000126 substance Substances 0.000 description 5
- 239000013589 supplement Substances 0.000 description 5
- 101150111620 AQP1 gene Proteins 0.000 description 4
- 101150047512 AQP2 gene Proteins 0.000 description 4
- 102000007469 Actins Human genes 0.000 description 4
- 108010085238 Actins Proteins 0.000 description 4
- 208000009304 Acute Kidney Injury Diseases 0.000 description 4
- 102100026609 Aldehyde dehydrogenase family 3 member B1 Human genes 0.000 description 4
- 101150115490 Aldh1a3 gene Proteins 0.000 description 4
- 101150012645 Aldh3b1 gene Proteins 0.000 description 4
- 102000004888 Aquaporin 1 Human genes 0.000 description 4
- 108090001004 Aquaporin 1 Proteins 0.000 description 4
- 101150059114 CAPN8 gene Proteins 0.000 description 4
- 102000014273 Calpain-8 Human genes 0.000 description 4
- 108050003158 Calpain-8 Proteins 0.000 description 4
- 241000282472 Canis lupus familiaris Species 0.000 description 4
- 241000283707 Capra Species 0.000 description 4
- 102000012272 Cholestanetriol 26-monooxygenase Human genes 0.000 description 4
- 108010022102 Cholestanetriol 26-monooxygenase Proteins 0.000 description 4
- 101100372758 Danio rerio vegfaa gene Proteins 0.000 description 4
- 102000010834 Extracellular Matrix Proteins Human genes 0.000 description 4
- 108010037362 Extracellular Matrix Proteins Proteins 0.000 description 4
- 241000282412 Homo Species 0.000 description 4
- 101000717973 Homo sapiens Aldehyde dehydrogenase family 3 member B1 Proteins 0.000 description 4
- 101000620589 Homo sapiens Ras-related protein Rab-17 Proteins 0.000 description 4
- 101000819111 Homo sapiens Trans-acting T-cell-specific transcription factor GATA-3 Proteins 0.000 description 4
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 4
- 206010020772 Hypertension Diseases 0.000 description 4
- 108091028049 Mir-221 microRNA Proteins 0.000 description 4
- 101100382351 Mus musculus Capn10 gene Proteins 0.000 description 4
- 101100013967 Mus musculus Gata3 gene Proteins 0.000 description 4
- KPKZJLCSROULON-QKGLWVMZSA-N Phalloidin Chemical compound N1C(=O)[C@@H]([C@@H](O)C)NC(=O)[C@H](C)NC(=O)[C@H](C[C@@](C)(O)CO)NC(=O)[C@H](C2)NC(=O)[C@H](C)NC(=O)[C@@H]3C[C@H](O)CN3C(=O)[C@@H]1CSC1=C2C2=CC=CC=C2N1 KPKZJLCSROULON-QKGLWVMZSA-N 0.000 description 4
- 102000013566 Plasminogen Human genes 0.000 description 4
- 108010051456 Plasminogen Proteins 0.000 description 4
- 102100022292 Ras-related protein Rab-17 Human genes 0.000 description 4
- 208000001647 Renal Insufficiency Diseases 0.000 description 4
- 208000033626 Renal failure acute Diseases 0.000 description 4
- 108091006647 SLC9A1 Proteins 0.000 description 4
- 101150104588 Slc9a4 gene Proteins 0.000 description 4
- 241000282887 Suidae Species 0.000 description 4
- 241000282898 Sus scrofa Species 0.000 description 4
- 101100497132 Sus scrofa CYP2D25 gene Proteins 0.000 description 4
- 102100021386 Trans-acting T-cell-specific transcription factor GATA-3 Human genes 0.000 description 4
- 108010053099 Vascular Endothelial Growth Factor Receptor-2 Proteins 0.000 description 4
- 102100033177 Vascular endothelial growth factor receptor 2 Human genes 0.000 description 4
- 101150030763 Vegfa gene Proteins 0.000 description 4
- PNNCWTXUWKENPE-UHFFFAOYSA-N [N].NC(N)=O Chemical compound [N].NC(N)=O PNNCWTXUWKENPE-UHFFFAOYSA-N 0.000 description 4
- 201000011040 acute kidney failure Diseases 0.000 description 4
- 230000033115 angiogenesis Effects 0.000 description 4
- 238000003556 assay Methods 0.000 description 4
- 201000004995 autoimmune glomerulonephritis Diseases 0.000 description 4
- 230000004071 biological effect Effects 0.000 description 4
- 230000033228 biological regulation Effects 0.000 description 4
- 238000012512 characterization method Methods 0.000 description 4
- 230000003247 decreasing effect Effects 0.000 description 4
- 239000003085 diluting agent Substances 0.000 description 4
- 210000002919 epithelial cell Anatomy 0.000 description 4
- 239000012634 fragment Substances 0.000 description 4
- 239000008187 granular material Substances 0.000 description 4
- 230000036541 health Effects 0.000 description 4
- 238000000703 high-speed centrifugation Methods 0.000 description 4
- 239000001257 hydrogen Substances 0.000 description 4
- 229910052739 hydrogen Inorganic materials 0.000 description 4
- NOESYZHRGYRDHS-UHFFFAOYSA-N insulin Chemical compound N1C(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(NC(=O)CN)C(C)CC)CSSCC(C(NC(CO)C(=O)NC(CC(C)C)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CCC(N)=O)C(=O)NC(CC(C)C)C(=O)NC(CCC(O)=O)C(=O)NC(CC(N)=O)C(=O)NC(CC=2C=CC(O)=CC=2)C(=O)NC(CSSCC(NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2C=CC(O)=CC=2)NC(=O)C(CC(C)C)NC(=O)C(C)NC(=O)C(CCC(O)=O)NC(=O)C(C(C)C)NC(=O)C(CC(C)C)NC(=O)C(CC=2NC=NC=2)NC(=O)C(CO)NC(=O)CNC2=O)C(=O)NCC(=O)NC(CCC(O)=O)C(=O)NC(CCCNC(N)=N)C(=O)NCC(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC=CC=3)C(=O)NC(CC=3C=CC(O)=CC=3)C(=O)NC(C(C)O)C(=O)N3C(CCC3)C(=O)NC(CCCCN)C(=O)NC(C)C(O)=O)C(=O)NC(CC(N)=O)C(O)=O)=O)NC(=O)C(C(C)CC)NC(=O)C(CO)NC(=O)C(C(C)O)NC(=O)C1CSSCC2NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(CCC(N)=O)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)C(C)C)CC1=CN=CN1 NOESYZHRGYRDHS-UHFFFAOYSA-N 0.000 description 4
- 230000003834 intracellular effect Effects 0.000 description 4
- 238000001155 isoelectric focusing Methods 0.000 description 4
- 201000006370 kidney failure Diseases 0.000 description 4
- 230000004807 localization Effects 0.000 description 4
- 108091035591 miR-23a stem-loop Proteins 0.000 description 4
- 108091076732 miR-99a stem-loop Proteins 0.000 description 4
- 108091064318 miR-99a-1 stem-loop Proteins 0.000 description 4
- 108091086202 miR-99a-2 stem-loop Proteins 0.000 description 4
- 239000013610 patient sample Substances 0.000 description 4
- 229920000573 polyethylene Polymers 0.000 description 4
- 239000011148 porous material Substances 0.000 description 4
- 239000013641 positive control Substances 0.000 description 4
- 108700042226 ras Genes Proteins 0.000 description 4
- 238000003753 real-time PCR Methods 0.000 description 4
- 238000011084 recovery Methods 0.000 description 4
- QFJCIRLUMZQUOT-HPLJOQBZSA-N sirolimus Chemical compound C1C[C@@H](O)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 QFJCIRLUMZQUOT-HPLJOQBZSA-N 0.000 description 4
- 241000894007 species Species 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- 230000001052 transient effect Effects 0.000 description 4
- 210000005233 tubule cell Anatomy 0.000 description 4
- 238000005406 washing Methods 0.000 description 4
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 3
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 3
- 102000029816 Collagenase Human genes 0.000 description 3
- 108060005980 Collagenase Proteins 0.000 description 3
- 108020004414 DNA Proteins 0.000 description 3
- 230000004568 DNA-binding Effects 0.000 description 3
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 3
- 229920002683 Glycosaminoglycan Polymers 0.000 description 3
- 239000012981 Hank's balanced salt solution Substances 0.000 description 3
- 206010019280 Heart failures Diseases 0.000 description 3
- 102000007513 Hemoglobin A Human genes 0.000 description 3
- 108010085682 Hemoglobin A Proteins 0.000 description 3
- 102000002704 Leucyl aminopeptidase Human genes 0.000 description 3
- 108010004098 Leucyl aminopeptidase Proteins 0.000 description 3
- 241000124008 Mammalia Species 0.000 description 3
- 108091028080 MiR-132 Proteins 0.000 description 3
- 108091036422 MiR-296 Proteins 0.000 description 3
- 108091028066 Mir-126 Proteins 0.000 description 3
- 101100352764 Mus musculus Podn gene Proteins 0.000 description 3
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 3
- 239000004721 Polyphenylene oxide Substances 0.000 description 3
- 241000243142 Porifera Species 0.000 description 3
- 239000013614 RNA sample Substances 0.000 description 3
- 206010061481 Renal injury Diseases 0.000 description 3
- 229920004890 Triton X-100 Polymers 0.000 description 3
- 239000013504 Triton X-100 Substances 0.000 description 3
- 239000004480 active ingredient Substances 0.000 description 3
- 229940121375 antifungal agent Drugs 0.000 description 3
- 238000004820 blood count Methods 0.000 description 3
- 210000001124 body fluid Anatomy 0.000 description 3
- 239000010839 body fluid Substances 0.000 description 3
- 229910052791 calcium Inorganic materials 0.000 description 3
- 239000011575 calcium Substances 0.000 description 3
- 230000021164 cell adhesion Effects 0.000 description 3
- 230000003915 cell function Effects 0.000 description 3
- HVYWMOMLDIMFJA-DPAQBDIFSA-N cholesterol Chemical compound C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HVYWMOMLDIMFJA-DPAQBDIFSA-N 0.000 description 3
- 229960002424 collagenase Drugs 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 230000001086 cytosolic effect Effects 0.000 description 3
- 230000006378 damage Effects 0.000 description 3
- 238000000151 deposition Methods 0.000 description 3
- 210000001755 duct epithelial cell Anatomy 0.000 description 3
- 238000005516 engineering process Methods 0.000 description 3
- 230000007705 epithelial mesenchymal transition Effects 0.000 description 3
- 210000002744 extracellular matrix Anatomy 0.000 description 3
- 230000003176 fibrotic effect Effects 0.000 description 3
- 238000001914 filtration Methods 0.000 description 3
- 238000009472 formulation Methods 0.000 description 3
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 3
- 210000004565 granule cell Anatomy 0.000 description 3
- 230000005484 gravity Effects 0.000 description 3
- 210000002865 immune cell Anatomy 0.000 description 3
- 238000010166 immunofluorescence Methods 0.000 description 3
- 230000002055 immunohistochemical effect Effects 0.000 description 3
- 238000003364 immunohistochemistry Methods 0.000 description 3
- 239000003018 immunosuppressive agent Substances 0.000 description 3
- 239000007943 implant Substances 0.000 description 3
- 238000001802 infusion Methods 0.000 description 3
- 239000003112 inhibitor Substances 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 230000005764 inhibitory process Effects 0.000 description 3
- 239000010410 layer Substances 0.000 description 3
- 108091007423 let-7b Proteins 0.000 description 3
- 108091033753 let-7d stem-loop Proteins 0.000 description 3
- 108091024449 let-7e stem-loop Proteins 0.000 description 3
- 108091044227 let-7e-1 stem-loop Proteins 0.000 description 3
- 108091071181 let-7e-2 stem-loop Proteins 0.000 description 3
- 108091042844 let-7i stem-loop Proteins 0.000 description 3
- 150000002632 lipids Chemical class 0.000 description 3
- 235000020121 low-fat milk Nutrition 0.000 description 3
- 210000004962 mammalian cell Anatomy 0.000 description 3
- 108091064218 miR-1224 stem-loop Proteins 0.000 description 3
- 108091044988 miR-125a stem-loop Proteins 0.000 description 3
- 108091084619 miR-125b-1 stem-loop Proteins 0.000 description 3
- 108091063409 miR-125b-2 stem-loop Proteins 0.000 description 3
- 108091029379 miR-139 stem-loop Proteins 0.000 description 3
- 108091060382 miR-140 stem-loop Proteins 0.000 description 3
- 108091026495 miR-148b stem-loop Proteins 0.000 description 3
- 108091091751 miR-17 stem-loop Proteins 0.000 description 3
- 108091059199 miR-200a stem-loop Proteins 0.000 description 3
- 108091049679 miR-20a stem-loop Proteins 0.000 description 3
- 108091039792 miR-20b stem-loop Proteins 0.000 description 3
- 108091085564 miR-25 stem-loop Proteins 0.000 description 3
- 108091080167 miR-25-1 stem-loop Proteins 0.000 description 3
- 108091083056 miR-25-2 stem-loop Proteins 0.000 description 3
- 108091083275 miR-26b stem-loop Proteins 0.000 description 3
- 108091039812 miR-28 stem-loop Proteins 0.000 description 3
- 108091088477 miR-29a stem-loop Proteins 0.000 description 3
- 108091029716 miR-29a-1 stem-loop Proteins 0.000 description 3
- 108091092089 miR-29a-2 stem-loop Proteins 0.000 description 3
- 108091066559 miR-29a-3 stem-loop Proteins 0.000 description 3
- 108091047189 miR-29c stem-loop Proteins 0.000 description 3
- 108091054490 miR-29c-2 stem-loop Proteins 0.000 description 3
- 108091043187 miR-30a stem-loop Proteins 0.000 description 3
- 108091057431 miR-30d stem-loop Proteins 0.000 description 3
- 108091023108 miR-30e stem-loop Proteins 0.000 description 3
- 108091046551 miR-324 stem-loop Proteins 0.000 description 3
- 108091023968 miR-330 stem-loop Proteins 0.000 description 3
- 108091065159 miR-339 stem-loop Proteins 0.000 description 3
- 108091055145 miR-342 stem-loop Proteins 0.000 description 3
- 108091088856 miR-345 stem-loop Proteins 0.000 description 3
- 108091090583 miR-34c stem-loop Proteins 0.000 description 3
- 108091082133 miR-34c-1 stem-loop Proteins 0.000 description 3
- 108091039521 miR-363 stem-loop Proteins 0.000 description 3
- 108091056495 miR-363-1 stem-loop Proteins 0.000 description 3
- 108091025820 miR-363-2 stem-loop Proteins 0.000 description 3
- 108091028761 miR-409 stem-loop Proteins 0.000 description 3
- 108091059135 miR-429 stem-loop Proteins 0.000 description 3
- 108091056921 miR-532 stem-loop Proteins 0.000 description 3
- 108091091333 miR-542 stem-loop Proteins 0.000 description 3
- 108091072761 miR-598 stem-loop Proteins 0.000 description 3
- 108091088163 miR-760 stem-loop Proteins 0.000 description 3
- 108091053257 miR-99b stem-loop Proteins 0.000 description 3
- 239000003607 modifier Substances 0.000 description 3
- 238000010172 mouse model Methods 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 201000008383 nephritis Diseases 0.000 description 3
- 229910052698 phosphorus Inorganic materials 0.000 description 3
- 239000011574 phosphorus Substances 0.000 description 3
- 239000004626 polylactic acid Substances 0.000 description 3
- 201000002793 renal fibrosis Diseases 0.000 description 3
- 230000002441 reversible effect Effects 0.000 description 3
- 230000019491 signal transduction Effects 0.000 description 3
- 239000000725 suspension Substances 0.000 description 3
- 230000009885 systemic effect Effects 0.000 description 3
- 238000013518 transcription Methods 0.000 description 3
- 230000035897 transcription Effects 0.000 description 3
- 230000009466 transformation Effects 0.000 description 3
- 210000004926 tubular epithelial cell Anatomy 0.000 description 3
- 239000011534 wash buffer Substances 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 2
- FHVDTGUDJYJELY-UHFFFAOYSA-N 6-{[2-carboxy-4,5-dihydroxy-6-(phosphanyloxy)oxan-3-yl]oxy}-4,5-dihydroxy-3-phosphanyloxane-2-carboxylic acid Chemical compound O1C(C(O)=O)C(P)C(O)C(O)C1OC1C(C(O)=O)OC(OP)C(O)C1O FHVDTGUDJYJELY-UHFFFAOYSA-N 0.000 description 2
- 102000052030 Aldehyde Dehydrogenase 1 Family Human genes 0.000 description 2
- 108700041701 Aldehyde Dehydrogenase 1 Family Proteins 0.000 description 2
- 102100039075 Aldehyde dehydrogenase family 1 member A3 Human genes 0.000 description 2
- 108090000672 Annexin A5 Proteins 0.000 description 2
- 102000004121 Annexin A5 Human genes 0.000 description 2
- CIWBSHSKHKDKBQ-JLAZNSOCSA-N Ascorbic acid Chemical compound OC[C@H](O)[C@H]1OC(=O)C(O)=C1O CIWBSHSKHKDKBQ-JLAZNSOCSA-N 0.000 description 2
- 208000023275 Autoimmune disease Diseases 0.000 description 2
- 208000037157 Azotemia Diseases 0.000 description 2
- 102100032912 CD44 antigen Human genes 0.000 description 2
- 102100025805 Cadherin-1 Human genes 0.000 description 2
- 102000019034 Chemokines Human genes 0.000 description 2
- 108010012236 Chemokines Proteins 0.000 description 2
- 208000017667 Chronic Disease Diseases 0.000 description 2
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 2
- 102100027286 Fanconi anemia group C protein Human genes 0.000 description 2
- 101000959046 Homo sapiens Aldehyde dehydrogenase family 1 member A3 Proteins 0.000 description 2
- 101000868273 Homo sapiens CD44 antigen Proteins 0.000 description 2
- 101000914680 Homo sapiens Fanconi anemia group C protein Proteins 0.000 description 2
- 101000975496 Homo sapiens Keratin, type II cytoskeletal 8 Proteins 0.000 description 2
- 102000004877 Insulin Human genes 0.000 description 2
- 108090001061 Insulin Proteins 0.000 description 2
- XEEYBQQBJWHFJM-UHFFFAOYSA-N Iron Chemical compound [Fe] XEEYBQQBJWHFJM-UHFFFAOYSA-N 0.000 description 2
- 102100023972 Keratin, type II cytoskeletal 8 Human genes 0.000 description 2
- 206010024291 Leukaemias acute myeloid Diseases 0.000 description 2
- 239000007987 MES buffer Substances 0.000 description 2
- 108091007773 MIR100 Proteins 0.000 description 2
- 108091007700 MIR543 Proteins 0.000 description 2
- 108091007772 MIRLET7C Proteins 0.000 description 2
- TWRXJAOTZQYOKJ-UHFFFAOYSA-L Magnesium chloride Chemical compound [Mg+2].[Cl-].[Cl-] TWRXJAOTZQYOKJ-UHFFFAOYSA-L 0.000 description 2
- 108091007780 MiR-122 Proteins 0.000 description 2
- 108091093073 MiR-134 Proteins 0.000 description 2
- 108091034054 MiR-138 Proteins 0.000 description 2
- 108091046841 MiR-150 Proteins 0.000 description 2
- 108091033433 MiR-191 Proteins 0.000 description 2
- 108091028141 MiR-203 Proteins 0.000 description 2
- 108091028108 MiR-212 Proteins 0.000 description 2
- 108091026807 MiR-214 Proteins 0.000 description 2
- 108091028695 MiR-224 Proteins 0.000 description 2
- 108091093085 MiR-338 Proteins 0.000 description 2
- 108091028076 Mir-127 Proteins 0.000 description 2
- 108091027966 Mir-137 Proteins 0.000 description 2
- 108091028232 Mir-184 Proteins 0.000 description 2
- 108091080933 Mir-192/215 microRNA precursor Proteins 0.000 description 2
- 108091062154 Mir-205 Proteins 0.000 description 2
- 108091062140 Mir-223 Proteins 0.000 description 2
- 108091060585 Mir-31 Proteins 0.000 description 2
- 108091093189 Mir-375 Proteins 0.000 description 2
- 108091061758 Mir-433 Proteins 0.000 description 2
- 108091080995 Mir-9/mir-79 microRNA precursor family Proteins 0.000 description 2
- 108091027559 Mir-96 microRNA Proteins 0.000 description 2
- 208000034486 Multi-organ failure Diseases 0.000 description 2
- 208000010718 Multiple Organ Failure Diseases 0.000 description 2
- JJIHLJJYMXLCOY-BYPYZUCNSA-N N-acetyl-L-serine Chemical compound CC(=O)N[C@@H](CO)C(O)=O JJIHLJJYMXLCOY-BYPYZUCNSA-N 0.000 description 2
- 208000008589 Obesity Diseases 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 101150044441 PECAM1 gene Proteins 0.000 description 2
- 108010009711 Phalloidine Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 239000004793 Polystyrene Substances 0.000 description 2
- 238000010240 RT-PCR analysis Methods 0.000 description 2
- 101500027601 Rattus norvegicus Neurokinin A Proteins 0.000 description 2
- 108091006207 SLC-Transporter Proteins 0.000 description 2
- 102000037054 SLC-Transporter Human genes 0.000 description 2
- 108010071390 Serum Albumin Proteins 0.000 description 2
- 102000007562 Serum Albumin Human genes 0.000 description 2
- 108020004459 Small interfering RNA Proteins 0.000 description 2
- 108010009583 Transforming Growth Factors Proteins 0.000 description 2
- 102000009618 Transforming Growth Factors Human genes 0.000 description 2
- 102000004243 Tubulin Human genes 0.000 description 2
- 108090000704 Tubulin Proteins 0.000 description 2
- 108700042768 University of Wisconsin-lactobionate solution Proteins 0.000 description 2
- 108010073929 Vascular Endothelial Growth Factor A Proteins 0.000 description 2
- 102000005789 Vascular Endothelial Growth Factors Human genes 0.000 description 2
- 108010019530 Vascular Endothelial Growth Factors Proteins 0.000 description 2
- 229930003316 Vitamin D Natural products 0.000 description 2
- QYSXJUFSXHHAJI-XFEUOLMDSA-N Vitamin D3 Natural products C1(/[C@@H]2CC[C@@H]([C@]2(CCC1)C)[C@H](C)CCCC(C)C)=C/C=C1\C[C@@H](O)CCC1=C QYSXJUFSXHHAJI-XFEUOLMDSA-N 0.000 description 2
- 238000009825 accumulation Methods 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 230000001154 acute effect Effects 0.000 description 2
- 208000012998 acute renal failure Diseases 0.000 description 2
- 235000010443 alginic acid Nutrition 0.000 description 2
- 229920000615 alginic acid Polymers 0.000 description 2
- 230000000735 allogeneic effect Effects 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 230000000840 anti-viral effect Effects 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 230000006907 apoptotic process Effects 0.000 description 2
- 238000003491 array Methods 0.000 description 2
- 238000011888 autopsy Methods 0.000 description 2
- 230000009286 beneficial effect Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- 229920000249 biocompatible polymer Polymers 0.000 description 2
- 230000008512 biological response Effects 0.000 description 2
- 230000036772 blood pressure Effects 0.000 description 2
- 238000010804 cDNA synthesis Methods 0.000 description 2
- 238000010805 cDNA synthesis kit Methods 0.000 description 2
- 239000002775 capsule Substances 0.000 description 2
- 229910002092 carbon dioxide Inorganic materials 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 230000012292 cell migration Effects 0.000 description 2
- 230000009087 cell motility Effects 0.000 description 2
- 229920002678 cellulose Polymers 0.000 description 2
- 238000002512 chemotherapy Methods 0.000 description 2
- OSASVXMJTNOKOY-UHFFFAOYSA-N chlorobutanol Chemical compound CC(C)(O)C(Cl)(Cl)Cl OSASVXMJTNOKOY-UHFFFAOYSA-N 0.000 description 2
- 238000004140 cleaning Methods 0.000 description 2
- 238000003501 co-culture Methods 0.000 description 2
- 238000010835 comparative analysis Methods 0.000 description 2
- 239000002131 composite material Substances 0.000 description 2
- 238000012136 culture method Methods 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 206010012601 diabetes mellitus Diseases 0.000 description 2
- 230000009274 differential gene expression Effects 0.000 description 2
- 238000010790 dilution Methods 0.000 description 2
- 239000012895 dilution Substances 0.000 description 2
- 150000002016 disaccharides Chemical group 0.000 description 2
- BVTBRVFYZUCAKH-UHFFFAOYSA-L disodium selenite Chemical compound [Na+].[Na+].[O-][Se]([O-])=O BVTBRVFYZUCAKH-UHFFFAOYSA-L 0.000 description 2
- 208000035475 disorder Diseases 0.000 description 2
- 108010007093 dispase Proteins 0.000 description 2
- 238000001962 electrophoresis Methods 0.000 description 2
- 230000007368 endocrine function Effects 0.000 description 2
- 239000004744 fabric Substances 0.000 description 2
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 2
- 238000005194 fractionation Methods 0.000 description 2
- 238000010230 functional analysis Methods 0.000 description 2
- 238000011194 good manufacturing practice Methods 0.000 description 2
- 239000003102 growth factor Substances 0.000 description 2
- 230000035876 healing Effects 0.000 description 2
- 239000008241 heterogeneous mixture Substances 0.000 description 2
- 230000013632 homeostatic process Effects 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 108010003425 hyaluronan-mediated motility receptor Proteins 0.000 description 2
- 229940124589 immunosuppressive drug Drugs 0.000 description 2
- 229940125396 insulin Drugs 0.000 description 2
- 230000010354 integration Effects 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 238000001361 intraarterial administration Methods 0.000 description 2
- 238000001990 intravenous administration Methods 0.000 description 2
- 108091063986 let-7f stem-loop Proteins 0.000 description 2
- 208000019423 liver disease Diseases 0.000 description 2
- 231100000053 low toxicity Toxicity 0.000 description 2
- 238000007898 magnetic cell sorting Methods 0.000 description 2
- 230000014759 maintenance of location Effects 0.000 description 2
- 210000002901 mesenchymal stem cell Anatomy 0.000 description 2
- 108091079786 miR-105 stem-loop Proteins 0.000 description 2
- 108091045790 miR-106b stem-loop Proteins 0.000 description 2
- 108091051828 miR-122 stem-loop Proteins 0.000 description 2
- 108091050014 miR-125b-3 stem-loop Proteins 0.000 description 2
- 108091070946 miR-128 stem-loop Proteins 0.000 description 2
- 108091040501 miR-129 stem-loop Proteins 0.000 description 2
- 108091045757 miR-129-3 stem-loop Proteins 0.000 description 2
- 108091090758 miR-129-4 stem-loop Proteins 0.000 description 2
- 108091065139 miR-129-5 stem-loop Proteins 0.000 description 2
- 108091028466 miR-130b stem-loop Proteins 0.000 description 2
- 108091079016 miR-133b Proteins 0.000 description 2
- 108091043162 miR-133b stem-loop Proteins 0.000 description 2
- 108091030496 miR-138 stem-loop Proteins 0.000 description 2
- 108091046685 miR-139-1 stem-loop Proteins 0.000 description 2
- 108091058688 miR-141 stem-loop Proteins 0.000 description 2
- 108091043612 miR-146b stem-loop Proteins 0.000 description 2
- 108091037426 miR-152 stem-loop Proteins 0.000 description 2
- 108091059964 miR-154 stem-loop Proteins 0.000 description 2
- 108091031326 miR-15b stem-loop Proteins 0.000 description 2
- 108091041042 miR-18 stem-loop Proteins 0.000 description 2
- 108091031103 miR-181a stem-loop Proteins 0.000 description 2
- 108091046591 miR-181a-4 stem-loop Proteins 0.000 description 2
- 108091049627 miR-181a-5 stem-loop Proteins 0.000 description 2
- 108091064825 miR-181c stem-loop Proteins 0.000 description 2
- 108091044400 miR-181c-1 stem-loop Proteins 0.000 description 2
- 108091048818 miR-181c-2 stem-loop Proteins 0.000 description 2
- 108091032779 miR-181c-3 stem-loop Proteins 0.000 description 2
- 108091073628 miR-181d stem-loop Proteins 0.000 description 2
- 108091023796 miR-182 stem-loop Proteins 0.000 description 2
- 108091029500 miR-183 stem-loop Proteins 0.000 description 2
- 108091047758 miR-185 stem-loop Proteins 0.000 description 2
- 108091047641 miR-186 stem-loop Proteins 0.000 description 2
- 108091028751 miR-188 stem-loop Proteins 0.000 description 2
- 108091062221 miR-18a stem-loop Proteins 0.000 description 2
- 108091081505 miR-190 stem-loop Proteins 0.000 description 2
- 108091086834 miR-190-2 stem-loop Proteins 0.000 description 2
- 108091065212 miR-190b stem-loop Proteins 0.000 description 2
- 108091086416 miR-192 stem-loop Proteins 0.000 description 2
- 108091064378 miR-196b stem-loop Proteins 0.000 description 2
- 108091025686 miR-199a stem-loop Proteins 0.000 description 2
- 108091083769 miR-199a-1 stem-loop Proteins 0.000 description 2
- 108091047470 miR-199a-2 stem-loop Proteins 0.000 description 2
- 108091048350 miR-199a-3 stem-loop Proteins 0.000 description 2
- 108091056793 miR-199a-4 stem-loop Proteins 0.000 description 2
- 108091050874 miR-19a stem-loop Proteins 0.000 description 2
- 108091086850 miR-19a-1 stem-loop Proteins 0.000 description 2
- 108091088468 miR-19a-2 stem-loop Proteins 0.000 description 2
- 108091037787 miR-19b stem-loop Proteins 0.000 description 2
- 108091074450 miR-200c stem-loop Proteins 0.000 description 2
- 108091031479 miR-204 stem-loop Proteins 0.000 description 2
- 108091032382 miR-204-1 stem-loop Proteins 0.000 description 2
- 108091085803 miR-204-2 stem-loop Proteins 0.000 description 2
- 108091089766 miR-204-3 stem-loop Proteins 0.000 description 2
- 108091073500 miR-204-4 stem-loop Proteins 0.000 description 2
- 108091053626 miR-204-5 stem-loop Proteins 0.000 description 2
- 108091063796 miR-206 stem-loop Proteins 0.000 description 2
- 108091048308 miR-210 stem-loop Proteins 0.000 description 2
- 108091050113 miR-211 stem-loop Proteins 0.000 description 2
- 108091053935 miR-212 stem-loop Proteins 0.000 description 2
- 108091028397 miR-212-1 stem-loop Proteins 0.000 description 2
- 108091028945 miR-212-2 stem-loop Proteins 0.000 description 2
- 108091088730 miR-215 stem-loop Proteins 0.000 description 2
- 108091074368 miR-216 stem-loop Proteins 0.000 description 2
- 108091086642 miR-216a stem-loop Proteins 0.000 description 2
- 108091090052 miR-219-1 stem-loop Proteins 0.000 description 2
- 108091073060 miR-219-2 stem-loop Proteins 0.000 description 2
- 108091061917 miR-221 stem-loop Proteins 0.000 description 2
- 108091063489 miR-221-1 stem-loop Proteins 0.000 description 2
- 108091055391 miR-221-2 stem-loop Proteins 0.000 description 2
- 108091031076 miR-221-3 stem-loop Proteins 0.000 description 2
- 108091080321 miR-222 stem-loop Proteins 0.000 description 2
- 108091092825 miR-24 stem-loop Proteins 0.000 description 2
- 108091048857 miR-24-1 stem-loop Proteins 0.000 description 2
- 108091047483 miR-24-2 stem-loop Proteins 0.000 description 2
- 108091032978 miR-24-3 stem-loop Proteins 0.000 description 2
- 108091064025 miR-24-4 stem-loop Proteins 0.000 description 2
- 108091070404 miR-27b stem-loop Proteins 0.000 description 2
- 108091091533 miR-291a stem-loop Proteins 0.000 description 2
- 108091023455 miR-297 stem-loop Proteins 0.000 description 2
- 108091060033 miR-298 stem-loop Proteins 0.000 description 2
- 108091025088 miR-29b-2 stem-loop Proteins 0.000 description 2
- 108091040861 miR-300 stem-loop Proteins 0.000 description 2
- 108091029203 miR-301 stem-loop Proteins 0.000 description 2
- 108091026505 miR-301a stem-loop Proteins 0.000 description 2
- 108091030637 miR-301b stem-loop Proteins 0.000 description 2
- 108091039462 miR-301b-1 stem-loop Proteins 0.000 description 2
- 108091028786 miR-301b-2 stem-loop Proteins 0.000 description 2
- 108091055059 miR-30c stem-loop Proteins 0.000 description 2
- 108091072917 miR-30c-1 stem-loop Proteins 0.000 description 2
- 108091066131 miR-30c-2 stem-loop Proteins 0.000 description 2
- 108091024082 miR-32 stem-loop Proteins 0.000 description 2
- 108091059493 miR-322 stem-loop Proteins 0.000 description 2
- 108091062225 miR-323 stem-loop Proteins 0.000 description 2
- 108091042879 miR-326 stem-loop Proteins 0.000 description 2
- 108091029997 miR-328 stem-loop Proteins 0.000 description 2
- 108091091696 miR-331 stem-loop Proteins 0.000 description 2
- 108091031484 miR-335 stem-loop Proteins 0.000 description 2
- 108091023791 miR-339-1 stem-loop Proteins 0.000 description 2
- 108091049667 miR-340 stem-loop Proteins 0.000 description 2
- 108091051052 miR-345-1 stem-loop Proteins 0.000 description 2
- 108091049311 miR-345-2 stem-loop Proteins 0.000 description 2
- 108091073301 miR-346 stem-loop Proteins 0.000 description 2
- 108091029119 miR-34a stem-loop Proteins 0.000 description 2
- 108091079013 miR-34b Proteins 0.000 description 2
- 108091084018 miR-34b stem-loop Proteins 0.000 description 2
- 108091063470 miR-34b-1 stem-loop Proteins 0.000 description 2
- 108091049916 miR-34b-2 stem-loop Proteins 0.000 description 2
- 108091057222 miR-34b-3 stem-loop Proteins 0.000 description 2
- 108091092639 miR-34b-4 stem-loop Proteins 0.000 description 2
- 108091068963 miR-361 stem-loop Proteins 0.000 description 2
- 108091068952 miR-362 stem-loop Proteins 0.000 description 2
- 108091074983 miR-3663 stem-loop Proteins 0.000 description 2
- 108091062637 miR-367 stem-loop Proteins 0.000 description 2
- 108091080781 miR-3671 stem-loop Proteins 0.000 description 2
- 108091041011 miR-3675 stem-loop Proteins 0.000 description 2
- 108091057188 miR-369 stem-loop Proteins 0.000 description 2
- 108091036633 miR-370 stem-loop Proteins 0.000 description 2
- 108091079007 miR-376b stem-loop Proteins 0.000 description 2
- 108091055954 miR-377 stem-loop Proteins 0.000 description 2
- 108091027983 miR-378-1 stem-loop Proteins 0.000 description 2
- 108091089716 miR-378-2 stem-loop Proteins 0.000 description 2
- 108091079015 miR-379 Proteins 0.000 description 2
- 108091086215 miR-379 stem-loop Proteins 0.000 description 2
- 108091079008 miR-380 Proteins 0.000 description 2
- 108091025212 miR-380 stem-loop Proteins 0.000 description 2
- 108091032985 miR-382 Proteins 0.000 description 2
- 108091050135 miR-382 stem-loop Proteins 0.000 description 2
- 108091029369 miR-410 stem-loop Proteins 0.000 description 2
- 108091023805 miR-411 stem-loop Proteins 0.000 description 2
- 108091047664 miR-421 stem-loop Proteins 0.000 description 2
- 108091037240 miR-423 stem-loop Proteins 0.000 description 2
- 108091090987 miR-425 stem-loop Proteins 0.000 description 2
- 108091028100 miR-431 stem-loop Proteins 0.000 description 2
- 108091026812 miR-4329 stem-loop Proteins 0.000 description 2
- 108091072779 miR-455 stem-loop Proteins 0.000 description 2
- 108091056879 miR-455-2 stem-loop Proteins 0.000 description 2
- 108091080309 miR-483 stem-loop Proteins 0.000 description 2
- 108091080700 miR-484 stem-loop Proteins 0.000 description 2
- 108091062429 miR-487b stem-loop Proteins 0.000 description 2
- 108091041063 miR-488 stem-loop Proteins 0.000 description 2
- 108091050850 miR-489 stem-loop Proteins 0.000 description 2
- 108091087492 miR-490 stem-loop Proteins 0.000 description 2
- 108091031190 miR-495 stem-loop Proteins 0.000 description 2
- 108091085103 miR-496 stem-loop Proteins 0.000 description 2
- 108091063340 miR-497 stem-loop Proteins 0.000 description 2
- 108091056170 miR-499 stem-loop Proteins 0.000 description 2
- 108091050885 miR-499-1 stem-loop Proteins 0.000 description 2
- 108091038523 miR-499-2 stem-loop Proteins 0.000 description 2
- 108091048782 miR-501 stem-loop Proteins 0.000 description 2
- 108091033331 miR-503 stem-loop Proteins 0.000 description 2
- 108091064134 miR-504 stem-loop Proteins 0.000 description 2
- 108091031110 miR-539 stem-loop Proteins 0.000 description 2
- 108091052540 miR-542-1 stem-loop Proteins 0.000 description 2
- 108091093086 miR-542-2 stem-loop Proteins 0.000 description 2
- 108091088868 miR-542-3 stem-loop Proteins 0.000 description 2
- 108091076271 miR-543 stem-loop Proteins 0.000 description 2
- 108091072629 miR-544 stem-loop Proteins 0.000 description 2
- 108091049798 miR-544-2 stem-loop Proteins 0.000 description 2
- 108091069934 miR-598-1 stem-loop Proteins 0.000 description 2
- 108091037724 miR-598-2 stem-loop Proteins 0.000 description 2
- 108091050734 miR-652 stem-loop Proteins 0.000 description 2
- 108091050528 miR-653 stem-loop Proteins 0.000 description 2
- 108091092761 miR-671 stem-loop Proteins 0.000 description 2
- 108091072838 miR-674 stem-loop Proteins 0.000 description 2
- 108091089534 miR-708 stem-loop Proteins 0.000 description 2
- 108091050357 miR-711 stem-loop Proteins 0.000 description 2
- 108091080296 miR-758 stem-loop Proteins 0.000 description 2
- 108091050446 miR-761 stem-loop Proteins 0.000 description 2
- 108091023650 miR-764 stem-loop Proteins 0.000 description 2
- 108091056169 miR-770 stem-loop Proteins 0.000 description 2
- 108091065429 miR-871 stem-loop Proteins 0.000 description 2
- 108091084058 miR-874 stem-loop Proteins 0.000 description 2
- 108091080557 miR-875 stem-loop Proteins 0.000 description 2
- 108091058491 miR-877 stem-loop Proteins 0.000 description 2
- 108091047084 miR-9 stem-loop Proteins 0.000 description 2
- 108091034121 miR-92a stem-loop Proteins 0.000 description 2
- 108091041519 miR-92a-3 stem-loop Proteins 0.000 description 2
- 108091038507 miR-92b stem-loop Proteins 0.000 description 2
- 108091081014 miR-92b-1 stem-loop Proteins 0.000 description 2
- 108091032846 miR-92b-2 stem loop Proteins 0.000 description 2
- 108091032902 miR-93 stem-loop Proteins 0.000 description 2
- 108091071148 miR-935 stem-loop Proteins 0.000 description 2
- 108091089336 miR-942 stem-loop Proteins 0.000 description 2
- 108091086713 miR-96 stem-loop Proteins 0.000 description 2
- 108091070961 miR-96-3 stem-loop Proteins 0.000 description 2
- 108091081987 miR384 stem-loop Proteins 0.000 description 2
- 239000011325 microbead Substances 0.000 description 2
- 238000012544 monitoring process Methods 0.000 description 2
- 208000029744 multiple organ dysfunction syndrome Diseases 0.000 description 2
- 102000039446 nucleic acids Human genes 0.000 description 2
- 108020004707 nucleic acids Proteins 0.000 description 2
- 150000007523 nucleic acids Chemical class 0.000 description 2
- 235000020824 obesity Nutrition 0.000 description 2
- 238000005457 optimization Methods 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- 150000003904 phospholipids Chemical class 0.000 description 2
- 229920003023 plastic Polymers 0.000 description 2
- 239000004033 plastic Substances 0.000 description 2
- 229920000747 poly(lactic acid) Polymers 0.000 description 2
- 229920001610 polycaprolactone Polymers 0.000 description 2
- 239000004632 polycaprolactone Substances 0.000 description 2
- 229920000570 polyether Polymers 0.000 description 2
- 229920001282 polysaccharide Polymers 0.000 description 2
- 239000005017 polysaccharide Substances 0.000 description 2
- 150000004804 polysaccharides Chemical class 0.000 description 2
- 229920002223 polystyrene Polymers 0.000 description 2
- 239000005077 polysulfide Substances 0.000 description 2
- 229920001021 polysulfide Polymers 0.000 description 2
- 150000008117 polysulfides Polymers 0.000 description 2
- 238000004321 preservation Methods 0.000 description 2
- 230000001737 promoting effect Effects 0.000 description 2
- 201000001474 proteinuria Diseases 0.000 description 2
- ZAHRKKWIAAJSAO-UHFFFAOYSA-N rapamycin Natural products COCC(O)C(=C/C(C)C(=O)CC(OC(=O)C1CCCCN1C(=O)C(=O)C2(O)OC(CC(OC)C(=CC=CC=CC(C)CC(C)C(=O)C)C)CCC2C)C(C)CC3CCC(O)C(C3)OC)C ZAHRKKWIAAJSAO-UHFFFAOYSA-N 0.000 description 2
- 230000001850 reproductive effect Effects 0.000 description 2
- 230000000717 retained effect Effects 0.000 description 2
- 239000012146 running buffer Substances 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- 230000011664 signaling Effects 0.000 description 2
- 229960002930 sirolimus Drugs 0.000 description 2
- 239000002002 slurry Substances 0.000 description 2
- 239000004055 small Interfering RNA Substances 0.000 description 2
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 2
- 239000011781 sodium selenite Substances 0.000 description 2
- 229960001471 sodium selenite Drugs 0.000 description 2
- 235000015921 sodium selenite Nutrition 0.000 description 2
- 239000002904 solvent Substances 0.000 description 2
- 230000000087 stabilizing effect Effects 0.000 description 2
- 239000011550 stock solution Substances 0.000 description 2
- 238000013517 stratification Methods 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 238000003786 synthesis reaction Methods 0.000 description 2
- 229920001059 synthetic polymer Polymers 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- 230000002103 transcriptional effect Effects 0.000 description 2
- 238000001890 transfection Methods 0.000 description 2
- 208000037978 tubular injury Diseases 0.000 description 2
- 230000010024 tubular injury Effects 0.000 description 2
- 230000010245 tubular reabsorption Effects 0.000 description 2
- 208000001072 type 2 diabetes mellitus Diseases 0.000 description 2
- 210000003556 vascular endothelial cell Anatomy 0.000 description 2
- 235000019166 vitamin D Nutrition 0.000 description 2
- 239000011710 vitamin D Substances 0.000 description 2
- 150000003710 vitamin D derivatives Chemical class 0.000 description 2
- 229940046008 vitamin d Drugs 0.000 description 2
- 239000002759 woven fabric Substances 0.000 description 2
- SFGFYNXPJMOUHK-PKAFTLKUSA-N (2r)-2-[[(2r)-2-amino-5-(diaminomethylideneamino)pentanoyl]amino]-n-[(2r)-1-[[(2r)-1-[[(2r)-1-[[(2r)-1-[[(2r)-1-[[(2r)-1-[[2-[[(2r)-1-amino-3-(4-hydroxyphenyl)-1-oxopropan-2-yl]amino]-2-oxoethyl]amino]-1-oxohexan-2-yl]amino]-1-oxohexan-2-yl]amino]-1-oxohe Chemical compound NC(N)=NCCC[C@@H](N)C(=O)N[C@H](CCCC)C(=O)N[C@H](CCCC)C(=O)N[C@H](CCCC)C(=O)N[C@H](CCCN=C(N)N)C(=O)N[C@H](CCCC)C(=O)N[C@H](CCCC)C(=O)N[C@H](CCCC)C(=O)NCC(=O)N[C@@H](C(N)=O)CC1=CC=C(O)C=C1 SFGFYNXPJMOUHK-PKAFTLKUSA-N 0.000 description 1
- PKZFFNJDFBBUIS-ZSCHJXSPSA-N (2s)-2-amino-4-methylpentanoic acid;4-nitroaniline Chemical compound CC(C)C[C@H](N)C(O)=O.NC1=CC=C([N+]([O-])=O)C=C1 PKZFFNJDFBBUIS-ZSCHJXSPSA-N 0.000 description 1
- GMVPRGQOIOIIMI-UHFFFAOYSA-N (8R,11R,12R,13E,15S)-11,15-Dihydroxy-9-oxo-13-prostenoic acid Natural products CCCCCC(O)C=CC1C(O)CC(=O)C1CCCCCCC(O)=O GMVPRGQOIOIIMI-UHFFFAOYSA-N 0.000 description 1
- FPVKHBSQESCIEP-UHFFFAOYSA-N (8S)-3-(2-deoxy-beta-D-erythro-pentofuranosyl)-3,6,7,8-tetrahydroimidazo[4,5-d][1,3]diazepin-8-ol Natural products C1C(O)C(CO)OC1N1C(NC=NCC2O)=C2N=C1 FPVKHBSQESCIEP-UHFFFAOYSA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- GSSLQBZZKKOMAF-UHFFFAOYSA-N 2-cyano-n-(4-cyanophenyl)-3-cyclopropyl-3-oxopropanamide Chemical compound C1CC1C(=O)C(C#N)C(=O)NC1=CC=C(C#N)C=C1 GSSLQBZZKKOMAF-UHFFFAOYSA-N 0.000 description 1
- KXGFMDJXCMQABM-UHFFFAOYSA-N 2-methoxy-6-methylphenol Chemical compound [CH]OC1=CC=CC([CH])=C1O KXGFMDJXCMQABM-UHFFFAOYSA-N 0.000 description 1
- HVCOBJNICQPDBP-UHFFFAOYSA-N 3-[3-[3,5-dihydroxy-6-methyl-4-(3,4,5-trihydroxy-6-methyloxan-2-yl)oxyoxan-2-yl]oxydecanoyloxy]decanoic acid;hydrate Chemical compound O.OC1C(OC(CC(=O)OC(CCCCCCC)CC(O)=O)CCCCCCC)OC(C)C(O)C1OC1C(O)C(O)C(O)C(C)O1 HVCOBJNICQPDBP-UHFFFAOYSA-N 0.000 description 1
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 1
- LSZNZLWBRQAWNH-UHFFFAOYSA-N 4-[5-(4,7-dimethyl-1-benzofuran-2-yl)-1h-pyrrol-2-yl]benzoic acid Chemical compound C=1C=2C(C)=CC=C(C)C=2OC=1C(N1)=CC=C1C1=CC=C(C(O)=O)C=C1 LSZNZLWBRQAWNH-UHFFFAOYSA-N 0.000 description 1
- WSSSPWUEQFSQQG-UHFFFAOYSA-N 4-methyl-1-pentene Chemical compound CC(C)CC=C WSSSPWUEQFSQQG-UHFFFAOYSA-N 0.000 description 1
- 102100025908 5-oxoprolinase Human genes 0.000 description 1
- 108010005465 AC133 Antigen Proteins 0.000 description 1
- 102000005908 AC133 Antigen Human genes 0.000 description 1
- 101150043214 AGXT2 gene Proteins 0.000 description 1
- 101150040698 ANGPT2 gene Proteins 0.000 description 1
- 101150058497 ANPEP gene Proteins 0.000 description 1
- 101150027946 Acsm2 gene Proteins 0.000 description 1
- 208000030090 Acute Disease Diseases 0.000 description 1
- 101710167788 Acyl-coenzyme A synthetase ACSM2, mitochondrial Proteins 0.000 description 1
- 101710092790 Acyl-coenzyme A synthetase ACSM2A, mitochondrial Proteins 0.000 description 1
- 102100030362 Acyl-coenzyme A synthetase ACSM2B, mitochondrial Human genes 0.000 description 1
- 206010067484 Adverse reaction Diseases 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- 102100026790 Alanine-glyoxylate aminotransferase 2, mitochondrial Human genes 0.000 description 1
- 108010033918 Alanine-glyoxylate transaminase Proteins 0.000 description 1
- 102000004400 Aminopeptidases Human genes 0.000 description 1
- 108090000915 Aminopeptidases Proteins 0.000 description 1
- 102100034608 Angiopoietin-2 Human genes 0.000 description 1
- 108010048036 Angiopoietin-2 Proteins 0.000 description 1
- 101150073415 Aqp4 gene Proteins 0.000 description 1
- 101150005351 Aqp7 gene Proteins 0.000 description 1
- 108010036280 Aquaporin 4 Proteins 0.000 description 1
- 108050006915 Aquaporin 7 Proteins 0.000 description 1
- 102100037276 Aquaporin-4 Human genes 0.000 description 1
- 102100029406 Aquaporin-7 Human genes 0.000 description 1
- 102000015790 Asparaginase Human genes 0.000 description 1
- 108010024976 Asparaginase Proteins 0.000 description 1
- 108010081099 BMS 205820 Proteins 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 108091032955 Bacterial small RNA Proteins 0.000 description 1
- 102000004506 Blood Proteins Human genes 0.000 description 1
- 108010017384 Blood Proteins Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- VYLJAYXZTOTZRR-BTPDVQIOSA-N CC(C)(O)[C@H]1CC[C@@]2(C)[C@H]1CC[C@]1(C)[C@@H]2CC[C@@H]2[C@@]3(C)CCCC(C)(C)[C@@H]3[C@@H](O)[C@H](O)[C@@]12C Chemical compound CC(C)(O)[C@H]1CC[C@@]2(C)[C@H]1CC[C@]1(C)[C@@H]2CC[C@@H]2[C@@]3(C)CCCC(C)(C)[C@@H]3[C@@H](O)[C@H](O)[C@@]12C VYLJAYXZTOTZRR-BTPDVQIOSA-N 0.000 description 1
- 101150012094 CDH11 gene Proteins 0.000 description 1
- 101150098097 CLTRN gene Proteins 0.000 description 1
- 101150066398 CXCR4 gene Proteins 0.000 description 1
- 102100024155 Cadherin-11 Human genes 0.000 description 1
- 102100029761 Cadherin-5 Human genes 0.000 description 1
- UXVMQQNJUSDDNG-UHFFFAOYSA-L Calcium chloride Chemical compound [Cl-].[Cl-].[Ca+2] UXVMQQNJUSDDNG-UHFFFAOYSA-L 0.000 description 1
- 102000000584 Calmodulin Human genes 0.000 description 1
- 108010041952 Calmodulin Proteins 0.000 description 1
- 241001164374 Calyx Species 0.000 description 1
- 229920002134 Carboxymethyl cellulose Polymers 0.000 description 1
- 206010007559 Cardiac failure congestive Diseases 0.000 description 1
- 108091007854 Cdh1/Fizzy-related Proteins 0.000 description 1
- PTOAARAWEBMLNO-KVQBGUIXSA-N Cladribine Chemical compound C1=NC=2C(N)=NC(Cl)=NC=2N1[C@H]1C[C@H](O)[C@@H](CO)O1 PTOAARAWEBMLNO-KVQBGUIXSA-N 0.000 description 1
- 101150032944 Cnn1 gene Proteins 0.000 description 1
- 102100033635 Collectrin Human genes 0.000 description 1
- 101710138990 Collectrin Proteins 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- PMATZTZNYRCHOR-CGLBZJNRSA-N Cyclosporin A Chemical compound CC[C@@H]1NC(=O)[C@H]([C@H](O)[C@H](C)C\C=C\C)N(C)C(=O)[C@H](C(C)C)N(C)C(=O)[C@H](CC(C)C)N(C)C(=O)[C@H](CC(C)C)N(C)C(=O)[C@@H](C)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)N(C)C(=O)[C@H](C(C)C)NC(=O)[C@H](CC(C)C)N(C)C(=O)CN(C)C1=O PMATZTZNYRCHOR-CGLBZJNRSA-N 0.000 description 1
- 108010036949 Cyclosporine Proteins 0.000 description 1
- 102000004127 Cytokines Human genes 0.000 description 1
- 108090000695 Cytokines Proteins 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- QXNVGIXVLWOKEQ-UHFFFAOYSA-N Disodium Chemical compound [Na][Na] QXNVGIXVLWOKEQ-UHFFFAOYSA-N 0.000 description 1
- 101150001833 EDNRB gene Proteins 0.000 description 1
- 102000016942 Elastin Human genes 0.000 description 1
- 108010014258 Elastin Proteins 0.000 description 1
- YHFUPQNXMNMQAK-UHFFFAOYSA-N Elsibucol Chemical compound CC(C)(C)C1=C(O)C(C(C)(C)C)=CC(SC(C)(C)SC=2C=C(C(OCCCC(O)=O)=C(C=2)C(C)(C)C)C(C)(C)C)=C1 YHFUPQNXMNMQAK-UHFFFAOYSA-N 0.000 description 1
- 102100040611 Endothelin receptor type B Human genes 0.000 description 1
- 101710194572 Endothelin receptor type B Proteins 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 229940087983 Erythropoietin receptor agonist Drugs 0.000 description 1
- HKVAMNSJSFKALM-GKUWKFKPSA-N Everolimus Chemical compound C1C[C@@H](OCCO)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 HKVAMNSJSFKALM-GKUWKFKPSA-N 0.000 description 1
- 101150010122 FBP1 gene Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 102000008857 Ferritin Human genes 0.000 description 1
- 108050000784 Ferritin Proteins 0.000 description 1
- 238000008416 Ferritin Methods 0.000 description 1
- 108010080379 Fibrin Tissue Adhesive Proteins 0.000 description 1
- 102100037181 Fructose-1,6-bisphosphatase 1 Human genes 0.000 description 1
- 101710179254 Fructose-1,6-bisphosphatase 1 Proteins 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 108091006027 G proteins Proteins 0.000 description 1
- 101150098511 GPX3 gene Proteins 0.000 description 1
- 102000030782 GTP binding Human genes 0.000 description 1
- 108091000058 GTP-Binding Proteins 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 229920002148 Gellan gum Polymers 0.000 description 1
- 102000006395 Globulins Human genes 0.000 description 1
- 108010044091 Globulins Proteins 0.000 description 1
- 102000042092 Glucose transporter family Human genes 0.000 description 1
- 108091052347 Glucose transporter family Proteins 0.000 description 1
- 102100033053 Glutathione peroxidase 3 Human genes 0.000 description 1
- 101710119049 Glutathione peroxidase 3 Proteins 0.000 description 1
- 229930186217 Glycolipid Natural products 0.000 description 1
- 241001289753 Graphium sarpedon Species 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 208000031886 HIV Infections Diseases 0.000 description 1
- 208000037357 HIV infectious disease Diseases 0.000 description 1
- 208000027761 Hepatic autoimmune disease Diseases 0.000 description 1
- 101000720962 Homo sapiens 5-oxoprolinase Proteins 0.000 description 1
- 101000762236 Homo sapiens Cadherin-11 Proteins 0.000 description 1
- 101000987586 Homo sapiens Eosinophil peroxidase Proteins 0.000 description 1
- 101001046870 Homo sapiens Hypoxia-inducible factor 1-alpha Proteins 0.000 description 1
- 101000975502 Homo sapiens Keratin, type II cytoskeletal 7 Proteins 0.000 description 1
- 101000967920 Homo sapiens Left-right determination factor 1 Proteins 0.000 description 1
- 101000645293 Homo sapiens Metalloproteinase inhibitor 3 Proteins 0.000 description 1
- 101001000104 Homo sapiens Myosin-11 Proteins 0.000 description 1
- 102000003918 Hyaluronan Synthases Human genes 0.000 description 1
- 108090000320 Hyaluronan Synthases Proteins 0.000 description 1
- 102100027735 Hyaluronan mediated motility receptor Human genes 0.000 description 1
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 1
- 206010020880 Hypertrophy Diseases 0.000 description 1
- 208000013016 Hypoglycemia Diseases 0.000 description 1
- 102100022875 Hypoxia-inducible factor 1-alpha Human genes 0.000 description 1
- 238000012404 In vitro experiment Methods 0.000 description 1
- 102100023974 Keratin, type II cytoskeletal 7 Human genes 0.000 description 1
- 206010023421 Kidney fibrosis Diseases 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- 108010070919 LJP 1082 Proteins 0.000 description 1
- 102100040508 Left-right determination factor 1 Human genes 0.000 description 1
- 108091007775 MIR1-1 Proteins 0.000 description 1
- 108091007777 MIR106B Proteins 0.000 description 1
- 108091007774 MIR107 Proteins 0.000 description 1
- 108091008060 MIR10A Proteins 0.000 description 1
- 108091008058 MIR10B Proteins 0.000 description 1
- 108091007781 MIR124-1 Proteins 0.000 description 1
- 108091007778 MIR124-2 Proteins 0.000 description 1
- 108091007702 MIR1260B Proteins 0.000 description 1
- 108091007690 MIR208B Proteins 0.000 description 1
- 108091008051 MIR27A Proteins 0.000 description 1
- 108091007701 MIR3162 Proteins 0.000 description 1
- 108091008055 MIR449A Proteins 0.000 description 1
- 108091007685 MIR541 Proteins 0.000 description 1
- 108091008063 MIR618 Proteins 0.000 description 1
- 108091007699 MIR665 Proteins 0.000 description 1
- 108091007771 MIRLET7A1 Proteins 0.000 description 1
- 102100026261 Metalloproteinase inhibitor 3 Human genes 0.000 description 1
- 206010027476 Metastases Diseases 0.000 description 1
- 108091093142 MiR-144 Proteins 0.000 description 1
- 108091033773 MiR-155 Proteins 0.000 description 1
- 108091081013 MiR-33 Proteins 0.000 description 1
- 108091061943 Mir-218 microRNA precursor family Proteins 0.000 description 1
- 108091060302 Mir-320 Proteins 0.000 description 1
- 108091064430 Mir-48 Proteins 0.000 description 1
- 108091029866 Mir-672 microRNA precursor family Proteins 0.000 description 1
- 241000713333 Mouse mammary tumor virus Species 0.000 description 1
- 241001529936 Murinae Species 0.000 description 1
- 101100503236 Mus musculus Folr1 gene Proteins 0.000 description 1
- 101100347983 Mus musculus Napsa gene Proteins 0.000 description 1
- 101000996033 Mus musculus Nodal Proteins 0.000 description 1
- 101100372299 Mus musculus Utf1 gene Proteins 0.000 description 1
- 101150054386 Myh11 gene Proteins 0.000 description 1
- 102100036639 Myosin-11 Human genes 0.000 description 1
- OVRNDRQMDRJTHS-UHFFFAOYSA-N N-acelyl-D-glucosamine Natural products CC(=O)NC1C(O)OC(CO)C(O)C1O OVRNDRQMDRJTHS-UHFFFAOYSA-N 0.000 description 1
- OVRNDRQMDRJTHS-FMDGEEDCSA-N N-acetyl-beta-D-glucosamine Chemical compound CC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-FMDGEEDCSA-N 0.000 description 1
- MBLBDJOUHNCFQT-LXGUWJNJSA-N N-acetylglucosamine Natural products CC(=O)N[C@@H](C=O)[C@@H](O)[C@H](O)[C@H](O)CO MBLBDJOUHNCFQT-LXGUWJNJSA-N 0.000 description 1
- 238000011887 Necropsy Methods 0.000 description 1
- 206010028980 Neoplasm Diseases 0.000 description 1
- 102100023195 Nephrin Human genes 0.000 description 1
- 102100027341 Neutral and basic amino acid transport protein rBAT Human genes 0.000 description 1
- 101710159376 Neutral and basic amino acid transport protein rBAT Proteins 0.000 description 1
- 239000000020 Nitrocellulose Substances 0.000 description 1
- 108020003217 Nuclear RNA Proteins 0.000 description 1
- 102000043141 Nuclear RNA Human genes 0.000 description 1
- 102000011931 Nucleoproteins Human genes 0.000 description 1
- 108010061100 Nucleoproteins Proteins 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 239000002033 PVDF binder Substances 0.000 description 1
- 229930040373 Paraformaldehyde Natural products 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 102000009658 Peptidylprolyl Isomerase Human genes 0.000 description 1
- 108010020062 Peptidylprolyl Isomerase Proteins 0.000 description 1
- BELBBZDIHDAJOR-UHFFFAOYSA-N Phenolsulfonephthalein Chemical group C1=CC(O)=CC=C1C1(C=2C=CC(O)=CC=2)C2=CC=CC=C2S(=O)(=O)O1 BELBBZDIHDAJOR-UHFFFAOYSA-N 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 229920003171 Poly (ethylene oxide) Polymers 0.000 description 1
- 239000004696 Poly ether ether ketone Substances 0.000 description 1
- 239000004952 Polyamide Substances 0.000 description 1
- 239000004695 Polyether sulfone Substances 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- 239000004642 Polyimide Substances 0.000 description 1
- 239000004734 Polyphenylene sulfide Substances 0.000 description 1
- 229920001213 Polysorbate 20 Polymers 0.000 description 1
- 239000004372 Polyvinyl alcohol Substances 0.000 description 1
- ZLMJMSJWJFRBEC-UHFFFAOYSA-N Potassium Chemical compound [K] ZLMJMSJWJFRBEC-UHFFFAOYSA-N 0.000 description 1
- 108010067787 Proteoglycans Proteins 0.000 description 1
- 102000016611 Proteoglycans Human genes 0.000 description 1
- 238000002123 RNA extraction Methods 0.000 description 1
- 101000595189 Rattus norvegicus Podocin Proteins 0.000 description 1
- 206010063897 Renal ischaemia Diseases 0.000 description 1
- 101150061614 Rgs4 gene Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- 101150091821 SLC3A1 gene Proteins 0.000 description 1
- 101150059736 SRY gene Proteins 0.000 description 1
- 108010013296 Sericins Proteins 0.000 description 1
- 101150116689 Slc2a2 gene Proteins 0.000 description 1
- 108020004688 Small Nuclear RNA Proteins 0.000 description 1
- 102000039471 Small Nuclear RNA Human genes 0.000 description 1
- 108020003224 Small Nucleolar RNA Proteins 0.000 description 1
- 102000042773 Small Nucleolar RNA Human genes 0.000 description 1
- 101150112740 Srgn gene Proteins 0.000 description 1
- 238000000692 Student's t-test Methods 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- 108010044281 TATA-Box Binding Protein Proteins 0.000 description 1
- 102100040296 TATA-box-binding protein Human genes 0.000 description 1
- ZIAXNZCTODBCKW-UHFFFAOYSA-N TMC-95 C Natural products C12=CC=CC3=C2NC(=O)C3(O)C(O)C(C(=O)NC=CC)NC(=O)C(CC(N)=O)NC(=O)C(NC(=O)C(=O)C(C)CC)CC2=CC=C(O)C1=C2 ZIAXNZCTODBCKW-UHFFFAOYSA-N 0.000 description 1
- 108010065317 TMC-95A Proteins 0.000 description 1
- ZIAXNZCTODBCKW-BOYGTWLISA-N TMC-95A Chemical compound O[C@@H]([C@]1(O)C(=O)NC2=C1C=CC=C21)[C@@H](C(=O)N\C=C/C)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](NC(=O)C(=O)[C@@H](C)CC)CC2=CC=C(O)C1=C2 ZIAXNZCTODBCKW-BOYGTWLISA-N 0.000 description 1
- QJJXYPPXXYFBGM-LFZNUXCKSA-N Tacrolimus Chemical compound C1C[C@@H](O)[C@H](OC)C[C@@H]1\C=C(/C)[C@@H]1[C@H](C)[C@@H](O)CC(=O)[C@H](CC=C)/C=C(C)/C[C@H](C)C[C@H](OC)[C@H]([C@H](C[C@H]2C)OC)O[C@@]2(O)C(=O)C(=O)N2CCCC[C@H]2C(=O)O1 QJJXYPPXXYFBGM-LFZNUXCKSA-N 0.000 description 1
- 101150079992 Timp3 gene Proteins 0.000 description 1
- 102000005406 Tissue Inhibitor of Metalloproteinase-3 Human genes 0.000 description 1
- 102000005747 Transcription Factor RelA Human genes 0.000 description 1
- 108010031154 Transcription Factor RelA Proteins 0.000 description 1
- 108091023040 Transcription factor Proteins 0.000 description 1
- 102000040945 Transcription factor Human genes 0.000 description 1
- 102000004338 Transferrin Human genes 0.000 description 1
- 108090000901 Transferrin Proteins 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- 102000004142 Trypsin Human genes 0.000 description 1
- 108090000631 Trypsin Proteins 0.000 description 1
- 229920001807 Urea-formaldehyde Polymers 0.000 description 1
- 208000036142 Viral infection Diseases 0.000 description 1
- 206010047626 Vitamin D Deficiency Diseases 0.000 description 1
- 101150010310 WNT-4 gene Proteins 0.000 description 1
- 101150084041 WT1 gene Proteins 0.000 description 1
- 208000008383 Wilms tumor Diseases 0.000 description 1
- 102000052548 Wnt-4 Human genes 0.000 description 1
- 108700020984 Wnt-4 Proteins 0.000 description 1
- PZDPVSFZTKORSP-OAHLLOKOSA-N [2-[6-(diaminomethylideneamino)hexylamino]-2-oxoethyl] n-[4-[[(3r)-3-aminobutyl]amino]butyl]carbamate Chemical compound C[C@@H](N)CCNCCCCNC(=O)OCC(=O)NCCCCCCN=C(N)N PZDPVSFZTKORSP-OAHLLOKOSA-N 0.000 description 1
- DYTKVFHLKPDNRW-UHFFFAOYSA-N [4-(2-amino-4-methyl-thiazol-5-yl)-pyrimidin-2-yl]-(3-nitro-phenyl)-amine Chemical compound N1=C(N)SC(C=2N=C(NC=3C=C(C=CC=3)[N+]([O-])=O)N=CC=2)=C1C DYTKVFHLKPDNRW-UHFFFAOYSA-N 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- VYTBPJNGNGMRFH-UHFFFAOYSA-N acetic acid;azane Chemical compound N.N.CC(O)=O.CC(O)=O.CC(O)=O.CC(O)=O VYTBPJNGNGMRFH-UHFFFAOYSA-N 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 239000000853 adhesive Substances 0.000 description 1
- 230000001070 adhesive effect Effects 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 230000006838 adverse reaction Effects 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 229940072056 alginate Drugs 0.000 description 1
- 239000000783 alginic acid Substances 0.000 description 1
- 229960001126 alginic acid Drugs 0.000 description 1
- 150000004781 alginic acids Chemical class 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- 229960000711 alprostadil Drugs 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 239000003708 ampul Substances 0.000 description 1
- 150000001450 anions Chemical class 0.000 description 1
- 229950005999 anisperimus Drugs 0.000 description 1
- 230000000843 anti-fungal effect Effects 0.000 description 1
- 230000000141 anti-hypoxic effect Effects 0.000 description 1
- 230000003110 anti-inflammatory effect Effects 0.000 description 1
- 229940030600 antihypertensive agent Drugs 0.000 description 1
- 239000002220 antihypertensive agent Substances 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- 206010003246 arthritis Diseases 0.000 description 1
- 229960005070 ascorbic acid Drugs 0.000 description 1
- 235000010323 ascorbic acid Nutrition 0.000 description 1
- 239000011668 ascorbic acid Substances 0.000 description 1
- 229960003272 asparaginase Drugs 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-M asparaginate Chemical compound [O-]C(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-M 0.000 description 1
- 229950003462 atiprimod Drugs 0.000 description 1
- SERHTTSLBVGRBY-UHFFFAOYSA-N atiprimod Chemical compound C1CC(CCC)(CCC)CCC11CN(CCCN(CC)CC)CC1 SERHTTSLBVGRBY-UHFFFAOYSA-N 0.000 description 1
- 239000012298 atmosphere Substances 0.000 description 1
- 239000007640 basal medium Substances 0.000 description 1
- 210000002469 basement membrane Anatomy 0.000 description 1
- 230000027455 binding Effects 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 230000005540 biological transmission Effects 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- OWMVSZAMULFTJU-UHFFFAOYSA-N bis-tris Chemical compound OCCN(CCO)C(CO)(CO)CO OWMVSZAMULFTJU-UHFFFAOYSA-N 0.000 description 1
- 229920001400 block copolymer Polymers 0.000 description 1
- 230000000903 blocking effect Effects 0.000 description 1
- 210000000988 bone and bone Anatomy 0.000 description 1
- 210000002798 bone marrow cell Anatomy 0.000 description 1
- 229940098773 bovine serum albumin Drugs 0.000 description 1
- 239000007853 buffer solution Substances 0.000 description 1
- 239000007975 buffered saline Substances 0.000 description 1
- 239000008366 buffered solution Substances 0.000 description 1
- 108010018828 cadherin 5 Proteins 0.000 description 1
- 239000001110 calcium chloride Substances 0.000 description 1
- 229910001628 calcium chloride Inorganic materials 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 235000014633 carbohydrates Nutrition 0.000 description 1
- 229940026110 carbon dioxide / nitrogen Drugs 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 235000010948 carboxy methyl cellulose Nutrition 0.000 description 1
- 239000008112 carboxymethyl-cellulose Substances 0.000 description 1
- 235000010418 carrageenan Nutrition 0.000 description 1
- 239000000679 carrageenan Substances 0.000 description 1
- 229920001525 carrageenan Polymers 0.000 description 1
- 229940113118 carrageenan Drugs 0.000 description 1
- 210000000845 cartilage Anatomy 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 230000001364 causal effect Effects 0.000 description 1
- 229950006754 cedelizumab Drugs 0.000 description 1
- 230000006727 cell loss Effects 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 230000017455 cell-cell adhesion Effects 0.000 description 1
- 239000001913 cellulose Substances 0.000 description 1
- 229920003086 cellulose ether Polymers 0.000 description 1
- JCKYGMPEJWAADB-UHFFFAOYSA-N chlorambucil Chemical compound OC(=O)CCCC1=CC=C(N(CCCl)CCCl)C=C1 JCKYGMPEJWAADB-UHFFFAOYSA-N 0.000 description 1
- 229960004630 chlorambucil Drugs 0.000 description 1
- 229960004926 chlorobutanol Drugs 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 230000001684 chronic effect Effects 0.000 description 1
- 229960001265 ciclosporin Drugs 0.000 description 1
- 229960002436 cladribine Drugs 0.000 description 1
- 239000012459 cleaning agent Substances 0.000 description 1
- 229960000928 clofarabine Drugs 0.000 description 1
- WDDPHFBMKLOVOX-AYQXTPAHSA-N clofarabine Chemical compound C1=NC=2C(N)=NC(Cl)=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@@H]1F WDDPHFBMKLOVOX-AYQXTPAHSA-N 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 229960005188 collagen Drugs 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 230000009850 completed effect Effects 0.000 description 1
- 150000001875 compounds Chemical class 0.000 description 1
- 238000007906 compression Methods 0.000 description 1
- 230000006835 compression Effects 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 238000004624 confocal microscopy Methods 0.000 description 1
- 239000000470 constituent Substances 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 230000001276 controlling effect Effects 0.000 description 1
- 229920001577 copolymer Polymers 0.000 description 1
- 238000005138 cryopreservation Methods 0.000 description 1
- 230000001186 cumulative effect Effects 0.000 description 1
- 238000005520 cutting process Methods 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 229930182912 cyclosporin Natural products 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 230000009089 cytolysis Effects 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000034994 death Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 238000000432 density-gradient centrifugation Methods 0.000 description 1
- 230000008021 deposition Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 238000000502 dialysis Methods 0.000 description 1
- 239000010432 diamond Substances 0.000 description 1
- 230000035487 diastolic blood pressure Effects 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 238000009792 diffusion process Methods 0.000 description 1
- 230000001079 digestive effect Effects 0.000 description 1
- 208000010643 digestive system disease Diseases 0.000 description 1
- 230000006806 disease prevention Effects 0.000 description 1
- 239000006185 dispersion Substances 0.000 description 1
- 239000002612 dispersion medium Substances 0.000 description 1
- 238000010494 dissociation reaction Methods 0.000 description 1
- 230000005593 dissociations Effects 0.000 description 1
- 239000002552 dosage form Substances 0.000 description 1
- 238000002651 drug therapy Methods 0.000 description 1
- 230000009977 dual effect Effects 0.000 description 1
- 239000000975 dye Substances 0.000 description 1
- 230000004064 dysfunction Effects 0.000 description 1
- 229960000284 efalizumab Drugs 0.000 description 1
- 229920002549 elastin Polymers 0.000 description 1
- 239000003792 electrolyte Substances 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 210000002308 embryonic cell Anatomy 0.000 description 1
- 210000002257 embryonic structure Anatomy 0.000 description 1
- 239000000839 emulsion Substances 0.000 description 1
- 201000000523 end stage renal failure Diseases 0.000 description 1
- 230000012202 endocytosis Effects 0.000 description 1
- 230000003511 endothelial effect Effects 0.000 description 1
- 210000003989 endothelium vascular Anatomy 0.000 description 1
- 230000006862 enzymatic digestion Effects 0.000 description 1
- 210000000981 epithelium Anatomy 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005167 everolimus Drugs 0.000 description 1
- 210000001723 extracellular space Anatomy 0.000 description 1
- 210000001508 eye Anatomy 0.000 description 1
- 229950001488 faralimomab Drugs 0.000 description 1
- 229960000556 fingolimod Drugs 0.000 description 1
- KKGQTZUTZRNORY-UHFFFAOYSA-N fingolimod Chemical compound CCCCCCCCC1=CC=C(CCC(N)(CO)CO)C=C1 KKGQTZUTZRNORY-UHFFFAOYSA-N 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 238000002825 functional assay Methods 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 229950004792 gavilimomab Drugs 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 239000000216 gellan gum Substances 0.000 description 1
- 235000010492 gellan gum Nutrition 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 210000001707 glomerular endothelial cell Anatomy 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 239000010931 gold Substances 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 238000000227 grinding Methods 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 238000010562 histological examination Methods 0.000 description 1
- VYLJAYXZTOTZRR-UHFFFAOYSA-N hopane-6alpha,7beta,22-triol Natural products C12CCC3C4(C)CCCC(C)(C)C4C(O)C(O)C3(C)C1(C)CCC1C2(C)CCC1C(C)(O)C VYLJAYXZTOTZRR-UHFFFAOYSA-N 0.000 description 1
- 102000044890 human EPO Human genes 0.000 description 1
- 208000033519 human immunodeficiency virus infectious disease Diseases 0.000 description 1
- 229920001477 hydrophilic polymer Polymers 0.000 description 1
- 230000002218 hypoglycaemic effect Effects 0.000 description 1
- 229950001392 ilodecakin Drugs 0.000 description 1
- MTNDZQHUAFNZQY-UHFFFAOYSA-N imidazoline Chemical compound C1CN=CN1 MTNDZQHUAFNZQY-UHFFFAOYSA-N 0.000 description 1
- 230000003832 immune regulation Effects 0.000 description 1
- 238000003018 immunoassay Methods 0.000 description 1
- 238000003125 immunofluorescent labeling Methods 0.000 description 1
- 238000002991 immunohistochemical analysis Methods 0.000 description 1
- 238000013115 immunohistochemical detection Methods 0.000 description 1
- 229960003444 immunosuppressant agent Drugs 0.000 description 1
- 238000007901 in situ hybridization Methods 0.000 description 1
- 238000000099 in vitro assay Methods 0.000 description 1
- 230000036512 infertility Effects 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 229910052500 inorganic mineral Inorganic materials 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 150000002497 iodine compounds Chemical class 0.000 description 1
- 229910052742 iron Inorganic materials 0.000 description 1
- 208000018937 joint inflammation Diseases 0.000 description 1
- 210000002510 keratinocyte Anatomy 0.000 description 1
- 208000022013 kidney Wilms tumor Diseases 0.000 description 1
- 230000006651 lactation Effects 0.000 description 1
- 238000002357 laparoscopic surgery Methods 0.000 description 1
- 238000002350 laparotomy Methods 0.000 description 1
- 230000002045 lasting effect Effects 0.000 description 1
- 108010034897 lentil lectin Proteins 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 108091091807 let-7a stem-loop Proteins 0.000 description 1
- 108091047626 let-7a-2 stem-loop Proteins 0.000 description 1
- 108091047557 let-7a-3 stem-loop Proteins 0.000 description 1
- 108091057746 let-7a-4 stem-loop Proteins 0.000 description 1
- 108091028376 let-7a-5 stem-loop Proteins 0.000 description 1
- 108091024393 let-7a-6 stem-loop Proteins 0.000 description 1
- 108091091174 let-7a-7 stem-loop Proteins 0.000 description 1
- 108091029710 let-7f-1 stem-loop Proteins 0.000 description 1
- 108091041587 let-7f-2 stem-loop Proteins 0.000 description 1
- 108091007427 let-7g Proteins 0.000 description 1
- 210000004185 liver Anatomy 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 239000003589 local anesthetic agent Substances 0.000 description 1
- 229910001629 magnesium chloride Inorganic materials 0.000 description 1
- 238000002826 magnetic-activated cell sorting Methods 0.000 description 1
- 230000035800 maturation Effects 0.000 description 1
- PSGAAPLEWMOORI-PEINSRQWSA-N medroxyprogesterone acetate Chemical compound C([C@@]12C)CC(=O)C=C1[C@@H](C)C[C@@H]1[C@@H]2CC[C@]2(C)[C@@](OC(C)=O)(C(C)=O)CC[C@H]21 PSGAAPLEWMOORI-PEINSRQWSA-N 0.000 description 1
- 208000030159 metabolic disease Diseases 0.000 description 1
- 239000002207 metabolite Substances 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 108091028606 miR-1 stem-loop Proteins 0.000 description 1
- 108091044592 miR-1-1 stem-loop Proteins 0.000 description 1
- 108091045542 miR-1-2 stem-loop Proteins 0.000 description 1
- 108091092072 miR-100 stem-loop Proteins 0.000 description 1
- 108091068974 miR-101 stem-loop Proteins 0.000 description 1
- 108091053561 miR-101-1 stem-loop Proteins 0.000 description 1
- 108091093015 miR-101-2 stem-loop Proteins 0.000 description 1
- 108091027857 miR-101a stem-loop Proteins 0.000 description 1
- 108091031328 miR-101a-1 stem-loop Proteins 0.000 description 1
- 108091085105 miR-101a-2 stem-loop Proteins 0.000 description 1
- 108091087432 miR-101b stem-loop Proteins 0.000 description 1
- 108091037473 miR-103 stem-loop Proteins 0.000 description 1
- 108091050170 miR-103-1 stem-loop Proteins 0.000 description 1
- 108091063231 miR-103-2 stem-loop Proteins 0.000 description 1
- 108091059450 miR-105-1 stem-loop Proteins 0.000 description 1
- 108091024075 miR-105-2 stem-loop Proteins 0.000 description 1
- 108091064157 miR-106a stem-loop Proteins 0.000 description 1
- 108091050539 miR-107 stem-loop Proteins 0.000 description 1
- 108091064399 miR-10b stem-loop Proteins 0.000 description 1
- 108091029615 miR-1178 stem-loop Proteins 0.000 description 1
- 108091044709 miR-1179 stem-loop Proteins 0.000 description 1
- 108091026955 miR-1180 stem-loop Proteins 0.000 description 1
- 108091090911 miR-1181 stem-loop Proteins 0.000 description 1
- 108091070518 miR-1182 stem-loop Proteins 0.000 description 1
- 108091074709 miR-1183 stem-loop Proteins 0.000 description 1
- 108091064410 miR-1184-1 stem-loop Proteins 0.000 description 1
- 108091043011 miR-1184-2 stem-loop Proteins 0.000 description 1
- 108091090941 miR-1184-3 stem-loop Proteins 0.000 description 1
- 108091038449 miR-1185-1 stem-loop Proteins 0.000 description 1
- 108091026347 miR-1185-2 stem-loop Proteins 0.000 description 1
- 108091028227 miR-1193 stem-loop Proteins 0.000 description 1
- 108091056404 miR-1197 stem-loop Proteins 0.000 description 1
- 108091084079 miR-12 stem-loop Proteins 0.000 description 1
- 108091066616 miR-1200 stem-loop Proteins 0.000 description 1
- 108091023403 miR-1202 stem-loop Proteins 0.000 description 1
- 108091061661 miR-1203 stem-loop Proteins 0.000 description 1
- 108091079787 miR-1204 stem-loop Proteins 0.000 description 1
- 108091048739 miR-1205 stem-loop Proteins 0.000 description 1
- 108091064942 miR-1206 stem-loop Proteins 0.000 description 1
- 108091054042 miR-1207 stem-loop Proteins 0.000 description 1
- 108091025237 miR-1208 stem-loop Proteins 0.000 description 1
- 108091089762 miR-1225 stem-loop Proteins 0.000 description 1
- 108091068881 miR-1226 stem-loop Proteins 0.000 description 1
- 108091038228 miR-1228 stem-loop Proteins 0.000 description 1
- 108091062185 miR-1229 stem-loop Proteins 0.000 description 1
- 108091026639 miR-1231 stem-loop Proteins 0.000 description 1
- 108091056695 miR-1233-1 stem-loop Proteins 0.000 description 1
- 108091029546 miR-1233-2 stem-loop Proteins 0.000 description 1
- 108091076486 miR-1234 stem-loop Proteins 0.000 description 1
- 108091040341 miR-1236 stem-loop Proteins 0.000 description 1
- 108091029235 miR-1237 stem-loop Proteins 0.000 description 1
- 108091044581 miR-1238 stem-loop Proteins 0.000 description 1
- 108091092839 miR-124-1 stem-loop Proteins 0.000 description 1
- 108091045380 miR-124-2 stem-loop Proteins 0.000 description 1
- 108091048120 miR-124-3 stem-loop Proteins 0.000 description 1
- 108091038601 miR-1243 stem-loop Proteins 0.000 description 1
- 108091024545 miR-1244-1 stem-loop Proteins 0.000 description 1
- 108091085131 miR-1244-2 stem-loop Proteins 0.000 description 1
- 108091046603 miR-1244-3 stem-loop Proteins 0.000 description 1
- 108091026053 miR-1245 stem-loop Proteins 0.000 description 1
- 108091043184 miR-1246 stem-loop Proteins 0.000 description 1
- 108091063365 miR-1247 stem-loop Proteins 0.000 description 1
- 108091049440 miR-1248 stem-loop Proteins 0.000 description 1
- 108091059647 miR-1248-1 stem-loop Proteins 0.000 description 1
- 108091087721 miR-1248-2 stem-loop Proteins 0.000 description 1
- 108091057905 miR-1249 stem-loop Proteins 0.000 description 1
- 108091036056 miR-1249-1 stem-loop Proteins 0.000 description 1
- 108091092614 miR-1249-2 stem-loop Proteins 0.000 description 1
- 108091070774 miR-1250 stem-loop Proteins 0.000 description 1
- 108091037074 miR-1251 stem-loop Proteins 0.000 description 1
- 108091063774 miR-1252 stem-loop Proteins 0.000 description 1
- 108091031350 miR-1253 stem-loop Proteins 0.000 description 1
- 108091051344 miR-1255a stem-loop Proteins 0.000 description 1
- 108091056875 miR-1256 stem-loop Proteins 0.000 description 1
- 108091053270 miR-1257 stem-loop Proteins 0.000 description 1
- 108091025483 miR-1258 stem-loop Proteins 0.000 description 1
- 108091049513 miR-125a-1 stem-loop Proteins 0.000 description 1
- 108091040046 miR-125a-2 stem-loop Proteins 0.000 description 1
- 108091091360 miR-125b stem-loop Proteins 0.000 description 1
- 108091045215 miR-1261 stem-loop Proteins 0.000 description 1
- 108091091214 miR-1262 stem-loop Proteins 0.000 description 1
- 108091071846 miR-1263 stem-loop Proteins 0.000 description 1
- 108091057630 miR-1264 stem-loop Proteins 0.000 description 1
- 108091062133 miR-1265 stem-loop Proteins 0.000 description 1
- 108091023935 miR-1266 stem-loop Proteins 0.000 description 1
- 108091082516 miR-1267 stem-loop Proteins 0.000 description 1
- 108091074816 miR-1268 stem-loop Proteins 0.000 description 1
- 108091051366 miR-1269 stem-loop Proteins 0.000 description 1
- 108091047499 miR-1271 stem-loop Proteins 0.000 description 1
- 108091032011 miR-1272 stem-loop Proteins 0.000 description 1
- 108091065148 miR-1273c stem-loop Proteins 0.000 description 1
- 108091046573 miR-1275 stem-loop Proteins 0.000 description 1
- 108091071866 miR-1276 stem-loop Proteins 0.000 description 1
- 108091040870 miR-1277 stem-loop Proteins 0.000 description 1
- 108091056018 miR-1278 stem-loop Proteins 0.000 description 1
- 108091031034 miR-1279 stem-loop Proteins 0.000 description 1
- 108091059786 miR-128-1 stem-loop Proteins 0.000 description 1
- 108091084874 miR-128-2 stem-loop Proteins 0.000 description 1
- 108091024426 miR-1281 stem-loop Proteins 0.000 description 1
- 108091043233 miR-1282 stem-loop Proteins 0.000 description 1
- 108091083785 miR-1282-1 stem-loop Proteins 0.000 description 1
- 108091050002 miR-1282-2 stem-loop Proteins 0.000 description 1
- 108091029111 miR-1283-1 stem-loop Proteins 0.000 description 1
- 108091090307 miR-1283-2 stem-loop Proteins 0.000 description 1
- 108091047943 miR-1284 stem-loop Proteins 0.000 description 1
- 108091068978 miR-1285-1 stem-loop Proteins 0.000 description 1
- 108091042798 miR-1285-2 stem-loop Proteins 0.000 description 1
- 108091029664 miR-1286 stem-loop Proteins 0.000 description 1
- 108091029137 miR-1287 stem-loop Proteins 0.000 description 1
- 108091089195 miR-1288 stem-loop Proteins 0.000 description 1
- 108091073573 miR-1289-1 stem-loop Proteins 0.000 description 1
- 108091036184 miR-1289-2 stem-loop Proteins 0.000 description 1
- 108091038720 miR-129-1 stem-loop Proteins 0.000 description 1
- 108091064138 miR-129-2 stem-loop Proteins 0.000 description 1
- 108091082518 miR-1290 stem-loop Proteins 0.000 description 1
- 108091074094 miR-1291 stem-loop Proteins 0.000 description 1
- 108091063361 miR-1292 stem-loop Proteins 0.000 description 1
- 108091054928 miR-1293 stem-loop Proteins 0.000 description 1
- 108091031304 miR-1294 stem-loop Proteins 0.000 description 1
- 108091027538 miR-1295 stem-loop Proteins 0.000 description 1
- 108091087161 miR-1296 stem-loop Proteins 0.000 description 1
- 108091048147 miR-1297 stem-loop Proteins 0.000 description 1
- 108091081029 miR-1298 stem-loop Proteins 0.000 description 1
- 108091039008 miR-1299 stem-loop Proteins 0.000 description 1
- 108091073241 miR-1301 stem-loop Proteins 0.000 description 1
- 108091047756 miR-1302-1 stem-loop Proteins 0.000 description 1
- 108091057640 miR-1302-2 stem-loop Proteins 0.000 description 1
- 108091062125 miR-1302-3 stem-loop Proteins 0.000 description 1
- 108091024526 miR-1302-4 stem-loop Proteins 0.000 description 1
- 108091043231 miR-1302-5 stem-loop Proteins 0.000 description 1
- 108091067109 miR-1302-6 stem-loop Proteins 0.000 description 1
- 108091038333 miR-1302-7 stem-loop Proteins 0.000 description 1
- 108091038893 miR-1302-8 stem-loop Proteins 0.000 description 1
- 108091030674 miR-1302-9 stem-loop Proteins 0.000 description 1
- 108091032604 miR-1303 stem-loop Proteins 0.000 description 1
- 108091059824 miR-1304 stem-loop Proteins 0.000 description 1
- 108091044266 miR-1305 stem-loop Proteins 0.000 description 1
- 108091071567 miR-1307 stem-loop Proteins 0.000 description 1
- 108091080353 miR-1321 stem-loop Proteins 0.000 description 1
- 108091048790 miR-1322 stem-loop Proteins 0.000 description 1
- 108091057983 miR-1323 stem-loop Proteins 0.000 description 1
- 108091055372 miR-1324 stem-loop Proteins 0.000 description 1
- 108091047757 miR-133a-1 stem-loop Proteins 0.000 description 1
- 108091057540 miR-133a-2 stem-loop Proteins 0.000 description 1
- 108091047111 miR-135a-1 stem-loop Proteins 0.000 description 1
- 108091047375 miR-135a-2 stem-loop Proteins 0.000 description 1
- 108091026375 miR-135b stem-loop Proteins 0.000 description 1
- 108091086065 miR-135b-2 stem-loop Proteins 0.000 description 1
- 108091047467 miR-136 stem-loop Proteins 0.000 description 1
- 108091047498 miR-138-1 stem-loop Proteins 0.000 description 1
- 108091031925 miR-138-2 stem-loop Proteins 0.000 description 1
- 108091049185 miR-139-2 stem-loop Proteins 0.000 description 1
- 108091079658 miR-142-1 stem-loop Proteins 0.000 description 1
- 108091071830 miR-142-2 stem-loop Proteins 0.000 description 1
- 108091062895 miR-144 stem-loop Proteins 0.000 description 1
- 108091030660 miR-1468 stem-loop Proteins 0.000 description 1
- 108091025226 miR-1469 stem-loop Proteins 0.000 description 1
- 108091059191 miR-1470 stem-loop Proteins 0.000 description 1
- 108091063763 miR-1471 stem-loop Proteins 0.000 description 1
- 108091039164 miR-147b stem-loop Proteins 0.000 description 1
- 108091027034 miR-148a stem-loop Proteins 0.000 description 1
- 108091047577 miR-149 stem-loop Proteins 0.000 description 1
- 108091035696 miR-149-1 stem-loop Proteins 0.000 description 1
- 108091031096 miR-149-2 stem-loop Proteins 0.000 description 1
- 108091033783 miR-153 stem-loop Proteins 0.000 description 1
- 108091051380 miR-153-1 stem-loop Proteins 0.000 description 1
- 108091053261 miR-153-2 stem-loop Proteins 0.000 description 1
- 108091091281 miR-1537 stem-loop Proteins 0.000 description 1
- 108091076618 miR-1538 stem-loop Proteins 0.000 description 1
- 108091072808 miR-1539 stem-loop Proteins 0.000 description 1
- 108091027943 miR-16 stem-loop Proteins 0.000 description 1
- 108091074057 miR-16-1 stem-loop Proteins 0.000 description 1
- 108091056204 miR-16-2 stem-loop Proteins 0.000 description 1
- 108091044046 miR-17-1 stem-loop Proteins 0.000 description 1
- 108091069239 miR-17-2 stem-loop Proteins 0.000 description 1
- 108091065423 miR-17-3 stem-loop Proteins 0.000 description 1
- 108091049641 miR-181-1 stem-loop Proteins 0.000 description 1
- 108091053227 miR-181a-1 stem-loop Proteins 0.000 description 1
- 108091092591 miR-181a-2 stem-loop Proteins 0.000 description 1
- 108091043222 miR-181b stem-loop Proteins 0.000 description 1
- 108091074194 miR-181b-1 stem-loop Proteins 0.000 description 1
- 108091038599 miR-181b-2 stem-loop Proteins 0.000 description 1
- 108091035084 miR-1825 stem-loop Proteins 0.000 description 1
- 108091045446 miR-1827 stem-loop Proteins 0.000 description 1
- 108091058104 miR-187 stem-loop Proteins 0.000 description 1
- 108091023683 miR-187-1 stem-loop Proteins 0.000 description 1
- 108091046933 miR-18b stem-loop Proteins 0.000 description 1
- 108091083606 miR-1908 stem-loop Proteins 0.000 description 1
- 108091073513 miR-1909 stem-loop Proteins 0.000 description 1
- 108091044438 miR-1910 stem-loop Proteins 0.000 description 1
- 108091063397 miR-1911 stem-loop Proteins 0.000 description 1
- 108091050061 miR-1912 stem-loop Proteins 0.000 description 1
- 108091058197 miR-1913 stem-loop Proteins 0.000 description 1
- 108091092658 miR-1914 stem-loop Proteins 0.000 description 1
- 108091023567 miR-1915 stem-loop Proteins 0.000 description 1
- 108091063348 miR-193 stem-loop Proteins 0.000 description 1
- 108091036762 miR-193a stem-loop Proteins 0.000 description 1
- 108091039097 miR-193b stem-loop Proteins 0.000 description 1
- 108091054642 miR-194 stem-loop Proteins 0.000 description 1
- 108091080253 miR-194-1 stem-loop Proteins 0.000 description 1
- 108091089177 miR-194-2 stem-loop Proteins 0.000 description 1
- 108091054189 miR-196a stem-loop Proteins 0.000 description 1
- 108091062444 miR-196a-1 stem-loop Proteins 0.000 description 1
- 108091092367 miR-196a-2 stem-loop Proteins 0.000 description 1
- 108091045069 miR-196c stem-loop Proteins 0.000 description 1
- 108091088515 miR-197 stem-loop Proteins 0.000 description 1
- 108091057662 miR-1972-1 stem-loop Proteins 0.000 description 1
- 108091063863 miR-1972-2 stem-loop Proteins 0.000 description 1
- 108091045878 miR-1973 stem-loop Proteins 0.000 description 1
- 108091056197 miR-1976 stem-loop Proteins 0.000 description 1
- 108091046261 miR-198 stem-loop Proteins 0.000 description 1
- 108091092012 miR-199b stem-loop Proteins 0.000 description 1
- 108091028067 miR-19b-1 stem-loop Proteins 0.000 description 1
- 108091091434 miR-19b-2 stem-loop Proteins 0.000 description 1
- 108091089775 miR-200b stem-loop Proteins 0.000 description 1
- 108091041313 miR-201 stem-loop Proteins 0.000 description 1
- 108091045665 miR-202 stem-loop Proteins 0.000 description 1
- 108091023892 miR-2052 stem-loop Proteins 0.000 description 1
- 108091027856 miR-2053 stem-loop Proteins 0.000 description 1
- 108091044346 miR-2054 stem-loop Proteins 0.000 description 1
- 108091090020 miR-207 stem-loop Proteins 0.000 description 1
- 108091071651 miR-208 stem-loop Proteins 0.000 description 1
- 108091084446 miR-208a stem-loop Proteins 0.000 description 1
- 108091062547 miR-208a-1 stem-loop Proteins 0.000 description 1
- 108091055375 miR-208a-2 stem-loop Proteins 0.000 description 1
- 108091055878 miR-20b-1 stem-loop Proteins 0.000 description 1
- 108091027746 miR-20b-2 stem-loop Proteins 0.000 description 1
- 108091031554 miR-2110 stem-loop Proteins 0.000 description 1
- 108091046612 miR-2113 stem-loop Proteins 0.000 description 1
- 108091091547 miR-2114 stem-loop Proteins 0.000 description 1
- 108091068709 miR-2115 stem-loop Proteins 0.000 description 1
- 108091056747 miR-2116 stem-loop Proteins 0.000 description 1
- 108091056091 miR-2117 stem-loop Proteins 0.000 description 1
- 108091051988 miR-216b stem-loop Proteins 0.000 description 1
- 108091035328 miR-217 stem-loop Proteins 0.000 description 1
- 108091039135 miR-217-1 stem-loop Proteins 0.000 description 1
- 108091029206 miR-217-2 stem-loop Proteins 0.000 description 1
- 108091040176 miR-218 stem-loop Proteins 0.000 description 1
- 108091065218 miR-218-1 stem-loop Proteins 0.000 description 1
- 108091054980 miR-218-2 stem-loop Proteins 0.000 description 1
- 108091063841 miR-219 stem-loop Proteins 0.000 description 1
- 108091079034 miR-219-3 stem-loop Proteins 0.000 description 1
- 108091054264 miR-219-4 stem-loop Proteins 0.000 description 1
- 108091026068 miR-2276 stem-loop Proteins 0.000 description 1
- 108091025853 miR-2277 stem-loop Proteins 0.000 description 1
- 108091089977 miR-2278 stem-loop Proteins 0.000 description 1
- 108091036050 miR-2355 stem-loop Proteins 0.000 description 1
- 108091044738 miR-23c stem-loop Proteins 0.000 description 1
- 108091061970 miR-26a stem-loop Proteins 0.000 description 1
- 108091052996 miR-26a-1 stem-loop Proteins 0.000 description 1
- 108091023402 miR-26a-2 stem-loop Proteins 0.000 description 1
- 108091087639 miR-2861 stem-loop Proteins 0.000 description 1
- 108091033928 miR-2909 stem-loop Proteins 0.000 description 1
- 108091090335 miR-293 stem-loop Proteins 0.000 description 1
- 108091072810 miR-294 stem-loop Proteins 0.000 description 1
- 108091076076 miR-295 stem-loop Proteins 0.000 description 1
- 108091007432 miR-29b Proteins 0.000 description 1
- 108091057475 miR-29b-1 stem-loop Proteins 0.000 description 1
- 108091084454 miR-302a stem-loop Proteins 0.000 description 1
- 108091050195 miR-302b stem-loop Proteins 0.000 description 1
- 108091056763 miR-302c stem-loop Proteins 0.000 description 1
- 108091053185 miR-302d stem-loop Proteins 0.000 description 1
- 108091025923 miR-302e stem-loop Proteins 0.000 description 1
- 108091042976 miR-302f stem-loop Proteins 0.000 description 1
- 108091043104 miR-3065 stem-loop Proteins 0.000 description 1
- 108091047945 miR-3074 stem-loop Proteins 0.000 description 1
- 108091028014 miR-3115 stem-loop Proteins 0.000 description 1
- 108091083302 miR-3116-1 stem-loop Proteins 0.000 description 1
- 108091051080 miR-3116-2 stem-loop Proteins 0.000 description 1
- 108091044623 miR-3117 stem-loop Proteins 0.000 description 1
- 108091055820 miR-3118-1 stem-loop Proteins 0.000 description 1
- 108091026876 miR-3118-2 stem-loop Proteins 0.000 description 1
- 108091090809 miR-3118-3 stem-loop Proteins 0.000 description 1
- 108091041004 miR-3118-4 stem-loop Proteins 0.000 description 1
- 108091079622 miR-3119-1 stem-loop Proteins 0.000 description 1
- 108091042833 miR-3119-2 stem-loop Proteins 0.000 description 1
- 108091066897 miR-3120 stem-loop Proteins 0.000 description 1
- 108091054492 miR-3121 stem-loop Proteins 0.000 description 1
- 108091056717 miR-3122 stem-loop Proteins 0.000 description 1
- 108091023154 miR-3123 stem-loop Proteins 0.000 description 1
- 108091082601 miR-3124 stem-loop Proteins 0.000 description 1
- 108091077997 miR-3125 stem-loop Proteins 0.000 description 1
- 108091049491 miR-3126 stem-loop Proteins 0.000 description 1
- 108091042190 miR-3127 stem-loop Proteins 0.000 description 1
- 108091028013 miR-3128 stem-loop Proteins 0.000 description 1
- 108091025232 miR-3129 stem-loop Proteins 0.000 description 1
- 108091055023 miR-3130-1 stem-loop Proteins 0.000 description 1
- 108091057528 miR-3130-2 stem-loop Proteins 0.000 description 1
- 108091024450 miR-3131 stem-loop Proteins 0.000 description 1
- 108091043220 miR-3132 stem-loop Proteins 0.000 description 1
- 108091067044 miR-3133 stem-loop Proteins 0.000 description 1
- 108091068934 miR-3134 stem-loop Proteins 0.000 description 1
- 108091038870 miR-3135 stem-loop Proteins 0.000 description 1
- 108091029773 miR-3136 stem-loop Proteins 0.000 description 1
- 108091084637 miR-3137 stem-loop Proteins 0.000 description 1
- 108091080559 miR-3138 stem-loop Proteins 0.000 description 1
- 108091050034 miR-3139 stem-loop Proteins 0.000 description 1
- 108091045113 miR-3140 stem-loop Proteins 0.000 description 1
- 108091049132 miR-3141 stem-loop Proteins 0.000 description 1
- 108091040536 miR-3142 stem-loop Proteins 0.000 description 1
- 108091035428 miR-3143 stem-loop Proteins 0.000 description 1
- 108091031099 miR-3144 stem-loop Proteins 0.000 description 1
- 108091087197 miR-3145 stem-loop Proteins 0.000 description 1
- 108091089551 miR-3146 stem-loop Proteins 0.000 description 1
- 108091068867 miR-3147 stem-loop Proteins 0.000 description 1
- 108091056721 miR-3148 stem-loop Proteins 0.000 description 1
- 108091064121 miR-3149 stem-loop Proteins 0.000 description 1
- 108091026030 miR-3151 stem-loop Proteins 0.000 description 1
- 108091025842 miR-3152 stem-loop Proteins 0.000 description 1
- 108091087793 miR-3153 stem-loop Proteins 0.000 description 1
- 108091050341 miR-3154 stem-loop Proteins 0.000 description 1
- 108091029336 miR-3156-1 stem-loop Proteins 0.000 description 1
- 108091044501 miR-3156-2 stem-loop Proteins 0.000 description 1
- 108091048089 miR-3156-3 stem-loop Proteins 0.000 description 1
- 108091023667 miR-3157 stem-loop Proteins 0.000 description 1
- 108091048639 miR-3158-1 stem-loop Proteins 0.000 description 1
- 108091080926 miR-3158-2 stem-loop Proteins 0.000 description 1
- 108091045887 miR-3159 stem-loop Proteins 0.000 description 1
- 108091063806 miR-3160-1 stem-loop Proteins 0.000 description 1
- 108091047018 miR-3160-2 stem-loop Proteins 0.000 description 1
- 108091089859 miR-3161 stem-loop Proteins 0.000 description 1
- 108091049091 miR-3162 stem-loop Proteins 0.000 description 1
- 108091056542 miR-3163 stem-loop Proteins 0.000 description 1
- 108091053718 miR-3164 stem-loop Proteins 0.000 description 1
- 108091026328 miR-3165 stem-loop Proteins 0.000 description 1
- 108091046424 miR-3166 stem-loop Proteins 0.000 description 1
- 108091044050 miR-3167 stem-loop Proteins 0.000 description 1
- 108091051715 miR-3168 stem-loop Proteins 0.000 description 1
- 108091080927 miR-3169 stem-loop Proteins 0.000 description 1
- 108091067527 miR-3170 stem-loop Proteins 0.000 description 1
- 108091055013 miR-3171 stem-loop Proteins 0.000 description 1
- 108091063801 miR-3173 stem-loop Proteins 0.000 description 1
- 108091080572 miR-3174 stem-loop Proteins 0.000 description 1
- 108091049028 miR-3175 stem-loop Proteins 0.000 description 1
- 108091050138 miR-3176 stem-loop Proteins 0.000 description 1
- 108091056920 miR-3177 stem-loop Proteins 0.000 description 1
- 108091024889 miR-3178 stem-loop Proteins 0.000 description 1
- 108091046919 miR-3179-1 stem-loop Proteins 0.000 description 1
- 108091048797 miR-3179-2 stem-loop Proteins 0.000 description 1
- 108091047540 miR-3179-3 stem-loop Proteins 0.000 description 1
- 108091057228 miR-3180-1 stem-loop Proteins 0.000 description 1
- 108091059828 miR-3180-2 stem-loop Proteins 0.000 description 1
- 108091082971 miR-3180-3 stem-loop Proteins 0.000 description 1
- 108091074280 miR-3180-4 stem-loop Proteins 0.000 description 1
- 108091071422 miR-3180-5 stem-loop Proteins 0.000 description 1
- 108091045430 miR-3181 stem-loop Proteins 0.000 description 1
- 108091041555 miR-3182 stem-loop Proteins 0.000 description 1
- 108091066558 miR-3183 stem-loop Proteins 0.000 description 1
- 108091054011 miR-3184 stem-loop Proteins 0.000 description 1
- 108091058499 miR-3185 stem-loop Proteins 0.000 description 1
- 108091060380 miR-3186 stem-loop Proteins 0.000 description 1
- 108091082217 miR-3187 stem-loop Proteins 0.000 description 1
- 108091084051 miR-3188 stem-loop Proteins 0.000 description 1
- 108091075059 miR-3189 stem-loop Proteins 0.000 description 1
- 108091049661 miR-3190 stem-loop Proteins 0.000 description 1
- 108091063274 miR-3191 stem-loop Proteins 0.000 description 1
- 108091081425 miR-3192 stem-loop Proteins 0.000 description 1
- 108091030511 miR-3193 stem-loop Proteins 0.000 description 1
- 108091064411 miR-3194 stem-loop Proteins 0.000 description 1
- 108091041315 miR-3195 stem-loop Proteins 0.000 description 1
- 108091065418 miR-3196 stem-loop Proteins 0.000 description 1
- 108091072864 miR-3197 stem-loop Proteins 0.000 description 1
- 108091090919 miR-3198-1 stem-loop Proteins 0.000 description 1
- 108091069781 miR-3198-2 stem-loop Proteins 0.000 description 1
- 108091026478 miR-3199-1 stem-loop Proteins 0.000 description 1
- 108091088478 miR-3199-2 stem-loop Proteins 0.000 description 1
- 108091074599 miR-3200 stem-loop Proteins 0.000 description 1
- 108091066961 miR-3201 stem-loop Proteins 0.000 description 1
- 108091039526 miR-3202-1 stem-loop Proteins 0.000 description 1
- 108091031544 miR-3202-2 stem-loop Proteins 0.000 description 1
- 108091059501 miR-320a stem-loop Proteins 0.000 description 1
- 108091088570 miR-320a-1 stem-loop Proteins 0.000 description 1
- 108091070041 miR-320a-2 stem-loop Proteins 0.000 description 1
- 108091065447 miR-320a-3 stem-loop Proteins 0.000 description 1
- 108091054114 miR-320a-4 stem-loop Proteins 0.000 description 1
- 108091026895 miR-320b-1 stem-loop Proteins 0.000 description 1
- 108091040992 miR-320b-2 stem-loop Proteins 0.000 description 1
- 108091054873 miR-320c-1 stem-loop Proteins 0.000 description 1
- 108091025794 miR-320c-2 stem-loop Proteins 0.000 description 1
- 108091072780 miR-320d-1 stem-loop Proteins 0.000 description 1
- 108091040523 miR-320d-2 stem-loop Proteins 0.000 description 1
- 108091058314 miR-320e stem-loop Proteins 0.000 description 1
- 108091047549 miR-323b stem-loop Proteins 0.000 description 1
- 108091072797 miR-325 stem-loop Proteins 0.000 description 1
- 108091054466 miR-327 stem-loop Proteins 0.000 description 1
- 108091089005 miR-329 stem-loop Proteins 0.000 description 1
- 108091073504 miR-329-1 stem-loop Proteins 0.000 description 1
- 108091054657 miR-329-2 stem-loop Proteins 0.000 description 1
- 108091061632 miR-336 stem-loop Proteins 0.000 description 1
- 108091090692 miR-337 stem-loop Proteins 0.000 description 1
- 108091044951 miR-339-2 stem-loop Proteins 0.000 description 1
- 108091049902 miR-33a stem-loop Proteins 0.000 description 1
- 108091037290 miR-33b stem-loop Proteins 0.000 description 1
- 108091051540 miR-340-1 stem-loop Proteins 0.000 description 1
- 108091057189 miR-340-2 stem-loop Proteins 0.000 description 1
- 108091031067 miR-343 stem-loop Proteins 0.000 description 1
- 108091039165 miR-344-1 stem-loop Proteins 0.000 description 1
- 108091055445 miR-344-2 stem-loop Proteins 0.000 description 1
- 108091034281 miR-347 stem-loop Proteins 0.000 description 1
- 108091060489 miR-349 stem-loop Proteins 0.000 description 1
- 108091056360 miR-350 stem-loop Proteins 0.000 description 1
- 108091061985 miR-350-1 stem-loop Proteins 0.000 description 1
- 108091086625 miR-350-2 stem-loop Proteins 0.000 description 1
- 108091029214 miR-351 stem-loop Proteins 0.000 description 1
- 108091089612 miR-352 stem-loop Proteins 0.000 description 1
- 108091088742 miR-3605 stem-loop Proteins 0.000 description 1
- 108091068706 miR-3606 stem-loop Proteins 0.000 description 1
- 108091024557 miR-3609 stem-loop Proteins 0.000 description 1
- 108091038230 miR-3610 stem-loop Proteins 0.000 description 1
- 108091025394 miR-3611 stem-loop Proteins 0.000 description 1
- 108091043295 miR-3612 stem-loop Proteins 0.000 description 1
- 108091065175 miR-3613 stem-loop Proteins 0.000 description 1
- 108091049660 miR-3614 stem-loop Proteins 0.000 description 1
- 108091057452 miR-3615 stem-loop Proteins 0.000 description 1
- 108091059198 miR-3616 stem-loop Proteins 0.000 description 1
- 108091023111 miR-3617 stem-loop Proteins 0.000 description 1
- 108091044002 miR-3618 stem-loop Proteins 0.000 description 1
- 108091070943 miR-3619 stem-loop Proteins 0.000 description 1
- 108091082685 miR-3620 stem-loop Proteins 0.000 description 1
- 108091047629 miR-3621 stem-loop Proteins 0.000 description 1
- 108091049175 miR-3622a stem-loop Proteins 0.000 description 1
- 108091058195 miR-3622b stem-loop Proteins 0.000 description 1
- 108091083268 miR-3646 stem-loop Proteins 0.000 description 1
- 108091074917 miR-3648-1 stem-loop Proteins 0.000 description 1
- 108091068682 miR-3648-2 stem-loop Proteins 0.000 description 1
- 108091055773 miR-3649 stem-loop Proteins 0.000 description 1
- 108091030670 miR-365 stem-loop Proteins 0.000 description 1
- 108091043604 miR-365-1 stem-loop Proteins 0.000 description 1
- 108091076262 miR-365-2 stem-loop Proteins 0.000 description 1
- 108091036688 miR-365-3 stem-loop Proteins 0.000 description 1
- 108091051358 miR-3650 stem-loop Proteins 0.000 description 1
- 108091032603 miR-3651 stem-loop Proteins 0.000 description 1
- 108091081482 miR-3652 stem-loop Proteins 0.000 description 1
- 108091086569 miR-3654 stem-loop Proteins 0.000 description 1
- 108091069694 miR-3655 stem-loop Proteins 0.000 description 1
- 108091064236 miR-3658 stem-loop Proteins 0.000 description 1
- 108091029955 miR-3659 stem-loop Proteins 0.000 description 1
- 108091031325 miR-3660 stem-loop Proteins 0.000 description 1
- 108091025139 miR-3661 stem-loop Proteins 0.000 description 1
- 108091089360 miR-3662 stem-loop Proteins 0.000 description 1
- 108091033645 miR-3664 stem-loop Proteins 0.000 description 1
- 108091031765 miR-3665 stem-loop Proteins 0.000 description 1
- 108091061759 miR-3666 stem-loop Proteins 0.000 description 1
- 108091047115 miR-3667 stem-loop Proteins 0.000 description 1
- 108091041629 miR-3668 stem-loop Proteins 0.000 description 1
- 108091055411 miR-3670-1 stem-loop Proteins 0.000 description 1
- 108091039169 miR-3670-2 stem-loop Proteins 0.000 description 1
- 108091024959 miR-3670-3 stem-loop Proteins 0.000 description 1
- 108091045722 miR-3670-4 stem-loop Proteins 0.000 description 1
- 108091049021 miR-3677 stem-loop Proteins 0.000 description 1
- 108091031631 miR-3678 stem-loop Proteins 0.000 description 1
- 108091063862 miR-3679 stem-loop Proteins 0.000 description 1
- 108091064231 miR-3681 stem-loop Proteins 0.000 description 1
- 108091030599 miR-3682 stem-loop Proteins 0.000 description 1
- 108091080131 miR-3683 stem-loop Proteins 0.000 description 1
- 108091065283 miR-3684 stem-loop Proteins 0.000 description 1
- 108091055930 miR-3685 stem-loop Proteins 0.000 description 1
- 108091057236 miR-3686 stem-loop Proteins 0.000 description 1
- 108091041028 miR-3688 stem-loop Proteins 0.000 description 1
- 108091056171 miR-3689a stem-loop Proteins 0.000 description 1
- 108091023723 miR-3689b stem-loop Proteins 0.000 description 1
- 108091063076 miR-3690-1 stem-loop Proteins 0.000 description 1
- 108091056452 miR-3690-2 stem-loop Proteins 0.000 description 1
- 108091041626 miR-3691 stem-loop Proteins 0.000 description 1
- 108091069294 miR-3692 stem-loop Proteins 0.000 description 1
- 108091031054 miR-37 stem-loop Proteins 0.000 description 1
- 108091086855 miR-3713 stem-loop Proteins 0.000 description 1
- 108091086250 miR-3714 stem-loop Proteins 0.000 description 1
- 108091062109 miR-372 stem-loop Proteins 0.000 description 1
- 108091043953 miR-373 stem-loop Proteins 0.000 description 1
- 108091041286 miR-374 stem-loop Proteins 0.000 description 1
- 108091047175 miR-374a stem-loop Proteins 0.000 description 1
- 108091048101 miR-374b stem-loop Proteins 0.000 description 1
- 108091056144 miR-374c stem-loop Proteins 0.000 description 1
- 108091087125 miR-376a stem-loop Proteins 0.000 description 1
- 108091080263 miR-376a-1 stem-loop Proteins 0.000 description 1
- 108091089006 miR-376a-2 stem-loop Proteins 0.000 description 1
- 108091073138 miR-376a-3 stem-loop Proteins 0.000 description 1
- 108091071616 miR-376c stem-loop Proteins 0.000 description 1
- 108091092015 miR-378b stem-loop Proteins 0.000 description 1
- 108091050584 miR-378c stem-loop Proteins 0.000 description 1
- 108091041657 miR-381 stem-loop Proteins 0.000 description 1
- 108091048468 miR-383 stem-loop Proteins 0.000 description 1
- 108091062184 miR-3907 stem-loop Proteins 0.000 description 1
- 108091082912 miR-3908 stem-loop Proteins 0.000 description 1
- 108091083505 miR-3909 stem-loop Proteins 0.000 description 1
- 108091064838 miR-3910-1 stem-loop Proteins 0.000 description 1
- 108091034077 miR-3910-2 stem-loop Proteins 0.000 description 1
- 108091044258 miR-3911 stem-loop Proteins 0.000 description 1
- 108091076258 miR-3912 stem-loop Proteins 0.000 description 1
- 108091038081 miR-3913-1 stem-loop Proteins 0.000 description 1
- 108091026052 miR-3913-2 stem-loop Proteins 0.000 description 1
- 108091070452 miR-3914-1 stem-loop Proteins 0.000 description 1
- 108091053798 miR-3914-2 stem-loop Proteins 0.000 description 1
- 108091029229 miR-3915 stem-loop Proteins 0.000 description 1
- 108091043146 miR-3916 stem-loop Proteins 0.000 description 1
- 108091089984 miR-3917 stem-loop Proteins 0.000 description 1
- 108091071608 miR-3918 stem-loop Proteins 0.000 description 1
- 108091057329 miR-3919 stem-loop Proteins 0.000 description 1
- 108091073269 miR-3920 stem-loop Proteins 0.000 description 1
- 108091040938 miR-3921 stem-loop Proteins 0.000 description 1
- 108091028354 miR-3922 stem-loop Proteins 0.000 description 1
- 108091030860 miR-3923 stem-loop Proteins 0.000 description 1
- 108091087798 miR-3924 stem-loop Proteins 0.000 description 1
- 108091050719 miR-3925 stem-loop Proteins 0.000 description 1
- 108091065419 miR-3926-1 stem-loop Proteins 0.000 description 1
- 108091053922 miR-3926-2 stem-loop Proteins 0.000 description 1
- 108091032008 miR-3927 stem-loop Proteins 0.000 description 1
- 108091023797 miR-3928 stem-loop Proteins 0.000 description 1
- 108091043415 miR-3929 stem-loop Proteins 0.000 description 1
- 108091057549 miR-3934 stem-loop Proteins 0.000 description 1
- 108091055157 miR-3935 stem-loop Proteins 0.000 description 1
- 108091025823 miR-3936 stem-loop Proteins 0.000 description 1
- 108091044896 miR-3937 stem-loop Proteins 0.000 description 1
- 108091045030 miR-3938 stem-loop Proteins 0.000 description 1
- 108091062526 miR-3939 stem-loop Proteins 0.000 description 1
- 108091067045 miR-3940 stem-loop Proteins 0.000 description 1
- 108091068702 miR-3941 stem-loop Proteins 0.000 description 1
- 108091056794 miR-3942 stem-loop Proteins 0.000 description 1
- 108091092877 miR-3943 stem-loop Proteins 0.000 description 1
- 108091059492 miR-3944 stem-loop Proteins 0.000 description 1
- 108091046695 miR-3945 stem-loop Proteins 0.000 description 1
- 108091043221 miR-412 stem-loop Proteins 0.000 description 1
- 108091044721 miR-422a stem-loop Proteins 0.000 description 1
- 108091030938 miR-424 stem-loop Proteins 0.000 description 1
- 108091024000 miR-4251 stem-loop Proteins 0.000 description 1
- 108091042975 miR-4252 stem-loop Proteins 0.000 description 1
- 108091077948 miR-4253 stem-loop Proteins 0.000 description 1
- 108091037581 miR-4254 stem-loop Proteins 0.000 description 1
- 108091032607 miR-4255 stem-loop Proteins 0.000 description 1
- 108091059806 miR-4256 stem-loop Proteins 0.000 description 1
- 108091083981 miR-4257 stem-loop Proteins 0.000 description 1
- 108091087307 miR-4258 stem-loop Proteins 0.000 description 1
- 108091069792 miR-4259 stem-loop Proteins 0.000 description 1
- 108091083430 miR-4260 stem-loop Proteins 0.000 description 1
- 108091047611 miR-4261 stem-loop Proteins 0.000 description 1
- 108091039988 miR-4262 stem-loop Proteins 0.000 description 1
- 108091027989 miR-4263 stem-loop Proteins 0.000 description 1
- 108091032417 miR-4264 stem-loop Proteins 0.000 description 1
- 108091091088 miR-4265 stem-loop Proteins 0.000 description 1
- 108091063235 miR-4266 stem-loop Proteins 0.000 description 1
- 108091073485 miR-4267 stem-loop Proteins 0.000 description 1
- 108091056965 miR-4268 stem-loop Proteins 0.000 description 1
- 108091058683 miR-4269 stem-loop Proteins 0.000 description 1
- 108091040106 miR-4270 stem-loop Proteins 0.000 description 1
- 108091029740 miR-4271 stem-loop Proteins 0.000 description 1
- 108091030480 miR-4272 stem-loop Proteins 0.000 description 1
- 108091083041 miR-4273 stem-loop Proteins 0.000 description 1
- 108091047366 miR-4274 stem-loop Proteins 0.000 description 1
- 108091057333 miR-4275 stem-loop Proteins 0.000 description 1
- 108091055855 miR-4276 stem-loop Proteins 0.000 description 1
- 108091023472 miR-4277 stem-loop Proteins 0.000 description 1
- 108091090850 miR-4278 stem-loop Proteins 0.000 description 1
- 108091046306 miR-4279 stem-loop Proteins 0.000 description 1
- 108091085616 miR-4280 stem-loop Proteins 0.000 description 1
- 108091090476 miR-4281 stem-loop Proteins 0.000 description 1
- 108091070710 miR-4282 stem-loop Proteins 0.000 description 1
- 108091063357 miR-4283-1 stem-loop Proteins 0.000 description 1
- 108091055518 miR-4283-2 stem-loop Proteins 0.000 description 1
- 108091054004 miR-4284 stem-loop Proteins 0.000 description 1
- 108091023365 miR-4285 stem-loop Proteins 0.000 description 1
- 108091045749 miR-4286 stem-loop Proteins 0.000 description 1
- 108091042852 miR-4286-1 stem-loop Proteins 0.000 description 1
- 108091026483 miR-4286-2 stem-loop Proteins 0.000 description 1
- 108091047633 miR-4287 stem-loop Proteins 0.000 description 1
- 108091051925 miR-4288 stem-loop Proteins 0.000 description 1
- 108091036596 miR-4289 stem-loop Proteins 0.000 description 1
- 108091074482 miR-4290 stem-loop Proteins 0.000 description 1
- 108091035179 miR-4291 stem-loop Proteins 0.000 description 1
- 108091059247 miR-4292 stem-loop Proteins 0.000 description 1
- 108091064083 miR-4293 stem-loop Proteins 0.000 description 1
- 108091044119 miR-4294 stem-loop Proteins 0.000 description 1
- 108091069786 miR-4295 stem-loop Proteins 0.000 description 1
- 108091042706 miR-4296 stem-loop Proteins 0.000 description 1
- 108091053586 miR-4297 stem-loop Proteins 0.000 description 1
- 108091029967 miR-4298 stem-loop Proteins 0.000 description 1
- 108091089445 miR-4299 stem-loop Proteins 0.000 description 1
- 108091054300 miR-4300 stem-loop Proteins 0.000 description 1
- 108091052745 miR-4301 stem-loop Proteins 0.000 description 1
- 108091026047 miR-4302 stem-loop Proteins 0.000 description 1
- 108091088869 miR-4303 stem-loop Proteins 0.000 description 1
- 108091044582 miR-4304 stem-loop Proteins 0.000 description 1
- 108091049131 miR-4305 stem-loop Proteins 0.000 description 1
- 108091040351 miR-4306 stem-loop Proteins 0.000 description 1
- 108091036125 miR-4307 stem-loop Proteins 0.000 description 1
- 108091031460 miR-4308 stem-loop Proteins 0.000 description 1
- 108091041869 miR-4309 stem-loop Proteins 0.000 description 1
- 108091059259 miR-4310 stem-loop Proteins 0.000 description 1
- 108091086208 miR-4311 stem-loop Proteins 0.000 description 1
- 108091078123 miR-4312 stem-loop Proteins 0.000 description 1
- 108091071655 miR-4313 stem-loop Proteins 0.000 description 1
- 108091052976 miR-4314 stem-loop Proteins 0.000 description 1
- 108091039849 miR-4315-1 stem-loop Proteins 0.000 description 1
- 108091036436 miR-4315-2 stem-loop Proteins 0.000 description 1
- 108091029838 miR-4316 stem-loop Proteins 0.000 description 1
- 108091087061 miR-4317 stem-loop Proteins 0.000 description 1
- 108091049130 miR-4318 stem-loop Proteins 0.000 description 1
- 108091047337 miR-4319 stem-loop Proteins 0.000 description 1
- 108091029445 miR-432 stem-loop Proteins 0.000 description 1
- 108091069252 miR-4320 stem-loop Proteins 0.000 description 1
- 108091037903 miR-4321 stem-loop Proteins 0.000 description 1
- 108091035337 miR-4322 stem-loop Proteins 0.000 description 1
- 108091031316 miR-4323 stem-loop Proteins 0.000 description 1
- 108091086430 miR-4324 stem-loop Proteins 0.000 description 1
- 108091089409 miR-4325 stem-loop Proteins 0.000 description 1
- 108091074132 miR-4326 stem-loop Proteins 0.000 description 1
- 108091057166 miR-4327 stem-loop Proteins 0.000 description 1
- 108091053719 miR-4328 stem-loop Proteins 0.000 description 1
- 108091029446 miR-4330 stem-loop Proteins 0.000 description 1
- 108091048162 miR-434 stem-loop Proteins 0.000 description 1
- 108091062761 miR-448 stem-loop Proteins 0.000 description 1
- 108091056281 miR-449b stem-loop Proteins 0.000 description 1
- 108091059726 miR-449c stem-loop Proteins 0.000 description 1
- 108091036674 miR-450a stem-loop Proteins 0.000 description 1
- 108091091563 miR-450a-1 stem-loop Proteins 0.000 description 1
- 108091071218 miR-450a-2 stem-loop Proteins 0.000 description 1
- 108091027743 miR-450b stem-loop Proteins 0.000 description 1
- 108091059056 miR-452 stem-loop Proteins 0.000 description 1
- 108091044133 miR-454 stem-loop Proteins 0.000 description 1
- 108091051576 miR-463 stem-loop Proteins 0.000 description 1
- 108091031557 miR-465 stem-loop Proteins 0.000 description 1
- 108091061720 miR-466 stem-loop Proteins 0.000 description 1
- 108091061791 miR-466b stem-loop Proteins 0.000 description 1
- 108091046904 miR-466c stem-loop Proteins 0.000 description 1
- 108091076178 miR-48 stem-loop Proteins 0.000 description 1
- 108091084599 miR-48-1 stem-loop Proteins 0.000 description 1
- 108091074333 miR-48-2 stem-loop Proteins 0.000 description 1
- 108091035982 miR-485 stem-loop Proteins 0.000 description 1
- 108091052738 miR-486-1 stem-loop Proteins 0.000 description 1
- 108091030654 miR-486-2 stem-loop Proteins 0.000 description 1
- 108091058273 miR-487a stem-loop Proteins 0.000 description 1
- 108091069917 miR-491 stem-loop Proteins 0.000 description 1
- 108091068970 miR-492 stem-loop Proteins 0.000 description 1
- 108091080238 miR-492-1 stem-loop Proteins 0.000 description 1
- 108091076986 miR-492-2 stem-loop Proteins 0.000 description 1
- 108091053306 miR-493 stem-loop Proteins 0.000 description 1
- 108091092564 miR-494 stem-loop Proteins 0.000 description 1
- 108091049470 miR-498 stem-loop Proteins 0.000 description 1
- 108091087529 miR-500 stem-loop Proteins 0.000 description 1
- 108091036496 miR-500-2 stem-loop Proteins 0.000 description 1
- 108091043157 miR-500a stem-loop Proteins 0.000 description 1
- 108091050398 miR-500b stem-loop Proteins 0.000 description 1
- 108091058133 miR-502 stem-loop Proteins 0.000 description 1
- 108091041309 miR-505 stem-loop Proteins 0.000 description 1
- 108091090896 miR-506 stem-loop Proteins 0.000 description 1
- 108091069048 miR-507 stem-loop Proteins 0.000 description 1
- 108091037417 miR-508 stem-loop Proteins 0.000 description 1
- 108091091880 miR-509-1 stem-loop Proteins 0.000 description 1
- 108091051359 miR-509-2 stem-loop Proteins 0.000 description 1
- 108091038238 miR-509-3 stem-loop Proteins 0.000 description 1
- 108091053329 miR-510 stem-loop Proteins 0.000 description 1
- 108091053449 miR-511 stem-loop Proteins 0.000 description 1
- 108091077010 miR-512-1 stem-loop Proteins 0.000 description 1
- 108091032013 miR-512-2 stem-loop Proteins 0.000 description 1
- 108091032623 miR-513a-1 stem-loop Proteins 0.000 description 1
- 108091080923 miR-513a-2 stem-loop Proteins 0.000 description 1
- 108091050453 miR-513b stem-loop Proteins 0.000 description 1
- 108091047492 miR-513b-1 stem-loop Proteins 0.000 description 1
- 108091048017 miR-513b-2 stem-loop Proteins 0.000 description 1
- 108091033398 miR-513c stem-loop Proteins 0.000 description 1
- 108091046680 miR-513c-1 stem-loop Proteins 0.000 description 1
- 108091087486 miR-513c-2 stem-loop Proteins 0.000 description 1
- 108091031465 miR-514-1 stem-loop Proteins 0.000 description 1
- 108091086203 miR-514-2 stem-loop Proteins 0.000 description 1
- 108091086843 miR-514-3 stem-loop Proteins 0.000 description 1
- 108091028375 miR-514b stem-loop Proteins 0.000 description 1
- 108091057485 miR-515-1 stem-loop Proteins 0.000 description 1
- 108091029052 miR-515-2 stem-loop Proteins 0.000 description 1
- 108091026498 miR-516a-1 stem-loop Proteins 0.000 description 1
- 108091086918 miR-516a-2 stem-loop Proteins 0.000 description 1
- 108091084940 miR-516a-3 stem-loop Proteins 0.000 description 1
- 108091056518 miR-516b-1 stem-loop Proteins 0.000 description 1
- 108091092864 miR-516b-2 stem-loop Proteins 0.000 description 1
- 108091032275 miR-517a stem-loop Proteins 0.000 description 1
- 108091086245 miR-517b stem-loop Proteins 0.000 description 1
- 108091043460 miR-517b-1 stem-loop Proteins 0.000 description 1
- 108091044088 miR-517b-2 stem-loop Proteins 0.000 description 1
- 108091089950 miR-517c stem-loop Proteins 0.000 description 1
- 108091040524 miR-518a-1 stem-loop Proteins 0.000 description 1
- 108091028747 miR-518a-2 stem-loop Proteins 0.000 description 1
- 108091077027 miR-518b stem-loop Proteins 0.000 description 1
- 108091065658 miR-518b-1 stem-loop Proteins 0.000 description 1
- 108091053758 miR-518b-2 stem-loop Proteins 0.000 description 1
- 108091028859 miR-518b-3 stem-loop Proteins 0.000 description 1
- 108091083761 miR-518b-4 stem-loop Proteins 0.000 description 1
- 108091035240 miR-518c stem-loop Proteins 0.000 description 1
- 108091024531 miR-518d stem-loop Proteins 0.000 description 1
- 108091062441 miR-518e stem-loop Proteins 0.000 description 1
- 108091044445 miR-518f stem-loop Proteins 0.000 description 1
- 108091036604 miR-518f-1 stem-loop Proteins 0.000 description 1
- 108091080798 miR-518f-2 stem-loop Proteins 0.000 description 1
- 108091044743 miR-519a-1 stem-loop Proteins 0.000 description 1
- 108091067493 miR-519a-2 stem-loop Proteins 0.000 description 1
- 108091025836 miR-519b stem-loop Proteins 0.000 description 1
- 108091091625 miR-519c stem-loop Proteins 0.000 description 1
- 108091026826 miR-519c-2 stem-loop Proteins 0.000 description 1
- 108091041877 miR-519d stem-loop Proteins 0.000 description 1
- 108091050363 miR-519e stem-loop Proteins 0.000 description 1
- 108091060330 miR-520a stem-loop Proteins 0.000 description 1
- 108091024613 miR-520b stem-loop Proteins 0.000 description 1
- 108091086222 miR-520c stem-loop Proteins 0.000 description 1
- 108091073864 miR-520d stem-loop Proteins 0.000 description 1
- 108091070778 miR-520e stem-loop Proteins 0.000 description 1
- 108091052995 miR-520f stem-loop Proteins 0.000 description 1
- 108091025782 miR-520g stem-loop Proteins 0.000 description 1
- 108091029517 miR-520h stem-loop Proteins 0.000 description 1
- 108091043430 miR-521-1 stem-loop Proteins 0.000 description 1
- 108091044256 miR-521-2 stem-loop Proteins 0.000 description 1
- 108091050172 miR-522 stem-loop Proteins 0.000 description 1
- 108091051536 miR-523 stem-loop Proteins 0.000 description 1
- 108091042838 miR-524 stem-loop Proteins 0.000 description 1
- 108091027598 miR-525 stem-loop Proteins 0.000 description 1
- 108091056507 miR-526a-1 stem-loop Proteins 0.000 description 1
- 108091029129 miR-526a-2 stem-loop Proteins 0.000 description 1
- 108091063385 miR-526b stem-loop Proteins 0.000 description 1
- 108091086170 miR-527 stem-loop Proteins 0.000 description 1
- 108091028769 miR-540 stem-loop Proteins 0.000 description 1
- 108091023526 miR-541 stem-loop Proteins 0.000 description 1
- 108091045812 miR-544b stem-loop Proteins 0.000 description 1
- 108091076617 miR-544b-1 stem-loop Proteins 0.000 description 1
- 108091050743 miR-544b-2 stem-loop Proteins 0.000 description 1
- 108091040490 miR-545 stem-loop Proteins 0.000 description 1
- 108091069948 miR-548a-1 stem-loop Proteins 0.000 description 1
- 108091073707 miR-548a-2 stem-loop Proteins 0.000 description 1
- 108091034066 miR-548a-3 stem-loop Proteins 0.000 description 1
- 108091034304 miR-548b stem-loop Proteins 0.000 description 1
- 108091029118 miR-548c stem-loop Proteins 0.000 description 1
- 108091092453 miR-548c-1 stem-loop Proteins 0.000 description 1
- 108091030775 miR-548c-2 stem-loop Proteins 0.000 description 1
- 108091033339 miR-548d-1 stem-loop Proteins 0.000 description 1
- 108091081018 miR-548d-2 stem-loop Proteins 0.000 description 1
- 108091068733 miR-548e stem-loop Proteins 0.000 description 1
- 108091089725 miR-548f-1 stem-loop Proteins 0.000 description 1
- 108091074082 miR-548f-2 stem-loop Proteins 0.000 description 1
- 108091074112 miR-548f-3 stem-loop Proteins 0.000 description 1
- 108091052977 miR-548f-4 stem-loop Proteins 0.000 description 1
- 108091025312 miR-548f-5 stem-loop Proteins 0.000 description 1
- 108091054834 miR-548g stem-loop Proteins 0.000 description 1
- 108091037006 miR-548h-1 stem-loop Proteins 0.000 description 1
- 108091028705 miR-548h-2 stem-loop Proteins 0.000 description 1
- 108091024683 miR-548h-3 stem-loop Proteins 0.000 description 1
- 108091041881 miR-548h-4 stem-loop Proteins 0.000 description 1
- 108091063105 miR-548i-1 stem-loop Proteins 0.000 description 1
- 108091052473 miR-548i-2 stem-loop Proteins 0.000 description 1
- 108091058402 miR-548i-3 stem-loop Proteins 0.000 description 1
- 108091028345 miR-548i-4 stem-loop Proteins 0.000 description 1
- 108091080668 miR-548j stem-loop Proteins 0.000 description 1
- 108091049014 miR-548k stem-loop Proteins 0.000 description 1
- 108091051371 miR-548l stem-loop Proteins 0.000 description 1
- 108091056908 miR-548m stem-loop Proteins 0.000 description 1
- 108091024672 miR-548n stem-loop Proteins 0.000 description 1
- 108091085840 miR-548n-1 stem-loop Proteins 0.000 description 1
- 108091063358 miR-548n-2 stem-loop Proteins 0.000 description 1
- 108091089002 miR-548o stem-loop Proteins 0.000 description 1
- 108091059788 miR-548o-1 stem-loop Proteins 0.000 description 1
- 108091086597 miR-548p stem-loop Proteins 0.000 description 1
- 108091040004 miR-548s stem-loop Proteins 0.000 description 1
- 108091029588 miR-548t stem-loop Proteins 0.000 description 1
- 108091045674 miR-548u stem-loop Proteins 0.000 description 1
- 108091092026 miR-548v stem-loop Proteins 0.000 description 1
- 108091067295 miR-548w stem-loop Proteins 0.000 description 1
- 108091058019 miR-548x stem-loop Proteins 0.000 description 1
- 108091064086 miR-548y stem-loop Proteins 0.000 description 1
- 108091049952 miR-548y-1 stem-loop Proteins 0.000 description 1
- 108091058297 miR-548y-2 stem-loop Proteins 0.000 description 1
- 108091063900 miR-549-1 stem-loop Proteins 0.000 description 1
- 108091046291 miR-549-2 stem-loop Proteins 0.000 description 1
- 108091052902 miR-550a-1 stem-loop Proteins 0.000 description 1
- 108091024065 miR-550a-2 stem-loop Proteins 0.000 description 1
- 108091069221 miR-550b-1 stem-loop Proteins 0.000 description 1
- 108091039385 miR-550b-2 stem-loop Proteins 0.000 description 1
- 108091049915 miR-551a stem-loop Proteins 0.000 description 1
- 108091057017 miR-551b stem-loop Proteins 0.000 description 1
- 108091032786 miR-552 stem-loop Proteins 0.000 description 1
- 108091038400 miR-553 stem-loop Proteins 0.000 description 1
- 108091030941 miR-554 stem-loop Proteins 0.000 description 1
- 108091043167 miR-555 stem-loop Proteins 0.000 description 1
- 108091063333 miR-556 stem-loop Proteins 0.000 description 1
- 108091071062 miR-557 stem-loop Proteins 0.000 description 1
- 108091057720 miR-558 stem-loop Proteins 0.000 description 1
- 108091061974 miR-559 stem-loop Proteins 0.000 description 1
- 108091028291 miR-561 stem-loop Proteins 0.000 description 1
- 108091029205 miR-562 stem-loop Proteins 0.000 description 1
- 108091084213 miR-563 stem-loop Proteins 0.000 description 1
- 108091047658 miR-564 stem-loop Proteins 0.000 description 1
- 108091092520 miR-567 stem-loop Proteins 0.000 description 1
- 108091043228 miR-568 stem-loop Proteins 0.000 description 1
- 108091046865 miR-569 stem-loop Proteins 0.000 description 1
- 108091084017 miR-570 stem-loop Proteins 0.000 description 1
- 108091086764 miR-571 stem-loop Proteins 0.000 description 1
- 108091075061 miR-571-1 stem-loop Proteins 0.000 description 1
- 108091051054 miR-571-2 stem-loop Proteins 0.000 description 1
- 108091071822 miR-572 stem-loop Proteins 0.000 description 1
- 108091058526 miR-573 stem-loop Proteins 0.000 description 1
- 108091055140 miR-574 stem-loop Proteins 0.000 description 1
- 108091058485 miR-574-1 stem-loop Proteins 0.000 description 1
- 108091055064 miR-574-2 stem-loop Proteins 0.000 description 1
- 108091023843 miR-575 stem-loop Proteins 0.000 description 1
- 108091045118 miR-576 stem-loop Proteins 0.000 description 1
- 108091048173 miR-577 stem-loop Proteins 0.000 description 1
- 108091047998 miR-578 stem-loop Proteins 0.000 description 1
- 108091033741 miR-579 stem-loop Proteins 0.000 description 1
- 108091073657 miR-580 stem-loop Proteins 0.000 description 1
- 108091056770 miR-581 stem-loop Proteins 0.000 description 1
- 108091059757 miR-582 stem-loop Proteins 0.000 description 1
- 108091059494 miR-583 stem-loop Proteins 0.000 description 1
- 108091046601 miR-584 stem-loop Proteins 0.000 description 1
- 108091041487 miR-584-1 stem-loop Proteins 0.000 description 1
- 108091091335 miR-584-2 stem-loop Proteins 0.000 description 1
- 108091072614 miR-584-3 stem-loop Proteins 0.000 description 1
- 108091042285 miR-584-4 stem-loop Proteins 0.000 description 1
- 108091035690 miR-584-5 stem-loop Proteins 0.000 description 1
- 108091059440 miR-584-6 stem-loop Proteins 0.000 description 1
- 108091082246 miR-584-7 stem-loop Proteins 0.000 description 1
- 108091088867 miR-584-8 stem-loop Proteins 0.000 description 1
- 108091072773 miR-585 stem-loop Proteins 0.000 description 1
- 108091064931 miR-586 stem-loop Proteins 0.000 description 1
- 108091052481 miR-587 stem-loop Proteins 0.000 description 1
- 108091030944 miR-588 stem-loop Proteins 0.000 description 1
- 108091088512 miR-589 stem-loop Proteins 0.000 description 1
- 108091024411 miR-590 stem-loop Proteins 0.000 description 1
- 108091043161 miR-591 stem-loop Proteins 0.000 description 1
- 108091091687 miR-592 stem-loop Proteins 0.000 description 1
- 108091050647 miR-593 stem-loop Proteins 0.000 description 1
- 108091031990 miR-595 stem-loop Proteins 0.000 description 1
- 108091059192 miR-596 stem-loop Proteins 0.000 description 1
- 108091089359 miR-597 stem-loop Proteins 0.000 description 1
- 108091076246 miR-599 stem-loop Proteins 0.000 description 1
- 108091023451 miR-600 stem-loop Proteins 0.000 description 1
- 108091079765 miR-601 stem-loop Proteins 0.000 description 1
- 108091075997 miR-602 stem-loop Proteins 0.000 description 1
- 108091073318 miR-603 stem-loop Proteins 0.000 description 1
- 108091040857 miR-604 stem-loop Proteins 0.000 description 1
- 108091025230 miR-605 stem-loop Proteins 0.000 description 1
- 108091031030 miR-606 stem-loop Proteins 0.000 description 1
- 108091088135 miR-607 stem-loop Proteins 0.000 description 1
- 108091051628 miR-608 stem-loop Proteins 0.000 description 1
- 108091047885 miR-609 stem-loop Proteins 0.000 description 1
- 108091065430 miR-610 stem-loop Proteins 0.000 description 1
- 108091070410 miR-611 stem-loop Proteins 0.000 description 1
- 108091038867 miR-612 stem-loop Proteins 0.000 description 1
- 108091030661 miR-613 stem-loop Proteins 0.000 description 1
- 108091080596 miR-614 stem-loop Proteins 0.000 description 1
- 108091090051 miR-615 stem-loop Proteins 0.000 description 1
- 108091049934 miR-616 stem-loop Proteins 0.000 description 1
- 108091057639 miR-617 stem-loop Proteins 0.000 description 1
- 108091054673 miR-618 stem-loop Proteins 0.000 description 1
- 108091026050 miR-619 stem-loop Proteins 0.000 description 1
- 108091033192 miR-620 stem-loop Proteins 0.000 description 1
- 108091028939 miR-621 stem-loop Proteins 0.000 description 1
- 108091041759 miR-622 stem-loop Proteins 0.000 description 1
- 108091066725 miR-623 stem-loop Proteins 0.000 description 1
- 108091069023 miR-624 stem-loop Proteins 0.000 description 1
- 108091056454 miR-625 stem-loop Proteins 0.000 description 1
- 108091092842 miR-626 stem-loop Proteins 0.000 description 1
- 108091062477 miR-627 stem-loop Proteins 0.000 description 1
- 108091046673 miR-628 stem-loop Proteins 0.000 description 1
- 108091049896 miR-629 stem-loop Proteins 0.000 description 1
- 108091086737 miR-630 stem-loop Proteins 0.000 description 1
- 108091048795 miR-631 stem-loop Proteins 0.000 description 1
- 108091069770 miR-631-1 stem-loop Proteins 0.000 description 1
- 108091042247 miR-631-2 stem-loop Proteins 0.000 description 1
- 108091036346 miR-631-3 stem-loop Proteins 0.000 description 1
- 108091079010 miR-632 stem-loop Proteins 0.000 description 1
- 108091036526 miR-633 stem-loop Proteins 0.000 description 1
- 108091024127 miR-634 stem-loop Proteins 0.000 description 1
- 108091044560 miR-635 stem-loop Proteins 0.000 description 1
- 108091091900 miR-636 stem-loop Proteins 0.000 description 1
- 108091069529 miR-637 stem-loop Proteins 0.000 description 1
- 108091038240 miR-638 stem-loop Proteins 0.000 description 1
- 108091056064 miR-639 stem-loop Proteins 0.000 description 1
- 108091057009 miR-640 stem-loop Proteins 0.000 description 1
- 108091053731 miR-641 stem-loop Proteins 0.000 description 1
- 108091056346 miR-642a stem-loop Proteins 0.000 description 1
- 108091037880 miR-642b stem-loop Proteins 0.000 description 1
- 108091044471 miR-643 stem-loop Proteins 0.000 description 1
- 108091049434 miR-644 stem-loop Proteins 0.000 description 1
- 108091063323 miR-645 stem-loop Proteins 0.000 description 1
- 108091035662 miR-646 stem-loop Proteins 0.000 description 1
- 108091030787 miR-647 stem-loop Proteins 0.000 description 1
- 108091064383 miR-648 stem-loop Proteins 0.000 description 1
- 108091092048 miR-649 stem-loop Proteins 0.000 description 1
- 108091063911 miR-650 stem-loop Proteins 0.000 description 1
- 108091046103 miR-651 stem-loop Proteins 0.000 description 1
- 108091052964 miR-654 stem-loop Proteins 0.000 description 1
- 108091026036 miR-655 stem-loop Proteins 0.000 description 1
- 108091089548 miR-656 stem-loop Proteins 0.000 description 1
- 108091086086 miR-657 stem-loop Proteins 0.000 description 1
- 108091079544 miR-658 stem-loop Proteins 0.000 description 1
- 108091081208 miR-659 stem-loop Proteins 0.000 description 1
- 108091063151 miR-660 stem-loop Proteins 0.000 description 1
- 108091055076 miR-661 stem-loop Proteins 0.000 description 1
- 108091057613 miR-662 stem-loop Proteins 0.000 description 1
- 108091041014 miR-663-1 stem-loop Proteins 0.000 description 1
- 108091050799 miR-663-10 stem-loop Proteins 0.000 description 1
- 108091038412 miR-663-11 stem-loop Proteins 0.000 description 1
- 108091062556 miR-663-2 stem-loop Proteins 0.000 description 1
- 108091036836 miR-663-3 stem-loop Proteins 0.000 description 1
- 108091056984 miR-663-4 stem-loop Proteins 0.000 description 1
- 108091064096 miR-663-5 stem-loop Proteins 0.000 description 1
- 108091089306 miR-663-6 stem-loop Proteins 0.000 description 1
- 108091074126 miR-663-7 stem-loop Proteins 0.000 description 1
- 108091074471 miR-663-8 stem-loop Proteins 0.000 description 1
- 108091053580 miR-663-9 stem-loop Proteins 0.000 description 1
- 108091056912 miR-663b stem-loop Proteins 0.000 description 1
- 108091074443 miR-665 stem-loop Proteins 0.000 description 1
- 108091039613 miR-667 stem-loop Proteins 0.000 description 1
- 108091025542 miR-668 stem-loop Proteins 0.000 description 1
- 108091090951 miR-67 stem-loop Proteins 0.000 description 1
- 108091030489 miR-670 stem-loop Proteins 0.000 description 1
- 108091041927 miR-672 stem-loop Proteins 0.000 description 1
- 108091078328 miR-673 stem-loop Proteins 0.000 description 1
- 108091040808 miR-675 stem-loop Proteins 0.000 description 1
- 108091061799 miR-676 stem-loop Proteins 0.000 description 1
- 108091037014 miR-7-1 stem-loop Proteins 0.000 description 1
- 108091030520 miR-7-2 stem-loop Proteins 0.000 description 1
- 108091025113 miR-7-3 stem-loop Proteins 0.000 description 1
- 108091072577 miR-718 stem-loop Proteins 0.000 description 1
- 108091038036 miR-742 stem-loop Proteins 0.000 description 1
- 108091064831 miR-743a stem-loop Proteins 0.000 description 1
- 108091084078 miR-744 stem-loop Proteins 0.000 description 1
- 108091049937 miR-759 stem-loop Proteins 0.000 description 1
- 108091076478 miR-762 stem-loop Proteins 0.000 description 1
- 108091066925 miR-762-1 stem-loop Proteins 0.000 description 1
- 108091069941 miR-762-2 stem-loop Proteins 0.000 description 1
- 108091042292 miR-762-3 stem-loop Proteins 0.000 description 1
- 108091043776 miR-765 stem-loop Proteins 0.000 description 1
- 108091045794 miR-766 stem-loop Proteins 0.000 description 1
- 108091066091 miR-767 stem-loop Proteins 0.000 description 1
- 108091056126 miR-769 stem-loop Proteins 0.000 description 1
- 108091029509 miR-802 stem-loop Proteins 0.000 description 1
- 108091039708 miR-872 stem-loop Proteins 0.000 description 1
- 108091049543 miR-876 stem-loop Proteins 0.000 description 1
- 108091054790 miR-878 stem-loop Proteins 0.000 description 1
- 108091053417 miR-885 stem-loop Proteins 0.000 description 1
- 108091092836 miR-887 stem-loop Proteins 0.000 description 1
- 108091046579 miR-888 stem-loop Proteins 0.000 description 1
- 108091049552 miR-889 stem-loop Proteins 0.000 description 1
- 108091088997 miR-890 stem-loop Proteins 0.000 description 1
- 108091050094 miR-891 stem-loop Proteins 0.000 description 1
- 108091023716 miR-891a stem-loop Proteins 0.000 description 1
- 108091060217 miR-891b stem-loop Proteins 0.000 description 1
- 108091056720 miR-891b-1 stem-loop Proteins 0.000 description 1
- 108091064151 miR-891b-2 stem-loop Proteins 0.000 description 1
- 108091076514 miR-892 stem-loop Proteins 0.000 description 1
- 108091043360 miR-892a stem-loop Proteins 0.000 description 1
- 108091062872 miR-892b stem-loop Proteins 0.000 description 1
- 108091089992 miR-9-1 stem-loop Proteins 0.000 description 1
- 108091071572 miR-9-2 stem-loop Proteins 0.000 description 1
- 108091076838 miR-9-3 stem-loop Proteins 0.000 description 1
- 108091092043 miR-920 stem-loop Proteins 0.000 description 1
- 108091090841 miR-921 stem-loop Proteins 0.000 description 1
- 108091069965 miR-922 stem-loop Proteins 0.000 description 1
- 108091033202 miR-924 stem-loop Proteins 0.000 description 1
- 108091028159 miR-92a-1 stem-loop Proteins 0.000 description 1
- 108091025616 miR-92a-2 stem-loop Proteins 0.000 description 1
- 108091030513 miR-933 stem-loop Proteins 0.000 description 1
- 108091091067 miR-934 stem-loop Proteins 0.000 description 1
- 108091078413 miR-936 stem-loop Proteins 0.000 description 1
- 108091036381 miR-937 stem-loop Proteins 0.000 description 1
- 108091024098 miR-938 stem-loop Proteins 0.000 description 1
- 108091062136 miR-939 stem-loop Proteins 0.000 description 1
- 108091030569 miR-940 stem-loop Proteins 0.000 description 1
- 108091073613 miR-941-1 stem-loop Proteins 0.000 description 1
- 108091039878 miR-941-2 stem-loop Proteins 0.000 description 1
- 108091035558 miR-941-3 stem-loop Proteins 0.000 description 1
- 108091031187 miR-941-4 stem-loop Proteins 0.000 description 1
- 108091047488 miR-943 stem-loop Proteins 0.000 description 1
- 108091057102 miR-944 stem-loop Proteins 0.000 description 1
- 108091023525 miR-95 stem-loop Proteins 0.000 description 1
- 108091007422 miR-98 Proteins 0.000 description 1
- 108091072565 miR-98 stem-loop Proteins 0.000 description 1
- 108091024443 miRa-135-1 stem-loop Proteins 0.000 description 1
- 238000001531 micro-dissection Methods 0.000 description 1
- 238000010208 microarray analysis Methods 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 239000011859 microparticle Substances 0.000 description 1
- 239000004005 microsphere Substances 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 235000010755 mineral Nutrition 0.000 description 1
- 239000011707 mineral Substances 0.000 description 1
- 239000011259 mixed solution Substances 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000007479 molecular analysis Methods 0.000 description 1
- 210000004877 mucosa Anatomy 0.000 description 1
- RTGDFNSFWBGLEC-SYZQJQIISA-N mycophenolate mofetil Chemical compound COC1=C(C)C=2COC(=O)C=2C(O)=C1C\C=C(/C)CCC(=O)OCCN1CCOCC1 RTGDFNSFWBGLEC-SYZQJQIISA-N 0.000 description 1
- 229960004866 mycophenolate mofetil Drugs 0.000 description 1
- PWDYHMBTPGXCSN-VCBMUGGBSA-N n,n'-bis[3,5-bis[(e)-n-(diaminomethylideneamino)-c-methylcarbonimidoyl]phenyl]decanediamide Chemical compound NC(N)=N/N=C(\C)C1=CC(C(=N/N=C(N)N)/C)=CC(NC(=O)CCCCCCCCC(=O)NC=2C=C(C=C(C=2)C(\C)=N\N=C(N)N)C(\C)=N\N=C(N)N)=C1 PWDYHMBTPGXCSN-VCBMUGGBSA-N 0.000 description 1
- FGGSNQOBRJVAKL-UHFFFAOYSA-N n-[4-(2,4-dimethyl-thiazol-5-yl)-pyrimidin-2-yl]-n',n'-dimethyl-benzene-1,4-diamine Chemical compound C1=CC(N(C)C)=CC=C1NC1=NC=CC(C2=C(N=C(C)S2)C)=N1 FGGSNQOBRJVAKL-UHFFFAOYSA-N 0.000 description 1
- 229950006780 n-acetylglucosamine Drugs 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 108010027531 nephrin Proteins 0.000 description 1
- 201000008026 nephroblastoma Diseases 0.000 description 1
- 231100000637 nephrotoxin Toxicity 0.000 description 1
- 238000006386 neutralization reaction Methods 0.000 description 1
- 229920001220 nitrocellulos Polymers 0.000 description 1
- QJGQUHMNIGDVPM-UHFFFAOYSA-N nitrogen group Chemical group [N] QJGQUHMNIGDVPM-UHFFFAOYSA-N 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 239000004745 nonwoven fabric Substances 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 239000002773 nucleotide Substances 0.000 description 1
- 125000003729 nucleotide group Chemical group 0.000 description 1
- 210000002747 omentum Anatomy 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- AHLBNYSZXLDEJQ-FWEHEUNISA-N orlistat Chemical compound CCCCCCCCCCC[C@H](OC(=O)[C@H](CC(C)C)NC=O)C[C@@H]1OC(=O)[C@H]1CCCCCC AHLBNYSZXLDEJQ-FWEHEUNISA-N 0.000 description 1
- 230000036407 pain Effects 0.000 description 1
- 229920002866 paraformaldehyde Polymers 0.000 description 1
- 238000005192 partition Methods 0.000 description 1
- 230000001575 pathological effect Effects 0.000 description 1
- 229960002340 pentostatin Drugs 0.000 description 1
- FPVKHBSQESCIEP-JQCXWYLXSA-N pentostatin Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(N=CNC[C@H]2O)=C2N=C1 FPVKHBSQESCIEP-JQCXWYLXSA-N 0.000 description 1
- 230000002572 peristaltic effect Effects 0.000 description 1
- 239000000546 pharmaceutical excipient Substances 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 229960003742 phenol Drugs 0.000 description 1
- 239000005011 phenolic resin Substances 0.000 description 1
- 229920001568 phenolic resin Polymers 0.000 description 1
- 229960003531 phenolsulfonphthalein Drugs 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000000704 physical effect Effects 0.000 description 1
- 239000006069 physical mixture Substances 0.000 description 1
- 229960005330 pimecrolimus Drugs 0.000 description 1
- KASDHRXLYQOAKZ-ZPSXYTITSA-N pimecrolimus Chemical compound C/C([C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@]2(O)O[C@@H]([C@H](C[C@H]2C)OC)[C@@H](OC)C[C@@H](C)C/C(C)=C/[C@H](C(C[C@H](O)[C@H]1C)=O)CC)=C\[C@@H]1CC[C@@H](Cl)[C@H](OC)C1 KASDHRXLYQOAKZ-ZPSXYTITSA-N 0.000 description 1
- 231100000614 poison Toxicity 0.000 description 1
- 229920001643 poly(ether ketone) Polymers 0.000 description 1
- 229920002492 poly(sulfone) Polymers 0.000 description 1
- 229920000058 polyacrylate Polymers 0.000 description 1
- 229920002239 polyacrylonitrile Polymers 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920002647 polyamide Polymers 0.000 description 1
- 229920002577 polybenzoxazole Polymers 0.000 description 1
- 239000004417 polycarbonate Substances 0.000 description 1
- 229920000515 polycarbonate Polymers 0.000 description 1
- 229920000728 polyester Polymers 0.000 description 1
- 229920006393 polyether sulfone Polymers 0.000 description 1
- 229920002530 polyetherether ketone Polymers 0.000 description 1
- 229920001223 polyethylene glycol Polymers 0.000 description 1
- 229920001721 polyimide Polymers 0.000 description 1
- 239000002861 polymer material Substances 0.000 description 1
- ODGAOXROABLFNM-UHFFFAOYSA-N polynoxylin Chemical compound O=C.NC(N)=O ODGAOXROABLFNM-UHFFFAOYSA-N 0.000 description 1
- 229920000098 polyolefin Polymers 0.000 description 1
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 1
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 1
- 229920006380 polyphenylene oxide Polymers 0.000 description 1
- 229920000069 polyphenylene sulfide Polymers 0.000 description 1
- 229920001451 polypropylene glycol Polymers 0.000 description 1
- 229920001343 polytetrafluoroethylene Polymers 0.000 description 1
- 239000004810 polytetrafluoroethylene Substances 0.000 description 1
- 239000004814 polyurethane Substances 0.000 description 1
- 229920002635 polyurethane Polymers 0.000 description 1
- 229920002451 polyvinyl alcohol Polymers 0.000 description 1
- 229920002981 polyvinylidene fluoride Polymers 0.000 description 1
- 239000011591 potassium Substances 0.000 description 1
- 229910052700 potassium Inorganic materials 0.000 description 1
- 239000002243 precursor Substances 0.000 description 1
- 230000003449 preventive effect Effects 0.000 description 1
- 230000037452 priming Effects 0.000 description 1
- 238000007639 printing Methods 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- QQONPFPTGQHPMA-UHFFFAOYSA-N propylene Natural products CC=C QQONPFPTGQHPMA-UHFFFAOYSA-N 0.000 description 1
- 125000004805 propylene group Chemical group [H]C([H])([H])C([H])([*:1])C([H])([H])[*:2] 0.000 description 1
- GMVPRGQOIOIIMI-DWKJAMRDSA-N prostaglandin E1 Chemical compound CCCCC[C@H](O)\C=C\[C@H]1[C@H](O)CC(=O)[C@@H]1CCCCCCC(O)=O GMVPRGQOIOIIMI-DWKJAMRDSA-N 0.000 description 1
- 239000012268 protein inhibitor Substances 0.000 description 1
- 229940121649 protein inhibitor Drugs 0.000 description 1
- 210000000512 proximal kidney tubule Anatomy 0.000 description 1
- 238000011158 quantitative evaluation Methods 0.000 description 1
- 230000011514 reflex Effects 0.000 description 1
- 239000003507 refrigerant Substances 0.000 description 1
- 239000004627 regenerated cellulose Substances 0.000 description 1
- 230000009719 regenerative response Effects 0.000 description 1
- 230000008439 repair process Effects 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 206010039073 rheumatoid arthritis Diseases 0.000 description 1
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 1
- 229920002477 rna polymer Polymers 0.000 description 1
- 239000010979 ruby Substances 0.000 description 1
- 229910001750 ruby Inorganic materials 0.000 description 1
- 239000008535 sairei-to Substances 0.000 description 1
- 210000000582 semen Anatomy 0.000 description 1
- 239000012679 serum free medium Substances 0.000 description 1
- 230000020509 sex determination Effects 0.000 description 1
- 230000001568 sexual effect Effects 0.000 description 1
- 230000008054 signal transmission Effects 0.000 description 1
- 229920002050 silicone resin Polymers 0.000 description 1
- 229950003804 siplizumab Drugs 0.000 description 1
- 210000003491 skin Anatomy 0.000 description 1
- 239000001488 sodium phosphate Substances 0.000 description 1
- 229910000162 sodium phosphate Inorganic materials 0.000 description 1
- 239000007787 solid Substances 0.000 description 1
- 238000000807 solvent casting Methods 0.000 description 1
- 238000000638 solvent extraction Methods 0.000 description 1
- 238000004611 spectroscopical analysis Methods 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 230000003068 static effect Effects 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 239000008227 sterile water for injection Substances 0.000 description 1
- 238000003756 stirring Methods 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 210000002536 stromal cell Anatomy 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 229920002994 synthetic fiber Polymers 0.000 description 1
- 230000009897 systematic effect Effects 0.000 description 1
- 230000001839 systemic circulation Effects 0.000 description 1
- 230000035488 systolic blood pressure Effects 0.000 description 1
- 229960001967 tacrolimus Drugs 0.000 description 1
- JGVWCANSWKRBCS-UHFFFAOYSA-N tetramethylrhodamine thiocyanate Chemical compound [Cl-].C=12C=CC(N(C)C)=CC2=[O+]C2=CC(N(C)C)=CC=C2C=1C1=CC=C(SC#N)C=C1C(O)=O JGVWCANSWKRBCS-UHFFFAOYSA-N 0.000 description 1
- RTKIYNMVFMVABJ-UHFFFAOYSA-L thimerosal Chemical compound [Na+].CC[Hg]SC1=CC=CC=C1C([O-])=O RTKIYNMVFMVABJ-UHFFFAOYSA-L 0.000 description 1
- 229940033663 thimerosal Drugs 0.000 description 1
- 230000009974 thixotropic effect Effects 0.000 description 1
- 239000003440 toxic substance Substances 0.000 description 1
- 239000012581 transferrin Substances 0.000 description 1
- 238000002054 transplantation Methods 0.000 description 1
- 150000003626 triacylglycerols Chemical class 0.000 description 1
- UFTFJSFQGQCHQW-UHFFFAOYSA-N triformin Chemical compound O=COCC(OC=O)COC=O UFTFJSFQGQCHQW-UHFFFAOYSA-N 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 239000003656 tris buffered saline Substances 0.000 description 1
- RYFMWSXOAZQYPI-UHFFFAOYSA-K trisodium phosphate Chemical compound [Na+].[Na+].[Na+].[O-]P([O-])([O-])=O RYFMWSXOAZQYPI-UHFFFAOYSA-K 0.000 description 1
- 239000012588 trypsin Substances 0.000 description 1
- 238000000539 two dimensional gel electrophoresis Methods 0.000 description 1
- 238000002604 ultrasonography Methods 0.000 description 1
- 238000000870 ultraviolet spectroscopy Methods 0.000 description 1
- 210000003954 umbilical cord Anatomy 0.000 description 1
- 208000009852 uremia Diseases 0.000 description 1
- 238000005353 urine analysis Methods 0.000 description 1
- 230000028973 vesicle-mediated transport Effects 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 230000009385 viral infection Effects 0.000 description 1
- 230000009278 visceral effect Effects 0.000 description 1
- BICRTLVBTLFLRD-PTWUADNWSA-N voclosporin Chemical compound CC[C@@H]1NC(=O)[C@H]([C@H](O)[C@H](C)C\C=C\C=C)N(C)C(=O)[C@H](C(C)C)N(C)C(=O)[C@H](CC(C)C)N(C)C(=O)[C@H](CC(C)C)N(C)C(=O)[C@@H](C)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)N(C)C(=O)[C@H](C(C)C)NC(=O)[C@H](CC(C)C)N(C)C(=O)CN(C)C1=O BICRTLVBTLFLRD-PTWUADNWSA-N 0.000 description 1
- 239000002699 waste material Substances 0.000 description 1
- 230000003313 weakening effect Effects 0.000 description 1
- 230000004584 weight gain Effects 0.000 description 1
- 235000019786 weight gain Nutrition 0.000 description 1
- 230000004580 weight loss Effects 0.000 description 1
- 230000036642 wellbeing Effects 0.000 description 1
- 230000037314 wound repair Effects 0.000 description 1
- 229940002552 xenical Drugs 0.000 description 1
- UHVMMEOXYDMDKI-JKYCWFKZSA-L zinc;1-(5-cyanopyridin-2-yl)-3-[(1s,2s)-2-(6-fluoro-2-hydroxy-3-propanoylphenyl)cyclopropyl]urea;diacetate Chemical compound [Zn+2].CC([O-])=O.CC([O-])=O.CCC(=O)C1=CC=C(F)C([C@H]2[C@H](C2)NC(=O)NC=2N=CC(=CC=2)C#N)=C1O UHVMMEOXYDMDKI-JKYCWFKZSA-L 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P7/00—Drugs for disorders of the blood or the extracellular fluid
- A61P7/06—Antianaemics
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0684—Cells of the urinary tract or kidneys
- C12N5/0686—Kidney cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K35/00—Medicinal preparations containing materials or reaction products thereof with undetermined constitution
- A61K35/12—Materials from mammals; Compositions comprising non-specified tissues or cells; Compositions comprising non-embryonic stem cells; Genetically modified cells
- A61K35/22—Urine; Urinary tract, e.g. kidney or bladder; Intraglomerular mesangial cells; Renal mesenchymal cells; Adrenal gland
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P13/00—Drugs for disorders of the urinary system
- A61P13/12—Drugs for disorders of the urinary system of the kidneys
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering N.A.
- C12N2310/141—MicroRNAs, miRNAs
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/30—Special therapeutic applications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/30—Special therapeutic applications
- C12N2320/32—Special delivery means, e.g. tissue-specific
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2509/00—Methods for the dissociation of cells, e.g. specific use of enzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/106—Pharmacogenomics, i.e. genetic variability in individual responses to drugs and drug metabolism
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/178—Oligonucleotides characterized by their use miRNA, siRNA or ncRNA
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Biomedical Technology (AREA)
- Organic Chemistry (AREA)
- Genetics & Genomics (AREA)
- Zoology (AREA)
- Biotechnology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- General Health & Medical Sciences (AREA)
- Urology & Nephrology (AREA)
- Cell Biology (AREA)
- General Engineering & Computer Science (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Veterinary Medicine (AREA)
- Public Health (AREA)
- Animal Behavior & Ethology (AREA)
- Pharmacology & Pharmacy (AREA)
- Medicinal Chemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Molecular Biology (AREA)
- Immunology (AREA)
- Analytical Chemistry (AREA)
- Biophysics (AREA)
- Physics & Mathematics (AREA)
- Virology (AREA)
- Developmental Biology & Embryology (AREA)
- Epidemiology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Plant Pathology (AREA)
- Pathology (AREA)
- Diabetes (AREA)
- Hematology (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
本發明涉及生物活性腎細胞群、腎細胞構建體及其製備和使用方法。
Description
本發明涉及到相比于健康個體還保留治療特性的缺乏細胞成分的生物活性腎細胞群或組分及其分離和培養方法,以及採用所述細胞群治療需要個體的方法。此外,本發明涉及到採用生物活性腎細胞群向自體腎提供再生效果的方法。
慢性腎病(CKD)在美國影響1900萬人以上,而且往往是涉及肥胖、糖尿病和高血壓的代謝障礙的結果。資料研究顯示,該增長率是由於繼發於高血壓和非胰島素依賴型糖尿病(NIDDM)的腎衰竭的發展(United States Renal Data System: Costs of CKD and ESRD. ed. Bethesda, MD, National Institutes of Health, National Institute of Diabetes and Digestive and Kidney Diseases, 2007 pp 223-238)-兩種疾病還在世界範圍內呈上升趨勢。肥胖、高血壓和血糖控制不佳都被證明是腎損傷的獨立風險因素,引起腎小球及腎小管病變,導致腎過濾功能的蛋白尿和其他全身檢測的變化(Aboushwareb, et al., World J Urol, 26: 295-300, 2008; Amann, K. et al., Nephrol Dial Transplant, 13: 1958-66, 1998)。階段1-3中CKD患者的進展是由針對於控制潛在疾病狀態的生活方式變化和藥物幹預進行控制,而階段4-5中患者是由透析和通常包括抗高血壓劑、紅細胞生成劑(ESA)、鐵和維生素D補充物的藥物療法進行控制。再生醫學技術可提供用於CKD的下一代治療劑選擇。Presnell et al. WO/2010/056328描述了包括腎小管和紅血球生成素(EPO)-產生腎細胞群的分離腎細胞及其分離和培養方法,以及採用所述細胞群治療需要個體的方法。需要一種提供實質性和持久增強腎功能的新治療形式,以減緩進展和改善這類患者的生活品質。
一方面,本發明提供了一種向自體腎提供再生效果的方法。在一個實例中,所述方法包括將自體腎與富集腎細胞群分泌的產物體內接觸的步驟。在另一個實例中,所述產物是由不是此處所述構建體一部分的富集腎細胞群分泌,例如,所述細胞群並不是接種於支架上。在另一個實例中,所述產物是由包含直接接種於支架上或支架內的富集腎細胞群的腎細胞構建體分泌。在另一個實例中,所述產物的分泌對氧水準有生物反應。分泌可由低於大氣氧水準誘導。在另一個實例中,所述更低氧水準比約5%氧低。 在一個實例中,再生效果為上皮細胞間質轉化(EMT)的減少。EMT的減少可通過減弱TGF-β信號和/或減弱纖溶酶原啟動物抑制劑1(PAI-1)信號達到。在另一個實例中,所述再生效果為腎纖維化的減少和/或腎炎症的減少。在一些實例中,所述炎症的減少可由NFκB介導。在另一個實例中,再生效果的特徵為自體腎中幹細胞標記的差異表達。相對於非接觸自體腎表達,所述表達可是體內接觸的自體腎標記的上調表達。 一方面,所述富集腎細胞群包括一種或多種群,即此處所述的混合物。在一個實例中,所述群包括第一細胞群、含有富集腎小管細胞群的B2。在另一個實例中,所述群包括具有第一細胞群、B2和第二細胞群的人類腎細胞的混合物,所述第二細胞群含有紅血球生成素(EPO)產生細胞、腎小球細胞和血管細胞中的一種或多種。在另一個實例中,所述第二細胞群為B4細胞群。在又一個實例中,所述第二細胞群為B3細胞群。 在一個實例中,所述混合物進一步包括具有紅血球生成素(EPO)產生細胞、腎小球細胞和血管細胞中的一種或多種的第三細胞群。在另一個實例中,所述第三細胞群為B4細胞群。在另一個實例中,所述第三細胞群為B3細胞群。 在所有實例中,所述B2細胞群具有介於約1.045克/毫升和約1.052克/毫升之間的密度。在所有實例中,所述B4細胞群具有介於約1.063克/毫升和約1.091克/毫升之間的密度。在所有實例中,所述B3細胞群具有介於約1.052克/毫升和約1.063克/毫升之間的密度。 在所有實例中,所述富集腎細胞群可對自體腎是非自體的。在所有實例中,所述富集腎細胞群可對自體腎是自體的。 在所有實例中,所述產物包括旁分泌因數、內分泌因數、鄰分泌因數、RNA、囊泡、微囊泡、外泌體及其任何組合。在另一個實例中,所述囊泡包括選自由旁分泌因數、內分泌因數、鄰分泌因數和RNA組成的組的一種或多種分泌產物。在另一個實例中,所述產物是由包含直接接種於支架上或在支架內的富集腎細胞群的腎細胞構建體分泌。 在所有實例中,所述支架可含有生物相容性材料。在所有實例中,所述生物相容性材料可為水凝膠。 在一個實例中,本發明提供了一種評價腎病(KD)患者是否對採用治療劑治療有反應的方法。所述方法可包括:相比於或相對於對照樣本中囊泡量,測定或檢測從採用治療劑治療的KD患者獲得的測試樣本中的囊泡或它們的腔內容物的量的步驟,其中,相比於對照樣本中囊泡或它們的腔內容物的量,測試樣本中囊泡或腔內容物的更高量或更低量表明治療患者對採用治療劑治療有反應。所述囊泡可為腎源性囊泡。所述測試樣本可含有尿液。所述囊泡可含有生物標記物,其可為miRNA。所述治療劑可含有富集腎細胞群。
本發明涉及生物活性腎細胞(BRC)的異質混合物或片段及其分離和培養方法,以及採用此處所述的接種有BRC的支架形成的BRC和/或含BRC-構建體治療需要個體的方法。生物活性腎細胞可是包括腎小管細胞和紅血球生成素(EPO)產生腎細胞的分離腎細胞。BRC細胞群可包括富集腎小管細胞和EPO產生細胞群。BRC可來自健康個體或本身是健康個體腎細胞組分。此外,當與健康個體的相應腎細胞組分相比,本發明提供來自可缺乏細胞組分的不健康個體的腎細胞組分,但仍然保留治療特性。本發明還提供了相比于健康個體缺乏細胞組分的治療上具有活性的細胞群,在一個實例中,細胞群可以是從各種疾病狀態下自體來源分離和擴展的。 本發明還涉及到通過在體內將自體腎與腎細胞分泌的產物接觸提供一種再生效果的方法,以及製備分泌產物的方法。本發明進一步涉及到標記的用途,以測定採用此處所述的方法治療後腎再生的存在。 定義 除非另有定義,此處所用的技術和科學術語,具有本發明所屬技術領域的普通技術人員所公知的相同含義。Principles of Tissue Engineering
, 3rd
Ed. (Edited by R Lanza, R Langer, & J Vacanti), 2007為本領域的普通技術人員提供了本申請中所用的很多術語的基本指南。本領域的普通技術人員將確認類似或等價於那些此處所述可用於本發明實際應用的方法和材料。事實上,本發明決不限制於所述的方法和材料。 此處所用的術語“細胞群”是指直接從合適的組織(通常是從哺乳動物)來源分離得到的許多細胞。所分離的細胞群隨後可在體外培養。所屬技術領域的普通技術人員領會到採用本發明分離和培養以及適用於本發明的細胞群細胞的各種細胞。細胞群可是來自腎臟的普通異質細胞群。例如,異質細胞群可是從腎活檢或整個腎組織分離的。此外,異質細胞群可來自於腎活檢或整個腎組織建立的哺乳動物細胞體外培養。普通異質細胞群也被稱為非富集細胞群。 術語“自體腎”應指活個體的腎。所述個體可是健康或不健康的。不健康的個體可具有腎病。 術語“再生效果”應指向自體腎提供益處的效果。所述效果可包括,但不限制於,自體腎損傷程度的減少或自體腎功能的改善、恢復或穩定。腎損傷可以個體中的纖維化、發炎、腎小球肥大等相關腎疾病的形式存在。 此處所用的術語“混合物”是指來自普通異質細胞群的兩種或多種分離的富集細胞群。根據某些實例,本發明的細胞群為腎細胞群。 “富集的”細胞群或製劑是指來自含有大於起始群中細胞類型比例的特定細胞類型的起始腎細胞群(例如,普通異質細胞群)的細胞群。例如,起始腎細胞群可富集第一、第二、第三、第四、第五等目標細胞群。此處所用的術語“細胞群”、“細胞製劑”和“細胞原型”可以互換使用。 一方面,此處所用的術語“富集的”細胞群來自含有大於起始群中能夠產生EPO的細胞比例的能夠產生EPO的細胞比例的起始腎細胞群(例如,來自腎活檢或培養哺乳動物腎細胞的細胞懸液)的細胞群。例如,術語“B4”是指來自含有較大比例EPO產生細胞、腎小球細胞和血管細胞的起始腎細胞群的細胞群。本發明的細胞群可富集一種或多種細胞類型,並去除一種或多種細胞其他類型。例如,相對于非富集細胞群中的間質成纖維細胞和腎小管細胞,富集的EPO產生細胞群可富集間質成纖維細胞,並去除腎小管細胞和收集導管上皮細胞,即起始細胞群來自富集的細胞群。在引用EPO富集或“B4”群的所有實例中,所述富集的細胞群為含有以氧氣調節方式可產生EPO細胞的異質細胞群,如本地內源性自體EPO基因的氧可調EPO表達所表明。 在另一方面,大於起始群中細胞類型比例的特定細胞類型(例如,血管、腎小球或內分泌細胞)的富集細胞群,相比于來自健康個人或個體的起始腎細胞群可還缺乏或欠缺一種或多種特定細胞類型,如血管、腎小球或內分泌細胞。例如,在一方面,術語“B4”或“B4初期”,是缺乏或欠缺一種或多種細胞類型(例如,血管、腎小球或內分泌細胞)的起始腎細胞群的細胞群,與健康個體相比這取決於起始樣本的疾病狀態。在一個實例中,B4細胞群來自患有慢性腎疾病的個體。在一個實例中,B4細胞群來自患有局灶性節段性腎小球硬化(FSGS)的個體。在另一個實例中,B4細胞群來自一個患有自身免疫性腎小球腎炎的個體。在另一方面,B4是來自包括所有細胞類型(例如,血管、腎小球或內分泌細胞)的起始細胞群的細胞群,然後除去或使其缺乏一種或多種細胞類型,例如,血管、腎小球或內分泌細胞。在又一方面,B4是來自包括所有細胞類型(例如,血管、腎小球或內分泌細胞)的起始細胞群的細胞群,其中一種或多種特定類型,例如,血管、腎小球或內分泌細胞隨後被富集。例如,在一個實例中,B4細胞群可富集血管細胞,但除去腎小球和/或內分泌細胞。在另一個實例中,B4細胞群可富集腎小球細胞,但除去血管和/或內分泌細胞。在另一個實例中,B4細胞群可富集內分泌細胞,但除去血管和/或腎小球細胞。在另一個實例中,B4細胞群可富集血管和內分泌細胞,但除去腎小球細胞。在優選實例中,B4細胞群單獨或與另一種富集細胞群(例如,B2和/或B3)混合,保留治療特性。B4細胞群,例如,在此處的實例(例如,例7-9)中有所描述。 在另一方面,富集細胞群也可指來自以上所討論的起始腎細胞群的細胞群,含有大於起始群中表達一種或多種腎小管細胞標記的細胞比例的表達一種或多種腎小管細胞標記的一部分細胞。例如,術語“B2”是指來源於含有較大比例腎小管細胞的起始腎細胞群的細胞群(相比於起始群)。此外,富集表達一種或多種腎小管細胞標記(或“B2”)的細胞的細胞群可含有收集管系統的一些上皮細胞。雖然富集表達一種或多種腎小管細胞標記(或“B2”)的細胞的細胞群相對缺乏EPO產生細胞,腎小球細胞和血管細胞,相比於起始群,富集群可含有較小比例的這些(EPO生成、腎小球和血管)細胞。一般而言,異質細胞群被除去一種或多種細胞類型,使得在除去之前相對於異質細胞群中所含的細胞類型除去的細胞群中含有較小比例的細胞類型。可被除去的細胞類型是腎細胞的任何類型。例如,在某些實例中,可被除去的細胞類型包括大粒度的集合管和密度為約1.045克/毫升以上管狀系統的細胞,簡稱為“B1”。在某些其他實例中,可被除去的細胞類型包括碎片和密度為約1.095克/毫升以上低粒度和存活率的小細胞,被稱為“B5”。在一些實例中,富集腎小管細胞的細胞群被相對地除去以下的所有:“B1”、“B5”、氧可調EPO表達細胞,腎小球細胞和血管細胞。 此處所用的術語“缺氧的”培養條件是指相對於在大氣氧水準(約21%)培養細胞的標準培養條件使細胞在培養體系中有效氧水準下減少的培養條件。非缺氧條件是指正常或常氧培養條件。 此處所用的術語“氧可調的”是指調節基於細胞有效供氧量的基因表達(向上或向下)的細胞能力。“缺氧誘導的”是指對氧含量減少的基因表達上調的反應(無論是誘導前或啟動氧含量)。 此處所用的術語“生物材料”是指適於介入活體組織的天然或合成生物相容性材料。天然生物材料為由生命系統制得的材料。合成生物材料為並非由生命系統制得的材料。此處所公開的生物材料可是天然和合成生物相容性材料的組合。此處所用的生物材料包括,例如,聚合物基質和支架。本領域的普通技術人員將領會到,生物材料可以不同的形式配置,例如,作為水凝膠懸浮液、多孔泡沫,還可包括一種或多種天然或合成生物相容性材料。 此處所用的術語“貧血症”是指缺乏紅細胞數和/或血紅蛋白水準,這是由於通過個體的EPO產生細胞產生的功能EPO蛋白不足,和/或EPO蛋白質不充分釋放到體循環,和/或骨髓中成紅細胞細胞無力應對EPO蛋白質。貧血個體不能維持紅細胞的動態平衡。一般而言,貧血可發生腎功能(如慢性腎功能衰竭)、相對EPO不足相關性貧血症、充血性心力衰竭相關性貧血症、骨髓抑制治療相關性貧血症(如化療損失或抗病毒治療相關性貧血症(例如,AZT))、非骨髓癌相關性貧血症、病毒感染相關性貧血症(如HIV)、慢性病貧血症(如自身免疫性疾病(例如,類風濕關節炎))、肝臟疾病和多器官功能衰竭的下降或喪失。 術語“EPO缺乏症”是指採用紅血球生成素受體激動劑(例如,EPO或EPO類似物重組體)能治療的任何病症或疾病,其中包括貧血症。 此處所用的術語“腎病”是指與導致執行血液過濾功能以及過多液體、電解質和血液廢物排除的腎臟能力喪失的急性或慢性腎功能衰竭的任何階段或程度相關的疾病。腎疾病還包括內分泌功能障礙,如貧血症(紅血球生成素缺乏症)和礦物不平衡(維生素D缺乏症)。腎疾病可源於腎臟或可繼發於各種病症,包括(但不限於)心臟衰竭、高血壓、糖尿病、自身免疫性疾病或肝臟疾病。腎臟疾病可是腎急性損傷後發展的慢性腎功能衰竭病症。例如,局部缺血和/或接觸有毒物質的腎損傷可會引起急性腎功能衰竭;急性腎損傷後的不完全恢復可會導致慢性腎功能衰竭的發展。 術語“治療”是指腎疾病、貧血、EPO缺乏症、腎小管運輸缺乏症或缺乏腎小球過濾的治療劑治療和預防疾病或預防性措施,其中目標是扭轉、防止或減慢(減少)目標障礙。那些治療需要的個體,包括那些已患有腎臟疾病、貧血症、EPO缺乏症、腎小管運輸缺乏症或缺乏腎小球過濾以及那些易患有腎疾病、貧血症、EPO缺乏症、腎小管運輸缺乏症或缺乏腎小球過濾或者那些可以預防腎疾病、貧血症、EPO缺乏症、腎小管運輸缺乏症或缺乏腎小球過濾的個體。此處所用的術語“治療”包括腎功能的穩定和/或改善。 此處所用的術語“體內接觸”是指腎細胞(或含有腎細胞/腎細胞組分的混合物或構建體)和自體腎富集群分泌的產物間的體內直接接觸。體內直接接觸本質上可是旁分泌、內分泌或近泌分泌。所分泌的產物可是此處所述不同產物的異質群。 此處所用的術語“核糖核酸”或“ RNA” 是指核苷酸單位鏈,其中每個單位由一個含氮堿基、核糖和磷酸酯組成。RNA可是單鏈或雙鏈形式。RNA可成為囊泡的一部分,在囊泡內部或與囊泡相關。囊泡可是外泌體。RNA包括但不限於,mRNA、rRNA、小RNA、snRNA、snoRNA、microRNA(miRNA)、小幹擾RNA(siRNA)和非編碼RNA。RNA優選為人類RNA。 術語“構建體”是指沉積於或在由一種或多種合成或天然存在的生物相容性材料組成的支架或基質表面上的一種或多種細胞群。一種或多種細胞群可包覆、沉積、嵌入、結合、接種或或包埋由一種或多種合成或天然存在的生物相容性聚合物、蛋白質或多肽。一種或多種細胞群可與體外或體內的生物材料或支架或基質結合。一般而言,用於形成支架/生物材料的一種或多種生物相容性材料被選定,以疏導、促進或允許沉積於其上的至少一種細胞群的多細胞三維排列。用於產生構建體的一種或多種生物材料也被選定,以疏導、促進或允許構建體的分散和/或整合或內源性宿主組織構建體的細胞組分,或疏導、促進或允許構建體的生存、植入、接受或功能性能或構建體的細胞組分。 術語“標記物”或“生物標記物”一般是指DNA、RNA、蛋白質,碳水化合物或糖脂分子標記物,其在培養細胞群中的表達或存在可以通過標準的方法(或此處所公開的方法)檢測,並與特定細胞類型的培養細胞群中的一種或多種細胞一致。該標記物可是由細胞或染色體上可識別的物理位置(如基因、限制性內切酶識別位點或由自體細胞表達的多肽(例如,mRNA)編碼的核酸)表達的多肽。該標記物可是被稱為“基因表達標記物”的基因的表達區,或一些沒有已知編碼功能的DNA片段。生物標記物可是細胞來源的,例如,分泌的產物。 術語“差異表達基因”、”基因差異表達”和可交替使用的其同義詞是指相對於第二細胞或細胞群中的表達水準,第一細胞或細胞群中被啟動到較高或較低水準的基因表達。術語還包括第一或第二細胞培養過程中隨著時間的推移在不同階段基因的表達被啟動到較高或較低水準的基因表達。這也是瞭解,差異表達基因可是在核酸水準或蛋白水準被啟動或抑制,或可受到可變剪接,以導致不同的多肽產物。這種差異可會以(例如)mRNA水準的變化、表面表達、多肽的分泌或其他分區證明。差異基因表達可包括兩種或多種基因或其基因產物,或兩種或多種基因或其基因產物之間的表達比比較,或甚至是相同基因的兩種不同加工產物的比較,其中第一和第二細胞間不同。差異表達包括基因或其表達產物中的暫態或細胞表達模式的定量以及定性差異,例如,在第一和第二細胞間。對於本發明的目的,當第一和第二細胞中的給定基因表達存在差異時,“差異基因表達”被認為是存在的。標記物的差異表達可是在施用細胞群、混合物或構建體(第一細胞)前患者的細胞相對于施用後患者(第二細胞)細胞的表達。 術語“抑制”、“下調”、“下表達”和“降低”可互換使用,而且表示相對於減少一種或多種對照(例如,一種或多種陽性和/或陰性對照),基因表達或編碼一種或多種蛋白質或蛋白質亞組分的RNA分子水準或同等RNA分子水準或一種或多種蛋白質或蛋白質亞組分的活性降低。下表達可是在施用細胞群、混合物或構建體前患者細胞相對于施用後患者細胞的表達。 術語“上調”或“過表達”是用來表示相對於減少一種或多種對照(例如,一種或多種陽性和/或陰性對照),基因表達或編碼一種或多種蛋白質或蛋白質亞組分的RNA分子水準或同等RNA分子水準或一種或多種蛋白質或蛋白質亞組分的活性升高。過表達可是在施用細胞群、混合物或構建體前患者細胞相對于施用後患者細胞的表達。 術語“個體”是指任何人包括可以獲得治療的患者的單一人類個體,其正經歷或已經歷一種或多種跡象、症狀或腎疾病、貧血症或EPO缺乏症。這些個體包括但不限於最近診斷或以前診斷,並正經歷復發或再發或存在腎疾病、貧血症或EPO缺乏症風險個體,不管原因。該個體可此前已接受腎疾病、貧血症或EPO缺乏症治療或沒有這樣治療。 術語“患者”是指任何單一的動物,較優選為被預計治療的哺乳動物(包括非人類動物,例如狗、貓、馬、兔動物園動物、牛、豬、羊和非人類靈長類動物)。最優選地,此處的患者是人類。 術語“樣本”或“患者樣本”或“生物樣本”一般應當是指從個體或患者、體液、人體組織、細胞系、組織培養或其他來源獲得的任何生物樣本。該術語包括組織切片,例如,腎活檢。該術語包括培養細胞,例如,培養的哺乳動物腎細胞。從哺乳動物獲得組織切片和培養細胞方法是本領域眾所周知的。如果術語“樣本”是單獨使用時,仍應指“樣本”是“生物樣本”或“患者樣本”,即術語可以互換使用。 術語“測試樣本”是指來自已接受本發明方法治療的個體的樣本。測試樣本可來自於哺乳動物個體的不同來源,包括但不限於血液、精液、血清、尿液、骨髓、粘膜和組織等。 術語“對照”或“對照樣本”是指陰性或陽性對照,其中陰性或陽性結果預計將幫助使測試樣品的結果相關。控制,適用於本發明的對照包括但不限於,正常紅細胞動態平衡特有的顯示指標已知的樣本、貧血症特有的顯示指標已知的樣本、從公知非貧血症個體獲得的樣本和從公知貧血症個體獲得的樣本。適用於本發明的其他對照包括但不限於,來自已採用調節紅細胞生成(例如,重組EPO或EPO類似物)的已知藥物製劑治療的個體的樣本。此外,該對照可是從通過本發明的方法治療前的個體獲得的樣本。其他合適的對照可是從已知患有任何類型或階段的腎疾病的個體獲得的測試樣本,並從已知的,或者是從已知不患有任何類型或階段的腎疾病的個體獲得的測試樣本。對照可是正常的健康對照組。本領域的普通技術人員將領會到其他對照適用於本發明。 “再生預後”、“再生的預後”或“用於再生的預後”一般是指此處所述的細胞群、混合物或構建體給藥或植入的可再生過程或結果的預示或預測。對於再生預後,預示或預測可由以下的一種或多種告知:植入或給藥後腎功能的改善、植入或給藥後腎功能的發展、植入或給藥後改善的腎功能或能力的發展以及植入或給藥後通過自體腎的某些標記物表達。 “再生腎”是指此處所述的細胞群、混合物或構建體植入或給藥後的自體腎。再生腎的特徵為包括但不限於,自體腎功能或能力的發展、自體腎功能或能力的改善以及自體腎某些標記物表達的各項指標。本領域的普通技術人員將領會到其他指標可適於表徵再生腎。 細胞群 腎細胞及其混合物的分離異質群,富集用於治療腎疾病的特定生物活性成分和/或除去用於治療腎疾病的特定失活或不需要成分或細胞類型,即提供腎功能的穩定和/或改善和/或再生,先前在於2009年11月12提交的第12/617721號美國專利中有所描述,其中它們的全部內容通過引用併入本文。本發明提供了相比於仍保留治療特性的健康個體缺乏細胞組分的分離的腎細胞組分,即提供腎功能的穩定和/或改善和/或再生。此處所述的細胞群、細胞組分和/或細胞混合物可來自健康個體、患有腎臟病的個體或此處所述的個體。 生物活性細胞群 一方面,本發明基於富集生物活性成分和除去失活或不需要成分或細胞類型的異質腎細胞群亞組分的驚人發現,提供比起始群更好的治療和再生結果。例如,本發明的生物活性成分,例如,除去失活或不需要成分的B2、B4和B3,例如,單獨或混合的B1和B5,提供意想不到的腎功能的穩定和/或改善和/或再生。 在另一方面,本發明基於除去或缺乏一種或多種細胞類型(例如,血管、內分泌或內皮)的特定亞組分B4的驚人發現,即B4保留治療特性,例如,當單獨或與其他生物活性亞組分(例如,B2和/或B3)混合時,腎功能的穩定和/或改善和/或再生。在優選實例中,生物活性細胞群為B2。在某些實例中, B2細胞群中混合有B4或B4'。在其他實例中,B2細胞群中混合有B3。在其他實例中,B2細胞群中混合有B3和B4兩者,或B3和/或B4的特定細胞成分。 B2細胞群的特徵為選自以下一種或多種組成的組的腎小管細胞標記物的表達:並轉運給陰離子蛋白、cubilin、透明質酸酶2(HAS2),維生素D3 25-羥化酶(CYP2D25)和N-鈣粘蛋白(Ncad)、E-鈣粘蛋白(Ecad)、水通道蛋白1(Aqp1)、水通道蛋白2 (Aqp2)、RAS致癌基因家族成員RAB17(Rab17)、GATA結合蛋白3(Gata3)、含FXYD結構域的離子調節劑4(Fxyd4)、溶質載體家族9(鈉/氫交換器)成員4(Slc9a4)、醛脫氫酶3家族成員B1(Aldh3b1),醛脫氫酶1家族成員A3(Aldh1a3)和鈣蛋白酶-8(Capn8)以及和集合管標記物水通道蛋白-4 (Aqp4)。B2是比B3和/或B4更大的顆粒,因而具有介於約1.045克/毫升和約1.063克/毫升(齧齒動物)之間、約1.045克/毫升和約1.052克/毫升(人類)以及約1.045克/毫升和約1.058克/毫升(犬科動物)之間的浮力密度。 B3細胞群的特徵為血管、腎小球和臨近腎小管標記物的一些EPO產生細胞的表達,與B2和B4相比,為中間粒度和顆粒,從而具有介於約1.063克/毫升和1.073克/毫升(齧齒動物)、約1.052克/毫升和約1.063克/毫升(人類)以及約1.058克/毫升和約1.063克/毫升(犬科動物)之間的浮力密度。B3的特徵為選自以下一種或多種組成的組的標記物的表達:水通道蛋白7(Aqp7)、含FXYD結構域的離子調節劑4(Fxyd4)、溶質載體家族17(磷酸鈉)成員3(Slc17a3)、溶質載體家族3成員1(Slc3a1)、水閘蛋白2(Cldn2)、napsin A天冬氨肽酶(Napsa),溶質載體家族2(促葡萄糖轉運蛋白)成員2(Slc2a2)、丙氨醯(膜)氨基肽酶(Anpep)、跨膜蛋白27(Tmem27)、醯基輔酶A合成酶中鏈家族成員2(Acsm2),谷胱甘肽過氧化物酶3(Gpx3)、果糖-1,6-二磷酸酶1(Fbp1)和丙氨酸-乙醛酸氨基轉移酶2(Agxt2)。B3還有一個特徵為血管表達標記物血小板內皮細胞粘附分子(Pecam)和腎小球表達標記物podocin(Podn)。 B4細胞群的特徵為含有以下一種或多種的血管標記物組的表達:PECAM、VEGF、KDR、HIF1a、CD31、CD146;含有以下一種或多種的腎小球標記物組的表達:Podocin (Podn)和腎病蛋白(Neph);以及相比於普通 (UNFX)、B2和B3的氧可調EPO富集群。B4還有一個特徵為以下一種或多種標記物的表達:趨化因數(C-X-C基序)受體4 (Cxcr4)、內皮素受體B型(Ednrb)、膠原蛋白V型、α2(Col5a2)、鈣粘蛋白5(Cdh5)、纖溶酶原啟動物、組織(Plat)、血管生成素2(Angpt2)、激酶插入域蛋白受體(Kdr)、分泌蛋白、酸性富半胱氨酸(骨粘連蛋白)(SPARC)、絲甘蛋白聚糖(Srgn)、TIMP金屬肽酶抑制劑3(Timp3)、腎母細胞瘤1(WT1基因)、無翅型MMTV整合位點家族成員4(Wnt4),G-蛋白信號調節劑4(Rgs4)、血小板內皮細胞粘附分子(Pecam)和紅血球生成素(EPO)。B4還有一個特徵為比B2或B3更小的小顆粒細胞,具有介於約1.073克/毫升和約1.091克/毫升(齧齒動物)、約1.063克/毫升和約1.091克/毫升(人類和犬科動物)之間的浮力密度。 B4'細胞群被定義為具有介於1.063克/毫升和1.091克/毫升之間的浮力密度,並表示以下標記物的一種或多種:PECAM、vEGF、KDR、HIF1a、podocin、nephrin、EPO、CK7、CK8/18/19。在一個實例中,所述B4'細胞群的特徵為含有以下一種或多種的血管標記物組的表達:PECAM、vEGF、KDR、HIF1a、CD31、CD146。在另一個實例中,所述B4'細胞群的特徵為內分泌標記物EPO的表達。在一個實例中,B4'細胞群的特徵為含有以下一種或多種的腎小球標記物組的表達:Podocin (Podn)和腎病蛋白(Neph)。在某些實例中,所述B4'細胞群的特徵為含有以下一種或多種的血管標記物組的表達:PECAM、vEGF、KDR、HIF1a和通過內分泌標記物EPO的表達。在另一個實例中,B4'的另一個特徵為比B2或B3更小的小顆粒細胞,具有介於約1.073克/毫升和約1.091克/毫升之間(齧齒動物)、約1.063克/毫升和約1.091克/毫升(人類和犬科動物)之間的浮力密度。 一方面,本發明提供了一種包含密度介於1.063克/毫升和1.091克/毫升之間的至少一種紅血球生成素(EPO)產生細胞、血管細胞和腎小球細胞的分離的人類腎細胞富集B4’群。在一個實例中,所述B4'細胞群的特徵為血管標記物的表達。在某些實例中,所述B4'細胞群的特徵不是腎小球標記物的表達。在一些實例中,所述B4'細胞群能夠氧可調紅血球生成素(EPO)表達。 在一個實例中,B4'細胞群不包括,包含有密度介於1.045克/毫升和1.052克/毫升之間的的腎小管細胞的B2細胞群。在另一個實例中,B4'細胞群不包括,包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群。在又一個實例中,B4'細胞群不包括,包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。 在一個實例中,B4'細胞群不包括,包含有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞的B2細胞群;包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群;包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。在一些實例中,B4'細胞群可來自患有腎病的個體。 一方面,本發明提供了包含第一細胞群的人類腎細胞、包含具有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞分離富集群的B2、第二細胞群以及包含紅血球生成素(EPO)產生細胞和血管細胞但除去密度介於約1.063克/毫升和1.091克/毫升之間的腎小球細胞的B4’的混合物,其中所述混合物不包括:包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群,或包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。在某些實例中,所述B4'細胞群的特徵為血管標記物的表達。在一個實例中,所述B4'細胞群的特徵不是腎小球標記物的表達。在某些實例中,B2進一步包含集合管上皮細胞。在一個實例中,所述細胞混合物能夠受體介導白蛋白吸收。在另一個實例中,所述細胞混合物能夠氧可調紅血球生成素(EPO)表達。在一個實例中,所述混合物含有能夠產生和/或促進高分子量透明質酸(HA)在體外和體內產生的HAS-2表達細胞。在所有實例中,所述第一和第二細胞群可來自腎組織或培養的腎細胞。 在一個實例中,所述混合物能夠提供一種體內傳送的再生促進。在其他實例中,所述混合物能夠減少下降、穩定或改善腎小球過濾、腎小管再吸收、尿產生和/或體內傳送的內分泌功能。在一個實例中,所述B4'細胞群來源於患有腎病的個體。 一方面,本發明提供了一種包含密度介於1.063克/毫升和1.091克/毫升之間的至少一種紅血球生成素(EPO)產生細胞、血管細胞和腎小球細胞的分離的人類腎細胞富集B4’群。在一個實例中,所述B4'細胞群的特徵為血管標記物的表達。在某些實例中,所述B4'細胞群的特徵不是腎小球標記物的表達。不表達的腎小球標記物可是podocin(見例7)。在一些實例中,所述B4'細胞群能夠氧可調紅血球生成素(EPO)表達。 在一個實例中,所述B4'細胞群不包括,包含有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞的B2細胞群。在另一個實例中,所述B4'細胞群不包括,包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群。在又一個實例中,所述B4'細胞群不包括,包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。 在一個實例中,所述B4'細胞群不包括,包含有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞的B2細胞群;包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群;以及包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。 在一些實例中,所述B4'細胞群可來源於患有腎病的個體。一方面,本發明提供了包含第一細胞群的人類腎細胞、包含具有密度介於1.045克/毫升 和 1.052克/毫升之間的腎小管細胞分離富集群的B2、第二細胞群以及包含紅血球生成素(EPO)產生細胞和血管細胞但除去密度介於約1.063克/毫升和1.091克/毫升之間的腎小球細胞的B4’的混合物,其中所述混合物不包括:包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群,或包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。在某些實例中,所述B4'細胞群的特徵為血管標記物的表達。在一個實例中,所述B4'細胞群的特徵不是腎小球標記物的表達。在某些實例中,所述B2進一步包含集合管上皮細胞。在一個實例中,所述細胞混合物能夠受體介導白蛋白吸收。在另一個實例中,所述細胞混合物能夠氧可調紅血球生成素(EPO)表達。在一個實例中,所述混合物含有能夠產生和/或促進高分子量透明質酸(HA)在體外和體內產生的HAS-2表達細胞。在所有實例中,所述第一和第二細胞群可來源於腎組織或培養的腎細胞。 在一個實例中,所述混合物能夠提供一種體內傳送的再生促進。在其他實例中,所述混合物能夠減少下降、穩定或改善腎小球過濾、腎小管再吸收、尿產生和/或體內傳送的內分泌功能。在一個實例中,所述B4'細胞群來源於患有腎病的個體。 在一個優選實例中,所述混合物包含B2與B3和/或B4的組合。在另一個優選實例中,所述混合物包含B2與B3和/或B4’的組合。在其他優選實例中,所述混合物組成或基本組成為(i) B2與B3和/或B4的組合;或(ii) B2與B3和/或B4’的組合。 含有B4’細胞群的混合物可含有也從非健康個體獲得的B2和/或B3細胞群。非健康個體可與獲得B4’組分的個體相同。與B4’細胞群相反,從非健康個體獲得的B2和B3細胞群相比于來自健康個體的起始腎細胞群基本上不缺乏一種或多種特定細胞類型。 由B2和B4產生透明質酸 透明質酸(也稱為玻尿酸或玻璃酸)是一種糖胺聚糖(GAG),其組成為非硫酸雙糖單元(特別是N-乙醯氨基葡萄糖和葡萄糖醛酸)的規則重複序列。它的分子量範圍為400道爾頓(二糖)至100萬道爾頓以上。它在所有組織中(如皮膚、軟骨和眼睛)的含量不同,如果不是所有成年動物的液體,也是多數。它在早期胚胎中特別豐富。透明質酸產生的空間,一般而言,GAG,允許它在創面修復、器官形成、免疫細胞粘附、細胞內信號啟動以及腫瘤轉移過程中發揮細胞遷移、細胞粘附的作用。這些作用是由特異性蛋白和蛋白多糖結合透明質酸介導的。細胞運動性和免疫細胞粘附是由細胞表面受體RHAMM (Receptor for Hyaluronan-Mediated Motility; Hardwick et al., 1992)和CD44 (Jalkenan et al., 1987; Miyake et al., 1990)介導的。透明質酸是通過其正在合成的細胞外面的膜在不斷增長的外擠聚合物細胞表面內膜上直接合成。合成是由單一蛋白質酶、其基因家族組成為至少3個成員的透明質酸合成酶介導的。 最近已表明,透明質酸與CD44發生相互作用,這種相互作用可,(除其他作用之外)將非儲備細胞(如骨髓間質幹細胞(MSC))吸納入受損傷腎組織,並增強腎再生作用(Kidney International (2007) 72, 430–441)。 出乎意料地,研究發現,B2和B4細胞製劑通過透明質酸合成酶-2 (HAS-2)(更具體地說,B2細胞群中富集的標記物)的作用能夠在體外和體內表達高分子量的透明質酸(HA)。5/6 Nx模型中B2的治療結果表明,可減少伴隨體內HAS-2強表達和治療組織內高分子量HA預計產生的纖維化。值得注意的是,5/6 Nx模型不及時治療,導致有限檢測HAS-2和高分子量HA產生的纖維化。如果不希望被理論束縛,假設這種主要通過B2產生的抗炎高分子量的HA(在一定的B4的程度)與細胞製劑協同地在減少腎纖維化和協助腎再生中發揮作用。因此,本發明包括本發明細胞原型在包含透明質酸的生物材料中的傳送。本發明還要考慮的是通過直接產生或通過植入細胞促進產生的再生促進生物材料成分的規定。 一方面,本發明提供了用於治療需要個體的腎病、貧血症和/或EPO缺乏症的EPO產生腎細胞的分離異質群。在一個實例中,所述細胞群來源於腎活檢。在另一個實例中,所述細胞群來源於整個腎組織。在另一個實例中,所述細胞群來源於腎組織腎活檢或整個腎組織建立的哺乳動物腎細胞的體外培養。在所有實例中,這些群都是普通細胞群,並在此被稱為非富集細胞群。 在另一方面,本發明提供了進一步富集的紅血球生成素(EPO)產生腎細胞的分離群,以使富集亞群中的EPO產生細胞比例相對於起始或初始細胞群中的EPO產生細胞較大。在一個實例中,相對于非富集初始群中所含的間質成纖維細胞和腎小管細胞,所述富集EPO產生細胞組分含有較大比例的間質成纖維細胞與較小比例的腎小管細胞。在某些實例中,相對于非富集初始群中所含的腎小球細胞、血管細胞和集合管細胞,所述富集EPO產生細胞組分含有較大比例的腎小球細胞和血管細胞以及較小比例的集合管細胞。在這些實例中,這些群在此被稱為“B4”細胞群。 在另一方面,本發明提供了一種混有一種或多種其他腎細胞群的EPO產生腎細胞群。在一個實例中,所述EPO產生細胞群是富集EPO產生細胞(例如,B4)的第一細胞群。在另一個實例中,所述EPO產生細胞群是不富集EPO產生細胞(例如,B2)的第一細胞群。在另一個實例中,所述第一細胞群混有第二腎細胞群。在一些實例中,所述第二細胞群富集腎小管細胞,這可是通過腎小管細胞表型的存在證明。在另一個實例中,所述腎小管細胞表型可是通過腎小管細胞標記物的存在顯示。在另一個實例中,所述腎小管細胞表型可是通過一個或多個腎小管細胞標記物的存在顯示。所述腎小管細胞標記物包括但不限於並轉運給陰離子蛋白、cubilin、透明質酸酶2(HAS2)、維生素D3 25-羥化酶(CYP2D25)、N-鈣粘蛋白(Ncad)、E-鈣粘蛋白(Ecad)、水通道蛋白1(Aqp1)、水通道蛋白2(Aqp2)、RAS致癌基因家族成員RAB17(Rab17)、GATA結合蛋白3(Gata3)、含FXYD結構域的離子調節劑4(Fxyd4)、溶質載體家族9(鈉/氫交換器)成員4(Slc9a4)、醛脫氫酶3家族成員B1(Aldh3b1),醛脫氫酶1家族成員A3(Aldh1a3)和鈣蛋白酶-8(Capn8)。在另一個實例中,所述第一細胞群混有幾個腎細胞類型中的至少一種,包括但不限於間質組織源性細胞、腎小管細胞、集合管源性細胞、腎小球源性細胞和/或血液或血管源性細胞。EPO產生腎細胞群可含有與B2和/或B3或富集細胞群(例如,B2+B3+B4/B4’)形成混合物的B4或B4'。 一方面,本發明EPO產生腎細胞群的特徵為EPO表達和對氧有生物反應,以使培養體系中氧含量的降低導致EPO表達的誘導。在一個實例中,EPO產生細胞群富集EPO產生細胞。在一個實例中,相比于有效氧正常大氣(〜21%)水準下培養的細胞群,當細胞群在有效氧水準培養體系中細胞減少條件下培養時,EPO表達被誘導。在一個實例中,相比于正常氧條件下培養的EPO產生細胞,較低氧條件下培養的EPO產生細胞表達較高水準的EPO。一般而言,有效氧降低水準(也被稱為缺氧培養條件)下的細胞培養是指減氧水準相對於有效氧大氣水準下的細胞培養(也被稱為正常或常氧培養條件)降低。在一個實例中,缺氧細胞培養條件包括在約低於1%氧、約低於2%氧、約低於3%氧、約低於4%氧或約低於5%氧條件下培養細胞。在另一個實例中,正常或常氧培養條件包括在約10%氧、約12%氧、約13%氧、約14%氧、約15%氧、約16%氧、約17%氧、約18 %氧、約19%氧、約20%氧或約21%氧條件下培養細胞。 在另一個實例中,得到EPO的誘導或增加表達,並且可以通過在低於約5%有效氧下培養以及將大氣(約21%)氧下培養的細胞與EPO表達水準比較進行觀察。在另一個實例中,通過一種包括細胞在大氣氧(約21%)下培養一段時間的第一培養階段以及有效氧水準降低和相同細胞在低於約5%有效氧下培養的第二階段的方法,在能夠表達EPO的細胞培養中得到EPO誘導。在另一個實例中,對缺氧條件有反應的EPO表達是受HIF1a調控的。本領域的普通技術人員將領會到,本領域已知的其他氧操縱培養條件可用於此處所述的細胞。 一方面,EPO產生哺乳動物細胞富集群的特徵為對灌注條件有生物反應(例如,EPO表達)。在一個實例中,所述灌注條件包括短暫、間歇或連續流體流動(灌注)。在一個實例中,當細胞培養基被間歇地或連續地流通或攪動,以這種方式通過流動使動力轉移到細胞時,EPO表達被機械地誘導。在一個實例中,對經過短暫、間歇或連續流體流動的細胞進行培養,以這種方式使它們作為三維結構存在于提供形成這種三維結構的框架和/或空間的材料上。在一個實例中,所述細胞在多孔珠上培養,並通過搖擺平臺、軌道平臺或旋轉瓶經過間歇或連續流體流動。在另一個實例中,所述細胞培養於三維支架上,並置於設備上,據此,支架被固定,流體通過或橫過支架定向流動。本領域的普通技術人員將領會到,本領域已知的其他灌注條件可用於此處所述的細胞。 失活細胞群 正如此處所述,本發明在某種程度上基於富集生物活性成分和除去失活或不需要成分的某些異質腎細胞群亞組分的驚人發現,提供比起始群更好的治療和再生結果。在優選實例中,本發明的細胞群被除去B1和/或B5細胞群。例如,以下可被除去B1和/或B5:B2、B3和B4中的兩種或多種混合物;B2、B3和B4'的富集細胞群。 B1細胞群包含集合管和浮力密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群。B5細胞群是由碎片和浮力密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞組成。 分離和培養細胞群的方法 一方面,本發明提供了用於治療(包括腎病、貧血症、EPO缺乏症、腎小管運輸缺乏症和腎小球過濾缺乏症的治療)的分隔和分離腎細胞成分(例如,富集細胞群)的方法。在一個實例中,所述細胞群是從新鮮消化的組織分離,即機械或酶消化的腎組織或異質性哺乳動物腎細胞體外培養。 出乎意料地發現,在密度梯度分離前於缺氧培養條件下對腎細胞異質混合物進行培養提供了包括B4'和B2和/或B3組分的B4的增強分佈和組成。從患病和非患病腎分離的腎細胞觀察到B2-B4中的氧依賴性細胞富集。如果不希望被理論束縛,這可是由於以下現象的一種或多種:1)特定細胞成分在缺氧培養期間的選擇性生存、死亡或增殖;2)為了應對缺氧培養,改變細胞粒度和/或大小,從而在密度梯度分離期間影響浮力密度和隨後定位的改變;和3)為了應對缺氧培養,改變細胞基因/蛋白表達,從而導致任何在給定梯度成分內細胞的差別特性。因此,在一個實例中,富集腎小管細胞的細胞群,例如,B2是抗缺氧的。 用於分隔和分離本發明細胞群的典型方法包括基於目標群內所含不同細胞類型的差異比重的密度梯度分離。任何給定細胞類型的比重可受到細胞內粒度範圍、細胞內水量以及其他因素的影響。一方面,本發明提供了用於分離本發明細胞製劑(例如,包括B4'的B2和B4)的最佳梯度條件,跨越多個物種,包括但不限於人類、犬科動物和齧齒動物。在一個優選實例中,密度梯度被用於獲得一種腎小管細胞組分的新富集群,即,來自腎細胞異質群的B2細胞群。在一個實例中,密度梯度被用於獲得一種EPO產生細胞組分的新富集群,即來自腎細胞異質群的B4細胞群。在其他實例中,密度梯度被用於獲得腎小管細胞、腎小球細胞和腎內皮細胞的富集亞群。在一個實例中,EPO產生細胞和腎小管細胞都是從紅細胞和細胞碎片中分離。在一個實例中,EPO產生細胞、腎小球細胞和血管細胞是從其他細胞類型和紅細胞以及細胞碎片中分離,而腎小管細胞和集合管細胞亞群附隨地從其他細胞類型和紅細胞以及細胞碎片中分離。在另一個實例中,內分泌細胞、腎小球細胞和/或血管細胞是從其他細胞類型和紅血細胞以及細胞碎片中分離,而腎小管細胞和集合管細胞亞群附隨地從其他細胞類型和紅細胞以及細胞碎片中分離。 通過某種程度上採用OPTIPREP®((Axis-Shield)密度梯度培養基的新細胞群產生的本發明,其中包括60%的非離子型碘化合物碘克沙醇水溶液,基於下述某些重要特徵。然而,本領域的普通技術人員將認識到,任何密度梯度或其他方法,例如,根據本發明包含分離本發明細胞群必要特性的本領域已知細胞表面標記物的免疫分離可用到。本領域的普通技術人員還應該認識到,通過密度梯度(大小和粒度)有助於細胞亞群分離的相同細胞特性可以通過流式細胞術(前散射=通過流式細胞術的大小反射,側散射=粒度反射)被用來分離。重要的是,密度梯度培養基應該對特定目標細胞的毒性低。而密度梯度培養基應該對特定目標細胞的毒性低,本發明應考慮在目標細胞選擇過程中起作用的梯度培養基的用途。如果不希望被理論束縛,通過包含碘克沙醇的梯度回收本發明的細胞群似乎是抗碘克沙醇的,因為負載和回收步驟間有明顯的細胞損失,這表明在梯度條件下接觸碘克沙醇導致某些細胞的除去。出現于碘克沙醇梯度後特異帶中的細胞對碘克沙醇和/或密度梯度接觸的任何不良反應產生抗性。因此,本發明還應考慮本發明細胞群分離和/選擇中輕度至中度腎毒素的其他對照培養基的用途。此外,密度梯度培養基應該也不會結合人類血漿或不利地影響目標細胞的主要功能。 在另一方面,本發明提供了採用螢光啟動細胞分選(FACS)富集和/或除去腎細胞類型的方法。在一個實例中,腎細胞類型可富集和/或除去BD FACSAria™或等價物。 在另一方面,本發明提供了採用磁性細胞分選富集和/或除去腎細胞類型的方法。在一個實例中,腎細胞類型可富集和/或除去Miltenyi autoMACS®系統或等價物。 在另一方面,本發明提供了腎細胞群的三維培養方法。一方面,本發明提供了通過連續灌注培養細胞群的方法。在一個實例中,當與靜態培養的細胞群相比時,通過三維培養和連續灌注培養的細胞群表現出更大的細胞性和相互連接性。在另一個實例中,當與這些細胞群的靜態培養相比時,通過三維培養和連續灌注培養的細胞群表現出EPO的較高表達以及腎小管相關性基因(如E-鈣粘蛋白)的增強表達。 在又一個實例中,當與靜態培養的細胞群相比時,通過連續灌注培養的細胞群表現出更高水準的葡萄糖和穀氨醯胺消耗量。 如此處所述(包括例3),低氧或缺氧條件可用於本發明細胞群的製備方法。然而,本發明的方法可被使用,而不需要低氧條件的步驟。在一個實例中,常氧條件可被使用。 本領域的普通技術人員將領會到,本領域已知的其他分離和培養方法可用於此處所述的細胞。 生物材料(聚合物基質或支架) 如Bertram et al.第20070276507號美國公佈專利申請(在此其全部內容通過引用併入本文)所述,聚合物基質或支架可被製成任何數量的可取配置,以滿足任何數量的整體系統、幾何形狀和空間限制。在一個實例中,本發明的基質或支架可是三維的,並被製成為符合器官或組織結構的尺寸和形狀。例如,在採用聚合物支架治療腎病、貧血症、EPO缺乏症、腎小管運輸缺乏症或腎小球過濾缺乏症中,可使用三維(3-D)基質。可使用多種不同形狀的3-D支架。自然地,聚合物基質可被製成不同大小和形狀,以符合不同大小的患者。所述聚合物基質形狀還可以其他方式製成,以滿足患者的特殊需要。在另一個實例中,所述聚合物基質或支架可是生物相容性多孔聚合物支架。所述支架可由各種合成或天然存在的材料構成,包括但不限於,開放式細胞聚乳酸(OPLA®)、纖維素醚、纖維素、纖維素酯、氟化聚乙烯、酚醛樹脂、聚-4-甲基戊烯、聚丙烯腈、聚醯胺、聚醯胺醯亞胺、聚丙烯酸酯、聚苯並噁唑、聚碳酸酯、聚醚腈、聚酯、聚酯碳酸酯、聚醚、聚醚醚酮、聚醚醯亞胺、聚醚酮、聚醚碸、聚乙烯、聚氟代烯、聚醯亞胺、聚烯烴、聚噁二唑、聚苯醚、聚苯硫醚、聚丙烯、聚苯乙烯、聚硫化物、聚碸、聚四氟乙烯、聚硫醚、聚三唑、聚氨酯、聚乙烯化合物、聚偏二氟乙烯、再生纖維素、矽樹脂、脲醛、膠原、層粘連蛋白、纖連蛋白、絲綢、彈性蛋白、海藻酸鹽、透明質酸、瓊脂糖或其共聚物或物理混合物。支架配置範圍可為水凝膠懸浮液至軟性多孔支架或剛性成形多孔支架。 水凝膠可是由各種聚合物材料,並用於各種生物醫學應用中。水凝膠可以被描述為三維網狀親水性聚合物。根據水凝膠類型的不同,它們含有不同比例的水,但完全不溶于水。儘管他們的含水量高,水凝膠能夠另外結合大量的液體,這是由於親水性殘基的存在。水凝膠膨脹很大,但不改變其凝膠狀結構。根據所用聚合物和其他特殊產品設備的性能,水凝膠的基本物理特性可以被特定地改變。 優選地,所述水凝膠是由聚合物、生物源性材料、合成源性材料或其組合組成,即與哺乳動物組織形成生物學上惰性和生理學上相容性。所述水凝膠材料優選為不引起炎症反應。可用于形成水凝膠的其他材料實例包括:(a)改性海藻酸鹽,(b)通過與單價陽離子接觸膠化的多糖(例如,結冷膠和角叉菜膠),(c)多糖(例如,透明質酸)是非常粘稠的液體或者是觸變的,並隨著時間推移通過結構的緩慢變化形成凝膠,和(d)聚合水凝膠前體(例如,聚乙烯氧化聚丙二醇嵌段共聚物和蛋白質)。第62/24893 B1號美國專利提供了各種聚合物以及根據本發明適用於製備水凝膠的這些聚合物的化學性質的詳細描述。 支架或生物材料特性可使細胞能夠與支架或生物材料結合和相互作用,和/或可提供細胞可以包埋的多孔空間。在一個實例中,本發明的多孔支架或生物材料允許細胞的一種或多種群或混合物添加或沉積於被配置為多孔支架的生物材料上(例如,通過細胞粘附)和/或支架氣孔內(例如,通過細胞包埋)。在另一個實例中,所述支架或生物材料允許或促進支架內細胞與細胞和/或細胞與生物材料之間的相互作用,以形成此處所述的構建體。 在一個實例中,根據本發明的所用生物材料是由水凝膠形式的透明質酸(HA)組成,其中含有大小為5.1 kDA至>2 x 106
kDa的HA分子。在另一個實例中,根據本發明的所用生物材料是由多孔泡沫形式的透明質酸組成,其中也含有大小為5.1 kDA至>2 x 106
kDa的HA分子。在又一個實例中,根據本發明的所用生物材料是由聚乳酸(PLA)基泡沫組成,具有開放的細胞結構和約50微米至約300微米的孔徑。在又一個實例中,特定細胞群,優選為B2且B4,通過透明質酸合酶-2(HAS-2)直接提供和/或促進高分子量透明質酸的合成,特別是在腎內植入後。 本領域的普通技術人員將領會到,本領域已知的其他合成或天然存在的材料可用于形成此處所述的支架。 一方面,本發明提供了由上面提到的支架或生物材料製成的此處所述的構建體。 構建體 一方面,本發明提供了具有用於治療需要個體的腎病、貧血症或EPO缺乏症的此處所述的一種或多種細胞群的植入式構建體。在一個實例中,所述構建體是由生物相容性材料或生物材料、一種或多種合成或天然存在的生物相容性材料組成的支架或基質以及通過粘附和/或包埋沉積或嵌入於表面的此處所述的一種或多種細胞群或混合物構成。在某些實例中,所述構建體是由生物材料和此處所述包覆、沉積、粘附、包埋、嵌入或結合于生物材料成分的一種或多種細胞群或混合物構成。此處所述包括富集細胞群或其混合物的任何細胞群,可用於與基質結合,以形成構建體。 在另一個實例中,所述構建體的沉積細胞群或細胞成分是富集氧可調EPO產生細胞的第一腎細胞群。在另一個實例中,所述第一腎細胞群含有除氧可調EPO產生細胞之外的腎小球和血管細胞。在一個實例中,所述第一腎細胞群為B4'細胞群。在另一個實例中,所述構建體的沉積細胞群或細胞成分包括第一富集腎細胞群和第二腎細胞群。在一些實例中,所述第二細胞群並不富集氧可調EPO產生細胞。在另一個實例中,所述第二細胞群富集腎小管細胞。在另一個實例中,所述第二細胞群富集腎小管細胞,並含有集合管上皮細胞。在其他實例中,所述腎小管細胞的特徵為一種或多種腎小管細胞標記物的表達,可包括但不限於並轉運給陰離子蛋白、cubilin、透明質酸酶2(HAS2)、維生素D3 25-羥化酶(CYP2D25)、N-鈣粘蛋白(Ncad)、E-鈣粘蛋白(Ecad)、水通道蛋白1(Aqp1)、水通道蛋白2 (Aqp2)、RAS致癌基因家族成員RAB17(Rab17)、GATA結合蛋白3(Gata3)、含FXYD結構域的離子調節劑4(Fxyd4)、溶質載體家族9(鈉/氫交換器)成員4(Slc9a4)、醛脫氫酶3家族成員B1(Aldh3b1)、醛脫氫酶1家族成員A3(Aldh1a3)和鈣蛋白酶-8(Capn8)。 在一個實例中,沉積於生物材料或支架上或與生物材料或支架結合以形成本發明構建體的細胞群來自各種來源,如自體來源。非自體來源也適合使用,包括但不限於,異基因或同基因(自體或同系)來源。 本領域的普通技術人員將領會到,有幾種將細胞群沉積於生物材料上或與生物材料結合以形成構建體的合適方法。 一方面,本發明的構建體適用於此處所述的方法。在一個實例中,所述構建體適合於向患有任何病因的腎病、貧血症或任何病因的EPO缺乏症的需要治療的個體給藥。在其他實例中,所述構建體適合於向需要改善或恢復紅細胞平衡的個體給藥。在另一個實例中,所述構建體適合於向需要改善腎功能的個體給藥。 在又一方面,本發明提供了一種向需要改善腎功能的個體植入的構建體,其中包含: a)一個包含一種或多種生物相容性合成聚合物或天然存在蛋白質或多肽的生物材料;和 b)來源於患有腎病個體的哺乳動物腎細胞的混合物,其中包含第一細胞群、包含具有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞富集群的B2、第二細胞群和包含紅血球生成素(EPO)產生細胞和血管細胞但除去具有密度介於1.063克/毫升和1.091克/毫升之間的腎小球細胞的B4’,包覆、沉積、包埋、懸浮、嵌入和/或其反面結合有生物材料。在某些實例中,所述混合物不包括:包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群或者包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。 在一個實例中,所述構建體包括以血管標記物表達為特徵的B4'細胞群。在一些實例中,所述B4'細胞群不以腎小球標記物的表達為特徵。在某些實例中,所述混合物能夠氧可調紅血球生成素(EPO)表達。在所有實例中,所述混合物可來源於哺乳動物腎組織或培養的腎細胞。 在一個實例中,所述構建體包括被配置為適合於混合物包埋和/或粘附的三維(3-D)多孔性生物材料的生物材料。在另一個實例中,所述構建體包括被配置為適合於植入、粘附、懸浮或包覆哺乳動物細胞的半液體凝膠的生物材料。在又一個實例中,所述構建體包括主要是由水凝膠形式的高分子量透明質酸(HA)組成配置的生物材料。在另一個實例中,所述構建體包括主要是由多孔泡沫形式的高分子量透明質酸(HA)組成配置的生物材料。在又一個實例中,所述構建體包括具有孔徑介於約50微米和約300微米之間的聚乳酸基泡沫組成的的生物材料。在又一個實例中,所述構建體包括可來源於需要改善腎功能個體的自體腎樣本的一種或多種細胞群。在一些實例中,所述樣本為腎活檢。在一些實例中,所述個體患有腎病。在其他實例中,所述細胞群來源於非自體腎樣本。在一個實例中,所述構建體提供了紅細胞動態平衡。 分泌產物 在另一方面,本發明涉及到此處所述的富集腎細胞群或富集腎細胞群的混合物分泌的產物。在一個實例中,所述產物包括以下的一種或多種:旁分泌因數、內分泌因數、鄰分泌因數和囊泡。所述囊泡可包括以下的一種或多種:旁分泌因數、內分泌因數、鄰分泌因數、微囊泡、外泌體和RNA。分泌產物還可包括但不限於微囊泡的產物,其中包括但不限於旁分泌因數、內分泌因數、鄰分泌因數和RNA。例如,已在囊泡外發現胞外miRNA (Wang et al., Nuc Acids Res 2010, 1-12 doi:10.1093/nar/gkq601, July 7, 2010)。所述分泌產物還被稱為細胞源性產物,例如,細胞源性囊泡。 在另一個實例中,所述分泌產物可是來源於腎細胞的一部分。所述囊泡可能夠將因數傳送到其他目的地。在一個實例中,所述囊泡為分泌囊泡。正考慮的幾種分泌囊泡包括但不限於,外泌體、微囊泡、核外顆粒體、膜顆粒,外泌體狀囊泡和細胞凋亡囊泡(Thery et al. 2010. Nat. Rev. Immunol. 9:581-593)。在一個實例中,所述分泌囊泡為外泌體。在另一個實例中,所述分泌囊泡為微囊泡。在另一個實例中,所述分泌囊泡含有或包含一種或多種細胞成分。所述成分可為以下的一種或幾種:脂質膜、RNA、蛋白質、代謝物、細胞內成分及其任何組合。在一個優選實例中,所述分泌囊泡包含、組成或基本組成為微型RNA。優選地,所述微型RNA為人類miRNA。在一個實例中,一種或多種miRNA選自由miR-30b-5p、miR-449a、miR-146a、miR-130a、miR-23b、miR-21、miR-124和miR-151組成的組。在另一個實例中,所述一種或多種miRNA可選自由let-7a-1; let-7a-2; let-7a-3; let-7b; let-7c; let-7d; let-7e; let-7f-1; let-7f-2; let-7g; let-7i; mir-1-1; mir-1-2; mir-7-1; mir-7-2; mir-7-3; mir-9-1; mir-9-2; mir-9-3; mir-10a; mir-10b; mir-15a; mir-15b; mir-16-1; mir-16-2; mir-17; mir-18a; mir-18b; mir-19a; mir-19b-1; mir-19b-2; mir-20a; mir-20b; mir-21; mir-22; mir-23a; mir-23b; mir-23c; mir-24-1; mir-24-2; mir-25; mir-26a-1; mir-26a-2; mir-26b; mir-27a; mir-27b; mir-28; mir-29a; mir-29b-1; mir-29b-2; mir-29c; mir-30a; mir-30b; mir-30c-1; mir-30c-2; mir-30d; mir-30e; mir-31; mir-32; mir-33a; mir-33b; mir-34a; mir-34b; mir-34c; mir-92a-1; mir-92a-2; mir-92b; mir-93; mir-95; mir-96; mir-98; mir-99a mir-99b; mir-100; mir-101-1; mir-101-2; mir-103-1; mir-103-1-as; mir-103-2; mir-103-2-as; mir-105-1; mir-105-2; mir-106a; mir-106b; mir-107; mir-122; mir-124-1; mir-124-2; mir-124-3; mir-125a; mir-125b-1; mir-125b-2; mir-126; mir-127; mir-128-1; mir-128-2; mir-129-1; mir-129-2; mir-130a; mir-130b; mir-132; mir-132; mir-133a-1; mir-133a-2; mir-133b; mir-134; mir-135a-1; mir-135a-2; mir-135b; mir-136 MI101351120; mir-137; mir-138-1; mir-138-2; mir-139; mir-140; mir-141; mir-142; mir-143; mir-144; mir-145; mir-146a; mir-146b; mir-147; mir-147b; mir-148a; mir-148b; mir-149; mir-150; mir-151; mir-152; mir-153-1; mir-153-2; mir-154; mir-155; mir-181a-1; mir-181a-2; mir-181b-1; mir-181b-2; mir-181c; mir-181d; mir-182; mir-183; mir-184; mir-185; mir-186; mir-187; mir-188; mir-190; mir-190b; mir-191; mir-192; mir-193a; mir-193b; mir-194-1; mir-194-2; mir-195; mir-196a-1; mir-196a-2; mir-196b; mir-197; mir-198; mir-199a-1; mir-199a-2; mir-199b; mir-200a; mir-200b; mir-200c; mir-202; mir-203; mir-204; mir-205; mir-206; mir-208a; mir-208b; mir-210; mir-211; mir-212; mir-214; mir-215; mir-216a; mir-216b; mir-217; mir-218-1; mir-218-2; mir-219-1; mir-219-2; mir-221; mir-222; mir-223; mir-224; mir-296; mir-297; mir-298; mir-299; mir-300; mir-301a; mir-301b; mir-302a; mir-302b; mir-302c; mir-302d; mir-302e; mir-302f; mir-320a; mir-320b-1; mir-320b-2; mir-320c-1; mir-320c-2; mir-320d-1; mir-320d-2; mir-320e; mir-323; mir-323b; mir-324; mir-325; mir-326; mir-328; mir-329-1; mir-329-2; mir-330; mir-331; mir-335; mir-337; mir-338; mir-339; mir-340; mir-342; mir-345; mir-346; mir-361; mir-362; mir-363; mir-365-1; mir-365-2; mir-367; mir-369; mir-370; mir-37; mir-372; mir-373; mir-374a; mir-374b; mir-374c; mir-375; mir-376a-1; mir-376a-2; mir-376b; mir-376c; mir-377; mir-378; mir-378b; mir-378c; mir-379; mir-380; mir-381; mir-382; mir-383; mir-384; mir-409; mir-410; mir-411; mir-412; mir-421; mir-422a; mir-423; mir-424; mir-425; mir-429; mir-431; mir-432; mir-433; mir-448; mir-449a; mir-449b; mir-449c; mir-450a-1; mir-450a-2; mir-450b; mir-451; mir-452; mir-454; mir-455; mir-466; mir-483; mir-484; mir-485; mir-486; mir-487a; mir-487b; mir-488; mir-489; mir-490; mir-491; mir-492; mir-493; mir-494; mir-495; mir-496; mir-497; mir-498; mir-499; mir-500a; mir-500b; mir-501; mir-502; mir-503; mir-504; mir-505; mir-506; mir-507; mir-508; mir-509-1; mir-509-2; mir-509-3; mir-510; mir-511-1; mir-511-2; mir-512-1; mir-512-2; mir-513a-1; mir-513a-2; mir-513b; mir-513c; mir-514-1; mir-514-2; mir-514-3; mir-514b; mir-515-1; mir-515-2; mir-516a-1; mir-516a-2; mir-516b-1; mir-516b-2; mir-517a; mir-517b; mir-517c; mir-518a-1; mir-518a-2; mir-518b; mir-518c; mir-518d; mir-518e; mir-518f; mir-519a-1; mir-519a-2; mir-519b; mir-519c; mir-519d; mir-519e; mir-520a; mir-520b; mir-520c; mir-520d; mir-520e; mir-520f; mir-520g; mir-520h; mir-521-1; mir-521-2; mir-522; mir-523; mir-524; mir-525; mir-526a-1; mir-526a-2; mir-526b; mir-527; mir-532; mir-539; mir-541; mir-542; mir-543; mir-544; mir-544b; mir-545; mir-548a-1; mir-548a-2; mir-548a-3; mir-548aa-1; mir-548aa-2; mir-548b; mir-548c; mir-548d-1; mir-548d-2; mir-548e; mir-548f-1; mir-548f-2; mir-548f-3; mir-548f-4; mir-548f-5; mir-548g; mir-548h-1; mir-548h-2; mir-548h-3; mir-548h-4; mir-548i-1; mir-548i-2; mir-548i-3; mir-548i-4; mir-548j; mir-548k; mir-548l; mir-548m; mir-548n; mir-548o; mir-548p; mir-548s; mir-548t; mir-548u; mir-548v; mir-548w; mir-548x; mir-548y; mir-548z; mir-549; mir-550a-1; mir-550a-2; mir-550b-1; mir-550b-2; mir-551a; mir-551b; mir-552; mir-553; mir-554; mir-555; mir-556; mir-557; mir-558; mir-559; mir-561; mir-562; mir-563; mir-564; mir-566; mir-567; mir-568; mir-569; mir-570; mir-571; mir-572; mir-573; mir-574; mir-575; mir-576; mir-577; mir-578; mir-579; mir-580; mir-581; mir-582; mir-583; mir-584; mir-585; mir-586; mir-587; mir-588; mir-589; mir-590; mir-591; mir-592; mir-593; mir-595; mir-596; mir-597; mir-598; mir-599; mir-600; mir-601; mir-602; mir-603; mir-604; mir-605; mir-606; mir-607; mir-608; mir-609; mir-610; mir-611; mir-612; mir-613; mir-614; mir-615; mir-616; mir-617; mir-618; mir-619; mir-620; mir-621; mir-622; mir-623; mir-624; mir-625; mir-626; mir-627; mir-628; mir-629; mir-630; mir-631; mir-632; mir-633; mir-634; mir-635; mir-636; mir-637; mir-638; mir-639; mir-640; mir-641; mir-642a; mir-642b; mir-643; mir-644; mir-645; mir-646; mir-647; mir-648; mir-649; mir-650; mir-651; mir-652; mir-653; mir-654; mir-655; mir-656; mir-657; mir-658; mir-659; mir-660; mir-661; mir-662; mir-663; mir-663b; mir-664; mir-665; mir-668; mir-670; mir-671; mir-675; mir-676; mir-708; mir-711; mir-718; mir-720; mir-744; mir-758; mir-759; mir-760; mir-761; mir-762; mir-764; mir-765; mir-766; mir-767; mir-769; mir-770; mir-802; mir-873; mir-874; mir-875; mir-876; mir-877; mir-885; mir-887; mir-888; mir-889; mir-890; mir-891a; mir-891b; mir-892a; mir-892b; mir-920; mir-921; mir-922; mir-924; mir-933; mir-934; mir-935; mir-936; mir-937; mir-938; mir-939; mir-940; mir-941-1; mir-941-2; mir-941-3; mir-941-4; mir-942; mir-942; mir-943; mir-944; mir-1178; mir-1179; mir-1180; mir-1181; mir-1182; mir-1183; mir-1184-1; mir-1184-2; mir-1184-3; mir-1185-1; mir-1185-2; mir-1193; mir-1197; mir-1200; mir-1202; mir-1203; mir-1204; mir-1205; mir-1206; mir-1207; mir-1208; mir-1224; mir-1225; mir-1226; mir-1227; mir-1228; mir-1229; mir-1231; mir-1233-1; mir-1233-2; mir-1234; mir-1236; mir-1237; mir-1238; mir-1243; mir-1244-1; mir-1244-2; mir-1244-3; mir-1245; mir-1246; mir-1247; mir-1248; mir-1249; mir-1250; mir-1251; mir-1252; mir-1253; mir-1254; mir-1255a; mir-1255b-1; mir-1255b-2; mir-1256; mir-1257; mir-1258; mir-1260; mir-1260b; mir-1261; mir-1262; mir-1263; mir-1264; mir-1265; mir-1266; mir-1267; mir-1268; mir-1269; mir-1270-1; mir-1270-2; mir-1271; mir-1272; mir-1273; mir-1273c; mir-1273d; mir-1273e; mir-1274a; mir-1274b; mir-1275; mir-1276; mir-1277; mir-1278; mir-1279; mir-1280; mir-1281; mir-1282; mir-1283-1; mir-1283-2; mir-1284; mir-1285-1; mir-1285-2; mir-1286; mir-1287; mir-1288; mir-1289-1; mir-1289-2; mir-1290; mir-1291; mir-1292; mir-1293; mir-1294; mir-1295; mir-1296; mir-1297; mir-1298; mir-1299; mir-1301; mir-1302-1; mir-1302-10; mir-1302-11; mir-1302-2; mir-1302-3; mir-1302-4; mir-1302-5; mir-1302-6; mir-1302-7; mir-1302-8; mir-1302-9; mir-1303; mir-1304; mir-1305; mir-1306; mir-1307; mir-1321; mir-1322; mir-1323; mir-1324; mir-1468; mir-1469; mir-1470; mir-1471; mir-1537; mir-1538; mir-1539; mir-1825; mir-1827; mir-1908; mir-1909; mir-1910; mir-1911; mir-1912; mir-1913; mir-1914; mir-1915; mir-1972-1; mir-1972-2; mir-1973; mir-1976; mir-2052; mir-2053; mir-2054; mir-2110; mir-2113; mir-2114; mir-2115; mir-2116; mir-2117; mir-2276; mir-2277; mir-2278; mir-2355; mir-2861; mir-2909; mir-3065; mir-3074; mir-3115; mir-3116-1; mir-3116-2; mir-3117; mir-3118-1; mir-3118-2; mir-3118-3; mir-3118-4; mir-3118-5; mir-3118-6; mir-3119-1;mir-3119-2; mir-3120; mir-3121; mir-3122; mir-3123; mir-3124; mir-3125; mir-3126; mir-3127; mir-3128; mir-3129; mir-3130-1; mir-3130-2; mir-3131; mir-3132; mir-3133; mir-3134; mir-3135; mir-3136; mir-3137; mir-3138; mir-3139; mir-3140; mir-3141; mir-3142; mir-3143; mir-3144; mir-3145; mir-3146; mir-3147; mir-3148; mir-3149; mir-3150; mir-3151; mir-3152; mir-3153; mir-3154; mir-3155; mir-3156-1; mir-3156-2; mir-3156-3; mir-3157; mir-3158-1; mir-3158-2; mir-3159; mir-3160-1; mir-3160-2; mir-3161; mir-3162; mir-3163; mir-3164; mir-3165; mir-3166; mir-3167; mir-3168; mir-3169; mir-3170; mir-3171; mir-3173; mir-3174; mir-3175; mir-3176; mir-3177; mir-3178; mir-3179-1; mir-3179-2; mir-3179-3; mir-3180-1; mir-3180-2; mir-3180-3; mir-3180-4; mir-3180-5; mir-3181; mir-3182; mir-3183; mir-3184; mir-3185; mir-3186; mir-3187; mir-3188; mir-3189; mir-3190; mir-3191; mir-3192; mir-3193; mir-3194; mir-3195; mir-3196; mir-3197; mir-3198; mir-3199-1; mir-3199-2; mir-3200; mir-3201; mir-3202-1; mir-3202-2; mir-3605; mir-3606; mir-3607; mir-3609; mir-3610; mir-3611; mir-3612; mir-3613; mir-3614; mir-3615; mir-3616; mir-3617; mir-3618; mir-3619; mir-3620; mir-3621; mir-3622a; mir-3622b; mir-3646; mir-3647; mir-3648; mir-3649; mir-3650; mir-3651; mir-3652; mir-3653; mir-3654; mir-3655; mir-3656mir-3657; mir-3658; mir-3659; mir-3660; mir-3661; mir-3662; mir-3663; mir-3664; mir-3665; mir-3666; mir-3667; mir-3668; mir-3669; mir-3670; mir-3670; mir-3671; mir-3671; mir-3673; mir-3673; mir-3675; mir-3675; mir-3676; mir-3663; mir-3677; mir-3678; mir-3679; mir-3680; mir-3681; mir-3682; mir-3683; mir-3684; mir-3685; mir-3686; mir-3687; mir-3688; mir-3689a; mir-3689b; mir-3690; mir-3691; mir-3692; mir-3713; mir-3714; mir-3907; mir-3908; mir-3909; mir-3910-1; mir-3910-2; mir-3911; mir-3912; mir-3913-1; mir-3913-2; mir-3914-1; mir-3914-2; mir-3915; mir-3916; mir-3917; mir-3918; mir-3919; mir-3920; mir-3921; mir-3922; mir-3923; mir-3924; mir-3925; mir-3926-1; mir-3926-2; mir-3927; mir-3928; mir-3929; mir-3934; mir-3935; mir-3936; mir-3937; mir-3938; mir-3939; mir-3940; mir-3941; mir-3942; mir-3943; mir-3944; mir-3945; mir-4251; mir-4252; mir-4253; mir-4254; mir-4255; mir-4256; mir-4257; mir-4258; mir-4259; mir-4260; mir-4261; mir-4262; mir-4263; mir-4264; mir-4265; mir-4266; mir-4267; mir-4268; mir-4269; mir-4270; mir-4271; mir-4272; mir-4273; mir-4274; mir-4275; mir-4276; mir-4277; mir-4278; mir-4279; mir-4280; mir-4281; mir-4282; mir-4283-1; mir-4283-2; mir-4284; mir-4285; mir-4286; mir-4287; mir-4288; mir-4289; mir-4290; mir-4291; mir-4292; mir-4293; mir-4294; mir-4295; mir-4296; mir-4297; mir-4298; mir-4299; mir-4300; mir-4301; mir-4302; mir-4303; mir-4304; mir-4305; mir-4306; mir-4307; mir-4308; mir-4309; mir-4310; mir-4311; mir-4312; mir-4313; mir-4314; mir-4315-1; mir-4315-2; mir-4316; mir-4317; mir-4318; mir-4319; mir-4320; mir-4321; mir-4322; mir-4323; mir-4324; mir-4325; mir-4326; mir-4327; mir-4328;mir-4329; mir-4329; 和mir-4330組成的組。 本發明涉及到從此處所述的細胞群或構建體獲得的細胞源性或分泌性miRNA。在一個實例中,一種或多種個體miRNA可用於提供一種自體腎的再生效果。個體miRNA的組合可適合於提供這種效果。典型組合包括以下的兩種或多種:miR-21;miR-23a;miR-30c;miR-1224;miR-23b;miR-92a;miR-100;miR-125b-5p;miR-195;miR-10a-5p及其任何組合。另一個典型組合包括以下的兩種或多種:miR-30b-5p、miR-449a、miR-146a、miR-130a、miR-23b、miR-21、miR-124、miR-151及其任何組合。在一個實例中,miRNA的組合可包括2、3、4、5、6、7、8、9、10或多種個體miRNA。本領域的普通技術人員將領會到,其他miRNA和miRNA組合可適用於本發明。其他miRNA的來源包括英國曼徹斯特大學生命科學學院主頁https://mirbase.org上的miRBase。 在一個實例中,所述分泌產物是由旁分泌因數組成。一般而言,旁分泌因數是由在小距離旁內擴散以誘導或改變鄰近細胞(即,旁分泌作用)的細胞合成的分子。所述擴散分子被稱為旁分泌因數。 在又一個實例中,本發明涉及此處所述的一種或多種分離的腎細胞源性分泌囊泡的組合物。本領域的普通技術人員將會明白含有分泌囊泡的各種組合物將是合適的。 在另一方面,本發明提供了製備腎細胞分泌產物(例如,囊泡)的方法。在一個實例中,所述方法包括提供腎細胞群的步驟,所述腎細胞群包括一種或多種富集腎細胞群。在另一個實例中,所述方法進一步包括在適當的條件下培養群的步驟。所述條件可為低氧條件。在另一個實例中,所述方法進一步包括從腎細胞群分離分泌產物的步驟。所述分泌囊泡可是從細胞群的細胞培養基獲得。在另一個實例中,所述腎細胞的特徵為對氧水準有生物反應的囊泡產生和/或分泌,這樣導致誘導囊泡產生和/或分泌的培養體系的氧含量減少。在一個實例中,相比于有效氧正常大氣(〜21%)水準下培養的細胞群,當細胞群在有效氧水準培養體系中細胞減少條件下培養時,囊泡產生和/或分泌被誘導。在一個實例中,相比于正常氧條件下培養的細胞群,較低氧條件下培養的細胞群產生和/或分泌較高水準的囊泡。一般而言,有效氧降低水準(也被稱為缺氧培養條件)下的細胞培養是指減氧水準相對於有效氧大氣水準下的細胞培養(也被稱為正常或常氧培養條件)降低。在一個實例中,所述缺氧細胞培養條件包括在約低於1%氧、約低於2%氧、約低於3%氧、約低於4%氧或約低於5%氧條件下培養細胞。在另一個實例中,正常或常氧培養條件包括在約10%氧、約12%氧、約13%氧、約14%氧、約15%氧、約16%氧、約17%氧、約18%氧、約19%氧、約20%氧或約21%氧條件下培養細胞。在一個優選實例中,所述方法為從腎細胞分離外泌體和/或微囊泡作準備。 在一個實例中,所述產物是由腎細胞分泌的。所述產物可由不在支架上的腎細胞分泌,例如,所述細胞不屬於此處所述的構建體。 在另一個實例中,所述產物是由已接種於支架上(例如,構建體)的腎細胞分泌的。所述構建體包括一種或多種富集腎細胞群或其直接接種於支架上或支架內的混合物。 在另一方面,本發明提供了體外篩選/優化/監控一種或多種富集腎細胞群及其所含混合物或構建體的生物療效的方法。在一個實例中,所述方法包括提供一種或多種測試群、測試混合物或測試構建體(“測試物”)的步驟。在另一個實例中,所述方法包括在適當條件下培養測試物的步驟,如此處所述。在另一個實例中,所述方法包括從培養測試物收集細胞培養基的步驟。這種培養基可被稱為“條件培養基”,預計將含有測試物的腎細胞分泌的產物。 在另一方面,所述條件培養基可用於進行一種或多種體外實驗,以檢測測試物的生物療效。在一個實例中,所述條件培養基進行了上皮細胞間質轉化(EMT)檢測。所述實驗可檢測TGFβ1誘導的EMT。例15提供了本實驗的典型協定。 在另一個實例中,所述條件培養基進行了RNA檢測,例如,通過PCR檢測和/或囊泡或外泌體,例如,通過流式細胞術。在另一個實例中,所述條件培養基進行了信號通路檢測,例如,免疫反應(例如,NFκB)、纖維化反應(PAI-1)和血管生成。例12-14提供了這些實驗的典型協議。 使用方法 一方面,本發明提供了採用此處所述的腎細胞群和腎細胞混合物治療需要個體的腎病、貧血症或EPO缺乏症的方法。在一個實例中,所述方法包括向個體施用包括富集EPO產生細胞的第一腎細胞群的組合物。在另一個實例中,所述第一細胞群富集EPO產生細胞、腎小球細胞和血管細胞。在一個實例中,所述第一腎細胞群為B4'細胞群。在另一個實例中,所述組合物可進一步包括一種或多種其他腎細胞群。在一個實例中,所述其他細胞群為不富集EPO產生細胞的第二細胞群。在另一個實例中,所述其他細胞群為不富集EPO產生細胞、腎小球細胞或血管細胞的第二細胞群。在另一個實例中,所述組合物還包括沉積、連接、嵌入、包覆或包埋於生物材料以形成此處所述構建體的腎細胞群或腎細胞混合物,用於治療此處所述的疾病或障礙。在一個實例中,所述細胞群是單獨使用或與其他細胞或生物材料(例如,水凝膠、多孔支架或自體或人工合成肽或蛋白質)結合使用,以促進急性或慢性疾病狀態下的再生。 在另一方面,通過本發明方法對個體腎病、貧血症或EPO缺乏症的有效治療可以通過紅細胞生成和/或腎功能的各種指標觀察到。在一個實例中,紅細胞動態平衡的指標包括但不限於,紅細胞壓積(HCT)、血紅蛋白(HB)、平均紅細胞血紅蛋白量(MCH)、紅細胞計數(RBC)、網織紅細胞數、網織紅細胞%、平均紅細胞體積(MCV)和紅血細胞分佈寬度(RDW)。在另一個實例中,腎功能的指標包括但不限於,血清白蛋白、白蛋白與球蛋白比值(A / G比值),血清磷、血清鈉、腎臟大小(通過超聲波測量)、血清鈣、磷與鈣比值、血清鉀、蛋白尿、尿肌酐、血清肌酐、血液尿素氮(BUN)、膽固醇水準、甘油三酯水準和腎小球濾過率(GFR)。此外,一般健康和幸福感,包括但不限於,體重增加或減少、生存、血壓(平均全身血壓、舒張壓或收縮壓)和身體忍耐力的幾個指標。 在另一個實例中,有效治療是通過腎功能的一種或多種指標的穩定進行證明。相比於未通過本發明方法治療個體的相同指標,通過本發明方法治療的個體指標變化觀察表明了腎功能的穩定。另外,相比於治療前相同個體的相同指標,通過本發明方法治療的個體指標變化觀察表明了腎功能的穩定。第一指標的變化可是值的增加或減少。在一個實例中,由本發明所提供的治療可包括個體血液尿素氮(BUN)水準的穩定,其中觀察到個體的BUN水準相比於未通過本發明方法治療的具有類似疾病狀態的個體比較低。在另一個實例中,所述治療可包括個體血清肌酐水準的穩定,其中觀察到個體的血清肌酐水準相比於未通過本發明方法治療的具有類似疾病狀態的個體比較低。在另一個實例中,所述治療可包括個體紅細胞壓積(HCT)水準的穩定,其中觀察到個體的HCT水準相比於未通過本發明方法治療的具有類似疾病狀態的個體比較高。在另一個實例中,所述治療可包括個體紅細胞(RBC)水準的穩定,其中觀察到個體的RBC水準相比於未通過本發明方法治療的具有類似疾病狀態的個體比較高。本領域的普通技術人員將領會到,此處所述或本領域已知的一種或多種指標可被測量,以確定個體腎病的有效治療。 在另一方面,本發明涉及一種提供需要個體紅細胞平衡的方法。在一個實例中,所述方法包括以下步驟:(a)向個體施用腎細胞群,例如,此處所述的B2或B4',或腎細胞混合物,例如,B2/B4'和/或B2/B3;及(b)測定從相比於對照指標水準不同的紅細胞生成指標水準的個體得到的生物樣本,其中,指標水準的不同(i)表示個體對步驟(a)有反應,或(ii)表示個體的紅細胞動態平衡。在另一個實例中,所述方法包括以下步驟:(a)向個體施用此處所述的包含腎細胞群或腎細胞混合物的組合物;及(b)測定從相比於對照指標水準不同的紅細胞生成指標水準的個體得到的生物樣本,其中,指標水準的不同(i) 表示個體對步驟(s)有反應,或(ii)表示個體的紅細胞動態平衡。在另一個實例中,所述方法包括以下步驟:(a)提供生物材料或生物相容性聚合物支架;(b)將本發明的腎細胞群或腎細胞混合物沉積於生物材料或支架上或生物材料或支架內,以形成植入式構建體; (c)將構建體植入個體中;及(d)測定從相比於對照指標水準不同的紅細胞生成指標水準的個體得到的生物樣本,其中,指標水準的不同(i)表示個體對步驟(a)有反應,或(ii)表示個體的紅細胞動態平衡。 在另一方面,本發明涉及一種同時向需要個體提供腎功能穩定和恢復紅細胞動態平衡的方法,所述個體同時患有腎功能和貧血症和/或EPO缺乏症。在一個實例中,所述方法包括施用含有以下至少一種細胞類型的此處所述的腎細胞群或腎細胞混合物的步驟:腎小管源性細胞、腎小球源性細胞、間質源性細胞、收集管源性細胞、基質組織源性細胞或血管源性細胞。在另一個實例中,所述群或混合物含有EPO產生細胞及腎小管上皮細胞,腎小管細胞已被以下至少一個標記物確定:並轉運給陰離子蛋白、cubilin、透明質酸酶2(HAS2),維生素D3 25-羥化酶(CYP2D25)和N-鈣粘蛋白(Ncad)、E-鈣粘蛋白(Ecad)、水通道蛋白1(Aqp1)、水通道蛋白2 (Aqp2)、RAS致癌基因家族成員RAB17(Rab17)、GATA結合蛋白3(Gata3)、含FXYD結構域的離子調節劑4(Fxyd4)、溶質載體家族9(鈉/氫交換器)成員4(Slc9a4)、醛脫氫酶3家族成員B1(Aldh3b1),醛脫氫酶1家族成員A3(Aldh1a3)和鈣蛋白酶-8(Capn8)。在這個實例中,相比於未治療個體或預治療個體的指標,個體的治療將通過腎功能至少一個指標的改善伴隨紅細胞生成至少一個指標的改善進行證明。 一方面,本發明提供了以下方法:(i)治療腎病、貧血症或EP缺乏症;(ii)穩定腎功能,(iii) 恢復紅細胞動態平衡,或(iv)通過施用富集EPO產生細胞的腎細胞群或含有富集此處所述的EPO產生細胞的腎細胞混合物的任何組合,其中,所述施用的有益效果大於未富集EPO產生細胞的細胞群。在另一個實例中,所述富集細胞群提供了高水準的血清尿素氮(BUN)。在另一個實例中,所述富集細胞群提供了高保持能力的血清蛋白質。在另一個實例中,所述富集細胞群提供了高水準的血清膽固醇和/或甘油三酯。 在另一個實例中,所述富集細胞群提供了高水準的維生素D。在一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的磷/鈣比。在另一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的血紅蛋白A。在又一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的血清肌酐。在又一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的血細胞壓積。在又一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的紅血細胞數(RBC#)。在一個實例中,高水準的紅細胞壓積被恢復到95%的正常健康水準。在一個進一步實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的網織紅細胞數。在其他實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的網織紅細胞比例。在其他實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的紅細胞容積分佈寬度(RDW)。在另一個實例中,相比于未富集細胞群,所述富集細胞群提供了高水準的血紅蛋白A。在另一個實例中,所述富集細胞群提供了骨髓中的紅細胞生成反應,使得骨髓細胞結構接近正常,骨髓與網織紅細胞之比接近正常。 在另一方面,本發明提供了以下方法:(i)治療腎病、貧血症或EP缺乏症;(ii)穩定腎功能,(iii) 恢復紅細胞動態平衡,或(iv)通過施用富集細胞群的任何組合,其中當與施用重組EPO (rEPO)相比時,施用此處所述的腎細胞群或腎細胞混合物的有益效果以改善的紅細胞動態平衡為特徵。在一個實例中,當與施用重組EPO蛋白相比時,向需要個體施用群或混合物提供了改善的紅動態平衡(通過紅細胞壓積、血紅蛋白或RBC#測定)。在一個實例中,相比於低於或高於對照中紅細胞壓積不超過約10%的重組EPO,向需要個體施用群或混合物提供了高水準的紅細胞壓積、RBC或血紅蛋白A。在又一個實例中,向所治療個體施用單劑量或傳送群或混合物提供改善紅細胞動態平衡(通過紅細胞壓積、血紅蛋白或RBC#測定)的一段時間顯著超過施用單劑量或傳送重組EPO蛋白供改善紅細胞動態平衡的時間。在另一個實例中,施用此處所述的單劑量群或混合物並不會導致紅細胞、血紅蛋白或RBC#大於對應健康對照正常水準的約110%。在又一個實例中,相比於傳送此處所述劑量的重組EPO蛋白,施用此處所述的單劑量群或混合物提供較佳的紅細胞動態平衡(通過紅細胞壓積、血紅蛋白或RBC#測定)。在另一個實例中,所述重組EPO是以約100 IU/kg,、約200 IU/kg、約300 IU/kg、約400 IU/kg或約500 IU/kg的劑量傳送。本領域的普通技術人員將領會到,本領域已知重組EPO的其他劑量可是合適的。 本發明的另一個實例涉及包括此處所述的富集細胞群及其混合物或植入式構建體或分泌產物的至少一種細胞群的用途,用於治療需要個體的腎病、貧血症或EPO缺乏症的藥物製劑、提供需要個體的紅細胞動態平衡、改善需要個體的的腎功能或提供自體腎的再生效果。 本發明的另一個實例涉及到用於治療特定病因腎病的富集細胞群(此處所述),可根據基於特定已證實治療屬性的特定細胞亞群選擇。 在又一方面,本發明提供了一種治療需要個體腎病的方法,其中包括:向個體施用包含第一細胞群的哺乳動物腎細胞、包含具有密度介於1.045克/毫升和1.052克/毫升之間的腎小管細胞分離富集群的B2、第二細胞群以及包含紅血球生成素(EPO)產生細胞和血管細胞但除去密度介於約1.063克/毫升和1.091克/毫升之間的腎小球細胞的B4’的混合物,其中所述混合物不包括:包含有集合管和密度小於1.045克/毫升的管系統的大顆粒細胞的B1細胞群,或包含有碎片和密度大於1.091克/毫升、低粒度和存活率的小顆粒細胞的B5細胞群。在某些實例中,所述方法包括測定從腎功能指標水準相對於對照指標水準不同的個體得到的測試樣本,其中,所述指標水準的差異表示個體一種或多種腎功能下降、穩定或改善的減少。在一個實例中,本方法所用B4'細胞群的特徵為血管標記物的表達。在某些實例中,本方法所用B4'細胞群的特徵不是腎小球標記物的表達。在一個實例中,本方法所用混合物能夠氧可調紅血球生成素(EPO)表達。在某些實例中,通過本方法治療的腎病伴隨有紅血球生成素(EPO)缺乏症。在某些實例中,所述EPO缺乏症為貧血症。在一些實例中,所述EPO缺乏症或貧血症繼發於個體的腎功能衰竭。在其他一些實例中,所述EPO缺乏症或貧血症繼發於慢性腎功能衰竭、原發性EPO缺乏症、化療或抗病毒治療、非骨髓癌、HIV感染、肝病、心力衰竭、類風濕關節炎或多器官功能衰竭。在某些實例中,本方法所用的組合物進一步包含:包含有一種或多種生物相容性合成聚合物/或天然產生蛋白質或多肽的生物材料,其中所述混合物包覆、沉積、包埋、懸浮、嵌入和/或相反結合於生物材料。在某些實例中,本方法所用的混合物來源於哺乳動物腎組織或培養的哺乳動物腎細胞。在其他實例中,所述混合物來源於需要個體自體的腎樣本。在一個實例中,所述樣本為腎活檢。在其他實例中,本發明方法所用的混合物來源於非自體腎樣本。 在又一方面,本發明提供了細胞製劑或其混合物或其用於治療需要個體的腎病、貧血症或EPO缺乏的藥物製劑的本發明的植入式構建體的用途。 在另一個方面,本發明提供了需要個體的自體腎再生的方法。在一個實例中,所述方法包括向個體施用或植入此處所述的細胞群、混合物或構建體的步驟。再生的自體腎可具有的特徵為許多指標,包括但不限於,自體腎功能或能力的發展、自體腎功能或能力的改善以及自體腎某些標記物的表達。在一個實例中,發展或改善的功能或能力可根據上述紅細胞動態平衡和腎臟功能的各項指標觀察到。在另一個實例中,再生腎的特徵為一種或多種幹細胞標記物的表達。所述幹細胞標記物可為以下的一種或多種:SRY基因(性別決定區Y)的盒2(SOX2);未分化胚胎細胞轉錄因數(UTF1);小鼠Nodal同源 (NODAL);Prominin 1(PROM1)或CD133(CD133);CD24;以及其任何組合。在另一個實例中,所述幹細胞標記物的表達相比於對照有所上調。 包括富集細胞群及其混合物,以及含有富集細胞群及其混合物的構建體的此處所述的細胞群,可用於提供自體腎的再生效果。所述效果可通過細胞自身和/或細胞分泌的產物提供。所述再生效果可以以下的一種或多種為特徵:上皮細胞間質轉化(可是通過TGF-β信號的減弱)減少;腎纖維化減少;腎臟發炎減少;自體腎幹細胞標記物的差異表達;植入細胞和/或自體細胞向腎損傷部位(例如,腎小管損傷)的遷移;腎臟損傷部位(例如,腎小管損傷)植入細胞的移植;腎功能一種或多種指標的穩定(此處所述);紅細胞動態平衡的恢復(此處所述)及其任何組合。 檢測再生的方法 在另一方面,本發明提供了在向個體施用或植入此處所述的細胞群、混合物或構建體後檢測自體腎再生的方法。在一個實例中,所述方法包括檢測得自個體的測試樣本和對照樣本中標記物表達的水準的步驟,其中,相比於對照樣本,測試樣本中標記物的表達水準較高,為個體中自體腎再生的預後。在另一個實例中,所述方法包括檢測樣本中一種或多種幹細胞標記物的表達。所述幹細胞標記物可選自SOX2;UTF1;NODAL;CD133;CD24;以及其任何組合。所述檢測步驟可包括確定,所述幹細胞標記物的表達相對於對照樣本在測試樣本中為上調或較高,其中較高的表達水準為個體自體腎再生的預後。在另一個實例中,對幹細胞標記物的mRNA表達進行檢測。在其他實例中,可通過PCR的方法(例如,qRT-PCR)對mRNA表達進行檢測。原位雜交也可用于基因表達的檢測。 在另一個實例中,對幹細胞標記物的多肽表達進行檢測。在另一個實例中,採用防幹細胞標記物劑對多肽表達進行檢測。在另一個實例中,所述藥劑是針對該標記物的抗體。在另一個實例中,採用免疫組織化學檢測或蛋白質印跡對幹細胞標記物多肽表達進行檢測。 本領域的普通技術人員將領會到用於檢測mRNA和/或標記物多肽表達的其他方法。 在一個實例中,所述檢測步驟之前為從個體獲得測試樣本的步驟。在另一個實例中,所述測試樣本為腎組織。 在另一方面,本發明提供了標記物的用途,如幹細胞作為自體腎再生的替代標記物。這種標記物可以單獨或結合基於功能或能力已發展或改善(例如,紅細胞動態平衡和腎功能的指標)的再生評價使用。在再生過程中檢測替代標記物還可作為再生的預後指標。 在另一方面,本發明提供了植入或施用此處所述的細胞群、混合物或構建體後患者的預後評價方法。在一個實例中,所述方法包括以下步驟:(a)檢測從所述個體獲得測試樣本的標記物表達水準;(b)相對於對照樣本(或對照參考值)的標記物表達水準,測定測試樣本的表達水準;及(c)基於標記物表達水準的測定,預測患者的再生預後,其中測試樣本相比於對照樣本(或對照參考值)的高水準表達,為個體中自體腎再生的預後。 在另一方面,本發明提供了植入或施用此處所述的細胞群、混合物或構建體後患者的預後評價方法。在一個實例中,所述方法包括以下步驟:(a)獲得患者的生物樣本;及(b)檢測生物樣本的幹細胞標記物表達,其中幹細胞標記物表達為患者自體腎的再生預後。在一些實例中,患者生物樣本中增強的幹細胞標記物表達相對於對照樣本(或對照參考值)為主體個體的再生預後。在一些實例中,患者生物樣本中降低的幹細胞標記物表達相對於對照樣本(或對照參考值)不是個體的再生預後。該患者樣本可為包括活檢的測試樣本。該患者樣本可為體液,如血液或尿液。 在另一方面,本發明提供了向個體植入或施用此處所述的細胞群、混合物或構建體後檢測自體腎再生的預後方法,其中採用非侵入性方法。作為組織活檢的替代,個體接受治療後的再生結果可以通過體液(例如,尿液)檢查進行評價。據發現,從個體尿液來源獲得的微囊泡含有某些成分包括但不限於,最終來源於通過採用本發明的細胞群治療影響的腎細胞群的特定蛋白質和miRNA。這些成分可包括涉及幹細胞複製和分化、凋亡、發炎和免疫調節的因數。微囊泡相關性miRNA/蛋白表達譜的暫態分析允許接收本發明的細胞群、混合物或構建體的個體的腎內再生結果的連續監控。例17描述了個體尿液分析的典型協議。 這些脫落到個體尿液的腎源性囊泡和/或腎分化囊泡腔內容物可通過表示再生結果的生物標記物進行分析。 在一個實例中,本發明提供了評價腎病(KD)患者是否對採用治療劑治療有反應的方法。所述方法可包括:相比於對照樣本中的囊泡量,測定或檢測從採用治療劑治療的KD患者獲得的測試樣本中的囊泡量或它們的腔內容物的步驟,其中,相比於對照樣本中囊泡量或它們的腔內容物,測試樣本中囊泡的較高或較低量或它們的腔內容物表示是經治療的患者對治療劑有反應。 本發明還提供了一種採用治療劑治療KD患者療效的檢測方法。在一個實例中,所述方法包括:相比於對照樣本中囊泡量或它們的腔內容物,測定或檢測從採用治療劑治療的KD患者獲得的測試樣本中的囊泡量或它們的腔內容物的步驟,其中,相比於對照樣本中囊泡量或它們的腔內容物,測試樣本中囊泡的較高或較低量或它們腔內容物表示KD患者採用治療劑的療效。 本發明還提供了一種藥劑作為治療劑有效治療患者亞群的腎病(KD)的鑒定方法。在一個實例中,所述方法包括:相比於對照樣本中囊泡量或它們的腔內容物,測定藥劑療效和從患者亞群獲得樣本中囊泡的存在量之間的相關性的步驟,其中,相比於對照樣本中囊泡量或它們的腔內容物,從患者亞群獲得樣本中囊泡的較高或較低量或它們腔內容物表示藥劑有效治療患者亞群的KD。 本發明提供了一種藥劑有效治療腎病(KD)的患者亞群的鑒定方法。在一個實例中,所述方法包括:相比於對照樣本中囊泡量或它們的腔內容物,測定藥劑療效和從患者亞群獲得樣本中囊泡的存在量之間的相關性的步驟,其中,相比於對照樣本中囊泡量或它們的腔內容物,從患者亞群獲得樣本中囊泡的較高或較低量或它們腔內容物表示藥劑有效治療患者亞群的KD。 探測步驟可包括分析miRNA或可存在於測試樣本中的其他分泌產物的量(見例17)。 非侵入性預後方法可包括:植入或施用此處所述的細胞群、混合物或構建體前和/或後從個體獲得尿液樣本的步驟。囊泡和其他分泌產物可採用標準方法從尿液中分離,包括但不限於,離心以除去不需要的碎片(Zhou et al. 2008. Kidney Int. 74(5):613-621; Skog et al.第20110053157號美國專利申請公佈, 其中它們的全部內容通過引用併入本文)。 本發明涉及檢測治療後個體再生結果的非侵入性方法。所述方法包括檢測未治療個體生物尿液中的囊泡或它們的腔內容物。所述腔內容物可是一種或多種miRNA。個體miRNA組合或板的檢測可適合於這種預後方法。典型的組合包括以下的兩種或多種:miR-24; miR-195; miR-871; miR-30b-5p; miR-19b; miR-99a; miR-429; let-7f; miR-200a; miR-324-5p; miR-10a-5p;以及任何組合。在一個實例中,miRNA的組合可包括2、3、4、5、6、7、8、9、10、11或多個個體miRNA。本領域的普通技術人員將領會到其他miRNA與miRNA的組合可適用於這種預後方法。其他miRNA的來源包括英國曼徹斯特大學生命科學學院主頁https://mirbase.org上的miRBase。 本領域的普通技術人員將領會到檢測再生的預後方法可適合於除此處所述的細胞群和構建體之外的採用已知的其他治療劑治療的個體。 在一些實施中,所述檢測步驟包括為了以下目的通過合適的處理器執行的軟體程式:(i)測量測試樣本和對照中標記物(囊泡或/囊泡內容物)表達的差異水準;和/或(ii)分析從測量測試樣本和對照中標記物表達的差異水準中獲得資料。合適的軟體和處理器是本領域眾所周知且是商用的。所述程式可體現於有形介質(如CD-ROM、軟碟、硬碟驅動器、DVD或與處理器相關的記憶體),但本領域的普通技術人員將很容易領會到,整個程式或其部分可以選擇性地由處理器以外的設備執行,和/或以眾所周知的方式體現於固件和/或專用硬體。 繼探測步驟後,測量結果、調查結果、診斷、預測和/或治療建議通常是被記錄和傳達給(例如)技術人員,醫生和/或患者。在某些實例中,電腦將被用於將這種資訊傳遞給相關各方,如患者和/或主診醫生。在一些實例中,進行實驗或將與國家或司法管轄區不同的的實驗結果傳達給國家或司法管轄區。 在一個優選實例中,基於具有標記物表達差異水準的測試個體中測量的標記物表達水準,預後、預測和/或治療建議在實驗完成和預後和/或預測產生後儘快地傳達給個體。所述結果和/或相關資訊,可通過個體的主治醫生傳達給個體。另外,所述結果可通過包括書寫、電子通信方式(如電子郵件或電話)的任何通信方法直接傳達給測試個體。使用電腦可促進通訊,例如在電子郵件通信情況下。在某些實例中,基於測試的含有預後測試和/或得出結論和/或治療建議結果的通信,可採用電信領域技術人員熟悉的電腦硬體和軟體結合自動生成並傳遞給個體。一個醫療保健面向通信系統的實例在第62/83761號美國專利中有所描述;然而,本發明不僅限於利用這種特定通信系統的方法。在本發明方法的某些實例中,包括分析樣本、再生預後和/或預測以及分析結果或預測通信的全部或部分方法步驟,可在不同的司法管轄區(例如,外國)進行。 在另一方面,此處所述的預後方法提供資訊給涉及植入或施用再生成功的相關方。 在所有實例中,向需要此處所述的這種治療的個體提供再生腎的方法,可包括如上所述再生的預後評價的植入後步驟。 給藥方法和路徑 本發明的細胞製劑和/或構建體可以單獨施用或與其他活性成分組合施用。 此處所述的腎細胞群或腎細胞混合物的治療有效量範圍可為個體安全接收的最大細胞數到治療腎病所必需的最小細胞數,例如,穩定、下降的減少率或一種或多種腎功能的改善。在某些實例中,本發明方法提供了此處所述的腎細胞群或腎細胞群混合物以約10,000細胞/千克、約20,000細胞/千克、約30,000細胞/千克、約40,000細胞/千克、約50,000細胞/千克、約100,000細胞/千克、約200,000細胞/千克、約300,000細胞/千克、約400,000細胞/千克、約500,000細胞/千克、約600,000細胞/千克、約700,000細胞/千克、約800,000細胞/千克、約900,000細胞/千克、約1.1x106
細胞/千克、約1.2x106
細胞/千克、約1.3x106
細胞/千克、約1.4x106
細胞/千克、約1.5x106
細胞/千克、約1.6x106
細胞/千克、約1.7x106
細胞/千克、約1.8x106
細胞/千克、約1.9x106
細胞/千克、約2.1x106
細胞/千克、約2.1x106
細胞/千克、約1.2x106
細胞/千克、約2.3x106
細胞/千克、約2.4x106
細胞/千克、約2.5x106
細胞/千克、約2.6x106
細胞/千克、約2.7x106
細胞/千克、約2.8x106
細胞/千克、約2.9x106
細胞/千克、約3x106
細胞/千克、約3.1x106
細胞/千克、約3.2x106
細胞/千克、約3.3x106
細胞/千克、約3.4x106
細胞/千克、約3.5x106
細胞/千克、約3.6x106
細胞/千克、約3.7x106
細胞/千克、約3.8x106
細胞/千克、約3.9x106
細胞/千克、約4x106
細胞/千克、約4.1x106
細胞/千克、約4.2x106
細胞/千克、約4.3x106
細胞/千克、約4.4x106
細胞/千克、約4.5x106
細胞/千克、約4.6x106
細胞/千克、約4.7x106
細胞/千克、約4.8x106
細胞/千克、約4.9x106
細胞/千克或約5x106
細胞/千克的劑量給藥。在另一個實例中,向個體施用的細胞劑量可是單劑量或單劑量附加其他劑量。在其他實例中,所述劑量可由此處所述的構建體提供。在其他實例中,向個體施用的細胞劑量可以根據估計腎品質或功能腎品質計算。 此處所述的腎細胞群或其混合物的治療有效量,可懸浮於藥學上可接受的載體或賦形劑中。這種載體包括但不限於,基本培養基中加1%血清白蛋白、鹽水、緩衝鹽水、葡萄糖、水、膠原蛋白、海藻酸、透明質酸、纖維蛋白膠、聚乙二醇、聚乙烯醇、羧甲基纖維素及其組合。所述配方應與給藥方式適應。因此,本發明提供了一種腎細胞群或其混合物的用途,例如,B2細胞群單獨或混合有B3和/或B4或B4’細胞群,用於治療個體腎病的藥物製備。在一些實例中,所述藥物進一步包含重組多肽,如生長因數、趨化因數或細胞因數。在進一步的實例中,所述藥物包含人類腎源性細胞群。用於製備藥物的細胞可以採用此處所述的方法所提供的任何變化分離、衍生或富集。 根據適合於人類給藥的藥物組合物的常規操作製成腎細胞製劑或其混合物或組合物。通常情況下,靜脈內給藥、動脈內給藥或腎囊內給藥(例如)為無菌等滲緩衝溶液。如有需要,所述組合物還可以包括局部麻醉劑,以減輕注射部位的任何疼痛。一般而言,這些成分是以單獨或單元劑型混合的方式提供,例如,作為密封容器中的冷凍濃縮,如表明活性劑量的安瓿。當組合物通過灌注給藥時,可以免除含無菌輸液藥品級的水或生理鹽水。其中所述組合物是通過注射給藥,可以提供無菌注射用水或鹽水的安瓿,使得這些成分可以在給藥前混合。 藥學上可接受的載體在某種程度上取決於所施用的特定組合物,以及施用所述組合物的特定方法。因此,有許多合適配方的藥物組合物(見,例如,Alfonso R Gennaro (ed), Remington: The Science and Practice of Pharmacy, formerly Remington's Pharmaceutical Sciences 20th ed., Lippincott, Williams & Wilkins, 2003,其中它們的全部內容通過引用併入本文)。所述藥物組合物通常被製成無菌基本等滲的,並完全符合美國食品和藥物管理局的良好產生規範(GMP)規定。 本發明的一個方面進一步提供了一種包括本發明腎細胞製劑的藥物配方,例如,B2細胞群單獨或與B3和/或B4或B4’細胞群結合,以及藥學上可接受的載體。在一些實例中,所述配方包含104至109個哺乳動物腎源性細胞。 一方面,本發明提供了向需要個體供給此處所述的一種或多種細胞群(包括混合物)的方法。在一個實例中,所述細胞群的來源可為自體或異體、同系(自體或同基因)及其任何組合。在來源不是自體的情況下,所述方法可包括免疫抑制劑的給藥。合適的免疫抑制劑藥物,包括但不限於,咪唑硫嘌呤、環磷醯胺、咪唑立賓、環孢素、他克莫司水合物、苯丁酸氮芥、氯苯紮利二鈉、金諾芬、前列地爾、鹽酸胍立莫司、biosynsorb、muromonab、阿法賽特、噴司他丁、賽尼呱,西羅莫司、黴酚酸酯、leflonomide、巴厘昔單抗、阿法鏈道酶α,bindarid、克拉屈濱、吡美莫司、ilodecakin、cedelizumab、依法珠單抗、依維莫司、anisperimus、gavilimomab、faralimomab、氯法拉濱、雷帕黴素、siplizumab、saireito、LDP-03、CD4、SR-43551、SK&F-106615、IDEC-114、IDEC-131、FTY-720、TSK-204、LF-080299、A-86281、A-802715、GVH-313、HMR-1279、ZD-7349、IPL-423323、CBP-1011、MT-1345、CNI-1493、CBP-2011、J-695、LJP-920、L-732531、ABX-RB2、AP-1903、IDPS、BMS-205820、BMS-224818、CTLA4-1g、ER-49890、ER-38925、ISAtx-247、RDP-58、PNU-156804、LJP-1082、TMC-95A、TV-4710、PTR-262-MG和AGI-1096(見第75/63822號美國專利)。本領域的普通技術人員將領會到其他合適的免疫抑制劑藥物。 本發明個體的治療方法涉及將分離的腎細胞群或其混合物的傳送給個體。在一個實例中,優選為將細胞直接施用到預期益處的位點。在一個實例中,本發明的細胞製劑或其混合物在傳送載體中被傳送個體。 需要的個體還可通過自體腎在體內與一種或多種富集腎細胞群和/或其含有的混合物或構建體分泌的產物接觸被治療。自體腎在體內與分泌產物接觸的步驟可通過施用一種或多種細胞培養基(例如,條件培養基)的分泌產物群或植入富集細胞群和混合物或能夠在體內分泌產物的構建體通過實現。體內接觸步驟提供了自體腎的再生效果。 考慮到本說明書,施用細胞和/或分泌產物的不同方法將對本領域的普通技術人員將是顯而易見的。這些方法包括將細胞注射入個體的靶位點。細胞和/或分泌產物可被插入傳送裝置或載體,這有利於通過注射或植入個體進行介入。在某些實例中,所述傳送載體可以包括天然材料。在某些其他實例中,所述傳送載體可以包括合成材料。在一個實例中,所述傳送載體提供了模擬或適當嵌入器官框架的結構。在其他實例中,所述傳送載體本質上是流動性的。這些傳送載體可以包括管,例如,導管,用於將細胞和流體注射入接受個體的體內。在一個優選實例中,所述管另外具有一個針,例如,注射器,通過本發明的細胞可以被介入指定位置的個體。在一些實例中,哺乳動物腎源性細胞群被製成通過導管(其中術語“導管”意欲包括將物質傳遞到血管的各種管狀系統)注射入血管。此外,所述細胞可被插入生物材料或支架內或生物材料或支架上,包括但不限於織物,如編織物、針織物、紡織物、網狀物、無紡布、打孔膜、海綿和泡沫以及珠,如固體或多孔珠、微粒和納米顆粒等(例如,Cultispher-S明膠珠-Sigma)。所述細胞可以為各種不同的形式給藥做準備。例如,所述細胞可以懸浮於溶液或凝膠中。該細胞可與藥學上可接受的載體或稀釋劑混合,其中本發明的細胞仍然可存活。藥學上可接受的載體和稀釋劑包括鹽水、水緩衝溶液、溶劑和/或分散培養基。這些載體和稀釋劑的使用是本領域眾所周知的。所述溶液優選為無菌流體,而且往往是等滲的。優選地,所述溶液在產生和貯存條件下是穩定的,並通過使用(例如)苯甲酸酯、氯丁醇、苯酚、抗壞血酸和硫柳汞等防止微生物(如細菌和真菌)作用污染被保藏。本領域的普通技術人員將領會到用於本發明的細胞群及其混合物的傳送載體可以包括上述特性的組合。 分離的腎細胞群的給藥方法,例如,B2腎細胞群單獨或混合有B4'和/或B3,包括但不限於,全身的、腎內的(例如,薄壁組織的)、靜脈內或動脈內注射,並直接注射入組織的活性位元點。根據本發明所用的其他給藥方法,包括通過直接注射剖腹手術、直接腹腔鏡、經腹或經皮的單一或多種注射。根據本發明所用的其他給藥方法包括(例如),逆行和輸尿管灌注。給藥的手術方法包括單步作業,例如但不限於,局部腎切除術和構建體注入、局部腎切除術、局部腎盂造口術、網膜±腹膜血管生成、多灶性活檢針跡、圓錐或角錐體到圓柱體、腎極樣替換以及兩步操作,包括例如用於重植的類器官內生物反應器。在一個實例中,細胞的混合物在同一時間通過同一路徑被傳送。在另一個實例中,包含對照混合物的每種細胞組合物被單獨傳送給特定位置或通過特定方法傳送(同時或以時間控制的方式通過此處所述的一種或多種)。 人體內適當的細胞植入劑量可以由現有的細胞活性相關資訊(例如EPO產生)確定或從臨床前研究實施的劑量研究推斷得出。體外培養和體內動物實驗,細胞數可被量化,並被用於計算植入材料的適當劑量。此外,所述患者可被檢測,以確定是否還可相應地進行其他植入或減少植入材料。 一種或多種其他成分可以被添加到本發明的細胞群及其混合物中,包括選定的細胞外基質成分,如本領域已知的一種或多種膠原蛋白或透明質酸和/或生長因數、富血小板的血漿和藥物。 本領域的普通技術人員將領會到給藥的不同配方和方法適合於此處所述的分泌產物。 試劑盒 本發明進一步包括:包含本發明的聚合物基質和支架以及相關材料,和/或細胞培養基和使用說明書的試劑盒。使用說明書可含有(例如)細胞培養或細胞和/或細胞產物施用說明書。在一個實例中,本發明提供了一種包含此處所述的支架和說明書的試劑盒。在另一個實例中,所述試劑盒包括用於標記物表達的檢測劑,藥劑所使用的試劑以及使用說明書。這種試劑盒可用於測定植入或施用此處所述的細胞群、混合物或構建體後個體的自體腎再生預後的目的。所述試劑盒還可用於測定此處所述的細胞群、混合物或構建體的療效。 報告 當為了實施商業目的時,本發明的方法一般會產生一份報告或再生預後的概要。本發明方法,將產生一份報告,包括此處所述的細胞群、混合物或構建體的任何植入或施用之前和之後可過程或再生結果的預測。所述報告可包括與預後相關的任何指標資訊。本發明的方法和報告可以進一步包括存儲於資料庫中的報告。另外,所述方法可以在個體資料庫中產生記錄,並將記錄填充入資料。在一個實例中,所述報告為紙件報告,在另一個實例中,所述報告為可聽報告,在另一個實例中,所述報告為電子記錄。據設想,所訴報告被提供給醫生和/或患者。所述報告的接收可進一步包括:與包括資料和報告的伺服器電腦建立網路連接,並從伺服器電腦索取資料和報告。本發明提供的方法還可為全部或部分自動化的。 所有專利、專利申請以及本說明書所引用的參考文獻的全部內容通過引用併入本文 以下實例僅提供說明目的之用,而不是以任何方式限制本發明的範圍。實例 實例 1- 生物活性腎細胞的分離和表徵
成年雄性豬(野豬)中一種自發性與進展性慢性腎臟病(CKD)伴隨有貧血症的實例提供了用於細胞組合物評價以及直接與年齡匹配的正常豬腎組織對比表徵的新鮮病變腎組織細胞。收穫時腎組織的組織學檢查證實了腎病的特徵為嚴重擴散慢性間質纖維化和多灶性纖維化新月體腎炎。臨床化學證實了氮質血症(血尿素氮和血清肌酐升高)以及輕度貧血症(紅細胞壓積和抑制血紅蛋白水準的輕微下降)。細胞被分離、擴增,並具有病變和正常腎組織的特徵。如Presnell et al. WO/2010/056328 (它們的全部內容通過引用併入本文)的圖1所示,Gomori的三色染色使得病變腎組織中的纖維化相比于正常腎組織(用箭頭表示藍染色)突出。表達cubulin並轉運給陰離子蛋白並能夠受體介導白蛋白運輸的功能性腎小管細胞,是由正常和病變腎臟組織增殖的。紅血球生成素(EPO)表達細胞還存在於培養基中,並通過多種通道,凍結/解凍迴圈保存。此外,分子分析證實,來自正常和病變組織的EPO表達細胞對EPO和其他缺氧調節基因靶(包括vEGF)的HIF1 a驅動誘導的體外缺氧條件有反應。細胞是通過膠原酶+分散酶的酶消化從豬腎組織分離,並通過簡單的機械分離消化和外植體培養在不同實驗中分離。在兩個通道中,含有紅血球生成素表達細胞的外植體源細胞培養都經受大氣(21%)和不同缺氧(<5%)培養條件,以確定接觸低氧是否EPO基因表達上調達到了頂點。正如齧齒動物培養(見例3)指出,正常豬顯示了氧依賴性表達及EPO基因調控。出人意料的是,儘管尿毒癥/貧血症狀態的CKD豬(紅細胞壓積<34,肌酐>9.0)EPO表達細胞很容易被分離,並通過組織增殖,EPO基因表達保持缺氧調控,如Presnell et al. WO/2010/056328 (它們的全部內容通過引用併入本文)的圖2所示。如Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖3所示,增殖培養中的細胞,顯示了自我組織成管狀結構的能力。如Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖4所示,培養基(在通道3)中功能性腎小管細胞的存在是通過觀察經過培養細胞的FITC標記物白蛋白的受體介導吸收證實。綠點(由細白箭頭表示)表示由腎小管細胞特異性受體、並轉運給陰離子蛋白和Cubilin介導的內吞螢光素標記物白蛋白,說明通過功能性腎小管細胞再吸附蛋白質。藍著色(由粗白箭頭表示)為Hoescht染色細胞核。總之,這些資料表明,功能性腎小管和內分泌細胞可以從豬腎組織分離和增殖,即使在已經受到嚴重損害的CKD腎組織中。此外,這些調查結果支持了用於CKD治療的自體細胞治療性產物的發展。 此外,採用酶法從正常成人腎中分離EPO產生細胞(如以上例1所述)。如Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖5所示,分離操作導致分離後比最初組織更多的EPO表達。如Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖6所示,維持EPO基因表達保留的人類EPO產生細胞培養是可的。對人類細胞普通組織培養處理的塑膠或包覆有一些細胞外基質的塑膠上進行培養/增殖,例如,纖連蛋白或膠原均被發現隨著時間的推移支持EPO的表達。實例 2- 特異性生物活性腎細胞的分離和富集
腎細胞隔離:簡言之,10批,2周齡雄性Lewis大鼠腎得自商業供應商(Hilltop Lab Animals Inc.),並於約4°C下保存在Viaspan保存培養基中連夜發貨。此處所述的所有步驟在生物安全櫃(BSC)中進行,以保持無菌。採用漢克氏平衡鹽溶液(HBSS中)洗滌腎臟3次,以洗滌掉Viaspan保存培養基。在第3次洗滌後,除去剩餘腎囊以及任何剩餘基質組織。採用微解剖方法除去大腎萼。然後採用無菌手術刀將腎精細地剁碎成漿液。然後將漿液轉移到一個50毫升錐形離心管中,並稱重。收集RNA的小樣本,並置於收集到1.5毫升無RNAse的無菌離心微型離心管中,並在液氮中捕捉冷凍。一旦冷凍,然後將它轉移到-80℃冰箱中,直到分析。10個初生腎的組織重量相當於約1克。基於批次的重量,消化培養基被調整為每1克組織提供20毫升消化。本操作的消化緩衝液含有4單位分散酶1的HBSS、5毫摩爾CaCl2
(Sigma)的300單位/毫升IV型膠原酶(Worthington)。 向管中加入適量的預熱消化緩衝液,然後密封並置於搖杆上的37℃培養箱中20分鐘。第一消化步驟除去了許多紅細胞,並增強了剩餘組織的消化。20分鐘後,移除該管,並置於BSC中。該組織被允許沉澱於試管底部,然後將上清液除去。再向剩餘組織中補充相當於初始量新鮮消化緩衝液。然後將該管置於搖杆上的37℃培養箱中另外30分鐘。 30分鐘後,通過70μm細胞篩檢程式(BD Falcon)將消化混合物吸取到等體積的中和緩衝液(DMEM w/ 10% FBS)中,以終止消化反應。然後通過300xg離心5分鐘對該細胞懸液進行洗滌。離心後,然後將該顆粒重新懸浮於20毫升KSFM培養基中,並得到細胞計數和存活率評價的樣本。一旦計算出細胞計數,收集到用於RNA的100萬細胞,在PBS中洗滌,並在液氮下捕捉冷凍。採用50毫升KSFM對剩餘的細胞懸液進行培養,然後通過300xg離心5分鐘再次洗滌。洗滌後,將細胞顆粒重新懸浮於濃度為1500萬細胞/每毫升KSFM中。 然後將5毫升腎細胞懸液加入5毫升30%(w/v) Optiprep®的15毫升錐形離心管(BD Falcon)中,並通過倒置6次混合。這形成15% (w/v) Optiprep®的最終混合物。倒置後,採用1毫升PBS對該管精心地分層。該管在800 x g下離心15分鐘而不停止。離心後,將該管移除。還有含有紅細胞、死細胞和活細胞小群的顆粒,其中活細胞小群包括一些小顆粒細胞、一些EPO產生細胞、一些腎小管細胞以及一些內皮細胞。採用吸管小心地取出細胞帶,並轉移至另一個15毫升的錐形管中。通過吸氣去除梯度培養基,通過重新懸浮於1毫升KSFM收集顆粒。然後將細胞帶和細胞顆粒組合,並採用KSFM重新懸浮於收集窄帶量的至少3個稀釋液中,並通過300xg離心5分鐘洗滌。洗滌後,將細胞重新懸浮於20毫升KSFM中,並收集細胞計數的樣本。一旦台盼藍排斥計算出細胞計數,收集RNA樣品的100萬細胞,在PBS中洗滌,並在液氮下捕捉冷凍。 採用密度梯度分離預培養'清理',以提高特定生物活性腎細胞的存活和培養性能:為了產生培養細胞的一個乾淨可存活群,首先在上述“腎細胞分離”中產生細胞懸液。作為可選步驟以及作為清理前期製備的方法,將懸浮於無菌等滲緩衝液中的高達100萬全部細胞以1:1與等體積室溫下由60%(w/v)的碘克沙醇原液(從而產生最終15%w/v Optiprep溶液)制得的30%Optiprep®通過倒置6次完全混合。混合後,對1毫升PBS緩衝液在混合細胞懸液的頂部進行仔細地分層。然後將梯度管小心裝入離心機中,以確保適當的平衡。將梯度管在800 x g下離心15分鐘(25˚C)而不中止。清理的細胞群(含有存活和功能性集合管、管、內分泌、腎小球和血管細胞)分割為介於6%至8%(w/v) Optiprep®之間,與1.025 – 1.045克/毫升的密度一致。其他細胞和碎片在管底形成顆粒。 腎細胞培養:然後將細胞帶和顆粒接種於三次組織培養處理的瓶(培養瓶T500)或相當於細胞濃度為30000細胞每平方釐米的150毫升含有抗生素/抗真菌藥的5%(v/v)FBS、2.5微克EGF、25毫克BPE、1X ITS(胰島素/轉鐵蛋白/亞硒酸鈉補充物)的DMEM(高糖)/ KSFM(50:50)混合物。這些細胞在濕度為5%CO2的培養箱中培養2-3天,以為細胞提供21%的大氣氧水準。兩天后,改變培養基,將該培養置於提供二氧化碳/氮氣多氣體加濕培養箱(Sanyo)的2%氧水準環境中24小時。經過24小時培養後,採用60毫升1XPBS洗滌細胞,然後採用40毫升0.25%(w/v)的胰蛋白酶/乙二胺四乙酸(Gibco)除去。在除去後,採用含有10% FBS的等體積KSFM中和細胞懸液。然後通過在300xg下離心10分鐘對細胞進行洗滌。洗滌後,將細胞重新懸浮於20毫升的KSFM中,並轉移至50毫升錐形管中,然後收集用於細胞計數的樣本。一旦採用台盼藍排斥測定活細胞數,收集RNA樣本的100萬細胞,在PBS中洗滌,並在液氮中捕捉冷凍。將該細胞在PBS再洗滌一次,通過在300xg下離心5分鐘收集。洗滌的細胞顆粒重新懸浮於濃度為3750萬細胞/毫升的KSFM中。 採用密度梯度分離步驟的富集特定生物活性腎細胞:採用碘克沙醇(Optiprep)的多濃度w/v製成的密度不連續梯度將主要由腎小管細胞組成但含有其他細胞類型(集合管、腎小球、血管、內分泌)小亞群的培養腎細胞亞群分離成它們的成分亞群。在收穫和應用於梯度前,培養被置於缺氧環境中長達24小時。通過對15毫升無菌錐形管頂部的四個不同密度培養基進行分層,將最高密度的溶液置於底部,並在頂部分層為最稠密溶液,產生不連續分層梯度。細胞應用於不連續梯度,並離心,這將導致群層化為基於大小和粒度的多個帶。 簡言之,採用KFSM培養基作為稀釋劑制得密度為7、11、13和16%Optiprep®(60%w/v碘克沙醇)。例如:對於50毫升7%(w/v)的Optiprep®,將60%5.83毫升(w/v)的碘克沙醇原液加入44.17毫升的KSFM培養基中,並通過倒置混合均勻。將配備有無菌L/S 16聚乙烯管連接無菌毛細管的蠕動泵(主軟硬度L/S)設置為每分鐘2毫升的流速,將4種溶液中每一種的2毫升裝入無菌15毫升錐形管中,以16%溶液開始,接著是13%溶液、11%溶液以及7%溶液。最後,將含有7500萬培養的齧齒動物腎細胞的2毫升細胞懸液負載於不連續梯度('腎細胞培養'中已如上所述產生)的頂部。重要的是,因為泵開始傳送梯度溶液到管,液體小心被帶到允許流下的管45°角的側面,以確保各梯度層之間形成適當的介面。然後將負載有細胞的不連續梯度在800 x g下離心20分鐘而不中止。離心後,小心地除去管,以免打擾各個介面。形成5個不同的細胞組分(4個帶和1個顆粒)(B1 – B4, +顆粒)(見圖1A,左錐管)。採用一個一次性球狀無菌移液管或5毫升吸管收集各個組分,並通過顯型和功能表型(見Presnell et al. WO/2010/056328的例10)。當在分離後對齧齒動物腎細胞懸液進行不連續梯度分餾時,富集腎小管細胞(以及含有一些得自集合管的細胞)的組分分段為密度介於1.062-1.088克/毫升之間。相反,當間接體內後進行培養密度梯度分離時,富集腎小管細胞(以及含有一些得自集合管的細胞)的組分分段為密度介於1.051-1.062克/毫升之間。同樣地,當在分離後對齧齒動物腎細胞懸液進行不連續梯度立分餾時,富集EPO產生細胞、腎小球細胞和血管細胞(“B4”)的組分分段為密度介於1.025-1.035克/毫升之間。相反,當間接體內後進行培養密度梯度分離時,富集EPO產生細胞,腎小球細胞和血管細胞(“B4”)的組分分段為密度介於1.073 – 1.091克/毫升之間。重要的是,細胞培養後分佈到的“B2”和“B4”組分通過收穫和不連續梯度(例3提供了關於缺氧對帶狀分佈影響的其他細節)操作前將培養接觸(為約1小時至約24小時內)缺氧培養環境(缺氧被定義為<21%(大氣)中分別增加。 通過3倍KSFM量的稀釋洗滌每個帶,混合均勻,並在300 x g下離心5分鐘。將顆粒重新懸浮於2毫升KSFM中,並採用台盼藍排斥和血細胞計數器對活細胞進行計數。收集RNA樣本的100萬細胞,在PBS中洗滌,並在液氮下捕捉冷凍。B2和B4的細胞用於植入通過查理斯河實驗室中兩步5/6腎切除術產生尿毒癥和貧血症雌性大鼠的研究。通過包括氧調節紅血球生成素和VEGF表達、腎小球標記物(腎病蛋白、podocin)表達以及血管標記物(PECAM)表達的定量即時PCR技術確定B4的特徵。通過E-鈣粘蛋白、N-鈣粘蛋白和水通道蛋白-2的表達確認'B2'成分的表型。見Presnell et al. WO/2010/056328圖49a及49b。 因此,使用不連續梯度策略,不僅可以富集EPO產生細胞(B4)的罕見群,同時也是產生功能腎小管細胞(B2)(見Presnell et al. WO/2010/056328的圖50和51)相對富集組分一種方法。不連續梯度策略還可使EPO產生細胞從紅細胞、細胞碎片及其它潛在的不良細胞類型(如大細胞團塊和某些類型的免疫細胞)中分離。 不連續梯度操作可需要對提供細胞成分的良好分離所用的相關特定密度進行調整。調整梯度的優選方法包括1)運行一個連續密度梯度,其中從梯度底部的高密度(例如,16-21%Optiprep)到頂部的相對較低密度(例如,5-10%)。根據標準方法(Axis Shield)採用任何標準密度梯度溶液(聚蔗糖、細胞分離液、蔗糖、碘克沙醇)製備連續梯度。將目標細胞負載於連續梯度,並在800x g下離心20分鐘而不中止。類似大小和粒度的細胞往往在梯度中一起分開,這樣可以測量梯度中的相對位置,同時測量溶液在該位置的比重。因此,隨後,確定的不連續梯度可以得出,側重於特定細胞群分離基於它們能夠在特定條件下橫斷密度梯度。當從不健康及健康組織分離細胞或從不同物種分離特定細胞時,可需要採用這種優化。例如,同時犬科動物和人類腎細胞培養進行優化,以確保大鼠中鑒定的特定B2和B4亞群從其他物種得到分離。齧齒動物B2和B4亞群分離的最優梯度組成為7%、11%、13%和16%Optiprep(w/v)。犬科動物B2和B4亞群分離的最優梯度組成為7%、10%、11%和16%Optiprep(w/v)。人類B2和B4亞群分離的最優梯度組成為7%、9%、11%和16%(w/v)。因此,表2.1提供了得自齧齒動物、犬科動物和人類腎細胞培養的B2和B4定位的密度範圍。 表2.1. 物種密度範圍。 實例 3- 梯度前的低氧培養影響了帶分佈、組成以及基因表達
為了確定氧條件對原型B2和B4的分佈及組合物的影響,將不同的新腎細胞製劑在梯度步驟之前暴露於不同氧條件下。如上所述採用齧齒動物細胞分離和初始培養的標準操作,確定齧齒動物新腎增強(NKA)細胞製劑(RK069)。在21%(大氣)氧條件下對所有瓶培養2-3天。改變培養基,然後將一半瓶轉移到一個設定為2%氧的氧控培養箱中,而剩餘的瓶存放於21%氧條件下另外24小時。然後採用如上所述的標準酶收穫操作從各組條件收穫細胞。根據標準操作制得不連續梯度,分別收穫“常氧的”(21%氧)和“缺氧的”(2%氧)培養,並行地應用於相同的不連續梯度(圖2)。而在兩種條件下產生4個帶和顆粒,整個梯度的細胞分佈在21%和2%的氧培養批次(表1)中不同。具體來說,缺氧條件下B2的產量增加,伴隨有B3的同時減少。此外,B4特異性基因(如紅血球生成素)在缺氧培養細胞所產生梯度中的表達增強(Presnell et al. WO/2010/056328的圖73)。 採用如上所述狗細胞分離和培養(類似於齧齒動物分離和培養操作)的標準操作確認犬科動物NKA細胞製劑(DK008)。所有瓶在21%(大氣)氧條件下培養4天,然後將一組瓶轉移入缺氧(2%)下24小時,而另一組瓶分別維持在21%氧。隨後,收穫各組瓶,並採用相同的不連續梯度(圖3)。類似於大鼠的結果(例1),缺氧培養的狗細胞梯度在貫穿不同的梯度中分佈不同於大氣氧培養狗細胞(表3.1)。梯度之前,B2的產量在缺氧條件下再次提高,伴隨有B3分佈的減少。 表3.1.
上述資料表明,接觸缺氧的預梯度增強了B2組成以及特定細胞(紅血球生成素產生細胞、血管細胞以及腎小球細胞)到B4的分佈。因此,缺氧培養,接著採用如上所述的密度梯度分離,是一種產生跨物種‘B2’和‘B4’細胞群的有效方法。實例 4- 人類腎小管細胞 / 腎小球細胞的分離
通過貫穿所述的酶分離方法從正常的人類腎組織分離和增殖腎小管細胞和腎小球細胞。通過上述方法的梯度,如Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖6所示,表型特徵均在分離和增殖中保持。通過標記物白蛋白吸收評價的腎小管細胞功能,也在重複的通道的低溫貯藏後保留。Presnell et al. WO/2010/056328(它們的全部內容通過引用併入本文)的圖69表明,當腎小管細胞富集和腎小管細胞除去群在三維動態培養時,腎小管細胞標記物的表達顯著增加,鈣粘蛋白在腎小管細胞富集群中表達。這證實,當細胞培養於三維動態培養環境時,腎小管細胞的富集可超出初始富集。實例 5- EPO 產生細胞通過流式細胞術的進一步分離
對以上例2中所述腎細胞的相同培養群進行流式細胞術分析,以研究前散射和側散射。少量小顆粒EPO產生細胞可被辨別(8.15%),並通過少量小顆粒群採用流式細胞術的分選能力進行分離(見Presnell et al. WO/2010/056328的圖70(它們的全部內容通過引用併入本文))。實例 6- 從自身免疫性腎小球腎炎患者樣本分離的腎細胞普通混合物的表徵
如上所述從自身免疫性腎小球腎炎患者樣本中分離腎細胞普通混合物。為了確定從腎組織分離和擴增的腎細胞特定亞群的無偏基因型組合物,進行即時定量PCR(qrtpcr)分析(Brunskill et al., 上文2008),以在細胞亞組份之間鑒定出不同的細胞類型特異性和路徑特異性基因表達模式。如表6.1所示,HK20為自身免疫性腎小球腎炎患者的樣本。表6.2顯示,HK20產生的細胞缺乏腎小球細胞,如qRTPCR測定。實例 7- 從局灶節段性腎小球硬化的情況分離的治療相關性腎生物活性細胞群的遺傳識別
為了確定從腎組織分離和擴展的腎細胞特定亞群的無偏基因型組合物,進行即時定量PCR(qrtpcr)分析(Brunskill et al., 上文2008),以在細胞亞組份之間鑒定出不同的細胞類型特異性和路徑特異性基因表達模式。對得自大部分腎小球被破壞的局灶節段性腎小球硬化(FSGS)的人類製劑HK023在收穫時B4組分中腎小球細胞的存在進行了評價。簡言之,採用標準活檢操作從腎中取出4個核中的每個獨立地產生和保持普通(UNFX)培養(Aboushwareb et al., 上文2008)。在UNFX間接體外的(2)通道之後,收穫細胞,並採用密度梯度方法(如例8)產生包括亞組分B4的亞組分,這是眾所周知的基於在齧齒動物、犬科動物和其他人類標本中進行工作的內分泌、血管和腎小球細胞富集。 表6.1
表6.2培養的人、豬和鼠腎細胞的隔室分析。 從看作介於1.063-1.091克/毫升之間浮力密度的不同帶的HK023每個獨立UNFX樣本中收集B4組分。從每個樣本分離RNA,並通過即時定量PCR研究podocin(腎小球細胞標記物)和PECAM(內皮細胞標記物)的表達。正如嚴重FSGS情況下活檢產生樣品的預期,B4組分中podocin(+)腎小球細胞的存在與2/4樣本中探測不到的podocin不一致。與此相反,PECAM+血管細胞一致地存在於4/4活檢初始培養的B4組分中。因此,B4組分可以在1.063-1.091克/毫升的密度範圍內分離,即使得自嚴重疾病狀態的人類腎。 表7.1從FSGS情況分離的亞組分B4中檢測腎小球和血管細胞的podocin和PECAM表達。
此外,如表7.2所示,人類樣本(HK018)顯示密度梯度離心後通過qRTPCR未檢測到的大鼠podocin(腎小球標記物)。 表7.2. B2及B4’的HK018梯度後基因表達表徵 實例 8- 採用螢光啟動細胞分選 (FACS) 富集 / 除去存活腎細胞類型
採用螢光啟動細胞分選(FACS)可富集一種或多種分離的腎細胞,和/或除去一種或多種特定的腎臟細胞類型。試劑:70%乙醇;洗滌緩衝液(PBS);50:50腎臟細胞培養基(50%DMEM高葡萄糖):50%角化細胞-SFM;台盼藍0.4%;靶腎細胞群(如腎血管內皮細胞CD31和腎小球細胞的腎病蛋白)的初級抗體。匹配的同型特異性螢光二級抗體;染色緩衝液(PBS中的0.05%BSA)。 操作:在清潔生物安全櫃(BSC)標準操作後,初步分離或培養細胞的腎細胞單一懸液可從一個T500 T/C處理瓶獲得,並重新懸浮於腎細胞培養基中,然後置於冰上。然後採用台盼藍排斥法測定細胞計數和存活率。對於(例如)來自異質群的腎小球細胞或內皮細胞的腎細胞富集/除去,得到至少70%存活率的10到50×106
個活細胞。然後採用染色緩衝液/1×106
細胞(滴度如有必要)起始濃度為1微克/0.1毫升的靶細胞類型的特定初級抗體對腎細胞異質群進行染色。靶抗體可與如CD31 PE(腎血管內皮細胞特異性)或非共軛如腎病蛋白(腎小球細胞特異性)結合。 然後在冰上或4℃避光對細胞染色30分鐘。經30分鐘培養後,通過300xg離心5分鐘對細胞進行洗滌。然後將顆粒懸浮於PBS或染色緩衝液,這取決於是否需要結合同型特異性二級抗體。如果採用螢光物共軛結合初級抗體對細胞進行標記物,細胞懸浮於2毫升PBS中(每10e7細胞),並繼續採用FACS或等價細胞分選儀進行細胞分類。如果沒有採用螢光物共軛結合抗體對細胞進行標記物,然後採用起始濃度為1微克/0.1毫升/1×106
細胞的結合同型特異性二級抗體進行標記物。 然後,細胞染色30分鐘。在冰上或4℃避光。經過30分鐘的培養,細胞沖洗300xg離心5分鐘。離心後,將顆粒重新懸浮於5×106
/毫升的PBS中,然後將4毫升每12x75毫米的轉移到無菌試管。 按照產生商的說明(BD FACs Aria用戶手冊),FACs Aria流式細胞術是為活細胞無菌分選作準備。門被採用特定波長的螢光強度選擇吸引腎特異性細胞類型。另一個門被選擇吸引陰性群。一旦所需的門被吸引到封裝陽性靶群和陰性群,採用製造商的說明對細胞進行分選。 陽性靶群被收集於一個15毫升錐形管中,另一個15毫升錐形管中的陰性群充滿1毫升腎細胞培養基中。收集之後,通過流式細胞術分析得自每個管的樣品,以確定純度。通過在300xg下離心5分鐘洗滌收集的細胞。而顆粒懸浮於用於進一步分析和實驗的腎細胞培養基中。實例 9- 採用磁性細胞分選的腎細胞類型的富集 / 除去
從分離的初級腎組織可富集一種或多種分離的腎細胞和/或可除去一種或多種特定的腎臟細胞類型。 試劑:70%乙醇,洗滌緩衝液(PBS),50:50腎細胞培養基(50%DMEM高糖):50%角質細胞-SFM,台盼藍0.4%,運行緩衝液(PBS,2毫摩爾乙二胺四乙酸,0.5%BSA),沖洗緩衝液(PBS,2毫摩爾EDTA),清潔劑(70%v /v乙醇),Miltenyi FCR阻斷劑,IgG、靶抗體(如CD31(PECAM)或腎病蛋白,或二級抗體)特異性的Miltenyi微球。 操作:按照清潔生物安全櫃(BSC)的標準操作,從初步分離或培養得到腎細胞的單一細胞懸液,並重新懸浮於腎細胞培養基中。採用台盼藍排斥法測定細胞計數和存活率。 對於(例如)異質群的腎小球細胞或血管內皮細胞的腎細胞富集/除去,得到存活率至少為70%的至少10e×106
-多達4×109
個活細胞。 富集/除去的最佳分離方法是根據靶細胞測定的。對於小於10%目標頻率的富集,例如,採用腎病蛋白抗體、Miltenyi autoMACS或等價物、儀器操作POSSELDS(敏感模式中的雙正選擇)的腎小球細胞被用到。對於大於10%目標頻率的除去,Miltenyi autoMACS或等價物,儀器操作除去(敏感模式中的除去)被使用。 採用靶向特異性抗體標記物活細胞,例如,腎小球細胞的腎病蛋白rb多克隆抗體,向含有0.05%BSA的15毫升錐形離心管中加入1微克/×106
細胞rb /0.1毫升PBS,接著在4℃下培養15分鐘。 標記物後,通過向每107
細胞加入1-2毫升緩衝液對細胞進行洗滌,以除去未結合的初級抗體,接著在300xg離心5分鐘。洗滌後,加入同型特異性二級抗體(如含有0.05%BSA的1微克/×106
/0.1毫升PBS的雞抗兔PE),接著在4℃下培養15分鐘。 培養後,通過向每107
細胞加入1-2毫升緩衝液對細胞進行洗滌,以除去未結合的初級抗體,接著在300xg離心5分鐘。除去上清液,將細胞顆粒重新懸浮於每107
細胞總數60微升的緩衝液中,接著向每107
總細胞加入20微升FCR封閉液,然後將其混合均勻。 直接加入20微升MACS微珠(如抗PE微珠)並攪拌,然後在4℃下培養15分鐘。 培養後,通過加入10-20x標記物量的緩衝液以及在300xg下離心5分鐘對細胞進行洗滌。然後將細胞顆粒重新懸浮於500µl-2毫升/108
細胞的緩衝液中。 按照產生商的指示,對autoMACS系統進行清理,並採用autoMACS裝填磁性細胞分離的製劑。將新無菌收集管置於電源插座埠下。對該autoMACS細胞分離方案進行選擇。對於選擇,POSSELDS方案被選中。對於除去,DEPLETES方案被選中。 將標記物細胞在吸收埠插入,然後開始該方案。 細胞選擇或除去後,收集樣本,並置於冰上備用。除去或選擇抽選樣本的純度是由流式細胞術驗證。實例 10- 具有治療潛力的細胞可以正常和慢性腎臟病變組織得到分離和增殖
本研究的目的是通過高含量分析(HCA)測定人類NKA細胞的功能特性。高含量成像(HCI)提供了多種亞細胞使用兩種或多種螢光探針(多工)跨越許多樣本事件的同時成像。高含量分析(HCA)可同時提供高含量成像中捕獲的多種細胞參數的定量測量。簡言之,普通(UNFX)培養生成(Aboushwareb et al., 上文2008),並且使用活檢操作從5個發展性慢性腎臟病(CKD)的人類腎和三個非CKD人類腎採取的核活檢保持獨立。在UNFX間接體內的(2)通道之後,收穫細胞,並採用密度梯度方法(如例2)產生包括亞組分B2、B3和/或B4的亞組分。 從非CKD與CKD器官捐獻者獲得人類腎組織總結於表10.1。圖4顯示了HK17和HK19樣本的病理特徵。對所有非CKD(3/3)和CKD(5/5)腎建立了間接體內培養。人類NKA細胞定義目標區域(ROI)中白蛋白運輸的高含量分析(HCA)顯示於圖5(人類NKA細胞中的白蛋白運輸HCA)。非CKD與CKD的源性NKA細胞中白蛋白運輸的定量比較顯示於圖6。如圖6所示,白蛋白運輸並不損害CKD源性NKA培養。腎小管富集B2和腎小管細胞除去B4亞組分之間標記物表達的比較分析顯示於圖7(CK8/18/19)。 表10.1
腎小管富集B2和腎小管細胞除去B4亞組分之間白蛋白運輸的比較功能分析顯示於圖8。亞組分B2富集於近端腎小管細胞,從而表現出白蛋白運輸功能的增加。 白蛋白的吸收:24孔,膠原IV板(BD Biocoat™)中生長克隆的細胞培養基被替代為含有1X抗真菌藥/抗生素和2毫摩爾穀氨醯胺的無酚紅、無血清、低血糖DMEM(pr-/s-/lg DMEM)培養18-24小時。試驗之前,洗滌細胞,並採用pr-/s-/lg DMEM + 10毫摩爾HEPES、2毫摩爾穀氨醯胺、1.8毫摩爾CaCl2
和1毫摩爾MgCl2
培養30分鐘。將細胞接觸羅丹明共軛結合牛血清白蛋白(Invitrogen)25微克/毫升30分鐘,用冰冷PBS洗滌,以停止內吞作用,並採用含有25微克/毫升赫斯特核染料的2%多聚甲醛對其立即進行固定。對於抑制實驗,在白蛋白加入前10分鐘將1微摩爾受體相關性蛋白(RAP)(Ray Biotech, Inc., Norcross GA)加入。採用BD Pathway™ 855高含量生物成像儀(Becton Dickinson)進行顯微成像和分析(見Kelley et al. Am J Physiol Renal Physiol. 2010 Nov;299(5):F1026-39. Epub Sep 8, 2010))。 總之,HCA產生細胞水準資料,並可以揭示由其他實驗未檢測到的種群動態,即基因或蛋白質表達。測量白蛋白運輸((HCA-AT)功能的HCA可量化間接體內實驗可以被用來將人類腎小管細胞描述為人類NKA原型成分。HCA-AT能夠使細胞功能進行比較評價,顯示出白蛋白運輸成分細胞被保留於人類CKD腎源性NKA培養中。還表明,NKA培養、B2和B4的特異性亞組分,在表型和功能方面不同,B2表示白蛋白運輸能力增強的腎小管細胞富集組分。得自人類CKD的B2細胞亞群在表型和功能上類似於表明體內療效的齧齒動物B2細胞(如上所示)。實例 11- 標記物表達作為腎再生的預測器
本研究涉及到幹細胞標記物表達和祖細胞標記物表達作為採用治療上生物活性的原發性腎細胞亞群治療的5/6腎切除大鼠中腎再生預測器。NKA治療改善腎功能以其基本機制為特徵。我們對NKA治療作用機制研究涉及到細胞間傳遞信號、植入和纖維化機路徑。目前的工作重點是NKA治療如何會增加器官內在的再生能力-也許通過移動腎幹細胞。我們猜測,NKA治療的5/6 NX大鼠中觀察到腎功能的延長存活和改善與特異性幹細胞標記物的分子表達相關。 採用大鼠CKD的5/6腎切除模型,本研究利用分子實驗,以對直接注射明確的治療上生物活性原發性腎細胞群有所反應的大鼠5/6腎切除腎臟內儲備幹細胞和祖細胞的移動進行評價。據觀察到,這種基於細胞的療法特別地與轉錄和蛋白水準上的關鍵幹細胞標記物CD24、CD133、UTF1、SOX2、LEFTY1和NODAL的上調相關。注射後1周檢測到上調,注射後12周達到高峰。幹細胞和祖細胞標記物的啟動與相對於未經腎切除處理的對照組存活率增加和明顯改善的血清生物標記物相關。 材料和方法 大鼠原發性腎細胞群的分離。如前所述對大鼠原發性腎細胞群進行分離(Aboushwareb et al., 上文2008; Presnell et al., 2009 FASEB J 23: LB143)。 體內研究設計和分析。原發性腎細胞群(Presnell et al. Tissue Eng Part C Methods. 2010 Oct 27. [Epub在印刷之前])分離和體內研究的詳細描述,對CKD 5 / 6腎切除鼠模型(Kelley et al. 上文2010)中原發性腎細胞亞群的生物活性進行了評價(Kelley et al. 上文2010)。原發性腎細胞的腎小管細胞亞群改善了存活,並增強了別處出版的慢性腎病鼠模型中的腎功能。在目前研究中,從與B2(NKA #1)或B2+B4混合物(NKA #2)治療的大鼠屍檢時分離得到組織,並和腎切除(NX)和假手術、非腎切除大鼠(對照)比較。在圖9和11和表11.1中,將NKA #1和NKA #2治療大鼠的資料進行合併。通過對大鼠每週和屍檢前得到的血液樣本分析進行研究得到系統性資料。 表11.1顯示了假治療動物(對照組)的生存資料,n=3;nx對照(Nx),n+3;B2細胞治療的動物(NKA#1), n= 7; B2+B4細胞((NKA#2); n= 7。在研究末期(23-24周),Nx動物沒有剩下。NKA治療動物相比於未經治療的Nx 對照具有較高的存活率。 表11.1
*用於組織的預定時間點處死的1個動物 **用於組織的預定時間點處死的2個動物 RNA分離、cDNA合成和qRT-PCR。從嵌入以下最佳切削溫度(OCT)冷凍劑的組織分離RNA:將組織塊置於室溫下,並去除多餘的OCT,然後將組織置於PBS中,以便徹底解凍,並除去剩餘OCT,將組織在PBS中洗滌3次,然後進行粗碎並分裝離心管。然後用研磨棒對分裝組織進行粉碎,並採用RNeasy Plu微型試劑盒(Qiagen, Valencia CA)進行提取。RNA的完整性,通過光譜測定cDNA,採用SuperScript® VILO™ cDNA合成試劑盒(Invitrogen, Carlsbad CA)由相當於1.4 µg量的RNA產生cDNA。cDNA合成之後,通過加入200微升diH2O將每個樣本稀釋至1:6,使最終體積達到240微升。通過使用編目引物和來自ABI和ABI-Prism 7300即時PCR系統(Applied Biosystems, Foster City CA)的探針的定量即時PCR (qRT-PCR)對目標轉錄的表達水準進行了研究。採用TaqMan®基因表達的混合液(ABI, Cat# 4369016) 進行擴增,肽醯脯氨醯異構酶B(PPIB)被用作內源性對照。qRT-PCR反應:10微升混合液(2X)、1微升引物和探針(20X)、9微升cDNA、20微升反應總體積。以下採用TaqMan®引物和探針設置每次反應。
蛋白質印跡。利用嵌入OCT冷凍劑冷凍整腎組織進行蛋白質樣本收集。如上所述除去OCT,將各組織裂解於50毫摩爾Tris (pH 8.0)、120毫摩爾NaCl、0.5% NP40和蛋白質抑制劑雞尾酒 (Roche Applied Science, Indianapolis IN)組成的緩衝液中。裂解在室溫下搖動進行15分鐘,接著在13,000 RPM下離心10分鐘。收集所有上清液,並採用Bradford實驗測定蛋白濃度。通過向NuPage® Novex 10% Bis-Tris Gels (Invitrogen)的每個孔中加入30微克蛋白/樣本進行SDS Page凝膠。將凝膠在200V的MES運行緩衝液(Invitrogen)中進行電泳40分鐘。然後採用I-Blot系統(Invitrogen)將蛋白質轉移到硝酸纖維素膜,並採用溶解於含0.1%Tween-20(TBS-T) (Sigma, St. Louis, MO) Tris緩衝鹽水的4% w/v低脂奶15毫升在室溫下阻斷2小時。採用以下抗體對該膜在室溫下過夜進行研究探:每個採用2% w/v低脂奶在5毫升TBS-T中稀釋(抗人LEFTY-長和短亞型 (R&D systems MAB7461); 抗人, 小鼠和大鼠 CD133 (Abcam AB19898); 抗人和小鼠UTF1 (Millipore MAB4337); 抗人NODAL (Abcam AB55676); 抗人和大鼠 CDH11 (OB Cadherin) (Thermo Scientific MA1-06306); 抗大鼠CD24 (Becton Dickinson))。採用TBS-T對膜進行洗滌(3次/ 10分鐘),然後採用在室溫下稀釋於2% w/v低脂奶(1:60,000)的TBS-T 1.5小時的適當HRP共軛結合次級抗體(Vector Labs PI-2000; PI-1000)進行研究。採用TBS-T對膜進行洗滌(3次/ 10分鐘),接著用diH2
O洗滌兩個10分鐘。採用ECL Advance化學發光試劑(GE Healthcare Life Sciences, Piscataway NJ)產生印跡,並使用ChemiDoc XRS分子成像儀和Quantity One®軟體(BioRad, Hercules CA)使其視覺化。 結果。開發出評價5/6 NX大鼠中儲備幹細胞和祖細胞轉移的分子實驗,並用於研究這些標記物對NKA治療的時間反應。據觀察到,NKA治療特別地與mRNA轉錄和蛋白水準上的關鍵幹細胞標記物CD24、CD133、UTF1、SOX2、LEFTY和NODAL的上調相關。注射後1周檢測到上調,注射後12周達到高峰。幹細胞和祖細胞標記物的啟動與相對於未經處理的5/6 NX對照組的存活率增加和明顯改善的血清生物標記物(即腎小球濾過改善)相關。 圖9顯示了採用NKA治療5/6 NX大鼠後宿主組織中SOX2 mRNA的表達。SOX2 mRNA表達的時間分析表明:植入後12周Nx對照組上面的NKA 治療組內SOX2 mRNA增加1.8倍。植入後24周Nx 對照組上面的NKA 治療組內SOX2 mRNA增加2.7倍。(1周:對於對照(假)、Nx (對照)和NKA治療中的每一個n= 3)(12周:對於對照(假)和Nx (對照)中的每一個n= 1;NKA治療n=4)(24周:對於對照(假)和Nx (對照)中的每一個n= 1;NKA治療n=4)。*表示p值=0.023或<0.05。 圖10 –蛋白質印跡顯示了假對照(對照)、Nx對照 (Nx)以及NKA #1和NKA #2治療後第1、12和24周的CD24、CD133、UTF1、SOX2、NODAL和LEFTY表達隨時間的變化過程。嵌入OCT冷凍劑的冷凍整腎組織(每個樣本N=1)被用於蛋白質樣本收集。通過負載的總品質蛋白質對泳道進行標準化。NKA治療組織中CD133、UTF1、NODAL、LEFTY和SOX2蛋白質水準SOX2的蛋白水準相對於各時間點的對照或Nx大鼠升高。 圖11描述了再生反應指數(RRI)隨時間的變化過程。個體蛋白表達(圖10)的光密度分析被用於產生再生標記物蛋白表達的定量指數或再生反應指數(RRI)。採用Image J v1.4軟體(NIH)計算出帶強度,並使每個蛋白質的每單位面積值標準化。通過編譯用於每個時間點蛋白質印跡的5個標記物測定假、Nx和NKA治療組的平均強度。圖顯示了第1、12和24周時間點產生的平滑線的xy散點。對每個組的平均強度隨時間的關係進行繪製,以突出幹細胞標記物蛋白表達的宿主組織中反應的趨勢。採用每個樣本的標準雙尾學生t-檢驗假設方差進行統計分析。95%的置信區間(p值<0.05)被用於確定統計學意義。(NKA治療組,n=2;對照(假)n=1;NX(對照)n=1)。在假對照動物中,RRI僅顯示從治療後1周(90.47)到治療後24周(81.89)略有減少。與此相反,得自5/6 Nx對照的腎基本上呈現相反的反應,RRI從治療後1周的82.26到治療後18周增加到140.56,而此時動物死亡。在NKA治療動物中,RRI從治療後1周的62.89大圖上升至治療後12周的135.61,並到治療後24周下降至112.61。 NKA治療被觀察到與宿主組織中轉錄和蛋白水準上的幹細胞標記物CD24、CD133、UTF1、SOX2、LEFTY和NODAL的上調相關。注射後1周檢測到上調,注射後12周達到高峰。宿主組織中幹細胞和祖細胞標記物的啟動與相對於未經腎切除手術處理的對照組的存活率增加和明顯改善的血清生物標記物(即腎小球濾過改善)相關。 對NKA治療有反應的儲備幹細胞和祖細胞群可有助於通過再生損傷腎組織和器官結構的5/6 NX動物中腎功能的修復。因此,本研究中所使用的分子實驗可提供一種評價CKD組織工程和再生醫學治療的再生結果的快速簡單的預測實驗。實例 12 - 原發性腎細胞源外泌體含有微型 RNA
我們尋求特定外泌體源性miRNA與體外靶細胞中功能相關性結果的相關性,以瞭解用於闡明再生結果機制的體內研究設計。 方法:採用商用細胞(HK-2(人近端腎小管細胞系)、原發性人腎系膜細胞(HRMC)和人臍帶內皮細胞(HUVEC))研究了再生培養基對與再生癒合反應相關信號路徑的影響。通過設計為檢測已知miRNA的PCR陣列對人和大鼠原發性腎細胞培養(UNFX)的條件培養基中外泌體的RNA含量進行篩選。據報導低氧影響外泌體脫落;因此,將一組培養基在培養基收集之前暴露於低氧(2%氧)下24小時。通過FACS從細胞碎片分離得到外泌體。 圖12提供了UNFX條件培養基製備和分析的原理圖。 結果:UNFX條件培養基被發現影響與再生癒合反應相關的信號路徑,這些反應並非在使用非條件培養基的中對照觀察到。具體來說,NFκB(免疫反應)和上皮細胞間質轉化(纖維化反應)在HK-2細胞中減弱,PAI-1(纖維化反應)在HRMC細胞中減弱,血管生成在內皮細胞中提升。得自UNFX條件培養基的外泌體含量的PCR陣列篩選初步資料表明,UNFX產生含有與觀察到對UNFX條件培養基反應一致的miRNA序列的外泌體。 圖13 A-C顯示,得自UNFX培養的條件培養基影響與再生結果潛在相關的多種細胞體外過程。NFkB信號被作為腎病中炎症過程的一種關鍵調節因數(Rangan et al., 2009. Front Biosci 12:3496-3522; Sanz et al., 2010. J Am Soc Nephrol 21:1254-1262),並且可以被腫瘤壞死因數(TNF)啟動。HK-2細胞與非條件培養基(左)或UNFX條件培養基(右)在37℃下預培養1小時,然後用或不用10納克/毫升腫瘤壞死因數啟動。 圖13A顯示UNFX條件培養基減弱NF-kB的TNF-a介導啟動。通過RelA/p65NFkB免疫螢光染色法(綠色)測定NFkB啟動。Hoechst反染核(藍色)和phalloidin染絲狀肌動蛋白(紅色)便於RelA/p65核定位(白色箭頭)的評價。 圖13B顯示UNFX條件培養基,促進了HUVEC細胞培養的血管形成作用。將HUVEC細胞(每孔10萬)覆蓋於培養基200加0.5%BSA的聚合基底膜上。添加無條件培養基(左)或UNFX條件培養基(右),採用圖像捕獲對細胞組織反應進行視覺化監測3-6小時。對細胞遷移(白色箭頭),對齊(黑色箭頭)、形成小管(紅色箭頭)以及形成封閉多邊形(星號)的細胞組織進行記錄。相比於無條件培養基,UNFX條件培養基誘發更多的小管和封閉多邊形,表明血管形成因數存在於培養基中。 圖13C顯示UNFX條件培養基抑制了上皮細胞的纖維化路徑。當在體外與轉化生長因數(TGF)接觸並複製與腎纖維化發展相關的上皮細胞間質轉化(EMT)(Zeisberg et al. 2003 Nat Med 9:964-968)時,HK-2細胞失去上皮細胞特徵,並獲得間質表型。HK-2細胞在無條件培養基(CTRL)、含有10納克/毫升TGFβ1(TGFβ1)的無條件培養基或含有10納克TGFβ1 (TGFβ1+CM)的UNFX條件培養基中培養72小時。通過CDH1(上皮標記物)、CNN1(間質標記物)和MYH11(間質標記物)的定量RT-PCR測定細胞。通過CDH1、CNN1和MYH11基因表達測量,條件培養基減少了TGFβ1誘導EMT度。誤差線表示三次實驗重複平均值(SEM)的標準誤差。 圖13D描述了通過TGFβ1和纖溶酶原啟動物抑制劑1 (PAI-1)建立的正回饋迴圈,當任其發展時,可導致細胞外基質蛋白的逐步積累(Seo et al., 2009. Am J Nephrol 30:481-490)。 圖14A-B顯示了系膜細胞纖維化路徑的減弱。HRMC在對照(CTRL)或有(+)或沒有(-)添加5納克/毫升TGFβ1的UNFX條件培養基(UNFX CM)中培養24小時。PAI-1的蛋白質印跡分析表明,UNFX CM抑制了TGFβ1誘導的PAI-1蛋白水準增加。bActin顯示為負載對照。在5納克/毫升TGFb1存在下,人腎系膜細胞(HRMC)表達增加(+)的PAI-1水準。與人生物活性腎細胞源條件培養基(CM)的共培養抑減弱TGFb1誘導的PAI-1蛋白表達。PAI-1的基因在mRNA水準的表達通過CM不變(資料未顯示)。 圖14B顯示,得自大鼠生物活性腎細胞的CM對採用TGFb1培養的HRMC誘導(+)和非誘導(-)具有類似的影響。離心後收集的CM上清液(除去大鼠CM)對抑制PAI-1表達不太有效,這表明導致PAI-1蛋白觀察到抑制的CM成分可與大鼠生物活性腎細胞分泌的囊泡相關。 圖15顯示了UNFX條件培養基含有分泌囊泡。圖145A描述了分泌囊泡(包括外泌體),其是包括細胞質源內部成分(綠色)的雙脂質結構(紅色)。磷脂醯絲氨酸(藍色三角形)都是在囊泡生物起源過程中暴露於胞外空間的膜成分(Thery et al., 2010. Nat Rev ImMunol 9:581-593)。 PKH26和CFSE分別標記物脂質膜和分泌囊泡的細胞質(Aliotta et al., 2010. Exp Hematol 38:233-245),而膜聯蛋白V結合磷脂醯絲氨酸。 圖15 B-C顯示了FACS分選。UNFX條件培養基標記物有PKH26、CFSE和APC共軛結合膜聯蛋白V,然後通過螢光輔助細胞分選(FACS)進行分選。收集表示分泌囊泡的三重陽性顆粒,並採用試劑盒試劑提取總RNA。採用商用基於RT-PCR的陣列篩選已知序列的微型RNA含量。 表12.1顯示了具有預測治療效果的含有微型RNA的分泌囊泡。UNFX細胞脫落含有已知miRNA序列的外泌體。UNFX條件培養基影響人細胞系中功能相關的再生反應。檢測的miRNA與觀察的再生反應之間的的因果關係正在積極調查;但迄今取得的結果表明,UNFX細胞有可通過miRNA的外泌體介導轉移對靶細胞和組織產生治療上相關的旁分泌效果。 表12.1
*Taganov et al, 2006. Proc Natl Acad Sci USA 103:12481-12486. **Chen and Gorski, 2008. Blood 111:1217-1226. ***Rogler et al., 2009. Hepatology 50:575-584. 這些資料支援了得自生物活性腎細胞培養的分泌囊泡含有減弱通過TGFb1/PAI-1反饋回路誘導的PAI-1成分的結論。 微陣列和RT-PCR分析. 得自Lewis大鼠的普通(UNFX)生物活性腎細胞於低氧條件下(2% O2
)在基礎培養基(不含有血清或補充物的DMEM和KSFM混合物(50:50))中培養24小時。收集條件培養基,並於4C下在100,000 xg下超高速離心2小時,以使分泌囊泡(例如,微囊泡、外泌體)成粒。從所得顆粒提取總RNA,並通過即時RT-PCR對已知的微型RNA進行測定 (大鼠微型RNA基因組V2.0 PCR 陣列; Qiagen #MAR-100A)。以下miRNA是可檢測的。 miR-21 miR-23a miR-30c miR-1224 miR-23b miR-92a miR-100 miR-125b-5p miR-195 miR-10a-5p miR-370 miR-24 miR-30a miR-16 miR-126* miR-30b-5p miR-27a miR-20a let-7c miR-26a miR-17-5p miR-30e miR-25 let-7b miR-20b-5p miR-29a let-7d miR-22 miR-322 let-7e miR-191 miR-99b miR-19b miR-10b miR-27b miR-125a-5p miR-30d miR-31 miR-93 miR-182 miR-99a miR-320 miR-664 miR-30e* let-7i miR-196a miR-26b miR-200a miR-126 miR-29c miR-200c miR-151 miR-429 miR-103 let-7a miR-322* miR-15b miR-378 miR-127 miR-199a-5p miR-181b miR-106b miR-196c miR-196b miR-19a miR-145 let-7f miR-181d miR-181a miR-221 miR-30a* miR-351 miR-218 miR-210 miR-98 miR-18a miR-342-3p miR-203 miR-352 miR-181c miR-222 miR-219-1-3p miR-708 miR-652 let-7d* miR-503 miR-138 miR-450a miR-365 miR-874 miR-345-5p miR-374 miR-872 miR-186 miR-130a miR-140* miR-28* miR-212 miR-139-3p miR-347 miR-151* miR-328 miR-185 miR-28 miR-192 miR-92b miR-672 miR-150 miR-425 miR-146a miR-107 miR-330* miR-409-3p miR-877 miR-760-3p miR-770 miR-152 miR-106b* miR-675 miR-423 miR-194 miR-490 miR-128 miR-497 miR-301a miR-130b miR-199a-3p miR-326 miR-132 miR-375 miR-25* miR-22* miR-96 miR-34a miR-223 miR-301b miR-505 miR-532-3p miR-7a miR-451 miR-34c* miR-339-3p miR-190 miR-671 miR-465 miR-674-3p miR-21* miR-99b* miR-125a-3p miR-183 miR-143 miR-324-5p miR-674-5p miR-760-5p miR-361 miR-296 miR-148b-3p miR-542-3p miR-667 miR-935 miR-24-2* miR-433 miR-295 miR-140 miR-9 miR-871 miR-29a* miR-542-5p miR-129 miR-214 miR-29c* miR-489 miR-141 miR-500 miR-17-3p miR-339-5p miR-7b miR-501 miR-206 miR-193* miR-350 miR-466b miR-205 miR-598-5p miR-532-5p miR-411 miR-188 miR-331 miR-124 miR-431 miR-154 miR-30d* miR-466c miR-142-3p miR-7a* miR-298 let-7b* miR-338 miR-449a miR-125b* miR-34c miR-346 miR-147 miR-9* miR-146b miR-219-5p miR-653 miR-340-3p miR-224 miR-330 miR-544 miR-193 miR-761 miR-181a* miR-27a* miR-34b miR-455 miR-20a* miR-488 miR-26b* miR-342-5p miR-543 miR-105 miR-291a-5p miR-138* miR-190b let-7i* miR-484 miR-30c-2* miR-125b-3p miR-764 miR-421 miR-485 miR-296* miR-29b miR-382 miR-511 miR-99a* miR-499 miR-101a* miR-20b-3p miR-434 miR-139-5p miR-10a-3p miR-148b-5p miR-598-3p miR-29b-2* miR-335 miR-215 miR-219-2-3p miR-327 miR-204 miR-504 let-7e* miR-129* miR-101b miR-122 miR-539 miR-30b-3p miR-184 miR-673 miR-463 miR-297 miR-363* miR-384-5p miR-344-5p miR-133a miR-582 miR-541 miR-344-3p miR-345-3p miR-362 miR-380 miR-878 miR-377 miR-409-5p miR-216a miR-496 miR-379 miR-711 miR-32 miR-291a-3p miR-487b miR-23a* miR-294 miR-207 miR-343 miR-292-3p miR-376a miR-540 miR-336 miR-134 miR-24-1* miR-363 miR-329 miR-133b miR-875 miR-201 miR-483 miR-742 miR-1 miR-293 miR-349 miR-30c-1* miR-153 miR-743a miR-323 miR-495 miR-410 miR-137 miR-300-3p miR-369-5p miR-376b-3p miR-211 miR-33 miR-758 miR-802實例 13- 生物活性腎細胞源旁分泌因數
在本研究中,我們採用體外細胞測定,以研究生物活性腎細胞通過調節劑(如纖溶酶原啟動物抑制劑-1(PAI-1))可以調控纖維化的潛在旁分泌機制。 材料和方法: 在血清和無補充條件下,從生物活性腎細胞(Aboushwareb et al., World J Urol 26, 295, 2008; Presnell et al. 2010上文)的大鼠和人培養收集條件培養基,並用於體外測定。商用大鼠和人源系膜細胞被用作體外測定中宿主回應組織的替代物,因為系膜細胞是一種對受傷或患病腎中PAI-1產生的來源(Rerolle et al., Kidney Int 58, 1841, 2000)。通過定量RT-PCR和蛋白質印跡分別測定PAI-1基因和蛋白表達。通過高速離心(Wang et al., Nuc Acids Res 2010, 1-12 doi:10.1093/nar/gkq601, July 7, 2010)收集由細胞脫落進入培養基的囊泡顆粒(例如,外泌體),並採用試劑盒試劑(Invitrogen)提取總RNA。採用已知RNA序列(Qiagen)的PCR陣列對囊泡的微型RNA內容物進行篩選。 結果:得自生物活性腎細胞培養的條件培養基減弱了系膜細胞中TGFβ1誘導PAI-1穩態蛋白水準的增加,但不影響穩態mRNA水準;觀察與微型RNA調控靶基因的機制一致。根據微型RNA可以通過外囊泡運輸在細胞間轉移的假說(Wang et al., 上文2010),我們分析了微型RNA內容物的條件培養基,並確認微型RNA 30b-5p (miR-30b-5p)的存在(PAI-1的假定抑制劑)。 此處提供的資料表明,生物活性腎細胞可直接通過外泌體靶向系膜細胞的miR-30b-5p的細胞間轉移對髒纖維化進行調控。由於通過系膜細胞吸收miR-30b-5p,減弱了穩態PAI-1蛋白水準的TGFβ1誘導增加,腎組織的反應可最終降低腎小球內細胞外基質空間的沉積。當前工作正在進行,以確認PAI-1確實是miR-30b-5p直接靶。 圖14A-B顯示了對照 (CTRL)或有(+)或沒有向培養基(-)添加TGFβ1的生物活性腎細胞條件培養基(CM)中培養24小時的人系膜細胞PAI-1和α-肌動蛋白(對照)蛋白表達的蛋白質印跡。在CTRL培養中,TGFβ1增加了PAI-1蛋白表達。在CM培養中,TGFβ1誘導的反應減弱。 對可是PAI-1的假定抑制因數的微型RNA的分泌囊泡進行分析。通過高速離心收集得自人和大鼠生物活性腎細胞CM的分泌囊泡,並採用已知序列的基於PCR陣列的微型RNA含量對其進行測定。miR-449a(PAI-1 (6)的推定調節劑)被確定。將miR-449a或非(CTRL)暫態轉染HRMC。轉染細胞後24小時,接觸5納克/毫升TGFb1(+)或不接觸(-)另外24個小時。 圖16A顯示了制得總蛋白的蛋白質印跡並對PAI-1和肌動蛋白進行測定。miR-449a降低的穩態PAI-1蛋白水準(將泳道1和3作比較)和誘導的PAI-11蛋白水準在miR-449a轉染培養(將泳道2和4作比較)中比較低。這些資料支援了分泌囊泡含有miR-449a和吸收入系膜細胞中的miR-449a降低PAI-1表達的結論。 圖16B描述了微型RNA miR-30b-5p在PCR陣列中被確定,並且是基於預測演算法的PAI-1的假定調節劑(https://mirbase.org – miRBase為曼徹斯特大學生命科學學院主頁)。 採用生物活性腎細胞治療5/6腎切除引起的CKD後,對腎小球的PAI-1蛋白水準進行了體內研究。 圖17A-C顯示了經歷單腎切除術(A)、5/6腎切除術(B)或生物活性腎細胞腎內傳送的5/6腎切除術(C)的Lewis大鼠腎中PAI-1 (A-C)的典型免疫組織化學成像。由於5/6腎切除操作(B)的腎小球(箭頭)中PAI-1的累積減少為治療(C)的結果。 在單獨研究中,對屍檢時收穫的腎組織進行qRT-PCR檢測,並在研究當天後繪製出相關基因的表達值。 圖17D顯示5/6(紅色方塊)腎切除大鼠相對於生物活性腎細胞(藍鑽石)及假手術對照(綠色三角形)治療的那些大鼠表現出PAI-1的更強表達。 圖17E顯示了治療後第3和6個月採取的腎樣本的典型蛋白質印跡。5/6(NX)腎切除大鼠(Nx)的治療組織(Nx+Tx)已經減少了PAI-1和纖連蛋白(FN)的累積(Kelley et al. 2010 上文)。 這些資料支援了採用生物活性腎細胞治療5/6腎切除誘導的CKD後腎小球中體內PAI-1蛋白水準降低的結論。 總之,例12-13支援了生物活性腎細胞的腎內運輸可通過調控儲備腎細胞的纖維化路徑的細胞間轉移改善腎功能的這一機制假設。實例 14- 得自生物活性腎細胞的分泌因數減弱了 NFκB 信號路徑
在本研究中,我們調查了5/6腎切除模型中疾病發展的NKA介導減弱中的NFκB路徑作用,並確定可有助於通過NFκB啟動的直接調控的再生結果的生物活性腎臟細胞特性。圖17G描述了通過TNFα的NFkB路徑的典型啟動。 材料與方法:從Lewis大鼠收穫殘存腎,其中兩步5/6腎切除術在採用PBS中的B2+B4(NKA原型)治療之前6周進行。通過免疫組織化學、RT-PCR、蛋白質印跡分析以及電泳遷移率變動檢測(EMSA)對NFκB啟動的NKA治療(TX)或未經治療(UNTX)組織進行測定。從生長於血清無補充培養基的間接體內NKA細胞培養收集的條件培養基(CM)用於體外功能測定。人類近端腎小管細胞系(HK-2)被用作分子和免疫螢光檢測資料分析的靶細胞類型。通過高速離心收集由細胞脫落進入培養基的囊泡顆粒(外泌體)。從外泌體分離的總RNA是採用已知微型RNA序列(Qiagen)的PCR陣列篩選的。 結果:從得自5/6腎切除大鼠的殘餘腎中觀察到NFκB亞基(RelA/p65)的核定位,這表明在UNTX組織炎症路徑的啟動。以RT-PCR檢測TX組織的初步分析結果顯示,RelA基因表達下降,這表明NKA治療可通過抑制RelA/p65表達影響NFκB路徑的啟動。這個假設是被CM抑制TNFα誘導的NFκB體外啟動的觀察支援的,相對於腫瘤壞死因數-α(TNF α)觀察到有所反應的CM接觸HK-2細胞(圖17F)中RelA/p65核定位的減少就是證明。NKA外泌體微型RNA的不間斷RT-PCR分析,正在研究已知的序列是否影響NFκB路徑存在。 圖17 F顯示:相比於螢光免疫檢測中TNFα預處理的對照培養所觀察,接觸NKA CM2小時減少了HK-2中NFκB p65的核定位(綠色)。在HK-2中,NFkB p65(綠色)在接觸TNFα (對照培養基) 30分鐘後使核定位。然而,在添加TNFα前,NKA條件培養基對HK-2細胞預處理2小時減弱了NFkB p65核定位的反應。DAPI(藍色)和絲狀肌動蛋白染色的核經過Alexa594-鬼筆環肽(紅色)染色,以有助於定性地評價NFκB核定位的穩健性(注意:最下麵一行合併面板中TNFα治療的對照細胞中鬼筆環肽邊界略有減少)。該反染色提供了合併圖像中NFkB定位的參考。 Lewis大鼠腎組織中NFkB p65亞基的免疫組織化學顯示,相對於對照動物(圖A)中單側腎切除引起的非進展性腎功能不全,5/6腎切除(圖B)引起的進展性CKD動物具有更穩健的NFkB p65亞基核定位,特別是在腎小管上皮細胞(黑箭頭)。腎切除術後6周收穫組織。放大倍數為200倍。 C圖:經歷5/6腎切除術的Lewis大鼠腎組織細胞質(‘C’)和核(‘N’)蛋白中NFkB p65的蛋白質印跡分析。比較第1和13周,其中微管蛋白水準(負載對照)相對一致,核NFkB p65隨著時間的推移而增加,與免疫組織化學結果相一致。 圖D:核提取物的電泳遷移率檢測(EMSA)證實了DNA結合啟動5/6腎切除術後定位於核的NFkB。泳道表示兩種動物在各時間點制得的核提取物。 NFkB路徑是在慢性腎病5/6腎切除模型中逐步啟動的。對Lewis大鼠腎組織中的NFkB p65亞基進行免疫組織化學分析。 圖18A-D顯示,相對於對照動物(圖A)中單側腎切除引起的非進展性腎功能不全,5/6腎切除術(圖B)引起的進展性CKD動物具有更穩健的NFkB p65亞基核定位,特別是在腎小管細胞(黑箭頭)。腎切除術後6周收穫組織。放大倍數為200倍。 圖18C顯示了經歷5/6腎切除術的Lewis大鼠腎組織細胞質(‘C’)和核(‘N’)蛋白提取物中NFkB p65的蛋白質印跡分析。比較第1和13周,其中微管蛋白水準(負載對照)相對一致,核NFkB p65隨著時間的推移而增加,與免疫組織化學結果相一致。 圖18D顯示,核提取物的電泳遷移率檢測(EMSA)證實DNA結合啟動5/6腎切除術後定位於核的NFkB。泳道表示兩種動物在各時間點制得的核提取物。1毫克核蛋白與5納克NFkB DNA結合位點在一起培養,並進行6%DNA凝膠電泳,隨後採用溴化乙錠染色。 NKA細胞的腎內傳送減少NFkB核定位。多重定義的腎細胞亞群已被分離,並對CKD的5/6腎切除模型中腎功能改善的生物活性進行體內測定(Presnell et al. 2010上文)。NKA細胞表現出生物活性,而其他亞群沒有(Kelley et al. 2010上文)。 圖18E顯示,接受NKA (A)或非活性腎細胞(B)腎內注射的CKD建立的Lewis大鼠。在治療後6個月,我們通過NFkB p65亞基的免疫組織化學收穫和檢測組織。相比於非生物活性腎細胞治療動物的組織,得自NKA治療動物的組織表現出NFkB p65的較少核定位,特別是在近端腎小管中,這表明NKA治療參與減弱NFkB路徑的體內活性。 通過採用已知序列的PCR陣列的高速離心從人和大鼠NKA條件培養基分離的分泌囊泡的微型RNA含量分析,確定了通過基於文獻報告(Marquez RT et al. (2010) Am J Physiol Gastrointest Liver Physiol 298:G535; Taganov KD et al. (2006) Proc Natl Acad Sci USA 103:12481)或預測演算法(https://mirbase.org – miRBase為英國曼徹斯特大學生命科學學院的主頁)的NFkB可影響免疫反應的幾種微型RNA。
體內和體外研究發現提供了生物活性腎細胞(NKA)如何通過調控免疫反應路徑(如通過NFkB啟動影響)可提高慢性腎病腎功能的知識。啟動NFkB(p65核定位,特別是在近端腎小管細胞中)與5/6腎切除鼠模型中慢性腎病的建立相關,並通過NKA治療減弱。近端腎小管細胞(HK-2)對NKA條件培養基的體外反應模擬了對NKA治療有所反應的NFkB核定位的體內減弱。NFkB啟動(微型RNA)細胞間抑制的推定調節劑在NKA條件培養基中被確定。總之,這些資料支援了生物活性腎細胞的腎內傳送可通過成分(例如,調控儲備腎細胞免疫反應的核RNA)的細胞間轉移改善腎功能這一機制的假設。實例 15- NKA 構建體的功能評價
接種於明膠或HA水凝膠的腎細胞群是能活的,並在體外成熟的3天期間保持腎小管上皮細胞的功能表型,以轉錄組學、蛋白質組學、分泌組學和共聚焦免疫螢光檢測衡量。為了研究NKA構建體可影響疾病狀態的潛在機制,評價了條件培養基對涉及CKD進展相關性腎小管間質纖維化的TGF-β信號路徑的影響。觀察到條件培養基可減緩TGF-β誘導的人近端腎小管細胞系(HK2)的體外皮間質轉化(EMT)。 材料和方法 生物材料。生物材料被製備為珠(均質,球形配置)或微粒(鋸齒狀邊緣的異質群)。Percell Biolytica (Åstorp, Sweden)產生的明膠珠(Cultispher S and Cultispher GL)分別購自Sigma-Aldrich (St. Louis, MO)和Fisher Scientific (Pittsburgh, PA)。交聯HA和HA/明膠(HyStem™和Extracel™購自Glycosan BioSystems, Salt Lake City, UT)微粒是由根據製造商說明制得的凍幹海綿構成。明膠(Sigma) 微粒是由交聯凍幹海綿構成。 PCL購自Sigma-Aldrich (St. Louis, MO)。PLGA 50:50購自Durect Corp. (Pelham, AL)。PCL和PLGA珠是採用改性雙乳液(W/O/W)溶劑萃取法制得。PLGA顆粒是採用溶劑澆鑄粒子濾出法制得。當在乾燥狀態測量時,所有珠和顆粒介於65至355微米之間。 細胞分離、製備和培養。按照用於研究目的人體組織的所有NIH使用指南,通過國家疾病研究所(NDRI)購得人屍的腎。犬腎購自合同研究組織(Integra)。大鼠腎(21日齡年老Lewis大鼠)得自查理斯河實(Aboushwareb et al. World J Urol 26(4):295-300; 2008; Kelley et al. 上文2010; Presnell et al. WO/2010/056328)。簡而言之,採用酶法在含有4.0單位/毫升分散酶(Stem Cell Technologies, Inc., Vancouver BC, Canada)和300單位/毫升膠原酶(Worthington Biochemical, Lakewood NJ)的緩衝液中對腎組織進行離解,然後通過15%碘克沙醇(Optiprep®, Axis Shield, Norton, MA)離心除去紅血細胞和碎片,以產生UNFX。將UNFX細胞接種於組織培養處理的聚苯乙烯板(NUNC, Rochester NY)上,並培養於50:50培養基,其為含有5%FBS、2.5微克EGF、25毫克BPE、1X ITS(胰島素/轉鐵蛋白/亞硒酸鈉補充物)和抗生素/抗真菌藥(全部來自Invitrogen, Carlsbad CA)的比例為1:1的高糖DMEM:角化細胞無血清培養基(KSFM)混合物。通過齧齒動物(16%、13%、11%和7%)、犬科動物(16%、11%、0%和7%)或人類(16%、11%、9%和7%)(Presnell et al. WO/2010/056328; Kelley et al. 上文2010)特異性分層的四步碘克沙醇(OptiPrep;未補充KSFM中60%w/v)。密度梯度在室溫、800 x g下離心20分鐘。通過吸管除去目標帶,並在無菌磷酸鹽緩衝液(PBS)中洗滌兩次。 細胞/生物材料複合材料(NKA構建體)。對於生物材料的體外細胞功能分析,將生物材料的均勻層(如上所述制得)在6孔低附著板(Costar #3471, Corning)的一個孔進行分層。將人類UNFX或B2細胞(每孔2.5 x 105
)直接接種於生物材料上。對於犬科動物細胞與生物材料的粘附研究,將2.5 x 106 UNFX細胞和50微升壓積的生物材料接種於非粘附24孔板(Costar #3473, Corning)上。在搖擺平臺4小時後,犬科動物NKA構建體在37℃,5%CO2
培養箱中過夜熟化。第二天,採用根據製造商說明的活/死染色檢測試劑盒(Invitrogen)進行活/死染色。大鼠NKA構建體是在一個轉速為1 RPM的滾子搖瓶裝置上的60 cc注射器中制得。 對於以下所述的轉錄組學、分泌組學和蛋白組學分析,NKA構建體熟化為3天。然後收穫用於轉錄組學或蛋白質組學分析的細胞,並收集用於分泌組學的條件培養基。 與酶活性相關的腎小管細胞的功能分析。採用改編自以前公佈方法(Tate et al. Methods Enzymol 113:400-419; 1985)的亮氨酸氨基肽酶(LAP)活性測定對24孔板中的犬科動物NKA構建體(10微升鬆散壓積)進行了評價。簡言之,在室溫下將PBS中0.5毫升的0.3毫摩爾L-亮氨酸p-硝基苯胺 (Sigma)加入NKA構建體1小時。從孔取樣為一式兩份,並將405納米的吸光度記錄為LAP活性測量。LLC-PK1細胞裂解液(美國菌種保藏中心或ATCC)作為陽性對照。 轉錄組學分析。採用RNeasy Plus微型試劑盒(Qiagen, CA)提取聚腺苷RNA。通過UV分光光度法測定濃度和完整性。採用SuperScript VILO cDNA合成試劑盒(Invitrogen)從1.4微克分離的RNA產生cDNA。通過採用商用引物和探針(表15.1)和ABI-Prism 7300即時PCR系統(Applied Biosystems, CA)的即時定量聚合酶鏈反應(qRT-PCR)對靶向轉錄表達水準進行測定。通過TaqMan基因表達Master混合液(ABI, Cat# 4369016)和TATA盒結合蛋白基因(TBP)作為內源性對照進行擴增。每個反應是由10微升Master混合液(2X)、1微升引物和探針(20X)和9微升基因組成。採取樣本為一式三份。 表15.1分泌組學分析。對得自人類NKA構建體的條件培養基進行收集,並在-80℃下冷凍。對樣本的生物標記物濃度定量進行評價。相對於對照培養(無生物材料的二維培養)的條件培養基中相同的生物標記物,對條件培養基中給定生物標記物濃度的的結果進行標準化,並表示為無單位的比率。 蛋白質組學分析。從細胞/生物材料複合材料提取三份獨立複製的蛋白,並通過二維凝膠電泳進行合併分析。所有試劑來自於Invitrogen。通過向pH 4-7 ZOOM IEF條 (Cat# ZM0012)中加入再懸浮於200微升ZOOM 2D蛋白增溶劑#1(Cat# ZS10001)、ZOOM兩性電解質pH值4-7(Cat# ZM0022)和2M DTT (Cat# 15508-013)的30微克蛋白質進行等電點聚焦(IEF)分析。在500V下電泳18小時後,將IEF條負載到用於SDS-Page分離的NuPage Novex 4-12%二-三ZOOM IPG孔膠(Cat# NP0330BOX)上,並在200V的MES緩衝液(Cat# NP0002)進行電泳45分鐘。根據製造商說明採用SYPRO Ruby蛋白凝膠染色(Cat# S-12000)使蛋白視覺化。 共聚焦顯微術。從人類或大鼠UNFX或B2細胞制得的NKA構建體熟化為3天,然後在2%多聚甲醛固定30分鐘。固定的NKA構建體被封閉,並通過在室溫(RT)下於D- PBS(Invitrogen) + 0.2% Triton X-100 (Sigma)的10%山羊血清(Invitrogen)中培養1小時使其浸透。對於免疫螢光,NKA構建體標記物有在室溫下過夜的最終濃度為5微克/毫升的初級抗體(表15.2)。採用2%山羊血清/D-PBS + 0/2% Triton X-100洗滌標記物NKA構建體兩次,並和5微克/毫升的山羊或兔TRITC共軛結合抗鼠IgG2A (Invitrogen)二級抗體共培養。對於DBA的雙標記物(雙花扁豆凝集素),備選NKA構建體進一步與2%山羊血清/D-PBS + 0.2% Triton X-100中稀釋為2毫克/毫升的FITC共軛結合DBA (Vector Labs)在RT水準共培養2小時。 表15.2採用D-PBS對樣本進行兩次洗滌,並用運行LSM成像軟體(Zeiss)或通路855共聚焦顯微鏡(BD Biosciences)的Zeiss LSM510鐳射掃描共聚焦系統(Cellular Imaging Core, Wake Forest Baptist Medical Center)進行光學切片。 HK2細胞中TGF-β介導EMT的分析。HK2細胞(ATCC)培養於纖連蛋白氮或膠原(IV)塗層培養皿((BD Biosciences)中的50:50培養基中。對於EMT檢測,將HK2細胞接種於培養基收集之前3天成熟的人類UNFX制得的二維(2D)人類UNFX培養或NKA構建體所收集的50:50培養基或條件培養基的24孔膠原(IV)塗層板(克隆率為70-80%)。在從EMT測定的細胞中分離RNA前3天,通過向培養基加入10納克/毫升啟動TGF-β誘導。通過在三天保溫培養期結束時分析E-鈣粘蛋白(上皮細胞標記物)和調寧蛋白(間質標記物)的相對表達的qRT-PCR對EMT進行監測。從以上所述TaqMan qRT-PCR分析收穫的HK2細胞製備核糖核酸準備RNA。採用每個樣本的標準雙尾學生t-檢驗假設方差進行統計分析。95%(p值0.05)和99%(p值<0.01)的置信區間被用於確定統計學意義。 非細胞生物材料和NKA構建體的體內植入。Lewis大鼠(6〜8周齡)均購自查理斯河(Kalamazoo, MI)。所有實驗操作都在Carolinas醫學中心的PHS和IACUC指南下進行。在異氟醚麻醉下,雌性Lewis大鼠(約2至3個月大)經受了正中切口,左腎暴露。通過微注射到腎實質介入35微升的包覆生物材料(非細胞生物材料或NKA構建體)。採用兩種注射軌跡:(i)對每極指向皮質(被稱為皮質注射),或(ii)從腎中線指向盂腎(被稱為髓注射)。在注射後第1、4或8周處死大鼠。沒有早期死亡發生。非細胞植入研究的研究設計存在於表15.3(ND =未完成)中。 腎組織學。收集典型腎樣本,並置於10%福馬林緩衝液24小時。將切片在升級的乙醇中脫水,並在石蠟中包埋。對切片(5微米)進行切割,安裝於帶電載玻片上,並根據標準染色協定(Prophet et al., Armed Forces Institute of Pathology: Laboratory methods in histotechnology. Washington, DC: American Registry of Pathology; 1992)採用蘇木精和伊紅(H&E染色)、Masson三色和高碘酸Schiff(PAS)進行處理。採用配備有Digital Sight (DS-U1)攝像機的Nikon Eclipse 50i顯微鏡在總放大倍率為40倍、100倍和400倍時捕獲數位顯微照片。通過常用(Shackelford et al. Toxicol Pathol 30(1):93-96; 2002)的強度等級方案(等級1、2、3、4)對腎形態變化進行評價,其中描述性術語(最小、輕度、中度、顯著/重度)分別用於描述觀察到的腎小球硬化、腎小管萎縮和擴張、上皮細胞管型、間質纖維化和炎症。 表15.3 結果
哺乳動物腎組織對生物材料注射入腎實質的反應。通過直接注射入健康大鼠腎(表15.3)的腎細胞/生物材料複合材料的潛在用途對生物材料進行了分析。通過在注射後第1和4周測量組織病理學參數(炎症、纖維化、壞死、鈣化/礦化)和生物相容性參數(生物材料降解、新血管生成和新組織形成)的程度對組織反應進行了評價。 圖19A-B顯示了植入後1周生物材料的體內評價。三色X10降低了顯示生物材料聚集的腎斷面的能量圖像。H&E X400:評價細胞/組織浸潤程度的高放大倍率生物材料聚集。如材料與方法所述對每個腎的兩個位置進行注射。在植入後1周,每個測試生物材料引起的宿主組織反應大致相似;然而,明膠凝膠似乎引起較少的強烈病理和較多的生物相容性反應。 圖19C顯示了植入後4周生物材料的體內評價。在植入後4周,採用HA或明膠顆粒注射的組織中病理學參數的嚴重程度相比於植入後1周定性地減少。明膠顆粒幾乎完全再吸收,接受明膠顆粒的組織中觀察到較少的巨細胞反應。在大多數情況下,通過脊髓注射軌跡(例如,深入到髓質/骨盆)對生物材料進行注射,觀察到包括導致腎盂積水的梗阻、更大程度的炎症反應以及導致心肌梗死的腎小動脈和毛細血管微栓塞的不良後果(資料未顯示)。 評價治療上與生物材料有關的腎細胞群的功能表型。直接注射入腎實質後慢性腎病的鼠模型中存活時間延長和腎功能增加的治療上相關的腎細胞群(UNFX)已被鑒定(Presnell et al. WO/2010/056328; Kelley et al. 上文2010),並已開發出它們的分離、鑒定和擴增方法以及翻譯跨越多個物種(Presnell et al. 2010上文)。當納入NKA構建體時,為了評價評價UNFX細胞是否粘附於仍然存活並保留主要腎小管上皮細胞表型,對UNFX細胞和各種生物材料產生的NKA構建體進行轉錄組學、分泌組學、蛋白質組學以及免疫螢光共聚焦顯微分析。 粘附和存活性。將犬源性UNFX細胞接種於所述的明膠珠、PCL珠、PLGA珠、HA顆粒以及HA/明膠顆粒(每個生物材料3個NKA構建體)。通過活/死染色接種後1天對細胞的分佈和存活性進行評價。 圖20A-D顯示了接種有犬科動物UNFX細胞(A =明膠珠; B = PCL珠; C =羥基磷灰石/明膠顆粒; D = HA顆粒)的NKA構建體的活/死染色。綠色表示活細胞;紅色表示死細胞。(A)明膠珠;(B) PCL珠;(C) HA/明膠顆粒;及(D) HA顆粒。所有水凝膠NKA構建體可觀察到活細胞。 UNFX細胞穩健地粘附于天然源水凝膠生物材料,如明膠珠和HA/明膠顆粒(A,D中的黑色箭頭),但顯示出最小粘附于合成PCL(B)或PLGA珠(未顯示)。細胞沒有粘附於HA顆粒(C),但表明有生物反應(即球體形成)。通過測定氨基肽酶(一種近端腎小管相關的水解酶(資料未顯示)),對接種到基於水凝膠的NKA構建體的UNFX細胞的功能存活性進行證實。 轉錄組譜。通過轉錄定量分析對基於水凝膠的NKA構建體和UNFX細胞的並行二維培養中人類UNFX細胞的基因表達譜(每3個神經激肽生物材料結構)進行了比較。 圖20E-G顯示了NKA構建體的轉錄組譜。TC:培養于2D的原發性人類UNFX細胞。明膠:NKA構建體由人類UNFX細胞和明膠凝膠組成。HA凝膠:NKA構建體由人類UNFX細胞和HA/明膠顆粒組成。定量RT- PCR資料以圖形和表格的格式提供。研究的轉錄屬於四個主要類別:(i)腎小管:水通道蛋白2(AQ2)、E-鈣粘蛋白(ECAD)、紅血球生成素(EPO)、N-鈣粘蛋白(NCAD)、細胞色素P450、24科、亞科A、多肽1-又名維生素D24-羥化酶(CYP)、cubilin、腎病蛋白;(ii)間質:調寧蛋白(CNN1)、平滑肌肌球蛋白重鏈(SMMHC);(iii)內皮:血管內皮生長因數(VEGF)、血小板內皮細胞粘附分子(PECAM);和(iv)腎小球:podocin。總體而言,基於水凝膠的NKA構建體和2D UNFX培養中的腎小管標記物表達均相似。同樣地,內皮標記物(血管內皮生長因數和粘附分子)相似。與此相反,NKA構建體之間的腎小球標記物表現出明顯的變化。HA/基於水凝膠的NKA構建體與2D UNFX培養中的podocin水準最相似。有趣的是,基於水凝膠的NKA構建體中的間質標記物(CNN1和SMMHC)表達相比於2D UNFX培養顯著下調(p < 0.05),表明腎培養基配方中基於水凝膠的NKA構建體中的UNFX成纖維細胞亞群可也不會增殖。 分泌組譜。採用人類UNFX細胞、B2細胞、明膠或HA/明膠凝膠產生NKA構建體(1個NKA構建體/每個生物材料/每個細胞類型=總共4個NKA構建體)。 圖21A- B顯示了NKA構建體的分泌組譜。資料被作為三維:二維比例顯示。如材料和方法中所述的人類UNFX或B2細胞和明膠(水凝膠1)或HA/明膠(水凝膠2)水凝膠產生NKA構建體。對得自3天成熟的NKA構建體的條件培養基進行分泌組譜分析,並通過計算NKA構建體(三維或3D培養)與二維培養的分析物表達之比(3D與2D之比)與人類UNFX或B2細胞的2D培養相比較。對於接種有UNFX細胞的3個NKA構建體中每一個,3D與2D之比分別達到或接近1,表明這些生物材料的接種過程和3天成熟對UNFX細胞的分泌組譜的影響不大。對於接種有B2細胞的NKA構建體,觀察到3D與2D之比分別達到或接近1的類似結果,這提供更多的證據表明,這些生物材料的接種過程和3天成熟對治療上相關的腎細胞的分泌組譜的影響不大。 蛋白質組譜。通過採用2D凝膠電泳分離總細胞蛋白產生給定細胞或組織的蛋白質組譜,並已用於鑒定與與腎病相關的特異性生物標記物(Vidal et al. Clin Sci (Lond) 109(5):421-430; 2005)。 圖22A-B顯示了NKA構建體的蛋白質組譜。如文采用人類UNFX細胞和生物材料產生NKA構建體。如材料和方法中所述的2D凝膠電泳分離人類總蛋白提取物中的蛋白質。在本實驗中,蛋白質組譜被用於比較NKA構建體和2D組織培養中人類UNFX細胞的蛋白表達(明膠或HA/基於明膠凝膠,3個NKA構建體/每生物材料)。從UNFX細胞的NKA構建體或2D組織培養分離的總蛋白的蛋白質組譜基本相同,這提供更多的證據表明,這些生物材料的接種過程和3天成熟對UNFX細胞表達的蛋白質組的影響不大。 共聚焦顯微術。通過建立生物標記物的共焦成像,對NKA構建體中大鼠和人類B2細胞(Presnell et al. 2010上文)的腎小管上皮細胞表型保留進行評價。圖23A-C顯示了NKA構建體的共聚焦顯微術。採用人類(A)或大鼠(B, C)B2細胞和明膠凝膠產生NKA構建體的共聚焦顯微術。(A)採用DIC光學器件使E-鈣黏蛋白(紅-白色實線箭頭)、DBA(綠-綠色虛線箭頭)和明膠凝膠珠視覺化。(B) DAPI染色視覺化的DNA(藍色-白色實線箭頭)和以下綠色標記物(白色虛線箭頭)的每個:IgG對照、N-鈣粘蛋白、E-鈣粘蛋白、細胞角蛋白8/18/18、DBA。(C)如文標記物和顏色的雙標記物。人類NKA構建體中的E-鈣粘蛋白和DBA以及大鼠NKA構建體中E-鈣粘蛋白、DBA、N-鈣粘蛋白、細胞角蛋白8/18/18、γ穀氨醯轉移酶(GGT-1)和並轉運給陰離子蛋白。共焦成像光學切片還對接種或3天成熟後細胞滲入生物材料的浸潤程度進行評價。人類和大鼠NKA構建體中B2細胞表現出多個腎小管上皮細胞標記物的表達。光學切片揭示了水凝膠構建體(一般只局限於生物材料表面細胞的)的最小細胞浸潤。 NKA構建體原型植入體內的反應。基於上述生物材料注射入腎實質的體內反應以及NKA構建體中UNFX和B2細胞的體外表型和功能特性,選用明膠水凝膠評價了NKA構建體注射入健康Lewis大鼠腎實質的體內反應。NKA構建體是由同系B2細胞產生,並植入兩個動物,其在植入後第1、4和8周被處死。當對腎組織切片進行收穫、切片以及採用三色、蘇木精和伊紅(H&E染色)和高碘酸Schiff(PAS)染色時,所有動物存活至預定屍檢。 圖24A-B顯示在了植入後第1和4周NKA構建體的體內評價。三色X10降低了顯示生物材料聚集的腎斷面的能量圖像。三色X400:生物材料聚集的放大圖。H&E/PAS X400: 評價細胞/組織浸潤程度的生物材料聚集的高放大倍率圖像。如上材料與方法所述對每個腎的兩個位置進行注射。 圖24A顯示了植入後第1周後NKA構建體的體內評價。在注射後1周,明膠珠被作為球形和多孔材料嗜鹼性染色的焦點聚集(左圖,圓圈區域),並通過標記物的成纖維血管組織、吞噬多核巨噬細胞和巨細胞包圍。纖維血管組織在珠內進行了整合,並顯示出表明新腎臟組織形成的腎小管上皮細胞部分。此外,通過形態學對腎小管和血管小球結構進行了鑒定(PAS圖)。 圖24B顯示了植入後第4周後NKA構建體的體內評價。在注射後4周,水凝膠被完全再吸收,該空間進展性腎再生和最低纖維化修復取代(注意,4周的三色圖區域內的許多功能性小管)。 圖25A-D顯示了植入後第8周後NKA構建體的體內評價。三色X10降低了顯示生物材料聚集的腎斷面的能量圖像。三色X400:生物材料聚集的放大圖。H&E/PAS X400: 評價細胞/組織浸潤程度的生物材料聚集的高放大倍率圖像。(A)中度慢性炎症(巨噬細胞,血漿細胞和淋巴細胞),具有顯著纖維血管反應(通過Masson三色進行藍染色-黑箭頭)的中等數量含鐵血黃素巨噬細胞(因注射的慢性出血);(B)與新腎組織形成一致的(A)顯示再生反應誘導的加框面積的高放大倍率(三色染色,X400)(C)表示呈現典型皮質腎小球形態學的相鄰(正常)腎實質,X400);(D)HE染色部分,X400比較治療面積中觀察到的新腎小球形態學與圖154C。 圖第25A-D顯示了植入後第8周後NKA構建體的體內評價。在植入後8周,觀察到新腎樣組織形成的跡象,與血管形成早期作用的誘導相一致。再生誘導的面積(B, D)與鄰近皮層薄壁(C)的比較顯示多個S形結構和新形成腎小球的存在。 得自NKA構建體的條件培養基對HK2細胞中TGF-β誘導EMT的影響。CKD進展過程中腎小管間質纖維化的發展與腎小管上皮細胞的TGF-β介導EMT相關(Zeisberg et al. Am J Pathol 160(6):2001-2008; 2002)。另外,觀察到進展性CKD的鼠模型在體內的TGF-β路徑減弱,其中生存期延長,並採用UNFX和B2細胞(Presnell et al. WO/2010/056328)治療改善腎功能。人類近端腎小管細胞系HK2已被充分地確立為體外模型系統,以測試小分子或蛋白質對TGF-β誘導的EMT的的促進或抑制效果(Dudas et al. Nephrol Dial Transplant 24(5):1406-1416; 2009; Hills et al. Am J Physiol Renal Physiol 296(3):F614-621; 2009)。為了研究潛在的機制,NKA構建體可影響植入後腎組織反應,對HK2 EMT檢測系統中從UNFX細胞和凝膠產生的NKA構建體收集的條件培養基進行了評價。 圖26顯示了得自NKA構建體的條件培養基減弱了體外HK2細胞中TGF-β誘導的EMT。通過定量測定ECAD(上皮)和CNN1(間質)標記物的相對表達對EMT進行監測。HK2細胞培養於50:50培養基(對照和TGFB對照樣本)或得自如上人類UNF X細胞或明膠或HA/明膠產生的人類UNFX細胞(TC)或NKA構建體的2D培養的條件培養基(CM)。為了誘導EMT,在檢測之前3天將10納克/毫升轉化生長因數-β添加到每個樣本(除對照外)中。當HK2細胞培養於50:50培養基(對照)時,ECAD(上皮標記物)表達的水準高於CNN1(間質標記物)。當將TGF-β添加到培養基3天(TGFB對照)時,ECAD表達顯著,伴隨有CNN1的上調,與EMT的事件誘導一致。得自2D UNFX細胞培養的條件培養基顯著減弱了HK2細胞對TGF-β(TC CM)的EMT反應(ECAD和CNN1的p<0.05)。得自NKA構建體(明膠CM和HA/明膠CM)的條件培養基同樣也減弱了對TGF-β EMT的反應;但總體效果明顯少於得自2D UNFX細胞培養的條件培養基的觀察情況(NKA構建體和趨向雖然CNN1統計學上顯著的對照的ECAD,顯著地-p < 0.05-)。對其他間質細胞標記物進行篩選,取得了類似的結果(資料未顯示)。這些資料表明,NKA構建體可以通過基於細胞治療觀察到的相似方式潛在地影響與體內腎小管間質纖維化路徑相關的TGF-β(Presnell et al. WO/2010/056328)。這些資料還表明,如果體內反應可以表明具有與體外EMT反應統計相關的統計學意義,體外EMT測定具有篩選/優化/檢測NKA構建體生物療效的應用潛力,從而有可減少耗時的需要和體內測定的花費。 本研究調查了哺乳動物腎實質對合成與天然生物材料(非細胞和生物活性腎細胞/生物材料複合材料(即NKA構建體))植入的反應。體外功能測定和體內再生結果相結合進行分析,以功能地篩選潛在納入NKA構建體的備選生物材料原型。基於非細胞凝膠的生物材料植入到腎實質(圖19)通常與植入後4周最少纖維化或慢性炎症以及沒有壞死跡象相關。觀察到生長的中度細胞/組織和新血管,具有最少的殘餘生物材料。根據這些體內資料,選用基於水凝膠的生物材料產生NKA構建體,用以評價體外生物功能和體內再生潛力。通過NKA構建體的活/死分析提供了材料生物相容性的體外確認(圖20)。含有明膠的水凝膠與原發性腎細胞群的強大粘附有關。生物活性原發性腎細胞群(UNFX或B2)和水凝膠生物材料產生的NKA構建體表型和功能分析與腎小管上皮細胞表型的持續保持相一致。NKA構建體的轉錄組學、分泌組學、蛋白質組學和共聚焦顯微鏡分析證實了相對於接種到2D培養的原發性腎細胞沒有顯著差異。最後,基於水凝膠的NKA構建體植入到健康成年鼠腎實質,與植入後8周新腎樣組織的最少炎症、纖維化反應及再生相關。 總之,這些資料提供了顯示通過NKA構建體內誘導再生反應的的證據。這些研究代表了哺乳動物腎對植入治療上相關的原發性腎細胞/生物材料複合材料的體內生物反應的第一腎內調查。觀察結果顯示NKA構建體有可既促進新腎組織的再生,又減弱非再生(例如,修復癒合)反應。 哺乳動物腎對植入聚合物材料的生物反應。在另一項研究中,調查了鼠腎內宿主組織對天然和合成生物材料腎內注射的反應,以評價形成含有生物活性腎細胞群細胞/生物材料複合材料的備用生物材料。方法:天然生物材料包括明膠和透明質酸(HA)。合成生物材料包括聚己酸內酯(PCL)的和聚乳酸乙醇酸共聚物(PLGA)。對兩種離散的物理構想(均勻球珠或異構和非均勻顆粒)的備用生物材料進行了評價。 PCL和PLGA珠是採用改性雙乳液(水/油/水)溶劑萃取法制得。購買明膠珠(Cultispher-S®, Sigma-Aldrich, St. Louis, MO)。PLGA顆粒是採用溶劑澆鑄粒子濾出法制得;明膠和HA顆粒是由交聯凍幹泡沫制得。將35微升結構鬆散的生物材料兩次注射到3個月大Lewis大鼠的左腎實質。在植入後1周和4周,採用炎症、組織/細胞生長、新血管、材料降解、成纖細胞反應的0(不存在)至4(明顯)的半定量分級嚴重程度量表對腎組織的福馬林固定切片進行病理組織學評價。總體分數被計算為陽性反應%與陰性反應%的比值(總得分越高結果越好)。 結果。對備用生物材料進行組織病理學評價 –表示植入後1周收穫的腎的40X圖像,用Masson三色(資料未顯示)對切片進行染色。當與合成生物材料(PLGA和PCL(組織纖維封裝))相比時,由天然來源聚合物組成的材料,如明膠和HA與組織較輕成纖細胞反應、慢性炎症、更大的細胞生長、新血管生成、生物材料降解以及癒合和整合需要的必要炎症相關。病理評價得分的綜述。材料組成(平均值±SD)為平均得分。合成材料(PLGA和PCL)的得分最低,明膠材料的得分一般比HA材料高。這種趨勢在4周時間點時最為明顯。由於材料注射無關的各種因素,不是第1周測試的所有樣本可用于第4周的分析。包括入明膠、HA和合成組的樣本數在第1周分別是3、4、3,第4周分別是2、3、1。 將傳送的天然來源生物材料(例如,明膠或HA)注射到健康腎實質引起的組織反應在注射後4周比合成來源生物材料的少,通過半定量組織病理學評價測量。實例 16- 缺氧培養的人腎細胞誘導細胞遷移和粘附的調節劑以及促進體外腎小管細胞單層修復
對氧含量在表現出慢性腎病模型(CKD)治療功能(B2)的選定腎小管上皮細胞分離和功能的作用進行了研究。本研究是調查過程中低氧接觸是否會改變選定的人腎細胞(SRC)或生物活性腎細胞(BRC)的組成和功能。在接觸2%氧時,觀察到以下內容:跨越密度梯度的細胞分佈改變(見Presnell et al. WO 10/056328,它們的全部內容通過引用併入本文)、整體梯度後產量的提高、氧調節基因表達的調控(以前在Kelley et al. 上文(2010)中報導)、紅血球生成素、VEGF、HIF1-α和KDR(VEGFR2)的表達增加。過程中接觸低氧增強了選定的生物活性腎細胞修復/再生損傷的腎小管的能力。 圖27描述了在過程中細胞接觸低氧的操作。圖28顯示,在接觸2%氧時,觀察到以下內容:改變跨越密度梯度的細胞分佈,提高了整體梯度後產量。相對於大氣氧含量(21%)(96%對比74%),低氧接觸(<3%)增加了得自碘克沙醇密度梯度的CKD源性腎細胞的回收,並增加了選定細胞(B2)向高密度(>9%碘克沙醇)組分的相對分佈分數(21.6%對比11.2%)。 競爭的體外實驗表明,缺氧條件下預接觸24小時的B2細胞比具有損傷兩小時內發生58.6% ± 3%修復的培養的B2細胞(21%氧含量)更善於修復腎近端腎小管細胞單層培養。 圖29A描述了體外觀察腎小管細胞單層修復的發展實驗。1.細胞,用螢光染料標記物細胞(2%氧、21%氧以及HK2腎小管細胞)。2.建立腎小管細胞單層,並使其受傷。3.添加氧接觸標記物細胞(2%和21%氧接觸細胞)。將它們同樣地以20,000/平方釐米接種。在5%O2
的無血清培養基中培養24小時。4.對修復受傷的細胞進行量化。圖29B-定量成像分析(BD PATHWAY855生物成像儀)的-紅色圓圈=2%氧培養的細胞,藍色圓圈=21%氧。圖29C-觀察到2%氧誘導細胞粘附更迅速(2小時),持續24小時溫和的優勢。2%氧誘導的細胞更善於腎小管上皮細胞單層的修復。 圖30A描述了體外觀察腎小管細胞單層修復的發展實驗。1.細胞,用螢光染料標記物細胞。2.在8微米孔徑的半透膜和受傷的底部建立腎小管細胞單層。3.翻轉半透膜,並添加氧接觸標記物細胞(2%和21%氧接觸細胞)。將它們同樣地以50,000/平方釐米接種。在5%O2
的無血清培養基中培養24小時。4.對修復受傷的細胞進行量化。 圖30B顯示了用2%氧細胞的誘導相比於未誘導(21%氧)提高了遷移和創面修復。圖30C繪製了遷移細胞與遷移時間的%。表16.1提供了細胞平均數和細胞平均比例。 缺氧還誘導CXCR4、MMP9、ICAM1和Dystroglycan的mRNA表達;基因介導細胞的遷移和粘附。通過免疫細胞化學法證實細胞質膜上MMP9的灶性累積和連接蛋白43聚集的增加。 圖31A顯示了骨橋蛋白是由腎小管細胞分泌以及對損傷有所反應而上調(骨橋蛋白免疫細胞化學:赫斯特核染色(藍色),骨橋蛋白(紅色),10倍)。骨橋蛋白是一種分泌磷酸化糖蛋白(Kelly et al. J Am Soc Soc Nephrol, 1999)。骨橋蛋白在腎小管中表達,並涉及黏附和遷移。骨橋蛋白是由免疫螢光(圖31A)和酶聯免疫吸附試驗(圖31B)所示的建立腎小管細胞膜單層的損傷上調的。 表16.1圖32A顯示了細胞遷移反應在某種程度上是由骨橋蛋白(綠色=遷移細胞(5倍))介導的。圖32B顯示了骨橋蛋白的中和抗體(NAb)減少了50%的腎細胞遷移反應。 圖33顯示了細胞低氧誘導調控了組織重構基因的表達。小窩蛋白1是涉及整合蛋白信號調控的支架蛋白。MMP9是一種通過細胞外基質降解促進遷移的金屬蛋白酶。ICAM1是與上皮細胞運動相關的細胞間粘附分子。CXCR4是介導細胞遷移的趨化因數表面受體。 圖34描述了細胞生物活性低氧增加導致腎再生的推定機制。 總之,這些結果表明,缺氧環境促進了具有體外腎小管損傷修復的已證明生物活性的特異性腎細胞亞群的分離,從而可會潛在地提高這些細胞在體內傳送後遷移並植入病變組織的能力。SRC表現出能夠穩定腎功能,並提高進展性CKD齧齒類動物模型的存活率。低氧水準(2%氧)提供如下:提高選定的再生細胞的培養後恢復;增強對腎小管損傷有所反應的細胞粘附及單層修護;以及促進對腎小管損傷有所反應的細胞遷移。此外,細胞遷移和粘附在某種程度上是由體外骨橋蛋白、低氧氣上調整合蛋白、分泌蛋白以及調控組織重構、遷移和細胞訊息傳遞的細胞粘附分子介導的。實例 17- 尿源性微囊泡
作者:對脫落到尿液的包含於腎源性微囊泡的腔內容物的miRNA與蛋白質進行分析,以確定它們是否可用作評價再生結果的生物標記物。過量的微囊泡脫落到細胞外空間,一些與鄰近細胞融合,而有些則是排泄到尿液(Zhou et al. 2008. Kidney Int. 74(5):613-621)。這些尿液微囊泡現在成為用於檢測發展的極好生物標記物,以便更好地瞭解治療效果。 採用慢性進展性腎衰竭與代謝性疾病的ZSF1齧齒動物模型。將B2與B4細胞注射入ZSF1動物的腎實質。健康動物和PBS載體被用作對照。以下總結了在不同時間點進行分析的尿源性微囊泡。 1: ZSF1 動物- PBS 載體注射; 在注射後第197天收集的尿液 2: ZSF1 動物- PBS 載體注射; 在注射後第253天收集的尿液 3: ZSF1 動物- B2 + B4組分注射; 在注射後第197天收集的尿液 4: ZSF1 動物- B2 + B4組分 注射; 在注射後第253天收集的尿液 5. ZSF1 動物-無注射; 在研究第197天收集的尿液 6. ZSF1 動物-無注射; 在研究第253天收集的尿液 7. 健康動物-無注射; 在研究第197天收集的尿液 8. 健康動物-無注射; 在研究第197天收集的尿液 治療後第197天和253天從測試動物收集尿液。通過本領域已知的標準方法從尿液回收微囊泡(例如,見Zhou et al. Kidney Int. 2008 September; 74(5): 613-621)。如按照圖35的標準蛋白質印跡所示,從治療動物(泳道3-4)尿液回收的微囊泡顯示,當與載體治療(泳道1-2)或非治療對照(通道5-8)相比時,與祖細胞(CD133 & WNT7A)相關的蛋白質增加。事實上,微囊泡只是從患病動物(泳道1-6)而不是健康對照(泳道7-8)的尿液回收,如微囊泡特異性蛋白CD63(圖35)的表達所示。含有CD133的微囊泡似乎是從腎臟細胞脫落的prominosome。CD133和WNT7A都與再生和幹細胞分裂相關(Romagnani P and Kalluri R. 2009. Fibrogenesis Tissue Repair. 2(1):3; Lie et al. 2005. Nature. 437(7063):1370-5; Willert et al. 2003. Nature. 423(6938):448-52; Li et al. 2009. Am J Physiol Renal Physiol. 297(6):F1526-33)。總之,這支持了靶蛋白質表達為微囊泡中旨在監測再生的實驗發展的生物標記物。 miRNA微陣列和RT-PCR。通過本領域已知的標準方法對得自尿源性囊泡的miRNA進行微陣列和RT- PCR分析(例如,見Wang et al. 上文2010)。除了蛋白質,發現微RNA在分離的微囊泡內容物中。表17.1提供了被發現採用治療增加的miRNA的實例。 表 17.1
對採用B2+ B4(第197天和第253天)治療的ZSF1動物中的miRNA水準隨時間的變化進行分析。觀察到以下miRNA的差異倍數: miR-370 miR-362 miR-1224 miR-22 miR-598-5p miR-540 miR-300-3p miR-206 miR-222 miR-433 let-7b let-7c miR-497 let-7b* miR-24-2* miR-770 miR-124 miR-485 miR-760-5p miR-296 miR-764 miR-221 miR-883 miR-15b miR-143 let-7a miR-339-3p miR-484 miR-742 miR-101b miR-425 miR-31 miR-29b let-7e* miR-345-3p miR-193 miR-207 miR-501 miR-125b-3p miR-154 miR-375 miR-298 miR-671 miR-21 let-7e let-7f miR-125a-5p miR-194 miR-152 miR-128 miR-423 miR-92b miR-139-3p miR-667 miR-98 miR-181d miR-500 miR-103 miR-99b* miR-935 miR-674-5p miR-743a miR-352 miR-326 miR-181c miR-504 miR-322* miR-7a miR-532-3p miR-29a miR-130b miR-328 miR-345-5p miR-29c miR-215 miR-138* miR-182 miR-147 miR-664 miR-339-5p miR-743b miR-96 miR-203 miR-130a miR-346 miR-674-3p miR-140* miR-192 miR-409-3p miR-106b* let-7d miR-28* miR-219-2-3p miR-148b-3p miR-23b miR-195 miR-30d miR-301b miR-320 miR-652 miR-322 miR-429 miR-291a-5p miR-26a miR-141 miR-185 miR-27b miR-200c miR-431 miR-16 miR-23a miR-330* miR-344-5p miR-347 miR-125b* miR-30b-5p miR-218 miR-99a miR-488 miR-493 miR-9* miR-92a miR-125b-5p miR-183 miR-708 miR-191 miR-181b miR-186 miR-872 miR-188 miR-22* miR-27a miR-7b miR-598-3p miR-490 miR-675 miR-10a-5p miR-184 miR-100 miR-874 miR-200a miR-30c miR-99b miR-212 let-7i miR-30a miR-199a-5p miR-25 miR-99a* miR-28 miR-30d* miR-151* miR-344-3p miR-126* miR-145 miR-20b-5p miR-294 miR-489 miR-483 miR-132 miR-505 miR-7a* let-7i* miR-21* miR-331 miR-434 miR-106b miR-30e miR-350 miR-34a miR-219-1-3p miR-543 miR-210 miR-30a* miR-365 miR-378 miR-196b miR-27a* miR-361 miR-10b miR-466c miR-342-3p miR-216a miR-101a* miR-24 miR-151 miR-205 miR-17-5p miR-134 miR-296* miR-26b* miR-30e* miR-382 miR-410 miR-327 miR-133b miR-142-3p miR-324-5p miR-125a-3p let-7d* miR-19b miR-223 miR-374 miR-127 miR-26b miR-25* miR-330 miR-146a miR-295 miR-196a miR-34b miR-129 miR-196c miR-17-3p miR-133a miR-19a miR-211 miR-20a miR-34c miR-871 miR-93 miR-9 miR-532-5p miR-181a miR-760-3p miR-140 miR-29a* miR-138 miR-193* miR-10a-3p miR-877 miR-29c* miR-503 miR-148b-5p miR-107 miR-34c* miR-672 miR-873 miR-351 miR-292-5p miR-343 miR-449a miR-323 miR-29b-2* miR-539 miR-465 miR-214 miR-541 miR-761 對採用B2+ B4(第253天)治療的ZSF1動物中的miRNA水準進行分析,並與採用PBS載體(第253天)治療的ZSF1動物中的miRNA水準相比。觀察到以下miRNA的差異倍數: miR-24 miR-195 miR-871 miR-30b-5p miR-19b miR-99a miR-429 let-7f miR-200a miR-324-5p miR-10a-5p miR-148b-3p miR-100 miR-16 let-7d* miR-30e miR-200c miR-292-5p miR-152 let-7i miR-351 miR-434 miR-26b miR-489 miR-186 miR-191 miR-10b miR-365 miR-431 miR-29c miR-15b miR-21 miR-125b-5p miR-30c miR-30a miR-503 miR-26a miR-30d miR-743b miR-9 let-7e miR-23a miR-322* miR-96 miR-106b miR-23b miR-27b miR-29a let-7a miR-425 miR-20a miR-22* miR-98 miR-25 miR-194 let-7d miR-141 miR-7a* miR-107 miR-93 miR-742 miR-505 miR-19a miR-17-5p miR-743a miR-182 miR-378 miR-761 miR-103 miR-221 miR-212 miR-27a miR-30d* miR-148b-5p miR-138* miR-465 miR-129 miR-344-3p miR-330 miR-374 miR-10a-3p miR-352 miR-181a let-7i* miR-134 miR-196a miR-203 let-7c miR-151 miR-20b-5p miR-26b* miR-140 miR-128 miR-222 miR-29a* miR-872 miR-328 miR-760-3p miR-295 miR-151* miR-138 miR-211 miR-339-5p miR-382 miR-497 miR-184 miR-375 let-7b miR-133a miR-345-5p miR-342-3p miR-34a miR-7a miR-291a-5p miR-34c miR-300-3p miR-219-1-3p miR-30a* miR-125a-5p miR-323 miR-327 miR-883 miR-541 miR-708 miR-196c miR-216a miR-146a miR-27a* miR-223 miR-106b* miR-466c miR-490 miR-326 miR-664 miR-29b miR-30e* miR-532-3p miR-199a-5p miR-154 miR-127 miR-667 miR-92a miR-140* miR-322 miR-181b miR-296* miR-339-3p miR-28* miR-24-2* miR-345-3p miR-330* miR-504 miR-449a miR-210 miR-188 miR-410 miR-101a* miR-147 miR-192 miR-296 miR-196b miR-433 miR-17-3p miR-350 miR-142-3p miR-423 miR-101b miR-219-2-3p let-7e* miR-484 miR-1224 miR-320 miR-675 miR-105 miR-874 miR-500 miR-501 miR-347 miR-338 miR-671 miR-34c* miR-92b miR-29b-2* miR-298 miR-139-3p miR-342-5p miR-877 miR-99a* miR-129* miR-181c miR-21* miR-25* miR-143 miR-770 miR-652 miR-183 miR-22 miR-485 miR-935 miR-532-5p miR-764 miR-193 miR-672 miR-325-5p miR-760-5p miR-346 miR-301a miR-488 miR-539 miR-132 miR-126* miR-99b miR-125a-3p miR-181d miR-543 let-7b* miR-99b* miR-463 miR-146b miR-343 miR-205 miR-23a* miR-674-5p miR-214 miR-540 miR-133b miR-29c* miR-758 miR-362 miR-34b miR-409-3p miR-185 miR-344-5p miR-294 miR-31 miR-125b-3p miR-204* miR-361 miR-711 miR-215 miR-301b miR-206 miR-370 miR-130a miR-193* miR-873 miR-218 miR-124 miR-130b 對採用B2+ B4(第197天)治療的ZSF1動物中的miRNA水準進行分析,並與採用PBS載體(第197天)治療的ZSF1動物中的miRNA水準相比。觀察到以下miRNA的差異倍數: miR-143 miR-370 miR-351 let-7a miR-152 miR-141 let-7c miR-222 miR-362 miR-200a miR-188 miR-429 miR-505 miR-21 let-7e miR-182 let-7b let-7i miR-200c miR-99a miR-221 miR-30b-5p let-7d miR-103 miR-148b-3p miR-26a miR-186 miR-24-2* miR-26b miR-375 let-7f miR-206 miR-29a miR-100 miR-29c miR-16 miR-96 miR-151 miR-125a-3p miR-195 miR-210 miR-742 miR-30d miR-194 miR-433 miR-23b miR-124 miR-101b miR-497 miR-425 miR-347 miR-19a miR-431 miR-17-5p miR-98 miR-434 miR-339-5p miR-296 miR-667 miR-181b miR-324-5p miR-30e miR-10a-5p miR-125a-5p miR-29b miR-28* miR-106b* miR-30a miR-423 miR-19b miR-500 miR-92a miR-291a-5p miR-181d miR-320 miR-345-3p miR-764 miR-191 miR-10b miR-298 miR-92b miR-22 miR-330* miR-484 miR-339-3p miR-106b miR-25 miR-326 miR-129 miR-31 miR-34a miR-652 miR-15b miR-130a miR-378 miR-30c let-7d* miR-874 miR-485 miR-93 miR-671 miR-99b* miR-139-3p miR-27b miR-21* miR-328 miR-185 miR-743b miR-127 miR-345-5p miR-140* miR-20a miR-374 miR-664 miR-877 miR-24 miR-205 miR-196b miR-126* miR-20b-5p miR-128 miR-22* miR-196c miR-192 miR-151* miR-134 miR-214 miR-674-5p miR-125b-5p miR-365 miR-532-3p miR-29c* miR-7a miR-147 miR-27a miR-181c miR-99b miR-125b-3p miR-193 miR-342-3p miR-215 miR-132 miR-532-5p miR-203 miR-130b miR-449a miR-7a* miR-219-2-3p miR-23a miR-322 miR-181a miR-219-1-3p miR-30d* miR-301b let-7e* miR-196a miR-9 miR-27a* miR-488 miR-183 miR-26b* miR-138 miR-382 miR-760-3p let-7i* miR-184 miR-25* miR-34c miR-30a* miR-466c miR-28 miR-142-3p miR-107 miR-148b-5p miR-331 miR-218 miR-30e* miR-330 miR-344-5p miR-493 miR-17-3p miR-7b miR-34b miR-193* miR-216a miR-99a* miR-483 miR-223 miR-361 miR-503 miR-873 表17.1列出的miRNA提供了已涉及相對于組織再生過程的miRNA實例。miR-15b已涉及通過Bcl-2和細胞凋亡蛋白酶(Guo et al. 2009. J Hepatol. 50(4):766-78)調控的細胞凋亡調控以及通過細胞週期蛋白調控(Xia et al. 2009. Biochem Biophys Res Commun. 380(2):205-10)的細胞週期進展。miR-21的結果表明,通過調控存活路徑MAPK/ERK抑制細胞凋亡。miRNA的miR-30家族是足細胞結構和功能的關鍵,表明增加可對腎小球發生是必須的。miR-141、200a、200c和429都參與調控對TGF-β信號可減少纖維化有所反應的上皮細胞間質轉化(EMT)(Saal et al. 2009. Curr. Opin. Nephrol. Hypertens. 18:317-323)。miR-146a和151已涉及NFκB調控,從而潛在地減少了體內炎症反應(Taganov et al. 2006. Proc Natl Acad Sci U S A. 103(33):12481-6; Griffiths-Jones et al. 2006. NAR. 34 Database Issue: D140-D144)。總的來說,這些miRNA調控與成功的再生結果的相關過程;因此使它們成為檢測發展的備用生物標記物。總體而言,這一資料支援的概念,尿微囊泡和/或它們的腔內容物是再生檢測的活靶,因為它們含有能夠調控包括以下多種路徑的蛋白質和miRNA:除了為從業人員提供監測治療的非侵入性方法之外的TGF β-1、NFκB、細胞凋亡、細胞分裂和多潛能。
圖1顯示了採用多層不連續梯度法(A-左圖)或單層混合梯度法(B-右圖)由於新鮮分離腎組織富集EPO產生細胞組分。這兩種方法都導致來自EPO帶的非EPO產生細胞組分(主要是腎小管細胞)的部分除去,其表現為介於1.025克/毫升和1.035克/毫升之間。 圖2顯示了分別收穫和並行應用於同一不連續梯度的“常氧的”(21%氧)和“缺氧的”(2%氧)齧齒動物培養的不連續梯度。 圖3顯示了分別收穫和並行應用於同一不連續梯度的“常氧的”(21%氧)和“缺氧的”((2%氧)犬科動物培養的不連續梯度。 圖4顯示了HK17和HK19樣本的組織病理學特徵。 圖5顯示了人類NKA細胞界定目標區域(ROI)中白蛋白運輸的高含量分析(HCA)。 圖6顯示了非CKD和CKD腎源性NKA細胞中白蛋白運輸的定量比較。 圖7描述了腎小管富集的B2和腎小管細胞去除的B4亞組分之間標記表達的比較分析。 圖8描述了腎小管富集的B2和腎小管細胞去除的B4亞組分之間白蛋白運輸的比較功能分析。 圖9顯示了5/6 NX大鼠治療後宿主組織中SOX2 mRNA的表達。 圖10顯示了CD24、CD133、UTF1、SOX2、NODAL和LEFTY表達隨時間變化的蛋白質印跡。 圖11描述了再生反應指數(RRI)隨時間的變化。 圖12提供了UNFX條件培養基製備和分析的原理圖。 圖13A-C顯示了得自 UNFX培養的條件培養基影響與再生結果潛在相關的多種細胞體外過程。圖13A顯示了UNFX條件培養基減弱NF-kB 的TNF-a介導啟動。圖13B顯示了UNFX條件培養基提高HUVEC細胞培養的促血管形成性能。圖13C顯示了UNFX條件培養基減弱上皮細胞中的纖維化路徑。圖13D描述了通過TGFβ1和纖溶酶原啟動物抑制劑1(PAI-1)建立的正反饋回路。 圖14A-B顯示了表明系膜細胞中纖維化路徑減弱的蛋白質印跡。 圖15 A-C顯示了UNFX條件培養基含有分泌囊泡。圖15A描述了分泌囊泡,其為包括細胞質源性內部成分(綠色)的雙脂質結構(紅色)。圖15B-C顯示了FACS分選。 圖16A顯示了用於PAI-1和肌動蛋白製備及檢測的總蛋白的蛋白質印跡。圖16B描述了microRNA,miR-30b-5p。 圖17A-C顯示了經受腎切除術後接著傳送生物活性腎細胞的Lewis大鼠腎中PAI-1的典型免疫組織化學圖像。圖17D顯示了未經治療的腎切除大鼠(紅色方格)、經治療的腎切除大鼠(藍色方塊)和對照動物(綠色三角形)中的PAI-1表達的比較。圖17E顯示了治療後第3和6個月採取腎樣本的典型蛋白質印跡分析。圖17F顯示了接觸NKA培養基2小時減少NFκB p65的核定位。圖17G描述了通過TNFα的典型NFkB路徑啟動。 圖18A-B顯示了 (A)通過5/6腎切除術啟動的進展性CKD和(B)通過單側腎切除術啟動的非進展性腎功能不全的動物中NFkB p65亞基的核定位。圖18C-D顯示了(C)已經受5/6腎切除術的Lewis大鼠腎組織提取物中NFkB p65的蛋白質印跡分析;和(D)提取物的電泳遷移率檢測(EMSA)。圖18E顯示了由接受NKA(A圖)或非活性腎細胞(B圖)腎內注射的建立CKD的Lewis大鼠獲得的組織中NFκB p65亞基的免疫組織化學檢測。 圖19A-C顯示了生物材料在植入術後第1周和4周的的體內評價。 圖20A- D顯示了NKA構建體的活/死染色。圖20E-G顯示了NKA構建的轉錄組譜。 圖21A-B顯示了NKA構建體的分泌組譜。 圖22A-B顯示了NKA構建體的蛋白質組譜。 圖23A-C顯示了NKA構建體的共焦顯微術。 圖24A-B顯示了NKA構建體在植入後第1周和4周的體內評價。 圖25A-D顯示了NKA構建體在植入後第8周的體內評價。 圖26顯示了得自NKA構建體的條件培養基減弱體外HK2細胞中TGF-β誘導的EMT。 圖27描述了在過程中細胞與低氧接觸的操作。 圖28顯示了在接觸2%氧時,觀察到以下內容:改變跨越密度梯度的細胞分佈,提高整體梯度後產量。 圖29A描述了發展為觀察體外腎小管單層修復的檢測。圖29B顯示了定量圖像分析(BD PATHWAY 855成像儀)的結果。圖29C顯示了更善於腎小管上皮細胞單層修復的2%氧誘導的細胞。 圖30A描述了發展為觀察體外腎小管單層修復的檢測。圖30B顯示了相比於未經誘導(21%氧)採用2%氧誘導的細胞增強遷移和創面修復。圖30 C繪製了相對於遷移時間遷移細胞的比例。 圖31A顯示,骨橋蛋白是由腎小管細胞分泌,並在對損傷作出反應中表達上調(骨橋蛋白免疫細胞化學:Hoechst核染色(藍色),骨橋蛋白(紅色),10倍)。骨橋蛋白是通過如免疫螢光(圖31A)和ELISA(圖31B)所示建立的腎小管細胞單層中的損傷而表達上調。 圖32A顯示,細胞遷移反應在某種程度上是由骨橋蛋白(綠色=遷移細胞(5倍))介導。圖32B顯示,抗體(NAb)中和為骨橋蛋白減少了50%的腎細胞遷移反應。 圖33顯示了細胞低氧誘導調控了組織重構基因的表達。 圖34描述了細胞生物活性的低氧增強導致腎再生的推定機制。 圖35顯示了通過蛋白質印跡的微囊泡檢測。
Claims (11)
- 一種包含分離之由富集之腎細胞群所產生之人類分泌之囊泡的組合物之用途,其係用於製備治療人類個體之腎臟疾病的藥物,其中該囊泡包含選自以下中至少兩者之微RNA(micro RNA):miR-30b-5p、miR-449a、miR-146a、miR-130a、miR-23b、miR-21、miR-124、miR-151、miR-145、miR-22、miR-7a、miR-7b、miR-10a-5p、miR-143及let7b;且其中該藥物係供體內投予,且向人類個體之自體腎提供再生效果。
- 如請求項1之用途,其中該微RNA係選自以下中至少兩者:miR-30b-5p、miR-449a、miR-146a、miR-130a、miR-23b、miR-21、miR-124及miR-151。
- 如請求項2之用途,其中該微RNA係選自以下中至少兩者:miR-146a、miR-21、miR-124及miR-151。
- 如請求項3之用途,其中該再生效果係炎症減少。
- 如請求項4之用途,其中該炎症減少係核因子κ B(Nuclear Factor kappa B,NFκB)信號減弱。
- 如請求項5之用途,其中該NFκB信號減弱係由腫瘤壞死因數α (TNF α)介導。
- 如請求項1至6中任一項之用途,其中該囊泡包含外來體(exosome)。
- 如請求項1至6中任一項之用途,其中該囊泡包含微囊泡。
- 如請求項1之用途,其中該富集之腎細胞群包含:富集之腎小管細胞群;或富集之腎小管細胞群以及紅血球生成素產生細胞、腎小球細胞和血管細胞中的一種或多種。
- 如請求項1之用途,其中該富集之腎細胞群對該人類個體而言係自體。
- 如請求項1之用途,其中該富集之腎細胞群對該人類個體而言係非自體。
Applications Claiming Priority (20)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US33403210P | 2010-05-12 | 2010-05-12 | |
| US61/334,032 | 2010-05-12 | ||
| US35389510P | 2010-06-11 | 2010-06-11 | |
| US61/353,895 | 2010-06-11 | ||
| US37188810P | 2010-08-09 | 2010-08-09 | |
| US37207710P | 2010-08-09 | 2010-08-09 | |
| US61/372,077 | 2010-08-09 | ||
| US61/371,888 | 2010-08-09 | ||
| US37658610P | 2010-08-24 | 2010-08-24 | |
| US61/376,586 | 2010-08-24 | ||
| US38876510P | 2010-10-01 | 2010-10-01 | |
| US61/388,765 | 2010-10-01 | ||
| US41293310P | 2010-11-12 | 2010-11-12 | |
| US41338210P | 2010-11-12 | 2010-11-12 | |
| US61/413,382 | 2010-11-12 | ||
| US61/412,933 | 2010-11-12 | ||
| US201161441423P | 2011-02-10 | 2011-02-10 | |
| US61/441,423 | 2011-02-10 | ||
| US201161473111P | 2011-04-07 | 2011-04-07 | |
| US61/473,111 | 2011-04-07 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| TW201902494A TW201902494A (zh) | 2019-01-16 |
| TWI690320B true TWI690320B (zh) | 2020-04-11 |
Family
ID=44121234
Family Applications (5)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| TW107113584A TWI690320B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW105117487A TWI629990B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW111113839A TWI840787B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW109109511A TWI762890B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW100116779A TWI542690B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
Family Applications After (4)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| TW105117487A TWI629990B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW111113839A TWI840787B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW109109511A TWI762890B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
| TW100116779A TWI542690B (zh) | 2010-05-12 | 2011-05-12 | 生物活性腎細胞 |
Country Status (15)
| Country | Link |
|---|---|
| US (3) | US10077442B2 (zh) |
| EP (3) | EP3190177B8 (zh) |
| JP (6) | JP5974215B2 (zh) |
| KR (5) | KR102428776B1 (zh) |
| CN (2) | CN106214700B (zh) |
| AU (5) | AU2011252915B2 (zh) |
| CA (1) | CA2798900C (zh) |
| DK (1) | DK3190177T3 (zh) |
| ES (1) | ES2906750T3 (zh) |
| HK (1) | HK1232143A1 (zh) |
| HU (1) | HUE058155T2 (zh) |
| NZ (1) | NZ603482A (zh) |
| PL (1) | PL3190177T3 (zh) |
| TW (5) | TWI690320B (zh) |
| WO (1) | WO2011143499A1 (zh) |
Families Citing this family (46)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| ITRM20030376A1 (it) | 2003-07-31 | 2005-02-01 | Univ Roma | Procedimento per l'isolamento e l'espansione di cellule staminali cardiache da biopsia. |
| US11660317B2 (en) | 2004-11-08 | 2023-05-30 | The Johns Hopkins University | Compositions comprising cardiosphere-derived cells for use in cell therapy |
| US20090104649A1 (en) | 2007-06-11 | 2009-04-23 | Garovic Vesna D | Markers for preeclampsia |
| BR112012017767A2 (pt) | 2009-12-21 | 2019-09-24 | Mayo Found Medical Education & Res | marcador precoce de proteinúria em pacientes tratados com um tratamento anti-vegf |
| US9845457B2 (en) | 2010-04-30 | 2017-12-19 | Cedars-Sinai Medical Center | Maintenance of genomic stability in cultured stem cells |
| US9249392B2 (en) | 2010-04-30 | 2016-02-02 | Cedars-Sinai Medical Center | Methods and compositions for maintaining genomic stability in cultured stem cells |
| TWI690320B (zh) | 2010-05-12 | 2020-04-11 | 開曼群島商英瑞金公司 | 生物活性腎細胞 |
| CA3065694A1 (en) | 2010-11-10 | 2012-05-18 | Inregen | Methods of forming injectable formulations for providing regenerative effects to an organ such as a kidney |
| WO2013170146A1 (en) | 2012-05-10 | 2013-11-14 | Uab Research Foundation | Methods and compositions for modulating mir-204 activity |
| EP2861238A4 (en) | 2012-06-05 | 2016-03-16 | Capricor Inc | OPTIMIZED METHODS FOR GENERATING CARDIAC STEM CELLS FROM CARDIAC TISSUE AND THEIR USE IN CARDIAC THERAPY |
| US9828603B2 (en) | 2012-08-13 | 2017-11-28 | Cedars Sinai Medical Center | Exosomes and micro-ribonucleic acids for tissue regeneration |
| AU2013334276B2 (en) * | 2012-10-24 | 2019-07-04 | Prokidney | Renal cell populations and uses thereof |
| DK2994528T3 (da) * | 2013-05-08 | 2024-02-05 | Prokidney | Organoider, der omfatter isolerede renale celler og anvendelser deraf |
| WO2015080838A1 (en) * | 2013-11-27 | 2015-06-04 | Mayo Foundation For Medical Education And Research | Detecting podocyte injury in diabetic nephropathy and glomerulonephritis |
| CA2942384C (en) | 2014-03-12 | 2024-01-23 | Icahn School Of Medicine At Mount Sinai | Method for identifying kidney allograft recipients at risk for chronic injury |
| EP3161165B1 (en) | 2014-06-26 | 2020-11-18 | Icahn School of Medicine at Mount Sinai | Method for diagnosing subclinical and clinical acute rejection by analysis of predictive gene sets, therapeutic agent for use in the treatment and kits for determining the expression |
| CN106661634B (zh) | 2014-06-26 | 2021-03-12 | 西奈山伊坎医学院 | 用于诊断肾异体移植物纤维化和排异风险的方法 |
| CN104195107B (zh) * | 2014-06-30 | 2017-12-15 | 中国人民解放军军事医学科学院野战输血研究所 | 微囊泡在诱导干细胞巨核分化中的用途 |
| US11357799B2 (en) | 2014-10-03 | 2022-06-14 | Cedars-Sinai Medical Center | Cardiosphere-derived cells and exosomes secreted by such cells in the treatment of muscular dystrophy |
| US11180810B2 (en) | 2015-09-29 | 2021-11-23 | Research Institute At Nationwide Children's Hospital | Methods for detecting hepatic fibrosis and responsiveness to therapy |
| US11253551B2 (en) | 2016-01-11 | 2022-02-22 | Cedars-Sinai Medical Center | Cardiosphere-derived cells and exosomes secreted by such cells in the treatment of heart failure with preserved ejection fraction |
| BR112018072797A2 (pt) | 2016-05-06 | 2019-03-12 | Unicyte Ev Ag | veículos farmacêuticos contendo mirnas para uso no tratamento de doenças fibróticas causadas por hiperglicemia |
| US11351200B2 (en) | 2016-06-03 | 2022-06-07 | Cedars-Sinai Medical Center | CDC-derived exosomes for treatment of ventricular tachyarrythmias |
| US11123372B2 (en) | 2016-07-29 | 2021-09-21 | Prokidney | Bioactive renal cells for the treatment of chronic kidney disease |
| EP3515459A4 (en) | 2016-09-20 | 2020-08-05 | Cedars-Sinai Medical Center | CELLS DERIVED FROM CARDIOSPHERES AND THEIR EXTRACELLULAR VESICLES TO DELAY OR REVERSE AGING AND AGE-RELATED DISORDERS |
| KR101725025B1 (ko) * | 2016-11-25 | 2017-04-10 | 서울대학교산학협력단 | 무형성골질환 진단용 miRNA 마커 |
| US10767164B2 (en) | 2017-03-30 | 2020-09-08 | The Research Foundation For The State University Of New York | Microenvironments for self-assembly of islet organoids from stem cells differentiation |
| WO2018195210A1 (en) | 2017-04-19 | 2018-10-25 | Cedars-Sinai Medical Center | Methods and compositions for treating skeletal muscular dystrophy |
| CN107236799B (zh) * | 2017-06-15 | 2020-06-09 | 昆明医科大学第六附属医院 | 一种肾纤维化miRNA标记物 |
| US11890395B2 (en) | 2017-06-16 | 2024-02-06 | Avery Therapeutics, Inc. | Three dimensional tissue compositions and methods of use |
| AU2018290250A1 (en) * | 2017-06-21 | 2019-12-19 | Timothy A. Bertram | Immunoprivileged bioactive renal cells for the treatment of kidney disease |
| CN111601890B (zh) * | 2017-12-14 | 2024-04-19 | 联合细胞Ev股份公司 | 用于治疗肾癌的包含miRNA的药物载体 |
| US11660355B2 (en) | 2017-12-20 | 2023-05-30 | Cedars-Sinai Medical Center | Engineered extracellular vesicles for enhanced tissue delivery |
| CA3095198A1 (en) | 2018-04-16 | 2019-10-24 | Icahn School Of Medicine At Mount Sinai | Method and kits for prediction of acute rejection and renal allograft loss using pre-transplant transcriptomic signatures in recipient blood |
| TWI698640B (zh) * | 2018-04-23 | 2020-07-11 | 長庚醫療財團法人高雄長庚紀念醫院 | 用於檢測狼瘡性腎炎或評估狼瘡性腎炎風險的方法及其應用 |
| US20210386786A1 (en) * | 2018-08-31 | 2021-12-16 | Timothy A. Bertram | Compositions comprising cell-delivered vesicles and uses thereof |
| JP2022506212A (ja) * | 2018-10-29 | 2022-01-17 | エイブリィ セラピューティクス インコーポレイテッド | 改変組織の凍結保存及び再構成のための組成物及び方法 |
| CN111733234A (zh) * | 2019-03-25 | 2020-10-02 | 深圳大学 | 一种食管癌分子标志物及其应用 |
| TW202122572A (zh) * | 2019-08-09 | 2021-06-16 | 日商宇部興產股份有限公司 | 胞外體的產生方法 |
| EP4015635A4 (en) * | 2019-08-16 | 2023-10-25 | Public University Corporation Nagoya City University | MARKER FOR THE DIAGNOSIS OF COLORECTAL CARCINOMA, METHOD FOR AIDING THE DIAGNOSIS OF COLORECTAL CARCINOMA, METHOD FOR COLLECTING DATA FOR THE DIAGNOSIS OF COLORECTAL CARCINOMA, DIAGNOSTIC KIT FOR COLORECTAL CARCINOMA, THERAPEUTIC FOR COLORECTAL CARCINOMA, METHOD FOR TREATING COLORECTAL CARCINOMA AND METHOD FOR DIAGNOSING COLORECTAL CARCINOMA |
| CN110734972B (zh) * | 2019-11-28 | 2023-06-16 | 河北仁博科技有限公司 | miR-181c-3p作为肾纤维化标志物的应用 |
| CA3174551A1 (en) | 2020-03-04 | 2021-09-10 | Metcela Inc. | Fibroblast having enhanced erythropoietin production ability |
| CN112263587B (zh) * | 2020-10-23 | 2022-08-30 | 河北仁博科技有限公司 | miR-3065-5p在制备预防和/或治疗肾纤维化的药物中的应用 |
| CN112760376B (zh) * | 2020-12-30 | 2021-12-07 | 陈晓熠 | miRNA-3918在非小细胞肺癌检测中的应用 |
| WO2023107698A1 (en) * | 2021-12-09 | 2023-06-15 | Encellin, Inc. | Methods and devices for pre-vascularization of implantation sites and/or cell encapsulation devices |
| CN116240161B (zh) * | 2023-02-27 | 2024-05-10 | 四川大学华西医院 | 具有肾脏损伤修复作用的细胞外囊泡制备方法及应用 |
Family Cites Families (44)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5032508A (en) | 1988-09-08 | 1991-07-16 | Marrow-Tech, Inc. | Three-dimensional cell and tissue culture system |
| US5863531A (en) | 1986-04-18 | 1999-01-26 | Advanced Tissue Sciences, Inc. | In vitro preparation of tubular tissue structures by stromal cell culture on a three-dimensional framework |
| CN1053698C (zh) | 1989-01-19 | 2000-06-21 | 南京大学 | 细胞培养生产促红细胞生成素的方法 |
| WO1990012604A1 (en) | 1989-04-25 | 1990-11-01 | Vacanti Joseph P | Method for implanting large volumes of cells on polymeric matrices |
| WO1993007913A1 (en) | 1991-10-24 | 1993-04-29 | Children's Medical Center Corporation | Neomorphogenesis of urological structures in vivo from cell culture |
| US5549674A (en) | 1992-03-02 | 1996-08-27 | The Regents Of The University Of Michigan | Methods and compositions of a bioartificial kidney suitable for use in vivo or ex vivo |
| US6283761B1 (en) | 1992-09-08 | 2001-09-04 | Raymond Anthony Joao | Apparatus and method for processing and/or for providing healthcare information and/or healthcare-related information |
| US6224893B1 (en) | 1997-04-11 | 2001-05-01 | Massachusetts Institute Of Technology | Semi-interpenetrating or interpenetrating polymer networks for drug delivery and tissue engineering |
| JP2004155788A (ja) * | 1998-09-24 | 2004-06-03 | Tanabe Seiyaku Co Ltd | 腎疾患治療薬およびそのスクリーニング方法 |
| DK1180686T3 (da) | 1999-05-27 | 2010-01-04 | Cmic Co Ltd | Lægemidler til nyresygdomme og fremgangsmåde til screening deraf |
| KR100369788B1 (ko) | 1999-09-03 | 2003-01-29 | 동아제약 주식회사 | 재조합 인간 에리트로포이에틴의 제조방법 |
| WO2002061053A1 (en) | 2001-01-31 | 2002-08-08 | The General Hospital Corporation | Renal stem cells and uses thereof |
| AU2002340463A1 (en) | 2001-11-16 | 2003-06-10 | Children's Medical Center Corporation | Augmentation of organ function |
| MXPA04009234A (es) | 2002-03-22 | 2005-01-25 | Kissei Pharmaceutical | Agente preventivo o terapeutico para enfermedades renales. |
| JP2004309186A (ja) | 2003-04-02 | 2004-11-04 | Tokai Univ | 小胞体ストレスの改善剤およびその評価方法 |
| JP4750697B2 (ja) * | 2003-06-25 | 2011-08-17 | オタワ ヘルス リサーチ インスティテュート | 幹細胞の成長及び分化を調節する方法及び組成物 |
| WO2005059133A2 (en) * | 2003-12-19 | 2005-06-30 | Antisense Pharma Gmbh | Combination therapy associating a tgf-breta antagonist with a chemiotherapeutic agent |
| US20070128174A1 (en) | 2005-09-21 | 2007-06-07 | Kleinsek Donald A | Methods and compositions for organ and tissue functionality |
| US20070104799A1 (en) * | 2005-11-09 | 2007-05-10 | Shire International Licensing B.V. | Treatment of chronic kidney disease (CKD) subjects using lanthanum compounds |
| CA3019254C (en) | 2006-02-07 | 2021-01-19 | Spinalcyte, Llc | Use of chondrocyte-like cells to treat a joint in need of repair |
| CA2641733C (en) | 2006-02-10 | 2015-03-31 | Tengion, Inc. | Scaffolds for organ reconstruction and augmentation |
| CA2676546A1 (en) | 2006-04-24 | 2007-11-08 | Stemcell Institute Inc. | Method for preparing an organ for transplantation |
| WO2007127848A1 (en) * | 2006-04-26 | 2007-11-08 | University Of Louisville Research Foundation, Inc | Isolation of membrane vesicles from biological fluids and methods of using same |
| US10500230B2 (en) | 2006-10-12 | 2019-12-10 | DePuy Synthes Products, Inc. | Kidney-derived cells and methods of use in tissue repair and regeneration |
| ES2374954T3 (es) | 2006-11-16 | 2012-02-23 | Genentech, Inc. | Variaciones genéticas asociadas con tumores. |
| US20110008347A1 (en) | 2006-12-01 | 2011-01-13 | Agency For Science ,Technology And Research | Cancer-related protein kinases |
| US7924332B2 (en) * | 2007-05-25 | 2011-04-12 | The Trustees Of The University Of Pennsylvania | Current/voltage mode image sensor with switchless active pixels |
| US10590391B2 (en) | 2007-06-08 | 2020-03-17 | Wake Forest University Health Sciences | Selective cell therapy for the treatment of renal failure |
| EP2162529B1 (en) | 2007-06-08 | 2019-03-27 | Wake Forest University Health Sciences | Selective cell therapy for the treatment of renal failure |
| DE102007033074A1 (de) * | 2007-07-13 | 2009-01-15 | Pac Tech-Packaging Technologies Gmbh | Verfahren und Vorrichtung zur Entfernung von Lotmaterialdepots von einem Substrat |
| EP2211875B1 (en) | 2007-10-29 | 2013-05-29 | Fresenius Medical Care Deutschland GmbH | Use of microvesicles (mvs) derived from stem cells for preparing a medicament for endo/epithelial regeneration of damaged or injured tissues or organs, and related in vitro and in vivo methods |
| EP2067785A1 (en) | 2007-12-03 | 2009-06-10 | Fresenius Medical Care Deutschland GmbH | Human CD154-binding synthetic peptide and uses thereof |
| SG10201609507TA (en) | 2008-02-01 | 2017-01-27 | Gen Hospital Corp | Use of microvesicles in diagnosis, prognosis and treatment of medical diseases and conditions |
| EP2186883A1 (en) | 2008-11-04 | 2010-05-19 | Fresenius Medical Care | An isolated multipotent mesenchymal stem cell from human adult glomeruli (hGL-MSC), a method of preparing thereof and uses thereof in the regenerative medicine of the kidney |
| US8318484B2 (en) | 2008-11-12 | 2012-11-27 | Tengion, Inc. | Isolated renal cells and uses thereof |
| WO2010057015A1 (en) | 2008-11-14 | 2010-05-20 | Wake Forest University Health Sciences | Kidney structures and methods of forming the same |
| JP5565607B2 (ja) | 2009-07-15 | 2014-08-06 | 国立大学法人 東京大学 | 敗血症又は多臓器不全の予後診断方法及び予後診断用キット |
| EP2333047A1 (en) | 2009-12-09 | 2011-06-15 | Fresenius Medical Care Deutschland GmbH | Adult stem cell derived conditioned medium and/or adult stem cells for use in the therapeutic treatment of a tumor disease |
| EP2363136A1 (en) | 2010-03-02 | 2011-09-07 | Fresenius Medical Care Deutschland GmbH | Microvesicles (MVs) derived from adult stem cells for use in the therapeutic treatment of a tumor disease |
| TWI690320B (zh) | 2010-05-12 | 2020-04-11 | 開曼群島商英瑞金公司 | 生物活性腎細胞 |
| CA3065694A1 (en) | 2010-11-10 | 2012-05-18 | Inregen | Methods of forming injectable formulations for providing regenerative effects to an organ such as a kidney |
| CN111635883A (zh) | 2013-12-16 | 2020-09-08 | 弗雷森纽斯医疗护理德国有限责任公司 | 胰岛样细胞结构及其制备方法 |
| WO2017178472A1 (en) | 2016-04-12 | 2017-10-19 | Unicyte Ev Ag | Isolation of extracellular vesicles (evs) from biological fluid samples |
| BR112018072797A2 (pt) | 2016-05-06 | 2019-03-12 | Unicyte Ev Ag | veículos farmacêuticos contendo mirnas para uso no tratamento de doenças fibróticas causadas por hiperglicemia |
-
2011
- 2011-05-12 TW TW107113584A patent/TWI690320B/zh active
- 2011-05-12 TW TW105117487A patent/TWI629990B/zh active
- 2011-05-12 KR KR1020217005873A patent/KR102428776B1/ko active IP Right Grant
- 2011-05-12 TW TW111113839A patent/TWI840787B/zh active
- 2011-05-12 WO PCT/US2011/036347 patent/WO2011143499A1/en active Application Filing
- 2011-05-12 KR KR1020197012275A patent/KR102223564B1/ko active IP Right Grant
- 2011-05-12 DK DK16196757.5T patent/DK3190177T3/da active
- 2011-05-12 KR KR1020187005695A patent/KR101975212B1/ko active IP Right Grant
- 2011-05-12 US US13/697,206 patent/US10077442B2/en active Active
- 2011-05-12 HU HUE16196757A patent/HUE058155T2/hu unknown
- 2011-05-12 KR KR1020127032373A patent/KR101834830B1/ko active IP Right Grant
- 2011-05-12 PL PL16196757T patent/PL3190177T3/pl unknown
- 2011-05-12 CN CN201610617264.0A patent/CN106214700B/zh active Active
- 2011-05-12 EP EP16196757.5A patent/EP3190177B8/en active Active
- 2011-05-12 NZ NZ603482A patent/NZ603482A/en unknown
- 2011-05-12 TW TW109109511A patent/TWI762890B/zh active
- 2011-05-12 AU AU2011252915A patent/AU2011252915B2/en active Active
- 2011-05-12 EP EP11722663.9A patent/EP2569418B1/en active Active
- 2011-05-12 KR KR1020227026491A patent/KR102539589B1/ko active IP Right Grant
- 2011-05-12 JP JP2013510320A patent/JP5974215B2/ja active Active
- 2011-05-12 CA CA2798900A patent/CA2798900C/en active Active
- 2011-05-12 ES ES16196757T patent/ES2906750T3/es active Active
- 2011-05-12 EP EP21212108.1A patent/EP4083191A1/en active Pending
- 2011-05-12 TW TW100116779A patent/TWI542690B/zh active
- 2011-05-12 CN CN201180034688.XA patent/CN103154236B/zh active Active
-
2016
- 2016-04-13 JP JP2016080335A patent/JP6385976B2/ja active Active
- 2016-08-18 AU AU2016216646A patent/AU2016216646B2/en active Active
-
2017
- 2017-06-13 HK HK17105838.6A patent/HK1232143A1/zh unknown
-
2018
- 2018-02-01 AU AU2018200761A patent/AU2018200761A1/en not_active Abandoned
- 2018-03-19 US US15/925,172 patent/US11066666B2/en active Active
- 2018-08-08 JP JP2018149199A patent/JP6744368B2/ja active Active
-
2020
- 2020-06-05 AU AU2020203727A patent/AU2020203727B2/en active Active
- 2020-07-30 JP JP2020128752A patent/JP7123426B2/ja active Active
-
2021
- 2021-02-09 US US17/171,943 patent/US20210238600A1/en active Pending
-
2022
- 2022-08-03 JP JP2022124203A patent/JP7442852B2/ja active Active
-
2023
- 2023-05-02 AU AU2023202707A patent/AU2023202707A1/en active Pending
-
2024
- 2024-02-14 JP JP2024019878A patent/JP2024056860A/ja active Pending
Non-Patent Citations (1)
| Title |
|---|
| Maria Luisa S et al.,「The MicroRNA-Processing Enzyme Dicer Maintains Juxtaglomerular Cells」 J Am Soc Nephrol 21:460-467,March 2010.(如附件) * |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7442852B2 (ja) | 生理活性腎臓細胞 |