JP7197584B2 - Methods for storing and retrieving digital pathology analysis results - Google Patents
Methods for storing and retrieving digital pathology analysis results Download PDFInfo
- Publication number
- JP7197584B2 JP7197584B2 JP2020530584A JP2020530584A JP7197584B2 JP 7197584 B2 JP7197584 B2 JP 7197584B2 JP 2020530584 A JP2020530584 A JP 2020530584A JP 2020530584 A JP2020530584 A JP 2020530584A JP 7197584 B2 JP7197584 B2 JP 7197584B2
- Authority
- JP
- Japan
- Prior art keywords
- image
- regions
- pixels
- sub
- derived
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 238000000034 method Methods 0.000 title claims description 121
- 238000004458 analytical method Methods 0.000 title description 73
- 230000007170 pathology Effects 0.000 title description 20
- 210000004027 cell Anatomy 0.000 claims description 107
- 238000010186 staining Methods 0.000 claims description 90
- 210000002950 fibroblast Anatomy 0.000 claims description 54
- 238000004422 calculation algorithm Methods 0.000 claims description 47
- 239000012472 biological sample Substances 0.000 claims description 32
- 238000013459 approach Methods 0.000 claims description 23
- 230000014509 gene expression Effects 0.000 claims description 23
- 238000010191 image analysis Methods 0.000 claims description 22
- 108090000623 proteins and genes Proteins 0.000 claims description 17
- 238000003064 k means clustering Methods 0.000 claims description 15
- 210000002540 macrophage Anatomy 0.000 claims description 13
- 102000004169 proteins and genes Human genes 0.000 claims description 12
- 238000005070 sampling Methods 0.000 claims description 6
- 230000004913 activation Effects 0.000 claims description 5
- 210000004940 nucleus Anatomy 0.000 description 99
- 210000001519 tissue Anatomy 0.000 description 82
- 206010028980 Neoplasm Diseases 0.000 description 44
- 239000000090 biomarker Substances 0.000 description 36
- 239000000523 sample Substances 0.000 description 31
- 239000012528 membrane Substances 0.000 description 29
- 210000004379 membrane Anatomy 0.000 description 29
- 238000003384 imaging method Methods 0.000 description 24
- 230000011218 segmentation Effects 0.000 description 23
- 238000000926 separation method Methods 0.000 description 23
- 230000000875 corresponding effect Effects 0.000 description 21
- 238000003860 storage Methods 0.000 description 19
- 230000003595 spectral effect Effects 0.000 description 18
- 238000012549 training Methods 0.000 description 18
- 210000004881 tumor cell Anatomy 0.000 description 18
- 230000002055 immunohistochemical effect Effects 0.000 description 15
- 238000012545 processing Methods 0.000 description 15
- 230000000877 morphologic effect Effects 0.000 description 14
- 230000003287 optical effect Effects 0.000 description 14
- 201000011510 cancer Diseases 0.000 description 13
- 238000001514 detection method Methods 0.000 description 13
- WZUVPPKBWHMQCE-UHFFFAOYSA-N Haematoxylin Chemical compound C12=CC(O)=C(O)C=C2CC2(O)C1C1=CC=C(O)C(O)=C1OC2 WZUVPPKBWHMQCE-UHFFFAOYSA-N 0.000 description 12
- 238000004590 computer program Methods 0.000 description 12
- 230000001788 irregular Effects 0.000 description 12
- 230000008569 process Effects 0.000 description 12
- 239000013598 vector Substances 0.000 description 12
- 238000003556 assay Methods 0.000 description 11
- 238000000701 chemical imaging Methods 0.000 description 11
- 238000002372 labelling Methods 0.000 description 11
- 238000001228 spectrum Methods 0.000 description 11
- 230000001086 cytosolic effect Effects 0.000 description 9
- 238000003709 image segmentation Methods 0.000 description 9
- 239000000427 antigen Substances 0.000 description 8
- 239000000834 fixative Substances 0.000 description 8
- 239000012491 analyte Substances 0.000 description 7
- 210000000170 cell membrane Anatomy 0.000 description 7
- 210000003855 cell nucleus Anatomy 0.000 description 7
- 230000006870 function Effects 0.000 description 7
- 230000004044 response Effects 0.000 description 7
- 230000003044 adaptive effect Effects 0.000 description 6
- 102000036639 antigens Human genes 0.000 description 6
- 108091007433 antigens Proteins 0.000 description 6
- 210000000805 cytoplasm Anatomy 0.000 description 6
- 210000004698 lymphocyte Anatomy 0.000 description 6
- 239000000126 substance Substances 0.000 description 6
- 238000012360 testing method Methods 0.000 description 6
- 101100117236 Drosophila melanogaster speck gene Proteins 0.000 description 5
- 238000004891 communication Methods 0.000 description 5
- 201000010099 disease Diseases 0.000 description 5
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 5
- 238000009826 distribution Methods 0.000 description 5
- 210000002865 immune cell Anatomy 0.000 description 5
- 238000007901 in situ hybridization Methods 0.000 description 5
- 239000011159 matrix material Substances 0.000 description 5
- 210000004882 non-tumor cell Anatomy 0.000 description 5
- 108020004707 nucleic acids Proteins 0.000 description 5
- 102000039446 nucleic acids Human genes 0.000 description 5
- 150000007523 nucleic acids Chemical class 0.000 description 5
- 238000007637 random forest analysis Methods 0.000 description 5
- 241001465754 Metazoa Species 0.000 description 4
- 230000009471 action Effects 0.000 description 4
- 238000003491 array Methods 0.000 description 4
- 238000012303 cytoplasmic staining Methods 0.000 description 4
- 238000001914 filtration Methods 0.000 description 4
- 230000003993 interaction Effects 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- -1 methacan Substances 0.000 description 4
- 238000000386 microscopy Methods 0.000 description 4
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- 206010006187 Breast cancer Diseases 0.000 description 3
- 208000026310 Breast neoplasm Diseases 0.000 description 3
- 201000009030 Carcinoma Diseases 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 206010016654 Fibrosis Diseases 0.000 description 3
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 3
- SXEHKFHPFVVDIR-UHFFFAOYSA-N [4-(4-hydrazinylphenyl)phenyl]hydrazine Chemical compound C1=CC(NN)=CC=C1C1=CC=C(NN)C=C1 SXEHKFHPFVVDIR-UHFFFAOYSA-N 0.000 description 3
- 230000003321 amplification Effects 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- 239000003153 chemical reaction reagent Substances 0.000 description 3
- 238000002059 diagnostic imaging Methods 0.000 description 3
- 238000011143 downstream manufacturing Methods 0.000 description 3
- 230000007717 exclusion Effects 0.000 description 3
- 230000004761 fibrosis Effects 0.000 description 3
- 239000012530 fluid Substances 0.000 description 3
- 239000007850 fluorescent dye Substances 0.000 description 3
- 239000011521 glass Substances 0.000 description 3
- 230000000873 masking effect Effects 0.000 description 3
- 238000005259 measurement Methods 0.000 description 3
- 239000000203 mixture Substances 0.000 description 3
- 238000003199 nucleic acid amplification method Methods 0.000 description 3
- 238000002360 preparation method Methods 0.000 description 3
- CSCPPACGZOOCGX-UHFFFAOYSA-N Acetone Chemical compound CC(C)=O CSCPPACGZOOCGX-UHFFFAOYSA-N 0.000 description 2
- 206010009944 Colon cancer Diseases 0.000 description 2
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 2
- 102000004190 Enzymes Human genes 0.000 description 2
- 108090000790 Enzymes Proteins 0.000 description 2
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 2
- 206010033128 Ovarian cancer Diseases 0.000 description 2
- 206010061535 Ovarian neoplasm Diseases 0.000 description 2
- 102000007066 Prostate-Specific Antigen Human genes 0.000 description 2
- 108010072866 Prostate-Specific Antigen Proteins 0.000 description 2
- 230000006399 behavior Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 230000027455 binding Effects 0.000 description 2
- 210000000481 breast Anatomy 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 238000004364 calculation method Methods 0.000 description 2
- 230000001413 cellular effect Effects 0.000 description 2
- 238000006243 chemical reaction Methods 0.000 description 2
- 239000002131 composite material Substances 0.000 description 2
- 238000004195 computer-aided diagnosis Methods 0.000 description 2
- 230000003750 conditioning effect Effects 0.000 description 2
- 230000001276 controlling effect Effects 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 230000002380 cytological effect Effects 0.000 description 2
- 238000009795 derivation Methods 0.000 description 2
- 238000010586 diagram Methods 0.000 description 2
- YQGOJNYOYNNSMM-UHFFFAOYSA-N eosin Chemical compound [Na+].OC(=O)C1=CC=CC=C1C1=C2C=C(Br)C(=O)C(Br)=C2OC2=C(Br)C(O)=C(Br)C=C21 YQGOJNYOYNNSMM-UHFFFAOYSA-N 0.000 description 2
- 210000002919 epithelial cell Anatomy 0.000 description 2
- 239000007789 gas Substances 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 201000010536 head and neck cancer Diseases 0.000 description 2
- 208000014829 head and neck neoplasm Diseases 0.000 description 2
- 238000007490 hematoxylin and eosin (H&E) staining Methods 0.000 description 2
- 230000001744 histochemical effect Effects 0.000 description 2
- 230000028993 immune response Effects 0.000 description 2
- 238000011532 immunohistochemical staining Methods 0.000 description 2
- 238000003364 immunohistochemistry Methods 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 201000007270 liver cancer Diseases 0.000 description 2
- 208000014018 liver neoplasm Diseases 0.000 description 2
- 230000004807 localization Effects 0.000 description 2
- 210000004072 lung Anatomy 0.000 description 2
- 210000005265 lung cell Anatomy 0.000 description 2
- 230000003211 malignant effect Effects 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 201000001441 melanoma Diseases 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000013188 needle biopsy Methods 0.000 description 2
- 239000013642 negative control Substances 0.000 description 2
- GYHFUZHODSMOHU-UHFFFAOYSA-N nonanal Chemical compound CCCCCCCCC=O GYHFUZHODSMOHU-UHFFFAOYSA-N 0.000 description 2
- 210000000633 nuclear envelope Anatomy 0.000 description 2
- 239000002853 nucleic acid probe Substances 0.000 description 2
- 238000012634 optical imaging Methods 0.000 description 2
- 238000005192 partition Methods 0.000 description 2
- 230000001575 pathological effect Effects 0.000 description 2
- 238000012805 post-processing Methods 0.000 description 2
- 108090000765 processed proteins & peptides Proteins 0.000 description 2
- 238000003672 processing method Methods 0.000 description 2
- 238000004393 prognosis Methods 0.000 description 2
- 230000000644 propagated effect Effects 0.000 description 2
- 238000013442 quality metrics Methods 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 238000012552 review Methods 0.000 description 2
- 238000013515 script Methods 0.000 description 2
- 239000004065 semiconductor Substances 0.000 description 2
- 210000002966 serum Anatomy 0.000 description 2
- 210000002536 stromal cell Anatomy 0.000 description 2
- 239000000758 substrate Substances 0.000 description 2
- 238000012706 support-vector machine Methods 0.000 description 2
- 230000007704 transition Effects 0.000 description 2
- 239000010981 turquoise Substances 0.000 description 2
- 230000000007 visual effect Effects 0.000 description 2
- 238000005406 washing Methods 0.000 description 2
- HSTOKWSFWGCZMH-UHFFFAOYSA-N 3,3'-diaminobenzidine Chemical compound C1=C(N)C(N)=CC=C1C1=CC=C(N)C(N)=C1 HSTOKWSFWGCZMH-UHFFFAOYSA-N 0.000 description 1
- WEVYNIUIFUYDGI-UHFFFAOYSA-N 3-[6-[4-(trifluoromethoxy)anilino]-4-pyrimidinyl]benzamide Chemical compound NC(=O)C1=CC=CC(C=2N=CN=C(NC=3C=CC(OC(F)(F)F)=CC=3)C=2)=C1 WEVYNIUIFUYDGI-UHFFFAOYSA-N 0.000 description 1
- 241000243818 Annelida Species 0.000 description 1
- 241000239290 Araneae Species 0.000 description 1
- 208000032791 BCR-ABL1 positive chronic myelogenous leukemia Diseases 0.000 description 1
- 108700020463 BRCA1 Proteins 0.000 description 1
- 102000036365 BRCA1 Human genes 0.000 description 1
- 101150072950 BRCA1 gene Proteins 0.000 description 1
- 102000052609 BRCA2 Human genes 0.000 description 1
- 108700020462 BRCA2 Proteins 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 239000011547 Bouin solution Substances 0.000 description 1
- 101150008921 Brca2 gene Proteins 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 208000005623 Carcinogenesis Diseases 0.000 description 1
- 206010050337 Cerumen impaction Diseases 0.000 description 1
- 208000010833 Chronic myeloid leukaemia Diseases 0.000 description 1
- 102000008186 Collagen Human genes 0.000 description 1
- 108010035532 Collagen Proteins 0.000 description 1
- 241000938605 Crocodylia Species 0.000 description 1
- 239000004971 Cross linker Substances 0.000 description 1
- 108020003215 DNA Probes Proteins 0.000 description 1
- 239000003298 DNA probe Substances 0.000 description 1
- 102000018651 Epithelial Cell Adhesion Molecule Human genes 0.000 description 1
- 108010066687 Epithelial Cell Adhesion Molecule Proteins 0.000 description 1
- 241000283086 Equidae Species 0.000 description 1
- 102000010834 Extracellular Matrix Proteins Human genes 0.000 description 1
- 108010037362 Extracellular Matrix Proteins Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 206010051066 Gastrointestinal stromal tumour Diseases 0.000 description 1
- 206010018338 Glioma Diseases 0.000 description 1
- SXRSQZLOMIGNAQ-UHFFFAOYSA-N Glutaraldehyde Chemical compound O=CCCCC=O SXRSQZLOMIGNAQ-UHFFFAOYSA-N 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 1
- 206010062016 Immunosuppression Diseases 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 241000289419 Metatheria Species 0.000 description 1
- 241000699666 Mus <mouse, genus> Species 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 208000033761 Myelogenous Chronic BCR-ABL Positive Leukemia Diseases 0.000 description 1
- 108091034117 Oligonucleotide Proteins 0.000 description 1
- 108700020796 Oncogene Proteins 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 1
- 229930040373 Paraformaldehyde Natural products 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 108020004518 RNA Probes Proteins 0.000 description 1
- 239000003391 RNA probe Substances 0.000 description 1
- 241000700159 Rattus Species 0.000 description 1
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 210000001744 T-lymphocyte Anatomy 0.000 description 1
- 230000005856 abnormality Effects 0.000 description 1
- 230000006978 adaptation Effects 0.000 description 1
- 150000001299 aldehydes Chemical class 0.000 description 1
- WLDHEUZGFKACJH-UHFFFAOYSA-K amaranth Chemical compound [Na+].[Na+].[Na+].C12=CC=C(S([O-])(=O)=O)C=C2C=C(S([O-])(=O)=O)C(O)=C1N=NC1=CC=C(S([O-])(=O)=O)C2=CC=CC=C12 WLDHEUZGFKACJH-UHFFFAOYSA-K 0.000 description 1
- 210000003484 anatomy Anatomy 0.000 description 1
- 230000033115 angiogenesis Effects 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 230000000692 anti-sense effect Effects 0.000 description 1
- 210000003719 b-lymphocyte Anatomy 0.000 description 1
- 230000002902 bimodal effect Effects 0.000 description 1
- 239000011230 binding agent Substances 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 239000013060 biological fluid Substances 0.000 description 1
- 238000001574 biopsy Methods 0.000 description 1
- 210000000601 blood cell Anatomy 0.000 description 1
- 210000004204 blood vessel Anatomy 0.000 description 1
- 230000009702 cancer cell proliferation Effects 0.000 description 1
- 230000036952 cancer formation Effects 0.000 description 1
- 230000009400 cancer invasion Effects 0.000 description 1
- 235000014633 carbohydrates Nutrition 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 231100000504 carcinogenesis Toxicity 0.000 description 1
- 210000003850 cellular structure Anatomy 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 210000001175 cerebrospinal fluid Anatomy 0.000 description 1
- 210000002939 cerumen Anatomy 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000005081 chemiluminescent agent Substances 0.000 description 1
- KRVSOGSZCMJSLX-UHFFFAOYSA-L chromic acid Substances O[Cr](O)(=O)=O KRVSOGSZCMJSLX-UHFFFAOYSA-L 0.000 description 1
- 230000008045 co-localization Effects 0.000 description 1
- 229920001436 collagen Polymers 0.000 description 1
- 208000030499 combat disease Diseases 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 210000002808 connective tissue Anatomy 0.000 description 1
- 239000000470 constituent Substances 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 239000003431 cross linking reagent Substances 0.000 description 1
- 239000003398 denaturant Substances 0.000 description 1
- 238000004925 denaturation Methods 0.000 description 1
- 230000036425 denaturation Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- 238000003708 edge detection Methods 0.000 description 1
- 230000005670 electromagnetic radiation Effects 0.000 description 1
- 238000005538 encapsulation Methods 0.000 description 1
- 102000052116 epidermal growth factor receptor activity proteins Human genes 0.000 description 1
- 108700015053 epidermal growth factor receptor activity proteins Proteins 0.000 description 1
- 230000001973 epigenetic effect Effects 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 210000003722 extracellular fluid Anatomy 0.000 description 1
- 210000002744 extracellular matrix Anatomy 0.000 description 1
- 230000002550 fecal effect Effects 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 238000011049 filling Methods 0.000 description 1
- 239000010408 film Substances 0.000 description 1
- 238000000799 fluorescence microscopy Methods 0.000 description 1
- 239000012634 fragment Substances 0.000 description 1
- AWJWCTOOIBYHON-UHFFFAOYSA-N furo[3,4-b]pyrazine-5,7-dione Chemical compound C1=CN=C2C(=O)OC(=O)C2=N1 AWJWCTOOIBYHON-UHFFFAOYSA-N 0.000 description 1
- 201000011243 gastrointestinal stromal tumor Diseases 0.000 description 1
- 210000004907 gland Anatomy 0.000 description 1
- 230000005484 gravity Effects 0.000 description 1
- 210000004251 human milk Anatomy 0.000 description 1
- 235000020256 human milk Nutrition 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 238000003711 image thresholding Methods 0.000 description 1
- 230000001506 immunosuppresive effect Effects 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 230000009545 invasion Effects 0.000 description 1
- 238000012804 iterative process Methods 0.000 description 1
- 239000004816 latex Substances 0.000 description 1
- 229920000126 latex Polymers 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 239000004973 liquid crystal related substance Substances 0.000 description 1
- 230000007762 localization of cell Effects 0.000 description 1
- 238000010801 machine learning Methods 0.000 description 1
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 229960002523 mercuric chloride Drugs 0.000 description 1
- LWJROJCJINYWOX-UHFFFAOYSA-L mercury dichloride Chemical compound Cl[Hg]Cl LWJROJCJINYWOX-UHFFFAOYSA-L 0.000 description 1
- 229910021645 metal ion Inorganic materials 0.000 description 1
- 229910044991 metal oxide Inorganic materials 0.000 description 1
- 150000004706 metal oxides Chemical class 0.000 description 1
- WSFSSNUMVMOOMR-NJFSPNSNSA-N methanone Chemical compound O=[14CH2] WSFSSNUMVMOOMR-NJFSPNSNSA-N 0.000 description 1
- 238000001531 micro-dissection Methods 0.000 description 1
- 238000004452 microanalysis Methods 0.000 description 1
- 238000001000 micrograph Methods 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 238000012544 monitoring process Methods 0.000 description 1
- 210000003097 mucus Anatomy 0.000 description 1
- 238000007837 multiplex assay Methods 0.000 description 1
- YOHYSYJDKVYCJI-UHFFFAOYSA-N n-[3-[[6-[3-(trifluoromethyl)anilino]pyrimidin-4-yl]amino]phenyl]cyclopropanecarboxamide Chemical compound FC(F)(F)C1=CC=CC(NC=2N=CN=C(NC=3C=C(NC(=O)C4CC4)C=CC=3)C=2)=C1 YOHYSYJDKVYCJI-UHFFFAOYSA-N 0.000 description 1
- 239000002547 new drug Substances 0.000 description 1
- 210000002445 nipple Anatomy 0.000 description 1
- 239000012454 non-polar solvent Substances 0.000 description 1
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 1
- 238000005457 optimization Methods 0.000 description 1
- 210000003463 organelle Anatomy 0.000 description 1
- 239000012285 osmium tetroxide Substances 0.000 description 1
- 229910000489 osmium tetroxide Inorganic materials 0.000 description 1
- 230000002611 ovarian Effects 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 239000007800 oxidant agent Substances 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 208000008443 pancreatic carcinoma Diseases 0.000 description 1
- 238000009595 pap smear Methods 0.000 description 1
- 239000012188 paraffin wax Substances 0.000 description 1
- 229920002866 paraformaldehyde Polymers 0.000 description 1
- 230000002688 persistence Effects 0.000 description 1
- 230000035790 physiological processes and functions Effects 0.000 description 1
- OXNIZHLAWKMVMX-UHFFFAOYSA-N picric acid Chemical compound OC1=C([N+]([O-])=O)C=C([N+]([O-])=O)C=C1[N+]([O-])=O OXNIZHLAWKMVMX-UHFFFAOYSA-N 0.000 description 1
- 239000002798 polar solvent Substances 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 238000007781 pre-processing Methods 0.000 description 1
- 102000004196 processed proteins & peptides Human genes 0.000 description 1
- 230000035755 proliferation Effects 0.000 description 1
- 210000004915 pus Anatomy 0.000 description 1
- 239000002096 quantum dot Substances 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 210000003935 rough endoplasmic reticulum Anatomy 0.000 description 1
- 102200055464 rs113488022 Human genes 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 238000013077 scoring method Methods 0.000 description 1
- 210000000582 semen Anatomy 0.000 description 1
- 230000001953 sensory effect Effects 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 210000004243 sweat Anatomy 0.000 description 1
- 210000001138 tear Anatomy 0.000 description 1
- 239000010409 thin film Substances 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000004614 tumor growth Effects 0.000 description 1
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 238000012800 visualization Methods 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 230000029663 wound healing Effects 0.000 description 1
Images
Classifications
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06V—IMAGE OR VIDEO RECOGNITION OR UNDERSTANDING
- G06V20/00—Scenes; Scene-specific elements
- G06V20/60—Type of objects
- G06V20/69—Microscopic objects, e.g. biological cells or cellular parts
- G06V20/695—Preprocessing, e.g. image segmentation
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F18/00—Pattern recognition
- G06F18/20—Analysing
- G06F18/21—Design or setup of recognition systems or techniques; Extraction of features in feature space; Blind source separation
- G06F18/214—Generating training patterns; Bootstrap methods, e.g. bagging or boosting
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F18/00—Pattern recognition
- G06F18/20—Analysing
- G06F18/24—Classification techniques
- G06F18/241—Classification techniques relating to the classification model, e.g. parametric or non-parametric approaches
- G06F18/2411—Classification techniques relating to the classification model, e.g. parametric or non-parametric approaches based on the proximity to a decision surface, e.g. support vector machines
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06V—IMAGE OR VIDEO RECOGNITION OR UNDERSTANDING
- G06V10/00—Arrangements for image or video recognition or understanding
- G06V10/40—Extraction of image or video features
- G06V10/50—Extraction of image or video features by performing operations within image blocks; by using histograms, e.g. histogram of oriented gradients [HoG]; by summing image-intensity values; Projection analysis
Landscapes
- Engineering & Computer Science (AREA)
- Theoretical Computer Science (AREA)
- General Physics & Mathematics (AREA)
- Physics & Mathematics (AREA)
- Life Sciences & Earth Sciences (AREA)
- Data Mining & Analysis (AREA)
- Multimedia (AREA)
- Molecular Biology (AREA)
- Computer Vision & Pattern Recognition (AREA)
- General Health & Medical Sciences (AREA)
- Biomedical Technology (AREA)
- Artificial Intelligence (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Bioinformatics & Computational Biology (AREA)
- Health & Medical Sciences (AREA)
- Evolutionary Biology (AREA)
- Evolutionary Computation (AREA)
- General Engineering & Computer Science (AREA)
- Investigating Or Analysing Biological Materials (AREA)
- Image Analysis (AREA)
- Investigating, Analyzing Materials By Fluorescence Or Luminescence (AREA)
Description
[0001]本願は、2017年12月6日に出願された米国仮特許出願第62/595,143号の出願日の利益を主張するものであり、そのすべての開示内容が参照により本明細書に組み込まれる。 [0001] This application claims the benefit of the filing date of U.S. Provisional Patent Application No. 62/595,143, filed December 6, 2017, the entire disclosure of which is incorporated herein by reference. incorporated into.
本願発明の一実施例は、例えば、デジタル病理学分析結果の格納および読み出し方法に関する。 One embodiment of the present invention, for example, relates to a method for storing and retrieving digital pathology analysis results.
[0002]デジタル病理学は、コンピュータ画面上で解釈可能なデジタル画像へと病理組織または病理細胞のガラススライド全体をスキャンすることを伴う。これらの画像は後で、画像処理アルゴリズムによる処理または病理学者による解釈がなされることになる。組織切片(実質的に透明)を検査するため、細胞成分に選択的に結び付く色付き組織化学的ステインを用いて組織切片が作成される。臨床医またはコンピュータ支援診断(CAD)アルゴリズムによる着色または染色細胞構造の使用によって、疾患の形態学的マーカを識別するとともに、これに応じた治療を進める。アッセイを観察することによって、疾患の診断、処置への反応の評価、および疾患と闘う新たな医薬品の開発といった多様なプロセスが可能になる。 [0002] Digital pathology involves scanning an entire glass slide of pathological tissue or cells into a digital image interpretable on a computer screen. These images will later be processed by image processing algorithms or interpreted by a pathologist. To examine tissue sections (substantially transparent), tissue sections are prepared using colored histochemical stains that selectively bind to cellular components. The use of staining or stained cell structures by clinicians or computer-aided diagnosis (CAD) algorithms identifies morphological markers of disease and guides treatment accordingly. Monitoring assays enables processes as diverse as diagnosing disease, evaluating response to treatment, and developing new drugs to combat disease.
[0003]免疫組織化学的(IHC)スライド染色は、組織切片の細胞中のタンパク質を識別するのに利用可能であるため、生物組織中のがん細胞および免疫細胞等、さまざまな種類の細胞の研究に広く用いられている。このため、免疫反応研究の場合に、がん細胞中の免疫細胞(T細胞またはB細胞等)の異なる発現のバイオマーカの分布および局在を理解する研究においては、IHC染色が使用され得る。たとえば、腫瘍は、免疫細胞の浸潤物を含むことが多く、これが腫瘍の成長を防止する場合もあれば、腫瘍の増殖を優先させる場合もある。 [0003] Immunohistochemical (IHC) slide staining can be used to identify proteins in cells of tissue sections, thus reducing the presence of various cell types, such as cancer cells and immune cells in biological tissues. Widely used in research. Thus, in the case of immune response studies, IHC staining can be used in studies to understand the distribution and localization of differentially expressed biomarkers of immune cells (such as T cells or B cells) in cancer cells. For example, tumors often contain an infiltrate of immune cells, which may prevent or favor tumor growth.
[0004]特に顕微鏡下で観察した場合に形態学的に悪性と見られる細胞中の遺伝学的異常または発がん遺伝子の増幅等の状態の有無の確認には、in-situハイブリダイゼーション(ISH)を使用可能である。ISHでは、標的遺伝子配列または転写物に対してアンチセンスの標識DNAまたはRNAプローブ分子を採用することにより、細胞または組織サンプル内の目標とする核酸標的遺伝子を検出または位置特定する。ISHは、ガラススライド上に固定された細胞または組織サンプル中の所与の標的遺伝子へと特にハイブリダイズ可能な標識核酸プローブに対して、当該細胞または組織サンプルを曝露することにより実行される。複数の異なる核酸タグで標識済みの複数の核酸プローブに対して細胞または組織サンプルを曝露することにより、複数の標的遺伝子を同時に分析可能である。発光波長が異なる標識を利用することにより、単一の標的細胞または組織サンプルに対して、1回のステップで同時多色分析が実行され得る。 [0004] In-situ hybridization (ISH) is used to confirm the presence or absence of conditions such as genetic abnormalities or amplification of oncogenes in cells that appear morphologically malignant, particularly when observed under a microscope. Available. ISH detects or localizes a target nucleic acid target gene in a cell or tissue sample by employing labeled DNA or RNA probe molecules that are antisense to the target gene sequence or transcript. ISH is performed by exposing a cell or tissue sample immobilized on a glass slide to a labeled nucleic acid probe specifically hybridizable to a given target gene in the sample. Multiple target genes can be analyzed simultaneously by exposing a cell or tissue sample to multiple nucleic acid probes that have been labeled with multiple different nucleic acid tags. By utilizing labels with different emission wavelengths, simultaneous multicolor analysis can be performed in one step on a single target cell or tissue sample.
本願発明の一実施例は、例えば、デジタル病理学分析結果の格納および読み出し方法に関する。 One embodiment of the present invention, for example, relates to a method for storing and retrieving digital pathology analysis results.
[0005]本開示は、とりわけ、不規則形状を有する生体(たとえば、線維芽細胞またはマクロファージ)と関連付けられたデータを分析および格納する自動化システムおよび方法に関する。また、本開示は、中解像度(mid-resolutionまたはmedium-resolution)分析手法すなわち類似特性(たとえば、染色強度、染色有無、および/または質感)を有するピクセルを「副領域」にグループ化する手法を用いて、生体と関連付けられたデータを分析および格納する自動化システムおよび方法に関する。 [0005] The present disclosure relates, among other things, to automated systems and methods for analyzing and storing data associated with irregularly shaped organisms (eg, fibroblasts or macrophages). The present disclosure also provides a mid-resolution or medium-resolution analysis technique, ie, a technique that groups pixels with similar characteristics (e.g., staining intensity, presence or absence of staining, and/or texture) into "sub-regions." Automated systems and methods for using to analyze and store data associated with living organisms.
[0006]デジタル病理学においては、ガラススライドに搭載され、バイオマーカの識別のために染色された生物標本(たとえば、組織標本)から画像が取得される。生物学的サンプルは、高倍率の顕微鏡下で評価すること、または、関心生体を検出して分類するデジタル病理学アルゴリズムによって自動的に分析することが可能である。たとえば、関心物体としては、細胞、血管、腺、組織領域等が可能である。如何なる導出情報も、データベースに格納されて後で読み出されるようになっていてもよく、当該データベースは、関心生物学的構造の有無、空間的関係、および/または染色の特性の統計値を含んでいてもよい。当業者には当然のことながら、明確に区別された細胞(たとえば、腫瘍細胞または免疫細胞)の分析結果の格納および読み出しは、比較的容易である。このような細胞は、各細胞の中心位置の点により表され、データベースに格納され得るためである(たとえば、図4参照)。同様に、サイズおよび形状が明確に定義された生体(たとえば、血管)は、簡単な外形により表され、この外形の座標がデータベースに格納されて、後で読み出しおよび/または別途分析が行われ得る(本明細書においては、「ポリゴン」または「ポリゴン輪郭」とも称する)。 [0006] In digital pathology, images are acquired from biological specimens (eg, tissue specimens) mounted on glass slides and stained for identification of biomarkers. Biological samples can be evaluated under a high power microscope or automatically analyzed by digital pathology algorithms that detect and classify organisms of interest. For example, objects of interest can be cells, blood vessels, glands, tissue regions, and the like. Any derived information may be stored in a database for later retrieval, including statistics of the presence or absence of biological structures of interest, spatial relationships, and/or staining properties. You can As will be appreciated by those skilled in the art, storing and retrieving analysis results for distinct cells (eg, tumor cells or immune cells) is relatively easy. Such cells can be represented by a point at the center position of each cell and stored in a database (see, eg, FIG. 4). Similarly, a living body of well-defined size and shape (e.g., a blood vessel) can be represented by a simple outline, the coordinates of which can be stored in a database for later retrieval and/or further analysis. (Also referred to herein as a "polygon" or "polygon contour").
[0007]一方、一部の関心生物学的構造(たとえば、線維芽細胞またはマクロファージ)は、不規則形状を有する。この種の細胞のグループは、互いの周りまたは他の細胞の周りに延びている場合がある(図5参照)。結果として、観察者または自動化アルゴリズムによって個別に、これら不規則形状の細胞を正確に識別するのは、困難な場合が多い。その代わりに、これらの細胞は、個々の細胞の識別なく、それぞれの染色細胞質または膜の局在のみによって識別されることが非常に多い。 [0007] On the other hand, some biological structures of interest (eg, fibroblasts or macrophages) have irregular shapes. Groups of such cells may extend around each other or around other cells (see Figure 5). As a result, it is often difficult to accurately identify these irregularly shaped cells individually by an observer or by an automated algorithm. Instead, these cells are very often distinguished only by their respective staining cytoplasmic or membrane localizations, without identification of individual cells.
[0008]このような不規則形状の構造は、高解像度分析を用いて分析および格納可能と考えられるが、このような手法では、相当のコンピュータリソース(演算時間および/または格納のリソース)が必要になることが多い。実際、関心生物学的構造のすべてのピクセル情報(たとえば、すべてのピクセルの分析結果)を格納する高解像度分析手法は、消費するソフトウェアおよびハードウェアリソース(たとえば、情報を処理または表示するメモリおよびプロセッサ)があまりにも多く、結局のところ、特定の生体に関して意味ある結果を提供できないと考えられる。 [0008] Although such irregularly shaped structures could be analyzed and stored using high resolution analysis, such techniques require substantial computational resources (computation time and/or storage resources). often become Indeed, high-resolution analytical techniques that store all pixel information of the biological structure of interest (e.g., analysis results for all pixels) consume software and hardware resources (e.g., memory and processors to process or display the information). ) are too numerous to ultimately provide meaningful results for a particular organism.
[0009]このような不規則構造は、低解像度分析を用いて分析することも可能と考えられ、このような低解像度データ表現では、複数の個々の細胞を単一のオブジェクトへと「一括化」して、データベースに格納するようにしてもよい。一例として、図6Aおよび図6Bは、望ましくない領域の「孔」(青緑色、640)を除外して一群の関連細胞を囲む大きなポリゴン外形(赤色、630)により表される腫瘍(黄色、620)および線維芽細胞(紫色、610)について染色されたIHC画像の一例を示している。本例においては、さまざまな特徴(たとえば、形状、サイズ、染色強度等)を有する多くの個々の細胞を含み得る大きな領域(赤色外形、630)に対して、分析結果が平均化される。たとえば、図6Bに関しては、外形規定されたFAP陽性面積が928.16μm2で、計算された線維芽細胞活性化タンパク質(FAP)陽性平均強度は0.26を有する。このように大きなピクセル面積領域における平均強度を所与として、0.26という平均強度は、この画像においてFAP陽性全体を示すとともに代表するには粗すぎる。任意特定の理論に縛られることを望むことなく、この低解像度分析手法は、格納結果が下流処理において後で利用される場合には、精度の低下につながり得ると考えられる。このため、このような染色細胞の不均質性により、この方法は、このような関心生物学的構造の領域の実際の詳細を局所的に提示しないと考えられる。 [0009] Such irregular structures could also be analyzed using low-resolution analysis, where such low-resolution data representation "lumps" multiple individual cells into a single object. ” and stored in the database. As an example, FIGS. 6A and 6B show a tumor (yellow, 620) represented by a large polygonal outline (red, 630) surrounding a group of related cells to the exclusion of undesirable area “pores” (turquoise, 640). ) and an example of an IHC image stained for fibroblasts (purple, 610). In this example, the analysis results are averaged over a large area (red outline, 630) that may contain many individual cells with varying characteristics (eg, shape, size, staining intensity, etc.). For example, with respect to FIG. 6B, the contoured FAP-positive area is 928.16 μm 2 with a calculated fibroblast activation protein (FAP)-positive average intensity of 0.26. Given an average intensity in such a large pixel area, an average intensity of 0.26 is too coarse to be indicative and representative of FAP positivity overall in this image. Without wishing to be bound by any particular theory, it is believed that this low-resolution analysis approach can lead to reduced accuracy if stored results are later utilized in downstream processing. Thus, due to the heterogeneity of such stained cells, the method may not locally present real details of such regions of biological structures of interest.
[00010]上述の高解像度および低解像度分析方法とは対照的に、本開示は、中解像度分析手法を用いて、画像特性(たとえば、質感、強度、または色のうちの少なくとも1つ)が類似する複数の副領域へと画像をセグメント化することにより、不規則形状の細胞に対応するデータを導出するシステムおよび方法を提供する。 [00010] In contrast to the high-resolution and low-resolution analysis methods described above, the present disclosure uses a medium-resolution analysis technique to analyze images with similar image characteristics (eg, at least one of texture, intensity, or color). Systems and methods are provided for deriving data corresponding to irregularly shaped cells by segmenting an image into a plurality of sub-regions that correspond to each other.
[00011]上記を考慮して、本開示の一態様は、少なくとも1つのステインを有する生物標本の画像から導出された画像分析データを格納する方法であって、(a)画像から、1つまたは複数の特徴測定基準を導出するステップと、(b)画像を複数の副領域にセグメント化するステップであり、各副領域が、染色有無、染色強度、または局所質感のうちの少なくとも1つにおいて実質的に均一なピクセルを含む、ステップと、(c)複数のセグメント化副領域に基づいて、複数の表現オブジェクトを生成するステップと、(d)複数の表現オブジェクトそれぞれを導出特徴測定基準と関連付けるステップと、(e)関連付け導出特徴測定基準と併せて、各表現オブジェクトの座標をデータベースに格納するステップと、を含む、方法である。当業者には当然のことながら、少なくともステップ(a)および(b)は、如何なる順序で実行されるようになっていてもよい。いくつかの実施形態において、画像を複数の副領域にセグメント化するステップは、スーパーピクセルを導出することを含む。いくつかの実施形態において、スーパーピクセルは、ピクセルを局所k平均クラスタリングでグループ化し、(ii)連結成分アルゴリズムを用いて小さな分離領域を大きな最近接スーパーピクセルへと統合することにより導出される。任意特定の理論に縛られることを望むことなく、(副領域としての)スーパーピクセルは、それぞれが知覚的に矛盾のない単位となるように、すなわち、スーパーピクセル中のすべてのピクセルの色および質感が均一となり得るように、知覚的に意味あるものと考えられる。いくつかの実施形態において、連結成分標識化では、画像をスキャンし、ピクセル連結度に基づいて、そのピクセルを成分へとグループ化する。すなわち、連結成分のすべてのピクセルが類似のピクセル強度値を共有し、何らかの方法で互いに連結されている。 [00011] In view of the above, one aspect of the present disclosure is a method of storing image analysis data derived from an image of a biological specimen having at least one stain, comprising: (a) from the image, one or (b) segmenting the image into a plurality of sub-regions, each sub-region being substantially different in at least one of staining presence/absence, staining intensity, or local texture; (c) generating a plurality of representation objects based on the plurality of segmented sub-regions; and (d) associating each of the plurality of representation objects with a derived feature metric. and (e) storing the coordinates of each representation object in a database along with the association-derived feature metrics. It will be appreciated by those skilled in the art that at least steps (a) and (b) may be performed in any order. In some embodiments, segmenting the image into subregions includes deriving superpixels. In some embodiments, superpixels are derived by grouping pixels with local k-means clustering and (ii) merging small isolated regions into large nearest neighbor superpixels using a connected component algorithm. Without wishing to be bound by any particular theory, superpixels (as subregions) are each perceptually consistent units, i.e., the color and texture of all pixels in the superpixel. can be uniform, it is considered perceptually meaningful. In some embodiments, connected component labeling scans an image and groups its pixels into components based on pixel connectivity. That is, all pixels of a connected component share similar pixel intensity values and are connected to each other in some way.
[00012]いくつかの実施形態において、画像を複数の副領域にセグメント化するステップは、サンプリンググリッドを画像に重ね合わせることであり、サンプリンググリッドが、所定のサイズおよび形状を有する非重畳エリアを規定する、ことを含む。いくつかの実施形態において、副領域は、M×Nのサイズを有し、Mが、50ピクセル~100ピクセルの範囲であり、Nが50ピクセル~およそ100ピクセルの範囲である。 [00012] In some embodiments, the step of segmenting the image into a plurality of subregions is superimposing a sampling grid over the image, the sampling grid defining non-overlapping areas having a predetermined size and shape. to do, including In some embodiments, the subregions have a size of M×N, where M ranges from 50 pixels to 100 pixels and N ranges from 50 pixels to approximately 100 pixels.
[00013]いくつかの実施形態において、表現オブジェクトは、所定の染色強度閾値を満たす副領域の外形を含む。いくつかの実施形態において、表現オブジェクトは、種子点を含む。いくつかの実施形態において、種子点は、複数の副領域それぞれの重心を演算することにより導出される。いくつかの実施形態において、導出特徴測定基準は、染色強度であり、各生成表現オブジェクト外形内のすべてのピクセルの平均染色強度が演算される。いくつかの実施形態において、導出特徴測定基準は、発現スコアであり、各生成副領域内のエリアに対応する平均発現スコアが複数の生成表現オブジェクトと関連付けられる。いくつかの実施形態において、この方法は、データベースから、格納座標および関連付け特徴測定基準データを読み出すステップと、読み出しデータを画像に投影するステップとをさらに含む。いくつかの実施形態において、対応する副領域内の分析結果(たとえば、強度、面積)は、当該副領域のピクセルデータを表す平均ピクセル測定結果の形態で格納可能である。 [00013] In some embodiments, the representational object includes outlines of subregions that meet a predetermined staining intensity threshold. In some embodiments, representation objects include seed points. In some embodiments, the seed points are derived by computing centroids of each of the multiple sub-regions. In some embodiments, the derived feature metric is staining intensity, and the average staining intensity of all pixels within each generated representational object outline is computed. In some embodiments, the derived feature metric is an expression score, and an average expression score corresponding to an area within each generated subregion is associated with a plurality of generated representational objects. In some embodiments, the method further includes retrieving the stored coordinates and associated feature metric data from the database and projecting the retrieved data onto the image. In some embodiments, analysis results (eg, intensity, area) within a corresponding sub-region can be stored in the form of average pixel measurements representing pixel data for that sub-region.
[00014]いくつかの実施形態において、生物学的サンプルは、膜ステインにより染色される。いくつかの実施形態において、生物学的サンプルは、膜ステインおよび核ステインにより染色される。いくつかの実施形態において、生物学的サンプルは、少なくともFAPにより染色され、1つまたは複数の導出特徴測定基準が、FAP染色強度またはFAP百分率正値性の少なくとも一方を含む。いくつかの実施形態においては、副領域内のすべてのピクセルに関して、平均FAP百分率正値性が計算される。いくつかの実施形態においては、副領域内のすべてのピクセルに関して、平均FAP染色強度が計算される。いくつかの実施形態において、サンプルは、FAPおよびH&Eにより染色される。いくつかの実施形態において、サンプルは、FAPならびに別の核もしくは膜ステインにより染色される。 [00014] In some embodiments, the biological sample is stained with a membrane stain. In some embodiments, biological samples are stained with membrane stains and nuclear stains. In some embodiments, the biological sample is stained with at least FAP and the one or more derived feature metrics comprise at least one of FAP staining intensity or FAP percent positivity. In some embodiments, the average FAP percent positivity is calculated for all pixels in the sub-region. In some embodiments, the average FAP staining intensity is calculated for all pixels within the sub-region. In some embodiments, samples are stained with FAP and H&E. In some embodiments, the sample is stained with FAP and another nuclear or membrane stain.
[00015]いくつかの実施形態において、入力として受信された画像は最初、画像チャネル画像(たとえば、特定のステインの画像チャネル画像)へと分離される。いくつかの実施形態においては、画像分析に先立って、関心領域が選択される。 [00015] In some embodiments, an image received as input is first separated into image channel images (eg, image channel images of a particular stain). In some embodiments, a region of interest is selected prior to image analysis.
[00016]本開示の別の態様は、少なくとも1つのステインを含む生物学的サンプルの画像から、不規則形状の細胞に対応するデータを導出するシステムであって、(i)1つまたは複数のプロセッサと、(ii)1つまたは複数のプロセッサに結合されたメモリであり、1つまたは複数のプロセッサにより実行された場合に、(a)画像から、1つまたは複数の特徴測定基準を導出することと、(b)画像内の複数の副領域を生成することであり、各副領域が、色、輝度、および/または質感から選択される類似特質を備えたピクセルを有する、ことと、(c)複数の生成副領域に基づいて、一連の表現オブジェクトを演算することと、(d)画像からの1つまたは複数の導出特徴測定基準を一連の演算表現オブジェクトそれぞれの計算座標と関連付けることと、を含む動作を1つまたは複数のプロセッサに実行させるコンピュータ実行可能命令を格納した、メモリと、を備えた、システムである。いくつかの実施形態において、副領域は、(i)隣り合うピクセル、(ii)知覚的に意味ある類似の特性(たとえば、色、輝度、および/または質感)を有するピクセル、および(iii)生物学的特性(たとえば、生物学的構造、生物学的構造の染色特性、細胞特性、細胞のグループ)に関して実質的に均質なピクセルをグループ化することによって形成される。いくつかの実施形態において、副領域のピクセルは、関心生体(たとえば、不規則形状の細胞であり、線維芽細胞およびマクロファージが挙げられるが、これらに限定されない)に関して、類似の特性および記述統計値を有する。 [00016] Another aspect of the present disclosure is a system for deriving data corresponding to irregularly shaped cells from an image of a biological sample comprising at least one stain, comprising: (i) one or more (ii) a memory coupled to the one or more processors for (a) deriving one or more feature metrics from an image when executed by the one or more processors; (b) generating a plurality of sub-regions within the image, each sub-region having pixels with similar attributes selected from color, brightness, and/or texture; c) computing a set of representation objects based on the plurality of generated sub-regions; and (d) associating one or more derived feature metrics from the image with the calculated coordinates of each of the set of computed representation objects. and a memory storing computer-executable instructions that cause one or more processors to perform operations including: In some embodiments, the sub-regions are (i) neighboring pixels, (ii) pixels with perceptually meaningful similar properties (e.g., color, brightness, and/or texture), and (iii) biological formed by grouping pixels that are substantially homogenous with respect to their biological properties (eg, biological structures, staining properties of biological structures, cellular properties, groups of cells). In some embodiments, the subregion pixels have similar properties and descriptive statistics with respect to the organism of interest (e.g., irregularly shaped cells, including but not limited to fibroblasts and macrophages). have
[00017]いくつかの実施形態において、画像を複数の副領域にセグメント化することは、スーパーピクセルを導出することを含む。いくつかの実施形態において、スーパーピクセルは、グラフベースの手法または勾配上昇ベースの手法の一方を用いて導出される。いくつかの実施形態において、スーパーピクセルは、ピクセルを局所k平均クラスタリングでグループ化し、(ii)連結成分アルゴリズムを用いて小さな分離領域を大きな最近接スーパーピクセルへと統合することにより導出される。 [00017] In some embodiments, segmenting the image into a plurality of subregions includes deriving superpixels. In some embodiments, superpixels are derived using either a graph-based approach or a gradient ascent-based approach. In some embodiments, superpixels are derived by grouping pixels with local k-means clustering and (ii) merging small isolated regions into large nearest neighbor superpixels using a connected component algorithm.
[00018]いくつかの実施形態において、表現オブジェクトは、所定の染色強度閾値を満たす副領域の外形を含む。いくつかの実施形態において、表現オブジェクトは、種子点を含む。いくつかの実施形態において、このシステムは、1つまたは複数の導出特徴測定基準および関連付け計算表現オブジェクトの座標をデータベースに格納する命令をさらに含む。いくつかの実施形態において、1つまたは複数の導出特徴測定基準は、百分率正値性、Hスコア、または染色強度から選択される少なくとも1つの発現スコアを含む。いくつかの実施形態において、不規則形状の細胞に対応するデータは、画像内の関心領域に対して導出される。いくつかの実施形態において、関心領域は、医療専門家によりアノテーションされた画像のエリアである。 [00018] In some embodiments, the representational object includes outlines of sub-regions that meet a predetermined staining intensity threshold. In some embodiments, representation objects include seed points. In some embodiments, the system further includes instructions for storing the coordinates of the one or more derived feature metrics and association computational representation objects in a database. In some embodiments, the one or more derived feature metrics comprise at least one expression score selected from percent positivity, H-score, or staining intensity. In some embodiments, data corresponding to irregularly shaped cells is derived for a region of interest within the image. In some embodiments, the region of interest is an area of the image annotated by a medical professional.
[00019]本開示の別の態様は、不規則形状を有する生体と関連付けられたデータを分析する命令を格納した非一時的コンピュータ可読媒体であって、命令が、(a)生物学的サンプルの画像から1つまたは複数の特徴測定基準を導出する命令であり、生物学的サンプルが、少なくとも1つのステインを含む、命令と、(b)類似特質を有するピクセルのグループ化によって画像を一連の副領域に分割する命令であり、特質が、色、輝度、および/または質感から選択される、命令と、(c)一連の分割副領域に基づいて、複数の表現オブジェクトを演算する命令と、(d)画像からの1つまたは複数の導出特徴測定基準を複数の演算表現オブジェクトそれぞれの計算座標と関連付ける命令と、を含む、非一時的コンピュータ可読媒体である。 [00019] Another aspect of the disclosure is a non-transitory computer-readable medium storing instructions for analyzing data associated with an irregularly shaped living body, the instructions comprising: (a) a biological sample; instructions for deriving one or more feature metrics from an image, the biological sample comprising at least one stain; (c) instructions for computing a plurality of representational objects based on a sequence of partitioned sub-regions; d) associating one or more derived feature metrics from the image with calculated coordinates of each of the plurality of computational representation objects.
[00020]いくつかの実施形態において、画像を一連の副領域に分割することは、スーパーピクセルを演算することを含む。いくつかの実施形態において、スーパーピクセルは、正規化カットアルゴリズム、凝集型クラスタリングアルゴリズム、クイックシフトアルゴリズム、ターボピクセルアルゴリズム、または単純線形反復クラスタリングアルゴリズムのうちの1つを用いて演算される。いくつかの実施形態において、スーパーピクセルは、単純反復クラスタリングを用いて生成され、スーパーピクセルサイズパラメータが、およそ40ピクセル~およそ400ピクセルに設定され、稠密度パラメータが、およそ10~およそ100に設定される。いくつかの実施形態において、スーパーピクセルは、ピクセルを局所k平均クラスタリングでグループ化し、(ii)連結成分アルゴリズムを用いて小さな分離領域を大きな最近接スーパーピクセルへと統合することにより演算される。 [00020] In some embodiments, dividing the image into a series of subregions includes computing superpixels. In some embodiments, superpixels are computed using one of a normalized cut algorithm, an agglomerative clustering algorithm, a quick shift algorithm, a turbo pixel algorithm, or a simple linear iterative clustering algorithm. In some embodiments, the superpixels are generated using simple iterative clustering, with the superpixel size parameter set from about 40 pixels to about 400 pixels, and the density parameter set from about 10 to about 100. be. In some embodiments, superpixels are computed by grouping pixels with local k-means clustering and (ii) merging small isolated regions into large nearest neighbor superpixels using a connected component algorithm.
[00021]いくつかの実施形態において、生物学的サンプルは、少なくともFAPにより染色され、1つまたは複数の導出特徴測定基準が、FAP染色強度またはFAP百分率正値性の少なくとも一方を含む。いくつかの実施形態においては、副領域内のすべてのピクセルに関して、平均FAP百分率正値性が計算される。いくつかの実施形態においては、副領域内のすべてのピクセルに関して、平均FAP染色強度が計算される。いくつかの実施形態において、表現オブジェクトは、ポリゴン外形および種子点の少なくとも一方を含む。いくつかの実施形態において、メモリは、1つまたは複数の導出特徴測定基準および関連付け計算表現オブジェクトの座標をデータベースに格納する命令を含む。いくつかの実施形態において、メモリは、格納情報を生物学的サンプルの画像に投影する命令をさらに含む。 [00021] In some embodiments, the biological sample is stained with at least FAP and the one or more derived feature metrics comprise at least one of FAP staining intensity or FAP percent positiveness. In some embodiments, the average FAP percent positivity is calculated for all pixels in the sub-region. In some embodiments, the average FAP staining intensity is calculated for all pixels within the sub-region. In some embodiments, the representation object includes at least one of a polygon outline and seed points. In some embodiments, the memory includes instructions for storing the coordinates of the one or more derived feature metrics and association computation representation objects in a database. In some embodiments, the memory further includes instructions for projecting the stored information onto an image of the biological sample.
[00022]本出願人らは、本明細書に記載のシステムおよび方法が、関心物体ごとに単一の位置または外形では規定不可能な生体の分析結果を格納する改良されたソリューションを提供することを示した。さらに、本出願人らは、本明細書に記載のシステムおよび方法が、ピクセルレベルの高解像度分析手法と比較して、分析結果を格納する格納空間を低減可能であると考える。特定のピクセルおよびその周りのピクセルの分析結果が一体的に副領域に格納され、副領域中のピクセルが類似の特性または特質(たとえば、色、輝度、質感)を有するためである。さらに、本出願人らは、数千個のピクセルから、分析結果の読み出しおよび報告の大幅な高速化を可能にするより少数で扱いやすい数の副領域へと、生成副領域が画像の複雑性を低減可能であることから、上記システムおよび方法が演算上効率的であると考える。また、本出願人らは、分析結果の格納および表現に対して、副領域が小さ過ぎることも大き過ぎることもないため、表現上効率的であるとも考える。最後に、本出願人らは、特に低解像度分析手法と比較して、本明細書に開示のシステムおよび方法が精度を向上可能であると考える。生成副領域は、大領域表現からの情報の格納と比較して、生物学的に関連する関心物体の特性または統計学的情報を記述するためである(すなわち、副領域は、染色有無、染色強度、および質感が可能な限り均一なピクセルを含む)。上記および他の利点については、本明細書において別途説明される。 [00022] Applicants believe that the systems and methods described herein provide an improved solution for storing biological analysis results that cannot be defined in a single location or geometry for each object of interest. showed that. Further, applicants believe that the systems and methods described herein can reduce storage space for storing analysis results compared to pixel-level high-resolution analysis techniques. This is because the analysis results for a particular pixel and its surrounding pixels are collectively stored in sub-regions, and pixels in sub-regions have similar characteristics or attributes (eg, color, brightness, texture). Furthermore, Applicants have demonstrated that the generated sub-regions can scale from image complexity from thousands of pixels to a smaller and more manageable number of sub-regions that allow for significantly faster readout and reporting of analytical results. We believe that the above system and method are computationally efficient because we can reduce . Applicants also believe that the sub-regions are neither too small nor too large for the storage and representation of the analysis results, and thus are expressively efficient. Finally, Applicants believe that the systems and methods disclosed herein can improve accuracy, especially when compared to low-resolution analytical techniques. The generated sub-region is to describe the biologically relevant properties or statistical information of the object of interest as compared to storing information from the large-region representation (i.e. the sub-regions are stained including pixels that are as uniform in intensity and texture as possible). These and other advantages are discussed elsewhere herein.
[00023]本開示の特徴の全般的な理解のため、図面を参照する。図面全体を通して、同一要素の識別には、同じ参照番号を使用している。 [00023] For a general understanding of the features of the present disclosure, reference is made to the drawings. The same reference numbers are used throughout the drawings to identify the same elements.
[00045]別段の明確な指定のない限り、2つ以上のステップまたは動作を含む本明細書に請求の如何なる方法においても、そのステップまたは動作の順序は、当該ステップまたは動作が列挙された順序に必ずしも限定されないことが了解されるものとする。 [00045] In any method claimed herein involving more than one step or action, unless expressly stated otherwise, the order of the steps or actions is the order in which the steps or actions are listed. It is to be understood that this is not necessarily so.
[00046]本明細書において、単数の用語「a」、「an」、および「the」は、文脈上の別段の明確な指定のない限り、複数の指示対象を含む。同様に、単語「または(or)」は、文脈上の別段の明確な指定のない限り、「および(and)」を含むことが意図される。用語「含む(includes)」は、「AまたはBを含む(includes A or B)」がA、B、またはAおよびBを含むことを意味するように、包含的に定義される。 [00046] As used herein, the singular terms "a," "an," and "the" include plural referents unless the context clearly dictates otherwise. Similarly, the word "or" is intended to include "and" unless the context clearly dictates otherwise. The term "includes" is defined inclusively such that "includes A or B" means including A, B, or A and B.
[00047]本明細書および特許請求の範囲において、「または(or)」は、上記定義の「および/または(and/or)」と同じ意味を有することが了解されるものとする。たとえば、リストにおいて項目を分離する場合、「または(or)」または「および/または(and/or)」は、包含的であるものと解釈され、多数または一覧の要素と、任意選択として付加的なリスト外の項目と、のうちの少なくとも1つを包含し、また、2つ以上を包含することも可能であるものとする。「~のうちの1つだけ(only one of)」もしくは「~のうちのちょうど1つだけ(exactly one of)」等の別段の明確な指定がなされた用語または特許請求の範囲において使用される「~から成る(consisting of)」という用語のみ、多数または一覧の要素のうちのちょうど1つの要素の包含を表す。一般的に、本明細書における用語「または(or)」は、「either」、「one of」、「only one of」、または「exactly one of」等の排他的な用語が先行する場合に排他的な選択肢(すなわち、「一方または他方であるが、両方ではない(one or the other but not both)」)を示す旨の解釈のみがなされるものとする。特許請求の範囲において使用される「~から本質的に成る(consisting essentially of)」は、特許法の分野において使用される場合の通常の意味を有するものとする。 [00047] As used herein and in the claims, "or" shall be understood to have the same meaning as "and/or" as defined above. For example, when separating items in a list, "or" or "and/or" is interpreted as being inclusive, and multiple or list elements and optionally additional unlisted items, and may include more than one. otherwise expressly specified terms such as "only one of" or "exactly one of" or when used in a claim The term "consisting of" alone indicates the inclusion of exactly one element of a plurality or list of elements. Generally, the term "or" herein is exclusive when preceded by an exclusive term such as "either," "one of," "only one of," or "exactly one of." are to be construed only as indicating alternatives (ie, "one or the other but not both"). As used in the claims, "consisting essentially of" shall have its ordinary meaning as used in the field of patent law.
[00048]用語「備える(comprising)」、「含む(including)」、「有する(having)」等は、区別なく使用され、同じ意味を有する。同様に、「備える(comprises)」、「含む(includes)」、「有する(has)」等は、区別なく使用され、同じ意味を有する。具体的に、これらの用語はそれぞれ、「備える(comprising)」の米国特許法における一般的な定義と矛盾なく定義されているため、「少なくとも以下の(at least the following)」を意味するオープンな用語として解釈され、また、付加的な特徴、限界、態様等を除外しないものと解釈される。このため、たとえば「構成要素a、b、およびcを有するデバイス(a device having components a, b, and c)」は、当該デバイスが少なくとも構成要素a、b、およびcを具備することを意味する。同様に、表現「ステップa、b、およびcを含む方法(a method involving steps a, b, and c)」は、当該方法が少なくともステップa、b、およびcを含むことを意味する。さらに、本明細書においては、ステップおよびプロセスが特定の順序で説明される可能性があるものの、当業者であれば、ステップおよびプロセスの順序が変動し得ることが認識されよう。 [00048] The terms "comprising," "including," "having," etc. are used interchangeably and have the same meaning. Similarly, the terms "comprises," "includes," "has," etc. are used interchangeably and have the same meaning. Specifically, each of these terms is defined consistently with the general definition in U.S. patent law of "comprising," and thus an open definition meaning "at least the following." It is to be interpreted as a term and not to exclude additional features, limitations, aspects, and the like. Thus, for example, "a device having components a, b, and c" means that the device comprises at least components a, b, and c. . Similarly, the phrase "a method involving steps a, b, and c" means that the method includes at least steps a, b, and c. Further, although steps and processes may be described in a particular order herein, those skilled in the art will recognize that the order of steps and processes may vary.
[00049]本明細書および特許請求の範囲において、表現「少なくとも1つ(at least one)」は、一覧の1つまたは複数の要素を参照して、要素一覧における要素のうちのいずれか1つまたは複数から選択される少なくとも1つの要素を意味するが、要素一覧に具体的に掲載されたありとあらゆる要素のうちの少なくとも1つを必ずしも含まず、また、要素一覧における要素の如何なる組み合わせも除外しないことが了解されるものとする。また、この定義によれば、表現「少なくとも1つ(at least one)」が参照する要素一覧内で具体的に識別された要素と関係するか否かに関わらず、これらの要素以外の要素が任意選択として存在し得る。このため、非限定的な一例として、「AおよびBのうちの少なくとも1つ(at least one of A and B)」(または同等に、「AまたはBのうちの少なくとも1つ(at least one of A or B)」もしくは「Aおよび/またはBのうちの少なくとも1つ(at least one of A and/or B)」)は、一実施形態において、少なくとも1つ(任意選択として、2つ以上)のA(Bは存在せず(任意選択として、B以外の要素を含む))、別の実施形態において、少なくとも1つ(任意選択として、2つ以上)のB(Aは存在せず(任意選択として、A以外の要素を含む))、さらに別の実施形態において、少なくとも1つ(任意選択として、2つ以上)のAおよび少なくとも1つ(任意選択として、2つ以上)のB(任意選択として、他の要素を含む)等を表し得る。 [00049] As used herein and in the claims, the phrase "at least one" refers to one or more elements of a list and any one of the elements in the list of elements. or at least one element selected from a plurality, but not necessarily including at least one of every and every element specifically listed in the list of elements, nor excluding any combination of elements in the list of elements shall be understood. Also, according to this definition, elements other than those specifically identified in the list of elements to which the phrase "at least one" refers may or may not be related. May be present as an option. Thus, as one non-limiting example, "at least one of A and B" (or equivalently, "at least one of A or B"). A or B" or "at least one of A and/or B"), in one embodiment, at least one (optionally two or more) of A (B is absent (optionally including elements other than B)), and in another embodiment, at least one (optionally two or more) B (A is absent (optionally optionally including elements other than A)), and in yet another embodiment, at least one (optionally two or more) A and at least one (optionally two or more) B (optionally optionally including other elements) and the like.
[00050]本明細書において、用語「生物学的サンプル(biological sample)」(本明細書においては、用語「生物標本(biological specimen)」もしくは「標本(specimen)」と区別なく使用される)または「組織サンプル(tissue sample)」(本明細書においては、用語「組織標本(tissue specimen)」と区別なく使用される)は、ウイルスを含む任意の有機体から得られた生体分子(タンパク質、ペプチド、核酸、脂質、糖質、またはこれらの組み合わせ等)を含む任意のサンプルを表す。有機体の他の例としては、哺乳類(たとえば、ヒト、猫、犬、馬、牛、および豚等の家畜動物、ならびにマウス、ラット、および霊長類等の実験動物)、昆虫、環形動物、クモ形類動物、有袋動物、爬虫類、両生類、バクテリア、および菌類が挙げられる。生物学的サンプルには、組織サンプル(たとえば、組織切片および組織の針生検標本)、細胞サンプル(たとえば、パップ塗抹標本もしくは血液塗抹標本等の細胞学的塗抹標本または顕微解剖により得られた細胞のサンプル)、または細胞分画、細胞片、もしくは細胞小器官(たとえば、細胞を溶解させ、遠心分離等によって細胞成分を分離したもの)を含む。生物学的サンプルの他の例としては、血液、血清、尿、精液、糞便物質、脳脊髄液、間質液、粘液、涙、汗、膿、(たとえば、外科生検もしくは針生検により得られた)生検組織、乳頭吸引液、耳垢、母乳、膣液、唾液、スワブ(口腔スワブ等)、または第1の生物学的サンプルから導出された生体分子を含む任意の物質が挙げられる。特定の実施形態において、本明細書における用語「生物学的サンプル(biological sample)」は、被験者から得られた腫瘍またはその一部から作成されたサンプル(均質化または液化サンプル)を表す。 [00050] As used herein, the term "biological sample" (used interchangeably herein with the terms "biological specimen" or "specimen") or A "tissue sample" (used interchangeably herein with the term "tissue specimen") is a biomolecule (protein, peptide , nucleic acids, lipids, carbohydrates, or combinations thereof). Other examples of organisms include mammals (e.g., domestic animals such as humans, cats, dogs, horses, cows, and pigs, and laboratory animals such as mice, rats, and primates), insects, annelids, spiders. Including morphs, marsupials, reptiles, amphibians, bacteria and fungi. Biological samples include tissue samples (e.g., tissue sections and needle biopsies of tissues), cell samples (e.g., cytological smears such as Pap smears or blood smears, or cells obtained by microdissection). sample), or cell fractions, cell debris, or organelles (eg, cells lysed and cellular components separated by centrifugation, etc.). Other examples of biological samples include blood, serum, urine, semen, fecal material, cerebrospinal fluid, interstitial fluid, mucus, tears, sweat, pus (obtained, for example, by surgical or needle biopsy). d) biopsy tissue, nipple aspirate, earwax, breast milk, vaginal fluid, saliva, swabs (such as buccal swabs), or any material containing biomolecules derived from a first biological sample. In certain embodiments, the term "biological sample" herein refers to a sample (homogenized or liquefied sample) made from a tumor or portion thereof obtained from a subject.
[00051]本明細書において、用語「バイオマーカ(biomarker)」または「マーカ(marker)」は、何らかの生物学的状態または条件の測定可能なインジケータを表す。特に、バイオマーカは、特に染色可能で、細胞の生物学的特徴(たとえば、細胞の種類または細胞の生理学的状態)を示すタンパク質またはペプチド(たとえば、表面タンパク質)であってもよい。免疫細胞マーカは、哺乳類の免疫反応に関する特徴を選択的に示すバイオマーカである。バイオマーカは、疾患もしくは病気の処置に対する身体の反応の決定または被験者が疾患もしくは病気に罹りやすくされているかの判定に用いられるようになっていてもよい。がんに関して、バイオマーカは、体内のがんの有無を示す生物学的物質を表す。バイオマーカは、腫瘍から分泌される分子またはがんの存在に対する身体の具体的反応であってもよい。がんの診断、予後診断、および疫学には、遺伝子バイオマーカ、後成的バイオマーカ、プロテオームバイオマーカ、グリコミックバイオマーカ、および撮像バイオマーカを使用可能である。このようなバイオマーカは、血液または血清等の非侵襲的に収集された生体液において検査され得る。複数の遺伝子およびタンパク質ベースのバイオマーカが患者ケアにおいてすでに使用されており、AFP(肝臓がん)、BCR-ABL(慢性骨髄性白血病)、BRCA1/BRCA2(乳がん/卵巣がん)、BRAF V600E(黒色腫/大腸がん)、CA-125(卵巣がん)、CA19.9(膵臓がん)、CEA(大腸がん)、EGFR(非小細胞肺がん)、HER-2(乳がん)、KIT(消化管間質系腫瘍)、PSA(前立腺特異抗原)、S100(黒色腫)、その他多くが挙げられるが、これらに限定されない。バイオマーカは、(早期がんを識別する)診断法および/または(がんの進行の予想ならびに/または特定の処置に対する被験者の反応および/もしくはがんの再発の可能性の予測を行う)予後診断法として有用と考えられる。 [00051] As used herein, the term "biomarker" or "marker" refers to a measurable indicator of some biological state or condition. In particular, biomarkers may be proteins or peptides (eg, surface proteins) that are particularly stainable and indicative of a cell's biological characteristics (eg, cell type or cell physiological state). Immune cell markers are biomarkers that selectively characterize mammalian immune responses. Biomarkers may be adapted for use in determining the body's response to a disease or disease treatment or determining whether a subject is predisposed to a disease or illness. With respect to cancer, biomarkers represent biological substances that indicate the presence or absence of cancer in the body. A biomarker may be a molecule secreted by a tumor or a specific response of the body to the presence of cancer. Genetic, epigenetic, proteomic, glycomic, and imaging biomarkers can be used for cancer diagnosis, prognosis, and epidemiology. Such biomarkers can be tested in non-invasively collected biological fluids such as blood or serum. Multiple gene- and protein-based biomarkers are already in use in patient care, including AFP (liver cancer), BCR-ABL (chronic myeloid leukemia), BRCA1/BRCA2 (breast/ovarian cancer), BRAF V600E ( melanoma/colorectal cancer), CA-125 (ovarian cancer), CA19.9 (pancreatic cancer), CEA (colorectal cancer), EGFR (non-small cell lung cancer), HER-2 (breast cancer), KIT ( gastrointestinal stromal tumors), PSA (prostate specific antigen), S100 (melanoma), and many others. Biomarkers are diagnostic (to identify early-stage cancer) and/or prognostic (to predict cancer progression and/or predict a subject's response to a particular treatment and/or the likelihood of cancer recurrence) It is considered useful as a diagnostic method.
[00052]本明細書において、用語「画像データ(image data)」は、本明細書における理解の通り、光学センサまたはセンサアレイ等によって生物学的組織サンプルから取得された生の画像データまたは前処理された画像データを含む。特に、画像データは、ピクセル行列を含んでいてもよい。本明細書において、用語「免疫組織化学(immunohistochemistry)」は、抗体等の特定の結合剤との抗原の相互作用を検出してサンプル中の抗原の有無または分布を決定する方法を表す。サンプルは、抗体-抗原結合を可能にする条件下で抗体と接触される。抗体-抗原結合は、抗体に接合された検出可能な標識による検出(直接検出)または主抗体と特異的に結合する副抗体に接合された検出可能な標識による検出(間接検出)が可能である。本明細書において、「マスク(mask)」は、デジタル画像の派生語であって、マスクの各ピクセルが2進値(たとえば、「1」もしくは「0」(または、「真」もしくは「偽」))として表される。デジタル画像に前記マスクを重ねることにより、デジタル画像に適用される別途処理ステップにおいては、2進値のうちの特定の1つのマスクピクセルにマッピングされたデジタル画像のすべてのピクセルが隠蔽、削除、あるいは無視またはフィルタリング除去される。たとえば、真あるいは偽の閾値を上回る強度値を元の画像のすべてのピクセルに割り当て、「偽」でマスクされたピクセルが重なるすべてのピクセルを除去することによって、元のデジタル画像からマスクが生成され得る。「マルチチャネル画像」は、本明細書における理解の通り、異なるスペクトル帯における蛍光あるいは検出可能性によってマルチチャネル画像のチャネルのうちの1つを構成する特定の蛍光染料、量子ドット、色原体等によって、核および組織構造等の異なる生物学的構造が同時に染色される生物学的組織サンプルから得られたデジタル画像を含む。 [00052] As used herein, the term "image data", as understood herein, refers to raw image data acquired from a biological tissue sample, such as by an optical sensor or sensor array, or pre-processed contains the processed image data. In particular, the image data may include pixel matrices. As used herein, the term "immunohistochemistry" refers to methods that detect the interaction of antigens with specific binding agents, such as antibodies, to determine the presence or distribution of antigens in a sample. The sample is contacted with the antibody under conditions that allow antibody-antigen binding. Antibody-antigen binding can be detected by a detectable label conjugated to the antibody (direct detection) or by a detectable label conjugated to a secondary antibody that specifically binds to the primary antibody (indirect detection). . As used herein, "mask" is a derivative of digital image in which each pixel of the mask has a binary value (e.g., "1" or "0" (or "true" or "false"). )). By superimposing the mask on the digital image, in a separate processing step applied to the digital image, all pixels of the digital image mapped to a particular one of the binary value mask pixels are obscured, deleted, or Ignored or filtered out. For example, a mask is generated from the original digital image by assigning an intensity value above the true or false threshold to all pixels in the original image, and removing all pixels that overlap the "false" masked pixels. obtain. A "multi-channel image", as understood herein, is defined as the specific fluorescent dyes, quantum dots, chromogens, etc. that make up one of the channels of the multi-channel image due to their fluorescence or detectability in different spectral bands. includes digital images obtained from biological tissue samples in which different biological structures, such as nuclei and tissue structures, are simultaneously stained by .
[00053]概要
[00054]本出願人らは、たとえば線維芽細胞またはマクロファージ等、不規則形状を有する生体の分析結果をデータベース等の非一時的メモリに格納するシステムおよび方法を開発した。分析結果は、データベースまたはメモリから後で読み出され、他の下流プロセスにおいてさらに分析または使用されるようになっていてもよい。また、分析結果は、入力画像または他の導出画像に投影されるようになっていてもよいし、他の手段により視覚化されるようになっていてもよい。また、本開示は、(たとえば、単純形状のサイズの増減またはスーパーピクセルアルゴリズムのパラメータの調整によって)生成副領域のサイズを調整可能とすることによって、調整可能な詳細レベルでの分析結果の格納および報告を容易化し得る。このことは、関心グローバル領域からの平均的な分析結果が保存される本明細書に記載の低解像度分析手法と比較して、効率および精度を向上可能であると考えられる。
[00053] Overview
[00054] Applicants have developed a system and method for storing analytical results of irregularly shaped organisms, such as fibroblasts or macrophages, in non-transitory memory, such as a database. Analysis results may later be retrieved from a database or memory for further analysis or use in other downstream processes. Analysis results may also be projected onto the input image or other derived image, or visualized by other means. The present disclosure also provides for storing analysis results at an adjustable level of detail and It can facilitate reporting. It is believed that this can improve efficiency and accuracy compared to the low-resolution analysis approach described herein, where average analysis results from global regions of interest are saved.
[00055]本明細書に別途記載の通り、開示のシステムおよび方法は、局所的に類似する小さな領域(副領域)を用いて分析結果を格納する中解像度分析手法に基づく。副領域としては、単純形状(たとえば、円形、正方形)も可能であるし、複雑形状(たとえば、スーパーピクセル)も可能であり、スライド全体にまたがる各小領域の局所的な分析結果を格納するのに利用される。本中解像度手法により規定される副領域は、類似(または、均質)の特性(たとえば、染色有無(すなわち、特定のステインの有無)、染色強度(すなわち、ステインの相対強度(または、量))、局所質感(すなわち、画像または画像の選択領域における色または強度の空間的配置に関する情報))を有するピクセルをグループ化して、不規則形状の物体の識別を可能にする。いくつかの実施形態において、中解像度手法における副領域は、およそ50~およそ100ピクセルの範囲のサイズまたはおよそ2,500ピクセル2~およそ10,000ピクセル2のピクセル面積を有する。当然のことながら、副領域は、如何なるサイズを有していてもよく、このサイズは、実施される分析の種類および/または調査される細胞の種類に基づいていてもよい。 [00055] As described elsewhere herein, the disclosed systems and methods are based on a medium-resolution analysis approach that stores analysis results using small locally similar regions (sub-regions). Subregions can be simple (e.g., circle, square) or complex (e.g., superpixel) and are used to store local analysis results for each subregion across the entire slide. used for Sub-regions defined by the present medium-resolution approach have similar (or homogenous) properties (e.g., presence or absence of staining (ie, presence or absence of a particular stain), staining intensity (ie, relative intensity (or amount) of staining) , pixels with local texture (ie, information about the spatial distribution of color or intensity in an image or selected regions of an image) are grouped to allow identification of irregularly shaped objects. In some embodiments, the subregions in the medium resolution approach have sizes ranging from approximately 50 to approximately 100 pixels or pixel areas from approximately 2,500 pixels2 to approximately 10,000 pixels2. Of course, the subregions may have any size, and this size may be based on the type of analysis to be performed and/or the type of cells investigated.
[00056]当業者には当然のことながら、中レベル手法は、本明細書に記載の高解像度分析手法と低解像度分析手法との間にあるため、データが副領域レベルで収集されるとともに、副領域は、低解像度手法における関心領域よりも比例的に小さく、また、高解像度分析手法におけるピクセルよりも明らかに大きい。「高解像度分析」は、画像データがピクセルレベルまたは実質的にピクセルレベルで取り込まれることを意味する。一方、「低解像度分析」は、少なくとも500ピクセル×500ピクセルのサイズを有する領域または250,000ピクセル2より大きなサイズを有するエリア等、領域レベル分析を表す。当業者には当然のことながら、低解像度分析手法は、多くの生体(たとえば、複数の不規則形状の細胞)を包含することになる。 [00056] It will be appreciated by those skilled in the art that the mid-level approach lies between the high-resolution and low-resolution analysis approaches described herein, so that data are collected at the subregion level and The sub-regions are proportionally smaller than the regions of interest in the low-resolution approach and clearly larger than the pixels in the high-resolution analysis approach. "High resolution analysis" means that image data is captured at the pixel level or substantially at the pixel level. On the other hand, "low-resolution analysis" refers to region-level analysis, such as regions having a size of at least 500 pixels by 500 pixels or areas having a size greater than 250,000 pixels2 . As will be appreciated by those skilled in the art, low resolution analytical techniques will encompass many organisms (eg, multiple irregularly shaped cells).
[00057]本開示は、線維芽細胞またはマクロファージ等、不規則な形状および/またはサイズを有する生体の分析および格納を表し得る。本開示は、線維芽細胞またはマクロファージに限定されず、サイズまたは形状が十分に規定されていない任意の生体へと拡張可能であることが了解されるものとする。 [00057] The present disclosure may represent analysis and storage of living organisms having irregular shapes and/or sizes, such as fibroblasts or macrophages. It is to be understood that the present disclosure is not limited to fibroblasts or macrophages, but is extendable to any living organism whose size or shape is not well defined.
[00058]線維芽細胞に関して、これは、動物の組織において細胞外基質およびコラーゲンで構成された構造骨組または間質を構成する細胞である。これらの細胞は、動物において最も一般的な種類の結合組織であり、創傷治癒に重要である。線維芽細胞は、さまざまな形状およびサイズのほか、活性化および非活性化の形態となる(たとえば、図5A~図5D参照)。線維芽細胞が活性化形態である(接尾辞「blast」が代謝的に活性な細胞を表す)一方、線維化関連細胞は、低活性と考えられる。ただし、線維芽細胞および線維化関連細胞の両者が異なるものとして指定されず、単に線維芽細胞と称する場合もある。線維芽細胞は、粗面小胞体の豊富さおよび相対的に大きなサイズによって、線維化関連細胞から形態学的に区別可能である。さらに、線維芽細胞は、それぞれの隣接細胞と接触すると考えられ、これらの接触は、分離細胞の形態を歪ませ得る付着と考えられる。本明細書に提示の中解像度分析手法は、これらの形態学的差異を考慮することができ、線維芽細胞、マクロファージ、および他の不規則な生体に関する情報を格納するのに最適と考えられる。 [00058] With respect to fibroblasts, these are cells that make up the structural framework or stroma composed of extracellular matrix and collagen in animal tissues. These cells are the most common type of connective tissue in animals and are important for wound healing. Fibroblasts come in a variety of shapes and sizes, as well as activated and non-activated forms (see, eg, FIGS. 5A-5D). Fibroblasts are the activated form (the suffix "blast" denotes metabolically active cells), whereas fibrosis-associated cells are considered less active. However, both fibroblasts and fibrosis-associated cells are not designated as distinct and are sometimes simply referred to as fibroblasts. Fibroblasts are morphologically distinguishable from fibrosis-associated cells by the abundance and relatively large size of the rough endoplasmic reticulum. In addition, fibroblasts are thought to make contact with their respective neighboring cells, and these contacts are thought to be attachments that can distort the morphology of the detached cells. The medium-resolution analysis approach presented here can account for these morphological differences and is considered optimal for storing information about fibroblasts, macrophages, and other irregular organisms.
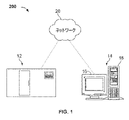
[00059]図1には、いくつかの実施形態に係る、標本を撮像および分析するデジタル病理学システム200を示している。デジタル病理学システム200は、撮像装置12(たとえば、標本を載せた顕微鏡スライドをスキャンする手段を有する装置)およびコンピュータ14を備えることにより、撮像装置12およびコンピュータが一体的に(たとえば、ネットワーク20を介して直接または間接的に)通信可能に結合されていてもよい。コンピュータシステム14には、デスクトップコンピュータ、ラップトップコンピュータ、タブレット等、デジタル電子回路、ファームウェア、ハードウェア、メモリ、コンピュータ記憶媒体、コンピュータプログラムもしくは命令セット(たとえば、プログラムがメモリまたは記憶媒体に格納されている場合)、1つもしくは複数のプロセッサ(プログラムされたプロセッサを含む)、ならびにその他任意のハードウェア、ソフトウェア、もしくはファームウェアモジュール、またはこれらの組み合わせを含み得る。たとえば、図1に示されるコンピュータシステム14は、表示装置16および筐体18を有するコンピュータを備えていてもよい。コンピュータは、2進形態のデジタル画像を(メモリ内など、ローカルに、サーバ、または別のネットワーク接続デバイスに)格納可能である。また、デジタル画像は、行列状のピクセルへと分割可能である。ピクセルは、ビット深度により規定された1つまたは複数のビットのデジタル値を含み得る。当業者には当然のことながら、他のコンピュータデバイスまたはシステムが利用されるようになっていてもよく、また、本明細書に記載のコンピュータシステムは、付加的な構成要素(たとえば、標本分析装置、顕微鏡、他の撮像システム、自動化スライド作成機器等)に対して通信可能に結合されていてもよい。これらの付加的な構成要素および利用可能な種々コンピュータ、ネットワーク等の一部については、本明細書において別途説明される。
[00059] FIG. 1 illustrates a
[00060]一般的に、撮像装置12(または、予備スキャン画像がメモリに格納された他の画像源)としては、1つまたは複数の画像取り込み装置が挙げられるが、これに限定されない。画像取り込み装置には、カメラ(たとえば、アナログカメラ、デジタルカメラ等)、光学素子(たとえば、1つまたは複数のレンズ、センサ焦点レンズ群、顕微鏡対物レンズ等)、撮像センサ(たとえば、電荷結合素子(CCD)、相補型金属酸化膜半導体(CMOS)画像センサ等)、写真フィルム等を含み得るが、これらに限定されない。デジタルの実施形態において、画像取り込み装置は、オンザフライフォーカシングを証明するように協働する複数のレンズを具備し得る。画像センサ(たとえば、CCDセンサ)が標本のデジタル画像を取り込み可能である。いくつかの実施形態において、撮像装置12は、明視野撮像システム、マルチスペクトル撮像(MSI)システム、または蛍光顕微鏡システムである。デジタル化された組織データは、たとえばVENTANA MEDICAL SYSTEMS,Inc.(Tucson、Arizona)によるVENTANA iScan HTスキャナ等の画像スキャンシステムまたは他の好適な撮像機器により生成されるようになっていてもよい。付加的な撮像デバイスおよびシステムについては、本明細書において別途説明される。当業者には当然のことながら、撮像装置12により取得されるデジタルカラー画像は従来、原色ピクセルで構成され得る。各色付きピクセルは、それぞれ同数のビットを含む3つのデジタル成分上でコード化可能であり、各成分が原色(一般的には、赤色、緑色、または青色で、用語「RGB」成分としても表される)に対応する。
[00060] In general, imager 12 (or other image source with pre-scan images stored in memory) includes, but is not limited to, one or more image capture devices. Image capture devices include cameras (e.g., analog cameras, digital cameras, etc.), optical elements (e.g., one or more lenses, sensor focusing lens groups, microscope objectives, etc.), imaging sensors (e.g., charge-coupled devices (CCD ), complementary metal oxide semiconductor (CMOS) image sensors, etc.), photographic film, and the like. In digital embodiments, the image capture device may include multiple lenses that cooperate to demonstrate on-the-fly focusing. An image sensor (eg, a CCD sensor) can capture a digital image of the specimen. In some embodiments,
[00061]図2は、上記開示のデジタル病理学システムにおいて利用されるさまざまなモジュールの概要を示している。いくつかの実施形態において、デジタル病理学システムは、1つもしくは複数のプロセッサ203ならびに少なくとも1つのメモリ201を有するコンピュータデバイス200またはコンピュータ実装方法を採用しており、少なくとも1つのメモリ201は、1つまたは複数のプロセッサによる実行によって、1つまたは複数のモジュール(たとえば、モジュール202および205~209)における命令(または、格納データ)を1つまたは複数のプロセッサに実行させる非一時的コンピュータ可読命令を格納している。
[00061] Figure 2 provides an overview of the various modules utilized in the digital pathology system disclosed above. In some embodiments, the digital pathology system employs a
[00062]図2および図3を参照して、本開示は、たとえば線維芽細胞またはマクロファージ等、不規則形状を有する生体の分析および/またはデータベース等の非一時的メモリへの分析結果の格納を行うコンピュータ実装方法を提供する。この方法は、たとえば(a)画像取得モジュール202を動作させて、マルチチャネル画像データ(たとえば、1つまたは複数のステインにより染色された生物学的サンプルの取得画像)を生成または受信するステップ(ステップ300)と、(b)画像分析モジュール205を動作させて、取得画像内の特徴から1つまたは複数の測定基準を導出するステップ(ステップ310)と、(c)セグメント化モジュール206を動作させて、取得画像を複数の副領域にセグメント化するステップ(ステップ320)と、(d)表現オブジェクト生成モジュール207を動作させて、副領域を識別するポリゴン、中心種子、または他のオブジェクトを生成するステップ(ステップ330)と、(e)標識化モジュール208を動作させて、1つまたは複数の導出測定基準を生成表現オブジェクトと関連付けるステップ(ステップ340)と、(f)表現オブジェクトおよび関連する測定基準をデータベース209に格納するステップ(ステップ350)とを含んでいてもよい。当業者には当然のことながら、ワークフローには、付加的なモジュールまたはデータベースが組み込まれていてもよい。たとえば、画像処理モジュールの動作によって、特定のフィルタを取得画像に適用するようにしてもよいし、組織サンプル内の特定の組織学的および/または形態学的構造を識別するようにしてもよい。また、関心領域選択モジュールの利用によって、分析する画像の特定の部分を選択するようにしてもよい。同様に、分離モジュールの動作によって、特定のステインまたはバイオマーカに対応する画像チャネル画像を提供するようにしてもよい。
[00062] Referring to FIGS. 2 and 3, the present disclosure provides analysis of irregularly shaped organisms, such as fibroblasts or macrophages, and/or storage of analysis results in non-transitory memory, such as a database. A computer-implemented method is provided. The method includes, for example, (a) operating the
[00063]画像取得モジュール
[00064]いくつかの実施形態において、デジタル病理学システム200は、最初のステップとして、図2を参照するに、画像取得モジュール202を動作させて、1つまたは複数のステインを有する生物学的サンプルの画像または画像データを取り込む。いくつかの実施形態において、受信または取得画像は、RGB画像またはマルチスペクトル画像(たとえば、多重化明視野および/または暗視野画像)である。いくつかの実施形態において、取り込まれた画像は、メモリ201に格納される。
[00063] Image Acquisition Module
[00064] In some embodiments, as a first step, referring to FIG. 2, the
[00065]画像または画像データ(本明細書においては、区別なく使用される)は、実時間等で撮像装置12により取得されるようになっていてもよい。いくつかの実施形態において、画像は、本明細書に記載の通り、標本を載せた顕微鏡スライドの画像データを取り込み可能な顕微鏡等の器具から取得される。いくつかの実施形態において、画像は、画像タイルをスキャンし得るような2DスキャナまたはVENTANA DP 200スキャナ等のラインごとに画像をスキャン可能なラインスキャナを用いて取得される。あるいは、画像は、事前に取得(たとえば、スキャン)され、メモリ201に格納された(または、この点に関して、ネットワーク20経由でサーバから読み出された)画像であってもよい。
[00065] An image or image data (used interchangeably herein) may be captured by imaging
[00066]生物学的サンプルは、1つまたは複数のステインの適用により染色されていてもよく、結果として画像または画像データは、1つまたは複数のステインそれぞれに対応する信号を含む。このため、本明細書に記載のシステムおよび方法は、単一のステイン(たとえば、ヘマトキシリン)の推定または正規化が可能であるが、生物学的サンプル内のステインの数に制限はない。実際、生物学的サンプルは、任意のカウンタステインの追加または包含として、2つ以上のステインの多重アッセイにおいて染色されていてもよい。 [00066] A biological sample may be stained by application of one or more stains, and as a result the image or image data includes signals corresponding to each of the one or more stains. Thus, the systems and methods described herein are capable of estimating or normalizing a single stain (eg, hematoxylin), but not limiting the number of stains within a biological sample. In fact, a biological sample may be stained in multiplex assays for two or more stains in addition or inclusion of any counterstain.
[00067]当業者には当然のことながら、生物学的サンプルは、異なる種類の核および/または細胞膜バイオマーカに対して染色されるようになっていてもよい。さまざまな目的に適したステインの選定において組織構造を染色するとともにガイドする方法は、たとえばSambrook et al.「Molecular Cloning:A Laboratory Manual(分子クローニング:実験室マニュアル)」,Cold Spring Harbor Laboratory Press(1989)およびAusubel et al.「Current Protocols in Molecular Biology(分子生物学における現在の慣習)」,Greene Publishing Associates and Wiley-Intersciences(1987)に記載されており、それぞれの開示内容が参照により本明細書に組み込まれる。 [00067] It will be appreciated by those skilled in the art that the biological sample may be stained for different types of nuclear and/or cell membrane biomarkers. Methods for staining tissue structures and guiding in the selection of suitable stains for various purposes are described, for example, in Sambrook et al. See "Molecular Cloning: A Laboratory Manual", Cold Spring Harbor Laboratory Press (1989) and Ausubel et al. "Current Protocols in Molecular Biology", Greene Publishing Associates and Wiley-Intersciences (1987), the disclosures of each of which are incorporated herein by reference.
[00068]非限定的な一例として、いくつかの実施形態においては、線維芽細胞活性化タンパク質(FAP)を含む1つまたは複数のバイオマーカの存在に対して、組織サンプルがIHCアッセイにおいて染色される。線維芽細胞の細胞株におけるFAPの過剰発現は、悪性挙動を促進すると考えられる。腫瘍微小環境の必須成分であり、がん関連線維芽細胞(CAF)として指定されることが多い間質線維芽細胞は、増殖、血管形成、侵入、残存、および免疫抑制等の複数のメカニズムによって、腫瘍形成および進行を促進し得ることが示されている。任意特定の理論に縛られることを望むことなく、がん細胞が間質線維芽細胞を活性化させるとともにFAPの発現を誘発し、これががん細胞の増殖、侵入、および移動に影響を及ぼすと考えられる。FAPは、乳房、肺、結腸直腸、卵巣、膵臓、および頭頚部を含む人間の上皮細胞がんの90%にて、反応性間質線維芽細胞に激しく発現すると考えられる。このため、腫瘍の臨床的挙動に対して、FAPの量が重要な予後診断を提示する可能性が最も高い(これは、導出後に生成副領域または表現オブジェクトと関連付け可能なある種の測定基準の一例である)。 [00068] As a non-limiting example, in some embodiments, tissue samples are stained in an IHC assay for the presence of one or more biomarkers, including fibroblast activation protein (FAP). be. Overexpression of FAP in fibroblast cell lines is thought to promote malignant behavior. Stromal fibroblasts, an essential component of the tumor microenvironment and often designated as cancer-associated fibroblasts (CAFs), are controlled by multiple mechanisms such as proliferation, angiogenesis, invasion, persistence, and immunosuppression. , can promote tumorigenesis and progression. Without wishing to be bound by any particular theory, it is believed that cancer cells activate stromal fibroblasts and induce expression of FAP, which affects cancer cell proliferation, invasion and migration. Conceivable. FAP is thought to be heavily expressed in reactive stromal fibroblasts in 90% of human epithelial cell carcinomas, including breast, lung, colorectal, ovarian, pancreatic, and head and neck. For this reason, the amount of FAP most likely presents an important prognostic value for the clinical behavior of tumors (it is some kind of metric that can be associated with generated subregions or representational objects after derivation). is an example).
[00069]色原体ステインとしては、ヘマトキシリン、エオシン、ファストレッド、または3,3’-ジアミノベンジジン(DAB)が挙げられる。また、当業者には当然のことながら、任意の生物学的サンプルが1つまたは複数のフルオロフォアにより染色されるようになっていてもよい。いくつかの実施形態において、組織サンプルは、主ステイン(たとえば、ヘマトキシリン)により染色される。いくつかの実施形態においては、特定のバイオマーカについて、組織サンプルがIHCアッセイにおいて染色される。また、サンプルは、1つまたは複数の蛍光染料により染色されるようになっていてもよい。 [00069] Chromogenic stains include hematoxylin, eosin, fast red, or 3,3'-diaminobenzidine (DAB). It will also be appreciated by those skilled in the art that any biological sample may be stained with one or more fluorophores. In some embodiments, tissue samples are stained with a primary stain (eg, hematoxylin). In some embodiments, tissue samples are stained in IHC assays for particular biomarkers. The sample may also be stained with one or more fluorescent dyes.
[00070]通常の生物学的サンプルは、ステインを当該サンプルに適用する自動染色/アッセイプラットフォームにおいて処理される。市場には、染色/アッセイプラットフォームとしての使用に適した多様な商品が存在しており、一例として、Ventana Medical Systems,Inc.(Tucson、AZ)のDiscovery(商標)製品がある。また、カメラプラットフォームとしては、明視野顕微鏡(Ventana Medical Systems,Inc.のVENTANA iScan HTまたはVENTANA DP 200スキャナ等)または1つもしくは複数の対物レンズおよびデジタル撮像装置を有する任意の顕微鏡が挙げられる。さまざまな波長で画像を取り込む他の技術が用いられるようになっていてもよい。当技術分野においては、染色された生物標本の撮像に適した別のカメラプラットフォームが知られており、Zeiss、Canon、Applied Spectral Imaging等の企業から市販されている。また、このようなプラットフォームは、本開示のシステム、方法、および装置における使用に対して容易に適応可能である。
[00070] A typical biological sample is processed in an automated staining/assay platform that applies a stain to the sample. There are a variety of commercial products on the market suitable for use as a staining/assay platform, one example being Ventana Medical Systems, Inc.; (Tucson, AZ) Discovery™ product. Camera platforms also include brightfield microscopes (such as the Ventana iScan HT or
[00071]いくつかの実施形態において、入力画像は、組織領域のみが画像中に存在するようにマスクされる。いくつかの実施形態においては、非組織領域を組織領域からマスクするように組織領域マスクが生成される。いくつかの実施形態においては、組織領域を識別するとともに、背景領域(たとえば、撮像源からの白色光のみが存在する場所等、サンプルのないガラスに対応する全スライド画像の領域)を自動的または半自動的に(すなわち、最小限のユーザ入力で)除外することによって、組織領域マスクが形成されるようになっていてもよい。当業者には当然のことながら、非組織領域の組織領域からのマスキングのほか、組織マスキングモジュールは、特定の組織種または腫瘍が疑われる領域に属するものとして識別された組織の一部等、必要に応じて他の関心エリアをマスクするようにしてもよい。いくつかの実施形態においては、入力画像において非組織領域から組織領域をマスクすることにより組織領域マスキング画像を生成するのにセグメント化技術が用いられる。好適なセグメント化技術としては、先行技術から既知のものがある(「Digital Image Processing(デジタル画像処理)」,Third Edition,Rafael C.Gonzalez,Richard E.Woods,chapter 10,page 689および「Handbook of Medical Imaging(医用画像ハンドブック)」,Processing and Analysis,Isaac N.Bankman Academic Press,2000,chapter 2参照)。いくつかの実施形態においては、画像中のデジタル化組織データとスライドとの識別に画像セグメント化技術が利用されるが、この場合、組織が前景に、スライドが背景に対応する。いくつかの実施形態においては、構成要素が全スライド画像中の関心エリア(AoI)を演算することにより、分析される背景の非組織エリアの量を制限しつつ、AoI中のすべての組織領域を検出する。たとえば、組織データと非組織または背景データとの境界を決定するのに、広範な画像セグメント化技術(たとえば、HSVカラーベース画像セグメント化、Lab画像セグメント化、平均シフトカラー画像セグメント化、領域拡張、レベル設定法、高速マーチング法等)が使用され得る。また、構成要素は、セグメント化に少なくとも部分的に基づいて、組織データに対応するデジタル化スライドデータの部分の識別に使用可能な組織前景マスクを生成可能である。あるいは、構成要素は、組織データに対応しないデジタル化スライドデータの部分の識別に用いられる背景マスクを生成可能である。 [00071] In some embodiments, the input image is masked so that only tissue regions are present in the image. In some embodiments, a tissue region mask is generated to mask non-tissue regions from tissue regions. Some embodiments automatically or A tissue region mask may be formed by semi-automatic (ie, with minimal user input) exclusion. Those skilled in the art will appreciate that, in addition to masking non-tissue regions from tissue regions, the tissue masking module may also mask any necessary tissue, such as a portion of tissue identified as belonging to a particular tissue type or suspected tumor region. Other areas of interest may be masked depending on . In some embodiments, a segmentation technique is used to generate a tissue region masked image by masking tissue regions from non-tissue regions in the input image. Suitable segmentation techniques include those known from the prior art ("Digital Image Processing", Third Edition, Rafael C. Gonzalez, Richard E. Woods, chapter 10, page 689 and "Handbook of Medical Imaging", Processing and Analysis, Isaac N. Bankman Academic Press, 2000, chapter 2). In some embodiments, image segmentation techniques are utilized to distinguish between the digitized tissue data in the image and the slide, where the tissue corresponds to the foreground and the slide to the background. In some embodiments, the component computes an area of interest (AoI) in the entire slide image, thereby limiting the amount of background non-tissue area analyzed, while all tissue regions in the AoI are To detect. For example, a wide range of image segmentation techniques (e.g., HSV color-based image segmentation, Lab image segmentation, mean-shift color image segmentation, region growing, level setting method, fast marching method, etc.) may be used. Also, based at least in part on the segmentation, the component can generate a tissue foreground mask that can be used to identify portions of the digitized slide data corresponding to the tissue data. Alternatively, the component can generate a background mask that is used to identify portions of the digitized slide data that do not correspond to tissue data.
[00072]この識別は、エッジ検出等の画像分析動作によって可能となり得る。画像(たとえば、非組織領域)中の非組織背景ノイズを除去するのに、組織領域マスクが用いられるようになっていてもよい。いくつかの実施形態において、組織領域マスクの生成には、輝度が所与の閾値を上回るピクセルが1に設定され、閾値を下回るピクセルが0に設定されて、組織領域マスクが生成されるように、低分解能入力画像の輝度の演算、輝度画像の生成、輝度画像への標準偏差フィルタの適用、フィルタリング輝度画像の生成、およびフィルタリング輝度画像への閾値の適用といった動作のうちの1つまたは複数を含む(ただし、これらの動作に限定されない)。組織領域マスクの生成に関する付加的な情報および例については、「An Image Processing Method and System for Analyzing a Multi-Channel Image Obtained from a Biological Tissue Sample Being Stained by Multiple Stains(複数のステインにより染色される生物学的組織サンプルから得られたマルチチャネル画像を分析する画像処理方法およびシステム)」という名称のPCT/EP/2015/062015に開示されており、そのすべての開示内容が参照により本明細書に組み込まれる。 [00072] This identification may be enabled by image analysis operations such as edge detection. A tissue region mask may be used to remove non-tissue background noise in an image (eg, non-tissue regions). In some embodiments, the tissue region mask is generated such that pixels whose luminance is above a given threshold are set to 1 and pixels below the threshold are set to 0 to generate the tissue region mask. , computing the luminance of the low-resolution input image, generating the luminance image, applying a standard deviation filter to the luminance image, generating a filtered luminance image, and applying a threshold to the filtered luminance image. Including (but not limited to) these actions. For additional information and examples regarding the generation of tissue region masks, see "An Image Processing Method and System for Analyzing a Multi-Channel Image Obtained from a Biological Tissue Sample Being Stained by Multi-Stains." PCT/EP/2015/062015 titled PCT/EP/2015/062015 entitled Image Processing Methods and Systems for Analyzing Multichannel Images Obtained from Targeted Tissue Samples, the entire disclosure of which is incorporated herein by reference. .
[00073]いくつかの実施形態においては、画像または画像データを取得すべき生物学的サンプルの部分(たとえば、線維芽細胞の濃度が高い関心領域)を選択するのに、関心領域識別モジュールが用いられるようになっていてもよい。図13は、いくつかの実施形態に係る、領域選択のステップを示したフローチャートである。ステップ420において、領域選択モジュールは、識別された関心領域または視野を受信する。いくつかの実施形態において、関心領域は、本開示のシステムまたは本開示のシステムに対して通信可能に結合された別のシステムのユーザにより識別される。あるいは、他の実施形態において、領域選択モジュールは、ストレージ/メモリから関心領域の箇所または識別情報を読み出す。いくつかの実施形態において、ステップ430に示されるように、領域選択モジュールは、たとえばPCT/EP2015/062015に記載の方法によって、視野(FOV)または関心領域(ROI)を自動的に生成するが、そのすべての開示内容が参照により本明細書に組み込まれる。いくつかの実施形態において、関心領域は、画像中または画像のいくつかの所定基準または特質に基づいてシステムにより自動的に決定される(たとえば、3つ以上のステインにより染色された生物学的サンプルの場合は、ステインを2つだけ含む画像のエリアを識別する)。ステップ440において、領域選択モジュールは、ROIを出力する。
[00073] In some embodiments, a region of interest identification module is used to select a portion of a biological sample for which an image or image data is to be acquired (eg, a region of interest with a high concentration of fibroblasts). It may be possible to FIG. 13 is a flowchart illustrating steps for region selection, according to some embodiments. At
[00074]画像分析モジュール
[00075]いくつかの実施形態においては、入力として受信された画像内の特徴から、特定の測定基準(たとえば、FAP陽性面積、FAP陽性強度)が導出される(ステップ300)(図3参照)。導出測定基準は、本明細書において生成される副領域と相関されるようになっていてもよく(ステップ320、330、および340)、また、測定基準(または、その平均、標準偏差等)および副領域の場所が一体としてデータベースに格納され(ステップ350)、後々の読み出しおよび/または下流処理が行われるようになっていてもよい。本明細書に記載の手順およびアルゴリズムは、線維芽細胞および/もしくはマクロファージからの測定基準の導出を含めた、さまざまな種類の細胞もしくは細胞核からの測定基準の導出、および/もしくはさまざまな種類の細胞もしくは細胞核の分類を行うように構成されていてもよい。
[00074] Image Analysis Module
[00075] In some embodiments, certain metrics (eg, FAP-positive area, FAP-positive intensity) are derived from features in the image received as input (step 300) (see Figure 3). . The derived metric may be adapted to be correlated with the subregions generated herein (
[00076]いくつかの実施形態において、測定基準は、入力画像内の核の検出ならびに/または検出核(検出核の周りの画像パッチ等)および/もしくは細胞膜(当然のことながら、入力画像内で利用されるバイオマーカによって決まる)からの特徴の抽出によって導出される。他の実施形態においては、細胞膜染色、細胞質染色、および/または(たとえば、膜染色エリアと非膜染色エリアとを識別するための)強調染色を分析することによって、測定基準が導出される。本明細書において、用語「細胞質染色(cytoplasmic staining)」は、細胞の細胞質領域の形態学的特質を有するパターンに配置されたピクセル群を表す。本明細書において、用語「膜染色(membrane staining)」は、細胞膜の形態学的特質を有するパターンに配置されたピクセル群を表す。本明細書において、用語「強調染色(punctate staining)」は、細胞の膜エリアに散らばったスポット/ドットとして現れる局在化した染色強度が格納されたピクセル群を表す。当業者には当然のことながら、細胞の核、細胞質、および膜は、特質が異なるため、異なる染色の組織サンプルが異なる生物学的特徴を明らかにし得る。実際、当業者には当然のことながら、細胞表面の特定の受容体は、膜または細胞質に対して局在化した染色パターンを有し得る。このため、「膜」染色パターンは、「細胞質」染色パターンとは分析的に異なる。同様に、「細胞質」染色パターンおよび「核」染色パターンも分析的に異なる。たとえば、間質細胞はFAPにより強く染色され得るが、腫瘍上皮細胞はEpCAMにより強く染色され、一方、サイトケラチンはPanCKにより染色され得る。このため、画像分析中は、異なるステインの利用によって、異なる細胞腫が差別化および区別されるようになっていてもよく、また、異なる測定基準が導出されるようになっていてもよい。 [00076] In some embodiments, the metric is the detection of nuclei in the input image and/or the detected nuclei (such as image patches around the detected nuclei) and/or cell membranes (of course the (depending on the biomarkers utilized). In other embodiments, the metric is derived by analyzing plasma membrane staining, cytoplasmic staining, and/or enhanced staining (eg, to distinguish between membranous and non-membrane stained areas). As used herein, the term "cytoplasmic staining" refers to groups of pixels arranged in a pattern having morphological characteristics of the cytoplasmic region of a cell. As used herein, the term "membrane staining" refers to groups of pixels arranged in a pattern having the morphological characteristics of a cell membrane. As used herein, the term "punctate staining" refers to groups of pixels that store localized staining intensities that appear as scattered spots/dots on the membrane area of the cell. Those skilled in the art will appreciate that the nucleus, cytoplasm, and membrane of cells have different characteristics, so that differently stained tissue samples may reveal different biological characteristics. Indeed, it will be appreciated by those skilled in the art that certain receptors on the cell surface may have localized staining patterns to the membrane or cytoplasm. For this reason, the 'membrane' staining pattern analytically differs from the 'cytoplasmic' staining pattern. Similarly, 'cytoplasmic' and 'nuclear' staining patterns are analytically different. For example, stromal cells can be stained strongly with FAP, while tumor epithelial cells can be stained strongly with EpCAM, while cytokeratins can be stained with PanCK. Thus, during image analysis, the use of different stains may allow different cell tumors to be differentiated and differentiated, and different metrics may be derived.
[00077]1つまたは複数のステインを有する生物学的サンプルの画像中の核、細胞膜、および細胞質の識別および/またはスコアリングを行う方法については、米国特許第7,760,927号(「’927特許」)に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。たとえば、’927特許は、バイオマーカにより染色された生物組織の入力画像中の複数のピクセルを同時に識別する自動化方法であって、細胞質および細胞膜ピクセルの同時識別のための入力画像の前景における複数のピクセルの第1の色平面を考慮するステップであり、入力画像が、当該入力画像の背景部分を除去するとともに当該入力画像のカウンタ染色成分を除去するように処理済みである、ステップと、デジタル画像の前景において細胞質と細胞膜のピクセル間の閾値レベルを決定するステップと、である場合に、前景からの選択ピクセルおよびその8つの隣接ピクセルを用いて、同時に、選択ピクセルがデジタル画像における細胞質ピクセルであるか、細胞膜ピクセルであるか、または遷移ピクセルであるかを、決定閾値レベルを用いて決定するステップと、を含む、方法を記載する。さらに、’927特許は、選択ピクセルおよびその8つの隣接ピクセルを用いて同時に決定するステップが、選択ピクセルとその8つの隣接ピクセルとの積の平方根を決定することと、積を決定閾値レベルと比較することと、比較に基づいて、細胞膜用の第1のカウンタ、細胞質用の第2のカウンタ、または遷移ピクセル用の第3のカウンタをインクリメントすることと、第1のカウンタ、第2のカウンタ、または第3のカウンタが所定の最大値を超えているかを判定し、超えている場合は、所定の最大値を超えたカウンタに基づいて、選択ピクセルを分類することと、を含む旨を記載する。核のスコアリングのほか、’927特許は、それぞれ演算細胞質ピクセル体積指標、細胞質ピクセル中央値強度、膜ピクセル体積、および膜ピクセル中央値強度等に基づく細胞質および膜のスコアリングに関する例を提供する。 [00077] U.S. Patent No. 7,760,927 ("' '927 patent), the entire disclosure of which is incorporated herein by reference. For example, the '927 patent describes an automated method for simultaneously identifying multiple pixels in an input image of biomarker-stained biological tissue, comprising multiple pixels in the foreground of the input image for simultaneous identification of cytoplasmic and membrane pixels. considering a first color plane of pixels, wherein the input image has been processed to remove a background portion of the input image and to remove a counter-staining component of the input image; and a digital image. and using a selected pixel from the foreground and its eight neighboring pixels, at the same time the selected pixel is a cytoplasmic pixel in the digital image. and determining whether it is a cell membrane pixel or a transition pixel using a decision threshold level. Further, the '927 patent states that the step of determining simultaneously using the selected pixel and its eight neighboring pixels includes determining the square root of the product of the selected pixel and its eight neighboring pixels; comparing the product to a decision threshold level; incrementing a first counter for the cell membrane, a second counter for the cytoplasm, or a third counter for the transition pixel based on the comparison; the first counter, the second counter; or determining whether the third counter exceeds a predetermined maximum value, and if so, classifying the selected pixel based on the counter exceeding the predetermined maximum value. . In addition to nuclear scoring, the '927 patent provides examples for cytoplasmic and membrane scoring based on computational cytoplasmic pixel volume index, cytoplasmic pixel median intensity, membrane pixel volume, and membrane pixel median intensity, etc., respectively.
[00078]膜、核、および他の関心細胞特徴の識別および/またはスコアリングを行う別の方法がPCT公開WO2017/037180(「’180公開」)に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。さらに、’180公開は、膜染色の領域が細胞質染色および/または強調染色と混合された場合の生物学的サンプル中の関心検体の膜染色を定量化する方法を記載する。これを実現するため、’180公開は、(A)検体染色パターンに基づいて、組織または細胞学的サンプルのデジタル画像を複数の異なる領域にセグメント化するステップであり、複数の領域が、少なくとも1つの複合染色領域すなわち少なくとも1つの第2の生物学的区画中の検体陽性染色と混合された第1の生物学的区画中の検体陽性染色を有する画像の領域を含み、前記第1の生物学的区画および前記少なくとも1つの第2の生物学的区画が、分析的に異なる、ステップと、(B)ステップ(A)とは別個に、候補生物学的区画すなわち少なくとも第1の生物学的区画に対応するデジタル画像中のピクセルクラスタを識別するステップと、(C)ステップ(A)および(B)とは別個に、検体染色に対応するピクセルクラスタを高強度ビン、低強度ビン、および背景強度ビンにセグメント化することによって検体強度マップを生成するステップと、(D)複合染色領域内の候補生物学的区画を検体強度マップからの適当なビンと整合させて、各複合染色領域の分析的に関連する部分を識別するステップと、(E)複合染色領域の分析的に関連する部分における検体染色を定量化するステップと、によって、分析的に異なる生物学的区画の検体染色と染色が混合された領域(たとえば、(i)瀰漫性膜染色が細胞質染色と混合された領域または(ii)瀰漫性膜染色が強調染色と混合された領域)における生物学的区画の検体染色を定量化する方法を記載する。そして、区画または染色強度定量化のエリアが決定され得るように、任意の識別区画中のピクセルを定量化可能である。また、’180公開は、膜固有発現レベルのスコアリングを記載する。 [00078] Another method for identifying and/or scoring membrane, nuclear, and other cell features of interest is described in PCT Publication WO2017/037180 ("'180 Publication"), the entire disclosure of which is hereby incorporated by reference. incorporated herein by reference. Additionally, the '180 publication describes a method for quantifying membrane staining of a specimen of interest in a biological sample when areas of membrane staining are mixed with cytoplasmic staining and/or enhanced staining. To accomplish this, the '180 publication describes (A) segmenting a digital image of a tissue or cytological sample into multiple distinct regions based on specimen staining patterns, wherein the multiple regions comprise at least one two composite staining regions, i.e. regions of the image having analyte positive staining in the first biological compartment mixed with analyte positive staining in at least one second biological compartment; (B) separately from step (A), a candidate biological compartment, i.e. at least the first biological compartment; and (C) separately from steps (A) and (B), categorizing pixel clusters corresponding to analyte staining into high-intensity bins, low-intensity bins, and background-intensity bins. generating an analyte intensity map by segmenting into bins; and (E) quantifying analyte staining in analytically relevant portions of the composite staining region to mix analyte staining and staining of analytically distinct biological compartments. quantify the analyte staining of the biological compartment in the marked areas (e.g., areas where (i) diffuse membrane staining was mixed with cytoplasmic staining or (ii) areas where diffuse membrane staining was mixed with enhanced staining) Describe the method. Pixels in any identified compartment can then be quantified so that the compartment or area of staining intensity quantification can be determined. The '180 publication also describes scoring membrane-specific expression levels.
[00079]いくつかの実施形態において、スコアリングは、分類された核に関して実行され、特定のバイオマーカの百分率正値性測定基準またはHスコア測定基準が得られる。核を識別することによって、対応する細胞が識別され得る。他の実施形態においては、関連する核それぞれを周囲の染色膜と関連付けることによって、細胞がスコアリングされる。核の周りの染色膜の有無に基づいて、たとえば非染色(核の周りに染色膜が見つからない)、部分染色(細胞の核の一部が染色膜に囲まれる)、または完全染色(核の全体が染色膜に囲まれる)として細胞が分類されるようになっていてもよい。 [00079] In some embodiments, scoring is performed on the classified nuclei to obtain a percentage positivity metric or H-score metric for a particular biomarker. By identifying the nuclei, the corresponding cells can be identified. In other embodiments, cells are scored by associating each relevant nucleus with the surrounding stained membrane. Based on the presence or absence of a staining membrane around the nucleus, for example, no staining (no staining membrane found around the nucleus), partial staining (a portion of the cell's nucleus is surrounded by a staining membrane), or complete staining (nuclear Entirely surrounded by a staining membrane) cells may be classified.
[00080]いくつかの実施形態においては、最初に候補核を識別した後、腫瘍核と非腫瘍核とを自動的に区別することによって、腫瘍核が自動的に識別される。当技術分野においては、組織の画像中の候補核を識別する多くの方法が知られている。たとえば、放射対称に基づく方法であって、本明細書に記載の通り、Ruifrok et al.により記述されたカラーデコンボリューションを用いて取得されたヘマトキシリン画像チャネルまたはバイオマーカ画像チャネル等に対して、同じく本明細書に記載の通り、Parvin et al.の放射対称に基づく方法を適用することにより自動候補核検出が実行され得る。例示的な一実施形態においては、本発明の譲受人に譲渡された同時係属特許出願WO2014/140085A1に記載の通り、放射対称に基づく核検出動作が使用され、そのすべての内容が参照により本明細書に組み込まれる。他の方法が米国特許出願公開第2017/0140246号に記載されており、その開示内容が参照により本明細書に組み込まれる。 [00080] In some embodiments, tumor nuclei are automatically identified by first identifying candidate nuclei and then automatically distinguishing between tumor nuclei and non-tumor nuclei. Many methods are known in the art for identifying candidate nuclei in images of tissue. For example, methods based on radial symmetry, as described in Ruifrok et al. As also described herein, for hematoxylin image channels or biomarker image channels acquired using color deconvolution as described by Parvin et al. Automatic candidate nucleus detection can be performed by applying a method based on the radial symmetry of In one exemplary embodiment, nuclear detection operation based on radial symmetry is used, as described in co-pending patent application WO2014/140085A1, assigned to the assignee of the present invention, the entire contents of which are incorporated herein by reference. incorporated into the book. Other methods are described in US Patent Application Publication No. 2017/0140246, the disclosure of which is incorporated herein by reference.
[00081]候補核は、識別された後、腫瘍核を他の候補核から区別するように別途分析される。他の候補核は、(たとえば、リンパ球核および間質核の識別によって)さらに分類されるようになっていてもよい。いくつかの実施形態においては、学習済みの教師あり分類器が腫瘍核の識別に適用される。たとえば、学習済み教師あり分類器は、核の特徴に関するトレーニングによって腫瘍核を識別した後、テスト画像において候補核を腫瘍核または非腫瘍核として分類するように適用される。任意選択として、学習済み教師あり分類器は、別途トレーニングによって、リンパ球核および間質核等、異なるクラスの非腫瘍核を区別するようにしてもよい。いくつかの実施形態において、腫瘍核の識別に用いられる学習済み教師あり分類器は、ランダムフォレスト分類器である。たとえば、ランダムフォレスト分類器のトレーニングは、(i)腫瘍および非腫瘍核のトレーニングセットを生成し、(ii)それぞれの核の特徴を抽出し、(iii)抽出した特徴に基づいて腫瘍核と非腫瘍核とを区別するようにランダムフォレスト分類器をトレーニングすることによって行われ得る。そして、トレーニングしたランダムフォレスト分類器の適用により、テスト画像において、核を腫瘍核および非腫瘍核に分類するようにしてもよい。任意選択として、ランダムフォレスト分類器は、別途トレーニングによって、リンパ球核および間質核等、異なるクラスの非腫瘍核を区別するようにしてもよい。 [00081] After candidate nuclei are identified, they are analyzed separately to distinguish tumor nuclei from other candidate nuclei. Other candidate nuclei may be further classified (eg, by distinguishing between lymphocyte and interstitial nuclei). In some embodiments, a trained supervised classifier is applied to identify tumor nuclei. For example, a trained supervised classifier is applied to classify candidate nuclei as tumor nuclei or non-tumor nuclei in test images after identifying tumor nuclei by training on nuclear features. Optionally, the trained supervised classifier may be trained separately to distinguish different classes of non-tumor nuclei, such as lymphocyte nuclei and stromal nuclei. In some embodiments, the trained supervised classifier used to identify tumor nuclei is a random forest classifier. For example, the training of a random forest classifier consists of (i) generating a training set of tumor and non-tumor nuclei, (ii) extracting features for each nucleus, and (iii) based on the extracted features, It can be done by training a random forest classifier to distinguish between tumor nuclei. Application of the trained random forest classifier may then classify the nuclei into tumor and non-tumor nuclei in the test image. Optionally, the random forest classifier may be separately trained to distinguish different classes of non-tumor nuclei, such as lymphocyte nuclei and stromal nuclei.
[00082]いくつかの実施形態において、入力として受信された画像は、核中心(種子)の検出および/または核のセグメント化を行うように処理される。たとえば、当業者に共通して既知の技術を用いた放射対称投票に基づいて核中心を検出する命令が与えられるようになっていてもよい(Parvin,Bahram,et al.「Iterative voting for inference of structural saliency and characterization of subcellular events(構造的特徴の推定および細胞内事象の特性化のための反復投票)」,Image Processing,IEEE Transactions on 16.3(2007):615-623(すべての開示内容が参照により本明細書に組み込まれる)参照)。いくつかの実施形態においては、放射対称を用いた核の検出によって核の中心が検出された後、細胞中心の周りのステインの強度に基づいて核が分類される。たとえば、画像内で画像の大きさが演算されるようになっていてもよく、選択領域内の大きさの合計を加算することによって、各ピクセルにおける1つまたは複数の投票が蓄積される。核の実際の場所を表す領域中の局所中心を見つけるのに、平均シフトクラスタリングが用いられるようになっていてもよい。放射対称投票に基づく核検出は、カラー画像強度データに対して実行され、核が、サイズおよび偏心度が変動する楕円形状の斑点であるという推測的な領域知識を明示的に使用する。これを実現するため、入力画像中の色強度と併せて、画像勾配情報も放射対称投票に使用され、適応セグメント化プロセスとの組み合わせによって、細胞核の正確な検出および位置特定を行う。本明細書において、「勾配(gradient)」は、たとえば特定のピクセルを囲むピクセル集合の強度値勾配を考慮することにより前記特定ピクセルに対して計算されたピクセルの強度勾配である。各勾配は、デジタル画像の2つの直交エッジによってx軸およびy軸が規定される座標系に対して、特定の「配向」を有していてもよい。たとえば、核種子検出には、細胞核の内側に存在すると仮定され、細胞核の位置特定の開始点として機能する点として種子を規定することを伴う。第1のステップでは、放射対称に基づく高堅牢な手法を用いて細胞核に類似する楕円形状の斑点、構造を検出することにより、各細胞核と関連付けられた種子点を検出する。放射対称手法は、カーネルベースの投票手順を用いることにより、勾配画像上で動作する。投票カーネルを通じて投票を蓄積した各ピクセルを処理することによって、投票応答行列が生成される。カーネルは、当該特定ピクセルにおいて演算された勾配方向、最大および最小核サイズの予想範囲、ならびに投票カーネル角度(通常、[π/4,π/8]の範囲)に基づく。結果としての投票空間において、所定の閾値よりも高い投票値を有する極大の場所は、種子点として保存される。無関係な種子については、後続のセグメント化または分類プロセスにおいて破棄されるようになっていてもよい。 [00082] In some embodiments, images received as input are processed to perform nuclear center (seed) detection and/or nucleus segmentation. For example, one may be instructed to detect the nuclear center based on radially symmetric voting using techniques commonly known to those skilled in the art (Parvin, Bahram, et al. "Iterative voting for influence of Structural saliency and characterization of subcellular events,” Image Processing, IEEE Transactions on 16.3 (2007): 615-623 (all disclosures are incorporated herein by reference) see). In some embodiments, after the center of the nucleus is detected by detecting nuclei using radial symmetry, the nuclei are classified based on the intensity of the stain around the cell center. For example, image dimensions may be computed within the image, and one or more votes at each pixel are accumulated by adding the sum of the dimensions within the selected region. Mean shift clustering may be used to find the local centers in the regions representing the actual locations of the nuclei. Nucleus detection based on radially symmetric voting is performed on color image intensity data and explicitly uses a priori domain knowledge that nuclei are oval-shaped patches of varying size and eccentricity. To achieve this, along with the color intensity in the input image, image gradient information is also used for radial symmetry voting, and in combination with the adaptive segmentation process, provides accurate detection and localization of cell nuclei. As used herein, a "gradient" is a pixel intensity gradient calculated for a particular pixel, for example by considering the intensity value gradients of a set of pixels surrounding the particular pixel. Each gradient may have a particular "orientation" with respect to a coordinate system whose x-axis and y-axis are defined by two orthogonal edges of the digital image. For example, nuclear seed detection involves defining the seed as a point that is assumed to reside inside the cell nucleus and serves as the starting point for localizing the cell nucleus. In the first step, the seed points associated with each cell nucleus are detected by using a robust method based on radial symmetry to detect oval-shaped spots, structures resembling cell nuclei. Radial symmetric methods operate on gradient images by using a kernel-based voting procedure. A vote response matrix is generated by processing each pixel that has accumulated votes through a voting kernel. The kernel is based on the gradient direction computed at that particular pixel, the expected range of maximum and minimum kernel sizes, and the voting kernel angle (typically in the range [π/4, π/8]). In the resulting vote space, local maxima with vote values higher than a predetermined threshold are saved as seed points. Irrelevant seeds may be discarded in subsequent segmentation or classification processes.
[00083]当業者に既知の他の技術を用いて核が識別されるようになっていてもよい。たとえば、H&EまたはIHC画像の一方の特定の画像チャネルから画像の大きさが演算されるようになっていてもよく、また、特定された大きさの周りの各ピクセルには、当該ピクセルの周りの領域内の大きさの合計に基づく多くの投票が割り当てられていてもよい。あるいは、平均シフトクラスタリング演算の実行によって、核の実際の場所を表す投票画像内の局所中心を見つけるようにしてもよい。他の実施形態においては、核セグメント化の使用により、形態学的演算および局所閾値化を介した当該核の現在既知の中心に基づいて、核全体をセグメント化するようにしてもよい。さらに他の実施形態においては、モデルベースのセグメント化の利用によって、核を検出するようにしてもよい(すなわち、トレーニングデータセットから核の形状モデルを学習し、これを先行知識として用いることにより、テスト画像において核をセグメント化する)。 [00083] Nuclei may be identified using other techniques known to those skilled in the art. For example, the image dimensions may be computed from a particular image channel of one of the H&E or IHC images, and each pixel around the specified dimensions may include the A number of votes may be assigned based on the total size within the region. Alternatively, a mean shift clustering operation may be performed to find local centers within the voting image that represent the actual locations of the nuclei. In other embodiments, the use of nucleus segmentation may segment the entire nucleus based on the currently known center of that nucleus via morphological operations and local thresholding. In still other embodiments, the kernels may be detected through the use of model-based segmentation (i.e., by learning a shape model of the kernels from the training data set and using this as prior knowledge, segment the nuclei in the test image).
[00084]いくつかの実施形態において、核はその後、それぞれについて個別に演算された閾値を用いてセグメント化される。たとえば、核領域のピクセル強度が変動すると考えられることから、識別された核の周りの領域におけるセグメント化には、大津法が用いられるようになっていてもよい。当業者には当然のことながら、大津法は、クラス内分散を最小限に抑えることにより最適な閾値を決定するのに用いられるが、当業者には既知である。より具体的に、大津法は、クラスタリングベースの画像閾値化すなわちグレーレベル画像の2値画像への減縮を自動的に実行するのに用いられる。アルゴリズムは、画像が二峰性ヒストグラムに従う2クラスのピクセル(前景ピクセルおよび背景ピクセル)を含むものと仮定する。そして、(ペアワイズ2乗距離の合計が一定であるため)2つのクラスの結合散布(クラス内分散)が最小または等価になるように分離する最適な閾値を計算することによって、それぞれのクラス内分散が最大となる。 [00084] In some embodiments, the nuclei are then segmented using separately computed thresholds for each. For example, Otsu's method may be used for segmentation in the region around the identified nucleus, since pixel intensities in the nucleus region are expected to vary. Those skilled in the art will appreciate that Otsu's method, which is used to determine the optimal threshold by minimizing the within-class variance, is known to those skilled in the art. More specifically, Otsu's method is used to automatically perform clustering-based image thresholding, ie, reduction of gray-level images to binary images. The algorithm assumes that the image contains two classes of pixels (foreground and background pixels) following a bimodal histogram. Then, by calculating the optimal threshold that separates the two classes such that the joint scatter (within-class variance) of the two classes is minimized or equal (because the sum of the pairwise squared distances is constant), each within-class variance is maximum.
[00085]いくつかの実施形態において、これらのシステムおよび方法は、非腫瘍細胞の核を識別する画像中の識別核のスペクトルおよび/または形状の特徴を自動的に分析することを含む。たとえば、第1のステップにおいては、第1のデジタル画像中で斑点が識別されるようになっていてもよい。本明細書において、「斑点(blob)」としては、たとえば一部の特性(たとえば、強度またはグレー値)が一定であるか、または、所定の値範囲内で変動するデジタル画像の領域が可能である。ある意味では、斑点中のすべてのピクセルが互いに類似すると考えられる。たとえば、デジタル画像上の位置の関数の導関数に基づく差分法および極値に基づく方法を用いて斑点が識別されるようになっていてもよい。核斑点は、おそらくは第1のステインにより染色された核によって生成されたことをピクセルおよび/または外形状が示す斑点である。たとえば、斑点の放射対称の評価によって、当該斑点を核斑点として識別すべきか、その他任意の構造(たとえば、染色アーチファクト)として識別すべきかを判定することも可能である。たとえば、斑点が非常に長い形状であり、放射対称ではない場合、前記斑点は、核斑点としてではなく、染色アーチファクトとして識別されるようになっていてもよい。本実施形態によれば、「核斑点」と識別された斑点は、候補核として識別され、前記核斑点が核を表すかを判定するようにさらに分析可能なピクセル集合を表していてもよい。いくつかの実施形態においては、任意の種類の核斑点が「識別核」として直接使用される。いくつかの実施形態においては、識別核または核斑点にフィルタリング演算が適用されて、バイオマーカ陽性腫瘍細胞に属さない核を識別するとともに、識別済みの核一覧から前記識別非腫瘍核を除去するか、または、最初から前記核を識別核一覧に追加しない。たとえば、識別核斑点の付加的なスペクトルおよび/または形状の特徴の分析によって、核または核斑点が腫瘍細胞の核であるか否かを判定するようにしてもよい。たとえば、リンパ球の核は、他の組織細胞(たとえば、肺細胞)の核よりも大きい。腫瘍細胞が肺組織に由来する場合は、正常な肺の細胞核の平均的なサイズまたは直径よりもはるかに大きな最小サイズまたは直径のすべての核斑点を識別することによって、リンパ球の核が識別される。リンパ球の核に関する識別核斑点は、識別済みの核の集合から除去(すなわち、「フィルタリング除去」)されるようになっていてもよい。非腫瘍細胞の核のフィルタリング除去によって、この方法の精度は向上し得る。バイオマーカによれば、ある程度までは非腫瘍細胞もバイオマーカを表し得るため、腫瘍細胞に由来しない第1のデジタル画像において強度信号を生成可能である。腫瘍細胞に属さない核を識別して、識別済みの核全体からフィルタリング除去することにより、バイオマーカ陽性腫瘍細胞を識別する精度が向上し得る。上記および他の方法が米国特許出願公開第2017/0103521号に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。いくつかの実施形態においては、種子が検出されたら、局所適応閾値化方法が用いられるようになっていてもよく、検出中心周りの斑点が生成される。いくつかの実施形態においては、他の方法が具現化されるようになっていてもよく、たとえば、マーカベースの分水嶺アルゴリズムの使用によって、検出核中心周りの核斑点を識別することも可能である。上記および他の方法が同時係属出願PCT/EP2016/051906(WO2016/120442として公開)に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。 [00085] In some embodiments, these systems and methods include automatically analyzing spectral and/or shape features of identified nuclei in images that identify nuclei of non-tumor cells. For example, in a first step, a speckle may be identified in the first digital image. As used herein, a "blob" can be, for example, a region of a digital image in which some characteristic (e.g., intensity or gray value) is constant or varies within a given range of values. be. In a sense, all pixels in a speckle are considered similar to each other. For example, speckles may be identified using a derivative-based method and an extremum-based method of a function of position on the digital image. A nuclear speck is a speck whose pixel and/or outline indicates that it was probably produced by a nucleus stained by the first stain. For example, evaluation of the radial symmetry of a speckle can determine whether the speckle should be identified as a nuclear speckle or any other structure (eg staining artifact). For example, if the speckle has a very long shape and is not radially symmetrical, the speckle may be identified as a staining artifact rather than as a nuclear speckle. According to this embodiment, a speckle identified as a "nuclear speckle" may represent a set of pixels that may be identified as a candidate nucleus and further analyzed to determine if the nuclear speckle represents a nucleus. In some embodiments, any kind of nuclear speck is used directly as a "identifying nucleus." In some embodiments, a filtering operation is applied to the identified nuclei or nuclear speckles to identify nuclei that do not belong to biomarker-positive tumor cells and remove said identified non-tumor nuclei from the list of identified nuclei , or do not add the nucleus to the list of identified nuclei from the beginning. For example, analysis of additional spectral and/or shape features of the discriminating nuclear speck may determine whether the nucleus or nuclear speck is the nucleus of a tumor cell. For example, the nuclei of lymphocytes are larger than those of other tissue cells (eg lung cells). If the tumor cells are derived from lung tissue, lymphocyte nuclei are identified by identifying all nuclear patches of minimal size or diameter that are much larger than the average size or diameter of normal lung cell nuclei. be. Identified nuclear patches for lymphocyte nuclei may be removed (ie, "filtered out") from the set of identified nuclei. Filtering out the nuclei of non-tumor cells may improve the accuracy of this method. Biomarkers allow the generation of intensity signals in the first digital image that are not derived from tumor cells, since non-tumor cells may also represent biomarkers to some extent. By identifying nuclei that do not belong to tumor cells and filtering out all identified nuclei, the accuracy of identifying biomarker-positive tumor cells may be improved. These and other methods are described in US Patent Application Publication No. 2017/0103521, the entire disclosure of which is incorporated herein by reference. In some embodiments, once a seed is detected, a locally adaptive thresholding method may be used to generate a speckle around the detection center. In some embodiments, other methods may be implemented, for example, the use of marker-based watershed algorithms may identify nuclear speckles around detected nuclear centers. . These and other methods are described in co-pending application PCT/EP2016/051906 (published as WO2016/120442), the entire disclosure of which is incorporated herein by reference.
[00086]このシステムは、少なくとも1つの画像特質測定基準および少なくとも1つの形態測定基準を使用して、画像内の特徴が関心構造に対応するかを判定することができる(「特徴測定基準」と総称する)。画像特質測定基準(画像内の特徴から導出)としては、たとえば色、色バランス、強度等が挙げられる。形態測定基準(画像内の特徴から導出)としては、たとえば特徴サイズ、特徴色、特徴配向、特徴形状、特徴(たとえば、隣り合う特徴)間の関係または距離、別の解剖学的構造に対する特徴の関係または距離等が挙げられる。本明細書に記載の通り、分類器のトレーニングには、画像特質測定基準、形態測定基準、および他の測定基準を使用可能である。画像特徴から導出される測定基準の具体例が以下に示される。 [00086] The system can use at least one image attribute metric and at least one morphology metric to determine whether a feature in an image corresponds to a structure of interest (referred to as a "feature metric"). collectively). Image attribute metrics (derived from features in the image) include, for example, color, color balance, intensity, and the like. Morphological metrics (derived from features in the image) include, for example, feature size, feature color, feature orientation, feature shape, relationship or distance between features (e.g., adjacent features), feature relative to different anatomy Relation or distance etc. are mentioned. As described herein, image attribute metrics, morphological metrics, and other metrics can be used for classifier training. Specific examples of metrics derived from image features are given below.
[00087](A)形態特徴から導出される測定基準
[00088]本明細書において、「形態特徴(morphology feature)」は、たとえば核の形状または寸法を示す特徴である。任意特定の理論に縛られることを望むことなく、形態特徴は、細胞またはその核のサイズおよび形状に関する何らかの不可欠な情報を提供すると考えられる。たとえば、核斑点もしくは種子に含まれるピクセルまたは核斑点もしくは種子を囲むピクセルに対してさまざまな画像分析アルゴリズムを適用することにより、形態特徴が演算され得る。いくつかの実施形態において、形態特徴としては、面積、短軸および長軸の長さ、周長、半径、中実性等が挙げられる。
[00087] (A) Metrics derived from morphological features
[00088] As used herein, a "morphology feature" is a feature indicative of, for example, the shape or size of the nucleus. Without wishing to be bound by any particular theory, morphological features are believed to provide some essential information regarding the size and shape of a cell or its nucleus. For example, morphological features can be computed by applying various image analysis algorithms to pixels contained in or surrounding a nuclear spot or seed. In some embodiments, morphological features include area, minor and major axis lengths, perimeter, radius, solidity, and the like.
[00089](B)外観特徴から導出される測定基準
[00090]本明細書において、「外観特徴(appearance feature)」は、たとえば核の識別に用いられる核斑点もしくは種子に含まれるピクセルまたは核斑点もしくは種子を囲むピクセルのピクセル強度値を比較することにより特定の核に対して演算された特徴であって、比較されるピクセル強度が異なる画像チャネル(たとえば、背景チャネル、バイオマーカの染色のためのチャネル等)から導出される。いくつかの実施形態において、外観特徴から導出される測定基準は、異なる画像チャネルから演算されたピクセル強度および勾配の大きさの百分位数値(たとえば、10番目、50番目、および95番目の百分位数値)から演算される。たとえば、まず初めに、関心核を表す核斑点内の画像チャネル(たとえば、HTX、DAB、輝度という3つのチャネル)の複数のICそれぞれのピクセル値のX百分位数値(X=10、50、95)の数Pが識別される。外観特徴測定基準を演算することは、導出された測定基準が核領域の特性を表すほか、核の周りの膜領域も表し得るため、好都合と考えられる。
[00089] (B) Metrics Derived from Appearance Features
[00090] As used herein, an "appearance feature" is defined, for example, by comparing pixel intensity values of pixels contained in or surrounding a nuclear speckle or seed used to identify a nucleus. Features computed for a particular nucleus, where pixel intensities to be compared are derived from different image channels (eg, background channel, channel for biomarker staining, etc.). In some embodiments, metrics derived from appearance features are pixel intensities and gradient magnitude percentile values (e.g., 10th, 50th, and 95th percentiles) computed from different image channels. quantile value). For example, first, the X-percentile values (X=10, 50, 95) is identified. Computing an appearance feature metric is considered advantageous because the derived metric represents properties of the nucleus area, as well as the membrane area around the nucleus.
[00091](C)背景特徴から導出される測定基準
[00092]「背景特徴(background feature)」は、たとえば細胞質中の外観および/もしくはステイン有無を示す特徴ならびに当該背景特徴が画像から抽出された核を含む細胞の細胞膜特徴である。たとえば、核斑点または核を表す種子を識別し、識別さされた細胞集合に直接隣り合うピクセルエリア(たとえば、核斑点境界周りの20ピクセル(およそ9ミクロン)厚のリボン)を分析することにより、この核を有する細胞に直接隣り合うエリアと一体的に、当該細胞の細胞質および膜中の外観およびステイン有無を取り込むことによって、デジタル画像に示される核および対応する細胞の背景特徴および対応する測定基準が演算され得る。これらの測定基準は、核外観特徴に類似するが、それぞれの核境界周りのおよそ20ピクセル(およそ9ミクロン)厚のリボンにおいて演算されるため、識別核を有する細胞に直接隣り合うエリアと一体的に、当該細胞の細胞質および膜中の外観およびステイン有無が取り込まれる。任意特定の理論に縛られることを望むことなく、リボンサイズが選択されるのは、核の識別に有用な情報の提供に使用可能な核周りの十分な量の背景組織エリアを当該リボンが取り込むと考えられるためである。これらの特徴は、J.Kong,et al.「A comprehensive framework for classification of nuclei in digital microscopy imaging: An application to diffuse gliomas(デジタル顕微鏡撮像における核の分類のための包括的枠組み:瀰漫性グリオーマへの適用)」,ISBI,2011,pp.2128-2131に開示の特徴と類似しており、そのすべての開示内容が参照により本明細書に組み込まれる。これらの特徴は、周囲の組織が(H&E染色組織サンプル等における)間質であるか上皮であるかを判定するのに使用可能と考えられる。また、任意特定の理論に縛られることを望むことなく、これらの背景特徴は、組織サンプルが適当な膜染色剤により染色される場合に有用な膜染色パターンを取り込むと考えられる。
[00091] (C) Metrics Derived from Background Features
[00092] A "background feature" is, for example, a feature that indicates appearance and/or presence or absence of stain in the cytoplasm and a cell membrane feature of the cell containing the nucleus from which the background feature was extracted from the image. For example, by identifying seeds representing nuclear spots or nuclei and analyzing pixel areas directly adjacent to identified cell clusters (e.g., 20 pixel (approximately 9 micron) thick ribbons around nuclear spot borders), Background features and corresponding metrics of the nucleus and corresponding cells shown in the digital image by capturing the appearance and presence of stains in the cytoplasm and membrane of the cell, together with the area directly adjacent to this nucleated cell. can be computed. These metrics are similar to nuclear appearance features, but are computed on ribbons approximately 20 pixels (approximately 9 microns) thick around each nuclear boundary, and thus integral with areas immediately adjacent to cells with discriminating nuclei. , incorporates the appearance and presence of stains in the cytoplasm and membrane of the cell. Without wishing to be bound by any particular theory, the ribbon size is chosen so that it captures a sufficient amount of background tissue area around the nuclei that can be used to provide useful information for identifying nuclei. This is because it is considered that These features are described in J. Am. Kong, et al. "A comprehensive framework for classification of nuclei in digital microscopy imaging: Application to diffuse gliomas", ISBI, p. 2, p. 2128-2131, the entire disclosure of which is incorporated herein by reference. These features could be used to determine whether the surrounding tissue is stromal or epithelial (such as in H&E stained tissue samples). Also, without wishing to be bound by any particular theory, it is believed that these background features capture useful membrane staining patterns when the tissue sample is stained with a suitable membrane stain.
[00093](D)色から導出される測定基準
[00094]いくつかの実施形態において、色から導出される測定基準としては、色比率R/(R+G+B)または色主成分が挙げられる。他の実施形態において、色から導出される測定基準としては、各色の局所統計値(平均/中央値/分散/標準偏差)および/または局所画像ウィンドウ中の色強度相関が挙げられる。
[00093] (D) Metrics Derived from Color
[00094] In some embodiments, color-derived metrics include color ratios R/(R+G+B) or color principal components. In other embodiments, color-derived metrics include local statistics (mean/median/variance/standard deviation) for each color and/or color intensity correlations within the local image window.
[00095](E)強度特徴から導出される測定基準
[00096]ある特定の特性値を有する隣り合う細胞群は、病理組織学的スライド画像において表されるグレーの色付き細胞の黒白の色調間に設定される。色特徴の相関がサイズクラスのインスタンスを規定するため、これら色付き細胞の強度は、その周囲の暗色細胞のクラスタから影響を受ける細胞を決定する。質感特徴の例がPCT公開WO2016/075095に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。
[00095] (E) Metrics Derived from Intensity Features
[00096] Neighboring cell groups with certain characteristic values are set between the black and white tones of gray colored cells represented in the histopathological slide image. Since the correlation of color features defines an instance of a size class, the intensity of these colored cells determines which cells are affected by clusters of dark cells around them. Examples of texture features are described in PCT Publication WO2016/075095, the entire disclosure of which is incorporated herein by reference.
[00097](F)空間特徴
[00098]いくつかの実施形態において、空間特徴としては、細胞の局所密度、2つの隣り合う検出細胞間の平均距離、および/または細胞からセグメント化領域までの距離が挙げられる。
[00097] (F) Spatial features
[00098] In some embodiments, the spatial features include the local density of cells, the average distance between two neighboring detected cells, and/or the distance from the cell to the segmented region.
[00099](G)核特徴から導出される測定基準
[000100]当業者には当然のことながら、測定基準は、核特徴からも導出され得る。このような核特徴の演算は、Xing et al.「Robust Nucleus/Cell Detection and Segmentation in Digital Pathology and Microscopy Images: A Comprehensive Review(デジタル病理学・顕微鏡画像における堅牢な核/細胞検出およびセグメント化:包括的レビュー)」,IEEE Rev Biomed Eng 9,234-263,January 2016に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。当然のことながら、特徴の演算の基礎としては、当業者に既知の他の特徴も考慮および使用され得る。
[00099] (G) Metrics Derived from Nuclear Features
[000100] Those skilled in the art will appreciate that metrics can also be derived from nuclear features. Computation of such kernel features is described in Xing et al. "Robust Nucleus/Cell Detection and Segmentation in Digital Pathology and Microscopy Images: A Comprehensive Review",
[000101]特徴測定基準の導出後は、特徴の単独での使用またはトレーニングデータとの併用によって(たとえば、トレーニング中は、当業者に既知の手順に従って観察専門家により与えられるグランドトゥルース識別情報と併せて、例示的な細胞が提示される)、核または細胞を分類するようにしてもよい。いくつかの実施形態において、このシステムは、バイオマーカごとのトレーニングセットまたは基準スライドセットに少なくとも部分的に基づいてトレーニングされた分類器を具備し得る。当業者には当然のことながら、バイオマーカごとの分類器のトレーニングには、さまざまなスライドセットを使用可能である。したがって、単一のバイオマーカに対して、トレーニング後に単一の分類器が得られる。また、当業者には当然のことながら、異なるバイオマーカから得られる画像データ間にはばらつきがあるため、異なるバイオマーカごとに異なる分類器をトレーニングすることによって、未知のテストデータに対するより優れた性能を保証することができ、テストデータのバイオマーカ種が把握されることになる。トレーニング対象の分類器は、スライド解釈に対して、たとえば組織の種類、染色プロトコル、および他の関心特徴におけるトレーニングデータのばらつきの最良の取り扱い方法に少なくとも部分的に基づいて選択可能である。 [000101] After derivation of feature metrics, the features may be used alone or in combination with training data (e.g., during training, in conjunction with ground truth discriminators provided by observing experts according to procedures known to those skilled in the art). example cells are presented), nuclei or cells may be classified. In some embodiments, the system may comprise a classifier trained based at least in part on a training set or reference slide set for each biomarker. Those skilled in the art will appreciate that different slide sets can be used for training classifiers for each biomarker. Thus, for a single biomarker, we get a single classifier after training. It should also be appreciated by those skilled in the art that there is variability between image data obtained from different biomarkers, so training a different classifier for each different biomarker may result in better performance on unknown test data. can be guaranteed, and the biomarker species of the test data will be grasped. Classifiers to be trained can be selected based, at least in part, on how best to handle training data variability in, for example, tissue type, staining protocol, and other features of interest for slide interpretation.
[000102]いくつかの実施形態において、分類モジュールは、サポートベクターマシン(「SVM」)である。一般的に、SVMは分類技術であって、非線形の場合のカーネルにより非線形入力データセットが高次元線形特徴空間に変換される統計的学習理論に基づく。任意特定の理論に縛られることを望むことなく、サポートベクターマシンは、カーネル関数Kによって、2つの異なるクラスを表すトレーニングデータセットEを高次元空間に投影すると考えられる。この変換データ空間においては、クラス分離を最大化するようにクラスを分離する平坦線(識別超平面)が生成され得るように、非線形データが変換される。その後、Kによってテストデータが高次元空間に投影され、超平面に対する位置に基づいて分類される。カーネル関数Kは、データが高次元空間に投影される方法を規定する。 [000102] In some embodiments, the classification module is a support vector machine ("SVM"). In general, SVM is a classification technique, based on statistical learning theory, in which a nonlinear input dataset is transformed into a high-dimensional linear feature space by kernels in the nonlinear case. Without wishing to be bound by any particular theory, it is believed that a support vector machine projects a training data set E representing two different classes by a kernel function K into a high dimensional space. In this transformed data space, the nonlinear data is transformed so that flat lines (discrimination hyperplanes) can be generated that separate classes to maximize class separation. Then K projects the test data into a higher dimensional space and sorts them based on their position relative to the hyperplane. A kernel function K defines how the data is projected into a high-dimensional space.
[000103]他の実施形態においては、AdaBoostアルゴリズムを用いて分類が実行される。AdaBoostは、多くの弱い分類器を組み合わせて強い分類器を生成する適応アルゴリズムである。弱い分類器と考えられる個々の質感特徴Φj(j∈{1,・・・,K})それぞれについて確率密度関数を生成するのに、トレーニング段階において病理学者により識別された画像ピクセル(たとえば、特定のステインを有するピクセルまたは特定の組織種に属するピクセル)が用いられる。その後、ベイズの定理の使用によって、Φjごとに、弱い学習器を構成する尤度シーンLj=(Cj,1j∈{1,・・・,K})を生成する。これらがAdaBoostアルゴリズムにより結合されて、強い分類器Πj=ΣTi=1αjiljiとなるが、すべてのピクセルcj∈Cjについて、Πj(cj)は、ピクセルcjがクラスωTに属する結合尤度であり、αjiは、特徴Φiのトレーニング中に決定される重みであり、Tは、反復回数である。 [000103] In another embodiment, the classification is performed using the AdaBoost algorithm. AdaBoost is an adaptive algorithm that combines many weak classifiers to generate a strong classifier. To generate a probability density function for each individual texture feature Φj (j∈{1, . pixels with stains of 1000 psi or belonging to a specific tissue type) are used. Then, for each Φj, we generate a likelihood scene Lj=(Cj, 1jε{1, . These are combined by the AdaBoost algorithm to give a strong classifier Πj=ΣTi=1αjilji, where for every pixel cjεCj Πj(cj) is the joint likelihood that pixel cj belongs to class ωT and αji is , are the weights determined during training of the features Φi, and T is the number of iterations.
[000104]いくつかの実施形態においては、導出ステイン強度値、特定の核の数、または他の分類結果の使用によって、百分率正値性またはHスコア等のさまざまなマーカ発現スコア(本明細書においては、用語「発現スコア」と区別なく使用される)を決定するようにしてもよい(すなわち、分類特徴から、発現スコアが計算されるようになっていてもよい)。スコアリング方法は、本発明の譲受人に譲渡された2013年12月19日出願の同時係属特許出願WO2014/102130A1「Image analysis for breast cancer prognosis(乳がん予知のための画像分析)」および2014年3月12日出願のWO2014/140085A1「Tissue object-based machine learning system for automated scoring of digital whole slides(デジタル・ホール・スライドの自動採点のための組織物体に基づく機械学習システム)」においてさらに詳しく記載されており、それぞれのすべての内容が参照により本明細書に組み込まれる。たとえば、バイオマーカ陽性腫瘍細胞/バイオマーカ陽性非腫瘍細胞の数に少なくとも部分的に基づいて、スコア(たとえば、ホールスライドスコア)を決定可能である。いくつかの実施形態においては、検出された核斑点ごとに、平均斑点強度、色、および幾何学的特徴(検出核斑点の面積および形状等)が演算されるようになっていてもよく、核斑点が腫瘍核および非腫瘍細胞の核に分類される。識別核出力の数は、腫瘍核の計数により示される通り、FOVにおいて検出されたバイオマーカ陽性腫瘍細胞の総数に対応する。 [000104] In some embodiments, the use of derived stain intensity values, the number of specific nuclei, or other classification results allows various marker expression scores, such as percentage positivity or H-scores (herein is used interchangeably with the term "expression score") (ie, from the classification features, the expression score may be calculated). The scoring method is described in co-pending patent application WO2014/102130A1 "Image analysis for breast cancer prognosis," filed Dec. 19, 2013, assigned to the assignee of the present invention and 3/2014. Further described in WO 2014/140085 A1 "Tissue object-based machine learning system for automated scoring of digital whole slides" filed on May 12. and the entire contents of each are incorporated herein by reference. For example, a score (eg, whole slide score) can be determined based at least in part on the number of biomarker-positive tumor cells/biomarker-positive non-tumor cells. In some embodiments, for each detected nuclear speckle, the average speckle intensity, color, and geometric features (such as the area and shape of the detected nuclear speckle) may be computed. Plaques are classified into tumor nuclei and non-tumor cell nuclei. The number of discriminating nuclei output corresponds to the total number of biomarker-positive tumor cells detected in the FOV, as indicated by the tumor nuclei count.
[000105]いくつかの実施形態においては、この場合もFAPによる染色に関して、特徴測定基準が導出されるとともに、FAP陽性または陰性細胞(たとえば、ポジティブまたはネガティブ染色の間質細胞)の割合(たとえば、百分率正値性発現スコア)が明らかとなり得るように分類器がトレーニングされる。いくつかの実施形態においては、腫瘍細胞の10%以下の染色エリアにスコア0が割り当てられ、腫瘍細胞の11%超~25%以下のエリアに1が割り当てられ、腫瘍細胞の26%超~50%以下のエリアに2が割り当てられ、腫瘍細胞の51%超のエリアに3が割り当てられるようになっていてもよい。染色強度に関しては、ゼロ/弱染色(ネガティブ制御)にスコア0が割り当てられ、ネガティブ制御レベルよりも明らかに強い弱染色に1が割り当てられ、中程度の強度の染色に2が割り当てられ、強染色に3が割り当てられるようになっていてもよい。いくつかの実施形態においては、3以上の最終スコアがFAPの陽性発現を示すものと認識されていてもよい。 [000105] In some embodiments, again for staining with FAP, a characteristic metric is derived and the percentage of FAP positive or negative cells (e.g., positively or negatively stained stromal cells) (e.g., A classifier is trained such that a percentage positive expression score) can be revealed. In some embodiments, staining areas of 10% or less of the tumor cells are assigned a score of 0, areas of greater than 11% to 25% or less of the tumor cells are assigned a score of 1, and areas of greater than 26% to 50% of the tumor cells are assigned a score of 0. A 2 may be assigned to areas below 51% of tumor cells and a 3 to areas above 51% of tumor cells. Regarding staining intensity, zero/weak staining (negative control) was assigned a score of 0, weak staining clearly stronger than the negative control level was assigned a score of 1, moderately intense staining was assigned a score of 2, and strong staining was assigned. may be assigned 3. In some embodiments, a final score of 3 or higher may be recognized as indicative of positive expression of FAP.
[000106]セグメント化モジュール
[000107]中解像度分析手法では、生物学的に意味ある関心領域を取り込むように規定された入力画像内の副領域を生成するセグメント化アルゴリズムを採用する。画像分析モジュール205による入力画像からの測定基準の導出(ステップ310)の後には、セグメント化生成モジュール206の利用によって、入力画像を複数の副領域にセグメント化する(ステップ320)。
[000106] Segmentation module
[000107] The medium resolution analysis approach employs a segmentation algorithm that generates sub-regions within the input image defined to capture biologically meaningful regions of interest. After the
[000108]いくつかの実施形態においては、単一のチャネル画像(たとえば、分離FAP画像中の「紫色」チャネル)上でセグメント化が実行される。分離方法は、当業者に既知である(たとえば、Zimmermann「Spectral Imaging and Linear Unmixing in Light Microscopy(光学顕微鏡法におけるスペクトル撮像および線形分離)」,Adv Biochem Engin/Biotechnol(2005)95:245-265およびC.L.Lawson and R.J.Hanson「Solving least squares Problems(最小2乗問題の解法)」,Prentice Hall,1974,Chapter 23,p.161に線形分離が記載されており、そのすべての開示内容が参照により本明細書に組み込まれる)。本明細書においては、他の分離方法が開示される(Ruifok et. al.「Quantification of histochemical staining by color deconvolution(カラーデコンボリューションによる組織化学的染色の定量化)」,Anal Quant Cytol Histol.2001 Aug;23(4):291-9(すべての開示内容が参照により本明細書に組み込まれる)も参照)。 [000108] In some embodiments, segmentation is performed on a single channel image (eg, the "purple" channel in the separated FAP image). Separation methods are known to those skilled in the art (see, for example, Zimmermann "Spectral Imaging and Linear Unmixing in Light Microscopy", Adv Biochem Engin/Biotechnol (2005) 95:245-265 and C. L. Lawson and R. J. Hanson, "Solving least squares Problems", Prentice Hall, 1974, Chapter 23, p. the contents of which are incorporated herein by reference). Other separation methods are disclosed herein (Ruifok et. al. "Quantification of histochemical staining by color deconvolution", Anal Quant Cytol Histol. 2001 Aug. ;23(4):291-9, the entire disclosure of which is incorporated herein by reference).
[000109]いくつかの実施形態において、生成された副領域は、所定のサイズまたは画像処理アルゴリズム内に指定の範囲(たとえば、本明細書に記載の通り、SLICスーパーピクセル生成アルゴリズムのパラメータ)内のサイズを有する入力画像のエリアの情報を取り込む。 [000109] In some embodiments, the generated subregions are of a predetermined size or within a specified range within an image processing algorithm (e.g., parameters of the SLIC superpixel generation algorithm as described herein). Capture information of an area of the input image that has a size.
[000110]いくつかの実施形態において、入力画像は、所定の形状、サイズ、面積、および/または間隔を有する副領域にセグメント化される。たとえば、副領域(710)は、図7に示されるように、長円形、円形、正方形、長方形等であってもよい。いくつかの実施形態において、長円形、円形、正方形、または長方形の副領域は、50ピクセル~およそ100ピクセルの範囲のサイズを有していてもよいし、類似の特性または特質(たとえば、色、輝度、および/または質感)を有するピクセル群が選択されるように、その他何らかのサイズを有していてもよい。いくつかの実施形態において、副領域は、重なり合わず、サンプリンググリッドを介して生成されるようになっていてもよい。本明細書において、用語「サンプリンググリッド(sampling grid)」は、均一間隔で画像に重ね合わされ、最終的には画像内の点に重ならない点を位置特定するのに用いられる水平線および垂直線のネットワークに関する。いくつかの実施形態においては、水平線および垂直線により確立された任意数の隣り合う位置が画像セグメントの規定に用いられるようになっていてもよい。いくつかの実施形態において、副領域は、分析用の関連領域の代表サンプル(たとえば、不規則形状の細胞が支配的な特徴であるエリア)を取り込むように画像全体に分布する。 [000110] In some embodiments, an input image is segmented into subregions having a predetermined shape, size, area, and/or spacing. For example, subregions (710) may be oval, circular, square, rectangular, etc., as shown in FIG. In some embodiments, the oval, circular, square, or rectangular subregions may have sizes ranging from 50 pixels to approximately 100 pixels and have similar properties or characteristics (e.g., color, It may also have some other size so that groups of pixels with (brightness, and/or texture) are selected. In some embodiments, the subregions may be non-overlapping and generated via a sampling grid. As used herein, the term "sampling grid" refers to a network of horizontal and vertical lines that are uniformly spaced and superimposed on an image and are ultimately used to locate points that do not overlap points in the image. Regarding. In some embodiments, any number of adjacent positions established by horizontal and vertical lines may be used to define image segments. In some embodiments, subregions are distributed across the image to capture a representative sample of relevant regions for analysis (eg, areas where irregularly shaped cells are the dominant feature).
[000111]他の実施形態において、入力画像は、グローバル閾値化フィルタ、局所適応閾値化フィルタ、形態学的演算、および分水嶺変換等、一連のアルゴリズムを画像に適用することによってセグメント化される。これらのフィルタは、順次動作するようになっていてもよいし、当業者が必要と考える任意の順序で動作するようになっていてもよい。当然のことながら、所望の結果が実現されるまで、任意のフィルタが反復的に適用されるようになっていてもよい。いくつかの実施形態においては、入力画像への第1のフィルタの適用によって、(無染色または略無染色の組織サンプル中の領域に対応する)白色の画像領域の除去等、核を有する可能性が低い領域を除去する。いくつかの実施形態において、これは、グローバル閾値化フィルタを適用することによって実現される。いくつかの実施形態において、グローバル閾値化は、(たとえば、グレースケールチャネルに類似する)第1の主成分チャネル上で演算された中央値および/または標準偏差に基づく。グローバル閾値を求めることによって、核が存在しない可能性が高い無染色または略無染色の領域を表す任意の白色画像領域が破棄され得ると考えられる。その後、画像へのフィルタの適用によって、アーチファクト(たとえば、小さな斑点、小さな切れ目、他の小さな物体)の選択的な除去および/または孔の充填を行う。いくつかの実施形態においては、形態学的演算子の適用によって、アーチファクトの除去および/または孔の充填を行う。いくつかの実施形態においては、入力として導入される2値画像(たとえば、先行するフィルタリングステップの結果としての2値画像)に基づいて、距離に基づく分水嶺が適用される。 [000111] In other embodiments, the input image is segmented by applying a series of algorithms to the image, such as global thresholding filters, locally adaptive thresholding filters, morphological operations, and watershed transforms. These filters may be operated sequentially or in any order deemed necessary by those skilled in the art. Of course, any filter may be applied iteratively until the desired result is achieved. In some embodiments, application of the first filter to the input image results in the possibility of having nuclei, such as removing white image areas (corresponding to areas in unstained or nearly unstained tissue samples). Remove areas with low . In some embodiments, this is achieved by applying a global thresholding filter. In some embodiments, global thresholding is based on the median and/or standard deviation computed on the first principal component channel (eg, analogous to the grayscale channel). By determining a global threshold, any white image areas representing unstained or near-unstained areas where nuclei are likely to be absent could be discarded. A filter is then applied to the image to selectively remove artifacts (eg, small speckles, small discontinuities, other small objects) and/or fill holes. In some embodiments, the application of morphological operators removes artifacts and/or fills holes. In some embodiments, a distance-based watershed is applied based on a binary image introduced as input (eg, a binary image resulting from a previous filtering step).
[000112]いくつかの実施形態において、入力画像は、スーパーピクセルにセグメント化される。スーパーピクセルアルゴリズムは、知覚的に意味あるエンティティを表す多くのセグメント(ピクセル群)へと画像を分割すると考えられる。各スーパーピクセルは、低レベルグループ化プロセスによって得られ、知覚的に矛盾のない単位を有する。すなわち、スーパーピクセルに含まれる生体中のすべてのピクセルが染色有無(たとえば、スーパーピクセルに存在するピクセルが特定種のステインのものである)、染色強度(たとえば、ピクセルが一定の相対強度値または値の範囲を有する)、および質感(たとえば、ピクセルが色または強度に関する特定の空間配置を有する)について可能な限り均一である。各スーパーピクセルの局所分析結果は、デジタル病理学画像上に分析結果を表すように格納および報告可能である。 [000112] In some embodiments, the input image is segmented into superpixels. A superpixel algorithm is thought to divide an image into many segments (groups of pixels) that represent perceptually meaningful entities. Each superpixel is obtained by a low-level grouping process and has perceptually consistent units. That is, all pixels in the organism included in the superpixel are stained or not (e.g., the pixels present in the superpixel are of a particular type of stain), staining intensity (e.g., the pixels have a constant relative intensity value or value). ), and texture (eg, pixels have a particular spatial arrangement of color or intensity). Local analysis results for each superpixel can be stored and reported to represent the analysis results on the digital pathology image.
[000113]スーパーピクセルは、色、輝度、および質感等の特質が類似するピクセルの集まりである。画像は、ピクセルの複数の組み合わせ特質を含み、元画像のエッジ情報を保存可能な一定数のスーパーピクセルで構成可能である。単一のピクセルと比較して、スーパーピクセルは、豊富な特質情報を含み、画像の後処理の複雑性を大幅に低減するとともに、画像セグメント化の速度を大幅に向上可能である。また、スーパーピクセルは、小さな近傍モデルによる確率の推定および決定にも有用である。 [000113] A superpixel is a collection of pixels with similar attributes such as color, brightness, and texture. An image can be composed of a fixed number of superpixels that contain multiple combinatorial features of pixels and that can preserve the edge information of the original image. Compared to single pixels, superpixels contain rich characteristic information and can greatly reduce the complexity of image post-processing and greatly speed up image segmentation. Superpixels are also useful for estimating and determining probabilities with small neighborhood models.
[000114]スーパーピクセルアルゴリズムは、ピクセルを類似サイズの意味ある原子領域へとグループ化する方法である。任意特定の理論に縛られることを望むことなく、スーパーピクセルは、画像内の重要な境界に位置することが多く、顕著な物体特徴を含む場合には基準外または一意の形状を帯びる傾向にあることから、効果的と考えられる。中解像度分析における情報の取得および格納の要望と矛盾することなく、スーパーピクセルは、ピクセルレベルとオブジェクトレベルとの間に位置付けられ、画像オブジェクトを包括的に表すことなく、知覚的に意味あるピクセル群を表すことによって、ピクセルよりも多くの情報をもたらす。スーパーピクセルは、短い演算時間で画像を過剰セグメント化する画像セグメント化の一形態と理解され得る。スーパーピクセルの外形は、画像中のほとんどの構造が保護されるため、自然な画像境界に十分従うことが分かっている。各ピクセルではなく各スーパーピクセルについて画像特徴が演算されることから、後続の処理タスクは、複雑性および演算時間が抑えられる。このため、スーパーピクセルは、画像セグメント化等のオブジェクトレベルでの分析の前処理ステップとして有用と考えられる。 [000114] A superpixel algorithm is a method of grouping pixels into meaningful atomic regions of similar size. Without wishing to be bound by any particular theory, superpixels are often located at important boundaries in images and tend to take on non-standard or unique shapes when they contain prominent object features. Therefore, it is considered effective. Consistent with the desire for information acquisition and storage in medium-resolution analysis, superpixels are positioned between the pixel level and the object level and are perceptually meaningful groups of pixels that do not comprehensively represent an image object. gives us more information than pixels. A superpixel may be understood as a form of image segmentation that over-segments an image with a short computation time. Superpixel contours have been found to follow natural image boundaries well, as most structures in the image are preserved. Subsequent processing tasks are of reduced complexity and computation time because the image features are computed for each superpixel instead of each pixel. For this reason, superpixels may be useful as a preprocessing step for object-level analysis such as image segmentation.
[000115]任意特定の理論に縛られることを望むことなく、スーパーピクセルは、たとえば色または形状が類似する特質を有する稠密かつ均一なピクセル群を形成することによって、画像を過剰セグメント化すると考えられる。過去には、複数のスーパーピクセル手法が開発されている。これらは、(i)グラフベースの手法および(ii)勾配上昇ベースの手法に分類可能である。グラフベースの手法においては、各ピクセルがグラフ中のノードと考えられる。すべてのノード対間には、それぞれの類似性に比例するエッジ重みが規定される。そして、グラフ上に規定されるコスト関数の公式化および最小化によって、スーパーピクセルセグメントを抽出する。勾配上昇ベースの手法においては、特徴空間へのピクセルの反復的なマッピングによって、クラスタを表す高密度領域を描く。各反復は、各クラスタの精緻化によって、収束までにより優れたセグメント化を得る。 [000115] Without wishing to be bound by any particular theory, superpixels are believed to over-segment an image by forming dense and uniform groups of pixels that have similar characteristics, for example in color or shape. . Several superpixel approaches have been developed in the past. These can be classified into (i) graph-based approaches and (ii) gradient ascent-based approaches. In graph-based approaches, each pixel is considered a node in the graph. Edge weights are defined between all node pairs that are proportional to their similarities. The superpixel segments are then extracted by formulating and minimizing a cost function defined on the graph. In the gradient ascent-based approach, iterative mapping of pixels into feature space delineates dense regions representing clusters. Each iteration obtains a better segmentation until convergence by refining each cluster.
[000116]正規化カット、凝集型クラスタリング、クイックシフト、およびターボピクセルアルゴリズム等、多くのスーパーピクセルアルゴリズムが開発されている。正規化カットアルゴリズムは、輪郭および質感キューを使用し、分割境界のエッジ上に規定されるコスト関数を最小化することによって、全ピクセルのグラフを再帰的に分割する。これにより、非常に規則的で、視覚的に快いスーパーピクセルが得られる(Jianbo Shi and Jitendra Malik「Normalized cuts and image segmentation(正規化カットおよび画像セグメント化)」,IEEE Transactions on Pattern Analysis and Machine Intelligence,(PAMI),22(8):888-905,Aug 2000(すべての開示内容が参照により本明細書に組み込まれる)参照)。Alastair Moore,Simon Prince,Jonathan Warrell,Umar Mohammed,and Graham Jones「Superpixel Lattices(スーパーピクセル格子)」,IEEE Computer Vision and Pattern Recognition (CVPR),2008は、より小さな垂直または水平領域へと画像を分割する最適経路またはシームを見つけることによって、グリッドに従うスーパーピクセルを生成する方法を記載する。最適経路は、グラフカット法を用いることにより見つかる。(Shai Avidan and Ariel Shamir「Seam carving for content-aware image resizing(内容を意識した画像サイズ調整のためのシームカービング)」,ACM Transactions on Graphics (SIGGRAPH),26(3),2007(開示内容が参照により本明細書に組み込まれる)参照)。クイックシフト(A.Vedaldi and S.Soatto「Quick shift and kernel methods for mode seeking(モード探索のためのクイックシフトおよびカーネル方法)」,European Conference on Computer Vision(ECCV),2008(開示内容が参照により本明細書に組み込まれる)参照)は、モード探索セグメント化方式を使用する。これは、medoid shift手順を用いてセグメント化を初期化する。その後、Parzen密度推定値を増大させる最も近い隣接点へと特徴空間の各点を移動させる。ターボピクセル法では、レベルセットベースの幾何学的フローを用いて、一組の種子位置を徐々に拡張させる(A.Levinshtein,A.Stere,K.Kutulakos,D.Fleet,S.Dickinson,and K.Siddiqi「Turbopixels: Fast superpixels using geometric flows(ターボピクセル:幾何学的フローを用いた高速スーパーピクセル)」,IEEE Transactions on Pattern Analysis and Machine Intelligence(PAMI),2009(開示内容が参照により本明細書に組み込まれる)参照)。幾何学的フローは、画像平面上でスーパーピクセルを規則的に分布させることを目的として、局所的な画像勾配に依拠する。ターボピクセルスーパーピクセルは、他の方法と異なり、均一なサイズ、稠密性、および境界追従を有するように制約される。スーパーピクセルを生成するさらに他の方法については、Radhakrishna Achanta「SLIC Superpixels Compared to State-of-the-art(最新技術との比較によるSLICスーパーピクセル)」,Journal of Latex Class Files,Vol.6,No.1,December 2011に記載されている(そのすべての開示内容が参照により本明細書に組み込まれる)。 [000116] Many superpixel algorithms have been developed, such as normalized cut, agglomerative clustering, quickshift, and turbopixel algorithms. The normalized cut algorithm recursively partitions the graph of all pixels by using contour and texture cues and minimizing a cost function defined on the edge of the partition boundary. This results in very regular and visually pleasing superpixels (Jianbo Shi and Jitendra Malik, "Normalized cuts and image segmentation", IEEE Transactions on Pattern Analysis and Machine Intelligence, (PAMI), 22(8):888-905, Aug 2000 (the entire disclosure of which is incorporated herein by reference)). Alastair Moore, Simon Prince, Jonathan Warrell, Umar Mohammed, and Graham Jones "Superpixel Lattices", IEEE Computer Vision and Pattern Recognition (CVPR), 2008. Splitting an image vertically into smaller regions or horizontally We describe a method to generate grid-following superpixels by finding the optimal path or seam. The optimal path is found by using the graph cut method. (Shai Avidan and Ariel Shamir, "Seam carving for content-aware image resizing," ACM Transactions on Graphics (SIGGRAPH), 26(3), 2007 (disclosure see incorporated herein by reference). Quickshift, A. Vedaldi and S. Soatto, "Quick shift and kernel methods for mode seeking," European Conference on Computer Vision (ECCV), 2008, the disclosure of which is incorporated herein by reference. ) uses a mode search segmentation scheme. This initializes the segmentation using the medoid shift procedure. We then move each point in the feature space to the nearest neighbor that increases the Parzen density estimate. Turbo-pixel methods use a level-set-based geometric flow to gradually extend a set of seed positions (A. Levinshtein, A. Stereo, K. Kutulakos, D. Fleet, S. Dickinson, and K. Siddiqi, "Turbopixels: Fast superpixels using geometric flows," IEEE Transactions on Pattern Analysis and Machine Intelligence (PAMI), 2009, the disclosure of which is incorporated herein by reference. incorporated) see). Geometric flow relies on local image gradients for the purpose of regular distribution of superpixels on the image plane. Turbopixel superpixels are constrained to have uniform size, density, and boundary following unlike other methods. For yet another method of generating superpixels, see Radhakrishna Achanta, "SLIC Superpixels Compared to State-of-the-art," Journal of Latex Class Files, Vol. 6, No. 1, December 2011, the entire disclosure of which is incorporated herein by reference.
[000117]単純線形反復クラスタリング(SLIC)と称するスーパーピクセルアルゴリズムが導入されているが、これは、最先端のスーパーピクセル法と比較して、境界追従および効率の両者において優れている。SLICには、2つのステップがある。第1には、局所k平均クラスタリング(KMC)法によってピクセルをグループ化することによりスーパーピクセルを生成するが、その距離は、データおよび空間距離と統合されたユークリッド距離として測定される。第2には、連結成分アルゴリズム(CCA)の使用によって、生成された微小分離領域を最も近い大きなスーパーピクセルとして統合することにより除去する。 [000117] A superpixel algorithm called simple linear iterative clustering (SLIC) has been introduced, which outperforms state-of-the-art superpixel methods in both boundary following and efficiency. SLIC has two steps. First, superpixels are generated by grouping pixels by a local k-means clustering (KMC) method, whose distances are measured as Euclidean distances integrated with data and spatial distances. Second, through the use of the Connected Component Algorithm (CCA), the generated small isolated regions are eliminated by merging them as the nearest large superpixel.
[000118]k平均クラスタリングは、n個の観察結果をk個のクラスタに分割することを目的としており、各観察結果は、平均に最も近いクラスタに属して、クラスタのプロトタイプとして機能する。連結成分標識化は、画像をピクセルごとに(上から下、左から右に)スキャンし、連結ピクセル領域すなわち同じ強度値Vの集合を共有する隣り合うピクセルの領域を識別することによって機能する(2値画像の場合はV={1}であるが、グレーレベル画像において、Vは、ある範囲の値を取る(たとえば、V={51、52、53、・・・、77、78、79、80}))。連結成分標識化は、2値画像またはグレーレベル画像に対して機能し、連結度の異なる尺度が可能である。ただし、以下では、2値入力画像および8連結度を仮定する。連結成分標識化の演算子は、V={1}となる点p(ここで、pは、スキャンプロセスの任意の段階で標識化されるピクセルを示す)まで、行に沿って移動することにより画像をスキャンする。これが真の場合は、スキャン中に通過済みのpの4つの隣接ピクセル(すなわち、(i)pの左方、(ii)上方、ならびに(iiiおよびiv)2つの上側対角関係の隣接ピクセル)を検査する。この情報に基づいて、pの標識化を以下のように行う。4つの隣接ピクセルがすべて0の場合は、新たなラベルをpに割り当て、隣接ピクセルが1つだけV={1}の場合は、そのラベルをpに割り当て、隣接ピクセルが2つ以上V={1}の場合は、ラベルのうちの1つをpに割り当てて、同等物を書き留める。 [000118] K-means clustering aims to divide n observations into k clusters, with each observation belonging to the cluster closest to the mean and serving as a cluster prototype. Connected component labeling works by scanning an image pixel by pixel (top to bottom, left to right) and identifying connected pixel regions, i.e. regions of adjacent pixels that share the same set of intensity values V ( For binary images V={1}, but in gray-level images V takes a range of values (eg V={51, 52, 53, . . . , 77, 78, 79 , 80})). Connected component labeling works on binary or gray-level images and allows for different measures of connectivity. However, in the following, we assume a binary input image and 8-connectivity. The connected component labeling operator is by moving along the row to a point p where V={1}, where p denotes the pixel to be labeled at any stage of the scanning process. Scan an image. If this is true, then the 4 neighbors of p passed through during the scan (i.e., (i) the left, (ii) above, and (iii and iv) the two upper diagonal neighbors of p) to inspect. Based on this information, the labeling of p is done as follows. If 4 neighbors are all 0 then assign a new label to p, if only 1 neighbor is V={1} then assign that label to p and if there are 2 or more neighbors V={ 1}, assign one of the labels to p and note the equivalent.
[000119]スキャンの完了後は、同等ラベル対が同等物クラスに格納され、一意のラベルが各クラスに割り当てられる。最終ステップとして、画像に第2のスキャンがなされるが、この間は、その同等物クラスに割り当てられたラベルによって各ラベルが置き換えられる。表示のため、各ラベルは、異なるグレーレベルまたは色であってもよい。 [000119] After the scan is complete, the equivalence label pairs are stored in equivalence classes and a unique label is assigned to each class. As a final step, the image undergoes a second scan, during which each label is replaced by the label assigned to its equivalence class. For display purposes, each label may be a different gray level or color.
[000120]SLICは、スーパーピクセルの生成のためのk平均の適応であるが、2つの重要な差異がある。(i)スーパーピクセルのサイズに比例した領域に探索空間を制限することによって、最適化における距離計算の回数が劇的に少なくなる(これによって、スーパーピクセルの数kとは無関係に、ピクセル数の複雑性が線形に小さくなると考えられる)。(ii)重み付け距離尺度が色と空間近接性とを組み合わせると同時に、スーパーピクセルのサイズおよび稠密度を制御する(Achanta,et al.「SLIC Superpixels Compared to State-of-the-Art Superpixel Methods(最先端のスーパーピクセル法との比較によるSLICスーパーピクセル)」,IEEE Transactions on Pattern Analysis and Machine Intelligence,Vol.34,No.l1,November 2012(すべての開示内容が参照により本明細書に組み込まれる)参照)。 [000120] SLIC is an adaptation of k-means for superpixel generation, but with two important differences. (i) By limiting the search space to an area proportional to the superpixel size, the number of distance calculations in the optimization is dramatically reduced (this allows the number of pixels complexity is expected to decrease linearly). (ii) A weighted distance measure combines color and spatial proximity while controlling superpixel size and density (Achanta, et al., SLIC Superpixels Compared to State-of-the-Art Superpixel Methods). SLIC Super-Pixel by Comparison with Advanced Super-Pixel Method,” IEEE Transactions on Pattern Analysis and Machine Intelligence, Vol. ).
[000121]SLICでは、CIELAB色空間のL*a*b値ならびにそれぞれのx座標およびy座標により規定される5D空間における画像ピクセルを考慮する。5D空間におけるピクセルは、画像平面における色類似性および近接性を統合した適応k平均クラスタリングに基づいてクラスタリングされる。このクラスタリングは、L*a*b空間における色類似性(dc)およびx、y空間におけるピクセル近接性(ds)を測定した距離尺度Dに基づく。後者は、スーパーピクセルの数(k)で除した画像ピクセルの総数の平方根を規定するグリッド間隔(S)によって正規化される。スーパーピクセルの稠密性および規則性は、定数mによって制御される。このパラメータは、空間距離(dc)とスペクトル距離(ds)との間の重み付け基準として機能する。mが大きいほど空間近接性の重みが増え、画像中のスペクトル外形に対する境界の追従が低下したより稠密なスーパーピクセルが得られる。 [000121] SLIC considers image pixels in 5D space defined by their L*a*b values in the CIELAB color space and their respective x and y coordinates. Pixels in 5D space are clustered based on adaptive k-means clustering that integrates color similarity and proximity in the image plane. This clustering is based on a distance measure D that measures color similarity (dc) in L*a*b space and pixel proximity (ds) in x,y space. The latter is normalized by the grid spacing (S) which defines the square root of the total number of image pixels divided by the number of superpixels (k). Superpixel density and regularity are controlled by a constant m. This parameter serves as a weighting criterion between spatial distance (dc) and spectral distance (ds). Larger m gives more spatial proximity weighting, resulting in denser superpixels with less boundary tracking to spectral contours in the image.
[000123]SLICアルゴリズムは、以下のように適用され得る。Npを所与の画像(または、その関心部分もしくは領域)のピクセル数とし、kを生成するスーパーピクセル数とする。次に、SLICアルゴリズムの主要なステップは、以下の通りである。 [000123] The SLIC algorithm may be applied as follows. Let N p be the number of pixels in a given image (or portion or region of interest thereof) and let k be the number of superpixels to generate. The main steps of the SLIC algorithm are then as follows.
[000124](1)クラスタ中心を初期化する。規則的なグリッド上のk個のクラスタ中心を [000124] (1) Initialize the cluster centers. Let k cluster centers on a regular grid be
ピクセルだけ離隔させた後、3×3近傍において勾配が最小の位置まで、これらのクラスタ中心を移動させる。任意特定の理論に縛られることを望むことなく、これは、スーパーピクセルの中心をエッジに設定しないようにするとともに、ノイズの多いピクセルにスーパーピクセルの種子を設定する機会を減らすためと考えられる。 After being spaced by a pixel, move these cluster centers to the location with the lowest gradient in the 3x3 neighborhood. Without wishing to be bound by any particular theory, it is believed that this is to avoid setting superpixel centers on edges and to reduce the chance of setting superpixel seeds on noisy pixels.
[000125](2)ピクセルを割り当てる。局所KMCによって、局所探索空間における最も近いクラスタ中心に各ピクセルを指定する。
[000126](3)クラスタ中心を更新する。対応するクラスタにおける全ピクセルの平均として、各クラスタ中心を設定する。
[000125] (2) Allocate pixels. A local KMC designates each pixel to the nearest cluster center in the local search space.
[000126] (3) Update cluster centers. Set each cluster center as the average of all pixels in the corresponding cluster.
[000127](4)クラスタが変化しなくなるまで、または、別の所与の基準が満たされるまで、ステップ(2)および(3)を繰り返す。
[000128](5)後処理。分離領域のサイズが最小サイズSmin未満の場合は、CCAの使用により、分離領域を近くのスーパーピクセルに再度割り当てる。
[000127] (4) Repeat steps (2) and (3) until no clusters change, or until another given criterion is met.
[000128] (5) Post-processing. If the size of the isolation region is less than the minimum size S min , use CCA to reassign the isolation region to a nearby superpixel.
[000129]SLIC法のステップ(2)においては、局所KMCが適用されるが、この場合は、探索エリアがその場所を網羅する最も近いクラスタ中心と各ピクセルが関連付けられる。従来のKMCにおいて、各クラスタ中心の探索エリアは、全画像であるため、画像中の各クラスタ中心からすべてのピクセルまでの距離が計算される。ただし、局所KMCにおいては、クラスタ中心の探索空間が局所的な2S×2S正方形領域に限定される。したがって、SLICでは、各クラスタ中心からその探索エリア内のピクセルまでの距離しか演算しない。 [000129] In step (2) of the SLIC method, local KMC is applied, where each pixel is associated with the closest cluster center whose location the search area covers. In conventional KMC, the search area for each cluster center is the entire image, so the distance from each cluster center to every pixel in the image is calculated. However, in local KMC, the cluster center search space is limited to a local 2S×2S square region. Therefore, SLIC only computes the distance from each cluster center to pixels within its search area.
[000130]局所KMCにおいては、クラスタリングにおいてユークリッド距離が使用される。ziを空間位置が(xi,yi)のi番目のクラスタ中心のデータとする。zjを当該中心の探索エリア内のピクセルの強度とする。そして、このピクセルと中心との間の統合距離は、以下の通りである。 [000130] In local KMC, Euclidean distance is used in clustering. Let z i be the data of the i-th cluster center at spatial position (x i , y i ). Let zj be the intensity of the pixel in the central search area. Then the integrated distance between this pixel and the center is:
[000132]ここで、df=|zi-zj|および [000132] where d f = |z i −z j | and
はそれぞれ、強度およびピクセルと中心との間の距離であり、mは、統合距離DIに対するdfおよびdsの相対的な寄与を重み付けする正則化パラメータである。mが大きくなることは、dsがdfよりも有意であることを示す。これら2つの距離の寄与を直接記述する同等の統合距離DIは、以下により与えられ得る。 are the intensity and the distance between the pixel and the center, respectively, and m is a regularization parameter that weights the relative contribution of d f and d s to the integrated distance DI . A larger m indicates that ds is more significant than df . An equivalent integrated metric DI that directly describes the contribution of these two metrics can be given by:
[000134]ここで、Nfは全画面の平均強度であり、w∈[0,1]は正則化パラメータである。これに関連して、wおよび(1-w)はそれぞれ、正規化強度とDIにおける各空間距離との比である。 [000134] where N f is the average intensity of the whole screen and wε[0,1] is the regularization parameter. In this context, w and ( 1 -w) are the ratios of the normalized intensity and each spatial distance in DI, respectively.
[000135]いくつかの実施形態において、SLICアルゴリズムのパラメータkは、略等しいサイズのスーパーピクセルの数を指定する。いくつかの実施形態において、稠密度パラメータmは、スーパーピクセルの均質性と境界追従とのトレードオフを制御するように設定可能である。任意特定の理論に縛られることを望むことなく、稠密度パラメータを変更することによって、規則的な形状のスーパーピクセルが非質感領域に生成され、高度に不規則なスーパーピクセルが質感領域に生成され得ると考えられる。この場合も、任意特定の理論に縛られることを望むことなく、パラメータmは、色類似性と空間近接性との間の相対的重要性の重み付けを可能にすると考えられる。mが大きい場合は、空間近接性がより重要であり、結果としてのスーパーピクセルがより稠密となる(すなわち、面積対周長比が小さくなる)。mが小さい場合は、結果としてのスーパーピクセルが画像境界に対してより緊密に追従するものの、サイズおよび形状は不規則になる。 [000135] In some embodiments, the parameter k of the SLIC algorithm specifies the number of approximately equally sized superpixels. In some embodiments, the density parameter m can be set to control the trade-off between superpixel homogeneity and boundary following. Without wishing to be bound by any particular theory, by varying the density parameter, regularly shaped superpixels are generated in non-textured regions and highly irregular superpixels are generated in textured regions. considered to be obtained. Again, without wishing to be bound by any particular theory, it is believed that the parameter m allows weighting of the relative importance between color similarity and spatial proximity. When m is large, spatial proximity is more important and the resulting superpixels are denser (ie, smaller area-to-perimeter ratio). If m is small, the resulting superpixels will follow the image boundaries more closely, but will be irregular in size and shape.
[000136]いくつかの実施形態においては、スーパーピクセルのサイズおよび稠密度の両パラメータが調整される。いくつかの実施形態においては、およそ40ピクセル~およそ400ピクセルの範囲のスーパーピクセルサイズが使用される。他の実施形態においては、およそ60ピクセル~およそ300ピクセルの範囲のスーパーピクセルサイズが使用される。さらに他の実施形態においては、およそ70ピクセル~およそ250ピクセルの範囲のスーパーピクセルサイズが使用される。さらに別の実施形態においては、およそ80ピクセル~およそ200ピクセルの範囲のスーパーピクセルサイズが使用される。 [000136] In some embodiments, both the size and density parameters of the superpixel are adjusted. In some embodiments, superpixel sizes ranging from approximately 40 pixels to approximately 400 pixels are used. In other embodiments, superpixel sizes ranging from approximately 60 pixels to approximately 300 pixels are used. In still other embodiments, superpixel sizes ranging from approximately 70 pixels to approximately 250 pixels are used. In yet another embodiment, superpixel sizes ranging from approximately 80 pixels to approximately 200 pixels are used.
[000137]いくつかの実施形態において、稠密度パラメータは、およそ10~およそ100の範囲である。他の実施形態において、稠密度パラメータは、およそ20~およそ90の範囲である。他の実施形態において、稠密度パラメータは、およそ40~およそ80の範囲である。他の実施形態において、稠密度パラメータは、およそ50~およそ80の範囲である。 [000137] In some embodiments, the compactness parameter ranges from approximately 10 to approximately 100. In other embodiments, the compactness parameter ranges from approximately 20 to approximately 90. In other embodiments, the compactness parameter ranges from approximately 40 to approximately 80. In other embodiments, the compactness parameter ranges from approximately fifty to approximately eighty.
[000138]図8Aは、本明細書に記載の通り、SLICを用いて生成されたスーパーピクセルの一例を示しており、重畳および間隙なく、関心領域の位置特定された特質に適するようにスーパーピクセルがセグメント化されている。さらに、各スーパーピクセル副領域は、バイオマーカ発現の存在の局所強度(810)および方向(820)に応じて、特定の最終形状を有する。このため、スーパーピクセルは、このような関心生物学的構造に対して知覚的に意味がある。図8B、図8C、および図8Dはそれぞれ、高倍率の元のIHC画像、スーパーピクセル生成プロセスの初期化、および局所的に均質な最終スーパーピクセルを示しており、それぞれの形状の規則性は、上述の通り、SLICアルゴリズムの技術的パラメータによって調整済みである。 [000138] FIG. 8A shows an example of superpixels generated using SLIC, as described herein, where the superpixels are aligned to suit the localized features of the region of interest without overlap and gaps. is segmented. Furthermore, each superpixel subregion has a specific final shape depending on the local intensity (810) and direction (820) of the presence of biomarker expression. Thus, superpixels are perceptually meaningful to such biological structures of interest. Figures 8B, 8C, and 8D show, respectively, the original IHC image at high magnification, the initialization of the superpixel generation process, and the locally homogeneous final superpixels, whose shape regularity is As mentioned above, it has been adjusted by the technical parameters of the SLIC algorithm.
[000139]表現オブジェクト生成モジュール
[000140]副領域生成モジュールによって副領域が生成された(ステップ320)後は、モジュール207を用いて、副領域ごとに表現オブジェクトまたは関心点が決定される(ステップ330)。いくつかの実施形態において、表現オブジェクトは、関心細胞または関心細胞群(たとえば、線維芽細胞またはマクロファージ)に関する副領域またはスーパーピクセルの外形である。他の実施形態において、表現オブジェクトは、種子点である。本明細書に記載の通り、本開示の目的は、類似の染色有無、染色強度、および/または局所質感を有する副領域に基づいて関心細胞(不規則形状の細胞)を特性化するとともに、これらの均質特性の副領域を自動的にデータベースに保存することである。表現オブジェクトまたはその座標は、生成副領域を格納する1つの方法である。図9Aおよび図9Bは、関心生体を含むスーパーピクセルのポリゴン外形および中心種子の例を提供する。
[000139] representation object generation module
[000140] After the sub-regions have been generated by the sub-region generation module (step 320), the representational objects or points of interest are determined for each sub-region (step 330) using
[000141]いくつかの実施形態においては、色または質感が異なる副領域を分離するとともに、画像中の支配的なエッジと一致する境界を形成するアルゴリズムが利用されるため、関心生体(たとえば、線維芽細胞またはマクロファージ等、サイズまたは形状が不規則な細胞)を表す境界が生成される。いくつかの実施形態においては、ステインを有さない副領域が除外され、閾値量のステインを含む副領域のみが表現オブジェクトとして提供されるように、閾値化アルゴリズム(たとえば、大津法、平均クラスタリング等)がステインチャネル画像に適用されるようになっていてもよい。いくつかの実施形態においては、閾値パラメータ(たとえば、病理学専門家により提供される閾値染色パラメータ)を用いて、副領域の2値マスクが生成されるようになっていてもよい。いくつかの実施形態においては、(i)関心物体を表す可能性が低い副領域が(ii)関心物体を有する細胞を表す副領域から分離されるように画像を強化するように設計された一連のフィルタを適用することによって、セグメント化が実現される。アーチファクトの除去、小さな斑点の除去、小さな切れ目の除去、孔の充填、および大きな斑点の分割のため、付加的なフィルタが選択的に適用されるようになっていてもよい。 [000141] In some embodiments, algorithms are utilized that separate sub-regions of different color or texture and form boundaries that coincide with dominant edges in the image so that the organism of interest (e.g., fiber Boundaries representing irregularly sized or shaped cells, such as blasts or macrophages, are generated. In some embodiments, a thresholding algorithm (e.g., Otsu's method, mean clustering, etc.) is applied such that subregions with no stain are excluded and only subregions with a threshold amount of stain are provided as representation objects. ) may be applied to the stain channel image. In some embodiments, a threshold parameter (eg, a threshold staining parameter provided by a pathologist) may be used to generate a binary mask of sub-regions. In some embodiments, a sequence designed to enhance the image such that (i) sub-regions unlikely to represent objects of interest are separated from (ii) sub-regions representing cells with objects of interest Segmentation is achieved by applying a filter of Additional filters may be selectively applied for artifact removal, small speckle removal, small discontinuity removal, hole filling, and large speckle segmentation.
[000142]いくつかの実施形態においては、ステインチャネルの2値画像中の(無染色または略無染色の組織サンプル中の領域に対応する)白色の画像領域の除去等によって、不規則形状の細胞を識別する副領域を有する可能性が低い領域が除去される。いくつかの実施形態において、これは、グローバル閾値化フィルタを適用することによって実現される。閾値化は、強度が何らかの閾値(ここでは、グローバル閾値)を上回るか下回る場合に、値1または0をすべてのピクセルに割り当てることによって、強度画像(I)を2値画像(I’)へと変換するのに用いられる方法である。言い換えると、強度値に応じてピクセルを分割するのに、グローバル閾値化が適用される。いくつかの実施形態において、グローバル閾値化は、(たとえば、グレースケールチャネルに類似する)第1の主成分チャネル上で演算された中央値および/または標準偏差に基づく。グローバル閾値を求めることによって、不規則形状の細胞が存在しない可能性が高い無染色または略無染色の領域を表す任意の白色画像領域が破棄され得ると考えられる。
[000142] In some embodiments, irregularly shaped cells are removed, such as by removing white image areas (corresponding to areas in unstained or nearly unstained tissue samples) in the stain channel binary image. Regions that are unlikely to have subregions that identify are removed. In some embodiments, this is achieved by applying a global thresholding filter. Thresholding converts the intensity image (I) into a binary image (I′) by assigning the
[000143]いくつかの実施形態においては、FAPステインに関して、1)紫色チャネルを分離し、2)紫色チャネルの閾値化によってFAP陽性領域を識別し、3)紫色チャネルに対してスーパーピクセルセグメント化を適用し、4)特徴測定基準をスーパーピクセルオブジェクトに付与することによって、境界が形成され得る。いくつかの実施形態において、FAP陽性領域の有無は、病理学者から得られたグランドトゥルースに基づいてトレーニングされた教師あり生成ルールを用いて識別されるようになっていてもよい。いくつかの実施形態においては、画像のトレーニングセット上での閾値の識別等によって、FAP陽性閾値パラメータが病理学者により供給されるようになっていてもよい。そして、この閾値パラメータを用いることにより、2値マスクが生成されるようになっていてもよい。これらの方法は、Auranuch Lorsakul et al.「Automated whole-slide analysis of multiplex-brightfield IHC images for cancer cells and carcinoma-associated fibroblasts(がん細胞およびがん関連線維芽細胞に関する多重化-明視野IHC画像の自動全スライド分析)」,Proc.SPIE 10140,Medical Imaging 2017:Digital Pathology,1014007(2017/03/01)に詳しく記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。 [000143] In some embodiments, for the FAP stain, 1) isolate the violet channel, 2) identify FAP-positive regions by thresholding the violet channel, and 3) perform superpixel segmentation on the violet channel. 4) applying a feature metric to the superpixel object, a boundary can be formed. In some embodiments, the presence or absence of FAP-positive regions may be identified using supervised generation rules trained on ground truth obtained from pathologists. In some embodiments, the FAP positive threshold parameter may be supplied by the pathologist, such as by identifying a threshold on a training set of images. A binary mask may be generated by using this threshold parameter. These methods are described by Auranuch Lorsakul et al. "Automated whole-slide analysis of multiplex-brightfield IHC images for cancer cells and carcinoma-associated fibroblasts", Proc. SPIE 10140, Medical Imaging 2017: Digital Pathology, 1014007 (03/01/2017), the entire disclosure of which is incorporated herein by reference.
[000144]いくつかの実施形態においては、副領域の境界がトレースされる。たとえば、副領域の外部境界のほか、副領域の内側または間の「孔」の外部境界をトレースするアルゴリズムが提供されていてもよい。いくつかの実施形態において、副領域の境界は、bwboundariesと称するmatlab関数を用いて境界トレースを形成することにより生成される(https://www.mathworks.com/help/images/ref/bwboundaries.html)。 [000144] In some embodiments, boundaries of subregions are traced. For example, an algorithm may be provided that traces the outer boundaries of sub-regions as well as "holes" inside or between sub-regions. In some embodiments, subregion boundaries are generated by forming boundary traces using a matlab function called bwboundaries (https://www.mathworks.com/help/images/ref/bwboundaries. html).
[000145]境界形成の後、境界トレースは、x、y座標から成るポリゴン外形に変換される。トレース境界のx、y座標は、メモリまたはデータベースに格納されるようになっていてもよく、たとえば、副領域オブジェクトのトレース境界の全ピクセルの行および列座標が決定および格納されるようになっていてもよい。 [000145] After bounding, the bounding trace is converted to a polygonal outline consisting of x,y coordinates. The x, y coordinates of the trace boundaries may be stored in memory or in a database, e.g. the row and column coordinates of all pixels of the trace boundaries of the subregion object are determined and stored. may
[000146]いくつかの実施形態においては、各副領域の重心または質量中心の計算または演算によって、種子点が導出される。当業者には、不規則なオブジェクトの重心を決定する方法が既知である。計算後は、副領域の重心の標識化ならびに/または種子のx、y座標のメモリもしくはデータベースへの格納が行われる。いくつかの実施形態において、重心または質量中心の位置は、入力画像に重ね合わされていてもよい。 [000146] In some embodiments, the seed points are derived by calculating or calculating the center of gravity or center of mass of each sub-region. A person skilled in the art knows how to determine the centroid of an irregular object. After calculation, the centroids of the subregions are labeled and/or the x,y coordinates of the seeds are stored in a memory or database. In some embodiments, the location of the centroid or center of mass may be superimposed on the input image.
[000147]標識化モジュール
[000148]セグメント化モジュール206を用いて副領域が生成され、モジュール207を用いて表現オブジェクトが演算された後は、標識化モジュール208を用いて、画像分析モジュール202から導出された測定基準等(ステップ310)、表現オブジェクトのアノテーション、標識化、またはデータとの関連付けがなされる(ステップ330)。標識化モジュール208は、本明細書に記載の通り、データを格納する非一時的メモリであるデータベース209を生成するようにしてもよい。いくつかの実施形態において、データベース209は、入力として受信された画像、任意のポリゴンおよび/もしくは種子点の座標、ならびに画像分析による任意の関連データもしくはラベルを格納する(図11参照)。
[000147] Labeling Module
[000148] After the
[000149]この点に関しては、画像のセグメント化副領域ごとに、データベクトルが格納されるようになっていてもよい。たとえば、任意の表現オブジェクトおよび関連する画像分析データを含めて、副領域ごとに、データベクトルが格納されるようになっていてもよい。一例として、データ点「a」、「b」、および「c」が表現オブジェクトの座標であり、「x」、「y」、および「z」が画像分析により導出された測定基準(または、特定の副領域に対応する測定基準の平均)である場合、データベースは、[a,b,c,x,y,z]1、[a,b,c,x,y,z]2、[a,b,c,x,y,z]Nといったデータベクトルを格納することになる。ここで、Nは、セグメント化モジュール206により生成された副領域の数である。
[000149] In this regard, a data vector may be stored for each segmented sub-region of the image. For example, a data vector may be stored for each subregion, including any representational objects and associated image analysis data. As an example, the data points 'a', 'b', and 'c' are the coordinates of the represented object, and 'x', 'y', and 'z' are the metrics derived by image analysis (or specific , then the database contains [a, b, c, x, y, z] 1 , [a, b, c, x, y, z] 2 , [a , b, c, x, y, z] N . where N is the number of subregions generated by
[000150]いくつかの実施形態において、画像分析モジュールからのデータは、画像内の個々のピクセルを表す。当業者には当然のことながら、特定の副領域内のすべてのピクセルのデータは、平均化によって、副領域内のピクセルデータの平均値を与え得る。たとえば、個々のピクセルはそれぞれ、一定の強度を有していてもよい。特定の副領域におけるすべてのピクセルの強度は、平均化によって、当該副領域の平均ピクセル強度を与え得る。当該副領域の当該平均ピクセルは、当該副領域の表現オブジェクトと関連付けられていてもよく、また、当該データが一体としてメモリに格納されていてもよい。 [000150] In some embodiments, the data from the image analysis module represents individual pixels within the image. Those skilled in the art will appreciate that the data for all pixels within a particular sub-region can be averaged to give the average value of the pixel data within the sub-region. For example, each individual pixel may have a constant intensity. The intensities of all pixels in a particular sub-region may be averaged to give the average pixel intensity for that sub-region. The average pixel of the sub-region may be associated with the representational object of the sub-region, and the data may be stored together in memory.
[000151]FAPによる染色に関して、FAP陽性面積は、別の特徴/測定結果がスーパーピクセルオブジェクトに付与され得る。FAP陽性面積は、設定閾値を上回るFAP強度を有するピクセルの合計を参照する。閾値の選択については、Auranuch Lorsakul et al.「Automated whole-slide analysis of multiplex-brightfield IHC images for cancer cells and carcinoma-associated fibroblasts(がん細胞およびがん関連線維芽細胞に関する多重化-明視野IHC画像の自動全スライド分析)」,Proc.SPIE 10140,Medical Imaging 2017:Digital Pathology,1014007(2017/03/01)に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。 [000151] With respect to staining with FAP, the FAP-positive area may be given another feature/measurement to the superpixel object. FAP positive area refers to the sum of pixels with FAP intensity above a set threshold. For threshold selection, see Auranuch Lorsakul et al. "Automated whole-slide analysis of multiplex-brightfield IHC images for cancer cells and carcinoma-associated fibroblasts", Proc. SPIE 10140, Medical Imaging 2017: Digital Pathology, 1014007 (03/01/2017), the entire disclosure of which is incorporated herein by reference.
[000152]標識化モジュールにより格納されるデータの一例として、また、FAPバイオマーカによる生物学的サンプルの染色に関して、副領域内のFAPステインの平均強度は、特定の副領域の画像分析により導出されるようになっていてもよく、また、当該FAPステイン強度は、当該副領域の任意の表現オブジェクトの座標と併せて、データベースに格納されるようになっていてもよい。同様に、画像分析によって、FAP発現スコア等、副領域の特定の発現スコアが導出されるようになっていてもよく、また、当該副領域の当該FAP発現スコアは、当該特定の副領域の表現オブジェクトと併せて格納されるようになっていてもよい。任意の副領域内の画像部分の平均強度スコアおよび平均発現スコアのほか、他のパラメータが格納されるようになっていてもよく、種子点間の距離、識別腫瘍細胞と不規則形状の細胞との間の距離(たとえば、腫瘍細胞と線維芽細胞との間の距離)、およびFAP陽性面積が挙げられるが、これらに限定されない。 [000152] As an example of the data stored by the labeling module, and for staining a biological sample with a FAP biomarker, the average intensity of FAP stain within a sub-region is derived by image analysis of a particular sub-region. and the FAP stain intensity may be stored in a database along with the coordinates of any representational object in the sub-region. Similarly, the image analysis may be adapted to derive a particular expression score for a subregion, such as a FAP expression score, and the FAP expression score for that subregion is a representation of that particular subregion. It may be stored together with the object. Besides the average intensity score and average expression score of image portions within any sub-region, other parameters may be stored, such as the distance between seed points, distinguishing between tumor cells and irregularly shaped cells. (eg, distance between tumor cells and fibroblasts), and FAP-positive area.
[000153]いくつかの実施形態において、一例としては、対応するスーパーピクセル内で演算された分析結果(たとえば、平均局所強度、ポジティブ染色面積)がそれぞれの対応するポリゴン外形および種子に付与される。全スライド画像の場合は、それぞれの分析結果が付与されたこれら表現オブジェクト(たとえば、ポリゴン外形および種子)がxy座標にてデータベースに格納される。図10Aは、線維芽細胞(1010)用の線維芽細胞活性化タンパク質(FAP)により紫色に染色され、上皮腫瘍(1020)用のパンサイトケラチン(PanCK)により黄色に染色された頭頚部がん組織の全スライドIHC画像の一例を示している。図10Bおよび図11はそれぞれ、データベースに格納可能な線維芽細胞領域に属するスーパーピクセルの分析結果が付与されたポリゴン外形および種子の例を示している。 [000153] In some embodiments, by way of example, analysis results (eg, average local intensity, positive staining area) computed within the corresponding superpixel are applied to each corresponding polygon outline and seed. For full slide images, these representational objects (eg, polygon outlines and seeds) with their respective analysis results are stored in the database in xy coordinates. FIG. 10A Head and neck cancer stained purple with fibroblast activation protein (FAP) for fibroblasts (1010) and pancytokeratin (PanCK) for epithelial tumors (1020) yellow. An example of a whole-slide IHC image of tissue is shown. Figures 10B and 11 show examples of polygonal outlines and seeds, respectively, annotated with analysis results of superpixels belonging to fibroblast regions that can be stored in the database.
[000154]データ読み出しまたは投影モジュール
[000155]当業者には当然のことながら、格納された分析結果および関連する生物学的特徴は、後で読み出し可能であり、データは、さまざまなフォーマット(たとえば、分析結果のヒストグラムプロット)で報告または視覚化されるようになっていてもよい。より具体的に、表現オブジェクトの座標データおよび関連する画像分析データは、データベース209から読み出され、別途分析に用いられるようになっていてもよい。いくつかの実施形態において、また、一例として、表現オブジェクトは、全スライド画像内またはユーザアノテーション領域中の分析結果の視覚化または報告のため、データベースから読み出し可能である。図12に示されるように、全スライドスーパーピクセルから読み出されたFAP強度のヒストグラム中のプロットによって、関連付けまたは付与された画像分析結果を報告可能である。あるいは、全スライド画像、視野画像、または別途精査用に医療専門家によりアノテーションされた画像の一部においてデータを視覚化可能である。
[000154] Data Readout or Projection Module
[000155] Those skilled in the art will appreciate that the stored analytical results and associated biological features can be later retrieved and the data reported in a variety of formats (e.g., histogram plots of analytical results). Or it may be visualized. More specifically, representational object coordinate data and associated image analysis data may be retrieved from
[000156]本開示の実施形態を実現する他の構成要素
[000157]本開示のシステム200は、組織標本に対して1つまたは複数の作成プロセスを実行可能な標本処理装置に接続されていてもよい。作成プロセスとしては、標本の脱パラフィン化、標本の調節(たとえば、細胞調節)、標本の染色、抗原読み出しの実行、免疫組織化学染色(標識化を含む)もしくは他の反応の実行、ならびに/またはin-situハイブリダイゼーション(たとえば、SISH、FISH等)染色(標識化を含む)もしくは他の反応の実行のほか、顕微鏡法、微量分析法、質量分光法、または他の分析方法のために標本を作成する他のプロセスが挙げられるが、これらに限定されない。
[000156] Other components that implement embodiments of the present disclosure
[000157] The
[000158]処理装置は、固定剤を標本に塗布可能である。固定剤としては、架橋剤(アルデヒド(たとえば、ホルムアルデヒド、パラホルムアルデヒド、およびグルタルアルデヒド)のほか、非アルデヒド架橋剤等)、酸化剤(たとえば、四酸化オスミウムおよびクロム酸等の金属イオンおよび錯体)、タンパク質変性剤(たとえば、酢酸、メタノール、およびエタノール)、メカニズムが未知の固定剤(たとえば、塩化第二水銀、アセトン、およびピクリン酸)、組み合わせ試薬(たとえば、カルノワ固定剤、メタカン、ブアン液、B5固定剤、ロスマン液、およびジャンドル液)、マイクロ波、および混合固定剤(たとえば、排除体積固定剤および蒸気固定剤)が挙げられる。 [000158] The processing device is capable of applying a fixative to the specimen. Fixatives include cross-linking agents (such as aldehydes (e.g., formaldehyde, paraformaldehyde, and glutaraldehyde), as well as non-aldehyde cross-linkers), oxidizing agents (e.g., metal ions and complexes such as osmium tetroxide and chromic acid), protein denaturants (e.g., acetic acid, methanol, and ethanol), fixatives of unknown mechanism (e.g., mercuric chloride, acetone, and picric acid), combination reagents (e.g., Carnoys fixative, methacan, Bouin's solution, B5 fixatives, Rothmann's solution, and Jandl's solution), microwave, and mixed fixatives (eg, excluded volume fixatives and vapor fixatives).
[000159]標本がパラフィンに埋め込まれたサンプルの場合、このサンプルは、適当な脱パラフィン化液を用いることにより脱パラフィン化され得る。パラフィンが除去された後は、任意数の物質が連続して標本に適用され得る。これらの物質としては、前処理用(たとえば、タンパク質架橋の逆転、核酸の曝露等)、変性用、ハイブリダイゼーション用、洗浄用(たとえば、ストリンジェンシ洗浄)、検出用(たとえば、視覚またはマーカ分子のプローブへのリンク)、増幅用(たとえば、タンパク質、遺伝子等の増幅)、カウンタ染色用、封入用等が可能である。 [000159] If the specimen is a paraffin-embedded sample, the sample may be deparaffinized by using a suitable deparaffinizing solution. Any number of substances may be applied to the specimen in succession after the paraffin is removed. These substances include pretreatment (e.g., reversal of protein cross-links, exposure of nucleic acids, etc.), denaturation, hybridization, washing (e.g., stringency washing), detection (e.g., visual or marker molecules). probes), for amplification (eg, amplification of proteins, genes, etc.), for counter staining, for encapsulation, and the like.
[000160]標本処理装置は、広範な物質を標本に適用可能である。これらの物質としては、ステイン、プローブ、試薬、洗浄液、および/または調節剤が挙げられるが、これらに限定されない。これらの物質としては、流体(たとえば、気体、液体、または気体/液体混合物)等が可能である。流体としては、溶媒(たとえば、極性溶媒、非極性溶媒等)、溶液(たとえば、水溶液または他種の溶液)等が可能である。試薬としては、ステイン、湿潤剤、抗体(たとえば、モノクローナル抗体、ポリクローナル抗体等)、抗原回復液(たとえば、水性または非水性の抗原緩衝液、抗原回復緩衝剤等)が挙げられるが、これらに限定されない。プローブとしては、検出可能な標識またはレポータ分子に付着した単離核酸または単離合成オリゴヌクレオチドが可能である。標識としては、放射性同位体、酵素基質、補因子、リガンド、化学発光または蛍光剤、ハプテン、および酵素が挙げられる。 [000160] The specimen processor is capable of applying a wide range of substances to the specimen. These substances include, but are not limited to, stains, probes, reagents, washes, and/or modifiers. These substances can be fluids (eg, gases, liquids, or gas/liquid mixtures), and the like. Fluids can be solvents (eg, polar solvents, non-polar solvents, etc.), solutions (eg, aqueous or other types of solutions), and the like. Reagents include, but are not limited to, stains, wetting agents, antibodies (eg, monoclonal antibodies, polyclonal antibodies, etc.), antigen retrieval solutions (eg, aqueous or non-aqueous antigen buffers, antigen retrieval buffers, etc.). not. A probe can be an isolated nucleic acid or an isolated synthetic oligonucleotide attached to a detectable label or reporter molecule. Labels include radioisotopes, enzyme substrates, cofactors, ligands, chemiluminescent or fluorescent agents, haptens, and enzymes.
[000161]標本処理装置としては、Ventana Medical Systems,Inc.が販売するBENCHMARK XT器具およびSYMPHONY器具のような自動化装置が可能である。Ventana Medical Systems,Inc.は、自動分析を実行するシステムおよび方法を開示した多くの米国特許の譲受人であり、米国特許第5,650,327号、第5,654,200号、第6,296,809号、第6,352,861号、第6,827,901号、および第6,943,029号、ならびに米国特許出願公開第2003/0211630号および第2004/0052685号を含み、それぞれのすべての内容が参照により本明細書に組み込まれる。あるいは、標本は、手動で処理され得る。 [000161] Specimen processors include Ventana Medical Systems, Inc.; Automated devices such as the BENCHMARK XT instrument and the SYMPHONY instrument sold by Co., Inc. are possible. Ventana Medical Systems, Inc. is the assignee of a number of U.S. patents that disclose systems and methods for performing automated analysis, including U.S. Patent Nos. 5,650,327; 6,352,861, 6,827,901, and 6,943,029, and U.S. Patent Application Publication Nos. 2003/0211630 and 2004/0052685, the entire contents of each of which are incorporated by reference. incorporated herein by. Alternatively, specimens can be processed manually.
[000162]標本が処理された後、ユーザは、標本支持スライドを撮像装置に移送することができる。いくつかの実施形態において、撮像装置は、明視野撮像スライドスキャナである。明視野撮像装置の1つとして、Ventana Medical Systems,Inc.が販売するiScan HT and DP200(Griffin)明視野スキャナがある。自動化された実施形態において、撮像装置は、「IMAGING SYSTEM AND TECHNIQUES(撮像システムおよび技法)」という名称の国際特許出願PCT/US2010/002772号(特許公開WO2011/049608)または2011年9月9日に出願された「IMAGING SYSTEMS,CASSETTES, AND METHODS OF USING THE SAME(撮像システム、カセット、およびこれらの使用方法)」という名称の米国特許出願第61/533,114号に開示されるようなデジタル病理学デバイスである。国際特許出願PCT/US2010/002772および米国特許出願第61/533,114号のすべてが参照により本明細書に組み込まれる。 [000162] After the specimen has been processed, the user can transfer the specimen support slide to the imaging device. In some embodiments, the imaging device is a bright field imaging slide scanner. As one of the bright field imaging devices, Ventana Medical Systems, Inc. There are iScan HT and DP200 (Griffin) bright field scanners sold by . In an automated embodiment, the imaging device is disclosed in International Patent Application No. PCT/US2010/002772 entitled "IMAGING SYSTEM AND TECHNIQUES" (Patent Publication No. WO2011/049608) or dated September 9, 2011. Digital Pathology as disclosed in U.S. Patent Application Serial No. 61/533,114 entitled "IMAGING SYSTEMS, CASSETTES, AND METHODS OF USING THE SAME" filed. Device. International Patent Application PCT/US2010/002772 and US Patent Application No. 61/533,114 are all incorporated herein by reference.
[000163]撮像システムまたは装置は、マルチスペクトル撮像(MSI)システムまたは蛍光顕微鏡システムであってもよい。ここで用いられる撮像システムは、MSIである。MSIは一般的に、ピクセルレベルで画像のスペクトル分布にアクセス可能とすることにより、コンピュータ化顕微鏡ベースの撮像システムを病理標本の分析に備える。多様なマルチスペクトル撮像システムが存在するが、これらのシステムすべてに共通する動作的態様は、マルチスペクトル画像を構成する能力である。マルチスペクトル画像とは、電磁スペクトル全体の特定の波長または特定のスペクトル帯域幅において画像データを取り込んだものである。これらの波長は、光学フィルタまたは赤外線(IR)等の可視光範囲を超える波長における電磁放射光線を含む所定のスペクトル成分を選択可能な他の器具の使用により選別されるようになっていてもよい。 [000163] The imaging system or device may be a multispectral imaging (MSI) system or a fluorescence microscopy system. The imaging system used here is MSI. MSI generally equips a computerized microscope-based imaging system for the analysis of pathological specimens by making the spectral distribution of the image accessible at the pixel level. Although there are a variety of multispectral imaging systems, a common operational aspect of all these systems is the ability to construct multispectral images. A multispectral image is one that captures image data at specific wavelengths or spectral bandwidths across the electromagnetic spectrum. These wavelengths may be filtered out through the use of optical filters or other instruments capable of selecting predetermined spectral components, including electromagnetic radiation at wavelengths beyond the visible light range, such as infrared (IR). .
[000164]MSIシステムとしては、光学撮像システムが挙げられ、その一部が、所定数Nの離散光帯域を規定するように調節可能なスペクトル選択システムを含む。この光学システムは、光検出器上に広帯域光源で透過照射される組織サンプルを撮像するように構成されていてもよい。光学撮像システムは、一実施形態において、たとえば顕微鏡等の拡大システムを含んでいてもよく、当該光学システムの1つの光出力と空間的に大略位置合わせされた1本の光軸を有する。このシステムは、異なる離散スペクトル帯において画像が取得されるように(たとえば、コンピュータプロセッサによって)スペクトル選択システムが調整または調節されるように、組織の一連の画像を構成する。また、この装置は、ディスプレイも含んでいてもよく、このディスプレイにおいて、取得された一連の画像から少なくとも1つの視覚的に知覚可能な組織の画像が現れる。スペクトル選択システムは、回折格子、薄膜干渉フィルタ等の一群の光学フィルタ、またはユーザ入力もしくは予めプログラムされたプロセッサのコマンドのいずれかに応答して、光源からサンプルを通じて検出器へと透過した光のスペクトルから特定の通過帯域を選択するように構成されたその他任意のシステム等、光分散要素を含んでいてもよい。 [000164] MSI systems include optical imaging systems, some of which include spectral selection systems that are adjustable to define a predetermined number N of discrete optical bands. The optical system may be configured to image a tissue sample transilluminated with a broadband light source onto a photodetector. The optical imaging system, in one embodiment, may include a magnifying system, such as a microscope, having a single optical axis generally spatially aligned with a single optical output of the optical system. The system constructs a series of images of tissue such that the spectral selection system is tuned or adjusted (eg, by a computer processor) such that images are acquired in different discrete spectral bands. The device may also include a display on which at least one visually perceptible image of tissue appears from the sequence of acquired images. The spectral selection system selects the spectrum of light transmitted from the source through the sample to the detector in response to either a diffraction grating, a group of optical filters such as thin film interference filters, or user input or preprogrammed processor commands. It may also include an optical dispersive element, such as any other system configured to select a particular passband from .
[000165]代替実施態様において、スペクトル選択システムは、N個の離散スペクトル帯に対応する複数の光出力を規定する。この種のシステムは、光学システムからの透過光出力を取り込み、識別されたスペクトル帯において、この識別されたスペクトル帯に対応する光路に沿ってサンプルを検出システム上で撮像するように、N本の空間的に異なる光路に沿って、この光出力の少なくとも一部を空間的に方向転換させる。 [000165] In an alternative embodiment, the spectral selection system defines a plurality of light outputs corresponding to N discrete spectral bands. A system of this type captures the transmitted light output from the optical system and, in an identified spectral band, images the sample onto the detection system along an optical path corresponding to the identified spectral band. At least a portion of this light output is spatially redirected along a spatially distinct optical path.
[000166]本明細書に記載の主題および動作の実施形態は、デジタル電子回路またはコンピュータソフトウェア、ファームウェア、もしくはハードウェア(本明細書に開示の構造およびその構造的同等物を含む)、あるいはこれらのうちの1つまたは複数の組み合わせにて実装され得る。本明細書に記載の主題の実施形態は、1つまたは複数のコピュータプログラム、すなわち、データ処理装置による実行またはデータ処理装置の動作の制御ためにコンピュータ記憶媒体上に符号化されたコンピュータプログラム命令の1つまたは複数のモジュールとして実装され得る。本明細書に記載のモジュールはいずれも、プロセッサにより実行されるロジックを含んでいてもよい。本明細書において、「ロジック(logic)」は、プロセッサの動作に影響を及ぼすように適用され得る命令信号および/またはデータの形態を有する如何なる情報をも表す。ソフトウェアは、ロジックの一例である。 [000166] Embodiments of the subject matter and operations described herein may be digital electronic circuits or computer software, firmware, or hardware (including the structures disclosed herein and their structural equivalents), or any of these. can be implemented in one or more combinations of Embodiments of the subject matter described herein comprise one or more computer programs, i.e., computer program instructions encoded on a computer storage medium for execution by or controlling operation of a data processing apparatus. May be implemented as one or more modules. Any of the modules described herein may include logic that is executed by a processor. As used herein, "logic" refers to any information in the form of instruction signals and/or data that can be applied to affect the operation of a processor. Software is an example of logic.
[000167]コンピュータ記憶媒体としては、コンピュータ可読記憶装置、コンピュータ可読記憶基板、ランダムもしくは順次アクセス・メモリアレイもしくはデバイス、またはこれらのうちの1つまたは複数の組み合わせも可能であるし、これらに含まれることも可能である。さらに、コンピュータ記憶媒体は、伝搬信号ではないが、人工的に生成された伝搬信号として符号化されたコンピュータプログラム命令の供給源または宛先が可能である。また、コンピュータ記憶媒体としては、1つまたは複数の別個の物理的構成要素または媒体(たとえば、複数のCD、ディスク、または他の記憶装置)も可能であるし、これらに含まれることも可能である。本明細書に記載の動作は、1つまたは複数のコンピュータ可読記憶装置に格納されたデータまたは他の供給源から受信されたデータに対してデータ処理装置により実行される動作として実装され得る。 [000167] Computer storage media can also be and include computer readable storage devices, computer readable storage substrates, random or sequential access memory arrays or devices, or combinations of one or more of these. is also possible. Moreover, a computer storage medium can be a source or destination of computer program instructions encoded in an artificially generated propagated signal, rather than a propagated signal. A computer storage medium can also be or be included in one or more separate physical components or media (eg, multiple CDs, disks, or other storage devices). be. The operations described herein may be implemented as operations performed by a data processing apparatus on data stored in one or more computer readable storage devices or received from other sources.
[000168]用語「プログラムされたプロセッサ」は、データを処理するためのあらゆる種類の装置、デバイス、および機械を含み、一例として、プログラム可能なマイクロプロセッサ、コンピュータ、システム・オン・チップ、またはこれらのうちの複数、もしくは組み合わせが挙げられる。装置としては、専用論理回路(たとえば、FPGA(フィールドプログラマブルゲートアレイ)またはASIC(特定用途向け集積回路))が挙げられる。また、装置としては、ハードウェアのほか、対象のコンピュータプログラムのための実行環境を生成するコード(たとえば、プロセッサファームウェア、プロトコルスタック、データベース管理システム、オペレーティングシステム、クロスプラットフォーム・ランタイム環境、仮想機械、またはこれらのうちの1つもしくは複数の組み合わせを構成するコード)が挙げられる。この装置および実行環境は、ウェブサービス、分散型コンピューティング、およびグリッドコンピューティング・インフラストラクチャ等、種々異なるコンピューティングモデル・インフラストラクチャを実現することができる。 [000168] The term "programmed processor" includes all kinds of apparatus, devices, and machines for processing data, including, by way of example, programmable microprocessors, computers, systems-on-a-chip, or A plurality of them or a combination thereof can be mentioned. Devices include dedicated logic circuits, such as FPGAs (Field Programmable Gate Arrays) or ASICs (Application Specific Integrated Circuits). In addition to hardware, the device includes code that generates an execution environment for the target computer program (for example, processor firmware, protocol stack, database management system, operating system, cross-platform runtime environment, virtual machine, or A code that constitutes one or more combinations of these). The device and execution environment can implement different computing model infrastructures, such as web services, distributed computing, and grid computing infrastructures.
[000169]コンピュータプログラム(プログラム、ソフトウェア、ソフトウェアアプリケーション、スクリプト、またはコードとしても知られる)は、コンパイラ型またはインタープリタ型言語、宣言型または手続き型言語等、如何なる形態のプログラミング言語でも記述可能であり、また、単体プログラムまたはモジュール、コンポーネント、サブルーチン、オブジェクト、もしくはコンピューティング環境における使用に適した他のユニット等、如何なる形態でも展開可能である。コンピュータプログラムは、ファイルシステムのファイルに対応していてもよいが、必ずしもその必要はない。プログラムは、他のプログラムもしくはデータ(たとえば、マークアップ言語文書に格納された1つまたは複数のスクリプト)を保持するファイルの一部、対象のプログラムに専用の1つのファイル、または複数の調整されたファイル(たとえば、1つもしくは複数のモジュール、サブプログラム、もしくはコードの一部を格納したファイル)に格納され得る。コンピュータプログラムは、1つのコンピュータ上または1つのサイトに位置付けられた複数のコンピュータもしくは複数のサイトに分散され、通信ネットワークにより相互接続された複数のコンピュータ上で実行されるように展開され得る。 [000169] A computer program (also known as a program, software, software application, script, or code) can be written in any form of programming language, such as a compiled or interpreted language, declarative or procedural language, It may also be deployed in any form, such as a single program or module, component, subroutine, object, or other unit suitable for use in a computing environment. Computer programs may, but need not, correspond to files in a file system. A program may be part of a file holding other programs or data (e.g., one or more scripts stored in a markup language document), a single file dedicated to the program of interest, or multiple coordinated It may be stored in a file (eg, a file containing one or more modules, subprograms, or portions of code). A computer program can be deployed to be executed on one computer or on multiple computers located at one site or distributed across multiple sites and interconnected by a communications network.
[000170]本明細書に記載のプロセスおよびロジックフローは、1つまたは複数のコンピュータプログラムを実行し、入力データに対する動作および出力の生成によって動作を実行する1つまたは複数のプログラム可能なプロセッサにより実行され得る。これらのプロセスおよびロジックフローは、専用論理回路(たとえば、FPGA(フィールドプログラマブルゲートアレイ)またはASIC(特定用途向け集積回路))により実行され得る。また、装置は、専用論理回路(たとえば、FPGA(フィールドプログラマブルゲートアレイ)またはASIC(特定用途向け集積回路))として実装され得る。 [000170] The processes and logic flows described herein are performed by one or more programmable processors executing one or more computer programs and performing operations on input data and generating output. can be These processes and logic flows may be performed by dedicated logic circuits, such as FPGAs (Field Programmable Gate Arrays) or ASICs (Application Specific Integrated Circuits). Also, the apparatus may be implemented as dedicated logic circuits, such as FPGAs (Field Programmable Gate Arrays) or ASICs (Application Specific Integrated Circuits).
[000171]コンピュータプログラムの実行に適したプロセッサとしては、一例として、汎用および専用マイクロプロセッサの両者、ならびに任意の種類のデジタルコンピュータの任意の1つまたは複数のプロセッサが挙げられる。一般的に、プロセッサは、リードオンリーメモリ、ランダムアクセスメモリ、またはその両者から命令およびデータを受信することになる。コンピュータの必須要素は、命令に従って動作を実行するプロセッサならびに命令およびデータを格納する1つもしくは複数のメモリデバイスである。また、一般的に、コンピュータは、データを格納する1つまたは複数の大容量記憶装置(たとえば、磁気、光磁気ディスク、または光ディスク)を含むか、あるいは、1つまたは複数の大容量記憶装置に対するデータの受信、送信、または両者を行うように動作結合されることになる。ただし、コンピュータは、そのようなデバイスを必ずしも有する必要がない。さらに、コンピュータは、別のデバイス(たとえば、携帯電話、携帯情報端末(PDA)、モバイルオーディオもしくはビデオプレーヤ、ゲームコンソール、全地球測位システム(GPS)受信機、または携帯型記憶装置(たとえば、ユニバーサルシリアルバス(USB)フラッシュドライブ)、あるいはその他多くのデバイス)に埋め込まれ得る。コンピュータプログラム命令およびデータを記憶するのに適したデバイスとしては、あらゆる形態の不揮発性メモリ、媒体、およびメモリデバイスが挙げられ、一例として、半導体メモリデバイス(たとえば、EPROM、EEPROM、およびフラッシュメモリデバイス)、磁気ディスク(たとえば、内部ハードディスクもしくはリムーバブルディスク)、光磁気ディスク、ならびにCD-ROMおよびDVD-ROMディスクを含む。プロセッサおよびメモリは、専用論理回路による補完または専用論理回路への組み込みがなされ得る。 [000171] Processors suitable for the execution of a computer program include, by way of example, both general and special purpose microprocessors, and any one or more processors of any kind of digital computer. Generally, a processor will receive instructions and data from read-only memory, random-access memory, or both. The essential elements of a computer are a processor, which performs operations according to instructions, and one or more memory devices, which store instructions and data. Also, computers typically include, or have access to, one or more mass storage devices (e.g., magnetic, magneto-optical or optical discs) for storing data. It will be operatively coupled to receive data, transmit data, or both. However, a computer need not necessarily have such devices. Additionally, the computer may be connected to another device (e.g., a mobile phone, a personal digital assistant (PDA), a mobile audio or video player, a game console, a global positioning system (GPS) receiver, or a portable storage device (e.g., a universal serial It can be embedded in a bus (USB flash drive), or many other devices). Devices suitable for storing computer program instructions and data include all forms of non-volatile memory, media, and memory devices, including semiconductor memory devices (e.g., EPROM, EEPROM, and flash memory devices). , magnetic disks (eg, internal or removable disks), magneto-optical disks, and CD-ROM and DVD-ROM disks. The processor and memory may be supplemented by or embedded in dedicated logic circuitry.
[000172]ユーザとの相互作用を可能にするため、本明細書に記載の主題の実施形態は、ユーザに情報を表示する表示装置(たとえば、LCD(液晶ディスプレイ)、LED(発光ダイオード)ディスプレイ、もしくはOLED(有機発光ダイオード)ディスプレイ)ならびにユーザがコンピュータに入力を与え得るキーボードおよびポインティングデバイス(たとえば、マウスもしくはトラックボール)を有するコンピュータ上に実装され得る。いくつかの実施態様においては、情報の表示およびユーザからの入力の受け付けにタッチスクリーンが使用され得る。また、ユーザとの相互作用を可能にするため、他の種類のデバイスも同様に使用され得る。たとえば、ユーザに提供されるフィードバックとしては、任意の形態の感覚フィードバック(たとえば、視覚的フィードバック、聴覚的フィードバック、または触覚的フィードバック)が可能であり、ユーザからの入力は、音響入力、音声入力、または触覚入力等、任意の形態で受け取られ得る。また、コンピュータは、ユーザが使用するデバイスに対する文書の送信および受信(たとえば、ウェブブラウザから受けた要求に応じてユーザのクライアントデバイス上のウェブブラウザにウェブページを送ること)によって、ユーザと相互作用可能である。 [000172] To enable interaction with a user, embodiments of the subject matter described herein include display devices (e.g., LCD (Liquid Crystal Display), LED (Light Emitting Diode) displays, or on a computer having an OLED (organic light emitting diode) display) and a keyboard and pointing device (eg, mouse or trackball) through which the user may provide input to the computer. In some implementations, a touch screen may be used to display information and accept input from a user. Also, other types of devices may be used as well to allow interaction with the user. For example, the feedback provided to the user can be any form of sensory feedback (e.g., visual feedback, auditory feedback, or tactile feedback), and input from the user can be acoustic input, voice input, Or it can be received in any form, such as haptic input. Computers can also interact with users by sending and receiving documents to and from devices used by users (e.g., by sending web pages to a web browser on a user's client device in response to requests received from the web browser). is.
[000173]本明細書に記載の主題の実施形態は、バックエンドコンポーネント(たとえば、データサーバ)、ミドルウェアコンポーネント(たとえば、アプリケーションサーバ)、またはフロントエンドコンポーネント(たとえば、本明細書に記載の主題の一実施態様とユーザが相互作用し得るグラフィカルユーザインターフェースもしくはウェブブラウザを有するクライアントコンピュータ)を含むコンピュータシステム、あるいは1つまたは複数のこのようなバックエンド、ミドルウェア、またはフロントエンドコンポーネントの任意の組み合わせにて実現され得る。システムの構成要素は、如何なる形態または媒体のデジタルデータ通信(たとえば、通信ネットワーク)によっても相互接続され得る。通信ネットワークの例としては、ローカルエリアネットワーク(「LAN」)およびワイドエリアネットワーク(「WAN」)、相互接続ネットワーク(たとえば、インターネット)、ならびにピア・ツー・ピアネットワーク(たとえば、アドホック・ピア・ツー・ピアネットワーク)が挙げられる。たとえば、図1のネットワーク20は、1つまたは複数のローカルエリアネットワークを含み得る。
[000173] Embodiments of the subject matter described herein may include back-end components (eg, data servers), middleware components (eg, application servers), or front-end components (eg, one of the subject matter described herein). a client computer having a graphical user interface or web browser through which a user can interact with an embodiment), or any combination of one or more such back-end, middleware, or front-end components. can be The components of the system can be interconnected by any form or medium of digital data communication (eg, a communication network). Examples of communication networks include local area networks (“LAN”) and wide area networks (“WAN”), interconnected networks (eg, the Internet), and peer-to-peer networks (eg, ad-hoc peer-to-peer networks). peer network). For example,
[000174]コンピュータシステムは、如何なる数のクライアントおよびサーバをも含み得る。クライアントおよびサーバは一般的に、互いに遠隔であって、通常は、通信ネットワークを通じて相互作用する。クライアントおよびサーバの関係は、それぞれのコンピュータ上で実行され、互いにクライアント-サーバ関係を有するコンピュータプログラムによって生じる。いくつかの実施形態においては、サーバが(たとえば、クライアントデバイスと相互作用するユーザへのデータの表示およびユーザからのユーザ入力の受け付けを目的として)クライアントデバイスにデータ(たとえば、HTMLページ)を送信する。クライアントデバイスで生成されたデータ(たとえば、ユーザ相互作用の結果)は、サーバにおいてクライアントデバイスから受信され得る。 [000174] A computer system may include any number of clients and servers. A client and server are generally remote from each other and typically interact through a communication network. The relationship of client and server arises by virtue of computer programs running on the respective computers and having a client-server relationship to each other. In some embodiments, the server sends data (e.g., HTML pages) to the client device (e.g., for the purpose of displaying data to and accepting user input from users interacting with the client device). . Data generated at the client device (eg, results of user interactions) may be received from the client device at the server.
[000175]付加的な分離方法/任意選択的な分離モジュール
[000176]分離は、混合ピクセルの測定スペクトルが一群の構成スペクトルすなわち端成分および一組の対応する断片すなわち個体に分解されて、当該ピクセルに存在する各端成分の割合を示す手順である。具体的に、分離プロセスでは、標準型の組織およびステインの組み合わせに関して周知の基準スペクトルを用いて、ステイン固有のチャネルを抽出することにより、個々のステインの局所濃度を決定することができる。分離では、制御画像から読み出された基準スペクトルまたは観察中の画像から推定された基準スペクトルを使用するようにしてもよい。各入力ピクセルのコンポーネント信号を分離することによって、H&E画像におけるヘマトキシリンチャネルおよびエオシンチャネルまたはIHC画像におけるジアミノベンジジン(DAB)チャネルおよびカウンタステイン(たとえば、ヘマトキシリン)チャネル等、ステイン固有のチャネルを読み出しおよび分析可能となる。用語「分離(unmixing)」および「カラーデコンボリューション(color deconvolution)」(または、「デコンボリューション(deconvolution)」)等(たとえば、「deconvolving」、「unmixed」)は、当技術分野において区別なく使用される。いくつかの実施形態において、多重画像は、線形分離を用いる分離モジュールによって分離される。線形分離については、たとえばZimmermann「Spectral Imaging and Linear Unmixing in Light Microscopy(光学顕微鏡法におけるスペクトル撮像および線形分離)」,Adv Biochem Engin/Biotechnol(2005)95:245-265およびC.L.Lawson and R.J.Hanson「Solving least squares Problems(最小2乗問題の解法)」,PrenticeHall,1974,Chapter 23,p.161に記載されており、そのすべての開示内容が参照により本明細書に組み込まれる。線形ステイン分離においては、任意のピクセルにおける測定スペクトル(S(λ))がステインスペクトル成分の線形混合と考えられ、当該ピクセルにおいて表される個々のステインの色基準(R(λ))の割合または重み(A)の合計に等しい。
[000175] Additional Separation Methods/Optional Separation Modules
[000176] Separation is a procedure in which the measured spectrum of a blend pixel is decomposed into a set of constituent spectra, or endmembers, and a set of corresponding fragments, or individuals, to indicate the proportion of each endmember present in that pixel. Specifically, in the separation process, local concentrations of individual stains can be determined by extracting stain-specific channels using known reference spectra for standard tissue and stain combinations. Separation may use a reference spectrum read from the control image or estimated from the image under observation. By separating the component signals of each input pixel, stain-specific channels can be read out and analyzed, such as the hematoxylin and eosin channels in H&E images or the diaminobenzidine (DAB) and counterstain (e.g., hematoxylin) channels in IHC images becomes. The terms "unmixing" and "color deconvolution" (or "deconvolution") and the like (e.g., "deconvolving", "unmixed") are used interchangeably in the art. be. In some embodiments, multiple images are separated by a separation module using linear separation. For linear separation see, for example, Zimmermann "Spectral Imaging and Linear Unmixing in Light Microscopy", Adv Biochem Engineering/Biotechnol (2005) 95:245-265 and C.I. L. Lawson andR. J. Hanson, "Solving least squares Problems," Prentice Hall, 1974, Chapter 23, p. 161, the entire disclosure of which is incorporated herein by reference. In linear stain separation, the measured spectrum (S(λ)) at any pixel is considered a linear mixture of stain spectral components, and the proportion of the individual stain color references (R(λ)) represented at that pixel or Equal to the sum of the weights (A).
[000177]S(λ)=A1・R1(λ)+A2・R2(λ)+A3・R3(λ)・・・Ai・Ri(λ)
[000178]より一般的には、以下のような行列の形態で表し得る。
[000177] S(λ) = A 1 ·R 1 (λ) + A 2 ·R 2 (λ) + A 3 ·R 3 (λ) ... A i · R i (λ)
[000178] More generally, it can be represented in the form of a matrix as follows.
[000179]S(λ)=ΣAi・Ri(λ)またはS=R・A
[000180]取得されたM個のチャネル画像およびN個の個別ステインが存在する場合は、本明細書において導出される通り、M×N行列Rの列が最適色系であり、N×1ベクトルAが個別ステインの割合の未知数であり、M×1ベクトルSがピクセルにおいて測定されたマルチチャネルスペクトルベクトルである。これらの式において、各ピクセルの信号(S)は、多重画像および基準スペクトルすなわち最適色系の取得中に測定され、本明細書に記載の通りに導出される。さまざまなステインの寄与(Ai)は、測定スペクトルにおける各点への寄与を計算することによって決定可能である。いくつかの実施形態においては、以下一組の式を解くことにより測定スペクトルと計算スペクトルとの2乗差を最小化する逆最小2乗フィッティング手法を用いて解が得られる。
[000179] S(λ) = ΣA i R i (λ) or S = R A
[000180] If there are M acquired channel images and N individual stains, then the columns of the M×N matrix R are the optimal color system, and the N×1 vector A is the unknown of the percentage of individual stains and the M×1 vector S is the multichannel spectral vector measured at the pixel. In these equations, the signal (S) of each pixel is measured during the acquisition of multiple images and a reference spectrum or optimum color system and derived as described herein. The various stain contributions (A i ) can be determined by calculating the contribution to each point in the measured spectrum. In some embodiments, the solution is obtained using an inverse least-squares fitting technique that minimizes the squared difference between the measured and calculated spectra by solving the following set of equations.
[000181][∂Σj{S(λj)-ΣiAi・Ri(λj)}2]/∂Ai=0
[000182]この式において、jは検出チャネルの数を表し、iはステインの数に等しい。線形方程式の解では、条件付き分離によって、重み(A)の合計が1になることが多い。
[000181] [∂Σ j {S(λ j )−Σ i A i ·R i (λ j )} 2]/∂ A i =0
[000182] In this equation, j represents the number of detection channels and i equals the number of stains. In solutions of linear equations, the weights (A) often sum to 1 due to conditional separability.
[000183]他の実施形態においては、2014年5月28日に出願された「Image Adaptive Physiologically Plausible Color Separation(画像適応的な生理学的に妥当な色分離)」という名称のWO2014/195193に記載された方法を用いて分離が実現されるが、そのすべての開示内容が参照により本明細書に組み込まれる。一般的に、WO2014/195193は、反復的に最適化された基準ベクトルを用いて入力画像のコンポーネント信号を分離することによる分離方法を記載する。いくつかの実施形態においては、アッセイの特質に固有の予想結果または理想的結果に対して、アッセイからの画像データの相関によって、品質測定基準が決定される。低品質画像または理想的結果に対する相関が不十分な場合は、行列Rの1つまたは複数の基準列ベクトルが調整され、生理学的および解剖学的要件に整合する良質の画像を相関が示すまで、調整された基準ベクトルを用いて分離が反復的に繰り返される。測定画像データに適用されて品質測定基準を決定するルールを規定するのに、解剖学的情報、生理学的情報、およびアッセイ情報が用いられるようになっていてもよい。この情報には、組織の染色方法、染色を意図した組織内構造もしくは染色を意図しなかった組織内構造、ならびに処理対象のアッセイに固有の構造、ステイン、およびマーカ間の関係を含む。反復的プロセスによって、関心構造および生物学的に関連する情報を正確に識別し、ノイズも不要なスペクトルも一切ないため分析に適した画像を生成し得るステイン固有のベクトルが得られる。基準ベクトルは、探索空間内で調整される。探索空間は、基準ベクトルがステインを表すのに取り得る値の範囲を規定する。探索空間は、既知または一般に発生する問題を含む多様な代表的トレーニングアッセイをスキャンし、当該トレーニングアッセイに対して、高品質な基準ベクトル集合を決定することにより決定されるようになっていてもよい。 [000183] In another embodiment, the method described in WO 2014/195193 entitled "Image Adaptive Physiologically Plausible Color Separation" filed May 28, 2014. Separation is achieved using the method described in the entire disclosure of which is incorporated herein by reference. Generally, WO2014/195193 describes a separation method by separating component signals of an input image using iteratively optimized reference vectors. In some embodiments, the quality metric is determined by correlating image data from an assay against expected or ideal results inherent to the nature of the assay. For poor quality images or poor correlation for ideal results, one or more reference column vectors of matrix R are adjusted until the correlation indicates a good quality image that matches the physiological and anatomical requirements. Separation is iteratively repeated using the adjusted reference vector. Anatomical, physiological, and assay information may be used to define rules that are applied to the measured image data to determine quality metrics. This information includes how the tissue was stained, structures within the tissue intended to be stained or not intended to be stained, and relationships between structures, stains, and markers specific to the assay being processed. An iterative process yields stain-specific vectors that accurately identify the structures of interest and biologically relevant information, and that are free of noise and unwanted spectra and thus can produce images suitable for analysis. A reference vector is aligned within the search space. The search space defines the range of values that the reference vector can take to represent the stain. The search space may be determined by scanning a variety of representative training assays containing known or commonly occurring problems and determining a high quality reference vector set for the training assay. .
[000184]他の実施形態においては、2015年2月23日に出願された「Group Sparsity Model for Image Unmixing(画像分離のためのグループスパーシティモデル)」という名称のWO2015/124772に記載された方法を用いて分離が実現されるが、そのすべての開示内容が参照により本明細書に組み込まれる。一般的に、WO2015/124772は、グループスパーシティフレームワークを用いた分離を記載しており、複数のコロケーションマーカからのステイン寄与の割合が「同一グループ」内でモデル化され、複数の非コロケーションマーカからのステイン寄与の割合が異なるグループにおいてモデル化されて、複数のコロケーションマーカの共局在化情報をモデル化グループスパーシティフレームワークに与えるとともに、グループラッソを用いてモデル化フレームワークを解くことにより、各グループ内の最小2乗解を生み出す。この最小2乗解は、コロケーションマーカの分離に対応し、グループの中で、非コロケーションマーカの分離に対応するスパース解を生み出す。さらに、WO2015/124772は、生物学的組織サンプルから得られた画像データを入力し、複数のステインそれぞれのステイン色を記述した基準データを電子メモリから読み出し、生物学的組織サンプルにおいて局在化可能なステインをそれぞれ含み、グループラッソ基準のためのグループをそれぞれ構成し、少なくとも1つが2以上のサイズを有するステイン群を記述したコロケーションデータを電子メモリから読み出し、基準データを基準行列として用いることにより、分離画像を得るためのグループラッソ基準の解を計算することによる分離方法を記載する。いくつかの実施形態において、画像を分離する方法は、局在化マーカからのステイン寄与の割合が単一のグループ内で割り当てられ、非局在化マーカからのステイン寄与の割合が別個のグループ内で割り当てられるグループスパーシティモデルを生成するステップと、分離アルゴリズムを用いてグループスパーシティモデルを解くことにより、各グループ内で最小2乗解を生み出すステップとを含んでいてもよい。 [000184] In another embodiment, the method described in WO2015/124772 entitled "Group Sparsity Model for Image Unmixing" filed Feb. 23, 2015. is used to achieve the separation, the entire disclosure of which is incorporated herein by reference. In general, WO2015/124772 describes separation using a group sparsity framework, where the proportion of stain contributions from multiple collocation markers is modeled within the "same group" and multiple non-collocation markers are modeled in different groups to provide the colocalization information of multiple colocation markers to the modeling group sparsity framework, and by solving the modeling framework using the group lasso , yields the least-squares solution within each group. This least-squares solution produces a sparse solution corresponding to the separation of the collocation markers and the separation of the non-collocation markers within the group. Further, WO2015/124772 inputs image data obtained from a biological tissue sample and retrieves from an electronic memory reference data describing the stain color for each of a plurality of stains, which can be localized in the biological tissue sample. by reading from an electronic memory collocation data describing a group of stains each comprising a stain group, each comprising a group for group Lasso criteria, at least one of which has a size of 2 or more, and using the reference data as a reference matrix, A separation method is described by computing the solution of the group Lasso criterion to obtain separated images. In some embodiments, the method of separating images is such that the percentage of stain contribution from localized markers is assigned within a single group and the percentage of stain contribution from delocalized markers is assigned within a separate group. and solving the group sparsity model using a separation algorithm to yield a least-squares solution within each group.
[000185]実施例-高解像度分析法と中解像度分析法とのFAP陽性面積の比較
[000186]以下を用いた実験によって、FAP陽性面積の結果の精度を比較した。
[000187]1)FAP陽性高解像度分析。この測定のため、0.465マイクロメートルのピクセルサイズの空間解像度にて、高倍率(20X)で閾値化後のすべてのFAP陽性ピクセルが蓄積された。その後、関心領域のピクセルごとのFAP陽性面積として、予備アノテーション領域から選択された報告面積が求められた。
[000185] Example - Comparison of FAP positive area between high resolution and medium resolution methods
[000186] The precision of the FAP-positive area results was compared by experiments using:
[000187] 1) FAP positive high resolution analysis. For this measurement, all FAP-positive pixels after thresholding were accumulated at high magnification (20X) at a spatial resolution of 0.465 micrometer pixel size. The reported area selected from the pre-annotated regions was then determined as the FAP-positive area per pixel of the region of interest.
[000188]2)予備アノテーション領域において、FAPスーパーピクセルオブジェクト、種子、またはポリゴン外形のFAP陽性面積を合計することにより、本明細書に記載の中解像度分析手法を用いて測定されるFAP陽性面積が計算された。 [000188] 2) Summing the FAP-positive areas of the FAP superpixel objects, seeds, or polygon outlines in the pre-annotated region yields the FAP-positive areas measured using the medium-resolution analysis approach described herein. calculated.
[000189]両方法に従って、異なる形状(大小、円形、または変わった形状等)をそれぞれ有する6つの異なるアノテーションエリア(図14参照)が分析された。図15および以下の表に示される通り、これら2つの方法を用いて測定されたFAP陽性面積の比較結果に大きな差はなかった(R2=0.99、p<0.001)。 [000189] According to both methods, six different annotation areas (see Fig. 14), each with a different shape (large, small, circular, or odd shape, etc.) were analyzed. As shown in Figure 15 and the table below, there was no significant difference in the comparative results of the FAP-positive area measured using these two methods ( R2 = 0.99, p < 0.001).
[000190]結論として、特定のアノテーションにおいてスーパーピクセル内で演算された面積特徴を合計した場合、その面積の合計値は、当該アノテーション内で高解像度分析手法により直接計算した面積に等しい。FAP陽性面積の結果は、アノテーション領域が異なる形状での2つの方法(スーパーピクセルの有無)による演算には大きな差がないことを示している。 [000190] In conclusion, summing the area features computed within a superpixel in a particular annotation equals the area computed directly by the high-resolution analytical technique within that annotation. The FAP-positive area results show that there is no significant difference in the computation by the two methods (with and without superpixels) with different geometries of the annotation regions.
[000191]本明細書における言及および/または出願データシートにおける掲載の対象となる米国特許、米国特許出願公開、米国特許出願、外国特許、外国特許出願、および非特許刊行物はすべて、その全内容が参照により本明細書に組み込まれる。種々特許、出願、および公開の概念を採用する必要に応じて、さらに別の実施形態を提供するように、実施形態の態様が改良され得る。 [000191] All U.S. patents, U.S. patent application publications, U.S. patent applications, foreign patents, foreign patent applications, and non-patent publications referenced herein and/or published in application data sheets are is incorporated herein by reference. Aspects of the embodiments may be modified, as necessary to employ concepts of the various patents, applications, and publications, to provide yet further embodiments.
[000192]以上、多くの例示的実施形態を参照して本開示を説明したが、本開示の原理の主旨および範囲に含まれるその他多くの改良形態および実施形態が当業者により考案され得ることが了解されるものとする。より詳細には、本開示の主旨から逸脱することなく、上記開示内容、図面、および添付の特許請求の範囲内において、主題の組み合せ構成の構成要素部品および/または配置における合理的な変形および改良が可能である。構成要素部品および/または配置における変形および改良のほか、当業者には、代替的な使用も明らかとなるであろう。 [000192] Although the present disclosure has been described with reference to a number of exemplary embodiments, it is understood that many other modifications and embodiments within the spirit and scope of the principles of the present disclosure can be devised by those skilled in the art. shall be understood. More particularly, reasonable variations and modifications in the component parts and/or arrangements of the subject combination constructions may be made within the scope of the foregoing disclosure, drawings, and appended claims without departing from the spirit of the present disclosure. is possible. Variations and improvements in component parts and/or arrangements, as well as alternative uses, will become apparent to those skilled in the art.
Claims (32)
(a)前記画像から、1つまたは複数の特徴測定基準(feature metrics)を導出するステップと、
(b)前記画像を複数の副領域(sub-regions)にセグメント化するステップであり、各副領域が、染色有無、染色強度、または局所質感(local texture)のうちの少なくとも1つにおいて実質的に均一なピクセルを含み、副領域のセットの各副領域が、閾値を超えるステインの量を識別する、ステップと、
(c)前記複数の副領域の副領域のセットに対応する複数の表現オブジェクトを生成するステップであって、
前記複数の表現オブジェクトの各々の表現オブジェクトが、(i)特定の細胞の種類を識別し、(ii)前記副領域のセットの対応する副領域からトレースされた境界から生成されたポリゴンの外形によって定義される、
ステップと、
(d)前記複数の副領域を除外する前記画像の他の領域を除去するステップと、
(e)前記複数の表現オブジェクトそれぞれを、導出された、1つ又は複数の前記特徴測定基準と関連付けるステップと、
(f)前記関連付け導出特徴測定基準と併せて(along with)、前記複数の表現オブジェクトの各表現オブジェクトの座標をデータベースに格納するステップと、
を含む、方法。 1. A method of storing image analysis data derived from an image of a biological specimen having at least one stain, comprising:
(a) deriving one or more feature metrics from the image;
(b) segmenting the image into a plurality of sub-regions, each sub-region being substantially in at least one of the presence or absence of staining, staining intensity, or local texture; comprising uniform pixels in , each sub-region of the set of sub-regions identifying an amount of stain above a threshold ;
(c) generating a plurality of representation objects corresponding to a set of sub-regions of said plurality of sub-regions;
each representation object of the plurality of representation objects (i) identifies a particular cell type; and (ii) by a polygon outline generated from boundaries traced from corresponding subregions of the set of subregions . defined ,
a step;
(d) removing other regions of said image that exclude said plurality of sub-regions;
(e) associating each of the plurality of representational objects with one or more of the derived feature metrics;
(f) storing coordinates of each representation object of said plurality of representation objects in a database along with said association derived feature metric;
A method, including
(a)前記画像から、1つまたは複数の特徴測定基準を導出することと、
(b)前記画像内の複数の副領域を生成することであり、各副領域が、色、輝度、および/または質感から選択される類似特性を備えたピクセルを有し、副領域のセットの各副領域が、閾値を超えるステインの量を識別することと、
(c)前記複数の副領域の副領域のセットに対応する一連の表現オブジェクトを演算することであって、
前記一連の表現オブジェクトの各々の表現オブジェクトが、(i)特定の細胞の種類を識別し、(ii) 前記副領域のセットの対応する副領域からトレースされた境界から生成されたポリゴンの外形によって定義される、ことと、
(d)前記複数の副領域を除外する前記画像の他の領域を除去することと、
(e)前記一連の表現オブジェクトの各々に対して、前記画像から導出された前記1つ又は複数の特徴測定基準を、前記オブジェクトの位置を識別する計算座標と関連付けることと、
を含む動作を当該システムに実行させるコンピュータ実行可能命令を格納した、メモリと、を備えた、システム。 1. A system for deriving data corresponding to irregularly shaped cells from an image of a biological sample comprising at least one stain, comprising: (i) one or more processors; a memory coupled to a processor of, when executed by said one or more processors,
(a) deriving one or more feature metrics from the image;
(b) generating a plurality of sub-regions in said image, each sub-region having pixels with similar properties selected from color, brightness and/or texture, of a set of sub-regions; each subregion identifying an amount of stain above a threshold ;
(c) computing a sequence of representation objects corresponding to a set of sub-regions of said plurality of sub-regions;
each representation object of said series of representation objects (i) identifies a particular cell type; and (ii) by a polygon outline generated from boundaries traced from corresponding subregions of said set of subregions . be defined and
(d) removing other regions of the image that exclude the plurality of sub-regions;
(e) for each of said series of representational objects, associating said one or more feature metrics derived from said images with calculated coordinates that identify the location of said objects;
a memory containing computer-executable instructions that cause the system to perform operations including:
(a)生物学的サンプルの画像から1つまたは複数の特徴測定基準を導出する命令であり、前記生物学的サンプルが、少なくとも1つのステイン(stain)を含む、命令と、
(b)類似特性を有するピクセルのグループ化によって前記画像を一連の副領域に分割する命令であり、前記類似特性が、色、輝度、および/または質感から選択され、前記一連の副領域の各副領域が、閾値を超えるステインの量を識別する、命令と、
(c)前記一連の副領域の副領域のセットに対応する複数の表現オブジェクトを演算する命令と、
前記一連の表現オブジェクトの各々の表現オブジェクトが、(i)特定の細胞の種類を識別し、(ii) 前記副領域のセットの対応する副領域からトレースされた境界から生成されたポリゴンの外形によって定義され、
(d)前記複数の副領域を除外する前記画像の他の領域を除去するための命令と、
(e)前記複数の表現オブジェクトの各々に対して、前記画像から導出された前記1つまたは複数の特徴測定基準を、前記表現オブジェクトの位置を識別する計算座標と関連付ける命令と、
を含む、非一時的コンピュータ可読媒体。 A non-transitory computer-readable medium storing instructions for analyzing data associated with an irregularly shaped living body, the instructions comprising:
(a) instructions for deriving one or more feature metrics from an image of a biological sample, said biological sample comprising at least one stain;
(b) dividing said image into a series of sub- regions by grouping pixels having similar properties, said similar properties being selected from color, brightness and/or texture; instructions , wherein the subregion identifies an amount of stain above a threshold ;
(c) an instruction to compute a plurality of representation objects corresponding to a set of sub-regions of said sequence of sub-regions;
each representation object of said series of representation objects (i) identifies a particular cell type; and (ii) by a polygon outline generated from boundaries traced from corresponding subregions of said set of subregions . defined ,
(d) instructions for removing other regions of the image that exclude the plurality of sub-regions;
(e) for each of said plurality of representational objects, instructions for associating said one or more feature metrics derived from said image with calculated coordinates identifying a position of said representational object;
A non-transitory computer-readable medium, including
アルゴリズム、または単純線形反復クラスタリングアルゴリズムのうちの1つを用いて演算される、請求項23に記載の非一時的コンピュータ可読媒体。 23. The superpixels are computed using one of a normalized cuts algorithm, an agglomerative clustering algorithm, a quick shift algorithm, a turbo pixel algorithm, or a simple linear iterative clustering algorithm. The non-transitory computer-readable medium as described in .
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2022200094A JP7558242B2 (en) | 2017-12-06 | 2022-12-15 | Method for storing and retrieving digital pathology analysis results - Patents.com |
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201762595143P | 2017-12-06 | 2017-12-06 | |
| US62/595,143 | 2017-12-06 | ||
| PCT/EP2018/083434 WO2019110561A1 (en) | 2017-12-06 | 2018-12-04 | Method of storing and retrieving digital pathology analysis results |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2022200094A Division JP7558242B2 (en) | 2017-12-06 | 2022-12-15 | Method for storing and retrieving digital pathology analysis results - Patents.com |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2021506003A JP2021506003A (en) | 2021-02-18 |
| JP7197584B2 true JP7197584B2 (en) | 2022-12-27 |
Family
ID=64604651
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2020530584A Active JP7197584B2 (en) | 2017-12-06 | 2018-12-04 | Methods for storing and retrieving digital pathology analysis results |
| JP2022200094A Active JP7558242B2 (en) | 2017-12-06 | 2022-12-15 | Method for storing and retrieving digital pathology analysis results - Patents.com |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2022200094A Active JP7558242B2 (en) | 2017-12-06 | 2022-12-15 | Method for storing and retrieving digital pathology analysis results - Patents.com |
Country Status (4)
| Country | Link |
|---|---|
| EP (1) | EP3721372A1 (en) |
| JP (2) | JP7197584B2 (en) |
| CN (2) | CN117038018A (en) |
| WO (1) | WO2019110561A1 (en) |
Families Citing this family (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN112347823B (en) * | 2019-08-09 | 2024-05-03 | 中国石油天然气股份有限公司 | Deposition phase boundary identification method and device |
| EP4022286A1 (en) | 2019-08-28 | 2022-07-06 | Ventana Medical Systems, Inc. | Label-free assessment of biomarker expression with vibrational spectroscopy |
| WO2021167984A1 (en) * | 2020-02-17 | 2021-08-26 | 10X Genomics, Inc. | Systems and methods for machine learning features in biological samples |
| CN112070041B (en) * | 2020-09-14 | 2023-06-09 | 北京印刷学院 | Living body face detection method and device based on CNN deep learning model |
| CN112329765B (en) * | 2020-10-09 | 2024-05-24 | 中保车服科技服务股份有限公司 | Text detection method and device, storage medium and computer equipment |
| WO2022107435A1 (en) * | 2020-11-20 | 2022-05-27 | コニカミノルタ株式会社 | Image analysis method, image analysis system, and program |
| CN112785713B (en) * | 2021-01-29 | 2024-06-14 | 广联达科技股份有限公司 | Method, device, equipment and readable storage medium for arranging light source |
| CN113469939B (en) * | 2021-05-26 | 2022-05-03 | 透彻影像(北京)科技有限公司 | HER-2 immunohistochemical automatic interpretation system based on characteristic curve |
| US11830622B2 (en) | 2021-06-11 | 2023-11-28 | International Business Machines Corporation | Processing multimodal images of tissue for medical evaluation |
| CN113763370B (en) * | 2021-09-14 | 2024-09-06 | 佰诺全景生物技术(北京)有限公司 | Digital pathology image processing method and device, electronic equipment and storage medium |
| CN115201092B (en) * | 2022-09-08 | 2022-11-29 | 珠海圣美生物诊断技术有限公司 | Method and device for acquiring cell scanning image |
| KR102579826B1 (en) * | 2022-12-09 | 2023-09-18 | (주) 브이픽스메디칼 | Method, apparatus and system for providing medical diagnosis assistance information based on artificial intelligence |
| CN116188423B (en) * | 2023-02-22 | 2023-08-08 | 哈尔滨工业大学 | Super-pixel sparse and unmixed detection method based on pathological section hyperspectral image |
| CN117272393B (en) * | 2023-11-21 | 2024-02-02 | 福建智康云医疗科技有限公司 | Method for checking medical images across hospitals by scanning codes in regional intranet |
Citations (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2008104432A (en) | 2006-10-27 | 2008-05-08 | Mitsui Eng & Shipbuild Co Ltd | Flow cytometer having cell sorting and treating function and method for sorting and treating live cell |
| JP2009528835A (en) | 2006-03-06 | 2009-08-13 | ゼティク テクノロジーズ リミテッド | Methods and compositions for identifying cellular phenotypes |
| WO2012105281A1 (en) | 2011-01-31 | 2012-08-09 | 日本電気株式会社 | Information processing system, information processing method, information processing device and method for control of same, and recording medium storing control program for same |
| JP2013540991A (en) | 2010-08-27 | 2013-11-07 | ユニバーシティ・オブ・チューリッヒ | Novel diagnostic and therapeutic targets in inflammation and / or cardiovascular disease |
| JP2015516985A (en) | 2012-04-27 | 2015-06-18 | ミレニアム ファーマシューティカルズ, インコーポレイテッドMillennium Pharmaceuticals, Inc. | Anti-GCC antibody molecules and their use to test susceptibility to GCC targeted therapy |
| WO2016150873A1 (en) | 2015-03-20 | 2016-09-29 | Ventana Medical Systems, Inc. | System and method for image segmentation |
| JP2016503167A5 (en) | 2013-12-19 | 2016-12-28 | ||
| US20170091937A1 (en) | 2014-06-10 | 2017-03-30 | Ventana Medical Systems, Inc. | Methods and systems for assessing risk of breast cancer recurrence |
Family Cites Families (25)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5595707A (en) | 1990-03-02 | 1997-01-21 | Ventana Medical Systems, Inc. | Automated biological reaction apparatus |
| US6582962B1 (en) | 1998-02-27 | 2003-06-24 | Ventana Medical Systems, Inc. | Automated molecular pathology apparatus having independent slide heaters |
| CA2321836C (en) | 1998-02-27 | 2004-09-28 | Ventana Medical Systems, Inc. | Automated molecular pathology apparatus having independent slide heaters |
| US20030211630A1 (en) | 1998-02-27 | 2003-11-13 | Ventana Medical Systems, Inc. | Automated molecular pathology apparatus having independent slide heaters |
| US8050868B2 (en) * | 2001-03-26 | 2011-11-01 | Cellomics, Inc. | Methods for determining the organization of a cellular component of interest |
| US20050136549A1 (en) * | 2003-10-30 | 2005-06-23 | Bioimagene, Inc. | Method and system for automatically determining diagnostic saliency of digital images |
| US7760927B2 (en) | 2003-09-10 | 2010-07-20 | Bioimagene, Inc. | Method and system for digital image based tissue independent simultaneous nucleus cytoplasm and membrane quantitation |
| JP4496943B2 (en) * | 2004-11-30 | 2010-07-07 | 日本電気株式会社 | Pathological diagnosis support apparatus, pathological diagnosis support program, operation method of pathological diagnosis support apparatus, and pathological diagnosis support system |
| EP1978485A1 (en) * | 2005-05-13 | 2008-10-08 | Tripath Imaging, Inc. | Methods of chromogen separation-based image analysis |
| EP2491452A4 (en) | 2009-10-19 | 2015-03-11 | Ventana Med Syst Inc | Imaging system and techniques |
| US9076198B2 (en) | 2010-09-30 | 2015-07-07 | Nec Corporation | Information processing apparatus, information processing system, information processing method, program and recording medium |
| US20130130930A1 (en) | 2011-11-17 | 2013-05-23 | Bhairavi Parikh | Methods and devices for obtaining and analyzing cells |
| EP2847738B1 (en) | 2012-05-11 | 2017-07-12 | Dako Denmark A/S | Method and apparatus for image scoring and analysis |
| ES2639559T3 (en) * | 2012-12-28 | 2017-10-27 | Ventana Medical Systems, Inc. | Image analysis for breast cancer prognosis |
| CN105027165B (en) | 2013-03-15 | 2021-02-19 | 文塔纳医疗系统公司 | Tissue object-based machine learning system for automated scoring of digital whole slides |
| CN105283904B (en) | 2013-06-03 | 2018-09-07 | 文塔纳医疗系统公司 | The physiologically believable color separated of image adaptive |
| CN103426169B (en) * | 2013-07-26 | 2016-12-28 | 西安华海盈泰医疗信息技术有限公司 | A kind of dividing method of medical image |
| CA2932892C (en) * | 2014-01-28 | 2020-09-22 | Ventana Medical Systems, Inc. | Adaptive classification for whole slide tissue segmentation |
| WO2015124772A1 (en) | 2014-02-21 | 2015-08-27 | Ventana Medical Systems, Inc. | Group sparsity model for image unmixing |
| CA2935473C (en) | 2014-02-21 | 2022-10-11 | Ventana Medical Systems, Inc. | Medical image analysis for identifying biomarker-positive tumor cells |
| WO2016016125A1 (en) | 2014-07-28 | 2016-02-04 | Ventana Medical Systems, Inc. | Automatic glandular and tubule detection in histological grading of breast cancer |
| CA2965564C (en) | 2014-11-10 | 2024-01-02 | Ventana Medical Systems, Inc. | Classifying nuclei in histology images |
| WO2016087589A1 (en) * | 2014-12-03 | 2016-06-09 | Ventana Medical Systems, Inc. | Methods, systems, and apparatuses for quantitative analysis of heterogeneous biomarker distribution |
| WO2016120442A1 (en) | 2015-01-30 | 2016-08-04 | Ventana Medical Systems, Inc. | Foreground segmentation and nucleus ranking for scoring dual ish images |
| EP3345122B1 (en) | 2015-09-02 | 2021-06-30 | Ventana Medical Systems, Inc. | Automated analysis of cellular samples having intermixing of analytically distinct patterns of analyte staining |
-
2018
- 2018-12-04 CN CN202311034131.7A patent/CN117038018A/en active Pending
- 2018-12-04 CN CN201880079402.1A patent/CN111448569B/en active Active
- 2018-12-04 JP JP2020530584A patent/JP7197584B2/en active Active
- 2018-12-04 WO PCT/EP2018/083434 patent/WO2019110561A1/en unknown
- 2018-12-04 EP EP18814573.4A patent/EP3721372A1/en active Pending
-
2022
- 2022-12-15 JP JP2022200094A patent/JP7558242B2/en active Active
Patent Citations (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2009528835A (en) | 2006-03-06 | 2009-08-13 | ゼティク テクノロジーズ リミテッド | Methods and compositions for identifying cellular phenotypes |
| JP2008104432A (en) | 2006-10-27 | 2008-05-08 | Mitsui Eng & Shipbuild Co Ltd | Flow cytometer having cell sorting and treating function and method for sorting and treating live cell |
| JP2013540991A (en) | 2010-08-27 | 2013-11-07 | ユニバーシティ・オブ・チューリッヒ | Novel diagnostic and therapeutic targets in inflammation and / or cardiovascular disease |
| WO2012105281A1 (en) | 2011-01-31 | 2012-08-09 | 日本電気株式会社 | Information processing system, information processing method, information processing device and method for control of same, and recording medium storing control program for same |
| JP2015516985A (en) | 2012-04-27 | 2015-06-18 | ミレニアム ファーマシューティカルズ, インコーポレイテッドMillennium Pharmaceuticals, Inc. | Anti-GCC antibody molecules and their use to test susceptibility to GCC targeted therapy |
| JP2016503167A5 (en) | 2013-12-19 | 2016-12-28 | ||
| US20170091937A1 (en) | 2014-06-10 | 2017-03-30 | Ventana Medical Systems, Inc. | Methods and systems for assessing risk of breast cancer recurrence |
| WO2016150873A1 (en) | 2015-03-20 | 2016-09-29 | Ventana Medical Systems, Inc. | System and method for image segmentation |
Non-Patent Citations (1)
| Title |
|---|
| Radhakrishna Achanta et al.,SLIC Superpixels Compared to State-of-the-Art Superpixel Methods,IEEE Transactions on Pattern Analysis and Machine Intelligence,Volume: 34, Issue: 11,米国,IEEE,2012年05月29日,p.2274-p.2281,https://ieeexplore.ieee.org/stamp/stamp.jsp?tp=&arnumber=6205760,IEL Online IEEE Xplore |
Also Published As
| Publication number | Publication date |
|---|---|
| WO2019110561A1 (en) | 2019-06-13 |
| JP7558242B2 (en) | 2024-09-30 |
| JP2021506003A (en) | 2021-02-18 |
| CN117038018A (en) | 2023-11-10 |
| CN111448569B (en) | 2023-09-26 |
| JP2023030033A (en) | 2023-03-07 |
| EP3721372A1 (en) | 2020-10-14 |
| CN111448569A (en) | 2020-07-24 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7558242B2 (en) | Method for storing and retrieving digital pathology analysis results - Patents.com | |
| JP7250793B2 (en) | Deep Learning Systems and Methods for Joint Cell and Region Classification in Biological Images | |
| JP7273215B2 (en) | Automated assay evaluation and normalization for image processing | |
| US11842483B2 (en) | Systems for cell shape estimation | |
| JP7231631B2 (en) | Methods for calculating tumor spatial heterogeneity and intermarker heterogeneity | |
| JP7228688B2 (en) | Image enhancement enabling improved nuclear detection and segmentation | |
| CN110088804B (en) | Computer scoring based on primary color and immunohistochemical images | |
| CN112868024A (en) | System and method for cell sorting | |
| US11959848B2 (en) | Method of storing and retrieving digital pathology analysis results | |
| JP7214756B2 (en) | Quantification of signal in stain aggregates |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20200721 |
|
| A977 | Report on retrieval |
Free format text: JAPANESE INTERMEDIATE CODE: A971007 Effective date: 20210830 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20211001 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20211222 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20220519 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20220727 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20221118 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20221215 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 7197584 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |