JP3691055B2 - 特異的結合力をもつ固定化タンパク質並びに各種プロセス・物品におけるそれらの使用 - Google Patents
特異的結合力をもつ固定化タンパク質並びに各種プロセス・物品におけるそれらの使用 Download PDFInfo
- Publication number
- JP3691055B2 JP3691055B2 JP51767994A JP51767994A JP3691055B2 JP 3691055 B2 JP3691055 B2 JP 3691055B2 JP 51767994 A JP51767994 A JP 51767994A JP 51767994 A JP51767994 A JP 51767994A JP 3691055 B2 JP3691055 B2 JP 3691055B2
- Authority
- JP
- Japan
- Prior art keywords
- protein
- sequence
- binding
- cell wall
- plasmid
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Lifetime
Links
- 238000000034 method Methods 0.000 title claims abstract description 40
- 230000008569 process Effects 0.000 title claims abstract description 11
- 230000009870 specific binding Effects 0.000 title claims description 5
- 108010058683 Immobilized Proteins Proteins 0.000 title 1
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 108
- 210000002421 cell wall Anatomy 0.000 claims abstract description 54
- 102000004169 proteins and genes Human genes 0.000 claims abstract description 50
- 108020001507 fusion proteins Proteins 0.000 claims abstract description 49
- 102000037865 fusion proteins Human genes 0.000 claims abstract description 49
- 150000001875 compounds Chemical class 0.000 claims abstract description 44
- 108010076504 Protein Sorting Signals Proteins 0.000 claims abstract description 37
- 230000027455 binding Effects 0.000 claims abstract description 37
- 108091008324 binding proteins Proteins 0.000 claims abstract description 35
- 210000004027 cell Anatomy 0.000 claims abstract description 32
- 239000013598 vector Substances 0.000 claims abstract description 30
- 210000004899 c-terminal region Anatomy 0.000 claims abstract description 24
- 101710204899 Alpha-agglutinin Proteins 0.000 claims abstract description 22
- 108091033319 polynucleotide Proteins 0.000 claims abstract description 22
- 102000040430 polynucleotide Human genes 0.000 claims abstract description 22
- 239000002157 polynucleotide Substances 0.000 claims abstract description 22
- 101150054379 FLO1 gene Proteins 0.000 claims abstract description 18
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims abstract description 18
- 241000235070 Saccharomyces Species 0.000 claims abstract description 14
- 108010051210 beta-Fructofuranosidase Proteins 0.000 claims abstract description 14
- 239000001573 invertase Substances 0.000 claims abstract description 14
- 235000011073 invertase Nutrition 0.000 claims abstract description 14
- 101100083069 Candida albicans (strain SC5314 / ATCC MYA-2876) PGA62 gene Proteins 0.000 claims abstract description 12
- 101100106993 Candida albicans (strain SC5314 / ATCC MYA-2876) YWP1 gene Proteins 0.000 claims abstract description 12
- 238000002955 isolation Methods 0.000 claims abstract description 11
- 230000028327 secretion Effects 0.000 claims abstract description 8
- 230000003100 immobilizing effect Effects 0.000 claims abstract description 4
- 108020004511 Recombinant DNA Proteins 0.000 claims abstract description 3
- 239000002773 nucleotide Substances 0.000 claims description 15
- 125000003729 nucleotide group Chemical group 0.000 claims description 15
- 108010021625 Immunoglobulin Fragments Proteins 0.000 claims description 14
- 102000008394 Immunoglobulin Fragments Human genes 0.000 claims description 14
- 238000004519 manufacturing process Methods 0.000 claims description 8
- 210000005253 yeast cell Anatomy 0.000 claims description 8
- 210000000349 chromosome Anatomy 0.000 claims description 4
- 230000001939 inductive effect Effects 0.000 claims description 4
- 239000000910 agglutinin Substances 0.000 claims description 3
- 238000005516 engineering process Methods 0.000 claims description 2
- 102000014914 Carrier Proteins Human genes 0.000 claims 9
- 102000023732 binding proteins Human genes 0.000 abstract description 26
- 241000894006 Bacteria Species 0.000 abstract description 18
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 abstract description 18
- 108091005804 Peptidases Proteins 0.000 abstract description 17
- 241000206602 Eukaryota Species 0.000 abstract description 16
- 102000035195 Peptidases Human genes 0.000 abstract description 16
- 235000019833 protease Nutrition 0.000 abstract description 16
- 244000005700 microbiome Species 0.000 abstract description 12
- SBKVPJHMSUXZTA-MEJXFZFPSA-N (2S)-2-[[(2S)-2-[[(2S)-1-[(2S)-5-amino-2-[[2-[[(2S)-1-[(2S)-6-amino-2-[[(2S)-2-[[(2S)-5-amino-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-amino-3-(1H-indol-3-yl)propanoyl]amino]-3-(1H-imidazol-4-yl)propanoyl]amino]-3-(1H-indol-3-yl)propanoyl]amino]-4-methylpentanoyl]amino]-5-oxopentanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]pyrrolidine-2-carbonyl]amino]acetyl]amino]-5-oxopentanoyl]pyrrolidine-2-carbonyl]amino]-4-methylsulfanylbutanoyl]amino]-3-(4-hydroxyphenyl)propanoic acid Chemical compound C([C@@H](C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)NC(=O)[C@@H](N)CC=1C2=CC=CC=C2NC=1)C1=CNC=N1 SBKVPJHMSUXZTA-MEJXFZFPSA-N 0.000 abstract description 10
- 235000014655 lactic acid Nutrition 0.000 abstract description 9
- 239000004310 lactic acid Substances 0.000 abstract description 9
- 108010038049 Mating Factor Proteins 0.000 abstract description 8
- 241000193830 Bacillus <bacterium> Species 0.000 abstract description 5
- 241000235649 Kluyveromyces Species 0.000 abstract description 5
- 238000004873 anchoring Methods 0.000 abstract description 4
- 238000005189 flocculation Methods 0.000 abstract description 4
- 230000016615 flocculation Effects 0.000 abstract description 4
- 108010090785 inulinase Proteins 0.000 abstract description 4
- 102000004139 alpha-Amylases Human genes 0.000 abstract description 3
- 108090000637 alpha-Amylases Proteins 0.000 abstract description 3
- 229940024171 alpha-amylase Drugs 0.000 abstract description 3
- 239000012634 fragment Substances 0.000 description 108
- 239000013612 plasmid Substances 0.000 description 107
- 108020004414 DNA Proteins 0.000 description 68
- 102000039446 nucleic acids Human genes 0.000 description 38
- 108020004707 nucleic acids Proteins 0.000 description 38
- 150000007523 nucleic acids Chemical class 0.000 description 38
- 239000013615 primer Substances 0.000 description 23
- 239000000203 mixture Substances 0.000 description 22
- 108091033380 Coding strand Proteins 0.000 description 21
- 238000010276 construction Methods 0.000 description 21
- 150000001413 amino acids Chemical group 0.000 description 19
- 108010089254 Cholesterol oxidase Proteins 0.000 description 17
- 238000002360 preparation method Methods 0.000 description 17
- 241000282836 Camelus dromedarius Species 0.000 description 14
- 102000004190 Enzymes Human genes 0.000 description 14
- 108090000790 Enzymes Proteins 0.000 description 14
- 229940088598 enzyme Drugs 0.000 description 14
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 12
- 239000003446 ligand Substances 0.000 description 12
- 108091026890 Coding region Proteins 0.000 description 10
- HVYWMOMLDIMFJA-DPAQBDIFSA-N cholesterol Chemical compound C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HVYWMOMLDIMFJA-DPAQBDIFSA-N 0.000 description 10
- 102100039555 Galectin-7 Human genes 0.000 description 7
- 101000608772 Homo sapiens Galectin-7 Proteins 0.000 description 7
- 244000057717 Streptococcus lactis Species 0.000 description 7
- 235000014897 Streptococcus lactis Nutrition 0.000 description 7
- 108060006698 EGF receptor Proteins 0.000 description 6
- 102000001301 EGF receptor Human genes 0.000 description 6
- 108010014251 Muramidase Proteins 0.000 description 6
- 102000016943 Muramidase Human genes 0.000 description 6
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 6
- 101100066910 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) FLO1 gene Proteins 0.000 description 6
- 239000000427 antigen Substances 0.000 description 6
- 102000036639 antigens Human genes 0.000 description 6
- 108091007433 antigens Proteins 0.000 description 6
- 230000029087 digestion Effects 0.000 description 6
- 239000004325 lysozyme Substances 0.000 description 6
- 229960000274 lysozyme Drugs 0.000 description 6
- 235000010335 lysozyme Nutrition 0.000 description 6
- 239000002609 medium Substances 0.000 description 6
- 108090000765 processed proteins & peptides Proteins 0.000 description 6
- 108020004705 Codon Proteins 0.000 description 5
- 108091028043 Nucleic acid sequence Proteins 0.000 description 5
- 241000235648 Pichia Species 0.000 description 5
- 235000012000 cholesterol Nutrition 0.000 description 5
- 239000000284 extract Substances 0.000 description 5
- 230000004927 fusion Effects 0.000 description 5
- 102000005962 receptors Human genes 0.000 description 5
- 108020003175 receptors Proteins 0.000 description 5
- 108091008146 restriction endonucleases Proteins 0.000 description 5
- 241000186311 Brevibacterium sterolicum Species 0.000 description 4
- 101800003838 Epidermal growth factor Proteins 0.000 description 4
- 102400001368 Epidermal growth factor Human genes 0.000 description 4
- 241000402754 Erythranthe moschata Species 0.000 description 4
- 241000233866 Fungi Species 0.000 description 4
- 101150078127 MUSK gene Proteins 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 101150114506 choB gene Proteins 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 229940116977 epidermal growth factor Drugs 0.000 description 4
- 210000003527 eukaryotic cell Anatomy 0.000 description 4
- 238000002703 mutagenesis Methods 0.000 description 4
- 231100000350 mutagenesis Toxicity 0.000 description 4
- 102000004196 processed proteins & peptides Human genes 0.000 description 4
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- 241000228212 Aspergillus Species 0.000 description 3
- 101100059995 Brevibacterium sterolicum choB gene Proteins 0.000 description 3
- 102000011022 Chorionic Gonadotropin Human genes 0.000 description 3
- 108010062540 Chorionic Gonadotropin Proteins 0.000 description 3
- 241000196324 Embryophyta Species 0.000 description 3
- 241000192125 Firmicutes Species 0.000 description 3
- 101500025419 Homo sapiens Epidermal growth factor Proteins 0.000 description 3
- 101000851181 Homo sapiens Epidermal growth factor receptor Proteins 0.000 description 3
- 101150007280 LEU2 gene Proteins 0.000 description 3
- 125000000539 amino acid group Chemical group 0.000 description 3
- 238000006243 chemical reaction Methods 0.000 description 3
- 238000007796 conventional method Methods 0.000 description 3
- 238000012136 culture method Methods 0.000 description 3
- 239000013613 expression plasmid Substances 0.000 description 3
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 3
- 229940084986 human chorionic gonadotropin Drugs 0.000 description 3
- 229940116978 human epidermal growth factor Drugs 0.000 description 3
- 210000004408 hybridoma Anatomy 0.000 description 3
- 230000002209 hydrophobic effect Effects 0.000 description 3
- 230000035772 mutation Effects 0.000 description 3
- GVUGOAYIVIDWIO-UFWWTJHBSA-N nepidermin Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)NC(=O)CNC(=O)[C@@H](NC(=O)[C@@H](NC(=O)[C@H](CS)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CS)NC(=O)[C@H](C)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CC(C)C)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C(C)C)C(C)C)C1=CC=C(O)C=C1 GVUGOAYIVIDWIO-UFWWTJHBSA-N 0.000 description 3
- 230000010076 replication Effects 0.000 description 3
- 238000006467 substitution reaction Methods 0.000 description 3
- 101100301559 Bacillus anthracis repS gene Proteins 0.000 description 2
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 2
- 101100247969 Clostridium saccharobutylicum regA gene Proteins 0.000 description 2
- 108010047041 Complementarity Determining Regions Proteins 0.000 description 2
- 241000235035 Debaryomyces Species 0.000 description 2
- 101710121765 Endo-1,4-beta-xylanase Proteins 0.000 description 2
- 102100031780 Endonuclease Human genes 0.000 description 2
- ULGZDMOVFRHVEP-RWJQBGPGSA-N Erythromycin Chemical compound O([C@@H]1[C@@H](C)C(=O)O[C@@H]([C@@]([C@H](O)[C@@H](C)C(=O)[C@H](C)C[C@@](C)(O)[C@H](O[C@H]2[C@@H]([C@H](C[C@@H](C)O2)N(C)C)O)[C@H]1C)(C)O)CC)[C@H]1C[C@@](C)(OC)[C@@H](O)[C@H](C)O1 ULGZDMOVFRHVEP-RWJQBGPGSA-N 0.000 description 2
- 241000588724 Escherichia coli Species 0.000 description 2
- 101100412434 Escherichia coli (strain K12) repB gene Proteins 0.000 description 2
- 241001465754 Metazoa Species 0.000 description 2
- 108091034117 Oligonucleotide Proteins 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 241000228143 Penicillium Species 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 2
- 101100114425 Streptococcus agalactiae copG gene Proteins 0.000 description 2
- 241000187747 Streptomyces Species 0.000 description 2
- 239000011543 agarose gel Substances 0.000 description 2
- 239000000556 agonist Substances 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 230000003698 anagen phase Effects 0.000 description 2
- 239000012876 carrier material Substances 0.000 description 2
- 239000013611 chromosomal DNA Substances 0.000 description 2
- 238000010494 dissociation reaction Methods 0.000 description 2
- 230000005593 dissociations Effects 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 238000002474 experimental method Methods 0.000 description 2
- 235000013305 food Nutrition 0.000 description 2
- 239000003205 fragrance Substances 0.000 description 2
- 229930182830 galactose Natural products 0.000 description 2
- 230000013595 glycosylation Effects 0.000 description 2
- 238000006206 glycosylation reaction Methods 0.000 description 2
- 229940088597 hormone Drugs 0.000 description 2
- 239000005556 hormone Substances 0.000 description 2
- 238000003125 immunofluorescent labeling Methods 0.000 description 2
- 238000000338 in vitro Methods 0.000 description 2
- 230000003993 interaction Effects 0.000 description 2
- 230000001404 mediated effect Effects 0.000 description 2
- 239000012528 membrane Substances 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 238000000329 molecular dynamics simulation Methods 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 229920001184 polypeptide Polymers 0.000 description 2
- 101150063028 prtP gene Proteins 0.000 description 2
- 101150044854 repA gene Proteins 0.000 description 2
- 238000000926 separation method Methods 0.000 description 2
- 238000002741 site-directed mutagenesis Methods 0.000 description 2
- 239000000758 substrate Substances 0.000 description 2
- VBEQCZHXXJYVRD-GACYYNSASA-N uroanthelone Chemical compound C([C@@H](C(=O)N[C@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CS)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)C(C)C)[C@@H](C)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CCSC)NC(=O)[C@H](CS)NC(=O)[C@@H](NC(=O)CNC(=O)CNC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CS)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CS)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O)C(C)C)[C@@H](C)CC)C1=CC=C(O)C=C1 VBEQCZHXXJYVRD-GACYYNSASA-N 0.000 description 2
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- IMRYETFJNLKUHK-SJCJKPOMSA-N (S,S)-traseolide Chemical compound CC1=C(C(C)=O)C=C2[C@@H](C(C)C)[C@H](C)C(C)(C)C2=C1 IMRYETFJNLKUHK-SJCJKPOMSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- 101710129138 ATP synthase subunit 9, mitochondrial Proteins 0.000 description 1
- 101710168506 ATP synthase subunit C, plastid Proteins 0.000 description 1
- 101710114069 ATP synthase subunit c Proteins 0.000 description 1
- 101710197943 ATP synthase subunit c, chloroplastic Proteins 0.000 description 1
- 101710187091 ATP synthase subunit c, sodium ion specific Proteins 0.000 description 1
- 108010025188 Alcohol oxidase Proteins 0.000 description 1
- 235000002198 Annona diversifolia Nutrition 0.000 description 1
- 244000063299 Bacillus subtilis Species 0.000 description 1
- 235000014469 Bacillus subtilis Nutrition 0.000 description 1
- 241000186146 Brevibacterium Species 0.000 description 1
- 125000001433 C-terminal amino-acid group Chemical group 0.000 description 1
- 102100037084 C4b-binding protein alpha chain Human genes 0.000 description 1
- 241000282832 Camelidae Species 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 244000303965 Cyamopsis psoralioides Species 0.000 description 1
- 102000053602 DNA Human genes 0.000 description 1
- 239000003155 DNA primer Substances 0.000 description 1
- 108090000204 Dipeptidase 1 Proteins 0.000 description 1
- 102000002322 Egg Proteins Human genes 0.000 description 1
- 108010000912 Egg Proteins Proteins 0.000 description 1
- 102100037642 Elongation factor G, mitochondrial Human genes 0.000 description 1
- 108010042407 Endonucleases Proteins 0.000 description 1
- 108010067770 Endopeptidase K Proteins 0.000 description 1
- 108010058643 Fungal Proteins Proteins 0.000 description 1
- 229920001503 Glucan Polymers 0.000 description 1
- 108010051696 Growth Hormone Proteins 0.000 description 1
- 101150105574 HCG gene Proteins 0.000 description 1
- 102100024023 Histone PARylation factor 1 Human genes 0.000 description 1
- 101000880344 Homo sapiens Elongation factor G, mitochondrial Proteins 0.000 description 1
- 241000223198 Humicola Species 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- 241000194036 Lactococcus Species 0.000 description 1
- 241000194034 Lactococcus lactis subsp. cremoris Species 0.000 description 1
- 241000282838 Lama Species 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 102000004882 Lipase Human genes 0.000 description 1
- 239000004367 Lipase Substances 0.000 description 1
- 108090001060 Lipase Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 101150113367 PRT1 gene Proteins 0.000 description 1
- 101100342162 Porphyromonas gingivalis kgp gene Proteins 0.000 description 1
- 101710136733 Proline-rich protein Proteins 0.000 description 1
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 1
- 241000235527 Rhizopus Species 0.000 description 1
- 101000718529 Saccharolobus solfataricus (strain ATCC 35092 / DSM 1617 / JCM 11322 / P2) Alpha-galactosidase Proteins 0.000 description 1
- 101000821983 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) Alpha-agglutinin Proteins 0.000 description 1
- XUIMIQQOPSSXEZ-UHFFFAOYSA-N Silicon Chemical compound [Si] XUIMIQQOPSSXEZ-UHFFFAOYSA-N 0.000 description 1
- 102100038803 Somatotropin Human genes 0.000 description 1
- 235000014962 Streptococcus cremoris Nutrition 0.000 description 1
- 241000187180 Streptomyces sp. Species 0.000 description 1
- 101100226845 Strongylocentrotus purpuratus EGF2 gene Proteins 0.000 description 1
- 108090000787 Subtilisin Proteins 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 101001099217 Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) Triosephosphate isomerase Proteins 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- NNISLDGFPWIBDF-MPRBLYSKSA-N alpha-D-Gal-(1->3)-beta-D-Gal-(1->4)-D-GlcNAc Chemical compound O[C@@H]1[C@@H](NC(=O)C)C(O)O[C@H](CO)[C@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O2)O)[C@@H](O)[C@@H](CO)O1 NNISLDGFPWIBDF-MPRBLYSKSA-N 0.000 description 1
- 108010030291 alpha-Galactosidase Proteins 0.000 description 1
- 102000005840 alpha-Galactosidase Human genes 0.000 description 1
- 238000004458 analytical method Methods 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 230000000844 anti-bacterial effect Effects 0.000 description 1
- 230000001130 anti-lysozyme effect Effects 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- 102000006635 beta-lactamase Human genes 0.000 description 1
- 230000003197 catalytic effect Effects 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 101150057842 choA gene Proteins 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 239000000539 dimer Substances 0.000 description 1
- 235000013345 egg yolk Nutrition 0.000 description 1
- 210000002969 egg yolk Anatomy 0.000 description 1
- 238000009585 enzyme analysis Methods 0.000 description 1
- 229960003276 erythromycin Drugs 0.000 description 1
- 239000012847 fine chemical Substances 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 239000000122 growth hormone Substances 0.000 description 1
- 239000001963 growth medium Substances 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 108091006058 immobilized fusion proteins Proteins 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 230000008105 immune reaction Effects 0.000 description 1
- 238000010820 immunofluorescence microscopy Methods 0.000 description 1
- 230000000415 inactivating effect Effects 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 238000007373 indentation Methods 0.000 description 1
- 235000019421 lipase Nutrition 0.000 description 1
- 102000019758 lipid binding proteins Human genes 0.000 description 1
- 230000004576 lipid-binding Effects 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 108010005335 mannoproteins Proteins 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 238000005374 membrane filtration Methods 0.000 description 1
- 108020004999 messenger RNA Proteins 0.000 description 1
- 238000003032 molecular docking Methods 0.000 description 1
- 238000012856 packing Methods 0.000 description 1
- 238000000053 physical method Methods 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 108020001775 protein parts Proteins 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 238000002708 random mutagenesis Methods 0.000 description 1
- 239000013606 secretion vector Substances 0.000 description 1
- 230000006258 serine-glycosylation Effects 0.000 description 1
- 210000002966 serum Anatomy 0.000 description 1
- 229910052710 silicon Inorganic materials 0.000 description 1
- 239000010703 silicon Substances 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 230000006262 threonine-glycosylation Effects 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 238000004448 titration Methods 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
- C07K14/36—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria from Actinomyces; from Streptomyces (G)
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
- C07K14/32—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria from Bacillus (G)
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/37—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N11/00—Carrier-bound or immobilised enzymes; Carrier-bound or immobilised microbial cells; Preparation thereof
- C12N11/16—Enzymes or microbial cells immobilised on or in a biological cell
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/62—DNA sequences coding for fusion proteins
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/02—Fusion polypeptide containing a localisation/targetting motif containing a signal sequence
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/70—Fusion polypeptide containing domain for protein-protein interaction
- C07K2319/74—Fusion polypeptide containing domain for protein-protein interaction containing a fusion for binding to a cell surface receptor
- C07K2319/75—Fusion polypeptide containing domain for protein-protein interaction containing a fusion for binding to a cell surface receptor containing a fusion for activation of a cell surface receptor, e.g. thrombopoeitin, NPY and other peptide hormones
Landscapes
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- Biomedical Technology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Zoology (AREA)
- Biophysics (AREA)
- Wood Science & Technology (AREA)
- Gastroenterology & Hepatology (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Medicinal Chemistry (AREA)
- Microbiology (AREA)
- Physics & Mathematics (AREA)
- Mycology (AREA)
- Plant Pathology (AREA)
- Peptides Or Proteins (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Immobilizing And Processing Of Enzymes And Microorganisms (AREA)
- Saccharide Compounds (AREA)
- Enzymes And Modification Thereof (AREA)
Description
医薬品産業、ファインケミカル産業及び食品産業では、動物・植物組織の抽出物や培地のような複雑な混合物から単離する必要のある化合物が数多く必要とされている。このような単離プロセスによって製品の価格が決定されることが多い。従来の単離プロセスは特異性が余り高くなく、単離プロセスの間に単離すべき化合物がなかり稀釈され、その結果、水その他の溶媒を除去するため経費のかさむ諸工程を設ける必要があった。
幾つかの特定の化合物の単離にはアフィニティー技術が利用されている。かかる技術の利点は、それらの化合物がある種のリガンドに極めて特異的に結合することである。しかし、このようなリガンドは非常に高価である場合が極めて多い。このような高価なリガンドが無駄に使われることのないように、かかるリガンドを不溶性担体に連結することも可能である。しかし、こうしたリガンドの連結処理も経費のかさむ場合が多く、しかもかかる処理によってリガンドの機能が悪影響を受けることも多い。したがって、有効性の高い固定化リガンドを調製するための経済的な方法を開発することが望まれている。
発明の概要
本発明は、特定の化合物に対する結合能力を有する「結合タンパク質」を固定化するための方法にして、上記結合タンパク質又は上記の特異的結合能力を保持したその機能的部分であって宿主細胞の外部に連結した結合タンパク質又は機能的部分の生産に組換えDNA技術を利用して、宿主細胞の細胞壁に固着し得るアンカータンパク質のアンカー部分(当該アンカー部分は上記アンカータンパク質のC末端部分から導かれる)のN末端に上記結合タンパク質又はその機能的部分がそのC末端で結合しているようなキメラタンパク質を宿主細胞が産生及び分泌できるようにすることによって上記結合タンパク質又はその機能的部分が細胞壁中又は細胞壁の外側に局在化するようにさせることを含んでなる方法を提供する。
宿主は好ましくはグラム陽性細菌及び菌類である。これらはその細胞外部に細胞壁を有しており、細胞外部に膜をもつグラム陰性細菌及び動物・植物細胞などの高等真核生物とは対照的である。好適なグラム陽性細菌には、乳酸菌やBacillus属及びStreptomyces属の細菌が含まれる。好適な菌類には、Candida属、Debaryomyces属、Hansenula属、Kluyveromyces属、Pichia属及びSaccharomyces属の酵母並びにRhizopus属、Aspergillus属及びPenicillium属のカビが含まれる。本明細書中では、菌類は酵母の群とカビの群を含んでなるが、これらは下等真核生物としても知られている。植物及び動物の細胞との対比において、細菌及び下等真核生物の群を本明細書では微生物と記載することもある。
本発明は、上記の方法に使用し得る組換えポリヌクレオチドにして、(i)上記結合タンパク質又は特異的結合能力を保持したその機能的部分をコードする構造遺伝子、及び(ii)グラム陽性細菌又は菌類の細胞壁に固着し得るアンカータンパク質をコードする遺伝子の少なくとも一部分であって、当該遺伝子部分がアンカータンパク質のアンカー部分(当該アンカー部分は上記アンカータンパク質のC末端部分に由来する)をコードしているもの、を含んでなるポリヌクレオチドをも提供する。
アンカータンパク質は、α-アグルチニン、a-アグルチニン、FLO1、下等真核生物の主要細胞壁タンパク質及び乳酸菌のプロテイナーゼから選択することができる。好ましくは、かかるポリヌクレオチドは当該ポリヌクレオチドの発現産物の分泌を確実にするためのシグナルペプチドをコードするヌクレオチド配列をさらに含んでなるが、そのようなシグナルペプチドは酵母のα-接合因子、酵母のα-アグルチニン、Saccharomycesのインベルターゼ、Kluyveromycesのイヌリナーゼ、Bacillusのα-アミラーゼ及び乳酸菌のプロテイナーゼから選択されるタンパク質から得ることができる。本発明のポリヌクレオチドはプロモーターに機能的に連結させることもできるが、プロモーターは誘導型プロモーターであるのが好ましい。
本発明は、さらに、本発明のポリヌクレオチドを含んでなる組換えベクター、本発明のポリヌクレオチドにコードされたキメラタンパク質、並びに細胞の外部に細胞壁をもつ宿主細胞にして本発明のポリヌクレオチドを少なくとも1つ保有する宿主細胞をも提供する。好ましくは、少なくとも1つのポリヌクレオチドが宿主細胞の染色体に組込まれている。これに関連した本発明の別の具体的態様は、本発明のキメラタンパク質が細胞壁に固定化されていてそのキメラタンパク質の結合タンパク質部分が細胞壁中又は細胞壁の外側に局在化している宿主細胞である。
本発明の別の具体的態様は、特定化合物に対する結合能力を保持している固定化された結合タンパク質又はその機能的部分を用いて単離プロセスを行う方法であるが、この方法では、上記特定化合物を含んだ媒質を本発明の宿主細胞に、特定化合物と固定化結合タンパク質との複合体が形成されるような条件下で、接触させ、特定化合物を当初含んでいた上記媒質から当該複合体を分離し、所望により、結合タンパク質又はその機能的部分から特定化合物を解離させる。
【図面の簡単な説明】
図1には、pEMBL9から誘導されたプラスミドであるpUR4122の組成が示してあるが、その調製法については実施例1に記載されている。
図2には、プラスミドpUR2741の組成が示してあるが、このプラスミドは既報のプラスミドpUR2740から誘導されたものである。実施例1参照。
図3には、pEMBL9から誘導されたプラスミドであるpUR2968の組成が示してある。その調製法については実施例1に記載されている。
図4には、プラスミドpUR2741とpUR2968とpUR4122とを出発材料としたプラスミドpUR4174の調製法、並びにプラスミドpSY16とpUR2968とpUR4122とを出発材料としたプラスミドpUR4175の調製法が示してある。これらの調製法は実施例1に記載されている。
図5には、プラスミドpUR2743.4の組成を示した。その調製法は実施例2に記載されている。このプラスミドは配列番号:12に示した714bpのPstI-XhoIフラグメントを含んでいるが、このフラグメントには、抗トラセオリド抗体02/01/01のscFv-TRASフラグメントがコードされている(トラセオリド(traseolide)は登録商標である)。
図6には、プラスミドpUR4178の組成を示した。その調製法は実施例2に記載されている。このプラスミドは配列番号:12に示す上述の714bpのPstI-XhoIフラグメントを含んでいる。このフラグメントは、インベルターゼのシグナル配列(SUC2)で先導されたscFv-TRASとαAGGの融合タンパク質の発現に適している。
図7には、プラスミドpUR4179の組成を示した。その調製法は実施例2に記載されている。このプラスミドは配列番号:12に示す上述の714bpのPstI-XhoIフラグメントを含んでいる。このフラグメントは、プレプロα-接合因子のシグナル配列で先導されたscFv-TRASとαAGGの融合タンパク質の発現に適している。
図8は分子設計図であり、実施例3に記載のムスク系香料分子のトラセオリド(登録商標)と修飾ムスク抗原が示してある。
図9には、プラスミドpUR4177の組成が示してある。その調製法は実施例4に記載されている。プラスミドpUR4177は、ヒト絨毛性性腺刺激ホルモンに対するモノクローナル抗体の重鎖及び軽鎖フラグメントの可変領域(scFv-HCGフラグメント)をコードした配列番号:13に示す734bpのEagI-XhoI DNAフラグメントを含んでおり、GAL7プロモーター調節下でのインベルターゼのシグナル配列で先導されたキメラなscFv-HCG-αAGG融合タンパク質の生産に適した基本鎖長2μmのベクターである。
図10には、プラスミドpUR4180の組成が示してある。その調製法は実施例4に記載されている。プラスミドpUR4177は、配列番号:13に示した上述の734bpのEagI-XhoI DNAフラグメントを含んでおり、GAL7プロモーター調節下でのプレプロα-接合因子のシグナル配列で先導されたキメラなscFv-HCG-αAGG融合タンパク質の生産に適した基本鎖長2μmのベクターである。
図11には、基本鎖長2μmのベクターであるプラスミドpUR2990の組成が示してあり、このプラスミドは、実施例5では、プラスミドpUR4196を調製する際の出発ベクターとして示した(図12参照)。プラスミドpUR2990はキメラなリパーゼ-FLO1タンパク質をコードするDNAフラグメントを含んでいるが、このタンパク質は下等真核生物の細胞壁につなぎとめられ、脂質の加水分解を触媒し得る。
図12には、プラスミドpUR4196の組成が示してある。その調製法は実施例5に記載されている。このプラスミドは、scFv-HCGとそれに続くFLO1タンパク質のC末端部分からなるキメラタンパク質をコードするDNAフラグメントを含んでおり、宿主生物の細胞壁につなぎとめられていてHCGを結合し得るキメラタンパク質の生産に適したベクターである。
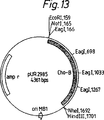
図13には、プラスミドpUR2985が組成が示してある。その調製法は実施例6に記載されている。このプラスミドには、Brevibacterium sterolicumの染色体からPCR法で得られたコレステロールオキシダーゼ(EC 1.1.3.6)の成熟部分をコードするchoB遺伝子が含まれている。
図14には、プラスミドpUR2987の組成を示した。プラスミドpUR2985からのその調製法は実施例6に記載されている。このプラスミドには、コレステロールオキシダーゼの成熟部分をコードするchoB遺伝子と、その前と後にそれぞれ位置するプレプロα-接合因子のシグナル配列をコードするDNAとα-アグルチニンのC末端部分をコードするDNAとからなるDNA配列が含まれている。
図15には、既報のプラスミドpGKV550の組成が示してある。このプラスミドは実施例7に記載されており、Lactococcus lactis subsp.cremoris Wg2株の細胞壁プロテイナーゼのオペロンを、プロモーター、リボソーム結合部位及びprtP遺伝子を含めて、完全に含んでいる。
図16には、プラスミドpUR2988の組成が示してある。その調製法は実施例7に記載されている。このプラスミドは別のプラスミドpUR2989の調製に使用できると期待されるが、そのpUR2989を乳酸菌に導入すれば、乳酸菌の外表面につなぎとめられていてコレステロールに結合し得るキメラタンパク質が生産されるはずである。
図17には、プラスミドpUR2993の組成が示してある。その調製法は実施例8に記載されている。このプラスミドは酵母細胞の形質転換に使用でき、その形質転換酵母細胞は、EGFレセプターをその一部として有する固定化キメラタンパク質を介してヒト上皮成長因子(EGF)に結合し得る。
図18には、プラスミドpUR4482及びpUR4483の組成が示してある。それらの調製法は実施例9に記載されている。プラスミドpUR4482は、インベルターゼのシグナル配列、ラクダ抗体のCHv09可変領域とMycテールと「X-P-X-P」ヒンジ領域及びαアグルチニンの細胞壁アンカー領域をもつ融合タンパク質を発現させるための、酵母のエピソーム型発現プラスミドである。プラスミドpUR4483は「X-P-X-P」ヒンジ領域を含んでいない点でpUR4482とは異なる。
図19には、プラスミドpUR4424、pUR4482及びpUR4483上に存在するラクダ抗体遺伝子を発現する対数増殖期(OD530=0.5)のSU10細胞の免疫蛍光標識実験(抗Myc抗体)の結果を示す。Ph=位相差。Fl=蛍光。
図20には、プラスミドpUR4424、pUR4482及びpUR4483上に存在するラクダ抗体遺伝子を発現する対数増殖期(OD530=0.5)のSU10細胞の免疫蛍光標識実験(抗ヒトIgG抗体)の結果を示す。Ph=位相差。Fl=蛍光。
図面で用いた略号
α-gal: グアーのα-ガラクトシダーゼをコードする遺伝子;
AG-alpha-1/AGα1: S.cerevisiaeのα-アグルチニンを発現する遺伝子;
AGα1 cds/α-AGG: α-アグルチニンのコーディング配列;
Amp/amp r: β-ラクタマーゼ耐性遺伝子;
CHv09: ラクダ重鎖可変09フラグメント;
EmR: エリスロマイシン耐性遺伝子;
f1: f1ファージ複製配列;
FLO1/FLO(C-part): フロキュレーションタンパク質のFLO1コーディング配列のC末端側部分;
Hinge: ラクダ「X-P-X-P」ヒンジ領域,実施例9参照;
LEU2: LEU2遺伝子;
LEU2d/leu2d: トランケート型LEU2遺伝子;
Leu2d cs: LEU2d遺伝子のコーディング配列;
MycT: ラクダMycテール;
Ori MB1: 大腸菌プラスミド由来のMB1複製起点;
Pgal7/pGAL7: GAL7プロモーター;
Tpgk: ホスホグリセリン酸キナーゼ遺伝子のターミネーター;
ppα-MF/MFα1ss: α接合因子のプレプロ部分(=シグナル配列);
repA: 複製に必要なタンパク質repAをコードする遺伝子(図15/16);
ScFv(Vh-V1): VH鎖とVL鎖を含む単鎖抗体フラグメント;
ss: シグナル配列;
SUC2: インベルターゼのシグナル配列;
2u/2micron: 2μm配列.
発明の詳細な説明
本発明は、固定化リガンドを利用して、複雑な混合物から貴重な化合物を単離することに関する。この固定化リガンドは、遺伝子工学によって得られるタンパク質であって、「アンカータンパク質」又はその機能的部分と「結合タンパク質」又はその機能的部分との2つの部分で構成される。
「アンカータンパク質」は微生物、好ましくは酵母やカビのような下等真核生物の細胞壁に固着する。この種のタンパク質は長いC末端部分を有していることが多く、そのC末端部分が細胞壁につなぎとめられる。そのようなC末端部分は非常に特殊なアミノ酸配列を有している。この典型的な例がプロリンに富んだタンパク質のC末端配列を介した細胞壁への固着である。Kok(1990)の報文参照。
これらのアンカータンパク質のC末端部分は、相当数の潜在的セリン及びトレオニン-グリコシル化部位を含んでいる可能性がある。これらの部位がO-グリコシル化されると、タンパク質のC末端部分が桿状のコンホメーションをとるようになる。
細胞壁固着マンノタンパク質の場合、ドデシル硫酸ナトリウム(SDS)を用いてもそれらを細胞壁から抽出することができないのに、グルカナーゼ処理すればそれらを遊離させることができることから、それらは下等真核生物の細胞壁中のグルカンに結合しているらしい。本出願人の係属中の特許出願で1994年1月20日に公開されたWO94/01567号並びにSchreuder他(1993)の報文参照。両文献はいずれも本出願の優先日以降に公開されたものである。タンパク質を細胞の外側につなぎとめるもう一つの機構は、グルコシル-ホスファチジルイノシトール(GPI)基を含んだタンパク質がそのGPIを介して細胞表面に固着する性質を利用したものである。Conzelmann他(1990)の報文参照。
「結合タンパク質」は、単離すべき特定の化合物に連結又は結合するので、そう呼ばれる。アンカータンパク質のN末端部分が特定の化合物に結合する能力を十分に有していれば、そのアンカータンパク質自身をその特定化合物の単離プロセスに使用することができる。結合タンパク質として好適なものの例には、抗体、抗体フラグメント、複数の抗体フラグメントの組合せ、レセプタータンパク質、それぞれの基質に対する結合能力を保持している不活性化酵素及び応用分子進化法(Applied Molecular Evolution)で得られるペプチド(Lewin(1990)の報文参照)、並びにこれらのタンパク質の一部分で単離すべき特定の化合物に対する結合能力を保持しているものが含まれる。これらの結合タンパク質はすべて、単離すべき化合物又は関連化合物群を特異的に認識する能力をもっていることを特徴とする。単離すべき特定の化合物と結合タンパク質の間の結合速度と解離速度は(したがって、当然に、結合定数も)、その化合物の存在する抽出液の組成を変化させるか或いはタンパク質工学によって結合タンパク質を変化させることによって調節することができるが、後者の方法が好ましい。
結合タンパク質とアンカータンパク質(或いはそれらの機能的部分)を含んでなる「キメラタンパク質をコードする遺伝子」は、構成型、誘導型又は抑制解除型プロモーターの調節下に置くことができ、通常はキメラタンパク質の効率的な分泌を図るためにシグナル配列をコードするDNAフラグメントで先導される。分泌によってキメラタンパク質が微生物の細胞壁に固着して、微生物の表面がそのキメラタンパク質によって覆われるようになる。これらの微生物は普通の培養法で得ることができ、その菌体の単離も遠心や膜濾過法などの物理的分離手段を用いれば経済的なプロセスとなる。
こうして単離した微生物を、洗浄後、貴重な特定化合物又は化合物群を含んだ抽出液に添加すればよい。ある程度時間が経過すれば、結合状態にある特定化合物(群)と遊離状態にある特定化合物(群)との間で平衡が成立し、特定の化合物又は関連化合物群の結合した微生物を抽出液から簡単な物理的技術で分離することができる。別法として、リガンドで覆われた微生物を担体材料の表面にくっつけて、その被覆担体材料をカラムに詰めて使用することもできる。そのカラムに、特定の化合物又は化合物群を含んだ抽出液を流し込み、その後で、溶出液の組成か温度又はそれらを共に変化させることによってリガンドから上記化合物(群)を解離させることができる。これら2通りの可能性の他にも、数々の変法を用いて、特定化合物とリガンドとの結合、それらの単離及び/又は特定化合物(群)の解離を行い得ることは当業者には明らかであろう。
特に、本発明は下等真核生物の細胞壁に結合したキメラタンパク質に関する。好適な下等真核生物には、Candida、Debaryomyces、Hansenula、Kluyveromyces、Pichia及びSaccharomycesなどの酵母、並びにAspergillus、Penicillium及びPhizopus等のカビが含まれる。用途によっては、特にグラム陽性細菌などの原核生物を用いることもでき、その具体例には、乳酸菌やBacillus属及びStreptomyces属に属する細菌が含まれる。
「下等真核生物」に関しては、本発明は、キメラタンパク質をコードする遺伝子にして下記のa及びbからなる遺伝子を提供する。
a.下等真核生物宿主において機能し得るシグナル配列、例えばα-接合因子、インベルターゼ、α-アグルチニン、イヌリナーゼのような酵母タンパク質由来のもの或いはキシラナーゼのようなカビタンパク質由来のものなど、をコードするDNA配列。
b.細胞壁タンパク質のC末端をコードする構造遺伝子と、その前に位置する構造遺伝子にして特定の化合物又は化合物群に対する結合能力を有するタンパク質をコードする構造遺伝子。後者のタンパク質の例としては、
・抗体;
・単鎖抗体フラグメント(scFv)(Bird及びWebb Walker(1991)の報文参照);
・抗体の重鎖可変領域(VH)又は軽鎖可変領域(VL)或いはこれらの可変領域の一部分であって1〜3つの相補性決定領域(CDR)を保持した部分;
・レセプタータンパク質のアゴニスト認識部分又はアゴニスト結合能力を保持したその一部分;
・触媒活性を喪失した酵素又はかかる酵素の一部分であって酵素の基質結合部位を保持した部分;
・特異的脂質結合タンパク質又はかかるタンパク質の一部分であって脂質結合部位を保持した部分(Ossendorf(1992)の報文参照);並びに
・応用分子進化法で得られるペプチド(Lewin(1990)の報文参照)、
が挙げられる。
これらの遺伝子の発現産物はすべてシグナル配列と2つのタンパク質部分(すなわち単離すべき1種類以上の化合物に対する結合能力を有する部分と通常は細胞壁結合タンパク質のC末端)で構成されていることを特徴とし、後者(細胞壁結合タンパク質)の具体例としては、α-アグルチニン(Lipke他(1989)の報文参照);a-アグルチニン(Roy他(1991)の報文参照);FLO1(実施例5及び配列番号:14参照);並びに下等真核生物の主要細胞壁タンパク質が挙げられるが、これらのタンパク質のC末端は下等真核生物宿主の細胞壁に発現産物をつなぎとめることができる。
キメラタンパク質をコードするこれらの遺伝子の発現は構成型プロモーターの制御下に置くこともできるが、誘導型プロモーターが好ましく、その好適な具体例としては、SaccharomycesのGAL7プロモーター、Kluyveromycesのイヌリナーゼプロモーター、Hansenulaのメタノールオキシダーゼプロモーター及びAspergillusのキシラナーゼプロモーターが挙げられる。かかる構築体は新たな遺伝情報が宿主細胞の染色体に安定に組込まれるような方法で作製するのが好ましい。WO91/00920号(UNILEVER)参照。
上記の遺伝子で形質転換した下等真核生物は普通の培養法、連続培養又は流加培養法で増殖させることができる。適当な微生物増殖法は、使用する遺伝子とプロモーターの構成並びに物理的分離後の菌体の所望精製度に応じて選択される。
「細菌」に関しては、本発明は、キメラタンパク質をコードする遺伝子にして下記のa及びbからなる遺伝子を提供する。
a.特定の細菌で機能し得るシグナル配列、例えばBacillusのα-アミラーゼ、Bacillus subtilisのズブチリシン又はLactococcus lactis subsp.cremorisのプロテイナーゼ由来のもの、をコードするDNA配列。
b.細胞壁タンパク質のC末端をコードする構造遺伝子と、その前に位置する構造遺伝子にして特定の化合物又は化合物群に対する結合能力を有するタンパク質をコードする構造遺伝子。後者のタンパク質の例は、下等真核生物に関して既に列挙した。
これらの遺伝子の発現産物はすべてシグナル配列と2つのタンパク質(単離すべき特定の化合物又は化合物群に対する結合能力を有する部分と通常は細胞壁結合タンパク質のC末端)で構成されていることを特徴とし、後者(細胞壁結合タンパク質)の具体例には、Lactococcus lactis subsp.cremoris Wg2株のプロテイナーゼが含まれるが、そのC末端は宿主細菌の細胞壁に発現産物をつなぎとめることができる。
本発明を以下の実施例で例示するが、これらの実施例は本発明を限定するものではない。最初に、実施例で挙げるエンドヌクレアーゼの制限部位を示しておく。
実施例1 下等真核生物の細胞壁に固定化され、複雑な混合物中のリゾチームに対して高い特異性で結合し得るキメラタンパク質をコードする遺伝子の構築
リゾチームは抗菌酵素であって医薬品産業及び食品産業において数多くの用途を有する。卵黄やリゾチーム産生微生物含有培地など、幾つかのリゾチーム源が知られている。リゾチームに対するモノクローナル抗体が得られており(Ward他(1989)の報文参照)、かかる抗体の軽鎖及び重鎖をコードするmRNAもハイブリドーマから単離されていて、逆転写酵素を用いたcDNA合成の鋳型として用いられている。Ward他(1989)の報文に記載されたプラスミドを出発材料として、我々はpUR4122と名付けたpEMBL由来プラスミドを構築した。このpUR4122においては、pEMBLベクターのEcoRIからHindIII部位までの範囲のマルチクローニング部位が231bpのDNAフラグメントで置換されているが、そのDNAフラグメントのヌクレオチド配列は配列番号:1に示したもので、ヌクレオチド1〜6にEcoRI部位(GAATTC)、ヌクレオチド105〜110にPstI部位(CTGCAG)、ヌクレオチド122〜128にBstEII部位(GGTCACC)、ヌクレオチド207〜212にXhoI部位(CTCGAG)及びヌクレオチド226〜231にHindIII部位(AAGCTT)を有している。
pUR4122の構築
プラスミドpEMBL9(Dente他(1983)の報文参照)をEcoRI及びHindIIIで消化し、得られた大フラグメントを配列番号:1に示す合成二本鎖DNAと連結した。後段でのDNAフラグメントの連結(それによって、リゾチームに対する単鎖抗体フラグメントのコーディング配列が最終的に得られる)のために、pEMBL9ベクターに挿入した上記231bpのDNAフラグメント(配列番号:1)の中には、以下の要素:GAL7プロモーターの3′部分;インベルターゼのシグナル配列(SUC2);PstI制限部位;BstEII制限部位;VHフラグメントとVLフラグメントとを繋ぐ(GGGGS)×3ペプチドリンカーをコードする配列;SacI制限部位XhoI制限部位;HindIII制限部位が組込まれており、プラスミドpUR4119が得られる。VHとGGGGSリンカーがインフレーム(in frame)で(すなわち、フレームシフトを起こさずに)融合するように、プラスミドpSW1-VHD1.3-VKD1.3-TAG1(Ward他(1989)の報文参照)をPstI及びBstEIIで消化し、その0.35kbpのDNAフラグメントを同様に消化したpUR4119と連結して、プラスミドpUR4119Aを得た。続いて、プラスミドpSW1-VHD1.3-VKD1.3-TAG1をSacI及びXhoIで消化し、得られたVLのコーディング部分を含むフラグメントをpUR4119AのSacI/XhoI部位に連結して、プラスミドpUR4122(図1参照)を得た。
pUR4174の構築,図4参照
α-アグルチニンのC末端部由来の細胞壁アンカーのコードされたDNAを有するS.cerevisiaeエピソーム型発現プラスミドを得るため、プラスミドpUR2741(図2参照)を出発ベクターとして選んだ。このプラスミドは基本的にpUR2740の誘導体であり、pUR2740はWO91/19782号及びVerbakel(1991)の報文に記載されている通りプラスミドpUR2730の誘導体である。pUR2730の作製法は欧州特許出願公開EP-A1-0255153(UNILEVER)の例9に明瞭に記載されている。プラスミドpUR2741は、既に不活性化されているテトラサイクリン(tet)耐性遺伝子の残存部分にあるEagI制限部位がNruI/SalI消化によって欠失している点でpUR2740とは異なる。そのSalI部位の一本鎖突出端は再連結前に埋められている。
pUR4122をSacI及びHindIIIで消化(SacIは部分的消化)しておよそ800bpのフラグメントを単離し、これを、同じ酵素でpUR2741を消化して得たpUR2741ベクターフラグメントにクローニングした。得られたプラスミドはpUR4125と名付けた。
pUR2968と名付けたプラスミド(図3参照)を、(1)Lipke他(1989)の報文に記載されたAgα1含有プラスミドpLα21をHindIIIで消化し、(2)約6.1kbpフラグメントを単離し、(3)このフラグメントをHindIII処理pEMBL9と当該6.1kbpフラグメントがpEMBL9ベクターのマルチクローニング部位に存在するHindIII部位に導入されるように連結することによって、作製した。
プラスミドpUR4125をXhoI及びHindIIIで消化して、その約8kbpフラグメントを、下記の配列のXhoI/NheIアダプターを用いて、pUR2968の約1.4kbpのNheI-HindIIIフラグメントに連結した。
プラスミドpUR2968及びpUR4125の適当な部分及びXhoI/NheIアダプターをこのように連結して得たプラスミドをpUR4174と名付けたが、このプラスミドpUR4174は、アミノ末端にインベルターゼのシグナル(プレ)ペプチド、続いてscFv-LYSポリペプチド、最後にα-アグルチニンのC末端からなるキメラ融合タンパク質をコードしている(図4参照)。
pUR4175の構築,図4参照
pUR4122(上記参照)をPstI及びHindIIIで消化しておよそ700bpのフラグメントを単離し、これを、EagI及びHindIIIで消化したpSY16(Harmsen他(1993)の報文参照)のベクターフラグメントに、下記配列のEagI-PstIアダプターを用いて連結した。
こうして得られたプラスミドpUR4132をXhoI及びHindIIIで消化して、上記のXhoI/NheIアダプターを用いて、pUR2968(上記参照)の約1.4kbpのNheI-HindIIIフラグメントに連結して、プラスミド4175を得た(図4参照)。このプラスミドは、α-接合因子のプレプロペプチド、続いてscFv-LYSポリペプチド、最後にα-アグルチニンのC末端からなるキメラ融合タンパク質をコードしている。
実施例2 下等真核生物の細胞壁に固定化され、複雑な混合物中のトラセオリドに対して高い特異性で結合し得る同族キメラタンパク質群をコードする遺伝子群の構築
ハイブリドーマ細胞系統からのRNAの単離、cDNAの調製並びに抗体可変領域をコードする遺伝子フラグメントのPCR法による増幅は、文献記載の常法にしたがって行った。例えばOrlandi他(1989)の報文参照。PCR増幅では、様々なオリゴヌクレオチドプライマーを使用した。
重鎖フラグメントに対しては、下記のプライマーを使用した。
軽鎖フラグメント(κ鎖)に対しては、下記のプライマーを使用した。
pUR4143の構築
将来の構築作業を簡単にするために、pUR4122(上記参照)のインベルターゼシグナル配列とscFv-LYSの連結部にEagI制限部位を導入した。これは、配列番号:1に示す合成フラグメント内の約110bpのEcoRI-PstIフラグメントを下記配列の合成アダプターで置換することによって達成した。
こうして得たプラスミドをpUR4122.1と名付けたが、このpUR4122.1は、EagI部位の後にインフレームで続く単鎖Fvをプレプロα-接合因子配列又はSUC2インベルターゼシグナル配列のいずれかに引き続いて発現させるための構築ベクターである。
重鎖PCRフラグメントをPstI及びBstEIIで消化して、約230bpのPstIフラグメントと約110bpのPstI/BstEIIフラグメントの2種類のフラグメントを得た。後者のフラグメントを、PstI及びBstEIIで消化しておいたベクターpUR4122.1にクローニングした。こうして新たに得られたプラスミド(pUR4122.2)をSacI及びXhoIで消化し、次いでこのベクターに軽鎖PCRフラグメント(同じ制限酵素で消化しておいたもの)をクローニングしてpUR4122.3を得た。このプラスミドをPstIで消化し、しかる後にこのプラスミドベクターに上記の約230bpのPstIフラグメントをクローニングして、pUR4143と呼ばれるプラスミドを得た。2通りに配向している可能性があるが、通例の制限酵素分析によって選択することができる。pUR4122に元々存在していたscFv-LYS遺伝子の代わりに、この新たなプラスミドpUR4143には抗トラセオリド抗体02/01/01のscFv-TRASフラグメントをコードする遺伝子が含まれている(714bpのPstI-XhoIフラグメントのヌクレオチド配列に関しては配列番号:12参照)。
pUR4178及びpUR4179の構築
pUR4143をEagI及びHindIIIで消化すれば、約715bpのフラグメントが単離できる。このフラグメントは、同じ制限酵素で消化しておいたpUR2741及びpUR4175のベクター主鎖にクローニングすることができる。pUR2741の場合、そのようにしてプラスミドpUR2743.4が得られた(図5参照)。このプラスミドはXhoI及びHindIIIで消化してpUR4174の8kbpのXhoI-HindIIIフラグメントに連結することができ、pUR4178を与える(図8)。
pUR4175を出発ベクターとして用いた場合に得たプラスミドはpUR4179と名付けた(図9)。
プラスミドpUR4178及びpUR4179を共にS.cerevisiaeに導入した。
実施例3 ある種の条件下でトリセオリドの結合又は解離を改良するための、トラセオリドに結合し得るキメラタンパク質の結合部分の修飾
免疫反応の際の抗体の結合特性が、抗原の分子的性質に応じた抗体の結合領域中の相補性決定配列の微調整に起因して変化することは周知の免疫学的現象である。抗体フラグメントの抗原結合領域を抗原接触領域の分子モデルに基づいて適合させることによってこの現象をインヴィトロ(in vitro)で真似ることができる。
その一つの具体例が、抗ムスク抗体M02/01/01をもっと強く結合する変異体M02O501iへとタンパク質工学的に改変することである。
まず、抗リゾチーム抗体HYHEL10の原子座標(ブルックヘブン・プロテイン・データバンク(Brookhaven Protein Data Bank)に収録;3HFM)を鋳型として利用したホモロジーモデリングにより、M02/01/01可変フラグメント(Fv)の分子モデルを構築した。この分子モデルを、Silicon Graphics 4D240ワークステーション上でBiosym製のプログラムDISCOVERに基づいて、モレキュラー・メカニクス(Molecular Mechanics)法及びモレキュラー・ダイナミクス(Molecular Dynamics)法を用いて改良した。次に、こうして得たFvの結合部位のマッピングをCDR領域にムスク抗原を視覚的にドッキングさせて行った後、再度モレキュラー・ダイナミクス法を用いて改良した。得られたモデルを充填効率(ファン・デル・ワールスの接触面積)について検討したところ、ALA H96をVALで置換すればリガンドとFvの間の(疎水性)接触面積が増えて相互作用が強まるものと結論できた(図8)。
この変異を実施例2で得たcDNA由来scFvのM02/01/01に導入したとき、得られるものはFv M020501iであり、少なくとも5倍増大した親和性をもつ変異体であると予想できたが、その増大した親和性は上記ムスク香料分子に対するFvの蛍光滴定法を利用して測定することができた。
実施例4 下等真核生物の細胞壁に固定化され、HCGのようなホルモンに結合し得るキメラタンパク質をコードする遺伝子の構築
ヒト絨毛性性腺刺激ホルモンに対するモノクローナル抗体由来の重鎖及び軽鎖フラグメントの可変領域をコードする遺伝子フラグメントを、実施例2に記載した方法と同様の方法でハイブリドーマから得た。
次に、これらのHCG VH及びVL遺伝子フラグメントを対応PstI-bstEII及びSacI-XhoI遺伝子フラグメントの置換によってプラスミドpUR4143にクローニングして、プラスミドpUR4146を得た。
実施例2に記載した方法と同様に、ヒト絨毛性性腺刺激ホルモンに対するモノクローナル抗体由来の重鎖及び軽鎖フラグメントの可変領域(scFv-HCGフラグメント)をコードする734bpのEagI-XhoIフラグメント(配列番号:13に示すヌクレオチド配列)をpUR4166から単離して、同一の制限酵素で消化しておいたpUR4178(実施例2参照)のベクター主鎖に実際に導入したが、同一の制限酵素で消化しておいたpUR4175(実施例1参照)のベクター主鎖にも導入できるはずである。こうしてプラスミドpUR4177(図9参照)及びpUR4180(図10参照)が得られるが、前者については実際にS.cerevisiaeのSU10株に導入し、後者もS.cerevisiaeのSU10株に導入できるはずである。
実施例5 下等真核生物の細胞壁に固定化され、HCGのようなホルモンに結合し得るキメラタンパク質scFv-FLO1をコードする遺伝子の構築
S.cerevisiaeにおけるフロキュレーション表現型に関連した遺伝子の一つがFLO1遺伝子である。FLO1遺伝子の主要部分を含んだクローンのDNA配列を今回決定した(2685bpのFLO1遺伝子を示す配列番号:14参照)。このクローン化フラグメントは、サザーンハイブリダイゼーション及びノーザンハイブリダイゼーションの結果ではゲノムコピーよりも約2kb短かったが、FLO1遺伝子の両端を含んでいた。DNA配列データの解析から、推定タンパク質には、分泌のためのシグナル配列と認められるN末端疎水性領域と、GPIアンカーの付着のためのシグナルとして機能していると思われる疎水性C末端と、多数のグリコシル化部位とが含まれていたが、グリコシル化部位は特にC末端側に多く含まれており、恣意的に決定したC末端(アミノ酸271〜894)には46.6%のセリンとトレオニンが含まれている。したがって、FLO1遺伝子産物は酵母細胞壁の中に配向して位置している可能性が高く、隣接細胞間の相互作用に直接関与しているらしい。
クローン化したFLO1配列は、したがって、種類の細胞壁アンカーによる細胞壁へのタンパク質又はペプチドの固定化に適している。scFv-HCGとFLO1タンパク質のC末端部分とからなるキメラタンパク質の生産には、出発ベクターとしてプラスミドpUR2990(図11参照)を使用できる。エピソーム型プラスミドpUR2990の調製法は、本出願人の係属中の特許出願で1994年1月20日(本願の優先期間中)に公開されたWO94/01567(UNILEVER)に記載されている。プラスミドpUR2990には、Humicolaのリパーゼをコードする遺伝子とFLO1遺伝子産物の推定C末端細胞壁アンカードメインをコードする遺伝子とからなるキメラ遺伝子が含まれており、そのキメラ遺伝子に先行してインベルターゼのシグナル配列(SUC2)及びGAL7プロモーターが存在している。さらに、このプラスミドには、酵母2μm配列、Eckard及びHollenberg(1983)の報文に記載された不完全Leu2プロモーター、及びLeu2遺伝子(Roy他(1991)の報文参照)も含まれている。実施例4記載のプラスミドpUR4146をPstI及びXhoIで消化すれば、scFv-GCGコーディング配列を含んだ約0.7kbpのPstI-XhoIフラグメントが得られる。このDNA配列をFLO1のC末端部分とSUC2シグナル配列との間にインフレームで融合させるには、このフラグメントをプラスミドpUR2990の9.3kbp EagI/NheI(部分)主鎖に直接連結すればよく、その結果、プラスミドpUR4196(図12参照)が得られる。このプラスミドは、SUC2シグナル配列とscFv-HCGの開始部分との間にAlaをコードする追加トリプレットと、FLO1タンパク質のC末端の最初のアミノ酸(Ser)の前にE-I-K-G-Gアミノ酸配列をコードする追加トリプレットを含んでいる。
以上の実施例1〜実施例5において抗体フラグメントの露出度が低すぎる場合には、抗体フラグメントのフレームワーク領域に変異を導入することによって生産レベルを上げることができる。このような変異導入は、文献に記載された公知技術を用いて、部位特異的変異導入法で行うこともできるし、(標的)ランダム変異誘発法で行うこともできる。
実施例6 下等真核生物の細胞壁に固定化され、コレステロールに結合し得るキメラタンパク質をコードする遺伝子の構築
文献には、コレステロールオキシダーゼについて2種類のDNA配列:すなわち、Brevibacterium sterolicum由来のchoB遺伝子(Ohta他(1991)の報文参照)とStreptomyces sp.SA-COO由来のchoA遺伝子(Ishizaka他(1989)の報文参照)が記載されている。コレステロールオキシダーゼ(EC 1.1.3.6)をコードするchoB遺伝子とAG-α1遺伝子の3′部分との融合DNAの構築には、染色体DNA上でのPCRを利用すればよい。染色体DNAはBrevibacterium sterolicumから常法にしたがって単離することができ、コレステロールオキシダーゼの成熟部分をコードするDNA部分の増幅は、下記のPCRプライマーcho01pcr及びcho02pcrによって行うことができる。
これらのプライマーは共に標的配列に特異的にハイブリダイズし得るので、上記遺伝子のコーディング部分が増幅されて、その特異的PCR産物は、プロテイナーゼK処理及びEcoRI及びHindIIIでの消化後、適当なベクター(ここでは、pTZ19R(Mead他(1986)の報文参照)が好ましい)に直接クローニングすることができるようになる。その結果、プラスミドpUR2985(図13参照)が得られる。
上記の制限部位の他に、これらのPCRプライマーはいずれも1.5kbpのDNAフラグメントの5′末端及び3′末端に別の制限部位を生じるので、それらの制限部位を利用すれば後段で上記フラグメントをその一端でSUC2シグナル配列又はプレプロ-α-接合因子シグナル配列と融合させ、かつもう一方の末端でα-アグルチニンのC末端コーディング部分と融合させることができる。プレプロMF配列の後での連結を容易にするには、PCRオリゴヌクレオチドプライマーcho01pcrの5′末端にNotI部位を導入して、例えばpUR4175中のscFv-Lysコーディング配列を含んだ731bpのEagI/NheIフラグメントでchoBコーディング配列を置換できるようにすればよい。
コレステロールオキシダーゼとα-アグルチニンとの酵素活性をもたない融合タンパク質を作るには、上述したpTZ19Rへのサブクローニング法を用いればよい。コレステロールオキシダーゼはFAD依存性酵素であり、Brevibacterium sterolicumの酵素については結晶構造にも既に決定されている(Vrielink他(1991)の報文参照)。この酵素はGly-X-Gly-X-X-Glyという配列のFAD結合ドメインの典型的なパターンに対する相同性をN末端付近(アミノ酸18〜23)に示す。プラスミドpUR2985に対する部位特異的インヴィトロ変異導入を製造業者のプロトコル(Bio-Rad社製のMuta-Geneキット)に基づいて利用すれば、Gly残基(群)をコードするトリプレット(群)を他のアミノ酸をコードするトリプレットで置換してFAD結合部位を不活性化し、ひいては上記酵素を不活性化できるはずである。例えば、保存されたGly残基のうちの2つに部位特異的な変異を導入するには、下記のプライマーを使用することができる。
このプライマーを用いて変異導入を行うと、プラスミドpUR2986が得られるはずである。このプラスミドからは、NotI/NheI消化によって、不活性化されていると推定されるコレステロールオキシダーゼをコードしたDNAが1527bpフラグメントとして放出されるはずであり、このDNAフラグメントをpUR4175中のscFv-Lysコーディング配列との置換に直接使用すれば、プラスミドpUR2987(図14参照)が得られる。分泌がSUC2シグナル配列で支配されているような、酵母分泌ベクターの変異体を得るには、例えばプラスミドpUR2986の1823bp長のSacI/NheIセグメントを用いてpUR4174中のSacI/NheIフラグメントを置換すればよい。こうしたFAD結合部位の不活性化は他の変異体に対しても好ましいと考えられるが、それは、活性中心を変えないでおくとコレステロールオキシダーゼのコレステロールに対する結合特性が変わらないと予想されるからである。上述した成熟コーディング配列の18位及び20位でのGly→Alaへの置換以外にも、他のどのような適当なアミノ酸への変更を行うこともできる。
酵素を不活性化するには、例えば正にプロトン受容体として機能する部位に位置する残基であるGlu331の置換などによって、活性部位のくぼみ構造部において部位特異的変異導入を行うこともでき、そうすることによって酵素活性をもたない固定化融合タンパク質の新たな変異体を得ることができる。
実施例7 乳酸菌の細胞壁に固定化され、コレステロールに結合し得るキメラタンパク質をコードする遺伝子の構築
Lactococcus lactis subsp.cremorisのプロテイナーゼが127アミノ酸残基からなる長いC末端を介して細胞壁に固着していることが報告されている。Kok他(1988)並びにKok(1990)の報文参照。実施例6に記載した方法と同様の方法で、Brevibacterium sterolicumのコレステロールオキシダーゼ(choB)をLactococcus lactisの細胞壁に固定化することができる。choB構造遺伝子とN末端シグナル配列とLactococcus lactisのプロテイナーゼのC末端アンカーとの間で融合を行うことができる。プラスミドpGKV550(図15参照)は、プロモーター、リボソーム結合部位及び上述のシグナル配列とアンカー配列をコードしたDNAフラグメントを含め、Lactococcus lactis subsp.cremoris Wg2株の全プロテイナーゼオペロンを含んでいる(Kok(1990)の報文参照)。最初に、以下に示すようなpGKV550上でのPCRによって、シグナル配列の主要部とその両側にClaI部位とEagI部位の含まれたDNAフラグメントを構築することができる。
実施例6で述べたPCR反応の後で、98bpのPCRフラグメントを単離してClaI及びHindIIIで消化すればよい。次に、pGKV550をClaIで部分的に切断し、HindIIIで完全に切断すれば、かかる消化の後、上記プロテイナーゼ遺伝子のプロモーター、リボソーム結合部位、N末端の8個のアミノ酸と127個のC末端アミノ酸の細胞壁結合フラグメントをコードしたDNAフラグメントを含んだベクターフラグメントをゲル上で単離することができる。
prtPアンカードメインとの融合に適したコレステロールオキシダーゼ遺伝子のコピーは、pUR2985(実施例6)を鋳型として用い、プライマーcho02pcrの代わりに下記のプライマーcho03pcrをプライマーcho01pcr(実施例6)と組合わせて利用したPCR反応によって作製することができる。
この反応によって生じる約1.53kbpフラグメントをNotI及びHindIIIで消化して得られる分子は、次いでpUR2988(図16参照)由来のEagI/HindIII大フラグメントと連結することができる。こうして得られるプラスミドpUR2989は、プロテイナーゼ遺伝子のシグナル配列とC末端細胞壁アンカードメインとの間に挿入されたコレステロールオキシダーゼのコーディング配列を含んでいるはずである。このプラスミドは、電気穿孔法でLactococcus lactis subsp.lactis MG1363株に導入すればプロテイナーゼプロモーターの制御下でコレステロールオキシダーゼを発現するはずである。膜を通しての輸送はプロテイナーゼのシグナル配列によって媒介されるはずであり、コレステロールオキシダーゼの固定化はプロテイナーゼのアンカーによって媒介される。LactococcusがFADを同時に分泌する可能性は低いので、コレステロールオキシダーゼはコレステロールに結合し得るものの、活性ではないはずである。
実施例8 下等真核生物の細胞壁に固定化され、上皮成長因子のような成長ホルモンに結合し得るキメラタンパク質をコードする遺伝子の構築
ヒト上皮成長因子(EGF)を大量に単離するため、結合ドメインが細胞壁アンカーとしてのα-アグルチニンのC末端部分と融合した形の対応レセプターを利用することができる。ヒト上皮成長因子の全cDNA配列がクローニングされ、配列が決定されている。EGF結合能力をもつ融合タンパク質の構築には、成熟型レセプターのN末端部分から中央の23個のアミノ酸残基からなる膜貫通領域までを利用することができる。
こうした構築にはプラスミドpUR4175を利用することができる。EagI及びNheI(部分)での消化によって、scFvのコードされた配列を含む731bpのDNAフラグメントが放出されるが、このフラグメントは、ヒト上皮成長因子レセプターの最初の621個のアミノ酸残基をコードするDNAフラグメントで置換することができる。既存のヒトcDNAライブラリーを出発点とするか、さもなければ例えばA431ガン腫細胞(Ullrich他(1984)の報文参照)のような優先的にEGFレセプターを過剰産生する細胞から常法によってcDNAライブラリーを作成しておいて、PCRをさらに行えばヒト上皮成長因子レセプターの細胞外結合ドメイン(アミノ酸1〜622)とα-アグルチニンのC末端部分とのインフレーム連結体を作成することができる。
ヒト上皮成長因子レセプターとα-アグルチニンのC末端とのインフレーム連結のためのPCRオリゴヌクレオチド
この融合によって、シグナル配列とEGFレセプターの成熟N末端の間に2つのAla残基が追加されることになる。こうして新たに得られる1.9kbpのPCRフラグメントをNotI及びNheIで消化すれば、同じ酵素で消化しておいたベクターpUR4175に直接連結することができ、GAL7プロモーターと、プレプロα-接合因子配列と、EGFレセプター結合ドメイン遺伝子/α-アグルチニンの融合遺伝子と、酵母2μm配列と、不完全LEU2プロモーターと、LEU2遺伝子とを含んだプラスミドpUR2993(図17参照)を得ることができる。このプラスミドはS.cerevisiaeを形質転換することができ、形質転換細胞はYP培地中で培養することができ、キメラタンパク質の発現はこの培地にガラクトースを添加することによって誘導し得る。
実施例9 酵母の細胞壁に固定化され、「ラクダ科」重鎖抗体の結合ドメインを含んでなるキメラタンパク質をコードする遺伝子の構築
最近、ラクダ類並びにその近縁種(例えばラマ)が重鎖のダイマーだけで構成されたIgG抗体分子をかなりの量で含んでいることが報告されている(Hamers-Casterman他(1993)の報文参照)。このような「重鎖」抗体は軽鎖を欠いているが、それにもかかわらず広い抗原結合範囲を有していることが実証されている。この種の抗体の可変領域を生産し、酵母の細胞壁の外側に連結できることを示すために、下記の構築体を作製した。
pUR2997,pUR2998及びpUR2999の構築
pUR4177(実施例4、図9参照)の約2.1kbpのEagI-HindIIIフラグメントを単離した。PCR法を利用して、EagI部位のすぐ上流にEcoRI部位を導入したが、そうして得られたEcoRI部位の塩基CはEagI部位の最初の塩基Cと同一である。こうして得たEcoRI-HindIIIフラグメントを、EcoRI及びHindIIIで消化しておいたプラスミドpEMBL9に連結して、pUR4177.Aを得た。
プラスミドpUR4177.AのEcoRI/NheIフラグメントを、3種類の異なる合成DNAフラグメント(配列番号:32,配列番号:33及び配列番号:34)のEcoRI/NheIフラグメントで置換して、それぞれ、pUR2997、pUR2998及びpUR2999を得た。pUR2997とpUR2998の約1.5kbpのBstEII-HindIIIフラグメントを単離した。
pUR4421の構築
プラスミドpEMBL9(Dente他(1983)の報文参照)のマルチクローニング部位(EcoRI部位からHindIII部位までの範囲)を、下記に示すヌクレオチド配列をもつ合成DNAフラグメント(コーディング鎖を示す配列番号:35並びに非コーディング鎖を示す配列番号:36参照)で置換した。このヌクレオチド配列の5′部分は、EagI部位、ラクダVH遺伝子フラグメントの最初の4つのコドン(ヌクレオチド16〜27)、及び5番目及び6番目のコドン(ヌクレオチド28〜33)に一致するXhoI部位(CTCGAG)を含んでいる。その3′部分は、ラクダVH遺伝子の最後の5つのコドン(ヌクレオチド46〜60)(その一部はBstEII部位に一致する)、Mycテールの11個のコドン(ヌクレオチド61〜93)(これら11個のコドンを含んだ配列番号:35及びアミノ酸配列を示す配列番号:37を参照)、及びEcoRI部位(GAATTC)を含んでいる。このEcoRI部位はpEMBL9に元々存在していたもので、ヌクレオチド配列の5′末端が下記の(EcoRI)として示す通りAATTCの代わりにAATTTを含んでいるので、機能しなくなっている。得られたプラスミドはpUR4421と呼ばれる。ラクダVHフラグメントはQ-V-Kというアミノ酸で始まり、V-S-Sというアミノ酸で終わる。
pUR4424の構築
プラスミドpB09をXhoI及びBstEIIで消化して、アガロースゲルから約0.34kbpのDNAフラグメントを単離した。このフラグメントは、ラクダVHフラグメントの最初の4つのアミノ酸と最後の5つのアミノ酸の欠けたトランケート型VHフラグメントをコードしている。プラスミドpB09は大腸菌JM109 pB09として1993年4月20日にオランダのバールン(Baarn)のCentraal Buresu voor Schimmelcultures(CBS)に寄託番号CBS271.93として寄託された。ラクダVHフラグメントのDNA及びアミノ酸配列の後には、欧州特許出願第93201239.6号(未公開)の図6Bに記載された通り、プラスミドpB09中に存在するFlag配列が続いている。この欧州特許出願の開示内容は文献の援用によって本明細書の内容の一部をなす。得られた約0.34kbpのフラグメントをpUR4421にクローニングした。そのため、プラスミドpUR4421をXhoI及びHindIIIで消化した後、アガロースゲルから約4kbのベクターフラグメントを単離した。得られたベクターを約0.34kbpのXhoI/BstEIIフラグメント及び下記の配列の合成DNAリンカーと連結して、プラスミドpUR4421-09を得た。
プラスミドpSY16をEagI及びHindIIIで消化して、約6.5kbp長のベクター鎖を単離して、それをpUR4421-09由来の約0.38kbpのEagI/HindIIIフラグメントと連結して、pUR4424を得た。
pUR4482及びpUR4483の構築
pUR4424からインベルターゼのシグナル配列とラクダ重鎖可変09(CHV09)フラグメントのコードされた約0.44kbpのSacI-BstEIIフラグメントを単離するとともに、約6.3kbpのSacI-HindIIIベクターフラグメントも単離した。約6.3kbpフラグメントとpUR4424の約0.44kbpフラグメントをpUR2997又はpUR2998のBstEII-HindIIIフラグメントと連結してそれぞれpUR4482とpUR4483を得た。
プラスミドpUR4482は、インベルターゼシグナル配列とCHV09可変領域とMycテールとラクダ「X-P-X-P」ヒンジ領域(Hamers-Casterman他(1993)の報文参照)とα-アグルチニン細胞壁アンカー領域との融合タンパク質の発現のための酵母エピソーム型発現プラスミドである。pUR4483はMycテールを含んではいるが「X-P-X-P」ヒンジ領域を含んでいない点でpUR4482とは異なる。同様に、pUR2999のBstEII-HindIIIフラグメントを、上記の約6.3kbpベクターフラグメント及びpUR4424の約0.44kbpフラグメントと連結することもでき、pUR4497が得られるが、このプラスミドは「X-P-X-P」ヒンジ領域を含んではいるがMycテールを含んでいない点でpUR4482とは異なる。
プラスミドpUR4424、pUR4482及びpUR4483を電気穿孔法でSaccharomyces cerevisiaeSU10株に導入し、ロイシンを欠く平板上で各形質転換体を選択した。pUR4424、pUR4482及びpUR4483の各々で形質転換したSU10株由来の各形質転換体を、5%ガラクトース添加YP培地上で増殖させ、本出願人の係属中の特許出願で1994年1月20日に公開されたWO94/01567号(UNILEVER)の例1に記載された免疫蛍光顕微鏡法で分析した。細胞表面に存在する、ラクダ抗体とMycテールを共に含んだキメラタンパク質を検出するため、この文献記載の方法に若干の修正を加えた。
一つの方法では、マウスのモノクローナル抗Myc抗体をキメラタンパク質のMycテールに結合させるための第1抗体として使用し、続いて結合マウス抗体を検出するためにフルオレセンイソチオシアネート(FITC)標識ポリクローナル抗マウスIg抗血清(Sigma社製,製品番号F0527)を使用し、陽性シグナルの有無を蛍光顕微鏡で測定した。
もう一つの方法では、ラクダ抗体と交差反応することが既に実証されているウサギのポリクローナル抗ヒトIgG血清をキメラタンパク質のラクダ抗体に結合させるための第1抗体として使用し、続いて結合ウサギ抗体を検出するためにFITC標識ポリクローナル抗ウサギIg抗血清(Sigma社製,製品番号F0382)を使用し、陽性シグナルの有無を蛍光顕微鏡で測定した。
図19及び図20に示す結果から、CHV09フラグメントとα-アグルチニン細胞壁アンカー領域の融合タンパク質を産生する細胞(pUR4482及びpUR4483)で蛍光が観察されることが分かる。しかし、このようなアンカーのないCHV09フラグメントを産生する細胞(pUR4424)では、同じ状況下で観察しても、蛍光は全く認められなかった。
ブダペスト条約に基づく微生物の寄託に関する情報は本明細書第27頁3〜6行に記載されている。欧州特許条約(EPC)規則28(4)又はEPCの締約国以外の国での同様の規定に基づき、請求のあったときはこれらの寄託菌株の試料が専門家のみに分譲されることを要求する。
配 列 表
(1) 一般情報
(i) 出願人
(A) 名称:ユニリーバー・エヌ・ヴイ
(B) 通りの住所:ヴェーナ455
(C) 都市名:ロッテルダム
(E) 国名:オランダ
(F) 郵便番号(ZIP):NL-3013 AL
(A) 名称:ユニリーバー・ピー・エル・シー
(B) 通りの住所:ブラックフライヤーズ、ユニリーバー・ハウス
(C) 都市名:ロンドン
(E) 国名:英国
(F) 郵便番号(ZIP):EC4P 4BQ
(A) 名称:レオン・ゲラルダス・ジェイ・フランケン
(B) 通りの住所:ゲルダースストラート 90
(C) 都市名:ロッテルダム
(E) 国名:オランダ
(F) 郵便番号(ZIP):NL-3011 MP
(A) 名称:ピーター・ディ・イー・ゲウス
(B) 通りの住所:ボエイエ 24
(C) 都市名:バーレンドレヒト
(E) 国名:オランダ
(F) 郵便番号(ZIP):NL-2991 KB
(A) 名称:フランシスカス・マリア・クリス
(B) 通りの住所:ベネデンラングス 102
(C) 都市名:アムステルダム
(E) 国名:オランダ
(F) 郵便番号(ZIP):NL-1025 KL
(A) 名称:ホルガー・ヨーク・トシュカ;c/o ラングネーズ・イグロ,BR3
(B) 通りの住所:アエケルン 1
(C) 都市名:レケン
(E) 国名:ドイツ
(F) 郵便番号(ZIP):D-48734
(A) 名称:コルネリス・テオドラス・ヴェルリップス
(B) 通りの住所:ハーグドルン 18
(C) 都市名:マースルイス
(E) 国名:オランダ
(F) 郵便番号(ZIP):NL-3142 KB
(ii) 発明の名称:
(iii) 配列の数:40
(iv) コンピューター可読形式:
(A) 記録媒体:フロッピーディスク
(B) コンピューター:IBM PCコンパチブル
(C) オペレーションシステム(OS):PC-DOS/MS-DOS
(D) ソフトウェア:PatentIn Release ♯1.0,ヴァージョン♯1.25(EPO)
(2) 配列番号:1に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:231塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:pUR4119中のフラグメント
(xi) 配列:配列番号:1:
(2) 配列番号:2に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:21塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:XhoI−NheIリンカーのコーディング鎖
(xi) 配列:配列番号:2:
(2) 配列番号:3に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:XhoI−NheIリンカーの非コーディング鎖
(xi) 配列:配列番号:3:
(2) 配列番号:4に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:21塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EagI−PstIリンカーのコーディング鎖
(xi) 配列:配列番号:4:
(2) 配列番号:5に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:13塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EagI−PstIリンカーの非コーディング鎖
(xi) 配列:配列番号:5:
(2) 配列番号:6に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:22塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:PCRプライマーA(重鎖)
(xi) 配列:配列番号:6:
(2) 配列番号:7に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:32塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:PCRプライマーB(重鎖)
(xi) 配列:配列番号:7:
(2) 配列番号:8に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:24塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:PCRプライマーC(軽鎖)
(xi) 配列:配列番号:8:
(2) 配列番号:9に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:22塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:PCRプライマーD(軽鎖)
(xi) 配列:配列番号:9:
(2) 配列番号:10に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:26塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EcoRI−PstIリンカーのコーディング鎖
(xi) 配列:配列番号:10:
(2) 配列番号:11に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:18塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EcoRI−PstIリンカーの非コーディング鎖
(xi) 配列:配列番号:11:
(2) 配列番号:12に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:714塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ScFv 抗トラセオリド02/01/01
(xi) 配列:配列番号:12:
(2) 配列番号:13に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:734塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ScFv 抗HCG
(xi) 配列:配列番号:13:
(2) 配列番号:14に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:2685塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(v) 起源
(A) 生物名:Saccharomyces cerevisieae
(vii) 直接の起源:
(B) クローン:pYY105
(ix) 配列の特徴:
(A) 特徴を表わす記号:CDS
(B) 存在位置:1...2685
(D) 他の情報:/遺伝子産物=“フロキュレーションタンパク質”/遺伝子=“FLO1”
(xi) 配列:配列番号:14:
(2) 配列番号:15に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:894アミノ酸
(B) 配列の型:アミノ酸
(D) トポロジー:直鎖状
(ii) 配列の種類:タンパク質
(xi) 配列:配列番号:15:
(2) 配列番号:16に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:19塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ChoB鋳型コーディング鎖
(xi) 配列:配列番号:16:
(2) 配列番号:17に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:19塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ChoB鋳型非コーディング鎖
(xi) 配列:配列番号:17:
(2) 配列番号:18に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:37塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:Cho01pcrプライマー
(xi) 配列:配列番号:18:
(2) 配列番号:19に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:42塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:cho02pcrプライマー
(xi) 配列:配列番号:19:
(2) 配列番号:20に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:21塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ChoB鋳型コーディング鎖
(xi) 配列:配列番号:20:
(2) 配列番号:21に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:21塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ChoB鋳型コーディング鎖
(xi) 配列:配列番号:21:
(2) 配列番号:22に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:42塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:変異導入プライマーChoB
(xi) 配列:配列番号:22:
(2) 配列番号:23に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:42塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ChoB鋳型コーディング鎖
(xi) 配列:配列番号:23:
(2) 配列番号:24に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:29塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:プライマーprt1
(xi) 配列:配列番号:24:
(2) 配列番号:25に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:32塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:プロテイナーゼ鋳型非コーディング鎖
(xi) 配列:配列番号:25:
(2) 配列番号:26に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:27塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:プロテイナーゼ鋳型コーディング鎖
(xi) 配列:配列番号:26:
(2) 配列番号:27に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:39塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:prt2プライマー
(xi) 配列:配列番号:27:
(2) 配列番号:28に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:33塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EGF1プライマー
(xi) 配列:配列番号:28:
(2) 配列番号:29に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:33塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EGFレセプター鋳型非コーディング鎖
(xi) 配列:配列番号:29:
(2) 配列番号:30に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:30塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EGFレセプター鋳型コーディング鎖
(xi) 配列:配列番号:30:
(2) 配列番号:31に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:40塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:EGF2プライマー
(xi) 配列:配列番号:31:
(2) 配列番号:32に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:177塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:MycT及びヒンジ部とのAGα1リンカー
(xi) 配列:配列番号:32:
(2) 配列番号:33に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:63塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:MycTとのAGα1リンカー
(xi) 配列:配列番号:33:
(2) 配列番号:34に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:144塩基対
(B) 配列の型:核酸
(C) 鎖の数:二本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:ヒンジ部とのAGα1リンカー
(xi) 配列:配列番号:34:
(2) 配列番号:35に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:119塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:pUR4421コーディング鎖中のフラグメント
(xi) 配列:配列番号:35:
(2) 配列番号:36に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:119塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:pUR4421非コーディング鎖中のフラグメント
(xi) 配列:配列番号:36:
(2) 配列番号:37に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:11アミノ酸
(B) 配列の型:アミノ酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:ペプチド
(vii) 直接の起源:
(B) クローン:Mycテール
(xi) 配列:配列番号:37:
(2) 配列番号:38に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:21塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:BstEII−HindIIIリンカーのコーディング鎖
(xi) 配列:配列番号:38:
(2) 配列番号:39に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:20塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:BstEII−HindIIIリンカーの非コーディング鎖
(xi) 配列:配列番号:39:
(2) 配列番号:40に関する情報:
(i) 配列の特徴:
(A) 配列の長さ:28塩基対
(B) 配列の型:核酸
(C) 鎖の数:一本鎖
(D) トポロジー:直鎖状
(ii) 配列の種類:DNA(genomic)
(vii) 直接の起源:
(B) クローン:プライマーcho03pcr
(xi) 配列:配列番号:40:
Claims (12)
- 特定の化合物に対する結合能力を有する、抗体、抗体フラグメント、及び複数の抗体フラグメントの組み合わせからなる群から選択される結合タンパク質を固定化するための方法にして、上記結合タンパク質又は上記の特異的結合能力を保持したその機能的部分であってSaccharomyces属に属する酵母の外部に連結した上記結合タンパク質又は機能的部分の生産に組換えDNA技術を利用して、Saccharomyces属に属する酵母の細胞壁に固着し得るアンカータンパク質のアンカー部分(当該アンカー部分は上記アンカータンパク質のC末端部分から導かれる)のN末端に上記結合タンパク質又はその機能的部分がそのC末端で結合しているようなキメラタンパク質をSaccharomyces属に属する酵母の細胞が産生及び分泌できるようにすることによって上記タンパク質又はその機能的部分が細胞壁又は細胞壁の外側に局在化するようにさせることを含んでなる方法。
- 下記の(i)及び(ii)を含んでなる組換えポリヌクレオチド。
(i)抗体、抗体フラグメント、及び複数の抗体フラグメントの組み合わせからなる群から選択される結合タンパク質又は特異的結合能力を保持したその機能的部分をコードする構造遺伝子、及び
(ii)Saccharomyces属に属する酵母の細胞壁に固着し得るアンカータンパク質をコードする遺伝子の少なくとも一部分であって、当該遺伝子部分がアンカータンパク質のアンカー部分(当該アンカー部分は上記アンカータンパク質のC末端部分から導かれる)をコードしている遺伝子部分。 - 請求項2記載のポリヌクレオチドにおいて、上記アンカータンパク質が、Saccharomyces由来のα−アグルチニン、a‐アグルチニン、FLO1からなる群から選択されることを特徴とするポリヌクレオチド。
- 請求項2記載のポリヌクレオチドにおいて、当該ポリヌクレオチドが、当該ポリヌクレオチドの発現産物の分泌を確実にするためのシグナルペプチドをコードするヌクレオチド配列をさらに含んでいることを特徴とするポリヌクレオチド。
- 請求項4記載のポリヌクレオチドにおいて、上記シグナルペプチドが、Saccharomycesのインベルターゼに由来することを特徴とするポリヌクレオチド。
- 請求項2乃至請求項5のいずれか1項記載のポリヌクレオチドにして、誘導型プロモーターとすることができるプロモーターに機能的に連結しているポリヌクレオチド。
- 請求項2乃至請求項6のいずれか1項記載のポリヌクレオチドを含んでなる組換えベクター。
- 請求項2乃至請求項6のいずれか1項記載のポリヌクレオチドにコードされたキメラタンパク質。
- 細胞の外部に細胞壁をもつSaccharomyces属に属する酵母の細胞にして、請求項2乃至請求項6のいずれか1項記載のポリヌクレオチドを少なくとも1つ保有する宿主細胞。
- 請求項9記載の細胞にして、その染色体に請求項2乃至請求項6のいずれか1項記載のポリヌクレオチドが少なくとも1つ組込まれていることを特徴とする細胞。
- 請求項8記載のキメラタンパク質がその細胞壁に固定化されているSaccharomyces属に属する酵母の細胞にして、当該キメラタンパク質の結合タンパク質部分がその細胞壁中又は細胞壁の外側に局在化している細胞。
- 特定化合物に対する結合能力を保持している固定化された、抗体、抗体フラグメント、及び複数の抗体フラグメントの組み合わせからなる群から選択される結合タンパク質又はその機能的部分を用いて単離プロセスを行う方法にして、上記特定化合物を含んだ媒質を請求項9乃至請求項11のいずれか1項記載のSaccharomyces属に属する酵母の細胞に、特定化合物と固定化結合タンパク質との複合体が形成されるような条件下で、接触させ、特定化合物を当初含んでいた上記媒質から当該複合体を分離し、所望により、結合タンパク質又はその機能的部分から特定化合物を解離させることを特徴とする方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| EP93200350 | 1993-02-10 | ||
| NL93200350.2 | 1993-02-10 | ||
| PCT/EP1994/000427 WO1994018330A1 (en) | 1993-02-10 | 1994-02-10 | Immobilized proteins with specific binding capacities and their use in processes and products |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JPH08506484A JPH08506484A (ja) | 1996-07-16 |
| JP3691055B2 true JP3691055B2 (ja) | 2005-08-31 |
Family
ID=8213623
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP51767994A Expired - Lifetime JP3691055B2 (ja) | 1993-02-10 | 1994-02-10 | 特異的結合力をもつ固定化タンパク質並びに各種プロセス・物品におけるそれらの使用 |
Country Status (12)
| Country | Link |
|---|---|
| US (1) | US6114147A (ja) |
| EP (1) | EP0682710B1 (ja) |
| JP (1) | JP3691055B2 (ja) |
| AT (1) | ATE253118T1 (ja) |
| AU (1) | AU6139794A (ja) |
| DE (1) | DE69433279T2 (ja) |
| DK (1) | DK0682710T3 (ja) |
| ES (1) | ES2210248T3 (ja) |
| FI (1) | FI953792A0 (ja) |
| PT (1) | PT682710E (ja) |
| WO (1) | WO1994018330A1 (ja) |
| ZA (1) | ZA94916B (ja) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US9873746B2 (en) | 2003-10-22 | 2018-01-23 | Keck Graduate Institute | Methods of synthesizing heteromultimeric polypeptides in yeast using a haploid mating strategy |
Families Citing this family (88)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20030036092A1 (en) * | 1991-11-15 | 2003-02-20 | Board Of Regents, The University Of Texas System | Directed evolution of enzymes and antibodies |
| US5866344A (en) * | 1991-11-15 | 1999-02-02 | Board Of Regents, The University Of Texas System | Antibody selection methods using cell surface expressed libraries |
| CN1070533C (zh) * | 1993-02-26 | 2001-09-05 | 三宝乐啤酒株式会社 | 酵母凝集基因及含这些基因的酵母 |
| US5766914A (en) * | 1995-01-26 | 1998-06-16 | Michigan State University | Method of producing and purifying enzymes |
| AU724621B2 (en) * | 1996-01-29 | 2000-09-28 | Georgetown University | Amplification of response from expressed recombinant protein |
| US6699658B1 (en) | 1996-05-31 | 2004-03-02 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| US6696251B1 (en) | 1996-05-31 | 2004-02-24 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| US6300065B1 (en) | 1996-05-31 | 2001-10-09 | Board Of Trustees Of The University Of Illinois | Yeast cell surface display of proteins and uses thereof |
| EP0879878A1 (en) * | 1997-05-23 | 1998-11-25 | Quest International B.V. | Beer and similar light-sensitive beverages with increased flavour stability and process for producing them |
| EP0916726A1 (en) | 1997-11-13 | 1999-05-19 | Rijksuniversiteit te Groningen | Attaching substances to micro-organisms |
| US6759243B2 (en) | 1998-01-20 | 2004-07-06 | Board Of Trustees Of The University Of Illinois | High affinity TCR proteins and methods |
| ATE535154T1 (de) | 1998-03-12 | 2011-12-15 | Vhsquared Ltd | Produkten die inaktivierte hefen oder schimmel enthalten, die auf ihrer aussenoberfläche aktive antikörper haben |
| EP0954978B1 (en) | 1998-03-12 | 2011-11-30 | VHsquared Limited | New products comprising inactivated yeasts or moulds provided with active antibodies |
| US7060669B1 (en) | 1999-03-25 | 2006-06-13 | Valtion Teknillinen Tutkimuskeskus | Process for partitioning of proteins |
| US7132273B1 (en) | 2000-07-26 | 2006-11-07 | Korea Research Institute Of Bioscience And Biotechnology | Cell wall anchor proteins derived from yeast, genes thereof and cell surface expression systems using the same |
| WO2002046400A2 (en) * | 2000-12-08 | 2002-06-13 | The Board Of Trustees Of The University Of Illinois | Mutated class ii major histocompatibility proteins |
| WO2002048193A2 (en) * | 2000-12-13 | 2002-06-20 | Unilever N.V. | Camelidae antibody arrays |
| KR100714670B1 (ko) * | 2001-04-19 | 2007-05-07 | 간사이가가쿠기카이세이사쿠가부시키가이샤 | 세포 표층 결합 단백질 및 이의 용도 |
| EP1395648B1 (en) | 2001-06-11 | 2009-04-22 | Applied NanoSystems B.V. | Methods for binding acma-type protein anchor fusions to cell-wall material of micro-organisms |
| US20030186374A1 (en) | 2001-10-01 | 2003-10-02 | Hufton Simon E. | Multi-chain eukaryotic display vectors and uses thereof |
| WO2003064608A2 (en) * | 2002-01-25 | 2003-08-07 | The Board Of Trustees Of The Leland Stanford Junior University | Surface based translation system |
| US20040067532A1 (en) | 2002-08-12 | 2004-04-08 | Genetastix Corporation | High throughput generation and affinity maturation of humanized antibody |
| US7425620B2 (en) | 2002-08-14 | 2008-09-16 | Scott Koenig | FcγRIIB-specific antibodies and methods of use thereof |
| US8193318B2 (en) * | 2002-08-14 | 2012-06-05 | Macrogenics, Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US8946387B2 (en) * | 2002-08-14 | 2015-02-03 | Macrogenics, Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US8530627B2 (en) * | 2002-08-14 | 2013-09-10 | Macrogenics, Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US8187593B2 (en) | 2002-08-14 | 2012-05-29 | Macrogenics, Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US8968730B2 (en) | 2002-08-14 | 2015-03-03 | Macrogenics Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US8044180B2 (en) * | 2002-08-14 | 2011-10-25 | Macrogenics, Inc. | FcγRIIB specific antibodies and methods of use thereof |
| US7563600B2 (en) | 2002-09-12 | 2009-07-21 | Combimatrix Corporation | Microarray synthesis and assembly of gene-length polynucleotides |
| US7355008B2 (en) | 2003-01-09 | 2008-04-08 | Macrogenics, Inc. | Identification and engineering of antibodies with variant Fc regions and methods of using same |
| US7960512B2 (en) | 2003-01-09 | 2011-06-14 | Macrogenics, Inc. | Identification and engineering of antibodies with variant Fc regions and methods of using same |
| WO2005055944A2 (en) | 2003-12-05 | 2005-06-23 | Cincinnati Children's Hospital Medical Center | Oligosaccharide compositions and use thereof in the treatment of infection |
| KR101297146B1 (ko) | 2004-05-10 | 2013-08-21 | 마크로제닉스, 인크. | 인간화 FcγRIIB 특이적 항체 및 그의 사용 방법 |
| WO2007024249A2 (en) | 2004-11-10 | 2007-03-01 | Macrogenics, Inc. | Engineering fc antibody regions to confer effector function |
| EP1674479A1 (en) * | 2004-12-22 | 2006-06-28 | Memorial Sloan-Kettering Cancer Center | Modulation of Fc Gamma receptors for optimizing immunotherapy |
| US9284375B2 (en) | 2005-04-15 | 2016-03-15 | Macrogenics, Inc. | Covalent diabodies and uses thereof |
| US11254748B2 (en) | 2005-04-15 | 2022-02-22 | Macrogenics, Inc. | Covalent diabodies and uses thereof |
| ES2971647T3 (es) | 2005-04-15 | 2024-06-06 | Macrogenics Inc | Diacuerpos covalentes y usos de los mismos |
| US9963510B2 (en) | 2005-04-15 | 2018-05-08 | Macrogenics, Inc. | Covalent diabodies and uses thereof |
| EP1726650A1 (en) * | 2005-05-27 | 2006-11-29 | Universitätsklinikum Freiburg | Monoclonal antibodies and single chain antibody fragments against cell-surface prostate specific membrane antigen |
| WO2006138700A2 (en) * | 2005-06-17 | 2006-12-28 | Biorexis Pharmaceutical Corporation | Anchored transferrin fusion protein libraries |
| CA2615057A1 (en) * | 2005-07-12 | 2007-01-18 | Codon Devices, Inc. | Compositions and methods for biocatalytic engineering |
| EP1746103A1 (en) * | 2005-07-20 | 2007-01-24 | Applied NanoSystems B.V. | Bifunctional protein anchors |
| JP5105485B2 (ja) | 2005-08-10 | 2012-12-26 | マクロジェニクス,インコーポレーテッド | 変異型Fc領域を有する抗体の同定および工学的改変およびその使用方法 |
| SG170110A1 (en) * | 2006-03-10 | 2011-04-29 | Macrogenics Inc | Identification and engineering of antibodies with variant heavy chains and methods of using same |
| US20070231805A1 (en) * | 2006-03-31 | 2007-10-04 | Baynes Brian M | Nucleic acid assembly optimization using clamped mismatch binding proteins |
| EP1918302A3 (en) * | 2006-05-18 | 2009-11-18 | AvantGen, Inc. | Methods for the identification and the isolation of epitope specific antibodies |
| WO2008105886A2 (en) | 2006-05-26 | 2008-09-04 | Macrogenics, Inc. | HUMANIZED FCγRIIB-SPECIFIC ANTIBODIES AND METHODS OF USE THEREOF |
| EP2032159B1 (en) | 2006-06-26 | 2015-01-07 | MacroGenics, Inc. | Combination of fcgammariib antibodies and cd20-specific antibodies and methods of use thereof |
| PT2029173T (pt) | 2006-06-26 | 2016-11-02 | Macrogenics Inc | Anticorpos específicos fc rib e seus métodos de uso |
| US20080112961A1 (en) * | 2006-10-09 | 2008-05-15 | Macrogenics, Inc. | Identification and Engineering of Antibodies with Variant Fc Regions and Methods of Using Same |
| WO2008140603A2 (en) | 2006-12-08 | 2008-11-20 | Macrogenics, Inc. | METHODS FOR THE TREATMENT OF DISEASE USING IMMUNOGLOBULINS HAVING FC REGIONS WITH ALTERED AFFINITIES FOR FCγR ACTIVATING AND FCγR INHIBITING |
| US8822639B2 (en) | 2006-12-12 | 2014-09-02 | Biorexis Pharmaceutical Corporation | Transferrin fusion protein libraries |
| AU2008216418A1 (en) * | 2007-02-09 | 2008-08-21 | Medimmune, Llc | Antibody libray display by yeast cell plasma membrane |
| EP2617827A1 (en) | 2007-03-26 | 2013-07-24 | Celexion, LLC | Method for displaying engineered proteins on a cell surface |
| US8877688B2 (en) | 2007-09-14 | 2014-11-04 | Adimab, Llc | Rationally designed, synthetic antibody libraries and uses therefor |
| MX2010002661A (es) | 2007-09-14 | 2010-05-20 | Adimab Inc | Bancos de anticuerpos sinteticos, designados racionalmente y usos para los mismos. |
| US8637435B2 (en) * | 2007-11-16 | 2014-01-28 | Merck Sharp & Dohme Corp. | Eukaryotic cell display systems |
| CN104645329A (zh) | 2007-11-30 | 2015-05-27 | Abbvie公司 | 蛋白制剂及其制备方法 |
| US8883146B2 (en) | 2007-11-30 | 2014-11-11 | Abbvie Inc. | Protein formulations and methods of making same |
| US8795667B2 (en) * | 2007-12-19 | 2014-08-05 | Macrogenics, Inc. | Compositions for the prevention and treatment of smallpox |
| WO2009111183A1 (en) * | 2008-03-03 | 2009-09-11 | Glycofi, Inc. | Surface display of recombinant proteins in lower eukaryotes |
| ITUD20080055A1 (it) * | 2008-03-13 | 2009-09-14 | Transactiva S R L | Procedimento per la produzione di una proteina umana in pianta, in particolare un enzima lisosomiale umano ricombinante in endosperma di cereali |
| AU2009231991B2 (en) | 2008-04-02 | 2014-09-25 | Macrogenics, Inc. | HER2/neu-specific antibodies and methods of using same |
| EP3045475B1 (en) | 2008-04-02 | 2017-10-04 | MacroGenics, Inc. | Bcr-complex-specific antibodies and methods of using same |
| CA2726845C (en) * | 2008-06-04 | 2017-09-26 | Macrogenics, Inc. | Antibodies with altered binding to fcrn and methods of using same |
| US8067339B2 (en) | 2008-07-09 | 2011-11-29 | Merck Sharp & Dohme Corp. | Surface display of whole antibodies in eukaryotes |
| MX2011006483A (es) | 2008-12-16 | 2011-07-13 | Novartis Ag | Sistemas de despliegue en levadura. |
| PT2486141T (pt) | 2009-10-07 | 2018-05-09 | Macrogenics Inc | Polipéptidos contendo uma região fc que apresenta uma função efectora melhorada, devido a modificações do grau de fucosilação, e métodos para a sua utilização |
| US8802091B2 (en) | 2010-03-04 | 2014-08-12 | Macrogenics, Inc. | Antibodies reactive with B7-H3 and uses thereof |
| ME03447B (me) | 2010-03-04 | 2020-01-20 | Macrogenics Inc | Anтitela reakтivna sa b7-нз, njihovi imunološki akтivni fragmenтi i upotreba |
| KR102024922B1 (ko) | 2010-07-16 | 2019-09-25 | 아디맵 엘엘씨 | 항체 라이브러리 |
| EP2601216B1 (en) | 2010-08-02 | 2018-01-03 | MacroGenics, Inc. | Covalent diabodies and uses thereof |
| EP2670779B1 (en) | 2011-02-01 | 2017-08-02 | Bac Ip B.V. | Antigen-binding protein directed against epitope in the ch1 domain of human igg antibodies |
| EP2710382B1 (en) | 2011-05-17 | 2017-10-18 | Bristol-Myers Squibb Company | Improved methods for the selection of binding proteins |
| EP2714079B2 (en) | 2011-05-21 | 2019-08-28 | MacroGenics, Inc. | Deimmunized serum-binding domains and their use for extending serum half-life |
| KR20140084253A (ko) | 2011-10-24 | 2014-07-04 | 애브비 인코포레이티드 | Tnf에 대한 면역결합제 |
| US9487587B2 (en) | 2013-03-05 | 2016-11-08 | Macrogenics, Inc. | Bispecific molecules that are immunoreactive with immune effector cells of a companion animal that express an activating receptor and cells that express B7-H3 and uses thereof |
| US9908938B2 (en) | 2013-03-14 | 2018-03-06 | Macrogenics, Inc. | Bispecific molecules that are immunoreactive with immune effector cells that express an activating receptor and an antigen expressed by a cell infected by a virus and uses thereof |
| UA116479C2 (uk) | 2013-08-09 | 2018-03-26 | Макродженікс, Інк. | БІСПЕЦИФІЧНЕ МОНОВАЛЕНТНЕ Fc-ДІАТІЛО, ЯКЕ ОДНОЧАСНО ЗВ'ЯЗУЄ CD32B I CD79b, ТА ЙОГО ЗАСТОСУВАННЯ |
| US11384149B2 (en) | 2013-08-09 | 2022-07-12 | Macrogenics, Inc. | Bi-specific monovalent Fc diabodies that are capable of binding CD32B and CD79b and uses thereof |
| EP2840091A1 (en) | 2013-08-23 | 2015-02-25 | MacroGenics, Inc. | Bi-specific diabodies that are capable of binding gpA33 and CD3 and uses thereof |
| EP2839842A1 (en) | 2013-08-23 | 2015-02-25 | MacroGenics, Inc. | Bi-specific monovalent diabodies that are capable of binding CD123 and CD3 and uses thereof |
| CA2963991A1 (en) | 2014-09-29 | 2016-04-07 | Duke University | Bispecific molecules comprising an hiv-1 envelope targeting arm |
| WO2016176573A1 (en) | 2015-04-30 | 2016-11-03 | Board Of Regents, The University Of Texas System | Antibacterial polypeptide libraries and methods for screening the same |
| US10961311B2 (en) | 2016-04-15 | 2021-03-30 | Macrogenics, Inc. | B7-H3 binding molecules, antibody drug conjugates thereof and methods of use thereof |
| WO2018104893A1 (en) | 2016-12-06 | 2018-06-14 | Glaxosmithkline Intellectual Property Development Limited | Alpha4-beta7 antibodies with incrased fcrn binding and/or half-life |
Family Cites Families (9)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4859609A (en) * | 1986-04-30 | 1989-08-22 | Genentech, Inc. | Novel receptors for efficient determination of ligands and their antagonists or agonists |
| CA1340772C (en) * | 1987-12-30 | 1999-09-28 | Patricia Tekamp-Olson | Expression and secretion of heterologous protiens in yeast employing truncated alpha-factor leader sequences |
| US5223409A (en) * | 1988-09-02 | 1993-06-29 | Protein Engineering Corp. | Directed evolution of novel binding proteins |
| CA2090969C (en) * | 1990-09-04 | 2001-08-21 | Russell Arthur Brierley | Production of insulin-like growth factor-1 in methylotrophic yeast cells |
| EP0474891A1 (en) * | 1990-09-08 | 1992-03-18 | BEHRINGWERKE Aktiengesellschaft | Vectors for expression of malarial antigens on the surface of Salmonella vaccine strains |
| SE9101433D0 (sv) * | 1991-05-13 | 1991-05-13 | Marianne Hansson | Recombinant dna sequence and its use |
| US5223408A (en) * | 1991-07-11 | 1993-06-29 | Genentech, Inc. | Method for making variant secreted proteins with altered properties |
| US6027910A (en) * | 1992-07-08 | 2000-02-22 | Unilever Patent Holdings B.V. | Process for immobilizing enzymes to the cell wall of a microbial cell by producing a fusion protein |
| US5618290A (en) * | 1993-10-19 | 1997-04-08 | W.L. Gore & Associates, Inc. | Endoscopic suture passer and method |
-
1994
- 1994-02-10 DE DE69433279T patent/DE69433279T2/de not_active Expired - Lifetime
- 1994-02-10 ZA ZA94916A patent/ZA94916B/xx unknown
- 1994-02-10 WO PCT/EP1994/000427 patent/WO1994018330A1/en active IP Right Grant
- 1994-02-10 EP EP94908307A patent/EP0682710B1/en not_active Expired - Lifetime
- 1994-02-10 ES ES94908307T patent/ES2210248T3/es not_active Expired - Lifetime
- 1994-02-10 AU AU61397/94A patent/AU6139794A/en not_active Abandoned
- 1994-02-10 AT AT94908307T patent/ATE253118T1/de active
- 1994-02-10 JP JP51767994A patent/JP3691055B2/ja not_active Expired - Lifetime
- 1994-02-10 PT PT94908307T patent/PT682710E/pt unknown
- 1994-02-10 DK DK94908307T patent/DK0682710T3/da active
- 1994-02-10 US US08/971,692 patent/US6114147A/en not_active Expired - Lifetime
-
1995
- 1995-08-10 FI FI953792A patent/FI953792A0/fi unknown
Cited By (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US9873746B2 (en) | 2003-10-22 | 2018-01-23 | Keck Graduate Institute | Methods of synthesizing heteromultimeric polypeptides in yeast using a haploid mating strategy |
| US10155819B2 (en) | 2003-10-22 | 2018-12-18 | Alderbio Holdings Llc | Methods of synthesizing heteromultimeric polypeptides in yeast using a haploid mating strategy |
| US10259883B2 (en) | 2003-10-22 | 2019-04-16 | Keck Graduate Institute | Methods of synthesizing heteromultimeric polypeptides in yeast using a haploid mating strategy |
| US11447560B2 (en) | 2003-10-22 | 2022-09-20 | Keck Graduate Institute | Methods of synthesizing heteromultimeric polypeptides in yeast using a haploid mating strategy |
Also Published As
| Publication number | Publication date |
|---|---|
| ES2210248T3 (es) | 2004-07-01 |
| EP0682710A1 (en) | 1995-11-22 |
| DE69433279T2 (de) | 2004-07-15 |
| PT682710E (pt) | 2004-03-31 |
| AU6139794A (en) | 1994-08-29 |
| FI953792A (fi) | 1995-08-10 |
| JPH08506484A (ja) | 1996-07-16 |
| WO1994018330A1 (en) | 1994-08-18 |
| ZA94916B (en) | 1995-08-10 |
| DE69433279D1 (de) | 2003-12-04 |
| FI953792A0 (fi) | 1995-08-10 |
| ATE253118T1 (de) | 2003-11-15 |
| EP0682710B1 (en) | 2003-10-29 |
| DK0682710T3 (da) | 2004-03-01 |
| US6114147A (en) | 2000-09-05 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP3691055B2 (ja) | 特異的結合力をもつ固定化タンパク質並びに各種プロセス・物品におけるそれらの使用 | |
| CA2139670C (en) | Process for immobilizing enzymes to the cell wall of a microbial cell by producing a fusion protein | |
| EP0702721B1 (en) | Process for producing fusion proteins comprising scfv fragments by a transformed mould | |
| US7794981B2 (en) | Prosuction of antibodies or (functionalized) fragments thereof derived from heavy chain immunoglobulins of camelidae | |
| US5024941A (en) | Expression and secretion vector for yeast containing a glucoamylase signal sequence | |
| JPH06500006A (ja) | ユビキチン特異的プロテアーゼ | |
| Fellinger et al. | Expression of the α‐galactosidase from Cyamopsis tetragonoloba (guar) by Hansenula polymorpha | |
| Günther et al. | The Saccharomyces cerevisiae TRG1 gene is essential for growth and encodes a lumenal endoplasmic reticulum glycoprotein involved in the maturation of vacuolar carboxypeptidase. | |
| US5674712A (en) | Recombinant vector and use thereof for exocellular preparation of antibodies in single molecule form from Bacillus subtilis | |
| TW562858B (en) | Actinomycete promoter | |
| EP0698097B1 (en) | Production of antibodies or (functionalized) fragments thereof derived from heavy chain immunoglobulins of camelidae | |
| CA2146240C (en) | Increased production of secreted proteins by recombinant eukaryotic cells | |
| JP2000501617A (ja) | 酵母細胞におけるn末端を伸長されたタンパクの発現のためのベクター | |
| Piesecki et al. | Immobilization of β‐galactosidase for application in organic chemistry using a chelating peptide | |
| JP4357116B2 (ja) | 酵素活性組換えヒトβ−トリプターゼ及びその生産方法 | |
| JPH0638771A (ja) | ヒトプロテインジスルフィドイソメラーゼ遺伝子の発現方法および該遺伝子との共発現によるポリペプチドの製造方法 | |
| Yong et al. | Secretion of heterologous cyclodextrin glycosyltransferase of Bacillus sp. E1 from Escherichia coli | |
| JPH04229188A (ja) | 融合タンパク質の試験管内プロセシング | |
| JP2001509392A (ja) | 組換え酵母細胞による分泌タンパク質の産生増加 | |
| Kriel | Development of synthetic signal sequences for heterologous protein secretion from Saccharomyces cerevisiae | |
| JPH10506787A (ja) | アスペルギルス・ニゲルのシグナル認識粒子をコードする遺伝子 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20041012 |
|
| RD02 | Notification of acceptance of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7422 Effective date: 20041025 |
|
| RD04 | Notification of resignation of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: A7424 Effective date: 20041101 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20050111 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20050517 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20050615 |
|
| R150 | Certificate of patent or registration of utility model |
Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20090624 Year of fee payment: 4 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20090624 Year of fee payment: 4 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313113 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20090624 Year of fee payment: 4 |
|
| R350 | Written notification of registration of transfer |
Free format text: JAPANESE INTERMEDIATE CODE: R350 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20090624 Year of fee payment: 4 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20100624 Year of fee payment: 5 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20110624 Year of fee payment: 6 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20110624 Year of fee payment: 6 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20120624 Year of fee payment: 7 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20130624 Year of fee payment: 8 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| EXPY | Cancellation because of completion of term |