DE69736247T2 - Verfahren zur herstellung von proteinhydrolysate - Google Patents
Verfahren zur herstellung von proteinhydrolysate Download PDFInfo
- Publication number
- DE69736247T2 DE69736247T2 DE69736247T DE69736247T DE69736247T2 DE 69736247 T2 DE69736247 T2 DE 69736247T2 DE 69736247 T DE69736247 T DE 69736247T DE 69736247 T DE69736247 T DE 69736247T DE 69736247 T2 DE69736247 T2 DE 69736247T2
- Authority
- DE
- Germany
- Prior art keywords
- strain
- substrate
- protein
- carboxypeptidase
- bacillus
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
- 238000000034 method Methods 0.000 title claims description 66
- 239000003531 protein hydrolysate Substances 0.000 title description 8
- 108010009736 Protein Hydrolysates Proteins 0.000 title description 7
- 239000000758 substrate Substances 0.000 claims description 62
- 102000035195 Peptidases Human genes 0.000 claims description 36
- 108091005804 Peptidases Proteins 0.000 claims description 36
- 102000004169 proteins and genes Human genes 0.000 claims description 34
- 108090000623 proteins and genes Proteins 0.000 claims description 34
- 235000018102 proteins Nutrition 0.000 claims description 33
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 claims description 31
- 102000005593 Endopeptidases Human genes 0.000 claims description 21
- 108010059378 Endopeptidases Proteins 0.000 claims description 21
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 claims description 20
- 102000018389 Exopeptidases Human genes 0.000 claims description 19
- 108010091443 Exopeptidases Proteins 0.000 claims description 19
- 235000013922 glutamic acid Nutrition 0.000 claims description 15
- 239000004220 glutamic acid Substances 0.000 claims description 15
- 108010068370 Glutens Proteins 0.000 claims description 13
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 13
- 108060008539 Transglutaminase Proteins 0.000 claims description 12
- 239000000203 mixture Substances 0.000 claims description 12
- 102000003601 transglutaminase Human genes 0.000 claims description 12
- 108010051815 Glutamyl endopeptidase Proteins 0.000 claims description 11
- 230000000694 effects Effects 0.000 claims description 11
- 235000021312 gluten Nutrition 0.000 claims description 11
- 108010053229 Lysyl endopeptidase Proteins 0.000 claims description 9
- 102000056251 Prolyl Oligopeptidases Human genes 0.000 claims description 9
- 241000193830 Bacillus <bacterium> Species 0.000 claims description 8
- 241000193752 Bacillus circulans Species 0.000 claims description 8
- 102000005367 Carboxypeptidases Human genes 0.000 claims description 8
- 108010006303 Carboxypeptidases Proteins 0.000 claims description 8
- 108010004098 Leucyl aminopeptidase Proteins 0.000 claims description 8
- 102000002704 Leucyl aminopeptidase Human genes 0.000 claims description 8
- 241000187747 Streptomyces Species 0.000 claims description 8
- 108010008256 glutaminyl-peptide glutaminase Proteins 0.000 claims description 8
- 108010086105 peptidyl-glutaminase Proteins 0.000 claims description 8
- 241000228212 Aspergillus Species 0.000 claims description 7
- 241000194108 Bacillus licheniformis Species 0.000 claims description 7
- 244000063299 Bacillus subtilis Species 0.000 claims description 7
- 235000014469 Bacillus subtilis Nutrition 0.000 claims description 7
- 108030007165 Leucyl endopeptidases Proteins 0.000 claims description 7
- 108010092515 glycyl endopeptidase Proteins 0.000 claims description 7
- 102100030988 Angiotensin-converting enzyme Human genes 0.000 claims description 6
- 108090000087 Carboxypeptidase B Proteins 0.000 claims description 6
- 102000003670 Carboxypeptidase B Human genes 0.000 claims description 6
- 108090000018 Carboxypeptidase D Proteins 0.000 claims description 6
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 claims description 6
- 108090000882 Peptidyl-Dipeptidase A Proteins 0.000 claims description 6
- 108030006244 Protein-glutamine glutaminases Proteins 0.000 claims description 6
- 108090000631 Trypsin Proteins 0.000 claims description 6
- 102000004142 Trypsin Human genes 0.000 claims description 6
- 125000000291 glutamic acid group Chemical group N[C@@H](CCC(O)=O)C(=O)* 0.000 claims description 6
- 108010075409 Alanine carboxypeptidase Proteins 0.000 claims description 5
- 241000235035 Debaryomyces Species 0.000 claims description 5
- 108010048963 Glutamate carboxypeptidase Proteins 0.000 claims description 5
- 102100033320 Lysosomal Pro-X carboxypeptidase Human genes 0.000 claims description 5
- 108030001566 Peptidyl-Asp metalloendopeptidases Proteins 0.000 claims description 5
- 101710178372 Prolyl endopeptidase Proteins 0.000 claims description 5
- 108010073771 Soybean Proteins Proteins 0.000 claims description 5
- 108010070926 Tripeptide aminopeptidase Proteins 0.000 claims description 5
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 claims description 5
- 108010003914 endoproteinase Asp-N Proteins 0.000 claims description 5
- 108010057284 lysosomal Pro-X carboxypeptidase Proteins 0.000 claims description 5
- 239000004475 Arginine Substances 0.000 claims description 4
- 240000006439 Aspergillus oryzae Species 0.000 claims description 4
- 235000002247 Aspergillus oryzae Nutrition 0.000 claims description 4
- 240000006432 Carica papaya Species 0.000 claims description 4
- 108010073324 Glutaminase Proteins 0.000 claims description 4
- 102000009127 Glutaminase Human genes 0.000 claims description 4
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 claims description 4
- 239000004472 Lysine Substances 0.000 claims description 4
- 108700015930 Prolyl Oligopeptidases Proteins 0.000 claims description 4
- 241000589516 Pseudomonas Species 0.000 claims description 4
- 235000013311 vegetables Nutrition 0.000 claims description 4
- 241001147825 Actinomyces sp. Species 0.000 claims description 3
- 108090000915 Aminopeptidases Proteins 0.000 claims description 3
- 102000004400 Aminopeptidases Human genes 0.000 claims description 3
- 102100032407 Carboxypeptidase D Human genes 0.000 claims description 3
- 102100026678 Carboxypeptidase N catalytic chain Human genes 0.000 claims description 3
- 108010080937 Carboxypeptidases A Proteins 0.000 claims description 3
- 102000000496 Carboxypeptidases A Human genes 0.000 claims description 3
- 108010059081 Cathepsin A Proteins 0.000 claims description 3
- 102000005572 Cathepsin A Human genes 0.000 claims description 3
- 108700033740 EC 3.4.17.1 Proteins 0.000 claims description 3
- 108030000059 Gly-Xaa carboxypeptidases Proteins 0.000 claims description 3
- 108010085169 Lysine carboxypeptidase Proteins 0.000 claims description 3
- 102100028524 Lysosomal protective protein Human genes 0.000 claims description 3
- 102100030612 Mast cell carboxypeptidase A Human genes 0.000 claims description 3
- 241001443590 Naganishia albida Species 0.000 claims description 3
- 235000013339 cereals Nutrition 0.000 claims description 3
- 108010083141 dipeptidyl carboxypeptidase Proteins 0.000 claims description 3
- 239000012588 trypsin Substances 0.000 claims description 3
- 241000590020 Achromobacter Species 0.000 claims description 2
- 241000590035 Achromobacter lyticus Species 0.000 claims description 2
- 235000017060 Arachis glabrata Nutrition 0.000 claims description 2
- 244000105624 Arachis hypogaea Species 0.000 claims description 2
- 235000010777 Arachis hypogaea Nutrition 0.000 claims description 2
- 235000018262 Arachis monticola Nutrition 0.000 claims description 2
- 241000228245 Aspergillus niger Species 0.000 claims description 2
- 235000007319 Avena orientalis Nutrition 0.000 claims description 2
- 241000209763 Avena sativa Species 0.000 claims description 2
- 235000007558 Avena sp Nutrition 0.000 claims description 2
- 241000193744 Bacillus amyloliquefaciens Species 0.000 claims description 2
- 241000193422 Bacillus lentus Species 0.000 claims description 2
- 240000002791 Brassica napus Species 0.000 claims description 2
- 235000004977 Brassica sinapistrum Nutrition 0.000 claims description 2
- 235000009467 Carica papaya Nutrition 0.000 claims description 2
- 241001337994 Cryptococcus <scale insect> Species 0.000 claims description 2
- 102000004860 Dipeptidases Human genes 0.000 claims description 2
- 108090001081 Dipeptidases Proteins 0.000 claims description 2
- 241000589565 Flavobacterium Species 0.000 claims description 2
- 239000004471 Glycine Substances 0.000 claims description 2
- 244000020551 Helianthus annuus Species 0.000 claims description 2
- 235000003222 Helianthus annuus Nutrition 0.000 claims description 2
- 240000005979 Hordeum vulgare Species 0.000 claims description 2
- 235000007340 Hordeum vulgare Nutrition 0.000 claims description 2
- 241000863031 Lysobacter Species 0.000 claims description 2
- 241000863030 Lysobacter enzymogenes Species 0.000 claims description 2
- 240000004658 Medicago sativa Species 0.000 claims description 2
- 235000017587 Medicago sativa ssp. sativa Nutrition 0.000 claims description 2
- 241000233654 Oomycetes Species 0.000 claims description 2
- 240000007594 Oryza sativa Species 0.000 claims description 2
- 235000007164 Oryza sativa Nutrition 0.000 claims description 2
- 108010084695 Pea Proteins Proteins 0.000 claims description 2
- 244000046052 Phaseolus vulgaris Species 0.000 claims description 2
- 241000233614 Phytophthora Species 0.000 claims description 2
- 241001149949 Phytophthora cactorum Species 0.000 claims description 2
- 241000589517 Pseudomonas aeruginosa Species 0.000 claims description 2
- 241000589538 Pseudomonas fragi Species 0.000 claims description 2
- 241000233639 Pythium Species 0.000 claims description 2
- 241001622909 Pythium intermedium Species 0.000 claims description 2
- 241001622892 Pythium periilum Species 0.000 claims description 2
- 241000918584 Pythium ultimum Species 0.000 claims description 2
- 235000007238 Secale cereale Nutrition 0.000 claims description 2
- 235000003434 Sesamum indicum Nutrition 0.000 claims description 2
- 244000000231 Sesamum indicum Species 0.000 claims description 2
- 241000191940 Staphylococcus Species 0.000 claims description 2
- 241000520730 Streptomyces cinnamoneus Species 0.000 claims description 2
- 241000218483 Streptomyces lydicus Species 0.000 claims description 2
- 240000000359 Triticum dicoccon Species 0.000 claims description 2
- 240000008042 Zea mays Species 0.000 claims description 2
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 claims description 2
- 235000002017 Zea mays subsp mays Nutrition 0.000 claims description 2
- 229920002494 Zein Polymers 0.000 claims description 2
- 230000001580 bacterial effect Effects 0.000 claims description 2
- 235000005822 corn Nutrition 0.000 claims description 2
- 235000012343 cottonseed oil Nutrition 0.000 claims description 2
- 125000003978 glutamoyl group Chemical group [H]N([H])[C@]([H])(C(=O)[*])C([H])([H])C([H])([H])C(N([H])[H])=O 0.000 claims description 2
- 235000019702 pea protein Nutrition 0.000 claims description 2
- 235000020232 peanut Nutrition 0.000 claims description 2
- 125000001151 peptidyl group Chemical group 0.000 claims description 2
- 235000009566 rice Nutrition 0.000 claims description 2
- 239000000126 substance Substances 0.000 claims description 2
- 239000005019 zein Substances 0.000 claims description 2
- 229940093612 zein Drugs 0.000 claims description 2
- 241001622893 Pythium periplocum Species 0.000 claims 1
- 241001385948 Pythium sp. Species 0.000 claims 1
- 241000209056 Secale Species 0.000 claims 1
- 230000009615 deamination Effects 0.000 claims 1
- 238000006481 deamination reaction Methods 0.000 claims 1
- 230000001788 irregular Effects 0.000 claims 1
- 235000019710 soybean protein Nutrition 0.000 claims 1
- 102000004190 Enzymes Human genes 0.000 description 29
- 108090000790 Enzymes Proteins 0.000 description 29
- 229940088598 enzyme Drugs 0.000 description 29
- 230000006240 deamidation Effects 0.000 description 23
- 238000006460 hydrolysis reaction Methods 0.000 description 22
- 230000007062 hydrolysis Effects 0.000 description 20
- 238000002360 preparation method Methods 0.000 description 17
- QGZKDVFQNNGYKY-UHFFFAOYSA-N Ammonia Chemical compound N QGZKDVFQNNGYKY-UHFFFAOYSA-N 0.000 description 12
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 12
- 230000002255 enzymatic effect Effects 0.000 description 12
- 238000011534 incubation Methods 0.000 description 12
- 239000004365 Protease Substances 0.000 description 11
- 229930195712 glutamate Natural products 0.000 description 11
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 8
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 8
- 235000019419 proteases Nutrition 0.000 description 8
- 241000209140 Triticum Species 0.000 description 7
- 235000021307 Triticum Nutrition 0.000 description 7
- 239000003153 chemical reaction reagent Substances 0.000 description 7
- ZWLUXSQADUDCSB-UHFFFAOYSA-N phthalaldehyde Chemical compound O=CC1=CC=CC=C1C=O ZWLUXSQADUDCSB-UHFFFAOYSA-N 0.000 description 7
- 230000002797 proteolythic effect Effects 0.000 description 7
- 229910021529 ammonia Inorganic materials 0.000 description 6
- 238000006243 chemical reaction Methods 0.000 description 6
- 229940024999 proteolytic enzymes for treatment of wounds and ulcers Drugs 0.000 description 6
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 5
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 5
- 239000000796 flavoring agent Substances 0.000 description 5
- 235000019634 flavors Nutrition 0.000 description 5
- 235000013305 food Nutrition 0.000 description 5
- 239000000243 solution Substances 0.000 description 5
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 4
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 4
- 238000002835 absorbance Methods 0.000 description 4
- 239000002253 acid Substances 0.000 description 4
- 239000005018 casein Substances 0.000 description 4
- BECPQYXYKAMYBN-UHFFFAOYSA-N casein, tech. Chemical compound NCCCCC(C(O)=O)N=C(O)C(CC(O)=O)N=C(O)C(CCC(O)=N)N=C(O)C(CC(C)C)N=C(O)C(CCC(O)=O)N=C(O)C(CC(O)=O)N=C(O)C(CCC(O)=O)N=C(O)C(C(C)O)N=C(O)C(CCC(O)=N)N=C(O)C(CCC(O)=N)N=C(O)C(CCC(O)=N)N=C(O)C(CCC(O)=O)N=C(O)C(CCC(O)=O)N=C(O)C(COP(O)(O)=O)N=C(O)C(CCC(O)=N)N=C(O)C(N)CC1=CC=CC=C1 BECPQYXYKAMYBN-UHFFFAOYSA-N 0.000 description 4
- 235000021240 caseins Nutrition 0.000 description 4
- 229940001941 soy protein Drugs 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- 239000000725 suspension Substances 0.000 description 4
- 238000012360 testing method Methods 0.000 description 4
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 4
- 241000196324 Embryophyta Species 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 108010054147 Hemoglobins Proteins 0.000 description 3
- 102000001554 Hemoglobins Human genes 0.000 description 3
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 3
- 241000191967 Staphylococcus aureus Species 0.000 description 3
- 238000004458 analytical method Methods 0.000 description 3
- 239000000872 buffer Substances 0.000 description 3
- 230000015556 catabolic process Effects 0.000 description 3
- 238000006731 degradation reaction Methods 0.000 description 3
- 235000021245 dietary protein Nutrition 0.000 description 3
- 230000007071 enzymatic hydrolysis Effects 0.000 description 3
- 238000006047 enzymatic hydrolysis reaction Methods 0.000 description 3
- 238000000855 fermentation Methods 0.000 description 3
- 230000004151 fermentation Effects 0.000 description 3
- 108010007119 flavourzyme Proteins 0.000 description 3
- 238000010438 heat treatment Methods 0.000 description 3
- 239000012139 lysis buffer Substances 0.000 description 3
- 230000007065 protein hydrolysis Effects 0.000 description 3
- 229960001153 serine Drugs 0.000 description 3
- AXZJHDNQDSVIDR-NSHDSACASA-N 4178-93-2 Chemical compound CC(C)C[C@H](N)C(=O)NC1=CC=C([N+]([O-])=O)C=C1 AXZJHDNQDSVIDR-NSHDSACASA-N 0.000 description 2
- 241001480052 Aspergillus japonicus Species 0.000 description 2
- 108030006828 D-glutaminases Proteins 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- 102000016943 Muramidase Human genes 0.000 description 2
- 108010014251 Muramidase Proteins 0.000 description 2
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 2
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 241000187177 Streptomyces thermovulgaris Species 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 239000007983 Tris buffer Substances 0.000 description 2
- 229940009098 aspartate Drugs 0.000 description 2
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 2
- 230000007073 chemical hydrolysis Effects 0.000 description 2
- 229940066758 endopeptidases Drugs 0.000 description 2
- 238000005187 foaming Methods 0.000 description 2
- 229960000274 lysozyme Drugs 0.000 description 2
- 235000010335 lysozyme Nutrition 0.000 description 2
- 239000004325 lysozyme Substances 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 238000010979 pH adjustment Methods 0.000 description 2
- 235000019629 palatability Nutrition 0.000 description 2
- 102000004196 processed proteins & peptides Human genes 0.000 description 2
- 239000000047 product Substances 0.000 description 2
- 239000011541 reaction mixture Substances 0.000 description 2
- 230000035484 reaction time Effects 0.000 description 2
- YNJBWRMUSHSURL-UHFFFAOYSA-N trichloroacetic acid Chemical compound OC(=O)C(Cl)(Cl)Cl YNJBWRMUSHSURL-UHFFFAOYSA-N 0.000 description 2
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 2
- 230000001810 trypsinlike Effects 0.000 description 2
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 2
- NPWMTBZSRRLQNJ-VKHMYHEASA-N (3s)-3-aminopiperidine-2,6-dione Chemical compound N[C@H]1CCC(=O)NC1=O NPWMTBZSRRLQNJ-VKHMYHEASA-N 0.000 description 1
- NHJVRSWLHSJWIN-UHFFFAOYSA-N 2,4,6-trinitrobenzenesulfonic acid Chemical compound OS(=O)(=O)C1=C([N+]([O-])=O)C=C([N+]([O-])=O)C=C1[N+]([O-])=O NHJVRSWLHSJWIN-UHFFFAOYSA-N 0.000 description 1
- 241000251468 Actinopterygii Species 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 101000950981 Bacillus subtilis (strain 168) Catabolic NAD-specific glutamate dehydrogenase RocG Proteins 0.000 description 1
- BTBUEUYNUDRHOZ-UHFFFAOYSA-N Borate Chemical compound [O-]B([O-])[O-] BTBUEUYNUDRHOZ-UHFFFAOYSA-N 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 239000004278 EU approved seasoning Substances 0.000 description 1
- 108010000196 Factor XIIIa Proteins 0.000 description 1
- 241000223221 Fusarium oxysporum Species 0.000 description 1
- 102000016901 Glutamate dehydrogenase Human genes 0.000 description 1
- 102100025961 Glutaminase liver isoform, mitochondrial Human genes 0.000 description 1
- 101710138819 Glutaminase liver isoform, mitochondrial Proteins 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 241001505297 Pythium irregulare Species 0.000 description 1
- 244000082988 Secale cereale Species 0.000 description 1
- 229920005654 Sephadex Polymers 0.000 description 1
- 239000012507 Sephadex™ Substances 0.000 description 1
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 1
- 241000499056 Streptomyces griseocarneus Species 0.000 description 1
- 241000187392 Streptomyces griseus Species 0.000 description 1
- 108010056079 Subtilisins Proteins 0.000 description 1
- 102000005158 Subtilisins Human genes 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 150000001408 amides Chemical class 0.000 description 1
- 235000001014 amino acid Nutrition 0.000 description 1
- 229940024606 amino acid Drugs 0.000 description 1
- 150000001413 amino acids Chemical class 0.000 description 1
- 235000019730 animal feed additive Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 235000013361 beverage Nutrition 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 238000009835 boiling Methods 0.000 description 1
- 239000007853 buffer solution Substances 0.000 description 1
- 239000006227 byproduct Substances 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 238000004140 cleaning Methods 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- YNPKJCSIKJCODK-UHFFFAOYSA-N disodium boric acid hydrogen borate decahydrate Chemical compound O.O.O.O.O.O.O.O.O.O.[Na+].[Na+].OB(O)O.OB(O)O.OB(O)O.OB([O-])[O-] YNPKJCSIKJCODK-UHFFFAOYSA-N 0.000 description 1
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 1
- 238000004945 emulsification Methods 0.000 description 1
- 230000001804 emulsifying effect Effects 0.000 description 1
- 239000000706 filtrate Substances 0.000 description 1
- 235000012041 food component Nutrition 0.000 description 1
- 239000005417 food ingredient Substances 0.000 description 1
- 235000011194 food seasoning agent Nutrition 0.000 description 1
- 125000000404 glutamine group Chemical group N[C@@H](CCC(N)=O)C(=O)* 0.000 description 1
- 108010036302 hemoglobin AS Proteins 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 230000004048 modification Effects 0.000 description 1
- 238000012986 modification Methods 0.000 description 1
- 238000006386 neutralization reaction Methods 0.000 description 1
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 1
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical compound C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 1
- 108010076761 pantothenase Proteins 0.000 description 1
- 229920000728 polyester Polymers 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 235000015067 sauces Nutrition 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 1
- 235000019333 sodium laurylsulphate Nutrition 0.000 description 1
- 235000014347 soups Nutrition 0.000 description 1
- 239000012086 standard solution Substances 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 230000009967 tasteless effect Effects 0.000 description 1
- 238000010998 test method Methods 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000002087 whitening effect Effects 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/11—Aminopeptidases (3.4.11)
- C12Y304/11001—Leucyl aminopeptidase (3.4.11.1)
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23J—PROTEIN COMPOSITIONS FOR FOODSTUFFS; WORKING-UP PROTEINS FOR FOODSTUFFS; PHOSPHATIDE COMPOSITIONS FOR FOODSTUFFS
- A23J3/00—Working-up of proteins for foodstuffs
- A23J3/30—Working-up of proteins for foodstuffs by hydrolysis
- A23J3/32—Working-up of proteins for foodstuffs by hydrolysis using chemical agents
- A23J3/34—Working-up of proteins for foodstuffs by hydrolysis using chemical agents using enzymes
- A23J3/341—Working-up of proteins for foodstuffs by hydrolysis using chemical agents using enzymes of animal proteins
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23J—PROTEIN COMPOSITIONS FOR FOODSTUFFS; WORKING-UP PROTEINS FOR FOODSTUFFS; PHOSPHATIDE COMPOSITIONS FOR FOODSTUFFS
- A23J3/00—Working-up of proteins for foodstuffs
- A23J3/30—Working-up of proteins for foodstuffs by hydrolysis
- A23J3/32—Working-up of proteins for foodstuffs by hydrolysis using chemical agents
- A23J3/34—Working-up of proteins for foodstuffs by hydrolysis using chemical agents using enzymes
- A23J3/346—Working-up of proteins for foodstuffs by hydrolysis using chemical agents using enzymes of vegetable proteins
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23K—FODDER
- A23K20/00—Accessory food factors for animal feeding-stuffs
- A23K20/10—Organic substances
- A23K20/142—Amino acids; Derivatives thereof
- A23K20/147—Polymeric derivatives, e.g. peptides or proteins
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23L—FOODS, FOODSTUFFS, OR NON-ALCOHOLIC BEVERAGES, NOT COVERED BY SUBCLASSES A21D OR A23B-A23J; THEIR PREPARATION OR TREATMENT, e.g. COOKING, MODIFICATION OF NUTRITIVE QUALITIES, PHYSICAL TREATMENT; PRESERVATION OF FOODS OR FOODSTUFFS, IN GENERAL
- A23L27/00—Spices; Flavouring agents or condiments; Artificial sweetening agents; Table salts; Dietetic salt substitutes; Preparation or treatment thereof
- A23L27/20—Synthetic spices, flavouring agents or condiments
- A23L27/21—Synthetic spices, flavouring agents or condiments containing amino acids
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23L—FOODS, FOODSTUFFS, OR NON-ALCOHOLIC BEVERAGES, NOT COVERED BY SUBCLASSES A21D OR A23B-A23J; THEIR PREPARATION OR TREATMENT, e.g. COOKING, MODIFICATION OF NUTRITIVE QUALITIES, PHYSICAL TREATMENT; PRESERVATION OF FOODS OR FOODSTUFFS, IN GENERAL
- A23L27/00—Spices; Flavouring agents or condiments; Artificial sweetening agents; Table salts; Dietetic salt substitutes; Preparation or treatment thereof
- A23L27/20—Synthetic spices, flavouring agents or condiments
- A23L27/21—Synthetic spices, flavouring agents or condiments containing amino acids
- A23L27/22—Synthetic spices, flavouring agents or condiments containing amino acids containing glutamic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/11—Aminopeptidases (3.4.11)
- C12Y304/11004—Tripeptide aminopeptidase (3.4.11.4)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/15—Peptidyl-dipeptidases (3.4.15)
- C12Y304/15001—Peptidyl-dipeptidase A (3.4.15.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/15—Peptidyl-dipeptidases (3.4.15)
- C12Y304/15005—Peptidyl-dipeptidase Dcp (3.4.15.5)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/16—Serine-type carboxypeptidases (3.4.16)
- C12Y304/16002—Lysosomal Pro-Xaa carboxypeptidase (3.4.16.2)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/16—Serine-type carboxypeptidases (3.4.16)
- C12Y304/16005—Carboxypeptidase C (3.4.16.5), i.e. carboxypeptidase Y
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/16—Serine-type carboxypeptidases (3.4.16)
- C12Y304/16006—Carboxypeptidase D (3.4.16.6)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17001—Carboxypeptidase A (3.4.17.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17002—Carboxypeptidase B (3.4.17.2)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17003—Lysine carboxypeptidase (3.4.17.3)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17004—Gly-Xaa carboxypeptidase (3.4.17.4)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17006—Alanine carboxypeptidase (3.4.17.6)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/17—Metallocarboxypeptidases (3.4.17)
- C12Y304/17011—Glutamate carboxypeptidase (3.4.17.11)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/21—Serine endopeptidases (3.4.21)
- C12Y304/21004—Trypsin (3.4.21.4)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/21—Serine endopeptidases (3.4.21)
- C12Y304/21019—Glutamyl endopeptidase (3.4.21.19)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/21—Serine endopeptidases (3.4.21)
- C12Y304/21026—Prolyl oligopeptidase (3.4.21.26), i.e. proline-specific endopeptidase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/21—Serine endopeptidases (3.4.21)
- C12Y304/2105—Lysyl endopeptidase (3.4.21.50)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/21—Serine endopeptidases (3.4.21)
- C12Y304/21057—Leucyl endopeptidase (3.4.21.57)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/22—Cysteine endopeptidases (3.4.22)
- C12Y304/22025—Glycyl endopeptidase (3.4.22.25)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y304/00—Hydrolases acting on peptide bonds, i.e. peptidases (3.4)
- C12Y304/24—Metalloendopeptidases (3.4.24)
- C12Y304/24033—Peptidyl-Asp metalloendopeptidase (3.4.24.33)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y305/00—Hydrolases acting on carbon-nitrogen bonds, other than peptide bonds (3.5)
- C12Y305/01—Hydrolases acting on carbon-nitrogen bonds, other than peptide bonds (3.5) in linear amides (3.5.1)
- C12Y305/01043—Peptidyl-glutaminase (3.5.1.43)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y305/00—Hydrolases acting on carbon-nitrogen bonds, other than peptide bonds (3.5)
- C12Y305/01—Hydrolases acting on carbon-nitrogen bonds, other than peptide bonds (3.5) in linear amides (3.5.1)
- C12Y305/01044—Protein-glutamine glutaminase (3.5.1.44)
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23V—INDEXING SCHEME RELATING TO FOODS, FOODSTUFFS OR NON-ALCOHOLIC BEVERAGES AND LACTIC OR PROPIONIC ACID BACTERIA USED IN FOODSTUFFS OR FOOD PREPARATION
- A23V2002/00—Food compositions, function of food ingredients or processes for food or foodstuffs
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Genetics & Genomics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Polymers & Plastics (AREA)
- Food Science & Technology (AREA)
- Nutrition Science (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Animal Husbandry (AREA)
- Enzymes And Modification Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Peptides Or Proteins (AREA)
- Fodder In General (AREA)
- Seasonings (AREA)
Description
- Fachgebiet
- Die vorliegende Erfindung betrifft ein Verfahren zum Erhalt von Protein-Hydrolysaten, die als Aromastoffe geeignet sind. Insbesondere stellt die Erfindung ein Verfahren zum Erhalt eines Hydrolysats, das mit glutaminfreier Säure und/oder peptidgebundenen Glutaminsäure-Resten aus proteinartiger Substanz angereichert ist, bereit, wobei das Verfahren die Schritte umfasst, wobei das Substrat einem Deamidierungsverfahren unterzogen und das Substrat der Wirkung eines spezifisch wirkenden proteolytischen Enzyms unterworfen wird.
- Technischer Hintergrund
- Verschiedene Lebensmittelprodukte, z.B. Suppen, Saucen und Würzmittel, umfassen Aromastoffe, die durch Hydrolyse von proteinartigen Materialien erhalten werden. Herkömmlichweise wird diese Hydrolyse durch die Verwendung starker Chlorwasserstoffsäure und anschließende Neutralisation mit Natriumhydroxid bewirkt. Jedoch führt eine derartige chemische Hydrolyse zu einem starken Abbau der Aminosäuren, die während der Hydrolyse erhalten werden und auch zu gesundheitsschädlichen Nebenprodukten, die sich im Verlauf dieser chemischen Reaktion bilden. Daher hat die wachsende Besorgnis bezüglich der Verwendung von Aromastoffen, die durch chemische Hydrolyse erhalten werden, zur Entwicklung enzymatischer Hydrolyseverfahren geführt.
- Der Zweck enzymatischer Hydrolyseverfahren ist der Erhalt eines hohen Hydrolysegrades (DH), und dieser wird im allgemeinen durch die Verwendung eines Komplexes aus unspezifisch wirkenden proteolytischen Enzymen (d.h. unspezifisch wirkende Endo- und Exopeptidasen) erreicht. So beschreibt z.B. WO 94/25580 ein Verfahren zur Hydrolyse von Proteinen durch die Verwendung einer aus Aspergillus oryzae erhaltenen unspezifisch wirkenden Enzymzubereitung. Spezifisch wirkende proteolytische Enzyme wurden für diesen Zweck bisher noch nicht verwendet, da solche Enzyme nur zu einem unzureichenden Hydrolysegrad führen.
- Bisher wurden nur relativ wenige spezifisch wirkende proteolytische Enzyme beschrieben. Somit wurde von proteolytischen Enzymen berichtet, die vorzugsweise Peptide an Glutamoyl-Peptid-Bindungen (Glu-spezifische Proteasen) spalten, erhalten aus Staphylococcus aureus [Drapeau G. R., Boily Y. and Houmard J., J. Biol. Chem. 1972, 247 (20)6720–6726], aus Actinomyces sp. [Mosolova O. V., Rudenskaya G. N.,Stepanav V. M., Khodava O. M, Tsaplina I. A.; Biokhimiya 1987, 52(3)414–422)], aus Streptomyces griseus [Yoshida N., Tsuruyama S., Nagata K., Hirayma K., Noda K. and Makisumi S., J. Biochem. 1988, 104,451–456], aus Streptomyces thermovulgaris [Khaldarova N. Y, Rudenskaya G. N., Revina L. P., Stephanov V. M. and Ergorov N. S. Biochimiia Moscow Eng. 1989, 54 (1),32–38], aus Bacillus subtilis [Niidome T., Yoshida N. Ogata F. Ito A. and Noda K., J. Biochem. 1990, 108,965–970] und aus Bacillus licheniformis [WO 91/13554A1].
- Glu-spezifische Proteasen finden hauptsächlich bei Studien der Proteinstrukturen Anwendungen. Jedoch wurden die Verwendung einer Glu-spezifischen Staphylococcus aureus-Protease zur Modifizierung der Löslichkeit und die strukturellen Eigenschaften von Casein beschrieben [Chobert J.-M., Sitohy M.Z. and Whitaker J. R., J.Argric. Food Chem. 1988, 36,220–224], und WO 91/13554 A1 beschreibt die Verwendung des Bacillus licheniformis, um eine begrenzte spezifische Hydrolyse von Proteinen zu erhalten.
- Während Glutamin (Gln) nahezu geschmacklos ist, spielt Glutaminsäure (Glu), gleich ob frei oder peptidgebunden, für das Aroma und die Schmackhaftigkeit der Proteinhydrolysate eine bedeutende Rolle. Glutaminsäure kann durch die Umwandlung von freiem Glutamin zu Glutaminsäure gebildet werden. Diese Umwandlung (d.h. Deamidierung) erfolgt schon von Natur aus während der herkömmlichen Hydrolyse unter Verwendung starker Chlorwasserstoffsäure.
- Alternativ kann Glutaminsäure aus freiem Glutamin durch die Einwirkung einer Glutaminase (L-Glutaminase,
EC 3.5.1.22 EC 3.5.1.35 - Im Gegensatz zu Glutaminase wirkt Peptidoglutaminase (PGasen) auf peptidgebundenes Glutamin. Es sind zwei Typen von Peptidoglutaminasen bekannt. Peptidylgutaminase (
EC 3.5.1.43 EC 3.5.1.44 - Peptidoglutaminasen sind zur Modifizierung von Nahrungsmittel-Proteinen bekannt und geeignet. Strukturelle Modifikationen von Proteinen verbessern ihre funktionellen Eigenschaften und weiten ihre Verwendung als Lebensmittelbestandteile aus. Daher beschreibt
US 5,082,672 ein Verfahren zur Behandlung von Nahrungsmittel-Proteinen durch Deamidierung mit einer Peptidoglutaminase, wobei bei dem Verfahren die Löslichkeit, das Emulgieren und Aufschäumen und weitere funktionelle Eigenschaften von Sojaprotein verbessert werden.US 5,082,672 berichtet jedoch auch, dass die Fähigkeit der Peptidoglutaminase, intaktes Soja-Protein zu deamidieren, aufgrund seiner großen Molekülgröße und/oder einzigartigen Konformation beschränkt ist. Daher lehrtUS 5,082,672 den anfänglichen Abbau des Substrats durch Hydrolyse mit AlcalaseTM, ein aus Bacillus licheniformis erhaltener unspezifisch wirkender Endo/Exopeptidase-Komplex und/oder durch Wärmebehandlung und stellt fest, dass die Wärmevor- und Wärmenachbehandlung des hydrolysierten Sojaproteins den größten Deamidierungsgrad bewirkte. -
US 3,857,967 beschreibt ein Verfahren zur Herstellung von Nahrungsmitteln und Getränken mit einer Peptidoglutaminase, erhalten aus Bacillus circulans. Um den größten Deamidierungsgrad zu erhalten, lehrtUS 3,857,976 auch den anfänglichen Abbau des proteinartigen Substrats durch die Verwendung unspezifisch wirkender Endo/Exopeptidasen. - Mimouni et al. [Mimouni B., Raymond J., Marie-Desnoyers AM., Azanza JL. & Ducastaing A.; Journal of Cereal Science, 1994, 21, 153–165] beschreiben eine Kombination aus Säure-Deamidierung und enzymatischer Hydrolyse zur Verbesserung der funktionellen Eigenschaften des Wei zenglutens. Insbesondere beschreiben Mimouni et al. die Säure-Deamidierung in Kombination mit der Verwendung von unspezifisch wirkenden Endopeptidasen.
- Zusammenfassung der Erfindung
- Es wurde nun gefunden, dass Proteinhydrolysate mit hervorragendem Aroma und Funktionalität erhalten werden können, indem das proteinartige Material der kombinierten Wirkung eines Deamidierungsverfahrens und der Wirkung eines spezifisch wirkenden proteolytischen Enzyms unterzogen wird.
- Demnach stellt die vorliegende Erfindung ein Verfahren zum Erhalt eines Hydrolysats, das mit freier Glutaminsäure und/oder peptidgebundenen Glutaminsäureresten angereichert ist, aus vegetabilem proteinartigem Substrat bereit, wobei das Verfahren die Schritte umfasst, wobei das Substrat mit einer Peptidoglutaminase oder einer Transglutaminase einer Deamidierung unterzogen und das Substrat der Aktivität eines spezifisch wirkenden proteolytischen Enzyms unterworfen wird.
- Bei einer bevorzugten Ausführungsform stellt die Erfindung ein Verfahren zum Erhalt eines Hydrolysats, das mit Glutaminsäure angereichert ist, aus einem proteinartigen Substrat bereit, wobei das Verfahren die Schritte umfasst, wobei das Substrat der Einwirkung einer Peptidoglutaminase unterzogen und das Substrat der Einwirkung eines proteolytischen Enzyms, das bevorzugt Glutamyol-Peptid-Bindungen spaltet, unterworfen wird, gefolgt von der Einwirkung von einer oder mehreren unspezifisch wirkenden Endo- und/oder Exopeptidase-Enzymen.
- Ausführliche Offenbarung der Erfindung
- Die vorliegende Erfindung betrifft ein Verfahren zum Erhalt eines Hydrolysats, das mit freier Glutaminsäure und/oder peptidgebundenen Glutaminsäureresten aus proteinartigem Substrat vegetabilen Ursprungs angereichert ist, wobei das Verfahren die Schritte umfasst:
- (i) Durchführung eines Deamidierungsverfahrens mit dem Substrat durch Unterziehen des Substrats der Wirkung einer Peptidoglutaminase oder einer Transglutaminase; und
- (ii) Unterziehen des Substrats der Wirkung eines spezifisch wirkenden proteolytischen Enzyms.
- Durch das erfindungsgemäße Verfahren können Proteinhydrolysate mit hervorragendem Aroma und Funktionalität erhalten werden. Insbesondere können Proteinhydrolysate mit hervorragendem Aroma erhalten werden, da Glutaminsäure (Glu), gleich ob frei oder peptidgebunden, eine wichtige Rolle für das Aroma und die Schmackhaftigkeit von Proteinhydrolysaten spielt. Durch das erfindungsgemäße Verfahren können jedoch auch Proteinhydrolysate mit verbesserter Funktionalität erhalten werden, insbesondere in Bezug auf verbesserte Löslichkeit, verbesserte emulgierende Eigenschaften, erhöhter Hydrolysegrad und verbesserte Schaumbildungseigenschaften.
- Deamidierungsverfahren
- Die Überführung von Amiden (Glutamin oder Asparagin) in geladene Säuren (Glutaminsäure oder Asparaginsäure) über die Freisetzung von Ammoniak ist als Deamidierung bekannt. Die Deamidierung kann als nicht-enzymatisches oder enzymatisches Deamidierungsverfahren stattfinden.
- Bei der vorliegenden Erfindung wird die enzymatische Deamidierung durchgeführt, indem das Substrat der Wirkung einer Transglutaminase oder der Wirkung einer Peptidoglutaminase unterzogen wird.
- Transglutamiasen
- Bei einer bevorzugten Ausführungsform wird die Deamidierung als ein enzymatisches Deamidierungsverfahren durchgeführt, wobei das Substrat der Wirkung einer Transglutaminase unterzogen wird. Die Transglutaminase kann aus jeder zweckmäßigen Quelle stammen, einschließlich von denjenigen, die aus Säugern stammen, siehe z.B.
JP 1050382 JP 5023182 EP 555.649 EP 379.606 - Bei einer weiteren spezifischen Ausführungsform ist die Transglutaminase von einer Oomycete, einschließlich eines Stammes von Phytophthora, insbesondere Phytophthora cactorum, eines Stammes von Pythium, insbesondere Pythium irregulare, Pytium sp., Pythium intermedium, Pythium ultimum, und Pythium periilum (oder P. perilocum) abgeleitet.
- Bei noch einer weiteren spezifischen Ausführungsform ist Transglutaminase bakteriellen Ursprungs und ist vorzugsweise von einem Bacillus-Stamm, insbesondere Bacillus subtilis, einem Streptoverticillium-Stamm, insbesondere Streptoverticillium mobaraensis, Streptoverticillium griseocarneum, Streptoverticillium cinnamoneum, und einem Stamm von Streptomyces, insbesondere Streptomyces lydicus abgeleitet.
- Die Transglutaminase soll in wirksamen Mengen zum proteinartigen Substrat zugegeben werden. Die Transglutaminase kann in den Mengen, die zweckmäßigerweise bei einem Deamidierungsverfahren verwendet werden, zugefügt werden. Es wird derzeit davon ausgegangen, dass eine wirksame Menge Transglutaminase im Bereich von etwa 0,01 bis etwa 5 % (Gew./Gew.) Enzymzubereitung bezüglich der Substratmenge, vorzugsweise im Bereich von etwa 0,1 bis etwa 1 % (Gew./Gew.) Enzymzubereitung bezüglich der Substratmenge liegt.
- Die enzymatische Behandlung (Inkubation) kann bei jeder zweckmäßigen Temperatur, bei der die Enzymzubereitung nicht inaktiviert wird, vorzugsweise in einem Bereich von etwa 20°C bis etwa 70°C liegen.
- Gemäß gängiger Praxis kann die Enzymzubereitung zweckmäßigerweise inaktiviert werden, indem die Temperatur des Inkubationsgemischs auf eine Temperatur erhöht wird, bei der die Enzyme inaktiviert werden, zum Beispiel auf über etwa 70°C, oder auch durch die Erniedrigung des pH- Wertes des Inkubationsgemisches auf einen Wert, bei dem die Enzyme inaktiviert werden, z.B. unter etwa 4.0.
- Peptidoglutaminasen
- Die Deamidierung wird als enzymatisches Deamidierungsverfahren durchgeführt, wobei das Substrat der Wirkung einer Peptidoglutaminase unterzogen wird.
- Bei einer spezielleren Ausführungsform kann die Peptidoglutaminase, die bei dem erfindungsgemäßen Verfahren verwendet wird, eine Peptidoglutaminase I (Peptidyl-Glutaminase;
EC 3.5.1.43 EC 3.5.1.44 - Die Peptidoglutaminase soll dem proteinartigen Substrat in wirksamen Mengen zugegeben werden. Die Peptidoglutaminase kann in den Mengen, die zweckmäßigerweise bei Deamidierungsverfahren verwendet werden, zugefügt werden. Es wird derzeit davon ausgegangen, dass eine wirksame Menge von Peptidoglutaminase im Bereich von etwa 0,01 bis etwa 100000 PGase-Einheiten pro 100 g Substrat, insbesondere im Bereich von etwa 0,1 bis etwa 10000 PGase-Einheiten pro 100 g Substrat liegt.
- Die enzymatische Behandlung (Inkubation) kann bei jeder zweckmäßigen Temperatur, bei der die Enzymzubereitung nicht inaktiviert wird, vorzugsweise im Bereich von etwa 20°C bis etwa 70°C stattfinden.
- Gemäß gängiger Praxis kann die Enzymzubereitung zweckmäßigerweise inaktiviert werden, indem die Temperatur des Inkubationsgemisches auf eine Temperatur erhöht wird, bei der die Enzyme inaktiviert werden, z.B. auf über etwa 70°C, oder auch indem der pH-Wert des Inkubationsgemischs auf einen Wert erniedrigt wird, bei dem die Enzyme inaktiviert werden, z.B. unter etwa 4,0, Spezifisch wirkende proteolytische Enzyme Das erfindungsgemäße Verfahren umfasst den Schritt, wobei das Substrat der Einwirkung eines spezifisch wirkenden proteolytischen Enzyms unterzogen wird. Insbesondere kann das spezifisch wirkende proteolytische Enzym eine spezifisch wirkende Endopeptidase oder eine spezifisch wirkende Exopeptidase sein. Das spezifisch wirkende proteolytische Enzym kann jeden verfügbaren Ursprungs sein.
- Endopeptidasen
- Bei einer bevorzugten Ausführungsform ist das spezifisch wirkende proteolytische Enzym eine Endopeptidase, wie
- (i) eine Glutamyl-Endopeptidase (
EC 3.4.21.19 - (ii) eine Lysyl-Endopeptidase (
EC 3.4.21.50 - (iii) eine Leucyl-Endopeptidase (
EC 3.4.21.57 - (iv) eine Glycyl-Endopeptidase (
EC 3.4.22.25 - (v) eine Prolyl-Endopeptidase (
EC 3.4.21.26 - (vi) Trypsin (
EC 3.4.21.4 - (vii) eine Peptidyl-Asp-Metalloendopeptidase (
EC 3.4.24.33 - Die Glutamyl-Endopeptidase (
EC 3.4.21.19 - Die Lysyl-Endopeptidase (
EC 3.4.21.50 - Die Leucyl-Endopeptidase (
EC 3.4.21.57 - Die Glycyl-Endopeptidase (
EC 3.4.22.25 - Die Prolyl-Endopeptidase (
EC 3.4.21.26 - Die Trypsin-ähnliche Endopeptidase von Punkt (vi) kann vorzugsweise von einem Stamm von Frisarium, insbesondere Fusarium oxysporum stammen, z.B. wie in WO 89/06270 oder WO 94/25583 beschrieben.
- Die Peptidyl-Asp-Metalloendopeptidase (
EC 3.4.24.33 - Exopeptidasen
- Bei einer weiteren bevorzugten Ausführungsform ist das spezifisch wirkende proteolytische Enzym ein Exopeptidaseenzym, das von beiden Enden des Peptids wirken kann, d.h. es kann eine Aminopeptidase oder eine Carboxypeptidase sein.
- Bei einer bevorzugten Ausführungsform ist das spezifisch wirkende proteolytische Enzym eine Aminopeptidase, wie
- (i) eine Leucylaminopeptidase (
EC 3.4.11.1 - (ii) eine Tripeptidaminopeptidase (
EC 3.4.11.4 - Bei einer weiteren bevorzugten Ausführungsform ist das spezifisch wirkende proteolytische Enzym eine Carboxypeptidase, wie
- (i) eine Prolincarboxypeptidase (
EC 3.4.16.2 - (ii) eine Carboxypeptidase A (
EC 3.4.17.1 - (iii) eine Carboxypeptidase B (
EC 3.4.17.2 - (iv) eine Carboxypeptidase C (
EC 3.4.16.5 - (v) eine Carboxypeptidase D (
EC 3.4.16.6 - (vi) eine Lysin(arginin)carboxypeptidase (
EC 3.4.17.3 - (vii) eine Glycincarboxypeptidase (
EC 3.4.17.4 - (viii) eine Alanincarboxypeptidase (
EC 3.4.17.6 - (ix) eine Glutamatcarboxypeptidase (
EC 3.4.17.11 - (x) eine Peptidyldipeptidase A (
EC 3.4.15.1 - (xi) eine Peptidyldipeptidase (
EC 3.4.15.5 - Das spezifisch wirkende Endo- oder Exopeptidaseenzym soll dem proteinartigen Substrat in wirksamen Mengen zugegeben werden. Das proteolytische Enzym kann in Mengen zugegeben werden, die bei herkömmlichen Proteinhydrolyseverfahren verwendet werden. Es wird derzeit davon ausgegangen, dass eine wirksame Menge der Endopeptidase im Bereich von etwa 0,05 bis etwa 15 CPU/100 g Substrat, insbesondere im Bereich von etwa 0,1 bis etwa 5 CPU/100 g Substrat liegt und eine wirksame Menge der Exopeptidase im Bereich von etwa 0,001 bis etwa 0,5 AU/100 g Substrat, insbesondere im Bereich von etwa 0,01 bis etwa 0,1 AU/100 g Substrat liegt.
- Die enzymatische Behandlung (Inkubation) kann bei jeder zweckmäßigen Temperatur stattfinden, bei der die Enzymzubereitung nicht inaktiviert wird, vorzugsweise in einem Bereich von etwa 20°C bis etwa 70°C.
- Gemäß gängiger Praxis kann die Enzymzubereitung zweckmäßigerweise inaktiviert werden, indem die Temperatur des Inkubationsgemischs auf eine Temperatur erhöht wird, bei der die Enzyme inaktiviert werden, z.B. bis über etwa 70°C, oder auch durch die Erniedrigung des pH-Werts des Inkubationsgemisches auf einem Wert, bei dem die Enzyme inaktiviert werden, z.B. unter etwa 4,0.
- Unspezifisch wirkende proteolytische Enzyme
- Bei einer spezielleren Ausführungsform umfasst das erfindungsgemäße Verfahren den zusätzlichen Schritt
- (iii) des Unterziehens des Substrats der Einwirkung einer oder mehrerer unspezifisch wirkender Endo- und/oder Exopeptidaseenzyme.
- Bei diesem Schritt wird das Substrat einem herkömmlichen Hydrolyseschritt unterzogen. Dieser zusätzliche Schritt kann gleichzeitig mit den Schritten (i) und (ii) stattfinden oder kann im Anschluss an die Schritte (i) und (ii) durchgeführt werden. Insbesondere kann das Substrat das Reaktionsgemisch von Schritt (i) und (ii) sein.
- Bei einer bevorzugten Ausführungsform stammt das unspezifisch wirkende Endo- und/oder Exopeptidaseenzym von einem Stamm von Aspergillus, insbesondere von einem Stamm von Aspergillus niger, einem Stamm von Aspergillus oryzae, oder einem Stamm von Aspergillus soyae, oder einem Stamm von Bacillus, insbesondere einem Stamm von Bacillus amyloliquefaciens, einem Stamm von Bacillus lentus, einem Stamm von Bacillus licheniformis oder einem Stamm von Bacillus subtilis.
- Das unspezifisch wirkende Endo- und/oder Exopeptidaseenzym wird dem Substrat in Mengen im Bereich von etwa 0,05 bis etwa 15 CPU/100 g Substrat, insbesondere im Bereich von etwa 0,1 bis etwa 5 CPU/100 g Substrat zugefügt.
- Die enzymatische Behandlung (Inkubation) kann bei jeder zweckmäßigen Temperatur, bei der die Enzymzubereitung nicht inaktiviert wird, stattfinden, vorzugsweise im Bereich von etwa 20°C bis etwa 70°C.
- Gemäß gängiger Praxis kann die Enzymzubereitung zweckmäßigerweise inaktiviert werden, indem die Temperatur des Reaktionsgemisches auf eine Temperatur erhöht wird, bei der die Enzyme inaktiviert werden, z.B. auf etwa über 70°C, oder auch durch Erniedrigung des pH-Werts des Inkubationsgemisches auf einen Wert, bei dem die Enzyme inaktiviert werden, z.B. unter etwa 4,0.
- Proteinartige Substrate
- Das proteinartige Substrat, das dem erfindungsgemäßen Verfahren unterzogen wird, kann aus intakten Proteinen bestehen, oder es kann aus vorhydrolysierten Proteinen (Peptiden) bestehen, oder es kann ein Gemisch davon sein.
- Das proteinartige Substrat ist pflanzlichen Ursprungs und kann insbesondere Sojaprotein, Getreideprotein, z.B. Weizengluten, Maisgluten, Gerste-, Roggen-, Hafer-, Reis-, Zein-, Baumwollsamenprotein, Rapssamenprotein, Erdnuss-, Alfalfaprotein, Erbsenprotein, bohnenartiges Bohnenprotein, Sesamsamenprotein oder Sonnenblumenprotein sein.
- Industrielle Anwendungsmöglichkeiten
- Die an freier Glutaminsäure und/oder peptidgebundenen Glutaminsäureresten angereicherten Hydrolysate, die durch das erfindungsgemäße Verfahren erhalten werden, können bei verschiedenen industriellen Anwendungen eingesetzt werden, insbesondere wobei es der Einarbeitung funktioneller Proteine bedarf.
- Darum stellt bei einem weiteren Aspekt die vorliegende Erfindung ein Lebensmittelprodukt bereit, das ein an freier Glutaminsäure und/oder peptidgebundenen Glutaminsäureresten angereichertes Hydrolysat, das durch das erfindungsgemäße Verfahren erhalten wird, umfasst.
- Bei noch einem weiteren Aspekt stellt die Erfindung einen Tierfutter-Zusatzstoff bereit, der ein mit freier Glutaminsäure und/oder peptidgebundenen Glutaminsäureresten angereichertes Hydrolysat, das durch das erfindungsgemäße Verfahren erhalten wird, umfasst.
- Enzymatischer Aktivitätstest
- Peatidoglutaminaseaktivität
- Die Peptidoglutaminaseaktivität kann gemäß dem Verfahren von Cedrangoro et al. [Cedrangoro et al.; Enzymolgia 1965, 29 143] und wie in
US 3,857,967 beschrieben bestimmt werden. - Nach diesem Verfahren werden 0,5 ml einer Enzymprobe, die mit 1 N NaOH auf pH 6,5 eingestellt ist, in einem kleinen Gefäß vorgelegt. Nach dem Verfahren von Cedrangoro et al. wird 1 ml Boratpufferlösung, pH 10,8, in das Gefäß gegeben, das abgegebene Ammoniak wird durch 5 N Schwefelsäure absorbiert, und eine Farbbildung des Gemisches durch die Verwendung von Nessler's Reagenz wird ermöglicht und die Lichtextinktion der gebildeten Farbe bei 420 nm gemessen.
- 1 PGase-Einheit ist die Enzymmenge, bei der unter diesen Bedingungen 1 μmol/min Ammoniak gebildet wird.
- Alternativ kann die Peptidoglutaminaseaktivität gemäß dem in Beispiel 2 beschriebenen Verfahren bestimmt werden.
- Proteolytische Aktivität: Caseinproteaseeinheiten (CPU)
- Die proteolytische Aktivität kann durch die Verwendung von Casein als Substrat bestimmt werden. Eine Casein-Proteaseeinheit (CPU) wird als die Enzymmenge definiert, die 1 mM der primären Aminogruppen (bestimmt durch Vergleich mit einem Serin-Standard) pro Minute unter Standardbedingungen freisetzt, d.h. Inkubation für 30 min bei 25°C und pH 9,5.
- Ein Ordner AF 228/1, der dieses analytisches Verfahren genauer beschreibt, ist auf Anfrage bei Novo Nordisk A/S, Dänemark, verfügbar, dessen Dokument hiermit als Referenz eingeschlossen wird.
- Proteolytische Aktivität: Anson-Einheiten (AU)
- Die proteolytische Aktivität kann alternativ mit denaturiertem Hämoglobin als Substrat bestimmt werden. Bei dem Anson-Hämoglobin-Verfahren zur Bestimmung der proteolytischen Aktivität wird denaturiertes Hämoglobin verdaut und das unverdaute Hämoglobin mit Trichloressigsäure (TCA) ausgefällt. Die Menge an TCA-löslichem Produkt wird mit Phenolreagenz bestimmt, das mit Tyrosin und Tryptophan eine blaue Farbe ergibt.
- 1 Anson-Einheit (AU) wird als die Enzymmenge definiert, wobei unter Standardbedingungen (d.h. 25°C, pH 7,5 und 10 min. Reaktionszeit) Hämoglobin mit einer Anfangsgeschwindigkeit verdaut wird, so dass eine Menge an TCA-löslichem Produkt pro Minute freigesetzt wird, welche mit Phenolreagenz die gleiche Farbe ergibt, wie 1 Milliäquivalent Tyrosin.
- Ein Ordner AF 4/5, der das analytische Verfahren genauer beschreibt, ist auf Anfrage bei Novo Nordisk A/S, Dänemark, erhältlich, wobei der Ordner hiermit durch Bezugnahme eingeschlossen ist.
- Proteolytische Aktivität: Leucinaminopeptidase-Einheiten (LAPU)
- Alternativ kann die proteolytische Aktivität mit Leucin-p-nitroanilid als Substrat bestimmt werden. Bei der Hydrolyse wird p-Nitroanilid freigesetzt und macht die Lösung gelb. Die Aktivität wird bestimmt, indem p-Nitroanilid als Chromophor verwendet und die gebildete gelbe Farbe bei 405 nm im Photometer gemessen wird.
- 1 Leucinaminopeptidase-Einheit (LAPU) ist die Enzymmenge, die 1 μ(micro)M Substrat pro Minute unter folgenden Bedingungen zersetzt: 26 mM Leucin-p-nitroanilid als Substrat, 0,1 M Trispuffer (pH 8,0), 40°C, 10 min Reaktionszeit.
- BEISPIELE
- Die Erfindung wird in den folgenden Beispielen weiter erläutert.
- Beispiel 1
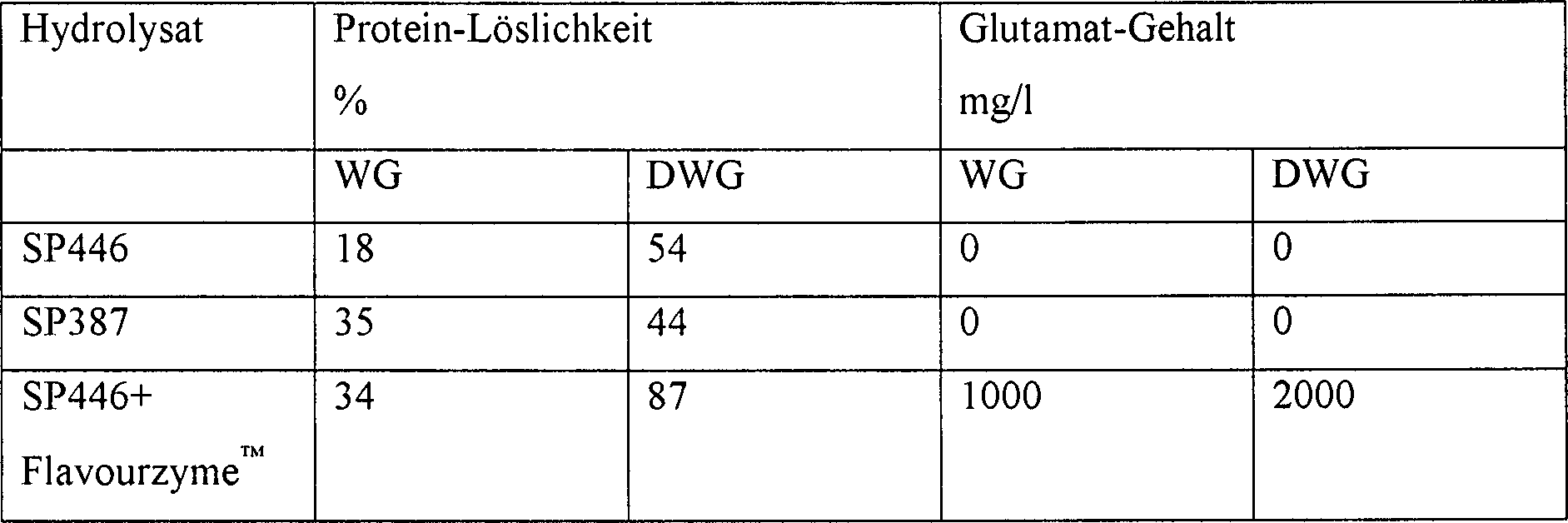
- Erhöhte Proteinlöslichkeit und Freisetzung von Glutamat durch Deamidierung
- Hydrolyse
- Weizengluten (WG) wurde von Cargill (JOB 5141) erhalten, und deamidiertes Weizengluten (DWG) wurde von StaPro Consultancy B. V., Lemdijk 32, 9422 TH Smilde, NL, erhalten. Suspensionen von 8 % Protein wurden hergestellt, indem 11 g Gluten mit 89 g Wasser gemischt wurden. Der p.H-Wert wurde mit NaOH auf 6,5 eingestellt. Den Suspensionen wurde Glutamat/Aspartatspezifische Protease (SP446), erhältlich wie in WO 91/13554 beschrieben, oder Lysin/Argininspezifische Protease (SP387), erhältlich wie in WO 89/06270 beschrieben, zugefügt. Die Dosierung betrug 0,01 AU/g Protein für SP446 und 0,006 AU/g Protein für SP387. FlavourzymeTM (eine unspezifisch wirkende Proteasezubereitung, erhältlich von Novo Nordisk A/S, Dänemark, die Endo- und Exopeptidaseaktivität enthält, und durch Fermentierung von Aspergillus oryzae erhalten wurde), wurde einigen der Hydrolysate in einer Dosis von 20 LAPU/g Protein zugefügt. Die Hydrolysen wurden bei 50°C ohne weitere pH-Einstellung für 18 h durchgeführt. Die Enzyme wurden durch Erhitzen auf 85°C für 15 min inaktiviert. Der pH-Wert wurde auf 5 eingestellt und die Hydrolysate zentrifugiert. Der Gehalt an Protein und freiem Glutamat im Überstand wurde bestimmt.
- Proteinbestimmung
- Protein wurde durch Kjeldahl-Analyse unter Verwendung eines Kjeldahl-Faktor 6,25 bestimmt.
- Glutamatbestimmung
- Die Gehalt an freiem Glutamat wurde unter Verwendung des Boehringer-Mannheim-Testsatzes zur Glutamatbestimmung (Katalog-Nr. 139 092) bestimmt. Das Verfahren wurde der Verwendung auf Mikrotiterplatten angepasst.
- Ergebnisse
- Beim Vergleich von Weizengluten (WG) mit deamidiertem Weizengluten (DWG) zeigen die Ergebnisse, dass die Deamidierung die Suszeptibilität von Gluten gegenüber spezifischen Proteasen erhöht, so dass mehr Protein löslich wird. Bei Zugabe von FlavourzymeTM, zusätzlich zu einer spezifischen Protease, wird die Freisetzung von Glutamat aufgrund von Deamidierung verdoppelt.
- Beispiel 2
- Enzymatische Deamidierung und Freisetzung von Glutamat
- Fermentierung von Bacilluss circulans
- Bacillus circulans-Kulturen des Stammes ATCC 21590 wurden in Schüttelkolben mit 400 ml Fassungsvermögen gezüchtet, die 200 ml eines Mediums enthielten, bestehend aus 1 % Polypepton; 0,5 % Lactose; 0,025 % MgSO4·7H2O; 0,005 % FeSO4·7H2O; 0,025 % KH2PO4 und 17 % Na2HPO4·12H2O, wobei der pH-Wert auf 7,2 eingestellt war. Die Fermentation erfolgte 20 h bei 30°C unter Verwendung eines Rührers mit 270 U/min. Die Zellen wurden durch Zentrifugation bei 4000 U/min in 1-1-Kolben geerntet. Die Zellen wurden eingefroren.
- Reinigung von Peptidoglutaminase II aus Bacillus circulans
- Alle Schritte wurden bei Raumtemperatur durchgeführt.
- Die gefrorenen Bacillus circulans-Zellen wurden aufgetaut und in Lysepuffer (50 mM Tris/HCl; 25 % (Gew./Vol.) Saccharose; 1 mM EDTA, pH 8,0) suspendiert, bis eine homogene Suspension erhalten wurde – 100 g nasse Zellen pro Liter Lysepuffer. LysozymTM (Fluka 62971, 10 mg/ml) und DNAse I (Sigma DN-25, 10 mg/ml) wurden in Lysepuffer gelöst. 100 ml LysozymTM-Lösung, 10 ml 1,0 M MgCl2 und 1 ml DNAse-I-Lösung wurden pro Liter Zensuspension zugefügt. Die Enzyme wurde 1 h einwirken gelassen.
- Die Suspension wurde über ein Seitz-Tiefenfilter filtriert und das Filtrat in 10 mM KH2PO4/NaOH, pH 8,0 (Puffer A) auf eine Sephadex G25-Säule (Pharmacia) übergeführt. Die Enzymlösung wurde auf eine mit Puffer A äquilibrierte SOURCE-Q-Säule (Pharmacia) aufgetragen und mit einem linearen Natriumchloridgradienten (0 → 500 mM) in Puffer A eluiert. Die Fraktionen der Säule wurden wie unten beschrieben auf die Peptidoglutaminase-II-Aktivität analysiert und Fraktionen mit Aktivität vereinigt. Die Extinktion der vereinigten Fraktionen bei 280 nm betrug 1,78, also wurde der Proteingehalt auf 1,8 mg/ml geschätzt.
- Die Reinheit des Proteins in dem Peptidoglutaminase-II-Pool war ungefähr 25 %, wie durch ein SDS-PAGE-Gel geschätzt wurde. Daher enthielt die Zubereitung etwa 0,5 mg/ml reine Peptidoglutaminase II.
- Peptidoglutaminase-Testverfahren
- Die enzymatische Aktivität wird bestimmt, indem das bei der Hydrolyse von γ-Carboxyamid von N-tert-Butoxycarbonyl (N-t-BOC-Gln-Pro; SIGMA Nr, B-4403) gebildete Ammoniak unter Verwendung des Boehringer-Mannheim-Testsatzes zur Ammoniakbestimmung (Katalog-Nr. 1112732) gemessen wird. In diesem Testsatz wird Ammoniak gemessen, indem der NADH-Verbrauch durch Glutamatdehydrogenase bestimmt wird und auch Leerproben ohne den Zusatz von N-t-BOC-Gln-Pro verwendet werden, um die Wirkung der anderen NADH-verbrauchenden Enzyme zu substrahieren.
- Weizengluten-Hydrolyse
- 200 mg Weizenglutenprotein wurden 9 ml kochendem Wasser zugefügt, und nach Abkühlung wurde der pH auf 7,0 eingestellt. Dann wurden 250 μl der Peptidoglutaminase-II-Zubereitung (PEP), oben beschrieben, zugefügt. Die Glutamat/Aspartat-spezifische Protease (SP446) aus Beispiel 1 wurde in einer Menge von 0,04 AU/g Protein zugefügt, und FlavourzymeTM aus Beispiel 1 wurde in einer Menge von 20 LAPU/g Protein zugefügt.
- Die Hydrolyse verlief 18 h ohne pH-Einstellung bei 50°C. Leerproben wurden ohne die Zugabe von Peptidoglutaminase hergestellt. Die Hydrolysate wurden zentrifugiert, und Glutamat wurde, wie in Beispiel 1 beschrieben, gemessen.
- Der Hydrolysegrad (DH) des Weizenglutenproteins wurde wie unten beschrieben bestimmt.
- Bestimmung des Hydrolysegrades (DH)
- Der DH des Proteins, definiert wie von Adler-Nissen [Adler-Nissen J; Enzymic Hydrolyse of Food Proteins; Elsevier Applied Science Publishers, 1986] beschrieben, wurde durch die Reaktion des Überstandes mit OPA (ortho-Phthaldialdehyd, Sigma) bestimmt. Für das OPA-Reagenz wurden 160 mg OPA in 4 ml Ethanol gelöst und in einen 200-ml-Messkolben übergeführt, der eine Lösung von 7,62 g Dinatriumtetraboratdecahydrat und 200 mg Natriumdodecylsulphat und 176 mg Dithiothreitol enthielt, und der Kolben wurde bis zur Markierung mit Wasser aufgefüllt. Das Reagenz ist lichtempfindlich.
- 25 μl des zweckmäßigerweise verdünnten Überstands wurden mit 200 μl OPA-Reagenz in einem Mikrotiterplattengefäß vermischt und eine Reaktion für genau 2 min bei 25°C ermöglicht. Die Extinktion bei 340 nm wurde in einem Mikrotiterplattenleser gemessen und mit der Extinktion einer 95 mM L-Serin-Standardlösung nach Abzug des Leerwertes (Wasser, das mit OPA-Reagenz reagiert hat), verglichen. Zur Feststellung des wahren DH, wurden die gemessenen Serin-Äquivalente in den Überständen mit den Faktoren korrigiert, die von Adler-Nissen für das Trinitrobenzolsulfonsäure-Verfahren vorgeschlagen wurden [Adler-Nissen J; Agricultural and Food Chemistry, 1979 27 (6), 1256], was zu der gleichen Reaktion wie bei der beschriebenen OPA-Methode führt. Der Hydrolysegrad wurde auf Basis der Gesamtproteinmenge in dem Hydrolysegemisch berechnet (nicht auf der Basis von löslichem Protein).
- Ergebnisse
- Wie von der Tabelle ersichtlich ist, erhöht die Hydrolyse mit der Peptidoglutaminase-Zubereitung den DH sowie die Freisetzung von Glutamat.
Claims (25)
- Verfahren zum Erhalt eines Hydrolysats, das an freier Glutaminsäure und/oder an Peptid-gebundenen Glutaminsäure-Resten angereichert ist, aus einer proteinartigen Substanz vegetabilen Ursprungs, wobei das Verfahren die Schritte umfasst: (i) Durchführung eines Deaminierungsverfahrens mit dem Substrat durch Unterziehen des Substrats der Wirkung einer Peptidoglutaminase oder einer Transglutaminase; und (ii) Unterziehen des Substrats der Wirkung eines spezifisch wirkenden proteolytischen Enzyms.
- Verfahren nach Anspruche 1, wobei die Glutaminase aus einer Oomycete stammt, einschließlich eines Stamms von Phytophthora, insbesondere Phytophthora cactorum, eines Stamms von Pythium, insbesondere Phythium irregulare, Pythium sp., Pythium intermedium, Pythium ultimum und Pythium periilum (oder Pythium periplocum).
- Verfahren nach Anspruche 1, wobei die Glutaminase bakteriellen Ursprungs ist und vorzugsweise aus einem Stamm von Bacillus, insbesondere Bacillus subtilis, einem Stamm von Streptoverticillium, insbesondere Streptoverticillium mobaraensis, Streptoverticillium griseocameum und Streptoverticillium cinnamoneum, und einem Stamm von Streptomyces, insbesondere Streptomyces lydicus stammt.
- Verfahren nach Anspruche 1, wobei die Peptidoglutaminase eine Peptidoglutaminase I (Peptidylglutaminase;
EC 3.5.1.43 EC 3.5.1.44 - Verfahren nach einem der Ansprüche 1 oder 4, wobei die Peptidoglutaminase aus einem Stamm von Aspergillus, insbesondere einem Stamm von Aspergillus, japonicus, einem Stamm von Bacillus, insbesondere einem Stamm von Bacillus circulans, einem Stamm von Cryptococcus, insbesondere einem Stamm von Cryptococcus albidus oder einem Stamm von Debaryomyces, insbesondere einem Stamm von Debaryomyces kloecheri stammt.
- Verfahren nach einem der Ansprüche 1, 4 und 5, wobei die Peptidoglutaminase dem Substrat in Mengen im Bereich von 0,01 bis 100.000 PGase-Einheiten pro 100 g Substrat, insbesondere im Bereich von 0,1 bis 10.000 PGase-Einheiten pro 100 g Substrat zugesetzt wird.
- Verfahren nach einem der Ansprüche 1 bis 6, wobei das spezifisch wirkende proteolytische Enzym eine Endopeptidase ist, wie (i) eine Glutamylendopeptidase (
EC 3.4.21.19 EC 3.4.21.50 EC 3.4.21.57 EC 3.4.22.25 EC 3.4.21.26 EC 3.4.21.4 EC 3.4.24.33 - Verfahren nach Anspruch 7, wobei die Glutamylendopeptidase von (i) aus einem Stamm von Bacillus, insbesondere Bacillus licheniformis und Bacillus subtilis, einem Stamm von Staphylococcus, insbesondere Stauhylococcus aurerus, einem Stamm von Streutomyces, insbesondere Streutomyces thermovulgaris und Streutomyces griseus oder einem Stamm von Actinomyces sp. stammt.
- Verfahren nach Anspruch 7, wobei die Lysylendopeptidase von (ii) aus einem Stamm von Achromobacter, insbesondere Achromobacter lyticus, einem Stamm von Lysobacter, insbesondere Lysobacter enzymogenes, oder einem Stamm von Pseudomonas, insbesondere Pseudomonas aeruginosa stammt.
- Verfahren nach Anspruch 7, wobei die Glycylendopeptidase aus (iv) aus der Papayapflanze (Carica Papaya) stammt.
- Verfahren nach Anspruch 7, wobei die Prolylendopeptidase aus (v) aus einem Stamm von Flavobacterium stammt.
- Verfahren nach Anspruch 7, wobei die Peptidyl-Asp-Metalloendopeptidase (
EC 3.4.24.33 - Verfahren nach einem der Ansprüche 1 bis 6, wobei das spezifisch wirkende proteolytische Enzym ein Exopeptidaseenzym ist.
- Verfahren nach Anspruch 13, wobei die Exopeptidase eine Aminopeptidase ist, wie (i) eine Leucylaminopeptidase (
EC 3.4.11.1 EC 3.4.11.4 - Verfahren nach Anspruch 13, wobei die Exopeptidase eine Carboxypeptidase ist, wie (i) eine Prolincarboxypeptidase (
EC 3.4.16.2 EC 3.4.17.1 EC 3.4.17.2 EC 3.4.16.5 EC 3.4.16.6 EC 3.4.17.3 EC 3.4.17.4 EC 3.4.17.6 (EC 3.4.17.11 EC 3.4.15.1 EC 3.4.15.5 - Verfahren nach einem der Ansprüche 1 zum Erhalt eines Hydrolysats, das an Glutaminsäure angereichert ist, aus einem proteinartigen Substrat, wobei das Verfahren die Schritte umfasst: (i) Unterziehen des Substrats der Wirkung einer Peptidoglutaminase; und (ii) Unterziehen des Substrats der Wirkung eines proteolytischen Enzyms, das vorzugsweise Glutamoyl-Peptidbindungen spaltet (d.h. eine Glutamylendopeptidase;
EC 3.4.21.19 - Verfahren nach einem der Ansprüche 1–16, wobei das spezifisch wirkende proteolytische Enzym dem Substrat in Mengen im Bereich von 0,05 bis 15 CPU/100 g Substrat, insbesondere im Bereich von 0,1 bis 5 CPU/100 g Substrat zugesetzt wird.
- Verfahren nach einem der Ansprüche 1–17, wobei Schritt (i) und (ii) von Anspruch 1 gleichzeitig durchgeführt werden.
- Verfahren nach einem der Ansprüche 1–17, wobei Schritt (ii) von Anspruch 1 im Anschluss an Schritt (i) von Anspruch 1 durchgeführt wird.
- Verfahren nach einem der Ansprüche 1–19, wobei das Verfahren den zusätzlichen Schritt umfasst (iii) Unterziehen des Substrats der Wirkung von einer oder mehreren unspezifisch wirkenden Endo- und/oder Exopeptidaseenzymen.
- Verfahren nach Anspruch 20, wobei die Schritte (i), (ii) und (iii) gleichzeitig durchgeführt werden.
- Verfahren nach Anspruch 20, wobei Schritt (iii) nach den Schritten (i)–(ii) durchgeführt wird.
- Verfahren nach einem der Ansprüche 20–22, wobei das unspezifisch wirkende Endo- und/oder Exopeptidaseenzym von Schritt (iii) aus einem Stanzen von Aspergillus, insbesondere einem Stamm von Aspergillus niger, einem Stamm von Aspergillus oryzae, oder einem Stamm von Aspergillus soyae oder einem Stamm von Bacillus, insbesondere einem Stamm von Bacillus amyloliquefaciens, einem Stamm von Bacillus Lentus, einem Stamm von Bacillus licheniformis oder einem Stamm von Bacillus subtilis stammt.
- Verfahren nach einem der Ansprüche 20–23, wobei das unspezifisch wirkende Endo- und/oder Exopeptidaseenzym dem Substrat in Mengen im Bereich von 0,05 bis 15 CPU/100 g Substrat, insbesondere im Bereich von 0,1 bis 5 CPU/100 g Substrat oder in Mengen im Bereich von 0,001 bis 0,5 AU/100 g Substrat, insbesondere im Bereich von 0,01 bis 0,1 AU/100 g Substrat zugesetzt wird.
- Verfahren nach einem der Ansprüche 1–24, wobei das proteinartige Substrat vegetabilen Ursprungs Sojaprotein, Getreideprotein, z.B. Weizengluten, Maisgluten, Gerste-, Roggen-, Hafer-, Reis-, Zein-, Baumwollsamenprotein, Rapssamenprotein, Erdnuss-, Alfalfaprotein, Erbsenprotein, bohnenartiges Bohnenprotein, Sesamsamenprotein oder Sonnenblumenprotein ist.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| DK58596 | 1996-05-20 | ||
| DK58596 | 1996-05-20 | ||
| PCT/DK1997/000230 WO1997043910A1 (en) | 1996-05-20 | 1997-05-20 | A method of obtaining protein hydrolysates |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| DE69736247D1 DE69736247D1 (de) | 2006-08-10 |
| DE69736247T2 true DE69736247T2 (de) | 2007-05-24 |
Family
ID=8095235
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| DE69736247T Expired - Fee Related DE69736247T2 (de) | 1996-05-20 | 1997-05-20 | Verfahren zur herstellung von proteinhydrolysate |
Country Status (6)
| Country | Link |
|---|---|
| EP (1) | EP0969736B1 (de) |
| JP (1) | JP2000515003A (de) |
| CN (2) | CN1099839C (de) |
| BR (1) | BR9709006A (de) |
| DE (1) | DE69736247T2 (de) |
| WO (1) | WO1997043910A1 (de) |
Families Citing this family (30)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO1998051803A1 (en) * | 1997-05-16 | 1998-11-19 | Novo Nordisk Biotech, Inc. | Polypeptides having prolyl pipeptidyl aminopeptidase activity and nucleic acids encoding same |
| DE69837475T2 (de) | 1997-05-16 | 2007-12-13 | Novozymes, Inc., Davis | Polypeptide mit aminopeptidaseaktivität und dafür kodierende nukleinsäuren |
| US6251651B1 (en) | 1998-06-04 | 2001-06-26 | Amano Pharmaceutical Co., Ltd. | Protein-deamidating enzyme, gene encoding the same, production process therefor, and use thereof |
| US6756221B1 (en) | 1999-06-03 | 2004-06-29 | Amano Enzyme Inc. | Protein-deamidating enzyme, microorganism producing the same, gene encoding the same, production process therefor, and use thereof |
| US6974597B2 (en) * | 1999-11-29 | 2005-12-13 | Kyowa Hakko Kogyo Co., Ltd. | Method of enhancing salty taste, salty taste enhancer, salty taste seasoning agent and salty taste-enhanced foods |
| JP3696500B2 (ja) * | 1999-12-03 | 2005-09-21 | 天野エンザイム株式会社 | 新規蛋白質脱アミド酵素、それを生産する微生物、それをコードする遺伝子、その製造法及び用途 |
| EP1312268A1 (de) * | 2001-11-19 | 2003-05-21 | Société des Produits Nestlé S.A. | Geschmackstoffkompositionen |
| DK175501B1 (da) | 2002-12-02 | 2004-11-15 | Green Earth | Anlæg og fremgangsmåde til kontinuerlig hydrolyse af et proteinholdigt animalsk eller vegetabilsk råmateriale |
| CN101090644A (zh) * | 2005-01-13 | 2007-12-19 | 味之素株式会社 | 肉制品或水产糜制品及其制造方法 |
| US7485323B2 (en) | 2005-05-31 | 2009-02-03 | Gelita Ag | Process for making a low molecular weight gelatine hydrolysate and gelatine hydrolysate compositions |
| US8222372B2 (en) * | 2007-04-16 | 2012-07-17 | Novozymes A/S | Whey protein hydrolysate |
| WO2009011253A1 (ja) | 2007-07-13 | 2009-01-22 | Fuji Oil Company, Limited | グルテン用分散性改良剤及びグルテンの分散液 |
| CN101513218B (zh) * | 2009-03-10 | 2011-09-07 | 周靖波 | 从小麦胚芽中提取抗氧化活性小肽营养粉的方法 |
| NZ595062A (en) * | 2009-04-02 | 2012-10-26 | Novozymes As | Process for making a milk-based protein hydrolysate |
| PE20120532A1 (es) | 2009-04-27 | 2012-05-18 | Novartis Ag | ANTICUERPOS ANTI-ActRIIB |
| CN102379359B (zh) * | 2010-08-30 | 2013-09-25 | 内蒙古伊利实业集团股份有限公司 | 酶法水解酪蛋白的方法及水解产物 |
| EP2648537B1 (de) * | 2010-12-06 | 2019-01-23 | Cargill, Incorporated | Verfahren zur verflüssigung von getreideproteinen |
| JP6574384B2 (ja) | 2013-01-22 | 2019-09-11 | マース インコーポレーテッドMars Incorporated | フレーバ組成物及びそれを含む食用組成物 |
| JP6511400B2 (ja) * | 2013-02-05 | 2019-05-15 | オートリー エービー | 液状のエンバクベース |
| CN103549113A (zh) * | 2013-10-30 | 2014-02-05 | 深圳市伟崇科技发展有限公司 | 一种改性植物蛋白的制备方法及其用途 |
| RU2611378C2 (ru) * | 2015-05-08 | 2017-02-21 | Федеральное государственное автономное образовательное учреждение высшего профессионального образования "Казанский (Приволжский) федеральный университет" (ФГАОУВПО КФУ) | Применение рекомбинантного штамма bacillus subtilis тв-06 в качестве продуцента глутамилэндопептидазы, вещества с тромболитическими и антикоагулянтными свойствами |
| CN104982891A (zh) * | 2015-06-11 | 2015-10-21 | 广东厨邦食品有限公司 | 一种酱油原油的制备方法 |
| CN105248835B (zh) * | 2015-09-02 | 2019-09-20 | 广东美味鲜调味食品有限公司 | 玉米蛋白水解物及其制备方法与在纯粮酿造食醋中的应用 |
| AU2017230091B2 (en) | 2016-03-10 | 2022-04-07 | Acceleron Pharma Inc. | Activin type 2 receptor binding proteins and uses thereof |
| CN106434799A (zh) * | 2016-09-28 | 2017-02-22 | 华南理工大学 | 一种蛋白质纳米颗粒及其制备方法 |
| FR3057180B1 (fr) * | 2016-12-12 | 2018-10-12 | Constellium Issoire | Procede d'amelioration du mouillage d'une surface d'un substrat solide par un metal liquide |
| WO2021254943A1 (en) * | 2020-06-17 | 2021-12-23 | Dsm Ip Assets B.V. | Umami flavour composition |
| CN113801908B (zh) * | 2021-08-23 | 2024-04-09 | 杭州娃哈哈科技有限公司 | 一种乳清蛋白源缓解体力疲劳功能肽及其制备方法 |
| CN115381083A (zh) * | 2022-08-09 | 2022-11-25 | 武汉新华扬生物股份有限公司 | 一种复合酶制剂及其在提高荔枝汁蛋白含量中的应用 |
| CN116235895B (zh) * | 2023-03-14 | 2024-09-27 | 西南科技大学 | 一种基于棉籽分离蛋白制备食品乳化剂的方法 |
Family Cites Families (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US3857967A (en) * | 1971-05-10 | 1974-12-31 | Kikkoman Shoyu Co Ltd | Preparation of food and beverages with peptidoglutaminase |
| US5082672A (en) * | 1989-06-21 | 1992-01-21 | The United States Of American As Represented By The Secretary Of Agriculture | Enzymatic deamidation of food proteins for improved food use |
| NL194258C (nl) * | 1989-09-01 | 2001-11-05 | Protein Technologies Internat | Werkwijze voor het behandelen van een eiwithoudend materiaal. |
| EP0518999B1 (de) * | 1990-03-09 | 1995-08-30 | Novo Nordisk A/S | Proteinhydrolysate |
-
1997
- 1997-05-20 JP JP09541414A patent/JP2000515003A/ja active Pending
- 1997-05-20 CN CN97194838A patent/CN1099839C/zh not_active Expired - Fee Related
- 1997-05-20 EP EP97921649A patent/EP0969736B1/de not_active Expired - Lifetime
- 1997-05-20 BR BR9709006A patent/BR9709006A/pt active Search and Examination
- 1997-05-20 DE DE69736247T patent/DE69736247T2/de not_active Expired - Fee Related
- 1997-05-20 WO PCT/DK1997/000230 patent/WO1997043910A1/en active IP Right Grant
-
2001
- 2001-05-31 CN CN01119457A patent/CN1326687A/zh active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| DE69736247D1 (de) | 2006-08-10 |
| JP2000515003A (ja) | 2000-11-14 |
| CN1219106A (zh) | 1999-06-09 |
| AU722915B2 (en) | 2000-08-17 |
| CN1099839C (zh) | 2003-01-29 |
| WO1997043910A1 (en) | 1997-11-27 |
| BR9709006A (pt) | 1999-08-03 |
| CN1326687A (zh) | 2001-12-19 |
| AU2765097A (en) | 1997-12-09 |
| EP0969736B1 (de) | 2006-06-28 |
| EP0969736A1 (de) | 2000-01-12 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| DE69736247T2 (de) | Verfahren zur herstellung von proteinhydrolysate | |
| US6036983A (en) | Method of obtaining protein hydrolysates | |
| JP3153237B2 (ja) | タンパク質加水分解物 | |
| US20160316786A1 (en) | Method for Producing a Wheat Protein Hydrolysate | |
| US20090297661A1 (en) | Methods For Flavor Enhancement | |
| US6589574B2 (en) | Process for preparation of protein-hydrolysate from milk protein | |
| WO2002068671A1 (fr) | Désamidation et dénaturation d'une protéine du lait | |
| Triki-Ellouz et al. | Biosynthesis of protease by Pseudomonas aeruginosa MN7 grown on fish substrate | |
| JP2958801B2 (ja) | 脱苦味された機能性ペプチドの製造法 | |
| JP2001061445A (ja) | 蛋白酵素分解物 | |
| Lei et al. | An arginine aminopeptidase from marine Bacillus axarquiensis SWJSX8 and its application in improving pumpkin seed protein hydrolysis | |
| JPH0360480B2 (de) | ||
| AU722915C (en) | A method of obtaining protein hydrolysates | |
| WO1998017127A1 (en) | Method of using enzymes and yeast fermentation for production of a fermentation-type savory flavored product | |
| JP2502531B2 (ja) | 改質蛋白の製造法 | |
| JP2799352B2 (ja) | コーングルテンミール加水分解物の製造方法 | |
| Gill et al. | An assessment of the potential of peptidoglutaminases I and II in modifying the charge characteristics of casein and whey proteins | |
| CN101002612B (zh) | 蛋白质水解物的制备方法 | |
| WO2002069734A1 (en) | Process for the preparation of protein hydrolysate from milk protein | |
| Nakadai et al. | Hydrolysis of acid-treated protein by a preparation of proteases | |
| Chaijamrus | Enzymatic hydrolysis of renewable vegetable proteins to amino acids | |
| JPH04297500A (ja) | 着色劣化性の小さいペプチド及びその製造方法 | |
| JP2004089050A (ja) | タンパク質酵素分解物の呈味改善方法 | |
| Arai | Improving the supply, quality, and utility of proteins | |
| JP2001017165A (ja) | 新規グリシンアミノペプチダーゼ |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| 8364 | No opposition during term of opposition | ||
| 8339 | Ceased/non-payment of the annual fee |